Перекрестная ссылка на родственные заявки
Данная заявка испрашивает приоритет согласно 35 U.S.C. §119 USSN 62/118208, поданной 19 февраля, 2015 г., и USSN 62/141120, поданной 31 марта 2015 г., и USSN 62/235823, поданной 1 октября 2015 г., которые все явным образом и в полном объемы включены в данный документ посредством ссылки.
Уровень техники
Для продуктивной активации наивные Т-клетки должны получить два независимых сигнала от антигенпредставляющих клеток (АПК). Первый Сигнал 1 является антигенспецифическим и возникает, когда антигенные рецепторы Т-клеток сталкиваются с подходящим комплексом антиген-ГКГС на АПК. Судьба иммунного ответа определяется вторым, антигеннезависимым сигналом (Сигнал 2), который доставляется посредством Т-клеточной ко-стимуляторной молекулы, которая задействует экспрессируемый АПК лиганд. Этот второй сигнал может быть как стимуляторным (положительная ко-стимуляция), так и ингибиторным (отрицательная ко-стимуляция или ко-ингибирование). В отсутствие ко-стимуляторного сигнала или в присутствии ко-ингибиторного сигнала нарушается или прекращается активация Т-клеток, что может привести к состоянию антигенспецифической неотвечаемости (известной как Т-клеточная толерантность) или может привести к апоптотической гибели Т-клеток.
Пары ко-стимуляторных молекул обычно состоят из лигандов, экспрессируемых на АПК, и их когнатных рецепторов, экспрессируемых на Т-клетках. Прототипными парами лиганд/рецептор ко-стимуляторных молекул являются B7/CD28 и CD40/CD40L. Семейство B7 состоит из структурно подобных лигандов белков клеточной поверхности, которые могут обеспечивать стимуляторный или ингибиторный вклад в иммунный ответ. Представители семейства В7 являются структурно подобными с внеклеточным доменом, содержащим по меньшей мере один вариабельный или константный домен иммуноглобулина.
Как положительные, так и отрицательные ко-стимуляторные сигналы играют важную роль в регуляции клеточноопосредованных иммунных ответов, и было доказано, что молекулы, которые опосредуют эти сигналы, являются эффективными мишенями для иммуномодуляции. На основании этой информации было разработано несколько терапевтических подходов, которые включают нацеливание на ко-стимуляторные молекулы, и было показано, что они полезны для предотвращения и лечения рака путем включения или предотвращения выключения иммунных ответов у раковых пациентов и для предотвращения и лечения аутоиммунных заболеваний и воспалительных заболеваний, а также отторжения аллогенной трансплантации путем выключения неконтролируемых иммунных ответов или путем индукции «сигнала выключения» посредством отрицательной ко-стимуляции (или ко-ингибирования) у субъектов с этими патологическими состояниями.
Было показано, что управление сигналами, доставляемыми лигандами В7, является эффективным при лечении аутоиммунных, воспалительных заболеваний и отторжения трансплантатов. Терапевтические стратегии включают блокирование ко-стимуляции с помощью моноклональных антител к лиганду или к рецептору ко-стимуляторной пары или с помощью растворимых слитых белков, состоящих из ко-стимуляторного рецептора, который может связывать и блокировать соответствующий ему лиганд. Другим подходом является индукция ко-ингибирования с помощью растворимого слитого белка ингибиторного лиганда. Эти подходы основаны, по меньшей мере частично, на случайном удалении ауто- или аллореактивных Т-клеток (которые отвечают за патогенные процессы при аутоиммунных заболеваниях или трансплантации соответственно), главным образом потому, что вследствие отсутствия ко-стимуляции (которая индуцирует гены клеточного выживания) Т-клетки становятся очень восприимчивыми в индукции апоптоза. Таким образом, новые агенты, способные модулировать ко-стимуляторные сигналы, не нарушая способности иммунной системы защищать от патогенов, имеют большое преимущество для лечения и предотвращения таких патологических состояний.
Ко-стимуляторные пути играют важную роль в развитии опухолей. Интересно, что было показано, что опухоли уклоняются от иммунного разрушения, препятствуя активации Т-клеток посредством ингибирования ко-стимуляторных факторов в семействах B7-CD28 и TNF, а также привлекая регуляторные Т-клетки, которые ингибируют противоопухолевые ответы Т-клеток (смотрите Wang (2006), “Immune Suppression by Tumor Specific CD4+ Regulatory T cells in Cancer”, Semin. Cancer. Biol. 16:73-79; Greenwald, et al. (2005), “The B7 Family Revisited”, Ann. Rev. Immunol. 23:515-48; Watts (2005), “TNF/TNFR Family Members in Co-stimulation of T Cell Responses”, Ann. Rev. Immunol. 23:23-68; Sadum, et al., (2007) “Immune Signatures of Murine and Human Cancers Reveal Unique Mechanisms of Tumor Escape and New Targets for Cancer Immunotherapy”, Clin. Canc. Res. 13(13): 4016-4025). Такие экспрессируемые опухолями ко-стимуляторные молекулы стали привлекательными раковыми биомаркерами и могут служить в качестве опухолеассоциированных антигенов (ОАА). Более того, было определено, что ко-стимуляторные пути являются иммунологическими контрольными точками, которые ослабляют зависимые от Т-клеток иммунные ответы как на уровне инициации, так и эффекторной функции в опухолевых метастазах. По мере улучшения создаваемых противораковых вакцин становится ясно, что такие иммунологические контрольные точки являются основными барьерами для возможности индукции вакциной терапевтических противоопухолевых ответов. В связи с этим ко-стимуляторные молекулы могут служить в качестве адъювантов для активной (вакцинации) и пассивной (антителоопосредованной) раковой иммунотерапии, обеспечивая стратегии для преодоления иммунной толерантности и стимуляции иммунной системы.
За последнее десятилетие были разработаны агонисты и/или антагонисты различных ко-стимуляторных белков для лечения аутоиммунных заболеваний, отторжения трансплантатов, аллергии и рака. Например, CTLA4-Ig (абатасепт, Оренсия®) разрешен для лечения РА, мутированный CTLA4-Ig (белатацепт, Нулоджикс®) - для предотвращения острого отторжения почечного трансплантата, а анти-CTLA4 антитело (ипилимумаб, Ервой®), недавно разрешено для лечения меланомы. Другие регуляторы ко-стимуляции, такие как анти-PD-1 антитела Merck (Кейтруда®) и BMS (Опдиво®), были разрешены для лечения рака и также проходят испытания в отношении вирусных инфекций.
Соответственно, целью изобретения является обеспечение иммуномодулирующих антител к PVRIG.
Краткое описание сущности изобретения
Соответственно, целью изобретения является обеспечение способов активации цитотоксических Т-клеток (ЦТЛ) пациента, включающих введение пациенту анти-PVRIG антитела, причем происходит активация субпопуляции ЦТЛ пациента.
Дополнительной целью изобретения является обеспечение способов активации NK-клеток пациента, включающих введение пациенту анти-PVRIG антитела, причем происходит активация субпопуляции NK-клеток пациента.
Дополнительной целью изобретения является обеспечение способов активации γδ Т-клеток пациента, включающих введение пациенту анти-PVRIG антитела, причем происходит активация субпопуляции γδ Т-клеток пациента.
Дополнительной целью изобретения является обеспечение способов активации Th1-клеток пациента, включающих введение пациенту анти-PVRIG антитела, причем происходит активация субпопуляции Th1-клеток пациента.
Дополнительной целью изобретения является обеспечение способов ингибирования взаимодействия PVRIG и PVLR2 в организме пациента, имеющего патологическое состояние, связанное с этим взаимодействием, включающих введение пациенту анти-PVRIG антитела.
Дополнительной целью изобретения является обеспечение способов лечения рака у пациента, включающих введение пациенту анти-PVRIG антитела, причем происходит лечение указанного рака.
Дополнительной целью изобретения является обеспечение вышеуказанных способов, причем анти-PVRIG антитело содержит последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3 из антитела, выбранного из группы, состоящей из CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение вышеуказанных способов, причем анти-PVRIG антитело конкурирует за связывание с антителом, содержащим последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3 из антитела, выбранного из группы, состоящей из CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение вышеуказанных способов, причем анти-PVRIG антитело выбрано из группы, состоящей из CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение вышеуказанных способов, причем анти-PVRIG антитело конкурирует за связывание с антителом, выбранным из группы, состоящей из CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение вышеуказанных способов, причем анти-PVRIG антитело содержит последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3 из антитела, выбранного из группы, состоящей из CHA.7.502, CHA.7.503, CHA.7.506, CHA.7.508, CHA.7.510, CHA.7.512, CHA.7.514, CHA.7.516, CHA.7.518, CHA.7.520.1, CHA.7.520.2, CHA.7.522, CHA.7.524, CHA.7.526, CHA.7.527, CHA.7.528, CHA.7.530, CHA.7.534, CHA.7.535, CHA.7.537, CHA.7.538.1, CHA.7.538.2, CHA.7.543, CHA.7.544, CHA.7.545, CHA.7.546, CHA.7.547, CHA.7.548, CHA.7.549, CHA.7.550, CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение вышеуказанных способов, причем указанное анти-PVRIG антитело конкурирует за связывание с антителом, выбранным из группы, состоящей из анти-PVRIG антитела, содержащего последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3 из антитела, выбранного из группы, состоящей из CHA.7.502, CHA.7.503, CHA.7.506, CHA.7.508, CHA.7.510, CHA.7.512, CHA.7.514, CHA.7.516, CHA.7.518, CHA.7.520.1, CHA.7.520.2, CHA.7.522, CHA.7.524, CHA.7.526, CHA.7.527, CHA.7.528, CHA.7.530, CHA.7.534, CHA.7.535, CHA.7.537, CHA.7.538.1, CHA.7.538.2, CHA.7.543, CHA.7.544, CHA.7.545, CHA.7.546, CHA.7.547, CHA.7.548, CHA.7.549, CHA.7.550, CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение способов диагностирования рака, включающих a) приведение ткани пациента в контакт с анти-PVRIG антителом; и b) определение наличия сверхэкспрессии PVRIG в ткани, как показателя наличия рака. Анти-PVRIG антитело может представлять собой описанное в данном документе и приведенное выше.
Дополнительной целью изобретения является обеспечение антигенсвязывающих доменов, включая антитела, которые представляют собой анти-PVRIG антитела, содержащие последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3 из антитела, выбранного из группы, состоящей из CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение композиций анти-PVRIG антигенсвязывающих доменов (включая антитела), которые представляют собой анти-PVRIG антитела, выбранные из группы, состоящей из CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение композиций анти-PVRIG антигенсвязывающих доменов (включая антитела), которые представляют собой анти-PVRIG антитела, выбранные из группы, состоящей из h518-1, h518-2, h518-3, h518-4, h518-5, h524-1, h524-2, h524-3, h524-4, h530-1, h530-2, h530-3, h530-4, h530-5, h538.1-1, h538.1-2, h538.1-3, h538.1-4, h538.2-1, h538.2-2 и h538.2-3 (как изображено на фиг. 90).
Дополнительной целью изобретения является обеспечение антигенсвязывающих доменов, включая антитела, которые представляют собой анти-PVRIG антитела, содержащие последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3 из антитела, выбранного из группы, состоящей из CHA.7.502, CHA.7.503, CHA.7.506, CHA.7.508, CHA.7.510, CHA.7.512, CHA.7.514, CHA.7.516, CHA.7.518, CHA.7.520.1, CHA.7.520.2, CHA.7.522, CHA.7.524, CHA.7.526, CHA.7.527, CHA.7.528, CHA.7.530, CHA.7.534, CHA.7.535, CHA.7.537, CHA.7.538.1, CHA.7.538.2, CHA.7.543, CHA.7.544, CHA.7.545, CHA.7.546, CHA.7.547, CHA.7.548, CHA.7.549, CHA.7.550, CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение композиций нуклеиновых кислот, содержащих: a) первую нуклеиновую кислоту, кодирующую вариабельный домен тяжелой цепи, содержащий vhCDR1, vhCDR2 и vhCDR3 из антитела; и b) вторую нуклеиновую кислоту, кодирующую вариабельный домен легкой цепи, содержащий vlCDR1, vlCDR2 и vlCDR3 из антитела. Антитело выбрано из группы, состоящей из CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049, CPA.7.050, CHA.7.502, CHA.7.503, CHA.7.506, CHA.7.508, CHA.7.510, CHA.7.512, CHA.7.514, CHA.7.516, CHA.7.518, CHA.7.520.1, CHA.7.520.2, CHA.7.522, CHA.7.524, CHA.7.526, CHA.7.527, CHA.7.528, CHA.7.530, CHA.7.534, CHA.7.535, CHA.7.537, CHA.7.538.1, CHA.7.538.2, CHA.7.543, CHA.7.544, CHA.7.545, CHA.7.546, CHA.7.547, CHA.7.548, CHA.7.549, CHA.7.550, CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050.
Дополнительной целью изобретения является обеспечение композиций экспрессионных векторов, содержащих вышеуказанные первую и вторую нуклеиновые кислоты.
Дополнительной целью изобретения является обеспечение клеток-хозяев, содержащих композиции экспрессионных векторов, как в виде одиночных экспрессионных векторов, так и двух экспрессионных векторов.
Дополнительной целью изобретения является обеспечение способов создания-PVRIG антитела, включающих a) культивирование клетки-хозяина согласно изобретению с экспрессионным(и) вектором(ами) в условиях, в которых происходит выработка антитела; и b) выделение антитела.
Краткое описание графических материалов
Фиг. 1 является схематическим представлением механизмов действия согласно изобретению.
На фиг. 2 представлена экспрессия мРНК PVRIG в различных нормальных человеческих тканях.
На фиг. 3 представлена экспрессия мРНК PVRIG в различных иммунных популяциях, полученных из периферической крови и костного мозга (на основании GSE49910).
На фиг. 4 представлена экспрессия мРНК PVRIG в различных популяциях лимфоцитов CD3+ (на основании GSE47855).
На фиг. 5 A, 5B и 5C представлена экспрессия мРНК PVRIG в специфических клеточных популяциях. На фиг. 5A представлена экспрессия мРНК PVRIG в специфических клеточных популяциях, полученная с помощью лазерной захватывающей микроскопии (на основании GSE39397). На фиг. 5B представлена экспрессия мРНК PVRIG в Т-клетках CD4 от нормального и ракового пациента, а так же экспрессия в Т-клетках CD4 в дренирующих лимфатических узлах и ОИЛ от пациентов с раком молочной железы (на основании GSE36765). На фиг. 5C представлена экспрессия мРНК PVRIG в Т-клетках CD8 и CD4, полученных их опухоли фолликулярной лимфомы и миндалевидной железы (на основании GSE27928).
На фиг. 6 представлена экспрессия PVRIG в нормальных тканях на основании GTEx. Уровни экспрессии приведены в значениях log2(RPKM) (фрагменты, определенные на миллион ридов на тысячу оснований). Значения, превышающие 1, считаются высокой экспрессией. Ткани упорядочены по возрастанию по медианным значениям экспрессии. Каждая точка на графике представляет один образец.
На фиг. 7 представлена экспрессия PVRIG в раковых тканях на основании TCGA. Уровни экспрессии приведены в значениях log2(RPKM) (фрагменты, определенные на миллион ридов на тысячу оснований). Значения, превышающие 1, считаются высокой экспрессией. Ткани упорядочены по возрастанию по медианным значениям экспрессии. Каждая точка на графике представляет один образец.
На фиг. 8 приведена тепловая карта, представляющая результаты анализа представленности в трех категориях: белковые взаимодействия, пути и связи с заболеваниями. Результаты упорядочены в порядке возрастания по среднему p-значению на ряд. Приведены только 10 наивысших результатов из каждой категории. Серые квадраты указывают на p-значения < 0,05. Каждая колонка на тепловой карте соответствует нормальной или раковой ткани, из которой был получен набор высококоррелирующих генов (r > 0,55 при применении по меньшей мере 50 образцов). Как показано на тепловой карте, PVRIG коррелирует с Т-клеточным профилем экспрессии генов, который прочно связан с иммунным ответом и иммунными заболеваниями.
На фиг. 9 представлена экспрессия PVRIG в нормальной коже и меланоме (анализ GTEx и TCGA). Такую сверхэкспрессию наблюдали в дополнительных солидных опухолях, а ее причиной являются инфильтрирующие лимфоциты и NK-клетки в опухолевом микроокружении. В нормальных условиях инфильтрирующие иммунные клетки отсутствуют и, следовательно, уровни экспрессии PVRIG являются очень низкими.
На фиг. 10 представлена корреляция PVRIG и PD1 в меланоме из образцов TCGA с несколькими Т-клеточными маркерами в аденокарциноме легкого, аденокарциноме толстой кишки и меланоме. Маркер CD3 является обычным маркером для Т-клеток и также экспрессируется в NKT-клетках. Маркеры CD4 и CD8 используют для получения характеристик субпопуляций Т-клеток.
На фиг. 11 приведена экспрессия PVRIG в человеческих ЛПК. Человеческие ЛПК, полученные от двух доноров, оценивали в отношении экспрессии PVRIG. Как донор 61, так и донор 40 демонстрировали значительное окрашивание анти-PVRIG специфическим Ab.
На фиг. 12 показано, что PVRIG-Ig демонстрирует сильное связывание со всеми четырьмя исследуемыми клеточными линиями человеческой меланомы MEL-23, Mel-624 и Mel-624.38 и mel-888. На связывание не влияет совместное культивирование со сконструированными специфическими в отношении меланомы Т-клетками. Серая линия соответствует изотипическому контролю, непрерывная черная линия соответствует PVRIG-ECD-Ig.
На фиг. 13 представлена корреляция PVRIG с Т-клетками и субпопуляциями Т-клеток. CD3G является компонентом комплекса рецепторов Т-клеток, CD4 является маркером хелперных Т-клеток, а CD8A является компонентом белка CD8, используемого для идентификации цитотоксических Т-клеток. PVRIG сильно коррелирует с Т-клетками во многих типах опухолей, включая приведенные здесь аденокарциному легкого, аденокарциному толстой кишки и меланому.
На фиг. 14 представлены типовые изображения анализа подтверждения/специфичности. Все хиты из первичного анализа и EGFR-экспрессирующий вектор (отрицательный контроль) повторно упорядочивали/экспрессировали в двух повторностях и зондировали PVRIG при 20 мкг/мл. Специфический хит с сильной интенсивностью показан зеленым цветом (PVRL2). Неспецифические хиты показаны черным цветом. Позже было показано, что другой слабый хит (MAG) связывается также с другими лигандами, что позволяет предположить, что он не является специфическим.
На фиг. 15A-15E показано действие различных белков PVRIG-ECD-Ig M:M на активацию мышиных Т-клеток CD4. Планшеты покрывали анти-CD3 mAb (2 мкг/мл) в присутствии 10 мкг/мл PVRIG-ECD Ig (партия #198) или контрольного mIgG2a, как описано в разделе материалов и методов. В лунки высевали 1x105 мышиных Т-клеток CD4+CD25- на лунку в присутствии 2 мкг/мл растворимого анти-CD28. (A) Экспрессию CD69 анализировали методом проточной цитометрии через 48 ч после стимуляции, приведены типовые гистограммы. Каждый столбик представляет собой среднее по двум параллельным культурам, планки погрешностей указывают стандартное отклонение. (B-C) Культуральные супернатанты собирали через 48 ч после стимуляции, а уровни мышиных IL-2 и IFNγ анализировали методом ELISA. Результаты показаны в виде среднего ± среднеквадратические погрешности по двум параллельным образцам. (D) Представлен дозозависимый эффект для PVRIG-ECD Ig (фиг. 92BB на поверхности CD69 (D)) и секреции IFNγ (E). Каждый столбик представляет собой среднее по трем параллельным культурам, планки погрешностей указывают стандартное отклонение.
На фиг. 16 представлен анализ FACS для трансдуцированных PVRIG ЛПК с применением специфического антитела. Приведен процент клеток, демонстрирующих положительное окрашивание (по сравнению с трансдуцированными пустым вектором) в отношении белка.
На фиг. 17 представлен анализ FACS для трансдуцированных PVRIG (коэкспрессируемого с F4 РТК или в двухцистронном векторе с F4 РТК и NGFR) ЛПК с применением специфического антитела. Приведен процент клеток, демонстрирующих положительное окрашивание (по сравнению с трансдуцированными пустым вектором) в отношении белка.
На фиг. 17A-18B представлен анализ FACS, проводимый для РТК-трансдуцированных стимулированных ЛПК из эксперимента 1 (на фиг. 17А) и из эксперимента 2 (на фиг. 17В) с применением специфического моноклонального антитела, которое распознает внеклеточный домен бета-цепи из трансдуцированных специфических РТК. Приведено процентное содержание клеток, демонстрирующих положительное окрашивание.
На фиг. 18 показано, что экспрессия PVRIG в F4-экспрессирующих ЛПК приводит к снижению секреции IFNγ после совместного культивирования с SK-MEL23, MEL-624 и MEL-624.38 по сравнению с экспрессией пустого вектора.
На фиг. 19A-20B показано, что экспрессия PVRIG и F4 в ЛПК путем совместной трансдукции (на фиг. 19А) не влияет на секрецию IFNγ при совместном культивировании с клеточными линиями меланомы. Экспрессия PVRIG и F4 в ЛПК с применением двухцистронного вектора (на фиг. 19В) приводит к снижению секреции IFNγ после совместного культивирования с SK-MEL23, MEL-624 и MEL-624.38 по сравнению с экспрессией пустого вектора.
На фиг. 20 показано, что экспрессия PVRIG и F4 в ЛПК с применением двухцистронного вектора приводит к снижению опосредованной Т-клетками цитотоксичности после совместного культивирования с клеточными линиями меланомы.
На фиг. 21 приведена экспрессия PVRIG в 3 подгруппах с низким, неизмененным и высоким уровнями истощенных Т-клеток. Истощенные Т-клетки отбирали на основании высокого уровня экспрессии 4 маркеров: CD8A, PD-1, TIM-3 и TIGIT. Образцы с низкой экспрессией не показаны, так как ни в одном не было выявляемых уровней PVRIG.
Фиг. 22A-23B. Вестерн-блоттинг анализ эктопически экспрессируемого человеческого белка PVRIG. Экстракты цельных клеток из пулов клеток HEK293, ранее трансфицированных экспрессионной конструкцией, кодирующей человеческий PVRIG-flag (дорожка 2), или пустым вектором (дорожка 1), анализировали методом ВБ, используя анти-flag антитело (23A) или анти-PVRIG антитела (23B).
Фиг. 23. Экспрессия эктопически экспрессируемого человеческого белка PVRIG-flag на клеточной поверхности клеток HEK293, определенная анализом FACS. Анти-PVRIG pAb (Abnova) использовали для анализа клеток HEK293, стабильно экспрессирующих человеческий белок PVRIG-flag. Клетки, экспрессирующие пустой вектор, использовали в качестве отрицательного контроля. Выявление проводили с помощью козьего антимышиного PE-конъюгированного вторичного Ab и анализировали методом FACS.
Фиг. 24 изображает полноразмерную последовательность человеческого PVRIG (показаны стартовые точки в двух разных метионинах) и Fc-слитого белка PVRIG, используемых в Примерах. Сигнальный пептид выделен подчеркиванием, ВКД выделен двойным подчеркиванием, а Fc-домен выделен пунктирным подчеркиванием.
Фиг. 25 изображает последовательность человеческого полиовирусного рецептор-связанного белка 2 (PVLR2, также известного как нектин-2, CD112 или медиатор проникновения вируса герпеса B, (HVEB)), партнера по связыванию PVRIG, как показано в Пример 5. PVLR2 представляет собой человеческий гликопротеин плазматической мембраны.
Фиг. 26 изображает специфичность антитела PVRIG в отношении клеток HEK, сконструированных так, чтобы сверхэкспрессировать PVRIG. Данные представляют измерения абсолютной геометрической СИФ (гСИФ) в виде функции возрастающей концентрации антитела. Ломанная черная линия с квадратами иллюстрирует окрашивание клеток HEK hPVRIG типовым антителом против человеческого PVRIG (CPA.7.021), а сплошная черная линия с кругами иллюстрирует окрашивание родительских клеток HEK тем же антителом.
Фиг. 27 изображает оценку РНК PVRIG в различных раковых клеточных линиях методом кПЦР. Приведенные данные представляют собой относительную экспрессию РНК PVRIG в клеточных линиях в виде кратности изменения по сравнению с уровнями в клетках expi согласно оценке методом 2(-ΔΔCt).
Фиг. 28 изображает оценку РНК PVRIG в отсортированных субпопуляциях МКПК методом кПЦР. Приведенные данные представляют собой относительную экспрессию РНК PVRIG в каждой субпопуляции в виде кратности изменения по сравнению с уровнями в клетках HEK ЗФБ согласно оценке методом 2(-ΔΔCt). Три отдельных донора обозначены D47-D49. CD4 означает Т-клетки CD4, CD8 означает Т-клетки CD8, CD14 означает моноциты, а CD56 означает NK-клетки.
Фиг. 29A-30B. Фиг. 30A: РНК PVRIG оценивали в отсортированных Т-клетках CD4 (CD4) и NK-клетках (NK) в наивных и активированных условиях методом кПЦР. Т-клетки CD4 стимулировали с помощью гранул dynabead для стимуляции человеческих Т-клеток и 50 Е/мл IL-2 в течение 3 дней. NK-клетки стимулировали в 50 Е/мл IL-2 в течение 3 дней. Приведенные данные представляют собой относительную экспрессию РНК PVRIG в каждой субпопуляции в виде кратности изменения по сравнению с уровнями в клетках expi согласно оценке методом 2(-ΔΔCt). Jurkat включены в качестве положительного контроля. Три отдельных донора обозначены D47-D49. Фиг. 30B РНК PVRIG оценивали в отсортированных Т-клетках CD8 в наивных и активированных условиях методом кПЦР. Т-клетки CD8 стимулировали с помощью гранул dynabead для стимуляции человеческих Т-клеток и 100 Е/мл IL-2 в течение 3 дней. Приведенные данные представляют собой относительную экспрессию РНК PVRIG в каждой субпопуляции в виде кратности изменения по сравнению с уровнями в клетках expi согласно оценке методом 2(-ΔΔCt). Jurkat включены в качестве положительного контроля. D49, 70 и 71 обозначают трех отдельных доноров.
На фиг. 30A-31B приведены характеристики связывания PVRIG со сконструированными клеточными линиями HEK hPVRIG, родительскими клетками HEK, клетками CA46 и клетками Jurkat. HEK OE означает клетки HEK hPVRIG, HEK par означает родительские клетки HEK. В случае данных по Jurkat и CA46 сгСИФ указывает кратность разницы в геометрической СИФ для окрашивания антителом к PVRIG по сравнению с контролями. Указанная концентрация является той, при которой рассчитывали сгСИФ. Отсутствие надежной аппроксимации указывает на то, что связывающие характеристики антитела не соответствуют математическим требованиям аппроксимации. Некоторые антитела не исследовали в некоторых условиях вследствие слабых характеристик связывания, специфичности или возможности получения.
На фиг. 31A-32B приведены характеристики связывания PVRIG с первичными человеческими МКПК, клетками яванского макака со временной сверхэкспрессией и первичными МКПК яванского макака. Expi cyno OE означает клетки expi, временно трансфицированные cPVRIG, expi par означает родительские клетки expi. сгСИФ указывает кратность разницы в геометрической СИФ для окрашивания антителом к PVRIG по сравнению с контролями. Указанная концентрация является той, при которой рассчитывали сгСИФ. Некоторые антитела не исследовали в некоторых условиях вследствие слабых характеристик связывания, специфичности или возможности получения, как на фиг. 30. Кроме того, отобранные антитела были отсортированы для проведения скрининга с субпопуляциями МКПК яванского макака на основании их способности связывать временные клетки cPVRIG или функциональности. Экспрессия PVRIG в Т-клетках CD4 аналогична экспрессии, описанной в таблице для Т-клеток CD8.
На фиг. 32 приведена специфичность антитела к PVRIG в отношении клеток CA46 и Jurkat. Данные представляют измерения абсолютной геометрической СИФ (гСИФ) методом FACS в виде функции возрастающей концентрации антитела. Сплошная черная линия с треугольниками иллюстрирует окрашивание клеток CA46 антителом против человеческого PVRIG (CPA.7.021), сплошная черная линия с квадратами иллюстрирует окрашивание клеток Jurkat. OV-90 (ломанная линия с перевернутыми треугольниками) и NCI-H4411 (ломанная линия с ромбами) представляют отрицательные контроли.
На фиг. 33A-34D приведена перекрестная реактивность антитела к PVRIG в отношении временных клеток cPVRIG. Данные представляют пример антитела, которое проявляет отрицательное связывание (a-b, CPA.7.021) и положительное связывание (c-d, CPA.7.024) на временных клетках cPVRIG. Сплошные серые столбики соответствуют контрольному антителу, открытые черные столбики соответствуют представляющему интерес антителу. Клетки окрашивали каждым антителом в концентрации 5 мкг/мл.
На фиг. 34 приведена оценка РНК cPVRIG в субпопуляциях отсортированных методом кПЦР МКПК яванского макака. Данные представляют средние значения Ct от трех яванских макаков-доноров, полученные с помощью двух групп праймеров, направленных на два разных участка гена cPVRIG.
На фиг. 35A-36C приведена оценка белка cPVRIG в a) лимфоцитах CD16+ (NK-клетки), b) миелоидных клетках CD14+ CD56+ (моноциты) и c) лимфоцитах CD3+ (Т-клетки) методом FACS. Данные представляют абсолютную геометрическую СИФ, при этом сплошная черная линия соответствует фоновым уровням флуоресценции. Данные представляют один образец из панели антител против человеческого PVRIG, исследуемых для трех яванских макаков-доноров.
Фиг. 36A-37B иллюстрирует последовательности CDR для Fab, которые были определены как успешно блокирующие взаимодействие PVRIG со своим контрпартнером PVRL2, как описано в Пример 5.
Фиг. 37A-38AA изображает аминокислотные последовательности вариабельных доменов тяжелой и легкой цепей, полноразмерных тяжелой и легкой цепей и CDR вариабельных доменов тяжелой и легкой цепей для пронумерованных человеческих анти-PVRIG последовательностей CPA согласно изобретению, которые как связывают PVRIG, так и блокируют связывание PVRIG и PVLR2.
Фиг. 38A-39H изображает аминокислотные последовательности вариабельных доменов тяжелой и легкой цепей, полноразмерных тяжелой и легкой цепей и CDR вариабельных доменов тяжелой и легкой цепей для восьми человеческих анти-PVRIG последовательностей CPA согласно изобретению, которые связывают PVRIG, но не блокируют связывание PVRIG и PVLR2.
Фиг. 39A-40D изображает CDR для всех созданных последовательностей анти-PVRIG антител CPA, которые связывают PVRIG, включая те, которые не блокируют связывание PVRIG и PVLR2.
Фиг. 40A-41DD изображает вариабельные домены тяжелой и легкой цепей, а также последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3 каждого из пронумерованных антител CHA согласно изобретению, CHA.7.502, CHA.7.503, CHA.7.506, CHA.7.508, CHA.7.510, CHA.7.512, CHA.7.514, CHA.7.516, CHA.7.518, CHA.7.520.1, CHA.7.520.2, CHA.7.522, CHA.7.524, CHA.7.526, CHA.7.527, CHA.7.528, CHA.7.530, CHA.7.534, CHA.7.535, CHA.7.537, CHA.7.538.1, CHA.7.538.2, CHA.7.543, CHA.7.544, CHA.7.545, CHA.7.546, CHA.7.547, CHA.7.548, CHA.7.549 и CHA.7.550 (они включают вариабельные последовательности тяжелой и легкой цепей из мышиных последовательностей (из гибридом).
Фиг. 41 изображает результаты сортировки из Пример 11. Не отсортированы: CPA.7.029 и CPA.7.026 (отсутствие связывания с антигеном).
На фиг. 42 приведена бинарная матрица попарного блокирования («0», красный бокс) или заключения антигена в сандвич-структуру («1», зеленый бокс) для 35 анти-PVRIG mAb. MAb, перечисленные в вертикальном направлении с левой стороны матрицы, представляют собой mAb, ковалентно иммобилизованные на матрице ProteOn. MAb, перечисленные в горизонтальном направлении вверху матрицы, были аналитами, инъецируемыми с предварительно смешанным антигеном. Клон CPA.7.041 исследовали только в качестве аналита. Черными боксами обозначены четыре группы эпитопов в соответствии с вертикальными профилями блокирования mAb.
На фиг. 43 приведена древовидная схема иерархической группировки вертикальных профилей связывания каждого mAb в бинарной матрице на фиг. 42. Существует четыре группы mAb с идентичными профилями блокирования эпитопов в каждой группе. Единственной разницей между группами 1 и 2 является то, что mAb в группе 1 блокируют связывание антигена с клоном CPA.7.039, тогда как mAb в группе 2 могут заключать антиген в сандвич-структуру с CPA.7.039. Клон CPA.7.050 может заключать антиген в сандвич-структуру со всеми остальными клонами.
На фиг. 44A-45JJ приведены сенсограммы, иллюстрирующие профиль блокирования антигена для CPA.7.036 с другими иммобилизованными mAb, которые представляют репрезентативные данные для группы #1. Каждая панель представляет разные участки матричного чипа ProteOn, содержащие разные иммобилизованные mAb. Синие ответы представляют содержащие только антиген контроли. Черные ответы представляют предварительно смешанные растворы CPA.7.036 с молярным избытком антигена. Серые ответы представляют содержащие только mAb контрольные инъекции. CPA.7.36 блокирует связывание антигена с другими mAb за исключением CPA.7.050 (JJ).
На фиг. 45A-46JJ приведены сенсограммы, иллюстрирующие профиль блокирования антигена для CPA.7.034 с другими иммобилизованными mAb, которые представляют репрезентативные данные для группы #2. Каждая панель представляет разные участки матричного чипа ProteOn, содержащие разные иммобилизованные mAb. Синие ответы представляют содержащие только антиген контроли. Черные ответы представляют предварительно смешанные растворы CPA.7.34 с молярным избытком антигена. Серые ответы представляют содержащие только mAb контрольные инъекции. CPA.7.34 блокирует связывание антигена с другими mAb за исключением CPA.7.039 (DD) и CPA.7.050 (JJ).
На фиг. 46A-47JJ приведены сенсограммы, иллюстрирующие профиль блокирования антигена для CPA.7.039 с другими иммобилизованными mAb. CPA.7.039 является единственным mAb в группе #3. Каждая панель представляет разные участки матричного чипа ProteOn, содержащие разные иммобилизованные mAb. Синие ответы представляют содержащие только антиген контроли. Черные ответы представляют предварительно смешанные растворы CPA.7.039 с молярным избытком антигена. Серые ответы представляют содержащие только mAb контрольные инъекции. Панели C, F, H, J, L, N, R, S, Z, EE, GG, HH, II и JJ демонстрируют заключение антигена в сандвич-структуру.
На фиг. 47A-48JJ приведены сенсограммы, иллюстрирующие профиль блокирования антигена для CPA.7.050 с другими иммобилизованными mAb. CPA.7.050 является единственным mAb в группе #4. Каждая панель представляет разные участки матричного чипа ProteOn, содержащие разные иммобилизованные mAb. Синие ответы представляют содержащие только антиген контроли. Черные ответы представляют предварительно смешанные растворы CPA.7.50 с молярным избытком антигена. Серые ответы представляют содержащие только mAb контрольные инъекции. Только панель JJ демонстрирует блокирование антигена, то есть случай, когда CPA.7.050 инъецировали без антигена через себя само.
На фиг. 48 приведены результаты экспериментов ППР из Пример 12.
На фиг. 49A-50Q приведены сенсограммы для некоторого количества концентраций анти-PVRIG fab в супернатанте, инъецируемом через захваченный человеческий слитый белок PVRIG (черные линии). Красными линиями показана 1:1 глобальная кинетическая аппроксимация некоторого количества концентраций fab для оценки kaсс. и kдисс. взаимодействий. Буквами указан клон, приведенный в таблице 1, в которой также приведены полученные в результате константы скорости и рассчитанные значения KД.
На фиг. 50A-51C приведены сенсограммы ППР для связывания клонов CPA.7.009 (A), CPA.7.003 (B) и CPA.7.014 (C) с иммобилизованным человеческим слитым белком PVRIG. В этих примерах сенсограммы демонстрируют сложную многофазовую кинетику и, следовательно, константы скорости не подлежат достоверной оценке.
Фиг. 51A-52B изображены результаты исследований блокирования из «Дополнительного проверочного исследования 4» в Пример 5.
На фиг. 52 показано, что после алло-активации повышался уровень экспрессии PVRIG в Т-клетках CD4+, а также в Т-клетках CD8+ и вдвойне отрицательных гамма-дельта Т-клетках. Это повышение наблюдали в МКПК одного из двух исследуемых доноров.
Фиг. 53 изображает человеческие клеточные линии, исследуемые в Пример 1G.
Фиг. 54 изображает мышиные клеточные линии, исследуемые в Пример 1G.
На фиг. 55A-56C приведена экспрессия транскрипта человеческого PVRIG в различных человеческих раковых клеточных линиях. Подтверждение человеческого транскрипта в нескольких клеточных линиях проводили методом кПЦР-ОТ, используя зонд TaqMan. На гистограмме представлены данные, наблюдаемые при применении зонда TaqMan Hs04189293_g1. Значения Ct уточнены в таблице. Анализ свидетельствует о высоких уровнях транскрипта в клеточных линиях Jurkat, HUT78 и HL60 и более низких уровнях в THP1 и RPMI8226.
На фиг. 56A-57B приведена экспрессия транскрипта мышиного PVRIG в различных мышиных клеточных линиях. Подтверждение мышиного транскрипта в нескольких клеточных линиях проводили методом кПЦР-ОТ, используя зонд TaqMan. На гистограмме представлены данные, наблюдаемые при применении зонда TaqMan CC70L8H. Значения Ct уточнены в таблице. Анализ свидетельствует о высоких уровнях транскрипта в клеточных линиях NIH/3T3, Renca, SaI/N и J774A.1 и более низких уровнях в CT26 и B104-1-1.
На фиг. 57 эндогенную экспрессию белка PVRIG анализировали методом ВБ с коммерческим кроличьим поликлональным антителом против человеческого PVRIG (Sigma, кат. # HPA047497), используя экстракты цельных клеток различных клеточных линий. В качестве положительного и отрицательного контролей использовали экстракты клеток HEK293, эктопически сверхэкспрессирующих человеческий PVRIG (дорожка 2), или клеток, трансфицированных пустым вектором (дорожка 1), соответственно.
На фиг. 58 приведен анализ кПЦР-ОТ транскрипта человеческого PVRIG в клеточной линии Jurkat, трансфицированной миРНК PVRIG. Человеческую раковую клеточную линию Jurkat, трансфицированную миРНК человеческого PVRIG или рандомизированной миРНК, анализировали методом кПЦР-ОТ, используя зонд TaqMan # Hs04189293_g1 для человеческого PVRIG, и нормализовали со средним геометрическим по двум конститутивным генам, указанным в таблице ниже. Значения Ct уточнены в таблице. Указано стандартное отклонение для трех технических повторностей реакции ПЦР.
На фиг. 59 приведена мембранная экспрессия белка человеческого PVRIG в человеческой клеточной линии Jurkat, трансфицированной миРНК человеческого PVRIG. Клетки Jurkat, трансфицированные миРНК человеческого PVRIG, окрашивали моноклональным анти-PVRIG Ab Inc, CPA.7.021 (левая панель, зеленая линия) или изотипическим контрольным антителом IgG2 (левая панель, синяя линия) и Sigma Ab (правая панель, красная линия) или IgG (правая панель, синяя линия). Клетки, трансфицированные рандомизированной миРНК, окрашивали таким же анти-PVRIG (оранжевая) или изотипическим контролем (левая панель, красная линия для окрашивания mAb; правая панель, зеленая линия для Sigma Ab). После промывки клеток PE-конъюгированное козье антимышиное вторичное Ab добавляли только к Sigma Ab.
На фиг. 60 приведены обобщенные результаты открытий, описанных в данном документе, выделены клеточные линии, демонстрирующие корреляцию между данными кПЦР и FACS, подтвержденную нокдауном, КГ - конститутивный ген, + - положительный, НИ - не исследовано, X - отрицательный, НД - нокдаун.
На фиг. 61 приведены обобщенные результаты открытий, описанных в данном документе, выделены клеточные линии, демонстрирующие корреляцию между данными кПЦР и FACS, подтвержденную нокдауном. КГ - конститутивный ген, + - положительный, НИ - не исследовано, X - отрицательный, НД - нокдаун.
Фиг. 62A-63D изображает последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3 для каждого из пронумерованных антител CPA согласно изобретению, последовательности от CPA.7.001 до CPA.7.050 представляют собой человеческие последовательности (из фагового дисплея).
На фиг. 63A-64B приведены результаты скрининга из Примера 1B.
На фиг. 64 приведены специфические антитела и концентрации для окрашивания, применяемые в Пример 1I.
Фиг. 65A-66C изображает последовательности человеческих IgG1, IgG2, IgG3 и IgG4.
Фиг. 66 изображает число ВКД фрагментов человеческого PVRIG.
Фиг. 67 изображает кривую связывания для CPA.7.021, как показано в Пример 13.
На фиг. 68A-69C приведены данные выявления поверхностной экспрессии CD137 и PD-1. Т-клетки CD8+, Т-клетки CD4+ и ОИЛ активировали и отслеживали в течение времени в 4 момента времени, как описано в разделе Материалы и методы. Сначала проводили гейтинг покоящихся или активированных клеток в отношении лимфоцитов (FSC-A и SSC-A), после чего проводили гейтинг живых клеток, дополнительно проводили гейтинг для синглетов (FSC-H и FSC-A), CD4/CD8 положительных клеток и дополнительно проводили гейтинг для CD137 и PD1. Поверхностную экспрессию PD-1 (слева) и CD137 (справа) на (A) Т-клетках CD8+, (B) Т-клетках CD4+ и (C) ОИЛ в разные моменты времени нормализовали относительно изотипического контроля в течение времени активации.
На фиг. 69A-70C приведена экспрессия PVRIG в покоящихся и активированных Т-клетках CD4+ и Т-клетках CD8+. Т-клетки CD4+ и CD8+ активировали и отслеживали в течение времени в 4 момента времени, как описано в разделе Материалы и методы. Клетки окрашивали красителем в отношении жизнеспособности, затем инкубировали с анти-PVRIG и изотипическим контролем (7,5 мкг/мл) и оценивали методом проточной цитометрии. (A) Экспрессия на Т-клетках CD4+. Экспрессию PVRIG на живых покоящихся (время 0) и активированных Т-клетках CD4+ после синглетного гейтинга через 24, 48, 72 ч и 144 ч сравнивали с изотипическим контролем. (B) Экспрессия на Т-клетках CD8+. Экспрессию PVRIG на живых покоящихся (время 0) и активированных Т-клетках CD8+ после синглетного гейтинга через 24, 48, 72 ч и 144 ч сравнивали с изотипическим контролем. Показаны средние геометрические значения полученной интенсивности флуоресценции. (C) Нормализацию кратности индукции окрашивания анти-PVRIG-CPA.7.021 ab сравнивали с человеческим изотипическим IgG2 в течение времени активации.
На фиг. 70A-71C приведена экспрессия PVRIG на покоящихся или активированных ОИЛ. ОИЛ Mart1 и 209 активировали и отслеживали в течение времени в 4 момента времени, как описано в разделе Материалы и методы. Клетки окрашивали красителем в отношении жизнеспособности, затем инкубировали с анти-PVRIG и изотипическим контролем (7,5 мкг/мл) и оценивали методом проточной цитометрии. (A) Экспрессию PVRIG на живых покоящихся (время 0) и активированных ОИЛ после синглетного гейтинга через 24, 48, 72 ч и 144 ч сравнивали с изотипическим контролем. (B) Экспрессия на ОИЛ 209. Экспрессию PVRIG на живых покоящихся (время 0) и активированных ОИЛ после синглетного гейтинга через 24, 48, 72 ч и 144 ч сравнивали с изотипическим контролем. Показаны средние геометрические значения полученной интенсивности флуоресценции. (C) Нормализацию кратности индукции окрашивания анти-PVRIG-CPA.7.021 ab сравнивали с человеческим изотипическим IgG2 в течение времени активации.
На фиг. 71 приведена экспрессия PVRL2 на моноцитарных ДК. Показана экспрессия PVRL2 (треугольники с ломанной линией) в виде функции от времени (дней) по сравнению с изотипическим контролем (круги со сплошной линией). День после дифференцировки указывает на время после добавления ГМ-КСФ и IL-4 к моноцитам.
На фиг. 72A-73B приведена экспрессия PVRIG на Т-клетках CD4 и CD8 в РСКЛ. Показана экспрессия PVRIG на пролиферирующих (низкое содержание CFSE) и непролиферирующих Т-клетках (высокое содержание CFSE). Данные получены от трех отдельных доноров Т-клеток CD3 и для ряда антител к PVRIG. Содержание CFSE отложено по оси Х, а экспрессия PVRIG отложена по оси Y. 3 верхних ряда графиков рассеяния относятся к экспрессии PVRIG на Т-клетках CD4, а 3 нижних ряда относятся к экспрессии на Т-клетках CD8.
На фиг. 73A-74B приведена нормализованная экспрессия PVRIG на Т-клетках CD4 и CD8 в РСКЛ. Показана экспрессия PVRIG по сравнению с изотипическим контрольным mIgG1 от трех отдельных доноров Т-клеток CD3 среди всех анализируемых антител.
На фиг. 74A-75B показано, что антитела к PVRIG повышают пролиферацию Т-клеток в РСКЛ. Процентное содержание клеток с низким CFSE приведено для анализа РСКЛ с обработкой указанными антителами к PVRIG. На каждом графике представлен один отдельный донор Т-клеток CD3.
На фиг. 75 приведен FACS-анализ эпитопов антител к PVRIG на Т-клетках. Уровень связывания конъюгированного CPA.7.021 (полученного в фаговой кампании) указан после предварительной инкубации Т-клеток с неконъюгированными антителами к PVRIG, полученными в гибридомной кампании, а также релевантными контролями. Анализ проводили на Т-клетках с низким содержанием CFSE, полученными из РСКЛ.
На фиг. 76 приведена специфичность антитела к PVRIG в отношении клеток HEK, сконструированных так, чтобы сверхэкспрессировать PVRIG. Данные представляют измерения абсолютной геометрической СИФ (гСИФ) в виде функции возрастающей концентрации антитела. Черная ломанная линия с квадратами иллюстрирует окрашивание клеток HEK hPVRIG типовым антителом против человеческого PVRIG (CHA.7.518), а сплошная черная линия с кругами иллюстрирует окрашивание родительских клеток HEK тем же антителом.
На фиг. 77 показано, что антитела к PVRIG демонстрируют специфичность в отношении клеток Jurkat. Данные представляют измерения абсолютной геометрической СИФ (гСИФ) методом FACS в виде функции возрастающей концентрации антитела. Черная ломанная линия с квадратами иллюстрирует окрашивание клеток Jurkat антителом против человеческого PVRIG (CHA.7.518), а сплошная черная линия с кругами иллюстрирует окрашивание контрольным антителом mIgG1.
На фиг. 78A-79B приведены характеристики связывания гибридомного антитела к PVRIG со сконструированными клеточными линиями HEK hPVRIG, родительскими клетками HEK и клетками Jurkat. HEK OE означает клетки HEK hPVRIG, HEK par означает родительские клетки HEK. В случае данных по Jurkat сгСИФ указывает кратность разницы в геометрической СИФ для окрашивания антителом к PVRIG по сравнению с контролями. Указанная концентрация является той, при которой рассчитывали сгСИФ. Отсутствие связывания указывает на то, что антитело не связывается с исследуемой клеточной линией. Выделенные антитела представляют собой «четыре лучших» представляющих интерес антитела.
На фиг. 79A-80B приведены характеристики связывания гибридомного антитела к PVRIG с первичными человеческими МКПК, сверхэкспрессирующими клетками яванского макака и первичными МКПК яванского макака. Expi cyno OE означает клетки expi, временно трансфицированные cPVRIG, expi par означает родительские клетки expi. сгСИФ указывает кратность разницы в геометрической СИФ для окрашивания антителом к PVRIG по сравнению с контролями. Указанные концентрации являются теми, при которых рассчитывали сгСИФ. Не исследовано указывает на то, что антитела не исследовали вследствие отсутствия связывания с человеческими HEK hPVRIG, клетками expi cPVRIG или несоответствия требования связывания для субпопуляций МКПК. Выделенные антитела представляют собой «четыре лучших» представляющих интерес антитела.
На фиг. 80A-81B приведены обобщенные результаты блокирующей способности антител к PVRIG в конкурентном анализе на основе FACS. Указаны значения IC50 ингибирования. Отсутствие IC50 указывает на то, что эти антитела не являются блокаторами. Выделенные антитела представляют собой «четыре лучших» представляющих интерес антитела.
На фиг. 81 показаны данные подтверждения НД, проводимого в ОИЛ через 24 ч после электропорации миРНК. ОИЛ окрашивали анти-PVRIG или анти-PD-1 и анализировали методом FACS. Процентное содержание НД-популяции рассчитывали сравнению с рандомизированным контролем (SCR), окрашенным релевантным Ab.
На фиг. 82A-83C показано, что ОИЛ с НД (специфические к MART-1) совместно культивировали с клетками меланомы 624 при 1:1 Э:М в течение 18 ч и окрашивали анти-CD8a антителом, а также анти-CD137 антителом, и анализировали методом FACS. На график нанесены средние геометрические значения интенсивности флуоресценции (A). Также собирали супернатант совместной культуры и исследовали в цитометрическом матричном анализе с гранулами для Th1 Th2 Th17, чтобы выявить секретируемые цитокины. Были выявлены уровни IFNγ и TNF (B, C). Процентное действие обработки рассчитывали путем сравнения каждой обработки с рандомизированным контролем (SCR). На фигуре представлены репрезентативные данные по 2 независимым экспериментам. Обработки сравнивали, используя t-критерий Стьюдента (*P ≤ 0,05, **P ≤ 0,01) для трех параллельных образцов.
На фиг. 83A-84B показано, что ОИЛ с НД (специфические к F4 gp100) совместно культивировали с клетками меланомы 624 при 1:1 Э:М в течение 18 ч и окрашивали анти-CD8a антителом, а также анти-CD137 антителом, и анализировали методом FACS. На график нанесены средние геометрические значения интенсивности флуоресценции (A). Также собирали супернатант совместной культуры и исследовали в цитометрическом матричном анализе с гранулами для Th1 Th2 Th17, чтобы выявить секретируемые цитокины. Были выявлены уровни IFNγ (B). Процентное действие обработки рассчитывали путем сравнения каждой обработки с рандомизированным контролем (SCR). На фигуре представлены репрезентативные данные по 2 независимым экспериментам. Обработки сравнивали, используя t-критерий Стьюдента (*P ≤ 0,05, **P ≤ 0,01) для трех параллельных образцов.
На фиг. 84A-85B показано, что ОИЛ совместно культивировали с клетками меланомы 624 при 1:1 Э:М в течение 18 ч в присутствии анти-PVRIG Ab (CPA.7.021; 10 мкг/мл), анти-TIGIT (клон 10A7; 10 мкг/мл) или в комбинации. Собирали супернатант и исследовали в цитометрическом матричном анализе с гранулами для Th1 Th2 Th17, чтобы выявить секретируемые цитокины. Были выявлены уровни IFNγ (A) и TNF (B). Обработки сравнивали, используя t-критерий Стьюдента (*P ≤ 0,05, **P ≤ 0,01) для трех параллельных образцов.
На фиг. 85A-86F показано, что MART-1 или 209 ОИЛ совместно культивировали с клетками меланомы 624 при 1:1 Э:М в течение 18 ч в присутствии анти-PVRIG Ab (CPA.7.021; 10 мкг/мл), анти-DNAM1 (клон DX11; 10 мкг/мл) или в комбинации. Собирали супернатант и исследовали в цитометрическом матричном анализе с гранулами для Th1 Th2 Th17, чтобы выявить секретируемые цитокины. Были выявлены уровни IFNγ (A, D) и TNF (B,E). ОИЛ окрашивали в отношении поверхностной экспрессии CD137 (C, F).
На фиг. 86A-87B показано, что ОИЛ (F4) совместно культивировали с клетками меланомы 624 при 1:1 Э:М в течение 18 ч в присутствии анти-PVRIG Ab (CPA.7.021; 10 мкг/мл), анти-TIGIT (клон 10A7; 10 мкг/мл), анти-PD1 (mAb 1B8, Merck; 10 мкг/мл) или в комбинации. Собирали супернатант и исследовали в цитометрическом матричном анализе с гранулами для Th1 Th2 Th17, чтобы выявить секретируемые цитокины. Были выявлены уровни IFNγ (A) и TNF (B).
Фиг. 87A-88I I изображает четыре гуманизированные последовательности для каждого из CHA.7.518, CHA.7.524, CHA.7.530, CHA.7.538_1 и CHA.7.538_2. Следует отметить, что легкая цепь CHA.7.538_2 является такой же, как и в случае CHA.7.538_1. «H1» каждой представляет собой «замену CDR» без изменений в человеческой каркасной области. В последующих последовательностях изменения в каркасных областях выделены более крупным жирным шрифтом. Последовательности CDR выделены жирным. Определения CDR соответствуют AbM с веб-страницы www.bioinf.org.uk/abs/. Человеческие последовательности зародышевой линии и соединительные последовательности получены из IMGT®, международной иммуногенетической® информационной системы, www.imgt.org (основатель и директор: Marie-Paule Lefranc, Montpellier, France). Нумерация остатков является последовательной (seq) или соответствует Chothia с веб-страницы www.bioinf.org.uk/abs/ (AbM). «b» обозначает утопленную боковую цепь; «p» обозначает частично утопленную; «i» обозначает боковую цепь на поверхности раздела между доменами VH и VL. Различия в последовательностях между человеческими и мышиными зародышевыми линиями отмечены звездочкой (*). Потенциальные дополнительные мутации в каркасных областях приведены под последовательностью. Потенциальные изменения в последовательностях CDR приведены под каждой последовательностью CDR, как отмечено на фигуре (# замены, связанные с деамидированием: Q/S/A; могут предотвращать деамидирование аспарагина (N). @ замены, связанные с окислением триптофана: Y/F/H; могут предотвращать окисление триптофана; @ замены, связанные с окислением метионина: L/F/A).
Фиг. 88A-E изображает сравнение гуманизированных последовательностей пяти CHA-антител.
Фиг. 89 изображает схемы комбинирования CHA-антител с гуманизированными VH и VL CHA с фиг. 88 и фиг. 89. «chimVH» и «chimVL» представляют мышиные вариабельные последовательности тяжелой и легкой цепей, присоединенные к константному домену человеческого IgG.
На фиг. 91 приведены характеристики связывания гибридомного антитела к PVRIG с первичными человеческими МКПК, сверхэкспрессирующими клетками яванского макака и первичными МКПК яванского макака. Expi cyno OE означает клетки expi, временно трансфицированные cPVRIG, expi par означает родительские клетки expi. сгСИФ указывает кратность разницы в геометрической СИФ для окрашивания антителом к PVRIG по сравнению с контролями. Указанные концентрации являются теми, при которых рассчитывали сгСИФ. «Не исследовано» указывает на то, что антитела не исследовали вследствие отсутствия связывания с человеческими HEK hPVRIG, клетками expi cPVRIG или несоответствия требования связывания для субпопуляций МКПК. Выделенные антитела представляют собой четыре антитела, для которых проводили гуманизацию (смотрите фиг. 90).
На фиг. 90 приведены обобщенные результаты блокирующей способности антител к PVRIG в конкурентном анализе на основе FACS. Указаны значения IC50 ингибирования. Отсутствие IC50 указывает на то, что эти антитела не являются блокаторами. Выделенные антитела представляют собой четыре антитела, для которых проводили гуманизацию (смотрите фиг. 90).
На фиг. 91A-93C изображено влияние антител к PVRIG на блокирование взаимодействия между PVRIG и PVRL2. (a-b) Данные представляют изменения в абсолютной гСИФ, соответствующие изменениям в связывании растворимого PVRIG с клетками HEK при добавлении четырех антител к PVRIG для разрушения взаимодействия. Также указаны значения IC50 для каждого антитела в каждом анализе. A) Данные демонстрируют разрушение связи растворимого PVRIG с клетками HEK в случае предварительной инкубации антител с антигеном B) Данные демонстрируют разрушение связи растворимого PVRIG с клетками HEK в случае добавления антител одновременно с антигеном. C) Данные демонстрируют изменения в абсолютной гСИФ, соответствующие изменениям в связывании растворимого PVRL2 Fc с клетками HEK hPVRIG в случае добавления четырех антител к PVRIG для разрушения взаимодействия. Указаны значения IC50 для каждого антитела. НО означает не определено.
На фиг. 92A-94H изображена экспрессия рецепторов и лигандов NK-клеток на клетках Reh. Показана экспрессия рецепторов NK-клеток, таких как a) PVRIG, b) DNAM-1, c) TIGIT. Показана экспрессия лигандов рецепторов NK-клеток, таких как d) PVR, e) PVRL2, f) ULBP2/5/6, g) ULBP3 и h) MICA/B. Сплошные серые столбики соответствуют изотипическим контролям, а черные столбики соответствуют представляющему интерес антителу.
На фиг. 93 изображено влияние антител к PVRIG на усиление опосредованной NK-клетками цитотоксичности против клеток Reh. Исследовали влияние 5 мкг/мл CPA.7.002 (a), CPA.7.005 (b), CPA.7.021 (a-c) и CPA.7.050 (c) в анализе NK-клеточной цитотоксичности против клеток Reh, в котором число NK-клеток титровали против постоянного числа клеток Reh. d) Исследовали влияние изменения концентрации CPA.7.002 и CPA.7.021 на опосредованную NK-клетками цитотоксичность с постоянным соотношением NK-клеток и клеток Reh (5:1). DNAM-1 (e) и TIGIT (f) исследовали в анализе в условиях, приведенных на панелях a-c.
На фиг. 94A-96H изображена экспрессия рецепторов и лигандов NK-клеток на клетках MOLM-13. Показана экспрессия рецепторов NK-клеток, таких как a) PVRIG, b) DNAM-1, c) TIGIT. Показана экспрессия лигандов рецепторов NK-клеток, таких как d) PVR, e) PVRL2, f) ULBP2/5/6, g) ULBP3 и h) MICA/B. Сплошные серые столбики соответствуют изотипическим контролям, а черные столбики соответствуют представляющему интерес антителу.
На фиг. 95A-97B изображено влияние антител к PVRIG на усиление опосредованной NK-клетками цитотоксичности против клеток MOLM-13. a) Исследовали влияние 5 мкг/мл CPA.7.002, CPA.7.005 и CPA.7.021 в анализе NK-клеточной цитотоксичности против клеток MOLM-13, в котором число NK-клеток титровали против постоянного числа клеток MOLM-13. b) TIGIT исследовали аналогично панели a.
На фиг. 96 приведены обобщенные результаты блокирующей способности антител к PVRIG в клеточном биохимическом анализе. Указаны варианты и последовательность анализа, а также IC50 ингибирования. (P) указывает на вариант анализа, когда антитела к PVRIG предварительно инкубировали с антигеном PVRIG перед добавлением к клеткам HEK. (NP) указывает на одновременное добавление антител к PVRIG и антигена PVRIG к клеткам HEK. Повышенное связывание указывает на то, что связывание PVRL2 Fc с клетками HEK hPVRIG было усилено, а не ингибировалось.
Фиг. 97. Обобщенные данные по активности отобранных антител к PVRIG в анализе цитотоксичности NK-клеток против клеток Reh и MOLM-13. Кратность изменения в цитотоксичности по сравнению с контролем рассчитывали, деля абсолютный уровень уничтожения (%) в условиях наличия антитела к PVRIG на абсолютный уровень уничтожения (%) с контрольным антителом. Кратность изменения рассчитывали для соотношения эффектора и мишени 5:1.
На фиг. 98 изображено выравнивание последовательностей ортологов PVRIG. Выровненные последовательности внеклеточного домена PVRIG человека, яванского макака, мармозетки и макака-резуса. Различия между человеком и яванским макаком выделены желтым.
На фиг. 99 изображено связывание анти-PVRIG с вариантами PVRIG человека, яванского макака и гибрида яванский макак/человек. Показано связывание антител с PVRIG дикого типа яванского макака (●), H61R PVRIG яванского макака, P67S PVRIG яванского макака, L95R/T97I PVRIG яванского макака и PVRIG дикого типа человека. Сигналы ELISA нанесены на график в виде функции концентрации антитела.
На фиг. 100 изображена корреляция группы эпитопов и перекрестной реактивности с яванским макаков антител против человеческого PVRIG.
На фиг. 101A-103BX изображено число использованных в изобретении последовательностей.
Подробное описание сущности изобретения
Введение
Рак можно рассматривать как неспособность организма пациента распознавать и уничтожать раковые клетки. Во многих случаях эти трансформированные (например, раковые) клетки противодействуют иммунологическому надзору. Существуют природные механизмы регуляции, которые ограничивают активацию Т-клеток в организме для предотвращения неограниченной активности Т-клеток, что может быть использовано раковыми клетками для избегания или подавления иммунного ответа. Восстановление способности иммунных эффекторных клеток, в особенности Т-клеток, распознавать и уничтожать рак является целью иммунотерапии. Область иммуно-онкологии, иногда называемой «иммунотерапией», быстро развивается, и недавно были одобрены несколько ингибиторных антител против Т-клеточных контрольных точек, такие как Ервой, Кейтруда и Опдиво. Эти антитела в общем случае называются «ингибиторами контрольных точек», так как они блокируют обычно отрицательные регуляторы Т-клеточного иммунитета. В общем случае понятно, что для организации оптимального антигенспецифического иммунного ответа можно использовать множество иммуномодулирующих сигналов, как ко-стимуляторных, так и ко-ингибиторных. В общем случае эти антитела связываются с белками ингибиторов контрольных точек, такими как CTLA-4 и PD-1, которые в обычных условиях предотвращают или подавляют активацию цитотоксических Т-клеток (ЦТЛ). Ингибируя белок контрольной точки, например, посредством применения антител, которые связывают эти белки, можно добиться повышенного Т-клеточного ответа против опухолей. Это означает, что эти белки раковых контрольных точек подавляют иммунный ответ; когда белки блокируют, например, применяя антитела против белка контрольной точки, происходит активация иммунной системы, приводя к иммунной стимуляции, что приводит к лечению патологических состояний, таких как рак и инфекционные заболевания.
Настоящее изобретение относится к применению антител к родственному рецептору человеческого полиовируса белку, содержащему домен иммуноглобулина, или «PVRIG», иногда также называемому в данном документе «белком PV». PVRIG экспрессируется на клеточной поверхности NK- и Т-клеток и имеет несколько сходных черт с другими известными иммунными контрольными точками.
С целью определения новых иммунных контрольных точек использовали компьютерные алгоритмы для анализа человеческого генома. Были определены гены, которые прогнозировано относятся к белкам клеточной поверхности, имеют домен Ig и экспрессируются на иммунных клетках в опухолевом микроокружении, в частности на опухоль-инфильтрирующих лимфоцитах (ОИЛ), которые предположительно являются рецепторами. Были определены белки, имеющие один домен IgV и имеющие внутриклеточный ITIM-подобный мотив, что позволяет предположить, что они действуют как иммунная контрольная точка и оказывают ингибиторное действие на Т-клетки и/или NK-клетки. После компьютерного определения были проведены различные проверочные эксперименты, включая: исследования экспрессии, демонстрирующие, что PVRIG экспрессируется на лимфоцитах и на лимфоцитах в опухолевом микроокружении и оказывает ингибиторное действие на Т-клетки и NK-клетки (что было продемонстрировано как экспериментами с нокдауном, так и с антителами, направленными на PVRIG). Было определено/подтверждено, что PVRL2 является контрпартнером PVRIG. Были созданы антитела, которые связываются с PVRIG, а затем среди них была определена субпопуляция, которая как связывается с PVRIG, так и блокирует взаимодействие PVRIG и PVLR2.
Соответственно, когда происходит связывание PVRIG его лигандом (PVRL2), это вызывает ингибиторный сигнал, который действует на ослабление иммунного ответа NK- и Т-клеток против клетки-мишени (т.е. аналогично PD-1/PDL1). Блокирование связывания PVRL2 с PVRIG выключает этот ингибиторный сигнал PVRIG и в результате модулирует иммунный ответ NK- и Т-клеток. Применение антитела против PVRIG, которое блокирует связывание с PVRL2, представляет собой терапевтический подход, который может усилить уничтожение раковых клеток NK- и Т-клетками. Были созданы блокирующие антитела, которые связывают PVRIG и блокируют связывание его лиганда PVRL2.
Как показано в разделе Примеров, экспрессия PVRIG положительно коррелировала с экспрессией PD-1 - известным белком иммунной контрольной точки. Кроме того, было показано, что внесение PVRIG (в виде слитого белка внеклеточного домена (ВКД)) ингибирует активацию Т-клеток и, таким образом, применение анти-PVRIG антител приводит к активации Т-клеток. Соответственно, анти-PVRIG антитела можно применять для лечения патологических состояний, при которых необходима активация Т-клеток или NK-клеток, таких как рак.
Функциональное действие PVRIG-блокирующих антител на NK- и Т-клетки можно оценить in vitro (и в некоторых случаях in vivo, как более подробно описано ниже) путем определения изменений в следующих параметрах: пролиферация, высвобождение цитокинов и маркеры клеточной поверхности. В случае NK-клеток повышение клеточной пролиферации, цитотоксичности (способности уничтожать клетки-мишени, определяемой по повышению количества CD107a, гранзимов и экспрессии перфорина или путем прямого определения уничтожения клеток-мишеней), выработки цитокинов (например, IFN-γ и TNF) и экспрессии рецепторов клеточной поверхности (например, CD25) свидетельствует об иммунной модуляции, например, усиленном уничтожении раковых клеток. В случае Т-клеток повышение пролиферации, повышение экспрессии маркеров активации клеточной поверхности (например, CD25, CD69, CD137 и PD1), цитотоксичности (способности уничтожать клетки-мишени) и выработки цитокинов (например, IL-2, IL-4, IL-6, IFNγ, TNF-a, IL-10, IL-17A) свидетельствуют об иммунной модуляции, например, усиленном уничтожении раковых клеток.
Соответственно, в настоящем изобретении предложены антитела, включая антигенсвязывающие домены, которые связываются с человеческим PVRIG, и способы активировать Т-клетки и/или NK-клетки для лечения заболеваний, таких как рак и инфекционные заболевания, а также других патологических состояний, при которых повышение иммунной активности приводит к лечению.
Белки PVRIG
В настоящем изобретении предложены антитела, которые специфически связываются с белком PVRIG. «Белок» в этом контексте взаимозаменяемо употребляется с «полипептидом» и также включает пептиды. В настоящем изобретении предложены антитела, которые специфически связываются с белками PVRIG. PVRIG представляет собой трансмембранный доменный белок длиной 326 аминокислот с сигнальным пептидом (простирающимся от аминокислоты 1 до 40), внеклеточным доменом (простирающимся от аминокислоты 41 до 171), трансмембранным доменом (простирающимся от аминокислоты 172 до 190) и цитоплазматическим доменом (простирающимся от аминокислоты 191 до 326). Полноразмерный человеческий белок PVRIG приведен на фиг. 25. Существует два метионина, которые могут быть стартовыми кодонами, но зрелые белки являются идентичными.
Соответственно, в контексте данного документа термин «PVRIG» или «белок PVRIG» или «полипептид PVRIG», необязательно, может включать любой такой белок, или его варианты, конъюгаты или фрагменты, включая, но не ограничиваясь этим, известный или дикого типа PVRIG, описанный в данном документе, а также любые встречающиеся в природе сплайс-варианты, аминокислотные варианты или изоформы и, в частности, ВКД-фрагмент PVRIG. Термин «растворимая» форма PVRIG также взаимозаменяемо употребляется с терминами «растворимый эктодомен (ЭКД)» или «эктодомен», или «внеклеточный домен (ВКД)», а также «фрагменты полипептидов PVRIG», которые в широком смысле могут относиться к одному или более из следующих необязательных полипептидов:
Белки PVRIG содержат домен иммуноглобулина (Ig) во внеклеточном домене, который представляет собой PVR-подобный структурный домен Ig. PVR-подобный структурный домен Ig может отвечать за связывание функционального контрпартнера по аналогии с другими представителями семейства В7. PVR-подобный структурный домен Ig внеклеточного домена содержит одну дисульфидную связь, образуемую между внутридоменными остатками цистеина, что типично для этой структуры и может быть важно для структурной функции. Эти цистеины расположены в остатках 22 и 93 (или 94). В одном варианте реализации изобретения предложен растворимый фрагмент PVRIG, который можно использовать для исследования антител к PVRIG.
В определение белков PVRIG включены ВКД-фрагменты PVRIG. Необязательно, ВКД-фрагменты PVRIG также относятся к любой из полипептидных последовательностей, приведенных на фиг. 67, которые с достаточной вероятностью содержат функциональные области белка PVRIG. Эта вероятность основана на системном анализе набора белковых комплексов с разрешенными 3D-структурами, которые включают комплексы белков Ig (например, PDB ID 1i85, которые описывают комплекс CTLA4 и CD86). Брали межмолекулярные контактные остатки из каждой «совместной структуры» из каждого PDB и проектировали на последовательность PVRIG. Определили несколько областей с кластерами взаимодействующих остатков, подтверждаемых несколькими контактными картами, и синтезировали их в виде серии пептидов, которые с достаточной вероятностью имитируют структуру интактного полноразмерного белка и, следовательно, модулируют одно или более действий PVRIG на иммунитет и определенные типы иммунных клеток. В соответствии по меньшей мере с некоторыми вариантами реализации изобретения ВКД-фрагменты PVRIG, представленные полипептидными последовательностями, приведенными на фиг. 67, расположены следующим образом (по сравнению с человеческим ВКД PVRIG с фиг. 25, начиная с первой аминокислоты ВКД): фрагмент А PVRIG расположен в позициях от 46 до 66; фрагмент В PVRIG расположен в позициях от 46 до 79; фрагмент С PVRIG расположен в позициях от 63 до 79; фрагмент D PVRIG расположен в позициях от 91 до 106; фрагмент Е PVRIG расположен в позициях от 91 до 114; фрагмент F PVRIG расположен в позициях от 11 до 25; фрагмент G PVRIG расположен в позициях от 3 до 24; фрагмент Н PVRIG расположен в позициях от 18 до 36; фрагмент PVRIG I расположен в позициях от 29 до 52; фрагмент J PVRIG расположен в позициях 73-98.
Как отмечалось в данном документе и более подробно описано ниже, анти-PVRIG антитела (включая антигенсвязывающие фрагменты), которые связываются с PVRIG и предотвращают активацию посредством PVRL2 (например, наиболее обычно путем блокирования взаимодействия PVRIG и PVLR2), применяют для усиления активации Т-клеток и/или NK-клеток и могут применяться для лечения заболеваний, таких как рак и патогенные инфекции.
Антитела
Соответственно, в изобретении предложены анти-PVRIG антитела. PVRIG, также называемый родственным рецептору человеческого полиовируса белком, содержащим домен иммуноглобулина, Q6DKI7 или C7orf15, относится к аминокислотным и нуклеотидным последовательностям, приведенным в RefSeq под кодом доступа NP_076975, приведенным на фиг. 25. Антитела согласно изобретению специфичны в отношении внеклеточного домена PVRIG, как более подробно описано в данном документе.
Как обсуждается ниже, термин «антитело» употребляется в широком смысле. Антитела, применимые в настоящем изобретении, могут быть представлены большим числом форматов, описанных в данном документе, включая традиционные антитела, а также производные, фрагменты и миметики антител, описанные ниже. В общем случае термин «антитело» включает любой полипептид, который содержит по меньшей мере один антигенсвязывающий домен, как более подробно описано ниже. Антитела могут быть поликлональными, моноклональными, ксеногенными, аллогенными, сингенными или являться их модифицированными формами, как описано в данном документе, при этом моноклональные антитела, в частности, применимы во многих вариантах реализации изобретения. В некоторых вариантах реализации антитела согласно изобретению специфически или в значительной степени специфически связываются с молекулами PVRIG. В контексте данного документа термины «моноклональные антитела» и «композиция моноклональных антител» относятся к популяции молекул антител, которая содержит только один тип антигенсвязывающего участка, способного к иммунной реакции с конкретным эпитопом антигена, тогда как «поликлональные антитела» и «композиция поликлональных антител» относятся к популяции молекул антител, которая содержит несколько типов антигенсвязывающих участков, способных взаимодействовать с конкретным антигеном. Композиция моноклональных антител, как правило, демонстрирует единичную аффинность связывания в отношении конкретного антигена, с которым происходит иммунная реакция.
Структурные единицы традиционного полноразмерного антитела, как правило, образуют тетрамер. Каждый тетрамер, как правило, состоит из двух идентичных пар полипептидных цепей, при этом в каждой паре содержится одна «легкая» (как правило, имеющая молекулярную массу около 25 кДа) и одна «тяжелая» цепь (как правило, имеющая молекулярную массу около 50-70 кДа). Человеческие легкие цепи классифицируют как каппа и лямбда легкие цепи. Настоящее изобретение относится к классу IgG, который имеет несколько подклассов, включая, но не ограничиваясь этим, IgG1, IgG2, IgG3 и IgG4. Таким образом, в контексте данного документа, «изотип» обозначает любой из подклассов иммуноглобулинов, определяемый химическими и антигенными характеристиками константных областей. Хотя типовые антитела, обозначаемые в данном документе «CPA», имеют в основе константные области тяжелой цепи IgG1, как показано на фиг. 38, анти-PVRIG антитела согласно изобретению включают те, которые имеют последовательности IgG2, IgG3 и IgG4 или их комбинации. Например, как известно в данной области техники, разные изотипы IgG имеют разные эффекторные функции, которые могу быть или могут не быть необходимыми. Соответственно, в CPA-антителах согласно изобретению константные домены IgG1 могут быть заменены константными доменами IgG2, IgG3 или IgG4 (как проиллюстрировано на фиг. 66), в частности, во многих ситуациях применимы IgG2 и IgG4, например, для облегчения получения или в случае необходимости эффекторной функции, что необходимо в некоторых ситуациях.
В случае пронумерованных антител, обозначаемых CHA, они представляют собой мышиные антитела, созданные в гибридомах (обозначение «Н»), и, таким образом, в общем случае они являются гуманизированными, как известно в данной области техники, в общем случае в каркасных областях (от F1 до F4 для каждой из вариабельных областей тяжелой и легкой цепей), а затем привитыми в константные домены тяжелой и легкой цепей человеческих IgG1, IgG2, IgG3 или IgG4 (как проиллюстрировано на фиг. 66), в частности, применим IgG4, как более подробно описано ниже.
Амино-концевая часть каждой цепи содержит вариабельную область от около 100 до 110 или более аминокислот, отвечающую, в первую очередь, за распознавание антигена, в общем случае называемую в данной области техники и в данном документе «Fv-доменом» или «Fv-областью». В вариабельной области сгруппированы три петли для каждого из V-доменов тяжелой цепи и легкой цепи, образуя антигенсвязывающий участок. Каждая из петель называется определяющей комплементарность областью (далее называемой в данном документе «CDR»), в которой наибольшее значение имеет вариация аминокислотной последовательности. «Вариабельный» относится к тому факту, что определенные сегменты вариабельной области существенно отличаются по последовательности среди антител. Вариабельность в пределах вариабельной области распределена неравномерно. Скорее, V-области состоят из относительно инвариантных участков, называемых каркасными областями (FR) из 15-30 аминокислот, разделенных более короткими областями сильной вариабельности, называемыми «гипервариабельными областями».
Каждая VH и VL состоит из трех гипервариабельных областей («определяющих комплементарность областей», «CDR») и четырех FR, расположенных от амино-конца в карбокси-концу в следующем порядке: FR1-CDR1-FR2-CDR2-FR3-CDR3-FR4.
Гипервариабельная область в общем случае включает аминокислотные остатки приблизительно от аминокислотных остатков 24-34 (LCDR1; «L» обозначает легкую цепь), 50-56 (LCDR2) и 89-97 (LCDR3) в вариабельной области легкой цепи и приблизительно 31-35B (HCDR1; «H» обозначает тяжелую цепь), 50-65 (HCDR2) и 95-102 (HCDR3) в вариабельной области тяжелой цепи, хотя иногда нумерация может быть немного сдвинута, как известно специалистам в данной области техники; Kabat et al., SEQUENCES OF PROTEINS OF IMMUNOLOGICAL INTEREST, 5 th Ed. Public Health Service, National Institutes of Health, Bethesda, Md. (1991), и/или остатки, образующие гипервариабельную петлю (например, остатки 26-32 (LCDR1), 50-52 (LCDR2) и 91-96 (LCDR3) в вариабельной области легкой цепи и 26-32 (HCDR1), 53-55 (HCDR2) и 96-101 (HCDR3) в вариабельной области тяжелой цепи; Chothia and Lesk (1987) J. Mol. Biol. 196:901-917. Конкретные CDR согласно изобретению описаны ниже и приведены на фиг. 39.
Карбокси-концевая часть каждой цепи определяет константную область, отвечающую, в первую очередь, за эффекторную функцию. Kabat et al. собрали большое количество первичных последовательностей вариабельных областей тяжелых цепей и легких цепей. На основании степени консервативности последовательностей они классифицировали отдельные первичные последовательности на CDR и каркасные области и составили их список (смотрите SEQUENCES OF IMMUNOLOGICAL INTEREST, 5 th edition, NIH publication, No. 91-3242, E. A. Kabat et al., в полном объеме включенную посредством ссылки).
В подклассе иммуноглобулинов IgG тяжелая цепь состоит из нескольких доменов иммуноглобулина. Под «доменом иммуноглобулина (Ig)» в данном документе подразумевается область иммуноглобулина, имеющая характерную третичную структуру. Интерес для настоящего изобретения представляют домены тяжелой цепи, включая константные домены тяжелой цепи (СН) и шарнирные домены. В контексте антител IgG каждый из изотипов IgG содержит три СН-области. Соответственно, «CH»-домены в контексте IgG определяются следующим образом: «CH1» относится к позициям 118-220 в соответствии с индексом EU согласно Kabat. «CH2» относится к позициям 237-340 в соответствии с индексом EU согласно Kabat, а «CH3» относится к позициям 341-447 в соответствии с индексом EU согласно Kabat.
Соответственно, в изобретении предложены вариабельные домены тяжелой цепи, вариабельные домены легкой цепи, константные домены тяжелой цепи, константные домены легкой цепи и Fc-домены для указанных в данном документе применений. В контексте данного документа под «вариабельной областью» подразумевается область иммуноглобулина, которая содержит один или более доменов Ig, кодируемые в значительной степени любым из генов Vκ или Vλ и/или VH, которые образуют генные локусы каппа, лямбда и тяжелой цепей иммуноглобулина соответственно. Соответственно, вариабельный домен тяжелой цепи содержит vhFR1-vhCDR1-vhFR2-vhCDR2-vhFR3-vhCDR3-vhFR4, а вариабельный домен легкой цепи содержит vlFR1-vlCDR1-vlFR2-vlCDR2-vlFR3-vlCDR3-vlFR4. Под «константной областью тяжелой цепи» в данном документе подразумевается часть CH1-шарнир-CH2-CH3 антитела. В контексте данного документа под «Fc» или «Fc-областью» или «Fc-доменом» подразумевается полипептид, содержащий константную область антитела, за исключением первого домена константной области иммуноглобулина и, в некоторых случаях, части шарнирной области. Таким образом, Fc относится к последним двум доменам константной области иммуноглобулинов IgA, IgD и IgG, последним трем доменам константной области иммуноглобулинов IgE и IgM и гибкой шарнирной области в N-концевом положении относительно этих доменов. В случае IgA и IgM Fc может включать цепь J. В случае IgG Fc-домен содержит домены иммуноглобулина Cγ2 и Cγ3 (Cγ2 и Cγ3) и нижнюю шарнирную область между Cγ1 (Cγ1) и Cγ2 (Cγ2). Хотя границы Fc-области могут варьироваться, Fc-область тяжелой цепи человеческого IgG по определению обычно включает остатки C226 или P230 в направлении карбокси-конца, причем нумерация соответствует индексу EU согласно Kabat. В некоторых вариантах реализации изобретения, как более подробно описано ниже, в Fc-области проведены аминокислотные модификации, например, для изменения связывания с одним или более FcγR-рецепторами или с FcRn-рецептором.
Таким образом, в контексте данного документа «Fc-вариант» или «вариантный Fc» обозначает белок, содержащий аминокислотную модификацию в Fc-домене. Fc-варианты согласно настоящему изобретению определены в соответствии с входящими в их состав аминокислотными модификациями. Таким образом, например, N434S или 434S представляет собой Fc-вариант с заменой серина в позиции 434 по сравнению с родительским Fc-полипептидом, причем нумерация соответствует индексу EU. Аналогично, M428L/N434S определяет Fc-вариант с заменами M428L и N434S по сравнению с родительским Fc-полипептидом. Идентичность аминокислоты ДТ может быть неустановлена, в случае чего вышеуказанный вариант называется 428L/434S. Следует отметить, что порядок, в котором проводят замены, является произвольным, что означает, например, что 428L/434S представляет такой же Fc-вариант, что и M428L/N434S, и т.д. В случае всех позиций, обсуждаемых в настоящем изобретении, которые относятся к антителу, если не указано иное, нумерация аминокислотных позиций соответствует индексу EU.
В контексте данного документа под «Fab» или «Fab-областью» подразумевается полипептид, который содержит домены VH, CH1, VL и CL иммуноглобулина. Fab может относиться к этой области в случае ее выделения или к этой области в контексте полноразмерного антитела, фрагмента антитела или слитого белка Fab. В контексте данного документа под «Fv» или «фрагментом Fv» или «Fv-областью» подразумевается полипептид, который содержит домены VL и VH одного антитела. Как понятно специалистам в данной области техники, они в общем случае состоят из двух цепей.
В тексте описания настоящего изобретения в общем случае используется система нумерации IMTG или система нумерации Kabat при определении остатка в вариабельном домене (приблизительно остатки 1-107 вариабельной области легкой цепи и остатки 1-113 вариабельной области тяжелой цепи) (например, Kabat et al., выше (1991)). Нумерацию EU согласно Kabat в общем случае используют для константных доменов и/или Fc-доменов.
CDR участвуют в образовании антигенсвязывающего, или, конкретнее, эпитопсвязывающего участка антител. «Эпитоп» относится к детерминанте, которая взаимодействует с конкретным антигенсвязывающим участком в вариабельной области молекулы антитела, известным как паратоп. Эпитопы представляют собой группы молекул, таких как аминокислоты или боковые цепи сахаров, и обычно обладают определенными структурными характеристиками, а также определенными зарядовыми характеристиками. Один антиген может содержать более одного эпитопа.
Эпитоп может содержать аминокислотные остатки, напрямую вовлеченные в связывание (также называемые иммунодоминантным компонентом эпитопа), и другие аминокислотные остатки, которые напрямую не вовлечены в связывание, такие как аминокислотные остатки, которые эффективно блокируются специфическим антигенсвязывающим пептидом; другими словами, аминокислотный остаток находится в отпечатке специфического антигенсвязывающего пептида.
Эпитопы могут быть конформационными или линейными. Конформационный эпитоп образуется вследствие пространственного совмещения аминокислот из разных сегментов линейной полипептидной цепи. Линейный эпитоп образуется смежными аминокислотными остатками в полипептидной цепи. Конформационные и неконформационные эпитопы можно различать по отсутствию связывания с первыми, но не последними, в присутствии денатурирующих растворителей.
Эпитоп, как правило, содержит по меньшей мере 3 и чаще по меньшей мере 5 или 8-10 аминокислот в уникальной пространственной конформации. Антитела, которые распознают один эпитоп, можно проверять с помощью простого иммуноанализа, демонстрирующего способность антитела блокировать связывание другого антитела с антигеном-мишенью, например, «сортировки». Конкретные сортировочные группы описаны ниже.
В определение «антитела» включена «антигенсвязывающая область» антитела (взаимозаменяемо употребляемая с «антигенсвязывающим фрагментом», «фрагментом антитела» и «производным антитела»). В контексте изобретения это означает, что антитело согласно изобретению имеет минимальные функциональные требования, состоящие в том, что оно должно связываться с антигеном PVRIG. Как понятно специалистам в данной области техники, существует большое количество фрагментов и производных антител, которые сохраняют способность связывать антиген и при этом имеют альтернативные структуры, включая, но не ограничиваясь этим, (i) фрагмент Fab, состоящий из доменов VL, VH, CL и CH1, (ii) фрагмент Fd, состоящий из доменов VH и CH1, (iii) фрагмент F(ab')2, бивалентный фрагмент, содержащий два связанных фрагмента Fab (vii) одноцепочечные молекулы Fv (scFv), в которых домен VH и домен VL связаны пептидным линкером, который делает возможной ассоциацию двух доменов с образованием антигенсвязывающего участка (Bird et al., 1988, Science 242:423-426, Huston et al., 1988, Proc. Natl. Acad. Sci. U.S.A. 85:5879-5883, в полном объеме включенная посредством ссылки), (iv) «диатела» или «триатела», мультивалентные или мультиспецифические фрагменты, сконструированные путем генного слияния (Tomlinson et. al., 2000, Methods Enzymol. 326:461-479; WO94/13804; Holliger et al., 1993, Proc. Natl. Acad. Sci. U.S.A. 90:6444-6448, в полном объеме включенные посредством ссылки), (v) «доменные антитела» или «dAb» (иногда называемые «одиночным вариабельным доменом иммуноглобулина», включая одиночные вариабельные домены антител от других видов, такие как V-HH dAb грызунов (например, как раскрыто в WO 00/29004), акул-нянек и верблюдовых, (vi) SMIP (низкомолекулярные иммунофармацевтические средства), верблюжьи антитела, нанотела и IgNAR.
Кроме того, антитело или его антигенсвязывающая часть (антигенсвязывающий фрагмент, фрагмент антитела, часть антитела) может быть частью более крупных иммуноадгезивных молекул (иногда также называемых «слитыми белками»), образуемые путем ковалентной или нековалентной ассоциации антитела или части антитела с одним или более другими белками или пептидами. Примеры иммуноадгезивных молекул включают использование центральной области стрептавидина для получения тетрамерной молекулы scFv и использование остатка цистеина, маркерного пептида и С-концевой полигистидиновой метки для получения бивалентных или биотинилированных молекул scFv. Части антител, такие как фрагменты Fab и F(ab')2, можно получать из цельных антител, используя традиционные методики, такие как расщепление соответственно папаином или пепсином цельных антител. Кроме того, антитела, части антител и иммуноадгезивные молекулы можно получать, используя описанные в данном документе стандартные технологии рекомбинантных ДНК.
В общем случае анти-PVRIG антитела согласно изобретению являются рекомбинантными. В контексте данного документа термин «рекомбинантный», в широком смысле употребляемый по отношению к продукту, например, клетке или нуклеиновой кислоте, белку или вектору, указывает на то, что клетка, нуклеиновая кислота, белок или вектор были модифицированы путем внесения гетерологичной нуклеиновой кислоты или белка или изменения нативной нуклеиновой кислоты или белка, или что клетка получена из модифицированной подобным образом клетки. Таким образом, например, рекомбинантные клетки экспрессируют гены, которые отсутствуют в нативной (нерекомбинантной) форме клетки, или экспрессируют нативные гены, которые в ином случае характеризуются аномальной экспрессией, недостатком экспрессии или вообще не экспрессируются.
В контексте данного документа термин «рекомбинантное антитело» включает все антитела, которые были получены, экспрессированы, созданы или выделены рекомбинантными способами, такие как (a) антитела, выделенные из организма животного (например, мыши), которое является трансгенным или трансхромосомным в отношении генов человеческого иммуноглобулина, или полученной из него гибридомы (дополнительно описано ниже), (b) антитела, выделенные из клетки-хозяина, трансформированной так, чтобы экспрессировать человеческое антитело, например, из трансфектомы, (c) антитела, выделенные из рекомбинантной, комбинаторной библиотеки человеческих антител, и (d) антитела, которые были получены, экспрессированы, созданы или выделены любыми другими способами, которые включают сплайсинг генных последовательностей человеческого иммуноглобулина или других последовательностей ДНК. Такие рекомбинантные человеческие антитела имеют вариабельные области, в которых каркасные области и области CDR получены из последовательностей иммуноглобулина человеческой зародышевой линии. Однако в определенных вариантах реализации изобретения такие рекомбинантные человеческие антитела можно подвергать in vitro мутагенезу (или, в случае использования животного, трансгенного в отношении последовательностей человеческого Ig, in vivo соматическому мутагенезу) и, таким образом, аминокислотные последовательности областей VH и VL рекомбинантных антител представляют собой последовательности, которые, хотя получены из и родственны последовательностям VH и VL человеческой зародышевой линии, могут отсутствовать в природном репертуаре антител человеческой зародышевой линии in vivo.
Опциональное конструирование антител
Антитела согласно изобретению могут быть модифицированы или сконструированы с целью изменения аминокислотных последовательностей аминокислотными заменами.
Под «аминокислотной заменой» или «заменой» в данном документе подразумевается замещение аминокислоты в конкретной позиции в родительской полипептидной последовательности другой аминокислотой. В частности, в некоторых вариантах реализации изобретения, проводят замену на аминокислоту, которая в природе не встречается в этой конкретной позиции, как в пределах организма, так и в любом организме. Например, замена E272Y относится к вариантному полипептиду, в данном случае к Fc-варианту, в котором глутаминовая кислота в позиции 272 замещена тирозином. Для ясности, белок, который был сконструирован так, чтобы изменить кодирующую нуклеотидную последовательность, но не изменить стартовую аминокислоту (например, путем замены CGG (кодирующего аргинин) на CGA (тоже кодирующего аргинин) для повышения уровней экспрессии организма-хозяина), не является «аминокислотной заменой»; что означает, что, несмотря на создание нового гена, кодирующего тот же самый белок, если белок содержит такую же аминокислоту в конкретной позиции, которая была изначально, это не является аминокислотной заменой.
Как обсуждается в данном документе, аминокислотные замены можно проводить для изменения аффинности CDR в отношении белка PVRIG (включая как повышение, так и снижение связывания, как более подробно описано ниже), а также для изменения дополнительных функциональных свойств антител. Например, антитела можно сконструировать так, чтобы они содержали модификации в Fc-области, как правило, для изменения одного или более функциональных свойств антитела, таких как сывороточное время полужизни, фиксация комплемента, связывание Fc-рецептора и/или антигензависимая клеточная цитотоксичность. Кроме того, антитело в соответствии по меньшей мере с некоторыми вариантами реализации изобретения может быть химически модифицировано (например, к антителу могут быть присоединены один или более химических компонентов) или быть модифицировано для изменения состояния его гликозилирования, снова для изменения одного или более функциональных свойств антитела. Такие варианты реализации изобретения дополнительно описаны ниже. Нумерация остатков в Fc-области соответствует индексу EU согласно Kabat.
В одном варианте реализации изобретения шарнирная область CH1 модифицирована так, чтобы изменить число остатков цистеина в шарнирной области, например, увеличить или уменьшить. Этот подход дополнительно описан в патенте США № 5677425 авторства Bodmer et al. Число остатков цистеина в шарнирной области CH1 изменяют, например, чтобы облегчить сборку легких и тяжелых цепей или повысить или снизить стабильность антитела.
В другом варианте реализации изобретения Fc-шарнирная область антитела мутирована, чтобы уменьшить биологическое время полужизни антитела. В частности, одна или более аминокислотных мутаций внесены в область поверхности раздела доменов CH2-CH3 фрагмента Fc-шарнирная область так, что антитело характеризуется нарушением связывания протеина А Staphylococcyl (SpA) по сравнению со связыванием SpA нативного Fc-шарнирного домена. Этот подход более подробно описан в патенте США № 6165745 авторства Ward et al.
В некоторых вариантах реализации изобретения аминокислотные замены можно проводить в Fc-области, в общем случае для изменения связывания с FcγR-рецепторами. В контексте данного документа под «Fc-гамма-рецептором», «FcγR» или «FcgammaR» подразумевается любой представитель семейства белков, которые связывают Fc-область антитела IgG и кодируются геном FcγR. Для людей это семейство включает, но не ограничивается этим, FcγRI (CD64), включая изоформы FcγRIa, FcγRIb и FcγRIc; FcγRII (CD32), включая изоформы FcγRIIa (включая аллотипы H131 и R131), FcγRIIb (включая FcγRIIb-1 и FcγRIIb-2), и FcγRIIc; и FcγRIII (CD16), включая изоформы FcγRIIIa (включая аллотипы V158 и F158) и FcγRIIIb (включая аллотипы FcγRIIIb-NA1 и FcγRIIIb-NA2) (Jefferis et al., 2002, Immunol Lett 82:57-65, в полном объеме включенная посредством ссылки), а также любые неоткрытые человеческие FcγR или изоформы или аллотипы FcγR. FcγR может происходить из любого организма, включая, но не ограничиваясь этим, людей, мышей, крыс, кроликов и обезьян. Мышиные FcγR включают, но не ограничиваются этим, FcγRI (CD64), FcγRII (CD32), FcγRIII-1 (CD16) и FcγRIII-2 (CD16-2), а также любые неоткрытые мышиные FcγR или изоформы или аллотипы FcγR.
Существует большое количество подходящих Fc-замен, которые можно проводить для изменения связывания с одним или более FcγR-рецепторами. Могут подходить замены, которые приводят к повышению связывания, а также снижению связывания. Например, известно, что повышение связывания с FcγRIIIa в общем случае приводит к повышенной АЗКЦ (антителозависимой клеточноопосредованной цитотоксичности; клеточноопосредованной реакции, в которой неспецифические цитотоксические клетки, которые экспрессируют FcγR, распознают связанное антитело на клетке-мишени и впоследствии вызывают лизис клетки-мишени). Аналогично, снижение связывания с FcγRIIb (ингибиторным рецептором) также может быть полезно в некоторых обстоятельствах. Аминокислотные замены, применимые в настоящем изобретении, включают приведенные в патенте США № 11/124620 (в частности, фиг. 41) и патенте США № 6737056, которые оба явным образом и в полном объеме включены в данный документ посредством ссылки, и, в частности, для раскрытых в данном документе вариантов. Конкретные применимые варианты включают, но не ограничиваются этим, 236A, 239D, 239E, 332E, 332D, 239D/332E, 267D, 267E, 328F, 267E/328F, 236A/332E, 239D/332E/330Y, 239D, 332E/330L, 299T и 297N.
Кроме того, антитела согласно изобретению модифицируют для увеличения биологического времени полужизни. Возможны различные подходы. Например, можно вносить одну или более следующих мутаций: T252L, T254S, T256F, описанных в патенте США № 6277375 авторства Ward. В альтернативном варианте чтобы увеличить биологическое время полужизни, можно изменить CH1 или CL область антитела так, чтобы она содержала эпитоп связывания рецептора реутилизации, полученный из двух петель СН2-домена Fc-области IgG, как описано в патентах США № 5869046 и 6121022 авторства Presta et al. Дополнительные мутации для увеличения биологического времени полужизни раскрыты в патентах США № 8883973, 6737056 и 7371826, и включают 428L, 434A, 434S и 428L/434S.
В других вариантах реализации изобретения изменяют Fc-область путем замены по меньшей мере одного аминокислотного остатка другим аминокислотным остатком для изменения эффекторных функций антитела. Например, один или более аминокислотных остатков, выбранных из аминокислотных остатков 234, 235, 236, 237, 297, 318, 320 и 322, могут быть заменены другим аминокислотным остатком так, чтобы антитело имело измененную аффинность в отношении эффекторного лиганда, но сохраняло антигенсвязывающую способность родительского антитела. Эффекторный лиганд, в отношении которого меняют аффинность, может представлять собой, например, Fc-рецептор или компонент C1 комплемента. Этот подход более подробно описан в патентах США № 5624821 и 5648260, оба авторства Winter et al.
В другом примере одна или более аминокислот, выбранных из аминокислотных остатков 329, 331 и 322, могут быть заменены другим аминокислотным остатком так, чтобы антитело характеризовалось измененным связыванием C1q и/или сниженной или отсутствующей комплементзависимой цитотоксичностью (КЗЦ). Этот подход более подробно описан в патенте США № 6194551 авторства Idusogie et al.
В другом примере изменяют один или более аминокислотных остатков в пределах аминокислотных позиций 231 и 239, чтобы таким образом изменить способность антитела фиксировать комплемент. Этот подход дополнительно описан в публикации согласно РСТ WO 94/29351 авторства Bodmer et al.
В другом примере модифицируют Fc-область, чтобы повысить способность антитела опосредовать антителозависимую клеточную цитотоксичность (АЗКЦ) и/или повысить аффинность антитела в отношении Fcγ-рецептора путем модификации одной или более аминокислот в следующих позициях: 238, 239, 248, 249, 252, 254, 255, 256, 258, 265, 267, 268, 269, 270, 272, 276, 278, 280, 283, 285, 286, 289, 290, 292, 293, 294, 295, 296, 298, 301, 303, 305, 307, 309, 312, 315, 320, 322, 324, 326, 327, 329, 330, 331, 333, 334, 335, 337, 338, 340, 360, 373, 376, 378, 382, 388, 389, 398, 414, 416, 419, 430, 434, 435, 437, 438 или 439. Этот подход дополнительно описан в публикации согласно РСТ WO 00/42072 авторства Presta. Кроме того, были картированы связывающие участки IgG1 в отношении FcγRI, FcγRII, FcγRIII и FcRn и описаны варианты с улучшенным связыванием (смотрите Shields, R. L. et al. (2001) J. Biol. Chem. 276:6591-6604). Показано, что конкретные мутации в позициях 256, 290, 298, 333, 334 и 339 улучшают связывание с FcγRIII. Кроме того, было показано, что следующие комбинации мутантов улучшают связывание FcγRIII: T256A/S298A, S298A/E333A, S298A/K224A и S298A/E333A/K334A. Более того, такие мутации, как M252Y/S254T/T256E или M428L/N434S, улучшают связывание с FcRn и увеличивают время полужизни антитела в циркуляции (смотрите Chan CA and Carter PJ (2010) Nature Rev Immunol 10:301-316).
В другом варианте реализации изобретения антитело можно модифицировать, чтобы прекратить in vivo обмен Fab-плеча. В частности, этот процесс включает обмен полумолекул IgG4 (одна тяжелая цепь плюс одна легкая цепь) с другими антителами IgG4, что эффективно приводит к получению функционально моновалентных биспецифических антител. Мутации в шарнирной области и константных доменах тяжелой цепи могут прекратить этот обмен (смотрите Aalberse, RC, Schuurman J., 2002, Immunology 105:9-19).
В другом варианте реализации изобретения модифицируют состояние гликозилирования антитела. Например, можно получать агликозилированное антитело (т.е. антитело с отсутствием гликозилирования). Состояние гликозилирования можно изменять, например, для повышения аффинности антитела в отношении антигена или снижения эффекторной функции, такой как АЗКЦ. Такие углеводные модификации можно осуществлять, например, изменяя один или более участков гликозилирования в последовательности антитела, например, N297. Например, можно провести одну или более аминокислотных замен, которые приводят к удалению одного или более участков гликозилирования в каркасной области вариабельной области, чтобы тем самым устранить гликозилирование в этом участке.
В дополнительном или альтернативном варианте можно получить антитело с измененным типом гликозилирования, такое как гипофукозилированное антитело, содержащее сниженное количество фукозильных остатков, или антитело, содержащее биссекторные структуры GlcNac. Было продемонстрировано, что такие измененные профили гликозилирования повышают АЗКЦ-способность антител. Такие углеводные модификации можно осуществлять, например, путем экспрессии антитела в клетке-хозяине с измененным аппаратом гликозилирования. Клетки с измененным аппаратом гликозилирования были описаны в данной области техники и могут использоваться в качестве клеток-хозяев, предназначенных для экспрессии рекомбинантных антител в соответствии по меньшей мере с некоторыми вариантами реализации изобретения, чтобы таким образом получить антитело с измененным состоянием гликозилирования. Например, в клеточных линиях Ms704, Ms705 и Ms709 отсутствует ген фукозилтрансферазы, FUT8 (α (1,6) фукозилтрансфераза), поэтому в антителах, экспрессируемых в клеточных линиях Ms704, Ms705 и Ms709 отсутствует фукоза в углеводах. Клеточные линии Ms704, Ms705 и Ms709 FUT8 создают путем нацеленного разрушения гена FUT8 в клетках CHO/DG44, используя два вектора замещения (смотрите публикацию патента США № 20040110704 авторства Yamane et al. и Yamane-Ohnuki et al. (2004) Biotechnol Bioeng 87:614-22). В качестве другого примера в EP 1176195 авторства Hanai et al. описана клеточная линия с функционально разрушенным геном FUT8, который кодирует фукозилтрансферазу, поэтому антитела, экспрессируемые в такой клеточной линии демонстрируют гипофукозилирование вследствие снижения количества или устранения связанного с α-1,6-связью фермента. В Hanai et al. также описаны клеточные линии, имеющие низкую ферментативную активность в отношении добавления фукозы к N-ацетилглюкозамину, который связывается с Fc-областью антитела, или не имеющие ферментативной активности, например, как клеточная линия крысиной меланомы YB2/0 (ATCC CRL 1662). В публикации согласно РСТ WO 03/035835 авторства Presta описана вариантная клеточная линия CHO, клетки Lec13 со сниженной способностью присоединять фукозу к Asn(297)-связанным углеводам, что также приводит к гипофукозилированию антител, экспрессируемых в таких клетках-хозяевах (также смотрите Shields, R. L. et al. (2002) J. Biol. Chem. 277:26733-26740). В публикации согласно РСТ WO 99/54342 авторства Umana et al. описаны клеточные линии, сконструированные для экспрессии модифицирующих гликопротеины гликозилтрансфераз (например, β(1,4)-N-ацетилглюкозаминилтрансферазы III (GnTIII)), поэтому антитела, экспрессируемые в сконструированных клеточных линиях демонстрируют повышенное содержание биссекторных структур GlcNac, что приводит к повышенной АЗКЦ-активности антител (смотрите также Umana et al. (1999) Nat. Biotech. 17:176-180). В альтернативном варианте можно отщеплять остатки фукозы антитела с помощью фермента фукозидазы. Например, α-L-фукозидаза удаляет фукозильные остатки из антител (Tarentino, A. L. et al. (1975) Biochem. 14:5516-23).
Другой подразумеваемой изобретением модификацией антител является пэгилирование или добавление других водорастворимых компонентов, как правило, полимеров, например, с целью увеличения времени полужизни. Антитело можно пэгилировать, например, чтобы увеличить биологическое (например, сывороточное) время полужизни антитела. Чтобы пэгилировать антитело, антитело или его фрагмент, как правило, приводят в реакцию с полиэтиленгликолем (ПЭГ), таким как реактивный сложный эфир или альдегидное производное ПЭГ, в условиях, в которых одна или более групп ПЭГ присоединяются к антителу или фрагменту антитела. Предпочтительно пэгилирование проводят посредством реакции ацилирования или реакции алкилирования с реактивной молекулой ПЭГ (или аналогичным реактивным водорастворимым полимером). В контексте данного документа подразумевается, что термин «полиэтиленгликоль» включает любые формы ПЭГ, которые используют для дериватизации других белков, такие как моно(C1-C10) алкокси- или арилокси-полиэтиленгликоль или полиэтиленгликоль-малеимид. В определенных вариантах реализации изобретения предназначенное для пэгилирования антитело представляет собой агликозилированное антитело. Способы пэгилирования белков известны в данной области техники и могут применяться к антителам в соответствии по меньшей мере с некоторыми вариантами реализации изобретения. Смотрите, например, EP 0 154 316 авторства Nishimura et al. и EP 0 401 384 авторства Ishikawa et al.
Кроме замен, проводимых для изменения аффинности связывания с FcγR и/или FcRn и/или повышения in vivo сывороточного времени полужизни, можно проводить дополнительные модификации, как более подробно описано ниже.
В некоторых случаях проводят созревание аффинности. Аминокислотные модификации в CDR иногда называются «созреванием аффинности». Антитело с «созревшей аффинностью» представляет собой антитело, имеющее одно или более изменение(ий) в одной или более CDR, которые приводят к улучшению аффинности антитела в отношении антигена по сравнению с родительским антителом, которое не обладает таким(и) изменением(ями). В некоторых случаях, хоть и редко, может возникать необходимость снизить аффинность антитела в отношении его антигена, но в общем случае это не является предпочтительным.
В некоторых вариантах реализации изобретения одну или более аминокислотных модификаций проводят в одной или более CDR антител VISG1 согласно изобретению. В общем случае в каждой CDR замещают только 1 или 2 или 3 аминокислоты, а в пределах группы CDR делают не более 1, 2, 3, 4, 5, 6, 7, 8 9 или 10 изменений. Однако следует понимать, что любую комбинацию из отсутствия замен, 1, 2 или 3 замен в любой CDR можно независимо и необязательно комбинировать с любой другой заменой.
Созревание аффинности можно проводить для того, чтобы повысить аффинность связывания антитела в отношении антигена PVRIG по меньшей мере на от около 10% до 50-100-150% или более или в 1-5 раз по сравнению с «родительским» антителом. Предпочтительные антитела с созревшей аффинностью имеют наномолярную или даже пикомолярную аффинность в отношении антигена PVRIG. Антитела с созревшей аффинностью получают с помощью известных процедур. Смотрите, например, Marks et al., 1992, Biotechnology 10:779-783, в которой описано созревание аффинности, осуществляемое путем перетасовки доменов вариабельной области тяжелой цепи (VH) и вариабельной области легкой цепи (VL). Случайный мутагенез остатков CDR и/или каркасных областей описан в: Barbas, et al. 1994, Proc. Nat. Acad. Sci, USA 91:3809-3813; Shier et al., 1995, Gene 169:147-155; Yelton et al., 1995, J. Immunol. 155:1994-2004; Jackson et al., 1995, J. Immunol. 154(7):3310-9; и Hawkins et al, 1992, J. Mol. Biol. 226:889-896, например.
В альтернативном варианте в одной или более CDR антител согласно изобретению можно проводить аминокислотные модификации, которые являются «молчащими», например, которые существенно не меняют аффинность антитела в отношении антигена. Их можно проводить по большому количеству причин, включая оптимизацию экспрессии (что можно осуществлять для нуклеиновых кислот, кодирующих антитела согласно изобретению).
Таким образом, в определение CDR и антител согласно изобретению входят вариантные CDR и антитела; что означает, что антитела согласно изобретению могут содержать аминокислотные модификации в одной или более CDR пронумерованных антител согласно изобретению. Кроме того, как описано ниже, аминокислотные модификации также можно независимо и необязательно проводить в любой области за пределами CDR, включая каркасные и константные области.
Антитела к PVRIG
В настоящем изобретении предложены анти-PVRIG антитела. (Для удобства «анти-PVRIG антитела» и «антитела к PVRIG» употребляются взаимозаменяемо). Анти-PVRIG антитела согласно изобретению специфически связываются с человеческим PVRIG и предпочтительно ВКД человеческого VISG1, как проиллюстрировано на фиг. 24
Специфическое связывание в отношении PVRIG или эпитопа PVRIG может демонстрироваться, например, антителом, имеющим KД, составляющую по меньшей мере около 10-4 M, по меньшей мере около 10-5 M, по меньшей мере около 10-6 M, по меньшей мере около 10-7 M, по меньшей мере около 10-8 M, по меньшей мере около 10-9 M, в альтернативном варианте по меньшей мере около 10-10 M, по меньшей мере около 10-11 M, по меньшей мере около 10-12 M, или более, где KД относится к скорости диссоциации конкретного взаимодействия антитело-антиген. Как правило, антитело, которое специфически связывает антиген, имеет KД в отношении антигена или эпитопа PVRIG, в 20, 50, 100, 500, 1000, 5000, 10000 или более раз превышающую значение для контрольной молекулы.
Однако, как показано в Примерах, для оптимального связывания с PVRIG, экспрессируемым на поверхности NK- и Т-клеток, антитела предпочтительно имеют KД менее 50 нМ и наиболее предпочтительно менее 1 нМ, при этом значения менее 0,1 нМ и менее 1 пМ и 0,1 пМ применимы в способах согласно изобретению.
Также специфическое связывание в отношении конкретного антигена или эпитопа может демонстрироваться, например, антителом, имеющим KA или Kа в отношении антигена или эпитопа PVRIG, в 20, 50, 100, 500, 1000, 5000, 10000 или более раз превышающую значение для контроля, где KA или Ka относится к скорости ассоциации конкретного взаимодействия антитело-антиген.
В некоторых вариантах реализации изобретения анти-PVRIG антитела согласно изобретению связываются с человеческим PVRIG с KД 100 нМ или менее, 50 нМ или менее, 10 нМ или менее или 1 нМ или менее (что означает более высокую аффинность связывания), или 1 пМ или менее, при этом KД определяют известными способами, например, методом поверхностного плазмонного резонанса (ППР, например, анализ Biacore), ELISA, KINEXA и наиболее предпочтительно ППР при 25ο или 37οC.
Специфические анти-PVRIG антитела
В изобретении предложены антигенсвязывающие домены, включая полноразмерные антитела, которые содержат некоторое количество специфических пронумерованных групп из 6 CDR.
Антитела, описанные в данном документе, обозначают следующим образом. Антитела имеют ссылочные номера, например, «CPA.7.013». Это представляет комбинацию вариабельной области тяжелой и вариабельной области легкой цепей, как проиллюстрировано на фиг. 37 и фиг. 38, например. «CPA.7.013.VH» относится к части вариабельной области тяжелой цепи CPA.7.013, тогда как «CPA.7.013.VL» представляет вариабельную область легкой цепи. «CPA.7.013.vhCDR1», «CPA.7.013.vhCDR2», «CPA.7.013.vhCDR3», «CPA.7.013.vlCDR1», «CPA.7.013.vlCDR2» и «CPA.7.013.vlCDR3» относятся к CDR. «CPA.7.013.HC» относится к целой тяжелой цепи (например, вариабельному и константному домену) этой молекулы, а «CPA.7.013.LC» относится к целой легкой цепи (например, вариабельному и константному домену) той же молекулы. «CPA.7.013.H1» относится к полноразмерному антителу, содержащему домены вариабельных областей тяжелой и легкой цепей, включая константный домен человеческого IgG1 (следовательно, последовательности H1; IgG1, IgG2, IgG3 и IgG4 приведены на фиг. 65). Соответственно, «CPA.7.013.H2» представляет вариабельные домены CPA.7.013, связанные с человеческим IgG2. «CPA.7.013.H3» представляет вариабельные домены CPA.7.013, связанные с человеческим IgG3, а «CPA.7.013.H4» представляет вариабельные домены CPA.7.013, связанные с человеческим IgG4.
В изобретении дополнительно предложены вариабельные домены тяжелых и легких цепей, а также полноразмерные тяжелые и легкие цепи.
Во многих вариантах реализации антитела согласно изобретению являются человеческими (полученными из фага) и блокируют связывание PVRIG и PVLR2. Как показано на фиг. 51, CPA-антитела, которые как связывают, так и блокируют взаимодействие рецептор-лиганд, представляют собой приведенные ниже, причем также указаны их компоненты:
CPA.7.001, CPA.7.001.VH, CPA.7.001.VL, CPA.7.001.HC, CPA.7.001.LC и CPA.7.001.H1, CPA.7.001.H2, CPA.7.001.H3, CPA.7.001.H4; CPA.7.001.vhCDR1, CPA.7.001.vhCDR2, CPA.7.001.vhCDR3, CPA.7.001.vlCDR1, CPA.7.001.vlCDR2 и CPA.7.001.vlCDR3;
CPA.7.003, CPA.7.003.VH, CPA.7.003.VL, CPA.7.003.HC, CPA.7.003.LC, CPA.7.003.H1, CPA.7.003.H2, CPA.7.003.H3, CPA.7.003.H4; CPA.7.003.vhCDR1, CPA.7.003.vhCDR2, CPA.7.003.vhCDR3, CPA.7.003.vlCDR1, CPA.7.003.vlCDR2 и CPA.7.003.vlCDR3;
CPA.7.004, CPA.7.004.VH, CPA.7.004.VL, CPA.7.004.HC, CPA.7.004.LC, CPA.7.004.H1, CPA.7.004.H2, CPA.7.004.H3 CPA.7.004.H4; CPA.7.004.vhCDR1, CPA.7.004.vhCDR2, CPA.7.004.vhCDR3, CPA.7.004.vlCDR1, CPA.7.004.vlCDR2 и CPA.7.004.vlCDR3;
CPA.7.006, CPA.7.006.VH, CPA.7.006.VL, CPA.7.006.HC, CPA.7.006.LC, CPA.7.006.H1, CPA.7.006.H2, CPA.7.006.H3 CPA.7.006.H4; CPA.7.006.vhCDR1, CPA.7.006.vhCDR2, CPA.7.006.vhCDR3, CPA.7.006.vlCDR1, CPA.7.006.vlCDR2 и CPA.7.006.vlCDR3;
CPA.7.008, CPA.7.008.VH, CPA.7.008.VL, CPA.7.008.HC, CPA.7.008.LC, CPA.7.008.H1, CPA.7.008.H2, CPA.7.008.H3 CPA.7.008.H4; CPA.7.008.vhCDR1, CPA.7.008.vhCDR2, CPA.7.008.vhCDR3, CPA.7.008.vlCDR1, CPA.7.008.vlCDR2 и CPA.7.008.vlCDR3;
CPA.7.009, CPA.7.009.VH, CPA.7.009.VL, CPA.7.009.HC, CPA.7.009.LC, CPA.7.009.H1, CPA.7.009.H2, CPA.7.009.H3 CPA.7.009.H4; CPA.7.009.vhCDR1, CPA.7.009.vhCDR2, CPA.7.009.vhCDR3, CPA.7.009.vlCDR1, CPA.7.009.vlCDR2 и CPA.7.009.vlCDR3;
CPA.7.010, CPA.7.010.VH, CPA.7.010.VL, CPA.7.010.HC, CPA.7.010.LC, CPA.7.010.H1, CPA.7.010.H2, CPA.7.010.H3 CPA.7.010.H4; CPA.7.010.vhCDR1, CPA.7.010.vhCDR2, CPA.7.010.vhCDR3, CPA.7.010.vlCDR1, CPA.7.010.vlCDR2 и CPA.7.010.vlCDR3;
CPA.7.011, CPA.7.011.VH, CPA.7.011.VL, CPA.7.011.HC, CPA.7.011.LC, CPA.7.011.H1, CPA.7.011.H2, CPA.7.011.H3 CPA.7.011.H4; CPA.7.011.vhCDR1, CPA.7.011.vhCDR2, CPA.7.011.vhCDR3, CPA.7.011.vlCDR1, CPA.7.011.vlCDR2 и CPA.7.011.vlCDR3;
CPA.7.012, CPA.7.012.VH, CPA.7.012.VL, CPA.7.012.HC, CPA.7.012.LC, CPA.7.012.H1, CPA.7.012.H2, CPA.7.012.H3 CPA.7.012.H4; CPA.7.012.vhCDR1, CPA.7.012.vhCDR2, CPA.7.012.vhCDR3, CPA.7.012.vlCDR1, CPA.7.012.vlCDR2 и CPA.7.012.vlCDR3;
CPA.7.013, CPA.7.013.VH, CPA.7.013.VL, CPA.7.013.HC, CPA.7.013.LC, CPA.7.013.H1, CPA.7.013.H2, CPA.7.013.H3 CPA.7.013.H4; CPA.7.013.vhCDR1, CPA.7.013.vhCDR2, CPA.7.013.vhCDR3, CPA.7.013.vlCDR1, CPA.7.013.vlCDR2 и CPA.7.013.vlCDR3;
CPA.7.014, CPA.7.014.VH, CPA.7.014.VL, CPA.7.014.HC, CPA.7.014.LC, CPA.7.014.H1, CPA.7.014.H2, CPA.7.014.H3 CPA.7.014.H4; CPA.7.014.vhCDR1, CPA.7.014.vhCDR2, CPA.7.014.vhCDR3, CPA.7.014.vlCDR1, CPA.7.014.vlCDR2 и CPA.7.014.vlCDR3;
CPA.7.015, CPA.7.015.VH, CPA.7.015.VL, CPA.7.015.HC, CPA.7.015.LC, CPA.7.015.H1, CPA.7.015.H2, CPA.7.015.H3 CPA.7.015.H4; CPA.7.015.vhCDR1, CPA.7.015.vhCDR2, CPA.7.015.vhCDR3, CPA.7.015.vlCDR1, CPA.7.015.vlCDR2 и CPA.7.015.vlCDR3;
CPA.7.017, CPA.7.017.VH, CPA.7.017.VL, CPA.7.017.HC, CPA.7.017.LC, CPA.7.017H1, CPA.7.017.H2, CPA.7.017.H3 CPA.7.017.H4; CPA.7.017.vhCDR1, CPA.7.000171.vhCDR2, CPA.7.017.vhCDR3, CPA.7.017.vlCDR1, CPA.7.017.vlCDR2 и CPA.7.017.vlCDR3;
CPA.7.018, CPA.7.018.VH, CPA.7.018.VL, CPA.7.018.HC, CPA.7.018.LC, CPA.7.018.H1, CPA.7.018.H2, CPA.7.018.H3 CPA.7.018.H4; CPA.7.017.vhCDR1, CPA.7.017.vhCDR2, CPA.7.017.vhCDR3, CPA.7.017.vlCDR1, CPA.7.017.vlCDR2 и CPA.7.017.vlCDR3;
CPA.7.019, CPA.7.019.VH, CPA.7.019.VL, CPA.7.019.HC, CPA.7.019.LC, CPA.7.019.H1, CPA.7.019.H2, CPA.7.019.H3 CPA.7.019.H4; CPA.7.019.vhCDR1, CPA.7.019.vhCDR2, CPA.7.019.vhCDR3, CPA.7.019.vlCDR1, CPA.7.019.vlCDR2 и CPA.7.019.vlCDR3;
CPA.7.021, CPA.7.021.VH, CPA.7.021.VL, CPA.7.021.HC, CPA.7.021.LC, CPA.7.021.H1, CPA.7.021.H2, CPA.7.021.H3 CPA.7.021.H4; CPA.7.021.vhCDR1, CPA.7.021.vhCDR2, CPA.7.021.vhCDR3, CPA.7.021.vlCDR1, CPA.7.021.vlCDR2 и CPA.7.021.vlCDR3;
CPA.7.022, CPA.7.022.VH, CPA.7.022.VL, CPA.7.022.HC, CPA.7.022.LC, CPA.7.022.H1, CPA.7.022.H2, CPA.7.022.H3 CPA.7.022.H4; CPA.7.022.vhCDR1, CPA.7.022.vhCDR2, CPA.7.002201.vhCDR3, CPA.7.022.vlCDR1, CPA.7.022.vlCDR2 и CPA.7.022.vlCDR3;
CPA.7.023, CPA.7.023.VH, CPA.7.023.VL, CPA.7.023.HC, CPA.7.023.LC, CPA.7.023.H1, CPA.7.023.H2, CPA.7.023.H3 CPA.7.023.H4; CPA.7.023.vhCDR1, CPA.7.023.vhCDR2, CPA.7.023.vhCDR3, CPA.7.023.vlCDR1, CPA.7.023.vlCDR2 и CPA.7.023.vlCDR3;
CPA.7.024, CPA.7.024.VH, CPA.7.024.VL, CPA.7.024.HC, CPA.7.024.LC, CPA.7.024.H1, CPA.7.024.H2, CPA.7.024.H3 CPA.7.024.H4; CPA.7.024.vhCDR1, CPA.7.024.vhCDR2, CPA.7.024.vhCDR3, CPA.7.024.vlCDR1, CPA.7.024.vlCDR2 и CPA.7.024.vlCDR3;
CPA.7.033, CPA.7.033.VH, CPA.7.033.VL, CPA.7.033.HC, CPA.7.033.LC, CPA.7.033.H1, CPA.7.033.H2, CPA.7.033.H3 CPA.7.033.H4; CPA.7.033.vhCDR1, CPA.7.033.vhCDR2, CPA.7.033.vhCDR3, CPA.7.033.vlCDR1, CPA.7.033.vlCDR2 и CPA.7.033.vlCDR3;
CPA.7.034, CPA.7.034.VH, CPA.7.034.VL, CPA.7.034.HC, CPA.7.034.LC, CPA.7.034.H1, CPA.7.034.H2, CPA.7.034.H3 CPA.7.034.H4; CPA.7.034.vhCDR1, CPA.7.034.vhCDR2, CPA.7.034.vhCDR3, CPA.7.034.vlCDR1, CPA.7.034.vlCDR2 и CPA.7.034.vlCDR3;
CPA.7.036, CPA.7.036.VH, CPA.7.036.VL, CPA.7.036.HC, CPA.7.036.LC, CPA.7.036.H1, CPA.7.036.H2, CPA.7.036.H3 CPA.7.036.H4; CPA.7.036.vhCDR1, CPA.7.036.vhCDR2, CPA.7.036.vhCDR3, CPA.7.036.vlCDR1, CPA.7.036.vlCDR2 и CPA.7.036.vlCDR3;
CPA.7.040, CPA.7.040.VH, CPA.7.040.VL, CPA.7.040.HC, CPA.7.040.LC, CPA.7.040.H1, CPA.7.040.H2, CPA.7.040.H3 and CPA.7.040.H4; CPA.7.040.vhCDR1, CPA.7.040.vhCDR2, CPA.7.040.vhCDR3, CPA.7.040.vlCDR1, CPA.7.040.vlCDR2 и CPA.7.040.vlCDR3;
CPA.7.046, CPA.7.046.VH, CPA.7.046.VL, CPA.7.046.HC, CPA.7.046.LC, CPA.7.046.H1, CPA.7.046.H2, CPA.7.046.H3 CPA.7.046.H4; CPA.7.046.vhCDR1, CPA.7.046.vhCDR2, CPA.7.046.vhCDR3, CPA.7.046.vlCDR1, CPA.7.046.vlCDR2 и CPA.7.046.vlCDR3;
CPA.7.047, CPA.7.047.VH, CPA.7.047.VL, CPA.7.047.HC, CPA.7.047.LC, CPA.7.047.H1, CPA.7.047.H2, CPA.7.047.H3 CPA.7.047.H4; CPA.7.047.vhCDR1, CPA.7.047.vhCDR2, CPA.7.047.vhCDR3, CPA.7.047.vlCDR1, CPA.7.004701.vlCDR2 и CPA.7.047.vlCDR3;
CPA.7.049, CPA.7.049.VH, CPA.7.049.VL, CPA.7.049.HC, CPA.7.049.LC, CPA.7.049.H1, CPA.7.049.H2, CPA.7.049.H3 CPA.7.049.H4; CPA.7.049.vhCDR1, CPA.7.049.vhCDR2, CPA.7.049.vhCDR3, CPA.7.049.vlCDR1, CPA.7.049.vlCDR2 и CPA.7.049.vlCDR3; and
CPA.7.050, CPA.7.050.VH, CPA.7.050.VL, CPA.7.050.HC, CPA.7.050.LC, CPA.7.050.H1, CPA.7.050.H2, CPA.7.050.H3 CPA.7.050.H4, CPA.7.050.vhCDR1, CPA.7.050.vhCDR2, CPA.7.050.vhCDR3, CPA.7.050.vlCDR1, CPA.7.050.vlCDR2 и CPA.7.050.vlCDR3.
Кроме того, существует большое количество CPA-антител, созданных согласно данному документу, которые связывались с PVRIG, но не блокировали взаимодействие PVRIG и PVLR2, как показано на фиг. 51, последовательности только восьми из них включены в данный документ на фиг. 39, а их компонентами являются:
CPA.7.028, CPA.7.028.VH, CPA.7.028.VL, CPA.7.028.HC, CPA.7.028.LC, CPA.7.028.H1, CPA.7.028.H2, CPA.7.028.H3 и CPA.7.028.H4; CPA.7.028.vhCDR1, CPA.7.028.vhCDR2, CPA.7.028.vhCDR3, CPA.7.028.vlCDR1, CPA.7.028.vlCDR2 и CPA.7.028.vlCDR3.
CPA.7.030, CPA.7.030.VH, CPA.7.030.VL, CPA.7.030.HC, CPA.7.030.LC, CPA.7.030.H1, CPA.7.030.H2, CPA.7.030.H3 и CPA.7.030.H4; CPA.7.030.vhCDR1, CPA.7.030.vhCDR2, CPA.7.030.vhCDR3, CPA.7.030.vlCDR1, CPA.7.030.vlCDR2 и CPA.7.030.vlCDR3.
CPA.7.041, CPA.7.041.VH, CPA.7.041.VL, CPA.7.041.HC, CPA.7.041.LC, CPA.7.041.H1, CPA.7.041.H2, CPA.7.041.H3 и CPA.7.041.H4; CPA.7.041.vhCDR1, CPA.7.041.vhCDR2, CPA.7.041.vhCDR3, CPA.7.041.vlCDR1, CPA.7.041.vlCDR2 и CPA.7.041.vlCDR3.
CPA.7.016, CPA.7.016.VH, CPA.7.016.VL, CPA.7.016.HC, CPA.7.016.LC, CPA.7.016.H1, CPA.7.016.H2, CPA.7.016.H3 и CPA.7.016.H4; CPA.7.016.vhCDR1, CPA.7.016.vhCDR2, CPA.7.016.vhCDR3, CPA.7.016.vlCDR1, CPA.7.016.vlCDR2 и CPA.7.016.vlCDR3.
CPA.7.020, CPA.7.020.VH, CPA.7.020.VL, CPA.7.020.HC, CPA.7.020.LC, CPA.7.020.H1, CPA.7.020.H2, CPA.7.020.H3 и CPA.7.020.H4; CPA.7.020.vhCDR1, CPA.7.020.vhCDR2, CPA.7.020.vhCDR3, CPA.7.020.vlCDR1, CPA.7.020.vlCDR2 и CPA.7.020.vlCDR3.
CPA.7.038, CPA.7.038.VH, CPA.7.038.VL, CPA.7.038.HC, CPA.7.038.LC, CPA.7.038.H1, CPA.7.038.H2, CPA.7.038.H3 и CPA.7.038.H4; CPA.7.038.vhCDR1, CPA.7.038.vhCDR2, CPA.7.038.vhCDR3, CPA.7.038.vlCDR1, CPA.7.038.vlCDR2 и CPA.7.038.vlCDR3.
CPA.7.044, CPA.7.044.VH, CPA.7.044.VL, CPA.7.044.HC, CPA.7.044.LC, CPA.7.044.H1, CPA.7.044.H2, CPA.7.044.H3 и CPA.7.044.H4; CPA.7.044.vhCDR1, CPA.7.044.vhCDR2, CPA.7.044.vhCDR3, CPA.7.044.vlCDR1, CPA.7.044.vlCDR2 и CPA.7.044.vlCDR3.
CPA.7.045, CPA.7.045.VH, CPA.7.045.VL, CPA.7.045.HC, CPA.7.045.LC, CPA.7.045.H1, CPA.7.045.H2, CPA.7.045.H3 и CPA.7.045.H4; CPA.7.045.vhCDR1, CPA.7.045.vhCDR2, CPA.7.045.vhCDR3, CPA.7.045.vlCDR1, CPA.7.045.vlCDR2 и CPA.7.045.vlCDR3.
Как обсуждается в данном документе, в изобретении дополнительно предложены варианты вышеуказанных компонентов, включая варианты CDR, указанные выше. Кроме того, вариабельные области тяжелых цепей могут быть на 80%, 90%, 95%, 98% или 99% идентичными последовательностям «VH» согласно данному документу и/или содержать от 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислотных изменений в случае применения Fc-вариантов. Предложены вариабельные области легких цепей, которые могут быть на 80%, 90%, 95%, 98% или 99% идентичными последовательностям «VL» согласно данному документу и/или содержать от 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислотных изменений в случае применения Fc-вариантов. Аналогично, предложены тяжелые и легкие цепи, которые на 80%, 90%, 95%, 98% или 99% идентичны последовательностям «HC» и «LC» согласно данному документу и/или содержат от 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислотных изменений в случае применения Fc-вариантов.
Более того, в настоящем изобретении предложено некоторое количество СНА-антител, которые представляют собой мышиные антитела, полученные из гибридом. Как хорошо известно в данной области техники, шесть CDR функциональны, когда они вставлены между каркасными областями человеческих вариабельных областей тяжелой и легкой цепей или когда вариабельные домены тяжелой и легкой цепей гуманизированы.
Соответственно, в настоящем изобретении предложены антитела, обычно полноразмерные или scFv-домены, которые содержат следующие CHA-группы CDR, последовательности которых приведены на фиг. 40:
CHA.7.502.vhCDR1, CHA.7.502.vhCDR2, CHA.7.502.vhCDR3, CHA.7.502.vlCDR1, CHA.7.502.vlCDR2 и CHA.7.502.vlCDR3.
CHA.7.503.vhCDR1, CHA.7.503.vhCDR2, CHA.7.503.vhCDR3, CHA.7.503.vlCDR1, CHA.7.503.vlCDR2 и CHA.7.503.vlCDR3.
CHA.7.506.vhCDR1, CHA.7.506.vhCDR2, CHA.7.506.vhCDR3, CHA.7.506.vlCDR1, CHA.7.506.vlCDR2 и CHA.7.506.vlCDR3.
CHA.7.508.vhCDR1, CHA.7.508.vhCDR2, CHA.7.508.vhCDR3, CHA.7.508.vlCDR1, CHA.7.508.vlCDR2 и CHA.7.508.vlCDR3.
CHA.7.510.vhCDR1, CHA.7.510.vhCDR2, CHA.7.510.vhCDR3, CHA.7.510.vlCDR1, CHA.7.510.vlCDR2 и CHA.7.510.vlCDR3.
CHA.7.512.vhCDR1, CHA.7.512.vhCDR2, CHA.7.512.vhCDR3, CHA.7.512.vlCDR1, CHA.7.512.vlCDR2 и CHA.7.512.vlCDR3.
CHA.7.514.vhCDR1, CHA.7.514.vhCDR2, CHA.7.514.vhCDR3, CHA.7.514.vlCDR1, CHA.7.514.vlCDR2 и CHA.7.514.vlCDR3.
CHA.7.516.vhCDR1, CHA.7.516.vhCDR2, CHA.7.516.vhCDR3, CHA.7.516.vlCDR1, CHA.7.516.vlCDR2 и CHA.7.516.vlCDR3.
CHA.7.518.vhCDR1, CHA.7.518.vhCDR2, CHA.7.518.vhCDR3, CHA.7.518.vlCDR1, CHA.7.518.vlCDR2 и CHA.7.518.vlCDR3.
CHA.7.520_1.vhCDR1, CHA.7.520_1.vhCDR2, CHA.7.520_1.vhCDR3, CHA.7.520_1.vlCDR1, CHA.7.520_1.vlCDR2 и CHA.7.520_1.vlCDR3.
CHA.7.520_2.vhCDR1, CHA.7.520_2.vhCDR2, CHA.7.520_2.vhCDR3, CHA.7.520_2.vlCDR1, CHA.7.520_2.vlCDR2 и CHA.7.520_2.vlCDR3.
CHA.7.522.vhCDR1, CHA.7.522.vhCDR2, CHA.7.522.vhCDR3, CHA.7.522.vlCDR1, CHA.7.522.vlCDR2 и CHA.7.522.vlCDR3.
CHA.7.524.vhCDR1, CHA.7.524.vhCDR2, CHA.7.524.vhCDR3, CHA.7.524.vlCDR1, CHA.7.524.vlCDR2 и CHA.7.524.vlCDR3.
CHA.7.526.vhCDR1, CHA.7.526.vhCDR2, CHA.7.526.vhCDR3, CHA.7.526.vlCDR1, CHA.7.526.vlCDR2 и CHA.7.526.vlCDR3.
CHA.7.527.vhCDR1, CHA.7.527.vhCDR2, CHA.7.527.vhCDR3, CHA.7.527.vlCDR1, CHA.7.527.vlCDR2 и CHA.7.527.vlCDR3.
CHA.7.528.vhCDR1, CHA.7.528.vhCDR2, CHA.7.528.vhCDR3, CHA.7.528.vlCDR1, CHA.7.528.vlCDR2 и CHA.7.528.vlCDR3.
CHA.7.530.vhCDR1, CHA.7.530.vhCDR2, CHA.7.530.vhCDR3, CHA.7.530.vlCDR1, CHA.7.530.vlCDR2 и CHA.7.530.vlCDR3.
CHA.7.534.vhCDR1, CHA.7.534.vhCDR2, CHA.7.534.vhCDR3, CHA.7.534.vlCDR1, CHA.7.534.vlCDR2 и CHA.7.534.vlCDR3.
CHA.7.535.vhCDR1, CHA.7.535.vhCDR2, CHA.7.535.vhCDR3, CHA.7.535.vlCDR1, CHA.7.535.vlCDR2 и CHA.7.535.vlCDR3.
CHA.7.537.vhCDR1, CHA.7.537.vhCDR2, CHA.7.537.vhCDR3, CHA.7.537.vlCDR1, CHA.7.537.vlCDR2 и CHA.7.537.vlCDR3.
CHA.7.538_1.vhCDR1, CHA.7.538_1.vhCDR2, CHA.7.538_1.vhCDR3, CHA.7.538_1.vlCDR1, CHA.7.538_1.vlCDR2 и CHA.7.538_1.vlCDR3.
CHA.7.538_2.vhCDR1, CHA.7.538_2.vhCDR2, CHA.7.538_2.vhCDR3, CHA.7.538_2.vlCDR1, CHA.7.538_2.vlCDR2 и CHA.7.538_2.vlCDR3.
CHA.7.543.vhCDR1, CHA.7.543.vhCDR2, CHA.7.543.vhCDR3, CHA.7.543.vlCDR1, CHA.7.543.vlCDR2 и CHA.7.543.vlCDR3.
CHA.7.544.vhCDR1, CHA.7.544.vhCDR2, CHA.7.544.vhCDR3, CHA.7.544.vlCDR1, CHA.7.544.vlCDR2 и CHA.7.544.vlCDR3.
CHA.7.545.vhCDR1, CHA.7.545.vhCDR2, CHA.7.545.vhCDR3, CHA.7.545.vlCDR1, CHA.7.545.vlCDR2 и CHA.7.545.vlCDR3.
CHA.7.546.vhCDR1, CHA.7.546.vhCDR2, CHA.7.546.vhCDR3, CHA.7.546.vlCDR1, CHA.7.546.vlCDR2 и CHA.7.546.vlCDR3.
CHA.7.547.vhCDR1, CHA.7.547.vhCDR2, CHA.7.547.vhCDR3, CHA.7.547.vlCDR1, CHA.7.547.vlCDR2 и CHA.7.547.vlCDR3.
CHA.7.548.vhCDR1, CHA.7.548.vhCDR2, CHA.7.548.vhCDR3, CHA.7.548.vlCDR1, CHA.7.548.vlCDR2 и CHA.7.548.vlCDR3.
CHA.7.549.vhCDR1, CHA.7.549.vhCDR2, CHA.7.549.vhCDR3, CHA.7.549.vlCDR1, CHA.7.549.vlCDR2 и CHA.7.549.vlCDR3.
CHA.7.550.vhCDR1, CHA.7.550.vhCDR2, CHA.7.550.vhCDR3, CHA.7.550.vlCDR1, CHA.7.550.vlCDR2 и CHA.7.550.vlCDR3.
Как и выше, эти группы CDR также могут представлять собой аминокислотные варианты, как описано выше.
Кроме того, каркасные области вариабельных областей тяжелой и легкой цепей могут быть гуманизированы, как известно в данной области техники (со случайными вариантами, создаваемыми в CDR в случае необходимости), и таким образом могут быть созданы гуманизированные варианты VH и VL цепей с фиг. 40. Более того, гуманизированные вариабельные домены тяжелой и легкой цепей можно затем сливать с человеческими константными областями, такими как константные области из IgG1, IgG2, IgG3 и IgG4.
В частности, как известно в данной области техники, мышиные VH и VL можно гуманизировать, например, используя программу IgBLAST с веб-страницы NCBI, как описано для способов гуманизации в Ye et al. Nucleic Acids Res. 41:W34-W40 (2013), в полном объеме включенной в данный документ посредством ссылки. IgBLAST берет мышиную VH и/или VL последовательность и сравнивает ее с библиотекой известных последовательностей человеческой зародышевой линии. Как показано в данном документе для созданных гуманизированных последовательностей, используемыми базами данных были IMGT человеческих VH-генов (F+ORF, 273 последовательности зародышевой линии) и IMGT человеческих VL-каппа генов (F+ORF, 74 последовательности зародышевой линии). Было отобрано пять типовых СНА-последовательностей: CHA.7.518, CHA.7.530, CHA.7.538_1, CHA.7.538_2 и CHA.7.524 (смотрите фиг. 40 для VH и VL последовательностей). В случае этого варианта реализации гуманизации в качестве акцепторной последовательности для всех 5 была выбрана человеческая зародышевая линия IGHV1-46 (аллель 1) и соединительная область (ген J) человеческой тяжелой цепи IGHJ4 (аллель 1). Для трех из четырех (CHA.7.518, CHA.7.530, CHA.7.538_1 и CHA.7.538_2) в качестве акцепторной последовательности была выбрана человеческая зародышевая линия IGKV1-39 (аллель 1) и соединительная область (ген J) человеческой легкой цепи IGKJ2 (аллель 1). Ген J был выбран из человеческих последовательностей соединительной области, собранных на странице IMGT®, международной иммуногенетической информационной системы, www.imgt.org. CDR определены в соответствии с определение AbM (смотрите www.bioinfo.org.uk/abs/). Фиг. 87 иллюстрирует гуманизированные последовательности, а также некоторые потенциальные изменения для оптимизации связывания с PVRIG.
Конкретные гуманизированные антитела из СНА-антител включают приведенные на фиг. 87, фиг. 88 и фиг. 89. Как понятно специалистам в данной области техники, каждую последовательность вариабельной области тяжелой (гуманизированная тяжелая; HH) и вариабельной области легкой (гуманизированная легкая, HL) цепей можно комбинировать с константными областями человеческих IgG1, IgG2, IgG3 и IgG4. Это означает, что CHA.7.518.HH1 является первой гуманизированной вариабельной областью тяжелой цепи, а CHA.7.518.HH1.1 является полноразмерной тяжелой цепью, содержащей гуманизированную последовательность «HH1» с константной областью IgG1 (CHA.7.518.HH1.2 представляет собой CHA.7.518.HH1 с IgG2, и т.д.).
В некоторых вариантах реализации изобретения анти-PVRIG антитела согласно настоящему изобретению включают анти-PVRIG антитела, в которых VH и VL последовательности разных анти-PVRIG антител могут быть «смешаны и сопоставлены» для создания других анти-PVRIG антител. Связывание PVRIG таких «смешанных и сопоставленных» антител можно исследовать, используя методы анализа связывания, описанные выше (например, ELISA). В некоторых вариантах реализации изобретения, когда VH и VL цепи смешаны и сопоставлены, последовательность VH из конкретной пары VH/VL замещена структурно сходной последовательностью VH. Аналогично, в некоторых вариантах реализации изобретения последовательность VL из конкретной пары VH/VL замещена структурно сходной последовательностью VL. Например, VH и VL последовательности гомологичных антител в особенности подходят для смешивания и сопоставления.
Соответственно, антитела согласно изобретению содержат аминокислотные последовательности CDR, выбранные из группы, состоящей из (a) последовательностей, приведенных в данном документе; (b) последовательностей, которые отличаются от аминокислотных последовательностей CDR, определенных в (a) на 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислотных замен; (c) аминокислотных последовательностей, имеющих 90% или более, 95% или более, 98% или более или 99% или более идентичности последовательностей с последовательностями, определенными в (a) или (b); (d) полипептида, имеющего аминокислотную последовательность, кодируемую полинуклеотидом, имеющим нуклеотидную последовательность, кодирующую приведенные в данном документе аминокислоты.
Кроме того, в определение антител к PVRIG включены антитела, которые обладают идентичностью с антителами к PVRIG, пронумерованными в данном документе. Это означает, что в определенных вариантах реализации анти-PVRIG антитело согласно изобретению содержит вариабельные области тяжелой и легкой цепи, содержащие аминокислотные последовательности, гомологичные выделенным анти-PVRIG аминокислотным последовательностям предпочтительных анти-PVRIG иммунных молекул соответственно, при этом антитела сохраняют необходимые функциональные свойства родительских анти-PVRIG антител. Процент идентичности между двумя последовательностями является функцией числа идентичных позиций в последовательностях (т.е. % гомологии = # идентичных позиций/общее # число позиций X 100) с учетом количества гэпов и длины каждого гэпа, которые необходимо вносить для оптимального выравнивания двух последовательностей. Сравнение последовательностей и определение процента идентичности между двумя последовательностями можно осуществлять, используя математический алгоритм, как описано в неограничивающих примерах ниже.
Процент идентичности между двумя аминокислотными последовательностями можно определить, используя алгоритм E. Meyers и W. Miller (Comput. Appl. Biosci., 4:11-17 (1988)), который был включен в программу ALIGN (версия 2.0), используя таблицу весов замен остатков PAM120, штраф за длину гэпа 12 и штраф за гэп 4. Кроме того, процент идентичности между двумя аминокислотными последовательностями можно определить, используя алгоритм Needleman и Wunsch (J. Mol. Biol. 48:444-453 (1970)), который был включен в программу GAP из пакета программного обеспечения GCG (коммерчески доступного), используя матрицу Blossum 62 или матрицу PAM250 и штраф за открытие гэпа 16, 14, 12, 10, 8, 6 или 4 и штраф за продолжение гэпа 1, 2, 3, 4, 5 или 6.
В дополнительном или альтернативном варианте белковые последовательности согласно настоящему изобретению можно дополнительно использовать в качестве «запрашиваемой последовательности» для проведения поиска в открытых базах данных, например, чтобы определить родственные последовательности. Такой поиск можно проводить, используя программу XBLAST (версия 2.0) по Altschul, et al. (1990) J Mol. Biol. 215:403-10. Белковый поиск BLAST можно проводить с помощью программы XBLAST, оценка = 50, длина слова = 3, для получения аминокислотных последовательностей, гомологичных молекулам антител в соответствии по меньшей мере с некоторыми вариантами реализации изобретения. Для получения в целях сравнения выравнивания с гэпами можно использовать Gapped BLAST, как описано в Altschul et al., (1997) Nucleic Acids Res. 25(17):3389-3402. При применении программ BLAST и Gapped BLAST можно использовать параметры по умолчанию соответствующих программ (например, XBLAST и NBLAST).
В общем случае процент идентичности для сравнения антител к PVRIG составляет по меньшей мере 75%, по меньшей мере 80%, по меньшей мере 90%, при этом предпочтительно составляет по меньшей мере около 95, 96, 97, 98 или 99%. Процент идентичности можно определять для всей аминокислотной последовательности, например, полной тяжелой или легкой цепи, или для части цепей. Например, в определение анти-PVRIG антител согласно изобретению включены антитела, которые обладают идентичностью на протяжении всей вариабельной области (например, когда идентичность составляет 95 или 98% на протяжении вариабельных областей) или на протяжении полной константной области, или только на протяжении Fc-домена.
Кроме того, также включены последовательности, которые могут иметь идентичные CDR, но содержать изменения в вариабельном домене (или полной тяжелой или легкой цепи). Например, антитела к PVRIG включают антитела с CDR, идентичными приведенным на фиг. 62, но чья идентичность на протяжении вариабельной области может быть ниже, например, 95 или 98%.
Антитела к PVRIG, которые конкурируют за связывание с пронумерованными антителами
В настоящем изобретении предложены не только пронумерованные антитела, но также дополнительные антитела, которые конкурируют с пронумерованными антителами (с номерами СРА и СНА, пронумерованными в данном документе, которые специфически связываются с PVRIG) за специфическое связывание с молекулой PVRIG. Как показано в Пример 11, антитела к PVRIG согласно изобретению, «попадают» в разные эпитопные группы. В данном документе указаны четыре отдельные группы; 1) эпитопная группа, в которую попадают CPA.7.002, CPA.7.003, CPA.7.005, CPA.7.007, CPA.7.010, CPA.7.012, CPA.7.015, CPA.7.016, CPA.7.017, CPA.7.019, CPA.7.020, CPA.7.021, CPA.7.024, CPA.7.028, CPA.7.032, CPA.7.033, CPA.7.036, CPA.7.037, CPA.7.038, CPA.7.043, CPA.7.046 и CPA.7.041; 2) эпитопная группа, в которую попадают CPA.7.004, CPA.7.009, CPA.7.011, CPA.7.014, CPA.7.018, CPA.7.022, CPA.7.023, CPA.7.034, CPA.7.040, CPA.7.045 и CPA.7.047; 3) CPA.7.039, который определяет различие между группой 1 и группой 2, состоящее в том, что группа 1 блокирует связывание CPA.7.039, а группа 2 заключает лиганд в сандвич-структуру с CPA.7.039, и группа 4) с CPA.7.050.
Таким образом, в изобретении предложены анти-PVRIG антитела, которые конкурируют за связывание с антителами из группы 1, с антителами из группы 2, с антителами из группы 3 и/или с антителами из группы 4.
Дополнительные антитела, которые конкурируют с пронумерованными антителами, создают так, как известно в данной области техники, и в общем случае описано ниже. Исследования конкурентного связывания можно проводить так, как известно в данной области техники, используя в общем случае анализ связывания ППР/Biacore®, а так же ELISA и клеточный анализ.
Создание дополнительных антител
Дополнительные антитела к человеческому PVRIG можно получать, как известно в данной области техники, используя известные способы, такие, как описаны в примерах. Таким образом, анти-PVRIG антитела можно создавать традиционными способами, такими как иммунизация мышей (иногда используя ДНК-иммунизацию, например, которую использовал Aldevron) с последующим скринингом против человеческого белка PVRIG и создание гибридом с очисткой и выделением антитела.
Композиции нуклеиновых кислот
Также предложены композиции нуклеиновых кислот, кодирующие анти-PVRIG антитела согласно изобретению, а также экспрессионные векторы, содержащие нуклеиновые кислоты, и клетки-хозяева, трансформированные композициями нуклеиновых кислот и/или экспрессионных векторов. Как понятно специалистам в данной области техники, приведенные в данном документе белковые последовательности могут кодироваться любым числом возможных нуклеотидных последовательностей вследствие вырожденности генетического кода.
Композиции нуклеиновых кислот, которые кодируют антитела к PVRIG, зависят от формата антитела. Традиционные тетрамерные антитела, содержащие две тяжелые цепи и две легкие цепи, кодируются двумя разными нуклеиновыми кислотами, одна из которых кодирует тяжелую цепь, а вторая кодирует легкую цепь. Их можно помещать в один экспрессионный вектор или два экспрессионных вектора, как известно в данной области техники, трансформировать в клетки-хозяев, в которых они экспрессируются с образованием антител согласно изобретению. В некоторых вариантах реализации изобретения, например, при применении конструкций scFv, в общем случае используют одну нуклеиновую кислоту, кодирующую вариабельную область тяжелой цепи-линкер-вариабельную область легкой цепи, которую можно вставлять в экспрессионный вектор для трансформации в клетки-хозяев. Нуклеиновые кислоты можно вставлять в экспрессионные векторы, которые содержат подходящие транскрипционные и трансляционные контрольные последовательности, включая, но не ограничиваясь этим, сигнальные и секреторные последовательности, регуляторные последовательности, промоторы, точки начала репликации, селекционные гены и т.д.
Предпочтительные клетки-хозяева млекопитающих для экспрессии рекомбинантных антител в соответствии по меньшей мере с некоторыми вариантами реализации изобретения включают клетки яичника китайского хомяка (клетки CHO), PER.C6, HEK293 и другие, как известно в данной области техники.
Нуклеиновые кислоты могут находиться в цельных клетках, в клеточном лизате или в частично очищенной или в значительной степени чистой форме. Нуклеиновая кислота является «выделенной» или «в значительной степени чистой» при очистке от других клеточных компонентов или других загрязнителей, например, других клеточных нуклеиновых кислот или белков, стандартными методами, включая обработку щелочью/ДСН, бэндинг CsCl, колоночную хроматографию, электрофорез в агарозном геле и другик, хорошо известные в данной области техники методы.
Для создания гена scFv VH- и VL-кодирующие фрагменты ДНК функционально связывают с другим фрагментом, кодирующим гибкий линкер, например, кодирующим аминокислотную последовательность (Gly4-Ser)3, так, что VH и VL последовательности могут экспрессироваться в виде непрерывного одноцепочечного белка, в котором области VL и VH соединены гибким линкером (смотрите, например, Bird et al. (1988) Science 242:423-426; Huston et al. (1988) Proc. Natl. Acad. Sci. USA 85:5879-5883; McCafferty et al., (1990) Nature 348:552-554).
Составы анти-PVRIG антител
Терапевтические композиции, применяемые для практической реализации вышеуказанных способов, можно смешивать в виде фармацевтических композиций, содержащих носитель, подходящий для желаемого способа доставки. Подходящие носители включают любой материал, который в комбинации с терапевтической композицией сохраняет противоопухолевую функцию терапевтической композиции и в общем случае не вступает в реакцию с иммунной системой пациента. Примеры включают, но не ограничиваются этим, любое число стандартных фармацевтических носителей, таких как стерильные фосфатно-солевые буферные растворы, бактериостатическая вода и тому подобное (смотрите, в общем случае, Remington's Pharmaceutical Sciences 16th Edition, A. Osal., Ed., 1980). Приемлемые носители, вспомогательные вещества или стабилизаторы являются нетоксичными для реципиентов в применяемых дозировках и концентрациях и включают буферы, такие как фосфатный, цитратный, ацетатный и другие органические кислоты; антиоксиданты, включая аскорбиновую кислоту и метионин; консерванты (такие как хлорид октадецилдиметилбензиламмония; хлорид гексаметония; хлорид бензалкония, хлорид бензетония; фенол, бутиловый или бензиловый спирт; алкилпарабены, такие как метил- или пропилпарабен; катехол; резорцин; циклогексанол; 3-пентанол; и м-крезол); низкомолекулярные полипептиды (менее чем около 10 остатков); белки, такие как сывороточный альбумин, желатин или иммуноглобулины; гидрофильные полимеры, такие как поливинилпирролидон; аминокислоты, такие как глицин, глутамин, аспарагин, гистидин, аргинин или лизин; моносахариды, дисахариды и другие углеводы, включая глюкозу, маннозу или декстрины; хелатирующие агенты, такие как ЭДТА; сахара, такие как сахароза, манит, трегалоза или сорбит; подсластители и другие ароматизаторы; наполнители, такие как микрокристаллическая целлюлоза, лактоза, кукурузный и другие крахмалы; связующие агенты; добавки; красители; солеобразующие противоионы, такие как натрий; комплексы металлов (например, комплексы Zn-белок); и/или неионные поверхностно-активные вещества, такие как ТВИН.TM., ПЛЮРОНИКИ.TM. или полиэтиленгликоль).
В предпочтительном варианте реализации изобретения фармацевтическая композиция, которая содержит антитела согласно изобретению, может находиться в водорастворимой форме, например, присутствовать в виде фармацевтически приемлемых солей, которые включают соли присоединения как кислоты, так и основания. «Фармацевтически приемлемые соли присоединения кислоты» относятся к тем солям, которые сохраняют биологическую эффективность свободных оснований и не являются биологически или каким-либо другим образом нежелательными, образуемым с неорганическими кислотами, такими как хлористоводородная кислота, бромистоводородная кислота, серная кислота, азотная кислота, фосфорная кислота и тому подобные, и органическими кислотами, такими как уксусная кислота, пропионовая кислота, гликолевая кислота, пировиноградная кислота, щавеленая кислота, малеиновая кислота, малоновая кислота, янтарная кислота, фумаровая кислота, винная кислота, лимонная кислота, бензойная кислота, коричная кислота, миндальная кислота, метансульфоновая кислота, этансульфоновая кислота, п-толуолсульфоновая кислота, салициловая кислота и тому подобные. «Фармацевтически приемлемые соли присоединения основания» включают соли, полученные из неорганических оснований, такие как соли натрия, калия, лития, аммония, кальция, магния, железа, цинка, меди, марганца, алюминия и тому подобные. В частности, предпочтительными являются соли аммония, калия, натрия, кальция и магния. Соли, полученные из фармацевтически приемлемых органических нетоксических оснований, включают соли первичных, вторичных и третичных аминов, замещенных аминов, включая встречающиеся в природе замещенные амины, циклических аминов и основных ионообменных смол, таких как изопропиламин, триметиламин, диэтиламин, триэтиламин, трипропиламин и этаноламин. Составы для применения для in vivo введения предпочтительно являются стерильными. Это легко осуществить путем фильтрации через стерильные фильтровальные мембраны или другими способами.
Введение фармацевтической композиции, содержащей антитела согласно изобретению, предпочтительно в форме стерильного водного раствора, можно осуществлять множеством путей, включая, но не ограничиваясь этим, подкожное и внутривенное. Подкожное введение может быть предпочтительным в некоторых обстоятельствах, так как пациент может сам вводить фармацевтическую композицию. Многие белковые терапевтические средства являются недостаточно сильными, чтобы обеспечить возможность состава терапевтически эффективной дозы в максимально приемлемом объеме для подкожного введения. Эту проблему можно частично решить путем применения белковых составов, содержащих аргинин-HCl, гистидин и полисорбат (смотрите WO 04091658). Fc-полипептиды согласно настоящему изобретению могут больше подходить для подкожного введения благодаря, например, повышенной эффективности, улучшенному сывороточному времени полужизни или повышенной растворимости.
Как известно в данной области техники, белковые терапевтические средства часто доставляют путем ВВ инфузии или болюса. Антитела согласно настоящему изобретению также можно доставлять, используя подобные способы. Например, введение может происходить путем внутривенной инфузии с 0,9% хлоридом натрия в качестве базового инфузионного раствора.
Кроме того, в данной области техники известно большое число систем доставки, которые можно использовать для введения Fc-вариантов согласно настоящему изобретению. Примеры включают, но не ограничиваются этим, инкапсулирование в липосомах, микрочастицах, микросферах (например, микросферах PLA/PGA) и тому подобное. В альтернативном варианте можно использовать имплантат из пористого, непористого или желеобразного материала, включая мембраны или волокна. Системы с замедленным высвобождением могут содержать полимерные материалы или матрицы, такие как сложные полиэфиры, гидрогели, поли(виниловый спирт), полилактиды, сополимеры L-глутаминовой кислоты и этил-L-глутамата, этиленвинилацетат, сополимеры молочной и гликолевой кислоты, такие как LUPRON DEPOT.RTM., и поли-D-(-)-3-гидроксимасляную кислоту. Раскрытые в данном документе антитела также могут быть получены в виде иммунолипосом. Липосома представляет собой небольшую везикулу, содержащую различные типы липидов, фосфолипидов и/или поверхностно-активное вещество, которая подходит для доставки терапевтического агента млекопитающему. Липосомы, содержащие антитело, получают способами, известными в данной области техники, такими как описанные в Epstein et al., 1985, Proc Natl Acad Sci USA, 82:3688; Hwang et al., 1980, Proc Natl Acad Sci USA, 77:4030; патенте США № 4485045; патенте США № 4544545; и PCT WO 97/38731. Липосомы с увеличенным временем нахождения в циркуляции описаны в патенте США № 5013556. Компоненты липосомы обычно упорядочены в бислоевом формате, аналогично упорядочению липидов биологических мембран. Особенно подходящие липосомы можно получить методом обращенно-фазового выпаривания с липидной композицией, содержащей фосфатидилхолин, холестерин и ПЭГ-дериватизированный фосфатидилэтаноламин (ПЭГ-ФЭ). Липосомы пропускают через фильтры с определенным размером пор, чтобы получить липосомы с желаемым диаметром. В липосоме необязательно содержится химиотерапевтический агент или другой терапевтически активный агент (Gabizon et al., 1989, J National Cancer Inst 81:1484).
Антитела также могут быть заключены в микрокапсулы, получаемые способами, включающими, но не ограничивающимися этим, методы коацервации, межфазную полимеризацию (например, с использованием микрокапсул из гидроксиметилцеллюлозы или желатина или микрокапсул из поли(метилметакрилата)), коллоидные системы доставки лекарственных препаратов (например, липосомы, альбуминовые микросферы, микроэмульсии, наночастицы и нанокапсулы) и макроэмульсии. Такие методы раскрыты в Remington's Pharmaceutical Sciences 16th edition, Osol, A. Ed., 1980. Можно готовить препараты с замедленным высвобождением. Подходящие примеры препаратов с замедленным высвобождением включают полупроницаемые матрицы из твердых гидрофобных полимеров в форме формованных изделий, например, пленок или микрокапсул. Примеры матриц с замедленным высвобождением включают сложные полиэфиры, гидрогели (например, поли(2-гидроксиэтилметакрилат) или поли(виниловый спирт), полилактиды (патент США № 3773919), сополимеры L-глутаминовой кислоты и гамма этил-L-глутамата, неразлагаемый этиленвинилацетат, разлагаемые сополимеры молочной и гликолевой кислоты, такие как LUPRON DEPOT.RTM. (которые представляют собой инъецируемые микросферы, состоящие из сополимера молочной кислоты и гликолевой кислоты и лейпролида ацетата), поли-D-(-)-3-гидроксимасляную кислоту и ProLease.RTM. (коммерчески доступный от Alkermes), который представляет собой систему доставки, состоящую из необходимой биоактивной молекулы, внедренной в матрицу поли-DL-лактида-ко-гликолида (ПЛГ).
Объем дозирования и частоту введения в предпочтительном варианте реализации изобретения выбирают так, чтобы они были терапевтически или профилактически эффективными. Как известно в данной области техники, могут понадобиться корректировки, учитывающие разложение белка, выбор между системной и местной доставкой и скорость синтеза новых протеаз, а также возраст, массу тела, общее состояние здоровья, пол, режим питания, время введения, взаимодействие между лекарственными препаратами и тяжесть патологического состояния, которые специалисты в данной области техники могут выяснить в ходе простых экспериментов.
Концентрация антитела в составе может варьироваться от около 0,1 до 100% масс. В предпочтительном варианте реализации изобретения концентрация Fc-варианта находится в молярном диапазоне от 0,003 до 1,0. Для лечения пациента можно вводить терапевтически эффективную дозу Fc-варианта согласно настоящему изобретению. Под «терапевтически эффективной дозой» подразумевается доза, которая оказывает то действие, ради которого ее вводили. Точная доза зависит от цели лечения и выясняется специалистом в данной области техники известными методами. Дозировки могут находиться в диапазоне от 0,0001 to 100 мг/кг массы тела или более, например, 0,1, 1, 10 или 50 мг/кг массы тела, предпочтительно от 1 до 10 мг/кг.
Способы применения анти-PVRIG антител
Полученные анти-PVRIG антитела согласно изобретению находят разные применения.
Терапевтические применения
Анти-PVRIG антитела согласно изобретению находят применение в лечение пациентов, таких как люди, имеющие в общем случае патологическое состояние, связанное с PVRIG. В контексте данного документа термин «лечение» относится как к терапевтическому лечению, так и к профилактическим или превентивным мерам, которые в этом примере связаны с лечением рака; при этом, как описано ниже, также предложены применения антител и фармацевтических композиций для лечения инфекционных заболеваний, например, сепсиса, и/или аутоиммунных патологических состояний, и/или для ингибирования нежелательной иммунной активации, которая следует за генной терапией. Нуждающиеся в лечении включают тех, у кого уже есть рак, а также тех, у кого необходимо предотвратить рак. Следовательно, млекопитающее, требующее лечения согласно данному документу, может быть диагностировано как имеющее рак или может быть предрасположено или восприимчиво к раку. В контексте данного документа термин «лечение» относится к предотвращению, задержке начала, излечению, обращению, ослаблению, облегчению, минимизации, подавлению, прекращению вредного действия или стабилизации видимых симптомов вышеописанных раковых заболеваний, нарушений или патологических состояний. Также он включает принятие мер в отношении рака, как описано выше. Под «принятием мер» подразумевается снижение тяжести заболевания, снижение частоты приступов заболевания, снижение продолжительности таких приступов, снижение тяжести таких приступов, замедление/снижение роста или пролиферации раковых клеток, замедление прогрессирования по меньшей мере одного симптома, улучшение по меньшей мере одного определяемого физического параметра и тому подобное. Например, иммуностимулирующие анти-PVRIG иммунные молекулы должны стимулировать активность Т-клеток или NK-клеток или цитокиновый иммунитет против клеток-мишеней, например, рака, инфицированных или патогенных клеток, и таким образом лечить рак или инфекционные заболевания путем снижения числа клеток, вовлеченных в болезненное состояние. В противоположном случае иммуностимулирующие анти-PVRIG иммунные молекулы должны снижать активность Т-клеток или NK-клеток и/или секрецию провоспалительных цитокинов, которые вовлечены в патологию заболевания в случае некоторых иммунных заболеваний, таких как аутоиммунные, воспалительные или аллергические патологические состояния, и таким образом лечить или облегчать патологию заболевания и разрушение тканей, которое может быть связано с такими патологическими состояниями (например, разрушение суставов, связанное с патологическими состояниями ревматоидного артрита).
Антитела к PVRIG согласно изобретению предложены в терапевтически эффективных дозировках. «Терапевтически эффективная дозировка» анти-PVRIG иммунной молекулы в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения предпочтительно приводит к снижению тяжести симптомов заболевания, увеличению частоты и длительности бессимптомных периодов, увеличению продолжительности жизни, ремиссии заболевания или предотвращению или снижению нарушений или недееспособности вследствие поражения заболеванием. Например, в случае лечения PVRIG-положительных опухолей «терапевтически эффективная дозировка» предпочтительно ингибирует рост клеток или рост опухоли по меньшей мере на около 20%, более предпочтительно по меньшей мере на около 40%, даже более предпочтительно по меньшей мере на около 60% и еще более предпочтительно по меньшей мере на около 80% по сравнению с не проходящими лечение субъектами. Способность соединения ингибировать рост опухоли можно оценить на животной модели, прогностической в отношении эффективности в случае человеческих опухолей. В альтернативном варианте это свойство композиции можно оценить путем исследования способности соединения к ингибированию in vitro с помощью методов анализа, известных специалистам-практикам. Терапевтически эффективное количество терапевтического соединения может снижать размер опухоли или каким-либо другим образом облегчать симптомы у субъекта.
Специалист в данной области техники сможет определить терапевтически эффективное количество на основании таких факторов, как масса субъекта, тяжесть симптомов у субъекта и конкретные выбранные композиция или путь введения.
Лечение рака
Антитела к PVRIG согласно изобретению находят применение, в частности, в лечении рака. В общем случае антитела согласно изобретению являются иммуномодулирующими в том смысле, что вместо прямой атаки раковых клеток анти-PVRIG антитела согласно изобретению стимулируют иммунную систему, в общем случае путем ингибирования действия PVRIG. Таким образом, в отличие от нацеленных на опухоль вариантов терапии, целью которых является ингибирование молекулярных путей, играющих важную роль для роста и развития опухоли, и/или снижение количества опухолевых клеток, целью иммунотерапии рака является стимуляция уничтожения раковых клеток собственной иммунной системой пациента, что обеспечивает долговременное разрушение опухоли. В иммунотерапии рака можно использовать различные подходы, среди которых терапевтические вакцины против рака для индукции опухолеспецифических Т-клеточных ответов и иммуностимулирующие антитела (т.е. антагонисты ингибиторных ответов = иммунных контрольных точек) для разрушения иммуносупрессивных путей.
Клинические результаты в случае нацеленной терапии или традиционных вариантов противораковой терапии имеют тенденцию к временному эффекту, так как раковые клетки развивают устойчивость и происходит повторное появление опухоли. При этом клиническое применение иммунотерапии рака в последние несколько лет показало, что этот тип терапии может приводить к длительным клиническим результатам, демонстрируя сильное влияние на долгосрочную выживаемость. Однако, хотя результаты являются долгосрочными, ответ наблюдали только у небольшого количества пациентов (в противоположность традиционной или нацеленной терапии, в случае которые ответ наблюдается у большого количества пациентов, но результаты являются временными).
Ко времени клинического выявления опухоли она уже успевает преодолеть систему иммунной защиты, приобретая иммунорезистентные и иммуносупрессивные свойства и создавая иммуносупрессивное опухолевое микроокружение посредством различных механизмов и множества иммунных клеток.
Соответственно, анти-PVRIG антитела согласно изобретению применимы в лечении рака. Из-за природы иммуно-онкологического механизма действия PVRIG не обязательно должен сверхэкспрессироваться или коррелировать с конкретным типом рака; это означает, что целью является наличие анти-PVRIG антител, чтобы снять подавление активации Т-клеток и NK-клеток так, чтобы иммунная система продолжала работать после рака.
В контексте данного документа «рак» в широком смысле относится к любому неопластическому заболеванию (как инвазивному, так и метастатическому), характеризуемой аномальным и неконтролируемым делением клеток, приводящим к злокачественному росту или опухоли (например, нерегулируемому клеточному росту). В контексте данного документа термин «рак» или «раковый» следует понимать, как включающий любое неопластическое заболевание (как инвазивное, так и неинвазивное или метастатическое), характеризуемое аномальным и неконтролируемым делением клеток, приводящим к злокачественному росту или опухоли, неограничивающие примеры которого описаны в данном документе. Он включает любое физиологическое состояние у млекопитающих, которое, как правило, характеризуется повышенной регуляцией клеточного роста. Примеры рака проиллюстрированы в рабочих примерах и также описаны в тесте заявки.
Неограничивающие примеры видов рака, которые можно лечить с помощью анти-PVRIG антител, включают, но не ограничиваются этим, карциному, лимфому, бластому, саркому и лейкоз. Более конкретные примеры таких видов рака включают плоскоклеточный рак, рак легкого (включая мелкоклеточный рак легкого, немелкоклеточный рак легкого, аденокарциному легкого и плоскоклеточную карциному легкого), рак брюшной полости, гепатоцеллюлярный рак, рак желудка (включая рак желудочно-кишечного тракта), рак поджелудочной железы, глиобластому, рак шейки матки, рак яичника, рак печени, рак мочевого пузыря, гепатому, рак молочной железы, рак толстой кишки, колоректальный рак, карциному эндометрия или матки, карциному слюнной железы, рак почки, рак печени, рак простаты, рак вульвы, рак щитовидной железы, гепатокарциному и различные типы рака головы и шеи, а также В-клеточную лимфому (включая высокодифференцированную/фолликулярную неходжкинскую лимфому (НХЛ); мелкоклеточную лимфоцитарную (МЛ) НХЛ; среднедифференцированную/фолликулярную НХЛ; среднедифференцированную диффузную НХЛ; низкодифференцированную иммунобластную НХЛ; низкодифференцированную лимфобластную НХЛ; низкодифференцированную мелкоклеточную НХЛ с нерассеченными ядрами; НХЛ с генерализованной лимфаденопатией; мантийноклеточную лимфому; СПИД-ассоциированную лимфому; и макроглобулинемию Вальденстрема); хронический лимфоцитарный лейкоз (ХЛЛ); острый лимфобластный лейкоз (ОЛЛ); лейкоз ворсистых клеток; хронический миелобластный лейкоз; множественную миелому и посттрансплантационное лимфопролиферативное расстройство (ПТЛР).
Другие виды рака, подлежащие лечению согласно настоящему изобретению, включают, но не ограничиваются этим, карциному, лимфому, бластому, саркому и лейкоз или лимфоидные злокачественные образования. Более конкретные примеры таких видов рака включают колоректальный рак, рак мочевого пузыря, рак яичника, меланому, плоскоклеточный рак, рак легкого (включая мелкоклеточный рак легкого, немелкоклеточный рак легкого, аденокарциному легкого и плоскоклеточную карциному легкого), рак брюшной полости, гепатоцеллюлярный рак, рак желудка (включая рак желудочно-кишечного тракта), рак поджелудочной железы, глиобластому, рак шейки матки, рак яичника, рак печени, рак мочевого пузыря, гепатому, рак молочной железы, рак толстой кишки, колоректальный рак, карциному эндометрия или матки, карциному слюнной железы, рак почки, рак печени, рак простаты, рак вульвы, рак щитовидной железы, гепатокарциному и различные типы рака головы и шеи, а также В-клеточную лимфому (включая высокодифференцированную/фолликулярную неходжкинскую лимфому (НХЛ); мелкоклеточную лимфоцитарную (МЛ) НХЛ; среднедифференцированную/фолликулярную НХЛ; среднедифференцированную диффузную НХЛ; низкодифференцированную иммунобластную НХЛ; низкодифференцированную лимфобластную НХЛ; низкодифференцированную мелкоклеточную НХЛ с нерассеченными ядрами; НХЛ с генерализованной лимфаденопатией; мантийноклеточную лимфому; СПИД-ассоциированную лимфому; и макроглобулинемию Вальденстрема); хронический лимфоцитарный лейкоз (ХЛЛ); острый лимфобластный лейкоз (ОЛЛ); лейкоз ворсистых клеток; хронический миелобластный лейкоз; и посттрансплантационное лимфопролиферативное расстройство (ПТЛР), а также аномальную пролиферацию сосудов, связанную с факоматозами, отек (например, связанный с опухолями головного мозга) и синдром Мейгса. Предпочтительно рак выбран из группы, состоящей из колоректального рака, рака молочной железы, рака прямой кишки, немелкоклеточного рака легкого, неходжкинской лимфомы (НХЛ), почечно-клеточного рака, рака простаты, рака печени, рака поджелудочной железы, саркомы мягких тканей, саркомы Капоши, карциноидной карциному, рака головы и шеи, меланомы, рака яичника, мезотелиомы и множественной миеломы. В типовом варианте реализации изобретения рак представляет собой ранний или запущенный (включая метастатический) рак мочевого пузыря, яичника или меланому. В другом варианте реализации изобретения рак представляет собой колоректальный рак. Раковые состояния, подлежащие лечению согласно настоящему изобретению, включают раки, которые экспрессируют или не экспрессируют PVRIG, и дополнительно включают неметастатические или неинвазивные, а также инвазивные или метастатические раки, при которых экспрессия PVRIG иммунными, стромальными или пораженными заболеванием клетками подавляет противоопухолевые ответы и противоинвазивные иммунные ответы. Способ согласно настоящему изобретению в особенности подходит для лечения васкуляризированных опухолей.
Как показано в Примерах, PVRIG сверхэкспрессируется и/или коррелирует с инфильтрацией опухолевыми лимфоцитами (как продемонстрировано корреляцией с экспрессией CD3, CD4, CD8 и PD-1) в случае большого числа разных опухолей различного происхождения и, следовательно, применим при лечении любого рака, включая, но не ограничиваясь этим, рак простаты, рак печени (ГЦК), колоректальный рак, рак яичника, рак эндометрия, рак молочной железы, рак поджелудочной железы, рак желудка, рак шейки матки, рак головы и шеи, рак щитовидной железы, рак яичка, рак уротелия, рак легкого, меланому, немеланомный рак кожи (плоскоклеточную и базальноклеточную карциному), глиому, рак почки (ПКК), лимфому (неходжкинскую лимфому (НХЛ) и лимфому Ходжкина (ЛХ)), острый миелоцитарный лейкоз (ОМЛ), Т-клеточный острый лимфобластный лейкоз (T-ОЛЛ), диффузную крупноклеточную В-клеточную лимфому, опухоль половых клеток яичка, мезотелиому и рак пищевода.
В данном документе «терапия рака» относится к любому способу, который предотвращает или лечит рак или облегчает один или более симптомов рака. Как правило, такие виды терапии включают введение иммуностимулирующих анти-PVRIG антител (включая антигенсвязывающие фрагменты), как одних, так и в комбинации с химиотерапией или лучевой терапией или другими биопрепаратами для повышения их активности, т.е. в случае индивидов, у которых экспрессия PVRIG подавляет противоопухолевые ответы и эффективность химиотерапии или лучевой терапии или эффективность биопрепаратов.
Комбинированная терапия при раке
Как известно в данной области техники, комбинированная терапия, включающая применение нацеливания антитела на иммунотерапевтическую мишень и дополнительного терапевтического агента, специфического в отношении болезненного состояния, является очень перспективной. Например, в области иммунотерапии существует большое число перспективных вариантов комбинированной терапии, включающих применение химиотерапевтического агента (низкомолекулярного лекарственного препарата или противоопухолевого антитела) с иммуно-онкологическими антителами, такими как анти-PD-1, и, следовательно, описанные в данном документе анти-PVRIG антитела можно использовать таким же образом. В соответствии с настоящим изобретением можно использовать любой химиотерапевтический агент, проявляющий противораковую активность; различные неограничивающие примеры описаны в тексте заявки.
Лежащее в основе научное обоснование резкого повышения эффективности комбинированной терапии состоит в том, что блокада иммунных контрольных точек в виде монотерапии индуцирует регрессию опухолей только в случае ранее существовавшего сильного противоопухолевого иммунного ответа, который «начинает работать» после блокирования пути. Однако для большинства пациентов и типов опухолей эндогенные противоопухолевые иммунные ответы являются слабыми и, как следствие, для эффективной блокады иммунных контрольных точек требуется индукция противоопухолевого иммунитета, как показано на фиг. 1. В соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения PVRIG-специфические антитела, фрагменты антител, содержащие их конъюгаты и композиции применяют для лечения всех типов рака в иммунотерапии рака в комбинированной терапии.
Термины «в комбинации с» и «совместное введение» не ограничиваются введением указанных профилактических или терапевтических агентов точно в одно время. Вместо этого подразумевается, что анти-PVRIG антитело и другой агент или агенты вводят последовательно и в течение некоторого периода времени так, чтобы они могли действовать вместе и обеспечивать благоприятный эффект, который повышен по сравнению с лечением только анти-PVRIG антителом согласно настоящему изобретению или другим агентом или агентами. Предпочтительно, чтобы анти-PVRIG антитело и другой агент или агенты действовали аддитивно и, в особенности предпочтительно, чтобы они действовали синергетически. Удобно, чтобы такие молекулы присутствовали в комбинации в количествах, эффективных для предполагаемой цели. Медицинский специалист-практик может эмпирически или, учитывая фармакокинетику и механизм действия агентов, определить подходящую дозу или дозы каждого терапевтического агента, а также подходящие сроки и способы введения.
Соответственно, антитела согласно настоящему изобретению можно вводить одновременно с одним или более другими терапевтическими режимами или агентами. Дополнительные терапевтические режимы или агенты можно использовать для улучшения эффективности или безопасности анти-PVRIG антитела. Также дополнительные терапевтические режимы или агенты можно использовать для лечения того же заболевания или сопутствующего заболевания, а не для изменения действия антитела к PVRIG. Например, антитело к PVRIG согласно настоящему изобретению можно вводить пациенту вместе с химиотерапией, лучевой терапией или как химиотерапией, так и лучевой терапией.
Антитела к PVRIG согласно настоящему изобретению можно вводить в комбинации с одним или более другими профилактическими или терапевтическими агентами, включая, но не ограничиваясь этим, цитотоксические агенты, химиотерапевтические агенты, цитокины, агенты-ингибиторы роста, антигормональные агенты, ингибиторы киназ, антиангиогенные агенты, кардиопротекторы, иммуностимулирующие агенты, иммуносупрессивные агенты, агенты, которые стимулируют пролиферацию гематологических клеток, ингибиторы ангиогенеза, ингибиторы протеинтирозинкиназ (ПТК) или другие терапевтические агенты.
В соответствии по меньшей мере с некоторыми вариантами реализации изобретения анти-PVRIG иммунные молекулы можно применять в комбинации с любым известным в данной области техники стандартом лечения рака (информацию можно найти, например, на http://www.cancer.gov/cancertopics).
Например, комбинированная терапия может включать применение анти-PVRIG антитела в комбинации по меньшей мере с одним другим терапевтическим или иммуномодулирующим агентом, другими соединениями или вариантами иммунотерапии, или иммуностимулирующей стратегией, описанными в данном документе, включая, но не ограничиваясь этим, противоопухолевые вакцины, адоптивную Т-клеточную терапию, снижение количества Tрег, антитела (например, бевацизумаб, эрбитукс), пептиды, пептитела, небольшие молекулы, химиотерапевтические агенты, такие как цитотоксические и цитостатические агенты (например, паклитаксел, цисплатин, винорелбин, доцетаксел, гемцитабин, темозоломид, иринотекан, 5FU, карбоплатин), иммунологические модификаторы, такие как интерфероны и интерлейкины, иммуностимулирующие антитела, гормоны роста или другие цитокины, фолиевую кислоту, витамины, минералы, ингибиторы ароматазы, РНКи, ингибиторы гистондеацетилазы, ингибиторы протеасом, доксорубицин (адриамицин), цисплатин, блеомицина сульфат, кармустин, хлорамбуцил и циклофосфамид, гидроксимочевина, которые сами по себе эффективны только при уровнях, токсичных или субтоксичных для пациента. Цисплатин вводят внутривенно в количестве 100 мг/дозу один раз в четыре недели, а адриамицин вводят внутривенно в количестве 60-75 мг/мл на дозу один раз каждый 21 день.
В соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения терапевтические агенты, которые можно применять в комбинации с анти-PVRIG антителами, представляют собой другие усиливающие агенты, которые усиливают противоопухолевые ответы, например, другие антитела против иммунных контрольных точек или другие усиливающие агенты, которые главным образом направлены на повышение эндогенных противоопухолевых ответов, такие как лучевая терапия, криотерапия, традиционная/классическая химиотерапия, усиливающая противоопухолевые иммунные ответы, нацеленная терапия, усиливающая противоопухолевые иммунные ответы, антиангиогенная терапия, терапевтические агенты, нацеленные на иммуносупрессорные клетки, такие как Tрег и СКМП, иммуностимулирующие антитела, цитокиновая терапия, терапевтические противораковые вакцины, адоптивный перенос клеток.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с бифосфонатами, в особенности амино-бифосфонатами (АБФ), которые продемонстрировали наличие противораковой активности. Некоторые виды активности, связанные с АБФ, состоят в действии на человеческие γδ T-клетки, которые находятся по обе стороны границы врожденного и адаптивного иммунитета и обладают сильной противораковой активностью.
Нацеленная терапия также может стимулировать опухолеспецифический иммунный ответ, индуцируя иммуногенную гибель опухолевых клеток или задействуя иммунные эффекторные механизмы (Galluzzi et al, 2012, Nature Reviews - Drug discovery, том 11, страницы 215-233).
В соответствии по меньшей мере с некоторыми вариантами реализации изобретения нацеленная терапия, применяемая в виде агентов для комбинирования с анти-PVRIG иммунными молекулами для лечения рака, соответствует описанной в данном документе.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с терапевтическими агентами, нацеленными на регуляторные иммуносупрессорные клетки, такие как регуляторные Т-клетки (Tрег) и супрессорные клетки миелоидного происхождения (СКМП). Большое число обычно применяемых химиотерапевтических препаратов проявляют неспецифическое нацеливание на Tрег и снижают число или иммуносупрессивную способность Tрег или СКМП (Facciabene A. et al 2012 Cancer Res; 72(9) 2162-71; Byrne WL. et al 2011, Cancer Res. 71:691520; Gabrilovich DI. and Nagaraj S, Nature Reviews 2009 том 9, страницы 162-174). В связи с этим метрономная терапия некоторыми химиотерапевтическими препаратами приводит к иммуностимулирующему, а не иммуносупрессивному действию, посредством модуляции регуляторных клеток. Таким образом, в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения, анти-PVRIG иммунную молекулу для иммунотерапии рака применяют в комбинации с лекарственными препаратами, выбранными, без ограничений, из циклофосфамида, гемцитабина, митоксантрона, флударабина, доцетаксела, паклитаксела, талидомида и производных талидомида.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с новыми Трег-специфическими нацеленными агентами, включая: 1) антитела, снижающие число клеток или уничтожающие клетки, которые прямо нацелены на Трег посредством распознавания рецепторов клеточной поверхности Трег, такие как анти-CD25 mAb даклизумаб, базиликсимаб, или 2) лиганд-направленные токсины, такие как денилейкин дифтитокс (Онтак) - слитый белок человеческого IL-2 и дифтерийного токсина, или LMB-2 - слитый белок scFv против CD25 и экзотоксина синегнойной палочки, и 3) антитела, нацеленные на рецепторы клеточной поверхности Трег, такие как CTLA4, PD-1, OX40 и GITR, или 4) антитела, небольшие молекулы или слитые белки, нацеленные на другие NK-рецепторы, такие, как указанные ранее.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с любыми из описанных ниже методов для нарушения индукции и/или функции Трег, включая агонисты ТПР (толл-подобных рецепторов); агенты, затрагивающие аденосинергический путь, такие как ингибиторы эктонуклеотидаз или ингибиторы рецептора аденозина A2A; ингибиторы TGF-β, такие как фрезолимумаб, лерделимумаб, метелимумаб, трабедерсен, LY2157299, LY210976; блокирование привлечения Трег в опухолевые ткани, включая ингибиторы рецепторов хемокинов, таких как путь CCR4/CCL2/CCL22.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с любыми из описанных ниже методов для ингибирования иммуносупрессивного опухолевого микроокружения, включая ингибиторы цитокинов и ферментов, которые проявляют иммуносупрессивную активность, такие как ингибиторы ИДО (индоламин-2,3-диоксигеназы); ингибиторы противовоспалительных цитокинов, которые стимулируют иммуносупрессивное микроокружение, таких как IL-10, IL-35, IL-4 и IL-13; бевацизумаб®, который снижает количество Трег и способствует дифференцировке ДК.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с любыми из описанных ниже методов для нацеливания на СКМП (супрессорные клетки миелоидного происхождения), включая стимуляцию их дифференцировки в зрелые миелоидные клетки, которые не обладают супрессивными функциями, витамином D3 или метаболитами витамина А, такими как ретиноевая кислота, полностью транс-ретиноевая кислота (ПТРК); ингибирование супрессивной активности СКМП ингибиторами COX2, подобным ингибиторам фосфодиэстеразы 5 силденафилом, ингибиторами АФК, такими как нитроаспирин.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с иммуностимулирующими антителами или другими агентами, которые усиливают противоопухолевые иммунные ответы (Pardoll J Exp Med. 2012; 209(2): 201-209). Иммуностимулирующие антитела стимулируют противоопухолевый иммунитет посредством прямой модуляции иммунных функций, т.е. блокирования других ингибиторных мишеней или усиления иммуностимулирующих белков. В соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения анти-PVRIG иммунные молекулы для иммунотерапии рака применяют в комбинации с антагонистическими антителами, нацеленными на дополнительные иммунные контрольные точки, включая анти-CTLA4 mAb, такие как ипилимумаб, тремелимумаб; анти-PD-1, такими как ниволумаб BMS-936558/ MDX-1106/ONO-4538, AMP224, CT-011, MK-3475, антагонистами анти-PDL-1, такими как BMS-936559/ MDX-1105, MEDI4736, RG-7446/MPDL3280A; анти-LAG-3, такими как IMP-321), анти-TIM-3, анти-BTLA, анти-B7-H4, анти-B7-H3, анти-VISTA; агонистическими антителами, нацеленными на иммуностимулирующие белки, включая анти-CD40 mAb, такими как CP-870,893, лукатумумаб, дацетузумаб; анти-CD137 mAb, такими как BMS-663513 урелумаб, PF-05082566; анти-OX40 mAb, такими как анти-OX40; анти-GITR mAb, такими как TRX518; анти-CD27 mAb, такими как CDX-1127; и анти-ICOS mAb.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с цитокинами. Большое количество цитокинов находятся в доклинической или клинической разработке в качестве агентов для иммунотерапии рака, усиливающих противоопухолевые иммунные ответы, и включают, помимо прочего: IL-2, IL-7, IL-12, IL-15, IL-17, IL-18 и IL-21, IL-23, IL-27, ГМ-КСФ, IFNα (интерферон α), IFNβ и IFNγ. Однако терапевтическая эффективность часто нивелируется тяжелыми побочными эффектами и слабыми фармакокинетическими свойствами. Следовательно, кроме системного введения цитокинов можно применять множество стратегий для доставки терапевтических цитокинов и их локализации в месте опухоли с целью улучшения их фармакокинетики, а также их эффективности и/или токсичности, включая слитые молекулы антитело-цитокин (иммуноцитокины), химическую конъюгацию с полиэтиленгликолем (ПЭГилирование), трансгенную экспрессию цитокинов в аутологичных цельных опухолевых клетках, внесение генов цитокинов в ДНК-вакцины, рекомбинантные вирусные векторы для доставки генов цитокинов и т.д. В случае иммуноцитокинов слияние цитокинов с опухолеспецифическими антителами или фрагментами антител обеспечивает нацеленную доставку и, следовательно, улучшает эффективность и фармакокинетику и снижает побочные эффекты.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с противораковыми вакцинами. Терапевтические противораковые вакцины обеспечивают улучшенное примирование Т-клеток и улучшенное представление антигенов и могут применяться в качестве терапевтических агентов для усиления противоопухолевых иммунных ответов (Mellman I. et al., 2011, Nature, 480:22-29; Schlom J, 2012, J Natl Cancer Inst; 104:599-613).
Несколько типов терапевтических противораковых вакцин находятся в доклинической или клинической разработке. Они включают, например:
1) Вакцины на основе цельных опухолевых клеток, в которых раковые клетки, удаленные во время хирургической операции, обрабатывают так, чтобы усилить их иммуногенность, и инъецируют пациенту для индукции иммунных ответов против антигенов в опухолевых клетках. Вакцина на основе опухолевых клеток может быть аутологичной, т.е. полученной из собственной опухоли пациента, или аллогенной, которая, как правило, содержит две или три стабильные и охарактеризованные опухолевые клеточные линии данного типа опухоли, такой как вакцинные платформы GVAX.
2) Вакцины на основе опухолевых антигенов, в которых опухолевый антиген (или комбинацию нескольких опухолевых антигенов), обычно белков или пептидов, вводят для того, чтобы стимулировать иммунную систему (возможно, с адъювантом и/или с иммунными модуляторами или аттрактантами дендритных клеток, такими как ГМ-КСФ). Опухолевые антигены могут быть специфическими в отношении определенного типа рака, но они не создаются для определенного пациента.
3) Векторные вакцины на основе опухолевых антигенов и ДНК-вакцины можно применять в качестве способа обеспечения постоянного источника антигенов, чтобы стимулировать противоопухолевый иммунный ответ. Векторы, кодирующие опухолевые антигены, инъецируются пациенту (возможно с провоспалительными или другими аттрактантами, такими как ГМ-КСФ), поглощаются клетками in vivo для получения специфических антигенов, которые впоследствии спровоцируют необходимый иммунный ответ. Векторы можно применять для доставки более чем одного опухолевого антигена за один раз для повышения иммунного ответа. Кроме того, векторы на основе рекомбинантных вирусов, бактерий или дрожжей должны запускать свои собственные иммунные ответы, которые также могут усиливать общий иммунный ответ.
4) Вакцины на основе онколитических вирусов, такие как OncoVex/T-VEC, которые включают внутриопухолевую инъекцию вируса простого герпеса с зависимой от условий репликацией, который инфицирует преимущественно раковые клетки. Вирус, который сконструирован так, чтобы экспрессировать ГМ-КСФ, способен к репликации внутри раковой клетки, вызывая ее лизис, высвобождая новые вирусы и ряд опухолевых антигенов и секретируя в процессе ГМ-КСФ. Таким образом, такие вакцины на основе онколитических вирусов усиливают функцию ДК в опухолевом микроокружении для стимуляции противоопухолевых иммунных ответов.
5) Вакцины на основе дендритных клеток (Palucka and Banchereau, 2102, Nat. Rev. Cancer, 12(4):265-277): Дендритные клетки фагоцитируют опухолевые клетки и представляют опухолевые антигены опухолеспецифическим Т-клеткам. В этом подходе ДК выделяют из организма ракового пациента и примируют для представления опухолеспецифическим Т-клеткам. Для этого можно использовать несколько способов: ДК нагружают опухолевыми клетками или лизатами; ДК нагружают слитыми белками или пептидами опухолевых антигенов; опухолевые антигены связывают с нацеленными на ДК mAb. ДК обрабатывают в присутствии стимулирующего фактора (такого как ГМ-КСФ), активируют и обеспечивают созревание ex vivo, а затем повторно инфузируют пациенту, чтобы спровоцировать иммунный ответ на раковые клетки. Дендритные клетки также можно примировать in vivo путем инъекции пациенту облученных цельных опухолевых клеток, сконструированных так, чтобы секретировать стимулирующие цитокины (такие как ГМ-КСФ). Подобные подходы можно осуществлять с моноцитами. Сипулеуцел-T (Провендж), терапевтическая противораковая вакцина, которая была одобрена для лечения запущенного рака простаты, является примером вакцины на основе дендритных клеток.
В некоторых вариантах реализации изобретения анти-PVRIG антитела применяют в комбинации с адоптивной Т-клеточной терапией или адоптивным переносом клеток (АПК), который включает ex vivo определение и размножение аутологичных опухолеспецифических Т-клеток природного происхождения, которые затем адоптивно переносят назад в организм ракового пациента (Restifo et al, 2013, Cancer Immunol. Immunother.62(4):727-36 (2013) Epub Dec 4 2012). Клетки, которые инфузируют обратно пациенту после ex vivo размножения, могут попадать в опухоль и опосредовать ее разрушение. Перед этим адоптивным переносом иммунитет хозяев может быть снижен посредством лучевой терапии и/или химиотерапии. Комбинация лимфоистощения, адоптивного переноса клеток и фактора роста Т-клеток (такого как IL-2) может приводить к продолжительному уничтожению опухолей у опухолевых пациентов. Более новый подход включает ex vivo генетическую модификацию нормальных Т-клеток периферической крови для придания специфичности в отношении опухолеспецифических антигенов. Например, клоны РТК или Т-клеток с особенно хорошими противоопухолевыми ответами можно вставлять в вирусные экспрессионные векторы и использовать для инфицирования аутологичных Т-клеток от подлежащего лечению пациента. Другим вариантом является применение химерных антигенных рецепторов (ХАГ), которые по существу представляют собой химерную молекулу иммуноглобулин-РТК, также известную как Т-тело. ХАГ обладают антитело-подобной специфичностью и распознают нерестриктированные по ГКГС структуры на поверхности клеток-мишеней (внеклеточный связывающий мишень модуль), привитые во внутриклеточные домены РТК, способные активировать Т-клетки (Restifo et al Cancer Immunol. Immunother.62(4):727-36 (2013) Epub Dec 4 2012; и Shi et al, Nature 493:111-115 2013).
Антитела к PVRIG и один или более других терапевтических агентов можно вводить в любом порядке или одновременно. Композиция может быть связана с агентом (в виде иммунокомплекса) или ее можно вводить отдельно от агента. В последнем случае (раздельное введение) композицию можно вводить до, после или одновременно с агентом или можно применять совместно с другими известными видами терапии, например, противораковой терапией, например, облучением.
Совместное введение гуманизированных анти-PVRIG иммунных молекул в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения с химиотерапевтическими агентами обеспечивает наличие двух противораковых агентов с разными механизмами действия, что приводит к цитотоксическому действию в отношении человеческих опухолевых клеток. Такое совместное введение может решить проблемы, связанные с развитием резистентности к лекарственным препаратам или изменением антигенности опухолевых клеток, которые делают их невосприимчивыми к антителу. В других вариантах реализации изобретения субъекта можно дополнительно лечить агентом, который модулирует, например, усиливает или ингибирует, экспрессию или активность рецепторов Fcγ или Fcγ, путем, например, лечения субъекта цитокином.
Также в качестве терапевтических агентов можно использовать специфические в отношении мишени эффекторные клетки, например, эффекторные клетки, связанные с композициями (например, человеческими антителами, мультиспецифическими и биспецифическими молекулами), в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения. Эффекторные клетки для нацеливания могут представлять собой человеческие лейкоциты, такие как макрофаги, нейтрофилы или моноциты. Другие клетки включают эозинофилы, естественные клетки-убийцы и другие клетки, несущие рецепторы IgG или IgA. При желании эффекторные клетки можно получать от подлежащего лечению субъекта. Специфические в отношении мишени эффекторные клетки можно вводить в виде суспензии клеток в физиологически приемлемом растворе. Число вводимых клеток может быть порядка от 108 до 109, но будет варьироваться в зависимости от терапевтической цели. В общем случае количество будет достаточным для достижения локализации вблизи клетки-мишени, например, опухолевой клетки, экспрессирующей белки PVRIG, и для осуществления уничтожения клеток, например, путем фагоцитоза. Пути введения также могут варьироваться.
Терапию специфическими в отношении мишени эффекторными клетками можно проводить в сочетании с другими методами уничтожения клеток-мишеней. Например, противоопухолевую терапию композициями (например, человеческими антителами, мультиспецифическими и биспецифическими молекулами) в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения и/или эффекторными клетками, усиленными этими композициями, можно применять в сочетании с химиотерапией. Кроме того, можно применять комбинированную иммунотерапию для направления двух разных цитотоксических эффекторных популяций на отторжение опухолевых клеток. Например, анти-PVRIG иммунные молекулы, связанные с анти-Fc-γRI или анти-CD3, можно использовать в сочетании со специфическими к рецепторам IgG или IgA связывающими агентами.
Биспецифические и мультиспецифические молекулы в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения также можно применять для модуляции уровней FcγR или FcγR на эффекторных клетках, например, путем кэпирования и удаления рецепторов на клеточной поверхности. В этих целях также можно применять смеси анти-Fc рецепторов.
Терапевтические композиции (например, человеческие антитела, мультиспецифические и биспецифические молекулы с альтернативными каркасами и иммуноконъюгаты) в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения, которые содержат участки связывания комплемента, такие как части из IgG1, -2 или -3 или IgM, которые связывают комплемент, также можно применять в присутствии комплемента. В одном варианте реализации изобретения ex vivo обработку популяции клеток, содержащей клетки-мишени, связывающим агентом в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения и соответствующими эффекторными клетками можно дополнить путем добавления комплемента или сыворотки, содержащей комплемент. Фагоцитоз клеток-мишеней, покрытых связывающим агентом в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения, можно улучшить путем связывания белков комплемента. В другом варианте реализации изобретения клетки-мишени, покрытые композициями (например, человеческими антителами, мультиспецифическими и биспецифическими молекулами) в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения, также можно лизировать с помощью комплемента. В другом варианте реализации изобретения композиции в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения не активируют комплемент.
Терапевтические композиции (например, человеческие антитела, мультиспецифические и биспецифические молекулы с альтернативными каркасами и иммуноконъюгаты) в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения также можно вводить вместе с комплементом. Таким образом, в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения существуют композиции, содержащие человеческие антитела, мультиспецифические или биспецифические молекулы и сыворотку или комплемент. Преимуществом этих композиций является то, что комплемент расположен в непосредственной близости к человеческие антителам, мультиспецифическим или биспецифическим молекулам. В альтернативном варианте человеческие антитела, мультиспецифические или биспецифические молекулы в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения и комплемент или сыворотку можно вводить раздельно.
Анти-PVRIG иммунные молекулы в соответствии по меньшей мере с некоторыми вариантами реализации настоящего изобретения можно применять в качестве нейтрализующих антител. Нейтрализующее антитело (Nab) представляет собой антитело, которое способно связывать и нейтрализовать или ингибировать специфический антиген, тем самым ингибируя биологическое действие. NAb частично или полностью прекращают биологическое действие антигена, блокируя важную поверхностную молекулу, необходимую для его активности, или препятствуя связыванию антигена с его рецептором на клетке-мишени.
В соответствии с дополнительным аспектом настоящего изобретения терапевтические агенты можно применять для предотвращения патологического ингибирования активности Т-клеток, такой как активность, направленная против раковых клеток.
Таким образом, в соответствии с дополнительным аспектом настоящего изобретения предложен способ лечения рака из перечисленных в данном документе и/или осуществления иммунной стимуляции путем введения нуждающемуся в этом субъекту эффективного количества любого из терапевтических агентов и/или фармацевтической композиции, содержащей любой из терапевтических агентов и дополнительно содержащей фармацевтически приемлемый разбавитель или носитель.
В соответствии по меньшей мере с некоторыми вариантами реализации изобретения иммунные клетки, предпочтительно Т-клетки, можно приводить в контакт in vivo или ex vivo с терапевтическими агентами для модуляции иммунных ответов. Приводимые в контакт с терапевтическими агентами Т-клетки могут представлять собой любую клетку, которая экспрессирует рецептор Т-клеток, включая рецепторы α/β и γ/δ Т-клеток. Т-клетки включают все клетки, которые экспрессируют CD3, включая Т-клетки, подгруппы которых также экспрессируют CD4 и CD8. Т-клетки включают как наивные клетки, так и клетки памяти и эффекторные клетки, такие как цитотоксические Т-лимфоциты (ЦТЛ) CD8+. Т-клетки также включают клетки, такие как Th1, Tc1, Th2, Tc2, Th3, Th9, Th17, Th22, Трег, фолликулярные хелперные клетки (TФХ) и клетки Tr1. Т-клетки также включают NKT-клетки iNKT, α/β NKT и γ/δ NKT-клетки и сходные уникальные классы Т-клеточной линии дифференцировки.
Блокирующие PVRIG антитела также можно применять в комбинации с биспецифическими антителами, которые нацеливают экспрессирующие Fcα- или Fcγ-рецепторы эффекторные клетки на опухолевые клетки (смотрите, например, патенты США № 5922845 и 5837243). Биспецифические антитела можно применять для нацеливания на два разных антигена. Например, биспецифические антитела против Fc-рецептора/опухолевого антигена (например, Her-2/neu) применяли для нацеливания макрофагов на участки опухоли. Такое нацеливание может более эффективно активировать опухолеспецифические ответы. Т-клеточная часть этих ответов будет усилена применением блокирования PVRIG. В альтернативном варианте антиген можно доставлять непосредственно к ДК, применяя биспецифические антитела, которые связываются с опухолевым антигеном и специфическим в отношении дендритных клеток маркером клеточной поверхности.
Опухоли уклоняются от иммунологического надзора посредством множества механизмов. Многие из этих механизмов можно преодолеть, инактивируя белки, которые экспрессируются опухолями и являются иммуносупрессивными. Они включают, помимо прочего, TGF-β (Kehrl, J. et al. (1986) J. Exp. Med. 163: 1037-1050), IL-10 (Howard, M. & O'Garra, A. (1992) Immunology Today 13: 198-200) и лиганд Fas (Hahne, M. et al. (1996) Science 274: 1363-1365). Антитела к каждому из этих белков можно применять в комбинации с анти-PVRIG для противодействия иммуносупрессивному агенту и содействия противоопухолевым иммунным ответам со стороны организма-хозяина.
Другие антитела, которые можно применять для активации иммунной отвечаемости организма-хозяина, можно использовать в комбинации с анти-PVRIG. Они включают молекулы на поверхности дендритных клеток, которые активируют функцию ДК и представление антигена. Анти-CD40 антитела способны эффективно замещать активность Т-хелперов (Ridge, J. et al. (1998) Nature 393: 474-478) и могут применяться в сочетании с антителами к PVRIG (Ito, N. et al. (2000) Immunobiology 201 (5) 527-40). Активация антител к Т-клеточным ко-стимуляторным молекулам, таким как OX-40 (Weinberg, A. et al. (2000) Immunol 164: 2160-2169), 4-1BB (Melero, I. et al. (1997) Nature Medicine 3: 682-685 (1997) и ICOS (Hutloff, A. et al. (1999) Nature 397: 262-266), а также антител, которые блокируют активность отрицательных ко-стимуляторных молекул, таких как CTLA-4 (например, патент США № 5811097, ипилимумаб) или BTLA (Watanabe, N. et al. (2003) Nat Immunol 4:670-9), B7-H4 (Sica, G L et al. (2003) Immunity 18:849-61), PD-1, также может обеспечить повышенные уровни активации Т-клеток. В настоящее время трансплантация костного мозга используется для лечения большого количества опухолей гемопоэтического происхождения. Хотя следствием такого варианта лечения является болезнь «трансплантат против хозяина», можно добиться благоприятного терапевтического эффекта посредством ответов трансплантат против опухоли. Блокировку PVRIG можно использовать для повышения эффективности привитых донорных опухолеспецифических клеток.
Также существует несколько экспериментальных протоколов лечения, которые включают ex vivo активацию и размножение антигенспецифических Т-клеток и адоптивный перенос этих клеток в организм реципиентов для нацеливания антигенспецифических Т-клеток против опухоли (Greenberg, R. & Riddell, S. (1999) Science 285: 546-51). Эти способы также можно использовать для активации Т-клеточных ответов на инфекционные агенты, такие как ЦМВ. Можно ожидать, что ex vivo активация в присутствии анти-PVRIG иммунных молекул будет повышать частоту и активность адоптивно перенесенных Т-клеток.
Необязательно, антитела к PVRIG можно комбинировать с иммуногенным агентом, таким как раковые клетки, очищенные опухолевые антигены (включая рекомбинантные белки, пептиды и углеводные молекулы), клетки и клетки, трансфицированные генами, кодирующими иммуностимулирующие цитокины (He et al (2004) J. Immunol. 173:4919-28). Неограничивающие примеры противоопухолевых вакцин, которые можно применять, включают пептиды MUC1 для лечения рака толстой кишки, пептиды MUC-1/CEA/TRICOM для лечения рака яичника или опухолевые клетки, трансфицированные так, чтобы экспрессировать цитокин ГМ-КСФ (дополнительно обсуждается ниже).
Было показано, что некоторые опухоли у людей, такие как ПКК, являются иммуногенными. Предполагается, что повышая порог активации Т-клеток путем блокирования PVRIG, можно ожидать активации противоопухолевых ответов в организме-хозяине.
Вероятно, блокирование PVRIG будет наиболее эффективно в комбинации с протоколом вакцинации. Было разработано много экспериментальных стратегий вакцинации против опухолей (смотрите Rosenberg, S., 2000, Development of Cancer Vaccines, ASCO Educational Book Spring: 60-62; Logothetis, C., 2000, ASCO Educational Book Spring: 300-302; Khayat, D. 2000, ASCO Educational Book Spring: 414-428; Foon, K. 2000, ASCO Educational Book Spring: 730-738; также смотрите Restifo, N. and Sznol, M., Cancer Vaccines, Ch. 61, pp. 3023-3043 in DeVita, V. et al. (eds.), 1997, Cancer: Principles and Practice of Oncology. Fifth Edition). В одной из этих стратегий вакцину готовят, используя аутологичные или аллогенные опухолевые клетки. Было показано, что эти клеточные вакцины являются наиболее эффективными в случае трансдукции опухолевых клеток так, чтобы они экспрессировали ГМ-КСФ. Было показано, что ГМ-КСФ является сильным активатором представления антигенов для противоопухолевой вакцинации (Dranoff et al. (1993) Proc. Natl. Acad. Sci U.S.A. 90: 3539-43).
Исследование генной экспрессии и крупномасштабных профилей генной экспрессии в различных опухолях привело к определению так называемых опухолеспецифических антигенов (Rosenberg, S A (1999) Immunity 10: 281-7). Во многих случаях эти опухолеспецифические антигены представляют собой дифференцировочные антигены, экспрессируемые в опухолях и в клетках, из которых происходит опухоль, например, меланоцитарные антигены gp100, антигены MAGE и Trp-2. Более важно то, что многие из этих антигенов могут быть мишенями опухолеспецифических Т-клеток организма-хозяина. Блокирование PVRIG можно применять в сочетании с набором рекомбинантных белков и/или пептидов, экспрессируемых в опухоли, с целью генерации иммунного ответа на эти белки. Обычно иммунная система видит эти белки как собственные антигены и, следовательно, не отвечает на них. Опухолевый антиген также может содержать белок теломеразу, который необходим для синтеза теломер в хромосомах и который экспрессируется в более чем 85% человеческих раков и только в ограниченном количестве соматических тканей (Kim, N et al. (1994) Science 266: 2011-2013). (Эти соматические ткани можно защищать от иммунной атаки различными способами). Опухолевый антиген также может представлять собой «нео-антигены», экспрессируемые в раковых клетках вследствие соматических мутаций, меняющих белковую последовательность или создающих белки слияния между двумя неродственными последовательностями (т.е. bcr-ab1 в филадельфийской хромосоме), или идиотип из В-клеточных опухолей.
Другие противоопухолевые вакцины могут содержать белки из вирусов, присутствующие в человеческих раках, таких как вирусы папилломы человека (ВПЧ), вирусы гепатипа (ВГВ и ВГС) и вирус герпеса, ассоциированный с саркомой Капоши (ВГСК). Другой формой опухолеспецифических антигенов, которую можно применять в сочетании с блокированием PVRIG, являются очищенные белки теплового шока (БТШ), выделенные из самой опухолевой ткани. Эти белки теплового шока содержат фрагменты белков из опухолевых клеток и эти БТШ очень эффективны в отношении доставки к антигенпредставляющим клеткам для развития противоопухолевого иммунитета (Suot, R & Srivastava, P (1995) Science 269:1585-1588; Tamura, Y. et al. (1997) Science 278:117-120).
Дендритные клетки (ДК) являются эффективными антигенпредставляющими клетками, которые можно использовать, чтобы примировать антигенспецифические ответы. ДК можно получать ex vivo и нагружать различными белковыми и пептидными антигенами, а также экстрактами опухолевых клеток (Nestle, F. et al. (1998) Nature Medicine 4: 328-332). ДК также можно трансдуцировать генетическими способами, чтобы они экспрессировали эти опухолевые антигены. Также ДК напрямую сливали с опухолевыми клетками в целях иммунизации (Kugler, A. et al. (2000) Nature Medicine 6:332-336). В качестве способа вакцинации иммунизацию ДК можно эффективно комбинировать с блокированием PVRIG, чтобы активировать более сильные противоопухолевые ответы.
Применение терапевтических агентов в соответствии по меньшей мере с некоторыми вариантами реализации изобретения в качестве адъювантов для противораковой вакцинации:
Иммунизация против опухолеассоциированных антигенов (ОАА) является перспективным подходом для терапии и предотвращения рака, но сталкивается с несколькими проблемами и ограничениями, такими как механизмы толерантности, связанный с собственными антигенами, экспрессируемыми опухолевыми клетками. Ко-стимуляторные молекулы, такие как B7.1 (CD80) и B7.2 (CD86), улучшили эффективность генных и клеточных вакцин в животных моделях и изучаются в качестве адъювантов в клинических исследованиях. Этой адъювантной активности можно достичь путем усиления ко-стимуляторного сигнала или путем блокирования ингибиторного сигнала, который передается отрицательными ко-стимуляторами, экспрессируемыми опухолевыми клетками (Neighbors et al., 2008 J Immunother.; 31(7):644-55).
В соответствии по меньшей мере с некоторыми вариантами реализации изобретения в качестве адъюванта для противораковой вакцинации можно использовать любое из поликлональных или моноклональных антител, и/или антигенсвязывающих фрагментов, и/или содержащих их конъюгатов, и/или альтернативных каркасов, специфических к любому из белков PVRIG. В соответствии по меньшей мере с некоторыми вариантами реализации в изобретении предложены способы улучшения иммунизации против ОАА, включающие введение пациенту эффективного количества любого из поликлональных или моноклональных антител, и/или антигенсвязывающих фрагментов, и/или содержащих их конъюгатов, и/или альтернативных каркасов, специфических к любому из белков PVRIG.
В некоторых вариантах реализации в изобретении предложено применение антител к PVRIG для осуществления одной или более функций в организме нуждающегося в этом субъекта: (a) повышения регуляции провоспалительных цитокинов; (b) повышения пролиферации и/или размножения Т-клеток; (c) повышения выработки интерферона-γ или TNF-α Т-клетками; (d) повышения секреции IL-2; (e) стимуляции ответов антител; (f) ингибирования роста раковых клеток; (g) стимуляции антигенспецифического Т-клеточного иммунитета; (h) стимуляции активации Т-клеток CD4+ и/или CD8+; (i) уменьшения супрессии Т-клеток; (j) стимуляции активности NK-клеток; (k) стимуляции апоптоза или лизиса раковых клеток; и/или (l) цитотоксического или цитостатического действия на раковые клетки.
В других вариантах реализации в изобретении предложено применение иммуностимулирующего антитела, его антигенсвязывающего фрагмента или конъюгата в соответствии по меньшей мере с некоторыми вариантами реализации изобретения (необязательно в фармацевтической композиции) для антагонизации по меньшей мере одного ингибирующего иммунитет действия PVRIG.
Такое антитело, его антигенсвязывающий фрагмент или конъюгат необязательно и предпочтительно опосредует одно из следующих действий:
(i) повышение иммунного ответа, (ii) повышение активации αβ и/или γδ Т-клеток, (iii) повышение цитотоксической Т-клеточной активности, (iv) повышение активности NK- и/или NKT-клеток, (v) уменьшение супрессии αβ и/или γδ Т-клеток, (vi) повышение секреции провоспалительных цитокинов, (vii) повышение секреции IL-2; (viii) повышение выработки интерферона-γ, (ix) повышение ответа Th1, (x) снижения ответа Th2, (xi) снижение числа или уничтожение клеток и/или активности по меньшей мере одной из регуляторных Т-клеток (Трег).
Оценка лечения
В общем случае анти-PVRIG антитела согласно изобретению вводят пациентам с раком, а эффективность оценивают с помощью большого количества способов, как описано в данном документе. Таким образом, хотя можно проводить стандартные оценки эффективности, такие как оценка раковой нагрузки, размера опухоли, оценка наличия или степени метастазов и т.д., иммуно-онкологические виды лечения можно оценивать также на основании оценки иммунного статуса. Это можно делать с помощью большого количества способов, включая in vitro и in vivo методы анализа. Например, можно проводить оценку изменений иммунного статуса (например, наличия Т-клеток ICOS+ CD4+ после лечения ипилимумабом) наряду со «старомодными» измерениями, такими как опухолевая нагрузка, размер, инвазивность, поражение лимфатических узлов, метастазы и т.д. Таким образом, можно оценить любые или все показатели: ингибиторное действие PVRIG на активацию или пролиферацию Т-клеток активацию или пролиферацию Т-клеток CD4+, CD8+ (ЦТЛ), опосредованную Т-клетками CD8+ цитотоксическую активность и/или опосредованное ЦТЛ снижение числа клеток, активность NK-клеток и опосредованное NK-клетками снижение числа клеток, усиливающее действие PVRIG на дифференцировку и пролиферацию клеток Трег и опосредованную Трег или супрессорными клетками миелоидного происхождения (СКМП) иммуносупрессию или иммунную толерантность, и/или действие PVRIG на выработку иммунными клетками провоспалительных цитокинов, например, выработку IL-2, IFN-γ или TNF-α Т-клетками или другими иммунными клетками.
В некоторых вариантах реализации изобретения оценку лечения проводят путем оценивания пролиферации иммунных клеток, используя, например, метод растворения CFSE, внутриклеточное окрашивание Ki67 иммунных эффекторных клеток и метод включения 3Н-тимидина.
В некоторых вариантах реализации изобретения оценку лечения проводят путем оценки повышения генной экспрессии или повышения белковых уровней связанных с активацией маркеров, включая один или более из: CD25, CD69, CD137, ICOS, PD1, GITR, OX40, и клеточной дегрануляции, определяемой по поверхностной экспрессии CD107A.
В общем случае анализ генной экспрессии проводят так, как известно в данной области техники. Смотрите, например, Goodkind et al., Computers and Chem. Eng. 29(3):589 (2005), Han et al., Bioinform. Biol. Insights 11/15/15 9(Suppl. 1):29-46, Campo et al., Nod. Pathol. 2013 Jan; 26 suppl. 1:S97-S110, методы измерения генной экспрессии из которых явным образом включены в данный документ посредством ссылки.
В общем случае определение белковой экспрессии также проводят, как известно в данной области техники, смотрите, например, Wang et al., Recent Advances in Capillary Electrophoresis-Based Proteomic Techniques for Biomarker Discovery, Methods. Mol. Biol. 2013:984:1-12; Taylor et al, BioMed Res. Volume 2014, Article ID 361590, 8 pages, Becerk et al., Mutat. Res 2011 June 17:722(2): 171-182, методы измерения из которых явным образом включены в данный документ посредством ссылки.
В некоторых вариантах реализации изобретения оценку лечения проводят путем оценивания цитотоксической активности, определяемой по жизнеспособности клеток-мишеней посредством оценки многочисленных клеточных параметров, таких как ферментативная активность (включая активность протеаз), проницаемость клеточной мембраны, адгезивность клеток, выработка АТФ, выработка коферментов и активность поглощения нуклеотидов. Конкретные примеры этих методов анализа включают, но не ограничиваются этим, окрашивание трипановым синим или PI, метод высвобождения 51Cr или 35S, активность ЛДН, MTT- и/или WST-тесты, анализ с кальцеином-AM, анализ на основе люминесценции и другие.
В некоторых вариантах реализации изобретения оценку лечения проводят путем оценивания Т-клеточной активности, определяемой по выработке цитокинов, проводимой внутриклеточно в культуральном супернатанте, с использованием цитокинов, включающих, но не ограничивающихся этим, IFNγ, TNFα, ГМ-КСФ, IL2, IL6, IL4, IL5, IL10, IL13, с помощью хорошо известных методов.
Соответственно, оценку лечения можно проводить, используя методы анализа, которые оценивают один или более из следующих показателей: (i) повышение иммунного ответа, (ii) повышение активации αβ и/или γδ Т-клеток, (iii) повышение цитотоксической Т-клеточной активности, (iv) повышение активности NK- и/или NKT-клеток, (v) уменьшение супрессии αβ и/или γδ Т-клеток, (vi) повышение секреции провоспалительных цитокинов, (vii) повышение секреции IL-2; (viii) повышение выработки интерферона-γ, (ix) повышение ответа Th1, (x) снижение ответа Th2, (xi) снижение числа или уничтожение клеток и/или активности по меньшей мере одной из регуляторных Т-клеток (Трег).
Методы анализа для определения эффективности
В некоторых вариантах реализации изобретения активацию Т-клеток оценивают, используя анализ реакции смешанной культуры лимфоцитов (РСКЛ), описанный в Пример 22. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение иммунных ответов, определяемое, например, по фосфорилированию или дефосфорилированию разных факторов или по другим посттрансляционным модификациям. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение активации αβ и/или γδ Т-клеток, определяемое, например, по секреции цитокинов или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD137, CD107a, PD1 и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение цитотоксической Т-клеточной активности, определяемое, например, по непосредственному уничтожению клеток-мишеней, таких как, например, раковые клетки, или по секреции цитокинов, или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD137, CD107a, PD1 и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение активности NK- и/или NKT-клеток, определяемое, например, по непосредственному уничтожению клеток-мишеней, таких как, например, раковые клетки, или по секреции цитокинов, или по изменениям в экспрессии активационных маркеров, таких как, например, CD107a и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение супрессии αβ и/или γδ Т-клеток, определяемое, например, по секреции цитокинов или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD137, CD107a, PD1 и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение секреции провоспалительных цитокинов, определяемое, например, методом ELISA или методами с применением гранул Luminex или Multiplex, или методом анализа внутриклеточного окрашивания и FACS, или методом Alispot и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение секреции IL-2, определяемое, например, методом ELISA или методами с применением гранул Luminex или Multiplex, или методом анализа внутриклеточного окрашивания и FACS, или методом Alispot и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение выработки интерферона-γ, определяемое, например, методом ELISA или методами с применением гранул Luminex или Multiplex, или методом анализа внутриклеточного окрашивания и FACS, или методом Alispot и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение ответа Th1, определяемое, например, по секреции цитокинов или по изменениям в экспрессии активационных маркеров. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение ответа Th2, определяемое, например, по секреции цитокинов или по изменениям в экспрессии активационных маркеров. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение числа клеток и/или активности по меньшей мере одной из регуляторных Т-клеток (Трег), определяемое, например, методом проточной цитометрии или методом ИГХ. Снижение ответа свидетельствует об иммуностимулирующей активности. Соответствующие значения снижения, как и повышения, описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение числа клеток макрофагов M2, определяемое, например, методом проточной цитометрии или методом ИГХ. Снижение ответа свидетельствует об иммуностимулирующей активности. Соответствующие значения снижения, как и повышения, описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение проонкогенной активности макрофагов M2, определяемое, например, по секреции цитокинов или по изменениям в экспрессии активационных маркеров. Снижение ответа свидетельствует об иммуностимулирующей активности. Соответствующие значения снижения, как и повышения, описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение числа нейтрофилов N2, определяемое, например, методом проточной цитометрии или методом ИГХ. Снижение ответа свидетельствует об иммуностимулирующей активности. Соответствующие значения снижения, как и повышения, описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение проонкогенной активности нейтрофилов N2, определяемое, например, по секреции цитокинов или по изменениям в экспрессии активационных маркеров. Снижение ответа свидетельствует об иммуностимулирующей активности. Соответствующие значения снижения, как и повышения, описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение ингибирования активации Т-клеток, определяемое, например, по секреции цитокинов или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD137, CD107a, PD1 и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение ингибирования активации ЦТЛ, определяемое, например, по непосредственному уничтожению клеток-мишеней, таких как, например, раковые клетки, или по секреции цитокинов, или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD137, CD107a, PD1 и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение истощения αβ и/или γδ Т-клеток, определяемое, например, по изменениям в экспрессии активационных маркеров. Снижение ответа свидетельствует об иммуностимулирующей активности. Соответствующие значения снижения, как и повышения, описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение ответа αβ и/или γδ Т-клеток, определяемое, например, по секреции цитокинов или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD137, CD107a, PD1 и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение стимуляции антигенспецифических вторичных ответов, определяемое, например, по секреции цитокинов или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD45RA, CCR7 и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение апоптоза или лизиса раковых клеток, определяемое, например, методами анализа цитотоксичности, такими как, например, методы MTT, высвобождения Cr, кальцеина-АМ, или методами на основе проточной цитометрии, такими как, например, растворение CFSE или окрашивание пропидиум иодидом, и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение стимуляции цитотоксического и цитостатического действия на раковые клетки, определяемое, например, методами анализа цитотоксичности, такими как, например, методы MTT, высвобождения Cr, кальцеина-АМ, или методами на основе проточной цитометрии, такими как, например, растворение CFSE или окрашивание пропидиум иодидом, и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение непосредственного уничтожения раковых клеток, определяемое, например, методами анализа цитотоксичности, такими как, например, методы MTT, высвобождения Cr, кальцеина-АМ, или методами на основе проточной цитометрии, такими как, например, растворение CFSE или окрашивание пропидиум иодидом, и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение активности Th17, определяемое, например, по секреции цитокинов или по пролиферации, или по изменениям в экспрессии активационных маркеров. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения в анализе сигнального пути измеряют повышение или снижение индукции комплементзависимой цитотоксичности и/или антителозависимой клеточноопосредованной цитотоксичности, определяемое, например, методами анализа цитотоксичности, такими как, например, методы MTT, высвобождения Cr, кальцеина-АМ, или методами на основе проточной цитометрии, такими как, например, растворение CFSE или окрашивание пропидиум иодидом, и т.д. Повышение активности свидетельствует об иммуностимулирующей активности. Соответствующие значения повышения активности описаны ниже.
В одном варианте реализации изобретения активацию Т-клеток определяют, например, по непосредственному уничтожению клеток-мишеней, таких как, например, раковые клетки, или по секреции цитокинов, или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD137, CD107a, PD1 и т.д. В случае Т-клеток повышение пролиферации, числа активационных маркеров клеточной поверхности (например, CD25, CD69, CD137, PD1), цитотоксичности (способности уничтожать клетки-мишени) и выработки цитокинов (например, IL-2, IL-4, IL-6, IFNγ, TNF-a, IL-10, IL-17A) свидетельствовало бы об иммунной модуляции, которая согласуется с повышенным уничтожением раковых клеток.
В одном варианте реализации изобретения активацию NK-клеток определяют, например, по непосредственному уничтожению клеток-мишеней, таких как, например, раковые клетки, или по секреции цитокинов, или по пролиферации, или по изменениям в экспрессии активационных маркеров, таких как, например, CD107a и т.д. В случае NK-клеток повышение пролиферации, цитотоксичности (способности уничтожать клетки-мишени и повышения экспрессии CD107a, гранзимов и перфорина), выработки цитокинов (например, IFNγ и TNF) и экспрессии рецепторов клеточной поверхности (например, CD25) свидетельствовало бы об иммунной модуляции, которая согласуется с повышенным уничтожением раковых клеток.
В одном варианте реализации изобретения активацию γδ Т-клеток определяют, например, по секреции цитокинов или по пролиферации, или по изменениям в экспрессии активационных маркеров.
В одном варианте реализации изобретения активацию Th1-клеток определяют, например, по секреции цитокинов или по изменениям в экспрессии активационных маркеров.
Соответствующие значения повышения активности или ответа (или снижения, в зависимости от обстоятельств, как указано выше) составляют повышение на от 10%, 20%, 30%, 40%, 50%, 60%, 70%, 80%, 90%, 95% или 98 до 99% по сравнению с сигналом в эталонном образце или контрольных образцах, например, в исследуемых образцах, которые не содержат анти-PVRIG антитело согласно изобретению. Аналогично, значение повышения, по меньшей мере в один, два, три, четыре или пять раз превышающее значения для эталонных или контрольных образцов, демонстрирует эффективность.
Лечение патогенных инфекций
В соответствии по меньшей мере с некоторыми вариантами реализации изобретения анти-PVRIG антитела, необязательно, можно применять для лечения инфекционных заболеваний по тем же причинам, по которым возможно лечение рака: хронические инфекции часто характеризуются разной степенью функционального нарушения вирус-специфических Т-клеточных ответов, и этот недостаток является основной причиной неспособности организма-хозяина уничтожать персистирующий патоген. Хотя функциональные эффекторные Т-клетки изначально появляются на ранних стадиях инфицирования, во время хронической инфекции они постепенно теряют функциональность в результате постоянного воздействия чужеродного антигена, что приводит к истощению Т-клеток. Истощенные Т-клетки экспрессируют высокие уровни нескольких ко-ингибиторных рецепторов, таких как CTLA-4, PD-1 и LAG3 (Crawford et al., Curr Opin Immunol. 2009;21:179-186; Kaufmann et al., J Immunol 2009;182:5891-5897, Sharpe et al., Nat Immunol 2007;8:239-245). Сверхэкспрессию PD-1 истощенными Т-клетками наблюдали в клинических условиях у пациентов, страдающих от хронических вирусных инфекций, включая ВИЧ, ВГС и ВТВ (Crawford et al., Curr Opin Immunol 2009;21:179-186; Kaufmann et al., J Immunol 2009;182:5891-5897, Sharpe et al., Nat Immunol 2007;8:239-245). Проводили исследования этого пути для дополнительных патогенов, включая другие вирусы, бактерии и паразитов (Hofmeyer et al., J Biomed Biotechnol. Vol 2011, Art. ID 451694, Bhadra et al., Proc Natl. Acad Sci. 2011;108(22):9196-201). Например, используя модель сепсиса, индуцированного стандартным методом лигирования и пункции слепой кишки, было показано, что путь PD-1 вовлечен в регуляцию бактериальной инфекции. Отсутствие PD-1 у мышей с нокаутом защищало от индуцированной сепсисом гибели в этой модели (Huang et al., PNAS 2009: 106; 6303-6308).
Истощение Т-клеток можно обратить вспять, блокируя ко-ингибиторные пути, такие как PD-1 или CTLA-4 (Rivas et al., J Immunol. 2009;183:4284-91; Golden-Mason et al., J Virol. 2009;83:9122-30; Hofmeyer et al., J Biomed Biotechnol. Vol 2011, Art. ID 451694), обеспечивая, таким образом, восстановление противовирусной иммунной функции. Терапевтический потенциал блокирования ко-ингибирования для лечения вирусных инфекций подробно изучали путем блокирования пути PD-1/PD-L1, которое оказалось эффективным в нескольких животных моделях инфекции, включая острую и хроническую инфекцию, вызываемую вирусом иммунодефицита обезьян (ВИО), у макаков резус (Valu et al., Nature 2009;458:206-210), и в мышиных моделях хронической вирусной инфекции, например, вызываемой вирусом лимфоцитарного хориоменингита (ВЛХМ) (Barber et al., Nature. 2006;439:682-7), и в модели инфекции вирусом энцефаломиелита мышей Тейлера (ВЭМТ) у мышей SJL/J (Duncan and Miller PLoS One. 2011;6:e18548). В этих моделях блокирование PD-1/PD-L1 улучшало противовирусные ответы и стимулировало выведение персистирующих вирусов. Кроме того, блокирование PD-1/PD-L1 повышает гуморальный иммунитет, который проявляется в виде повышенной выработки специфических противовирусных антител в плазме крови, что в комбинации с улучшенными клеточными ответами приводит к снижению вирусной нагрузки в плазме крови и повышению выживаемости.
В контексте данного документа термины «инфекционное нарушение и/или заболевание» и/или «инфекция», употребляемые взаимозаменяемо, включают любое нарушение, заболевание и/или патологическое состояние, вызываемое присутствием и/или ростом патогенного биологического агента в отдельном организме-хозяине. В контексте данного документа термин «инфекция» включают нарушение, заболевание и/или патологическое состояние, как указано выше, проявляющееся в виде клинически очевидного недомогания (т.е. характерных медицинских признаков и/или симптомов заболевания) и/или являющееся асимптоматическим в течение большей части или всего курса. В контексте данного документа термин «инфекция» также включают нарушение, заболевание и/или патологическое состояние, вызываемое персистенцией чужеродного антигена, которая приводит к истощенному Т-клеточному фенотипу, характеризуемому нарушением функциональности, которое проявляет себя в виде сниженной пролиферации и выработки цитокинов. В контексте данного документа термины «инфекционное нарушение и/или заболевание» и/или «инфекция» дополнительно включают любое из приведенных ниже инфекционных нарушений, заболеваний и/или патологических состояний, вызываемых бактериальными инфекциями, вирусными инфекциями, грибковыми инфекциями и/или паразитарными инфекциями.
Анти-PVRIG антитела можно вводить одни или в комбинации с одним или более дополнительными терапевтическими агентами, применяемыми для лечения бактериальных инфекций, вирусных инфекций, грибковых инфекций, необязательно, как описано в данном документе.
Это означает, что инфицированному субъекту вводят анти-PVRIG антитела, которые антагонизируют по меньшей мере одно опосредованное PVRIG действие на иммунитет, например, его ингибиторное действие на активность цитотоксических Т-клеток или NK-клеток и/или его ингибиторное действие на выработку провоспалительных цитокинов, или ингибируют стимуляторное действие PVRIG на Tрег, тем самым инициируя снижение числа или уничтожение инфицированных клеток или патогена и потенциально приводя к ремиссии заболевания вследствие повышенного уничтожения патогена или инфицированных клеток иммунными клетками субъекта.
Лечение сепсиса
В соответствии по меньшей мере с некоторыми вариантами реализации изобретения анти-PVRIG антитела можно применять для лечения сепсиса. В контексте данного документа термин «сепсис» или «связанное с сепсисом состояние» включает сепсис, острый сепсис, септический шок, синдром системной воспалительной реакции (ССВР), бактериемию, септицемию, токсемию, септический синдром.
Недавно выяснилось, что повышенная регуляция ингибиторных белков является одним из основных механизмов, лежащих в основе иммуносупрессии при сепсисе. Например, оказалось, что путь PD-1/PDL-1 является решающим фактором исхода сепсиса, регулируя хрупкое равновесие между эффективностью и вредом со стороны антибактериального иммунного ответа. Во время сепсиса в экспериментальной модели перитонеальные макрофаги и моноциты крови заметно повышали уровни PD-1, что было связано с развитием клеточной дисфункции (Huang et al 2009 PNAS 106:6303-6308). Аналогично, у пациентов с септическим шоком наблюдалось резкое повышение регуляции экспрессии PD-1 на периферических Т-клетках и PDL-1 на моноцитах (Zhang et al 2011 Crit. Care 15:R70). Недавние исследования на животных показали, что блокирование пути PD-1/PDL-1 анти-PD1 или анти-PDL1 антителами улучшало выживаемость при сепсисе (Brahmamdam et al 2010 J. Leukoc. Biol. 88:233-240; Zhang et al 2010 Critical Care 14:R220; Chang et al 2013 Critical Care 17:R85). Аналогично, блокирование CTLA-4 анти-CTLA4 антителами улучшало выживаемость при сепсисе (Inoue et al 2011 Shock 36:38-44; Chang et al 2013 Critical Care 17:R85). Вместе эти открытия позволяют предположить, что блокирование ингибиторных белков, включая отрицательные ко-стимуляторные молекулы, является эффективным терапевтическим подходом для предотвращения вредного действия сепсиса (Goyert and Silver, J Leuk. Biol., 88(2): 225-226, 2010).
В соответствии с некоторыми вариантами реализации в изобретении предложено лечение сепсиса с применением анти-PVRIG антител, одних или в комбинации с известными терапевтическими агентами, эффективными в лечении сепсиса, такими как терапевтические средства, блокирующие цитокиновую бурю на начальной гипервоспалительной стадии сепсиса, и/или терапевтическими средствами, которые обладают иммуностимулирующим действием, с целью преодоления индуцированной сепсисом фазы иммуносупрессии.
Комбинация со стандартами лечения сепсиса, рекомендуемыми “International Guidelines for Management of Severe Sepsis and Septic Shock” (Dellinger et al 2013 Intensive Care Med 39:165-228), некоторые из которых описаны ниже.
Широкий спектр антибиотиков, обладающих активностью против всех возможных патогенов (бактериальных и/или грибковых - лечение начинается, когда сепсис диагностирован, но конкретный патоген не определен), например, цефотаксим (Клафоран®), тикарциллин и клавуланат (Тиментин®), пиперациллин и тазобактам (Зосин®), имипенем и циластатин (Примаксин®), меропенем (Меррем®), клиндамицин (Клеоцин), метронидазол (Флагил®), цефтриаксон (Роцефин®), ципрофлоксацин (Ципро®), цефепим (Максипим®), левофлоксацин (Леваквин®), ванкомицин или любая комбинация перечисленных препаратов.
Вазопрессоры: например, норэпинефрин, допамин, эпинефрин, вазопрессин.
Стероиды: например, гидрокортизон, дексаметазон или флудрокортизон, внутривенно или каким-либо другим способом.
Инотропная терапия: например, добутамин для септических пациентов с дисфункцией миокарда.
Рекомбинантный человеческий активированный протеин C (rhAPC), такой как дротрекогин альфа (активированный) (DrotAA).
β-блокаторы дополнительно снижают местное и системное воспаление.
Метаболические вмешательства, такие как замещение пируватом, сукцинатом или высокой дозой инсулина.
Комбинация с новыми эффективными видами терапии сепсиса:
Избирательные ингибиторы sPLA2-IIA (такие как LY315920NA/S-5920). Обоснование: Секреторная фосфолипаза А2 группы IIA (sPLA2-IIA), высвобождаемая во время воспаления, повышена при остром сепсисе, а ее плазменные уровни обратно пропорциональны выживаемости.
Фосфолипидная эмульсия (такая как GR270773). Обоснование: Доклинические и ex vivo исследования показали, что липопротеины связывают и нейтрализуют эндотоксин, а экспериментальные исследования на животных продемонстрировали защиту от смерти вследствие сепсиса при введении липопротеинов. Нейтрализация эндотоксина коррелирует с количеством фосфолипида в липопротеиновых частицах.
Анти-TNF-α антитело: Обоснование: Фактор некроза опухолей α (TNF-α) индуцирует появление многих патофизиологических признаков и симптомов, наблюдаемых при сепсисе.
Анти-CD14 антитело (такое как IC14). Обоснование: Молекулы первичного узнавания, такие как CD14, играют ключевую роль в патогенезе. Компоненты стенки бактериальной клетки связываются с CD14 и ко-рецепторами на миелоидных клетках, приводя к клеточной активации и выработке провоспалительных медиаторов. Было показано, что анти-CD14 моноклональное антитело (IC14) снижает индуцированные липополисахаридами ответы в животной и человеческой моделях эндотоксемии.
Ингибиторы толл-подобных рецепторов (ТПР) и их нисходящие сигнальные пути. Обоснование: Инфицирующие микробы демонстрируют наличие высококонсервативных макромолекул (например, липополисахаридов, пептидогликанов) на своей поверхности. Когда эти молекулы распознаются паттерн-распознающими рецепторами (называемыми толл-подобными рецепторами [ТПР]) на поверхности иммунных клеток, инициируется иммунный ответ организма-хозяина. Это может давать свой вклад в избыточный системный воспалительный ответ, который характеризует сепсис. Ингибирование нескольких ТПР было оценено в качестве потенциальной терапии сепсиса, в частности, ТПР4, рецептора липополисахаридов или эндотоксинов внешней мембраны грамотрицательных бактерий. Различные лекарственные препараты, нацеленные на экспрессию и путь ТПР4, имеют терапевтический потенциал в отношении сепсиса (Wittebole et al 2010 Mediators of Inflammation Vol 10 Article ID 568396). Среди них антитела, нацеленные на ТПР4, растворимый ТПР4, статины (такие как розувастатин®, симвастатин®), кетамин, никотиновые аналоги, эриторан (E5564), ресаторвид (TAK242). Кроме того, применимы антагонисты других ТПР, такие как хлорохин, ингибирование ТПР-2 нейтрализующим антителом (анти-ТПР-2).
Лансопразол посредством его действия на SOCS1 (супрессор секреции цитокинов).
Талактоферрин или рекомбинантный человеческий лактоферрин. Обоснование: Лактоферрин представляет собой гликопротеин с противоинфекционными и противовоспалительными свойствами, обнаруживаемый в секретах и иммунных клетках. Талактоферрин альфа, рекомбинантная форма человеческого лактоферрина, обладает сходными свойствами и играет важную роль в поддержании целостности мукозального барьера желудочно-кишечного тракта. Талактоферрин продемонстрировал эффективность в животных моделях сепсиса и в клинических исследованиях на пациентах с острым сепсисом (Guntupalli et al Crit Care Med. 2013;41(3):706-716).
EGF фактор VIII жировых глобул молока (MFG-E8) - соединительная молекула между апоптотическими клетками и фагоцитами, которая стимулирует фагоцитоз апоптотических клеток.
Агонисты «холинергического противовоспалительного пути», такие как никотин и аналоги. Обоснование: Стимуляция блуждающего нерва снижает выработку цитокинов или медиаторов иммунной системы и блокирует воспаление. Эта нервная «схема», называемая «воспалительным рефлексом» осуществляется посредством специфического действия ацетилхолина, высвобождаемого нервными окончаниями, на субъединицу α7 никотинового холинорецептора (α7nAChR), экспрессируемого на макрофагах, механизма, называемого «холинергическим противовоспалительным путем». Активация этого пути посредством стимуляции блуждающего нерва или фармакологических агонистов или α7 предотвращает повреждение тканей во многих моделях системного воспаления, шока и сепсиса (Matsuda et al 2012 J Nippon Med Sch.79:4-18; Huston 2012 Surg. Infect. 13:187-193).
Терапевтические агенты, нацеленные на инфламмасомные пути. Обоснование: Инфламмасомные пути вносят большой вклад в воспалительный ответ при сепсисе и являются критическими элементами, ответственными за переход от локализованного воспаления к вредному гипервоспалительному ответу организма (Cinel and Opal 2009 Crit. Care Med. 37:291-304; Matsuda et al 2012 J Nippon Med Sch.79:4-18).
Терапия стволовыми клетками. Обоснование: Мезенхимальные стволовые клетки (МСК) демонстрируют множество полезных свойств вследствие своей способности попадать в поврежденные ткани, активировать стволовые клетки резидента, секретировать паракринные сигналы для ограничения системного и местного воспалительного ответа, благоприятным образом модулировать иммунные клетки, стимулировать заживление тканей путем снижения апоптоза в находящихся под угрозой тканях и стимуляции неоангиогенеза, и проявлять непосредственную противомикробную активность. Эти действия связаны со снижением органной дисфункции и улучшением выживаемости при сепсисе в животных моделях, которые обеспечили подтверждение того, что МСК могут быть полезными терапевтическими вспомогательными средствами (Wannemuehler et al 2012 J. Surg. Res. 173:113-26).
Комбинация анти-PVRIG антитела с другими иммуномодулирующими агентами, такими как иммуностимулирующие антитела, цитокиновая терапия, иммуномодулирующие лекарственные препараты. Такие агенты вызывают повышенную иммунную реактивность, особенно в ситуациях, когда иммунная защита (врожденная и/или адаптивная) была нарушена, например, как при индуцированном сепсисом гиповоспалительном и иммуносупрессивном состояниях. Обращение индуцированного сепсисом иммунного паралича с помощью терапевтических агентов, которые повышают иммунитет организма-хозяина, может снизить вероятность возникновения вторичных инфекций и улучшить результаты у пациентов с диагностированной иммунной супрессией (Hotchkiss et al 2013 Lancet Infect. Dis. 13:260-268; Payen et al 2013 Crit Care. 17:118).
Иммуностимулирующие антитела стимулируют иммунные ответы посредством прямой модуляции иммунных функций, т.е. блокирования других ингибиторных белков, или посредством усиления ко-стимуляторных белков. Экспериментальные модели сепсиса продемонстрировали, что иммуностимуляция посредством блокирования антителами ингибиторных белков, таких как PD-1, PDL-1 или CTLA-4, улучшала выживаемость при сепсисе (Brahmamdam et al 2010 J. Leukoc. Biol. 88:233-240; Zhang et al 2010 Critical Care 14:R220; Chang et al 2013 Critical Care 17:R85; Inoue et al 2011 Shock 36:38-44), что свидетельствует о том, что такие иммуностимулирующие агенты могут быть потенциальными вариантами терапии для предотвращения вредного действия индуцированной сепсисом иммуносупрессии (Goyert and Silver J Leuk. Biol. 88(2):225-226, 2010). Иммуностимулирующие антитела включают: 1) Антагонистические антитела, нацеленные на ингибиторные иммунные контрольные точки, включают анти-CTLA4 mAb (такие как ипилимумаб, тремелимумаб), анти-PD-1 (такие как ниволумаб BMS-936558/ MDX-1106/ONO-4538, AMP224, CT-011, ламбролизумаб MK-3475), антагонисты анти-PDL-1 (такие как BMS-936559/ MDX-1105, MEDI4736, RG-7446/MPDL3280A); анти-LAG-3, такие как IMP-321), анти-TIM-3, анти-BTLA, анти-B7-H4, анти-B7-H3, анти-VISTA. 2) Агонистические антитела, усиливающие иммуностимулирующие белки, включают анти-CD40 mAb (такие как CP-870,893, лукатумумаб, дацетузумаб), анти-CD137 mAb (такие как BMS-663513 урелумаб, PF-05082566), анти-OX40 mAb (такие как анти-OX40), анти-GITR mAb (такие как TRX518), анти-CD27 mAb (такие как CDX-1127) и анти-ICOS mAb.
Цитокины, которые напрямую стимулируют иммунные эффекторные клетки и усиливают иммунные ответы, можно применять в комбинации с анти-GEN антителом для терапии сепсиса: IL-2, IL-7, IL-12, IL-15, IL-17, IL-18 и IL-21, IL-23, IL-27, ГМ-КСФ, IFNα (интерферон α), IFNβ, IFNγ. Обоснование: Терапия на основе цитокинов реализует прямую попытку стимуляции собственной иммунной системы пациента. Экспериментальные модели сепсиса продемонстрировали, что введение цитокинов, таких как IL-7 и IL-15, способствует жизнеспособности Т-клеток и приводит к улучшению выживаемости при сепсисе (Unsinger et al 2010 J. Immunol. 184:3768-3779; Inoue et al 2010 J. Immunol. 184:1401-1409). Интерферон-γ (IFNγ) обращает индуцированный сепсисом иммунный паралич моноцитов in vitro. In vivo исследования продемонстрировали, что IFNγ частично обращает иммунный паралич in vivo у людей. IFNγ и гранулоцитарно-макрофагальный колониестимулирующий фактор (ГМ-КСФ) восстанавливают иммунокомпетентность ex vivo стимулируемых лейкоцитов пациентов с сепсисом (Mouktaroudi et al Crit Care. 2010; 14: P17; Leentjens et al Am J Respir Crit Care Med Vol 186, pp 838-845, 2012).
Иммуномодулирующие препараты, такие как тимозин α1. Обоснование: Тимозин α 1 (Tα1) представляет собой тимический пептид природного происхождения, который действует как эндогенный регулятор как врожденной, так и адаптивной иммунной системы. Во всем мире его используют для лечения заболеваний, связанных с иммунной дисфункцией, включая вирусные инфекции, такие как гепатит В и С, некоторые виды рака, и для усиления вакцин. Следует отметить, что недавние разработки в иммуномодулирующих исследованиях свидетельствуют о благоприятном действии лечения Ta1 на пациентов с сепсисом (Wu et al. Critical Care 2013, 17:R8).
В случае вышеописанных вариантов терапии сепсиса субъекту с сепсисом или подверженному риску развития сепсиса в связи с вирулентной инфекцией, например, устойчивой к антибиотикам или другим лекарственным препаратам, предпочтительно вводят иммуностимулирующее анти-PVRIG антитело или антигенсвязывающий фрагмент в соответствии с изобретением, при этом антитело антагонизирует по меньшей мере одно опосредованное PVRIG действие на иммунитет, например, его ингибиторное действие на активность цитотоксических Т-клеток или NK-клеток и/или его ингибиторное действие на выработку провоспалительных цитокинов, или ингибирует стимуляторное действие PVRIG на Tрег, тем самым инициируя снижение числа или уничтожение инфицированных клеток или патогена и потенциально приводя к ремиссии заболевания вследствие повышенного уничтожения патогена или инфицированных клеток эндогенными иммунными клетками субъекта. Так как сепсис может быстро приводить к органной недостаточности, в этом варианте реализации изобретения может быть целесообразно вводить фрагменты анти-PVRIG антитела, такие как Fab, вместо интактных антител, так как они могут достигать участков сепсиса или инфекции быстрее, чем интактные антитела. При таких схемах лечения время полужизни антитела может иметь меньшее значение вследствие быстрого, в некоторых случаях, наступления смерти при этом заболевании.
Диагностические применения
Предложенные анти-PVRIG антитела также находят применение в in vitro или in vivo диагностике, включая визуализацию, опухолей, которые сверхэкспрессируют PVRIG. Однако следует отметить, что, как обсуждается в данном документе, PVRIG, как иммуно-онкологический белок-мишень, не обязательно сверхэкспрессируется на раковых клетках, а скорее в иммунных инфильтратах в раке. В некоторых случаях скорее механизм действия, активация иммунных клеток, таких как Т-клетки и NK-клетки, приводит к диагностике рака. Соответственно, анти-PVRIG антитела можно применять для диагностики рака.
В частности, иммунные клетки, инфильтрирующие опухоли, которые сверхэкспрессируют PVRIG, и, таким образом, могут быть диагностированы с помощью анти-PVRIG антител, включают, но не ограничиваются этим, рак простаты, рак печени (ГЦК), колоректальный рак, рак яичника, рак эндометрия, рак молочной железы, рак поджелудочной железы, рак желудка, рак шейки матки, рак головы и шеи, рак щитовидной железы, рак яичка, рак уротелия, рак легкого, меланому, немеланомный рак кожи (плоскоклеточную и базальноклеточную карциному), глиому, рак почки (ПКК), лимфому (НХЛ или ЛХ), острый миелоцитарный лейкоз (ОМЛ), Т-клеточный острый лимфобластный лейкоз (T-ОЛЛ), диффузную крупноклеточную В-клеточную лимфому, опухоль половых клеток яичка, мезотелиому и рак пищевода.
В общем случае диагностику можно осуществлять несколькими путями. В одном варианте реализации изобретения ткань пациента, такую как образец биопсии, приводят в контакт с антителом к PVRIG, в общем случае меченым, так, что антитело связывается с эндогенным PVRIG. Уровень сигнала сравнивают с уровнем нормальной нераковой ткани, полученной от того же пациента или из эталонного образца, чтобы определить наличие или отсутствие рака. Образец биопсии может быть взят из солидной опухоли, образца крови (в случае лимфом и лейкозов, таких как ОЛЛ, Т-клеточная лимфома и т.д.).
В общем случае в этом варианте реализации изобретения анти-PVRIG метят, например, флуорофором или другой оптической меткой, которую можно выявить, используя флуорометр или другую оптическую систему выявления, как хорошо известно в данной области техники. В альтернативном варианте реализации изобретения вторичное меченое антитело приводят в контакт с образцом, используя антитело к человеческому IgG от другого млекопитающего (мыши, крысы, кролика, козы и т.д.) для проведения сандвич-анализа, как известно в данной области техники. В альтернативном варианте анти-PVRIG mAb можно напрямую метить (т.е. биотином) и проводить выявление с помощью вторичного Ab, направленного на метящий агент, как известно в данной области техники.
При наличии сверхэкспрессии PVRIG можно продолжать лечение путем введения анти-PVRIG антитела в соответствии с изобретением, как описано в данном документе.
В других вариантах реализации изобретения проводят in vivo диагностику. В общем случае в этом варианте реализации изобретения анти-PVRIG антитело (включая фрагменты антител) инъецируют пациенту и проводят визуализацию. В этом варианте реализации изобретения, например, антитело в общем случае метят оптической меткой или МРТ-меткой, такой как хелат гадолиния, радиоактвиной меткой для mAb (включая фрагменты).
В некоторых вариантах реализации изобретения описанные в данном документе антитела применяют как для диагностики, так и для лечения, или только для диагностики. Когда анти-PVRIG антитела применяют как для диагностики, так и для лечения, некоторые варианты реализации базируются на двух разных анти-PVRIG антителах к двум разным эпитопам так, что диагностическое антитело не конкурирует за связывание с терапевтическим антителом, хотя в некоторых случаях одно и то же антитело можно использовать в обеих целях. Например, это можно осуществлять, используя антитела, находящиеся в разных группах, например, которые связываются с разными эпитопами на PVRIG, как описано в данном документе. Такие образом, в изобретение включены композиции, содержащие диагностическое антитело и терапевтическое антитело, и, в некоторых вариантах реализации, диагностическое антитело метят, как описано в данном документе. Кроме того, композицию диагностического и терапевтического антител также можно вводить вместе с другими лекарственными препаратами, как описано в данном документе.
В частности, подходящие антитела для применения в диагностике включают, но не ограничиваются этим, эти пронумерованные антитела или антитела, в которых используются CDR с вариантными последовательностями, или те, которые конкурируют за связывание с любым из CPA.7.001-CPA.7.050, и, в частности, те, которые как связывают PVRIG, так и блокируют связывание рецептора, включая CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049 и CPA.7.050. Кроме того, можно применять те, которые связывают, но не блокируют, включая CPA.7.016, CAP.7.020, CPA.7.028, CPA.7.030, CPA.7.038, CPA.7.044 и CPA.7.045.
Как понятно специалистам в данной области техники, для ex vivo или in vitro методов анализа можно использовать мышиные антитела и, таким образом, вариабельные домены тяжелой и легкой цепей из любого из следующих антител можно применять для диагностики, включая наборы CDR в других форматах, из CHA.7.502, CHA.7.503, CHA.7.506, CHA.7.508, CHA.7.510, CHA.7.512, CHA.7.514, CHA.7.516, CHA.7.518, CHA.7.520.1, CHA.7.520.2, CHA.7.522, CHA.7.524, CHA.7.526, CHA.7.527, CHA.7.528, CHA.7.530, CHA.7.534, CHA.7.535, CHA.7.537, CHA.7.538.1, CHA.7.538.2, CHA.7.543, CHA.7.544, CHA.7.545, CHA.7.546, CHA.7.547, CHA.7.548, CHA.7.549 и CHA.7.550.
Во многих вариантах реализации изобретения диагностическое антитело метят. Под «меченым» подразумевается то, что раскрытые в данном документе антитела содержат один или более элементов, изотопов или химических соединений, присоединенных к ним, чтобы сделать возможным выявление во время процедуры скрининга или диагностирования. В общем случае метки делятся на несколько классов: a) иммунные метки, которые могут представлять собой эпитоп, включенный в виде партнера по слиянию, который распознается антителом, b) изотопные метки, которые могут представлять собой радиоактивные или тяжелые изотопы, c) низкомолекулярные метки, которые могут включать флуоресцентные и колориметрические красители, или молекулы, такие как биотин, которые делают возможным осуществление других способов мечения, и d) метки, такие как частицы (включая пузырьки для ультразвукового мечения) или парамагнитные метки, которые делают возможной визуализацию организма. Метки могут быть включены в антитела в любой позиции и могут быть включены in vitro или in vivo во время белковой экспрессии, как известно в данной области техники.
Диагностику можно проводить как in vivo, путем введения диагностического антитела, которое позволяет проводить визуализацию всего организма, как описано ниже, так и in vitro, на образцах, полученных от пациента. В этом контексте «образец» включает любое количество элементов, включая, но не ограничиваясь этим, жидкости организма (включая, но не ограничиваясь этим, кровь, мочу, сыворотку, лимфу, слюну, анальные и вагинальные выделения, пот и сперму), а также образцы тканей, такие как получают в результате биопсии соответствующих тканей.
В некоторых вариантах реализации изобретения проводят in vivo визуализацию, включая, но не ограничиваясь этим, ультразвуковое исследование, КТ-исследование, рентген, МРТ и ПЭТ-исследования, а также оптические методы, такие как методы с использованием оптических меток для опухолей вблизи поверхности тела.
In vivo визуализацию заболеваний, связанных с PVRIG, можно проводить любым подходящим методом. Например, можно использовать 99Tc-мечение или мечение другим испускающим бета-излучение изотопом, чтобы пометить анти-PVRIG антитела. Вариации этого метода могут включать применение магнито-резонансной томографии (МРТ) для улучшения визуализации по сравнению с методами с использованием гамма-камеры.
В одном варианте реализации в настоящем изобретении предложен метод in vivo визуализации, при этом анти-PVRIG антитело конъюгировано с облегчающим выявление агентом, при этом конъюгированное антитело вводят в организм-хозяин, например, путем инъекции в кровоток, и анализируют наличие и расположение меченого антитела в организме-хозяине. Посредством этого метода и любого другого диагностического метода, предложенного в данном документе, в настоящем изобретении предложен метод проведения скрининга в отношении наличия связанных с заболеванием клеток в организме пациента-человека или биологическом образце, полученном от пациента-человека.
Для диагностической визуализации радиоактивные изотопы можно связывать с анти-PVRIG антителом как прямо, так и непрямо, используя промежуточную функциональную группу. Применимые промежуточные функциональные группы включают хелаторы, такие как этилендиаминтетрауксусная кислота и диэтилентриаминпентауксусная кислота (смотрите, например, патент США № 5057313); в таких диагностических методах анализа, включающих применение конъюгированных с радиоактивными изотопами анти-PVRIG антител, дозировку конъюгированного анти-PVRIG антитела, доставляемого пациенту, как правило, поддерживают на максимально возможном низком уровне путем выбора изотопа с наилучшей комбинацией минимального времени полужизни, минимального удержания в организме и минимального количества изотопа, что сделает возможным выявление и точное определение.
Кроме радиоактивных изотопов и рентгеноконтрастных агентов диагностические способы можно осуществлять, используя анти-PVRIG антитела, конъюгированные с красителями (например, с комплексом биотин-стрептавидин), контрастными агентами, флуоресцентными соединениями или молекулами и усиливающими агентами (например, парамагнитными ионами) для магнито-резонансной томографии (МРТ) (смотрите, например, патент США № 6331175, в котором описаны методы МТР и получение антител, конъюгированных с усиливающим агентом для МРТ). Такие диагностические/выявляющие агенты можно выбрать из агентов для применения в магнито-резонансной томографии и флуоресцентных соединений.
Чтобы нагрузить анти-PVRIG антитело радиоактивными металлами или парамагнитными ионами, может возникнуть необходимость приведения его в реакцию с реагентом, имеющим длинный хвост, к которому присоединено множество хелатирующих групп для связывания ионов. Такой хвост может представлять собой полимер, такой как полилизин, полисахарид или другую дериватизированную или дериватизируемую цепь, содержащую боковые подвешенные группы, с которыми можно связать хелатирующие группы, такие как, например, порфирины, полиамины, краун-эфиры, бистиосемикарбазоны, полиоксимы и тому подобные группы, применимые в этих целях.
Хелаты можно связывать с анти-PVRIG антителами, используя стандартные химические вещества. Хелат обычно связан с анти-PVRIG антителом группой, которая делает возможным образование связи с молекулой с минимальной потерей иммунореактивности и минимальной агрегацией и/или внутренним перекрестным сшиванием.
Примеры потенциально применимых комбинаций металл-хелат включают 2-бензил-ДТПА и его монометильные и циклогексильные аналоги, применяемые с диагностическими изотопами с энергией в диапазоне от 60 до 4000 кэВ, такими как 125I, 123I, 124I, 62Cu, 64Cu, 18F, 111In, 67Ga, 99Tc, 94Tc, 11C, 13N, 5O и 76Br, для радиоактивной визуализации.
Метки включают радионуклид, радиологический контрастный агент, парамагнитный ион, металл, флуоресцентную метку, хемилюминесцентную метку, ультразвуковой контрастный агент и фотоактивный агент. Такие диагностические агенты хорошо известны, и можно использовать любой такой известный диагностический агент. Неограничивающие примеры диагностических агентов могут включать радионуклиды, такие как 110In, 111In, 177Lu, 18F, 52Fe, 62Cu, 64Cu, 67Cu, 67Ga, 68Ga, 86Y, 90Y, 89Zr, 94mTc, 94Tc, 99mTc, 120I, 123I, 124I, 125I, 131I, 154-158Gd, 32P, 11C, 13N, 15O, 186Re, 188Re, 51Mn, 52мМn, 55Co, 72As, 75Br, 76Br, 82mRb, 83Sr или другие гамма-, бета- или позитронные излучатели.
Применимые парамагнитные ионы могут включать хром (III), марганец (II), железо (III), железо (II), кобальт (II), никель (III), медь (III), неодим (III), самарий (III), иттербий (III), гадолиний (III), ванадий (II), тербий (III), диспрозий (III), гольмий (III) или эрбий (III). Металлические контрастные агенты могут включать лантан (III), золото (III), свинец (II) или висмут (III).
Ультразвуковые контрастные агенты могут включать липосомы, такие как заполненные газом липосомы. Рентгеноконтрастные диагностические агенты можно выбрать из соединений, таких как соединения бария, соединения галлия и соединения таллия.
Эти и сходные хелаты, комплексированные с нерадиоактивными металлами, такими как марганец, железо и гадолиний, можно применять в МРТ-диагностических способах вместе с анти-PVRIG антителами. Макроциклические хелаты, такие как NOTA, DOTA и TETA применяют с различными металлами и радиоактивными металлами, в частности, с радионуклидами галлия, иттрия и меди соответственно. Такие комплексы металл-хелат можно делать очень стабильными, подгоняя размер кольца под представляющий интерес металл. Другие хелаты кольцевого типа, такие как макроциклические сложные полиэфиры, которые представляют интерес для стабильно связывающих нуклидов, таких как 223Ra, также могут подходить для диагностических способов.
Таким образом, в настоящем изобретении предложены диагностические конъюгаты анти-PVRIG антитела, при этом конъюгат анти-PVRIG антитела конъюгирован с контрастным агентом (например, для магнито-резонансной томографии, компьютерной томографии, или ультразвуковым контрастным усиливающим агентом) или радионуклидом, который может представлять собой, например, гамма-, бета-, альфа-, Оже-электрон- или позитрон-излучающий изотоп.
Анти-PVRIG антитела также можно применять, например, для выявления экспрессии представляющего интерес антигена в конкретных клетках, тканях или сыворотке. Для диагностических применений антитело, как правило, метят выявляемым компонентом для in vitro анализа. Как понятно специалисту в данной области техники, существует широкий спектр подходящих меток для применения в in vitro исследованиях. Подходящие красители для применения в этом аспекте изобретения включают, но не ограничиваются этим, флуоресцентные комплексы лантаноидов, включая комплексы европия и тербия, флуоресцеин, родамин, тетраметилродамин, эозин, эритрозин, кумарин, метилкумарины, квантовые точки (также называемые «нанокристаллами»; смотрите США сер. № 09/315584, включенный в данный документ посредством ссылки), пирен, малахитовый зеленый, стильбен, люцифер желтый, каскад синий.TM., техасский красный, красители Cy (Cy3, Cy5 и т.д.), красители alexa (включая Alexa, фикоэритрин, bodipy и другие, описанные в 6-м издании Molecular Probes Handbook авторства Richard P. Haugland, явным образом включенном в данный документ посредством ссылки).
Затем окрашенные ткани можно оценивать в отношении радиоактивности, принимая ее за показатель количества PVRIG-ассоциированных пептидов в опухоли. Изображения, полученные с помощью таких методов, можно использовать для оценки биораспределения PVRIG в организме пациента, млекопитающего или ткани, например, в контексте применения PVRIG в качестве биомаркера наличия инвазивных раковых клеток.
Примеры
Настоящим ссылаемся на USSN XX, поданную 19 февраля 2016 г., под названием «PVRIG POLYPEPTIDES AND METHODS OF TREATMENT» («ПОЛИПЕПТИДЫ PVRIG И СПОСОБЫ ЛЕЧЕНИЯ»), заявляющей приоритет USSN 62/118235, поданной 19 февраля 2015 г., и USSN 62/141168, поданной 31 марта 2015 г., которые все явным образом включены в данный документ посредством ссылки.
Пример 1. Анализ экспрессии белков PVRIG
Пример 1A
Анализировали набор данных GDS3113 (http://www.ncbi.nlm.nih.gov/sites/GDSbrowser?acc=GDS3113), чтобы определить гены со специфическим в отношении лимфоидных органов профилем. PVRIG был идентифицирован как лимфоцит-специфический благодаря высокой экспрессии в первичных и вторичных лимфоидных органах, которые включают периферическую кровь, костный мозг, селезенку, лимфатические узлы, миндалевидную железу и вилочковую железу (на фиг. 2). Другие типы тканей были отрицательными или демонстрировали экспрессию на фоновых уровнях. Чтобы исследовать, какие конкретно типы клеток в общей популяции иммунных клеток экспрессируют PVRIG, анализировали дополнительные наборы данных из Gene Expression Omnibus (www.ncbi.nlm.nih.gov/GEO), как описано в разделе «методологии». Анализ проводили для популяций иммунных клеток, полученных из периферической крови и костного мозга. PVRIG экспрессировался в лимфоцитах как В-клеточной линии дифференцировки, так и Т-клеточной линии дифференцировки, включая наивные, эффекторные Т-клетки CD8 и клетки памяти (на фиг. 3). Кроме того, PVRIG экспрессировался в NK-клетках и характеризовался наивысшей экспрессией в популяции iNKT (на фиг. 4). В экспериментальных моделях iNKT-популяция лимфоцитов действует в качестве эффективных активаторов противоопухолевого иммунитета при стимуляции синтетическим агонистом. Однако в некоторых случаях iNKT-клетки могут действовать как супрессоры и регуляторы противоопухолевого иммунитета (Clin Dev Immunol. 2012;2012:720803). Более того, ранние клинические исследования иммунотерапии на основе iNKT продемонстрировали, что инфузия активированных лигандами антигенпредставляющих клеток и/или in vitro активированных iNKT-клеток была безопасной и хорошо переносимой при раке легкого и раке головы и шеи (Clin Immunol. 2011 Aug;140(2):167-76.).
Ключевой вопрос в отношении экспрессии PVRIG состоял в том, сохраняют ли опухоль-инфильтрирующие лимфоциты (ОИЛ) экспрессию PVRIG в опухолевом микроокружении. Анализ данных по экспрессии ОИЛ из фолликулярной лимфомы, рака молочной железы и рака толстой кишки показал наличие четкой экспрессии PVRIG в ОИЛ, инфильтрирующих опухоль. В примере с раком толстой кишки специфичность в отношении иммунных инфильтрирующих клеток проявлялась как экспрессия только в CD45-положительной популяции (специфический в отношении лейкоцитов маркер), и отсутствие экспрессии в EPCAM-положительной популяции (специфический в отношении эпителия маркер) или в CD45-отрицательной EPCAM-отрицательной популяции (популяция стромальных клеток). Хотя CD45 не является специфическим в отношении лимфоцитов маркером, из другого описания экспрессии можно сделать вывод, что он экспрессируется в популяции лимфоцитов (на фиг. 5А - рак толстой кишки, на фиг. 5В - рак молочной железы и на фиг. 5С - фолликулярная лимфома).
Приведенные в данном документе данные по экспрессии мРНК свидетельствуют о том, что PVRIG экспрессируется в лимфоцитах и в опухоль-инфильтрирующих лимфоцитах (ОИЛ). Эти результаты вместе с ингибиторной активностью PVRIG предполагают ингибиторную роль этой молекулы в отношении Т-клеток, что позволяет предположить, что ингибиторные антитела к PVRIG повышают супрессивное влияние PVRIG на ОИЛ и, таким образом, позволяют ОИЛ индуцировать иммунный ответ против рака. Так как предложенный механизм действия направлен на инфильтрирующие опухоль ОИЛ, а не на прямое действие на опухолевые клетки, любой рак с иммунной инфильтрацией является кандидатом для лечения с помощью ингибиторных антител к PVRIG.
Методология: Исходные данные загружены со страницы GEO в формате SOFT. В случаях, когда исходные данные имеют формат MAS5, данные были получены без обработки. Если данные имеют формат Log MAS5, то данные конвертируют в линейные данные. Если данные имеют формат RMA, файлы CEL (исходные данные) были загружены и повторно проанализированы с применением MAS5. Если исходные файлы CEL не были доступны, использовали формат RMA.
Затем данные нормализовали с помощью мультипликативного алгоритма в соответствии с 95-м процентилем для данных Affy. Анализируемые наборы данных: GSE49910, GSE47855, GSE39397, GSE36765, GSE27928.
Пример 1B
Эталонная транскриптома была создана на основании известных генных моделей UCSC (http://hgdownload.cse.ucsc.edu/goldenPath/hg19/database/knownGene.txt.gz). Сначала все считанные фрагменты последовательностей РНК выравнивали относительно последовательностей транскриптомы. Это выравнивание позволило получить неоднозначное картирование, так как изоформы имеют много общих экзонов. Затем каждому считанному фрагменту присваивали геномные координаты и области соединения экзонов на основании совпадений с транскриптомой. Оставшиеся некартированные считанные фрагменты выравнивали непосредственно относительно генома с учетом одной или более областей соединений экзонов. В конце некоторое количество считанных фрагментов нормализовали, как описано в Bo et al. (Bioinformatics 2010, 26 (4): 493-500), и преобразовывали в значения генной экспрессии, как описано в Trapnell et al (Nat Biotechnol. 2010 May;28(5):511-5).
Как показано на фиг. 6, учитывая данные Genotype-Tissue Expression (GTEx) (http://www.nature.com/ng/journal/v45/n6/full/ng.2653.html; http://www.gtexportal.org/home/), PVRIG экспрессируется главным образом в клетках крови и, в меньшей степени, в различных нормальных тканях. Такие же результаты наблюдали для раковых тканей из The Cancer Genome Atlas (TCGA) (http://cancergenome.nih.gov/), где высокая экспрессия наблюдается в раках крови, таких как В-клеточные лимфомы и ОМЛ (на фиг. 7). Профили генной экспрессии создавали для ряда раков и нормальных тканей, используя данные GTEx и TCGA, путем определения генов с высокой корреляцией профиля экспрессии с PVRIG.
Корреляционный анализ проводили в расчете на тип опухоли, при этом учитывали только те корреляции, где оба гена экспрессировались выше 0 RPKM с учетом по меньшей мере 50 образцов для одного типа опухоли. Эти профили генной экспрессии исследовали в отношении представленности взаимодействующих белков, путей и генов заболевания. Р-значения для представленности рассчитывали для каждого типа опухоли, а среднее -log(p-значение) использовали для упорядочения результатов для наборов генов. Для ряда раков наблюдали четкий профиль лимфоцитов и Т-клеток, как показано на фиг. 8. Например, геном с самыми высокими результатами в отношении белкового взаимодействия был IL2, что означает, что гены, взаимодействующие с IL2, сильнее коррелируют с PVRIG, чем случайно ожидалось для большинства раков. Дополнительный анализ показал, что экспрессия PVRIG в раковых тканях превышает нормальную. Тогда как на фиг. 5 медианное значение уровня экспрессии PVRIG ниже 1 для большинства нормальных солидных тканей, на фиг. 6 оно явно превышает 1 для многих раков. Например, при параллельном сравнении, как на фиг. 7, PVRIG в меланоме экспрессировался сильнее, чем в нормальной коже (на фиг. 9). Мы дополнительно получили характеристики источника сверхэкспрессии в раке. PVRIG сильно экспрессируется в Т-клетках и сильно коррелирует маркерами Т-клеток при раке. На фиг. 10 в качестве примера приведена корреляция PVRIG с CD3, CD4 и CD8 в трех типах рака, а именно, аденокарциноме легкого, аденокарциноме толстой кишки и меланоме. Кроме того, PVRIG сильно коррелирует с PD1, проверенной мишенью для иммунотерапии при раке, которая экспрессируется на Т-клетках (на фиг. 10).
Эти профили генной экспрессии исследовали в отношении представленности взаимодействующих белков, путей и генов заболевания. Для ряда раков наблюдали четкий профиль лимфоцитов и Т-клеток, как показано на фиг. 8. Мы дополнительно проанализировали корреляцию PVRIG с PD1 и продемонстрировали сильную корреляцию между их экспрессией в ряде опухолей, включая опухоли молочной железы, легкого, поджелудочной железы и почки (таблица 2). Как PD-1, так и PVRIG сильно экспрессируются на активированных Т-клетках. PVRIG продемонстрировал сильную корреляцию с маркерами Т-клеток, а именно, CD8A, CD4 и CD3G (
На фиг. 13. Вместе эти данные демонстрируют, что экспрессия PVRIG при раке связана с опухоль-инфильтрирующими лимфоцитами.
Методы: Корреляция генов: Значения FPKM преобразовывали в log2 (FPKM+0,1). Образцы со значением, которое удовлетворяет log2 (FPKM+0,1)< log2(0,1) по меньшей мере для одного из генов, не учитывали. Коэффициент корреляции Пирсона (ККП) и оценки наименьших квадратов для регрессионной прямой рассчитывали для 2 перечней (один перечень на ген). ККП со значением менее 0,5 не учитывали, как и ККП, которые не демонстрировали значимой величины при исследовании линейной корреляции между уровнями экспрессии 2-х генов.
Анализ представленности генов: Данные по пути, взаимодействию и заболеванию получали из GeneGo Metacore (https://portal.genego.com), Reactome (http://www.reactome.org) и KEGG Pathways (http://www.genome.jp/kegg). Для определения путей и процессов, представленных в пределах заданного перечня генов, применяли анализ представленности на основе гипергеометрического распределения. Гипергеометрическое р-значение рассчитывали с помощью программы (http://www.R-project.org) со следующей командой: phyper(x − 1, m, n-m, k and lower.tail = FALSE), где x представляет собой число генов из перечня генов, являющихся представителями указанного пути, m представляет собой число генов в пути, n представляет собой общее число генов во всех путях, а k представляет собой число генов из перечня, которые присутствовали по меньшей мере в одном пути. Получаемое в результате р-значение указывает на вероятность случайной представленности для конкретного пути при заданном размере перечня генов. Такую же аналитическую процедуру применяли для генных взаимодействий, где все гены, взаимодействующие с заданным геном, рассматривались как путь; или генов, связанных с заболеванием, где все связанные гены рассматривались как путь. Смотрите фиг. 63.
Экспрессия PVRIG была связана с истощением Т-клеток при раке. Были отобраны образцы рака из TCGA, которые характеризовались высокой (4-й квартиль) экспрессией следующих 4 маркеров: CD8, PD-1, TIM-3 и TIGIT. Затем образцы рака делили на образцы с высокими, неизменными и низкими уровнями комбинированной экспрессии 4 маркеров. PVRIG не был выявлен ни в одном из образцов с низкой экспрессией маркеров (низкое количество или отсутствие истощенных Т-клеток). Подавляющее большинство опухолей, ассоциируемых с высокими уровнями истощенных Т-клеток, экспрессировали высокие уровни PVRIG (на фиг. 21).
Пример 1C
Оценивали экспрессию РНК и белка PVRIG в клеточных линиях и первичных лейкоцитах человека и отличного от человека примата.
Протоколы
Анализ FACS сконструированных сверхэкспрессирующих клеток: Следующие клеточные линии использовали для оценки специфичности антител к человеческому PVRIG: родительские клетки HEK и сверхэкспрессирующие hPVRIG клетки HEK. Эти клетки культивировали в DMEM (Gibco) + 10% фетальная телячья сыворотка (Gibco) + глутамакс (Gibco). В случае сверхэкспрессирующих hPVRIG клеток HEK для положительной селекции в среду также добавляли 0,5 мкг/мл пуромицина (Gibco). Для проведения анализа FACS все клеточные линии собирали в log-фазе роста и высевали 50000-100000 клеток на лунку в 96-луночные планшеты. Антитела к человеческому PVRIG (человеческие IgG1, hIgG1) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или в виде 8-точечных титрований, начиная с 30 мкг/мл, на льду в течение 30 мин-1 ч. Титрование проводили в виде 1:3 или 1:3,3 серийных разведений. Данные получали с помощью FACS Canto II (BD Biosciences) и анализировали с помощью программного обеспечения FlowJo (Treestar) и Prism (Graphpad).
Анализ FACS клеточных линий человека: Следующие клеточные линии использовали для оценки экспрессии и специфичности антител к человеческому PVRIG: Jurkat, CA46, NK-92, OV-90, HepG2 и NCI-H441. Клетки Jurkat, CA46 и NCI-H441 культивировали в среде RPMI + 10% фетальная телячья сыворотка, глутамакс, заменимые аминокислоты (Gibco), пируват натрия (Gibco) и пенициллин/стрептомицин (Gibco). Клетки NK-92 культивировали в среде RPMI + 25% фетальная телячья сыворотка, глутамакс, заменимые аминокислоты, пируват натрия, пенициллин/стрептомицин и 500 Е/мл IL-2 (R&D systems). Клетки OV-90 культивировали в 1:1 смеси среды MCDB 105 (Sigma), содержащей конечную концентрацию 1,5 г/л бикарбоната натрия (Life Technologies), и среды 199 (Sigma), содержащей конечную концентрацию 2,2 г/л бикарбоната натрия с конечной концентрацией 15% фетальной телячьей сыворотки. Клетки HepG2 культивировали в DMEM + 10% фетальная телячья сыворотка + глутамакс. Для проведения анализа FACS все клеточные линии собирали в log-фазе роста и высевали 50000-100000 клеток на лунку в 96-луночные планшеты. Антитела к человеческому PVRIG (hIgG1) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или в виде 8-точечных титрований, начиная с 30 мкг/мл, на льду в течение 30 мин-1 ч. Титрование проводили в виде 1:3 или 1:3,3 серийных разведений. Данные получали с помощью FACS Canto II и анализировали с помощью программного обеспечения FlowJo и Prism.
Анализ FACS наивных первичных лейкоцитов человека: Первичные лейкоциты получали с помощью выделения в градиенте фиколла (GE Healthcare) периферической крови (Stanford Blood Bank). Лейкоциты в виде выделенных мононуклеарных клеток периферической крови (МКПК) замораживали в жидком азоте с плотностью от 1x107 до 5x107 клеток/мл в смеси 10% ДМСО (Sigma), 90% фетальной телячьей сыворотки. Чтобы оценить белковую экспрессию PVRIG в МКПК, составляли коктейли антител против основных иммунных субпопуляций, которые содержали человеческие анти-PVRIG антитела. Антитела к человеческому PVRIG (hIgG1) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или, в некоторых случаях, в виде 8-точечных титрований, начиная с 30 мкг/мл, на льду в течение 30 мин-1 ч.
Вкратце, коктейльные смеси антител добавляли к восстановленным МКПК, которые были высеяны при 5x105-1x106 клеток/лунку после предварительной блокировки Fc-рецептора, и окрашивания в отношении живых/мертвых клеток (Aqua Live/Dead, Life Technologies). Коктейли антител инкубировали с МКПК в течение 30 мин-1 ч на льду. Затем МКПК промывали и получали данные методом FACS с помощью FACS Canto II. Данные анализировали с помощью программного обеспечения FlowJo и Prism. Анализируемые иммунные субпопуляции включали NK-клетки CD56 dim, NK-клетки CD56 bright, Т-клетки CD4+, Т-клетки CD8+, нетрадиционные Т-клетки (например, NKТ-клетки и γδ Т-клетки), В-клетки и моноциты.
Анализ FACS активированных эффекторных лимфоцитов человека: В некоторых случаях оценивали экспрессию PVRIG в субпопуляциях активированных эффекторных лимфоцитов, выделенных из цельных МКПК или в препаратах цельных МКПК. Эффекторные лимфоциты стимулировали комбинациями цитокинов, комбинациями антител и цитокинов или патогенными продуктами. Анализ FACS экспрессии PVRIG на активированных клетках проводили аналогично вышеописанному анализу для наивных первичных лейкоцитов.
Чтобы исследовать экспрессию PVRIG на стимулированных NK-клетках, клетки CD56+ выделяли и культивировали в различных коктейлях из цитокинов в течение 1-3 дней в NK-клеточной среде (RPMI + 10% фетальная телячья сыворотка, глутамакс, пенициллин/стрептомицин, заменимые аминокислоты, пируват натрия и бета-меркаптоэтанол [Gibco]). Сортировку NK-клеток проводили, используя микрогранулы против человеческого CD56+ (Miltenyi Biotec) или набор для выделения человеческих NK-клеток (Miltenyi Biotec) в соответствии с инструкциями производителя. Коктейли из цитокинов, применяемые для стимуляции NK-клеток, содержали IL-2, IL-12, IL-15, IL-2/IL-12, IL-2/IL-15, IL-12/IL-15 (R&D systems).
Чтобы исследовать экспрессию PVRIG на стимулированных Т-клетках, CD4+ или CD8+, выделяли Т-клетки, используя микрогранулы CD4+ или CD8+ (Miltenyi Biotec). Выделенные клетки культивировали в течение 3 дней в присутствии ряда активационных условий в Т-клеточной среде (RPMI + 10% фетальная телячья сыворотка, глутамакс, пенициллин/стрептомицин, заменимые аминокислоты, пируват натрия). Условия, применяемые для стимуляции выделенных Т-клеток, включали стимуляцию человеческими гранулами dynabead (гранулы, сопряженные с антителами к CD3/CD28, Life Technologies) с IL-2 или коктейлем цитокинов, который обеспечивал определенный фенотип Т-клеток (например, фенотипы Th1, Th2, Th17 и регуляторные Т-клетки). Цитокины, обеспечивающие Th1, представляют собой рекомбинантный IL-12 (R&D systems) и анти-IL-4 нейтрализующее антитело (Biolegend). Условия, обеспечивающие Th2, включают рекомбинантный IL-4 (R&D systems) и анти-IFN-гамма нейтрализующее антитело (Biolegend). Условия, обеспечивающие Th17, включают рекомбинантный IL-6 (R&D systems), TGF-бета (R&D systems), IL-23 (R&D systems) и анти-IL-4 и анти-IFNγ нейтрализующие антитела. Условия, обеспечивающие Т-регуляторный фенотип, включают рекомбинантные TGF-бета и IL-2, и анти-IL-4 и анти-IFNγ нейтрализующие антитела.
В альтернативном варианте Т-клетки анализировали также в культурах цельных стимулированных МКПК с антигеном стафилококкового энтеротоксина B (СЭВ) (List Biological Laboratories) в течение 3 дней, или в реакции смешанной культуры лимфоцитов (РСКЛ), в которой Т-клетки CD4+ культивировали совместно с аллогенными дендритными клетками в течение 2 или 5 дней.
Анализ FACS поляризованных моноцитов человека: Оценивали экспрессию PVRIG на дендритных клетках, полученных из поляризованных моноцитов. В этом случае клетки CD14+ обогащали с помощью коктейля для обогащения человеческих моноцитов RosetteSep в соответствии с инструкциями производителя. После обогащения клеток CD14+ моноциты поляризовали к дендритным клеткам после культивирования с ГМ-КСФ (R&D systems) и IL-4 (R&D systems) а течение 4 дней в RPMI + 10% фетальная телячья сыворотка, глутамакс, пенициллин/стрептомицин, заменимые аминокислоты, пируват натрия и бета-меркаптоэтанол.
Анализ экспрессии РНК человеческих клеточных линий и лейкоцитов методом кПЦР: Клеточными линиями, которые оценивали в отношении экспрессии РНК методом кПЦР, были клетки Jurkat, CA46, Daudi, Raji и expi 293. Клетки Jurkat, CA46, Raji и Daudi культивировали в среде RPMI + 10% фетальная телячья сыворотка, глутамакс, заменимые аминокислоты, пируват натрия и пенициллин/стрептомицин. Клетки Expi 293 культивировали в DMEM + 10% ФТС + глутамакс. РНК OV-90, HepG2 и NCI-H441 анализировали путем проведения биоинформационного скрининга атласа раковых клеточных линий. В случае тех клеточных линий, которые оценивали в отношении экспрессии РНК методом кПЦР, клетки собирали в log-фазе роста, собирали 1000000 клеток, промывали в ФСБ и лизировали в 350 мкл буфера RLT (Qiagen). Лизированные клетки в буфере RLT хранили при -80oС до применения.
Первичными лейкоцитами, которые оценивали в отношении экспрессии РНК, были NK-клетки CD56+, Т-клетки CD4+, Т-клетки CD8+ и моноциты CD14+. Популяции клеток выделяли, используя наборы для положительной селекции человеческих CD56+, CD4+, CD8+ и CD14+ в соответствии с инструкциями производителя (Miltenyi Biotec). После сортировки клетки лизировали в 350 мкл буфера RLT и хранили при -80oС до применения. В некоторых случаях из культуры собирали субпопуляции активированных МКПК (условия активации приведены выше) и лизировали в 350 мкл буфера RLT и хранили при -80oС до применения.
Через день применения из лизированных клеток получали РНК, используя мининабор Qiagen в соответствии с инструкциями производителя. кДНК получали с помощью набора для обратной транскрипции кДНК Applied Biosystems с большой емкостью. кПЦР с кДНК проводили, используя праймеры Taqman (ThermoFisher) и быстрый усовершенствованный мастер-микс Applied Biosystems Taqman. Набор используемых праймеров PVRIG соответствовал номеру в каталоге Taqman: Hs04189293_g1. Набор используемых базовых праймеров бета-актина соответствовал номеру в каталоге Taqman: Hs01060665_g1. Экспрессию транскрипта оценивали путем подсчета значений Ct, а относительную экспрессию рассчитывали по методу 2(-ΔΔCt). Данные получали с помощью прибора Applied Biosystems Step One Plus.
Анализ FACS сконструированных сверхэкспрессирующих PVRIG клеток яванского макака: Следующие клеточные линии использовали для оценки перекрестной реактивности антител к человеческому PVRIG с PVRIG яванского макака (cPVRIG): родительские клетки expi и сверхэкспрессирующие cPVRIG клетки expi. Эти клетки культивировали в DMEM + 10% фетальная телячья сыворотка + глутамакс. Клетки expi, временно сверхэкспрессирующие cPVRIG, получали путем электропорации ДНК cPVRIG в родительские клетки expi с помощью системы для трансфекции Neon. Для анализа FACS использовали клетки expi cPVRIG через 1-3 дня после трансфекции. Родительские клетки expi собирали в log-фазе роста. 50000-100000 клеток каждого типа на лунку высевали в 96-луночные планшеты. Антитела к человеческому PVRIG (hIgG1) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или в виде 8-точечных титрований, начиная с 100 мкг/мл, на льду в течение 30 мин-1 ч. Титрование проводили в виде 1:3 или 1:3,3 серийных разведений. Данные получали с помощью FACS Canto II и анализировали с помощью программного обеспечения FlowJo и Prism.
Анализ FACS наивных первичных лейкоцитов яванского макака: Первичные лейкоциты яванского макака (cyno) получали из свежей крови, которую брали не более чем за 24 часа до анализа экспрессии. Источником крови служил Bioreclamation. Чтобы оценить экспрессию белка PVRIG в МКПК яванского макака, составляли коктейли антител против основных иммунных субпопуляций, которые содержали человеческие анти-PVRIG антитела. Антитела к человеческому PVRIG (hIgG1) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл).
Вкратце, коктейльные смеси антител добавляли к МКПК, которые были высеяны при 5x105-1x106 клеток/лунку после предварительной блокировки Fc-рецептора, и окрашивания в отношении живых/мертвых клеток. Коктейли антител инкубировали с МКПК в течение 30 мин-1 ч на льду. Затем МКПК промывали и получали данные методом FACS с помощью FACS Canto II. Данные анализировали с помощью программного обеспечения Prism. Анализируемые иммунные субпопуляции включали лимфоциты CD16+, моноциты/миелоидные клетки CD14+/CD56+ и Т-клетки CD3+.
Анализ экспрессии РНК в первичных лейкоцитах яванского макака: Первичными лейкоцитами, которые оценивали в отношении экспрессии РНК, были субпопуляции CD56+, CD16+ и CD56-/CD16. Популяции клеток выделяли, используя наборы для положительной селекции CD56 и CD16 отличных от человека приматов в соответствии с инструкциями производителя (Miltenyi Biotec). После сортировки клетки лизировали в 350 мкл буфера RLT и хранили при -80oС до применения.
Через день применения из лизированных клеток получали РНК, используя мининабор в соответствии с инструкциями производителя. кДНК получали с помощью набора для обратной транскрипции кДНК Applied Biosystems с большой емкостью. кПЦР с кДНК проводили, используя праймеры Taqman и быстрый усовершенствованный мастер-микс Applied Biosystems Taqman. Два набора праймеров для выявления PVRIG яванского макака были разработаны Compugen USA, Inc., и произведены Genscript. Последовательности и обозначения праймеров следующие:
Набор праймеров 1
Прямой: CTTGTGTTCACCACCTCTGG
Обратный: TGTTCTCATCGCAGGAGGTC
Набор праймеров 2
Прямой: TTGGCTGTGGATACCTCCTT
Обратный: ATAAGGGTCGTGGAGAGCAG
Праймеры бета-актина использовали как базовые, а используемый набор праймеров соответствовал номеру в каталоге Taqman: Mf04354341_g1. Экспрессию транскриптов оценивали путем подсчета значений Ct, а относительную экспрессию рассчитывали по методу 2(-ΔΔCt). Также для продуктов, полученных с помощью праймеров PVRIG и праймеров бета-актина проводили анализ размера методом традиционной ПЦР-ОТ, используя 2,5% агарозный гель. Данные кПЦР получали с помощью прибора Applied Biosystems Step One Plus.
Результаты
Антитела к PVRIG распознают PVRIG на сверхэкспрессирующих клетках: Чтобы провести скрининг в отношении антител, специфических к PVRIG, мы оценивали способность антител, созданных с помощью фагового подхода, связывать клеточные линии HEK, сконструированные так, чтобы сверхэкспрессировать PVRIG. Большинство антител из этого подхода после переформатирования в человеческий IgG1 связывались с клетками HEK hPVRIG, хотя и с разной аффинностью. Более того, большинство этих антител также демонстрировали низкое фоновое связывание с родительскими клеточными линиями HEK, что свидетельствует о высокой специфичности в отношении PVRIG. На фиг. 26 приведен один пример специфичности антител к PVRIG. Обобщенные результаты по всем характеристикам связывания созданных в этой фаговой кампании антител с клетками HEK hPVRIG по сравнению с контролем приведены на фиг. 30.
РНК человеческого PVRIG экспрессируется в ряде раковых клеточных линий: Для проведения первичного скрининга в отношении клеточных линий, которые можно использовать для оценки белковой экспрессии PVRIG антителами, мы изучали атлас раковых клеточных линий с высоким содержанием РНК PVRIG согласно биоинформационной оценке. Мы обнаружили четыре клеточных линии, легко доступные на коммерческой основе, которые характеризовались высокой экспрессией РНК PVRIG, которые мы отобрали для проверки методом кПЦР-анализа. Этими клеточными линиями были Jurkat, CA46, Raji и Daudi.
При проведении кПЦР-анализа мы выявили РНК PVRIG во всех четырех клеточных линиях, что согласуется с биоинформационным анализом (фиг. 27). В качестве отрицательного контроля мы включили клетки expi с относительно низкой экспрессией РНК PVRIG.
РНК человеческого PVRIG экспрессируется в Т-клетках и NK-клетках: Для проведения первичного скрининга МКПК в отношении субпопуляций, вероятно положительных в отношении белка PVRIG, согласно выявлению с помощью антител, мы провели сортировку субпопуляций МКПК и исследовали экспрессию РНК PVRIG методом кПЦР. Уровни РНК PVRIG в NK-клетках CD56+, Т-клетках CD4+, Т-клетках CD8+ и моноцитах CD14+ сравнивали с уровнями в клеточных линиях Jurkat, родительских HEK и HEK hPVRIG. Как показано на Фиг. 28, РНК PVRIG была выявлена в наибольшем количестве, и ее количество было в до 50 раз выше в Т-клетках CD4+, Т-клетках CD8+ и NK-клетках CD56+ при нормализации относительно клеток HEK ЗФБ. Аналогично с фиг. 27, клетки Jurkat также демонстрировали положительную экспрессию. В противоположность этому, моноциты CD14+ не демонстрировали высокой экспрессии PVRIG по сравнению с клетками HEK ЗФБ, что свидетельствует об очень низкой экспрессии РНК PVRIG.
Кроме анализа наивных МКПК, также в различных стимуляторных условиях активировали выбранные популяции (эффекторные лимфоциты) и оценивали экспрессию РНК PVRIG. В частности, NK-клетки активировали различными комбинациями стимуляторных цитокинов, тогда как Т-клетки поликлонально активировали человеческими активирующими гранулами dynabead или стафилококковым энтеротоксином B (СЭБ) в присутствии или отсутствие поляризующих цитокинов (подробности смотрите в описании протокола). Как показано на фиг. 29А и B, экспрессия РНК PVRIG в общем случае возросла как в NK-клетках, так и в Т-клетках после применения различных условий стимуляции, а ее степень зависела от индивидуального донора. В частности, фиг. 29А изображает экспрессию РНК PVRIG в наивных и активированных Т-клетках CD4 и NK-клетках. Фиг. 29В изображает экспрессию РНК PVRIG в наивных и активированных Т-клетках CD8.
Антитела к PVRIG наиболее сильно распознают белок PVRIG на NK-клетках в наивных и активированных первичных иммунных субпопуляциях: После подтверждения профилей РНК-экспрессии для РНК PVRIG в субпопуляциях наивных и активированных МКПК мы использовали панель антител к PVRIG для оценки белковой экспрессии. Сначала мы оценивали экспрессию PVRIG в субпопуляциях наивных МКПК. Популяцией, продемонстрировавшей самый высокий уровень PVRIG, были NK-клетки. Т-клетки CD4+ и CD8+ демонстрировали низкие уровни PVRIG, тогда как В-клетки и моноциты не имели выявляемой экспрессии. Обобщенные данные по экспрессии на NK-клетках и Т-клетках CD8+ согласно выявлению с помощью антител приведены на фиг. 31. Другие небольшие субпопуляции также демонстрировали экспрессию PVRIG и включали нетрадиционные Т-клетки, такие как NKT-клетки и γδ Т-клетки. Профиль экспрессии в популяциях МКПК был очень сходным среди всех служивших источниками и анализируемых доноров.
При оценке белка PVRIG после применения различных условий стимуляции (включая поликлональную стимуляцию, стимуляцию цитокинами и РСКЛ) не наблюдали сильной повышающей регуляции PVRIG в любых субпопуляциях МКПК, включая NK-клетки и Т-клетки CD4+ и CD8+. Более того, моноциты, поляризованные in vitro в дендритные клетки с ГМ-КСФ и IL-4, не демонстрировали выявляемой экспрессии PVRIG, что согласуется с наблюдениями для неполяризованных моноцитов.
PVRIG выявляется в клеточных линиях частью антител к PVRIG: Кроме скрининга МКПК в отношении экспрессии белка PVRIG, мы хотели понять, экспрессировался ли он также в раковых клеточных линиях. Используя положительные клеточные линии, определенные по экспрессии РНК (фиг. 27), мы решили провести скрининг антител на клетках Jurkat и CA46, так как они демонстрировали самые низкие абсолютные значения Ct по сравнению с конститутивным геном. Также мы отобрали ряд отрицательных клеточных линий для дополнительной проверки специфичности антител, которые включали OV-90, NCI-H441 и HepG2. Часть антител выявляли экспрессию белка PVRIG в клетках Jurkat и CA46 (на фиг. 30), но не в отрицательных клеточных линиях. Пример выявления PVRIG в Jurkat и CA46 приведен на фиг. 32 с репрезентативным антителом CPA.7.021. Экспрессия в Jurkat и CA46 полностью соответствовали друг другу, а интенсивность экспрессии была сходной для двух клеточных линий.
Антитела к PVRIG выявляют PVRIG яванского макака, временно экспрессируемый клетками expi: Чтобы оценить доклиническую пригодность антител к человеческому PVRIG для фармакологических исследований на яванских макаках, мы хотели понять, способны ли антитела перекрестно реагировать с PVRIG яванского макака (cPVRIG). Часть антител были способны выявлять cPVRIG, который был временно трансфицирован в клетки expi (фиг. 28). Пример антитела, которое давало отрицательное окрашивание (CPA.7.021), и антитела, которое давало положительное окрашивание (CPA.7.024), приведены на фиг. 33.
РНК PVRIG выявляется в МКПК яванского макака: Перед оценкой белка PVRIG в МКПК яванского макака мы хотели сначала определить профиль экспрессии РНК PVRIG в субпопуляциях МКПК яванского макака. Ввиду отсутствия наборов праймеров cPVRIG, мы сконструировали два набора, которые были направлены на два разных участка гена cPVRIG. Один набор праймеров был специфическим в отношении варианта Х2 cPVRIG, тогда как другой набор был способен подхватывать оба варианта, X1 и X2. Как показано на фиг. 34, оба набора праймеров были способны выявлять РНК cPVRIG на одинаковом уровне по сравнению друг с другом. Более того, в отличие от человеческих МКПК, для которых профиль РНК PVRIG в эффекторных лимфоцитах (NK- и Т-клетках) отличался от моноцитов, РНК cPVRIG экспрессировалась на сходном уровне среди всех оцениваемых субпопуляций МКПК от всех доноров.
Экспрессия белка PVRIG в МКПК яванского макака является очень низкой или отрицательной: После установления профиля РНК cPVRIG для МКПК яванского макака мы провели скрининг в отношении присутствия белка cPVRIG в МКПК яванского макака, используя выбранную панель антител к человеческому PVRIG. Антитела для скрининга МКПК выбирали на основании их способности связывать временно экспрессирующие cPVRIG клетки и/или функциональной активности. Как показано на фиг. 35, мы смогли выявить низкий уровень экспрессии cPVRIG в субпопуляции лимфоцитов CD16+ (NK-клетки) для ряда антител, но не в субпопуляции лимфоцитов CD3+ (Т-клетки) или субпопуляции миелоидных клеток CD14+ CD56+ (моноциты). Несмотря на эти данные, те антитела, которые демонстрировали положительное выявление по сравнению с контролем (как показано непрерывной черной линией), не коррелировали с теми, которые были способны связывать временно экспрессирующие cPVRIG клетки. Например, уровень окрашивания CPA.7.021 был больше, чем для CPA.7.024, несмотря на то, что первое не связывало временно экспрессирующие cPVRIG клетки (смотрите фиг. 35).
Выводы и заключения: Используя фаговую платформу для антител, нам удалось успешно создать моноклональные антитела против человеческого антигена PVRIG. Используя сконструированные сверхэкспрессирующие клетки, а также набор раковых клеточных линий, мы показали, что наши антитела являются высокоспецифическими в отношении антигена PVRIG и способны выявлять экспрессию белка, которая коррелировала с экспрессией РНК. После анализа субпопуляций человеческих МКПК мы показали, что белок PVRIG наиболее сильно экспрессируется на NK-клетках с низкой экспрессией на традиционных Т-клетках CD3+ и невыявляемой на В-клетках и миелоидных клетках. Экспрессия сильно не менялась после применения к этим типам клеток различных условий стимуляции. Также мы показали, что панель наших антител перекрестно реагируют с антигеном PVRIG яванского макака (cyno), путем оценки их связывания со сверхэкспрессирующими клетками. Однако комбинация низкого уровня связывания этой панели антител с МКПК яванского макака, отсутствие корреляции белка с РНК и несоответствие их способности связываться со сверхэкспрессирующими клетками (по сравнению с МКПК) свидетельствует, что антиген PVRIG в МКПК яванского макака может присутствовать в очень малом количестве/быть отрицательным или он экспрессируется в отличной/более сложной форме по сравнению со сверхэкспрессирующими клетками.
Пример 1D
Экспрессия PVRIG в субпопуляциях МКПК от здоровых доноров: Исследовали экспрессию PVRIG в субпопуляциях МКПК от здоровых доноров (стратегия гейтинга приведена на фиг. 1a). Было показано, что в исследуемых образцах PVRIG экспрессируется на Т-клетках CD8+ (данные не показаны), γδ Т-клетках CD8α+ (данные не показаны), вдвойне отрицательных γδ Т-клетках (данные не показаны) и в меньшей степени также на Т-клетках CD4+ (данные не показаны) здоровых доноров МКПК (n = 5).
Пример 1E
Коэкспрессия PVRIG с PD1, TIGIT и HLA-DR в асцитах рака яичника, МПК из СМС, КРР и в покоящихся и алло-активированных здоровых МКПК: PVRIG коэкспрессируется с TIGIT на Т-клетках CD8+ в асцитах рака яичника (данные не показаны). В этом образце наблюдали смешанный уровень экспрессии PVRIG, которая перекрывалась с экспрессией PD-1. Низкий уровень HLA-DR коррелировал с низким уровнем экспрессии PVRIG. Очень низкий уровень PVRIG наблюдали на Т-клетках CD4+ в этом конкретном образце, что свидетельствует об отсутствии корреляции с PD1, TIGIT и HLA-DR.
В МПК пациентов с СМС и КРР PVRIG коэкспрессируется с TIGIT на Т-клетках CD8+ (данные не показаны). В этом образце наблюдали низкие уровни экспрессии PVRIG, что коррелирует с низкими уровнями TIGIT и HLA-DR. ОИЛ от этого пациента имели небольшую популяцию CD8+, которая демонстрировала положительное окрашивание в отношении поверхностного PVRIG, которое также было положительным в отношении PD1 и TIGIT (данные не показаны). Внутриклеточное окрашивание выявило ярко выраженное окрашивание PVRIG, которое отображало профиль экспрессии PD-1, демонстрируя две разные популяции, которые представляли собой PD1-PVRIG- и PD1+PVRIG+ (данные не показаны). PVRIG+ Т-клетки CD8+ согласно данным внутриклеточного окрашивания лучше коррелируют с HLA-DR+ и TIGIT+. PVRIG не выявлялся на поверхности Т-клеток CD4+, и только небольшая часть Т-клеток CD4+ демонстрировала положительное внутриклеточное окрашивание PVRIG в популяции PD1+. Из-за очень небольшой внутриклеточной популяции PVRIG+ трудно определить, коэкспрессируется ли PVRIG с TIGIT и HLA-DR.
В здоровых МКПК окрашивание PVRIG на Т-клетках CD8 отображало профиль экспрессии PD-1 и TIGIT, демонстрируя наличие разных популяций PD1-PVRIG- и PD1+PVRIG+ и разных популяций TIGIT-PVRIG- и TIGIT+PVRIG+ (данные не показаны). PVRIG не был выявлен на Т-клетках CD4+. Интересно, что после алло-активации коэкспрессию PVRIG и PD-1 наблюдали на CD4+ (но не CD8+) (данные не показаны).
В целом, было показано, что PVRIG коэкспрессируется с TIGIT в Т-клетках CD8+ из асцитов рака яичника, МПК пациентов с СМС и КРР и с PD-1 в МКПК здоровых доноров, и с PD1 в Т-клетках CD4+ алло-активированных МКПК от здоровых доноров.
Пример 1F
Экспрессия PVRIG в популяциях лимфоцитов из здоровых МКПК, рака мочевого протока, колоректального рака, асцитов рака яичника и рака легкого: Результаты: Экспрессию PVRIG на Т-клетках CD4+ и CD8+, NK-клетках и на NKT-клетках CD4+ и CD8+ анализировали в МКПК и миндалевидных железах здоровых доноров и в ОИЛ из рака мочевого протока, колоректального рака, асцитов рака яичника, рака легкого и меланомы.
В МКПК здоровых доноров (n = 5) и в ОИЛ асцитов рака яичника (n = 1) выявили высокие уровни экспрессии PVRIG на NK-клетках (данные не показаны) и NKT-клетках CD8+ (данные не показаны) и в меньшей степени также на Т-клетках CD8+ (данные не показаны) и NKT-клетках CD4+ (данные не показаны). Т-клетки CD4+ также демонстрировали положительное окрашивание в отношении PVRIG в некоторых МКПК, однако уровни экспрессии были довольно низкими (данные не показаны).
Кроме того, экспрессию PVRIG выявили на Т-клетках CD4+ для двух из 6 исследуемых ОИЛ колоректального рака и в ОИЛ рака легкого (n = 3) (данные не показаны), а также в NK-клетках из ОИЛ рака мочевого протока (n = 1).
В ОИЛ меланомы не было выявлено экспрессии PVRIG вследствие отсутствия ОИЛ в исследуемом образце.
Пример 1G
Проводили дополнительные оценки, чтобы определить дополнительные ткани, сверхэкспрессирующие PVRIG в мышиных и человеческих клеточных линиях.
Реагенты: Зонды TaqMan для человеческого PVRIG (Life technologies) Hs04189293_g1, Кат. # 4331182, зонд TaqMan для конститутивного гена (HSKG) (Life technologies) человеческого RPL19 Mm 01577060_gH, человеческого HPRT1 Hs02800695_m1, человеческого SDHA Hs00417200_m1, человеческого PBGD Hs00609296_g1 и человеческого TATA-бокса Hs00375874_g1. Зонды TaqMan для мышиного PVRIG (Life technologies) специальные зонды TaqMan CC70L8H, CC6RN19. Зонды TaqMan для конститутивного гена (HSKG) (Life technologies) мышиного RPL19: Mm02601633_g1. Быстрый усовершенствованный мастер-микс ABI TaqMan, партия № 4444557, Applied Biosystem. Коммерческие человеческие и мышиные раковые клеточные линии от Американской коллекции типовых культур (ATCC) и CLS (Cell line service) уточнены в таблице 1. Экстракцию РНК из человеческих и мышиных клеточных линий проводили с помощью мини-набора RNAeasy (Qiagen кат. # 74014). кДНК получали, используя набор для обратной транскрипции кДНК высокой емкости (Applied Biosystems кат. #4368814). Коммерческое мышиное поликлональное анти-PVRIG Ab MaxPab (B01), Abnova, Кат. #H00079037-B01, разведенное 1:200. Мышиный IgG1, Life Technologies, Кат. #MG100, разведенный 1:200. Коммерческое мышиное поликлональное анти-PVRIG Ab, Sigma, Кат. #SAB1407935, 10 мкг/мл. Хроматографически очищенный мышиный IgG, цельная молекула, Jackson, Кат. #015-000-003, 10 мкг/мл. Козье анти мышиное-PE антитело, Jackson, Кат. #115-116-146, разведенное 1:100. Специальное поликлональное крысиное антитело против мышиного PVRIG, Партия #20153456C.1, Aldevron, 10 мкг/мл. Специальный крысиный общий IgG, Партия #GV20884.1, Aldevron, 10 мкг/мл. Козье антикрысиное-PE антитело, Jackson, кат. # 112-116-143, разведенное 1:100. Античеловеческий PVRIG-CPA.7.024 mIgG1, конъюгированный с AF647, 10 мкг/мл. Античеловеческий PVRIG-CPA.7.050 mIgG1, конъюгированный с AF647, 10 мкг/мл. Античеловеческий PVRIG-CPA.7.005 mIgG1, конъюгированный с AF647, 10 мкг/мл. Античеловеческий PVRIG-CPA.7.002 mIgG1, конъюгированный с AF647, 10 мкг/мл. IgG1 Синагис, конъюгированный с A647, 10 мкг/мл. Античеловеческий PVRIG-CPA.7.021 mIgG1, конъюгированный с AF647, 10 мкг/мл. IgG2 Синагис, конъюгированный с A647, 10 мкг/мл. Кроличье поликлональное анти-PVRIG Ab, Sigma, Кат. #HPA047497, разведенное 1:300. Козье антикроличье-HRP, Jackson, Кат. # 111-035-003, разведенное 1:100. VioBlue, фиксируемый краситель для анализа жизнеспособности 450, BD Bioscience, кат. # 562247, разведенный 1:1000. Человеческий краситель Trustain FcX, Biolegend, Кат. #422302. Крысиный антимышиный CD16/CD32 Fc-блокатор, BD, Кат. #553142. Раствор для электропорации Ingenio, Mirus, Кат. #MIR50114. ON-TARGETplus SMARTpool для миРНК человеческого PVRIG, Dharmacon, Кат. # L-032703-02. ON TARGET plus, не нацеленный на миРНК, Dharmacon, Кат. # D-001810-01-05. Человеческие клеточные линии, применяемые в исследовании, приведены на фиг. 53.
Экспрессия транскрипта. Количественная ПЦР-ОТ (кПЦР-ОТ): Экстракцию РНК (1-5 мкг) из человеческих и мышиных клеточных линий (подробное описание приведено выше в таблицах 1 и 2) проводили в соответствии с протоколами производителя. кДНК получали в соответствии с протоколами производителя (1 мкг РНК, разведенной в 20 мкл смешанной реакции кДНК). кДНК, полученную, как описано выше, разведенную 1:10 (что соответствует 25 нг РНК на реакцию), использовали в качестве матрицы для реакций ПЦР-ОТ, используя генноспецифические зонды TaqMan (подробное описание приведено в материалах и методах, 1.1 1-4). Выявление проводили, используя устройство QuantStudio 12k. Регистрировали цикл, на котором реакции достигали порогового уровня флуоресценции (Ct = Пороговый цикл), и использовали для расчета относительного количества транскрипта в ОТ-реакциях. Абсолютное количество рассчитывали, используя уравнение Q=2^-Ct. Полученные в результате относительные количества нормализовали к относительным количествам конститутивных генов, mRPL19 или hRPL19.
Определение экспрессии белка методом вестерн-блоттинга (ВБ): Экспрессию человеческого PVRIG в человеческих клеточных линиях анализировали методом ВБ, используя экстракты цельных клеток (45 мкг для раковых клеточных линий и 30 мкг для сверхэкспрессирующей клеточной линии и отрицательной контрольной клеточной линии). Применяли коммерческое кроличье поликлональное pAb к человеческому PVRIG, Sigma, кат. # HPA047497, разведенное 1:300 в 5% БСА/ТБСТ, а после этого - вторичное козье антикроличье Ab, конъюгированное с пероксидазой (Jackson, кат. # 111-035-003), разведенное 1:20000 в 5% молочном ТБСТ.
Анализ экспрессии белка методом проточной цитометрии (FACS): Экспрессию белка PVRIG на поверхности клеток анализировали методом FACS. Человеческие или мышиные клеточные линии окрашивали реагентом VioBlue, разведенным 1:1000 в ФСБ. Клетки инкубировали 15 мин при КТ, а затем один раз промывали ФСБ. Клеточные линии для анализа эндогенного белка предварительно инкубировали с блокирующими Fc-рецепторы растворами, приведенными выше в разделе материалов (использовали 2,5 мкл/реакцию человеческого блокатора и 1 мкл/реакцию мышиного блокатора в соответствии с процедурами производителя). Чтобы выявить человеческий белок PVRIG, клетки окрашивали коммерческим поликлональным антителом к человеческому PVRIG или специальными моноклональными mAb к человеческому PVRIG (Inc production, подробно описано выше в разделе материалов и методов), разведенными до концентрации 10 мкг/мл или 1:200 (для Sigma Ab и для mAb или для Abnova Ab соответственно), или изотипическим контрольным IgG1 в той же концентрации, после чего применяли козье антимышиное РЕ-конъюгированное Ab.
Чтобы выявить мышиный белок PVRIG, клетки окрашивали специальным крысиным поликлональным pAb против мышиного PVRIG (Aldevron,) разведенным до концентрации 10 мкг/мл, или цельными молекулами крысиного IgG в качестве изотипических контролей в той же концентрации, после чего применяли ослиное антикрысиное РЕ-конъюгированное Ab, разведенное 1:100.
Нокдаун PVRIG: Нокдаун эндогенного человеческого PVRIG осуществляли путем временной трансфекции миРНК. Трансфекцию 100 пмоль пула миРНК PVRIG или рандомизированной миРНК проводили путем электропорации, используя нуклеофектор Amaxa и раствор для электропорации MI ЕОS Ingenio, как описано выше в разделе материалов и методов и в соответствии с процедурами производителя. Через 48 часов после трансфекции клетки собирали для дополнительного анализа методом кПЦР-ОТ и FACS.
Результаты: Эндогенная экспрессия транскрипта PVRIG в человеческих и мышиных клеточных линиях методом кПЦР-ОТ
Человеческие клеточные линии: Чтобы удостовериться в наличии транскрипта PVRIG в человеческих клеточных линиях (приведенных на фиг. 53), проводили кПЦР-ОТ, используя специфический зонд TaqMan, как описано выше в разделе Материалы и методы. Как показано на фиг. 55 транскрипт человеческого PVRIG наблюдали, используя зонд TaqMan Hs04189293_g1, на относительно высоких уровнях в клеточных линиях Jurkat (A, B), HUT78 (A, B) и HL60 (B). Более низкий уровень транскрипта наблюдали в клеточных линиях THP1, RPMI8226 (B). Все другие клеточные линии демонстрируют очень низкий уровень транскрипта или его отсутствие.
Эндогенная экспрессия транскрипта PVRIG в мышиных клеточных линиях методом кПЦР-ОТ: Чтобы удостовериться в наличии транскрипта PVRIG в мышиных клеточных линиях (приведенных на фиг. 53), проводили кПЦР-ОТ, используя специфический зонд TaqMan, как описано выше в разделе Материалы и методы. Как показано на фиг. 56, транскрипт мышиного PVRIG наблюдали, используя зонд TaqMan CC70L8H, на относительно высоких уровнях в клеточных линиях NIH/3T3, Renca, SaI/N и J774A.1 (A). Более низкий уровень транскрипта наблюдали в клеточных линиях CT26 (A) и B-104-1-1(B). Все другие клеточные линии демонстрируют очень низкий уровень транскрипта.
Эндогенная экспрессия белков PVRIG в человеческих клеточных линиях методом ВБ: ВБ-анализ эндогенной экспрессии белка PVRIG проводили для лизатов различных человеческих раковых клеточных линий, уточненных на фиг. 53, используя коммерческое pAb против человеческого PVRIG (Sigma, HPA047497), как описано выше в разделе Материалы и методы. В качестве положительного контроля использовали экстракт из цельных клеток стабильного пула клеток HEK293, сверхэкспрессирующих PVRIG, тогда как клетки, трансцфицированные пустым вектором, служили в качестве отрицательного контроля. Как показано на фиг. 57, полоса белка, соответствующая ~35 кДа, была выявлена для положительных контрольных сверхэкспрессирующих клеток HEK293 (дорожка 2), а также для клеточной линии Jurkat (дорожка 3). Отсутствие экспрессии человеческого PVRIG было зарегистрировано в клетках с пустым вектором (дорожка 1), которые служили в качестве отрицательного контроля, и в клеточной линии ZR75-1 (дорожка 4).
Эндогенная экспрессия белков PVRIG в человеческих и мышиных клеточных линиях методом FACS:
Человеческая клеточная линия: Чтобы проверить наличие эндогенной экспрессии человеческого PVRIG на поверхности клеток, исследовали различные клеточные линии (уточненные на Фиг. 53), как описано выше в разделе Материалы и методы. Клеточные линии окрашивали коммерческим Ab (Abnova) или изотипическим контролем с последующим применением вторичного козьего антимышиного PE Ab. Анализ проводили методом FACS. Связывание антитела Abnova наблюдали в человеческой раковой клеточной линии Jurkat по сравнению со связыванием изотипического контроля. Отсутствие связывания Abnova Ab наблюдали в других исследуемых клеточных линиях: для Capan2 и ZR75-1 по сравнению со связыванием изотипического контроля, дополнительный анализ FACS проводили с применением коммерческого Ab от Sigma для различных человеческих клеточных линий (Jurkat, HUT78, Karpas299 и NK-YTS), связывание наблюдали только в клетках Jurkat, но в других клеточных линиях связывание не наблюдали (данные не показаны).
Дополнительный анализ для подтверждения наличия эндогенного человеческого PVRIG в клеточной линии Jurkat проводили, исследуя связывание различных моноклональных антител согласно изобретению. Клеточную линию Jurkat окрашивали пятью специальными mAb против человеческого PVRIG (CPA.7.024, CPA.7.050, CPA.7.005, CPA.7.002 и CPA.7.021), конъюгированными с AF647, или родственным изотипическим контрольным Ab, конъюгированным с AF647. Анализ проводили методом FACS. Экспрессию человеческого PVRIG в человеческой клеточной линии Jurkat наблюдали только с помощью CPA.7.021 и CPA.7.050 по сравнению с экспрессией изотипического контроля. Отсутствие связывания в отношении человеческого PVRIG наблюдали в клеточной линии Jurkat при применении других трех mAb.
Мышиная клеточная линия: Чтобы проверить наличие эндогенной экспрессии мышиного PVRIG на поверхности клеток, исследовали различные клеточные линии: J774A.1, NIH/3T3, SaI/N и Renca (уточненные на фиг. 54), как описано выше в разделе Материалы и методы. Клеточные линии окрашивали специальным поликлональным крысиным Ab против мышиного PVRIG (Aldevron) или изотипическим контролем (Aldevron) с последующим применением вторичного козьего антимышиного PE Ab. Анализ проводили методом FACS. Отсутствие связывания в отношении мышиного белка PVRIG наблюдали во всех исследуемых клеточных линиях с помощью поликлонального Ab от Aldevron (данные не показаны).
Нокдаун человеческого PVRIG в человеческих клеточных линиях: Чтобы дополнительно подтвердить наличие эндогенной экспрессии белка PVRIG в клеточной линии Jurkat, использовали пул миРНК человеческого PVRIG для нокдауна, как описано в разделе Материалы и методы. Через 48 часов после трансфекции миРНК клетки собирали для дополнительного анализа методом кПЦР-ОТ и методом FACS.
Нокдаун человеческого PVRIG в человеческих клеточных линиях, исследуемый методом кПЦР: Как показано На фиг. 58, уровень транскрипта человеческого PVRIG в клетках Jurkat, трансфицированных миРНК человеческого PVRIG, значительно снижен (правый столбик гистограммы) по сравнению с клетками, трансфицированными рандомизированной миРНК (левый столбик гистограммы), согласно анализу методом кПЦР-ОТ, как описано в разделе Материалы и методы.
Нокдаун человеческого PVRIG в человеческих клеточных линиях, исследуемый методом FACS: Дополнительный анализ мембранной экспрессии человеческого PVRIG в таких же трансфицированных миРНК клетках проводили методом FACS. Как показано на фиг. 59, мембранная экспрессия белка человеческого PVRIG снижена в клетках, трансфицированных миРНК PVRIG (зеленый цвет в случае CPA.7.021 mAb или красный в случае Sigma Ab), по сравнению с клетками, трансфицированными рандомизированной миРНК (оранжевый). Кратность изменения (анти-PVRIG против изотипического контроля) в клеточной линии Jurkat снижена от 8 раз до 3,3 раза при применении Sigma Ab или от 15,3 раза до 2,8 раза при применении CPA.7.021 mAb.
В этот отчет включены предварительные данные по эндогенной экспрессии PVRIG в клеточных линиях, как на уровне РНК, так и на уровне белка в человеческих и мышиных клеточных линиях.
Различные человеческие раковые клеточные линии исследовали методами кПЦР-ОТ, ВБ и FACS в отношении эндогенной экспрессии PVRIG.
Экспрессию человеческого PVRIG на клеточной поверхности наблюдали в клеточной линии Jurkat при применении коммерческих поликлональных Ab (Sigma и Abnova) и мышиных моноклональных Ab (Inc), как показано на фиг. 4A и 4B соответственно. Эти наблюдения коррелируют с уровнями РНК-транскрипта, как показано на фиг. 1 A и B, и с результатами ВБ, как показано на фиг. 3.
Дополнительное подтверждение наличия эндогенного человеческого PVRIG в клеточных линиях Jurkat получили с помощью эксперимента с нокдауном, подтверждающим четкое снижение уровня РНК-транскрипта после трансфекции миРНК PVRIG, как показано на фиг. 5, и так же наблюдали снижение в экспрессии белка на клеточной поверхности в клеточных линиях Jurkat, как показано на фиг. 6, с помощью коммерческого Ab и моноклонального Ab.
Различные мышиные клеточные линии исследовали методами кПЦР-ОТ и FACS в отношении эндогенной экспрессии PVRIG. На уровне транскрипта наличие PVRIG наблюдали в клеточных линиях J774A.1, NIH/3T3, SaI/N и Renca, как показано на фиг. 2A и B. Хотя при применении поликлонального Ab (Aldevron) мембранную экспрессию мышиного PVRIG в этих исследуемых клеточных линиях не наблюдали (данные не показаны). На фиг. 60 и на фиг. 61 приведены обобщающие результаты наблюдений, описанных в этом отчете, и указаны клеточные линии, демонстрирующие корреляцию между кПЦР и FACS, подтверждаемую нокдауном.
Пример 1H
Задачей этих экспериментов была оценка экспрессии белка PVRIG на покоящихся или активированных человеческих ОИЛ (опухоль-инфильтрирующих лимфоцитах), выделенных из образцов человеческой меланомы и размноженных в присутствии специфических к меланоме антигенов и IL2. Получали человеческие mAb, направленные против внеклеточного домена (ВКД) человеческого PVRIG. Эти Ab напрямую метили Alexa flour 647, чтобы исследовать экспрессию PVRIG на клетках методом FACS-анализа.
Материалы и методы
ОИЛ: В этой серии экспериментов использовали три разных опухоль-инфильтрирующих лимфоцита (ОИЛ) из резецированных метастазов от трех пациентов с меланомой: 1) TIL-412, HLA-A2-Mart1-специфический; 2) TIL-F4, HLA-A2-gp100-специфический, и 3) TIL-209, HLA-A2-gp100-специфический. Человеческие ОИЛ (>90% CD8+) размораживали за 24 часа до начала эксперимента. Клетки размораживали в 12 мл среды ОИЛ (IMDM + 10% человеческая сыворотка + 1% глутамакс + 1% Na-пируват + 1% заменимые аминокислоты + 1% пен.-стреп.), дополненной 300 Е/мл rhIL2 (Biolegend 509129). Клетки оставляли для восстановления после заморозки на 24 часа.
Аналитические условия: После восстановления исследовали ОИЛ в четырех разных условиях: 1) Покоящиеся, с применением 300 Е/мл IL2 (Biolegend, кат.-589106), 2) с поликлональной активацией Т-клеток, с использованием 1 мкг/мл связанного с планшетом анти-CD3 антитела (клон OKT3 от eBioscience, кат.-16-0037-85) + 2 мкг/мл анти-CD28 ab (клон CD28.2 от eBioscience, кат.-16-0289-85) + 300 Е/мл IL2. 3) совместно культивируемые (1:1) с клетками меланомы Mel888 (LIMS ID: CL-216) (HLA-A-отрицательными) и 4) совместно культивируемые (1:1) с клетками меланомы Mel624 (LIMS ID CL-218) (HLA-A2 + Mart1/gp100-положительными).
Через 12 часов покоя/активации/совместного культивирования клетки исследовали методом FACS в отношении экспрессии PVRIG, а также экспрессии представителей пути PVRIG и других поверхностных маркеров.
Окрашивание клеток: Через 12 часов клетки собирали и дважды промывали ФСБ. Клетки окрашивали при комнатной температуре в течение 20 минут ФСБ, дополненным 1/1000 фиксируемого красителя в отношении жизнеспособности efluor 450 (BD horizon, кат.-562247). После окрашивания клетки дважды промывали ФСБ и окрашивали в течение 15 минут на льду буфером FACS (ФСБ + 0,5% БСА + 2 мМ ЭДТА + 0,05% азид), дополненным 1/25 человеческого FC-блокатора Truestain (Biolegend, 422302). После FC-блокирования клетки окрашивали на льду в течение 30 минут с помощью Ab в концентрациях, приведенных в таблице 1.
После окрашивания клетки промывали один раз и пересуспендировали в буфере FACS для анализа. Компенсационную калибровку, проводили, используя компенсационные гранулы (BD, 552843). Одну каплю гранул окрашивали в течение 30 минут вышеуказанными антителами. Окрашивание гранул проводили, используя те же концентрации, что и для окрашивания клеток. После окрашивания гранул проводили компенсацию на устройстве MacsQuant FACS в соответствии со стандартной процедурой. Все образцы исследовали на анализаторе MACSQuant (Miltenyi), а данные анализировали с помощью программного обеспечения Tree Star FlowJo (v10.0.8).
PVRIG экспрессируется на человеческих покоящихся ОИЛ: Покоящиеся ОИЛ, культивируемые в течение 12 часов только с 300 Е/мл IL2, окрашивали в отношении экспрессии PVRIG и анализировали методом FACS. Стратегия гейтинга для ОИЛ: Сначала проводили гейтинг лимфоцитов в соответствии с размером и степенью детализации на графике FCS:SSC, затем проводили гейтинг одиночных клеток в соответствии с FSC-H и FSC-A, затем проводили гейтинг живых клеток в соответствии окрашиванием в отношении жизнеспособности на графике Vioblue:FSC, затем проводили гейтинг клеток CD8+ в соответствии окрашиванием CD8 на графике CD8:FSC. Затем уровни экспрессии PVRIG наносили на график в соответствии окрашиванием PVRIG на гистограммах.
Наблюдается понижение регуляции экспрессии PVRIG на человеческих ОИЛ после активации анти-CD3 + анти-CD28 ab: Человеческие ОИЛ, культивируемые в течение 12 часов с анти-CD3 + анти-CD28 ab + IL2, окрашивали в отношении экспрессии PVRIG и анализировали методом FACS. После активации наблюдалось понижение регуляции экспрессии PVRIG на поверхности всех трех исследуемых ОИЛ по сравнению с покоящимися ОИЛ (данные не показаны).
Наблюдается небольшое понижение регуляции экспрессии PVRIG на человеческих ОИЛ после совместного культивирования с Mel888: Человеческие ОИЛ, культивируемые в течение 12 часов с клетками Mel888, окрашивали в отношении экспрессии PVRIG и анализировали методом FACS. После совместного культивирования с Mel888 наблюдалось небольшое понижение регуляции экспрессии PVRIG на поверхности всех трех исследуемых ОИЛ по сравнению с покоящимися ОИЛ.
Наблюдается понижение регуляции экспрессии PVRIG на человеческих ОИЛ после совместного культивирования с Mel624: Человеческие ОИЛ, культивируемые в течение 12 часов с клетками Mel624, окрашивали в отношении экспрессии PVRIG и анализировали методом FACS. После совместного культивирования с Mel624 наблюдалось небольшое понижение регуляции экспрессии PVRIG на поверхности всех трех исследуемых ОИЛ по сравнению с покоящимися ОИЛ.
Экспрессия других представителей пути на покоящихся ОИЛ: Человеческие ОИЛ, культивируемые в течение 12 часов только с IL2, окрашивали в отношении экспрессии CD96, PVR, PVRL2, TIGIT и DNAM1 и анализировали методом FACS. CD96, TIGIT и DNAM1 экспрессируются на всех трех исследуемых ОИЛ. PVR также экспрессируется на поверхности всех трех ОИЛ, но на относительно низких уровнях. PVRL2 не выявляется ни в одном из ОИЛ.
Экспрессия других представителей пути на ОИЛ, активированных анти-CD3 и анти-CD28 ab: Человеческие ОИЛ, культивируемые в течение 12 часов с анти-CD3 и анти-CD28 ab, окрашивали в отношении экспрессии CD96, PVR, PVRL2, TIGIT и DNAM1 и анализировали методом FACS. После активации анти-CD3 + анти-CD28 ab наблюдалось понижение регуляции CD96, небольшое повышение регуляции PVR, небольшое повышение регуляции TIGIT и также повышение регуляции DNAM1.
Экспрессия других представителей пути на ОИЛ, совместно культивируемых с Mel888: Человеческие ОИЛ, совместно культивируемые в течение 12 часов с клетками Mel888, окрашивали в отношении экспрессии CD96, PVR, PVRL2, TIGIT и DNAM1 и анализировали методом FACS. После совместного культивирования с Mel888 наблюдалось понижение регуляции CD96, сильное повышение регуляции PVR, понижение регуляции TIGIT и DNAM1, а также небольшая индукция PVRL2.
Экспрессия других представителей пути на ОИЛ, совместно культивируемых с Mel624: Человеческие ОИЛ, совместно культивируемые в течение 12 часов с клетками Mel624, окрашивали в отношении экспрессии CD96, PVR, PVRL2, TIGIT и DNAM1 и анализировали методом FACS. Стратегию гейтинга осуществляли в соответствии с фиг. 1. После совместного культивирования с Mel624 наблюдалось понижение регуляции CD96, сильное повышение регуляции PVR, стабильная регуляция или небольшое повышение регуляции TIGIT, понижение регуляции DNAM1 и небольшая индукция PVRL2.
Экспрессия PD1 на ОИЛ: Человеческие ОИЛ, культивируемые в течение 12 часов только с IL2 или активированные анти-CD3 + анти-CD28 ab, или совместно культивируемые с клетками Mel888 или Mel624, окрашивали в отношении экспрессии PD1 и анализировали методом FACS. Как видно на фиг. 16 и фиг. 17, PD1 экспрессируется только на покоящихся TIL412. Никаких изменений в экспрессии PD1 не наблюдали после совместного культивирования с Mel888, но наблюдалось повышение регуляции PD1 во всех трех ОИЛ после совместного культивирования с Mel624 или после активации анти-CD3 + анти-CD28 ab.
Выводы и заключения: Для всех трех исследованных ОИЛ:
CPA.7.021 ab против PVRIG окрашивает ОИЛ (до 2,6 раза)
Наблюдается понижение регуляции экспрессии PVRIG после 12 часов активации анти-CD3 + анти-CD28 ab или после совместного культивирования с Mel624 (практически до фонового уровня).
Покоящиеся ОИЛ экспрессируют CD96, TIGIT и DNAM1(до 35, 12 и 79 раз соответственно)
Наблюдается понижение регуляции экспрессии CD96 после активации (от 35 до ~11 раз) или совместного культивирования с нерелевантной (HLA-A2) меланомой
Наблюдается повышение регуляции экспрессии DNAM1 после активации αCD3/CD28 ab (от 79 до 102 раз), но сильное понижение регуляции после совместного культивирования ОИЛ с Mel (до 8 раз меньше).
Наблюдается небольшое понижение регуляции экспрессии TIGIT после совместного культивирования ОИЛ с клеточной линией mel888, и стабильная регуляция с небольшим повышением регуляции после совместного культивирования с Mel624 или активации анти-CD3 + анти-CD28 ab.
Наблюдается повышение регуляции экспрессии PD1 после активации (от 0 до 18 раз)
Высокие уровни PVR были выявлены после совместного культивирования ОИЛ с меланомами (от <2 до 18 раз).
Покоящиеся TIL-412 демонстрируют положительное окрашивание в отношении PD1. TIL-F4 также является немного положительным в отношении PD1, тогда как TIL-209 является отрицательным. Обобщенные результаты по изменениям в уровнях экспрессии всех исследуемых компонентов в разных условиях можно найти в таблице 2.
Таблица 2
Пример 1I. Экспрессия PVRIG на покоящихся и активированных человеческих Т-клетках и ОИЛ
Задачей этого примера была оценка экспрессии белка PVRIG на покоящихся и активированных человеческих выделенных первичных Т-клетках CD4+ и CD8+, а также ОИЛ (опухоль-инфильтрирующих лимфоцитах), выделенных из образцов человеческой меланомы и размноженных в присутствии специфических к меланоме антигенов и IL2. Получали человеческие mAb, направленные против внеклеточного домена (ВКД) человеческого PVRIG. Эти Ab напрямую метили Alexa flour 647, чтобы исследовать экспрессию PVRIG на клетках методом FACS-анализа.
Материалы и методы
ОИЛ: В этой серии экспериментов использовали два ОИЛ из резецированных метастазов от трех пациентов с меланомой:
(ОИЛ) TIL-Mart1, HLA-A2-Mart1-специфический
(ОИЛ) TIL-209, HLA-A2-gp100-специфический
Человеческие ОИЛ (>95% CD8+) размораживали за 24 часа до начала эксперимента. Клетки размораживали в 12 мл среды ОИЛ (IMDM + 10% человеческая сыворотка + 1% глутамакс + 1% Na-пируват + 1% заменимые аминокислоты + 1% пен.-стреп.), дополненной 300 Е/мл rhIL2 (Biolegend 509129). Клетки оставляли для восстановления на 24 часа.
Первичные Т-клетки: В этой серии экспериментов использовали клетки от двух разных доноров:
CD4+ и CD8+ от донора #147
CD4+ и CD8+ от донора #186
Человеческие первичные клетки (>95% очистки) размораживали за 24 часа до начала эксперимента. Клетки размораживали в полной среде RPMI (RPMI + 10% ФБС + 1% глутамакс + 1% Na-пируват + 1% пен.-стреп.), дополненной 300 Е/мл rhIL2 (Biolegend 509129). Клетки оставляли для восстановления на 24 часа.
Аналитические условия: После восстановления клетки активировали, используя поликлональную активацию Т-клеток, с помощью 1 мкг/мл связанного с планшетом анти-CD3 антитела (клон Ucht-1 от BD-pharmingen, кат.-555329), 2 мкг/мл анти-CD28 ab (клон CD28.2 от eBioscience, кат.-16-0289-85) и 300 Е/мл IL2.
Активацию проводили в течение 24 ч, 48 ч, 72 ч и 144 ч.
Окрашивание клеток: Клетки собирали и промывали ФСБ. Клетки окрашивали при комнатной температуре в течение 10 минут ФСБ, дополненным 1/1000 фиксируемого красителя в отношении жизнеспособности efluor 450 (BD horizon, кат.-562247). После окрашивания клетки дважды промывали ФСБ и окрашивали с помощью Ab в концентрациях, приведенных на фиг. 64, в течение 30 минут на льду в буфере FACS (ФСБ + 0,5% БСА + 2 мМ ЭДТА + 0,05% азид), и в концентрациях, приведенных на фиг. 64.
После окрашивания клетки один раз промывали и пересуспендировали в буфере FACS для анализа. 8 H #138) to Human T cell lines.
Результаты: Человеческие Т-клетки от двух разных доноров и ОИЛ оставляли необработанными (покоящимися) или поликлонально стимулировали в течение разных отрезков времени, как описано в разделе Материалы и методы. Состояние клеточной активации оценивали по определению поверхностной экспрессии CD137 и PD-1 в каждый момент времени по сравнению с изотипическим контролем (FMO), как показано для активированных Т-клеток CD8+, CD4+ и ОИЛ (на фиг. 69А, B и C соответственно). Как и ожидалось, после активации была выявлена повышенная экспрессия PD-1 и CD137 (на фиг. 69А, B и C).
Экспрессия PVRIG наблюдалась в покоящихся Т-клетках CD4+ и CD8+ с более высокой экспрессией в клетках CD8+ (6-8 раз) по сравнению с клетками CD4+ (3 раза), и снижалась после активации (на фиг. 70А, B и C). На 3-6 дни после активации экспрессия PVRIG была повышена на Т-клетках CD8+ (4-5 раз) и CD4+ (2-3 раза), как видно на фиг. 70.
Кроме того, экспрессию PVRIG также наблюдали на покоящихся ОИЛ Mart1 и 209, при этом после активации экспрессия снижалась (на фиг. 71А, B и C). На 3-6 дни после активации экспрессия PVRIG была повышена, как видно на фиг. 71, по сравнению с 1-2 днем активации.
Пример 2. Создание и получение характеристик пулов PVRIG-экспрессирующих стабильно трансфицированных клеток
Были созданы рекомбинантные стабильные пулы клеточных линий, сверхэкспрессирующих человеческие и мышиные белки PVRIG, для применения в определении действия PVRIG на иммунитет, для получения характеристик PVRIG и для определения иммунорегуляторных терапевтических агентов на основе PVRIG.
Материалы и методы:
Реагенты: ДНК-конструкции:
Человеческий PVRIG flag pUC57
Человеческий PVRIG flag pCDNA3.1
Человеческий PVRIG flag pMSCV
Рекомбинантные клетки:
HEK293 pCDNA3.1 Человеческий PVRIG flag
HEK293 pMSCV Человеческий PVRIG flag
Коммерческие:
Анти-PVRIG, Sigma кат. HPA047497 - кроличье поликлональное
Анти-PVRIG, Abnova кат. H00079037-B01 - мышиное поликлональное
Полноразмерное подтверждение мышиного PVRIG проводили с помощью ПЦР-реакций и секвенирования ПЦР-продуктов.
Использовали три пары праймеров (таблица 3).
Таблица 3. Последовательность праймеров, применяемых для подтверждения мышиной полноразмерной последовательности
В качестве матрицы для ПЦР-реакции использовали кДНК клеточной линии NIH 3T3 или смесь трех коммерческих панелей кДНК:
1. панель кДНК I, мышиная, Biochain, кат. № C8334501 (сердце, головной мозг, почки, печень).
2. панель кДНК II, мышиная, Biochain, кат. № C8334502 (легкие, поджелудочная железа, селезенка, скелетные мышцы).
3. кДНК, Clontech, кат. № 637301, (головной мозг, сердце, 7-дневный эмбрион, яички, селезенка).
Экспрессионные конструкции
Полноразмерное клонирование человеческого и мышиного PVRIG-flag проводили с помощью генного синтеза (GenScript), используя кодон-оптимизированную последовательность в векторе pUC57 для человеческого транскрипта и неоптимизированную для мышиного транскрипта, которую субклонировали в экспрессионный вектор млекопитающих pcDNA3.1 или в pMSCV, чтобы создать экспрессионную плазмиду.
Последовательность человеческого PVRIG, субклонированную в pcDNA3.1, инициировали, начиная со второго метионина человеческого белка PVRIG, тогда как последовательность человеческого PVRIG, субклонированную в pMSCV, инициировали, начиная с первого метионина человеческого белка PVRIG.
Конструкция, кодирующая человеческий PVRIG-flag.
Ген полноразмерного человеческого PVRIG, синтезированный с помощью GenScript, субклонировали в pcDNA3.1, используя рестрикционные ферменты BamI и NheI.
Конструкции, кодирующие мышиные белки PVRIG:
Следующие четыре конструкции, кодирующие мышиную последовательность, были синтезированы с помощью GenScript:
1. Первый метионин без метки
2. Первый метионин с меткой Flag
3. Второй метионин без метки
4. Второй метионин с меткой Flag
Синтезированный ген субклонировали в pCDNA3.1
Создание стабильных трансфектантов, сверхэкспрессирующих белки PVRIG
Полученную в результате экспрессионную конструкцию проверяли в отношении последовательности и впоследствии использовали для трансфекции и создания стабильных пулов, как описано ниже. Белковые последовательности, кодируемые экспрессионными конструкциями, приведены на фиг. 101.
Создание стабильных пулов трансфектантов, экспрессирующих человеческий белок PVRIG-flag
Клетки HEK293 (ATCC, номер в каталоге: CRL-1573) трансфицировали плазмидой pCDNA3.1+ человеческий PVRIG-flag или пустым вектором (pCDNA3.1+ в качестве отрицательного контроля), используя реагент FUGENE 6 (Roch, номер в каталоге 11-988-387). Устойчивые к генетицину, G418 (Gibco, номер в каталоге: 11811-031), колонии отбирали для создания стабильного пула.
Пакующую клеточную линию GP2-293 (Clontech, кат. #631458) трансфицировали pMSCV-человеческий PVRIG или пустым вектором pMSCV, используя трансфекционный реагент липофектамин 2000 (Invitrogen, номер в каталоге 11668019). Через 48 ч после трансфекции собирали супернатанты, содержащие вирионы, и непосредственно использовали их для инфицирования человеческой клеточной линии следующим образом:
Клетки HEK-293 (ATCC, CRL- CRL-1573) инфицировали вирионами, экспрессирующими человеческий PVRIG, или вирионами пустого вектора pMSCV в качестве отрицательного контроля; устойчивые к пуромицину (Invivogen, номер в каталоге: 58-58-2) колонии отбирали для создания стабильного пула.
Подтверждение экспрессии
Подтверждение экспрессии методом вестерн-блоттинга
Экстракты цельных клеток или пула клеток (30 мкг общего белка) анализировали методом вестерн-блоттинга. В качестве отрицательного контроля использовали экстракты цельных клеток из стабильных пулов клеток, трансфицированных пустым вектором. Для выявления человеческого PVRIG-flag использовали анти-flag и анти-PVRIG антитела следующим образом:
• Мышиное анти-Flag M2-пероксидаза, Sigma, кат. A8592, разведенное 1:1000 в ТТБС/5% БСА;
• Анти-PVRIG, Sigma, кат. HPA047497- кроличье поликлональное, разведенное 1:200 в ТТБС/5% БСА. После этого козье антикроличье-HRP, Jackson, кат.: 111-035-003, разведенное 1:20000 в 5%молоке/ТТБС-растворе.
Подтверждение экспрессии методом проточной цитометрии (FACS)
Чтобы подтвердить экспрессию человеческого белка PVRIG на клеточной поверхности в рекомбинантных стабильных пулах, 1x105 клеток окрашивали фиксируемым красителем в отношении жизнеспособности 450 (BD, 562247), разведенным 1:1000 в ФСБ, в течение 10 мин при КТ. Затем к клеткам добавляли мышиное поликлональное анти-PVRIG, (Abnova, кат. H00079037-B01), разведенное 1:200, или мышиный изотипический контрольный IgG1 (Life Technologies) и после этого окрашивали козьим антимышиным-PE (Jackson, кат. 115-116-146).
Результаты подтверждения экспрессии стабильных пулов клеток HEK293, сверхэкспрессирующих человеческий белок PVRIG-Flag
Чтобы подтвердить экспрессию белка PVRIG в пулах стабильно трансфицированных клеток HEK293, анализировали экстракты цельных клеток методом вестерн-блоттинга, используя анти-flag антитело или анти-PVRIG антитела (Abnova), как описано в разделе Материалы и методы. Результаты, приведенные на фиг. 24, демонстрируют наличие полосы, соответствующей ожидаемому размеру белка ~33 кДа для экстрактов пулов клеток HEK293, экспрессирующих человеческий PVRIG, но не для клеток, трансфицированных пустым вектором.
Чтобы подтвердить экспрессию белка PVRIG на клеточной поверхности, стабильно трансфицированные клетки HEK293, сверхэкспрессирующие вектор PVRIG-flag pCDNA3.1, анализировали методом FACS, используя мышиное анти-PVRIG pAb (Abnova), как описано в разделе Материалы и методы. Результаты, представленные на фиг. 25, показывают, что связывание мышиного анти-PVRIG pAb с клетками, стабильно экспрессирующими человеческий PVRIG-flag (серый цвет), выше, чем наблюдаемое для клеток, трансфицированных пустым вектором (светло-серый цвет).
Пример 3. Получение слитого белка PVRIG-ECD Ig
Слитый белок PVRIG mECD-mIg (смотрите фиг. 101, состоящий из двух ВКД мышиного PVRIG, слитых с Fc мышиного IgG2a, получали с помощью ProBioGen (Germany) в клетках CHO-DG44 путем культивирования стабильных пулов клеток в течение 12 дней с последующей очисткой собранных клеток с помощью протеина А и препаративной ЭХ-очисткой для удаления агрегатов. Конечный продукт смешивали с 5 мМ цитрата Na, 5 мМ фосфата Na/K, 140 мМ NaCl, 0,01% Твин, pH 5,5.
Применяемым экспрессионным вектором был PBG-GPEX6 от ProBioGen. Ген PVRIG находится под управлением гибридного промотора CMV/EF1, за которым следует сигнал полиаденилирования pA-1. Вектор содержит ген пуромицин-N-ацетилтрансферазы, который позволяет осуществлять отбор трансфицированных клеток, используя пуромицин, а также ген дигидрофолатредуктазы, который позволяет осуществлять отбор трансфицированных клеток, используя метотрексат (МТХ).
Слитый белок PVRIG hECD-hIg (смотрите фиг. 101), состоящий из ВКД человеческого PVRIG, слитого с Fc человеческого IgG1, несущего мутации C220, C226 и C229 на S в шарнирной области, получали с помощью GenScript (China) путем временной трансфекции в клетки CHO-3E7, которые культивировали в течение 6 дней, с последующей очисткой собранных клеток с помощью протеина А. Конечный продукт смешивали с ФСБ, pH 7,2.
Применяемым экспрессионным вектором был экспрессионный вектор млекопитающих pTT5, в котором ген PVRIG находится под управлением промотора CMV.
Пример 4. Экспрессия PVRIG на человеческих ЛПК и связывание PVRIG-Fc с клеточными линиями меланомы
PVRIG представляет собой новый белок иммунной контрольной точки, который, не ограничиваясь какой-либо одной теорией, функционирует в качестве CD28-подобного рецептора на Т-клетках. В этом исследовании оценивали экспрессию PVRIG на лимфоцитах человеческой периферической крови и связывание PVRIG-ECD-Ig (состоящего из внеклеточного домена человеческого PVRIG, слитого с человеческим IgG1) с клеточными линиями меланомы.
Материалы и методы
Три клеточные линии человеческой меланомы, которые представляют антиген MART-1 в контексте HLA-A2 (SK-MEL-23, Mel-624 и Mel-624.38), использовали в качестве мишеней для ЦТЛ. Mel-888, который не экспрессирует HLA-A2, служил в качестве отрицательного контроля.
Лейкоцитарные пленки от здоровых человеческих доноров получали из банка крови Tel Hashomer. Мононуклеарные клетки периферической крови стимулировали ФГА и культивировали в течение 3 дней, а после этого трансдуцировали ретровирусным вектором на основе MSCV (pMSGV1). После трансдукции клетки дополнительно выращивали в лимфоцитарной среде (среда Bio target, фетальная бычья сыворотка (10%), L-глутамин, пенициллин/стрептомицин (100 единиц/мл), IL-2 300 МЕ) в течение дополнительных 5 дней.
Чтобы оценить экспрессию PVRIG на ЛПК, клетки окрашивали специфическим антителом к PVRIG (мышиным поликлональным) в концентрации 5 мкг/мл в течение 30 минут при 4 градусах. После промывания клетки окрашивали ФИТЦ-конъюгированным козьим антимышиным mAb (1:250) (Invitrogen, Кат. # A10667) в буфере FACS в темноте в течение 30 минут при 4 градусах. После двух промывок в буфере FACS образцы считывали на BD Bioscience FACS Calibur с Cytek HTS.
Чтобы оценить связывание PVRIG-Ig с клеточными линиями меланомы, клетки SK-MEL-23, Mel-624, Mel-624.38 и mel-888 совместно культивировали с F4-трансдуцированными или нетрансдуцированными (обозначенными «без») ЛПК, а после этого окрашивали 20 мкг/мл слитого белка PVRIG-Ig, HH партия #125. После двух промывок в буфере FACS образцы окрашивали вторичным козьим античеловеческим PE (Jackson, кат. # 109-116-098).
Результаты
Чтобы оценить эндогенную экспрессию PVRIG на первичных человеческих лейкоцитах, ЛПК стимулировали ФГА, а после этого трансдуцировали пустым вектором и окрашивали анти-PVRIG специфическим антителом. Как показано на фиг. 11, для двух разных доноров наблюдается окрашивание анти-PVRIG по сравнению с изотипически совпадающим контролем.
Чтобы оценить эндогенную экспрессию PVRIG в клеточных линиях меланомы и определить, влияет ли на эндогенную экспрессию совместное культивирование с антигенспецифическими Т-клетками, 4 разных клеточных линии меланомы (SK-MEL-23, Mel-624, Mel-624.38 и mel-888) совместно культивировали с ЛПК, экспрессирующими или не экспрессирующими F4 (gp100-специфический РТК). После этого клетки окрашивали слитым белком, состоящим из внеклеточного домена человеческого PVRIG, слитого с Fc-частью человеческого IgG1. Как показано на фиг. 12, все 4 исследуемые клеточные линии меланомы демонстрировали связывание с PVRIG-Ig. На интенсивность связывания не влияет Т-клеточнозависимая активация после совместного культивирования с реактивными в отношении меланомы сконструированными Т-клетками.
Обобщенные результаты: Результаты, представленные в данном документе, позволяют предположить, что PVRIG экспрессируется на ФГА-активированных человеческих первичных лейкоцитах периферической крови (ЛПК). Кроме того, 4 клеточные линии меланомы, которые изучали в данном исследовании, связываются со слитым белком, состоящим из внеклеточного домена человеческого PVRIG, слитого с Fc-частью человеческого IgG1, что позволяет предположить, что эти клеточные линии экспрессируют контрпартнера PVRIG.
Пример 5. Определение и проверка рецепторов и лигандов
Первое проверочное исследование проводили, используя клеточную микроматричную технологию для исследования взаимодействия PVRIG с 3559 полноразмерными белками человеческой плазматической мембраны, которые индивидуально экспрессировались в человеческих клетках HEK293.
Человеческие клетки HEK293 выращивали на слайдах с нанесенными пятнами экспрессионными векторами, кодирующими 3559 полноразмерные белки человеческой плазматической мембраны. Экспрессионный вектор (pIRES-hEGFR-IRES-ZsGreen1) наносили пятнами в четырех экземплярах на каждый слайд и использовали, чтобы удостовериться, чтобы на каждом слайде был достигнут или превышен минимальный порог эффективности трансфекции. Человеческие клетки HEK293 использовали для обратной трансфекции/экспрессии. Слитый белок, состоящий из ВКД PVRIG, слитого с человеческим IgG1, добавляли в концентрации 20 мкг/мл на каждый слайд после фиксации клеток. Определение связывания проводили, используя подходящее флуоресцентное вторичное антитело. Наборы слайдов исследовали в двух повторностях. Анализ и количественное определение флуоресцентных изображений (в отношении эффективности трансфекции) проводили с помощью программного обеспечения ImageQuant (GE).
Белковый «хит» определяется как двойное пятно, демонстрирующее повышенный сигнал по сравнению с фоновыми уровнями. Это осуществляли путем визуального изучения, используя изображения с координатной сеткой, полученные с помощью программного обеспечения ImageQuant. Хиты делятся на «сильные, средние, слабые или очень слабые» в зависимости от интенсивности двойных пятен. Для подтверждения хитов все кодирующие хиты векторы, идентифицированные при первичном скрининге, помещали на новые слайды. Скрининг и анализ в отношении подтверждения/специфичности проводили, как и в случае первичного скрининга (n = 2 параллельных слайда на образец) за исключением того, что идентичные слайды также зондировали соответствующими отрицательными контролями. Кроме того, проводили секвенирование всех кодирующих хиты векторов. Проводили секвенирование векторов, кодирующих каждый из первичных хитов, подтверждая их идентичность.
Фоновый скрининг показал пренебрежимо малое связывание с нетрансфицированными клетками HEK293 при 2, 5 и 20 мкг/мл (фиг. 13).
На основании фоновых данных для полного профилирования была выбрана концентрация 20 мкг/мл. Результатом первичного скрининга стало множество двойных хитов (клонов), при этом большинство из них имело слабую или очень слабую интенсивность. Все идентифицированные первичные хиты и контрольный вектор EGFR-ZsGreen1 наносили и повторно экспрессировали в двух повторностях и зондировали PVRIG в концентрации 20 мкг/мл для скрининга в отношении подтверждения/специфичности.
Был идентифицирован один специфический хит, PVRL2, с высокой интенсивностью (на фиг. 14). Позже было показано, что другой слабый хит, MAG, связывается также другие исследуемые слитые белки (данные не показаны), что позволяет предположить, что он не является специфическим. Эти результаты согласуются с недавно опубликованным абстрактом https://www.yumpu.com/en/document/view/7263720/sunday-december-4-late-abstracts-1-molecular-biology-of-the-/133 авторства G. Quinones в New Technologies & Frontlers. Известно, что PVRL2 играет роль лиганда для TIGIT и DNAM1, которые оба являются модуляторами Т-клеточной и NK-клеточной активации. Недавно сообщалось, что TIGIT является ключевым фактором в ингибировании иммунного ответа, направленного против опухолевых клеток (Noa Stanietsky, journal of immunology, vol. 106 no. 42, 17858-17863; Robert J Johnston, Cancer cell, Volume 26, Issue 6, p923-937, 8 December 2014). Результаты, представленные в Примере 5, демонстрирующие взаимодействие PVRIG с тем же контрпартнером, что и TIGIT, позволяют предположить вовлеченность PVRIG в важный регуляторный путь, который регулирует раковый иммунологический надзор, и таким образом, делает PVRIG потенциальной мишенью для лечения рака.
Дополнительное проверочное исследование 2
Материалы и методы
Материалы
Fc-слитые белки, His-меченые белки и контрольный Ig: Fc-слитый белок PVRIG-Fc M:M использовали для исследования связывания. Мышиный IgG2a использовали в качестве изотипического контроля. Другими коммерческими мышиными белками, используемыми в исследовании, были PVRL2-his (R&D, 3869-N2) и PVRL2-his (Sino Biological, 50318-M08H).
Клетки: Были получены HEK293, сверхэкспрессирующие (СЭ) мышиный PVRIG и PVRIG-FLAG (RC-287 и RC-286 соответственно), а связывание PVRL2 с этими клетками сравнивали с клетками HEK293, экспрессирующими пустой вектор (ПВ) (RC-83). Были созданы HEK293, сверхэкспрессирующие сплайс-варианты мышиного PVRL2 1 и 2 (sv1 и sv2) (RC-334 и RC-335 соответственно), а связывание PVRIG с этими клетками сравнивали с клетками HEK293, экспрессирующими ПВ. Также использовали клетки B16-F10 (CL-161, клетки мышиной кожной меланомы, эндогенно экспрессирующие mPVRL2) для исследования взаимодействия между PVRIG и PVRL2.
Антитела: Антимышиное PVRL2-PE Ab (R&D, FAB3869P, 25 мкг/мл, 1:100) использовали для выявления PVRL2. Крысиное IgG2A-PE (R&D, IC006P, 25 мкг/мл, 1:100) использовали в качестве изотипического контроля. Антимышиное-PE (Jackson Immunoresearch, 115-115-206, 0,5 мг/мл, 1:200) и анти-his Ab (Abcam, ab72467, 0,1 мг/мл, 1:300) использовали для выявления связывания рекомбинантных белков. Ab против метки DYKDDDDK (анти-FLAG) (BioLegend, 637302, 0,5 мг/мл, 1:300) использовали для выявления экспрессии PVRIG на HEK293, сверхэкспрессирующих мышиный PVRIG-FLAG. Для мечения PVRIG использовали набор для мечения антител Alexa Fluor® 647 (Molecular Probes, A-20186) в соответствии с протоколом производителя. Для биотинилирования PVRIG использовали набор для мечения белков биотином DSB-X™ (Molecular Probes, D-20655) в соответствии с протоколом производителя. Биотинилированный PVRIG выявляли с помощью стрептавидина-PE (SA-PE) (Jackson Immunoresearch, 016-110-084, 0,5 мг/мл, 1:300).
Методы
Анализ FACS связывания мышиного PVRIG-Fc со стабильными клетками HEK293, сверхэкспрессирующими (СЭ) мышиный PVRL2, или с клетками B16-F10: Клетки HEK293, сверхэкспрессирующие PVRL2 (sv1 или sv2), или клетки B16-F10 суспендировали до 106 клеток/мл в ФСБ. На каждый 1 мл клеток добавляли 1 мкл маточного раствора с красителем для определения жизнеспособности (устраняемый краситель для определения жизнеспособности от BD Horizon 450, кат. 562247, BD Bioscience). Клетки инкубировали в течение 10 мин с защитой от света при комнатной температуре. Затем клетки дважды промывали ФСБ и суспендировали до 3x106 клеток/мл в присутствии 1:50 человеческого красителя TruStain FcXTM (BioLegend 422302) в буфере FACS (ФСБ, дополненный 2% ФБС и 0,5 мМ ЭДТА) при комнатной температуре в течение 15 мин для блокирования Fcγ-рецепторов. Затем, не промывая, высевали 1x105 клеток/лунку в V-образные 96-луночные планшеты (Costar #3357). Экспрессию PVRL2 исследовали с помощью анти-PVRL2 антитела (смотрите выше). Связывание PVRIG-Fc с клетками исследовали, используя разные партии (смотрите выше), в общем случае в концентрации 60 мкг/мл или в нескольких концентрациях. Клетки инкубировали с антителами или PVRIG-Fc в течение 40 мин при комнатной температуре, затем дважды промывали. Вторичное антитело (антимышиное-PE) добавляли в течение 15 мин при комнатной температуре, дважды промывали клетки и собирали для анализа на FACS-анализаторе MACSQuant® (Miltenyi Biotec) с последующим анализом данных с помощью программного обеспечения Flow-Jo 10.
Анализ FACS связывания мышиного PVRL2-his со стабильными клетками HEK293, сверхэкспрессирующими мышиный PVRIG: Уровни PVRIG исследовали с помощью анти-FLAG антитела. Связывание PVRL2-his отслеживали с помощью анти-his антитела. Анализ FACS проводили, как описано выше.
Биофизический ППР-анализ взаимодействия PVRIG/PVRL2 с помощью Biacore: Взаимодействие между мышиным PVRIG и PVRL2 анализировали на ППР-анализаторе биомолекулярных взаимодействий Biacore T100 в университете им. Бар-Илана. Белки разводили до 100 нM в ацетатном буфере, pH 4,0, и проводили ковалентное сопряжение с поверхностью проточной ячейки чипа CM5 Series S Biacore, используя стандартную реакцию аминного сопряжения. Поверхности активировали EDC-NHS и позже блокировали, инъецируя 1 M этаноламина (pH 8,5). Подвижный буфер состоял из 10 мМ Гэпэс, pH 7,3, 150 мМ NaCl, 3 мМ ЭДТА и 0,05% Твин-20 (HBS-EP+). Конечные уровни иммобилизации составляли ~1000 ЕО. Белки, используемые в качестве аналитов, разводили до 2500 нМ, 500 нМ и 100 нМ. В каждом эксперименте одна пробирка содержала только подвижный буфер для сравнения. После каждого эксперимента проводили этап восстановления с помощью 4 M MgCl2 в течение 30 с при 20 мкл/с.
Результаты
Связывание мышиного PVRIG с клетками HEK293, сверхэкспрессирующими PVRL2 sv1: Чтобы подтвердить взаимодействие между мышиным PVRIG и мышиным PVRL2, мы сначала исследовали связывание PVRIG-Fc с клетками, сверхэкспрессирующими (СЭ) PVRL2. Уровень экспрессии PVRL2 на HEK293, сверхэкспрессирующих PVRL2 sv1, определяли, используя специфические антитела к мышиному PVRL2. Экспрессия мышиного PVRL2 была в 10 раз выше по сравнению с клетками HEK293, экспрессирующими пустой вектор (данные не показаны). Четыре партии PVRIG-Fc исследовали в отношении связывания со сверхэкспрессирующими PVRL2 клетками. Все партии PVRIG-Fc демонстрировали 6-11-кратное связывание с клетками, сверхэкспрессирующими PVRL2, по сравнению с клетками с пустым вектором (данные не показаны). Связывание PVRIG-Fc со сверхэкспрессирующими PVRL2 клетками также исследовали, используя биотинилированные и флуоресцентно меченые (Alexa Fluor 647) белки PVRIG. Тогда как биотинилированные белки проявляли немного более сильное связывание со сверхэкспрессирующими PVRL2 клетками по сравнению с немеченым PVRIG-Fc (данные не показаны), флуоресцентно меченый PVRIG демонстрировал намного более низкое связывание (данные не показаны). Эти результаты демонстрируют, что PVLR2 выявляется на мембране клеток HEK2930, сверхэкспрессирующих PVRL2; связывание мышиного PVRIG-Fc со сверхэкспрессирующими PVRL2 клетками выявляется с помощью антител к мышиному IgG2A; связывание биотинилированного мышиного PVRIG-Fc со сверхэкспрессирующими PVRL2 клетками выявляется с помощью стрептавидина-PE, как и связывание меченого Alexa Fluor 647 PVRIG-Fc со сверхэкспрессирующими PVRL2 клетками.
Связывание мышиного PVRL2 с клетками HEK293, сверхэкспрессирующими PVRIG: Чтобы дополнительно подтвердить взаимодействие между мышиным PVRIG и мышиным PVRL2, мы исследовали связывание PVRL2 с клетками, сверхэкспрессирующими PVRIG, в присутствии или отсутствие FLAG-метки. Мембранную экспрессию мышиного PVRIG на клетках HEK293, сверхэкспрессирующих PVRIG, с FLAG-меткой подтверждали, используя анти-FLAG антитело (данные не показаны). Как и ожидалось, клетки HEK293, сверхэкспрессирующие PVRIG, без FLAG-метки демонстрировали отсутствие экспрессии при применении анти-FLAG антитела. При применении анти-PVRIG супернатантов (Aldeveron) эти клетки демонстрировали более низкую экспрессию PVRIG по сравнению с клетками, сверхэкспрессирующими PVRIG, с FLAG-меткой. Коммерческий мышиный рекомбинантный белок PVRL2 был доступен только в виде His-меченого белка. Следовательно, для получения подходящего анти-His антитела и условий для выявления требовалось проведение масштабной калибровки. His-меченый PVRL2 из двух разных источников был исследован в отношении связывания со сверхэкспрессирующими PVRIG клетками в концентрации 60 мкг/мл и демонстрировал 2-кратное (данные не показаны) и 3-4-кратное (данные не показаны) связывание по сравнению с клетками HEK293, экспрессирующими пустой вектор. Это означает, что his-меченый мышиный PVLR2 связывает HEK293, сверхэкспрессирующие мышиный PVRIG, а мышиный PVRIG экспрессируется на мембранах клеток HEK293, сверхэкспрессирующих PVRIG.
Исследование взаимодействия мышиного PVRIG и мышиного PVRL2 с помощью ППР-Biacore: Чтобы оценить взаимодействие между мышиным PVRIG-Fc и мышиным His-меченым PVRL2, оба белка иммобилизовали на чипе Biacore. После иммобилизации оба белка, а также PVRIG-Fc (данные не показаны) исследовали в качестве аналитов в трех концентрациях: 2500, 500 и 100 нМ (PVRIG из партии #480 и PVRL2 дважды исследовали в качестве аналитов). Взаимодействие между двумя белками было выявлено в обоих направлениях и для обеих партий PVRIG (данные не показаны). Из-за сложной кинетики невозможно было определить точное значение KД по результатам Biacore.
Дозозависимое связывание мышиного PVRIG с клетками HEK293, сверхэкспрессирующими PVRL2 sv2, и клетками B16-F10: Как показано выше, связывание мышиного PVRL2 со сверхэкспрессирующими мышиный PVRIG клетками было относительно низким. Для определения метода скрининга антител к мышиному PVRIG, способных блокировать взаимодействие между мышиным PVRIG и мышиным PVRL2, выбрали связывание PVRIG-Fc со сверхэкспрессирующими PVRL2 клетками. Сначала строили дозозависимую кривую связывания мышиного IgG2A и мышиного PVRIG-Fc с клетками, сверхэкспрессирующими мышиный PVRL2, и сравнивали с клетками, экспрессирующими пустой вектор (ПВ). Исследование дозозависимости проводили для двукратных серийных разведений (1:2) от 50 мкг/мл до 0,1 мкг/мл. Тогда как в связывании мышиного IgG2A не наблюдали различий (данные не показаны), PVRIG-Fc демонстрировал насыщение связывания при 12,5 мкг/мл снижение связывания, коррелирующее со снижением концентрации белка (данные не показаны). Аналогичные результаты были получены для PVRIG-Fc (данные не показаны). Эти результаты позволяют предположить, что этот анализ связывания можно рассматривать как вариант для скрининга блокирующих антител.
Чтобы рассмотреть также эндогенную систему для скрининга антител к мышиному PVRIG, оценивали экспрессию PVRL2 на клетках B16-F10, используя анти-PVRL2 антитело. Результаты показывают, что PVRL2 на высоком уровне экспрессируется в клетках B16-F10 (данные не показаны). Следовательно, получали аналогичную дозозависимую кривую связывания мышиного IgG2A и мышиного PVRIG-Fc с клетками B16-F10. Аналогично результатам, полученным с клетками HEK293, сверхэкспрессирующими PVRL2, мышиный PVRIG-Fc демонстрировал дозозависимое связывание с клетками B16-F10, достигающее насыщения при 12,5 мкг/мл, тогда как в связывании мышиного IgG2A никаких изменений выявлено не было (данные не показаны).
Обсуждение и выводы: Взаимодействие человеческого PVRIG с человеческим PVRL2 определяли, используя клеточную микроматричную технологию, с помощью Retrogenix. Чтобы подтвердить это взаимодействие также в мышином организме, было предпринято несколько подходов. Среди них - применение клеток, сверхэкспрессирующих PVRIG или PVRL2, и биофизические измерения с помощью ППР-Biacore. Все подходы свидетельствовали о том, что мышиный PVRIG взаимодействует с мышиным PVRL2. Однако связывание мышиного PVRL2 со сверхэкспрессирующими PVRIG клетками было относительно низким по сравнению со связыванием PVRIG со сверхэкспрессирующими PVRL2 клетками. Причиной этого может быть тот факт, что коммерческий PVRL2 доступен только в виде мономерного His-меченого белка, но не в виде Fc-слитого белка (как в случае PVRIG). В связи с этим с помощью GenScript получали специальный Fc-слитый мышиный PVRL2. Однако, согласно предварительным данным, для этого белка наблюдали только небольшое повышение связывания (~5-кратное по сравнению с 2-3 кратным в случае PVRL2-his). Следовательно, некоторые другие факторы могут влиять на это относительно слабое связывание.
Из-за слабого связывания PVRL2 со сверхэкспрессирующими PVRIG клетками было решено разработать анализ связывания анти-PVRIG антитела, используя связывание PVRIG-Fc с клетками, сверхэкспрессирующими PVRL2. В соответствии с наблюдаемыми дозозависимыми кривыми мы предложили три рабочие концентрации: 0,1, 0,2 и 0,4 мкг/мл. После аналогичных результатов, полученных для связывания PVRIG с эндогенно экспрессирующими PVRL2 клетками B16-F10, мы предложили провести анализ блокирования антителами также для этих клеток при следующих концентрациях: 0,2, 0,4, 0,8 мкг/мл.
PVRIG является предполагаемым рецептором, следовательно, анализ блокирования антителами предпочтительно проводить с PVRL2 в качестве растворимого белка и PVRIG, экспрессируемым на клетках. Таким образом, следует исследовать антитела к мышиному PVRIG, которые демонстрируют блокирующую активность в текущем формате, также в этой системе.
Дополнительное проверочное исследование 3
Задачей этого исследования было подтверждение партнеров по связыванию PVRIG, новой иммуно-онкологической мишени. Предварительные исследования указывают на то, что одним из этих лигандов является PVRL2. В этом исследовании изучали связывание рекомбинантного белка PVRIG с несколькими потенциальными лигандами оси PVRIG методом ELISA.
Протоколы
Перечень реагентов: Современные литературные данные по белкам PVRIG предполагают существование трех потенциальных лигандов: PVR (CD155), PVRL2 (CD112) и PVRL3 (CD113). Чтобы исследовать их способность связывать рецептор PVRIG, эти три лиганда были приобретены на коммерческой основе, а именно: PVR и PVRL3 от Sino Biologicals Inc. и PVRL2 от R&D Systems и Sino Biologicals Inc. Человеческий рекомбинантный белок PVRIG был получен с помощью Compugen в виде внеклеточного домена (ВКД) PVRIG, слитого с Fc-доменом человеческого IgG1 (PVRIGHH).
ELISA для определения взаимодействия рецептор-лиганд: Приобретенные на коммерческой основе His-меченые лиганды PVR, PVRL2 и PVRL3 наносили на стенки лунок планшета с высокой степенью связывания EIA/RIA (Costar 9018) в течение ночи при 4°C. Нерелевантный His-меченый белок был включен в качестве отрицательного контроля. Покрытые лунки планшета дважды промывали ФСБ и инкубировали с 300 мкл блокирующего буфера (5% сухое обезжиренное молоко в ФСБ, pH 7,4) при комнатной температуре (КТ) в течение 1 ч. Блокирующий буфер удаляли, а планшеты дополнительно дважды промывали ФСБ. Связанные с планшетом лиганды инкубировали с разными концентрациями PVRIGHH в растворе (линейный диапазон от 0,1 мкг/мл до 4 мкг/мл в объеме 50 мкл/лунку) при КТ в течение 1 ч. Планшеты три раза промывали ФСБ-Т (ФСБ 7,4, 0,05% Твин20), затем три раза ФСБ и добавляли 50 мкл/лунку HRP-конъюгированного вторичного антитела (Специфического к Fc-домену человеческого IgG, Jackson ImmunoResearch). Реакцию инкубировали при КТ в течение 1 ч и снова промывали планшеты. Сигналы ELISA инициировали во всех лунках путем добавления 50 мкл ТМБ-субстрата Sureblue (KPL Inc) и инкубации в течение 5-20 мин. Реакцию HRP останавливали путем добавления 50 мкл 2N H2SO4 (VWR), а сигналы поглощения на 450 нм считывали на спектрофотометре SpectraMax (Molecular Devices) или EnVision (PerkinElmer). Данные экспортировали в Excel (Microsoft) и строили графики в GraphPad Prism (GraphPad Software, Inc.).
Результаты: PVRIG предпочтительно связывается с PVRL2: Fc-слитый белок человеческого PVRIG анализировали в отношении связывания с PVR, PVRL2 и PVRL3, иммобилизованными на планшете EIA/RIA. Разные концентрации рецептора PVRIG в фазе раствора инкубировали с иммобилизованным лигандом. Данные четко указывают на дозозависимое связывание PVRIGHH с PVRL2, но отсутствие связывания с лигандами PVR, PVRL3 или отрицательным контрольным белком (данные не показаны). Сигнал ELISA A450 наносили на график в виде функции концентрации рецептора, используя уравнение односайтового связывания, которое позволило определить равновесную константу связывания (KД), составляющую 13 ± 1 нМ.
Выводы и заключения: PVRIG представляет собой новую иммуно-онкологическую мишень, биология которой не до конца понятна. В попытке пролить больше света на эту биологию мы исследовали его связывание с несколькими потенциальными лигандами. PVRL2 был четко определен как партнер по связыванию PVRIG. Количественный анализ позволяет предположить, что это взаимодействие является очень сильным с KД 13±1 нМ. Наши результаты также позволяют предположить, что человеческий PVRIG не связывает человеческий PVR и PVRL3, или же связывание является слишком слабым для выявления методом ELISA.
Дополнительное проверочное исследование 4:
В этом примере оценивали экспрессию PVRIG в клеточных субпопуляциях МКПК до и после алло-активации. После алло-активации экспрессия PVRIG была повышена на Т-клетках CD4+, а также на Т-клетках CD8+ и вдвойне отрицательных гамма-дельта Т-клетках. Это повышение наблюдалось в МКПК одного из двух исследуемых доноров (смотрите фиг. 51).
Пример 6. Исследование связывания PVR, PVRL2 и PVRL3 с PVRIG, DNAM и TIGIT методом поверхностного плазмонного резонанса
Материалы и методы
Все эксперименты проводили на приборе ProteOn XPR 36 при 22°C.
Этап 1: Готовили поверхность с высокой плотностью козьего поликлонального антитела против человеческого fc (Invitrogen H10500) на всех шести дорожках чипа GLC, используя биосенсор ProteOn XPR 36. Этап активации поверхности против человеческого fc происходил в направлении горизонтального потока, тогда как этап иммобилизации pAb высокой плотности происходил в направлении вертикального потока. Этап блокирования происходил как в вертикальном, так и в горизонтальном положениях так, что горизонтальные «промежутки» можно было использовать в качестве эталонных поверхностей. На каждой дорожке было иммобилизовано в среднем ~4400 ЕО козьего античеловеческого pAb.
Этап 2: Для каждого цикла три разных партии человеческого слитого белка PVRIG (человеческий fc, GenScript, партии 451, 448, 125), человеческого слитого белка DNAM-1 (человеческий fc, R&D Systems), человеческого слитого белка TIGIT (человеческий fc, R&D Systems) и контрольного человеческого IgG (Synagis) иммобилизовали на разных вертикальных дорожках в течение двух минут при концентрации 2 мкг/мл. PVR, две партии PVRL2 и PVRL3 инъецировали в направлении горизонтального потока при шести разных концентрациях через шесть иммобилизованных лигандов при разных циклах иммобилизации лигандов. Инъекции длились по две минуты с последующей 10-минутной диссоциацией при скорости потока 50 мкл/мин. Диапазон концентраций PVR составлял 1,4 нМ - 332 нМ в 3-кратном серийном разведении, обе партии PVRL2 инъецировали в диапазоне концентраций 1,3 нМ - 322 нМ в 3-кратном серийном разведении, а PVRL3 инъецировали в диапазоне концентраций 1,4 нМ - 334 нМ в 3-кратном серийном разведении. Все белковые реагенты готовили в подвижном буфере, который представлял собой дегазированный буфер ФСБ с добавлением 0,05% Твин 20 и 0,01% БСА. После каждого цикла поверхности с иммобилизованным антителом против человеческого fc восстанавливали двумя 30-секундными впрыскиваниями 146 мМ фосфорной кислоты.
Этап 3: Данные сенсограмм по связыванию аналитов с каждым иммобилизованным лигандом обрабатывали и проводили двойной контроль с помощью ProteOn Manager версия 3.1.0.6, используя промежуточные контроли и инъекцию пустой пробы, идентичную инъекциям аналитов.
Результаты
a) PVR: Слабо связывается с иммобилизованными DNAM-1 и TIGIT и не проявляет связывания со всеми тремя партиями PVRIG и контрольным IgG. Для оценки KД взаимодействия PVR с DNAM-1 и TIGIT было недостаточно информации (данные не показаны).
b) PVRL2: Обе партии PVRL2 проявляли связывание со всеми тремя партиями PVRIG и с DNAM-1, но минимум или отсутствие связывания с TIGIT и отсутствие связывания с контрольным IgG. Сенсограммы продемонстрировали сложную кинетику, следовательно, оценить константы связывания было невозможно (данные не показаны).
c) PVRL3: Проявлял минимальное связывание с TIGIT и не связывался с другими белками (данные не показаны).
Пример 7. In-vitro иммуномодулирующая активность PVRIG ECD-IG в отношении мышиных Т-клеток
В этих экспериментах исследовали иммуномодулирующую активность рекомбинантного слитого белка PVRIG-ECD-Ig в отношении активации мышиных Т-клеток. Действие PVRIG-ECD-Ig на активацию мышиных Т-клеток CD4 исследовали, используя некоторое количество in-vitro показателей Т-клеточной активации: маркеры клеточной активации, секрецию цитокинов и пролиферацию.
Чтобы оценить активность белка pvrig в отношении активации Т-клеток, получали рекомбинантный белок, содержащий мышиный внеклеточный домен (ВКД) мышиного pvrig, слитый с fc мышиного igg2a (обозначаемый pvrig-ecd ig m:m) (seq id no:29). Исследовали действие fc-слитого белка, иммобилизованного совместно с анти-cd3, на функции мышиных Т-клеток, проявляемое по маркерам активации и секреции цитокинов.
Материалы и методы
Fc-слитый белок и контрольный Ig: Исследовали Fc-слитый белок, PVRIG-ECD-Ig (партия #198). Мышиный IgG2a (клон MOPC-173; Biolegend или C1.18.4; BioXcell) использовали в качестве изотипического контроля.
Выделение мышиных Т-клеток CD4: Нетронутые Т-клетки CD4+CD25- выделяли из селезеночных пулов мышей BALB/C, используя набор для выделения Т-клеток (Miltenyi, Кат. # 130-093-227) в соответствии с инструкциями производителя. Получаемая степень очистки составляла > 90%.
Активация мышиных Т-клеток CD4: Антимышиное CD3-ε mAb (клон 145-2C11; BD Biosciences) в концентрации 2 мкг/мл вместе с белком PVRIG-ECD-Ig или контрольным Ig в разных концентрациях (1, 3 или 10 мкг/мл) иммобилизовали в течение 3 ч при 37°C на 96-луночных плоскодонных планшетах для тканевого культивирования (Sigma, Кат. # Z707910). В каждую лунку добавляли контрольный Ig, чтобы получить общую концентрацию белка 12 мкг/мл на лунку. Лунки три раза промывали ФСБ и высевали 1x105 очищенных Т-клеток CD4+CD25- на лунку, и держали в увлажненном инкубаторе с 5% CO2, 37°C. В некоторых экспериментах добавляли растворимое анти-CD28 (клон: 37.51; eBioscience; 1 мкг/мл). В указанные моменты времени после стимуляции собирали культуральные супернатанты и анализировали в отношении секреции мышиного IFNγ или IL-2 с помощью наборов для ELISA (R&D Systems). Действие белка PVRIG-ECD-Ig (смотрите фиг. 101) на экспрессию маркера активации CD69 на мышиных Т-клетках CD4+ анализировали методом проточной цитометрии. Через 48 ч после стимуляции клетки окрашивали коктейлем из антител, содержащим PerCP-анти-CD4 (клон G41.5; Biolegend), ФИТЦ- или PE-анти-CD69 (клон H1.2F3; Biolegend), в присутствии анти-CD16/32 (клон 2.4g2; BD Biosciences) для блокирования Fcγ-рецепторов. Клетки оценивали с помощью анализатора MACSQuant 9 (Miltenyi), а данные анализировали с помощью программного обеспечения BD CellQuest или MACSQuantify TM. Данные анализировали с помощью программного обеспечения Excel или Prism4.
Результаты и заключение
Действие PVRIG-ECD Ig M:M (смотрите
На фиг. 101) на функцию мышиных Т-клеток CD4+: фиг. 15 изображает in-vitro иммуномодулирующую активность PVRIG-ECD-Ig (смотрите фиг. 101) в отношении выделенных из мышиной селезенки Т-клеток (CD4+, степень очистки > 95%), которые стимулировали, применяя микропланшеты с совместно иммобилизованным одним анти-CD3 (2 мкг/мл) или совместно иммобилизованным контрольным Ig (mIgG2a) или PVRIG-ECD-Ig (смотрите фиг. 101) (10 мкг/мл) в присутствии растворимого анти-CD28 (1 мкг/мл). PVRIG-ECD-Ig (смотрите фиг. 101) подавлял активацию мышиных Т-клеток CD4 дозозависимым образом, что проявлялось в снижении регуляции CD69 (фиг. 15A, D) и снижении секреции РТК-индуцируемых цитокинов (IL-2 и IFNγ) (фиг. 15B-C, E). Величина ингибиторного действия PVRIG-ECD-Ig (смотрите фиг. 101) находилась в диапазоне 30-100%. Ингибиторное действие PVRIG-ECD-Ig (смотрите фиг. 101) на секрецию IFNγ наблюдали для концентраций всего лишь 3 мкг/мл (~60% ингибирование по сравнению с контрольным Ig).
PVRIG-ECD-Ig (смотрите фиг. 101) ингибирует активацию Т-клеток зависимым от концентрации образом, когда Fc-слитый белок иммобилизован на планшетах с анти-CD3. Максимальное ингибиторное действие наблюдали при 10 мкг/мл PVRIG-ECD-Ig (смотрите фиг. 101).
Результаты демонстрируют ингибиторное действие PVRIG-ECD-Ig на активацию мышиных Т-клеток, проявляющееся в снижении секреции цитокинов и подавлении регуляции маркера активации CD69. Это ингибирование активации Т-клеток подтверждает терапевтический потенциал иммуномодулирующих белков PVRIG (полипептидов и слитых белков PVRIG) в соответствии с настоящим изобретением для лечения Т-клеточных аутоиммунных заболеваний, таких как ревматоидный артрит, множественный склероз, псориаз и воспалительное заболевание кишечника, а также других иммунных заболеваний, и/или для снижения нежелательной иммунной активации, которая происходит после генной терапии. Кроме того, эти результаты также подтверждают терапевтический потенциал иммуномодулирующих белков PVRIG (полипептидов и слитых белков PVRIG), которые уменьшают ингибиторную активность PVRIG, для лечения патологических состояний, при которых может быть полезен усиленный иммунный ответ, в частности, повышение ЦТЛ-иммунитета и уровня провоспалительных цитокинов, таких как рак, инфекционные заболевания, в частности, хронические инфекции и сепсис, когда опосредованное Т-клетками снижение числа больных клеток является терапевтически выгодным.
Пример 8. In-vitro иммуномодулирующая активность PVRIG в отношении человеческих цитотоксических Т-клеток (ЦТЛ)
В экспериментах, описанных в этом примере, оценивали действие эктопической экспрессии человеческого PVRIG в разных клеточных линиях меланомы на их способность активировать ЦТЛ (цитотоксические Т-лимфоциты) и служить мишенями для уничтожения этими клетками.
Материалы и методы:
Три человеческие клеточные линии меланомы, которые представляют антиген MART-1 в контексте HLA-A2 (SK-MEL-23, Mel-624 и Mel-624.38), использовали в качестве мишеней для ЦТЛ. Mel-888, которая не экспрессирует HLA-A2, служила в качестве отрицательного контроля.
Эктопическая экспрессия человеческого PVRIG на цитотоксических Т-лимфоцитах (ЦТЛ): Чтобы экспрессировать человеческий PVRIG в культурах лейкоцитов периферической крови (ЛПК), кДНК, кодирующую PVRIG, амплифицировали, используя специфические праймеры, и клонировали в ретровирусный вектор на основе MSCV (pMSGV1) или в трехкомпонентные векторы: CD8-зависимые α- и β-цепи РТК F4 были связаны с последовательностью P2A и клонированы в вектор pMSGV1, с расположенным за ним участком внутренней посадки рибосомы (IRES) и PVRIG. Ретровирусный вектор, кодирующий NGFR1, в качестве отрицательного контроля или в трехкомпонентные векторы: CD8-зависимые α- и β-цепи РТК F4 были связаны с последовательностью P2A и клонированы в вектор pMSGV1, с расположенным за ним участком внутренней посадки рибосомы (IRES) и NGFR. Подтверждение клонирования проводили, используя сначала расщепление рестрикционным ферментом, а после этого - секвенирование. После подтверждения последовательности получали большие количества ретровирусного вектора (Maxi-prep) для последующего применения.
Лейкоциты периферической крови здоровых человеческих доноров трансдуцировали ретровирусными конструкциями, кодирующими PVRIG, или ретровирусными векторами, кодирующими NGFR1, или пустым вектором в качестве отрицательного контроля. Трансдукцию проводили, используя протокол на основе ретронектина; вкратце, в клетках 293GP (ретровирусная пакующая клеточная линия) получали ретровирусный супернатант после трансфекции ретровирусным вектором и амфотропным оболочечным геном (VSV-G). Ретровирусный супернатант высевали перед трансдукцией на покрытые ретронектином планшеты, чтобы обеспечить связывание вирионов с планшетом, и добавляли в планшет ЛПК в течение 6 часов. После этого клетки переносили в новый культуральный сосуд. Эффективность трансдукции и экспрессию белка определяли посредством окрашивания трансдуцированных ЛПК коммерческим PVRIG-специфическим кроличьим поликлональным антителом или коммерческим анти-NGFR (Кат. № 345108; BioLegend). Кроличий IgG (Sigma Кат. № I5006) использовали в качестве изотипического контроля, а в качестве вторичного антитела использовали APC-конъюгированный антикроличий IgG (Jackson, Кат. № 711-136-152).
Эктопическая экспрессия рецептора Т-клеток F4 на цитотоксических Т-лимфоцитах (ЦТЛ): Чтобы получить эффекторные лимфоциты, которые экспрессируют MART-1-специфический РТК F4, специфически распознающий пептидно-ГКГС комплекс MART-126-35-/HLA-A2, недавно выделенные человеческие ЦТЛ, ранее трансдуцированные так, чтобы экспрессировать PVRIG, NGFR или пустой вектор, стимулировали ФГА и культивировали в течение 5-10 дней, а после этого трансдуцировали in vitro транскрибируемой мРНК, кодирующей α и β цепи из MART-1-специфического РТК F4. Трансдуцированные лимфоциты культивировали в лимфоцитарной среде (среда Bio target, фетальная бычья сыворотка (10%), L-глутамин, пенициллин/стрептомицин (100 единиц/мл), IL-2, 300 МЕ), меняемой каждый 2-3 дня. Уровни экспрессии РТК F4 подтверждали посредством окрашивания FACS, используя специфическое моноклональное антитело, которое распознает внеклеточный домен бета-цепи из трансдуцированного специфического РТК. (TCR-Vb12-PE, (Кат. № IM2291; Beckman Coulter).
Секреция цитокинов из трансдуцированных PVRIG, NGFR или пустым вектором и F4-РТК лимфоцитов после совместного культивирования с клетками меланомы: ЛПК, экспрессирующие PVRIG или NGFR наряду с F4-РТК, культивировали совместно с необработанными клетками меланомы. 105 трансдуцированных ЛПК культивировали совместно с 105 клетками-мишенями меланомы в течение 16 часов. Чтобы оценить ответ эффекторных Т-клеток CD8 на разные опухолевые клеточные линии, определяли секрецию цитокинов (IFNγ, IL-2 и TNF-α) методом ELISA в культуральных супернатантах (IFNγ (Кат. № DY285E), IL-2 (Кат. № DY202E), TNF-α (Кат. № DY210E) R&D SYSTEMS), разведенных, чтобы соответствовать линейному диапазону анализа ELISA.
Анализ клеточноопосредованной цитотоксичности: Этот анализ проводили, чтобы оценить уничтожение клеток-мишеней после совместного культивирования. PVRIG и F4 экспрессировали в ЛПК, используя двухцистронный вектор, и культивировали совместно с CFSE-мечеными клетками-мишенями меланомы (мечеными 2 мМ CFSE (eBioscience) в течение 6 мин) при 37°C в течение 18 ч, при соотношении Э:М 3:1. Через 18 ч клетки собирали и добавляли 1 мМ пропидиум иодида (Sigma-Aldrich) для определения доли погибших клеток. Образцы исследовали на проточном цитометре CyAn-ADP (Beckman Coulter).
Результаты:
Общий дизайн экспериментальной системы: В описанной в данном документе экспериментальной системе PVRIG сверхэкспрессируется на человеческих ЛПК, которые затем обрабатывают так, чтобы они экспрессировали MART1-специфический и HLA-A2-ограниченный РТК F4. Затем сверхэкспрессирующие клетки культивируют совместно с HLA-A2-положительными (назовите их) и HLA-A2-отрицательными (названия) клеточными линиями меланомы (ссылка). РТК F4 недавно использовали в клинических исследованиях на пациентах с меланомой в терминальной стадии, чтобы обеспечить специфическое распознавание опухоли аутологичными лимфоцитами из периферической крови, используя ретровирус, кодирующий РТК (Morgan et al, 2006 Science, 314:126-129). Действие экспрессии PVRIG на антигенспецифическую активацию Т-клеток CD8 путем совместного культивирования с когнатными клетками меланомы оценивали по секреции цитокинов.
Сверхэкспрессия PVRIG на человеческих ЛПК - эксперимент 1: Человеческие ЛПК трансдуцировали ретровирусным вектором, кодирующим PVRIG, или пустым вектором в качестве отрицательного контроля, как описано в параграфе Материалы и методы. Уровни PVRIG оценивали методом проточной цитометрии через 48 ч после трансдукции и сравнивали с клетками, трансдуцированными пустым вектором. Процентное содержание экспрессирующих трансген клеток составило 62,4%, как показано на фиг. 16.
Сверхэкспрессия PVRIG на человеческих ЛПК - эксперимент 2: Человеческие ЛПК трансдуцировали ретровирусным вектором, кодирующим PVRIG или NGFR, или пустым вектором в качестве отрицательного контроля, как описано в параграфе Материалы и методы. Уровни PVRIG оценивали методом проточной цитометрии через 48 ч после трансдукции и сравнивали с клетками, трансдуцированными пустым вектором. Процентное содержание экспрессирующих PVRIG клеток находилось в диапазоне 20%. Экспрессия NGFR составляла 63%, как показано на фиг. 16. Несколько дополнительных попыток добиться сверхэкспрессии PVRIG на ЛПК были неудачными. Одной из причин является то, что трудность экспрессии PVRIG в первичных ЛПК связана с основным уровнем эндогенной экспрессии в этих клетках.
Сверхэкспрессия РТК F4 на человеческих ЛПК: Чтобы провести функциональный анализ с человеческими ЦТЛ, мы использовали ЛПК, сконструированные для экспрессии РТК F4, который распознает клетки меланомы HLA-A2+/MART1+, как описано в разделе Материалы и методы. На фиг. 18A приведены уровни экспрессии РТК F4, полученные после РТК-трансдукции лейкоцитов, применяемых в эксперименте 1, на фиг. 18B приведены уровни экспрессии РТК F4, полученные после РТК-трансдукции лейкоцитов, применяемых в эксперименте 2.
Действие экспрессии PVRIG на секрецию IFNγ - эксперимент 1: Трансдуцированные PVRIG или пустым вектором и F4 ЛПК совместно культивировали с клеточными линиями меланомы. Уровни секреции IFNγ определяли через 16 ч после совместного культивирования. Как показано на фиг. 18, величина ингибирования секреции IFNγ вследствие сверхэкспрессии PVRIG составила более 90%. Совместное культивирование с HLA-A2-отрицательной клеточной линией Mel-888, которая служила в качестве отрицательного контроля, привело только к незначительной зависимой от активации секреции IFNγ из F4-трансдуцированных лимфоцитов. ЛПК, не экспрессирующие РТК F4 (обозначаемые «без»), служили в качестве дополнительного отрицательного контроля.
Действие экспрессии PVRIG на секрецию IFNγ - эксперимент 2: PVRIG, NGFR или пустой вектор и F4 трансдуцировали в ЛПК путем совместной трансдукции (фиг. 20A) или используя двухцистронный вектор (фиг. 20B). Трансдуцированные ЛПК совместно культивировали с клеточными линиями меланомы. Уровни секреции IFNγ определяли через 16 ч после совместного культивирования. Как показано на фиг. 19А, величина ингибирования секреции цитокинов вследствие сверхэкспрессии PVRIG находилась в диапазоне 30%. Совместное культивирование с HLA-A2-отрицательной клеточной линией Mel-888, которая служила в качестве отрицательного контроля, привело только к незначительной зависимой от активации секреции IFNγ из F4-трансдуцированных лимфоцитов. ЛПК, не экспрессирующие РТК F4 (обозначаемые «без»), служили в качестве дополнительного отрицательного контроля. Как показано на фиг. 19В, при совместной трансдукции PVRIG с РТК F4 ингибирование IFNγ не наблюдалось.
Действие PVRIG на ЦТЛ-опосредованную активность уничтожения - эксперимент 2: PVRIG или NGFR и F4 трансдуцировали в ЛПК, используя двухцистронный вектор, и культивировали совместно с CFSE-мечеными клеточными линиями меланомы. Как показано на фиг. 21, процентное содержание пропидиум иодид-положительных событий (отображающее интенсивность активности уничтожения) было снижено на ~50% вследствие экспрессии PVRIG по сравнению с отрицательными контрольными NGFR-трансдуцированными клетками. Активность уничтожения экспрессирующих PVRIG клеток сходна с активностью совместной культуры меланомы и ЛПК, не экспрессирующей РТК F4 (обозначаемой «без»).
Заключение: Не ограничиваясь одной гипотезой, представленные в данном документе результаты свидетельствуют о том, что сверхэкспрессия на первичных лимфоцитах приводит к снижению секреции цитокинов ЦТЛ, что позволяет предположить, что PVRIG оказывает ингибиторное действие на ЦТЛ.
Пример 9. Человеческие анти-PVRIG антитела
Задачей этого исследования было выделение человеческих антител, которые связываются с иммуно-онкологической мишенью PVRIG с высокой аффинностью и специфичностью и блокируют взаимодействие PVRIG с его партнером по связыванию PVRL2. Этого достигали путем пэннинга библиотеки фагового дисплея человеческого фрагмента fab против рекомбинантного белка, содержащего внеклеточный домен (ВКД) человеческого PVRIG, слитый с Fc-областью человеческого IgG1, и скрининга полученных в результате антител в отношении их способности блокировать взаимодействие PVRIG с PVRL2.
Протоколы
Функциональный КК реагентов: Степень очистки реагента для пэннинга, ВКД PVRIG, слитого с Fc-доменом человеческого IgG1 (PVRIGHH), определяли методом микрофлюидного капиллярного электрофореза с применением системы LabChip (PerkinElmer). Активность реагента для пэннинга подтверждали по его способности связывать свой лиганд PVRL2.
ELISA для выявления белок-белкового взаимодействия: His-меченый рекомбинантный белок PVRL2 разводили до 2 мкг/мл в фосфатно-солевом буфере (ФСБ) и покрывали аликвотами по 50 мкл лунки планшета с высокой степенью связывания EIA/RIA (Costar) в течение ночи при 4°C. Покрытые лунки планшета дважды промывали ФСБ и инкубировали с 300 мкл блокирующего буфера (5% сухое обезжиренное молоко в ФСБ, pH 7,4) при комнатной температуре (КТ) в течение 1 ч. Блокирующий буфер удаляли, а планшеты дополнительно дважды промывали ФСБ. Связанный с планшетом PVRL2 инкубировали с разными концентрациями PVRIGHH в растворе (линейный диапазон от 0,1 мкг/мл до 4 мкг/мл в объеме 50 мкл/лунку) при КТ в течение 1 ч. Планшеты три раза промывали ФСБ-Т (ФСБ 7,4, 0,05% Твин20), затем три раза ФСБ и добавляли 50 мкл/лунку HRP-конъюгированного вторичного антитела (Специфического к Fc-домену человеческого IgG). Реакцию инкубировали при КТ в течение 1 ч и снова промывали планшеты. Сигналы ELISA инициировали во всех лунках путем добавления 50 мкл ТМБ-субстрата Sureblue (KPL Inc) и инкубации в течение 5-20 мин. Реакцию HRP останавливали путем добавления 50 мкл 2N H2SO4 (VWR), а сигналы поглощения на 450 нм считывали на спектрофотометре SpectraMax (Molecular Devices) или EnVision (PerkinElmer).
Получение биотинилированного PVRIG: Чтобы облегчить фаговый пэннинг в растворе с применением покрытых стрептавидином магнитных гранул, PVRIGHH и нерелевантный изотипический контрольный человеческий IgG1 Fc биотинилировали, используя набор с биотином Lightning-Link® (Innova Biosciences). Реакции биотинилирования проводили, следуя протоколу производителя, а биотинилированные реагенты хранили при 4°C для проведения дополнительного КК и биопэннинга. Степень очистки и активность меченых биотином белков оценивали с помощью LabChip и функционального анализа ELISA, как описано в Разделе 2.1. Кроме того, степень биотинилирования оценивали методом ELISA, используя два подхода: 1) биотинилированные реагенты адсорбировали на планшете с высокой степенью связывания EIA/RIA и проводили выявление белков, используя HRP-конъюгированный стрептавидин, и 2) биотинилированные белки инкубировали на планшете EIA/RIA, предварительно покрытом стрептавидином, и выявляли связывание, используя HRP-конъюгированное специфическое к Fc-домену человеческого IgG вторичное антитело.
Фаговый пэннинг библиотеки человеческих антител: Реакции пэннинга проводили в растворе, используя покрытые стрептавидином магнитные гранулы для захвата биотинилированных антигенов. Следует отметить, что все этапы промывки и элюирования проводили, используя магнитную подставку для захвата гранул (Promega). Все этапы инкубации проводили при комнатной температуре с аккуратным перемешиванием на шейкере для пробирок (BioExpress). Проводили четыре подкампании пэннинга, каждая с разной комбинацией концентраций антигена, этапов промывки и элиминации Fc-связывающих компонентов (таблица 1).
Все подкампании пэннинга проводили, используя биотинилированный антиген PVRIGHH. Для каждого цикла пэннинга осуществляли элиминацию фаговых библиотек против 100 пмолей белка нерелевантного человеческого IgG1 Fc на двух последовательных этапах. После элиминации подкампании A и B включали пэннинг против 50 нМ антигена на каждом цикле, в условиях низкой и высокой жесткости промывки соответственно. Подкампании С и D были идентичны подкампании B за исключением того, что во время подкампании С библиотеку блокировали 10-кратным избытком нерелевантного белка IgG1 Fc на 2 и 3 циклах пэннинга. Подкампания D отличалась тем, что на 3 цикле использовали 5 нМ антигена.
Таблица 1. Антиген и условия промывки, применяемые для фагового пэннинга против PVRIGHH.
Подкампания Цикл Антиген Концентрация Промывки Элиминация Fc
A 1 50 нМ 3x ФСБ-T + 3x ФСБ 2X 100 пмоль
2 50 нМ 3x ФСБ-T + 3x ФСБ 2X 100 пмоль
3 50 нМ 3x ФСБ-T + 3x ФСБ 2X 100 пмоль
B 1 50 нМ 3x ФСБ-T + 3x ФСБ 2X 100 пмоль
2 50 нМ 6x ФСБ-T + 6x ФСБ 2X 100 пмоль
3 50 нМ 6x ФСБ-T + 6x ФСБ 2X 100 пмоль
C 1 50 нМ 3x ФСБ-T + 3x ФСБ 2X 100 пмоль
2 50 нМ 6x ФСБ-T + 6x ФСБ 2X 100 пмоль + блок 1 нмоль
3 50 нМ 6x ФСБ-T + 6x ФСБ 2X 100 пмоль
блок 1 нмоль
D 1 50 нМ 3x ФСБ-T + 3x ФСБ 2X 100 пмоль
2 50 нМ 6x ФСБ-T + 6x ФСБ 2X 100 пмоль
3 5 нМ 6x ФСБ-T + 6x ФСБ 2X 100 пмоль
2.4. Получение фаговой библиотеки для пэннинга: Во всех экспрериментах по фаговому пэннингу использовали библиотеку фагового дисплея человеческих fab-антител XOMA031 (XOMA Corporation, Berkeley, CA). Достаточное количество фага для 50-кратного преобладания над библиотекой блокировали путем смешивания 1:1 с 10% сухим обезжиренным молоком в ФСБ (конечная концентрация обезжиренного молока 5%) и инкубации в течение 1 ч.
2.4.1. Сопряжение антигена со стрептавидиновыми гранулами: Для каждой подкампании три 100 мкл аликвоты покрытых стрептавидином магнитных гранул Dynal (Life Technologies) блокировали суспензией в 1 мл блокирующего буфера (5% сухое обезжиренное молоко в ФСБ) и инкубировали в течение 30 мин. Одну аликвоту заблокированных гранул смешивали с 100 пмолями биотинилированного PVRIGHH. Две другие аликвоты смешивали со 100 пмолями нерелевантного антигена для элиминации только Fc-связывающих компонентов. Проводили сопряжение меченых биотином антигенов с гранулами в течение 30 мин при КТ. Суспензии гранул дважды промывали ФСБ для удаления свободного антигена и пересуспендировали в 100 мкл блокирующего буфера.
2.4.2. Элиминация человеческого IgG1 Fc и компонентов, связывающих стрептавидиновые гранулы, из фаговой библиотеки: Перед началом пэннинга было необходимо удалить нежелательные компоненты, связывающиеся со стрептавидиновыми гранулами и Fc-областью PVRIGHH. Для достижения этого заблокированный фаг смешивали с одной 100 мкл аликвотой несопряженных стрептавидиновых гранул и инкубировали в течение 45 мин. Гранулы (и предположительно нежелательные компоненты, связывающие гранулы и человеческий IgG1 Fc) удаляли. Этот этап повторяли один раз, а истощенные супернатанты фаговой библиотеки оставляли для пэннинга.
2.5. Фаговый пэннинг, цикл 1: Заблокированную и истощенную фаговую библиотеку смешивали с биотинилированным PVRIGHH, сопряженным с магнитными гранулами, описанными выше. Эту суспензию инкубировали в течение 1 ч при КТ с аккуратным перемешиванием для обеспечения связывания специфического к PVRIGHH фага. Неспецифические связывающие компоненты удаляли в соответствии с протоколом в Таблице 1. После промывки связанный фаг элюировали путем инкубации с 500 мкл 100 мМ триэтиламина (ТЭА) (EMD) в течение 15 мин при КТ. Элюат нейтрализовали путем добавления 500 мкл 1 M Трис-HCl, pH 8,0 (Teknova).
2.5.1. Определение фагового титра: 10 мкл исходной фаговой библиотеки (входной титр) или пэннингового элюата (выходной титр) серийно разводили (10-кратно) в ФСБ. 90 мкл аликвоту каждого фагового разведения смешивали с 500 мкл клеток E. coli TG1, выращенных до оптической плотности ~0,5 на 600 нм (ОП 600 нм). Фагу позволяли инфицировать клетки путем стационарной инкубации в течение 30 мин, затем инкубацию встряхивали (250 об/мин) в течение 30 мин, все при 37°C. 10 мкл аликвоту каждой инфицированной клеточной культуры пятнами наносили на агаровый планшет 2YT, дополненный 2% глюкозой и 100 мкг/мл карбенициллина (2YTCG, Teknova). Планшеты инкубировали в течение ночи при 30°C. Подсчитывали колонии, растущие из каждого 10 мкл пятна, и использовали их для расчета входных и выходных титров.
2.5.2. Освобождение фага: Оставшийся фаговый элюат (~1 мл) смешивали с 10 мл клеток E. coli TG1, выращенных до ОП 600 нм 0,5. Фаги инфицировали в клетки, как подробно описано в разделе 2.5.1. Инфицированные клетки осаждали путем центрифугирования при 2500xG, пересуспендировали в 750 мкл среды 2YT (Teknova) и наносили на агаровые планшеты 2YTCG. Планшеты инкубировали в течение ночи при 37°C, а оставшиеся участки E. coli соскребали и пересуспендировали в ~20 мл жидкой 2YTCG (Teknova). Небольшую аликвоту пересуспендированных клеток инокулировали в 50 мл 2YTCG для достижения ОП 600 нм 0,05, а затем выращивали при 37°C при 250 об/мин, встряхивая, пока ОП не достигала 0,5. Полученную в результате культуру инфицировали хелперным фагом M13K07 (New England Biolabs) и инкубировали в течение ночи при 25°C, встряхивая для обеспечения упаковки фага. Культуральный супернатант, содержащий освобожденные фаговые частицы, очищали центрифугированием при 2500xG, а 1 мл переносили для a) следующего цикла пэннинга или b) исследования связывания fab.
Фаговый пэннинг, циклы 2-3: Второй и третий циклы пэннинга проводили, как и вышеописанные этапы, за исключением того, что освобожденный фаговый супернатант с предыдущего цикла использовали вместо фаговой библиотеки. Применяемые условия промывки, элиминации и концентрации антигена приведены в таблице 1.
2.6. Исследование связывания с применением fab, полученных из периплазматических экстрактов
2.6.1. Экспрессионные векторы Fab: В основе библиотеки XOMA031 лежат фагмидные конструкции, которые функционируют также как экспрессионные векторы fab. Эти векторы содержат экспрессионные кассеты тяжелой цепи и легкой цепи fab, промотор lac для управления экспрессией генов антитела и ген устойчивости к ампициллину. Цепи антитела дополнены N-концевыми сигнальными пептидами для управления их секрецией в периплазматическое пространство. С-конец тяжелой цепи содержит усеченную белковую последовательность гена III для внедрения в фаговые частицы. Тяжелая цепь также содержит аффинные метки гекса-гистидин, c-myc и V5. Трансформация этих векторов в E. coli и индукция изопропил-β-D-1-тиогалактопиранозидом (ИПТГ) приводит к периплазматической экспрессии растворимых молекул fab.
2.6.2. Получение ППЭ Fab: Элюированные фаговые пулы с 3-го цикла пэннинга разводили и инфицировали клетки E. coli TG1 (Lucigen) так, что при нанесении на агаровый планшет 2YTCG получались одиночные колонии. Это приводило к тому, что каждая колония содержала один клон fab. Индивидуальные колонии инокулировали в 1 мл стартовые культуры 2YTCG в 96-луночные блоки с глубокими лунками (VWR), используя прибор Qpix2 (Molecular Devices). Эти стартовые культуры выращивали в течение ночи в 3 мм инкубаторе Multitron (Infors) при 37°C со встряхиванием при 700 об/мин. Для экспрессии fab 20 мкл из 1 мл стартовых культур переносили во второй набор планшетов с глубокими лунками, содержащими 1 мл 2YT с 0,1% глюкозой и 100 мкг/мл ампициллина. Культуры выращивали до достижения средней ОП 600 нм 0,5-1,0, а экспрессию белка индуцировали добавлением ИПТГ (Teknova) до конечной концентрации 1 мМ. Экспрессионные культуры инкубировали в течение ночи в приборе Multitron при 25°C со встряхиванием при 700 об/мин.
Белки Fab, секретируемые в периплазму E. coli, экстрагировали для анализа. Клетки собирали путем центрифугирования при 2500xG, удаляли супернатанты, а осадок пересуспендировали в 75 мкл охлажденного буфера КФБ (Teknova). Экстракты инкубировали в течение 10 мин при 4°C со встряхиванием при 1000 об/мин, добавляли 225 мкл охлажденной ddH2O и дополнительно инкубировали в течение 1 ч. Полученный в результате периплазматический экстракт (ППЭ) очищали центрифугированием при 2500xG и переносили в отдельные планшеты или пробирки для анализа ELISA и FACS. Все экстракционные буферы содержали не содержащий ЭДТА полный коктейль ингибиторов протеаз (Roche).
Каждый планшет с образцами также содержал по две лунки с «пустым ППЭ», служащие в качестве отрицательного контроля. Их создавали, намеренно оставляя две 1 мл культуры неинокулированными, а затем обрабатывая их таким же способом, как и ППЭ fab, тем самым создавая образец с отсутствием бактериального роста и, следовательно, отсутствием экспрессии fab.
2.6.3. Первичный скрининг методом ELISA: По два 96-луночных планшета с ППЭ-экстрактами на подкампанию исследовали в отношении связывания с PVRIGHH методом ELISA. Следует отметить, что для скрининга ELISA использовали небиотинилированную версию белка, чтобы избежать селекции компонентов, связывающих биотин или стрептавидин. Рекомбинантный белок PVRIGHH разводили до 2 мкг/мл в фосфатно-солевом буфере (ФСБ) и наносили 50 мкл аликвоты на стенки лунок планшета с высокой степенью связывания EIA/RIA (Costar) в течение ночи при 4°C. Покрытые лунки планшета дважды промывали ФСБ и инкубировали с 300 мкл блокирующего буфера (5% сухое обезжиренное молоко в ФСБ, pH 7,4) при комнатной температуре (КТ) в течение 1 ч. Блокирующий буфер удаляли, а планшеты дополнительно дважды промывали ФСБ. Связанный с планшетом PVRIG инкубировали с ППЭ, предварительно заблокированными 3% обезжиренным молоком, при КТ в течение 1 ч. Планшеты три раза промывали ФСБ-Т (ФСБ 7,4, 0,05% Твин20), затем три раза ФСБ и добавляли 50 мкл/лунку HRP-конъюгированного вторичного антитела против человеческого Fab (Jackson ImmunoResearch) в разведении 1:2000 в 5% молоке в ФСБ. Реакцию инкубировали при КТ в течение 1 ч и снова промывали планшеты. Сигналы ELISA инициировали во всех лунках путем добавления 50 мкл ТМБ-субстрата Sureblue (KPL Inc) и инкубации в течение 5-20 мин. Реакцию HRP останавливали путем добавления 50 мкл 2N H2SO4 (VWR), а сигналы поглощения на 450 нм считывали на спектрофотометре SpectraMax (Molecular Devices) или EnVision (PerkinElmer). Лунки, которые демонстрировали отношение сигнала к фону (пустому ППЭ) > 3, отбирали как положительные хиты.
2.6.4. Анализ последовательностей ELISA-положительных fab: Отбирали положительные хиты из скрининга ELISA и переносили в новый 96-луночнй планшет. Клоны выращивали в течение ночи при 37°C и проводили секвенирование плазмидной ДНК, используя специфические к тяжелой цепи и легкой цепи праймеры. Сборку и анализ последовательностей проводи, используя программное обеспечение Xabtracker (XOMA). Клоны считали уникальными по последовательности при наличии более одной неконсервативной замены в CDR3 тяжелой цепи. Клоны с такой же или сходной тяжелой цепью, но с существенными различиями в легких цепях, отмечали как родственные исходному клону последовательности.
2.6.5. Скрининг FACS для fab в виде ППЭ: Отбирали уникальные по последовательности ELISA-положительные клоны fab и анализировали в отношении их способности связывать сверхэкспрессирующие PVRIG клетки методом сортировки флуоресцентно-активированных клеток (FACS). Анализ проводили, используя клетки HEK293, сверхэкспрессирующие человеческий антиген PVRIG. В параллельном эксперименте использовали нетрансфицированные клетки HEK293 в качестве отрицательного контроля для каждого образца fab.
ППЭ для уникальных по последовательности ELISA-положительных клонов fab получали, как описано выше. Все анализы проводили, используя буфер FACS (1% БСА и 0,1% азид натрия в ФСБ). Собирали человеческий PVRIG и нетрансфицированные клетки HEK293, дважды промывали и пересуспендировали с плотностью 2x106 клеток/мл. 25 мкл аликвоту клеток смешивали с 25 мкл каждого ППЭ-образца и инкубировали в течение 1 ч при 4°C, аккуратно встряхивая. Также в анализ были включены два пустых ППЭ-контроля. Планшеты один раз промывали в 200 мкл буфера FACS и добавляли в каждую лунку 50 мкл 2 мкг/мл разведенного мышиного анти-C-myc антитела (Roche). После инкубации в течение 30 мин при 4°C клетки снова промывали и в каждую лунку с ППЭ и отрицательным контролем добавляли 25 мкл 5 мкг/мл разведенного козьего антимышиного fab-AF647 (Jackson Immunoresearch). Все вторичные антитела инкубировали в течение 30 мин при 4°C. После двух промывок клетки пересуспендировали в конечном объеме 50 мкл буфера для фиксации (2% параформальдегид в буфере FACS). Образцы считывали на приборе для скрининга Intellicyt HTFC, записывая приблизительно 5000 событий на лунку в обозначенной живой области. Данные анализировали с помощью FlowJo (De Novo Software, CA, USA) и экспортировали в Excel. Отношение средней интенсивности флуоресценции (СИФ) для сверхэкспрессирующих человеческий PVRIG клеток HEK и нетрансфицированных клеток 293 рассчитывали с помощью программного обеспечения Xabtracker (XOMA). Положительные хиты на каждом планшете определяли как такие, которые давали отношение СИФ в 5 раз превосходящее средний сигнал для пустого ППЭ-контроля.
Переформатирование хитов fab и получение в виде молекул человеческого IgG: Потенциальные PVRIG-связывающие fab преобразовывали в полноразмерные человеческие IgG для получения дополнительных характеристик. Конструкции для экспрессии белка получали путем ПЦР-амплификации генов вариабельных доменов тяжелой цепи, лямбда и каппа доменов, которые субклонировали в векторы pFUSE-CHIg-hG1 (тяжелая цепь человеческого IgG1), pFUSE2-CLIg-hK (человеческая каппа легкая цепь) или pFUSE2-CLIg-hL2 (человеческая лямбда 2 легкая цепь) соответственно (все экспрессионные векторы были получены от Invivogen).
Клетки еxpi293 (Life Technologies) высевали при 6x105 клеток/мл в среду Expi293 (Life Technologies) и инкубировали в течение 72 ч при 37°C во влажной атмосфере 8% CO2, встряхивая при 125 об/мин. Эту исходную массу клеток использовали для посева экспрессионных культур при 2,0 x106 клеток/мл в среду Expi293. Эти культуры инкубировали, как и ранее, в течение 24 ч, встряхивая при 135 об/мин.
Для трансфекции клетки снова разводили до 2,5x106 клеток/мл в среде Expi293. Конструкции для экспрессии белка тяжелой цепи и легкой цепи антитела смешивали в соотношении 1:2. Для каждых 30 мл объема экспрессионной культуры отдельно разводили 30 мкг ДНК и 81 мкл Expifectamine (Life Technologies) до 1,5 мл средой Opti-MEM (Life Technologies) и инкубировали в течение пяти минут. Затем разведенные ДНК и Expifectamine смешивали и инкубировали при КТ в течение 20 мин. Затем эту смесь добавляли в экспрессионную культуру в смесительной колбе и инкубировали, как описано выше, встряхивая при 125 об/мин.
Приблизительно через 20 ч после трансфекции в каждую колбу добавляли 150 мкл усилителя трансфекции 293 ExpiFectamine 1 и 1,5 мл усилителя трансфекции 293 ExpiFectamine 2. Культуры инкубировали в течение дополнительных пяти дней (всего шесть дней после трансфекции) и собирали супернатанты путем центрифугирования. IgG очищали от супернатантов с помощью аффинных колонок AKTA Pure FPLC (GE Healthcare Bio-Sciences) и HiTrap MabSelect Sure (GE Healthcare Bio-Sciences) в соответствии с инструкциями производителя.
Скрининг FACS переформатированных антител IgG1: Скрининг FACS переформатированных антител проводили аналогично описанному в данном документе скринингу ППЭ, за исключением того, что проводили дозозависимое титрование очищенных антител. Сверхэкспрессирующие человеческий PVRIG клетки HEK293 или нетрансфицированные клетки HEK293 инкубировали с разными концентрациями (0-10 мкг/мл) анти-PVRIG антител или изотипических контролей в буфере FACS при 4°C в течение 60 мин. Клетки один раз промывали в буфере FACS, пересуспендировали в 50 мкл конъюгированного с Alexa Fluor 647 антитела против человеческого IgG (специфического в отношении фрагмента Fab), разведенного 1:200, и инкубировали в течение 30 мин при 4°C в темноте. Клетки дважды промывали и пересуспендировали в конечном объеме 80 мкл буфера FACS и пропидиум иодида (Biolegend кат. # 421301), разведенном 1:1000. Образцы анализировали с помощью системы для скрининга Intellicyt HTFC (Intellicyt). Данные анализировали с помощью FlowJo (DeNovo), экспортировали в Excel (Microsoft) и строили график в GraphPad Prism (GraphPad Software, Inc.).
Результаты
Функциональный КК рекомбинантного белка PVRIGHH: Степень очистки белка PVRIGHH оценивали методом микрофлюидного капиллярного электрофореза, используя систему LabChip. В восстановительных условиях рекомбинантный белок сместился на 80 кДа, что согласуется с рассчитанной молекулярной массой 80,4 кДа, и демонстрировал степень очистки 99% (данные не показаны). В невосстановительных условиях наблюдали один дополнительный пик, который, вероятно, является результатом наличия димерной формы белка вследствие Fc-Fc взаимодействия.
Функциональную целостность рекомбинантного белка определяли, оценивая его связывание с PVRL2 (известным лигандом для PVRIG) методом ELISA. Для связывания PVRIGHH с PVRL2 наблюдали дозозависимый ответ (данные не показаны). Для сравнения, в случае нерелевантного человеческого контрольного IgG1 Fc связывание не наблюдали. Вместе это свидетельствует о том, что рекомбинантный белок PVRIGHH имеет высокую степень очистки и является функционально активным и, таким образом, подходит для биопэннинга.
КК биотинилированного рекомбинантного белка PVRIGHH: Степень очистки биотинилированного белка PVRIGHH оценивали методом микрофлюидного капиллярного электрофореза, используя систему LabChip. Между небиотинилированным и биотинилированным рекомбинантными белками существенной разницы не наблюдали (данные не показаны). Следует отметить, что для биотинилированного образца наблюдали дополнительный 44,3 кДа пик. Этот пик может появляться благодаря мономерной форме белка PVRIGHH или может являться артефактом реакции гашения набора для биотинилирования.
Успешное биотинилирование подтверждали путем инкубации биотинилированного белка в покрытом стрептавидином планшете EIA и выявления связанного белка с помощью HRP-конъюгированного вторичного антитела против человеческого IgG1 Fc. Связывание биотинилированного PVRIGHH с покрытым стрептавидином планшетом EIA было сравнимо с приобретенным на коммерческой основе нерелевантным биотинилированным белком (данные не показаны).
Фаговый пэннинг: Биотинилированный белок PVRIGHH использовали для фагового пэннинга против библиотеки фагового дисплея человеческих fab-антител XOMA031 (XOMA Corporation, Berkeley, CA). Было проведено три цикла биопэннинга с 4 разными комбинациями условий жесткости промывки, концентрации антигена и элиминации связывающих Fc компонентов (подкампании A-D). Успешность каждого цикла оценивали, используя выходные фаговые титры. Для определения успешности подкампаний пэннинга использовали качественные критерии, такие как значительное снижение фаговых титров после 1-го цикла, повышение или сохранение фаговых титров после 2-го и 3-го циклов и снижение фаговых титров после повышения жесткости промывки или снижения концентрации антигена. Результатом всех 4 подкампаний были фаговые титры в ожидаемом диапазоне, согласованные между подкампаниями (данные не показаны).
Скрининг фагового выхода в виде ППЭ fab: Проводили скрининг двух 96-луночных планшетов с клонами fab (в виде ППЭ) для каждой из четырех подкампаний для оценки успешности биопэннинга. Результаты обобщены в таблице 3 и более подробно обсуждаются ниже. В целом, все 4 подкампании привели к получению значительного числа PVRIGHH-специфических fab. Всего было идентифицировано 49 мишень-специфических уникальных fab. Подкампании В и D продемонстрировали наивысшие уровни хитов ELISA и корреляцию FACS и были отобраны для расширенного скрининга.
Таблица 3: Обобщенные данные по первичному скринингу ППЭ fab. Для каждой подкампании приведены общее число исследуемых клонов, хитов ELISA, хитов FACS и уникальность последовательности. Открытые рамки считывания (ОРС) представляют клоны, которые были успешно секвенированы как полноразмерные fab. Специфичность основана на отсутствии неспецифического связывания с нерелевантными белками в анализе ELISA. Корреляция FACS представляет процент хитов ELISA, которые были также FACS-положительными (специфически связывались со сверхэкспрессирующими PVRIG клетками HEK293).
Подкампания A Подкампания B Подкампания C Подкампания D Всего
Исследовано клонов 182 182 182 182 728
ELISA-положительные (>3 С/Ш) 48 51 44 68 211
Уровень хитов ELISA 26% 28% 24% 37% 29%
ОРС 36 (75%) 45 (88%) 35 (80%) 63 (93%) 179 (85%)
Уникальные последовательности 25 21 17 31 73
Разнообразие 69% 47% 49% 49% 41%
Специфичность по ELISA* 100% 100% 100% 100% 100%
Связывающие компоненты по FACS (>5 С/Ш) 14 17 14 24 49**
Корреляция FACS 56% 81% 82% 77% 67%
*Отсутствие неспецифического связывания с нерелевантными Fc-конъюгатами или PVRL2; **35 уникальных HC, 14 родственных последовательностей
Первичный скрининг fab (ELISA): Скрининг двух 96-луночных планшетов (182 клонов fab) с ППЭ для каждой подкампании проводили методом ELISA против рекомбинантного белка PVRIGHH. Следует отметить, что хотя для пэннинга использовали биотинилированный белок, для скрининга ELISA использовали небиотинилированную версию, что позволило избежать выявления специфических в отношении биотина или стрептавидина связывающих компонентов. Результатом 4 подкампаний стали уровни хитов ELISA в диапазоне 24-37%, при этом порог для «положительного» сигнала был установлен как 3-кратное превышение мишень-специфического связывания по сравнению с пустым контрольным ППЭ-сигналом.
Вторичный скрининг (анализ последовательности ДНК, ELISA и FACS) fab: Секвенировали ELISA-положительные клоны, чтобы отобрать недублирующие fab. Было идентифицировано семьдесят три уникальных по последовательности клонов fab. 19 клонов были уникальными для подкампании A, 13 клонов были уникальными для подкампании B, 10 клонов были уникальными для подкампании C, 18 клонов были уникальными для подкампании D, тогда как оставшиеся 23 клона были общими для разных подкампаний. Уникальные по последовательности ELISA-положительные клоны fab повторно экспрессировали в виде ППЭ и исследовали в отношении специфического связывания методом FACS. Всего 49 из 73 уникальных клонов были идентифицированы как PVRIG-специфические связывающие компоненты по данным ELISA и FACS (согласно критериям, установленным в 2.6.5). 49 связывающих компонентов по данным FACS соответствовали 35 антителам с уникальными тяжелыми цепями и 14 родственным последовательностям, которые имеют уникальные легкие цепи, но одинаковые тяжелые цепи с одним из уникальных клонов. Обобщенные данные по связыванию FACS представлены в Таблице 4.
Уникальные по последовательности fab также исследовали в отношении неспецифического связывания. Все анализируемые ППЭ fab связывались с рекомбинантным белком PVRIGHH с аналитическим сигналом более чем в 3 раза превышающим пустой ППЭ-контроль. В параллельном анализе ППЭ fab исследовали в отношении связывания с двумя нерелевантными белками с такой же Fc-областью IgG1, а также рекомбинантным белком PVRL2. Ни один из клонов не демонстрировал существенное неспецифическое связывание с контролями, что позволяет предположить, что отобранные fab специфичны в отношении PVRIG.
Таблица 4. Обобщенные данные по связыванию FACS для PVRIG fab. Все уникальные ELISA-положительные fab анализировали методом FACS. Для сверхэкспрессирующих PVRIG клеток HEK293, а также нетрансфицированных клеток HEK293 измеряли среднюю интенсивность флуоресценции (СИФ). Рассчитывали отношение СИФ между мишень-специфическим и нецелевым связыванием. Клоны с отношением СИФ > 5 были отобраны как хиты и приведены ниже.
клон fab отношение СИФ клон fab отношение СИФ
CPA.7.001 11 CPA.7.026 5.3
CPA.7.002 8.9 CPA.7.027 9.2
CPA.7.003 9.5 CPA.7.028 17
CPA.7.004 9.3 CPA.7.029 6.7
CPA.7.005 6.5 CPA.7.030 15
CPA.7.006 9.6 CPA.7.031 8.5
CPA.7.007 14 CPA.7.032 7.6
CPA.7.008 14 CPA.7.033 22
CPA.7.009 10 CPA.7.034 7.7
CPA.7.010 7.6 CPA.7.035 14
CPA.7.011 10 CPA.7.036 5
CPA.7.012 19 CPA.7.037 5.3
CPA.7.013 12 CPA.7.038 6.3
CPA.7.014 14 CPA.7.039 12
CPA.7.015 15 CPA.7.040 12
CPA.7.016 7.6 CPA.7.041 7.6
CPA.7.017 13 CPA.7.042 5.4
CPA.7.018 7.8 CPA.7.043 13
CPA.7.019 16 CPA.7.044 7.9
CPA.7.020 6.9 CPA.7.045 7.8
CPA.7.021 15 CPA.7.046 10
CPA.7.022 7.5 CPA.7.047 8.4
CPA.7.023 12 CPA.7.049 10
CPA.7.024 9.8 CPA.7.050 22
CPA.7.025 6
Переформатирование ELISA- и FACS-положительных fab в hIgG1: Все уникальные связывающие компоненты по данным ELISA и FACS переформатировали для экспрессии в виде молекул человеческого IgG1 в клетках Expi293. Из исходных 49 антител 44 успешно экспрессировались в виде полноразмерных антител. Эти переформатированные антитела исследовали в отношении сохранения связывания со сверхэкспрессирующими PVRIG клетками HEK293 наряду с изотипическим контрольным нерелевантным человеческим IgG1. Все антитела также исследовали против нетрансфицированных клеток HEK293. Полученные результаты по связыванию использовали, чтобы продемонстрировать специфичность антител, а также строили графики, чтобы рассчитать равновесную константу связывания (KД). Девять из оставшихся 44 антител демонстрировали слабое связывание или существенное неспецифическое связывание. Оставшиеся 35 антител были отобраны для дополнительного анализа с помощью клеточного функционального анализа. KД этих антител на основе данных FACS приведены в таблице 6. Значения KД находятся в диапазоне от 0,30 нМ до 96 нМ, с медианным значением 9,4 нМ, что позволяет предположить, что большинство антител, полученных после кампании пэннинга являются очень специфическими и связываются с PVRIG с высокой аффинностью.
Таблица 5. Обобщенные данные по экспрессии и связыванию переформатированных антител. Все уникальные ELISA- и FACS-положительные fab были переформатированы в структуру человеческого IgG1. Значения KД по данным FACS определяли путем титрования дозы против сверхэкспрессирующих PVRIG клеток HEK293. Нецелевое связывание определяли путем титрования дозы против нетрансфицированных клеток HEK293.
Антитело KД FACS (нМ) Антитело KД FACS (нМ)
CPA.7.001 Отсутствие экспрессии CPA.7.026 Отсутствие связывания
CPA.7.002 44.35 CPA.7.027 Отсутствие связывания
CPA.7.003 Неспецифическое связывание CPA.7.028 7.14
CPA.7.004 21.71 CPA.7.029 Слабое связывание
CPA.7.005 95.56 CPA.7.030 Отсутствие экспрессии
CPA.7.006 Отсутствие экспрессии CPA.7.031 Отсутствие связывания
CPA.7.007 0.73 CPA.7.032 8.78
CPA.7.008 Отсутствие экспрессии CPA.7.033 12.8
CPA.7.009 33.00 CPA.7.034 14.2
CPA.7.010 21.89 CPA.7.035 Отсутствие связывания
CPA.7.011 66.02 CPA.7.036 6.0
CPA.7.012 0.30 CPA.7.037 Неспецифическое связывание
CPA.7.013 Отсутствие экспрессии CPA.7.038 20.26
CPA.7.014 2.04 CPA.7.039 3.76
CPA.7.015 1.34 CPA.7.040 0.79
CPA.7.016 22.02 CPA.7.041 52.2
CPA.7.017 1.82 CPA.7.042 24.26
CPA.7.018 9.29 CPA.7.043 13.2
CPA.7.019 0.45 CPA.7.044 9.4
CPA.7.020 86.97 CPA.7.045 3.73
CPA.7.021 11.22 CPA.7.046 Неспецифическое связывание
CPA.7.022 4.17 CPA.7.047 5.36
CPA.7.023 4.08 CPA.7.049 19.9
CPA.7.024 9.08 CPA.7.050 68.3
CPA.7.025 Отсутствие связывания
Выводы и заключения
Проводили кампанию по выявлению антител с применением фагового дисплея для выделения связывающих компонентов против иммуно-онкологической мишени PVRIG, используя рекомбинантную Fc-меченую версию антигена. Анализ контроля качества показал, что полученный в результате пэннинга антиген был чистым и функционально активным. Подход на основе пэннинга привел к получению 49 уникальных клонов fab, которые специфически связывались с мишенью PVRIG, как в виде рекомбинантного белка, так и на клеточной поверхности. Из них 35 были успешно получены в виде человеческих антител IgG1, и было показано, что они сохраняют способность специфически связываться с PVRIG. Этот пул антител демонстрировал высокую аффинность в анализе FACS, при этом 18 из 35 антител связывались с KД < 10 нМ.
Пример 10. Демонстрация способности fab против человеческого PVRIG блокировать взаимодействие между PVRIG и PVRL2 методом ELISA
Метод: Человеческим PVRL2-His (Каталог #2229-N2-050/CF, R&D Systems) покрывали планшет ELISA. Периплазматические экстракты (ППЭ) fab, разведенные 1:1 в 5% обезжиренном молоке, предварительно инкубировали с 1 мкг/мл (конечная концентрация) человеческого PVRIG-Fc в течение 15 мин при КТ. Смесь fab-рецептор оставляли для связывания PVRL2-His, нанесенного на планшет ELISA. Взаимодействие PVRIG-Fc/PVRL2-His проверяли, используя антитело против человеческого Fc, конъюгированное с HRP (Jackson Immuno Research, каталог #709-035-098). В отсутствие ППЭ (отрицательные лунки) ожидалось появление сильного положительного сигнала. Для блокирующих fab сигнал должен быть существенно снижен. Клоны fab с >5-кратно меньшим сигналом по сравнению с отрицательными лунками (>80% блокирования) можно отбирать как блокирующие fab.
Протокол:
Планшеты ELISA (Costar 9018) покрывали 50 мкл 2 мкг/мл антигена и хранили при 4 С в течение ночи. Покрытые антигеном планшеты 3 раза промывали 1Х ФСБ. Планшет блокировали 200 мкл 5% обезжиренного молока в ФСБ и инкубировали 1 ч при КТ (комнатной температуре). После этого планшет промывали 1Х ФСБ.
После добавления 50 мкл/лунку ППЭ Fab (разведенных в 5% обезжиренном молоке) планшет предварительно инкубировали с 1 мкг/мл человеческого PVRIG-Fc, который добавляли в соответствующие лунки. В 2 лунках находился контроль «без fab».
Планшет инкубировали 1 ч при КТ.
Планшеты промывали 3 раза 1Х ФСБТ и 3 раза 1Х ФСБ.
После добавления 50 мкл/лунку HRP-конъюгированного вторичного антитела (Jackson Immuno Research, 709-035-098), разведенного в 5% молоке в ФСБ, планшет инкубировали 1 ч при КТ.
Планшеты промывали 3 раза 1Х ФСБТ и 3 раза 1Х ФСБ.
После добавления 50 мкл/лунку ТМБ-субстрата и ожидания до появления цвета реакцию останавливали, добавляя 50 мкл/лунку 2N H2SO4. Поглощение измеряли на 450 нм.
Результаты
На фиг. 51 приведены результаты исследования анти-PVRIG антител в отношении их способности блокировать по меньшей мере 80% связывания PVRL2 с PVRIG. Как показано, большое количество таких антител были способны успешно блокировать по меньшей мере 80% связывания. В частности, антитела, которые демонстрировали успешное блокирование, обозначены следующим образом:
CPA.7.001, CPA.7.003, CPA.7.004, CPA.7.006, CPA.7.008, CPA.7.009, CPA.7.010, CPA.7.011, CPA.7.012, CPA.7.013, CPA.7.014, CPA.7.015, CPA.7.017, CPA.7.018, CPA.7.019, CPA.7.021, CPA.7.022, CPA.7.023, CPA.7.024, CPA.7.033, CPA.7.034, CPA.7.036, CPA.7.040, CPA.7.046, CPA.7.047, CPA.7.049, CPA.7.050,
Пример 11. Исследование методом поверхностного плазмонного резонанса эпитоп-специфической сортировки для 37 анти-PVRIG IgG антител, связывающихся со слитым белком человеческого PVRIG
Материалы и методы
Эксперименты проводили, используя прибор ProteOn XPR 36 при 22ºC, при этом все образцы хранили при 4ºC во время эксперимента.
Этап 1: Каждое из следующих анти-PVRIG mAb разводили до ~10 мкг/мл в 10 мМ ацетате натрия, pH 5,0, и ковалентно иммобилизовали отдельными пятнами на биосенсорном чипе ProteOn GLC, используя процедуру стандартного аминного сопряжения:
CPA.7.002 CPA.7.017 CPA.7.033
CPA.7.003 CPA.7.018 CPA.7.034
CPA.7.004 CPA.7.019 CPA.7.036
CPA.7.005 CPA.7.020 CPA.7.037
CPA.7.007 CPA.7.021 CPA.7.038
CPA.7.009 CPA.7.022 CPA.7.039
CPA.7.010 CPA.7.023 CPA.7.040
CPA.7.011 CPA.7.024 CPA.7.043
CPA.7.012 CPA.7.026 CPA.7.045
CPA.7.014 CPA.7.028 CPA.7.046
CPA.7.015 CPA.7.029 CPA.7.047
CPA.7.016 CPA.7.032 CPA.7.050
Этап активации происходил в направлении горизонтального потока в течение пяти минут, тогда как этап иммобилизации происходил в направлении вертикального потока. MAb инъецировали в течение четырех минут после активации поверхности. Этап блокирования происходил как в вертикальном, так и в горизонтальном положениях, по пять минут в каждом, так, что горизонтальные «промежутки» можно было использовать в качестве эталонных поверхностей. MAb иммобилизовали в диапазоне ~450 ЕО-5000 ЕО. Также в этом исследовании сортировке подвергали дополнительное mAb CPA.7.041, но только в качестве аналита в растворе. Смотрите ниже.
Этап 2: Предварительные эксперименты включали несколько циклов инъецирования ~20 нМ антигена PVRIG (PVRIGHH-2-1-1 #448, GenScript) через все иммобилизованные mAb в течение трех минут при скорости потока 25 мкл/мин с последующим восстановлением 30-секундным впрыскиванием 10 мМ глицина-HCl с pH 2,0 или pH 2,5 в зависимости от горизонтального ряда mAb в матричном чипе GLC. Образцы антигена готовили в дегазированном подвижном буфере ФСБТ (ФСБ с 0,05% Твин 20) с 100 мкг/мл БСА. Эти предварительные эксперименты показали, что клоны CPA.7.026 и CPA.7.029 не связывают антиген и, следовательно, были исключены из сортировки. Оставшиеся mAb на матрице ProteOn демонстрировали воспроизводимое связывание с антигеном.
Этап 3: Осуществляли протокол эпитоп-специфической сортировки с «предварительным смешиванием» из-за бивалентности fc-слитого антигена PVRIG. В этом протоколе каждое mAb, приведенное на Этапе 1, плюс mAb CPA.7.041, предварительно смешивали с антигеном PVRIG, а затем инъецировали в течение трех минут через все иммобилизованные mAb. Молярная концентрация связывающих участков каждого mAb превышала молярную концентрацию связывающих участков антигена. Конечная концентрация связывающих участков каждого mAb составила ~400 нМ, а конечная концентрация связывающих участков антигена составила ~20 нМ. После каждого восьмого цикла инъецирования mAb проводили контрольный цикл с одним антигеном, чтобы отследить активность иммобилизованных mAb в течение эксперимента. Также приблизительно после каждого восьмого цикла инъецирования mAb проводили инъекцию пустого буфера для двойного контроля. Дополнительные контроли включали отдельное инъецирование каждого mAb через все иммобилизованные mAb в концентрациях, идентичных с циклами инъецирования с предварительным смешиванием. Все поверхности восстанавливали 30-секундным впрыскиванием 10 мМ глицина-HCl с pH 2,0 или pH 2,5 в зависимости от восстанавливаемого ряда mAb в матрице при скорости потока 25 мкл/мин. Образцы mAb и антигена готовили в дегазированном подвижном буфере ФСБТ с 100 мкг/мл БСА.
Этап 4: Данные сенсограмм обрабатывали и проводили соотнесение, используя ProteOn Manager версию 3.1.0.6 с применением промежутков и пустого буфера для двойного контроля. Контрольные инъекции одного mAb использовали в качестве эталонных инъекций, при этом для инъекций одного mAb наблюдалось существенное связывание. Пару антител определяли, как имеющую общий антигенсвязывающий эпитоп (обозначение красным «0» в матрице на фиг. 42), если не наблюдалось связывание после инъекции смеси mAb и антигена через иммобилизованное mAb или если связывание было значительно снижено по сравнению с контрольной инъекцией одного антигена через то же иммобилизованное mAb. Пару антител определяли, как связывающуюся с разными эпитопами антигена или заключающую антигена в «сандвич-структуру» (обозначение зеленой «1» в матрице на фиг. 42), если инъекция смеси mAb и антигена демонстрировала связывание с иммобилизованным mAb, сходное или превышающее контроль с одним антигеном через то же иммобилизованное mAb.
Этап 5: Профиль блокирования для mAb CPA.7.041 (#37) исследовали только как для аналита, так как матричный чип GLC имеет только 36 мест. Поэтому для согласованности проводили иерархическую группировку профилей связывания бинарной матрицы для каждого mAb, предварительно смешанного с антигеном (вертикальные профили на фиг. 41), используя программное обеспечение JMP, версия 11.0.0. Профили блокирования иммобилизованных mAb (горизонтальные профили на фиг. 41) также группировали в качестве сравнения с профилями блокирования mAb, предварительно смешанных в растворе (данные не показаны, смотрите Результаты для обсуждения).
Результаты: фиг. 41 изображает бинарную матрицу блокирования («0») или заключения в сандвич-структуру («1») между каждой парой mAb, при этом mAb приведены в одном и том же порядке в вертикальном (mAb на поверхности - «лиганды») и горизонтальном (mAb в растворе - «аналиты») направлениях. Идентичные «группы» профилей блокирования для всех mAb в виде аналитов выделены на Фиг. 41 черным прямоугольником вокруг каждой группы одинаковых вертикальных профилей. На фиг. 42 приведена древовидная схема вертикальных (аналитических) профилей блокирования в матрице на фиг. 41. Для точного определения эпитопной «группы», в которой технически собраны только те mAb, которые демонстрируют идентичные профили блокирования, получено всего 4 дискретные группы. В частности, 33 из 35 mAb, которые проходили сортировку, составляют две группы, причем единственной разницей между этими двумя группами является то, заключают ли mAb в сандвич-структуру (группа 2, смотрите фиг. 41 и фиг. 42) или блокируют (группа 1, смотрите фиг. 41 и фиг. 42) связывание с CPA.7.039. Это означает, что CPA.7.039 находится в своей отдельной группе. Четвертая группа состоит только из mAb CPA.7.050, которое неспособно блокировать связывание антигена с любым из остальных 34 mAb. Иерархическая группировка профилей связывания mAb в виде лигандов (горизонтальные профили на фиг. 41) показала, что mAb CPA.7.016 заключает антиген в сандвич-структуру с mAb CPA.7.039, тогда как в виде аналита оно блокирует связывание антигена с CPA.7.039. Следовательно, клон CPA.7.016 следует поместить в группу 2, а не в группу 1. mAb из каждой группы приведены на фиг. 42. Обработанные данные сенсограмм для каждой группы приведены на фиг. 43 - фиг. 46.
Заключение: Методом ППР провели сортировку 35 анти-PVRIG IgG mAb в соответствии с их попарными профилями блокирования с fc-слитым человеческим PVRIG. В ходе точного определения эпитопной группы получили всего четыре дискретные группы. 33 из 35 mAb составляют две группы, которые отличаются только тем, что соответствующее mAb блокирует или заключает антиген в сандвич-структуру с клоном CPA.7.039.
Пример 12. Исследование кинетики 50 анти-PVRIG человеческих FAB, полученных в периплазматических экстрактах, методом поверхностного плазмонного резонанса
Материалы и методы
Все эксперименты проводили, используя прибор Biacore 3000 и прибор ProteOn XPR 36, при 22°C.
Этап 1: Количественную оценку молярной концентрации всех 52 fab в супернатанте периплазматического экстракта проводили с помощью прибора Biacore 3000 при 22ºC. Каждый fab 20-кратно разводили, а затем в течение 2 минут инъецировали при 5 мкл/мин через поверхности с высокой плотностью антител против человеческого fab (GE Healthcare 28-9583-25), полученные путем стандартного аминного сопряжения с чипом CM5 Biacore (GE Healthcare). Затем инъецировали стандартный человеческий fab (Bethyl P80-115) в известной концентрации через анти-fab поверхности в тех же условиях, что и супернатанты fab. Образцы готовили в подвижном буфере, который представлял собой дегазированный HBSP (0,01 M ГЭПЭС, 0,15 M NaCl, 0,005% P20, pH 7,4) с добавлением 0,01% БСА. Наклон кривой ассоциации каждой ППР-сенсограммы для каждого супернатанта fab аппроксимировали относительно наклона кривой ассоциации стандартного человеческого fab с известной концентрацией, используя программное обеспечение CLAMP 3.40 для оценки молярных концентраций каждого fab в супернатанте.
Этап 2: Поверхность с высокой плотностью козьего поликлонального антитела против человеческого fc (Invitrogen H10500) готовили с помощью стандартного аминного сопряжения на двух дорожках чипа GLC, используя биосенсор ProteOn XPR 36. Поверхность с высокой плотностью поликлонального антитела против мышиного fc (GE Healthcare BR-1008-38) готовили с помощью стандартного аминного сопряжения на двух разных дорожках одного чипа GLC. Этапы активации и блокирования для всех четырех захватывающих поверхностей происходили в направлении вертикального потока. Каждый fab в супернатанте затем инъецировали в трех концентрациях через fc-слитый человеческий PVRIG (PVRIG-HH-2-1-1 #448, GenScript) и fc-слитый мышиный PVRIG (PVRIG-MM-2-1-1 #198, GenScript), которые были иммобилизованы на одной поверхности с высокой плотностью антител против человеческого fc и одной поверхности с антителами против мышиного fc (соответственно) в среднем при ~200 ЕО и ~290 ЕО на цикл соответственно. Каждую концентрационную серию fab инъецировали в течение двух минут с последующей 10-минутной диссоциацией со скоростью потока 50 мкл/мин. Начальный концентрационный диапазон (определенный на этапе 1) составил ~20 нМ-~400 нМ с двумя трехкратными разведениями наибольшей концентрации для каждого fab. Fab разводили в подвижном буфере, который представлял собой дегазированный ФСБ с добавлением 0,05% Твин 20 и 0,01% БСА. Захватывающие поверхности из антител против человеческого fc восстанавливали 30-секундными впрыскиваниями 146 мМ фосфорной кислоты после каждого цикла, а поверхности из антител против мышиного fc восстанавливали 30-секундными впрыскиваниями 10 мМ глицина, pH 1,7, после каждого цикла.
Этап 3: Данные сенсограмм по связыванию fab в супернатанте с захваченным PVRIG обрабатывали и подвергали двойному контролю с помощью ProteOn Manager, версия 3.1.0.6. Двойной контроль сенсограмм проводили, используя соответствующие захватывающие поверхности против данных молекул без иммобилизованного PVRIG в качестве контрольных поверхностей и пустую инъекцию через иммобилизованный PVRIG в условиях, идентичных с инъекциями fab. При возможности сенсограммы для трех разных концентраций каждого fab затем подвергали глобальной аппроксимации 1:1 кинетической моделью (с термином для переноса массы) для оценки констант скоростей ассоциации и диссоциации. Сенсограммы, которые не демонстрировали простое 1:1 связывание, не аппроксимировались кинетической моделью и, следовательно, для них не были оценены kасс. и kдисс..
Результаты
Ни один из включенных в это исследование fab не продемонстрировал активность в отношении связывания с мышиным PVRIG (данные не показаны). Сенсограммы для 17 из 50 fab, исследуемых против человеческого PVRIG, можно было аппроксимировать для достоверного определения их констант скорости. Двадцать восемь клонов продемонстрировали сложную кинетику, пять fab не продемонстрировали никакого связывания с иммобилизованным человеческим слитым белком PVRIG (CPA.7.025, CPA.7.026, CPA.7.027, CPA.7.029, CPA.7.035), а один клон (CPA.7.035) продемонстрировал отсутствие титра при определении концентрации на этапе 1. Константы скорости и их соответствующие сенсограммы приведены ниже на фиг. 48 и на фиг. 49. Перечисленные ниже клоны демонстрировали сложную кинетику. На фиг. 50 приведены некоторые примеры этих данных.
CPA.7.001 CPA.7.006 CPA.7.013 CPA.7.045
CPA.7.030 CPA.7.036 CPA.7.014 CPA.7.046
CPA.7.031 CPA.7.037 CPA.7.041 CPA.7.017
CPA.7.032 CPA.7.009 CPA.7.042 CPA.7.018
CPA.7.033 CPA.7.038 CPA.7.043 CPA.7.047
CPA.7.034 CPA.7.039 CPA.7.016 CPA.7.023
CPA.7.003 CPA.7.011 CPA.7.044 CPA.7.024
Пример 13. Определение аффинности связывания клона IGG CPA.7.021 С PVRIG, экспрессируемым в клетках HEK, методом проточной цитометрии
Материалы и методы
Метод проточной цитометрии использовали для определения аффинности связывания CPA.7.021 IgG с человеческим PVRIG, экспрессируемым в клетках HEK 293. CPA.7.021, конъюгированное с Alexa 647, добавляли в двух повторностях при концентрации связывающих участков в диапазоне 3 пM-101 нМ в 2-кратном серийном разведении до постоянного числа клеток (100000 клеток/лунку) в 17 лунок в 96-луночном планшете. Одна лунка содержала клетки без добавления IgG, чтобы служить контрольной чистой лункой. Клетки уравновешивали в течение 4 часов IgG при 4ºC. Клетки дважды промывали, а затем записывали среднюю интенсивность флуоресценции (СИФ) приблизительно для 10000 «событий», используя проточный цитометр Intellicyte. Ниже приведены полученные в результате значения СИФ в виде функции концентрации связывающих участков CPA.7.021 IgG. KД связывания CPA.7.021 с клетками HEK 293, экспрессирующими человеческий PVRIG, оценивали путем аппроксимации кривой зависимости СИФ от концентрации связывающих участков IgG с помощью 1:1 равновесной модели, как подробно описано в Drake and Klakamp, Journal of Immunol Methods, 318 (2007) 147-152.
Результаты: Меченое Alexa647 CPA.7.021 IgG титровали с клетками HEK 293, экспрессирующими человеческий PVRIG, а сигнал связывания измеряли методом проточной цитометрии. Ниже представлена полученная в результате изотерма связывания, показывающая, в двух повторностях, зависимость СИФ от концентрации связывающих участков CPA.7.021. Красная линия представляет 1:1 равновесную аппроксимацию кривой, которая позволила получить оценку KД в 2,5 нМ ± 0,5 нМ (95% доверительный интервал аппроксимации, N = 1).
Пример 14. Влияние нокдауна (НД) PVRIG и анти-PVRIG антитела на функцию специфических к человеческой меланоме ОИЛ
Целью этого анализа была оценка функциональной способности PVRIG в человеческих ОИЛ по активационным маркерам и секреции цитокинов после совместного культивирования с клетками-мишенями меланомы. PD1 использовали в качестве референтной иммунной контрольной точки для исследования нокдауна (миРНК). Оценивали влияние анти-PVRIG антитела (CPA.7.21), которое блокирует взаимодействие PVRIG и PVRL2, одно или в комбинации с другими антителами (например, TIGIT, DNAM1).
Материалы и методы
ОИЛ
Использовали опухоль-инфильтрирующие лимфоциты (ОИЛ) от трех пациентов с меланомой:
- (ОИЛ) TIL-412 HLA-A2-Mart1-специфический
- (ОИЛ) TIL-F4 HLA-A2-gp100-специфический
- (ОИЛ) TIL-209 HLA-A2-gp100-специфический
ОИЛ размораживали в полной среде IMDM (BI, 01-058-1A), дополненной 10% человеческой сывороткой (Sigma, H3667) + 1% глутамаксом (Life technologies, 35050-038) + 1% Na-пируватом (Biological Industries, 03-042-1B) + 1% заменимыми аминокислотами (Biological Industries, 01-340-1B) + 1% пен.-стрепт. (Biological Industries, 03-031-1B) + 300 Е/мл rhIL2 (Biolegend, 509129).
Опухолевые клеточные линии: Клетки человеческой меланомы Mel-624 экспрессируют антигены MART-1 и gp-100 в контексте гаплотипа ГКГС-I HLA-A2. Клетки культивировали в полной среде DMEM (Biological Industries, 01-055-1A), дополненной 10% ФБС (BI, 04-127-1A), 25 мМ буфера ГЭПЭС (BI, 03-025-1B), 1% глутамаксом (Life technologies, 35050-038) и 1% пен.-стрепт. (Biological Industries, 03-031-1B).
Нокдаун в ОИЛ: Нокдаун (НД) человеческого PVRIG и человеческого PD1 в ОИЛ проводили, используя 100 пмолей SMARTpool миРНК человеческого PVRIG (L-032703-02) или SMARTpool миРНК человеческого PD1 (L-004435) Dharmacon ON-TARGETplus или ненацеленную миРНК (D-001810-01-05). миРНК электропорировали в ОИЛ (AMAXA, программа X-005). Электропорацию проводили с покоящимися ОИЛ, культивируемыми в полной среде IMDM, дополненной IL-2, через 24 ч после размораживания. После электропорации ОИЛ высевали в 96-луночный планшет ТС для восстановления в течение 24 ч. Через 24 ч клетки собирали и окрашивали красителем в отношении жизнеспособности (BD Horizon; Кат. # 562247, BD biosciences), промывали ФСБ и окрашивали антителом против человеческого PVRIG CPA.7.021 (CPA.7.021 IgG2 A647, 7,5 мкг/мл) или антителом против человеческого PD-1 (Biolegend, #329910 AF647, 5 мкг/мл) при комнатной температуре в течение 30 мин. Применяемыми изотипическими контролями были синагис (IgG2 A647, 7,5 мкг/мл) и мышиный IgG1 (Biolegend #400130 A647, 5 мкг/мл) соответственно. Все образцы исследовали на анализаторе MACSQuant (Miltenyi), а данные анализировали с помощью программного обеспечения FlowJo (v10.0.8).
Совместное культивирование ОИЛ с клетками меланомы 624: ОИЛ с электропорированной миРНК собирали и высевали в 96-луночный планшет ТС при 5x104/лунку. Клетки Mel-624 также собирали и высевали при соотношении Э:М 1:1/1:3 в совместной культуре. Планшет инкубировали в течение ночи (18 ч) при 37°C, 5% CO2.
Чтобы оценить действие анти-PVRIG антитела (CPA.7.021), анти-TIGIT (клон 10A7) и анти-DNAM1 (клон DX11) на специфическую в отношении меланомы активность ОИЛ, ОИЛ (1x105 клеток/лунку) предварительно инкубировали с исследуемыми антителами или релевантными изотипическими контролями в виде моно-обработки (10 мкг/мл) или комбинированной обработки (конечная концентрация 10 мкг/мл для каждого) перед добавлением клеток-мишеней меланомы 624 при соотношении эффекторные клетки:клетки-мишени 1:1. Планшет инкубировали в течение ночи (18 ч) при 37°C, 5% CO2.
Оценка активации ОИЛ: через 16 часов после совместного культивирования клетки окрашивали красителем в отношении жизнеспособности (BD Horizon; Кат. # 562247, BD biosciences), промывали ФСБ и подвергали действию Fc-блокирующего раствора (кат. # 309804, Biolegend) с последующим поверхностным окрашиванием анти-CD8a (Кат. #301048, Biolegend) и анти-CD137 (Кат. #309804, Biolegend) при 4°C в течение 30 мин. Все образцы исследовали на анализаторе MACSQuant (Miltenyi), а данные анализировали с помощью программного обеспечения FlowJo (v10.0.8). Собирали культуральные супернатанты и анализировали в отношении секреции цитокинов с помощью набора CBA (Кат. #560484, BD).
Результаты
Нокдаун PVRIG в ОИЛ: ОИЛ MART-1 и TIL F4 культивировали в течение 24 ч с IL-2. 100 пмолей SMARTpool миРНК человеческого PVRIG (L-032703-02) или SMARTpool миРНК человеческого PD1 (L-004435) ON-TARGETplus или ненацеленную миРНК (D-001810-01-05) электропорировали в ОИЛ (AMAXA, программа X-005). Выявление PVRIG или PD-1 проводили через 24 ч после электропорации (и перед совместным культивированием). Клетки окрашивали красителем в отношении жизнеспособности с последующей 30-минутной инкубацией при КТ с анти-PVRIG или анти-PD-1. Процентное содержание НД-популяции приведено на фиг. 81.
Функциональный анализ с применением ОИЛ с нокдауном: Человеческие ОИЛ, культивируемые в течение 24 ч с IL2, электропорировали миРНК, кодирующей человеческий PVRIG или PD-1, или рандомизированной последовательностью в качестве контроля. ОИЛ исследовали в отношении экспрессии PVRIG и PD-1 через 24 ч после электропорации. Наблюдали ~80% нокдаун PVRIG и ~50% нокдаун PD-1 по сравнению с электропорированными рандомизированной последовательностью ОИЛ (на фиг. 81).
ОИЛ с НД культивировали с клетками Mel-624 при соотношении Э:М 1:1 или 1:3 в течение 18 ч и окрашивали в отношении экспрессии CD137. Повышенные уровни маркера активации CD137 наблюдались в ОИЛ MART-1, электропорированных миРНК PVRIG, аналогично с ОИЛ, которые были электропорированы миРНК PD-1, по сравнению с контрольной рандомизированной миРНК (на фиг. 82А). Собирали супернатант совместной культуры и исследовали в отношении наличия секретируемых цитокинов. ОИЛ, которые электропорировали миРНК PVRIG, демонстрировали значительное повышение уровней IFNγ и TNF по сравнению с контрольной рандомизированной миРНК. Аналогичное действие было продемонстрировано для ОИЛ, которые были электропорированы миРНК PD-1 (На фиг. 82B-C).
Такую же тенденцию к повышению уровней активации наблюдали в ОИЛ F4. Совместное культивирование электропорированных миРНК PVRIG ОИЛ F4 с Mel-624 при соотношении Э:М 1:3 привело к повышению уровней поверхностной экспрессии CD137 (на фиг. 83А), а также повышению секреции IFNγ в совместном культуральном супернатанте (на фиг. 83В). Аналогичные тенденции наблюдали в ОИЛ, электропорированных миРНК PD-1.
Функциональный анализ с блокирующими Ab:
In vitro монотерапия и комбинированная терапия анти-PVRIG и анти-TIGIT: ОИЛ 209 культивировали с клетками Mel-624 при соотношении Э:М 1:1 в течение 18 ч. Собирали супернатант совместной культуры и исследовали в отношении наличия секретируемых цитокинов. Обработка анти-TIGIT не влияла на уровни секреции IFNγ или TNF. При этом наблюдали повышение уровней IFNγ и TNF, когда анти-TIGIT и анти-PVRIG добавляли в совместную культуру в комбинации (фиг. 84А-В).
In vitro монотерапия и комбинированная терапия анти-PVRIG и анти-TIGIT: ОИЛ 209 культивировали с клетками Mel-624 при соотношении Э:М 1:1 в течение 18 ч. ОИЛ были окрашены в отношении поверхностной экспрессии маркера активации CD137 и демонстрировали сниженный уровень экспрессии после обработки анти-DNAM-1. Собирали супернатант совместной культуры и исследовали в отношении наличия секретируемых цитокинов. Обработка анти-DNAM-1 опосредовала тенденцию к возрастанию секреции цитокинов IFNγ и TNF. Обработка анти-DNAM-1 и анти-PVRIG в комбинации частично обращала действие на экспрессию CD137 (фиг. 85С) и усиливала действие на секрецию цитокинов IFNγ и TNF (фиг. 5A-B). ОИЛ MART-1 культивировали с клетками Mel-624 при соотношении Э:М 1:1 в течение 18 ч. Собирали супернатант совместной культуры и исследовали в отношении наличия секретируемых цитокинов. Обработка анти-DNAM-1 снижала поверхностную экспрессию CD137 на ОИЛ, а также секрецию цитокинов IFNγ и TNF. Обработка анти-DNAM-1 и анти-PVRIG в комбинации частично обращала эти действия (фиг. 85D-F).
Выводы и заключения
НД PD1 улучшал активность ОИЛ согласно измерениям IFNγ и секреции в ОИЛ F4 и MART-1. Повышение секреции (~20%) IFNγ и TNF наблюдали после НД PVRIG в ОИЛ MART-1 по сравнению с контрольной миРНК. Такую же тенденцию наблюдали для экспрессии CD137 после совместного культивирования с клетками меланомы 624 для ОИЛ F4.
Обработка анти-TIGIT не влияла на уровни секреции IFNγ или TNF из ОИЛ, культивируемых с 624 Mel, однако повышение уровней IFNγ и TNF наблюдали, когда анти-TIGIT и анти-PVRIG (CPA.7.021) добавляли в совместную культуру в комбинации.
Обработка анти-TIGIT снижала активацию ОИЛ MART-1, что проявлялось в снижении экспрессии CD137 и цитокинов, а анти-PVRIG (CPA.7.21) частично могло обращать это действие в случае комбинированной обработки DNAM-1 Ab. В ОИЛ 209 уровни секреции IFNγ и TNF были немного повышены (~10%) при обработке анти-DNAM-1, также повышение уровней IFNγ и TNF (~40% и 30% соответственно) наблюдали, когда анти-DNAM1 и анти-PVRIG (CPA.7.021) добавляли в совместную культуру в комбинации. Вместе наши результаты позволяют предположить, что PVRIG представляет собой новый ко-ингибиторный рецептор для PVRL2.
Пример 15. Действие анти-PVRIG на функцию специфических к человеческой меланоме ОИЛ в комбинации с анти-TIGIT И анти-PD1 антителами
Материалы и методы
ОИЛ: Использовали опухоль-инфильтрирующие лимфоциты (ОИЛ) от трех пациентов с меланомой:
- (ОИЛ) TIL-412 HLA-A2-Mart1-специфический
- (ОИЛ) TIL-F4 HLA-A2-gp100-специфический
- (ОИЛ) TIL-209 HLA-A2-gp100-специфический
ОИЛ размораживали в полной среде IMDM (BI, 01-058-1A), дополненной 10% человеческой сывороткой (Sigma, H3667) + 1% глутамаксом (Life technologies, 35050-038) + 1% Na-пируватом (Biological Industries, 03-042-1B) + 1% заменимыми аминокислотами (Biological Industries, 01-340-1B) + 1% пен.-стрепт. (Biological Industries, 03-031-1B) + 300 Е/мл rhIL2 (Biolegend, 509129).
Опухолевые клеточные линии: Клетки человеческой меланомы Mel-624 экспрессируют антигены MART-1 и gp-100 в контексте гаплотипа ГКГС-I HLA-A2. Клетки культивировали в полной среде DMEM (Biological Industries, 01-055-1A), дополненной 10% ФБС (BI, 04-127-1A), 25 мМ буфера ГЭПЭС (BI, 03-025-1B), 1% глутамаксом (Life technologies, 35050-038) и 1% пен.-стрепт. (Biological Industries, 03-031-1B).
Совместное культивирование ОИЛ с клетками меланомы 624 в присутствии анти-PVRIG, анти-TIGIT и PD1 блокирующих антител: Чтобы оценить действие анти-PVRIG антитела (CPA.7.021), анти-TIGIT (клон 10A7) и анти-PD1 (mAb 1B8, Merck) на специфическую в отношении меланомы активность ОИЛ, ОИЛ (3x104 клеток/лунку) предварительно инкубировали с исследуемыми антителами или релевантными изотипическими контролями в виде моно-обработки (10 мкг/мл) или комбинированной обработки (конечная концентрация 10 мкг/мл для каждого) перед добавлением клеток-мишеней меланомы 624 при соотношении эффекторные клетки:клетки-мишени 1:3. Планшет инкубировали в течение ночи (18 ч) при 37°C, 5% CO2.
Оценка активации ОИЛ: Собирали культуральные супернатанты и анализировали в отношении секреции цитокинов с помощью набора CBA (Кат. #560484, BD).
In vitro монотерапия анти-PVRIG и комбинированная терапия с анти-TIGIT и PD1 блокирующими антителами: ОИЛ F4 (специфические к gp100) культивировали с клетками Mel-624 при соотношении Э:М 1:3 в течение 18 ч. Собирали супернатант совместной культуры и исследовали в отношении наличия секретируемых цитокинов. Обработка анти-TIGIT или анти-PD1 не влияла на уровни секреции IFNγ или TNF. При этом наблюдали повышение уровней IFNγ и TNF, когда анти-TIGIT или анти-PD1 добавляли в совместную культуру в комбинации с анти-PVRIG (на фиг. 86А-В).
Обработка одними анти-PVRIG, анти-TIGIT и PD1 не влияла на уровни секреции IFNγ или TNF из совместной культуры ОИЛ и 624 Mel, однако наблюдали повышение уровней IFNγ и TNF, когда анти-TIGIT или анти-PD1 антитела добавляли в комбинации с анти-PVRIG (CPA.7.021). Представленные данные позволяют предположить наличие синергетического действия в случае комбинированной терапии анти-TIGIT или анти-PD1 антителами.
Пример 16. Изучение действия анти-PVRIG антител на сигнализацию РТК с помощью анализа репортерного гена
Использовали систему для анализа репортерного гена для РТК-сигнализации, такую как клеточная линия Jurkat-NFAT-Luc, чтобы исследовать действие анти-PVRIG антител на опосредованную РТК сигнализацию. Это производное клеточной линии Jurkat экспрессирует репортерный ген люциферазы под управлением элемента ответа NFAT. Эти клетки трансфицированы вектором, кодирующим полноразмерный человеческий PVRIG. В качестве отрицательного контроля использовали клетки, трансфицированные пустым вектором. Трансфектанты с векторами, кодирующими ко-стимуляторные или ко-ингибиторные эталонные молекулы, такие как CD28 и PD-1, служили в качестве положительного контроля. Трансфектанты стимулировали добавлением антитела против человеческого CD3 (например, OKT3) в отсутствие или присутствии анти-PVRIG антител. Изотипический контроль служил в качестве отрицательного контроля. Известные функциональные антитела против эталонных молекул служили в качестве положительных контролей. Ожидается, что функциональное агонистическое перекрестно связывающееся антитело будет демонстрировать ингибиторное действие на активность люциферазы.
Пример 17. Изучение действия анти-PVRIG антител на активацию Т-клеток с помощью PVRL2-FC
Чтобы исследовать действие анти-PVRIG антител на активацию, пролиферацию Т-клеток и секрецию цитокинов, используют анализ со связыванием с планшетом. Общее количество очищенных Т-клеток стимулируют, используя 1 мкг/мл связанного с планшетом антитела против человеческого CD3 (например, OKT3) и 5 мкг/мл PVRL2-Fc (рекомбинантный слитый белок, состоящий из ВКД PVRL2, контрпартнера PVRIG) или отрицательного контроля. Активацию Т-клеток оценивают по экспрессии маркеров активации, например, CD137, или по клеточному делению, оцениваемому путем растворения красителя CFSE (перед стимуляцией Т-клетки метят CFSE). Также оценивают выработку цитокинов (например, IFNg, IL-2) как дополнительный показатель активации Т-клеток. Маркеры Т-клеточного подтипа используют, чтобы различать специфическое действие на Т-клетки CD4 или CD8. Совместно иммобилизованный PVRL2-Fc может оказывать базовое стимуляторное действие на активацию Т-клеток, опосредованную эндогенным DNAM1 - известным ко-стимуляторным рецептором контрпартнера PVRL2 на Т-клетках. В присутствии антагонистических анти-PVRIG Ab ожидается, что это стимуляторное базовое действие PVRL2-Fc будет дополнительно усилено благодаря блокированию ими ингибиторного влияния эндогенного PVRIG на активацию Т-клеток. Соответственно, ожидается, что агонистические анти-PVRIG Ab будут демонстрировать ингибирование активации Т-клеток.
Пример 18. Изучение действия анти-PVRIG антител на активацию Т-клеток с помощью эктопически экспрессирующих PVRL2 клеток
Чтобы исследовать действие анти-PVRIG антител на активацию, пролиферацию Т-клеток и секрецию цитокинов, используют клеточный анализ. Общее количество очищенных человеческих Т-клеток CD4 или CD8 стимулируют после совместного культивирования со стимуляторными клетками СНО (клетками СНО, экспрессирующими мембраносвязанное анти-CD3), эктопически экспрессирующими PVRL2 или пустой вектор. Активацию Т-клеток оценивают по экспрессии маркеров активации, например, CD137, или по клеточному делению, оцениваемому путем растворения красителя CFSE (перед стимуляцией Т-клетки метят CFSE). Также оценивают выработку цитокинов (например, IFNg, IL-2) как дополнительный показатель активации Т-клеток. Маркеры Т-клеточного подтипа используют, чтобы различать специфическое действие на Т-клетки CD4 или CD8. Ожидается, что экспрессирующие PVRL2 СНО-стимуляторы, как и в Примере 19, будут оказывать базовое стимуляторное действие на активацию Т-клеток, опосредованную эндогенным DNAM1 - известным ко-стимуляторным рецептором контрпартнера PVRL2 на Т-клетках. В присутствии антагонистических анти-PVRIG Ab ожидается, что это стимуляторное базовое действие поверхностно экспрессируемого PVRL2 будет дополнительно усилено благодаря блокированию ими ингибиторного влияния эндогенного PVRIG на активацию Т-клеток. Соответственно, ожидается, что агонистические анти-PVRIG Ab будут демонстрировать ингибирование активации Т-клеток.
Пример 19. Изучение действия анти-PVRIG антител на активацию Т-клеток с помощью анализа СЭВ
Анти-PVRIG антитела исследуют в отношении их действия на активность Т-клеток, используя клетки крови от здоровых добровольцев и суперантиген СЭВ (стафилококковый энтеротоксин B), чтобы задействовать и активировать все Т-клетки, экспрессирующие Vβ3 и Vβ8 цепь рецептора Т-клеток. Человеческие МКПК культивируют в 96-луночных круглодонных планшетах и предварительно инкубируют в течение 30-60 мин с исследуемыми антителами. Затем добавляют СЭВ в разных концентрациях в диапазоне от 10 нг/мл до 10 мкг/мл. Супернатанты собирают через 2-4 дня культивирования, а количество вырабатываемых цитокинов (например, IL-2, IFNγ) оценивают методом ELISA или используют стандартный набор CBA. СЭВ стимулирует выработку цитокинов цельными клетками крови дозозависимым образом. Действие анти-PVRIG mAb на выработку цитокинов исследуют для нескольких доз Ab. Ожидается, что блокирующие анти-PVRIG mAb будут усиливать выработку IL-2 по сравнению с контрольным IgG. Кроме IL-2, в этом анализе можно исследовать действие Ab на уровни других цитокинов, таких как TNFα, IL-17, IL-7, IL-6 и IFNγ, используя набор CBA.
Пример 20. Изучение действия анти-PVRIG антител в Ag-специфическом анализе
Анализ, который используют для профилирования функционального действия антител к человеческому PVRIG на Ag-специфическую стимуляцию предсуществующих Т-клеток памяти в крови здорового донора, представляет собой анализ со столбнячным анатоксином (СА). С этой целью свежеприготовленные МКПК (2 x 105 клеток) высевают в 96-луночные круглодонные планшеты в полную среду RPMI 1640 (содержащую 5% термоинактивированную человеческую сыворотку), предварительно инкубируют с исследуемыми антителами в разной концентрации и стимулируют СА (Astarte Biologics) в концентрации 100 нг/мл. Клетки инкубируют в течение 3-7 дней при 37°C, после чего собирают супернатанты. Концентрацию цитокинов (например, IL-2, IFN-γ) определяют с помощью ELISA и/или набора CBA. Ожидается, что блокирующие анти-PVRIG Ab будут усиливать пролиферацию Т-клеток и выработку цитокинов по сравнению с одним антигеном СА.
Аналогично способу, описанному выше, в котором используется СА для стимуляции человеческих Т-клеток памяти, можно исследовать действие анти-PVRIG Ab на активацию Т-клеток после ответов на другие антигены, такие как антигены ЦМВ, ВЭБ, гриппа, ВИЧ, свинки и туберкулеза, используя аналогичные описанным выше экспериментальные условия. Также их можно использовать для исследования действия анти-PVRIG антител на стимуляцию наивных клеток, используя нео-антигены, такие как ГЛУ.
Кроме того, действие анти-PVRIG Ab исследуют на антигенспецифических ответах тетрамер-сортированных Ag-специфических Т-клеток CD8 из периферической крови пациентов, страдающих от вирусных инфекций, таких как ВГС и ВИЧ. Тетрамер-сортированные Т-клетки CD8 культивируют совместно с нагруженными пептидами аутологичными МКПК в течение 5 дней. Оценивают пролиферацию Ag-специфических Т-клеток CD8 и секрецию цитокинов (например, IFNγ, IL2, TNF-α). Мы ожидаем, что анти-PVRIG антитела будут усиливать пролиферацию и выработку цитокинов по сравнению с одним антигеном.
Пример 21. Связывание и функциональный анализ полученных из гибридомы антител против PVRIG
В этом примере проиллюстрировано определение характеристик связывания полученных из гибридомы антител (СНА-антител) с белком PVRIG человека и яванского макака в клеточных линиях и первичных лейкоцитах, а также определение характеристик способности полученных из гибридомы антител блокировать взаимодействие между PVRIG и PVRL2.
Протоколы
Анализ FACS сверхэкспрессирующих hPVRIG клеток: Следующие клеточные линии использовали для оценки специфичности антител к человеческому PVRIG: родительские клетки HEK и сверхэкспрессирующие hPVRIG клетки HEK. Эти клетки культивировали в DMEM (Gibco) + 10% фетальная телячья сыворотка (Gibco) + глутамакс (Gibco). В случае сверхэкспрессирующих hPVRIG клеток HEK для положительной селекции в среду также добавляли 0,5 мкг/мл пуромицина (Gibco). Для проведения анализа FACS все клеточные линии собирали в log-фазе роста и высевали 50000-100000 клеток на лунку в 96-луночные планшеты. Антитела к человеческому PVRIG (mIgG1 или mIgG2a) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или в виде 8-точечных титрований, начиная с 10 мкг/мл, на льду в течение 30 мин-1 ч. Титрование проводили в виде 1:3 или 1:3,3 серийных разведений. Данные получали с помощью FACS Canto II (BD Biosciences) или IntelliCyt (IntelliCyt Corporation) и анализировали с помощью программного обеспечения FlowJo (Treestar) и Prism (Graphpad).
Анализ FACS клеточных линий человека для hPVRIG: Следующие клеточные линии использовали для оценки экспрессии и специфичности антител к человеческому PVRIG: Jurkat и HepG2. Клетки Jurkat культивировали в среде RPMI + 10% фетальная телячья сыворотка, глутамакс, заменимые аминокислоты (Gibco), пируват натрия (Gibco) и пенициллин/стрептомицин (Gibco). Клетки HepG2 культивировали в DMEM + 10% фетальная телячья сыворотка + глутамакс. Для проведения анализа FACS все клеточные линии собирали в log-фазе роста и высевали 50000-100000 клеток на лунку в 96-луночные планшеты. Антитела к человеческому PVRIG (mIgG1 или mIgG2a) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или в виде 8-точечных титрований, начиная с 10 мкг/мл, на льду в течение 30 мин-1 ч. Титрование проводили в виде 1:3 или 1:3,3 серийных разведений. Данные получали с помощью FACS Canto II или IntelliCyte и анализировали с помощью программного обеспечения FlowJo и Prism.
Анализ FACS наивных первичных лейкоцитов человека для hPVRIG: Первичные лейкоциты получали с помощью выделения в градиенте фиколла (GE Healthcare) периферической крови (Stanford Blood Bank). Лейкоциты в виде выделенных мононуклеарных клеток периферической крови (МКПК) замораживали в жидком азоте с плотностью от 1x107 до 5x107 клеток/мл в смеси 10% ДМСО (Sigma), 90% фетальной телячьей сыворотки. Чтобы оценить экспрессию белка PVRIG в МКПК, составляли коктейли антител против основных иммунных субпопуляций, которые содержали человеческие анти-PVRIG антитела. Антитела к человеческому PVRIG (mIgG1 или mIgG2a) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или, в некоторых случаях, в виде 4-точечных титрований, начиная с 10 мкг/мл, на льду в течение 30 мин-1 ч.
Вкратце, коктейльные смеси антител добавляли к восстановленным МКПК, которые были высеяны при 5x105-1x106 клеток/лунку после предварительной блокировки Fc-рецептора и окрашивания в отношении живых/мертвых клеток (Aqua Live/Dead, Life Technologies). Коктейли антител инкубировали с МКПК в течение 30 мин-1 ч на льду. Затем МКПК промывали и получали данные методом FACS с помощью FACS Canto II. Данные анализировали с помощью программного обеспечения FlowJo и Prism. Анализируемые иммунные субпопуляции включали NK-клетки CD56 dim, NK-клетки CD56 bright, Т-клетки CD4+, Т-клетки CD8+, нетрадиционные Т-клетки (например, NKТ-клетки и Т-клетки), В-клетки и моноциты.
Анализ FACS сконструированных сверхэкспрессирующих PVRIG клеток яванского макака: Следующие клеточные линии использовали для оценки перекрестной реактивности антител к человеческому PVRIG с PVRIG яванского макака (cPVRIG): родительские клетки expi и сверхэкспрессирующие cPVRIG клетки expi. Эти клетки культивировали в DMEM + 10% фетальная телячья сыворотка + глутамакс. Клетки expi, временно сверхэкспрессирующие cPVRIG, получали путем электропорации ДНК cPVRIG в родительские клетки expi с помощью системы для трансфекции Neon. Для анализа FACS использовали клетки expi cPVRIG через 1-3 дня после трансфекции. Родительские клетки expi собирали в log-фазе роста. 50000-100000 клеток каждого типа на лунку высевали в 96-луночные планшеты. Антитела к человеческому PVRIG (mIgG1 или mIgG2a) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или в виде 8-точечных титрований, начиная с 10 мкг/мл, на льду в течение 30 мин-1 ч. Титрование проводили в виде 1:3 или 1:3,3 серийных разведений. Данные получали с помощью FACS Canto II или IntelliCyte и анализировали с помощью программного обеспечения FlowJo и Prism.
Анализ FACS наивных первичных лейкоцитов яванского макака: Первичные лейкоциты яванского макака (cyno) получали из свежей крови, которую брали не более чем за 24 часа до анализа экспрессии. Источником крови служил Bioreclamation. Чтобы оценить экспрессию белка PVRIG в МКПК яванского макака, составляли коктейли антител против основных иммунных субпопуляций, которые содержали человеческие анти-PVRIG антитела. Антитела к человеческому PVRIG (mIgG1 или mIgG2a) и их соответствующие контроли добавляли как одноточечные разведения (5 мкг/мл) или в виде 8-точечных титрований, начиная с 10 мкг/мл, на льду в течение 30 мин-1 ч.
Вкратце, коктейльные смеси антител добавляли к МКПК, которые были высеяны при 5x105-1x106 клеток/лунку после предварительной блокировки Fc-рецептора и окрашивания в отношении живых/мертвых клеток. Коктейли антител инкубировали с МКПК в течение 30 мин-1 ч на льду. Затем МКПК промывали и получали данные методом FACS с помощью FACS Canto II. Данные анализировали с помощью программного обеспечения Prism. Анализируемые иммунные субпопуляции включали лимфоциты CD16+, моноциты/миелоидные клетки CD14+/CD56+ и Т-клетки CD3+.
Клеточный конкурентный анализ: Способность антител к PVRIG ингибировать взаимодействие PVRIG со своим лигандом PVRL2 оценивали в клеточном конкурентном анализе. В этом анализе лиганд PVRL2 эндогенно экспрессируется на необработанных клетках HEK и добавляется растворимый Fc-меченый PVRIG (произведенный по запросу Genscript). В этом случае способность антител к PVRIG блокировать связывание растворимого PVRIG с клетками HEK оценивали посредством одновременного добавления 33 нМ растворимого белка PVRIG и антител к PVRIG (0,066-66 нМ) к 100000 клеткам HEK и инкубации в течение 1 часа на льду. Степень связывания PVRIG Fc определяли путем добавления античеловеческого Fc Alexa 647 (Jackson Laboratories) в течение 20-30 минут на льду. Клетки дважды промывали ФСБ, используя для получения данных FACS Canto II. Данные анализировали с помощью FlowJo (Treestar), Excel (Microsoft) и Prism (GraphPad).
Результаты
Гибридомные антитела к PVRIG распознают PVRIG на сверхэкспрессирующих клетках: Чтобы провести скрининг в отношении антител, специфических к PVRIG, мы оценивали способность антител, созданных в двух гибридомных кампаниях, связывать клеточные линии HEK, сконструированные так, чтобы сверхэкспрессировать PVRIG. Большинство антител из этих кампаний связывались с клетками HEK hPVRIG, хотя и с разной аффинностью. Более того, большинство этих антител также демонстрировали низкое фоновое связывание с родительскими клеточными линиями HEK, что свидетельствует о высокой специфичности в отношении PVRIG. На фиг. 76 приведен один пример специфичности антител к PVRIG. Обобщенные результаты по всем характеристикам связывания созданных в гибридомных кампаниях антител с клетками HEK hPVRIG по сравнению с контролем приведены на фиг. 79.
Антитела к PVRIG распознают белок PVRIG на наивных NK- и Т-клетках: Популяциями, которые демонстрировали наивысший уровень PVRIG в субпопуляциях наивных МКПК, были NK-клетки и Т-клетки CD8, а абсолютный уровень экспрессии для этих двух субпопуляций клеток был одинаковым (гСИФ). Т-клетки CD4 демонстрировали более низкие уровни PVRIG, тогда как В-клетки и моноциты проявляли очень низкую/не выявляемую экспрессию. Обобщенные данные по экспрессии на наивных NK-клетках и Т-клетках CD8, выявляемой с помощью антител, приведены на фиг. 80. Другие малочисленные субпопуляции также демонстрировали экспрессию PVRIG и включали нетрадиционные Т-клетки, такие как NKT-клетки и Т-клетки. Профиль экспрессии в субпопуляциях МКПК был очень сходным среди всех анализируемых доноров.
PVRIG выявляется в клеточных линиях Jurkat с помощью полученных из гибридомы антител к PVRIG: Кроме исследований МКПК в отношении экспрессии белка PVRIG, мы хотели понять, экспрессировался ли он также в раковых клеточных линиях. Мы решили провести скрининг антител на клетках Jurkat, учитывая их высокую экспрессию РНК PVRIG. Также мы выбрали HepG2 в качестве отрицательной контрольной клеточной линии для дополнительной проверки специфичности антител. Большинство полученных из гибридомы антител выявляли экспрессию белка PVRIG в клетках Jurkat (фиг. 78). Характеристики связывания полученного из гибридомы антитела к PVRIG с первичными человеческими МКПК, сверхэкспрессирующими клетками яванского макака и первичными МКПК яванского макака. Expi cyno OE обозначает клетки expi, временно трансфицированные cPVRIG, expi par обозначает родительские клетки expi. сгСИФ показывает кратность разницы геометрической СИФ окрашивания антитела к PVRIG по сравнению с контролями. Указаны концентрации, для которых рассчитывали сгСИФ. Не исследовано указывает на антитела, которые не исследовали вследствие отсутствия связывания с человеческими клетками HEK hPVRIG, клетками expi cPVRIG или несоответствия требованиям связывания с субпопуляциями МКПК. Выделенные антитела представляют собой четыре антитела, для которых была проведена гуманизация (смотрите фиг. 90). На фиг. 91 приведены характеристики связывания гибридомного антитела к PVRIG с первичными человеческими МКПК, сверхэкспрессирующими клетками яванского макака и первичными МКПК яванского макака. Expi cyno OE означает клетки expi, временно трансфицированные cPVRIG, expi par означает родительские клетки expi. сгСИФ указывает кратность разницы в геометрической СИФ для окрашивания антителом к PVRIG по сравнению с контролями. Указанные концентрации являются теми, при которых рассчитывали сгСИФ. «Не исследовано» указывает на то, что антитела не исследовали вследствие отсутствия связывания с человеческими HEK hPVRIG, клетками expi cPVRIG или несоответствия требования связывания для субпопуляций МКПК. Выделенные антитела представляют собой четыре антитела, для которых проводили гуманизацию (смотрите фиг. 90).
На фиг. 90На фиг. 90, но не клетки HepG2 (данные не показаны). Пример выявления PVRIG на Jurkat приведен на фиг. 77 с типовым антителом CHA.7.518.
Клеточный биохимический анализ: После скрининга 29 гибридомных антител в клеточном биохимическом анализе мы обнаружили, что среди них было 20 четких блокаторов и 9 не-блокаторов взаимодействия PVRIG-PVRL2. Все блокирующие антитела были способны ингибировать взаимодействие PVRIG Fc с клетками HEK по меньшей мере на 50%, причем большинство этих антител полностью прекращали связывание PVRIG Fc. Значения IC50, соответствующие тем антителам, которые демонстрировали блокирующую способность, приведены на фиг. 90. Большинство значений IC50 составляли 20-60 нМ.
Выводы и заключения
Используя гибридомную платформу, нам удалось успешно создать моноклональные антитела против человеческого антигена PVRIG. Используя сконструированные сверхэкспрессирующие клетки, а также набор раковых клеточных линий, мы показали, что наши антитела являются высокоспецифическими в отношении антигена PVRIG и способны выявлять экспрессию белка, которая коррелирует с экспрессией РНК. После анализа субпопуляций человеческих МКПК мы показали, что белок PVRIG наиболее сильно экспрессируется на NK- и Т-клетках с низкой/отрицательной экспрессией на В-клетках и миелоидных клетках. Также мы показали, что часть этих антител перекрестно реагируют с антигеном PVRIG яванского макака (cyno), путем оценки их связывания со сверхэкспрессирующими клетками. Более того, профиль экспрессии в МКПК яванского макака сходен с человеческими МКПК. И наконец, с помощью конкурентного анализа FACS нам удалось показать, что часть гибридомных антител способны ингибировать взаимодействие PVRIG со своим лигандом PVRL2. Антителами, которые демонстрировали наилучшие характеристики в отношении всех вышеуказанных данных, были CHA-7-518, CHA-7-524, CHA-7-530 и CHA-7-538.
Пример 22. Действие CHA анти-PVRIG антител в анализе РСКЛ
Методом анализа, используемым для профилирования функционального действия антител к человеческому PVRIG на алло-антигенные ответы, является пролиферация человеческих Т-клеток CD8+ в анализе реакции в смешанной культуре лимфоцитов (РСКЛ). Как известно в данной области техники, РСКЛ представляет собой ex vivo клеточный иммунный анализ, который обеспечивает in vitro корреляцию Т-клеточной функции.
Ожидается, что анти-PVRIG антитела будут усиливать пролиферацию человеческих Т-клеток CD4 и CD8 в ответ на клетки от ГКГС-несовпадающего донора. Человеческие Т-клетки обогащают из цельной крови одного донора (например, донора А), используя RosetteSep RTM для человеческих Т-клеток (StemCell Technologies) в соответствии с инструкциями производителя. После сепарации клетки флуоресцентно метят красителем CFSE (Molecular Probes). Для того чтобы они служили аллогенными антигенпредставляющими клетками (АПК), мононуклеарные клетки сначала выделяют из цельной крови ГКГС-несовпадающего донора (например, донора В), а затем освобождают от Т-клеток CD3+. Затем АПК облучают 2500 рад в цезиевом облучателе.
В общем случае анализ РСКЛ проводят следующим образом. Человеческие Т-клетки и 150000 алогенных АПК совместно культивируют в 96-луночном плоскодонном планшете с 150000 Т-клетками CD8+ и АПК в течение 5 дней с анти-PVRIG антителами в разных концентрациях. На 5 день клетки собирают, промывают и окрашивают анти-CD8-биотином с последующим применением стрептавидина-PerCp. Затем образцы анализируют методом FACS, чтобы оценить степень пролиферации, которую отображает растворение CFSE. Ожидается, что функциональные блокирующие анти-PVRIG антитела будут усиливать пролиферацию Т-клеток и секрецию цитокинов в ответ на клетки от ГКГС-несовпадающего донора.
Анализ РСКЛ применяли, чтобы охарактеризовать биохимическое действие СНА-антител согласно изобретению на покоящиеся и активированные человеческие Т-клетки и чтобы охарактеризовать способность полученных из гибридомы антител модулировать пролиферацию Т-клеток в условиях РСКЛ.
Протоколы
Реакция в смешанной культуре лимфоцитов (РСКЛ): Реакцию в смешанной культуре лимфоцитов проводили путем совместного культивирования дендритных клеток (ДК) и Т-клеток, полученных от разных доноров, в аллогенных условиях. ДК получали путем культивирования очищенных моноцитов с 100 нг/мл ГМ-КСФ (R&D systems) и 100 нг/мл IL-4 (R&D systems) в течение 7 дней. Через 7 дней очищенные CFSE-меченые Т-клетки CD3 смешивали с ДК в соотношении 10:1 и культивировали в бессывороточной среде X vivo-20 (Lonza) в течение 5 дней. В таких же условиях в планшеты добавляли неконъюгированные анти-PVRIG антитела или изотипические контрольные антитела в концентрации 10 мкг/мл. Осуществляли три комбинации РСКЛ-анализа, в которых ДК от одного донора культивировали совместно с Т-клетками CD3 от 3 разных аллогенных доноров. Все продукты крови были получены от Stanford Blood Bank.
Экспрессия и функциональный анализ: Через 5 дней РСКЛ-культуру, уровень и степень активации и пролиферации Т-клеток оценивали по растворению CFSE и экспрессии маркеров активации, таких как CD25 и PD-1. Внутрилабораторные анти-PVRIG антитела из фаговой и гибридомной кампаний использовали для оценки экспрессии PVRIG. Также кинетическим образом оценивали экспрессию лиганда PVRIG, PVRL2, на ДК. Все данные были получены с помощью проточной цитометрии, а анализ данных проводили с помощью программного обеспечения FlowJo (Treestar) и Prism (Graphpad).
FACS-анализ эпитопов: Так как мы исследовали панель антител в РСКЛ, мы были заинтересованы определить, можно ли провести эпитопную «сортировку» этих антител на основании данных связывания FACS и будет ли эта «сортировка» коррелировать с изменениями Т-клеточной активации и пролиферации в анализе. Чтобы это выполнить, собранные в анализе Т-клетки предварительно инкубировали с неконъюгированными антителами к PVRIG, а затем проводили контрокрашивание конъюгированным антителом к PVRIG из другого клона. Степень, в которой конъюгированное антитело к PVRIG давало сигнал на Т-клетках, указывает на степень, в которой это антитело конкурирует с неконъюгированным антителом за связывание PVRIG на Т-клетках. Отрицательный или низкий сигнал указывал бы на сильную конкуренцию, что указывает, что два антитела находятся в одной и той же эпитопной «группе». Высокий сигнал указывал бы на низкую или отсутствующую конкуренцию и, таким образом, считалось бы, что антитела находятся в разных «группах».
Результаты
Экспрессия PVRL2 на моноцитарных ДК: Чтобы определить, будет ли PVRL2 экспрессироваться на ДК в РСКЛ-анализе, из моноцитов получали ДК и оценивали экспрессию PVRL2 кинетическим образом с суточными интервалами после добавления ГМ-КСФ и IL-4. Как указано на фиг. 71, экспрессия PVRL2 повышалась от 0 дня по 5 день, когда наблюдался пик экспрессии. На 6 день экспрессия была немного снижена по сравнению с 5 днем. На 7 день экспрессия была такой же, как и на 6 день, что свидетельствует о стабилизации экспрессии PVRL2 в эти моменты времени. Таким образом, ДК экспрессировали PVRL2 в подходящий момент времени для применения в РСКЛ-анализе.
Экспрессия PVRIG на Т-клетках после РСКЛ-культуры: Многие рецепторы Т-клеток, которые модулируют функцию в РСКЛ, экспрессируются на пролиферирующих Т-клетках. Таким образом, мы хотели определить, экспрессируется ли также PVRIG. Мы анализировали пролиферирующие Т-клетки на 5 день после начала совместного культивирования с РСКЛ и определяли характеристики по растворению CFSE (например, низкое содержание CFSE). Как показано на фиг. 72 и на фиг. 73, по сравнению с изотипическим контролем (mIgG1) PVRIG экспрессировался в клетках с низким содержанием CFSE согласно определению с помощью некоторого количества антител к PVRIG на Т-клетках CD4 и CD8 для всех трех анализируемых доноров. На фиг. 72 приведены графики FACS, указывающие на наличие PVRIG в клетках с низким содержанием CFSE, а гистограммы на фиг. 73 указывают на уровень экспрессии PVRIG по сравнению с mIgG1.
Антитела к PVRIG усиливают пролиферацию Т-клеток: Показав, что PVRIG экспрессируется на пролиферирующих Т-клетках в РСКЛ, мы хотели определить, может ли обработка антителами к PVRIG влиять на уровни пролиферации Т-клеток. Как показано на фиг. 4, добавление антител к PVRIG в анализ РСКЛ приводило к повышению процентной доли клеток с низким содержанием CFSE для всех исследуемых гибридомных антител по сравнению с контролем. Это наблюдали для всех анализируемых доноров.
Антитела к PVRIG связываются с несколькими эпитопами на PVRIG: Чтобы сравнить способность антител к PVRIG связывать разные эпитопы на PVRIG, мы провели конкурентный эксперимент, в котором Т-клетки из РСКЛ в течение 5 дней культивировали с немечеными анти-PVRIG антителами, полученными в гибридомной кампании. На 5 день Т-клетки собирали и проводили контрокрашивание конъюгированным анти-PVRIG антителом, которое было получено в фаговой кампании (CPA.7.021). Как показано на фиг. 75, полное или практически полное снижение связывания CPA.7.021 по сравнению с фоновыми уровнями флуоресценции наблюдали в условиях, которые включали применение CHA.7.516-M1, CHA.7.518-M1, CHA.7.524-M1, CHA.7.530-M1 и CHA.7.538-M1, что позволяет предположить наличие для этих антител перекрытия при распознавании эпитопа. Частичное снижение связывания CPA.7.021 наблюдали при применении CHA.7.537-M1, CHA.7.528-M1 и CHA.7.548-M1, что позволяет предположить наличие частичного перекрытия при распознавании эпитопа. Отсутствие снижения в связывании CPA.7.021 наблюдали в клетках, которые предварительно культивировали с CHA.7.543-M1, что позволяет предположить отсутствие распознавания эпитопа. Вместе эти данные указывают на то, что антитела к PVRIG из наших кампаний при оценке в сравнении с CPA.7.021 могли распознавать по меньшей мере 3 разных эпитопа на PVRIG.
Выводы. Мы охарактеризовали антитела к PVRIG в отношении их способности связываться с пролиферирующими и покоящимися Т-клетками, а также их функциональной активности в РСКЛ. Было выявлено связывание некоторого количества антител к PVRIG на пролиферирующих Т-клетках, и оно было выше на пролиферирующих Т-клетках по сравнению с покоящимися клетками, в особенности в субпопуляции CD8+. Эти данные демонстрируют, что экспрессия PVRIG повышается после активации Т-клеток. Более того, несколько антител к PVRIG повышали пролиферацию Т-клеток по сравнению с изотипическим mIgG1, что указывает на то, что они также могут модулировать Т-клеточную функцию. Как и выше, все эти антитела обладают способностью блокировать взаимодействие PVRIG со своим лигандом PVRL2. На основании этого мы заключили, что, блокируя взаимодействие PVRIG-PVRL2, эти антитела приводят к повышению активации и пролиферации Т-клеток, что является ярким показателем желаемого действия для ингибитора иммунных контрольных точек, который можно применять для лечения рака. И наконец, мы провели конкурентные эксперименты, сравнивая связывание некоторого числа полученных из гибридомы антител к PVRIG с активированными Т-клетками по сравнению с фаговым антителом. Из этой серии экспериментов мы получили свидетельства разнообразия в отношении эпитопов для фаговых и гибридомных антител.
Пример 23. Действие анти-PVRIG антител на активацию Т-клеток в комбинации с блокированием иммунных контрольных точек
Ожидается, что комбинация блокирования PVRIG с блокирующими Ab для известных иммунных контрольных точек (например, PD1, PDL-1 или TIGIT) дополнительно повысит стимуляторное действие на активацию Т-клеток в вышеописанных анализах.
Пример 24. Функциональный анализ антител к PVRIG
Антитела к человеческому PVRIG согласно изобретению были охарактеризованы в отношении способности ингибировать взаимодействие PVRIG со своим лигандом PVRL2 и способности модулировать функцию эффекторных лимфоцитов в анализе первичных клеток.
Протоколы
Клеточный биохимический анализ
Способность антител к PVRIG ингибировать взаимодействие PVRIG со своим лигандом PVRL2 оценивали в формате клеточного биохимического анализа в двух последовательностях.
В первой последовательности лиганд PVRL2 эндогенно экспрессируют в необработанных клетках HEK и добавляют растворимый биотинилированный Fc-меченый PVRIG (произведенный по запросу Genscript). В этом случае способность антител к PVRIG блокировать связывание растворимого PVRIG с клетками HEK оценивали в двух вариантах. В первом варианте разные концентрации антител к PVRIG (в диапазоне 0,066-66 нМ) предварительно инкубировали с 33 нМ растворимого PVRIG в фосфатно-солевом буфере (ФСБ, Gibco) в течение 30 минут на льду. Впоследствии этот комплекс добавляли к 100000 клеток HEK и инкубировали еще в течение 1 часа на льду. Через 1 час клетки HEK дважды промывали в ФСБ, а количество PVRIG, связанного с клетками HEK, определяли путем добавления стрептавидина, конъюгированного с Alexa 647 (Jackson Laboratories), в течение 30 минут на льду. Клетки HEK дважды промывали в ФСБ и пересуспендировали в 100 мкл ФСБ для получения данных на FACS Canto II (BD Biosciences). Данные анализировали с помощью программного обеспечения FlowJo (Treestar) и Prism (Graphpad). Во втором варианте 33 нМ растворимого белка PVRIG и антитела к PVRIG (0,066-66 нМ) одновременно добавляли к 100000 клеток HEK и инкубировали в течение 1 часа на льду. Последующие этапы для анализа этого варианта эквивалентны первому варианту.
Во второй последовательности конструировали клетки HEK, сверхэкспрессирующие PVRIG, и добавляли растворимый биотинилированный Fc-меченый PVRL2 (CD Biosciences). В этом случае разные концентрации антител к PVRIG (в диапазоне 0-200 нМ) с 160 нМ растворимого PVRL2 одновременно добавляли к 100000 клеток HEK hPVRIG или родительских клеток HEK и инкубировали в ФСБ + 1% БСА + 0,1% азиде натрия (буфер FACS) в течение 1 ч на льду. Связывание растворимого PVRL2 определяли путем добавления стрептавидина-Alexa 647 в буфер FACS в течение 30 минут на льду. Клетки дважды промывали в буфере FACS и пересуспендировали в 50 мкл ФСБ для получения данных на Intellicyt HTFC (Intellicyt). Данные анализировали с помощью FlowJo (Treestar), Excel (Microsoft) и Prism (GraphPad).
Анализ первичных NK-клеток
Субпопуляцией МКПК с наиболее стабильным профилем экспрессии в отношении PVRIG были NK-клетки. Следовательно, мы разработали анализ на основе совместного культивирования NK-клеток с PVRL2-экспрессирующими опухолевыми клетками, чтобы определить, могут ли наши антитела модулировать опосредованную NK-клетками цитотоксичность в отношении этих мишеней. В качестве мишеней мы выбрали клеточную линию острого В-клеточного лимфоцитарного лейкоза, Reh (банк клеток ATCC), и клеточную линию острого миелоидного лейкоза, MOLM-13 (банк клеток DSMZ). Клетки Reh и MOLM-13 выращивали в среде RPMI (Gibco) + 20% фетальная телячья сыворотка (Gibco), глутамакс (Gibco), пенициллин/стрептомицин (Gibco), заменимые аминокислоты (Gibco), пируват натрия (Gibco), ГЭПЭС (Gibco) и бета-меркаптоэтанол (Gibco).
За два для до анализа совместной культуры выделяли первичные NK-клетки, используя набор для выделения человеческих NK-клеток (Miltenyi Biotec), и культивировали в среде RPMI + 20% фетальная телячья сыворотка, глутамакс, пенициллин/стрептомицин, заменимые аминокислоты, пируват натрия, ГЭПЭС, бета-меркаптоэтанол и 250 Е/мл IL-2 (R&D systems). В день анализа NK-клетки собирали, подсчитывали и предварительно инкубировали с антителами к PVRIG в течение 15-30 минут при комнатной температуре. Во время этой инкубации клетки-мишени собирали из культуры, метили кальцеином-AM (Life Technologies) в течение 30 минут при 37oc, промывали в среде и подсчитывали для анализа. Проводили анализ опосредованной NK-клетками цитотоксичности, в котором постоянное число клеток-мишеней (50000) культивировали совместно с возрастающими концентрациями NK-клеток, которые предварительно инкубировали с 5 мкг/мл антител к PVRIG (изменяя, таким образом, соотношение между NK-клетками и мишенями). В альтернативном варианте использовали фиксированное соотношение между NK-клетками и мишенями, но NK-клетки предварительно инкубировали с разными концентрациями антител к PVRIG (в диапазоне 3,9 нг/мл-5 мкг/мл) с титрованием дозы. После добавления NK-клеток и мишеней планшеты центрифугировали при 1400 об/мин в течение 1 минуты и помещали при 37oC в атмосферу 5% CO2 на 4 часа. Через 4 часа планшеты центрифугировали при 1400 об/мин в течение 4 минут и отбирали 80 мкл супернатанта для количественной оценки высвобождения кальцеина-АМ из клеток-мишеней. Количество кальцеина-АМ, высвобождаемого из мишеней, оценивали с помощью флуорометра Spectramax Gemini XS (Molecular Devices). В качестве контролей для высвобождения кальцеина-АМ оценивали общее и спонтанное высвобождение путем обработки клеток-мишеней 70% этанолом или только средой в течение анализа. Уровни уничтожения (в виде процентной доли) NK-клетками рассчитывали по следующей формуле:
(Высвобождение образца - спонтанное высвобождение) / (общее высвобождение - спонтанное высвобождение)*100
Кроме антител к PVRIG в некоторых случаях для сравнения также добавляли другие антитела против рецепторов NK-клеток, такие как TIGIT (Genentech, клон 10A7, патент номер: WO2009126688 A2) и DNAM-1 (Biolegend, клон 11A8).
Результаты
Клеточный биохимический анализ: После скрининга панели наших антител к PVRIG в клеточном биохимическом анализе мы обнаружили, что среди исследуемых антител наблюдались разные уровни ингибирования, а уровень ингибирования зависел от варианта и последовательности анализа (на фиг. 96). В частности, на фиг. 91 приведены четыре антитела, иллюстрирующие эти моменты. Последовательность и вариант анализа, которые давали наиболее сильный ингибиторный эффект по сравнению с контролем, включали условия, когда растворимый PVRIG, который предварительно инкубировали с антителами к PVRIG, добавляли к клеткам HEK (на фиг. 91а). В этом варианте CPA.7.021 продемонстрировало наилучшую блокирующую способность по сравнению с другими тремя антителами (CPA.7.002, CPA.7.005 и CPA.7.050). Несмотря на разницу в уровне блокирования, все антитела в этом варианте демонстрировали одинаковые значения IC50, находящиеся в низком наномолярном диапазоне, при этом при более высоких концентрациях блокирующая способность выходила на плато.
Когда после этого определяли абсолютный уровень ингибирования, вызываемый четырьмя антителами к PVRIG, когда растворимый PVRIG и антитела к PVRIG одновременно добавляли к клеткам HEK, в этом анализе наблюдали больший разброс в блокировании (на фиг. 91В). CPA.7.021 оставалось наилучшим блокирующим антителом. Однако CPA.7.002 и CPA.7.005 продемонстрировали заметно меньшую способность ингибировать связывание растворимого PVRIG с клетками HEK по сравнению с контрольным антителом. CPA.7.050 продемонстрировало средний уровень блокирования по сравнению с CPA.7.021, CPA.7.002 и CPA.7.005. Эта разница в абсолютном уровне ингибирования также соответствует разнице в значениях IC50 каждого антитела. CPA.7.021 и CPA.7.050 снова продемонстрировали низкие наномолярные значения IC50, хотя они оба были выше, чем в первом варианте анализа. В противоположность этому, значения IC50 для CPA.7.002 и CPA.7.005 значительно возросли, для CPA.7.002 приблизительно в 20 раз, а для CPA.7.005 приблизительно в 30 раз. Эти данные говорят о том, что то, насколько антитело должно конкурировать за связывание с PVRIG со своим когнатным лигандом, указывает на эффективность, с которой антитело может блокировать это взаимодействие.
При обратной последовательности биохимического анализа (т.е. оценивали связывание PVRL2Fc с клетками HEK hPVRIG) способность четырех антител к PVRIG блокировать взаимодействие PVRL2 Fc была разной (На фиг. 91c). В соответствии с биохимическим анализом, в котором клетки HEK использовали как мишени (на фиг. 91a-b), CPA.7.021 и CPA.7.050 ингибировали связывание PVRL2 Fc с клетками HEK hPVRIG, а их способность блокировать связывание была одинаковой. Неожиданно наблюдалось повышение связывания PVRL2 Fc в присутствии антител CPA.7.002 и CPA.7.005, которое не наблюдалось, когда клетки HEK использовали в качестве мишеней.
Анализ цитотоксичности NK-клеток с клетками Reh: Первой мишенью, которую мы исследовали в анализе цитотоксичности NK-клеток, была линия Reh. Изначально Reh выбрали потому, что она демонстрировала высокие уровни PVRL2 по данным проточной цитометрии, но низкую частоту других активирующих лигандов, таких как лиганды NKG2D, и низкую экспрессию PVR (на фиг. 92). Традиционные мишени NK-клеток, такие как K562, не использовали из-за их высокочастотной экспрессии лигандов NKG2D и высокой экспрессии PVR, которые могут экранировать функциональное действие антител к PVRIG. Важно, что клетки Reh не экспрессируют никакие рецепторы NK-клеток, взаимодействующие с PVRL2 и PVR, такие как TIGIT, DNAM-1 и PVRIG.
После скрининга панели наших антител к PVRIG в этом анализе мы обнаружили четыре антитела, которые были способны модулировать опосредованную NK-клетками цитотоксичность (фиг. 97). Этими четырьмя антителами были те, которые мы обсуждали в результатах биохимического анализа, - CPA.7.002, CPA.7.005, CPA.7.021 и CPA.7.050. Во всех случаях добавление этих антител усиливало опосредованную NK-клетками цитотоксичность против клеток Reh (на фиг. 93a-c). Добавление CPA.7.002 и CPA.7.005 усиливало цитотоксичность наиболее сильно (На фиг. 93a-b), далее шли CPA.7.021 и CPA.7.050, которые демонстрировали одинаковые уровни усиления (На фиг. 93c). На фиг. 93d приведен концентрационно-зависимый анализ усиления опосредованной NK-клетками цитотоксичности со стороны CPA.7.002 и CPA.7.021. Блокирующие антитела к рецепторам, которые также связывают PVRL2, такие как TIGIT и DNAM-1, добавляли в анализ с клетками Reh для сравнения. Как показано наНа фиг. 93e-f, добавление антител TIGIT и DNAM-1 не продемонстрировало функциональное действие в этом анализе.
Анализ NK-клеток с клетками MOLM-13: Чтобы оценить, способны ли антитела к PVRIG модулировать опосредованную NK-клетками цитотоксичность против второй мишени, использовали клетки MOLM-13. MOLM-13 также экспрессируют PVRL2, аналогично клеткам Reh, но также характеризуются сильной экспрессией PVR (На фиг. 92). Как и клетки Reh, MOLM-13 не экспрессируют никаких рецепторов NK-клеток. Применение этой клеточной линии в дополнение к клеткам Reh покажет, могут ли антитела к PVRIG модулировать опосредованную NK-клетками цитотоксичность в контексте отличных взаимодействий рецептор-лиганд, в частности, при экспрессии PVR.
После скрининга наших антител к PVRIG в этом анализе мы обнаружили, что функциональное действие CPA.7.021 было снижено и не демонстрировало значительного усиления опосредованной NK-клетками цитотоксичности выше контрольных уровней (На фиг. 95a). В противоположность этому, CPA.7.002 и CPA.7.005 были способный усиливать опосредованную NK-клетками цитотоксичность в этом анализе (На фиг. 95a). При применении сравнительного антитела блокирование TIGIT в этом анализе не продемонстрировало функциональное действие по сравнению с контролем (На фиг. 95b).
Выводы и заключения
Используя фаговую платформу для наших антител мы создали панель антител против человеческого антигена PVRIG, которые продемонстрировали способность блокировать взаимодействие PVRIG со своим лигандом PVRL2 и усиливать опосредованную NK-клетками цитотоксичность против двух гематологических клеточных линий. На способность антител к PVRIG ингибировать взаимодействие PVRIG и PVRL2 влияла последовательность анализа, а также этапы предварительной инкубации, представляющие потенциальную динамику антитела с PVRIG в физиологических условиях, таких как рак. Четыре антитела продемонстрировали способность усиливать опосредованную NK-клетками цитотоксичность против клеточной линии Reh, но только два антитела продемонстрировали способность усиливать цитотоксичность против клеток MOLM-13. Эта разница может быть связана с альтернативными взаимодействиями рецептор-лиганд, вовлеченными в опосредованное NK-клетками распознавание каждой клеточной линии, и/или с разными свойствами антител и их эффективностью в модулировании функции PVRIG.
Пример 25. Изучение влияния анти-PVRIG антител на активацию ГД Т-клеток с помощью клеток, эктопически или естественным образом экспрессирующих PVRL2
Клеточный анализ используют для исследования действия анти-PVRIG антител на активацию, пролиферацию гамма-дельта Т-клеток и секрецию цитокинов. Очищенные человеческие гамма-дельта Т-клетки активируют с помощью HMBPP или IPP и культивируют совместно с клетками-мишенями (например, REH, MOLM-13), которые естественным образом экспрессируют PVRL2, или с клетками-мишенями, эктопически экспрессирующими PVRL2 или пустой вектор (например, CHO, Raji, 721.221). Функцию гамма-дельта Т-клеток оценивают по выработке цитокинов (например, IFN-γ, IL-17) в культуральных супернатантах или цитотоксической активности в отношении клеток-мишеней. Ожидается, что экспрессия PVLR2 будет оказывать базовое стимуляторное действие на активацию гамма-дельта Т-клеток, опосредованную эндогенным DNAM1 - известным ко-стимуляторным рецептором контрпартнера PVRL2 на гамма-дельта Т-клетках. Ожидается, что в присутствии антагонистических анти-PVRIG Ab выработка цитокинов или цитотоксическая активность будут дополнительно усилены благодаря блокированию ингибиторной функции эндогенного PVRIG в отношении активации гамма-дельта Т-клеток. Соответственно, ожидается, что агонистические анти-PVRIG Ab продемонстрируют ингибирование активации гамма-дельта Т-клеток.
Пример 26. Действие белков на человеческие Т-клетки, активированные с помощью анти-CD3 и анти-CD28 в присутствии аутологичных МКПК
Материалы и методы
В этих экспериментах оценивали действие PVRIG на человеческие Т-клетки, которые были активированы с помощью-CD3 и анти-CD28 в присутствии аутологичных МКПК. И наоборот, этот анализ также можно использовать, чтобы оценить действие анти-PVRIG антител на активацию Т-клеток.
Слитый белок PVRIG hECD-hIg (фиг. 92BA), состоящий из ВКД человеческого PVRIG, слитого с Fc человеческого IgG1, несущий мутации C220, C226 и C229 на S в шарнирной области, получали с помощью GenScript (China) путем временной трансфекции в клетки CHO-3E7, которые культивировали в течение 6 дней, с последующей очисткой собранных клеток с помощью протеина А. Конечный продукт смешивали в ФСБ, pH 7,2. Применяемым экспрессионным вектором был экспрессионный вектор млекопитающих pTT5, в котором ген PVRIG находится под управлением промотора CMV.
Набор для выделения человеческих Т-клеток CD4+ II приобретают у Miltenyi (Кат. #130-094-131). Контрольный hIgG1 (Synagis®) приобретают у Medimmune Inc. Ab против человеческого CD3 (OKT3, Кат. # 16-0037) и Ab против человеческого CD28 (клон CD28. 2; Кат. # 16-0289) приобретают у eBioscience. Гранулы Dynabead M-450 Epoxy (Кат. # 140. 11) приобретают у Invitrogen. Лейкоцитарные пленки из человеческой крови приобретают у LifeSource. Ficoll-Paque Plus (Кат. #17-1440-02) приобретают у GE HealthCare.
Выделение МКПК из лейкоцитарных пленок методом фиколл-сепарации: Общее количество МКПК суспендируют в среде Ex-Vivo 20 и облучают при 3000 рад. Наивные Т-клетки CD4+ выделяют из лейкоцитарных пленок из крови трех здоровых человеческих доноров, используя набор для выделения человеческих Т-клеток CD4+ II (Miltenyi) в соответствии с инструкциями производителя, и культивируют совместно с облученными аутологичными МКПК в соотношении 1:1 (1,5x105 Т-клеток с 1,5x105 облученных МКПК на лунку). Культуры активируют анти-CD3 (0,5 мкг/мл) и анти-CD28 (0,5 мкг/мл) антителами. Анти-PVRIG антитело или белок ВКД PVRIG добавляют в культуру в указанных концентрациях. Через 24 ч в культуре клетки активируют Н3-тимидином. Клетки собирают через 72 часа в культуре.
В случае ВКД-эксперимента ожидается, что результаты покажут дозозависимое ингибирование пролиферации и/или активации Т-клеток, подтверждая терапевтический потенциал терапевтических агентов на основе иммуноингибиторного PVRIG (например, полипептидов PVRIG или слитых белков PVRIG в соответствии по меньшей мере с некоторыми вариантами реализации изобретения) для лечения Т-клеточных аутоиммунных заболеваний, таких как ревматоидный артрит, множественный склероз, псориаз и воспалительное заболевание кишечника, а также для лечения других иммунных заболеваний и/или для снижения нежелательной иммунной активации после генной или клеточной терапии. По большому счету, иммуноингибиторные белки PVRIG, которые агонизируют PVRIG, должны предотвращать или снижать активацию Т-клеток и выработку провоспалительных цитокинов, вовлеченных в патологию таких заболеваний.
Кроме того, также ожидается, что эти результаты подтвердят терапевтический потенциал иммуностимулирующих анти-PVRIG антител, которые снижают ингибиторную активность PVRIG, для лечения патологических состояний, при которых усиление иммунных ответов будет благоприятным, например, для иммунотерапии рака, инфекционных заболеваний, в частности, хронических инфекций и сепсиса. По большому счету, иммуностимулирующие анти-PVRIG антитела будут способствовать активации Т-клеток и вызывать выработку провоспалительных цитокинов, тем самым способствуя снижению количества раковых или инфицированных клеток или инфекционных агентов.
Пример 27. Анализ ингибирования активации Т-клеток.
В этих экспериментах оценивают действие ВКД PVRIG или анти-PVRIG антител на активацию Т-клеток в анализе с гранулами.
Материалы и методы
Выделение человеческих Т-клеток: Лейкоцитарные пленки получают от Stanford Blood Bank от здоровых человеческих доноров. Т-клетки CD3+ выделяют из лейкоцитарных пленок, используя набор RosetteSep (StemCell Technologies), следуя инструкциям производителя. Клетки анализируют с помощью анти-CD45 и анти-CD3 методом проточной цитометрии, чтобы оценить % полученных клеток CD3+. Жизнеспособность оценивают после размораживания перед анализом.
Покрытие гранул и КК: Тозил-активируемые гранулы (Invitrogen, Кат. # 14013) при 500x106/мл покрывают анти-CD3 mAb и белками ВКД PVRIG или анти-PVRIG антителами в соответствии с двухэтапным протоколом: с 50 мкг/мл клона UTCH1 человеческого анти-CD3 (R&D systems, Кат. # mab 100) в натрий-фосфатном буфере при 37°C в течение ночи, а после этого с 0-320 мкг/мл белков ВКД PVRIG или анти-PVRIG антител для еще одной инкубации в течение ночи при 37°C.
Анализируют количество белка PVRIG (ВКД или антитела), связанного с гранулами.
Условия анализа с гранулами: 100 тыс. человеческих Т-клеток CD3+ культивируют с 100 тыс. или 200 тыс. гранул, покрытых разными концентрациями белка PVRIG в течение 5 дней в полной среде IMDM (Gibco, Кат. #12440-053), дополненной 2% AB человеческой сывороткой (Gibco, Кат. # 34005-100), глутамаксом (Gibco, Кат. #35050-061), пируватом натрия (Gibco, Кат. #11360-070), раствором MEM заменимых аминокислот (Gibco, Кат. #11140-050) и 2-меркаптоэтанолом (Gibco, Кат. #21985). В конце 5 дня культивирования клетки окрашивают анти-CD25, анти-CD4, анти-CD8 и фиксируемым красителем в отношении живых и мертвых клеток для определения уровней экспрессии CD25 в каждой субпопуляции клеток. Собирают супернатанты и анализируют в отношении секреции IFNγ методом ELISA (Human IFNγ duoset, R&D systems, DY285).
В этих примерах человеческие Т-клетки CD3 культивируют совместно с гранулами, покрытыми разными концентрациями белка PVRIG и анализируют в отношении уровня экспрессии CD25. Ожидается, что клетки CD4+ и CD8+ будут демонстрировать дозозависимое ингибирование со стороны слитого белка PVRIG-ВКД или, наоборот, ожидается, что клетки CD4+ и CD8+ будут демонстрировать дозозависимую активацию антителом к PVRIG.
Пример 29. Картирование эпитопов антител к человеческому PVRIG на основании перекрестной реактивности с яванским макаком
Обоснование и задачи
Задачей этого исследования является идентификация эпитопов на белке PVRIG, которые определяют перекрестную реактивность антител к человеческому PVRIG с ортологом яванского макака (cyno). Многие из основных антител против человеческого PVRIG демонстрируют разную степень перекрестной реактивности с яванским макаком, несмотря на тот факт, что многие из этих антител принадлежат одной эпитопной группе. Чтобы пролить свет на молекулярную подоплеку перекрестной реактивности человек/яванский макак (или ее отсутствия), было разработано несколько мутаций яванский макак-человек в рекомбинантных белках PVRIG, которые затем экспрессировали, очищали и исследовали в отношении связывания с панелью антител против человеческого PVRIG методом ELISA.
Методы
Разработка вариантов PVRIG яванский макак-человек: Выравнивание последовательностей человеческого и внеклеточных доменов (ВКД) PVRIG показало 90% идентичности последовательностей и 93% гомологии последовательностей между ортологами человека и яванского макака (На фиг. 98). С учетом природы мутаций (консервативных или неконсервативных) и прогнозирования вторичной структуры (спиральная или развернутая) области мутации было разработано три сайт-направленных мутанта PVRIG яванского макака для исследования сфокусированного на перекрестной реактивности с яванским макаком картирования эпитопов. Эти мутанты включают H61R, P67S и L95R/T97I в PVRIG яванского макака. Также получали PVRIG человека и яванского макака дикого типа.
Экспрессия и очистка вариантов PVRIG яванского макака, человека и гибридных вариантов: Все варианты PVRIG экспрессировали в виде слияния ВКД с С-концевой 6XHis-меткой в клетках млекопитающих. Белки очищали методом аффинной очистки, ионообменной хроматографии и эксклюзионной хроматографии. Для очищенных белков проводили замену буфера на буфер ФСБ (pH 7,4) и хранили при 4°C.
ELISA для определения взаимодействия PVRIG-антитело: Функциональный анализ ELISA проводили следующим образом: рекомбинантные белки PVRIG яванского макака, человека и гибрида яванский макак/человек (His-меченые) адсорбировали на IA-планшете в течение ночи при 4°C. Покрытые лунки планшета дважды промывали ФСБ и инкубировали с 300 мкл блокирующего буфера (5% сухое обезжиренное молоко в ФСБ, pH 7,4) при комнатной температуре (КТ) в течение 1 ч. Блокирующий буфер удаляли, а планшеты дополнительно дважды промывали ФСБ. Связанные с планшетом варианты PVRIG инкубировали с mAb против человеческого PVRIG (человеческий IgG1 изотип) в растворе (линейный диапазон от 0,1 мкг/мл до 8 мкг/мл в объеме 50 мкл/лунку) при КТ в течение 1 ч. Планшеты три раза промывали ФСБ-Т (ФСБ 7,4, 0,05% Твин20), затем три раза ФСБ и добавляли 50 мкл/лунку HRP-конъюгированного вторичного антитела (Специфического к Fc-домену человеческого IgG, Jackson ImmunoResearch). Реакцию инкубировали при КТ в течение 1 ч и снова промывали планшеты. Сигналы ELISA инициировали во всех лунках путем добавления 50 мкл ТМБ-субстрата Sureblue (KPL Inc) и инкубации в течение 5-20 мин. Реакцию HRP останавливали путем добавления 50 мкл 2N H2SO4 (VWR), а сигналы поглощения на 450 нм считывали на спектрофотометре SpectraMax (Molecular Devices) или EnVision (PerkinElmer). Данные экспортировали в Excel (Microsoft) и строили графики в GraphPad Prism (GraphPad Software, Inc.).
Результаты
Остатки S67, R95 и I97 как детерминанты перекрестной реактивности яванского макака: Данные по связыванию, приведенные на фиг. 99, четко показывают, что остатки S67, R95 и I97 влияют на перекрестную реактивность с яванским макаком разных антител. Тогда как мутация P67S яванский макак-человек отрицательно влияет на связывание CPA.7.002 и CPA.7.041, мутация L95R/T97I яванский макак-человек значительно улучшает связывание CPA.7.002, CPA.7.021, CPA.7.028 и CPA.7.041. С одной стороны, мутация H61R яванский макак-человек не влияет на связывание исследуемых антител.
Относительное связывание с вариантами яванский макак-человек предполагает наличие трех эпитопных групп: Относительное связывание антител с вариантами PVRIG яванского макака, человека и гибридными вариантами предполагает наличие 3 разных эпитопных групп: Группа 1 связывается с остатками R95/I97 (CPA.7.021 и CPA.7.028). Группа 2 связывается с остатками S67 и R95/I97 (CPA.7.002 и CPA.7.041). Группа 3 не связывается с остатками S67 или R95/I97 (CPA.7.024 и CPA.7.050). Эпитопные группы демонстрируют сильную корреляцию со степенью перекрестной реактивности с яванским макаком этих антител (на фиг. 100).
Выводы и заключения
Ограниченное картирование эпитопов на основании вариаций яванский макак-человек в ВКД PVRIG позволило идентифицировать остатки S67, R95 и I97 как детерминанты перекрестной реактивности с яванским макаком антител к человеческому PVRIG. Полное восстановление связывания с L95R/T97I PVRIG яванского макака в случае антител CPA.7.021 и CPA.7.028 и улучшение связывания CPA.7.002 с этим мутантом позволяют с большой вероятностью предположить, что остатки R95 и I97 представляют собой важные эпитопы человеческого PVRIG для этих антител. Эти открытия также позволяют предположить возможный способ прогнозировать перекрестную реактивность с ортологами PVRIG отличных от человека приматов на основании их первичной аминокислотной последовательности.
--->
Список последовательностей
<110> COMPUGEN, LTD.
<120> ANTI-PVRIG ANTIBODIES AND METHODS OF USE
<130> 114386-5002-US
<140> 15/048,967
<141> 2016-02-19
<150> 62/235,823
<151> 2015-10-01
<150> 62/141,120
<151> 2015-03-31
<150> 62/118,208
<151> 2015-02-19
<160> 1543
<170> PatentIn version 3.5
<210> 1
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 2
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 2
cttgtgttca ccacctctgg 20
<210> 3
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 3
tgttctcatc gcaggaggtc 20
<210> 4
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 4
ttggctgtgg atacctcctt 20
<210> 5
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 5
ataagggtcg tggagagcag 20
<210> 6
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 6
Asp Tyr Lys Asp Asp Asp Asp Lys
1 5
<210> 7
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
6xHis tag
<400> 7
His His His His His His
1 5
<210> 8
<211> 204
<212> PRT
<213> Homo sapiens
<400> 8
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly
35 40 45
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
50 55 60
Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp
65 70 75 80
Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu
85 90 95
Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe
100 105 110
Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
115 120 125
Ser Leu Pro Pro Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro
130 135 140
Ala Pro Ile Leu Arg Ala Asp Leu Ala Gly Ile Leu Gly Val Ser Gly
145 150 155 160
Val Leu Leu Phe Gly Cys Val Tyr Leu Leu His Leu Leu Arg Arg His
165 170 175
Lys His Arg Pro Ala Pro Arg Leu Gln Pro Ser Arg Thr Ser Pro Gln
180 185 190
Ala Pro Arg Ala Arg Ala Trp Ala Pro Ser Gln Ala
195 200
<210> 9
<211> 224
<212> PRT
<213> Homo sapiens
<400> 9
Met Arg Thr Glu Ala Gln Val Pro Ala Leu Gln Pro Pro Glu Pro Gly
1 5 10 15
Leu Glu Gly Ala Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu
20 25 30
Leu Thr Leu Cys Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val
35 40 45
Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe
50 55 60
Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro
65 70 75 80
Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly
85 90 95
Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser
100 105 110
Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala
115 120 125
Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp
130 135 140
Glu Ala Cys Gly Ser Leu Pro Pro Ser Ser Asp Pro Gly Leu Ser Ala
145 150 155 160
Pro Pro Thr Pro Ala Pro Ile Leu Arg Ala Asp Leu Ala Gly Ile Leu
165 170 175
Gly Val Ser Gly Val Leu Leu Phe Gly Cys Val Tyr Leu Leu His Leu
180 185 190
Leu Arg Arg His Lys His Arg Pro Ala Pro Arg Leu Gln Pro Ser Arg
195 200 205
Thr Ser Pro Gln Ala Pro Arg Ala Arg Ala Trp Ala Pro Ser Gln Ala
210 215 220
<210> 10
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 10
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser
<210> 11
<211> 382
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 11
Met Gly Trp Ser Cys Ile Ile Leu Phe Leu Val Ala Thr Ala Thr Gly
1 5 10 15
Val His Ser Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr
20 25 30
Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser
35 40 45
Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly
50 55 60
Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala
65 70 75 80
Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile
85 90 95
Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys
100 105 110
Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser
115 120 125
Leu Pro Pro Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala
130 135 140
Pro Ile Leu Arg Ala Asp Glu Pro Lys Ser Ser Asp Lys Thr His Thr
145 150 155 160
Ser Pro Pro Ser Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe
165 170 175
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
180 185 190
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
195 200 205
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
210 215 220
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
225 230 235 240
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
245 250 255
Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
260 265 270
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
275 280 285
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
290 295 300
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
305 310 315 320
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
325 330 335
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
340 345 350
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
355 360 365
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375 380
<210> 12
<211> 479
<212> PRT
<213> Homo sapiens
<400> 12
Met Ala Arg Ala Ala Ala Leu Leu Pro Ser Arg Ser Pro Pro Thr Pro
1 5 10 15
Leu Leu Trp Pro Leu Leu Leu Leu Leu Leu Leu Glu Thr Gly Ala Gln
20 25 30
Asp Val Arg Val Gln Val Leu Pro Glu Val Arg Gly Gln Leu Gly Gly
35 40 45
Thr Val Glu Leu Pro Cys His Leu Leu Pro Pro Val Pro Gly Leu Tyr
50 55 60
Ile Ser Leu Val Thr Trp Gln Arg Pro Asp Ala Pro Ala Asn His Gln
65 70 75 80
Asn Val Ala Ala Phe His Pro Lys Met Gly Pro Ser Phe Pro Ser Pro
85 90 95
Lys Pro Gly Ser Glu Arg Leu Ser Phe Val Ser Ala Lys Gln Ser Thr
100 105 110
Gly Gln Asp Thr Glu Ala Glu Leu Gln Asp Ala Thr Leu Ala Leu His
115 120 125
Gly Leu Thr Val Glu Asp Glu Gly Asn Tyr Thr Cys Glu Phe Ala Thr
130 135 140
Phe Pro Lys Gly Ser Val Arg Gly Met Thr Trp Leu Arg Val Ile Ala
145 150 155 160
Lys Pro Lys Asn Gln Ala Glu Ala Gln Lys Val Thr Phe Ser Gln Asp
165 170 175
Pro Thr Thr Val Ala Leu Cys Ile Ser Lys Glu Gly Arg Pro Pro Ala
180 185 190
Arg Ile Ser Trp Leu Ser Ser Leu Asp Trp Glu Ala Lys Glu Thr Gln
195 200 205
Val Ser Gly Thr Leu Ala Gly Thr Val Thr Val Thr Ser Arg Phe Thr
210 215 220
Leu Val Pro Ser Gly Arg Ala Asp Gly Val Thr Val Thr Cys Lys Val
225 230 235 240
Glu His Glu Ser Phe Glu Glu Pro Ala Leu Ile Pro Val Thr Leu Ser
245 250 255
Val Arg Tyr Pro Pro Glu Val Ser Ile Ser Gly Tyr Asp Asp Asn Trp
260 265 270
Tyr Leu Gly Arg Thr Asp Ala Thr Leu Ser Cys Asp Val Arg Ser Asn
275 280 285
Pro Glu Pro Thr Gly Tyr Asp Trp Ser Thr Thr Ser Gly Thr Phe Pro
290 295 300
Thr Ser Ala Val Ala Gln Gly Ser Gln Leu Val Ile His Ala Val Asp
305 310 315 320
Ser Leu Phe Asn Thr Thr Phe Val Cys Thr Val Thr Asn Ala Val Gly
325 330 335
Met Gly Arg Ala Glu Gln Val Ile Phe Val Arg Glu Thr Pro Arg Ala
340 345 350
Ser Pro Arg Asp Val Gly Pro Leu Val Trp Gly Ala Val Gly Gly Thr
355 360 365
Leu Leu Val Leu Leu Leu Leu Ala Gly Gly Ser Leu Ala Phe Ile Leu
370 375 380
Leu Arg Val Arg Arg Arg Arg Lys Ser Pro Gly Gly Ala Gly Gly Gly
385 390 395 400
Ala Ser Gly Asp Gly Gly Phe Tyr Asp Pro Lys Ala Gln Val Leu Gly
405 410 415
Asn Gly Asp Pro Val Phe Trp Thr Pro Val Val Pro Gly Pro Met Glu
420 425 430
Pro Asp Gly Lys Asp Glu Glu Glu Glu Glu Glu Glu Glu Lys Ala Glu
435 440 445
Lys Gly Leu Met Leu Pro Pro Pro Pro Ala Leu Glu Asp Asp Met Glu
450 455 460
Ser Gln Leu Asp Gly Ser Leu Ile Ser Arg Arg Ala Val Tyr Val
465 470 475
<210> 13
<211> 518
<212> PRT
<213> Homo sapiens
<400> 13
Met Ala Arg Ala Ala Ala Leu Leu Pro Ser Arg Ser Pro Pro Thr Pro
1 5 10 15
Leu Leu Trp Pro Leu Leu Leu Leu Leu Leu Leu Glu Thr Gly Ala Gln
20 25 30
Asp Val Arg Val Gln Val Leu Pro Glu Val Arg Gly Gln Leu Gly Gly
35 40 45
Thr Val Glu Leu Pro Cys His Leu Leu Pro Pro Val Pro Gly Leu Tyr
50 55 60
Ile Ser Leu Val Thr Trp Gln Arg Pro Asp Ala Pro Ala Asn His Gln
65 70 75 80
Asn Val Ala Ala Phe His Pro Lys Met Gly Pro Ser Phe Pro Ser Pro
85 90 95
Lys Pro Gly Ser Glu Arg Leu Ser Phe Val Ser Ala Lys Gln Ser Thr
100 105 110
Gly Gln Asp Thr Glu Ala Glu Leu Gln Asp Ala Thr Leu Ala Leu His
115 120 125
Gly Leu Thr Val Glu Asp Glu Gly Asn Tyr Thr Cys Glu Phe Ala Thr
130 135 140
Phe Pro Lys Gly Ser Val Arg Gly Met Thr Trp Leu Arg Val Ile Ala
145 150 155 160
Lys Pro Lys Asn Gln Ala Glu Ala Gln Lys Val Thr Phe Ser Gln Asp
165 170 175
Pro Thr Thr Val Ala Leu Cys Ile Ser Lys Glu Gly Arg Pro Pro Ala
180 185 190
Arg Ile Ser Trp Leu Ser Ser Leu Asp Trp Glu Ala Lys Glu Thr Gln
195 200 205
Val Ser Gly Thr Leu Ala Gly Thr Val Thr Val Thr Ser Arg Phe Thr
210 215 220
Leu Val Pro Ser Gly Arg Ala Asp Gly Val Thr Val Thr Cys Lys Val
225 230 235 240
Glu His Glu Ser Phe Glu Glu Pro Ala Leu Ile Pro Val Thr Leu Ser
245 250 255
Val Arg Tyr Pro Pro Glu Val Ser Ile Ser Gly Tyr Asp Asp Asn Trp
260 265 270
Tyr Leu Gly Arg Thr Asp Ala Thr Leu Ser Cys Asp Val Arg Ser Asn
275 280 285
Pro Glu Pro Thr Gly Tyr Asp Trp Ser Thr Thr Ser Gly Thr Phe Pro
290 295 300
Thr Ser Ala Val Ala Gln Gly Ser Gln Leu Val Ile His Ala Val Asp
305 310 315 320
Ser Leu Phe Asn Thr Thr Phe Val Cys Thr Val Thr Asn Ala Val Gly
325 330 335
Met Gly Arg Ala Glu Gln Val Ile Phe Val Arg Glu Thr Pro Asn Thr
340 345 350
Ala Gly Ala Gly Ala Thr Gly Gly Ile Ile Gly Gly Ile Ile Ala Ala
355 360 365
Ile Ile Ala Thr Ala Val Ala Ala Thr Gly Ile Leu Ile Cys Arg Gln
370 375 380
Gln Arg Lys Glu Gln Thr Leu Gln Gly Ala Glu Glu Asp Glu Asp Leu
385 390 395 400
Glu Gly Pro Pro Ser Tyr Lys Pro Pro Thr Pro Lys Ala Lys Leu Glu
405 410 415
Ala Gln Glu Met Pro Ser Gln Leu Phe Thr Leu Gly Ala Ser Glu His
420 425 430
Ser Pro Leu Lys Thr Pro Tyr Phe Asp Ala Gly Ala Ser Cys Thr Glu
435 440 445
Gln Glu Met Pro Arg Tyr His Glu Leu Pro Thr Leu Glu Glu Arg Ser
450 455 460
Gly Pro Leu His Pro Gly Ala Thr Ser Leu Gly Ser Pro Ile Pro Val
465 470 475 480
Pro Pro Gly Pro Pro Ala Val Glu Asp Val Ser Leu Asp Leu Glu Asp
485 490 495
Glu Glu Gly Glu Glu Glu Glu Glu Tyr Leu Asp Lys Ile Asn Pro Ile
500 505 510
Tyr Asp Ala Leu Ser Tyr
515
<210> 14
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 14
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 15
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 15
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 16
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 16
Ala Arg Glu Glu Val Ser Ser Pro Tyr Gly Met Asp Val
1 5 10
<210> 17
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 17
Thr Gly Ala Val Thr Ser Gly His Tyr
1 5
<210> 18
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 18
Asp Thr Gly
1
<210> 19
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 19
Leu Leu Ser Tyr Ser Gly Ala Ser Trp Val
1 5 10
<210> 20
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 20
Gly Phe Ser Leu Ser His Phe Ser
1 5
<210> 21
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 21
Phe Asp Pro Glu Glu Gly Gly Thr
1 5
<210> 22
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 22
Ala Thr Gly Ile Trp Tyr Ser Ser Gly Trp Pro Val Asp Tyr
1 5 10
<210> 23
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 23
Gln Ser Leu Leu Asp Ser Ser Gly Tyr Asn Tyr
1 5 10
<210> 24
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 24
Leu Gly Ser
1
<210> 25
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 25
Met Gln Ala Leu Gln Thr Pro Ile Thr
1 5
<210> 26
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 26
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 27
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 27
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 28
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 28
Ala Thr Val Ser Arg Val Arg Gly Val Ile Asn Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210> 29
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 29
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 30
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 30
Leu Gly Ser
1
<210> 31
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 31
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 32
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 32
Gly Gly Thr Phe Gly Thr Tyr Ala
1 5
<210> 33
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 33
Ile Thr Pro Ile Ser Ala Thr Ile
1 5
<210> 34
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 34
Ala Arg Gly Phe Glu Tyr Ser Asp Gly Leu Leu Asp Asp
1 5 10
<210> 35
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 35
Gln Ser Leu Phe Tyr Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 36
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 36
Arg Leu Ser
1
<210> 37
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 37
Met Gln His Met Glu Phe Pro Leu Thr
1 5
<210> 38
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 38
Ser Gly Ser Ile Ser Ser Thr Asn Trp
1 5
<210> 39
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 39
Ile Tyr His Ser Gly Ser Thr
1 5
<210> 40
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 40
Ala Arg Val Gly Pro Ala Ala Ile Tyr Tyr
1 5 10
<210> 41
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 41
Ser Asn Asn Val Gly Tyr Glu Gly
1 5
<210> 42
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 42
Arg Asn Asn
1
<210> 43
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 43
Ser Ala Trp Asp Ser Ser Leu Asn Ala Val Val
1 5 10
<210> 44
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 44
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 45
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 45
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 46
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 46
Ala Thr Ala Lys Pro Gly Ile Ala Val Ala Gly Gln Asn Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Met Asp Val
20
<210> 47
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 47
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 48
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 48
Leu Gly Ser
1
<210> 49
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 49
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 50
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 50
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 51
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 51
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 52
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 52
Ala Ser Ser Pro Ile Gly Tyr Ser Tyr Gly Tyr Trp Gly Gly Met Asp
1 5 10 15
Val
<210> 53
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 53
Ser Gly Ile Asp Val Arg Thr Asn Lys
1 5
<210> 54
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 54
Phe Gln Ser Asp Ser Asp Lys
1 5
<210> 55
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 55
Leu Ile Trp His Thr Ser Gly Trp Val
1 5
<210> 56
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 56
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 57
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 57
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 58
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 58
Ala Thr Gly Pro Ala Ala Ala Gly Val Gly Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 59
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 59
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 60
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 60
Leu Gly Ser
1
<210> 61
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 61
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 62
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 62
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 63
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 63
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 64
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 64
Ala Arg Asp Val Met Val Tyr Cys Ser Ser Thr Ser Cys Tyr Phe Tyr
1 5 10 15
Gly Met Asp Val
20
<210> 65
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 65
Gln Asp Ile Arg Asp Tyr
1 5
<210> 66
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 66
Asp Ala Ser
1
<210> 67
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 67
Gln Gln Phe Glu Asn Leu Pro Ile Thr
1 5
<210> 68
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 68
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 69
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 69
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 70
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 70
Ala Thr Gly Gly Tyr Ser Ser Gly Phe Asn Tyr Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210> 71
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 71
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 72
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 72
Leu Gly Ser
1
<210> 73
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 73
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 74
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 74
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 75
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 75
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 76
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 76
Ala Thr Gly Val Thr Thr Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10
<210> 77
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 77
Gln Ser Leu Leu Tyr Ser Asn Gly Asn Asn Phe
1 5 10
<210> 78
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 78
Leu Gly Ser
1
<210> 79
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 79
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 80
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 80
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 81
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 81
Ile Arg Tyr Asp Gly Ser Asn Lys
1 5
<210> 82
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 82
Ala Arg Asp Leu Phe Asp Phe Trp Trp Asp Gly Met Asp Val
1 5 10
<210> 83
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 83
Gln Ser Val Ser Ser Met Tyr
1 5
<210> 84
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 84
Gly Ala Ser
1
<210> 85
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 85
Gln Gln Tyr Val Ser Ser Pro Met Tyr Thr
1 5 10
<210> 86
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 86
Gly Gly Thr Phe Asn Asn Tyr Gly
1 5
<210> 87
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 87
Ile Ile Pro Leu Phe Gly Thr Thr
1 5
<210> 88
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 88
Ala Arg Asp Arg Met Ala Ala Asp Gly Met Ala Val Phe Asp Tyr
1 5 10 15
<210> 89
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 89
Ser Ser Asn Ile Gly Arg His Phe
1 5
<210> 90
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 90
Lys Asn Asp
1
<210> 91
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 91
Ser Ser Trp Asp Ala Ala Leu Asn Gly Val Val
1 5 10
<210> 92
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 92
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 93
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 93
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 94
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 94
Ala Thr Glu Val Pro Met Val Arg Gly Ala Arg Arg Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 95
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 95
Gln Thr Leu Leu Tyr Ile Asn Glu Asn Asn Tyr
1 5 10
<210> 96
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 96
Leu Gly Ser
1
<210> 97
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 97
Met Gln Gly Leu Gln Thr Pro Pro Thr
1 5
<210> 98
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 98
Gly Gly Ser Ile Ser Asn Ser Asn Tyr Tyr
1 5 10
<210> 99
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 99
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 100
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 100
Ala Arg Gly Ala Trp Glu Leu Ser Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 101
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 101
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 102
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 102
Gly Asn Asn
1
<210> 103
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 103
Gln Ser Tyr Asp Ser Ser Leu Ser Val Tyr Val Val
1 5 10
<210> 104
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 104
Gly Phe Thr Phe Gly Thr Ser Ser
1 5
<210> 105
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 105
Ile Ser Phe Asp Gly Thr Glu Ile
1 5
<210> 106
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 106
Ala Lys Gly Ser Gly Asn Ile Tyr Phe Tyr Ser Gly Met Asp Val
1 5 10 15
<210> 107
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 107
Gln Ser Ile Ser Gly Trp
1 5
<210> 108
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 108
Glu Thr Ser
1
<210> 109
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 109
Gln Gln Tyr Tyr Ser Tyr Pro Leu Thr
1 5
<210> 110
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 110
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 111
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 111
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 112
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 112
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 113
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 113
Gln Ser Leu Leu Tyr Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 114
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 114
Leu Gly Ser
1
<210> 115
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 115
Met Gln Ala Leu Gln Ser Pro Val Thr
1 5
<210> 116
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 116
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 117
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 117
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 118
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 118
Ala Thr Asp Ser Arg Asp Gly Pro Ala Ala Arg Gly Gly Tyr Tyr Tyr
1 5 10 15
Tyr Met Asp Val
20
<210> 119
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 119
Gln Ser Leu Leu Tyr Ile Asn Gly Tyr Asn Tyr
1 5 10
<210> 120
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 120
Leu Gly Ser
1
<210> 121
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 121
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 122
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 122
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 123
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 123
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 124
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 124
Ala Arg Asp Ala Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr Asn Pro Asp
1 5 10 15
Ala Phe Asp Ile
20
<210> 125
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 125
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 126
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 126
Leu Gly Ser
1
<210> 127
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 127
Met Gln Gly Leu Gln Thr Pro Arg Thr
1 5
<210> 128
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 128
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 129
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 129
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 130
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 130
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 131
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 131
Gln Asp Ile Asp Asp Asp
1 5
<210> 132
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 132
Glu Ala Ser
1
<210> 133
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 133
Leu Gln His Asp Asn Leu Pro Leu Thr
1 5
<210> 134
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 134
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 135
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 135
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 136
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 136
Ala Thr Glu Asp Pro Gly Pro Val Ala Gly Pro Tyr Tyr Tyr Tyr Gly
1 5 10 15
Met Asp Val
<210> 137
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 137
Gln Ser Leu Leu Tyr Ile Asn Gly Tyr His Tyr
1 5 10
<210> 138
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 138
Leu Gly Ser
1
<210> 139
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 139
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 140
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 140
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 141
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 141
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 142
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 142
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 143
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 143
Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 144
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 144
Thr Leu Ser
1
<210> 145
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 145
Met Gln Arg Leu Gln Phe Pro Leu Thr
1 5
<210> 146
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 146
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 147
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 147
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 148
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 148
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 149
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 149
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 150
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 150
Trp Gly Ser
1
<210> 151
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 151
Met Gln Ala Val Gln Asn Pro Pro Thr
1 5
<210> 152
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 152
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 153
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 153
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 154
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 154
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 155
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 155
Gln Thr Met Asn Asn Tyr
1 5
<210> 156
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 156
Asp Ala Ser
1
<210> 157
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 157
Gln Gln Tyr Gly Asp Trp Leu Pro Ile Thr
1 5 10
<210> 158
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 158
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 159
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 159
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 160
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 160
Ala Thr Ala Phe Pro Glu Ala Thr Ile Ser Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 161
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 161
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 162
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 162
Trp Gly Ser
1
<210> 163
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 163
Met Gln Ala Val Gln Asn Pro Pro Thr
1 5
<210> 164
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 164
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 165
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 165
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 166
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 166
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 167
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 167
Arg Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr His
1 5 10
<210> 168
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 168
Ser Leu Ser
1
<210> 169
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 169
Met Gln Arg Lys Glu Phe Pro Leu Thr
1 5
<210> 170
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 170
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 171
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 171
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 172
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 172
Ala Arg Gly Pro Trp Tyr Tyr Asp Ser Ser Gly Tyr Ser Ser Tyr Ala
1 5 10 15
Tyr Tyr Met Asp Val
20
<210> 173
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 173
Gln Ser Leu Leu His Ser Asp Gly Tyr Asn Tyr
1 5 10
<210> 174
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 174
Leu Gly Ser
1
<210> 175
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 175
Met Gln Ala Leu His Thr Pro Gly Val Thr
1 5 10
<210> 176
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 176
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Glu Val Ser Ser Pro Tyr Gly Met Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 177
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 177
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 178
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 178
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 179
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 179
Ala Arg Glu Glu Val Ser Ser Pro Tyr Gly Met Asp Val
1 5 10
<210> 180
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 180
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Glu Val Ser Ser Pro Tyr Gly Met Asp Val Trp Gly Gln
100 105 110
Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 181
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 181
Gln Ala Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Thr Ser Thr Gly Ala Val Thr Ser Gly
20 25 30
His Tyr Pro Tyr Trp Phe Gln Gln Lys Pro Gly Gln Ala Pro Lys Thr
35 40 45
Leu Ile Tyr Asp Thr Gly Asn Lys His Ser Trp Thr Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Ala
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Leu Leu Ser Tyr Ser Gly
85 90 95
Ala Ser Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 182
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 182
Thr Gly Ala Val Thr Ser Gly His Tyr
1 5
<210> 183
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 183
Asp Thr Gly
1
<210> 184
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 184
Leu Leu Ser Tyr Ser Gly Ala Ser Trp Val
1 5 10
<210> 185
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 185
Gln Ala Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Thr Ser Thr Gly Ala Val Thr Ser Gly
20 25 30
His Tyr Pro Tyr Trp Phe Gln Gln Lys Pro Gly Gln Ala Pro Lys Thr
35 40 45
Leu Ile Tyr Asp Thr Gly Asn Lys His Ser Trp Thr Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Ala
65 70 75 80
Gln Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Leu Leu Ser Tyr Ser Gly
85 90 95
Ala Ser Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 186
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 186
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Phe Ser Leu Ser His Phe
20 25 30
Ser Met His Trp Val Arg Gln Val Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Gly Phe Asp Pro Glu Glu Gly Gly Thr Ile Pro Ala Gln Lys Phe
50 55 60
Gln Gly Arg Leu Thr Met Thr Glu Asp Thr Ser Thr Glu Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Ile Trp Tyr Ser Ser Gly Trp Pro Val Asp Tyr Trp Gly
100 105 110
Pro Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 187
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 187
Gly Phe Ser Leu Ser His Phe Ser
1 5
<210> 188
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 188
Phe Asp Pro Glu Glu Gly Gly Thr
1 5
<210> 189
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 189
Ala Thr Gly Ile Trp Tyr Ser Ser Gly Trp Pro Val Asp Tyr
1 5 10
<210> 190
<211> 458
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 190
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro Trp Tyr Tyr Asp Ser Ser Gly Tyr Ser Ser Tyr Ala
100 105 110
Tyr Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
130 135 140
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
145 150 155 160
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
165 170 175
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
180 185 190
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
195 200 205
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
210 215 220
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
225 230 235 240
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
245 250 255
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
260 265 270
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
275 280 285
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
290 295 300
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
305 310 315 320
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
325 330 335
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
340 345 350
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu
355 360 365
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
370 375 380
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
385 390 395 400
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
405 410 415
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
420 425 430
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
435 440 445
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 191
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 191
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Gly Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser
20 25 30
Ser Gly Tyr Asn Tyr Val Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Ser Leu Gly Ser Asp Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
Arg
<210> 192
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 192
Gln Ser Leu Leu Asp Ser Ser Gly Tyr Asn Tyr
1 5 10
<210> 193
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 193
Leu Gly Ser
1
<210> 194
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 194
Met Gln Ala Leu Gln Thr Pro Ile Thr
1 5
<210> 195
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 195
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Gly Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Asp Ser
20 25 30
Ser Gly Tyr Asn Tyr Val Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Ser Leu Gly Ser Asp Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 196
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 196
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Val Ser Arg Val Arg Gly Val Ile Asn Tyr Tyr Tyr Tyr Met
100 105 110
Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 197
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 197
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 198
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 198
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 199
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 199
Ala Thr Val Ser Arg Val Arg Gly Val Ile Asn Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210> 200
<211> 455
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 200
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Val Ser Arg Val Arg Gly Val Ile Asn Tyr Tyr Tyr Tyr Met
100 105 110
Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
130 135 140
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
195 200 205
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
210 215 220
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 201
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 201
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Asn Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg
<210> 202
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 202
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 203
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 203
Leu Gly Ser
1
<210> 204
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 204
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 205
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 205
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Asn Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 206
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 206
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Arg Arg Pro Gly Ser
1 5 10 15
Ser Val Arg Val Ser Cys Lys Pro Ser Gly Gly Thr Phe Gly Thr Tyr
20 25 30
Ala Phe Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Thr Pro Ile Ser Ala Thr Ile Asn Arg Ala Gln Asn Leu
50 55 60
Gln Asp Arg Leu Thr Ile Thr Ala Asp Glu Ser Thr Thr Thr Val His
65 70 75 80
Met Asp Leu Thr Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Phe Glu Tyr Ser Asp Gly Leu Leu Asp Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 207
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 207
Gly Gly Thr Phe Gly Thr Tyr Ala
1 5
<210> 208
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 208
Ile Thr Pro Ile Ser Ala Thr Ile
1 5
<210> 209
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 209
Ala Arg Gly Phe Glu Tyr Ser Asp Gly Leu Leu Asp Asp
1 5 10
<210> 210
<211> 450
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 210
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Arg Arg Pro Gly Ser
1 5 10 15
Ser Val Arg Val Ser Cys Lys Pro Ser Gly Gly Thr Phe Gly Thr Tyr
20 25 30
Ala Phe Thr Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Thr Pro Ile Ser Ala Thr Ile Asn Arg Ala Gln Asn Leu
50 55 60
Gln Asp Arg Leu Thr Ile Thr Ala Asp Glu Ser Thr Thr Thr Val His
65 70 75 80
Met Asp Leu Thr Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Phe Glu Tyr Ser Asp Gly Leu Leu Asp Asp Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val
115 120 125
Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala
130 135 140
Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser
145 150 155 160
Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val
165 170 175
Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro
180 185 190
Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys
195 200 205
Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp
210 215 220
Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly
225 230 235 240
Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile
245 250 255
Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu
260 265 270
Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His
275 280 285
Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg
290 295 300
Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys
305 310 315 320
Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu
325 330 335
Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr
340 345 350
Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu
355 360 365
Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp
370 375 380
Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val
385 390 395 400
Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp
405 410 415
Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His
420 425 430
Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro
435 440 445
Gly Lys
450
<210> 211
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 211
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ile Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Phe Tyr Ser
20 25 30
Asp Asp Gly Asn Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Gln Leu Leu Ile Tyr Arg Leu Ser His Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys
65 70 75 80
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln
85 90 95
His Met Glu Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg
<210> 212
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 212
Gln Ser Leu Phe Tyr Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 213
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 213
Arg Leu Ser
1
<210> 214
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 214
Met Gln His Met Glu Phe Pro Leu Thr
1 5
<210> 215
<211> 220
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 215
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ile Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Phe Tyr Ser
20 25 30
Asp Asp Gly Asn Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Gln Leu Leu Ile Tyr Arg Leu Ser His Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys
65 70 75 80
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln
85 90 95
His Met Glu Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 216
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 216
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gly
1 5 10 15
Thr Leu Ser Leu Thr Cys Val Val Ser Ser Gly Ser Ile Ser Ser Thr
20 25 30
Asn Trp Trp Thr Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Tyr His Ser Gly Ser Thr Ser Tyr Asn Ser Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Glu Asp Lys Ser Lys Asn Gln Ile Ser
65 70 75 80
Leu Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Gly Pro Ala Ala Ile Tyr Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 217
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 217
Ser Gly Ser Ile Ser Ser Thr Asn Trp
1 5
<210> 218
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 218
Ile Tyr His Ser Gly Ser Thr
1 5
<210> 219
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 219
Ala Arg Val Gly Pro Ala Ala Ile Tyr Tyr
1 5 10
<210> 220
<211> 447
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 220
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gly
1 5 10 15
Thr Leu Ser Leu Thr Cys Val Val Ser Ser Gly Ser Ile Ser Ser Thr
20 25 30
Asn Trp Trp Thr Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Glu Ile Tyr His Ser Gly Ser Thr Ser Tyr Asn Ser Ser Leu
50 55 60
Lys Ser Arg Val Thr Ile Ser Glu Asp Lys Ser Lys Asn Gln Ile Ser
65 70 75 80
Leu Arg Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Val Gly Pro Ala Ala Ile Tyr Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
115 120 125
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
130 135 140
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
145 150 155 160
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
165 170 175
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
180 185 190
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
195 200 205
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
210 215 220
Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val
225 230 235 240
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
245 250 255
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
260 265 270
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
275 280 285
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
290 295 300
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
305 310 315 320
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
325 330 335
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
340 345 350
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
355 360 365
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
370 375 380
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
385 390 395 400
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
405 410 415
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
420 425 430
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
435 440 445
<210> 221
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 221
Gln Ala Gly Leu Thr Gln Pro Pro Ser Val Ser Lys Gly Leu Arg Gln
1 5 10 15
Thr Ala Thr Leu Thr Cys Thr Gly Asn Ser Asn Asn Val Gly Tyr Glu
20 25 30
Gly Ala Ala Trp Leu Gln Gln His Gln Gly His Ala Pro Lys Leu Leu
35 40 45
Leu Tyr Arg Asn Asn Asn Arg Pro Ser Gly Ile Ser Lys Arg Phe Ser
50 55 60
Ala Ser Arg Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Leu Gln
65 70 75 80
Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ala Trp Asp Ser Ser Leu
85 90 95
Asn Ala Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 222
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 222
Ser Asn Asn Val Gly Tyr Glu Gly
1 5
<210> 223
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 223
Arg Asn Asn
1
<210> 224
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 224
Ser Ala Trp Asp Ser Ser Leu Asn Ala Val Val
1 5 10
<210> 225
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 225
Gln Ala Gly Leu Thr Gln Pro Pro Ser Val Ser Lys Gly Leu Arg Gln
1 5 10 15
Thr Ala Thr Leu Thr Cys Thr Gly Asn Ser Asn Asn Val Gly Tyr Glu
20 25 30
Gly Ala Ala Trp Leu Gln Gln His Gln Gly His Ala Pro Lys Leu Leu
35 40 45
Leu Tyr Arg Asn Asn Asn Arg Pro Ser Gly Ile Ser Lys Arg Phe Ser
50 55 60
Ala Ser Arg Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Leu Gln
65 70 75 80
Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ala Trp Asp Ser Ser Leu
85 90 95
Asn Ala Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 226
<211> 128
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 226
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Ala Lys Pro Gly Ile Ala Val Ala Gly Gln Asn Tyr Tyr Tyr
100 105 110
Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 227
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 227
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 228
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 228
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 229
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 229
Ala Thr Ala Lys Pro Gly Ile Ala Val Ala Gly Gln Asn Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Met Asp Val
20
<210> 230
<211> 458
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 230
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Ala Lys Pro Gly Ile Ala Val Ala Gly Gln Asn Tyr Tyr Tyr
100 105 110
Tyr Tyr Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
130 135 140
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
145 150 155 160
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
165 170 175
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
180 185 190
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
195 200 205
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
210 215 220
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
225 230 235 240
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
245 250 255
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
260 265 270
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
275 280 285
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
290 295 300
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
305 310 315 320
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
325 330 335
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
340 345 350
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu
355 360 365
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
370 375 380
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
385 390 395 400
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
405 410 415
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
420 425 430
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
435 440 445
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 231
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 231
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Asn Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg
<210> 232
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 232
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 233
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 233
Leu Gly Ser
1
<210> 234
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 234
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 235
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 235
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Asn Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 236
<211> 124
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 236
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Ser Pro Ile Gly Tyr Ser Tyr Gly Tyr Trp Gly Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 237
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 237
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 238
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 238
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 239
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 239
Ala Ser Ser Pro Ile Gly Tyr Ser Tyr Gly Tyr Trp Gly Gly Met Asp
1 5 10 15
Val
<210> 240
<211> 454
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 240
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Ser Pro Ile Gly Tyr Ser Tyr Gly Tyr Trp Gly Gly Met Asp
100 105 110
Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 241
<211> 116
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 241
Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser Ala Ser Pro Gly Ala
1 5 10 15
Ser Ala Ser Leu Thr Cys Thr Leu Arg Ser Gly Ile Asp Val Arg Thr
20 25 30
Asn Lys Ile Phe Trp Tyr Gln Val Lys Pro Gly Ser Pro Pro Gln His
35 40 45
Leu Leu Thr Phe Gln Ser Asp Ser Asp Lys Gln Gln Gly Ser Gly Val
50 55 60
Pro Ser Arg Phe Ser Gly Ser Lys Asp Ala Ser Ala Asn Ala Gly Ile
65 70 75 80
Leu Ile Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys
85 90 95
Leu Ile Trp His Thr Ser Gly Trp Val Phe Gly Gly Gly Thr Gln Leu
100 105 110
Thr Val Leu Gly
115
<210> 242
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 242
Ser Gly Ile Asp Val Arg Thr Asn Lys
1 5
<210> 243
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 243
Phe Gln Ser Asp Ser Asp Lys
1 5
<210> 244
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 244
Leu Ile Trp His Thr Ser Gly Trp Val
1 5
<210> 245
<211> 221
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 245
Gln Ala Val Leu Thr Gln Pro Ala Ser Leu Ser Ala Ser Pro Gly Ala
1 5 10 15
Ser Ala Ser Leu Thr Cys Thr Leu Arg Ser Gly Ile Asp Val Arg Thr
20 25 30
Asn Lys Ile Phe Trp Tyr Gln Val Lys Pro Gly Ser Pro Pro Gln His
35 40 45
Leu Leu Thr Phe Gln Ser Asp Ser Asp Lys Gln Gln Gly Ser Gly Val
50 55 60
Pro Ser Arg Phe Ser Gly Ser Lys Asp Ala Ser Ala Asn Ala Gly Ile
65 70 75 80
Leu Ile Ile Ser Gly Leu Gln Ser Glu Asp Glu Ala Asp Tyr Tyr Cys
85 90 95
Leu Ile Trp His Thr Ser Gly Trp Val Phe Gly Gly Gly Thr Gln Leu
100 105 110
Thr Val Leu Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro
115 120 125
Pro Ser Ser Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu
130 135 140
Ile Ser Asp Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp
145 150 155 160
Ser Ser Pro Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln
165 170 175
Ser Asn Asn Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu
180 185 190
Gln Trp Lys Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly
195 200 205
Ser Thr Val Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215 220
<210> 246
<211> 124
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 246
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Pro Ala Ala Ala Gly Val Gly Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 247
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 247
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 248
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 248
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 249
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 249
Ala Thr Gly Pro Ala Ala Ala Gly Val Gly Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 250
<211> 454
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 250
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Pro Ala Ala Ala Gly Val Gly Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 251
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 251
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg
<210> 252
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 252
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 253
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 253
Leu Gly Ser
1
<210> 254
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 254
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 255
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 255
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 256
<211> 127
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 256
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Val Met Val Tyr Cys Ser Ser Thr Ser Cys Tyr Phe Tyr
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 257
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 257
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 258
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 258
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 259
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 259
Ala Arg Asp Val Met Val Tyr Cys Ser Ser Thr Ser Cys Tyr Phe Tyr
1 5 10 15
Gly Met Asp Val
20
<210> 260
<211> 457
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 260
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Val Met Val Tyr Cys Ser Ser Thr Ser Cys Tyr Phe Tyr
100 105 110
Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 261
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 261
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Val Thr Cys Gln Ala Ser Gln Asp Ile Arg Asp Tyr
20 25 30
Leu Asn Trp Tyr Gln Lys Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Ala Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Gly Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Gln Phe Glu Asn Leu Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg
100 105
<210> 262
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 262
Gln Asp Ile Arg Asp Tyr
1 5
<210> 263
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 263
Asp Ala Ser
1
<210> 264
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 264
Gln Gln Phe Glu Asn Leu Pro Ile Thr
1 5
<210> 265
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 265
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Val Thr Cys Gln Ala Ser Gln Asp Ile Arg Asp Tyr
20 25 30
Leu Asn Trp Tyr Gln Lys Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Leu Glu Ala Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Gly Leu Gln Pro
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Gln Phe Glu Asn Leu Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 266
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 266
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Gly Tyr Ser Ser Gly Phe Asn Tyr Tyr Tyr Tyr Tyr Met
100 105 110
Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 267
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 267
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 268
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 268
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 269
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 269
Ala Thr Gly Gly Tyr Ser Ser Gly Phe Asn Tyr Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210> 270
<211> 455
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 270
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Gly Tyr Ser Ser Gly Phe Asn Tyr Tyr Tyr Tyr Tyr Met
100 105 110
Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
115 120 125
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
130 135 140
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
145 150 155 160
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
165 170 175
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
180 185 190
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
195 200 205
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
210 215 220
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
325 330 335
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 271
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 271
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Asn Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg
<210> 272
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 272
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 273
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 273
Leu Gly Ser
1
<210> 274
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 274
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 275
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 275
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Asn Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 276
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 276
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Thr Thr Tyr Tyr Tyr Tyr Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 277
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 277
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 278
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 278
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 279
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 279
Ala Thr Gly Val Thr Thr Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10
<210> 280
<211> 451
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 280
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Thr Thr Tyr Tyr Tyr Tyr Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 281
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 281
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Asn Asn Phe Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Arg Leu Leu Ile Ser Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg
<210> 282
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 282
Gln Ser Leu Leu Tyr Ser Asn Gly Asn Asn Phe
1 5 10
<210> 283
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 283
Leu Gly Ser
1
<210> 284
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 284
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 285
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 285
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Asn Asn Phe Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Arg Leu Leu Ile Ser Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 286
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 286
Gln Ile Thr Leu Lys Glu Ser Gly Gly Gly Val Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Phe Ile Arg Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Phe Asp Phe Trp Trp Asp Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 287
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 287
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 288
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 288
Ile Arg Tyr Asp Gly Ser Asn Lys
1 5
<210> 289
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 289
Ala Arg Asp Leu Phe Asp Phe Trp Trp Asp Gly Met Asp Val
1 5 10
<210> 290
<211> 451
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 290
Gln Ile Thr Leu Lys Glu Ser Gly Gly Gly Val Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Phe Ile Arg Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Leu Phe Asp Phe Trp Trp Asp Gly Met Asp Val Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu Gly
225 230 235 240
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
245 250 255
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
260 265 270
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
275 280 285
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
290 295 300
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
305 310 315 320
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
325 330 335
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
340 345 350
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
355 360 365
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
370 375 380
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
385 390 395 400
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
405 410 415
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
420 425 430
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
435 440 445
Pro Gly Lys
450
<210> 291
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 291
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Lys Ala Thr Leu Ser Cys Arg Val Ser Gln Ser Val Ser Ser Met
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Tyr Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Asn Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Val Ser Ser Pro
85 90 95
Met Tyr Thr Phe Gly Leu Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
<210> 292
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 292
Gln Ser Val Ser Ser Met Tyr
1 5
<210> 293
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 293
Gly Ala Ser
1
<210> 294
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 294
Gln Gln Tyr Val Ser Ser Pro Met Tyr Thr
1 5 10
<210> 295
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 295
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Lys Ala Thr Leu Ser Cys Arg Val Ser Gln Ser Val Ser Ser Met
20 25 30
Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu
35 40 45
Ile Tyr Gly Ala Ser Tyr Arg Ala Thr Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Asn Leu Thr Ile Ser Arg Leu Glu
65 70 75 80
Pro Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Val Ser Ser Pro
85 90 95
Met Tyr Thr Phe Gly Leu Gly Thr Lys Leu Glu Ile Lys Arg Thr Val
100 105 110
Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys
115 120 125
Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg
130 135 140
Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn
145 150 155 160
Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser
165 170 175
Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys
180 185 190
Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr
195 200 205
Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 296
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 296
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Asn Asn Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gln Ile Ile Pro Leu Phe Gly Thr Thr Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Ala Ala Asp Glu Pro Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Gly Ser Leu Arg Ser Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Met Ala Ala Asp Gly Met Ala Val Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 297
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 297
Gly Gly Thr Phe Asn Asn Tyr Gly
1 5
<210> 298
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 298
Ile Ile Pro Leu Phe Gly Thr Thr
1 5
<210> 299
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 299
Ala Arg Asp Arg Met Ala Ala Asp Gly Met Ala Val Phe Asp Tyr
1 5 10 15
<210> 300
<211> 452
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 300
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Asn Asn Tyr
20 25 30
Gly Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gln Ile Ile Pro Leu Phe Gly Thr Thr Lys Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Ala Ala Asp Glu Pro Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Gly Ser Leu Arg Ser Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Met Ala Ala Asp Gly Met Ala Val Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 301
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 301
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Thr Pro Gly Gln
1 5 10 15
Lys Val Ile Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Arg His
20 25 30
Phe Val Phe Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Lys Asn Asp Glu Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Val Ser Gly Leu Arg
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Trp Asp Ala Ala Leu
85 90 95
Asn Gly Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 302
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 302
Ser Ser Asn Ile Gly Arg His Phe
1 5
<210> 303
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 303
Lys Asn Asp
1
<210> 304
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 304
Ser Ser Trp Asp Ala Ala Leu Asn Gly Val Val
1 5 10
<210> 305
<211> 216
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 305
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Thr Pro Gly Gln
1 5 10 15
Lys Val Ile Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Arg His
20 25 30
Phe Val Phe Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Lys Asn Asp Glu Arg Pro Ser Gly Val Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Val Ser Gly Leu Arg
65 70 75 80
Ser Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ser Trp Asp Ala Ala Leu
85 90 95
Asn Gly Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gln
100 105 110
Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser Glu Glu
115 120 125
Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp Phe Tyr
130 135 140
Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro Val Lys
145 150 155 160
Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn Lys Tyr
165 170 175
Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys Ser His
180 185 190
Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val Glu Lys
195 200 205
Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 306
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 306
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Glu Val Pro Met Val Arg Gly Ala Arg Arg Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 307
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 307
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 308
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 308
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 309
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 309
Ala Thr Glu Val Pro Met Val Arg Gly Ala Arg Arg Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 310
<211> 456
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 310
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Glu Val Pro Met Val Arg Gly Ala Arg Arg Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
130 135 140
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
195 200 205
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
210 215 220
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
245 250 255
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
260 265 270
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
275 280 285
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
290 295 300
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
305 310 315 320
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
325 330 335
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
340 345 350
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
355 360 365
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
370 375 380
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
385 390 395 400
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
405 410 415
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
420 425 430
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
435 440 445
Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 311
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 311
Asp Val Val Met Thr Gln Ser Pro Leu Phe Leu Ala Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Thr Leu Leu Tyr Ile
20 25 30
Asn Glu Asn Asn Tyr Leu Asp Trp Tyr Val Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Gly Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Leu Tyr Tyr Cys Met Gln Gly
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
Arg
<210> 312
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 312
Gln Thr Leu Leu Tyr Ile Asn Glu Asn Asn Tyr
1 5 10
<210> 313
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 313
Leu Gly Ser
1
<210> 314
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 314
Met Gln Gly Leu Gln Thr Pro Pro Thr
1 5
<210> 315
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 315
Asp Val Val Met Thr Gln Ser Pro Leu Phe Leu Ala Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Thr Leu Leu Tyr Ile
20 25 30
Asn Glu Asn Asn Tyr Leu Asp Trp Tyr Val Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Thr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Gly Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Leu Tyr Tyr Cys Met Gln Gly
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 316
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 316
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Ser Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Ser Gly Gly Ser Ile Ser Asn Ser
20 25 30
Asn Tyr Tyr Trp Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Gly Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Glu Ser Arg Val Thr Ile Ser Glu Asp Thr Ser Lys Asn Gln Ile
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Gly Ala Trp Glu Leu Ser Leu Gly Asp Trp Phe Asp Pro
100 105 110
Trp Gly Pro Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 317
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 317
Gly Gly Ser Ile Ser Asn Ser Asn Tyr Tyr
1 5 10
<210> 318
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 318
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 319
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 319
Ala Arg Gly Ala Trp Glu Leu Ser Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 320
<211> 453
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 320
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Ser Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Val Ser Gly Gly Ser Ile Ser Asn Ser
20 25 30
Asn Tyr Tyr Trp Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Gly Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Glu Ser Arg Val Thr Ile Ser Glu Asp Thr Ser Lys Asn Gln Ile
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Gly Ala Trp Glu Leu Ser Leu Gly Asp Trp Phe Asp Pro
100 105 110
Trp Gly Pro Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
225 230 235 240
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 321
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 321
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Ile Gln Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Arg Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Val Tyr Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu
100 105 110
Gly
<210> 322
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 322
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 323
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 323
Gly Asn Asn
1
<210> 324
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 324
Gln Ser Tyr Asp Ser Ser Leu Ser Val Tyr Val Val
1 5 10
<210> 325
<211> 218
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 325
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Ile Gln Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Asn Asn Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Arg Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Val Tyr Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 326
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 326
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Gly Thr Ser
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Phe Asp Gly Thr Glu Ile His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Ser Thr Val Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Asp Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Gly Ser Gly Asn Ile Tyr Phe Tyr Ser Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 327
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 327
Gly Phe Thr Phe Gly Thr Ser Ser
1 5
<210> 328
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 328
Ile Ser Phe Asp Gly Thr Glu Ile
1 5
<210> 329
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 329
Ala Lys Gly Ser Gly Asn Ile Tyr Phe Tyr Ser Gly Met Asp Val
1 5 10 15
<210> 330
<211> 452
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 330
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Val Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Gly Thr Ser
20 25 30
Ser Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Phe Asp Gly Thr Glu Ile His Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Ser Thr Val Phe
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Asp Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Lys Gly Ser Gly Asn Ile Tyr Phe Tyr Ser Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 331
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 331
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Gly Gln Ser Ile Ser Gly Trp
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Glu Thr Ser Thr Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 332
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 332
Gln Ser Ile Ser Gly Trp
1 5
<210> 333
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 333
Glu Thr Ser
1
<210> 334
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 334
Gln Gln Tyr Tyr Ser Tyr Pro Leu Thr
1 5
<210> 335
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 335
Asp Ile Gln Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Gly Gln Ser Ile Ser Gly Trp
20 25 30
Leu Ala Trp Phe Gln Gln Lys Pro Gly Lys Ala Pro Asn Leu Leu Ile
35 40 45
Tyr Glu Thr Ser Thr Leu Glu Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 336
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 336
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 337
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 337
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 338
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 338
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 339
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 339
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 340
<211> 456
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 340
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
130 135 140
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
195 200 205
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
210 215 220
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
245 250 255
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
260 265 270
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
275 280 285
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
290 295 300
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
305 310 315 320
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
325 330 335
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
340 345 350
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
355 360 365
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
370 375 380
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
385 390 395 400
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
405 410 415
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
420 425 430
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
435 440 445
Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 341
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 341
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Ser Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Ser Pro Val Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg
<210> 342
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 342
Gln Ser Leu Leu Tyr Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 343
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 343
Leu Gly Ser
1
<210> 344
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 344
Met Gln Ala Leu Gln Ser Pro Val Thr
1 5
<210> 345
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 345
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Ser Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Ser Pro Val Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 346
<211> 127
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 346
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asp Ser Arg Asp Gly Pro Ala Ala Arg Gly Gly Tyr Tyr Tyr
100 105 110
Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 347
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 347
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 348
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 348
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 349
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 349
Ala Thr Asp Ser Arg Asp Gly Pro Ala Ala Arg Gly Gly Tyr Tyr Tyr
1 5 10 15
Tyr Met Asp Val
20
<210> 350
<211> 457
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 350
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Asp Ser Arg Asp Gly Pro Ala Ala Arg Gly Gly Tyr Tyr Tyr
100 105 110
Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 351
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 351
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ile
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg
<210> 352
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 352
Gln Ser Leu Leu Tyr Ile Asn Gly Tyr Asn Tyr
1 5 10
<210> 353
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 353
Leu Gly Ser
1
<210> 354
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 354
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 355
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 355
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Leu Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ile
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 356
<211> 127
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 356
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Ala Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr Asn Pro Asp
100 105 110
Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120 125
<210> 357
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 357
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 358
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 358
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 359
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 359
Ala Arg Asp Ala Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr Asn Pro Asp
1 5 10 15
Ala Phe Asp Ile
20
<210> 360
<211> 457
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 360
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Ala Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr Asn Pro Asp
100 105 110
Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala
115 120 125
Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser
130 135 140
Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe
145 150 155 160
Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly
165 170 175
Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu
180 185 190
Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr
195 200 205
Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys
210 215 220
Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
225 230 235 240
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
245 250 255
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
260 265 270
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
275 280 285
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
290 295 300
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
305 310 315 320
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
325 330 335
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
340 345 350
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
355 360 365
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
370 375 380
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
385 390 395 400
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
405 410 415
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
420 425 430
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
435 440 445
Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 361
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 361
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Ser Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Val Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Gly
85 90 95
Leu Gln Thr Pro Arg Thr Phe Gly Arg Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg
<210> 362
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 362
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 363
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 363
Leu Gly Ser
1
<210> 364
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 364
Met Gln Gly Leu Gln Thr Pro Arg Thr
1 5
<210> 365
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 365
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Ser Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Val Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Gly
85 90 95
Leu Gln Thr Pro Arg Thr Phe Gly Arg Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 366
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 366
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 367
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 367
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 368
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 368
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 369
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 369
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 370
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 370
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 371
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 371
Glu Thr Thr Leu Thr Gln Ser Pro Ala Phe Met Ser Ala Thr Pro Gly
1 5 10 15
Asp Glu Val Asn Ile Ser Cys Lys Ala Ser Gln Asp Ile Asp Asp Asp
20 25 30
Val Ser Trp Tyr Gln Gln Lys Pro Gly Gly Ala Pro Ile Phe Leu Ile
35 40 45
Gln Glu Ala Ser Thr Leu Val Pro Gly Ile Pro Pro Arg Phe Ser Gly
50 55 60
Ser Gly Phe Gly Thr Asp Phe Thr Leu Thr Ile Lys Asn Met Glu Ser
65 70 75 80
Glu Asp Ala Ala Tyr Tyr Phe Cys Leu Gln His Asp Asn Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Asp Ile Lys Arg
100 105
<210> 372
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 372
Gln Asp Ile Asp Asp Asp
1 5
<210> 373
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 373
Glu Ala Ser
1
<210> 374
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 374
Leu Gln His Asp Asn Leu Pro Leu Thr
1 5
<210> 375
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 375
Glu Thr Thr Leu Thr Gln Ser Pro Ala Phe Met Ser Ala Thr Pro Gly
1 5 10 15
Asp Glu Val Asn Ile Ser Cys Lys Ala Ser Gln Asp Ile Asp Asp Asp
20 25 30
Val Ser Trp Tyr Gln Gln Lys Pro Gly Gly Ala Pro Ile Phe Leu Ile
35 40 45
Gln Glu Ala Ser Thr Leu Val Pro Gly Ile Pro Pro Arg Phe Ser Gly
50 55 60
Ser Gly Phe Gly Thr Asp Phe Thr Leu Thr Ile Lys Asn Met Glu Ser
65 70 75 80
Glu Asp Ala Ala Tyr Tyr Phe Cys Leu Gln His Asp Asn Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Asp Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 376
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 376
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Glu Asp Pro Gly Pro Val Ala Gly Pro Tyr Tyr Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 377
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 377
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 378
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 378
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 379
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 379
Ala Thr Glu Asp Pro Gly Pro Val Ala Gly Pro Tyr Tyr Tyr Tyr Gly
1 5 10 15
Met Asp Val
<210> 380
<211> 456
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 380
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Glu Asp Pro Gly Pro Val Ala Gly Pro Tyr Tyr Tyr Tyr Gly
100 105 110
Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
130 135 140
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
195 200 205
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
210 215 220
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
245 250 255
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
260 265 270
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
275 280 285
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
290 295 300
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
305 310 315 320
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
325 330 335
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
340 345 350
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
355 360 365
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
370 375 380
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
385 390 395 400
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
405 410 415
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
420 425 430
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
435 440 445
Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 381
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 381
Asp Val Val Met Thr Gln Ser Pro Leu Val Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ile
20 25 30
Asn Gly Tyr His Tyr Leu Asp Trp Tyr Val Gln Arg Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Phe Leu Gly Ser Thr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Glu Ile
65 70 75 80
Ser Lys Val Glu Ala Glu Asp Val Gly Ile Tyr Phe Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg
<210> 382
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 382
Gln Ser Leu Leu Tyr Ile Asn Gly Tyr His Tyr
1 5 10
<210> 383
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 383
Leu Gly Ser
1
<210> 384
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 384
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 385
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 385
Asp Val Val Met Thr Gln Ser Pro Leu Val Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Ile
20 25 30
Asn Gly Tyr His Tyr Leu Asp Trp Tyr Val Gln Arg Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Phe Leu Gly Ser Thr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Glu Ile
65 70 75 80
Ser Lys Val Glu Ala Glu Asp Val Gly Ile Tyr Phe Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 386
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 386
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 387
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 387
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 388
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 388
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 389
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 389
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 390
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 390
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 391
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 391
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Pro Ser Gln Ser Leu Leu Asp Ser
20 25 30
Asp Asp Gly Asn Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Gln Leu Leu Ile His Thr Leu Ser Tyr Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys
65 70 75 80
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln
85 90 95
Arg Leu Gln Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg
<210> 392
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 392
Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 393
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 393
Thr Leu Ser
1
<210> 394
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 394
Met Gln Arg Leu Gln Phe Pro Leu Thr
1 5
<210> 395
<211> 220
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 395
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Pro Ser Gln Ser Leu Leu Asp Ser
20 25 30
Asp Asp Gly Asn Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Gln Leu Leu Ile His Thr Leu Ser Tyr Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys
65 70 75 80
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln
85 90 95
Arg Leu Gln Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 396
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 396
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 397
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 397
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 398
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 398
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 399
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 399
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 400
<211> 456
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 400
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
100 105 110
Met Asp Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser
115 120 125
Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr
130 135 140
Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro
145 150 155 160
Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val
165 170 175
His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser
180 185 190
Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile
195 200 205
Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val
210 215 220
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
225 230 235 240
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
245 250 255
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
260 265 270
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
275 280 285
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
290 295 300
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
305 310 315 320
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
325 330 335
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
340 345 350
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr
355 360 365
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
370 375 380
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
385 390 395 400
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
405 410 415
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
420 425 430
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
435 440 445
Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 401
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 401
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Trp Gly Ser Tyr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Val Gln Asn Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Asp Ile Lys
100 105 110
Arg
<210> 402
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 402
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 403
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 403
Trp Gly Ser
1
<210> 404
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 404
Met Gln Ala Val Gln Asn Pro Pro Thr
1 5
<210> 405
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 405
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Trp Gly Ser Tyr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Val Gln Asn Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Asp Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 406
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 406
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 407
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 407
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 408
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 408
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 409
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 409
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 410
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 410
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 411
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 411
Asp Val Val Met Thr Gln Ser Pro Ala Phe Leu Ser Val Thr Pro Gly
1 5 10 15
Glu Arg Val Thr Leu Ser Cys Lys Ala Ser Gln Thr Met Asn Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Thr Arg Ala Thr Asp Thr Pro Pro Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Val Gln Ser
65 70 75 80
Glu Asp Phe Ala Leu Tyr Tyr Cys Gln Gln Tyr Gly Asp Trp Leu Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg
100 105
<210> 412
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 412
Gln Thr Met Asn Asn Tyr
1 5
<210> 413
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 413
Asp Ala Ser
1
<210> 414
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 414
Gln Gln Tyr Gly Asp Trp Leu Pro Ile Thr
1 5 10
<210> 415
<211> 215
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 415
Asp Val Val Met Thr Gln Ser Pro Ala Phe Leu Ser Val Thr Pro Gly
1 5 10 15
Glu Arg Val Thr Leu Ser Cys Lys Ala Ser Gln Thr Met Asn Asn Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Thr Arg Ala Thr Asp Thr Pro Pro Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Val Gln Ser
65 70 75 80
Glu Asp Phe Ala Leu Tyr Tyr Cys Gln Gln Tyr Gly Asp Trp Leu Pro
85 90 95
Ile Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala
100 105 110
Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser
115 120 125
Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu
130 135 140
Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser
145 150 155 160
Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu
165 170 175
Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val
180 185 190
Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys
195 200 205
Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 416
<211> 124
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 416
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Ala Phe Pro Glu Ala Thr Ile Ser Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 417
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 417
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 418
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 418
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 419
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 419
Ala Thr Ala Phe Pro Glu Ala Thr Ile Ser Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 420
<211> 454
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 420
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Ala Phe Pro Glu Ala Thr Ile Ser Tyr Tyr Tyr Tyr Met Asp
100 105 110
Val Trp Gly Lys Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
115 120 125
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
130 135 140
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
145 150 155 160
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
165 170 175
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
180 185 190
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
195 200 205
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
210 215 220
Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro
325 330 335
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Pro Gly Lys
450
<210> 421
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 421
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Trp Gly Ser Tyr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Val Gln Asn Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg
<210> 422
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 422
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 423
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 423
Trp Gly Ser
1
<210> 424
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 424
Met Gln Ala Val Gln Asn Pro Pro Thr
1 5
<210> 425
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 425
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Trp Gly Ser Tyr Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Val Gln Asn Pro Pro Thr Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 426
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 426
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 427
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 427
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 428
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 428
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 429
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 429
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 430
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 430
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 431
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 431
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Met Ser Cys Arg Ser Ser Arg Ser Leu Leu Asp Ser
20 25 30
Asp Asp Gly Asn Thr His Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Gln Leu Leu Ile Gln Ser Leu Ser Tyr Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Glu
65 70 75 80
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln
85 90 95
Arg Lys Glu Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg
<210> 432
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 432
Arg Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr His
1 5 10
<210> 433
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 433
Ser Leu Ser
1
<210> 434
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 434
Met Gln Arg Lys Glu Phe Pro Leu Thr
1 5
<210> 435
<211> 220
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 435
Asp Ile Val Met Thr Gln Thr Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Met Ser Cys Arg Ser Ser Arg Ser Leu Leu Asp Ser
20 25 30
Asp Asp Gly Asn Thr His Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln
35 40 45
Ser Pro Gln Leu Leu Ile Gln Ser Leu Ser Tyr Arg Ala Ser Gly Val
50 55 60
Pro Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Glu
65 70 75 80
Ile Ser Arg Val Glu Ala Glu Asp Val Gly Ile Tyr Tyr Cys Met Gln
85 90 95
Arg Lys Glu Phe Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 436
<211> 128
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 436
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro Trp Tyr Tyr Asp Ser Ser Gly Tyr Ser Ser Tyr Ala
100 105 110
Tyr Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
<210> 437
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 437
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 438
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 438
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 439
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 439
Ala Arg Gly Pro Trp Tyr Tyr Asp Ser Ser Gly Tyr Ser Ser Tyr Ala
1 5 10 15
Tyr Tyr Met Asp Val
20
<210> 440
<211> 458
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 440
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Phe Gly Thr Ala Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gly Pro Trp Tyr Tyr Asp Ser Ser Gly Tyr Ser Ser Tyr Ala
100 105 110
Tyr Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
130 135 140
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
145 150 155 160
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
165 170 175
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
180 185 190
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
195 200 205
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
210 215 220
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
225 230 235 240
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
245 250 255
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
260 265 270
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
275 280 285
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
290 295 300
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
305 310 315 320
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
325 330 335
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
340 345 350
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu
355 360 365
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
370 375 380
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
385 390 395 400
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
405 410 415
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
420 425 430
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
435 440 445
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 441
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 441
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu His Thr Pro Gly Val Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg
<210> 442
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 442
Gln Ser Leu Leu His Ser Asp Gly Tyr Asn Tyr
1 5 10
<210> 443
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 443
Leu Gly Ser
1
<210> 444
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 444
Met Gln Ala Leu His Thr Pro Gly Val Thr
1 5 10
<210> 445
<211> 220
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 445
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asp Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu His Thr Pro Gly Val Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
100 105 110
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
115 120 125
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
130 135 140
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
145 150 155 160
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
165 170 175
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
180 185 190
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
195 200 205
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215 220
<210> 446
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 446
Gln Leu Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Thr Gly Gly Ser Ile Ser Ser Ser
20 25 30
Ser Tyr Tyr Trp Ala Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Gly Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Val Ser
50 55 60
Leu Glu Ser Arg Val Thr Ile Ser Gln Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Thr Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Gly Ala Trp Glu Leu Arg Leu Gly Asp Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 447
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 447
Gly Gly Ser Ile Ser Ser Ser Ser Tyr Tyr
1 5 10
<210> 448
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 448
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 449
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 449
Ala Arg Gly Ala Trp Glu Leu Arg Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 450
<211> 453
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 450
Gln Leu Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Thr Gly Gly Ser Ile Ser Ser Ser
20 25 30
Ser Tyr Tyr Trp Ala Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Gly Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Val Ser
50 55 60
Leu Glu Ser Arg Val Thr Ile Ser Gln Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Thr Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Gly Ala Trp Glu Leu Arg Leu Gly Asp Trp Phe Asp Pro
100 105 110
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly
115 120 125
Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly
130 135 140
Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val
145 150 155 160
Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe
165 170 175
Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val
180 185 190
Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val
195 200 205
Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys
210 215 220
Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu
225 230 235 240
Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr
245 250 255
Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val
260 265 270
Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val
275 280 285
Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser
290 295 300
Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu
305 310 315 320
Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala
325 330 335
Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro
340 345 350
Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln
355 360 365
Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala
370 375 380
Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr
385 390 395 400
Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu
405 410 415
Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser
420 425 430
Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser
435 440 445
Leu Ser Pro Gly Lys
450
<210> 451
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 451
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Ile Gln Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Tyr Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Val Tyr Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu
100 105 110
Gly Gln Pro Lys Ala Ala
115
<210> 452
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 452
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 453
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 453
Gly Tyr Ser
1
<210> 454
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 454
Gln Ser Tyr Asp Ser Ser Leu Ser Val Tyr Val Val
1 5 10
<210> 455
<211> 218
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 455
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Ile Gln Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Tyr Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Val Tyr Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 456
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 456
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Arg Phe Thr Phe Glu Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Lys Ser Gly Gly Ile Asn Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Gln Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Val Lys Asp Pro Thr Leu Val Ala Thr Asp Arg Ala Phe Asn Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 457
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 457
Arg Phe Thr Phe Glu Asp Tyr Ala
1 5
<210> 458
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 458
Ile Ser Trp Lys Ser Gly Gly Ile
1 5
<210> 459
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 459
Val Lys Asp Pro Thr Leu Val Ala Thr Asp Arg Ala Phe Asn Ile
1 5 10 15
<210> 460
<211> 452
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 460
Gln Val Gln Leu Gln Glu Ser Gly Gly Gly Val Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Arg Phe Thr Phe Glu Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Trp Lys Ser Gly Gly Ile Asn Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Gln Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Val Lys Asp Pro Thr Leu Val Ala Thr Asp Arg Ala Phe Asn Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro
115 120 125
Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr
130 135 140
Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr
145 150 155 160
Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro
165 170 175
Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr
180 185 190
Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn
195 200 205
His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser
210 215 220
Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Leu Leu
225 230 235 240
Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
245 250 255
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
260 265 270
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
275 280 285
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
290 295 300
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
305 310 315 320
Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro
325 330 335
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
340 345 350
Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val
355 360 365
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
370 375 380
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
385 390 395 400
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
405 410 415
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
420 425 430
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
435 440 445
Ser Pro Gly Lys
450
<210> 461
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 461
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Tyr
85 90 95
Leu Gln Thr Pro Asp Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Ala Ala Pro Ser
115
<210> 462
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 462
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 463
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 463
Leu Gly Ser
1
<210> 464
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 464
Met Gln Tyr Leu Gln Thr Pro Asp Thr
1 5
<210> 465
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 465
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu His Ser
20 25 30
Asn Gly Tyr Asn Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Tyr
85 90 95
Leu Gln Thr Pro Asp Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 466
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 466
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 467
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 467
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 468
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 468
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 469
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 469
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 470
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 470
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 471
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 471
Glu Ile Val Leu Thr Gln Ser Pro Asp Phe Gln Ser Val Thr Pro Lys
1 5 10 15
Glu Lys Val Thr Ile Thr Cys Arg Ala Ser Gln Asn Ile Asp Ser Ser
20 25 30
Leu His Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile
35 40 45
Asn Tyr Ala Ser Gln Ser Phe Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Asp Ser Leu Glu Pro
65 70 75 80
Glu Asp Ala Ala Thr Tyr Phe Cys His Gln Ser Ser Ser Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Arg Arg Thr Val Ala Ala
100 105 110
Pro Ser
<210> 472
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 472
Gln Asn Ile Asp Ser Ser
1 5
<210> 473
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 473
Tyr Ala Ser
1
<210> 474
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 474
His Gln Ser Ser Ser Leu Pro Leu Thr
1 5
<210> 475
<211> 218
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 475
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Gly Ala Pro Gly Gln
1 5 10 15
Arg Val Thr Ile Ser Cys Thr Gly Ser Ser Ser Asn Ile Gly Ala Gly
20 25 30
Tyr Asp Ile Gln Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu
35 40 45
Leu Ile Tyr Gly Tyr Ser Asn Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Thr Ser Ala Ser Leu Ala Ile Thr Gly Leu
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp Ser Ser
85 90 95
Leu Ser Val Tyr Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu
100 105 110
Gly Gln Pro Lys Ala Ala Pro Ser Val Thr Leu Phe Pro Pro Ser Ser
115 120 125
Glu Glu Leu Gln Ala Asn Lys Ala Thr Leu Val Cys Leu Ile Ser Asp
130 135 140
Phe Tyr Pro Gly Ala Val Thr Val Ala Trp Lys Ala Asp Ser Ser Pro
145 150 155 160
Val Lys Ala Gly Val Glu Thr Thr Thr Pro Ser Lys Gln Ser Asn Asn
165 170 175
Lys Tyr Ala Ala Ser Ser Tyr Leu Ser Leu Thr Pro Glu Gln Trp Lys
180 185 190
Ser His Arg Ser Tyr Ser Cys Gln Val Thr His Glu Gly Ser Thr Val
195 200 205
Glu Lys Thr Val Ala Pro Thr Glu Cys Ser
210 215
<210> 476
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 476
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 477
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 477
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 478
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 478
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 479
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 479
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 480
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 480
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 481
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 481
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Asp Asp Trp Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser
<210> 482
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 482
Gln Ser Val Ser Ser Tyr
1 5
<210> 483
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 483
Asp Ala Ser
1
<210> 484
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 484
Gln Gln Tyr Asp Asp Trp Pro Gln Thr
1 5
<210> 485
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 485
Glu Ile Val Leu Thr Gln Ser Pro Gly Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Tyr Asp Asp Trp Pro Gln
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 486
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 486
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 487
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 487
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 488
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 488
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 489
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 489
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 490
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 490
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 491
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 491
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Thr Gly
1 5 10 15
Glu Arg Ala Thr Leu Phe Cys Arg Thr Ser Gln Asn Val Tyr Gly Glu
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Thr Phe Glu Arg Ala Ala Gly Ile Pro Ala Lys Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Val Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Arg Asp Trp Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser
<210> 492
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 492
Gln Asn Val Tyr Gly Glu
1 5
<210> 493
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 493
Asp Thr Phe
1
<210> 494
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 494
Gln Gln Arg Arg Asp Trp Pro Ile Thr
1 5
<210> 495
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 495
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Thr Gly
1 5 10 15
Glu Arg Ala Thr Leu Phe Cys Arg Thr Ser Gln Asn Val Tyr Gly Glu
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Thr Phe Glu Arg Ala Ala Gly Ile Pro Ala Lys Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Arg Val Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Arg Asp Trp Pro Ile
85 90 95
Thr Phe Gly Gln Gly Thr Arg Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 496
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 496
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 497
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 497
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 498
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 498
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 499
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 499
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 500
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 500
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 501
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 501
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Arg Asp Ile Ser Asp Ser
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Phe Asp Ala Ser Asn Leu Lys Thr Gly Val Ser Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys His Gln Tyr Asp Asn Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser
<210> 502
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 502
Arg Asp Ile Ser Asp Ser
1 5
<210> 503
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 503
Asp Ala Ser
1
<210> 504
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 504
His Gln Tyr Asp Asn Leu Pro Leu Thr
1 5
<210> 505
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 505
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Arg Asp Ile Ser Asp Ser
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Phe Asp Ala Ser Asn Leu Lys Thr Gly Val Ser Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys His Gln Tyr Asp Asn Leu Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 506
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 506
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 507
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 507
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 508
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 508
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 509
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 509
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 510
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 510
Gln Met Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ser
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Gly Thr Phe Ser Ser Ser
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Gln Gly Phe Glu Trp Met
35 40 45
Gly Gly Ile Ile Pro Ile Tyr Gly Ile Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Thr Asp Glu Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
100 105 110
Tyr Tyr Ala Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 511
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 511
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Glu Ser Val Thr Thr Phe
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Thr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Gly Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys His Gln His Thr Asn Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser
<210> 512
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 512
Glu Ser Val Thr Thr Phe
1 5
<210> 513
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 513
Asp Ala Ser
1
<210> 514
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 514
His Gln His Thr Asn Trp Pro Leu Thr
1 5
<210> 515
<211> 214
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 515
Glu Ile Val Met Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Glu Ser Val Thr Thr Phe
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Thr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Gly Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys His Gln His Thr Asn Trp Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 516
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 516
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Glu Val Gly Tyr Cys Ser Gly Gly Ser Cys Tyr Ile Ser Tyr
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser
<210> 517
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 517
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 518
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 518
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 519
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 519
Ala Thr Glu Val Gly Tyr Cys Ser Gly Gly Ser Cys Tyr Ile Ser Tyr
1 5 10 15
Tyr Tyr Gly Met Asp Val
20
<210> 520
<211> 459
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 520
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Val Ser Gly Tyr Thr Leu Thr Glu Leu
20 25 30
Ser Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Gly Phe Asp Pro Glu Asp Gly Glu Thr Ile Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Glu Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Glu Val Gly Tyr Cys Ser Gly Gly Ser Cys Tyr Ile Ser Tyr
100 105 110
Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
115 120 125
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
130 135 140
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
145 150 155 160
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
165 170 175
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
180 185 190
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
195 200 205
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
210 215 220
Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro
225 230 235 240
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
245 250 255
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
260 265 270
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
275 280 285
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
290 295 300
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
305 310 315 320
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
325 330 335
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
340 345 350
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu
355 360 365
Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
370 375 380
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
385 390 395 400
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
405 410 415
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
420 425 430
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
435 440 445
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
450 455
<210> 521
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 521
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly His Asn Phe Leu Asp Trp Tyr Val Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser
115
<210> 522
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 522
Gln Ser Leu Leu Tyr Arg Asn Gly His Asn Phe
1 5 10
<210> 523
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 523
Leu Gly Ser
1
<210> 524
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 524
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 525
<211> 219
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 525
Asp Val Val Met Thr Gln Ser Pro Leu Ser Leu Pro Val Thr Pro Gly
1 5 10 15
Glu Pro Ala Ser Ile Ser Cys Arg Ser Ser Gln Ser Leu Leu Tyr Arg
20 25 30
Asn Gly His Asn Phe Leu Asp Trp Tyr Val Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Gly Ser Asn Arg Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln Ala
85 90 95
Leu Gln Thr Pro Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105 110
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
115 120 125
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
130 135 140
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
145 150 155 160
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
165 170 175
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
180 185 190
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
195 200 205
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
210 215
<210> 526
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 526
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 527
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 527
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 528
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 528
Ala Arg Glu Glu Val Ser Ser Pro Tyr Gly Met Asp Val
1 5 10
<210> 529
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 529
Thr Gly Ala Val Thr Ser Gly His Tyr
1 5
<210> 530
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 530
Asp Thr Gly
1
<210> 531
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 531
Leu Leu Ser Tyr Ser Gly Ala Ser Trp Val
1 5 10
<210> 532
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 532
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 533
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 533
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 534
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 534
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 535
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 535
Gln Gly Ile Gly Asn Tyr
1 5
<210> 536
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 536
Ser Ala Ser
1
<210> 537
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 537
Gln Gln Leu Lys Asp Tyr Pro Ile Thr
1 5
<210> 538
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 538
Gly Phe Ser Leu Ser His Phe Ser
1 5
<210> 539
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 539
Phe Asp Pro Glu Glu Gly Gly Thr
1 5
<210> 540
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 540
Ala Thr Gly Ile Trp Tyr Ser Ser Gly Trp Pro Val Asp Tyr
1 5 10
<210> 541
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 541
Gln Ser Leu Leu Asp Ser Ser Gly Tyr Asn Tyr
1 5 10
<210> 542
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 542
Leu Gly Ser
1
<210> 543
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 543
Met Gln Ala Leu Gln Thr Pro Ile Thr
1 5
<210> 544
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 544
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 545
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 545
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 546
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 546
Ala Thr Val Ser Arg Val Arg Gly Val Ile Asn Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210> 547
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 547
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 548
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 548
Leu Gly Ser
1
<210> 549
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 549
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 550
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 550
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 551
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 551
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 552
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 552
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 553
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 553
Gln Ser Val Asp Ser Ser
1 5
<210> 554
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 554
Asp Ala Ser
1
<210> 555
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 555
Gln Gln Tyr Lys Asp Trp Pro Phe Thr
1 5
<210> 556
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 556
Gly Gly Thr Phe Gly Thr Tyr Ala
1 5
<210> 557
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 557
Ile Thr Pro Ile Ser Ala Thr Ile
1 5
<210> 558
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 558
Ala Arg Gly Phe Glu Tyr Ser Asp Gly Leu Leu Asp Asp
1 5 10
<210> 559
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 559
Gln Ser Leu Phe Tyr Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 560
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 560
Arg Leu Ser
1
<210> 561
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 561
Met Gln His Met Glu Phe Pro Leu Thr
1 5
<210> 562
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 562
Gly Gly Ser Ile Ser Ser Ser Ser Tyr Tyr
1 5 10
<210> 563
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 563
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 564
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 564
Ala Arg Gly Ala Trp Glu Leu Arg Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 565
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 565
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 566
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 566
Gly Asn Asn
1
<210> 567
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 567
Gln Ser Tyr Asp Ser Ser Leu Ser Ile Tyr Val Val
1 5 10
<210> 568
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 568
Ser Gly Ser Ile Ser Ser Thr Asn Trp
1 5
<210> 569
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 569
Ile Tyr His Ser Gly Ser Thr
1 5
<210> 570
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 570
Ala Arg Val Gly Pro Ala Ala Ile Tyr Tyr
1 5 10
<210> 571
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 571
Ser Asn Asn Val Gly Tyr Glu Gly
1 5
<210> 572
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 572
Arg Asn Asn
1
<210> 573
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 573
Ser Ala Trp Asp Ser Ser Leu Asn Ala Val Val
1 5 10
<210> 574
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 574
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 575
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 575
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 576
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 576
Ala Thr Ala Lys Pro Gly Ile Ala Val Ala Gly Gln Asn Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Met Asp Val
20
<210> 577
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 577
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 578
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 578
Leu Gly Ser
1
<210> 579
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 579
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 580
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 580
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 581
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 581
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 582
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 582
Ala Ser Ser Pro Ile Gly Tyr Ser Tyr Gly Tyr Trp Gly Gly Met Asp
1 5 10 15
Val
<210> 583
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 583
Ser Gly Ile Asp Val Arg Thr Asn Lys
1 5
<210> 584
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 584
Phe Gln Ser Asp Ser Asp Lys
1 5
<210> 585
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 585
Leu Ile Trp His Thr Ser Gly Trp Val
1 5
<210> 586
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 586
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 587
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 587
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 588
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 588
Ala Thr Gly Pro Ala Ala Ala Gly Val Gly Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 589
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 589
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 590
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 590
Leu Gly Ser
1
<210> 591
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 591
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 592
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 592
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 593
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 593
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 594
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 594
Ala Arg Asp Val Met Val Tyr Cys Ser Ser Thr Ser Cys Tyr Phe Tyr
1 5 10 15
Gly Met Asp Val
20
<210> 595
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 595
Gln Asp Ile Arg Asp Tyr
1 5
<210> 596
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 596
Asp Ala Ser
1
<210> 597
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 597
Gln Gln Phe Glu Asn Leu Pro Ile Thr
1 5
<210> 598
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 598
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 599
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 599
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 600
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 600
Ala Thr Gly Gly Tyr Ser Ser Gly Phe Asn Tyr Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210> 601
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 601
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 602
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 602
Leu Gly Ser
1
<210> 603
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 603
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 604
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 604
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 605
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 605
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 606
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 606
Ala Thr Gly Val Thr Thr Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10
<210> 607
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 607
Gln Ser Leu Leu Tyr Ser Asn Gly Asn Asn Phe
1 5 10
<210> 608
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 608
Leu Gly Ser
1
<210> 609
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 609
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 610
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 610
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 611
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 611
Ile Arg Tyr Asp Gly Ser Asn Lys
1 5
<210> 612
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 612
Ala Arg Asp Leu Phe Asp Phe Trp Trp Asp Gly Met Asp Val
1 5 10
<210> 613
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 613
Gln Ser Val Ser Ser Met Tyr
1 5
<210> 614
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 614
Gly Ala Ser
1
<210> 615
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 615
Gln Gln Tyr Val Ser Ser Pro Met Tyr Thr
1 5 10
<210> 616
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 616
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 617
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 617
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 618
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 618
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 619
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 619
Gln Ser Val Ser Ser Tyr
1 5
<210> 620
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 620
Asp Ala Ser
1
<210> 621
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 621
Gln Gln Tyr Asp Asp Trp Pro Gln Thr
1 5
<210> 622
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 622
Gly Gly Thr Phe Asn Asn Tyr Gly
1 5
<210> 623
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 623
Ile Ile Pro Leu Phe Gly Thr Thr
1 5
<210> 624
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 624
Ala Arg Asp Arg Met Ala Ala Asp Gly Met Ala Val Phe Asp Tyr
1 5 10 15
<210> 625
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 625
Ser Ser Asn Ile Gly Arg His Phe
1 5
<210> 626
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 626
Lys Asn Asp
1
<210> 627
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 627
Ser Ser Trp Asp Ala Ala Leu Asn Gly Val Val
1 5 10
<210> 628
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 628
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 629
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 629
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 630
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 630
Ala Thr Glu Val Pro Met Val Arg Gly Ala Arg Arg Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 631
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 631
Gln Thr Leu Leu Tyr Ile Asn Glu Asn Asn Tyr
1 5 10
<210> 632
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 632
Leu Gly Ser
1
<210> 633
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 633
Met Gln Gly Leu Gln Thr Pro Pro Thr
1 5
<210> 634
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 634
Gly Gly Ser Ile Ser Asn Ser Asn Tyr Tyr
1 5 10
<210> 635
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 635
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 636
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 636
Ala Arg Gly Ala Trp Glu Leu Ser Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 637
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 637
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 638
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 638
Gly Asn Asn
1
<210> 639
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 639
Gln Ser Tyr Asp Ser Ser Leu Ser Val Tyr Val Val
1 5 10
<210> 640
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 640
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 641
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 641
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 642
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 642
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 643
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 643
Gln Asn Val Tyr Gly Glu
1 5
<210> 644
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 644
Asp Thr Phe
1
<210> 645
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 645
Gln Gln Arg Arg Asp Trp Pro Ile Thr
1 5
<210> 646
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 646
Gly Phe Thr Phe Gly Thr Ser Ser
1 5
<210> 647
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 647
Ile Ser Phe Asp Gly Thr Glu Ile
1 5
<210> 648
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 648
Ala Lys Gly Ser Gly Asn Ile Tyr Phe Tyr Ser Gly Met Asp Val
1 5 10 15
<210> 649
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 649
Gln Ser Ile Ser Gly Trp
1 5
<210> 650
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 650
Glu Thr Ser
1
<210> 651
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 651
Gln Gln Tyr Tyr Ser Tyr Pro Leu Thr
1 5
<210> 652
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 652
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 653
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 653
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 654
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 654
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 655
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 655
Gln Ser Leu Leu Tyr Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 656
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 656
Leu Gly Ser
1
<210> 657
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 657
Met Gln Ala Leu Gln Ser Pro Val Thr
1 5
<210> 658
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 658
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 659
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 659
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 660
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 660
Ala Thr Asp Ser Arg Asp Gly Pro Ala Ala Arg Gly Gly Tyr Tyr Tyr
1 5 10 15
Tyr Met Asp Val
20
<210> 661
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 661
Gln Ser Leu Leu Tyr Ile Asn Gly Tyr Asn Tyr
1 5 10
<210> 662
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 662
Leu Gly Ser
1
<210> 663
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 663
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 664
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 664
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 665
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 665
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 666
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 666
Ala Arg Asp Ala Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr Asn Pro Asp
1 5 10 15
Ala Phe Asp Ile
20
<210> 667
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 667
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 668
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 668
Leu Gly Ser
1
<210> 669
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 669
Met Gln Gly Leu Gln Thr Pro Arg Thr
1 5
<210> 670
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 670
Gly Phe Ser Leu Thr Ser Gly Gly Met Ser
1 5 10
<210> 671
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 671
Ile Asp Trp Asn Asp Asp Lys
1 5
<210> 672
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 672
Ala Arg Ile Arg Gly Met Thr Trp Gly Phe Asp Ser
1 5 10
<210> 673
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 673
Gln Ser Val Ser Ser Ser Tyr
1 5
<210> 674
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 674
Gly Ala Ser
1
<210> 675
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 675
Gln Gln Tyr Gly Thr Thr Pro Phe Ala
1 5
<210> 676
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 676
Gly Tyr Thr Phe Thr Ala Tyr Tyr
1 5
<210> 677
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 677
Ile Asn Pro Asn Ser Gly Gly Thr
1 5
<210> 678
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 678
Ala Arg Asp Gly Ala Phe Tyr Tyr Gly Ser Glu Asn Tyr Tyr Asn Ala
1 5 10 15
Gly Trp Phe Asp Pro
20
<210> 679
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 679
Gln Ser Leu Leu Ser Gly Asn Gly Tyr Asn Tyr
1 5 10
<210> 680
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 680
Leu Gly Ser
1
<210> 681
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 681
Met Gln Ala Leu Lys Ser Pro Leu Thr
1 5
<210> 682
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 682
Gly Tyr Thr Phe Thr Asn Tyr Tyr
1 5
<210> 683
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 683
Ile Asn Pro Ser Gly Gly Ile Thr
1 5
<210> 684
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 684
Ala Arg Ala Gly Leu Gly Tyr Asn Trp Asn Tyr Ala Pro Ser Gly Met
1 5 10 15
Asp Val
<210> 685
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 685
Ser Ser Asp Val Gly Gly Tyr Asn Tyr
1 5
<210> 686
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 686
Glu Val Ser
1
<210> 687
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 687
Ser Ser Tyr Ala Gly Ser Asn Asn Leu Val
1 5 10
<210> 688
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 688
Gly Gly Ser Ile Ser Ser Ser Ser Tyr Tyr
1 5 10
<210> 689
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 689
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 690
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 690
Ala Arg Gly Ala Trp Glu Leu Arg Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 691
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 691
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 692
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 692
Gly Tyr Ser
1
<210> 693
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 693
Gln Ser Tyr Asp Ser Ser Leu Ser Val Tyr Val Val
1 5 10
<210> 694
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 694
Gly Phe Thr Phe Asp Asp Tyr Ala
1 5
<210> 695
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 695
Ile Ser Trp Asn Ser Gly Ser Ile
1 5
<210> 696
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 696
Ala Lys Asp Val Asn Arg Ile Leu Val Ala Gly Met Val Asp Tyr
1 5 10 15
<210> 697
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 697
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 698
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 698
Leu Gly Ser
1
<210> 699
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 699
Met Gln Gly Leu Gln Thr Pro Ile Thr
1 5
<210> 700
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 700
Arg Phe Thr Phe Glu Asp Tyr Ala
1 5
<210> 701
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 701
Ile Ser Trp Lys Ser Gly Gly Ile
1 5
<210> 702
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 702
Val Lys Asp Pro Thr Leu Val Ala Thr Asp Arg Ala Phe Asn Ile
1 5 10 15
<210> 703
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 703
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 704
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 704
Leu Gly Ser
1
<210> 705
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 705
Met Gln Tyr Leu Gln Thr Pro Asp Thr
1 5
<210> 706
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 706
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 707
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 707
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 708
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 708
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 709
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 709
Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Ile His
1 5 10
<210> 710
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 710
Thr Leu Ser
1
<210> 711
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 711
Met Gln Arg Lys Glu Phe Pro Leu Thr
1 5
<210> 712
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 712
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 713
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 713
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 714
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 714
Ala Lys Glu Asp Arg Leu Arg Phe Leu Glu Trp Leu Phe Tyr Gly Met
1 5 10 15
Asp Val
<210> 715
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 715
His Asp Ile Tyr Thr Tyr
1 5
<210> 716
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 716
Asp Ala Ser
1
<210> 717
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 717
Gln Gln Tyr Asp Asn His Pro Pro Glu Val Thr
1 5 10
<210> 718
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 718
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 719
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 719
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 720
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 720
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 721
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 721
Gln Asp Ile Asp Asp Asp
1 5
<210> 722
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 722
Glu Ala Ser
1
<210> 723
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 723
Leu Gln His Asp Asn Leu Pro Leu Thr
1 5
<210> 724
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 724
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 725
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 725
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 726
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 726
Ala Thr Glu Asp Pro Gly Pro Val Ala Gly Pro Tyr Tyr Tyr Tyr Gly
1 5 10 15
Met Asp Val
<210> 727
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 727
Gln Ser Leu Leu Tyr Ile Asn Gly Tyr His Tyr
1 5 10
<210> 728
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 728
Leu Gly Ser
1
<210> 729
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 729
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 730
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 730
Gly Tyr Met Phe Thr Asn Tyr Pro
1 5
<210> 731
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 731
Ile Asn Ala Gly Thr Gly Asn Thr
1 5
<210> 732
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 732
Ala Arg Glu Gly Met Phe Tyr Tyr Gly Leu Glu Ser Tyr Tyr Lys Gly
1 5 10 15
Gly Trp Phe Asp Pro
20
<210> 733
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 733
Gln Ser Leu Leu His Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 734
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 734
Leu Ala Ser
1
<210> 735
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 735
Met Gln Ala Leu Gln Thr Pro Leu Thr
1 5
<210> 736
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 736
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 737
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 737
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 738
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 738
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 739
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 739
Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 740
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 740
Thr Leu Ser
1
<210> 741
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 741
Met Gln Arg Leu Gln Phe Pro Leu Thr
1 5
<210> 742
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 742
Thr Tyr Thr Phe Thr Thr Tyr Tyr
1 5
<210> 743
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 743
Ile Tyr Pro Ser Gly Gly Asn Thr
1 5
<210> 744
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 744
Val Arg Asp Gln Asn Tyr Tyr Tyr Ser Ala Met Asp Val
1 5 10
<210> 745
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 745
Gln Asp Ile Arg Asn Tyr
1 5
<210> 746
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 746
Asp Ala Ser
1
<210> 747
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 747
Gln Gln Phe Glu Asn Leu Pro Ile Thr
1 5
<210> 748
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 748
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 749
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 749
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 750
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 750
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 751
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 751
Arg Asp Ile Ser Asp Ser
1 5
<210> 752
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 752
Asp Ala Ser
1
<210> 753
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 753
His Gln Tyr Asp Asn Leu Pro Leu Thr
1 5
<210> 754
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 754
Gly Phe Asn Phe Arg Gly Tyr Ala
1 5
<210> 755
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 755
Ile Ser Gly Ser Gly Gly Thr Thr
1 5
<210> 756
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 756
Ala Gln Ser Tyr Ala Gln Ile Gly Tyr Gly Gly His Ile Asp His
1 5 10 15
<210> 757
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 757
Ser Gly Ile Asp Val Ala Thr Tyr Met
1 5
<210> 758
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 758
Tyr Lys Ser Asp Ser Asp Lys
1 5
<210> 759
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 759
Leu Ile Trp His Gly Ser His Tyr Val
1 5
<210> 760
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 760
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 761
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 761
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 762
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 762
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 763
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 763
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 764
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 764
Trp Gly Ser
1
<210> 765
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 765
Met Gln Ala Val Gln Asn Pro Pro Thr
1 5
<210> 766
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 766
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 767
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 767
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 768
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 768
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 769
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 769
Gln Asn Ile Asp Ser Ser
1 5
<210> 770
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 770
Tyr Ala Ser
1
<210> 771
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 771
His Gln Ser Ser Ser Leu Pro Leu Thr
1 5
<210> 772
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 772
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 773
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 773
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 774
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 774
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 775
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 775
Gln Ser Val Tyr Asn Asn Tyr
1 5
<210> 776
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 776
Asp Ala Ser
1
<210> 777
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 777
Gln Gln Tyr Asn Ser Trp Pro Pro Tyr Thr
1 5 10
<210> 778
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 778
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 779
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 779
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 780
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 780
Ala Arg Asp Ala Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr Asn Pro Asp
1 5 10 15
Ala Phe Asp Ile
20
<210> 781
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 781
Gln Ser Leu Leu Tyr Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 782
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 782
Leu Gly Ser
1
<210> 783
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 783
Met Gln Ala Arg Gln Thr Pro Tyr Thr
1 5
<210> 784
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 784
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 785
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 785
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 786
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 786
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 787
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 787
Glu Ser Val Thr Thr Phe
1 5
<210> 788
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 788
Asp Ala Ser
1
<210> 789
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 789
His Gln His Thr Asn Trp Pro Leu Thr
1 5
<210> 790
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 790
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 791
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 791
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 792
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 792
Ala Thr Glu Val Gly Tyr Cys Ser Gly Gly Ser Cys Tyr Ile Ser Tyr
1 5 10 15
Tyr Tyr Gly Met Asp Val
20
<210> 793
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 793
Gln Ser Leu Leu Tyr Arg Asn Gly His Asn Phe
1 5 10
<210> 794
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 794
Leu Gly Ser
1
<210> 795
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 795
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 796
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 796
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 797
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 797
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 798
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 798
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 799
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 799
Gln Thr Met Asn Asn Tyr
1 5
<210> 800
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 800
Asp Ala Ser
1
<210> 801
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 801
Gln Gln Tyr Gly Asp Trp Leu Pro Ile Thr
1 5 10
<210> 802
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 802
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 803
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 803
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 804
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 804
Ala Thr Ala Phe Pro Glu Ala Thr Ile Ser Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 805
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 805
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 806
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 806
Trp Gly Ser
1
<210> 807
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 807
Met Gln Ala Val Gln Asn Pro Pro Thr
1 5
<210> 808
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 808
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 809
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 809
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 810
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 810
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 811
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 811
Arg Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr His
1 5 10
<210> 812
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 812
Ser Leu Ser
1
<210> 813
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 813
Met Gln Arg Lys Glu Phe Pro Leu Thr
1 5
<210> 814
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 814
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 815
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 815
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 816
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 816
Ala Arg Gly Pro Trp Tyr Tyr Asp Ser Ser Gly Tyr Ser Ser Tyr Ala
1 5 10 15
Tyr Tyr Met Asp Val
20
<210> 817
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 817
Gln Ser Leu Leu His Ser Asp Gly Tyr Asn Tyr
1 5 10
<210> 818
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 818
Leu Gly Ser
1
<210> 819
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 819
Met Gln Ala Leu His Thr Pro Gly Val Thr
1 5 10
<210> 820
<211> 121
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 820
Asp Val Gln Leu Val Glu Ser Gly Gly Asp Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Arg Lys Leu Ser Cys Thr Ala Ser Gly Phe Thr Phe Ser Asn Phe
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Glu Lys Gly Leu Glu Trp Val
35 40 45
Ala Tyr Ile Ser Ser Gly Ser Ser Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Pro Glu Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Thr Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Thr Arg Leu Asp Tyr Tyr Thr Asn Ser Tyr Ser Met Asp His Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 821
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 821
Gly Phe Thr Phe Ser Asn Phe Gly
1 5
<210> 822
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 822
Ile Ser Ser Gly Ser Ser Thr Ile
1 5
<210> 823
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 823
Thr Arg Leu Asp Tyr Tyr Thr Asn Ser Tyr Ser Met Asp His
1 5 10
<210> 824
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 824
Gln Ile Val Leu Thr Gln Ser Pro Ala Leu Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Leu Thr Cys Ser Ala Ser Ser Ser Leu Pro Tyr Ile
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Ser Ser Pro Lys Pro Trp Ile Tyr
35 40 45
Leu Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Arg Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Ser Ser Asn Pro Phe Thr
85 90 95
Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 825
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 825
Ser Ser Leu Pro Tyr
1 5
<210> 826
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 826
Leu Thr Ser
1
<210> 827
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 827
Gln Gln Trp Ser Ser Asn Pro Phe Thr
1 5
<210> 828
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 828
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Ala Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Asn
20 25 30
Trp Met His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Asn Pro Ser Asn Gly Tyr Thr Glu Cys Asn Gln Lys Phe
50 55 60
Arg Asp Lys Ala Thr Leu Ser Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Asn Ser Leu Thr Ser Ala Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Leu Met Ile Ser Ala Trp Leu Pro Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ala
115
<210> 829
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 829
Gly Tyr Thr Phe Thr Ser Asn Trp
1 5
<210> 830
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 830
Ile Asn Pro Ser Asn Gly Tyr Thr
1 5
<210> 831
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 831
Ala Leu Met Ile Ser Ala Trp Leu Pro Tyr
1 5 10
<210> 832
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 832
Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Ile Ser Leu Gly
1 5 10 15
Gln Arg Ala Thr Ile Ser Cys Arg Ala Ser Gln Ser Val Ser Ala Ser
20 25 30
Ser Tyr Ser Tyr Val His Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Lys Leu Leu Ile Lys Tyr Ala Ser Ser Leu Glu Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Asn Ile His
65 70 75 80
Pro Val Glu Glu Glu Asp Thr Ala Thr Tyr Tyr Cys Leu His Thr Trp
85 90 95
Glu Ile Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 833
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 833
Gln Ser Val Ser Ala Ser Ser Tyr Ser Tyr
1 5 10
<210> 834
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 834
Tyr Ala Ser
1
<210> 835
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 835
Leu His Thr Trp Glu Ile Pro Tyr Thr
1 5
<210> 836
<211> 116
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 836
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Thr Arg Pro Gly Ala
1 5 10 15
Ser Val Asn Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Trp Met Gln Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Tyr Pro Gly Asp Gly Asp Thr Arg Phe Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Glu Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Ala Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Thr Tyr Tyr Arg Tyr Asp Asp Tyr Trp Gly Gln Gly Thr Thr Leu
100 105 110
Thr Val Ser Ser
115
<210> 837
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 837
Gly Tyr Thr Phe Thr Ser Tyr Trp
1 5
<210> 838
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 838
Ile Tyr Pro Gly Asp Gly Asp Thr
1 5
<210> 839
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 839
Ala Thr Tyr Tyr Arg Tyr Asp Asp Tyr
1 5
<210> 840
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 840
Gln Ile Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Ser Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
His Trp Tyr Gln Gln Lys Ser Gly Thr Ser Pro Lys Arg Trp Ile Tyr
35 40 45
Asp Thr Ser Lys Leu Ala Ser Gly Val Pro Thr Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Met Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Ser Ser Asn Pro Tyr Thr
85 90 95
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 841
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 841
Ser Ser Val Ser Tyr
1 5
<210> 842
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 842
Asp Thr Ser
1
<210> 843
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 843
Gln Gln Trp Ser Ser Asn Pro Tyr Thr
1 5
<210> 844
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 844
Glu Val Gln Leu Gln Gln Ser Gly Pro Asp Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Met Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Ile Asp Tyr
20 25 30
Asn Ile His Trp Val Lys Gln Ser Gln Gly Lys Ser Leu Asp Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Asn Gly Gly Thr Gly Tyr Asn Gln Lys Phe
50 55 60
Lys Asn Lys Ala Thr Leu Thr Val Asp Ser Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Val Arg Ser Leu Thr Phe Glu Asp Ser Ala Val Tyr Phe Cys
85 90 95
Ala Arg Glu Ala Asp Tyr Tyr Gly Asn Arg Gly Gln Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ala
115 120
<210> 845
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 845
Gly Tyr Thr Phe Ile Asp Tyr Asn
1 5
<210> 846
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 846
Ile Tyr Pro Tyr Asn Gly Gly Thr
1 5
<210> 847
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 847
Ala Arg Glu Ala Asp Tyr Tyr Gly Asn Arg Gly Gln Phe Asp Tyr
1 5 10 15
<210> 848
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 848
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Val Ser Val Gly
1 5 10 15
Glu Thr Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Phe Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Gly Glu Ala Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Asn Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Gly Asn Tyr Tyr Cys Gln His Phe Trp Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Thr Leu Glu Ile Lys
100 105
<210> 849
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 849
Glu Asn Ile Phe Ser Asn
1 5
<210> 850
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 850
Gly Glu Ala
1
<210> 851
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 851
Gln His Phe Trp Gly Thr Pro Tyr Thr
1 5
<210> 852
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 852
Gln Val Thr Leu Lys Glu Ser Gly Pro Gly Ile Leu Gln Pro Ser Gln
1 5 10 15
Thr Leu Ser Leu Thr Cys Ser Phe Ser Gly Phe Ser Leu Asn Thr Ser
20 25 30
Gly Thr Gly Val Gly Trp Ile Arg Gln Pro Ser Gly Lys Gly Leu Glu
35 40 45
Trp Leu Thr His Ile Trp Trp Asn Asp Asn Lys Phe Tyr Asn Thr Phe
50 55 60
Leu Lys Ser Arg Leu Thr Ile Ser Lys Glu Thr Ser Asn Asn Gln Val
65 70 75 80
Phe Leu Lys Ile Ala Ser Val Asp Thr Ala Asp Ala Ala Thr Tyr Tyr
85 90 95
Cys Ala Arg Met Ala Tyr Gly Asn Leu Trp Phe Val Asn Trp Gly Gln
100 105 110
Gly Thr Leu Val Ala Val Ser Thr
115 120
<210> 853
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 853
Gly Phe Ser Leu Asn Thr Ser Gly Thr Gly
1 5 10
<210> 854
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 854
Ile Trp Trp Asn Asp Asn Lys
1 5
<210> 855
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 855
Ala Arg Met Ala Tyr Gly Asn Leu Trp Phe Val Asn
1 5 10
<210> 856
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 856
Asp Ile Val Leu Thr Gln Ser Pro Ala Ser Leu Ala Val Ser Leu Gly
1 5 10 15
Gln Arg Ala Ser Ile Ser Cys Arg Ala Ser Arg Ser Val Thr Ile Ser
20 25 30
Gly Tyr Ser Tyr Met Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro
35 40 45
Arg Leu Leu Phe Tyr Leu Ala Ser Asn Leu Ala Ser Gly Val Pro Ala
50 55 60
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Asn Ile His
65 70 75 80
Pro Val Glu Glu Glu Asp Ala Ala Ile Tyr Tyr Cys Gln His Ser Arg
85 90 95
Glu Leu Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 857
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 857
Arg Ser Val Thr Ile Ser Gly Tyr Ser Tyr
1 5 10
<210> 858
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 858
Leu Ala Ser
1
<210> 859
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 859
Gln His Ser Arg Glu Leu Pro Tyr Thr
1 5
<210> 860
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 860
Gln Ile Gln Leu Val Gln Ser Gly Pro Glu Leu Lys Lys Pro Arg Glu
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Ser Met His Trp Val Lys Gln Ala Pro Gly Lys Gly Leu Lys Trp Met
35 40 45
Gly Trp Ile Asn Thr Glu Thr Gly Glu Pro Thr Tyr Ala Asp Asp Phe
50 55 60
Lys Gly Arg Phe Ala Phe Ser Leu Glu Ala Ser Ala Ser Ser Ala Tyr
65 70 75 80
Leu Gln Ile Asn Ile Leu Lys Asp Glu Asp Thr Ala Thr Tyr Phe Cys
85 90 95
Ala Arg Ser Arg Gly Gly Tyr Tyr Glu Asp Tyr Tyr Ala Leu Asp Tyr
100 105 110
Trp Gly Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 861
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 861
Gly Tyr Thr Phe Thr Asp Tyr Ser
1 5
<210> 862
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 862
Ile Asn Thr Glu Thr Gly Glu Pro
1 5
<210> 863
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 863
Ala Arg Ser Arg Gly Gly Tyr Tyr Glu Asp Tyr Tyr Ala Leu Asp Tyr
1 5 10 15
<210> 864
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 864
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Glu Ser Val Thr Ile Thr Cys Arg Ala Ser Gly Asn Ile His Tyr Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Asn Ala Lys Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Phe Ser Leu Lys Ile Asn Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His Phe Trp Ile Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 865
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 865
Gly Asn Ile His Tyr Tyr
1 5
<210> 866
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 866
Asn Ala Lys
1
<210> 867
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 867
Gln His Phe Trp Ile Ser Pro Pro Thr
1 5
<210> 868
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 868
Glu Val Gln Leu Arg Gln Ser Gly Ala Asp Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Thr
20 25 30
Tyr Ile Asp Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Arg Ile Asp Pro Ala Asn Gly Asn Thr Lys Tyr Asp Pro Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Ile Thr Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Asn Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Gly Ser Tyr Pro Tyr Phe Asp Tyr Trp Gly Arg Gly Thr
100 105 110
Thr Leu Ala Val Ser Ser
115
<210> 869
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 869
Gly Phe Asn Ile Lys Asp Thr Tyr
1 5
<210> 870
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 870
Ile Asp Pro Ala Asn Gly Asn Thr
1 5
<210> 871
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 871
Ala Arg Tyr Gly Ser Tyr Pro Tyr Phe Asp Tyr
1 5 10
<210> 872
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 872
Ser Ile Val Met Thr Gln Thr Pro Lys Phe Leu Leu Ile Ser Ala Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Arg Asn Asp
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Met
35 40 45
Tyr Tyr Ala Ser Asn Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Tyr Gly Thr Asp Phe Thr Phe Thr Ile Ser Thr Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Val Tyr Phe Cys Gln Gln Asp Tyr Ser Ser Pro Pro
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 873
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 873
Gln Ser Val Arg Asn Asp
1 5
<210> 874
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 874
Tyr Ala Ser
1
<210> 875
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 875
Gln Gln Asp Tyr Ser Ser Pro Pro Thr
1 5
<210> 876
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 876
Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Arg Pro Gly Val
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Val Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Val Met His Trp Val Lys Gln Ser His Ala Lys Ser Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Pro Tyr Ser Gly Asn Thr Asn Tyr Asn Gln Asn Phe
50 55 60
Lys Gly Lys Ala Thr Met Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Ala Leu Ala Arg Leu Thr Ser Glu Asp Ser Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Met Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ala
115
<210> 877
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 877
Gly Tyr Thr Phe Thr Asp Tyr Val
1 5
<210> 878
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 878
Ile Ser Pro Tyr Ser Gly Asn Thr
1 5
<210> 879
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 879
Ala Arg Glu Gly Asp Leu Pro Met Phe Ala Tyr
1 5 10
<210> 880
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 880
Gln Ile Val Leu Thr Gln Ser Pro Thr Ile Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Ser Ala Ser Ser Ser Val Ser Tyr Ile
20 25 30
Tyr Trp Tyr Gln Gln Asn Pro Gly Ser Ser Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Ile Leu Ala Ser Gly Val Pro Phe Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Arg Met Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Thr Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Ser Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 881
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 881
Ser Ser Val Ser Tyr
1 5
<210> 882
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 882
Asp Thr Ser
1
<210> 883
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 883
Gln Gln Trp Thr Ser Tyr Pro Leu Thr
1 5
<210> 884
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 884
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Asn Ile Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Asn Gln Lys Phe
50 55 60
Lys Ser Lys Ala Thr Leu Ser Ala Asp Asn Pro Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Asn Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Pro Val Thr Val Ser Ser
115 120
<210> 885
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 885
Gly Tyr Thr Phe Thr Asp Tyr Asn
1 5
<210> 886
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 886
Ile Tyr Pro Tyr Ile Gly Gly Ser
1 5
<210> 887
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 887
Ala Arg Glu Asp Lys Thr Ala Arg Asn Ala Met Asp Tyr
1 5 10
<210> 888
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 888
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Val Ser Val Gly
1 5 10 15
Glu Thr Val Thr Ile Ile Cys Arg Val Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Glu Ala Thr Asn Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Asn Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His Phe Trp Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 889
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 889
Glu Asn Ile Tyr Ser Asn
1 5
<210> 890
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 890
Glu Ala Thr
1
<210> 891
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 891
Gln His Phe Trp Gly Thr Pro Tyr Thr
1 5
<210> 892
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 892
Asp Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Ser Leu Ser Leu Thr Cys Thr Val Thr Gly Tyr Ser Ile Thr Ser Asp
20 25 30
Tyr Ala Trp Asn Trp Ile Arg Gln Phe Pro Gly Asn Lys Leu Glu Trp
35 40 45
Met Gly Tyr Ile Ser Tyr Ser Gly Ser Thr Ser Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Ile Ser Ile Thr Arg Asp Thr Ser Lys Asn Gln Phe Phe
65 70 75 80
Leu Gln Leu Asn Ser Val Thr Thr Glu Asp Thr Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg His Tyr Arg Tyr Pro Pro Tyr Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 893
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 893
Gly Tyr Ser Ile Thr Ser Asp Tyr Ala
1 5
<210> 894
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 894
Ile Ser Tyr Ser Gly Ser Thr
1 5
<210> 895
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 895
Ala Arg His Tyr Arg Tyr Pro Pro Tyr Ala Met Asp Tyr
1 5 10
<210> 896
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 896
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Met Ser Val Gly
1 5 10 15
Gln Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Leu Asn Ser
20 25 30
Ser Asn Gln Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Val Tyr Phe Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ile Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Leu Ala Asp Tyr Phe Cys Gln Gln
85 90 95
His Tyr Ser Thr Pro Phe Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile
100 105 110
Lys
<210> 897
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 897
Gln Ser Leu Leu Asn Ser Ser Asn Gln Lys Asn Tyr
1 5 10
<210> 898
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 898
Phe Ala Ser
1
<210> 899
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 899
Gln Gln His Tyr Ser Thr Pro Phe Thr
1 5
<210> 900
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 900
Gln Val Gln Leu Lys Gln Ser Gly Pro Gly Leu Val Gln Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Ser Phe
20 25 30
Gly Val His Trp Val Arg Gln Ser Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Ser Gly Gly Thr Thr Val Tyr Asp Ala Ala Phe Ile
50 55 60
Ser Arg Leu Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Phe
65 70 75 80
Lys Met Asn Ser Leu Gln Thr Asn Asp Thr Ala Ile Tyr Tyr Cys Ala
85 90 95
Arg Lys Arg Gly Asn Phe Tyr Val Met Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Ser Val Thr Val Ser Ser
115
<210> 901
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 901
Gly Phe Ser Leu Thr Ser Phe Gly
1 5
<210> 902
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 902
Ile Trp Ser Gly Gly Thr Thr
1 5
<210> 903
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 903
Ala Arg Lys Arg Gly Asn Phe Tyr Val Met Asp Tyr
1 5 10
<210> 904
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 904
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ala Met Ser Val Gly
1 5 10 15
Gln Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Leu Asn Ser
20 25 30
Ser Asn Gln Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Val Tyr Phe Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Ile Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Leu Ala Asp Tyr Phe Cys Gln Gln
85 90 95
His Tyr Ser Thr Pro Phe Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile
100 105 110
Lys
<210> 905
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 905
Gln Ser Leu Leu Asn Ser Ser Asn Gln Lys Asn Tyr
1 5 10
<210> 906
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 906
Phe Ala Ser
1
<210> 907
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 907
Gln Gln His Tyr Ser Thr Pro Phe Thr
1 5
<210> 908
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 908
Gln Val Gln Leu Gln Gln Pro Gly Ser Glu Leu Val Arg Pro Gly Thr
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Trp Val His Trp Val Arg Gln Arg His Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Asn Val Tyr Pro Gly Ser Gly Ser Thr Asn Tyr Asp Glu Lys Phe
50 55 60
Lys Ser Lys Gly Thr Leu Thr Val Asp Thr Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met His Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Gly Val Leu Arg Phe Pro Leu Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Leu Thr Val Ser Ser
115
<210> 909
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 909
Gly Tyr Thr Phe Thr Ser Tyr Trp
1 5
<210> 910
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 910
Val Tyr Pro Gly Ser Gly Ser Thr
1 5
<210> 911
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 911
Thr Arg Gly Val Leu Arg Phe Pro Leu Asp Tyr
1 5 10
<210> 912
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 912
Asp Ile Val Met Thr Gln Ala Ala Pro Ser Val Pro Val Thr Pro Gly
1 5 10 15
Glu Ser Val Ser Ile Ser Cys Arg Ser Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu Tyr Trp Phe Leu Gln Arg Pro Gly Gln Ser
35 40 45
Pro His Leu Leu Ile Tyr Arg Met Ser Asn Leu Ala Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Ala Phe Thr Leu Arg Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln His
85 90 95
Leu Glu Tyr Pro Leu Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105 110
<210> 913
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 913
Lys Ser Leu Leu His Ser Asn Gly Asn Thr Tyr
1 5 10
<210> 914
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 914
Arg Met Ser
1
<210> 915
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 915
Met Gln His Leu Glu Tyr Pro Leu Thr
1 5
<210> 916
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 916
Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Val Val Arg Pro Gly Val
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Gly Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Lys Gln Ser His Ala Lys Ser Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Asn Thr Asn Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Met Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ala Arg Leu Thr Ser Glu Asp Ser Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Met Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ala
115
<210> 917
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 917
Gly Tyr Lys Phe Pro Asp Tyr Val
1 5
<210> 918
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 918
Ile Ser Ile Tyr Ser Gly Asn Thr
1 5
<210> 919
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 919
Ala Arg Glu Gly Asp Leu Pro Met Phe Ala Tyr
1 5 10
<210> 920
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 920
Gln Ile Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Asn Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Ile Ser Ser Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Asn Leu Ala Ser Gly Val Pro Val Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Gly Arg Met Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Trp Ser Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Ala Gly Thr Lys Val Glu Val Lys
100 105
<210> 921
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 921
Ser Ser Val Ser Tyr
1 5
<210> 922
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 922
Asp Thr Ser
1
<210> 923
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 923
Gln Gln Trp Ser Ser Tyr Pro Leu Thr
1 5
<210> 924
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 924
Gln Val Gln Leu Lys Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Ala Tyr
20 25 30
Asn Ile Asn Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Met Ile Trp Gly Asp Gly Asn Thr Asp Tyr Asn Ser Pro Leu Lys
50 55 60
Ser Arg Leu Thr Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asp Ser Leu Gln Thr Asp Asp Thr Ala Arg Tyr Tyr Cys Ala
85 90 95
Arg Asp Leu Lys Val Arg Arg Asp Ser Pro Tyr Thr Met Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 925
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 925
Gly Phe Ser Leu Thr Ala Tyr Asn
1 5
<210> 926
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 926
Ile Trp Gly Asp Gly Asn Thr
1 5
<210> 927
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 927
Ala Arg Asp Leu Lys Val Arg Arg Asp Ser Pro Tyr Thr Met Asp Tyr
1 5 10 15
<210> 928
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 928
Asn Ile Met Met Thr Gln Ser Pro Ser Ser Leu Ala Val Ser Ala Gly
1 5 10 15
Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Val Leu Tyr Ser
20 25 30
Ser Asn Gln Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Asn Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys His Gln
85 90 95
Tyr Leu Ser Ser Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 929
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 929
Gln Ser Val Leu Tyr Ser Ser Asn Gln Lys Asn Tyr
1 5 10
<210> 930
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 930
Trp Ala Ser
1
<210> 931
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 931
His Gln Tyr Leu Ser Ser Tyr Thr
1 5
<210> 932
<211> 124
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 932
Asp Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Ser Leu Ser Leu Thr Cys Thr Val Thr Gly Tyr Ser Leu Thr Ser Asp
20 25 30
Tyr Ala Trp Asn Trp Ile Arg Gln Phe Pro Gly Asn Lys Leu Glu Trp
35 40 45
Met Gly Tyr Ile Thr Tyr Ser Gly Gly Thr Thr Tyr Asn Pro Ser Leu
50 55 60
Lys Ser Arg Ile Ser Ile Thr Arg Asp Thr Ser Lys Asn Gln Phe Phe
65 70 75 80
Leu Gln Leu Thr Ser Val Thr Thr Glu Asp Thr Ala Thr Tyr Tyr Cys
85 90 95
Ala Arg Arg Gly Ser Gly Thr Thr Val Val Gly Asp Trp Tyr Phe Asp
100 105 110
Val Trp Gly Ala Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 933
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 933
Gly Tyr Ser Leu Thr Ser Asp Tyr Ala
1 5
<210> 934
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 934
Ile Thr Tyr Ser Gly Gly Thr
1 5
<210> 935
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 935
Ala Arg Arg Gly Ser Gly Thr Thr Val Val Gly Asp Trp Tyr Phe Asp
1 5 10 15
Val
<210> 936
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 936
Asp Ile Val Met Ser Gln Ser Pro Ser Ser Leu Ala Val Ser Val Gly
1 5 10 15
Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Leu Tyr Ser
20 25 30
Phe Asn Gln Lys Tyr Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Ser Pro Lys Leu Leu Ile Tyr Trp Ala Ser Thr Arg Glu Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Pro Thr
65 70 75 80
Ile Ser Ser Val Thr Ala Glu Asp Leu Ala Val Tyr Tyr Cys Gln Gln
85 90 95
Phe Tyr Thr Tyr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Met
100 105 110
Lys
<210> 937
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 937
Gln Ser Leu Leu Tyr Ser Phe Asn Gln Lys Tyr Tyr
1 5 10
<210> 938
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 938
Trp Ala Ser
1
<210> 939
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 939
Gln Gln Phe Tyr Thr Tyr Pro Tyr Thr
1 5
<210> 940
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 940
Glu Val Gln Val Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Lys Ser
20 25 30
Asn Met His Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Asn Gly Gly Thr Gly Tyr Asn Gln Asn Phe
50 55 60
Lys Ser Lys Ala Thr Leu Thr Val Asp Ile Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Leu Glu Asp Ser Ala Val Tyr Leu Cys
85 90 95
Ala Arg Glu Ala Asp Tyr Tyr Gly Asn Arg Gly Gln Phe Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Leu Val Thr Val Ser Ala
115 120
<210> 941
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 941
Gly Tyr Thr Phe Thr Lys Ser Asn
1 5
<210> 942
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 942
Ile Tyr Pro Tyr Asn Gly Gly Thr
1 5
<210> 943
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 943
Ala Arg Glu Ala Asp Tyr Tyr Gly Asn Arg Gly Gln Phe Asp Tyr
1 5 10 15
<210> 944
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 944
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Val Ser Val Gly
1 5 10 15
Glu Thr Val Thr Ile Thr Cys Arg Ala Ser Asp Asn Ile Phe Ser Asn
20 25 30
Leu Ala Trp Tyr His Gln Lys Gln Gly Lys Ser Pro His Leu Leu Val
35 40 45
Tyr Gly Ala Thr Asn Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Asn Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Gly Asp Tyr Tyr Cys Gln His Phe Trp Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 945
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 945
Asp Asn Ile Phe Ser Asn
1 5
<210> 946
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 946
Gly Ala Thr
1
<210> 947
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 947
Gln His Phe Trp Gly Thr Pro Tyr Thr
1 5
<210> 948
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 948
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Met Arg Pro Gly Thr
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Asn His
20 25 30
Leu Ile Glu Trp Ile Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Ser Asp Ser Thr Asp Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Asp Asp Ser Ala Val Tyr Phe Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Asn Ser Trp Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ala
115
<210> 949
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 949
Gly Tyr Ala Phe Thr Asn His Leu
1 5
<210> 950
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 950
Ile Asn Pro Gly Ser Asp Ser Thr
1 5
<210> 951
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 951
Ala Arg Ser Leu Tyr Tyr Asn Ser Trp Phe Val Tyr
1 5 10
<210> 952
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 952
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Glu Thr Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Arg Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Asn Ala Lys Thr Leu Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Phe Ser Leu Lys Ile Asn Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His His Tyr Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 953
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 953
Glu Asn Ile Tyr Ser Tyr
1 5
<210> 954
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 954
Asn Ala Lys
1
<210> 955
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 955
Gln His His Tyr Gly Thr Pro Tyr Thr
1 5
<210> 956
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 956
Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Ile Arg Asp Thr
20 25 30
Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Lys Ile Asp Pro Ala Asn Gly Lys Ser Glu Tyr Asp Pro Lys Phe
50 55 60
Gln Gly Arg Ala Thr Met Thr Thr Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Tyr Gly Tyr Tyr Pro Tyr Phe Asp Val Trp Gly Ala Gly Thr
100 105 110
Thr Val Thr Val Phe Ser
115
<210> 957
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 957
Gly Phe Ser Ile Arg Asp Thr Tyr
1 5
<210> 958
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 958
Ile Asp Pro Ala Asn Gly Lys Ser
1 5
<210> 959
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 959
Thr Arg Tyr Gly Tyr Tyr Pro Tyr Phe Asp Val
1 5 10
<210> 960
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 960
Ser Ile Val Met Thr Gln Thr Pro Lys Phe Leu Leu Val Ser Ala Gly
1 5 10 15
Asp Arg Val Ala Ile Thr Cys Lys Ala Ser Gln Ser Val Arg His Asp
20 25 30
Val Val Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Ala Ser Ser Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Tyr Gly Thr Asp Phe Thr Phe Thr Ile Ser Thr Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Leu Tyr Phe Cys Leu Gln Asp Phe Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 961
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 961
Gln Ser Val Arg His Asp
1 5
<210> 962
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 962
Tyr Ala Ser
1
<210> 963
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 963
Leu Gln Asp Phe Ser Ser Pro Trp Thr
1 5
<210> 964
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 964
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Thr Ser Gly Tyr Thr Phe Thr Lys Asn
20 25 30
Thr Met His Trp Val Arg Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Gly Ile Asn Pro Asn Ser Gly Gly Ala Ser Phe Asn Gln Lys Phe
50 55 60
Met Gly Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Gly Tyr Asp Gly Asp Trp Phe Phe Asp Val Trp Gly Ala
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 965
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 965
Gly Tyr Thr Phe Thr Lys Asn Thr
1 5
<210> 966
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 966
Ile Asn Pro Asn Ser Gly Gly Ala
1 5
<210> 967
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 967
Ala Arg Asp Gly Tyr Asp Gly Asp Trp Phe Phe Asp Val
1 5 10
<210> 968
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 968
Asp Ile Gln Met Asn Gln Ser Pro Phe Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Thr Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Asn Ile Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asp Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Asn Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Ser Tyr Pro Arg
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 969
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 969
Gln Asn Ile Tyr Val Trp
1 5
<210> 970
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 970
Lys Ala Ser
1
<210> 971
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 971
Gln Gln Gly Gln Ser Tyr Pro Arg Thr
1 5
<210> 972
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 972
Gln Val Gln Leu Lys Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ile Gly His
20 25 30
Gly Val Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Gly Asp Gly Asn Thr Asp Tyr Asn Ser Ala Leu Lys
50 55 60
Ser Arg Leu Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Arg Tyr Phe Cys Ala
85 90 95
Val Asn Ser Ala Met Asp Tyr Trp Gly Gln Gly Thr Ala Val Thr Val
100 105 110
Ser Ser
<210> 973
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 973
Gly Phe Ser Leu Ile Gly His Gly
1 5
<210> 974
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 974
Ile Trp Gly Asp Gly Asn Thr
1 5
<210> 975
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 975
Ala Val Asn Ser Ala Met Asp Tyr
1 5
<210> 976
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 976
Asn Ile Val Met Thr Gln Ser Pro Lys Ser Met Ser Met Ser Val Gly
1 5 10 15
Glu Arg Val Thr Leu Asn Cys Thr Ala Ser Glu Asn Val Ala Ser Phe
20 25 30
Val Ser Trp Tyr Gln Gln Lys Pro Glu Gln Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Thr Ser Asn Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Ala Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Ala
65 70 75 80
Glu Asp Leu Gly Asp Tyr His Cys Gly Gln Ser Tyr Asn Tyr Pro Phe
85 90 95
Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Glu
100 105
<210> 977
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 977
Glu Asn Val Ala Ser Phe
1 5
<210> 978
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 978
Gly Thr Ser
1
<210> 979
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 979
Gly Gln Ser Tyr Asn Tyr Pro Phe Thr
1 5
<210> 980
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 980
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Ala Phe Thr Asn Tyr
20 25 30
Leu Ile Glu Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Ser Gly Gly Ile Tyr Tyr Asn Asp Lys Phe
50 55 60
Lys Val Lys Thr Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Asp Asp Ser Ala Val Tyr Phe Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Trp Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ala
115
<210> 981
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 981
Gly Tyr Ala Phe Thr Asn Tyr Leu
1 5
<210> 982
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 982
Ile Asn Pro Gly Ser Gly Gly Ile
1 5
<210> 983
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 983
Ala Arg Ser Glu Thr His Asp Thr Trp Phe Ala Tyr
1 5 10
<210> 984
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 984
Asp Ile Val Met Thr Gln Ser Gln Lys Phe Ile Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Ser Ile Thr Cys Lys Ala Ser Gln Ser Val Arg Ile Ala
20 25 30
Val Ala Trp Phe Gln Gln Lys Pro Gly Gln Ser Pro Lys Ala Leu Ile
35 40 45
Tyr Leu Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asn Val Gln Ser
65 70 75 80
Glu Asp Leu Ala Asp Tyr Phe Cys Leu Gln His Trp Asn Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 985
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 985
Gln Ser Val Arg Ile Ala
1 5
<210> 986
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 986
Leu Ala Ser
1
<210> 987
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 987
Leu Gln His Trp Asn Tyr Pro Tyr Thr
1 5
<210> 988
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 988
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Thr
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ala Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Lys Gln Arg Pro Gly His Gly Leu Glu Trp Ile
35 40 45
Gly Asp Ile Tyr Pro Gly Gly Gly Tyr Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Thr Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Ile Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ala
115 120
<210> 989
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 989
Gly Tyr Thr Phe Thr Asn Tyr Trp
1 5
<210> 990
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 990
Ile Tyr Pro Gly Gly Gly Tyr Thr
1 5
<210> 991
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 991
Ala Ser Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe
1 5 10
<210> 992
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 992
Asp Ile Val Met Thr Gln Ser Gln Lys Phe Ile Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Ser Ile Thr Cys Lys Ala Ser Gln Ser Val Arg Ile Ala
20 25 30
Val Ala Trp Phe Gln Gln Lys Pro Gly Gln Ser Pro Lys Ala Leu Ile
35 40 45
Tyr Leu Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asn Val Gln Ser
65 70 75 80
Glu Asp Leu Ala Asp Tyr Phe Cys Leu Gln His Trp Asn Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys Arg
100 105
<210> 993
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 993
Gln Ser Val Arg Ile Ala
1 5
<210> 994
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 994
Leu Ala Ser
1
<210> 995
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 995
Leu Gln His Trp Asn Tyr Pro Tyr Thr
1 5
<210> 996
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 996
Gln Val Gln Leu Lys Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Ser Gly Tyr
20 25 30
Gly Ile Lys Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Thr Ile Trp Gly Asp Gly Ser Thr Asp Tyr Asn Ser Ala Leu Lys
50 55 60
Ser Arg Leu Ser Ile Ser Lys Asp Asn Ser Lys Ser Gln Val Phe Leu
65 70 75 80
Lys Met Thr Ser Leu Gln Thr Asp Asp Thr Ala Arg Tyr Tyr Cys Ala
85 90 95
Ser Asp Ser Leu Gly Ile Thr Phe Gly Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ala
115
<210> 997
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 997
Gly Phe Ser Leu Ser Gly Tyr Gly
1 5
<210> 998
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 998
Ile Trp Gly Asp Gly Ser Thr
1 5
<210> 999
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 999
Ala Ser Asp Ser Leu Gly Ile Thr Phe Gly Tyr
1 5 10
<210> 1000
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1000
Asp Ile Gln Met Thr Gln Thr Thr Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Asp Arg Val Thr Ile Ser Cys Arg Ala Ser Gln Asp Ile Ser Asn Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Asp Gly Thr Val Lys Leu Leu Ile
35 40 45
Tyr Tyr Thr Ser Arg Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Ser Leu Thr Ile Ser Asn Leu Glu Gln
65 70 75 80
Glu Asp Ile Ala Thr Tyr Phe Cys Gln Gln Gly Asn Thr Leu Pro Leu
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 1001
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1001
Gln Asp Ile Ser Asn Tyr
1 5
<210> 1002
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1002
Tyr Thr Ser
1
<210> 1003
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1003
Gln Gln Gly Asn Thr Leu Pro Leu Thr
1 5
<210> 1004
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1004
Gln Val Gln Leu Lys Glu Ser Gly Pro Gly Leu Val Ala Pro Ser Gln
1 5 10 15
Ser Leu Ser Ile Thr Cys Thr Val Ser Gly Phe Ser Leu Thr Ser Tyr
20 25 30
Ala Ile Thr Trp Val Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp Leu
35 40 45
Gly Val Ile Trp Pro Gly Gly Gly Thr Asn Tyr Asn Ser Ala Leu Lys
50 55 60
Ser Arg Leu Ser Ile Ser Lys Asp Asn Ser Lys Ser His Leu Phe Leu
65 70 75 80
Lys Met Asn Ser Leu Gln Thr Asp Asp Thr Ala Arg Tyr Tyr Cys Val
85 90 95
Arg Ser Tyr Asp Gly Tyr Leu Asp Trp Tyr Phe Asp Val Trp Gly Thr
100 105 110
Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 1005
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1005
Gly Phe Ser Leu Thr Ser Tyr Ala
1 5
<210> 1006
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1006
Ile Trp Pro Gly Gly Gly Thr
1 5
<210> 1007
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1007
Val Arg Ser Tyr Asp Gly Tyr Leu Asp Trp Tyr Phe Asp Val
1 5 10
<210> 1008
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1008
Asn Ile Val Met Thr Gln Ser Pro Lys Ser Met Ser Met Ser Val Gly
1 5 10 15
Glu Arg Val Thr Leu Ser Cys Lys Ala Ser Glu Asn Val Gly Thr Tyr
20 25 30
Val Ser Trp Tyr Gln Gln Lys Pro Asp Gln Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Ser Asn Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Ala Thr Asp Phe Thr Leu Ile Ile Ser Ser Val Gln Ala
65 70 75 80
Glu Asp Leu Ser Asp Tyr His Cys Gly Gln Ser Tyr Ser Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Ile
100 105
<210> 1009
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1009
Glu Asn Val Gly Thr Tyr
1 5
<210> 1010
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1010
Gly Ala Ser
1
<210> 1011
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1011
Gly Gln Ser Tyr Ser Tyr Pro Tyr Thr
1 5
<210> 1012
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1012
Gln Ile Gln Leu Val Gln Ser Gly Pro Asp Leu Lys Lys Pro Gly Glu
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Thr Tyr
20 25 30
Gly Met Ser Trp Val Lys Gln Ala Pro Gly Arg Gly Leu Lys Trp Met
35 40 45
Gly Trp Ile Asn Thr Tyr Ser Gly Val Ser Thr Phe Pro Asp Asp Phe
50 55 60
Lys Gly Arg Phe Ala Phe Ser Leu Glu Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Asn Asn Leu Lys Asn Glu Asp Ser Ala Thr Tyr Phe Cys
85 90 95
Ala Arg Leu Gly Met Gly Ser Thr Thr Gly Ala Gly Tyr Phe Asp Val
100 105 110
Trp Gly Thr Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 1013
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1013
Gly Tyr Thr Phe Thr Thr Tyr Gly
1 5
<210> 1014
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1014
Ile Asn Thr Tyr Ser Gly Val Ser
1 5
<210> 1015
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1015
Ala Arg Leu Gly Met Gly Ser Thr Thr Gly Ala Gly Tyr Phe Asp Val
1 5 10 15
<210> 1016
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1016
Asp Ile Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Ser Ala Ser Ser Ser Val Ser Ser Trp
20 25 30
Tyr Leu His Trp Tyr Gln Gln Lys Ser Gly Ala Ser Pro Lys Leu Trp
35 40 45
Ile Tyr Gly Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu
65 70 75 80
Ala Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr Arg Ser Asp Pro
85 90 95
Tyr Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1017
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1017
Ser Ser Val Ser Ser Trp Tyr
1 5
<210> 1018
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1018
Gly Thr Ser
1
<210> 1019
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1019
Gln Gln Tyr Arg Ser Asp Pro Tyr Thr
1 5
<210> 1020
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1020
Gln Ile Gln Leu Val Gln Ser Gly Pro Glu Leu Lys Lys Pro Gly Glu
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Thr Tyr
20 25 30
Gly Met Ser Trp Val Lys Gln Ala Pro Gly Lys Gly Leu Lys Trp Met
35 40 45
Gly Trp Ile Asn Thr Tyr Ser Gly Val Ser Thr Tyr Ala Asp Asp Phe
50 55 60
Glu Gly Arg Phe Ala Phe Ser Leu Glu Thr Ser Val Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Asn Asn Leu Lys Asn Glu Asp Thr Ala Thr Tyr Phe Cys
85 90 95
Ala Arg Leu Gly Arg Gly Ser Thr Thr Gly Ala Gly Tyr Leu Asp Val
100 105 110
Trp Gly Thr Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 1021
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1021
Gly Tyr Thr Phe Thr Thr Tyr Gly
1 5
<210> 1022
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1022
Ile Asn Thr Tyr Ser Gly Val Ser
1 5
<210> 1023
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1023
Ala Arg Leu Gly Arg Gly Ser Thr Thr Gly Ala Gly Tyr Leu Asp Val
1 5 10 15
<210> 1024
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1024
Asp Ile Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Ser Met Thr Cys Ser Ala Ser Ser Ser Val Ser Ser Trp
20 25 30
Tyr Leu His Trp Tyr Gln Gln Lys Ser Gly Ala Ser Pro Lys Leu Trp
35 40 45
Ile Tyr Gly Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu
65 70 75 80
Ala Glu Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Tyr His Ser Asp Pro
85 90 95
Tyr Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1025
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1025
Ser Ser Val Ser Ser Trp Tyr
1 5
<210> 1026
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1026
Gly Thr Ser
1
<210> 1027
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1027
Gln Gln Tyr His Ser Asp Pro Tyr Thr
1 5
<210> 1028
<211> 123
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1028
Gln Ile Gln Leu Val Gln Ser Gly Pro Asp Leu Lys Lys Pro Gly Glu
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Thr Tyr
20 25 30
Gly Met Ser Trp Val Lys Gln Ala Pro Gly Arg Gly Leu Lys Trp Met
35 40 45
Gly Trp Ile Asn Thr Tyr Ser Gly Val Ser Thr Phe Pro Asp Asp Phe
50 55 60
Lys Gly Arg Phe Ala Phe Ser Leu Glu Thr Ser Ala Ser Thr Ala Tyr
65 70 75 80
Leu Gln Ile Asn Asn Leu Lys Asn Glu Asp Ser Ala Thr Tyr Phe Cys
85 90 95
Ala Arg Leu Gly Met Gly Ser Thr Thr Gly Ala Gly Tyr Phe Asp Val
100 105 110
Trp Gly Thr Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 1029
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1029
Gly Tyr Thr Phe Thr Thr Tyr Gly
1 5
<210> 1030
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1030
Ile Asn Thr Tyr Ser Gly Val Ser
1 5
<210> 1031
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1031
Ala Arg Leu Gly Met Gly Ser Thr Thr Gly Ala Gly Tyr Phe Asp Val
1 5 10 15
<210> 1032
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1032
Glu Asn Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Leu Gly
1 5 10 15
Glu Lys Val Thr Leu Ser Cys Arg Ala Ser Ser Ser Val Asn Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Ser Asp Ala Ser Pro Lys Leu Trp Ile Tyr
35 40 45
Tyr Thr Ser Asn Leu Ala Pro Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Asn Ser Tyr Ser Leu Thr Ile Ser Ser Val Glu Gly Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Phe Thr Ser Ser Pro Trp Thr
85 90 95
Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1033
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1033
Ser Ser Val Asn Tyr
1 5
<210> 1034
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1034
Tyr Thr Ser
1
<210> 1035
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1035
Gln Gln Phe Thr Ser Ser Pro Trp Thr
1 5
<210> 1036
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1036
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Ser Met Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Asp Ile Thr Pro Asn Asn Gly Ser Pro Asn Tyr Asn Gln Lys Phe
50 55 60
Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Leu Phe Phe Asp Tyr Trp Gly His Gly Thr Thr Leu Thr Val
100 105 110
Ser Ser
<210> 1037
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1037
Gly Tyr Thr Phe Thr Asp Tyr Ser
1 5
<210> 1038
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1038
Ile Thr Pro Asn Asn Gly Ser Pro
1 5
<210> 1039
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1039
Ala Ser Leu Phe Phe Asp Tyr
1 5
<210> 1040
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1040
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Val Ser Ala Gly
1 5 10 15
Glu Lys Val Thr Met Ser Cys Lys Ser Ser Gln Ser Leu Leu Asn Ser
20 25 30
Gly Asn Gln Lys Asn Tyr Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln
35 40 45
Pro Pro Lys Leu Leu Ile Tyr Gly Ala Ser Thr Arg Asp Ser Gly Val
50 55 60
Pro Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr
65 70 75 80
Ile Thr Ser Val Gln Ala Glu Asp Leu Ala Val Tyr Tyr Cys Gln Asn
85 90 95
Asp His Thr Tyr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile
100 105 110
Lys
<210> 1041
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1041
Gln Ser Leu Leu Asn Ser Gly Asn Gln Lys Asn Tyr
1 5 10
<210> 1042
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1042
Gly Ala Ser
1
<210> 1043
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1043
Gln Asn Asp His Thr Tyr Pro Tyr Thr
1 5
<210> 1044
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1044
Gln Val Gln Leu Gln Gln Pro Gly Thr Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Phe Thr Phe Thr Thr His
20 25 30
Trp Met His Trp Val Arg Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Asn Ile Tyr Pro Ser Asn Gly Gly Ser Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Thr Lys Ala Thr Leu Thr Val Asp Arg Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met His Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Val Asn Trp Asp Gly Tyr Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Thr Val Ser Ser
115 120
<210> 1045
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1045
Gly Phe Thr Phe Thr Thr His Trp
1 5
<210> 1046
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1046
Ile Tyr Pro Ser Asn Gly Gly Ser
1 5
<210> 1047
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1047
Ala Arg Arg Val Asn Trp Asp Gly Tyr Tyr Phe Asp Tyr
1 5 10
<210> 1048
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1048
Asp Ile Val Met Thr Gln Ser Gln Lys Phe Met Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Ser Val Thr Cys Lys Ala Ser Gln Asn Val Gly Thr Asn
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Ser Ala Ser Tyr Arg Tyr Ser Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asn Val Gln Ser
65 70 75 80
Glu Asp Leu Ala Asp Tyr Phe Cys Gln Gln Tyr Asn Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1049
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1049
Gln Asn Val Gly Thr Asn
1 5
<210> 1050
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1050
Ser Ala Ser
1
<210> 1051
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1051
Gln Gln Tyr Asn Ser Tyr Pro Leu Thr
1 5
<210> 1052
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1052
Gln Val Gln Leu Gln Glu Ser Gly Thr Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Trp Ile His Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Asn Ile Asn Pro Ser Asn Gly Gly Thr Asn Tyr Asn Glu Lys Phe
50 55 60
Lys Ser Lys Ala Ala Leu Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Gly Leu Pro Tyr Phe Phe Asp Tyr Trp Gly Gln Gly Thr
100 105 110
Thr Leu Thr Val Ser Ser
115
<210> 1053
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1053
Gly Tyr Thr Phe Thr Ser Tyr Trp
1 5
<210> 1054
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1054
Ile Asn Pro Ser Asn Gly Gly Thr
1 5
<210> 1055
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1055
Ala Arg Arg Gly Leu Pro Tyr Phe Phe Asp Tyr
1 5 10
<210> 1056
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1056
Asp Ile Val Met Thr Gln Ser Gln Lys Phe Met Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Ser Val Thr Cys Lys Gly Ser Gln Asn Val Gly Tyr Asn
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Ala Leu Val
35 40 45
Tyr Ser Ala Ser Asp Arg His Ser Gly Val Pro Asp Arg Phe Ala Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asn Val Gln Ser
65 70 75 80
Glu Asp Leu Ala Glu Tyr Phe Cys Gln Gln Tyr Asn Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 1057
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1057
Gln Asn Val Gly Tyr Asn
1 5
<210> 1058
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1058
Ser Ala Ser
1
<210> 1059
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1059
Gln Gln Tyr Asn Ser Tyr Pro Leu Thr
1 5
<210> 1060
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1060
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 1061
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1061
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 1062
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1062
Ala Arg Glu Glu Val Ser Ser Pro Tyr Gly Met Asp Val
1 5 10
<210> 1063
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1063
Thr Gly Ala Val Thr Ser Gly His Tyr
1 5
<210> 1064
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1064
Asp Thr Gly
1
<210> 1065
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1065
Leu Leu Ser Tyr Ser Gly Ala Ser Trp Val
1 5 10
<210> 1066
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1066
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1067
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1067
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1068
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1068
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1069
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1069
Gln Gly Ile Gly Asn Tyr
1 5
<210> 1070
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1070
Ser Ala Ser
1
<210> 1071
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1071
Gln Gln Leu Lys Asp Tyr Pro Ile Thr
1 5
<210> 1072
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1072
Gly Phe Ser Leu Ser His Phe Ser
1 5
<210> 1073
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1073
Phe Asp Pro Glu Glu Gly Gly Thr
1 5
<210> 1074
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1074
Ala Thr Gly Ile Trp Tyr Ser Ser Gly Trp Pro Val Asp Tyr
1 5 10
<210> 1075
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1075
Gln Ser Leu Leu Asp Ser Ser Gly Tyr Asn Tyr
1 5 10
<210> 1076
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1076
Leu Gly Ser
1
<210> 1077
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1077
Met Gln Ala Leu Gln Thr Pro Ile Thr
1 5
<210> 1078
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1078
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1079
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1079
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1080
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1080
Ala Thr Val Ser Arg Val Arg Gly Val Ile Asn Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210> 1081
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1081
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 1082
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1082
Leu Gly Ser
1
<210> 1083
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1083
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 1084
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1084
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1085
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1085
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1086
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1086
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1087
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1087
Gln Ser Val Asp Ser Ser
1 5
<210> 1088
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1088
Asp Ala Ser
1
<210> 1089
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1089
Gln Gln Tyr Lys Asp Trp Pro Phe Thr
1 5
<210> 1090
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1090
Gly Gly Thr Phe Gly Thr Tyr Ala
1 5
<210> 1091
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1091
Ile Thr Pro Ile Ser Ala Thr Ile
1 5
<210> 1092
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1092
Ala Arg Gly Phe Glu Tyr Ser Asp Gly Leu Leu Asp Asp
1 5 10
<210> 1093
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1093
Gln Ser Leu Phe Tyr Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 1094
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1094
Arg Leu Ser
1
<210> 1095
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1095
Met Gln His Met Glu Phe Pro Leu Thr
1 5
<210> 1096
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1096
Gly Gly Ser Ile Ser Ser Ser Ser Tyr Tyr
1 5 10
<210> 1097
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1097
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 1098
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1098
Ala Arg Gly Ala Trp Glu Leu Arg Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 1099
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1099
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 1100
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1100
Gly Asn Asn
1
<210> 1101
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1101
Gln Ser Tyr Asp Ser Ser Leu Ser Ile Tyr Val Val
1 5 10
<210> 1102
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1102
Ser Gly Ser Ile Ser Ser Thr Asn Trp
1 5
<210> 1103
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1103
Ile Tyr His Ser Gly Ser Thr
1 5
<210> 1104
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1104
Ala Arg Val Gly Pro Ala Ala Ile Tyr Tyr
1 5 10
<210> 1105
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1105
Ser Asn Asn Val Gly Tyr Glu Gly
1 5
<210> 1106
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1106
Arg Asn Asn
1
<210> 1107
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1107
Ser Ala Trp Asp Ser Ser Leu Asn Ala Val Val
1 5 10
<210> 1108
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1108
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1109
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1109
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1110
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1110
Ala Thr Ala Lys Pro Gly Ile Ala Val Ala Gly Gln Asn Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Met Asp Val
20
<210> 1111
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1111
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 1112
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1112
Leu Gly Ser
1
<210> 1113
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1113
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 1114
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1114
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 1115
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1115
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 1116
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1116
Ala Ser Ser Pro Ile Gly Tyr Ser Tyr Gly Tyr Trp Gly Gly Met Asp
1 5 10 15
Val
<210> 1117
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1117
Ser Gly Ile Asp Val Arg Thr Asn Lys
1 5
<210> 1118
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1118
Phe Gln Ser Asp Ser Asp Lys
1 5
<210> 1119
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1119
Leu Ile Trp His Thr Ser Gly Trp Val
1 5
<210> 1120
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1120
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1121
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1121
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1122
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1122
Ala Thr Gly Pro Ala Ala Ala Gly Val Gly Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 1123
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1123
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 1124
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1124
Leu Gly Ser
1
<210> 1125
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1125
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 1126
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1126
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 1127
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1127
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 1128
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1128
Ala Arg Asp Val Met Val Tyr Cys Ser Ser Thr Ser Cys Tyr Phe Tyr
1 5 10 15
Gly Met Asp Val
20
<210> 1129
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1129
Gln Asp Ile Arg Asp Tyr
1 5
<210> 1130
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1130
Asp Ala Ser
1
<210> 1131
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1131
Gln Gln Phe Glu Asn Leu Pro Ile Thr
1 5
<210> 1132
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1132
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1133
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1133
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1134
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1134
Ala Thr Gly Gly Tyr Ser Ser Gly Phe Asn Tyr Tyr Tyr Tyr Tyr Met
1 5 10 15
Asp Val
<210> 1135
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1135
Gln Ser Leu Leu Tyr Arg Asn Gly Asn Asn Tyr
1 5 10
<210> 1136
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1136
Leu Gly Ser
1
<210> 1137
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1137
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 1138
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1138
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1139
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1139
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1140
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1140
Ala Thr Gly Val Thr Thr Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10
<210> 1141
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1141
Gln Ser Leu Leu Tyr Ser Asn Gly Asn Asn Phe
1 5 10
<210> 1142
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1142
Leu Gly Ser
1
<210> 1143
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1143
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 1144
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1144
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 1145
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1145
Ile Arg Tyr Asp Gly Ser Asn Lys
1 5
<210> 1146
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1146
Ala Arg Asp Leu Phe Asp Phe Trp Trp Asp Gly Met Asp Val
1 5 10
<210> 1147
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1147
Gln Ser Val Ser Ser Met Tyr
1 5
<210> 1148
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1148
Gly Ala Ser
1
<210> 1149
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1149
Gln Gln Tyr Val Ser Ser Pro Met Tyr Thr
1 5 10
<210> 1150
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1150
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1151
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1151
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1152
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1152
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1153
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1153
Gln Ser Val Ser Ser Tyr
1 5
<210> 1154
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1154
Asp Ala Ser
1
<210> 1155
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1155
Gln Gln Tyr Asp Asp Trp Pro Gln Thr
1 5
<210> 1156
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1156
Gly Gly Thr Phe Asn Asn Tyr Gly
1 5
<210> 1157
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1157
Ile Ile Pro Leu Phe Gly Thr Thr
1 5
<210> 1158
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1158
Ala Arg Asp Arg Met Ala Ala Asp Gly Met Ala Val Phe Asp Tyr
1 5 10 15
<210> 1159
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1159
Ser Ser Asn Ile Gly Arg His Phe
1 5
<210> 1160
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1160
Lys Asn Asp
1
<210> 1161
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1161
Ser Ser Trp Asp Ala Ala Leu Asn Gly Val Val
1 5 10
<210> 1162
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1162
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1163
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1163
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1164
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1164
Ala Thr Glu Val Pro Met Val Arg Gly Ala Arg Arg Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 1165
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1165
Gln Thr Leu Leu Tyr Ile Asn Glu Asn Asn Tyr
1 5 10
<210> 1166
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1166
Leu Gly Ser
1
<210> 1167
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1167
Met Gln Gly Leu Gln Thr Pro Pro Thr
1 5
<210> 1168
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1168
Gly Gly Ser Ile Ser Asn Ser Asn Tyr Tyr
1 5 10
<210> 1169
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1169
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 1170
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1170
Ala Arg Gly Ala Trp Glu Leu Ser Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 1171
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1171
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 1172
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1172
Gly Asn Asn
1
<210> 1173
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1173
Gln Ser Tyr Asp Ser Ser Leu Ser Val Tyr Val Val
1 5 10
<210> 1174
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1174
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1175
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1175
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1176
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1176
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1177
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1177
Gln Asn Val Tyr Gly Glu
1 5
<210> 1178
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1178
Asp Thr Phe
1
<210> 1179
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1179
Gln Gln Arg Arg Asp Trp Pro Ile Thr
1 5
<210> 1180
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1180
Gly Phe Thr Phe Gly Thr Ser Ser
1 5
<210> 1181
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1181
Ile Ser Phe Asp Gly Thr Glu Ile
1 5
<210> 1182
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1182
Ala Lys Gly Ser Gly Asn Ile Tyr Phe Tyr Ser Gly Met Asp Val
1 5 10 15
<210> 1183
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1183
Gln Ser Ile Ser Gly Trp
1 5
<210> 1184
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1184
Glu Thr Ser
1
<210> 1185
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1185
Gln Gln Tyr Tyr Ser Tyr Pro Leu Thr
1 5
<210> 1186
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1186
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1187
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1187
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1188
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1188
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 1189
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1189
Gln Ser Leu Leu Tyr Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 1190
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1190
Leu Gly Ser
1
<210> 1191
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1191
Met Gln Ala Leu Gln Ser Pro Val Thr
1 5
<210> 1192
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1192
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1193
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1193
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1194
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1194
Ala Thr Asp Ser Arg Asp Gly Pro Ala Ala Arg Gly Gly Tyr Tyr Tyr
1 5 10 15
Tyr Met Asp Val
20
<210> 1195
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1195
Gln Ser Leu Leu Tyr Ile Asn Gly Tyr Asn Tyr
1 5 10
<210> 1196
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1196
Leu Gly Ser
1
<210> 1197
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1197
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 1198
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1198
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 1199
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1199
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 1200
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1200
Ala Arg Asp Ala Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr Asn Pro Asp
1 5 10 15
Ala Phe Asp Ile
20
<210> 1201
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1201
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 1202
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1202
Leu Gly Ser
1
<210> 1203
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1203
Met Gln Gly Leu Gln Thr Pro Arg Thr
1 5
<210> 1204
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1204
Gly Phe Ser Leu Thr Ser Gly Gly Met Ser
1 5 10
<210> 1205
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1205
Ile Asp Trp Asn Asp Asp Lys
1 5
<210> 1206
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1206
Ala Arg Ile Arg Gly Met Thr Trp Gly Phe Asp Ser
1 5 10
<210> 1207
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1207
Gln Ser Val Ser Ser Ser Tyr
1 5
<210> 1208
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1208
Gly Ala Ser
1
<210> 1209
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1209
Gln Gln Tyr Gly Thr Thr Pro Phe Ala
1 5
<210> 1210
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1210
Gly Tyr Thr Phe Thr Ala Tyr Tyr
1 5
<210> 1211
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1211
Ile Asn Pro Asn Ser Gly Gly Thr
1 5
<210> 1212
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1212
Ala Arg Asp Gly Ala Phe Tyr Tyr Gly Ser Glu Asn Tyr Tyr Asn Ala
1 5 10 15
Gly Trp Phe Asp Pro
20
<210> 1213
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1213
Gln Ser Leu Leu Ser Gly Asn Gly Tyr Asn Tyr
1 5 10
<210> 1214
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1214
Leu Gly Ser
1
<210> 1215
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1215
Met Gln Ala Leu Lys Ser Pro Leu Thr
1 5
<210> 1216
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1216
Gly Tyr Thr Phe Thr Asn Tyr Tyr
1 5
<210> 1217
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1217
Ile Asn Pro Ser Gly Gly Ile Thr
1 5
<210> 1218
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1218
Ala Arg Ala Gly Leu Gly Tyr Asn Trp Asn Tyr Ala Pro Ser Gly Met
1 5 10 15
Asp Val
<210> 1219
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1219
Ser Ser Asp Val Gly Gly Tyr Asn Tyr
1 5
<210> 1220
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1220
Glu Val Ser
1
<210> 1221
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1221
Ser Ser Tyr Ala Gly Ser Asn Asn Leu Val
1 5 10
<210> 1222
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1222
Gly Gly Ser Ile Ser Ser Ser Ser Tyr Tyr
1 5 10
<210> 1223
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1223
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 1224
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1224
Ala Arg Gly Ala Trp Glu Leu Arg Leu Gly Asp Trp Phe Asp Pro
1 5 10 15
<210> 1225
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1225
Ser Ser Asn Ile Gly Ala Gly Tyr Asp
1 5
<210> 1226
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1226
Gly Tyr Ser
1
<210> 1227
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1227
Gln Ser Tyr Asp Ser Ser Leu Ser Val Tyr Val Val
1 5 10
<210> 1228
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1228
Gly Phe Thr Phe Asp Asp Tyr Ala
1 5
<210> 1229
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1229
Ile Ser Trp Asn Ser Gly Ser Ile
1 5
<210> 1230
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1230
Ala Lys Asp Val Asn Arg Ile Leu Val Ala Gly Met Val Asp Tyr
1 5 10 15
<210> 1231
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1231
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 1232
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1232
Leu Gly Ser
1
<210> 1233
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1233
Met Gln Gly Leu Gln Thr Pro Ile Thr
1 5
<210> 1234
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1234
Arg Phe Thr Phe Glu Asp Tyr Ala
1 5
<210> 1235
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1235
Ile Ser Trp Lys Ser Gly Gly Ile
1 5
<210> 1236
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1236
Val Lys Asp Pro Thr Leu Val Ala Thr Asp Arg Ala Phe Asn Ile
1 5 10 15
<210> 1237
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1237
Gln Ser Leu Leu His Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 1238
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1238
Leu Gly Ser
1
<210> 1239
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1239
Met Gln Tyr Leu Gln Thr Pro Asp Thr
1 5
<210> 1240
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1240
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1241
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1241
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1242
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1242
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1243
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1243
Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Ile His
1 5 10
<210> 1244
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1244
Thr Leu Ser
1
<210> 1245
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1245
Met Gln Arg Lys Glu Phe Pro Leu Thr
1 5
<210> 1246
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1246
Gly Phe Thr Phe Ser Ser Tyr Gly
1 5
<210> 1247
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1247
Ile Ser Tyr Asp Gly Ser Asn Lys
1 5
<210> 1248
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1248
Ala Lys Glu Asp Arg Leu Arg Phe Leu Glu Trp Leu Phe Tyr Gly Met
1 5 10 15
Asp Val
<210> 1249
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1249
His Asp Ile Tyr Thr Tyr
1 5
<210> 1250
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1250
Asp Ala Ser
1
<210> 1251
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1251
Gln Gln Tyr Asp Asn His Pro Pro Glu Val Thr
1 5 10
<210> 1252
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1252
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1253
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1253
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1254
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1254
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1255
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1255
Gln Asp Ile Asp Asp Asp
1 5
<210> 1256
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1256
Glu Ala Ser
1
<210> 1257
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1257
Leu Gln His Asp Asn Leu Pro Leu Thr
1 5
<210> 1258
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1258
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1259
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1259
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1260
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1260
Ala Thr Glu Asp Pro Gly Pro Val Ala Gly Pro Tyr Tyr Tyr Tyr Gly
1 5 10 15
Met Asp Val
<210> 1261
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1261
Gln Ser Leu Leu Tyr Ile Asn Gly Tyr His Tyr
1 5 10
<210> 1262
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1262
Leu Gly Ser
1
<210> 1263
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1263
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 1264
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1264
Gly Tyr Met Phe Thr Asn Tyr Pro
1 5
<210> 1265
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1265
Ile Asn Ala Gly Thr Gly Asn Thr
1 5
<210> 1266
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1266
Ala Arg Glu Gly Met Phe Tyr Tyr Gly Leu Glu Ser Tyr Tyr Lys Gly
1 5 10 15
Gly Trp Phe Asp Pro
20
<210> 1267
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1267
Gln Ser Leu Leu His Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 1268
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1268
Leu Ala Ser
1
<210> 1269
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1269
Met Gln Ala Leu Gln Thr Pro Leu Thr
1 5
<210> 1270
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1270
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1271
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1271
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1272
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1272
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1273
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1273
Gln Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr Tyr
1 5 10
<210> 1274
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1274
Thr Leu Ser
1
<210> 1275
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1275
Met Gln Arg Leu Gln Phe Pro Leu Thr
1 5
<210> 1276
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1276
Thr Tyr Thr Phe Thr Thr Tyr Tyr
1 5
<210> 1277
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1277
Ile Tyr Pro Ser Gly Gly Asn Thr
1 5
<210> 1278
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1278
Val Arg Asp Gln Asn Tyr Tyr Tyr Ser Ala Met Asp Val
1 5 10
<210> 1279
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1279
Gln Asp Ile Arg Asn Tyr
1 5
<210> 1280
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1280
Asp Ala Ser
1
<210> 1281
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1281
Gln Gln Phe Glu Asn Leu Pro Ile Thr
1 5
<210> 1282
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1282
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1283
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1283
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1284
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1284
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1285
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1285
Arg Asp Ile Ser Asp Ser
1 5
<210> 1286
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1286
Asp Ala Ser
1
<210> 1287
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1287
His Gln Tyr Asp Asn Leu Pro Leu Thr
1 5
<210> 1288
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1288
Gly Phe Asn Phe Arg Gly Tyr Ala
1 5
<210> 1289
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1289
Ile Ser Gly Ser Gly Gly Thr Thr
1 5
<210> 1290
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1290
Ala Gln Ser Tyr Ala Gln Ile Gly Tyr Gly Gly His Ile Asp His
1 5 10 15
<210> 1291
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1291
Ser Gly Ile Asp Val Ala Thr Tyr Met
1 5
<210> 1292
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1292
Tyr Lys Ser Asp Ser Asp Lys
1 5
<210> 1293
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1293
Leu Ile Trp His Gly Ser His Tyr Val
1 5
<210> 1294
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1294
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1295
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1295
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1296
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1296
Ala Thr Gly Val Pro Ala Ala Ile Gly Val Tyr Tyr Tyr Tyr Tyr Tyr
1 5 10 15
Met Asp Val
<210> 1297
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1297
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 1298
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1298
Trp Gly Ser
1
<210> 1299
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1299
Met Gln Ala Val Gln Asn Pro Pro Thr
1 5
<210> 1300
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1300
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1301
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1301
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1302
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1302
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1303
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1303
Gln Asn Ile Asp Ser Ser
1 5
<210> 1304
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1304
Tyr Ala Ser
1
<210> 1305
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1305
His Gln Ser Ser Ser Leu Pro Leu Thr
1 5
<210> 1306
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1306
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1307
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1307
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1308
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1308
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1309
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1309
Gln Ser Val Tyr Asn Asn Tyr
1 5
<210> 1310
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1310
Asp Ala Ser
1
<210> 1311
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1311
Gln Gln Tyr Asn Ser Trp Pro Pro Tyr Thr
1 5 10
<210> 1312
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1312
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 1313
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1313
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 1314
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1314
Ala Arg Asp Ala Tyr Tyr Tyr Asp Ser Ser Gly Tyr Tyr Asn Pro Asp
1 5 10 15
Ala Phe Asp Ile
20
<210> 1315
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1315
Gln Ser Leu Leu Tyr Ser Asn Gly Tyr Asn Tyr
1 5 10
<210> 1316
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1316
Leu Gly Ser
1
<210> 1317
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1317
Met Gln Ala Arg Gln Thr Pro Tyr Thr
1 5
<210> 1318
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1318
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1319
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1319
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1320
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1320
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1321
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1321
Glu Ser Val Thr Thr Phe
1 5
<210> 1322
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1322
Asp Ala Ser
1
<210> 1323
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1323
His Gln His Thr Asn Trp Pro Leu Thr
1 5
<210> 1324
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1324
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1325
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1325
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1326
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1326
Ala Thr Glu Val Gly Tyr Cys Ser Gly Gly Ser Cys Tyr Ile Ser Tyr
1 5 10 15
Tyr Tyr Gly Met Asp Val
20
<210> 1327
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1327
Gln Ser Leu Leu Tyr Arg Asn Gly His Asn Phe
1 5 10
<210> 1328
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1328
Leu Gly Ser
1
<210> 1329
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1329
Met Gln Ala Leu Gln Thr Pro Pro Thr
1 5
<210> 1330
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1330
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1331
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1331
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1332
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1332
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1333
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1333
Gln Thr Met Asn Asn Tyr
1 5
<210> 1334
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1334
Asp Ala Ser
1
<210> 1335
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1335
Gln Gln Tyr Gly Asp Trp Leu Pro Ile Thr
1 5 10
<210> 1336
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1336
Gly Tyr Thr Leu Thr Glu Leu Ser
1 5
<210> 1337
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1337
Phe Asp Pro Glu Asp Gly Glu Thr
1 5
<210> 1338
<211> 17
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1338
Ala Thr Ala Phe Pro Glu Ala Thr Ile Ser Tyr Tyr Tyr Tyr Met Asp
1 5 10 15
Val
<210> 1339
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1339
Gln Ser Leu Leu Tyr Arg Asn Gly Tyr Asn Tyr
1 5 10
<210> 1340
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1340
Trp Gly Ser
1
<210> 1341
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1341
Met Gln Ala Val Gln Asn Pro Pro Thr
1 5
<210> 1342
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1342
Gly Gly Thr Phe Ser Ser Ser Ala
1 5
<210> 1343
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1343
Ile Ile Pro Ile Tyr Gly Ile Thr
1 5
<210> 1344
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1344
Ala Arg Asp Asp Thr Ala Arg Arg Val Arg Gly Val Pro Tyr Tyr Tyr
1 5 10 15
Tyr Tyr Ala Met Asp Val
20
<210> 1345
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1345
Arg Ser Leu Leu Asp Ser Asp Asp Gly Asn Thr His
1 5 10
<210> 1346
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1346
Ser Leu Ser
1
<210> 1347
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1347
Met Gln Arg Lys Glu Phe Pro Leu Thr
1 5
<210> 1348
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1348
Gly Gly Thr Phe Ser Ser Tyr Ala
1 5
<210> 1349
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1349
Ile Ile Pro Ile Phe Gly Thr Ala
1 5
<210> 1350
<211> 21
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1350
Ala Arg Gly Pro Trp Tyr Tyr Asp Ser Ser Gly Tyr Ser Ser Tyr Ala
1 5 10 15
Tyr Tyr Met Asp Val
20
<210> 1351
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1351
Gln Ser Leu Leu His Ser Asp Gly Tyr Asn Tyr
1 5 10
<210> 1352
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1352
Leu Gly Ser
1
<210> 1353
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1353
Met Gln Ala Leu His Thr Pro Gly Val Thr
1 5 10
<210> 1354
<211> 330
<212> PRT
<213> Homo sapiens
<400> 1354
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
225 230 235 240
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 1355
<211> 326
<212> PRT
<213> Homo sapiens
<400> 1355
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Asn Phe Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Thr Val Glu Arg Lys Cys Cys Val Glu Cys Pro Pro Cys Pro Ala Pro
100 105 110
Pro Val Ala Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
115 120 125
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
130 135 140
Val Ser His Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
145 150 155 160
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
165 170 175
Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Val His Gln Asp Trp
180 185 190
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
195 200 205
Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln Pro Arg Glu
210 215 220
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn
225 230 235 240
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
245 250 255
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
260 265 270
Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
275 280 285
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
290 295 300
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
305 310 315 320
Ser Leu Ser Pro Gly Lys
325
<210> 1356
<211> 377
<212> PRT
<213> Homo sapiens
<400> 1356
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Thr Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Leu Lys Thr Pro Leu Gly Asp Thr Thr His Thr Cys Pro
100 105 110
Arg Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg
115 120 125
Cys Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys
130 135 140
Pro Glu Pro Lys Ser Cys Asp Thr Pro Pro Pro Cys Pro Arg Cys Pro
145 150 155 160
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
165 170 175
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
180 185 190
Val Val Asp Val Ser His Glu Asp Pro Glu Val Gln Phe Lys Trp Tyr
195 200 205
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
210 215 220
Gln Tyr Asn Ser Thr Phe Arg Val Val Ser Val Leu Thr Val Leu His
225 230 235 240
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
245 250 255
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Thr Lys Gly Gln
260 265 270
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
275 280 285
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
290 295 300
Ser Asp Ile Ala Val Glu Trp Glu Ser Ser Gly Gln Pro Glu Asn Asn
305 310 315 320
Tyr Asn Thr Thr Pro Pro Met Leu Asp Ser Asp Gly Ser Phe Phe Leu
325 330 335
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Ile
340 345 350
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn Arg Phe Thr Gln
355 360 365
Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375
<210> 1357
<211> 327
<212> PRT
<213> Homo sapiens
<400> 1357
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Cys Ser Arg
1 5 10 15
Ser Thr Ser Glu Ser Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Lys Thr
65 70 75 80
Tyr Thr Cys Asn Val Asp His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Arg Val Glu Ser Lys Tyr Gly Pro Pro Cys Pro Ser Cys Pro Ala Pro
100 105 110
Glu Phe Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
115 120 125
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
130 135 140
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
145 150 155 160
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
165 170 175
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
180 185 190
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
195 200 205
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
210 215 220
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
225 230 235 240
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
245 250 255
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
260 265 270
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
275 280 285
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
290 295 300
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
305 310 315 320
Leu Ser Leu Ser Leu Gly Lys
325
<210> 1358
<211> 21
<212> PRT
<213> Homo sapiens
<400> 1358
Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala
1 5 10 15
Pro Ala Arg Gln Ala
20
<210> 1359
<211> 34
<212> PRT
<213> Homo sapiens
<400> 1359
Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala
1 5 10 15
Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile
20 25 30
Leu Glu
<210> 1360
<211> 17
<212> PRT
<213> Homo sapiens
<400> 1360
Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu
1 5 10 15
Glu
<210> 1361
<211> 16
<212> PRT
<213> Homo sapiens
<400> 1361
Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala
1 5 10 15
<210> 1362
<211> 24
<212> PRT
<213> Homo sapiens
<400> 1362
Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala
1 5 10 15
Cys Gly Ser Leu Pro Pro Ser Ser
20
<210> 1363
<211> 15
<212> PRT
<213> Homo sapiens
<400> 1363
Glu Ala Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu
1 5 10 15
<210> 1364
<211> 22
<212> PRT
<213> Homo sapiens
<400> 1364
Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe
1 5 10 15
Thr Ile Arg Cys Gly Phe
20
<210> 1365
<211> 19
<212> PRT
<213> Homo sapiens
<400> 1365
Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val
1 5 10 15
Thr Val Ser
<210> 1366
<211> 24
<212> PRT
<213> Homo sapiens
<400> 1366
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
1 5 10 15
Gly Thr Thr Leu Ala Val Leu His
20
<210> 1367
<211> 26
<212> PRT
<213> Homo sapiens
<400> 1367
Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala
1 5 10 15
Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser
20 25
<210> 1368
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1368
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1369
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1369
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10 15
<210> 1370
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Asn, Gln, Ser or Ala
<400> 1370
Glu Val Gln Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Xaa Ile Asn Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Asn Gln Lys Phe
50 55 60
Lys Ser Lys Ala Thr Leu Ser Ala Asp Asn Pro Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Xaa Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Pro Val Thr Val Ser Ser
115 120
<210> 1371
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Tyr, Gln, Ser or Ala
<400> 1371
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Xaa Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1372
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Asn, Gln, Ser or Ala
<400> 1372
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Xaa Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Xaa Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1373
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Asn, Gln, Ser or Ala
<400> 1373
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Xaa Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ala Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Xaa Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1374
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Asn, Gln, Ser or Ala
<400> 1374
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Xaa Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Xaa Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1375
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Asn, Gln, Ser or Ala
<400> 1375
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Xaa Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Asn Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Xaa Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1376
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1376
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro
85 90 95
<210> 1377
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1377
Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
1 5 10
<210> 1378
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (32)..(32)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (53)..(53)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Trp, Tyr, Phe or His
<400> 1378
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Val Ser Val Gly
1 5 10 15
Glu Thr Val Thr Ile Ile Cys Arg Val Ser Glu Xaa Ile Tyr Ser Xaa
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Gln Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Glu Ala Thr Xaa Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Tyr Ser Leu Lys Ile Asn Ser Leu Gln Ser
65 70 75 80
Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His Phe Xaa Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1379
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (32)..(32)
<223> Tyr, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (53)..(53)
<223> Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Tyr, Phe or His
<400> 1379
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Xaa Ile Ser Ser Xaa
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Xaa Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Xaa Ser Thr Pro Pro
85 90 95
<210> 1380
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (32)..(32)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (53)..(53)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Trp, Tyr, Phe or His
<400> 1380
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Val Ser Glu Xaa Ile Tyr Ser Xaa
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Thr Xaa Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Xaa Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1381
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (32)..(32)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (53)..(53)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Trp, Tyr, Phe or His
<400> 1381
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Val Ser Glu Xaa Ile Tyr Ser Xaa
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Thr Xaa Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Xaa Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1382
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (32)..(32)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (53)..(53)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Trp, Tyr, Phe or His
<400> 1382
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Val Ser Glu Xaa Ile Tyr Ser Xaa
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Val
35 40 45
Tyr Glu Ala Thr Xaa Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Thr Tyr Tyr Cys Gln His Phe Xaa Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1383
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1383
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1384
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1384
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10 15
<210> 1385
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (57)..(57)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (104)..(104)
<223> Met, Leu, Phe or Ala
<400> 1385
Gln Val Gln Leu Gln Gln Ser Gly Pro Glu Val Val Arg Pro Gly Val
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Gly Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Lys Gln Ser His Ala Lys Ser Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Xaa Thr Xaa Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Thr Met Thr Val Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ala Arg Leu Thr Ser Glu Asp Ser Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Xaa Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ala
115
<210> 1386
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (57)..(57)
<223> Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Gln, Ser or Ala
<400> 1386
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Xaa Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1387
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (57)..(57)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (104)..(104)
<223> Met, Leu, Phe or Ala
<400> 1387
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Xaa Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Xaa Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1388
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (57)..(57)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (104)..(104)
<223> Met, Leu, Phe or Ala
<400> 1388
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Xaa Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Xaa Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1389
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (57)..(57)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (104)..(104)
<223> Met, Leu, Phe or Ala
<400> 1389
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Gly Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Xaa Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Met Thr Val Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Xaa Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1390
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (57)..(57)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (104)..(104)
<223> Met, Leu, Phe or Ala
<400> 1390
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Gly Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Xaa Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Met Thr Val Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Xaa Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1391
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1391
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Asn Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Arg Ser Asn Trp Pro Pro
85 90 95
<210> 1392
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1392
Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
1 5 10
<210> 1393
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (24)..(24)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (90)..(90)
<223> Trp, Tyr, Phe or His
<400> 1393
Gln Ile Val Leu Thr Gln Ser Pro Ala Ile Met Ser Ala Ser Pro Gly
1 5 10 15
Glu Lys Val Thr Met Thr Cys Xaa Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Ile Ser Ser Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Xaa Leu Ala Ser Gly Val Pro Val Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Ser Tyr Ser Leu Thr Ile Gly Arg Met Glu Ala Glu
65 70 75 80
Asp Ala Ala Thr Tyr Tyr Cys Gln Gln Xaa Ser Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Ala Gly Thr Lys Val Glu Val Lys
100 105
<210> 1394
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (24)..(24)
<223> Arg, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (53)..(53)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (91)..(91)
<223> Arg, Tyr, Phe or His
<400> 1394
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Xaa Ala Ser Gln Ser Val Ser Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Xaa Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Gln Xaa Ser Asn Trp Pro Pro
85 90 95
<210> 1395
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (24)..(24)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (90)..(90)
<223> Trp, Tyr, Phe or His
<400> 1395
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Xaa Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Xaa Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln Gln Xaa Ser Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1396
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (24)..(24)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (90)..(90)
<223> Trp, Tyr, Phe or His
<400> 1396
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Xaa Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Xaa Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln Gln Xaa Ser Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1397
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (24)..(24)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (90)..(90)
<223> Trp, Tyr, Phe or His
<400> 1397
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Val Thr Met Ser Cys Xaa Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Xaa Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Met Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln Gln Xaa Ser Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 1398
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1398
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1399
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1399
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10 15
<210> 1400
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (103)..(103)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1400
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Met Arg Pro Gly Thr
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Xaa His
20 25 30
Leu Ile Glu Trp Ile Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Xaa Pro Gly Ser Asp Ser Thr Asp Tyr Asn Glu Lys Phe
50 55 60
Lys Asp Lys Ala Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Asp Asp Ser Ala Val Tyr Phe Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Xaa Ser Xaa Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ala
115
<210> 1401
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<400> 1401
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Xaa Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Xaa Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1402
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (103)..(103)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1402
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Xaa His
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Xaa Pro Gly Ser Asp Ser Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Xaa Ser Xaa Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1403
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (103)..(103)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1403
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Xaa His
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Xaa Pro Gly Ser Asp Ser Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ala Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Xaa Ser Xaa Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1404
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (103)..(103)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1404
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Xaa His
20 25 30
Leu Ile Glu Trp Ile Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Xaa Pro Gly Ser Asp Ser Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Xaa Ser Xaa Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1405
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (103)..(103)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1405
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Xaa His
20 25 30
Leu Ile Glu Trp Ile Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Xaa Pro Gly Ser Asp Ser Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Xaa Ser Xaa Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1406
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1406
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro
85 90 95
<210> 1407
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1407
Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
1 5 10
<210> 1408
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (50)..(50)
<223> Asn, Gln, Ser, Ala or Asp
<400> 1408
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Glu Thr Val Thr Ile Thr Cys Arg Ala Ser Glu Xaa Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Arg Gly Lys Ser Pro Gln Leu Leu Val
35 40 45
Tyr Xaa Ala Lys Thr Leu Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Gln Phe Ser Leu Lys Ile Asn Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Ser Tyr Tyr Cys Gln His His Tyr Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1409
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (50)..(50)
<223> Gln, Ser, Ala or Asp
<400> 1409
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Xaa Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Xaa Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro
85 90 95
<210> 1410
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (50)..(50)
<223> Asn, Gln, Ser, Ala or Asp
<400> 1410
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Xaa Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Xaa Ala Lys Thr Leu Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His His Tyr Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1411
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (28)..(28)
<223> Asn, Gln, Ser, Ala or Asp
<220>
<221> MOD_RES
<222> (50)..(50)
<223> Asn, Gln, Ser, Ala or Asp
<400> 1411
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Xaa Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Val
35 40 45
Tyr Xaa Ala Lys Thr Leu Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Thr Tyr Tyr Cys Gln His His Tyr Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1412
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1412
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1413
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1413
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10 15
<210> 1414
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1414
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Ala Phe Thr Xaa Tyr
20 25 30
Leu Ile Glu Trp Val Lys Gln Arg Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Xaa Pro Gly Ser Gly Gly Ile Tyr Tyr Asn Asp Lys Phe
50 55 60
Lys Val Lys Thr Thr Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Asp Asp Ser Ala Val Tyr Phe Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Xaa Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ala
115
<210> 1415
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<400> 1415
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Xaa Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Xaa Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1416
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1416
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Xaa Tyr
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Xaa Pro Gly Ser Gly Gly Ile Tyr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Xaa Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1417
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1417
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Xaa Tyr
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Xaa Pro Gly Ser Gly Gly Ile Tyr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ala Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Xaa Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1418
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1418
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Ala Phe Thr Xaa Tyr
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Xaa Pro Gly Ser Gly Gly Ile Tyr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Leu Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Xaa Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1419
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (52)..(52)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (105)..(105)
<223> Trp, Tyr, Phe or His
<400> 1419
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Ala Phe Thr Xaa Tyr
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Xaa Pro Gly Ser Gly Gly Ile Tyr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Xaa Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1420
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1420
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Tyr Ser Thr Pro Pro
85 90 95
<210> 1421
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1421
Tyr Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
1 5 10
<210> 1422
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1422
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Gly Ile Arg Asn Asp
20 25 30
Leu Gly Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Asn Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Asn Ser Tyr Pro Pro
85 90 95
<210> 1423
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Trp, Tyr, Phe or His
<220>
<221> MOD_RES
<222> (93)..(93)
<223> Asn, Gln, Ser, Ala or Asp
<400> 1423
Asp Ile Val Met Thr Gln Ser Gln Lys Phe Ile Ser Thr Ser Val Gly
1 5 10 15
Asp Arg Val Ser Ile Thr Cys Lys Ala Ser Gln Ser Val Arg Ile Ala
20 25 30
Val Ala Trp Phe Gln Gln Lys Pro Gly Gln Ser Pro Lys Ala Leu Ile
35 40 45
Tyr Leu Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Asn Val Gln Ser
65 70 75 80
Glu Asp Leu Ala Asp Tyr Phe Cys Leu Gln His Xaa Xaa Tyr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1424
<211> 96
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Tyr, Phe or His
<220>
<221> MOD_RES
<222> (93)..(93)
<223> Gln, Ser, Ala or Asp
<400> 1424
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Ser Xaa Xaa Thr Pro Pro
85 90 95
<210> 1425
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Trp, Tyr, Phe or His
<220>
<221> MOD_RES
<222> (93)..(93)
<223> Asn, Gln, Ser, Ala or Asp
<400> 1425
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Arg Ile Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Leu Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Xaa Xaa Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1426
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (92)..(92)
<223> Trp, Tyr, Phe or His
<220>
<221> MOD_RES
<222> (93)..(93)
<223> Asn, Gln, Ser, Ala or Asp
<400> 1426
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Arg Ile Ala
20 25 30
Val Ala Trp Phe Gln Gln Lys Pro Gly Lys Ala Pro Lys Ala Leu Ile
35 40 45
Tyr Leu Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Xaa Xaa Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1427
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1427
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Ser Tyr
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Ser Thr Ser Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1428
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
peptide
<400> 1428
Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10 15
<210> 1429
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Trp, Tyr, Phe or His
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<400> 1429
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Thr
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ala Gly Tyr Thr Phe Thr Xaa Tyr
20 25 30
Xaa Ile Gly Trp Val Lys Gln Arg Pro Gly His Gly Leu Glu Trp Ile
35 40 45
Gly Asp Ile Tyr Pro Gly Gly Gly Tyr Thr Xaa Tyr Asn Glu Lys Phe
50 55 60
Lys Gly Lys Ala Thr Leu Thr Ala Asp Thr Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Ile Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ala
115 120
<210> 1430
<211> 98
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Tyr, Phe or His
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Gln, Ser or Ala
<400> 1430
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Xaa Tyr
20 25 30
Xaa Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Asn Pro Ser Gly Gly Ser Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg
<210> 1431
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Trp, Tyr, Phe or His
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<400> 1431
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Xaa Tyr
20 25 30
Xaa Ile Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Asp Ile Tyr Pro Gly Gly Gly Tyr Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1432
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Trp, Tyr, Phe or His
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<400> 1432
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Xaa Tyr
20 25 30
Xaa Ile Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Asp Ile Tyr Pro Gly Gly Gly Tyr Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ala Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1433
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<220>
<221> MOD_RES
<222> (31)..(31)
<223> Asn, Gln, Ser or Ala
<220>
<221> MOD_RES
<222> (33)..(33)
<223> Trp, Tyr, Phe or His
<220>
<221> MOD_RES
<222> (59)..(59)
<223> Asn, Gln, Ser or Ala
<400> 1433
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Xaa Tyr
20 25 30
Xaa Ile Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Asp Ile Tyr Pro Gly Gly Gly Tyr Thr Xaa Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1434
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1434
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Asn Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Asn Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1435
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1435
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Asn Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ala Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Asn Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1436
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1436
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Asn Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Asn Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1437
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1437
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Asn Ile Asn Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Tyr Ile Tyr Pro Tyr Ile Gly Gly Ser Gly Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Asn Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Asp Lys Thr Ala Arg Asn Ala Met Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1438
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1438
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Met Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1439
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1439
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Val Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Met Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1440
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1440
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Gly Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Met Thr Val Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Met Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1441
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1441
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Ser Cys Lys Gly Ser Gly Tyr Lys Phe Pro Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Ser Ile Tyr Ser Gly Asn Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Met Thr Val Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Glu Gly Asp Leu Pro Met Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 1442
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1442
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Asn His
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Ser Asp Ser Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Asn Ser Trp Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1443
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1443
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Asn His
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Ser Asp Ser Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ala Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Asn Ser Trp Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1444
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1444
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Asn His
20 25 30
Leu Ile Glu Trp Ile Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Ser Asp Ser Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Asn Ser Trp Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1445
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1445
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Asn His
20 25 30
Leu Ile Glu Trp Ile Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Ser Asp Ser Thr Asp Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Leu Tyr Tyr Asn Ser Trp Phe Val Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1446
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1446
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Asn Tyr
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Val Ile Asn Pro Gly Ser Gly Gly Ile Tyr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Trp Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1447
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1447
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Ala Phe Thr Asn Tyr
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Ser Gly Gly Ile Tyr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ala Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Trp Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1448
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1448
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Ala Phe Thr Asn Tyr
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Ser Gly Gly Ile Tyr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Leu Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Trp Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1449
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1449
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Thr Ser Gly Tyr Ala Phe Thr Asn Tyr
20 25 30
Leu Ile Glu Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Asn Pro Gly Ser Gly Gly Ile Tyr Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Leu Thr Ala Asp Lys Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Glu Thr His Asp Thr Trp Phe Ala Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 1450
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1450
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Met
35 40 45
Gly Asp Ile Tyr Pro Gly Gly Gly Tyr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Arg Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1451
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1451
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Asp Ile Tyr Pro Gly Gly Gly Tyr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Val Thr Met Thr Ala Asp Thr Ser Thr Ser Thr Val Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1452
<211> 120
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1452
Gln Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Met Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Asp Ile Tyr Pro Gly Gly Gly Tyr Thr Asn Tyr Ala Gln Lys Phe
50 55 60
Gln Gly Arg Ala Thr Leu Thr Ala Asp Thr Ser Thr Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Pro Tyr Tyr Gly Ser Ser Tyr Gly Phe Ala Phe Trp Gly Gln
100 105 110
Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 1453
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1453
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Val Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Thr Asn Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Trp Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1454
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1454
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Val Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Glu Ala Thr Asn Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His Phe Trp Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1455
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1455
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Val Ser Glu Asn Ile Tyr Ser Asn
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Val
35 40 45
Tyr Glu Ala Thr Asn Leu Ala Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Thr Tyr Tyr Cys Gln His Phe Trp Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1456
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1456
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Asn Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln Gln Trp Ser Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1457
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1457
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Asn Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Asn Leu Ala Ser Gly Ile Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln Gln Trp Ser Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1458
<211> 106
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1458
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Val Thr Met Ser Cys Asn Ala Ser Ser Ser Val Ser Tyr Met
20 25 30
Tyr Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Arg Leu Leu Ile Tyr
35 40 45
Asp Thr Ser Asn Leu Ala Ser Gly Val Pro Ala Arg Phe Ser Gly Ser
50 55 60
Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Met Glu Pro Glu
65 70 75 80
Asp Phe Ala Val Tyr Tyr Cys Gln Gln Trp Ser Ser Tyr Pro Leu Thr
85 90 95
Phe Gly Gln Gly Thr Lys Val Glu Ile Lys
100 105
<210> 1459
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1459
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Asn Ala Lys Thr Leu Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln His His Tyr Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1460
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1460
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Glu Asn Ile Tyr Ser Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Val
35 40 45
Tyr Asn Ala Lys Thr Leu Val Glu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Gly Thr Tyr Tyr Cys Gln His His Tyr Gly Thr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1461
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1461
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Arg Ile Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Leu Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Trp Asn Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1462
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1462
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Arg Ile Ala
20 25 30
Val Ala Trp Phe Gln Gln Lys Pro Gly Lys Ala Pro Lys Ala Leu Ile
35 40 45
Tyr Leu Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Val Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln His Trp Asn Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 1463
<211> 131
<212> PRT
<213> Homo sapiens
<400> 1463
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala Asp
130
<210> 1464
<211> 131
<212> PRT
<213> Macaca fascicularis
<400> 1464
Thr Pro Glu Val Trp Val Gln Val Gln Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Val His Cys Gly Phe Leu Gly Pro Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asp Gly Ala Gly Gly Thr Lys Leu
35 40 45
Ala Val Leu His Pro Glu Leu Gly Thr Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ala Leu Glu Asp
65 70 75 80
Ser Gly Ala Ser Ser Pro Phe Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ser Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Val Pro Ile Leu
115 120 125
Arg Ala Asp
130
<210> 1465
<211> 126
<212> PRT
<213> Callithrix jacchus
<400> 1465
Thr Pro Glu Val Trp Val Gln Val Gln Met Lys Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Ile Val His Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asp Gly Ala Gly Gly Thr Arg Leu
35 40 45
Ala Val Leu His Pro Glu Leu Gly Thr Arg Gln Trp Ala Pro Ala His
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Val Leu Glu Glu
65 70 75 80
Pro Gly Ala Ser Ser Pro Ser Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Ile Leu Arg Ala Asp
115 120 125
<210> 1466
<211> 131
<212> PRT
<213> Macaca mulatta
<400> 1466
Thr Pro Glu Val Trp Val Gln Val Gln Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Val His Cys Gly Phe Leu Gly Pro Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asp Gly Ala Gly Gly Thr Lys Leu
35 40 45
Ala Val Leu His Pro Glu Leu Gly Thr Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ala Leu Glu Asp
65 70 75 80
Ser Gly Ala Ser Ser Pro Phe Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ser Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Val Pro Ile Leu
115 120 125
Arg Ala Asp
130
<210> 1467
<211> 131
<212> PRT
<213> Homo sapiens
<400> 1467
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala Asp
130
<210> 1468
<211> 151
<212> PRT
<213> Homo sapiens
<400> 1468
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly
35 40 45
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
50 55 60
Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp
65 70 75 80
Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu
85 90 95
Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe
100 105 110
Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
115 120 125
Ser Leu Pro Pro Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro
130 135 140
Ala Pro Ile Leu Arg Ala Asp
145 150
<210> 1469
<211> 104
<212> PRT
<213> Homo sapiens
<400> 1469
Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe
1 5 10 15
Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr
20 25 30
Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val
35 40 45
Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala
50 55 60
Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly
65 70 75 80
Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser
85 90 95
Phe Pro Glu Gly Ser Trp Glu Ala
100
<210> 1470
<211> 179
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1470
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly
35 40 45
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
50 55 60
Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp
65 70 75 80
Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu
85 90 95
Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe
100 105 110
Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
115 120 125
Ser Leu Pro Pro Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro
130 135 140
Ala Pro Ile Leu Arg Ala Asp Leu Ala Gly Ile Leu Gly Val Ser Gly
145 150 155 160
Val Leu Leu Phe Gly Cys Val Tyr Leu Leu His Leu Leu Arg Arg His
165 170 175
Lys His Arg
<210> 1471
<211> 191
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1471
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly
35 40 45
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
50 55 60
Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp
65 70 75 80
Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu
85 90 95
Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe
100 105 110
Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
115 120 125
Ser Leu Pro Pro Ser Ser Asp Pro Gly Leu Ser Val Pro Pro Thr Pro
130 135 140
Ala Pro Ile Leu Arg Ala Asp Leu Ala Gly Ile Leu Gly Val Ser Gly
145 150 155 160
Val Leu Leu Phe Asp Cys Gly Tyr Leu Leu His Leu Leu Cys Arg Gln
165 170 175
Lys His Arg Pro Ala Pro Arg Leu Gln Pro Ser His Thr Ser Ser
180 185 190
<210> 1472
<211> 211
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1472
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Trp Leu Ser Ala Pro Pro Thr
35 40 45
Pro Ala Pro Ile Leu Arg Ala Asp Leu Ala Gly Ile Leu Gly Val Ser
50 55 60
Gly Val Leu Leu Phe Gly Cys Val Tyr Leu Leu His Leu Leu Arg Arg
65 70 75 80
His Lys His Arg Pro Ala Pro Arg Leu Gln Pro Ser Arg Thr Ser Pro
85 90 95
Gln Ala Pro Arg Ala Arg Ala Trp Ala Pro Ser Gln Ala Ser Gln Ala
100 105 110
Ala Leu His Val Pro Tyr Ala Thr Ile Asn Thr Ser Cys Arg Pro Ala
115 120 125
Thr Leu Asp Thr Ala His Pro His Gly Gly Pro Ser Trp Trp Ala Ser
130 135 140
Leu Pro Thr His Ala Ala His Arg Pro Gln Gly Pro Ala Ala Trp Ala
145 150 155 160
Ser Thr Pro Ile Pro Ala Arg Gly Ser Phe Val Ser Val Glu Asn Gly
165 170 175
Leu Tyr Ala Gln Ala Gly Glu Arg Pro Pro His Thr Gly Pro Gly Leu
180 185 190
Thr Leu Phe Pro Asp Pro Arg Gly Pro Arg Ala Met Glu Gly Pro Leu
195 200 205
Gly Val Arg
210
<210> 1473
<211> 189
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1473
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu Arg
20 25 30
Ala Asp Leu Ala Gly Ile Leu Gly Val Ser Gly Val Leu Leu Phe Gly
35 40 45
Cys Val Tyr Leu Leu His Leu Leu Arg Arg His Lys His Arg Pro Ala
50 55 60
Pro Arg Leu Gln Pro Ser Arg Thr Ser Pro Gln Ala Pro Arg Ala Arg
65 70 75 80
Ala Trp Ala Pro Ser Gln Ala Ser Gln Ala Ala Leu His Val Pro Tyr
85 90 95
Ala Thr Ile Asn Thr Ser Cys Arg Pro Ala Thr Leu Asp Thr Ala His
100 105 110
Pro His Gly Gly Pro Ser Trp Trp Ala Ser Leu Pro Thr His Ala Ala
115 120 125
His Arg Pro Gln Gly Pro Ala Ala Trp Ala Ser Thr Pro Ile Pro Ala
130 135 140
Arg Gly Ser Phe Val Ser Val Glu Asn Gly Leu Tyr Ala Gln Ala Gly
145 150 155 160
Glu Arg Pro Pro His Thr Gly Pro Gly Leu Thr Leu Phe Pro Asp Pro
165 170 175
Arg Gly Pro Arg Ala Met Glu Gly Pro Leu Gly Val Arg
180 185
<210> 1474
<211> 74
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1474
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Leu Ser Val Pro Pro Thr Pro Ala Pro Ile Leu Arg
20 25 30
Ala Asp Leu Ala Gly Ile Leu Gly Val Ser Gly Val Leu Leu Phe Asp
35 40 45
Cys Gly Tyr Leu Leu His Leu Leu Cys Arg Gln Lys His Arg Pro Ala
50 55 60
Pro Arg Leu Gln Pro Ser His Thr Ser Ser
65 70
<210> 1475
<211> 203
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1475
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly
35 40 45
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
50 55 60
Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp
65 70 75 80
Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu
85 90 95
Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe
100 105 110
Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
115 120 125
Ser Leu Pro Pro Ser Ser Asp Pro Gly Gly Ala Gly Ala Arg Gly Pro
130 135 140
Gly Gly Gln Gly Gly Ala Arg Glu Leu Ala Thr His Leu Ile Leu Val
145 150 155 160
Ser Val Pro Arg Ala Leu Cys Pro Ala Asp Ser Cys Pro His Ser Ala
165 170 175
Gly Arg Pro Gly Arg Asp Leu Gly Gly Leu Arg Ser Pro Pro Leu Trp
180 185 190
Leu Cys Leu Pro Pro Ser Ser Ala Ala Pro Thr
195 200
<210> 1476
<211> 230
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1476
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly
35 40 45
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
50 55 60
Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ala Gln Ile Ser
65 70 75 80
Pro Arg Gly Pro Lys Ile Gly Gly Pro Pro Cys Pro Arg Pro Gly Pro
85 90 95
Ala Gly Lys Pro Arg Ala Ala Ser Leu Ser Ser Trp Lys Ala Leu Gly
100 105 110
Pro Ala Ala Pro Ala Pro Thr Pro Pro Ser Ala Ala Ser Leu Arg Pro
115 120 125
Ser Gln Ala Ala Leu His Val Pro Tyr Ala Thr Ile Asn Thr Ser Cys
130 135 140
Arg Pro Ala Thr Leu Asp Thr Ala His Pro His Gly Gly Pro Ser Trp
145 150 155 160
Trp Ala Ser Leu Pro Thr His Ala Ala His Arg Pro Gln Gly Pro Ala
165 170 175
Ala Trp Ala Ser Thr Pro Ile Pro Ala Arg Gly Ser Phe Val Ser Val
180 185 190
Glu Asn Gly Leu Tyr Ala Gln Ala Gly Glu Arg Pro Pro His Thr Gly
195 200 205
Pro Gly Leu Thr Leu Phe Pro Asp Pro Arg Gly Pro Arg Ala Met Glu
210 215 220
Gly Pro Leu Gly Val Arg
225 230
<210> 1477
<211> 182
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1477
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly
35 40 45
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
50 55 60
Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp
65 70 75 80
Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu
85 90 95
Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe
100 105 110
Cys Cys Lys Phe Ala Ser Phe Pro Gly Cys Ser Ser Arg Pro Leu Cys
115 120 125
His Tyr Gln His Gln Leu Pro Pro Ser Tyr Phe Gly His Ser Ser Pro
130 135 140
Pro Trp Gly Ala Val Leu Val Gly Val Thr Pro His Pro Arg Cys Thr
145 150 155 160
Pro Ala Pro Gly Pro Cys Arg Leu Gly Leu His Thr His Pro Cys Thr
165 170 175
Trp Gln Leu Cys Leu Cys
180
<210> 1478
<211> 183
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1478
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Gly Ala Gly Ala Arg Gly Pro Gly Gly Gln Gly
115 120 125
Gly Ala Arg Glu Leu Ala Thr His Leu Ile Leu Val Ser Val Pro Arg
130 135 140
Ala Leu Cys Pro Ala Asp Ser Cys Pro His Ser Ala Gly Arg Pro Gly
145 150 155 160
Arg Asp Leu Gly Gly Leu Arg Ser Pro Pro Leu Trp Leu Cys Leu Pro
165 170 175
Pro Ser Ser Ala Ala Pro Thr
180
<210> 1479
<211> 210
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1479
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ala Gln Ile Ser Pro Arg Gly Pro
50 55 60
Lys Ile Gly Gly Pro Pro Cys Pro Arg Pro Gly Pro Ala Gly Lys Pro
65 70 75 80
Arg Ala Ala Ser Leu Ser Ser Trp Lys Ala Leu Gly Pro Ala Ala Pro
85 90 95
Ala Pro Thr Pro Pro Ser Ala Ala Ser Leu Arg Pro Ser Gln Ala Ala
100 105 110
Leu His Val Pro Tyr Ala Thr Ile Asn Thr Ser Cys Arg Pro Ala Thr
115 120 125
Leu Asp Thr Ala His Pro His Gly Gly Pro Ser Trp Trp Ala Ser Leu
130 135 140
Pro Thr His Ala Ala His Arg Pro Gln Gly Pro Ala Ala Trp Ala Ser
145 150 155 160
Thr Pro Ile Pro Ala Arg Gly Ser Phe Val Ser Val Glu Asn Gly Leu
165 170 175
Tyr Ala Gln Ala Gly Glu Arg Pro Pro His Thr Gly Pro Gly Leu Thr
180 185 190
Leu Phe Pro Asp Pro Arg Gly Pro Arg Ala Met Glu Gly Pro Leu Gly
195 200 205
Val Arg
210
<210> 1480
<211> 162
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1480
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Gly Cys Ser Ser Arg Pro Leu Cys His Tyr Gln His
100 105 110
Gln Leu Pro Pro Ser Tyr Phe Gly His Ser Ser Pro Pro Trp Gly Ala
115 120 125
Val Leu Val Gly Val Thr Pro His Pro Arg Cys Thr Pro Ala Pro Gly
130 135 140
Pro Cys Arg Leu Gly Leu His Thr His Pro Cys Thr Trp Gln Leu Cys
145 150 155 160
Leu Cys
<210> 1481
<211> 66
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1481
Gly Ala Gly Ala Arg Gly Pro Gly Gly Gln Gly Gly Ala Arg Glu Leu
1 5 10 15
Ala Thr His Leu Ile Leu Val Ser Val Pro Arg Ala Leu Cys Pro Ala
20 25 30
Asp Ser Cys Pro His Ser Ala Gly Arg Pro Gly Arg Asp Leu Gly Gly
35 40 45
Leu Arg Ser Pro Pro Leu Trp Leu Cys Leu Pro Pro Ser Ser Ala Ala
50 55 60
Pro Thr
65
<210> 1482
<211> 154
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1482
Ala Gln Ile Ser Pro Arg Gly Pro Lys Ile Gly Gly Pro Pro Cys Pro
1 5 10 15
Arg Pro Gly Pro Ala Gly Lys Pro Arg Ala Ala Ser Leu Ser Ser Trp
20 25 30
Lys Ala Leu Gly Pro Ala Ala Pro Ala Pro Thr Pro Pro Ser Ala Ala
35 40 45
Ser Leu Arg Pro Ser Gln Ala Ala Leu His Val Pro Tyr Ala Thr Ile
50 55 60
Asn Thr Ser Cys Arg Pro Ala Thr Leu Asp Thr Ala His Pro His Gly
65 70 75 80
Gly Pro Ser Trp Trp Ala Ser Leu Pro Thr His Ala Ala His Arg Pro
85 90 95
Gln Gly Pro Ala Ala Trp Ala Ser Thr Pro Ile Pro Ala Arg Gly Ser
100 105 110
Phe Val Ser Val Glu Asn Gly Leu Tyr Ala Gln Ala Gly Glu Arg Pro
115 120 125
Pro His Thr Gly Pro Gly Leu Thr Leu Phe Pro Asp Pro Arg Gly Pro
130 135 140
Arg Ala Met Glu Gly Pro Leu Gly Val Arg
145 150
<210> 1483
<211> 52
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1483
Ala Gln Ile Ser Pro Arg Gly Pro Lys Ile Gly Gly Pro Pro Cys Pro
1 5 10 15
Arg Pro Gly Pro Ala Gly Lys Pro Arg Ala Ala Ser Leu Ser Ser Trp
20 25 30
Lys Ala Leu Gly Pro Ala Ala Pro Ala Pro Thr Pro Pro Ser Ala Ala
35 40 45
Ser Leu Arg Pro
50
<210> 1484
<211> 62
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1484
Gly Cys Ser Ser Arg Pro Leu Cys His Tyr Gln His Gln Leu Pro Pro
1 5 10 15
Ser Tyr Phe Gly His Ser Ser Pro Pro Trp Gly Ala Val Leu Val Gly
20 25 30
Val Thr Pro His Pro Arg Cys Thr Pro Ala Pro Gly Pro Cys Arg Leu
35 40 45
Gly Leu His Thr His Pro Cys Thr Trp Gln Leu Cys Leu Cys
50 55 60
<210> 1485
<211> 129
<212> PRT
<213> Homo sapiens
<400> 1485
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg
<210> 1486
<211> 130
<212> PRT
<213> Homo sapiens
<400> 1486
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala
130
<210> 1487
<211> 132
<212> PRT
<213> Homo sapiens
<400> 1487
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala Asp Leu
130
<210> 1488
<211> 133
<212> PRT
<213> Homo sapiens
<400> 1488
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala Asp Leu Ala
130
<210> 1489
<211> 104
<212> PRT
<213> Homo sapiens
<400> 1489
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp
100
<210> 1490
<211> 105
<212> PRT
<213> Homo sapiens
<400> 1490
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu
100 105
<210> 1491
<211> 106
<212> PRT
<213> Homo sapiens
<400> 1491
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala
100 105
<210> 1492
<211> 107
<212> PRT
<213> Homo sapiens
<400> 1492
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys
100 105
<210> 1493
<211> 108
<212> PRT
<213> Homo sapiens
<400> 1493
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
100 105
<210> 1494
<211> 103
<212> PRT
<213> Homo sapiens
<400> 1494
Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser
1 5 10 15
Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val
20 25 30
Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala
35 40 45
Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln
50 55 60
Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser
65 70 75 80
Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala
85 90 95
Ser Phe Pro Glu Gly Ser Trp
100
<210> 1495
<211> 104
<212> PRT
<213> Homo sapiens
<400> 1495
Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser
1 5 10 15
Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val
20 25 30
Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala
35 40 45
Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln
50 55 60
Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser
65 70 75 80
Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala
85 90 95
Ser Phe Pro Glu Gly Ser Trp Glu
100
<210> 1496
<211> 105
<212> PRT
<213> Homo sapiens
<400> 1496
Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser
1 5 10 15
Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val
20 25 30
Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala
35 40 45
Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln
50 55 60
Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser
65 70 75 80
Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala
85 90 95
Ser Phe Pro Glu Gly Ser Trp Glu Ala
100 105
<210> 1497
<211> 106
<212> PRT
<213> Homo sapiens
<400> 1497
Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser
1 5 10 15
Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val
20 25 30
Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala
35 40 45
Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln
50 55 60
Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser
65 70 75 80
Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala
85 90 95
Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys
100 105
<210> 1498
<211> 107
<212> PRT
<213> Homo sapiens
<400> 1498
Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser
1 5 10 15
Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val
20 25 30
Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala
35 40 45
Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln
50 55 60
Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser
65 70 75 80
Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala
85 90 95
Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
100 105
<210> 1499
<211> 102
<212> PRT
<213> Homo sapiens
<400> 1499
Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe
1 5 10 15
Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr
20 25 30
Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val
35 40 45
Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala
50 55 60
Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly
65 70 75 80
Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser
85 90 95
Phe Pro Glu Gly Ser Trp
100
<210> 1500
<211> 103
<212> PRT
<213> Homo sapiens
<400> 1500
Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe
1 5 10 15
Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr
20 25 30
Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val
35 40 45
Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala
50 55 60
Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly
65 70 75 80
Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser
85 90 95
Phe Pro Glu Gly Ser Trp Glu
100
<210> 1501
<211> 105
<212> PRT
<213> Homo sapiens
<400> 1501
Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe
1 5 10 15
Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr
20 25 30
Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val
35 40 45
Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala
50 55 60
Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly
65 70 75 80
Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser
85 90 95
Phe Pro Glu Gly Ser Trp Glu Ala Cys
100 105
<210> 1502
<211> 106
<212> PRT
<213> Homo sapiens
<400> 1502
Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe
1 5 10 15
Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr
20 25 30
Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val
35 40 45
Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala
50 55 60
Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly
65 70 75 80
Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser
85 90 95
Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
100 105
<210> 1503
<211> 101
<212> PRT
<213> Homo sapiens
<400> 1503
Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr
1 5 10 15
Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val
20 25 30
Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu
35 40 45
His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg
50 55 60
Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala
65 70 75 80
Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe
85 90 95
Pro Glu Gly Ser Trp
100
<210> 1504
<211> 102
<212> PRT
<213> Homo sapiens
<400> 1504
Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr
1 5 10 15
Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val
20 25 30
Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu
35 40 45
His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg
50 55 60
Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala
65 70 75 80
Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe
85 90 95
Pro Glu Gly Ser Trp Glu
100
<210> 1505
<211> 103
<212> PRT
<213> Homo sapiens
<400> 1505
Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr
1 5 10 15
Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val
20 25 30
Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu
35 40 45
His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg
50 55 60
Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala
65 70 75 80
Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe
85 90 95
Pro Glu Gly Ser Trp Glu Ala
100
<210> 1506
<211> 104
<212> PRT
<213> Homo sapiens
<400> 1506
Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr
1 5 10 15
Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val
20 25 30
Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu
35 40 45
His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg
50 55 60
Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala
65 70 75 80
Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe
85 90 95
Pro Glu Gly Ser Trp Glu Ala Cys
100
<210> 1507
<211> 105
<212> PRT
<213> Homo sapiens
<400> 1507
Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr
1 5 10 15
Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val
20 25 30
Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu
35 40 45
His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg
50 55 60
Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala
65 70 75 80
Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe
85 90 95
Pro Glu Gly Ser Trp Glu Ala Cys Gly
100 105
<210> 1508
<211> 100
<212> PRT
<213> Homo sapiens
<400> 1508
Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr Ile
1 5 10 15
Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val Ser
20 25 30
Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu His
35 40 45
Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg Trp
50 55 60
Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala Ser
65 70 75 80
Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro
85 90 95
Glu Gly Ser Trp
100
<210> 1509
<211> 101
<212> PRT
<213> Homo sapiens
<400> 1509
Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr Ile
1 5 10 15
Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val Ser
20 25 30
Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu His
35 40 45
Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg Trp
50 55 60
Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala Ser
65 70 75 80
Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro
85 90 95
Glu Gly Ser Trp Glu
100
<210> 1510
<211> 102
<212> PRT
<213> Homo sapiens
<400> 1510
Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr Ile
1 5 10 15
Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val Ser
20 25 30
Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu His
35 40 45
Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg Trp
50 55 60
Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala Ser
65 70 75 80
Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro
85 90 95
Glu Gly Ser Trp Glu Ala
100
<210> 1511
<211> 103
<212> PRT
<213> Homo sapiens
<400> 1511
Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr Ile
1 5 10 15
Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val Ser
20 25 30
Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu His
35 40 45
Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg Trp
50 55 60
Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala Ser
65 70 75 80
Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro
85 90 95
Glu Gly Ser Trp Glu Ala Cys
100
<210> 1512
<211> 104
<212> PRT
<213> Homo sapiens
<400> 1512
Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr Ile
1 5 10 15
Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val Ser
20 25 30
Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu His
35 40 45
Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg Trp
50 55 60
Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala Ser
65 70 75 80
Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro
85 90 95
Glu Gly Ser Trp Glu Ala Cys Gly
100
<210> 1513
<211> 234
<212> PRT
<213> Mus musculus
<400> 1513
Met Arg Thr Gly Asn Thr Gln Ala Ala His Ala Thr Asn Met Gly Gln
1 5 10 15
Met Gln Thr Leu Val Leu Phe Ser Thr Leu Leu Thr Leu Cys Val Ser
20 25 30
Glu Ala Ser Pro Glu Val Trp Val Gln Val Gln Met Glu Ala Thr Asn
35 40 45
Leu Ser Ser Phe Ser Val His Cys Gly Val Leu Gly Tyr Ser Leu Ile
50 55 60
Ser Leu Val Thr Val Ser Cys Glu Gly Phe Val Asp Ala Gly Arg Thr
65 70 75 80
Lys Leu Ala Val Leu His Pro Glu Phe Gly Thr Gln Gln Trp Ala Pro
85 90 95
Ala Arg Gln Ala His Trp Glu Thr Pro Asn Ser Val Ser Val Thr Leu
100 105 110
Thr Met Gly Gln Ser Lys Ala Arg Ser Ser Leu Ala Asn Thr Thr Phe
115 120 125
Cys Cys Glu Phe Val Thr Phe Pro His Gly Ser Arg Val Ala Cys Arg
130 135 140
Asp Leu His Arg Ser Asp Pro Gly Leu Ser Ala Pro Thr Pro Ala Leu
145 150 155 160
Asn Leu Gln Ala Asp Leu Val Arg Ile Leu Gly Thr Ser Gly Val Phe
165 170 175
Leu Phe Gly Phe Ile Phe Ile Leu Cys Leu Arg Trp Gln Gln Arg His
180 185 190
Trp Cys Leu Ser Lys Ser Gln Pro Ser Leu Thr Ser Thr Gln Ala Gln
195 200 205
Val Glu Thr Gln Pro Pro His Leu Ala Ser Thr His Ser Ser Phe Ile
210 215 220
Ser Met Glu Asn Gly Leu Tyr Ala Leu Ala
225 230
<210> 1514
<211> 221
<212> PRT
<213> Mus musculus
<400> 1514
Met Gly Gln Met Gln Thr Leu Val Leu Phe Ser Thr Leu Leu Thr Leu
1 5 10 15
Cys Val Ser Glu Ala Ser Pro Glu Val Trp Val Gln Val Gln Met Glu
20 25 30
Ala Thr Asn Leu Ser Ser Phe Ser Val His Cys Gly Val Leu Gly Tyr
35 40 45
Ser Leu Ile Ser Leu Val Thr Val Ser Cys Glu Gly Phe Val Asp Ala
50 55 60
Gly Arg Thr Lys Leu Ala Val Leu His Pro Glu Phe Gly Thr Gln Gln
65 70 75 80
Trp Ala Pro Ala Arg Gln Ala His Trp Glu Thr Pro Asn Ser Val Ser
85 90 95
Val Thr Leu Thr Met Gly Gln Ser Lys Ala Arg Ser Ser Leu Ala Asn
100 105 110
Thr Thr Phe Cys Cys Glu Phe Val Thr Phe Pro His Gly Ser Arg Val
115 120 125
Ala Cys Arg Asp Leu His Arg Ser Asp Pro Gly Leu Ser Ala Pro Thr
130 135 140
Pro Ala Leu Asn Leu Gln Ala Asp Leu Val Arg Ile Leu Gly Thr Ser
145 150 155 160
Gly Val Phe Leu Phe Gly Phe Ile Phe Ile Leu Cys Leu Arg Trp Gln
165 170 175
Gln Arg His Trp Cys Leu Ser Lys Ser Gln Pro Ser Leu Thr Ser Thr
180 185 190
Gln Ala Gln Val Glu Thr Gln Pro Pro His Leu Ala Ser Thr His Ser
195 200 205
Ser Phe Ile Ser Met Glu Asn Gly Leu Tyr Ala Leu Ala
210 215 220
<210> 1515
<211> 131
<212> PRT
<213> Mus musculus
<400> 1515
Ser Pro Glu Val Trp Val Gln Val Gln Met Glu Ala Thr Asn Leu Ser
1 5 10 15
Ser Phe Ser Val His Cys Gly Val Leu Gly Tyr Ser Leu Ile Ser Leu
20 25 30
Val Thr Val Ser Cys Glu Gly Phe Val Asp Ala Gly Arg Thr Lys Leu
35 40 45
Ala Val Leu His Pro Glu Phe Gly Thr Gln Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala His Trp Glu Thr Pro Asn Ser Val Ser Val Thr Leu Thr Met
65 70 75 80
Gly Gln Ser Lys Ala Arg Ser Ser Leu Ala Asn Thr Thr Phe Cys Cys
85 90 95
Glu Phe Val Thr Phe Pro His Gly Ser Arg Val Ala Cys Arg Asp Leu
100 105 110
His Arg Ser Asp Pro Gly Leu Ser Ala Pro Thr Pro Ala Leu Asn Leu
115 120 125
Gln Ala Asp
130
<210> 1516
<211> 152
<212> PRT
<213> Mus musculus
<400> 1516
Met Gly Gln Met Gln Thr Leu Val Leu Phe Ser Thr Leu Leu Thr Leu
1 5 10 15
Cys Val Ser Glu Ala Ser Pro Glu Val Trp Val Gln Val Gln Met Glu
20 25 30
Ala Thr Asn Leu Ser Ser Phe Ser Val His Cys Gly Val Leu Gly Tyr
35 40 45
Ser Leu Ile Ser Leu Val Thr Val Ser Cys Glu Gly Phe Val Asp Ala
50 55 60
Gly Arg Thr Lys Leu Ala Val Leu His Pro Glu Phe Gly Thr Gln Gln
65 70 75 80
Trp Ala Pro Ala Arg Gln Ala His Trp Glu Thr Pro Asn Ser Val Ser
85 90 95
Val Thr Leu Thr Met Gly Gln Ser Lys Ala Arg Ser Ser Leu Ala Asn
100 105 110
Thr Thr Phe Cys Cys Glu Phe Val Thr Phe Pro His Gly Ser Arg Val
115 120 125
Ala Cys Arg Asp Leu His Arg Ser Asp Pro Gly Leu Ser Ala Pro Thr
130 135 140
Pro Ala Leu Asn Leu Gln Ala Asp
145 150
<210> 1517
<211> 363
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1517
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala Asp Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro
130 135 140
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
145 150 155 160
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
165 170 175
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
180 185 190
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
195 200 205
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
210 215 220
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
225 230 235 240
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
245 250 255
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
260 265 270
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
275 280 285
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
290 295 300
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
305 310 315 320
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
325 330 335
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
340 345 350
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
355 360
<210> 1518
<211> 363
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1518
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Phe Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Ser Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala Asp Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro
130 135 140
Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
145 150 155 160
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
165 170 175
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
180 185 190
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
195 200 205
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
210 215 220
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
225 230 235 240
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
245 250 255
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
260 265 270
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
275 280 285
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
290 295 300
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
305 310 315 320
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
325 330 335
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
340 345 350
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
355 360
<210> 1519
<211> 363
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1519
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala Asp Glu Pro Lys Ser Ser Asp Lys Thr His Thr Ser Pro Pro
130 135 140
Ser Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
145 150 155 160
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
165 170 175
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
180 185 190
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
195 200 205
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
210 215 220
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
225 230 235 240
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
245 250 255
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
260 265 270
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
275 280 285
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
290 295 300
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
305 310 315 320
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
325 330 335
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
340 345 350
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
355 360
<210> 1520
<211> 363
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1520
Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala Thr Glu Leu Ser
1 5 10 15
Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly Ser Ile Ser Leu
20 25 30
Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly Gly Thr Thr Leu
35 40 45
Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu Ile Leu Glu Gly
65 70 75 80
Ser Gly Ala Ser Ser Pro Phe Ala Asn Thr Thr Phe Cys Cys Lys Phe
85 90 95
Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly Ser Leu Pro Pro
100 105 110
Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro Ala Pro Ile Leu
115 120 125
Arg Ala Asp Glu Pro Lys Ser Ser Asp Lys Thr His Thr Ser Pro Pro
130 135 140
Ser Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro
145 150 155 160
Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr
165 170 175
Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn
180 185 190
Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg
195 200 205
Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val
210 215 220
Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser
225 230 235 240
Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys
245 250 255
Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp
260 265 270
Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe
275 280 285
Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu
290 295 300
Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe
305 310 315 320
Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly
325 330 335
Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr
340 345 350
Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
355 360
<210> 1521
<211> 364
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1521
Ser Pro Glu Val Trp Val Gln Val Gln Met Glu Ala Thr Asn Leu Ser
1 5 10 15
Ser Phe Ser Val His Cys Gly Val Leu Gly Tyr Ser Leu Ile Ser Leu
20 25 30
Val Thr Val Ser Cys Glu Gly Phe Val Asp Ala Gly Arg Thr Lys Leu
35 40 45
Ala Val Leu His Pro Glu Phe Gly Thr Gln Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala His Trp Glu Thr Pro Asn Ser Val Ser Val Thr Leu Thr Met
65 70 75 80
Gly Gln Ser Lys Ala Arg Ser Ser Leu Ala Asn Thr Thr Phe Cys Cys
85 90 95
Glu Phe Val Thr Phe Pro His Gly Ser Arg Val Ala Cys Arg Asp Leu
100 105 110
His Arg Ser Asp Pro Gly Leu Ser Ala Pro Thr Pro Ala Leu Asn Leu
115 120 125
Gln Ala Asp Glu Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys
130 135 140
Lys Cys Pro Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe
145 150 155 160
Pro Pro Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val
165 170 175
Thr Cys Val Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile
180 185 190
Ser Trp Phe Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr
195 200 205
His Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro
210 215 220
Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val
225 230 235 240
Asn Asn Lys Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro
245 250 255
Lys Gly Ser Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu
260 265 270
Glu Glu Met Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp
275 280 285
Phe Met Pro Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr
290 295 300
Glu Leu Asn Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser
305 310 315 320
Tyr Phe Met Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu
325 330 335
Arg Asn Ser Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His
340 345 350
His Thr Thr Lys Ser Phe Ser Arg Thr Pro Gly Lys
355 360
<210> 1522
<211> 364
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1522
Ser Pro Glu Val Trp Val Gln Val Gln Met Glu Ala Thr Asn Leu Ser
1 5 10 15
Ser Phe Ser Val His Cys Gly Val Leu Gly Tyr Ser Leu Ile Ser Leu
20 25 30
Val Thr Val Ser Cys Glu Gly Phe Val Asp Ala Gly Arg Thr Lys Leu
35 40 45
Ala Val Leu His Pro Glu Phe Gly Thr Gln Gln Trp Ala Pro Ala Arg
50 55 60
Gln Ala His Trp Glu Thr Pro Asn Ser Val Ser Val Thr Leu Thr Met
65 70 75 80
Gly Gln Ser Lys Ala Arg Ser Ser Leu Ala Asn Thr Thr Phe Cys Cys
85 90 95
Glu Phe Val Thr Phe Pro His Gly Ser Arg Val Ala Cys Arg Asp Leu
100 105 110
His Arg Ser Asp Pro Gly Leu Ser Ala Pro Thr Pro Ala Leu Asn Leu
115 120 125
Gln Ala Asp Glu Pro Arg Gly Pro Thr Ile Lys Pro Ser Pro Pro Ser
130 135 140
Lys Ser Pro Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe
145 150 155 160
Pro Pro Lys Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val
165 170 175
Thr Cys Val Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile
180 185 190
Ser Trp Phe Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr
195 200 205
His Arg Glu Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro
210 215 220
Ile Gln His Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val
225 230 235 240
Asn Asn Lys Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro
245 250 255
Lys Gly Ser Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu
260 265 270
Glu Glu Met Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp
275 280 285
Phe Met Pro Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr
290 295 300
Glu Leu Asn Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser
305 310 315 320
Tyr Phe Met Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu
325 330 335
Arg Asn Ser Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His
340 345 350
His Thr Thr Lys Ser Phe Ser Arg Thr Pro Gly Lys
355 360
<210> 1523
<211> 232
<212> PRT
<213> Homo sapiens
<400> 1523
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 1524
<211> 232
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1524
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 1525
<211> 232
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1525
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
Tyr Ala Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 1526
<211> 217
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1526
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
1 5 10 15
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
20 25 30
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
35 40 45
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
50 55 60
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
65 70 75 80
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
85 90 95
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
100 105 110
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu
115 120 125
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
130 135 140
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
145 150 155 160
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
165 170 175
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
180 185 190
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
195 200 205
Lys Ser Leu Ser Leu Ser Pro Gly Lys
210 215
<210> 1527
<211> 233
<212> PRT
<213> Mus musculus
<400> 1527
Glu Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys Cys Pro
1 5 10 15
Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys
20 25 30
Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val
35 40 45
Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe
50 55 60
Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu
65 70 75 80
Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His
85 90 95
Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys
100 105 110
Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser
115 120 125
Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met
130 135 140
Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro
145 150 155 160
Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn
165 170 175
Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met
180 185 190
Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser
195 200 205
Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr
210 215 220
Lys Ser Phe Ser Arg Thr Pro Gly Lys
225 230
<210> 1528
<211> 233
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1528
Glu Pro Arg Gly Pro Thr Ile Lys Pro Cys Pro Pro Cys Lys Cys Pro
1 5 10 15
Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys
20 25 30
Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val
35 40 45
Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe
50 55 60
Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu
65 70 75 80
Asp Tyr Ala Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His
85 90 95
Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys
100 105 110
Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser
115 120 125
Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met
130 135 140
Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro
145 150 155 160
Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn
165 170 175
Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met
180 185 190
Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser
195 200 205
Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr
210 215 220
Lys Ser Phe Ser Arg Thr Pro Gly Lys
225 230
<210> 1529
<211> 217
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1529
Ala Pro Asn Leu Leu Gly Gly Pro Ser Val Phe Ile Phe Pro Pro Lys
1 5 10 15
Ile Lys Asp Val Leu Met Ile Ser Leu Ser Pro Ile Val Thr Cys Val
20 25 30
Val Val Asp Val Ser Glu Asp Asp Pro Asp Val Gln Ile Ser Trp Phe
35 40 45
Val Asn Asn Val Glu Val His Thr Ala Gln Thr Gln Thr His Arg Glu
50 55 60
Asp Tyr Asn Ser Thr Leu Arg Val Val Ser Ala Leu Pro Ile Gln His
65 70 75 80
Gln Asp Trp Met Ser Gly Lys Glu Phe Lys Cys Lys Val Asn Asn Lys
85 90 95
Asp Leu Pro Ala Pro Ile Glu Arg Thr Ile Ser Lys Pro Lys Gly Ser
100 105 110
Val Arg Ala Pro Gln Val Tyr Val Leu Pro Pro Pro Glu Glu Glu Met
115 120 125
Thr Lys Lys Gln Val Thr Leu Thr Cys Met Val Thr Asp Phe Met Pro
130 135 140
Glu Asp Ile Tyr Val Glu Trp Thr Asn Asn Gly Lys Thr Glu Leu Asn
145 150 155 160
Tyr Lys Asn Thr Glu Pro Val Leu Asp Ser Asp Gly Ser Tyr Phe Met
165 170 175
Tyr Ser Lys Leu Arg Val Glu Lys Lys Asn Trp Val Glu Arg Asn Ser
180 185 190
Tyr Ser Cys Ser Val Val His Glu Gly Leu His Asn His His Thr Thr
195 200 205
Lys Ser Phe Ser Arg Thr Pro Gly Lys
210 215
<210> 1530
<211> 232
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1530
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Ser Pro Pro Ser Pro Ala
1 5 10 15
Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly Lys
225 230
<210> 1531
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1531
ccaccaacct ctcgtctttc 20
<210> 1532
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1532
tcatgccaga gcatacag 18
<210> 1533
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1533
cagtgcctct aactgctgac 20
<210> 1534
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1534
tcactgttac cagggagatg ag 22
<210> 1535
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1535
cacaggctgc ccatgcaac 19
<210> 1536
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1536
tgcctgggtg ctagtgagag 20
<210> 1537
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1537
ccaccaacct ctcgtctttc 20
<210> 1538
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
primer
<400> 1538
gaccctgtta cctgtcattg 20
<210> 1539
<211> 314
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1539
Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu Leu Thr Leu Cys
1 5 10 15
Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val Arg Met Glu Ala
20 25 30
Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe Leu Gly Ser Gly
35 40 45
Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro Asn Gly Ala Gly
50 55 60
Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly Ile Arg Gln Trp
65 70 75 80
Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser Ser Ile Ser Leu
85 90 95
Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala Asn Thr Thr Phe
100 105 110
Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp Glu Ala Cys Gly
115 120 125
Ser Leu Pro Pro Ser Ser Asp Pro Gly Leu Ser Ala Pro Pro Thr Pro
130 135 140
Ala Pro Ile Leu Arg Ala Asp Leu Ala Gly Ile Leu Gly Val Ser Gly
145 150 155 160
Val Leu Leu Phe Gly Cys Val Tyr Leu Leu His Leu Leu Arg Arg His
165 170 175
Lys His Arg Pro Ala Pro Arg Leu Gln Pro Ser Arg Thr Ser Pro Gln
180 185 190
Ala Pro Arg Ala Arg Ala Trp Ala Pro Ser Gln Ala Ser Gln Ala Ala
195 200 205
Leu His Val Pro Tyr Ala Thr Ile Asn Thr Ser Cys Arg Pro Ala Thr
210 215 220
Leu Asp Thr Ala His Pro His Gly Gly Pro Ser Trp Trp Ala Ser Leu
225 230 235 240
Pro Thr His Ala Ala His Arg Pro Gln Gly Pro Ala Ala Trp Ala Ser
245 250 255
Thr Pro Ile Pro Ala Arg Gly Ser Phe Val Ser Val Glu Asn Gly Leu
260 265 270
Tyr Ala Gln Ala Gly Glu Arg Pro Pro His Thr Gly Pro Gly Leu Thr
275 280 285
Leu Phe Pro Asp Pro Arg Gly Pro Arg Ala Met Glu Gly Pro Leu Gly
290 295 300
Val Arg Asp Tyr Lys Asp Asp Asp Asp Lys
305 310
<210> 1540
<211> 242
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1540
Met Arg Thr Gly Asn Thr Gln Ala Ala His Ala Thr Asn Met Gly Gln
1 5 10 15
Met Gln Thr Leu Val Leu Phe Ser Thr Leu Leu Thr Leu Cys Val Ser
20 25 30
Glu Ala Asp Tyr Lys Asp Asp Asp Asp Lys Ser Pro Glu Val Trp Val
35 40 45
Gln Val Gln Met Glu Ala Thr Asn Leu Ser Ser Phe Ser Val His Cys
50 55 60
Gly Val Leu Gly Tyr Ser Leu Ile Ser Leu Val Thr Val Ser Cys Glu
65 70 75 80
Gly Phe Val Asp Ala Gly Arg Thr Lys Leu Ala Val Leu His Pro Glu
85 90 95
Phe Gly Thr Gln Gln Trp Ala Pro Ala Arg Gln Ala His Trp Glu Thr
100 105 110
Pro Asn Ser Val Ser Val Thr Leu Thr Met Gly Gln Ser Lys Ala Arg
115 120 125
Ser Ser Leu Ala Asn Thr Thr Phe Cys Cys Glu Phe Val Thr Phe Pro
130 135 140
His Gly Ser Arg Val Ala Cys Arg Asp Leu His Arg Ser Asp Pro Gly
145 150 155 160
Leu Ser Ala Pro Thr Pro Ala Leu Asn Leu Gln Ala Asp Leu Val Arg
165 170 175
Ile Leu Gly Thr Ser Gly Val Phe Leu Phe Gly Phe Ile Phe Ile Leu
180 185 190
Cys Leu Arg Trp Gln Gln Arg His Trp Cys Leu Ser Lys Ser Gln Pro
195 200 205
Ser Leu Thr Ser Thr Gln Ala Gln Val Glu Thr Gln Pro Pro His Leu
210 215 220
Ala Ser Thr His Ser Ser Phe Ile Ser Met Glu Asn Gly Leu Tyr Ala
225 230 235 240
Leu Ala
<210> 1541
<211> 218
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1541
Met Gln Thr Leu Val Leu Phe Ser Thr Leu Leu Thr Leu Cys Val Ser
1 5 10 15
Glu Ala Ser Pro Glu Val Trp Val Gln Val Gln Met Glu Ala Thr Asn
20 25 30
Leu Ser Ser Phe Ser Val His Cys Gly Val Leu Gly Tyr Ser Leu Ile
35 40 45
Ser Leu Val Thr Val Ser Cys Glu Gly Phe Val Asp Ala Gly Arg Thr
50 55 60
Lys Leu Ala Val Leu His Pro Glu Phe Gly Thr Gln Gln Trp Ala Pro
65 70 75 80
Ala Arg Gln Ala His Trp Glu Thr Pro Asn Ser Val Ser Val Thr Leu
85 90 95
Thr Met Gly Gln Ser Lys Ala Arg Ser Ser Leu Ala Asn Thr Thr Phe
100 105 110
Cys Cys Glu Phe Val Thr Phe Pro His Gly Ser Arg Val Ala Cys Arg
115 120 125
Asp Leu His Arg Ser Asp Pro Gly Leu Ser Ala Pro Thr Pro Ala Leu
130 135 140
Asn Leu Gln Ala Asp Leu Val Arg Ile Leu Gly Thr Ser Gly Val Phe
145 150 155 160
Leu Phe Gly Phe Ile Phe Ile Leu Cys Leu Arg Trp Gln Gln Arg His
165 170 175
Trp Cys Leu Ser Lys Ser Gln Pro Ser Leu Thr Ser Thr Gln Ala Gln
180 185 190
Val Glu Thr Gln Pro Pro His Leu Ala Ser Thr His Ser Ser Phe Ile
195 200 205
Ser Met Glu Asn Gly Leu Tyr Ala Leu Ala
210 215
<210> 1542
<211> 226
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic
polypeptide
<400> 1542
Met Gln Thr Leu Val Leu Phe Ser Thr Leu Leu Thr Leu Cys Val Ser
1 5 10 15
Glu Ala Asp Tyr Lys Asp Asp Asp Asp Lys Ser Pro Glu Val Trp Val
20 25 30
Gln Val Gln Met Glu Ala Thr Asn Leu Ser Ser Phe Ser Val His Cys
35 40 45
Gly Val Leu Gly Tyr Ser Leu Ile Ser Leu Val Thr Val Ser Cys Glu
50 55 60
Gly Phe Val Asp Ala Gly Arg Thr Lys Leu Ala Val Leu His Pro Glu
65 70 75 80
Phe Gly Thr Gln Gln Trp Ala Pro Ala Arg Gln Ala His Trp Glu Thr
85 90 95
Pro Asn Ser Val Ser Val Thr Leu Thr Met Gly Gln Ser Lys Ala Arg
100 105 110
Ser Ser Leu Ala Asn Thr Thr Phe Cys Cys Glu Phe Val Thr Phe Pro
115 120 125
His Gly Ser Arg Val Ala Cys Arg Asp Leu His Arg Ser Asp Pro Gly
130 135 140
Leu Ser Ala Pro Thr Pro Ala Leu Asn Leu Gln Ala Asp Leu Val Arg
145 150 155 160
Ile Leu Gly Thr Ser Gly Val Phe Leu Phe Gly Phe Ile Phe Ile Leu
165 170 175
Cys Leu Arg Trp Gln Gln Arg His Trp Cys Leu Ser Lys Ser Gln Pro
180 185 190
Ser Leu Thr Ser Thr Gln Ala Gln Val Glu Thr Gln Pro Pro His Leu
195 200 205
Ala Ser Thr His Ser Ser Phe Ile Ser Met Glu Asn Gly Leu Tyr Ala
210 215 220
Leu Ala
225
<210> 1543
<211> 326
<212> PRT
<213> Artificial Sequence
<220>
<223> NCBI Reference Sequence: NP 076975
<400> 1543
Met Arg Thr Glu Ala Gln Val Pro Ala Leu Gln Pro Pro Glu Pro Gly
1 5 10 15
Leu Glu Gly Ala Met Gly His Arg Thr Leu Val Leu Pro Trp Val Leu
20 25 30
Leu Thr Leu Cys Val Thr Ala Gly Thr Pro Glu Val Trp Val Gln Val
35 40 45
Arg Met Glu Ala Thr Glu Leu Ser Ser Phe Thr Ile Arg Cys Gly Phe
50 55 60
Leu Gly Ser Gly Ser Ile Ser Leu Val Thr Val Ser Trp Gly Gly Pro
65 70 75 80
Asn Gly Ala Gly Gly Thr Thr Leu Ala Val Leu His Pro Glu Arg Gly
85 90 95
Ile Arg Gln Trp Ala Pro Ala Arg Gln Ala Arg Trp Glu Thr Gln Ser
100 105 110
Ser Ile Ser Leu Ile Leu Glu Gly Ser Gly Ala Ser Ser Pro Cys Ala
115 120 125
Asn Thr Thr Phe Cys Cys Lys Phe Ala Ser Phe Pro Glu Gly Ser Trp
130 135 140
Glu Ala Cys Gly Ser Leu Pro Pro Ser Ser Asp Pro Gly Leu Ser Ala
145 150 155 160
Pro Pro Thr Pro Ala Pro Ile Leu Arg Ala Asp Leu Ala Gly Ile Leu
165 170 175
Gly Val Ser Gly Val Leu Leu Phe Gly Cys Val Tyr Leu Leu His Leu
180 185 190
Leu Arg Arg His Lys His Arg Pro Ala Pro Arg Leu Gln Pro Ser Arg
195 200 205
Thr Ser Pro Gln Ala Pro Arg Ala Arg Ala Trp Ala Pro Ser Gln Ala
210 215 220
Ser Gln Ala Ala Leu His Val Pro Tyr Ala Thr Ile Asn Thr Ser Cys
225 230 235 240
Arg Pro Ala Thr Leu Asp Thr Ala His Pro His Gly Gly Pro Ser Trp
245 250 255
Trp Ala Ser Leu Pro Thr His Ala Ala His Arg Pro Gln Gly Pro Ala
260 265 270
Ala Trp Ala Ser Thr Pro Ile Pro Ala Arg Gly Ser Phe Val Ser Val
275 280 285
Glu Asn Gly Leu Tyr Ala Gln Ala Gly Glu Arg Pro Pro His Thr Gly
290 295 300
Pro Gly Leu Thr Leu Phe Pro Asp Pro Arg Gly Pro Arg Ala Met Glu
305 310 315 320
Gly Pro Leu Gly Val Arg
325
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| АНТИ-PVRIG АНТИТЕЛА И СПОСОБЫ ПРИМЕНЕНИЯ | 2016 |
|
RU2827926C2 |
| Антитела против CXCR2 и их применение | 2019 |
|
RU2807067C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИГЕНСВЯЗЫВАЮЩИЕ КОНСТРУКЦИИ ПРОТИВ HER2 | 2014 |
|
RU2737882C2 |
| ГИБРИДНЫЕ БЕЛКИ ВАРИАНТА sPD-1—FC | 2019 |
|
RU2785993C2 |
| IL-12 ГЕТЕРОДИМЕРНЫЕ СЛИТЫЕ БЕЛКИ FC | 2019 |
|
RU2819097C2 |
| АНТИТЕЛА ПРОТИВ PD-L1 И ИХ ВАРИАНТЫ | 2017 |
|
RU2770590C2 |
| НОВЫЕ ФЕРМЕНТЫ CRISPR И СИСТЕМЫ | 2016 |
|
RU2771826C2 |
| ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ И ПУТИ ИХ ПРИМЕНЕНИЯ | 2020 |
|
RU2841244C2 |
| НОВЫЕ ФЕРМЕНТЫ И СИСТЕМЫ CRISPR | 2016 |
|
RU2777988C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИГЕНСВЯЗЫВАЮЩИЕ ПОЛИПЕПТИДЫ | 2015 |
|
RU2723940C2 |
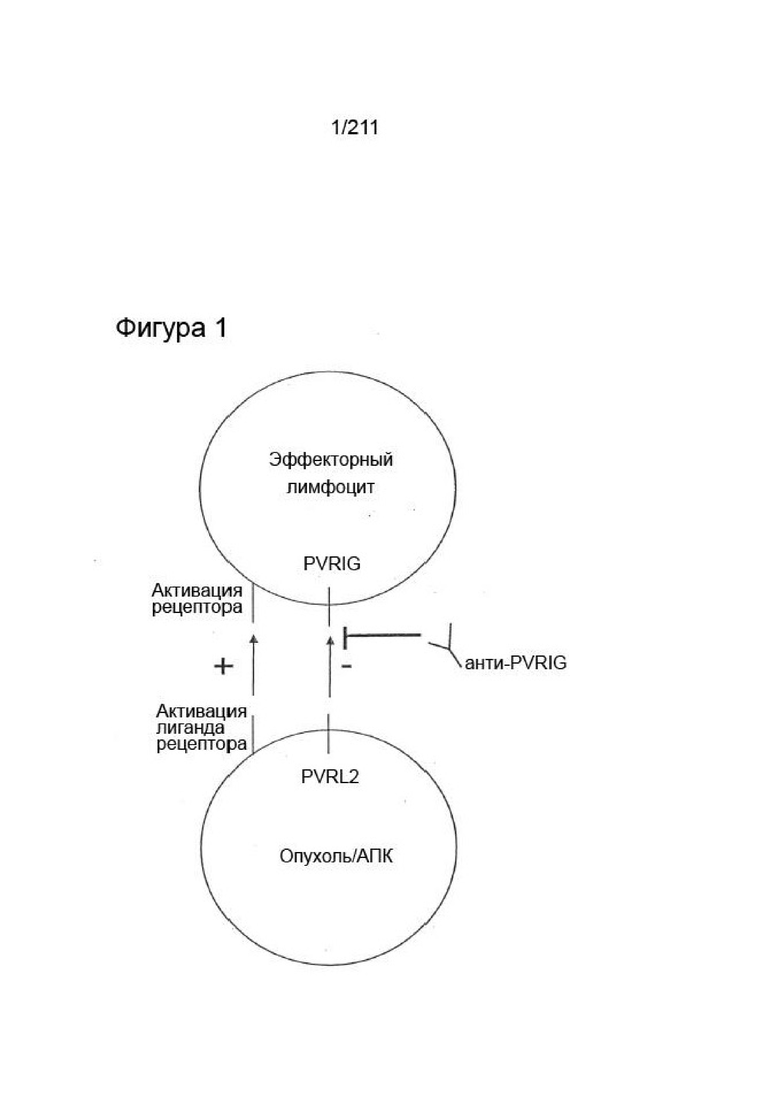
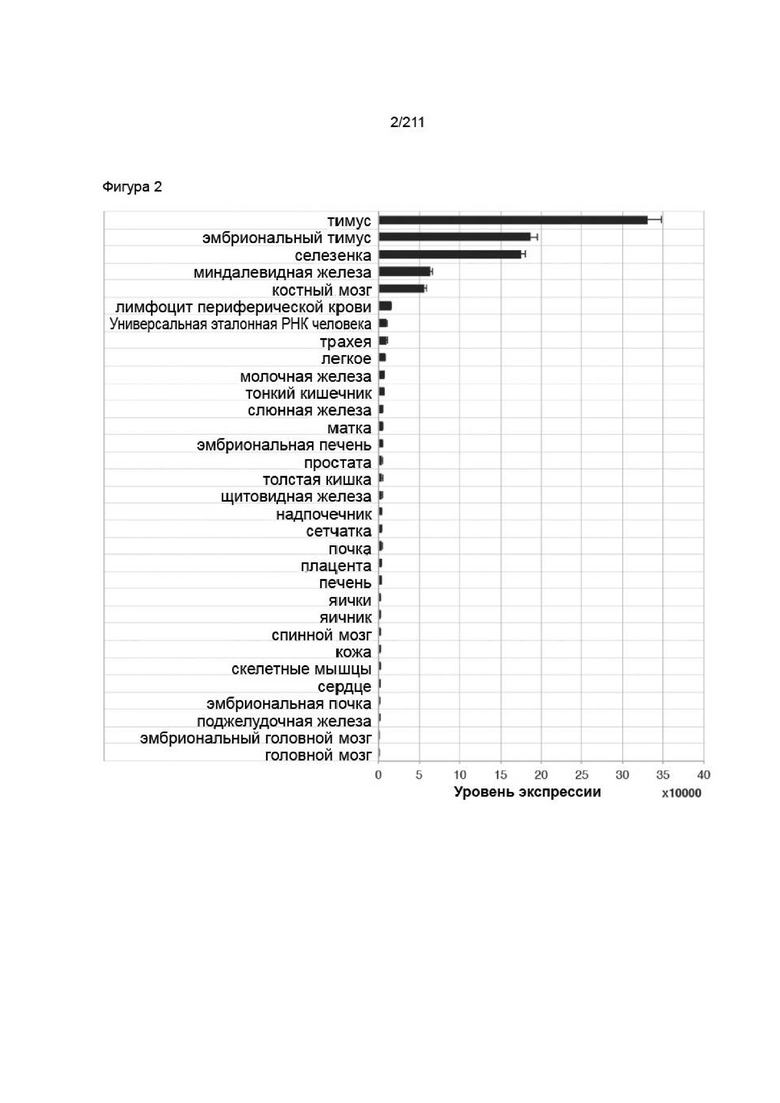

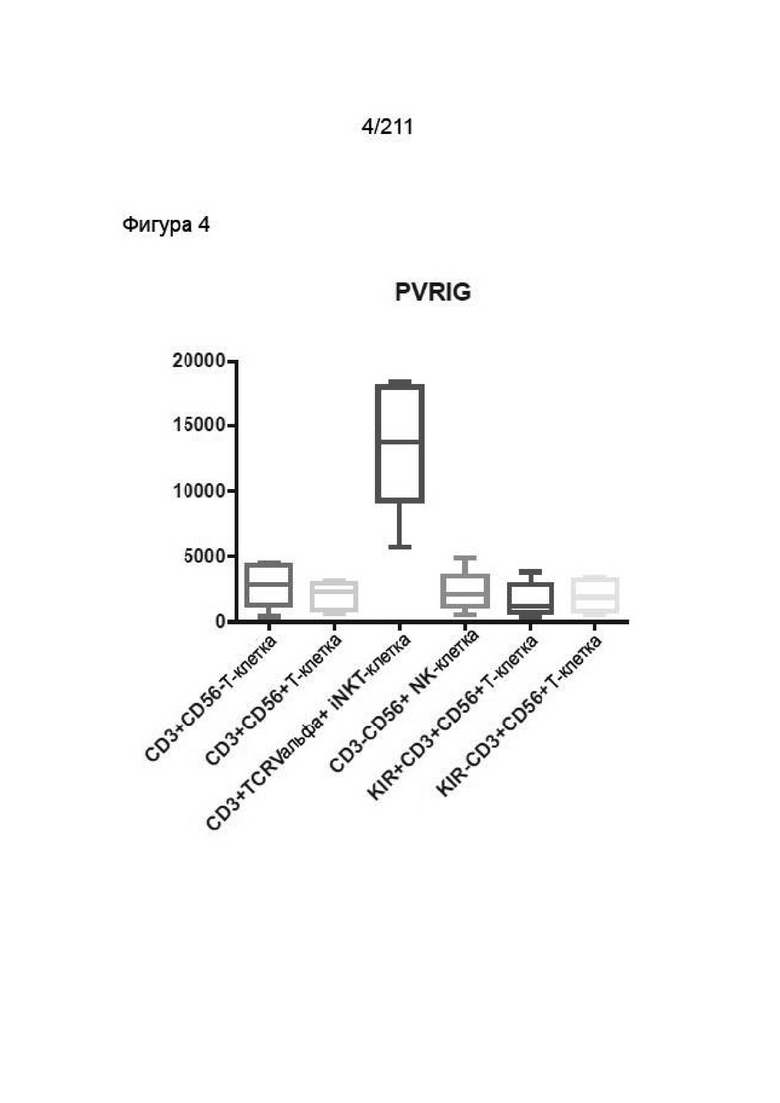
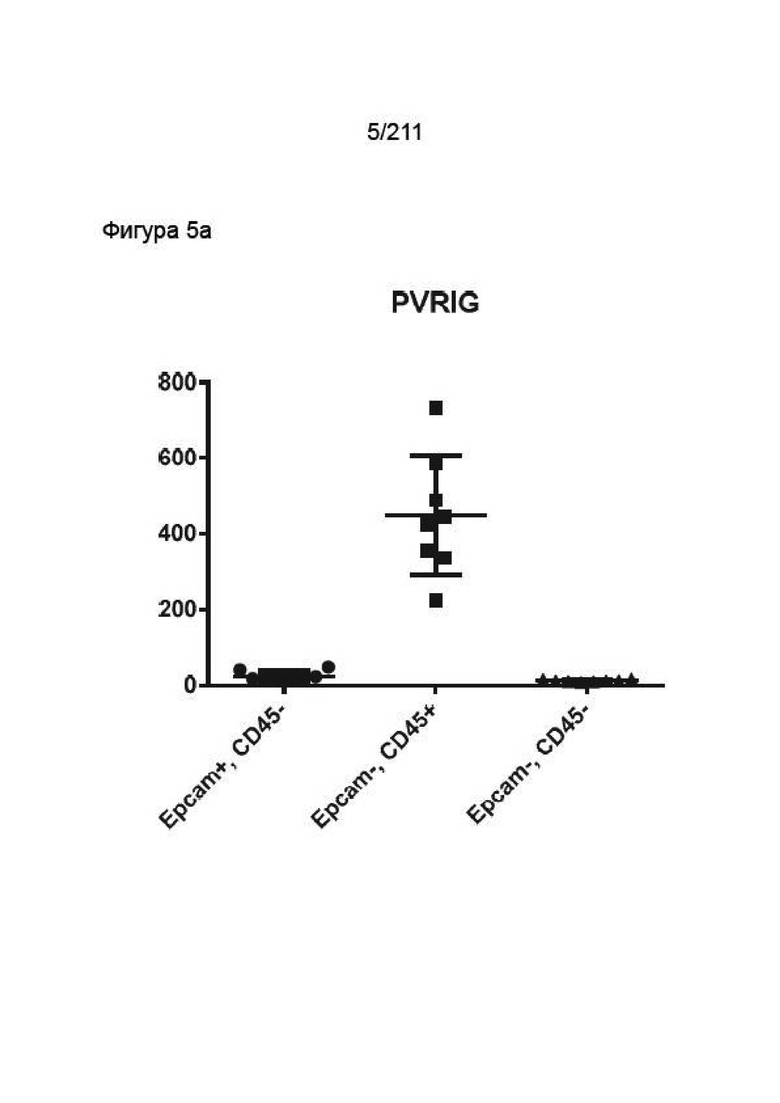
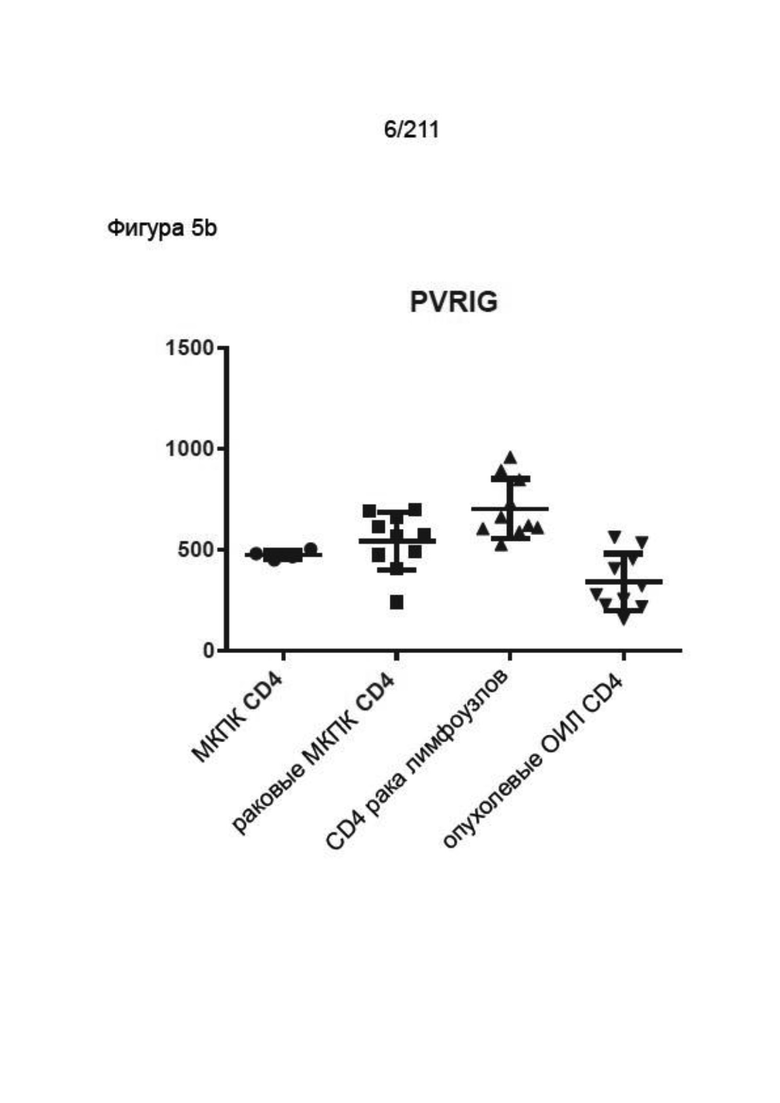
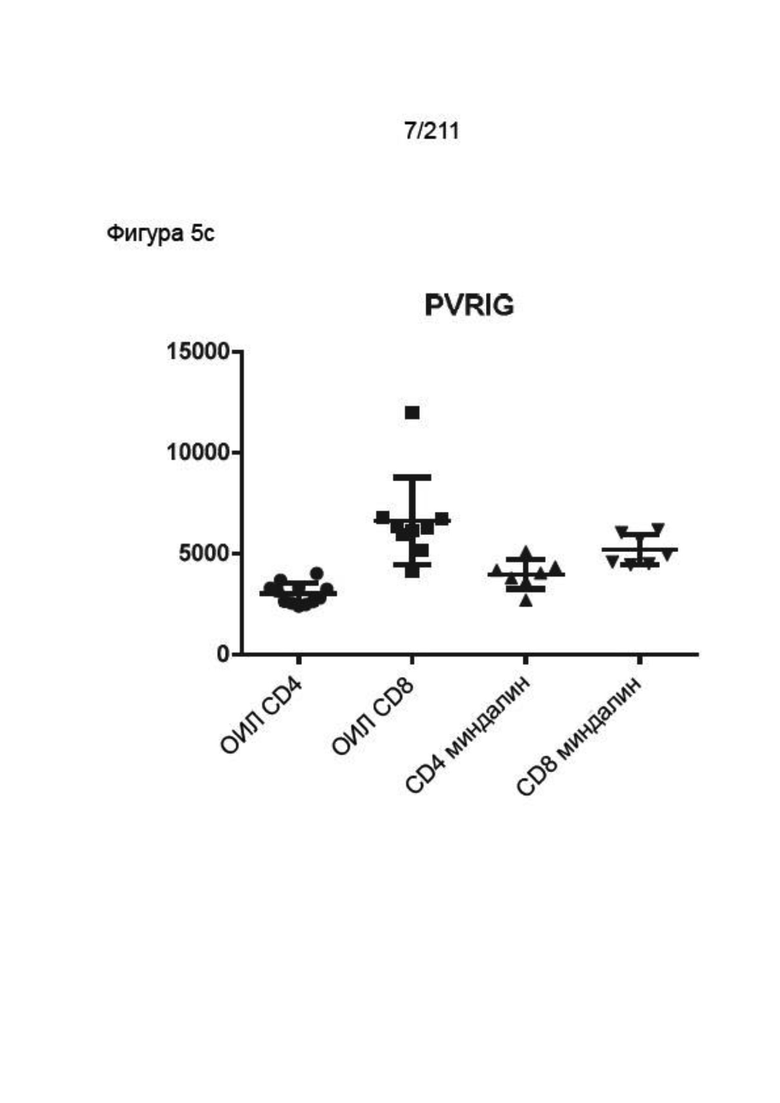


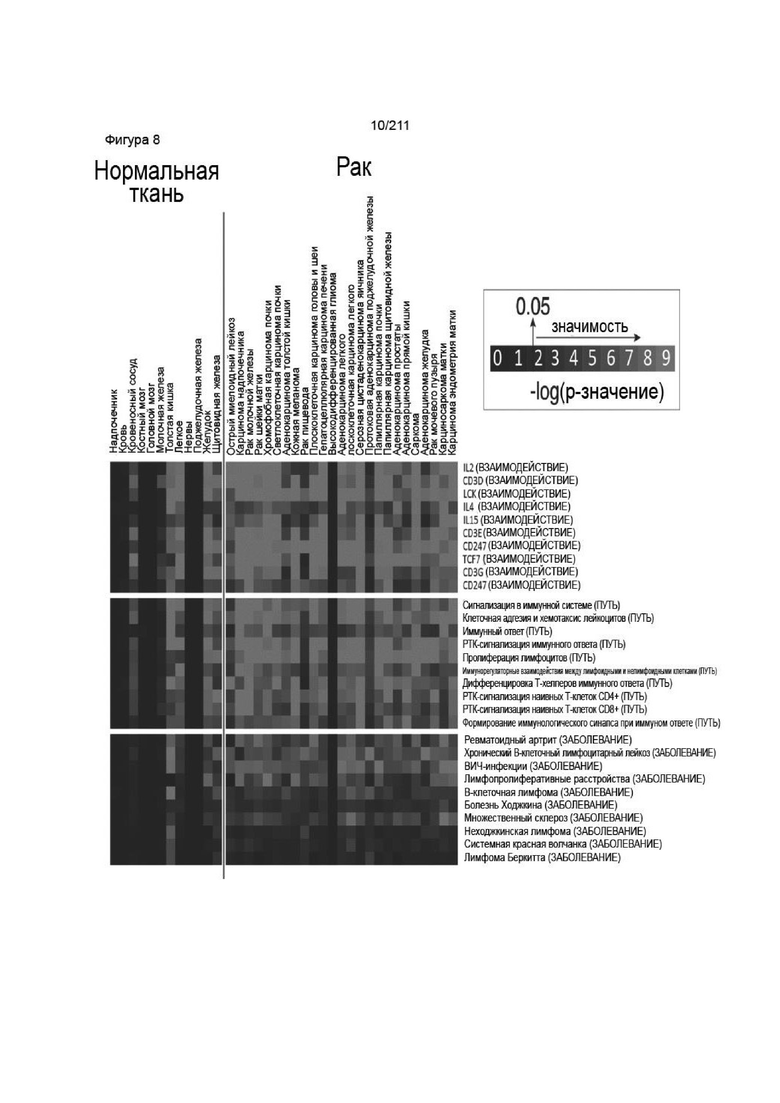
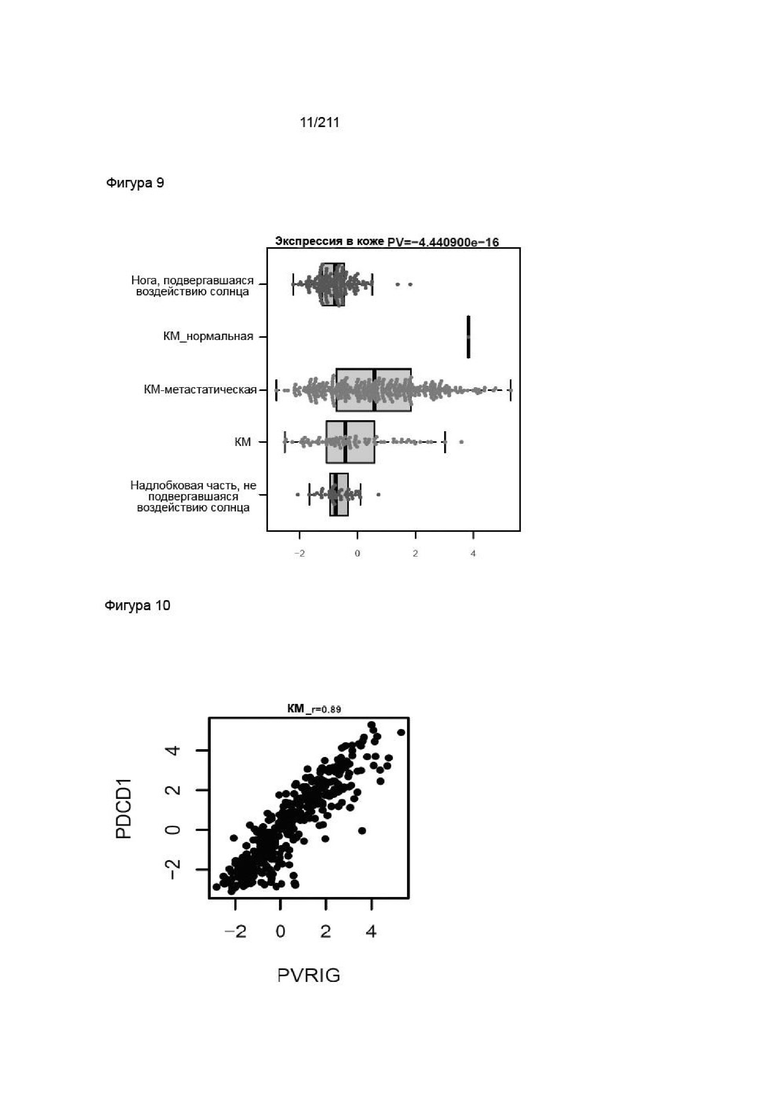
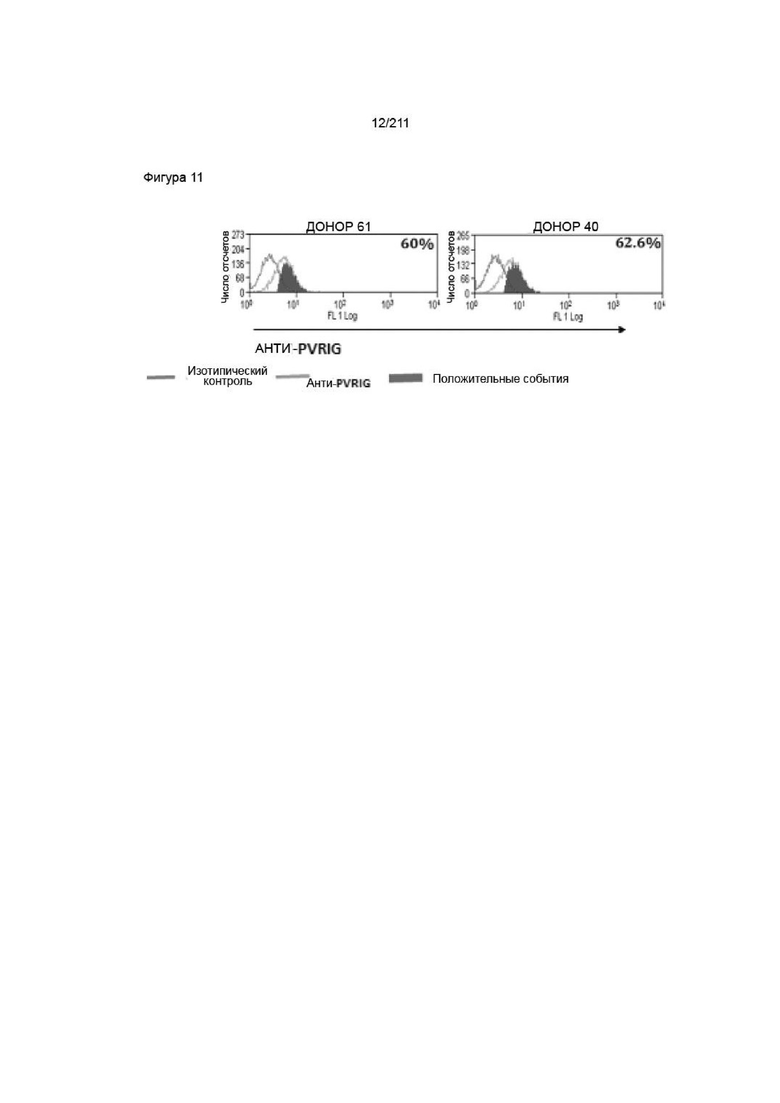
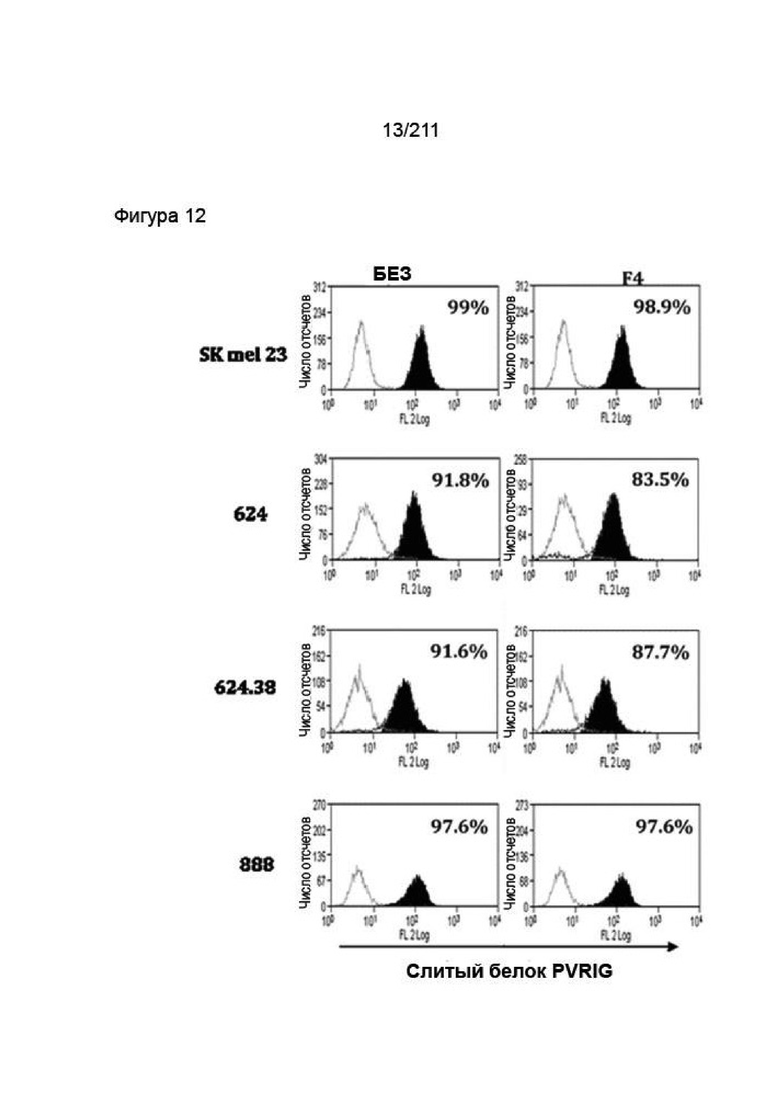
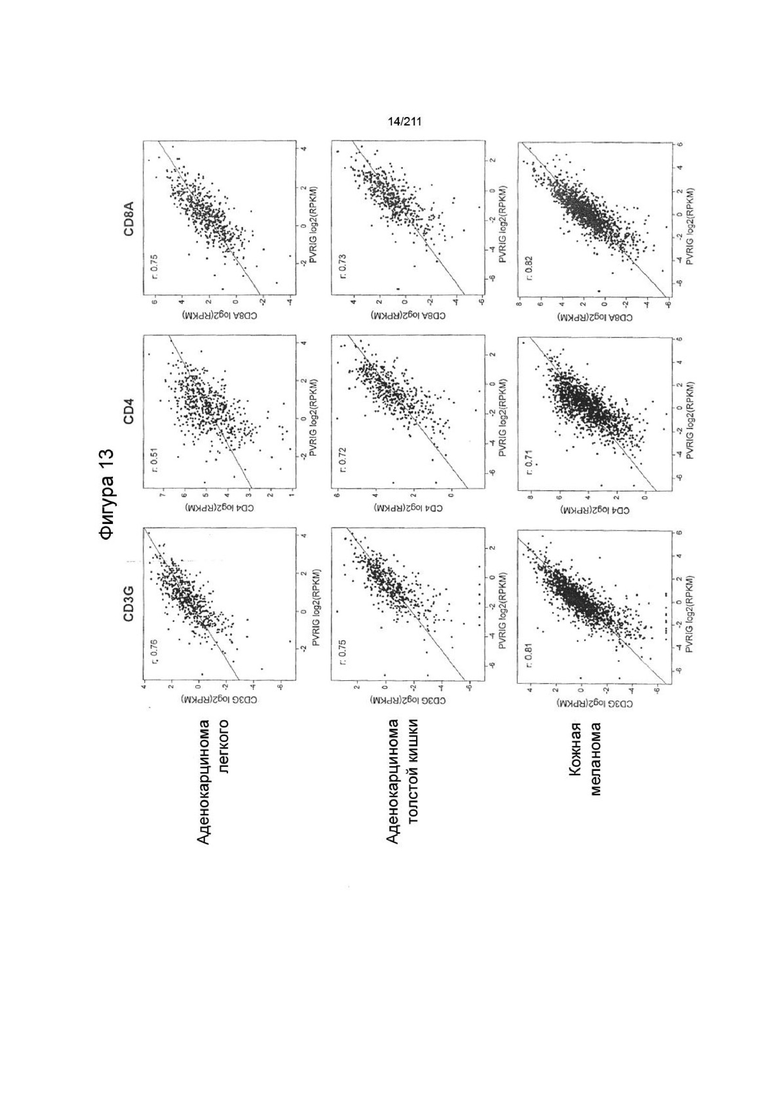

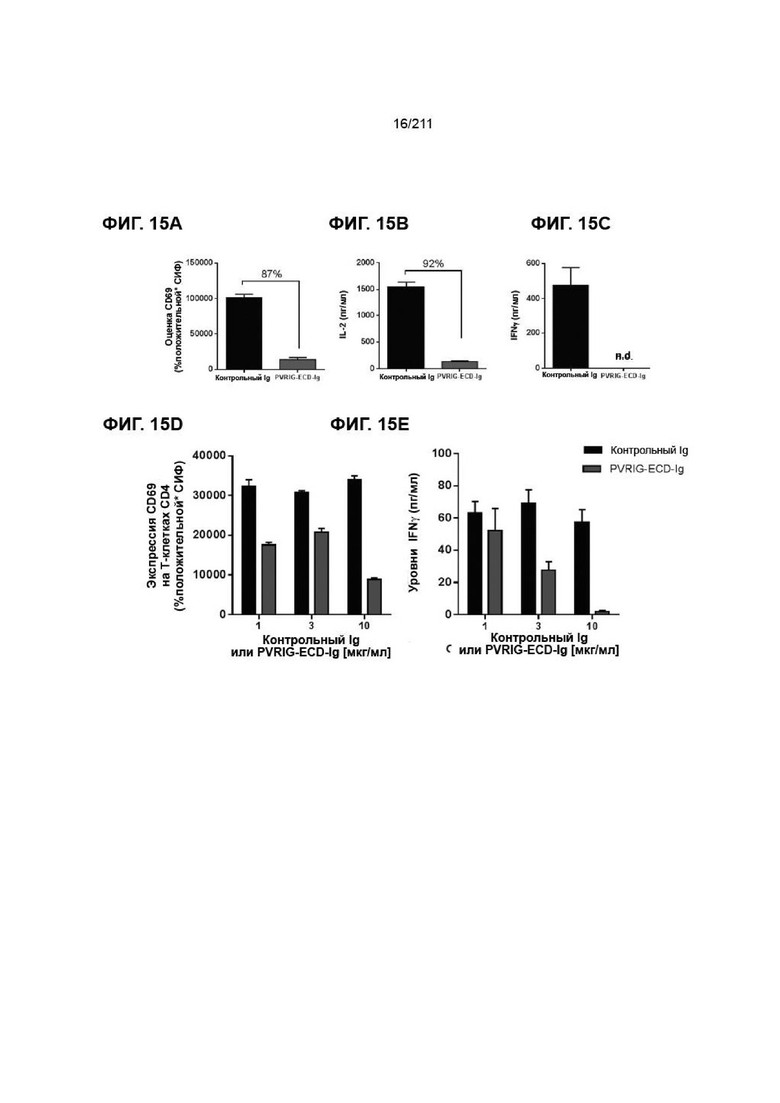
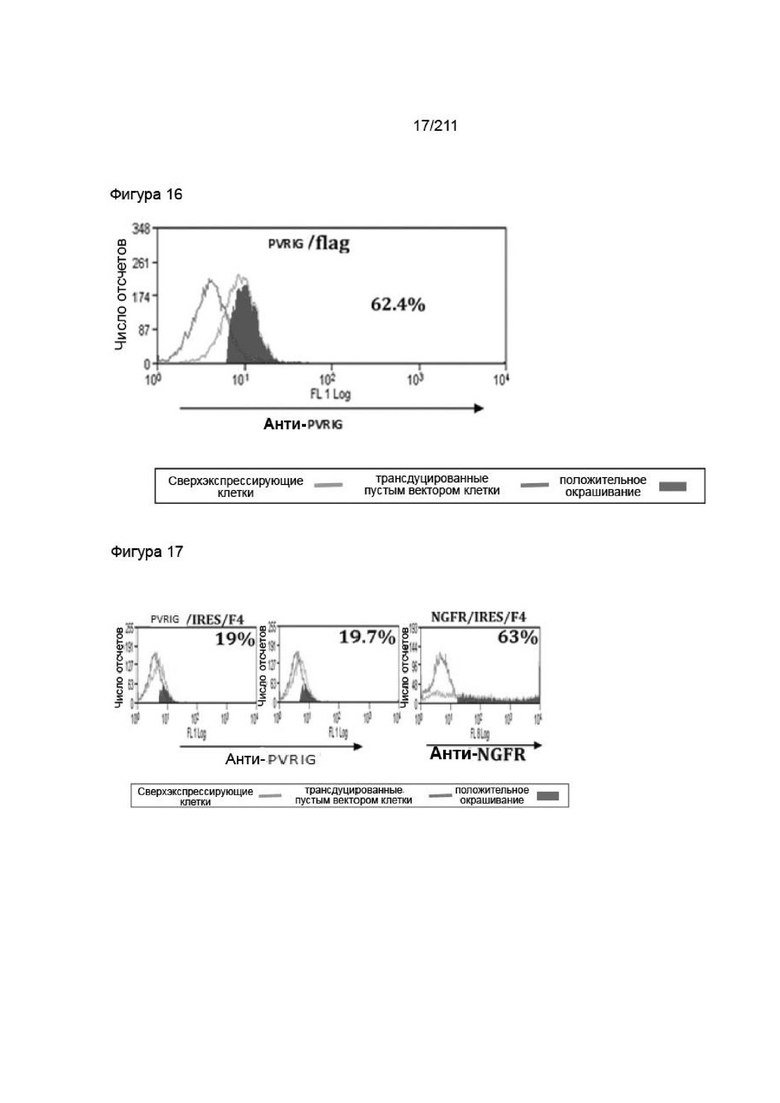
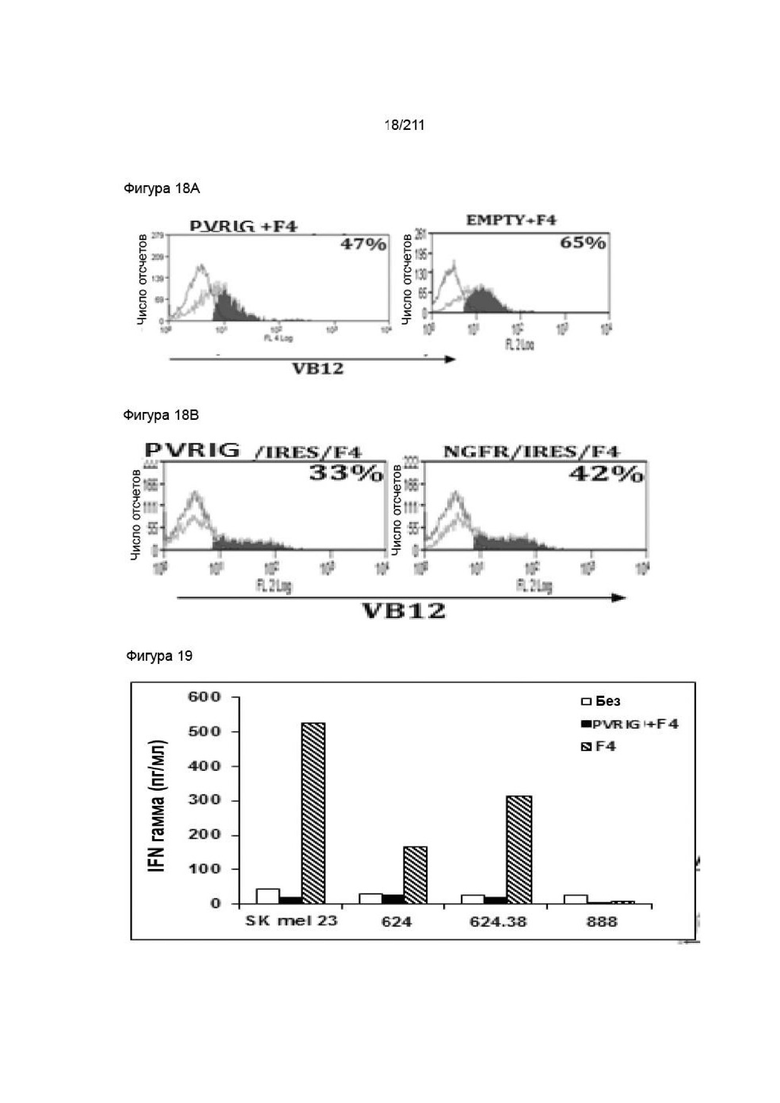
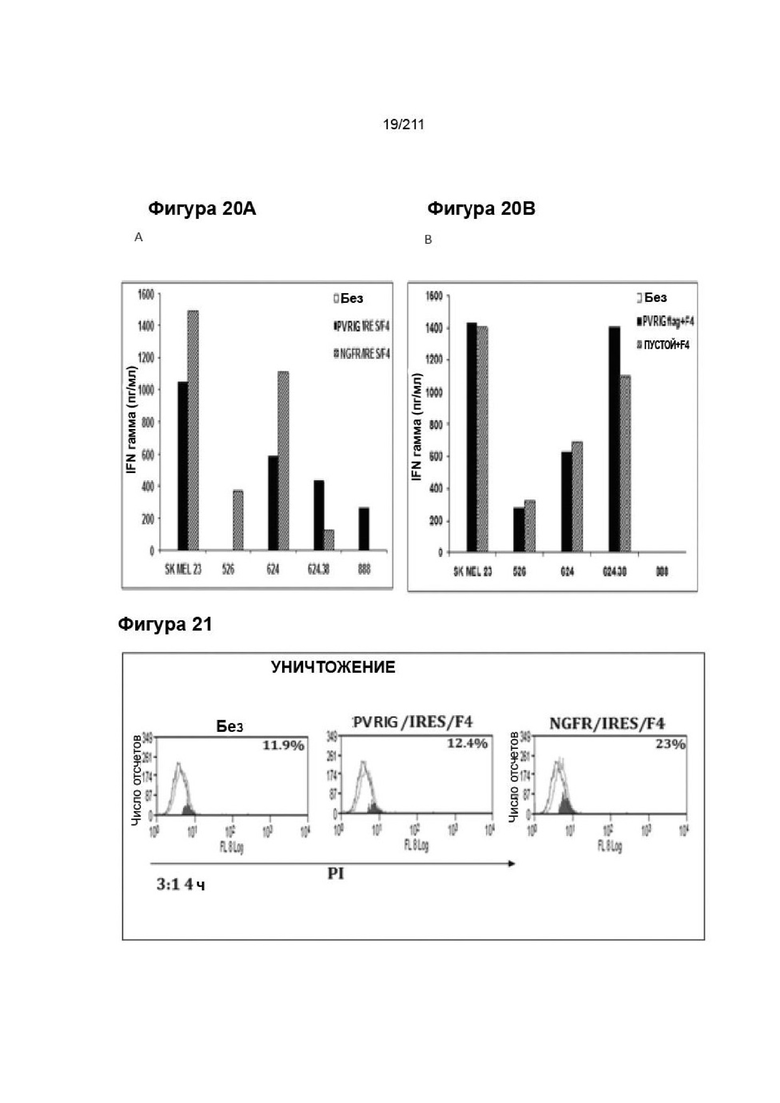

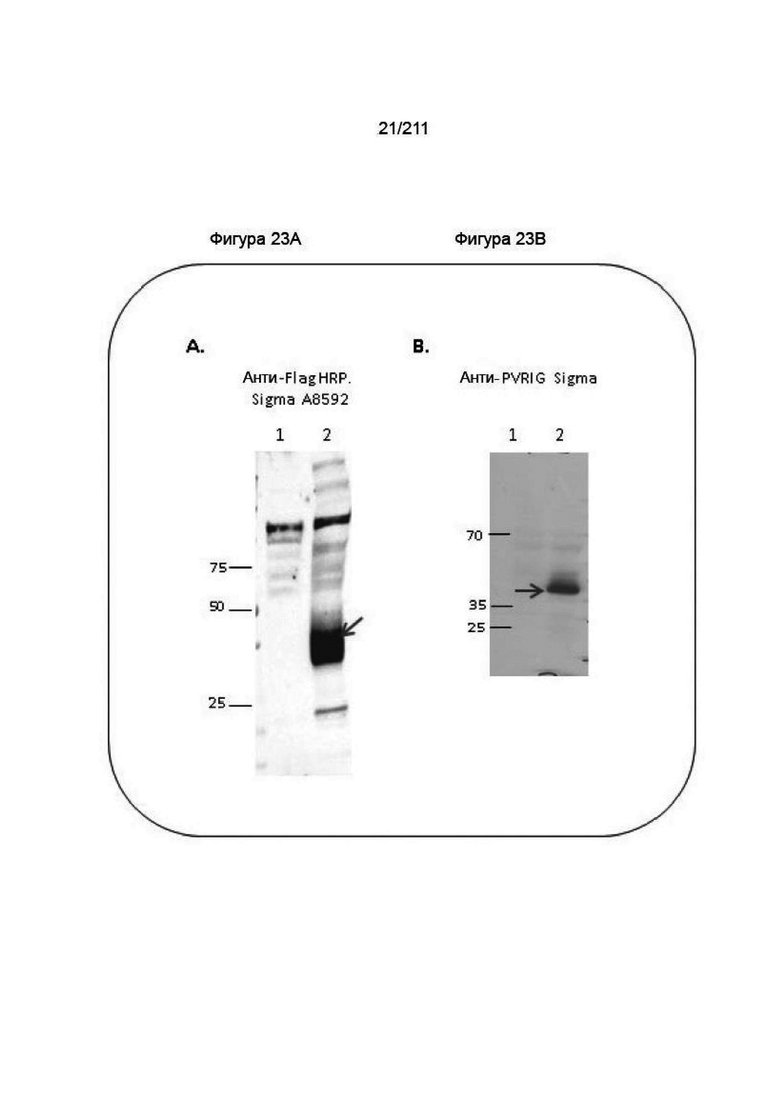
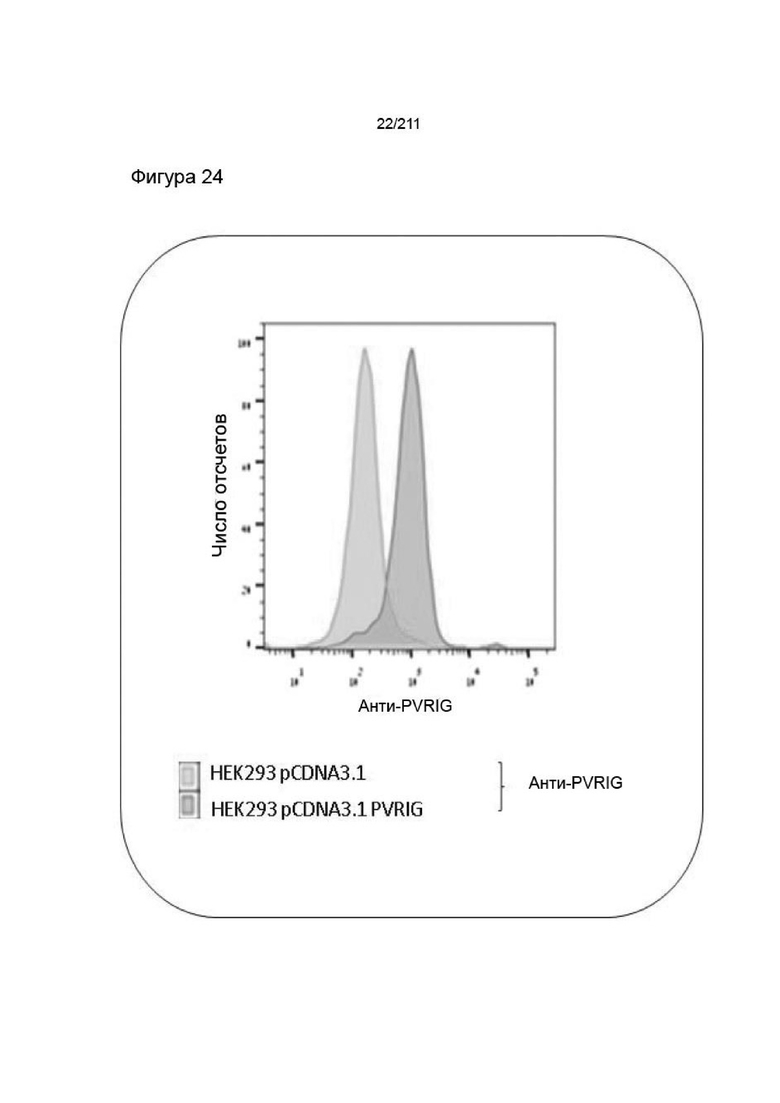


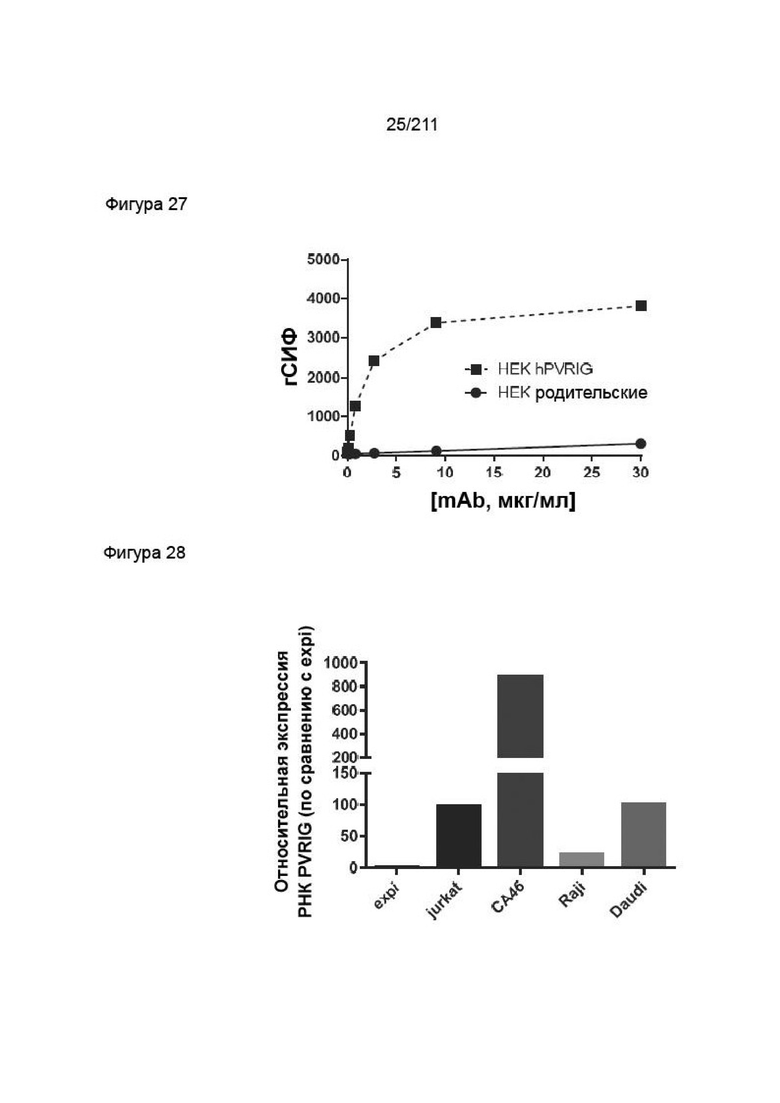
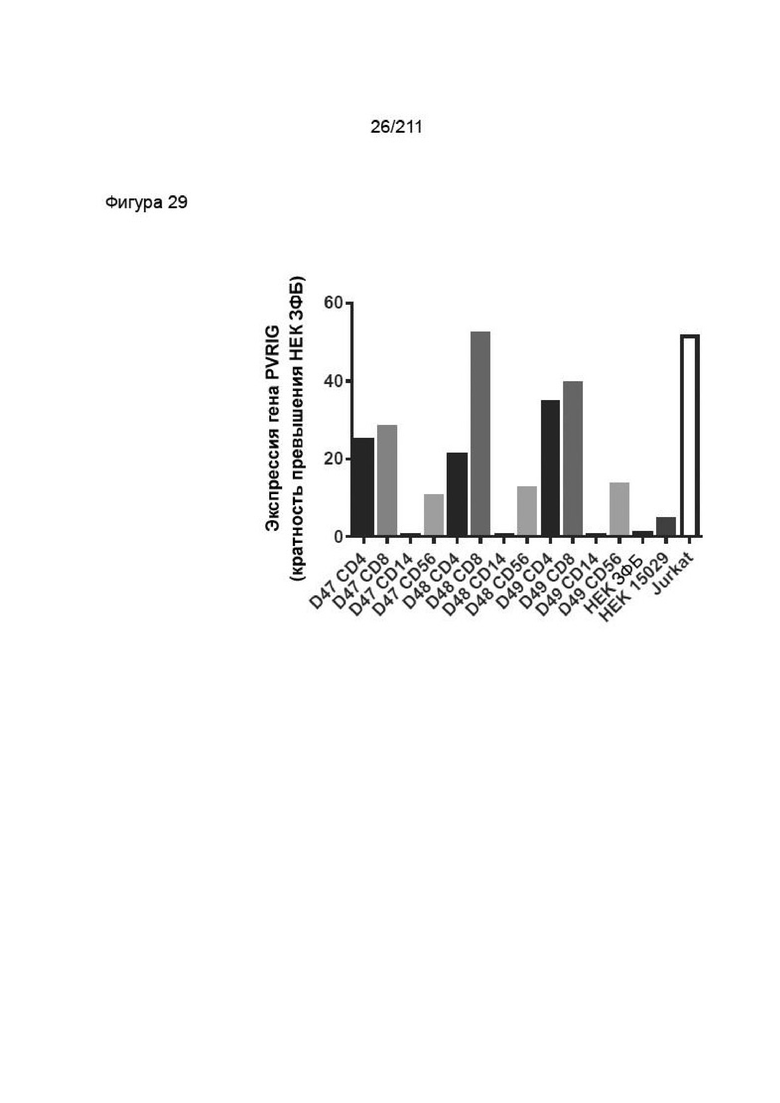
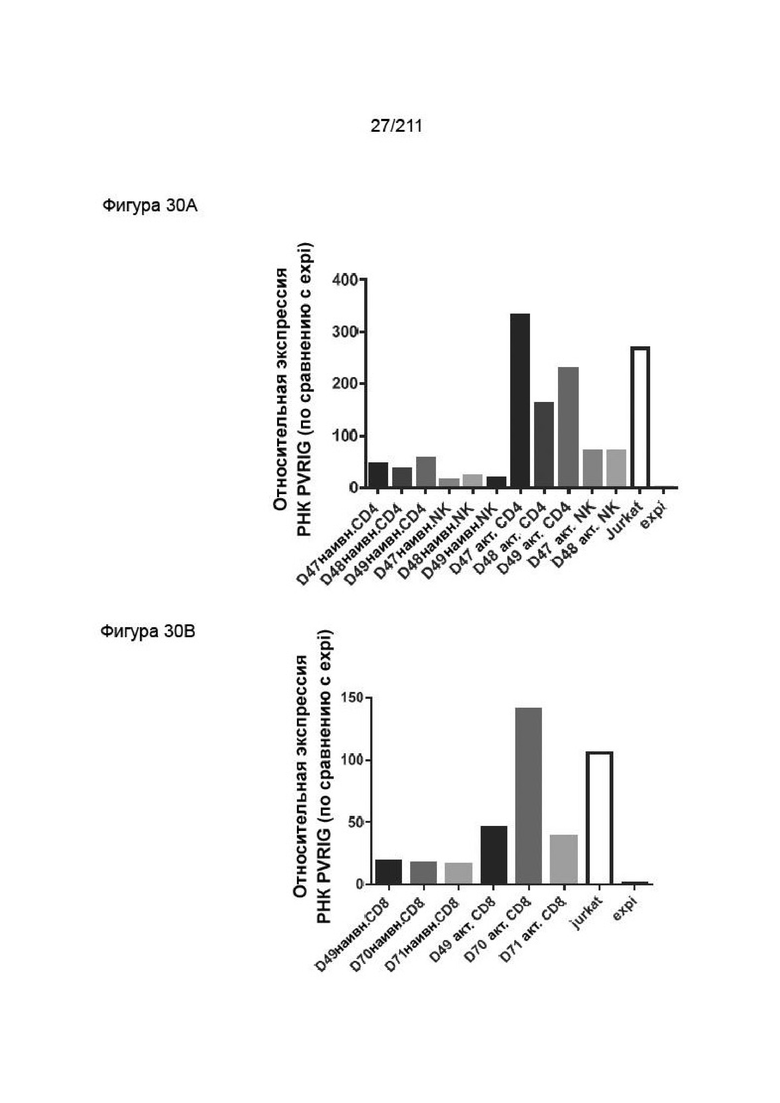
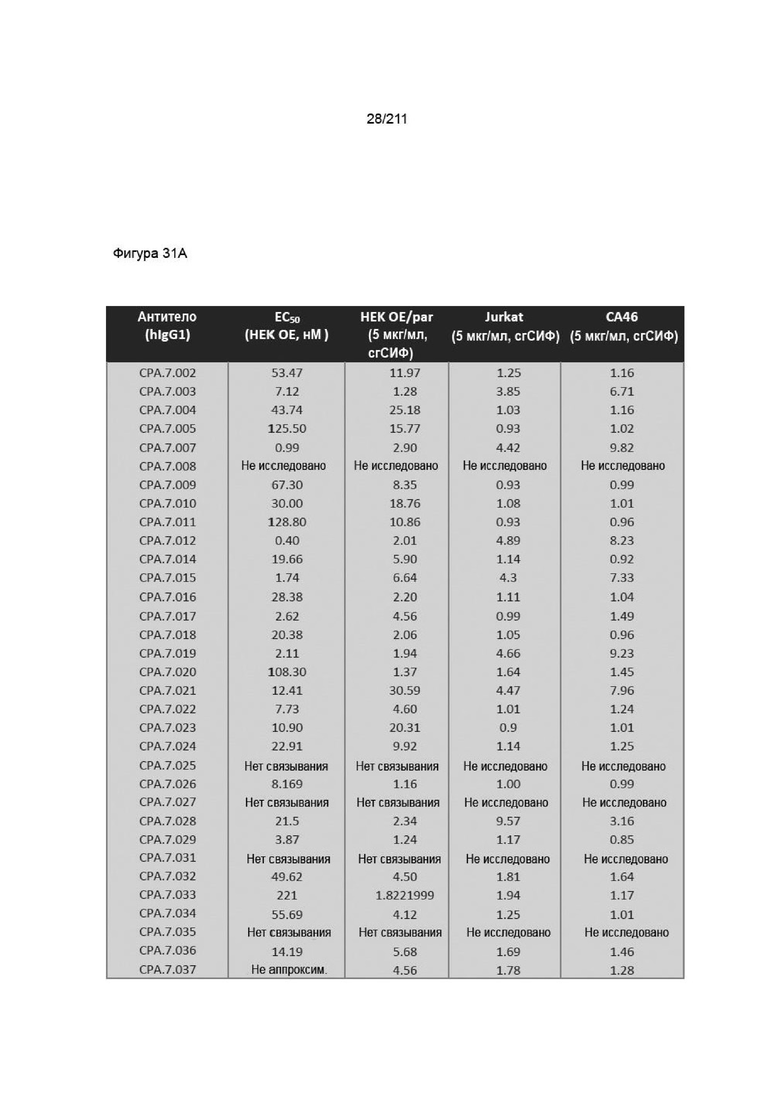
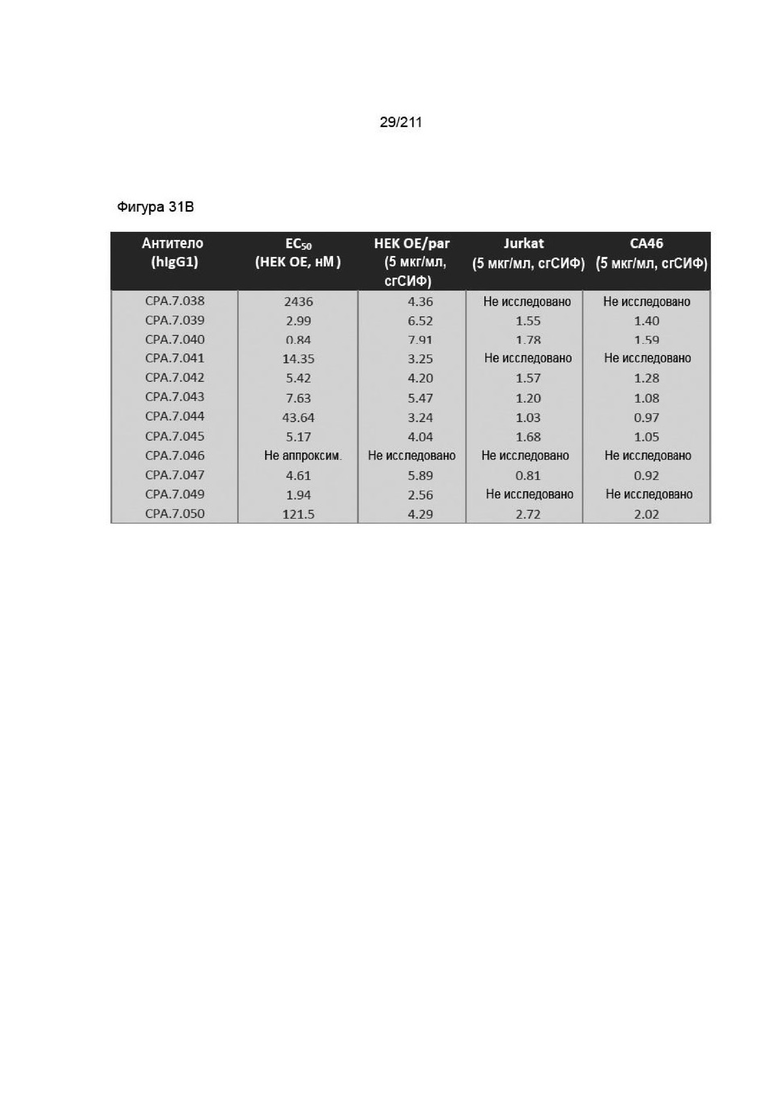
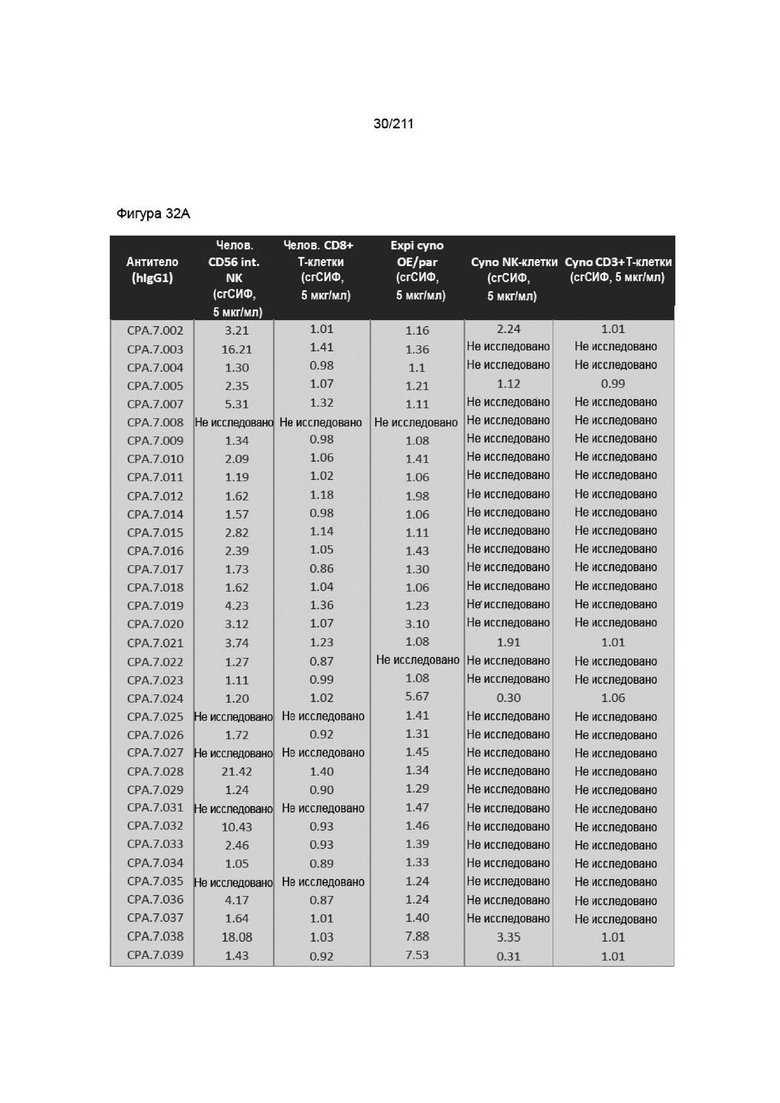
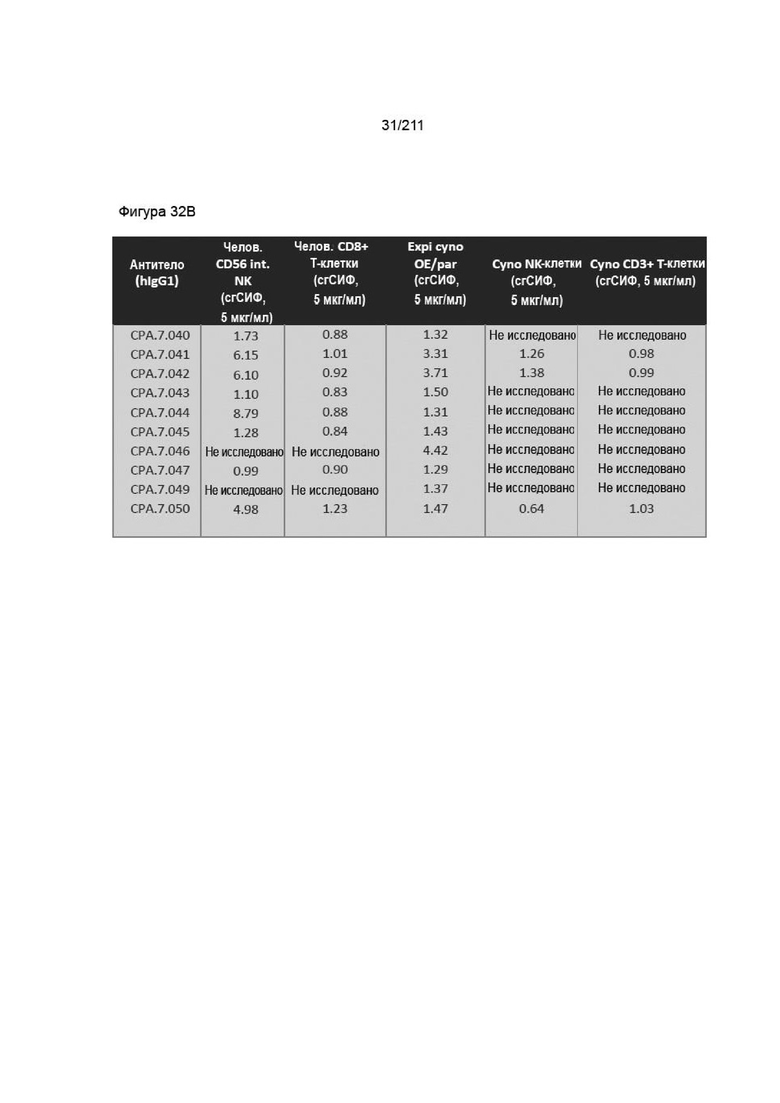
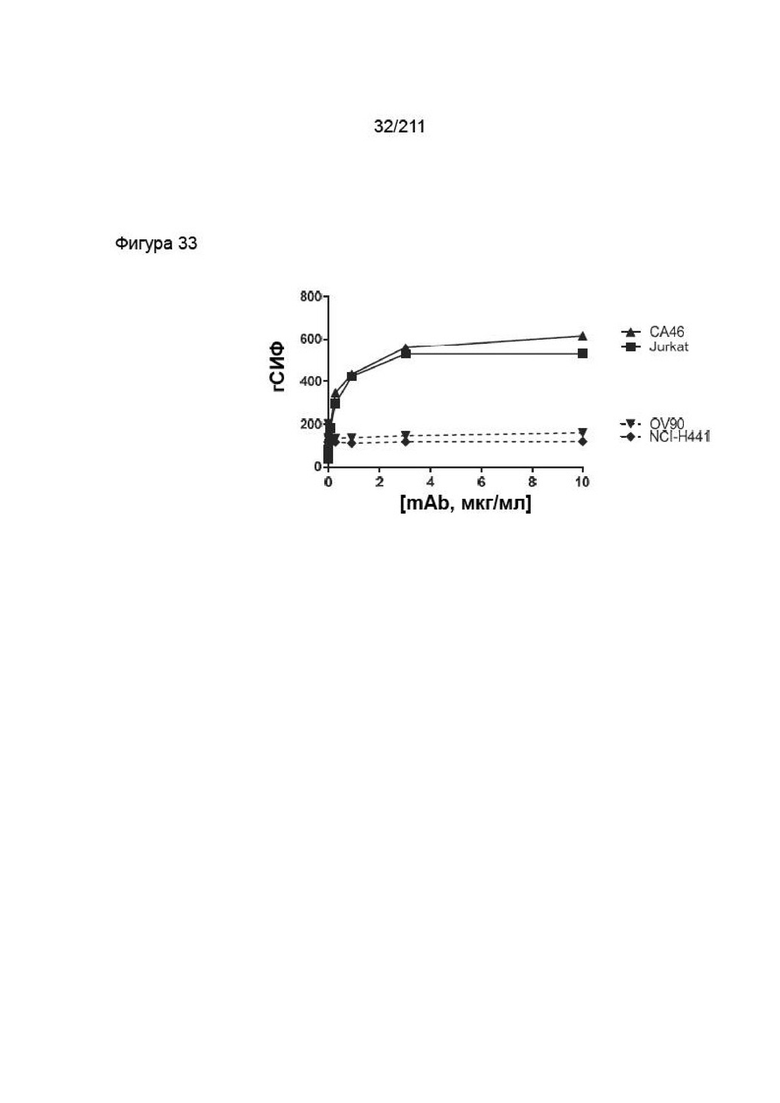
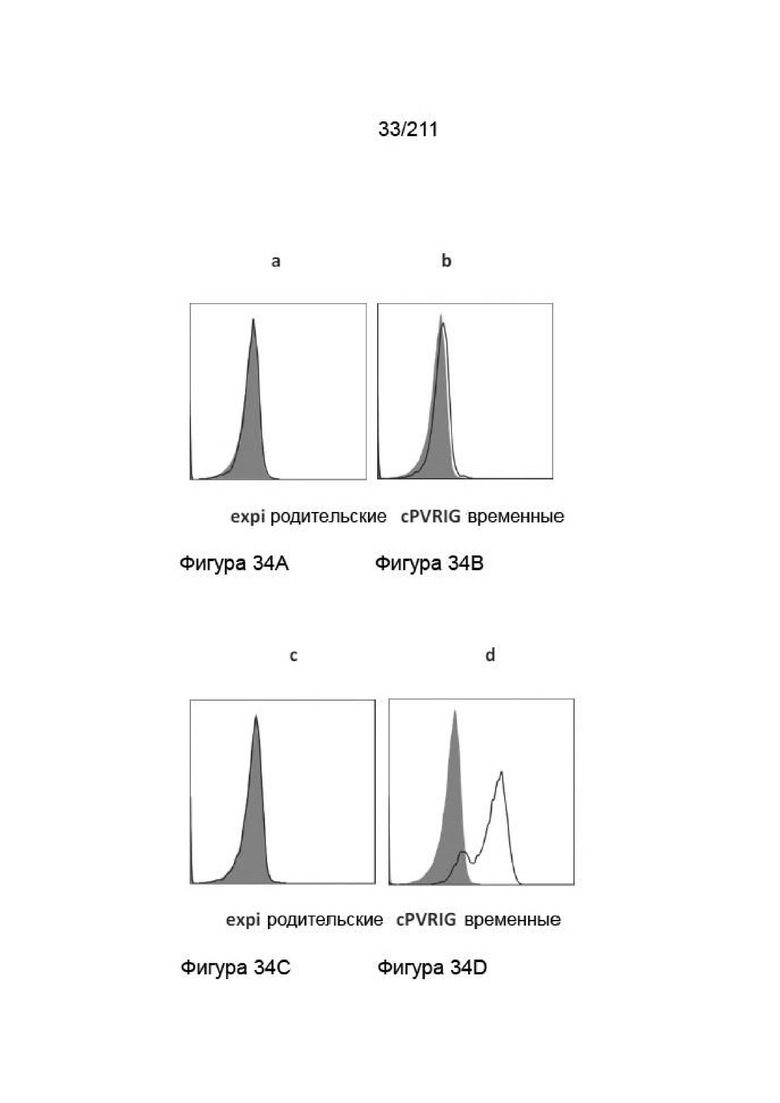
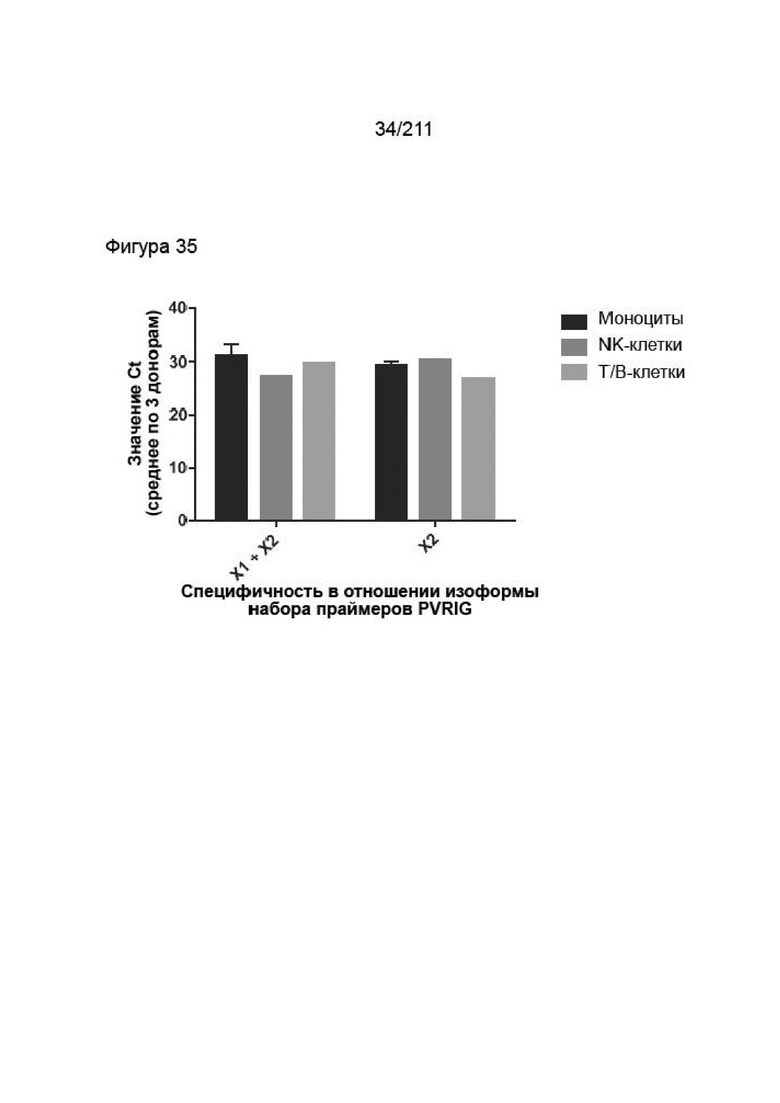
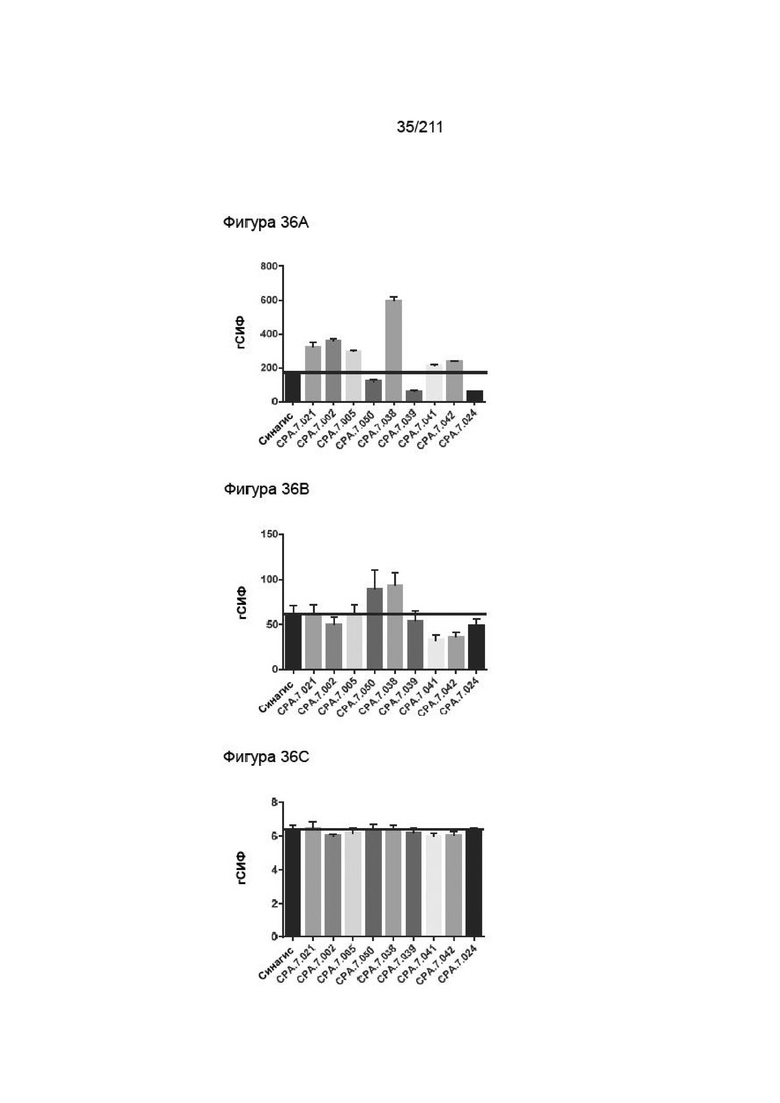
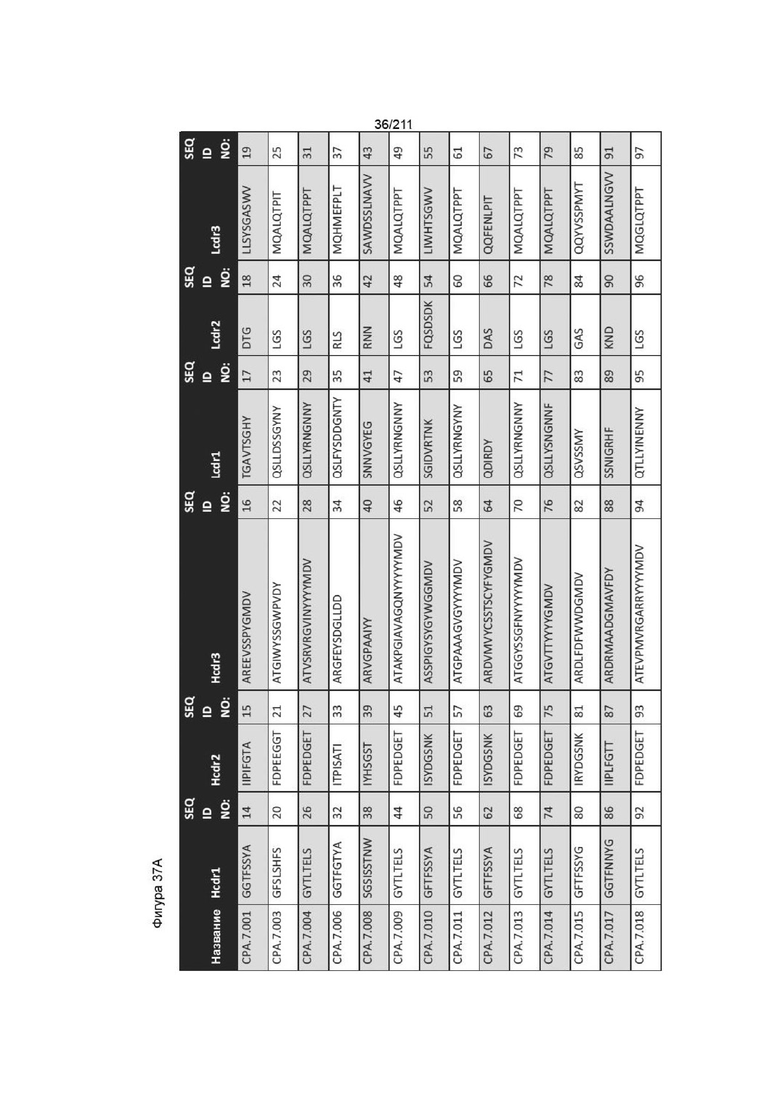








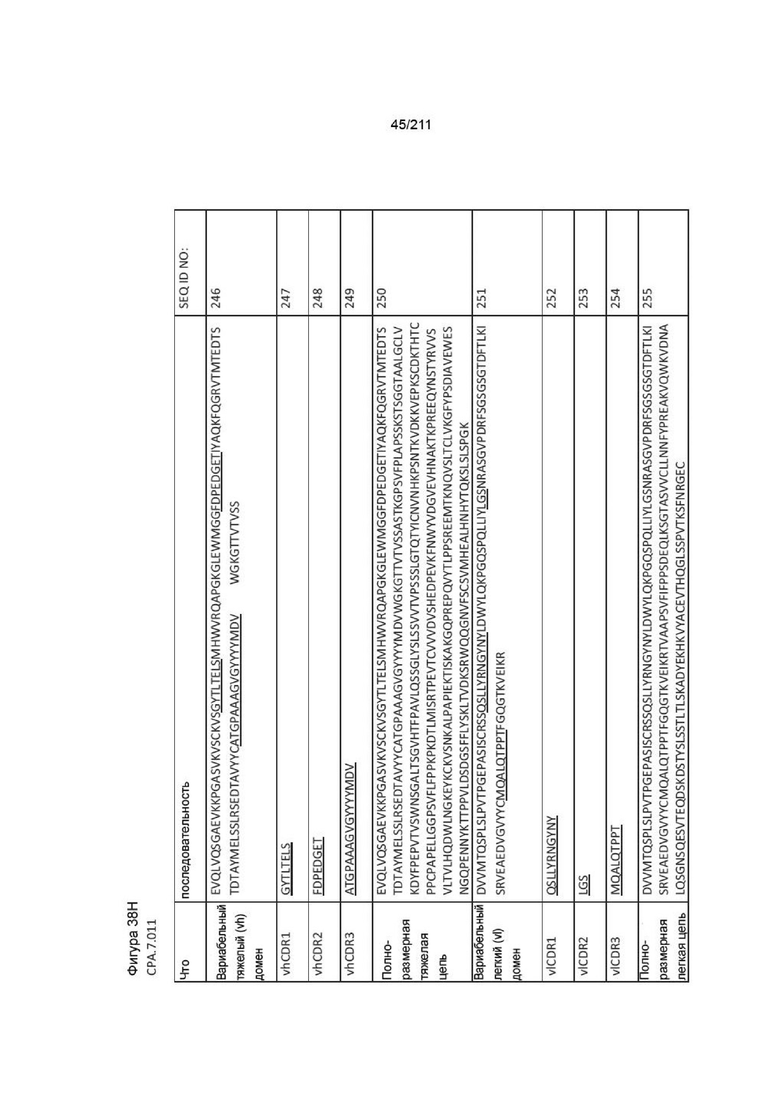
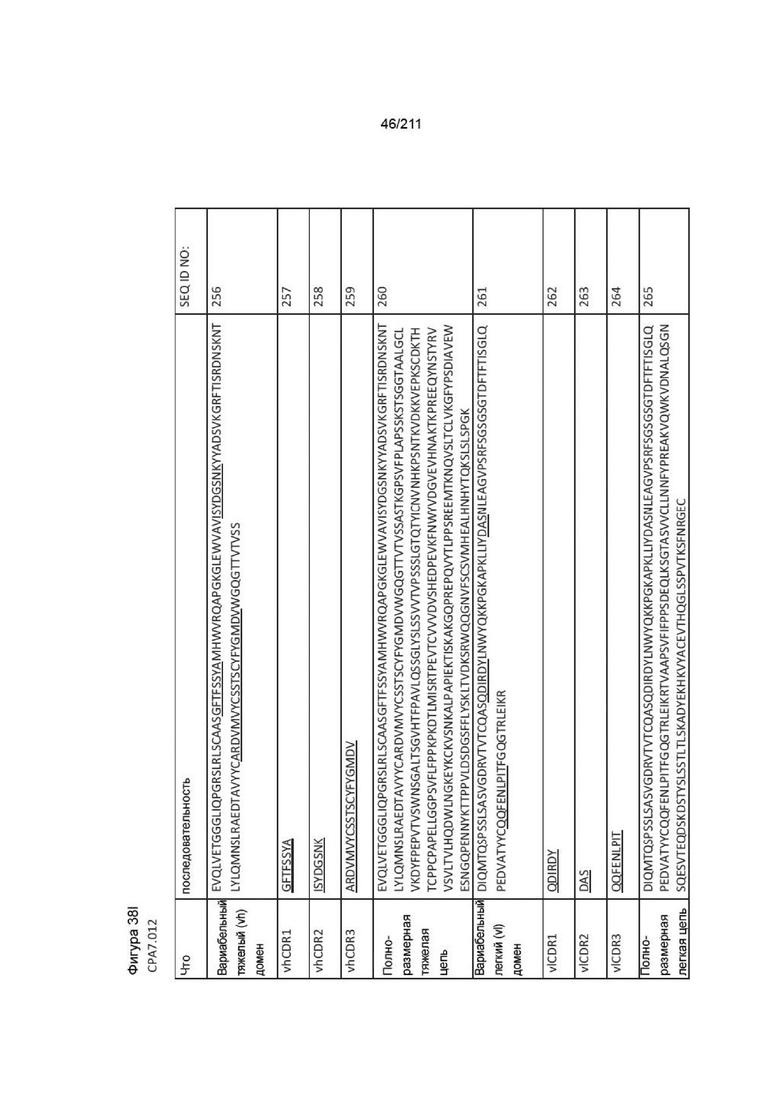













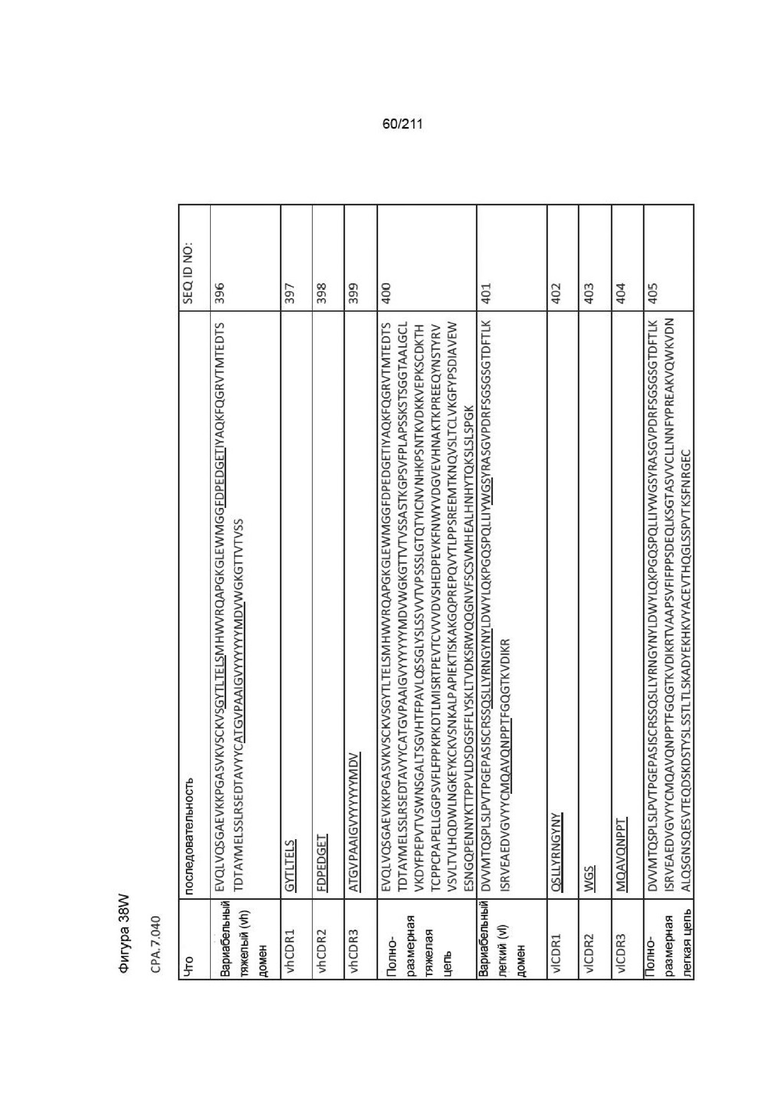
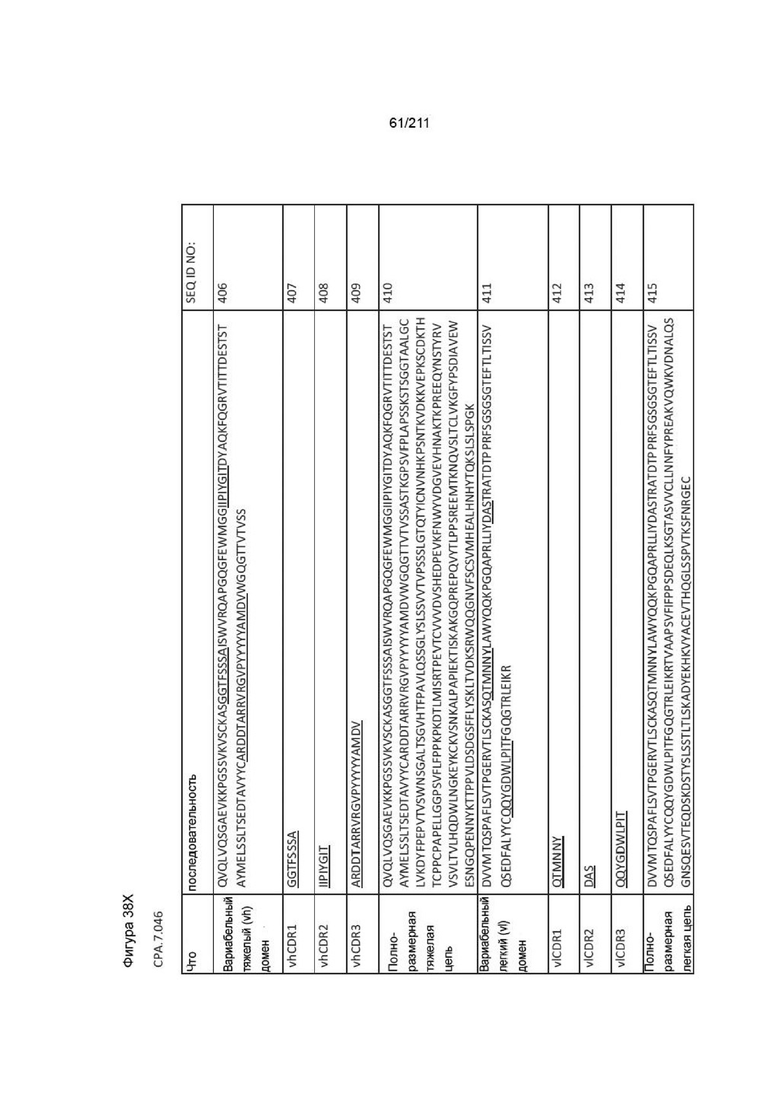


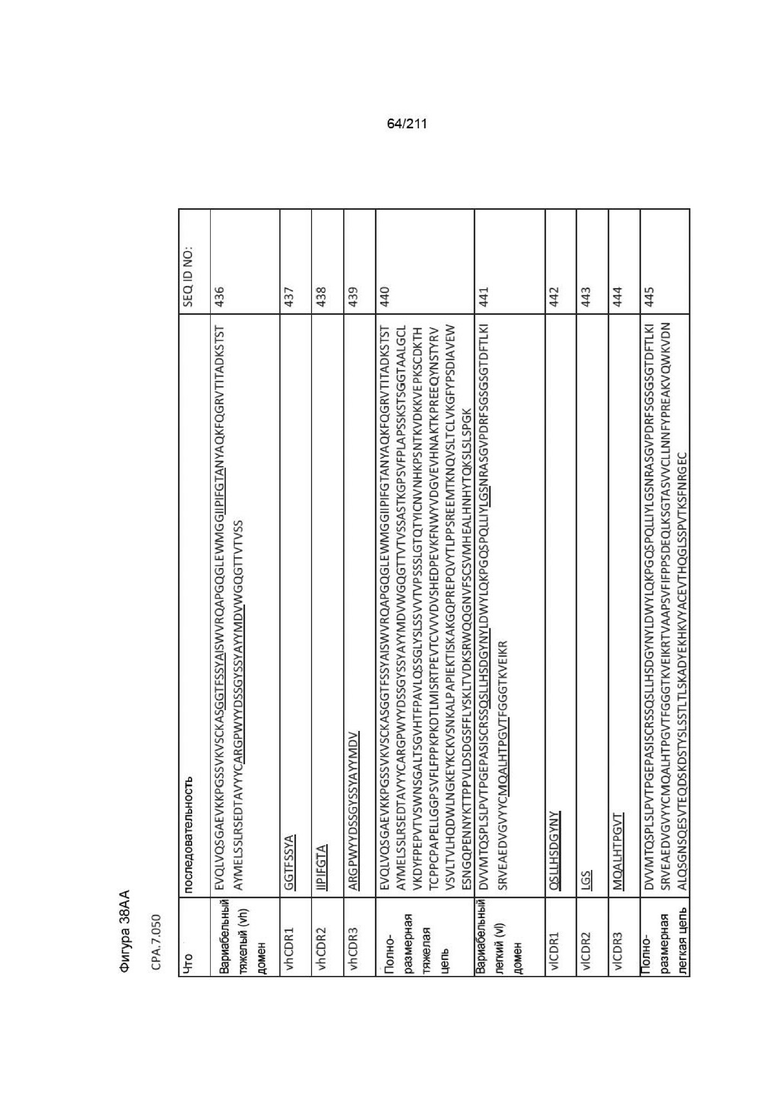


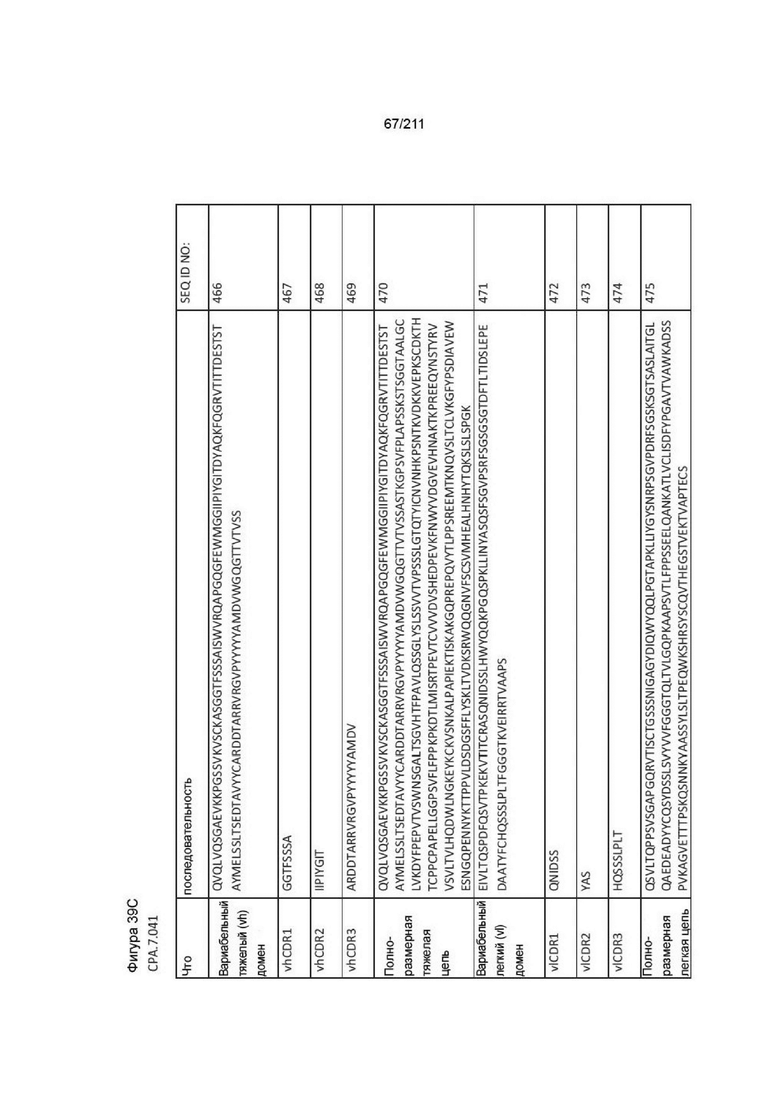
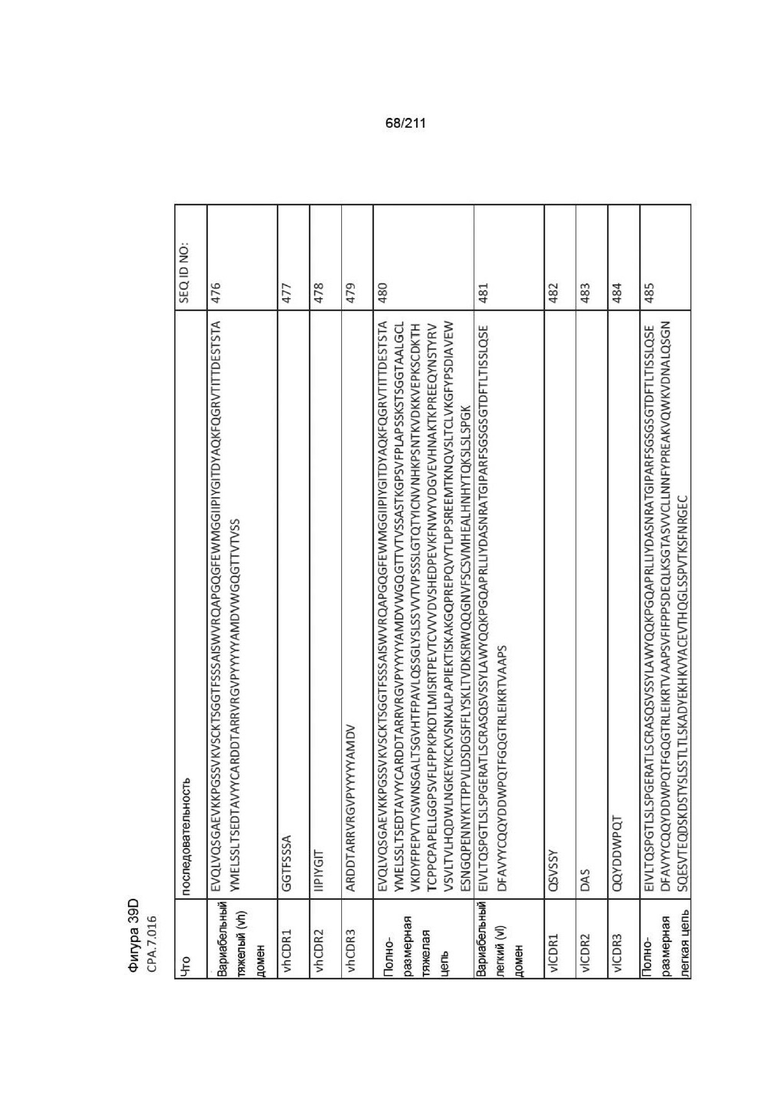


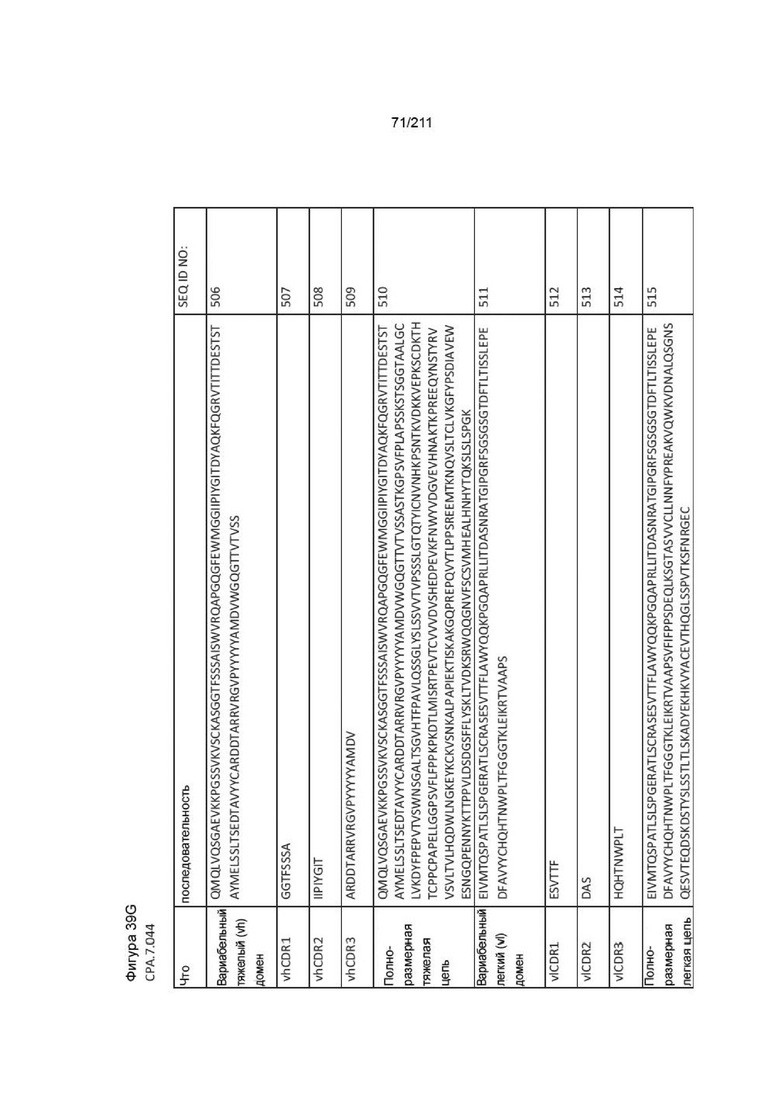


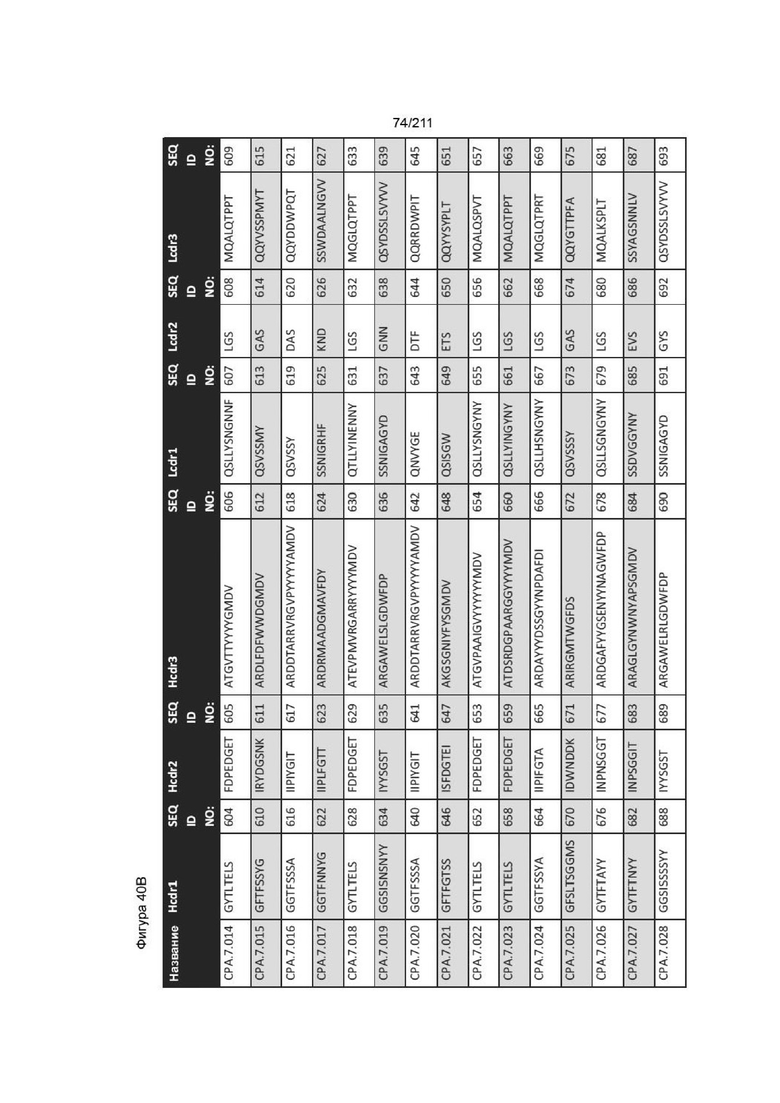
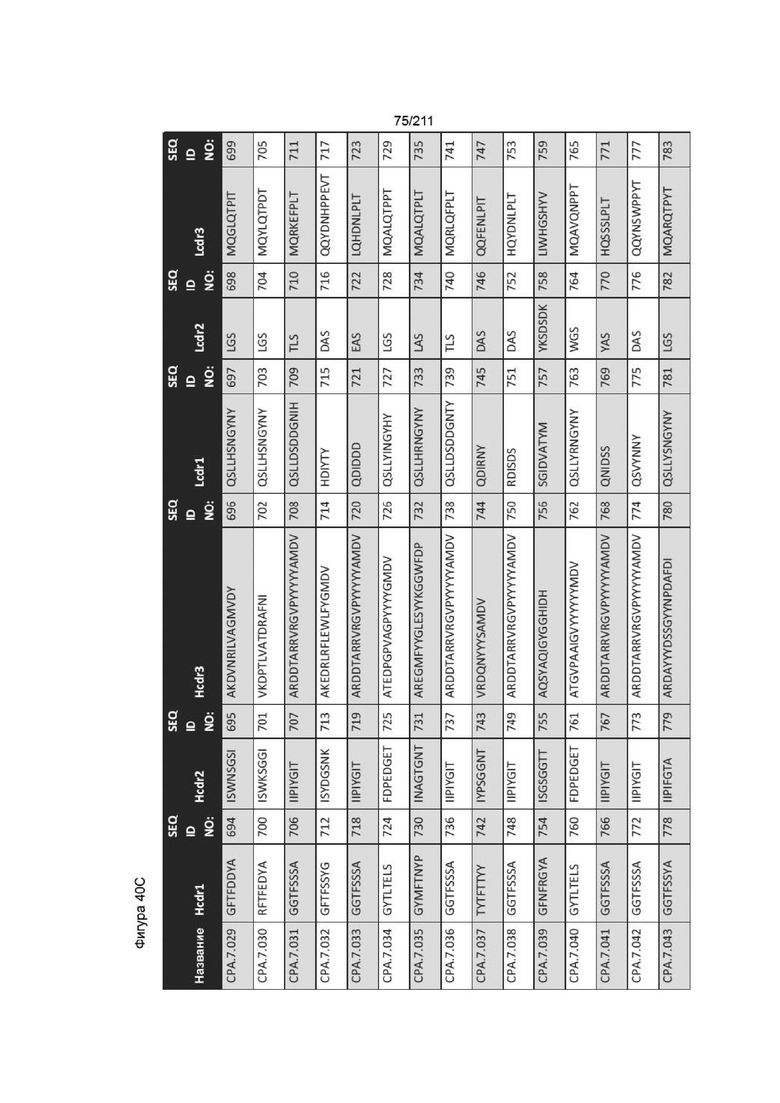
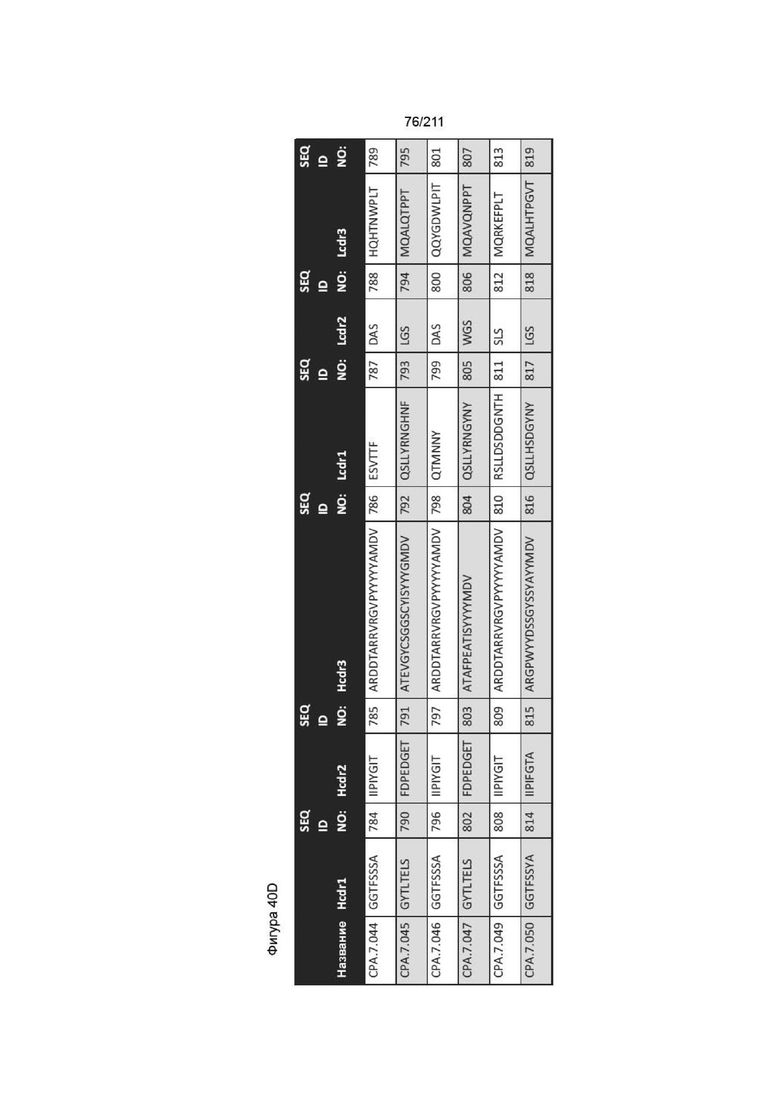

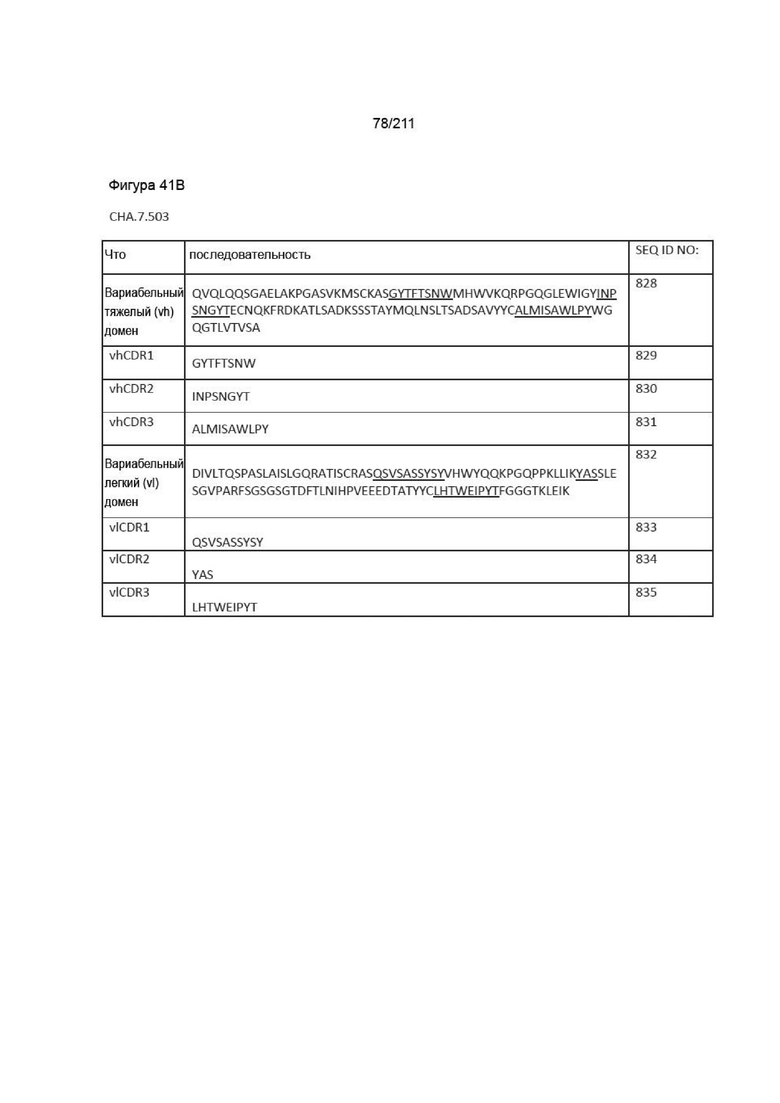
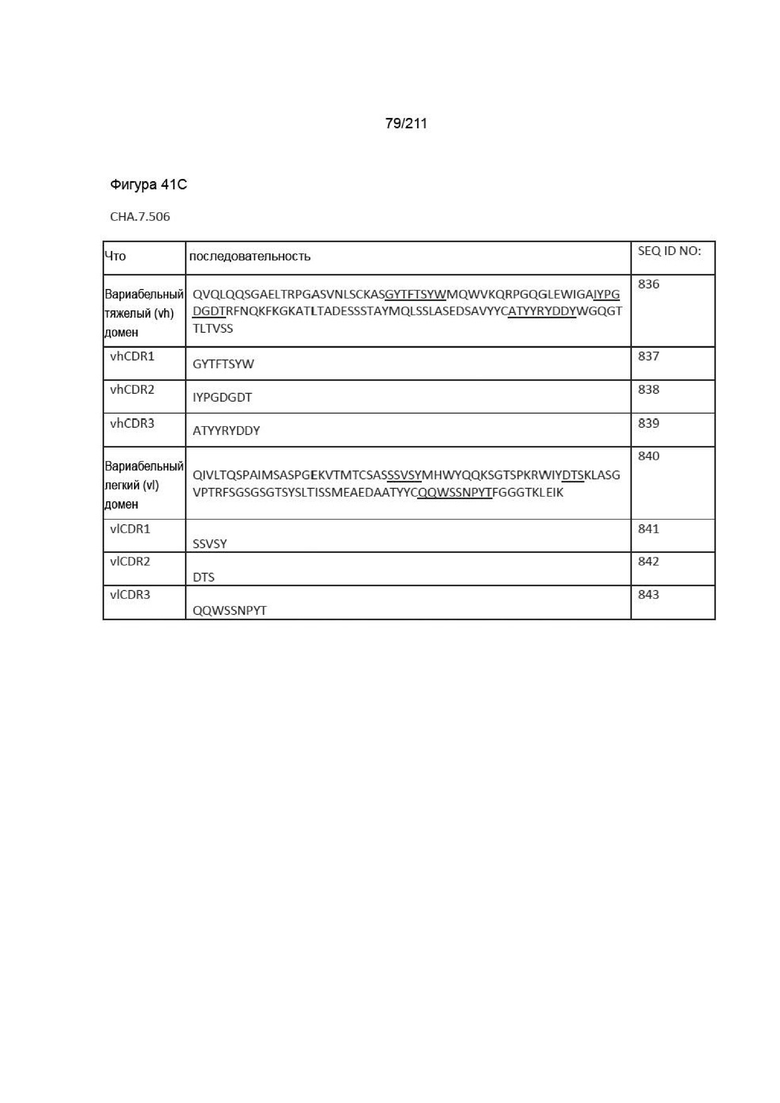




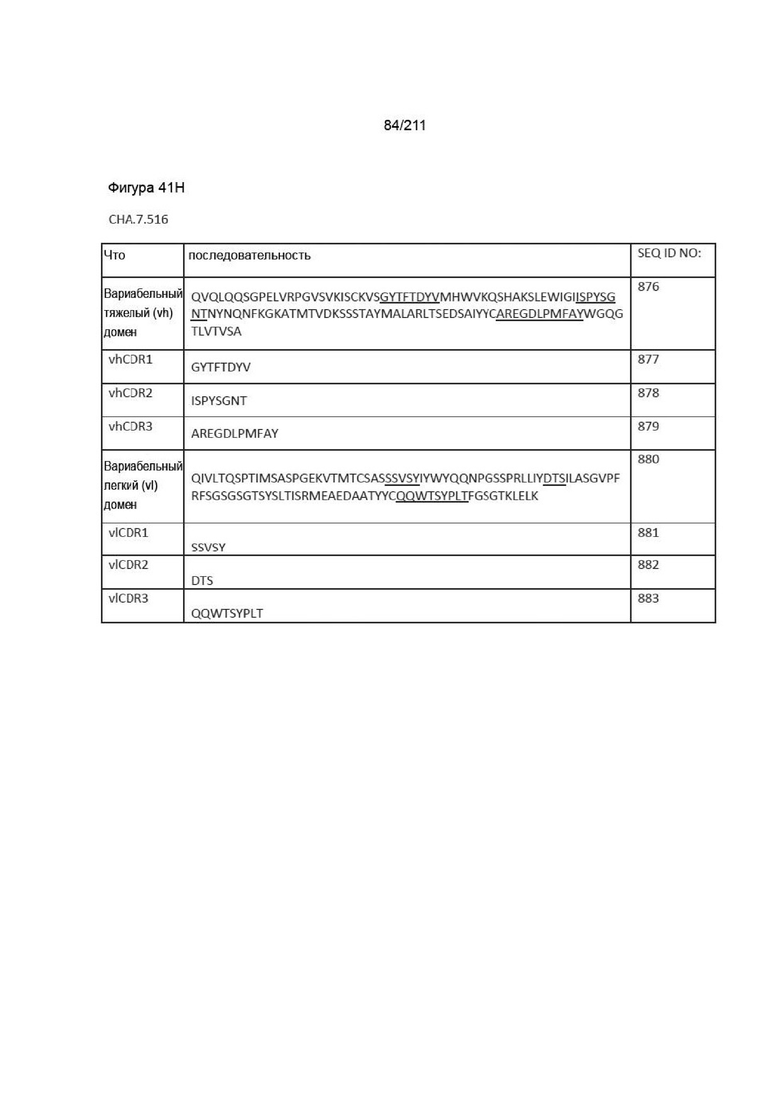
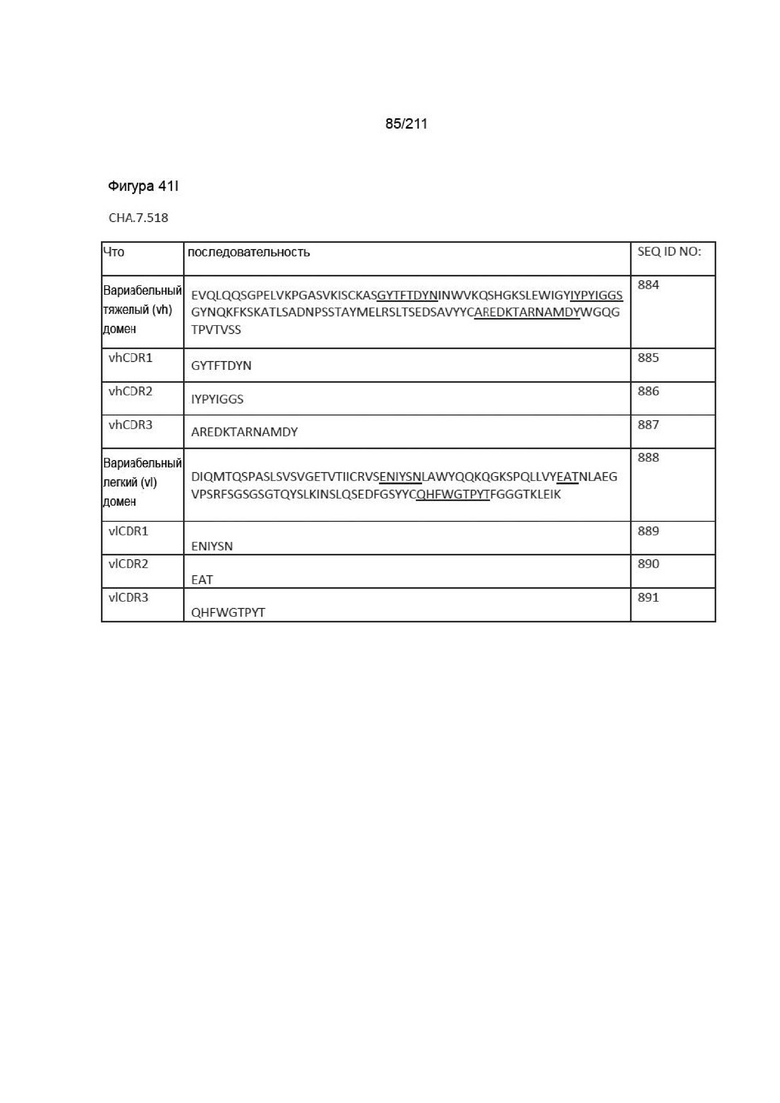
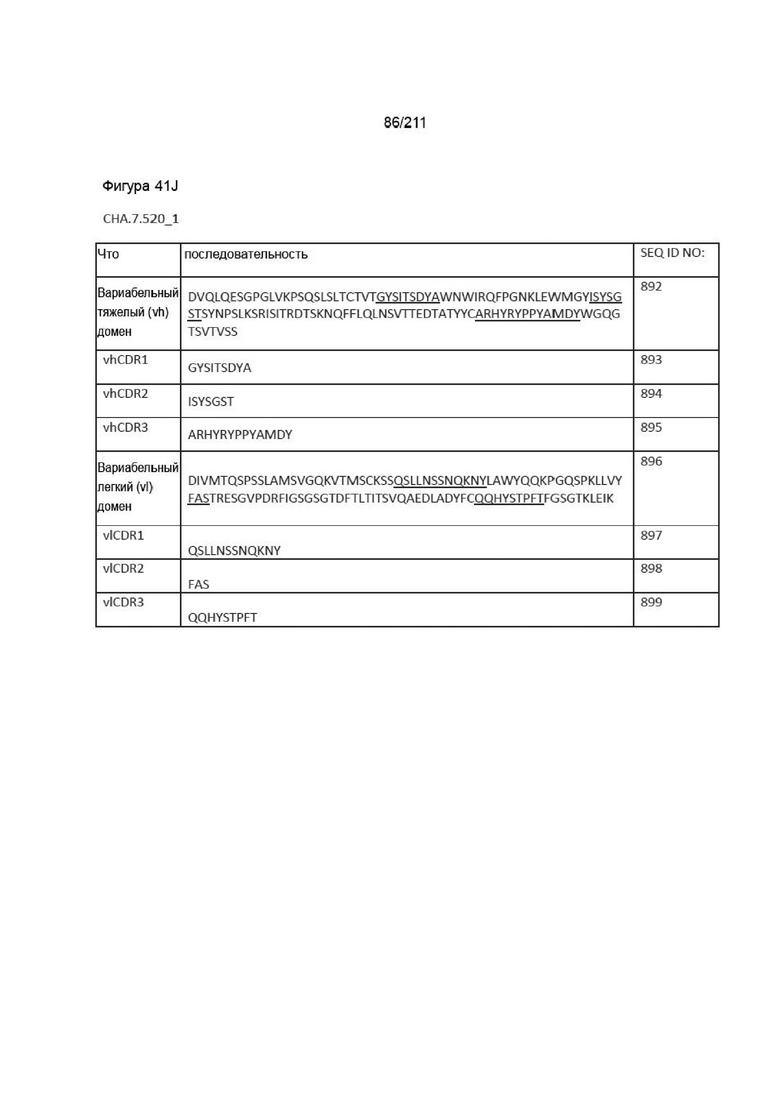
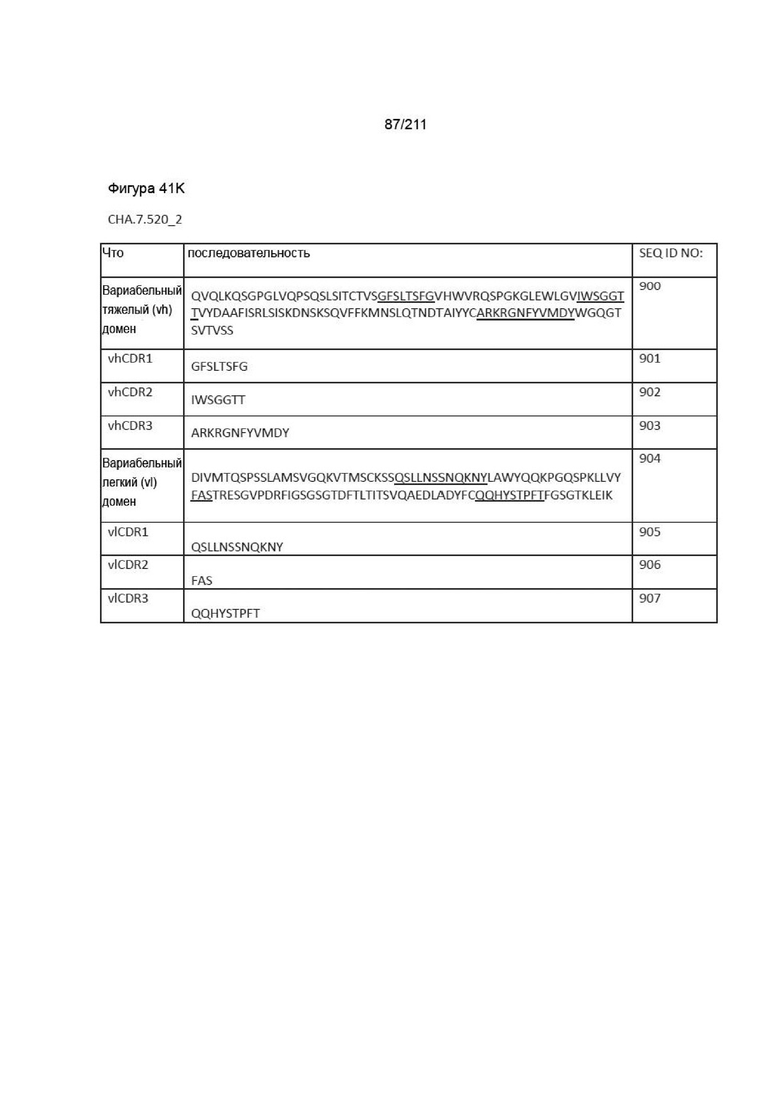




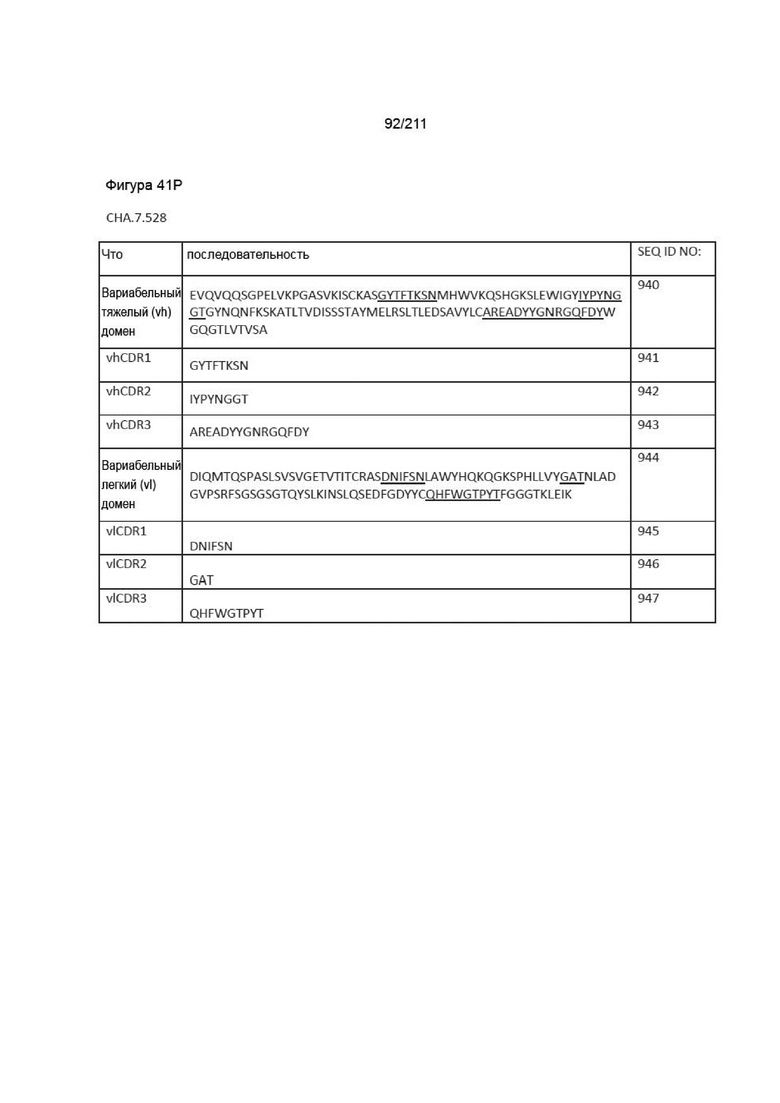
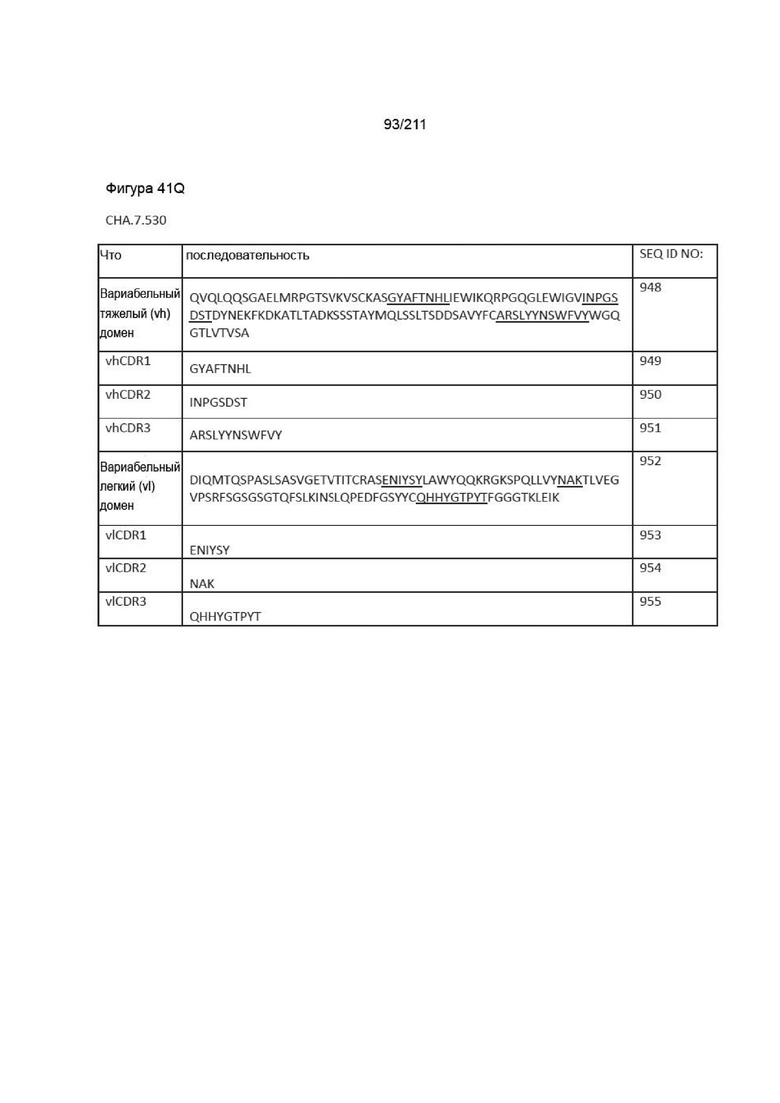
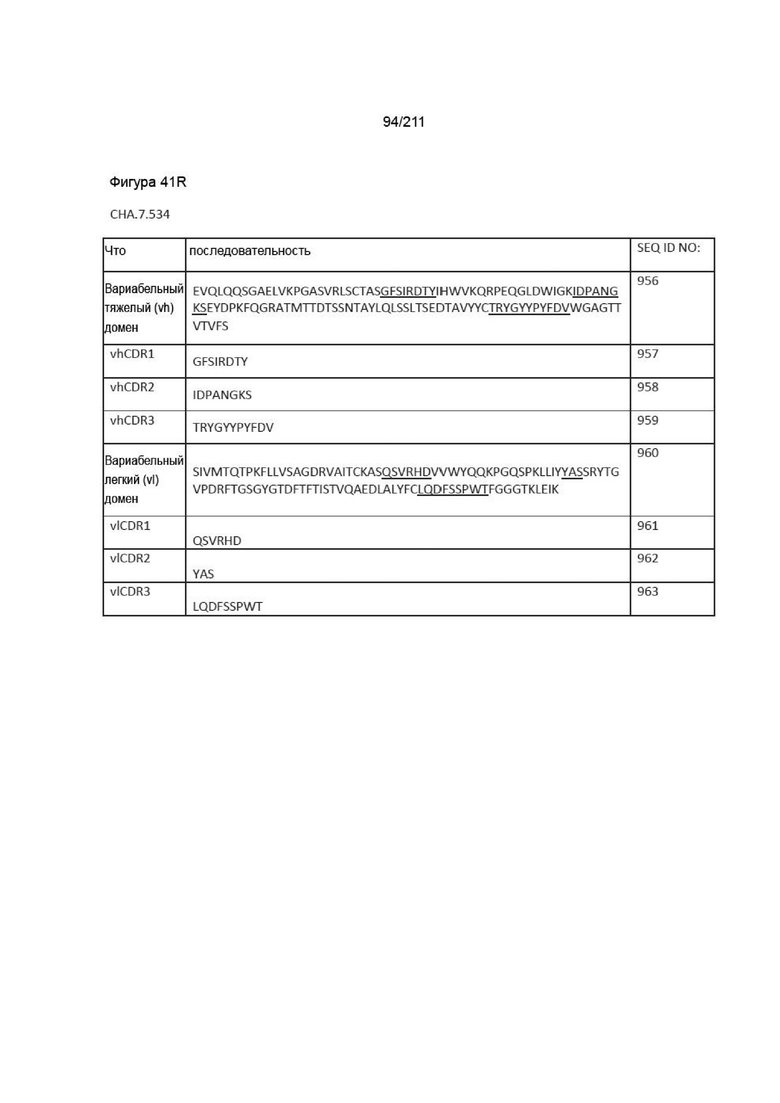
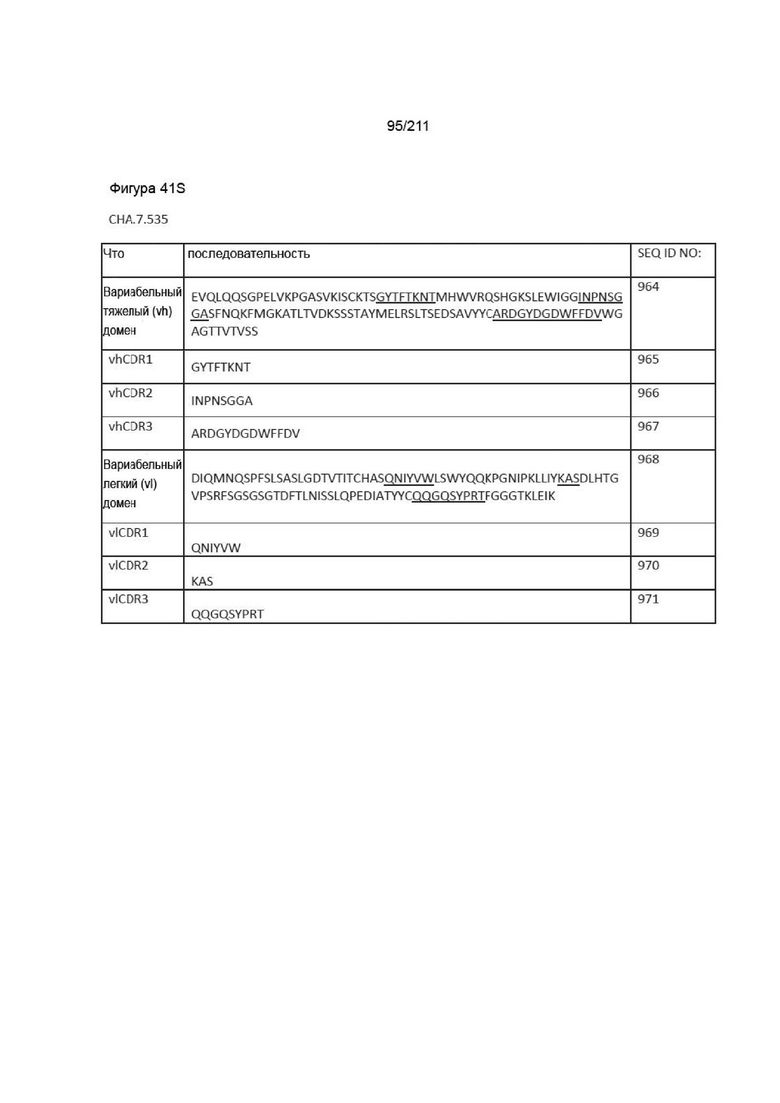
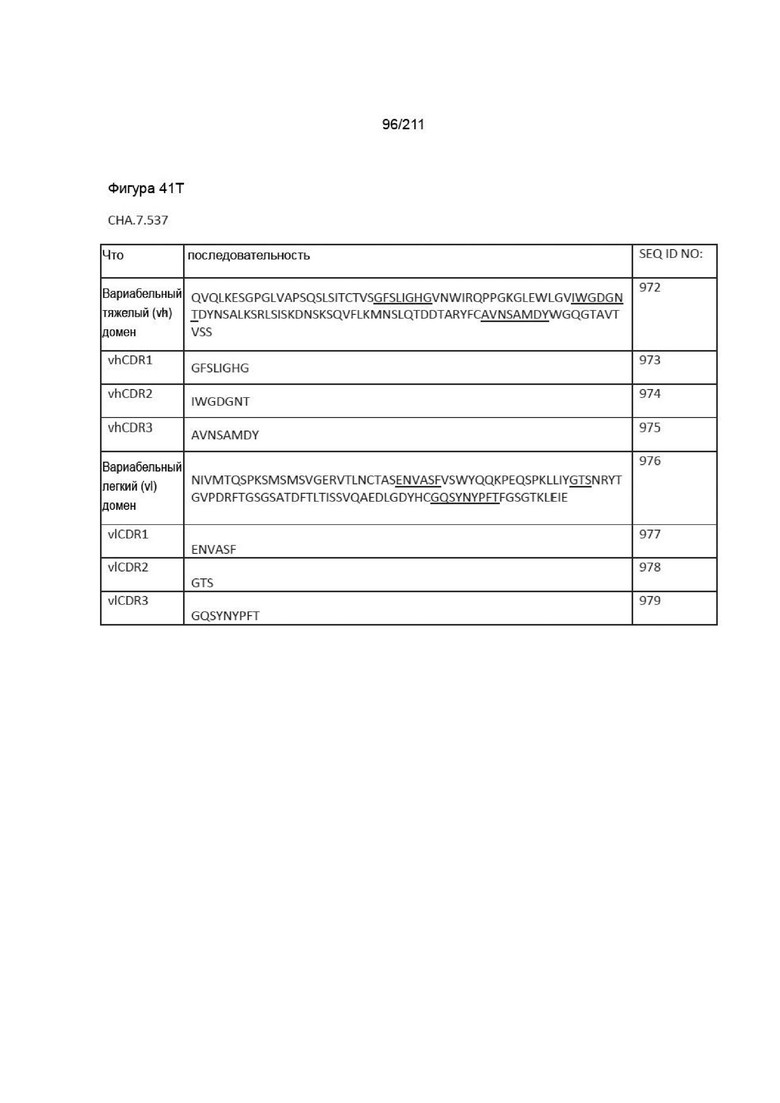
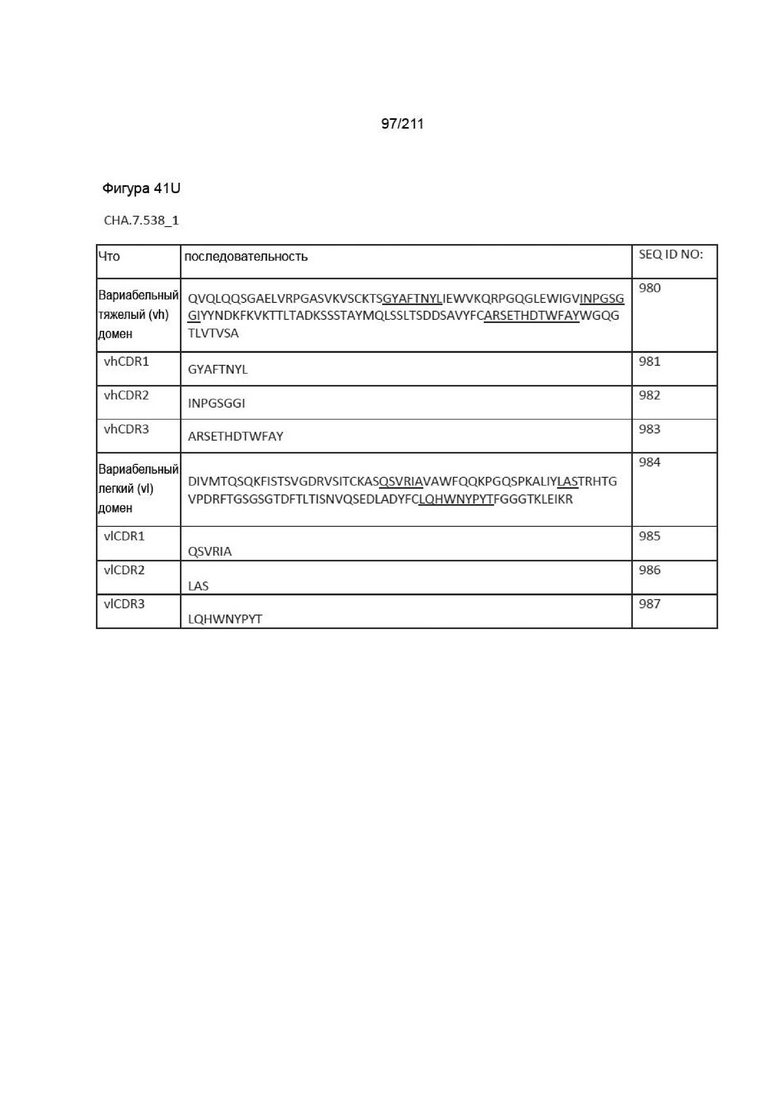

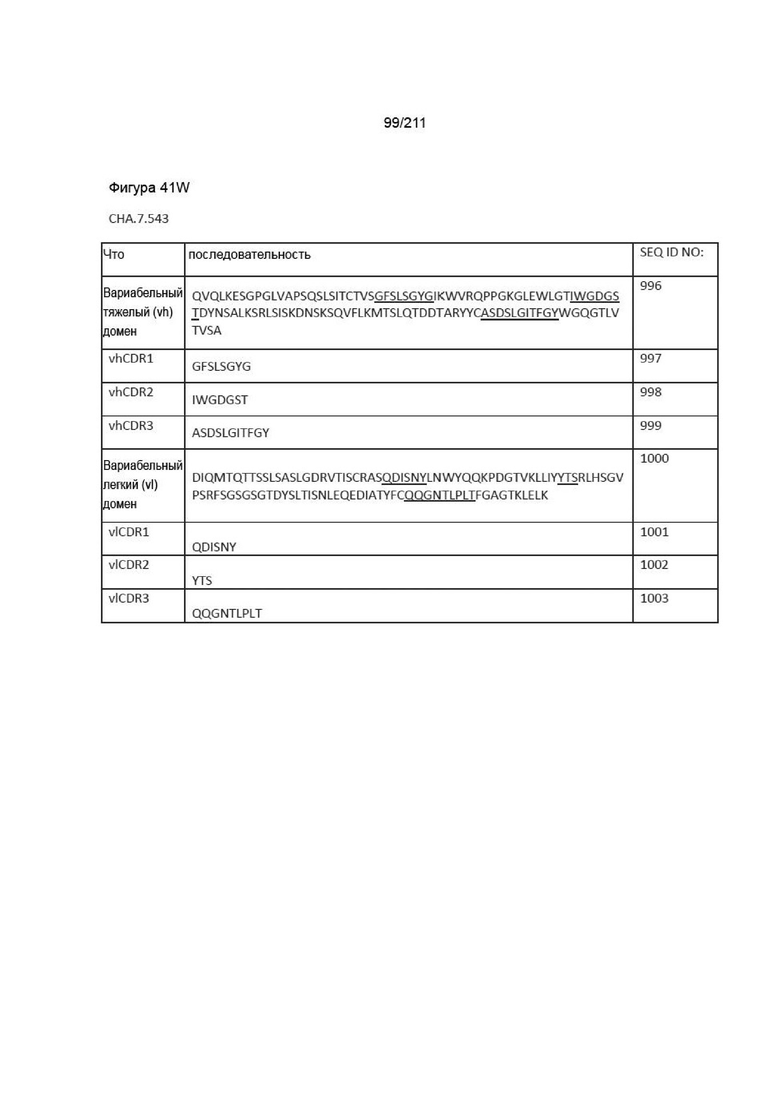
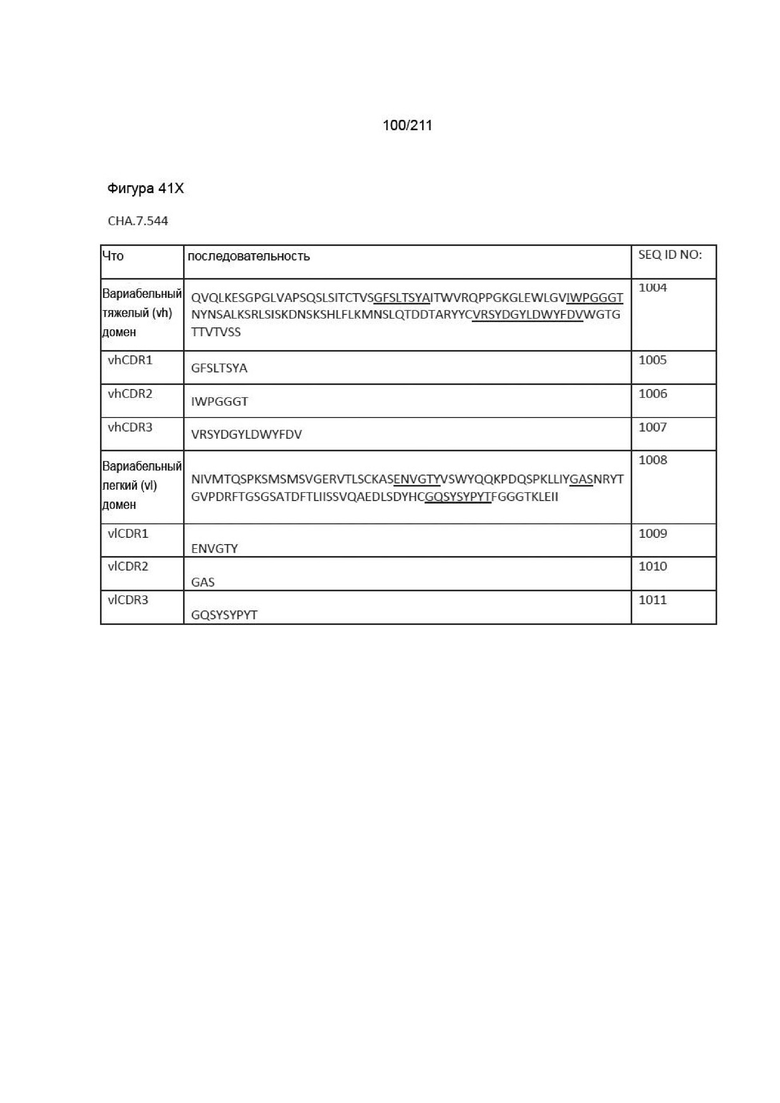

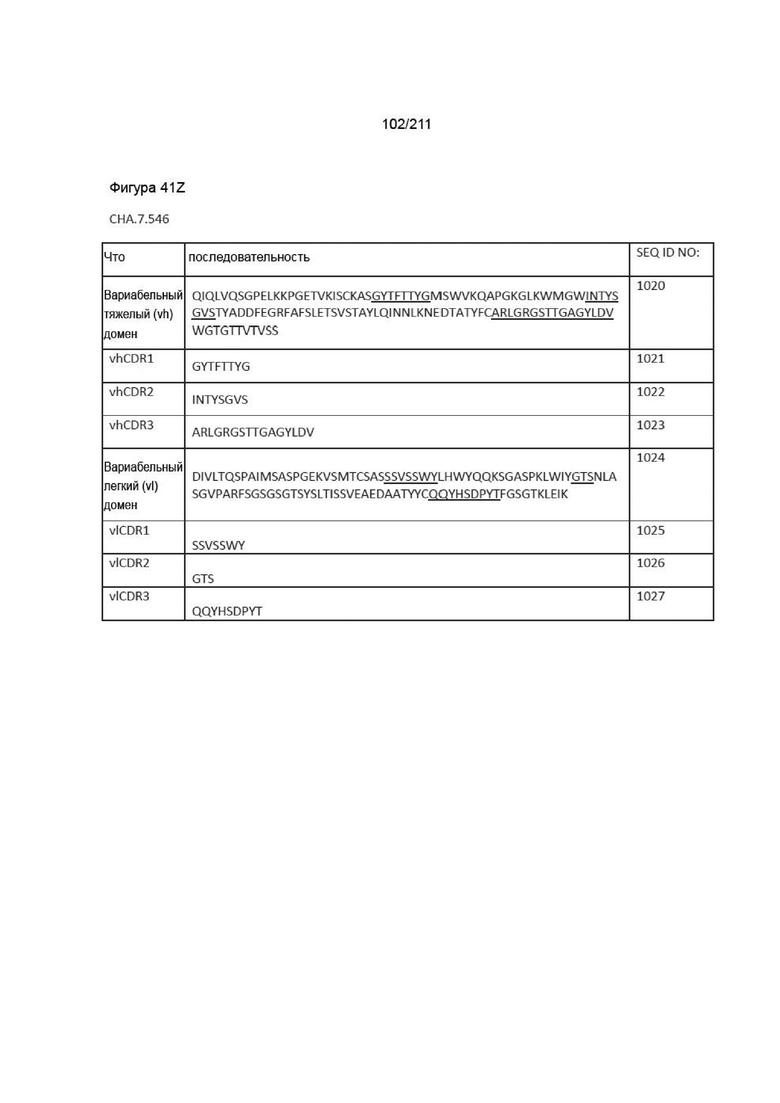
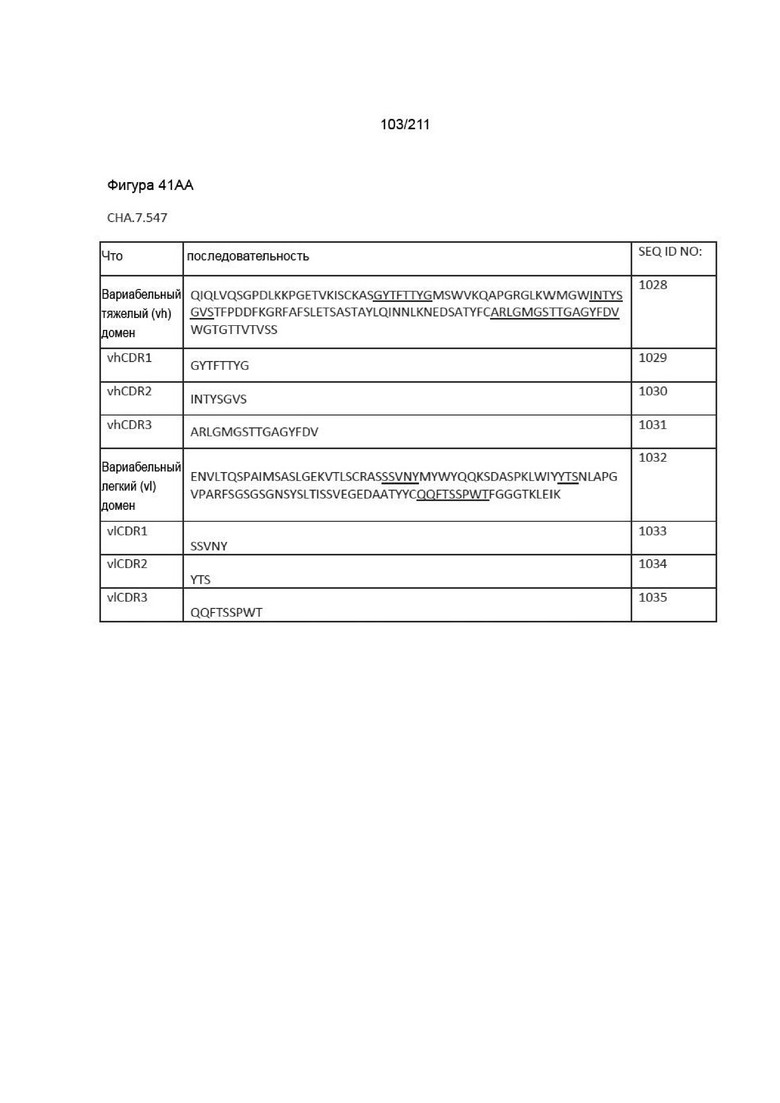
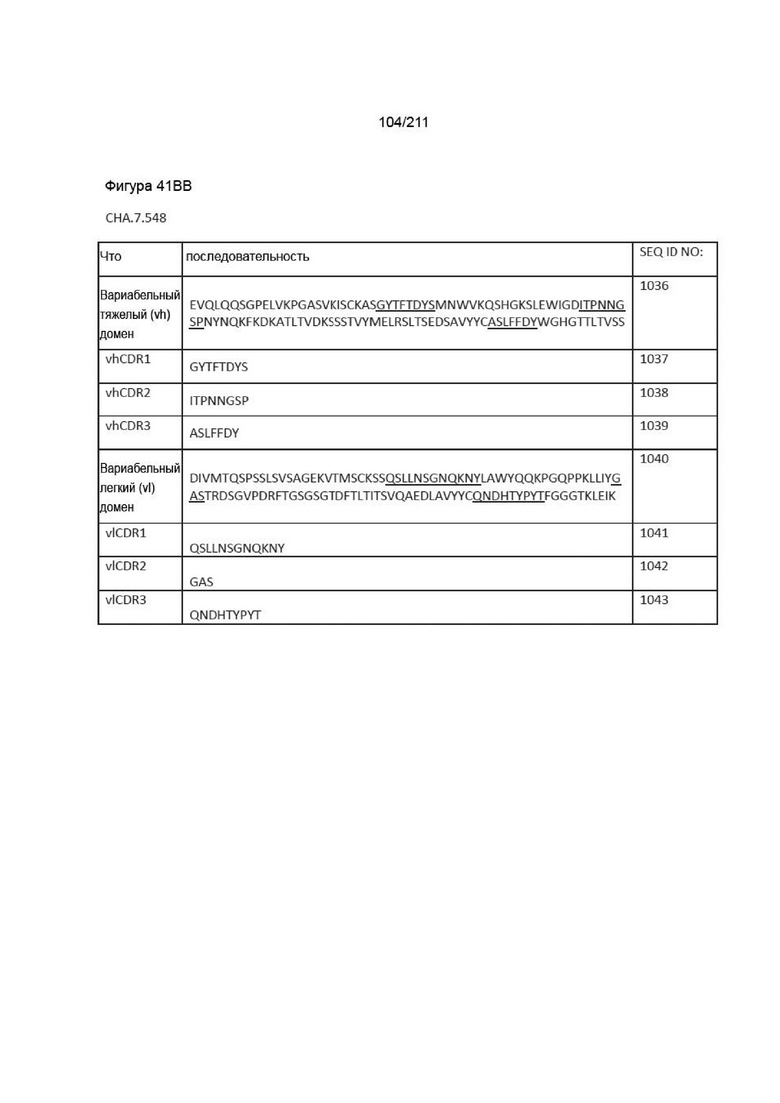
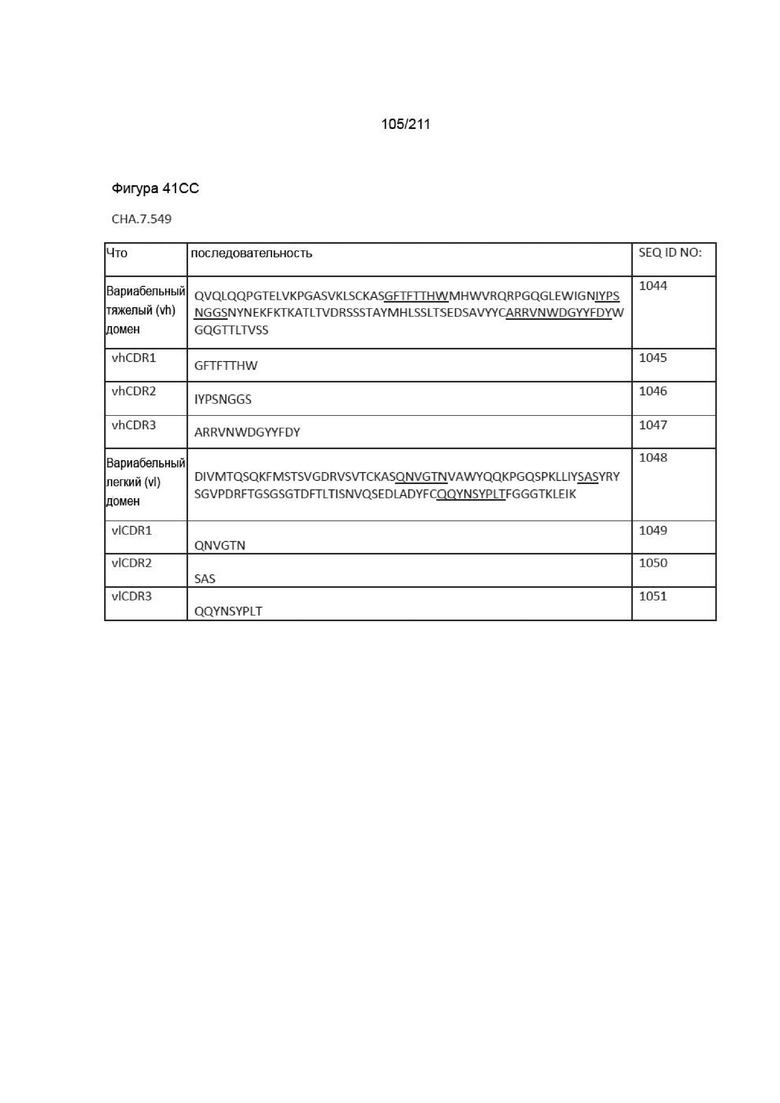
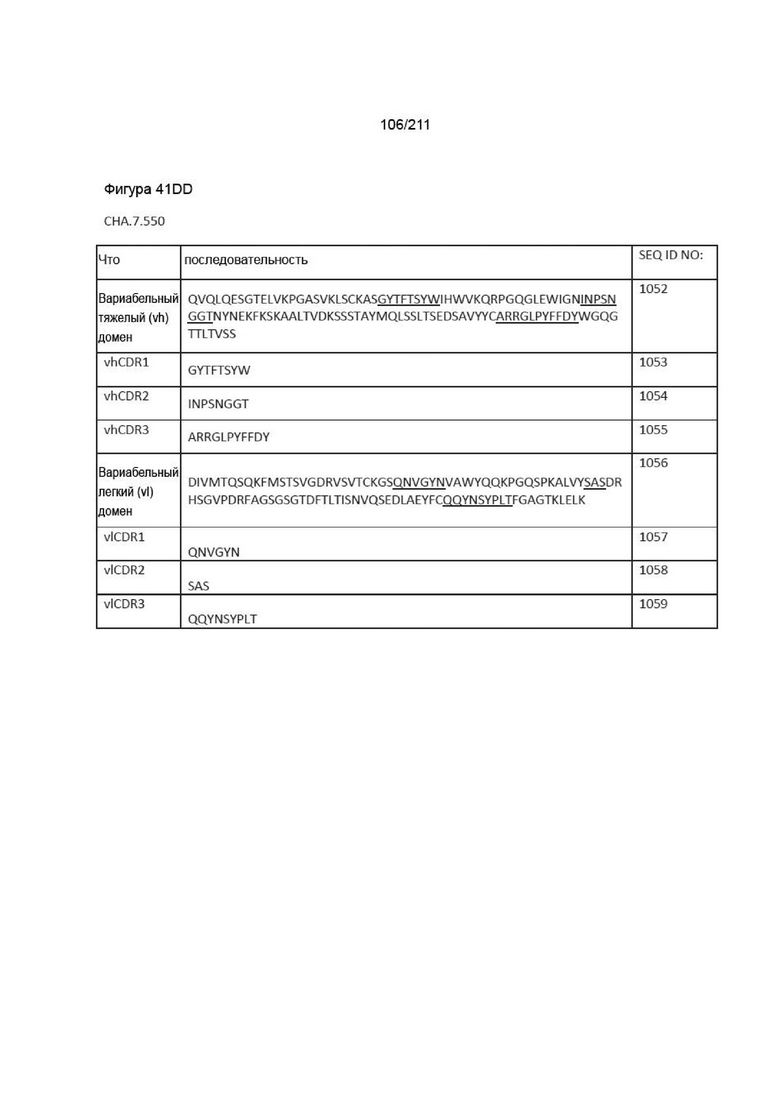
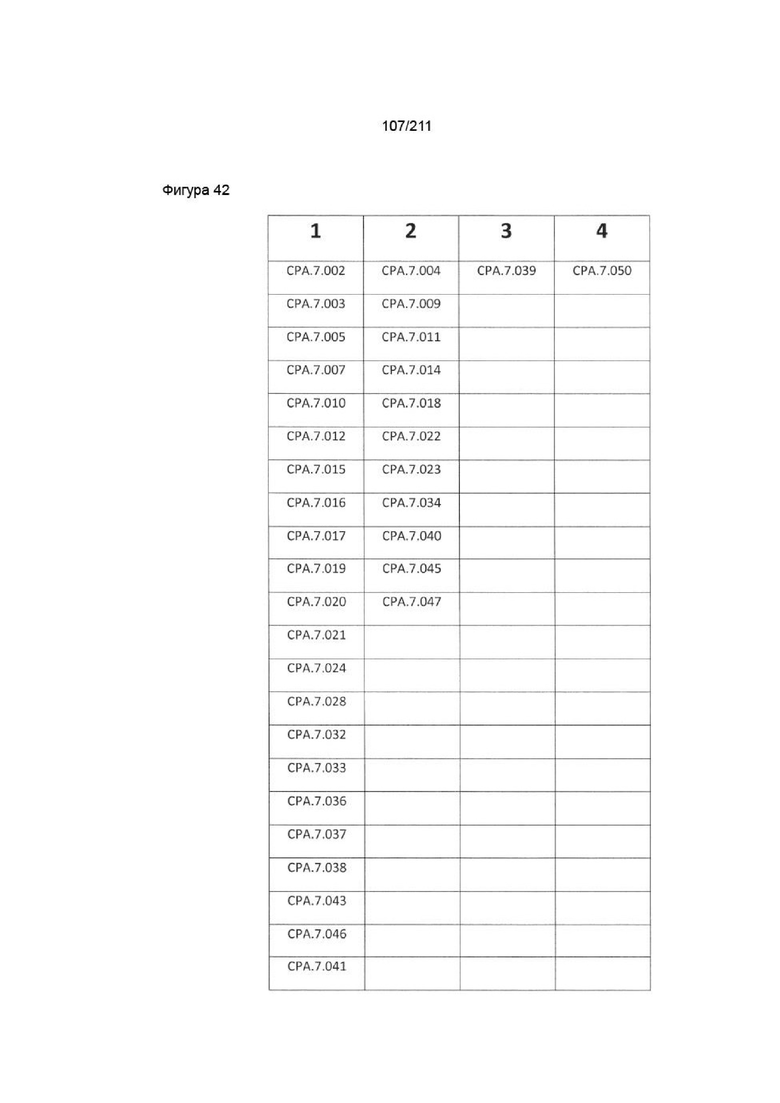

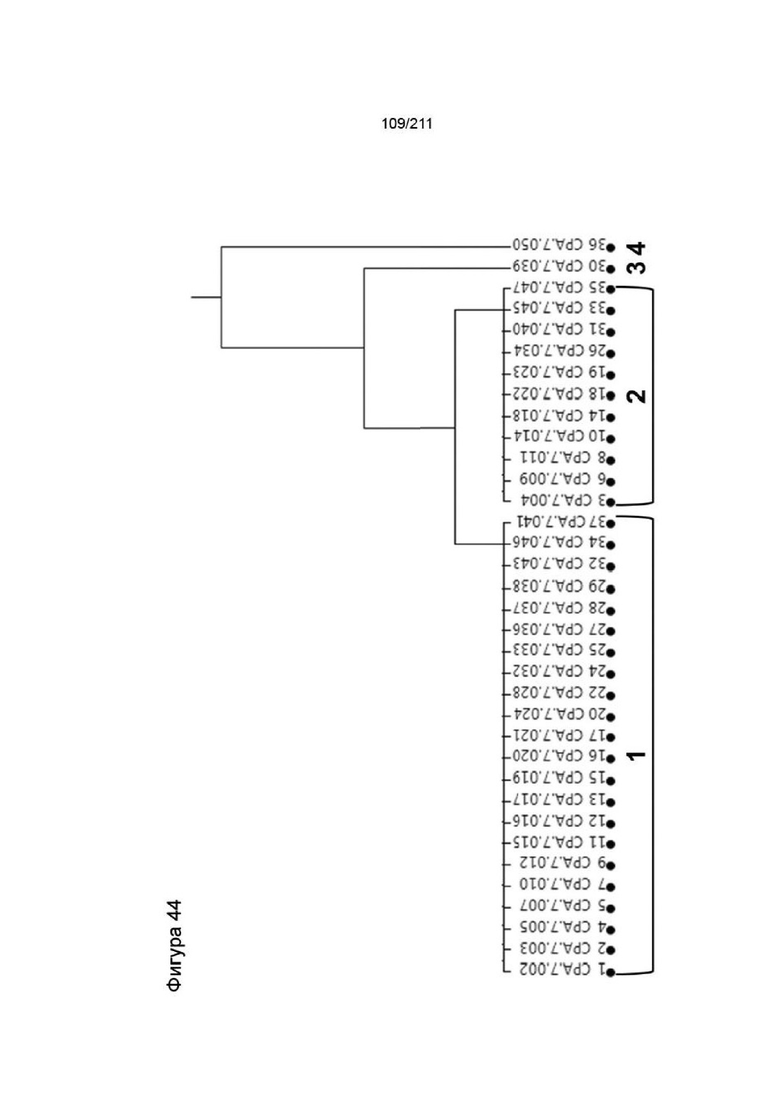

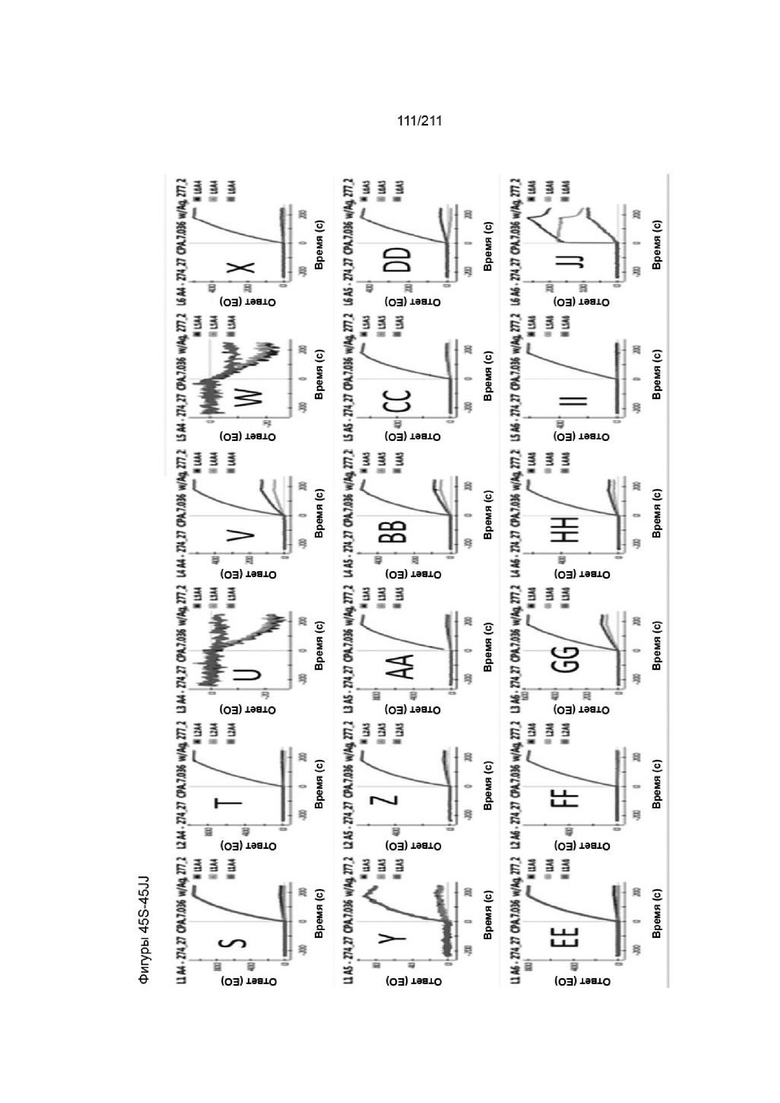
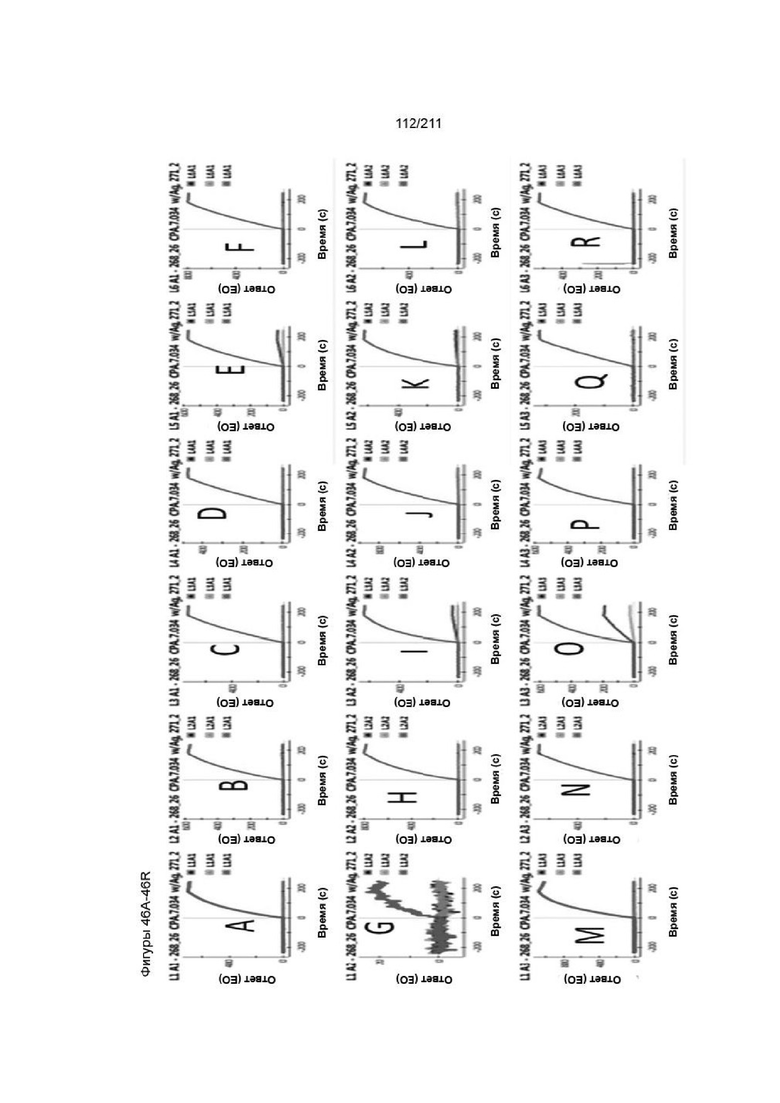
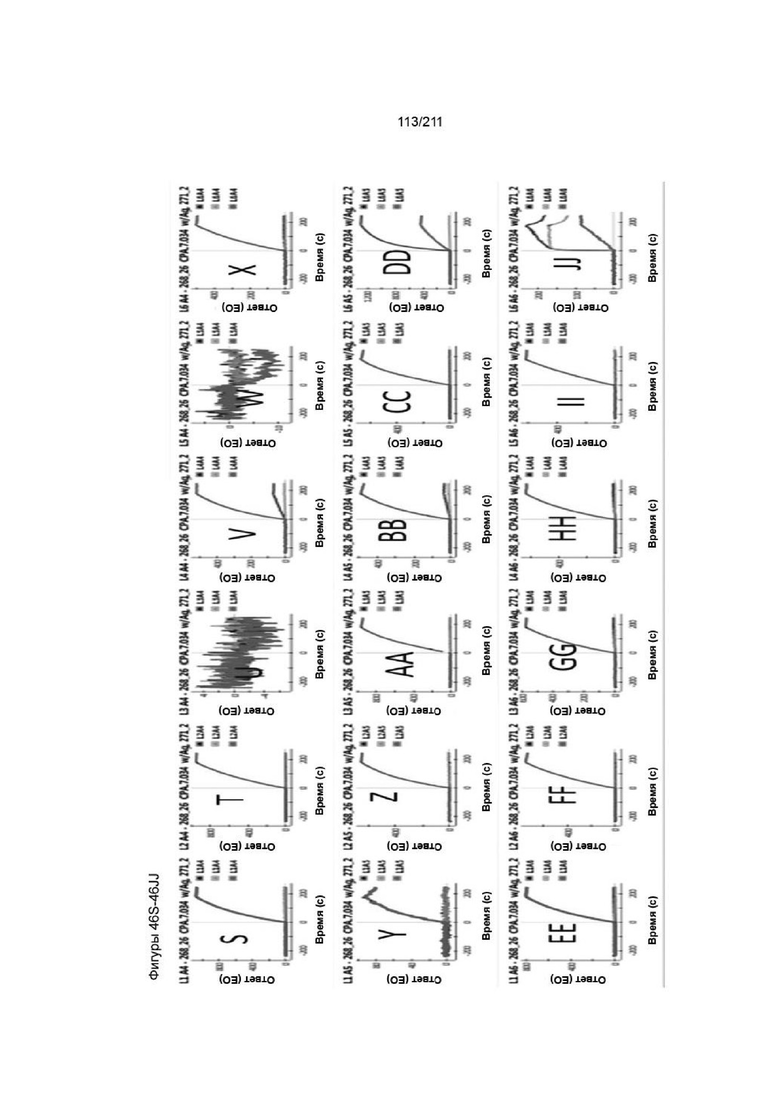
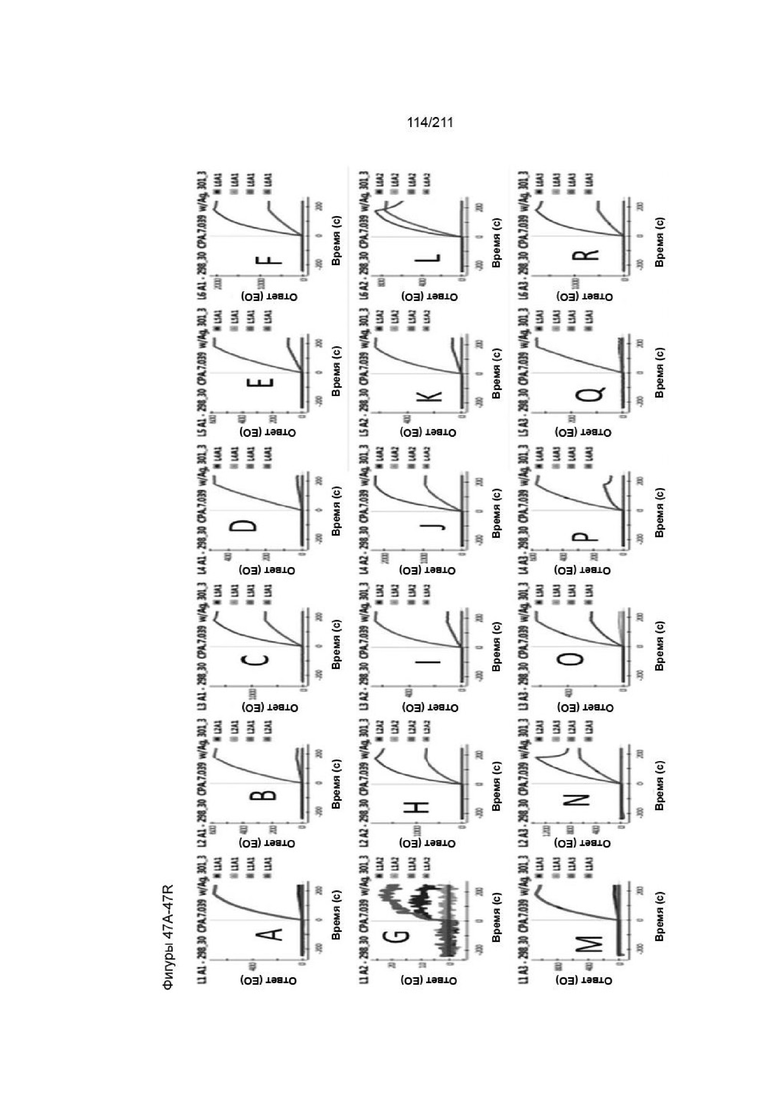

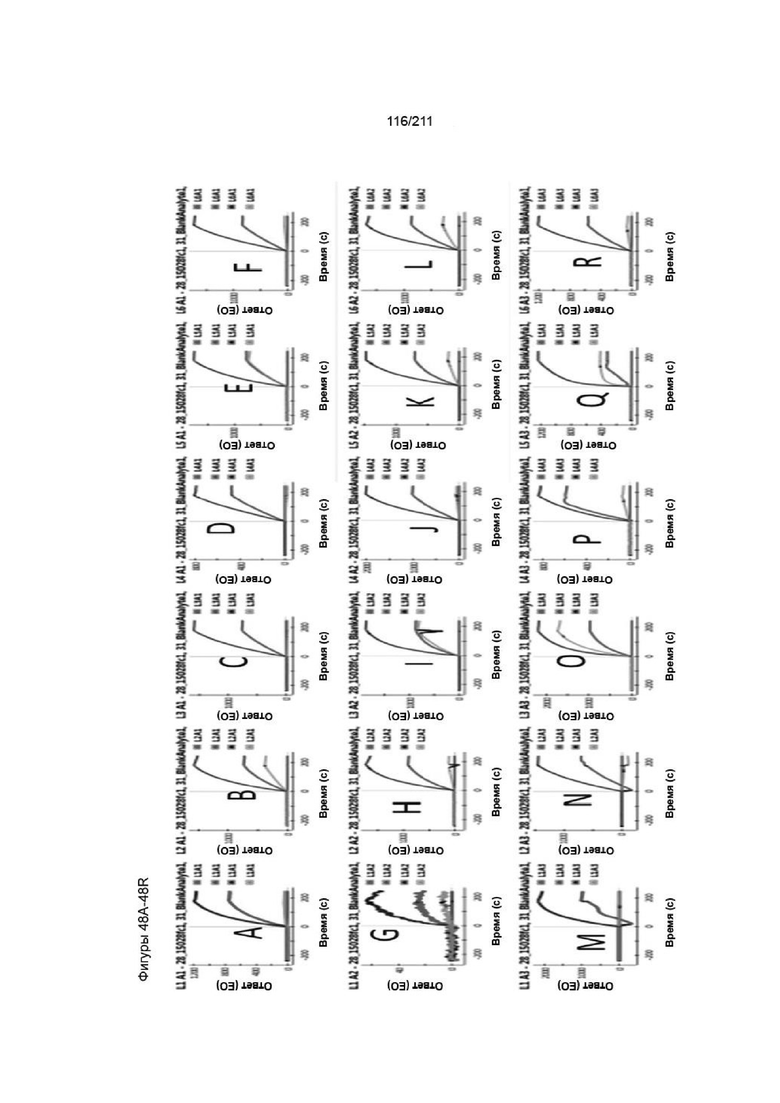

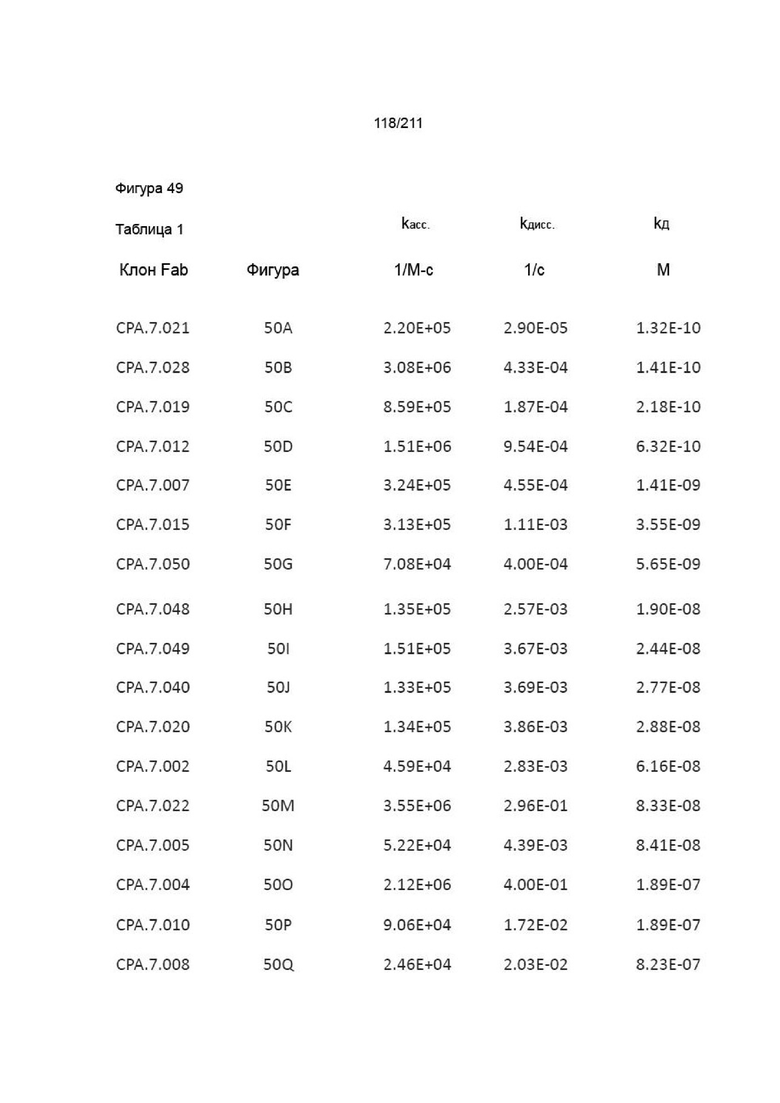
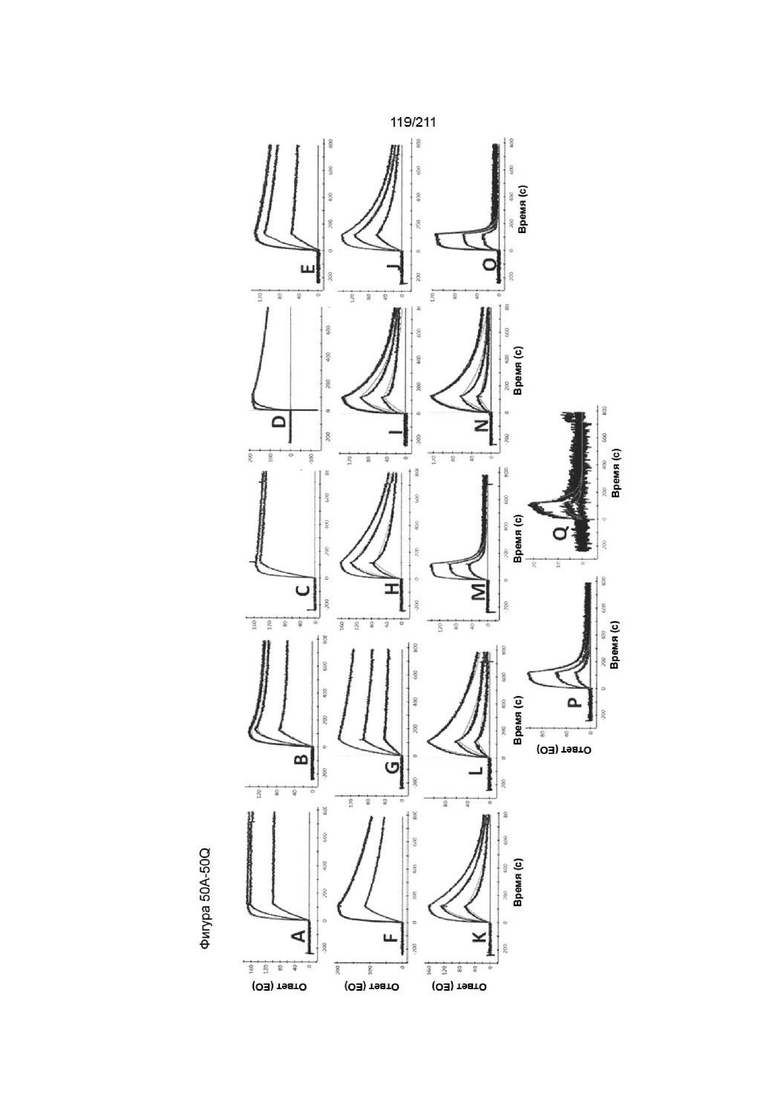
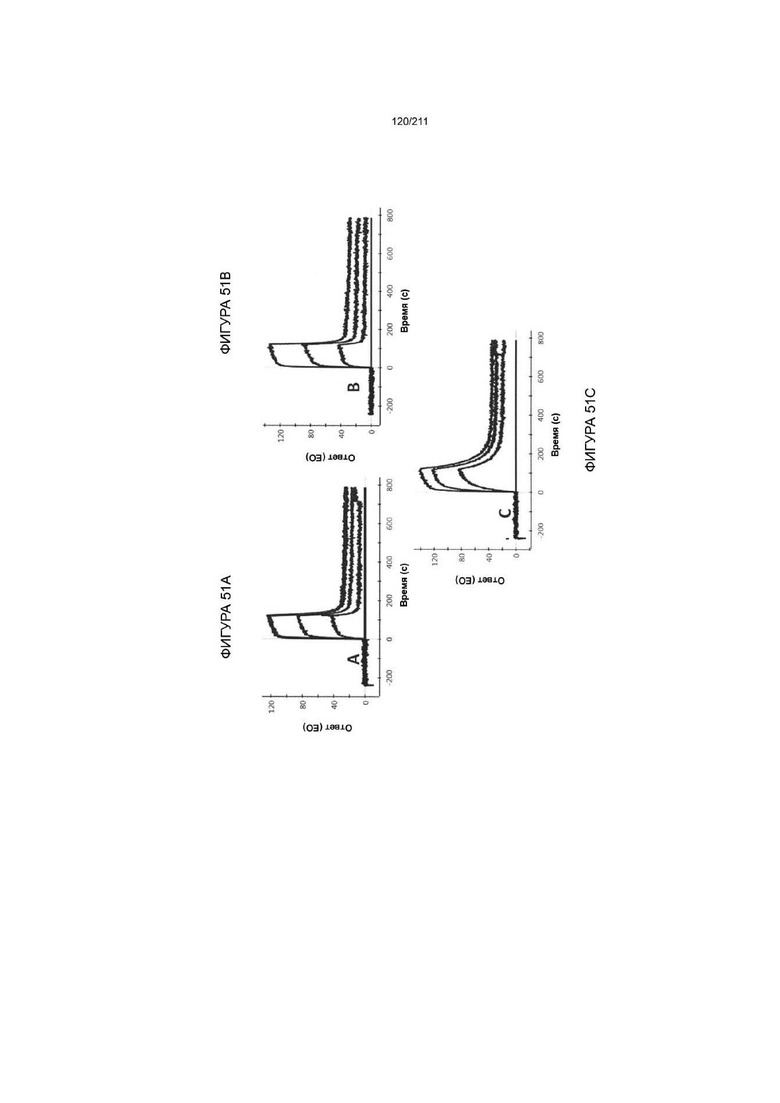
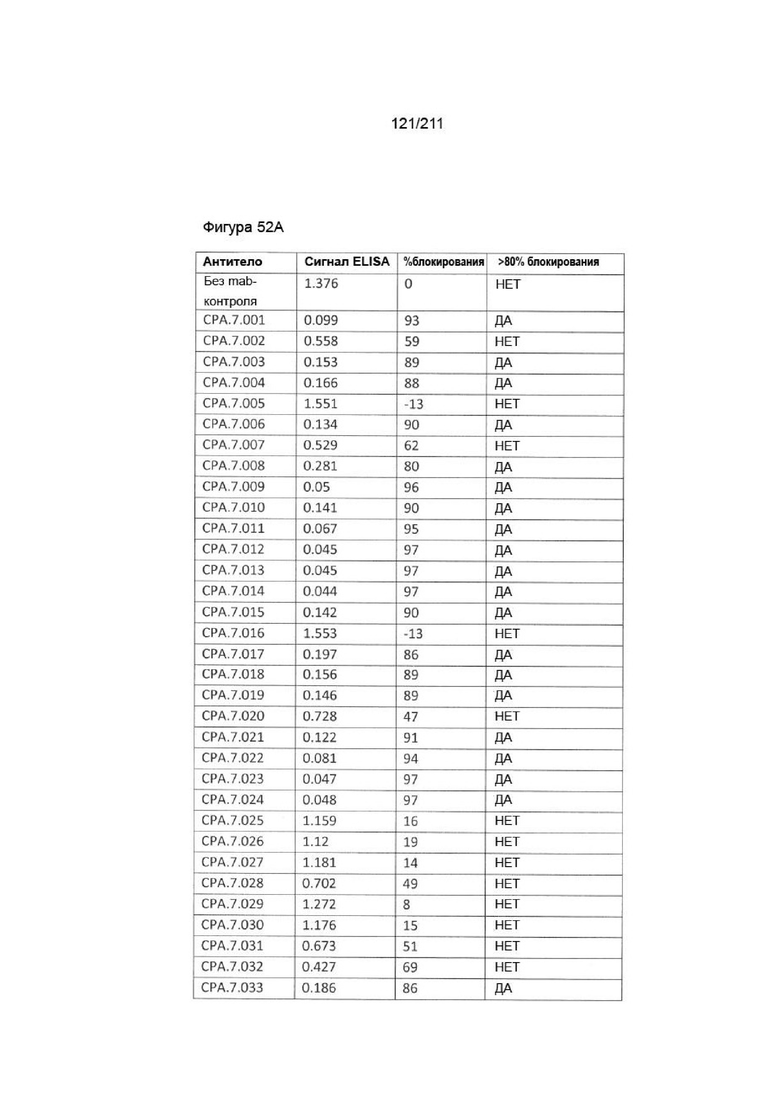
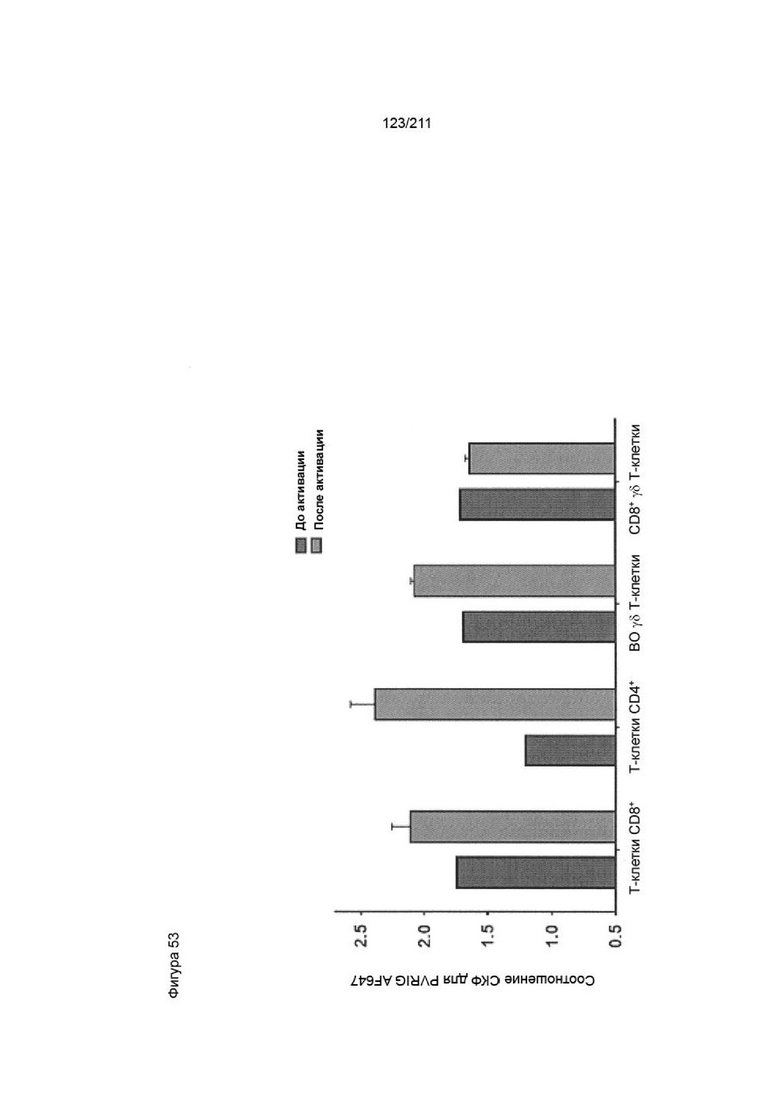

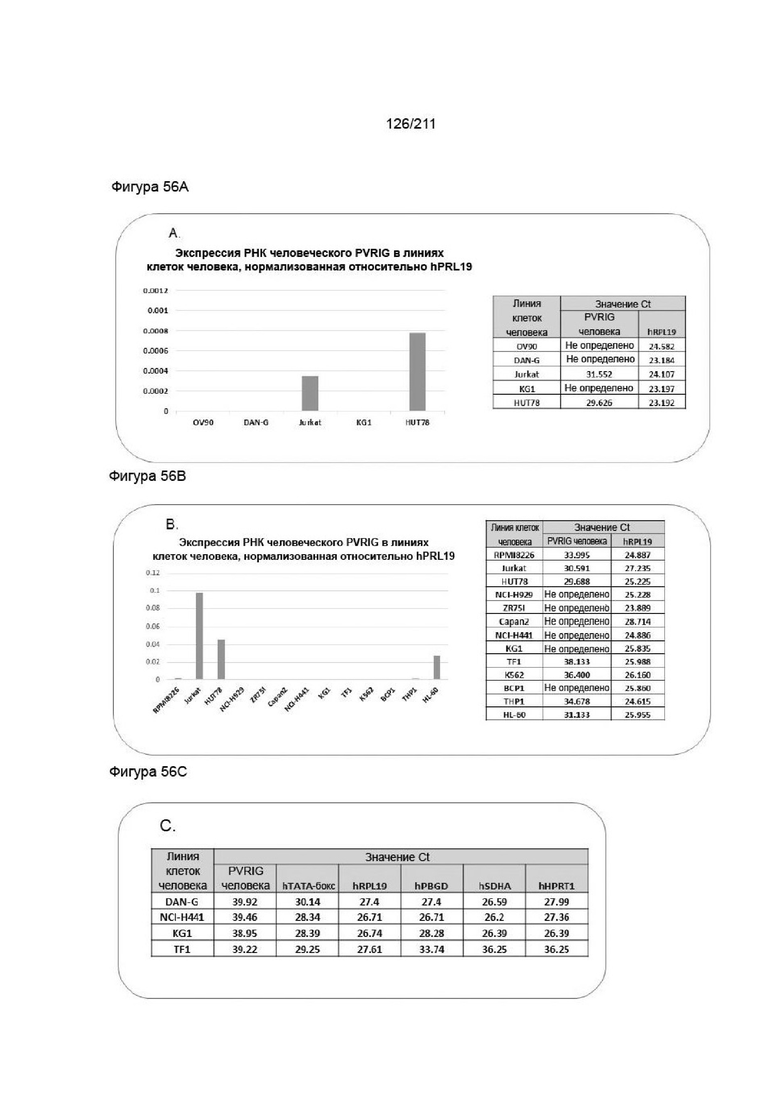
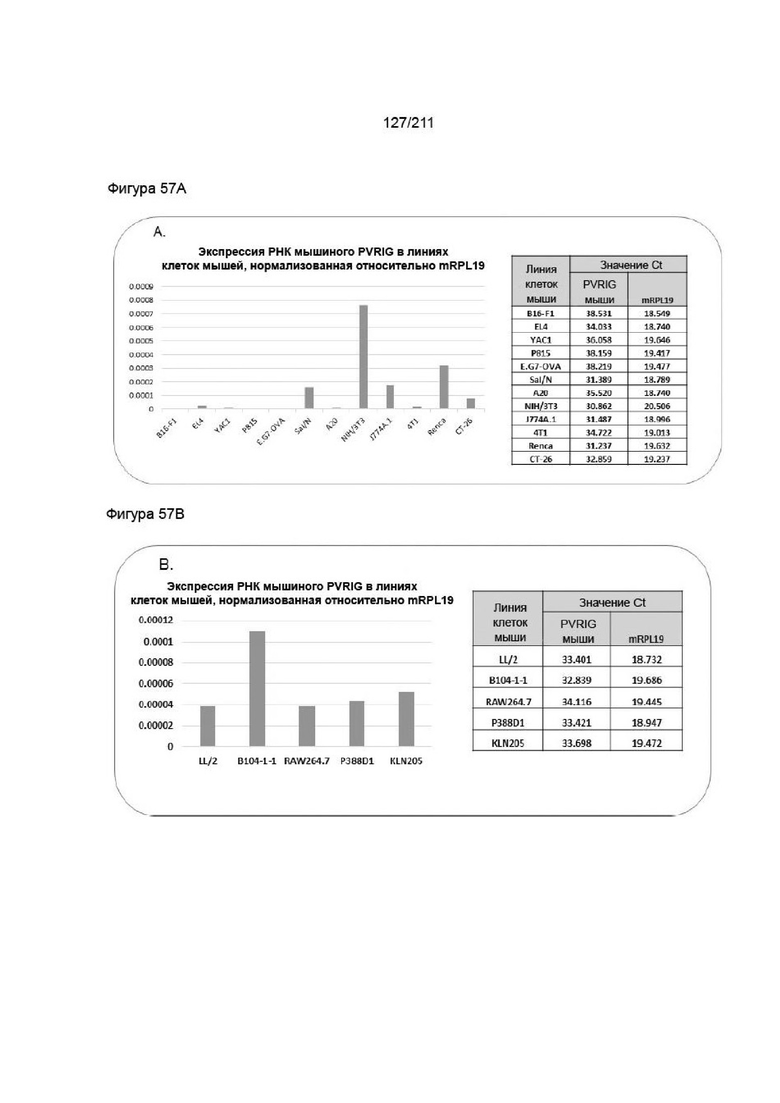
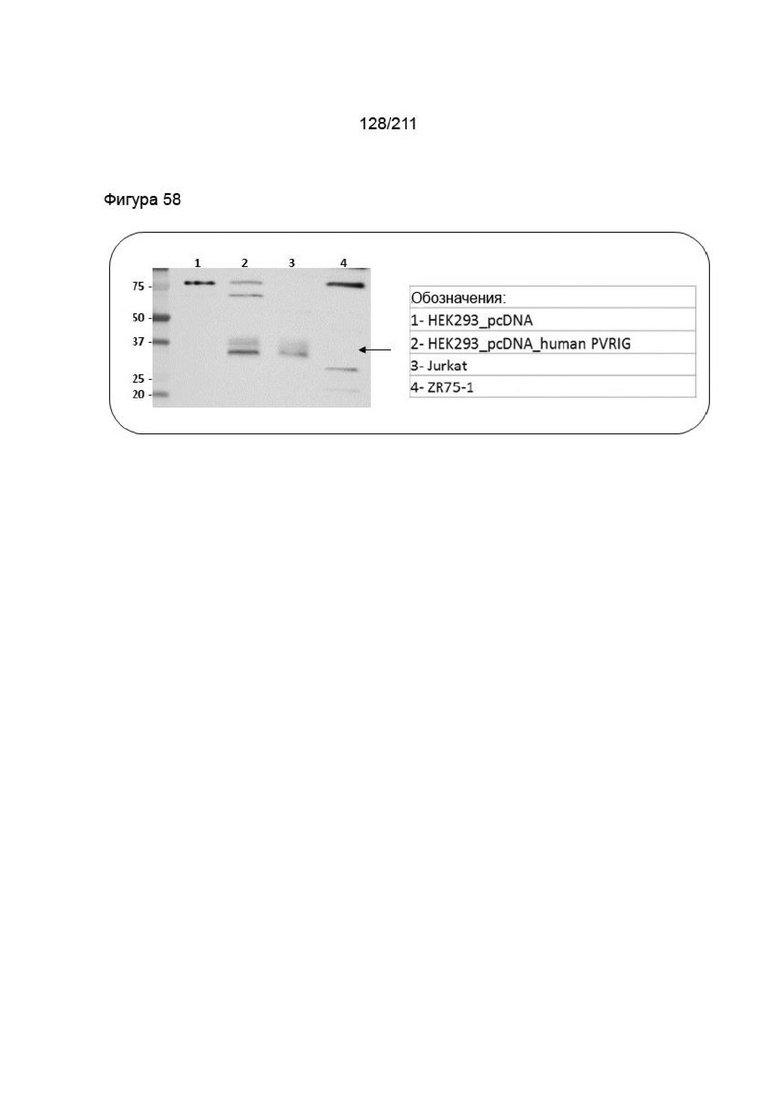
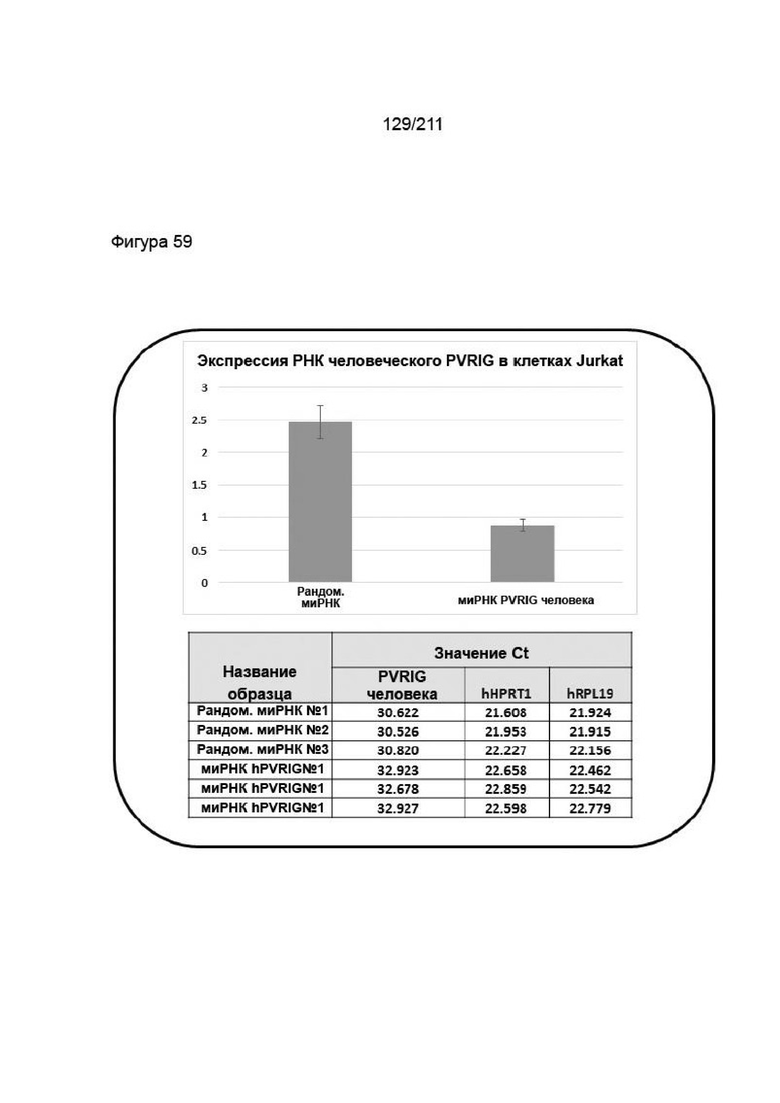
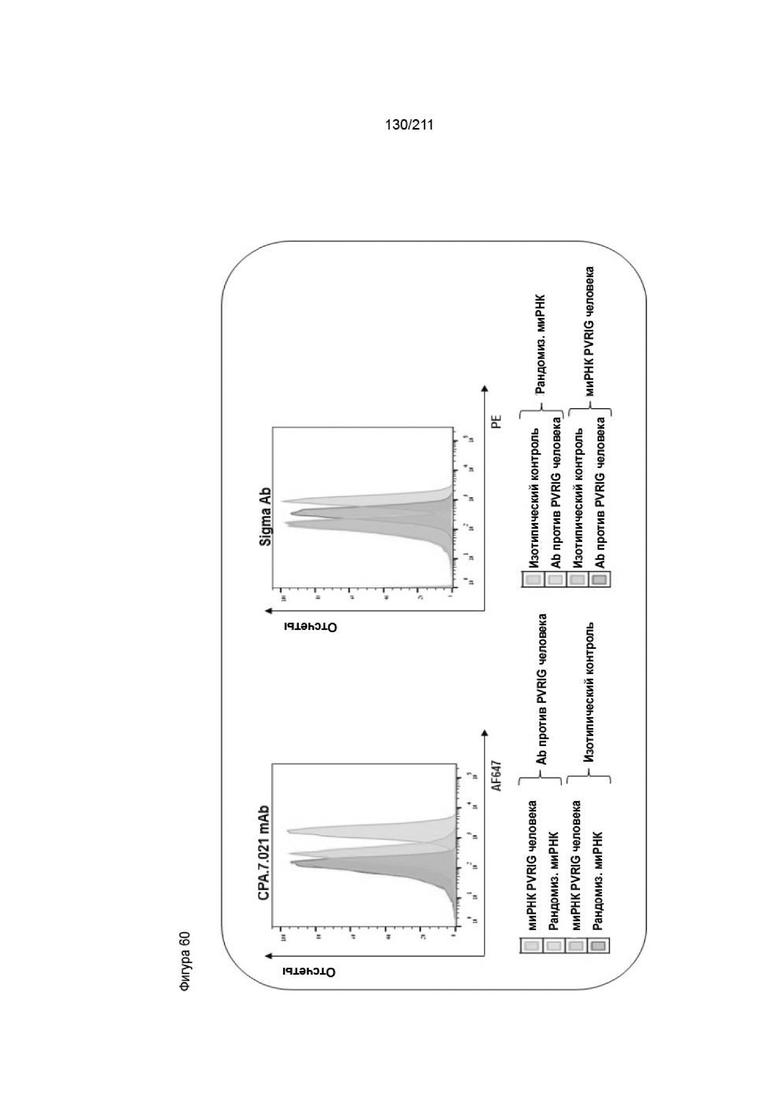
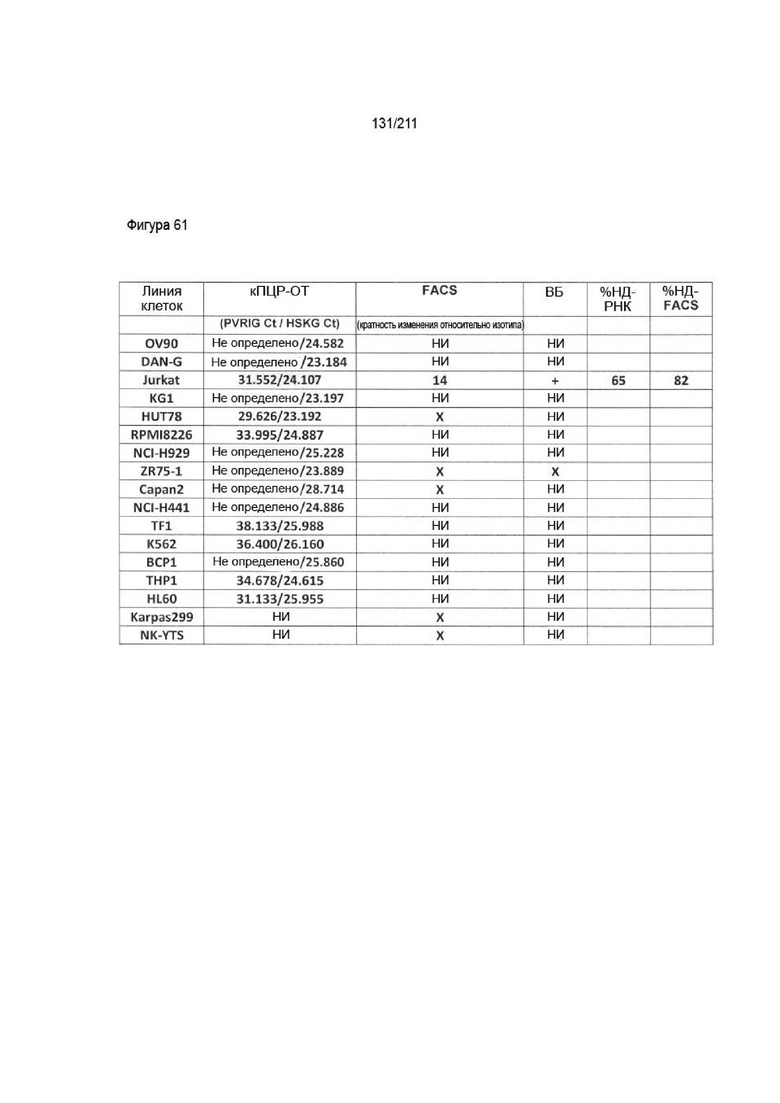
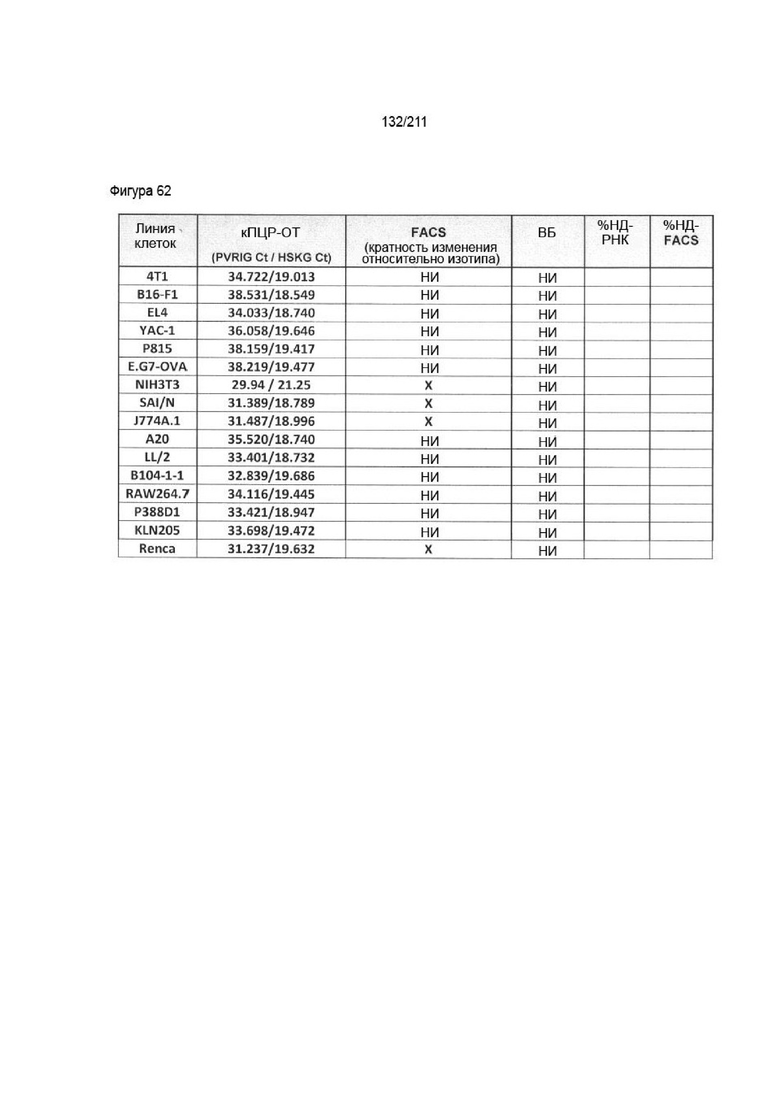

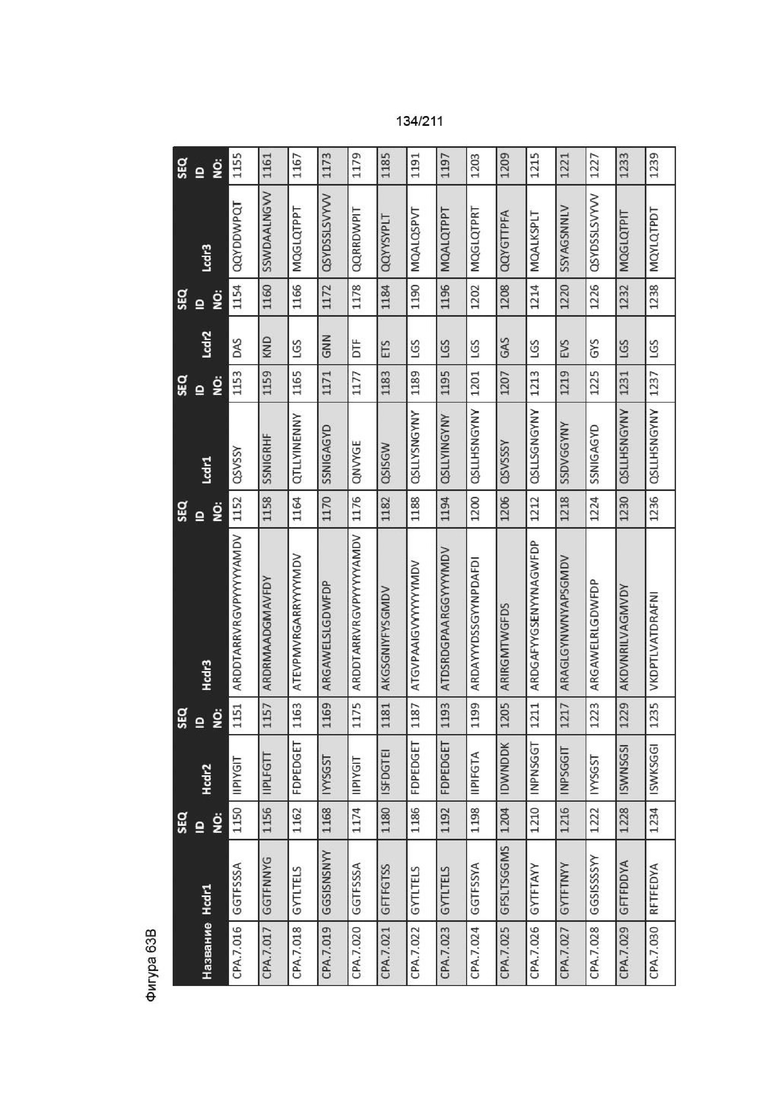

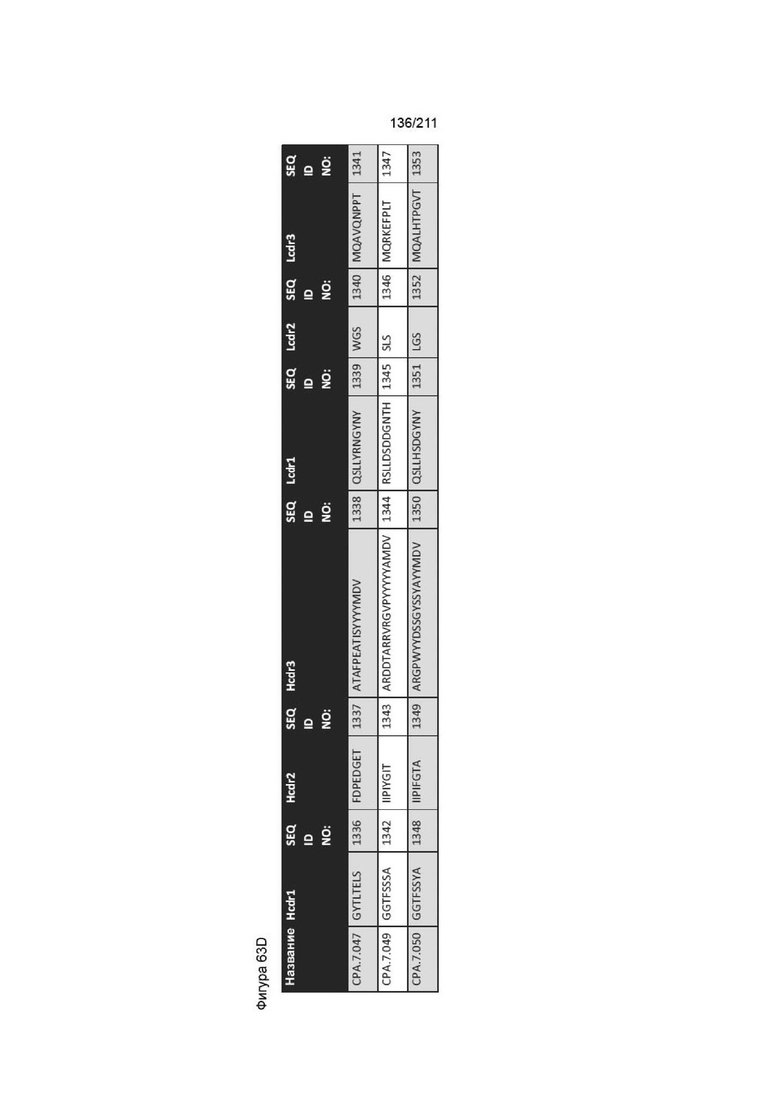


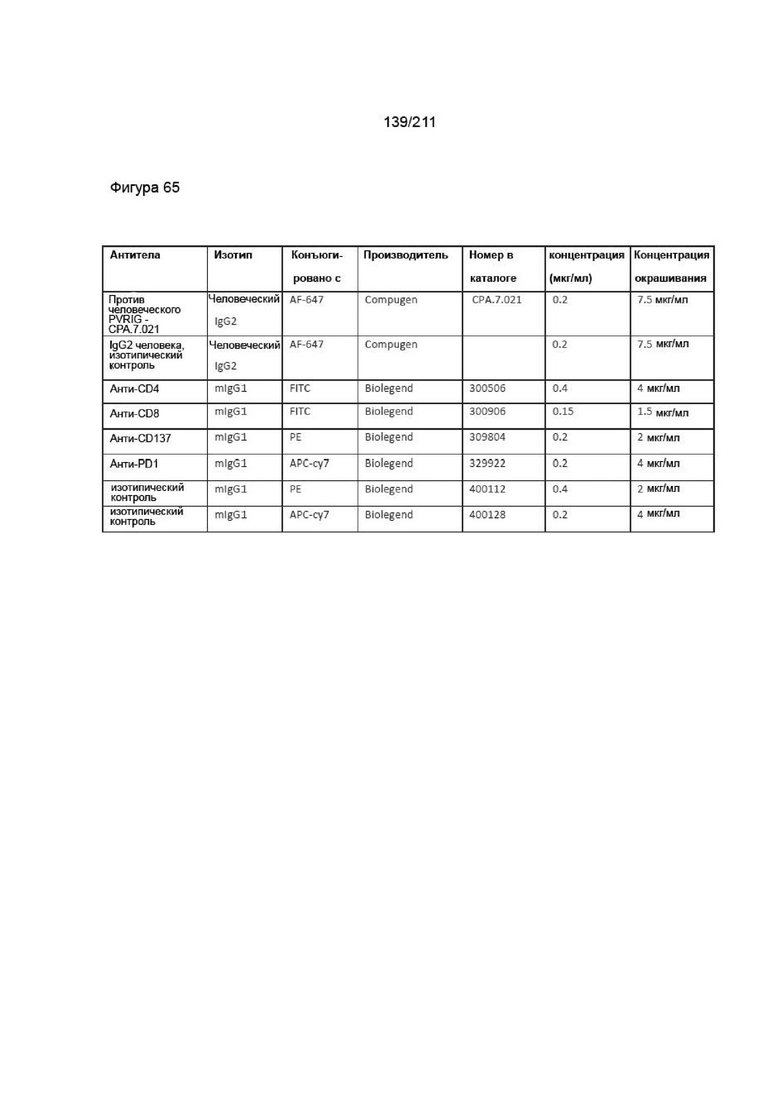




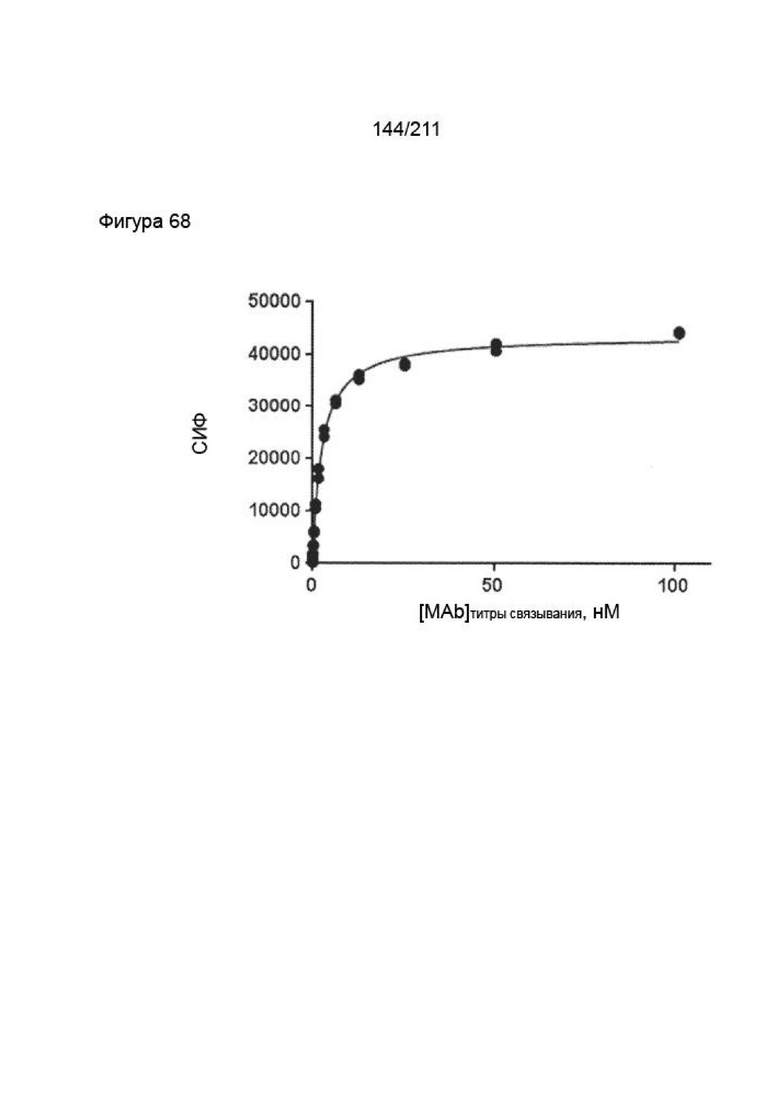
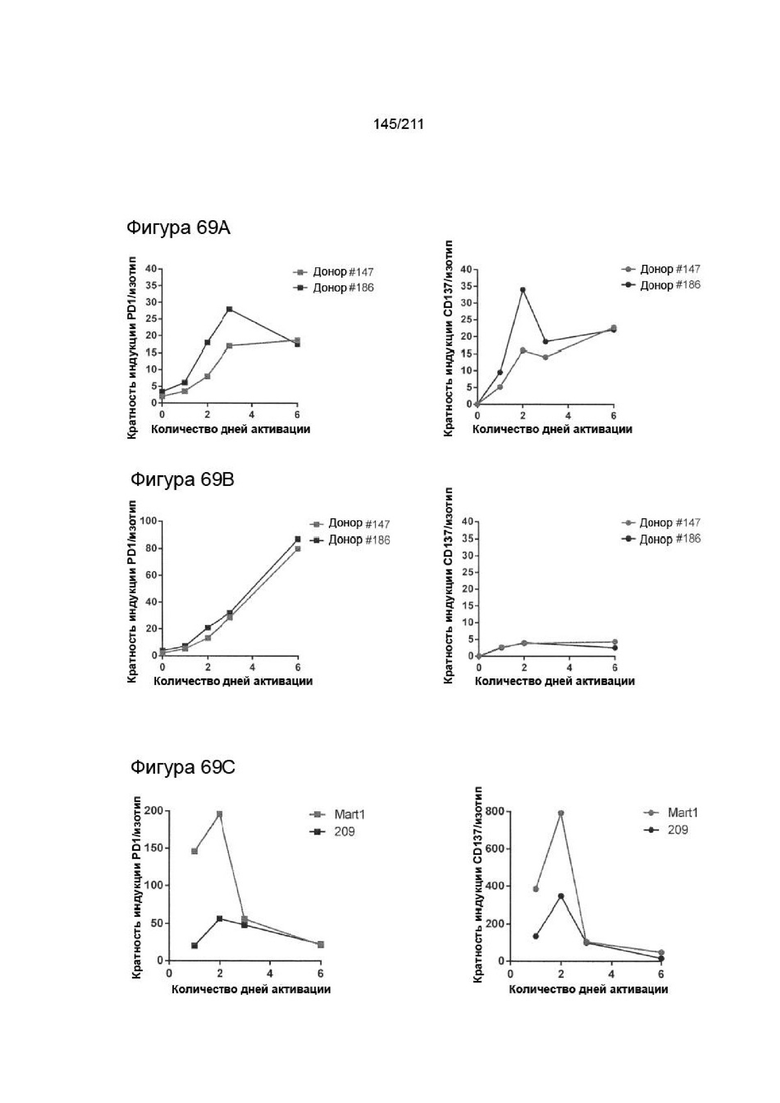
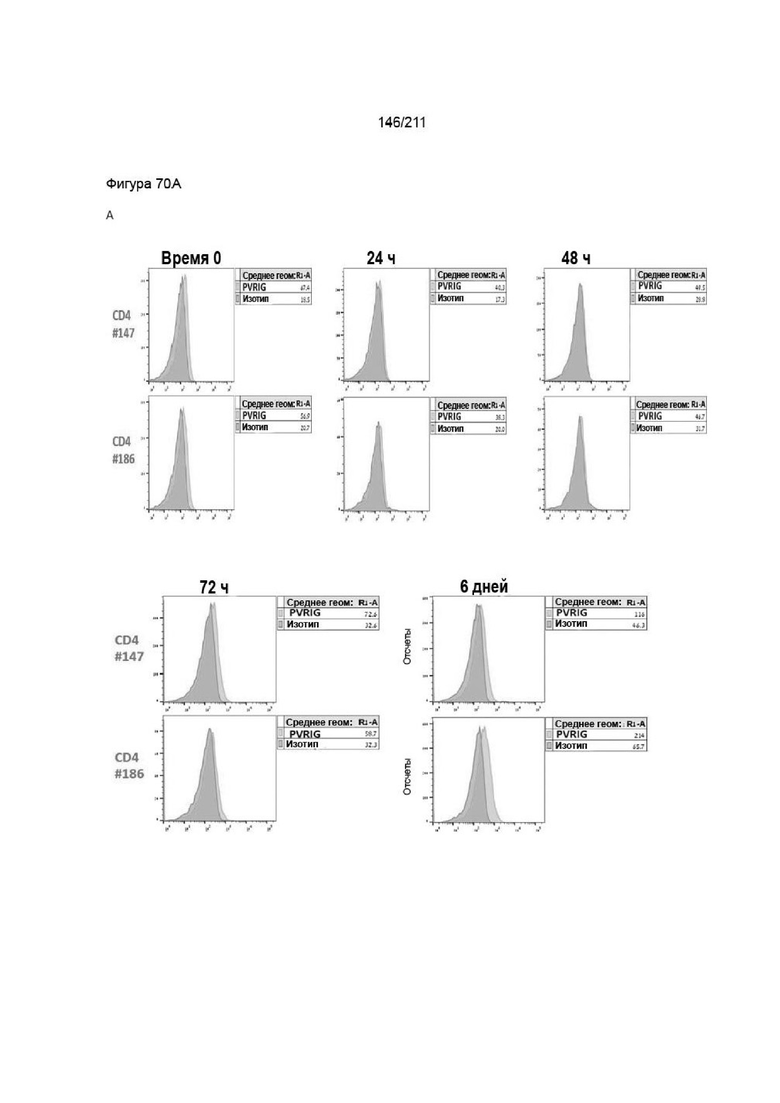
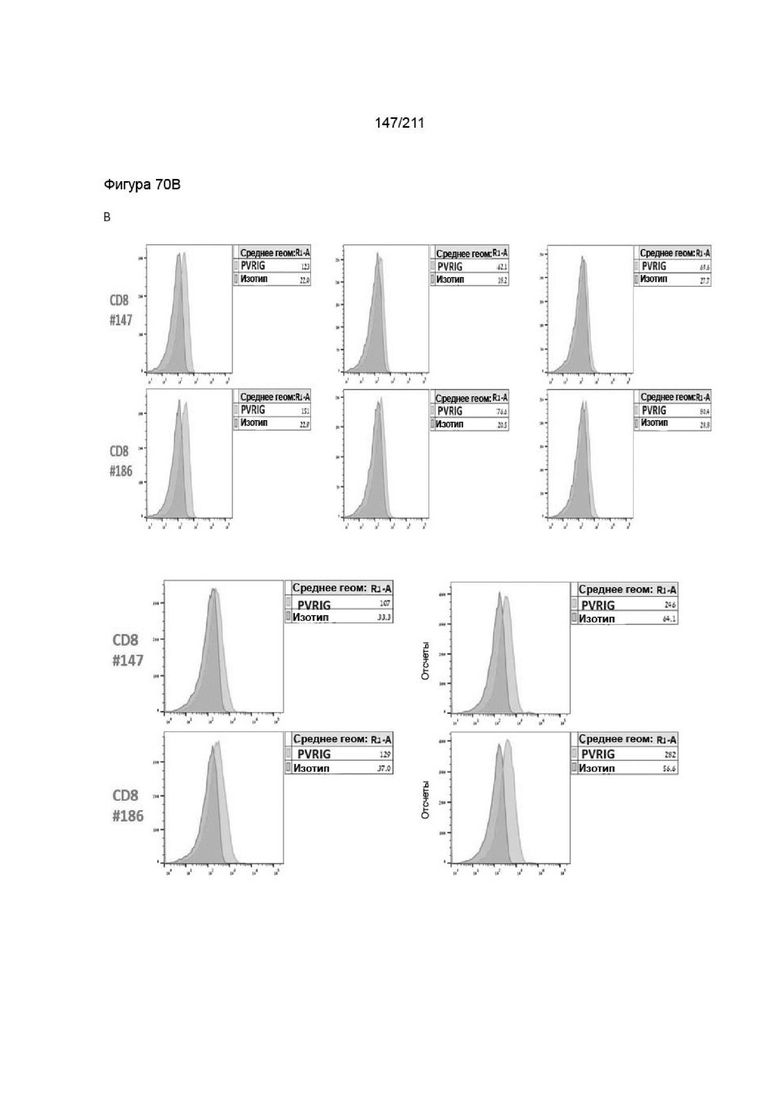
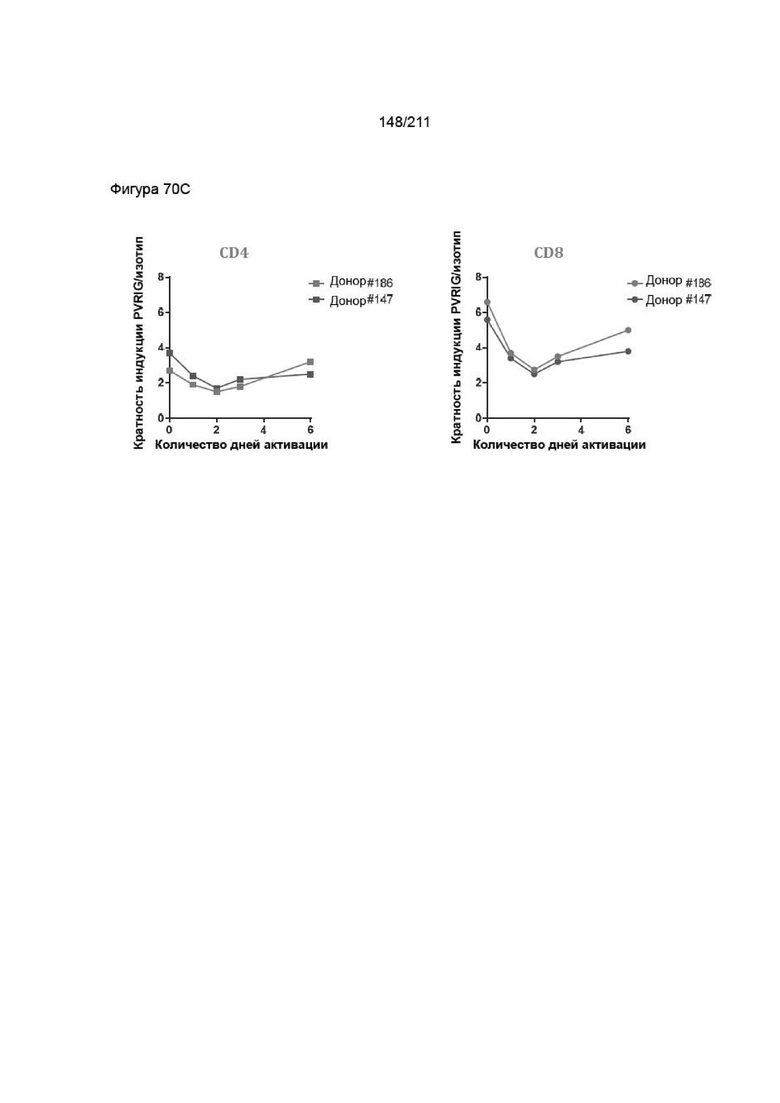
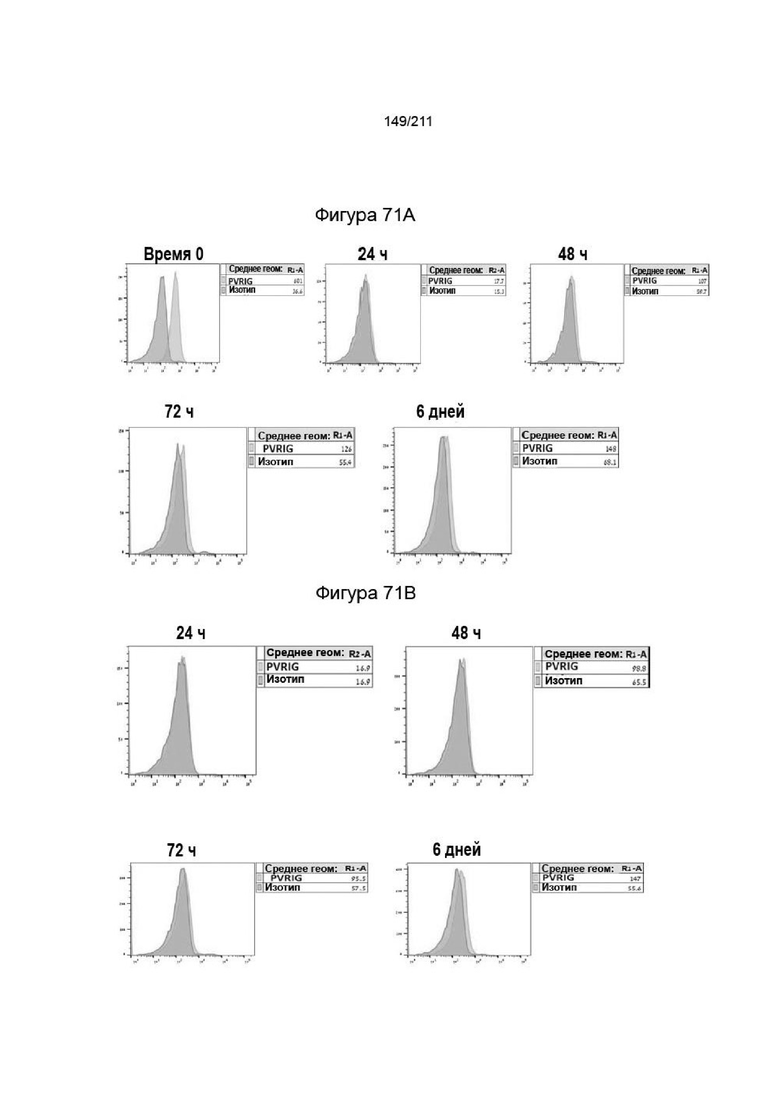
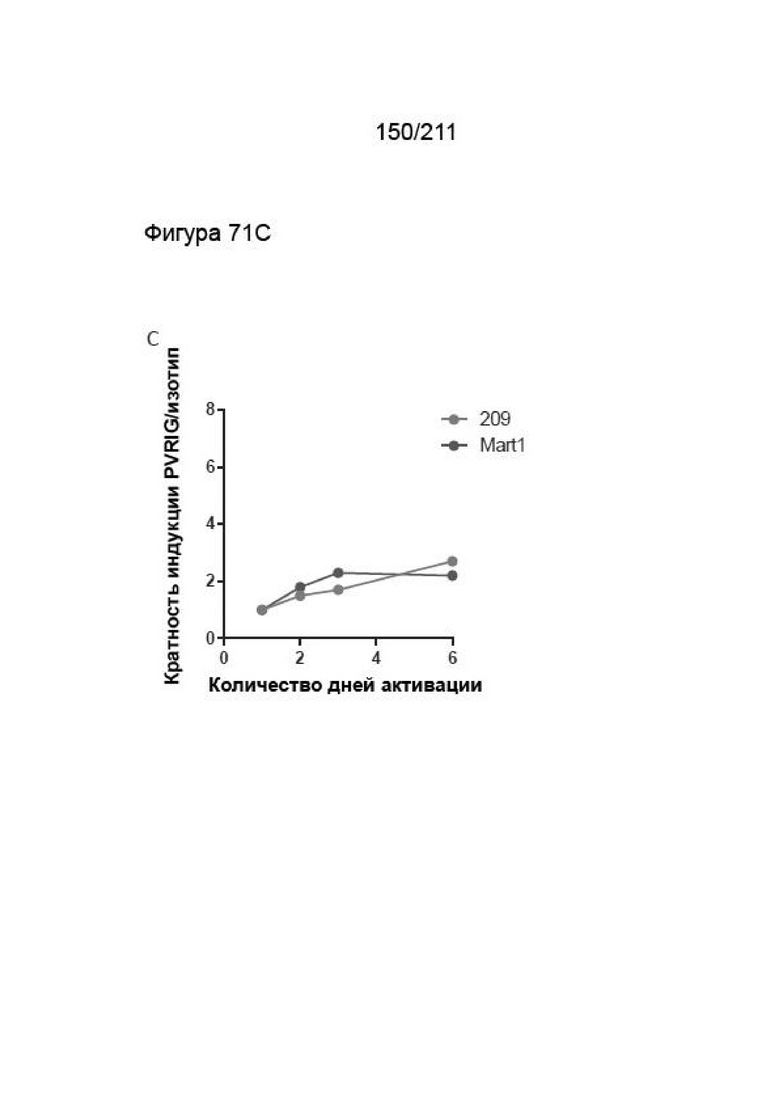
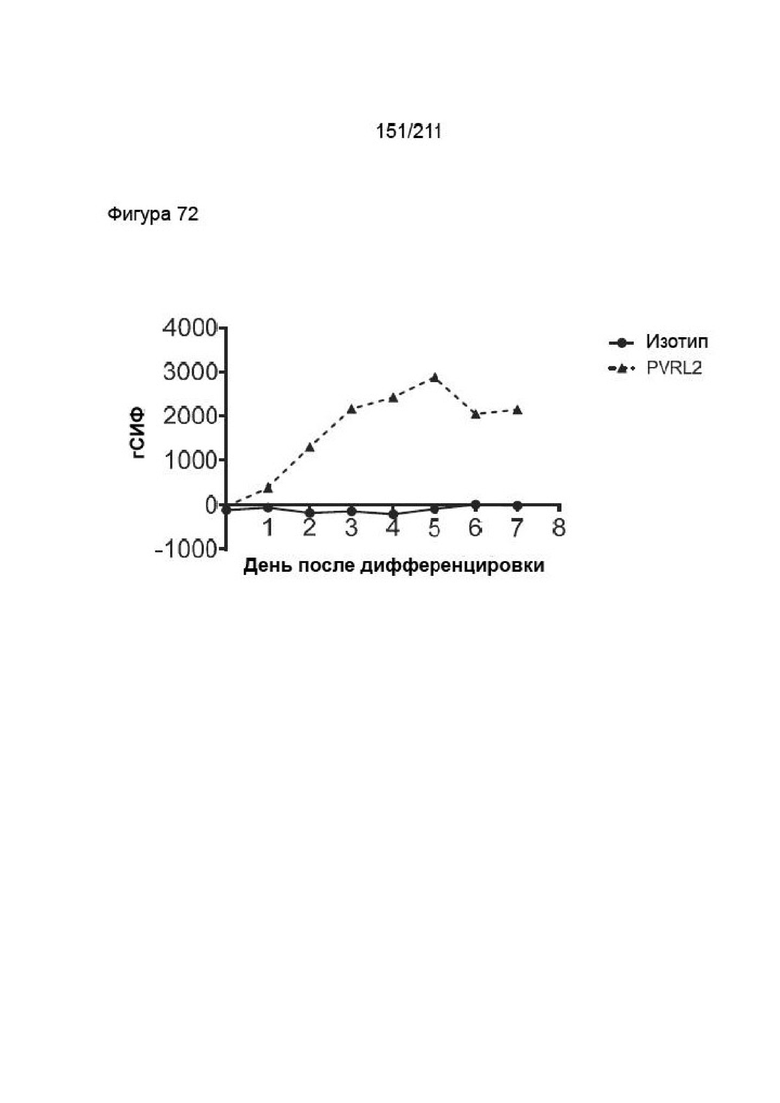


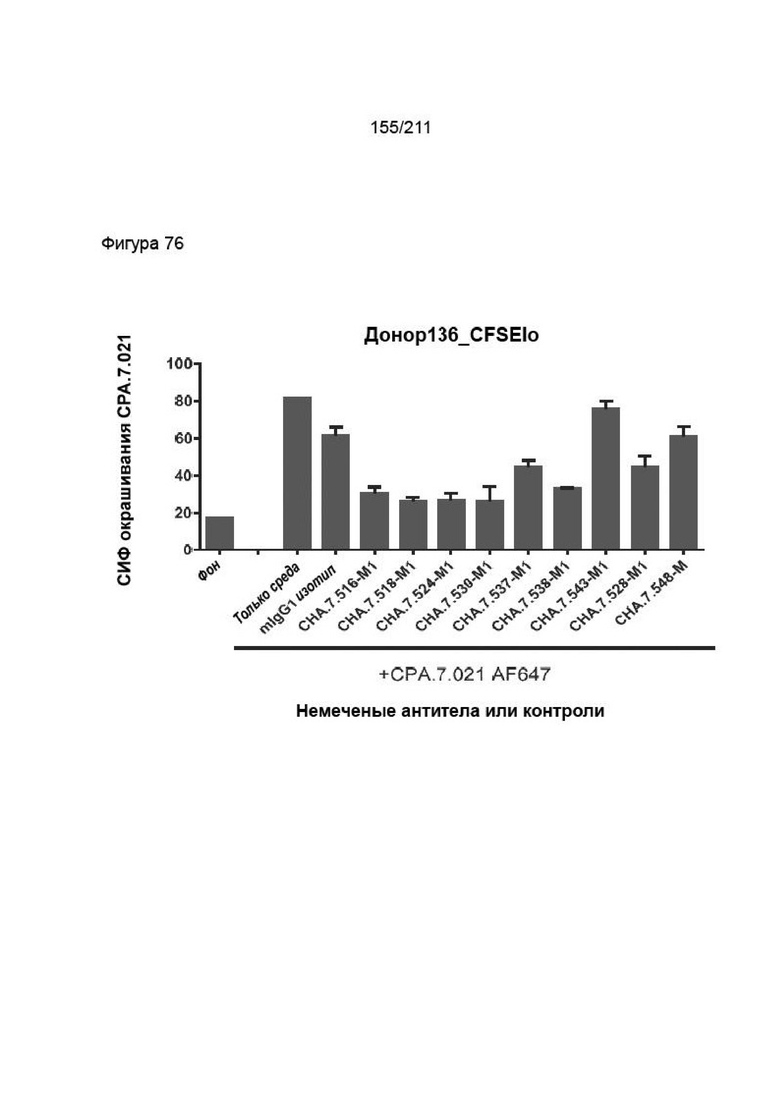
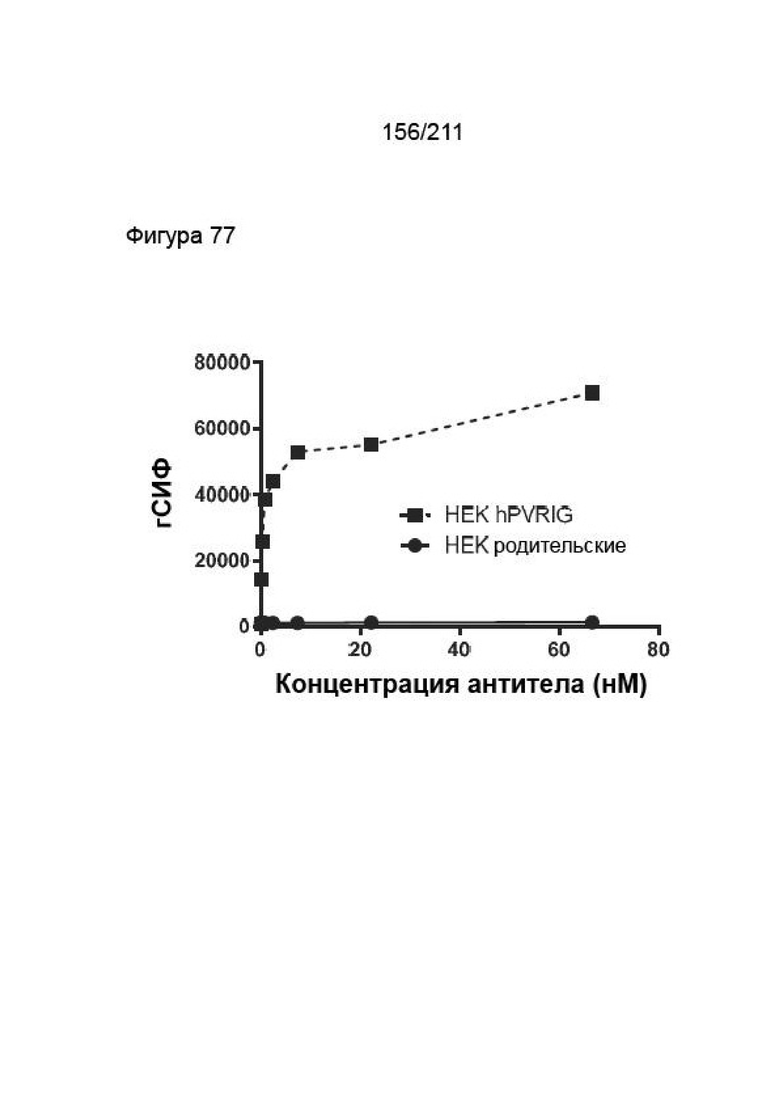
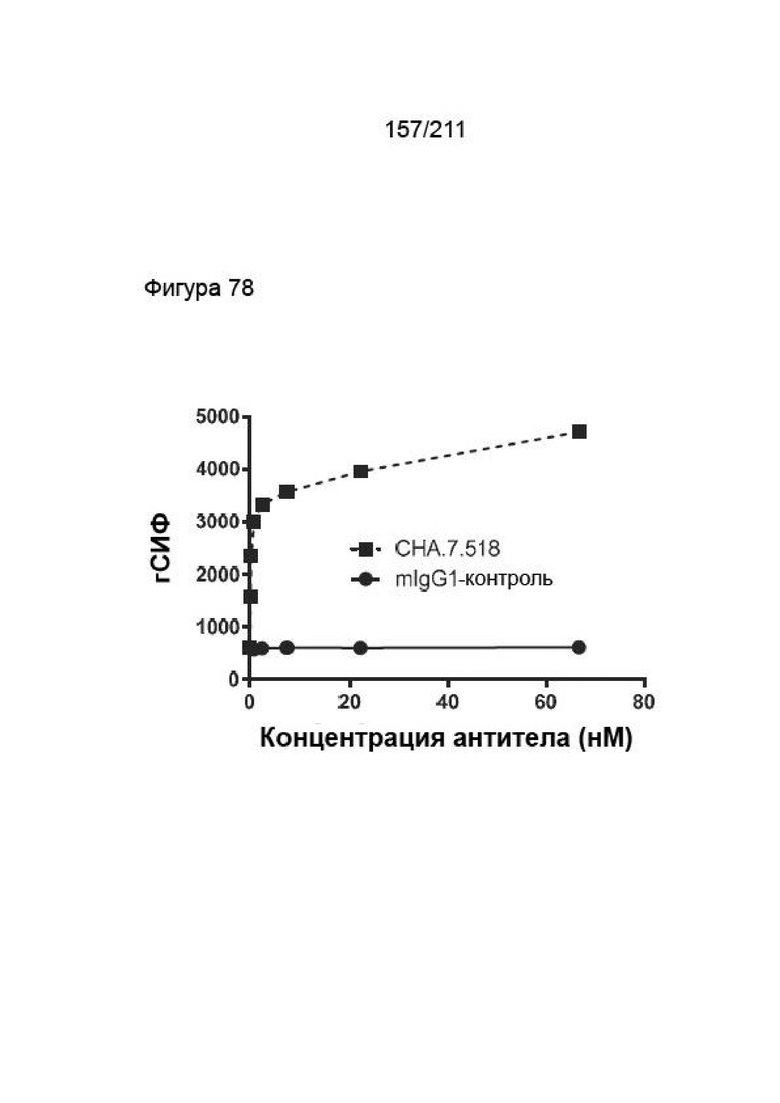
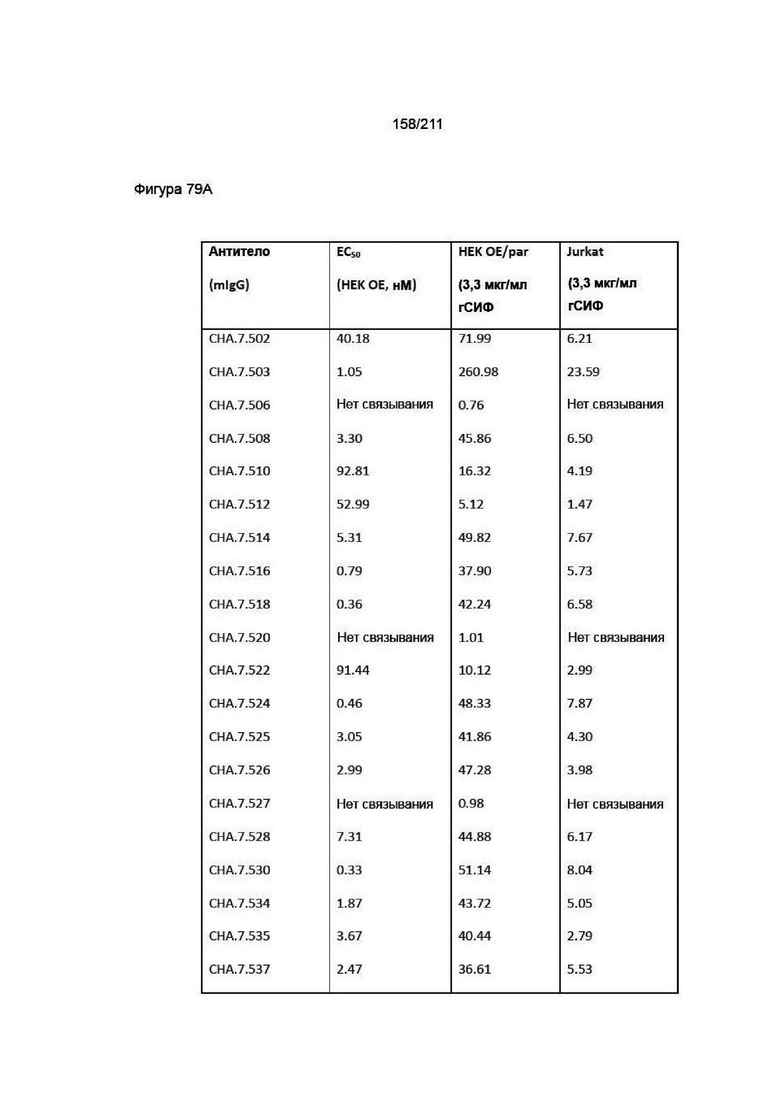
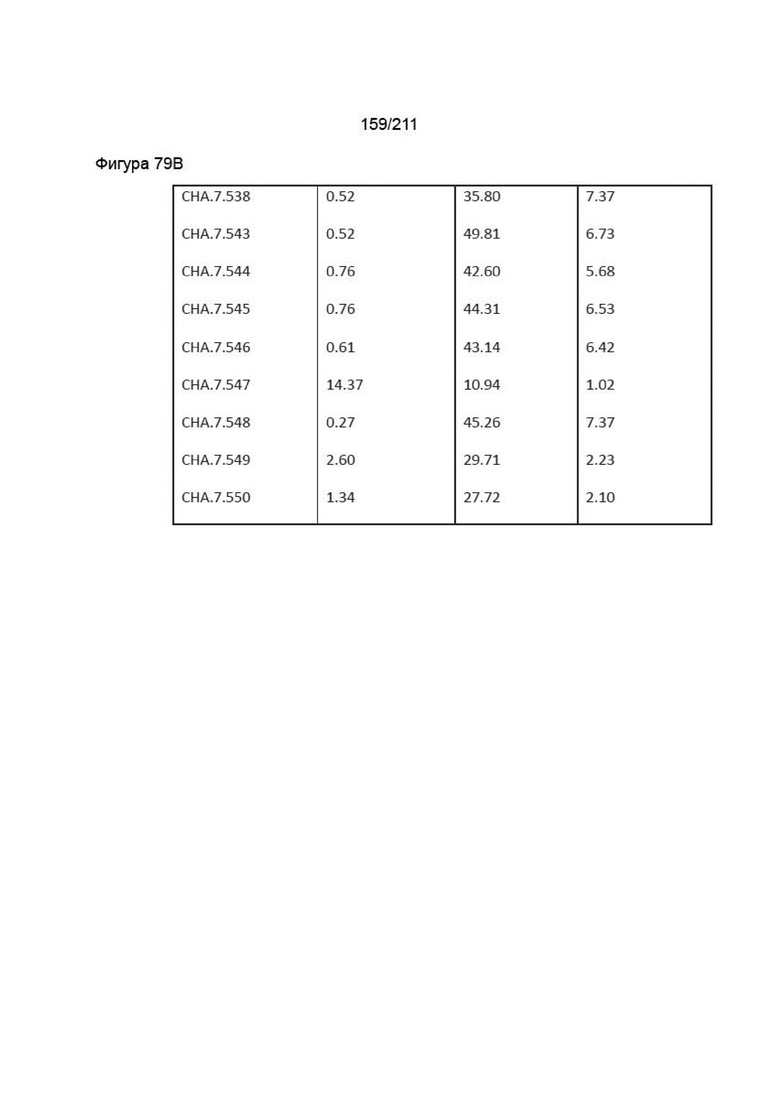
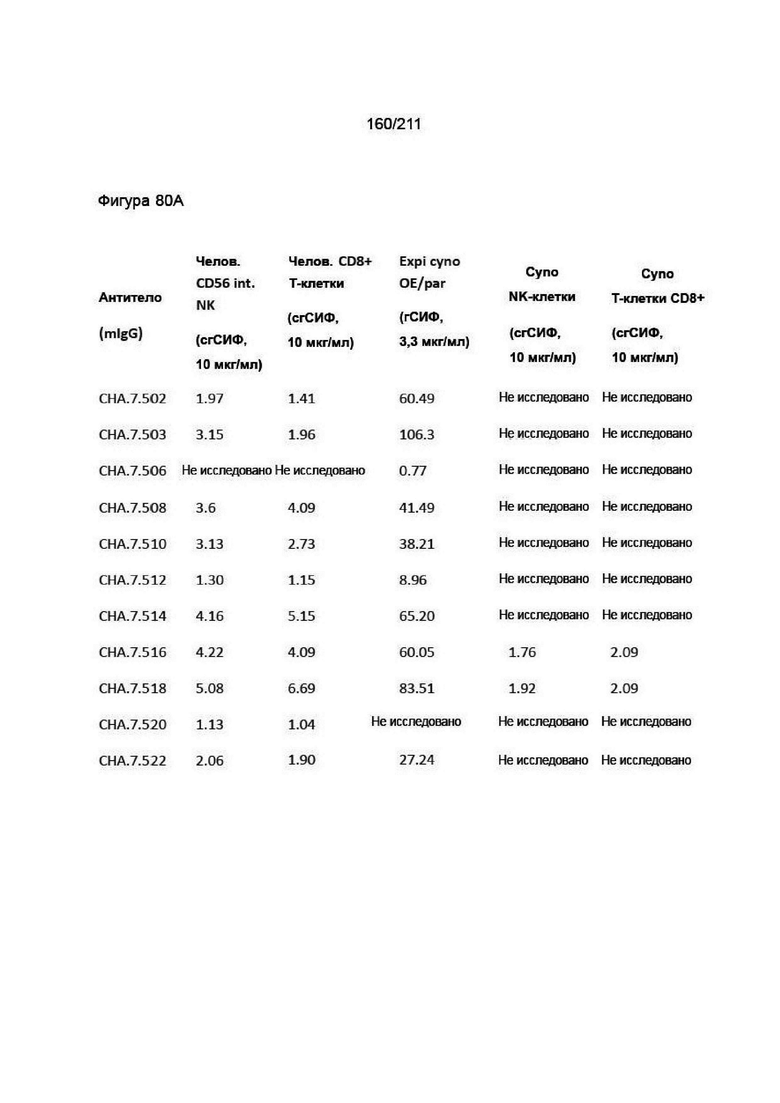
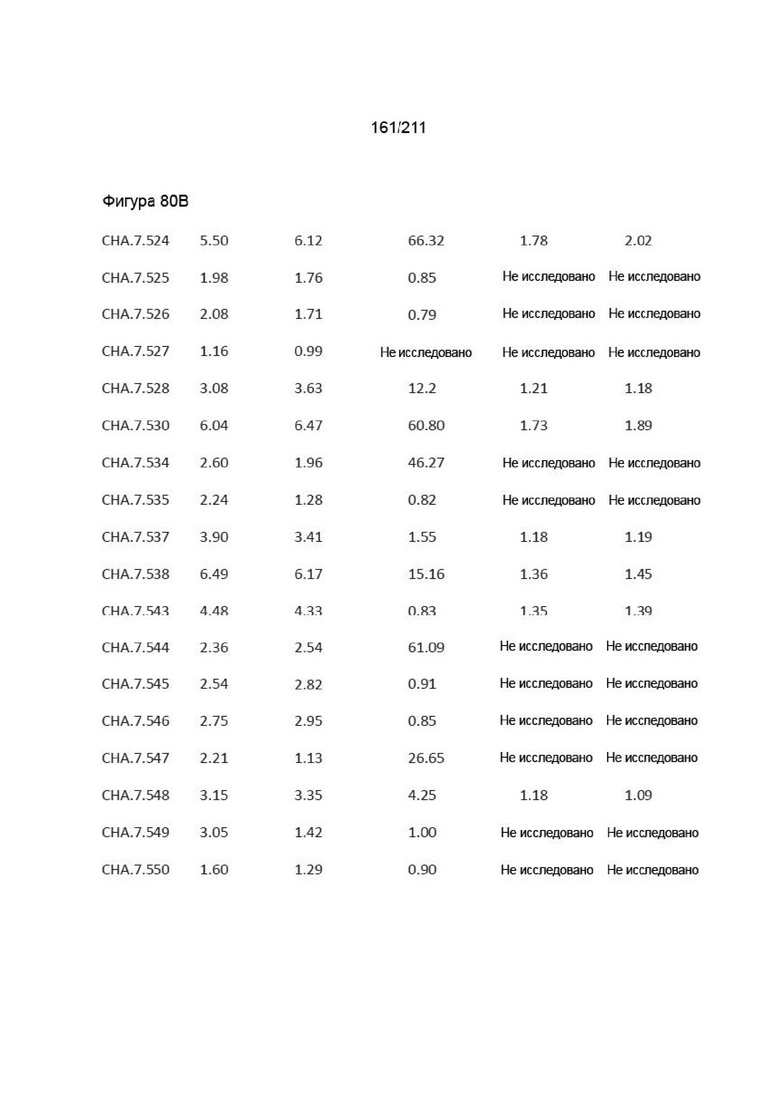
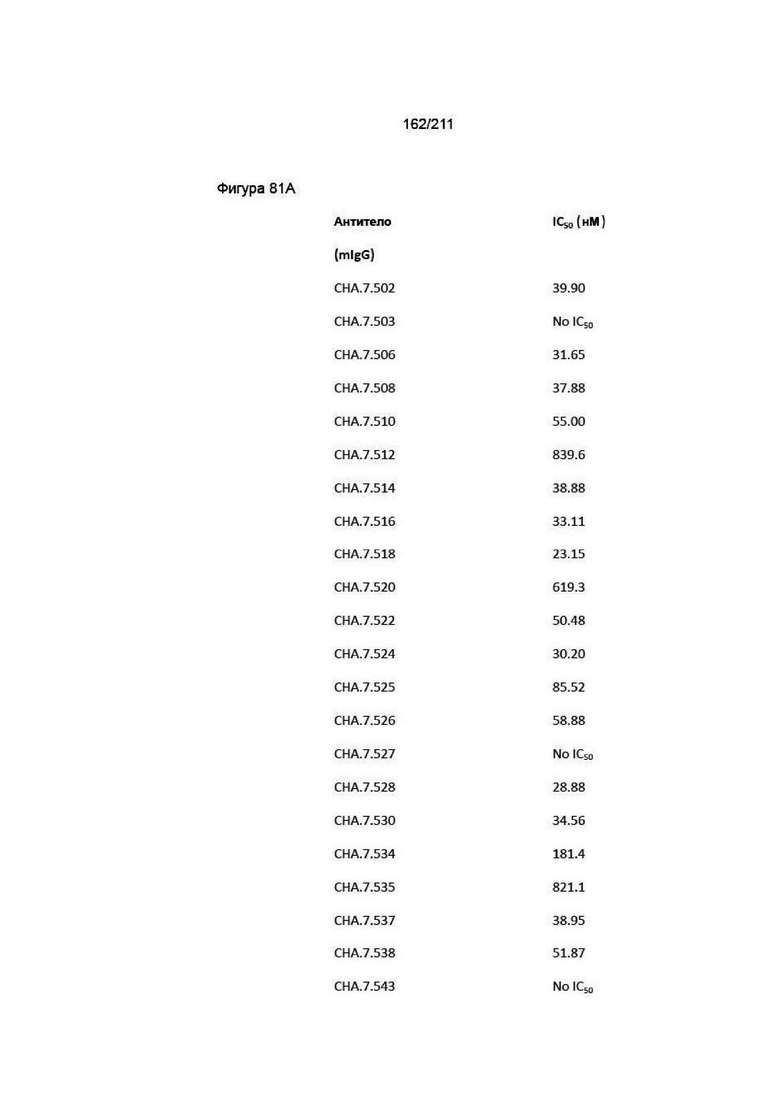

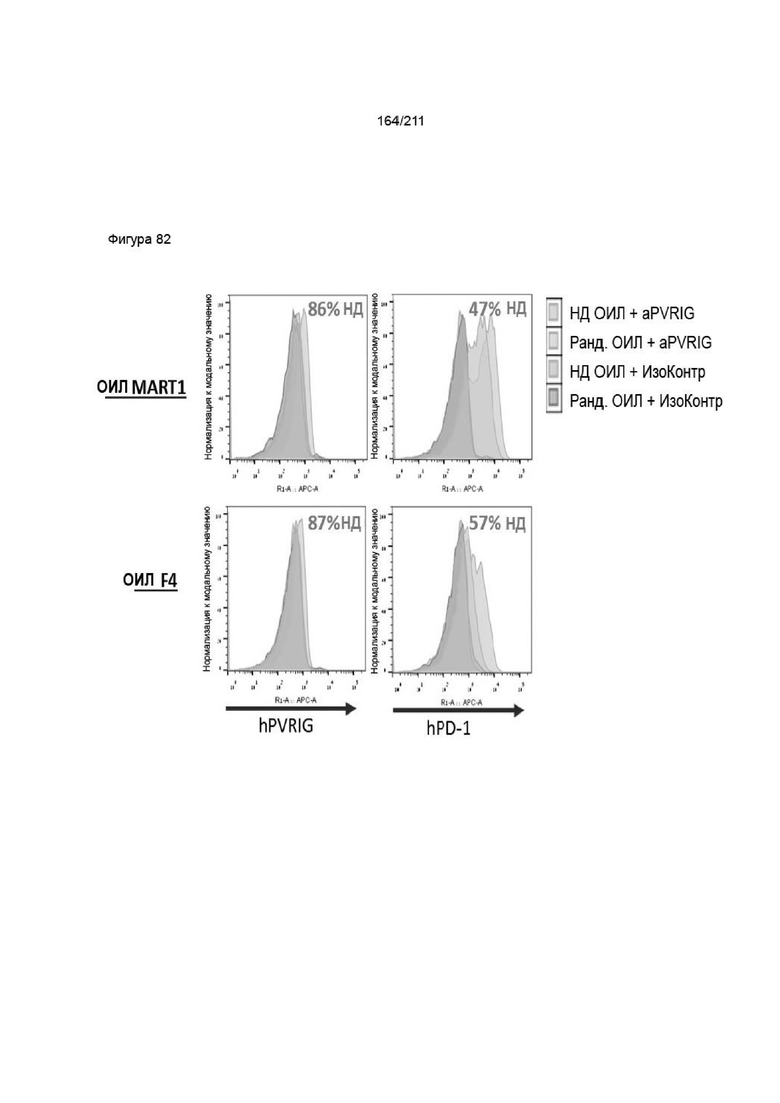
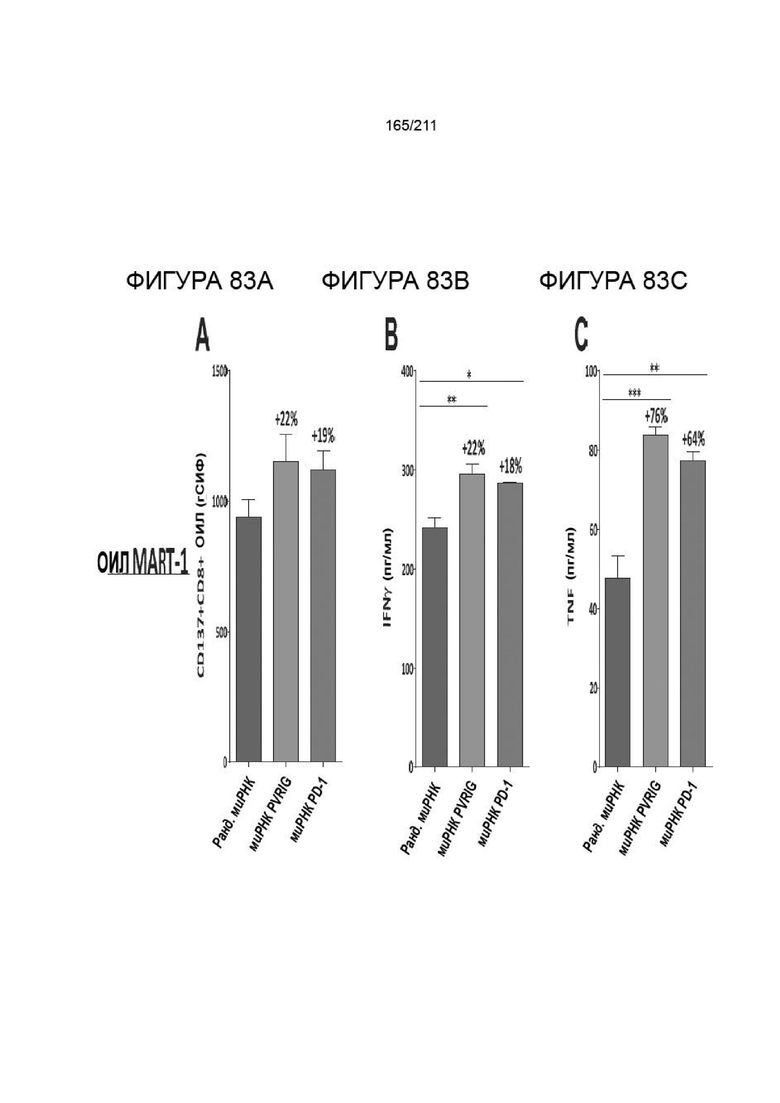
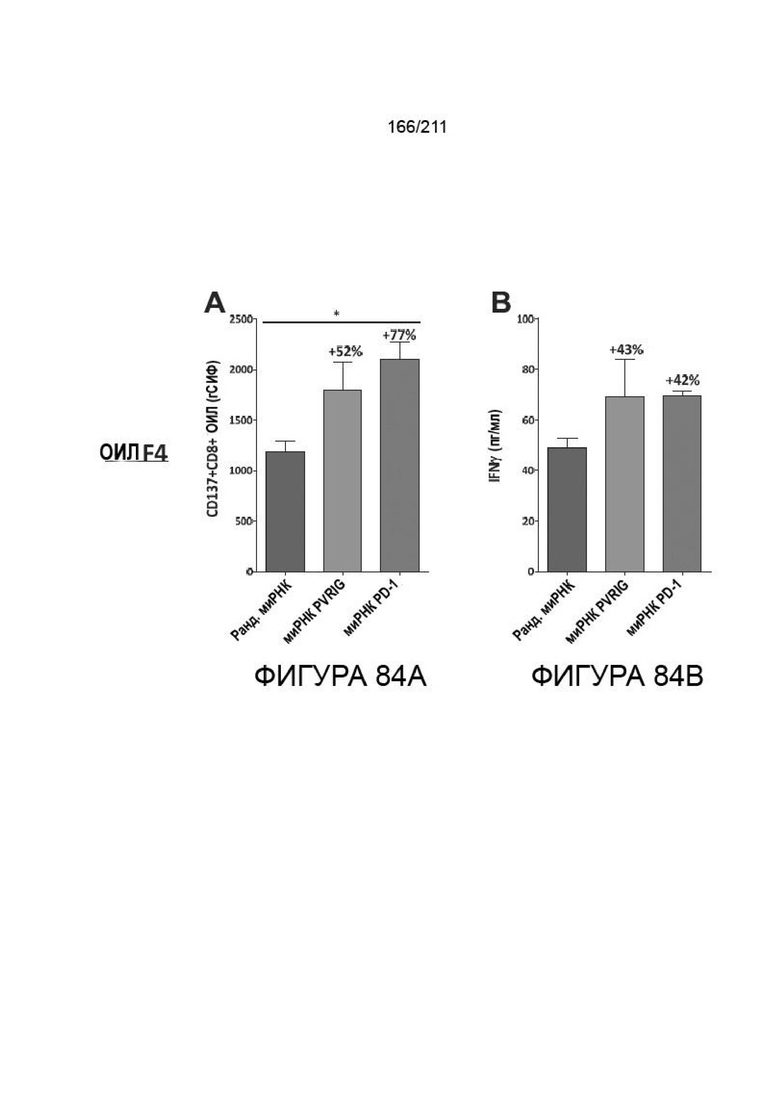
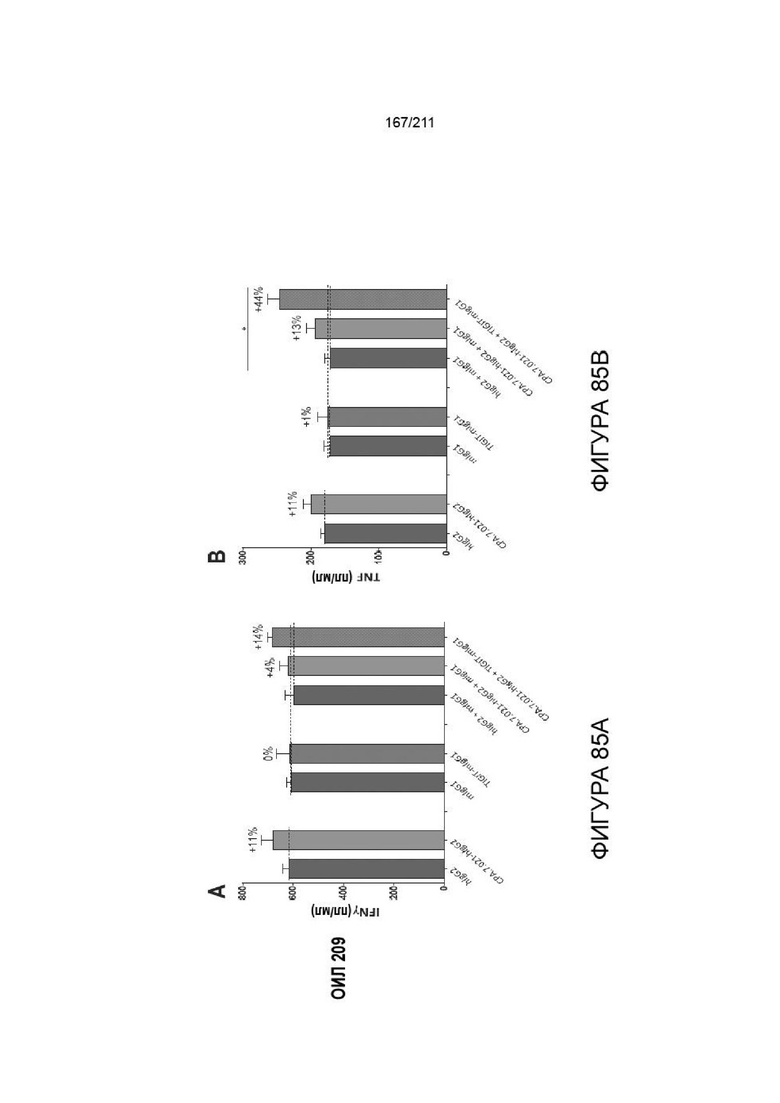

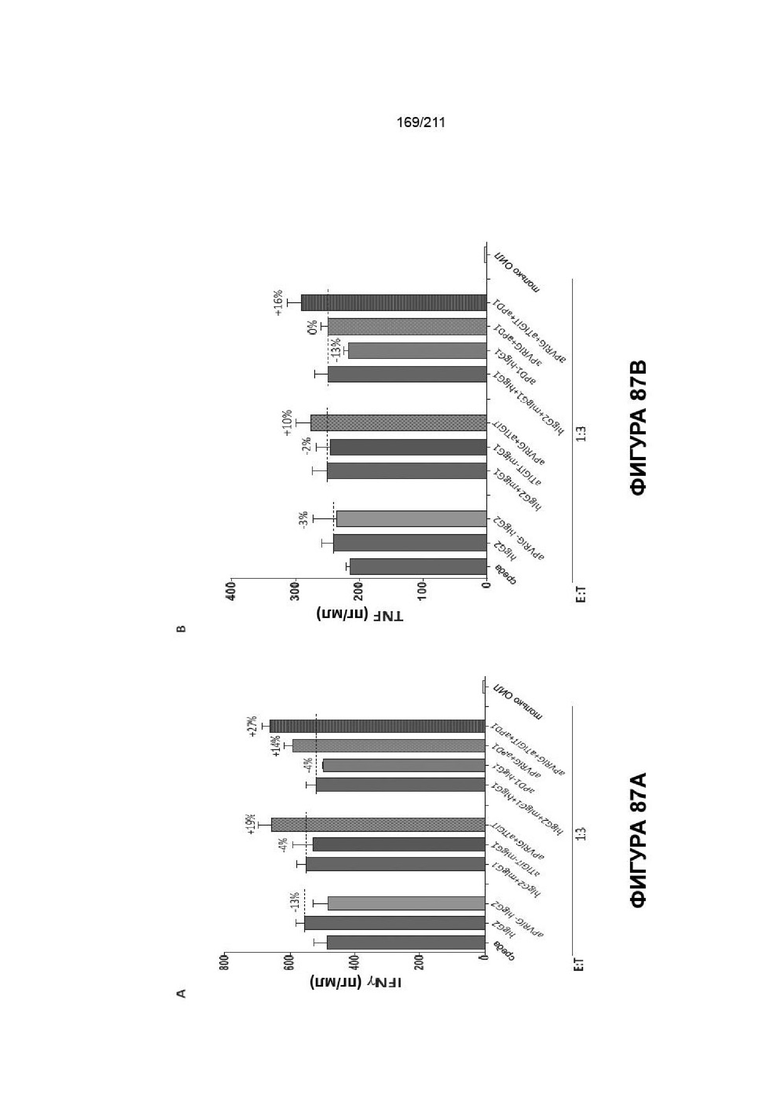















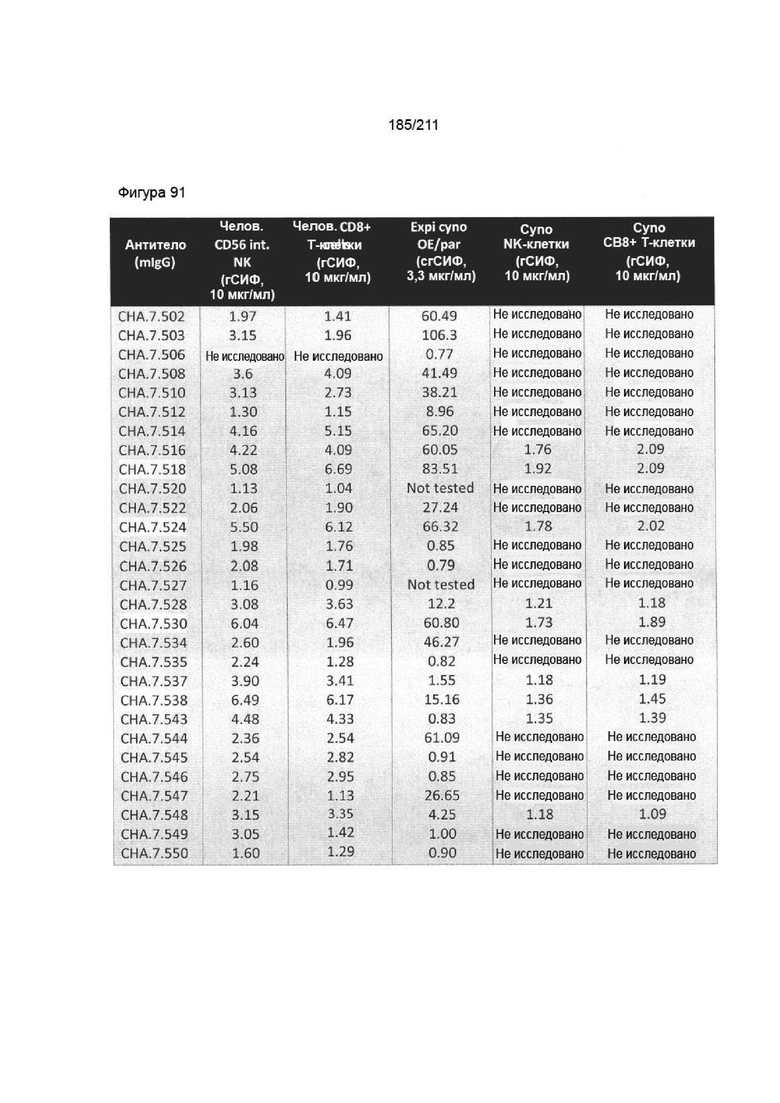
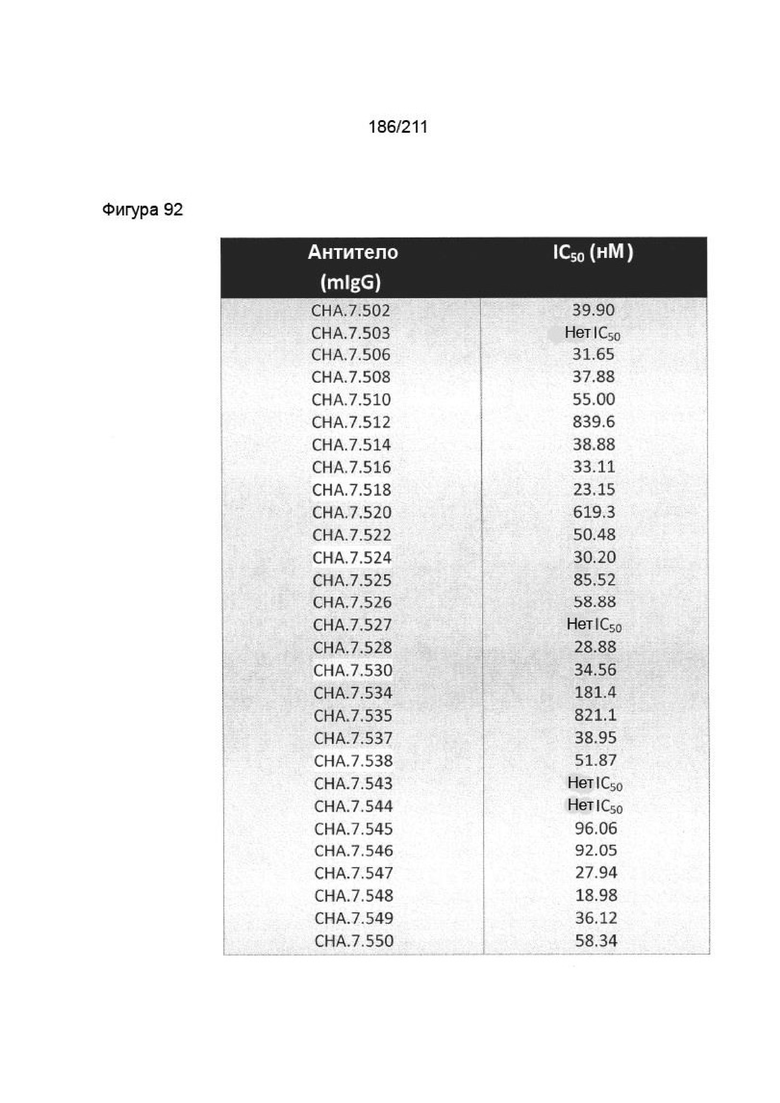
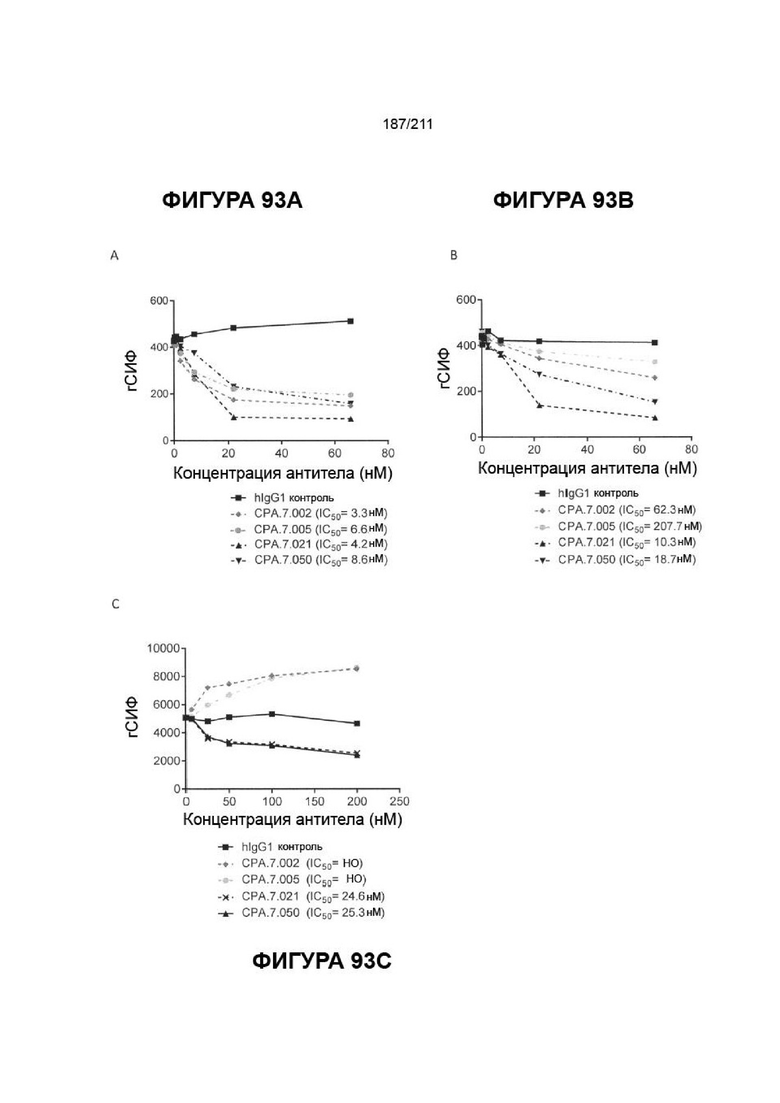
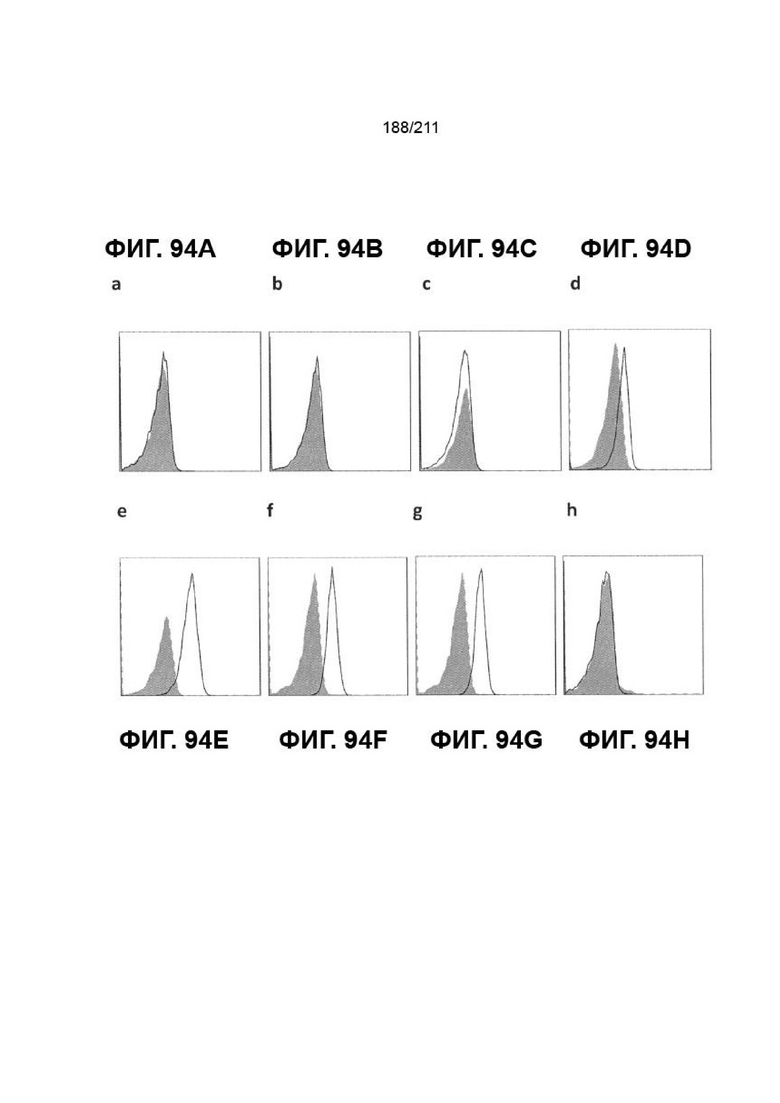
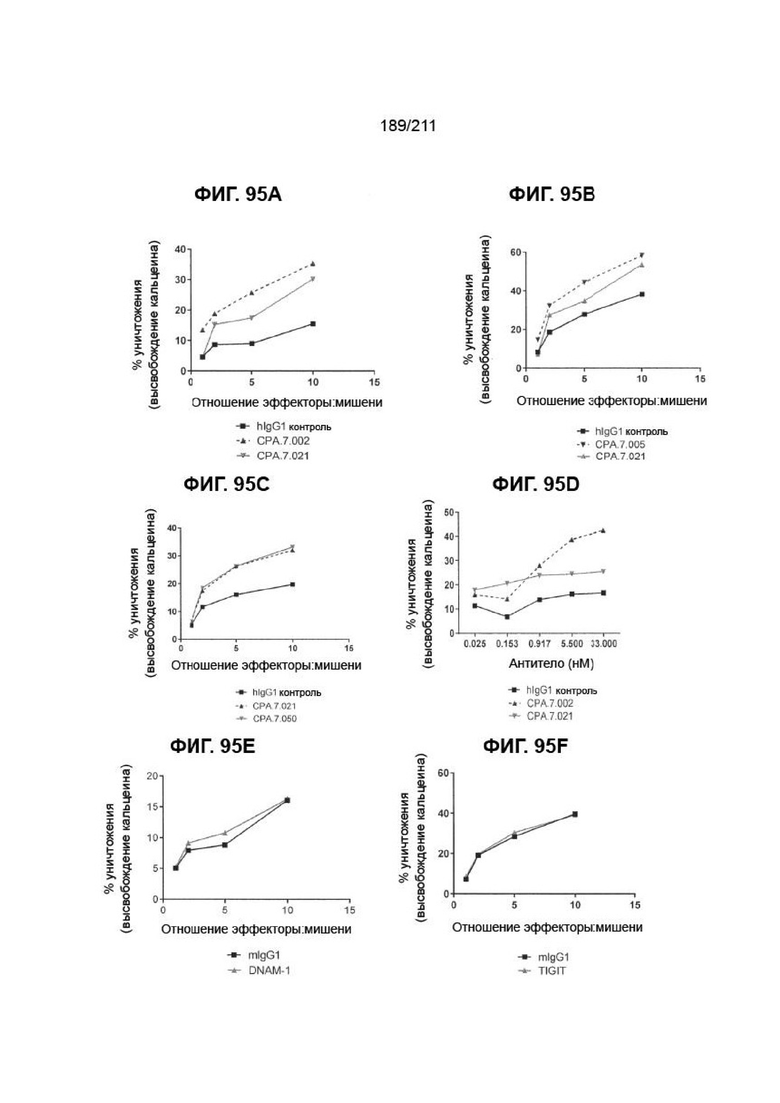
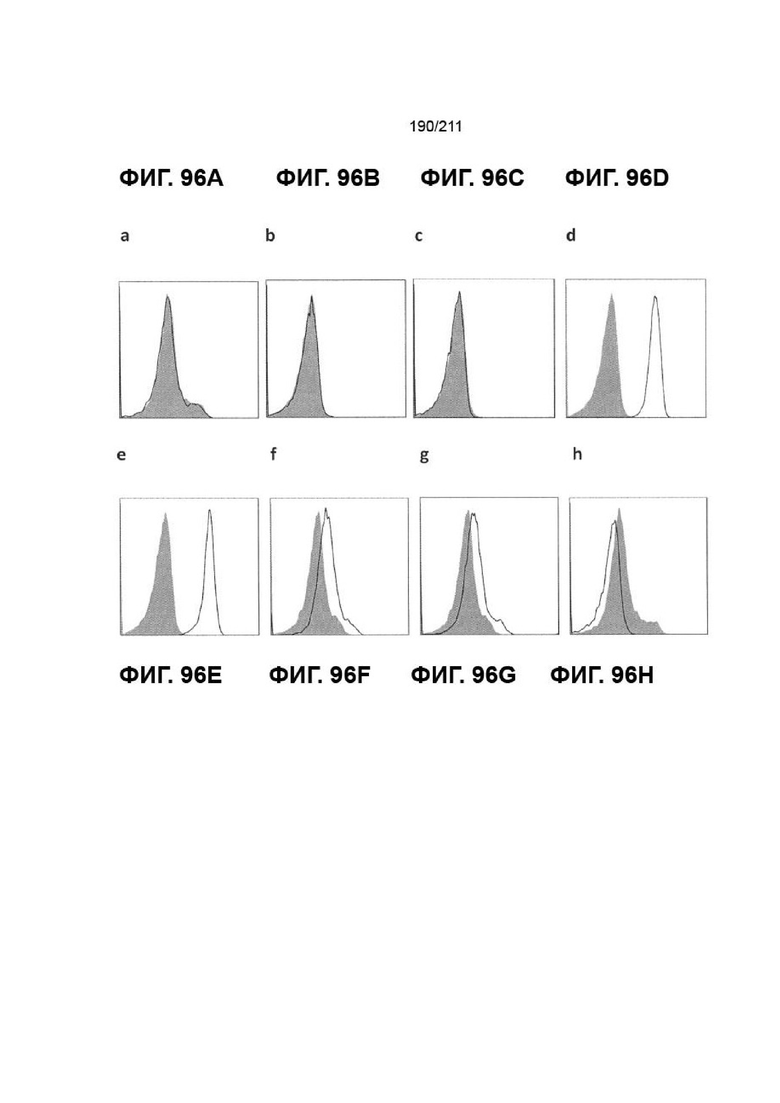
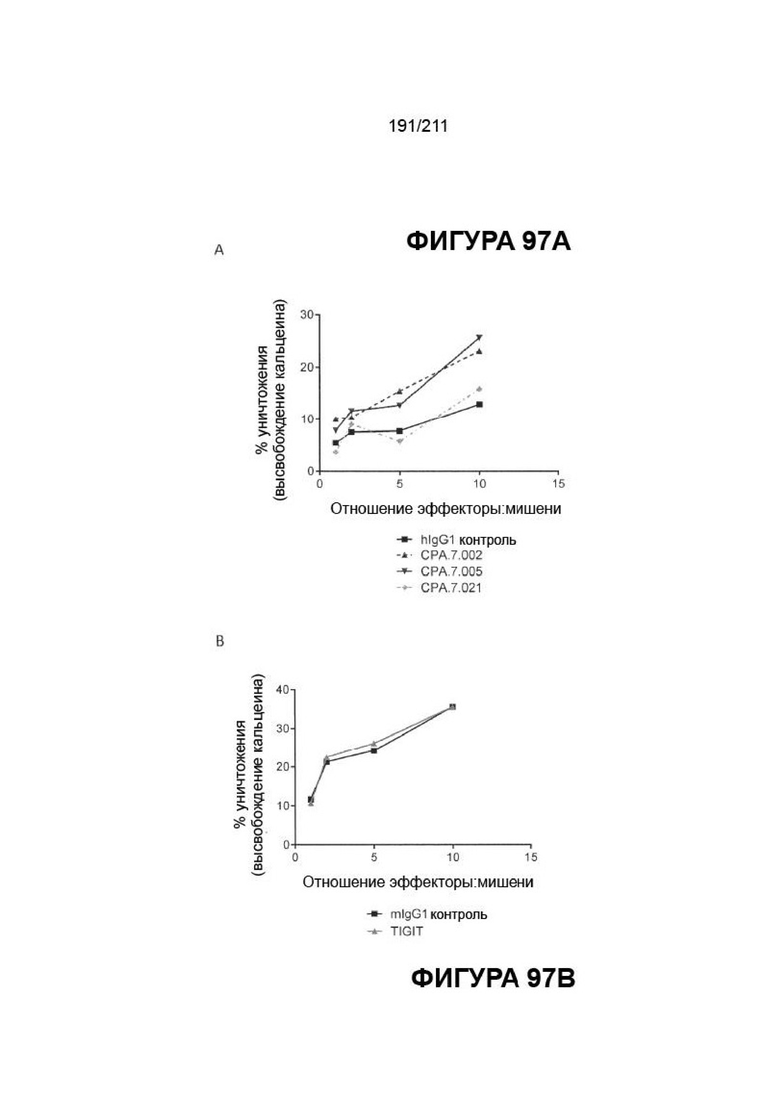
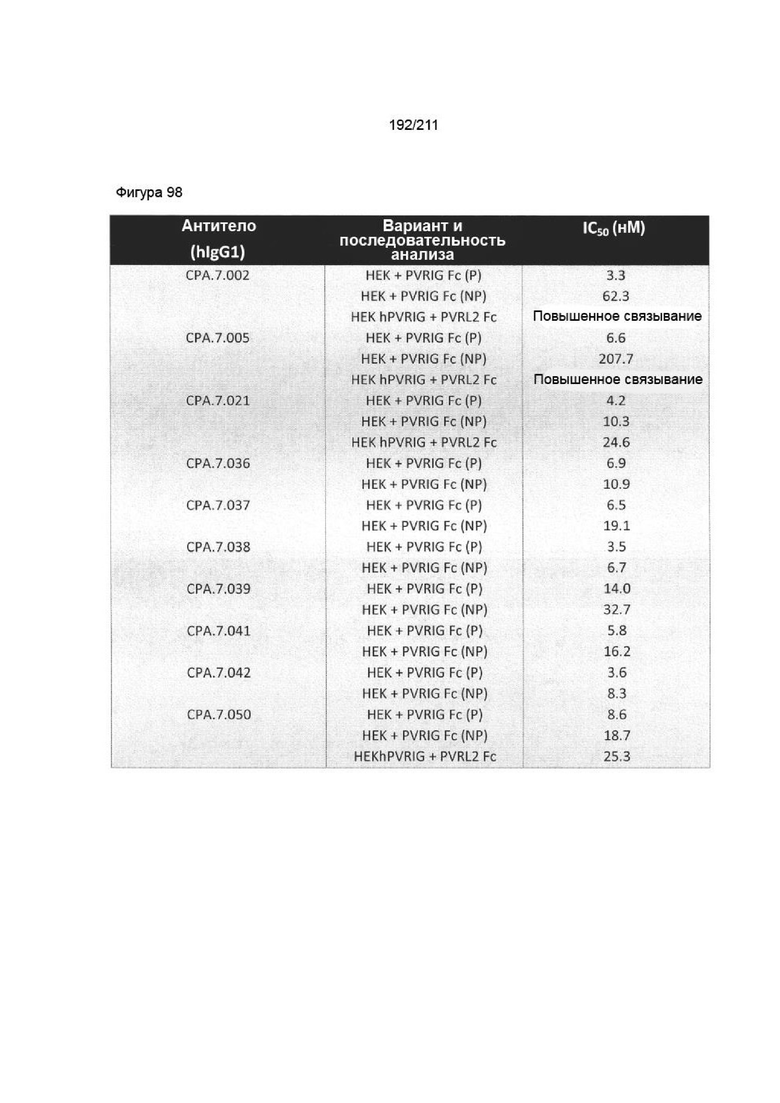
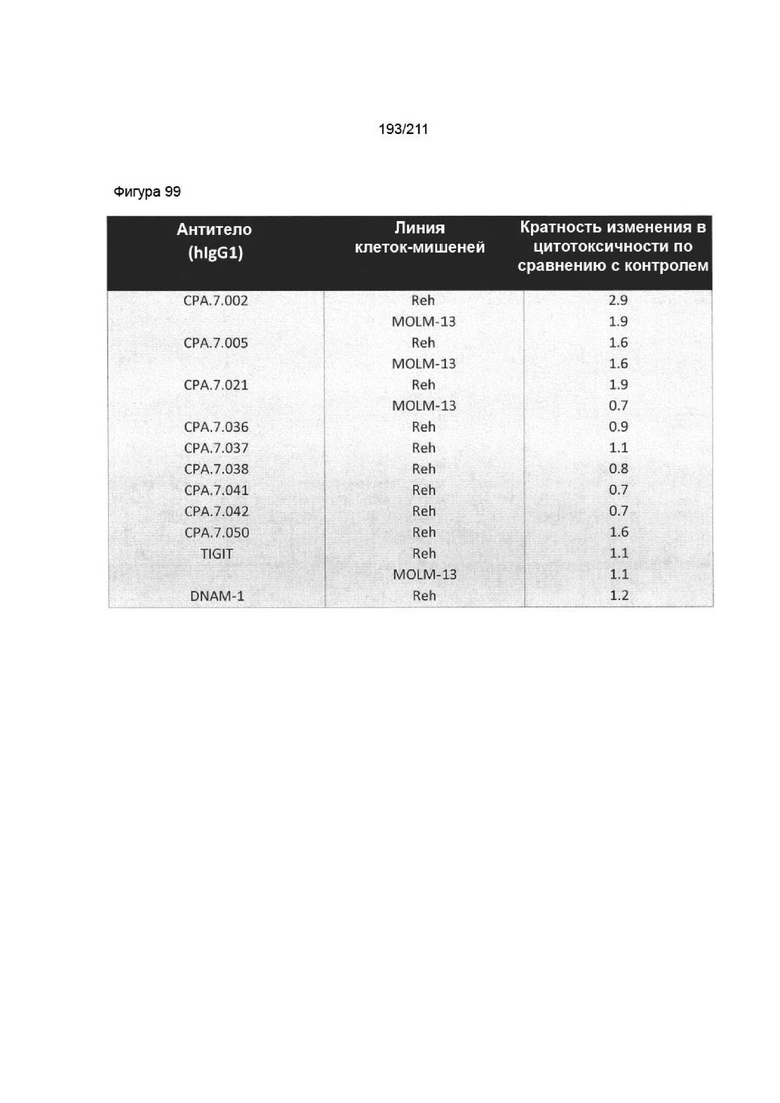
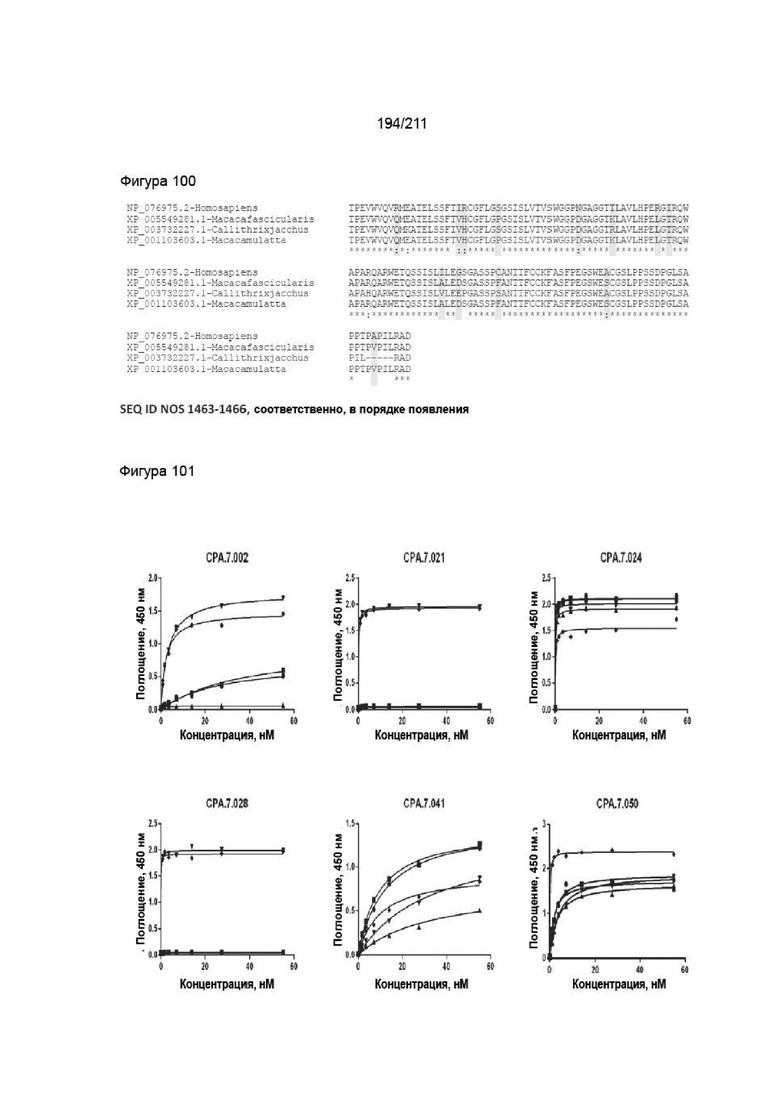
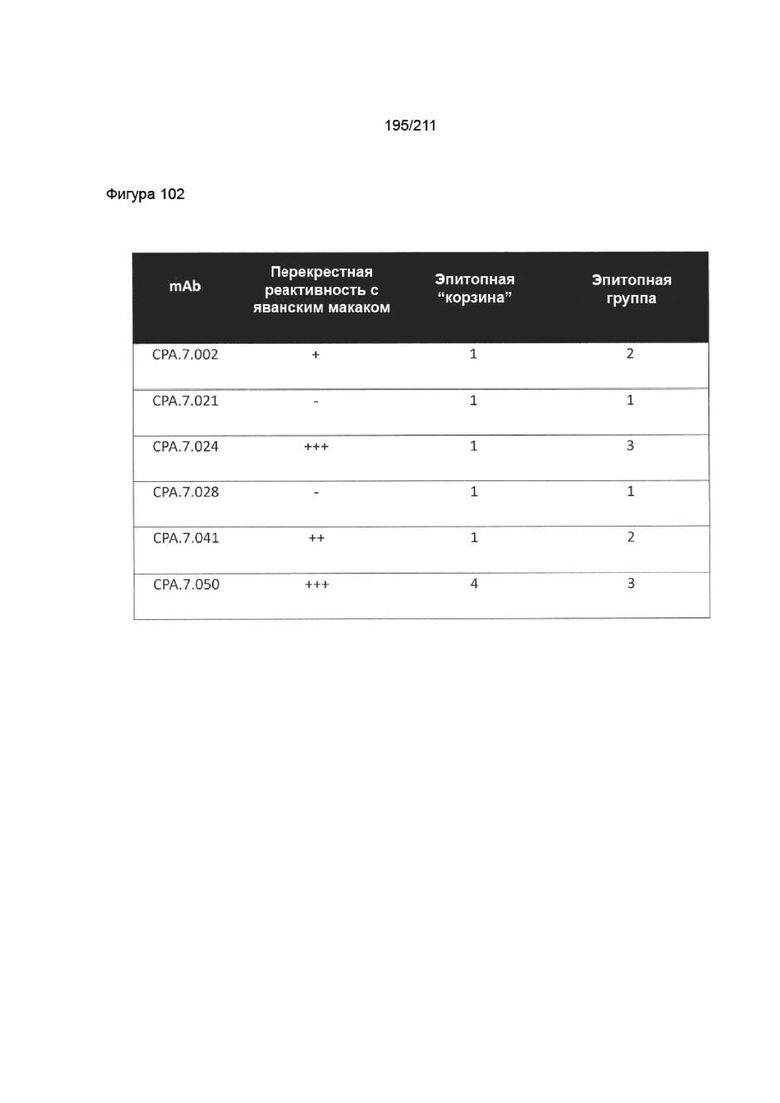
















Группа изобретений относится к медицине и касается применения антитела против PVRIG (родственный рецептору человеческого полиовируса белок, содержащий домен иммуноглобулина), активирующего T-клетки и/или NK-клетки, для лечения рака, характеризующегося экспрессией PVRIG и/или PVRL2. Группа изобретений также касается способа активации Т-клеток пациента, больного раком, характеризующимся экспрессией PVRIG и/или PVRL2, включающего введение указанному пациенту антитела против PVRIG; композиции экспрессионных векторов для экспрессии антитела против PVRIG; клетки-хозяина для экспрессии антитела против PVRIG, содержащей указанную композицию экспрессионных векторов. Группа изобретений обеспечивает лечение рака, характеризующегося экспрессией PVRIG и/или PVRL2. 18 н. и 40 з.п. ф-лы, 103 ил., 5 табл., 29 пр.
1. Применение антитела против PVRIG (родственный рецептору человеческого полиовируса белок, содержащий домен иммуноглобулина), активирующего T-клетки и/или NK-клетки, для лечения рака, характеризующегося экспрессией PVRIG и/или PVRL2.
2. Применение по п. 1, в котором указанное антитело против PVRIG включает последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3, выбранные из группы, состоящей из:
I. vhCDR1, как представлено в SEQ ID NO: 877, vhCDR2, как представлено в SEQ ID NO: 878, vhCDR3, как представлено в SEQ ID NO: 879, vlCDR1, как представлено в SEQ ID NO: 881, vlCDR2, как представлено в SEQ ID NO: 882, и vlCDR3, как представлено в SEQ ID NO:883;
II. vhCDR1, как представлено в SEQ ID NO: 885, vhCDR2, как представлено в SEQ ID NO: 886, vhCDR3, как представлено в SEQ ID NO: 887, vlCDR1, как представлено в SEQ ID NO: 889, vlCDR2, как представлено в SEQ ID NO: 890, и vlCDR3, как представлено в SEQ ID NO: 891;
III. vhCDR1, как представлено в SEQ ID NO: 917, vhCDR2, как представлено в SEQ ID NO: 918, vhCDR3, как представлено в SEQ ID NO: 919, vlCDR1, как представлено в SEQ ID NO: 921, vlCDR2, как представлено в SEQ ID NO: 922, и vlCDR3, как представлено в SEQ ID NO: 923;
IV. vhCDR1, как представлено в SEQ ID NO: 941, vhCDR2, как представлено в SEQ ID NO: 942, vhCDR3, как представлено в SEQ ID NO: 943, vlCDR1, как представлено в SEQ ID NO: 945, vlCDR2, как представлено в SEQ ID NO: 946, и vlCDR3, как представлено в SEQ ID NO: 947;
V. vhCDR1, как представлено в SEQ ID NO: 949, vhCDR2, как представлено в SEQ ID NO: 950, vhCDR3, как представлено в SEQ ID NO: 951, vlCDR1, как представлено в SEQ ID NO: 953, vlCDR2, как представлено в SEQ ID NO: 954, и vlCDR3, как представлено в SEQ ID NO: 955;
VI. vhCDR1, как представлено в SEQ ID NO: 973, vhCDR2, как представлено в SEQ ID NO: 974, vhCDR3, как представлено в SEQ ID NO: 975, vlCDR1, как представлено в SEQ ID NO: 977, vlCDR2, как представлено в SEQ ID NO: 978, и vlCDR3, как представлено в SEQ ID NO: 979;
VII. vhCDR1, как представлено в SEQ ID NO: 997, vhCDR2, как представлено в SEQ ID NO: 998, vhCDR3, как представлено в SEQ ID NO: 999, vlCDR1, как представлено в SEQ ID NO: 1001, vlCDR2, как представлено в SEQ ID NO: 1002, и vlCDR3, как представлено в SEQ ID NO: 1003; и
VIII. vhCDR1, как представлено в SEQ ID NO: 1037, vhCDR2, как представлено в SEQ ID NO: 1038, vhCDR3, как представлено в SEQ ID NO: 1039, vlCDR1, как представлено в SEQ ID NO: 1041, vlCDR2, как представлено в SEQ ID NO: 1042, и vlCDR3, как представлено в SEQ ID NO: 1043.
3. Применение по любому из пп. 1 и 2, в котором указанное антитело содержит вариабельный домен тяжелой цепи, как указано в SEQ ID NO: 884, и вариабельный домен легкой цепи, как указано в SEQ ID NO: 888.
4. Применение по любому из пп. 1-3, в котором указанное антитело содержит вариабельный домен тяжелой цепи, как указано в SEQ ID NO: 980, и вариабельный домен легкой цепи, как указано в SEQ ID NO: 984.
5. Применение по любому из пп. 1-4, в котором антитело против PVRIG используют в комбинации с иммуностимулирующим антителом, терапией цитокинами или иммуномодулирующим лекарственным средством.
6. Применение по п. 1, в котором антитело против PVRIG содержит либо:
I. i) вариабельный домен тяжелой цепи, содержащий vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1434, и ii) вариабельный домен легкой цепи, содержащий vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1453, или
II. i) вариабельный домен тяжелой цепи SEQ ID NO: 1434 и ii) вариабельный домен легкой цепи SEQ ID NO: 1453.
7. Применение по п. 1, где антитело против PVRIG содержит либо:
I. i) vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1447 и ii) vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1462 или
II. i) вариабельный домен тяжелой цепи SEQ ID NO: 1447 и ii) вариабельный домен легкой цепи SEQ ID NO: 1462.
8. Применение по любому из предыдущих пунктов, где указанные Т-клетки представляют собой цитотоксические Т-клетки (ЦТЛ).
9. Применение по любому из предыдущих пунктов, где указанные Т-клетки выбраны из группы, состоящей из CD4 + Т-клеток и CD8 + Т-клеток.
10. Применение по любому из предыдущих пунктов, где указанную активацию измеряют как увеличение продукции интерферона-γ и/или увеличение секреции провоспалительных цитокинов.
11. Применение по любому из предыдущих пунктов, в котором указанный рак выбран из группы, состоящей из рака простаты, рака печени (ГЦК), колоректального рака, рака яичника, рака эндометрия, рака молочной железы, рака поджелудочной железы, рака желудка, рака шейки матки, рака головы и шеи, рака щитовидной железы, рака яичка, рака уротелия, рака легкого, меланомы, немеланомного рака кожи (плоскоклеточной и базальноклеточной карциномы), глиомы, рака почки (ПКК), лимфомы (НХЛ или ЛХ), острого миелоцитарного лейкоза (ОМЛ), Т-клеточного острого лимфобластного лейкоза (T-ОЛЛ), диффузной крупноклеточной В-клеточной лимфомы, опухоли половых клеток яичка, мезотелиомы и рака пищевода.
12. Применение по любому из предыдущих пунктов, в котором экспрессия PVRIG и/или PVRL2 включает экспрессию PVRIG и/или PVRL2 на лимфоцитах внутри микроокружения опухоли.
13. Способ активации Т-клеток пациента, больного раком, характеризующимся экспрессией PVRIG и/или PVRL2, включающий введение указанному пациенту антитела против PVRIG, где указанное антитело содержит одно из следующего:
I. i) вариабельный домен тяжелой цепи, содержащий vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1434, и ii) вариабельный домен легкой цепи, содержащий vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1453;
II. i) вариабельный домен тяжелой цепи SEQ ID NO: 1434 и ii) вариабельный домен легкой цепи SEQ ID NO: 1453;
III. i) vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1447 и ii) vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1462;
IV. i) вариабельный домен тяжелой цепи SEQ ID NO: 1447 и ii) вариабельный домен легкой цепи SEQ ID NO: 1462 или
V. а) вариабельный домен тяжелой цепи, содержащий: i) vhCDR1, содержащий SEQ ID NO: 885; ii) vhCDR2, содержащий SEQ ID NO: 886; iii) vhCDR3, содержащий SEQ ID NO: 887; и b) вариабельный домен легкой цепи, содержащий: i) vlCDR1, содержащий SEQ ID NO: 889; ii) vlCDR2, содержащий SEQ ID NO: 890; iii) vlCDR3, содержащий SEQ ID NO: 891;
причем происходит активация субпопуляции Т-клеток указанного пациента.
14. Способ по п. 13, в котором указанное антитело дополнительно содержит область CH1-шарнир-CH2-CH3 из IgG1, IgG2, IgG3 или IgG4, где указанная шарнирная область необязательно содержит мутации.
15. Способ по п. 13, в котором указанное антитело дополнительно содержит область CL легкой цепи каппа-2 человека.
16. Способ по п. 14 или 15, в котором указанное антитело содержит:
I. a) тяжелую цепь, содержащую: i) VH-CH1-шарнир-CH2-CH3, где VH представляет собой SEQ ID NO: 1434 и где область CH1-шарнир-CH2-CH3 происходит из IgG4; и
b) легкую цепь, содержащую: i) VL-CL, где VL представляет собой SEQ ID NO: 1453 и где область CL происходит из легкой цепи каппа-2 человека; или
II. а) тяжелую цепь, содержащую: i) VH-CH1-шарнир-CH2-CH3, где VH представляет собой SEQ ID NO: 1447 и где область CH1-шарнир-CH2-CH3 происходит из IgG4; и
b) легкую цепь, содержащую: i) VL-CL, где VL представляет собой SEQ ID NO: 1462 и где область CL происходит из легкой цепи каппа-2 человека.
17. Способ по п. 13, в котором указанные Т-клетки представляют собой цитотоксические Т-клетки (ЦТЛ).
18. Способ по п. 13, в котором указанные Т-клетки выбраны из группы, состоящей из Т-клеток CD4 + и CD8 +.
19. Способ по п. 13, в котором указанная активация измеряется по повышению выработки интерферона-γ и/или секреции цитокинов.
20. Способ по любому из пп. 13-19, отличающийся тем, что указанный рак выбран из группы, состоящей из рака простаты, рака печени (ГЦК), колоректального рака, рака яичника, рака эндометрия, рака молочной железы, рака поджелудочной железы, рака желудка, рака шейки матки, рака головы и шеи, рака щитовидной железы, рака яичка, рака уротелия, рака легкого, меланомы, немеланомного рака кожи (плоскоклеточной и базальноклеточной карциномы), глиомы, рака почки (ПКК), лимфомы (НХЛ или ЛХ), острого миелоцитарного лейкоза (ОМЛ), Т-клеточного острого лимфобластного лейкоза (T-ОЛЛ), диффузной крупноклеточной В-клеточной лимфомы, опухоли половых клеток яичка, мезотелиомы и рака пищевода.
21. Способ по любому из пп. 13-20, в котором экспрессия PVRIG и/или PVRL2 включает экспрессию PVRIG и/или PVRL2 на лимфоцитах внутри микроокружения опухоли.
22. Применение комбинации антитела против PD-1 и антитела против PVRIG для активации Т-клеток у пациента, больного раком, характеризующимся экспрессией PVRIG и/или PVRL2, где указанное антитело против PVRIG включает одно из следующего:
I. i) вариабельный домен тяжелой цепи, содержащий vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1434, и ii) вариабельный домен легкой цепи, содержащий vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1453;
II. i) вариабельный домен тяжелой цепи SEQ ID NO: 1434 и ii) вариабельный домен легкой цепи SEQ ID NO: 1453;
III. i) vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1447 и ii) vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1462;
IV. i) вариабельный домен тяжелой цепи SEQ ID NO: 1447 и ii) вариабельный домен легкой цепи SEQ ID NO: 1462; или
V. а) вариабельный домен тяжелой цепи, содержащий: i) vhCDR1, содержащий SEQ ID NO: 885; ii) vhCDR2, содержащий SEQ ID NO: 886; iii) vhCDR3, содержащий SEQ ID NO: 887; и b) вариабельный домен легкой цепи, содержащий: i) vlCDR1, содержащий SEQ ID NO: 889; ii) vlCDR2, содержащий SEQ ID NO: 890; iii) vlCDR3, содержащий SEQ ID NO: 891;
причем происходит активация субпопуляции Т-клеток указанного пациента.
23. Применение по п. 22, где указанное антитело против PVRIG содержит область CH1-шарнир-CH2-CH3 из IgG1, IgG2, IgG3 или IgG4, где указанная шарнирная область необязательно содержит мутации.
24. Применение по п. 22, где указанное антитело против PVRIG содержит область CL легкой цепи каппа-2 человека.
25. Применение по п. 23 или 24, где указанное антитело содержит:
I. a) тяжелую цепь, содержащую: i) VH-CH1-шарнир-CH2-CH3, где VH представляет собой SEQ ID NO: 1434 и где область CH1-шарнир-CH2-CH3 происходит из IgG4; и b) легкую цепь, содержащую: i) VL-CL, где VL представляет собой SEQ ID NO: 1453 и где область CL происходит из легкой цепи каппа-2 человека; или
II. а) тяжелую цепь, содержащую: i) VH-CH1-шарнир-CH2-CH3, где VH представляет собой SEQ ID NO: 1447 и где область CH1-шарнир-CH2-CH3 происходит из IgG4; и b) легкую цепь, содержащую: i) VL-CL, где VL представляет собой SEQ ID NO: 1462 и где область CL происходит из легкой цепи каппа-2 человека.
26. Применение по п. 22, где указанные Т-клетки представляют собой цитотоксические Т-клетки (ЦТЛ).
27. Применение по п. 22, в котором указанные Т-клетки выбраны из группы, состоящей из Т-клеток CD4 + и CD8 +.
28. Применение по п. 22, в котором указанную активацию измеряют как увеличение продукции интерферона-γ и/или увеличение секреции цитокинов.
29. Применение по п. 22, где указанное антитело против PD-1 представляет собой пембролизумаб.
30. Применение по п. 22, где указанное антитело против PD-1 представляет собой ниволумаб.
31. Применение по любому из пп. 22-30, в котором указанный рак выбран из группы, состоящей из рака простаты, рака печени (ГЦК), колоректального рака, рака яичника, рака эндометрия, рака молочной железы, рака поджелудочной железы, рака желудка, рака шейки матки, рака головы и шеи, рака щитовидной железы, рака яичка, рака уротелия, рака легкого, меланомы, немеланомного рака кожи (плоскоклеточной и базальноклеточной карциномы), глиомы, рака почки (ПКК), лимфомы (НХЛ или ЛХ), острого миелоцитарного лейкоза (ОМЛ), Т-клеточного острого лимфобластного лейкоза (T-ОЛЛ), диффузной крупноклеточной В-клеточной лимфомы, опухоли половых клеток яичка, мезотелиомы и рака пищевода.
32. Применение по любому из пп. 22-31, в котором экспрессия PVRIG и/или PVRL2 включает экспрессию PVRIG и/или PVRL2 на лимфоцитах внутри микроокружения опухоли.
33. Способ активации Т-клеток пациента, больного раком, характеризующимся экспрессией PVRIG и/или PVRL2, включающий введение указанному пациенту антитела против PD-1 и антитела против PVRIG, где антитело против PVRIG включает одно из следующего:
I. i) вариабельный домен тяжелой цепи, содержащий vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1434, и ii) вариабельный домен легкой цепи, содержащий vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1453;
II. i) вариабельный домен тяжелой цепи SEQ ID NO: 1434 и ii) вариабельный домен легкой цепи SEQ ID NO: 1453;
III. i) vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1447 и ii) vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1462;
IV. i) вариабельный домен тяжелой цепи SEQ ID NO: 1447 и ii) вариабельный домен легкой цепи SEQ ID NO: 1462; или
V. a) вариабельный домен тяжелой цепи, содержащий: i) vhCDR1, содержащий SEQ ID NO: 885; ii) vhCDR2, содержащий SEQ ID NO: 886; iii) vhCDR3, содержащий SEQ ID NO: 887; и b) вариабельный домен легкой цепи, содержащий: i) vlCDR1, содержащий SEQ ID NO: 889; ii) vlCDR2, содержащий SEQ ID NO: 890; iii) vlCDR3, содержащий SEQ ID NO: 891,
причем происходит активация субпопуляции Т-клеток указанного пациента.
34. Способ по п. 33, в котором указанное антитело дополнительно содержит область CH1-шарнир-CH2-CH3 из IgG1, IgG2, IgG3 или IgG4, где указанная шарнирная область необязательно содержит мутации.
35. Способ по п. 33, в котором указанное антитело против PVRIG дополнительно содержит область CL легкой цепи каппа-2 человека.
36. Способ по п. 34 или 35, в котором указанное антитело содержит:
I. a) тяжелую цепь, содержащую: i) VH-CH1-шарнир-CH2-CH3, где VH представляет собой SEQ ID NO: 1434 и где область CH1-шарнир-CH2-CH3 происходит из IgG4; и
b) легкую цепь, содержащую: i) VL-CL, где VL представляет собой SEQ ID NO: 1453 и где область CL происходит из легкой цепи каппа-2 человека; или
II. а) тяжелую цепь, содержащую: i) VH-CH1-шарнир-CH2-CH3, где VH представляет собой SEQ ID NO: 1447 и где область CH1-шарнир-CH2-CH3 происходит из IgG4; и b) легкую цепь, содержащую: i) VL-CL, где VL представляет собой SEQ ID NO: 1462 и где область CL происходит из легкой цепи каппа-2 человека.
37. Способ по любому из пп. 33-36, в котором указанные Т-клетки представляют собой цитотоксические Т-клетки (ЦТЛ).
38. Способ по п. 33, в котором указанные Т-клетки выбраны из группы, состоящей из Т-клеток CD4 + и CD8 +.
39. Способ по п. 33, в котором указанная активация измеряется по повышению выработки интерферона-γ и/или секреции цитокинов.
40. Способ по п. 33, где указанное антитело против PD-1 представляет собой пембролизумаб.
41. Способ по п. 33, где указанное антитело против PD-1 представляет собой ниволумаб.
42. Способ по любому из пп. 33-41, в в котором указанный рак выбран из группы, состоящей из рака простаты, рака печени (ГЦК), колоректального рака, рака яичника, рака эндометрия, рака молочной железы, рака поджелудочной железы, рака желудка, рака шейки матки, рака головы и шеи, рака щитовидной железы, рака яичка, рака уротелия, рака легкого, меланомы, немеланомного рака кожи (плоскоклеточной и базальноклеточной карциномы), глиомы, рака почки (ПКК), лимфомы (НХЛ или ЛХ), острого миелоцитарного лейкоза (ОМЛ), Т-клеточного острого лимфобластного лейкоза (T-ОЛЛ), диффузной крупноклеточной В-клеточной лимфомы, опухоли половых клеток яичка, мезотелиомы и рака пищевода.
43. Способ по любому из пп. 33-42, в котором экспрессия PVRIG и/или PVRL2 включает экспрессию PVRIG и/или PVRL2 на лимфоцитах внутри микроокружения опухоли.
44. Антитело против PVRIG, активирующее Т-клетки пациента, больного раком, экспрессирующим PVRIG, содержащее последовательности vhCDR1, vhCDR2, vhCDR3, vlCDR1, vlCDR2 и vlCDR3, выбранные из группы, состоящей из:
I. vhCDR1, как представлено в SEQ ID NO: 877, vhCDR2, как представлено в SEQ ID NO: 878, vhCDR3, как представлено в SEQ ID NO: 879, vlCDR1, как представлено в SEQ ID NO: 881, vlCDR2, как представлено в SEQ ID NO: 882, и vlCDR3, как представлено в SEQ ID NO: 883;
II. vhCDR1, как представлено в SEQ ID NO: 885, vhCDR2, как представлено в SEQ ID NO: 886, vhCDR3, как представлено в SEQ ID NO: 887, vlCDR1, как представлено в SEQ ID NO: 889, vlCDR2, как представлено в SEQ ID NO: 890, и vlCDR3, как представлено в SEQ ID NO: 891;
III. vhCDR1, как представлено в SEQ ID NO: 917, vhCDR2, как представлено в SEQ ID NO: 918, vhCDR3, как представлено в SEQ ID NO: 919, vlCDR1, как представлено в SEQ ID NO: 921, vlCDR2, как представлено в SEQ ID NO: 922, и vlCDR3, как представлено в SEQ ID NO: 923;
IV. vhCDR1, как представлено в SEQ ID NO: 941, vhCDR2, как представлено в SEQ ID NO: 942, vhCDR3, как представлено в SEQ ID NO: 943, vlCDR1, как представлено в SEQ ID NO: 945, vlCDR2, как представлено в SEQ ID NO: 946, и vlCDR3, как представлено в SEQ ID NO: 947;
V. vhCDR1, как представлено в SEQ ID NO: 949, vhCDR2, как представлено в SEQ ID NO: 950, vhCDR3, как представлено в SEQ ID NO: 951, vlCDR1, как представлено в SEQ ID NO: 953, vlCDR2, как представлено в SEQ ID NO: 954, и vlCDR3, как представлено в SEQ ID NO: 955;
VI. vhCDR1, как представлено в SEQ ID NO: 973, vhCDR2, как представлено в SEQ ID NO: 974, vhCDR3, как представлено в SEQ ID NO: 975, vlCDR1, как представлено в SEQ ID NO: 977, vlCDR2, как представлено в SEQ ID NO: 978, и vlCDR3, как представлено в SEQ ID NO: 979;
VII. vhCDR1, как представлено в SEQ ID NO: 997, vhCDR2, как представлено в SEQ ID NO: 998, vhCDR3, как представлено в SEQ ID NO: 999, vlCDR1, как представлено в SEQ ID NO: 1001, vlCDR2, как представлено в SEQ ID NO: 1002, и vlCDR3, как представлено в SEQ ID NO: 1003; и
VIII. vhCDR1, как представлено в SEQ ID NO: 1037, vhCDR2, как представлено в SEQ ID NO: 1038, vhCDR3, как представлено в SEQ ID NO: 1039, vlCDR1, как представлено в SEQ ID NO: 1041, vlCDR2, как представлено в SEQ ID NO: 1042, и vlCDR3, как представлено в SEQ ID NO: 1043.
45. Антитело против PVRIG, активирующее Т-клетки у пациента, больного раком, экспрессирующим PVRIG и/или PVRL2, содержащее:
i) вариабельный домен тяжелой цепи SEQ ID NO: 1434 и
ii) вариабельный домен легкой цепи SEQ ID NO: 1453.
46. Композиция нуклеиновых кислот, кодирующая антитело против PVRIG по п. 45, содержащая:
a) первую нуклеиновую кислоту, кодирующую вариабельный домен тяжелой цепи из SEQ ID NO: 1434; и
b) вторую нуклеиновую кислоту, кодирующую вариабельный домен легкой цепи из SEQ ID NO: 1453.
47. Композиция экспрессионных векторов для экспрессии антитела против PVRIG по п. 45, содержащая:
a) первый экспрессионный вектор, содержащий указанную первую нуклеиновую кислоту композиции по п. 46; и
b) второй экспрессионный вектор, содержащий указанную вторую нуклеиновую кислоту композиции по п. 46.
48. Композиция экспрессионных векторов для экспрессии антитела против PVRIG по п. 45, содержащая экспрессионный вектор, содержащий указанную первую нуклеиновую кислоту композиции по п. 46 и указанную вторую нуклеиновую кислоту композиции по п. 46.
49. Клетка-хозяин для экспрессии антитела против PVRIG по п. 45, содержащая композицию экспрессионных векторов по п. 47 или 48.
50. Способ получения антитела против PVRIG, включающий:
a) культивирование клетки-хозяина по п. 49 в условиях, в которых экспрессируется указанное антитело; и
b) выделение указанного антитела.
51. Антитело против PVRIG, активирующее Т-клетки пациента, больного раком, экспрессирующим PVRIG, включающее:
i) вариабельный домен тяжелой цепи SEQ ID NO: 1447 и
ii) вариабельный домен легкой цепи SEQ ID NO: 1462.
52. Композиция нуклеиновых кислот, кодирующая антитело против PVRIG по п. 51, содержащая:
а) первую нуклеиновую кислоту, кодирующую вариабельный домен тяжелой цепи SEQ ID NO: 1447; и
б) вторую нуклеиновую кислоту, кодирующую вариабельный домен легкой цепи SEQ ID NO: 1462.
53. Композиция экспрессионных векторов для экспрессии антитела против PVRIG по п. 51, содержащая:
а) первый вектор экспрессии, содержащий указанную первую нуклеиновую кислоту композиции по п. 52; и
б) второй вектор экспрессии, содержащий указанную вторую нуклеиновую кислоту композиции по п. 52.
54. Композиция экспрессионных векторов для экспрессии антитела против PVRIG по п. 51, содержащая вектор экспрессии, включающий указанную первую нуклеиновую кислоту композиции по п. 52 и указанную вторую нуклеиновую кислоту композиции по п. 52.
55. Клетка-хозяин для экспрессии антитела против PVRIG по п. 51, содержащая композицию экспрессионных векторов по п. 53 или 54.
56. Способ получения антитела против PVRIG по п. 51, включающий:
а) культивирование клетки-хозяина по п. 55 в условиях, в которых указанное антитело экспрессируется; и
б) выделение указанного антитела.
57. Композиция для лечения рака, характеризующегося экспрессией PVRIG и/или PVRL2, содержащая антитело против PVRIG, активирующее Т-клетки и/или NK-клетки, где указанное антитело против PVRIG содержит одно из следующего:
I. i) вариабельный домен тяжелой цепи, содержащий vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1434, и ii) вариабельный домен легкой цепи, содержащий vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1453;
II. i) вариабельный домен тяжелой цепи SEQ ID NO: 1434 и ii) вариабельный домен легкой цепи SEQ ID NO: 1453;
III. i) vhCDR1, vhCDR2 и vhCDR3 из SEQ ID NO: 1447 и ii) vlCDR1, vlCDR2 и vlCDR3 из SEQ ID NO: 1462;
IV. i) вариабельный домен тяжелой цепи SEQ ID NO: 1447 и ii) вариабельный домен легкой цепи SEQ ID NO: 1462; или
V. а) вариабельный домен тяжелой цепи, содержащий: i) vhCDR1, содержащий SEQ ID NO: 885; ii) vhCDR2, содержащий SEQ ID NO: 886; iii) vhCDR3, содержащий SEQ ID NO: 887; и b) вариабельный домен легкой цепи, содержащий: i) vlCDR1, содержащий SEQ ID NO: 889; ii) vlCDR2, содержащий SEQ ID NO: 890; iii) vlCDR3, содержащий SEQ ID NO: 891.
58. Композиция по п. 57, в которой экспрессия PVRIG и/или PVRL2 включает экспрессию PVRIG и/или PVRL2 на лимфоцитах внутри микроокружения опухоли.
| WO2013184912 A2, 12.12.2013 | |||
| WO2009017679 A2, 05.02.2009 | |||
| WO2012178128 A1, 27.12.2012 | |||
| CHEN Y., et al., Development and characterization of monoclonal antibodies against pancreatic cancer marker hippocalcin-like 1 protein.Monoclon Antib Immunodiagn Immunother | |||
| Способ защиты переносных электрических установок от опасностей, связанных с заземлением одной из фаз | 1924 |
|
SU2014A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
Авторы
Даты
2020-09-10—Публикация
2016-02-19—Подача