ОБЛАСТЬ ТЕХНИКИ
Настоящее изобретение относится к области медицины и вычислительной техники, в частности, к способу идентификации участков связывания белковых комплексов с низкомолекулярными химическими соединениями, на основе структурной и временной информации с использованием машинного обучения.
УРОВЕНЬ ТЕХНИКИ
Из уровня техники известен источник информации WO2002031510A1, опубл. 18.04.2002, который относится к области молекулярного распознавания или обнаружения участков прерывистого или конформационного связывания или эпитопов, соответствующих связывающей молекуле, в частности, в отношении взаимодействий белок-белок, белок-нуклеиновая кислота, нуклеиновая кислота-нуклеиновая кислота или биомолекула-лиганд. Способ обеспечивает синтетическую молекулярную библиотеку, позволяющую тестировать, идентифицировать, характеризовать или обнаруживать прерывающийся участок связывания, способный взаимодействовать со связывающей молекулой, Указанная библиотека включает множество тестовых объектов, причем каждый тестовый объект содержит по крайней мере один первый сегмент, отмеченный рядом со вторым сегментом, каждый сегмент может быть потенциальной единственной частью участка прерывистого связывания.
Из уровня техники известен источник информации WO2007148130A1, опубл. 27.12.2007, раскрывающий способ и систему для итеративного синтеза de novo, автоматизированный итеративный способ обнаружения лекарств и систему, обеспечивающую быструю идентификацию и синтез новых соединений. Способ итеративного синтеза de novo, включает стадии:
a) выбор соединения-кандидата, имеющего желаемое фармакофорное соответствие начальной структуре;
б) синтез соединения-кандидата;
c) анализ синтезированного соединения и сравнение синтезированного соединения с начальной структурой, чтобы определить, обладает ли синтезированное соединение синтетически желательными свойствами, при этом, если синтезированное соединение не имеет синтетически желаемых свойств, стадию a) повторяют для нового соединения-кандидата, и если синтезированное соединение действительно имеет синтетически желаемые свойства, то выполняется стадия d);
d) повторение этапов с а) по в), на которых синтезированное соединение используется в качестве начальной структуры этапа а) до исчерпания.
Предлагаемое изобретение отличается от известных из уровня техники тем, что использует методы машинного обучения для обнаружения объектов, предлагаемый способ представляет трехмерную структуру белка в виде трехмерного изображения с каналами, соответствующими плотностям атомов различных типов. Предлагаемый способ исследует динамику и гибкость белков с помощью крупномасштабного анализа конформационных ансамблей. Обнаруженные конформации с наблюдаемым интересующим участком связывания затем могут быть использованы для подходов к разработке лекарств на основе структуры, таких как молекулярный стыковка и скрининг виртуального лиганда, а также для разработки лекарств de novo на основе структуры.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Технической проблемой, на решение которой направлено заявленное изобретение, является создание способа идентификации новых участков связывания белковых комплексов (состоящих из одной или более молекул различной природы, но не менее одной белковой молекулы) с низкомолекулярными химическими соединениями, на основе структурной и временной информации с использованием машинного обучения в независимом пункте формулы. Дополнительные варианты реализации настоящего изобретения представлены в зависимых пунктах изобретения.
Технический результат заключается в идентификации участков связывания белковых комплексов с низкомолекулярными химическими соединениями. Технический результат также заключается в реализации предлагаемого способа.
Заявленный результат достигается за счет осуществления способа идентификации участков связывания белковых комплексов с низкомолекулярными химическими соединениями, на основе структурной информации, с использованием методов машинного обучения, содержащий этапы, на которых:
получают тензорное представление пространственной структуры белкового комплекса;
осуществляют предобработку пространственной структуры белкового комплекса, в результате которой получают трехмерную воксельную сетку структуры белкового комплекса, где каналы данных вокселей соответствуют плотностям атомов, входящих в состав белкового комплекса;
трехмерную воксельную сетку структуры белкового комплекса разбивают на кубические сетки меньшего размера, покрывающие исходную трехмерную воксельную сетку;
каждую кубическую сетку анализируют с помощью алгоритма машинного обучения для прогнозирования центра участка связывания низкомолекулярного соединения в ячейках кубической сетки;
каждой ячейке в кубических сетках ставят в соответствие действительное число в диапазоне от 0 до 1, где 1 соответствует максимальной оценке достоверности предсказания;
получают результаты предсказания центров участков связывания для каждой кубической сетки с оценками достоверности предсказания, используя кластеризацию предсказаний для ячеек, соответствующих одному участку белкового комплекса;
осуществляют идентификацию аминокислотных остатков участков связывания в заданном расстоянии относительно предсказанного центра участка связывания;
полученные предсказания и идентифицированные аминокислотные остатки для набора различных конформаций белковых комплексов группируют с использованием алгоритмов кластеризации по предсказаниям по каждой входной структуре белкового комплекса.
В частном варианте реализации предлагаемого способа, входные данные представляют собой набор упорядоченных по времени конформаций, при этом реализована кластеризация предсказаний от каждой конформации. Таким образом, получают тензорное представление нескольких атомарных структур белкового комплекса, полученных в разные промежутки времени, и для каждой такой структуры реализуют кластеризацию предсказаний.
В частном варианте реализации предлагаемого способа низкомолекулярное соединение является лекарственным средством.
ОПИСАНИЕ ЧЕРТЕЖЕЙ
Реализация изобретения будет описана в дальнейшем в соответствии с прилагаемыми чертежами, которые представлены для пояснения сути изобретения и никоим образом не ограничивают область изобретения. К заявке прилагаются следующие чертежи:
Фиг.1 иллюстрирует пример осуществления способа.
Фиг. 2 иллюстрирует пример общей схемы вычислительного устройства.
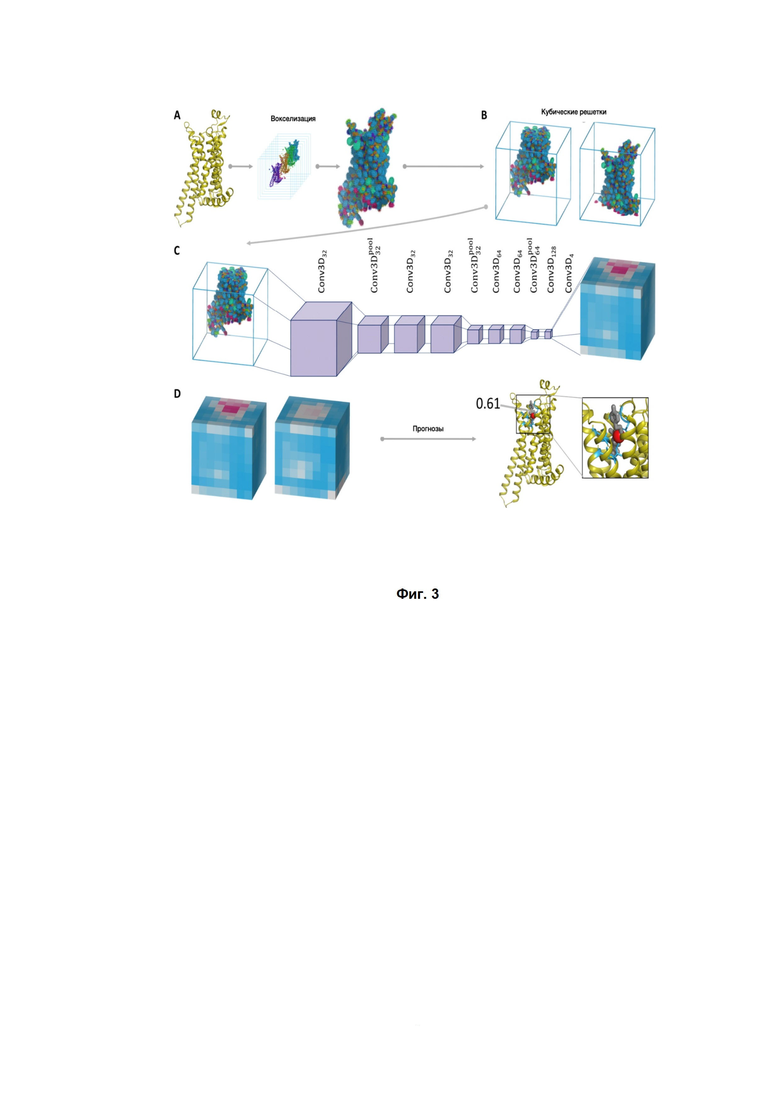
Фиг. 3. Схематическое изображение рабочего процесса BiteNet. (A) Входная трехмерная структура белка представлена сеткой вокселей, где каналы соответствуют атомным плотностям. (B) Воксельная сетка разбита на кубические сетки фиксированного размера, которые будут загружены в нейронную сеть. (C) Каждая кубическая сетка обрабатывается трехмерной сверточной нейронной сетью для прогнозирования сайтов привязки в ячейках фиксированного размера. Ячейки в кубической сетке окрашены в соответствии с доверительной вероятностью от синего до красного. (D) Прогнозы, полученные для каждой кубической сетки и затем обработанные для вывода центра сайта связывания (красная сфера), его вероятностной оценки и аминокислотных остатков в пределах 6Å от прогнозируемого центра (синие палочки). Совместно кристаллизованный лиганд показан серыми полосками.
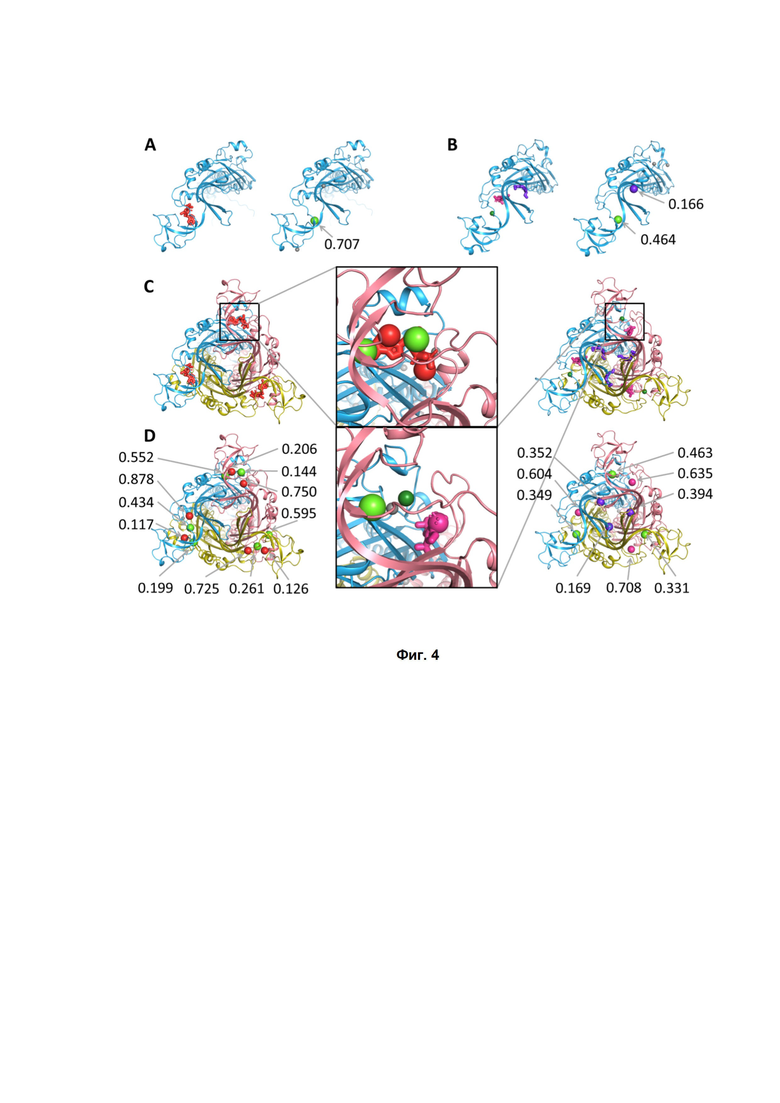
Фиг. 4. Прогнозы BiteNet для мономерной и олигомерной структуры рецептора P2X3. (A) Структура мономера с ортостерическим лигандом и катион-ионом (слева) и прогнозы BiteNet для структуры мономера (справа). (B) Структура мономера с аллостерическим лигандом, катион-ионом и этиленгликолем (слева) и прогнозы BiteNet для этой структуры (справа). (C) Связанная с агонистом (слева) и связанная с антагонистом (справа) структуры тримера P2X3. (D) Прогнозы BiteNet для связанных с агонистами (слева) и антагонистических (справа) структур тримера P2X3. Ортостерические и аллостерические лиганды показаны красными и пурпурными палочками соответственно. катионные ионы показаны темно-зелеными сферами, а молекулы этиленгликоля показаны фиолетовыми полосками. Прогнозы BiteNet для этих молекул показаны в виде сфер с соответствующим цветом.
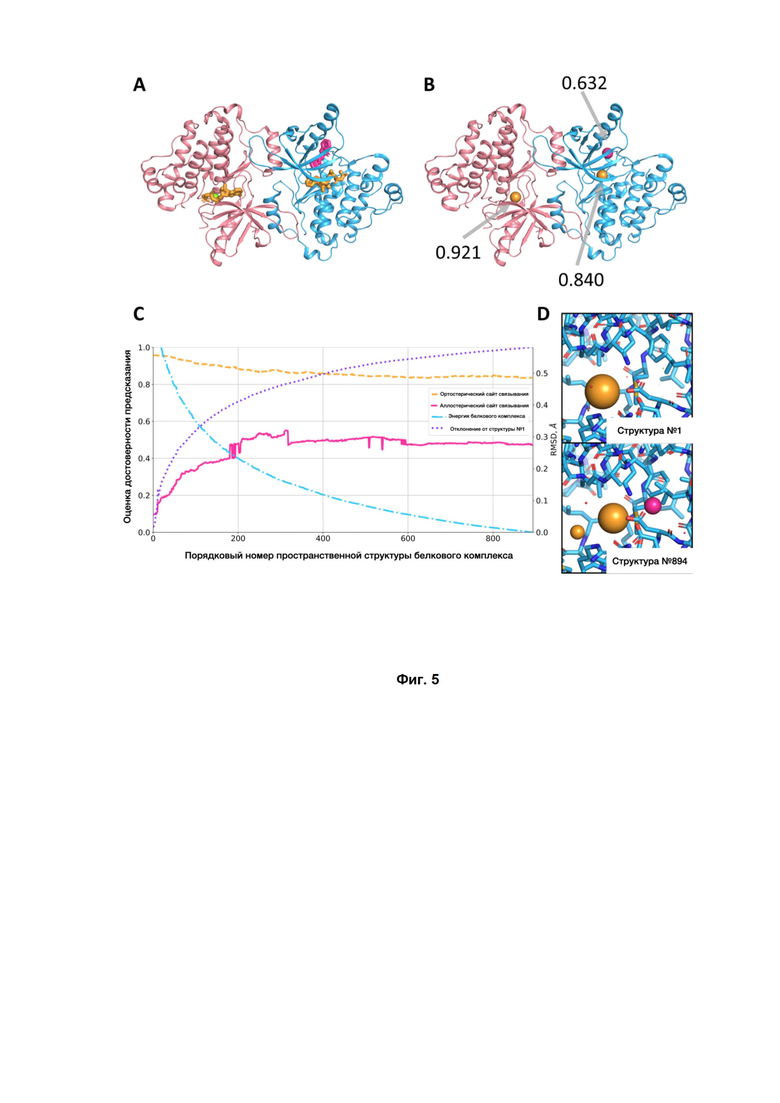
Фиг. 5. (A) Ассиметрическая димерная структура киназного домена EGFR. Ортостерические и аллостерические лиганды показаны желтыми и пурпурными палочками соответственно, ион Mg показан зеленой сферой. (B) Прогнозы BiteNet для асимметричного димера, предсказанные центры для лигандов показаны в виде сфер с соответствующим цветом. (C) Прогнозы BiteNet, полученные для траектории минимизации энергии. Нормализованная энергия показана синей штрихпунктирной линией, среднеквадратичное отклонение по отношению к несвязанной конформации сайта аллотерического связывания показано фиолетовой пунктирной линией, оценка вероятности BiteNet для ортостерических и аллостерических сайтов связывания показаны пунктирным оранжевым и сплошным пурпурным цветом. линии соответственно. Нормированная энергия для 1 и 0 соответствует --7,76969e+5 кДж / моль и -8,80655e+5 кДж / моль соответственно. (D) Начальная и конечная конформации траектории минимизации вместе с прогнозами BiteNet.
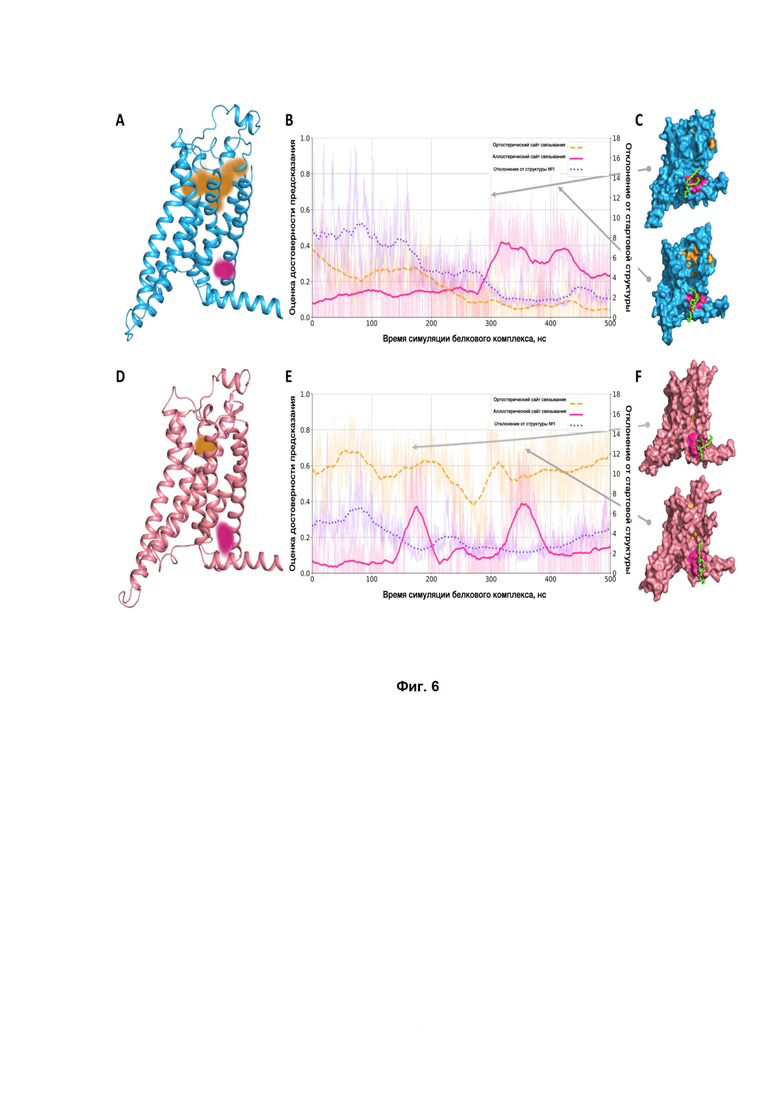
Фиг. 6. Прогнозы BiteNet для моделирования молекулярной динамики аденозинового рецептора A2A. (A, D) Исходные конформации A2A без лиганда и связанные с агонистом соответственно. Оранжевые облака точек соответствуют предсказаниям BiteNet канонического ортостерического сайта связывания в A2A, в то время как пурпурное облако точек соответствует предсказаниям BiteNet гипотетического сайта связывания, наблюдаемым во время моделирования. (B, E) Оценки вероятности BiteNet для ортостерического сайта связывания (пунктирная оранжевая линия), аллостерического сайта связывания (пурпурная сплошная линия) и RMSD по отношению к средней конформации липидного хвоста на основе окна (пунктирная фиолетовая линия), вычисленные для траектории молекулярной динамики. (C, F) Конформации A2A, соответствующие наивысшим оценкам вероятности BiteNet для гипотетического сайта связывания. Связывание липидного хвоста с гипотетическим сайтом связывания показано зелеными палочками.
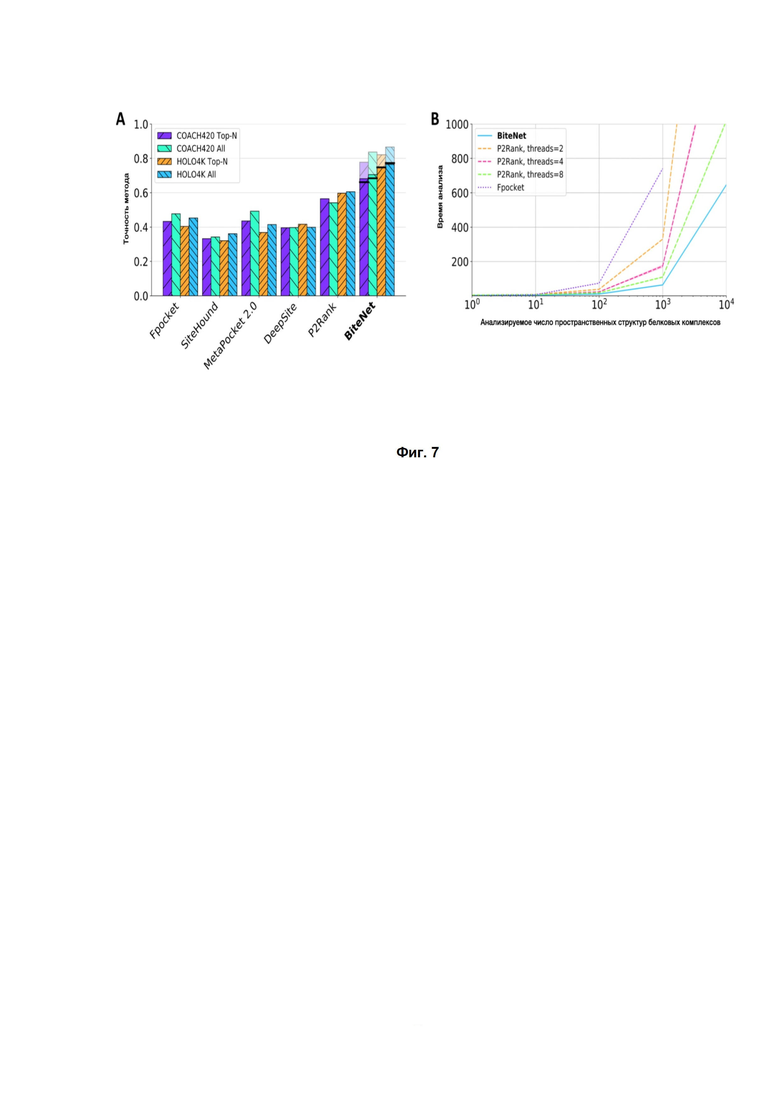
Фиг. 7. (А) Производительность методов прогнозирования сайтов связывания в тестах COACH420 и HOLO4K. All и Top - N соответствуют средней точности, рассчитанной с учетом всех прогнозов и N верхних прогнозов, соответственно, где N - количество истинных сайтов связывания в белке. Светлые столбики соответствуют характеристикам BiteNet, когда истинно положительный сайт связывания определен, как при обучении. Черные линии соответствуют производительности BiteNet по всем тестам. (B) Время, затраченное fpocket, P2Rank и BiteNet на анализ конформаций 1, 10, 1000 и 10000 белка, имеющего около 2000 атомов. Вычисленное затраченное время представляет собой среднее значение 10 независимых прогонов.
ДЕТАЛЬНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
В приведенном ниже подробном описании реализации изобретения приведены многочисленные детали реализации, призванные обеспечить отчетливое понимание настоящего изобретения. Однако, квалифицированному в предметной области специалисту, будет очевидно каким образом можно использовать настоящее изобретение, как с данными деталями реализации, так и без них. В других случаях хорошо известные методы, процедуры и компоненты не были описаны подробно, чтобы не затруднять излишне понимание особенностей настоящего изобретения. Кроме того, из приведенного изложения будет ясно, что изобретение не ограничивается приведенной реализацией. Многочисленные возможные модификации, изменения, вариации и замены, сохраняющие суть и форму настоящего изобретения, будут очевидными для квалифицированных в предметной области специалистов. Используя преимущества методов машинного обучения для обнаружения объектов, предлагаемый способ представляет трехмерную структуру белка в виде трехмерного изображения с каналами, соответствующими плотностям атомов, входящих в состав белковых молекул из белкового комплекса. Предлагаемый способ позволяет идентифицировать участки связывания низкомолекулярных соединений в белковых комплексах с учетом гибкой и динамической природы белковых комплексов посредством анализа конформационных ансамблей. Обнаруженные конформации с наблюдаемым интересующим участком связывания могут быть использованы для подходов к разработке лекарств на основе структуры, включая, но не ограничиваясь такими методами как молекулярный докинг и виртуальный скрининг молекул, а также для разработки лекарств de novo на основе структурной информации.
Под белковым комплексом понимают одну или несколько молекул, находящихся друг относительно друга на расстоянии физического взаимодействия. Типы молекул, входящих в такой комплекс, могут быть различными (одна или несколько молекул полипептидов, нуклеиновые кислоты (ДНК, РНК), низкомолекулярные химические соединения, вода, липиды, ионы и пр.), при этом в методе анализируются молекулы только белковой природы.
Предлагаемый способ идентификации участков связывания белковых комплексов с низкомолекулярными химическими соединениями, на основе структурной и временной информации выполняется на вычислительном устройстве и представлен на фиг.1.
Для реализации предлагаемого изобретения была обучена нейронная сеть.
получают тензорное представление пространственных структур белкового комплекса (101).
осуществляют предобработку пространственных структур белкового комплекса, в результате которой получают трехмерную воксельную сетку структур белкового комплекса, где каналы данных вокселей соответствуют плотностям атомов, входящих в состав белкового комплекса (102).
Изображения белковых комплексов представляют, как 3D-изображения с тремя измерениями (шириной, высотой и длиной) и 11 каналами для каждого вокселя, где каналы соответствуют плотностям атомов, входящих в состав белкового комплекса (процесс вокселизации).
трехмерную воксельную сетку структуры белкового комплекса разбивают на кубические сетки меньшего размера, покрывающие исходную трехмерную воксельную сетку (103).
Для реализации способа использовали кубическую сетку вокселей из 64х64х64 вокселей размером 1Åх1Åх1Å (1Å - ангстрем). Если белок превышает 64 Å в любом из измерений, то используют несколько кубических сеток вокселей, чтобы представить его.
Каждую кубическую сетку анализируют с помощью алгоритма машинного обучения для прогнозирования центра участка связывания низкомолекулярного соединения в ячейках кубической сетки; (104).
Каждой ячейке в кубических сетках ставят в соответствие действительное число в диапазоне от 0 до 1, где 1 соответствует максимальной оценке достоверности предсказания участка связывания белкового комплекса с низкомолекулярным соединением;, а 0 - отсутствию предсказания (105).
Результатом обработки кубических сеток являются 4-ех мерные тензоры (в реализации способа тензоры размерности 8х8х8х4), где первые три измерения соответствуют координатам ячейки относительно кубической сетки вокселей (в реализации способа область 8х8х8 вокселей), а четыре числа последнего измерения соответствуют оценке достоверности предсказания участка связывания в ячейке и его декартовым координатам.
Для получения более точного результата были выбраны следующие гиперпараметры модели сверточной нейронной сети. Размер кубической сетки равен 64 вокселям, 1: 0Å для размера вокселя, 4: 0Å для ограничения плотности, 48 для параметра шага, 16 для размера мини-пакета, 1e-5 и 10.0 для параметров и соответственно. Среди этих параметров размер вокселя имеет большое влияние на скорость вычислений, требуется в 2 раза больше времени для обучения и применения модели с размером вокселя 0: 8 Å по сравнению с размером вокселя 1: 0 Å. С другой стороны, модель, соответствующая размеру вокселя 2: 0 Å, работает быстрее, хотя и менее точна.
В реализации способа используется нейронная сеть, которая состоит из десяти трехмерных сверточных слоев: Conv3D32, Conv3Dpool32, Conv3D32, Conv3D32, Conv3Dpool32, Conv3D64, Conv3D64, Conv3Dpool64, Conv3D128, Conv3D4, где номер индекса обозначает количество конволюционных фильтров. Были использованы ядра размером (3; 3; 3) для каждого слоя, для слоев объединения был использован шаг 2, а также функцию пакетной нормализации и функции активации выпрямленного линейного блока (ReLu) для всех слоев, кроме последнего. Наконец, использовали сигмоидную функцию активации, чтобы получить оценку достоверности предсказания ^s в диапазоне (0,1) и относительные координаты ^ x, ^ y, ^ z предсказанного центра участка связывания относительно клетки.
Затем декартовы координаты вычисляются согласно формуле (2):

где csize и vsize соответствуют размеру ячейки и вокселя соответственно, а Ox, Oy, Oz - декартовы координаты начала кубической сетки.
В реализации способа используется настраиваемая функция потерь, которая содержит три термина (3):
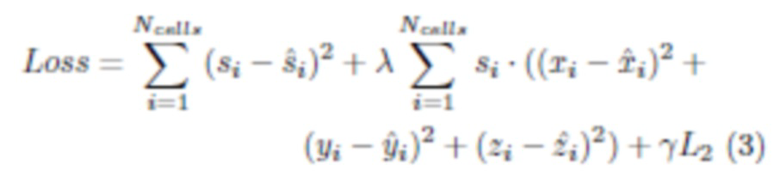
где Ncells - количество ячеек в единой кубической сетке, si и ^si - истинные (0 или 1) и прогнозируемые оценки вероятности ячейки, xi; уi; zi и ^xi; ^ yi ^ zi - истинная и предсказанная координаты i-й ячейки соответственно, а L2 соответствует члену регуляризации. Следовательно, первый и второй члены направлены на штрафы за неправильное предсказание оценки вероятности и центра участка связывания соответственно. Второй член умножается на истинную оценку вероятности (0 или 1), чтобы учесть только релевантные прогнозы. Третий член - это член регуляризации L2 для параметров нейронной сети. Коэффициенты λ= 5 и γ= 1e-5 являются весами штрафных членов.
На следующем этапе шаге применяется постобработка полученных результатов. Сначала отбрасывают все прогнозы с вероятностью ^s <sthreshold. Остальные прогнозы затем обрабатываются методом подавления немаксимумов. Выбирается лучший прогноз с точки зрения оценки достоверности предсказания в качестве начального элемента кластера и помещаются все прогнозы с центрами участка связывания ближе, чем d<dthreshold к центру лучшего прогноза. Затем выбирается второй лучший прогноз в качестве начального элемента для следующего кластера и повторяется описанную выше процедуру до тех пор, пока все прогнозы не будут кластеризованы.
Наконец, в качестве окончательных прогнозов оставляют только начальные элементы Ntop с точки зрения оценок достоверности предсказания.
Получают результаты предсказания центров участков связывания для каждой кубической сетки с оценками достоверности предсказания, используя кластеризацию предсказаний для ячеек, соответствующих одному участку белкового комплекса (106).
Таким образом, входом является пространственные структуры белкового комплекса, а на выходе - центры предсказанных участков связывания вместе с оценками достоверности предсказаний.
Осуществляют идентификацию аминокислотных остатков участков связывания в заданном расстоянии относительно предсказанного центра участка связывания (107).
В реализации способа идентифицируют аминокислотные остатки участка связывания в пределах 6Å соседства по отношению к предсказанному центру.
полученные предсказания и идентифицированные аминокислотные остатки для набора различных конформаций белковых комплексов группируют с использованием алгоритмов кластеризации по предсказаниям по каждой входной структуре белкового комплекса (108).
Используют методы кластеризации в трехмерном пространстве. В реализации способа демонстрируется три различных подхода кластеризации: алгоритм кластеризации среднего сдвига (MSCA), алгоритм кластеризации на основе плотности (DBSCAN) и агломеративный иерархический алгоритм. Первые два подхода в основном применяются для набора точек в евклидовом пространстве, последний подход может применяться также для набора аминокислотных остатков, образующих предсказанный участок связывания.
Присваивают по две оценки каждому кластеру. Первая оценка - это сумма максимальной оценки вероятности кластера в каждой конформации, усредненная по конформационному ансамблю. Для второй оценки средняя сумма оценок вероятностей (больше, чем cluster_score_threshold_step = 0.1) кластера вычисляется для каждой конформации. Затем полученные суммы усредняются по общему количеству конформаций в ансамбле. Использование нескольких подходов к кластеризации обусловлено тем, что результаты кластеризации могут сильно различаться в зависимости от алгоритма кластеризации и различных параметров для них.
На Фиг. 2 далее будет представлена общая схема вычислительного устройства (200), обеспечивающего обработку данных, необходимую для реализации заявленного решения.
В общем случае устройство (200) содержит такие компоненты, как: один или более процессоров (201), по меньшей мере одну память (202), средство хранения данных (203), интерфейсы ввода/вывода (204), средство В/В (205), средства сетевого взаимодействия (206).
Процессор (201) устройства выполняет основные вычислительные операции, необходимые для функционирования устройства (200) или функциональности одного или более его компонентов. Процессор (201) исполняет необходимые машиночитаемые команды, содержащиеся в оперативной памяти (202).
Память (202), как правило, выполнена в виде ОЗУ и содержит необходимую программную логику, обеспечивающую требуемый функционал.
Средство хранения данных (203) может выполняться в виде HDD, SSD дисков, рейд массива, сетевого хранилища, флэш-памяти, оптических накопителей информации (CD, DVD, MD, Blue-Ray дисков) и т.п. Средство (203) позволяет выполнять долгосрочное хранение различного вида информации, например, вышеупомянутых файлов с наборами данных пользователей, базы данных, содержащих записи измеренных для каждого пользователя временных интервалов, идентификаторов пользователей и т.п.
Интерфейсы (204) представляют собой стандартные средства для подключения и работы с серверной частью, например, USB, RS232, RJ45, LPT, COM, HDMI, PS/2, Lightning, FireWire и т.п.
Выбор интерфейсов (204) зависит от конкретного исполнения устройства (200), которое может представлять собой персональный компьютер, мейнфрейм, серверный кластер, тонкий клиент, смартфон, ноутбук и т.п.
В качестве средств В/В данных (205) в любом воплощении системы, реализующей описываемый способ, должна использоваться клавиатура. Аппаратное исполнение клавиатуры может быть любым известным: это может быть, как встроенная клавиатура, используемая на ноутбуке или нетбуке, так и обособленное устройство, подключенное к настольному компьютеру, серверу или иному компьютерному устройству. Подключение при этом может быть, как проводным, при котором соединительный кабель клавиатуры подключен к порту PS/2 или USB, расположенному на системном блоке настольного компьютера, так и беспроводным, при котором клавиатура осуществляет обмен данными по каналу беспроводной связи, например, радиоканалу, с базовой станцией, которая, в свою очередь, непосредственно подключена к системному блоку, например, к одному из USB-портов. Помимо клавиатуры, в составе средств В/В данных также может использоваться: джойстик, дисплей (сенсорный дисплей), проектор, тачпад, манипулятор мышь, трекбол, световое перо, динамики, микрофон и т.п.
Средства сетевого взаимодействия (206) выбираются из устройства, обеспечивающий сетевой прием и передачу данных, например, Ethernet карту, WLAN/Wi-Fi модуль, Bluetooth модуль, BLE модуль, NFC модуль, IrDa, RFID модуль, GSM модем и т.п. С помощью средств (205) обеспечивается организация обмена данными по проводному или беспроводному каналу передачи данных, например, WAN, PAN, ЛВС (LAN), Интранет, Интернет, WLAN, WMAN или GSM.
Компоненты устройства (200) сопряжены посредством общей шины передачи данных (210).
Нижеследующие примеры осуществления способа приведены в целях раскрытия характеристик настоящего изобретения и их не следует рассматривать как каким-либо образом ограничивающие объем изобретения.
Чтобы продемонстрировать применимость разработанного способа (реализация которого далее обозначается как BiteNet), авторы рассмотрели несколько наиболее сложных задач обнаружения сайтов связывания, включающие три фармакологические мишени: рецептор P2X3 из семейства АТФ-управляемых катионных каналов, рецептор эпидермального фактора роста из семейства киназ и рецептор аденозина A2A из семейства рецепторов, сопряженных с G белком.
Схематическое изображение рабочего процесса BiteNet приведено на Фиг. 3.
Пример 1. Пространственно-временное прогнозирование сайтов связывания в катионном канале, управляемом АТФ.
Катионный канал, управляемый АТФ, образованный рецептором P2X3, опосредует различные физиологические процессы и представляет собой фармакологическую мишень для модулирования гипертонии, воспаления, восприятия боли и других состояний. Канал состоит из трех одинаковых мономеров, пересекающих мембрану, а ортостерический АТФ-связывающий сайт состоит из аминокислотных остатков двух мономеров (см. Фиг. 4С). Создание лекарств, нацеленных на ортостерический сайт связывания, затруднено из-за сильно поляризованного АТФ-специфического интерфейса, с другой стороны, аллостерические лиганды, нацеленные на межбелковые взаимодействия, формируют многообещающую возможность для открытия лекарств. Недавно для рецепторов P2X3 и P2X7 был открыт аллостерический сайт связывания, образованный двумя мономерами канала. Авторы применили BiteNet к АТФ-связанным и (AF-219)-связанным структурам тримерного комплекса, образованного мономерами P2X3 (идентификаторы PDB: 5SVK, 5YVE), а также к структурам одиночных мономеров. BiteNet правильно идентифицировал ортостерический сайт связывания в АТФ-связанной структуре и аллостерический сайт связывания в (AF-219) -связанной структуре тримера, но не в структурах мономеров (см. Фиг. 4). Интересно, что BiteNet также предсказал центр для сайта связывания АТФ, расположенный на противоположном конце молекулы АТФ, с более низкой оценкой вероятности. Чтобы убедиться, что это не артефакт вращательной дисперсии модели, было сгенерировано 50 копий при помощи вращения мономера вокруг 10 осей на углы π/3, 2 π /3, π , 4 π /3 и 5 π /3, и далее усредняли полученные прогнозы. Как видно из Фиг.4D, хотя абсолютные значения оценок вероятности различаются в зависимости от мономеров, во всех случаях BiteNet правильно определяет аллостерический сайт связывания для тримерного комплекса, а не для мономера. Обратите внимание, что АТФ является эндогенным агонистом, а AF-219 - антагонистом тримерного P2X. Связанные с агонистом и связанные с антагонистом конформации различаются, особенно в областях ортостерических и аллостерических участков связывания (Фиг. 4 C,D). Следовательно, BiteNet чувствителен к конформационным изменениям, так как не предсказывает сайт связывания АТФ в (AF-219) -связанной структуре и наоборот. Интересно, что, несмотря на отсутствие сайта связывания в структуре мономера, BiteNet предсказал различные сайты связывания с относительно высокими показателями в структурах мономера. Более пристальный взгляд на доступные трехмерные структуры рецепторов P2X3 выявил катионные ионы (Mg, Na, Ca) и молекулы этиленгликоля, соответствующие этим прогнозам (идентификаторы PDB: 5YVE, 5SVS, 5SVT, 5SVJ, 5SVR, 5SVQ, 5SVP, 5SVM, 5SVL, 6AH4, 6AH5).
Пример 2. Пространственно-временное прогнозирование сайтов связывания в рецепторе эпидермального фактора роста (EGFR).
EGFR представляет собой трансмембранный белок из семейства тирозинкиназ. Сверхэкспрессия EGFR ассоциирована с различными типами опухолей. Хотя существуют ингибиторы EGFR, нацеленные на ортостерический сайт связывания киназного домена, белки, обнаруженные в раковых клетках, часто имеют аминокислотные замены, которые делает их нечувствительными к таким ингибиторам. Существуют также мутантно-селективные необратимые ингибиторы, которые ковалентно связываются с аминокислотным остатком Cys797, однако некоторые рецепторы мутантного типа также обладают другим аминокислотным остатком в 797 положении. Недавно была обнаружена трехмерная структура варианта киназного домена L858R / T790M EGFR, связанного с мутант-селективным аллостерическим ингибитором EAI001 (PDB ID: 5d41). Было показано, что EAI001 связывается только с одним мономером, что приводит к неполному ингибированию, но и снижению аутофосфорилирования в клетке. Соответственно, трехмерная структура представляет собой асимметричный димер с одним мономером, связанным как с ортостерическими, так и с аллостерическими лигандами (аналог АТФ аденилимидодифосфат (AMP-PNP) и EAI001, соответственно), в то время как другой мономер связывается только с AMP-PNP. BiteNet успешно идентифицировал как ортостерические, так и аллостерические сайты связывания в одном мономере (цепь A) и только первый в другом мономере (цепь B). Отметим, что в обучающей выборке была еще одна структура киназного домена EGFR (PDB ID: 5UG9), однако она содержит только ортостерический лиганд вдали от аллостерического сайта связывания. Хотя этот и предыдущие примеры ясно демонстрируют способность BiteNet обнаруживать сайты связывания в holo конформациях, на практике такие конформации могут быть неизвестны, особенно когда есть задача обнаружить новые сайты связывания. Чтобы оценить способность BiteNet обнаруживать сайты связывания, начиная с несвязанной конформации, авторы смоделировали конформационный переход от несвязанного к связанному состоянию, как показано ниже. Сначала авторы смоделировали недостающие остатки в цепи B и поместили EAI001, как это наблюдается в цепи A.
Затем авторы подготовили систему молекулярной динамики, содержащую цепь B, AMP-PNP и EAI001, встроенную в водяной ящик с ионами с помощью веб-сервера CHARMM-GUI. Затем авторы запустили полную минимизацию атомарной энергии подготовленной системы до сходимости, используя Gromacs [37], в результате чего получили траекторию минимизации, состоящую из 900 конформаций. Наконец, авторы удалили лиганды, ионы и воду и применили BiteNet к каждому кадру траектории минимизации вместе с его 50 репликами. Фиг. 5 показывает, что оценка вероятности для сайта аллостерического связывания неуклонно увеличивается, в то время как энергия системы снижается, а среднеквадратичное отклонение (RMSD) относительно сайта аллостерического связывания в исходной (несвязанной) конформации увеличивается. Обратите внимание, что оценка вероятности для сайта ортостерического связывания остается высокой во время минимизации.
Также, что авторы использовали 4 Å для порогового значения расстояния подавления, отличного от максимального, чтобы избежать слияния прогнозов для ортостерических и аллостерических сайтов связывания на этапе постобработки BiteNet. Следовательно, BiteNet может применяться для крупномасштабных пространственно-временных траекторий для обнаружения конформаций белков, которые обладают сайтами связывания, невидимыми в исходной структуре.
Пример 3. Пространственно-временное прогнозирование сайтов связывания в рецепторах, связанных с G-белками (GPCR).
Рецепторы, связанные с G-белками (GPCR), опосредуют многочисленные физиологические процессы в организме, что делает их важными мишенями для лекарств. Большинство одобренных FDA препаратов связываются с ортостерическими сайтами связывания GPCR. Однако такие препараты могут быть неселективными по отношению к подтипам высокогомологичных рецепторов.
В таких случаях существует потребность в разработке лекарств, нацеленных на аллостерические сайты связывания, которые менее консервативны, чем ортостерические. Трехмерные структуры GPCR обнаруживают аллостерические сайты связывания, охватывающие внеклеточные, трансмембранные и внутриклеточные области белкового комплекса; идентификация новых аллостерических сайтов в GPCR может предоставить альтернативные варианты для открытия лекарств. Чтобы продемонстрировать использование BiteNet для пространственно-временной идентификации сайтов связывания GPCR, авторы проанализировали траектории молекулярной динамики человеческого аденозинового рецептора A2A (A2A), полученные из репозитория GPCRmd.
А именно, авторы рассмотрели траектории A2A, встроенного в липидный бислой POPC, окруженного молекулами воды, натрия и хлорид-иона, начиная с активно-подобной конформации (PDB ID: 5G53) в комплексе с агонистом NECA и без лиганда (GPCRMD ID: 48:10498 и 47:10488 соответственно). В общей сложности каждая симуляция длилась 500 нс с временным шагом 4 фс и интервалом между кадрами 2 нс, что дало 2500 конформаций A2A. Затем, авторы применили BiteNet для каждого кадра траектории. Как и ожидалось, на обеих траекториях моделирования авторы наблюдали кластер предсказаний, соответствующий каноническому ортостерическому сайту связывания в GPCR. Кластер был более плотный и с более высоким средним баллом на траектории моделирования связанного лиганда, что можно объяснить более низкой гибкостью белка из-за взаимодействий белок-лиганд. Удивительно, на обеих траекториях моделирования авторы также наблюдали кластер предсказаний в окрестности конца TM1, TM7 и спирали 8, начиная с 300 нс при моделировании без лиганда, и от 150 до 200 нс и от 320 до 370 нс при моделировании комплекса со связанным лигандом. Более пристальный взгляд на конформации с наивысшими оценками вероятности, соответствующими этому кластеру, выявил липидный хвост, скрытый в полости, образованной гидрофобными аминокислотными остатками. Важно отметить, что, хотя GPCR плотно окружены липидами, BiteNet не давал прогнозов по всей области, контактирующей с мембраной, поскольку он был специально обучен на участках связывания, пригодных для лекарств. Чтобы исследовать, связывается ли липидный хвост с полостью, для каждого кадра f авторы рассчитали его подвижность в терминах RMSD между конформацией липидного хвоста в этом кадре и конформацией липидного хвоста, усредненной по [f 100; f +100] кадров. Как видно из Фиг.6, вычисленное RMSD ниже для кадров с высокими оценками вероятности, соответствующими предполагаемому сайту связывания. Насколько известно авторам, в литературе нет доступных структур для GPCR с лигандом, связанным с этой областью. При применении BiteNet к траекториям молекулярной динамики, полученным для других рецепторов из GPCRmd, авторы также наблюдали аналогичный кластер в мускариновом рецепторе M2, опять же, начиная с активной подобной конформации. Таким образом, на предсказанную область стоит обратить внимание, поскольку она может соответствовать новому аллостерическому сайту связывания в GPCR.
Подводя итог, авторы показали применимость BiteNet для обнаружения сайтов связывания для трех различных фармакологических мишеней и проблемных сайтов связывания, наблюдаемых как в растворимых, так и в трансмембранных доменах белков. BiteNet способен обнаруживать конформационно-специфические и олигомер-специфические аллостерические сайты связывания, и может применяться для крупномасштабного пространственно-временного анализа белковых структур. На примере A2A авторы продемонстрировали, как BiteNet можно использовать на практике для исследования новых сайтов связывания. Также нужно отметить, что использованные трехмерные структуры не были представлены BiteNet в процессе обучения.
Пример 4. Сравнение вычислительной эффективности BiteNet.
Чтобы сравнить BiteNet с другими подходами, авторы оценили его производительность на наборах данных COACH420 (A. Roy, J. Yang, and Y. Zhang, Cofactor: an accurate comparative algorithm for structure-based protein function annotation, Nucleic acids research 40, W471 (2012)) и HOLO4K (P. Schmidtke, C. Souaille, F. Estienne, N. Baurin, and R. T. Kroemer, Large-scale comparison of four binding site detection algorithms, Journal of chemical information and modeling 50, 2191 (2010)), которые содержат 420 и 4542 белка соответственно. Для корректного сравнения авторы рассматривали только белки, не представленные в наборах последовательностей метода, для которых все методы успешно предсказывают истинные сайты связывания в соответствии с критерием P2Rank [28], что приводит к подмножествам белков 230 и 2305 из COACH420 и HOLO4K, соответственно. В качестве показателя производительности авторы рассчитали среднюю точность (AP), то есть площадь под кривой точности-отзыва, для прогнозов All и TopN, где N - количество истинных сайтов связывания, присутствующих в структуре белка. Как видно из Фиг.7, BiteNet статистически превосходит классические методы прогнозирования сайтов привязки, такие как fpocket (V. Le Guilloux, P. Schmidtke, and P. Tuffery, Fpocket: an open source platform for ligand pocket detection, BMC bioinformatics 10, 168 (2009)); SiteHound (M. Hernandez, D. Ghersi, and R. Sanchez, Sitehoundweb: a server for ligand binding site identification in protein structures, Nucleic acids research 37, W413 (2009)); MetaPocket (Z. Zhang, Y. Li, B. Lin, M. Schroeder, and B. Huang, Identification of cavities on protein surface using multiple computational approaches for drug binding site prediction, Bioinformatics 27, 2083 (2011)), а также современные методы машинного обучения, такие как DeepSite (J. Jiménez, S. Doerr, G. Martínez-Rosell, A. S. Rose, and G. De Fabritiis, Deepsite: protein-binding site predictor using 3d-convolutional neural networks, Bioinformatics 33, 3036 (2017)) и P2Rank (R. Krivák and D. Hoksza, P2rank: machine learning based tool for rapid and accurate prediction of ligand binding sites from protein structure, Journal of cheminformatics 10, 39 (2018)), p-value 1.2e-6.
BiteNet также эффективен с точки зрения вычислений. На Фиг.7B показано время, затраченное BiteNet вместе с fpocket и P2Rank, которые являются одними из самых быстрых методов с точки зрения количества конформаций обработанного белка. BiteNet, работающий на одном графическом процессоре (GeForce GTX 1080 Ti), превосходит P2Rank, работающий на нескольких процессорах (Intel (R) Core (TM) i7-8700K CPU @ 3,70 ГГц). В среднем BiteNet требуется приблизительно 0,1 секунды для обработки конформации одного белка. Дальнейшая оптимизация взаимодействия CPU-GPU и реализация BiteNet с несколькими графическими процессорами приведет к еще большей производительности.
В настоящих материалах заявки было представлено предпочтительное раскрытие осуществление заявленного изобретения, которое не должно использоваться как ограничивающее иные, частные воплощения его реализации, которые не выходят за рамки испрашиваемого объема правовой охраны и являются очевидными для специалистов в соответствующей области техники.
| название | год | авторы | номер документа |
|---|---|---|---|
| ВНЕКЛЕТОЧНЫЙ ДОМЕН ТИРОЗИНКИНАЗНОГО РЕЦЕПТОРА, СВЯЗЫВАЮЩИЙ АЛЛОСТЕРИЧЕСКИЙ ИНГИБИТОР | 2010 |
|
RU2604805C2 |
| Модифицированная генетическая конструкция для рекомбинантной экспрессии и кристаллизации человеческого S1P5 рецептора | 2022 |
|
RU2792893C1 |
| АНТАГОНИСТЫ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2005 |
|
RU2401842C2 |
| УЛУЧШЕННЫЕ СЕЛЕКТИВНЫЕ В ОТНОШЕНИИ ПРОТОФИБРИЛЛ АНТИТЕЛА И ИХ ПРИМЕНЕНИЕ | 2007 |
|
RU2429244C2 |
| Способ разметки лиганд-белковых сайтов связывания | 2024 |
|
RU2838984C1 |
| Сконструированный пептид, направленный на белок c-FLIP и индуцирующий апоптоз | 2023 |
|
RU2811497C1 |
| ПЕПТИД, ОБЛАДАЮЩИЙ РОСТОСТИМУЛИРУЮЩЕЙ АКТИВНОСТЬЮ ГРАНУЛОЦИТАРНОГО КОЛОНИЕСТИМУЛИРУЮЩЕГО ФАКТОРА ЧЕЛОВЕКА | 2008 |
|
RU2385875C2 |
| СПОСОБЫ ВЫЯВЛЕНИЯ В БЕЛКАХ ОБЛАСТЕЙ, ОТВЕТСТВЕННЫХ ЗА СВЯЗЫВАНИЕ МАКРОМОЛЕКУЛ, И ОБЛАСТЕЙ, СКЛОННЫХ К АГРЕГАЦИИ, И ИХ ПРИМЕНЕНИЕ | 2009 |
|
RU2571217C2 |
| ОСНОВАННОЕ НА СТРУКТУРЕ ПРОГНОЗНОЕ МОДЕЛИРОВАНИЕ | 2014 |
|
RU2694321C2 |
| СПОСОБ ОПТИМИЗАЦИИ УСЛОВИЙ КРИСТАЛЛИЗАЦИИ БЕЛКОВ С ПРИМЕНЕНИЕМ МЕТОДА МОЛЕКУЛЯРНОЙ ДИНАМИКИ | 2021 |
|
RU2781051C1 |

Изобретение относится к способу идентификации участков связывания белкового комплекса с низкомолекулярным химическим соединением на основе структурной информации. Технический результат заключается в идентификации участков связывания белковых комплексов. В способе получают тензорное представление пространственной структуры белкового комплекса, для которой получают трехмерную воксельную сетку структуры белкового комплекса, где каналы данных вокселей соответствуют плотностям атомов, входящих в состав белкового комплекса, которую разбивают на кубические сетки меньшего размера, каждую из которых анализируют с помощью алгоритма машинного обучения для прогнозирования центра участка связывания низкомолекулярного соединения в ячейках кубической сетки, каждой ячейке ставят в соответствие действительное число от 0 до 1, где 1 соответствует максимальной оценке достоверности предсказания, получают результаты предсказания центров участков связывания для каждой кубической сетки с оценками достоверности предсказания, осуществляют идентификацию аминокислотных остатков участков связывания в заданном расстоянии относительно предсказанного центра участка связывания, полученные данные группируют с использованием алгоритмов кластеризации по предсказаниям по каждой входной структуре белкового комплекса. 3 з.п. ф-лы, 7 ил.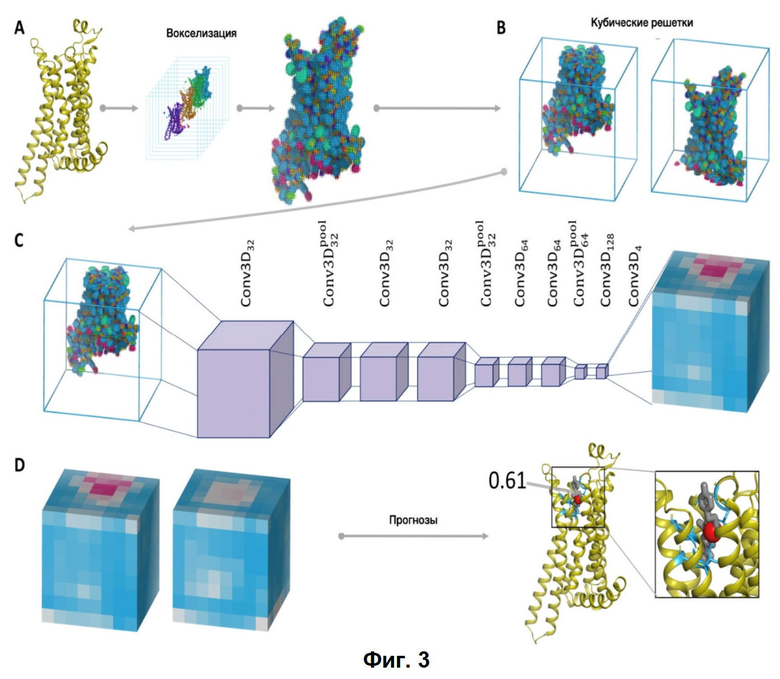
1. Способ идентификации участков связывания белкового комплекса с низкомолекулярным химическим соединением на основе структурной информации с использованием машинного обучения, содержащий этапы, на которых
получают тензорное представление пространственной структуры белкового комплекса;
осуществляют предобработку пространственной структуры белкового комплекса, в результате которой получают трехмерную воксельную сетку структуры белкового комплекса, где каналы данных вокселей соответствуют плотностям атомов, входящих в состав белкового комплекса;
трехмерную воксельную сетку структуры белкового комплекса разбивают на кубические сетки меньшего размера, покрывающие исходную трехмерную воксельную сетку;
каждую кубическую сетку анализируют с помощью алгоритма машинного обучения для прогнозирования центра участка связывания низкомолекулярного соединения в ячейках кубической сетки;
каждой ячейке в кубических сетках ставят в соответствие действительное число в диапазоне от 0 до 1, где 1 соответствует максимальной оценке достоверности предсказания;
получают результаты предсказания центров участков связывания для каждой кубической сетки с оценками достоверности предсказания, используя кластеризацию предсказаний для ячеек, соответствующих одному участку белкового комплекса;
осуществляют идентификацию аминокислотных остатков участков связывания в заданном расстоянии относительно предсказанного центра участка связывания;
полученные предсказания и идентифицированные аминокислотные остатки для набора различных конформаций белковых комплексов группируют с использованием алгоритмов кластеризации по предсказаниям по каждой входной структуре белкового комплекса.
2. Способ по п.1, характеризующийся тем, что низкомолекулярное соединение является лекарственным средством.
3. Способ по п.1, характеризующийся тем, что получают тензорное представление нескольких пространственных структур белкового комплекса, полученных в разные промежутки времени, и для каждой такой структуры реализуют кластеризацию предсказаний с учетом хронологического порядка пространственных структур белкового комплекса.
4. Способ по п. 1, характеризующийся тем, осуществляют предобработку пространственной структуры белкового комплекса, в результате которой получают трехмерную воксельную сетку структуры белкового комплекса, где каналы данных вокселей соответствуют плотностям атомарных групп.
| Державка для пластинчатых клинков типа "Жиллет" | 1928 |
|
SU10258A1 |
| Пресс для выдавливания из деревянных дисков заготовок для ниточных катушек | 1923 |
|
SU2007A1 |
| Топчак-трактор для канатной вспашки | 1923 |
|
SU2002A1 |
| CN 111210870 A, 29.05.2020 | |||
| Способ восстановления спиралей из вольфрамовой проволоки для электрических ламп накаливания, наполненных газом | 1924 |
|
SU2020A1 |
| Станок для придания концам круглых радиаторных трубок шестигранного сечения | 1924 |
|
SU2019A1 |
| CN 111243668 B, 07.08.2020. | |||
Авторы
Даты
2021-02-17—Публикация
2020-08-14—Подача