Настоящее изобретение относится к гуманизированным антителам к ИЛ-1-бета, в частности к одновалентным высокоэффективным фрагментам антител к ИЛ-1-бета. Настоящее изобретение также относится к нуклеиновым кислотам, кодирующим такие антитела, к векторам, клеткам-хозяевам, содержащим такие последовательности, фармацевтическим и диагностическим композициям, содержащим антитела или нуклеиновые кислоты, и к их применению.
Предшествующий уровень техники
Интерлейкин-1-бета (ИЛ-1-бета) является про воспалительным цитокином, продуцируемым в качестве предшественника активированными макрофагами. При протеолитическом расщеплении трансдукция сигнала инициируется путем связывания активной формы с рецептором ИЛ-1 I типа (ИЛ-1Р1), который, в свою очередь, ассоциирован с трансмембранным акцессорным белком рецептора ИЛ-1 (IL-1RAP). Образованный комплекс обладает способностью к трансдукции сигнала. Являясь ключевым медиатором воспалительного ответа, цитокин влияет на ряд видов клеточной активности, таких как клеточная пролиферация, дифференцировка и апоптоз. Таким образом, ИЛ-1-бета считается важной мишенью для множества фармацевтических средств.
Имеется потребность в данной области техники в антителах с терапевтическим потенциалом против ИЛ-1-бета человека. Для терапевтического эффекта важно, чтобы такое антитело демонстрировало необходимые биофизические и биохимические характеристики. Например, поскольку целевой ИЛ-бета является высокоэффективным интерлейкином, активным при очень низких концентрациях, и таким образом, его нужно полностью блокировать, такое антитело должно быть высокоэффективным, а также обладать высокой стабильностью и растворимостью.
Краткое изложение сущности настоящего изобретения
В первом аспекте настоящее изобретение обеспечивает одновалентный фрагмент антитела, направленный против интерлейкина 1-бета, обладающий активностью при концентрации менее 50 пикомоль (пМ), как установлено по результатам определения полу-максимальной ингибирующей концентрации IC50 в отношении подавления биологического эффекта ИЛ-1-бета человека.
Одновалентные фрагменты антител, гуманизированные или не гуманизированные, обладающие значениями активности в пикомолярном диапазоне, являются особенными, редкими и не получаемыми рутинными способами. В дополнение к этому и как правило, антитело теряет аффинность к своей мишени при гуманизации по сравнению с исходным антителом, не являющимся антителом человека. Таким образом, гуманизация антитела таким образом, чтобы параметры аффинности были близкими или эквивалентными параметрам исходного антитела, является сложной задачей. Это в особенности касается одновалентных фрагментов антител, содержащих только одну вариабельную легкую цепь и тяжелую цепь, и таким образом, связывающегося с мишенью менее прочно, чем двухвалентные антитела, содержащие две легких цепи и тяжелых цепи.
Кроме того, при превращении полноразмерного антитела в меньший фрагмент его активность обычно уменьшается. Это обусловлено не только соответствующим изменением валентности (например, фрагмент антител может быть только одновалентным, в то время как полноразмерный иммуноглобулин является двух- или поливалентным), но также может быть обусловлено стерическими факторами.
Высокоэффективное антитело особенно полезно, поскольку оно позволяет вводить пациенту сниженные количества лекарства, уменьшая таким образом общие затраты на лечение. Кроме того, это позволяет достичь более полной нейтрализации молекулярной мишени, связанной с заболеванием.
Далее, можно применять различные пути введения в моделях на животных, а также при лечении человека при использовании антител с наивысшей активностью. Например, для лекарств местного применения, хотя доставка может быть ограничена из-за барьерной функции эпителиального слоя, эффективность лечения восстанавливается за счет высокой эффективности ограниченного количества молекул лекарства, проходящих через физиологический барьер.
Часто большое количество менее активного лекарства, которое нужно вводить для достижения схожих фармакодинамических эффектов, соответствует гораздо большим объемам для внутривенного или подкожного применения, чем у более эффективного лекарства. Такие более значительные объемы введения являются недостатком для применения у животных и людей по двум причинам: во-первых, непрактичность лечения пациентов большими объемами лекарства и, во-вторых, высокая стоимость антител на единицу массы.
Таким образом, сниженные количества антител, используемые для лечения, соответствуют уменьшенным затратам на производство лекарства. В частности, фрагменты антител пригодны для производства с использованием, например, систем культивирования бактериальных или дрожжевых клеток, которое связано со сравнительно меньшими затратами, чем использование систем экспрессии млекопитающих, обычно используемых для производства полноразмерных иммуноглобулинов, таких как IgG. Комбинация меньших количеств вводимого лекарства и более дешевых производственных процессов открывает возможность получения более экономичных медикаментов для пациентов. Таким образом, такое лекарство может быть использовано для большего числа пациентов.
Параметры стабильности и растворимости являются другими факторами, критическими для обеспечения более жизнеспособного медикамента. Чем более стабильно и растворимо лекарство в виде антитела, тем меньше вводимый объем и дольше срок хранения. Антитела, обеспечиваемые в настоящей заявке, являются высокостабильными и хорошо растворимыми, т.е. они остаются мономерными в течение длительного периода времени, а также при высоких концентрациях.
В одном аспекте обеспечивается антитело, в частности, одновалентный фрагмент антитела, содержащий:
(a) по меньшей мере одну из последовательностей гипервариабельного участка (CDR) вариабельной тяжелой цепи (VH) CDR-H1, CDR-H2 или CDR-H3, указанных в SEQ ID No: 1, 2 и 3, соответственно, или их варианты; и/или
(b) по меньшей мере одну из последовательностей гипервариабельного участка (CDR) вариабельной легкой цепи (VL) CDR-L1, CDR-L2 или CDR-L3, указанных в SEQ ID No: 4, 5, и 6, соответственно, или их варианты.
В другом варианте осуществления антитело, и в частности, указанный одновалентный фрагмент антитела, содержит:
(a) по меньшей мере одну из последовательностей гипервариабельного участка (CDR) вариабельной тяжелой цепи (VH) CDR-H1, CDR-H2 или CDR-H3, указанных в SEQ ID No: 155, 156 и 157, соответственно, или их варианты; и/или
(b) по меньшей мере одну из последовательностей гипервариабельного участка (CDR) вариабельной легкой цепи (VL) CDR-L1, CDR-L2 или CDR-L3,
(i) указанную в SEQ ID No: 158, 159 и 160, соответственно, или их варианты, или
(ii) указанную в SEQ ID No: 161, 162 и 163, соответственно, или их варианты.
В некоторых вариантах осуществления антитело содержит: (a) VH участок, обладающий по меньшей мере 85% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID No: 7 и SEQ ID No: 146; и/или (b) VL участок, обладающий по меньшей мере 85% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID No: 8, SEQ ID No: 136 и SEQ ID No: 145.
Антитело может содержать линкерную последовательность, являющуюся последовательностью SEQ ID No: 9 или последовательностью, производной от нее. В некоторых вариантах осуществления такое антитело является фрагментом антитела, последовательность которой обладает по меньшей мере 85% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID No: 10, SEQ ID No: 73 и SEQ ID No: 82.
В одном аспекте настоящее изобретение обеспечивает связывающие элементы, которые связываются с ИЛ-1-бета и конкурируют с антителами, описанными в настоящей заявке, за связывание. Указанный связывающий элемент может быть одновалентным или поливалентным. Предпочтительный поливалентный связывающий элемент является двухвалентным. Поливалентный связывающий элемент может быть биспецифическим.
В одном аспекте настоящее изобретение обеспечивает выделенную последовательность нуклеиновых кислот, кодирующую антитело или связывающий элемент, раскрытый в настоящей заявке.
В одном аспекте обеспечивается вектор, содержащий указанную последовательность нуклеиновых кислот.
В одном аспекте настоящее изобретение обеспечивает клетку-хозяина, содержащую вышеуказанную последовательность нуклеиновых кислот или вышеуказанный вектор.
В одном аспекте композиция содержит вышеуказанное антитело, вышеуказанный связывающий элемент, вышеуказанную последовательность нуклеиновых кислот, вышеуказанный вектор или вышеуказанную клетку-хозяина; и кроме того, подходящий носитель, разбавитель или наполнитель. Композиция предпочтительно является фармацевтической композицией, содержащей фармацевтически пригодный носитель, разбавитель или наполнитель. Такая фармацевтическая композиция предпочтительно находится в форме, пригодной для местного, интрадермального, трансдермального, внутривенного, подкожного, внутримышечного, парентерального, сублингвального, буккального, перорального, назального, интраназального, ректального, локального или окулярного введения.
Кроме того, обеспечивается способ лечения ИЛ-1-бета-зависимого заболевания, включающий введение субъекту, нуждающемуся в лечении, вышеуказанной фармацевтической композиции.
Также обеспечивается вышеуказанное антитело, вышеуказанный элемент связывания, вышеуказанная последовательность нуклеиновых кислот, вышеуказанный вектор или клетка-хозяин, раскрытая в настоящей заявке, (i) для применения в лечении ИЛ-1-бета-зависимого заболевания; (ii) для применения в диагностике; (iii) для применения в косметике; и/или (iv) для целей детекции.
В еще одном аспекте настоящее изобретение обеспечивает способ получения антитела или связывающего агента, описанного в настоящей заявке, включающий (i) этапы культивирования вышеуказанной клетки-хозяина, и извлечения и очистки фрагмента антитела или связывающего элемента, соответственно; или (ii) применение системы, не содержащей клеток. В дополнение или альтернативно, указанный способ может включать по меньшей мере один этап химического синтеза белка.
Краткое описание чертежей
Фигура 1 демонстрирует результаты ИФА для определения связывания DLX2323 с рекомбинантным ИЛ-1-бета человека (рч) при различных концентрациях. На график нанесены значения разницы поглощения при длине волны 450 нм в зависимости от концентрации scFv (одноцепочечного вариабельного фрагмента) в нг/мл. Квадраты: DLX2323; круги: контроль.
Фигура 2 является графиком, изображающим результаты связывания DLX2323 с природным ИЛ-1-бета человека по сравнению со связыванием с рчИЛ-1-бета. Природный ИЛ-1-бета человека был получен из надосадочной жидкости от активированных ТНР-клеток. На график нанесены значения разницы поглощения при длине волны 450 нм в зависимости от концентрации ИЛ-1-бета в нг/мл. Квадраты: рИЛ-1-бета; круги: природный ИЛ-1-бета.
Фигура 3 демонстрирует сравнение DLX2323 с некоторыми коммерческими ингибиторами ИЛ-1-бета для нейтрализации рчИЛ-1-бета в тесте на фибробластах человека в двух независимых экспериментах. Фигура 3А демонстрирует результаты для DLX2323 и МАВ201. По оси Y показано высвобождение ИЛ-6 из фибробластов человека в пг/мл; по оси X указана концентрация антител в пМ. Квадраты: DLX2323; круги: контроль МАВ201. Фигура 3В обобщает данные для DLX2323, антагониста рецептора рчИЛ-1 (ра) канакинумаба (Ilaris®). По оси Y указано высвобождение ИЛ-6 из фибробластов человека в пг/мл; по оси X показана концентрация антител или рчИЛ-1 ra в пМ. Квадраты: DLX2323; круги: канакинумаб; треугольники: рчИЛ-1ра.
Фигура 4 демонстрирует эффективность DLX2323 in vivo на мышиной модели воспаления, индуцированного ИЛ-1-бета человека. Количество ИЛ-6 (пг/мл) определяли в сыворотке после применения ИЛ-1-бета человека и (a) DLX2323 при 5 мг/мл; (b) DLX2323 при 15 мг/мл; (с) канакинумаба; (d) контроля scFv; или (d) ФБР.
Фигура 5 иллюстрирует определение CDR-H1, как описано в настоящей заявке. Стрелками указаны остатки CDR-H1 в соответствии с нумерацией Kabat (вверху) или как описано в настоящей заявке (внизу).
Фигура 6 иллюстрирует результаты ИФА, где очищенные клеточные лизаты вариантов DLX2323, экспрессированных в E.coli, связывались с поверхностью, покрытой рчИЛ-1-бета. На графике нанесены значения разницы поглощения при длине волны 450 нм, как наблюдалось для указанных образцов белка scFv. Анализировали разведения образца 1:2 (серые столбцы) или 1:10 (черные столбцы) в буферном растворе для анализа.
Подробное описание настоящего изобретения
Чтобы настоящее изобретение стало более понятным, вначале определены некоторые термины. Если в описании не указано иное, все технические и научные термины, используемые в заявке, имеют значение, принятое в данной области техники. Хотя схожие с описанными в настоящей заявке, или эквивалентные способы и материалы можно применять для осуществления или тестирования настоящего изобретения, подходящие способы и материалы описаны ниже. Все публикации, патентные заявки, патенты и другие ссылки, упомянутые в настоящей заявке, включены посредством ссылки целиком. В случае конфликта преобладает настоящее описание, включая определения. Материалы, методы и примеры являются исключительно иллюстративными и не предназначены для ограничения.
В рамках настоящего изобретения термин «антитело» относится к полноразмерным иммуноглобулинам, а также к их фрагментам. Такие полноразмерные иммуноглобулины могут быть моноклональными, поликлональными, химерными, гуманизированными, венированными антителами или антителами человека.
«Фрагменты антител» включают в себя части полноразмерного иммуноглобулина, сохраняющие направленную специфичность указанного иммуноглобулина. Во многих, но не во всех фрагментах антител отсутствует, по меньшей мере частично, константная область (Fc область) полноразмерного иммуноглобулина. В некоторых вариантах осуществления фрагменты антител получают путем протеолиза полноразмерного иммуноглобулина. Фрагмент антитела может также быть синтетической или рекомбинантной конструкцией, содержащей части иммуноглобулина или иммуноглобулиновые цепи (см., например, HOLLIGER, P. and Hudson, J. «Engineered antibody fragments and the rise of single domains». Nature Biotechnology 2005, vol. 23, no. 9, p. 1126-1136 («Фрагменты сконструированных антител и рост отдельных доменов»)). Примеры фрагментов антител включают scFv, Fab, Fv, Fab', F(ab')2 фрагменты, dAb, VHH, нанотела, V(NAR) или минимальные единицы распознавания, но не ограничиваются ими.
«Одноцепочечные вариабельные фрагменты» или «одноцепочечные антитела» или «scFv» являются одним типом фрагментов антител. scFv являются гибридными белками, содержащими VH и VL иммуноглобулинов, соединенные линкером. Они не содержат константной области Fc, присутствующей в полноразмерных иммуноглобулинах, но сохраняют специфичность исходного иммуноглобулина.
«Связывающий элемент», используемый в настоящей заявке, относится к полноразмерным иммуноглобулинам, фрагментам антител, не-антительным каркасам, и/или другим связывающим соединениям. Такой связывающий элемент может быть одновалентным или поливалентным, т.е. содержащим один или несколько участков связывания антигена. Не ограничивающие рамки настоящего изобретения примеры одновалентных связывающих элементов включают scFv (одноцепочечные вариабельные фрагменты), Fab фрагменты, dAb (доменные антитела), VHH (вариабельный домен тяжелой цепи), дарпины (сконструированные белки с анкириновым повтором), аффилины и нанотела. Поливалентный связывающий элемент может иметь два, три, четыре или больше участков связывания антигена, где может распознаваться один или несколько различных антигенов. Полноразмерные иммуноглобулины, F(ab')2 фрагменты, bis-scFv и диатела являются не ограничивающими рамки настоящего изобретения примерами поливалентных связывающих элементов; в указанных поливалентных связывающих элементах, используемых в качестве примеров, присутствуют два участка связывания, т.е. связывающий элемент является двухвалентным.
В одном варианте осуществления поливалентный связывающий элемент является биспецифическим, т.е. связывающий элемент направлен против двух различных мишеней или двух различных целевых участков на одной целевой молекуле. Биспецифические антитела, например, описаны в MÜLLER, D. and Kontermann, R.E. «Bispecific antibodies». Edited by DÜBEL, S. Weinheim: Wiley-VCH, 2007. ISBN 3527314539. p. 345-378 («Биспецифические антитела»). В другом варианте осуществления поливалентный связывающий элемент содержит больше двух, например, три или четыре различных участка связывания для трех или четырех, соответственно, различных антигенов. Такой связывающий элемент является поливалентным и полиспецифическим, в частности, три- или тетра-специфическим, соответственно.
«Не-антительные каркасы» являются антигенсвязывающими полипептидами, которые, например, описаны в FIELDER, M. and Skerra, A. «Non-antibody scaffolds». Edited by DÜBEL, S. Weinheim: Wiley-VCH, 2007. ISBN 3527314539. p. 467-500 («Не-антительные каркасы»); или GILBRETH, R.N. and Koide, S. «Structural insights for engineering binding proteins based on 10 non-antibody scaffolds». Current Opinion in Structural Biology2012, vol. 22, p. 413-420 («Структурные оценки для конструирования связывающих белков на основе 10 не-антительных каркасов»). Не ограничивающие рамки настоящего изобретения примеры включают аффитела, аффилиновые молекулы, аднектин, антикалин, дарпины, кноттин, домен типа Кунитца, авимер, тетранектин и транстело.
«Связывающие соединения» являются химическими или биологическими молекулами, которые связываются с мишенью и не принадлежат к классу полноразмерных иммуноглобулинов, фрагментов антител и не-антительных каркасов, как определено выше. Примеры связывающих соединений включают макролиды (GUNDLURU, М.K. et al. «Design, synthesis and initial biological evaluation of a novel pladienolide analog scaffold». Medchemcomm. 2011, vol. 2, p. 904-908 («Конструирование, синтез и первоначальная биологическая оценка нового пладиенолидного аналогового каркаса»); PATERSON, I. et al. «Total synthesis and biological evaluation of a series of macrocyclic hybrids and analogies of the antimitotic natural products dictyostatin, discodermolide and taxol». Chem Asian J. 2011, vol. 6, p. 459-473 («Общий синтез и биологическая оценка серий макроциклических гибридов и аналогов антимитотических натуральных продуктов диктиостатина, дискодермолида и таксола»); MORITA, H. et al. «Synthesis of unnatural alkaloid scaffolds by exploiting plant polyketide synthase». PNAS2011, vol. 108, p. 13504-13509 («Синтез ненатуральных алкалоидных каркасов с применением растительной поликетид-синтазы»)), полимеры молекулярного импринта (HOSHINO, Y. et al. «Recognition, neutralization and clearance of target peptides in the blood stream of living mice by molecular imprinted polymer nanoparticles: a plastic antibody». Journal of the American Chemical Society, 2010, vol. 19, p. 664-6645 («Распознавание, нейтрализация и клиренс целевых пептидов в кровотоке живых мышей посредством наночастиц полимера молекулярного импринта: пластическое антитело»)), аптамеры (STREHLITZ, В., et al. «Aptamers for pharmaceuticals and their application in environmental analytics». Bioanalytical reviews, 2012, vol. 4, p. 1-30 («Аптамеры для фармацевтических препаратов и их применение в исследовании окружающей среды»); YE, M. et al. «Generating Aptamers by Cell-SELEX for Applications in Molecular Medicine». International Journal of Molecular Sciences, 2012, vol. 13, p. 3341-3353 («Генерация аптамеров посредством Cell-SELEX для применения в молекулярной медицине»)), шпигельмеры (см., например, MAASCH, С. et al. «Polyethylenimine-Polyplexes of Spiegelmer NOX-A50 directed against intracellular high mobility group protein A1 (HMGA1) reduce tumor growth in vivo». JBC2010, vol. 285, p. 40012-40018 («Полиэтиленимин-полиплексы из шпигельмера NOX-A50, направленные против внутриклеточного белка группы с высокой подвижностью A1 (HMGA1) снижают рост опухоли in vivo»)), или пептиды (циклические или линейные; см., например, GOULD, A. et al. «Cyclotides, а novel ultrastable polypeptide scaffold for drug discovery». Curr Pharm Des. 2011, vol. 17, p. 4294-4307 («Циклотиды, новый сверхстойкий полипептидный каркас для открытия лекарств»)), но не ограничиваются ими.
«IC50» или «концентрация полумаксимального ингибирования» является мерой активности лекарства-антагониста и количественно описывает эффективность соединения в ингибировании биологической или биохимической функции. Эта мера указывает, сколько соединения необходимо для ингибирования определенного биологического или биохимического процесса на 50%. Хотя и не являясь прямым индикатором аффинности, обе величины коррелируют, и могут быть определены посредством уравнения Ченга-Прусоффа (CHENG Y. and Prusoff W.H. «Relationship between the inhibition constant (Ki) and the concentration of inhibitor which causes 50 per cent inhibition (150) of an enzymatic reaction». Biochemical Pharmacology, 1973, vol. 22, p. 3099-3108 («Взаимосвязь между константой ингибирования (Ki) и концентрацией ингибитора, вызывающей 50% ингибирование (I50) ферментативной реакции»); RAMMES, G., et al. «Identification of a domain which affects kinetics and antagonistic potency of clozapine at 5-HT3 receptora». PLOS one 2009, vol. 4, p. 1-14 («Идентификация домена, влияющего на кинетику и антагонистическую активность клозапина в отношении 5-НТ3 рецепторы»); ZHEN, J., et al. «Concentration of receptor and ligand revisited in a modified receptor binding protocol for high-affinity radioligands: [3H] spiperone binding to D2 and D3 dopamine receptors». Journal of Neuroscience Methods, 2010, vol. 188, p. 32-38 («Концентрация рецептора и лиганда пересматривается в протоколе связывания с модифицированным рецептором высокоаффинных радиолигандов: связывание [3Н] спиперона с D2 и D3 дофаминовыми рецепторами»).
Термин «ИЛ-1-бета специфическое связывание», используемый в настоящей заявке, указывает, что связывающий элемент связывается с ИЛ-1-бета с более высокой аффинностью, чем со структурно отличающимся антигеном, который не содержит эпитопа ИЛ-1-бета, с которым связывается анти-ИЛ-1-бета-связывающий элемент. Специфическое связывание отражается константой равновесия при диссоциации (KD) меньше 1 микромоль. Эту константу можно определить, например, с применением кварцевых микровесов на приборе Attana, или методики поверхностного плазмонного резонанса (SPR) на приборе BIACORE.
Использующийся в настоящей заявке термин «ИЛ-1-бета» означает молекулу, описанную, например, в Dinarello С.А., «Treating inflammation by blocking interleukin-1 in a broad spectrum of diseases». Nature reviews, 2012, vol. 11, p. 633-652 («Лечение воспаления путем блокирования интерлейкина-1 при широком спектре заболеваний»). «чИЛ-1-бета», использующийся в настоящей заявке, означает ИЛ-1-бета человека. «рИЛ-1-бета» означает рекомбинантный ИЛ-1-бета. Рекомбинантный ИЛ-1-бета может иметь или не иметь аминоконцевой метиониновый остаток, в зависимости от способа приготовления. «рчИЛ-1-бета» означает рекомбинантный ИЛ-1-бета человека. рчИЛ-1-бета может, например, быть получен от Peprotech, США, кат. No: 200-01В. ИЛ-1-бета может также быть получен путем выделения из биологических образцов, полученных из человека или из объекта, не являющегося человеком.
«Гуманизированные» антитела означают антитела, включающие один или несколько, как правило, все шесть CDR участков исходного антитела объекта, не являющегося человеком, или их варианты, и скелет которых является, например, (i) скелетом антитела человека, по возможности содержащим один или несколько скелетных остатков из исходного антитела объекта, не являющегося человеком, или (ii) скелетом из антитела объекта, не являющегося человеком, модифицированного для повышения сходства с натурально продуцируемыми скелетами антител человека. Способы гуманизации антител известны в данной области техники, см., например, LEGER, О. and Saldanha, J. «Antibody Drug Discovery». Edited by WOOD, С. London: Imperial College Press, 2011. ISBN 1848166281. p. 1-23 («Открытие лекарственных антител»).
«Скелет» (FR) относится к каркасу вариабельного иммуноглобулинового домена, вариабельной легкой цепи (VL) или вариабельной тяжелой цепи (VH), несущим соответствующие CDR. Скелет VL и/или VH, как правило, содержит четыре скелетных секции, FR1, FR2, FR3 и FR4, обрамляющих CDR участки. Таким образом, как известно в данной области техники, VL имеет общую структуру: (FR-L1)-(CDR-L1)-(FR-L2)-(CDR-L2)-(FR-L3)-(CDR-L3)-(FR-L4), в то время как VH имеет общую структуру: (FR-H1)-(CDR-H1)-(FR-H2)-(CDR-H2)-(FR-H3)-(CDR-H3)-(FR-H4).
«CDR» означает гипервариабельные участки антитела, которые главным образом вносят вклад в связывание с антигеном. Как правило, участок связывания антигена содержит шесть CDR, встроенных в скелетный каркас. В настоящей заявке CDR из VL обозначаются как CDR-L1, CDR-L2 и CDR-L3, а CDR из VH обозначаются как CDRH1, CDR-H2 и CDR-H3. Они могут быть идентифицированы, как описано в KABAT, Е.А., et al. «Sequences of Proteins of Immunological Interest». 5th edition. Edited by U.S. DEPARTMENT OF HEALTH AND HUMAN SERVICES. NIH Publications, 1991. p. 91-3242 («Последовательности белков, представляющих интерес для иммунологии»). CDR-H1, использующийся в настоящей заявке, отличается от нумерации Kabat тем, что начинается с положения 27 и заканчивается перед положением 36 (см. Фигуру 5 для иллюстрации).
Использующаяся в настоящей заявке система нумерации для идентификации положений аминокислотных остатков в VH и VL антитела соответствует «AHo» системе, описанной HONEGGER, A. and Plückthun, A. «Yet another numbering scheme for immunoglobulin variable domains: An automatic modelling and analysis tool». Journal of Molecular Biology 2001, vol. 309, p. 657-670 («Еще одна схема нумерации для вариабельных доменов иммуноглобулинов: инструмент для автоматического моделирования и анализа»). Указанная публикация также обеспечивает таблицы преобразования между AHo и нумерацией Kabat (KABAT, Е.А., et al. «Sequences of Proteins of Immunological Interest». 5th edition. Edited by U.S. DEPARTMENT OF HEALTH AND HUMAN SERVICES. NIH Publications, 1991. p. 91-3242 («Последовательности белков, представляющих интерес для иммунологии»)).
«Выделенное» антитело или нуклеиновая кислота являются идентифицированными и выделенными и/или извлеченными по меньшей мере из одного компонента из их натуральной среды.
Термин «идентичность», используемый в настоящей заявке, означает совпадение последовательностей двух белков или нуклеиновых кислот. Последовательности сравниваемых белков или нуклеиновых кислот выравнивают до получения максимальной идентичности, например, с помощью таких инструментов биоинформатики, как EMBOSS Needle (попарное выравнивание; доступно на www.ebi.ac.uk). Когда одно и то же положение сравниваемых последовательностей занято одним и тем же нуклеотидным основанием или аминокислотным остатком, то соответствующие молекулы являются идентичными в этом положении. Соответственно, «процент идентичности» является функцией числа совпадающих положений, разделенного на число сравниваемых положений, и умноженного на 100%. Например, если 6 или 10 положений последовательности являются идентичными, то идентичность составляет 60%. Процент идентичности между двумя последовательностями белков можно, например, определить с применением алгоритма Нидлмана-Вунша (NEEDLEMAN, S.B. and Wunsch, C.D. «A general method applicable to the search for similarities in the amino acid sequence of two proteins». Journal of Molecular Biology, 1970, vol. 48, p. 443-453 («Общий метод, пригодный для поиска сходства аминокислотной последовательности двух белков»)), который включен в EMBOSS Needle, с применением матрицы BLOSUM62, «штрафом за внесение пропуска» 10, «штрафом за продолжение пропуска» 0,5, ошибочным «штрафом за концевой пропуск», и «штрафом за открытый концевой пропуск» 10, и «штрафом за продолжение концевого пропуска» 0,5. Две молекулы, имеющие одну и туже первичную последовательность аминокислот или нуклеиновых кислот, являются идентичными, независимо от какой-либо химической и/или биологической модификации. Например, два антитела, имеющие одну и ту же первичную аминокислотную последовательность, но различные характеры гликозилирования, являются идентичными согласно этому определению. В случае нуклеиновых кислот, например, две молекулы, имеющие одну и ту же последовательность, но различные связывающие компоненты, такие как тиофосфат вместо фосфата, являются идентичными согласно этому определению.
«Схожими» последовательностями белков являются такие последовательности, которые при выравнивании обнаруживают схожие аминокислотные остатки и в частности, но не обязательно, идентичные аминокислотные остатки в одних и тех же положениях сравниваемых последовательностей. Схожие аминокислотные остатки группируются по химическим характеристикам боковых цепей в семейства. Указанные семейства описаны ниже для «консервативных аминокислотных замен». «Процентное сходство» между последовательностями является числом положений, содержащих идентичные или схожие остатки в одних и тех же положениях сравниваемых последовательностей, разделенным на общее число сравниваемых положений, и умноженным на 100%. Например, если 6 из 10 положений последовательности имеют идентичные аминокислотные остатки, а 2 из 10 положений содержат схожие остатки, то последовательности имеют 80% сходство. Сходство между двумя последовательностями можно определить, например, с применением EMBOSS Needle.
«Вариант» означает аминокислотную или нуклеотидную последовательность, которая отличается от исходной последовательности ввиду добавления (включая вставки), удаления и/или замены одного или нескольких аминокислотных остатков или нуклеотидных оснований при сохранении по меньшей мере одной необходимой активности исходной последовательности, раскрытой в настоящей заявке. В случае антител такая необходимая активность может включать специфическое связывание с антигеном. Подобным образом, вариант последовательности нуклеиновых кислот может быть модифицирован по сравнению с исходной последовательностью посредством добавления, удаления и/или замены одного или нескольких нуклеотидных оснований, но кодируемое антитело сохраняет необходимую активность, как описано выше. Варианты могут быть природными, такими как аллельные или сплайс-варианты, или могут быть искусственно сконструированными.
Использующийся в настоящей заявке термин «консервативные модификации» означает модификации, которые физически, биологически, химически или функционально подобны соответствующему образцу, например, имеют схожий размер, форму, электрический заряд, химические свойства, включая способность к образованию ковалентных или водородных связей, или тому подобному. Такие консервативные модификации включают замены, добавления и делеции одного или нескольких нуклеотидных оснований или аминокислот, но не ограничиваются ими.
Например, консервативные аминокислотные замены включают замены, в которых аминокислотный остаток замещен аминокислотным остатком, имеющим схожую боковую цепь. Например, аминокислотные остатки, являющиеся не существенными по отношению к связыванию с антигеном, можно заменить другим аминокислотным остатком из того же самого семейства, определяемого боковой цепью, например, серии может быть замещен треонином. Аминокислотные остатки обычно разделяют на семейства на основе общих, схожих свойств боковых цепей, таких как:
1) неполярные боковые цепи (например, глицин, аланин, валин, лейцин, изолейцин, метионин);
2) незаряженные полярные боковые цепи (например, аспарагин, глутамин, серии, треонин, тирозин, пролин, цистеин, триптофан);
3) основные боковые цепи (например, лизин, аргинин, гистидин, пролин);
4) кислые боковые цепи (например, аспарагиновая кислота, глутаминовая кислота);
5) бета-разветвленные боковые цепи (например, треонин, валин, изолейцин); и
6) ароматические боковые цепи (например, тирозин, фенилаланин, триптофан, гистидин).
Консервативная замена может также включать применение неприродной аминокислоты.
Неконсервативные замены, т.е. замены элементов из одного семейства элементами из другого семейства, могут приводить к существенным изменениям, например, по отношению к заряду, дипольному моменту, размеру, гидрофильности, гидрофобности, или конформации связывающего элемента, что может приводить к существенному падению связывающей активности, в частности, если затронуты аминокислоты, существенные для связывания с целевой молекулой. Неконсервативная замена может также включать применение неприродной аминокислоты.
Консервативные и неконсервативные модификации могут быть введены в исходные связывающие элементы с помощью множества стандартных методик, известных в данной области техники, таких как комбинаторная химия, сайт-специфический мутагенез ДНК, ПЦР-опосредованный и/или кассетный мутагенез, химический синтез пептидов/белков, химическая реакция, специфически модифицирующая реактивные группы в исходном связывающем элементе. Варианты могут быть протестированы рутинными способами в отношении их химических, биологических, биофизических и/или биохимических свойств.
Реакции гибридизации нуклеиновых кислот могут быть проведены в условиях различной жесткости. «Жесткие условия» широко известны и описаны в данной области техники. Как правило, во время реакции гибридизации можно применять буферный раствор на основе SSC, в котором SSC является 0,15 M NaCl и 15 мМ цитратным буферным раствором с pH 7,0. Повышенные концентрации буферного раствора и присутствие денатурирующего агента повышают жесткость этапа гибридизации. Например, условия гибридизации высокой жесткости могут включать применение (i) 50% (о/о) формамида, 5 × SSC (0,75 M NaCl, 0,075 M цитрата натрия), 50 мМ фосфата натрия (pH 6,8), 0,1% пирофосфата натрия, 5 × раствора Денхардта, обработанной ультразвуком ДНК из молок лососевых рыб (50 мкг/мл), 0,1 ДСН%, и 10% декстран сульфата при 42°C с отмыванием при 42°С в 0,2 × SSC и 0,1% ДСН; (ii) 50% (о/о) формамида с 0,1% бычьим сывороточным альбумином/0,1% фиколлом/0,1% поливинилпирролидоном/50 мМ натрий-фосфатным буферным раствором при pH 6,5 с 750 мМ хлоридом натрия, 75 мМ цитратом натрия при 42°С, или (iii) 10% декстран сульфатом, 2 × SSC, и 50% формамидом при 55°С, с последующим промыванием в жестких условиях, заключающихся в 0,1 × SSC с ЭДТА при 55°С. В дополнение или альтернативно, два или более этапов отмывания с применением отмывающих растворов низкой ионной силы и высокой температуры могут быть включены в протокол гибридизации, с использованием, например, 0,015 M хлорида натрия/0,0015 M цитрата натрия/0,1% додецилсульфата натрия при 50°С.
Различные аспекты настоящего изобретения описаны более подробно в следующих подразделах. Необходимо понимать, что различные варианты осуществления, предпочтения и диапазоны могут быть объединены, если необходимо. Далее, в зависимости от специфического варианта осуществления избранные определения, варианты осуществления или диапазоны могут не применяться.
В первом аспекте настоящее изобретение обеспечивает одновалентный фрагмент антитела, связывающий ИЛ-1-бета, который ингибирует биологический эффект ИЛ-1-бета человека с IC50 ниже 50 пМ. Указанное значение IC50 предпочтительно ниже 40 пМ, более предпочтительно ниже примерно 30, 20, 10, 5, 4, 3, 2 или 1 пМ.
Предпочтительно, указанный одновалентный фрагмент антитела имеет молекулярную массу примерно 50 кДа или ниже, такую как примерно 45 кДа, 40 кДа, 35 кДа или ниже, предпочтительно примерно 25 кДа, такую как 23, 24, 25, 26 или 27 кДа.
В одном аспекте настоящее изобретение обеспечивает антитело, содержащее (а) по меньшей мере одну из VH CDR последовательностей CDR-H1, CDR-H2 или CDR-Н3, указанных в SEQ ID No: 1, 2 и 3, соответственно, или их варианты; и/или (b) по меньшей мере одну из VL CDR последовательностей CDR-L1, CDR-L2 или CDR-L3, указанных в SEQ ID No: 4, 5 и 6, соответственно, или их варианты.
Такое антитело может содержать (а) по меньшей мере одну из VH CDR последовательностей CDR-H1, CDR-H2 или CDR-H3, указанных в SEQ ID No: 155, 156 и 157, соответственно, или их варианты; и/или (b) по меньшей мере одну из VL CDR последовательностей CDR-L1, CDR-L2 или CDR-L3 (i), указанных в SEQ ID No: 158, 159 и 160, соответственно, или их варианты, или (ii) указанных в SEQ ID No: 161, 162 и 163, соответственно, или их варианты.
Предпочтительно, антитело содержит по меньшей мере CDR-H3 из SEQ ID No: 3 и CDR-L3 из SEQ ID No: 6 или SEQ ID No: 157, или их вариант, соответственно. Еще более предпочтительно, указанное антитело содержит все шесть CDR из (i) SEQ ID No: 1-6 или их варианты; (ii) SEQ ID No: 155-160 или их варианты; или (iii) SEQ ID No: 155-157 и SEQ ID No: 161-163 или их варианты.
Указанное антитело обладает очень высокой ингибирующей активностью против ИЛ-1-бета человека с IC50 ниже 50 пМ, более предпочтительно ниже примерно 40 пМ, 30, 20, 10, и еще более предпочтительно ниже 5 пМ, и наиболее предпочтительно примерно 1 пМ и ниже.
Предпочтительно, антитело обладает ингибирующей активностью против ИЛ-1-бета человека с IC50 по меньшей мере 2 пМ, более предпочтительно по меньшей мере 1 пМ.
IC50 может быть определена с использованием анализа активности на основе клеток. В одном варианте осуществления значение IC50 определяют по ингибированию ИЛ-1-бета-индуцированного высвобождения ИЛ-6 из фибробластов человека. Такой анализ основан на том наблюдении, что фибробласты, стимулированные ИЛ-1-бета, высвобождают ИЛ-6. В присутствии антител, ингибирующих ИЛ-1-бета, концентрация высвобождаемого ИЛ-6 снижается. В предпочтительном варианте осуществления применяют фибробласты из здоровой кожи человека (NHDF-Neo, например, полученные от Lonza, Уолкерсвиль, США, кат. No: СС-2509). При инкубации со смесью чИЛ-1-бета и интересующего антитела, собирают надосадочную жидкость и исследуют с помощью ИФА на ИЛ-6, такого как ИФА набор для ИЛ-6 человека «R&D Systems Human IL-6 DuoSet ELISA kit» (R&D Systems, кат. No: DY206). В одном варианте осуществления анализ является анализом нейтрализации ИЛ-1-бета, как описано в Примере 3. Предпочтительно, значение IC50 является средним значением, полученным по результатам по меньшей мере трех повторов такого анализа.
Антитело, описанное в настоящей заявке, может быть полноразмерным иммуноглобулином или фрагментом антитела, таким как Fab, Fab', F(ab')2, scFv, Fv фрагмент, нанотело, VHH или минимальная единица распознавания.
В предпочтительном варианте осуществления антителом и, в частности, вышеуказанным одновалентным фрагментом антитела является scFv. VH и VL домены могут быть соединены в любой ориентации, VL-линкер-VH или VH-линкер-VL, посредством гибкого линкера. В предпочтительном варианте осуществления ориентацией является VL-линкер-VH, т.е. вариабельный участок легкой цепи находится на N-конце, а вариабельный участок тяжелой цепи находится на С-конце полипептида.
Антитело предпочтительно является гуманизированным. Такое гуманизированное антитело может, например, включать в вариабельной легкой цепи FR-L1 из SEQ ID No: 18, FR-L2 из SEQ ID No: 19, FR-L3 из SEQ ID No: 20 и/или FR-L4 из SEQ ID No: 21, или их варианты. В дополнение или альтернативно, гуманизированное антитело может содержать вариабельный каркасный участок тяжелой цепи FR-H1 из SEQ ID No: 22, 26 или 30; вариабельный каркасный участок тяжелой цепи FR-H2 из SEQ ID No: 23, 27 или 31; вариабельный каркасный участок тяжелой цепи FR-H3 из SEQ ID No: 24, 28 или 32; и/или вариабельный каркасный участок тяжелой цепи FR-H4 из SEQ ID No: 25, 29 или 33.
Таким образом, в предпочтительном варианте осуществления антитело содержит VH последовательность из SEQ ID No: 7 или SEQ ID No: 146, или их варианты, соответственно. Последовательность такого варианта имеет по меньшей мере 85%, более предпочтительно 90%, 91%,92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% и наиболее предпочтительно 100% идентичность с последовательностью SEQ ID No: 7 или SEQ ID No: 146. Примеры такого варианта VH последовательностей включают SEQ ID No: 121, SEQ ID No: 122, SEQ ID No: 124, SEQ ID No: 126, SEQ ID No: 128, SEQ ID No: 130, SEQ ID No: 132, SEQ ID No: 134, SEQ ID No: 142, SEQ ID No: 144, SEQ ID No: 146, SEQ ID No: 148, SEQ ID No: 150 или SEQ ID No: 152, но не ограничиваются ими.
В дополнение или в качестве альтернативы, антитело, раскрытое в настоящей заявке, включает VL последовательность, выбранную из группы, состоящей из SEQ ID No: 8, SEQ ID No: 136 и SEQ ID No: 145, или их варианта, соответственно. Последовательность такого варианта обладает по меньшей мере 85%, более предпочтительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, и наиболее предпочтительно 100% идентичностью с последовательностью SEQ ID No: 8, SEQ ID No: 136 или SEQ ID No: 145. Примеры такого варианта VL последовательностей включают SEQ ID No: 123, SEQ ID No: 125, SEQ ID No: 127, SEQ ID No: 129, SEQ ID No: 131, SEQ ID No: 133, SEQ ID No: 135, SEQ ID No: 136, SEQ ID No: 137, SEQ ID No: 139 или SEQ ID No: 153, но не ограничиваются ими.
В одном варианте осуществления антитело содержит VH последовательность, имеющую по меньшей мере 85%, более предпочтительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% и наиболее предпочтительно 100% идентичность с последовательностью SEQ ID No: 7 или SEQ ID No: 146. В дополнение или в качестве альтернативы, антитело содержит VL последовательность, имеющую по меньшей мере 85%, более предпочтительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% и наиболее предпочтительно, 100% идентичность с последовательностью SEQ ID No: 8, SEQ ID No: 136 или SEQ ID No: 145.
В наиболее предпочтительном варианте осуществления антитело содержит VH, как указано в SEQ ID No: 7, и VL, как указано в SEQ ID No: 8. Каркасные последовательности в обеих из SEQ ID No: 7 и SEQ ID No: 8 получены из иммуноглобулина человека, как описано в WO 03/097697 A (ESBATech AG). Его VH и VL каркасные последовательности были модифицированы для гуманизации и стабилизации антител кролика, см., например, WO 2009/155726 A (ESBATech, AN ALCON BIOMEDICAL RESEARCH UNIT LLC); BORRAS, L., et al. Generic approach for the generation of stable humanized single-chain Fv fragments from rabbit monoclonal antibodies. Journal of Biological Chemistry 2010, vol. 285, no. 12, p. 9054-9066 («Генетический подход к генерации стабильных гуманизированных одноцепочечных Fv фрагментов из кроличьих моноклональных антител»). В одном варианте осуществления VL каркас антитела, раскрытый в настоящей заявке, включает SEQ ID No: 18-21 или их варианты. В дополнение или в качестве альтернативы, VH каркас антитела включает SEQ ID No: 22-25, SEQ ID No: 26-29 или SEQ ID No: 30-33 или их варианты, соответственно.
В другом предпочтительном варианте осуществления антитело содержит VH, как указано в SEQ ID No: 146, и VL, как указано в SEQ ID No: 8 или в SEQ ID No: 145.
В другом предпочтительном варианте осуществления антитело содержит VH, как указано в SEQ ID No: 146, и VL, как указано в SEQ ID No: 136.
Антитело, в частности, в случае scFv, может содержать линкерную последовательность. Такая линкерная последовательность, как правило, имеет от 10 до примерно 25 аминокислот. Обычно такой линкерный пептид обогащен глицином, который обеспечивает гибкость, а также серином и/или треонином для улучшения растворимости. В предпочтительном варианте осуществления применяют (GGGGS)4 линкер (SEQ ID No: 9) или его вариант. Могут также применяться варианты указанного мотива, содержащего от трех до пяти повторов. Другие подходящие линкеры описаны, например, в ALFTHAN, K. «Properties of a single-chain antibody containing different linker peptides». Protein Engineering 1995, vol. 8, no. 7, p. 725-731 («Свойства одноцепочечного антитела, содержащего различные линкерные пептиды»).
В некоторых вариантах осуществления подразумеваются варианты антител, обеспеченных в настоящей заявке. Например, может быть необходимо улучшить связывание антигена, антитело-зависимую клеточно-опосредованную цитотоксичность (ADCC), комплемент-зависимую цитотоксичность (CDC) для повышения стабильности или растворимости, для снижения иммуногенности и/или изменения других биологических, биохимических или биофизических свойств антитела. В некоторых вариантах осуществления вариант не демонстрирует какого-либо улучшения по сравнению с исходным антителом.
Варианты антител, обеспеченные в настоящей заявке, могут быть приготовлены путем белковой и/или химической инженерии, введения подходящих модификаций в последовательность нуклеиновых кислот, кодирующую антитело, или путем белкового/пептидного синтеза. Любая комбинация(и) делеций, замен, добавлений и вставок может быть выполнена в каркасной области или в CDR, с тем условием, что полученное антитело обладает необходимыми характеристиками, для которых можно провести скрининг с применением подходящих способов. Особый интерес представляют замены, предпочтительно консервативные замены, как описано выше. Предпочтительные консервативные замены включают:
1. Замену аланина (А) на валин (V);
2. Замену аргинина (R) на лизин (K);
3. Замену аспарагина (N) на глутамин (Q);
4. Замену аспарагиновой кислоты (D) на глутаминовую кислоту (Е);
5. Замену цистеина (С) на серии (S);
6. Замену глутаминовой кислоты (Е) на аспарагиновую кислоту (D);
7. Замену глицина (G) на аланин (А);
8. Замену гистидина (Н) на аргинин (R) или лизин (K);
9. Замену изолейцина (I) на лейцин (L);
10. Замену метионина (М) на лейцин (L);
11. Замену фенилаланина (F) на тирозин (Y);
12. Замену пролина (Р) на аланин (А);
13. Замену серина (S) на треонин (Т);
14. Замену триптофана (W) на тирозин (Y);
15. Замену фенилаланина (F) на триптофан (W); и/или
16. Замену валина (V) на лейцин (L), и наоборот.
Антитело, описанное в настоящей заявке, может содержать одну или несколько, например, две, три, четыре, пять, шесть, семь, восемь, девять, десять, одиннадцать, двенадцать или больше таких консервативных замен.
Неконсервативные замены могут приводить к более существенным изменениям, например, в отношении заряда, дипольного момента, размера, гидрофильности, гидрофобности, или конформации полипептида. В одном варианте осуществления антитело содержит одну или несколько, например, две, три, четыре, пять, шесть, семь, восемь, девять, десять, одиннадцать, двенадцать или более таких неконсервативных замен.
Модификации могут присутствовать в CDR или в каркасных последовательностях. Например, CDR, обеспеченные в настоящей заявке, могут включать одну, две, три, четыре, пять или еще больше модификаций. Например, CDR-L1, CDR-L2 и CDR-L3 последовательности, взятые в целом, имеют по меньшей мере 75%, предпочтительно по меньшей мере 76%, 77%, 78%, 79%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или более предпочтительно 99% идентичности с CDR, обеспеченными в настоящей заявке, в частности, (i) с SEQ ID No: 4, 5 и 6, или (ii) с SEQ ID No: 161, 162 и 163. В дополнение или в качестве альтернативы, последовательности CDR-H1, CDR-H2 и CDRH3, взятые в целом, по меньшей мере на 80%, предпочтительно по меньшей мере на 81%, 82%, 83%, 84%, 95%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, или более предпочтительно на 99% идентичны CDR, обеспеченным в настоящей заявке, в частности, (i) SEQ ID No: 1, 2 и 3, или (ii) SEQ ID No: 155, 156 и 157.
В одном варианте осуществления CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2 и CDR-H3, взятые вместе, по меньшей мере на 85%, предпочтительно, на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, или более предпочтительно на 99% сходны с CDR, обеспеченным в настоящей заявке. В дополнение или в качестве альтернативы, CDR-L1, CDR-L2, CDR-L3, CDR-H1, CDR-H2 и CDR-H3, взятые в целом, по меньшей мере на 85%, предпочтительно на 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, или более предпочтительно на 99% сходны с CDR, обеспеченным в настоящей заявке.
Таким образом, вариант может, например, включать одну, две, три, четыре или пять замен в SEQ ID No: 4. Более предпочтительными являются замены в положениях, отмеченных X в SEQ ID No: 14. Вариант может, например, включать (i) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 32 вариабельной легкой цепи; (ii) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 33 вариабельной легкой цепи; и/или (iii) глутаминовую кислоту (Е), фенилаланин (F), глицин (G), метионин (М), аспарагин (N), глутамин (Q), серии (S), триптофан (W), тирозин (Y) в AHo положении 40 вариабельной легкой цепи.
В дополнение или в качестве альтернативы, вариант содержит одну, две, три или четыре замены в SEQ ID No: 5. Более предпочтительными являются замены в положениях, отмеченных X в SEQ ID No: 15. Такой вариант может, например, содержать (i) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), триптофан (W), тирозин (Y) в AHo положении 58 вариабельной легкой цепи; и/или (ii) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 69 вариабельной легкой цепи.
В дополнение или в качестве альтернативы, вариант содержит одну, две, три, четыре, пять или шесть замен в SEQ ID No: 6. Более предпочтительными являются замены в положениях, отмеченных X в SEQ ID No: 16. Такой вариант может, например, содержать (i) аланин (А), цистеин (С), изолейцин (I), аспарагин (N), серии (S), треонин (Т), валин (V) в AHo положении 109 вариабельной легкой цепи; (ii) аланин (А), глицин (G), пролин (Р), серии (S) в AHo положении 111 вариабельной легкой цепи; (iii) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 112 вариабельной легкой цепи; (iv) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 135 вариабельной легкой цепи; и/или (v) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лейцин (L), метионин (М), аспарагин (Ν), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Υ) в AHo положении 136 вариабельной легкой цепи.
В дополнение или в качестве альтернативы, вариант содержит одну, две, три или четыре замены в SEQ ID No: 1 или в SEQ ID No: 155. Более предпочтительными являются замены в SEQ ID No: 11. Такой вариант может, например, включать (i) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 33 вариабельной тяжелой цепи; и/или (ii) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 39 вариабельной тяжелой цепи.
В дополнение или в качестве альтернативы, вариант содержит одну, две, три, четыре, пять или шесть замен в SEQ ID No: 2 или в SEQ ID No: 156. Более предпочтительными являются замены в положениях, отмеченных X в SEQ ID No: 12. Такой вариант может, например, включать (i) аланин (А), цистеин (С), глицин (G), метионин (М) или тирозин (Y) в AHo положении 59 вариабельной тяжелой цепи; (ii) аспарагиновую кислоту (D), аспарагин (N) или пролин (Р) в AHo положении 60 вариабельной тяжелой цепи; и/или (iii) аланин (А), аспарагиновую кислоту (D), глутаминовую кислоту (Е), глицин (G), фенилаланин (F), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), серии (S), треонин (Т), триптофан (W) или тирозин (Y) в AHo положении 69 вариабельной тяжелой цепи.
В дополнение или в качестве альтернативы, вариант содержит одну, две, три, четыре, пять, шесть, семь, восемь, девять, десять или одиннадцать замен в SEQ ID No: 3 или в SEQ ID No: 157. Более предпочтительными являются замены в положениях, отмеченных X в SEQ ID No: 13. Такой вариант может, например, включать (i) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 110 вариабельной тяжелой цепи; (ii) аланин (А), цистеин (С), аспарагиновую кислоту (D), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 111 вариабельной тяжелой цепи; (iii) аланин (А), цистеин (С), фенилаланин (F), гистидин (Н), изолейцин (I), лейцин (L), метионин (М), аспарагин (N), глутамин (Q), серии (S), треонин (Т), валин (V), тирозин (Y) в AHo положении 112 вариабельной тяжелой цепи; (iv) фенилаланин (F) или изолейцин (I) в AHo положении 113 вариабельной тяжелой цепи; (v) аланин (А), цистеин (С), глутаминовую кислоту (Е), глицин (G), серии (S), треонин (Т), валин (V) в AHo положении 114 вариабельной тяжелой цепи; (vi) аланин (А), глицин (G), метионин (М) или аспарагин (N) в AHo положении 115 вариабельной тяжелой цепи; (vii) аланин (А), аспарагиновую кислоту (D), глутаминовую кислоту (Е), гистидин (Н), аспарагин (N), серии (S), треонин (Т) в AHo положении 135 вариабельной тяжелой цепи; (viii) аланин (А), цистеин (С), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лейцин (L), метионин (М), аспарагин (N), глутамин (Q), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 136 вариабельной тяжелой цепи; (ix) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 137 вариабельной тяжелой цепи; и/или (x) аланин (А), цистеин (С), аспарагиновую кислоту (D), глутаминовую кислоту (Е), фенилаланин (F), глицин (G), гистидин (Н), изолейцин (I), лизин (K), лейцин (L), метионин (М), аспарагин (N), пролин (Р), глутамин (Q), аргинин (R), серии (S), треонин (Т), валин (V), триптофан (W), тирозин (Y) в AHo положении 138 вариабельной тяжелой цепи.
Особо предпочтительным типом варианта является тот, в котором замещены один или несколько целых CDR. Как правило, CDR-H3 и CDR-L3 вносят наиболее значительный вклад в связывание антигена. Например, все CDR-L1, CDR-L2, CDR-H1 и/или CDR-H2 могут быть замещены отличающимся CDR природного или искусственного происхождения. В некоторых вариантах осуществления один или несколько CDR замещены аланиновой кассетой.
В одном варианте осуществления последовательность варианта, описанного в настоящей заявке, имеет по меньшей мере 85%, более предпочтительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, и наиболее предпочтительно 100% идентичность с последовательностью, выбранной из группы, состоящей из SEQ ID No: 10, SEQ ID No: 73 и SEQ ID No: 82.
В одном варианте осуществления последовательность варианта, описанного в настоящей заявке, имеет по меньшей мере 85%, более предпочтительно 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99%, и наиболее предпочтительно 100% сходство с последовательностью SEQ ID No: 10, SEQ ID No: 73 и SEQ ID No: 82.
В дополнение или в качестве альтернативы, VH из антитела включает точечные мутации, повышающие растворимость. WO 2009/155725 (ESBATech, Novartis company) описывает мотив, который, как было доказано, повышает общую растворимость антитела. Остатки размещены в положениях, находящихся в промежутке между вариабельным доменом и константным доменом антитела, и стабилизируют фрагменты антитела, в частности, scFv, не содержащие константный домен. В частности, присутствует один, предпочтительно все три из следующих остатков: (i) серии (S) в положении 12 аминокислоты тяжелой цепи (в соответствии с нумерацией AHo); (ii) серии (S) или треонин (Т) в положении 103 аминокислоты тяжелой цепи (в соответствии с нумерацией AHo); и/или (iii) серии (S) или треонин (Т) в положении 144 аминокислоты тяжелой цепи (в соответствии с нумерацией AHo).
В предпочтительном варианте осуществления антитело содержит серии в VH положении 12; серии в VH положении 103; и треонин в VH положении 144 (все в соответствии с нумерацией AHo).
Таким образом, в одном варианте осуществления антитело, раскрытое в настоящей заявке, содержит VH каркасные последовательности из SEQ ID No: 30-33, или их варианты.
Предпочтительно, вариант антитела, как применяется в настоящей заявке, (i) сохраняет специфическое связывание с ИЛ-1-бета, в частности, с чИЛ-1-бета; (ii) обладает активностью (IC50) в отношении ингибирования биологического эффекта ИЛ-1-бета человека ниже 500 пМ, предпочтительно ниже 400 пМ, 300 пМ, 200 пМ, 100 пМ, 50 пМ, более предпочтительно ниже 25 пМ; (iii) перекрестно реагирует с ИЛ-1-бета яванских макак, с ИЛ-1-бета макак-резус, и/или с ИЛ-1-бета крысы; и/или (iv) конкурирует с антителами, раскрытыми в настоящей заявке, за связывание с ИЛ-1-бета, предпочтительно ИЛ-1-бета человеке, ИЛ-1-бета яванских макак, ИЛ-1-бета макак-резус, и/или ИЛ-1-бета крысы, наиболее предпочтительно чИЛ-1-бета.
В одном варианте осуществления вариант содержит VL последовательность, выбранную из группы, состоящей из SEQ ID No: 96, SEQ ID No: 97, SEQ ID No: 98, SEQ ID No: 99, SEQ ID No: 100, SEQ ID No: 101, SEQ ID No: 102, SEQ ID No: 103, SEQ ID No: 104, SEQ ID No: 105, SEQ ID No: 123, SEQ ID No: 125, SEQ ID No: 127, SEQ ID No: 129, SEQ ID No: 131, SEQ ID No: 133, SEQ Ю No: 135, SEQ ID No: 136, SEQ ID No: 137, SEQ ID No: 139, SEQ ID No: 141, SEQ ID No: 143, SEQ ID No: 145, SEQ ID No: 147, SEQ ID No: 149, SEQ ID No: 151 и SEQ ID No: 153.
В дополнение или в качестве альтернативы, вариант включает VH последовательность, выбранную из группы, состоящей из SEQ ID No: 106, SEQ ID No: 107, SEQ ID No: 108, SEQ ID No: 109, SEQ ID No: 110, SEQ ID No: 111, SEQ ID No: 112, SEQ ID No: 113, SEQ ID No: 114, SEQ ID No: 115, SEQ ID No: 116, SEQ ID No: 117, SEQ ID No: 118, SEQ ID No: 119, SEQ ID No: 120, SEQ ID No: 121, SEQ ID No: 122, SEQ ID No: 124, SEQ ID No: 126, SEQ ID No: 128, SEQ ID No: 130, SEQ ID No: 132, SEQ ID No: 134, SEQ ID No: 138, SEQ ID No: 140, SEQ ID No: 142, SEQ ID No: 144, SEQ ID No: 146, SEQ ID No: 148, SEQ ID No: 150, SEQ ID No: 152.
Варианты могут также быть получены путем перестановки легкой и тяжелой цепей. Отдельную легкую цепь можно объединить с библиотекой из тяжелых цепей с получением библиотеки вариантов. В одном варианте осуществления указанная отдельная легкая цепь выбрана из группы из VL последовательностей, указанных выше, и/или указанная библиотека тяжелых цепей включает одну или несколько VH последовательностей, указанных выше. Подобным образом, отдельная тяжелая цепь может быть объединена с библиотекой из легких цепей. Предпочтительно, указанная отдельная тяжелая цепь выбрана из группы из VH последовательностей, указанных выше, и/или указанная библиотека из легких цепей включает одну или несколько из VL последовательностей, указанных выше.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 135 и/или VH из SEQ ID No: 7, SEQ ID No: 142, SEQ ID No: 146, SEQ ID No: 150 или SEQ ID No: 152. Предпочтительно, вариант включает SEQ ID No: 67, SEQ ID No: 85, SEQ ID No: 86, SEQ ID No: 87 или SEQ ID No: 88.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 136 и/или VH из SEQ ID No: 7, SEQ ID No: 142, SEQ ID No: 146, SEQ ID No: 150 или SEQ ID No: 152. Предпочтительно, вариант включает SEQ ID No: 68, SEQ ID No: 81, SEQ ID No: 82, SEQ ID No: 83 или SEQ ID No: 84.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 137 и/или VH из SEQ ID No: 7, SEQ ID No: 138, SEQ ID No: 142, SEQ ID No: 146, SEQ ID No: 150 или SEQ ID No: 152. Предпочтительно, вариант включает SEQ ID No: 69, SEQ ID No: 92, SEQ ID No: 93, SEQ ID No: 94 или SEQ ID No: 95.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 139 и/или VH из SEQ ID No: 140, SEQ ID No: 142, SEQ ID No: 146, SEQ ID No: 150 или SEQ ID No: 152. Предпочтительно, вариант включает SEQ ID No: 70, SEQ ID No: 77, SEQ ID No: 78, SEQ ID No: 79 или SEQ ID No: 80.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 141 и/или VH из SEQ ID No: 142. Предпочтительно, вариант включает SEQ ID No: 71.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 143 и/или VH из SEQ ID No: 144. Предпочтительно, вариант включает SEQ ID No: 72.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 145 и/или VH из SEQ ID No: 146. Предпочтительно, вариант включает SEQ ID No: 73.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 147 и/или VH из SEQ ID No: 148. Предпочтительно, вариант включает SEQ ID No: 74.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 149 и/или VH из SEQ ID No: 150. Предпочтительно, вариант включает SEQ ID No: 75.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 151 и/или VH из SEQ ID No: 152. Предпочтительно, вариант включает SEQ ID No: 76.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 8 и/или VH из SEQ ID No: 121 или SEQ ID No: 122. Предпочтительно, вариант включает SEQ ID No: 59 или SEQ ID No: 60.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 153 и/или VH из SEQ ID No: 142, SEQ ID No: 146 или SEQ ID No: 152. Предпочтительно, вариант включает SEQ ID No: 89, SEQ ID No: 90 или 91.
В одном варианте осуществления вариант содержит VL из SEQ ID No: 8 и/или VH из SEQ ID No: 121, SEQ ID No: 122, SEQ ID No: 142, SEQ ID No: 144, SEQ ID No: 146, SEQ ID No: 148, SEQ ID No: 150 или SEQ ID No: 152.
В одном варианте осуществления вариант содержит VH из SEQ ID No: 7 и/или VL из SEQ ID No: 135, SEQ ID No: 136, SEQ ID No: 137, SEQ ID No: 139 или SEQ ID No: 153.
В одном варианте осуществления вариант включает последовательность, выбранную из группы, состоящей из SEQ ID No: 34-95 и SEQ ID No: 154.
Связывающий элемент может содержать любую из VL и/или VH последовательностей, упомянутых выше. Связывающие элементы, имеющие формат единичного домена, такие как нанотело или VHH, содержат только одну из любой из VL или VH последовательностей, упомянутых выше, предпочтительно, VH последовательность. Поливалентные связывающие элементы, в частности, F(ab')2 фрагменты, бис-scFv или диатела, предпочтительно, биспецифические связывающие элементы, могут содержать одну или несколько из VL последовательностей, упомянутых выше, и/или одну или несколько из VH последовательностей, упомянутых выше.
Антитела из настоящего изобретения являются особенно стабильными. Использующийся в настоящей заявке термин «стабильность» относится к биофизическому свойству антитела сохранять мономерное состояние в растворе после длительной инкубации и/или после инкубации при повышенной температуре. Нестабильные антитела имеют тенденцию к димеризации или олигомеризации и даже к осаждению, что приводит к снижению срока хранения и снижению пригодности для фармацевтических приложений.
Антитела, обеспеченные в настоящей заявке, и в частности, одновалентный фрагмент антитела, остаются мономерными по меньшей мере на 75%, предпочтительно по меньшей мере на 80%, 85%, и наиболее предпочтительно до 93% после инкубации в течение 1 месяца при 37°С в концентрации 1 мг/мл в ФБР при pH 7,2. В дополнение или в качестве альтернативы, антитела остаются мономерными по меньшей мере на 90%, предпочтительно, по меньшей мере на 92%, 94%, 96%, 98%, более предпочтительно на 100% спустя 1 месяц при комнатной температуре при концентрации 1 мг/мл в ФБР при pH 7,2.
Содержание мономеров можно, например, определить с применением SEC-HPLC (гель-фильтрации - высокоэффективной жидкостной хроматографии). Подходящей мобильной фазой для такого анализа является, например, ФБР при pH 7,2. Содержание мономеров можно количественно определить путем интеграции пика UV280 сигнала, измеренного при хроматографии белков. Подходящей системой является, например, Dionex Summit HPLC, контролируемая программным обеспечением Chromeleon® 6.5, которая также позволяет проводить последующий анализ хроматограмм и количественный анализ пиков.
Антитела, раскрытые в настоящей заявке, и в частности, одновалентный фрагмент антитела, указанный выше, также являются стабильными при более высоких концентрациях, например, они остаются мономерными по меньшей мере до 50%, предпочтительно, по меньшей мере до 55%, 60%, 65%, 70%, и наиболее предпочтительно, до 75% после инкубации в течение 2 недель при комнатной температуре и/или при 4°С при концентрации около 50 мг/мл в ФБР при pH 7,2.
Далее, антитела, раскрытые в настоящей заявке, и в частности, вышеуказанный одновалентный фрагмент антитела является особенно растворимым, и таким образом, может иметь высокую концентрацию без осаждения из-за формирования агрегатов. Предпочтительно, антитела могут быть концентрированными в ФБР при pH 7,2 до концентрации более 20 мг/мл без осаждения, более предпочтительно до концентрации 30 мг/мл, 40 мг/мл, 50 мг/мл, 60 мг/мл, и наиболее предпочтительно 70 мг/мл в ФБР при pH 7,2.
В более предпочтительном варианте осуществления антитело имеет температуру плавления около 60°С, определяемой дифференциальной сканирующей калориметрией (ДСК), предпочтительно, 65°С, 70°С, 71°С, 72°С, 73°С, и наиболее предпочтительно 74°С. Этот способ основан на свойствах определенных красителей к флуоресценции только в гидрофобной среде. Например, можно выявить развертывание белка в виде повышения флуоресценции при связывании с красителем SYPRO®Orange с белком, денатурировавшим при нагревании (NIESEN F.H. et al. «The use of differential scanning fluorimetry to detect ligand interactions that promote protein stability)). Nature Protocols 2007, vol. 2, p. 2212-2221 («Применение дифференциальной сканирующей флуориметрии для детекции взаимодействий с лигандом, стимулирующих стабильность белка»)). Таким образом, стабильность белка можно анализировать посредством термической денатурации.
Антитело предпочтительно имеет теоретическую изоэлектрическую точку (pI) в диапазоне от 5 до 10, предпочтительно от 7 до 9, наиболее предпочтительно около 8,3. Теоретическое значение pI можно, например, рассчитать с применением ProtParam tool на сервере ExPASy (на http://web.expasy.org/protparam/; см. также GASTEIGER Е. et al. «Protein Identification and Analysis Tools on the ExPASy Server. (In) The Proteomics Protocols Handbook)). Edited by WALKER J.M. Totowa: Humana Press Inc., 2005. ISBN 9781588295934. р. 571-607 («Средства идентификации и анализа белков на сервере ExPASy. В «Справочнике протеомных протоколов»»)).
Антитело может обладать перекрестной реактивностью с ИЛ-1-бета из видов, не являющихся человеком, таких как ИЛ-1-бета яванских макак, ИЛ-1-бета макак-резус, ИЛ-1-бета крысы, ИЛ-1-бета мыши, ИЛ-1-бета собаки, ИЛ-1-бета кошки, ИЛ-1-бета мартышки, ИЛ-1-бета свиньи, и/или ИЛ-1-бета морской свинки, но не ограничивается ими. Предпочтительно, антитело обладает перекрестной реактивностью с ИЛ-1-бета яванских макак (например, рекомбинантным и доступным от Sino Biological Inc., кат. No: 90010-CNAE), ИЛ-1-бета макак-резус (например, рекомбинантным и доступным от R&D Systems, кат. No: 1318-RL/CF), и/или ИЛ-1-бета крысы (например, рекомбинантным и доступным от Peprotech, кат. No: 400-501В).
Предпочтительно, не остается остаточной активности ИЛ-1-бета при нейтрализации с антителом, раскрытым в настоящей заявке, в приложениях in vivo и/или in vitro, т.е. антитело полностью ингибирует активность ИЛ-1-бета. Термин «отсутствие остаточной активности», использующийся в настоящей заявке, означает менее 2% мощности сигнала, соответствующего высвобождению ИЛ-6 из фибробластов человека, индуцированному 10 пг/мл ИЛ-1-бета, предпочтительно в анализе, описанном в Примере 3, в присутствии 60 нг/мл антитела, описанного в настоящей заявке, по сравнению с антителами другой специфичности или с растворителем в качестве контроля в той же самой концентрации.
Настоящее изобретение также обеспечивает связывающий элемент, конкурирующий с антителами, раскрытыми в настоящей заявке, за связывание с ИЛ-1-бета человека.
Использующиеся в настоящей заявке термин «конкурирующий» относится к конкуренции между связывающими элементами за связывание с одной и той же мишенью. Конкуренцию можно определить посредством анализа конкурентного связывания, в котором исследуемый связывающий элемент предотвращает или подавляет, или снижает специфическое связывание антител, раскрытых в настоящей заявке, с общим антигеном (здесь чИЛ-1-бета или его фрагмент). Такие анализы конкурентного связывания известны в данной области техники и включают твердофазный прямой или непрямой радиоиммунный анализ (РИА) или твердофазный прямой или непрямой иммуноферментный анализ (ИФА), но не ограничиваются ими. Как правило, такие анализы включают применение очищенного антигена, связанного с твердой поверхностью, анализируемого связывающего элемента и контрольного антитела, как описано в настоящей заявке. Конкурентное ингибирование измеряют путем определения количества либо (i) контрольного антитела, связанного с твердой поверхностью в присутствии анализируемого связывающего элемента, или (ii) тестируемого связывающего элемента, связанного с твердой поверхностью в присутствии контрольного антитела. Конкурирующий связывающий элемент может связываться (i) с тем же самым эпитопом, что и антитело, (ii) с перекрывающимся эпитопом, или (iii) с другим эпитопом на той же самой целевой молекуле, но стерически затрудняющим связывание антитела с мишенью.
Как правило, если при конкуренции связывающий элемент присутствует в избытке, он снижает специфическое связывание антитела, как описано в настоящей заявке, с ИЛ-1-бета по меньшей мере на 40-45%, 45-50%, 50-55%, 55-60%, 60-65%, 65-70%, 70-75% или 75% или больше. Предпочтительно, связывание антитела снижается по меньшей мере на 80-85%, 85-90%, 90-95%, 95-97%, или 97% или больше.
В одном варианте осуществления конкурирующий связывающий элемент связывается с чИЛ-1-бета с аффинностью KD по меньшей мере примерно 1 пМ, 10 пМ, 100 пМ, 500 пМ, 1 нМ, 10 нМ.
В одном варианте осуществления связывающий элемент является одновалентным, таким как scFv или Fab фрагмент. В другом варианте осуществления связывающий элемент является поливалентным. Такая поливалентная молекула может быть двухвалентной (такой как полноразмерный иммуноглобулин или F(ab')2 фрагмент), или содержит по меньшей мере три целевых участка связывания.
Поливалентный связывающий элемент может быть биспецифическим антителом, таким как диатело, одноцепочечное диатело или тандемный scFv (см., например, KONTERMANN, R.E. «Methods in Molecular Biology». Edited by LO, В. Totowa, N.J.: Humana Press, 2004, ISBN 1588290921. p. 227-242 («Методы в молекулярной биологии»)). В указанных биспецифических антителах могут использоваться более короткие линкеры, чем те, которые описаны выше для scFv, т.е., имеющие только от одного до трех повторов основного мотива из SEQ ID No: 14 (см., например, HOLLIGER, P., et al. «Diabodies: small bivalent and bispecific antibody fragments». PNAS1993, vol. 90, no. 14, p. 6444-6448 («Диатела: малые двухвалентные и биспецифические фрагменты антител»)). В другом варианте осуществления поливалентный связывающий элемент является триателом, мини-телом или тетрателом.
Настоящее изобретение также обеспечивает Т-тела, содержащие антитела, раскрытые в настоящей заявке. Т-тела являются иммуноглобулиновыми Т-клеточными рецепторами (cIgTCR), объединяющими распознавание антигена антителами с сигнальными и эффекторными свойствами комплекса Т-клеточного рецептора. В таких конструкциях антитело предпочтительно является фрагментом антитела, таким как Fv, Fab, scFv или scFv-Fc, наиболее предпочтительно scFv. Для дополнительного описания общей конструкции Т-тел и их применения см., например, SCHIRRMANN, Т. and Pecher, G. «Handbook of Therapeutic Antibodies». Edited by DÜBEL, S. Weinheim: Wiley-VCH, 2009. ISBN 3527314539. p. 533-561 («Справочник по антителам»).
Настоящее изобретение также обеспечивает наивный (т.е. не модифицированный для повышения аффинности или активности) связывающий элемент для ИЛ-1-бета, обладающий одновалентной активностью (например, измеренной по аффинности (KD) или биологической активности в анализах на клеточной основе (IC50) в единицах в моль/л) при определенной молекулярной массе (в г/моль) после нормализации по числу мест связывания на связывающий элемент, как определено в уравнении: К = (одновалентная активность (моль/л) × молекулярная масса (г/моль))/ число участков связывания.
Как описано выше, одновалентные фрагменты антитела, обладающие значениями активности в пикомолярном диапазоне, являются специфическими, особенными и не полученными рутинным образом. Активность часто коррелирует с размером связывающего элемента: высокая активность в пикомолярном диапазоне может быть достигнута с помощью полноразмерных иммуноглобулинов, в то время как очень малые фрагменты антител, такие как нанотела или минимальные единицы распознавания, или малые не-антительные скелеты, такие как аффилины, часто демонстрируют низкие значения активности, т.е. в наномолярном диапазоне. По-видимому, имеется минимум для указанной функции К, обеспеченный scFv, как описано в настоящей заявке: чем меньше связывающий элемент, и чем выше его одновалентная активность или аффинность, и чем больше участков связывания на молекулу, тем меньше К. Например, для scFv, как описано в настоящей заявке, нижний предел К равен примерно 50 нг/л, в то время как верхний предел К равен примерно 12500 нг/л; для соответствующих полноразмерных иммуноглобулинов нижний предел равен примерно 150 нг/л, а верхний К предел равен примерно 37500 нг/мл, для других связывающих элементов с молекулярной массой меньше scFv, как описано в настоящей заявке, значение К составляет больше 500000 нг/л. В предпочтительном варианте осуществления значение К составляет примерно 50 нг/л, 100 нг/л, 200 нг/л, 500 нг/л, 750 нг/л, 1000 нг/л, 1250 нг/л, 1500 нг/л, 1750 нг/л, 2000 нг/л, 2250 нг/л, или 2500 нг/л.
Нуклеиновые кислоты, векторы, клетки-хозяева, и способ получения
Антитела, описанные в настоящей заявке, кодируются одной нуклеиновой кислотой или двумя или более нуклеиновыми кислотами, например, каждая из которых кодирует по меньшей мере один вариабельный участок. Зная последовательность антитела или его частей, можно сгенерировать кДНК, кодирующие полипептидную последовательность, посредством способов, известных в данной области техники, например, путем генного синтеза. Эти кДНК может быть клонирована посредством методик стандартного клонирования и мутагенеза в подходящий вектор, такой как вектор экспрессии или клонирующий вектор. Вариабельная легкая цепь необязательно кодируется отдельной нуклеиновой кислотой, иной, чем вариабельная тяжелая цепь антитела. Далее, дополнительные последовательности, такие как метки (например, гистидиновая метка), константные домены для получения Fab или полноразмерного иммуноглобулина, линкеры, кодирующая последовательность второй специфичности связывания, или другой функциональный полипептид, такой как фермент для генерации гибридной конструкции или биспецифической молекулы, могут быть включены в генетическую конструкцию.
На основе выбранной стратегии клонирования, генетические конструкции могут создавать антитело, имеющее один или несколько дополнительных остатков на N- или С-конце. Например, N-концевой метионин, полученный из исходного кодона, или дополнительный аланин может быть представлен в экспрессируемом полипептиде, если только они не вырезаны после трансляции. Таким образом, необходимо понимать, что антитела, раскрытые в настоящей заявке, скорее содержат раскрытые последовательности, чем состоят из них.
В одном варианте осуществления настоящее изобретение обеспечивает последовательность нуклеиновых кислот, содержащую по меньшей мере 300 нуклеотидных оснований, более предпочтительно по меньшей мере 350, 400, 450, или 500 нуклеотидных оснований, и имеющую по меньшей мере 85%, более предпочтительно по меньшей мере 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% идентичность с последовательностью SEQ ID No: 17. В более предпочтительном варианте осуществления последовательностью нуклеиновых кислот является SEQ ID No: 17.
В дополнение или в качестве альтернативы, настоящее изобретение обеспечивает последовательность нуклеиновых кислот, содержащую по меньшей мере 300 нуклеотидных оснований, более предпочтительно по меньшей мере 350, 400, 450 или 500 нуклеотидных оснований, которые гибридизуются с нуклеиновой кислотой из SEQ ID No: 17 в жестких условиях.
Основные протоколы методик стандартного клонирования, мутагенеза и молекулярной биологии описаны, например, в «Molecular Cloning, A Laboratory Manual» (GREEN, M. and Sambrook, J. Molecular Cloning: a Laboratory Manual. 4th edition. Cold Spring Harbor Laboratory, 2012. ISBN 1936113422) («Молекулярное клонирование, лабораторное руководство»).
Подходящие клетки-хозяева для экспрессии генетической конструкции могут быть прокариотическими или эукариотическими. Подходящими прокариотическими клетками-хозяевами являются грамотрицательные или грамположительные клетки, и включают виды семейств Escherichia, Erwinina, Enterobacter, Klebsiella, Pseudomonas или Bacillus. Наиболее предпочтительным видом является Escherichia coli, в частности, штаммы Е. coli BL21 (DE3) (Life Technologies ТМ, кат. No: С6000-03) и Origami™ 2(DE3) (Novagen, кат. No: 71345).
Если необходимы посттрансляционные модификации, такие как гликозилирование или фосфорилирование, предпочтительными являются эукариотические клетки-хозяева. Например, эукариотические микроорганизмы, такие как обычно применяемые штаммы Saccharomyces cerevisiae или Pichia pastoris, могут служить в качестве клеток-хозяев. Клетки-хозяева могут также включать растительные или животные клетки, в частности, клетки насекомых или млекопитающих. Подходящие клетки млекопитающих включают клетки яичников китайского хомячка (СНО), клетки почек эмбриона человека (НЕК), эндотелиальные клетки пуповинной вены человека (HUVEC) или клетки миеломы NS0.
Антитело может быть получено путем экспрессии в подходящей клетке-хозяине. Например, векторы экспрессии, описанные выше, вводят в клетку-хозяин путем стандартных методик, таких как электропорация или химическая трансформация. Трансформированные клетки затем культивируют в условиях, подходящих для экспрессии рекомбинантного белка, как правило, в подходящей питательной среде, необязательно модифицированной для индукции промоторов, селекции трансформантов, или амплификации необходимых кодирующих последовательностей. Антитело выделяют из культуры и необязательно очищают с применением стандартных методик из данной области техники. Выход рекомбинантного белка может быть повышен путем оптимизации сред и условий культивирования, таких как температура или подача кислорода. В прокариотах антитело можно получить в периплазме, внутриклеточно в виде телец включения или секретированными в среду. При выделении белок может быть очищен с применением способов, хорошо известных в данной области техники, таких как гель-фильтрация, ионообменная хроматография, обращено-фазовая хроматография, гидрофобное взаимодействие, хроматография комбинированного режима и/или аффинная хроматография.
В одном варианте осуществления антитело получают в системе, не содержащей клеток. Это, как правило, включает транскрипцию in vitro с последующей трансляцией in vitro продукционных матриц нуклеиновых кислот, кодирующих белок, описанный в настоящей заявке, например, плазмидной ДНК или ПЦР продукционных матриц. Например, применяют неочищенные лизаты из выращенных клеток, обеспечивающих необходимые ферменты, а также компоненты синтеза белков клетки. Необходимые строительные элементы, такие как аминокислоты или нуклеотидные основания, а также молекулы, доставляющие энергию и другие, могут поставляться экзогенно. Системы экспрессии, не содержащие клеток, могут, например, быть основаны на лизированных ретикулоцитах кролика (например, Система лизата ретикулоцитов кролика, Promega, кат. No:. L4540), клетках HeLa (например, 1-этапный набор для трансляции клеток человека in vitro, Thermo Scientific, кат. No: 88881), клетках насекомых (например, EasyXpress Insect Kit II, Qiagen, кат. No:. 32561), зародышах пшеницы (например, Экстракт зародышей пшеницы, кат. No: L4380), или клетках Е. coli (например, набор для синтеза белков in vitro PURExpress®, NEB, кат. No:. E6800S). Также для получения можно использовать оптимизированные не содержащие клеток системы экспрессии антитела для улучшения образования дисульфидных связей. Коммерческие наборы включают лизаты клеток насекомых (например, EasyXpress Disulfide Insect Kit, Qiagen, кат. No: 32582) или лизаты клеток Ε. coli (например, EasyXpress Disulfide Ε. coli Kit, Qiagen, кат. No:. 32572). Бесклеточный синтез белка обладает, например, тем преимуществом, что он является быстрым, обеспечивает высокий выход продукта, позволяет легко модифицировать условия реакции, с образованием малого количества побочных продуктов или даже при их отсутствии. Бесклеточный синтез белка может включать биологические и/или химические этапы, которые не могут быть проведены в чисто биологических или химических системах продукции. Например, неприродные или химически модифицированные аминокислоты могут быть встроены в белок в необходимых положениях. Гибридные белки ScFv-токсин были успешно получены в системах, не содержащих клеток (NICHOLLS, P.J., et al. «Characterization of single-chain antibody (sFv)-toxin fusion proteins produced in vitro in rabbit reticulocyte lysate». Journal of Biological Chemistry 1993, vol. 268, pp. 5302-5308 («Характеристика одноцепочечных антител (sFv)-токсин гибридных белков, полученных in vitro в лизате кроличьих ретикулоцитов»). Таким образом, в одном варианте осуществления обеспечивается способ получения антитела, описанного в настоящей заявке, вышеуказанного связывающего элемента или вышеуказанного Т-тела, включающий этапы (а) обеспечения системы, не содержащей клеток, (b) обеспечения продукционной матрицы нуклеиновых кислот, кодирующих антитело, описанное в настоящей заявке, вышеуказанный связывающий элемент или вышеуказанное Т-тело, (с) обеспечения транскрипции и трансляции указанной продукционной матрицы нуклеиновых кислот; (d) выделения; и необязательно (е) очистки указанного антитела, указанного связывающего элемента или указанного Т-тела, соответственно.
В дополнение или в качестве альтернативы, способ получения антитела, описанный в настоящей заявке, включает по меньшей мере один этап химического синтеза. Например, способ может быть полностью химическим. В другом варианте системы продукции на клеточной основе или без клеток, описанные выше, включают такой по меньшей мере один этап химического синтеза.
В предпочтительном варианте осуществления антитела, описанные в настоящей заявке, получают в системе на основе клеток, с применением вектора экспрессии для внутриклеточной экспрессии в Е. coli. При экспрессии полипептид образуется в качестве телец включения внутри клеток, которые отделяются от других клеточных частиц с последующей солюбилизацией в денатурирующем агенте, таком как гуанидина гидрохлорид (GndHCl), и подвергаются рефолдингу посредством процедур ренатурации, хорошо известных специалистам в данной области техники.
Необходимо понять, что нуклеиновые кислоты, векторы, клетки-хозяева и способ получения, описанный выше, также применяются к связывающим элементам (в той мере, насколько они являются белками) и/или к Т-телам, описанным в настоящей заявке.
Химические и/или биологические модификации
В одном аспекте антитело согласно настоящему изобретению является химически и/или биологически модифицированным. Такая модификация может включать гликозилирование, ПЭГилирование, ГЭКилирование, технологию гибридизации с альбумином, ПАСилирование, маркирование красителями и/или радиоизотопами, конъюгацию с ферментами и/или токсинами, фосфорилирование, гидроксилирование и/или сульфатирование, но не ограничивается ими. Подобным образом, любой связывающий элемент, последовательность нуклеиновых кислот, вектор и/или клетка-хозяин, описанная выше, могут быть модифицированы соответствующим образом.
Химические и/или биологические модификации могут быть проведены для оптимизации фармакодинамики или растворимости в воде белков, или для уменьшения их побочных эффектов. Например, ПЭГилирование, ПАСилирование и/или ГЭКилирование могут применяться для замедления почечного клиренса и таким образом, для повышения времени полужизни антитела в плазме крови. В дополнение или в качестве альтернативы, модификация может добавлять другую функциональность белку, например, токсин для более эффективного уничтожения раковых клеток, или молекулу детекции для диагностических целей.
Гликозилирование означает процесс присоединения углеводов к белкам. В биологических системах этот процесс осуществляется ферментативно внутри клетки в виде котрансляционной и/или посттрансляционной модификации. Белок, в данном случае антитело, может также быть химически гликозилирован. Как правило, гликозилирование бывает (i) N-связанным с азотом боковых цепей аспарагина или аргинина; (ii) O-связанным с кислородом гидрокси-группы боковых цепей серина, треонина, тирозина, гидрокислизина или гидроксипролина; (iii) включающим присоединение ксилозы, фукозы, маннозы и N-ацетилглюкозамина к фосфо-серину, или (iv) в форме C-маннозилирования, где сахар манноза присоединяется к остатку триптофана, находящемуся в специфическом участке распознавания, но не ограничивается ими. Характер гликозилирования может, например, контролироваться путем выбора подходящих линий клеток, питательных сред, способов инженерии белков и стратегий процесса (HOSSLER, P. «Optimal and consistent protein glycosylation in mammalian cell culture». Glycobiology 2009, vol. 19, no. 9, p. 936-949 («Оптимальное и согласованное гликозилирование белков в культуре клеток млекопитающих»).
Инженерия белков для контроля или изменения характера гликозилирования может включать удаление и/или добавление одного или нескольких участков гликозилирования. Создание участков гликозилирования может предпочтительно достигаться путем введения соответствующей последовательности распознавания ферментами в последовательность аминокислот антитела, или путем добавления или замены одного или нескольких вышеуказанных остатков аминокислот.
Может быть необходимым ПЭГилирование антитела. ПЭГилирование может изменять фармакодинамические и фармакокинетические свойства белка. Полиэтиленгликоль (ПЭГ) подходящей молекулярной массы ковалентно присоединяют к каркасу белков (см., например, PASUT, G. and Veronese, F. «State of the art in PEGylation: the great versatility achieved after forty years of research». Journal of Controlled Release 2012, vol. 161, no. 2, p. 461-472 («Современное ПЭГилирование: разносторонние достижения спустя сорок лет исследований»)). ПЭГилирование может дополнительно снижать иммуногенность путем экранирования ПЭГилированного белка от иммунной системы и/или изменения его фармакокинетики, например, путем повышения стабильности антитела in vivo, защиты от протеолитической деградации, увеличения времени полужизни, и путем изменения его распределения в организме.
Подобные эффекты могут достигаться путем миметиков ПЭГ, например, ГЭКилирования или ПАСилирования антитела. Для ГЭКилирования используют производные гидроксиэтилкрахмала («ГЭК»), в то время как при ПАСилировании антитело связывается с конформационно разупорядоченными полипептидными последовательностями, состоящими из аминокислот пролина, аланина и серина. Указанные миметики ПЭГ и родственные соединения описаны, например, в BINDER, U. and Skerra, A. «Half-Life Extension of Therapeutic Proteins via Genetic Fusion to Recombinant PEG Mimetics», in «Therapeutic Proteins: Strategies to Modulate Their Plasma Half-Lives». Edited by KONTERMANN, R., Weinheim, Germany: Wiley-VCH, 2012. ISBN: 9783527328499. p. 63-81 («Увеличение полужизни терапевтических белков посредством генетической гибридизации с рекомбинантными ПЭГ миметиками», в «Терапевтические белки: Стратегии модуляции времени их полужизни в плазме»).
Антитело может включать эпитоп, и в частности, эпитоп связывания с рецептором реутилизации. Такой эпитоп связывания с рецептором реутилизации, как правило, означает эпитоп Fc области молекулы IgG (например, IgG1, IgG2, IgG3 или IgG4), и оказывающий влияние на повышение времени полужизни молекулы in vivo.
В дополнение или в качестве альтернативы, антитело является маркированным или конъюгированным со вторым компонентом, которому приписываются дополнительные функции после связывания с мишенью. Указанный второй компонент может, например, выполнять дополнительную иммунологическую эффекторную функцию, быть эффективным для лекарственного таргетинга или пригодным для детекции. Второй компонент может, например, быть химически связанным или генетически гибридизованным с антителом с применением способов, известных в данной области техники.
Молекулы, которые могут служить в качестве второго компонента, включают радионуклиды, также называемые радиоизотопами (например, 35S, 32Р, 14С, 18F, 125I); апоферменты; ферменты (такие, как щелочная фосфатаза, пероксидаза хрена, бета-галактозидаза или ангиогенин); кофакторы; пептиды (например, гистидиновые метки); белки (включая лектины); углеводы (включая маннозо-6-фосфатную метку); флуорофоры (включая флуоресцеин изотиоцианат (ФИТЦ); фикоэритрин; зеленый/синий/красный и другие флуоресцентные белки; аллофикоцианин (АФЦ); хромофоры; витамины (включая биотин); хелаторы; антиметаболиты (например, метотрексат); липосомы; токсины, включая цитотоксические лекарства, такие как таксол, грамицидин D или колхицин; или радиотоксин, но не ограничиваются ими.
Маркированное антитело особо пригодно для детекции in vitro и in vivo, или диагностических целей. Например, антитело, маркированное подходящим радиоизотопом, ферментом, флуорофором или хромофором, может быть детектировано посредством радиоиммунного анализа (РИА), иммуноферментного анализа (ИФА), или анализа отдельных клеток на основе флуориметрии (например, FACS анализа), соответственно. Подобным образом, нуклеиновые кислоты и/или векторы, раскрытые в настоящей заявке, могут применяться для детекции или диагностических целей, например, с применением маркированных фрагментов в качестве зондов при гибридизационном анализе. Протоколы маркирования можно найти, например, в JOHNSON, I. and Spence, M. T.Z. «Molecular Probes Handbook, A Guide to Fluorescent Probes and Labeling Technologies». Life Technologies, 2010. ISBN: 0982927916 («Справочник по молекулярным зондам, Руководство по флуоресцентным зондам и технологиям маркировки»).
Композиции
Антитело согласно настоящему изобретению, любой связывающий элемент, последовательность нуклеиновых кислот или вектор, раскрытый в настоящей заявке, могут быть обеспечены в виде композиции, дополнительно включающей подходящий носитель, наполнитель или разбавитель. Наиболее предпочтительной является композиция, содержащая антитело, описанное в настоящей заявке.
Такая композиция может быть, например, диагностической, косметической или фармацевтической композицией. Для терапевтических или косметических целей указанная композиция является фармацевтической композицией, содержащей фармацевтический носитель, наполнитель или разбавитель, т.е. нетоксичной в применяемых дозировках и концентрации.
Подходящие «носители», «наполнители» или «разбавители» включают: (i) буферные агенты, такие как фосфат, цитрат или другие, органические кислоты; (ii) антиоксиданты, такие как аскорбиновая кислота и токоферол; (iii) консерванты, такие как 3-пентанол, гексаметония хлорид, бензалкония хлорид, бензиловый спирт, алкил-парабен, катехол или циклогексанол; (iv) аминокислоты, например, такие как гистидин, аргинин; (v) пептиды, предпочтительно до 10 остатков, такие как полилизин; (vi) белки, такие как бычий сывороточный альбумин или сывороточный альбумин человека; (vii) гидрофильные полимеры, такие как поливинилпирролидон; (viii) моносахариды, дисахариды, полисахариды и/или другие углеводы, включая глюкозу, маннозу, сахарозу, маннитол, трегалозу, сорбитол, аминодекстран или полиамидоамины; (ix) хелатирующие агенты, например, ЭДТА; (х) солеобразующие ионы, такие как натрий; (xi) комплексы металлов (например, комплексы цинка и белка); и/или (xii) ионные или неионные сурфактанты, такие как Твин™, Плюроники™ или полиэтиленгликоль (ПЭГ), но не ограничиваются ими.
Многие из указанных соединений, приведенных в качестве примеров, выполняют различные функции, и могут, например, действовать в качестве носителя и в качестве разбавителя. Также необходимо понимать, что композиция может содержать более одного из каждого из носителя, разбавителя или наполнителя. Антитело, связывающий элемент, последовательности нуклеиновых кислот или вектор могут быть обеспечены на твердых материалах подложки, таких как бусы и микрочастицы. Как правило, молекулы связаны с таким носителем ковалентной связью (необязательно, с применением линкера), нековалентно или в качестве присадки. Указанные бусы и микрочастицы могут содержать, например, крахмал, целлюлозу, полиакрилат, полиацетат, полигликолят, поли(лактид-со-гликолид), латекс или декстран.
Терапевтические применения
Молекулы, описанные в настоящей заявке, в частности, антитело, связывающий элемент, нуклеиновая кислота или вектор, пригодны в качестве медикамента. Как правило, такой медикамент содержит терапевтически эффективное количество молекул, обеспеченных в настоящей заявке. Соответственно, указанные молекулы могут применяться для производства медикамента, пригодного в лечении ИЛ-1-бета-зависимых заболеваний.
В одном аспекте обеспечивается способ лечения ИЛ-1-бета-зависимого заболевания, включающий этапы введения фармакологически эффективного количества молекул, описанных в настоящей заявке, в частности, антитела, субъекту, нуждающемуся в таком лечении. В одном варианте осуществления вышеуказанную фармацевтическую композиция (т.е. медикамент), содержащую такое фармацевтически эффективное количество антитела, вводят указанному субъекту.
Термин «лечить» или «лечение», использующийся в настоящей заявке, означает введение фармацевтически эффективного количества антитела, связывающего элемента, нуклеиновой кислоты, вектора или клетки-хозяина согласно настоящему изобретению, субъекту, нуждающемуся в нем, для профилактики, лечения, замедления проявления и/или прогрессирования, снижения тяжести, стабилизации, модуляции, излечения или облегчения одного или нескольких симптомов ИЛ-1-бета-зависимого заболевания. Как правило, антитело, связывающий элемент, нуклеиновая кислота, вектор или клетка-хозяин обеспечиваются в фармацевтической композиции, включая те, что описаны выше в настоящей заявке.
«Терапевтически эффективное количество» означает количество, которое в режиме применяемой дозировки дает необходимый терапевтический эффект, т.е. достигает целей лечения, как определено выше. Дозировка зависит от различных факторов, включая факторы, связанные с пациентом, и клинические факторы (например, возраст, массу тела, пол, историю болезни пациента, тяжесть заболевания и/или ответ на лечение), природу заболевания, подлежащего лечению, конкретную применяемую композицию, способ введения, и другие факторы.
Субъект, нуждающийся в таком лечении, может быть человеком или животным, не являющимся человеком, например, мышью, крысой, кроликом, обезьяной, собакой, лошадью, коровой, курицей, морской свинкой или свиньей. Как правило, субъект имеет диагноз ИЛ-1-бета-зависимого заболевания или может страдать таким заболеванием.
Примеры ИЛ-1-бета-зависимых заболеваний, при которых показаны терапевтические эффекты антагониста ИЛ-1-бета, включают пролиферативную диабетическую ретинопатию, подагрический артрит, синдром Шницлера, системный ювенильный идиопатический артрит, ревматоидный артрит, острый подагрический артрит, хронический подагрический артрит, крапивную лихорадку, васкулит, сахарный диабет 1 типа, сахарный диабет 2 типа, анкилозирующий спондилит, рецидивирующий множественный остеомиелит, рецидивирующий полихондрит, периодический синдром, ассоциированный с криопирином (CAPS), болезнь Бехчета, семейную средиземноморскую лихорадку, хроническое обструктивное заболевание легких, ревматическую полимиалгию, мутации NALP3, гангренозную пиодермию, хроническую идиопатическую крапивницу, остеоартрит, влажную возрастную макулярную дегенерацию желтого пятна, синдром сухого глаза, пустулезный псориаз, синдром синовита-акне-пустулеза-гиперостоза-остеита, синдром активации макрофагов, перемежающуюся лихорадку, афтозного стоматит, фарингит, синдром афтозного стоматита, синдром Стилла у совершеннолетних, дефицит мевалонат-киназы, атеросклероз, периодический синдром, ассоциированный с рецепторами ФНО (TRAPS), обыкновенные угри и/или гнойный гидраденит, но не ограничиваются ими.
Термин «CAPS» или «периодический синдром ассоциированный с криопирином» необходимо понимать как включающий каждое заболевание из семейного холодового аутовоспалительного синдрома (FCAS), синдрома Макла-Уэльса (MWS) и мультисистемного воспалительного заболевания неонатального возраста, также известного как хронический младенческий неврологический кожно-артикулярный (CINCA) синдром.
Фармацевтическая композиция может применяться с использованием различных способов введения. Введение может осуществляться парентерально, например, внутримышечно, подкожно, внутривенно в виде болюса или путем непрерывной инфузии, интраартикулярно, интрасиновиально, интрацеребрально, интрацереброспинально, интратекально, эпидурально или интраперитонеально; перорально; ректально; локально, урогенитально; местно, например, на кожу или в глаз; интравитреально; внутривенно; интраокулярно; внутриушным образом; интраназально; посредством ингаляции; дермально, например, интрадермально, подкожно или чрескожно; сублингвально; буккально, но не ограничиваясь этими способами. Предпочтительными являются местное, ректальное, локальное, интраназальное, внутривенное и/или интрадермальное введение.
Антитело согласно настоящему изобретению, связывающий элемент, последовательности нуклеиновых кислот, вектор или клетку-хозяин можно объединять с одним или несколькими дополнительными терапевтически эффективными соединениями. Указанное соединение может быть либо способным к прерыванию передачи сигнала через ИЛ-1-рецептор, или в качестве альтернативы, к ингибированию одной или нескольких различных мишеней, например, таких как другие медиаторы воспалительного ответа. Такое соединение(я) могут применяться одновременно или последовательно.
Для терапевтического применения антитело может быть радиоактивно маркированным или связанным с токсином, или связанным с другой эффекторной функцией, как описано выше.
Применение с целью диагностики и/или детекции
Антитело из настоящего изобретения может применяться с целью детекции или диагностики in vivo и/или in vitro. Например, широкий диапазон иммуноанализов, включающих антитела для детекции экспрессии специфических клеток или тканей известен специалисту в данной области техники. Подобным образом, любой связывающий элемент, последовательность нуклеиновых кислот, вектор и/или клетку-хозяин, описанную выше, может применяться, как описано в этом разделе.
Для такого применения антитело, связывающий элемент, последовательность нуклеиновых кислот, вектор или клетка-хозяин, раскрытые в настоящей заявке, могут быть маркированными или не маркированными. Например, может применяться не маркированное антитело и детектироваться вторичным антителом, распознающим эпитоп на антителе, описанном в настоящей заявке.
В другом варианте осуществления антитело, связывающий элемент, последовательность нуклеиновых кислот, вектор и/или клетка-хозяин конъюгированы с одним или несколькими веществами, которые могут распознаваться детекторным веществом (веществами), например, антитело, конъюгированное с биотином, может быть выявлено стрептавидином. Подобным образом, нуклеиновые кислоты и/или векторы, раскрытые в настоящей заявке, могут применяться с целью детекции и/или диагностики, например, с использованием их маркированных фрагментов в качестве зондов в гибридизационном анализе.
В некоторых вариантах осуществления любые из молекул, обеспеченных в настоящей заявке, в частности, антитело, пригодны для детекции наличия ИЛ-1-бета, предпочтительно, включая полноразмерный ИЛ-1-бета, его фрагменты и/или предшественники, в образце, предпочтительно в биологическом образце. Термин «детекция» охватывает количественное и/или качественное определение. В некоторых вариантах осуществления биологический образец содержит клетки или ткани от пациентов - людей. Не ограничивающие примеры биологических образцов включают кровь, мочу, цереброспинальную жидкость, биоптат, лимфу и/или ткани, не относящиеся к крови.
В некоторых вариантах осуществления способ включает обеспечение контакта биологического образца с антителом к ИЛ-1-бета, как описано в настоящей заявке, в условиях, пригодных для связывания антитела с ИЛ-1-бета, если он присутствует, и определение того, формируется ли комплекс между антителом и ИЛ-1-бета. Такой способ может быть способом in vitro или in vivo. В одном варианте осуществления антитело к ИЛ-1-бета используется для выбора субъектов, подходящих для лечения антителом, описанным в настоящей заявке, например, где ИЛ-1-бета является биомаркером для выбора пациентов. Подобным образом, вместо антитела такой способ может включать применение связывающего элемента, указанного выше, или Т-тела, описанного в настоящей заявке.
В еще одном аспекте антитело применяют в косметических целях, например, для улучшения эстетического внешнего вида кожи.
В другом аспекте обеспечивается набор, содержащий антитело, упакованную комбинацию реагентов с инструкциями по выполнению анализа для детекции или диагностики. Реагенты, как правило, обеспечиваются в виде сухих порошков с предварительно заданными количествами, обычно лиофилизированных, включая наполнители, которые после растворения обеспечивают раствор реагента, имеющий подходящую концентрацию. Могут быть также включены другие добавки, такие как стабилизаторы и/или буферные агенты. Если антитело маркировано ферментом, набор, как правило, включает соответствующие субстраты и кофакторы. Подобным образом, любой связывающий элемент, последовательность нуклеиновых кислот, вектор и/или клетку-хозяин, описанные выше, могут применяться, как подробно описано в данном разделе.
Список последовательностей
Последовательностями, описанными в настоящей заявке, являются:
SEQ ID No: 1 - VH CDR1
SEQ ID No: 2 - VH CDR2
SEQ ID No: 3 - VH CDR3
SEQ ID No: 4 - VL CDR1
SEQ ID No: 5 - VL CDR2
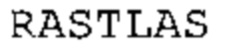
SEQ ID No: 6 - VL CDR3
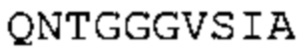
SEQ ID No: 7 - VH

SEQ ID No: 8 - VL
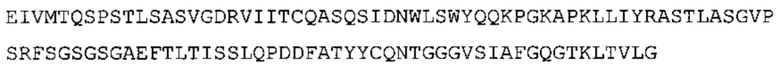
SEQ ID No: 9 - линкер
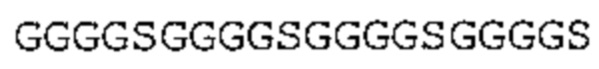
SEQ ID No: 10 - DLX2323

SEQ ID No: 11 - CDR вариант VH CDR1
SEQ ID No: 12 - CDR вариант VH CDR2
SEQ ID No: 13 - CDR вариант VH CDR3
SEQ ID No: 14 - CDR вариант VL CDR1
SEQ ID No: 15 - CDR вариант VL CDR2
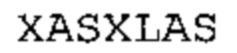
SEQ ID No: 16 - CDR вариант VL CDR3
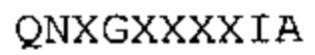
SEQ ID No: 17 - последовательность ДНК DLX2323
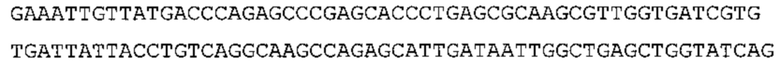

SEQ ID No: 18-FR-L1 из FW1.4
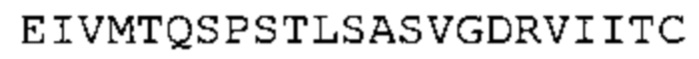
SEQ ID No: 19 - FR-L2 из FW1.4
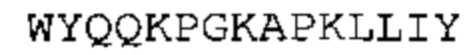
SEQ ID No: 20 - FR-L3 из FW1.4
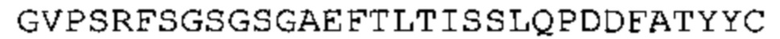
SEQ ID No: 21 - FR-L4 из FW1.4
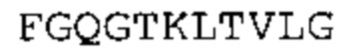
SEQ ID No: 22 - FR-H1 из rFW1.4
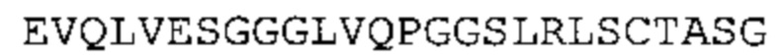
SEQ ID No: 23 - FR-H2 из rFW1.4
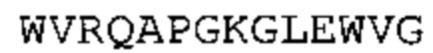
SEQ ID No: 24 - FR-H3 из rFW1.4
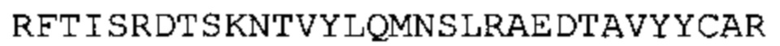
SEQ ID No: 25 - FR-H4 из rFW1.4
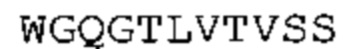
SEQ ID No: 26 - FR-H1 из rFW1.4(V2)
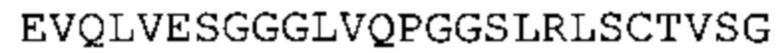
SEQ ID No: 27 - FR-H2 из rFW1.4(V2)
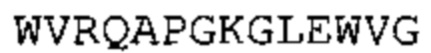
SEQ ID No: 28 - FR-H3 из rFW1.4(V2)
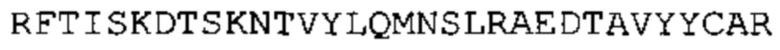
SEQ ID No: 29 - FR-H4 из rFW1.4(V2)
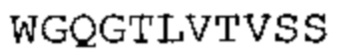
SEQ ID No: 30 - FR-H1 из rFW1.4-SST
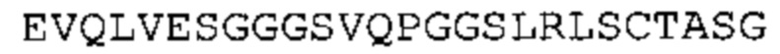
SEQ ID No: 31 - FR-H2 из rFW1.4-SST
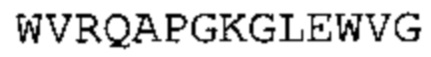
SEQ ID No: 32 - FR-H3 из rFW1.4-SST
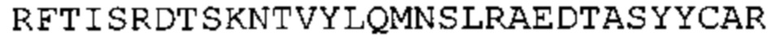
SEQ ID No: 33 - FR-H4 из rFW1.4-SST
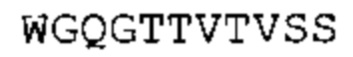
SEQ ID No: 34 - CDR-L1_D32X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 35 - CDR-L1_N33X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 36 - CDR-L1_W40X
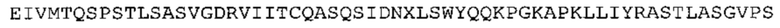

Предпочтительно, X выбран из группы, состоящей из глутаминовой кислоты (Е), фенилаланина (F), глицина (G), метионина (М), аспарагина (N), глутамина (Q), серина (S), триптофана (W) and тирозина (Y).
SEQ ID No: 37 - CDR-L2_R58X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), триптофана (W) и тирозина (Y).
SEQ ID No: 38 - CDR-L2_T69X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 39 - CDR-L3_T109X

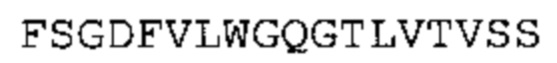
Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), изолейцина (I), аспарагина (N), серина (S), треонина (Т) и валина (V).
SEQ ID No: 40 - CDR-L3_G111X

Предпочтительно, X выбран из группы, состоящей из аланина (А), глицина (G), пролина (Р) и серина (S).
SEQ ID No: 41 - CDR-L3_G112X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 42 - CDR-L3_V135X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланин (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 43 - CDR-L3_S136X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 44 - CDR-H1_S33X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 45 - CDR-H1_A39X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 46 - CDR-H2_Y59X
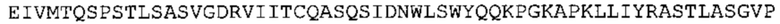

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), глицина (G), метионина (М) и тирозина (Y).
SEQ ID No: 47 - CDR-H2_D60X

Предпочтительно, X выбран из группы, состоящей из аспарагиновой кислоты (D), аспарагина (N) и пролина (Р).
SEQ ID No: 48 - CDR-H2_Y69X

Предпочтительно, X выбран из группы, состоящей из аланина (А), аспарагиновой кислоты (D), глутаминовой кислоты (Е), глицина (G), фенилаланина (F), гистидина (Н). изолейцина (I), лизина (K), лейцина (L), метионина (М), пролина (Р), аспарагина (N), серина (S), треонина (Т), триптофана (W) и тирозина (Y).
SEQ ID No: 49 - CDR-H3_R110X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 50 - CDR-H3_A111X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 51 - CDR-H3_I112X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), фенилаланина (F), гистидина (Н), изолейцина (I), лейцина (L), метионина (М), аспарагина (N), глутамина (Q), серина (S), треонина (Т), валина (V) и тирозина (Y).
SEQ ID No: 52 - CDR-H3_F113X

Предпочтительно, X выбран из группы, состоящей из аланина (F) и изолейцина (I).
SEQ ID No: 53 - CDR-H3_S114X
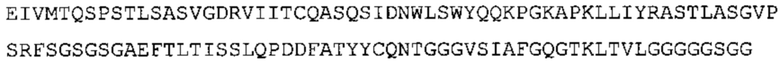

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), глутаминовой кислоты (Е), глицина (G), серина (S), треонина (Т) и валина (V).
SEQ ID No: 54 - CDR-H3_G115X

Предпочтительно, X выбран из группы, состоящей из аланина (А), глицина (G), метионина (М) и аспарагина (N).
SEQ ID No: 55 - CDR-H3_D135X

Предпочтительно, X выбран из группы, состоящей из аланина (А), аспарагиновой кислоты (D), глутаминовой кислоты (Е), гистидина (Н), аспарагина (N), серина (S) и треонина (Т).
SEQ ID No: 56 - CDR-H3_F136X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лейцина (L), метионина (М), аспарагина (N), глутамина (Q), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 57 - CDR-H3_V137X
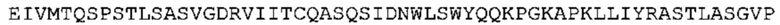

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланин (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 58 - CDR-H3_L138X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 59 - DLX2464

SEQ ID No: 60 - DLX2465

SEQ ID No: 61 - DLX2466
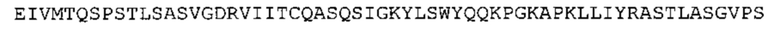

SEQ ID No: 62 - DLX2467

SEQ ID No: 63 - DLX2468

SEQ ID No: 64 - DLX2475

SEQ ID No: 65 - DLX2476

SEQ ID No: 66 - DLX2480

SEQ ID No: 67 - DLX2543
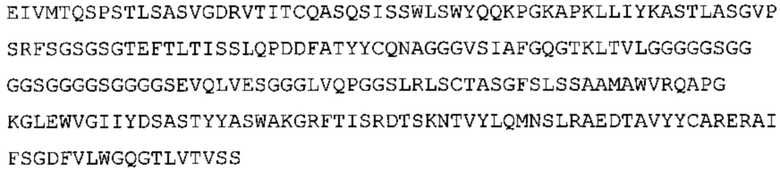
SEQ ID No: 68 - DLX2529

SEQ ID No: 69 - DLX2547

SEQ ID No: 70 - DLX2528

SEQ ID No: 71 - DLX2585

SEQ ID No: 72 - DLX2545

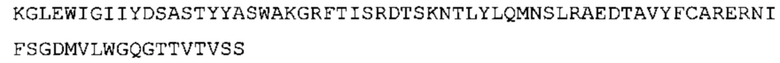
SEQ ID No:73 - DLX2531

SEQ ID No: 74 - DLX2586

SEQ ID No: 75 - DLX2530

SEQ ID No: 76 - DLX2548

SEQ ID No: 77 - DLX2676

SEQ ID No: 78 - DLX2677
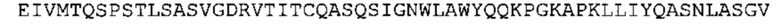

SEQ ID No: 79 - DLX2678

SEQ ID No: 80 - DLX2679

SEQ ID No: 81 - DLX2680

SEQ ID No: 82 - DLX2681

SEQ ID No: 83 - DLX2682

SEQ ID No: 84 - DLX2683

SEQ ID No: 85 - DLX2684

SEQ ID No: 86 - DLX2685

SEQ ID No: 87 - DLX2686

SEQ ID No: 88 - DLX2687

SEQ ID No: 89 - DLX2689


SEQ ID No: 90 - DLX2690

SEQ ID No: 91 - DLX2691

SEQ ID No: 92 - DLX2692

SEQ ID No: 93 - DLX2693

SEQ ID No: 94 - DLX2694

SEQ ID No: 95 - DLX2695
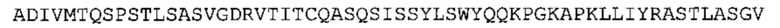

SEQ ID No: 96 - VL CDR-L1_D32X
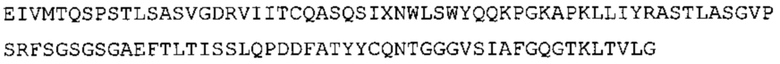
Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 97 - VL CDR-L1_N33X
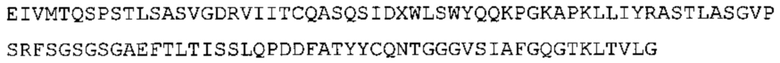
Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 98 - VL CDR-L1_W40X
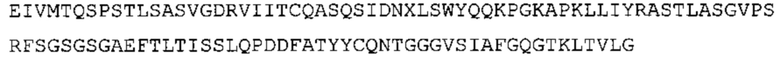
Предпочтительно, X выбран из группы, состоящей из глутаминовой кислоты (Е), фенилаланина (F), глицина (G), метионина (М), аспарагина (N), глутамина (Q), серина (S), триптофана (W) и тирозина (Y).
SEQ ID No: 99 - VL CDR-L2_R58X
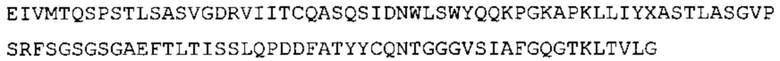
Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), триптофана (W) и тирозина (Y).
SEQ ID No: 100 - VL CDR-L2_T69X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 101 - VL CDR-L3_T109X
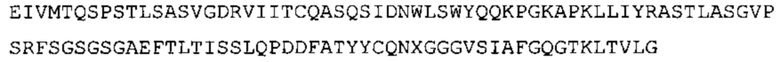
Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), изолейцина (I), аспарагина (N), серина (S), треонина (Т) и валина (V).
SEQ ID No: 102 - VL CDR-L3_G111X
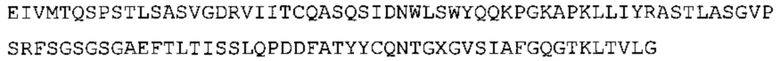
Предпочтительно, X выбран из группы, состоящей из аланина (А), глицина (G), пролина (Р) и серина (S).
SEQ ID No: 103 - VL CDR-L3_G112X
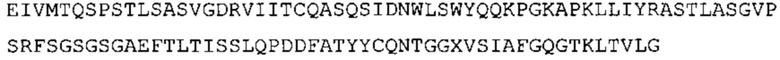
Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 104 - VL CDR-L3_V135X
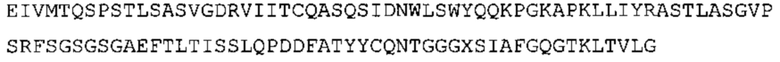
Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 105 - VL CDR-L3_S136X
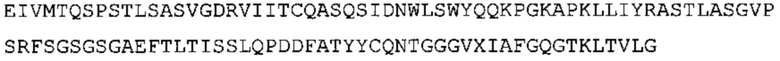
Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 106 - VH CDR-H1_S33X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 107 - VH CDR-H1_A39X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 108 - VH CDR-H2_Y59X

Предпочтительно X выбран из группы, состоящей из аланина (А), цистеина (С), глицина (G), метионина (М) и тирозина (Y).
SEQ ID No: 109 - VH CDR-H2_D60X
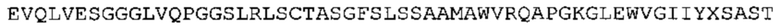
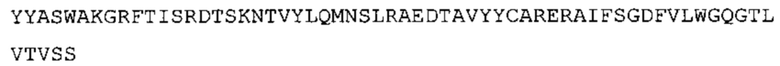
Предпочтительно, X выбран из группы, состоящей из аспарагиновой кислоты (D), аспарагина (N) и пролина (Р).
SEQ ID No: 110 - VH CDR-H2_Y69X

Предпочтительно, X выбран из группы, состоящей из аланина (А), аспарагиновой кислоты (D), глутаминовой кислоты (Е), глицина (G), фенилаланина (F), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), пролина (Р), аспарагина (N), серина (S), треонина (Т), триптофана (W) и тирозина (Y).
SEQ ID No: 111 - VH CDR-H3_R110X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 112 - VH CDR-H3_A111X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 113 - VH CDR-H3_I112X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), фенилаланина (F), гистидина (Н), изолейцина (I), лейцина (L), метионина (М), аспарагина (N), глутамина (Q), серина (S), треонина (Т), валина (V) и тирозина (Y).
SEQ ID No: 114 - VH CDR-H3_F113X

Предпочтительно, X выбран из группы, состоящей из фенилаланина (F) и изолейцина (I).
SEQ ID No: 115 - VH CDR-H3_S114X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), глутаминовой кислоты (Е), глицина (G), серина (S), треонина (Т) и валина (V).
SEQ ID No: 116 - VH CDR-H3_G115X

Предпочтительно, X выбран из группы, состоящей из аланина (А), глицина (G), метионина (М) и аспарагина (N).
SEQ ID No: 117 - VH CDR-H3_D135X

Предпочтительно, X выбран из группы, состоящей из аланина (А), аспарагиновой кислоты (D), глутаминовой кислоты (Е), гистидина (Н), аспарагина (N), серина (S) и треонина (Т).
SEQ ID No: 118 - VH CDR-H3_F136X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), фенилаланина (F), глицина (G), гистидина (Н),изолейцина (I), лейцина (L), метионина (М), аспарагина (N), глутамина (Q), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 119 - VH CDR-H3_V137X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 120 - VH CDR-H3_L138X

Предпочтительно, X выбран из группы, состоящей из аланина (А), цистеина (С), аспарагиновой кислоты (D), глутаминовой кислоты (Е), фенилаланина (F), глицина (G), гистидина (Н), изолейцина (I), лизина (K), лейцина (L), метионина (М), аспарагина (N), пролина (Р), глутамина (Q), аргинина (R), серина (S), треонина (Т), валина (V), триптофана (W) и тирозина (Y).
SEQ ID No: 121 - VH DLX2464

SEQ ID No: 122 - VH DLX2465

SEQ ID No: 123 - VL DLX2466
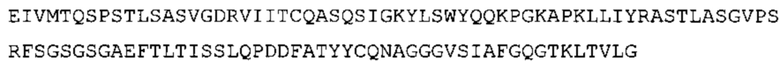
SEQ ID No: 124 - VH DLX2466

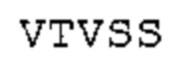
SEQ ID No: 125 - VL DLX2467
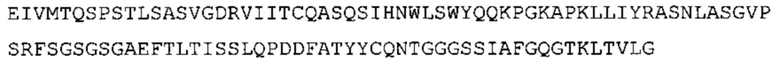
SEQ ID No: 126 - VH DLX2467

SEQ ID No: 127 - VL DLX2468
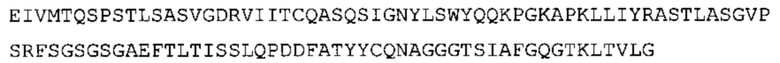
SEQ ID No: 128 - VH DLX2468

SEQ ID No: 129 - VL DLX2475

SEQ ID No: 130 - VH DLX2475

SEQ ID No: 131 - VL DLX2476
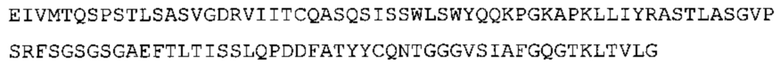
SEQ ID No: 132 - VH DLX2476

SEQ ID No: 133 - VL DLX2480

SEQ ID No: 134 - VH DLX2480
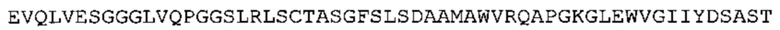

SEQ ID No: 135 - VL DLX2543

SEQ ID No: 136 - VL DLX2529

SEQ ID No: 137 - VL DLX2547
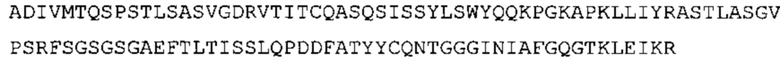
SEQ ID No: 138 - VH DLX2547

SEQ ID No: 139 - VL DLX2528

SEQ ID No: 140 - VH DLX2528

SEQ ID No: 141 - VL DLX2585
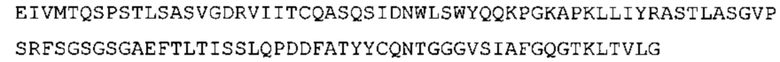
SEQ ID No: 142 - VH DLX2585

SEQ ID No: 143 - VL DLX2545

SEQ ID No: 144 - VH DLX2545
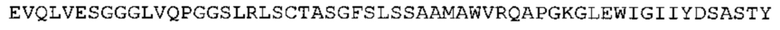
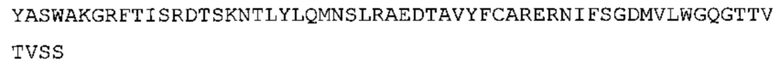
SEQ ID No: 145 - VL DLX2531
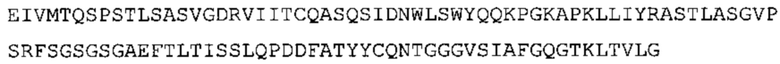
SEQ ID No: 146 - VH DLX2531

SEQ ID No: 147 - VL DLX2586
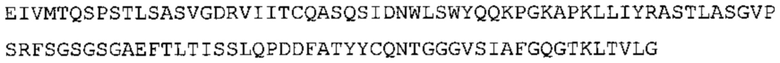
SEQ ID No: 148 - VH DLX2586

SEQ ID No: 149 - VL DLX2530
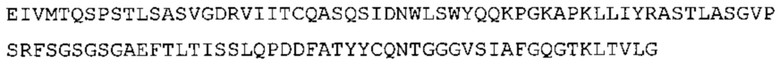
SEQ ID No: 150 - VH DLX2530

SEQ ID No: 151 - VL DLX2548
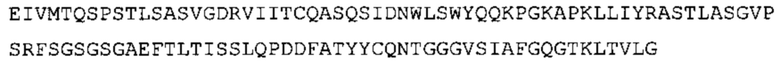
SEQ ID No: 152 - VH DLX2548

SEQ ID No: 153 - VL DLX2544
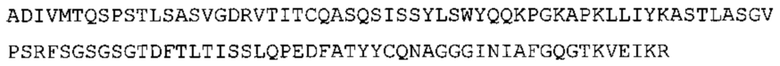
SEQ ID No: 154 - DLX2544

SEQ ID No: 155 - CDR-H1 of DLX2531
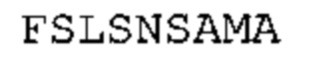
SEQ ID No: 156 - CDR-H2 of DLX2531
SEQ ID No: 157 - CDR-H3 of DLX2531
SEQ ID No: 158 - CDR-L1 of DLX2531
SEQ ID No: 159 - CDR-L2 of DLX2531
SEQ ID No: 160 - CDR-L3 of DLX2531
SEQ ID No: 161 - CDR-L1 of DLX2681
SEQ ID No: 162 - CDR-L2 of DLX2681
SEQ ID No: 163 - CDR-L3 of DLX2681
Примеры
Пример 1 - Идентификация scFv, нейтрализующего рчИЛ-1-бета
Иммунизация кроликов: Кроликов иммунизировали белком рекомбинантного ИЛ-1-бета человека (Peprotech, США, кат. No: 200-01В). Клетки лимфоузлов и селезенки выделяли после итогового обескровливания, и клетки криоконсервировали.
Сортинг В-клеток кролика посредством проточной цитометрии и культивирование: ИЛ-1-бета специфичные В-клетки памяти сортировали в виде единичных клеток в 96-луночных микропланшетах с применением FACSAria III (BD Biosciences). Клоны единичных В-клеток культивировали в присутствии фидерных клеток.
Скрининг клонов В-клеток: Надосадочную жидкость от клеточных культур анализировали посредством ИФА на наличие анти-ИЛ-1-бета специфичного IgG. Вкратце, рчИЛ-1-бета (Peprotech, кат. No: 200-01B) наносили в концентрации 2 мкг/мл в течение ночи при 4°С в 96-луночные микропланшеты Maxisorp в ФБР. После блокирования неспецифического связывания 5% обезжиренным сухим молоком, добавляли надосадочную жидкость клеточных культур. ИЛ-1-бета-специфичные IgG выявляли с помощью конъюгата антител к IgG кролика с пероксидазой хрена (Southern Biotech, кат. No: 4050-05). ИФА проявляли с ВМ Blue POD субстратом (Roche Applied Science). B-клеточные клоны, специфичные к рчИЛ-1-бета, дополнительно анализировали на их нейтрализующую способность в анализе с использованием фибробластов человека.
Определение последовательности ИЛ-1-бета-нейтрализующих IgG: все кроличьи В-клеточные клоны, продуцирующие нейтрализующие анти-ИЛ-1-бета антитела, подвергали выделению мРНК с применением RNeasy Mini Kit (Qiagen, Германия, кат. No: 74106). мРНК использовали в качестве матрицы для обратной транскрипции в соответствии с протоколом производителя (OneStep RT-PCR kit, Qiagen, Германия, кат. No: 210212). Затем проводили ПЦР с применением олигонуклеотидов для специфической амплификации последовательностей, кодирующих тяжелые и легкие цепи IgG кролика (Biometra Thermocycler Т3). ПЦР фрагменты тяжелых и легких цепей независимо секвенировали (ABI, Sanger3730xl; Microsynth AG, Balgach, Швейцария), и полученные последовательности ДНК транслировали в последовательности белков с применением EMBOSS Transeq (http://www.ebi.ac.uk/Tools/st/), и выравнивали с применением CLUSTALW2 (http://www.ebi.ac.uk/Tools/msa/clustalw2/).
Конструирование генов scFv анти-ИЛ-1-бета, и экспрессия белка scFv: идентифицировали CDR области легких и тяжелых цепей IgG кролика, как указано выше, и трансплантировали в акцепторные каркасы тяжелых и легких цепей человека, содержащие SEQ ID No: 18-21 и 22-25, соответственно. Генерировали бактериальные векторы экспрессии, кодирующие scFv белки, с N-концевой вариабельной легкой цепью, связанной последовательностью SEQ ID No: 9 с С-концевой вариабельной тяжелой цепью. ScFv белки экспрессировали в E. coli BL21 (DE3); (Novagen, США, кат. No: 69450-3) в качестве телец включения, которые очищали, солюбилизировали и подвергали белки рефолдингу. scFv, подвергшиеся рефолдингу, очищали путем стандартной гель-фильтрации, и собирали фракции пиков мономеров. Очищенные scFv анализировали на связывание с ИЛ-1-бета посредством ИФА. scFv, которые специфически связывались с рчИЛ-1-бета, тестировали на нейтрализацию ИЛ-1-бета в анализе на фибробластах человека. В ходе этой процедуре было установлено, что scFv DLX2323 и другие анти-ИЛ- 1-бета scFv являются мощными ингибиторами ИЛ-1-бета.
Пример 2 - Распознавание ИЛ-1-бета человека
Вначале специфическое распознавание рчИЛ-1-бета с DLX2323 подтверждали посредством ИФА (Фигура 1). Вкратце, рчИЛ-1-бета (Peprotech, кат. No: 200-01В) наносили в концентрации 2 мкг/мл в течение ночи при 4°С в 96-луночные микропланшеты Maxisorp в ФБР. После блокирования неспецифического связывания 5% обезжиренным сухим молоком, добавляли повышающиеся концентрации scFv (от 10 до 300 нг/мл), и выявляли scFv с помощью конъюгата Белка L с пероксидазой хрена ((Sigma-Aldrich, кат. No: Р3226). ИФА проявляли с ВМ Blue POD субстратом (Roche Applied Science). В качестве отрицательного контроля использовали scFv с неподходящей специфичностью. Этот результат показывает, что DLX2323 специфически связывается с рчИЛ- 1-бета.
Чтобы подтвердить, что DLX2323 и контрольный scFv распознаются конъюгатом Белка L с пероксидазой хрена, и таким образом, отсутствие сигнала о связывании рчИЛ-1-бета в вышеуказанном ИФА не обусловлено проблемами детекции, проводили другой эксперимент. Планшеты покрывали scFv непосредственно, и выявляли конъюгатом Белка L с пероксидазой хрена, как описано выше. Все scFv наносили в концентрации 2 мкг/мл в ФБР. Этот ИФА эксперимент показал, что DLX2323 и контрольный scFv распознавались агентом детекции конъюгатом Белка L с пероксидазой хрена.
В другом ИФА (Фигура 2) проводили распознавание природного ИЛ-1-бета DLX2323 человека. Как общеизвестно, экспрессия белков человека в клетках, отличных отклеток человека, может вызывать изменения, например, посттрансляционные модификации и/или изменения конформации. Это может приводить к различному распознаванию рекомбинантных и природных белков антителами. Природный ИЛ-1-бета человека секретировался ТНР-1 клетками (DSMZ, Германия, кат. No: АСС16) после стимуляции 10 нг/мл ФМА (Sigma-Aldrich, кат. No: Р1585), 1 мг/мл ЛПС (Sigma-Aldrich, кат. No: L4391) м 2 мМ АТФ (Sigma-Aldrich, кат. No: A6559-25UMO). Надосадочную жидкость от клеток собирали, и проводили количественное определение секретированного природного ИЛ-1-бета человека с применением ИФА набора для определения ИЛ-1-бета/ИЛ1Р2 ELISA DuoSet (R&D Systems, кат. No: DY201). DLX2323 наносили в 96-луночные микропланшеты (Maxisorp, Nunc) с плотностью 5 мкг/мл в DPBS (рН 7,4). ИЛ-1-бета человека в виде рекомбинантно экспрессированного варианта (Peprotech, кат. No: 200-01B) или нативной секретированной версии наносили в конечных концентрациях в диапазоне от 0,5 до 4 нг/мл. Связанный ИЛ-1-бета выявляли биотинилированными козьими антителами против чИЛ-1-бета (R&D Systems, кат. No: DY201) и конъюгатом стрептавидина с пероксидазой хрена (BD Pharmingen, кат. No: 554060). ИФА проявляли с применением субстрата ВМ Blue POD (Roche Applied Science). Для количественного определения измеряли поглощение при 450 нм с применением ридера для микропланшетов VersaMax (Molecular Devices, США). Результаты (см. Фигуру 2) показали, что и рекомбинантный, и природный ИЛ-1-бета человека распознаются DLX2323 на сопоставимых уровнях.
Пример 3 - Нейтрализация биологической активности рчИЛ-1-бета
Антитела и scFv тестировали на их способность к нейтрализации ИЛ-1-бета в анализе на дермальных фибробластах человека (NHDF-Neo, кат. No: СС-2509, Lonza Walkersville, США). Активация таких фибробластов с ИЛ-1-бета приводит к специфическому высвобождению ИЛ-6, который определяют посредством ИФА. Ингибирование ИЛ-1-бета специфичными антителами снижает количество ИЛ-6, высвобождаемое из таких фибробластов. Ингибирующую активность анти-ИЛ-1-бета антитела определяли путем измерения полумаксимального снижения (IC50) ИЛ-1-бета-индуцированного высвобождения ИЛ-6. Дермальные фибробласты человека засевали в 96-луночные микропланшеты в количестве 5000 клеток на ячейку за 16-20 часов до добавления ИЛ-1-бета. Фибробласты культивировали в основной среде для фибробластов (FBM; Lonza, кат. No: СС-3131) с добавками (чФРФ-В, инсулин, ЭТС, GA-1000), как описано поставщиком клеток (Lonza Walkersville, США: системы дермальных фибробластов человека Clonetics™). Затем удаляли FBM, и клетки промывали один раз Дульбекко модифицированной средой Игла (DMEM; Gibco, Life Technologies, кат. No: 11880) для удаления факторов роста. Затем клетки инкубировали в течение 7 часов в среде DMEM. Антитела или scFv и рчИЛ-1-бета предварительно инкубировали в DMEM в течение 1 часа при 37°С. Смесь добавляли к клеткам в конечной концентрации 10 пг/мл ИЛ-1-бета. В качестве отрицательного контроля 10 пг/мл ИЛ-1-бета добавляли к клеткам без антител к ИЛ-1-бета. В качестве положительного контроля наносили мышиное моноклональное антитело к ИЛ-1-бета (R&D systems, США, кат. No: MAB201). Клетки инкубировали со смесью ИЛ-1-бета/антитела к ИЛ-1-бета в течение 18024 часов, и надосадочную жидкость от клеточных культур анализировали на высвобождение ИЛ-6 с применением ИФА набора к ИЛ-6 человека «Human IL-6 DuoSet ELISA Kit» в соответствии с инструкциями производителя (R&D Systems, США, кат. No: DY206). Значение IC50 DLX2323 составило 3 пМ ± 1,05 в восьми независимых анализах.
Пример 4 - Сравнение нейтрализующей активности с коммерческими ингибиторами ИЛ-1-бета
DLX2323 был идентифицирован как ИЛ-1-бета-нейтрализующий scFv. Биологическую активность DLX2323 и других ингибиторов оценивали в анализе с дермальными фибробластами человека, как описано в Примере 3. Рекомбинантный ИЛ-1-бета человека предварительно инкубировали с повышающимися концентрациями scFv DLX2323, моноклональным IgG антителом МАВ201 к ИЛ-1-бета человека, антагонистом рецептора ИЛ-1-бета (рчИЛ-lpa) (R&D systems, кат. No: 280-RA-010/CF), или утвержденным Управлением по контролю за продуктами питания и лекарственными средствами США коммерческим канакинумабом IgG (Novartis, Ilaris®) перед добавлением в ячейки. Проводили два независимых эксперимента: на Фигуре 3А показано сравнение DLX2323 с МАВ201, в то время как на Ффигуре 3В показано сравнение DLX2323 с рчИЛ-1pa и канакинумабом. Определены следующие значения IC50: МАВ201: 2-3 пМ; DLX2323: 2-4 пМ; рчИЛ-1pa: 40 пМ и канакинумаб: 90 пМ. В заключение, одновалентный мономерный scFv DLX2323 является почти таким же мощным, как двухвалентное моноклональное мышиное антитело МАВ201. Он продемонстрировал явно более высокую активность в нейтрализации ИЛ-1-бета человека, чем коммерческий ингибитор Канакинумаб и рчИЛ-1pa. Далее, DLX2323 позволяет полностью блокировать индуцированное ИЛ-1-бета высвобождение ИЛ-6.
Пример 5 - Растворимость
DLX2323 хранили в ФБР буферном растворе с рН 7,2 (Фосфатный буферный солевой раствор 1X, Gibco, Life Technologies™, кат. No: 20012). Для определения его максимальной растворимости, DLX2323 концентрировали с применением центрифужных концентраторов Vivaspin 20 (Sartorius Stedim Biotech, кат. No: VS2001) при комнатной температуре. Процесс концентрирования останавливали на 71 мг/мл из за высокой вязкости образца. Полученный раствор белка DLX2323 был вязким, прозрачным, и не наблюдалось осадка при визуальном анализе.
Пример 6 - Стабильность
Что касается стабильности scFv, можно наблюдать два различных процесса, вносящих вклад в его нестабильность. Во-первых, scFv может быть склонным к димеризации, часто сопровождающейся олигомеризацией, и в итоге агрегацией. Во-вторых, со временем может происходить деградация scFv, приводящая к образованию малых фрагментов. Чтобы проверить, является ли DLX2323 стабильным, проводили ВЭЖХ (Dionex, Summit system) гель-фильтрационную хроматографию (Tosoh, TSKgel 73G2000SWxl, кат. No: 08540) для определения процентного содержания мономерного, не деградированного scFv белка в определенные моменты времени при различных концентрациях белка и температурах (например, 4°С, комнатной температуре и 37°С). Процентное содержание мономера определяли в исходной точке исследования (Т0) и спустя 1 месяц для 1 мг/мл DLX2323, а в другом эксперименте, спустя 2 недели для 50 мг/мл раствора DLX2323. Белок был растворен в ФБР, рН 7,2. Результаты анализа стабильности показаны в Таблицах 1 и 2.
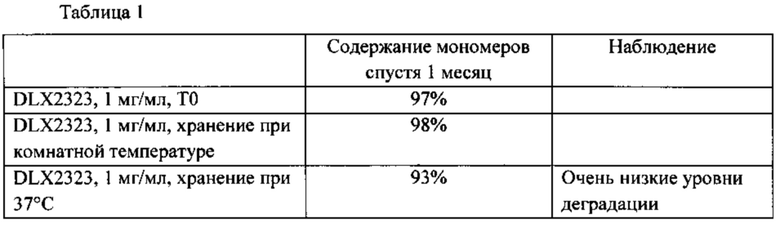
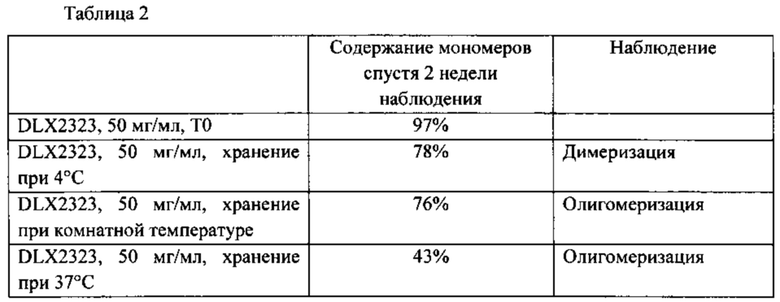
Термическую стабильность DLX2323 оценивали посредством дифференциальной сканирующей флуориметрии (ДСФ). Для этого анализа в устройстве ПЦР в режиме реального времени (Corbett, Rotor-Gene) нагревали DLX2323 в температурном градиенте от 30°С до 95°С (подъем с этапами 1°С, ожидание 5 секунд на этап). Образец белка содержал 0,5 мг/мл DLX2323 и 20х SYPRO® Orange (Sigma-Aldrich, кат. No: S5692, 5000x) в ФБР. Когда белок начинал плавиться, Sypro Orange становился флуоресцентным. Эту флуоресценцию измеряли он-лайн (длина волны возбуждения 470 нм; длина волны испускания 555 нм) при проходе градиента. С помощью программного обеспечения Rotor-Gene 6000 Series Software 1.7 рассчитывали среднюю температуру плавления (Tm) DLX2323, составившую 74°С.
Пример 7 - Перекрестная реактивность
Перекрестную реактивность DLX2323 с гомологами ИЛ-1-бета из видов, отличных от человека, оценивали в ИФА. Исследовали связывание с рекомбинантно экспрессируемыми белками ИЛ-1-бета следующих видов: яванских макак (Sino Biological Inc., США, кат. No: 90010-CNAE), макак-резус (R&D Systems, США, кат. No: 1318-RL/CF), свиньи (Kingfisher Biotech, США, кат. No: RP0297S-025), собаки (Kingfisher Biotech, США, кат. No: RP0085D-025), морской свинки (Kingfisher Biotech, кат. No: RP0343GP-025), крысы (Peprotech, кат. No: 400-01B) и мыши (BioLegend, кат. No: 575102). Связывание DLX2323 сравнивали с ИФА-положительными контрольными антителами (R&D Systems, США, поликлональные козьи IgG антитела против ИЛ-1-бета человека, кат. No: AB-201-NA; BioLegend, Inc., США, биотинилированные крысиные антитела против ИЛ-1-бета мыши, кат. No: 503505). Вкратце, белки наносили в концентрации 2 мкг/мл в течение ночи при 4°С в 96-луночные микропланшеты Maxisorp в ФБР. После блокирования неспецифических связей с 5% обезжиренным сухим молоком, в лунки добавляли повышающиеся концентрации (0,1 мкг/мл, 0,3 мкг/мл и 1,0 мкг/мл) DLX2323. Успешное нанесение каждого белка отдельно подтверждали с применением контрольных антител, специфичных к ИЛ-1-бета. В то время как DLX2323 выявляли конъюгатом Белка L с пероксидазой хрена (Sigma- Aldrich, США, кат. No: Р3226), контрольные антитела выявляли либо конъюгатом стрептавидина с пероксидазой хрена (BD Pharmingen, США, кат. No: 554060), либо другими пригодными вторичными антителами, маркированными пероксидазой хрена. ИФА проявляли с субстратом ВМ Blue POD (Roche Applied Science), и измеряли поглощение при 450 нм. DLX2323 распознавал ортологи четырех видов ИЛ-1-бета, а именно ИЛ-1-бета человека, яванских макак, макак-резус и крысы. Не отмечалось перекрестной реактивности для ИЛ-1-бета свиньи, морской свинки, собаки и мыши.
Помимо перекрестной реактивности DLX2323 с гомологами ИЛ-1-бета видов, отличных от человека, определяли характер распознавания DLX2323 в отношении различных членов семейства ИЛ-1 человека, и других цитокинов: рчИЛ-lpa (R&D systems, США, кат. No: 280-RA-010/CF), рчИЛ-1-альфа (PeproTech, кат. No: 200-01A), рчИЛ-18 (BioVision, кат. No: 4179-25), рчИЛ-33 (Peprotech, кат. No: 200-33), ИЛ-36ра (R&D Systems, кат. No: 1275-IL/CF), рчФНО-альфа (Peprotech, Гамбург, Германия, кат. No: 300-01A) рчИЛ-6 (Peprotech, кат. No: 200-06). В используемом ИФА анализе следующие антитела служили в качестве положительного контроля: биотинилированные антитела против ИЛ-1ра человека (BioLegend, кат. No: 509501), биотинилированные антитела к ИЛ-1-альфа человека (BioLegend, кат. No: 515703), поликлональные антитела против ИЛ-18 человека (BioVision, кат. Νο: 5179-100), биотинилированные антитела против ИЛ-33 человека (Peprotech, кат. No: 500-P261Bt), scFv DLX105 против ФНО-альфа человека (антитело, принадлежащее Delenex, описанное в WO 2006/131013 А), биотинилированные антитела против ИЛ-6 человека (R&D systems, DY206, кат. No: 840114), антитела против ИЛ-36ра человека (R&D systems, кат. No: AF1275). ИФА проводили по существу как описано выше. Не было выявлено перекрестной реактивности DLX2323 с каким-либо из тестированных членов семейства ИЛ-1 человека и цитокинов.
Пример 8 - Эффективность in vivo.
В этом примере продемонстрировано ингибирование активности ИЛ-1-бета человека in vivo с помощью DLX2323. ИЛ-1-бета человека может связываться и активировать рецептор ИЛ-1 мыши, таким образом, индуцируя воспалительный ответ у мыши in vivo. Воспаление приводит к повышению уровней цитокинов в сыворотке крови, включая ИЛ-6 мыши (мИЛ-6). Рекомбинантный ИЛ-1-бета человека (Peprotech, кат. No: 200-01B) вводили подкожно в дозе 1,5 мкг/кг массы тела мышам-самцам BALB/c в возрасте 8 недель (Charles River, Германия). Спустя 2 часа в сыворотке существенно повышались уровни мИЛ-6. Для анализа нейтрализующей активности DLX2323 in vivo его вводили интраперитонеально за два часа до введения ИЛ-1-бета. Одной группе мышей вводили 5 мг/кг DLX2323, второй группе вводили 15 мг/кг DLX2323. Группе отрицательного контроля вводили или ФБР интраперитонеально, или scFvs несоответствующей специфичности. Пятой группе мышей вводили внутривенно 10 мг/кг канакинумаба (Novartis, Ilaris®) в качестве положительного контроля. Спустя два часа после введения рчИЛ-1-бета отбирали образцы крови, и измеряли сывороточные уровни мИЛ-6 с применением ИФА набора для ИЛ-6 мыши DuoSet, в соответствии с инструкциями производителя (R&D Systems, кат. No: DY406). В группах мышей, которым вводили DLX2323 и канакинумаб, отмечались лишь очень низкие количества мИЛ-6, или даже не выявлялся мИЛ-6 (0,0-2 пг/мл мИЛ-6; Фигура 4). Мыши, получавшие ФБР или контрольный scFv, показали существенно повышенные уровни ИЛ-6 от 50 до 170 пг/мл. DLX2323 очень эффективно нейтрализовал ИЛ-1-бета in vivo, даже в дозе 5 мг/кг.
Пример 9 - Библиотеки CDR
Для лучшей характеристики ассоциации DLX2323 с ИЛ-1-бета человека, конструировали аминокислотные мутации и вставляли сайт-специфическим образом в CDR области DLX2323. CDR положения были выбраны для мутагенеза на основе их поверхностного размещения, ожидаемого взаимодействия с ИЛ-1-бета человека, как было установлено из моделей гомологии или сравнения последовательности с предшествующими кроличьими IgG. В легкой цепи мотив DNW в CDR-L1 (SEQ ID No: 14), аминокислотные остатки R и Τ в CDR-L2 (SEQ ID No: 15), треонин в CDR-L3, а также мотив GGVS были выбраны (SEQ ID No: 16) для конструкции вариантов DLX2323. В тяжелой цепи мотив SA был выбран в CDR-H1 (SEQ ID No: 11), в CDR-H2 мотив YD и следующий тирозин (SEQ ID No: 12). В CDR-H3 все положения были выбраны для замены (SEQ ID No: 13), за исключением N-концевой глутаминовой кислоты (Е). Все названные положения указаны X в SEQ ID No: 11-16, соответственно. Сайт-направленный мутагенез, секвенирование клонов и библиотечную сборку проводили посредством GeneArt (Life Technologies ТМ, Регенсбург, Германия).
Полученные scFv мутанты экспрессировали в 96-луночном формате в 1 мл культурах в Е. coli BL21 (DE3) (Novagen, США, кат. No: 69450-3). Во время экспрессии в этой системе большинство scFv белков формировало нерастворимые тельца включения внутри клеток, но значительное количество белка можно было извлечь из растворимых фракций после лизиса клеток, что, как было установлено, стало достаточным для анализа рчИЛ-1-бета посредством ИФА (подробности см. в примере 2). Клетки лизировали лизирующим буферным раствором (1 мМ ЭДТУК, 0,1 мг/мл лизоцима, ФБР, рН 7,2) в 96-луночном формате путем замораживания и последующего оттаивания. Полученные неочищенные экстракты осветляли центрифугированием. Надосадочную жидкость добавляли в лунки планшета для микротитрования, покрытые 50 нг/мл рчИЛ-1-бета (Peprotech) на лунку. После отмывания связавшийся scFv выявляли с помощью конъюгата Белка L с перксидазой хрена, и связывание с рчИЛ-1-бета количественно определяли с помощью субстрата ВМ Blue POD (Roche Applied Science).
В Таблице 3 перечислены единичные мутации, которые явно обеспечивают связывание рчИЛ-1-бета по результатам ИФА сигнала, по меньшей мере в два раза превышающего сигнал в отрицательном контроле, но не меньше 0,1 оптических единиц.
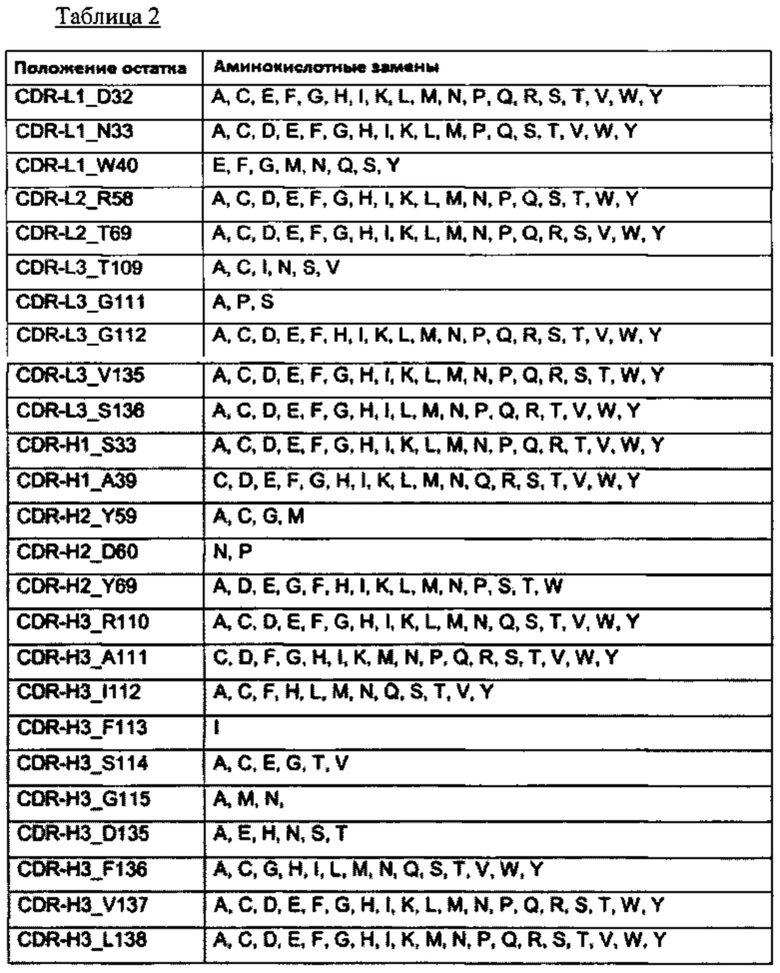
Эти данные по связыванию имеют прогностическое значение, и также касаются нейтрализации биологических функций рчИЛ-1-бета, как подтверждено экспрессией пяти примеров мутантов с единичными мутациями в крупномасштабном формате, очищенных до однородного состояния и подвергнутых анализу на клеточной основе вместе с подобным образом очищенным DLX2323 белком.
DLX2323_CDR-H1_A39N (SEQ ID No: 45 с X=N), DLX2323_CDRH3_A111N (SEQ ID No: 50 с X=N), DLX2323_CDR-H3_F136N (SEQ ID No: 56 с X=N), DLX2323_CDR-H3_V137D (SEQ ID No: 57 с X=D), и DLX2323_CDR-H3_L138D (SEQ ID No: 58 с X=D) и соответствующий исходный scFv, DLX2323, экспрессировали в клетках Е. coli BL21(DE3) (Novagen) в 2-литровом масштабе. После лизиса клеток посредством ультразвука, тельца включения обогащали с помощью центрифугирования, промывали, солюбилизировали белок в гуанидиновом буферном растворе (6 М гуанидин/HCl, 100 мМ Трис, 1 мМ ЭДТУК, рН 8,5), подвергали рефолдингу в буферном растворе с мочевиной (4 М мочевина, 50 мМ глицин, 2 мМ цистин, 2 мМ цистеин, рН 10), концентрировали в 50 мМ глицине, 50 мМ NaCl, pH 10, и очищали до однородного состояния посредством гель-фильтрации. Фракции пиков мономеров из этой процедуры концентрировали до итогового содержания белка примерно 0,5-5 мг/мл, пропускали через стерильный фильтрующий блок, и подвергали анализу на клеточной основе, как описано в Примере 3. В Таблице 4 показана нейтрализующая активность IC50 каждого из мутантов, определенная в двух независимых анализах.
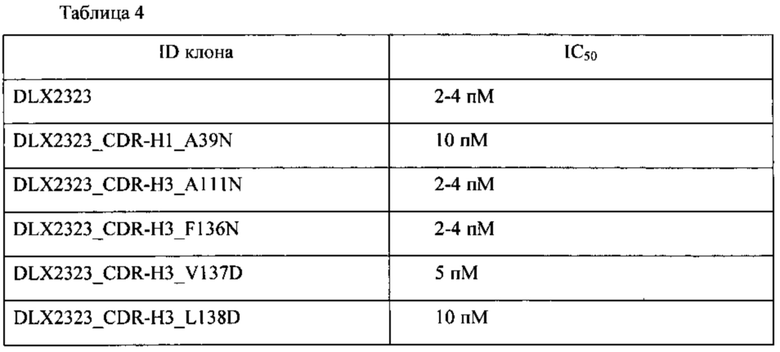
При общем хорошем соответствии с предшествующими результатами ИФА, значение IC50 в нижнем пМ диапазоне наблюдалось для всех пяти выбранных мутантов с единичной мутацией. Каждый член из выбранного поднабора мутантов обладал нейтрализующей активностью по отношению к рчИЛ-1-бета, сопоставимой с активностью исходного scFv DLX2323. Соответственно, связывание DLX2323 и его вариантов с рчИЛ-1-бета показало, что допустима значительная степень вариабельности последовательностей при сопутствующем сохранении ключевых функциональных характеристик.
Пример 10 - Получение комбинаторных вариантов
На основании вышеизложенного, были сконструированы комбинаторные мутанты DLX2323, объединяющие 3-9 из ранее исследованных изменений CDR с единичной мутацией. Изменения индивидуальных остатков были выбраны на основе (i) высоких результатов связывания в ИФА, сопоставимых или превышающих результаты исходного антитела DLX2323, (ii) комбинаций размещенных далеко друг от друга изменений с единичной мутацией, которые одновременно влияют на множество CDR областей и (iii) кумулятивной модуляции CDR-H3 по отдельности. Сконструированные мутанты экспрессировали в крупномасштабном формате и очищали, как описано выше для мутантов с единичными мутациями, перед оценкой нейтрализующей активности в анализе на фибробластах человека из Примера 3. Индивидуальные аминокислотные изменения в последовательности и данные IC50 для соответствующих scFv подробно приведены в Таблице 5. Все эти изолированные scFv белки обладали нейтрализующей активностью против рчИЛ-1-бета in vitro. Пять наилучших кандидатов показали значения IC50 в низком пикомолярном диапазоне, и таким образом, высоко сопоставимы с исходным scFv DLX2323.


Сконструировали варианты, несущие более 6 аминокислотных замен в CDR и фланкирующих каркасных областях вариабельной легкой и/или вариабельной тяжелой цепи. В целом были созданы 10 комбинаторных кандидатов (четыре VL и шесть VH мутантных последовательностей), экспрессированы и подвергнуты функциональному анализу на связывание с рчИЛ-1-бета (ИФА) и нейтрализации (анализ на фибробластах человека) in vitro, как описано в Примере 3. В Таблице 6 обобщены соответствующие точечные мутации, а также нейтрализующая активность, измеренная в двух независимых экспериментах для каждого клона.
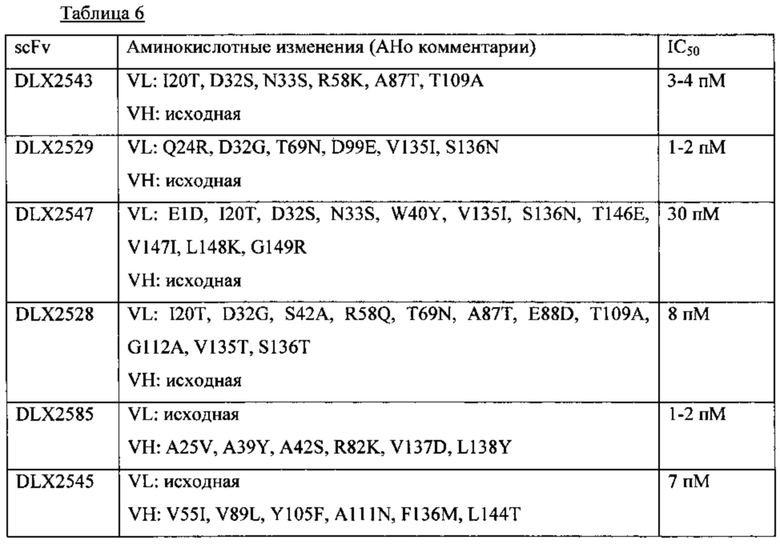

Было установлено, что все десять scFv белков эффективно нейтрализуют рчИЛ-1-бета в анализе на фибробластах человека in vitro со значениями IC50 в диапазоне от высоких фемтомолярных значений до низких пикомолярных значений. Клоны DLX2531, DLX2548, DLX2585, DLX2530, DLX2529 и DLX2586 воспроизводимо давали значения IC50 ниже значений для исходного scFv DLX2323.
В итоге, перетасовывали цепи вышеуказанных VL и VH последовательностей. Связывание с рчИЛ-1-бета подтверждали для осветленных лизатов оригами-клеток Е. coli BL21, трансформированных 19 мутантами с перетасованными цепями VL и VH. В Таблице 7 обобщены указанные замены.

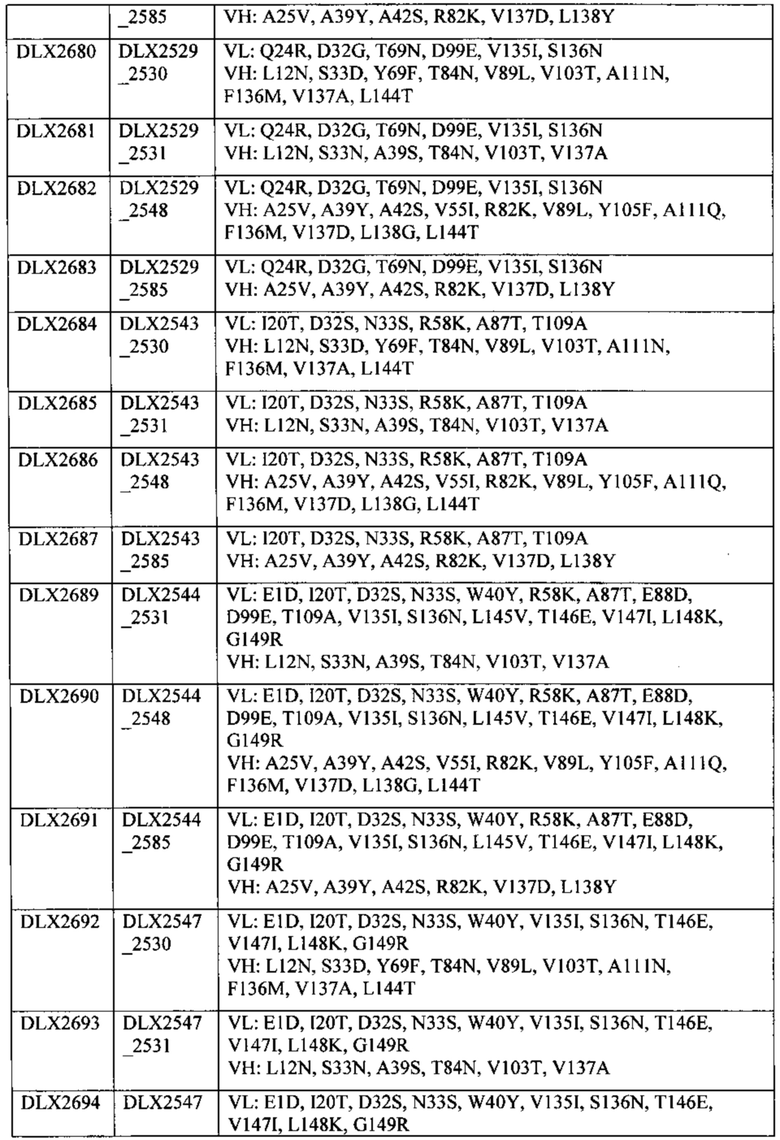

Связывание считали специфическим, если был получен сигнал ИФА ≥0,1 оптических единиц, что было по меньшей мере в два раза выше отрицательного контроля. Результаты показаны на Фигуре 6. Например, DLX2690 и DLX2691 едва проходили за отсекающий фильтр, но были признаны положительными. Оставшиеся совокупные мутанты с перетасовкой VL и VH цепей связывались с рчИЛ-1-бета с абсолютными ИФА сигналами, сопоставимыми, или даже превышающими DLX2323.
Значения IC50 для DLX2681 и DLX2693 определяли в тесте на фибробластах человека, как описано выше. Было установлено, что DLX2681 нейтрализует рчИЛ-1-бета со значением IC50 0,6 пМ, а значение IC50 для DLX2693 составило 12,5 пМ.
В то время как показаны и описаны предпочтительные варианты осуществления настоящего изобретения, необходимо понимать, что настоящее изобретение не ограничивается ими, но может быть воплощено иначе и осуществлено в объеме следующей формулы изобретения. Поскольку многочисленные модификации и альтернативные варианты осуществления настоящего изобретения легко понятны для специалиста в данной области техники, описание следует считать лишь иллюстративным и предназначенным для обучения специалистов в данной области техники наилучшему способу осуществления настоящего изобретения. Соответственно, все подходящие модификации и эквиваленты можно рассматривать как относящиеся к объему следующей формулы изобретения.
--->
SEQUENCE LISTING
<110> Delenex Therapeutics AG
<120> Binding members to IL-1 beta
<130> P109026PC00
<150> US 61/722532
<151> 2012-11-05
<150> EP 12007503.1
<151> 2012-11-05
<160> 163
<170> PatentIn version 3.5
<210> 1
<211> 9
<212> PRT
<213> Oryctolagus cuniculus
<400> 1
Phe Ser Leu Ser Ser Ala Ala Met Ala
1 5
<210> 2
<211> 16
<212> PRT
<213> Oryctolagus cuniculus
<400> 2
Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys Gly
1 5 10 15
<210> 3
<211> 11
<212> PRT
<213> Oryctolagus cuniculus
<400> 3
Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu
1 5 10
<210> 4
<211> 11
<212> PRT
<213> Oryctolagus cuniculus
<400> 4
Gln Ala Ser Gln Ser Ile Asp Asn Trp Leu Ser
1 5 10
<210> 5
<211> 7
<212> PRT
<213> Oryctolagus cuniculus
<400> 5
Arg Ala Ser Thr Leu Ala Ser
1 5
<210> 6
<211> 10
<212> PRT
<213> Oryctolagus cuniculus
<400> 6
Gln Asn Thr Gly Gly Gly Val Ser Ile Ala
1 5 10
<210> 7
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> humanized VH sequence
<400> 7
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 8
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> humanized VL sequence
<400> 8
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 9
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> linker sequence
<400> 9
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 10
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> humanized IL-1 beta scFv
<400> 10
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 11
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> variant of CDR-H1
<220>
<221> misc_feature
<222> (5)..(6)
<223> Xaa can be any naturally occurring amino acid
<400> 11
Phe Ser Leu Ser Xaa Xaa Ala Met Ala
1 5
<210> 12
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant of CDR-H2
<220>
<221> misc_feature
<222> (3)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (9)..(9)
<223> Xaa can be any naturally occurring amino acid
<400> 12
Ile Ile Xaa Xaa Ser Ala Ser Thr Xaa Tyr Ala Ser Trp Ala Lys Gly
1 5 10 15
<210> 13
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> variant of CDR-H3
<220>
<221> misc_feature
<222> (2)..(11)
<223> Xaa can be any naturally occurring amino acid
<400> 13
Glu Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 14
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant of CDR-L1
<220>
<221> misc_feature
<222> (7)..(9)
<223> Xaa can be any naturally occurring amino acid
<400> 14
Gln Ala Ser Gln Ser Ile Xaa Xaa Xaa Leu Ser
1 5 10
<210> 15
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant of CDR-L2
<220>
<221> misc_feature
<222> (1)..(1)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (4)..(4)
<223> Xaa can be any naturally occurring amino acid
<400> 15
Xaa Ala Ser Xaa Leu Ala Ser
1 5
<210> 16
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant of CDR-L3
<220>
<221> misc_feature
<222> (3)..(3)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (5)..(8)
<223> Xaa can be any naturally occurring amino acid
<400> 16
Gln Asn Xaa Gly Xaa Xaa Xaa Xaa Ile Ala
1 5 10
<210> 17
<211> 744
<212> DNA
<213> Artificial Sequence
<220>
<223> DNA sequence of DLX2323
<400> 17
gaaattgtta tgacccagag cccgagcacc ctgagcgcaa gcgttggtga tcgtgtgatt 60
attacctgtc aggcaagcca gagcattgat aattggctga gctggtatca gcagaaaccg 120
ggtaaagcac cgaaactgct gatttatcgt gcaagcaccc tggcaagcgg tgttccgagc 180
cgttttagcg gtagcggtag tggtgcagaa tttaccctga ccattagcag cctgcagccg 240
gatgattttg caacctatta ttgtcagaat accggtggtg gtgttagcat tgcatttggt 300
cagggcacca aactgaccgt tctgggtggt ggcggtggat ccggtggggg tggtagcgga 360
ggtggtggtt caggcggtgg tggcagcgaa gttcagctgg ttgaaagtgg tggtggtctg 420
gttcagcctg gtggtagcct gcgtctgagc tgtaccgcaa gcggttttag cctgagcagc 480
gcagcaatgg catgggttcg tcaggcacct ggtaaaggtc tggaatgggt tggtattatc 540
tatgatagcg caagcaccta ttatgcaagc tgggcaaaag gtcgttttac cattagccgt 600
gataccagta aaaataccgt ttacctgcag atgaatagtc tgcgtgcaga ggataccgca 660
gtgtattatt gtgcacgtga acgtgcaatt ttcagcggtg attttgttct gtggggtcag 720
ggaaccctgg ttaccgttag cagc 744
<210> 18
<211> 23
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence L1
<400> 18
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys
20
<210> 19
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence L2
<400> 19
Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr
1 5 10 15
<210> 20
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence L3
<400> 20
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Ala Glu Phe Thr
1 5 10 15
Leu Thr Ile Ser Ser Leu Gln Pro Asp Asp Phe Ala Thr Tyr Tyr Cys
20 25 30
<210> 21
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence L4
<400> 21
Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
1 5 10
<210> 22
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H1
<400> 22
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly
20 25
<210> 23
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H2
<400> 23
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
1 5 10
<210> 24
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H3
<400> 24
Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
20 25 30
<210> 25
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H4
<400> 25
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 26
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H1
<400> 26
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Val Ser Gly
20 25
<210> 27
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H2
<400> 27
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
1 5 10
<210> 28
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H3
<400> 28
Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg
20 25 30
<210> 29
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H4
<400> 29
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
1 5 10
<210> 30
<211> 26
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H1
<400> 30
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Ser Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly
20 25
<210> 31
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H2
<400> 31
Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Gly
1 5 10
<210> 32
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H3
<400> 32
Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu Gln
1 5 10 15
Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ser Tyr Tyr Cys Ala Arg
20 25 30
<210> 33
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> framework sequence H4
<400> 33
Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
1 5 10
<210> 34
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1_D32X
<220>
<221> misc_feature
<222> (30)..(30)
<223> Xaa can be any naturally occurring amino acid
<400> 34
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Xaa Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 35
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L1_N33X
<220>
<221> misc_feature
<222> (31)..(31)
<223> Xaa can be any naturally occurring amino acid
<400> 35
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Xaa Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 36
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L1_W40X
<220>
<221> misc_feature
<222> (32)..(32)
<223> Xaa can be any naturally occurring amino acid
<400> 36
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Xaa
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 37
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L2_R58X
<220>
<221> misc_feature
<222> (50)..(50)
<223> Xaa can be any naturally occurring amino acid
<400> 37
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Xaa Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 38
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L2_T69X
<220>
<221> misc_feature
<222> (53)..(53)
<223> Xaa can be any naturally occurring amino acid
<400> 38
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Xaa Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 39
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L3_T109X
<220>
<221> misc_feature
<222> (91)..(91)
<223> Xaa can be any naturally occurring amino acid
<400> 39
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Xaa Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 40
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L3_G111X
<220>
<221> misc_feature
<222> (93)..(93)
<223> Xaa can be any naturally occurring amino acid
<400> 40
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Xaa Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 41
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L3_G112X
<220>
<221> misc_feature
<222> (94)..(94)
<223> Xaa can be any naturally occurring amino acid
<400> 41
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Xaa Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 42
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L3_V135X
<220>
<221> misc_feature
<222> (95)..(95)
<223> Xaa can be any naturally occurring amino acid
<400> 42
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Xaa Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 43
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-L3_S136X
<220>
<221> misc_feature
<222> (96)..(96)
<223> Xaa can be any naturally occurring amino acid
<400> 43
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Xaa
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 44
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H1_S33X
<220>
<221> misc_feature
<222> (160)..(160)
<223> Xaa can be any naturally occurring amino acid
<400> 44
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Xaa
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 45
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H1_A39X
<220>
<221> misc_feature
<222> (161)..(161)
<223> Xaa can be any naturally occurring amino acid
<400> 45
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Xaa Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 46
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H2_Y59X
<220>
<221> misc_feature
<222> (181)..(181)
<223> Xaa can be any naturally occurring amino acid
<400> 46
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Xaa Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 47
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H2_D60X
<220>
<221> misc_feature
<222> (182)..(182)
<223> Xaa can be any naturally occurring amino acid
<400> 47
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Xaa Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 48
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H2_Y69X
<220>
<221> misc_feature
<222> (187)..(187)
<223> Xaa can be any naturally occurring amino acid
<400> 48
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Xaa Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 49
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_R110X
<220>
<221> misc_feature
<222> (228)..(228)
<223> Xaa can be any naturally occurring amino acid
<400> 49
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Xaa Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 50
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_A111X
<220>
<221> misc_feature
<222> (229)..(229)
<223> Xaa can be any naturally occurring amino acid
<400> 50
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Xaa Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 51
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_I112X
<220>
<221> misc_feature
<222> (230)..(230)
<223> Xaa can be any naturally occurring amino acid
<400> 51
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Xaa Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 52
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_F113X
<220>
<221> misc_feature
<222> (231)..(231)
<223> Xaa can be any naturally occurring amino acid
<400> 52
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Xaa Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 53
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_S114X
<220>
<221> misc_feature
<222> (232)..(232)
<223> Xaa can be any naturally occurring amino acid
<400> 53
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Xaa Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 54
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_G115X
<220>
<221> misc_feature
<222> (233)..(233)
<223> Xaa can be any naturally occurring amino acid
<400> 54
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Xaa Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 55
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_D135X
<220>
<221> misc_feature
<222> (234)..(234)
<223> Xaa can be any naturally occurring amino acid
<400> 55
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Xaa Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 56
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_F136X
<220>
<221> misc_feature
<222> (235)..(235)
<223> Xaa can be any naturally occurring amino acid
<400> 56
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Xaa Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 57
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_V137X
<220>
<221> misc_feature
<222> (236)..(236)
<223> Xaa can be any naturally occurring amino acid
<400> 57
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Xaa Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 58
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant CDR-H3_L138X
<220>
<221> misc_feature
<222> (237)..(237)
<223> Xaa can be any naturally occurring amino acid
<400> 58
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Xaa Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 59
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2464
<400> 59
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Ala Gly Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 60
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2465
<400> 60
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Asp Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 61
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2466
<400> 61
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Lys Tyr
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Asp Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Ala Gly Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 62
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2467
<400> 62
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile His Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ser Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Arg
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Met Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 63
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2468
<400> 63
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Asn Tyr
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Thr Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ser Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 64
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2475
<400> 64
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Lys Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val His
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Lys Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 65
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2476
<400> 65
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Asp Ile Phe Ser Gly Asp Phe Val Gly Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 66
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2480
<400> 66
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile Asn
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asp
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 67
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2543
<400> 67
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 68
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2529
<400> 68
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile Asn
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 69
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2547
<400> 69
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser
145 150 155 160
Ser Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
210 215 220
Cys Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 70
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2528
<400> 70
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Ala Thr Thr
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 71
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2585
<400> 71
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Asp Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 72
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2545
<400> 72
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Ile Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Leu Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
210 215 220
Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Val Leu Trp Gly Gln
225 230 235 240
Gly Thr Thr Val Thr Val Ser Ser
245
<210> 73
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2531
<400> 73
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asn
145 150 155 160
Ser Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Ala Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 74
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2586
<400> 74
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Ile Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
210 215 220
Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 75
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2530
<400> 75
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asp
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Phe Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Ala Leu Trp Gly Gln
225 230 235 240
Gly Thr Thr Val Thr Val Ser Ser
245
<210> 76
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2548
<400> 76
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Ile Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Leu Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
210 215 220
Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly Gln
225 230 235 240
Gly Thr Thr Val Thr Val Ser Ser
245
<210> 77
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2676
<400> 77
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Ala Thr Thr
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asp
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Phe Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Ala Leu Trp Gly Gln
225 230 235 240
Gly Thr Thr Val Thr Val Ser Ser
245
<210> 78
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2677
<400> 78
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Ala Thr Thr
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asn
145 150 155 160
Ser Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Ala Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 79
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2678
<400> 79
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Ala Thr Thr
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Ile Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Leu Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
210 215 220
Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly Gln
225 230 235 240
Gly Thr Thr Val Thr Val Ser Ser
245
<210> 80
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2679
<400> 80
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Ala Thr Thr
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Asp Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 81
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2680
<400> 81
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile Asn
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asp
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Phe Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Ala Leu Trp Gly Gln
225 230 235 240
Gly Thr Thr Val Thr Val Ser Ser
245
<210> 82
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2681
<400> 82
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile Asn
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asn
145 150 155 160
Ser Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Ala Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 83
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2682
<400> 83
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile Asn
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Ile Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Leu Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys
210 215 220
Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly Gln
225 230 235 240
Gly Thr Thr Val Thr Val Ser Ser
245
<210> 84
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2683
<400> 84
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile Asn
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Asp Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 85
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2684
<400> 85
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asp
145 150 155 160
Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Phe Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Ala Leu Trp Gly Gln
225 230 235 240
Gly Thr Thr Val Thr Val Ser Ser
245
<210> 86
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2685
<400> 86
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asn
145 150 155 160
Ser Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Ala Leu Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 87
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2686
<400> 87
Met Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Trp Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Val
85 90 95
Ser Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser
145 150 155 160
Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Ile Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Leu
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe
210 215 220
Cys Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly
225 230 235 240
Gln Gly Thr Thr Val Thr Val Ser Ser
245
<210> 88
<211> 248
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2687
<400> 88
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
130 135 140
Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser
145 150 155 160
Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
165 170 175
Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala
180 185 190
Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val Tyr
195 200 205
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Asp Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 89
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2689
<400> 89
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser
145 150 155 160
Asn Ser Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr
210 215 220
Cys Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Ala Leu Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 90
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2690
<400> 90
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser
145 150 155 160
Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Ile Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Leu
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe
210 215 220
Cys Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly
225 230 235 240
Gln Gly Thr Thr Val Thr Val Ser Ser
245
<210> 91
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2691
<400> 91
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser
145 150 155 160
Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
210 215 220
Cys Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Asp Tyr Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 92
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2692
<400> 92
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser
145 150 155 160
Asp Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Phe Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr
210 215 220
Cys Ala Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Ala Leu Trp Gly
225 230 235 240
Gln Gly Thr Thr Val Thr Val Ser Ser
245
<210> 93
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2693
<400> 93
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser
145 150 155 160
Asn Ser Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr
210 215 220
Cys Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Ala Leu Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 94
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2694
<400> 94
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser
145 150 155 160
Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Ile Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Leu
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe
210 215 220
Cys Ala Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly
225 230 235 240
Gln Gly Thr Thr Val Thr Val Ser Ser
245
<210> 95
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> Variant DLX2695
<400> 95
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser
145 150 155 160
Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
210 215 220
Cys Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Asp Tyr Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 96
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L1_D32X
<220>
<221> misc_feature
<222> (30)..(30)
<223> Xaa can be any naturally occurring amino acid
<400> 96
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Xaa Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 97
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L1_N33X
<220>
<221> misc_feature
<222> (31)..(31)
<223> Xaa can be any naturally occurring amino acid
<400> 97
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Xaa Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 98
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L1_W40X
<220>
<221> misc_feature
<222> (32)..(32)
<223> Xaa can be any naturally occurring amino acid
<400> 98
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Xaa
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 99
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L2_R58X
<220>
<221> misc_feature
<222> (50)..(50)
<223> Xaa can be any naturally occurring amino acid
<400> 99
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Xaa Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 100
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L2_T69X
<220>
<221> misc_feature
<222> (53)..(53)
<223> Xaa can be any naturally occurring amino acid
<400> 100
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Xaa Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 101
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L3_T109X
<220>
<221> misc_feature
<222> (91)..(91)
<223> Xaa can be any naturally occurring amino acid
<400> 101
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Xaa Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 102
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L3_G111X
<220>
<221> misc_feature
<222> (93)..(93)
<223> Xaa can be any naturally occurring amino acid
<400> 102
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Xaa Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 103
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L3_G112X
<220>
<221> misc_feature
<222> (94)..(94)
<223> Xaa can be any naturally occurring amino acid
<400> 103
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Xaa Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 104
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L3_V135X
<220>
<221> misc_feature
<222> (95)..(95)
<223> Xaa can be any naturally occurring amino acid
<400> 104
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Xaa Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 105
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL CDR-L3_S136X
<220>
<221> misc_feature
<222> (96)..(96)
<223> Xaa can be any naturally occurring amino acid
<400> 105
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Xaa
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 106
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H1_S33X
<220>
<221> misc_feature
<222> (31)..(31)
<223> Xaa can be any naturally occurring amino acid
<400> 106
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Xaa Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 107
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H1_A39X
<220>
<221> misc_feature
<222> (32)..(32)
<223> Xaa can be any naturally occurring amino acid
<400> 107
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Xaa
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 108
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H2_Y59X
<220>
<221> misc_feature
<222> (52)..(52)
<223> Xaa can be any naturally occurring amino acid
<400> 108
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Xaa Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 109
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H2_D60X
<220>
<221> misc_feature
<222> (53)..(53)
<223> Xaa can be any naturally occurring amino acid
<400> 109
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Xaa Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 110
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H2_Y69X
<220>
<221> misc_feature
<222> (58)..(58)
<223> Xaa can be any naturally occurring amino acid
<400> 110
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Xaa Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 111
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_R110X
<220>
<221> misc_feature
<222> (99)..(99)
<223> Xaa can be any naturally occurring amino acid
<400> 111
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Xaa Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 112
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_A111X
<220>
<221> misc_feature
<222> (100)..(100)
<223> Xaa can be any naturally occurring amino acid
<400> 112
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Xaa Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 113
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_I112X
<220>
<221> misc_feature
<222> (101)..(101)
<223> Xaa can be any naturally occurring amino acid
<400> 113
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Xaa Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 114
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_F113X
<220>
<221> misc_feature
<222> (102)..(102)
<223> Xaa can be any naturally occurring amino acid
<400> 114
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Xaa Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 115
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_S114X
<220>
<221> misc_feature
<222> (103)..(103)
<223> Xaa can be any naturally occurring amino acid
<400> 115
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Xaa Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 116
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_G115X
<220>
<221> misc_feature
<222> (104)..(104)
<223> Xaa can be any naturally occurring amino acid
<400> 116
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Xaa Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 117
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_D135X
<220>
<221> misc_feature
<222> (105)..(105)
<223> Xaa can be any naturally occurring amino acid
<400> 117
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Xaa Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 118
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_F136X
<220>
<221> misc_feature
<222> (106)..(106)
<223> Xaa can be any naturally occurring amino acid
<400> 118
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Xaa Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 119
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_V137X
<220>
<221> misc_feature
<222> (107)..(107)
<223> Xaa can be any naturally occurring amino acid
<400> 119
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Xaa Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 120
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH CDR-H3_L138X
<220>
<221> misc_feature
<222> (108)..(108)
<223> Xaa can be any naturally occurring amino acid
<400> 120
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Xaa Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 121
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2464
<400> 121
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Ala Gly Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 122
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2465
<400> 122
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Asp Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 123
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2466
<400> 123
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Lys Tyr
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 124
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2466
<400> 124
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Asp
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Ala Gly Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 125
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2467
<400> 125
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile His Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ser Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 126
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX 2467
<400> 126
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Arg Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Met Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 127
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2468
<400> 127
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Asn Tyr
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Thr Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 128
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2468
<400> 128
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ser
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 129
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2475
<400> 129
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Lys Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val His
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 130
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2475
<400> 130
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Tyr
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Lys Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 131
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2476
<400> 131
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 132
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2476
<400> 132
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Asp Ile Phe Ser Gly Asp Phe Val Gly Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 133
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2480
<400> 133
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile Asn
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 134
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2480
<400> 134
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asp Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Gln Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 135
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2543
<400> 135
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 136
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2529
<400> 136
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Arg Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile Asn
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 137
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2547
<400> 137
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg
100 105 110
<210> 138
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2547
<400> 138
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 139
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2528
<400> 139
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Gly Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gln Ala Ser Asn Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Ala Thr Thr
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 140
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2528
<400> 140
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 141
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2585
<400> 141
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 142
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2585
<400> 142
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 143
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2545
<400> 143
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 144
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2545
<400> 144
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Ser Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys Ala
85 90 95
Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Val Leu Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 145
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2531
<400> 145
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 146
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2531
<400> 146
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asn Ser
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Ala Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 147
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2586
<400> 147
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 148
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2586
<400> 148
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Val Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys Ala
85 90 95
Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 149
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2530
<400> 149
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 150
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2530
<400> 150
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Asn Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser Asp Ala
20 25 30
Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Phe Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Thr Tyr Tyr Cys Ala
85 90 95
Arg Glu Arg Asn Ile Phe Ser Gly Asp Met Ala Leu Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 151
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2548
<400> 151
Glu Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Ile Ile Thr Cys Gln Ala Ser Gln Ser Ile Asp Asn Trp
20 25 30
Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Arg Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Ala Glu Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Asp Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Thr Gly Gly Gly Val Ser
85 90 95
Ile Ala Phe Gly Gln Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 152
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> VH DLX2548
<400> 152
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Thr Val Ser Gly Phe Ser Leu Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys
50 55 60
Gly Arg Phe Thr Ile Ser Lys Asp Thr Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Phe Cys Ala
85 90 95
Arg Glu Arg Gln Ile Phe Ser Gly Asp Met Asp Gly Trp Gly Gln Gly
100 105 110
Thr Thr Val Thr Val Ser Ser
115
<210> 153
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> VL DLX2544
<400> 153
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg
100 105 110
<210> 154
<211> 249
<212> PRT
<213> Artificial Sequence
<220>
<223> DLX2544
<400> 154
Ala Asp Ile Val Met Thr Gln Ser Pro Ser Thr Leu Ser Ala Ser Val
1 5 10 15
Gly Asp Arg Val Thr Ile Thr Cys Gln Ala Ser Gln Ser Ile Ser Ser
20 25 30
Tyr Leu Ser Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Lys Ala Ser Thr Leu Ala Ser Gly Val Pro Ser Arg Phe Ser
50 55 60
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
65 70 75 80
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Asn Ala Gly Gly Gly Ile
85 90 95
Asn Ile Ala Phe Gly Gln Gly Thr Lys Val Glu Ile Lys Arg Gly Gly
100 105 110
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
115 120 125
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
130 135 140
Gly Gly Ser Leu Arg Leu Ser Cys Thr Ala Ser Gly Phe Ser Leu Ser
145 150 155 160
Ser Ala Ala Met Ala Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
165 170 175
Trp Val Gly Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp
180 185 190
Ala Lys Gly Arg Phe Thr Ile Ser Arg Asp Thr Ser Lys Asn Thr Val
195 200 205
Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
210 215 220
Cys Ala Arg Glu Arg Ala Ile Phe Ser Gly Asp Phe Val Leu Trp Gly
225 230 235 240
Gln Gly Thr Leu Val Thr Val Ser Ser
245
<210> 155
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H1 of DLX2531
<400> 155
Phe Ser Leu Ser Asn Ser Ala Met Ala
1 5
<210> 156
<211> 16
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H2 of DLX2531
<400> 156
Ile Ile Tyr Asp Ser Ala Ser Thr Tyr Tyr Ala Ser Trp Ala Lys Gly
1 5 10 15
<210> 157
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-H3 of DLX2531
<400> 157
Glu Arg Ala Ile Phe Ser Gly Asp Phe Ala Leu
1 5 10
<210> 158
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1 of DLX2531
<400> 158
Gln Ala Ser Gln Ser Ile Asp Asn Trp Leu Ser
1 5 10
<210> 159
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L2 of DLX2531
<400> 159
Arg Ala Ser Thr Leu Ala Ser
1 5
<210> 160
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3 of DLX2531
<400> 160
Gln Asn Thr Gly Gly Gly Val Ser Ile Ala
1 5 10
<210> 161
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L1 of DLX2681
<400> 161
Arg Ala Ser Gln Ser Ile Gly Asn Trp Leu Ser
1 5 10
<210> 162
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L2 of DLX2681
<400> 162
Arg Ala Ser Asn Leu Ala Ser
1 5
<210> 163
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> CDR-L3 of DLX2681
<400> 163
Gln Asn Thr Gly Gly Gly Ile Asn Ile Ala
1 5 10
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| АМАТОКСИНОВЫЕ КОНЪЮГАТЫ АНТИТЕЛА С ЛЕКАРСТВЕННЫМ СРЕДСТВОМ И ИХ ПРИМЕНЕНИЕ | 2020 |
|
RU2826004C2 |
| МОНОКЛОНАЛЬНЫЕ АГЕНТЫ, СВЯЗЫВАЮЩИЕ сМЕТ, ИХ КОНЪЮГАТЫ С ЛЕКАРСТВЕННЫМИ СРЕДСТВАМИ И ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2744914C2 |
| АНТИТЕЛО К TIGIT И ЕГО ИСПОЛЬЗОВАНИЕ | 2019 |
|
RU2786434C2 |
| CD131-СВЯЗЫВАЮЩИЕ БЕЛКИ И ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2773927C2 |
| АНТИТЕЛА ПРОТИВ HLA-G, КОМПОЗИЦИИ, СОДЕРЖАЩИЕ АНТИТЕЛА ПРОТИВ HLA-G, И СПОСОБЫ ПРИМЕНЕНИЯ АНТИТЕЛ ПРОТИВ HLA-G | 2019 |
|
RU2810326C2 |
| Антитела против ингибитора пути тканевого фактора и их применения | 2016 |
|
RU2815681C1 |
| АНТИТЕЛА, КОТОРЫЕ СВЯЗЫВАЮТСЯ С РАКОВЫМИ КЛЕТКАМИ И ОБЕСПЕЧИВАЮТ НАПРАВЛЕННЫЙ ПЕРЕНОС РАДИОНУКЛИДОВ К УКАЗАННЫМ КЛЕТКАМ | 2020 |
|
RU2833191C2 |
| МЕТОДЫ ПРОФИЛАКТИКИ И ЛЕЧЕНИЯ СЕРДЕЧНЫХ ЗАБОЛЕВАНИЙ | 2018 |
|
RU2830232C2 |
| ГУМАНИЗИРОВАННОЕ АНТИТЕЛО ПРОТИВ РЕЦЕПТОРА IGF-1 | 2021 |
|
RU2836869C1 |
| КОМБИНИРОВАННАЯ ТЕРАПИЯ ИЗ АНТИТЕЛА К ПРОГАСТРИНУ И ИММУНОТЕРАПИИ ДЛЯ ЛЕЧЕНИЯ РАКА | 2018 |
|
RU2784604C2 |
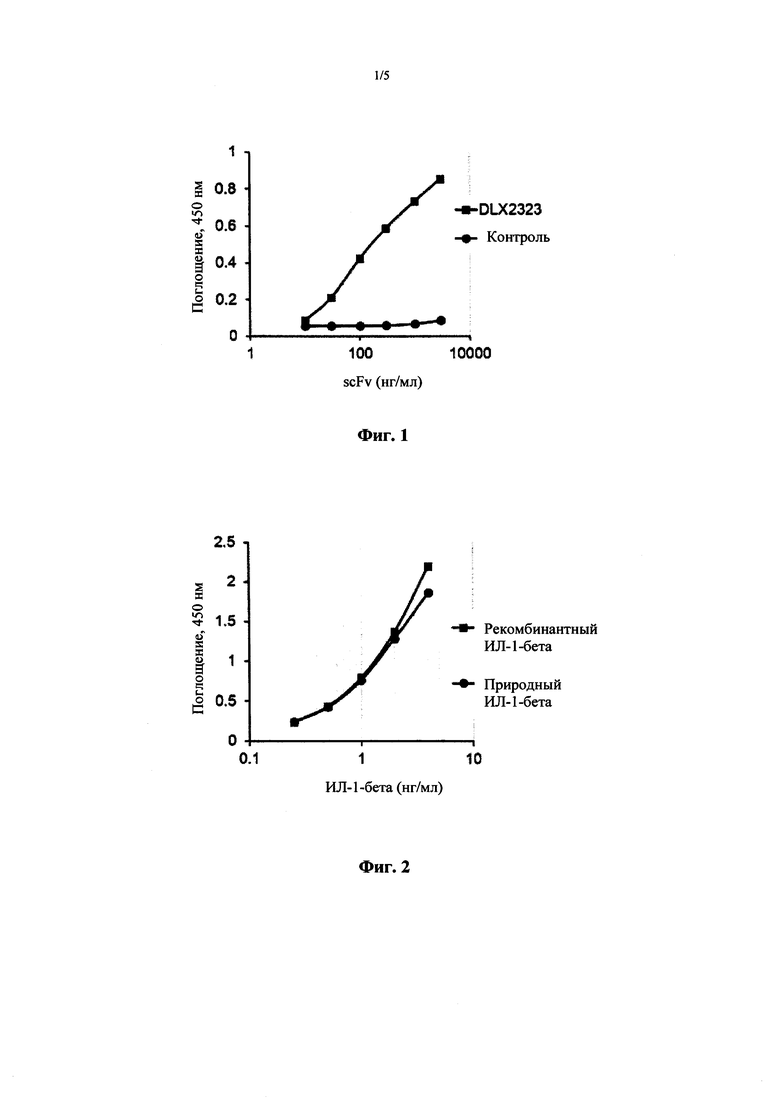
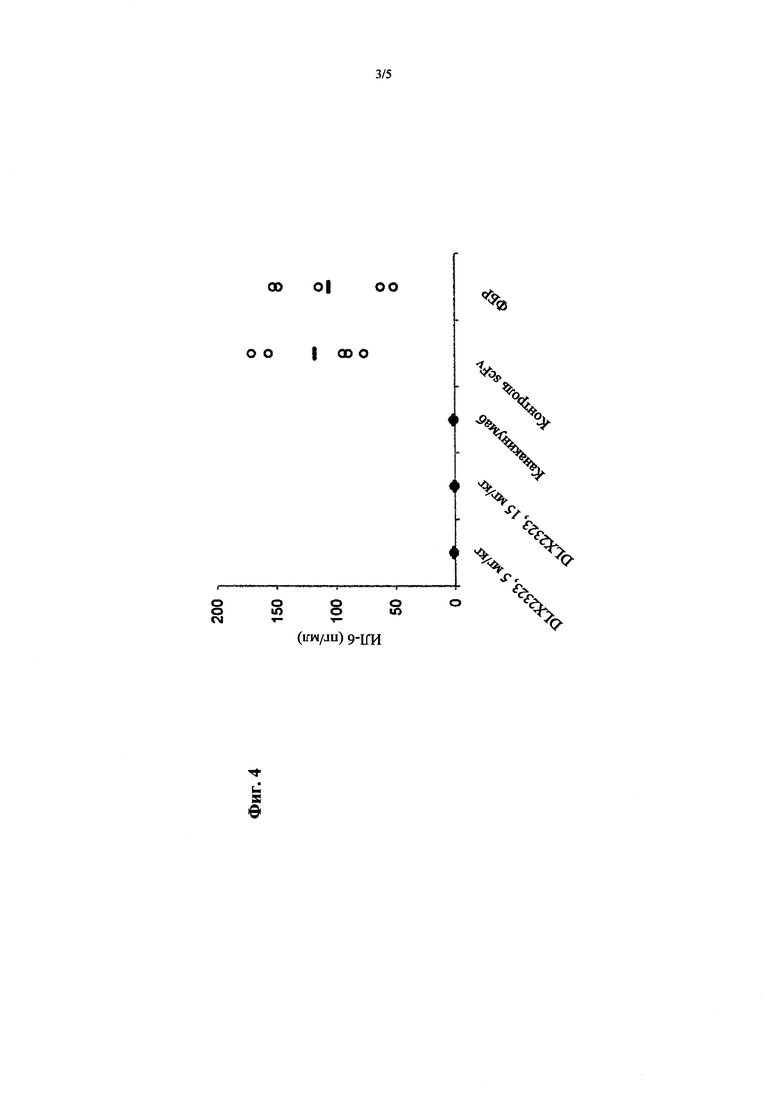

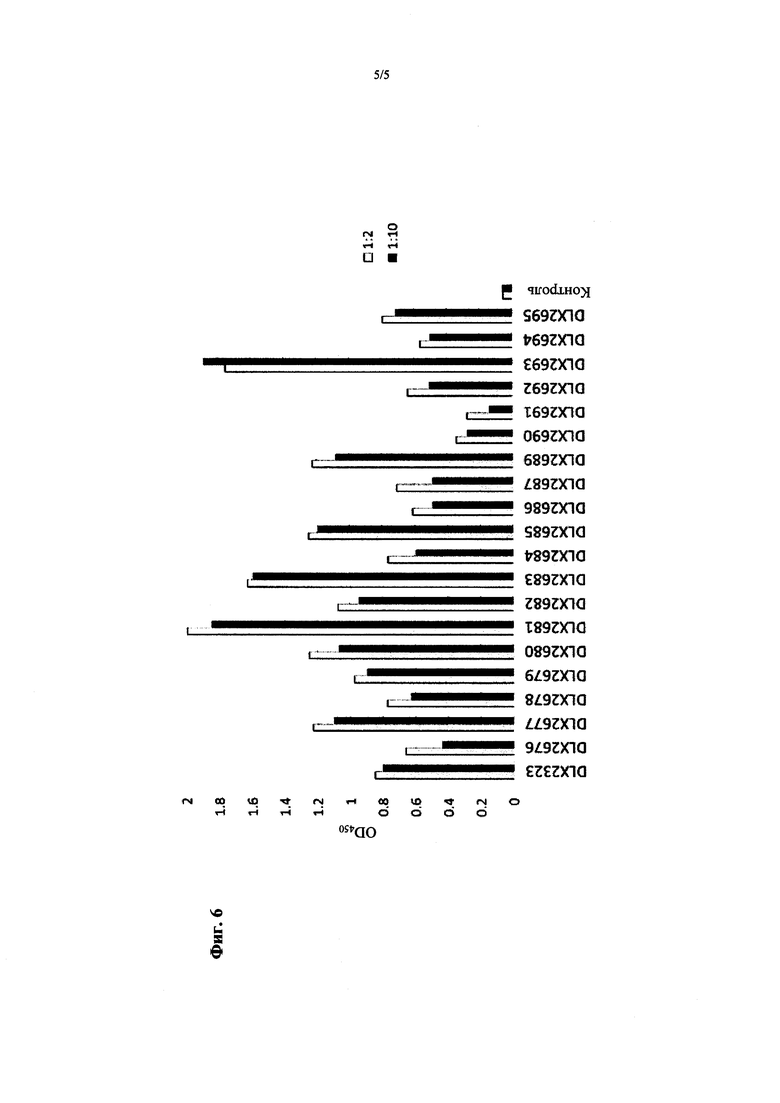
Настоящее изобретение относится к биотехнологии. Предложены антитела и их антиген-связывающие фрагменты против ИЛ-1-бета. Кроме того, описаны кодирующая нуклеиновая кислота, вектор, клетка-хозяин и способы получения антитела и его антиген-связывающего фрагмента. Также представлены фармацевтическая композиция и способ лечения заболевания, ассоциированного с ИЛ-1 бета; набор и способ детекции присутствия ИЛ-1 бета в биологическом образце. Данное изобретение может найти применение в лечении и диагностике различных воспалительных заболеваний. 11 н. и 34 з.п. ф-лы, 6 ил., 7 табл., 10 пр.
1. Антитело против ИЛ-1 бета или его антиген-связывающий фрагмент, содержащее
a) вариабельные последовательности CDR тяжелой цепи (VH) CDR-H1, CDR-H2 и CDR-H3, указанные в
(i) SEQ ID No.: 1, 2 и 3 соответственно, или
(ii) SEQ ID No.: 155, 156 и 157 соответственно; и
b) вариабельные последовательности CDR легкой цепи (VL) CDR-L1, CDR-L2 и CDR-L3, указанные в
(i) SEQ ID No.: 4, 5, и 6 соответственно, или
(ii) SEQ ID No.: 161, 162 и 163 соответственно.
2. Антитело или его антиген-связывающий фрагмент по п. 1, являющееся моновалентным фрагментом антитела.
3. Антитело или его антиген-связывающий фрагмент по п. 2, являющееся Fab, Fab', scFv или Fv фрагментом.
4. Антитело или его антиген-связывающий фрагмент по п. 1, обладающее активностью (IC50) в отношении ингибирования биологической активности ИЛ-1 бета человека менее 50 пМ, определяемой по ингибированию стимулированного ИЛ-1 бета высвобождения ИЛ-6 из фибробластов человека.
5. Антитело или его антиген-связывающий фрагмент по п. 4, отличающееся тем, что указанная IC50 меньше чем 30, 20, 10, 5 пМ.
6. Антитело или его антиген-связывающий фрагмент по п. 1, являющееся полноразмерным иммуноглобулином или бивалентным фрагментом антитела, предпочтительно F(ab')2.
7. Антитело или его антиген-связывающий фрагмент по любому из пп. 1-6, содержащее по меньшей мере один вариабельный каркасный участок легкой цепи FR-L1, представленный в SEQ ID No.: 18, вариабельный каркасный участок легкой цепи FR-L2, представленный в SEQ ID No.: 19, вариабельный каркасный участок легкой цепи FR-L3, представленный в SEQ ID No.: 20, и/или вариабельный каркасный участок легкой цепи FR-L4, представленный в SEQ ID No.: 21.
8. Антитело или его антиген-связывающий фрагмент по любому из пп. 1-6, содержащее по меньшей мере один вариабельный каркасный участок тяжелой цепи FR-H1, представленный в SEQ ID No.: 22, 26 или 30; вариабельный каркасный участок тяжелой цепи FR-H2, представленный в SEQ ID No.: 23, 27 или 31; вариабельный каркасный участок легкой цепи FR-H3, представленный в SEQ ID No.: 24, 28 или 32; и/или вариабельный каркасный участок легкой цепи FR-H4, представленный в SEQ ID No.: 25, 29 или 33.
9. Антитело или его антиген-связывающий фрагмент по любому из пп. 1-6, содержащее линкерную последовательность, представленную в SEQ ID No.: 9.
10. Антитело или его антиген-связывающий фрагмент по любому из пп. 1-6, являющееся гуманизированным.
11. Антитело или его антиген-связывающий фрагмент по любому из пп. 1-6, являющееся кросс-реактивным с ИЛ-1 бета яванского макака, ИЛ-1 бета макака-резуса и/или ИЛ-1 бета крысы.
12. Антитело или его антиген-связывающий фрагмент по любому из пп. 1-6, являющееся химически или биологически модифицированным.
13. Антитело или его антиген-связывающий фрагмент по п. 12, модифицированное для увеличения периода полувыведения in vivo в сыворотке.
14. Антитело или его антиген-связывающий фрагмент по любому из пп. 1-6
(i) для применения при лечении заболевания, ассоциированного с ИЛ-1 бета;
(ii) для применения в диагностических целях;
(iii) для применения в косметических целях; и/или
(iv) для целей детектирования.
15. Антитело или его антиген-связывающий фрагмент по п. 14, где заболеванием, ассоциированным с ИЛ-1 бета, является пролиферативная диабетическая ретинопатия, подагрический артрит, синдром Шницлера, системный юношеский идиопатический артрит, ревматоидный артрит, острый подагрический артрит, хронический подагрический артрит, крапивная лихорадка, васкулит, диабет первого типа, диабет второго типа, анкилозирующий спондилоартрит, рецидивирующий множественный остеомиелит, рецидивирующее воспаление нескольких хрящей, периодический синдром, ассоциированный с циропирином (CAPS), болезнь Бехчета, семейная средиземноморская лихорадка, хроническое обструктивное заболевание легких, ревматическая полимиалгия, мутации NALP3, гангренозная пиодермия, хроническая идиопатическая крапивная лихорадка, остеоартрит, влажная возрастная дегенерация желтого пятна, синдром сухого глаза, пустулезный псориаз, синдром синовита, акне, пустулеза, гиперостоза и остеита, синдром активации макрофагов, синдром лихорадки, афтозного стоматита, фарингита и шейного лимфаденита, болезнь Стилла у совершеннолетних, дефицит мевалонаткиназы, атеросклероз, периодический синдром, ассоциированный с рецепторами ФНО (TRAPS), обыкновенные угри и/или гнойный гидраденит.
16. Антитело или его антиген-связывающий фрагмент против ИЛ-1 бета, содержащее
a) последовательность VH, выбранную из группы, состоящей из SEQ ID No.: 7 и SEQ ID No.: 146; и
b) последовательность VL, выбранную из группы, состоящей из SEQ ID No.: 8, SEQ ID No.: 136 и SEQ ID No.: 145.
17. Антитело или его антиген-связывающий фрагмент по п. 16, содержащее линкерную последовательность, представленную в SEQ ID No.: 9.
18. Антитело или его антиген-связывающий фрагмент по любому из пп. 16 или 17, являющееся гуманизированным.
19. Антитело или его антиген-связывающий фрагмент по любому из пп. 16 или 17, являющееся кросс-реактивным с ИЛ-1 бета яванского макака, ИЛ-1 бета макака-резуса и/или ИЛ-1 бета крысы.
20. Антитело или его антиген-связывающий фрагмент по любому из пп. 16 или 17, являющееся химически или биологически модифицированным.
21. Антитело или его антиген-связывающий фрагмент по п. 20, модифицированное для увеличения периода полувыведения in vivo в сыворотке.
22. Антитело или его антиген-связывающий фрагмент по любому из пп. 16 или 17
(i) для применения при лечении заболевания, ассоциированного с ИЛ-1 бета;
(ii) для применения в диагностических целях;
(iii) для применения в косметических целях; и/или
(iv) для целей детектирования.
23. Антитело или его антиген-связывающий фрагмент по п. 22, где заболеванием, ассоциированным с ИЛ-1 бета, является пролиферативная диабетическая ретинопатия, подагрический артрит, синдром Шницлера, системный юношеский идиопатический артрит, ревматоидный артрит, острый подагрический артрит, хронический подагрический артрит, крапивная лихорадка, васкулит, диабет первого типа, диабет второго типа, анкилозирующий спондилоартрит, рецидивирующий множественный остеомиелит, рецидивирующее воспаление нескольких хрящей, периодический синдром, ассоциированный с циропирином (CAPS), болезнь Бехчета, семейная средиземноморская лихорадка, хроническое обструктивное заболевание легких, ревматическая полимиалгия, мутации NALP3, гангренозная пиодермия, хроническая идиопатическая крапивная лихорадка, остеоартрит, влажная возрастная дегенерация желтого пятна, синдром сухого глаза, пустулезный псориаз, синдром синовита, акне, пустулеза, гиперостоза и остеита, синдром активации макрофагов, синдром лихорадки, афтозного стоматита, фарингита и шейного лимфаденита, болезнь Стилла у совершеннолетних, дефицит мевалонаткиназы, атеросклероз, периодический синдром, ассоциированный с рецепторами ФНО (TRAPS), обыкновенные угри и/или гнойный гидраденит.
24. Изолированная нуклеиновая кислота, кодирующая антитело или его антиген-связывающий фрагмент по любому из пп. 1 или 16.
25. Нуклеиновая кислота по п. 24, содержащая SEQ ID No.: 17.
26. Вектор экспрессии, содержащий нуклеиновую кислоту по любому из пп. 24 или 25.
27. Клетка-хозяин, содержащая последовательность нуклеиновой кислоты по п. 24 или 25 или вектор по п. 26 для экспрессии антитела или его фрагмента.
28. Клетка-хозяин по п. 27, являющаяся прокариотической клеткой или эукариотической клеткой.
29. Фармацевтическая композиция для лечения заболевания, ассоциированного с ИЛ-1 бета, содержащая эффективное количество антитела или его антиген-связывающего фрагмента по любому из пп. 1 или 16 и дополнительно подходящий носитель, разбавитель или наполнитель.
30. Фармацевтическая композиция по п. 29, содержащая фармацевтически приемлемый носитель, разбавитель или наполнитель, предпочтительно изготовленный для парентерального, орального, ректального, урогенитального, местного, интравитреального, внутривенного. внутриглазного, ушного, внутриносового, ингаляторного, кожного, подъязычного или буккального введения.
31. Способ получения антитела или его антиген-связывающего фрагмента по любому из пп. 1 или 16, содержащий стадии
(i) культивирования клетки-хозяина по п. 27 для экспрессии антитела или его антиген-связывающего фрагмента;
(ii) выделения; и
(ii) очистки антитела или его антиген-связывающего фрагмента.
32. Способ получения антитела или его антиген-связывающего фрагмента по любому из пп. 1 или 16, содержащий стадии
(i) обеспечения бесклеточной системы,
(ii) обеспечения нуклеиновой кислоты по п. 24,
(iii) осуществления транскрипции и трансляции указанной нуклеиновой кислоты;
(iv) выделения; и
(v) необязательно, очистки антитела или его антиген-связывающего фрагмента.
33. Способ лечения заболевания, ассоциированного с ИЛ-1 бета, содержащий стадию введения терапевтически эффективного количества антитела или его антиген-связывающего фрагмента по любому из пп. 1 или 16 в субъект, нуждающийся в этом.
34. Способ по п. 33, где заболеванием, ассоциированным с ИЛ-1 бета, является пролиферативная диабетическая ретинопатия, подагрический артрит, синдром Шницлера, системный юношеский идиопатический артрит, ревматоидный артрит, острый подагрический артрит, хронический подагрический артрит, крапивная лихорадка, васкулит, диабет первого типа, диабет второго типа, анкилозирующий спондилоартрит, рецидивирующий множественный остеомиелит, рецидивирующее воспаление нескольких хрящей, периодический синдром, ассоциированный с циропирином (CAPS), болезнь Бехчета, семейная средиземноморская лихорадка, хроническое обструктивное заболевание легких, ревматическая полимиалгия, мутации NALP3, гангренозная пиодермия, хроническая идиопатическая крапивная лихорадка, остеоартрит, влажная возрастная дегенерация желтого пятна, синдром сухого глаза, пустулезный псориаз, синдром синовита, акне, пустулеза, гиперостоза и остеита, синдром активации макрофагов, синдром лихорадки, афтозного стоматита, фарингита и шейного лимфаденита, болезнь Стилла у совершеннолетних, дефицит мевалонаткиназы, атеросклероз, периодический синдром, ассоциированный с рецепторами ФНО (TRAPS), обыкновенные угри и/или гнойный гидраденит.
35. Способ по любому из пп. 33 или 34, в котором указанным субъектом является человек.
36. Способ по любому из пп. 33 или 34, в котором антитело или его антиген-связывающий фрагмент по любому из пп. 1 или 16 представлен в виде фармацевтической композиции по п. 29.
37. Способ по любому из пп. 33 или 34, в котором указанное антитело или его антиген-связывающий фрагмент вводят парентерально, орально, ректально, урогенитально, местно, интравитреально, внутривенно, внутрь глаза, внутрь уха, внутрь носа, путем ингаляции, на кожу, под язык или буккально.
38. Способ по любому из пп. 33 или 34, в котором указанное антитело или его антиген-связывающий фрагмент вводят с по меньшей мере одним дополнительным терапевтически эффективным соединением.
39. Способ по п. 38, в котором указанное соединение вводят одновременно с указанным антителом или его антиген-связывающим фрагментом или после них.
40. Способ детекции присутствия ИЛ-1 бета в биологическом образце, содержащий стадии
a) введения в контакт указанного биологического образца с антителом или его антиген-связывающим фрагментом по любому из пп. 1 или 16 в условиях, допускающих связывание с ИЛ-1 бета, и
b) определения факта, образовался ли комплекс с ИЛ-1 бета.
41. Способ по п. 40, являющийся способом in vitro или in vivo.
42. Способ по любому из пп. 40 или 41, в котором указанный биологический образец получен у человека.
43. Способ по любому из пп. 40 или 41, в котором указанным биологическим образцом является кровь, моча, спинномозговая жидкость, образец, полученный биопсией, и/или лимфа.
44. Способ по любому из пп. 40 или 41, где указанный способ применяют для отбора субъектов, пригодных для терапии с помощью антитела или его антиген-связывающего фрагмента по любому из пп. 1 или 16.
45. Набор для детектирования ИЛ-1 бета в биологическом образце, содержащий антитело или его антиген-связывающий фрагмент по любому из пп. 1 или 16 и упакованную комбинацию реагентов, и инструкцию для применения.
| Способ приготовления мыла | 1923 |
|
SU2004A1 |
| Пресс для выдавливания из деревянных дисков заготовок для ниточных катушек | 1923 |
|
SU2007A1 |
| OWYANG A.M | |||
| et al | |||
| Устройство для устранения мешающего действия зажигательной электрической системы двигателей внутреннего сгорания на радиоприем | 1922 |
|
SU52A1 |
| Пресс для выдавливания из деревянных дисков заготовок для ниточных катушек | 1923 |
|
SU2007A1 |
| Приспособление для суммирования отрезков прямых линий | 1923 |
|
SU2010A1 |
| АНТИТЕЛА К ЧЕЛОВЕЧЕСКОМУ IL-1БЕТА | 2001 |
|
RU2286351C2 |
Авторы
Даты
2020-01-15—Публикация
2013-11-05—Подача