ПРИОРИТЕТНЫЕ ЗАЯВКИ
[0001] Настоящая заявка испрашивает приоритет или преимущество на основании следующих заявок:
[0002] Предварительная заявка на патент США № 62/696,699, озаглавленная “DEEP LEARNING-BASED FRAMEWORK FOR IDENTIFYING SEQUENCE PATTERNS, которые вызывают последовательность- специфичные ошибки (SSEs)” (“ФРЕЙМВОРК НА ОСНОВЕ ГЛУБОКОГО ОБУЧЕНИЯ ДЛЯ ИДЕНТИФИКАЦИИ ПАТТЕРНОВ ПОСЛЕДОВАТЕЛЬНОСТИ, КОТОРЫЕ ВЫЗЫВАЮТ ПОСЛЕДОВАТЕЛЬНОСТЬ-СПЕЦИФИЧНЫЕ ОШИБКИ (SSEs)”), поданная 11 июля 2018, (№ дела поверенного ILLM 1006-1/IP-1650-PRV);
[0003] Заявка Нидеррландов № 2021473, озаглавленная entitled “DEEP LEARNING-BASED FRAMEWORK FOR IDENTIFYING SEQUENCE PATTERNS, которые вызывают последовательность- специфичные ошибки (SSEs)”(“ФРЕЙМВОРК НА ОСНОВЕ ГЛУБОКОГО ОБУЧЕНИЯ ДЛЯ ИДЕНТИФИКАЦИИ ПАТТЕРНОВ ПОСЛЕДОВАТЕЛЬНОСТИ, КОТОРЫЕ ВЫЗЫВАЮТ ПОСЛЕДОВАТЕЛЬНОСТЬ-СПЕЦИФИЧНЫЕ ОШИБКИ (SSEs)”), поданная 16 августа 2018 г., (№ дела поверенного ILLM 1006-4/IP-1650-NL); и
[0004] Непредварительная заявка на патент США №16/505,100, озаглавленная “DEEP LEARNING-BASED FRAMEWORK FOR IDENTIFYING SEQUENCE PATTERNS, которые вызывают последовательность- специфичные ошибки (SSEs)”(“ФРЕЙМВОРК НА ОСНОВЕ ГЛУБОКОГО ОБУЧЕНИЯ ДЛЯ ИДЕНТИФИКАЦИИ ПАТТЕРНОВ ПОСЛЕДОВАТЕЛЬНОСТИ, КОТОРЫЕ ВЫЗЫВАЮТ ПОСЛЕДОВАТЕЛЬНОСТЬ-СПЕЦИФИЧНЫЕ ОШИБКИ (SSEs)”), поданная 08 июля 2019 г., (№ дела поверенного ILLM 1006-2/IP-1650-US).
[0005] Приоритетные заявки включены в настоящий текст посредством ссылки для любых целей.
ВКЛЮЧЕНИЕ
[0006] Следующие документы полностью включены в настоящий текст посредством ссылки так как если бы они были приведены здесь полностью:
[0007] Приложение Strelka™, от компании Illumina Inc., размещенная по адресу https://github.com/Illumina/strelka и описанное в статье T Saunders, Christopher & Wong, Wendy & Swamy, Sajani & Becq, Jennifer & J Murray, Lisa & Cheetham, Keira. (2012). Strelka: Accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinformatics (Oxford, Англия). 28. 1811-7;
[0008] Приложение Strelka2™ , от компании Illumina Inc., размещенное по адресу https://github.com/Illumina/strelka и описанное в статье Kim, S., Scheffler, K., Halpern, A.L., Bekritsky, M.A., Noh, E.,  , M., Chen, X., Beyter, D., Krusche, P., and Saunders, C.T. (2017);
, M., Chen, X., Beyter, D., Krusche, P., and Saunders, C.T. (2017);
[0009] A. van den Oord, S. Dieleman, H. Zen, K. Simonyan, O. Vinyals, A. Graves, N. Kalchbrenner, A. Senior, and K. Kavukcuoglu, “WAVENET: A GENERATIVE MODEL FOR RAW AUDIO”, arXiv:1609.03499, 2016;
[0010] S.  . Arik, M. Chrzanowski, A. Coates, G. Diamos, A. Gibiansky, Y. Kang, X. Li, J. Miller, A. Ng, J. Raiman, S. Sengupta, M. Shoeybi, “DEEP VOICE: REAL-TIME NEURAL TEXT-TO-SPEECH”, arXiv:1702.07825, 2017;
. Arik, M. Chrzanowski, A. Coates, G. Diamos, A. Gibiansky, Y. Kang, X. Li, J. Miller, A. Ng, J. Raiman, S. Sengupta, M. Shoeybi, “DEEP VOICE: REAL-TIME NEURAL TEXT-TO-SPEECH”, arXiv:1702.07825, 2017;
[0011] F. Yu and V. Koltun, “MULTI-SCALE CONTEXT AGGREGATION BY DILATED CONVOLUTIONS”, arXiv:1511.07122, 2016;
[0012] K. He, X. Zhang, S. Ren, J. Sun, “DEEP RESIDUAL LEARNING FOR IMAGE RECOGNITION”, arXiv:1512.03385, 2015;
[0013] R.K. Srivastava, K. Greff, J. Schmidhuber, “HIGHWAY NETWORKS”, arXiv: 1505.00387, 2015;
[0014] G. Huang, Z. Liu, L. van der Maaten, K. Q. Weinberger, “DENSELY CONNECTED CONVOLUTIONAL NETWORKS”, arXiv:1608.06993, 2017;
[0015] C. Szegedy, W. Liu,Y. Jia, P. Sermanet, S. Reed, D. Anguelov, D. Erhan, V. Vanhoucke, A. Rabinovich, “GOING DEEPER WITH CONVOLUTIONS”, arXiv: 1409.4842, 2014;
[0016] S. Ioffe, C. Szegedy, “BATCH NORMALIZATION: ACCELERATING DEEP NETWORK TRAINING BY REDUCING INTERNAL COVARIATE SHIFT”, arXiv: 1502.03167, 2015;
[0017] Srivastava, Nitish, Hinton, Geoffrey, Krizhevsky, Alex, Sutskever, Ilya, and Salakhutdinov, Ruslan, “DROPOUT: A SIMPLE WAY TO PREVENT NEURAL NETWORKS FROM OVERFITTING”, The Journal of Machine Learning Research, 15 (1):1929-1958, 2014;
[0018] J. M. Wolterink, T. Leiner, M. A. Viergever, I.  , “DILATED CONVOLUTIONAL NEURAL NETWORKS FOR CARDIOVASCULAR MR SEGMENTATION IN CONGENITAL HEART DISEASE”, arXiv:1704.03669, 2017;
, “DILATED CONVOLUTIONAL NEURAL NETWORKS FOR CARDIOVASCULAR MR SEGMENTATION IN CONGENITAL HEART DISEASE”, arXiv:1704.03669, 2017;
[0019] L. C. Piqueras, “AUTOREGRESSIVE MODEL BASED ON A DEEP CONVOLUTIONAL NEURAL NETWORK FOR AUDIO GENERATION”, Tampere University of Technology (Технологиеский университет Тампере), 2016;
[0020] J. Wu, “Introduction to Convolutional Neural Networks”, Nanjing University (Нанкинский университет), 2017;
[0021] I. J. Goodfellow, D. Warde-Farley, M. Mirza, A. Courville, and Y. Bengio, “CONVOLUTIONAL NETWORKS”, Deep Learning, MIT Press, 2016;
[0022] J. Gu, Z. Wang, J. Kuen, L. Ma, A. Shahroudy, B. Shuai, T. Liu, X. Wang, G. Wang, “RECENT ADVANCES IN CONVOLUTIONAL NEURAL NETWORKS”, arXiv:1512.07108, 2017;
[0023] M. Lin, Q. Chen, and S. Yan, “Network in Network”, в Proc. of ICLR, 2014;
[0024] L. Sifre, “Rigid-motion Scattering for Image Classification, Ph.D. thesis, 2014;
[0025] L. Sifre and S. Mallat, “Rotation, Scaling and Deformation Invariant Scattering for Texture Discrimination”, в Proc. of CVPR, 2013;
[0026] F. Chollet, “Xception: Deep Learning with Depthwise Separable Convolutions”, в Proc. of CVPR, 2017;
[0027] X. Zhang, X. Zhou, M. Lin, and J. Sun, “ShuffleNet: An Extremely Efficient Convolutional Neural Network for Mobile Devices”, в arXiv:1707.01083, 2017;
[0028] K. He, X. Zhang, S. Ren, J. Sun, “Deep Residual Learning for Image Recognition”, в Proc. of CVPR, 2016;
[0029] S. Xie, R. Girshick, P. Dollár, Z. Tu, and K. He, “Aggregated Residual Transformations for Deep Neural Networks”, в Proc. of CVPR, 2017;
[0030] A. G. Howard, M. Zhu, B. Chen, D. Kalenichenko, W. Wang, T. Weyand, M. Andreetto, and H. Adam, “Mobilenets: Efficient Convolutional Neural Networks for Mobile Vision Applications”, в arXiv:1704.04861, 2017;
[0031] M. Sandler, A. Howard, M. Zhu, A. Zhmoginov, and L. Chen, “MobileNetV2: Inverted Residuals and Linear Bottlenecks”, в arXiv:1801.04381v3, 2018;
[0032] Z. Qin, Z. Zhang, X. Chen, and Y. Peng, “FD-MobileNet: Improved MobileNet with a Fast Downsampling Strategy”, в arXiv:1802.03750, 2018;
[0033] Международная патентная заявка РСТ № PCT/US17/61554, озаглавленная “Validation Methods and Systems for Sequence Variant Calls”, поданная 14 ноября 2017 г.;
[0034] Предварительная заявка на патент США 62/447,076, озаглавленная “Validation Methods and Systems for Sequence Variant Calls”, поданная 17 января 2017 г.;
[0035] Предварительная заявка на патент США 62/422,841, озаглавленная “Methods and Systems to Improve Accuracy in Variant Calling”, поданная 16 января 2016 г.; и
[0036] N. ten DIJKE, “Convolutional Neural Networks for Regulatory Genomics”, Диссертация на соискание магистерской степени, Universiteit Leiden Opleiding Informatica, 17 июня 2017 г.
ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ РАСКРЫТАЯ ТЕХНОЛОГИЯ
[0037] Раскрытая технология относится к компьютерам и цифровым системам обработки данных, относящихся к типу искусственного интеллекта, и соответствующим способам обработки данных и продуктам для эмуляции интеллекта (т.е. системам, основанным на знаниях, системам построения рассуждений и системам приобретения знаний); включая системы для логических рассуждений в условиях неопределенности (например, системы нечеткой логики), адаптивным системам, системам машинного обучения и искусственным нейронным сетям. В частности, раскрытая технология относится к применению глубоких нейронных сетей, таких как глубокие сверточные нейронные сети (CNN) и полностью связанные нейронные сети (FCNN), для анализа данных.
УРОВЕНЬ ТЕХНИКИ
[0038] Не следует полагать, что аспекты, обсуждаемые в этом разделе, составляют часть уровня техники только потому, что они упоминаются в этом разделе. Аналогичным образом, не следует полагать, что задача, упоминающаяся в этом разделе или связанная с объектом, указанным в качестве предпосылки, является признанным уровнем техники. Предмет этого раздела лишь представляет различные подходы, которые сами по себе также могут соответствовать вариантам реализации заявленной технологии.
[0039] Секвенирование следующего поколения сделало большое количество данных секвенирования доступным для фильтрации вариантов. Данные секвенирования высоко коррелированы и имеют сложные взаимные зависимости, что затрудняло применение традиционных классификаторов, таких как машина опорных векторов, для задачи фильтрации вариантов. Соответственно, существует потребность в более совершенных классификаторах, способных извлекать высокоуровневые признаки из секвенированных данных.
[0040] Глубокие нейросети - это тип искусственных нейронных сетей, которые используют множественные нелинейные и сложные преобразующие слои, чтобы последовательно моделировать высокоуровневые признаки. Глубокие нейросети обеспечивают обратную связь посредством алгоритма обратного распространения, который несет информацию о разнице между наблюдаемыми и ожидаемыми выходными данными, с целью коррекции параметров. Глубокие нейронные сети развивались по мере того, как становились доступны большие объемы данных для обучения, мощности параллельных и распределенных вычислений, и развитые алгоритмы обучения. Глубинные нейросети способствовали существенному развитию в множестве областей, таких как компьютерное зрение, распознавание речи и обработка естественных языков.
[0041] Сверточные нейронные сети (CNN) и рекуррентные нейронные сети (RNN) являются компонентами нейронных сетей глубокого обучения (глубоких нейронных сетей). Сверточные нейронные сети особенно успешно выполняют задачи по распознаванию образов и имеют архитектуру, которая включает слои свертки, нелинейные слои, слои пулинга (объединения). Рекуррентные нейронные сети созданы для использования последовательных входных данных с циклическими связями между строительными блоками, перцептронами, единицами долгосрочной и краткосрочной памяти, и управляемые рекуррентные блоки. В дополнение было предложено много других новейших нейросетей глубокого обучения для ограниченных контекстов, например глубокие пространственно-временные нейронные сети, многомерные рекуррентные нейронные сети, и сверточные автоэнкодеры.
[0042] Цель обучения глубоких нейронных сетей заключается в оптимизации веса параметров в каждом слое, который постепенно комбинирует более простые признаки в сложные, что позволяет получить из данных наиболее подходящие иерархические представления. Отдельный цикл процесса оптимизации организован следующим образом. Сначала, на тренировочном (обучающем) наборе данных, прямой проход алгоритма последовательно вычисляет выходные данные в каждом слое, и распространяет сигналы функции вперед по сети. В конечном выходном слое (слое выходных данных), целевая функция потерь измеряет погрешность между выходными данными работы обученной нейронные сети и данными метками. Для минимизации ошибок обучения, при обратном проходе используется правило сложной производной (цепное правило) для обратного распространения сигналов ошибки и вычисления градиентов по всем весам по всей нейронные сети. В конце весовые параметры обновляются посредством алгоритмов оптимизации, основанных на стохастическом градиентном спуске. В то время как градиентный спуск осуществляет обновление параметров для каждого полного набора данных, стохастический градиентный спуск обеспечивает стохастическую аппроксимацию, проводя обновление для каждого небольшого набора семплированных данных (данных в выборке). На принципе стохастического градиентного спуска основаны несколько алгоритмов оптимизации. Например, обучающий алгоритм Адаграда и Адама проводит стохастический градиентный спуск с адаптивным изменением скорости обучения на основе частоты обновления моментов градиентов для каждого параметра, соответственно.
[0043] Другим базовым элементом обучения глубокой нейронной сети является регуляризация, понятие, относящееся к стратегиям, направленным на то, чтобы избежать переобучения нейронные сети, и таким образом добиться хорошей производительности генерализации. Например, сокращение весов добавляет штрафные слагаемые к целевой функции потерь, так что весовые параметры сходятся к меньшим абсолютным значениям. Метод исключения (отсев, dropout) случайным образом убирает скрытые узлы из нейронной сети во время обучения, и может рассматриваться как ансамбль возможных подсетей. Чтобы улучшить возможности метода исключения, была предложена новая функция активации, maxout, и определен вариант метода исключения для рекуррентных нейросетей - rnnDrop. Кроме того, пакетная нормализация обеспечивает новый метод регуляризации посредством нормализации скалярных признаков для каждой активации внутри мини-пакета с обучением каждого среднего и отклонения в качестве параметров.
[0044] Кроме того, пакетная нормализация обеспечивает новый метод регуляризации посредством нормализации скалярных признаков для каждой активации внутри мини-пакета с обучением каждого среднего и отклонения в качестве параметров. Сверточные нейронные сети адаптированы для решения задач геномики, основанных на последовательностях, таких как обнаружение мотива, идентификация патогенных вариантов и исследование экспрессии генов. Характерной чертой сверточных нейросетей является использование сверточных фильтров. отличие от традиционных подходов к классификации, которые основаны на признаках, полученных в результате тщательной ручной работы, сверточные фильтры проводят адаптивное обучение признаков, аналогично процессу картирования необработанных входных данных на информативное представление знаний. В этом смысле, сверточные фильтры служат серией сканеров мотивов, поскольку набор таких фильтров способен опознать релевантные паттерны во входных данных, и адаптироваться в процессе обучения. Рекуррентные нейронные сети могут регистрировать дальномерные зависимости в последовательных данных различной длины, таких как белковые последовательности или ДНК.
[0045] Соответственно, возникает возможность применять такой фреймворк на основе глубокого обучения, который связывает паттерны последовательностей с ошибками секвенирования.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
[0046] На чертежах одинаковые ссылочные позиции обычно относятся к одинаковым деталям на разных видах. Кроме того, чертежи не обязательно выполнены в масштабе, вместо этого, как правило, делается акцент на иллюстрации принципов раскрытой технологии. В последующем описании различные реализации раскрытой технологии описаны со ссылкой на следующие чертежи, на которых:
[0047] ФИГ. 1 представляет собой блок-схему, которая показывает различные аспекты DeepPOLY, фреймворка на основе глубокого обучения для идентификации паттернов последовательности, которые вызывают последовательность-специфичные ошибки (SSEs). ФИГ. 1 включает модули, такие как фильтр вариантов, симулятор и анализатор. ФИГ. 1 также включает базы данных, которые хранят перекрывающиеся образцы, нуклеотидные последовательности и паттерны повторов.
[0048] ФИГ. 2 иллюстрирует пример архитектуры фильтра вариантов. Фильтр вариантов имеет иерархическую структуру, построенную на сверточной нейронной сети (CNN) и полностью связанной нейронной сети (FCNN). DeepPOLY использует фильтр вариантов для исследования влияния известных паттернов последовательности на фильтрацию вариантов.
[0049] ФИГ. 3 демонстрирует один вариант реализации пайплайна обработки фильтра вариантов.
[0050] На ФИГ. 4A показаны истинно и ложно положительные графики, которые графически иллюстрируют работу фильтра вариантов на отложенных данных.
[0051] ФИГ. 4B и 4C показывают наложенные изображения выровненных ридов, которые валидируют точность фильтра вариантов.
[0052] ФИГ. 5 демонстрирует один вариант реализации кодирования с одним активным состояние для кодирования перекрывающегося образца, который содержит определенный вариант в целевом положении, фланкированный 20-50 основаниями с каждой стороны.
[0053] ФИГ. 6 иллюстрирует примеры перекрывающихся образцов, образованных блоком подготовки входных данных путем накладывания повторяющихся паттернов (т.е. паттернов повторов) на нуклеотидные последовательности.
[0054] ФИГ. 7A применяет график «ящик с усами» для идентификации того, что ошибки обусловлены повторяющимися паттернами слева от вариантных нуклеотидов в целевом положении перекрытых образцов.
[0055] ФИГ. 7B применяет график «ящик с усами» для идентификации того, что последовательность-специфичных ошибки обуславливаются повторяющимися паттернами справа от вариантных нуклеотидов в целевом положении перекрытых образцов.
[0056] ФИГ. 7C применяет график «ящик с усами» для идентификации того, что последовательность-специфичных ошибки обуславливаются повторяющимися паттернами, включая вариантный нуклеотид в целевом положении перекрытых образцов.
[0057] ФИГ. 8A применяет график «ящик с усами» для идентификации того, что последовательность-специфичных ошибки обуславливаются повторяющимися паттернами гомополимеров одного основания “C”, перекрытых в различных участках нуклеотидной последовательности.
[0058] ФИГ. 8B применяет график «ящик с усами» для идентификации того, что последовательность-специфичных ошибки обуславливаются повторяющимися паттернами гомополимеров единственного основания “G” в различных участках нуклеотидной последовательности.
[0059] ФИГ. 8C применяет график «ящик с усами» для идентификации того, что последовательность-специфичных ошибки обуславливаются повторяющимися паттернами гомополимеров единственного основания “A” в различных участках нуклеотидной последовательности.
[0060] ФИГ. 8D применяет график «ящик с усами» для идентификации того, что последовательность-специфичных ошибки обуславливаются повторяющимися паттернами гомополимеров единственного основания “T” в различных участках нуклеотидной последовательности.
[0061] ФИГ. 9 отображает оценки классификации как распределение вероятности того, что вариантный нуклеотид является истинным вариантом или ложным вариантом, если повторяющиеся паттерны гомополимеров единственного основания размещены по одному “до” и “после” вариантного нуклеотида каждого из четырех оснований в целевом положении.
[0062] ФИГ. 10A - 10C отображают представление природных повторяющихся паттернов сопорлимеров в каждой нуклеотидной последовательности образца, которая вносит вклад в классификацию ложных вариантов.
[0063] ФИГ. 11 представляет собой упрощенную блок-схему компьютерной системы, которую можно применять для реализации фильтра вариантов.
[0064] ФИГ. 12 иллюстрирует один вариант реализации того как последовательность - специфичные ошибки (SSE) коррелируют с повторяющимися паттернами на основании классификации ложных вариантов.
ПОДРОБНОЕ ОПИСАНИЕ
[0065] Следующее обсуждение представлено для того, чтобы дать возможность любому специалисту в данной области техники создавать и применять раскрытую технологию, и предоставляется в контексте конкретного приложения и его требований. Различные модификации раскрытых вариантов реализации будут очевидны для специалистов в данной области техники, и общие принципы, определенные в данном документе, могут быть применены к другим вариантам реализации и приложениям без отступления от сущности и объема раскрытой технологии. Таким образом, не предполагается, что раскрытая технология ограничена показанными вариантами реализации, но должна соответствовать самому широкому объему, согласующемуся с принципами и признаками, раскрытыми в данном документе.
Введение
[0066] Последовательность-специфичные ошибки (SSE), представляют собой ошибки определения оснований, вызванные конкретными паттернами последовательности. Например, было обнаружено, что паттерны последовательностей «GGC» и «GGCNG» и их инвертированные повторы вызывают большое количество ошибочных определений. SSE приводят к пробелам в сборке и артефактам выравнивания. Кроме того, поскольку любое ошибочное определение может быть ошибочно принято за вариант, SSE приводят к ложным определениям вариантов и являются основным препятствием для точного определения варианта.
[0067] Мы раскрываем фреймворк на основе глубокого обучения DeepPOLY, который определяет паттерны последовательностей, вызывающие SSE. DeepPOLY обучает фильтр вариантов на крупномасштабных данных вариантов, чтобы узнать причинные зависимости между паттернами последовательности и ложными определениями вариантов. Фильтр вариантов имеет иерархическую структуру, построенную на глубоких нейронных сетях, которые оценивают входную последовательность в нескольких пространственных масштабах и выполняют фильтрацию вариантов, то есть предсказывают, является ли определенный вариант во входной последовательности истинным определением варианта или ложным определением варианта. Крупномасштабные данные вариантов включают варианты родословной, из которых наследуемые варианты применяются в качестве обучающих примеров истинных определений вариантов, а варианты de novo, наблюдаемые только у одного ребенка, используются в качестве обучающих примеров ложных определений вариантов. В некоторых вариантах реализации по меньшей мере некоторые из вариантов de novo, наблюдаемых только у одного ребенка, используются в качестве обучающих примеров истинных определений вариантов.
[0068] Во время обучения параметры глубоких нейронных сетей оптимизируются для максимальной точности фильтрации с использованием подхода градиентного спуска. Результирующий фильтр вариантов учится связывать ложные определения вариантов с паттернами последовательностей во входных последовательностях.
[0069] DeepPOLY затем реализует моделирование, которое использует фильтр вариантов для тестирования известных паттернов последовательности на предмет их влияния на фильтрацию вариантов. Известные паттерны последовательности представляют собой повторяющиеся паттерны (или сополимеры), которые различаются по составу оснований, длине паттерна и коэффициенту повтора. Повторяющиеся паттерны тестируются при различных отклонениях от определенных вариантов.
[0070] Предпосылка моделирования заключается в следующем: когда пара тестируемого повторяющегося паттерна и определенного варианта подается в фильтр вариантов как часть смоделированной входной последовательности, и фильтр вариантов классифицирует определенный вариант как ложный вариант, считается, что повторяющийся паттерн вызвал ложное определение варианта и идентифицирован как вызывающий SSE. Исходя из этого, DeepPOLY тестирует сотни и тысячи повторяющихся паттернов, чтобы определить, какие из них вызывают SSE, с чувствительностью к смещению.
[0071] DeepPOLY также обнаруживает природные паттерны последовательностей, которые вызывают SSE, путем обработки природных входных последовательностей через фильтр вариантов и анализа активаций параметров глубоких нейронных сетей во время обработки. Эти паттерны последовательности идентифицируются как вызывающие SSE, для которых входные нейроны глубоких нейронных сетей производят активацию самых высоких параметров, а выходные нейроны производят классификацию ложного определения вызова.
[0072] DeepPOLY подтверждает ранее известные паттерны последовательностей, вызывающих SSE, и сообщает о новых, более специфичных.
[0073] DeepPOLY не зависит от лежащей в основе химии секвенирования, платформы секвенирования и полимераз секвенирования и может создавать исчерпывающие профили паттернов последовательностей, вызывающих SSE, для различных химических реагентов секвенирования, платформ секвенирования и полимераз секвенирования. Эти профили можно применять для улучшения химического состава секвенирования, создания платформ для секвенирования более высокого качества и создания различных полимераз для секвенирования. Их также можно использовать для пересчета оценок качества определения оснований и повышения точности определения вариантов.
[0074] Фильтр вариантов имеет две глубокие нейронные сети: сверточную нейронную сеть (CNN), за которой следует полностью связанная нейронная сеть (FCNN). Тестируемый повторяющийся паттерн накладывается на нуклеотидную последовательность для получения перекрытого (наложенного) образца. Перекрытый образец имеет определенный вариант в целевом положении, окруженном 20-50 основаниями с каждой стороны. Мы рассматриваем перекрытый образец как изображение с несколькими каналами, которые численно кодируют четыре типа оснований, A, C, G и T. Перекрытый образец, покрывающий определенный вариант, кодируется с одним активным положением для сохранения информации, зависящей от положения. каждого отдельного основания в перекрытом образце.
[0075] Сверточная нейронная сеть принимает перекрытый с одним активным положением образец, потому что он способен сохранять отношения пространственного расположения в перекрытом образце. Сверточная нейронная сеть обрабатывает перекрытый образец несколькими сверточными слоями и создает один или несколько промежуточных свернутых признаков. Слои свертки используют фильтры свертки для идентификации паттернов последовательности в перекрытом образце. Сверточные фильтры действуют как детекторы мотивов, которые сканируют перекрытый образец на предмет низкоуровневых мотивов и создают сигналы разной силы в зависимости от лежащих в основе паттернов последовательности. Фильтры свертки автоматически обучаются после обучения на сотнях и тысячах обучающих примеров истинных и ложных определений вариантов.
[0076] Полносвязанная (полностью связанная, полностью соединенная) нейронная сеть затем обрабатывает промежуточные свернутые признаки через несколько полносвязных слоев. Плотно связанные нейроны полностью связанных слоев обнаруживают высокоуровневые паттерны последовательностей, закодированные в свернутых элементах. Наконец, слой классификации полностью связанной нейронной сети выводит вероятности того, что определенный вариант является истинным определением варианта или ложным определением варианта.
[0077] В дополнение к применению исключения, пары пакетной нормализации и нелинейности блока линейной ректификации расположены между сверточными слоями и полностью связными слоями для повышения скорости обучения и снижения переобучения.
Терминология
[0078] Все литературные источники и аналогичный материал, цитируемый в настоящей заявке, в том числе, но не ограничиваясь перечисленным, патенты, патентные заявки, статьи, книги, научные работы и веб-страницы, независимо от формата таких литературных источников и аналогичных материалов, явным образом и полностью включены в настоящий документ посредством ссылок. В тех случаях, когда один или более из включенных литературных источников и аналогичных материалов отличается от настоящей заявки или противоречит ей, в том числе, но не ограничиваясь перечисленным, определяемые термины, силу будет иметь настоящая заявка.
[0079] В настоящем документе следующие термины имеют указанные значения.
[0080] Основание относится к нуклеотидному основанию или нуклеотиду, A (аденину), C (цитозину), T (тимину) или G (гуанину).
[0081] Термин «хромосома» относится к носителю генов, передающих наследственные признаки, в живой клетке, происходящему из нитей хроматина, содержащих ДНК и белковые компоненты (в частности, гистоны). В настоящем документе используется стандартная международно признанная система нумерации индивидуальных хромосом генома человека.
[0082] Термин «сайт» относится к уникальному положению (например, идентификатору хромосомы, положению и ориентации хромосомы) на референсном геноме. В некоторых вариантах реализации сайт может представлять собой остаток, метку последовательности или положение сегмента в последовательности. Термин «локус» может применяться для обозначения специфической локализации последовательности нуклеиновой кислоты или полиморфизма на референсной хромосоме.
[0083] Термин «образец» в настоящем документе относится к образцу, как правило, происходящему из биологической жидкости, клетки, ткани, органа или организма, содержащего нуклеиновую кислоту или смесь нуклеиновых кислот, содержащую по меньшей мере одну последовательность нуклеиновой кислоты, подлежащую секвенированию и/или фазированию. Такие образцы включают, не ограничиваясь перечисленными, образцы мокроты/жидкости ротовой полости, амниотической жидкости, крови, фракции крови, тонкоигольной биопсии (например, хирургической биопсии, тонкоигольной биопсии и т.п.),мочи, жидкости брюшной полости, плевральной жидкости, эксплантата ткани, культуры органа и любого другого препарата ткани или клеток, или его фракции или производного, или выделенные из них образцы. Хотя образец часто получают от субъекта-человека (например, пациента), образцы могут быть взяты из любого организма, имеющего хромосомы, в том числе, но не ограничиваясь перечисленными, организма собак, кошек, лошадей, коз, овец, крупного рогатого скота, свиней и т.п. Образец может применяться непосредственно в полученном из биологического источника виде или после предварительной обработки для модификации характера образца. Например, такая предварительная обработка может включать получение плазмы из крови, разведение вязких текучих сред и т.д. Методы предварительной обработки могут также включать, не ограничиваясь перечисленными, фильтрацию, осаждение, разведение, дистилляцию, смешивание, центрифугирование, замораживание, лиофилизацию, концентрацию, амплификацию, фрагментацию нуклеиновых кислот, инактивацию мешающих компонентов, добавление реагентов, лизис и т.п.
[0084] Термин «последовательность» включает или обозначает цепь взаимно сопряженных нуклеотидов. Нуклеотиды могут быть основаны на ДНК или РНК. Следует понимать, что одна последовательность может включать несколько субпоследовательностей. Например, одна последовательность (например, ПЦР-ампликона) может содержать 350 нуклеотидов. Рид образца может включать несколько субпоследовательностей в пределах указанных 350 нуклеотидов. Например, рид образца может включать первую и вторую фланкирующие субпоследовательности, содержащие, например, 20-50 нуклеотидов. Указанные первая и вторая фланкирующие субпоследовательности могут быть локализованы на любой стороне повторяющегося сегмента, содержащего соответствующую субпоследовательность (например, 40-100 нуклеотидов). Каждая из фланкирующих субпоследовательностей может включать (или включать частично) субпоследовательность праймера (например, 10-30 нуклеотидов). Для простоты чтения вместо термина «субпоследовательность» используют «последовательность», но следует понимать, что две последовательности не обязательно отделены одна от другой на общей цепи. Для различения различных последовательностей, описанных в настоящем документе, в указанные последовательности могут быть включены разные метки (например, целевая последовательность, праймерная последовательность, фланкирующая последовательность, референсная последовательность и т.п.). В другие объекты, такие как описываемые термином «аллель», могут быть включены разные метки для дифференциации сходных объектов.
[0085] Термин «парно-концевое секвенирование» относится к способам секвенирования с секвенированием обоих концов целевого фрагмента. Парно-концевое секвенирование может облегчать детекцию геномных перестановок и повторяющихся сегментов, а также слитых генов и новых транскриптов. Методология парно-концевого секвенирования описана в PCT-публикации WO07010252, PCT-публикации сер. № PCTGB2007/003798 и опубликованной заявке на патент США US 2009/0088327, каждая из которых включена посредством ссылки в настоящий документ. Согласно одному примеру может быть выполнен следующий ряд операций; (a) генерация кластеров нуклеиновых кислот; (b) линеаризация указанных нуклеиновых кислот; (c) гибридизация первого праймера для секвенирования и проведение многократных циклов удлинения, сканирования и деблокирования согласно описанию выше; (d) “инверсия» целевых нуклеиновых кислот на поверхности проточной ячейки путем синтеза комплементарной копии; (e) линеаризация ресинтезированной цепи; и (f) гибридизация второго праймера для секвенирования и проведения многократных циклов удлинения, сканирования и деблокирования согласно описанию выше. Операция инверсии может быть проведена с доставкой реагентов согласно описанию выше для одного цикла мостиковой амплификации.
[0086] Термин «референсный геном» или «референсная последовательность» относится к любой конкретной известной последовательности генома, частичной или полной, любого организма, которая может быть использована в качестве референсной для идентифицированных последовательностей субъекта. Например, референсный геном, используемый для субъектов-людей, а также многих других организмов можно найти по ссылке ncbi.nlm.nih.gov от Национального центра биотехнологической информации. “Геном» относится к полной генетической информации организма или вируса, представленной в виде последовательностей нуклеиновых кислот. Геном включает как гены, так и некодирующие последовательности ДНК. Референсная последовательность может быть длиннее ридов, которые на нее выравнивают. Например, она может быть по меньшей мере приблизительно в 100 раз длиннее, или по меньшей мере приблизительно в 1000 раз длиннее, или по меньшей мере приблизительно в 10 000 раз длиннее, или по меньшей мере приблизительно в 105 раз длиннее, или по меньшей мере приблизительно 106 раз длиннее, или по меньшей мере приблизительно в 107 раз длиннее. В одном примере референсная последовательность генома представляет собой последовательность полноразмерного генома человека. В другом примере референсная последовательность генома ограничена специфической хромосомой человека, такой как хромосома 13. В некоторых вариантах реализации референсная хромосома представляет собой последовательность хромосомы из генома человек версии hg19. Такие последовательности могут называться референсными последовательностями хромосомы, хотя предполагается, что термин «референсный геном» охватывает такие последовательности. Другие примеры референсных последовательностей включают геномы других видов, а также хромосом, субхромосомных областей (например, цепей) и т.п., любых видов. В различных вариантах реализации референсный геном представляет собой консенсусную последовательность или другую комбинацию, полученную от нескольких индивидуумов. Однако в определенных вариантах применения референсная последовательность может быть получена от конкретного индивидуума.
[0087] Термин “рид” относится к набору данных о последовательности, который описывает нуклеотидный образец или референс (эталон). Термин «рид» может относиться к риду образца и/или референсному риду. Обычно, хотя не обязательно, рид представлен короткой последовательностью непрерывно расположенных пар оснований в образце или референсной последовательности. Рид может быть символически представлен последовательностью пар оснований (ATCG) образца или референсного фрагмента. Он может храниться в запоминающем устройстве и обрабатываться подходящим образом для определения того, совпадает ли рид с референсной последовательностью или отвечает ли другим критериям. Рид может быть получен непосредственно из аппарата для секвенирования или непрямо, из сохраненной информации о последовательности, касающейся указанного образца. В некоторых случаях рид представляет собой последовательность ДНК достаточной длины (например, по меньшей мере приблизительно 25 п.о.) которые могут применяться для идентификации последовательности или области большей длины, например, например, которая может быть выравнена и специфическим образом соотнесена с хромосомой, или геномной областью, или генов.
[0088] Методы секвенирования следующего поколения включают, например, технологию секвенирования путем синтеза (Illumina), пиросеквенирование (454), технологию ионного полупроводникового секвенирования (секвенирование Ion Torrent), одномолекулярное секвенирование в реальном времени (Pacific Biosciences) и секвенирование путем лигирования (секвенирование SOLiD). В зависимости от методов секвенирования длина каждого рида может варьировать от приблизительно 30 п.о. до более 10 000 п.о. Например, метод секвенирования Illumina с использованием секвенатора SOLiD генерирует риды нуклеиновых кислот длиной приблизительно 50 п.о. В другом примере секвенирование Ion Torrent генерирует риды нуклеиновых кислот длиной до 400 п.о., а пиросеквенирование 454 генерирует риды нуклеиновых кислот длиной приблизительно 700 п.о. В еще одном примере способы одномолекулярного секвенирования в реальном времени могут генерировать риды длиной от 10 000 п.о. до 15 000 п.о. Соответственно, в определенных вариантах реализации риды последовательностей нуклеиновых кислот имеют длину 30-100 п.о., 50-200 п.о. или 50-400 п.о.
[0089] Термины “рид образца», «последовательность образца» или «фрагмент образца» относятся к данным представляющей интерес геномной последовательности из образца. Например, рид образца содержит данные о последовательности из ПЦР-ампликона, содержащего последовательности прямого и обратного праймера. Данные о последовательности могут быть получены с применением любого выбранного метода секвенирования. Рид образца может быть получен, например, в результате реакции секвенирования путем синтеза (SBS), реакции секвенирования путем лигирования или любого другого подходящего метода секвенирования, для которого требуется определение длины и/или идентичности повторяющегося элемента. Рид образца может представлять собой консенсусную (например, усредненную или взвешенную) последовательность, полученную из нескольких ридов образца. В некоторых вариантах реализации получение референсной последовательности включает идентификацию представляющего интерес локуса на основании последовательности праймера из ПЦР-ампликона.
[0090] Термин “необработанный фрагмент» относится к данным о последовательности части представляющей интерес геномной последовательности, которая по меньшей мере частично перекрывает заданное положение или представляющее интерес вторичное положение в риде образца или фрагменте образца. Неограничивающие примеры необработанных фрагментов включают дуплексный фрагмент со сшивкой, симплексный фрагмент со сшивкой, дуплексный фрагмент без сшивки и симплексный фрагмент без сшивки. Термин “необработанный» используют, чтобы показать, что необработанный фрагмент включает данные о последовательности, определенным образом связанные с данными о последовательности в риде образца, независимо от того, демонстрирует ли необработанный фрагмент подтверждающий вариант, который соответствует и удостоверяет или подтверждает потенциальный вариант в риде образца. Термин “необработанный фрагмент» не указывает на то, что указанный фрагмент обязательно включает подтверждающий вариант, валидирующий распознанный вариант в риде образца. Например, если приложением для распознавания вариантов определено, что рид образца демонстрирует первый вариант, указанное приложение для распознавания вариантов может определить, что в одном или более необработанных фрагментах отсутствует соответствующий тип «подтверждающего» варианта, наличие которого в ином случае можно ожидать на основании варианта в риде образца.
[0091] Термины “картирование», «выравненный», «выравнивание» относятся к процессу сравнения рида или метки с референсной последовательностью, с определением таким образом того, содержит ли указанная референсная последовательность содержит последовательность рида. Если референсная последовательность содержит рид, указанный рид может быть картирован на указанную референсную последовательность или, в определенных вариантах реализации, на конкретное место в референсной последовательности. В некоторых случаях выравнивание просто показывает, входит ли рид в состав конкретной референсной последовательности (т.е. присутствует или отсутствует указанный рид в референсной последовательности). Например, выравнивание рида на референсную последовательность хромосомы 13 человека показывает, присутствует ли указанный рид в указанной референсной последовательности хромосомы 13. Инструмент, который обеспечивает получение указанной информации, может называться тестировщиком принадлежности множеству. В некоторых случаях выравнивание, кроме того, указывает на место в референсной последовательности, куда картируется рид или метка. Например, если референсная последовательность представляет собой полную последовательность генома человека, выравнивание может показать, что рид присутствует на хромосоме 13, и может дополнительно показать, что рид располагается в конкретной цепи и/или сайте хромосомы 13.
[0092] Термин “индел» относится к инсерции и/или делеции оснований в ДНК организма. Микроиндел представляет собой индел, который приводит к чистому изменению 1-50 нуклеотидов. В кодирующих областях генома, за исключением случаев, когда длина индела кратна 3, он дает мутацию со сдвигом рамки. Инделы могут быть противопоставлены точечным мутациям. Индел инсертирует и делетирует нуклеотиды в последовательности, тогда как точечная мутация представляет собой форму замены, при которой один из нуклеотидов заменяют без изменения общего числа в ДНК. Инделы могут также быть противопоставлены тандемной мутации оснований (TBM), которая может быть определена как замена нуклеотидов в смежных положениях (“вариант» относится к последовательности нуклеиновой кислоты, отличающейся от референсной нуклеиновой кислоты.
[0093] Термин “вариант” относится к нуклеиновой кислоте, которая отличается от референсной нуклеиновой кислоты. Типичный вариант последовательности нуклеиновой кислоты включает, без ограничения, однонуклеотидный полиморфизм (SNP), короткие делеционные и инсерционные полиморфизмы (индел), вариацию числа копий (CNV), микросателлитные маркеры или короткие тандемные повторы, и структурную вариацию. Распознавание соматических вариантов представляет собой попытку идентификации вариантов, присутствующих в образце ДНК с низкой частотой. Распознавание соматических вариантов представляет интерес в контексте лечения рака. Образец ДНК из опухоли обычно являются гетерогенным и включает некоторое число нормальных клеток, некоторое число клеток ранней стадии прогрессирования рака (с меньшим количеством мутаций) и некоторое число клеток поздней стадии (с большим количеством мутаций). Из-за указанной гетерогенности при секвенировании опухоли (например, из фиксированного формалином и залитого в парафин (FFPE) образца) соматические мутации часто появляется с низкой частотой. Например, однонуклеотидная вариация (SNV) наблюдается только в 10% ридов, захватывающих заданное основание. Вариант, который подлежит классификации как относящийся к соматической или зародышевой линии классификатором вариантов, также называется в настоящем документе «тестируемым вариантом».
[0094] Термин “шум» относится к ошибочно распознанному варианту, полученному в результате одной или более ошибок в процессе секвенирования и/или в приложении для распознавания вариантов.
[0095] Термин “частота варианта» относится к относительной частоте аллеля (варианта гена) в конкретном локусе в популяции, выраженной в виде доли или процента. Например, указанные доля или процент могут быть представлены долей всех хромосом в популяции, несущих указанный аллель. Например, частота варианта в образце представляет собой относительную частоту аллеля/варианта в конкретном локусе/положении вдоль представляющей интерес геномной последовательности в «популяции», соответствующей числу ридов и/или образцов, полученных для указанной представляющей интерес геномной последовательности от индивидуума. В другом примере исходная частота варианта представляет собой относительную частоту аллеля/варианта в конкретном локусе/положении вдоль одной или более исходных геномных последовательностей, где «популяция» соответствует числу ридов и/или образцов, полученных для одной или более исходных геномных последовательностей из популяции здоровых индивидуумов.
[0096] Термин “частота варианта аллеля (VAF)» относится к наблюдаемому проценту секвенированных ридов, совпадающих с указанным вариантом, разделенному на общее покрытие в целевом положении. VAF представляет собой показатель пропорции секвенированных ридов, несущих указанный вариант.
[0097] Термины положение», «заданное положение» и «локус» относятся к месту или координатам одного или более нуклеотидов в составе последовательности нуклеотидов. Термины “положение», «заданное положение» и «локус» также относятся к месту или координатам одной или более пар оснований в последовательности нуклеотидов.
[0098] Термин “гаплотип» относится к комбинации аллелей в смежных сайтах на хромосоме, наследуемых вместе. Гаплотип может быть представлен одним локусом, несколькими локусами или всей хромосомой в зависимости от числа событий рекомбинации, произошедших между локусами в определенном наборе локусов, если они вообще происходили.
[0099] Термин “порог» в настоящем документе относится к численному или не-численному значению, которое применяют в качестве значения отсечения для характеризации образца, нуклеиновой кислоты или их части (например, рида). Порог может варьировать на основании результатов эмпирического анализа. Порог можно сравнивать с измеренным или рассчитанным значением для определения того, должен ли источник таких предполагаемых значений быть классифицирован конкретным образом. Выбор порога зависит от уровня доверительности, с которым пользователь желает получить при осуществлении классификации. Порог может быть выбран с конкретной целью (например, для достижения баланса чувствительности и селективности). В настоящем документе порог» указывает на точку, в которой ход анализа может быть изменен, и/или точку, в которой может быть запущено действие. Порог не обязательно должен представлять собой заранее заданное число. Вместо этого порог может представлять собой, например, функцию, основанную на множестве факторов. Порог может быть адаптивно регулируемым с учетом обстоятельств. Кроме того, порог может задавать верхний предел, нижний предел или диапазон между пределами.
[00100] В некоторых вариантах реализации меру или оценку (балл, score), основанная(ый) на данных секвенирования, можно сравнивать с порогом. В настоящем документе термины «мера» или «оценка» могут включать значения или результаты, определенные исходя из данных секвенирования, или могут включать функции, основанные на значениях или результатах, определенных исходя из данных секвенирования. Как и порог, мера или оценка могут быть адаптивно регулироваться с учетом обстоятельств. Например, метрика или оценка может представлять собой нормированное значение. В качестве примера оценки или меры один или более вариантов реализации может задействовать показатели подсчитанных количеств при анализе данных. оценка подсчитанного количества может быть основан на числе ридов образца. Оценка может быть основана на числе ридов образца. Риды образца могут быть подвергнуты одной или более стадий фильтрации, таким образом, чтобы они обладали по меньшей мере одной общей характеристикой или одним общим качеством. Например, каждый из ридов образца, который используют для определения оценки подсчитанного количества, может быть выравнен по референсной последовательности или может быть определен как потенциальный аллель. Может быть подсчитано число ридов образца, обладающих общей характеристикой, для определения подсчитанного количества ридов. Счетные оценки могут быть основаны на подсчитанном количестве ридов. В некоторых вариантах реализации счетная оценка может представлять собой значение, равное подсчитанному количеству ридов. Согласно другим вариантам реализации счетная оценка может быть основана на подсчитанном количестве ридов и другой информации. Например, счетная оценка может быть основана на подсчитанном количестве ридов для конкретного аллеля генетического локуса и общего числа ридов для генетического локуса. В некоторых вариантах реализации счетные оценки могут быть основаны на подсчитанном количестве ридов и ранее полученных данных для генетического локуса. В некоторых вариантах реализации счетные оценки могут представлять собой нормированные показатели между заранее заданными значениями. Счетная оценка может также представлять собой функцию от подсчитанных количеств ридов из других локусов образца или функцию от подсчитанных количеств ридов из других образцов, которые анализировали одновременно с представляющим интерес образцом. Например, счетная оценка может представлять собой функцию от подсчитанного количества ридов конкретного аллеля и подсчитанных количеств ридов других локусов в образце, и/или подсчитанных количества ридов из других образцов. В одном примере подсчитанные количества ридов из других локусов и/или подсчитанные количества ридов из других образцов могут быть использованы для нормирования оценки подсчитанного количества для конкретного аллеля.
[00101] Термины “покрытие» или «покрытие фрагмента» относятся к подсчитанному количеству или другой мере ряда ридов образца для одного и того же фрагмента последовательности. Подсчитанное количество ридов может представлять собой подсчитанное количество ридов, покрывающих соответствующий фрагмент. Как вариант, покрытие может быть определено путем умножения подсчитанного количества ридов на заданный коэффициент, основанный на ретроспективной информации, информации об образце, информации о локусе и т.п.
[00102] Термин “глубина считывания» (обычно в виде числа с последующим символом «×») относится к числу секвенированных ридов, перекрывающихся при выравнивании в целевом положении. Его часто выражают через среднее значение или процент, превышающий значение отсечения на протяжении множества интервалов (таких как экзоны, гены или панели). Например, в клиническом заключении может быть сказано, что среднее покрытие панели составляет 1,105× при 98% покрытии целевых оснований >100×.
[00103] Термины “оценка качества распознавания оснований» или «оценка Q» относятся к вероятности по шкале PHRED в диапазоне от 0-20, обратно пропорциональной вероятности того, что отдельное секвенированное основание является корректным. Например, распознанное основание T с Q, равным 20, считают вероятно корректным с достоверностью, соответствующей P-значению 0,01. Любые распознанные основания с Q<20 должны считаться результатами низкого качества, и любой идентифицированный вариант с существенной пропорцией имеющих низкое качество секвенированных ридов, подтверждающих указанный вариант, должен считаться потенциально ложноположительным.
[00104] Термины «риды вариантов» или «число ридов вариантов» относятся к числу секвенированных ридов, свидетельствующих о присутствии указанного варианта.
DeepPOLY
[00105] Мы описываем DeepPOLY, a фреймворк на основе глубокого обучения для идентификации паттернов последовательности, которые вызывают последовательность-специфичные ошибки (SSEs). Система и процесс описаны со ссылкой на ФИГ. 1. Поскольку ФИГ. 1 представляет собой схему архитектуры, некоторые детали намеренно опущены для ясности описания. Обсуждение ФИГ. 1 организовано следующим образом. Вначале вводятся модули, представленные на фигуре, а затем из взаимные связи. После этого более подробно описано применение модулей.
[00106] ФИГ. 1 включает систему 100. Система 100 включает в себя фильтр вариантов 111 (также называемый здесь подсистемой фильтра вариантов), устройство 161 подготовки входных данных (также называемое здесь подсистемой подготовки входных данных), блок моделирования (симулятор) 116 (также называемый здесь подсистемой моделирования), анализатор 194 (также называемый здесь подсистемой анализа), база данных 196 повторяющихся паттернов, база данных последовательностей 169, база данных 119 наложенных образцов и устройство вывода повторяющихся паттернов 198 (также называемое здесь подсистемой вывода повторяющихся паттернов).
[00107] Блоги обработки и базы данных ФИГ. 1, обозначенные как модули, могут быть реализованы аппаратно или программно, и не обязательно разделены на точно такие же блоки, как показано на ФИГ. 1. Некоторые из модулей также могут быть реализованы на разных процессорах, компьютерах или серверах или распределены между несколькими различными процессорами, компьютерами или серверами. Кроме того, понятно, что некоторые из модулей могут быть объединены, работать параллельно или в другой последовательности, чем показано на ФИГ. 1, без влияния на осуществляемые функции. Модули в ФИГ. 1 можно также рассматривать как этапы блок-схемы этапов способа. Также весь код модуля не обязательно расположен непрерывно в памяти; некоторые части кода могут быть отделены от других частей кода кодом из других модулей или других функций, расположенных между ними.
[00108] Далее описываются взаимосвязи модулей среды 100. Сеть (сети) 114 объединяет блоки обработки и базы данных, все они имеют взаимные связи (обозначены сплошными линиями с двойной стрелкой). Фактический путь связи может быть двухточечным через общедоступные и / или частные сети. Связь может осуществляться через множество сетей, например, частные сети, VPN, канал MPLS или Интернет, и может использовать соответствующие программные интерфейсы приложений (API) и форматы обмена данными, например, передача репрезентативного состояния (REST), нотация объектов JavaScript. (JSON), расширяемый язык разметки (XML), простой протокол доступа к объектам (SOAP), служба сообщений Java (JMS) и / или система модулей платформы Java. Все сообщения могут быть зашифрованы. Связь обычно осуществляется по сети, такой как LAN (локальная сеть), WAN (глобальная сеть), телефонная сеть (коммутируемая телефонная сеть общего пользования (PSTN), протокол инициирования сеанса (SIP), беспроводная сеть, точка-точка. сеть, звездообразная сеть, сеть Token Ring, сеть-концентратор, Интернет, в том числе мобильный Интернет, через такие протоколы, как EDGE, 3G, 4G LTE, Wi-Fi и WiMAX. Кроме того, доступны различные методы авторизации и аутентификации, такие как имя пользователя / пароль, открытая авторизация (OAuth), Kerberos, SecureID, цифровые сертификаты и др. могут использоваться для защиты связи.
Процесс секвенирования
[00109] Варианты реализации, представленные в данном документе, могут быть применимы к анализу последовательностей нуклеиновых кислот для идентификации вариаций последовательностей. Варианты реализации могут применяться для анализа потенциальных вариантов / аллелей генетического положения / локуса и определения генотипа генетического локуса или, другими словами, обеспечения распознавания генотипа для локуса. В качестве примера, последовательности нуклеиновой кислоты могут быть проанализированы в соответствии со способами и системами, описанными в публикации заявки на патент США № 2016/0085910 и публикации заявки на патент США № 2013/0296175, полное содержащие которых в явном виде включено в настоящий документ в полном объеме посредством ссылки.
[00110] В одном варианте реализации процесс секвенирования включает получение образца, который содержит или предположительно содержит нуклеиновые кислоты, такие как ДНК. Образец может быть из известного или неизвестного источника, такого как животное (например, человек), растение, бактерии или гриб. Образец может быть взят непосредственно из источника. Например, кровь или слюна могут быть взяты непосредственно от индивидуума. Как вариант, образец может не быть получен непосредственно из источника. Затем один или более процессоров дают системе команду на подготовку образца к секвенированию. Подготовка может включать удаление постороннего материала и / или выделение определенного материала (например, ДНК). Биологический образец может быть подготовлен для включения признаков для конкретного анализа. Например, биологический образец может быть подготовлен для секвенирования путем синтеза (SBS). В некоторых вариантах реализации подготовка может включать амплификацию определенных областей генома. Например, подготовка может включать амплификацию заранее определенных генетических локусов, которые, как известно, включают STR (короткие тандемные повторы) и/или SNP (однонуклеотидные полиморфизмы). Генетические локусы могут быть амплифицированы с использованием предварительно определенных последовательностей праймеров.
[00111] Затем, указанные один или более процессоров передают системе инструкцию секвенировать образец. Секвенирование может осуществляться в соответствии с различными известными протоколами секвенирования. В частных вариантах реализации секвенирование включает SBS. В SBS множество флуоресцентно меченых нуклеотидов используется для последовательности множества кластеров амплифицированной ДНК (возможно, миллионов кластеров), присутствующих на поверхности оптического субстрата (например, поверхности, которая по меньшей мере частично ограничивает канал в проточной ячейке). Проточные ячейки могут содержать образцы нуклеиновых кислот для секвенирования, причем проточные ячейки размещены в соответствующих держателях проточных ячеек.
[00112] Нуклеиновые кислоты могут быть подготовлены таким образом, чтобы они содержали известную последовательность праймера, которая соседствует с неизвестной целевой последовательностью. Чтобы инициировать первый цикл секвенирования SBS, один или несколько нуклеотидов, меченных различным образом, ДНК-полимеразу и т. Д., можно подать в проточную ячейку или через нее посредством подсистемы потока жидкости. Можно добавлять либо по одному типу нуклеотида, либо нуклеотиды, используемые в процедуре секвенирования, могут быть специально сконструированы так, чтобы обладать свойством обратимой терминации, что дает возможность одновременного проведения каждого цикла реакции секвенирования в присутствии нескольких типов меченых нуклеотидов (например, A, C, T, G). Нуклеотиды могут включать обнаруживаемые фрагменты-метки, такие как флуорофоры. Когда четыре нуклеотида смешаны вместе, полимераза может выбрать правильное основание для включения, и каждая последовательность удлиняется на одно основание. Невключенные нуклеотиды можно отмывать потоком промывочного раствора через проточную ячейку. Один или несколько лазеров могут возбуждать нуклеиновые кислоты и вызывать флуоресценцию. Флуоресценция, испускаемая нуклеиновыми кислотами, основана на флуорофорах включенного основания, и разные флуорофоры могут излучать света с разными длинами волн. Деблокирующий реагент может быть добавлен в проточную ячейку для удаления обратимых терминаторных групп из удлиненных и детектированных цепей ДНК. Деблокирующий реагент затем можно отмыть, пропуская промывочный раствор через проточную ячейку. После этого проточная ячейка готова к следующему циклу секвенирования, начиная с введения меченого нуклеотида, как описано выше. Операции с текучей средой и обнаружением могут повторяться несколько раз для завершения последовательности операций. Примеры способов секвенирования описаны, например, в Bentley et al., Nature 456: 53-59 (2008), международной публикации № WO 04/018497, патенте США № 7,057,026, международной публикации № WO 91/06678, международной публикации № WO 07/123744, патенте США № 7,329,492, патенте США № 7,211,414, патенте США № 7,315,019, патенте США № 7,405,281и публикации заявки на патент США № 2008/0108082, каждый (ая) из которых включен(а) в настоящий документ посредством ссылки.
[00113] В некоторых проявлениях реализации нуклеиновые кислоты могут быть присоединены к поверхности и амплифицированы до или во время секвенирования. Например, амплификация может быть проведена с использованием мостиковой амплификации с образованием кластеров нуклеиновых кислот на поверхности. Применимые методы амплификации описаны, например, в Патенте США № 5,641,658, патентной публикации США № 2002/0055100, патенте США № 7,115,400, патентной публикации США № 2004/0096853, патентной публикации США № 2004/0002090, патентной публикации США № 2007/0128624и публикации заявки на патент США № 2008/0009420, каждый из этих документов полностью включен в настоящую заявку посредством ссылки. Другим полезным способом амплификации нуклеиновых кислот на поверхности является амплификация по типу катящегося кольца (RCA), например, как описано в Lizardi et al., Nat. Genet. 19:225-232 (1998) и в публикации заявки на патент США № 2007/0099208 A1, каждый (ая) из которых включен(а) в настоящий документ посредством ссылки.
[00114] Один пример протокола SBS использует модифицированные нуклеотиды, имеющие удаляемые 3'-блоки, например, как описано в международной публикации № WO 04/018497, публикации заявки на патент США № 2007/0166705A1 и патенте США № 7057026, каждый(ая) из которых включен(а) в настоящий документ посредством ссылки. Например, реагенты SBS могут доставляться повторяющимися циклами в проточную ячейку, к которой присоединены целевые нуклеиновые кислоты, например, по протоколу мостиковой амплификации. Кластеры нуклеиновых кислот могут быть преобразованы в одноцепочечную форму с использованием линеаризирующего раствора. Линеаризирующий раствор может содержать, например, эндонуклеазу рестрикции, способную расщеплять одну цепь каждого кластера. Линеаризирующий раствор может содержать, например, эндонуклеазу рестрикции, способную расщеплять одну цепь каждого кластера (например, расщепление диольной связи периодатом), расщепление сайтов без оснований путем расщепления эндонуклеазой (например, «USER», которая поставляется компанией NEB, Ипсвич, штат Массачусетс, США, номер компонента (M5505S), (путем воздействия тепла или щелочи, расщепления рибонуклеотидов, включенных в продукты амплификации, в остальном состоящих из дезоксирибонуклеотидов, фотохимического расщепления или расщепления пептидного линкера. После операции линеаризации праймер для секвенирования может быть подан в проточную ячейку в условиях гибридизации праймера для секвенирования с целевыми нуклеиновыми кислотами, которые должны быть секвенированы.
[00115] Затем проточную клетку можно привести в контакт с реагентом-удлинителем SBS, имеющим модифицированные нуклеотиды с удаляемыми 3'-блоками и флуоресцентными метками в условиях, позволяющих удлинить праймер, гибридизованный с каждой целевой нуклеиновой кислотой путем добавления одного нуклеотида. К каждому праймеру добавляется только один нуклеотид, поскольку включение модифицированного нуклеотида в растущую полинуклеотидную, комплементарную секвенируемой области матрицы, обуславливает отсутствие свободной группы 3'-ОН, доступной для направления дальнейшего удлинения последовательности и, следовательно, полимераза. не может добавить дополнительные нуклеотиды. Удлиняющий реагент SBS можно удалить и заменить сканирующим реагентом, содержащим компоненты, которые защищают образец при возбуждении излучением. Примеры компонентов сканирующего реагента описаны в публикации заявки на патент США № 2008/0280773 А1 и заявке на патент США № 13/018,255, каждый(ая) из которых включен(а) в настоящий документ посредством ссылки. Затем удлиненные нуклеиновые кислоты могут быть детектированы флуоресцентно в присутствии сканирующего реагента. После детектирования флуоресценции 3'-блок может быть удален с использованием деблокирующего реагента, который соответствует используемой блокирующей группе. Примеры деблокирующих реагентов, которые можно применять для соответствующих блокирующих групп, описаны в WO 004018497, US 2007 / 0166705A1 и патенте США № 7057026, каждый(ая) из которых включен(а) в настоящий документ посредством ссылки. Деблокирующий реагент можно смыть, оставляя целевые нуклеиновые кислоты гибридизованными с удлиненными праймерами, имеющими 3'-ОН-группы, к которым теперь можно присоединять другие нуклеотиды. Соответственно, циклы добавления удлиняющего реагента, сканирующего реагента и деблокирующего реагента с необязательными промываниями между одной или несколькими операциями могут повторяться до тех пор, пока не будет получена необходимая последовательность. Вышеуказанные циклы могут быть выполнены с использованием одной операции доставки удлиняющего реагента на цикл, когда к каждому из модифицированных нуклеотидов прикреплена отличная от других метка, о которой известно, что она соответствует конкретному основанию. Различные метки облегчают различение нуклеотидов, добавляемых во время каждой операции включения. В качестве альтернативы, каждый цикл может включать в себя отдельные операции доставки удлиняющего реагента, за которыми следуют отдельные операции доставки и детектирования сканирующего реагента, и в этом случае два или более нуклеотида могут иметь одинаковую метку и могут различаться на основании известного порядка доставки.
[00116] Хотя операция секвенирования обсуждалась выше в отношении конкретного протокола SBS, следует понимать, что при желании могут выполняться другие протоколы для секвенирования любого из множества других молекулярных анализов.
[00117] Затем указанные один или более процессоров системы получают данные секвенирования для последующего анализа. Данные секвенирования могут быть отформатированы различными способами, например, в файле .BAM. Данные секвенирования могут включать в себя, например, несколько ридов образцов. Данные секвенирования могут включать в себя множество ридов образцов, которые имеют соответствующие нуклеотидные последовательности образцов. Хотя обсуждается только один рид образца, следует понимать, что данные последовательности могут включать, например, сотни, тысячи, сотни тысяч или миллионы ридов образцов. Различные риды образцов могут содержать различное число нуклеотидов. Например, риды образцов может варьировать от 10 нуклеотидов до 500 нуклеотидов или более. Риды образцов могут охватывать весь геном источника (ов). В качестве одного примера, риды образцов направлены на заранее определенные генетические локусы, такие как генетические локусы, которые имеют подозрительные STR или предполагаемые SNP.
[00118] Каждый рид образца может включать последовательность нуклеотидов, которая может называться последовательностью образца, фрагментом образца или целевой последовательностью. Последовательность образца может включать, например, последовательности праймеров, фланкирующие последовательности и целевую последовательность. Количество нуклеотидов в последовательности образца может включать 30, 40, 50, 60, 70, 80, 90, 100 или более. В некоторых вариантах реализации один или более ридов образцов (или ридов последовательности) включают по меньшей мере 150 нуклеотидов, 200 нуклеотидов, 300 нуклеотидов, 400 нуклеотидов, 500 нуклеотидов или более. В некоторых вариантах реализации риды образцов могут включать более 1000 нуклеотидов, 2000 нуклеотидов или более. Риды образцов (или последовательности образцов) могут включать последовательности праймеров на одном или обоих концах.
[00119] Затем, указанные один или более процессоров анализируют данные секвенирования, чтобы получить потенциальные распознавание (ия) варианта (ов) образца и частоту варианта образца для указанных распознавания (ий) варианта (ов) образца. Эта операция также может называться приложением распознавания вариантов или распознавателем (определителем) вариантом. Таким образом, распознаватель вариантов идентифицирует или обнаруживает варианты, а классификатор вариантов классифицирует обнаруженные варианты как соматические или зародышевые. Могут применяться альтернативные распознаватели вариантов в соответствии с приведенным в настоящем документе вариантами реализации, причем могут применяться различные распознаватели вариантов в зависимости от типа выполняемой операции упорядочения, на основе характеристик образца, которые представляют интерес, и т.п. Одним из неограничивающих вариантов такого приложения для распознавания вариантов является приложение Pisces™ от компании Illumina Inc., (San Diego, CA, США), размещенное по адресу и https://github.com/Illumina/Pisces и описанное в статье Dunn, Tamsen & Berry, Gwenn & Emig-Agius, Dorothea & Jiang, Yu & Iyer, Anita & Udar, Nitin &  , Michael. (2017). Pisces: An Accurate and Versatile Single Sample Somatic and Germline Variant Caller. 595-595. 10.1145/3107411.3108203, полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки.
, Michael. (2017). Pisces: An Accurate and Versatile Single Sample Somatic and Germline Variant Caller. 595-595. 10.1145/3107411.3108203, полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки.
[00120] Такое приложение для распознавания вариантов содержит четыре выполняемых последовательно модуля:
[00121] ((1) Pisces Read Stitcher (cшиватель ридов Pisces): снижает шум путем сшивания парных ридов в BAM (рида один и рида два одной молекулы) в консенсусные. На выходе сшитый BAM.
[00122] (2) Pisces Variant Caller (определитель вариантов Pisces): определяет небольшие SNV, вставки (инсерции) и делеции. Pisces включают в себя алгоритм свертки вариантов для объединения вариантов, разбитых по границам ридов, основные алгоритмы фильтрации и простой алгоритм оценки достоверности вариантов на основе пуассоновского процесса. На выходе - VCF.
[00123] (3) Pisces Variant Quality Recalibrator (Рекалибратор качества вариантов Pisces, VQR): В случае, если определения (вызовы) вариантов в подавляющем большинстве случаев следуют некоторому паттерну, связанному с термическим повреждением или дезаминированием FFPE, шаг VQR будет понижать оценку Q варианта для подозрительных определений (вызовов). На выходе - откорректированный VCF.
[00124] (4) Pisces Variant Phaser (Фазировщик фариантов Pisces -Scylla): использует жадный метод кластеризации на основе ридов для сборки небольших вариантов в сложные аллели из клональных субпопуляций. Это позволяет более точно определять функциональные последствия последующими инструментами. На выходе - откорректированный VCF.
[00125] В качестве дополнения или альтернативы для этой операции можно применять приложение для определения вариантов Strelka™, от компании Illumina Inc., размещенное по адресу https://github.com/Illumina/strelka и описанное в статье T Saunders, Christopher & Wong, Wendy & Swamy, Sajani & Becq, Jennifer & J Murray, Lisa & Cheetham, Keira. (2012). Strelka: Accurate somatic small-variant calling from sequenced tumor-normal sample pairs. Bioinformatics (Oxford, Англия). 28. 1811-7. 10.1093/bioinformatics/bts271, полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки. Далее, в качестве дополнения или альтернативы, для этой операции можно применять приложение Strelka2™, от компании Illumina Inc., размещенное по адресу https://github.com/Illumina/strelka и описанное в статье Kim, S., Scheffler, K., Halpern, A.L., Bekritsky, M.A., Noh, E.,  , M., Chen, X., Beyter, D., Krusche, P., and Saunders, C.T. (2017). Strelka2: Fast and accurate variant calling for clinical sequencing applications, полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки. Более того, в качестве дополнения или альтернативы, для этой операции можно применять инструмент для аннотации/определения вариантов, такой как Nirvana™, от компании Illumina Inc., размещенное по адресу https://github.com/Illumina/Nirvana/wiki и описанная в статье Stromberg, Michael & Roy, Rajat & Lajugie, Julien & Jiang, Yu & Li, Haochen & Margulies, Elliott. (2017). Nirvana: Clinical Grade Variant Annotator. 596-596. 10.1145/3107411.3108204, полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки.
, M., Chen, X., Beyter, D., Krusche, P., and Saunders, C.T. (2017). Strelka2: Fast and accurate variant calling for clinical sequencing applications, полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки. Более того, в качестве дополнения или альтернативы, для этой операции можно применять инструмент для аннотации/определения вариантов, такой как Nirvana™, от компании Illumina Inc., размещенное по адресу https://github.com/Illumina/Nirvana/wiki и описанная в статье Stromberg, Michael & Roy, Rajat & Lajugie, Julien & Jiang, Yu & Li, Haochen & Margulies, Elliott. (2017). Nirvana: Clinical Grade Variant Annotator. 596-596. 10.1145/3107411.3108204, полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки.
[00126] Такой инструмент для аннотации/определения вариантов может применять различные алгоритмические методики, такие как описанные у Nirvana:
[00127] a. Идентификация всех перекрывающихся транскриптов с помощью массива интервалов: для функциональной аннотации мы можем идентифицировать все транскрипты, перекрывающие вариант, и можно применять дерево интервалов. Однако, поскольку набор (множество) интервалов может быть статическим, мы смогли дополнительно оптимизировать его в Массив Интервалов. Дерево интервалов возвращает все перекрывающиеся транскрипты за время O (min (n, k lg n)), где где n - количество интервалов в дереве, а k - количество перекрывающихся интервалов. На практике, поскольку k на самом деле мало по сравнению с n для большинства вариантов, эффективное время выполнения на дереве интервалов будет O (k lg n). Мы улучшили до O (lg n + k) за счет создания массива интервалов, в котором все интервалы хранятся в отсортированном массиве, так что нам нужно только найти первый перекрывающийся интервал, а затем пронумеровать оставшиеся (k-1).
[00128] b. CNV / SV (Yu): могут быть предоставлены аннотации для вариаций количества копий (CNV) и структурных вариантов (SV). Аналогично аннотациям небольших вариантов, транскрипты, перекрывающиеся с SV, а также ранее определенные структурные варианты могут быть аннотированы в онлайн-базах данных. В отличие от небольших вариантов, не обязательно все перекрывающиеся транскрипты аннотировать, так как слишком много транскриптов будут перекрываться с большими SV. Вместо этого могут быть аннотированы все перекрывающиеся транскрипты, относящиеся к частичному перекрывающемуся гену. В частности, для этих транскриптов могут выявляться (включаться в отчет) затронутые интроны, экзоны и последствия, обусловленные структурными вариантами. Доступна опция, позволяющая выводить все перекрывающиеся транскрипты, но может быть представлена основная информация для этих транскриптов, такая как символ гена, отметка, является ли это каноническим перекрыванием или частичным перекрыванием с транскриптами. Для каждого SV / CNV также интересно знать, были ли изучены эти варианты и их частота в разных популяциях. Соответственно, мы регистрировали перекрывающиеся SV во внешних базах данных, таких как “1000 геномов”, DGV и ClinGen. Чтобы избежать применения произвольного отсечения для определения того, какой SV перекрывается, вместо этого можно применять все перекрывающиеся транскрипты и вычислять взаимное перекрывание, вместо этого можно применять все перекрывающиеся транскрипты и вычислять взаимное, то есть длину перекрывания, деленную на минимум длины этих двух SV.
[00129] c. Регистрация дополнительных аннотаций: Дополнительные аннотации бывают двух типов: малые и структурные варианты (SV). SV можно моделировать как интервалы и использовать массив интервалов, описанный выше, для идентификации перекрывающихся SV. Небольшие варианты моделируются в виде точек и сопоставляются по положению и (необязательно) аллелю. Соответственно, их ищут с применением алгоритма, подобного бинарному поиску. Поскольку база данных дополнительных аннотаций может быть довольно большой, создают гораздо меньший индекс для картирования хромосомных положений на местоположения файлов, в которых находится дополнительная аннотация. Индекс - это отсортированный массив объектов (состоящих из хромосомного положения и расположения файла), по которым можно выполнять двоичный поиск с использованием положения. Чтобы размер индекса оставался небольшим, множество положений (до определенного максимального числа) сжимают в один объект, который хранит значения для первого положения и только дельты для последующих положений. Поскольку мы используем двоичный поиск, время выполнения - O (lg n), где n - количество элементов в базе данных.
[00130] d. Кэш-файлы VEP
[00131] e. База данных транскриптов: Файлы Transcript Cache (кэш транскриптов, кэш) и Supplementary database (дополнительная база данных, SAdb) представляют собой упорядоченное хранилище объектов данных, таких как транскрипты и дополнительные аннотации. Мы применяем кэш Ensembl VEP cache в качестве источника данных для кэша. Для создания кэша все транскрипты помещают в массив интервалов, а конечное состояние массива сохраняется в файлах кэша. Таким образом, в процессе аннотации нам нужно только загрузить предварительно вычисленный массив интервалов и выполнить поиск по нему. Поскольку кэш загружается в память, а поиск выполняется очень быстро (описано выше), поиск перекрывающихся транскриптов согласно Nirvana выполняется очень быстро (профилировано менее 1% от общего времени выполнения?).
[00132] f. Дополнительная база данных: источники данных для SAdb перечислены в дополнительных материалах. База данных SAdb для небольших вариантов создается путем k-направленного объединения всех источников данных, так что каждый объект в базе данных (идентифицируемый ссылочным именем и положением) содержит все соответствующие дополнительные аннотации. Проблемы, возникающие при парсировании файлов - источников данных, подробно описаны на домашней странице Nirvana. Чтобы ограничить использование памяти, в память загружается только индекс SA. Этот индекс позволяет осуществить быстрый поиск положения файла для дополнительной аннотации. Однако, поскольку данные должны быть извлечены с диска, добавление дополнительных аннотаций было определено как самое узкое место Nirvana (профилируется примерно как 30% от общего времени выполнения).
[00133] g. Последствия и онтология последовательности: Последствия и онтология последовательности. Иногда у нас была возможность выявить проблемы в текущей SO и сотрудничать с командой SO, чтобы улучшить состояние аннотации.
[00134] Такой инструмент вариантов аннотации может включать предварительную обработку. Например, Nirvana включала большое количество аннотаций из внешних источников данных, таких как ExAC, EVS, проект “1000 геномов”, dbSNP, ClinVar, Cosmic, DGV и ClinGen. Чтобы в полной мере использовать эти базы данных, мы должны очистить информацию из них. Мы реализовали разные стратегии для решения разных конфликтов, обусловленных разными источниками данных. Например, в случае нескольких записей dbSNP для одного и того же положения и другого аллеля, мы объединяем все идентификаторы в список идентификаторов, разделенных запятыми; если есть несколько записей с разными значениями CAF для одного и того же аллеля, мы используем первое значение CAF. Для конфликтующих записей ExAC и EVS мы учитываем количество образцов и используем запись с большим количеством образцов. В проекте “1000 геномов” мы удаляли частоту аллеля конфликтующего аллеля. Другая проблема - неточная информация. В основном мы брали информацию о частотах аллелей из проекта “1000 геномов”, однако мы заметили, что для GRCh38 частота аллелей, указанная в информационном поле, не исключала образцы с недоступным генотипом, что приводило к повышенным частотам для вариантов, которые доступны не для всех образцов. Чтобы гарантировать точность нашей аннотации, мы используем все генотипы индивидуального уровня для вычисления истинных частот аллелей. Как мы знаем, одни и те же варианты могут иметь разные представления на основе разных выравниваний. Чтобы быть уверенным, что мы можем точно получить (вывести) информацию об уже идентифицированных вариантах, мы должны предварительно обработать варианты из разных ресурсов, чтобы они имели единообразное представление. Для всех внешних источников данных мы удалили аллели, чтобы удалить дублированные нуклеотиды как в референсном аллеле, так и в альтернативных аллелях. Для ClinVar мы непосредственно парсировали xml-файл и выполнили пятизначное выравнивание для всех вариантов, которое часто используется в vcf-файле. Различные базы данных могут содержать одинаковый набор информации. Чтобы избежать ненужного дублирования, мы удалили часть повторяющейся информации. Чтобы избежать ненужного дублирования, мы удалили часть повторяющейся информации.
[00135] В соответствии с по меньшей мере некоторыми вариантами реализации, указанное приложение для определения вариантов выдает варианты с низкой частотой, определение зародышевой линии и т.п. В качестве неограничивающего примера, указанное приложение для определения вариантов может работать только с опухолевыми образцами и/или с парными образцами опухоль-норма. Приложение для определения вариантов может искать однонуклеотидные варианты(SNV), многонуклеотидные варианты (MNV), инделы и т.п. Приложение определения вариантов идентифицирует варианты, одновременно фильтруя несоответствия из-за ошибок секвенирования или подготовки образца. Для каждого варианта определитель вариантов идентифицирует референсную последовательность, положение варианта и потенциальную последовательность (и) варианта (например, SNV от A до C или делеция из AG в A). Приложение определения вариантов идентифицирует последовательность образца (или фрагмент образца), референсную последовательность / фрагмент и определение варианта как показатель присутствия варианта. Приложение определения вариантов может идентифицировать необработанные фрагменты и выводить обозначение исходных фрагментов, подсчет числа необработанных фрагментов, которые верифицируют возможное определение варианта, положение в исходном фрагменте, в котором присутствует подтверждающий вариант, и другую важную информацию. Неограничивающие примеры необработанных фрагментов включают дуплексных сшитый фрагмент, симплексный сшитый фрагмент, дуплексный несшитый фрагмент и симплексный несшитый фрагмент.
[00136] Приложение для определения вариантов может выводить определения (вызовы) в различных форматах, например, в файл .VCF или .GVCF. Только в качестве примера указанное приложение для определения вариантов может быть включено в пайплайн MiSeqReporter (например, когда оно реализовано в секвенаторе MiSeq®). При желании приложение может быть реализовано с различными рабочими процессами. Анализ может включать единый протокол или комбинацию протоколов, которые анализируют риды образца определенным образом для получения желаемой информации.
[00137] Затем указанные один или более процессоров осуществляют операцию валидации применительно к определению потенциальных вариантов. Операция валидации может быть основана на оценке качества и / или иерархии многоуровневых тестов, как объясняется ниже. Когда операция валидации (проверки) аутентифицирует или проверяет наличие потенциального определения варианта, операция проверки передает информацию об определенном варианте (из указанного приложения для определения вариантов) в генератор отчетов по образцам. В качестве альтернативы, когда операция проверки делает недействительным или дисквалифицирует потенциальное определение варианта, операция проверки передает соответствующий индикатор (например, отрицательный индикатор, индикатор отсутствия определения, индикатор недействительного определения) генератору отчетов по образцам. Операция проверки также может передавать оценку достоверности, связанную со степенью уверенности в том, что конкретное определение варианта правильно или определение варианта правильно обозначено как недействительное (невалидное).
[00138] Затем, указанные один или более процессоров генерируют и сохраняют отчет по образцу. Отчет по образцу может включать, например, информацию о множестве генетических локусов по отношению к образцу. Например, для каждого генетического локуса заранее определенного набора генетических локусов отчет по образцу может по меньшей мере одно из: определить генотип; указывать, что определение генотипа невозможно; предоставить оценку достоверности определения генотипа; или указать потенциальные проблемы с анализом в отношении одного или нескольких генетических локусов. В отчете по образцу также может быть указан пол человека, предоставившего образец, и / или указано, что образец включает несколько источников. В настоящем документе «отчет по образцу» («отчет об образце») может включать цифровые данные (например, файл данных) генетического локуса или заранее определенного набора генетических локусов и / или печатный отчет о генетическом локусе или наборе генетических локусов. Таким образом, создание или предоставление может включать в себя создание файла данных и / или печать отчета по образцу, или отображение отчета по образцу.
[00139] Отчет по образцу может указывать на то, что определение варианта было установлено, но не было подтверждено. Когда определение варианта определяется как недопустимое, отчет по образцу может указывать дополнительную информацию, касающуюся основания для решения не подтверждать определение варианта. Например, the дополнительная информация в отчете может включать описание необработанных фрагментов и степень (например, число), в которой эти необработанные фрагменты поддерживают определения вариантов или противоречат им. Дополнительно или в качестве альтернативы, дополнительная информация в отчете может включать оценку качества, полученную в соответствии с вариантами реализации, описанными в данном документе.
Применение определения вариантов
[00140] Варианты реализации, раскрытые в настоящем документе, включают анализ секвенированных данных для определения потенциальных вариаций. Распознавание вариантов может проводиться над сохраненными данными для выполненной ранее операции секвенирования. В качестве дополнения или альтернативы, его можно проводить в режиме реального времени одновременно с выполнением операции секвенирования. Каждый из ридов образцов ставится в соответствие соответствующим генетическим локусам. Риды образца могут быть поставлены в соответствие определенным генетическим локусам на основании последовательности нуклеотидов рида образца, или, другими словами, порядку нуклеотидов, входящих в рид (например, A, C, G, T). На основании этого анализа рид образца может быть охарактеризован как включающий возможную вариацию/аллель определенного генетического локуса. Рид образца можно собирать (или агрегировать или группировать) вместе с другими ридами образца, охарактеризованными как включающие возможную вариацию/аллель генетического локуса. Под операцией определения соответствия можно также понимать операцию распознавания, в которой рид образца определяется как как возможно ассоциированный с определенным генетическим положением/локусом. Риды образцов можно анализировать с целью локализовать идентифицирующие последовательности (например, последовательности праймеров) нуклеотидов, которые отличают данный рид образца от других ридов образца. Более конкретно, идентифицирующая последовательность(и) может идентифицировать рид образца среди других ридов образцов как ассоциированный с определенным генетическим локусом.
[00141] Операция определения соответствия (присваивания) может включать анализ серии n нуклеотидов идентифицирующей последовательности для определения, соответствует ли серия n нуклеотидов, идентифицирующих последовательности, одной или более выбранным последовательностям. В частных вариантах реализации, операция определения соответствия (присваивания) может включать анализ первых n нуклеотидов последовательности образца для определения, соответствуют ли первые n нуклеотидов последовательности образца одной или более выбранным последовательностям. Число n может принимать разнообразные значения, которые могут быть заложены в программу протокола или вводиться пользователем. Например, число n может быть определено как число нуклеотидов самой короткой выбранной последовательности в базе данных. Это заранее предопределенное число может составлять, например, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нуклеотидов. Однако, в других вариантах реализации может применяться меньшее или большее число нуклеотидов. Число n может также быть выбрано человеком, например пользователем системы. Выбор числа n может быть основан на одном или более условиях. Например, число n может быть определено как число нуклеотидов самой короткой последовательности праймера в базе данных, или определенное число, смотря какое из них меньше. В некоторых вариантах реализации a для n может быть использовано минимальное значение, такое как 15, такое что любую последовательность праймера короче 15 нуклеотидов можно считать исключением.
[00142] В некоторых случаях, серия n нуклеотидов идентифицирующей последовательности может не соответствовать точно нуклеотидам последовательности выборки. Тем не менее, идентифицирующая последовательность может эффективно соответствовать последовательности выборки, если идентифицирующая последовательность почти идентична последовательности выборки. Например, рид образца может быть определен для генетического локуса, если серия n нуклеотидов (например, первые n нуклеотидов) идентифицирующей последовательности совпадают с последовательностью выборки с не более чем установленным числом несоответствий (например, 3) и/или установленным числом сдвигов (например, 2). Правила можно установить так, что каждое несоответствие или сдвиг может считаться как различие между ридом образца и последовательностью праймера. Если число различий меньше установленного значения, операция присваивания может быть применена к риду образца для соответствующего генетического локуса (то есть, рид присвоен соответствующему локусу). В некоторых вариантах реализации, вводится оценка совпадения, которая основана на количестве различий между идентифицирующей последовательностью рида образца и последовательностью выборки, ассоциированной с генетическим локусом. Если оценка совпадения превосходит установленный порог совпадения, генетический локус, соответствующий выбранной последовательности, можно считать потенциальным локусом рида образца. В некоторых вариантах реализации, может проводиться последующий анализ с целью определить, действительно ли рид образца соответствует генетическому локусу.
[00143] Если рид образца эффективно совпадает с одной из выбранных последовательностей в базе данных (т.е., в точности совпадает или совпадает в пределах критериев, описанных выше), то риду образца назначают или ставят в соответствие генетический локус, который коррелирует с выбранной последовательностью. Это можно назвать определением локуса или предварительным определением локуса, где рид образца определен для генетического локуса, который коррелирует с выбранной последовательностью. Однако, как описано выше, рид образца может быть определен для более одного генетического локуса В таких вариантах осуществления, может проводиться последующий анализ для определения или присваивания рида образца только одному из потенциальных генетических локусов. В некоторых вариантах реализации рид последовательности, который сравнивают с базой данных референсных последовательностей, представляет собой первый рид из секвенирования спаренных концов. При осуществлении секвенирования спаренных концов, получают второй рид (представляющий фрагмент необработанных данных) который коррелирует с ридом образца. После присваивания, последующий анализ, который проводится с присвоенными ридами, может быть основан на типе генетического локуса, который был определен для этого рида.
[00144] Затем, риды образца анализируют для идентификации потенциальных вариантов. Среди прочего, результаты этого анализа идентифицируют потенциальный вариант, частоту последовательности варианта, референсную последовательность и положение в исследуемой генетической последовательности, в которой встретился вариант. Например, если известно, что генетический локус включает однонуклеотидные полиморфизмы, то присвоенные риды, которые были определены для генетического локуса можно подвергать дополнительному анализу для идентификации однонуклеотидных полиморфизмов присвоенных ридов. Если известно, что генетический локус включает полиморфные повторяющиеся элементы ДНК, то присвоенные риды можно анализировать для того, чтобы идентифицировать или охарактеризовать полиморфные повторяющиеся элементы ДНК в составе ридов образцов. В некоторых вариантах реализации В некоторых вариантах реализации если присвоенный рид эффективно совпадает с STR-локусом и SNP-локусом, риду образца может быть присвоено предупреждение или флаг. Рид образца может быть определен и как STR-локус и как SNP-локус. Анализ может включать выравнивание присвоенных ридов в соответствии с алгоритмом выравнивания с целью определить последовательности и/или длины присвоенных ридов. Протокол выравнивания может включать метод, описанный в Международной Патентной Заявке № PCT/US2013/030867 (№ публикации WO 2014/142831), поданной 15 марта 2013, которая в полном объеме включена в данную заявку посредством ссылки.
[00145] Затем один или более процессов анализируют необработанный фрагмент с целью определить, существуют ли поддерживающие варианты в соответствующих положениях необработанных фрагментов. Можно идентифицировать различные типы необработанных фрагментов. Например, определитель вариантов может идентифицировать тип необработанного фрагмента, который имеет вариант, валидирующий (подтверждающий) исходно найденный вариант. Например, тип необработанного фрагмента может представлять двунитевый сшитый фрагмент, однонитевый сшитый фрагмент, двунитевый несшитый фрагмент или однонитевой несшитый фрагмент. Опционально можно идентифицировать другие необработанные фрагменты вместо или в дополнение к приведенным примерам. Вместе с идентификацией каждого типа необработанных фрагментов, пользователь также определяет положение в этом фрагменте, в котором встретился поддерживающий вариант, а также число необработанных фрагментов, в которых этот выявили поддерживающий вариант. Например, определитель вариантов может вывести индикацию того, что 10 ридов необработанных фрагментов идентифицированы как представляющие собой двунитевые сшитые фрагменты, содержащие поддерживающий вариант в определенном положении X. Определитель вариантов может также выводить индикацию того, что пять ридов необработанных фрагментов представляют собой однонитевые несшитые фрагменты, имеющие поддерживающий вариант в определенном положении Y. Определитель вариантов может также выводить число необработанных фрагментов, которые соответствуют референсной последовательности, и таким образом не включают поддерживающий вариант, который в ином случае был бы свидетельством поддерживающим определение потенциального варианта в исследуемой генной последовательности.
[00146] Далее, сохраняется число необработанных фрагментов, которые включают поддерживающие варианты, а также положения, в которых встретились поддерживающие варианты. В качестве дополнения или альтернативы, можно сохранять число необработанных фрагментов, которые не включают поддерживающие варианты в представляющем интерес положении (относительно положения определения потенциального вариантов рида образца или фрагмента образца). В качестве дополнения или альтернативы, может сохраняться число необработанных фрагментов, которые соответствуют референсной последовательности и не удостоверяют/подтверждают определение потенциального варианта. Полученная информация выводится в приложение валидации определения вариантов, включая количество и тип необработанных фрагментов, которые поддерживают определение потенциального варианта, положения поддерживающих вариантов в необработанных фрагментах, число необработанных фрагментов, которые не поддерживают потенциального определение варианта и т.п.
[00147] Когда потенциальный вариант идентифицирован, в выходных данных процесса появляется индикация определения потенциального варианта, последовательность варианта, положение варианта и референсная последовательность, ассоциированная с ним. Вариант обозначается как “потенциальный”, поскольку ошибки могут приводить к идентификации ложного варианта. В соответствии с приведенными здесь вариантами осуществления определение потенциального варианта анализируют, чтобы уменьшить или исключить ложные варианты и ложные совпадения. В качестве дополнения или альтернативы, процесс (способ) анализирует один или более необработанных фрагментов, ассоциированных с ридом образца ,и дополняет выходные данные соответствующим вариантом, ассоциированным с необработанными фрагментами.
Фильтр вариантов
[00148] Фильтр вариантов 111 включает сверточную нейронную сеть (CNN) и полностью связанную нейронную сеть (FCNN). Входные данные фильтра вариантов 111 накладываются (перекрываются) на образцы нуклеотидных последовательностей из базы данных перекрывающихся образцов 119. На нуклеотидные последовательности из базы данных 169 нуклеотидных последовательностей накладываются повторяющиеся паттерны из базы данных повторяющихся паттернов 196 для генерации наложенных (перекрытых) образцов. Блок наложения 181 накладывает повторяющиеся паттерны на нуклеотидные последовательности из базы данных 169 для генерации наложенных образцов, которые хранятся в базе данных наложенных образцов 119. Блок моделирования 116 подает комбинации повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах для фильтр вариантов для анализа. Когда наложенные образцы с исследуемым повторяющимся паттерном подаются в качестве входных данных в фильтра вариантов 111, фильтр вариантов 111 выводит классификационные оценки для вероятности того, что вариантный нуклеотид в каждом из наложенных образцов является истинным вариантом или ложным вариантом. Наконец, анализатор 194 вызывает отображение классификационных оценок в виде распределения для каждого из факторов повтора чтобы поддерживать оценку того, что последовательность-специфичная ошибка обуславливаются этими повторяющимися паттернами.
Повторяющиеся паттерны
[00149] Генератор повторяющихся паттернов 171 генерирует повторяющиеся паттерны “rp”, используя гомополимерные или сополимерные паттерны длины “n” с различными факторами повторов “m”. Гомополимерные повторяющиеся паттерны содержат только одно основание (A, C, G или T), а сополимерные повторяющиеся паттерны содержат более одного основания. “Повторящийся паттерн” генерируется путем применения “фактора повтора (m)” к “паттерну”. Связь между паттерном (n), фактором повтора (m) и повторяющимся паттерном (rp) представлена уравнением (1) следующим образом:
паттерн * m = rp (1)
[00150] В Таблице 1 представлены примеры гомополимерных повторяющихся паттернов. Длина гомополимерных паттернов равна единице, т.е., “n = 1”.
n = паттерн m = повторяющейся паттерн (rp)
Длина Фактор
Паттерна Повтора
1 A 5 AAAAA (5 As)
1 A 9 AAAAAAAAA (9 As)
1 A 13 AAAAAAAAAAAAA (13 As)
1 A 17 AAAAAAAAAAAAAAAAA (17 As)
1 A 21 AAAAAAAAAAAAAAAAAAAAA (21 As)
1 A 25 AAAAAAAAAAAAAAAAAAAAAAAAA (25 As)
1 C 5 CCCCC (5 Cs)
1 C 9 CCCCCCCCC (9 Cs)
1 C 13 CCCCCCCCCCCCC (13 Cs)
1 C 17 CCCCCCCCCCCCCCCCC (17 Cs)
1 C 21 CCCCCCCCCCCCCCCCCCCCC (21 Cs)
1 C 25 CCCCCCCCCCCCCCCCCCCCCCCCC (25 Cs)
1 T 5 TTTTT (5 Cs)
1 T 9 TTTTTTTTT (9 Ts)
1 T 13 TTTTTTTTTTTTT (13 Ts)
1 T 17 TTTTTTTTTTTTTTTTT (17 Ts)
1 T 21 TTTTTTTTTTTTTTTTTTTTT (21 Ts)
1 T 25 TTTTTTTTTTTTTTTTTTTTTTTTT (25 Ts)
1 G 5 TTTTT (5 Cs)
1 G 9 TTTTTTTTT (9 Ts)
1 G 13 TTTTTTTTTTTTT (13 Ts)
1 G 17 TTTTTTTTTTTTTTTTT (17 Ts)
1 G 21 TTTTTTTTTTTTTTTTTTTTT (21 Ts)
1 G 25 TTTTTTTTTTTTTTTTTTTTTTTTT (25 Ts)
[00151] В таблице 2, представлен пример повторяющихся паттернов сополимеров. Длина сополимерных паттернов больше единицы, т.е. “n>1”.
n = паттерн m = повторяющейся паттерн (rp)
Длина Фактор
Паттерна Повтора
2 AC 1 AC (1 AC)
2 AC 3 ACACAC (3 ACs)
2 AC 5 ACACACACAC (5 ACs)
2 AC 7 ACACACACACACAC (7 ACs)
2 AC 9 ACACACACACACACACAC (9 ACs)
2 AC 11 ACACACACACACACACACACAC (11 ACs)
2 TA 1 TA (1 TA)
2 TA 3 TATATA (3 TAs)
2 TA 5 TATATATATA (5 TAs)
2 TA 7 TATATATATATATA (7 TAs)
2 TA 9 TATATATATATATATATA (9 TAs)
2 TA 11 TATATATATATATATATATATA (11 TAs)
3 AAT 1 AAT (1 AAT)
3 AAT 2 AATAAT (2 AATs)
3 AAT 3 AATAATAAT (3 AATs)
3 AAT 4 AATAATAATAAT (4 AATs)
3 AAT 5 AATAATAATAATAAT (5 AATs)
3 AAT 6 AATAATAATAATAATAAT (6 AATs)
4 CTAT 1 CTAT (1 CTAT)
4 CTAT 2 CTATCTAT (2 CTATs)
4 CTAT 3 CTATCTATCTAT (3 CTATs)
4 CTAT 4 CTATCTATCTATCTAT (4 CTATs)
4 CTAT 5 CTATCTATCTATCTATCTAT (5 CTATs)
4 CTAT 6 CTATCTATCTATCTATCTATCTAT (5 CTATs)
Фильтр вариантов
[00152] ФИГ. 2 иллюстрирует пример архитектуры 200 фильтра вариантов 111. Фильтр вариантов 111 имеет иерархическую структуру, построенную на сверточной нейронной сети (CNN) и полностью связанной нейронной сети (FCNN). DeepPOLY использует фильтр вариантов 111 для тестирования известных паттернов последовательностей на предмет их влияния на фильтрацию вариантов. Входные данные для фильтра 111 вариантов содержат нуклеотидные последовательности длиной 101, содержащие вариантный нуклеотид в центре и фланкированный слева и справа 50 нуклеотидами. Понятно, что в качестве входных данных в качестве входных данных для фильтра вариантов 111 могут использоваться нуклеотидные последовательности различной длины.
[00153] Сверточная нейронная сеть содержит слои свертки, которые выполняют операцию свертки между входными значениями и фильтрами свертки (матрица весов), которые обучаются в течение многих итераций обновления градиента в ходе обучения.
[00154] Пусть (m, n) - размер фильтра, а W - матрица весов, тогда сверточный слой выполняет свертку W с входом X, вычисляя скалярное произведение W ⋅ x + b, где x - это пример X а b - смещение. Размер шага, на который сверточные фильтры скользят по входу, называется шагом, а область фильтра (m × n) называется рецептивным полем. Один и тот же фильтр свертки применяется к разным положениям входных данных, что сокращает количество обученных весов. Он также обеспечивает возможность обучения без зависимости от положения, то есть, если во входных данных существует важный паттерн, фильтры свертки изучают его независимо от того, где он находится в последовательности. Дополнительные сведения о сверточной нейронной сети можно найти в I. J. Goodfellow, D. Warde-Farley, M. Mirza, A. Courville, and Y. Bengio, “CONVOLUTIONAL NETWORKS”, Deep Learning, MIT Press, 2016; J. Wu, “INTRODUCTION TO CONVOLUTIONAL NEURAL NETWORKS”, Nanjing University (Нанкинский университет), 2017; и N. ten DIJKE, “Convolutional Neural Networks for Regulatory Genomics”, Диссертация на соискание магистерской степени, Universiteit Leiden Opleiding Informatica, 17 июня 2017 г., полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки. Архитектура сверточной нейронной сети, показанной на ФИГ. 2, включает два сверточных слоя. Первый сверточный слой обрабатывает входные данные с использованием 64 фильтров, каждый из которых имеет размер 3. Выходные данные первого сверточного слоя проходят через слой пакетной нормализации.
[00155] Распределение каждого слоя сверточной нейронной сети меняется во время обучения и варьирует от одного слоя к другому. Это снижает скорость сходимости алгоритма оптимизации. Пакетная нормализация (Ioffe, Szegedy 2015) - это метод решения этой проблемы. Если мы обозначим вход слоя пакетной нормализации как x, а его как z, пакетная нормализация применяет следующее преобразование к x:
 .
.
[00156] Пакетная нормализация применяет нормализацию среднего отклонения на входе x с использованием μ и σ и линейно масштабирует и сдвигает его, используя γ и β. Параметры нормализации μ и σ вычисляются для текущего слоя по тренировочному (обучающему) набору с использованием метода, называемого экспоненциальным скользящим средним. Другими словами, это не обучаемые параметры. Напротив, γ и β являются обучаемыми параметрами. Значения μ и σ, вычисленные во время обучения, используются при прямом проходе во время вывода. Нелинейная функция блока линейной ректификации (ReLU) применяется к выходу слоя пакетной нормализации для получения нормированного выхода. Другие примеры нелинейных функций включают сигмоидную функцию, фунцию гиперболического тангенса (tanh) и функция «Leaky ReLU».
[00157] Второй сверточный слой использует 128 фильтров размера 5 на нормализованном выходе. Пример CNN, показанный в ФИГ. 2, включает в себя сглаживающий слой, который сглаживает выходные данные второго сверточного слоя до одномерного массива, который проходит через второй набор слоев пакетной нормализации и активации ReLU. Нормализованные выходные данные второго сверточного слоя подаются в полностью связанную нейронную сеть (FCNN). Полностью связанная нейронная сеть состоит из полностью связанных слоев - каждый нейрон получает входные данные от всех нейронов предыдущего слоя и отправляет свои выходные данные каждому нейрону следующего слоя. Это отличается от того как работают сверточные слои, когда нейроны отправляют свой вывод только некоторым из нейронов следующего слоя. Нейроны полностью связанных слоев оптимизируются в ходе многих итераций обновления градиента во время обучения. Дополнительные сведения о полностью связанной нейронной сети можно найти в работе I. J. Goodfellow, D. Warde-Farley, M. Mirza, A. Courville, and Y. Bengio, “CONVOLUTIONAL NETWORKS”, Deep Learning, MIT Press, 2016; J. Wu, “INTRODUCTION TO CONVOLUTIONAL NEURAL NETWORKS”, Nanjing University (Нанкинский университет), 2017; и N. ten DIJKE, “Convolutional Neural Networks for Regulatory Genomics”, Диссертация на соискание магистерской степени, Universiteit Leiden Opleiding Informatica, 17 июня 2017 г., полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки. Слой классификации (например, слой softmax), следующий за полностью связанными слоями, дает классификационные оценки для правдоподобия того, что каждый вариант-кандидат в целевом положении нуклеотида является истинным вариантом или ложным вариантом. Слой классификации может быть слоем softmax или сигмоидным слоем. Количество классов и их тип могут быть изменены в зависимости от реализации.
[00158] ФИГ. 3 демонстрирует один вариант реализации пайплайна обработки 300 фильтра вариантов 111. В показанном варианте реализации сверточная нейронная сеть (CNN) имеет два сверточных слоя, а полностью связная нейронная сеть (FCNN) имеет два полностью связных слоя. В других вариантах реализации фильтр фариантов 111 и его сверточная нейронная сеть и полностью связная нейронная сеть могут иметь дополнительные, меньшее количество или другие параметры и гиперпараметры. Частными римерами параметров являются количество слоев свертки, количество слоев пакетной нормализации и ReLU, количество полностью связанных слоев, количество сверточных фильтров в соответствующих сверточных слоях, количество нейронов в соответствующих полностью связных слоях, количество выходов, производимых окончательным уровнем классификации и остаточная связность. Некоторыми примерами гиперпараметров являются размер окна фильтров свертки, длина шага фильтров свертки, заполнение и разрежение. В приведенном ниже обсуждении термин «уровень» относится к алгоритму, реализованному в коде, как программная логика или модуль. Некоторые примеры слоев можно найти в документации Keras ™, доступной по адресу https://keras.io/layers/about-keras-layers/, полное содержание которой в явном виде полностью включено в настоящий документ посредством ссылки.
[00159] Закодированная с одним активным состоянием входная последовательность 302 подается на первый сверточный слой 304 сверточной нейронной сети (CNN). Размерность входной последовательности 302 составляет 101, 5, где 101 представляет 101 нуклеотид во входной последовательности 302 с конкретным вариантом в центральном положении-мишени, фланкированной 50 нуклеотидами с каждой стороны, а 5 представляет 5 каналов A, T, C, G, N, используемых для кодирования входной последовательности 302. Подготовка входных последовательностей 302 описана со ссылкой на ФИГ. 5.
[00162] Первый сверточный слой 304 содержит 64 фильтра, каждый из которых выполняет свертку по входной последовательности 302 с размером окна 3 и длиной шага 1. За сверткой следуют слои 306 пакетной нормализации и нелинейности ReLU. выходные данные (карта характеристик) 308 размерности 101, 64. Выходные данные 308 можно рассматривать как первый промежуточный свернутый элемент.
[00163] Выход 308 подается как вход во второй сверточный слой 310 сверточной нейронной сети. Второй сверточный слой 310 имеет 128 фильтров, каждый из которых выполняет свертку по выходному сигналу 308 с размером окна 5 и длиной шага 1. За сверткой следует пакетная нормализация и слои 312 нелинейности ReLU. Результатом является вывод (карта характеристик ) 314 размерностей 101, 128. Выход 314 можно рассматривать как второй промежуточный свернутый признак, а также как конечный результат сверточной нейронной сети.
[00164] Метод исключения (отсев, Dropout) представляет собой эффективный метод предотвращения переобучения нейронной сети. Он работает путем случайного удаления части нейронов из сети на каждой итерации обучения. Это означает, что выходной сигнал и градиенты выбранных нейронов устанавливаются на ноль, поэтому они не влияют на прямые и обратные проходы. В ФИГ. 3, выпадение выполняется на уровне выпадения 316 с вероятностью 0,5.
[00163] После обработки выходных данных слоем исключения выходные данные сглаживаются с помощью сглаживающим слоем 318, что обеспечивает возможность последующей обработкой полностью связанной нейронной сетью. Сглаживание включает векторизацию выходных данных 314, чтобы он имел либо одну строку, либо один столбец. Например, преобразование выходных данных 314 размерности 101, 128 в сглаженный вектор размерности 1, 12928 (1 строка и 101x128 = 12928 столбцов).
[00164] Сглаженные выходные данные размерности 1,12928 из сглаживающего слоя 318 затем подается в качестве входных данных в полностью связанную нейронную сеть (FCNN). Полностью связанная нейронная сеть имеет два полностью связанных слоя 320 и 328. Первый полностью связанный -связанный слой 320 имеет 128 нейронов, которые полностью связаны с 2 нейронами во втором полностью связанном слое 328. За первым полностью связанным слоем 320 следуют пакетная нормализация, нелинейность ReLU и слои исключения 322 и 326. За вторым полностью связанным слоем 328 следует уровень 330 пакетной нормализации. Уровень 332 классификации (например, softmax) имеет 2 нейрона, которые выводят 2 классификационных оценки или значения правдоподобия 334 того, что конкретный вариант является истинным вариантом или ложным вариантом.
Эффективность определителя вариантов на отложенных данных
[00165] На ФИГ. 4A показаны истинные и ложноположительные графики, которые графически иллюстрируют работу фильтра вариантов на отложенных данных. В наборе отложенных данных содержится 28 000 примеров для валидации, из которых около 14 000 примеров для валидации истинных вариантов (положительные примеры) и 14000 примеров для валидации ложных вариантов (отрицательные примеры). Два графика 410 и 416 показывают эффективность фильтра вариантов 111, когда 28000 примеров для валидации вводятся в качестве входных данных на этапе валидации. На графиках 410 и 416 классификационные баллы нанесены на ось х, которая показывает достоверность обученной модели в прогнозировании истинных вариантов и ложных вариантов как истинно положительных. Таким образом, ожидается, что обученная модель будет давать высокие классификационные оценки для истинных вариантов и низкие классификационные оценки для ложных вариантов. Высота вертикальных столбиков указывает количество валидационных примеров с каждой классификационной оценкой по оси х.
[00166] График 416 показывает, что фильтр вариантов 111 классифицировал более 7000 валидационных примеров ложных вариантов как «истинно положительные с низкой достоверностью» (т. е. классификационная оценка <0.5 (например, 426)), подтверждая, что модель успешно научилась классифицировать отрицательные примеры как ложные варианты. Фильтр вариантов 111 классифицировал некоторые валидационные примеры ложных вариантов как «истинно положительные с высокой степенью достоверности» (например, 468). Это произошло потому, что в данных обучения и / или в отложенных данных наблюдались присутствовали варианты de novo, присутствующие только у одного ребенка, которые были ошибочно помечены как ложные варианты, хотя на самом деле они были истинными.
[00167] График 410 показывает, что фильтр вариантов 111 классифицировал более 11000 валидационных примеров истинных вариантов как «истинно положительные с высокой степенью достоверности» (т.е. классификационная оценка> 0,5), подтверждая, что модель успешно научилась классифицировать положительные примеры как истинные варианты.
[00168] На ФИГ. 4B результаты классификации фильтра вариантов 111 сравниваются с результатами анализа, полученного из наложенного изображения, которое выравнивает риды, произведенные секвенатором, с референсной последовательностью 498. Референсная последовательность 498 содержит гомополимер повторяющихся паттернов, состоящий из единственного основания «Т», отмеченный меткой 494 на ФИГ. 4B. Наложенное изображение показывает, что по меньшей мере семь ридов (помеченных как 455) сообщили об основании «Т» в положении нуклеотида «G» по сравнению с референсным геном 498. Таким образом, есть два возможных результирующих варианта определения для определения основания в этом положении в последовательности: «G» или «T». Эталонная истина из «родословной платиновых геномов» показывает, что никто из родителей, бабушек и дедушек не имеет вариантного нуклеотида в этом положении в соответствующих эталонных последовательностях. Таким образом, определение основания «Т» определяется как «ложноположительный результат», возникший из-за ошибки секвенирования. Кроме того, изображение с наложением показывает, что символы «Ts» появляются только в конце рида 1, что еще раз подтверждает ложность варианта.
[00169] Эффективнсть фильтра вариантов 111 согласуется с приведенным выше анализом, поскольку фильтр вариантов 111 классифицировал нуклеотид в этом положении как ложный вариант с высокой степенью достоверности, как показано в ФИГ. 4B как «P (X is False) = 0,974398. ».
[00170] ФИГ. 4C демонстрирует наслоение 412 ридов секвенирования для примера, который содержит истинный вариант. Риды секвенирования для потомка (помеченные как «NA12881») имеют не более трех нуклеотидов «Т», отмеченных меткой 495. референсная последовательность имеет нуклеотид «C» в этом положении, отмеченный меткой 496. Однако результаты секвенирования матери показывают по меньшей мере семь нуклеотидов «T» в том же положении. Таким образом, это пример примера с истинным вариантом показан на графике 410 в верхнем левом углу. Фильтр вариантов 111 классифицировал этот пример как истинно положительный с низкой оценкой достоверности («P (X is True) = 0,304499»). То есть фильтр вариантов 111 классифицировал целевой нуклеотид как ложный вариант (или слабо классифицируемый как истинный вариант) из-за присутствия повторяющегося сополимерного паттерна «AC» перед положением целевого нуклеотида. Обученная последовательность рассматривает повторяющийся паттерн как потенциальную последовательность-специфичную ошибку (SSE) и там поэтому классифицировала вариант «Т» с низким показателем достоверности.
[00171] ФИГ. 5 демонстрирует пример подготовки входных данных блоком подготовки входных данных 161 с использованием кодирования с одним активным положением для кодирования наложенных нуклеотидных последовательностей, имеющих вариантный нуклеотид в целевом положении для ввода в фильтр вариантом 111. Нуклеотидная последовательность 514, содержащая по меньшей мере 50 нуклеотидов с обеих сторон (слева и справа) вариантного нуклеотида в целевом положении используется для подготовки ввода. Обратите внимание, что нуклеотидная последовательность 514 является частью референсного генома. При кодировании с одним активным положением каждая пара оснований в последовательности кодируется двоичным вектором из четырех битов, причем один из битов является активным (т. е. 1), а другой - 0. Например, T = (1, 0, 0, 0), G = (0, 1, 0, 0), C = (0, 0, 1, 0) и A = (0, 0, 0, 1). В одном из вариантов реализации неизвестный нуклеотид кодируется как N = (0, 0, 0, 0) На рисунке показан пример нуклеотидной последовательности из 101 нуклеотида, представленной с использованием кодирования с одним активным состоянием.
[00172] ФИГ. 6 иллюстрирует подготовку наложенных образцов, созданных блоком подготовки входных данных, путем наложения повторяющихся паттернов на нуклеотидные последовательности. Наложенные образцы хранятся в базе данных 119 наложенных образцов. Этот пример показывает наложенный образец 1, который создается путем наложения гомополимерного повторяющегося паттерна из 7 «А» слева от центрального нуклеотида в целевом положении в наложенном образце. Наложенный образец 2 создается путем наложения одного и того же повторяющегося паттерна из 7 «А» на нуклеотидную последовательность с покрытием центрального нуклеотида. Третий наложенный образец n генерируется путем наложения повторяющегося паттерна из 7 «A» справа от центрального нуклеотида в наложенных образцах.
[00173] Подсистема фильтра вариантов преобразует анализ, выполненный фильтром вариантов 111 в классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом. За подсистемой фильтрации вариантов следует подсистема анализа, в которой анализатор 194 обеспечивает отображение классификационных оценок как распределения для каждого из факторов повтора для поддержки оценку того, что последовательность-специфичные ошибки обуславливаются повторяющихся паттернами. На ФИГ. 7A-7C представлены примеры такого отображения с анализатора 194. ФИГ. 7A с использованием диаграммы «ящик с усами» для идентификации обусловленности последовательность-специфичной ошибки повторяющимся паттерном, наложенным слева от центрального нуклеотида в наложенных образцах.
[00174] Ось Y графического графика показывает распределение оценок классификации, выдаваемых фильтром вариантов, когда наложенные образцы, содержащие различные повторяющиеся паттерны, подавались в фильтр вариантов в качестве входных данных. Ось x показывает коэффициенты повтора (m), примененные к паттерну, порожденному повторяющимся паттерном, подаваемым в качестве входных данных. Рассматриваемые здесь повторяющиеся паттерны представляют собой гомополимеры, полученные с использованием факторов повтора, указанных на оси абсцисс. В примере показаны четыре диаграммы типа «ящик с усами» для каждого отдельного значения коэффициента повтора. Эти четыре графика соответствуют гомополимерным повторяющимся паттернам четырех типов нуклеотидов (G, A, T и C). Каждый повторяющийся паттерн размещается на по меньшей мере 100 нуклеотидных последовательностях для генерации 100 наложенных образцов, подаваемых в качестве входных данных в CNN-фильтра вариантов 111. В другом варианте реализации на один повторяющейся паттерн используется по меньшей мере 200 нуклеотидных последовательностей используются для создания не менее 200 наложенных образцов. Тот же процесс повторяется для создания гомополимерных повторяющихся паттернов для всех факторов повтора, показанных вдоль оси x.
[00175] Графический график в ФИГ. 7A показывает, что более короткие повторяющиеся паттерны (длиной менее 10 нуклеотидов) из единственного основания «G» могут вносить специфичные для последовательности ошибки в идентификацию вариантов. Аналогично, более короткие повторяющиеся паттерны из единственного основания «C» также могут вносить некоторые ошибки, в то время как повторяющиеся паттерны нуклеотидных оснований «A» и «T» с меньшей вероятностью вызовут последовательность-специфичные ошибки, в случае коротких повторяющихся паттернов. Однако более длинные повторяющиеся паттерны (длина более 10 нуклеотидов) всех четырех типов нуклеотидов вызывают больше последовательность-специфичных ошибок.
[00176] ФИГ. 7B представляет собой график типа «ящик с усами», отображающий классификационные оценки как распределение правдоподобия того, что вариантный нуклеотид является истинным вариантом или ложным вариантом, когда повторяющиеся паттерны накладываются на нуклеотидную последовательность справа от центрального нуклеотида в наложенных образцах. В сравнении с ФИГ. 7A, более короткие гомополимерные паттерны из одного нуклеотида «C» с большей вероятностью вызовут ошибку в идентификации истинного варианта. ФИГ. 7C представляет собой график «ящик с усами», отображающий классификационные оценки как распределение правдоподобия того, что вариантный является нуклеотид истинным вариантом или ложным является, когда повторяющиеся паттерны включают центральный нуклеотид (в целевом положении) в наложенных образцах. В сравнении с ФИГ. 7А и 7Б, ФИГ. 7C показывает, что более короткие повторяющиеся паттерны всех четырех типов нуклеотидов с меньшей вероятностью вызовут специфичную для последовательности ошибку в идентификации вариантов.
[00177] ФИГ. 8A - 8C представляют графические графики для идентификации обусловленности последовательность-специфичных ошибок, для случая, когда повторяющиеся паттерны из одного основания (A, C, G или T) накладываются в различных участках нуклеотидной последовательности с получением наложенных образцов. Варьирующие смещения варьируют положение, в котором повторяющиеся паттерны накладываются на нуклеотидные последовательности. Варьирующее смещение измеряется как смещение между исходным положением повторяющихся паттернов и исходным положением нуклеотидных последовательностей. В одном варианте реализации по меньшей мере используется по меньшей мере десять смещений с получением наложенных образцов. Десять - это разумный минимум для генерации наложенных образцов с повторяющимися паттернами при варьирующих смещениях для анализа обусловленности (причин) последовательность-специфичных ошибок.
[00178] ФИГ. 8A представляет собой график “ящик с усами” для идентификации обусловленности последовательность-специфичных ошибок повторяющимися паттернами гомополимеров единственного основания “C” в различных участках нуклеотидной последовательности. Фактор повтора m=15, что означает повторяющийся паттерн представляет собой гомополимер длины 15 из единственного основания “C”. Этот повторяющийся паттерн накладывается на нуклеотидные последовательности, состоящие из 101 нуклеотида для генерации наложенных образцов с различными смещениями. Для каждого значения смещения комбинации повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных , в по меньшей мере 100 наложенных образцов, подаются в на CNN фильтра вариантов ФИГ. 1 На ФИГ. 8A показаны графики «ящик с усами» для смещенных положений в 0, 2, 4, до 84, когда повторяющийся паттерн из 15 оснований одного вида «C» накладывается на нуклеотидные последовательности. Например, когда смещение равно «0», исходное положение повторяющегося паттерна совпадает с исходным положением нуклеотидных последовательностей. При смещении «2» исходное положение повторяющегося паттерна выравнивается с третьим положением (с индексом 2) чтобы наложить повторяющийся паттерн на нуклеотидные последовательности. По мере увеличения смещения накладывающийся повторяющийся паттерн становится ближе к вариантному нуклеотиду в целевом положении нуклеотидной последовательности. В примере, использованном для иллюстрации на ФИГ. 8А, целевой нуклеотид находится в положении с индексом «50», который является центром нуклеотидной последовательности, содержащей 101 нуклеотид. Когда значение смещения превышает 50, повторяющийся паттерн перемещается мимо вариантного нуклеотида и располагается справа от вариантного нуклеотида в целевом положении.
[00179] ФИГ. 8B, 8C и 8D представляют собой аналогичные диаграммы в виде «ящика с усами», как описанные выше, для идентификации обусловленности ошибок, вызываемых повторяющимися гомополимерными паттернами и оснований одного вида «G», «A» и «T», соответственно, в различных участках нуклеотидной последовательности. Фактор повтора m = 15 для каждого из трех повторяющихся паттернов.
[00181] На ФИГ. 9 показано отображение классификационных оценок как распределение правдоподобия того, что вариантный нуклеотид является истинным вариантом или ложным проявлением, когда повторяющиеся гомополимерные паттерны из единственного основания накладываются «перед» и «после» вариантного нуклеотида. Гомополимерные повторяющиеся паттерны накладываются один за другим перед и после вариантных нуклеотидов в целевом положении с получением наложенных образцов. График типа «ящик с усами» 932 показывает классификационные оценки для случая, когда гомополимерный повторяющийся паттерн с одним основанием «G» наложен слева от центрального нуклеотида на нуклеотидной последовательности. Результаты получены для четырех типов нуклеотидов (A, C, G и T) в качестве вариантного нуклеотида в целевом положении, за которым следует гомополимерный повторяющийся паттерн. Результаты показывают, что классификационные оценки различаются в большей степени, если целевой нуклеотид относится к типу «A» и «C».
[00183] Графический график 935 демонстрирует аналогичную визуализацию, но для гомополимерного повторяющегося паттерна с одним основанием «C», наложенным справа от центрального нуклеотида на нуклеотидной последовательности 912. Сравнение графиков типа «ящик с усами» показывает больший разброс классификационных оценок, когда целевой нуклеотид относится к типу «G».
[00184] ФИГ. 10A-10C представляют отображение природных повторяющихся паттернов сополимеров в каждой из последовательностей нуклеотидов, которые вносят вклад в классификацию ложных вариантов. Графические визуализации, представленные в ФИГ. 10A-10C, генерируются с использованием DeepLIFT, представленного Shrikumar et. El. в статье “Not Just a Black Box: Learning Important Features Through Propagating Activation Differences”, доступной по адресу https://arxiv.org/pdf/1605.01713.pdf (источник 1). Реализация модели DeepLIFT представлена по адресу http://github.com/kundajelab/deeplift (источник 2), а дополнительные подробности по реализации DeepLIFT представлены по адресу https://www.biorxiv.org/content/biorxiv/suppl/2017/10/05/105957.DC1/105957-6.pdf (reference 3). Один или несколько природных повторяющихся сополимерных паттернов, включающих вариантный нуклеотид в целевом положении, подаются в качестве входных данных в модель DeepLIFT для создания визуализаций, показанных на ФИГ. 10A - 10C. Выходные данные модели DeepLIFT - это массивы вкладов входных данных в классификацию варианта для вариантного нуклеотида в целевом положении.
[00183] Например, рассмотрим входную последовательность, показанную в графической визуализации 911. Вариантный нуклеотид 916 находится в положении 50 в нуклеотидной последовательности образца, состоящей из 101 нуклеотида. Вариантный нуклеотид в целевом положении фланкирован 50 нуклеотидами с каждой стороны в положениях от 0 до 49 и от 51 до 100 в нуклеотидной последовательности образца. Фильтр вариантов 111 ФИГ. 2 классифицировал вариантный нуклеотид («C») в целевом положении как ложный вариант. Результатом DeepLIFT является визуализация 911, показывающая, что природный повторяющийся паттерн 917 внес наибольший вклад в классификацию вариантного нуклеотида 916. Высоты нуклеотидов указывают их соответствующий вклад в классификацию вариантного нуклеотида. Как показано на графической визуализации 911, самый высокий вклад вносит последовательность нуклеотидов 917, которая представляет собой повторяющийся паттерн, содержащий одно основание «А».
[00184] Матрицы вкладов DeepLIFT имеют ту же форму, что и входные данные, т.е. входная последовательность нуклеотидов, умноженная на 4 для стандартного кодирования с одним активным состояние (представлена на ФИГ.5). Соответственно, DeepLIFT присваивает оценки каждому положении последовательности путем суммирования вкладов входных нейронов, связанных с фиксированным положением последовательности, и связывает эти суммарные вклады с нуклеотидом, присутствующим в этом положении во входной нуклеотидной последовательности образца. Суммарные вклады называются «оценками интерпретации DeepLIFT». В приложении DeepLIFT применяются следующие рекомендуемые передовые практики (представленные в источнике 3 выше). Рассчитывается вклад входных нейронов в предварительную активацию (активацию перед применением окончательной нелинейности) выходного нейрона. Когда выходной слой использует нелинейность softmax, веса, соединяющие фиксированный нейрон предпоследнего слоя с набором выходных нейронов, центрируются по среднему значению. Поскольку нуклеотидные последовательности образцов кодируются с одним активным состоянием, как показано в ФИГ. 5, метод «нормализации веса для ограниченных входов» используется перед преобразованием из Keras в DeepLIFT, как описано в источнике 3 выше.
[00185] Графические визуализации 921, 931 и 941 показывают повторяющиеся паттерны 927, 934 и 946, соответственно, которые вносят наибольший вклад в классификацию вариантного нуклеотида в нуклеотидных последовательностях образца. ФИГ. 10B включает графические визуализации 921, 931, 941 и 951. Обратите внимание, что в этих графических визуализациях повторяющиеся паттерны сополимеров содержат паттерны из двух и более нуклеотидов. Аналогичным образом, на ФИГ. 10C представлены дополнительные примеры графических визуализаций 931, 932, 933 и 934, иллюстрирующие различные повторяющиеся паттерны, которые вносят вклад в классификацию вариантного нуклеотида в целевом положении в соответствующей входной нуклеотидной последовательности.
Компьютерная система
[00186] ФИГ. 11 представляет собой упрощенную блок-схему компьютерной системы 1100, которая может применяться для реализации фильтра вариантов 111, показанного на ФИГ. 1, для идентификации повторяющихся паттернов, которые вызывают последовательность- специфичные ошибки. Компьютерная система 1100 включает по меньшей мере один центральный процессор (CPU) 1172 который взаимодействует с рядом периферийных устройств через подсистему шин 1155. Эти периферийные устройства могут включать в себя подсистему хранения 1110, включающую, например, запоминающее устройство и подсистему хранения файлов 1136, устройства ввода пользовательского интерфейса 1138, устройства вывода пользовательского интерфейса 1176 и подсистему сетевого интерфейса 1174. Устройства ввода и вывода позволяют пользователю взаимодействовать с компьютерной системой 1100. Подсистема сетевого интерфейса 1174 обеспечивает интерфейс с внешними сетями, включая интерфейс с соответствующими интерфейсными устройствами в других компьютерных системах.
[00187] В одном варианте реализации фильтр вариантов 111 на ФИГ. 1 связан с возможностью обмена данными с подсистемой хранения данных 1110 и устройствами ввода пользовательского интерфейса 1138.
[00189] Устройства ввода пользовательского интерфейса 1138 могут включать клавиатуру; указывающие устройства, такие как мышь, трекбол, тачпад или графический планшет; сканер; сенсорный экран (тач-скрин), встроенный в дисплей; устройства звукового ввода, такие как системы распознавания голоса и микрофоны; и другие типы устройств ввода. В общем, использование термина «устройство ввода» предназначено для включения всех возможных типов устройств и способов ввода информации в компьютерную систему 1100.
[00191] Устройства вывода пользовательского интерфейса 1176 могут включать подсистему дисплея, принтер, факсимильный аппарат или невизуальные дисплеи, такие как устройства звукового вывода. Подсистема дисплея может включать в себя электронно-лучевую трубку (ЭЛТ), устройство с плоской панелью, такое как жидкокристаллический дисплей (ЖКД), проекционное устройство или какой-либо другой механизм для создания видимого изображения. Подсистема дисплея может также обеспечивать невизуальный дисплей, такой как устройства звукового вывода. В общем, использование термина «устройство вывода» предназначено для включения всех возможных типов устройств и способов вывода информации из компьютерной системы 1100 пользователю, другой машине или компьютерной системе.
[00192] Подсистема хранения 1110 программы и конструкции данных, которые обеспечивают функциональные возможности некоторых или всех модулей и способов, описанных в данном документе. Подсистема 1178 может представлять собой графические процессоры (GPU) или матрицы, программируемые пользователем (FPGA).
[00193] Подсистема памяти 1122, используемая в подсистеме хранения 1110 может включать ряд запоминающих устройств, включая основную память с произвольным доступом (RAM) 1132 для хранения инструкций и данных во время выполнения программы, и постоянную память (ROM) 1134 , в которой хранятся фиксированные инструкции. Подсистема хранения файлов 1136 , в которой хранятся фиксированные инструкции. Подсистема хранения файлов 1136 в подсистеме хранения 1110, или на других машинах, доступных процессору.
[00194] Подсистема шин 1155 обеспечивает механизм, позволяющий различным компонентам и подсистемам компьютерной системы 1100 определенным образом связываться друг с другом. Хотя подсистема шин 1155 схематично показана как одна шина, альтернативные реализации подсистемы шины могут использовать множество шин.
[00193] Сама компьютерная система 1100 может быть различных типов, включая персональный компьютер, портативный компьютер, рабочую станцию, компьютерный терминал, сетевой компьютер, телевизор, мэйнфрейм, серверную ферму, распределенный набор слабо связанных в сеть компьютеров или любую другую систему обработки данных или пользовательское устройство. В связи с изменчивой природой компьютеров и сетей описание компьютерной системы1100, изображенной на ФИГ. 11 , приведено только в качестве конкретного примера с целью иллюстрации раскрытой технологии. Возможны многие другие конфигурации компьютерной системы, имеющие больше или меньше компонентов, чем компьютерная система 1100 , изображенная на ФИГ. 11.
Корреляция последовательность-специфичных ошибок (SSE)
[00194] ФИГ. 12 иллюстрирует один вариант реализации определения корреляции последовательность - специфичными ошибками (SSEs) с повторяющимися паттернами на основании классификации ложных вариантов.
[00195] Подсистема подготовки входных данных 161 вычислительно накладывает исследуемые повторяющиеся паттерны не множество нуклеотидных последовательностей и выдает наложенные образцы 119. Каждый повторяющийся паттерн представляет конкретный состав нуклеотидов, который имеет конкретную длину и присутствует в наложенном образце в определенном смещенном положении. Каждый наложенный образец содержит положение, считающееся вариантным нуклеотидом. Для каждой комбинации конкретных составов нуклеотидов, конкретной длины и конкретного смещенного положения, вычислительно генерируется набор наложенных образцов.
[00196] Заранее обученная подсистема фильтрации вариантов 111 обрабатывает наложенные образцы 119 сверточной нейронной сетью 200 и, на основании определения нуклеотидных паттернов в наложенных образцах и, на основании определения нуклеотидных паттернов в наложенных образцах 119 сверточными фильтрами сверточной нейронной сети 200, генерирует классификационные оценки 334 для правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом.
[00197] Система вывода повторяющихся паттернов 1202 выводит в качестве выходных данных распределения 1212 классификационных оценок 334, склонность заранее обученной подсистемы фильтрации вариантов 111 к ложным классификациям из-за присутствия повторяющихся паттернов.
[00198] Эта подсистема корреляции последовательность-специфичных ошибок 199 указывает, на основании порога 1222, подмножество классификационных оценок как указывающих на ложные классификации вариантов, а повторяющиеся паттерны 1232, которые ассоциированы с указанным множеством классификационных оценок, которые указывают на ложную классификацию вариантов, как вызывающие последовательность-специфичные ошибки. Подсистема корреляции последовательность-специфичных ошибок 199 классифицирует конкретные длины и конкретные смещенные положения повторяющихся паттернов 1232, классифицированных как вызывающие последовательность-специфичные ошибки, также как вызывающие последовательность-специфичные ошибки.
[00199] На фигурах 7A, 7B и 7C показан пример порога 702 (например, 0.6), который применяется к распределениям на выходе 1212 классификационных оценок 334 для идентификации подмножества классификационных оценок, которые превышают порог 702. Такие классификационные оценки указывают на ложные классификации вариантов, а повторяющиеся паттерны, ассоциированные с ними, классифицируются как вызывающие последовательность-специфичные ошибки.
Частные варианты реализации
[00200] Раскрытая технология относится к идентификации повторяющихся паттернов, которые вызывают последовательность- специфичные ошибки.
[00201] Раскрытая технология может быть реализована как система, способ, продукт, компьютерочитаемый носитель или изделие. Один или более признаков одного из вариантов реализации могут быть объединены с базовым вариантом реализации. Подразумевается, что варианты, которые не являются взаимоисключающими, совместимы. Один или более признаков варианта реализации можно комбинировать с другими вариантами реализации. Настоящее описание периодически напоминает пользователю об этих опциях. То, что в некоторых вариантах реализации пропущено упоминание об этих опциях, не следует воспринимать как ограничение комбинаций, описанных в предыдущих разделах - в настоящем тексте такое описание включается посредством ссылки в каждый их последующих вариантов реализации.
[00202] Первый вариант реализации системы согласно раскрытой технологии включает один или более процессоров, связанных с памятью. В память загружены компьютерные команды по идентификации повторяющихся паттернов, которые вызывают последовательность- специфичные ошибки. Система включает подсистему подготовки входных данных, реализуемую на множестве процессоров, работающих параллельно и связанных с памятью. Подсистема подготовки входных данных накладывает исследуемые повторяющиеся паттерны на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Повторяющиеся паттерны представляют собой гомополимеры единственного основания (A, C, G или T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанного единственного основания в повторяющихся паттернах. Система включает подсистему моделирования, которая подает каждую комбинацию повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа. Система включает систему фильтрации вариантов, которая преобразует анализ, выполненный фильтром вариантов в классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом. Наконец, система включает подсистему анализа, которая обеспечивает отображение классификационных оценок как распределения для каждого из факторов повторения для поддержки оценки обусловленности последовательность-специфичных ошибок повторяющимися паттернами.
[00203] Этот вариант реализации системы и другие раскрытые системы необязательно включают один или более из следующих признаков. Система может также включать признаки, описанные в связи с раскрытыми способами. Для краткости альтернативные комбинации признаков системы не приводятся по отдельности. Признаки, связанные с системами, способами и изделиями, не повторяются для каждого заявленного набора классов основных признаков. Читатель поймет, как признаки, идентифицированные в этом разделе, можно легко объединять с основными признаками в других заявленных классах.
[00204] В одном варианте реализации повторяющиеся паттерны находятся справа от центрального нуклеотида в перекрытых образцах и не перекрывают центральный нуклеотид. В другом варианте реализации повторяющиеся паттерны находятся слева от центрального нуклеотида в перекрытых образцах и не перекрывают центральный нуклеотид. В другом варианте реализации the повторяющиеся паттерны включают центральный нуклеотид в наложенных образцах.
[00205] Факторы повторов представляют собой целый числа в диапазоне от 5 до одной четверти от числа нуклеотидов в наложенных образцах. Система дополнительно сконфигурирована для применения к повторяющимся паттернам, которые представляют собой гомополимеры единственного основания для каждого из четырех оснований (A, C, G и T).
[00206] Подсистема подготовки входных данных дополнительно сконфигурирована чтобы выдавать повторяющиеся паттерны и наложенные образы для гомополимеров для каждого из четырех оснований, а подсистема анализа дополнительно сконфигурирована чтобы обеспечивать отображение распределения классификационных оценок для каждого из гомополимеров в смежном положении.
[00207] Повторяющиеся паттерны расположены справа от центрального нуклеотида в наложенных образцах и смежное положение применяется к гомополимерам, наложенным справа от центрального нуклеотида. Повторяющиеся паттерны расположены слева от центрального нуклеотида в наложенных образцах и смежное положение применяется к гомополимерам, наложенным слева от центрального нуклеотида. Нуклеотидные последовательности, на которые накладываются повторяющиеся паттерны, генерируются случайным образом. Нуклеотидные последовательности, на которые накладываются повторяющиеся паттерны, случайным образом выбираются из природных последовательностей ДНК. Подсистема анализа дополнительно сконфигурирована чтобы обеспечивать отображение классификационных оценок для каждого из факторов повтора при помощи графика “ящик с усами”.
[00208] Фильтр вариантов обучен (натренирован) на по меньшей мере 500000 тренировочных примеров истинных вариантов и по меньшей мере 50000 тренировочных примеров ложных вариантов. Каждый обучающий пример представляет собой нуклеотидную последовательность с вариантным нуклеотидом в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Фильтр вариантов представляет собой сверточную нейронную сеть (CNN) с двумя сверточными слоями и полностью связанным слоем.
[00209] Другие варианты осуществления могут включать компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению функций описанной выше системы. Еще один вариант реализации может включать способ реализации функций описанной выше системы.
[00210] Первый вариант реализации реализуемого с применением компьютера способа согласно раскрытой технологии включает идентификацию повторяющихся паттернов, которые вызывают последовательность- специфичные ошибки. Реализуемый с применением компьютера способ включает подготовку входных данных путем исследуемых накладывания повторяющихся паттернов на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Повторяющиеся паттерны представляют собой гомополимеры единственного основания (A, C, G или T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанного единственного основания в повторяющихся паттернах. Реализуемый с применением компьютера способ включает подачу каждой комбинации повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа. Реализуемый с применением компьютера способ включает преобразования анализа, выполненного фильтром вариантов, в оценки правдоподобия того, что каждый вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом в выходных данных. Наконец, реализуемый с применением компьютера способ включает обеспечение отображения классификационных оценок как распределения для каждого из факторов повторения для поддержки оценки обусловленности последовательность-специфичных ошибок повторяющимися паттернами.
[00211] Каждый из признаков, обсуждаемых в этом разделе этого конкретного варианта реализации, для первого варианта реализации подсистемы, равно применим к этому варианту реализации реализуемого с применением компьютера способа. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00212] Вариант реализации компьютерочитаемого носителя (CRM) включает Компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению реализуемого с использованием компьютера способа, описанного выше. Другой вариант реализации CRM может включать систему, включающую память и один или более процессоров, которые могут исполнять инструкции, хранящиеся в памяти, по исполнению реализуемого с использованием компьютера способа, описанного выше.
[00213] Каждый из признаков, обсуждаемых в этом разделе этого конкретного варианта реализации, для варианта реализации системы, равно применим к этому варианту реализации CRM. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00214] Второй вариант реализации системы согласно раскрытой технологии включает один или более процессоров, связанных с памятью. В память загружены компьютерные команды по идентификации повторяющихся паттернов, которые вызывают последовательность- специфичные ошибки. Система включает систему подготовки входных данных, которая накладывает исследуемые повторяющиеся паттерны с различными сдвигами на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Повторяющиеся паттерны представляют собой гомополимеры единственного основания (A, C, G или T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанного единственного основания в повторяющихся паттернах. Указанные варьирующие сдвиги варьируют положение, в котором повторяющиеся паттерны накладываются на нуклеотидные системы. Варьирующие сдвиги измеряется как смещение между исходным положением повторяющихся паттернов и исходным положением нуклеотидных последовательностей. В одном варианте реализации по меньшей мере десять сдвигов используют для получения наложенных образцов.
[00215] Система дополнительно содержит подсистему моделирования, которая подает каждую комбинацию повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа. Система включает подсистему фильтрации вариантов, которая преобразует анализ, выполненный фильтром вариантов, в классификационные оценки для правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом. Наконец, система включает подсистему анализа, которая обеспечивает отображение классификационных оценок для каждого из факторов повтора для поддержки оценки обусловленности последовательность-специфичных ошибок присутствием повторяющихся паттернов при различных смещениях.
[00216] Другие варианты осуществления могут включать компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению функций описанной выше системы. Еще один вариант реализации может включать способ реализации функций описанной выше системы.
[00217] Второй вариант реализации реализуемого с применением компьютера способа согласно раскрытой технологии включает идентификацию повторяющихся паттернов, которые вызывают последовательность- специфичные ошибки. Способ включает накладывание исследуемых повторяющихся паттернов с различными смещениями на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Повторяющиеся паттерны представляют собой гомополимеры единственного основания (A, C, G или T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанного единственного основания в повторяющихся паттернах. Варьирующие смещения варьируют положение, в котором повторяющиеся паттерны накладываются на нуклеотидные последовательности. Смещение измеряется как смещение между исходным положением повторяющихся паттернов и исходным положением нуклеотидных последовательностей. В одном варианте реализации по меньшей мере десять сдвигов используют для получения наложенных образцов.
[00218] Реализуемый с применением компьютера способ включает подачу каждой комбинации повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа. За этим следует преобразование анализа, выполненного фильтром вариантов, в классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом. Наконец, указанный реализуемый с применением компьютера способ обеспечивает отображение классификационных оценок как распределения для каждого из повторяющихся факторов для поддержки оценки обусловленности последовательность-специфичных оценок присутствием повторяющихся паттернов при различных смещениях.
[00219] Вариант реализации компьютерочитаемого носителя включает (CRM) Компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению реализуемого с использованием компьютера способа, описанного выше. Другой вариант реализации CRM может включать систему, включающую память и один или более процессоров, которые могут исполнять инструкции, хранящиеся в памяти, по исполнению реализуемого с использованием компьютера способа, описанного выше.
[00220] Третий вариант реализации системы согласно раскрытой технологии включает один или более процессоров, связанных с памятью. В память загружены компьютерные команды по идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки. Система включает систему подготовки входных данных, реализуемую на множестве процессоров, работающих параллельно и соединенных с памятью, которая накладывает исследуемые повторяющиеся паттерны на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Указанные повторяющиеся паттерны представляют собой сополимеры по меньшей мере двух оснований из четырех оснований (A, C, G и T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанных по меньшей мере двух оснований в повторяющихся паттернах. Система включает подсистему систему моделирования, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа. Система включает систему фильтрации вариантов, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию. Подсистема фильтрации вариантов преобразует анализ, выполненный фильтром вариантов в классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом. Наконец, система включает подсистему анализа, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию, которая обеспечивает отображение классификационных оценок как распределения для каждого из факторов повторения для поддержки оценки обусловленности последовательность-специфичных ошибок повторяющимися паттернами.
[00221] Этот вариант реализации системы и другие раскрытые системы необязательно включают один или более из следующих признаков. Система может также включать признаки, описанные в связи с раскрытыми способами. Для краткости альтернативные комбинации признаков системы не приводятся по отдельности. Признаки, связанные с системами, способами и изделиями, не повторяются для каждого заявленного набора классов основных признаков. Читатель поймет, как признаки, идентифицированные в этом разделе, можно легко объединять с основными признаками в других заявленных классах.
[00222] Повторяющиеся паттерны представляют комбинаторное перечисление совместных паттернов различных факторов повтора и варьирующих длин паттернов.
[00223] Другие варианты осуществления могут включать компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению функций описанной выше системы. Еще один вариант реализации может включать способ реализации функций описанной выше системы.
[00224] Третий вариант реализации реализуемого с применением компьютера способа согласно раскрытой технологии включает идентификацию повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки. Способ включает накладывание повторяющихся паттернов на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Указанные повторяющиеся паттерны представляют собой сополимеры по меньшей мере двух оснований из четырех оснований (A, C, G и T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанных по меньшей мере двух оснований в повторяющихся паттернах. Способ включает подачу каждой комбинации повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа. Способ включает преобразование анализа, выполненного фильтром вариантов, в классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом. Наконец, способ включает обеспечение отображения классификационных оценок как распределение для каждого из факторов повтора для поддержки оценки обусловленности последовательность-специфичных оценок указанным паттерном повторов.
[00225] Каждый из признаков, обсуждаемых в этом разделе этого конкретного варианта реализации, для третьего варианта реализации системы равно применим к варианту реализации реализуемого с применением компьютера способа. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00226] Вариант реализации компьютерочитаемого носителя включает (CRM) Компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению реализуемого с использованием компьютера способа, описанного выше. Другой вариант реализации CRM может включать систему, включающую память и один или более процессоров, которые могут исполнять инструкции, хранящиеся в памяти, по исполнению реализуемого с использованием компьютера способа, описанного выше.
[00227] Каждый из признаков, обсуждаемых в этом разделе этого конкретного варианта реализации, для третьего варианта реализации системы равно применим к варианту реализации to CRM. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00228] Четвертый вариант реализации системы согласно раскрытой технологии включает один или более процессоров, связанных с памятью. В память загружены компьютерные команды по идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки. Система включает систему подготовки входных данных, реализуемую на множестве процессоров, работающих параллельно и соединенных с памятью, которая накладывает исследуемые повторяющиеся паттерны с различными смещениями на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Указанные повторяющиеся паттерны представляют собой сополимеры по меньшей мере двух оснований из четырех оснований (A, C, G и T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанных по меньшей мере двух оснований в повторяющихся паттернах. Варьирующие смещения варьируют положение, в котором повторяющиеся паттерны накладываются на нуклеотидные последовательности. Варьирующие смещения измеряются как смещение между исходным положением повторяющихся паттернов и исходным положением нуклеотидных последовательностей. В одном варианте реализации по меньшей мере десять сдвигов используют для получения наложенных образцов.
[00229] Система включает подсистему моделирования, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию повторяющихся паттернов. Указанные повторяющиеся паттерны накладываются на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа. Система также включает систему фильтрации вариантов, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию ,, которая преобразует анализ, выполненный фильтром вариантов, в классификационные оценки для правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом. Наконец, система включает подсистему анализа, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию. Указанная подсистема анализа обеспечивает отображение классификационных оценок как распределения для каждого из повторяющихся факторов для поддержки оценки обусловленности последовательность-специфичных оценок присутствием повторяющихся паттернов при различных смещениях.
[00230] Другие варианты осуществления могут включать компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению функций описанной выше системы. Еще один вариант реализации может включать способ реализации функций описанной выше системы.
[00231] Четвертый вариант реализации реализуемого с применением компьютера способа согласно раскрытой технологии включает идентификацию повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки. Реализуемый с применением компьютера способ включает накладывание исследуемых повторяющихся паттернов на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Указанные повторяющиеся паттерны представляют собой сополимеры по меньшей мере двух оснований из четырех оснований (A, C, G и T) с по меньшей мере 6 факторами повторов. Факторы повторов указывают число повторов указанных по меньшей мере двух оснований в повторяющихся паттернах. Варьирующие смещения варьируют положение, в котором повторяющиеся паттерны накладываются на нуклеотидные последовательности. Факторы повторов измеряются как смещение между исходным положением повторяющихся паттернов и исходным положением нуклеотидных последовательностей. В одном варианте реализации по меньшей мере десять сдвигов используют для получения наложенных образцов. Реализуемый с применением компьютера способ включает подачу каждой комбинации повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа. Реализуемый с применением компьютера способ дополнительно включает преобразование анализа, выполненного фильтром вариантов, в оценки правдоподобия того, что каждый вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом. Наконец, реализуемый с применением компьютера способ включает обеспечение отображения классификационных оценок как распределение для каждого из факторов повтора для поддержки оценки обусловленности последовательность-специфичных оценок присутствием повторяющихся паттернов при различных смещениях.
[00232] Вариант реализации компьютерочитаемого носителя включает (CRM) Компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению реализуемого с использованием компьютера способа, описанного выше. Другой вариант реализации CRM может включать систему, включающую память и один или более процессоров, которые могут исполнять инструкции, хранящиеся в памяти, по исполнению реализуемого с использованием компьютера способа, описанного выше.
[00233] Пятый вариант реализации системы согласно раскрытой технологии включает один или более процессоров, связанных с памятью. В память загружены компьютерные команды по идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки. Система включает подсистему подготовки входных данных, реализуемую на множестве процессоров, работающих параллельно и связанных с памятью. Подсистема подготовки входных данных выбирает нуклеотидные последовательности образцов из природных нуклеотидных ДНК-последовательностей. Каждая из нуклеотидных последовательностей образцов содержит один или более природных повторяющихся паттернов сопорлимеров и вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Система включает подсистему моделирования, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию . Подсистема моделирования подает каждую из нуклеотидных последовательностей образцов в фильтр вариантов для анализа.
[00234] Система включает подсистему фильтрации вариантов, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию. Подсистема фильтрации вариантов преобразует анализ, выполненный фильтром вариантов в классификационные оценки правдоподобия того, что вариантный нуклеотид в каждой из последовательностей нуклеотидов образцов является истинным вариантом или ложным вариантом, и обеспечивает наличие активаций параметров фильтра вариантов с учетом анализа. Наконец, система включает подсистему анализа, реализуемую на указанном множестве процессоров, работающих параллельно и соединенных с памятью, которая подает каждую комбинацию. Указанная подсистема анализа анализирует активации параметров фильтра вариантов и обеспечивает отображение и представление природных повторяющихся паттернов сополимеров в каждой из последовательностей нуклеотидов образцов, которые вносят вклад в классификацию ложных вариантов.
[00235] Другие варианты осуществления могут включать компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению функций описанной выше системы. Еще один вариант реализации может включать способ реализации функций описанной выше системы.
[00236] Пятый вариант реализации реализуемого с применением компьютера способа согласно раскрытой технологии включает идентификацию повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки. Реализуемый с применением компьютера способ включает выбор нуклеотидных последовательностей образцов из природных нуклеотидных ДНК-последовательностей. Каждая из нуклеотидных последовательностей образцов содержит один или более природных повторяющихся паттернов сополимеров, и вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Реализуемый с применением компьютера способ включает подачу из нуклеотидных последовательностей образцов, в фильтр вариантов для анализа. Способ включает преобразование анализа, выполненного фильтром вариантов, в классификационные оценки правдоподобия того, что вариантный нуклеотид в каждой из последовательностей нуклеотидов образцов является истинным вариантом или ложным вариантом. Реализуемый с применением компьютера способ обеспечивает наличие активации параметров фильтра вариантов с учетом анализа. Наконец, реализуемый с применением компьютера способ включает активации параметров фильтра вариантов и обеспечение отображения представления природных повторяющихся паттернов сопорлимеров в каждой из последовательностей нуклеотидов образцов, которые вносят вклад в классификацию ложных вариантов.
[00237] Вариант реализации компьютерочитаемого носителя включает (CRM) Компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению реализуемого с использованием компьютера способа, описанного выше. Другой вариант реализации CRM может включать систему, включающую память и один или более процессоров, которые могут исполнять инструкции, хранящиеся в памяти, по исполнению реализуемого с использованием компьютера способа, описанного выше.
[00238] Раскрытая технология представляет систему для идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки.
[00239] Система содержит подсистему подготовки входных данных, которая реализуется на множестве процессоров, работающих параллельно и связанных с памятью. Подсистема подготовки входных данных накладывает исследуемые повторяющиеся паттерны на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Повторяющиеся паттерны включают по меньшей мере одно основание их четырех оснований (A, C, G и T) с по меньшей мере 6 факторами повторов.
[00240] Система содержит подсистему моделирования, реализуемую на множестве процессоров, работающих параллельно и соединенных с памятью. Подсистема моделирования подает каждую комбинацию повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа.
[00241] Система содержит подсистему фильтрации вариантов, реализованную на множестве процессоров, работающих параллельно и связанных с памятью. Подсистема фильтрации вариантов преобразует анализ, выполненный фильтром вариантов в классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом.
[00242] Система содержит подсистему анализа, реализованную на множестве процессоров, работающих параллельно и связанных с памятью. Подсистема анализа обеспечивает отображения классификационных оценок как распределения для каждого из факторов повторения для поддержки оценки обусловленности последовательность-специфичных ошибок повторяющимися паттернами.
[00243] Каждый признак, обсуждаемый в этом разделе, относящемся к конкретному варианту реализации, применительно к первому варианту реализации системы, равно применим к этому варианту реализации системы. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00244] В одном варианте реализации повторяющиеся паттерны представляют собой гомополимеры единственного основания (A, C, G или T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанного единственного основания в повторяющихся паттернах.
[00245] В другом варианте реализации указанные повторяющиеся паттерны представляют собой сополимеры по меньшей мере двух оснований из четырех оснований (A, C, G и T) с по меньшей мере 6 факторами повторов, которые указывают число повторов указанных по меньшей мере двух оснований в повторяющихся паттернах.
[00246] В некоторых вариантах реализации подсистема подготовки входных данных дополнительно сконфигурирована чтобы накладывать исследуемые повторяющиеся паттерны с различными смещениями на нуклеотидные последовательности с получением наложенных образцов. Варьирующие смещения варьируют положение, в котором повторяющиеся паттерны накладываются на нуклеотидные последовательности, что измеряется как смещение между исходным положением повторяющихся паттернов и исходным положением нуклеотидных последовательностей, и по меньшей мере десять сдвигов используют для получения наложенных образцов. В таких вариантах реализации подсистема анализа дополнительно сконфигурирована чтобы обеспечивать отображение классификационных оценок как распределения для каждого из повторяющихся факторов для поддержки оценки обусловленности последовательность-специфичных оценок присутствием повторяющихся паттернов при различных смещениях.
[00247] В одном варианте реализации повторяющиеся паттерны находятся справа от центрального нуклеотида в перекрытых образцах и не перекрывают центральный нуклеотид. В другом варианте реализации повторяющиеся паттерны находятся слева от центрального нуклеотида в перекрытых образцах и не перекрывают центральный нуклеотид. В другом варианте реализации повторяющиеся паттерны включают центральный нуклеотид в наложенных образцах.
[00248] Факторы повторов представляют собой целые числа в диапазоне от 5 до одной четверти от числа нуклеотидов в наложенных образцах. Система дополнительно сконфигурирована для применения к повторяющимся паттернам, которые представляют собой гомополимеры единственного основания для каждого из четырех оснований (A, C, G и T).
[00249] Подсистема подготовки входных данных дополнительно сконфигурирована, чтобы выдавать повторяющиеся паттерны и наложенные образы для гомополимеров для каждого из четырех оснований, а подсистема анализа дополнительно сконфигурирована, чтобы обеспечивать отображение распределения классификационных оценок для каждого из гомополимеров в смежном положении.
[00250] Повторяющиеся паттерны расположены справа от центрального нуклеотида в наложенных образцах и смежное положение применяется к гомополимерам, наложенным справа от центрального нуклеотида. Повторяющиеся паттерны расположены слева от центрального нуклеотида в наложенных образцах и смежное положение применяется к гомополимерам, наложенным слева от центрального нуклеотида. Нуклеотидные последовательности, на которые накладываются повторяющиеся паттерны, генерируются случайным образом. Нуклеотидные последовательности, на которые накладываются повторяющиеся паттерны, случайным образом выбираются из природных последовательностей ДНК. Подсистема анализа дополнительно сконфигурирована, чтобы обеспечивать отображение классификационных оценок для каждого из факторов повтора при помощи графика “ящик с усами”.
[00251] Фильтр вариантов обучен (натренирован) на по меньшей мере 500000 тренировочных примеров истинных вариантов и по меньшей мере 50000 тренировочных примеров ложных вариантов. Каждый обучающий пример представляет собой нуклеотидную последовательность с вариантным нуклеотидом в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны. Фильтр вариантов представляет собой сверточную нейронную сеть (CNN) с двумя сверточными слоями и полностью связанным слоем.
[00252] Раскрытая технология представляет реализуемый с применением компьютера способ идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки.
[00253] Реализуемый с применением компьютера способ включает накладывание исследуемых повторяющихся паттернов на нуклеотидные последовательности с получением наложенных образцов.
[00254] Реализуемый с применением компьютера способ включает подачу каждой комбинации повторяющихся паттернов, наложенных на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах, в фильтр вариантов для анализа.
[00255] Реализуемый с применением компьютера способ включает преобразования анализа, выполненного фильтром вариантов, в оценки правдоподобия того, что каждый вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом.
[00256] Реализуемый с применением компьютера способ включает обеспечение отображения классификационных оценок как распределения для каждого из факторов повторения для поддержки оценки обусловленности последовательность-специфичных ошибок повторяющимися паттернами.
[00257] Каждый из признаков, обсуждаемых в этом разделе этого конкретного варианта реализации, для первого варианта реализации подсистемы, равно применим к этому варианту реализации реализуемого с применением компьютера способа. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00258] Раскрытая технология представляет еще одну систему для идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования. Система содержит один или более процессоров и одно или более устройств хранения, хранящих инструкции, которые, при исполнении на указанных одном или более процессорах, заставляет указанные один или более процессоров реализовать подсистему подготовки входных данных, подсистему фильтра вариантов, и подсистему вывода повторяющихся паттернов.
[00261] Подсистема подготовки входных данных сконфигурирована чтобы накладывать исследуемые повторяющиеся паттерны на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид, а повторяющиеся паттерны включают по меньшей мере одно основание из четырех оснований (A, C, G и T).
[00262] Подсистема фильтрации вариантов сконфигурирована, чтобы обрабатывать каждую комбинацию повторяющихся паттернов, наложенных на нуклеотидные последовательности, в наложенных образцах с генерацией классификационных оценок правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом.
[00263] Система вывода повторяющихся паттернов сконфигурирован чтобы выводить конкретные повторяющиеся паттерны, которые вызывают последовательность- специфичные ошибки в данных нуклеотидного секвенирования на основании классификационных оценок.
[00264] Каждый признак, обсуждаемый в этом разделе, относящемся к конкретному варианту реализации, применительно к первому варианту реализации системы, равно применим к этому варианту реализации системы. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00263] Система дополнительно сконфигурирована таким образом, что она содержит подсистему анализа, которая сконфигурирована, чтобы отображать классификационные оценки как распределения для каждого из факторов повторения для поддержки оценки обусловленности последовательность-специфичных ошибок повторяющимися паттернами.
[00264] Вариант реализации компьютерочитаемого носителя включает (CRM) Компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению реализуемого с использованием компьютера способа, описанного выше. Другой вариант реализации CRM может включать систему, включающую память и один или более процессоров, которые могут исполнять инструкции, хранящиеся в памяти, по исполнению реализуемого с использованием компьютера способа, описанного выше.
[00265] Раскрытая технология представляет собой другую систему идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования. Система включает в себя один или более процессоров и одно или более устройств хранения, хранящих инструкции, которые при исполнении, заставляют один или более процессоров реализовать подсистему подготовки входных данных, подсистему фильтра вариантов, и подсистему вывода повторяющихся паттернов.
[00266] Подсистема подготовки входных данных сконфигурирована, чтобы накладывать исследуемые повторяющиеся паттерны на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид, и повторяющиеся паттерны включают по меньшей мере одно основание их четырех оснований (A, C, G и T).
[00267] Подсистема фильтрации вариантов сконфигурирована, чтобы обрабатывать каждую комбинацию повторяющихся паттернов, наложенных на нуклеотидные последовательности, в наложенных образцах чтобы генерировать классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом.
[00269] Система вывода повторяющихся паттернов сконфигурирована чтобы выводить конкретные повторяющиеся паттерны, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования на основании классификационных оценок.
[00271] Каждый признак, обсуждаемый в этом разделе, относящемся к конкретному варианту реализации, применительно к первому варианту реализации системы, равно применим к этому варианту реализации системы. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00272] Система дополнительно сконфигурирована таким образом, что она содержит подсистему анализа, которая сконфигурирована, чтобы обеспечивать отображение классификационных баллов как распределения для каждого из факторов повторения для поддержки оценки обусловленности последовательность-специфичных ошибок повторяющимися паттернами.
[00273] Раскрытая технология представляет реализуемый с применением компьютера способ идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования.
[00274] Реализуемый с применением компьютера способ включает накладывание исследуемых повторяющихся паттернов на нуклеотидные последовательности с получением наложенных образцов. Каждый из наложенных вариантов содержит вариантный нуклеотид, а повторяющиеся паттерны включают по меньшей мере одно из четырех оснований (A, C, G и T).
[00273] Реализуемый с применением компьютера способ включает обработку каждой комбинации повторяющихся паттернов, наложенных на нуклеотидные последовательности, в наложенных образцах подсистемой фильтра вариантов для генерации классификационных оценок правдоподобия того, что указанный вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом.
[00274] Реализуемый с применением компьютера способ включает преобразования анализа, выполненного фильтром вариантов, в оценки правдоподобия того, что каждый вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом.
[00275] Реализуемый с применением компьютера способ включает вывод определенных повторяющихся паттернов, которые вызывают последовательность- специфичные ошибки в данных нуклеотидного секвенирования на основании классификационных оценок.
[00276] Каждый из признаков, обсуждаемых в этом разделе этого конкретного варианта реализации, для первого варианта реализации подсистемы, равно применим к этому варианту реализации реализуемого с применением компьютера способа. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00277] Вариант реализации компьютерочитаемого носителя (CRM) включает Компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды по исполнению реализуемого с использованием компьютера способа, описанного выше. Другой вариант реализации CRM может включать систему, включающую память и один или более процессоров, которые могут исполнять инструкции, хранящиеся в памяти, по исполнению реализуемого с использованием компьютера способа, описанного выше.
[00278] Раскрытая технология представляет еще одну систему для идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования. Эта система содержит один или более процессоров и одно или более устройств хранения, хранящих инструкции, которые, при исполнении на указанных одном или более процессорах, заставляют указанные один или более процессоров реализовать подсистему подготовки входных данных, подсистему фильтра вариантов, и подсистему вывода повторяющихся паттернов.
[00279] Подсистема подготовки входных данных сконфигурирована чтобы из природных нуклеотидных ДНК-последовательностей. Каждая из нуклеотидных последовательностей образцов содержит один или более природных повторяющихся паттернов сополимеров и вариантный нуклеотид.
[00280] Подсистема фильтрации вариантов сконфигурирована чтобы обрабатывать каждую из нуклеотидных последовательностей образцов с генерацией классификационных оценок правдоподобия того, что вариантный нуклеотид в каждой из последовательностей нуклеотидов образцов является истинным вариантом или ложным вариантом.
[00281] Система вывода повторяющихся паттернов сконфигурирована для обеспечения наличия активации параметров подсистемы фильтра вариантов в учетом анализа и вывода определенных повторяющихся паттернов, которые вызывают последовательность- специфичные ошибки в данных нуклеотидного секвенирования на основании классификационных оценок.
[00282] Каждый признак, обсуждаемый в этом разделе, относящемся к конкретному варианту реализации, применительно к первому варианту реализации системы, равно применим к этому варианту реализации системы. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00283] Система дополнительно сконфигурирована таким образом, что она содержит подсистему анализа, которая сконфигурирована для анализа активации параметров подсистемы фильтра вариантов и обеспечения отображения и представления природных повторяющихся паттернов сопорлимеров в каждой из последовательностей нуклеотидов образцов, которые вносят вклад в классификацию ложных вариантов.
[00284] Раскрытая технология представляет реализуемый с применением компьютера способ идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования.
[00285] Реализуемый с применением компьютера способ включает выбор нуклеотидных последовательностей образцов из природных нуклеотидных ДНК-последовательностей. Каждая из нуклеотидных последовательностей образцов содержит один или более природных повторяющихся паттернов сополимеров и вариантный нуклеотид.
[00286] Реализуемый с применением компьютера способ включает обработку каждой из нуклеотидных последовательностей образцов подсистемой фильтра вариантов для генерации классификационных оценок правдоподобия того, что указанный вариантный нуклеотид в каждой из последовательностей нуклеотидов образцов является истинным вариантом или ложным вариантом.
[00287] Реализуемый с применением компьютера способ включает обеспечение активаций параметров подсистемы фильтра вариантов, с учетом анализа.
[00288] Реализуемый с применением компьютера способ включает вывод определенных повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования, но основании классификационных оценок.
[00291] Каждый из признаков, обсуждаемых в этом разделе этого конкретного варианта реализации, для первого варианта реализации подсистемы, равно применим к этому варианту реализации реализуемого с применением компьютера способа. Как указано выше, здесь не повторяются все признаки системы, подразумевается, что они повторяются посредством ссылки.
[00292] Вариант реализации компьютерочитаемого носителя включает (CRM) компьютерочитаемый носитель долговременного хранения информации, на котором хранятся исполняемые процессором команды для исполнения реализуемого с использованием компьютера способа, описанного выше. Другой вариант реализации CRM может включать систему, включающую память и один или более процессоров, которые могут исполнять инструкции, хранящиеся в памяти, по исполнению реализуемого с использованием компьютера способа, описанного выше.
[00293] Любые структуры данных и код, описанные или упомянутые выше, хранятся согласно многим вариантам реализации на машиночитаемом носителе данных, который может быть любым устройством или носителем, который может хранить код и / или данные для использования компьютерной системой. Это включает, помимо прочего, энергозависимую память, энергонезависимую память, специализированные интегральные схемы (ASIC), программируемые вентильные матрицы (FPGA), магнитные и оптические запоминающие устройства, такие как дисководы, магнитные ленты, компакт-диски ( компакт-диски), DVD (универсальные цифровые диски или цифровые видеодиски) или другие носители, способные хранить машиночитаемые носители, известные в настоящее время или разработанные позже.
[00294] Предшествующее описание приведено для того, чтобы сделать возможным создание и применение раскрытой технологии. Различные модификации раскрытых вариантов реализации будут очевидны, и общие принципы, определенные в данном документе, могут быть применены к другим вариантам реализации и приложениям без отступления от сущности и объема раскрытой технологии. Таким образом, не предполагается, что раскрытая технология ограничена показанными вариантами реализации, она должна соответствовать самому широкому объему, согласующемуся с принципами и признаками, раскрытыми в данном документе. Объем раскрытой технологии определяется прилагаемой формулой изобретения.
Притязания
[00293] Настоящее раскрытие также включает следующие притязания:
1. Система для идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования, включающая:
один или более процессоров и одно или более устройств хранения, хранящих инструкции, которые, при исполнении на указанных одном или более процессорах, обеспечивают реализацию указанными одним или более процессорами:
подсистемы подготовки входных данных, сконфигурированной чтобы:
вычислительно накладывать исследуемые повторяющиеся паттерны на множество нуклеотидных последовательностей и получать наложенные образцы,
причем каждый повторяющийся паттерн представляет конкретный состав нуклеотидов, который имеет конкретную длину и присутствует в наложенном образце в определенном смещенном положении,
причем каждый наложенный образец содержит положение, считающееся вариантным нуклеотидом, и
при этом для каждой комбинации конкретного состава нуклеотидов, конкретной длины и конкретного смещенного положения, вычислительно генерируется набор наложенных образцов;
заранее обученной подсистемы фильтра вариантов, сконфигурированной чтобы:
обрабатывать наложенные образцы сверточной нейронной сетью и, на основании выявления нуклеотидных паттернов в наложенных образцах сверточными фильтрами сверточной нейронной сети, генерировать классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом;
подсистему вывода повторяющихся паттернов, сконфигурированную чтобы:
выводить распределения классификационных оценок, которые показывают склонность заранее обученной системы фильтра вариантов к ложной классификации, обусловленной присутствием повторяющихся паттернов; и
подсистемы корреляции последовательность-специфичных ошибок, сконфигурированной чтобы:
указывать, на основании порога, подмножество классификационных оценок, как указывающих на ложную классификацию вариантов, и
классифицировать повторяющиеся паттерны, которые ассоциированы с этим поднабором классификационных оценок, которые указывают на ложную классификацию вариантов, как вызывающие последовательность-специфичные ошибки.
2. Система по п. 1, где указанная система корреляции последовательность-специфичных ошибок дополнительно сконфигурирована чтобы:
классифицировать конкретные длины и конкретные смешенные положения повторяющихся паттернов, классифицированных как вызывающие последовательность-специфичные ошибки, также как вызывающие последовательность-специфичные ошибки.
3. Система по любому из пп. 1-2, где указанный вариантный нуклеотид представляет собой целевое положение, фланкированное по меньшей мере 20 нуклеотидами с каждой стороны.
4. Система по любому из пп. 1-3, где указанная заранее обученная подсистема фильтра вариантов сконфигурирована чтобы обрабатывать каждую комбинацию повторяющихся паттернов, накладываемых на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах.
5. Система по любому из пп. 1-5, где повторяющиеся паттерны включают по меньшей мере одно основание из четырех оснований (A, C, G и T) с по меньшей мере 6 факторами повторов.
6. Система по п. 5, где повторяющиеся паттерны представляют собой гомополимеры единственного основания (A, C, G или T) с по меньшей мере 6 факторами повторов; и
где указанные по меньшей мере 6 факторов повторов указывают число повторов указанного единственного основания в повторяющихся паттернах.
7. Система по любому из пп. 1-6, где указанные повторяющиеся паттерны представляют собой сополимеры по меньшей мере двух оснований из четырех оснований (A, C, G и T) с по меньшей мере 6 факторами повторов; и
где указанные по меньшей мере 6 факторов повторов указывают число повторов указанных по меньшей мере двух оснований в повторяющихся паттернах.
8. Система по любому из пп. 1-7, где указанные смещенные положения варьируют в смысле положения, в котором указанные повторяющиеся паттерны накладываются на нуклеотидные последовательности, что измеряется как смещение между исходным положением повторяющихся паттернов и исходным положением нуклеотидных последовательностей, и по меньшей мере десять сдвигов используют для получения наложенных образцов.
9. Система по любому из пп. 1-8, где повторяющиеся паттерны находятся справа от центрального нуклеотида в перекрытых образцах и не перекрывают центральный нуклеотид.
10. Система по любому из пп. 1-9, где повторяющиеся паттерны находятся слева от центрального нуклеотида в перекрытых образцах и не перекрывают центральный нуклеотид.
11. Система по любому из пп. 1-10, где указанные повторяющиеся паттерны включают центральный нуклеотид в наложенных образцах.
12. Система по любому из пп. 1-11, где указанные факторы повтора представляют собой целые числа в диапазоне от 5 до одной четверти от числа нуклеотидов в наложенных образцах.
13. Система по п. 6, дополнительно сконфигурированная для применения к повторяющимся паттернам, которые представляют собой гомополимеры единственного основания для каждого из четырех оснований (A, C, G и T).
14. Система по п. 13, где указанная подсистема подготовки входных данных дополнительно сконфигурирована чтобы выдавать повторяющиеся паттерны и наложенные образы для гомополимеров для каждого из четырех оснований.
15. Система по п. 14, где повторяющиеся паттерны расположены справа от центрального нуклеотида в наложенных образцах и смежное положение применяется к гомополимерам, наложенным справа от центрального нуклеотида.
16. Система по п. 14, где повторяющиеся паттерны расположены слева от центрального нуклеотида в наложенных образцах и смежное положение применяется к гомополимерам, наложенным слева от центрального нуклеотида.
17. Система по любому из пп. 1-16, где нуклеотидные последовательности, на которые накладываются повторяющиеся паттерны, генерируются случайным образом.
18. Система по любому из пп. 1-17, где нуклеотидные последовательности, на которые накладываются повторяющиеся паттерны, случайным образом выбираются из природных последовательностей ДНК.
19. Система по любому из пп. 1-18, где подсистема анализа сконфигурирована чтобы отображать распределения классификационных оценок для каждого из факторов повтора.
20. Система по любому из пп. 1-19, где указанная заранее обученная система фильтрации вариантов обучена на по меньшей мере 500000 тренировочных примеров истинных вариантов и по меньшей мере 50000 тренировочных примеров ложных вариантов; и
где каждый обучающий пример представляет собой нуклеотидную последовательность с вариантным нуклеотидом в целевом положении, фланкированный по меньшей мере 20 нуклеотидами с каждой стороны.
21. Система по любому из пп. 1-20, где указанная заранее обученная подсистема фильтрации вариантов содержит сверточные слои, полностью связанные слои и слои классификации.
22. Реализуемый с применением компьютера способ идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования, включающий:
вычислительное накладывание исследуемых повторяющихся паттернов на множество нуклеотидных последовательностей и получение наложенных образцов, причем каждый повторяющийся паттерн представляет конкретный состав нуклеотидов, который имеет конкретную длину и присутствует в наложенном образце в определенном смещенном положении, причем каждый наложенный образец содержит положение, считающееся вариантным нуклеотидом, и при этом для каждой комбинации конкретного состава нуклеотидов, конкретной длины и конкретного смещенного положения, вычислительно генерируется набор наложенных образцов;
обработку указанных наложенных образцов сверточной нейронной сетью и, на основании выявления нуклеотидных паттернов в наложенных образцах сверточными фильтрами сверточной нейронной сети, генерацию классификационных оценок для правдоподобия того, что указанный вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом;
вывод распределений классификационных баллов, которые указывают склонность заранее обученной системы фильтра вариантов к ложной классификации, обусловленной присутствием повторяющихся паттернов; и
указание, на основании порога, подмножества классификационных оценок, как указывающих на ложную классификацию вариантов и классификацию повторяющихся паттернов, которые ассоциированы с этим поднабором классификационных оценок, которые указывают на ложную классификацию вариантов, как вызывающие последовательность-специфичные ошибки.
23. Реализуемый с применением компьютера способ по п. 22, применяющий каждый из объектов, который в конечном итоге зависит от объекта по п. 1.
24. Компьютерочитаемый носитель долговременного хранения информации, на который нанесены программные инструкции по идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования, причем указанные инструкции при исполнении процессором реализуют реализуемый с применением компьютера способ, включающий:
вычислительное накладывание исследуемых повторяющихся паттернов на множество нуклеотидных последовательностей и получение наложенных образцов, причем каждый повторяющийся паттерн представляет конкретный состав нуклеотидов, который имеет конкретную длину и присутствует в наложенном образце в определенном смещенном положении, причем каждый наложенный образец содержит положение, считающееся вариантным нуклеотидом, и при этом для каждой комбинации конкретного состава нуклеотидов, конкретной длины и конкретного смещенного положения, вычислительно генерируется набор наложенных образцов;
обработку указанных наложенных образцов сверточной нейронной сетью и, на основании выявления нуклеотидных паттернов в наложенных образцах сверточными фильтрами сверточной нейронной сети, генерацию классификационных оценок для правдоподобия того, что указанный вариантный нуклеотид в каждом из наложенных вариантов является истинным вариантом или ложным вариантом;
вывод распределений классификационных баллов, которые указывают склонность заранее обученной системы фильтра вариантов к ложной классификации, обусловленной присутствием повторяющихся паттернов; и
указание, на основании порога, подмножества классификационных оценок, как указывающих на ложную классификацию вариантов и классификацию повторяющихся паттернов, которые ассоциированы с этим поднабором классификационных оценок, которые указывают на ложную классификацию вариантов, как вызывающие последовательность-специфичные ошибки.
25. Компьютерочитаемый носитель долговременного хранения информации по п. 24, применяющий каждый из объектов, который в конечном итоге зависит от объекта по п. 1.
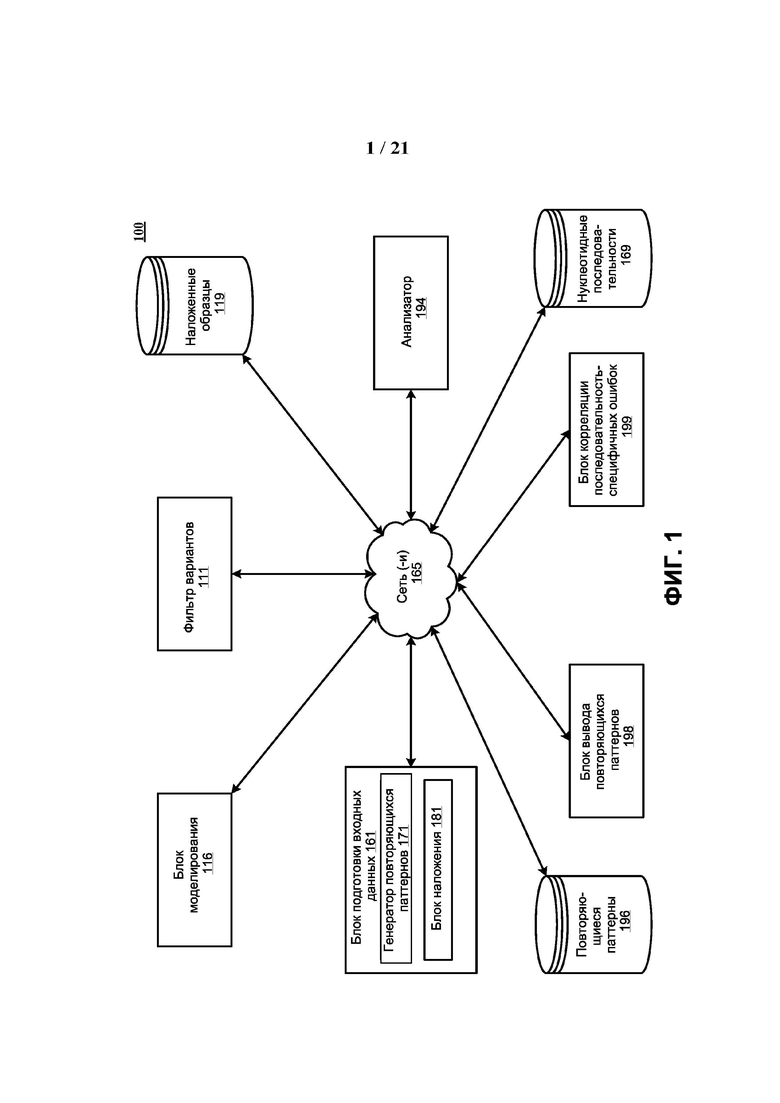

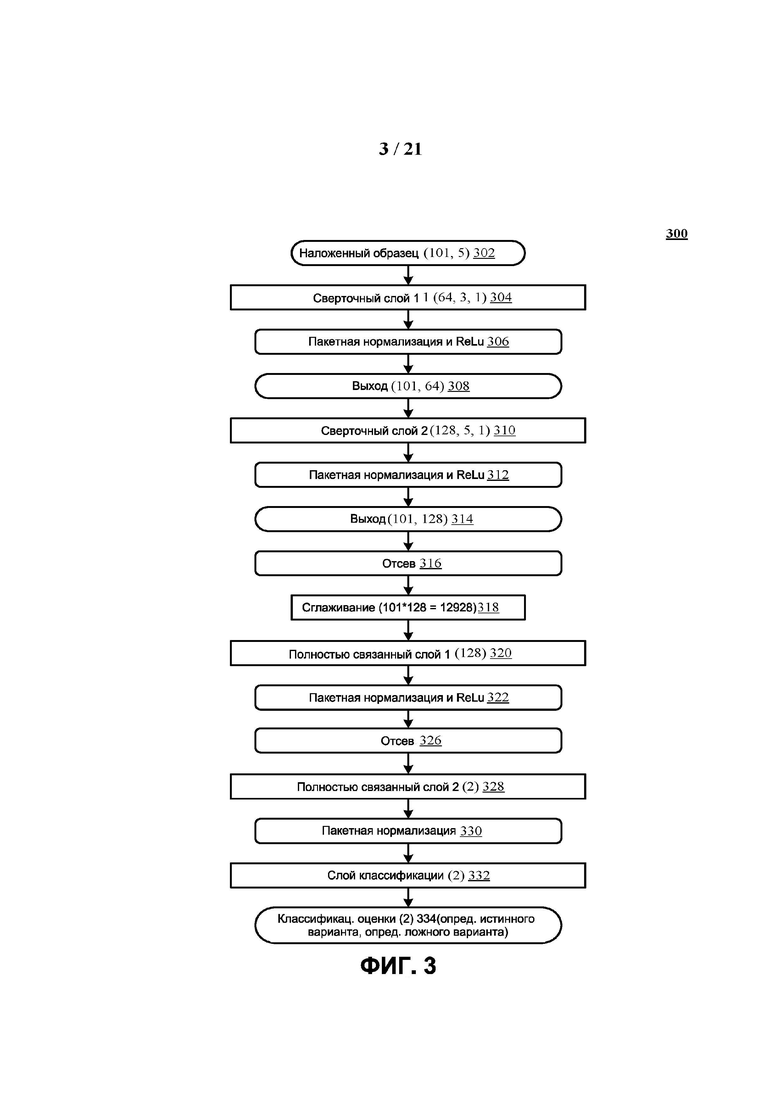




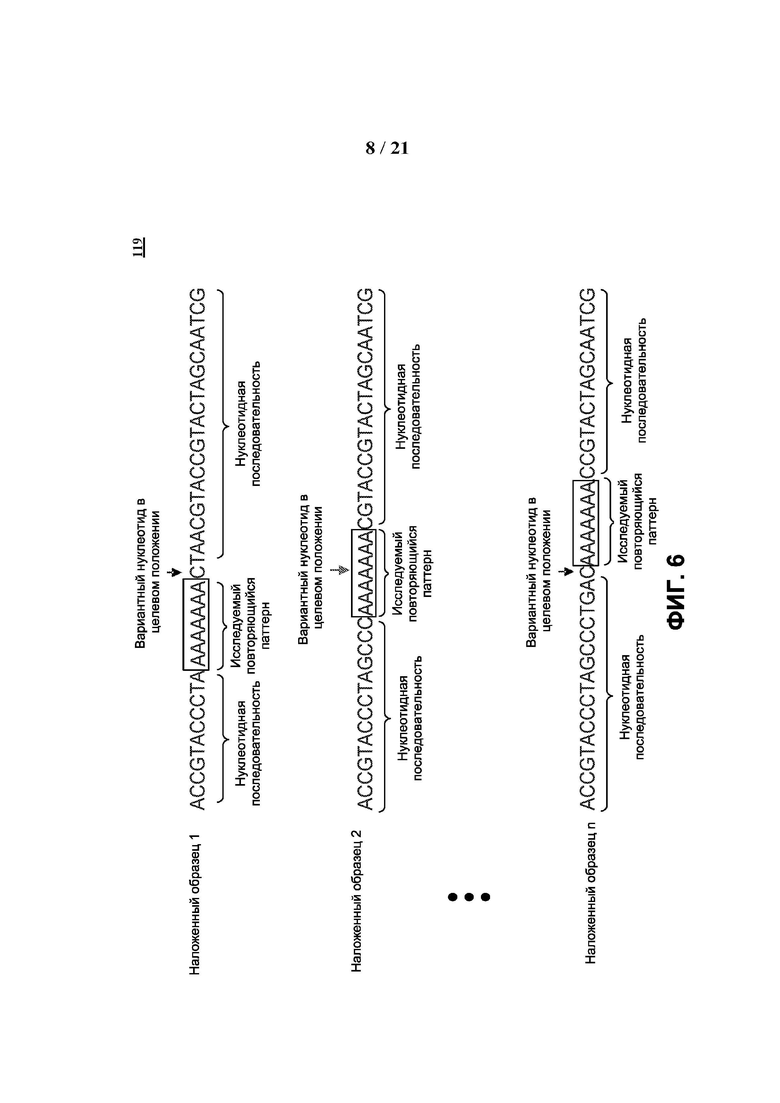






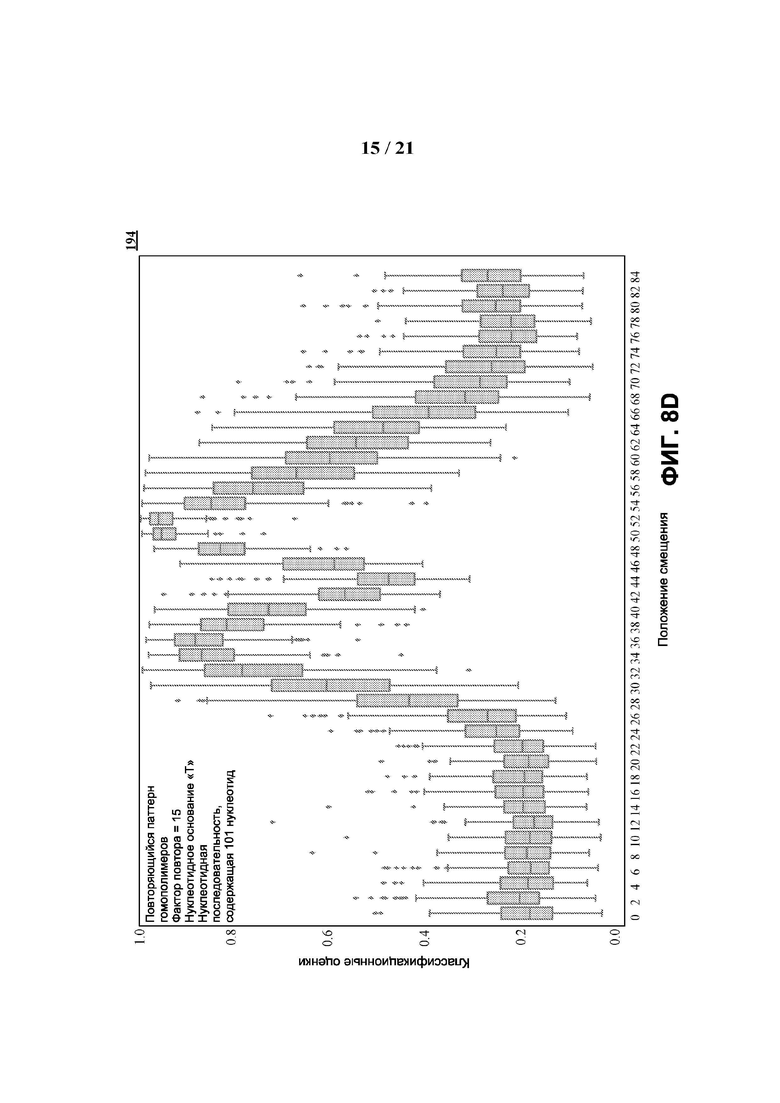

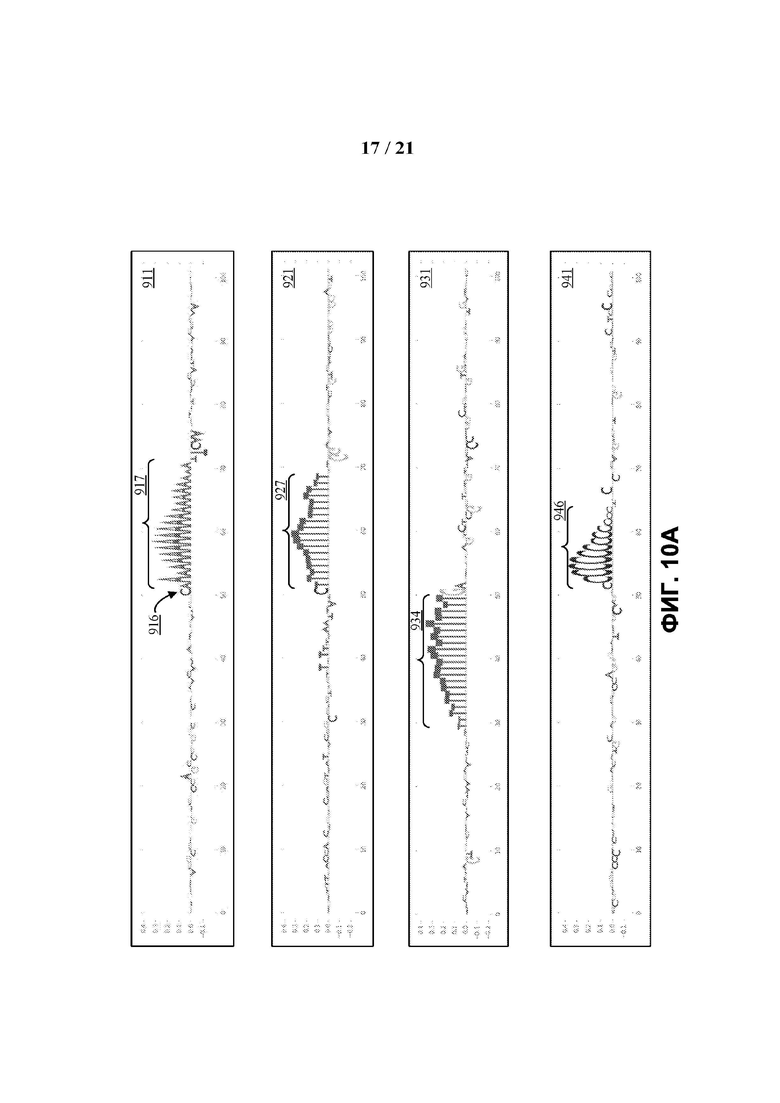
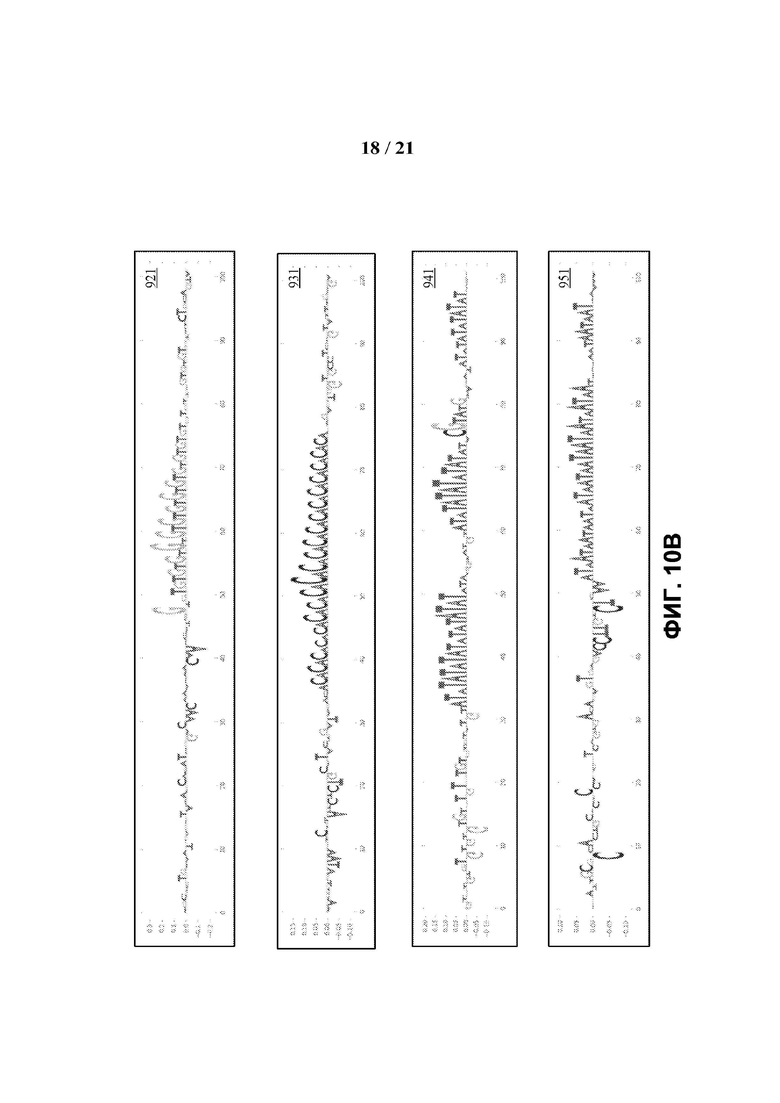

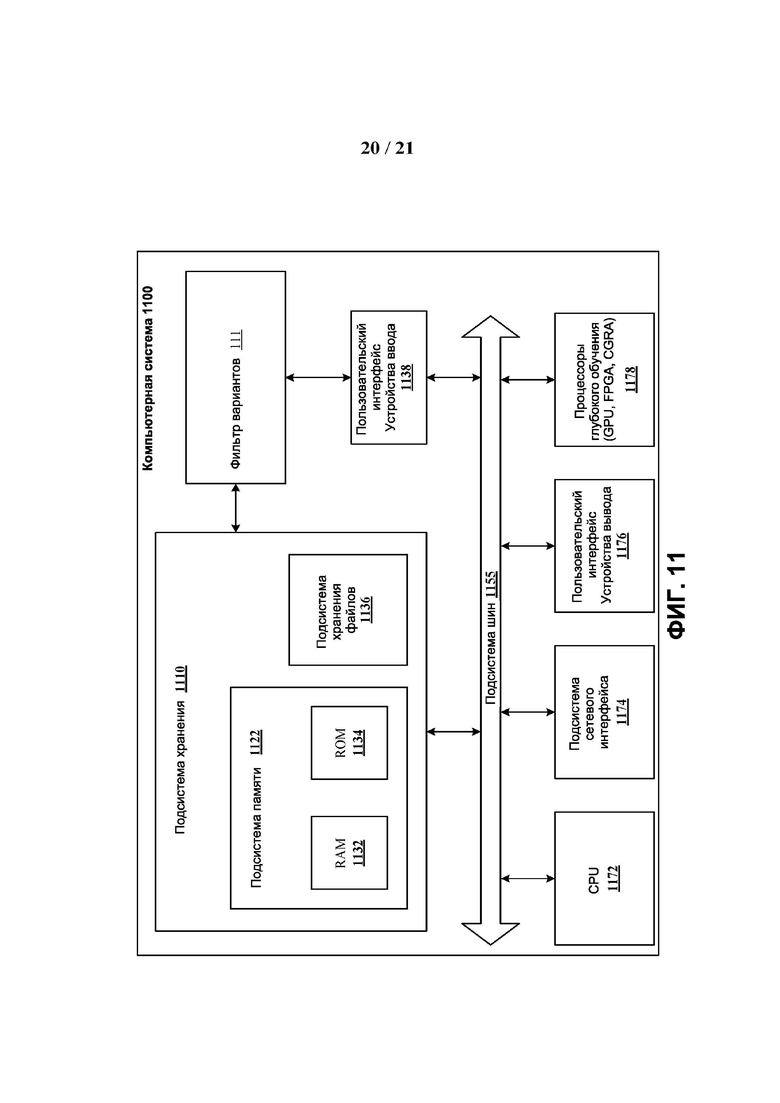
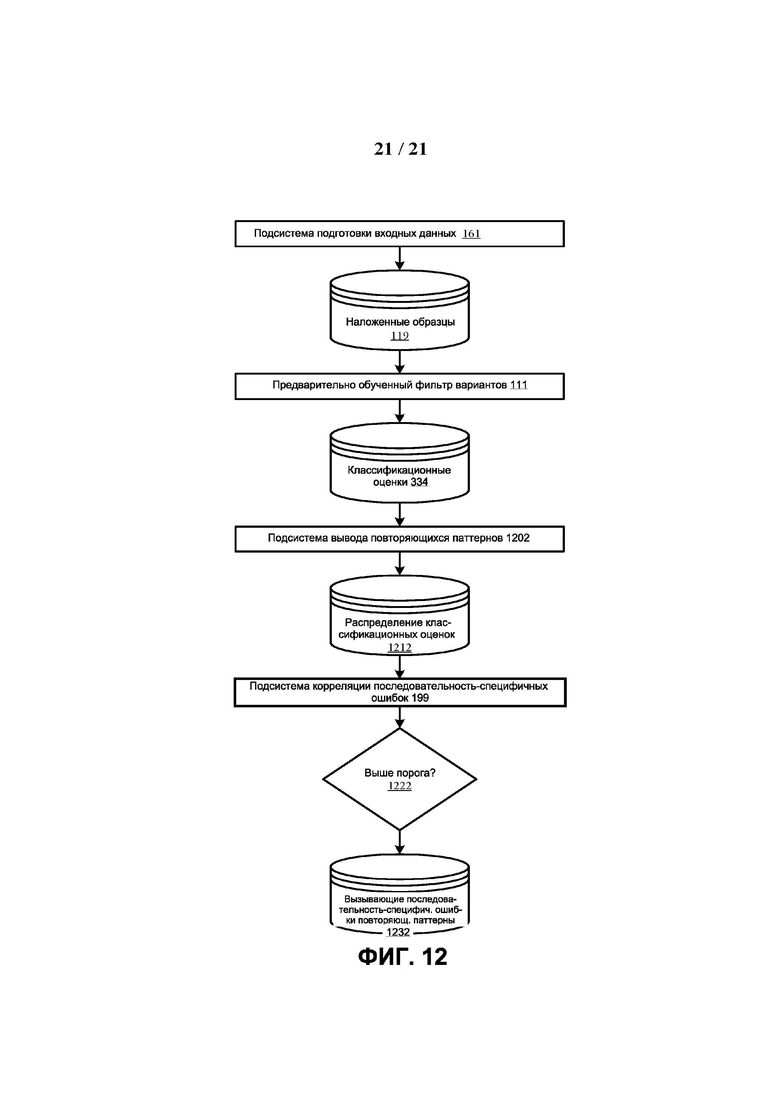
Изобретение относится к области вычислительной техники, в частности к компьютерам и цифровым системам обработки данных, относящихся к типу искусственного интеллекта. Технический результат заключается в минимизации ошибок обучения сверточной нейронной сети. Технический результат достигается за счет обработки наложенных образцов сверточной нейронной сетью и на основании выявления нуклеотидных паттернов в наложенных образцах сверточными фильтрами сверточной нейронной сети, генерации классификационных оценок для правдоподобия того, что указанный вариантный нуклеотид в каждом из наложенных образцов является истинным вариантом или ложным вариантом; вывода распределений классификационных баллов, сгенерированных заранее обученной подсистемой фильтра вариантов для факторов повтора соответствующих повторяющихся паттернов; и указания, на основании порога, подмножества классификационных оценок в указанных распределениях как указывающих на ложные классификации вариантов и классификации повторяющихся паттернов, которые ассоциированы с этим подмножеством классификационных оценок, которые указывают на ложные классификации вариантов, как вызывающих последовательность-специфичные ошибки. 3 н. и 22 з.п. ф-лы, 21 ил.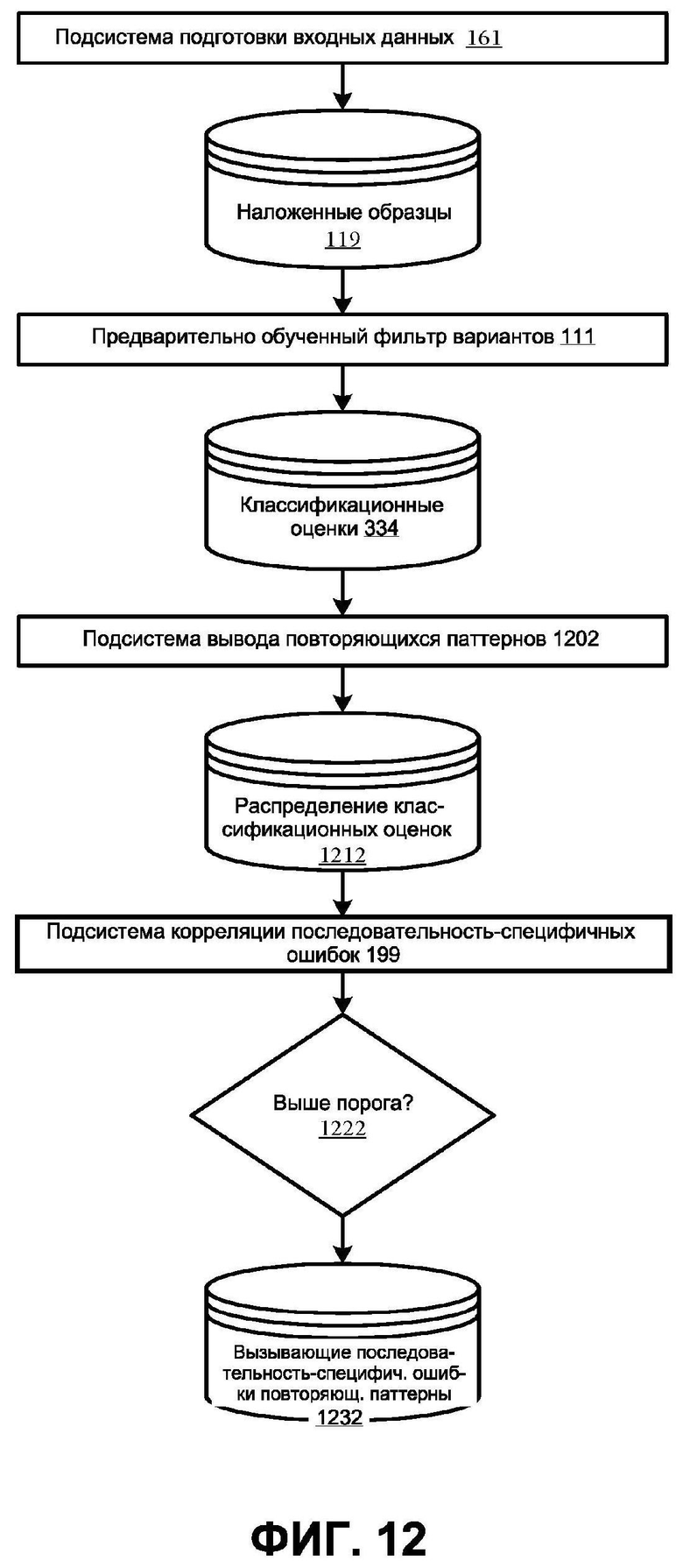
1. Система для идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования, содержащая:
один или несколько процессоров и одно или несколько устройств хранения, хранящих инструкции, которые, при исполнении на указанных одном или нескольких процессорах, обеспечивают реализацию указанными одним или несколькими процессорами:
подсистемы подготовки входных данных, сконфигурированной чтобы:
вычислительно накладывать исследуемые повторяющиеся паттерны на множество нуклеотидных последовательностей и получать наложенные образцы,
причем каждый повторяющийся паттерн представляет конкретный состав нуклеотидов, который имеет конкретную длину и присутствует в наложенном образце в определенном смещенном положении,
причем каждый наложенный образец содержит целевое положение, считающееся вариантным нуклеотидом, и
при этом для каждой комбинации конкретного состава нуклеотидов, конкретной длины и конкретного смещенного положения, вычислительно генерируется набор наложенных образцов;
заранее обученной подсистемы фильтра вариантов, сконфигурированной чтобы:
обрабатывать наложенные образцы сверточной нейронной сетью и, на основании выявления нуклеотидных паттернов в наложенных образцах сверточными фильтрами сверточной нейронной сети, генерировать классификационные оценки правдоподобия того, что вариантный нуклеотид в каждом из наложенных образцов является истинным вариантом или ложным вариантом;
подсистемы вывода повторяющихся паттернов, сконфигурированной чтобы:
выводить распределения классификационных оценок, сгенерированных заранее обученной подсистемой фильтра вариантов для факторов повтора соответствующих повторяющихся паттернов, причем указанные распределения указывают влияние на заранее обученную подсистему фильтра вариантов указанных повторяющихся паттернов; и
подсистемы корреляции последовательность-специфичных ошибок, сконфигурированной чтобы:
указывать, на основании порога, подмножество классификационных оценок в указанных распределениях как указывающих на ложные классификации вариантов и
классифицировать повторяющиеся паттерны, которые ассоциированы с этим подмножеством классификационных оценок, которые указывают на ложные классификации вариантов, как вызывающие последовательность-специфичные ошибки.
2. Система по п. 1, где указанная подсистема корреляции последовательность-специфичных ошибок дополнительно сконфигурирована чтобы
классифицировать длины и смещенные положения повторяющихся паттернов, которые ассоциированы с указанным подмножеством классификационных оценок, как вызывающие последовательность-специфичные ошибки.
3. Система по любому из пп. 1, 2, где вариантный нуклеотид находится в целевом положении, фланкированном по меньшей мере 20 нуклеотидами с каждой стороны.
4. Система по любому из пп. 1-3, где указанная заранее обученная подсистема фильтра вариантов сконфигурирована, чтобы обрабатывать каждую комбинацию повторяющихся паттернов, накладываемых на по меньшей мере 100 нуклеотидных последовательностей в по меньшей мере 100 наложенных образцах.
5. Система по любому из пп. 1-4, где повторяющиеся паттерны включают по меньшей мере одно основание из четырех оснований (A, C, G и T) с по меньшей мере шестью вариациями факторов повтора соответствующих повторяющихся паттернов.
6. Система по п. 5, где повторяющиеся паттерны представляют собой гомополимеры единственного основания (A, C, G или T) с по меньшей мере шестью вариациями факторов повтора соответствующих повторяющихся паттернов и
где указанные по меньшей мере шесть вариаций факторов повтора указывают число повторов указанного единственного основания в повторяющихся паттернах.
7. Система по любому из пп. 1-6, где указанные повторяющиеся паттерны представляют собой сополимеры по меньшей мере двух оснований из четырех оснований (A, C, G и T) с по меньшей мере шестью вариациями факторов повтора соответствующих повторяющихся паттернов и
где указанные по меньшей мере шесть вариаций факторов повтора указывают число повторов указанных по меньшей мере двух оснований в повторяющихся паттернах.
8. Система по любому из пп. 1-7, где указанные смещенные положения варьируют в смысле положения, в котором указанные повторяющиеся паттерны накладываются на нуклеотидные последовательности, что измеряется как смещение между исходным положением повторяющихся паттернов и исходным положением нуклеотидных последовательностей, и по меньшей мере десять смещений используют для получения наложенных образцов.
9. Система по любому из пп. 1-8, где повторяющиеся паттерны находятся справа от центрального нуклеотида в наложенных образцах и не накладываются на центральный нуклеотид.
10. Система по любому из пп. 1-8, где повторяющиеся паттерны находятся слева от центрального нуклеотида в наложенных образцах и не накладываются на центральный нуклеотид.
11. Система по любому из пп. 1-8, где повторяющиеся паттерны накладываются на центральный нуклеотид в наложенных образцах.
12. Система по любому из пп. 1-11, где указанные факторы повтора представляют собой целые числа в диапазоне от пяти до одной четверти от числа нуклеотидов в наложенных образцах.
13. Система по п. 6, дополнительно сконфигурированная для применения к повторяющимся паттернам, которые представляют собой гомополимеры единственного основания для каждого из четырех оснований (A, C, G и T).
14. Система по п. 13, где указанная подсистема подготовки входных данных дополнительно сконфигурирована, чтобы выдавать повторяющиеся паттерны и наложенные образы для гомополимеров для каждого из четырех оснований.
15. Система по п. 14, где повторяющиеся паттерны расположены справа от центрального нуклеотида в наложенных образцах и отображают распределения гомополимеров для указанных четырех оснований в смежных положениях.
16. Система по п. 14, где повторяющиеся паттерны расположены слева от центрального нуклеотида в наложенных образцах и отображают распределения гомополимеров для указанных четырех оснований в смежных положениях.
17. Система по любому из пп. 1-16, где нуклеотидные последовательности, на которые накладываются повторяющиеся паттерны, генерируются случайным образом.
18. Система по любому из пп. 1-17, где нуклеотидные последовательности, на которые накладываются повторяющиеся паттерны, случайным образом выбираются из природных нуклеотидных последовательностей ДНК.
19. Система по любому из пп. 1-18, где подсистема анализа сконфигурирована, чтобы отображать распределения классификационных оценок для каждого из факторов повтора.
20. Система по любому из пп. 1-19, где указанная заранее обученная подсистема фильтра вариантов обучена на по меньшей мере 500000 тренировочных примеров истинных вариантов и по меньшей мере 50000 тренировочных примеров ложных вариантов и
где каждый тренировочный пример представляет собой нуклеотидную последовательность с вариантным нуклеотидом в целевом положении, фланкированном по меньшей мере 20 нуклеотидами с каждой стороны.
21. Система по любому из пп. 1-20, где указанная заранее обученная подсистема фильтра вариантов содержит сверточные слои, полностью связанные слои и слои классификации.
22. Реализуемый с применением компьютера способ идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования, включающий:
вычислительное накладывание исследуемых повторяющихся паттернов на множество нуклеотидных последовательностей и получение наложенных образцов, причем каждый повторяющийся паттерн представляет конкретный состав нуклеотидов, который имеет конкретную длину и присутствует в наложенном образце в определенном смещенном положении, причем каждый наложенный образец содержит целевое положение, считающееся вариантным нуклеотидом, и при этом для каждой комбинации конкретного состава нуклеотидов, конкретной длины и конкретного смещенного положения вычислительно генерируется набор наложенных образцов;
обработку указанных наложенных образцов сверточной нейронной сетью и, на основании выявления нуклеотидных паттернов в наложенных образцах сверточными фильтрами сверточной нейронной сети, генерацию классификационных оценок для правдоподобия того, что указанный вариантный нуклеотид в каждом из наложенных образцов является истинным вариантом или ложным вариантом;
вывод распределений классификационных оценок, сгенерированных заранее обученной подсистемой фильтра вариантов для факторов повтора соответствующих повторяющихся паттернов, причем указанные распределения указывают влияние на заранее обученную подсистему фильтра вариантов указанных повторяющихся паттернов; и
указание, на основании порога, подмножества классификационных оценок в указанных распределениях как указывающих на ложные классификации вариантов и классификацию повторяющихся паттернов, которые ассоциированы с этим подмножеством классификационных оценок, которые указывают на ложные классификации вариантов, как вызывающих последовательность-специфичные ошибки.
23. Реализуемый с применением компьютера способ по п. 22, дополнительно реализующий любой из пп. 1-21 путем исполнения заявленных систем.
24. Компьютерочитаемый носитель долговременного хранения информации, на который нанесены программные инструкции по идентификации повторяющихся паттернов, которые вызывают последовательность-специфичные ошибки в данных нуклеотидного секвенирования, причем указанные инструкции при исполнении процессором реализуют реализуемый с применением компьютера способ, включающий:
вычислительное накладывание исследуемых повторяющихся паттернов на множество нуклеотидных последовательностей и получение наложенных образцов, причем каждый повторяющийся паттерн представляет конкретный состав нуклеотидов, который имеет конкретную длину и присутствует в наложенном образце в определенном смещенном положении, причем каждый наложенный образец содержит целевое положение, считающееся вариантным нуклеотидом, и при этом для каждой комбинации конкретного состава нуклеотидов, конкретной длины и конкретного смещенного положения вычислительно генерируется набор наложенных образцов;
обработку указанных наложенных образцов сверточной нейронной сетью и, на основании выявления нуклеотидных паттернов в наложенных образцах сверточными фильтрами сверточной нейронной сети, генерацию классификационных оценок для правдоподобия того, что указанный вариантный нуклеотид в каждом из наложенных образцов является истинным вариантом или ложным вариантом;
вывод распределений классификационных оценок, сгенерированных заранее обученной подсистемой фильтра вариантов для факторов повтора соответствующих повторяющихся паттернов, причем указанные распределения указывают влияние на заранее обученную подсистему фильтра вариантов указанных повторяющихся паттернов; и
указание, на основании порога, подмножества классификационных оценок в указанных распределениях как указывающих на ложные классификации вариантов и классификацию повторяющихся паттернов, которые ассоциированы с этим подмножеством классификационных оценок, которые указывают на ложные классификации вариантов, как вызывающих последовательность-специфичные ошибки.
25. Компьютерочитаемый носитель долговременного хранения информации по п. 24, дополнительно включающий признаки, которые, в комбинации с аппаратным обеспечением, реализуют любой из пп. 1-21.
| МОДЕЛЬ НЕЙРОННОЙ СЕТИ | 2006 |
|
RU2309457C1 |
| Способ защиты переносных электрических установок от опасностей, связанных с заземлением одной из фаз | 1924 |
|
SU2014A1 |
| Автомобиль-сани, движущиеся на полозьях посредством устанавливающихся по высоте колес с шинами | 1924 |
|
SU2017A1 |
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| Многоступенчатая активно-реактивная турбина | 1924 |
|
SU2013A1 |
Авторы
Даты
2021-03-31—Публикация
2019-07-09—Подача