ОБЛАСТЬ ИЗОБРЕТЕНИЯ
Данное изобретение относится к способу выявления остаточных, недифференцированных плюрипотентных стволовых клеток (PSC) в культуре клеток, дифференцировавшихся из PSC.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
Плюрипотентные стволовые клетки (PSC), в частности человеческие PSC, которые включают эмбриональные стволовые клетки (ESC) и индуцированные плюрипотентные стволовые клетки (iPSC), дают возможность разработки новых терапевтических продуктов на основе клеток.
Дифференцированные производные iPSC имеют терапевтическую эффективность в целом ряде приложений, связанных с заболеваниями (например, цирроз, болезнь Паркинсона, возрастная макулодегенерация, ишемическая болезнь сердца, диабет, болезнь «трансплантат против хозяина»).
Однако применение в клинических испытаниях терапевтических композиций, содержащих клетки, происходящие от PSC, создает несколько вызовов, в частности, присутствие остаточных недифференцированных PSC в конечном продукте, которые имеют известный потенциал в отношении образования опухолей. Такой вызов является особенно уместным в отношении терапий, для которых требуются высокие дозы дифференцированных клеток, происходящих из PSC.
Соответственно, существует потребность в способе или в улучшенном способе выявления остаточных, недифференцированных PSC в культуре клеток, происходящих от PSC, подлежащем применению в контроле качества при производстве терапевтических композиций, содержащих клетки, происходящие от PSC.
Любые публикации, упомянутые в данном описании изобретения, являются включенными сюда посредством ссылки. Однако, если здесь дается ссылка на любую публикацию, такая ссылка не составляет допущения того, что данная публикация образует часть обычного общего знания в данной области в Австралии или в любой другой стране.
КРАТКОЕ ИЗЛОЖЕНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
Авторы данного изобретения удовлетворили эту потребность контроля качества в изготовлении терапевтических композиций, содержащих клетки, происходящие от PSC, посредством разработки способа выявления остаточных, недифференцированных PSC в культуре клеток, происходящих от PSC. Данный способ основывается на протоколе культивирования клеток для размножения сделанных одиночными, недифференцированных PSC для увеличения чувствительности (истинно позитивные) и специфичности (истинно негативные) в отношении остаточных, недифференцированных PSC.
Соответственно, в первом аспекте предложен способ выявления остаточных, недифференцированных плюрипотентных стволовых клеток (PSC) в культуре клеток, дифференцировавшихся из PSC, включающий:
культивирование указанных клеток на субстрате, покрытом ламинином-521 и Е- кадгерином, в среде, содержащей ингибитор Rho-ассоциированной протеинкиназы, содержащей суперспираль (ROCK);
количественное измерение в культивируемых клетках экспрессии маркера остаточных, недифференцированных PSC; и
сравнение экспрессии маркера в культивируемых клетках с экспрессией маркера в контрольной культуре клеток, содержащей известную долю PSC,
где более низкая экспрессия маркера в культуре клеток, чем экспрессия маркера в контрольной культуре клеток, указывает на отсутствие остаточных, недифференцированных PSC в культивируемых клетках или на присутствие остаточных, недифференцированных PSC в культивируемых клетках в меньшей доле, чем известная доля PSC в контрольной культуре клеток.
Авторы данного изобретения предлагают то, что согласно данному способу предложено улучшенное средство контроля качества, и он представляет собой важный вклад в безопасность и, следовательно, прогресс терапий, основанных на клетках, происходящих от PSC.
Соответственно, в одном воплощении данный способ дополнительно включает приготовление культуры клеток, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, в виде терапевтической композиции. Данный способ может дополнительно включать лечение или предупреждение состояния у субъекта посредством введения данному субъекту терапевтической композиции.
Согласно второму аспекту предложен способ изготовления терапевтической композиции, включающий приготовление в виде композиции для терапевтического введения субъекту культуры клеток, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, при выявлении способом по первому аспекту.
Согласно третьему аспекту предложен способ лечения или предупреждения состояния у субъекта, включающий введение субъекту:
культуры клеток, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, при выявлении способом по первому аспекту; или
терапевтической композиции, изготовленной способом по второму аспекту. Согласно альтернативной форме третьего аспекта предложено применение культуры клеток, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля при изготовлении лекарственного средства, такого как терапевтическая композиция, для лечения или предупреждения состояния у субъекта, где остаточные, недифференцированные PSC в культуре клеток, дифференцированных из PSC, выявляют способом по первому аспекту.
Согласно другой альтернативной форме третьего аспекта предложена:
культура клеток, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, при выявлении способом по первому аспекту; или
терапевтическая композиция, изготовленная способом по второму аспекту, для применения в лечении или предупреждении состояния у субъекта.
Состояние, подлежащее предупреждению или лечению, может представлять собой следующие: кисты кости, новообразования кости, переломы, дефекты хряща, остеоартрит, повреждение связки, незавершенный остеогенез, остеонекроз, остеопороз, апластическая анемия, реакция «трансплантат против хозяина» (GvHD), миелодиспластический синдром, диабет типа 1, диабет типа 2, аутоиммунный гепатит, цирроз печени, печеночная недостаточность, дилатационная кардиомиопатия, сердечная недостаточность, инфаркт миокарда, ишемия миокарда, болезнь Крона, язвенный колит, ожоги, буллезный эпидермолиз, красная волчанка, ревматоидный артрит, болезнь Шегрена, системный склероз, бронхолегочная дисплазия, хроническое обструктивное заболевание дыхательных путей, эмфизема, легочный фиброз, боковой амиотрофический склероз (ALS), болезнь Альцгеймера, травма мозга, атаксия, остеохондроз, множественная системная атрофия, рассеянный склероз, болезнь Паркинсона, пигментная дистрофия сетчатки, болезнь Ромберга, травма спинного мозга, инсульт, мышечная дистрофия, ишемия конечности, повреждение почки, волчаночный нефрит, эндометриоз и осложнения трансплантации костного мозга или солидных органов.
Согласно четвертому аспекту предложен набор для выявления остаточных, недифференцированных PSC в культуре клеток, дифференцировавшихся из PSC, содержащий:
ламинин-521; и Е-кадгерин; и ингибитор ROCK.
В одном воплощении данный набор используют согласно способу по первому аспекту.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
Фиг. 1 представляет собой пример аминокислотной последовательности α цепи человеческого ламинина-521 (SEQ ID NO: 1).
Фиг. 2 представляет собой пример аминокислотной последовательности β цепи человеческого ламинина-521 (SEQ ID NO: 2).
Фиг. 3 представляет собой пример аминокислотной последовательности γ цепи человеческого ламинина-521 (SEQ ID NO: 3).
Фиг. 4 представляет собой пример аминокислотной последовательности человеческого Е-кадгерина (SEQ ID NO: 4).
Фиг. 5 представляет собой пример кодирующей нуклеотидной последовательности человеческого LIN28 (LIN28A) (SEQ ID NO: 5).
Фиг. 6 представляет собой пример кодирующей нуклеотидной последовательности человеческого ОСТ4 (POU5F1) (SEQ ID NO: 6).
Фиг. 7 представляет собой пример кодирующей нуклеотидной последовательности человеческого SOX2 (SEQ ID NO: 7).
Фиг. 8 представляет собой пример кодирующей нуклеотидной последовательности человеческого FOXD3 (SEQ ID NO: 8).
Фиг. 9 представляет собой пример кодирующей нуклеотидной последовательности человеческого NANOG (SEQ ID NO: 9).
Фиг. 10 представляет собой пример кодирующей нуклеотидной последовательности человеческого PODXL (SEQ ID NO: 10).
Фиг. 11 представляет собой пример кодирующей нуклеотидной последовательности человеческого REX1 (ZFP42) (SEQ ID NO: 11).
Фиг. 12 представляет собой пример кодирующей нуклеотидной последовательности человеческого SSEA1 (FUT4) (SEQ ID NO: 12).
Фиг. 13 представляет собой пример кодирующей нуклеотидной последовательости человеческого DPPA2 (SEQ ID NO: 13).
Фиг. 14 представляет собой пример кодирующей нуклеотидной последовательости человеческого DPPA3 (SEQ ID NO: 14).
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
В данном документе раскрыт способ выявления остаточных, недифференцированных PSC в культуре клеток, происходящих от PSC, где клетки, происходящие от PCS, предназначены для терапевтического введения субъекту. Соответственно, согласно изобретению предложен улучшенный способ контроля качества и минимизации риска, например, риска образования опухоли из таких остаточных, недифференцированных PSC.
Данный способ основывается на конкретных условиях культуры для размножения недифференцированных PSC при условиях, которые поддерживают клональный рост PSC, затем на количественном измерении экспрессии гена, который высоко экспрессируется в PSC, но не экспрессируется или только минимально экспрессируется в клетках, происходящих от PSC, которые подверглись дифференциации. Культуральные условия могут селективно поддерживать размножение PSC на уровне одной клетки. В одном воплощении PSC могут культивироваться при условиях, которые поддерживают рост PSC из суспензии одиночных клеток. Такие культуральные условия известны в данной области и включают ламинин-521 и Е-кадгерин, как здесь описано, а также имеющиеся в продаже системы, которые включают набор культуральой среды Cellartis DEF-CS для клонирования единичных клеток iPSC, среду Gibco™ StemFlex™ и среду для клонирования PluriQ™ G9™ в сочетании с ростом одиночных клеток на витронектине.
Экспрессия маркера в опытной культуре на уровне или ниже уровня экспрессии маркера в контрольной культуре с известной долей недифференцированных PSC указывает на то, что опытная культура содержит меньшую долю недифференцированных PSC, чем контрольная культура.
Важно то, что данный способ может выявлять остаточные недифференцированные PSC в культуре клеток, происходящих от PSC, при меньших долях общего числа клеток, например, 0,001% или 10 млн-1.
Если в данном описании изобретения не определено иначе, используемые здесь технические и научные термины, имеют такое же значение, которое обычно понятно специалисту в области, к которой принадлежит данное изобретение, и посредством ссылки на опубликованные тексты.
Следует отметить то, что термин в единственном числе относится к одному или более чем одному, например, понятно то, что «молекула» представляет собой одну или более чем одну молекулу. Термины в единственном числе, «один или более чем один» и «по меньшей мере один», как таковые, могут использоваться в данном документе взаимозаменяемо.
В формуле изобретения, которая следует далее, и в описании изобретения, за исключением случаев, когда контекст требует иного из-за прямо оговоренной формулировки или необходимого подразумеваемого, слово «содержать» или вариации, такие как «содержит» или «содержащий», используются во включительном смысле, т.е. для определения присутствия заявленной характеристики, но не для исключения присутствия или добавления дополнительных характеристик в разных воплощениях данного изобретения.
Термин «примерно» в том виде, в котором он здесь используется, рассматривает целый ряд значений для данного числа плюс/минус 25% от значения данного числа. В других воплощениях термин «примерно» рассматривает целый ряд значений для данного числа плюс/минус 20%, плюс/минус 15%, плюс/минус 10% или плюс/минус 5% от значения данного числа. Например, в одном воплощении фраза «примерно 3 грамма» указывает значение от 2,7 до 3,3 грамма (т.е. 3 грамма плюс/минус 10%) и тому подобное.
Аналогичным образом, в других воплощениях периоды времени могут варьировать на плюс/минус 25%, плюс/минус 20%, плюс/минус 15%, плюс/минус 10% или плюс/минус 5% от данного периода времени. Например, «одни сутки» могут включать период от примерно 18 до примерно 30 часов. Указанные периоды времени, которые представляют собой периоды в много суток, могут представлять собой кратные единицы «одних суток», как, например, двое суток могут охватывать период от примерно 36 до примерно 60 суток и тому подобное. В других воплощениях вариация времени может быть уменьшена, например, где: сутки 1 представляют собой 24 плюс/минус 3 часа от суток 0; сутки 2 представляют собой 48 плюс/минус 3 часа от суток 0; сутки 3 представляют собой 72 плюс/минус 3 часа от суток 0; сутки 4 представляют собой 96 плюс/минус 3 часа от суток 0; сутки 5 представляют собой 120 плюс/минус 3 часа от суток 0 и так далее. В некоторых воплощениях примерно 3 суток представляют собой 3 суток плюс/минус 1 сутки, и примерно 5 суток представляют собой 5 суток плюс/минус 1 сутки.
Термин «плюрипотентная стволовая клетка» или «PSC» в том виде, в котором он здесь используется, относится к клетке, которая имеет способность бесконечно воспроизводиться и дифференцироваться в любой другой тип клетки. Имеются два главных типа плюрипотентных стволовых клеток: эмбриональные стволовые клетки (ESC) и индуцированные плюрипотентные стволовые клетки (iPSC).
Термин «эмбриональная стволовая клетка» или «ЕSC» в том виде, в котором он здесь используется, относится к клетке, выделенной из пяти-семисуточного эмбриона, подаренного по согласию пациентов, которые завершили терапию на основе оплодотворения in vitro и имеют излишек эмбрионов. Применению ESC в некоторой степени препятствовали этические опасения о выделении клеток из человеческих эмбрионов.
Человеческие PSC, подходящие для изготовления терапевтической композиции, включают человеческие ESC H1 и Н9.
Термин «индуцированная плюрипотентная стволовая клетка» или «iPSC» в том виде, в котором он здесь используется, относится к ESC-подобной клетке, полученной из взрослых клеток. iPSC имеют очень сходные характеристики с ESC, но с ними избегаются этические опасения, связанные с ESC, так как iPSC не происходят из эмбрионов. Вместо этого iPSC типично происходят из полностью дифференцированных клеток взрослых, которые были «перепрограммированы» обратно в плюрипотентное состояние.
Человеческие iPSC, подходящие для изготовления терапевтической композиции, включают iPSC 19-9-7T, MIRJT6i-mND1-4 и MIRJT7i-mND2-0, происходящие от фибробластов, и iPSC BM119-9, происходящие от одноядерных клеток костного мозга. Другие подходящие iPSCs могут быть приобретены у Cellular Dynamics International (CDI; Nasdaq: ICEL) из Madison, WI, США.
Термин «осуществление дифференциации» в том виде, в котором он здесь используется, относится к процессу изменения клетки от одного типа клетки до другого, в частности, когда менее специализированный тип клетки становится более специализированным типом клетки.
Термин «происходящий из» в том виде, в котором он здесь используется, может охватывать «осуществление дифференциации» PSC до другого типа клетки.
Соответственно, «недифференцированная PSC» представляет собой PSC, которая не дифференцировалась до другого типа клетки. В том, что касается настоящего раскрытия, недифференцированные PSC присутствуют в популяции дифференцированных в иных отношениях PSC.
Термин «среда» или его множественное число - «среды» - в том виде, в котором он здесь используется, относится к жидкости или гелю, разработанным для поддержки роста, включающего размножение и дифференциацию клеток. Такой рост, включающий размножение и дифференциацию клеток in vitro, называется «культивированием» клеток или «культурой» клеток.
Однако клетки не могут бесконечно сохраняться в культуре, благодаря возрастающей концентрации токсичных метаболитов, снижающейся концентрации питательных веществ и, для делящихся клеток, возрастающему числу клеток. Термин «пассирование» в том виде, в котором он здесь используется, относится к способу получения новой клеточной культуры с освеженными концентрациями питательных веществ, отсутствием токсичных метаболитов и, возможно, меньшей плотностью клеток, чем в исходной культуре.
Число пассажей для культуры клеток, например, культуры клеток, происходящих от PSC, подлежащих тестированию настоящим способом, может составлять 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25. Предпочтительно число пассажей составляет 10 или меньше. Более предпочтительно число пассажей составляет 5 или 6. В одном воплощении клетки, происходящие от PSC, претерпели 5 пассажей перед тем, как быть подвергнутыми настоящему способу.
В одном воплощении клетка, происходящая из PSC, представляет собой мезенхимную стволовую (или стромальную) клетку (MSC).
В одном воплощении клетка, происходящая от ЕSC, представляет собой MSC.
В предпочтительном воплощении клетка, происходящая от iPSC, представляет собой MSC, которая также может называться iPSC-MSC.
Термин «мезенхимная стволовая клетка» или «MSC» в том виде, в котором он здесь используется, относится к конкретному типу стволовой клетки, которая может быть выделена из широкого спектра тканей, включающих костный мозг, жировую ткань (жир), плаценту и кровь пуповины. MSC могут дифференцироваться в клетки кости (остеоциты), клетки хряща (хондроциты), жировые клетки (адипоциты) и другие виды клеток соединительной ткани, такие как клетки в сухожилиях.
Согласно настоящему раскрытию MSC могут быть образованы из PSC посредством примитивных мезодермальных клеток EMHlin-KDR+APLNR+PDGFRальфа+ с потенциалом мезенхимоангиобластов (МСА). Термин «примитивные мезодермальные клетки EMHlin-KDR+APLNR+PDGFRальфа+ с потенциалом мезенхимоангиобластов (МСА)» относится к клетке, экспрессирующей типичные гены первичной полоски и боковой пластинки/внеэмбриональной мезодермы. Данные клетки имеют потенциал к образованию колоний мезенхимоангиобластов (МСА) и гемангиобластов в бессывороточной среде в ответ на FGF2. Термин EMHlin- обозначает отсутствие экспрессии CD31, VE-кадгериновых эндотелиальных маркеров, мезенхимных/эндотелиальных маркеров CD73 и Cd105 и маркеров гематопоэтической линии CD43 и CD45.
Термин «мезенхима» или «мезенхимный» в том виде, в котором он здесь используется, относится к эмбриональной соединительной ткани, которая происходит из мезодермы, и которая дифференцируется в гематопоэтическую ткань (лимфатическую и кровеносную системы) и соединительную ткань, такую как кость и хрящ. Однако MSC не дифференцируются в гематопоэтические клетки.
MSC секретируют биоактивные молекулы, такие как цитокины, хемокины и факторы роста, и имеют способность модулировать иммунную систему. Было показано то, что MSC облегчают регенерацию и осуществляют воздействия на иммунную систему, не полагаясь на пересадку. Другими словами, сами MSC не обязательно могут становиться включенными в хозяина - скорее они могут оказывать их эффекты и затем устраняться в пределах короткого периода времени. Однако MSC могут быть пересажены в поврежденные ткани после введения и миграции.
MSC в настоящее время находятся в клинических испытаниях для лечения многочисленных состояний, заболеваний и расстройств, включая болезнь «трансплантат против хозяина».
Считается, что способность MSC оказывать иммуномодулирующие/иммунодепрессивные эффекты, в частности, посредством подавления Т-клеток, является центральной для терапевтических эффектов MSC при широком спектре состояний, заболеваний и расстройств, включающих болезнь «трансплантат против хозяина», иммунные расстройства, включающие аутоиммунные расстройства, сердечно-сосудистые расстройства, ортопедические расстройства и отторжение трансплантированных солидных органов. Некоторые конкретные примеры включают следующие: кисты кости, новообразования кости, переломы, дефекты хряща, остеоартрит, повреждение связки, незавершенный остеогенез, остеонекроз, остеопороз, апластическая анемия, миелодиспластический синдром, диабет типа 1, диабет типа 2, аутоиммунный гепатит, цирроз печени, печеночная недостаточность, дилатационная кардиомиопатия, сердечная недостаточность, инфаркт миокарда, ишемия миокарда, болезнь Крона, язвенный колит, ожоги, буллезный эпидермолиз, красная волчанка, ревматоидный артрит, болезнь Шегрена, системный склероз, бронхолегочная дисплазия, хроническое обструктивное заболевание дыхательных путей, эмфизема, легочный фиброз, боковой амиотрофический склероз (ALS), болезнь Альцгеймера, травма мозга, атаксия, остеохондроз, множественная системная атрофия, рассеянный склероз, болезнь Паркинсона, пигментная дистрофия сетчатки, болезнь Ромберга, травма спинного мозга, инсульт, мышечная дистрофия, ишемия конечности, повреждение почки, волчаночный нефрит, эндометриоз и осложнения трансплантации костного мозга или солидного органа.
Полагают, что MSC выполняют критически важную роль в заживлении ран, и было показано то, что они являются эффективными в лечении повреждения ткани и дегенеративных заболеваний, включающих заболевания в пищеварительной системе, например, цирроз печени и печеночная недостаточность, в скелетно-мышечной системе, в периодонтальной ткани, при диабетической критической ишемии конечностей, при остеонекрозе, при заболеваниях, связанных с ожогами, при инфаркте миокарда, при повреждении роговицы, в мозге, в спинном мозге, в легких и при лечении последствий воздействия радиации.
MSC показали терапевтические результаты при иммунных расстройствах, включающих болезнь «трансплантат против хозяина», системную красную волчанку (SLE), болезнь Крона, множественную системную атрофию, рассеянный склероз, боковой амиотрофический склероз и инсульт.
Было показано то, что MSC оказывают иммунодепрессивные активности против Т-клеток, В-клеток, дендритных клеток, макрофагов и клеток-природных киллеров. Не желая быть связанными теорией, лежащие в основе механизмы могут включать иммунодепрессивные медиаторы, например, оксид азота, индоламин 2,3, диоксигеназу, простагландин Е2, белок гена 6, индуцируемого фактором некроза опухолей, CCL-2 и лиганд 1 программируемой смерти. Данные медиаторы экспрессируются на низком уровне, пока не происходит стимуляции, например, воспалительными цитокинами, такими как IFNγ (интерферон-гамма), TNFα (фактор некроза опухолей-альфа) и IL-17 (интерлейкин-17).
MSC, происходящие из iPSC (iPSC-MSC) имеют уникальное преимущество над MSC, полученными прямым способом, т.е. происходящими из таких тканей, как костный мозг, кровь пуповины, жировая ткань, так как размножение iPSC in vitro может обеспечивать фактически неограниченную поставку MSC.
iPSC-MSC могут быть получены согласно Примеру 1.
PSC способны дифференцироваться в любой тип клеток любой из эндодермы, эктодермы и мезодермы. PSC подвергаются дифференциации в мультипотентные клетки-предшественики, которые дифференцируются далее в функциональные клетки. Следовательно, приведенное выше описание MSC является примерным, и его не следует истолковывать как ограничивающее, и другие примеры клеток, происходящих от PSC, могут включать: гематопоэтические стволовые клетки, которые дают все типы клеток крови, включая эритроциты, В-лимфоциты, Т-лимфоциты, природные клетки- киллеры, нейтрофилы, базофилы, эозинофилы, моноциты, макрофаги и тромбоциты; природные стволовые клетки в мозгу, которые дают три его главных типа клеток, представляющих собой нервные клетки (нейроны) и две категории ненейронных клеток - астроциты и олигодендроциты; эпителиальные стволовые клетки в выстилке пищеварительного тракта, которые дают всасывающие клетки, бокаловидные клетки, клетки Панета и энтероэндокринные клетки; и стволовые клетки кожи, которые встречаются в базальном слое эпидермиса, давая кератиноциты, и которые встречаются у основания волосяных фолликулов и дают как волосяной фолликул, так и эпидермис.
Термин «субстрат» в том виде, в котором он здесь используется, представляет собой любое вещество, подходящее для культивирования PSC и/или клеток, происходящих от PSC. Примеры включают пластмассовую культуральную посуду, такую как чашки, многолуночные планшеты и флаконы.
Все белки, описанные в данном документе, известны специалисту в данной области и имеются в продаже.
Ламинин-521 представляет собой гетеротримерный белок, секретируемый человеческими PSC. Ламинин-521 содержит пять α цепей, две β цепи и одну γ цепь (т.е. α5β2γ1). В одном воплощении α цепь, β цепь и γ цепь имеют аминокислотные последовательности, представленные SEQ ID NO: 1, 2 и 3 (Фиг. 1, 2 и 3) соответственно.
Е-кадгерин представляет собой кальций-зависимый белок клеточной адгезии, ассоциированный с функцией эпителиальных клеток. В одном воплощении человеческий Е-кадгерин имеет аминокислотную последовательность, представленную SEQ ID NO: 4 (Фиг. 4).
При осуществлении настоящего способа субстрат может быть покрыт ламинином-521 с использованием примерно 0,1 мкг/мл, 0,2 мкг/мл, 0,3 мкг/мл, 0,4 мкг/мл, 0,5 мкг/мл, 0,6 мкг/мл, 0,7 мкг/мл, 0,8 мкг/мл, 0,9 мкг/мл, 1 мкг/мл, 2 мкг/мл, 3 мкг/мл, 4 мкг/мл, 5 мкг/мл, 6 мкг/мл, 7 мкг/мл, 8 мкг/мл, 9 мкг/мл или 10 мкг/мл, и/или примерно 0,1 мкг/см2, 0,2 мкг/см2, 0,3 мкг/см2, 0,4 мкг/см2, 0,5 мкг/см2, 0,6 мкг/см2, 0,7 мкг/см2, 0,8 мкг/см2, 0,9 мкг/см2, 1 мкг/см2, 2 мкг/см2, 3 мкг/см2, 4 мкг/см2, 5 мкг/см2, 6 мкг/см2, 7 мкг/см2, 8 мкг/см2, 9 мкг/см2 или 10 мкг/см2.
Субстрат может быть покрыт Е-кадгерином с использованием примерно 0,1 мкг/мл, 0,2 мкг/мл, 0,3 мкг/мл, 0,4 мкг/мл, 0,5 мкг/мл, 0,6 мкг/мл, 0,7 мкг/мл, 0,8 мкг/мл, 0,9 мкг/мл, 1 мкг/мл, 2 мкг/мл, 3 мкг/мл, 4 мкг/мл, 5 мкг/мл, 6 мкг/мл, 7 мкг/мл, 8 мкг/мл, 9 мкг/мл или 10 мкг/мл, и/или примерно 0,01 мкг/см2, 0,05 мкг/см2, 0,1 мкг/см2, 0,2 мкг/см2, 0,25 мкг/см2, 0,3 мкг/см2, 0,4 мкг/см2, 0,5 мкг/см2, 0,6 мкг/см2, 0,7 мкг/см2, 0,8 мкг/см2, 0,9 мкг/см2, 1 мкг/см2, 2 мкг/см2, 3 мкг/см2, 4 мкг/см2, 5 мкг/см2, 6 мкг/см2, 7 мкг/см2, 8 мкг/см2, 9 мкг/см2 или 10 мкг/см2.
В предпочтительном воплощении субстрат покрыт ламинином-521 с использованием 10 мкг/мл и 2 мкг/см2 и Е-кадгерином с использованием 1,1 мкг/мл и 0,22 мкг/см2.
Rho-ассоциированная протеинкиназа, содержащая суперспираль (ROCK), участвует, главным образом, в регуляции формы и движения клеток, посредством действия на цитоскелет. Таким образом, «ингибитор ROCK» ингибирует данную функцию, и для настоящих целей «ингибитор ROCK» увеличивает выживание iPSC.
В одном воплощении ингибитор ROCK представляет собой Y27632 (CAS No: 129830-38-2). Другими примерами ингибиторов ROCK, которые можно использовать в настоящем способе, являются AS 1892802 (CAS No: 928320-12-1), фасудила гидрохлорид (CAS No: 105628-07-7), GSK 269962 (CAS No: 850664-21-0), GSK 429286 (CAS No: 864082-47-3), H 1152 дигидрохлорид (CAS No: 871543-07-6), глицил-H 1152 дигидрохлорид (CAS No: 913844-45-8), HA 1100 гидрохлорид (CAS No: 155558-32-0), OXA 06 дигидрохлорид, RKI 1447 дигидрохлорид, SB 772077B дигидрохлорид (CAS No: 607373-46-6), SR 3677 дигидрохлорид (CAS No: 1072959-67-1) и TC-S 7001 (CAS No: 867017-68-3).
Концентрация ингибитора ROCK, в которой культивируются клетки, может составлять, например, примерно 0,1 мкМ, 0,5 мкМ, 1 мкМ, 2 мкМ, 3 мкМ, 4 мкМ, 5 мкМ, 6 мкМ, 7 мкМ, 8 мкМ, 9 мкМ, 10 мкМ, 11 мкМ, 12 мкМ, 13 мкМ, 14 мкМ, 15 мкМ, 16 мкМ, 17 мкМ, 18 мкМ, 19 мкМ, 20 мкМ, 25 мкМ, 30 мкМ, 40 мкМ, 50 мкМ, 60 мкМ, 70 мкМ, 80 мкМ, 90 мкМ или 100 мкМ.
Предпочтительно ингибитор ROCK представляет собой Y27632, и концентрация Y27632, в которой культивируются клетки, составляет примерно 10 мкМ.
Согласно настоящему способу для культивирования клеток на субстрате, покрытом ламинином-521 и Е-кадгерином, в среде, содержащей ингибитор Rho- ассоциированной протеинкиназы, содержащей суперспираль (ROCK), клетки должны быть посеяны на субстрат. Плотность посева клеток на субстрате может составлять примерно 1×102 клеток/см2, 2×102 клеток/см2, 3×102 клеток/см2, 4×102 клеток/см2, 5×102 клеток/см2, 6×102 клеток/см2, 7×102 клеток/см2, 8×102 клеток/см2, 9×102 клеток/см2, 1×103 клеток/см2, 2×103 клеток/см2, 3×103 клеток/см2, 4×103 клеток/см2, 5×103 клеток/см2, 6×103 клеток/см2, 7×103 клеток/см2, 8×103 клеток/см2, 9×103 клеток/см2, 1×104 клеток/см2, 2×104 клеток/см2, 3×104 клеток/см2, 4×104 клеток/см2, 5×104 клеток/см2, 6×104 клеток/см2, 7×104 клеток/см2, 8×104 клеток/см2, 9×104 клеток/см2, 1×105 клеток/см2, 2×105 клеток/см2, 3×105 клеток/см2, 4×105 клеток/см2, 5×105 клеток/см2, 6×105 клеток/см2, 7×105 клеток/см2, 8×105 клеток/см2, 9×105 клеток/см2, 1×106 клеток/см2, 2×106 клеток/см2, 3×106 клеток/см2, 4×106 клеток/см2, 5×106 клеток/см2, 6×106 клеток/см2, 7×106 клеток/см2, 8×106 клеток/см2, 9×106 клеток/см2. В некоторых воплощениях плотность посева может составлять примерно 1,1×104 клеток/см2, 1,2×104 клеток/см2, 1,3×104 клеток/см2, 1,4×104 клеток/см2, 1,5×104 клеток/см2, 1,6×104 клеток/см2, 1,7×104 клеток/см2, 1,8×104 клеток/см2 или 1,9×104 клеток/см2. В предпочтительном воплощении плотность посева составляет примерно 1,3×104 клеток/см2.
Как часть настоящего способа клетки могут культивироваться в течение примерно 1 суток, 2 суток, 3 суток, 4 суток, 5 суток, 6 суток, 7 суток, 8 суток, 9 суток, 10 суток или более перед количественным измерением экспрессии маркера в культивируемых клетках. Предпочтительно клетки культивируются в течение примерно 5 суток перед количественным измерением экспрессии маркера в культивируемых клетках.
Как часть настоящего способа клетки могут культивироваться в присутствии ингибитора ROCK в течение примерно 1 суток, 2 суток, 3 суток, 4 суток, 5 суток, 6 суток, 7 суток, 8 суток, 9 суток, 10 суток или более перед количественным измерением экспрессии маркера в культивируемых клетках.
После культивирования клеток в присутствии ингибитора ROCK клетки можно далее культивировать в отсутствие ингибитора ROCK перед количественным измерением экспрессии маркера в культивируемых клетках. В одном воплощении клетки далее культивируются в отсутствие ингибитора ROCK в течение примерно 1 суток, 2 суток, 3 суток, 4 суток, 5 суток, 6 суток, 7 суток, 8 суток, 9 суток, 10 суток или более перед количественным измерением экспрессии маркера в культивируемых клетках.
В предпочтительном воплощении клетки культивруются в присутствии ингибитора ROCK в течение примерно 3 суток, затем культивируются далее в отсутствие ингибитора ROCK в течение примерно 2 суток перед количественным измерением экспрессии маркера в культивируемых клетках.
В заключение, было продемонстрировано то, что размножение клеток, происходящих из PSC, таких как MSC, на субстрате, таком как пластмассовая культуральная посуда, покрытом ламинином-521 и Е-кадгерином, поддерживает размножение ставших единичными, недифференцированных PSC, которые находятся в популяции клеток, происходящих из PSC, тогда как ингибитор ROCK одновременно увеличивает выживание PSC.
Термин «экспрессия маркера» в том виде, в котором он здесь используется, относится к гену, который экспрессируется в остаточных, недифференцированных PSC, но не экспрессируется или экспрессируется только с более низкими уровнями в клетках, происходящих от PSC. Такой ген может быть кодирующим или некодирующим, например, мРНК, микроРНК или некодирующую РНК.
Маркеры, экспрессия которых может быть количественно измерена согласно настоящему способу, включают LIN28 (LIN28A), OCT4 (POU5F1), SOX2, FOXD3, NANOG, PODXL (изоформы белка которого, выявляют антителами TRA-1-60 и TRA-1-81), REX1 (ZFP42), SSEA1 (FUT4), SSEA4, DPPA2 и DPPA3. Предпочтительно экспрессией маркера является экспрессия LIN28. Ген LIN28 кодирует белок LIN28, который представляет собой РНК-связывающий белок, который стимулирует плюрипотентность и высоко экспрессируется в PSC, но демонстрирует понижающую регуляцию в ответ на дифференциацию.
«Экспрессию маркера» можно количественно измерять как экспрессию белка или мРНК. Предпочтительно экспрессия маркера количественно измеряется как экспрессия мРНК.
Белок-маркер может быть количественно измерен способами, включающими гель-электрофорез (например, вестерн-блоттинг или двухмерный гель-электрофорез), денситометрию, флюоресценцию, люминесценцию, радиоактивность, чипы и/или масс- спектрометрию.
Маркерную мРНК можно количественно измерять способами, включающими гель-электрофорез (например, нозерн-блоттинг), денситометрию, флуоресценцию, люминесценцию, радиоактивность, чипы и/или полимеразную цепную реакцию (ПЦР). ПЦР обычно основывается на обратной транскрипции с получением маркерной кДНК из маркерной мРНК.
Предпочтительно экспрессия маркера количественно измеряется с использованием ПЦР. Предпочтительно экспрессия маркера количественно измеряется с использованием количественной ПЦР, сопряженной с обратной транскрипцией (кПЦР-РВ). Предпочтительно кПЦР-РВ представляет собой кПЦР-РВ в реальном времени, в которой мРНК подвергается обратной транскрипции до матрицы - кДНК, и матрица - кДНК - экспоненциально амплифицируется и количественно измеряется посредством флуоресценции в реальном времени.
Количественное измерение экспрессии маркера может быть относительным или абсолютным.
В одном воплощении количественное измерение экспрессии маркера является относительным и основывается на измерении цикла количественного измерения (Cq), который представляет собой цикл, где флуоресцентный сигнал пересекает порог для анализов кПЦР-РВ. Cq обратно пропорционален экспрессии мРНК.
Относительное количественное измерение экспрессии маркера основывается на сравнении экспрессии маркера в опытной культуре клеток с экспрессией маркера в одной или более чем одной контрольной культуре (т.е. контроли).
«Контрольная культура» может представлять собой позитивный контроль или негативный контроль. Когда контрольная культура представляет собой позитивный контроль, данная контрольная культура клеток содержит PSC. Предпочтительно такая контрольная культура содержит известную долю PSC. То есть, в контрольную культуру «добавлен внутренний стандарт» с известной долей недифференцированных PSC, обеспечивая, посредством этого, сравнение между опытной культурой и контрольной культурой. В данном воплощении количественное измерение может быть и относительным (например, применение Cq в качестве единицы количественного измерения), и абсолютным (например, применение Cq к известному количеству). Это также может называться полуколичественным. Таким образом, меньшая экспрессия маркера в культуре тестируемых клеток, чем экспрессия маркера в контрольной культуре клеток показывает отсутствие остаточных, недифференцированных PSC в культивируемых клетках. В качестве альтернативы, меньшая экспрессия маркера в культуре тестируемых клеток, чем экспрессия маркера в контрольной культуре клеток показывает присутствие остаточных, недифференцированных PSC в культивируемых клетках, но присутствие в меньшей доле, чем известная доля PSC в контрольной культуре клеток.
Известная доля PSC в контрольной культуре клеток может составлять 0,000001%, 0,000005%, 0,00001%, 0,00005%, 0,0001%, 0,0005%, 0,0006%, 0,0007%, 0,0008%, 0,0009%, 0,001%, 0,002%, 0,003%, 0,004%, 0,005%, 0,006%, 0,007%, 0,008%, 0,009% или 0,01% от общего числа клеток. В предпочтительном воплощении известная доля PSC в контрольной культуре составляет 0,001% от общего числа клеток.
В некоторых воплощениях экспрессия маркера «нормируется», что компенсирует внутри- и межкинетическии вариации (вариации от образца к образцу и от измерения к измерению). Нормированные данные являются особенно полезными при количественном измерении экспрессии гена с использованием кПЦР-РВ. Специалисту в данной области будут известны разные способы нормирования, включающие нормирование к одному или более чем одному подвергающемуся повышающей регуляции или конститутивному гену «домашнего хозяйства», например, GAPDH (глицеральдегид-3-фосфатдегидрогеназа), ACTB (актин В), LDHA (лактатдегидрогеназа А), NONO (октамерсвязывающий белок, не содержащий домен POU), PGK1 (фосфоглицераткиназа) или PPIH (пептидилпролилизомераза Н), и нормирование к общей РНК. В одном воплощении экспрессия маркера нормируется к экспрессии GAPDH.
Для ПЦР требуются нуклеотидные праймеры. Специалисту в данной области будет понятно, как конструировать праймеры для ПЦР и кПЦР-РВ, и комбинации праймер/зонд для кПЦР-РВ. Праймеры для некоторых генов имеются в продаже. Комбинации праймер/зонд для некоторых генов также имеются в продаже. Например, имеются в продаже следующие анализы для экспрессии генов TaqMan®, все с каталожным № 4331182: анализ человеческого LIN28 (LIN28A) Hs00702808_s1; анализ человеческого OCT4 (POU5F1) Hs04260367_gH; анализ человеческого SOX2 Hs01053049_s1; анализ человеческого FOXD3 Hs00255287_s1; анализ человеческого NANOG Hs04399610_g1; анализ человеческого PODXL (изоформы белка которого выявляются антителами TRA-1-60 и TRA-1-81) Hs01574644; анализ человеческого REX1 (ZFP42) Hs01938187_s1; анализ человеческого SSEA1 (FUT4) Hs01106466_s1; анализ человеческого DPPA2 Hs00414515_m1 и анализ человеческого DPPA3 Hs01931905_g1.
Комбинации праймеров или праймеров/зондов могут быть легко сконструированы специалистом в данной области с использованием инструмента, доступного онлайн, например, комбинации праймеров/зондов могут быть сконструированы на заказ с использованием такого инструмента, как Custom TaqMan® Assay Design Tool или инструмент GenScript Real-time PCR (TaqMan®) Primer Design. Праймеры могут быть сконструированы с использованием таких инструментов как Primer3Plus или PrimerQuest Tool. Данные инструменты являются лишь примерами, и значительно больше легко доступно специалисту в данной области.
Альтернативно или дополнительно, комбинации праймеров или праймеров/зондов могут быть легко сконструированы на основе основных принципов, которые включают следующие соображения.
ПЦР включает цикл: денатурирования двухцепочечной ДНК-мишени; отжига праймеров с комплементарными областями одноцепочечной ДНК; элонгации ДНК посредством действия ДНК-полимеразы; и повторение часто около 50 циклов. Данные стадии являются температурочувствительными и обычно проводятся при 94°С, 60°С и 70°С соответственно. Хорошая конструкция праймеров является важной для успешных реакций. Важные соображения при конструировании включают:
1. длину праймера, часто 18-22 п.о.;
2. температуру плавления праймера (Tm), часто в интервале 52-58°С;
3. температуру отжига праймера (Tа);
4. содержание GC, часто 40-60%;
5. GC-кламп, т.е. основания G или С, расположенные в пределах последних пяти оснований от 3'-конца праймеров;
6. вторичные структуры праймера и матрицы, т.е. межмолекулярные или внутримолекулярные взаимодействия, например, шпильки, димеры с самим собой или перекрестные димеры;
7. двухнуклеотидные повторы;
8. длинные тяжи одного вида основания;
9. стабильность 3'-конца;
10. перекрестная гомология/специфичность;
11. длина ампликона, часто около 100 п.о. для кПЦР-РВ и около 500 п.о. для стандартной ПЦР;
12. Tm продукта;
13. Tm пары праймеров, часто меньше 5°С.
Tm праймера может быть рассчитана следующим образом:
Tm(K)={ΔH/ ΔS + R ln(C)}, или Tm(°C) = {ΔH/ ΔS + R ln(C)} - 273,15, где
ΔH (ккал/моль): H представляет собой энтальпию, и ΔH представляет собой изменение энтальпии
ΔS (ккал/моль): S представляет собой энтропию, и ΔS представляет собой изменение энтропии.
ΔS (солевая поправка) = ΔS (1M NaCl) + 0,368 × N × ln([Na+]), где
N представляет собой число пар нуклеотидов в праймере (длина праймера -1)
[Na+] представляет собой солевой эквивалент в мM
Расчет [Na+]:
[Na+] = концентрация одновалентного иона +4 × свободный Mg2+.
Та праймера может быть рассчитана следующим образом:
Та = 0,3 × Tm (праймер) + 0,7 Tm (продукт) - 14,9
Типичные кодирующие нуклеотидные последовательности для человеческого LIN28 (LIN28A), человеческого OCT4 (POU5F1), человеческого SOX2, человеческого FOXD3, человеческого NANOG, человеческого PODXL, человеческого REX1 (ZFP42), человеческого SSEA1 (FUT4), человеческого DPPA2 и человеческого DPPA3 предоставлены на Фиг. 5-14 и в SEQ ID NO: 5-14 соответственно. Данные последовательности могут быть использованы специалистом в данной области для конструирования праймеров и комбинаций праймер/зонд.
кПЦР-РВ основана на флуоресценции для количественного измерения экспрессии генов, например, экспрессии маркера. Такая флуоресценция может включать (1) неспецифичный флуоресцентный краситель, который внедряется в любую двухцепочечную ДНК, т.е. амплифицированный ПЦР-продукт, или (2) специфичный в отношении последовательности олигонуклеотидный зонд, меченный флуоресцентным репортером, который флуоресцирует только после гибридизации зонда с комплементарной ему последовательностью. Одним примером первого типа флуоресценции является SYBR® Green (CAS No. 163795-75-3). Обычно второй тип содержит гаситель, ковалентно присоединенный к 3'-концу зонда, который гасит флуоресцентный репортер, пока гаситель не высвобождается от зонда во время амплификации.
В одном воплощении способ выявления остаточных, недифференцированных PSC в культуре клеток, дифференцированных из PSC, раскрытый в данном документе, может дополнительно включать получение сообщения, описывающего долю остаточных, недифференцированных PSC, выявленных в культуре клеток, происходящих из PSC.
В одном воплощении способ выявления остаточных, недифференцированных PSC в культуре клеток, дифференцированных из PSC, раскрытый в данном документе, может дополнительно включать приготовление культуры клеток, происходящих от PSC, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля в терапевтической композиции для введения субъекту. Данный способ может дополнительно включать лечение или предупреждение состояния у субъекта посредством введения данному субъекту культуры клеток, происходящих от PSC, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, или терапевтической композиции, содержащей такие клетки, происходящих от PSC. Данное состояние может представлять собой следующие: кисты кости, новообразования кости, переломы, дефекты хряща, остеоартрит, повреждение связки, незавершенный остеогенез, остеонекроз, остеопороз, апластическая анемия, болезнь «трансплантат против хозяина», миелодиспластический синдром, диабет типа 1, диабет типа 2, аутоиммунный гепатит, цирроз печени, печеночная недостаточность, дилатационная кардиомиопатия, сердечная недостаточность, инфаркт миокарда, ишемия миокарда, болезнь Крона, язвенный колит, ожоги, буллезный эпидермолиз, красная волчанка, ревматоидный артрит, болезнь Шегрена, системный склероз, бронхолегочная дисплазия, хроническое обструктивное заболевание дыхательных путей, эмфизема, легочный фиброз, боковой амиотрофический склероз (ALS), болезнь Альцгеймера, травма мозга, атаксия, остеохондроз, множественная системная атрофия, рассеянный склероз, болезнь Паркинсона, пигментная дистрофия сетчатки, болезнь Ромберга, травма спинного мозга, инсульт, мышечная дистрофия, ишемия конечности, повреждение почки, волчаночный нефрит, эндометриоз и осложнения трансплантации костного мозга или солидного органа.
Термин «состояние» в том виде, в котором он здесь используется, включает состояние, заболевание или расстройство, и их симптом.
Термин «терапевтическая композиция» в том виде, в котором он здесь используется, относится к клеткам, происходящим от PSC, как здесь описано, которые были приготовлены для введения субъекту. Предпочтительно терапевтическая композиция является стерильной. Это легко осуществляется фильтрованием через мембраны для стерилизующего фильтрования. Предпочтительно терапевтическая композиция является апирогенной.
Термин «приготовление» в том виде, в котором он здесь используется, относится к смешиванию культуры клеток, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, с возможными фармацевтически приемлемыми носителями, эксципиентами или стабилизаторами и поддержанию жизнеспособности клеток. Приемлемые носители, эксципиенты или стабилизаторы являются нетоксичными для субъектов-реципиентов при используемых дозировках и концентрациях, и могут включать: буферы, такие как фосфатный, цитратный и на основе других органических кислот; антиоксиданты, включающие аскорбиновую кислоту и метионин; низкомолекулярные (меньше, чем примерно 10 остатков) полипептиды; белки, такие как сывороточный альбумин, желатин или иммуноглобулины; гидрофильные полимеры, такие как поливинилпирролидон; аминокислоты, такие как глицин, глутамин, аспарагин, гистидин, аргинин или лизин; моносахариды, дисахариды и другие углеводы, включающие глюкозу, маннозу или декстрины; хелаторы, такие как EDTA (этилендиаминтетрауксусная кислота); сахара, такие как сахароза, маннит, трегалоза или сорбит; солеобразующие противоионы, такие как натрий; комплексы металлов (например, комплексы Zn-белок); и/или неионные поверхностно-активные вещества, такие как TWEEN™, PLURONICS™ или полиэтиленгликоль (PEG).
Приготовленная в виде препарата фармацевтическая композиция также может содержать другие активные соединения, как необходимо для конкретного показания, которое лечат, предпочтительно соединения с дополняющими активностями, которые не оказывают вредного влияния друг на друга. Данная композиция может содержать цитотоксический агент, цитокин и/или агент, ингибирующий рост. Такие молекулы подходящим образом присутствуют в комбинации в количествах, которые являются эффективными для намеченной цели.
Клетки, происходящие от PSC, в которых остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, или терапевтическая композиция, содержащая такие клетки, происходящие от PSC, могут вводиться до, во время или после наличия состояния. В одном воплощении клетки, происходящие от PSC, или терапевтическая композиция вводятся во время воспаления. Клетки, происходящие из PSC, могут вводиться (а) в качестве предупредительной меры, (б) как только было диагностировано состояние, (в) когда другие лечения не имели успеха и/или (г) когда состояние развилось до заранее определенной степени тяжести.
В одном воплощении клетки, происходящие из PSC, в которых остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, или терапевтическая композиция, содержащая такие клетки, происходящие от PSC, предварительно обрабатывают до введения. Предварительная обработка может осуществляться фактором роста или посредством редактирования генов, например, где фактор роста может примировать клетки, происходящие от PSC, и редактирование генов может придавать новое терапевтическое свойство клеткам, происходящим из PSC.
Специалисту в данной области будет понятно то, что точный способ введения субъекту терапевтически эффективного количества клеток, происходящих из PSC, в которых остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, или терапевтической композиции, содержащей такие клетки, происходящие из PSC, будет осуществляться по решению лечащего врача со ссылкой на состояние, подлежащее лечению или предупреждению. На способ введения, включающий дозировку, комбинацию с другими агентами, расписание и частоту введения, и тому подобное может влиять диагноз вероятной восприимчивости субъекта на лечение клетками, происходящими из PSC, или терапевтической композицией, а также состояние и история заболевания субъекта.
Клетки, происходящие из PSC, в которых остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, будут приготовлены, дозированы и введены способом, согласующимся с надлежащей медицинской практикой. Факторы для рассмотрения в данном контексте включают конкретное состояние, которое лечат или предупреждают, конкретного субъекта, которого лечат, клинический статус субъекта, место введения, способ введения, схему введения, возможные побочные эффекты и другие факторы, известные практикующим врачам. Терапевтически эффективное количество клеток, происходящих из PSC, в которых остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, или терапевтической композиции, содержащей такие клетки, происходящие из PSC, подлежащее введению, будет определяться такими соображениями.
Клетки, происходящие из PSC, в которых остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, или терапевтическая композиция, содержащая такие клетки, происходящие из PSC, могут вводиться системно или периферически, например, путями, включающими внутривенный (в.в.), внутриартериальный, внутримышечный, внутрибрюшинный, интрацереброспинальный, подкожный (п.к.), внутрисуставной, интрасиновиальный, подоболочечный, внутрикоронарный, трансэндокардиальный, хирургическую имплантацию, местный и ингаляцию (например, внутрилегочную). Наиболее предпочтительно клетки, происходящие из PSC, или терапевтическая композиция вводится в.в. Клетки, происходящие из PSC, или терапевтическая композиция может вводиться в комбинации с каркасом биосовместимого вещества.
Термин «терапевтически эффективное количество» относится к количеству клеток, происходящих из PSC, в которых остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, или терапевтической композиции, содержащей такие клетки, происходящие от PSC, которое является эффективным для лечения состояния у субъекта.
Термины «лечить», «осуществление лечения» или «лечение» относятся как к терапевтическому лечению, так и к профилактическим или предупредительным мерам, где целью является предупреждение или уменьшение интенсивности состояния, заболевания или расстройства у субъекта, или замедление (ослабление) прогрессирования состояния, заболевания или расстройства у субъекта. Субъекты, нуждающиеся в лечении, включают субъектов уже с состоянием, заболеванием или расстройством, а также субъектов, у которых состояние, заболевание или расстройство подлежит предупреждению.
Термины «осуществление предупреждения», «предупреждение», «предупредительный» или «профилактический» относятся к предотвращению появления или к препятствованию, защите от появления состояния, заболевания или расстройства, включая ненормальность или симптом. Субъект, нуждающийся в предупреждении, может быть склонным к развитию состояния, заболевания или расстройства.
Термин «уменьшать интенсивность» или «уменьшение интенсивности» относится к уменьшению, ослаблению или устранению состояния, заболевания или расстройства, включая ненормальность или симптом. Субъект, нуждающийся в лечении, уже может иметь состояние, заболевание или расстройство, или может быть склонен к тому, чтобы иметь состояние, заболевание или расстройство, или может представлять собой того, у кого состояние, заболевание или расстройство подлежит предупреждению.
Термин «субъект» в том виде, в котором он здесь используется, относится к млекопитающему. Млекопитающее может представлять собой примата, в частности, человека, или может быть домашним, содержащимся в зоопарке животным или животным-спутником. Хотя, в частности, и рассматривается то, что способ и образующиеся в результате его MSC или популяция MSC, раскрытые в данном документе, подходят для медицинского лечения человека, они также применимы для ветеринарного лечения, включающего лечение домашних животных, таких как лошади, крупный рогатый скот и овцы, животных-спутников, таких как собаки и кошки, или животных, содержащихся в зоопарке, таких как кошачьи, псовые, полорогие жвачные и копытные.
Также здесь раскрыта культура клеток, в которой остаточные, недифференцированные PSC отсутствуют или присутствуют в меньшей доле, чем известная доля, при выявлении способом по первому аспекту.
Также здесь раскрыта терапевтическая композиция, изготовленная способом по второму аспекту.
Также здесь раскрыт набор для выявления остаточных, недифференцированных плюрипотентных стволовых клеток (PSC) в культуре клеток, дифференцированных из PSC, содержащий:
ламинин-521; и
Е-кадгерин; и
ингибитор ROCK.
В одном воплощении ингибитор ROCK представляет собой раскрытый здесь ингибитор ROCK.
В одном воплощении данный набор дополнительно содержит праймеры ПЦР и, возможно, зонд ПЦР для количественного измерения в культивируемых клетках экспрессии маркера остаточных, недифференцированных PSC. Праймеры и зонд ПЦР являются специфичными в отношении маркера остаточных, недифференцированных PSC. В одном воплощении праймеры и/или зонд ПЦР представляют собой раскрытые здесь праймеры и/или зонд ПЦР.
В одном воплощении данный набор содержит готовую к применению композицию для ПЦР, возможно кПЦР-РВ, именуемую «мастер-микс», содержащую компоненты для ПЦР, за исключением матрицы ДНК и праймеров, и, возможно, зонд. Данные компоненты могут включать буфер, дезоксинуклеотидтрифосфаты (дНТФ), Mg2+ и полимеразу. Мастер-миксы имеются в продаже, например, мастер-микс 2X TaqMan® GE.
В другом воплощении данный набор дополнительно содержит среду. В одном воплощении данная среда представляет собой раскрытую здесь среду. В одном воплощении данная среда содержит ингибитор ROCK.
В другом воплощении данный набор дополнительно содержит субстрат. Такой субстрат подходит для культивирования клеток, дифференцированных из PSC, в которых подлежат выявлению остаточные, недифференцированные PSC. Предпочтительно данным субстратом является пластмассовая культуральная посуда, такая как чашка, многолуночный планшет или колба. В одном воплощении субстрат покрыт ламинином-521 и Е-кадгерином.
В одном воплощении набор содержит инструкцию по применению данного набора в способе, включающем:
культивирование клеток на субстрате, покрытом ламинином-521 и Е-кадгерином, в среде, содержащей ингибитор ROCK;
количественное измерение экспрессии маркера в культивируемых клетках; и
сравнение экспрессии маркера в культивируемых клетках с экспрессией маркера в контрольной культуре клеток, содержащей известную долю PSC,
где меньшая экспрессия маркера в культуре клеток, чем экспрессия маркера в контрольной культуре клеток показывает отсутствие остаточных, недифференцированных PSC в культивируемых клетках или присутствие остаточных, недифференцированных PSC в культивируемых клетках в меньшей доле, чем известная доля PSC в контрольной культуре клеток.
В одном воплощении набор содержит инструкцию по применению данного набора согласно способу по первому аспекту.
В другом воплощении набор используют согласно способу по первому аспекту. Это может указываться термином «при использовании».
В предпочтительном воплощении iPSC-MSC культивируются в течение 3 суток в среде Е8, содержащей 10 мкМ Y27632, ингибитор ROCK, затем культивируются в течение 2 суток в отсутствие ингибитора ROCK, и экспрессия LIN28 количественно измеряется кПЦР-РВ и нормируется к экспрессии GAPDH, обеспечивая выявление 0,001% остаточных, недифференцированных iPSC.
Другие определения, которые могут помогать в понимании настоящего раскрытия, в частности, примеров, включают следующие.
а) Анализ экспрессии генов (GE) TaqMan® (20×): тип анализа кПЦР-РВ в реальном времени, доступный от Life Technologies, который разработан с использованием патентованных программного обеспечения и реактивов Applied Biosystems®. Данные анализы используют для количественного измерения специфических мРНК в образце и доступны в виде предварительно разработанных или сделанных на заказ анализов. Анализы GE TaqMan® предоставляют в виде 20× смеси геноспецифичных праймеров и зонда.
1) Зонд FAM™/MGB-NFQ: флуоресцентная репортерная молекула (FAM: 6- карбоксифлуоресцеин), ковалентно присоединенная к 5'-концу зонда TaqMan®. MGB- NFQ представляет собой связыватель малой бороздки-нефлуоресцентную молекулу- гаситель, ковалентно присоединенную к 3'-концу зонда TaqMan®. Во время амплификации зонд отщепляется, высвобождая репортер от гасителя. Образующийся флуоресцентный сигнал является пропорциональным концентрации мРНК в образце. Вместо FAM™ доступны другие флуоресцентные красители. VIC® обычно используется для эндогенных контрольных генов, используемых для нормирования, таких как GAPDH.
б) 2× мастер-микс TaqMan® GE: содержит ДНК-полимеразу AmpliTaq Gold®, UP (ультрачистая) для активации горячего старта, смесь дНТФ с дТТФ/дУТФ и урацил- ДНК-гликозилазой (UDG) для минимизации переходящего загрязнения ПЦР, и пассивный внутренний контроль на основе патентованного красителя ROX™.
в) кДНК: комплементарная ДНК, которая подвергается обратной транскрипции от мРНК с использованием обратной транскриптазы (ОТ).
г) мРНК: матричная РНК.
д) гДНК: геномная ДНК.
е) матрица: нуклеиновая кислота-мишень (например, кДНК), намеченная для амплификации посредством ПЦР.
ж) Общая РНК: сложная смесь видов РНК, включающая мРНК (~1-5%), рибосомальную РНК (рРНК) (~80%), транспортную РНК (тРНК) и микро РНК (миРНК). Точная композиция варьирует, в зависимости от типа клеток.
з) РНКаза: рибонуклеаза - класс ферментов, которые разрушают РНК, и которые очень трудно инактивировать.
и) Эндогенный контрольный ген: ген позитивного контроля, который экспрессируется во всех типах образцов. GAPDH: глицеральдегид-3-фосфатдегидрогеназа представляет собой обычный эндогенный контрольный ген, используемый для нормирования.
й) Ген-мишень: интересующий ген, который дифференциально экспрессируется в стволовых клетках по сравнению с дифференцированными клетками (например, в iPSC по сравнению с iPSC-MSC), такой как LIN28.
к) плюс ОТ и минус ОТ мастер-миксы: содержат необходимые реактивы для обратной транскрипции образцов общей РНК. Минус ОТ представляет собой негативный контроль, содержащий общую РНК, но не содержащий обратную транскриптазу. Образцы минус ОT анализируют на загрязнение образцов общей РНК посредством гДНК.
л) Реакционные смеси плюс/минус ОT: комбинация плюс/минус ОT мастер-микса с каждым образцом общей РНК или контролем для обратной транскрипции.
м) Смесь для амплификации кДНК: комбинация 2× мастер-микса TaqMan® GE с каждой реакционной смесью плюс/минус ОT.
н) Смесь для анализа GE (например, смесь для анализа LIN28 или GAPDH): комбинация 20× анализа TaqMan® GE с каждой смесью для амплификации кДНК для кПЦР-РВ в реальном времени.
о) Нематричный контроль (NTC): тип негативного контроля (NEG), в котором общая РНК заменяется водой в реакции кДНК плюс/минус ОT. Используется для выявления загрязнения реактивов матрицами нуклеиновых кислот.
п) SNP: однонуклеотидный полиморфизм.
р) Cq: цикл количественного измерения (например, Cq(X) представляет собой цикл, где флуоресцентный сигнал пересекает пороговое значение для анализов кПЦР в реальном времени и кПЦР-РВ. Cq обратно связан с концентрацией мРНК. По мере увеличения Cq концентрация мРНК снижается и наоборот. «Cq(50)» показывает то, что было проведено 50 циклов. «Нет Cq(50)» показывает то, что за 50 циклов не было получено значение Cq.
ПРИМЕРЫ Пример 1 - получение iPSC-MSC Материалы

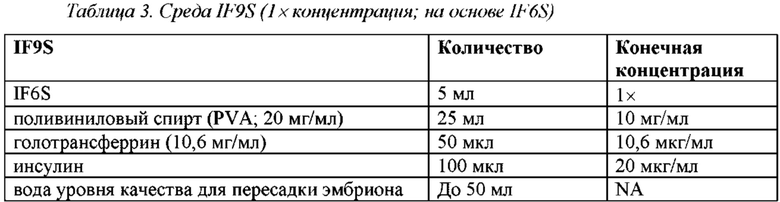
Реактивы, перечисленные в Таблице 1, известны специалисту в данной области и имеют принятые составы, например, IMDM и F12 Хэма. GLUTAMAX содержит дипептид L-аланил-L-глутамин, обычно поставляемый в концентрации 200 мМ в 0,85% NaCl. GLUTAMAX высвобождает L-глутамин при расщеплении культивируемыми клетками связи данного дипептида. Химически определенный липидный концентрат содержит 2 мг/л арахидоновой кислоты, 220 мг/л холестерина, 70 мг/л DL-альфа- токоферола ацетата, 10 мг/л линолевой кислоты, 10 мг/л линоленовой кислоты, 10 мг/л миристиновой кислоты, 10 мг/л олеиновой кислоты, 10 мг/л пальмитиновой кислоты, 10 мг/мл пальмитоолеиновой кислоты, 90 г/л плюроника F-68, 10 мг/л стеариновой кислоты, 2,2 г/л TWEEN 80® и этиловый спирт. Н-1152 и Y27632 являются высокоэффективными проницаемыми в клетку селективными ингибиторами ROCK (Rho-ассоциированная серин/треониновая протеинкиназа, образующая суперспираль).


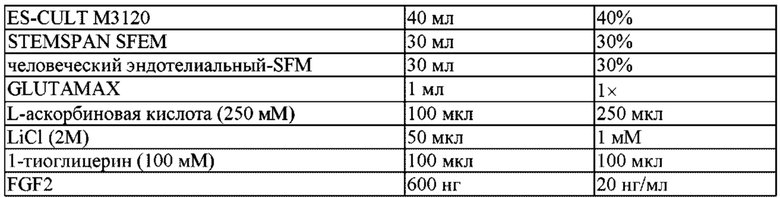
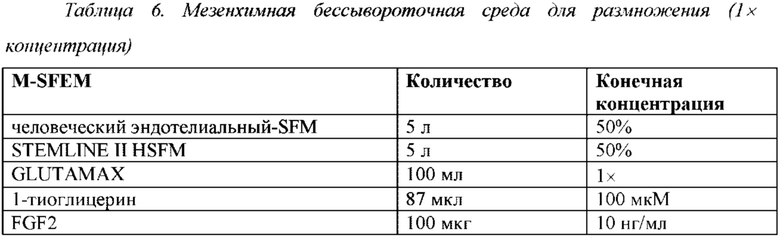
Способ
1. Размораживали iPSC в полной среде Е8 (базальная среда DMEM/F12 плюс добавка Е8) плюс 1 мкМ Н1152 на пластмассовой посуде, покрытой витронектином (0,5 мкг/см2). Инкубировали пересаженные на чашки iPSC при 37°С, 5% СО2, 20% О2 (нормоксические).
2. Проводили три пассажа размноженных iPSC в полной среде Е8 (без ингибитора ROCK) на пластмассовой посуде, покрытой витронектином (0,5 мкг/см2), и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) до инициации процесса дифференциации.
3. Отбирали и высевали iPSC в виде одиночных клеток/маленьких колоний в плотности 5×103 клеток/см2 на пластмассовую посуду, покрытую коллагеном IV (0,5 мкг/см2), в полной среде Е8 плюс 10 мкМ Y27632 и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) в течение 24 ч.
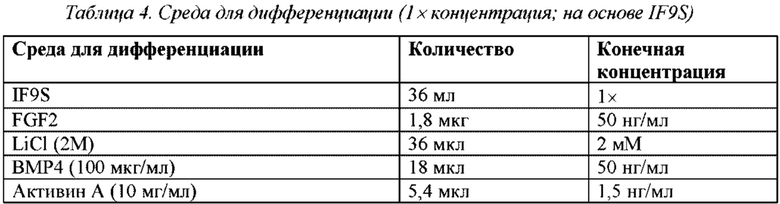
4. Заменяли полную среду Е8 плюс 10 мкМ Y27632 средой для дифференциации и инкубировали при 37°С, 5% СО2, 5% О2 (гипоксические) в течение 48 ч.
5. Отбирали колониеобразующие клетки из прикрепленной культуры в среде для дифференциации в виде суспензии одиночных клеток, переносили в суспензионную культуру с M-CFM и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) в течение 12 суток.
6. Отбирали и высевали колонии (пассаж 0) на пластмассовую посуду, покрытую фибронектином/коллагеном I (0,67 мкг/см2 фибронектина, 1,2 мкг/см2 коллагена I) в M-SFEM и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) в течение 3 суток.
7. Отбирали колонии и высевали в виде одиночных клеток (пассаж 1) в плотности 1,3×104 клеток/см2 на пластмассовую посуду, покрытую фибронектином/коллагеном I, в M-SFEM и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) в течение 3 суток.
8. Отбирали и высевали в виде одиночных клеток (пассаж 2) в плотности 1,3×104 клеток/см2 на пластмассовую посуду, покрытую фибронектином/коллагеном I, в M-SFEM и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) в течение 3 суток.
9. Отбирали и высевали в виде одиночных клеток (пассаж 3) в плотности 1,3×104 клеток/см2 на пластмассовую посуду, покрытую фибронектином/коллагеном I, в M- SFEM и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) в течение 3 суток.
10. Отбирали и высевали в виде одиночных клеток (пассаж 4) в плотности 1,3×104 клеток/см2 на пластмассовую посуду, покрытую фибронектином/коллагеном I, в M-SFEM и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) в течение 3 суток.
11. Отбирали и высевали в виде одиночных клеток (пассаж 5) в плотности 1,3×104 клеток/см2 на пластмассовую посуду, покрытую фибронектином/коллагеном I, в M-SFEM и инкубировали при 37°С, 5% СО2, 20% О2 (нормоксические) в течение 3 суток.
12. Отбирали пассаж 5 (Р5) iPSC-MSC в виде одиночных клеток и замораживали конечный продукт.
Пример 2 - количественное измерение остаточных недифференцированных стволовых клеток посредством кПЦР-РВ
1) ЦЕЛЬ
Данный типичный протокол описывает количественное измерение остаточных, недифференцированных iPSC посредством анализов экспрессии генов TagMan® (кПЦР-РВ). Несмотря на то, что в качестве примера приводится выявление недифференцированных iPSC среди iPSC-MSC с использованием экспрессии LIN28, данный протокол, в общем, применим к культуре клеток, происходящих от PSC, с использованием любого маркера PSC, дифференциально экспрессируемого в PSC, но не экспресируемого в клетках, происходящих от PSC. Данный протокол предназначен для контроля качества клеток, происходящих от PSC, предназначенных для приготовления в виде терапевтической композиции.
2) МАТЕРИАЛЫ
а) Материалы для протокола для культуры размножения iPSC-MSC c ламинином-521/Е-кадгерином:
i) Человеческий рекомбинантный ламинин-521 - LN521™, Biolamina, каталожный № LN521-03.
ii) Е-кадгерин, человеческий, рекомбинантный, Advanced BioMatrix, каталожный № 5085-0.1MG.
iii) Фосфатно-солевой буфер Дульбекко (1×) с Mg2+ и Са2+ (DPBS++) (6 × 500 мл), Corning/Mediatech, каталожный № 21-030-СV или эквивалентный.
iv) Фосфатно-солевой буфер Дульбекко HyClone™ без Mg2+ и Са2+, раствор, (DPBS--), GE Healthcare Life Sciences, каталожный № SH30028 или эквивалентный.
v) Ингибитор ROCK Y27632, Sigma-Aldrich, каталожный № Y0503-1MG или Y0503-5MG
vi) Питательная среда 8™ (набор), Thermo Fisher Scientific, каталожный № A1517001.
(1) Базовая питательная среда 8™ (500 мл)
(2) Питательная добавка 8™ (Витронектин) (10 мл)
vii) Фермент TrypLE™ (1×), без фенолового красного (100 мл), Thermo Fisher Scientific, каталожный № 12563011.
viii) Диметилсульфоксид (DMSO), Sigma-Aldrich, каталожный № D2650.
ix) 75 см2 прямоугольный флакон для культуры клеток со скошенным горлышком и вентилируемой крышкой Falcon®, Corning Life Sciences, каталожный №353136 или эквивалентный.
x) Скребок для клеток Falcon® с 25 см ручкой и 1,8 см лезвием, стерильный, Corning Life Sciences, каталожный № 353086 или эквивалентный.
xi) 0,4%-ный трипановый синий HyClone™, Thermo Fisher Scientific, каталожный № SV30084 или эквивалентный.
xii) Гемоцитометр с покровным стеклом, Reichert Right Line или эквивалентный.
б) Мининабор RNeasy® Protect Cell (50): QIAGEN®, каталожный № 74624. Состоит из двух продуктов QIAGEN®:
i) Реактив RNАрrotect® Cell (коробка 1 из 2): разработан для культивируемых или сортированных клеток. Может быть добавлен непосредственно к клеткам в культуральную среду. Останавливает картины экспрессии генов, давая немедленную стабилизацию общей РНК. Образцы можно хранить при 4°С, -20°С или архивировать при -80°С.
ii) Мининабор RNeasy® Plus (коробка 2 из 2): разработан для выделения общей РНК из целого ряда типов образцов. «Plus» указывает на то, что данный набор содержит колонки для устранения геномной ДНК (гДНК) и связанных с ней реактивов.
в) QIAshredder (50): каталожный № 79654 QIAGEN®. Требуется для гомогенизации клеточных лизатов для выделения РНК.
г) Набор ДНКазы, не содержащей РНКазы (50 препаратов), QIAGEN®, каталожный № 79254. Для обработки на мембране ДНКазой с использованием наборов для очистки РНК QIAGEN®.
д) Этанол (96-100%): Fisher BioReagents, уровень качества для молекулярной биологии, абсолютнный (мера крепости 200), 100 мл, каталожный № ВР2818100 Fisher Scientific или эквивалентный. Требуется для мининабора RNeasy® Plus от QIAGEN®.
е) ДНКаза UltraPure™/дистиллированная вода, не содержащая РНКазу, Life Technologies, каталожный № 10977-015 или эквивалентный.
ж) β-Меркаптоэтанол (β-МЕ) 14,3 М, Thermo Fisher Scientific, каталожный № ВР176-100 или эквивалентный. Требуется для мининабора RNeasy® Plus от QIAGEN® для инактивации РНКаз.
з) Порошок для контроля биологически опасных жидкостей Safetec Green-Z™, Thermo Fisher Scientific, каталожный № 19-023-901. Для удаления реактива RNAprotect® Cell, который является опасным для окружающей среды.
и) Реактив для удаления загрязнений RNase AWAY™ (250 мл), Life Technologies, каталожный № 10328-011 или эквивалентный. Нанести готовый для применения раствор на поверхности, такие как лабораторные столы, пипетки, стеклянная или пластмассовая посуда. Промыть водой Milli-Q для устранения загрязнения РНКазой и ДНК.
й) Высокоэффективный набор RNA-to-cDNA™ (50): Thermo Fisher Scientific, каталожный № 4387406.
к) Ингибитор РНКазы SUPERase⋅In™: Thermo Fisher Scientific, каталожный № AM2694.
л) Мастер-микс для экспрессии генов TaqMan® (2×): Thermo Fisher Scientific, каталожный № 4369016 (1 упаковка, 5 мл: 200 × 50 мкл реакции).
м) Вода уровня качества для ПЦР-РВ Ambion®: Thermo Fisher Scientific, каталожный № AM9935 (10 × 1,5 мл) (предпочтительная) или эквивалентная. Примечание: AM9935 является автоклавированной, фильтрованной на мембране и не обработанной DEPC. AM9935 тестируется на загрязнение прокариотической (16S рРНК) и эукариотической (18S рРНК) геномной ДНК посредством ПЦР в реальном времени. Она является сертифицированной, не содержащей РНКазы, не содержащей ДНКазы и не содержащей геномную ДНК.
н) Анализ(ы) экспрессии генов TaqMan®, который(рые) содержит(жат) 20×, готовую к применению смесь праймеров и зонда FAM/MGB-NFG в ТЕ. FAM представляет собой самый обычный флуоресцентный репортер. (1× - 250 нМ зонд TaqMan плюс 900 нМ каждый ПЦР-праймер (прямой и обратный)). Каталожные номера, приведенные ниже, относятся к размеру анализа, изготовленного на заказ. Светочувствительные. Предпочтительно хранить в виде аликвот для одноразового применения при -20°С в 1,5 мл антипригарных пробирках Ambion®.
i) Сделанные на заказ анализы экспрессии генов TaqMan® (20×, одна пробирка), Thermo Fisher Scientific, сделанные по заказу. (Заметьте то, что сделанные на заказ анализы могут быть заказаны только с зондами FAM-MGB).
(1) Каталожный № 4331348 (маленький - 360 реакций по 20 мкл для кПЦР-РВ).
(2) Каталожный № 4332078 (средний - 750 реакций по 20 мкл для кПЦР-РВ).
ii) Сделанные на заказ анализы экспрессии гена LIN28 TaqMan®, Thermo Fisher Scientific; каталожный №: 4331348; ID анализа: AIVI48S; название анализа: LIN28. кПЦР-РВ; масштаб: малый: 360 реакций; сделанные на заказ. Это сконструированный по заказу анализ с известными последовательностями праймеров и зондов, как раскрыто в Kuroda, et al. (PLoS ONE 7, 1-9 (2012) Highly Sensitive In Vitro Methods for Detection of Residual Undifferentiated Cells in Retinal Pigment Epithelial Cells Derived from Human iPS Cells). Последовательность зонда LIN28 (5'→3') CGCATGGGGTTCGGCTTCCTGTCC (SEQ ID NO: 14); последовательность прямого праймера LIN28 (5'→3') CACGGTGCGGGCATCTG (SEQ ID NO: 15); последовательность обратного праймера LIN28 (5'→3') CCTTCCATGTGCAGCTTACTC (SEQ ID NO: 16).
iii) Анализы эндогенного контроля TaqMan®: данные анализы были предварительно разработаны и могут быть инвентаризированы. Эндогенные контроли доступны с зондами FAM-MGB или VIC-MGB. Предпочтительно выбирают зонд VIC- MGB для отличения контрольного анализа от различающего анализа.
(1) Анализ экспрессии генов GAPDH (глицеральдегид-3-фосфат-дегидрогеназа) (символ гена: GAPDH, hCG2005673), Life Technologies; каталожный №: 4448489; ID анализа: Hs02758991_g1 (зонд VIC-MGB); сделан на заказ. Анализ «наилучшего покрытия» был выбран из многих предварительно разработанных анализов GAPDH. Данный анализ охватывает интрон и, следовательно, не должен выявлять геномную ДНК.
о) Антипригарные, не содержащие РНКаз микроцентрифужные пробирки Ambion® (1,5 мл): Life Technologies; каталожный № АМ12450. Примечание: 1,5 мл пробирки АМ12450 имеют поверхность со слабым связыванием и являются сертифицированными, не содержащими РНКаз и не содержащими ДНКаз. Предпочтительно используются для хранения аликвот сделанного на заказ анализа GE TaqMan® (20×) и получения кДНК.
п) 1,5 мл микроцентрифужные пробирки с закручивающейся крышкой (Sarstedt 72.692.005): Fisher Scientific, каталожный № 50809238 или эквивалентный; коническое дно, стерильные с собранной крышкой с О-кольцом. Предпочтительно используются для получения амплификационных смесей кДНК. Винтовая крышка с наружной резьбой предотвращает образование аэрозолей, которые, в противном случае, могли бы вызвать загрязнение ПЦР.
р) Белые, 96-луночные окантованные ПЦР-планшеты с твердыми стенками и низким профилем: Bio-Rad, каталожный № HSP-9655. Белый планшет будет уменьшать фоновый шум и увеличивать флуоресцентный сигнал по сравнению с прозрачными 96- луночными планшетами и пробирками. Для данной процедуры не следует использовать полосатые пробирки.
с) Адгезивные запечатывающие средства Microseal 'B' (100): Bio-Rad, каталожный № MSB-1001. Это рекомендованное оптически прозрачное запечатывающее средство для твердостенных 96-луночных планшетов.
т) Запечатывающий ролик: Bio-Rad, каталожный № MSR-1001. Используется для запечатывания твердостенного 96-луночного планшета адгезивным запечатывающим средством Microseal 'B'.
у) Выявляющая система ПЦР в реальном времени Bio-Rad CFX96 с программой Bio-Rad CFX (ID 0394) или эквивалентной.
ф) Микроцентрифуга Eppendorf MiniSpin Plus (ID 0569) или эквивалентная.
ц) Спектрофотометр, работающий в УФ (ультрафиолетовая) - видимой области Nanodrop 2000 (ID 0504), или эквивалентный.
3) МЕТОДИКА
а) Получение конечного продукта iPSC-MSC P5 (1 флакон) для анализа кПЦР-РВ с использованием селективного размножения остаточных недифференцированных iPSC
i) Данная методика селективно размножает недифференцированные iPSC на фоне iPSC-MSC для того, чтобы увеличивать чувствительность кПЦР-РВ в отношении остаточных недифференцированных iPSC.
ii) Получение сред для культуры клеток:
(1) Получение полной среды Е8 (Е8СМ):
(а) Разморозить добавку Е8 в течение ночи при 2-8°С.
(б) Удалить 10 мл базальной среды Е8 из 500 мл бутыли.
(в) Добавить 10 мл добавки Е8 к 490 мл базальной среды Е8. Хорошо смешать и хранить при 2-8°С. Срок хранения составляет 2 недели после приготовления.
(2) Получение Е8СМ плюс 10 мкМ среды Y27632:
(a) Приготовить 10 мМ Y27632 посредством добавления 312 мкл DPBS - (без Ca2+ и Mg2+) к 1 мг Y27632. Аликвотировать и заморозить при -20°С.
(б) Добавить 1 мкл 10 мМ Y27632 в каждый мл Е8СМ для достижения конечной концентрации 10 мкМ Y27632 (например, 75 мкл 10 мМ Y27632 в 75 мл Е8СМ). Хранить при 4°С. Срок хранения составляет 2 недели после приготовления.
iii) Приготовление замораживающей среды (Е8СМ плюс 20% DMSO):
(1) Приготовить 10 мл замораживающей среды следующим образом: объединить 8 мл Е8СМ с 2 мл DMSO. Перенести в 4°С для охлаждения перед применением.
(2) Добавить 1 мкл 10 мМ Y27632 в каждый мл Е8СМ для достижения конечной концентрации 10 мкМ Y27632 (например, 75 мкл 10 мМ Y27632 в 75 мл Е8СМ). Хранить при 4°С. Срок хранения составляет 2 недели после приготовления.
iv) Приготовление покрытия ламинин-521/Е-кадгерин (в DPBS++):
(1) Разморозить ламинин-521 и Е-кадгерин в течение ночи при 2-8°С.
(2) Приготовить вещество покрытия (30 мл, требующихся для 2 × флакона Т75):
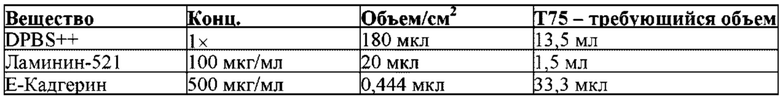
(3) Покрыть культуральную посуду (2 × флакона Т75) и хранить при 2-8°С в течение по меньшей мере ночи до применения (срок хранения 1 месяц). Убедиться в том, что раствор покрытия покрывает всю культуральную область.
v) Приготовление культуры для размножения
(1) Минимум за 1 час до посева удалить вещество покрытия из флаконов и добавить 15 мл E8CM плюс 10 мкМ Y27632/T75. Дать уравновеситься при 37°С.
(2) Разморозить iPSC-MSC P5 на водяной бане при 37°С.
(3) Перенести содержимое флакона в 15 мл пробирку, содержащую 9 мл E8CM плюс 10 мкМ Y27632.
(4) Центрифугировать при 200 g в течение 5 минут.
(5) Отсосать супернатант и ресуспендировать осадок в 5 мл E8CM плюс 10 мкМ Y27632.
(6) Подсчитать клетки с использованием гемоцитометра. Оценочная концентрация 5×106 клеток/5 мл - 1×106 клеток/мл. Предложенное разведение в трипановом синем составляет 1:2. Рассчитать процент жизнеспособности.
(7) Рассчитать число клеток, требующееся для посева в каждый флакон Т75 в плотности 1,3×104 клеток/см2 или 1×105 клеток/Т75).
(8) Засеять флаконы, покрытые ламинином-521/Е-кадгерином, рассчитанным числом клеток.
(9) Подпитывать клетки по следующей схеме: (а) Сутки 1 - нет подпитки.
(б) Сутки 2 - подпитать каждый флакон Т75 15 мл E8CM плюс 10 мкМ Y27632. (в) Сутки 3 - нет подпитки.
(г) Сутки 4 и 5 - подпитать каждый флакон Т75 15 мл E8CM (без Y27632).
(10) Ежесуточно проверять культуры на признаки микробного загрязнения (например, мутность). Отбрасывать любые загрязненные культуры и начинать заново со стадии 3) а).
vi) Сбор (сутки 6 после посева)
(1) Примечания: клетки трудно отделять от культуральной посуды, покрытой ламинином-521/Е-кадгерином. Применение клеточных скребков рекомендуется для отделения всех клеток. Средняя плотность сбора составляет 4×104 клеток/см2 или 3×106 клеток/флакон Т75. При сборе недостаточного числа клеток из одного флакона Т75 (меньше 2×106 клеток) может потребоваться сбор со второго Т75.
(2) Отсосать ростовую среду и промыть флакон 10 мл DPBS--.
(3) Добавить во флакон Т75 8 мл TrypLE. Инкубировать при 37°С в течение 15 минут.
(4) Решительно постучать по флакону для того, чтобы выбить клетки и перенести в 50 мл коническую пробирку (приблизительно 8 мл).
(5) Добавить во флакон 8 мл Е8СМ плюс 10 мкМ Y27632, всасывать в пипетку и выпускать из нее, и объединить с клеточной суспензией в 50 мл пробирке (всего 16 мл).
(6) Добавить во флакон 8 мл Е8СМ плюс 10 мкМ Y27632 и использовать стерильный одноразовый скребок для клеток для аккуратного соскабливания клеток. Собрать клетки посредством промывки средой соскобленной поверхности и объединить с клеточной суспензией (всего 24 мл).
(7) Повторить соскабливание после добавления во флакон дополнительных 8 мл Е8СМ плюс 10 мкМ Y27632. Собрать суспензию клеток, как описано выше (всего 32 мл).
(8) Центрифугировать клетки при 200 g в течение 10 минут. Отсосать супернатант.
(9) Ресуспендировать клеточный осадок в Е8СМ плюс 10 мкМ Y27632 и подсчитать клетки. Предложенный ресуспензионный объем составляет 2 мл с оценочной концентрацией 3×106 клеток/2 мл - 1,5×106 клеток/мл. Предложенное разведение в трипановом синем составляет 1:2. Для точного подсчета может потребоваться диссоциация на одиночные клетки с использованием наконечника 1 мл пипетки.
(а) Подсчитать клетки с использованием гемоцитометра.
(б) Требующаяся процентная доля жизнеспособности составляет 70% или более. Требующийся выход составляет 2×106 клеток или более.
(10) Центрифугировать клетки при 200 g в течение 10 минут. Отсосать супернатант.
(11) Ресуспендировать клеточный осадок в Е8СМ при 2×106 клеток/мл.
vii) Заморозить клетки до анализа
(1) Добавить равный объем замораживающей среды COLD (Е8СМ с 20% DMSO) для получения конечной концентрации 1×106 клеток/мл.
(2) Добавить 1 мл клеточной суспензии в каждый криофлакон.
(3) Немедленно перенести криофлаконы в холодильник на -80°С. (4) Перенести на следующие сутки в емкость с жидким азотом.
б) Получение реактивов для набора RNeasy® Plus Mini:
i) Получение промывочного раствора буфера RPE oт QIAGEN®:
(1) Добавить 100% этанол, как указано на бутыли, в концентрат буфера RPE (т.е. 44 мл для набора RNeasy® Protect Cell Mini на 50 препаратов).
ii) Приготовление 70%-ного этанола:
(1) Объединить 7 мл 100%-ного этанола с 3 мл воды, не содержащей нуклеаз. Срок хранения - один месяц после приготовления.
iii) Получение лизирующего буфера для выделения общей РНК:
(1) Добавить 10 мкл β-меркаптоэтанола (β-МЕ) на 1 мл буфера RLT Plus до применения, используя химический вытяжной шкаф.
iv) Получение ДНКазы, не содержащей РНКаз, для набора ДНКазы, не содержащей РНКаз, от QIAGEN®:
(1) Добавить указанное количество воды, не содержащей РНКаз (т.е. 550 мкл) к лиофилизированной ДНКазе с использованием стерильной иглы и шприца. Осторожно ресуспендировать ДНКазу посредством переворачивания. Не встряхивать. Хранить разведенную ДНКазу при -20°С в 20 мкл аликвотах, предпочтительно в антипригарных микроцентрифужных пробирках, не содержащих РНКаз, от Ambion®. Срок хранения разведенной ДНКазы составляет девять месяцев после приготовления. Хранить остальные компоненты набора при 2-8°С.
в) Получение культивируемых клеток для выделения общей РНК с использованием реактива для клеток RNAprotect®
i) Реактив для клеток RNAprotect® немедленно стабилизирует общую РНК в обработанных клетках, сохраняя профиль экспрессии генов. Данный реактив добавляют непосредственно к клеткам, в присутствии среды или без нее. Также могут быть приготовлены замороженные клетки в DMSO с использованием модифицированного протокола.
(1) Рекомендованное число клеток составляет 1-2×106 клеток на общий минипрепарат РНК. Оно может увеличиваться до максимум 5×106 клеток на минипрепарат без изменения объема реактивов. Для большего числа клеток обработайте многие минипрепараты или используйте методику для препаратов РНК среднего размера, следуя протоколу изготовителя.
(2) Используйте отношение 1 мл образца к 5 мл реактива для клеток RNAprotect®.
ii) Приготовьте подходящее число 15 мл пробирок, содержащих 5 мл реактива для клеток RNAprotect®.
iii) Замороженные клетки: поместить флаконы с замороженными клетками на сухой лед для предотвращения оттаивания. Не размораживать клетки до добавления реактивов. Обрабатывать один флакон за раз для максимизации защиты РНК.
(1) Перенести ~750 мкл реактива для клеток RNAprotect® из одной 15 мл пробирки в криофлакон с замороженными клетками, содержащий ~1 мл замораживающей среды. Довести объемы, как необходимо для других сценариев. Не переполнять.
(а) Использовать высококачественные наконечники пипеток с малым удерживанием для предотвращения потерь при пипетировании.
(2) Немедленно встряхнуть смесь в течение ~10 секунд. Быстро перенести частично оттаявшую смесь обратно в ту же самую 15 мл пробирку, содержащую реактив для клеток RNAprotect®.
(3) Повторять стадии 3) в) iii) (1) и 3) в) iii) (2), пока смесь полностью не оттает. На это уйдет 3-4 повтора.
(4) Хорошо перемешать посредством встряхивания, пока образец не станет гомогенным.
iv) Немедленно перейти к выделению общей РНК или хранить полученные образцы в реактиве для клеток RNAprotect®. Хранить образцы при 2-8°С в течение вплоть до 4 недель или архивировать при -20°С или -80°С.
г) Приготовление клеточных лизатов для выделения общей РНК.
i) При нахождении в замороженном состоянии разморозить клеточную смесь RNAprotect® при комнатной температуре без смешивания.
ii) Центрифугировать клеточную смесь RNAprotect® в течение 5 минут при 4000 об./мин (Sorvall ID 0018 или эквивалентная) для сбора клеток и любого осадка, который возможно образовался. Должен образоваться видимый белый осадок.
iii) Удалить столько супернатанта, сколько возможно, в контейнер для отходов. Постучать по пробирке или встряхнуть для разрыхления осадка. Это очень важно для полной солюбилизации на следующей стадии.
(1) Добавить 350 мкл свежеприготовленного буфера RLT Plus плюс β- меркаптоэтанол к осадку. Энергично встряхивать в течение 1-2 минут, пока осадок полностью не растворится. При необходимости данный лизат можно хранить при -80°С. Разморозить при комнатной температуре и встряхнуть перед применением.
д) Выделение общей РНК с использованием набора QIAGEN® RNeasy® Plus Mini.
Все стадии проводятся при комнатной температуре. Выделить общую РНК из следующих криоконсервированных образцов. Разводить в 30 мкл воды, не содержащей нуклеаз.
№1: iPSC-MSC (неразмноженные) (контрольная культура)
№2: iPSC-MSC (размноженные) (контрольная культура)
№3: iPSC-MSC плюс 0,001% внутреннего стандарта iPSC (неразмноженные) (контрольная культура)
№4: iPSC-MSC плюс 0,001% внутреннего стандарта iPSC (размноженные) (контрольная культура)
№5: iPSC-MSC Р5 конечный продукт (неразмноженные)
№6: iPSC-MSC Р5 конечный продукт (размноженные)
i) Перенести лизат в QIAshredder. Центрифугировать на полной скорости в течение 2 минут для приложения усилия сдвига и гомогенизации лизата. На дне пробирки для сбора может образоваться тонкий круглый осадок.
ii) Перенести гомогенизированный лизат в собранную центрифугируемую колонку gDNA Eliminator, избегая любого осажденного вещества. Центрифугировать на полной скорости в течение 30 секунд. При необходимости, повторять, пока весь лизат не пройдет через центрифугируемую колонку.
iii) Добавить 350 мкл 70%-ного этанола в элюат и хорошо перемешать пипетированием. Не центрифугировать. Немедленно перенести данную смесь в собранную центрифугируемую колонку RNeasy® с любым осадком, который возможно образовался.
iv) Осторожно закрыть крышку и центрифугировать на полной скорости в течение 15 секунд. Отбросить элюат из пробирки для сбора. РНК теперь связана с центрифугируемой колонкой RNeasy®.
v) Обработка ДНКазой на мембране:
(1) Для максимального удаления геномной ДНК связавшуюся РНК обрабатывают ДНКазой, не содержащей РНКаз.
(а) Добавить 350 мкл буфера RW1 в центрифугируемую колонку. Осторожно закрыть крышку и центрифугировать на полной скорости в течение 15 секунд. Отбросить элюат из пробирки для сбора.
(б) Для каждого образца объединить 20 мкл разведенной ДНКазы, 140 мкл буфера RDD и 2 мкл SUPERase. In™. Осторожно смешать пипетированием и объединить все в одной пробирке. Не встряхивать.
(в) Отобрать пипеткой 80 мкл смеси ДНКазы непосредственно в центр мембраны RNeasy®. Инкубировать при комнатной температуре в течение 20 минут.
(г) Добавить 350 мкл буфера RW1 в центрифугируемую колонку. Осторожно закрыть крышку и центрифугировать на полной скорости в течение 15 секунд. Отбросить элюат из пробирки для сбора.
vi) Добавить 500 мкл буфера RPE в центрифугируемую колонку. Осторожно закрыть крышку и центрифугировать на полной скорости в течение 15 секунд. Отбросить элюат из пробирки для сбора.
vii) Повторить промывку добавлением 500 мкл буфера RPE в центрифугируемую колонку. Осторожно закрыть крышку и центрифугировать на полной скорости в течение 2 минут.
viii) Перенести центрифугируемую колонку в свежую 2 мл пробирку для сбора. Центрифугировать на полной скорости в течение 1 минуты для удаления любого остаточного промывочного раствора.
ix) Перенести центрифугируемую колонку в 1,5 мл пробирку для сбора. Добавить 30 мкл воды, не содержащей РНКаз, непосредственно на мембрану. Осторожно закрыть крышку и центрифугировать на полной скорости в течение 1 минуты для элюции РНК.
x) Отбросить центрифугируемую колонку и закрыть крышкой пробирку. Пометить пробирку датой и ID (идентификационный номер) образца.
xi) Нормировать объем каждого образца общей РНК до 30 мкл.
(1) Установить пипетку 10-100 мкл на 30 мкл и измерить объем элюата.
(2) Если объем меньше, чем 30 мкл, добавить воду, не содержащую нуклеаз, из набора RNeasy® до конечного объема 30 мкл.
(3) Образец теперь нормирован по отношению к исходному числу клеток.
xii) Определить общую концентрацию РНК для каждого образца с использованием Nanodrop 2000.
(1) Очистить основание образца водой Milli-Q до запуска программы для удаления любого потенциального загрязнения от предыдущего использования.
(2) Как только запускается программа, снять отметку с поля поправка на фон 340 нм.
(3) Очистить прибор водой Milli-Q.
(4) Проанализировать образец воды Milli-Q для убеждения в том, что фон равен нулю. Если необходимо, повторно очистить основание образца водой Milli-Q.
(5) Определить общую концентрацию РНК каждого образца с использованием одного определения.
(6) Рассчитать общий выход РНК и выход на клетку.
(7) Рассчитать объем 1,0-2,0 мкг общей РНК для каждого образца. (Максимум 2,0 мкг общей РНК может быть подвергнуто обратной транскрипции на реакцию кДНК).
(8) Концентрация общей РНК должна составлять 100 нг/мкл или более для удовлетворения требования 1 мкг общей РНК на 10 мкл (максимальный объем) для обратной транскрипции. Повторно выделить общую РНК из свежего флакона с клетками, если необходимо.
Ожидаемый выход общей РНК на 1×106 криоконсервированных iPSC-MSC составляет ~5-10 мкг. Это эквивалентно ~5-10 пг общей РНК на клетку.
xiii) Перейти к синтезу кДНК или хранить общую РНК при -20°С в течение вплоть до 1 года.
е) Получение кДНК с использованием высокоэффективного набора RNA-to-cDNA™
i) Для каждого образца общей РНК приготовить кДНК плюс/минус ОТ с использованием 1 мкг общей РНК / 22 мкл ОТ. Включить контроли Н2О (NTC) плюс/минус ОТ (N равно 14).
ii) Разморозить компоненты набора на льду.
iii) Приготовить один плюс ОТ и один минус ОТ мастер-микс на льду, в зависимости от числа образцов общей РНК, подлежащих анализу. Образцы минус ОТ служат в качестве негативных контролей, не имеющих обратной транскриптазы. Включить контроли Н2О (NTC) плюс/минус ОТ посредством замены общей РНК водой уровня качества для ПЦР-ОТ.
iv) Получить индивидуальные реакционные смеси плюс/минус ОТ на льду для каждого образца общей РНК.
(1) Максимальный объем общей ДНК составляет 10 мкл на 22 мкл плюс/минус ОТ. Объем общей РНК будет варьировать, в зависимости от концентрации общей РНК.
(2) Рассчитать объем воды уровня качества для ПЦР-ОТ для каждой пробирки путем вычитания объема общей РНК из 10 мкл.
(3) Перенести пипеткой подходящий объем воды уровня качества для ПЦР-ОТ в каждую пробирку. Использовать 10 мкл для контролей с Н2О плюс/минус ОТ.
(4) Добавить подходящий объем общей РНК в каждую пробирку.
(5) Добавить 12 мкл плюс ОТ или минус ОТ мастер-микса в каждую пробирку и осторожно смешивать пипетированием.
v) Инкубировать при 37°С в течение 60 минут.
vi) Инактивировать нагреванием при 95°С в течение 5 минут.
vii) Кратковременно центрифугировать пробирки для сбора кДНК.
viii) Измерить объем кДНК с использованием калиброванной пипетки на 10-100 мкл.
(1) Установить объем пипетки на реакционный объем кДНК (20-22 мкл) и измерить объем кДНК.
(2) Довести объем кДНК до 100 мкл водой, не содержащей нуклеаз. Это разбавит ингибиторы и даст достаточное количество кДНК для многих анализов кПЦР-РВ.
ix) Смешать посредством встряхивания и кратковременно осадить образцы центрифугированием.
x) Перейти к кПЦР-РВ или хранить при -20°С.
ж) кПЦР-РВ (анализы экспрессии генов TaqMan®)
i) Для каждой кДНК (N равно 14) амплифицировать 50 нг кДНК (эквивалентов общей РНК) на 20 мкл кПЦР-РВ для LIN28 и GAPDH (N равно 3 для каждого). Провести кПЦР-РВ с использованием 50 циклов. (Файл протокола: TaqMan qRT-PCR Assays Cq(50).prcl).
ii) Приготовить смесь для амплификации кДНК для каждой кДНК плюс ОТ и минус ОТ, в зависимости от числа повторов и числа анализов GE TaqMan®, подлежащих проведению.
(1) Рекомендованное максимальное количество кДНК на реакцию составляет 100 нг (эквивалентов общей РНК).
iii) Приготовить смесь для анализа GE для каждой кДНК, в зависимости от числа анализов GE TaqMan® и повторов. Одна смесь для анализа GE будет получена для каждой кДНК и каждого анализа GE TaqMan®, подлежащего проведению.
iv) Перенести 20 мкл каждой смеси для анализа GE в белый планшет CFX96 и запечатать оптическим приспособлением для запечатывания планшетов.
v) Центрифугировать планшет при 400 об./мин в течение 1 минуты в центрифуге с адаптером для планшетов (ID #0018) для сбора содержимого на дне лунок.
vi) Осторожно поместить плашет в термоциклер, будучи осторожным, чтобы не встряхивать содержимое лунок.
vii) Запрограммировать систему выявления ПЦР в реальном времени Bio-Rad CFX96 с использованием программы Bio-Rad CFX Manager™ на присоединенном компьютере со следующими параметрами термоциклирования:
(1) 2 минуты при 50,0°С (инкубация UNG; требуется для оптимальной активности урацил-N-гликозилазы; деградирует ранее амплифицированные ПЦР- продукты с включением дУТФ с уменьшением загрязнения ПЦР).
(2) 10 минут при 95°С (горячий старт: AmpliTaq Gold®, активация поимеразы UP).
(3) [15 секунд при 95°С (денатурация) плюс 1 минута при 60°С (отжиг/элонгация) плюс считывание планшета] - повторить 49 раз за 50 циклов (амплификация и выявление в реальном времени).
viii) Установить объем образца 20 мкл.
ix) Оценочное время прогона должно составлять 01:59:00 ч.
х) Считать все лунки и все каналы во время прогона для убеждения в том, что во время прогона отбираются все данные.
(1) Установки предварительного просмотра, расположенные выше схемы планшета, должны быть следующими:
(а) Флуорофоры: FAM, HEX, техасский красный, Cy5, Quasar 705 (VIC может быть добавлен позднее)
(б) Тип планшета: BR белый
(в) Способ сканирования: все каналы
xi) Все лунки должны быть помечены «Unk», указывая то, что программа рассматривает все лунки как неизвестные во время прогона. Реальная схема планшета будет установлена после завершения прогона.
xii) Нажмите Next для перехода к вкладке Start Run. Введите идентификационную информацию о прогоне в разделе Notes.
xiii) Нажмите на кнопку Start Run для начала кПЦР-РВ.
з) Анализ данных
i) Окно анализа данных будет открыто в конце прогона.
(1) Данные кПЦР-РВ приводятся в виде значений Cq (т.е. Cq(50)).
ii) Нажмите на кнопку Plate Setup сверху, на правой стороне окна.
(1) Выберите View/Edit Plate. Следуйте руководству по загрузке планшета для установки для планшета информации об образце.
(а) Очистите любые пустые лунки.
(б) Не ставьте метку «Exclude Wells in Analysis». Должны быть проанализированы все данные.
(2) Нажмите на OK и сохраните файл.
iii) Нажмите на вкладку Quantification около верхней части окна.
iv) Отметьте следующие установки:
(1) Способ определения Cq: один порог
(2) Установка фонового уровня: аппроксимация кривой с вычитанием фона
(3) Способ анализа: флуорофор
v) Генерировать отчет из меню Tools со следующими разделами и ассоциированными подразделами: Header, Run Setup, Quantification и QC Parameters.
(1) Снимите выделение с опций Gene Expression, Allelic Discrimination и End Point, так как данные разделы не являются релевантными.
vi) Экспортировать данные для анализа посредством выбора Export > “Export All Data Sheets to Excel”. Это будет давать данные в формате крупноформатной таблицы.
vii) Анализировать данные с использованием крупноформатной таблицы.
(1) Сообщить «N/A» (не доступно) в виде «Нет Cq(50)».
(2) Рассчитать средн. Cq(50) и % относительного стандартного отклонения (RSD или коэффициент вариации (CV)) для каждого набора данных LIN28 и GAPDH плюс/минус ОТ.
(3) Нормировать средн. значения Cq(50) посредством вычитания средн. Cq(50) GAPDH (N равно 3) из средн. Cq(50) LIN28 (N равно 3).
(4) Рассчитать средн. Cq(50) GAPDH и %RSD для всех образцов.
(5) Сравнить нормированные средн. значения Cq(50) образцов №2 (iPSC-MSC (размноженные) и №4 (iPSC-MSC плюс 0,001% внутреннего стандарта iPSC (размноженные)). Сравнить нормированные средн. значения Cq(50) образцов №4 (iPSC-MSC плюс 0,001% внутреннего стандарта iPSC (размноженные)) и №6 (конечный продукт - iPSC-MSC Р5 (размноженные)).
4) КРИТЕРИИ ПРИНЯТИЯ
а) Нормированное средн. значение Cq(50) образца №2 (iPSC-MSC (размноженные)) должно быть больше, чем нормированное средн. значение Cq(50) образца №4 (iPSC-MSC плюс 0,001% внутреннего стандарта iPSC (размноженные)). Это демонстрирует то, что 0,001% внутреннего стандарта iPSC (размноженные) может быть выявлено выше фона.
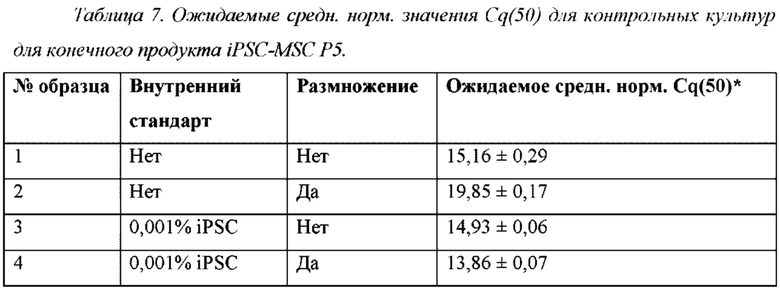
* Норм. Cq(50) LIN28 = (Cq(50) LIN28 - Cq(50) GAPDH) +/- станд. отклон. (N равно 3×3)
б) %RSD Cq(50) GAPDH для всех образцов должен составлять 5% или меньше. %RSD для всех средних значений Cq(50) и нормированных значений Cq(50) должен составлять 5% или меньше.
в) Контроли Н2О (NTC) (плюс/минус ОТ) не должны давать выявляемый амплификационный сигнал в пределах 50 циклов амплификации для всех повторностей.
i) Если любому из контролей Н2О (NTC) (плюс/минус ОТ) приписывается значение Cq(50), повторите анализы кПЦР-РВ TaqMan® со стадии 2) ж). Это указывает на загрязнение реактивов матричной ДНК.
Пример 3 - результаты протокола, проведенного согласно Примеру 2
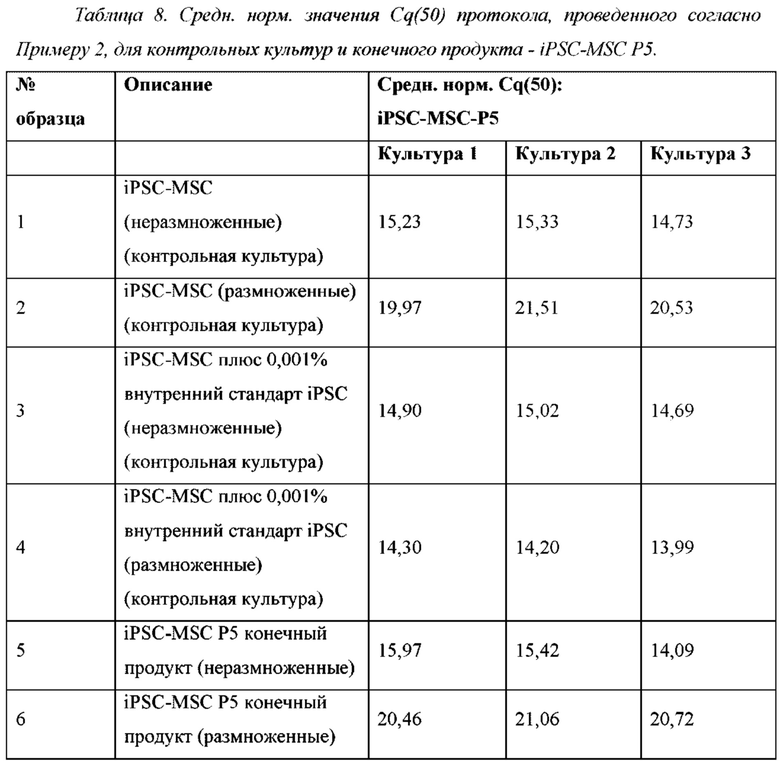
Пример 4 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют на ОСТ4 (POU5F1). Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 5 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют на SOX2. Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 6 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют на FOXD3. Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 7 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют NANOG. Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 8 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют PODXL. Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 9 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют на REX1 (ZFP42). Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 10 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют на SSEA1 (FUT4). Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 11 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют на SSEA4. Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 12 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют на DPPA2. Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
Пример 13 - количественное измерение остаточных, недифференцированных стволовых клеток посредством кПЦР-РВ
Данный пример описывает количественное измерение остаточных, недифференцированных iPSC по существу посредством протокола Примера 2, но LIN28 заменяют на DPPA3. Предполагаемые результаты согласуются с Примером 2, Таблицей 7 и Примером 3, Таблицей 8.
--->
СПИСОК ПОСЛЕДОВАТЕЛЬНОСТЕЙ
SEQUENCE LISTING
<110> Cynata Therapeutics Limited
<120> PLURIPOTENT STEM CELL ASSAY
<130> P104383.PCT
<150> AU 2016904679
<151> 2016-11-16
<160> 14
<170> PatentIn version 3.5
<210> 1
<211> 3695
<212> PRT
<213> Homo sapiens
<400> laminin-521 alpha chain
Met Ala Lys Arg Leu Cys Ala Gly Ser Ala Leu Cys Val Arg Gly Pro
1 5 10 15
Arg Gly Pro Ala Pro Leu Leu Leu Val Gly Leu Ala Leu Leu Gly Ala
20 25 30
Ala Arg Ala Arg Glu Glu Ala Gly Gly Gly Phe Ser Leu His Pro Pro
35 40 45
Tyr Phe Asn Leu Ala Glu Gly Ala Arg Ile Ala Ala Ser Ala Thr Cys
50 55 60
Gly Glu Glu Ala Pro Ala Arg Gly Ser Pro Arg Pro Thr Glu Asp Leu
65 70 75 80
Tyr Cys Lys Leu Val Gly Gly Pro Val Ala Gly Gly Asp Pro Asn Gln
85 90 95
Thr Ile Arg Gly Gln Tyr Cys Asp Ile Cys Thr Ala Ala Asn Ser Asn
100 105 110
Lys Ala His Pro Ala Ser Asn Ala Ile Asp Gly Thr Glu Arg Trp Trp
115 120 125
Gln Ser Pro Pro Leu Ser Arg Gly Leu Glu Tyr Asn Glu Val Asn Val
130 135 140
Thr Leu Asp Leu Gly Gln Val Phe His Val Ala Tyr Val Leu Ile Lys
145 150 155 160
Phe Ala Asn Ser Pro Arg Pro Asp Leu Trp Val Leu Glu Arg Ser Met
165 170 175
Asp Phe Gly Arg Thr Tyr Gln Pro Trp Gln Phe Phe Ala Ser Ser Lys
180 185 190
Arg Asp Cys Leu Glu Arg Phe Gly Pro Gln Thr Leu Glu Arg Ile Thr
195 200 205
Arg Asp Asp Ala Ala Ile Cys Thr Thr Glu Tyr Ser Arg Ile Val Pro
210 215 220
Leu Glu Asn Gly Glu Ile Val Val Ser Leu Val Asn Gly Arg Pro Gly
225 230 235 240
Ala Met Asn Phe Ser Tyr Ser Pro Leu Leu Arg Glu Phe Thr Lys Ala
245 250 255
Thr Asn Val Arg Leu Arg Phe Leu Arg Thr Asn Thr Leu Leu Gly His
260 265 270
Leu Met Gly Lys Ala Leu Arg Asp Pro Thr Val Thr Arg Arg Tyr Tyr
275 280 285
Tyr Ser Ile Lys Asp Ile Ser Ile Gly Gly Arg Cys Val Cys His Gly
290 295 300
His Ala Asp Ala Cys Asp Ala Lys Asp Pro Thr Asp Pro Phe Arg Leu
305 310 315 320
Gln Cys Thr Cys Gln His Asn Thr Cys Gly Gly Thr Cys Asp Arg Cys
325 330 335
Cys Pro Gly Phe Asn Gln Gln Pro Trp Lys Pro Ala Thr Ala Asn Ser
340 345 350
Ala Asn Glu Cys Gln Ser Cys Asn Cys Tyr Gly His Ala Thr Asp Cys
355 360 365
Tyr Tyr Asp Pro Glu Val Asp Arg Arg Arg Ala Ser Gln Ser Leu Asp
370 375 380
Gly Thr Tyr Gln Gly Gly Gly Val Cys Ile Asp Cys Gln His His Thr
385 390 395 400
Thr Gly Val Asn Cys Glu Arg Cys Leu Pro Gly Phe Tyr Arg Ser Pro
405 410 415
Asn His Pro Leu Asp Ser Pro His Val Cys Arg Arg Cys Asn Cys Glu
420 425 430
Ser Asp Phe Thr Asp Gly Thr Cys Glu Asp Leu Thr Gly Arg Cys Tyr
435 440 445
Cys Arg Pro Asn Phe Ser Gly Glu Arg Cys Asp Val Cys Ala Glu Gly
450 455 460
Phe Thr Gly Phe Pro Ser Cys Tyr Pro Thr Pro Ser Ser Ser Asn Asp
465 470 475 480
Thr Arg Glu Gln Val Leu Pro Ala Gly Gln Ile Val Asn Cys Asp Cys
485 490 495
Ser Ala Ala Gly Thr Gln Gly Asn Ala Cys Arg Lys Asp Pro Arg Val
500 505 510
Gly Arg Cys Leu Cys Lys Pro Asn Phe Gln Gly Thr His Cys Glu Leu
515 520 525
Cys Ala Pro Gly Phe Tyr Gly Pro Gly Cys Gln Pro Cys Gln Cys Ser
530 535 540
Ser Pro Gly Val Ala Asp Asp Arg Cys Asp Pro Asp Thr Gly Gln Cys
545 550 555 560
Arg Cys Arg Val Gly Phe Glu Gly Ala Thr Cys Asp Arg Cys Ala Pro
565 570 575
Gly Tyr Phe His Phe Pro Leu Cys Gln Leu Cys Gly Cys Ser Pro Ala
580 585 590
Gly Thr Leu Pro Glu Gly Cys Asp Glu Ala Gly Arg Cys Leu Cys Gln
595 600 605
Pro Glu Phe Ala Gly Pro His Cys Asp Arg Cys Arg Pro Gly Tyr His
610 615 620
Gly Phe Pro Asn Cys Gln Ala Cys Thr Cys Asp Pro Arg Gly Ala Leu
625 630 635 640
Asp Gln Leu Cys Gly Ala Gly Gly Leu Cys Arg Cys Arg Pro Gly Tyr
645 650 655
Thr Gly Thr Ala Cys Gln Glu Cys Ser Pro Gly Phe His Gly Phe Pro
660 665 670
Ser Cys Val Pro Cys His Cys Ser Ala Glu Gly Ser Leu His Ala Ala
675 680 685
Cys Asp Pro Arg Ser Gly Gln Cys Ser Cys Arg Pro Arg Val Thr Gly
690 695 700
Leu Arg Cys Asp Thr Cys Val Pro Gly Ala Tyr Asn Phe Pro Tyr Cys
705 710 715 720
Glu Ala Gly Ser Cys His Pro Ala Gly Leu Ala Pro Val Asp Pro Ala
725 730 735
Leu Pro Glu Ala Gln Val Pro Cys Met Cys Arg Ala His Val Glu Gly
740 745 750
Pro Ser Cys Asp Arg Cys Lys Pro Gly Phe Trp Gly Leu Ser Pro Ser
755 760 765
Asn Pro Glu Gly Cys Thr Arg Cys Ser Cys Asp Leu Arg Gly Thr Leu
770 775 780
Gly Gly Val Ala Glu Cys Gln Pro Gly Thr Gly Gln Cys Phe Cys Lys
785 790 795 800
Pro His Val Cys Gly Gln Ala Cys Ala Ser Cys Lys Asp Gly Phe Phe
805 810 815
Gly Leu Asp Gln Ala Asp Tyr Phe Gly Cys Arg Ser Cys Arg Cys Asp
820 825 830
Ile Gly Gly Ala Leu Gly Gln Ser Cys Glu Pro Arg Thr Gly Val Cys
835 840 845
Arg Cys Arg Pro Asn Thr Gln Gly Pro Thr Cys Ser Glu Pro Ala Arg
850 855 860
Asp His Tyr Leu Pro Asp Leu His His Leu Arg Leu Glu Leu Glu Glu
865 870 875 880
Ala Ala Thr Pro Glu Gly His Ala Val Arg Phe Gly Phe Asn Pro Leu
885 890 895
Glu Phe Glu Asn Phe Ser Trp Arg Gly Tyr Ala Gln Met Ala Pro Val
900 905 910
Gln Pro Arg Ile Val Ala Arg Leu Asn Leu Thr Ser Pro Asp Leu Phe
915 920 925
Trp Leu Val Phe Arg Tyr Val Asn Arg Gly Ala Met Ser Val Ser Gly
930 935 940
Arg Val Ser Val Arg Glu Glu Gly Arg Ser Ala Thr Cys Ala Asn Cys
945 950 955 960
Thr Ala Gln Ser Gln Pro Val Ala Phe Pro Pro Ser Thr Glu Pro Ala
965 970 975
Phe Ile Thr Val Pro Gln Arg Gly Phe Gly Glu Pro Phe Val Leu Asn
980 985 990
Pro Gly Thr Trp Ala Leu Arg Val Glu Ala Glu Gly Val Leu Leu Asp
995 1000 1005
Tyr Val Val Leu Leu Pro Ser Ala Tyr Tyr Glu Ala Ala Leu Leu
1010 1015 1020
Gln Leu Arg Val Thr Glu Ala Cys Thr Tyr Arg Pro Ser Ala Gln
1025 1030 1035
Gln Ser Gly Asp Asn Cys Leu Leu Tyr Thr His Leu Pro Leu Asp
1040 1045 1050
Gly Phe Pro Ser Ala Ala Gly Leu Glu Ala Leu Cys Arg Gln Asp
1055 1060 1065
Asn Ser Leu Pro Arg Pro Cys Pro Thr Glu Gln Leu Ser Pro Ser
1070 1075 1080
His Pro Pro Leu Ile Thr Cys Thr Gly Ser Asp Val Asp Val Gln
1085 1090 1095
Leu Gln Val Ala Val Pro Gln Pro Gly Arg Tyr Ala Leu Val Val
1100 1105 1110
Glu Tyr Ala Asn Glu Asp Ala Arg Gln Glu Val Gly Val Ala Val
1115 1120 1125
His Thr Pro Gln Arg Ala Pro Gln Gln Gly Leu Leu Ser Leu His
1130 1135 1140
Pro Cys Leu Tyr Ser Thr Leu Cys Arg Gly Thr Ala Arg Asp Thr
1145 1150 1155
Gln Asp His Leu Ala Val Phe His Leu Asp Ser Glu Ala Ser Val
1160 1165 1170
Arg Leu Thr Ala Glu Gln Ala Arg Phe Phe Leu His Gly Val Thr
1175 1180 1185
Leu Val Pro Ile Glu Glu Phe Ser Pro Glu Phe Val Glu Pro Arg
1190 1195 1200
Val Ser Cys Ile Ser Ser His Gly Ala Phe Gly Pro Asn Ser Ala
1205 1210 1215
Ala Cys Leu Pro Ser Arg Phe Pro Lys Pro Pro Gln Pro Ile Ile
1220 1225 1230
Leu Arg Asp Cys Gln Val Ile Pro Leu Pro Pro Gly Leu Pro Leu
1235 1240 1245
Thr His Ala Gln Asp Leu Thr Pro Ala Met Ser Pro Ala Gly Pro
1250 1255 1260
Arg Pro Arg Pro Pro Thr Ala Val Asp Pro Asp Ala Glu Pro Thr
1265 1270 1275
Leu Leu Arg Glu Pro Gln Ala Thr Val Val Phe Thr Thr His Val
1280 1285 1290
Pro Thr Leu Gly Arg Tyr Ala Phe Leu Leu His Gly Tyr Gln Pro
1295 1300 1305
Ala His Pro Thr Phe Pro Val Glu Val Leu Ile Asn Ala Gly Arg
1310 1315 1320
Val Trp Gln Gly His Ala Asn Ala Ser Phe Cys Pro His Gly Tyr
1325 1330 1335
Gly Cys Arg Thr Leu Val Val Cys Glu Gly Gln Ala Leu Leu Asp
1340 1345 1350
Val Thr His Ser Glu Leu Thr Val Thr Val Arg Val Pro Lys Gly
1355 1360 1365
Arg Trp Leu Trp Leu Asp Tyr Val Leu Val Val Pro Glu Asn Val
1370 1375 1380
Tyr Ser Phe Gly Tyr Leu Arg Glu Glu Pro Leu Asp Lys Ser Tyr
1385 1390 1395
Asp Phe Ile Ser His Cys Ala Ala Gln Gly Tyr His Ile Ser Pro
1400 1405 1410
Ser Ser Ser Ser Leu Phe Cys Arg Asn Ala Ala Ala Ser Leu Ser
1415 1420 1425
Leu Phe Tyr Asn Asn Gly Ala Arg Pro Cys Gly Cys His Glu Val
1430 1435 1440
Gly Ala Thr Gly Pro Thr Cys Glu Pro Phe Gly Gly Gln Cys Pro
1445 1450 1455
Cys His Ala His Val Ile Gly Arg Asp Cys Ser Arg Cys Ala Thr
1460 1465 1470
Gly Tyr Trp Gly Phe Pro Asn Cys Arg Pro Cys Asp Cys Gly Ala
1475 1480 1485
Arg Leu Cys Asp Glu Leu Thr Gly Gln Cys Ile Cys Pro Pro Arg
1490 1495 1500
Thr Ile Pro Pro Asp Cys Leu Leu Cys Gln Pro Gln Thr Phe Gly
1505 1510 1515
Cys His Pro Leu Val Gly Cys Glu Glu Cys Asn Cys Ser Gly Pro
1520 1525 1530
Gly Ile Gln Glu Leu Thr Asp Pro Thr Cys Asp Thr Asp Ser Gly
1535 1540 1545
Gln Cys Lys Cys Arg Pro Asn Val Thr Gly Arg Arg Cys Asp Thr
1550 1555 1560
Cys Ser Pro Gly Phe His Gly Tyr Pro Arg Cys Arg Pro Cys Asp
1565 1570 1575
Cys His Glu Ala Gly Thr Ala Pro Gly Val Cys Asp Pro Leu Thr
1580 1585 1590
Gly Gln Cys Tyr Cys Lys Glu Asn Val Gln Gly Pro Lys Cys Asp
1595 1600 1605
Gln Cys Ser Leu Gly Thr Phe Ser Leu Asp Ala Ala Asn Pro Lys
1610 1615 1620
Gly Cys Thr Arg Cys Phe Cys Phe Gly Ala Thr Glu Arg Cys Arg
1625 1630 1635
Ser Ser Ser Tyr Thr Arg Gln Glu Phe Val Asp Met Glu Gly Trp
1640 1645 1650
Val Leu Leu Ser Thr Asp Arg Gln Val Val Pro His Glu Arg Gln
1655 1660 1665
Pro Gly Thr Glu Met Leu Arg Ala Asp Leu Arg His Val Pro Glu
1670 1675 1680
Ala Val Pro Glu Ala Phe Pro Glu Leu Tyr Trp Gln Ala Pro Pro
1685 1690 1695
Ser Tyr Leu Gly Asp Arg Val Ser Ser Tyr Gly Gly Thr Leu Arg
1700 1705 1710
Tyr Glu Leu His Ser Glu Thr Gln Arg Gly Asp Val Phe Val Pro
1715 1720 1725
Met Glu Ser Arg Pro Asp Val Val Leu Gln Gly Asn Gln Met Ser
1730 1735 1740
Ile Thr Phe Leu Glu Pro Ala Tyr Pro Thr Pro Gly His Val His
1745 1750 1755
Arg Gly Gln Leu Gln Leu Val Glu Gly Asn Phe Arg His Thr Glu
1760 1765 1770
Thr Arg Asn Thr Val Ser Arg Glu Glu Leu Met Met Val Leu Ala
1775 1780 1785
Ser Leu Glu Gln Leu Gln Ile Arg Ala Leu Phe Ser Gln Ile Ser
1790 1795 1800
Ser Ala Val Phe Leu Arg Arg Val Ala Leu Glu Val Ala Ser Pro
1805 1810 1815
Ala Gly Gln Gly Ala Leu Ala Ser Asn Val Glu Leu Cys Leu Cys
1820 1825 1830
Pro Ala Ser Tyr Arg Gly Asp Ser Cys Gln Glu Cys Ala Pro Gly
1835 1840 1845
Phe Tyr Arg Asp Val Lys Gly Leu Phe Leu Gly Arg Cys Val Pro
1850 1855 1860
Cys Gln Cys His Gly His Ser Asp Arg Cys Leu Pro Gly Ser Gly
1865 1870 1875
Val Cys Val Asp Cys Gln His Asn Thr Glu Gly Ala His Cys Glu
1880 1885 1890
Arg Cys Gln Ala Gly Phe Val Ser Ser Arg Asp Asp Pro Ser Ala
1895 1900 1905
Pro Cys Val Ser Cys Pro Cys Pro Leu Ser Val Pro Ser Asn Asn
1910 1915 1920
Phe Ala Glu Gly Cys Val Leu Arg Gly Gly Arg Thr Gln Cys Leu
1925 1930 1935
Cys Lys Pro Gly Tyr Ala Gly Ala Ser Cys Glu Arg Cys Ala Pro
1940 1945 1950
Gly Phe Phe Gly Asn Pro Leu Val Leu Gly Ser Ser Cys Gln Pro
1955 1960 1965
Cys Asp Cys Ser Gly Asn Gly Asp Pro Asn Leu Leu Phe Ser Asp
1970 1975 1980
Cys Asp Pro Leu Thr Gly Ala Cys Arg Gly Cys Leu Arg His Thr
1985 1990 1995
Thr Gly Pro Arg Cys Glu Ile Cys Ala Pro Gly Phe Tyr Gly Asn
2000 2005 2010
Ala Leu Leu Pro Gly Asn Cys Thr Arg Cys Asp Cys Thr Pro Cys
2015 2020 2025
Gly Thr Glu Ala Cys Asp Pro His Ser Gly His Cys Leu Cys Lys
2030 2035 2040
Ala Gly Val Thr Gly Arg Arg Cys Asp Arg Cys Gln Glu Gly His
2045 2050 2055
Phe Gly Phe Asp Gly Cys Gly Gly Cys Arg Pro Cys Ala Cys Gly
2060 2065 2070
Pro Ala Ala Glu Gly Ser Glu Cys His Pro Gln Ser Gly Gln Cys
2075 2080 2085
His Cys Arg Pro Gly Thr Met Gly Pro Gln Cys Arg Glu Cys Ala
2090 2095 2100
Pro Gly Tyr Trp Gly Leu Pro Glu Gln Gly Cys Arg Arg Cys Gln
2105 2110 2115
Cys Pro Gly Gly Arg Cys Asp Pro His Thr Gly Arg Cys Asn Cys
2120 2125 2130
Pro Pro Gly Leu Ser Gly Glu Arg Cys Asp Thr Cys Ser Gln Gln
2135 2140 2145
His Gln Val Pro Val Pro Gly Gly Pro Val Gly His Ser Ile His
2150 2155 2160
Cys Glu Val Cys Asp His Cys Val Val Leu Leu Leu Asp Asp Leu
2165 2170 2175
Glu Arg Ala Gly Ala Leu Leu Pro Ala Ile His Glu Gln Leu Arg
2180 2185 2190
Gly Ile Asn Ala Ser Ser Met Ala Trp Ala Arg Leu His Arg Leu
2195 2200 2205
Asn Ala Ser Ile Ala Asp Leu Gln Ser Gln Leu Arg Ser Pro Leu
2210 2215 2220
Gly Pro Arg His Glu Thr Ala Gln Gln Leu Glu Val Leu Glu Gln
2225 2230 2235
Gln Ser Thr Ser Leu Gly Gln Asp Ala Arg Arg Leu Gly Gly Gln
2240 2245 2250
Ala Val Gly Thr Arg Asp Gln Ala Ser Gln Leu Leu Ala Gly Thr
2255 2260 2265
Glu Ala Thr Leu Gly His Ala Lys Thr Leu Leu Ala Ala Ile Arg
2270 2275 2280
Ala Val Asp Arg Thr Leu Ser Glu Leu Met Ser Gln Thr Gly His
2285 2290 2295
Leu Gly Leu Ala Asn Ala Ser Ala Pro Ser Gly Glu Gln Leu Leu
2300 2305 2310
Arg Thr Leu Ala Glu Val Glu Arg Leu Leu Trp Glu Met Arg Ala
2315 2320 2325
Arg Asp Leu Gly Ala Pro Gln Ala Ala Ala Glu Ala Glu Leu Ala
2330 2335 2340
Ala Ala Gln Arg Leu Leu Ala Arg Val Gln Glu Gln Leu Ser Ser
2345 2350 2355
Leu Trp Glu Glu Asn Gln Ala Leu Ala Thr Gln Thr Arg Asp Arg
2360 2365 2370
Leu Ala Gln His Glu Ala Gly Leu Met Asp Leu Arg Glu Ala Leu
2375 2380 2385
Asn Arg Ala Val Asp Ala Thr Arg Glu Ala Gln Glu Leu Asn Ser
2390 2395 2400
Arg Asn Gln Glu Arg Leu Glu Glu Ala Leu Gln Arg Lys Gln Glu
2405 2410 2415
Leu Ser Arg Asp Asn Ala Thr Leu Gln Ala Thr Leu His Ala Ala
2420 2425 2430
Arg Asp Thr Leu Ala Ser Val Phe Arg Leu Leu His Ser Leu Asp
2435 2440 2445
Gln Ala Lys Glu Glu Leu Glu Arg Leu Ala Ala Ser Leu Asp Gly
2450 2455 2460
Ala Arg Thr Pro Leu Leu Gln Arg Met Gln Thr Phe Ser Pro Ala
2465 2470 2475
Gly Ser Lys Leu Arg Leu Val Glu Ala Ala Glu Ala His Ala Gln
2480 2485 2490
Gln Leu Gly Gln Leu Ala Leu Asn Leu Ser Ser Ile Ile Leu Asp
2495 2500 2505
Val Asn Gln Asp Arg Leu Thr Gln Arg Ala Ile Glu Ala Ser Asn
2510 2515 2520
Ala Tyr Ser Arg Ile Leu Gln Ala Val Gln Ala Ala Glu Asp Ala
2525 2530 2535
Ala Gly Gln Ala Leu Gln Gln Ala Asp His Thr Trp Ala Thr Val
2540 2545 2550
Val Arg Gln Gly Leu Val Asp Arg Ala Gln Gln Leu Leu Ala Asn
2555 2560 2565
Ser Thr Ala Leu Glu Glu Ala Met Leu Gln Glu Gln Gln Arg Leu
2570 2575 2580
Gly Leu Val Trp Ala Ala Leu Gln Gly Ala Arg Thr Gln Leu Arg
2585 2590 2595
Asp Val Arg Ala Lys Lys Asp Gln Leu Glu Ala His Ile Gln Ala
2600 2605 2610
Ala Gln Ala Met Leu Ala Met Asp Thr Asp Glu Thr Ser Lys Lys
2615 2620 2625
Ile Ala His Ala Lys Ala Val Ala Ala Glu Ala Gln Asp Thr Ala
2630 2635 2640
Thr Arg Val Gln Ser Gln Leu Gln Ala Met Gln Glu Asn Val Glu
2645 2650 2655
Arg Trp Gln Gly Gln Tyr Glu Gly Leu Arg Gly Gln Asp Leu Gly
2660 2665 2670
Gln Ala Val Leu Asp Ala Gly His Ser Val Ser Thr Leu Glu Lys
2675 2680 2685
Thr Leu Pro Gln Leu Leu Ala Lys Leu Ser Ile Leu Glu Asn Arg
2690 2695 2700
Gly Val His Asn Ala Ser Leu Ala Leu Ser Ala Ser Ile Gly Arg
2705 2710 2715
Val Arg Glu Leu Ile Ala Gln Ala Arg Gly Ala Ala Ser Lys Val
2720 2725 2730
Lys Val Pro Met Lys Phe Asn Gly Arg Ser Gly Val Gln Leu Arg
2735 2740 2745
Thr Pro Arg Asp Leu Ala Asp Leu Ala Ala Tyr Thr Ala Leu Lys
2750 2755 2760
Phe Tyr Leu Gln Gly Pro Glu Pro Glu Pro Gly Gln Gly Thr Glu
2765 2770 2775
Asp Arg Phe Val Met Tyr Met Gly Ser Arg Gln Ala Thr Gly Asp
2780 2785 2790
Tyr Met Gly Val Ser Leu Arg Asp Lys Lys Val His Trp Val Tyr
2795 2800 2805
Gln Leu Gly Glu Ala Gly Pro Ala Val Leu Ser Ile Asp Glu Asp
2810 2815 2820
Ile Gly Glu Gln Phe Ala Ala Val Ser Leu Asp Arg Thr Leu Gln
2825 2830 2835
Phe Gly His Met Ser Val Thr Val Glu Arg Gln Met Ile Gln Glu
2840 2845 2850
Thr Lys Gly Asp Thr Val Ala Pro Gly Ala Glu Gly Leu Leu Asn
2855 2860 2865
Leu Arg Pro Asp Asp Phe Val Phe Tyr Val Gly Gly Tyr Pro Ser
2870 2875 2880
Thr Phe Thr Pro Pro Pro Leu Leu Arg Phe Pro Gly Tyr Arg Gly
2885 2890 2895
Cys Ile Glu Met Asp Thr Leu Asn Glu Glu Val Val Ser Leu Tyr
2900 2905 2910
Asn Phe Glu Arg Thr Phe Gln Leu Asp Thr Ala Val Asp Arg Pro
2915 2920 2925
Cys Ala Arg Ser Lys Ser Thr Gly Asp Pro Trp Leu Thr Asp Gly
2930 2935 2940
Ser Tyr Leu Asp Gly Thr Gly Phe Ala Arg Ile Ser Phe Asp Ser
2945 2950 2955
Gln Ile Ser Thr Thr Lys Arg Phe Glu Gln Glu Leu Arg Leu Val
2960 2965 2970
Ser Tyr Ser Gly Val Leu Phe Phe Leu Lys Gln Gln Ser Gln Phe
2975 2980 2985
Leu Cys Leu Ala Val Gln Glu Gly Ser Leu Val Leu Leu Tyr Asp
2990 2995 3000
Phe Gly Ala Gly Leu Lys Lys Ala Val Pro Leu Gln Pro Pro Pro
3005 3010 3015
Pro Leu Thr Ser Ala Ser Lys Ala Ile Gln Val Phe Leu Leu Gly
3020 3025 3030
Gly Ser Arg Lys Arg Val Leu Val Arg Val Glu Arg Ala Thr Val
3035 3040 3045
Tyr Ser Val Glu Gln Asp Asn Asp Leu Glu Leu Ala Asp Ala Tyr
3050 3055 3060
Tyr Leu Gly Gly Val Pro Pro Asp Gln Leu Pro Pro Ser Leu Arg
3065 3070 3075
Arg Leu Phe Pro Thr Gly Gly Ser Val Arg Gly Cys Val Lys Gly
3080 3085 3090
Ile Lys Ala Leu Gly Lys Tyr Val Asp Leu Lys Arg Leu Asn Thr
3095 3100 3105
Thr Gly Val Ser Ala Gly Cys Thr Ala Asp Leu Leu Val Gly Arg
3110 3115 3120
Ala Met Thr Phe His Gly His Gly Phe Leu Arg Leu Ala Leu Ser
3125 3130 3135
Asn Val Ala Pro Leu Thr Gly Asn Val Tyr Ser Gly Phe Gly Phe
3140 3145 3150
His Ser Ala Gln Asp Ser Ala Leu Leu Tyr Tyr Arg Ala Ser Pro
3155 3160 3165
Asp Gly Leu Cys Gln Val Ser Leu Gln Gln Gly Arg Val Ser Leu
3170 3175 3180
Gln Leu Leu Arg Thr Glu Val Lys Thr Gln Ala Gly Phe Ala Asp
3185 3190 3195
Gly Ala Pro His Tyr Val Ala Phe Tyr Ser Asn Ala Thr Gly Val
3200 3205 3210
Trp Leu Tyr Val Asp Asp Gln Leu Gln Gln Met Lys Pro His Arg
3215 3220 3225
Gly Pro Pro Pro Glu Leu Gln Pro Gln Pro Glu Gly Pro Pro Arg
3230 3235 3240
Leu Leu Leu Gly Gly Leu Pro Glu Ser Gly Thr Ile Tyr Asn Phe
3245 3250 3255
Ser Gly Cys Ile Ser Asn Val Phe Val Gln Arg Leu Leu Gly Pro
3260 3265 3270
Gln Arg Val Phe Asp Leu Gln Gln Asn Leu Gly Ser Val Asn Val
3275 3280 3285
Ser Thr Gly Cys Ala Pro Ala Leu Gln Ala Gln Thr Pro Gly Leu
3290 3295 3300
Gly Pro Arg Gly Leu Gln Ala Thr Ala Arg Lys Ala Ser Arg Arg
3305 3310 3315
Ser Arg Gln Pro Ala Arg His Pro Ala Cys Met Leu Pro Pro His
3320 3325 3330
Leu Arg Thr Thr Arg Asp Ser Tyr Gln Phe Gly Gly Ser Leu Ser
3335 3340 3345
Ser His Leu Glu Phe Val Gly Ile Leu Ala Arg His Arg Asn Trp
3350 3355 3360
Pro Ser Leu Ser Met His Val Leu Pro Arg Ser Ser Arg Gly Leu
3365 3370 3375
Leu Leu Phe Thr Ala Arg Leu Arg Pro Gly Ser Pro Ser Leu Ala
3380 3385 3390
Leu Phe Leu Ser Asn Gly His Phe Val Ala Gln Met Glu Gly Leu
3395 3400 3405
Gly Thr Arg Leu Arg Ala Gln Ser Arg Gln Arg Ser Arg Pro Gly
3410 3415 3420
Arg Trp His Lys Val Ser Val Arg Trp Glu Lys Asn Arg Ile Leu
3425 3430 3435
Leu Val Thr Asp Gly Ala Arg Ala Trp Ser Gln Glu Gly Pro His
3440 3445 3450
Arg Gln His Gln Gly Ala Glu His Pro Gln Pro His Thr Leu Phe
3455 3460 3465
Val Gly Gly Leu Pro Ala Ser Ser His Ser Ser Lys Leu Pro Val
3470 3475 3480
Thr Val Gly Phe Ser Gly Cys Val Lys Arg Leu Arg Leu His Gly
3485 3490 3495
Arg Pro Leu Gly Ala Pro Thr Arg Met Ala Gly Val Thr Pro Cys
3500 3505 3510
Ile Leu Gly Pro Leu Glu Ala Gly Leu Phe Phe Pro Gly Ser Gly
3515 3520 3525
Gly Val Ile Thr Leu Asp Leu Pro Gly Ala Thr Leu Pro Asp Val
3530 3535 3540
Gly Leu Glu Leu Glu Val Arg Pro Leu Ala Val Thr Gly Leu Ile
3545 3550 3555
Phe His Leu Gly Gln Ala Arg Thr Pro Pro Tyr Leu Gln Leu Gln
3560 3565 3570
Val Thr Glu Lys Gln Val Leu Leu Arg Ala Asp Asp Gly Ala Gly
3575 3580 3585
Glu Phe Ser Thr Ser Val Thr Arg Pro Ser Val Leu Cys Asp Gly
3590 3595 3600
Gln Trp His Arg Leu Ala Val Met Lys Ser Gly Asn Val Leu Arg
3605 3610 3615
Leu Glu Val Asp Ala Gln Ser Asn His Thr Val Gly Pro Leu Leu
3620 3625 3630
Ala Ala Ala Ala Gly Ala Pro Ala Pro Leu Tyr Leu Gly Gly Leu
3635 3640 3645
Pro Glu Pro Met Ala Val Gln Pro Trp Pro Pro Ala Tyr Cys Gly
3650 3655 3660
Cys Met Arg Arg Leu Ala Val Asn Arg Ser Pro Val Ala Met Thr
3665 3670 3675
Arg Ser Val Glu Val His Gly Ala Val Gly Ala Ser Gly Cys Pro
3680 3685 3690
Ala Ala
3695
<210> 2
<211> 1811
<212> PRT
<213> Homo sapiens
<400> laminin-521 beta chain
Met Glu Leu Thr Ser Arg Glu Arg Gly Arg Gly Gln Pro Leu Pro Trp
1 5 10 15
Glu Leu Arg Leu Gly Leu Leu Leu Ser Val Leu Ala Ala Thr Leu Ala
20 25 30
Gln Ala Pro Ala Pro Asp Val Pro Gly Cys Ser Arg Gly Ser Cys Tyr
35 40 45
Pro Ala Thr Gly Asp Leu Leu Val Gly Arg Ala Asp Arg Leu Thr Ala
50 55 60
Ser Ser Thr Cys Gly Leu Asn Gly Pro Gln Pro Tyr Cys Ile Val Ser
65 70 75 80
His Leu Gln Asp Glu Lys Lys Cys Phe Leu Cys Asp Ser Arg Arg Pro
85 90 95
Phe Ser Ala Arg Asp Asn Pro His Ser His Arg Ile Gln Asn Val Val
100 105 110
Thr Ser Phe Ala Pro Gln Arg Arg Ala Ala Trp Trp Gln Ser Glu Asn
115 120 125
Gly Ile Pro Ala Val Thr Ile Gln Leu Asp Leu Glu Ala Glu Phe His
130 135 140
Phe Thr His Leu Ile Met Thr Phe Lys Thr Phe Arg Pro Ala Ala Met
145 150 155 160
Leu Val Glu Arg Ser Ala Asp Phe Gly Arg Thr Trp His Val Tyr Arg
165 170 175
Tyr Phe Ser Tyr Asp Cys Gly Ala Asp Phe Pro Gly Val Pro Leu Ala
180 185 190
Pro Pro Arg His Trp Asp Asp Val Val Cys Glu Ser Arg Tyr Ser Glu
195 200 205
Ile Glu Pro Ser Thr Glu Gly Glu Val Ile Tyr Arg Val Leu Asp Pro
210 215 220
Ala Ile Pro Ile Pro Asp Pro Tyr Ser Ser Arg Ile Gln Asn Leu Leu
225 230 235 240
Lys Ile Thr Asn Leu Arg Val Asn Leu Thr Arg Leu His Thr Leu Gly
245 250 255
Asp Asn Leu Leu Asp Pro Arg Arg Glu Ile Arg Glu Lys Tyr Tyr Tyr
260 265 270
Ala Leu Tyr Glu Leu Val Val Arg Gly Asn Cys Phe Cys Tyr Gly His
275 280 285
Ala Ser Glu Cys Ala Pro Ala Pro Gly Ala Pro Ala His Ala Glu Gly
290 295 300
Met Val His Gly Ala Cys Ile Cys Lys His Asn Thr Arg Gly Leu Asn
305 310 315 320
Cys Glu Gln Cys Gln Asp Phe Tyr Arg Asp Leu Pro Trp Arg Pro Ala
325 330 335
Glu Asp Gly His Ser His Ala Cys Arg Lys Cys Glu Cys His Gly His
340 345 350
Thr His Ser Cys His Phe Asp Met Ala Val Tyr Leu Ala Ser Gly Asn
355 360 365
Val Ser Gly Gly Val Cys Asp Gly Cys Gln His Asn Thr Ala Gly Arg
370 375 380
His Cys Glu Leu Cys Arg Pro Phe Phe Tyr Arg Asp Pro Thr Lys Asp
385 390 395 400
Leu Arg Asp Pro Ala Val Cys Arg Ser Cys Asp Cys Asp Pro Met Gly
405 410 415
Ser Gln Asp Gly Gly Arg Cys Asp Ser His Asp Asp Pro Ala Leu Gly
420 425 430
Leu Val Ser Gly Gln Cys Arg Cys Lys Glu His Val Val Gly Thr Arg
435 440 445
Cys Gln Gln Cys Arg Asp Gly Phe Phe Gly Leu Ser Ile Ser Asp Arg
450 455 460
Leu Gly Cys Arg Arg Cys Gln Cys Asn Ala Arg Gly Thr Val Pro Gly
465 470 475 480
Ser Thr Pro Cys Asp Pro Asn Ser Gly Ser Cys Tyr Cys Lys Arg Leu
485 490 495
Val Thr Gly Arg Gly Cys Asp Arg Cys Leu Pro Gly His Trp Gly Leu
500 505 510
Ser His Asp Leu Leu Gly Cys Arg Pro Cys Asp Cys Asp Val Gly Gly
515 520 525
Ala Leu Asp Pro Gln Cys Asp Glu Gly Thr Gly Gln Cys His Cys Arg
530 535 540
Gln His Met Val Gly Arg Arg Cys Glu Gln Val Gln Pro Gly Tyr Phe
545 550 555 560
Arg Pro Phe Leu Asp His Leu Ile Trp Glu Ala Glu Asp Thr Arg Gly
565 570 575
Gln Val Leu Asp Val Val Glu Arg Leu Val Thr Pro Gly Glu Thr Pro
580 585 590
Ser Trp Thr Gly Ser Gly Phe Val Arg Leu Gln Glu Gly Gln Thr Leu
595 600 605
Glu Phe Leu Val Ala Ser Val Pro Lys Ala Met Asp Tyr Asp Leu Leu
610 615 620
Leu Arg Leu Glu Pro Gln Val Pro Glu Gln Trp Ala Glu Leu Glu Leu
625 630 635 640
Ile Val Gln Arg Pro Gly Pro Val Pro Ala His Ser Leu Cys Gly His
645 650 655
Leu Val Pro Lys Asp Asp Arg Ile Gln Gly Thr Leu Gln Pro His Ala
660 665 670
Arg Tyr Leu Ile Phe Pro Asn Pro Val Cys Leu Glu Pro Gly Ile Ser
675 680 685
Tyr Lys Leu His Leu Lys Leu Val Arg Thr Gly Gly Ser Ala Gln Pro
690 695 700
Glu Thr Pro Tyr Ser Gly Pro Gly Leu Leu Ile Asp Ser Leu Val Leu
705 710 715 720
Leu Pro Arg Val Leu Val Leu Glu Met Phe Ser Gly Gly Asp Ala Ala
725 730 735
Ala Leu Glu Arg Gln Ala Thr Phe Glu Arg Tyr Gln Cys His Glu Glu
740 745 750
Gly Leu Val Pro Ser Lys Thr Ser Pro Ser Glu Ala Cys Ala Pro Leu
755 760 765
Leu Ile Ser Leu Ser Thr Leu Ile Tyr Asn Gly Ala Leu Pro Cys Gln
770 775 780
Cys Asn Pro Gln Gly Ser Leu Ser Ser Glu Cys Asn Pro His Gly Gly
785 790 795 800
Gln Cys Leu Cys Lys Pro Gly Val Val Gly Arg Arg Cys Asp Leu Cys
805 810 815
Ala Pro Gly Tyr Tyr Gly Phe Gly Pro Thr Gly Cys Gln Ala Cys Gln
820 825 830
Cys Ser His Glu Gly Ala Leu Ser Ser Leu Cys Glu Lys Thr Ser Gly
835 840 845
Gln Cys Leu Cys Arg Thr Gly Ala Phe Gly Leu Arg Cys Asp Arg Cys
850 855 860
Gln Arg Gly Gln Trp Gly Phe Pro Ser Cys Arg Pro Cys Val Cys Asn
865 870 875 880
Gly His Ala Asp Glu Cys Asn Thr His Thr Gly Ala Cys Leu Gly Cys
885 890 895
Arg Asp His Thr Gly Gly Glu His Cys Glu Arg Cys Ile Ala Gly Phe
900 905 910
His Gly Asp Pro Arg Leu Pro Tyr Gly Gly Gln Cys Arg Pro Cys Pro
915 920 925
Cys Pro Glu Gly Pro Gly Ser Gln Arg His Phe Ala Thr Ser Cys His
930 935 940
Gln Asp Glu Tyr Ser Gln Gln Ile Val Cys His Cys Arg Ala Gly Tyr
945 950 955 960
Thr Gly Leu Arg Cys Glu Ala Cys Ala Pro Gly His Phe Gly Asp Pro
965 970 975
Ser Arg Pro Gly Gly Arg Cys Gln Leu Cys Glu Cys Ser Gly Asn Ile
980 985 990
Asp Pro Met Asp Pro Asp Ala Cys Asp Pro His Thr Gly Gln Cys Leu
995 1000 1005
Arg Cys Leu His His Thr Glu Gly Pro His Cys Ala His Cys Lys
1010 1015 1020
Pro Gly Phe His Gly Gln Ala Ala Arg Gln Ser Cys His Arg Cys
1025 1030 1035
Thr Cys Asn Leu Leu Gly Thr Asn Pro Gln Gln Cys Pro Ser Pro
1040 1045 1050
Asp Gln Cys His Cys Asp Pro Ser Ser Gly Gln Cys Pro Cys Leu
1055 1060 1065
Pro Asn Val Gln Gly Pro Ser Cys Asp Arg Cys Ala Pro Asn Phe
1070 1075 1080
Trp Asn Leu Thr Ser Gly His Gly Cys Gln Pro Cys Ala Cys His
1085 1090 1095
Pro Ser Arg Ala Arg Gly Pro Thr Cys Asn Glu Phe Thr Gly Gln
1100 1105 1110
Cys His Cys Arg Ala Gly Phe Gly Gly Arg Thr Cys Ser Glu Cys
1115 1120 1125
Gln Glu Leu His Trp Gly Asp Pro Gly Leu Gln Cys His Ala Cys
1130 1135 1140
Asp Cys Asp Ser Arg Gly Ile Asp Thr Pro Gln Cys His Arg Phe
1145 1150 1155
Thr Gly His Cys Ser Cys Arg Pro Gly Val Ser Gly Val Arg Cys
1160 1165 1170
Asp Gln Cys Ala Arg Gly Phe Ser Gly Ile Phe Pro Ala Cys His
1175 1180 1185
Pro Cys His Ala Cys Phe Gly Asp Trp Asp Arg Val Val Gln Asp
1190 1195 1200
Leu Ala Ala Arg Thr Gln Arg Leu Glu Gln Arg Ala Gln Glu Leu
1205 1210 1215
Gln Gln Thr Gly Val Leu Gly Ala Phe Glu Ser Ser Phe Trp His
1220 1225 1230
Met Gln Glu Lys Leu Gly Ile Val Gln Gly Ile Val Gly Ala Arg
1235 1240 1245
Asn Thr Ser Ala Ala Ser Thr Ala Gln Leu Val Glu Ala Thr Glu
1250 1255 1260
Glu Leu Arg Arg Glu Ile Gly Glu Ala Thr Glu His Leu Thr Gln
1265 1270 1275
Leu Glu Ala Asp Leu Thr Asp Val Gln Asp Glu Asn Phe Asn Ala
1280 1285 1290
Asn His Ala Leu Ser Gly Leu Glu Arg Asp Arg Leu Ala Leu Asn
1295 1300 1305
Leu Thr Leu Arg Gln Leu Asp Gln His Leu Asp Leu Leu Lys His
1310 1315 1320
Ser Asn Phe Leu Gly Ala Tyr Asp Ser Ile Arg His Ala His Ser
1325 1330 1335
Gln Ser Ala Glu Ala Glu Arg Arg Ala Asn Thr Ser Ala Leu Ala
1340 1345 1350
Val Pro Ser Pro Val Ser Asn Ser Ala Ser Ala Arg His Arg Thr
1355 1360 1365
Glu Ala Leu Met Asp Ala Gln Lys Glu Asp Phe Asn Ser Lys His
1370 1375 1380
Met Ala Asn Gln Arg Ala Leu Gly Lys Leu Ser Ala His Thr His
1385 1390 1395
Thr Leu Ser Leu Thr Asp Ile Asn Glu Leu Val Cys Gly Ala Pro
1400 1405 1410
Gly Asp Ala Pro Cys Ala Thr Ser Pro Cys Gly Gly Ala Gly Cys
1415 1420 1425
Arg Asp Glu Asp Gly Gln Pro Arg Cys Gly Gly Leu Ser Cys Asn
1430 1435 1440
Gly Ala Ala Ala Thr Ala Asp Leu Ala Leu Gly Arg Ala Arg His
1445 1450 1455
Thr Gln Ala Glu Leu Gln Arg Ala Leu Ala Glu Gly Gly Ser Ile
1460 1465 1470
Leu Ser Arg Val Ala Glu Thr Arg Arg Gln Ala Ser Glu Ala Gln
1475 1480 1485
Gln Arg Ala Gln Ala Ala Leu Asp Lys Ala Asn Ala Ser Arg Gly
1490 1495 1500
Gln Val Glu Gln Ala Asn Gln Glu Leu Gln Glu Leu Ile Gln Ser
1505 1510 1515
Val Lys Asp Phe Leu Asn Gln Glu Gly Ala Asp Pro Asp Ser Ile
1520 1525 1530
Glu Met Val Ala Thr Arg Val Leu Glu Leu Ser Ile Pro Ala Ser
1535 1540 1545
Ala Glu Gln Ile Gln His Leu Ala Gly Ala Ile Ala Glu Arg Val
1550 1555 1560
Arg Ser Leu Ala Asp Val Asp Ala Ile Leu Ala Arg Thr Val Gly
1565 1570 1575
Asp Val Arg Arg Ala Glu Gln Leu Leu Gln Asp Ala Arg Arg Ala
1580 1585 1590
Arg Ser Trp Ala Glu Asp Glu Lys Gln Lys Ala Glu Thr Val Gln
1595 1600 1605
Ala Ala Leu Glu Glu Ala Gln Arg Ala Gln Gly Ile Ala Gln Gly
1610 1615 1620
Ala Ile Arg Gly Ala Val Ala Asp Thr Arg Asp Thr Glu Gln Thr
1625 1630 1635
Leu Tyr Gln Val Gln Glu Arg Met Ala Gly Ala Glu Arg Ala Leu
1640 1645 1650
Ser Ser Ala Gly Glu Arg Ala Arg Gln Leu Asp Ala Leu Leu Glu
1655 1660 1665
Ala Leu Lys Leu Lys Arg Ala Gly Asn Ser Leu Ala Ala Ser Thr
1670 1675 1680
Ala Glu Glu Thr Ala Gly Ser Ala Gln Gly Arg Ala Gln Glu Ala
1685 1690 1695
Glu Gln Leu Leu Arg Gly Pro Leu Gly Asp Gln Tyr Gln Thr Val
1700 1705 1710
Lys Ala Leu Ala Glu Arg Lys Ala Gln Gly Val Leu Ala Ala Gln
1715 1720 1725
Ala Arg Ala Glu Gln Leu Arg Asp Glu Ala Arg Asp Leu Leu Gln
1730 1735 1740
Ala Ala Gln Asp Lys Leu Gln Arg Leu Gln Glu Leu Glu Gly Thr
1745 1750 1755
Tyr Glu Glu Asn Glu Arg Ala Leu Glu Ser Lys Ala Ala Gln Leu
1760 1765 1770
Asp Gly Leu Glu Ala Arg Met Arg Ser Val Leu Gln Ala Ile Asn
1775 1780 1785
Leu Gln Val Gln Ile Tyr Asn Thr Cys Gln Lys Ser Ser Trp Pro
1790 1795 1800
Gly Arg Ala Pro Asn Lys Pro Val
1805 1810
<210> 3
<211> 1609
<212> PRT
<213> Homo sapiens
<400> laminin-521 gamma chain
Met Arg Gly Ser His Arg Ala Ala Pro Ala Leu Arg Pro Arg Gly Arg
1 5 10 15
Leu Trp Pro Val Leu Ala Val Leu Ala Ala Ala Ala Ala Ala Gly Cys
20 25 30
Ala Gln Ala Ala Met Asp Glu Cys Thr Asp Glu Gly Gly Arg Pro Gln
35 40 45
Arg Cys Met Pro Glu Phe Val Asn Ala Ala Phe Asn Val Thr Val Val
50 55 60
Ala Thr Asn Thr Cys Gly Thr Pro Pro Glu Glu Tyr Cys Val Gln Thr
65 70 75 80
Gly Val Thr Gly Val Thr Lys Ser Cys His Leu Cys Asp Ala Gly Gln
85 90 95
Pro His Leu Gln His Gly Ala Ala Phe Leu Thr Asp Tyr Asn Asn Gln
100 105 110
Ala Asp Thr Thr Trp Trp Gln Ser Gln Thr Met Leu Ala Gly Val Gln
115 120 125
Tyr Pro Ser Ser Ile Asn Leu Thr Leu His Leu Gly Lys Ala Phe Asp
130 135 140
Ile Thr Tyr Val Arg Leu Lys Phe His Thr Ser Arg Pro Glu Ser Phe
145 150 155 160
Ala Ile Tyr Lys Arg Thr Arg Glu Asp Gly Pro Trp Ile Pro Tyr Gln
165 170 175
Tyr Tyr Ser Gly Ser Cys Glu Asn Thr Tyr Ser Lys Ala Asn Arg Gly
180 185 190
Phe Ile Arg Thr Gly Gly Asp Glu Gln Gln Ala Leu Cys Thr Asp Glu
195 200 205
Phe Ser Asp Ile Ser Pro Leu Thr Gly Gly Asn Val Ala Phe Ser Thr
210 215 220
Leu Glu Gly Arg Pro Ser Ala Tyr Asn Phe Asp Asn Ser Pro Val Leu
225 230 235 240
Gln Glu Trp Val Thr Ala Thr Asp Ile Arg Val Thr Leu Asn Arg Leu
245 250 255
Asn Thr Phe Gly Asp Glu Val Phe Asn Asp Pro Lys Val Leu Lys Ser
260 265 270
Tyr Tyr Tyr Ala Ile Ser Asp Phe Ala Val Gly Gly Arg Cys Lys Cys
275 280 285
Asn Gly His Ala Ser Glu Cys Met Lys Asn Glu Phe Asp Lys Leu Val
290 295 300
Cys Asn Cys Lys His Asn Thr Tyr Gly Val Asp Cys Glu Lys Cys Leu
305 310 315 320
Pro Phe Phe Asn Asp Arg Pro Trp Arg Arg Ala Thr Ala Glu Ser Ala
325 330 335
Ser Glu Cys Leu Pro Cys Asp Cys Asn Gly Arg Ser Gln Glu Cys Tyr
340 345 350
Phe Asp Pro Glu Leu Tyr Arg Ser Thr Gly His Gly Gly His Cys Thr
355 360 365
Asn Cys Gln Asp Asn Thr Asp Gly Ala His Cys Glu Arg Cys Arg Glu
370 375 380
Asn Phe Phe Arg Leu Gly Asn Asn Glu Ala Cys Ser Ser Cys His Cys
385 390 395 400
Ser Pro Val Gly Ser Leu Ser Thr Gln Cys Asp Ser Tyr Gly Arg Cys
405 410 415
Ser Cys Lys Pro Gly Val Met Gly Asp Lys Cys Asp Arg Cys Gln Pro
420 425 430
Gly Phe His Ser Leu Thr Glu Ala Gly Cys Arg Pro Cys Ser Cys Asp
435 440 445
Pro Ser Gly Ser Ile Asp Glu Cys Asn Ile Glu Thr Gly Arg Cys Val
450 455 460
Cys Lys Asp Asn Val Glu Gly Phe Asn Cys Glu Arg Cys Lys Pro Gly
465 470 475 480
Phe Phe Asn Leu Glu Ser Ser Asn Pro Arg Gly Cys Thr Pro Cys Phe
485 490 495
Cys Phe Gly His Ser Ser Val Cys Thr Asn Ala Val Gly Tyr Ser Val
500 505 510
Tyr Ser Ile Ser Ser Thr Phe Gln Ile Asp Glu Asp Gly Trp Arg Ala
515 520 525
Glu Gln Arg Asp Gly Ser Glu Ala Ser Leu Glu Trp Ser Ser Glu Arg
530 535 540
Gln Asp Ile Ala Val Ile Ser Asp Ser Tyr Phe Pro Arg Tyr Phe Ile
545 550 555 560
Ala Pro Ala Lys Phe Leu Gly Lys Gln Val Leu Ser Tyr Gly Gln Asn
565 570 575
Leu Ser Phe Ser Phe Arg Val Asp Arg Arg Asp Thr Arg Leu Ser Ala
580 585 590
Glu Asp Leu Val Leu Glu Gly Ala Gly Leu Arg Val Ser Val Pro Leu
595 600 605
Ile Ala Gln Gly Asn Ser Tyr Pro Ser Glu Thr Thr Val Lys Tyr Val
610 615 620
Phe Arg Leu His Glu Ala Thr Asp Tyr Pro Trp Arg Pro Ala Leu Thr
625 630 635 640
Pro Phe Glu Phe Gln Lys Leu Leu Asn Asn Leu Thr Ser Ile Lys Ile
645 650 655
Arg Gly Thr Tyr Ser Glu Arg Ser Ala Gly Tyr Leu Asp Asp Val Thr
660 665 670
Leu Ala Ser Ala Arg Pro Gly Pro Gly Val Pro Ala Thr Trp Val Glu
675 680 685
Ser Cys Thr Cys Pro Val Gly Tyr Gly Gly Gln Phe Cys Glu Met Cys
690 695 700
Leu Ser Gly Tyr Arg Arg Glu Thr Pro Asn Leu Gly Pro Tyr Ser Pro
705 710 715 720
Cys Val Leu Cys Ala Cys Asn Gly His Ser Glu Thr Cys Asp Pro Glu
725 730 735
Thr Gly Val Cys Asn Cys Arg Asp Asn Thr Ala Gly Pro His Cys Glu
740 745 750
Lys Cys Ser Asp Gly Tyr Tyr Gly Asp Ser Thr Ala Gly Thr Ser Ser
755 760 765
Asp Cys Gln Pro Cys Pro Cys Pro Gly Gly Ser Ser Cys Ala Val Val
770 775 780
Pro Lys Thr Lys Glu Val Val Cys Thr Asn Cys Pro Thr Gly Thr Thr
785 790 795 800
Gly Lys Arg Cys Glu Leu Cys Asp Asp Gly Tyr Phe Gly Asp Pro Leu
805 810 815
Gly Arg Asn Gly Pro Val Arg Leu Cys Arg Leu Cys Gln Cys Ser Asp
820 825 830
Asn Ile Asp Pro Asn Ala Val Gly Asn Cys Asn Arg Leu Thr Gly Glu
835 840 845
Cys Leu Lys Cys Ile Tyr Asn Thr Ala Gly Phe Tyr Cys Asp Arg Cys
850 855 860
Lys Asp Gly Phe Phe Gly Asn Pro Leu Ala Pro Asn Pro Ala Asp Lys
865 870 875 880
Cys Lys Ala Cys Asn Cys Asn Leu Tyr Gly Thr Met Lys Gln Gln Ser
885 890 895
Ser Cys Asn Pro Val Thr Gly Gln Cys Glu Cys Leu Pro His Val Thr
900 905 910
Gly Gln Asp Cys Gly Ala Cys Asp Pro Gly Phe Tyr Asn Leu Gln Ser
915 920 925
Gly Gln Gly Cys Glu Arg Cys Asp Cys His Ala Leu Gly Ser Thr Asn
930 935 940
Gly Gln Cys Asp Ile Arg Thr Gly Gln Cys Glu Cys Gln Pro Gly Ile
945 950 955 960
Thr Gly Gln His Cys Glu Arg Cys Glu Val Asn His Phe Gly Phe Gly
965 970 975
Pro Glu Gly Cys Lys Pro Cys Asp Cys His Pro Glu Gly Ser Leu Ser
980 985 990
Leu Gln Cys Lys Asp Asp Gly Arg Cys Glu Cys Arg Glu Gly Phe Val
995 1000 1005
Gly Asn Arg Cys Asp Gln Cys Glu Glu Asn Tyr Phe Tyr Asn Arg
1010 1015 1020
Ser Trp Pro Gly Cys Gln Glu Cys Pro Ala Cys Tyr Arg Leu Val
1025 1030 1035
Lys Asp Lys Val Ala Asp His Arg Val Lys Leu Gln Glu Leu Glu
1040 1045 1050
Ser Leu Ile Ala Asn Leu Gly Thr Gly Asp Glu Met Val Thr Asp
1055 1060 1065
Gln Ala Phe Glu Asp Arg Leu Lys Glu Ala Glu Arg Glu Val Met
1070 1075 1080
Asp Leu Leu Arg Glu Ala Gln Asp Val Lys Asp Val Asp Gln Asn
1085 1090 1095
Leu Met Asp Arg Leu Gln Arg Val Asn Asn Thr Leu Ser Ser Gln
1100 1105 1110
Ile Ser Arg Leu Gln Asn Ile Arg Asn Thr Ile Glu Glu Thr Gly
1115 1120 1125
Asn Leu Ala Glu Gln Ala Arg Ala His Val Glu Asn Thr Glu Arg
1130 1135 1140
Leu Ile Glu Ile Ala Ser Arg Glu Leu Glu Lys Ala Lys Val Ala
1145 1150 1155
Ala Ala Asn Val Ser Val Thr Gln Pro Glu Ser Thr Gly Asp Pro
1160 1165 1170
Asn Asn Met Thr Leu Leu Ala Glu Glu Ala Arg Lys Leu Ala Glu
1175 1180 1185
Arg His Lys Gln Glu Ala Asp Asp Ile Val Arg Val Ala Lys Thr
1190 1195 1200
Ala Asn Asp Thr Ser Thr Glu Ala Tyr Asn Leu Leu Leu Arg Thr
1205 1210 1215
Leu Ala Gly Glu Asn Gln Thr Ala Phe Glu Ile Glu Glu Leu Asn
1220 1225 1230
Arg Lys Tyr Glu Gln Ala Lys Asn Ile Ser Gln Asp Leu Glu Lys
1235 1240 1245
Gln Ala Ala Arg Val His Glu Glu Ala Lys Arg Ala Gly Asp Lys
1250 1255 1260
Ala Val Glu Ile Tyr Ala Ser Val Ala Gln Leu Ser Pro Leu Asp
1265 1270 1275
Ser Glu Thr Leu Glu Asn Glu Ala Asn Asn Ile Lys Met Glu Ala
1280 1285 1290
Glu Asn Leu Glu Gln Leu Ile Asp Gln Lys Leu Lys Asp Tyr Glu
1295 1300 1305
Asp Leu Arg Glu Asp Met Arg Gly Lys Glu Leu Glu Val Lys Asn
1310 1315 1320
Leu Leu Glu Lys Gly Lys Thr Glu Gln Gln Thr Ala Asp Gln Leu
1325 1330 1335
Leu Ala Arg Ala Asp Ala Ala Lys Ala Leu Ala Glu Glu Ala Ala
1340 1345 1350
Lys Lys Gly Arg Asp Thr Leu Gln Glu Ala Asn Asp Ile Leu Asn
1355 1360 1365
Asn Leu Lys Asp Phe Asp Arg Arg Val Asn Asp Asn Lys Thr Ala
1370 1375 1380
Ala Glu Glu Ala Leu Arg Lys Ile Pro Ala Ile Asn Gln Thr Ile
1385 1390 1395
Thr Glu Ala Asn Glu Lys Thr Arg Glu Ala Gln Gln Ala Leu Gly
1400 1405 1410
Ser Ala Ala Ala Asp Ala Thr Glu Ala Lys Asn Lys Ala His Glu
1415 1420 1425
Ala Glu Arg Ile Ala Ser Ala Val Gln Lys Asn Ala Thr Ser Thr
1430 1435 1440
Lys Ala Glu Ala Glu Arg Thr Phe Ala Glu Val Thr Asp Leu Asp
1445 1450 1455
Asn Glu Val Asn Asn Met Leu Lys Gln Leu Gln Glu Ala Glu Lys
1460 1465 1470
Glu Leu Lys Arg Lys Gln Asp Asp Ala Asp Gln Asp Met Met Met
1475 1480 1485
Ala Gly Met Ala Ser Gln Ala Ala Gln Glu Ala Glu Ile Asn Ala
1490 1495 1500
Arg Lys Ala Lys Asn Ser Val Thr Ser Leu Leu Ser Ile Ile Asn
1505 1510 1515
Asp Leu Leu Glu Gln Leu Gly Gln Leu Asp Thr Val Asp Leu Asn
1520 1525 1530
Lys Leu Asn Glu Ile Glu Gly Thr Leu Asn Lys Ala Lys Asp Glu
1535 1540 1545
Met Lys Val Ser Asp Leu Asp Arg Lys Val Ser Asp Leu Glu Asn
1550 1555 1560
Glu Ala Lys Lys Gln Glu Ala Ala Ile Met Asp Tyr Asn Arg Asp
1565 1570 1575
Ile Glu Glu Ile Met Lys Asp Ile Arg Asn Leu Glu Asp Ile Arg
1580 1585 1590
Lys Thr Leu Pro Ser Gly Cys Phe Asn Thr Pro Ser Ile Glu Lys
1595 1600 1605
Pro
<210> 4
<211> 555
<212> PRT
<213> Homo sapiens
<400> E-cadherin
Met Asp Trp Val Ile Pro Pro Ile Ser Cys Pro Glu Asn Glu Lys Gly
1 5 10 15
Pro Phe Pro Lys Asn Leu Val Gln Ile Lys Ser Asn Lys Asp Lys Glu
20 25 30
Gly Lys Val Phe Tyr Ser Ile Thr Gly Gln Gly Ala Asp Thr Pro Pro
35 40 45
Val Gly Val Phe Ile Ile Glu Arg Glu Thr Gly Trp Leu Lys Val Thr
50 55 60
Glu Pro Leu Asp Arg Glu Arg Ile Ala Thr Tyr Thr Leu Phe Ser His
65 70 75 80
Ala Val Ser Ser Asn Gly Asn Ala Val Glu Asp Pro Met Glu Ile Leu
85 90 95
Ile Thr Val Thr Asp Gln Asn Asp Asn Lys Pro Glu Phe Thr Gln Glu
100 105 110
Val Phe Lys Gly Ser Val Met Glu Gly Ala Leu Pro Gly Thr Ser Val
115 120 125
Met Glu Val Thr Ala Thr Asp Ala Asp Asp Asp Val Asn Thr Tyr Asn
130 135 140
Ala Ala Ile Ala Tyr Thr Ile Leu Ser Gln Asp Pro Glu Leu Pro Asp
145 150 155 160
Lys Asn Met Phe Thr Ile Asn Arg Asn Thr Gly Val Ile Ser Val Val
165 170 175
Thr Thr Gly Leu Asp Arg Glu Ser Phe Pro Thr Tyr Thr Leu Val Val
180 185 190
Gln Ala Ala Asp Leu Gln Gly Glu Gly Leu Ser Thr Thr Ala Thr Ala
195 200 205
Val Ile Thr Val Thr Asp Thr Asn Asp Asn Pro Pro Ile Phe Asn Pro
210 215 220
Thr Thr Tyr Lys Gly Gln Val Pro Glu Asn Glu Ala Asn Val Val Ile
225 230 235 240
Thr Thr Leu Lys Val Thr Asp Ala Asp Ala Pro Asn Thr Pro Ala Trp
245 250 255
Glu Ala Val Tyr Thr Ile Leu Asn Asp Asp Gly Gly Gln Phe Val Val
260 265 270
Thr Thr Asn Pro Val Asn Asn Asp Gly Ile Leu Lys Thr Ala Lys Gly
275 280 285
Leu Asp Phe Glu Ala Lys Gln Gln Tyr Ile Leu His Val Ala Val Thr
290 295 300
Asn Val Val Pro Phe Glu Val Ser Leu Thr Thr Ser Thr Ala Thr Val
305 310 315 320
Thr Val Asp Val Leu Asp Val Asn Glu Ala Pro Ile Phe Val Pro Pro
325 330 335
Glu Lys Arg Val Glu Val Ser Glu Asp Phe Gly Val Gly Gln Glu Ile
340 345 350
Thr Ser Tyr Thr Ala Gln Glu Pro Asp Thr Phe Met Glu Gln Lys Ile
355 360 365
Thr Tyr Arg Ile Trp Arg Asp Thr Ala Asn Trp Leu Glu Ile Asn Pro
370 375 380
Asp Thr Gly Ala Ile Ser Thr Arg Ala Glu Leu Asp Arg Glu Asp Phe
385 390 395 400
Glu His Val Lys Asn Ser Thr Tyr Thr Ala Leu Ile Ile Ala Thr Asp
405 410 415
Asn Gly Ser Pro Val Ala Thr Gly Thr Gly Thr Leu Leu Leu Ile Leu
420 425 430
Ser Asp Val Asn Asp Asn Ala Pro Ile Pro Glu Pro Arg Thr Ile Phe
435 440 445
Phe Cys Glu Arg Asn Pro Lys Pro Gln Val Ile Asn Ile Ile Asp Ala
450 455 460
Asp Leu Pro Pro Asn Thr Ser Pro Phe Thr Ala Glu Leu Thr His Gly
465 470 475 480
Ala Ser Ala Asn Trp Thr Ile Gln Tyr Asn Asp Pro Thr Gln Glu Ser
485 490 495
Ile Ile Leu Lys Pro Lys Met Ala Leu Glu Val Gly Asp Tyr Lys Ile
500 505 510
Asn Leu Lys Leu Met Asp Asn Gln Asn Lys Asp Gln Val Thr Thr Leu
515 520 525
Glu Val Ser Val Cys Asp Cys Glu Gly Ala Ala Gly Val Cys Arg Lys
530 535 540
Ala Gln Pro Val Glu Ala Gly Leu Gln Ile Pro
545 550 555
<210> 5
<211> 4024
<212> DNA
<213> Homo sapiens
<400> LIN28 (LIN28A) coding
caccgctatt gtgcggggga agatgtagca gcttcttctc cgaaccaacc ctttgccttc 60
ggacttctcc ggggccagca gccgcccgac caggggcccg gggccacggg ctcagccgac 120
gaccatgggc tccgtgtcca accagcagtt tgcaggtggc tgcgccaagg cggcagaaga 180
ggcgcccgag gaggcgccgg aggacgcggc ccgggcggcg gacgagcctc agctgctgca 240
cggtgcgggc atctgtaagt ggttcaacgt gcgcatgggg ttcggcttcc tgtccatgac 300
cgcccgcgcc ggggtcgcgc tcgacccccc agtggatgtc tttgtgcacc agagtaagct 360
gcacatggaa gggttccgga gcttgaagga gggtgaggca gtggagttca cctttaagaa 420
gtcagccaag ggtctggaat ccatccgtgt caccggacct ggtggagtat tctgtattgg 480
gagtgagagg cggccaaaag gaaagagcat gcagaagcgc agatcaaaag gagacaggtg 540
ctacaactgt ggaggtctag atcatcatgc caaggaatgc aagctgccac cccagcccaa 600
gaagtgccac ttctgccaga gcatcagcca tatggtagcc tcatgtccgc tgaaggccca 660
gcagggccct agtgcacagg gaaagccaac ctactttcga gaggaagaag aagaaatcca 720
cagccctacc ctgctcccgg aggcacagaa ttgagccaca atgggtgggg gctattcttt 780
tgctatcagg aagttttgag gagcaggcag agtggagaaa gtgggaatag ggtgcattgg 840
ggctagttgg cactgccatg tatctcaggc ttgggttcac accatcaccc tttcttccct 900
ctaggtgggg ggaaagggtg agtcaaagga actccaacca tgctctgtcc aaatgcaagt 960
gagggttctg ggggcaacca ggagggggga atcaccctac aacctgcata ctttgagtct 1020
ccatccccag aatttccagc ttttgaaagt ggcctggata gggaagttgt tttcctttta 1080
aagaaggata tataataatt cccatgccag agtgaaatga ttaagtataa gaccagattc 1140
atggagccaa gccactacat tctgtggaag gagatctctc aggagtaagc attgtttttt 1200
tttcacatct tgtatcctca tacccacttt tgggataggg tgctggcagc tgtcccaagc 1260
aatgggtaat gatgatggca aaaagggtgt ttgggggaac agctgcagac ctgctgctct 1320
atgctcaccc ccgccccatt ctgggccaat gtgattttat ttatttgctc ccttggatac 1380
tgcaccttgg gtcccacttt ctccaggatg ccaactgcac tagctgtgtg cgaatgacgt 1440
atcttgtgca ttttaacttt ttttccttaa tataaatatt ctggttttgt atttttgtat 1500
attttaatct aaggccctca tttcctgcac tgtgttctca ggtacatgag caatctcagg 1560
gatagccagc agcagctcca ggtctgcgca gcaggaatta ctttttgttg tttttgccac 1620
cgtggagagc aactatttgg agtgcacagc ctattgaact acctcatttt tgccaataag 1680
agctggcttt tctgccatag tgtcctcttg aaaccccctc tgccttgaaa atgttttatg 1740
ggagactagg ttttaactgg gtggccccat gacttgattg ccttctactg gaagattggg 1800
aattagtcta aacaggaaat ggtggtacac agaggctagg agaggctggg cccggtgaaa 1860
aggccagaga gcaagccaag attaggtgag ggttgtctaa tcctatggca caggacgtgc 1920
tttacatctc cagatctgtt cttcaccaga ttaggttagg cctaccatgt gccacagggt 1980
gtgtgtgtgt ttgtaaaact agagttgcta aggataagtt taaagaccaa tacccctgta 2040
cttaatcctg tgctgtcgag ggatggatat atgaagtaag gtgagatcct taacctttca 2100
aaattttcgg gttccaggga gacacacaag cgagggtttt gtggtgcctg gagcctgtgt 2160
cctgccctgc tacagtagtg attaatagtg tcatggtagc taaaggagaa aaagggggtt 2220
tcgtttacac gctgtgagat caccgcaaac ctaccttact gtgttgaaac gggacaaatg 2280
caatagaacg cattgggtgg tgtgtgtctg atcctgggtt cttgtctccc ctaaatgctg 2340
ccccccaagt tactgtattt gtctgggctt tgtaggactt cactacgttg attgctaggt 2400
ggcctagttt gtgtaaatat aatgtattgg tctttctccg tgttctttgg gggttttgtt 2460
tacaaacttc tttttgtatt gagagaaaaa tagccaaagc atctttgaca gaaggttctg 2520
caccaggcaa aaagatctga aacattagtt tggggggccc tcttcttaaa gtggggatct 2580
tgaaccatcc tttcttttgt attccccttc ccctattacc tattagacca gatcttctgt 2640
cctaaaaact tgtcttctac cctgccctct tttctgttca cccccaaaag aaaacttaca 2700
cacccacaca catacacatt tcatgcttgg agtgtctcca caactcttaa atgatgtatg 2760
caaaaatact gaagctagga aaaccctcca tcccttgttc ccaacctcct aagtcaagac 2820
cattaccatt tctttctttc tttttttttt ttttttaaaa tggagtctca ctgtgtcacc 2880
caggctggag tgcagtggca tgatcggctc actgcagcct ctgcctcttg ggttcaagtg 2940
attctcctgc ctcagcctcc tgagtagctg ggatttcagg cacccgccac actcagctaa 3000
tttttgtatt tttagtagag acggggtttc accatgttgt ccaggctggt ctggaactcc 3060
tgacctcagg tgatctgccc accttggctt cccaaagtgc tgggattaca ggcatgagcc 3120
accatgctgg gccaaccatt tcttggtgta ttcatgccaa acacttaaga cactgctgta 3180
gcccaggcgc ggtggctcac acctgtaatc ccagcacttt ggaaggctga ggcgggcgga 3240
tcacaaggtc acgagttcaa aactatcctg gccaacacag tgaaaccccg tctctactaa 3300
aatacaaaaa aattagccgg gtgtggtggt gcatgccttt agtcctagct attcaggagg 3360
ctgaggcagg ggaatcgctt gaacccgaga ggcagaggtt gcagtgagct gagatcgcac 3420
cactgcactc cagcctggtt acagagcaag actctgtctc aaacaaaaca aaacaaaaca 3480
aaaacacact actgtatttt ggatggatca aacctcctta attttaattt ctaatcctaa 3540
agtaaagaga tgcaattggg ggccttccat gtagaaagtg gggtcaggag gccaagaaag 3600
ggaatatgaa tgtatatcca agtcactcag gaacttttat gcaggtgcta gaaactttat 3660
gtcaaagtgg ccacaagatt gtttaatagg agacgaacga atgtaactcc atgtttactg 3720
ctaaaaacca aagctttgtg taaaatcttg aatttatggg gcgggagggt aggaaagcct 3780
gtacctgtct gtttttttcc tgatcctttt ccctcattcc tgaactgcag gagactgagc 3840
ccctttgggc tttggtgacc ccatcactgg ggtgtgttta tttgatggtt gattttgctg 3900
tactgggtac ttcctttccc attttctaat cattttttaa cacaagctga ctcttccctt 3960
cccttctcct ttccctggga aaatacaatg aataaataaa gacttattgg tacgcaaact 4020
gtca 4024
<210> 6
<211> 1430
<212> DNA
<213> Homo sapiens
<400> OCT4 (POU5F1) coding
agagaggggt tgagtagtcc cttcgcaagc cctcatttca ccaggccccc ggcttggggc 60
gccttccttc cccatggcgg gacacctggc ttcggatttc gccttctcgc cccctccagg 120
tggtggaggt gatgggccag gggggccgga gccgggctgg gttgatcctc ggacctggct 180
aagcttccaa ggccctcctg gagggccagg aatcgggccg ggggttgggc caggctctga 240
ggtgtggggg attcccccat gccccccgcc gtatgagttc tgtgggggga tggcgtactg 300
tgggccccag gttggagtgg ggctagtgcc ccaaggcggc ttggagacct ctcagcctga 360
gggcgaagca ggagtcgggg tggagagcaa ctccgatggg gcctccccgg agccctgcac 420
cgtcacccct ggtgccgtga agctggagaa ggagaagctg gagcaaaacc cggaggagtc 480
ccaggacatc aaagctctgc agaaagaact cgagcaattt gccaagctcc tgaagcagaa 540
gaggatcacc ctgggatata cacaggccga tgtggggctc accctggggg ttctatttgg 600
gaaggtattc agccaaacga ccatctgccg ctttgaggct ctgcagctta gcttcaagaa 660
catgtgtaag ctgcggccct tgctgcagaa gtgggtggag gaagctgaca acaatgaaaa 720
tcttcaggag atatgcaaag cagaaaccct cgtgcaggcc cgaaagagaa agcgaaccag 780
tatcgagaac cgagtgagag gcaacctgga gaatttgttc ctgcagtgcc cgaaacccac 840
actgcagcag atcagccaca tcgcccagca gcttgggctc gagaaggatg tggtccgagt 900
gtggttctgt aaccggcgcc agaagggcaa gcgatcaagc agcgactatg cacaacgaga 960
ggattttgag gctgctgggt ctcctttctc agggggacca gtgtcctttc ctctggcccc 1020
agggccccat tttggtaccc caggctatgg gagccctcac ttcactgcac tgtactcctc 1080
ggtccctttc cctgaggggg aagcctttcc ccctgtctcc gtcaccactc tgggctctcc 1140
catgcattca aactgaggtg cctgcccttc taggaatggg ggacaggggg aggggaggag 1200
ctagggaaag aaaacctgga gtttgtgcca gggtttttgg gattaagttc ttcattcact 1260
aaggaaggaa ttgggaacac aaagggtggg ggcaggggag tttggggcaa ctggttggag 1320
ggaaggtgaa gttcaatgat gctcttgatt ttaatcccac atcatgtatc acttttttct 1380
taaataaaga agcctgggac acagtagata gacacactta aaaaaaaaaa 1430
<210> 7
<211> 2520
<212> DNA
<213> Homo sapiens
<400> SOX2 coding
ggatggttgt ctattaactt gttcaaaaaa gtatcaggag ttgtcaaggc agagaagaga 60
gtgtttgcaa aagggggaaa gtagtttgct gcctctttaa gactaggact gagagaaaga 120
agaggagaga gaaagaaagg gagagaagtt tgagccccag gcttaagcct ttccaaaaaa 180
taataataac aatcatcggc ggcggcagga tcggccagag gaggagggaa gcgctttttt 240
tgatcctgat tccagtttgc ctctctcttt ttttccccca aattattctt cgcctgattt 300
tcctcgcgga gccctgcgct cccgacaccc ccgcccgcct cccctcctcc tctccccccg 360
cccgcgggcc ccccaaagtc ccggccgggc cgagggtcgg cggccgccgg cgggccgggc 420
ccgcgcacag cgcccgcatg tacaacatga tggagacgga gctgaagccg ccgggcccgc 480
agcaaacttc ggggggcggc ggcggcaact ccaccgcggc ggcggccggc ggcaaccaga 540
aaaacagccc ggaccgcgtc aagcggccca tgaatgcctt catggtgtgg tcccgcgggc 600
agcggcgcaa gatggcccag gagaacccca agatgcacaa ctcggagatc agcaagcgcc 660
tgggcgccga gtggaaactt ttgtcggaga cggagaagcg gccgttcatc gacgaggcta 720
agcggctgcg agcgctgcac atgaaggagc acccggatta taaataccgg ccccggcgga 780
aaaccaagac gctcatgaag aaggataagt acacgctgcc cggcgggctg ctggcccccg 840
gcggcaatag catggcgagc ggggtcgggg tgggcgccgg cctgggcgcg ggcgtgaacc 900
agcgcatgga cagttacgcg cacatgaacg gctggagcaa cggcagctac agcatgatgc 960
aggaccagct gggctacccg cagcacccgg gcctcaatgc gcacggcgca gcgcagatgc 1020
agcccatgca ccgctacgac gtgagcgccc tgcagtacaa ctccatgacc agctcgcaga 1080
cctacatgaa cggctcgccc acctacagca tgtcctactc gcagcagggc acccctggca 1140
tggctcttgg ctccatgggt tcggtggtca agtccgaggc cagctccagc ccccctgtgg 1200
ttacctcttc ctcccactcc agggcgccct gccaggccgg ggacctccgg gacatgatca 1260
gcatgtatct ccccggcgcc gaggtgccgg aacccgccgc ccccagcaga cttcacatgt 1320
cccagcacta ccagagcggc ccggtgcccg gcacggccat taacggcaca ctgcccctct 1380
cacacatgtg agggccggac agcgaactgg aggggggaga aattttcaaa gaaaaacgag 1440
ggaaatggga ggggtgcaaa agaggagagt aagaaacagc atggagaaaa cccggtacgc 1500
tcaaaaagaa aaaggaaaaa aaaaaatccc atcacccaca gcaaatgaca gctgcaaaag 1560
agaacaccaa tcccatccac actcacgcaa aaaccgcgat gccgacaaga aaacttttat 1620
gagagagatc ctggacttct ttttggggga ctatttttgt acagagaaaa cctggggagg 1680
gtggggaggg cgggggaatg gaccttgtat agatctggag gaaagaaagc tacgaaaaac 1740
tttttaaaag ttctagtggt acggtaggag ctttgcagga agtttgcaaa agtctttacc 1800
aataatattt agagctagtc tccaagcgac gaaaaaaatg ttttaatatt tgcaagcaac 1860
ttttgtacag tatttatcga gataaacatg gcaatcaaaa tgtccattgt ttataagctg 1920
agaatttgcc aatatttttc aaggagaggc ttcttgctga attttgattc tgcagctgaa 1980
atttaggaca gttgcaaacg tgaaaagaag aaaattattc aaatttggac attttaattg 2040
tttaaaaatt gtacaaaagg aaaaaattag aataagtact ggcgaaccat ctctgtggtc 2100
ttgtttaaaa agggcaaaag ttttagactg tactaaattt tataacttac tgttaaaagc 2160
aaaaatggcc atgcaggttg acaccgttgg taatttataa tagcttttgt tcgatcccaa 2220
ctttccattt tgttcagata aaaaaaacca tgaaattact gtgtttgaaa tattttctta 2280
tggtttgtaa tatttctgta aatttattgt gatattttaa ggttttcccc cctttatttt 2340
ccgtagttgt attttaaaag attcggctct gtattatttg aatcagtctg ccgagaatcc 2400
atgtatatat ttgaactaat atcatcctta taacaggtac attttcaact taagttttta 2460
ctccattatg cacagtttga gataaataaa tttttgaaat atggacactg aaaaaaaaaa 2520
<210> 8
<211> 2078
<212> DNA
<213> Homo sapiens
<400> FOXD3 coding
atgaccctct ccggcggcgg cagcgccagc gacatgtccg gccagacggt gctgacggcc 60
gaggacgtgg acatcgatgt ggtgggcgag ggcgacgacg ggctggaaga gaaggacagc 120
gacgcaggtt gcgatagccc cgcggggccg ccggagctgc gcctggacga ggcggacgag 180
gtgcccccgg cggcacccca tcacggacag cctcagccgc cccaccagca gcccctgaca 240
ttgcccaagg aggcggccgg agccggggcc ggaccggggg gcgacgtggg cgcgccggag 300
gcggacggct gcaagggcgg tgttggcggc gaggagggcg gcgcgagcgg cggcgggcct 360
ggcgcgggca gcggttcggc gggaggcctg gccccgagca agcccaagaa cagcctagtg 420
aagccgcctt actcgtacat cgcgctcatc accatggcca tcctgcagag cccgcagaag 480
aagctgaccc tgagcggcat ctgcgagttc atcagcaacc gcttccccta ctacagggag 540
aagttccccg cctggcagaa cagcatccgc cacaacctct cactcaacga ctgcttcgtc 600
aagatccccc gcgagccggg caacccgggc aagggcaact actggaccct ggacccgcag 660
tccgaggaca tgttcgacaa cggcagcttc ctgcggcgcc ggaaacgctt caagcgccac 720
cagcaggagc acctgcgcga gcagacggcg ctcatgatgc agagcttcgg cgcttacagc 780
ctggcggcgg cggccggcgc cgcgggaccc tacggccgcc cctacggcct gcaccctgcg 840
gcggcggccg gtgcctattc gcacccggca gcggcggcgg ccgcggctgc tgcggcggcg 900
ctccagtacc cgtacgcgct gccgccggtg gcaccggtgc tgcctcccgc tgtgccgctg 960
ctgccctcgg gcgagctggg ccgcaaagcg gccgccttcg gctcacagct cggcccgggc 1020
ctgcagctgc agctcaatag cctgggcgcc gccgcggccg ctgcgggcac agcgggcgcc 1080
gcgggcacca ccgcgtcgct catcaagtcc gagccaagcg cgcggccgtc gttcagcatc 1140
gagaacatca taggtggggg ccccgcggct cctgggggct cggcggtggg cgctggggtc 1200
gccggcggca ctgggggttc agggggcggc agcacggcgc agtcgtttct gcggccaccc 1260
gggaccgtgc agtcggcagc gctcatggcc acccaccaac cgctgtcgct gagccggacg 1320
actgccacca tcgcgcccat tcttagcgtg ccactctccg gacagtttct gcagcccgca 1380
gcctcggccg ccgccgctgc tgcggccgcc gctcaagcca aatggccggc gcaataggga 1440
cgcgccaatg gccgggaccc agggtccggc ggcggcctcg agcaacaaat gcacctccag 1500
gctgcgcgcc ctgtcccaag cccggtcccg gtcccgctgc ccaatcctgg actctgcctc 1560
tccccaattt cctttcccct gagcccccaa cgcctacctt ccgcggcctc catcccctcg 1620
cgcacaccta agctggtcga gcaaactcac cgcgcgcccg ccggggatag ctttccatac 1680
aggtaaaacc gaaaaccgaa ttttccaaaa atgcaccccg acggcgcctg ctcttagtac 1740
cgtggggatg ggagggaaat tctttgtata tatttgtaaa aaaattattg actttccttt 1800
tggggttttt atttttttaa gaaaaaacaa attccgtaga tttagagctc tgaactttca 1860
ttttttttga aggttcactc tccgaagttt tatctgagaa aagaatgtat agagacgttg 1920
ggagatttta aatataaaaa attttcaaaa aggcaaaaag tgtcattcta ttataaaagt 1980
ctgtttatat atgaatgaat atatatggta ttctaaatgt tattccatcg tgttgtacac 2040
aactttgtaa ataaattttt aaaatgccaa aaaaaaaa 2078
<210> 9
<211> 2103
<212> DNA
<213> Homo sapiens
<400> NANOG coding
ttcattataa atctagagac tccaggattt taacgttctg ctggactgag ctggttgcct 60
catgttatta tgcaggcaac tcactttatc ccaatttctt gatacttttc cttctggagg 120
tcctatttct ctaacatctt ccagaaaagt cttaaagctg ccttaacctt ttttccagtc 180
cacctcttaa attttttcct cctcttcctc tatactaaca tgagtgtgga tccagcttgt 240
ccccaaagct tgccttgctt tgaagcatcc gactgtaaag aatcttcacc tatgcctgtg 300
atttgtgggc ctgaagaaaa ctatccatcc ttgcaaatgt cttctgctga gatgcctcac 360
acggagactg tctctcctct tccttcctcc atggatctgc ttattcagga cagccctgat 420
tcttccacca gtcccaaagg caaacaaccc acttctgcag agaagagtgt cgcaaaaaag 480
gaagacaagg tcccggtcaa gaaacagaag accagaactg tgttctcttc cacccagctg 540
tgtgtactca atgatagatt tcagagacag aaatacctca gcctccagca gatgcaagaa 600
ctctccaaca tcctgaacct cagctacaaa caggtgaaga cctggttcca gaaccagaga 660
atgaaatcta agaggtggca gaaaaacaac tggccgaaga atagcaatgg tgtgacgcag 720
aaggcctcag cacctaccta ccccagcctt tactcttcct accaccaggg atgcctggtg 780
aacccgactg ggaaccttcc aatgtggagc aaccagacct ggaacaattc aacctggagc 840
aaccagaccc agaacatcca gtcctggagc aaccactcct ggaacactca gacctggtgc 900
acccaatcct ggaacaatca ggcctggaac agtcccttct ataactgtgg agaggaatct 960
ctgcagtcct gcatgcagtt ccagccaaat tctcctgcca gtgacttgga ggctgccttg 1020
gaagctgctg gggaaggcct taatgtaata cagcagacca ctaggtattt tagtactcca 1080
caaaccatgg atttattcct aaactactcc atgaacatgc aacctgaaga cgtgtgaaga 1140
tgagtgaaac tgatattact caatttcagt ctggacactg gctgaatcct tcctctcccc 1200
tcctcccatc cctcatagga tttttcttgt ttggaaacca cgtgttctgg tttccatgat 1260
gcccatccag tcaatctcat ggagggtgga gtatggttgg agcctaatca gcgaggtttc 1320
tttttttttt tttttcctat tggatcttcc tggagaaaat actttttttt tttttttttt 1380
tgaaacggag tcttgctctg tcgcccaggc tggagtgcag tggcgcggtc ttggctcact 1440
gcaagctccg tctcccgggt tcacgccatt ctcctgcctc agcctcccga gcagctggga 1500
ctacaggcgc ccgccacctc gcccggctaa tattttgtat ttttagtaga gacggggttt 1560
cactgtgtta gccaggatgg tctcgatctc ctgaccttgt gatccacccg cctcggcctc 1620
cctaacagct gggatttaca ggcgtgagcc accgcgccct gcctagaaaa gacattttaa 1680
taaccttggc tgccgtctct ggctatagat aagtagatct aatactagtt tggatatctt 1740
tagggtttag aatctaacct caagaataag aaatacaagt acaaattggt gatgaagatg 1800
tattcgtatt gtttgggatt gggaggcttt gcttattttt taaaaactat tgaggtaaag 1860
ggttaagctg taacatactt aattgatttc ttaccgtttt tggctctgtt ttgctatatc 1920
ccctaatttg ttggttgtgc taatctttgt agaaagaggt ctcgtatttg ctgcatcgta 1980
atgacatgag tactgcttta gttggtttaa gttcaaatga atgaaacaac tatttttcct 2040
ttagttgatt ttaccctgat ttcaccgagt gtttcaatga gtaaatatac agcttaaaca 2100
taa 2103
<210> 10
<211> 6007
<212> DNA
<213> Homo sapiens
<400> PODXL coding
agccgcgcag acgccgccca ggacgcagcc gccgccgccg ccgctcctct gccactggct 60
ctgcgcccca gcccggctct gctgcagcgg cagggaggaa gagccgccgc agcgcgactc 120
gggagccccg ggccacagcc tggcctccgg agccacccac aggcctcccc gggcggcgcc 180
cacgctccta ccgcccggac gcgcggatcc tccgccggca ccgcagccac ctgctcccgg 240
cccagaggcg acgacacgat gcgctgcgcg ctggcgctct cggcgctgct gctactgttg 300
tcaacgccgc cgctgctgcc gtcgtcgccg tcgccgtcgc cgtcgccctc ccagaatgca 360
acccagacta ctacggactc atctaacaaa acagcaccga ctccagcatc cagtgtcacc 420
atcatggcta cagatacagc ccagcagagc acagtcccca cttccaaggc caacgaaatc 480
ttggcctcgg tcaaggcgac cacccttggt gtatccagtg actcaccggg gactacaacc 540
ctggctcagc aagtctcagg cccagtcaac actaccgtgg ctagaggagg cggctcaggc 600
aaccctacta ccaccatcga gagccccaag agcacaaaaa gtgcagacac cactacagtt 660
gcaacctcca cagccacagc taaacctaac accacaagca gccagaatgg agcagaagat 720
acaacaaact ctggggggaa aagcagccac agtgtgacca cagacctcac atccactaag 780
gcagaacatc tgacgacccc tcaccctaca agtccactta gcccccgaca acccacttcg 840
acgcatcctg tggccacccc aacaagctcg ggacatgacc atcttatgaa aatttcaagc 900
agttcaagca ctgtggctat ccctggctac accttcacaa gcccggggat gaccaccacc 960
ctactagaga cagtgtttca ccatgtcagc caggctggtc ttgaactcct gacctcgggt 1020
gatctgccca ccttggcctc ccaaagtgct gggattacag cgtcatcggt tatctcgcaa 1080
agaactcaac agacctccag tcagatgcca gccagctcta cggccccttc ctcccaggag 1140
acagtgcagc ccacgagccc ggcaacggca ttgagaacac ctaccctgcc agagaccatg 1200
agctccagcc ccacagcagc atcaactacc caccgatacc ccaaaacacc ttctcccact 1260
gtggctcatg agagtaactg ggcaaagtgt gaggatcttg agacacagac acagagtgag 1320
aagcagctcg tcctgaacct cacaggaaac accctctgtg cagggggcgc ttcggatgag 1380
aaattgatct cactgatatg ccgagcagtc aaagccacct tcaacccggc ccaagataag 1440
tgcggcatac ggctggcatc tgttccagga agtcagaccg tggtcgtcaa agaaatcact 1500
attcacacta agctccctgc caaggatgtg tacgagcggc tgaaggacaa atgggatgaa 1560
ctaaaggagg caggggtcag tgacatgaag ctaggggacc aggggccacc ggaggaggcc 1620
gaggaccgct tcagcatgcc cctcatcatc accatcgtct gcatggcatc attcctgctc 1680
ctcgtggcgg ccctctatgg ctgctgccac cagcgcctct cccagaggaa ggaccagcag 1740
cggctaacag aggagctgca gacagtggag aatggttacc atgacaaccc aacactggaa 1800
gtgatggaga cctcttctga gatgcaggag aagaaggtgg tcagcctcaa cggggagctg 1860
ggggacagct ggatcgtccc tctggacaac ctgaccaagg acgacctgga tgaggaggaa 1920
gacacacacc tctagtccgg tctgccggtg gcctccagca gcaccacaga gctccagacc 1980
aaccacccca agtgccgttt ggatggggaa gggaaagact ggggagggag agtgaactcc 2040
gaggggtgtc ccctcccaat ccccccaggg ccttaatttt tcccttttca acctgaacaa 2100
atcacattct gtccagattc ctcttgtaaa ataacccact agtgcctgag ctcagtgctg 2160
ctggatgatg agggagatca agaaaaagcc acgtaaggga ctttatagat gaactagtgg 2220
aatcccttca ttctgcagtg agattgccga gacctgaaga gggtaagtga cttgcccaag 2280
gtcagagcca cttggtgaca gagccaggat gagaacaaag attccatttg caccatgcca 2340
cactgctgtg ttcacatgtg ccttccgtcc agagcagtcc cgggcagggg tgaaactcca 2400
gcaggtggct gggctggaaa ggagggcagg gctacatcct ggctcggtgg gatctgacga 2460
cctgaaagtc cagctcccaa gttttccttc tcctacccca gcctcgtgta cccatcttcc 2520
caccctctat gttcttaccc ctccctacac tcagtgtttg ttcccactta ctctgtcctg 2580
gggcctctgg gattagcaca ggttattcat aaccttgaac cccttgttct ggattcggat 2640
tttctcacat ttgcttcgtg agatgggggc ttaacccaca caggtctccg tgcgtgaacc 2700
aggtctgctt aggggacctg cgtgcaggtg aggagagaag gggacactcg agtccaggct 2760
ggtatctcag ggcagctgat gaggggtcag caggaacact ggcccattgc ccctggcact 2820
ccttgcagag gccacccacg atcttctttg ggcttccatt tccaccaggg actaaaatct 2880
gctgtagcta gtgagagcag cgtgttcctt ttgttgttca ctgctcagct gatgggagtg 2940
attccctgag acccagtatg aaagagcagt ggctgcagga gaggccttcc cggggccccc 3000
catcagcgat gtgtcttcag agacaatcca ttaaagcagc caggaaggac aggctttccc 3060
ctgtatatca taggaaactc agggacattt caagttgctg agagttttgt tatagttgtt 3120
ttctaaccca gccctccact gccaaaggcc aaaagctcag acagttggca gacgtccagt 3180
tagctcatct cactcactct gattctcctg tgccacagga aaagagggcc tggaaagcgc 3240
agtgcatgct gggtgcatga agggcagcct gggggacaga ctgttgtggg aacgtcccac 3300
tgtcctggcc tggagctagg ccttgctgtt cctcttctct gtgagcctag tggggctgct 3360
gcggttctct tgcagtttct ggtggcatct caggggaaca caaagctatg tctattcccc 3420
aatataggac ttttatgggc tcggcagtta gctgccatgt agaaggctcc taagcagtgg 3480
gcatggtgag gtttcatctg attgagaagg gggaatcctg tgtggaatgt tgaactttcg 3540
ccatggtctc catcgttctg ggcgtaaatt ccctgggatc aagtaggaaa atgggcagaa 3600
ctgcttaggg gaatgaaatt gccatttttc gggtgaaacg ccacacctcc agggtcttaa 3660
gagtcaggct ccggctgtag tagctctgat gaaataggct atccactcgg gatggcttac 3720
tttttaaaag ggtaggggga ggggctgggg aagatctgtc ctgcaccatc tgcctaattc 3780
cttcctcaca gtctgtagcc atctgatatc ctaggggaaa aggaaggcca ggggttcaca 3840
tagggcccca gcgagtttcc caggagttag agggatgcga ggctaacaag ttccaaaaac 3900
atctgccccg atgctctagt gtttggaggt gggcaggatg gagaacagtg cctgtttggg 3960
ggaaaacagg aaatcttgtt aggcttgagt gaggtgtttg cttccttctt gcccagcgct 4020
gggttctctc cacccagtag gttttctgtt gtggtcccgt gggagaggcc agactggatt 4080
attcctcctt tgctgatcct gggtcacact tcaccagcca gggcttttga cggagacagc 4140
aaataggcct ctgcaaatca atcaaaggct gcaaccctat ggcctcttgg agacagatga 4200
tgactggcaa ggactagaga gcaggagtgc ctggccaggt cggtcctgac tctcctgact 4260
ctccatcgct ctgtccaagg agaacccgga gaggctctgg gctgattcag aggttactgc 4320
tttatattcg tccaaactgt gttagtctag gcttaggaca gcttcagaat ctgacacctt 4380
gccttgctct tgccaccagg acacctatgt caacaggcca aacagccatg catctataaa 4440
ggtcatcatc ttctgccacc tttactgggt tctaaatgct ctctgataat tcagagagca 4500
ttgggtctgg gaagaggtaa gaggaacact agaagctcag catgacttaa acaggttgta 4560
gcaaagacag tttatcatca gctctttcag tggtaaactg tggtttcccc aagctgcaca 4620
ggaggccaga aaccacaagt atgatgacta ggaagcctac tgtcatgaga gtggggagac 4680
aggcagcaaa gcttatgaag gaggtacaga atattctttg cgttgtaaga cagaatacgg 4740
gtttaatcta gtctaggcac cagatttttt tcccgcttga taaggaaagc tagcagaaag 4800
tttatttaaa ccacttcttg agctttatct tttttgacaa tatactggag aaactttgaa 4860
gaacaagttc aaactgatac atatacacat atttttttga taatgtaaat acagtgacca 4920
tgttaaccta ccctgcactg ctttaagtga acatactttg aaaaagcatt atgttagctg 4980
agtgatggcc aagttttttc tctggacagg aatgtaaatg tcttactgga aatgacaagt 5040
ttttgcttga tttttttttt taaacaaaaa atgaaatata acaagacaaa cttatgataa 5100
agtatttgtc ttgtagatca ggtgttttgt tttgtttttt taattttaaa atgcaaccct 5160
gccccctccc cagcaaagtc acagctccat ttcagtaaag gttggagtca atatgctctg 5220
gttggcaggc aaccctgtag tcatggagaa aggtatttca agatctagtc caatcttttt 5280
ctagagaaaa agataatctg aagctcacaa agatgaagtg acttcctcaa aatcacatgg 5340
ttcaggacag aaacaagatt aaaacctgga tccacagact gtgcgcctca gaaggaataa 5400
tcggtaaatt aagaattgct actcgaaggt gccagaatga cacaaaggac agaattcctt 5460
tcccagttgt taccctagca aggctaggga gggcatgaac acaaacataa gaactggtct 5520
tctacacttt ctctgaatca tttaggttta agatgtaagt gaacaattct ttctttctgc 5580
caagaaacaa agttttggat gagcttttat atatggaact tactccaaca ggactgaggg 5640
accaaggaaa catgatgggg gaggcagaga gggcaagagt aaaactgtag catagctttt 5700
gtcacggtca ctagctgatc cctcaggtct gctgcaaaca cagcatggag gacacagatg 5760
actctttggt gttggtcttt ttgtctgcag tgaatgttca acagtttgcc caggaactgg 5820
gggatcatat atgtcttagt ggacaggggt ctgaagtaca ctggaattta ctgagaaact 5880
tgtttgtaaa aactatagtt aataattatt gcattttctt acaaaaatat attttggaaa 5940
attgtatact gtcaattaaa gtgtttttgt gtaaactggt tcaaaaaaaa aaaaaaaaaa 6000
aaaaaaa 6007
<210> 11
<211> 2660
<212> DNA
<213> Homo sapiens
<400> REX1 (ZFP42)
agtttctcct ttgttttacg tttgggagga ggtggcattg gaaatagcag agtgcttcgc 60
ggtaacaggg gttggagtgc aatggtgtga tctcagctca ctgcaacccc tgcctcccag 120
gctccagcga tcctcccacc tcagcctcct gaatagctga ccaccagcac actaggcaaa 180
cccaccccac tcacggcctc ccttgggaat tcagacctaa ccatcgctga gctgaaacaa 240
atgtactgag gctggagcct gtgtgaacag aacagaagag gccttcactc tagtagtgct 300
cacagtccag caggtgtttg ctgaagacag cttactcaga tcactactgc ctggaggtgg 360
ttgatatatc ctggtgtaaa ccttcaagaa gggcacaggc aggaaaacat gagccagcaa 420
ctgaagaaac gggcaaagac aagacaccag aaaggcctgg gtggaagagc ccccagtggg 480
gctaagccca ggcaaggcaa gtcaagccaa gacctgcagg cggaaataga acctgtcagc 540
gcggtgtggg ccttatgtga tggctatgtg tgctatgagc ctggccctca ggctctcgga 600
ggggatgatt tctcagactg ttacatagaa tgcgtcataa ggggtgagtt ttctcaaccc 660
atcctggaag aggactcact ttttgagtcc ttggaatacc taaagaaagg atcagaacaa 720
cagctttctc aaaaggtttt cgaagcaagc tcccttgaat gttctttgga atacatgaaa 780
aaaggggtaa agaaagagct tccacaaaag atagttggag agaattcgct tgagtattct 840
gagtacatga caggcaagaa gcttccgcct ggaggaatac ctggcattga cctatcagat 900
cctaaacagc tcgcagaatt tgctagaaag aagcccccca taaataaaga atatgacagt 960
ctgagcgcaa tcgcttgtcc tcagagtgga tgcactagga agttgaggaa tagagctgcc 1020
ctgagaaagc atctcctcat tcatggtccc cgagaccacg tctgtgcgga atgtgggaaa 1080
gcgttcgttg agagctcaaa actaaagaga catttcctgg ttcatactgg agagaagccg 1140
tttcggtgca cttttgaagg gtgcggaaag cgcttctctc tggactttaa tttgcgtacg 1200
cacgtgcgca tccacacggg ggagaaacgt ttcgtgtgtc cctttcaagg ctgcaacagg 1260
aggtttattc agtcaaataa cctgaaagcc cacatcctaa cgcatgcaaa tacgaacaag 1320
aatgaacaag agggaaagta gtcctccaac aggatgaagc agattaacag aagagtgatc 1380
agtgacaaac atgcctcatt gattattgtt tctaggaagg aatttctaaa tcaatattgc 1440
aaccccaaaa gcggttataa tttggtgtta ctaagatgct cctacacttt gtgataccgt 1500
tttaaggaca tggtgcattt ttttttcttt tatttgtttt atttagaact ttttttattt 1560
gttttattta gaactttgtg tgttcttaaa gtgtgcttcc aacaggaagg tcagtgataa 1620
attttcaaaa gcataacctt caatatatta tctgttggat tattggatat aagacttatt 1680
ttcatgtact ataaatatga aaataacttt gatttttaat tgtgtagttt ccatttctta 1740
gcttttgcct tttaaattta tacttcagcc aggcatagtg actgatgcct gtaatcccaa 1800
cactttgttg ggaggccaaa gcaggaggat agcttgaggc caggagttcc agaccagcct 1860
gggcaacata gtgagatcct gtctctacaa aaaaatttgt ttttatttgt atttatatat 1920
ttttattttt gtttttgttg gtaggcgtct cgctctgtca cccaggctgg agtctagtgt 1980
cgtgatcttg gctcactgca acctccacct cccgggttca agtgattctc tggcctcagc 2040
ctcccaagta gctgggacta caggtgtgtg tcaccacgcc cggctaattt ttgtattttt 2100
agtagagatg gggtttcacc atgttggcca ggctagtctc aaactcctga cctccagtga 2160
tctgcccacc tcggcctccc aaagtgctgg gattacaggt gtgagccact gtgcctggcc 2220
ccccacaaca tgtttaaact tagctaggcc tggttgcata cgcctgtgtt cccagctact 2280
caggaggctg aagcaggagg atagcttgag cccaggagtt tgaggctaca gtgagctgtg 2340
attgcaccac tgtactccag actggataac agcaagagcc catcttttaa aaaaagtaaa 2400
aattaaaaat atacttcatg gttcatgtca tagccctaga gaatgaaaaa tttgcagtag 2460
atagtcaata aatgaatcag tagttaaata ttccttaaag tcaactgtat ttcattgtga 2520
tttttgtttt ctttttatca ttgtatcaaa ctatatggaa atcatatggt tagatgtgat 2580
tatttgataa tgttagtcca tttgaatcca ttttagatat ttcacaatta aagaatatga 2640
aacttcagaa aaaaaaaaaa 2660
<210> 12
<211> 6063
<212> DNA
<213> Homo sapiens
<400> SSEA1 (FUT4) coding
aatccctccc tccggcgggc gtcgctggcg ggtggctagg cccaacggca ggaagccgac 60
gctatcctcc gttccgcggc gccgggtccg ccttccgtct gttctagggc ctgctcctgc 120
gcggcagctg ctttagaagg tctcgagcct cctgtacctt cccagggatg aaccgggcct 180
tccctctgga aggcgagggt tcgggccaca gtgagcgagg gccagggcgg tgggcgcgcg 240
cagagggaaa ccggatcagt tgagagagaa tcaagagtag cggatgaggc gcttgtgggg 300
cgcggcccgg aagccctcgg gcgcgggctg ggagaaggag tgggcggagg cgccgcagga 360
ggctcccggg gcctggtcgg gccggctggg ccccgggcgc agtggaagaa agggacgggc 420
ggtgcccggt tgggcgtcct ggccagctca ccttgccctg gcggctcgcc ccgcccggca 480
cttgggagga gcagggcagg gcccgcggcc tttgcattct gggaccgccc ccttccattc 540
ccgggccagc ggcgagcggc agcgacggct ggagccgcag ctacagcatg agagccggtg 600
ccgctcctcc acgcctgcgg acgcgtggcg agcggaggca gcgctgcctg ttcgcgccat 660
gggggcaccg tggggctcgc cgacggcggc ggcgggcggg cggcgcgggt ggcgccgagg 720
ccgggggctg ccatggaccg tctgtgtgct ggcggccgcc ggcttgacgt gtacggcgct 780
gatcacctac gcttgctggg ggcagctgcc gccgctgccc tgggcgtcgc caaccccgtc 840
gcgaccggtg ggcgtgctgc tgtggtggga gcccttcggg gggcgcgata gcgccccgag 900
gccgccccct gactgccggc tgcgcttcaa catcagcggc tgccgcctgc tcaccgaccg 960
cgcgtcctac ggagaggctc aggccgtgct tttccaccac cgcgacctcg tgaaggggcc 1020
ccccgactgg cccccgccct ggggcatcca ggcgcacact gccgaggagg tggatctgcg 1080
cgtgttggac tacgaggagg cagcggcggc ggcagaagcc ctggcgacct ccagccccag 1140
gcccccgggc cagcgctggg tttggatgaa cttcgagtcg ccctcgcact ccccggggct 1200
gcgaagcctg gcaagtaacc tcttcaactg gacgctctcc taccgggcgg actcggacgt 1260
ctttgtgcct tatggctacc tctaccccag aagccacccc ggcgacccgc cctcaggcct 1320
ggccccgcca ctgtccagga aacaggggct ggtggcatgg gtggtgagcc actgggacga 1380
gcgccaggcc cgggtccgct actaccacca actgagccaa catgtgaccg tggacgtgtt 1440
cggccggggc gggccggggc agccggtgcc cgaaattggg ctcctgcaca cagtggcccg 1500
ctacaagttc tacctggctt tcgagaactc gcagcacctg gattatatca ccgagaagct 1560
ctggcgcaac gcgttgctcg ctggggcggt gccggtggtg ctgggcccag accgtgccaa 1620
ctacgagcgc tttgtgcccc gcggcgcctt catccacgtg gacgacttcc caagtgcctc 1680
ctccctggcc tcgtacctgc ttttcctcga ccgcaacccc gcggtctatc gccgctactt 1740
ccactggcgc cggagctacg ctgtccacat cacctccttc tgggacgagc cttggtgccg 1800
ggtgtgccag gctgtacaga gggctgggga ccggcccaag agcatacgga acttggccag 1860
ctggttcgag cggtgaagcc gcgctcccct ggaagcgacc caggggaggc caagttgtca 1920
gctttttgat cctctactgt gcatctcctt gactgccgca tcatgggagt aagttcttca 1980
aacacccatt tttgctctat gggaaaaaaa cgatttacca attaatatta ctcagcacag 2040
agatgggggc ccggtttcca tattttttgc acagctagca attgggctcc ctttgctgct 2100
gatgggcatc attgtttagg ggtgaaggag ggggttcttc ctcaccttgt aaccagtgca 2160
gaaatgaaat agcttagcgg caagaagccg ttgaggcggt ttcctgaatt tccccatctg 2220
ccacaggcca tatttgtggc ccgtgcagct tccaaatctc atacacaact gttcccgatt 2280
cacgtttttc tggaccaagg tgaagcaaat ttgtggttgt agaaggagcc ttgttggtgg 2340
agagtggaag gactgtggct gcaggtggga ctttgttgtt tggattcctc acagccttgg 2400
ctcctgagaa aggtgaggag ggcagtccaa gaggggccgc tgacttcttt cacaagtact 2460
atctgttccc ctgtcctgtg aatggaagca aagtgctgga ttgtccttgg aggaaactta 2520
agatgaatac atgcgtgtac ctcactttac ataagaaatg tattcctgaa aagctgcatt 2580
taaatcaagt cccaaattca ttgacttagg ggagttcagt atttaatgaa accctatgga 2640
gaatttatcc ctttacaatg tgaatagtca tctcctaatt tgtttcttct gtctttatgt 2700
ttttctataa cctggatttt ttaaatcata ttaaaattac agatgtgaaa ataaagcaga 2760
agcaaccttt ttccctcttc ccagaaaacc agtctgtgtt tacagacaga agagaaggaa 2820
gccatagtgt cacttccaca caattattta tttcatgtct ttactggacc tgaaatttaa 2880
actgcaatgc cagtcctgca ggagtgctgg cattaccctc tgcagaacag tgaaaggtat 2940
tgcactacat tatggaatca tgcaaaagga aaaaaagttt catgatatct gttgttggca 3000
gtttttgttt atctctgaca gtttttagtt aaatgtttag atcctcagaa ctacattagt 3060
gcctactatt aacttactct gtctcttgtt aaaggctaaa tctgcgcttc tccctggtgc 3120
cagcaggttc ccctcacagt caatgcagtg gtatagcata tcctcacatt tctagtgccc 3180
ttgagactgt gctatggaac caatcttgaa catacatgca ttgacttgac aagttactga 3240
gtaagcagca tattcagcag gtgccactac atgcctactc tgccagacac tgagcttggg 3300
gccctaggga agatagagaa ttatacaagg caaagtcctt ctctttaggg ctcttacaat 3360
ctatcacttc caaaaagtaa atggtgactg ataaaacaat tggcagaacc tgtttgatta 3420
ctgtgacagt cttaatgata ccataaatca atattagaaa gctagttgac ttaaagcctg 3480
aaataatggg agttttctcc tccacttatt agaataagga ccctcagtga ctaattattg 3540
tgggtagggt caagattaac tagttttata cagagttctg ctgtaaatag tcattttgca 3600
tttgattagt gcagttctct gaatcataaa gcaagtttta cctctctgta catgtttttg 3660
cagacatact tgaaaagctc acttaaatct aggtgcttca attcactttc ttgagaggac 3720
aaatgaaaag ctgtggagaa aatgtcctca ttaaagtatt aaagtgtggg cagaattaca 3780
attacaaagt gccagccacc gaataaagat aaaagttcag ttcttaaaat gagtttttat 3840
gagataacag tcagtgatct tggtgttacc gggattccac atggggcagt gggaaagagt 3900
tcaggttttg aaggtaacct agtttagatt tgaattccag ctatgtgaca ttgggtaaat 3960
tagtagtagt cctgagcctc agcgtcctca tctataaaat gactggcgaa aatacttcac 4020
aagctcattt tgagcacttt aggaagtaag tgaaagtacc taaaatagca ggcacccaat 4080
tgatgatttt atatcttcct tctttgcttg cagtgatttc aggatgtcct catatctatt 4140
tataggtcta aaattatatc ttaaggtatg ttgtagaata aattaaaagg ataatctaaa 4200
tcaccattta gattaagctt gacttgcaaa ctaggaagaa gcacctaggc tttctttgaa 4260
aatatttttt tggttcgttt tggtaaagct ctataaattg gtatctatta ttttaccaat 4320
ttttttttag tattaagtcc atttagaact aaccatatta tttatggaat aattagcatg 4380
aggaaggtat aattgcattt tttttttttt gagacggagt cttgcactgt agccccagct 4440
ggactgcagt ggcgtgatct tggctcactg caacctccgc ctcccaggtt caagcgattc 4500
tcctgcctca gcctcccgag cagctgagac tacaggcgcc tgccaccacg cctggccaat 4560
tttttgtatt tttagtagag actgcgtttc accatgttgg gcaggctggt cttgaactcc 4620
tgaccttgtg atccacctgc ctcggcctct cagagagctg ggattacagg tgtgagccgc 4680
cgtgcccagc cattgcattt ttattcacat acacattgtt aatgtggaac aatttaacac 4740
taatctcatc agagagcgag atgaatgtgg caattgctca ttttattttg catatattaa 4800
attgagtagg ttcagctcta acatacctta agaaaaatgc atatcggtgc actgtatgta 4860
tttcaaaatg cctttcctat gattgtcatg tcctccttta aggcttttcc ctcaaattta 4920
ttacaaattt agtattttta gtacttgatg actctaatta catgaatgca cctggaatga 4980
catttgtaac agaagacggt ctgacttgct ttcagtattc acaagttctt tccagtttcc 5040
aagtcttttc ctagcagtaa tttaggggag acagaggagt ttcatgtaaa gagcatgcag 5100
tttggagtca gaacctgggt atgactctgt ggccttgatg aagcaagtta cttaaactct 5160
tgagttttag ctttctcctt tacaatgcat gaatgcctat ccccctacaa aacaaagatt 5220
aaatgtgatg atgtatgcca aggtgctttg tatattgtaa agtgctatat aattataaga 5280
tgttctaaat tttcaaggat ctaaaccagg gattggcaaa cgtttttcca gggagtaaat 5340
attttacgct ttgcatatat aatttatgga ggtgttgaga ggatagatta gacacttgaa 5400
gtactcagga tagtgcctgg catgtaggaa gcacctggaa aatattcgct gtgattacca 5460
tcagtccatt ttaccgagga aggagccaag gtccaggccc actgaaggac ttgcataaca 5520
ttacaatagc agtggcagaa ccagccatgc ttctgcaaat cacaacctct ttgagcctct 5580
gtcacctgaa ctgcaaaatg agtgggttag acaaaatcat ctgttgggac ctcctagttc 5640
cacgtgctat cattctacta actggcaccc taaggttgaa agtgcttatc tgctttccaa 5700
tgtggcttcc ttacagtctg gaactgacaa tatgcaggag cagtaaactg gcagaaaacc 5760
aggaatcaga gaaagaaaat ataatttaac tttaaagatg taaattatat atatagtata 5820
ttatatatat ttttaaagct ttatatgcct caaatatcag ggaaaggagc caagtccttg 5880
gtatttagtt tggtgaatac ttgcattgaa tacatgtcaa gatgtcaagt catttttgaa 5940
tgtgtctcag ggatttctat gctacacatt cttttaacaa atcaagtatt tatgtacaca 6000
tgttcagatt ttttgacaaa atgattaaaa taatgagatg gaaaatgaaa aaaaaaaaaa 6060
aaa 6063
<210> 13
<211> 1393
<212> DNA
<213> Homo sapiens
<400> DPPA2 coding
agccctttgt ttatggcctg atctagctaa ggcttctaga cttcaggagc ttaagaatcg 60
tccggagggc tgggcgtggc ggtgcaggcc tgtagtccca cccactccga aggctgcgga 120
gggaggatca acttgagtct gggaactcag ccaggaattc aagaccagcc tgggcaacac 180
agtgaggccc cctacccaca tcctctccgt ccccgcaatc tccttccatc ccagggtgtt 240
gctgaaaatg tcagatgcaa atttggatag cagcaagaag aatttcttgg agggggaagt 300
agatgatgag gaaagtgtga ttttgacact ggtgccagtt aaagatgacg caaatatgga 360
acaaatggaa ccaagcgttt cttcaacttc tgatgtcaaa ctggagaagc ctaagaaata 420
caatccaggt catctacttc aaacaaatga gcaatttaca gctccacaaa aagctagatg 480
caaaatacca gcccttccct tgccgaccat tttgcctccc attaataagg tgtgtcggga 540
cactttgcgg gactggtgtc aacaactcgg tttgagtact aatggcaaga aaatcgaagt 600
ttatctgagg cttcataggc atgcttaccc tgaacaacgg caagatatgc ctgaaatgtc 660
acaagagacc agattacagc gatgttcgag gaaacgcaag gcagtgacca agagagcaag 720
gcttcagaga agttatgaga tgaatgagag agcagaagag accaatacag ttgaagtgat 780
aacttcagca ccgggagcca tgttggcatc atgggcaaga attgctgcaa gagctgttca 840
gcctaaggct ttgaattcat gttccattcc tgtttctgtt gaggcctttt tgatgcaagc 900
ctctggcgtc aggtggtgtg tggtccatgg cagacttctc tcggcagaca caaagggttg 960
ggtacgcctg cagtttcatg caggtcaggc ctgggtgcct accactcaca ggaggatgat 1020
ttctctcttc ttgttacctg cctgcatttt cccatcccca ggcatagaag ataatatgtt 1080
atgccccgac tgtgctaaga ggaataagaa gatgatgaaa agattaatga cagtagagaa 1140
gtagcagcaa cctgtttgaa tacaatgtac taaaggaggg atgtactttc agatcatgta 1200
acctattacg aaggagtgga agaggagaca atttgaatga atcctcatga tctacaaaac 1260
aaaatcatag tgactaggac tccacagtga agatggttga ctagtgacac agccccatct 1320
aaagaatccc tttctgtatg tctgaaaacc cattaaaata aagtcactgc aattggcctt 1380
gtaaaaaaaa aaa 1393
<210> 14
<211> 1079
<212> DNA
<213> Homo sapiens
<400> DPPA3 coding
tggagctccg gttttcagcc tctttccggg ctacctggta gcaatttgag gctctgtcat 60
cagtttctgc tacgtttcaa agatcctgga gaagcctagt gttgtgtcaa gacgccgatg 120
gacccatcac agtttaatcc aacctacatc ccagggtctc cacaaatgct caccgaagaa 180
aattcccggg acgattcagg ggcctctcaa atctcctccg agacgttgat aaagaacctt 240
agtaacttga ctatcaacgc tagtagcgaa tctgtttccc ctctatcgga agctttactc 300
cgtcgagagt ctgtaggagc agcagtcctc agggaaatcg aagatgagtg gctttacagc 360
aggagaggag taagaacatt gctgtctgtg cagagagaaa agatggcaag attgagatac 420
atgttactcg gcggagttcg tacgcatgaa agaagaccaa caaacaagga gcctaaggga 480
gttaagaagg aatcaagacc attcaaatgt ccctgcagtt tctgcgtgtc taatggatgg 540
gatccttctg agaatgctag aatagggaat caagacacca agccacttca gccataaatc 600
ttattcttgc accttttttt cttggtagta attttatata gcaggttgag aaagctactc 660
tatgctagta tagactatac accaataatt ttgataatga gttctaggat gtatttttct 720
tgtatctttt tcttcctact atgatactag taattcataa gggatctgtg taatctgaat 780
gtatttgaat aactttagct ctactgtttg atttgaccca aagaagccaa gatgatataa 840
gtattcccat gtgtcttaga agcccaaagt cagtgagatg aaacccaaca tcaagaaatt 900
gaagcaaagt tacttatgga taaagaaagc attaggtagt tgggctatag cataattaga 960
ttttctggct ttcaaaaatt tggattgcaa tcacagcaaa ctttgttatt tttacagttt 1020
tcagtacaaa agtgtttata tagaaacaat aaagttgaca tttgagtacc ttttaaaaa 1079
<---










Настоящее изобретение относится к области клеточной биологии и медицины, в частности к способу выявления остаточных, недифференцированных индуцированных плюрипотентных стволовых клеток (iPSC) в культуре клеток, дифференцировавшихся из iPSC. Для осуществления способа согласно изобретению сначала культивируют указанные клетки на субстрате, покрытом ламинином-521 и Е-кадгерином, в среде, содержащей ингибитор Rho-ассоциированной протеинкиназы, содержащей суперспираль (ROCK). Затем проводят количественное измерение в культивируемых клетках экспрессии маркера остаточных, недифференцированных iPSC. Далее сравнивают экспрессию маркера в культивируемых клетках с экспрессией маркера в контрольной культуре клеток, содержащей известную долю iPSC. Делают вывод об отсутствии остаточных, недифференцированных iPSC в культивируемых клетках или на присутствие их в меньшей доле, чем известная доля iPSC в контрольной культуре клеток в случае, когда в культуре клеток обнаруживают более низкую экспрессию маркера, чем экспрессия маркера в контрольной культуре клеток. Настоящее изобретение также раскрывает набор для осуществления указанного способа, а также способ изготовления терапевтической композиции и способ лечения или предупреждения состояний, при которых полезен иммуномодулирующий эффект. Настоящее изобретение позволяет получить улучшенный способ выявления остаточных, недифференцированных PSC в культуре клеток, происходящих от iPSC, подлежащий применению в контроле качества при производстве терапевтических композиций, содержащих клетки, происходящие от iPSC. 4 н. и 18 з.п. ф-лы, 14 ил., 8 табл., 13 пр.
1. Способ выявления остаточных, недифференцированных индуцированных плюрипотентных стволовых клеток (iPSC) в культуре клеток, дифференцировавшихся из iPSC, включающий:
культивирование указанных клеток на субстрате, покрытом ламинином-521 и Е-кадгерином, в среде, содержащей ингибитор Rho-ассоциированной протеинкиназы, содержащей суперспираль (ROCK);
количественное измерение в культивируемых клетках экспрессии маркера остаточных, недифференцированных iPSC; и
сравнение экспрессии маркера в культивируемых клетках с экспрессией маркера в контрольной культуре клеток, содержащей известную долю iPSC,
где более низкая экспрессия маркера в культуре клеток, чем экспрессия маркера в контрольной культуре клеток, указывает на отсутствие остаточных, недифференцированных iPSC в культивируемых клетках или на присутствие остаточных, недифференцированных iPSC в культивируемых клетках в меньшей доле, чем известная доля iPSC в контрольной культуре клеток.
2. Способ по п. 1, где экспрессия маркера представляет собой экспрессию LIN28, OCT4, SOX2, FOXD3, NANOG, PODXL, REX1, SSEA1, SSEA4, DPPA2 или DPPA3.
3. Способ по п. 1 или 2, где ингибитор ROCK представляет собой Y27632.
4. Способ по п. 3, где клетки культивируют в примерно 10 мкМ Y27632.
5. Способ по любому из пп. 1-4, включающий культивирование клеток в среде, содержащей ингибитор ROCK, в течение примерно 3 суток.
6. Способ по п. 5, дополнительно включающий культивирование клеток в среде, не содержащей ингибитор ROCK, в течение примерно 2 суток после культивирования в среде, содержащей ингибитор ROCK.
7. Способ по любому из пп. 1-6, где экспрессию маркера количественно измеряют полимеразной цепной реакцией (ПЦР).
8. Способ по п. 7, где ПЦР представляет собой количественную ПЦР в реальном времени (кПЦР-РВ).
9. Способ по п. 8, где кПЦР-РВ включает зонд, состоящий из 5′→3′ последовательности CGCATGGGGTTCGGCTTCCTGTCC (SEQ ID NO: 15), праймер, состоящий из 5′→3′ последовательности CACGGTGCGGGCATCTG (SEQ ID NO: 16), и праймер, состоящий из 5′→3′ последовательности CCTTCCATGTGCAGCTTACTC (SEQ ID NO: 17).
10. Способ по любому из пп. 1-9, где экспрессия маркера нормализована.
11. Способ по п. 10, где экспрессия маркера нормализована по отношению к экспрессии GAPDH.
12. Способ по любому из пп. 1-11, где известная доля составляет 0,001%.
13. Способ по любому из пп. 1-12, где клетки представляют собой мезенхимные стволовые клетки (MSC).
14. Способ по п. 13, где MSC культивируют в полной среде Е8.
15. Способ по любому из пп. 1-14, дополнительно включающий приготовление в виде терапевтической композиции культуры клеток, в которой остаточные, недифференцированные iPSC отсутствуют или присутствуют в меньшей доле, чем известная доля.
16. Способ по любому из пп. 1-15, дополнительно включающий введение субъекту культуры клеток, в которой остаточные, недифференцированные iPSC отсутствуют или присутствуют в меньшей доле, чем известная доля, или терапевтической композиции.
17. Способ изготовления терапевтической композиции для терапевтического введения субъекту, содержащей культуру клеток, дифференцировавшихся из iPSC, в которой остаточные, недифференцированные iPSC отсутствуют или присутствуют в меньшей доле, чем известная доля, включающий
культивирование указанных клеток на субстрате, покрытом ламинином-521 и Е-кадгерином, в среде, содержащей ингибитор ROCK;
количественное измерение в культивируемых клетках экспрессии маркера остаточных, недифференцированных iPSC;
сравнение экспрессии маркера в культивируемых клетках с экспрессией маркера в контрольной культуре клеток, содержащей известную долю iPSC, где более низкая экспрессия маркера в культуре клеток, чем экспрессия маркера в контрольной культуре клеток, указывает на отсутствие остаточных, недифференцированных iPSC в культивируемых клетках или на присутствие остаточных, недифференцированных iPSC в культивируемых клетках в меньшей доле, чем известная доля iPSC в контрольной культуре клеток, и
приготовление культуры клеток, в которой остаточные, недифференцированные iPSC отсутствуют или присутствуют в меньшей доле, чем известная доля, в виде терапевтической композиции.
18. Способ лечения или предупреждения состояния, при котором полезны иммуномодулирующие эффекты, у субъекта, где состояние выбрано из кист кости, новообразований кости, переломов, дефектов хряща, остеоартрита, повреждения связки, незавершенного остеогенеза, остеонекроза, остеопороза, апластической анемии, реакции «трансплантат против хозяина» (GvHD), миелодиспластического синдрома, диабета типа 1, диабета типа 2, аутоиммунного гепатита, цирроза печени, печеночной недостаточности, дилатационной кардиомиопатии, сердечной недостаточности, инфаркта миокарда, ишемии миокарда, болезни Крона, язвенного колита, ожогов, буллезного эпидермолиза, красной волчанки, ревматоидного артрита, болезни Шегрена, системного склероза, бронхолегочной дисплазии, хронического обструктивного заболевания дыхательных путей, эмфиземы, легочного фиброза, бокового амиотрофического склероза (ALS), болезни Альцгеймера, травмы головного мозга, атаксии, остеохондроза, множественной системной атрофии, рассеянного склероза, болезни Паркинсона, пигментной дистрофии сетчатки, болезни Ромберга, травмы спинного мозга, инсульта, мышечной дистрофии, ишемии конечности, повреждения почки, волчаночного нефрита, эндометриоза и осложнений трансплантации костного мозга или солидных органов, включающий
культивирование клеток, дифференцировавшихся из iPSC, на субстрате, покрытом ламинином-521 и Е-кадгерином, в среде, содержащей ингибитор ROCK;
количественное измерение в культивируемых клетках экспрессии маркера остаточных, недифференцированных iPSC;
сравнение экспрессии маркера в культивируемых клетках с экспрессией маркера в контрольной культуре клеток, содержащей известную долю iPSC, где более низкая экспрессия маркера в культуре клеток, чем экспрессия маркера в контрольной культуре клеток, указывает на отсутствие остаточных, недифференцированных iPSC в культивируемых клетках или на присутствие остаточных, недифференцированных iPSC в культивируемых клетках в меньшей доле, чем известная доля iPSC в контрольной культуре клеток, и
введение указанному субъекту:
культуры клеток, дифференцировавшихся из iPSC, в которой остаточные, недифференцированные iPSC отсутствуют или присутствуют в меньшей доле, чем известная доля, или
культуры клеток, дифференцировавшихся из iPSC, в которой остаточные, недифференцированные iPSC отсутствуют или присутствуют в меньшей доле, чем известная доля, приготовленной в виде терапевтической композиции.
19. Набор для выявления остаточных, недифференцированных iPSC в культуре клеток, дифференцировавшихся из iPSC, содержащий:
ламинин-521; и
Е-кадгерин; и
ингибитор ROCK.
20. Набор по п. 19, дополнительно содержащий праймеры ПЦР и, возможно, зонд ПЦР для количественного измерения в культивируемых клетках экспрессии маркера остаточных, недифференцированных iPSC.
21. Набор по п. 19 или 20, дополнительно содержащий среду, возможно содержащую ингибитор ROCK.
22. Набор по любому из пп. 19-21, дополнительно содержащий субстрат, возможно покрытый ламинином-521 и Е-кадгерином.
| KURODA T., et al., Highly Sensitive In Vitro Methods for Detection of Residual Undifferentiated Cells in Retinal Pigment Epithelial Cells Derived from Human iPS Cells, 17.05.2012, PLoS ONE, Vol.7, Is.5, e37342 | |||
| TANO K., et al., A Novel In Vitro Method for Detecting Undifferentiated Human Pluripotent Stem Cells as Impurities in Cell Therapy |
Авторы
Даты
2021-10-25—Публикация
2017-11-15—Подача