Область техники
Изобретение относится к области генетики растений, в частности к способу молекулярно-генетической идентификации неизвестного образца древесины хвойных растений с использованием ПЦР. Изобретение позволяет проверить соответствие профилей фрагментов ДНК неизвестных образцов древесины из бревен с мест заготовки или после транспортировки с идентификационными фрагментами ДНК деревьев природных популяций с использованием одного или двух типов молекулярных маркеров. Способ может найти применение при определении места происхождения и заготовки древесины, что будет способствовать разработке мер борьбы с незаконными рубками леса и уменьшению ущерба лесным экосистемам, а также разработке рекомендаций охраны и восстановления природных популяций.
Уровень техники
В основе системы отслеживания происхождения древесины в России в настоящее время лежат принципы идентификации, раздельности и документирования [Пальчиков, Румянцев, 2009]. У видов хвойных растений определение мест произрастания срубленных деревьев возможно проводить на спиле ствола по годичным кольцам, но этот метод не всегда позволяет определить происхождение древесины [Хох, 2019]. Внешние признаки подвержены изменениям и их практически невозможно определить на разных стадиях переработки древесины, в том числе и на стадии пиломатериалов [Шейкина и др., 2012]. В связи с этим молекулярно-генетический метод, основанный на применении ДНК-маркеров, является единственным надежным методам для идентификации природных популяций, в которых произведена заготовка древесины [Рамазанова, 2008].
Для идентификации древесины хвойных растений используются разные типы ДНК-маркеров: STS (Sequence Tagged Site) и PCR-RFLP (Polymerase Chain Reaction Restriction Fragment Length Polymorphism) [Nowakowska, 2011], ISSR-(Inter Simple Sequence Repeats) [Шейкина и др., 2012], SSR-(Simple Sequence Repeats) [Крутовский и др., 2019].
Ядерные SSR (Simple Sequence Repeats)-маркеры изменчивы и их использование имеет множество недостатков, связанных с качеством данных, полученных на образцах древесины [Jolivet, Degen, 2012; Degen et al., 2013]. Технология секвенирования позволяет выявить SNP (Single Nucleotide Polymorphism), которые могут представлять интересную альтернативу использованию микросателлитных маркеров [Blanc-Jolivet, Liesebach, 2015; Jardine et al., 2016]. К настоящему времени разработаны различные методики выявления SNP в древесине видов хвойных растений [Blanc-Jolivet et al., 2018; Крутовский и др., 2019; Overview of current practices..., 2020], но они не ассоциированы с конкретной популяцией, поэтому не пригодны для идентификации древесины.
Молекулярно-генетическая идентификация на популяционном уровне видов хвойных растений представляют собой аналитический инструмент, в котором используются идентификационные генетические маркеры видов древесных растений, выявляемые в определенных популяциях в естественных лесах. При сравнении этих маркеров с молекулярными идентификационными маркерами, выявленными после молекулярно-генетического анализа с пробами ДНК, выделенными из древесины бревен, можно установить природную популяцию, в которой ранее произрастали срубленные деревья; а также выявить, соответствует ли место заготовки древесины тому, что указано в документах. Таким образом, можно определить географическое происхождение древесины и установить легальность ее заготовки. Эффективность разработанной системы молекулярно-генетической идентификации подтверждается ее апробацией на пробах ДНК, выделенных из анонимных образцов древесины срубленных деревьев, произрастающих в одной из популяций исследованных ранее видов хвойных растений на примере сосны обыкновенной и лиственницы сибирской. После проведения молекулярно-генетического анализа выявленные идентификационные маркеры (фрагменты ДНК), характерных для популяций мест произрастания, сравниваются с аналогичными показателями, полученными в ПЦР с пробами ДНК, выделенными из неизвестных образцов древесины, взятых у срубленных деревьев в регионе исследований. Ни в одной из публикаций или в иных источниках информации не описан способ молекулярно-генетической идентификации неизвестного образца древесины хвойных растений. Предлагаемое решение устраняет этот недостаток, так как предлагает относительно недорогой и доступный для выполнения в генетических лабораториях, способ молекулярно-генетической идентификации неизвестного образца древесины на основании сопоставления идентификационных фрагментов ДНК этих образцов с ранее выявленными фрагментами ДНК при проведении идентификации древесины в природных популяциях.
Наиболее близким по технической сущности к предлагаемому решению, взятым за прототип является способ, описанный в патенте РФ на изобретение Боронниковой С.В. и Бобошиной И.В. «Способ молекулярно-генетической идентификации популяций древесных видов растений» (Патент РФ №2505956, МПК А01Н 1/00, опубл. 10.02.2014 в Бюл. №4. 10 с.)
Изобретение относится к способу молекулярно-генетической идентификации природных популяций древесных видов растений, который включает выбор эффективных стабильных молекулярных маркеров, сбор материала, проведение молекулярно-генетического анализа с использованием ПЦР, анализ выявленных ISSR-маркеров и определение идентификационных (мономорфных и полиморфных), анализ полученных данных после секвенирования, составление молекулярно-генетической формулы, составление штрихкода, составление генетического паспорта. Способ характеризуется тем, что в молекулярно-генетическую формулу и штрихкод, помимо идентификационных фрагментов ДНК разного размера, амплифицированных в результате полимеразной цепной реакции (ПЦР), вносят и другие структурные изменения геномов, такие как делеции, дупликации, однонуклеотидные замены (SNP - Single Nucleotide Polymorphism), выявленные при сравнительном анализе нуклеотидных последовательностей после секвенирования геномной ДНК. Изобретение позволяет эффективно идентифицировать природные популяции древесных видов растений на нуклеотидном уровне.
Недостатки прототипа:
1. Прототип разработан на примере древесных растений, относящихся к отделу «Цветковые» или «Покрытосеменные растения», для которых характерны листья, поэтому они называются «лиственные древесные растения». Вследствие более высокой ступени эволюционного развития эти виды более генетически гетерогенны. На большей территории России распространены виды хвойных растений, относящиеся к отделу «Голосеменные растения», для которых характерна хвоя и эти популяции видов хвойных растений более генетически гомогенны.
2. В прототипе ДНК выделяется из листьев, которые являются живыми вегетативными органами растений и состоят из сложных живых тканей, а в предлагаемом способе - непосредственно из древесины, которая в своем составе имеет мертвые ткани, характеризуется меньшим генетическим разнообразием, а как следствие этого, содержит меньшее фрагментов ДНК (молекулярных маркеров).
3. Система разработана для более 65 фрагментов ДНК, то есть молекулярных маркеров, таких как ISSR-PCR и SNP-маркеры; в то время как в древесине у видов хвойных растений, как показано для сосны обыкновенной и лиственницы сибирской, обычно присутствуют меньшее число фрагментов ДНК (молекулярных маркеров).
4. В прототипе не представлен способ молекулярно-генетической идентификации неизвестного образца древесины хвойных растений, так как прототип разработан на примере природных популяций лиственных древесных растений.
Раскрытие изобретения
Изобретение призвано решить задачу создания способа, который с высокой точностью устанавливает тождество различных объектов; а также предусматривает сравнение (тождество) испытуемых образцов с уже имеющимися изученными объектами (в данном случае с родовыми и видовыми идентификационными фрагментами ДНК), а также с сочетаниями фрагментов ДНК (молекулярных маркеров), характерными для природных популяций, выявленных в процессе предварительного маркирования.
Предварительно в соответствии с патентом (Патент РФ №2505956, МПК А01Н 1/00, опубл. 10.02.2014 в Бюл. №4. 10 с.) проведена процедура молекулярно-генетической идентификации, при которой ДНК выделялась из хвои деревьев из природных популяций тестируемых видов хвойных растений в регионе заготовки древесины.
Средства и методы для осуществления заявленного способа.
Способ молекулярно-генетической идентификации неизвестных образцов древесины включает в себя 6 стадий:
1 стадия - сбор неизвестных образцов древесины срубленных деревьев или бревен.
Методы стадии 1. Сбор неизвестных образцов древесины (кернов) у бревен производится в любое время года, исключая зимний период, в как минимум в двух случайно выбранных популяциях испытуемых видов хвойных растений в регионе исследований. В популяциях одного или нескольких видов хвойных растений образцы древесины собираются, в идеальном случае, с не менее, чем с 30 срубленных деревьев, а для вида - с не менее, чем 60 деревьев, но при этом не указывается популяция, из которой взяты образцы, то есть образцы являются анонимными; помещаются в стерильные пакеты и нумеруются по порядку арабскими цифрами. Осуществление способа в случае острой необходимости возможно и на меньшем числе образцов древесины.
Средства стадии 1. возрастной бурав; стерильные бумажные пакеты по числу образцов древесины; пинцеты; 70% спирт для стерилизации инструментов; перчатки; маркер для нумерации пакетов; сумка для хранения образцов древесины.
2 стадия - выделение проб ДНК из неизвестных образцов древесины. Методы стадии 2. Выделение тотальной ДНК по методике с использованием СТАВ (Cetyl Trimethyammonium Bromide) и PVPP (Polyvinylpolypyrrolidone), в которой оптимизирован процесс осаждения ДНК путем добавления трехмолярного ацетата натрия на этапе осаждения ДНК изопропанолом и инкубированием при -20°С в течении 30 минут. Измерение концентрации выделенной тотальной ДНК и разведение каждой пробы ДНК до концентрации 10 нг/мкл в ТЕ (Tris-EDTA)-буфере.
Средства стадии 2: вортекс-микроцентрифуга FVL-2400 BioSan; твердотельный термостат Т-48 Биоком; спектрофотометр Spectrofotometr™ NanoDrop 2000 («Thermo scientific», USA), термостат «Binder»; холодильник Hotpoint-Ariston HBM 1201.4.
3 стадия - проведение ISSR (Inter Simple Sequence Repeats)-анализа полиморфизма ДНК с использованием ПЦР (полимеразной цепной реакции) Методы стадии 3: ПЦР-анализ проб тотальной ДНК из неизвестных образцов древесины индивидуально с использованием 5 эффективными ранее установленных для каждого вида растений ISSR-праймерами; проведение электрофореза в агарозном геле; фотографирование гелей; повтор ПЦР.
Средства стадии 3: термоциклер для амплификации нуклеиновых кислот MyCycler (Bio-Rad); камеры для горизонтального электрофореза для анализа большого количества образцов Sub-Cell 192; бокс для ПЦР работ BioSan UV-Cleaner box-1200 (UVT-S-AR); набор микродозаторов с изменяемым объемом дозирования Pipetman (Gilson), система гель-документации Gel Doc XR (Bio-Rad); камеры для горизонтального электрофореза для анализа большого количества образцов Sub-Cell 192, маркер молекулярной массы (100 bp+1.5+3 Kb DNA Ladder; ООО «СибЭнзим-М», Москва).
4 стадия - отбор наиболее генетически гетерогенных тестируемых проб ДНК для использования в стадии 5.
Методы стадии 4: подсчет на электрофореграммах ISSR-спектров, полученных на стадии 3 с использованием в ПЦР отдельно у каждой амплифицированной пробы ДНК, общего числа и числа полиморфных фрагментов ДНК (ISSR-PCR маркеров); отбор не менее одной четвертой части (не менее 8 из 30) проб ДНК с наибольшим числом полиморфных фрагментов.
Средства стадии 4: программа Quantity One («Bio-Rad», USA) системы гель-документации Gel Doc XR (Bio-Rad); персональный компьютер.
5 стадия - определение (секвенирование) нуклеотидных последовательностей полиморфного (ых) локуса(ов) у каждого вида хвойных растений; полиморфные локусы и их число определяются при молекулярно-генетической идентификации деревьев в природных популяциях, проводимой до процедуры молекулярно-генетической идентификации неизвестных образцов древесины.
Методы стадии 5: Секвенцовая реакция амплификации последовательности ДНК от одного до трех полиморфных локусов у каждого вида хвойных растений в регионе исследований; детекция продуктов амплификации электрофорезом в 2% агарозном геле, ферментативная очистка продуктов ПЦР смесью ExoI и FAST-АР в отношении 0,5:1; для реакции секвенирования нуклеотидных последовательностей набор BigDye® Terminator v3.1 Cycle Sequencing Kit («Applied Biosystems», USA), секвенирование (определение нуклеотидной последовательности) в капиллярном генетическом анализаторе 3500 XL («Applied Biosystems», USA); секвенированные последовательности выравнивали и анализировали в программе Unipro UGENE.
Средства стадии 5: термоциклер Gene Amp PCR System 9700 («Applied Biosystems», USA); набор BigDye® XTerminator™ Purification Kit («Applied Biosystems», USA); система гель-документации Gel Doc XR («Bio-Rad», USA); 24-капиллярный генетический анализатор Genetic Analyzer 3500xL («Applied Biosystems», USA).
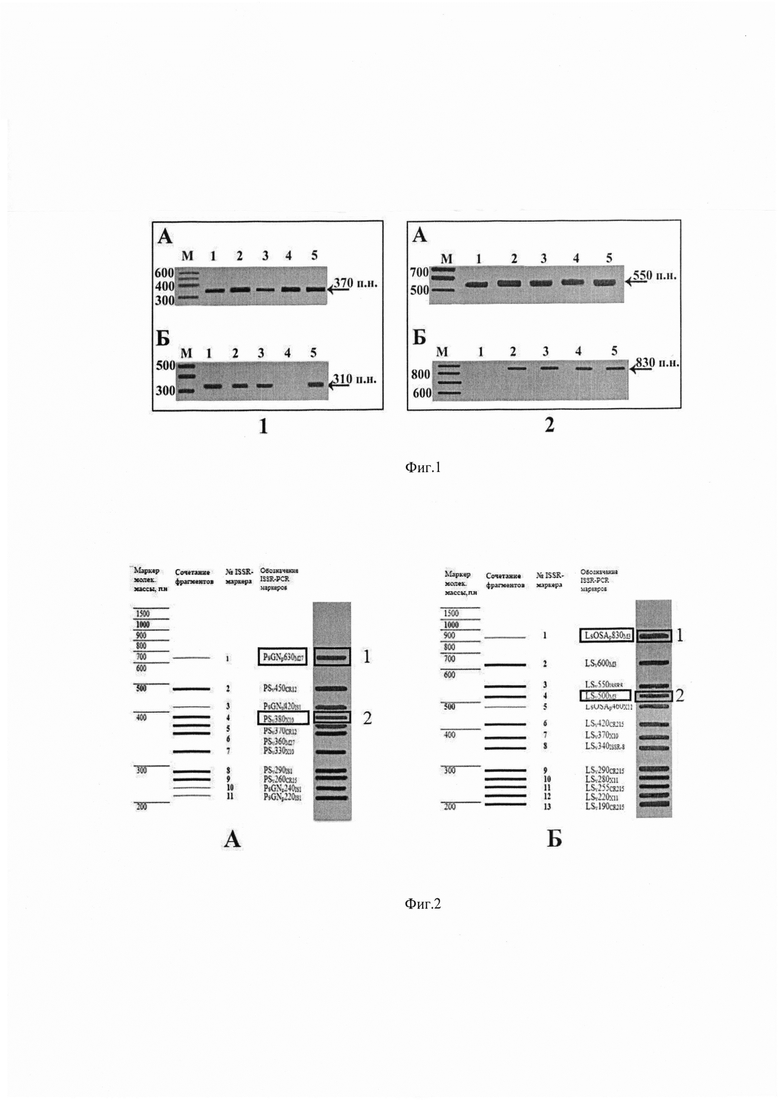
6 стадия - сопоставление с идентификационными родовыми, видовыми или сочетаниями популяционных фрагментов ДНК (фиг. 1), выявленными в процессе предварительного молекулярно-генетического анализа, фрагментов ДНК, полученных при амплификации неизвестных образцов древесины (фиг. 2).
Методы стадии 6: выявление в ISSR-спектрах и в нуклеотидных последовательностях, полученных при молекулярно-генетическом анализе тестируемых проб ДНК от неизвестных образцов древесины, характерных для рода (родовых) для вида (видовых) и сочетаний популяционных фрагментов ДНК (идентификационных ISSR-PCR, а у некоторых популяций и SNP-маркеров); сопоставление их с установленными ранее идентификационными фрагментами ДНК, характерными для природных популяциях тестируемых видов хвойных растений в регионе исследований.
Средства стадии 6: установленные ранее идентификационные фрагменты ДНК (ISSR-PCR и SNP-маркеры), характерные для природных популяциях тестируемых видов хвойных растений в регионе исследований; программы Quantity One («Bio-Rad», USA) системы гель-документации Gel Doc XR (Bio-Rad); Unipro UGENE; персональный компьютер.
Изобретение поясняется следующими фигурами.
Фиг. 1 - Идентификационные фрагменты ДНК природных популяций.
1 - Pinus sylvestris L.; 1А - видовой маркер PSv370CR12 на фореграмме популяции GN с праймером CR-212; 1Б - полиморфный маркер UKu310x10 популяции UK с праймером Х10;
2 - Larix sibirica Ledeb.; 2А - видовой маркер LSv550ISSR-8 на фореграмме популяции Osa с праймером ISSR-8; 2Б - полиморфный маркер Osau830M3 популяции Osa с праймером М3;
М - маркер молекулярного веса; 1-5 - номера проб ДНК, слева указан молекулярный вес фрагментов в п. н., стрелками обозначен идентификационный маркер
Фиг. 2 - Сравнение сочетания идентификационных фрагментов ДНК в популяции с фрагментами ДНК неизвестного образца древесины;
A - Pinus sulvestris L., популяция из Гаинского лесничества, GN; Б - Larix sibirica Ledeb., популяция из Осинского лесничества, Osa; 1 - полиморфный идентификационный фрагмент ДНК; 2 - видовой идентификационный фрагмент ДНК
Осуществление изобретения
Осуществление изобретения до состояния, в котором его функциональность может быть проверена, заключается в выполнении всех 6 стадий способа молекулярно-генетической идентификации неизвестных образцов древесины.
Пример 1. Использование способа молекулярно-генетической идентификации неизвестных образцов древесины P. sylvestris в регионе исследований.
На 1 стадии в летний период (июнь 2020 года) собраны образцы древесины (керны) в районе пос.Карагай Пермского края с двух штабелей бревен, один из которых расположенных в лесу, а второй штабель бревен - недалеко от пилорамы. Расстояние между штабелями около 600 м. Если бревна не ошкурены, то по коре срубленного дерева можно определить его принадлежность к роду. В данном случае определяется род сосна (Pinus). В каждом штабеле (выборке) с 30 разных бревен индивидуально в стерильные пакеты взяты образцы древесины. В связи с тем, что местонахождение природных популяций, из которых привезли бревна, неизвестно, образцы древесины являются неизвестными (анонимными) и нумеруются по порядку арабскими цифрами. На 2 стадии из каждого из 60 неизвестных образцов древесины из двух штабелей индивидуально выделены пробы тотальной ДНК по методике для выделения ДНК из древесины. Проведено измерение концентрации каждой выделенной пробы ДНК и ее разведение до концентрации 10 нг/мкл в ТЕ (Tris-EDTA)-буфере. На 3 стадии пробы ДНК из неизвестных образцов древесины амплифицированы в ПЦР с 5 эффективными ISSR-праймерами для P. sylvestris; проведены электрофорезы в агарозных гелях и их фотографирование. На 4 стадии из 60 проб ДНК двух выборок произведен отбор наиболее генетически гетерогенных 8 проб ДНК от каждой выборки на основании результатов ISSR-маркирования, проведенного на 3 стадии, по таким показателям как общее число и число полиморфных фрагментов ДНК. Полиморфные локусы и их число определяются при ранее проведенной молекулярно-генетической идентификации деревьев в природных популяциях. На 5 стадии у 16 проб ДНК из двух выборок проведена ПЦР для выявления целевых фрагментов изучаемых полиморфных локусов (trnV, rpl20-rps18, psbA-trnH), а также секвенцовая реакция и горизонтальный электрофорез в секвенаторе. Секвенированные нуклеотидные последовательности сохранены в формате FASTA.
На 6 стадии выявлены мономорфные ISSR-фрагменты с частотой, равной 1,000, соответствующие выявленным ранее видовым фрагментам P. sylvestris - PSv450CR212; PSv380X10; PSv370CR212; PSv360M27; PSv330X10; PSv290IS1; PSv260CR215.
Следовательно, можно сделать вывод, что тестируемые пробы ДНК неизвестных образцов древесины обеих выборок принадлежит именно виду сосна обыкновенная.
Проведено сравнение полиморфных фрагментов ДНК на популяционном уровне. На электрофореграмме неизвестного образца сосны обыкновенной с праймером Х10 присутствуют фрагменты 1120X10 (с частотой 0,634) и 200X10 (с частотой 0,566), которые встречаются только у выборки сосны обыкновенной, произрастающей на территории Большесосновского лесничества (BS). Кроме того, при выравнивании последовательностей ДНК неизвестного образца сосны обыкновенной с уже имеющимися последовательностями, были обнаружены SNP-маркеры, характерные для популяции сосны обыкновенной из Большесосновского лесничества (BS-trnV SNP358C/G; BS-trnH SNP289T/A; BS-rps18 SNP129A/T). Следовательно, данный неизвестный тестируемый образец древесины принадлежит выборке P. sylvestris с территории Большесосновского лесничества.
При сравнении второго неизвестного образца на электрофореграмме с праймерами CR-215 присутствуют фрагменты 900CR15 и 870CR15, с частотами 0,900 и 0,600, соответственно, которые встречаются только у популяции из Карагайского лесничества (KR). При выравнивании последовательностей ДНК у неизвестного образца так же обнаружились SNP-маркеры встречающиеся только у популяции из Карагайского лесничества (KR-trnH SNP402G/C; KR-trnV SNP207A/T). Следовательно, данный неизвестный тестируемый образец древесины принадлежит выборке из популяции KR из Карагайского лесничества.
Таким образом, при молекулярно-генетической идентификации неизвестных образцов древесины из штабелей около пос. Карагай подтверждено, что исследуемые неизвестные образцы древесины принадлежат виду сосна обыкновенная. Первый неизвестный образец древесины принадлежит бревну, срубленному в популяции этого вида в Большесосновском районе, а второй - в популяции из Карагайского лесничества Пермского края.
Пример 2. Использование способа молекулярно-генетической идентификации неизвестных образцов древесины L. sibirica на популяционном уровне в регионе исследований.
На 1 стадии в летний период (июль 2020 года) были собраны образцы древесины (керны) в районе пос.Гайны Пермского края с двух штабелей бревен. Расстояние между штабелями около 800 м. Если бревна не ошкурены, то по коре срубленного дерева можно определить его принадлежность к роду. В данном случае определяется род лиственница (Larix). В каждом штабеле (выборке) с 30 разных бревен индивидуально в стерильные пакеты взяты образцы древесины. В связи с тем, что местонахождение природных популяций, из которых привезли бревна, неизвестно, образцы древесины являются анонимными и нумеруются по порядку арабскими цифрами. На 2 стадии из каждого из 60 анонимных образцов древесины из двух штабелей индивидуально выделены пробы тотальной ДНК по методике для выделения ДНК из древесины. Проведено измерение концентрации каждой выделенной пробы ДНК и ее разведение до концентрации 10 нг/мкл в ТЕ (Tris-EDTA)-буфере. На 3 стадии пробы ДНК из неизвестных образцов древесины амплифицированы в ПЦР с 5 эффективными ISSR-праймерами для L. sibirica; проведены электрофорезы в агарозных гелях, их фотографирование. На 4 стадии из 60 проб ДНК двух выборок произведен отбор наиболее генетически гетерогенных 8 проб ДНК от каждой выборки на основании результатов ISSR-маркирования, проведенного на 3 стадии, по таким показателям как общее число и число полиморфных фрагментов ДНК. Полиморфные локусы и их число были определены при ранее проведенной молекулярно-генетической идентификации деревьев в природных популяциях. На 5 стадии у 16 проб ДНК из двух выборок проведена ПЦР для выявления целевых фрагментов изучаемых полиморфных локусов (4CL1-363, sSPcDFD040B03103-274), а также секвенцовая реакция и горизонтальный электрофорез в секвенаторе. Секвенированные нуклеотидные последовательности сохранены в формате FASTA.
На 6 стадии выявлены мономорфные ISSR-фрагменты с частотой, равной 1,000, соответствующие выявленным ранее видовым фрагментам L. sibirica - LSv420CR-215; LSv290CR-215; LSv255CR-215; LSv190CR-215; LSv550ISSR-8; LSv340ISSR-8; LSv600M3; LSv500M3; LSv370X10; LSv280X11; LSv220X11. Следовательно, можно сделать вывод, что тестируемая проба ДНК неизвестных образцов древесины обеих выборок принадлежит именно виду лиственница сибирская.
Проведено сравнение полиморфных ISSR-фрагментов на популяционном уровне. На электрофореграмме неизвестного образца лиственницы сибирской с праймером ISSR-8 присутствуют фрагменты 700IS8 (с частотой 0,523) и 460IS8 (с частотой 0,741), которые встречаются только у популяции лиственницы сибирской произрастающей на территории Чердынского лесничества (Che). При выравнивании секвенированных последовательностей ДНК неизвестного образца и ранее полученных последовательностей, выявлены SNP-маркер характерный для популяции лиственницы сибирской из Чердынского лесничества (Che-sSP SNP1185G/T). Следовательно, данный неизвестный тестируемый образец принадлежит выборке L. sibirica с территории Чердынского лесничества.
При сравнении второго неизвестного образца на электрофореграмме с праймерами X11 и Х10 присутствуют фрагменты 1240X11, 940X10 и 790X11, с частотами 0,811, 0,787 и 0,837, соответственно, которые встречаются только на уровне популяции из Гаинского лесничества (Gai). Выравнивание последовательности ДНК второго неизвестного образца, выявило наличие SNP-маркеров, встречающихся только в популяции из Гаинского лесничества (Gai-sSP Del269-271TCT; Gai-sSP SNP729C/T; Gai-4CL SNP722A/T). Следовательно, данный неизвестный тестируемый образец принадлежит выборке из популяции из Гаинского лесничества.
Таким образом, молекулярно-генетическая идентификация неизвестных образцов древесины из двух штабелей около пос. Гайны Пермского края подтвердила, что исследуемые анонимные образцы древесины принадлежат виду лиственница сибирская. Первый неизвестный образец древесины принадлежит бревну, срубленному в популяции этого вида в Чердынском районе, а второй - из популяции из Гайнского лесничества Пермского края.
Литература
1. Пальчиков СБ., Румянцев Д. Е. Контроль за законностью заготовки древесины на основе древесно-кольцевой информации // Устойчивое лесопользование. 2009. №2 (21). С. 13-16.
2. Хох А.Н. Подтверждение задекларированного места произрастания срубленной древесины сосны исследованием параметров радиального прироста при проведении судебно-ботанических экспертиз // Вопросы криминологии, криминалистики и судебной экспертизы. 2019. №1 (45). С. 149-154.
3. Шейкина О.В., Прохорова А.А., Новиков П.С, Криворотова Т.Н. Разработка методики идентификации клонов плюсовых деревьев ели обыкновенной (Picea abies L.) с использованием ISSR маркеров // Политематический сетевой электронный научный журнал кубанского государственного аграрного университета. 2012. №83. С. 56-69.
4. Рамазанова С.А., Гучетль С.З., Челюстникова Т.А., Антонова Т.С Идентификация сортов сои российской селекции на основе анализа микросателлитных (SSR) локусов ДНК. Масличные культуры // Научно-технический бюллетень Всероссийского научно-исследовательского института масличных культур. 2008. №2 (139). С. 56-58.
5. Nowakowska J.A. Application of DNA markers against illegal logging as a new tool for the Forest Guard Service // Folia Forestalia Polonica. Series A. Forestry. 2011. T. 53. №2. P. 142-149.
6. Крутовский K.B., Путинцева Ю.А., Орешкова H.B., Бондар Е.И., Шаров В.В., Кузьмин Д.А. Постгеномные технологии в практическом лесном хозяйстве: разработка полногеномных маркеров для идентификации происхождения древесины и других задач // Лесотехнический журнал. 2019. Т. 9. №1 (33). С. 9-16.
7. Jolivet С., Degen В. Use of DNA fingerprints to control the origin of sapelli timber (Entandrophragma cylindricum) at the forest concession level in Cameroon // Forensic Science International: Genetics. 2012. Vol. 6. №4. P. 487-493.
8. Degen В., Ward S.E., Lemes M.R., Navarro C, Cavers S., Sebbenn A.M. Verifying the geographic origin of mahogany (Swietenia macrophylla King) with DNA-fingerprints // Forensic Science International: Genetics. 2013. Vol. 7. №1. P. 55-66.
9. Blanc-Jolivet C, Liesebach M. Tracing the origin and species identity of Quercus robur and Quercuspetraea in Europe: a review // Silvae Genetica. 2015. Vol. 64. №4. P. 182-193.
10. Jardine D.I., Blanc-Jolivet С, M. Dixon R.R., Dormontt E.E, Dunker В., Gerlach J., Kersten В., van Dijk K.-J, Degen В., Lowe A.J. Development of SNP markers for Ayous (Triplochiton scleroxylon K. Schum) an economically important tree species from tropical West and Central Africa // Conservation Genet Resour. 2016. №8. P. 129-139.
11. Blanc-Jolivet C, Yanbaev Yu., Kersten В., Degen B. A set of SNP markers for timber tracking of Larix spp. in Europe and Russia // Forestry: An International Journal of Forest Research. 2018. Vol. 91. №5. P. 614-628.
12. Overview of current practices in data analysis for wood identification. A guide for the different timber tracking methods / Schmitz, N., ed. GTTN, Global Timber Tracking Network: GTTN Secretariat: European Forest Institute and Thunen Institute. 2020. 143 p.
13. Способ молекулярно-генетической идентификации популяций древесных видов растений» С.В. Боронникова, И.В. Бобошина. Патент РФ №2505956, МПК А01Н 1/00, опубл. 10.02.2014 в Бюл. №4. 10 с.
14. Боронникова С/В. Молекулярно-генетическая идентификация и паспортизация редких и находящихся под угрозой уничтожения видов растений / Перм. ун-т. Пермь, 2008. 120 с.
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ молекулярно-генетической паспортизации древесины популяций ели сибирской для определения ее географического происхождения | 2023 |
|
RU2825802C1 |
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ИДЕНТИФИКАЦИИ ПОПУЛЯЦИЙ ДРЕВЕСНЫХ ВИДОВ РАСТЕНИЙ | 2012 |
|
RU2505956C2 |
| СПОСОБ МОЛЕКУЛЯРНОГО МАРКИРОВАНИЯ, ОСНОВАННЫЙ НА МИКРОСАТЕЛЛИТНЫХ ЛОКУСАХ, ПРЕДНАЗНАЧЕННЫЙ ДЛЯ ИДЕНТИФИКАЦИИ ГЕНОТИПОВ ИВЫ | 2016 |
|
RU2630662C1 |
| СПОСОБ МОЛЕКУЛЯРНОГО МАРКИРОВАНИЯ, ОСНОВАННЫЙ НА МИКРОСАТЕЛЛИТНЫХ ЛОКУСАХ, ПРЕДНАЗНАЧЕННЫЙ ДЛЯ ИДЕНТИФИКАЦИИ ГЕНОТИПОВ БЕРЕЗЫ | 2016 |
|
RU2646112C1 |
| BpCW1 - молекулярный маркер для выявления генотипов карельской березы на ранних стадиях развития | 2023 |
|
RU2823809C1 |
| БИОЛОГИЧЕСКИЙ ДНК МАРКЕР ДЛЯ ОПРЕДЕЛЕНИЯ СОРТОВ КАРТОФЕЛЯ, НАБОР И СПОСОБ СОРТОВОЙ ИДЕНТИФИКАЦИИ КАРТОФЕЛЯ | 2009 |
|
RU2413774C1 |
| ДНК-маркер для селекции гибридов сорго на основе цитоплазматической мужской стерильности А1-типа | 2021 |
|
RU2792135C1 |
| СПОСОБ ДИФФЕРЕНЦИАЛЬНОЙ ДНК-ДИАГНОСТИКИ НА РАЗНЫХ СТАДИЯХ ЖИЗНЕННОГО ЦИКЛА ГЕЛЬМИНТОВ-ВОЗБУДИТЕЛЕЙ ФАСЦИОЛЕЗА ЧЕЛОВЕКА И ЖИВОТНЫХ | 2007 |
|
RU2360003C1 |
| Способ получения молекулярных однонуклеотидных маркеров для идентификации неизвестного индивида методом мультиплексной амплификации для работы с образцами деградированной ДНК, набор для получения молекулярных маркеров | 2021 |
|
RU2800083C2 |
| СПОСОБ ДНК-ИДЕНТИФИКАЦИИ И ГЕНЕТИЧЕСКОЙ ПАСПОРТИЗАЦИИ СОРТОВ РАЙГРАСА ПАСТБИЩНОГО И ОДНОЛЕТНЕГО НА ОСНОВЕ СИСТЕМ SSR- и SCoT-МАРКИРОВАНИЯ | 2023 |
|
RU2826148C1 |
Изобретение относится к области биотехнологии, в частности к способу молекулярно-генетической идентификации неизвестных образцов древесины у видов хвойных растений, при использовании в ПЦР проб ДНК, выделенных непосредственно из древесины бревен в месте ее заготовки или после транспортировки. Способ включает в себя 6 стадий: сбор неизвестных образцов древесины; выделение из них проб ДНК; проведение ISSR (Inter Simple Sequence Repeats)-анализа полиморфизма ДНК с использованием ПЦР; отбор наиболее генетически гетерогенных тестируемых проб ДНК; определение (секвенирование) нуклеотидных последовательностей от одного до трех полиморфных локусов у каждого тестируемого вида хвойных растений в регионе исследований; выявление у тестируемых образцов древесины фрагментов ДНК, характерных для рода (родовых), для вида (видовых) и сочетаний полиморфных популяционных ISSR-PCR, а у некоторых популяций и SNP-маркеров; сопоставление (определение тождества) выявленных фрагментов ДНК с установленными ранее идентификационными фрагментами ДНК для природных популяций тестируемых видов в регионе исследований. Изобретение позволяет эффективно идентифицировать неизвестные образцы древесины у видов хвойных растений и установить соответствие или его отсутствие фрагментов ДНК срубленных деревьев идентификационным фрагментам ДНК деревьев, произрастающих в природной популяции вида в регионе исследований. Изобретение позволяет с высокой точностью устанавливать тождество объектов предусматривает сравнение (тождество) испытуемых образцов, то сеть фрагментов ДНК, амплифицированных с пробами ДНК из древесины, с уже имеющимися изученными объектами, в данном случае с родовыми, видовыми и сочетаниями популяционных идентификационных фрагментов ДНК, выявленными в процессе предварительного молекулярно-генетического анализа природных популяций хвойных растений в регионе исследований. 2 з.п. ф-лы, 2 ил., 2 пр.
1. Способ молекулярно-генетической идентификации неизвестных образцов древесины Larix sibirica Ledeb. и Pinus sylvestris L., включающий сбор образцов древесины в местах ее заготовки или после транспортировки бревен; выделение из них проб ДНК; проведение ISSR (Inter Simple Sequence Rереаts)-анализа полиморфизма ДНК с использованием ПЦР; отбор наиболее генетически гетерогенных тестируемых проб ДНК с использованием программы для определения длин фрагментов ДНК; определение (секвенирование) нуклеотидных последовательностей от одного до трех полиморфных локусов у каждого вида хвойных растений в регионе исследований; выявление характерных для рода (родовых), для вида (видовых) и сочетаний популяционных ISSR-PCR, а у некоторых популяций и SNP (Single Nucleotide Polymorphism)-маркеров; их сопоставление с установленными ранее аналогичными идентификационными фрагментами ДНК (молекулярными маркерами), характерными для природных популяций тестируемых видов в регионе исследований.
2. Способ по п. 1, отличающийся тем, что неизвестные образцы древесины собираются не в природных популяциях, в которых растут деревья, а после заготовки древесины и, возможно, транспортировки.
3. Способ по п. 1, отличающийся тем, что пробы ДНК выделяют непосредственно из образцов древесины, которая в своем составе в отличие от хвои имеет мертвые ткани.
| СПОСОБ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОЙ ИДЕНТИФИКАЦИИ ПОПУЛЯЦИЙ ДРЕВЕСНЫХ ВИДОВ РАСТЕНИЙ | 2012 |
|
RU2505956C2 |
| BLANC-JOLIVET C, et al | |||
| Tracing the origin and species identity of Quercus robur and Quercuspetraea in Europe: a review, Silvae Genetica | |||
| Устройство для закрепления лыж на раме мотоциклов и велосипедов взамен переднего колеса | 1924 |
|
SU2015A1 |
| Vol | |||
| Нефтяной конвертер | 1922 |
|
SU64A1 |
| Очаг для массовой варки пищи, выпечки хлеба и кипячения воды | 1921 |
|
SU4A1 |
| Затвор для дверей холодильных камер | 1920 |
|
SU182A1 |
| NOWAKOWSKA J.A | |||
| Application of DNA markers against illegal logging as a new tool for the Forest Guard Service // Folia Forestalia Polonica | |||
| Series A | |||
Авторы
Даты
2021-11-26—Публикация
2020-12-09—Подача