Ссылка на родственные заявки
[0001] Согласно настоящей заявке испрашивается приоритет в соответствии с заявкой на выдачу патента США №62/449335, поданной 23 января 2017 года, заявкой на выдачу патента США №62/472972, поданной 17 марта 2017 года, и заявкой на выдачу патента США №62/581918, поданной 6 ноября 2017 года, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
Ссылка на перечень последовательностей, поданный в виде текстового файла через веб-систему электронной подачи
[0002] Перечень последовательностей, записанный в файле 507242SEQLIST.txt, имеет размер 507 килобайт, был создан 19 января 2018 года и настоящим включен посредством ссылки.
Предшествующий уровень техники настоящего изобретения
[0003] Хроническое заболевание печени и цирроз являются основными причинами заболеваемости и смертности в Соединенных Штатах, на которые пришлось 38170 смертей (1,5% от общего числа смертей) в 2014 году (Kochanek et al. (2016) Natl Vital Stat Rep 65:1-122, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Наиболее распространенной этиологией цирроза в США являются алкогольная болезнь печени, хронический гепатит С и неалкогольная жировая болезнь печени (NAFLD), на долю которых приходится ~80% пациентов, ожидавших трансплантации печени в период между 2004 и 2013 годами (Wong et al. (2015) Gastroenterology 148:547-555, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Оценочная доля NAFLD в США составляет от 19 до 46 процентов (Browning et al. (2004) Hepatology 40:1387-1395; Lazo et al. (2013) Am J Epidemiol 178:38-45; и Williams et al. (2011) Gastroenterology 140:124-131, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) и растет со временем (Younossi et al. (2011) Clin Gastroenterol Hepatol 9:524-530 el; quiz e60 (2011), включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), вероятно, в сочетании с повышенным уровнем ожирения, основным ее фактором риска (Cohen et al. (2011) Science 332:1519-1523, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Несмотря на значительные успехи в лечении гепатита С (Morgan et al. (2013) Ann Intern Med 158:329-337 и van der Meer et al. (2012) JAMA 308:2584-2593, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), в настоящее время не существует научно обоснованных способов лечения алкогольной или неалкогольной болезни печени и цирроза.
[0004] Предыдущие полногеномные поиски ассоциаций (GWAS) выявили ограниченное количество генов и вариантов, ассоциируемых с хроническим заболеванием печени. На сегодняшний день наиболее достоверно подтвержденная генетическая ассоциация обнаружена для распространенного миссенс-варианта гена белка 3, содержащего пататин-подобный фосфолипазный домен (PNPLA3 p.Ile148Met, rs738409), для которого ранее было обнаружено, что он ассоциирован с повышенным риском развития неалкогольной жировой болезни печени (NAFLD) (Romeo et al. (2008) Nat. Genet. 40:1461-1465 и Speliotes et al. (2011) PLoS Genet. 7:e1001324, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), и впоследствии было обнаружено, что он ассоциирован со степенью тяжести данного заболевания (Rotman et al. (2010) Hepatology 52:894-903 и Sookoian et al. (2009) J. Lipid Res. 50:2111-2116, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) и прогрессированием данного заболевания (Trepo et al. (2016) J. Hepatol. doi:10.1016/j.jhep.2016.03.011, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Также было показано, что изменение в гене белка 2, который является представителем суперсемейства 6 трансмебранных белков (TM6SF2), повышает риск развития NAFLD (Kozlitina et al. (2014) Nat. Genet. 46:352-356; Liu et al. (2014) Nat. Commun. 5:4309; и Sookoian et al. (2015) Hepatology 61:515-525, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Нормальные функции этих двух белков не совсем ясны, хотя было выдвинуто предположение, что оба они участвуют в метаболизме липидов у гепатоцитов. Как варианты PNPLA3 и TM6SF2 способствуют увеличению риска развития заболевания печени, все еще предстоит выяснить. В GWAS также было выявлено несколько генетических факторов, ассоциированных с сывороточной аланинаминотрансферазой (ALT) и аспартатаминотрансферазой (AST) (Chambers et al. (2011) Nat. Genet. 43:131-1138 и Yuan et al. (2008) Am. J. Hum. Genet. 83:520-528, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), количественных показателей повреждения гепатоцитов и накопления жира в печени, которые зачастую измеряют в клинических ситуациях. На сегодняшний день нет описанных генетических вариантов, защитных при хроническом заболевании печени. Обнаружение генетических вариантов, являющихся защитными при других состояниях, таких как варианты с потерей функции PCSK9, которые снижают риск развития сердечнососудистого заболевания, послужило катализатором для разработки новых классов терапевтических средств.
[0005] Знание генетических факторов, лежащих в основе развития и прогрессирования хронического заболевания печени, может улучшить стратификацию на группы риска и дать основу для новых терапевтических стратегий. Лучшее понимание лежащих в основе генетических факторов необходимо для улучшения стратификации на группы риска и создания новых способов терапии заболеваний печени.
Краткое раскрытие настоящего изобретения
[0006] Настоящее изобретение относится к способам и композициям, имеющим отношение к варианту rs72613567 гена HSD17B13, вариантам транскрипта HSD17B13 и вариантам изоформ белка HSD17B13.
[0007] Согласно одному аспекту, настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим мутантный остаток из варианта rs72613567 гена HSD17B13. Такие выделенные нуклеиновые кислоты могут содержать по меньшей мере 15 смежных нуклеотидов гена HSD17B13 и содержать тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании с SEQ ID NO: 1. Необязательно, смежные нуклеотиды по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%о или по меньшей мере на 99% идентичны соответствующей последовательности в SEQ ID NO: 2, включающей положение 12666 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. Необязательно, ген HSD17B13 представляет собой ген HSD17B13 человека. Необязательно, выделенная нуклеиновая кислота содержит по меньшей мере 15, по меньшей мере 20, по меньшей мере 25, по меньшей мере 30, по меньшей мере 35, по меньшей мере 40, по меньшей мере 45, по меньшей мере 50, по меньшей мере 60, по меньшей мере 70, по меньшей мере 80, по меньшей мере 90, по меньшей мере 100, по меньшей мере 200, по меньшей мере 300, по меньшей мере 400, по меньшей мере 500, по меньшей мере 600, по меньшей мере 700, по меньшей мере 800, по меньшей мере 900, по меньшей мере 1000, по меньшей мере 2000, по меньшей мере 3000, по меньшей мере 4000, по меньшей мере 5000, по меньшей мере 6000, по меньшей мере 7000, по меньшей мере 8000, по меньшей мере 9000, по меньшей мере 10000, по меньшей мере 11000, по меньшей мере 12000, по меньшей мере 13000, по меньшей мере 14000, по меньшей мере 15000, по меньшей мере 16000, по меньшей мере 17000, по меньшей мере 18000 или по меньшей мере 19000 смежных нуклеотидов из SEQ ID NO: 2.
[0008] Некоторые такие выделенные нуклеиновые кислоты содержат миниген HSD17B13, в котором были делетерованы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа. Необязательно, делетерованные сегменты содержат одну или несколько интронных последовательностей. Необязательно, выделенная нуклеиновая кислота дополнительно содержит интрон, соответствующий интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. Необязательно, интроном является интрон 6 из SEQ ID NO: 2.
[0009] Согласно другому аспекту, настоящее изобретение относится к выделенным нуклеиновым кислотам, соответствующим различным мРНК-транскриптам или кДНК HSD17B13. Некоторые такие выделенные нуклеиновые кислоты содержат по меньшей мере 15 смежных нуклеотидов, кодирующих весь или часть белка HSD17B13, причем смежные нуклеотиды составляют сегмент, который по меньшей мере на 90%, по меньшей мере на 95%), по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99%) или на 100%) идентичен сегменту, присутствующему в SEQ ID NO: 7 (транскрипте D HSD17B13), SEQ ID NO: 10 (транскрипте G HSD17B13) и SEQ ID NO: 11 (транскрипте Н HSD17B13) и отсутствующему в SEQ ID NO: 4 (транскрипте A HSD17B13). Необязательно, смежные нуклеотиды дополнительно содержат сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%), по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 7 (транскрипте D HSD17B13) и отсутствующему в SEQ ID NO: 11 (транскрипте Н HSD17B13), и при этом смежные нуклеотиды дополнительно содержат сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 7 (транскрипте D HSD17B13) и отсутствующему в SEQ ID NO: 10 (транскрипте G HSD17B13). Необязательно, смежные нуклеотиды дополнительно содержат сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 11 (транскрипте H HSD17B13) и отсутствующему в SEQ ID NO: 7 (транскрипте D HSD17B13). Необязательно, смежные нуклеотиды дополнительно содержат сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 10 (транскрипте G HSD17B13) и отсутствующему в SEQ ID NO: 7 (транскрипте D HSD17B13).
[0010] Некоторые такие выделенные нуклеиновые кислоты содержат по меньшей мере 15 смежных нуклеотидов, кодирующих весь или часть белка HSD17B13, причем смежные нуклеотиды составляют сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 8 (транскрипте Е HSD17B13) и отсутствующему в SEQ ID NO: 4 (транскрипте A HSD17B13). Необязательно, смежные нуклеотиды дополнительно содержат сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 8 (транскрипте Е HSD17B13) и отсутствующему в SEQ ID NO: 11 (транскрипте Н HSD17B13)
[0011] Некоторые такие выделенные нуклеиновые кислоты содержат по меньшей мере 15 смежных нуклеотидов, кодирующих весь или часть белка HSD17B13, причем смежные нуклеотиды составляют сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 9 (транскрипте F HSD17B13) и отсутствующему в SEQ ID NO: 4 (транскрипте A HSD17B13).
[0012] Некоторые такие выделенные нуклеиновые кислоты содержат по меньшей мере 15 смежных нуклеотидов, кодирующих весь или часть белка HSD17B13, причем смежные нуклеотиды составляют сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 6 (транскрипте С HSD17B13) и отсутствующему в SEQ ID NO: 4 (транскрипте A HSD17B13).
[0013] Необязательно, белок HSD17B13 представляет собой 6enoKHSD17B13 человека. Необязательно, выделенная нуклеиновая кислота содержит по меньшей мере 20, по меньшей мере 25, по меньшей мере 30, по меньшей мере 35, по меньшей мере 40, по меньшей мере 45, по меньшей мере 50, по меньшей мере 60, по меньшей мере 70, по меньшей мере 80, по меньшей мере 90, по меньшей мере 100, по меньшей мере 200, по меньшей мере 300, по меньшей мере 400, по меньшей мере 500, по меньшей мере 600, по меньшей мере 700, по меньшей мере 800, по меньшей мере 900, по меньшей мере 1000 или по меньшей мере 2000 смежных нуклеотидов, кодирующих весь или часть белка HSD17B13.
[0014] Некоторые такие выделенные нуклеиновые кислоты содержат последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична последовательности, изложенной под SEQ ID NO: 6, 7, 8, 9, 10 или 11 (транскрипт С, D, Е, F, G или Н HSD17B13) и кодирует белок HSD17B13, содержащий последовательность, изложенную соответственно под SEQ ID NO: 14, 15, 16, 17, 18 или 19 (изоформа С, D, Е, F, G или Н HSD17B13).
[0015] В любой из вышеуказанных нуклеиновых кислот смежные нуклеотиды могут необязательно содержать последовательность по меньшей мере из двух различных экзонов гена HSD17B13 без промежуточного интрона.
[0016] Согласно другому аспекту, настоящее изобретение относится к белкам, кодируемым любой из вышеуказанных выделенных нуклеиновых кислот.
[0017] Согласно другому аспекту, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются с мутантным остатком из варианта rs72613567 гена HSD17B13 или возле него. Такие выделенные нуклеиновые кислоты могут содержать по меньшей мере 15 смежных нуклеотидов, которые гибридизируются с геном HSD17B13 в сегменте, который включает или находится в пределах 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов положения, соответствующего положению 12666 в SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. Необязательно, сегменты по меньшей мере на 90%, по меньшей мере на 95%), по меньшей мере на 96%), по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны соответствующей последовательности в SEQ ID NO: SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. Необязательно, сегмент содержит по меньшей мере 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000 или 2000 смежных нуклеотидов из SEQ ID NO: 2. Необязательно, сегмент включает положение 12666 в SEQ ID NO: 2 или положение, соответствующего положению 12666 в SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. Необязательно, ген HSD17B13 представляет собой ген HSD17B13 человека. Необязательно, выделенная нуклеиновая кислота составляет до приблизительно 30, 40, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 нуклеотидов в длину. Необязательно, выделенная нуклеиновая кислота связана с гетерологичной нуклеиновой кислотой или содержит гетерологичную метку. Необязательно, гетерологичная метка представляет собой флуоресцентную метку.
[0018] Согласно другому аспекту, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются с различными мРНК-транскриптами или кДНК HSD17B13. Некоторые такие выделенные нуклеиновые кислоты гибридизируются по меньшей мере с 15 смежными нуклеотидами нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды составляют сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, присутствующему в SEQ ID NO: 7 (транскрипте D HSD17B13), SEQ ID NO: 10 (транскрипте G HSD17B13) и SEQ ID NO: 11 (транскрипте Н HSD17B13) и отсутствующему в SEQ ID NO: 4 (транскрипте А HSD17B13).
[0019] Некоторые такие выделенные нуклеиновые кислоты гибридизируются по меньшей мере с 15 смежными нуклеотидами нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды составляют сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или на 99% идентичен сегменту, присутствующему в SEQ ID NO: 8 (транскрипте Е HSD17B13) и SEQ ID NO: 11 (транскрипте Н HSD17B13) и отсутствующему в SEQ ID NO: 4 (транскрипте A HSD17B13).
[0020] Некоторые такие выделенные нуклеиновые кислоты гибридизируются по меньшей мере с 15 смежными нуклеотидами нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды составляют сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или на 99% идентичен сегменту в SEQ ID NO: 9 (транскрипте F HSD17B13), которого нет в SEQ ID NO: 4 (транскрипте A HSD17B13).
[0021] Некоторые такие выделенные нуклеиновые кислоты гибридизируются по меньшей мере с 15 смежными нуклеотидами нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды составляют сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или на 99% идентичен сегменту, присутствующему в SEQ ID NO: 6 (транскрипте С HSD17B13) и отсутствующему в SEQ ID NO: 4 (транскрипте A HSD17B13).
[0022] Необязательно, белок HSD17B13 представляет собой белок HSD17B13 человека. Необязательно, выделенная нуклеиновая кислота составляет до приблизительно 30, 40, 50, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 нуклеотидов в длину. Необязательно, выделенная нуклеиновая кислота связана с гетер о логичной нуклеиновой кислотой или содержит гетерологичную метку. Необязательно, гетерологичная метка представляет собой флуоресцентную метку.
[0023] Необязательно, любая из вышеуказанных выделенных нуклеиновых кислот содержит ДНК. Необязательно, любая из вышеуказанных выделенных нуклеиновых кислот содержит РНК. Необязательно, любая из вышеуказанных выделенных нуклеиновых кислот является антисмысловой РНК, короткой шпилечной РНК или малой интерферирующей РНК. Необязательно, любая из вышеуказанных выделенных нуклеиновых кислот может включать не встречающийся в естественной среде нуклеотид.
[0024] Согласно другому аспекту, настоящее изобретение относится к векторам и экзогенным донорным последовательностям, содержащим любую из вышеуказанных выделенных нуклеиновых кислот и гетерологичную последовательность нуклеиновой кислоты.
[0025] Согласно другому аспекту, настоящее изобретение относится к применению любой из вышеуказанных выделенных нуклеиновых кислот, векторов или экзогенных донорных последовательностей в способе детекции варианта rs72613567 HSD17B13 у субъекта, способу выявления наличия транскрипта С, D, Е, F, G или HHSD17B13 у субъекта, способе определения склонности субъекта к развитию хронического заболевания печени, способе диагностики субъекта с жировой болезнью печени или способе модификации гена HSD17B13 в клетке, способе изменения экспрессии гена HSD17B13 в клетке.
[0026] Согласно другому аспекту, настоящее изобретение относится к гидовым РНК, которые нацелены на ген HSD17B13. Такие гидовые РНК могут быть эффективны для направления фермента Cas для связывания или расщепления гена HSD17B13, при этом гидовая РНК содержит нацеливающий на ДНК сегмент, который гибридизируется с последовательностью распознания для гидовой РНК в гене HSD17B13. Говоря иными словами, такие гидовые РНК могут быть эффективными для направления фермента Cas для связывания или расщепления гена HSD17B13, при этом гидовая РНК содержит нацеливающий на ДНК сегмент, который нацеливает на целевую для гидовой РНК последовательность в гене HSD17B13. Такие гидовые РНК могут быть эффективными для направления фермента Cas для связывания или расщепления гена HSD17B13, при этом гидовая РНК содержит нацеливающий на ДНК сегмент, который нацеливает на целевую для гидовой РНК последовательность в гене HSD17B13, который включает или находится в непосредственной близости к положению, соответствующему положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность содержит, фактически состоит из или состоит из любой из SEQ ID NO: 226-239 и 264-268. Необязательно, нацеливающий на ДНК сегмент содержит, фактически состоит из или состоит из любой из SEQ ID NO: 1629-1642 и 1648-1652. Необязательно, гидовая РНК содержит, фактически состоит из или состоит из любой из SEQ ID NO: 706-719; 936-949; 1166-1179, 1396-1409, 725-729, 955-959, 1185-1189 и 1415-1419. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 226-239 или SEQ ID NO: 230 и 231. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 226-230 и 264-268. Необязательно, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 6 и/или интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 6, и/или интрону 6, и/или экзону 7 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность находится в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от положения, соответствующего положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность включает положение, соответствующее положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2.
[0027] Такие гидовые РНК могут быть эффективными для направления фермента Cas для связывания или расщепления гена HSD17B13, при этом гидовая РНК содержит нацеливающий на ДНК сегмент, который нацеливает целевую для гидовой РНК последовательность в гене HSD17B13, который включает или находится в непосредственной близости к старт-кодону гена HSD17B13. Необязательно, целевая для гидовой РНК последовательность содержит, фактически состоит из или состоит из любой из SEQ ID NO: 20-81 и 259-263. Необязательно, нацеливающий на ДНК сегмент содержит, фактически состоит из или состоит из любой из SEQ ID NO: 1423-1484 и 1643-1647. Необязательно, гидовая РНК содержит, фактически состоит из или состоит из любой из SEQ ID NO: 500-561, 730-791, 960-1021, 1190-1251,720-724, 950-954, 1180-1184 и 1410-1414. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 20-81 и 259-263. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 21-23, 33 и 35. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 33 и 35. Необязательно, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 1 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность находится в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от старт-кодона.
[0028] Такие гидовые РНК могут быть эффективными для направления фермента Cas для связывания или расщепления гена HSD17B13, при этом гидовая РНК содержит нацеливающий на ДНК сегмент, который нацеливает целевую для гидовой РНК последовательность в гене HSD17B13, который включает или находится в непосредственной близости к стоп-кодону гена HSD17B13. Необязательно, целевая для гидовой РНК последовательность содержит, фактически состоит из или состоит из любой из SEQ ID NO: 82-225. Необязательно, нацеливающий на ДНК сегмент содержит, фактически состоит из или состоит из любой из SEQ ID NO: 1485-1628. Необязательно, гидовая РНК содержит, фактически состоит из или состоит из любой из SEQ ID NO: 562-705, 792-935, 1022-1165 и 1252-1395. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 82-225. Необязательно, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 7 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность находится в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от стоп-кодона.
[0029] Необязательно, ген HSD17B13 представляет собой ген HSD17B13 человека. Необязательно, ген HSD17B13 содержит SEQ TD NO: 2.
[0030] Некоторые такие гидовые РНК содержат короткие полиндромные повторы, регулярно расположенными группами (CRISPR) (crRNA), содержащую нацеливающий на ДНК сегмент и транс-активирующую РНК CRISPR (tracrRNA). Необязательно, гидовая РНК представляет собой модульную гидовую РНК, в которой crRNA и tracrRNA являются отдельными молекулами, которые гибридизируются друг с другом. Необязательно, crRNA содержит, фактически состоит из или состоит из последовательности, изложенной под SEQ ID NO: 1421, и tracrRNA содержит, фактически состоит из или состоит из последовательности, изложенной под SEQ ID NO: 1422. Необязательно, гидовая РНК является одиночной гидовой РНК, в которой crRNA слита с tracrRNA посредством линкера. Необязательно, одиночная гидовая РНК содержит состоит фактически из или состоит из последовательности, изложенной под любым из SEQ ID NO: 1420 и 256-258.
[0031] Согласно другому аспекту, настоящее изобретение относится к антисмысловым РНК, siRNA или shRNA, которые гибридизируются с последовательностью в пределах раскрываемого в настоящем документе транскрипта HSD17B13. Некоторые такие антисмысловые РНК, siRNA или shRNA гибридизируются с последовательностью в пределах SEQ ID NO: 4 (транскрипта A HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA может уменьшать экспрессию транскрипта A HSD17B13 в клетке. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 4 (транскрипте AHSD17B13) и отсутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 4 (транскрипта А HSD17B13). Некоторые такие антисмысловые РНК, siRNA или shRNA гибридизируются с последовательностью в пределах SEQ ID NO: 7 (транскрипта D HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA может уменьшать экспрессию транскрипта D HSD17B13 в клетке. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13) и отсутствующей в SEQ ID NO: 4 (транскрипте A HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 7 (транскрипта D HSD17B13).
[0032] Согласно другому аспекту, настоящее изобретение относится к ДНК, кодирующим любую из вышеуказанных гидовых РНК, антисмысловых РНК, siRNA или shRNA. Согласно другому аспекту, настоящее изобретение относится к векторам, содержащим ДНК, кодирующую любую из вышеуказанных гидовых РНК, антисмысловых РНК, siRNA или shRNA, и гетерологичную нуклеиновую кислоту. Согласно другому аспекту, настоящее изобретение относится к применению любой из вышеуказанных гидовых РНК, антисмысловых РНК, siRNA или shRNA, молекул ДНК, кодирующих гидовые РНК, антисмысловые РНК, siRNA или shRNA, или векторов, содержащих ДНК, кодирующие гидовые РНК, антисмысловые РНК, siRNA или shRNA, в способе модификации гена HSD17B13 в клетке или в способе изменения экспрессии гена HSD17B13 в клетке.
[0033] Согласно другому аспекту, настоящее изобретение относится к композициям, содержащим любую из вышеуказанных выделенных нуклеиновых кислот, любую из вышеуказанных гидовых РНК, любой из вышеуказанных выделенных полипептидов, любую из вышеуказанных антисмысловых РНК, siRNA или shRNA, любой из вышеуказанных векторов или любую из вышеуказанных экзогенных донорных последовательностей. Необязательно, композиция содержит любую из вышеуказанных гидовых РНК и белок Cas, такой как белок Cas9. Необязательно, такие композиции содержат носитель, повышающий стабильность выделенного полипептида, гидовой РНК, антисмысловой РНК, siRNA, shRNA, выделенной нуклеиновой кислоты, вектора или экзогенной донорной последовательности. Необязательно, носитель содержит микросферу из полимолочной кислоты (PLA), микросферу из сополимера D,L-молочной и гликолевой кислот (PLGA), липосому, мицеллу, обратную мицеллу, липидный кохлеат или липидную микротрубочку.
[0034] Также настоящее изобретение относится к клеткам, содержащим любую из вышеуказанных выделенных нуклеиновых кислот, любую из вышеуказанных гидовых РНК, любую из вышеуказанных антисмысловых РНК, siRNA или shRNA, любой из вышеуказанных выделенных полипептидов или любой из вышеуказанных векторов. Необязательно, клетка представляет собой клетку человека, клетку грызуна, клетку мыши или клетку крысы. Необязательно, любая из вышеуказанных клеток представляют собой клетки печени или плюрипотентные клетки.
[0035] Также настоящее изобретение относится к применениям любой из вышеуказанных гидовых РНК в способе модификации гена HSD17B13 в клетке или способе изменения экспрессии гена HSD17B13 в клетке. Также настоящее изобретение относится к применениям любой из вышеуказанных антисмысловых РНК, siRNA или shRNA в способе изменения экспрессии гена HSD17B13 в клетке.
[0036] Также настоящее изобретение относится к способам модификации клетки, модификации гена HSD17B13 или изменения экспрессии гена HSD17B13. Некоторые такие способы предназначены для модификации гена HSD17B13 в клетке, при этом они предусматривают приведение в контакт генома клетки с: (а) белком Cas; и (b) гидовой РНК, которая образует комплекс с белком Cas и нацелена на целевую для гидовой РНК последовательность в гене HSD17B13, причем целевая для гидовой РНК последовательность включает положение или находится в непосредственной близости от него, которое соответствует положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2, причем белок Cas расщепляет ген HSD17В13. Необязательно, белок Cas представляет собой белок Cas9. Необязательно, целевая для гидовой РНК последовательность содержит, фактически состоит из или состоит из любой из SEQ ID NO: 226-239 и 264-268. Необязательно, нацеливающий на ДНК сегмент содержит, фактически состоит из или состоит из любой из SEQ ID NO: 1629-1642 и 1648-1652. Необязательно, гидовая РНК содержит, фактически состоит из или состоит из любой из SEQ ID NO: 706-719; 936-949; 1166-1179, 1396-1409, 725-729, 955-959, 1185-1189 и 1415-1419. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 226-239, или причем целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 230 и 231. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 226-239 и 264-268 или выбрана из SEQ ID NO: 264-268. Необязательно, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 6 и/или интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 6, и/или интрону 6, и/или экзону 7 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность находится в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от положения, соответствующего положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность включает положение, соответствующее положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2.
[0037] Некоторые такие способы дополнительно предусматривают приведение генома в контакт с экзогенной донорной последовательностью, содержащей 5' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 5' от положения, соответствующего положению 12666 из SEQ ID NO: 2, и 3' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 3' от положения, соответствующего положению 12666 из SEQ ID NO: 2, причем экзогенная донорная последовательность рекомбинирует с геном HSD17B13. Необязательно, экзогенная донорная последовательность дополнительно содержит вставку нуклеиновой кислоты, фланкированную 5' гомологичным плечом и 3' гомологичным плечом. Необязательно, вставка нуклеиновой кислоты содержит тимин, и причем при рекомбинации экзогенной донорной последовательности с геном HSD17B13 тимин вставляется между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1. Необязательно, экзогенная донорная последовательность составляет от приблизительно 50 нуклеотидов до приблизительно 1 т.о. в длину или от приблизительно 80 нуклеотидов до приблизительно 200 нуклеотидов в длину. Необязательно, экзогенная донорная последовательность является одноцепочечным олигодезоксинуклеотидом.
[0038] Некоторые такие способы предназначены для модификации гена HSD17B13 в клетке, при этом они предусматривают приведение в контакт генома клетки с: (а) белком Cas; и (b) первой гидовой РНК, которая образует комплекс с белком Cas и нацелена на первую целевую для гидовой РНК последовательность в гене HSD17B13, причем первая целевая для гидовой РНК последовательность содержит старт-кодон для гена HSD17B13, или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона, или выбрана из SEQ ID NO: 20-81, или выбрана из SEQ ID NO: 20-81 и 259-263, причем белок Cas расщепляет ген HSD17B13 или изменяет его экспрессию. Необязательно, первая целевая для гидовой РНК последовательность содержит, фактически состоит из или состоит из любой из SEQ ID NO: 20-81 и 259-263. Необязательно, первая целевая для гидовой РНК последовательность содержит, фактически состоит из или состоит из любой из SEQ ID NO: 20-41, любой из SEQ ID NO: 21-23, 33 и 35 или любой из SEQ ID NO: 33 и 35. Необязательно, первая гидовая РНК содержит, фактически состоит из или состоит из нацеливающего на ДНК сегмента, который содержит любую из SEQ ID NO: 1423-1484 и 1643-1647. Необязательно, первая гидовая РНК содержит, фактически состоит из или состоит из нацеливающего на ДНК сегмента, который содержит любую из SEQ ID NO: 1447-1468, любую из SEQ ID NO: 1448-1450, 1460 и 1462; или любую из SEQ ID NO: 1460 и 1462. Необязательно, первая гидовая РНК содержит, фактически состоит из или состоит из любой из SEQ ID NO: 500-561, 730-791, 960-1021, 1190-1251, 720-724, 950-954, 1180-1184 и 1410-1414. Необязательно, первая гидовая РНК содержит, фактически состоит из или состоит из любой из SEQ ID NO: 524-545, 754-775, 984-1005 и 1214-1235 или любой из SEQ ID NO: 295-297, 525-527, 755-757, 985-987, 1215-1217, 307, 309, 537, 539, 767, 769, 997, 999, 1227 и 1229 или любой из SEQ ID NO: 307, 309, 537, 539, 767, 769, 997, 999, 1227 и 1229. Необязательно, первая целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 20-41, выбрана из SEQ ID NO: 21-23, 33 и 35 или выбрана из SEQ ID NO: 33 и 35. Необязательно, белок Cas представляет собой белок Cas9. Необязательно, белок Cas является обладающим нуклеазной активностью белком Cas. Необязательно, белок Cas является не обладающим нуклеазной активностью белком Cas, слитым с доменом-активатором транскрипции, или не обладающим нуклеазной активностью белком Cas, слитым с доменом-репрессором транскрипции.
[0039] Некоторые такие способы дополнительно предусматривают приведение генома клетки в контакт со второй гидовой РНК, которая образует комплекс с белком Cas и нацеливает на вторую целевую для гидовой РНК последовательность в гене HSD17B13, причем вторая целевая для гидовой РНК последовательность содержит стоп-кодон для гена HSD17B13, или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от стоп-кодона, или выбрана из SEQ ID NO: 82-225, причем клетку модифицируют так, чтобы у нее была делеция между первой целевой для гидовой РНК последовательности до второй целевой для гидовой РНК последовательности. Необязательно, вторая целевая для гидовой РНК последовательность содержит, фактически состоит из или состоит из любой из SEQ ID NO: 82-225. Необязательно, вторая гидовая РНК содержит, фактически состоит из или состоит из нацеливающего на ДНК сегмента, который содержит любую из SEQ ID NO: 1485-1628. Необязательно, вторая гидовая РНК содержит, фактически состоит из или состоит из любой из SEQ ID NO: 562-705, 792-935, 1022-1165 и 1252-1395.
[0040] Некоторые такие способы предназначены для уменьшения экспрессии гена HSD17B13 в клетке или уменьшения экспрессии конкретного транскрипта HSD17B13 (например, транскрипта А или транскрипта D) в клетке. Некоторые такие способы предназначены для уменьшения экспрессии гена HSD17B13 в клетке, предусматривая приведение генома клетки в контакт с антисмысловой РНК, siRNA или shRNA, которая гибридизируется с последовательностью в пределах экзона 7 из SEQ ID NO: 4 (транскрипта А HSD17B13) и уменьшают экспрессию транскрипта A HSD17B13. Некоторые такие способы предназначены для уменьшения экспрессии гена HSD17B13 в клетке, предусматривая приведение генома клетки в контакт с антисмысловыми РНК, siRNA или shRNA, которые гибридизируются с последовательностью в пределах раскрываемого в настоящем документе транскрипта HSD17B13. В соответствии с некоторыми такими способами, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах SEQ ID NO: 4 (транскрипта A HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA может уменьшать экспрессию транскрипта A HSD17B13 в клетке. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 4 (транскрипте A HSD17B13) и отсутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 4 (транскрипта A HSD17B13). В соответствии с некоторыми такими способами, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах SEQ ID NO: 7 (транскрипта D HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA может уменьшать экспрессию транскрипта D HSD17B13 в клетке. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13) и отсутствующей в SEQ ID NO: 4 (транскрипте A HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 7 (транскрипта D HSD17B13).
[0041] Согласно любому из вышеуказанных способов модификации гена HSD17B13 или изменения экспрессии гена HSD17B13, способ может дополнительно предусматривать введение вектора экспрессии в клетку, причем вектор экспрессии содержит рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. Необязательно, рекомбинантный ген НSD17В13 является геном человека. Необязательно, рекомбинантный ген HSD17B13 является минигеном HSD17B13, в котором были делетерованы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа. Необязательно, делетерованные сегменты содержат одну или несколько интронных последовательностей. Необязательно, миниген HSD17B13 содержит интрон, соответствующий интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2.
[0042] В соответствии с любым из вышеуказанных способов модификации гена HSD17B13 или изменения экспрессии гена HSD17B13, способ может дополнительно предусматривать введение вектора экспрессии в клетку, причем вектор экспрессии содержит нуклеиновую кислоту, кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13). Необязательно, нуклеиновая кислота, кодирующая белок HSD17B13, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична SEQ ID NO: 7 (транскрипту D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7.
[0043] В соответствии с любым из вышеуказанных способов модификации гена HSD17B13 или изменения экспрессии гена HSD17B13, способ может дополнительно предусматривать введение белка HSD17B13 или его фрагмента в клетку. Необязательно, белок HSD17B13 или его фрагмент по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13).
[0044] Некоторые такие способы предназначены для модификации клетки, причем способы предусматривают введение вектора экспрессии в клетку, причем вектор экспрессии содержит рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. Необязательно, рекомбинантный ген HSD17B13 является геном человека. Необязательно, рекомбинантный ген HSD17B13 является минигеном HSD17B13, в котором были делетерованы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа. Необязательно, делетерованные сегменты содержат одну или несколько интронных последовательностей. Необязательно, миниген HSD17B13 содержит интрон, соответствующий интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2.
[0045] Некоторые такие способы предназначены для модификации клетки, причем такие способы предусматривают введение вектора экспрессии в клетку, при этом вектор экспрессии содержит нуклеиновую кислоту, кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13). Необязательно, нуклеиновая кислота, кодирующая белок HSD17B13, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична SEQ ID NO: 7 (транскрипту D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7.
[0046] Некоторые такие способы предназначены для модификации клетки, причем такие способы предусматривают введение белка HSD17B13 или его фрагмента в клетку. Необязательно, белок HSD17B13 или его фрагмент по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13).
[0047] В соответствии с любым из вышеуказанных способов модификации клетки, модификации гена HSD17B13 или изменения экспрессии гена HSD17B13, клетка может быть клеткой человека, клеткой грызуна, клеткой мыши или клеткой крысы. Любые из этих клеток могут быть плюрипотентными клетками или диффенцированными клетками. Любые из клеток могут быть клетками печени. В соответствии с любым из вышеуказанных способов модификации клетки, модификации гена HSD17B13 или изменения экспрессии гена HSD17B13, способ или клетка могут быть ex vivo или in vivo. Гидовые РНК, применяемые в соответствии с любым из вышеуказанных способов, могут быть модульными гидовыми РНК, содержащими отдельные молекулы crRNA и tracrRNA, которые гибридизируются друг с другом или одиночной гидовой РНК, в которой часть crRNA слита с частью tracrRNA (например, с помощью линкера).
[0048] Согласно другому аспекту, настоящее изобретение относится к способам лечения субъекта, который имеет хроническое заболевание печени или подвержен риску его развития. Согласно другому аспекту, настоящее изобретение относится к способам лечения субъекта, который имеет алкогольную или неалкогольную болезнь печени или подвержен риску ее развития. Такой субъект может быть, например, субъектом, который не является носителем варианта rs72613 567 HSD17B13, или субъектом, который не является гомозиготным носителем варианта rs72613567 HSD17B13. Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, который предусматривает введение субъекту: (а) белка Cas или нуклеиновой кислоты, кодирующей белок Cas; (b) гидовой РНК или нуклеиновой кислоты, кодирующей такую гидовую РНК, причем гидовая РНК образует комплекс с белком Cas и нацелена на целевую для гидовой РНК последовательность в гене HSD17B13, причем целевая для гидовой РНК последовательность включает положение или находится в непосредственной близости от него, которое соответствует положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2; и (с) экзогенной донорной последовательности, содержащей 5' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 5' от положения, соответствующего положению 12666 из SEQ ID NO: 2, 3' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 3' от положения, соответствующего положению 12666 из SEQ ID NO: 2, и содержащую тимин вставку нуклеиновой кислоты, фланкированную 5' гомологичным плечом и 3' гомологичным плечом, причем белок Cas расщепляет ген HSD17B13 в клетке печени у субъекта, а экзогенная донорная последовательность подвергается рекомбинации с геном HSD17B13 в клетке печени, причем при рекомбинации экзогенной донорной последовательности с геном HSD17В13 тимин вставляется между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1.
[0049] Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 226-239, или причем целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 230 и 231. Необязательно, целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 226-239 и 264-268. Необязательно, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 6 и/или интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 6, и/или интрону 6, и/или экзону 7 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность находится в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от положения, соответствующего положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Необязательно, целевая для гидовой РНК последовательность включает положение, соответствующее положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2.
[0050] Необязательно, экзогенная донорная последовательность находится составляет от приблизительно 50 нуклеотидов до приблизительно 1 т.о. в длину. Необязательно, экзогенная донорная последовательность составляет от приблизительно 80 нуклеотидов до приблизительно 200 нуклеотидов в длину. Необязательно, экзогенная донорная последовательность является одноцепочечным олигодезоксинуклеотидом.
[0051] Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, который предусматривает введение субъекту: (а) белка Cas или нуклеиновой кислоты, кодирующей белок Cas; (b) первой гидовой РНК или нуклеиновой кислоты, кодирующей первую гидовую РНК, причем первая гидовая РНК образует комплекс с белком Cas и нацелена на первую целевую для гидовой РНК последовательность в гене HSD17B13, причем первая целевая для гидовой РНК последовательность содержит старт-кодон для гена HSD17B13, или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона, или выбрана из SEQ ID NO: 20-81, или выбрана из SEQ ID NO: 20-81 и 259-263; и (с) вектора экспрессии, содержащего рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1, причем белок Cas расщепляет ген HSD17B13 или изменяет его экспрессию в клетке печени у субъекта, а вектор экспрессии экспрессирует рекомбинантный ген HSD17B13 в клетке печени у субъекта. Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, который предусматривает введение субъекту: (а) белка Cas или нуклеиновой кислоты, кодирующей белок Cas; (b) первой гидовой РНК или нуклеиновой кислоты, кодирующей первую гидовую РНК, причем первая гидовая РНК образует комплекс с белком Cas и нацелена на первую целевую для гидовой РНК последовательность в гене HSD17B13, причем первая целевая для гидовой РНК последовательность содержит старт-кодон для гена HSD17B13, или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона, или выбрана из SEQ ID NO: 20-81, или выбрана из SEQ ID NO: 20-81 и 259-263; и необязательно (с) вектора экспрессии, содержащего рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1, причем белок Cas расщепляет ген HSD17B13 или изменяет его экспрессию в клетке печени у субъекта, а вектор экспрессии экспрессирует рекомбинантный ген HSD17B13 в клетке печени у субъекта.
[0052] Необязательно, первая целевая для гидовой РНК последовательность выбрана из SEQ ID NO: 20-41, выбрана из SEQ ID NO: 21-23, 33 и 35 или выбрана из SEQ ID NO: 33 и 35. Необязательно, белок Cas является обладающим нуклеазной активностью белком Cas. Необязательно, белок Cas является не обладающим нуклеазной активностью белком Cas слитым с доменом-репрессором транскрипции.
[0053] Такие способы могут дополнительно предусматривать введение субъекту второй гидовой РНК, причем вторая гидовая РНК образовывает комплекс с белком Cas и нацелена вторую целевую для гидовой РНК последовательность в гене HSD17B13, причем вторая целевая для гидовой РНК последовательность содержит стоп-кодон для гена HSD17B13, или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от стоп-кодона, или выбрана из SEQ ID NO: 82-225, причем белок Cas расщепляет ген HSD17B13 в клетке печени в пределах как первой целевой для гидовой РНК последовательности, так и второй целевой для гидовой РНК последовательности, при этом клетку печени модифицируется так, чтобы у нее была делеция между первой целевой для гидовой РНК последовательности и второй целевой для гидовой РНК последовательности.
[0054] Необязательно, рекомбинантный ген HSD17B13 является минигеном HSD17B13, в котором были делетерованы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа. Необязательно, делетерованные сегменты содержат одну или несколько интронных последовательностей. Необязательно, миниген HSD17B13 содержит интрон, соответствующий интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2.
[0055] В соответствии с любым из вышеуказанных терапевтических или профилактических способов, белок Cas может представлять собой белок Cas9. В соответствии с любым из вышеуказанных терапевтических или профилактических способов, субъектом может быть человек. В соответствии с любым из вышеуказанных терапевтических или профилактических способов, хроническое заболевание печени может представлять собой жировую болезнь печени, неалкогольную жировую болезнь печени (NAFLD), алкогольную жировую болезнь печени, цирроз или гепатоклеточную карциному. Аналогичным образом, в соответствии с любым из вышеуказанных способов, терапевтический или профилактический способ может быть предназначен для терапии или профилактики заболевания печени, которое представляет собой алкогольную болезнь печени или неалкогольную болезнь печени.
[0056] Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту антисмысловой РНК, siRNA или shRNA, которая гибридизируется с последовательностью в пределах экзона 7 или с последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 4 (транскрипта A HSD17B13), и уменьшает экспрессию транскрипта A HSD17B13 в клетке печени у субъекта. Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту антисмысловой РНК, siRNA или shRNA, которая гибридизируется с последовательностью в пределах раскрываемого в настоящем документе транскрипта HSD17B13. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в SEQ ID NO: 4 (транскрипте A HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA может уменьшать экспрессию транскрипта A HSD17B13 в клетке. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 4 (транскрипте A HSD17B13) и отсутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 4 (транскрипта A HSD17B13).
[0057] Необязательно, такие способы дополнительно предусматривают введение вектора экспрессии субъекту, причем вектор экспрессии содержит рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1, причем вектор экспрессии экспрессирует рекомбинантный ген HSD17B13 в клетке печени у субъекта.
[0058] Необязательно, такие способы дополнительно предусматривают введение вектора экспрессии субъекту, причем вектор экспрессии содержит нуклеиновую кислоту, кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (HSD17B13, изоформа D), причем вектор экспрессии экспрессирует нуклеиновую кислоту, кодирующую белок HSD17B13, в клетке печени у субъекта. Необязательно, нуклеиновая кислота, кодирующая белок HSD17B13, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична SEQ ID NO: 7 (транскрипту D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7.
[0059] Необязательно, такие способы дополнительно предусматривают введение матричной РНК субъекту, причем матричная РНК кодирует белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13), причем мРНК экспрессирует белок HSD17B13 в клетке печени у субъекта. Необязательно, комплементарная ДНК, обратно транскрибированная с матричной РНК, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична SEQ ID NO: 7 (транскрипту D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7.
[0060] Необязательно, такие способы дополнительно предусматривают введение белка HSD17B13 или его фрагмента субъекту. Необязательно, белок HSD17B13 или его фрагмент по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13).
[0061] Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение вектора экспрессии субъекту, причем вектор экспрессии содержит рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1, причем вектор экспрессии экспрессирует рекомбинантный ген HSD17B13 в клетке печени у субъекта.
[0062] В соответствии с любым из вышеуказанных способов, рекомбинантный ген HSD17B13 может быть геном человека. В соответствии с любым из вышеуказанных способов, рекомбинантный ген HSD17B13 может быть по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. В соответствии с любым из вышеуказанных способов, рекомбинантный ген HSD17B13 может быть минигеном HSD17B13, в котором были делетерованы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа. Необязательно, делетерованные сегменты содержат одну или несколько интронных последовательностей. Необязательно, миниген HSD17B13 содержит интрон, соответствующий интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2.
[0063] Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение вектора экспрессии субъекту, причем вектор экспрессии содержит нуклеиновую кислоту, кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13), причем вектор экспрессии экспрессирует нуклеиновую кислоту, кодирующую белок HSD17B13, в клетке печени у субъекта. Необязательно, нуклеиновая кислота, кодирующая белок HSD17B13, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична SEQ ID NO: 7 (транскрипту D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7.
[0064] Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение матричной РНК субъекту, приче матричная РНК кодирует белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13), причем мРНК экспрессирует белок HSD17B13 в клетке печени у субъекта. Необязательно, комплементарная ДНК, обратно транскрибированная с матричной РНК, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична SEQ ID NO: 7 (транскрипту D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7.
[0065] Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение белка HSD17B13 или его фрагмента в печень субъекта. Необязательно, белок HSD17B13 или его фрагмент по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15 (изоформе D HSD17B13).
[0066] В соответствии с любым из вышеуказанных способов, субъектом может быть человек. В соответствии с любым из вышеуказанных способов, хроническое заболевание печени может представлять собой неалкогольную жировую болезнь печени (NAFLD), алкогольную жировую болезнь печени, цирроз или гепатоклеточную карциному. Аналогичным образом, в соответствии с любым из вышеуказанных способов, терапевтический или профилактический способ может быть предназначен для терапии или профилактики заболевания печени, которое представляет собой алкогольную болезнь печени или неалкогольную болезнь печени. В соответствии с любым из вышеуказанных способов, введение субъекту может предусматривать гидродинамическую доставку, опосредованную вирусом доставку, опосредованную липидными наночастицами доставку или внутривенную инфузию.
Краткое описание чертежей
[0067] На фиг. 1А и 1В показаны график Манхэттена (слева) и квантиль-квантиль график (справа) ассоциаций однонуклеотидного варианта с медианными уровнями аланинаминотрансферазы (ALT; фиг. 1А) и аспартатаминотрансферазы (AST; фиг. 1В) в группе исследования GHS. На фиг. 1А показано, что был обнаружен 31 вариант в 16 генах, значимо ассоциированный с уровнями ALT (N=41414) при Р<1,0×10-7. На фиг. 1В показано, что было обнаружено 12 вариантов в 10 генах, значимо ассоциированных с уровнями AST (N=40753) при Р<1,0×10-7. Все значимые ассоциации приведены в таблице 2. Было обнаружено тринадцать вариантов в девяти генах (обозначенных в настоящем документе по названию их гена), включая HSD17B13, которые оставались значимо ассоциированными с ALT или AST в репликационном мета-анализе трех отдельных групп европейского происхождения (таблица 3). Тесты на ассоциации были высокоточными, как видно из полноэкзомных квантиль-квантиль графиков и значений лямбда геномного контроля (фиг. 1А и фиг. 1В).
[0068] На фиг. 2А и 2В показано, что rs72613567:TA HSD17B13 ассоциирован со сниженным риском развития фенотипов алкогольной и неалкогольной болезни печени в группе исследования (фиг. 2А) и со сниженным риском прогрессирования от простого стеатоза до стеатогепатита и фиброза в группе бариатрической хирургии (фиг. 2В). Отношения шансов были рассчитаны с использованием логистической регрессии с коррекцией по возрасту, возрасту2, полу, BMI и основные компоненты происхождения. Также показаны генотипические отношения шансов для гетерозиготных (Het OR) и гомозиготных (Hom OR) носителей. В группе исследования GHS на фиг. 2А вариант HSD17B13 был ассоциирован со значимо сниженным риском развития неалкогольной и алкогольной болезни печени, цирроза и гепатоклеточной карциномы зависимым от дозы аллеля образом. В группе бариатрической хирургии GHS на фиг. 2В rs72613567 HSD17B13 был ассоциирован с более низкими на 13% и 52% шансами развития неалкогольного стеатогепатита (NASH) и с более низкими на 13% и 61% шансами развития фиброза у гетерозиготных и гомозиготных носителей ТА соответственно.
[0069] На фиг. 3A-3D показана экспрессия четырех транскриптов (A-D) HSD17B13 у гомозиготных эталонных (Т/Т), гетерозиготных (Т/ТА) и гомозиготных альтернативных (ТА/ТА) носителей с плайс-варианта rs72613567 HSD17B13. Каждый транскрипт проиллюстрирован с помощью соответствующей модели гена. Кодирующие участки в генных моделях показаны в заштрихованных прямоугольниках, а не транслируемые участки - в черных прямоугольных диаграммах. На фиг. 3А показано представление транскрипта А и данные экспрессии для транскрипта А. На фиг. 3В показано представление транскрипта В и данные экспрессии для транскрипта В. В транскрипте В экзон 2 пропущен. На фиг. 3С показано представление транскрипта С и данные экспрессии для транскрипта С. В транскрипте С экзон 6 пропущен. На фиг. 3D показано представление транскрипта D и данные экспрессии для транскрипта D. Звездочка в транскрипте D иллюстрирует вставку G из rs72613567 на 3'-конце экзона 6, что приводит к преждевременному укорочению белка. Транскрипт D становится доминантным транскриптом у гомозиготных носителей сплайс-варианта HSD17B13. Экспрессия гена отображена в единицах FPKM (количество фрагментов на тысячу оснований транскрипта на миллион картированных циклов считывания). На вставках на фиг. 3В и фиг. 3С показано увеличенное изображение.
[0070] На фиг. 4 показано, что в результате исследований по секвенированию РНК печени человека было выявлено восемь транскриптов HSD17B13, включая шесть новых транскриптов HSD17B13 (транскрипты С-Н). Экспрессия транскриптов отображена в единицах FPKM (количество фрагментов на тысячу оснований транскрипта на миллион картированных циклов считывания). Структуры транскриптов представлены в правой части фигуры.
[0071] На фиг. 5А и 5В показаны масштабированные графики локуса HSD17B13 (частичные графики ассоциаций в участке вокруг HSD17B13) в группе исследования GHS для ALT и AST соответственно. Не наблюдали никакой значимой рекомбинации по всему участку. Ромбами обозначен сплайс-вариант rs72613567. Каждый кружок обозначает однонуклеотидный вариант, цвет кружка указывает на дисбаланс связей (r2, рассчитанное в группе DiscovEHR) между этим вариантом и rs72613567. Линиями указаны оценочные значения частоты рекомбинации в НарМар. В нижних секциях показаны относительное положение и транскрибированная цепь каждого гена в локусе. Значимые ассоциации между ALT или AST и кодирующими вариантами или сплайс-вари антами участка в соседнем гене HSD17B11 отсутствовали (наиболее значимые Р-значения 1,4×10-1 и 4,3×10-2 для ALT и AST соответственно).
[0072] На фиг. 6A-6D показана экспрессия мРНК четырех дополнительных новых транскриптов (Е-Н) HSD17B13 у гомозиготных эталонных (Т/Т), гетерозиготных (Т/ТА) и гомозиготных альтернативных (ТА/ТА) носителей сплайс-варианта HSD17B13. Каждый транскрипт проиллюстрирован с помощью соответствующей модели гена. Кодирующие участки в генных моделях показаны в заштрихованных прямоугольниках, а нетранслируемые участки - в черных прямоугольных диаграммах. На фиг. 6А и 6D показано, что транскрипты Е и Н содержат дополнительный экзон между экзонами 3 и 4. На фиг. 6В показано, что транскрипт F включает участок считываняе от экзона 6 в интрон 6. На фиг. 6С показано, что в транскрипте G экзон 2 пропущен. Звездочкой в транскриптах G и Н (на фиг. 6С и 6D соответственно) проиллюстрирована вставка G из rs72613567 на 3'-конце экзона 6, что приводит к преждевременному укорочению белка. Транскрипты дифференциально экспрессируются в соответствии с генотипом HSD17B13, как показано на ящичковых диаграммах. Экспрессия мРНК отображена в единицах FPKM (количество фрагментов на тысячу оснований транскрипта на миллион картированных циклов считывания).
[0073] На фиг. 7А-7В показано выравнивание белковых последовательностей изоформ А-Н белка HSD17B13.
[0074] На фиг. 8 показано, что rs72613567:TA HSD17B13 ассоциирован со сниженным риском развития фенотипов алкогольной и неалкогольной болезни печени. В частности, на фиг. 8 показано, что в исследовании печени в Далласе rs72613567 HSD17B13 был ассоциирован с более низким шансом возникновения любого заболевания печени зависимым от дозы аллеля образом. Схожие зависимые от дозы аллеля эффекты наблюдали для всех подтипов заболевания печени. Отношения шансов были рассчитаны с помощью логистической регрессии с коррекцией по возрасту, возрасту2, полу, BMI и сообщенной участником этнической принадлежности.
[0075] На фиг. 9 показано, что rs72613567 HSD17B13 ассоциирован со сниженным риском прогрессирования от простого стеатоза до стеатогепатита и фиброза. В частности, на ней показана доля гистопатологически охарактеризованного заболевания печени в соответствии с генотипом rs72613567 HSD17B13 у 2391 индивидуума с биопсией печени из группы бариатрической хирургии GHS. Доля случаев с нормальной печенью, по-видимому, не различалась по генотипу (Р=0,5 по критерию хи-квадрат для тенденции в пропорциях), но доля случаев с NASH снижалась (Р=1,6×10-4), а доля случаев с простым стеатозом увеличивалась (Р=1,1×10-3) с каждым аллелем ТА.
[0076] На фиг. 10А-10Е показана экспрессия, субклеточная локализация и ферментативная активность нового транскрипта HSD17B13. На фиг. 10А показаны результаты вестерн-блоттинга, полученные от клеток HepG2 со сверхэкспрессией транскриптов А и D HSD17B13, и показано, что транскрипт D HSD17B13 транслировался в укороченный белок с более низкой молекулярной массой по сравнению с транскриптом A HSD17B13. На фиг. 10В показаны результаты вестерн-блоттинга HSD17B13 из свежезамороженных образцов клеток HEK293 и клеток печени человека. Образцы печени человека взяты у гомозиготных эталонных (Т/Т), гетерозиготных (Т/ТА) и гомозиготных альтернативных (ТА/ТА) носителей сплайс-варианта rs72613567 HSD17B13. Образцы клеток взяты из клеток HEK293, сверхэкспрессирующих немеченные транскрипты А и D HSD17B13. Транскрипт HSD17B13 транслировался в укороченный белок изоформы D (IsoD) с более низкой молекулярной массой, чем HSD17B13 изоформы A (IsoA). На фиг. 10С показано, что уровни белка изоформы D (IsoD) HSD17B13 были ниже уровней белка IsoA как из образцов печени человека (слева), так и из клеток (справа). Уровень белка, нормализованный к актину, показан в виде столбцов; **Р<0,001, *Р<0,05. На фиг. 10D показана ферментативная активность изоформ А и D HSD17B13 в отношении 17-бета-эстрадиола (эстрадиола), лейкотриена В4 (LTB4) и 13-гидроксиоктадекадиеновой кислоты (13(S)-HODE). В случае изоформы D HSD17B13 наблюдали <10% ферментативную активность относительно соответствующих значений для изоформы А. На фиг. 10Е показана изоформа D HSD17B13 при сверх экспрессии в клетках HEK293 не наблюдали значительного превращения эстрадиола (субстрата) в эстрон (продукт) при измерении в культуральной среде, в то время как при сверхэкспрессии изоформы А HSD17B13 наблюдали устойчивое превращение.
[0077] На фиг. 11А-11С показано, что белок изоформы D HSD17B13 имеет более низкую молекулярную массу и является нестабильным при сверхэкспрессии в клетках НЕК 293. На фиг. 11А показаны результаты RT-PCR HSD17B13 из клеток HEK 293, сверхэкспрессирующих транскрипты A (IsoA) и D (IsoD) HSD17B13, что указывает на то, что уровень РНК IsoD HSD17B13 был выше, чем уровень РНК IsoA. На фиг. 11В показаны результаты вестерн-блоттинга от тех же клеточных линий, из которых видно, что транскрипт D HSD17B13 транслировался в укороченный белок с более низкой молекулярной массой по сравнению с транскриптом A HSD17B13. На фиг. 11С показано, что уровни белка IsoD HSD17B13 были ниже, чем уровни белка IsoA, хотя уровень РНК был выше. Уровень белка HSD17B13 был нормализован к актину; *Р<0,05.
[0078] На фиг. 12 показаны схожие паттерны локализации изоформы А и изоформы D HSD17B13 для выделенных липидных капель (LD), полученных из стабильных клеточных линий HepG2. ADRP и TIP47 применяли в качестве маркеров липидных капель. LAMP1, кальретикулин и СОХ IV применяли в качестве маркеров для соответственно лизосомального компартмента, компартмента эндоплазматического ретикулума и митохондриального компартмента. GAPDH был включен в качестве цитозольного маркера, а актин применяли в качестве маркера цитоскелета. Этот эксперимент повторяли дважды на клетках HepG2, причем вышеизложенное является репрезентативным для обоих циклов. PNS = постядерная фракция; ТМ = общая мембрана.
[0079] На фиг. 13A-13D показаны уровни триглицеридов с повышенным содержанием олеиновой кислоты в клетках HepG2, сверхэкспрессирующих транскрипт А или D HSD17B13. На фиг. 13А показаны результаты обработки с повышением концентраций триглицеридов с повышенным содержанием олеиновой кислоты (TG) относительно аналогичной степени у контроля (клетки со сверхэкспрессией GFP) и клеточных линий транскрипта А и D HSD17B13. На фиг. 13В показаны уровни РНК транскриптов А и D HSD17B13, которые были схожими в клеточных линиях. Уровни РНК показаны в количестве считываний на тысячу оснований транскрипта на миллион картированных циклов считывания (RPKM). На фиг. 13С показаны результаты вестерн-блоттинга, полученные от клеток HepG2 со сверхэкспрессией транскриптов А и D HSD17B13. Транскрипт D HSD17B13 транслировался в укороченный белок с более низкой молекулярной массой по сравнению с транскриптом AHSD17B13. На фиг. 13D видно, что уровни белка IsoD HSD17B13 были ниже, чем уровни белка IsoA. Уровень белка, нормализованный к актину; **Р<0,01.
[0080] На фиг. 14 представлены значения Km и Vmax для эстрадиола, полученные с применением очищенного рекомбинантного белка HSD17B13. Для определения значений Km и Vmax были проведены анализы с диапазоном доз 17β-эстрадиола от 0,2 мкМ до 200 мкМ и моментами времени от 5 минут до 180 минут, с 500 мкМ NAD+ и 228 нМ HSD17B13. Затем Vmax и Km определяли с помощью модели Михаэлиса-Ментена и программного обеспечения Prism (GraphPad Software, США).
[0081] На фиг. 15 показан процент редактирования генома (общее количество вставок или делеций, наблюдаемых в интервале из 20 пар оснований по обе стороны от Cas9-индуцированного разрыва ДНК, относительно общего количества последовательностей, считанных в реакции ПЦР, из пула лизированных клеток) в локусе Hsd17b13 мыши по результатам определения с помощью секвенирования следующего поколения (NGS) на первичных гепатоцитах, выделенных у гибридных мышей дикого типа (75% C57BL/6NTac 25% 129S6/SvEvTac). Тестируемые образцы включали гепатоциты, обработанные рибонуклеопротеиновыми комплексами, содержащими Cas9 и гидовые РНК, предназначенные для нацеливания на локус Hsd17b13 мыши.
[0082] На фиг. 16 показан процент редактирования генома (общее количество вставок или делеций, наблюдаемое относительно общего количества последовательностей, считанных в реакции ПЦР, из пула лизированных клеток) в локусе Hsd17b13 мыши по результатам определения с помощью секвенирования следующего поколения (NGS) на выделенных образцах, полученных из печени мышей через три недели после инъекции AAV8, содержащего кассеты экспрессии sgRNA, предназначенные для нацеливания на Hsd17b13 мыши у Cas9-готовых мышей. Мышам дикого типа, у которых отсутствовала экспрессия какого-либо Cas9, вводили инъекцией AAV8, содержащий все кассеты экспрессии sgRNA, и использовали в качестве отрицательного контроля.
[0083] На фиг. 17А и 17В показана относительная экспрессия мРНК соответственно для Hsd17b13 мыши и нецелевого представителя семейства HSD по результатам определения с помощью RT-qPCR в образцах печени от Cas9-готовых мышей, обработанных AAV8, несущим кассеты экспрессии гидовой РНК, предназначенные для нацеливания на Hsd17b13 мыши. Мышам дикого типа, у которых отсутствовала экспрессия какого-либо Cas9, вводили инъекцией AAV8, несущий кассеты экспрессии гидовой РНК для всех гидовых РНК, и использовали в качестве отрицательного контроля.
Определения
[0084] Термины «белок», «полипептид» и «пептид», применяемые в контексте настоящего документа взаимозаменяемо, включают полимерные формы аминокислот любой длины, включая кодируемые и некодируемые аминокислоты и химически или биохимически модифицированные или дериватизированные аминокислоты. Термины также включают полимеры, которые были модифицированы, такие как полипептиды, имеющие модифицированные пептидные остовы.
[0085] Говорят, что у белков есть «N-конец» и «С-конец». Термин «N-конец» относится к началу белка или полипептида, оканчивающегося аминокислотой со свободной аминогруппой (-NH2). Термин «С-конец» относится к концу аминокислотной цепи (белка или полипептида), оканчивающемуся свободной карбоксильной группой (-СООН).
[0086] Термины «нуклеиновая кислота» и «полинуклеотид», применяемые в контексте настоящего документа взаимозаменяемо, включают полимерные формы нуклеотидов любой длины, включая рибонуклеотиды, дезоксирибонуклеотиды или их аналоги или модифицированные варианты. Они включают одно-, двух- и многоцепочечную ДНК или РНК, геномную ДНК, кДНК, гибриды ДНК-РНК и полимеры, содержащие пуриновые основания, пиримидиновые основания или другие природные, химически модифицированные, биохимически модифицированные, неприродные или дериватизированные нуклеотидные основания.
[0087] Говорят, что у нуклеиновых кислот есть «5'-концы» и «3'-концы», поскольку мононуклеотиды вступают в реакцию с образованием олигонуклеотидов таким образом, чтобы 5'-фосфат одного мононуклеотидпентозного кольца был присоединен к 3'-кислороду его соседа в одном направлении через фосфодиэфирную связь. Конец олигонуклеотида называют «5'-концом», если его 5'-фосфат не связан с 3'-кислородом мононуклеотидпентозного кольца. Конец олигонуклеотида называют «3'-концом», если его 3'-кислород не связан с 5'-фосфатом другого мононуклеотидпентозного кольца. Можно также сказать, что последовательность нуклеиновой кислоты, даже если она находится внутри более крупного олигонуклеотида, имеет 5'- и 3'-концы. В линейной или кольцевой молекуле ДНК дискретные элементы называют находящимися «выше» или 5'-элементами либо «ниже» или 3'-элементами.
[0088] Термин «дикий тип» включает объекты, имеющие структуру и/или активность, обнаруживаемую в нормальном (в отличие от мутантного, пораженного заболеванием, измененного и т.д.) состоянии или контексте. Гены и полипептиды дикого типа зачастую существуют в нескольких различных формах (например, аллелях).
[0089] Термин «выделенный» в отношении белков и нуклеиновых кислот включает белки и нуклеиновые кислоты, которые являются относительно очищенными по отношению к другим бактериальным, вирусным или клеточным компонентам, которые в норме могут присутствовать in situ, вплоть до практически чистого препарата белка и полинуклеотида. Термин «выделенный» также включает белки и нуклеиновые кислоты, которые не имеют встречающегося в естественной среде аналога, были химически синтезированы и, таким образом, практически не загрязнены другими белками или нуклеиновыми кислотами или были отделены или очищены от большинства других клеточных компонентов, которыми они в естественной среде сопровождаются (например, другие клеточные белки, полинуклеотиды или клеточные компоненты).
[0090] «Экзогенные» молекулы или последовательности включают молекулы или последовательности, которые в норме не присутствуют в клетке в такой форме. Присутствие в норме включает присутствие относительно конкретной стадии развития и условий окружающей среды клетки. Например, экзогенная молекула или последовательность может включать мутантную версию соответствующой эндогенной последовательности в клетке или может включать последовательность, соответствующую эндогенной последовательности в клетке, но в другой форме (т.е. не находящующся в хромосоме). Напротив, эндогенные молекулы или последовательности включают молекулы или последовательности, которые в норме присутствуют в такой форме в конкретной клетке на определенной стадии развития в определенных условиях окружающей среды.
[0091] Термин «гетерологичная» при его применении в контексте нуклеиновой кислоты или белка указывает на то, что нуклеиновая кислота или белок содержит по меньшей мере две части, которые совместно в норме не встречаются. Аналогичным образом, термин «гетерологичный» при его применении в контексте промотора, функционально связанного с нуклеиновой кислотой, кодирующей белок, указывает на то, что промотор и нуклеиновая кислота, кодирующая белок, совместно в норме не встречаются (т.е. в норме функционально не связаны). Например, термин «гетерологичный» при его применении в отношении частей нуклеиновой кислоты или частей белка, указывает, что нуклеиновая кислота или белок содержит две или более подпоследовательностей, которые не встречаются в такой же связи друг с другом (например, совместно связанными) в естественной среде. В качестве одного примера, «гетерологичный» участок вектора нуклеиновой кислоты представляет собой сегмент нуклеиновой кислоты, находящийся в или присоединенный к другой молекуле нуклеиновой кислоты, который в естественной среде не встречается в ассоциации с другой молекулой. Например, гетерологичный участок вектора нуклеиновой кислоты может включать кодирующую последовательность, фланкированную последовательностями, которые не встречаются в естественной среде в ассоциации с кодирующей последовательностью. Аналогичным образом, «гетерологичный» участок белка представляет собой сегмент из аминокислот, находящийся в или присоединенный к другой пептидной молекуле, который не встречается в естественной среде в ассоциации с другой пептидной молекулой (например, белок слияния или белок с маркером). Подобным образом, нуклеиновая кислота или белок могут содержать гетерологичную метку или гетерологичную последовательность секреции или локализации.
[0092] Термин «метка» относится к химическому фрагменту или белку, который можно прямо или опосредованно детектировать (например, благодаря его спектральным свойствам, конформации или активности) при его присоединении к целевому соединению. Метка может быть прямо детектируемой (флуорофор) или опосредованно детектируемой (гаптен, фермент или гаситель флуорофора). Такие метки можно детектировать с помощью спектроскопических, фотохимических, биохимических, иммунохимических или химических средств. Такие метки включают, например, радиоактивные метки, которые можно измерять с помощью приборов для подсчета радиации; пигменты, красители или других хромогены, которые можно визуально наблюдать или измерять с помощью спектрофотометра; спиновые метки, которые можно измерять с помощью анализатора спиновых меток; и флуоресцентные метки (флуорофоры), при этом выходной сигнал вырабатывается при возбуждении подходящего молекулярного аддукта, и его можно визуализировать при возбуждении светом, который поглощается красителем, или можно измерить с помощью стандартных флуорометров или систем визуализации. Метка также может быть, например, хемилюминесцентным веществом, при этом выходной сигнал вырабатывается при химической модификации сигнального соединения; металлсодержащим веществом; или ферментом, при этом происходит фермент-зависимая вторичная выработка сигнала, такая как образование окрашенного продукта из бесцветного субстрата. Термин «метка» может также относиться к «маркеру» или гаптену, который может селективно связываться с конъюгированной молекулой, так чтобы конъюгированная молекула при ее последующем добавлении к субстрату использовалась для выработки детектируемого сигнала. Например, в качестве маркера можно использовать биотин, а затем использовать конъюгат авидина или стрептавидина с пероксидазой хрена (HRP) для связывания с маркером, а затем использовать калориметрический субстрат (например, тетраметилбензидин (ТМВ)) или флуорогенный субстрат для детектирования наличия HRP. Термин «метка» может также относиться к маркеру, который можно применять, например, для облегчения очистки. Неограничивающие примеры таких маркеров включают myc, НА, FLAG или 3XFLAG, 6XHis или полигистидин, глутатион-S-трансферазу (GST), мальтозосвязывающий белок, эпитопный маркер или Fc-часть иммуноглобулина. Известны многочисленные метки, и к ним относятся, например, частицы, флуорофоры, гаптены, ферменты и их калориметрические, флуорогенные и хемилюминесцентные субстраты и другие метки.
[0093] «Оптимизация кодонов» учитывает вырожденность кодонов, что проявляется в множественности комбинаций кодонов из трех пар оснований, которые определяют аминокислоту, и, как правило, включает процесс модификации последовательности нуклеиновой кислоты для усиления экспрессии у конкретных клеток-хозяев путем замены по меньшей мере одного кодона нативной последовательности на кодон, который чаще или наиболее часто используют в генах клетки-хозяина при сохранении нативной аминокислотной последовательности. Например, полинуклеотид, кодирующий белок Cas9, может быть модифицирован с заменой кодонов, имеющих более высокую частоту использования в данной прокариотической или эукариотической клетке, включая бактериальную клетку, дрожжевую клетку, клетку человека, клетку отличного от человека животного, клетку млекопитающего, клетку грызуна, клетку мыши, клетку крысы, клетку хомячка или любую другую клетку-хозяина, по сравнению со встречающейся в естественной среде последовательностью нуклеиновой кислоты. Таблицы частот использования кодонов легко доступны, например, в «Базе данных по частоте использования кодонов». Такие таблицы можно приспособить несколькими способами. См. Nakamura et al. (2000) Nucleic Acids Research 28:292, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Также доступны компьютерные алгоритмы для оптимизации кодонов конкретной последовательности для экспрессии в конкретном хозяине (см, например, Gene Forge).
[0094] Термин «локус» относится к конкретному местоположению гена (или значимой последовательности), последовательности ДНК, последовательности, кодирующей полипептид, или положению в хромосоме генома организма. Например, «локус HSD17B13» может относиться к конкретному местоположению гена HSD17B13, последовательности ДНК гена HSD17B13, кодирующей HSD17B13 последовательности или положению HSD17B13 на хромосоме генома организма, которое было выявлено для местонахождения такой последовательности. «Локус HSD17B13» может содержать регуляторный элемент гена HSD17B13, включая, например, энхансер, промотор, 5' и/или 3' UTR или их комбинацию.
[0095] Термин «ген» относится к последовательности ДНК в хромосоме, которая кодирует продукт (например, РНК-продукт и/или полипептидный продукт) и включает кодирующий участок, прерываемый одним или несколькими некодирующими нитронами, и последовательность, расположенную рядом с кодирующим участком на 5'- и 3'-концах так, чтобы ген соответствовал полноразмерной мРНК (включая 5'- и 3'-нетранслируемые последовательности). Термин «ген» также включает другие некодирующие последовательности, включая регуляторные последовательности (например, промоторы, энхансеры и сайты связывания транскрипционных факторов), сигналы полиаденилирования, сайти внутренней посадки рибосомы, сайленсеры, изолирующая последовательность и участки присоединения к матрице. Эти последовательности могут находиться близко к кодирующему участку гена (например, в пределах 10 т.о.) или в удаленных сайтах, и они влияют на уровень или скорость транскрипции и трансляции гена. Термин «ген» также охватывает «минигены».
[0096] Термин «миниген» относится к гену, в котором были делетерованы один или несколько несущественных сегментов гена относительно соответствующего встречающегося в естественной среде гена зародышевой линии, но в котором остается по меньшей мере один интрон. Делетированные сегменты могут быть интронными последовательностями. Например, делетерованные сегменты могут быть интронными последовательностями длиной по меньшей мере от приблизительно 500 пар оснований до нескольких тысяч пар оснований. Как правило, можно удалять интронные последовательности, которые не охватывают основные регуляторные элементы. Генные сегменты гена, составляющие миниген, обычно расположены в том же линейном порядке, что и в гене зародышевой линии, но это не всегда так. Некоторые необходимые регуляторные элементы (например, энхансеры, сайленсеры) могут быть относительно нечувствительными к положению, так что регуляторный элемент функционирует надлежащим образом, даже если он иначе расположен в минигене, чем в соответствующем гене зародышевой линии. Например, энхансер может быть расположен на другом расстоянии от промотора, в другой ориентации и/или в другом линейном порядке. Например, энхансер, который расположен 3' к промотору в конфигурации зародышевой линии, в минигене может быть расположен 5' к промотору. Аналогично, некоторые гены могут иметь экзоны, которые подвергаются альтернативному сплайсингу на уровне РНК. Таким образом, миниген может иметь меньше экзонов и/или иметь экзоны в другом линейном порядке, чем у соответствующего гена зародышевой линии, и все же кодировать функциональный генный продукт. КДНК, кодирующую генный продукт, также можно использовать для построения минигена (например, гибридной молекулы слияния кДНК и геномной последовательности).
[0097] Термин «аллель» относится к вариантной форме гена. Некоторые гены имеют множество различных форм, которые расположены в одном и том же положении или генетическом локусе на хромосоме. Диплоидный организм имеет два аллеля в каждом генетическом локусе. Каждая пара аллелей представляет генотип определенного генетического локуса. Генотипы описывают как гомозиготные, если в конкретном локусе есть два идентичных аллеля, и как гетерозиготные, если эти два аллеля различаются.
[0098] Термин «вариант» или «генетический вариант» относится к нуклеотидной последовательности, отличающейся от последовательности, наиболее распространенной у населения (например, на один нуклеотид). Например, некоторые варианты или замены в нуклеотидной последовательности изменяют кодон так, чтобы кодировалась другая аминокислота, что приводит к получению генетического варианта полипептида. Термин «вариант» может также относиться к гену, отличающемуся по последовательности от последовательности, наиболее распространенной у населения в положении, которое не изменяет аминокислотную последовательность кодируемого полипептида (т.е. консервативная замена). Генетические варианты могут быть ассоциированы с риском, ассоциированы с защитой или могут быть нейтральными.
[0099] «Промотор» представляет собой регуляторный участок ДНК, обычно содержащий ТАТА-бокс, способный направлять РНК-полимеразу II для инициации синтеза РНК в соответствующем сайте инициации транскрипции для конкретной полинуклеотидной последовательности. Промотор может дополнительно содержать другие участки, которые влияют на скорость инициации транскрипции. Описанные в настоящем документе промоторные последовательности модулируют транскрипцию функционально связанного полинуклеотида. Промотор может быть активным в клетках одного или нескольких типов, раскрытых в настоящем документе (например, эукариотической клетке, клетке отличного от человека млекопитающего, клетке человека, клетке грызуна, плюрипотентной клетке, дифференцированной клетке или их комбинации). Промотор может быть, например, конститутивно активным промотором, условным промотором, индуцируемым промотором, ограниченным во времени промотором (например, регулируемым стадией развития промотором) или пространственно ограниченным промотором (например, специфичным для типа клеток или тканеспецифичным промотером). Примеры промоторов можно найти, например, в WO 2013/176772, включенной в настоящий документ посредством ссылки в полном ее объеме для всех целей.
[00100] Примеры индуцируемых промоторов включают, например, химически регулируемые промоторы и физически регулируемые промоторы. К химически регулируемым промоторам относятся, например, регулируемые спиртом промоторы (например, промотор гена алкогольдегидрогеназы (alcA)), регулируемые тетрациклином промоторы (например, чувствительный к тетрациклину промотор, последовательность тетрациклинового оператора (tetO), промотор tet-On или промотор tet-Off), регулируемые стероидами промоторы (например, промотор глюкокортикоидного рецептора крысы, промотор рецептора эстрогена или промотор рецептора экдизона) или регулируемые металлом промоторы (например, промотор металлосодержащего белка). К физически регулируемым промоторам относятся, например, регулируемые температурой промоторы (например, промотор теплового шока) и регулируемые светом промоторы (например, индуцируемый светом промотор или репрессируемый светом промотор).
[00101] Тканеспецифичными промоторами могут быть, например, нейронспецифичные промоторы, глияспецифичные промоторы, специфичные для мышечных клеток промоторы, специфичные для клеток сердца промоторы, специфичные для клеток почек промоторы, специфичные для костных клеток промоторы, специфичные для эндотелиальных клеток промоторы или специфичные для иммунных клеток промоторы (например, промотор В-клеток или промотор Т-клеток).
[00102] К регулируемым стадией развития промоторам относятся, например, промоторы, активные только во время эмбриональной стадии развития или только во взрослой клетке.
[00103] «Функциональная связь» или быть «функционально связанным» включает смежное положение двух или более компонентов (например, промотора и другого элемента последовательности) так, чтобы оба компонента нормально функционировали и обеспечивали возможность того, чтобы по меньшей мере один из компонентов мог опосредовать функцию, на которую оказывает воздействие по меньшей мере один из других компонентов. Например, промотор может быть функционально связан с кодирующей последовательностью, если промотор контролирует уровень транскрипции кодирующей последовательности в ответ на присутствие или отсутствие одного или нескольких регулирующих транскрипцию факторов. Функциональная связь может включать такие последовательности, которые являются смежными друг с другом или действуют в трансположении (например, регуляторная последовательность может действовать на расстоянии, управляя транскрипцией кодирующей последовательности).
[00104] Термин «праймер» относится к олигонуклеотиду, способному действовать в качестве точки инициации синтеза полинуклеотида вдоль комплементарной цепи при его нахождении в условиях, в которых катализируется синтез продукта элонгации праймера, комплементарного полинуклеотиду. Такие условия включают наличие четырех различных нуклеотидтрифосфатов или нуклеозидных аналогов и одного или нескольких средств для полимеризации, таких как ДНК-полимераза и/или обратная транскриптаза, в подходящем буфере (включающем заместители, которыми являются кофакторы, или которые влияют на рН, ионную силу и т.д.) и при подходящей температуре. Элонгация праймера специфичным для последовательности образом может включать, например, способы ПЦР, секвенирование ДНК, элонгацию ДНК, полимеризацию ДНК, транскрипцию РНК или обратную транскрипцию. Праймер должен быть достаточно длинным, чтобы запускать синтез продуктов элонгации в присутствии средства для полимеразы. Типичный праймер составляет по меньшей мере приблизительно 5 нуклеотидов в длину последовательности, практически комплементарной целевой последовательности, но предпочтительными являются более длинные праймеры. Как правило, праймеры составляют приблизительно 15-30 нуклеотидов в длину, но также можно использовать более длинные праймеры. Последовательность праймера не обязательно должна быть точно комплементарной матричной или целевой последовательности, но должна быть достаточно комплементарной для гибридизации с матричной или целевой последовательностью. Термин «пара праймеров» означает набор праймеров, включающий 5' верхний праймер, который гибридизируется с 5'-концом подлежащей амплификации последовательности ДНК, и 3' нижний праймер, который гибридизируется с комплементарной цепью 3'-конца подлежащей амплификации последовательности. Пары праймеров можно применять для амплификации целевого полинуклеотида (например, с помощью полимеразной цепной реакции (ПЦР) или других традиционных способов амплификации нуклеиновых кислот). «ПЦР» или «полимеразная цепная реакция» представляет собой методику, применяемую для амплификации конкретных сегментов ДНК (см. патенты США №№4683195 и 4800159, каждый из которых включен в настоящий документ посредством ссылки в полном своем объеме для всех целей).
[00105] Термин «зонд» относится к молекуле, которая позволяет детектируемым образом различать отличающиеся по структуре целевые молекулы. Детекцию можно осуществить различными способами в зависимости от типа применяемого зонда и типа целевой молекулы. Таким образом, например, детекция может быть основана на различении уровней активности целевой молекулы, но предпочтительно основана на детекции специфического связывания. Примеры такого специфического связывания включают связывание антител и гибридизацию зондов для нуклеиновых кислот. Так, к зондам можно отнести, например, субстраты для ферментов, антитела и фрагменты антител, а также зонды для гибридизации с нуклеиновыми кислотами. Например, зонд может представлять собой выделенный полинуклеотид, присоединенный к обычной детектируемой метке или репортерной молекуле, такой как радиоактивный изотоп, лиганд, хемилюминесцентное средство, фермент или тому подобное. Такой зонд комплементарен цепи целевого полинуклеотида, такого как полинуклеотид, содержащий вариант rs72613567 HSD17B13 или специфические мРНК-транскрипты HSD17B13. К зондам для дезоксирибонуклеиновой кислоты могут относиться зонды, полученные с помощью ПЦР с применением мРНК/кДНК-HSD17B13-специфических праймеров или rs72613567-HSD17B13-специфических праймеров, олигонуклеотидных зондов, синтезированных in vitro, или ДНК, полученной из библиотек бактериальных искусственных хромосом, фосмид или космид. Зонды включают не только дезоксирибонуклеиновую или рибонуклеиновую кислоты, но также полиамиды и другие материалы зондов, которые могут специфически детектировать наличие целевой последовательности ДНК. В случае зондов для нуклеиновых кислот детектирующие реагенты могут включать, например, меченые радиоактивным изотопом зонды, меченые ферментами зонды (например, пероксидазой хрена и щелочной фосфатазой), меченные аффинной меткой зонды (например, биотином, авидином и стрептавидином) и меченные флуоресцентной меткой зонды (например, 6-FAM, VIC, TAMRA, MGB, флуоресцеином, родамином и техасским красным). Описываемые в настоящем документе зонды для нуклеиновой кислоты можно легкой включить в один из общепринятых форматов набора, которые хорошо известны.
[00106] Термин «антисмысловая РНК» относится к одноцепочечной РНК, которая комплементарна цепи матричной РНК, транскрибированной в клетке.
[00107] Термин «малая интерферирующая РНК (siRNA)» относится, как правило, к двухцепочечной молекуле РНК, которая индуцирует путь РНК-интерференции (RNAi). Эти молекулы могут различаться по длине (обычно от 18 до 30 пар оснований) и иметь различную степень комплементарности своей целевой мРНК в антисмысловой цепи. Некоторые, но не все, молекулы siRNA имеют непарные выступающие основания на 5' или 3' конце смысловой цепи и/или антисмысловой цепи. Термин «siRNA» включает дуплексы из двух отдельных цепей, а также одиночные цепи, которые могут образовывать шпилечные структуры, содержащие дуплексный участок. Двухцепочечная структура может составлять, например, менее 20, 25, 30, 35, 40, 45 или 50 нуклеотидов в длину. Например, двухцепочечная структура может составлять от приблизительно 21 до 23 нуклеотидов в длину, от приблизительно 19 до 25 нуклеотидов в длину или от приблизительно 19 до 23 нуклеотидов в длину.
[00108] Термин «короткая шпилечная РНК (shRNA)» относится к одной цепи оснований РНК, которая самогибридизируется в шпилечную структуру шпильки и может индуцировать путь РНК-интерференции (RNAi) при процессинге. Такие молекулы могут различаться по длине (обычно приблизительно 50-90 нуклеотидов в длину или в некоторых случаях до более чем 250 нуклеотидов в длину, например, в случае микроРНК-адаптированной shRNA). Молекулы shRNA процессируются в клетке с образованием молекул siRNA, которые, в свою очередь, могут вызывать нокдаун экспрессии генов. Молекулы shRNA могут быть включены в векторы. Термин «shRNA» также относится к молекуле ДНК, с которой может быть транскрибирована молекула короткой шпилечной РНК.
[00109] «Комплементарность» нуклеиновых кислот означает, что нуклеотидная последовательность в одной цепи нуклеиновой кислоты, благодаря ориентации групп ее нуклеиновых оснований, образует водородные связи с другой последовательностью на противоположной цепи нуклеиновой кислоты. Комплементарными основаниями в ДНК обычно являются А и Т, а также С и G. В РНК это обычно С и G, а также U и А. Комплементарность может быть совершенной или существенной/достаточной. Совершенная комплементарность между двумя нуклеиновыми кислотами означает, что две нуклеиновые кислоты могут образовывать дуплекс, в котором каждое основание в дуплексе связано с комплементарным основанием в результате спаривания по Уотсону-Крику. «Существенно» или «достаточно» комплементарный означает, что последовательность в одной цепи не является полностью и/или совершенно комплементарной последовательности в противоположной цепи, но что между основаниями на двух цепях происходит достаточное связывание для образования стабильного гибридного комплекса в ряде условий гибридизации (например, концентрации солей и температуре). Такие условия можно спрогнозировать с помощью последовательностей и стандартных математических расчетов для прогнозирования Tm (температуры плавления) гибридизированных цепей или путем эмпирического определения Tm с помощью стандартных способов. Tm включает температуру, при которой группа гибридизационных комплексов, образованных между двумя цепями нуклеиновой кислоты, денатурируется на 50% (т.е. группа двухцепочечных молекул нуклеиновой кислоты становится наполовину диссоциированной на отдельные цепи). При температуре ниже Tm условия способствуют образованию гибридизационного комплекса, тогда как при температуре выше Tm условия способствуют плавлению или разделению цепей в гибридизационном комплексе. Tm можно оценить для нуклеиновой кислоты, имеющей известное содержание G+C в водном 1 М растворе NaCl, с помощью, например, Tm=81,5+0,41 (%G+C), хотя в других известных вычислениях Tm учитываются структурные характеристики нуклеиновой кислоты.
[00110] «Условие гибридизации» включает совокупные условия среды, при которых одна цепь нуклеиновой кислоты связывается со второй цепью нуклеиновой кислоты посредством взаимодействия комплементарных цепей и формирования водородных связей с образованием гибридизационного комплекса. Такие условия включают химические компоненты и их концентрации (например, солей, хелатирующих средств, формамида) в водном или органическом растворе, содержащем нуклеиновые кислоты, и температуру смеси. На условия среды могут влиять и другие факторы, такие как продолжительность инкубации или размеры реакционной камеры. См., например, Sambrook et al., Molecular Cloning, A Laboratory Manual, 2.sup.nd ed., pp. 1.90-1.91, 9.47-9.51, 11.47-11.57 (Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 1989), включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00111] При гибридизации необходимо, чтобы две нуклеиновые кислоты содержали комплементарные последовательности, хотя между основаниями возможны несоответствия. Условия, подходящие для гибридизации между двумя нуклеиновыми кислотами, зависят от длины нуклеиновых кислот и степени комплементарности, переменных, которые хорошо известны. Чем больше степень комплементарности между двумя нуклеотидными последовательностями, тем больше значение температуры плавления (Tm) для гибридов нуклеиновых кислот, имеющих такие последовательности. Для гибридизации между нуклеиновыми кислотами с короткими участками комплементарности (например, с комплементарностью на протяжении 35 или менее, 30 или менее, 25 или менее, 22 или менее, 20 или менее или 18 или менее нуклеотидов) важным становится положение несоответствий (см. Sambrook et al. al., ранее, 11.7-11.8). Как правило, длина гибридизирующейся нуклеиновой кислоты составляет по меньшей мере приблизительно 10 нуклеотидов. К иллюстративным минимальным значениям длины для гибридизирующейся нуклеиновой кислоты относятся по меньшей мере приблизительно 15 нуклеотидов, по меньшей мере приблизительно 20 нуклеотидов, по меньшей мере приблизительно 22 нуклеотидов, по меньшей мере приблизительно 25 нуклеотидов и по меньшей мере приблизительно 30 нуклеотидов. Кроме того, температуру и концентрацию солей в промывочном растворе можно корректировать по мере необходимости в соответствии с такими факторами, как длина участка комплементарности и степень комплементарности.
[00112] Последовательность полинуклеотида не должна быть на 100% комплементарной последовательности его целевой нуклеиновой кислоты, чтобы быть специфически гибридизирующейся. Кроме того, полинуклеотид может гибридизироваться с одним или несколькими сегментами так, чтобы промежуточные или смежные сегменты не участвовали в событии гибридизации (например, в случае петлевой структуры или шпилечной структуры). Полинуклеотид (например, gRNA) может характеризоваться, по меньшей мере 70%, по меньшей мере 80%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 99% или 100% комплементарностью последовательности к целевому участку в последовательности целевой нуклеиновой кислоты, на которую они нацелены. Например, gRNA, в которой 18 из 20 нуклеотидов являются комплементарными целевому участку и, следовательно, будет специфически гибридизироваться, будет характеризоваться 90% комплементарностью. В этом примере оставшиеся некомплементарные нуклеотиды могут образовывать кластеры или перемежаться с комплементарными нуклеотидами и не должны обязательно быть смежными друг с другом или с комплементарными нуклеотидами.
[00113] Процент комплементарности между отдельными участками последовательностей нуклеиновых кислот в нуклеиновых кислотах можно определить стандартным способом с помощью программ BLAST (средства поиска основного локального выравнивания) и программ PowerBLAST (Altschul et al. (1990) J. Mol. Biol. 215:403-410; Zhang and Madden (1997) Genome Res. 7:649-656) или с помощью программы Gap (Wisconsin Sequence Analysis Package, версия 8 для Unix, Genetics Computer Group, University Research Park, Мэдисон, Висконсин) с применением настроек по умолчанию, в которых используется алгоритм Смита и Уотермана (Adv. Appl. Math., 1981, 2, 482-489).
[00114] В способах и композициях, представленных в настоящем документе, используют множество различных компонентов. Некоторые компоненты по всему описанию могут иметь активные варианты и фрагменты. Такие компоненты включают, например, белки Cas9, РНК CRISPR, tracrRNA и гидовые РНК. Биологическая активность для каждого из этих компонентов описана в других разделах настоящего документа.
[00115] «Идентичность последовательности» или «идентичность» в контексте двух полинуклеотидных или полипептидных последовательностей относится к остаткам в двух последовательностях, которые являются одинаковыми при выравнивании для максимального соответствия на протяжении указанного промежутка сравнения. При использовании процента идентичности последовательности в отношении белков положения остатков, которые не являются идентичными, зачастую отличаются по консервативным аминокислотным заменам, причем аминокислотные остатки заменены другими аминокислотными остатками со сходными химическими свойствами (например, зарядом или гидрофобностью) и, следовательно, не изменяют функциональные свойства молекулы. Если последовательности отличаются по консервативным заменам, процент идентичности последовательностей можно скорректировать в большую сторону для поправки на консервативную природу замены. Говорят, что последовательности, которые отличаются такими консервативными заменами, имеют «схожесть последовательностей» или «схожесть». Средства для внесения такой корректировки хорошо известны. Как правило, она предусматривает оценку консервативной замены как частичного, а не полного несоответствия, что увеличивает процент идентичности последовательностей. Таким образом, например, если идентичной аминокислоте присваивают оценку, равную 1, а неконсервативной замене присваивают оценку, равную нулю, консервативной замене присваивают оценку между нулем и 1. Оценку консервативных замен рассчитывают, например, так, как реализовано в программе PC/GENE (Intelligenetics, Маунтин-Вью, Калифорния).
[00116] «Процент идентичности последовательностей» включает значение, определенное путем сравнения двух оптимально выровненных последовательностей (наибольшее количество идеально совпадающих остатков) в интервале сравнения, причем часть полинуклеотидной последовательности в интервале сравнения может содержать добавления или делеции (т.е. гэпы) в сравнении с эталонной последовательностью (которая не содержит добавления или делеции) для оптимального выравнивания данных двух последовательностей. Процент рассчитывают путем определения количества положений, в которых идентичное основание нуклеиновой кислоты или аминокислотный остаток встречается в обеих последовательностях, с получением количества совпадающих положений, деления количества совпадающих положений на общее количество положений в интервале сравнения и умножения результата на 100 с получением процента идентичности последовательностей. Если не указано иное (например, более короткая последовательность включает связанную гетерологичную последовательность), интервал сравнения является полноразмерной от более короткой из двух сравниваемых последовательностей.
[00117] Если не указано иное, значения идентичности/сходства последовательностей включают значение, полученное с помощью GAP версии 10 с применением следующих параметров: % идентичности и % сходства для нуклеотидной последовательности с применением штрафа за открытие гэпа 50 и штрафа за продолжение гэпа 3 и матрицы замен nwsgapdna.cmp; % идентичности и % сходства для аминокислотной последовательности с применением штрафа за открытие гэпа 8 и штрафа за продолжение гэпа 2 и матрицы замен BLOSUM62; или любой эквивалентной ей программы. «Эквивалентная программа» включает любую программу для сравнения последовательностей, которая для любых двух рассматриваемых последовательностей создает выравнивание, имеющее идентичные совпадения нуклеотидных или аминокислотных остатков и выдает процент идентичности последовательностей в сравнении с соответствующим выравниванием, созданным GAP версии 10.
[00118] Термин «консервативная аминокислотная замена» относится к замене аминокислоты, которая в норме присутствует в последовательности, на другую аминокислоту схожего размера, заряда или полярности. Примеры консервативных замен включают замену неполярного (гидрофобного) остатка, такого как изолейцин, валин или лейцин, на другой неполярный остаток. Аналогичным образом, примеры консервативных замен включают замену одного полярного (гидрофильного) остатка на другой, например, между аргинином и лизином, между глутамином и аспарагином или между глицином и серином. Кроме того, дополнительными примерами консервативных замен являются замена основного остатка, такого как лизин, аргинин или гистидин, на другой или замена одного кислотного остатка, такого как аспарагиновая кислота или глутаминовая кислота, на другой кислотный остаток. Примеры неконсервативных замен включают замену неполярного (гидрофобного) аминокислотного остатка, такого как изолейцин, валин, лейцин, аланин или метионин, на полярный (гидрофильный) остаток, такой как цистеин, глутамин, глутаминовая кислота или лизин, и/или полярного остатка на неполярный остаток. Ниже приведены типичные категории аминокислот.
[00119] Рассматриваемая нуклеиновая кислота, такая как праймер или гидовая РНК, гибридизируется, или нацелена на положение, или включает положение, находящееся в непосредственной близости к указанному положению нуклеотида в эталонной нуклеиновой кислоте, когда она находится в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от данного положения.
[00120] Термин «биологический образец» относится к образцу биологического материала, находящемуся внутри или полученному от субъекта, из которого выделают нуклеиновую кислоту или белок. Термин «биологический образец» также может охватывать любой материал, полученный путем обработки образца, такого как клетки или их потомство. Обработка биологического образца может включать одно или несколько из фильтрации, дистилляции, экстракции, концентрации, фиксации, инактивации мешающих компонентов и т.п. В соответствии с некоторыми вариантами осуществления, биологический образец содержит нуклеиновую кислоту, такую как геномная ДНК, кДНК или мРНК. В соответствии с некоторыми вариантами осуществления, биологический образец содержит белок. Субъектом может быть любой организм, в том числе, например, человек, отличное от человека млекопитающее, грызун, мышь или крыса. Биологический образец может быть получен из любой клетки, ткани или биологической жидкости, взятой у субъекта. Образец может содержать любую клинически значимую ткань, такую как образец костного мозга, биоптат опухоли, тонкоигольный пунктат или образец телесной жидкости, такой как кровь, плазма, сыворотка, лимфа, асцитическая жидкость, кистозная жидкость или моча. В некоторых случаях образец содержит щечный мазок. Образец, применяемый в раскрываемых в настоящем документе способах, будет варьировать в зависимости от формата анализа, природы способа детекции и тканей, клеток или экстрактов, которые применяют в качестве образца.
[00121] Термин «контрольный образец» относится к образцу, полученному от субъекта, который не имеет варианта rs72613567 HSD17B13 и предпочтительно является гомозиготным по аллелю дикого типа гена HSD17B13. Такие образцы можно получить одновременно с биологическим образцом или по другому случаю. Как биологический образец, так и контрольный образец могут быть получены из одной и той же ткани или жидкости организма.
[00122] «Гомологичная» последовательность (например, последовательность нуклеиновой кислоты) включает последовательность, которая либо идентична, либо обладает существенным сходством с известной эталонной последовательностью, так чтобы она была, например, по меньшей мере на 50%, по меньшей мере на 55%, по меньшей мере на 60%, по меньшей мере на 65%, по меньшей мере на 70%, по меньшей мере на 75%, по меньшей мере на 80%, по меньшей мере на 85%, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична известной эталонной последовательности. Гомологичные последовательности могут включать, например, ортологичные последовательности и паралогичные последовательности. Например, гомологичные гены обычно происходят от общей предковой последовательности ДНК либо посредством события видообразования (ортологичные гены), либо посредством события генетической дупликации (паралогичные гены). К «ортологичным» генам относятся гены у различных видов, которые эволюционировали от общего предкового гена путем видообразования. Ортологи обычно сохраняют одну и ту же функцию в ходе эволюции. К «паралогичным» генам относятся гены, связанные дупликацией в геноме. У паралогов могут в ходе эволюции развиться новые функции.
[00123] Термин «in vitro» включает искусственные условиях среды и относится к процессам или реакциям, которые происходят в искусственной среде (например, в пробирке). Термин «in vivo» включает естественные условия среды (например, клетку, или организм, или многоклеточный организм, такой как клетка в организме или теле) и относится к процессам или реакциям, которые происходят в естественной среде. Термин «ех vivo» включает клетки, которые были удалены из организма человека, а также относится к процессам или реакциям, которые происходят внутри таких клеток.
[00124] Композиции или способы, «содержащие» или «включающие» один или несколько перечисленных элементов, могут включать другие элементы, которые конкретно не указаны. Например, композиция, которая «содержит» или «включает» белок, может содержать белок отдельно или в комбинации с другими ингредиентами. Переходная фраза «фактически состоящий из» означает, что объем пункта формулы изобретения следует интерпретировать как охватывающий указанные элементы, перечисленные в данном пункте формулы изобретения, и элементы, которые не оказывают существенного влияния на основную(-ые) и новую(-ые) характеристику(-и) заявляемого изобретения. Таким образом, термин «фактически состоящий из» при использовании в пункте формулы изобретения не следует интерпретировать как эквивалентный термину «включающий».
[00125] «Необязательный» или «необязательно» означает, что описанное далее событие или обстоятельство может иметь место или может не иметь место, и что описание включает случаи, когда такое событие или обстоятельство имеет место, и случаи, в которых оно не имеет место быть.
[00126] Обозначение диапазона значений включает все целые числа в пределах или определяющие диапазон и все поддиапазоны, определяемые целыми числами в пределах диапазона.
[00127] Если иное не очевидно из контекста, термин «приблизительно» охватывает значения в пределах стандартной погрешности измерения (например, SEM) указанного значения.
[00128] Термин «и/или» относится и охватывает все возможные комбинации одного или нескольких связанных перечисленных элементов, а также отсутствие комбинаций при интерпретации в альтернативе («или»).
[00129] Термин «или» относится к любому одному членов из конкретного перечня, а также включает любую комбинацию членов из данного перечня.
[00130] Формы единственного числа включают ссылки и на формы множественного числа, если контекст явно не предписывает иное. Например, термин «белок Cas9» или «по меньшей мере один белок Cas9» может включать множество белков Cas9, включая их смеси.
[00131] Статистически значимое означает р≤0,05.
Подробное раскрытие настоящего изобретения
I. Обзор
[00132] Настоящее изобретение относится к варианту HSD17B13, который, как было обнаружено, ассоциирован со сниженными уровнями аланинтрансаминазы и аспартаттрансаминазы; сниженным риском развития хронических заболеваний печени, в том числе неалкогольной и алкогольной болезни печени, ожирения печени, цирроза и гепатоклеточной карциномы; и сниженным прогрессированием от простого стеатоза до более клинически запущенных стадий хронического заболевания печени. Также настоящее изобретение относится к ранее неизвестным транскриптам гена HSD17B13, ассоциированным с данным вариантом.
[00133] Настоящее изобретение относится к выделенным нуклеиновым кислотам и белкам, относящимся к вариантам HSD17B13, и клеткам, содержащим эти нуклеиновые кислоты и белки. Также настоящее изобретение относится к способам модификации клетки посредством применения любой комбинации из нуклеазных средств, экзогенных донорных последовательностей, активаторов транскрипции, репрессоров транскрипции и векторов экспрессии для экспрессии рекомбинантного гена HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13. Также настоящее изобретение относится к терапевтическим и профилактическим способам лечения субъекта, имеющего хроническое заболевание печени или риск его развития.
II. Варианты HSD17B13
[00134] Настоящее изобретение относится к выделенным нуклеиновым кислотам и белкам, относящимся к вариантам HSD17B13 (также известным как гидроксистероид-17-бета-дегидрогеназа 13, 17-бета-гидроксистероиддегидрогеназа 13, 17β-гидроксистероиддегидрогеназа-13, 17β-HSD13, короткоцепочечная дегидрогеназа/редуктаза 9, SCDR9, HMFN0376, NIIL497 и SDR16C3). Ген HSD17B13 человека составляет примерно 19 т.о. в длину и включает семь экзонов и шесть интронов, расположенных в 4q22.1 в геноме. Иллюстративным последовательностям белка HSD17B13 человека присвоен регистрационный номер в UniProt №Q7Z5P4 (SEQ ID NO: 240 и 241; Q7Z5P4-1 и Q7Z5P4-2 соответственно) и номера эталонных последовательностей в NCBI NP_835236 и NP_001129702 (SEQ ID NO: 242 и 243 соответственно). Иллюстративным мРНК HSD17B13 человека присвоены номера эталонных последовательностей в NCBI NM_178135 и NM_001136230 (SEQ ID NO: 244 и 245 соответственно).
[00135] Так, в частности, настоящее изобретение относится к сплайс-варианту HSD17B13 (rs72613567), имеющему вставку аденина рядом с донорным сплайс-сайтом в интроне 6. Аденин является вставкой на прямой (плюс) цепи хромосомы, которая соответствует вставленному тимину на обратной (минус) цепи хромосомы. Так как ген HSD17B13 человека транскрибируется в обратном направлении, эта нуклеотидная вставка отображена как вставленный тимин в иллюстративной последовательности варианта rs72613567 HSD17B13 под SEQ ID NO: 2 относительно иллюстративной последовательности гена HSD17B13 дикого типа, представленной под SEQ ID NO: 1. Поэтому вставка будет называться в настоящем документе тимином, вставленным между положениями 12665 и 12666 в SEQ ID NO: 1 или в положении 12666 в SEQ ID NO: 2.
[00136] Ранее было выявлено, что у субъектов с геном HSD17B13 дикого типа экспрессируются два мРНК-транскрипта (А и В; SEQ ID NO: 4 и 5 соответственно). Транскрипт А включает все семь экзонов гена HSD17B13, в то время как в транскрипте В пропущен экзон 2. Транскрипт А является доминантным транскриптом у субъектов с геном дикого типа. Настоящее изобретение, однако, относится к шести дополнительным, ранее неизвестным экспрессируемым транскриптам HSD17B13 (С-Н, SEQ ID NO: 6-11 соответственно). Эти транскрипты показаны на фиг. 4. В транскрипте С пропущен экзон 6 в сравнении с транскриптом А. В транскрипте D присутствует вставка гуанина 3' от экзона 6, что приводит в результате к сдвигу рамки считывания и преждевременному укорочению экзона 7 в сравнении с транскриптом А. В транскрипте Е присутствует дополнительной экзон между экзонами 3 и 4 в сравнении с транскриптом А. В транскрипте F, который экспрессируется только у носителей варианта rs72613567 HSD17B13, присутствует участок считывания от экзона 6 в интрон 6 в сравнении с транскриптом А. В транскрипте G пропущен экзон 2 и присутствует вставка гуанина 3' от экзона 6, что приводит в результате к сдвигу рамки считывания и преждевременному укорочению экзона 7 в сравнении с транскриптом А. В транскрипте Н присутствует дополнительный экзон между экзонами 3 и 4 и присутствует вставка гуанина 3' от экзона 6, что приводит в результате к сдвигу рамки считывания и преждевременному укорочению экзона 7 в сравнении с транскриптом А. Транскрипты С, D, F, G и Н являются доминантными у носителей варианта rs72613567 HSD17B13, при этом транскрипт D является наиболее распространенным транскриптом у носителей варианта rs72613567 HSD17B13. Также настоящее изобретение относится к одному дополнительному, ранее неизвестному транскрипту HSD17B13, который характеризуется низкими уровнями экспрессии (F', SEQ ID NO: 246). Как и транскрипт F, транскрипт F' также включает участок считывания от экзона 6 в интрон 6 в сравнении с транскриптом А, но, в отличие от транскрипта F, считывание не включает вставленный тимин, присутствующий в варианте rs72613567 гена HSD17B13. Ниже представлены нуклеотидные положения экзонов в генах HSD17B13 для каждого транскрипта.
[00137]
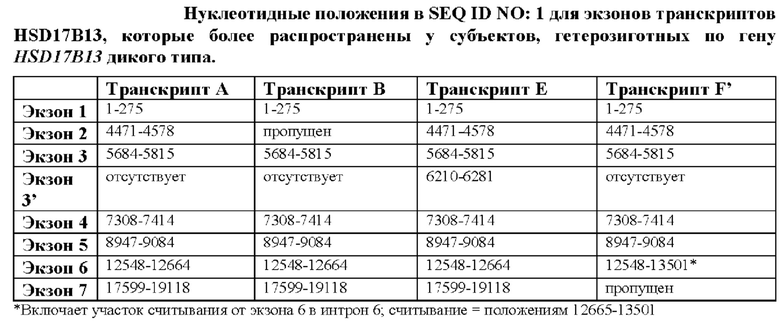
[00138]
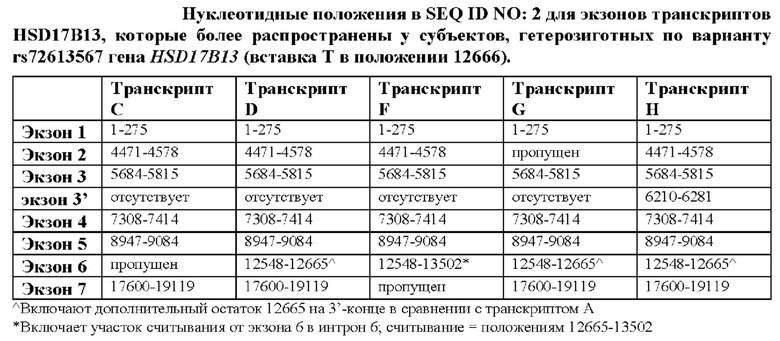
[00139] Как более подробно поясняется в других разделах настоящего документа, вариант rs72613567 HSD17B13 ассоциирован со сниженными уровнями аланинтрансаминазы и аспартаттрансаминазы и сниженным риском развития хронических заболеваний печени, в том числе неалкогольной и алкогольной жировой болезни печени, цирроза и гепатоклеточной карциномы. Вариант rs72613567 HSD17B13 также ассоциирован с уменьшенным прогрессированием от простого стеатоза до более клинически запущенных стадий хронического заболевания печени.
А. Нуклеиновые кислоты
[00140] В настоящем документе раскрыты выделенные нуклеиновые кислоты, относящиеся к вариантам HSD17B13 и вариантам транскриптов HSD17B13. Также раскрыты выделенные нуклеиновые кислоты, которые гибридизируются в жестких или умеренных условиях с любой из раскрываемых в настоящем документе нуклеиновых кислот. Такие нуклеиновые кислоты могут быть пригодны, например, для экспрессии вариантов белков HSD17B13 или в качестве праймеров, зондов, экзогенных донорных последовательностей, гидовых РНК, антисмысловых РНК, shRNA и siRNA, каждая из которых более подробно описана в других разделах настоящего документа.
[00141] Также раскрыты функциональные нуклеиновые кислоты, которые могут взаимодействовать с раскрытыми полинуклеотидами. Функциональные нуклеиновые кислоты представляют собой молекулы нуклеиновых кислот, которые выполняют определенную функцию, такую как связывание целевой молекулы или катализ конкретной реакции. Примеры функциональных нуклеиновых кислот включают антисмысловые молекулы, аптамеры, рибозимы, молекулы, образующие триплекс, и внешние гидовые последовательности. Функциональные молекулы нуклеиновой кислоты могут выполнять роль эффекторов, ингибиторов, модуляторов и стимуляторов специфической активности, которыми обладает целевая молекула, или функциональные молекулы нуклеиновой кислоты могут обладать de novo активностью, независящей от любых других молекул.
[00142] Антисмысловые молекулы сконструированы для взаимодействия с целевой молекулой нуклеиновой кислоты посредством либо канонического, либо неканонического спаривания оснований. Взаимодействие антисмысловой молекулы и целевой молекулы сконструирована для индукции нарушения структуры целевой молекулы посредством, например, опосредованного РНКазой-Н разложения гибрида РНК-ДНК. В качестве альтернативы, антисмысловая молекула сконструирована для прерывания функции процессирования, которая в норме происходила бы на целевой молекуле, такой как транскрипция или репликация. Антисмысловые молекулы можно сконструировать на основе последовательности целевой молекулы. Существуют многочисленные способы оптимизации антисмысловой эффективности путем поиска наиболее доступных существующих участков целевой молекулы. Иллюстративными способами могут быть эксперименты по отбору in vitro и исследования модификации ДНК с применением DMS и DEPC. Антисмысловые молекулы обычно связывают целевую молекулу с константой диссоциации (kd), меньшей или равной 10-6, 10-8, 10-10 или 10-12. Репрезентативную выборку способов и методик, которые помогают в конструировании и применении антисмысловых молекул, можно найти в следующем неограничивающем перечне патентов США: 5135917, 5294533, 5627158, 5641754, 5691317, 5780607, 5786138, 5849903, 5856103, 5919772, 5955590, 5990088, 5994320, 5998602, 6005095, 6007995,6013522,6017898, 6018042, 6025198, 6033910, 6040296, 6046004, 6046319 и 6057437, каждый из которых включен в настоящий документ посредством ссылки в полном своем объеме для всех целей. Примеры антисмысловых молекул включают антисмысловые РНК, малые интерферирующие РНК (siRNA) и короткие шпилечные РНК (shRNA), которые более подробно описаны в других разделах настоящего документа.
[00143] Раскрываемые в настоящем документе выделенные нуклеиновые кислоты могут содержать РНК, ДНК или как РНК, так и ДНК. Выделенные нуклеиновые кислоты также можно связать или слить с гетерологичной последовательностью нуклеиновой кислоты, например, в векторе или с гетерологичной меткой. Например, выделенные нуклеиновые кислоты, раскрываемые в настоящем документе, могут находиться в векторе или в экзогенных донорных последовательностях, содержащих выделенную нуклеиновую кислоту и гетерологичную последовательность нуклеиновой кислоты. Выделенные нуклеиновые кислоты также можно связать или слить с гетерологичной меткой, такой как флуоресцентная метка. Другие примеры меток раскрыты в других разделах настоящего документа.
[00144] Раскрываемые молекулы нуклеиновых кислот могут состоять, например, из нуклеотидов или не встречающихся в естественной среде или модифицированных нуклеотидов, таких как нуклеотидные аналоги или нуклеотидные заменители. Такие нуклеотиды включают нуклеотид, который содержит модифицированное основание, сахар или фосфатную группу или который включает в своей структуре не встречающийся в естественной среде фрагмент. Примеры не встречающихся в естественной среде нуклеотидов включают дидезоксинуклеотиды, биотинилированные, аминированные, дезаминированные, алкилированные, бензилированные и меченные флуорофором нуклеотиды.
[00145] Раскрываемые в настоящем документе молекулы нуклеиновых кислот могут содержать один или несколько нуклеотидных аналогов или заменителей. Нуклеотидный аналог представляет собой нуклеотид, который содержит модификацию определенного типа в любом из основного, сахарного или фосфатного фрагментов. Модификации в основном фрагменте будут включать природные и синтетические модификации А, С, G и T/U, а также различные пуриновые или пиримидиновые основания, такие как псевдоуридин, урацил-5-ил, гипоксантил-9-ил (I) и 2-аминоаденин-9-ил. Модифицированные основания включают, например, 5-метилцитозин (5-me-С), 5-гидроксиметилцитозин, ксантин, гипоксантин, 2-аминоаденин, 6-метильные и другие алкильные производные аденина и гуанина, 2-пропильные и другие алькильные производные аденина и гуанина, 2-тиоурацил, 2-тиотимин и 2-тиоцитозин, 5-галогенурацил и цитозин, 5-пропинилурацил и 5-пропинилцитозин, 6-азоурацил, 6-азоцитозин и 6-азотимин, 5-урацил (псевдоурацил), 4-тиоурацил, 8-галоген, 8-амино, 8-тиол, 8-тиоалкил, 8-гидроксил и другие 8-замещенные аденины и гуанины, 5-галоген, особенно 5-бром, 5-трифторметил и другие 5-замещенные урацилы и цитозины, 7-метилгуанин и 7-метиладенин, 8-азагуанин и 8-азааденин, 7-дезазагуанин и 7-дезазааденин и 3-дезазагуанин и 3-дезазааденин. Дополнительные модификации оснований можно найти, например, в патенте США №3687808; Englisch et al. (1991) Angewandte Chemie, International Edition 30:613; и Sanghvi, Y. S., Chapter 15, Antisense Research and Applications, pages 289-302, Crooke, S. T. and Lebleu, B. ed., CRC Press, 1993, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Определенные нуклеотидные аналоги, такие как 5-замещенные пиримидины, 6-азапиримидины и N-2-, N-6- и O-6-замещенные пурины, включая 2-аминопропиладенин, 5-пропинилурацил, 5-пропинилцитозин и 5-метилцитозин, могут повышать стабильность образования дуплекса. Зачастую модификации оснований можно совмещать, например, с модификацией сахара, такой как 2'-О-метоксиэтил, для достижения уникальных свойств, таких как увеличенная стабильность дуплекса. Есть множество патентов США, таких как 4845205, 5130302, 5134066, 5175273, 5367066, 5432272, 5457187, 5459255, 5484908, 5502177, 5525711, 5552540, 5587469, 5594121, 5596091, 5614617 и 5681941, в которых подробно описан спектр модификаций оснований. Каждый из них включен в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00146] Нуклеотидные аналоги также могут включать модификации сахарного фрагмента. Модификации в сахарном фрагмента могут включать, например, естественные модификации рибозы и дезоксирибозы, а также синтетические модификации. Модификации в сахаре включают, например, следующие модификации в 2'-положении: ОН; F; О-, S- или N-алкил; О-, S- или N-алкенил; О-, S- или N-алкинил; или О-алкил-О-алкил, при этом алкил, алкенил и алкинил могут быть замещенными или незамещенными С1-С10 алкилом или С2-С10 алкенилом и алкинилом. Иллюстративные 2' модификации в сахаре также включают, например, -O[(CH2)n O]m СН3, -O(CH2)nOCH3, -O(CH2)n NH2, -O(CH2)n СН3, -O(СН2)n -ONH2 и -O(CH2)nON[(CH2)nCH3)]2, где n и m равны от 1 до приблизительно 10.
[00147] Другие модификации в 2' положении включают, например, C1-С10 низший алкил, замещенный низший алкил, алкарил, аралкил, О-алкарил или О-аралкил, SH, SCH3, OCN, Cl, Br, CN, CF3, OCF3, SOCH3, SO2 СН3, ONO2, NO2, N3, NH2, гетероциклоалкил, гетероциклоалкарил, аминоалкиламино, полиалкиламино, замещенный силил, РНК-расщепляющую группу, репортерную группу, интеркалятор, группу для улучшения фармакокинетических свойств олигонуклеотида или группу для улучшения фармакодинамических свойств олигонуклеотида и другие заместители, имеющие схожие свойства. Схожие модификации также можно осуществить и в других положениях на сахаре, в частности, 3' положении сахара на 3'-концевом нуклеотиде или в 2'-5' связанных олигонуклеотидах и 5' положении 5'-концевого нуклеотида. К модифицированным сахарам также можно отнести сахара, которые содержат модификации в мостиковом кислороде кольца, такие как СН2 и S. Сахарные аналоги в нуклеотиде могут также содержать сахаромиметики, такие как циклобутильные фрагменты вместо пентофуранозильного сахара. Существует множество патентов США, в которых описано получение таких структур с модифицированным сахаром, такие как 4981957, 5118800, 5319080, 5359044, 5393878, 5446137, 5466786, 5514785, 5519134, 5567811, 5576427, 5591722, 5597909, 5610300, 5627053, 5639873, 5646265, 5658873, 5670633 и 5700920, каждый из которых включен в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00148] Нуклеотидные аналоги также могут быть модифицированы по фосфатному фрагменту. Модифицированные фосфатные фрагменты включают, например, фрагменты, которые могут быть модифицированы так, чтобы связь между двумя нуклеотидами включала фосфотиоат, хиральный фосфотиоат, фосфодитиоат, фосфотриэфир, аминоалкилфосфотриэфир, метил- и другие алкилфосфонаты, включая 3'-алкиленфосфонат и хиральные фосфонаты, фосфинаты, фосфоамидаты, включая 3'-аминофосфоамидат и аминоалкилфосфоамидаты, тионофосфоамидаты, тионоалкилфосфонаты, тионоалкилфосфотриэфиры и боранофосфаты. Такая фосфатная или модифицированная фосфатная связь между двумя нуклеотидами может проходить по 3'-5'-связи или 2'-5' -связи, и связь может иметь обратную полярность, такую как 3'-5' в 5'-3' или 2'-5' в 5'-2'. Также включены различные соли, смешанные соли и формы свободных кислот. Во многих патентах США раскрыто, каким образом получать и применять нуклеотиды, содержащие модифицированные фосфаты, и к ним относятся, например, 3687808, 4469863, 4476301, 5023243, 5177196, 5188897, 5264423, 5276019, 5278302, 5286717, 5321131, 5399676, 5405939, 5453496, 5455233, 5466677, 5476925, 5519126, 5536821, 5541306, 5550111, 5563253, 5571799, 5587361 и 5625050, каждый из которых включен в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00149] Нуклеотидные замены включают молекулы, имеющие функциональные свойства, схожие с нуклеотидами, но не содержащие фосфатный фрагмент, такие как пептид-нуклеиновая кислота (PNA). К нуклеотидным заменам относятся молекулы, которые будут распознавать нуклеиновые кислоты по принципу Уотсона-Крика или Хугстина, но которые связаны друг с другом через фрагмент, отличный от фосфатного фрагмента. Нуклеотидные замены могут соответствовать структуре типа двойной спирали при взаимодействии с соответствующей целевой нуклеиновой кислотой.
[00150] К нуклеотидным заменам также относятся нуклеотиды или нуклеотидные аналоги, в которых были заменены фосфатный фрагмент или сахарные фрагменты. Нуклеотидные замены могут не содержать стандартный атом фосфора. Заменами фосфата могут быть, например, короткоцепочечные алкильные или циклоалкильные межнуклеозидные связи, смешанные, содержащие гетероатом и алкильные или циклоалкильные межнуклеозидные связи или одна или несколько короткоцепочечных, содержащих гетероатом или гетероциклических межнуклеозидных связей. К ним относятся таковые с морфолиносвязями (образованные частично из сахарной части нуклеозида), силоксановые остовы, сульфидные, сульфоксидные и сульфоновые остовы, формацетильные и тиоформацетильные остовы, метиленформацетильные и тиоформацетильные остовы, алкенсодержащие остовы, сульфаматные остовы, метилениминовые и метиленгидразиновые остовы, сульфонатные и сульфонамидные остовы, амидные остовы и другие, имеющие смешанные N, О, S и СН2 составляющие части. В многих патентах США раскрыто, каким образом получить и применять фосфатные замены таких типов, и к ним относятся без ограничения 5034506, 5166315, 5185444, 5214134, 5216141, 5235033, 5264562, 5264564, 5405938, 5434257, 5466677, 5470967, 5489677, 5541307, 5561225, 5596086, 5602240, 5610289, 5602240, 5608046, 5610289, 5618704, 5623070, 5663312, 5633360, 5677437 и 5677439, каждый из которых включен в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00151] Также понятно, что в нуклеотидной замене как сахарный, так и фосфатный фрагменты нуклеотида могут быть заменены, например, связью амидного типа (аминоэтилглицином) (PNA). В патентах США 5539082, 5714331 и 5719262 раскрыто, как получить и применять молекулы PNA, каждый из которых включена в настоящее описание посредством ссылки во всей своей полноте для всех целей. См. также Nielsen et al. (1991) Science 254:1497-1500, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00152] Также с нуклеотидами или нуклеотидными аналогами можно связать молекулы и других типов (конъюгаты) для усиления, например, поглощения клеткой. Конъюгаты могут быть химически связаны с нуклеотидом или нуклеотидными аналогами. Такие конъюгаты включают, например, липидные фрагменты, такие как холестериновый фрагмент (Letsinger et al. (1989) Proc. Natl. Acad. Sci. USA 86:6553-6556, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей), холевую кислоту (Manoharan et al. (1994) Bioorg. Med. Chem. Let. 4:1053-1060, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей), тиоэфир, такой как гексил-S-тритилтиол (Manoharan et al. (1992) Ann. NY. Acad. Sci. 660:306-309; Manoharan et al. (1993) Bioorg. Med. Chem. Let. 3:2765-2770, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей), тиохолестерин (Oberhauser et al. (1992) Nucl. Acids Res. 20:533-538, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей), алифатическую цепь, такую как додекандиольные или ундецильные остатки (Saison-Behmoaras et al. (1991) EMBO J. 10:1111-1118; Kabanov et al. (1990) FEBS Lett. 259:327-330; Svinarchuk et al. (1993) Biochimie 75:49-54, каждая из которых включена в настоящий документ посредством ссылки в полном ее объеме для всех целей), фосфолипид, такой как рацемическая смесь дигексадецила-рац-глицерин или триэтиламмония 1,2-ди-О-гексадецил-рац-глицеро-3-Н-фосфонат (Manoharan et al. (1995) Tetrahedron Lett. 36:3651-3654; Shea et al. (1990) Nucl. Acids Res. 18:3777-3783, каждая из которых включена в настоящий документ посредством ссылки в полном ее объеме для всех целей), полиаминовую или полиэтиленгликолевую цепь (Manoharan et al. (1995) Nucleosides & Nucleotides 14:969-973, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей) или адамантануксусную кислоту (Manoharan et al. (1995) Tetrahedron Lett. 36:3651-3654, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей), пальмитиловый фрагмент (Mishra et al. (1995) Biochim. Biophys. Acta 1264:229-237) или октадециламиновый или гексиламинокарбонилоксихолистериновый фрагмент (Crooke et al. (1996) J. Pharmacol. Exp. Ther. 277:923-937, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей). Во многих патентах США описано получение таких конъюгатов, и к ним относятся, например, патенты США №№4828979, 4948882, 5218105, 5525465, 5541313, 5545730, 5552538, 5578717, 5580731, 5580731, 5591584, 5109124, 5118802, 5138045, 5414077, 5486603, 5512439, 5578718, 5608046, 4587044, 4605735, 4667025, 4762779, 4789737, 4824941, 4835263, 4876335, 4904582, 4958013, 5082830, 5112963, 5214136, 5082830, 5112963, 5214136, 5245022, 5254469, 5258506, 5262536, 5272250, 5292873, 5317098, 5371241, 5391723, 5416203, 5451463, 5510475, 5512667, 5514785, 5565552, 5567810, 5574142, 5585481, 5587371, 5595726, 5597696, 5599923, 5599928 и 5688941, каждый из которых включен в настоящий документ посредством ссылки в полном его объеме для всех целей.
[00153] Раскрываемые в настоящем документе выделенные нуклеиновые кислоты могут содержать нуклеотидную последовательность встречающегося в естественной среде гена HSD17B13 или мРНК-транскрипта или могут содержать не встречающуюся в естественной среде последовательность. В одном примере не встречающаяся в естественной среде последовательность может отличаться от не встречающейся в естественной среде последовательности вследствие синонимичных мутаций или мутаций, которые не влияют на кодируемый белок HSD17B13. Например, последовательность может быть идентичной, за исключением синонимичных мутаций или мутаций, которые не влияют на кодируемый белок HSD17B13. Синонимичной мутацией или заменой является такая замена одного нуклеотида другим в экзоне гена, кодирующего белок, чтобы полученная аминокислотная последовательность не модифицировалась. Это возможно из-за вырожденности генетического кода, поскольку некоторые аминокислоты кодируются более чем одним кодоном из трех пар оснований. Синонимичные замены используют, например, в процессе оптимизации кодонов.
[00154] Также в настоящем документе раскрыты белки, кодируемые раскрываемыми в настоящем документе нуклеиновыми кислотами, и композиции, содержащие раскрываемые в настоящем документе выделенную нуклеиновую кислоту или белок и носитель, повышающий стабильность выделенной нуклеиновой кислоты или белка (например, продлевающий указанный период при заданных условиях хранения (например, при -20°С, 4°С или температуре окружающей среды), в течение которого продукты разложения остаются ниже порогового уровня, такого как ниже 0,5% по массе исходной нуклеиновой кислоты или белка, или повышение стабильности in vivo). Неограничивающие примеры таких носителей включают микросферы из полимолочной кислоты (PLA), микросферы из сополимера D,L-молочной и когликолиевой кислот (PLGA), липосомы, мицеллы, инверсные мицеллы, липидные кохлеаты и липидные микротрубочки.
(1) Нуклеиновые кислоты, включающие мутантные остатки варианта rs72613567 HSD17B13
[00155] В настоящем документе раскрыты выделенные нуклеиновые кислоты, содержащие по меньшей мере 15 смежных нуклеотидов гена HSD17B13 и имеющие тимин в положении, соответствующем положению 12666 (или тимины в положениях, соответствующих положениям 12666 и 12667) варианта rs72613567 HSD17B13 (SEQ ID NO: 2) при оптимальном выравнивании с вариантом rs72613567 HSD17B13. То есть, в настоящем документе раскрыты выделенные нуклеиновые кислоты, содержащие по меньшей мере 15 смежных нуклеотидов гена HSD17B13 и имеющие тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 гена HSD17B13 дикого типа (SEQ ID NO: 1) при оптимальном выравнивании с геном HSD17B13 дикого типа. Такие выделенные нуклеиновые кислоты могут быть пригодны, например, для экспрессии транскриптов и белков варианта HSD17B13 или в качестве экзогенных донорных последовательностей. Такие выделенные нуклеиновые кислоты могут быть пригодны, например, в качестве гидовых РНК, праймеров и зондов.
[00156] Ген HSD17B13 может представлять собой ген HSD17B13, полученный из любого организма. Например, ген HSD17B13 может представлять собой ген HSD17B13 человека или ортолог из другого организма, такого как отличное от человека млекопитающее, грызун, мышь или крыса.
[00157] Понятно, что последовательности генов в популяции могут варьировать по причине полиморфизмов, таких как однонуклеотидные полиморфизмы. Представленные в настоящем документе примеры являются лишь иллюстративными последовательностями. Также возможны и другие последовательности. В качестве одного примера, по меньшей мере 15 смежных нуклеотидов могут быть по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны соответствующей последовательности в варианте rs72613567 HSD17B13 (SEQ ID NO: 2), включая положение 12666 или положения 12666 и 12667 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. Необязательно, выделенная нуклеиновая кислота содержит по меньшей мере 15 смежных нуклеотидов из SEQ ID NO: 2, включая положение 12666 или положения 12666 и 12667 из SEQ ID NO: 2. В качестве другого примера, по меньшей мере 15 смежных нуклеотидов могут быть по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичны соответствующей последовательности гена HSD17B13 дикого типа (SEQ ID NO: 1), включая положения 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании с SEQ ID NO: 1, при этом тимин находится между положениями, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1. Необязательно, выделенная нуклеиновая кислота содержит по меньшей мере 15 смежных нуклеотидов из SEQ ID NO: 1, включая положения 12665 и 12666 из SEQ ID NO: 1, при этом тимин находится между положениям, соответствующим положениям 12665 и 12666 из SEQ ID NO: 1.
[00158] Выделенная нуклеиновая кислота может содержать, например, по меньшей мере 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 смежных нуклеотидов гена HSD17B13. В качестве альтернативы, выделенная нуклеиновая кислота может содержать, например, по меньшей мере 1000, 2000, 3000, 4000, 5000,6000,7000,8000,9000,10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000 или 19000 смежных нуклеотидов гена HSD17B13.
[00159] В некоторых случаях выделенная нуклеиновая кислота может содержать миниген HSD17B13, в котором были делетерованы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа. В качестве одного примера, делетерованные сегменты содержат одну или несколько интронных последовательностей. Такие минигены HSD17B13 могут содержать, например, экзоны, соответствующие экзонам 1-7 из транскрипта D HSD17B13, и интрон, соответствующий интрону 6 в SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. В качестве одного примера, миниген HSD17B13 может содержать экзоны 1-7 и интрон 6 из SEQ ГО NO: 2. Минигены более подробно описаны в других разделах настоящего документа.
(2) Нуклеиновые кислоты, гибридизирующиеся с последовательностью, расположенной рядом с мутантным остатком варианта rs72613567 HSD17B13 или включающей его
[00160] Также в настоящем документе раскрыты выделенные нуклеиновые кислоты, содержащие по меньшей мере 15 смежных нуклеотидов, которые гибридизируются с геном HSD17B13 (например, минигеном HSD17B13) в сегменте, который включает или находится в пределах 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от положения, соответствующего положению 12666 или положениям 12666 и 12667 варианта rs72613567 HSD17B13 (SEQ ID NO: 2) при оптимальном выравнивании с вариантом rs72613567 HSD17B13. Такие выделенные нуклеиновые кислоты могут быть пригодны, например, в качестве гидовых РНК, праймеров, зондов или экзогенных донорных последовательностей.
[00161] Ген HSD17B13 может представлять собой ген HSD17B13, полученный из любого организма. Например, ген HSD17B13 может представлять собой ген HSD17B13 человека или ортолог из другого организма, такого как отличное от человека млекопитающее, мышь или крыса.
[00162] В качестве одного примера, по меньшей мере 15 смежных нуклеотидов могут гибридизироваться с сегментом гена HSD17B13 или минигена HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен соответствующей последовательности в варианте rs72613567 HSD17B13 (SEQ ID NO: 2) при оптимальном выравнивании с SEQ ID NO: 2. Необязательно, выделенная нуклеиновая кислота может гибридизироваться по меньшей мере с 15 смежными нуклеотидами из SEQ ID NO: 2. Необязательно, выделенная нуклеиновая кислота гибридизируется с сегментом, включающем положение 12666 или положения 12666 и 12667 в SEQ ID NO: 2 или положение, соответствующее положению 12666 или положениям 12666 и 12667 в SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2.
[00163] Сегмент, с которым выделенная нуклеиновая кислота может гибридизироваться, может содержать, например, по меньшей мере 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 75, 90, 95, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 смежных нуклеотидов гена HSD17B13. В качестве альтернативы, выделенная нуклеиновая кислота может содержать, например, по меньшей мере 1000, 2000, 3000, 4000, 5000, 6000, 7000, 8000, 9000, 10000, 11000, 12000, 13000, 14000, 15000, 16000, 17000, 18000 или 19000 смежных нуклеотидов гена HSD17B13. В качестве альтернативы, сегмент, с которым выделенная нуклеиновая кислота может гибридизироваться, может представлять собой, например, до 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 75, 90, 95, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 смежных нуклеотидов гена HSD17B13. Например, сегмент может составлять приблизительно 15-100 нуклеотидов в длину или приблизительно 15-35 нуклеотидов в длину.
(3) кДНК и варианты транскриптов, полученные с помощью варианта rs72613567 HSD17B13
[00164] Также настоящее изобретение относится к нуклеиновым кислотам, соответствующим всему или части мРНК-транскрипта или всей или части кДНК, которые соответствуют любому из транскриптов А-Н (SEQ ID NO: 4-11 соответственно) и, в частности, транскриптов С-Н при оптимальном выравнивании с любым из транскриптов А-Н. Понятно, что последовательности гена и в пределах популяции и мРНК-последовательности, транскрибированные с таких генов, могут варьировать по причине полиморфизмов, таких как однонуклеотидные полиморфизмы. Представленные в настоящем документе последовательности для каждого транскрипта являются лишь иллюстративными последовательностями. Также возможны и другие последовательности. Ниже представлены конкретные неограничивающие примеры. Такие выделенные нуклеиновые кислоты могут быть пригодны, например, для экспрессии транскриптов и белков варианта HSD17B13.
[00165] Выделенная нуклеиновая кислота может быть любой длины. Например, выделенная нуклеиновая кислота может содержать по меньшей мере 10, 15, 20, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000 или 2000 смежных нуклеотидов, кодирующих весь или часть белка HSD17B13. В некоторых случаях выделенные нуклеиновые кислоты содержат смежные нуклеотиды, кодирующие весь или часть белка HSD17B13, причем смежные нуклеотиды содержат последовательность по меньшей мере из двух различных экзонов гена HSD17B13 (например, охватывающую по меньшей мере одну границу между экзонами гена HSD17B13 без промежуточного интрона).
[00166] Транскрипт D (SEQ ID NO: 7), транскрипт G (SEQ ID NO: 10) и транскрипт Н (SEQ ID NO: 11) HSD17B13 включают вставку гуанина на 3'-конце экзона 6, что приводит в результате к сдвигу рамки считывания в экзоне 7 и преждевременному укорочению участка белка HSD17B13, кодируемого экзоном 7, в сравнении с транскриптом А. Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим сегмент (например, по меньшей мере из 15 смежных нуклеотидов), присутствующий в транскриптах D, G и Н (или их фрагментах или гомологах) и отсутствующий в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Например, настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов), кодирующих весь или часть белка HSD17B13, причем сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку, охватывающему границу между экзоном 6 и экзоном 7 в SEQ ID NO: 7 (транскрипте D HSD17B13), SEQ ID NO: 10 (транскрипте G HSD17B13) или SEQ ID NO: 11 (транскрипте Н HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7, 10 или 11 соответственно, и сегмент включает гуанин в остатке, соответствующем остатку 878 на 3'-конце экзона 6 в SEQ ID NO: 7 (т.е. вставку гуанина на 3'-конце экзона 6 относительно транскрипта А помимо гуанина в начале экзона 7), остатке, соответствующем остатку 770 на 3'-конце экзона 6 в SEQ ID NO: 10 (т.е. вставку гуанина на 3'-конце экзона 6 относительно транскрипта В помимо гуанина в начале экзона 7), или остатке, соответствующем остатку 950 на 3'-конце экзона 6 в SEQ ID NO: 11 (т.е. вставку гуанина на 3'-конце экзона 6 относительно транскрипта Е помимо гуанина в начале экзона 7). Понятно, что такая нуклеиновая кислота будет включать достаточное количество нуклеотидов в каждом из экзонов 6 и 7, что позволяет отличать вставленный гуанин от других признаков в транскриптах HSD17B13 (например, от гуанина в начале экзона 7, от участка считывания в интрон 6 в транскрипте F или от делетерованного экзона 6 в транскрипте С).
[00167] В качестве одного примера, выделенная нуклеиновая кислота может содержать по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов) из SEQ ID NO: 7, охватывающие границу между экзоном 6 и экзоном 7, необязательно содержащие экзоны 6 и 7 из SEQ ID NO: 7 и необязательно содержащие всю последовательность SEQ ID NO: 7.
[00168] Необязательно, выделенная нуклеиновая кислота дополнительно содержит сегмент, присутствующий в транскрипте D (или его фрагменте или гомологе) и отсутствующий в транскрипте G (или его фрагменте или гомологе), и выделенная нуклеиновая кислота дополнительно содержит сегмент, присутствующий в транскрипте D (или его фрагменте или гомологе) и отсутствующий в транскрипте Н (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Например, такие выделенные нуклеиновые кислоты могут содержать сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку, охватывающему границу экзонов 3 и 4 из SEQ ID NO: 7 (транскрипта D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7, что позволяет отличать от транскрипта Н. Аналогичным образом, такие выделенные нуклеиновые кислоты могут содержать сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку в пределах экзона 2 из SEQ ID NO: 7 (транскрипта D HSD17B13), участку, охватывающему границу между экзоном 1 и экзоном 2 из SEQ ID NO: 7, или участку, охватывающему границу между экзоном 2 и экзоном 3 из SEQ ID NO: 7 при оптимальном выравнивании с SEQ ID NO: 7, что позволяет отличать от транскрипта G. Необязательно, выделенная нуклеиновая кислота содержит последовательность по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичную последовательности, изложенной под SEQ ID NO: 7 (транскрипту D HSD17B13), и кодирует белок HSD17B13, содержащий последовательность, изложенную под SEQ ID NO: 15 (изоформу D HSD17B13). Как и транскрипт D, транскрипт Н (SEQ ID NO: 11) включает вставку гуанина 3' от экзона 6 по сравнению с транскриптом А. Транскрипт Н дополнительно включает дополнительный экзон (экзон 3') между экзонами 3 и 4 по сравнению с транскриптом А и транскриптом D. Соответственно, настоящее изобретение относится к описанным выше выделенным нуклеиновым кислотам, содержащим сегмент, присутствующий в транскриптах D, G и Н (или их фрагментах или гомологах) и отсутствующий в транскрипте А (или его фрагменте или гомологе), но дополнительно содержащим сегмент (например, по меньшей мере из 15 смежных нуклеотидов) из транскрипта Н (или его фрагмента или гомолога), которого нет в транскрипте D (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Например, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые описаны для транскрипта D, причем сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку в пределах экзона 3' из SEQ ID NO: 11 (транскрипта Н HSD17B13), участку, охватывающему границу между экзоном 3 и экзоном 3' из SEQ ID NO: 11, или участку, охватывающему границу между экзоном 3' и экзоном 4 из SEQ ID NO: 11 при оптимальном выравнивании с SEQ ID NO: 11. Понятно, что такая нуклеиновая кислота будет включать достаточное количество нуклеотидов в каждом из экзонов 3 и 3' или каждом из экзонов 3' и 4, что позволяет отличить ее от других признаков в транскриптах HSD17B13 (например, от границы экзонов 3 и 4). Например, участок экзона 3' может содержать весь экзон 3'. Необязательно, выделенная нуклеиновая кислота содержит последовательность, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%) или по меньшей мере на 99% идентичную последовательности, изложенной под SEQ ID NO: 11 (транскрипту Н HSD17B13), и кодирует белок HSD17B13, содержащий последовательность, изложенную под SEQ ID NO: 19 (изоформу Н HSD17B13).
[00169] В качестве одного примера, выделенная нуклеиновая кислота может содержать по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов) из SEQ ID NO: 11, включая участок в пределах экзона 3', участок, охватывающий границу между экзоном 3 и экзоном 3', или участок, охватывающий границу между экзоном 3' и экзоном 4, необязательно содержащий весь экзон 3' из SEQ ID NO: 11, и необязательно содержащий всю последовательность SEQ ID NO: 11.
[00170] Как и транскрипт D, транскрипт G (SEQ ID NO: 10) включает вставку гуанина 3' от экзона 6 по сравнению с транскриптом А. Однако, кроме того, в транскрипте G пропущен экзон 2 по сравнению с транскриптом А и транскриптом D (т.е. транскрипт G включает границу между экзоном 1 и экзоном 3, не присутствующую в транскриптах А и D). Соответственно, настоящее изобретение относится к описанным выше выделенным нуклеиновым кислотам, содержащим сегмент, присутствующий в транскриптах D, G и Н (или их фрагментах или гомологах) и отсутствующий в транскрипте А (или его фрагменте или гомологе), но дополнительно содержащим сегмент (например, по меньшей мере из 15 смежных нуклеотидов) из транскрипта G (или его фрагмента или гомолога), которого нет в транскрипте D (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Например, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые описаны для транскрипта D, причем сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку, охватывающему границу между экзоном 1 и экзоном 3 в SEQ ID NO: 10 (транскрипте D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 10. Понятно, что такая нуклеиновая кислота будет включать достаточное количество нуклеотидов в каждом из экзонов 1 и 3, что позволяет отличать от других признаков в транскриптах HSD17B13 (например, границы экзонов 1 и 2 или границы экзонов 2 и 3). Например, участок может содержать полностью экзоны 1 и 3 в SEQ ID NO: 10. Необязательно, выделенная нуклеиновая кислота содержит последовательность, по меньшей мере на 90%, по меньшей мере на 95%), по меньшей мере на 96%, по меньшей мере на 97%), по меньшей мере на 98%) или по меньшей мере на 99% идентичную последовательности, изложенной под SEQ ID NO: 10 (транскрипту G HSD17B13), и кодирует белок HSD17B13, содержащий последовательность, изложенную под SEQ ID NO: 18 (изоформу G HSD17B13).
[00171] В качестве одного примера, выделенная нуклеиновая кислота может содержать по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов) из SEQ ID NO: 10, включая участок, охватывающий границу между экзоном 1 и экзоном 3, необязательно содержащий экзоны 1 и 3 из SEQ ID NO: 10 и необязательно содержащий полную последовательность SEQ ID NO: 10.
[00172] Также настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим сегмент (например, по меньшей мере 15 смежных нуклеотидов), присутствующий в транскрипте Е (или его фрагменте или гомологе) и отсутствующий в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Транскрипт Е (SEQ ID NO: 8) включает дополнительный экзон между экзонами 3 и 4 по сравнению с транскриптом А. Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов), кодирующим весь или часть белка HSD17B13, причем сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку в пределах экзона 3' из SEQ ID NO: 8 (транскрипта Е HSD17B13), участку, охватывающему границу между экзоном 3 и экзоном 3' из SEQ ID NO: 8, или участку, охватывающему границу между экзоном 3' и экзоном 4 из SEQ ID NO: 8 при оптимальном выравнивании с SEQ ID NO: 8. Понятно, что такая нуклеиновая кислота будет включать достаточное количество нуклеотидов в каждом из экзонов 3 и 3' или каждом из экзонов 3' и 4, что позволяет отличить ее от других признаков в транскриптах HSD17B13 (например, от границы экзонов 3 и 4). Например, участок экзона 3' может содержать весь экзон 3'. Необязательно, выделенная нуклеиновая кислота дополнительно содержит сегмент (например, по меньшей мере 15 смежных нуклеотидов) из транскрипта Е (или его фрагмента или гомолога), которого нет в транскрипте Н (или их фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Например, настоящее изобретение относится к описанным выше выделенным нуклеиновым кислотам, причем сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку, охватывающему границу между экзоном 6 и экзоном 7 в SEQ ID NO: 8 (транскрипте E HSD17B13) при оптимальном выравнивании с SEQ ID NO: 8. Понятно, что такая нуклеиновая кислота будет включать достаточное количество нуклеотидов в каждом из экзонов 6 и 7, что позволяет отличать от других признаков в транскриптах HSD17B13 (в частности, дополнительный гуанин на 3'-конце экзона 6 в транскрипте Н)). Например, участок может содержать полностью экзоны 6 и 7 в SEQ ID NO: 8. Необязательно, выделенная нуклеиновая кислота содержит последовательность, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичную последовательности, изложенной под SEQ ID NO: 8 (транскрипту Е HSD17B13), и кодирует белок HSD17B13, содержащий последовательность, изложенную под SEQ ID NO: 16 (изоформу Е HSD17B13).
[00173] В качестве одного примера, выделенная нуклеиновая кислота может содержать по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов) из SEQ ID NO: 8, включая участок в пределах экзона 3', участок, охватывающий границу между экзоном 3 и экзоном 3', или участок, охватывающий границу между экзоном 3' и экзоном 4, необязательно содержащий весь экзон 3' из SEQ ID NO: 8 и необязательно содержащий полную последовательность SEQ ID NO: 8.
[00174] Также настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим сегмент (например, по меньшей мере 15 смежных нуклеотидов), присутствующий в транскрипте F (или его фрагменте или гомологе) и отсутствующий в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Транскрипт F (SEQ ID NO: 9) включает участок считывания от экзона 6 в интрон 6 по сравнению с транскриптом А, и участок считывания включает вставленный тимин, присутствующий в гене варианта rs72613567 HSD17B13. Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов), кодирующих весь или часть белка HSD17B13, причем сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку в пределах участка считывания в интрон 6 в SEQ ID NO: 9 (транскрипте F HSD17B13) или участку, охватывающему границу между участком считывания в интрон 6 и остальной частью экзона 6 в SEQ ID NO: 9 при оптимальном выравнивании с SEQ ID NO: 9. Понятно, что такая нуклеиновая кислота будет включать достаточное количество нуклеотидов в участке считывания, что позволяет отличать участок считывания от других признаков в транскриптах HSD17B13 (например, от границы экзонов 6 и 7 в других транскриптах HSD17B13). Необязательно, смежные нуклеотиды содержат последовательность, присутствующую в транскрипте F (т.е. вставленный тимин) и отсутствующую в транскрипте F' (SEQ ID NO: 246). Транскрипт F' также включает участок считывания от экзона 6 в интрон 6 по сравнению с транскриптом А, но участок считывания не включает вставленный тимин, присутствующий в гене варианта rs72613567 HSD17B13. Например, участок может быть полным участком считывания в интрон 6 в SEQ ID NO: 9. Необязательно, выделенная нуклеиновая кислота содержит последовательность, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичную последовательности, изложенной под SEQ ID NO: 9 (транскрипту F HSD17B13), и кодирует белок HSD17B13, содержащий последовательность, изложенную под SEQ ID NO: 17 (изоформу F HSD17B13).
[00175] В качестве одного примера, выделенная нуклеиновая кислота может содержать по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов) из SEQ ID NO: 9, включая участок в пределах участка считывания в интрон 6, или участок, охватывающий границу между участком считывания в интрон 6 и остальной частью экзона 6, необязательно содержащий полный участок считывания в интрон 6 и необязательно содержащий полную последовательность SEQ ID NO: 9.
[00176] Также настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим сегмент (например, по меньшей мере 15 смежных нуклеотидов), присутствующий в транскрипте F' (или его фрагменте или гомологе) и отсутствующий в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Транскрипт F' (SEQ ID NO: 246) включает участок считывания от экзона 6 в интрон 6 по сравнению с транскриптом А, а участок считывания не включает вставленный тимин, присутствующий в гене варианта rs72613567 HSD17B13. Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов), кодирующих весь или часть белка HSD17B13, причем сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку в пределах участка считывания в интрон 6 в SEQ ID NO: 246 (транскрипте F' HSD17B13) или участку, охватывающему границу между участком считывания в интрон 6 и остальной частью экзона 6 в SEQ ID NO: 246 при оптимальном выравнивании с SEQ ID NO: 246. Понятно, что такая нуклеиновая кислота будет включать достаточное количество нуклеотидов в участке считывания, что позволяет отличать участок считывания от других признаков в транскриптах HSD17B13 (например, от границы экзонов 6 и 7 в других транскриптах HSD17B13). Необязательно, смежные нуклеотиды содержат последовательность, присутствующую в транскрипте F' и отсутствующую в транскрипте F (SEQ ID NO: 9). Участок считывания в транскрипте F включает вставленный тимин, присутствующий в гене варианта rs72613567 HSD17B13, в то время как участок считывания в транскрипте F' его не включает. Например, участок может быть полным участком считывания в интрон 6 в SEQ ID NO: 246. Необязательно, выделенная нуклеиновая кислота содержит последовательность, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичную последовательности, изложенной под SEQ ID NO: 246 (транскрипту F' HSD17B13), и кодирует белок HSD17B13, содержащий, фактически состоящий из или состоящий из последовательности, изложенной под SEQ ID NO: 247 (изоформу F' HSD17B13).
[00177] В качестве одного примера, выделенная нуклеиновая кислота может содержать по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов) из SEQ ID NO: 246, включая участок в пределах участка считывания в интрон 6, или участок, охватывающий границу между участком считывания в интрон 6 и остальной частью экзона 6, необязательно содержащий полный участок считывания в интрон 6 и необязательно содержащий полную последовательность SEQ ID NO: 246.
[00178] Также настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим сегмент (например, по меньшей мере 15 смежных нуклеотидов), присутствующий в транскрипте С' (или его фрагменте или гомологе) и отсутствующий в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. В транскрипте С (SEQ ID NO: 6) пропущен экзон 6 по сравнению с транскриптом А (т.е. транскрипт С включает границу между экзоном 5 и экзоном 7, не присутствующую в транскрипте А). Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов), кодирующих весь или часть белка HSD17B13, причем сегмент из смежных нуклеотидов (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку, охватывающему границу между экзоном 5 и экзоном 7 в SEQ ID NO: 6 (транскрипте С HSD17B13) при оптимальном выравнивании с SEQ ID NO: 6. Понятно, что такая нуклеиновая кислота будет включать достаточное количество нуклеотидов в каждом из экзонов 5 и 7, что позволяет отличать от других признаков в транскриптах HSD17B13 (например, от границы экзонов 5 и 6 или экзонов 6 и 7 в других транскриптах HSD17B13). Например, участок может содержать полностью экзоны 5 и 7 в SEQ ID NO: 6. Необязательно, выделенная нуклеиновая кислота содержит последовательность, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичную последовательности, изложенной под SEQ ID NO: 6 (транскрипте С HSD17B13), и кодирует белок HSD17B13, содержащий последовательность, изложенную под SEQ ID NO: 14 (изоформу С HSD17B13).
[00179] В качестве одного примера, выделенная нуклеиновая кислота может содержать по меньшей мере 15 смежных нуклеотидов (например, по меньшей мере 20 смежных нуклеотидов или по меньшей мере 30 смежных нуклеотидов) из SEQ ID NO: 6, включая участок, охватывающий границу между экзоном 5 и экзоном 7, необязательно содержащий полностью экзоны 5 и 7 в SEQ ID NO: 6 и необязательно содержащий полную последовательность SEQ ID NO: 6.
(4) Нуклеиновые кислоты, гибридизирующиеся с кДНК и транскриптами варианта HSD17B13
[00180] Также настоящее изобретение относится к нуклеиновым кислотам, гибридизирующимся с сегментами мРНК-транскрипта или кДНК, которые соответствуют любому из транскриптов А-Н (SEQ ID NO: 4-11 соответственно) и, в частности, транскриптов С-Н при оптимальном выравнивании с любым из транскриптов А-Н. Ниже представлены конкретные неограничивающие примеры. Такие выделенные нуклеиновые кислоты могут быть полезными, например, праймерами, зондами, антисмысловыми РНК, siRNA или shRNA.
[00181] Сегмент, с которым выделенная нуклеиновая кислота может гибридизироваться, может содержать, например, по меньшей мере 5, по меньшей мере 10 или по меньшей мере 15 смежных нуклеотидов нуклеиновой кислоты, кодирующей белок HSD17B13. Сегмент, с которым выделенная нуклеиновая кислота может гибридизироваться, может содержать, например, по меньшей мере 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 75, 90, 95, 100, 200, 300, 400, 500, 600, 700, 800, 900, 1000 или 2000 смежных нуклеотидов нуклеиновой кислоты, кодирующей белок HSD17B13. В качестве альтернативы, сегмент, с которым выделенная нуклеиновая кислота может гибридизироваться, может представлять собой, например, до 20, 25, 30, 35, 40, 45, 50, 55, 60, 65, 70, 75, 80, 75, 90, 95, 100, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 смежных нуклеотидов нуклеиновой кислоты, кодирующей белок HSD17B13. Например, сегмент может составлять приблизительно 15-100 нуклеотидов в длину или приблизительно 15-35 нуклеотидов в длину.
[00182] Транскрипт D (SEQ ID NO: 7), транскрипт G (SEQ ID NO: 10) и транскрипт Н (SEQ ID NO: 11) HSD17B13 включают вставку гуанина на 3'-конце экзона 6, что приводит в результате к сдвигу рамки считывания и преждевременному укорочению экзона 7 по сравнению с транскриптом А. Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом, присутствующим в транскриптах D, G и Н (или их фрагментах или гомологах) и отсутствующим в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Например, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами из нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку, охватывающему границу между экзоном 6 и экзоном 7 в SEQ ID NO: 7 (транскрипте D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7, и сегмент включает гуанин в остатке, соответствующем остатку 878 на 3'-конце экзона 6 в SEQ ID NO: 7 (т.е. вставку гуанина на 3'-конце экзона 6 относительно транскрипта А помимо гуанина в начале экзона 7). В качестве альтернативы, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами из сегмента нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку, охватывающему границу между экзоном 6 и экзоном 7 в SEQ ID NO: 10 (транскрипте D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 10, и сегмент включает гуанин в остатке, соответствующем остатку 770 на 3'-конце экзона 6 в SEQ ID NO: 10 (т.е. вставку гуанина на 3'-конце экзона 6 относительно транскрипта В помимо гуанина в начале экзона 7). В качестве альтернативы, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами из нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку, охватывающему границу между экзоном 6 и экзоном 7 в SEQ ID NO: 11 (транскрипте Н HSD17B13) при оптимальном выравнивании с SEQ ID NO: 11, и сегмент включает гуанин в остатке, соответствующем остатку 950 на 3'-конце экзона 6 в SEQ ID NO: 11 (т.е. вставку гуанина на 3'-конце экзона 6 относительно транскрипта Е помимо гуанина в начале экзона 7). Понятно, что такие нуклеиновые кислоты будут сконструированы такими, чтобы они гибридизировались с достаточным количеством нуклеотидов в каждом из экзонов 6 и 7, что позволит отличить вставленный гуанин от других признаков в транскриптах HSD17B13 (например, от участка считывания в интрон 6 в транскрипте F или от делетерованного экзона 6 в транскрипте С).
[00183] В качестве одного примера, сегмент может содержать участок из SEQ ID NO: 7, охватывающий границу между экзоном 6 и экзоном 7 (т.е. включающий гуанин в остатке 878 из SEQ ID NO: 7). В качестве другого примера, сегмент может содержать участок из SEQ ID NO: 10, охватывающий границу между экзоном 6 и экзоном 7 (т.е. включающий гуанин в остатке 770 из SEQ ID NO: 10). В качестве другого примера, сегмент может содержать участок из SEQ ID NO: 11, охватывающий границу между экзоном 6 и экзоном 7 (т.е. включающий гуанин в остатке 950 из SEQ ID NO: 11).
[00184] Необязательно, выделенная нуклеиновая кислота дополнительно содержит участок (например, 15 смежных нуклеотидов), который гибридизируется с сегментом, присутствующим в транскрипте D (или его фрагменте или гомологе) и отсутствующий в транскрипте G (или его фрагменте или гомологе), и выделенная нуклеиновая кислота дополнительно содержит участок, который гибридизируется с сегментом, присутствующим в транскрипте D (или его фрагменте или гомологе) и отсутствующим в транскрипте Н (или его фрагменте или гомологе). Такие сегменты можно легко выявить путем сравнения последовательностей транскриптов. Например, сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), присутствующий в транскрипте D (или его фрагменте или гомологе) и отсутствующий в транскрипте Н (или его фрагменте или гомологе), может быть по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку, охватывающему границу экзонов 3 и 4 из SEQ ID NO: 7 (транскрипта D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7, что позволяет отличать от транскрипта Н. Аналогичным образом, сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), присутствующий в транскрипте D (или его фрагменте или гомологе) и отсутствующий в транскрипте Н (или его фрагменте или гомологе) может быть по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку в пределах экзона 2 из SEQ ID NO: 7 (транскрипта D HSD17B13), участку, охватывающему границу между экзоном 1 и экзоном 2 из SEQ ID NO: 7, или участку, охватывающему границу между экзоном 2 и экзоном 3 из SEQ ID NO: 7 при оптимальном выравнивании с SEQ ID NO: 7, что позволяет отличать от транскрипта G.
[00185] Как и транскрипт D, транскрипт Н (SEQ ID NO: 11) включает вставку гуанина на 3'-конце экзона 6 по сравнению с транскриптом А. Транскрипт Н дополнительно включает дополнительный экзон между экзонами 3 и 4 по сравнению с транскриптом А и транскриптом D. Соответственно, настоящее изобретение относится к описанным выше выделенным нуклеиновым кислотам, содержащим участок, который гибридизируется с сегментом, присутствующим в транскриптах D, G и Н (или их фрагментах или гомологах) и отсутствующим в транскрипте А (или его фрагменте или гомологе), но дополнительно содержащим участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом, который присутствует в транскрипте Н (или его фрагменте или гомологе), но не в транскрипте D (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Например, сегмент может быть по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку (например, по меньшей мере 5 смежным нуклеотидам, по меньшей мере 10 смежным нуклеотидам или по меньшей мере 15 смежным нуклеотидам) в пределах экзона 3' из SEQ ID NO: 11 (транскрипта Н HSD17B13), участку, охватывающему границу между экзоном 3 и экзоном 3' из SEQ ID NO: 11, или участку, охватывающему границу между экзоном 3' и экзоном 4 из SEQ ID NO: 11 при оптимальном выравнивании с SEQ ID NO: 11. Понятно, что такая нуклеиновая кислота будет сконструирована такой, чтобы гибридизироваться с достаточным количеством нуклеотидов в каждом из экзонов 3 и 3' или в каждом из экзонов 3' и 4, что позволяет отличать от других признаков в транскриптах HSD17B13 (например, от границы экзонов 3 и 4).
[00186] В качестве одного примера, сегмент может содержать участок из SEQ ID NO: 11 в пределах экзона 3', охватывающий границу между экзоном 3 и экзоном 3' или охватывающий границу между экзоном 3' и экзоном 4.
[00187] Как и транскрипт D, транскрипт G (SEQ ID NO: 10) включает вставку гуанина на 3'-конце экзона 6 по сравнению с транскриптом А. Однако, кроме того, в транскрипте G пропущен экзон 2 по сравнению с транскриптом А и транскриптом D (т.е. транскрипт G включает границу между экзоном 1 и экзоном 3, не присутствующую в транскриптах А и D). Соответственно, настоящее изобретение относится к описанным выше выделенным нуклеиновым кислотам, содержащим участок, который гибридизируется с сегментом, присутствующим в транскриптах D, G и Н (или их фрагментах или гомологах) и отсутствующим в транскрипте А (или его фрагменте или гомологе), но дополнительно содержащим участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом, присутствующим в транскрипте G (или его фрагменте или гомологе), но не в транскрипте D (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Например, сегмент может быть по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку (например, по меньшей мере 5 смежным нуклеотидам, по меньшей мере 10 смежным нуклеотидам или по меньшей мере 15 смежным нуклеотидам), охватывающему границу между экзоном 1 и экзоном 3 в SEQ ID NO: 10 (транскрипте D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 10. Понятно, что такая нуклеиновая кислота будет сконструирована такой, чтобы гибридизироваться с достаточным количеством нуклеотидов в каждом из экзонов 1 и 3, что позволяет отличать от других признаков в транскриптах HSD17B13 (например, границы экзонов 1 и 2 или границы экзонов 2 и 3).
[00188] В качестве одного примера, сегмент может содержать участок из SEQ ID NO: 10, охватывающий границу между экзоном 1 и экзоном 3.
[00189] Также настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом нуклеиновой кислоты, кодирующей белок HSD17B13, который присутствует в транскрипте Е (или его фрагменте или гомологе), но не в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Транскрипт Е (SEQ ID NO: 8) включает дополнительный экзон между экзонами 3 и 4 по сравнению с транскриптом А. Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку (например, по меньшей мере 5 смежным нуклеотидам, по меньшей мере 10 смежным нуклеотидам или по меньшей мере 15 смежным нуклеотидам) в пределах экзона 3' из SEQ ID NO: 8 (транскрипта Е HSD17B13), участку, охватывающему границу между экзоном 3 и экзоном 3' из SEQ ID NO: 8, или участку, охватывающему границу между экзоном 3' и экзоном 4 из SEQ ID NO: 8 при оптимальном выравнивании с SEQ ID NO: 8. Понятно, что такая нуклеиновая кислота будет сконструирована такой, чтобы гибридизироваться с достаточным количеством нуклеотидов в каждом из экзонов 3 и 3' или в каждом из экзонов 3' и 4, что позволяет отличать от других признаков в транскриптах HSD17B13 (например, от границы экзонов 3 и 4).
[00190] В качестве одного примера, сегмент может содержать участок из SEQ ID NO: 8 в пределах экзона 3', охватывающий границу между экзоном 3 и экзоном 3' из SEQ ID NO: 8 или охватывающий границу между экзоном 3' и экзоном 4.
[00191] Необязательно, выделенная нуклеиновая кислота дополнительно содержит участок (например, 15 смежных нуклеотидов), который гибридизируется с сегментом, присутствующим в транскрипте Е (или его фрагменте или гомологе) и отсутствующим в транскрипте Н (или его фрагменте или гомологе). Такие сегменты можно легко выявить путем сравнения последовательностей транскриптов. Например, сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), присутствующий в транскрипте Е (или его фрагменте или гомологе) и отсутствующий в транскрипте Н (или его фрагменте или гомологе), может быть по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен участку, охватывающему границу экзонов 6 и 7 из SEQ ID NO: 8 (транскрипта Е HSD17B13) при оптимальном выравнивании с SEQ ID NO: 8, что позволяет отличать от транскрипта G. Понятно, что такая нуклеиновая кислота будет сконструирована, чтобы гибридизироваться с достаточным количеством нуклеотидов в каждом из экзонов 6 и 7, что позволяет отличать от других признаков в транскриптах HSD17B13 (в частности, дополнительный гуанин на 3'-конце экзона 6 в транскрипте Н)).
[00192] Также настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом нуклеиновой кислоты, кодирующей белок HSD17B13, который присутствует в транскрипте F (или его фрагменте или гомологе), но не в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Транскрипт F (SEQ ID NO: 9) включает участок считывания от экзона 6 в интрон 6 по сравнению с транскриптом А. Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку в пределах участка считывания в интрон 6 в SEQ ID NO: 9 (транскрипте F HSD17B13) или участку, охватывающему границу между участком считывания в интрон 6 и остальной частью экзона 6 в SEQ ID NO: 9 при оптимальном выравнивании с SEQ ID NO: 9. Понятно, что такая нуклеиновая кислота будет сконструирована такой, чтобы гибридизироваться с достаточным количеством нуклеотидов в участке считывания, что позволяет отличить считывание от других признаков в транскриптах HSD17B13 (например, от границы экзонов 6 и 7 в других транскриптах HSD17B13). Необязательно, смежные нуклеотиды содержат последовательность, присутствующую в транскрипте F (т.е. вставленный тимин) и отсутствующую в транскрипте F' (SEQ ID NO: 246). Транскрипт F' также включает участок считывания от экзона 6 в интрон 6 по сравнению с транскриптом А, но участок считывания не включает вставленный тимин, присутствующий в гене варианта rs72613567 HSD17B13.
[00193] В качестве одного примера, сегмент может содержать участок из SEQ ID NO: 9 в пределах участка считывания в интрон 6 или охватывающий границы между участком считывания в интрон 6 и остальной частью экзона 6.
[00194] Также настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом нуклеиновой кислоты, кодирующей белок HSD17B13, который присутствует в транскрипте F' (или его фрагменте или гомологе), но не в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Транскрипт F' (SEQ ID NO: 246) включает участок считывания от экзона 6 в интрон 6 по сравнению с транскриптом А. Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку в пределах участка считывания в интрон 6 в SEQ ID NO: 246 (транскрипте F' HSD17B13) или участку, охватывающему границу между участком считывания в интрон 6 и остальной частью экзона 6 в SEQ ID NO: 246 при оптимальном выравнивании с SEQ ID NO: 246. Понятно, что такая нуклеиновая кислота будет сконструирована такой, чтобы гибридизироваться с достаточным количеством нуклеотидов в участке считывания, что позволяет отличить считывание от других признаков в транскриптах HSD17B13 (например, от границы экзонов 6 и 7 в других транскриптах HSD17B13). Необязательно, смежные нуклеотиды содержат последовательность, присутствующую в транскрипте F' и отсутствующую в транскрипте F (SEQ ID NO: 9). Участок считывания в транскрипте F включает вставленный тимин, присутствующий в гене варианта rs72613567 HSD17B13, в то время как участок считывания в транскрипте F' его не включает.
[00195] В качестве одного примера, сегмент может содержать участок из SEQ ID NO: 246 в пределах участка считывания в интрон 6 или охватывающий границы между участком считывания в интрон 6 и остальной частью экзона 6.
[00196] Также настоящее изобретение относится к выделенным нуклеиновым кислотам, содержащим участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом нуклеиновой кислоты, кодирующей белок HSD17B13, который присутствует в транскрипте С (или его фрагменте или гомологе), но не в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. В транскрипте С (SEQ ID NO: 6) пропущен экзон 6 по сравнению с транскриптом А (т.е. транскрипт С включает границу между экзоном 5 и экзоном 7, не присутствующую в транскрипте А). Соответственно, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами из нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку, охватывающему границу между экзоном 5 и экзоном 7 в SEQ ID NO: 6 (транскрипте С HSD17B13) при оптимальном выравнивании с SEQ ID NO: 6. Понятно, что такая нуклеиновая кислота будет сконструирована такой, чтобы гибридизироваться с достаточным количеством нуклеотидов в экзонах 5 и 7, что позволяет отличать от других признаков в транскриптов HSD17B13 (например, от границы экзонов 5 и 6 или экзонов 6 и 7 в других транскриптах HSD17B13).
[00197] В качестве одного примера, сегмент может содержать участок из SEQ ID NO: 6, охватывающий границу между экзоном 5 и экзоном 7.
[00198] Также настоящее изобретение относится к выделенным нуклеиновым кислотам (например, антисмысловым РНК, siRNA или shRNA), которые гибридизируются по меньшей мере с 15 смежными нуклеотидами из нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку из транскрипта D HSD17B13 (SEQ ID NO: 7). Выделенные нуклеиновые кислоты могут содержать участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом, присутствующим в транскрипте D (или его фрагментах или гомологах) и отсутствующим в транскрипте А (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Транскрипт D HSD17B13 (SEQ ID NO: 7) включает вставку гуанина на 3'-конце экзона 6, что приводит в результате к сдвигу рамки считывания и преждевременному укорочению экзона 7 по сравнению с транскриптом A (SEQ ID NO: 4). Например, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами из нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку, охватывающему границу между экзоном 6 и экзоном 7 в SEQ ID NO: 7 (транскрипте D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 7. Сегмент может включать гуанин в остатке, соответствующем остатку 878 на 3'-конце экзона 6 в SEQ ID NO: 7 (т.е. вставку гуанина на 3'-конце экзона 6 относительно транскрипта А помимо гуанина в начале экзона 7). Понятно, что такие нуклеиновые кислоты будут сконструированы такими, чтобы они гибридизировались с достаточным количеством нуклеотидов в каждом из экзонов 6 и 7, что позволит отличить вставленный гуанин от других признаков в транскриптах HSD17B13 (например, от участка считывания в интрон 6 в транскрипте F или от делетерованного экзона 6 в транскрипте С).
[00199] Также настоящее изобретение относится к выделенным нуклеиновым кислотам (например, антисмысловым РНК, siRNA или shRNA), которые гибридизируются по меньшей мере с 15 смежными нуклеотидами из нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 91%, по меньшей мере на 98%), по меньшей мере на 99% или на 100% идентичен участку из транскрипта A HSD17B13 (SEQ ID NO: 4). Выделенные нуклеиновые кислоты могут содержать участок (например, по меньшей мере 15 смежных нуклеотидов), который гибридизируется с сегментом, присутствующим в транскрипте А (или его фрагментах или гомологах) и отсутствующим в транскрипте D (или его фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей транскриптов. Транскрипт D HSD17B13 (SEQ ID NO: 7) включает вставку гуанина на 3'-конце экзона 6, что приводит в результате к сдвигу рамки считывания и преждевременному укорочению экзона 7 по сравнению с транскриптом A (SEQ ID NO: 4). Например, настоящее изобретение относится к выделенным нуклеиновым кислотам, которые гибридизируются по меньшей мере с 15 смежными нуклеотидами из нуклеиновой кислоты, кодирующей белок HSD17B13, причем смежные нуклеотиды содержат сегмент (например, по меньшей мере 5 смежных нуклеотидов, по меньшей мере 10 смежных нуклеотидов или по меньшей мере 15 смежных нуклеотидов), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен участку, охватывающему границу между экзоном 6 и экзоном 7 в SEQ ID NO: 4 (транскрипте A HSD17B13) при оптимальном выравнивании с SEQ ID NO: 4.
(5) Векторы
[00200] Также настоящее изобретение относится к векторам, содержащим любую из раскрываемых в настоящем документе нуклеиновых кислот и гетерологичную нуклеиновую кислоту. Векторы могут представлять собой вирусные или невирусные векторы, способные транспортировать нуклеиновую кислоту. В некоторых случаях вектор может представлять собой плазмиду (например, кольцевую двухцепочечную ДНК, в которую могут быть лигированы дополнительные сегменты ДНК). В некоторых случаях вектор может быть вирусным вектором, причем в вирусный геном могут быть лигированы дополнительные сегменты ДНК. В некоторых случаях вектор может автономно реплицироваться в клетке-хозяине, в которую он введен (например, бактериальные векторы, имеющие бактериальную точку начала репликации, и эписомальные векторы млекопитающих). В других случаях векторы (например, отличные от эписомальных векторов млекопитающих) могут встраиваться в геном клетки-хозяина при введении в клетку-хозяина и тем самым реплицироваться наряду с геномом хозяина. Кроме того, определенные векторы могут управлять экспрессией генов, с которыми они функционально связаны. Такие векторы могут называться «рекомбинантными векторами экспрессии» или «векторами экспрессии». Такие векторы также могут быть направленными векторами (т.е. экзогенными донорными последовательностями), как раскрыто в других разделах настоящего документа.
[00201] В некоторых случаях белки, кодируемые раскрываемыми генетическими вариантами, экспрессируются путем вставки нуклеиновых кислот, кодирующих раскрываемые генетические варианты, в векторы экспрессии так, чтобы гены были функционально связаны с необходимыми последовательностями для регуляции экспрессии, такими как последовательности для регуляции транскрипции и трансляции. К векторам экспрессии можно отнести, например, плазмиды, ретровирусы, аденовирусы, аденоассоциированные вирусы (AAV), вирусы растений, такие как вирус мозаики цветной капусты, вирус табачной мозаики, космиды, YAC, полученные из EBV эписомы и т.п. В отдельных случаях нуклеиновые кислоты, содержащие раскрываемые генетические варианты, можно лигировать в вектор так, чтобы последовательности для регуляции транскрипции и трансляции в пределах вектора выполняли свою предназначенную функцию регуляции транскрипции и трансляции генетического варианта. Вектор экспрессии и последовательности для регуляции экспрессии выбирают такие, чтобы они были совместимы с используемой для экспрессии клеткой-хозяином. Последовательности нуклеиновых кислот, содержащие раскрываемые генетические варианты, можно вставить в отдельные векторы или в один вектор экспрессии. Последовательность нуклеиновой кислоты, содержащую раскрываемые генетические варианты, можно вставить в вектор экспрессии стандартными способами (например, лигированием комплементарных сайтов рестрикции на нуклеиновой кислоте, содержащей раскрываемые генетические варианты, и векторе или лигированием дефосфолированного конца, если отсутствуют сайты рестрикции).
[00202] В дополнение к последовательности нуклеиновой кислоты, содержащей раскрываемые генетические варианты, рекомбинантные векторы экспрессии могут нести регуляторные последовательности, которые осуществляют регуляцию экспрессии генетического варианта в клетке-хозяине. Структура вектора экспрессии, включая выбор регуляторных последовательностей, может зависеть от таких факторов, как выбор подлежащей трансформации клетки-хозяина, необходимый уровень экспрессии белка и т.д. Предпочтительные регуляторные последовательности для экспрессии в клетке-хозяине млекопитающего могут включать, например, вирусные элементы, которые дают высокие уровни экспрессии белка в клетках млекопитающих, такие как промоторы и/или энхансеры, полученные из ретровирусных LTR, цитомегаловируса (CMV) (такие как промотор/энхансер CMV), вируса обезьян 40 (SV40) (такие как промотор/энхансер SV40), аденовируса (например, главный поздний промотор аденовируса (AdMLP)), полиомы и сильные промоторы млекопитающих, такие как нативные иммуноглобулиновые и актиновые промоторы. Дополнительное описание вирусных регуляторных элементов и их последовательностей представлено в патентах США №№5168062, 4510245 и 4968615, каждый из которых включен в настоящий документ посредством ссылки в полном своем объеме для всех целей. Также известны способы экспрессии полипептидов в бактериальных клетках или грибных клетках (например, дрожжевых клетках).
[00203] В дополнение к последовательности нуклеиновой кислоты, содержащей раскрываемые генетические варианты и регуляторные последовательности, рекомбинантные векторы экспрессии могут нести дополнительные последовательности, такие как последовательности, которые регулируют репликацию вектора в клетках-хозяевах (например, точку начала репликации) и селектируемые маркерные гены. Селектируемый маркерный ген может облегчить отбор клеток-хозяев, в которых был введен вектор (см., например, патенты США №№4399216, 4634665 и 5179017, каждый из которых включен в настоящий документ посредством ссылки в полном своем объеме для всех целей). Например, селектируемый маркерный ген может придавать устойчивость к лекарственным средствам, таким как G418, гигромицин или метотрексат, клетке-хозяину, в которую был введен вектор. К иллюстративным селектируемым маркерным генам относятся ген дигидрофолатредуктазы (DHFR) (для применения в клетках-хозяевах dhfr с отбором/амплификацией по метотрексату), ген neo (для отбора с помощью G418) и ген глутаматсинтетазы (GS).
В. Белки
[00204] В настоящем документе раскрыты выделенные белки HSD17B13 и их фрагменты, в частности, белки HSD17B13 и их фрагменты, произведенные вариантом rs72613567 HSD17B13.
[00205] Раскрываемые в настоящем документе выделенные белки могут содержать аминокислотную последовательность встречающегося в естественной среде белка HSD17B13 или могут содержать не встречающуюся в естественной среде последовательность. В одном примере не встречающаяся в естественной среде последовательность может отличаться от не встречающейся в естественной среде последовательности вследствие консервативных аминокислотных замен. Например, последовательность может быть идентичной, за исключением консервативных аминокислотных замен.
[00206] Раскрываемые в настоящем документе выделенные белки могут быть связаны или слиты с гетерологичными полипептидами или гетерологичными молекулами или метками, многочисленные примеры которых раскрыты в других разделах настоящего документа. Например, белки могут быть слиты с гетерологичным полипептидом, обеспечивающим повышенную или пониженную стабильность. Слитый домен или гетерологичный полипептид может быть расположен на N-конце, С-конце или внутри белка. Партнер слияния может, например, способствовать созданию Т-хелперных эпитопов (иммунологический партнер слияния) или может способствовать экспрессии белка (энхансер экспрессии) с более высокими выходами, чем нативный рекомбинантный белок. Некоторые партнеры по слиянию являются как иммунологическими, так и повышающими экспрессию партнерами по слиянию. Другие партнеры по слиянию могут быть подобраны таким образом, чтобы повысить растворимость полипептида или обеспечить нацеливание полипептида на необходимые внутриклеточные компартменты. Еще одни партнеры по слиянию включают аффинные маркеры, которые облегчают очистку полипептида.
[00207] Белок слияния может быть слит непосредственно с гетерологичной молекулой или может быть связан с гетерологичной молекулой через линкер, такой как пептидный линкер. Подходящие пептидные линкерные последовательности могут быть выбраны, например, на основании следующих факторов: (1) их способность принимать гибкую расширенную конформацию; (2) их неспособность принимать вторичную структуру, которая могла бы взаимодействовать с функциональными эпитопами на первом и втором полипептидах; и (3) отсутствие гидрофобных или заряженных остатков, которые могли бы вступать в реакцию с функциональными эпитопами полипептида. Например, пептидные линкерные последовательности могут содержать остатки Gly, Asn и Ser. Также в линкерной последовательности можно применять и другие почти нейтральные аминокислоты, такие как Thr и Ala. К аминокислотным последовательностям, которые можно эффективно использовать в качестве линкеров, относятся последовательности, раскрытые в Maratea et al. (1985) Gene 40:39-46; Murphy et al. (1986) Proc. Natl. Acad. Sci. USA 83:8258-8262; патенте США №4935233; и патенте США №4751180, каждый из которых включен в настоящий документ посредством ссылки в полном своем объеме. Линкерная последовательность обычно может иметь длину, например, от 1 до 50 аминокислот. Линкерные последовательности обычно не нужны, если первый и второй полипептиды имеют несущественные N-концевые аминокислотные участки, которые можно использовать для разделения функциональных доменов и предотвращения стерического влияния.
[00208] Белки также можно функционально связать с проникающим доменом. Например, проникающий домен можно получить из белка TAT HIV-1, проникающего мотива TLM из вируса гепатита В человека, MPG, Рер-1, VP22, проникающего пептида из вируса простого герпеса или полиаргининовой пептидной последовательности. См., например, WO 2014/089290, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Проникающий домен может быть локализован на N-конце, С-конце или в любом месте в белке Cas.
[00209] Белки также можно функционально связать с гетерологичным полипептидом для простоты отслеживания или очистки, таким как флуоресцентный белок, маркер для очистки или эпитопный маркер. Примеры флуоресцентных белков включают зеленые флуоресцентные белки (например, GFP, GFP-2, tagGFP, turboGFP, eGFP, Emerald, Azami Green, Monomeric Azami Green, CopGFP, AceGFP, ZsGreenl), желтые флуоресцентные белки (например, YFP, eYFP, Citrine, Venus, YPet, PhiYFP, ZsYellowl), синие флуоресцентные белки (например, eBFP, eBFP2, Azurite, mKalamal, GFPuv, Sapphire, T-sapphire), голубые флуоресцентные белки (например, eCFP, Cerulean, CyPet, AmCyanl, Midorii), красные флуоресцентные белки (например, mKate, mKate2, mPlum, мономер DsRed, mCherry, mRFP1, DsRed-Express, DsRed2, DsRed-Monomer, HcRed-Tandem, HcRedl, AsRed2, eqFP611, mRaspberry, mStrawberry, Jred) оранжевые флуоресцентные белки (например, mOrange, mKO, Kusabira-Orange, Monomeric Kusabira-Orange, mTangerine, tdTomato) и любой другой подходящий флуоресцентный белок. Примеры меток включают глутатион-S-трансферазу (GST), хитин-связывающий белок (СВР), мальтозосвязывающий белок, тиоредоксин (TRX), поли(NANP), маркер для тандемной аффинной очистки (ТАР), myc, AcV5, AU1, AU5, Е, ECS, Е2, FLAG, гемагглютинин (НА), nus, Softag 1, Softag 3, Strep, SBP, Glu-Glu, HSV, KT3, S, S1, T7, V5, VSV-G, гистидин (His), белок-носитель биотина и карбоксила (ВССР) и кальмодулин.
[00210] Выделенные белки по настоящему изобретению также могут содержать неприродные или модифицированные аминокислоты или пептидные аналоги. Например, существует множество D-аминокислот или аминокислот, которые имеют функциональный заместитель, отличный от встречающихся в естественной среде аминокислот. Раскрыты и противоположные стереоизомеры встречающихся в естественной среде пептидов, а также стереоизомеры пептидных аналогов. Эти аминокислоты можно легко включить в полипептидные цепи путем загрузки на молекулы тРНК выбранной аминокислоты и конструирования генетических конструкций, которые используют, например, амбер-кодоны для вставки аналога аминокислоты в пептидную цепь сайт-специфическим образом (Thorson et al. (1991) Methods Molec. Biol. 77:43-73; Zoller (1992) Current Opinion in Biotechnology 3:348-354; Ibba, (1995) Biotechnology & Genetic Engineering Reviews 13:197-216; Cahill et al. (1989) TIBS 14(10):400-403; Benner (1993) TIB Tech 12:158-163; и Ibba and Hennecke (1994) Biotechnology 12:678-682, каждая из которой включена в настоящий документ посредством ссылки в полном своем объеме для всех целей).
[00211] Можно получить молекулы, которые напоминают пептиды, но которые не присоединяются через природную пептидную связь. Например, связи для аминокислот или аналогов аминокислот могут включать CH2NH--, --CH2S--, --СН2-- --, --СН=СН-- (цис и транс), --СОСН2 --, --СН(ОН)СН2-- и --CHH2SO-- (см., например, Spatola, A.F. in Chemistry and Biochemistry of Amino Acids, Peptides, and Proteins, B. Weinstein, eds., Marcel Dekker, New York, p. 267 (1983); Spatola, A. F., Vega Data (March 1983), Vol. 1, Issue 3, Peptide Backbone Modifications (general review); Morley (1994) Trends Pharm Sci 15(12):463-468; Hudson et al. (1979) Int J Pept Prot Res 14:177-185; Spatola et al. (1986) Life Sci 38:1243-1249; Hann (1982) Chem. Soc Perkin Trans. I 307-314; Almquist et al. (1980) J. Med. Chem. 23:1392-1398; Jennings-White et al. (1982) Tetrahedron Lett 23:2533); Szelke et al., европейскую заявку ЕР 45665 CA (1982): 97:39405 (1982); Holladay et al. (1983) Tetrahedron. Lett 24:4401-4404; и Hruby (1982) Life Sci 31:189-199; каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Пептидные аналоги могут иметь более одного атома между участвующими в образовании связи атомами, такие как b-аланин, гаминомасляная кислота и тому подобное.
[00212] Аналоги аминокислот и пептидные аналоги зачастую имеют улучшенные или требуемые свойства, такие как более экономичное получение, более высокая химическая стабильность, улучшенные фармакологические свойства (период полужизни, всасываемость, активность, эффективность и т.д.), измененная специфичность (например, широкий спектр биологической активности), сниженная антигенность и другие требуемые свойства.
[00213] D-аминокислоты можно использовать для получения более стабильных пептидов, поскольку D-аминокислоты не распознаются пептидазами и т.п. Для получения более стабильных пептидов можно использовать системную замену одной или нескольких аминокислот в консенсусной последовательности на D-аминокислоту того же типа (например, D-лизин вместо L-лизина). Цистеиновые цистеина можно использовать для циклизации или соединения двух или более пептидов друг с другом. Это может быть полезным для ограничения пептидов в определенных конформациях (см., например, Rizo and Gierasch (1992) Ann. Rev. Biochem. 61:387, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей).
[00214] Также в настоящем документе раскрыты нуклеиновые кислоты, кодирующие любой из раскрываемых в настоящем документе белков. Сюда относятся все вырожденные последовательности, связанные с конкретной полипептидной последовательностью (т.е. все нуклеиновые кислоты, имеющие последовательность, которая кодирует одну отдельную полипептидную последовательность, а также все нуклеиновые кислоты, включая вырожденные нуклеиновые кислоты, кодирующие раскрываемые варианты и производные белковых последовательностей). Таким образом, несмотря на то, что каждая отдельная последовательность нуклеиновой кислоты не может быть записана в настоящей документе, все без исключения последовательности фактически раскрыты и описаны в настоящем документе посредством раскрытых полипептидных последовательностей.
[00215] Также в настоящем документе раскрыты композиции, содержащие раскрываемый в настоящем документе выделенный полипептид или белок и носитель, повышающий стабильность выделенного полипептида. Неограничивающие примеры таких носителей включают микросферы из полимолочной кислоты (PLA), микросферы из сополимера D,L-молочной и когликолиевой кислот (PLGA), липосомы, мицеллы, инверсные мицеллы, липидные кохлеаты и липидные микротрубочки.
(1) Белки и фрагменты HSD17B13
[00216] В настоящем документе раскрыты выделенные белки HSD17B13 и их фрагменты, в частности белок HSD17B13 и его фрагменты, полученный с помощью варианта rs72613567 HSD17B13, или, в частности, изоформы С, D, Е, F, F', G и Н HSD17B13. Такие белки могут включать, например, выделенный полипептид, содержащий по меньшей мере 5, 6, 8, 10, 12, 14, 15, 16, 18, 20, 22, 24, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 150, 200, 250 или 300 смежных аминокислот изоформы С, D, Е, F, F', G или Н HSD17B13 или ее фрагмента. Понятно, что последовательности генов в популяции и белки, кодируемые такими генами, могут варьировать по причине полиморфизмов, таких как однонуклеотидные полиморфизмы. Представленные в настоящем документе последовательности для каждой изоформы HSD17B13 являются лишь иллюстративными последовательностями. Также возможны и другие последовательности. Например, выделенный полипептид содержит аминокислотную последовательность (например, последовательность из смежных аминокислот), по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичную изоформе С, D, Е, F, F', G или Н HSD17B13 при оптимальном выравнивании с изоформой С, D, Е, F, F', G или Н соответственно. Необязательно, выделенный полипептид содержит последовательность, идентичную изоформе С, D, Е, F, F', G или Н HSD17B13.
[00217] В качестве одного примера, выделенный полипептид может содержать сегмент (например, по меньшей мере 8 смежных аминокислот), который присутствует в изоформах D, G и Н (или их фрагментах или гомологах) и которого нет в изоформе А (или ее фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей изоформ. Участок, кодируемый экзоном 7 в изоформах D, G и Н, имеет сдвинутую рамку считывания и укорочен по сравнению с участком, кодируемым экзоном 7 в изоформе А. Таким образом, такой выделенный полипептид может содержать по меньшей мере 5, 6, 8, 10, 12, 14, 15, 16, 18, 20, 22, 24, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 150 или 200 смежных аминокислот белка HSD17B13 (например, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот белка HSD17B13), причем сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислоты, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, включающему по меньшей мере часть участка, кодируемого экзоном 7 в SEQ ID NO: 15 (изоформе D HSD17B13), SEQ ID NO: 18 (изоформе G HSD17B13) или SEQ ID NO: 19 (изоформе Н HSD17B13) при оптимальном выравнивании выделенного полипептида с SEQ ID NO: 15, 18 или 19 соответственно.
[00218] Такие выделенные полипептиды могут дополнительно содержать сегмент, присутствующий в изоформе D (или ее фрагменте или гомологе) отсутствующий в изоформе G (или ее фрагменте или гомологе), и могут дополнительно содержать сегмент, присутствующий в изоформе D (или ее фрагменте или гомологе) и отсутствующий в изоформе Н (или ее фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей изоформ. Например, такие выделенные полипептиды могут содержать сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислот, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен сегменту, охватывающему границу участков, кодируемых экзонами 3 и 4 из SEQ ID NO: 15 (изоформы D HSD17B13) при оптимальном выравнивании с SEQ ID NO: 15, что позволяет отличать от изоформы Н. Аналогичным образом, такие выделенные полипептиды могут содержать сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислот, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен сегменту в пределах участка, кодируемого экзоном 2 из SEQ ID NO: 15 (изоформы D HSD17B13), сегменту, охватывающему границу участков, кодируемых экзонами 1 и 2 в SEQ ID NO: 15, или сегменту, охватывающему границу участков, кодируемых экзонами 2 и 3 в SEQ ID NO: 15 при оптимальном выравнивании с SEQ ID NO: 15, что позволяет отличать от изоформы G.
[00219] Как и изоформа D, участок, кодируемый экзоном 7 в изоформе Н (SEQ ID NO: 19), имеет сдвинутую рамку считывания и укорочен по сравнению с изоформой А. Тем не менее, изоформа Н дополнительно включает участок, кодируемый дополнительным экзоном (экзоном 3') между экзонами 3 и 4 по сравнению с изоформами А и D. Соответственно, такой выделенный полипептид может быть таким, как описано выше, и содержит сегмент, присутствующий в изоформах D, G и Н (или их фрагментах или гомологах) и отсутствующий в изоформе А (или ее фрагменте или гомологе), но дополнительно содержит сегмент (например, по меньшей мере 8 смежных аминокислот) из изоформы Н (или ее фрагмента или гомолога), который отсутствует в изоформе D (или ее фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей изоформ. Например, такой выделенный полипептид может дополнительно содержать сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислот, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или по меньшей мере на 100% идентичен сегменту, включающему по меньшей мере часть участка, кодируемого экзоном 3' из SEQ ID NO: 19 (изоформы H HSD17B13) при оптимальном выравнивании выделенного полипептида с SEQ ID NO: 19.
[00220] Как и изоформа D, участок, кодируемый экзоном 7 в изоформе G (SEQ ID NO: 18), имеет сдвинутую рамку считывания и укорочен по сравнению с изоформой А. Тем не менее, в изоформе G дополнительно отсутствует участок, кодируемый экзоном 2, по сравнению с изоформами А и D, и, следовательно, она включает границу между экзоном 1 и экзоном 3, отсутствующую в изоформах А и D. Соответственно, такой выделенный полипептид может быть таким, как описано выше, и содержит сегмент, присутствующий в изоформах D, G и Н (или их фрагментах или гомологах) и отсутствующий в изоформе А (или ее фрагменте или гомологе), но дополнительно содержит сегмент (например, по меньшей мере 8 смежных аминокислот) из изоформы G (или ее фрагмента или гомолога), который отсутствует в изоформе D (или ее фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей изоформ. Например, такой выделенный полипептид могут содержать сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислот, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, охватывающему границу участков, кодируемых экзонами 1 и 3 в SEQ ID NO: 18 (изоформе G HSD17B13) при оптимальном выравнивании выделенного полипептида с SEQ ID NO: 18.
[00221] Также настоящее изобретение относится к выделенным полипептидам, содержащим сегмент (например, по меньшей мере 8 смежных аминокислот), присутствующий в изоформе Е (или ее фрагменте или гомологе) и отсутствующий в изоформе А (или ее фрагменте или гомологе). Изоформа Е включает участок, кодируемый дополнительным экзоном (экзоном 3') между экзонами 3 и 4, которого нет в изоформе А. Такие участки можно легко выявить путем сравнения последовательностей изоформ. Следовательно, выделенный полипептид может содержать по меньшей мере 5, 6, 8, 10, 12, 14, 15, 16, 18, 20, 22, 24, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 150 или 200 смежных аминокислот белка HSD17B13 (например, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот белка HSD17B13), причем сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислоты, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, включающему по меньшей мере часть участка, кодируемого экзоном 3 в SEQ ID NO: 16 (изоформе Е HSD17B13) или SEQ ID NO: 19 (изоформе Н HSD17B13) при оптимальном выравнивании выделенного полипептида с SEQ ID NO: 16 или 19 соответственно. Необязательно, такой выделенный полипептид может дополнительно содержать сегмент (например, по меньшей мере 8 смежных аминокислот) из изоформы Е (или ее фрагмента или гомолога), отсутствующий в изоформе Н (или ее фрагменте или гомологе). Такие участки можно легко выявить путем сравнения последовательностей изоформ. Например, такой выделенный полипептид могут содержать сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислот, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот), который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, охватывающему границу участков, кодируемых экзонами 6 и 7 в SEQ ID NO: 16 (изоформе Е HSD17B13) при оптимальном выравнивании выделенного полипептида с SEQ ID NO: 16.
[00222] Также настоящее изобретение относится к выделенному полипептиду, содержащему сегмент (например, по меньшей мере 8 смежных аминокислот), присутствующий в изоформе F (или ее фрагменте или гомологе) и отсутствующий в изоформе А (или ее фрагменте или гомологе). Изоформа F включает участок, кодируемый участком считывания из экзона 6 в интрон 6, которого нет в изоформе А. Такие участки можно легко выявить путем сравнения последовательностей изоформ. Следовательно, выделенный полипептид может содержать по меньшей мере 5, 6, 8, 10, 12, 14, 15, 16, 18, 20, 22, 24, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 150 или 200 смежных аминокислот белка HSD17B13 (например, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот белка HSD17B13), причем сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислоты, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, включающему по меньшей мере часть участка, кодируемого участком считывания в интрон 6 в SEQ ID NO: 17 (изоформе F HSD17B13) при оптимальном выравнивании выделенного полипептида с SEQ ID NO: 17.
[00223] Также настоящее изобретение относится к выделенному полипептиду, содержащему сегмент (например, по меньшей мере 8 смежных аминокислот), присутствующий в изоформе С (или ее фрагменте или гомологе) и отсутствующий в изоформе А (или ее фрагменте или гомологе). В изоформе С пропущен участок, кодируемый экзоном 6, по сравнению с изоформой А, и она включает границу между экзоном 5 и экзоном 7, не присутствующую в изоформе А. Такие участки можно легко выявить путем сравнения последовательностей изоформ. Следовательно, выделенный полипептид может содержать по меньшей мере 5, 6, 8, 10, 12, 14, 15, 16, 18, 20, 22, 24, 25, 30, 35, 40, 45, 50, 60, 70, 80, 90, 100, 150 или 200 смежных аминокислот белка HSD17B13 (например, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот белка HSD17B13), причем сегмент из смежных аминокислот (например, по меньшей мере 3 смежных аминокислоты, по меньшей мере 5 смежных аминокислот, по меньшей мере 8 смежных аминокислот, по меньшей мере 10 смежных аминокислот или по меньшей мере 15 смежных аминокислот) по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен сегменту, охватывающему границу участков, кодируемых экзонами 5 и 7 в SEQ ID NO: 14 (изоформе С HSD17B13) при оптимальном выравнивании выделенного полипептида с SEQ ID NO: 14.
[00224] Любые из, выделенных полипептидов, раскрываемых в настоящем документе, можно связать с гетерологичной молекулой или гетерологичной меткой. Примеры таких гетерологичных молекул или меток раскрыты в других разделах настоящего документа. Например, гетерологичная молекула может быть Fc-доменом иммуноглобулина, пептидным маркером, как раскрыто в других разделах настоящего документа, полиэтиленгликолем, полисиаловой кислотой или гликолиевой кислотой.
(2) Способы получения белков или фрагментов HSD17B13
[00225] Также раскрыты способы получения любого из раскрываемых в настоящем документе белков HSD17B13 или их фрагментов. Такие белки HSD17B13 или их фрагменты можно получить любым подходящим способом. Например, белки HSD17B13 или их фрагменты можно получить из клеток-хозяев, содержащих нуклеиновые кислоты (например, рекомбинантные векторы экспрессии), кодирующие такие белки HSD17B13 или их фрагменты. Такие способы могут предусматривать культивирование клетки-хозяина, содержащей нуклеиновую кислоту (например, рекомбинантный вектор экспрессии), кодирующую белок HSD17B13 или его фрагмент, таким образом получая белок HSD17B13 или его фрагмент. Нуклеиновая кислота может быть функционально связанной с промотором, который активен в клетке-хозяине, и культивирование можно производить в условиях, при которых происходит экспрессия нуклеиновой кислоты. Такие способы могут дополнительно предусматривать выделение экспрессированного белка HSD17B13 или его фрагмента. Выделение может дополнительно предусматривать очистку белка HSD17B13 или его фрагмента.
[00226] Примеры подходящих систем для экспрессии белка включают системы экспрессии в бактериальных клетках (например, Escherichia coli, Lactococcus lactis), системы экспрессии в дрожжевых клетках (например, Saccharomyces cerevisiae, Pichia pastoris), системы экспрессии в клетках насекомых (например, опосредованная бакуловирусом экспрессия белка) и системы экспрессии в клетках млекопитающих.
[00227] Примеры нуклеиновых кислот, кодирующих белки HSD17B13 или их фрагменты, более подробно раскрыты в других разделах настоящего документа. Необязательно, такие нуклеиновые кислоты подвергнуты оптимизации кодона для экспрессии в клетке-хозяине. Необязательно, такие нуклеиновые кислоты функционально связаны с промотором, который активен в клетке-хозяине. Промотор может быть гетерологичным промотором (т.е. промотором, который не является встречающимся в естественной среде промотором HSD17B13). Примеры промоторов, подходящих для Escherichia coli, включают арабинозный промотор, промоторы lac, tac и Т7. Примеры промоторов, подходящих для Lactococcus lactis, включают промотор P170 и низиновый промотор. Примеры промоторов, подходящих для Saccharomyces cerevisiae, включают конститутивные промоторы, такие как алкогольдегидрогеназный (ADHI) или енолазный (ENO) промоторы, или индуцируемые промоторы, такие как РНО, CUP1, GAL1 и G10. Примеры промоторов, подходящих для Pichia pastoris, включают промотор гена алкогольоксидазы I (АОХ I), промотор гена глицеральдегид-3-фосфатдегидрогеназы (GAP) и промотор гена глутатионзависимой формальдегиддегидрогеназы (FLDI). Примером промотора, подходящего для опосредованной бакуловирусом системы, является поздний вирусный сильный полиэдриновый промотор.
[00228] Необязательно, нуклеиновая кислота дополнительно кодирует маркер, находящийся в одной в рамке считывания с белком HSD17B13 или его фрагментом, для облегчения очистки белка. Примеры маркеров раскрыты в других разделах настоящего документа. Такие маркеры могут, например, связываться с партнерским лигандом (например, иммобилизированный на смоле) так, чтобы меченный маркером белок можно выделить из всех других белков (например, белков клетки-хозяина). Аффинная хроматография, высокоэффективная жидкостная хроматография (ВЭЖХ) и эксклюзионная хроматография (SEC) являются примерами способов, которые можно применять для увеличения степени чистоты экспрессируемого белка.
[00229] Для получения белков HSD17B13 или их фрагментов также можно применять и другие способы. Например, два или более пептидов или полипептидов можно связать друг с другом методиками химии белков. Например, пептиды или полипептиды можно синтезировать химическими методами с применением либо Fmoc (9-флуоренилметилоксикарбонильной) или Вое (трет-бутилоксикарбоноильной) химии. Такие пептиды или полипептиды можно синтезировать с помощью стандартных химических реакций. Например, пептид или полипептид можно синтезировать и не отщепить его от синтетической смолы, в то же время другой фрагмент пептида или белка можно синтезировать и впоследствии отщепить от смолы, тем самым открывая концевую группу, которая функционально заблокирована на другом фрагменте. В результате реакций конденсации пептидов эти два фрагмента можно ковалентно связть через пептидную связь на их соответственно карбоксильном и аминоконце. (Grant GA (1992) Synthetic Peptides: A User Guide. W.H. Freeman and Co., NY. (1992); и Bodansky M and Trost В., Ed. (1993) Principles of Peptide Synthesis. Springer-Verlag Inc., NY, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). В качестве альтернативы, пептид или полипептид можно независимо синтезировать in vivo, как описано в настоящем документе. После выделения эти независимые пептиды или полипептиды можно связать с получением пептида или его фрагмента посредством аналогичных реакций конденсации пептидов.
[00230] Например, ферментативное лигирование клонированных или синтетических пептидных сегментов позволяет соединять относительно короткие пептидные фрагменты с получением более крупных пептидных фрагментов, полипептидов или целых белковых доменов (Abrahmsen L et al. (1991) Biochemistry 30:4151, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). В качестве альтернативы, можно использовать нативное химическое лигирование синтетических пептидов для синтетического построения больших пептидов или полипептидов из более коротких пептидных фрагментов. Этот способ может предусматривать двухэтапную химическую реакцию (Dawson et al. (1994) Science 266:116-119, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Первым этапом может быть хемоселективная реакция незащищенного синтетического пептида-тиоэфира с другим незащищенным пептидным сегментом, содержащим аминоконцевой остаток Cys, с получением связанного тиоэфирной связью промежуточного соединения в качестве исходного ковалентного продукта. Без изменения условий реакции это промежуточное соединение может подвергнуться спонтанной, быстрой внутримолекулярной реакции с образованием естественной пептидной связи в сайте лигирования (Baggiolini et al. (1992) FEBS Lett 307:97-101; Clark-Lewis et al. (1994) J Biol Chem 269:16075; Clark-Lewis et al. (1991) Biochemistry 30:3128; и Rajarathnam et al. (1994) Biochemistry 33:6623-6630, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей).
[00231] В качестве альтернативы, незащищенные пептидные сегменты можно связать химически, причем связь, образованная между пептидными сегментами в результате химического лигирования, представляет собой неестественную (непептидную) связь (Schnolzer et al. (1992) Science 256:221, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Эту методику используют для синтеза аналогов белковых доменов, а также большого количества относительно чистых белков с полной биологической активностью (deLisle Milton RC et al., Techniques in Protein Chemistry IV. Academic Press, New York, pp. 257-267 (1992), включена в настоящий документ посредством ссылки в полном своем объеме для всех целей).
С. Клетки
[00232] Также настоящее изобретение относится к клеткам (например, рекомбинантным клеткам-хозяевам), содержащим любые из раскрываемых в настоящем документе нуклеиновых кислот и белков. Клетки могут находиться в условиях in vitro, ex vivo или in vivo. Нуклеиновые кислоты можно связать с промоторными и другими регуляторными последовательностями так, чтобы происходила их экспрессия с образованием кодируемого белка. Возможна клетка любого типа.
[00233] Клетка может представлять собой, например, тотипотентную клетку или плюрипотентную клетку (например, эмбриональную стволовую (ES) клетку, такую как ES клетка грызуна, ES клетка мыши или ES клетка крысы). К тотипотентным клеткам относятся недифференцированные клетки, которые могут давать клетку любого типа, а к плюрипотентным клеткам относятся недифференцированные клетки, которые могут развиваться в несколько типов дифференцированных клеток. Такими плюрипотентными и/или тотипотентными клетками могут быть, например, ES клетки или ES-подобные клетки, такие как индуцированные плюрипотентные стволовые (iPS) клетки. К ES клеткам относятся эмбриональные тотипотентные или плюрипотентные клетки, которые способны принимать участие в формировании любой ткани у развивающегося эмбриона при введении в эмбрион. ES клетки можно получить из внутренней клеточной массы бластоцисты, и они могут дифференцироваться в клетки любого из трех зародышевых слоев позвоночных (энтодермы, эктодермы и мезодермы).
[00234] Клетка также может быть первичной соматической клеткой или клеткой, которая не является первичной соматической клеткой. Соматические клетки могут включать любую клетку, которая не является гаметой, зародышевой клеткой, гаметоцитом или недифференцированной стволовой клеткой. Клетка также может быть первичной клеткой. Первичные клетки включают клетки или культуры клеток, которые были выделены непосредственно из организма, органа или ткани. Первичные клетки включают клетки, которые не являются ни трансформированными, ни иммортализированными. Они включают любую клетку, полученную из организма, органа или ткани, которая ранее не была высеяна в культуре ткани или ранее была высеяна в культуре ткани, но не способна бесконечно пересеваться в культуре ткани. Такие клетки могут быть выделены общепринятыми методиками и включают, например, соматические клетки, гематопоэтические клетки, эндотелиальные клетки, эпителиальные клетки, фибробласты, мезенхимные клетки, кератиноциты, меланоциты, моноциты, мононуклеарные клетки, адипоциты, преадипоциты, нейроны, глиальные клетки, гепатоциты, скелетные миобласты и гладкомышечные клетки. Например, первичные клетки можно получить из соединительных тканей, мышечных тканей, тканей нервной системы или эпителиальных тканей.
[00235] Такие клетки также в норме не будут размножаться бесконечно, но, вследствие мутации или изменения, избежали нормального клеточного старения и вместо этого могут продолжать подвергаться делению. Такие мутации или изменения могут появляться естественным путем, или их можно индуцировать преднамеренно. Примеры иммортализированных клеток включают клетки яичника китайского хомячка (СНО), клетки эмбриональной почки человека (например, клетки HEK 293) и эмбриональные фибробласты мыши (например, клетки 3Т3). Хорошо известны многочисленные типы иммортализированных клеток. Иммортализированные или первичные клетки включают клетки, которые обычно применяют для культивирования или для экспрессии рекомбинантных генов или белков.
[00236] Клетка также может быть дифференцированной клеткой, такой как клетка печени (например, клетка печени человека).
[00237] Клетка может быть получена из любого источника. Например, клетка может быть эукариотической клеткой, клеткой животного, растительной клеткой или грибной (например, дрожжевой) клеткой. Такие клетки могут быть клетками рыб или клетками птиц, или такие клетки могут быть клетками млекопитающих, такими как клетки человека, клетки отличного от человека млекопитающего, клетки грызунов, клетки мыши или клетки крысы. К млекопитающим относят, например, людей, отличных от человека приматов, низших обезьян, высших обезьян, кошек, собак, лошадей, быков, оленя, бизона, овец, грызунов (например, мышей, крыс, хомяков, морских свинок), домашний скот (например, бычьи, такие как коровы, быки и т.д.; овечьи, такие как овцы, козы и т.д; и свиньи, такие как домашние свиньи и дикие кабаны). К птицам относят, например, кур, индеек, страуса, гусей, уток и т.д. Также включены домашние животные и сельскохозяйственные животные. Термин «отличное от человека животное» исключает людей.
[00238] В случае клеток мышей мышь может представлять собой любую линию, включая, например, линию 129, линию C57BL/6, линию BALB/c, линию Swiss Webster, смесь линий 129 и C57BL/6, смесь линий BALB/c и C57BL/6, смесь линий 129 и BALB/c и смесь линий BALB/c, C57BL/6 и 129. Например, мышь может быть получена, по крайней мере частично, от линии BALB/c (например, по меньшей мере приблизительно на 25%, по меньшей мере приблизительно на 50%, по меньшей мере приблизительно на 75% происходящей от линии BALB/c или приблизительно на 25%, приблизительно на 50%, приблизительно на 75% или приблизительно на 100% происходящей от линии BALB/c). В одном примере мышь представляет собой линию, содержащую 50% BALB/c, 25% C57BL/6 и 25% 129. В качестве альтернативы, мышь содержит линию или комбинацию линий, которая исключает BALB/c.
[00239] Примеры линий 129 включают 129Р1, 129Р2, 129Р3, 129X1, 129S1 (например, 129S1/SV, 129S1/Svlm), 129S2, 129S4, 129S5, 129S9/SvEvH, 129S6 (129/SvEvTac), 129S7, 129S8, 129Т1 и 129Т2. См., например, Festing et al. (1999) Mammalian Genome 10(8):836, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Примеры линий C57BL включают C57BL/A, C57BL/An, C57BL/GrFa, C57BL/Kal_wN, C57BL/6, C57BL/6J, C57BL/6ByJ, C57BL/6NJ, C57BL/10, C57BL/10ScSn, C57BL/10Cr и C57BL/Ola. Клетки мыши также происходят из смеси вышеупомянутой линии 129 и вышеупомянутой линии C57BL/6 (например, 50% 129 и 50% C57BL/6). Аналогичным образом, клетки мыши могут быть получены из смеси вышеупомянутых линий 129 или смеси вышеупомянутых линий BL/6 (например, линии 129S6 (129/SvEvTac)).
[00240] В случае клеток крысы крыса может представлять собой любую линию крыс, включая, например, линию крыс ACI, линию крыс Dark Agouti (DA), линию крыс Wistar, линию крыс LEA, линию крыс Sprague Dawley (SD) или линию крыс Fischer, такую как Fisher F344 или Fisher F6. Крысы также могут быть из линии, полученной из смеси двух или более указанных выше линий. Например, крыса может быть из линии DA или линии ACI. Линия крыс ACI характеризуется наличием цвета черный агути, белого брюшка и лап и гаплотипом RT1av1;. Такие линии доступны из различных источников, включая Harlan Laboratories. Линия крыс Dark Agouti (DA) характеризуется наличием покрова цвета агути и гаплотипом RT1av1. Такие крысы доступны из различных источников, включая Charles River и Harlan Laboratories. В некоторых случаях крысы получены от линии инбредных крыс. См., например, US 2014/0235933 A1, включенную в настоящий документ посредством ссылки в полном своем объеме для всех целей.
III. Способы модификации или изменения экспрессии HSD17B13
[00241] Настоящее изобретение относится к различным способам модификации клетки посредством применения любой комбинации нуклеазных средств, экзогенных донорных последовательностей, активаторов транскрипции, репрессоров транскрипции, антисмысловых молекул, таких как антисмысловая РНК, siRNA и shRNA, белков HSD17B13 или их фрагментов и векторов экспрессии для экспрессии рекомбинантного гена HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13. Способы можно осуществлять in vitro, ex vivo или in vivo. Нуклеазные средства, экзогенные донорные последовательности, активаторы транскрипции, репрессоры транскрипции, антисмысловые молекулы, такие как антисмысловая РНК, siRNA и shRNA, белки HSD17B13 или их фрагменты и векторы экспрессии можно вводить в клетку в любой форме и любыми способами, которые описаны в других разделах настоящего документа, и все или некоторые из них можно вводить одновременно или последовательно в любой комбинации. Некоторые способы предусматривают только изменение эндогенного гена HSD17B13 в клетке. Некоторые способы предусматривают только изменение экспрессии эндогенного гена HSD17B13 в результате применения активаторов или репрессоров транскрипции или в результате применения антисмысловых молекул, таких как антисмысловая РНК, siRNA и shРНК. Некоторые способы предусматривают только введение рекомбинантного гена HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13 или его фрагмент, в клетку. Некоторые способы предусматривают только введение белка HSD17B13 или его фрагмента в клетку (например, любой из или любую комбинацию из раскрытых в настоящем документе белков HSD17B13 или их фрагментов или любую из или любую комбинацию из раскрытых в настоящем документе изоформ А-Н HSD17B13 или их фрагментов). Например, такие способы могут предусматривать введение одной или нескольких изоформ С, D, F, G и Н HSD17B13 (или их фрагментов) в клетку или введение изоформы D HSD17B13 (или ее фрагмента) в клетку. В качестве альтернативы, такие способы могут предусматривать введение одной или нескольких изоформ А, В и Е или изоформ А, В, Е и F' HSD17B13 (или их фрагментов) в клетку или введение изоформы A HSD17B13 (или ее фрагмента) в клетку. Другие способы могут предусматривать как изменение эндогенного гена HSD17В13 в клетке, так и введение в клетку белка HSD17B13 или его фрагмента или рекомбинантного гена HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13 или его фрагмент. Еще одни способы могут предусматривать как изменение экспрессии эндогенного гена HSD17B13 в клетке, так и введение в клетку белка HSD17B13 или его фрагмента или рекомбинантного гена HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13 или его фрагмент.
А. Способы модификации нуклеиновых кислот HSD17B13
[00242] Настоящее изобретение относится к различным способам модификации гена HSD17B13 в геноме клетки (например, плюрипотентной клетки или дифференцированной клетки, такой как клетка печени) с применением нуклеазных средств и/или экзогенных донорных последовательностей. Способы можно осуществлять in vitro, ex vivo или in vivo. Нуклеазное средство можно применять отдельно или в комбинации с экзогенной донорной последовательностью. В качестве альтернативы, экзогенную донорную последовательность можно применять отдельно или в комбинации с нуклеазным средством.
[00243] Репарация в ответ на двухцепочечные разрывы (DSB) происходит в основном по двум консервативным путям репарации ДНК: негомологичное соединение концов (NHEJ) и гомологичная рекомбинация (HR). См. Kasparek & Humphrey (2011) Seminars in Cell & Dev. Biol. 22:886-897, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. NHEJ включает репарацию двух цепочечных разрывов в нуклеиновой кислоте путем прямого лигирования концов разрыва друг с другом или с экзогенной последовательностью без необходимости гомологичной матрицы. Лигирование несмежных последовательностей путем NHEJ часто может приводить к образованию делеций, вставок или транслокаций вблизи сайта двухцепочечного разрыва.
[00244] Репарация целевой нуклеиновой кислоты (например, гена HSD17B13), опосредованная экзогенной донорной последовательностью, может включать любой процесс обмена генетической информацией между двумя полинуклеотидами. Например, NHEJ также может приводить к целенаправленному встраиванию экзогенной донорной последовательности посредством прямого лигирования концов разрыва с концами экзогенной донорной последовательности (т.е. захват на основе NHEJ). Такая NHEJ-опосредованное целенаправленное встраивание может быть предпочтительным в случае вставки экзогенной донорной последовательности, когда сложно использовать пути гомологичной направленной репарации (HDR) (например, в неделящихся клетках, первичных клетках и клетках, у которых плохо реализуется гомологичная репарация ДНК). Кроме того, в отличие от гомологичной репарации, нет необходимости в сведениях касательно идентичности больших участков последовательностей, фланкирующих сайт расщепления (за пределами «липких» концов, создаваемых Cas-опосредованным расщеплением), что может быть полезным при попытке целенаправленной вставки в организмы, имеющие геномы, для которых доступны лишь ограниченные сведения о геномной последовательности. Встраивание может происходить посредством лигирования тупых концов между экзогенной донорной последовательностью и расщепленной геномной последовательностью или посредством лигирования липких концов (т.е. имеющих 5' или 3' выступов) с применением экзогенной донорной последовательности, которая фланкирована выступами, которые совместимы с выступами, созданными белком Cas в расщепленной геномной последовательности. См., например, US 2011/020722, WO 2014/033644, WO 2014/089290 и Maresca et al. (2013) Genome Res. 23(3):539-546, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Если лигируют тупые концы, может требоваться надрезание мишени и/или донора для создания участков с микрогомологией, необходимых для соединения фрагментов, что может привести к нежелательным изменениям в целевой последовательности.
[00245] Репарация также может происходить посредством гомологичной репарации (HDR) или гомологичной рекомбинации (HR). HDR или HR предусматривает форму репарации нуклеиновой кислоты, для которой может быть необходима гомология нуклеотидных последовательностей, использование «донорной» молекулы в качестве матрицы для репарации «целевой» молекулы (т.е. молекулы, которая была подвергнута двухцепочечному разрыву), и приводит к передаче генетической информации от донора к цели. Без желания привязываться к какой-либо конкретной теории, такой перенос может включать коррекцию несоответствий ДНК гетеродуплекса, которая образуется между поврежденной целевой и донорной последовательностью, и/или зависимую от синтеза гибридизацию цепей, при которой донорная цепь используется для повторного синтеза генетической информации, которая станет частью целевой последовательности, и/или аналогичные процессы. В некоторых случаях в целевую ДНК встраивается донорный полинуклеотид, часть донорного полинуклеотида, копия донорного полинуклеотида или часть копии донорного полинуклеотида. См. Wang et al. (2013) Cell 153:910-918; Mandalos et al. (2012) PLOS ONE 7:e45768:1-9; и Wang et al. (2013) Nat Biotechnol. 31:530-532, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00246] Целевые генетические модификации гена HSD17B13 в геноме можно осуществить путем приведения клетки в контакт с экзогенной донорной последовательностью, содержащей 5' гомологичное плечо, которое гибридизируется с 5' целевой последовательностью в целевом геномном локусе в гене HSD17B13, и 3' гомологичное плечо, которое гибридизируется с 3' целевой последовательностью в целевом геномном локусе в гене HSD17B13. Экзогенная донорная последовательность может подвергаться рекомбинации с целевым геномным локусом с образованием целевой генетической модификации в гене HSD17B13. В качестве одного примера, 5' гомологичное плечо может гибридизироваться с целевой последовательностью 5' от положения, соответствующего положению 12666 из SEQ ID NO: 2, а 3' гомологичное плечо может гибридизироваться с целевой последовательностью 3' от положения, соответствующего положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17В13 с SEQ ID NO: 2. Такие способы могут привести в результате, например, к созданию гена HSD17B13, в котором тимин вставлен между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ TD NO: 1 (или аденин вставлен в соответствующем положении на противоположной цепи). В качестве другого примера, 5' и 3' гомологичные плечи могут гибридизироваться соответственно с 5' и 3' целевыми последовательностями в положениях, соответствующих положениям, фланкирующим экзон 6 в SEQ ID NO: 1 при оптимальном выравнивании гена HSD17В13 с SEQ ID NO: 1. Такие способы могут приводить в результате, например, к созданию гена HSD17B13, в котором была делетерована последовательность, соответствующая экзону 6 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1. В качестве другого примера, 5' и 3' гомологичные плечи могут гибридизироваться соответственно с 5' и 3' целевыми последовательностями в положениях, соответствующих положениям, фланкирующим экзон 2 в SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1. Такие способы могут приводить в результате, например, к созданию гена HSD17В13, в котором была делетерована последовательность, соответствующая экзону 2 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17В13 с SEQ ID NO: 1. В качестве другого примера, 5' и 3' гомологичные плечи могут гибридизироваться соответственно с 5' и 3' целевыми последовательностями в положениях, соответствующих границе экзон 6/интрон 6 в SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1. В качестве другого примера, 5' и 3' гомологичные плечи могут гибридизироваться соответственно с 5' и 3' целевыми последовательностями в положениях, соответствующих экзону 6 и экзону 7 в SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1. Такие способы могут привести в результате, например, к созданию гена HSD17B13, в котором тимин вставлен между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1 (или аденин вставлен в соответствующем положении на противоположной цепи). В качестве другого примера, 5' и 3' гомологичные плечи могут гибридизироваться соответственно с 5' и 3' целевыми последовательностями в положениях, соответствующих положениям, фланкирующим участок или находящимся в участке, который соответствует донорному сплайс-сайту в интроне 6 из SEQ ID NO: 1 (т.е. в участку на 5'-конце интрона 6 в SEQ ID NO: 1). Такие способы могут привести в результате, например, к созданию гена HSD17B13, в котором нарушена структура донорного сплайс-сайта в интроне 6. Примеры экзогенных донорных последовательностей раскрыты в других разделах настоящего документа.
[00247] Целевые генетические модификации гена HSD17B13 в геноме также можно создать путем приведения клетки в контакт с нуклеазным средством, которое индуцирует один или несколько одноцепочечных или двухцепочечных разрывов в целевой последовательности в целевом геномном локусе в гене HSD17В13. Такие способы могут привести в результате, например, к созданию гена HSD17B13, в котором нарушена структура участка, соответствующего донорному сплайс-сайту в интроне 6 из SEQ ID NO: 1 (т.е. участка на 5'-конце интрона 6 в SEQ ID NO: 1). Примеры и варианты нуклеазных средств, которые можно применять в данных способах, описаны в других разделах настоящего документа.
[00248] Например, целевые генетические модификации гена HSD17B13 в геноме могут быть получены путем приведения клетки или генома клетки в контакт с белком Cas и одной или несколькими гидовыми РНК, которые гибридизируются с одной или несколькими последовательностями распознавания гидовой РНК в целевом геномном локусе в гене HSD17B13. То есть, целевые генетические модификации гена HSD17B13 в геноме могут быть получены путем приведения клетки или генома клетки в контакт с белком Cas и одной или несколькими гидовыми РНК, которые нацелены на одну или несколько целевых для гидовой РНК последовательностей в целевом геномном локусе в гене HSD17В13. Например, такие способы могут предусматривать приведение клетки в контакт с белком Cas и гидовой РНК, которые нацелены на целевую для гидовой РНК последовательность в гене HSD17B13. В качестве одного примера, целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 6 и/или интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. В качестве одного примера, целевая для гидовой РНК последовательность находится в участке, соответствующем экзону 6, и/или интрону 6, и/или экзону 7 (например, экзону 6 и/или интрону 6 либо экзону 6 и/или экзону 7) из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. В качестве другого примера, целевая для гидовой РНК последовательность может включать положение или находится в непосредственной близости от него, которое соответствует положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17В13 с SEQ ID NO: 2. Например, целевая для гидовой РНК последовательность может находиться в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от положения, соответствующего положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. В качестве еще одного примера, целевая для гидовой РНК последовательность может включать старт-кодон гена HSD17B13 или стоп-кодон гена HSD17B13 или находится в непосредственной близости от него. Например, целевая для гидовой РНК последовательность может находиться в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона или стоп-кодона. Белок Cas и гидовая РНК образуют комплекс, и белок Cas расщепляет целевую для гидовой РНК последовательность. Расщепление, производимое белком Cas, может создать двух цепочечный разрыв или одноцепочечный разрыв (например, если белок Cas является никазой). Такие способы могут привести в результате, например, к созданию гена HSD17B13, в котором нарушена структура участка, соответствующего донорному сплайс-сайту в интроне 6 из SEQ ID NO: 1 (т.е. участка на 5'-конце интрона 6 в SEQ ID NO: 1), нарушена структура старт-кодона, нарушена структура стоп-кодона или делетерована кодирующая последовательность. Примеры и варианты белков Cas (например, Cas9) и гидовых РНК, которые можно применять в данных способах, описаны в других разделах настоящего документа.
[00249] В некоторых способах можно применять два или более нуклеазных средств. Например, можно применять два нуклеазных средства, каждое из которых нацелено на целевую для нуклеазы последовательность в участке, соответствующем экзону 6 и/или интрону 6 или экзону 6 и/или экзону 7 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2, или включающем положение или находящемся в непосредственной близости от него, соответствующее положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2 (например, в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от положения, соответствующего положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2). Например, можно применять два нуклеазных средства, каждое из которых нацелено на целевую для нуклеазы последовательность в участке, соответствующем экзону 6, и/или интрону 6, и/или экзону 7 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. В качестве другого примера, можно применять два или более нуклеазных средств, каждое из которых нацелено на целевую для нуклеазы последовательность, включающую старт-кодон или находящуюся в непосредственной близости от него. В качестве другого примера, можно применять два нуклеазных средства, одно из которых нацелено на целевую для нуклеазы последовательность, включающую старт-кодон или находящуюся в непосредственной близости от него, а другое нацелено на целевую для нуклеазы последовательность, включающую стоп-кодон или находящуюся в непосредственной близости от него, причем расщепление, производимое нуклеазными средствами, может привести созданию делеции в кодирующем участке между двумя целевыми для нуклеазы последовательностями. В качестве еще одного примера, можно применять три или более нуклеазных средств, причем одно или несколько (например, два) нацелены на целевые для нуклеазы последовательности, включающие старт-кодон или находящиеся в непосредственной близости от него, а одно или несколько (например, два) нацелены на целевые для нуклеазы последовательности, включающие стоп-кодон или находящиеся в непосредственной близости от него, причем расщепление, производимое нуклеазными средствами, может приводить к делеции кодирующего участка между целевыми для нуклеазы последовательностями, включающими старт-кодон или находящимися в непосредственной близости от него, и целевой для нуклеазы последовательностью, включающей стоп-кодон или находящейся в непосредственной близости от него.
[00250] Необязательно, клетку можно дополнительно привести в контакт с одной или несколькими дополнительными гидовыми РНК, которые нацелены на дополнительные целевые для гидовой РНК последовательности в целевом геномном локусе в гене HSD17B13. При приведении клетки в контакт с одной или несколькими дополнительными гидовыми РНК (например, второй гидовой РНК, которые нацелены на вторую целевую для гидовой РНК последовательность) расщепление, выполняемое белком Cas, может создать два или более двухцепочечных разрывов или два или более о дно цепочечных разрывов (например, если белок Cas является никазой).
[00251] Необязательно, клетку можно дополнительно привести в контакт с одной или несколькими экзогенными донорными последовательностями, которые подвергаются рекомбинации с целевым геномным локусом в гене HSD17B13 с образованием целенаправленной генетической модификации. Примеры и варианты экзогенных донорных последовательностей, которые можно применять в данных способах, раскрыты в других разделах настоящего документа.
[00252] Белок Cas, гидовую(-ые) РНК и экзогенную(-ые) донорную(-ые) последовательность(-и) можно вводить в клетку в любой форме и любыми способами, как описано в других разделах настоящего документа, и все или несколько из белка(-ов) Cas, гидовой(-ых) РНК и экзогенной(-ых) донорной(-ых) последовательности(-ей) можно вводить одновременно или последовательно в любой комбинации.
[00253] В соответствии с некоторыми такими способами, репарация целевой нуклеиновой кислоты (например, гена HSD17B13) с участием экзогенной донорной последовательности происходит посредством гомологичной репарации (HDR). Гомологичная репарация может происходить, когда белок Cas расщепляет обе цепи ДНК в гене HSD17B13, создавая двухцепочечный разрыв, когда белок Cas представляет собой никазу, которая расщепляет одну цепь ДНК в целевой нуклеиновой кислоте, создавая одноцепочечный разрыв, или при применении никаз Cas для создания двухцепочечного разрыва, образованного двумя никазами, которые производят разрывы в смещенных друг относительно друга участках. В соответствии с такими способами, экзогенная донорная последовательность содержит 5' и 3' гомологичные плечи, соответствующие 5' и 3' целевым последовательностям. Целевая(-ые) для гидовой РНК последовательность(-и) или сайт(-ы) расщепления могут быть смежными с 5' целевой последовательности, смежными с 3' целевой последовательности, смежными как с 5' целевой последовательностью, так и к 3' целевой последовательностью или не быть смежными ни с 5' целевой последовательностью, ни с 3' целевой последовательностью. Необязательно, экзогенная донорная последовательность может дополнительно содержать вставку нуклеиновой кислоты, фланкированную 5' и 3' гомологичными плечами, и такая вставка нуклеиновой кислоты вставлена между 5' и 3' целевыми последовательностями. Если вставка нуклеиновой кислоты отсутствует, экзогенная донорная последовательность может выполнять свою функцию, удаляя геномную последовательности между 5' и 3' целевыми последовательностями. Примеры экзогенных донорных последовательностей раскрыты в других разделах настоящего документа.
[00254] В качестве альтернативы, репарация гена HSD17B13, опосредуемая экзогенной донорной последовательностью, может происходить путем сшивания, опосредованного негомологичным соединением концов (NHEJ). В соответствии с такими способами, по меньшей мере один конец экзогенной донорной последовательности содержит короткий одноцепочечный участок, который комплементарен по меньшей мере одному «липкому» концу, созданному путем Cas-опосредованного расщепления в гене HSD17B13. Комплементарный конец в экзогенной донорной последовательности может фланкировать вставку нуклеиновой кислоты. Например, каждый конец экзогенной донорной последовательности может содержать короткий одноцепочечный участок, который комплементарен «липкому» концу, созданному путем Cas-опосредованного расщепления в гене HSD17B13, и эти комплементарные участки в экзогенной донорной последовательности могут фланкировать вставку нуклеиновой кислоты.
[00255] «Липкие» концы (т.е. ступенчатые концы) можно создать путем надрезания тупых концов двухцепочечного разрыва, созданного в результате Cas-опосредованного расщепления. Такое надрезание может создавать участки с микрогомологией, необходимые для соединения фрагментов, но это может создавать нежелательные или неконтролируемые изменения в гене HSD17B13. В качестве альтернативы, такие «липкие» концы могут быть созданы с помощью парных никаз Cas. Например, клетку можно привести в контакт с первой и второй никазами, которые расщепляют противоположные цепи ДНК, в результате чего геном модифицируют посредством двойного одноцепочечного разреза. Это можно осуществить путем приведения клетки в контакта с первой никазой в форме белка Cas, первой гидовой РНК, которая нацелена на первую целевую для гидовой РНК последовательность в целевом геномном локусе в гене HSD17В13, второй никазой в форме белка Cas и второй гидовой РНК, которая нацелена на вторую целевую для гидовой РНК последовательность в целевом геномном локусе в гене HSD17B13. Первый белок Cas и первая гидовая РНК образуют первый комплекс, а второй белок Cas и вторая гидовая РНК образуют второй комплекс. Первая никаза в форме белка Cas расщепляет первую цепь геномной ДНК в первой целевой для гидовой РНК последовательности, вторая никаза в форме белка Cas расщепляет вторую цепь геномной ДНК во второй целевой для гидовой РНК последовательности, и, необязательно, экзогенная донорная последовательность подвергается рекомбинации с целевым геномным локусом в гене HSD17B13 с образованием целевой генетической модификации.
[00256] Первая никаза может расщеплять первую цепь геномной ДНК (т.е. комплементарную цепь), а вторая никаза может расщеплять вторую цепь геномной ДНК (т.е. не комплементарную цепь). Первую и вторую никазы можно создать, например, путем введения мутации в каталитический остаток в домене RuvC (например, мутации D10A, описанной в другом разделе настоящего документа) Cas9 или мутации каталитического остатка в домене HNH (например, мутации Н840А, описанной в другом разделе настоящего документа) Cas9. В соответствии с такими способами, можно использовать двойной одноцепочечный разрез для создания двухцепочечного разрыва, имеющего ступенчатые концы (т.е. «липкие» концы). Первую и вторую целевые для гидовой РНК последовательности можно расположить так, чтобы образовывался такой сайт расщепления, чтобы одноцепочечные разрезы, создаваемые первой и второй никазами на первой и второй цепях ДНК, создавали двухцепочечный разрыв. «Липкие» концы получаются при смещении одноцепочечных разрезов в первой и второй целевых последовательностей РНК CRISPR. Интервал смещения может составлять, например, по меньшей мере приблизительно 5 п.о., 10 п.о., 20 п.о., 30 п.о., 40 п.о., 50 п.о., 60 п.о., 70 п.о., 80 п.о., 90 п.о., 100 п.о. или более. См., например, Ran et al. (2013) Cell 154:1380-1389; Mali et al. (2013) Biotech 31:833-838; и Shen et al. (2014) Nat. Methods 11:399-404, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
(1) Типы целенаправленных генетических модификаций
[00257] С помощью описанных в настоящем документе способов можно вводить целенаправленные генетические модификации различных типов. Такие целенаправленные модификации могут включать, например, добавления одного или нескольких нуклеотидов, делеции одного или нескольких нуклеотидов, замены одного или нескольких нуклеотидов, точечную мутацию или их комбинацию. Например, можно изменить по меньшей мере 1, 2, 3, 4, 5, 7, 8, 9, 10 или более нуклеотидов (например, удалить, вставить или заменить) с получением целенаправленной геномной модификации. Делеции, вставки или замены могут быть любого размера, как раскрыто в других разделах настоящего документа. См., например, Wang et al. (2013) Cell 153:910-918; Mandalos et al. (2012) PLOS ONE 7:e45768:1-9; и Wang et al. (2013) Nat Biotechnol. 31:530-532, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00258] Такие целенаправленные генетические модификации могут привести в результате к нарушению структуры целевого геномного локуса. Нарушение структуры может предусматривать изменение регуляторного элемента (например, промотора или энхансера), миссенс-мутацию, нонсенс-мутацию, мутацию со сдвигом рамки считывания, мутацию укорочения, нуль-мутацию или вставку или делецию небольшого количества нуклеотидов (например, вызывая мутацию со сдвигом рамки считывания), и это может привести к инактивации (т.е. потере функции) или утрате аллеля. Например, целенаправленная модификация может включать нарушение структуры старт-кодона гена HSD17В13 так, чтобы старт-кодон больше не функционировал.
[00259] В конкретном примере целенаправленная модификация может включать делецию между первой и второй целевыми для гидовой РНК последовательностями или сайтами расщепления Cas. При использовании экзогенной донорной последовательность (например, репаративной матрицы или направленного вектор) модификация может включать делецию между первой и второй целевыми для гидовой РНК последовательностями или сайтами расщепления Cas, а также вставку нуклеиновой кислоты между 5' и 3' целевыми последовательностями.
[00260] В качестве альтернативы, при использовании экзогенной донорной последовательности отдельно или в комбинации с нуклеазным средством модификация может включать делецию между 5' и 3' целевыми последовательностями, а также вставку нуклеиновой кислоты между 5' и 3' целевыми последовательностями в паре первой и второй гомологичных хромосом, что приводит к получению гомозиготного модифицированного генома. В качестве альтернативы, если экзогенная донорная последовательность содержит 5' и 3' гомологичные плечи без вставки нуклеиновой кислоты, модификация может заключаться в делеции между 5' и 3' целевыми последовательностями.
[00261] Делеция между первой и второй целевыми для гидовой РНК последовательностями или делеция между 5' и 3' целевыми последовательностями может быть точной делецией, причем делетерованная нуклеиновая кислота состоит только из последовательности нуклеиновой кислоты между первым и вторым сайтами расщепления нуклеазой или только из последовательности нуклеиновой кислоты между 5' и 3' целевыми последовательностями, так, что в модифицированном целевом геномном локусе нет дополнительных делеций или вставок. Делеция между первой и второй целевыми для гидовой РНК последовательностями также может быть неточной делецией, выходящей за пределы первого и второго сайтов расщепления нуклеазой, что согласуется с неточной репарацией путем негомологичного соединения концов (NHEJ), что приводит в результате к созданию дополнительных делеций и/или вставок в модифицированном геномном локусе. Например, делеция может составлять в длину приблизительно 1 п.о., приблизительно 2 п.о., приблизительно 3 п.о., приблизительно 4 п.о., приблизительно 5 п.о., приблизительно 10 п.о., приблизительно 20 п.о., приблизительно 30 п.о., приблизительно 40 п.о., приблизительно 50 п.о., приблизительно 100 п.о., приблизительно 200 п.о., приблизительно 300 п.о., приблизительно 400 п.о., приблизительно 500 п.о., или более за пределами первого и второго сайтов расщепления белка Cas. Аналогичным образом, модифицированный геномный локус может содержать дополнительные вставки с учетом неточной репарации путем NHEJ, такие как вставки, составляющие приблизительно 1 п.о., приблизительно 2 п.о., приблизительно 3 п.о., приблизительно 4 п.о., приблизительно 5 п.о., приблизительно 10 п.о., приблизительно 20 п.о., приблизительно 30 п.о., приблизительно 40 п.о., приблизительно 50 п.о., приблизительно 100 п.о., приблизительно 200 п.о., приблизительно 300 п.о., приблизительно 400 п.о., приблизительно 500 п.о. или более.
[00262] Целенаправленная генетическая модификация может представлять собой, например, двухаллельную модификацию или одноаллельную модификацию. Двухаллельные модификации включают события, при которых одна и та же модификация производится в одном и том же локусе в соответствующих гомологичных хромосомах (например, в диплоидной клетке) или при которых разные модификации производятся в одном и том же локусе в соответствующих гомологичных хромосомах. В соответствии с некоторыми способами, целенаправленная генетическая модификация является одноаллельной модификацией. Одноаллельная модификация включает события, при которых модификация производится лишь в одном аллеле (т.е. модификация в гене HSD17B13 лишь в одной из двух гомологичных хромосом). Гомологичные хромосомы включают хромосомы, которые имеют одинаковые гены в одних и тех же локусах, но, возможно, разные аллели (например, хромосомы, которые образуют пару во время мейоза). Термин аллель включает любую из одной или нескольких альтернативных форм генетической последовательности. В диплоидной клетке или диплоидном организме два аллеля данной последовательности обычно занимают соответствующие локусы на паре гомологичных хромосом.
[00263] Одноаллельная мутация может привести к образованию клетки, гетерозиготной по целенаправленной модификации HSD17B13. Гетерозиготность включает ситуацию, при которой лишь один аллель гена HSD17В13 (т.е. соответствующие аллели на обеих гомологичных хромосомах) имеет целенаправленную модификацию.
[00264] Двухаллельная модификация может привести к гомозиготности по целенаправленной модификации. Гомозиготность включает ситуации, при которых оба аллеля гена HSD17B13 (т.е. соответствующие аллели на обеих гомологичных хромосомах) имеют целенаправленную модификацию. В качестве альтернативы, двухаллельная модификация может приводить к сложной гетерозиготности (например, гемизиготности) по целенаправленной модификации. Сложная гетерозиготность включает ситуации, при которых были модифицированы оба аллеля целевого локуса (т.е. аллели на обеих гомологичных хромосомах), но они были модифицированы различными способами (например, целенаправленная модификация в одном аллеле и инактивация или нарушение структуры в другом аллеле). Например, в аллеле без целенаправленной модификации двух цепочечный разрыв, создаваемый белком Cas, может быть репарирован путем репарации ДНК, опосредованной негомологичным соединением концов (NHEJ), что приводит к образованию мутантного аллеля, содержащего вставку или делецию последовательности нуклеиновой кислоты, и, следовательно, вызывает нарушение в данном геномном локусе. Например, двухаллельная модификация может привести к сложной гетерозиготности, если клетка имеет один аллель с целенаправленной модификацией и другой аллель, который не способен к экспрессии. Сложная гетерозиготность включает гемизиготность. Гемизиготность включает ситуации, при которых присутствует лишь один аллель (т.е. аллель на одной из двух гомологичных хромосом) целевого локуса. Например, двухаллельная модификация может привести к гемизиготности по целенаправленной модификации, если целенаправленная модификация происходит в одном аллеле с соответствующей утратой или делецией другого аллеля.
(2) Выявление клеток с целенаправленными генетическими модификациями
[00265] Раскрываемые в настоящем документе способы могут дополнительно предусматривать выявление клеток, имеющих модифицированный ген HSD17B13. Для выявления клеток, имеющих целевую генетическую модификацию, такую как делеция или вставка, можно применять различные способы. Такие способы могут предусматривать выявление одной клетки, имеющей целенаправленную генетическую модификацию в гене HSD17B13. Для выявления таких клеток с модифицированными геномными локусами можно провести скрининг.
[00266] Этап скрининга может включать количественный анализ для оценки модификации аллеля (МОА) (например, анализы утраты аллеля (LOA) и/или приобретения аллеля (GOA)) родительской хромосомы. Например, количественный анализ можно проводить с помощью количественной ПЦР, такой как ПЦР в режиме реального времени (qPCR). ПЦР в режиме реального времени может предусматривать использование первого набора праймеров, который распознает целевой геномный локус, и второго набора праймеров, который распознает нецелевой эталонный локус. Набор праймеров может содержать флуоресцентный зонд, который распознает амплифицированную последовательность. Анализ утраты аллеля (LOA) инвертирует традиционную логику скрининга и предусматривает количественное определение количество копий нативного локуса, на который была направлена мутация. В клоне клеток с правильной целенаправленной модификацией с помощью анализа LOA детектируют один из двух нативных аллелей (для генов, отсутствующих в хромосоме X или Y), причем у другого аллеля нарушена структура в результате целенаправленной модификации. Тот же принцип можно применить в обратном порядке в качестве анализа приобретения аллеля (GOA) для количественного определения количества копий вставленного направленного вектора. Например, комбинированное применение анализов GOA и LOA позволит выявить гетерозиготный клон с правильной целенаправленной модификацией, который утратил одну копию нативного целевого гена и приобрел одну копию гена устойчивости к лекарственному средству или другого вставленного маркера.
[00267] В качестве примера, в качестве способа количественного определения аллелей можно применять количественную полимеразную цепную реакцию (qPCR), но для разработки анализа МОА можно применять любой способ, который может надежно различать разницу между отсутствием, одной и двумя копиями целевого гена или между отсутствием, одной и двумя копиями вставки нуклеиновой кислоты. Например, можно применять TAQMAN® для определения количества копий ДНК-матрицы в образце геномной ДНК, особенно путем сравнения с эталонным геном (см., например, патент US 6596541, включен в настоящий документ посредством ссылки в полном его объеме для всех целей). Эталонный ген количественно определяют в той же геномной ДНК, что и целевой(-ые) ген(-ы) или локус(-ы). Поэтому проводят две амплификации TAQMAN® (каждая со своим соответствующим зондом). Один зонд TAQMAN® определяет «Ct» (пороговый цикл) эталонного гена, тогда как другой зонд определяет Ct участка целевого(-ых) гена(-ов) или локуса(-ов), который заменяется при успешной целенаправленной модификации (т.е. анализ LOA). Ct представляет собой количество, которое отражает количество исходной ДНК для каждого из зондов TAQMAN®, т.е. для менее представленной последовательности необходимо большее количество циклов ПНР для достижения порогового цикла. Уменьшение вдвое количества копий матричной последовательности для реакции TAQMAN® приведет к увеличению приблизительно на одну единицу Ct. Реакции TAQMAN® в клетках, в которых один аллель целевого(-ых) гена(-ов) или локуса(-ов) был заменен путем гомологичной рекомбинации, приведут к увеличению на одну Ct для целевой реакции TAQMAN® без увеличения Ct для эталонного гена по сравнению с ДНК из нецелевых клеток. В случае анализа GOA можно использовать другой зонд TAQMAN® для определения Ct вставки нуклеиновой кислоты, которая заменяет целевой(-ые) ген(-ы) или локус(-ы) при успешной целенаправленной модификации.
[00268] Другие примеры подходящих количественных анализов включают флуоресцентную гибридизацию in situ (FISH), сравнительную геномную гибридизацию, изотермическую амплификацию ДНК, количественную гибридизацию с иммобилизованным(-и) зондом(-ами), зонды INVADER®, зонды TaqMan® от Molecular Beacon или методику с зондом ECLIPSE™ (см., например, US 2005/0144655, включена в настоящий документ посредством ссылки в полном ее объеме для всех целей). Для скрининга целенаправленных модификаций также можно использовать традиционные анализы, такие как ПЦР длинных фрагментов, Саузерн-блоттинг или секвенирование по Сэнджеру. Такие анализы обычно применяют для получения доказательств связи между вставленным направленным вектором и целевым геномным локусом. Например, для анализа ПЦР длинных фрагментов один праймер может распознавать последовательность во вставленной ДНК, в то время как другой распознает последовательность целевого геномного локуса за пределами концов гомологичных плеч направленного вектора.
[00269] Для скрининга также можно применять секвенирование следующего поколения (NGS). Секвенирование следующего поколения также можно называть «NGS» или «массивно-параллельное секвенирование» или «высокопроизводительное секвенирование». В раскрываемых в настоящем документе способах нет необходимости проводить скрининг в отношении целевых клеток с применением маркеров отбора. Например, можно положиться на результаты описанных в настоящем документе анализов МОА и NGS без применения кассет отбора.
В. Способы изменения экспрессии нуклеиновых кислот HSD17B13
[00270] Предусмотрены различные способы изменения экспрессии нуклеиновых кислот, кодирующих белки HSD17B13. В соответствии с некоторыми способами, экспрессию изменяют посредством расщепления нуклеазным средством, вызывая нарушение структуры нуклеиновой кислоты, кодирующей белок HSD17B13, как описано более подробно в других разделах настоящего документа. В соответствии с некоторыми способами, экспрессию изменяют посредством применения ДНК-связывающего белка, слитого или связанного с доменом активации транскрипции или доменом репрессии транскрипции. В соответствии с некоторыми способами, экспрессию изменяют посредством применения композиций для РНК-интерференции, таких как антисмысловая РНК, shRNA или siRNA.
[00271] В одном примере экспрессию гена HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13, можно модифицировать путем приведения клетки или генома в клетке в контакт с нуклеазным средством, которое индуцирует один или несколько одноцепочечных разрывов или двухцепочечных разрывов в целевой последовательности в целевой геном в гене HSD17B13 или нуклеиновой кислоте, кодирующей белок HSD17B13. Такое расщепление может привести к нарушению экспрессии гена HSD17B13 и ли нуклеиновой кислоты, кодирующей белок HSD17B13. Например, целевая для нуклеазы последовательность может включать старт-кодон гена HSD17B13 или находится в непосредственной близости от него. Например, целевая последовательность может находиться в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона, и расщепление нуклеазным средством может нарушать структуру стоп-кодона. В качестве другого примера, можно применять два или более нуклеазных средств, каждое из которых нацелено на целевую для нуклеазы последовательность, включающую старт-кодон или находящуюся в непосредственной близости от него. В качестве другого примера, можно применять два нуклеазных средства, одно из которых нацелено на целевую для нуклеазы последовательность, включающую старт-кодон или расположенную в непосредственной близости от него, а другое нацелено на целевую для нуклеазы последовательность, включающую стоп-кодон или находящуюся в непосредственной близости от него, причем расщепление, производимое нуклеазными средствами, может привести к делеции кодирующего участка между двумя целевыми для нуклеазы последовательностями. В качестве еще одного примера, можно применять три или более нуклеазных средств, причем одно или несколько (например, два) нацелены на целевые для нуклеазы последовательности, включающие старт-кодон или находящиеся в непосредственной близости от него, а одно или несколько (например, два) нацелены на целевые для нуклеазы последовательности, включающие стоп-кодон или находящиеся в непосредственной близости от него, причем расщепление, производимое нуклеазными средствами, может приводить к делеции кодирующего участка между целевыми для нуклеазы последовательностями, включающими старт-кодон или находящимися в непосредственной близости от него, и целевой для нуклеазы последовательностью, включающей стоп-кодон или находящейся в непосредственной близости от него. Другие примеры модификации гена HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13, раскрыты в других разделах настоящего документа.
[00272] В другом примере экспрессия гена HSD17В13 или нуклеиновой кислоты, кодирующей белок HSD17B13, можно модифицировать путем приведения клетки или генома в клетке в контакт с ДНК-связывающим белком, который связывается с целевым геномным локусом в гене HSD17B13. ДНК-связыващий белок может быть, например, обладающим нуклеазной активностью белком Cas, слитым с доменом-активатором транскрипции или доменом-репрессором транскрипции. Другие примеры ДНК-связывающих белков включают белки с цинковыми пальцами, слитые с доменом-активатором транскрипции или доменом-репрессором транскрипции, или эффекторные белки, подобные активатору транскрипции (TALE), слитые с доменом-активатором транскрипции или доменом-репрессором транскрипции. Примеры таких белков раскрыты в других разделах настоящего документа. Например, в некоторых способах репрессор транскрипции можно применять для уменьшения экспрессии гена HSD17B13 дикого типа или гена HSD17B13, который не является вариантом rs72613567 (например, для уменьшения экспрессии транскрипта или изоформы A HSD17B13). Аналогичным образом, в некоторых способах можно применять активатор транскрипции для увеличения экспрессии варианта rs72613567 гена HSD17B13 (например, для увеличения экспрессии транскрипта или изоформы D HSD17B13).
[00273] Целевая последовательность (например, целевая для гидовой РНК последовательность) для ДНК-связывающего белка может находиться в любом подходящем для изменения экспрессии месте в гене HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13. В качестве одного примера, целевая последовательность может находиться в регуляторном элементе, таком как энхансер или промотор, или может находиться в непосредственной близости от регуляторного элемента. Например, целевая последовательность может включать старт-кодон гена HSD17B13 или находиться в непосредственной близости от него. Например, целевая последовательность может находиться в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона.
[00274] В другом примере антисмысловые молекулы можно применять для изменения экспрессии гена HSD17B13 или нуклеиновой кислоты, кодирующей белок HSD17B13. Примеры антисмысловых молекул включают антисмысловые РНК, малые интерферирующие РНК (siRNA) и короткие шпилечные РНК (shRNA). Такие антисмысловые РНК, siRNA или shRNA могут быть сконструированы для нацеливания на любой участок мРНК. Например, антисмысловые РНК, siRNA или shRNA могут быть сконструированы для нацеливания на участок, уникальный для одного или нескольких из раскрываемых в настоящем документе транскриптов HSD17B13 или участок, общий для одного или нескольких раскрываемых в настоящем документе транскриптов HSD17B13. Примеры нуклеиновых кислот, гибридизующихся с кДНК и транскриптами варианта HSD17B13, раскрыты более подробно в других разделах настоящего документа. Например, антисмысловая РНК, siRNA или shRNA может гибридизироваться с последовательностью в SEQ ID NO: 4 (транскрипте A HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA может уменьшать экспрессию транскрипта A HSD17B13 в клетке. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 4 (транскрипте A HSD17B13) и отсутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 4 (транскрипта A HSD17B13).
[00275] В качестве другого примера, антисмысловая РНК, siRNA или shRNA может гибридизироваться с последовательностью в SEQ ID NO: 7 (транскрипте D HSD17В13). Необязательно, антисмысловая РНК, siRNA или shRNA может уменьшать экспрессию транскрипта D HSD17B13 в клетке. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13) и отсутствующей в SEQ ID NO: 4 (транскрипте A HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 7 (транскрипта D HSD17B13).
С. Введение нуклеиновых кислот и белков в клетки
[00276] Раскрываемые в настоящем документе нуклеиновые кислоты и белки можно вводить в клетку любым способом. «Введение» предусматривает представление клетке нуклеиновой кислоты или белка таким образом, чтобы последовательность получала доступ к внутреннему содержимому клетки. Введение можно осуществлять любым способом, и один или несколько компонентов (например, два компонента или все компоненты) можно введены в клетку одновременно или последовательно в любой комбинации. Например, экзогенную донорную последовательность можно вводить до введения нуклеазного средства, или ее можно вводить после введения нуклеазного средства (например, экзогенную донорную последовательность можно вводить приблизительно через 1, 2, 3, 4, 8, 12, 24, 36, 48 или 72 часа до или после введения нуклеазного средства). См., например, US 2015/0240263 и US 2015/0110762, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Приведение генома клетки в контакт с нуклеазным средством или экзогенной донорной последовательностью может предусматривать введение в клетку одного или нескольких нуклеазных средств или нуклеиновых кислот, кодирующих нуклеазные средства (например, одного или нескольких белков Cas или нуклеиновых кислот, кодирующих один или несколько белков Cas, и одной или нескольких гидовых РНК или нуклеиновых кислот, кодирующих одну или несколько гидовых РНК (т.е. одну или несколько РНК CRISPR и одну или несколько tracrRNA)) и/или одной или нескольких экзогенных донорных последовательностей. Приведение в контакт с геномом клетки (т.е. приведение в контакт с клеткой) может предусматривать введение в клетку лишь одного из указанных выше компонентов, одного или нескольких компонентов или всех компонентов.
[00277] Нуклеазное средство можно вводить в клетку в форме белка или в форме нуклеиновой кислоты, кодирующей нуклеазное средство, такой как РНК (например, матричной РНК (мРНК)) или ДНК. При введении в форме ДНК она может быть функционально связана с активным в клетке промотором. Такие ДНК могут находиться в одной или нескольких экспрессирующих конструкциях.
[00278] Например, белок Cas можно вводить в клетку в форме белка, такого как белок Cas в комплексе с gRNA, или в форме нуклеиновой кислоты, кодирующей белок Cas, такой как РНК (например, матричная РНК (мРНК)) или ДНК. Гидовую РНК можно вводить в клетку в форме РНК или в форме ДНК, кодирующей гидовую РНК. При введении в форме ДНК ДНК, кодирующая белок Cas и/или гидовую РНК, может быть функционально связана с активным в клетке промотором. Такие ДНК могут находиться в одной или нескольких экспрессирующих конструкциях. Например, такие экспрессирующие конструкции могут быть компонентами одной молекулы нуклеиновой кислоты. В качестве альтернативы, они могут быть разделены в любой комбинации между двумя или более молекулами нуклеиновой кислоты (т.е. ДНК, кодирующими одну или несколько РНК CRISPR, ДНК, кодирующими одну или несколько tracrRNA, и ДНК, кодирующими белок Cas, могут быть компонентами отдельных молекул нуклеиновой кислоты).
[00279] В соответствии с некоторыми способами, ДНК, кодирующую нуклеазное средство (например, белок Cas и гидовую РНК), и/или ДНК, кодирующую экзогенную донорную последовательность, можно вводить в клетку с помощью миниколец ДНК. См., например, WO 2014/182700, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Миникольца ДНК представляют собой сверхспиральные молекулы ДНК, которые можно применять для невирусного переноса генов, которые не имеют ни точки начала репликации, ни маркера отбора с помощью антибиотиков. Поэтому миникольца ДНК обычно меньше по размеру, чем плазмидный вектор. Такие ДНК лишены бактериальной ДНК и, следовательно, лишены неметилированных CpG-мотивов, встречающихся в бактериальной ДНК.
[00280] Представленные в настоящем документе способы не зависят от конкретного способа введения нуклеиновой кислоты или белка в клетку, зависят лишь от того, чтобы нуклеиновая кислота или белок получили доступ во внутреннюю часть по меньшей мере одной клетки. Способы введения нуклеиновых кислот и белков в клетки различных типов известны и включают, например, способы стабильной трансфекции, способы временной трансфекции и опосредованные вирусом способы.
[00281] Протоколы трансфекции, а также протоколы введения нуклеиновых кислот или белков в клетки могут варьировать. Неограничивающие способы трансфекции включают химические способы трансфекции с применением липосом; наночастиц; фосфата кальция (Graham et al. (1973) Virology 52 (2): 456-67, Bacchetti et al. (1977) Proc. Natl. Acad. Sci. USA 74 (4): 1590-4, и Kriegler, M (1991). Transfer and Expression: A Laboratory Manual. New York: W.H. Freeman and Company, pp. 96-97); дендримеров или катионных полимеров, таких как DEAE-декстран или полиэтиленимин. Нехимические методы предусматривают электропорацию, сонопорацию и оптическую трансфекцию. Трансфекция на основе частиц предусматривает применение генной пушки или магнитной трансфекции (Bertram (2006) Current Pharmaceutical Biotechnology 7, 277-28). Для трансфекции также можно применять вирусные способы.
[00282] Введение нуклеиновых кислот или белков в клетку также может быть опосредовано электропорацией, внутрицитоплазматической инъекцией, вирусной инфекцией, аденовирусом, аденоассоциированным вирусом, лентивирусом, ретровирусом, трансфекцией, опосредованной липидами трансфекцией или нуклеофекцией. Нуклеофекция представляет собой усовершенствованную методику электропорации, которая позволяет доставлять субстраты нуклеиновых кислот не только в цитоплазму, но также через ядерную мембрану и в ядро. Кроме того, при применении нуклеофекции в раскрываемых в настоящем документе способах обычно необходимо гораздо меньше клеток, чем при обычной электропорации (например, лишь приблизительно 2 миллиона по сравнению с 7 миллионами при обычной электропорации). В одном примере нуклеофекцию производят с помощью системы LONZA® NUCLEOFECTOR™.
[00283] Введение нуклеиновых кислот или белков в клетку также можно осуществить путем микроинъекции. Микроинъекцию мРНК предпочтительно осуществляют в цитоплазму (например, для доставки мРНК непосредственно в механизм трансляции), тогда как микроинъекцию белка или ДНК, кодирующей белок Cas, предпочтительно осуществляют в ядро. В качестве альтернативы, микроинъекцию можно осуществлять путем инъекции как в ядро, так и в цитоплазму: сначала иглу можно ввести в ядро, и можно ввести первое количество, а при удалении иглы из клетки в цитоплазму можно ввести второе количество. Если в цитоплазму вводят нуклеазный белок, белок предпочтительно содержит сигнал ядерной локализации для обеспечения доставки в ядро/пронуклеус. Способы осуществления микроинъекции хорошо известны. См., например, Nagy et al. (Nagy A, Gertsenstein M, Vintersten K, Behringer R., 2003, Manipulating the Mouse Embryo. Cold Spring Harbor, New York: Cold Spring Harbor Laboratory Press); Meyer et al. (2010) Prac. Natl. Acad. Sci. USA 107:15022-15026 и Meyer et al. (2012) Proc. Natl. Acad. Sci. USA 109:9354-9359.
[00284] Другие способы введения нуклеиновой кислоты или белков в клетку могут предусматривать, например, векторную доставку, опосредованную частицами доставку, опосредованную экзосомами доставку, опосредованную липидными наночастицами доставку, доставку, опосредованную проникающими пептидами, или доставку, опосредованную имплантируемым устройством. Способы введения нуклеиновых кислот или белков субъекту для модификации клеток in vivo раскрыты в других разделах настоящего документа.
[00285] Введение нуклеиновых кислот и белков в клетки также можно осуществить с помощью гидродинамической доставки (HDD). Гидродинамическая доставка стала почти идеальным способом для внутриклеточной доставки ДНК in vivo. Для доставки генов в паренхиматозные клетки необходимо вводить инъекцией через выбранный кровеносный сосуд только необходимые последовательности ДНК, что устраняет проблемы безопасности, ассоциированные с современными вирусными и синтетическими векторами. При введении инъекцией в кровоток ДНК может достичь клетки в различных тканях, доступных для крови. В гидродинамической доставке используется сила, создаваемая быстрой инъекцией большого объема раствора в несжимаемую кровь в кровотоке для преодоления физических барьеров эндотелия и клеточных мембран, которые препятствуют проникновению в паренхиматозные клетки крупных и непроницаемых для мембран соединений. Помимо доставки ДНК этот способ полезен для эффективной внутриклеточной доставки РНК, белков и других небольших соединений in vivo. См., например, Bonamassa et al. (2011) Pharm. Res. 28(4):694-701, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00286] Другие способы введения нуклеиновой кислоты или белков в клетку могут предусматривать, например, векторную доставку, опосредованную частицами доставку, опосредованную экзосомами доставку, опосредованную липидными наночастицами доставку, доставку, опосредованную проникающими пептидами, или доставку, опосредованную имплантируемым устройством. В качестве конкретных примеров нуклеиновую кислоту или белок можно вводить в клетку в носителе, таком как микросфера из полимолочной кислоты (PLA), микросфера из сополимера D,L-молочной и гликолевой кислот (PLGA), липосома, мицелла, обратная мицелла, липидный кохлеат или липидная микротрубочка.
[00287] Введение нуклеиновых кислот или белков в клетку можно осуществлять один раз или несколько раз за период времени. Например, введение можно осуществлять по меньшей мере два раза за период времени, по меньшей мере три раза за период времени, по меньшей мере четыре раза за период времени, по меньшей мере пять раз за период времени, по меньшей мере шесть раз за период времени, по меньшей мере семь раз за период времени, по меньшей мере восемь раз за период времени, по меньшей мере девять раз за период времени, по меньшей мере десять раз за период времени, по меньшей мере одиннадцать раз, по меньшей мере двенадцать раз за период времени, по меньшей мере тринадцать раз за период времени, по меньшей мере четырнадцать раз за период времени, по меньшей мере пятнадцать раз за период времени, по меньшей мере шестнадцать раз за период времени, по меньшей мере семнадцать раз за период времени, по меньшей мере восемнадцать раз за период времени, по меньшей мере девятнадцать раз за период времени или по меньшей мере двадцать раз за период времени.
[00288] В некоторых случаях клетки, используемые в способах и композициях, имеют конструкцию ДНК, стабильно встроенную в их геном. В таких случаях приведение в контакт может включать обеспечение клетки конструкцией, уже стабильно встроенной в ее геном. Например, клетка, используемая в раскрываемых в настоящем документе способах, может иметь предсуществующий ген, кодирующий Cas, стабильно встроенный в ее геном (т.е. Cas-готовая клетка). «Стабильно встроенный» или «стабильно включенный» или «стабильно интегрированный» включает введение полинуклеотида в клетку таким образом, чтобы нуклеотидная последовательность интегрировалась в геном клетки и могла наследоваться ее потомством. Для стабильного включения конструкций ДНК или различных компонентов системы целевой интеграции в геном можно использовать любой протокол.
D. Нуклеазные средства и ДНК-связывающие белки
[00289] В раскрываемых в настоящем документе способах и композициях можно применять любое нуклеазное средство, которое индуцирует одноцепочечный или двухцепочечный разрыв в требуемой целевой последовательности, или любой ДНК-связывающий белок, который связывается с требуемой целевой последовательностью. Можно использовать встречающееся в естественной среде или природное нуклеазное средство при условии, что нуклеазное средство индуцирует одноцепочечный или двухцепочечный разрыв в требуемой целевой последовательности. Аналогичным образом, можно использовать встречающийся в естественной среде или природный ДНК-связывающий белок при условии, что такой ДНК-связывающий белок связывается с требуемой целевой последовательностью. В качестве альтернативы, можно использовать модифицированные или сконструированные нуклеазное средство или ДНК-связывающий белок. «Сконструированное нуклеазное средство или сконструированный ДНК-связывающий белок» включает нуклеазное средство или ДНК-связывающий белок, которые сконструированы (модифицированы или получены как производное) из его природной формы для специфического распознавания требуемой целевой последовательности. Таким образом, сконструированное нуклеазное средство или сконструированный ДНК-связывающий белок можно получить как производное из природного, встречающегося в естественной среде нуклеазного средства или ДНК-связывающего белка или их можно создать или синтезировать искусственно. Сконструированное нуклеазное средство или сконструированный ДНК-связывающий белок может распознавать целевую последовательность, например, если целевая последовательность не является последовательностью, которая распознавалась бы природным (несконструированным или немодифицированным) нуклеазным средством или ДНК-связывающим белком. Модификация нуклеазного средства или ДНК-связывающего белка может составлять всего одну аминокислоту в расщепляющим средство белке или один нуклеотид в расщепляющем нуклеиновую кислоту средстве. Создание одноцепочечного или двухцепочечного разрыва в целевой последовательности или другой ДНК в настоящем документе может называться «разрезанием» или «расщеплением» целевой последовательности или другой ДНК.
[00290] Также предусмотрены активные варианты и фрагменты нуклеазных средств или ДНК-связывающих белков (т.е. сконструированного нуклеазного средства или ДНК-связывающего белка). Такие активные варианты могут характеризоваться по меньшей мере 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или большей идентичностью последовательности с природным нуклеазным средством или ДНК-связывающим белком, причем активные варианты сохраняют способность производить разрез в требуемой целевой последовательности и, следовательно, сохраняют индуцирующую одноцепочечный или двухцепочечный разрыв активность или сохраняют способность связывать требуемую целевую последовательность. Например, любое из описываемых в настоящем документе нуклеазных средств может быть получено путем модификации последовательности природной эндонуклеазы и предназначено для распознавания и индукции одноцепочечных или двухцепочечных разрывов в целевой последовательности, которая не распознавалась природным нуклеазным средством. Таким образом, некоторые сконструированные нуклеазы обладают специфичностью индуцировать одноцепочечный или двухцепочечный разрыв в целевой последовательности, которая отличается от соответствующей целевой последовательности природного нуклеазного средства. Анализы в отношении индуцирующей одноцепочечные или двухцепочечные разрывы активности известны, и обычно с их помощью измеряют общую активность и специфичность эндонуклеазы на ДНК-субстратах, содержащих целевую последовательность.
[00291] Термин «целевая для нуклеазного средства последовательность» включает последовательность ДНК, в которой нуклеазное средство индуцирует одноцепочечный или двухцепочечный разрыв. Аналогичным образом, термин «целевая для ДНК-связывающего белка последовательность» включает последовательность ДНК, с которой будет связываться ДНК-связывающий белок. Целевая последовательность может быть экзогенной (или природной) для клетки, или же целевая последовательность может быть экзогенной для клетки. Целевая последовательность, которая является экзогенной для клетки, в естественной среде не встречается в геноме клетки. Целевая последовательность также может быть экзогенной для представляющих интерес полинуклеотидов, которые необходимо расположить в целевом локусе. В некоторых случаях целевая последовательность представлена лишь один раз в геноме клетки-хозяина.
[00292] Также предусмотрены активные варианты и фрагменты проиллюстрированных целевых последовательностей. Такие активные варианты могут характеризоваться по меньшей мере 65%, 70%, 75%, 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или большей идентичностью последовательности с заданной целевой последовательностью, причем активные варианты сохраняют биологическую активность и, следовательно, могут распознаваться и расщепляться нуклеазным средством специфичным для последовательности образом. Анализы по измерению двухцепочечного разрыва целевой последовательности нуклеазным средством известны из уровня техники (например, qPCR-анализ TAQMAN®, Frendewey et al. (2010) Methods in Enzymology 476:295-307, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей).
[00293] Длина целевой последовательности может варьировать и включает, например, целевые последовательности, которые составляют приблизительно 30-36 п.о. для пары белка с цинковыми пальцами или нуклеазы с цинковыми пальцами (ZFN) (т.е. приблизительно 15-18 п.о. для каждой ZFN), приблизительно 36 п.о. для эффекторного белка, подобного активаторам транскрипции (TALE), или эффекторной нуклеазы, подобной активатору транскрипции (TALEN), или приблизительно 20 п. о. для гидовой РНК системы CRISPR/Cas9.
[00294] Целевая последовательность ДНК-связывающего белка или нуклеазного средства может быть расположена где угодно в или возле целевого геномного локуса. Целевая последовательность может быть расположена внутри кодирующего участка гена (например, гена HSD17B13) или внутри регуляторных участков, которые влияют на экспрессию гена. Целевая последовательность ДНК-связывающего белка или нуклеазного средства может быть расположена в интроне, экзоне, промоторе, энхансере, регуляторном участке или любом небелковом кодирующем участке.
[00295] Одним типом ДНК-связывающего белка, который можно использовать в различных способах и композициях, раскрытых в настоящем документе, является эффектор, подобный активатору транскрипции (TALE). TALE можно слить или связать, например, с доменом эпигенетической модификации, доменом активации транскрипции или доменом-репрессором транскрипции. Примеры таких доменов описаны ниже в отношении белков Cas, а также их можно найти, например, в WO 2011/145121, включенной в настоящий документ посредством ссылки в полном своем объеме для всех целей. Соответственно, одним типом нуклеазного средства, который можно использовать в различных способах и композициях, раскрытых в настоящем документе, является эффекторная нуклеаза, подобная активатору транскрипции (TALEN). Эффекторные нуклеазы TAL представляют собой класс специфичных для последовательности нуклеаз, которые можно применять для создания двухцепочечных разрывов в конкретных целевых последовательностях в геноме прокариотического или эукариотического организма. Эффекторные нуклеазы TAL получают путем слияния природного или сконструированного эффектора, подобного активатору транскрипции (TAL), или его функциональной части с каталитическим доменом эндонуклеазы, такой как FokI. Уникальный модульный TAL-эффекторный ДНК-связывающий домен позволяет конструировать белки с потенциально любой специфичностью распознавания заданной ДНК. Таким образом, ДНК-связывающие домены эффекторных нуклеаз TAL можно сконструировать так, чтобы они распознавали конкретные целевые сайты ДНК, и, таким образом, их можно было бы использовать для создания двухцепочечных разрывов в требуемых целевых последовательностях. См. WO 2010/079430; Morbitzer et al. (2010) Proc. Natl. Acad. Sci. U.S.A. 107 (50:21617-21622; Scholze & Boch (2010) Virulence 1:428-432; Christian et al. (2010) Genetics 186:757-761; Li et al. (2011) Nucleic Acids Res. 39(1):359-372; и Miller et al. (2011) Nature Biotechnology 29:143-148, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00296] Примеры подходящих нуклеаз TAL и способов получения подходящих нуклеаз TAL раскрыты, например, в US 2011/0239315 A1, US 2011/0269234 A1, US 2011/0145940 A1, US 2003/0232410 A1, US 2005/0208489 A1, US 2005/0026157 A1, US 2005/0064474 A1, US 2006/0188987 A1 и US 2006/0063231 A1, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. В соответствии с различными вариантами осуществления, эффекторные нуклеазы TAL конструируют такими, чтобы они производили разрез в или вблизи целевой последовательности нуклеиновой кислоты, например, в представляющем интерес геномном локусе, причем целевая последовательность нуклеиновой кислоты находится в или вблизи последовательности, подлежащей модификации экзогенной донорной последовательности. Нуклеазы TAL, подходящие для применения с различными способами и композициями, представленными в настоящем документе, включают такие нуклеазы, которые специально сконструированы для связывания в или вблизи целевых последовательностей нуклеиновой кислоты, подлежащих модификации экзогенными донорными последовательностями, как описано в других разделах настоящего документа.
[00297] В некоторых TALEN каждый мономер TALEN содержит 33-35 повторов TAL, которые распознают одну пару оснований с помощью двух гипервариабельных остатков. В некоторых TALEN нуклеазное средство представляет собой химерный белок, содержащий ДНК-связывающий домен с TAL-повторами, функционально связанный с независимой нуклеазой, такой как эндонуклеаза FokI. Например, нуклеазное средство может содержать первый ДНК-связывающий домен с TAL-повторами и второй ДНК-связывающий домен с TAL-повторами, причем каждый из первого и второго ДНК-связывающих доменов с TAL-повторами функционально связан с нуклеазой FokI, первый и второй ДНК-связывающие домены с TAL-повторами распознают две смежные целевые последовательности ДНК в каждой цепи целевой последовательности ДНК, разделенные спейсерной последовательностью различной длины (12-20 п.о.), и при этом субъединицы нуклеазы FokI димеризируются с образованием активной нуклеазы, которая производит двухцепочечный разрыв в целевой последовательности.
[00298] Другим примером ДНК-связывающего белка является белок с цинковыми пальцами. Такие белки с цинковыми пальцами можно связать или слить с доменом эпигенетической модификации, доменом активации транскрипции или доменом-репрессором транскрипции. Примеры таких доменов описаны ниже в отношении белков Cas, а также их можно найти, например, в WO 2011/145121, включенной в настоящий документ посредством ссылки в полном своем объеме для всех целей. Соответственно, другим примером нуклеазного средства, который можно использовать в различных способах и композициях, раскрытых в настоящем документе, является нуклеаза с цинковыми пальцами (ZFN). У некоторых ZFN каждый мономер ZFN содержит три или более ДНК-связывающих домена с цинковыми пальцами, причем каждый ДНК-связывающий домен с цинковыми пальцами связывается с подсайтом размером 3 п.о. У некоторых ZFN ZFN представляет собой химерный белок, содержащий ДНК-связывающий домен с цинковыми пальцами, функционально связанный с независимой нуклеазой, такой как эндонуклеаза FokI. Например, нуклеазное средство может содержать первую ZFN и вторую ZFN, причем каждая из первой и второй ZFN функционально связана с субъединицей нуклеазы FokI, первая и вторая ZFN распознают две смежные целевые последовательности ДНК в каждой цепи целевой последовательности ДНК, разделенные спейсером размером приблизительно 5-7 п.о., и при этом субъединицы нуклеазы FokI димеризируются с образованием активной нуклеазы, которая производит двухцепочечный разрыв. См., например, US 2006/0246567, US 2008/0182332, US 2002/0081614, US 2003/0021776, WO 2002/057308 A2, US 2013/0123484, US 2010/0291048, WO 2011/017293 A2 и Gaj et al. (2013) Trends in Biotechnology 31(7):397-405, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00299] Другие подходящие ДНК-связывающие белки и нуклеазные средства для применения в описываемых в настоящем документе способах и композициях включают системы CRISPR-Cas, которые описаны в других разделах настоящего документа.
[00300] ДНК-связывающий белок или нуклеазное средство можно ввести в клетку с помощью любого известного средства. В клетку можно ввести непосредственно полипептид, кодирующий ДНК-связывающий белок или нуклеазное средство. В качестве альтернативы, в клетку можно ввести полинуклеотид, кодирующий ДНК-связывающий белок или нуклеазное средство. При введении полинуклеотида, кодирующего ДНК-связывающий белок или нуклеазное средство, ДНК-связывающий белок или нуклеазное средство может временно, условно или конститутивно экспрессироваться в клетке. Например, полинуклеотид, кодирующий ДНК-связывающий белок или нуклеазное средство, может содержаться в кассете экспрессии и быть функционально связанным с условным промотором, индуцируемым промотором, конститутивным промотором или тканеспецифичным промотором. Такие промоторы более подробно рассмотрены в других разделах настоящего документа. В качестве альтернативы, ДНК-связывающий белок или нуклеазное средство можно ввести в клетку в виде мРНК, кодирующей ДНК-связывающий белок или нуклеазное средство.
[00301] Полинуклеотид, кодирующий ДНК-связывающий белок или нуклеазное средство, можно стабильно интегрировать в геном клетки и функционально связать с активным в клетке промотором. В качестве альтернативы, полинуклеотид, кодирующий ДНК-связывающий белок или нуклеазное средство, может находиться в направленном векторе или в векторе или плазмиде, которые представлены отдельно от направленного вектора, содержащего полинуклеотидную вставку.
[00302] При введении ДНК-связывающего белка или нуклеазного средства в клетку путем введения полинуклеотида, кодирующего ДНК-связывающий белок или нуклеазное средство, такой полинуклеотид, кодирующий ДНК-связывающий белок или нуклеазное средство, может быть модифицирован с заменой кодонов, имеющих более высокую частоту использования в представляющей интерес клетке по сравнению со встречающейся в естественной среде полинуклеотидной последовательностью, кодирующей ДНК-связывающий белок или нуклеазное средство. Например, полинуклеотид, кодирующий ДНК-связывающий белок или нуклеазное средство, может быть модифицирован с заменой кодонов, имеющих более высокую частоту использования в данной представляющей интерес прокариотической или эукариотической клетке, включая бактериальную клетку, дрожжевую клетку, клетку человека, клетку отличного от человека животного, клетку млекопитающего, клетку грызуна, клетку мыши, клетку крысы или любую другую представляющую интерес клетку-хозяина, по сравнению со встречающейся в естественной среде полинуклеотидной последовательностью.
Е. Системы CRISPR-Cas
[00303] Раскрываемые в настоящем документе способы могут предусматривать использование систем с короткими полиндромными повторами, регулярно расположенными группами (CRISPR)/CRISPR-ассоциированных (Cas) систем или компонентов таких систем для модификации генома в клетке. Системы CRISPR-Cas включают в себя транскрипты и другие элементы, участвующие в экспрессии или управлении активностью генов Cas. Система CRISPR-Cas может быть системой I типа, II типа или III типа. В качестве альтернативы, система CRISPR/Cas может быть, например, системой V типа (например, подтипа V-A или подтипа V-B). Раскрываемые в настоящем документе способы и композиции могут предусматривать использование систем CRISPR-Cas с использованием комплексов CRISPR (содержащих гидовую РНК (gRNA) в комплексе с белком Cas) для сайт-направленного расщепления нуклеиновых кислот.
[00304] Системы CRISPR-Cas, применяемые в раскрываемых в настоящем документе способах, могут не встречаться в естественной среде. «Не встречающаяся в естественной среде» система включает в себя все, что указывает на произведенную человеком манипуляцию, например, один или несколько компонентов системы подвергнуты изменению или мутации из своего естественного состояния, по меньшей мере практически не содержат по меньшей мере один другой компонент, с которым они в норме ассоциированы в естественной среде, или ассоциированы по меньшей мере с одним другим компонентом, с которым они в норме не ассоциированы. Например, в не встречающихся в естественной среде системах CRISPR/Cas могут быть использованы комплексы CRISPR, содержащие gRNA и белок Cas, которые не встречаются в естественной среде вместе, белок Cas, который не встречается в естественной среде, или gRNA, которая не встречается в естественной среде.
(1) Белки Cas и полинуклеотиды, кодирующие белки Cos
[00305] Белки Cas обычно содержат по меньшей мере один домен распознавания или связывания РНК, который может взаимодействовать с гидовыми РНК (gRNA, которые более подробно описаны ниже). Белки Cas также могут содержать нуклеазные домены (например, ДНКазные или РНКазные домены), ДНК-связывающие домены, геликазные домены, домены межбелкового взаимодействия, домены димеризации и другие домены. Нуклеазный домен обладает каталитической активностью для расщепления нуклеиновой кислоты, которое включает разрыв ковалентных связей молекулы нуклеиновой кислоты. Расщепление может приводить к образованию тупых концов или ступенчатых концов, и оно может быть одноцепочечным или двухцепочечным. Например, белок Cas9 дикого типа обычно создает продукт расщепления с тупыми концами. В качестве альтернативы, белок Cpf1 дикого типа (например, FnCpf1) может приводить в результате к образованию продукта расщепления с 5-нуклеотидным 5' «липким» концом, причем расщепление происходит после 18-й пары оснований из последовательности РАМ на нецелевой цепи и после 23-го основания на целевой цепи. Белок Cas может обладать активностью полного расщепления с образованием двухцепочечного разрыва в гене HSD17B13 (например, двухцепочечного разрыва с тупыми концами), или это может быть никаза, которая создает одноцепочечный разрыв в гене HSD17B13.
[00306] Примеры белков Cas включают Cas1, Cas1B, Cas2, Cas3, Cas4, Cas5, Cas5e (CasD), Cas6, Cas6e, Cas6f, Cas7, Cas8a1, Cas8a2, Cas8b, Cas8c, Cas9 (Csn1 or Csx12), Cas10, Cas10d, CasF, CasG, CasH, Csy1, Csy2, Csy3, Cse1 (CasA), Cse2 (CasB), Cse3 (CasE), Cse4 (CasC), Csc1, Csc2, Csa5, Csn2, Csm2, Csm3, Csm4, Csm5, Csm6, Cmr1, Cmr3, Cmr4, Cmr5, Cmr6, Csb1, Csb2, Csb3, Csx17, Csx14, Csx10, Csx16, CsaX, Csx3, Csx1, Csx15, Csf1, Csf2, Csf3, Csf4 и Cu1966 и их гомологи и модифицированные версии.
[00307] Иллюстративный белок Cas представляет собой белок Cas9 или белок, полученный из белка Cas9 из системы II типа CRISPR/Cas. Белки Cas9 получены из системы II типа CRISPR/Cas и обычно имеют четыре ключевых мотива с консервативной архитектурой. Мотивы 1, 2 и 4 являются RuvC-подобными мотивами, а мотив 3 является мотивом HNH. Иллюстративные белки Cas9 получены из Streptococcus pyogenes, Streptococcus thermophilus, Streptococcus sp., Staphylococcus aureus, Nocardiopsis dassonvillei, Streptomyces pristinaespiralis, Streptomyces viridochromogenes, Streptomyces viridochromogenes, Streptosporangium roseum, Streptosporangium roseum, Alicyclobacillus acidocaldarius, Bacillus pseudomycoides, Bacillus selenitireducens, Exiguobacterium sibiricum, Lactobacillus delbrueckii, Lactobacillus salivarius, Microscilla marina, Burkholderiales bacterium, Polaromonas naphthalenivorans, Polaromonas sp., Crocosphaera watsonii, Cyanothece sp., Microcystis aeruginosa, Synechococcus sp., Acetohalobium arabaticum, Ammonifex degensii, Caldicelulosiruptor becscii, Candidatus Desulforudis, Clostridium botulinum, Clostridium difficile, Finegoldia magna, Natranaerobius thermophilus, Pelotomaculum thermopropionicum, Acidithiobacillus caldus, Acidithiobacillus ferrooxidans, Allochromatium vinosum, Marinobacter sp., Nitrosococcus halophilus, Nitrosococcus watsoni, Pseudoalteromonas haloplanktis, Ktedonobacter racemifer, Methanohalobium evestigatum, Anabaena variabilis, Nodularia spumigena, Nostoc sp., Arthrospira maxima, Arthrospira platensis, Arthrospira sp., Lyngbya sp., Microcoleus chthonoplastes, Oscillatoria sp., Petrotoga mobilis, Thermosipho africanus или Acaryochloris marina. Дополнительные примеры представителей семейства Cas9 описаны в WO 2014/131833, которая включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Иллюстративным белком Cas9 является Cas9 из S. pyogenes (SpCas9) (обозначен в SwissProt под регистрационным номером Q99ZW2). Другим иллюстративным белком Cas9 является Cas9 из S. aureus (SaCas9) (обозначен в UniProt под регистрационным номером J7RUA5).
[00308] Другим примером белка Cas является белок Cpf1 (CRISPR из Prevotella и Francisella 1). Cpf1 представляет собой большой белок (приблизительно 1300 аминокислот), который содержит RuvC-подобный нуклеазный домен, гомологичный соответствующему домену Cas9, наряду с аналогом характерного богатого аргинином кластера Cas9. Однако в Cpf1 отсутствует нуклеазный домен HNH, который присутствует в белках Cas9, а RuvC-подобный домен является смежным в последовательности Cpf1, в отличие от Cas9, где он содержит длинные вставки, включая домен HNH. См., например, Zetsche et al. (2015) Cell 163(3):759-771, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Иллюстративные белки Cpf1 получены из Francisella tularensis 1, Francisella tularensis subsp. novicida, Prevotella albensis, Lachnospiraceae bacterium MC2017 1, Butyrivibrio proteoclasticus, Peregrinibacteria bacterium GW2011_GWA2_33_10, Parcubacteria bacterium GW2011_GWC2_44_17, Smithella sp. SCADC, Acidaminococcus sp. BV3L6, Lachnospiraceae bacterium MA2020, Candidatus Methanoplasma termitum, Eubacterium eligens, Moraxella bovoculi 237, Leptospira inadai, Lachnospiraceae bacterium ND2006, Porphyromonas crevioricanis 3, Prevotella disiens и Porphyromonas macacae. Иллюстративным белком Cpf1 является Cpf1 из U112 Francisella novicida (FnCpf1; обозначен в UniProt под регистрационным номером A0Q7Q2).
[00309] Белки Cas могут быть белками дикого типа (т.е. белками, которые встречаются в естественной среде), модифицированными белками Cas (т.е. вариантами белков Cas) или фрагментами белков дикого типа или модифицированных белков Cas. Белки Cas также могут быть активными вариантами или фрагментами в отношении каталитической активности белков Cas дикого типа или модифицированных белков Cas. Активные варианты или фрагменты в отношении каталитической активности могут характеризоваться по меньшей мере 80%, 85%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или большей идентичностью последовательности с белком Cas дикого типа или модифицированным белком Cas или его частью, причем активные варианты сохраняют способность производить разрез в требуемом сайте расщепления и, следовательно, сохраняют индуцирующую одноцепочечный или двухцепочечный разрыв активность. Анализы в отношении индуцирующей одноцепочечные или двухцепочечные разрывы активности известны, и обычно с их помощью измеряют общую активность и специфичность белка Cas на ДНК-субстратах, содержащих сайт расщепления.
[00310] Белки Cas можно модифицировать для увеличения или уменьшения одной или нескольких из следующих: аффинности связывания нуклеиновой кислоты, специфичности связывания нуклеиновой кислоты и ферментативной активности. Белки Cas также можно модифицировать для изменения любой другой активности или любого другого свойства белка, такого как стабильность. Например, один или несколько нуклеазных доменов белка Cas можно модифицировать, удалить или инактивировать или белок Cas можно укоротить для удаления доменов, которые не являются необходимыми для функции белка или для оптимизации (например, для усиления или уменьшения) активности белка Cas.
[00311] Белки Cas могут содержать по меньшей мере один нуклеазный домен, такой как ДНКазный домен. Например, белок Cpf1 дикого типа обычно содержит RuvC-подобный домен, который расщепляет обе цепи целевой ДНК, возможно, в димерной конфигурации. Белки Cas также могут содержать по меньшей мере два нуклеазных домена, таких как ДНКазные домены. Например, белок Cas9 дикого типа обычно содержит RuvC-подобный нуклеазный домен и HNH-подобный нуклеазный домен. Каждый из доменов RuvC и HNH может разрезать разные цепи двухцепочечной ДНК с образованием двухцепочечного разрыва в ДНК. См., например, Jinek et al. (2012) Science 337:816-821, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00312] Один или несколько нуклеазных доменов можно удалить или подвергнуть мутации так, чтобы они более не были функциональными или обладали пониженной нуклеазной активностью. Например, если один из нуклеазных доменов удалить или подвергнуть мутации в белке Cas9, полученный белок Cas9 можно назвать никазой, и он может создавать одноцепочечный разрыв в целевой для гидовой РНК последовательности в двухцепочечной ДНК, но не двухцепочечный разрыв (т.е. он может расщеплять комплементарную цепь или некомплементарную цепь, но не обе одновременно). Если удалить или подвергнуть мутации оба нуклеазных домена, полученный белок Cas (например, Cas9) будет обладать пониженной способностью расщеплять обе цепи двухцепочечной ДНК (например, обладающий нуклеазной активностью белки Cas или не обладающий нуклеазной активностью белок Cas, или каталитически мертвый белок Cas (dCas)). Примером мутации, которая превращает Cas9 в никазу, является мутация D10A (аспартат в аланин в положении 10 в Cas9) в RuvC-домене Cas9 из S. pyogenes. Аналогичным образом, Н939А (гистидин на аланин в 839-м аминокислотном положении) или Н840А (гистидин на аланин в 840-м аминокислотном положении) в HNH-домене Cas9 из S. pyogenes могут превратить Cas9 в никазу. Другие примеры мутаций, которые превращают Cas9 в никазу, включают соответствующие мутации, вносимые в Cas9 из S. thermophilic. См., например, Sapranauskas et al. (2011) Nucleic Acids Research 39:9275-9282 и WO 2013/141680, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Такие мутации можно создать с помощью таких способов, как сайт-направленный мутагенез, ПЦР-опосредованный мутагенез или синтез всего гена. Примеры других мутаций, создающих никазы, можно найти, например, в WO 2013/176772 и WO 2013/142578, каждая из которых включена в настоящий документ посредством ссылки в полном ее объеме для всех целей.
[00313] Белки Cas (например, обладающие нуклеазной активностью белки Cas или не обладающие нуклеазной активностью белки Cas) также можно функционально связать с гетерологичными полипептидами в виде белков слияния. Например, белок Cas можно слить с доменом расщепления, доменом эпигенетической модификации, доменом активации транскрипции или доменом-репрессором транскрипции. См. WO 2014/089290, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Примеры доменов активации транскрипции включают домен активации VP16 вируса простого герпеса, VP64 (который является тетрамерным производным VP16), домен активации р65 NFκB, домены активации 1 и 2 р53, домен активации CREB (белка, связывающего элемент ответа цАМФ), домен активации Е2А и домен активации NFAT (ядерного фактора активированных Т-клеток). Другие примеры включают домены активации из Oct1, Oct-2A, SP1, АР-2, CTF1, Р300, СВР, PCAF, SRC1, PvALF, ERF-2, OsGAI, HALF-1, C1, AP1, ARF-5, ARF-6, ARF-7, ARF-8, CPRF1, CPRF4, MYC-RP/GP, TRAB1PC4 и HSF1. См., например, US 2016/0237456, EP3045537 и WO 2011/145121, каждая из которых включена посредством ссылки в полном своем объеме для всех целей. В некоторых случаях можно использовать систему активации транскрипции, содержащую белок слияния dCas9-VP64 в паре с MS2-p65-HSF1. Гидовые РНК в таких системах можно сконструировать с аптамерными последовательностями, присоединенными к тетра-петле sgRNA и стволовой петле 2, предназначенным для связывания димеризованных белков оболочки бактериофага MS2. См., например, Konermann et al. (2015) Nature 517(7536):583-588, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Примеры доменов-репрессоров транскрипции включают домены раннего индуцируемого цАМФ-репрессора (ICER), домены репрессора Kruppel-associated box A (KRAB-A), домены богатого глицином репрессора YY1, Sp1-подобные репрессоры, репрессоры E(spl), репрессор IκВ и МеСР2. Другие примеры включают домены-репрессоры транскрипции из А/В, КОХ, TGF-бета-индуцируемого раннего гена (TFEG), v-erbA, SID, SID4X, MBD2, MBD3, DNMT1, DNMG3A, DNMT3B, Rb, ROM2. См., например, ЕР3045537 и WO 2011/145121, каждая из которых включена посредством ссылки в своем полном объеме для всех целей. Белки Cas также можно слить с гетерологичным полипептидом, обеспечивающим повышенную или пониженную стабильность. Слитый домен или гетерологичный полипептид может быть расположен на N-конце, С-конце или внутри белка Cas.
[00314] В качестве одного примера, белок Cas можно слить с гетерологичным полипептидом, который обеспечивает субклеточную локализацию. Такие гетерологичные полипептиды могут включать, например, один или несколько сигналов ядерной локализации (NLS), таких как NLS SV40, для нацеливания на ядро, сигнал митохондриальной локализации для нацеливания на митохондрии, сигнал удерживания в ER и тому подобное. См, например, Lange et al. (2007) J. Biol. Chem. 282:5101-5105, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Такие сигналы субклеточной локализации могут быть расположены на N-конце, С-конце или в любом месте в белке Cas. NLS может содержать отрезок из основных аминокислот и может представлять собой последовательность из одной части или последовательность из двух частей.
[00315] Белки Cas также можно функционально связать с проникающим доменом. Например, проникающий домен можно получить из белка TAT HIV-1, проникающего мотива TLM из вируса гепатита В человека, MPG, Рер-1, VP22, проникающего пептида из вируса простого герпеса или полиаргининовой пептидной последовательности. См., например, WO 2014/089290, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Проникающий домен может быть локализован на N-конце, С-конце или в любом месте в белке Cas.
[00316] Белки Cas также можно функционально связать с гетерологичным полипептидом для простоты отслеживания или очистки, таким как флуоресцентный белок, маркер для очистки или эпитопный маркер. Примеры флуоресцентных белков включают зеленые флуоресцентные белки (например, GFP, GFP-2, tagGFP, turboGFP, eGFP, Emerald, Azami Green, Monomeric Azami Green, CopGFP, AceGFP, ZsGreenl), желтые флуоресцентные белки (например, YFP, eYFP, Citrine, Venus, YPet, PhiYFP, ZsYellowl), синие флуоресцентные белки (например, eBFP, eBFP2, Azurite, mKalamal, GFPuv, Sapphire, T-sapphire), голубые флуоресцентные белки (например, eCFP, Cerulean, CyPet, AmCyanl, Midorii), красные флуоресцентные белки (например, mKate, mKate2, mPlum, мономер DsRed, mCherry, mRFP1, DsRed-Express, DsRed2, DsRed-Monomer, HcRed-Tandem, HcRedl, AsRed2, eqFP611, mRaspberry, mStrawberry, Jred), оранжевые флуоресцентные белки (например, mOrange, mKO, Kusabira-Orange, Monomeric Kusabira-Orange, mTangerine, tdTomato) и любой другой подходящий флуоресцентный белок. Примеры меток включают глутатион-S-трансферазу (GST), хитин-связывающий белок (СВР), мальтозосвязывающий белок, тиоредоксин (TRX), поли(NANP), маркер для тандемной аффинной очистки (ТАР), myc, AcV5, AU1, AU5, Е, ECS, Е2, FLAG, гемагглютинин (НА), nus, Softag 1, Softag 3, Strep, SBP, Glu-Glu, HSV, KT3, S, S1, T7, V5, VSV-G, гистидин (His), белок-носитель биотина и карбоксила (ВССР) и кальмодулин.
[00317] Белки Cas также могут быть прикреплены к экзогенным донорным последовательностям или меченым нуклеиновым кислотам. Такое прикрепление (т.е. физическое связывание) может быть достигнуто посредством ковалентных взаимодействий или нековалентных взаимодействий, и такое прикрепление может быть прямым (например, посредством прямого слияния или химического конъюгирования, что может быть достигнуто путем модификации цистеиновых или лизиновых остатков на белке или путем модификации интеином) или может быть достигнуто с помощью одного или нескольких промежуточных линкеров или адаптерных молекул, таких как стрептавидин или аптамеры. См., например, Pierce et al. (2005) Mini Rev. Med. Chem. 5(1):41-55; Duckworth et al. (2007) Angew. Chem. Int. Ed. Engl. 46(46):8819-8822; Schaeffer and Dixon (2009) Australian J. Chem. 62(10):1328-1332; Goodman et al. (2009) Chembiochem. 10(9): 1551-1557; и Khatwani et al. (2012) Bioorg. Med. Chem. 20(14):4532-4539, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Нековалентные стратегии синтеза конъюгатов белка и нуклеиновой кислоты включают способы с применением биотина-стрептавидина и никеля-гистидина. Ковалентный конъюгаты белка и нуклеиновой кислоты можно синтезировать путем соответствующего присоединения функционализированных нуклеиновых кислот и белков с использованием широкого спектра химических реакций. Некоторые из этих химических реакций предусматривают прямое присоединение олигонуклеотида к аминокислотному остатку на поверхности белка (например, лизинаминовая или цистеинтиоловая), в то время как при других более сложных схемах необходима посттрансляционная модификация белка или участия каталитического или реакционноспособного домена белка. Способы ковалентного присоединения белков к нуклеиновым кислотам могут включать, например, химическое сшивание олигонуклеотидов с лизиновым или цистеиновым остатками, лигирование экспрессированного белка, хемоферментативные способы и применение фотоаптамеров. Экзогенную донорную последовательность или меченную нуклеиновую кислоту можно присоединить к С-концу, N-концу или внутреннему участку в белке Cas. Предпочтительно, экзогенную донорную последовательность или меченную нуклеиновую кислоту присоединяют к С-концу или N-концу белка Cas. Аналогичным образом, белок Cas можно присоединить к 5'-концу, 3'-концу или внутренней части в экзогенной донорной последовательности или меченой нуклеиновой кислоте. То есть, экзогенную донорную последовательность или меченную нуклеиновую кислоту можно присоединить в любой ориентации и полярности. Предпочтительно, белок Cas присоединен к 5'-концу или 3'-концу экзогенной донорной последовательности или меченой нуклеиновой кислоты.
[00318] Белки Cas могут быть представлены в любой форме. Например, белок Cas может быть представлен в форме белка, такого как белок Cas в комплексе с gRNA. В качестве альтернативы, белок Cas может быть представлен в форме нуклеиновой кислоты, кодирующей белок Cas, такой РНК (например, матричной РНК (мРНК)) или ДНК. Необязательно, нуклеиновая кислота, кодирующая белок Cas, может быть оптимизирована по кодону для эффективной трансляции в белок в конкретной клетке или конкретном организме. Например, нуклеиновая кислота, кодирующая белок Cas, может быть модифицирована с заменой ко донов, имеющих более высокую частоту использования в бактериальной клетке, дрожжевой клетке, клетке человека, клетке отличного от человека животного, клетке млекопитающего, клетке грызуна, клетке мыши, клетке крысы или любой другой представляющей интерес клетке-хозяине, по сравнению со встречающейся в естественной среде полинуклеотидной последовательностью. При введении нуклеиновой кислоты, кодирующей белок Cas, в клетку белок Cas может экспрессироваться в клетке временно, условно или конститутивно.
[00319] Нуклеиновые кислоты, кодирующие белки Cas, можно стабильно интегрировать в геном клетки и функционально связать с активным в клетке промотором. В качестве альтернативы, нуклеиновые кислоты, кодирующие белки Cas, можно функционально связать с промотором в экспрессирующей конструкции. Экспрессирующие конструкции включают любые конструкции нуклеиновых кислот, которые способны управлять экспрессией гена или другой представляющей интерес последовательности нуклеиновой кислоты (например, гена Cas) и могут переносить такую представляющую интерес последовательность нуклеиновой кислоты в целевую клетку. Например, нуклеиновая кислота, кодирующая белок Cas, может находиться в направленном векторе, содержащем вставку нуклеиновой кислоты, и/или векторе, содержащем ДНК, кодирующую gRNA. В качестве альтернативы, она может находиться в векторе или плазмиде, которые отделены от направленного вектора, содержащего вставку нуклеиновой кислоты, и/или отделены от вектора, содержащего ДНК, кодирующую такую gRNA. Промоторы, которые можно применять в экспрессирующей конструкции, включают промоторы, активные, например, в одной или нескольких из эукариотической клетки, клетки человека, клетки отличного от человека животного, клетки млекопитающего, клетки отличного от человека млекопитающего, клетки грызуна, клетки мыши, клетки крысы, клетки хомяка, клетки кролика, плюрипотентной клетки, эмбриональной стволовой (ES) клетки или зиготы. Такие промоторы могут быть, например, условными промоторами, индуцируемыми промоторами, конститутивными промоторами или тканеспецифичными промоторами. Необязательно, промотор может быть двунаправленным промотором, управляющим экспрессией как белка Cas в одном направлении, так и гидовой РНК в другом направлении. Такие двунаправленные промоторы могут состоять из (1) полного традиционного однонаправленного промотора Pol III, который содержит 3 внешних управляющих элемента: элемент периферической последовательности (DSE), элемент проксимальной последовательности (PSE) и ТАТА-бокс; и (2) второй основной промотор Pol III, который включает PSE и ТАТА-блок, слитый с 5'-концом DSE в обратной ориентации. Например, в промоторе H1 DSE находится рядом с PSE и ТАТА-блоком, и данный промотор можно сделать двунаправленным путем создания гибридного промотора, в котором транскрипцией в обратном направлении управляют путем добавления PSE и ТАТА-блока, полученного из промотера U6. См., например, US 2016/0074535, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Использование двунаправленного промотора для экспрессии генов, кодирующих одновременно белок Cas и гидовую РНК, позволяет создавать компактные кассеты экспрессии, что облегчает доставку.
(2) Гидовые РНК
[00320] «Гидовая РНК» или «gRNA» представляет собой молекулу РНК, которая связывается с белком Cas (например, белком Cas9) и нацеливает белок Cas на конкретное местоположение в целевой ДНК (например, ген HSD17B13). В частности, в настоящем документе раскрыты гидовые РНК, эффективные для направления фермента Cas на связывание или расщепление локуса HSD17B13 или гена HSD17B13. Одной иллюстративной гидовой РНК является гидовая РНК, эффективная для направления фермента Cas на связывание или расщепление гена HSD17B13, при этом гидовая РНК содержит нацеливающий на ДНК сегмент, который гибридизируется с последовательностью распознания гидовой у РНК (т.е. нацеливает на целевую для гидовой РНК последовательность) в гене HSD17B13, который включает, или находится в непосредственной близости от него, положение, соответствующее положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Под нацеливанием на целевую для гидовой РНК последовательность понимают гибридизацию с последовательностью комплементарной цепи, которая обратно комплементарна целевой для гидовой РНК последовательности на не комплементарной цепи. Например, целевая для гидовой РНК последовательность может находиться в пределах приблизительно 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от положения, соответствующего положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Другие иллюстративные гидовые РНК содержат нацеливающий на ДНК сегмент, который нацеливает целевую для гидовой РНК последовательность в гене HSD17B13, которая находится в пределах участка, соответствующего экзону 6 и/или интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Другие иллюстративные гидовые РНК содержат нацеливающий на ДНК сегмент, который нацеливает целевую для гидовой РНК последовательность в гене HSD17B13, которая находится в пределах участка, соответствующего экзону 6, и/или интрону 6, и/или экзону 7 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Другие иллюстративные гидовые РНК содержат нацеливающий на ДНК сегмент, гибридизируется с последовательностью распознания гидовой РНК (т.е. нацеливает на целевую для гидовой РНК последовательность) в гене HSD17B13, который включает старт-кодон гена HSD17B13 или находится в непосредственной близости от него или включает стоп-кодон гена HSD17B13 или находится в непосредственной близости от него. Например, целевая для гидовой РНК последовательность может находиться в пределах приблизительно 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона или в пределах приблизительно 5, 10, 15, 20, 25, 30, 35, 40, 45, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от стоп-кодона. Например, целевая для гидовой РНК последовательность может находиться в пределах участка, соответствующего экзону 1 из SEQ ID NO: 1 или 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1 или 2. Аналогичным образом, целевая для гидовой РНК последовательность может находиться в пределах участка, соответствующего экзону 7 из SEQ ID NO: 1 или 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1 или 2. Ген HSD17B13 может представлять собой ген HSD17B13 из любого организма. Например, ген HSD17B13 может представлять собой ген HSD17B13 человека или ортолог из другого организма, такого как отличное от человека млекопитающее, грызун, мышь или крыса.
[00321] Примеры целевых для гидовой РНК последовательностей на 5'-конце гена HSD17B13 человека содержат, фактически состоят или состоят из последовательностей, изложенных в SEQ ID NO: 20-81, и изложены в приведенной ниже таблице. Примеры нацеливающих на ДНК сегментов гидовой РНК, соответствующих SEQ ID NO: 20-81, изложены в приведенной ниже таблице и идентичны SEQ ID NO: 20-81 за исключением урацилов вместо тиминов. Нацеливающий на ДНК сегмент гидовой РНК может содержать, фактически состоять или состоять из любых последовательностей нацеливающих на ДНК сегментов, изложенных в представленной ниже таблице. Примеры целевых для гидовой РНК последовательностей, находящихся рядом с сайтом начала транскрипции (TSS) гена HSD17B13 человека, содержат, фактически состоят или состоят из последовательностей, изложенных в SEQ ID NO: 20-41, и изложены в приведенной ниже таблице. Иллюстративные целевые для гидовой РНК последовательности, находящиеся рядом с TSS, включают SEQ ID NO: 21-23, 33 и 35. SEQ ID NO: 33 и 35 находятся ближе всего к TSS. Иллюстративные crRNA и sgRNA (содержащие каркас версии 1, 2, 3 или 4), соответствующие целевым для гидовой РНК последовательностям на 5'-конце гена HSD17B13 человека, содержат, фактически состоят из или состоят из любой из последовательностей, изложенных в представленной ниже таблице.
[00322]
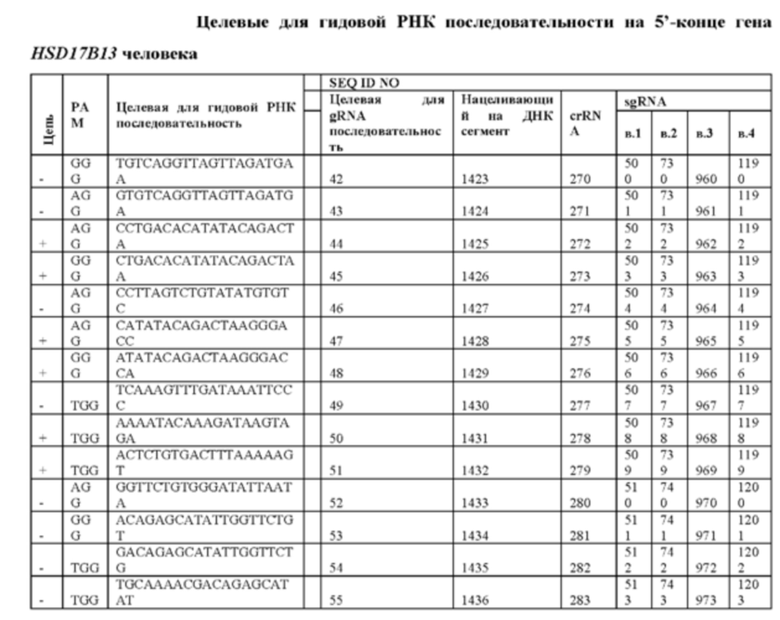
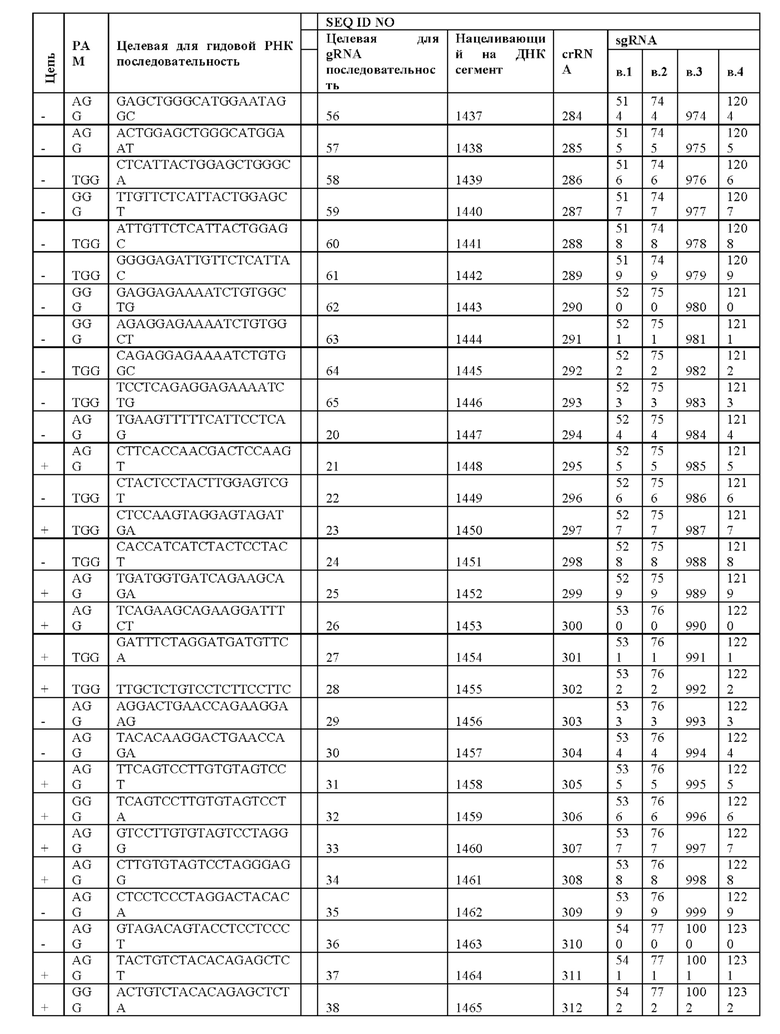

[00323] Примеры целевых для гидовой РНК последовательностей на 3'-конце гена HSD17B13 человека содержат, фактически состоят или состоят из последовательностей, изложенных в SEQ ID NO: 82-225, и изложены в приведенной ниже таблице. Примеры нацеливающих на ДНК сегментов гидовой РНК, соответствующих SEQ ID NO: 82-225, представлены соответственно под SEQ ID NO: 1485-1628, которые идентичны SEQ ID NO: 82-225 за исключением урацилов вместо тиминов. Нацеливающий на ДНК сегмент гидовой РНК может содержать, фактически состоять или состоять из любых последовательностей, изложенных под SEQ ID NO: 1485-1628. Иллюстративные crRNA и sgRNA (содержащие каркас версии 1, 2, 3 или 4), соответствующие целевым для гидовой РНК последовательностям на 3'-конце гена HSD17B13 человека, содержат, фактически состоят из или состоят из любой из последовательностей, изложенных в представленной ниже таблице.
[00324]
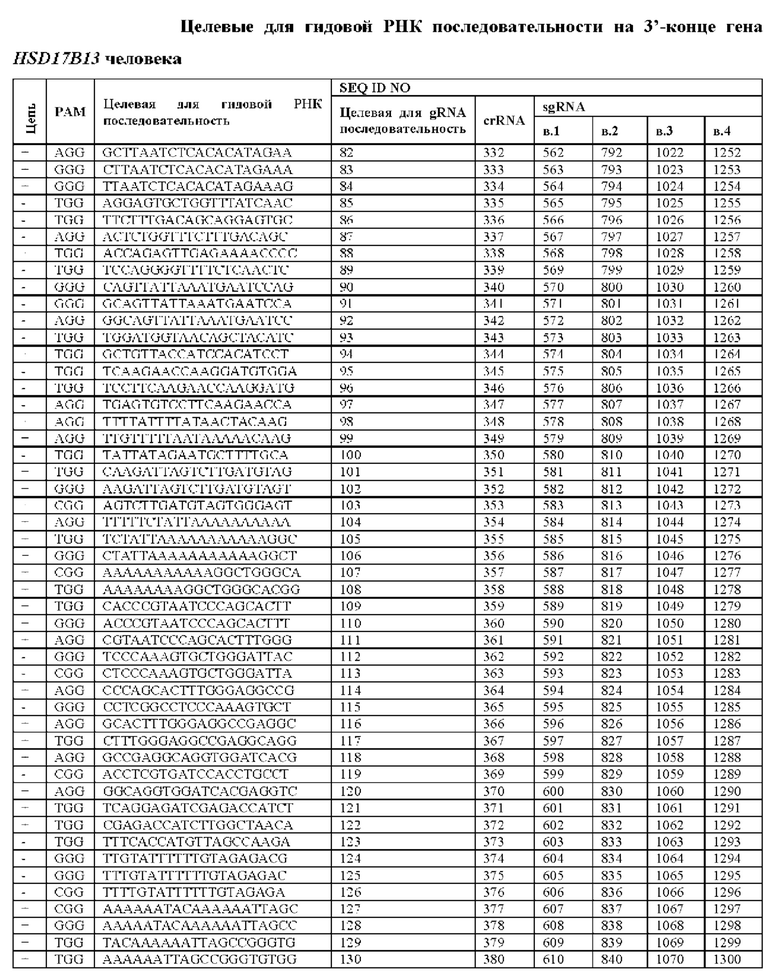
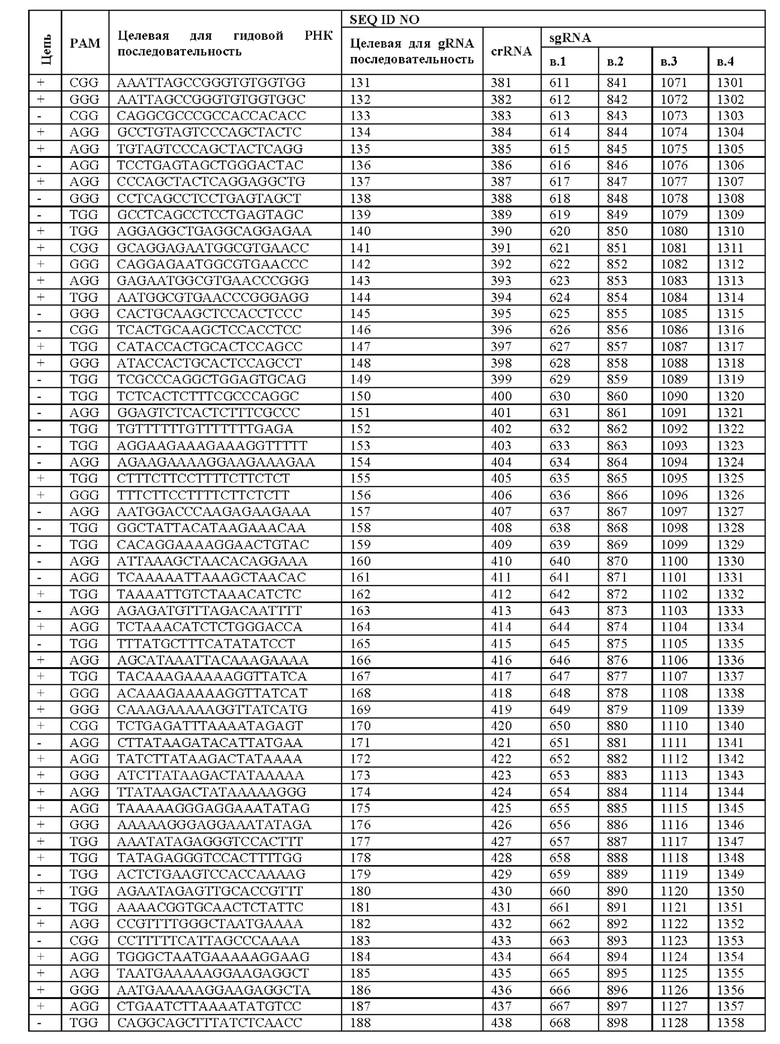
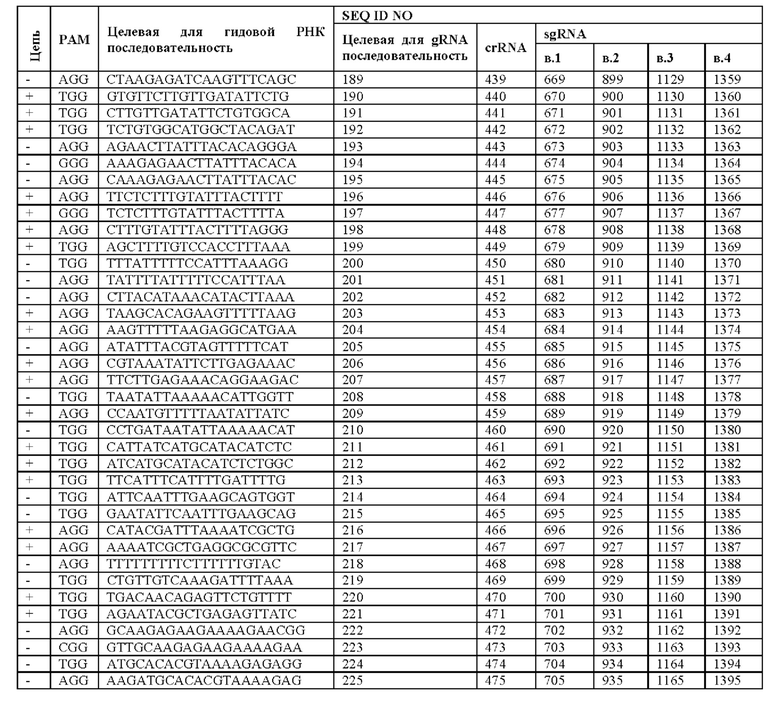
[00325] Примеры целевых для гидовой РНК последовательностей, находящихся в непосредственной близости к положению, соответствующему положению 12666 из SEQ ID NO: 2, содержат, фактически состоят из или состоят из последовательностей, изложенных под SEQ ID NO: 226-239, и изложены в приведенной ниже таблице. Примеры нацеливающих на ДНК сегментов гидовой РНК, соответствующих SEQ ID NO: 226-239, представлены соответственно под SEQ ID NO: 1629-1642, которые идентичны SEQ ID NO: 226-239 за исключением урацилов вместо тиминов. Нацеливающий на ДНК сегмент гидовой РНК может содержать, фактически состоять или состоять из любых последовательностей, изложенных под SEQ ID NO: 1629-1642. Иллюстративные целевые для гидовой РНК последовательности, находящиеся в непосредственной близости к положению, соответствующему положению 12666 из SEQ ID NO: 2, включают SEQ ID NO: 230 и 231. Иллюстративные crRNA и sgRNA (содержащие каркасы версии 1, 2, 3 или 4), соответствующие целевым для гидовой РНК последовательностям, находящимся в непосредственной близости к положению, соответствующему положению 12666 из SEQ ID NO: 2, содержат, фактически состоят из или состоят из любой из последовательностей, изложенных в приведенной ниже таблице.
[00326]
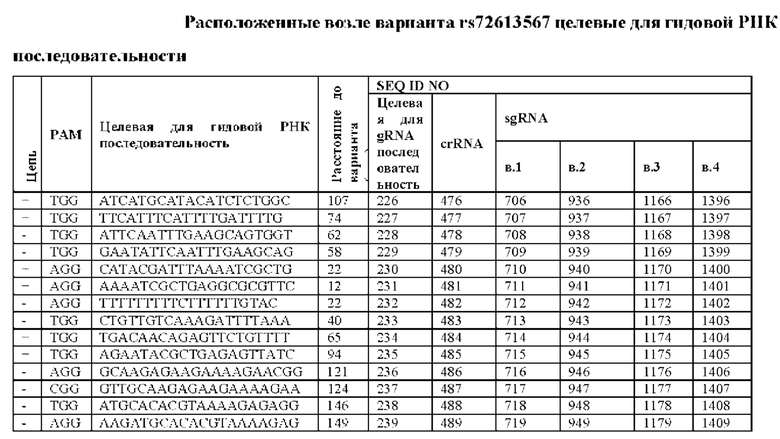
[00327] Примеры целевых для гидовой РНК последовательностей в гене Hsd17b13 мыши, находящихся в непосредственной близости к положению, соответствующему положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена Hsd17b13 мыши с SEQ ID NO: 2, содержат, фактически состоят из или состоят из последовательностей, изложенных в таблице 12 примера 4. Примеры целевых для гидовой РНК последовательностей на 5'-конце гена Hsd17b13 мыши содержат, фактически состоят или состоят из последовательностей, изложенных в таблице 12 примера 4. Примеры нацеливающих на ДНК сегментов гидовой РНК, соответствующих таковым последовательностям целевой для гидовой РНК, также изложены в таблице 12 примера 4. Нацеливающий на ДНК сегмент гидовой РНК может содержать, фактически состоять или состоять из любых таких последовательностей. Иллюстративные crRNA и sgRNA (содержащие каркасы версии 1, 2, 3 или 4), соответствующие целевым для гидовой РНК последовательностям из таблицы 12 примера 4, могут содержать, фактически состоять или состоять из последовательностей crRNA или sgRNA, изложенных в таблице 12 примера 4.
[00328] Гидовые РНК могут содержать два сегмента: «нацеливающий на ДНК сегмент» и «связывающий белок сегмент». «Сегмент» включает отрезок или участок молекулы, такой непрерывный интервал из нуклеотидов в РНК. Некоторые gRNA, такие как те, которые нацелены на Cas9, могут содержать две отдельные молекулы: «РНК-активатор» (например, tracrRNA) и «РНК-нацеливатель» (например, РНК CRISPR или crRNA). Другие gRNA представляют собой единственную молекулу РНК (единственный полинуклеотид РНК), которую также можно назвать «одномолекулярной gRNA», «одиночной гидовой РНК» или «sgRNA». См., например, WO 2013/176772, WO 2014/065596, WO 2014/089290, WO 2014/093622, WO 2014/099750, WO 2013/142578 и WO 2014/131833, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. В случае Cas9, например, одиночная гидовая РНК может содержать crRNA, слитую с tracrRNA (например, с помощью линкера). В случае Cpf1, например, для осуществления связывания и/или расщепления целевой последовательности необходима только crRNA. Термины «гидовая РНК» и «gRNA» включают как двухмолекулярные (т.е. модульные) gRNA, так и одномолекулярные gRNA.
[00329] Иллюстративная двух молекулярная gRNA содержит crRNA-подобную («РНК CRISPR», или «РНК-нацеливатель», или «crRNA» или «повтор crRNA») молекулу и соответствующую tracrRNA-подобную («транс-действующую РНК CRISPR», или «РНК-активатор», или «tracrRNA») молекулу. crRNA содержит как нацеливающий на ДНК сегмент (одноцепочечный) gRNA, так и интервал из нуклеотидов (т.е. хвост crRNA), который образует одну половину dsRNA-дуплекса связывающего белок сегмента gRNA. Пример хвоста crRNA, расположенного ниже (3') нацеливающего на ДНК сегмента, содержит, фактически состоит из или состоит из GUUUUAGAGCUAUGCU (SEQ ID NO: 1421). Любой из раскрытых в настоящем документе нацеливающих на ДНК сегментов можно присоединить к 5'-концу SEQ ID NO: 1421 с образованием crRNA.
[00330] Соответствующий tracrRNA (РНК-активатор) содержит интервал из нуклеотидов, который образует другую половину dsRNA-дуплекса связывающего белок сегмента gRNA. Интервал из нуклеотидов crRNA комплементарен и гибридизируется с интервалом из нуклеотидов tracrRNA с образованием dsRNA-дуплекса связывающего белок домена gRNA. Таким образом, можно сказать, что каждая crRNA имеет соответствующую tracrRNA. Пример последовательности tracrRNA содержит, фактически состоит из или состоит из AGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCUUU (SEQ ID NO: 1422).
[00331] В системах, в которых необходимы как crRNA, так и tracrRNA, crRNA и соответствующая tracrRNA гибридизируются с образованием gRNA. В системах, в которых необходима только crRNA, crRNA может представлять собой gRNA. Кроме того, crRNA обеспечивает одноцепочечный нацеливающий на ДНК сегмент, который нацеливает на целевую для гидовой РНК последовательность путем гибридизации с противоположной цепью (т.е. комплементарной цепью). При использовании для модификации в клетке можно разработать такую точную последовательность заданной молекулы crRNA или tracrRNA, чтобы она была специфичной для вида, в котором будут использоваться данные молекулы РНК. См., например, Mali et al. (2013) Science 339:823-826; Jinek et al. (2012) Science 337:816-821; Hwang et al. (2013) Nat. Biotechnol. 31:227-229; Jiang et al. (2013) Nat. Biotechnol. 31:233-239; и Cong et al. (2013) Science 339:819-823, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00332] Нацеливающий на ДНК сегмент (crRNA) заданной gRNA содержит нуклеотидную последовательность, которая комплементарна последовательности (т.е. комплементарной цепи последовательности распознавания для гидовой РНК на цепи, противоположной целевой для гидовой РНК последовательности) в целевой ДНК. Нацеливающий на ДНК сегмент в gRNA взаимодействует с целевой ДНК (например, гена HSD17B13) специфичным для последовательности образом посредством гибридизации (т.е. спаривания оснований). В связи с этим, нуклеотидная последовательность нацеливающего на ДНК сегмента может варьировать и определяет местоположение в целевой ДНК, с которым будут взаимодействовать gRNA и целевая ДНК. Нацеливающий на ДНК сегмент рассматриваемой gRNA можно модифицировать так, чтобы он гибридизировался с любой необходимой последовательностью в целевой ДНК. Встречающиеся в естественной среде crRNA различаются в зависимости от системы CRISPR/Cas и организма, но зачастую содержат нацеливающий сегмент длиной 21-72 нуклеотида, фланкированный двумя прямыми повторами (DR) длиной от 21 до 46 нуклеотидов (см., например, WO 2014/131833, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). В случае S. pyogenes, DR в длину составляют 36 нуклеотидов, а нацеливающий сегмент в длину составляет 30 нуклеотидов. 3'-расположенный DR комплементарен и гибридизируется с соответствующей tracrRNA, которая, в свою очередь, связывается с белком Cas.
[00333] Нацеливающий на ДНК сегмент может иметь длину, составляющую по меньшей мере приблизительно 12 нуклеотидов, по меньшей мере приблизительно 15 нуклеотидов, по меньшей мере приблизительно 17 нуклеотидов, по меньшей мере приблизительно 18 нуклеотидов, по меньшей мере приблизительно 19 нуклеотидов, по меньшей мере приблизительно 20 нуклеотидов, по меньшей мере приблизительно 25 нуклеотидов, по меньшей мере приблизительно 30 нуклеотидов, по меньшей мере приблизительно 35 нуклеотидов или по меньшей мере приблизительно 40 нуклеотидов. Такие нацеливающие на ДНК сегменты могут иметь длину от приблизительно 12 нуклеотидов до приблизительно 100 нуклеотидов, от приблизительно 12 нуклеотидов до приблизительно 80 нуклеотидов, от приблизительно 12 нуклеотидов до приблизительно 50 нуклеотидов, от приблизительно 12 нуклеотидов до приблизительно 40 нуклеотидов, от приблизительно 12 нуклеотидов до приблизительно 30 нуклеотидов, от приблизительно 12 нуклеотидов до приблизительно 25 нуклеотидов или от приблизительно 12 нуклеотидов до приблизительно 20 нуклеотидов. Например, нацеливающий на ДНК сегмент может составлять от приблизительно 15 нуклеотидов до приблизительно 25 нуклеотидов (например, от приблизительно 17 нуклеотидов до приблизительно 20 нуклеотидов или приблизительно 17 нуклеотидов, приблизительно 18 нуклеотидов, приблизительно 19 нуклеотидов или приблизительно 20 нуклеотидов). См., например, US 2016/0024523, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. В случае Cas9 от S. pyogenes, типичный нацеливающий на ДНК сегмент составляет от 16 до 20 нуклеотидов в длину или от 17 до 20 нуклеотидов в длину. В случае Cas9 от S. aureus, типичный нацеливающий на ДНК сегмент составляет от 21 до 23 нуклеотидов в длину. В случае Cpf1, типичный нацеливающий на ДНК сегмент составляет по меньшей мере 16 нуклеотидов в длину или по меньшей мере 18 нуклеотидов в длину.
[00334] TracrRNA могут быть представлены в любой форме (например, полноразмерные tracrRNA или активные частичные tracrRNA) и иметь различную длину. Они могут включать первичные транскрипты или процессированные формы. Например, tracrRNA (как часть одиночной гидовой РНК или как отдельная молекула как часть двухмолекулярной gRNA) могут содержать всю или часть последовательности tracrRNA дикого типа (например, приблизительно или более чем приблизительно 20, 26, 32, 45, 48, 54, 63, 67, 85 или более нуклеотидов последовательности tracrRNA дикого типа) или состоять из нее. Примеры последовательностей tracrRNA дикого типа от S. pyogenes включают 171-нуклеотидные, 89-нуклеотидные, 75-нуклеотидные и 65-нуклеотидные варианты. См., например, Deltcheva et al. (2011) Nature 471:602-607; WO 2014/093661, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Примеры tracrRNA в одиночных гидовых РНК (sgRNA) включают tracrRNA-сегменты, среди которых встречаются +48, +54, +67 и +85 варианты sgRNA, где «+n» обозначает, что в sgRNA включено до +n нуклеотида tracrRNA дикого типа. См. US 8697359, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00335] Процент комплементарности между нацеливающей на ДНК последовательностью и комплементарной цепью последовательности распознавания для гидовой РНК в целевой ДНК может составлять по меньшей мере 60% (например, по меньшей мере 65%, по меньшей мере 70%, по меньшей мере 75%, по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 97%, по меньшей мере 98%), по меньшей мере 99% или 100%). Процент комплементарности между нацеливающей на ДНК последовательностью и комплементарной цепью последовательности распознавания для гидовой РНК в целевой ДНК может составлять по меньшей мере 60% на протяжении приблизительно 20 смежных нуклеотидов. В качестве примера, процент комплементарности между нацеливающей на ДНК последовательностью и комплементарной цепью последовательности распознавания для гидовой РНК в целевой ДНК составляет 100%) на протяжении 14 смежных нуклеотидов на 5'-конце комплементарной цепи последовательности распознавания для гидовой РНК в комплементарной цепи целевой ДНК и всего 0% на протяжении остальной цепи. В таком случае, можно считать, что нацеливающая на ДНК последовательность в длину составляет 14 нуклеотидов. В качестве другого примера, процент комплементарности между нацеливающей на ДНК последовательностью и комплементарной цепью последовательности распознавания для гидовой РНК в целевой ДНК составляет 100% на протяжении семи смежных нуклеотидов на 5'-конце комплементарной цепи последовательности распознавания для гидовой РНК в комплементарной цепи целевой ДНК и всего 0% на протяжении остальной цепи. В таком случае, можно считать, что нацеливающая на ДНК последовательность в длину составляет 7 нуклеотидов. В некоторых гидовых РНК по меньшей мере 17 нуклеотидов в нацеливающей на ДНК последовательности комплементарны целевой ДНК. Например, нацеливающая на ДНК последовательность может составлять 20 нуклеотидов в длину и может содержать 1, 2 или 3 несоответствия с комплементарной цепью последовательности распознавания для гидовой РНК. Предпочтительно, несоответствия не являются смежными с последовательностью смежного с протоспейсером мотива (РАМ) (например, несоответствия находятся в 5'-конце нацеливающей на ДНК последовательности, или несоответствия находятся на расстоянии по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18 или 19 пар оснований от последовательности РАМ).
[00336] Связывающий белок сегмент gRNA может содержать два интервала из нуклеотидов, которые комплементарны друг другу. Комплементарные нуклеотиды связывающего белок сегмента гибридизируются с образованием двухцепочечного РНК-дуплекса (dsRNA). Связывающий белок сегмент рассматриваемой gRNA взаимодействует с белком Cas, a gRNA направляет связанный белок Cas к конкретной нуклеотидной последовательности в целевой ДНК с помощью нацеливающего на ДНК сегмента.
[00337] Одиночные гидовые РНК имеют нацеливающий на ДНК сегмент и остовную последовательность (т.е. связывающую белок или связывающую Cas последовательность гидовой РНК). Например, такие гидовые РНК имеют 5' нацеливающий на ДНК сегмент и 3' остовную последовательность. Иллюстративные остовные последовательности содержат, фактически состоят из или состоят из GUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGCU (вариант 1; SEQ ID NO: 1420), GUUGGAACCAUUCAAAACAGCAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (вариант 2; SEQ ID NO: 256), GUUUUAGAGCUAGAAAUAGCAAGUUAAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (вариант 3; SEQ ID NO: 257) и GUUUAAGAGCUAUGCUGGAAACAGCAUAGCAAGUUUAAAUAAGGCUAGUCCGUUAUCAACUUGAAAAAGUGGCACCGAGUCGGUGC (вариант 4; SEQ ID NO: 258). Гидовые РНК, нацеленные на любую из раскрываемых в настоящем документе целевых для гидовой РНК последовательностей (например, SEQ ID NO: 20-239 и 259-268) может включать, например, нацеливающий на ДНК сегмент на 5'-конце гидовой РНК, слитый с любой из иллюстративных остовных последовательностей гидовой РНК на 3'-конце гидовой РНК. То есть, любой из раскрытых в настоящем документе нацеливающих на ДНК сегментов можно присоединить к 5'-концу любой из SEQ ID NO: 1420, 256, 257 или 258 с образованием одной гидовой РНК (химерной гидовой РНК). Варианты 1, 2, 3 и 4 гидовой РНК, которые раскрыты в других разделах настоящего документа, относятся к нацеливающим на ДНК сегментам, соединенным соответственно с остовными версиями 1, 2, 3 и 4.
[00338] Гидовые РНК могут включать модификации или последовательности, которые обеспечивают дополнительные требуемые признаки (например, измененную или регулируемую стабильность; субклеточное нацеливание, отслеживание с помощью флуоресцентной метки; сайт связывания для белка или белкового комплекса и др.). Примеры таких модификаций включают, например, 5'-кэп (например, 7-метилгуанилатный кэп (m7G)); 3'-полиаденилатный хвост (т.е. 3'-поли(А)-хвост); последовательность рибосвитч (например, для обеспечения регулируемой стабильности и/или регулируемой доступности для белков и/или белковых комплексов); последовательность контроля стабильности; последовательность, которая образует dsRNA-дуплекс (т.е. шпильку); модификацию или последовательность, которая нацеливает РНК на субклеточную локализацию (например, ядро, митохондрии, хлоропласта и тому подобное); модификацию или последовательность, которая позволяет производить отслеживание (например, прямое конъюгирование с флуоресцентной молекулой, конъюгирование с фрагментом, который облегчает детектирование флуоресценции, последовательность, которая делает возможным детектирование флуоресценции и т.д.); модификацию или последовательность, которая обеспечивает сайт связывания для белков (например, белков, которые оказывают влияние на ДНК, в том числе активаторы транскрипции, репрессоры транскрипции, ДНК-метилтрансферазы, ДНК-деметилазы, гистондеацетилазы и т.д); и их комбинации. К другим примерам модификаций относятся сконструированные дуплексные структуры с петлей и стеблем, сконструированные участки выпетливания, сконструированные шпильки 3' от дуплексной структуры с петлей и стеблем или любая их комбинация. См., например, US 2015/0376586, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Выпетливание может представлять собой неспаренный участок нуклеотидов в дуплексе, состоящем из crRNA-подобного участка и минимального tracrRNA-подобного участка. Выпетливание может содержать на одной стороне дуплекса неспаренный 5'-XXXY-3', где X представляет собой любой пурин, a Y может представлять собой нуклеотид, который образует неустойчивую пару с нуклеотидом на противоположной цепи, и неспаренный участок на другой стороне дуплекса.
[00339] В некоторых случаях можно использовать систему активации транскрипции, содержащую белок слияния dCas9-VP64 в паре с MS2-p65-HSF1. Гидовые РНК в таких системах можно сконструировать с аптамерными последовательностями, присоединенными к тетра-петле sgRNA и стволовой петле 2, предназначенными для связывания димеризованных белков оболочки бактериофага MS2. См., например, Konermann et al. (2015) Nature 517(7536):583-588, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей.
[00340] Гидовые РНК могут быть представлены в любой форме. Например, gRNA может быть представлена в форме РНК, либо в виде двух молекул (отдельных crRNA и tracrRNA), либо в виде одной молекулы (sgRNA), и необязательно в форме комплекса с белком Cas. Например, молекулы gRNA можно получить путем транскрипции in vitro с применением, например, РНК-полимеразы Т7 (см., например, WO 2014/089290 и WO 2014/065596, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Гидовые РНК также можно получить с помощью химического синтеза.
[00341] GRNA также может быть представлена в форме ДНК, кодирующей gRNA. ДНК, кодирующая gRNA, может кодировать одну молекулу РНК (sgRNA) или отдельные молекулы РНК (например, отдельные crRNA и tracrRNA). В последнем случае ДНК, кодирующая gRNA, может быть представлена в виде одной молекулы ДНК или в виде раздельных молекул ДНК, кодирующих соответственно crRNA и tracrRNA.
[00342] Если gRNA представлена в форме ДНК, gRNA может временно, условно или конститутивно экспрессироваться в клетке. Молекулы ДНК, кодирующие молекулы gRNA, можно стабильно интегрировать в геном клетки и функционально связать с активным в клетке промотором. В качестве альтернативы, молекулы ДНК, кодирующие молекулы gRNA, можно функционально связать с промотором в экспрессирующей конструкции. Например, ДНК, кодирующая gRNA, может находиться в векторе, содержащем гетерологичную нуклеиновую кислоту. Вектор может дополнительно содержать экзогенную донорную последовательность, и/или вектор может дополнительно содержать нуклеиновую кислоту, кодирующую белок Cas. В качестве альтернативы, ДНК, кодирующая gRNA, может находиться в векторе или плазмиде, которая отделена от вектора, содержащего экзогенную донорную последовательность, и/или вектора, содержащего нуклеиновую кислоту, кодирующую белок Cas. Промоторы, которые можно применять в таких экспрессирующих конструкциях, включают промоторы, активные, например, в одной или нескольких из эукариотической клетки, клетки человека, клетки отличного от человека животного, клетки млекопитающего, клетки отличного от человека млекопитающего, клетки грызуна, клетки мыши, клетки крысы, клетки хомяка, клетки кролика, плюрипотентной клетки, эмбриональной стволовой (ES) клетки, стволовой клетки взрослого человека, клетки-предшественника, ограниченной стадией развития, индуцированной плюрипотентной стволовой клетки (iPS) или эмбриона на одноклеточной стадии. Такие промоторы могут быть, например, условными промоторами, индуцируемыми промоторами, конститутивными промоторами или тканеспецифичными промоторами. Такие промоторы также могут быть, например, двунаправленными промоторами. Конкретные примеры подходящих промоторов включают промотор РНК-полимеразы III, такой как промотор U6 человека, промотор полимеразы III U6 крысы или промотор полимеразы III U6 мыши.
[00343] Также в настоящем документе раскрыты композиции, содержащие одну или несколько раскрываемых в настоящем документе гидовых РНК (например, 1, 2, 3, 4 или более гидовых РНК) и носитель, повышающий стабильность выделенной нуклеиновой кислоты или белка (например, продлевающий указанный период при заданных условиях хранения (например, при -20°С, 4°С или температуре окружающей среды), в течение которого продукты разложения остаются ниже порогового уровня, такого как ниже 0,5% по массе исходной нуклеиновой кислоты или белка, или повышение стабильности in vivo). Неограничивающие примеры таких носителей включают микросферы из полимолочной кислоты (PLA), микросферы из сополимера D,L-молочной и когликолиевой кислот (PLGA), липосомы, мицеллы, инверсные мицеллы, липидные кохлеаты и липидные микротрубочки. Такие композиции могут дополнительно содержать белок Cas, такое как белок Cas9, или нуклеиновую кислоту, кодирующую белок Cas. Такие композиции могут дополнительно содержать одну или несколько (например, 1, 2, 3, 4 или более) экзогенных донорных последовательностей, и/или один или несколько (например, 1, 2, 3, 4 или более) направленных векторов, и/или один или несколько (например, 1, 2, 3, 4 или более) векторов экспрессии, которые раскрыты в других разделах настоящего документа.
(3) Последовательности распознавания для гидовой РНК и целевые для гидовой РНК последовательности
[00344] Термин «последовательность распознавания для гидовой РНК» охватывает последовательности нуклеиновой кислоты, присутствующие в целевой ДНК (например, гене HSD17B13), с которыми будет связываться нацеливающий на ДНК сегмент в gRNA, при условии существования достаточных условий для связывания. Используемый в настоящем документе термин «последовательность распознавания для гидовой РНК» охватывает обе цепи целевой двухцепочечной ДНК (т.е. последовательность на комплементарной цепи, с которой гибридизируется гидовая РНК, и соответствующую последовательность на некомплементарной цепи, находящейся рядом со смежным с протоспейсером мотивом (РАМ)). Используемый в настоящем документе термин «целевая для гидовой РНК последовательность» обозначает, в частности, последовательность на некомплементарной цепи рядом с РАМ (т.е. выше или 5' от РАМ). То есть, целевая для гидовой РНК последовательность обозначает последовательность на некомплементарной цепи, которая соответствует последовательности, с которой гидовая РНК гибридизируется на комплементарной цепи. Целевая для гидовой РНК последовательность эквивалентна нацеливающему на ДНК сегменту гидовой РНК, но с тиминами вместо урацилов. В качестве одного примера, целевая для гидовой РНК последовательность в случае фермента Cas9 будет обозначать последовательность на некомплементарной цепи рядом с 5'-NGG-3' РАМ. Последовательности распознавания для гидовой РНК включают последовательности, которые должны быть такими, чтобы гидовая РНК была комплементарна к ним, причем гибридизация между комплементарной цепью последовательности распознавания для гидовой РНК и нацеливающей на ДНК последовательностью гидовой РНК способствует образованию комплекса CRISPR. Полная комплементарность не является обязательной при условии, что существует достаточная комплементарность, обуславливающая гибридизацию и способствующая образованию комплекса CRISPR. Последовательности распознавания для гидовой РНК или целевые для гидовой РНК последовательности также включают сайты расщепления для белков Cas, которые более подробно описаны ниже. Последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность может содержать любой полинуклеотид, который может располагаться, например, в ядре или цитоплазме клетки или в органелле клетки, такой как митохондрия или хлоропласт.
[00345] Последовательность распознавания для гидовой РНК в целевой ДНК может служить целью для (т.е. ее может связывать, или с ней может гибридизироваться, или она является комплементарной для) белка Cas или gRNA. Подходящие условия связывания ДНК/РНК включают физиологические условия, в норме присутствующие в клетке. Известны и другие подходящие условия связывания ДНК/РНК (например, условия в бесклеточной системе) (см., например, Molecular Cloning: A Laboratory Manual, 3rd Ed. (Sambrook et al., Harbor Laboratory Press 2001), включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Цепь целевой ДНК, которая комплементарна и гибридизируется с белком Cas или gRNA, можно назвать «комплементарной цепью», а цепь целевой ДНК, которая комплементарна «комплементарной цепи» (и, следовательно, не является комплементарной белку Cas или gRNA), можно назвать «некомплементарной цепью» или «матричной цепью».
[00346] Белок Cas может расщеплять нуклеиновую кислоту в сайте, находящемся в пределах или за пределами последовательности нуклеиновой кислоты, присутствующей в целевой ДНК, с которой будет связываться нацеливающий на ДНК сегмент в gRNA. «Сайт расщепления» включает положение нуклеиновой кислоты, в котором белок Cas производит одноцепочечный разрыв или двухцепочечный разрыв. Например, образование комплекса CRISPR (содержащего gRNA, гибридизированную с комплементарной цепью последовательности распознавания для гидовой РНК и в комплексе с белком Cas) может привести к расщеплению одной или обеих цепей непосредственно в последовательности нуклеиновой кислоты или рядом с последовательностью нуклеиновой кислоты (например, в пределах 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 20, 50 или более пар оснований от последовательности нуклеиновой кислоты), присутствующей в целевой ДНК, с которой будет связываться нацеливающий на ДНК сегмент gRNA. Если сайт расщепления находится за пределами последовательности нуклеиновой кислоты, с которой будет связываться нацеливающий на ДНК сегмент gRNA, сайт расщепления по-прежнему считают находящимся в пределах «последовательности распознавания для гидовой РНК» или целевой для гидовой РНК последовательности. Сайт расщепления может находиться только на одной цепи или на обеих цепях нуклеиновой кислоты. Сайты расщепления могут находиться в одном и том же положении на обеих цепях нуклеиновой кислоты (что приводит к образованию тупых концов) или могут находиться в различных сайтах на каждой цепи (что приводит к образованию ступенчатых концов (т.е. «липких» концов)). Ступенчатые концы можно получить, например, с помощью двух белков Cas, каждый из которых производит одноцепочечный разрыв в ином сайте расщепления на иной цепи, таким образом получая двухцепочечный разрыв. Например, первая никаза может создавать одноцепочечный разрыв на первой цепи двухцепочечной ДНК (dsDNA), а вторая никаза может создавать одноцепочечный разрыв на второй цепи dsDNA так, чтобы создавались последовательности «липких» концов. В некоторых случаях последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность никазы на первой цепи отделена от последовательности распознавания для гидовой РНК или целевой для гидовой РНК последовательности никазы на второй цепи по меньшей мере 2, 3, 4, 5, 6, 7, 8, 9, 10, 15, 20, 25, 30, 40, 50, 75, 100, 250, 500 или 1000 парами оснований.
[00347] Сайт-специфическое связывание и/или расщепление целевой ДНК белками Cas может происходить в местоположениях, определяемых как (i) комплементарностью спаривания оснований между gRNA и целевой ДНК, так и (ii) коротким мотивом, называемым смежным к протоспейсеру мотивом (РАМ), в целевой ДНК. РАМ может фланкировать целевую для гидовой РНК последовательность на некомплементарной цепи, которая противоположна цепи, с которой гибридизируется гидовая РНК. Необязательно, целевая для гидовой РНК последовательность может быть фланкирована на 3'-конце посредством РАМ. В качестве альтернативы, целевая для гидовой РНК последовательность может быть фланкирована на 5'-конце посредством РАМ. Например, сайт расщепления белков Cas может находиться на расстоянии от приблизительно 1 до приблизительно 10 или от приблизительно 2 до приблизительно 5 пар оснований (например, 3 пар оснований) выше или ниже последовательности РАМ. В некоторых случаях (например, при использовании Cas9 от S. pyogenes или близкородственного Cas9) последовательность РАМ некомплементарной цепи может представлять собой 5'-N-1GG-3', где N-1 представляет собой любой нуклеотид ДНК и находится непосредственно 3' от последовательности распознавания для гидовой РНК некомплементарной цепи целевой ДНК (т.е. непосредственно 3' от целевой для гидовой РНК последовательности). Таким образом, последовательность РАМ комплементарной цепи будет 5'-CCN-2-3', где N-2 представляет собой любой нуклеотид ДНК, и находится непосредственно 5' от последовательности распознавания для гидовой РНК комплементарной цепи целевой ДНК. В некоторых таких случаях N1 и N2 могут быть комплементарны, а пара оснований N1-N2 может быть любой парой оснований (например, N1=C и N2=G; N1=G и N2=С; N1=A и N2=Т; или N1=T и N2=А). В случае Cas9 от S. aureus, РАМ может представлять собой NNGRRT или NNGRR, где N может представлять собой A, G, С или Т, a R может представлять собой G или А. В некоторых случаях (например, в случае FnCpf1) последовательность РАМ может находиться выше 5'-конца и имеет последовательность 5'-TTN-3'.
[00348] Ниже приведены примеры целевых для гидовой РНК последовательностей или целевых для гидовой РНК последовательностей в дополнение к последовательности РАМ. Например, целевая для гидовой РНК последовательность может представлять собой последовательность ДНК из 20 нуклеотидов, непосредственно предшествующую мотиву NGG, который распознается белком Cas9. Примерами такой целевой для гидовой РНК последовательности плюс последовательности РАМ являются GN19NGG (SEQ ID NO: 248) или N20NGG (SEQ ID NO: 249). См., например, WO 2014/165825, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Гуанин на 5'-конце может облегчать транскрипцию РНК-полимеразой в клетках. Другие примеры целевых для гидовой РНК последовательностей плюс последовательность РАМ может включать два гуаниновых нуклеотида на 5'-конце (например, GGN20NGG; SEQ ID NO: 250) для облегчения эффективной транскрипции с помощью полимеразы Т7 in vitro. См., например, WO 2014/065596, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Другие целевые для гидовой РНК последовательности плюс последовательность РАМ могут составлять 4-22 нуклеотида в длину из SEQ ID NO: 248-250, включая 5' G или GG и 3' GG или NGG. Еще одни целевые для гидовой РНК последовательности могут составлять 14-20 нуклеотидов в длину из SEQ ID NO: 248-250.
[00349] Последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность может быть любой последовательностью нуклеиновой кислоты, которая является эндогенной или экзогенной для клетки. Последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность может быть последовательностью, кодирующей генный продукт (например, белок), или некодирующей последовательностью (например, регуляторной последовательностью) или может включать обе из них.
[00350] В качестве одного примера, последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность может находиться в участке, соответствующем экзону 6 и/или интрону 6, экзону 6 и/или экзону 7 либо экзону 6, и/или интрону 6, и/или экзону 7 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. В качестве другого примера, последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность может включать положение или находится в непосредственной близости от него, которое соответствует положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. Например, последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность может находиться в пределах приблизительно 1000, 500, 400, 300, 200, 100, 50, 45, 40, 35, 30, 25, 20, 15, 10 или 5 нуклеотидов от положения, соответствующего положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2. В качестве еще одного примера, последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность может включать старт-кодон гена HSD17B13 или стоп-кодон гена HSD17B13 или находится в непосредственной близости от него. Например, последовательность распознавания для гидовой РНК или целевая для гидовой РНК последовательность может находиться в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона или стоп-кодона. Примеры таких целевых для гидовой РНК последовательностей и гидовых РНК, нацеленных на такие целевые для гидовой РНК последовательности, раскрыты в других разделах настоящего документа.
F. Экзогенные донорные последовательности или направленные векторы
[00351] Способы и композиции, раскрываемые в настоящем документе, могут предусматривать использование экзогенных донорных последовательностей (например, направленных векторов или матриц для репарации) для модификации гена HSD17B13 либо без расщепления гена HSD17В13, либо после расщепления гена HSD17В13 нуклеазным средством. Экзогенная донорная последовательность относится к любой нуклеиновой кислоте или любому вектору, которые включают элементы, которые необходимы для осуществления сайт-специфической рекомбинации с целевой последовательностью. Применение экзогенных донорных последовательностей в сочетании с нуклеазными средствами может привести к более точным модификациям в гене HSD17В13 путем облегчения гомологичной репарации.
[00352] В таких способах нуклеазное средство расщепляет ген HSD17B13, создавая одноцепочечный разрыв (надрез) или двухцепочечный разрыв, а экзогенная донорная последовательность подвергается рекомбинации с геном HSD17B13 путем сшивания, опосредованного негомологичным соединением концов (NHEJ), или посредством события гомологичной репарации. Необязательно, репарация с помощью экзогенной донорной последовательности удаляет или нарушает структуру сайта расщепления нуклеазой с тем, чтобы аллели, которые уже подверглись целевой модификации, нельзя было повторно подвергнуть целевой модификации нуклеазным средством.
[00353] Экзогенные донорные последовательности могут содержать дезоксирибонуклеиновую кислоту (ДНК) или рибонуклеиновую кислоту (РНК), они могут быть одноцепочечными или двухцепочечными, и они могут иметь линейную или кольцевую форму. Например, экзогенная донорная последовательность может представлять собой одноцепочечный олигодезоксинуклеотид (ssODN). См., например, Yoshimi et al. (2016) Nat. Commun. 7:10431, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Иллюстративная экзогенная донорная последовательность составляет от приблизительно 50 нуклеотидов до приблизительно 5 т.о. в длину, составляет от приблизительно 50 нуклеотидов до приблизительно 3 т.о. в длину или составляет от приблизительно 50 до приблизительно 1000 нуклеотидов в длину. Другие иллюстративные экзогенные донорные последовательности составляют от приблизительно 40 до приблизительно 200 нуклеотидов в длину. Например, экзогенная донорная последовательность может составлять от приблизительно 50 до приблизительно 60, от приблизительно 60 до приблизительно 70, от приблизительно 70 до приблизительно 80, от приблизительно 80 до приблизительно 90, от приблизительно 90 до приблизительно 100, от приблизительно 100 до приблизительно 110, от приблизительно 110 до приблизительно 120, от приблизительно 120 до приблизительно 130, от приблизительно 130 до приблизительно 140, от приблизительно 140 до приблизительно 150, от приблизительно 150 до приблизительно 160, от приблизительно 160 до приблизительно 170, от приблизительно 170 до приблизительно 180, от приблизительно 180 до приблизительно 190 или от приблизительно 190 до приблизительно 200 нуклеотидов в длину. В качестве альтернативы, экзогенная донорная последовательность может составлять от приблизительно 50 до приблизительно 100, от приблизительно 100 до приблизительно 200, от приблизительно 200 до приблизительно 300, от приблизительно 300 до приблизительно 400, от приблизительно 400 до приблизительно 500, от приблизительно 500 до приблизительно 600, от приблизительно 600 до приблизительно 700, от приблизительно 700 до приблизительно 800, от приблизительно 800 до приблизительно 900 или от приблизительно 900 до приблизительно 1000 нуклеотидов в длину. В качестве альтернативы, экзогенная донорная последовательность может составлять от приблизительно 1 т.о. до приблизительно 1,5 т.о., от приблизительно 1,5 т.о. до приблизительно 2 т.о., от приблизительно 2 т.о. до приблизительно 2,5 т.о., от приблизительно 2,5 т.о. до приблизительно 3 т.о., от приблизительно 3 т.о. до приблизительно 3,5 т.о., от приблизительно 3,5 т.о. до приблизительно 4 т.о., от приблизительно 4 т.о. до приблизительно 4,5 т.о. или от приблизительно 4,5 т.о. до приблизительно 5 т.о. в длину. В качестве альтернативы, экзогенная донорная последовательность может составлять, например, не более 5 т.о., 4,5 т.о., 4 т.о., 3,5 т.о., 3 т.о., 2,5 т.о., 2 т.о., 1,5 т.о., 1 т.о., 900 нуклеотидов, 800 нуклеотидов, 700 нуклеотидов, 600 нуклеотидов, 500 нуклеотидов, 400 нуклеотидов, 300 нуклеотидов, 200 нуклеотидов, 100 нуклеотидов или 50 нуклеотидов в длину.
[00354] В одном примере экзогенная донорная последовательность представляет собой ssODN, который составляет от приблизительно 80 нуклеотидов до приблизительно 200 нуклеотидов в длину (например, приблизительно 120 нуклеотидов в длину). В другом примере экзогенная донорная последовательность представляет собой ssODN, который составляет от приблизительно 80 нуклеотидов до приблизительно 3 т.о. в длину. Такой ssODN может иметь гомологичные плечи, например, каждое из которых составляет от приблизительно 40 нуклеотидов до приблизительно 60 нуклеотидов в длину. Такой ssODN также может иметь гомологичные плечи, например, каждое из которых составляет от приблизительно 30 нуклеотидов до 100 нуклеотидов в длину. Гомологичные плечи могут быть симметричными (например, каждое по 40 нуклеотидов или каждое по 60 нуклеотидов в длину), или же они могут быть асимметричными (например, одно гомологичное плечо, которое составляет 36 нуклеотидов в длину, и одно гомологичное плечо, которое составляет 91 нуклеотид в длину).
[00355] Экзогенные донорные последовательности могут включать модификации или последовательности, которые обеспечивают дополнительные требуемые признаки (например, измененную или регулируемую стабильность; отслеживание или детектирование с помощью флуоресцентной метки; сайт связывания для белка или белкового комплекса и т.д.). Экзогенные донорные последовательности могут содержать одну или несколько флуоресцентных меток, меток для очистки, эпитопных меток или их комбинацию. Например, экзогенная донорная последовательность может содержать одну или несколько флуоресцентных меток (например, флуоресцентных белков или других флуорофоров или красителей), таких как по меньшей мере 1, по меньшей мере 2, по меньшей мере 3, по меньшей мере 4 или по меньшей мере 5 флуоресцентных меток. К иллюстративным флуоресцентным меткам относятся флуорофоры, такие как флуоресцеин (например, 6-карбоксифлуоресцеин (6-FAM)), техасский красный, HEX, Су3, Су5, Су5.5, Pacific Blue, 5-(и-6)-карбокситетраметилродамин (TAMRA) и Су7. Для включения метки в олигонуклеотиды на рынке доступен широкий спектр флуоресцентных красителей коммерчески (например, от компании Integrated DNA Technologies). Такие флуоресцентные метки (например, внутренние флуоресцентные метки) можно применять, например, для детекции экзогенной донорной последовательности, которая была непосредственно встроена в расщепленный ген HSD17B13, имеющий выступающие концы, совместимые с концами экзогенной донорной последовательности. Метка или маркер могут располагаться на 5'-конце, на 3'-конце или внутри экзогенной донорной последовательности. Например, экзогенная донорная последовательность может быть конъюгирована на 5'-конце с флуорофором IR700 от Integrated DNA Technologies (5'IRDYE®700).
[00356] Экзогенные донорные последовательности также могут содержать вставки нуклеиновой кислоты, включая сегменты ДНК, подлежащие встраиванию в ген HSD17B13. Встраивание вставки нуклеиновой кислоты в ген HSD17B13 может привести к добавлению представляющей интерес последовательности нуклеиновой кислоты в ген HSD17B13, делеции представляющей интерес последовательности нуклеиновой кислоты в гене HSD17B13 или замене представляющей интерес последовательности нуклеиновой кислоты в гене HSD17B13 (т.е. делеции и вставке). Некоторые экзогенные донорные последовательности предназначены для включения вставки нуклеиновой кислоты в ген HSD17B13 без какой-либо соответствующей делеции в гене HSD17B13. Другие экзогенные донорные последовательности предназначены для делеции представляющей интерес последовательности нуклеиновой кислоты в гене HSD17B13 без какого-либо соответствующего включения вставки нуклеиновой кислоты. Еще одни экзогенные донорные последовательности предназначены для делеции представляющей интерес последовательности нуклеиновой кислоты в гене HSD17B13 и замене ее вставкой нуклеиновой кислоты.
[00357] Вставка нуклеиновой кислоты или соответствующая нуклеиновая кислота в гене HSD17B13, в котором необходимо произвести делецию и/или замену, может иметь различную длину. Иллюстративная вставка нуклеиновой кислоты или соответствующая нуклеиновая кислота в гене HSD17B13, в котором необходимо произвести делецию и/или замену, составляет от приблизительно 1 нуклеотида до приблизительно 5 т.о. в длину или составляет от приблизительно 1 нуклеотида до приблизительно 1000 нуклеотидов в длину. Например, вставка нуклеиновой кислоты или соответствующая нуклеиновая кислота в гене HSD17B13, в котором необходимо произвести делецию и/или замену, может составлять от приблизительно 1 до приблизительно 10, от приблизительно 10 до приблизительно 20, от приблизительно 20 до приблизительно 30, от приблизительно 30 до приблизительно 40, от приблизительно 40 до приблизительно 50, от приблизительно 50 до приблизительно 60, от приблизительно 60 до приблизительно 70, от приблизительно 70 до приблизительно 80, от приблизительно 80 до приблизительно 90, от приблизительно 90 до приблизительно 100, от приблизительно 100 до приблизительно 110, от приблизительно 110 до приблизительно 120, от приблизительно 120 до приблизительно 130, от приблизительно 130 до приблизительно 140, от приблизительно 140 до приблизительно 150, от приблизительно 150 до приблизительно 160, от приблизительно 160 до приблизительно 170, от приблизительно 170 до приблизительно 180, от приблизительно 180 до приблизительно 190 или от приблизительно 190 до приблизительно 200 нуклеотидов в длину. Аналогичным образом, вставка нуклеиновой кислоты или соответствующая нуклеиновая кислота в гене HSD17B13, в котором необходимо произвести делецию и/или замену, может составлять от приблизительно 1 до приблизительно 100, от приблизительно 100 до приблизительно 200, от приблизительно 200 до приблизительно 300, от приблизительно 300 до приблизительно 400, от приблизительно 400 до приблизительно 500, от приблизительно 500 до приблизительно 600, от приблизительно 600 до приблизительно 700, от приблизительно 700 до приблизительно 800, от приблизительно 800 до приблизительно 900 или от приблизительно 900 до приблизительно 1000 нуклеотидов в длину. Аналогичным образом, вставка нуклеиновой кислоты или соответствующая нуклеиновая кислота в гене HSD17B13, в котором необходимо произвести делецию и/или замену, может составлять от приблизительно 1 т.о. до приблизительно 1,5 т.о., от приблизительно 1,5 т.о. до приблизительно 2 т.о., от приблизительно 2 т.о. до приблизительно 2,5 т.о., от приблизительно 2,5 т.о. до приблизительно 3 т.о., от приблизительно 3 т.о. до приблизительно 3,5 т.о., от приблизительно 3,5 т.о. до приблизительно 4 т.о., от приблизительно 4 т.о. до приблизительно 4,5 т.о. или от приблизительно 4,5 т.о. до приблизительно 5 т.о. в длину.
[00358] Вставка нуклеиновой кислоты может содержать геномную ДНК или ДНК любого другого типа. Например, вставка нуклеиновой кислоты может содержать кДНК.
[00359] Вставка нуклеиновой кислоты может содержать последовательность, которая гомологична всему гену HSD17B13 или его части (например, доле гена, кодирующего конкретный мотив или участок белка HSD17B13). Например, вставка нуклеиновой кислоты может содержать последовательность, которая содержит одну или несколько точечных мутаций (например, 1, 2, 3, 4, 5 или более) или одну или несколько нуклеотидных вставок или делеций в сравнении с последовательностью, которая предназначена для замены в гене HSD17B13.
[00360] Вставка нуклеиновой кислоты или соответствующая нуклеиновая кислота в гене HSD17B13, в котором необходимо произвести делецию и/или замену, может представлять собой кодирующий участок, такой как экзон; некодирующий участок, такой как интрон, нетранслируемый участок или регуляторный участок (например, промотор, энхансер или транскрипционный репрессор-связывающий элемент); или любую их комбинацию.
[00361] Вставка нуклеиновой кислоты также может содержать условный аллель. Условный аллель может представлять собой многофункциональный аллель, который описан в US 2011/0104799, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Например, условный аллель может содержать: (а) инициирующую последовательность в смысловой ориентации относительно транскрипции целевого гена; (b) кассету для отбора с помощью лекарственных средств (DSC) в смысловой или антисмысловой ориентации; (с) представляющую интерес нуклеотидную последовательность (NSI) в антисмысловой ориентации; и (d) зависящий от условия инверсии модуль (COIN, в котором используется расщепляющий экзон интрон и модуль для обратимой вставки инверсии в ген) в обратной ориентации. См., например, US 2011/0104799. Условный аллель может дополнительно содержать рекомбинируемые единицы, которые подвергаются рекомбинации под воздействием первой рекомбиназы с образованием условного аллеля, который (i) не имеет инициирующей последовательности и DSC; и (ii) содержит NSI в смысловой ориентации и COIN в антисмысловой ориентации. См., например, US 2011/0104799.
[00362] Вставки с нуклеиновой кислотой также могут содержать полинуклеотид, кодирующий маркер для отбора. В качестве альтернативы, во вставках нуклеиновой кислоты может отсутствовать полинуклеотид, кодирующий маркер для отбора. Маркер для отбора может содержаться в кассете для отбора. Необязательно, кассета для отбора может быть самоделетирующейся кассетой. См., например, US 8697851 и US 2013/0312129, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. В качестве примера, самоделетирующаяся кассета может содержать ген Crei (содержит два экзона, кодирующих рекомбиназу Cre, которые разделены интроном), функционально связанный с промотором Prm1 мыши, и ген устойчивости к неомицину, функционально связанный с убиквитиновым промотором человека. К иллюстративным маркерам для отбора относятся гены неомицинфосфотрансферазы (neor), гигромицин-В-фосфотрансферазы (hygr), пуромицин-N-ацетилтрансферазы (puror), бластицидин-S-дезаминазы (bsrr), ксантин/гуанинфосфорибозилтрансферазы (gpt) или тимидинкиназы вирус простого герпеса (HSV-k) либо их комбинация. Полинуклеотид, кодирующий маркер для отбора, может быть функционально связан с промотором, активным в подвергаемой целевой модификации клетке. Примеры промоторов описаны в других разделах настоящего документа.
[00363] Вставка нуклеиновой кислоты также может содержать репортерный ген. К иллюстративным репортерным генам относятся гены, кодирующие люциферазу, β-галактозидазу, зеленый флуоресцентный белок (GFP), усиленный зеленый флуоресцентный белок (eGFP), голубой флуоресцентный белок (CFP), желтый флуоресцентный белок (YFP), усиленный желтый флуоресцентный белок (eYFP), синий флуоресцентный белок (BFP), усиленный синий флуоресцентный белок (eBFP), DsRed, ZsGreen, MmGFP, mPlum, mCherry, tdTomato, mStrawberry, J-Red, mOrange, mKO, mCitrine, Venus, YPet, Emerald, CyPet, Cerulean, T-Sapphire и щелочную фосфатазу. Такие репортерные гены могут быть функционально связаны с промотором, активным в подвергаемой целевой модификации клетке. Примеры промоторов описаны в других разделах настоящего документа.
[00364] Вставка нуклеиновой кислоты также может содержать одну или несколько кассет экспрессии или делеционных кассет. Данная кассета может содержать одну или несколько представляющих интерес нуклеотидных последовательностей, полинуклеотид, кодирующий маркер для отбора, и репортерный ген наряду с различными регуляторными компонентами, которые влияют на экспрессию. Примеры позволяющих производить отбор маркеров и репортерных генов, которые можно включить, подробно рассмотрены в других разделах настоящего документа.
[00365] Вставка нуклеиновой кислоты может содержать нуклеиновую кислоту, фланкированную целевыми последовательностями для сайт-специфической рекомбинации. В качестве альтернативы, вставка нуклеиновой кислоты может содержать одну или несколько целевых последовательностей для сайт-специфической рекомбинации. Несмотря на то, что вся вставка нуклеиновой кислоты может быть фланкирована такими целевыми последовательностями для сайт-специфической рекомбинации, также такими сайтами может быть фланкирован любой представляющий интерес участок или отдельный полинуклеотид внутри вставки нуклеиновой кислоты. К целевым последовательностям для сайт-специфической рекомбинации, которые могут фланкировать вставку нуклеиновой кислоты или любой представляющий интерес полинуклеотид во вставке нуклеиновой кислоты, можно отнести, например, loxP, lox511, lox2272, lox66, lox71, loxM2, lox5171, FRT, FRT11, FRT71, attp, att, FRT, rox или их комбинацию. В одном примере сайты для сайт-специфической рекомбинации фланкируют полинуклеотид, кодирующий маркер для отбора и/или репортерный ген, содержащийся во вставке нуклеиновой кислоты. После встраивания вставки нуклеиновой кислоты в ген HSD17B13 последовательности между сайтами для сайт-специфической рекомбинации могут быть удалены. Необязательно, можно использовать две экзогенные донорные последовательности, каждая из которых имеет вставку нуклеиновой кислоты, содержащую сайт для сайт-специфической рекомбинации. Экзогенные донорные последовательности могут быть нацелены на 5'- и 3'-участки, фланкирующие представляющую интерес нуклеиновую кислоту. После встраивания двух вставок нуклеиновой кислоты в целевой геномный локус представляющая интерес нуклеиновая кислота между двумя вставленными сайтами для сайт-специфической рекомбинации может быть удалена.
[00366] Вставки нуклеиновой кислоты также могут содержать один или несколько сайтов рестрикции для рестрикционных эндонуклеаз (т.е. рестриктаз), к которым относятся эндонуклеазы I типа, II типа, III типа и IV типа. Рестрикционные эндонуклеазы I типа и III типа распознают конкретные последовательности распознавания, но расщепляют обычно в ином положении относительно сайта связывания нуклеазы, который может находиться на расстоянии сотен пар оснований от сайта расщепления (последовательности распознавания). В системах II типа рестрикционная активность не зависит от какой-либо метилазной активности, и расщепление обычно происходит в конкретных сайтах внутри или вблизи сайта связывания. Большинство ферментов II типа разрезают палиндромные последовательности, однако ферменты IIa типа распознают непалиндромные последовательности распознавания и производят расщепление за пределами последовательности распознавания, ферменты IIb типа разрезают последовательности дважды, причем оба сайта находятся за пределами последовательности распознавания, а ферменты IIs типа распознают асимметричную последовательность распознавания и производят расщепление на одной стороне и на заданном расстоянии, составляющем приблизительно 1-20 нуклеотидов от последовательность распознавания. Рестриктазы IV типа нацелены на метилированную ДНК. Рестриктазы дополнительно описаны и классифицированы, например, в базе данных REBASE (веб-страница находится по адресу rebase.neb.com; Roberts et al., (2003) Nucleic Acids Res. 31:418-420; Roberts et al., (2003) Nucleic Acids Res. 31:1805-1812; и Belfort et al. (2002) in Mobile DNA II, pp. 761-783, Eds. Craigie et al., (ASM Press, Washington, DC)).
(1) Донорные последовательности для вставки, опосредованной негомологичным соединением концов
[00367] Некоторые экзогенные донорные последовательности имеют короткие одноцепочечные участки на 5'-конце и/или 3'-конце, которые комплементарны одному или нескольким «липким» концам, созданным путем нуклеазного Cas-белкового расщепления в целевом геномном локусе (например, в гене HSD17B13). Эти «липкие» концы также называют 5'- и 3'-гомологичными плечами. Например, некоторые экзогенные донорные последовательности имеют короткие одноцепочечные участки на 5'-конце и/или 3'-конце, которые комплементарны одному или нескольким «липким» концам, созданным путем Cas-белкового расщепления на 5'- и/или 3'-целевых последовательностях в целевом геномном локусе. Некоторые такие экзогенные донорные последовательности имеют комплементарный участок только на 5'-конце или только на 3'-конце. Например, некоторые такие экзогенные донорные последовательности имеют комплементарный участок только на 5'-конце, комплементарный «липкому» концу, созданному на 5' целевой последовательности в геномном локусе, или только на 3'-конце, комплементарный «липкому» концу, созданному на 3' целевой последовательности в целевом геномном локусе. Другие такие экзогенные донорные последовательности имеют комплементарный участок как на 5'-, так и на 3'-концах. Например, другие такие экзогенные донорные последовательности имеют комплементарные участки как на 5'-, так и на 3'-концах, например, комплементарные соответственно первому и второму «липкому» концу, созданному Cas-опосредованным расщеплением в целевом геномном локусе. Например, если экзогенная донорная последовательность является двухцепочечной, одноцепочечные комплементарные участки могут выступать от 5'-конца верхней цепи донорной последовательности и 5'-конца нижней цепи донорной последовательности, создавая 5' «липкие» концы на каждом конце. В качестве альтернативы, одноцепочечный комплементарный участок может выступать от 3'-конца верхней цепи донорной последовательности и от 3'-конца нижней цепи матрицы, создавая 3' «липкие» концы.
[00368] Комплементарные участки могут иметь любую длину, достаточную для стимуляции лигирования между экзогенной донорной последовательностью и геном HSD17B13. Иллюстративные комплементарные участки составляют от приблизительно 1 до приблизительно 5 нуклеотидов в длину, от приблизительно 1 до приблизительно 25 нуклеотидов в длину или от приблизительно 5 до приблизительно 150 нуклеотидов в длину. Например, комплементарный участок может составлять по меньшей мере приблизительно 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 нуклеотидов в длину. В качестве альтернативы, комплементарный участок может составлять от приблизительно 5 до приблизительно 10, от приблизительно 10 до приблизительно 20, от приблизительно 20 до приблизительно 30, от приблизительно 30 до приблизительно 40, от приблизительно 40 до приблизительно 50, от приблизительно 50 до приблизительно 60, от приблизительно 60 до приблизительно 70, от приблизительно 70 до приблизительно 80, от приблизительно 80 до приблизительно 90, от приблизительно 90 до приблизительно 100, от приблизительно 100 до приблизительно 110, от приблизительно 110 до приблизительно 120, от приблизительно 120 до приблизительно 130, от приблизительно 130 до приблизительно 140, от приблизительно 140 до приблизительно 150 нуклеотидов в длину или больше.
[00369] Такие комплементарные участки могут быть комплементарны «липким» концам, образованным двумя парами никаз. Два двухцепочечных разрыва со ступенчатыми концами можно создать с помощью первой и второй никаз, которые расщепляют противоположные цепи ДНК, создавая первый двухцепочечный разрыв, и третьей и четвертой никаз, которые расщепляют противоположные цепи ДНК, создавая второй двухцепочечный разрыв. Например, можно использовать белок Cas для создания одноцепочечного разреза в первой, второй, третьей и четвертой целевой для гидовой РНК последовательности, которые соответствуют первой, второй, третьей и четвертой гидовой РНК. Первую и вторую целевую для гидовой РНК последовательность можно расположить так, чтобы создать первый сайт расщепления с тем, чтобы одноцепочечные разрезы, создаваемые первой и второй никазами на первой и второй цепях ДНК, создавали двухцепочечный разрыв (т.е. первый сайт расщепления содержал одноцепочечные разрезы в первой и второй целевой для гидовой РНК последовательности). Аналогичным образом, третью и четвертую целевую для гидовой РНК последовательность можно расположить так, чтобы создать второй сайт расщепления с тем, чтобы одноцепочечные разрезы, создаваемые третьей и четвертой никазами на первой и второй цепях ДНК, создавали двухцепочечный разрыв (т.е. второй сайт расщепления содержал одноцепочечные разрезы в третьей и четвертой целевой для гидовой РНК последовательности). Предпочтительно, чтобы одноцепочечные разрезы в первой и второй целевой для гидовой РНК последовательности и/или в третьей и четвертой целевой для гидовой РНК последовательности могли быть смещенными одноцепочечными разрезами, которые создают «липкие» концы. Интервал смещения может составлять, например, по меньшей мере приблизительно 5 п.о., 10 п.о., 20 п.о., 30 п.о., 40 п.о., 50 п.о., 60 п.о., 70 п.о., 80 п.о., 90 п.о., 100 п.о. или более. См. Ran et al. (2013) Cell 154:1380-1389; Mali et al. (2013) Nat. Biotech. 31:833-838; и Shen et al. (2014) Nat. Methods 11:399-404, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. В таких случаях двухцепочечную экзогенную донорную последовательность можно сконструировать с одноцепочечными комплементарными участками, которые комплементарны «липким» концам, создаваемым посредством одноцепочечных разрезов в первой и второй целевой для гидовой РНК последовательности и посредством одноцепочечных разрезов в третьей и четвертой целевой для гидовой РНК последовательности. Такую экзогенную донорную последовательность затем можно вставить посредством сшивания, опосредованного негомологичным соединением концов.
(2) Донорные последовательности для вставки путем гомологичной репарации
[00370] Некоторые экзогенные донорные последовательности (т.е. направленные векторы) содержат гомологичные плечи. Если экзогенная донорная последовательность также содержит вставку нуклеиновой кислоты, гомологичные плечи могут фланкировать вставку нуклеиновой кислоты. Для удобства гомологичные плечи в настоящем документе называют 5' и 3' (т.е. вышележащим и нижележащим) гомологичными плечами. Данная терминология обозначает относительное положение гомологичных плеч к вставке нуклеиновой кислоты в экзогенной донорной последовательности. 5' и 3' гомологичные плечи соответствуют участкам в гене HSD17B13, которые в настоящем документе соответственно называют «5' целевой последовательностью» и «3' целевой последовательностью».
[00371] Гомологичное плечо и целевая последовательность «соответствуют» или являются «соответствующими» друг другу, если два участка имеют достаточный уровень идентичности последовательности друг с другом, чтобы играть роль субстратов для реакции гомологичной рекомбинации. Термин «гомология» охватывает последовательности ДНК, которые либо идентичны, либо имеют идентичность последовательности с соответствующей последовательностью. Идентичность последовательности между заданной целевой последовательностью и соответствующим гомологичным плечом, находящимся в экзогенной донорной последовательности, может быть любой степенью идентичности последовательности, которая позволяет происходить гомологичной рекомбинации. Например, величина идентичности последовательности между гомологичным плечом экзогенной донорной последовательности (или ее фрагмента) и целевой последовательностью (или ее фрагментом) может быть по меньшей мере 50%, 55%, 60%, 65%, 70%, 75%, 80%, 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% идентичностью последовательности, такой, чтобы последовательности подвергаются гомологичной рекомбинации. Более того, соответствующий участок гомологии между гомологичным плечом и соответствующей целевой последовательностью может иметь любую длину, которой достаточно для активации гомологичной рекомбинации. Иллюстративные гомологичные плечи составляют от приблизительно 25 нуклеотидов до приблизительно 2,5 т.о. в длину, составляют от приблизительно 25 нуклеотидов до приблизительно 1,5 т.о. в длину или составляют от приблизительно 25 до приблизительно 500 нуклеотидов в длину. Например, заданное гомологичное плечо (или каждое из гомологичных плеч) и/или соответствующая целевая последовательность может содержать соответствующие участки, которые составляют от приблизительно 25 до приблизительно 30, от приблизительно 30 до приблизительно 40, от приблизительно 40 до приблизительно 50, от приблизительно 50 до приблизительно 60, от приблизительно 60 до приблизительно 70, от приблизительно 70 до приблизительно 80, от приблизительно 80 до приблизительно 90, от приблизительно 90 до приблизительно 100, от приблизительно 100 до приблизительно 150, от приблизительно 150 до приблизительно 200, от приблизительно 200 до приблизительно 250, от приблизительно 250 до приблизительно 300, от приблизительно 300 до приблизительно 350, от приблизительно 350 до приблизительно 400, от приблизительно 400 до приблизительно 450 или от приблизительно 450 до приблизительно 500 нуклеотидов в длину так, чтобы гомологичные плечи имели достаточную гомологию для гомологичной рекомбинации с соответствующими целевыми последовательностями в гене HSD17B13. В качестве альтернативы, данное гомологичное плечо (или каждое гомологичное плечо) и/или соответствующая целевая последовательность могут содержать соответствующие участки гомологии, которые составляют от приблизительно 0,5 т.о. до приблизительно 1 т.о., от приблизительно 1 т.о. до приблизительно 1,5 т.о., от приблизительно 1,5 т.о. до приблизительно 2 т.о. или от приблизительно 2 т.о. до приблизительно 2,5 т.о. в длину. Например, каждое из гомологичных плеч может составлять приблизительно 750 нуклеотидов в длину. Гомологичные плечи могут быть симметричными (каждое приблизительно с одним и тем же размером в длину), или они могут быть асимметричными (одно длиннее другого).
[00372] Гомологичные плечи могут соответствовать локусу, который является нативным для клетки (например, целевому локусу). В качестве альтернативы, например, они могут соответствовать участку гетерологичного или экзогенного сегмента ДНК, которая была встроена в геном клетки, включая, например, трансгены, кассеты экспрессии или гетерологичные или экзогенные участки ДНК. В качестве альтернативы, гомологичные плечи направленного вектора могут соответствовать участку искусственной хромосомы дрожжей (YAC), бактериальной искусственной хромосомы (ВАС), искусственной хромосомы человека или любого другого сконструированного участка, содержащегося в соответствующей клетке-хозяине. Кроме того, гомологичные плечи направленного вектора могут соответствовать участку библиотеки ВАС, библиотеки космид или библиотеки фагов Р1 или быть получены из него или могут быть получены из синтетической ДНК.
[00373] При использовании нуклеазного средства в комбинации с экзогенной донорной последовательностью 5' и 3' целевые последовательности предпочтительно являются расположенными в достаточной близости к сайту расщепления нуклеазой с тем, чтобы активировать начало события гомологичной рекомбинации между целевыми последовательностями и гомологичными плечами при одноцепочечном разрыве (одноцепочечном разрезе) или двухцепочечном разрыве в сайте расщепления нуклеазой. Термин «сайт расщепления нуклеазой» охватывает последовательность ДНК, в которой нуклеазным средством создается одноцепочечный или двухцепочечный разрыв (например, белком Cas9 в комплексе с гидовой РНК). Целевые последовательности в гене HSD17B13, которые соответствуют 5' и 3' гомологичным плечам экзогенной донорной последовательности, являются «расположенными в достаточной близости» к сайту расщепления нуклеазой, если расстояние является таким, чтобы активировать начало события гомологичной рекомбинации между 5' и 3' целевыми последовательностями и гомологичными плечами при одноцепочечном разрыве или двухцепочечном разрые в сайте расщепления нуклеазой. Так, целевые последовательности, соответствующие 5' и/или 3' гомологичным плечам экзогенной донорной последовательности, могут располагаться, например, в пределах по меньшей мере 1 нуклеотида от заданного сайта расщепления нуклеазой или в пределах от по меньшей 10 нуклеотидов до приблизительно 1000 нуклеотидов от заданного сайта расщепления нуклеазой. В качестве примера, сайт расщепления нуклеазой может находиться непосредственно рядом по меньшей мере с одной или обеими целевыми последовательностями.
[00374] Пространственное взаимное расположение целевых последовательностей, которые соответствуют гомологичным плечам экзогенной донорной последовательности, и сайта расщепления нуклеазой может варьировать. Например, целевые последовательности могут быть расположены 5' относительно сайта расщепления нуклеазой, целевые последовательности могут быть расположены 3' относительно сайта расщепления нуклеазой, или целевые последовательности могут фланкировать сайт расщепления нуклеазой.
IV. Терапевтические и профилактические применения
[00375] Также настоящее изобретение относится к терапевтическим способам и способам лечения или профилактики хронического заболевания печени у субъекта, имеющего такое заболевание или подверженного риску развития такого заболевания, с помощью раскрываемых в настоящем документе способов модификации или изменения экспрессии эндогенного гена HSD17B13. Также настоящее изобретение относится к терапевтическим способам и способам лечения или профилактики заболевания печени, такого как алкогольная болезнь печени или неалкогольная болезнь печени, у субъекта, имеющего такое заболевание или подверженного риску развития такого заболевания, с помощью раскрываемых в настоящем документе способов модификации или изменения экспрессии эндогенного гена HSD17B13. Также настоящее изобретение относится к терапевтическим способам и способам лечения или профилактики хронического заболевания печени у субъекта, имеющего такое заболевание или подверженного риску развития такого заболевания, с помощью способов уменьшения экспрессии мРНК-транскриптов HSD17B13 или с помощью способов введения рекомбинантных нуклеиновых кислот, кодирующих белки HSD17B13, введения мРНК, кодирующих белки HSD17B13 или введения белков HSD17B13 субъекту. Также настоящее изобретение относится к терапевтическим способам и способам лечения или профилактики заболевания печени, такого как алкогольная болезнь печени или неалкогольная болезнь печени, у субъекта, имеющего такое заболевание или подверженного риску развития такого заболевания, с помощью способов уменьшения экспрессии мРНК-транскриптов HSD17B13 или с помощью способов введения рекомбинантных нуклеиновых кислот, кодирующих белки HSD17B13, введения мРНК, кодирующих белки HSD17B13 или введения белков HSD17B13 субъекту. Способы могут предусматривать введение одной или нескольких нуклеиновых кислот или белков субъекту, в печень субъекта или в клетку (например, клетку печени) субъекта (например, in vivo или ex vivo).
[00376] К хроническим заболеваниям печени относятся заболевания печени, которые длятся на протяжении шести месяцев и могут включать, например, заболевания печени, вовлекающие прогрессирующее разрушение и регенерацию паренхимы печени, что может привести к фиброзу и циррозу. Хроническими заболеваниями печени могут быть алкогольные болезни печени или неалкогольные болезни печени. Патологии печени, охватываемые хроническими заболеваниями печени, могут включать, например, воспаление (например, хронический гепатит), цирроз печени и гепатоклеточную карциному. Типы хронического заболевания печени раскрыты в других разделах настоящего документа и включают, например, жировую болезнь печени, неалкогольную жировую болезнь печени, алкогольную жировую болезнь печени, цирроз и гепатоклеточную карциному. Симптомы и признаки хронических заболеваний печени известны и могут включать в себя, например, увеличенную печень, усталость, боль в верхней правой части живота, вздутие живота (асцит), увеличенные кровеносные сосуды непосредственно под поверхностью кожи, увеличенные груди у мужчин, увеличенная селезенка, красные ладони и пожелтение кожи и глаз (желтуха). Тестирование на хронические заболевания печени может включать анализ крови, визуализацию печени и биопсию печени. Индивидуум подвергается повышенному риску развития хронического заболевания печени, если у субъекта имеется по меньшей мере один известный фактор риска (например, генетический фактор, такой как вызывающая заболевание мутация), ставящий индивидуумов с таким фактором риска на статистически значимый больший риск развития такого заболевания, чем у индивидуумов без фактора риска. Факторы риска для хронических заболеваний печени также хорошо известны и могут включать, например, чрезмерное употребление алкоголя, ожирение, высокий уровень холестерина, высокий уровень триглицеридов в крови, синдром поликистоза яичников, апноэ во сне, сахарный диабет 2-го типа, недостаточную активность щитовидной железы (гипотиреоз), недостаточную активность гипофиза (гипопитуитаризм) и метаболические синдромы, включая повышенные уровни липидов крови.
[00377] Термин «субъект» включает человека и других млекопитающих (например, кошек, собак, грызунов, мышей или крыс) или отличных от млекопитающих субъектов (например, домашнюю птицу), которые получают профилактическое или терапевтическое лечение. Такие субъекты могут представлять собой, например, субъекта (например, человека), который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития. Возможны различные способы детекции наличия варианта rs72613567 HSD17B13 в биологическом образце, содержащем геномную ДНК, детекции наличия или уровней любого из или комбинации транскриптов С, D, Е, F, F', G и Н, и особенно D, HSD17B13 в биологическом образце, содержащем мРНК или кДНК, или детекции наличия или уровней любой из или комбинации изоформ С, D, Е, F, F', G или Н, и особенно D, белка HSD17B13 в биологическом образце, содержащем белок. Способы детекции наличия последовательности в геномной ДНК и детекции наличия конкретного мРНК-транскрипта или конкретной изоформы белка хорошо известны. Понятно, что последовательности генов в популяции и мРНК и белки, кодируемые такими генами, могут варьировать по причине полиморфизмов, таких как однонуклеотидные полиморфизмы. Представленные в настоящем документе последовательности гена HSD17B13 и каждого из транскрипта HSD17B13 и изоформы HSD17B13 являются лишь иллюстративными последовательностями гена HSD17B13 и каждого из транскрипта HSD17B13 и изоформы HSD17B13. Также возможны и другие последовательности гена HSD17В13 и каждого из транскрипта HSD17В13 и изоформы HSD17B13.
[00378] Например, способ детекции варианта rs72613567 HSD17B13 в клетке или у субъекта, такого как субъект-человек, может предусматривать, например, получение биологического образца от субъекта, содержащего ген HSD17B13, и проведение анализа на биологическом образце, с помощью которого определяют, что положение гена HSD17B13, соответствующее положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 и SEQ ID NO: 2, занято тимином или что тимин вставлен между положениями, соответствующими положениям 12665 и 12666 при оптимальном выравнивании гена HSD17B13 и SEQ ID NO: 1. Понятно, что определение того, что положение гена HSD17B13, соответствующее положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 и SEQ ID NO: 2, занято тимином, означает, что определяют идентичность достаточного количества нуклеотидов в положениях, фланкирующих положения, соответствующие положениям 12665 и 12666 из SEQ ID NO: 1, чтобы можно было определить, что тимин вставлен между положениями, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1. Такие анализы могут предусматривать, например, определение идентичности положений гена HSD17B13, соответствующих положению 12666 из SEQ ID NO: 2 (или положениям 12665 и 12666 из SEQ ID NO: 1) и одному или нескольким окружающим положениям (например, по меньшей мере 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10 положениям, фланкирующим одну сторону или каждую сторону положения 12666 из SEQ ID NO: 2 или положений 12665 и 12666 из SEQ ID NO: 1) при оптимальном выравнивании гена HSD17B13 и SEQ ID NO: 2 (или SEQ ID NO: 1). Данный анализ в таком способе может предусматривать, например, секвенирование части гена HSD17B13, включающей положение, соответствующее положению 12666 или положениям 12666 и 12667 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 и SEQ ID NO: 2. Аналогичным образом, данный анализ может предусматривать секвенирование части гена HSD17B13, включающей положения, соответствующее положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 и SEQ ID NO: 1. В качестве альтернативы, данный анализ в таком способе может предусматривать приведение в контакт биологического образца с праймером или зондом, который специфично гибридизируется с вариантом rs72613567 HSD17B13 и не соответствует последовательности HSD17B13 дикого типа (например, в жестких условиях), и определение, произошла ли гибридизация.
[00379] Такие способы могут предусматривать редактирование генома или генную терапию. Например, эндогенный ген HSD17B13, который не является вариантом rs72613567 HSD17B13, можно модифицировать с включением варианта, ассоциированного с вариантом rs72613567 HSD17B13 (т.е. вставкой тимина между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1, или вставкой аденина в соответствующее положение на противоположной цепи). В качестве другого примера, эндогенный ген HSD17B13, который не является вариантом rs72613567 HSD17В13, можно подвергнуть нокауту или инактивировать. Аналогичным образом, эндогенный ген HSD17B13, который не является вариантом rs72613567 HSD17B13, можно подвергнуть нокауту или инактивировать и можно ввести и экспрессировать ген HSD17B13, содержащий модификацию, ассоциированную с вариантом rs72613567 HSD17B13 (например, полный вариант rs72613567 HSD17B13 или миниген, содержащий такую модификацию). Аналогично, эндогенный ген HSD17B13, который не является вариантом rs72613567 HSD17B13, можно подвергнуть нокауту или инактивировать и можно ввести и экспрессировать рекомбинантную ДНК, кодирующую любую из или любую комбинацию из изоформ С, D, F, G и Н (или их фрагментов) HSD17B13, можно ввести и экспрессировать мРНК, кодирующую любую из или любую комбинацию из изоформ С, D, F, G и Н (или их фрагментов) HSD17B13 (например, средство для внутриклеточной белок-заместительной терапии), или можно ввести любую из или любую комбинацию из изоформ С, D, F, G и Н (или их фрагментов) HSD17B13 (например, средство для белок-заместительной терапии). В соответствии с конкретными вариантами осуществления, комбинация изоформ HSD17B13 (или ДНК или мРНК, которые их кодируют) представляет собой комбинацию, включающую изоформу D HSD17B13 (например, D, DC, DF, DG, DH, DCF, DCG, DCH, DFG, DFH, DGH, DCFG, DCFH, DCGH, DFGH или DCFGH).
[00380] Другие такие способы могут предусматривать введение и экспрессию рекомбинантного гена HSD17В13, содержащего модификацию, ассоциированную с вариантом rs72613567 HSD17B13 (например, полного варианта HSD17B13 rs72613567 или минигена, содержащего такую модификацию), введение и экспрессию рекомбинантных нуклеиновых кислот (например, ДНК), кодирующих любую из или любую комбинацию из изоформ С, D, F, G и Н HSD17B13 или их фрагментов, введение и экспрессию одной или нескольких мРНК, кодирующих любую из или любую комбинацию из изоформ С, D, F, G и Н HSD17B13 или их фрагментов (например, средства для внутриклеточной белок-заместительной терапии), или введение любой из или любой комбинации из изоформ С, D, F, G и Н HSD17B13 или их фрагментов (например, средства для внутриклеточной белок-заместительной терапии) без нокаута или инактивации эндогенного гена HSD17B13, который не является вариантом rs72613567 HSD17B13. В соответствии с конкретными вариантами осуществления, комбинация изоформ HSD17B13 (или ДНК или мРНК, которые их кодируют) представляет собой комбинацию, включающую изоформу D HSD17B13 (например, D, DC, DF, DG, DH, DCF, DCG, DCH, DFG, DFH, DGH, DCFG, DCFH, DCGH, DFGH или DCFGH). Необязательно, такие способы также можно осуществлять в сочетании со способами, в которых на транскрипт HSD17B13, экспрессия которого уменьшается у носителей варианта rs72613567 HSD17B13 (например, транскрипты А, В, Е и F'), оказывают целенаправленное воздействие для снижения экспрессии, например, посредством применения антисмысловой РНК, siRNA или shRNA. В соответствии с конкретными вариантами осуществления, транскрипты HSD17B13, на которые оказывают целенаправленное воздействие для снижения экспрессии, являются комбинацией, включающей транскрипт А (например, А, АВ, АЕ, AF', ABE, ABF', AEF' или ABEF').
[00381] Ген HSD17 В13 или миниген или ДНК, кодирующие любую из или любую комбинацию из изоформ С, D, F, G и Н HSD17B13 или их фрагментов, можно ввести и экспрессировать в форме вектора экспрессии, который не модифицирует геном, его можно ввести в форме направленного вектора так, чтобы он встроился в геноме в локус HSD17B13, или его можно ввести так, чтобы он встроился в геноме в локус, отличный от локуса HSD17B13, такой как локус «безопасной гавани». Встроенный в геном ген HSD17B13 может быть функционально связан с промотором HSD17B13 или с другим промотором, таким как эндогенный промотор в сайте встраивания. Локусы «безопасной гавани» являются хромосомными сайтами, где трансгены могут стабильно и надежно экспрессироваться во всех представляющих интерес тканях без неблагоприятного влияния на структуру или экспрессию генов. Локусы «безопасной гавани» могут иметь, например, одну или несколько из или все следующие характеристики: (1) расстояние более 50 т.о. от 5'-конца любого гена; расстояние более 300 т.о. от любого связанного с раком гена; расстояние более 300 т.о. от любой микроРНК; быть за пределами транскрипционной единицы гена и за пределами ультраконсервативных участков. К примерам подходящих локусов «безопасной гавани» относятся сайт 1 аденоассоциированный вируса (AAVS1), локус гена хемокинового (мотив СС) рецептора 5 (CCR5) и локус человеческого ортолога ROSA26 мыши.
[00382] Комбинации изоформ белка HSD17B13 или нуклеиновых кислот, кодирующих изоформы белка HSD17B13, которые можно вводить и экспрессировать, включают, например, С, D, F, G, Н, CD, CF, CG, СН, DF, DG, DH, FG, FH, GH, CDF, CDG, CDH, CFG, CFH, CGH, DFG, DFH, DGH, FGH, CDFG, CDFH, CFGH, DFGH и CDFGH. В конкретных способах вводят или экспрессируют изоформу D HSD17B13 или нуклеиновую кислоту, кодирующую изоформу D (отдельно или в комбинации с другими изоформами). Иллюстративные последовательности для каждой из этих изоформ и транскриптов приведены в других разделах настоящего документа. Тем не менее, понятно, что последовательности генов и в популяции, последовательности мРНК, транскрибируемые с таких генов, и белки, транслируемые с таких мРНК, могут варьировать по причине полиморфизмов, таких как однонуклеотидные полиморфизмы. Представленные в настоящем документе последовательности для каждого транскрипта и изоформы являются лишь иллюстративными последовательностями. Также возможны и другие последовательности.
[00383] Комбинации транскриптов HSD17B13, на экспрессию которых можно оказывать целенаправленное воздействие с целью ее снижения посредством антисмысловой РНК, shRNA или siRNA, включают, например, А, В, Е, F', АВ, АЕ, AF', BE, BF', ABE, ABF', AEF', BEF' и ABEF'. В конкретных способах целенаправленное воздействие оказывают на транскрипт A HSD17B13 (отдельно или в комбинации с другими транскриптами). Например, антисмысловая РНК, siRNA или shRNA может гибридизироваться с последовательностью в SEQ ID NO: 4 (транскрипте A HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 4 (транскрипте A HSD17B13) и отсутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 4 (транскрипта A HSD17B13).
[00384] Например, некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту или введение в клетку печени субъекта: (а) нуклеазного средства (или нуклеиновой кислоты, которая его кодирует), которое связывается с целевой для нуклеазы последовательностью в гене HSD17B13, причем целевая для нуклеазы последовательность включает положение или находится в непосредственной близости от него, которое соответствует положению 12666 из SEQ ID NO: 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 2; и (b) экзогенной донорной последовательности, содержащей 5' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 5' от положения, соответствующего положению 12666 из SEQ ID NO: 2, 3' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 3' от положения, соответствующего положению 12666 из SEQ ID NO: 2, и содержащую тимин вставку нуклеиновой кислоты, фланкированную 5' гомологичным плечом и 3' гомологичным плечом. Нуклеазное средство может расщеплять ген HSD17B13 в клетке печени у субъекта, а экзогенная донорная последовательность может подвергаться рекомбинации с геном HSD17B13 в клетке печени, причем при рекомбинации экзогенной донорной последовательности с геном HSD17B13 тимин вставляется между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1. Примеры нуклеазных средств (например, белка Cas9 и гидовой РНК), которые можно применять в таких способах, раскрыты в других разделах настоящего документа. Примеры подходящих гидовых РНК и целевых для гидовой РНК последовательностей раскрыты в других разделах настоящего документа. Примеры экзогенных донорных последовательностей, которые можно применять в таких способах, раскрыты в других разделах настоящего документа.
[00385] В качестве другого примера, некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту или введение в клетку печени субъекта экзогенной донорной последовательности, содержащей 5' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 5' от положения, соответствующего положению 12666 из SEQ ID NO: 2, 3' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 3' от положения, соответствующего положению 12666 из SEQ ID NO: 2, и содержащую тимин вставку нуклеиновой кислоты, фланкированную 5' гомологичным плечом и 3' гомологичным плечом. Экзогенная донорная последовательность может подвергаться рекомбинации с геном HSD17B13 в клетке печени, причем при рекомбинации экзогенной донорной последовательности с геном HSD17B13 тимин вставляется между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1. Примеры экзогенных донорных последовательностей, которые можно применять в таких способах, раскрыты в других разделах настоящего документа.
[00386] Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту или введение в клетку печени субъекта: (а) нуклеазного средства (или нуклеиновой кислоты, которая его кодирует), которое связывается с целевой для нуклеазы последовательностью в гене HSD17B13, причем целевая для нуклеазы последовательность содержит старт-кодон для гена HSD17B13 или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона или выбрана из SEQ ID NO: 20-81. Нуклеазное средство может расщеплять ген HSD17B13 и нарушать его экспрессию в клетке печени у субъекта. Некоторые такие способы включают способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту или введение в клетку печени субъекта: (а) нуклеазного средства (или нуклеиновой кислоты, которая его кодирует), которое связывается с целевой для нуклеазы последовательностью в гене HSD17B13, причем целевая для нуклеазы последовательность содержит старт-кодон для гена HSD17B13 или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона или выбрана из SEQ ID NO: 20-81; и (b) вектора экспрессии, содержащего рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. Вектор экспрессии может быть вектором, который не встраивается в геном. В качестве альтернативы, можно ввести направленный вектор (т.е. экзогенную донорную последовательность), содержащий рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. Нуклеазное средство может расщеплять ген HSD17B13 и нарушать его экспрессию в клетке печени у субъекта, а вектор экспрессии может экспрессировать рекомбинантный ген HSD17B13 в клетке печени у субъекта. В качестве альтернативы, в интегрированном в геном состоянии рекомбинантный ген HSD17B13 может экспрессироваться в клетке печени у субъекта. Примеры нуклеазных средств (например, обладающего нуклеазной активностью белка Cas9 и гидовой РНК), которые можно применять в таких способах, раскрыты в других разделах настоящего документа. Примеры подходящих гидовых РНК и целевых для гидовой РНК последовательностей раскрыты в других разделах настоящего документа. Стадия (b) альтернативно может предусматривать введение вектора экспрессии или направленного вектора, содержащего нуклеиновую кислоту (например, ДНК), кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен изоформе С, D, F, G или Н HSD17B13 или ее фрагменту, и/или содержащего последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична транскрипту С, D, F, G или Н HSD17B13 или его фрагменту. Аналогичным образом, стадия (b) альтернативно может предусматривать введение мРНК, кодирующей белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен изоформе С, D, F, G или Н HSD17B13 или ее фрагменту, и/или обладающей комплементарностью к ДНК (или ее части), которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична транскрипту С, D, F, G или Н HSD17B13 или его фрагменту. Аналогичным образом, стадия (b) альтернативно может предусматривать введение белка, содержащего последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична изоформе С, D, F, G или Н HSD17B13 или ее фрагменту. В конкретных способах транскрипт может представлять собой транскрипт D HSD17B13 (например, SEQ ID NO: 7), или изоформа может представлять собой изоформу D HSD17B13 (например, SEQ ID NO: 15). В других конкретных способах можно вводить комбинацию изоформ HSD17B13 или векторов экспрессии или направленных векторов, кодирующих комбинацию изоформ HSD17B13, или мРНК, кодирующих комбинацию изоформ HSD17B13 (например, D, DC, DF, DG, DH, DCF, DCG, DCH, DFG, DFH, DGH, DCFG, DCFH, DCGH, DFGH или DCFGH).
[00387] В некоторых таких способах также вводят второе нуклеазное средство субъекту или в клетку печени субъекта, причем второе нуклеазное средство связывается со второй целевой для нуклеазы последовательностью в гене HSD17B13, причем вторая целевая для нуклеазы последовательность содержит стоп-кодон для гена HSD17B13, или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от стоп-кодона, или выбрана из SEQ ID NO: 82-225, причем нуклеазное средство расщепляет ген HSD17B13 в клетке печени как в первой целевой для нуклеазы последовательности, так и во второй целевой для нуклеазы последовательности, при этом клетка печени модифицируется так, чтобы у нее была делеция между первой целевой для нуклеазы последовательностью и второй целевой для нуклеазы последовательностью. Например, второе нуклеазное средство может представлять собой белок Cas9 и гидовую РНК. Подходящие гидовые РНК и целевые для гидовой РНК последовательности, расположенные вблизи стоп-кодона, раскрыты в других разделах настоящего документа.
[00388] Такие способы также могут включать способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту или введение в клетку печени субъекта: (а) ДНК-связывающего белка (или нуклеиновой кислоты, которая его кодирует), который связывается с целевой для ДНК-связывающего белка последовательностью в гене HSD17B13, причем целевая для ДНК-связывающего белка последовательность содержит старт-кодон для гена HSD17B13 или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона или выбрана из SEQ ID NO: 20-81. ДНК-связывающий белок может изменять (например, понижать) экспрессию гена HSD17B13 в клетке печени у субъекта. Такие способы также могут включать способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту или введение в клетку печени субъекта: (а) ДНК-связывающего белка (или нуклеиновой кислоты, которая его кодирует), который связывается с целевой для ДНК-связывающего белка последовательностью в гене HSD17B13, причем целевая для ДНК-связывающего белка последовательность содержит старт-кодон для гена HSD17B13 или находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона или выбрана из SEQ ID NO: 20-81; и (b) вектора экспрессии, содержащего рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. Вектор экспрессии может быть вектором, который не встраивается в геном. В качестве альтернативы, можно ввести направленный вектор (т.е. экзогенную донорную последовательность), содержащий рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. ДНК-связывающий белок может изменять (например, понижать) экспрессию гена HSD17B13 в клетке печени у субъекта, а вектор экспрессии может экспрессировать рекомбинантный ген HSD17B13 в клетке печени у субъекта. В качестве альтернативы, в интегрированном в геном состоянии рекомбинантный ген HSD17B13 может экспрессироваться в клетке печени у субъекта. Примеры ДНК-связывающих белков, подходящих для применения в таких способах, раскрыты в других разделах настоящего документа. Такие ДНК-связывающие белки (например, белок Cas9 и гидовая РНК) могут быть слиты или функционально связаны с доменом-репрессором транскрипции. Например, ДНК-связывающий белок может быть каталитически неактивным белком Cas9, слитым с доменом-репрессором транскрипции. Такой ДНК-связывающий белок, слитый с доменом-репрессором транскрипции, можно применять, например, для уменьшения экспрессии гена HSD17B13 дикого типа или гена HSD17B13, который не является вариантом rs72613567 (например, для снижения экспрессии транскрипта или изоформы A HSD17B13). Примеры подходящих гидовых РНК и целевых для гидовой РНК последовательностей раскрыты в других разделах настоящего документа. Стадия (b) альтернативно может предусматривать введение вектора экспрессии или направленного вектора, содержащего нуклеиновую кислоту (например, ДНК), кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен изоформе С, D, F, G или Н HSD17B13 или ее фрагменту, и/или содержащего последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична транскрипту С, D, F, G или Н HSD17B13 или его фрагменту. Аналогичным образом, стадия (b) альтернативно может предусматривать введение мРНК, кодирующей белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен изоформе С, D, F, G или Н HSD17B13 или ее фрагменту, и/или обладающей комплементарностью к ДНК (или ее части), которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична транскрипту С, D, F, G или Н HSD17B13 или его фрагменту. Аналогичным образом, стадия (b) альтернативно может предусматривать введение белка, содержащего последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична изоформе С, D, F, G или Н HSD17B13 или ее фрагменту. В конкретных способах транскрипт может представлять собой транскрипт D HSD17B13 (например, SEQ ID NO: 7), или изоформа может представлять собой изоформу D HSD17B13 (например, SEQ ID NO: 15). В других конкретных способах можно вводить комбинацию изоформ HSD17B13 или векторов экспрессии или направленных векторов, кодирующих комбинацию изоформ HSD17B13, или мРНК, кодирующих комбинацию изоформ HSD17B13 (например, D, DC, DF, DG, DH, DCF, DCG, DCH, DFG, DFH, DGH, DCFG, DCFH, DCGH, DFGH или DCFGH).
[00389] Такие способы также могут включать способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту или введение в клетку печени субъекта антисмысловой РНК, siRNA или shRNA, которая гибридизируется с последовательностью в пределах участка одного или нескольких из транскриптов А, В, Е и F' (и особенно A) HSD17B13, который необязательно отсутствует в одном или нескольких транскриптах С, D, F, G и Н (и особенно D) HSD17B13. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в SEQ ID NO: 4 (транскрипте А HSD17B13), и антисмысловая РНК, siRNA или shRNA может уменьшать экспрессию транскрипта A HSD17B13 в клетке. Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью, присутствующей в SEQ ID NO: 4 (транскрипте А HSD17B13) и отсутствующей в SEQ ID NO: 7 (транскрипте D HSD17B13). Необязательно, антисмысловая РНК, siRNA или shRNA гибридизируется с последовательностью в пределах экзона 7 или последовательностью, охватывающей границу между экзоном 6 и экзоном 7 из SEQ ID NO: 4 (транскрипта A HSD17B13). Например, антисмысловая РНК, siRNA или shRNA может гибридизироваться с последовательностью в пределах участка в экзоне 7 или участка, охватывающего границу между экзоном 6 и экзоном 7 из SEQ ID NO: 4 (транскрипта А HSD17B13), и уменьшать экспрессию транскрипта A HSD17B13 в клетке печени у субъекта. Необязательно, такие способы могут дополнительно предусматривать введение субъекту вектора экспрессии, содержащего рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. Вектор экспрессии может быть вектором, который не встраивается в геном. В качестве альтернативы, можно ввести направленный вектор (т.е. экзогенную донорную последовательность), содержащий рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. В способах, предусматривающих применение вектора экспрессии, вектор экспрессии может экспрессировать рекомбинантный ген HSD17B13 в клетке печени у субъекта. В качестве альтернативы, в способах, в которых рекомбинантный ген HSD17B13 встроен в геном, рекомбинантный ген HSD17B13 может экспрессироваться в клетке печени у субъекта. Такие способы альтернативно могут предусматривать введение вектора экспрессии или направленного вектора, содержащего нуклеиновую кислоту (например, ДНК), кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен изоформе С, D, F, G или Н HSD17B13 или ее фрагменту, и/или содержащего последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична транскрипту С, D, F, G или Н HSD17B13 или его фрагменту. Аналогичным образом, такие способы альтернативно могут предусматривать введение мРНК, кодирующей белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен изоформе С, D, F, G или Н HSD17B13 или ее фрагменту, и/или обладающей комплементарностью к ДНК (или ее части), которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична транскрипту С, D, F, G или Н HSD17B13 или его фрагменту. Аналогичным образом, такие способы альтернативно могут предусматривать введение белка, содержащего последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична изоформе С, D, F, G или Н HSD17B13 или ее фрагменту. В конкретных способах транскрипт может представлять собой транскрипт D HSD17B13 (например, SEQ ID NO: 7), или изоформа может представлять собой изоформу D HSD17B13 (например, SEQ ID NO: 15). В других конкретных способах можно вводить комбинацию изоформ HSD17B13 или векторов экспрессии или направленных векторов, кодирующих комбинацию изоформ HSD17B13, или мРНК, кодирующих комбинацию изоформ HSD17B13 (например, D, DC, DF, DG, DH, DCF, DCG, DCH, DFG, DFH, DGH, DCFG, DCFH, DCGH, DFGH или DCFGH).
[00390] Другие такие способы могут включать способ лечения субъекта, который не является носителем варианта rs72613567 HSD17B13 (или является лишь гетерозиготным носителем варианта rs72613567 HSD17B13) и имеет хроническое заболевание печени или подвержен риску его развития, предусматривающий введение субъекту или введение в клетку печени субъекта вектора экспрессии, причем вектор экспрессии содержит рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1, причем вектор экспрессии экспрессирует рекомбинантный ген HSD17B13 в клетке печени у субъекта. Вектор экспрессии может быть вектором, который не встраивается в геном. В качестве альтернативы, можно ввести направленный вектор (т.е. экзогенную донорную последовательность), содержащий рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1. В способах, предусматривающих применение вектора экспрессии, вектор экспрессии может экспрессировать рекомбинантный ген HSD17B13 в клетке печени у субъекта. В качестве альтернативы, в способах, в которых рекомбинантный ген HSD17B13 встроен в геном, рекомбинантный ген HSD17B13 может экспрессироваться в клетке печени у субъекта. Такие способы альтернативно могут предусматривать введение вектора экспрессии или направленного вектора, содержащего нуклеиновую кислоту (например, ДНК), кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен изоформе С, D, F, G или Н HSD17B13 или ее фрагменту, и/или содержащего последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична транскрипту С, D, F, G или Н HSD17B13 или его фрагменту. Аналогичным образом, такие способы альтернативно могут предусматривать введение мРНК, кодирующей белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен изоформе С, D, F, G или Н HSD17B13 или ее фрагменту, и/или обладающей комплементарностью к ДНК (или ее части), которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична транскрипту С, D, F, G или Н HSD17B13 или его фрагменту. Аналогичным образом, такие способы альтернативно могут предусматривать введение белка, содержащего последовательность, которая по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична изоформе С, D, F, G или Н HSD17B13 или ее фрагменту. В конкретных способах транскрипт может представлять собой транскрипт D HSD17B13 (например, SEQ ID NO: 7), или изоформа может представлять собой изоформу D HSD17B13 (например, SEQ ID NO: 15). В других конкретных способах можно вводить комбинацию изоформ HSD17B13 или векторов экспрессии или направленных векторов, кодирующих комбинацию изоформ HSD17B13, или мРНК, кодирующих комбинацию изоформ HSD17B13 (например, D, DC, DF, DG, DH, DCF, DCG, DCH, DFG, DFH, DGH, DCFG, DCFH, DCGH, DFGH или DCFGH).
[00391] Подходящие векторы экспрессии и рекомбинантные гены HSD17B13 для применения в любом из вышеуказанных способов раскрыты в других разделах настоящего документа. Например, рекомбинантный ген HSD17B13 может быть полным вариантом rs72613567 гена или может быть минигеном HSD17B13, в котором были делетерованы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа. В качестве примера, делетерованные сегменты могут содержать одну или несколько интронных последовательностей, а миниген может содержать интрон, соответствующий интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2. Примером полного варианта rs72613567 гена является такой, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичен SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2.
[00392] Некоторые такие способы включают способ модификации клетки (например, клетки печени) у субъекта, имеющего хроническое заболевание печени или подверженного риску его развития. В соответствии с такими способами, нуклеазные средства, и/или экзогенные донорные последовательности, и/или рекомбинантные векторы экспрессии можно вводить в клетку путем введения в эффективном режиме, означающем дозу, путь введения и частоту введения, который отсрочивает начало заболевания, уменьшает степень тяжести, ингибирует дальнейшее ухудшение и/или облегчает по меньшей мере один признак или симптом подвергаемого лечению хронического заболевания печени. Термин «симптом» обозначает субъективные свидетельства заболевания, воспринимаемые субъектом, а «признак» обозначает объективные свидетельства заболевания, наблюдаемые врачом. Если субъект уже страдает от заболевания, этот режим можно назвать терапевтически эффективным режимом. Если субъект подвергается повышенному риску развития заболевания относительно генеральной совокупности, но еще не испытывает симптомов, этот режим можно назвать профилактически эффективным режимом. В некоторых случаях терапевтическую или профилактическую эффективность можно наблюдать у индивидуального пациента в сравнении с историческими контролями или прошлым опытом у того же субъекта. В других случаях терапевтическую или профилактическую эффективность можно продемонстрировать в доклинических или клинических испытаниях у совокупности подвергаемых лечению субъектов относительно контрольной совокупности не подвергаемых лечению субъектов.
[00393] Доставку можно осуществлять любым подходящим способом, как раскрыто в других разделах настоящего документа. Например, нуклеазные средства, или экзогенные донорные последовательности, или рекомбинантные векторы экспрессии можно доставлять посредством векторной доставки, вирусной доставки, опосредованной частицами доставки, опосредованной наночастицами доставки, опосредованной липосомами доставки, опосредованной экзосомами доставки, опосредованной липидами доставки, опосредованной липидными наночастицами доставки, доставки, опосредованной проникающими пептидами, или доставки, опосредованной имплантируемым устройством. К некоторым конкретным примерам относятся гидродинамическая доставка, вирусная доставка и опосредованная липидными наночастицами доставка.
[00394] Введение можно осуществлять любым подходящим путем, включая, например, парентеральный, внутривенный, пероральный, подкожный, внутриартериальный, внутричерепной, интратекальный, внутрибрюшинный, местный, интраназальный или внутримышечный. Конкретным примером, который часто используют, например, для белок-заместительных форм терапии, является внутривенная инфузия. Частота введения и количество доз могут зависеть, среди прочих факторов, от периода полужизни нуклеазных средств, или экзогенных донорорных последовательностей, или рекомбинантных векторов экспрессии, общего состояния субъекта и пути введения. Фармацевтические композиции для введения предпочтительно являются стерильными и практически изотоническими и изготовленными в условиях GMP. Фармацевтические композиции могут быть предоставлены в стандартной лекарственной форме (т.е. с дозой для однократного введения). Фармацевтические композиции могут быть составлены с применением одного или нескольких физиологически и фармацевтически приемлемых носителей, разбавителей, наполнителей или вспомогательных веществ. Состав зависит от выбранного пути введения. Термин «фармацевтически приемлемый» означает, что носитель, разбавитель, наполнитель или вспомогательное вещество совместимы с другими ингредиентами состава и практически безвредны для принимающего их пациента.
[00395] Другие такие способы включают способ, осуществляемый ex vivo в клетке от субъекта, имеющего хроническое заболевание печени или подверженного риску его развития. Затем клетку с целевой генетической модификацией можно трансплантировать обратно субъекту.
[00396] Любой из раскрытых в настоящем документе терапевтических или профилактических способов может дополнительно предусматривать введение терапевтического средства, предназначенного для предупреждения или ослабления одного или нескольких симптомов, ассоциированных с прогрессированием до более клинически запущенных стадий хронического заболевания печени (например, с прогрессированием от простого стеатоза до более клинически запущенных стадий хронического заболевания печени или с прогрессированием от простого стеатоза до одного или нескольких из стеатогепатита, фиброза, цирроза и гепатоклеточной карциномы). Например, такие способы лечения могут быть направлены на предупреждение или уменьшение воспаления или предупреждение или уменьшение фиброза. Ниже приведены примеры таких разрабатываемых терапевтических средств.
[00397] Все упомянутые выше или ниже патентные заявки, веб-сайты, другие публикации, регистрационные номера и тому подобное включены посредством ссылки во всей своей полноте для всех целей в той же степени, как если бы каждый из них в отдельности был специально и индивидуально указан как включенный посредством ссылки. Если с регистрационным номером в разные моменты времени ассоциированы разные версии последовательности, подразумевают версию, ассоциированную с регистрационным номером на дату подачи настоящей заявки. Действительная дата подачи означает более раннюю от фактической даты подачи или дату подачи приоритетной заявки со ссылкой на регистрационный номер, если это необходимо. Аналогичным образом, если в разные моменты времени опубликованы различные версии публикации, веб-сайта или тому подобного, подразумевают последнюю версию, опубликованную на действительную дату подачи заявки, если не указано иное. Любые признак, стадию, элемент, вариант осуществления или аспект настоящего изобретения можно использовать в сочетании с любым другим, если специально не указано иное. Несмотря на то, что настоящее изобретение для ясности и понимания было подробно описано посредством иллюстрации и примера, будет очевидно, что в объем пунктов прилагаемой формулы изобретения можно внести определенные изменения и модификации.
Краткое описание последовательностей
[00398] Нуклеотидные и аминокислотные последовательности, перечисленные в прилагаемом перечне последовательностей, показаны с помощью стандартных буквенных сокращений для нуклеотидных оснований и трехбуквенного кода для аминокислот. Нуклеотидные последовательности следуют стандартному условию, начинаясь с 5'-конца последовательности и далее (т.е. слева направо в каждой строке) до 3'-конца. Показана лишь одна цепь каждой нуклеотидной последовательности, но при упоминании отображаемой цепи считается, что включена и комплементарная цепь. Аминокислотные последовательности следуют стандартному условию, начинаясь с аминоконца последовательности и далее (т.е. слева направо в каждой строке) до карбоксиконца.
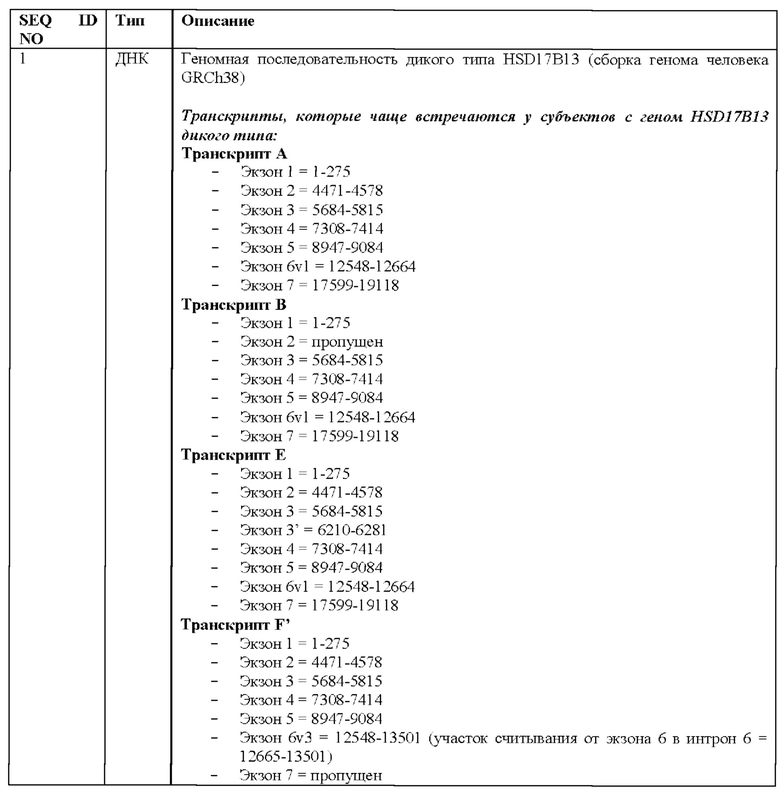
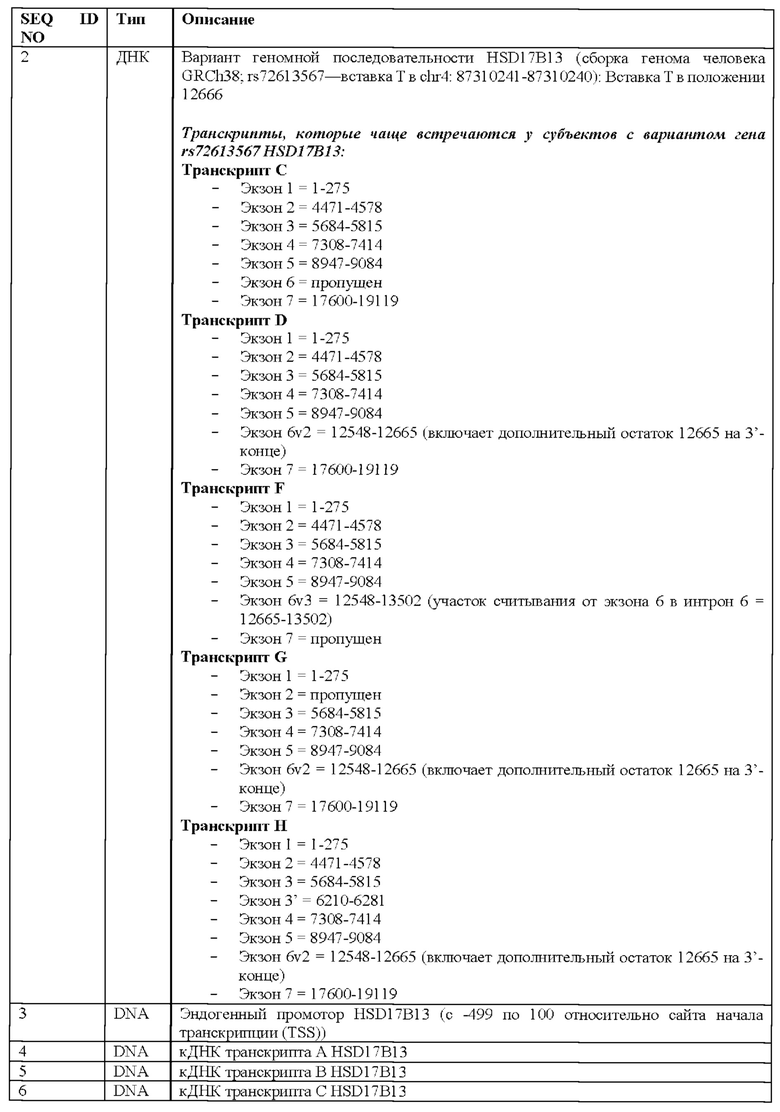
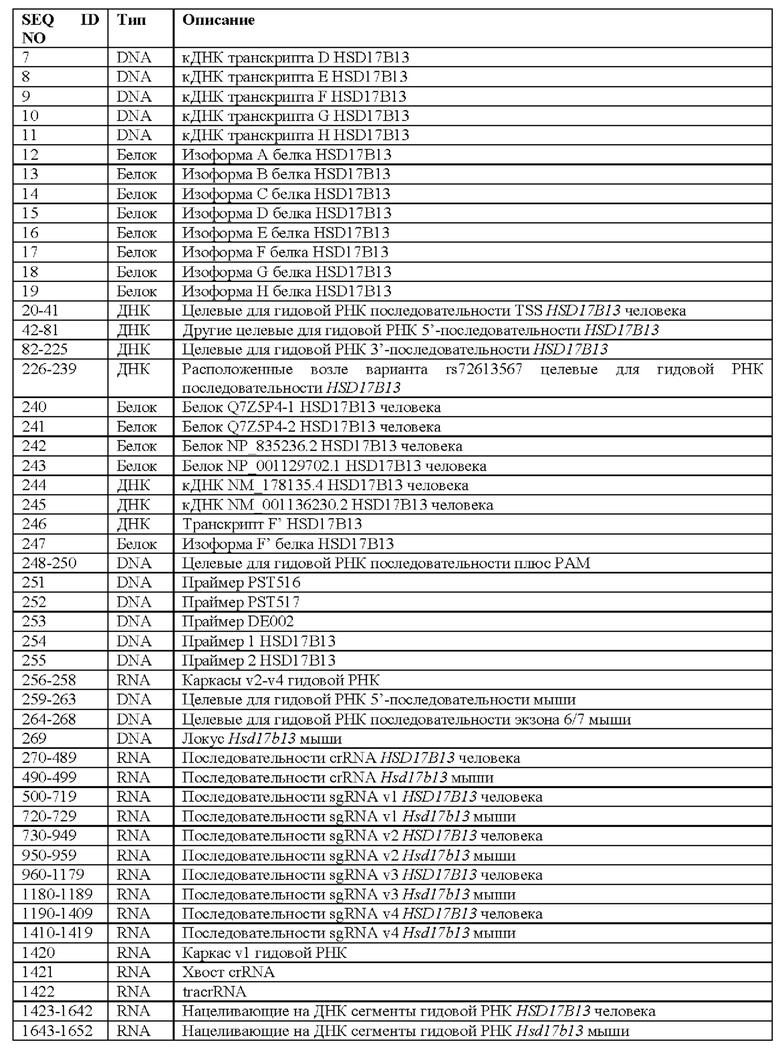
Примеры
Пример 1 Вариант 17бета-гидроксистероиддегидрогеназы 13 защищает от хронического заболевания печени
[00399] Хроническое заболевание печени и цирроз являются основными причинами заболеваемости и смертности в США (Kochanek et al. (2016) Natl Vital Stat Rep 65:1-122, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Наиболее распространенной этиологией цирроза являются алкогольная болезнь печени, хронический гепатит С и неалкогольная жировая болезнь печени (NAFLD), на долю которых приходится около ~80% пациентов, ожидающих трансплантации печени (Wong et al. (2015) Gastroenterology 148:547-555, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Следует отметить, что оценочная доля NAFLD в США составляет от 19 до 46 процентов (Browning et al. (2004) Hepatology 40:1387-1395; Lazo et al. (2013) Am J Epidemiol 178:38-45; и Williams et al. (2011) Gastroenterology 140:124-131, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) и растет со временем (Younossi et al. (2011) Clin Gastroenterol Hepatol 9:524-530 e1; quiz e60 (2011), включена в настоящий документ посредством в полном ее объеме для всех целей), вероятно, в сочетании с повышенным уровнем ожирения. На сегодняшний день остается большая неопределенность в отношении межиндивидуальной вариабельности в прогрессировании NAFLD и результатах ее лечения; информация об основных генетических факторах может облегчить выделение групп риска и обеспечить основу для создания новых стратегий терапии. В данном примере было показано, что носители сплайс-варианта в HSD17B13 (кодирующем гидроксистероид-17-бета-дегидрогеназу 13) снижают риск алкогольной и неалкогольной болезни печени и снижают риск прогрессирования NAFLD. В результате поисков ассоциаций в данных по всей последовательности экзома в связи с электронными медицинскими картами от 46544 участников европейского происхождения в исследовании DiscovEHR был выявлен сплайс-вариант в HSD17B13 (rs72613567), ассоциируемый со сниженными уровнями аланинтрансаминазы и аспартаттрансаминазы; эти результаты были воспроизведены на трех отдельных группах, включавших 12528 индивидуумов. В группе исследования вариант HSD17B13 был связан со сниженным риском развития алкогольной и неалкогольной болезни печени, цирроза и гепатоклеточной карциномы. В группе бариатрической хирургии вариант был связан с уменьшенным риском развития гистопатологического стеатогепатита у индивидуумов со стеатозом. Из результатов секвенирования РНК образцов печени человека из группы бариатрической хирургии было видно, что гомозиготные носители сплайс-варианта преимущественно экспрессировали новый транскрипт, кодирующий укороченную изоформу HSD17B13. Эти данные пролили новый свет на роль HSD17B13 в содействии прогрессированию заболевания печени и его потенциал в качестве терапевтической мишени при стеатогепатите и циррозе печени.
[00400] Ранее проведенные полногеномные поиски ассоциаций (GWAS) выявили ограниченное количество генов и вариантов, ассоциируемых с хроническим заболеванием печени. На сегодняшний день наиболее достоверно подтвержденная генетическая связь обнаружена для распространенного миссенс-варианта гена белка 3, содержащего пататин-подобный фосфолипазный домен (PNPLA3 p. Ile148Met, rs738409), для которого ранее было обнаружено, что он связан с повышенным риском развития неалкогольной жировой болезни печени (NAFLD) (Romeo et al. (2008) Nat Genet 40:1461-1465 и Speliotes et al. (2011) PLoS Genet 7:e1001324, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), и впоследствии было обнаружено, что он связан со степенью тяжести заболевания (Rotman et al. (2010) Hepatology 52:894-903 и Sookoian et al. (2009) J Lipid Res 50:2111-2116, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) и прогрессированием заболевания (Trepo et al. (2016) J Hepatol doi:10.1016/j.jhep.2016.03.011, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Также было показано, что изменение в гене белка 2, который является членом суперсемейства 6 трансмебранных белков (TM6SF2), повышает риск развития NAFLD (Kozlitina et al. (2014) Nat Genet 46:352-356, Liu et al. (2014) Nat Commun 5:4309, и Sookoian et al. (2015) Hepatology 61:515-525, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Нормальные функции этих двух белков не совсем понятны, хотя было выдвинуто предположение, что оба они участвуют в липидном метаболизме гепатоцитов. Как варианты в PNPLA3 и TM6SF2 способствуют увеличению риска развития заболевания печени, еще предстоит выяснить. В GWAS также было выявлено несколько генетических факторов, связанных с сывороточной аланинаминотрансферазой (ALT) и аспартатаминотрансферазой (AST) (Chambers et al. (2011) Nat Genet 43:1131-1138 и Yuan et al. (2008) Am J Hum Genet 83:520-528, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), количественные показатели повреждения гепатоцитов и накопления жира в печени, которые зачастую измеряют в клинических условиях. На сегодняшний день нет описанных защитных генетических вариантов при хроническом заболевании печени. Обнаружение защитных генетических вариантов в других условиях, таких как варианты с утратой функции в PCSK9, которые снижают риск сердечно-сосудистых заболеваний, послужило катализатором для разработки новых классов терапевтических средств.
[00401] В результате сотрудничества в DiscovEHR между Regeneron Genetics Center и Geisinger Health System (GHS) были объединены усилия по секвенированию экзома с целью деидентификации данных в электронных медицинских картах (EHR) для того, чтобы сделать возможными генетические открытия и прецизионную медицину (Dewey et al. (2016) Science 354(6319) doi:10.1126/science.aaf6814, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Группа DiscovEHR состояла из пациентов, набранных из групп первичной и специализированной медицинской помощи в рамках интегрированной системы здравоохранения GHS, включая пациентов с бариатрической хирургией с образцами биопсии печени (Gorden et al. (2013) Hum Hered 75:34-43, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). В этом исследовании нами был предпринят комплексный подход функциональной геномики для оценки вклада вариаций последовательности экзома в количественные признаки, диагнозы заболеваний и гистопатологические фенотипы, относящиеся к хроническому заболеванию печени и циррозу печени, у 49188 индивидуумов европейского происхождения из группы DiscovEHR с последующим проведением исследований с использованием секвенирования всего экзома 9883 индивидуумов европейского происхождения.
[00402] Используя данные по последовательности всего экзома, связанные с полученными из EHR фенотипами, сначала было проведено исследование ассоциаций показателей ALT и AST в сыворотке у 46544 лиц европейского происхождения из группы DiscovEHR («группа исследования GHS»). Клинические характеристики группы описаны в таблице 1А. Было задействовано 41908 индивидуумов с документально подтвержденными в EHR показателями трансаминаз (включая 40561 индивидуумов с показателями как ALT, так и AST). Использовали линейную смешанную модель (Yang et al. (2011) Am J Hum Genet 88:76-82, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) для обнаружения ассоциаций между log10-преобразованными медианными уровнями ALT и AST (скорректированными по полу, возрасту, возрасту2, индексу массы тела (BMI) и первыми четырьмя основными компонентами родства) и 502219 двухаллельными единичными вариантами с частотой минорного аллеля, превышавшей 0,1%. С помощью полногеномного порога значимости Р<1,0×10-7 было выявлено 35 вариантов в 19 генах, которые значимо ассоциированы с ALT или AST, в том числе восемь вариантов в семи генах, которые были ассоциированы как с ALT, так и с AST (фиг. 1 и таблица 2).
[00403]
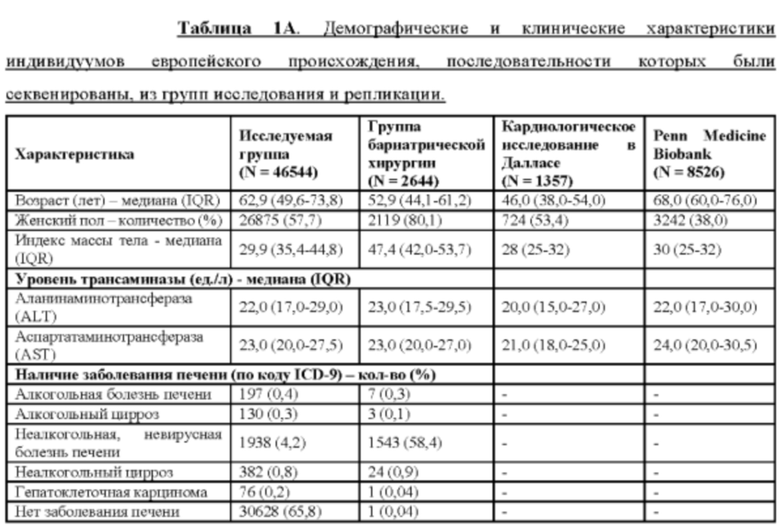
[00404] 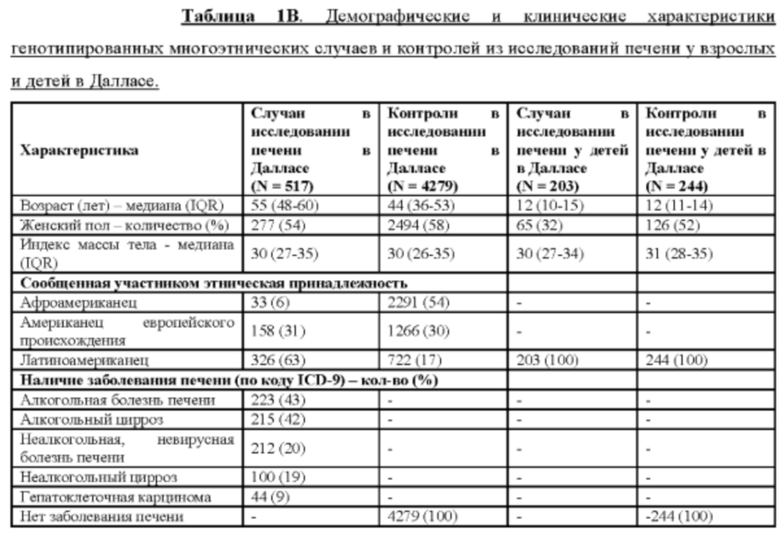
[00405]
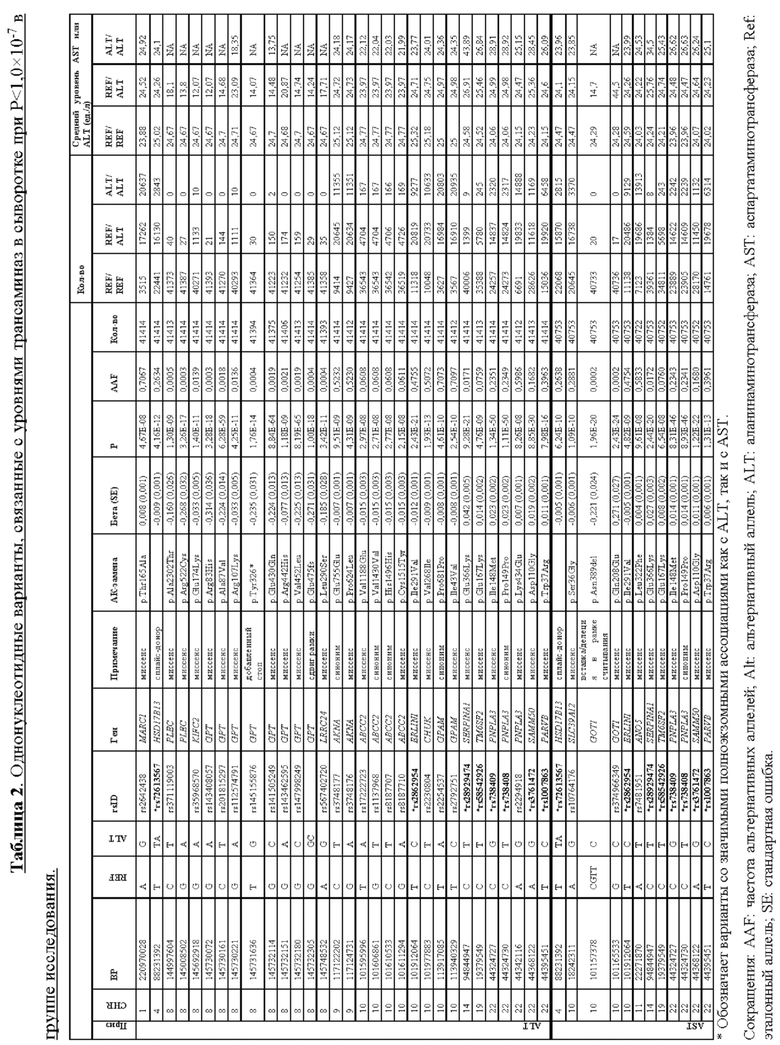
[00406] Для реплицирования этих ассоциаций были проанализированы 35 AST- или ALT-ассоциированных вариантов, выявленных посредством секвенирования всего экзома в трех отдельных группах лиц европейского происхождения: 2644 пациента с бариатрической хирургией из DiscovEHR («группа бариатрической хирургии GHS»), 1357 индивидуумов из кардиологического исследования в Далласе и 8526 индивидуумов из медицинского биобанка Penn Medicine Biobank (таблица 1А). В метаанализе репликационных групп значимо были ассоциированы тринадцать вариантов девяти генах (порог значимости по Бонферрони Р<1,43×10-3) с ALT или AST (таблица 3). В их число входили ранее раскрытые гены и варианты, ассоциированные с заболеванием печени, такие как PNPLA3 p. Ile148Met (Romeo et al. (2008) Nat Genet 40:1461-1465, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), TM6SF2 p. Glu167Lys (Kozlitina et al. (2014) Nat Genet 46:352-356, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) и SERPINA1 p. Glu366Lys (Z-аллель, связанный с дефицитом альфа-1-антитрипсина) (Brantly et al. (1988) Am J Med 84:13-31, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), SAMM50 и ERLIN1. SERPINA1 кодирует альфа-1-антитрипсин, функциональный дефицит которого, как известно, вызывает наследственное заболевание печени; ассоциация с SAMM50 может быть опосредована неравновесной связью с варьированием PNPLA3, а ERLIN1 участвует в отложении жира в печени. Несколько вариантов в GPT и GOT1, генах, кодирующих соответственно ALT и AST, были значимо связаны с уровнями ALT или AST, но об их связи с заболеванием печени ранее сообщений не было. SLC39A12 ранее не связывали с трансаминазами или заболеванием печени. Метаанализ также реплицировал новые ассоциации в нашей группе исследования между сниженными уровнями ALT (бета (SE) -0,009 (0,001); Р=4,16×10-12) и AST (бета (SE) -0,005 (0,001); Р=6,24×10-10) и сплайс-вариантом HSD17B13, гена, кодирующего гидроксистероид-17-бета-дегидрогеназу 13, неохарактеризованный член семейства 17-бета-гидроксистероиддегидрогеназ. Данный вариант, rs72613567, соответствует вставке нуклеотида А рядом с донорским сплайс-сайтом (аллель ТА). Р-значения в метаанализе репликации для этих ассоциаций составляли 3,85×10-5 и 9,38×10-5, а Р-значения для совместного метаанализа составляли 1,17×10-15 и 6,82×10-13 для соответственно ALT и AST (таблица 3). Ранее проведенные GWAS выявили находящийся рядом локус в 4q22 (rs6834314) как ассоциированный с уровнями ALT (Chambers et al. (2011) Nat Genet 43:1131-1138, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей); насколько нам известно, ранее не было проведено никаких исследований по описанию какой-либо ассоциации с rs72613567.
[00407]
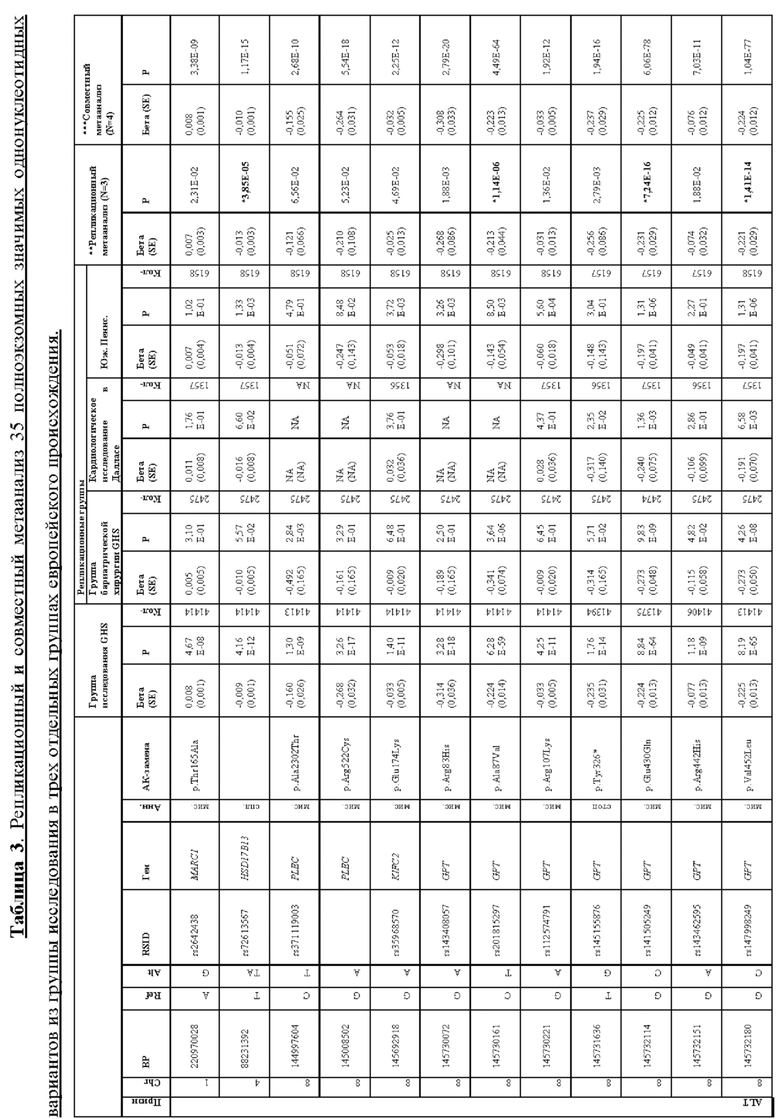
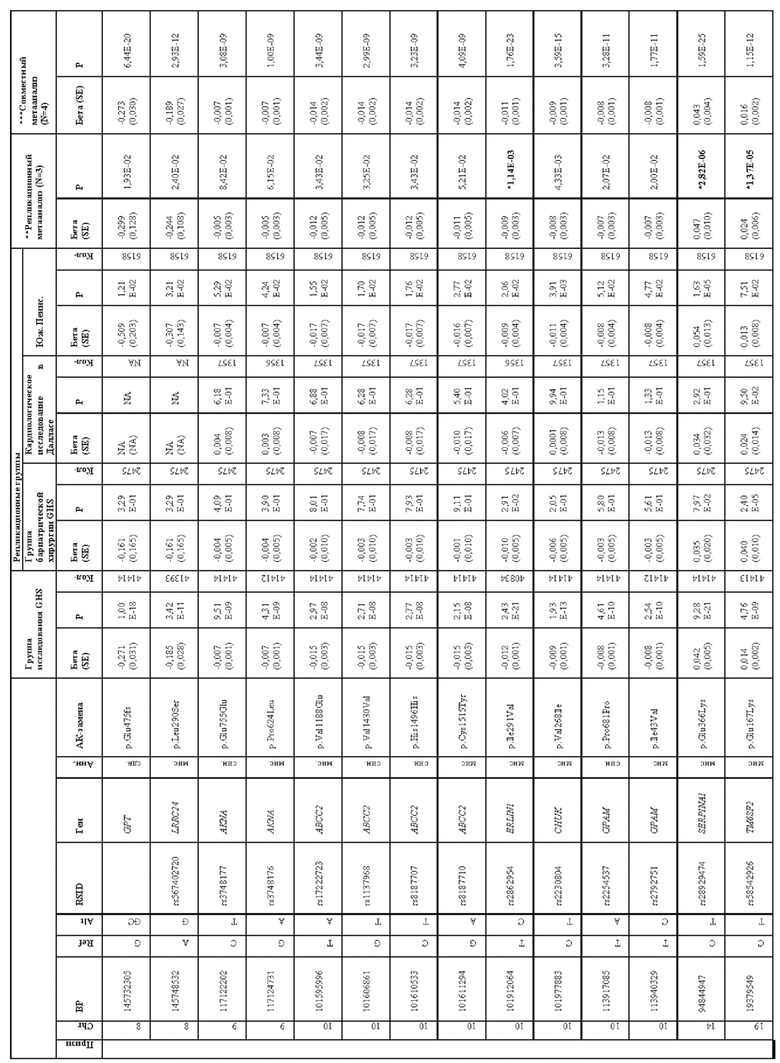
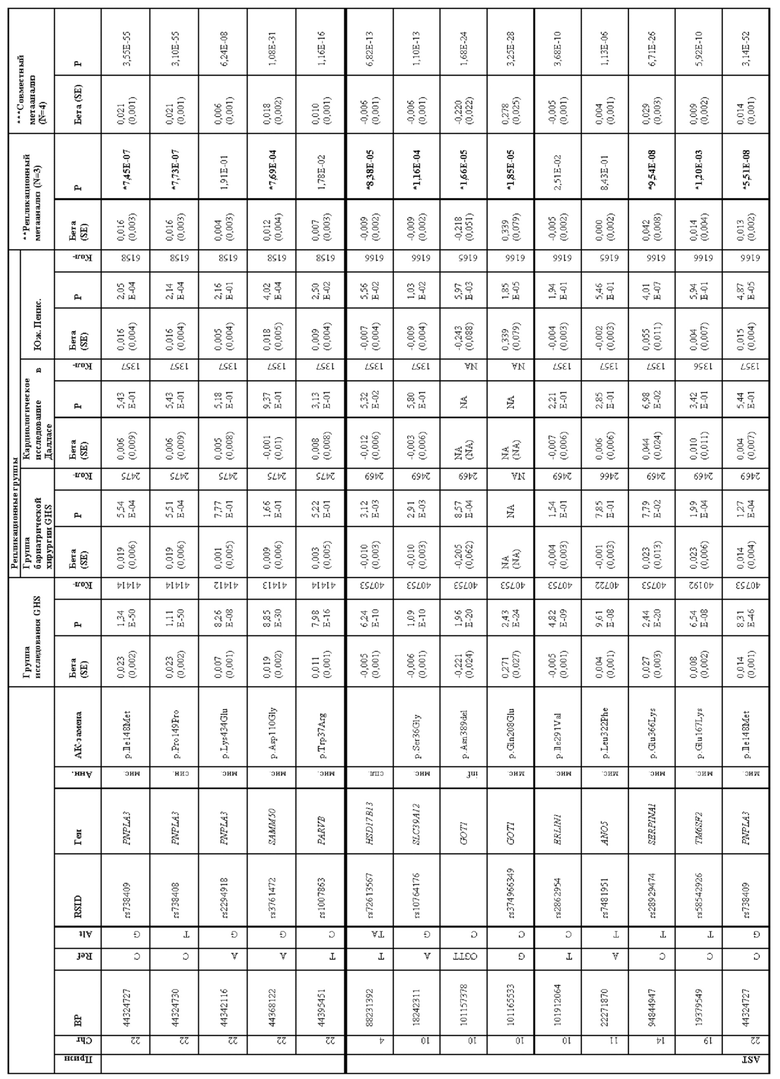
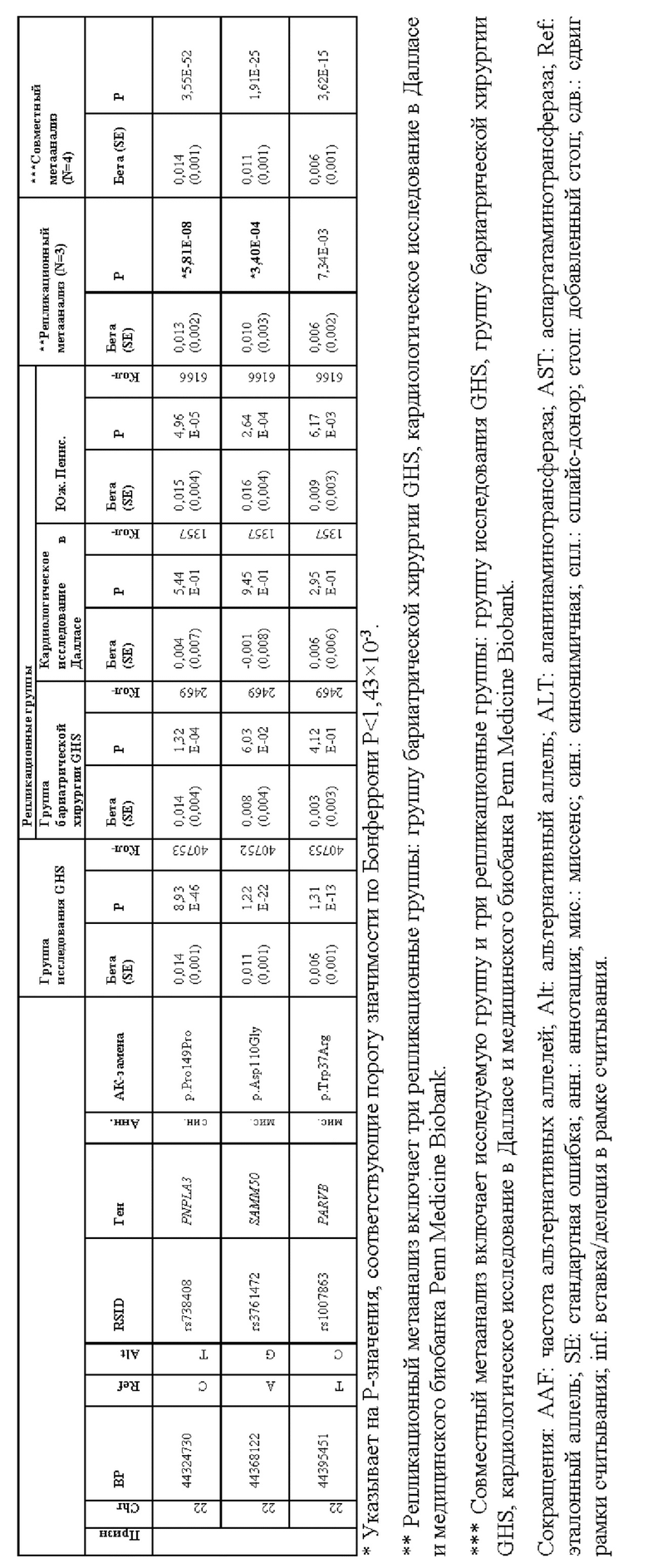
[00408] HSD17B13 находится на 30 т.о. выше HSD17B11, представителя того же семейства генов, и оба гена лежат у европейцев в пределах одного большого блока гаплотипов. Не наблюдали какой-либо ассоциации между кодирующими вариантами или сплайс-вариантами у HSD17B11 и уровней трансаминазы в группе исследования (фиг. 5А и 5В; наиболее значимые Р-значения в группе исследования были 1,36×10-1 для ALT и 4,32×10-2 для AST) или в совместном метаанализе исследуемой группы и трех репликационных групп (наиболее значимыми Р-значениями были 6,25×10-3 и 1,17×10-5 для ALT и AST соответственно). Кроме того, дисбаланс связи rs72613567 с вариантами HSD17B11 был умеренным во всех группах происхождения, в том числе у американцев европейского происхождения, которые в основном составляли нашу исследуемую группу, а также у латиноамериканцев и афроамериканцев, представленных в кардиологическом исследовании в Далласе (r2<0,4 со всеми установленными вариантами HSD17B11 во всех группах происхождения; данные не показаны). В совокупности эти результаты свидетельствуют о HSD17B13 в качестве гена в геномном участке, который наиболее вероятно функционально связан с уровнями трансаминаз.
[00409] Затем нами была предпринята попытка установить, ассоциированы варианты, ассоциируемые с уровнями ALT или AST, также и с хроническим заболеванием печени. В группе исследования были использованы диагностические коды EHR для широкого определения случаев алкогольной и неалкогольной (невирусной) болезни печени, а также следующих осложнений: алкогольный цирроз, неалкогольный цирроз и гепатоклеточная карцинома (НСС). Общую контрольную группу («нет заболевания печени») определяли как индивидуумов без диагностических кодов для любого типа заболевания печени (таблица 1). Было протестировано двенадцать проассоциированных с трансаминазой вариантов из исследуемой и репликационной групп в отношении ассоциации с хроническим заболеванием печени, используя порог значимости по Бонферрони Р<0,05/24 (Р<2,08×10-3) для учета протестированных тринадцати вариантов и двух широких категорий хронических заболеваний печени (алкогольных и неалкогольных) (таблица 4). В целом, были обнаружены значимые ассоциации между шестью вариантами у пяти генов (HSD17B13, SERPINA1, TM6SF2, PNPLA3 и SAMM50) и фенотипами хронического заболевания печени. Ассоциации SERPINA1, TM6SF2, PNPLA3 и SAMM50 подтверждали раскрытые ранее ассоциации. Варианты у GPTт. е. GOT1, ERLIN1 и SLC39A12, не имели значимых ассоциаций с каким-либо фенотипом заболевания печени. Ассоциация HSD17B13 с заболеванием печени, раскрываемая в данном документе, является новым и первым описанным потенциально защитным генетическим вариантом.
[00410]
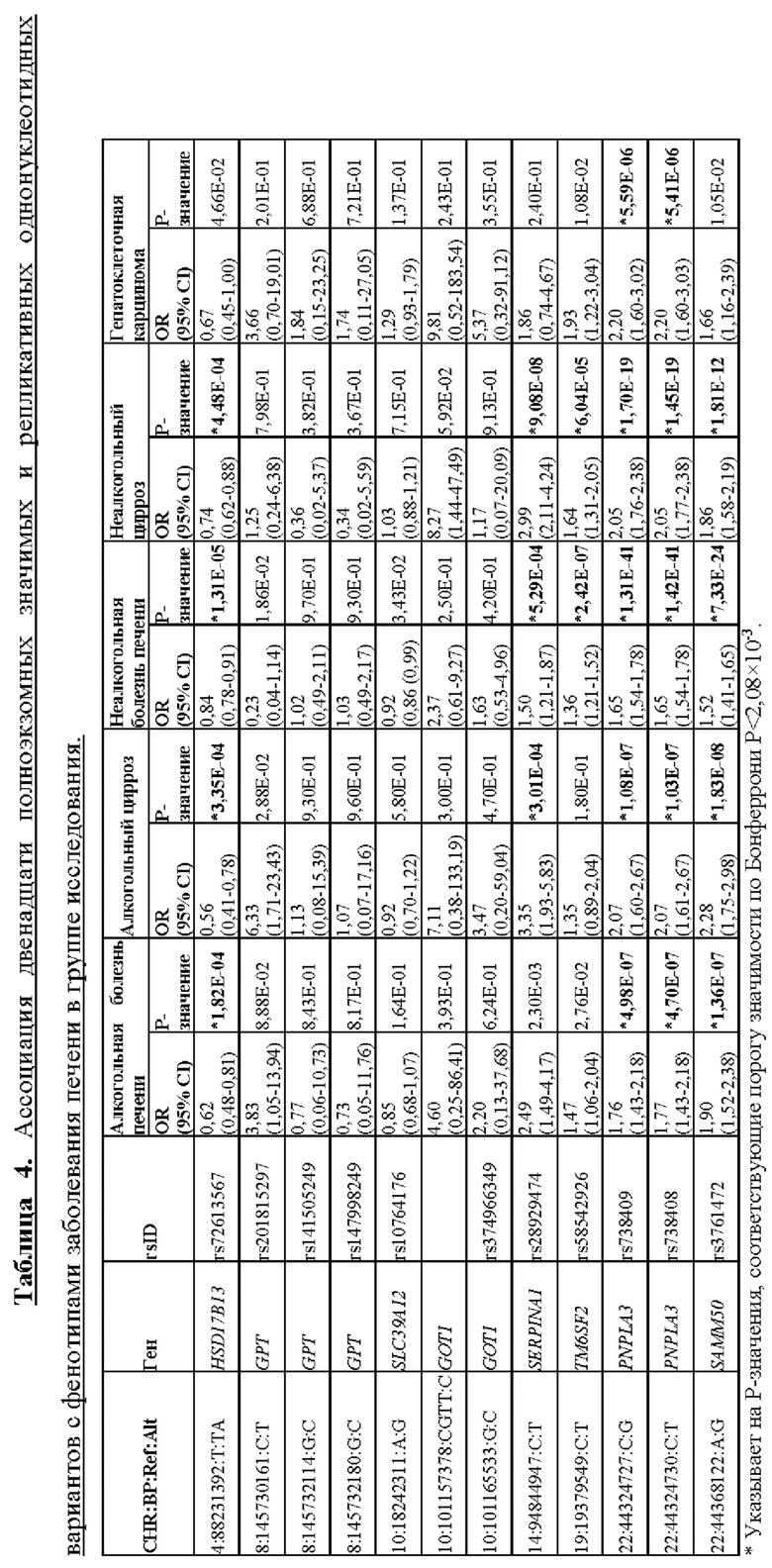
[00411] Альтернативный (ТА) аллель HSD17B13 rs72613567 наблюдали с более высокой частотой у контролей по сравнению с участниками с любым из оцениваемых фенотипов хронических заболеваний печени (фиг. 2А и таблица 5). После корректировки по возрасту, возраст2, полу, BMI и происхождению наблюдали 38% снижение шансов развития алкогольной болезни печени (отношение шансов [OR] 0,62; 95% доверительный интервал [CI] 0,48-0,81, Р=1,8×10-4) и 16% снижение шансов развития неалкогольной (невирусной) болезни печени (OR 0,84, 95% CI 0,78-0,91, Р=1,3×10-5) на аллель ТА. При ограничении до случаев с циррозом аллель ТА был ассоциирован с на 44% более низкими шансами развития алкогольного (OR 0,56, 95% CI 0,41-0,78, Р=3,4×10-4) и на 26% более низкими шансами развития неалкогольного (OR 0,74, 95% CI 0,62-0,88, Р=4,5×10-4) цирроза. Аллель ТА был номинально ассоциирован с на 33% более низкими шансами развития НСС на аллель (OR 0,67, 95% CI 0,45-1,00, Р=4,7×10-2). Нескорректированные генотипические OR свидетельствовали кодоминантном эффекте; например, в случае алкогольного цирроза OR составлял 0,59 (95% CI 0,40-0,86) для гетерозиготных носителей Т/ТА и 0,26 (95% CI 0,08-0,82) для гомозиготных носителей ТА/ТА, а также в случае неалкогольного цирроза OR составлял 0,75 (95% CI 0,61-0,93) для гетерозиготных и 0,55 (95% CI 0,34-0,91) для гомозиготных носителей.
[00412] Так, в группе исследования альтернативный (ТА) аллель HSD17B13 rs72613567 был ассоциирован с более низкими шансами развития всех оцениваемых полученных из EHR фенотипов хронических заболеваний печени зависимым от дозы совместимого аллеля образом (фиг. 2А): все категории алкогольной болезни печени, отношение шансов для гетерозигот (ORhet) [95% доверительный интервал] 0,58 [0,42-0,79], OR для гомозигот (ORhom) 0,46 [0,23-0,94], OR для аллельных вариантов (ORallelic) 0,62 [0,48-0,81], Р=1,82×10-4; все категории неалкогольной болезни печени: ORhet 0,84 [0,76-0,92], ORhom 0,73 [0,59-0,89], ORallelic 0,84 [0,78-0,91], Р=1,31×10-5. Аллель ТА также был ассоциирован с более низким шансом развития наиболее распространенных форм этих хронических заболеваний печени (что определяли с помощью диагностических кодов, полученных из EHR), а именно алкогольного и неалкогольного цирроза и НСС. Аллель ТА был ассоциирован с 42% и 73% снижением шансов развития алкогольного цирроза в случае соответственно гетерозигот и гомозигот (ORhet 0,59 [0,40-0,86], ORhom 0,26 [0,08-0,82], ORallelic 0,56 [0,41-0,78], Р=3,35×10-4), 26% и 49% снижением шансов развития неалкогольного цирроза в случае соответственно гетерозигот и гомозигот (ORhet 0,75 [0,61-0,93], ORhom 0,55 [0,34-0,91], ORallelic 0,74 [0,62-0,88], Р=4,48×10-4). Аллель ТА также был номинально ассоциирован с более низкими шансами развития ННС.
[00413] Затем была предпринята попытка подтвердить и расширить эти результаты в многоэтническом исследовании печени в Далласе (DLS) и исследовании печени у детей в Далласе (DPLS), включающем афроамериканцев, американцев европейского происхождения и латиноамериканцев, взрослых и детей (таблица 1В). В DLS аллель ТА был ассоциирован со снижением шансов развития какого-либо заболевания печени зависимым от дозы аллеля образом (ORhet 0,74 [0,57-0,97], ORhom 0,41 [0,21-0,83], ORallelic 0,70 [0,5-0,88], Р=1,77×10-3, фиг. 8). Схожие зависимые от дозы аллеля эффекты наблюдали для всех подтипов заболеваний печени, полученных из EHR, в том числе защитные ассоциации с распространенными циррозными формами алкогольной (ORallelic 0,72 [0,53-0,99], Р=4,37×10-2) и неалкогольной (ORallelic 0,65 [0,40-1,07], Р=8,96×10-2) болезни печени. В подгруппах анализов индивидуумов, сгруппированных по этнической принадлежности, о которой они сами сообщали, ассоциация с заболеванием печени оставалась значимой у латиноамериканцев, в частности, из-за высокого уровня заболеваемости печени в этой подгруппе населения (n=326 случаев и 722 контроля, ORallelic 0,51 [0,35-0,74], Р=3,98×10-4); аналогичные числовые тенденции, которые не достигали статистической значимости, были также отмечены в подгруппах афроамериканцев (n=33 случая и 2291 контроль, ORallelic 0,74 [0,25-2,47], Р=0,67) и американцев европейского происхождения (n=158 случаев и 1266 контролей, ORallelic 0,87 [0,65-1,15], Р=0,32) из DLS. В DPLS, отдельном исследовании детей-пациентов латиноамериканцев с заболеванием печени и страдающих избыточным весом контролей (таблица 1В), аллель ТА также был ассоциирован со снижением шансов развития заболевания печени (ORallelic 0,59 [0,36-0,97], Р=3,6×10-2). Так, аллель rs72613567:TA HSD17B13 был ассоциирован со сниженными шансами развития множественных форм хронического заболевания печени, в том числе цирроза, у взрослых и детей в трех независимых контингентах.
[00414] Под NAFLD описывают спектр заболеваний, начиная от жирового метаморфоза печени без проявления значительного воспаления (при гистопатологическом исследовании обозначают как «простой стеатоз») до более клинически значимых проявлений (обозначают как «неалкогольный стеатогепатит» (NASH), с гистопатологическим проявлением лобулярного воспаления, вспучивания гепатоцитов и/или фиброза). Для понимания взаимосвязи между аллелем ТА HSD17B13 и гистологически определенными NAFLD и NASH были проведены тесты ассоциации rs72613567 у 2391 индивидуума с общеэкзомным секвенированием с биоптатами печени из группы бариатрической хирургии GHS. Среди этих индивидуумов было 555 (23%) без проявления стеатоза, стеатогепатита или фиброза («нормальные»), 830 (35%) с простым стеатозом и 1006 (42%) с NASH (т.е. проявлением лобулярного воспаления, вспучиванием гепатоцитов или фиброзом). Аллель ТА HSD17B13 не имел значий ассоциации с простым стеатозом (OR 1,11, 95% CI 0,94-1,32, Р=0,21) или NASH (OR 0,86, 95% CI 0,72-1,02, Р=0,09) по сравнению с нормальной печенью (фиг. 2В и таблица 5). При сравнении доли случаев с нормальной печенью, простым стеатозом и NASH по генотипу наблюдали, что доля случаев с нормальной печенью, по-видимому, не отличалась по генотипу (23%, 24% и 23% соответственно для Т/Т, Т/ТА и ТА/ТА носителей, Р=0,5 по критерию хи-квадрат для тенденции в пропорциях), но что снижалась доля случаев с NASH (45%, 40% и 31% соответственно для Т/Т, Т/ТА и ТА/ТА носителей, Р=1,6×10-4) и что увеличивалась доля случаев с простым стеатозом (33%, 35% и 47% соответственно для Т/Т, Т/ТА и ТА/ТА носителей, Р=1,1×10-3) с каждым аллелем ТА (фиг. 9). Среди индивидуумов со стеатозом аллель ТА был ассоциирован со статистически значимыми более низкими шансами развития NASH, по сравнению с простым стеатозом, зависимым от дозы аллеля образом. На фоне простого стеатоза аллель ТА был ассоциирован с 23% снижением шансов NASH (OR 0,77, 95% CI 0,66-0,90, Р=6,5×10-4), что свидетельствовало о роли HSD17B13 в опосредовании прогрессирования NAFLD до более запущенных стадий NASH и фиброза. Результаты установления генотипических ассоциаций согласовывались с кодоминантным эффектом; при сравнении NASH относительно простого стеатоза OR составлял 0,84 (95% CI 0,69-1,02) для гетерозиготных Т/ТА носителей и 0,48 (95% CI 0,34-0,68) для гомозиготных ТА/ТА носителей.
[00415]
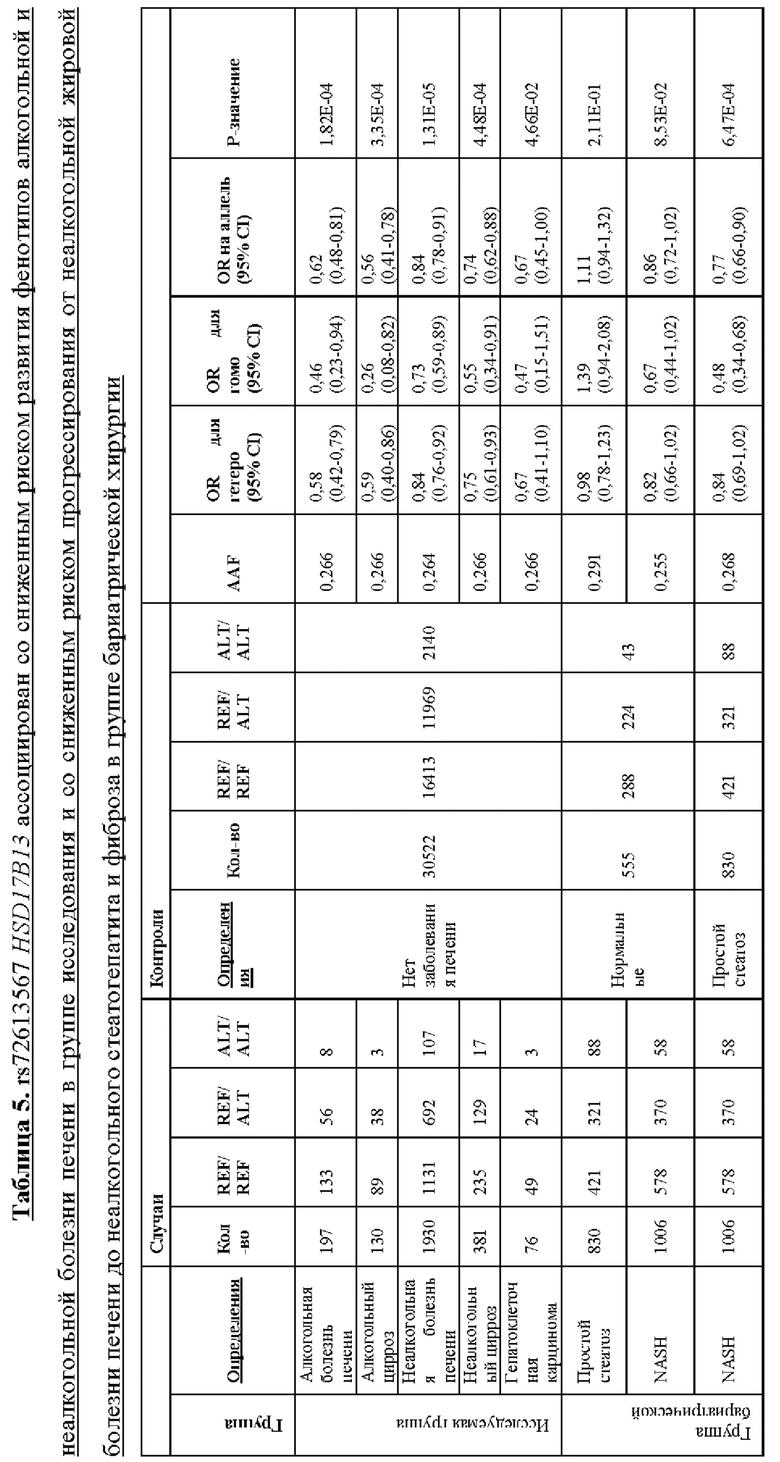
[00416] Далее была предпринята попытка понять, как аллель ТА HSD17B13 влияет на экспрессию известных и новых транскриптов гена. Использовали секвенирование РНК для оценки экспрессии мРНК HSD17B13 в нормальных с точки зрения гистологии образцах печени, полученных от 22 гомозиготных эталонных (Т/Т), 30 гетерозиготных (Т/ТА) и 17 гомозиготных альтернативных (ТА/ТА) носителей сплайс-варианта rs72613567 HSD17B13 (фиг. 3). Помимо двух известных транскриптов HSD17B13, А и В, были выявлены два новых транскрипта: транскрипт С, у которого отсутствовал экзон 6, и транскрипт D, характеризуемый вставкой нуклеотида G на 3'-конце экзона 6, что приводило к преждевременному укорочению белка. Новые транскрипты проверяли с помощью RT-PCR, а транскрипт D дополнительно проверяли путем секвенирования кДНК с длинными участками считывания. Уровни экспрессии этих транскриптов варьировали в соответствии с генотипом rs72613567 HSD17B13; уровни транскриптов А и В снижались,, в то время как количество транскриптов С и D увеличилось зависимыми от дозы аллеля образом у Т/ТА гетерозигот и ТА/ТА гомозигот (фиг. 3). Транскрипт А, кодирующий белок из 300 аминокислот, был преобладающим транскриптом у Т/Т гомозигот (фиг. 3А), тогда как транскрипт D, кодирующий преждевременно укороченный белок, был преобладающим транскриптом у ТА/ТА гомозигот (фиг. 3D). Эти паттерны экспрессии свидетельствуют о функциональной роли rs72613567 HSD17B13 в определении экспрессии изоформы HSD17B13. Также были выявлены четыре дополнительных транскрипта (Е-Н) с очень низкими уровнями экспрессии (фиг. 6A-6D). Результаты выравнивания белковых последовательностей у всех выявленных изоформ HSD17B13 показаны на фиг. 7А-7В.
[00417] HSD17B13 был ранее описан как ассоциированный с липидными каплями белок в гепатоцитах человека (Su et al. (2014) Proc Natl Acad Sci USA 111:11437-11442, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Была проведена оценка экспрессии и локализации изоформы белка в иммортализированной линии гепатоцитов человека (клеток гепатомы HepG2), стабильно трансдуцированной лентивирусом, экспрессирующим известные и новые изоформы A-D HSD17B13. Изоформа А HSD17B13 локализуется в липидных каплях в необработанных и обработанных олеиновой кислотой клетках. Изоформу А в основном детектировали на мембранах, окружающих меченные посредством BODIPY липидные капли, и она совместно локализовалась с перилипином, белком оболочки липидной капли (PLIN). Аналогичную субклеточную локализацию наблюдали в случае изоформы D HSD17B13 на поверхности липидных капель, однако после обработки олеиновой кислотой липидные капли на вид имели больший размер. Напротив, изоформы В и С совместно локализовались с калнексином, маркером эндоплазматического ретикулума.
[00418] Подводя итог всему вышесказанному, используя данные о последовательностях экзома в связи с данными из EHR и биопсии печени, полученными от 49188 индивидуумов из контингента, участвовавшего в исследовании DiscovEHR, и с помощью последующих исследований данных о последовательностях экзома, полученных от 9883 дополнительных индивидуумов с измерениями ALT и AST, была обнаружена новая ассоциация между сплайс-вариантом HSD17B13, уровнями трансаминаз и фенотипом хронического заболевания печени. В нашем исследовании вариант HSD17B13 снижал риск неалкогольной и алкогольной болезни печени и цирроза печени. Насколько нам известно, это первое сообщение об экзонном варианте с защитной ассоциацией с фенотипами хронического заболевания печени. Аллель ТА HSD17B13 не имел ассоциации с простым стеатозом, но снижал риск развития гистопатологического стеатогепатита у индивидуумов со стеатозом, что свидетельствовало о роли HSD17B13 в прогрессировании до более клинически запущенных стадий хронического заболевания печени. Согласованность защитных ассоциаций в четырех независимых группах (группе исследования GHS, группе бариатрической хирургии GHS, DLS и DPLS) по нескольким различным категориям заболеваний печени, которую характеризовали с использованием диагностических кодов EHR, а также гистопатологических определений заболевания печени, вместе с поразительной зависимостью ассоциаций от дозы аллеля, подтверждали вывод, что зарегистрированный вариант HSD17B13 защищает от прогрессирования до более клинически запущенных стадий хронического заболевания печени. Наблюдаемая зависимость от дозы аллеля также доказывает, что более глубокая регуляция функции HSD17B13 может привести к более сильным эффектам на риск появления и прогрессирование заболевания.
[00419] Известно, что и другие представители семейства 17-бета-гидроксистероиддегидрогеназ вовлечены в метаболизм половых стероидов и жирных кислот (Moeller and Adamski (2009) Mol Cell Endocrinol 301:7-19, полностью включена в настоящий документ посредством ссылки для всех целей), однако имеется лишь незначительная информация о функции HSD17B13. HSD17B13 экспрессируется преимущественно в печени (Liu et al. (2007) Acta Biochim Pol 54:213-218, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), где он локализуется в липидных каплях (Su et al. (2014) Proc Natl Acad Sci USA 111:11437-11442, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), что согласуется с ролью HSD17B13 в патогенезе жировой болезни печени. Наши данные согласовывались с недавно полученными результатами, что сверхэкспрессия HSD17B13 увеличивала липогенез в печени мыши и увеличивала количество и размер липидных капель в культивируемых гепатоцитах (Su et al. (2014) Proc Natl Acad Sci USA 111:11437-11442, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Из результатов двух ранее проведенных исследований также было видно, что экспрессия в печени белка HSD17B13 увеличивалась у пациентов с жировым метаморфозом печени (Su et al. (2014) Proc Natl Acad Sci USA 111:11437-11442 и Kampf et al. (2014) FASEB J 28:2901-2914, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Также в метаболизме липидов в гепатоцитах физиологическую роль играют два гена с вариантами, о которых сообщалось, что они ассоциированы с повышенным риском развития заболевания печени, т.е. гены PNPLA3 и TM6SF2. Вариант HSD17B13, описываемый в данном документе, является первым защитным вариантом для заболевания печени и может стать подходом для создания новых терапевтических стратегий для целенаправленного лечения хронического заболевания печени, подобно генетическим вариантам, которые привели к разработке новых терапевтических средств в других областях.
[00420] В целом, наши данные подтверждали, что HSD17B13 является новой терапевтической мишенью для уменьшения риска развития хронического заболевания печени у людей. Важно отметить, что наши данные указывали на то, что целенаправленное воздействие на HSD17B13 может снизить прогрессирование от NAFLD до более запущенных стадий NASH, фиброза и цирроза печени, которые ассоциированы со значительной заболеваемостью и смертностью и для которых в настоящее время нет эффективных средств лечения.
Способы
[00421] Участники исследования. Исследования в области генетики человека были проведены в рамках сотрудничества по программе DiscovEHR центра генетики Regeneron Genetics Center и Geisinger Health System (GHS). Исследование было одобрено экспертным советом организации GHS. Два контингента из исследования DiscovEHR (исследуемая группа и группа бариатрической хирургии) были созданы из первых 50726 участников, достигших возраста согласия ≥18 лет, из программы Community Health Initiative MYCODE® компании GHS (Dewey et al. (2016) Science 354(6319) doi:10.1126/science.aaf6814, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Группа исследования GHS состояла из 46544 европейцев, набранных из поликлиник и специализированных клиник в период между 2007 и 2016 годами, за исключением набранных в группу бариатрической хирургии. В группу бариатрической хирургии GHS вошли 2644 европейца, которые были направлены на бариатрическую хирургию.
[00422] Репликационные исследования включали 1357 европейцев из кардиологического исследования в Далласе и 8527 европейцев из медицинского биобанка Penn Medicine Biobank. Кардиологическое исследование в Далласе представляло собой вероятностное исследование групп контингента резидентов округа Даллас возрастом 30-65 лет (Victor et al. (2004) Am J Cardiol 93:1473-1480, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей. Penn Medicine Biobank включает участников, набранных из системы здравоохранения Университета Пенсильвании (University of Pennsylvania Health System) и давших согласие на хранение биологических образцов, доступ к данным из EHR и разрешение на повторный контакт.
[00423] Репликационные исследования ассоциаций с хроническим заболеванием печени включали 517 индивидуумов из исследования печени в Далласе (Dallas Liver Study - DLS) и 447 человек из исследования печени у детей в Далласе (Dallas Pediatric Liver Study - DPLS). DLS представляет собой биобанк пациентов с заболеванием печени невирусной этиологии. Набор участников начался в январе 2015 года и продолжается по сей день. Участников набирали из клиник по лечению заболеваний печени в системе UT Southwestern and Parkland Health and Hospital System, в Далласе. Биобанк был одобрен экспертным советом UT Southwestern. Участники предоставляли письменное информированное согласие. Участники заполняли опросник об этническом/расовом происхождении, истории болезни, факторах образа жизни и семейном анамнезе заболевания печени и других заболеваний. Дополнительная клиническая информация была получена из медицинских карт с помощью обученного специалиста. Были включены все пациенты, которые были афроамериканцами, американцами европейского происхождения и латиноамериканцами с ДНК, имеющейся на момент настоящего исследования (n=517). DPLS представляет собой биобанк детей, набранных из клиник по лечению заболеваний печени у детей в UT Southwestern and Parkland Health and Hospital System, Даллас, и из клиники по лечению ожирения в Детском медицинском центре, Даллас. Биобанк был одобрен экспертным советом UT Southwestern. Законные опекуны участников давали письменное информированное согласие. Клиническая информация была получена из медицинских карт с помощью обученного специалиста. Поскольку более 95% пациентов были латиноамериканцами, в настоящее исследование были включены только латиноамериканские пациенты и контрольные пациенты (n=203 пациента и 244 контрольных пациента).
[00424] Подготовка образцов и секвенирование. Подготовку образцов и секвенирование всего экзома проводили в Центре генетики компании Regeneron, как описано ранее (Dewey et al. (2016) Science 354(6319) doi:10.1126/science.aaf6814, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Вкратце: захват экзома проводили с помощью зондов NimbleGen в соответствии с рекомендованным производителем протоколом (Roche NimbleGen). Захваченную ДНК амплифицировали с помощью ПЦР и количественно оценивали с помощью qRT-PCR (Kapa Biosystems). Мультиплексированные образцы секвенировали с помощью секвенирования спаренных концов размером 75 п. о. на Illumina v4 HiSeq 2500 до глубины охвата, достаточной для обеспечения более чем 20-кратной глубины гаплоидного считывания более 85% целевых оснований у 96% образцов (примерно 80-кратная средняя глубина гаплоидного считывания целевых оснований). Необработанные данные о последовательностях каждого цикла Illumina Hiseq 2500 загружали в платформу DNAnexus (Reid et al. (2014) BMC Bioinformatics 15, 30 doi:10.1186/1471-2105-15-30) для выравнивания результатов считывания последовательностей и выявления вариантов. Вкратце: необработанные данные о последовательностях преобразованы из файлов BCL в специфичные по образцу FASTQ-файлы, для которых проводили выравнивание с человеческой эталонной сборкой GRCh37.p13 с BWA-mem (Li and Durbin (2009) Bioinformatics 25:1754-1760, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Однонуклеотидные варианты (SNV) и варианты последовательностей со вставками/делециями (indel) выявляли с помощью инструментария Genome Analysis Toolkit (McKenna et al. (2010) Genome Res 20:1297-1303, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей).
[00425] Целевое генотипирование rs72613567 в исследованиях печени у взрослых и детей в Далласе. Генотипирование rs72613567 HSD17B13 производили с помощью анализа TAQMAN® в ходе исследования печени в Далласе и исследования печени у детей в Далласе и с помощью секвенирования экзома в кардиологическом исследовании в Далласе. Результаты, полученные с помощью TAQMAN®, проверяли с помощью секвенирования по Сэнджеру у 5 индивидуумов с каждым генотипом.
[00426] Клинические измерения и определения хронического заболевания печени в группе исследования. Результаты клинических лабораторных измерений ALT и AST получали из EHR участников из группы исследования GHS и группы бариатрической хирургии. Медианные значения ALT и AST рассчитывали для всех участников с двумя или более результатами измерениями и преобразовывали в log10 для нормализации распределения перед проведением анализа ассоциаций.
[00427] Коды заболеваний по Международной классификации болезней девятой ревизии (ICD-9) получали из EHR и сводили в категории клинических заболеваний для определения невирусных, неалкогольных (ICD-9 571.40, 571.41, 571.49, 571.5, 571.8, 571.9) или алкогольных (ICD-9 571.0, 571.1, 571.2, 571.3) случаев заболевания печени. Дополнительные определения случаев, основанные на единичных диагностических кодах, включали: алкогольный цирроз (ICD-9 571.2), неалкогольный цирроз (ICD-9 571.5) и НСС (ICD-9 155.0). Для определения таких случаев общую контрольную группу без заболевания печени определяли как участников, не имеющих критериев такого случая или имеющих диагностический код однократного проявления или диагностический код перечня проблем, указывающий на любой тип заболевания печени.
[00428] Определения гистопатологического фенотипа печени в группе бариатрической хирургии. Группа бариатрической хирургии GHS состояла из 2644 индивидуумов европейского происхождения, при этом от 2391 из этих индивидуумов были получены образцы биоптата печени, взятые в ходе хирургического вмешательства. Образцы биоптата печени фиксировали формалином и окрашивали гематоксилином и эозином для стандартного гистологического исследования и трихромным окрашиванием по Массону для оценки фиброза, как описано ранее (Gerhard et al. (2011) Patient Saf Surg 5, 1, doi:10.1186/1754-9493-5-1, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Гистологические диагнозы были поставлены гепатопатологами с использованием ранее установленных критериев (Brunt et al. (1999) Am J Gastroenterol 94:2467-2474, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Гистологические диагнозы использовали для определения следующих фенотипов: 1) нормальный: нет признаков стеатоза, NASH или фиброза; 2) простой стеатоз: стеатоз (независимо от степени тяжести) без признаков NASH или фиброза; 3) NASH/фиброз: любое присутствие лобулярного воспаления или вспучивания гепатоцитов (независимо от степени тяжести) или любое наличие фиброза (независимо от стадии); 4) фиброз: любое наличие фиброза (независимо от стадии).
[00429] Анализ полноэкзомных ассоциаций ферментов печени. В группе исследования GHS было протестировано 502219 двухаллельных вариантов со степенью потерянной информации <1%, р-значением закона Харди-Вайнберга >1,0×10-6 и частотой минорного аллеля >0,1% для ассоциации с уровнями трансаминаз. Log10-преобразованные медианные ALT и AST корректировали по возрасту, возрасту2, полу, BMI и первым четырем основным компонентам происхождения. Для учета родства среди участников исследования также использовали матрицу генетического родства в качестве ковариаты случайных эффектов. Как основные компоненты, так и матрица генетического родства были сконструированы из 39858 не относящихся к МНС маркеров в примерном равновесии связей и с частотой минорного аллеля >0,1%. Использовали линейные смешанные модели, реализованные в пакете GCTA (Yang et al. (2011) Am J Hum Genet 88:76-82, включена в настоящий документ посредством ссылок во всей своей полноте для всех целей) для тестирования ассоциации между невязками признаков и однонуклеотидными вариантами. Тесты были высокоточными, как видно из полноэкзомных квантиль-квантиль графиков и значений лямбда геномного контроля (фиг. 1).
[00430] Репликационный метаанализ ассоциаций ферментов печени. Была предпринята попытка реплицировать ассоциации в группе исследования GHS, в трех отдельных группах с европейским происхождением: в группе бариатрической хирургии GHS, кардиологического исследования в Далласе и медицинского биобанка Penn Medicine Biobank (описано выше). Результаты измерений ALT и AST в группе бариатрической хирургии GHS и из медицинского биобанка Penn Medicine Biobank преобразовывали в log10 и корректировали по возрасту, возрасту2, полу, BMI и первым четырем основным компонентам происхождения. Результаты измерений ALT и AST из образцов медицинского биобанка Penn Medicine Biobank преобразовывали в log10 и корректировали по возрасту, возрасту2, полу, BMI и первым четырем основным компонентам происхождения. Включали матрицы генетического родства как ковариаты случайных эффектов и проводили анализ с использованием линейных смешанных моделей в GCTA. В кардиологическом исследовании в Далласе, преобразованные в log10 показатели ALT и AST корректировали по возрасту, возрасту2, полу и первым десяти основным компонентам происхождения и проводили анализ с использованием линейной регрессии, реализованной в PLINK. Сводная статистика для трех репликационных групп была подвергнута метаанализу с помощью METAL (репликационного метаанализа) (Willer et al. (2010) Bioinformatics 26:2190-2191, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Аналогичным образом была подвергнута метаанализу сводная статистика для исследуемой группы и трех репликационных групп (совместный метаанализ).
[00431] Анализ ассоциаций с фенотипами хронического заболевания печени. Анализировали девять значимых и реплицированных однонуклеотидных вариантов из ExWAS ферментов печени в отношении ассоциаций с бинарными фенотипами заболевания печени, определенными из группы исследования GHS, как описано выше. Использовали порог значимости Бонферрони Р<0,05/26 (Р<1,92×10-3) для учета протестированных тринадцати вариантов и двух обширных категорий хронических заболеваний печени (алкогольных и не алкогольных). Вариант HSD17B13 дополнительно тестировали в отношении ассоциации с определенными с точки зрения гистопатологии фенотипами печени из группы бариатрической хирургии GHS, как описано выше. Отношения шансов оценивали с помощью вероятностного способа логистической регрессии с поправкой Фирта после коррекции по возрасту, возрасту2, полу, BMI и первых четырех основных компонентов происхождения. Нескорректированные генотипические отношения шансов также анализировали для rs72613567 HSD17B13.
[00432] Отношения шансов для заболевания печени в DLS оценивали с помощью логистической регрессии с коррекцией по возрасту, возрасту2, полу, BMI и сообщенной участником этнической принадлежности. Участников кардиологического исследования в Далласе с имеющимися генотипами rs72613567 использовали в качестве нормальных контролей (n=4279). Отношения шансов в DPLS оценивали с помощью логистической регрессии.
[00433] Программное обеспечение. Анализ генетических ассоциаций проводили с использованием программного обеспечения GCTA, версии 1.25.0 (Yang et al. (2011) Am J Hum Genet 88:76-82, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), и PLINK, версии 1.9.0. Квантиль-квантиль график и график Манхэттена строили с использованием программного обеспечения R, версии 3.2.1 (R Project for Statistical Computing). Графики региональных ассоциаций строили с помощью LocusZoom (Pruim et al. (2010) Bioinformatics 26:2336-2337, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей).
[00434] Исследования по секвенированию РНК. Качество и концентрацию РНК оценивали путем прогона общей РНК на чипе Agilent RNA Nano Bioanalyzer; все образцы имели число целостности РНК (RTN), превышавшее 8. Полиаденилированные РНК-транскрипты выделяли с использованием двух циклов обогащения микроносителями с олиго(dT)25 (Thermo Fisher Scientific). Образцы очищали и концентрировали с микроносителями RNAclean ХР (Beckman Coulter) и подвергали тепловой фрагментации примерно до 140 пар оснований. Синтез первой цепи осуществляли с использованием обратной транскриптазы Superscript III (Thermo Fisher Scientific) с применением случайных гексамеров; во время синтеза второй цепи dTTP заменяли на dUTP. Образцы обрабатывали в соответствии с нашим стандартным способом получения библиотеки ДНК, упомянутой выше для экзомов, с добавлением стадии с использованием урацил-ДНК-гликозилазы для получения библиотек для специфичного по цепи секвенирования. Образцы объединяли в пул и секвенировали с помощью секвенирования спаренных концов размером 75 п. о. на Illumina v4 HiSeq2500.
[00435] Выявление новых транскриптов HSD17B13. Результаты считывания наносили на карту относительно Human.B38 с помощью программного обеспечения ARRAYSTUDIO® (OMICSOFT®, Кэри, Северная Каролина), допуская два несоответствия. Для выявления новых транскриптов HSD17B13 использовали два подхода. Новые соединения экзонов искали относительно Gencode v24. De novo сборку транскриптов проводили с помощью Trinity (v2.2.0) с настройками по умолчанию. Строили индивидуальные генные модели с включением новых транскриптов HSD17B13 и производили количественную оценку транскриптов путем выравнивания результатов считывания с индивидуальной генной моделью. Результаты выравнивания белковых последовательностей для всех выявленных изоформ HSD17B13 показаны на фиг. 7А и 7В.
[00436] RT-PCR-проверка новых транскриптов. RT-PCR на общей РНК из образцов печени человека проводили с использованием системы SUPERSCRIPT™ One-Step RT-PCR System с ДНК-полимеразой Taq DNA Polymerase PlatinumTM (Thermofisher). Каждые 50 мкл реакционной смеси для RT-PCR содержали 1 × реакционного микса, 500 нМ каждого из прямого и обратного праймеров (PST516: ATGAACATCATCCTAGAAATCCTTC (SEQ ID NO: 251) и PST517: ATCATGCATACATCTCTGGCTGGAG (SEQ ID NO: 252)), 1 мкл RT/Platinum Taq и 75 нг РНК. Условия циклирования: один цикл при 45°С в течение 30 минут; один цикл при 94°С в течение 2 минут; 40 циклов при 94°С в течение 20 с, 53°С в течение 30 с и 72°С в течение 90 с; один цикл при 72°С в течение 5 минут; затем удержание на уровне 10°С. Продукты очищали с помощью набора для очистки ПЦР QIAquick PCR Purification Kit (Qiagen) и подвергали прямому секвенированию по Сэнджеру с применением праймера DE002 (ATCAGAACTTCAGGCCTTGG (SEQ ID NO: 253)). Для выявления транскриптов В и С продукты RT-PCR прогоняли на 2% агарозном геле с окрашиванием посредством красителя SYBR GOLDSYBR® Gold Nucleic Acid Gel Stain (ThermoFisher) и бэнды с ожидаемой молекулярной массой вырезали и очищали с использованием набора для экстракции из геля QIAquick Gel Extraction Kit (Qiagen), затем подвергали клонированию с использованием набора для клонирования ТТОРО® ТА Cloning Kit (Thermofisher). Секвенирование клонов ТОРО проводили с применением праймеров для секвенирования M13F и M13R. Анализ последовательностей проводили с использованием программного обеспечения для анализа ДНК Sequencher (Gene Codes Corporation).
[00437] PacBio-проверка новых транскриптов. Полноразмерные транскрипты HSD17B13 амплифицировали непосредственно с 50 нг общей РНК с помощью системы SuperScript III One-step RT-PCR System с Platinum Taq High Fidelity (Thermo Fisher Scientific) с применением геноспецифических праймеров в первом (GCAAAGCCATGAACATCATCC (SEQ ID NO: 254) и последнем экзонах (TCTTGATGTAGTGGGAGTCGGATT (SEQ ID NO: 255)) для получения ампликона размером ~2,2 т.о. (транскрипта с максимальным прогнозируемым размером). Ампликоны проверяли на биоанализаторе Agilent Bioanalyzer. К ампликонам пришивали PacBio-совместимые бар кодированные адаптеры и очищали с микроносителями PacBio PB (Pacific Biosciences). Библиотеки объединяли в пул в равных количествах и секвенировали на одной ячейке для SMRT в течение 180 минут на платформе PacBio RSII. Данные подвергали демультиплексированию с использованием программного обеспечения PacBio software smrtanalysis v2.3 tool labeizmw, а затем анализировали с помощью ConsensusTools AmpliconAnalysis. Полученные в результате ампликоны сравнивали с генами HSD17B13 из базы данных RefSeq для определения статуса изоформы генотипа.
[00438] Субклеточная локализация изоформ HSD17B13. Клетки HepG2 культивировали в минимальной основной среде Игла, дополненной 10% эмбриональной бычьей сыворотки. Транскрипты А, В, С и D HSD17B13 субклонировали в остовные лентивирусные конструкции Myc-DDK и получали лентивирус. Клетки HepG2 инфицировали лентивирусом, несущим различные транскрипты HSD17B13. Стабильные клеточные линии, экспрессирующие каждый транскрипт HSD17B13, подвергали отбору с помощью 1-3 мг/мл сульфата генетицина G-418 в полной культуральной среде в течение двух недель. Отобранные клетки HepG2 обрабатывали или не обрабатывали 200 мкМ олеиновой кислоты в течение ночи, а затем фиксировали. Изоформы HSD17B13 метили мышиным антителом к Мус. Липидные капли метили красителем BODIPY FL (Sigma). Белок липидной оболочки и эндоплазматический ретикулум метили соответственно кроличьим антителом к PLIN (Sigma) и кроличьим антителом к кальнексину (Cell Signaling Technology). Вторичными антителами для иммунофлуоресценции были конъюгированные с Alexa Fluor 488 антитела осла к IgG кролика и конъюгированные с Alexa Fluor 594 антитела осла к IgG мыши (Jackson ImmunoResearch).
Пример 2 Влияние rs72613567:TA на экспрессию мРНК HSD17B13 и белка HSD17B13
[00439] Изучали влияние аллеля rs72613567:TA HSD17B13 на экспрессию известных и новых транскриптов гена. Использовали секвенирование РНК для оценки экспрессии мРНК HSD17B13 в нормальных с точки зрения гистологии образцах печени, полученных от 22 Т/Т гомозиготных, 30 Т/ТА гетерозиготных и 17 ТА/ТА гомозиготных носителей сплайс-варианта rs72613567 HSD17B13. В дополнение к двум известным транскриптам А и В HSD17B13 были выявлены два новых транскрипта: транскрипт С, у которого отсутствовал экзон 6, и транскрипт D, который содержал вставку гуанинового нуклеотида на 3'-конце экзона 6, что, как было спрогнозировано, приведет к преждевременному укорочению белка. Транскрипты проверяли с помощью RT-PCR и секвенирования по Сэнджеру (данные не показаны). Также транскрипт D проверяли с помощью секвенирования кДНК с длинными участками считывания. Уровни экспрессии этих транскриптов варьировали в соответствии с генотипом rs72613567 HSD17B13; уровни транскрипта А снижались, тогда как уровень транскриптов D возрастал зависимым от дозы аллеля образом с каждым аллелем ТА (см. фиг. 3А, 3D и 10В). Транскрипт А, кодирующий полноразмерный белок из 300 аминокислот, был преобладающим транскриптом у Т/Т гомозигот, тогда как транскрипт D, кодирующий преждевременно укороченный белок, был преобладающим транскриптом у ТА/ТА гомозигот. В ткани биоптата печени человека укороченный белок изоформы D минимально присутствовал у гетерозигот и ТА/ТА гомозигот, а содержание белка изоформы А снижалось зависимым от дозы аллеля образом (см. фиг. 10В и 10С). Эти данные указывали на rs72613567 HSD17B13, который изменял сплайсинг мРНК, что приводило к синтезу укороченной формы белка со значительно сниженной экспрессией в печени человека.
[00440] Обратимся к фиг. 10А-10Е, на которых показана экспрессия, субклеточная локализация и ферментативная активность нового транскрипта HSD17B13. На фиг. 3А и 3D показана экспрессия транскриптов А и D HSD17B13 у гомозиготных эталонных (Т/Т), гетерозиготных (Т/ТА) и гомозиготных альтернативных (ТА/ТА) носителей сплайс-варианта rs72613567 HSD17B13. Кодирующие участки в генных моделях показаны в заштрихованных прямоугольниках, а нетранслируемые участки - в черных прямоугольных диаграммах. Звездочкой в транскрипте D показана вставка G из rs72613567 на 3'-конце экзона 6, что приводит к преждевременному укорочению белка. Экспрессия мРНК отображена в единицах FPKM (количество фрагментов на тысячу оснований транскрипта на миллион картированных считываний). Из результатов вестерн-блоттинга с клеток HepG2, сверхэкспрессирующих транскрипты А и D HSD17B13, видно, что транскрипт D HSD17B13 транслировался в укороченный белок с более низкой молекулярной массой по сравнению с транскриптом А HSD17B13 (см. фиг. 10А). Схожие результаты наблюдали с вестерн-блоттингом HSD17B13 из образцов свежезамороженной печени человека и клеток HEK293 (см. фиг. 10В). Образцы печени человека были взяты у гомозиготных эталонных (Т/Т), гетерозиготных (Т/ТА) и гомозиготных альтернативных (ТА/ТА) носителей сплайс-варианта rs72613567 HSD17B13. Образцы клеток были взяты из клеток HEK293, сверхэкспрессирующих немеченые транскрипты А и D HSD17B13. Транскрипт HSD17B13 транслировался в укороченный белок изоформы D (IsoD) с более низкой молекулярной массой, чем HSD17B13 изоформы A (IsoA). Уровни белка IsoD HSD17B13 были ниже уровней белка IsoA как из образцов печени человека (слева), так и из клеток (справа) (см. фиг. 10С). Уровень белка, нормализованный к актину, показан в виде столбцов на фиг. 10С; ** Р<0,001, * Р<0,05. Как изоформа А, так и D HSD17B13 локализовались на мембране липидных капель у HepG2, стабильно сверхэкспрессирующих HSD17B13, транскрипты А или D метили BODIPY для демонстрации липидных капель и антителами к Мус для демонстрации локализации HSD17B13 (данные не показаны). Также оценивали ферментативную активность изоформ А и D HSD17B13 в отношении 17-бета-эстрадиола (эстрадиола), лейкотриена В4 (LTB4) и 13-гидроксиоктадекадиеновой кислоты (13(S)-HODE) (см. фиг. 10D). У изоформы D HSD17B13 наблюдали <10% ферментативной активности относительно соответствующих значений для изоформы А. В случае изоформы D HSD17B13 при сверхэкспрессии в клетках HEK293 не наблюдали значительного превращения эстрадиола (субстрата) в эстрон (продукт) при измерении в культуральной среде, в то время как при сверхэкспрессии изоформы А HSD17B13 наблюдали устойчивое превращение (см. фиг. 10Е).
[00441] HSD17B13 экспрессируется преимущественно в печени (Liu et al., Acta Biochim. Pol., 2007, 54, 213-8, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), где он локализуется в липидных каплях (Su et al., Proc. Natl. Acad. Sci. USA, 2014, 111, 11437-42, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), что согласуется с ролью в патогенезе жировой болезни печени. Экспрессию HSD171B3 и его локализацию оценивали в иммортализированной линии клеток печени человека, стабильно трансдуцированной лентивирусом, экспрессирующим транскрипты А и D HSD17B13. Изоформу А HSD17B13 преимущественно детектировали на мембранах, окружающих меченные посредством BODIPY липидные капли (данные не показаны). Схожую субклеточную локализацию наблюдали для изоформы D HSD17B13 на поверхности липидных капель (см. фиг. 10D).
[00442] Чтобы понять функциональные последствия преждевременного укорочения белка HSD17B13, вызванной rs72613567:ТА, оценивали ферментативную активность изоформ А и D in vitro с помощью рекомбинантного белка. Исследовали более 300 предполагаемых субстратов, из которых эстрадиол, лейкотриен В4 и 13-гидроксиоктадекадиеновая кислота подвергались ферментативному превращению посредством HSD17B13, что приводило к окислению гидроксила до кетонной группы. У изоформы D HSD17B13 наблюдали значительное снижение активности по отношению к данным трем субстратам (см. фиг. 10D).
[00443] По сравнению с контрольными клетками с GFP клетки со сверхэкспрессией транскрипта А HSD17B13 имели более низкую концентрацию эстрадиола, а также более высокую концентрацию эстрона в среде для культивирования клеток, что свидетельствовало о ферментативной активности в отношении эстрадиола (см. фиг. 10Е). Клетки со сверхэкспрессией транскрипта D HSD17B13 имели схожее соотношение эстрона/эстрадиола к контрольным клеткам с GFP, что свидетельствовало о том, что транскрипт D HSD17B13 характеризовался значимой потерей функции. По результатам масс-спектрометр ического анализа выявляли быстрое превращение эстрона в гидроксиэстрон и другие продукты, что объясняет низкое накопление эстрона по сравнению с потребляемым эстрадиолом.
[00444] Посредством крупномасштабного секвенирования экзома была выявлена новая ассоциация между сплайс-вариантами HSD17B13 и сниженными сывороточными уровнями трансаминаз, а также сниженным риском развития неалкогольных и алкогольных форм заболевания печени, в том числе запущенных циррозных форм заболевания печени и НСС. Насколько нам известно, это первое сообщение об изменяющем белок варианте, который характеризуется защитной ассоциацией с хроническим заболеванием. Аллель rs72613567:TA HSD17B13 не имел ассоциации с простым стеатозом, но уменьшал риск прогрессирования до NASH. Согласованность дозозависимых защитных ассоциаций в четырех независимых группах (DiscovEHR, независимая группа бариатрической хирургии в DiscovEHR, DLS и DPLS) по нескольким различным категориям заболеваний печени и этнической принадлежности подтверждала идею о том, что раскрываемый вариант HSD17B13 защищает от прогрессирования до более клинически запущенных стадий хронического заболевания печени. Наблюдаемая зависимость от дозы аллеля также доказывает, что более глубокая регуляция функции HSD17B13 может привести к более сильным эффектам на риск появления и прогрессирование заболевания.
[00445] Описываемые в настоящем документе результаты по ассоциациям в основном были основаны на наблюдениях, сделанных на американцах европейского и латиноамериканского происхождения, у которых был повышенный BMI. HSD17B13 находится в непосредственной близости от HSD17B11, представителя того же семейства генов, с высоким сходством последовательности с HSD17B13, но с более широким распределением в тканях. В целом, представленные в настоящем документе данные подтверждают точку зрения, что HSD17B13 является потенциальной терапевтической мишенью для предупреждения и лечения жировой болезни печени у людей. Представленные в настоящем документе данные свидетельствуют, что целенаправленное воздействие на HSD17B13 может снизить прогрессирование заболевания печени от стеатоза до более запущенных стадий NASH, фиброза и цирроза печени, которые ассоциированы со значительной заболеваемостью и смертностью и для которых в настоящее время нет эффективных средств лечения.
Пример 3 Вариант 17бета-гидроксистероиддегидрогеназы 13 защищает от хронического заболевания печени
[00446] Для выявления генетических факторов, вносящих вклад в развитие хронического заболевания печени, использовали данные о последовательности экзома и электронные медицинские записи от 46544 участников исследования DiscovEHR по генетике человека. Были выявлены генетические варианты, ассоциированные с известными биомаркерами повреждения печени (сывороточная аланинаминотрансфераза (ALT) и аспартатаминотрансфераза (AST)), для определения кандидатов, которые могут быть ассоциированы с хроническим заболеванием печени. Затем варианты-кандидаты, повторяющиеся в трех дополнительных группах (12527 индивидуммов), оценивали в отношении ассоциации с клиническими диагнозами хронического заболевания печени в DiscovEHR и в двух независимых группах (всего 37892 индивидумма). Также исследовали связь с гистопатологической тяжестью заболевания печени в независимой группе бариатрической хирургии (n=2391 образец печени человека).
[00447] Сплайс-вариант (rs72613567:TA) HSD17B13, кодирующий белок в липидной капле клеток печени под названием 17-бета-гидроксистероиддегидрогеназа 13, имел воспроизводимую ассоциацию со сниженными уровнями ALT (P=4,2×l0-12) и AST (P=6,2×10-10). В DiscovEHR данный вариант был ассоциирован со сниженным риском развития алкогольной и неалкогольной болезни печени (на 38%, 95% доверительный интервал (CI) 19%-52%; и на 16%, 95% CI 9%-22% соответственно для каждого аллеля rs72613567:TA) и цирроза (на 44%, 95% CI 22-59%; и на 26%, 95% CI 12%-38% для алкогольного и неалкогольного цирроза соответственно для каждого аллеля rs72613567:TA) зависимым от дозы аллеля образом; ассоциации были подтверждены в двух независимых группах. rs72613567:TA был ассоциирован с уменьшенной тяжестью гистологических признаков неалкогольного стеатогепатита (NASH) (23% снижение, 95% CI 10%-34% для каждого аллеля rs72613567:TA среди индивидуумов с жировой болезнью печени). rs72613567:TA давал в результате нестабильный и укороченный белок со сниженной ферментативной активностью в отношении стероидных субстратов.
[00448] Вариант с потерей функции HSD17B13 был ассоциирован со сниженным риском развития алкогольной и неалкогольной болезни печени и прогрессирования от стеатоза до NASH.
Схема исследования и участники
[00449] Исследования в области генетики человека были проведены в рамках сотрудничества по программе DiscovEHR центра генетики Regeneron Genetics Center и Geisinger Health System (GHS). Два контингента из исследования DiscovEHR (исследуемая группа и группа бариатрической хирургии) были созданы из первых 50726 участников, достигших возраста согласия ≥18 лет, из программы Community Health Initiative MyCode® компании GHS. Группа исследования GHS состояла из 46544 европейцев, набранных из поликлиник и специализированных клиник в период между 2007 и 2016 годами, за исключением набранных в группу бариатрической хирургии. В группу бариатрической хирургии GHS вошли 2644 европейца, которые были направлены на бариатрическую хирургию.
[00450] Репликационные исследования ассоциаций с печеночными трансаминазами включали 1357 европейцев из кардиологического исследования в Далласе и 8527 европейцев из медицинского биобанка Penn Medicine Biobank. Кардиологическое исследование в Далласе представляло собой вероятностное исследование групп контингента резидентов округа Даллас возрастом 30-65 лет (Victor et al., Am. J. Cardiol., 2004; 93, 1473-80, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Penn Medicine Biobank включает участников, набранных из системы здравоохранения Университета Пенсильвании (University of Pennsylvania Health System) и давших согласие на хранение биологических образцов, доступ к данным из EHR и разрешение на повторный контакт.
[00451] Репликационные исследования ассоциаций с хроническим заболеванием печени включали 517 индивидуумов из исследования печени в Далласе (Dallas Liver Study - DLS) и 447 человек из исследования печени у детей в Далласе (Dallas Pediatric Liver Study - DPLS). DLS представляет собой биобанк пациентов с заболеванием печени невирусной этиологии. Набор участников начался в январе 2015 года и продолжается по сей день. Участников набирали из клиник по лечению заболеваний печени в системе UT Southwestern and Parkland Health and Hospital System, в Далласе. Участники заполняли опросник об этническом/расовом происхождении, истории болезни, факторах образа жизни и семейном анамнезе заболевания печени и других заболеваний. Дополнительная клиническая информация была получена из медицинских карт с помощью обученного специалиста. Были включены все пациенты, которые были афроамериканцами, американцами европейского происхождения и латиноамериканцами с ДНК, имеющейся на момент настоящего исследования (n=517) с контролями из кардиологического исследования в Далласе. DPLS представляет собой биобанк латиноамериканских детей, набранных из клиник по лечению заболеваний печени у детей в UT Southwestern and Parkland Health and Hospital System, Даллас, и из клиники по лечению ожирения в Детском медицинском центре, Даллас. Клиническая информация была получена из медицинских карт с помощью обученного специалиста. Поскольку более 95% пациентов были латиноамериканцами, в настоящее исследование были включены только латиноамериканские пациенты и контрольные пациенты (n=205 пациентов и 234 контрольных пациентов).
Клинические измерения и определения хронического заболевания печени в группе исследования
[00452] Результаты клинических лабораторных измерений ALT и AST получали из EHR участников из группы исследования GHS и группы бариатрической хирургии. Медианные значения ALT и AST рассчитывали для всех участников с двумя или более результатами измерениями и преобразовывали в log10 для нормализации распределения перед проведением анализа ассоциаций.
[00453] Коды диагнозов заболеваний по Международной классификации болезней девятой ревизии (ICD-9) получали из EHR и сводили в категории клинических заболеваний для определения невирусных, неалкогольных (ICD-9 571.40, 571.41, 571.49, 571.5, 571.8, 571.9) или алкогольных (ICD-9 571.0, 571.1, 571.2, 571.3) случаев заболевания печени. Дополнительные определения случаев, основанные на единичных диагностических кодах, включали: алкогольный цирроз (ICD-9 571.2), неалкогольный цирроз (ICD-9 571.5) и НСС (ICD-9 155.0). Для определения таких случаев общую контрольную группу без заболевания печени («нет заболевания печени») определяли как участников, не имеющих критериев такого случая или имеющих диагностический код однократного проявления или диагностический код перечня проблем, указывающий на любой тип заболевания печени.
Определения гистопатологического фенотипа печени в группе бариатрической хирургии
[00454] Группа бариатрической хирургии GHS состояла из 2644 индивидуумов европейского происхождения. От 2391 из этих индивидуумов в ходе бариатрической операции были взяты клинообразные биоптаты печени. Каждый раз биоптаты брали на расстоянии 10 см слева от серповидной связки перед любой ретракцией печени или хирургической операцией на желудке. Биоптат разделяли на срезы, первичный срез передавали клиническим патоморфологам для проведения гистологии печени (фиксированный в 10% забуференном до нейтрального рН формалине и окрашенный гематоксилином и эозином для стандартной гистологии и трихромом Массона для оценки фиброза), а остальные срезы хранили в исследовательском биобанке (замороженными в реагенте для стабилизации РНК RNAlater и/или в жидком азоте). Гистологию печени проводил опытный патоморфолог, а затем повторный анализ проводил второй опытный патоморфолог с применением системы оценки the NASH Clinical Research Network (Kleiner et al., Hepatology, 2005, 41, 1313-21, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) следующим образом: степень стеатоза 0 (<5% вовлечение паренхимы), 1 (от 5 до <33%), 2 (от 34 до <66%) и 3 (>67%); степень лобулярного воспаления 0 (без очагов), степень 1 (слабое, <2 очагов на 200× поле), степень 2 (умеренное, 2-4 очага на 200× поле), степень 3 (тяжелое, >4 очагов на 200× поле); стадия фиброза 0 (нет), стадия 1 (перисинусоидальный или перипортальный фиброз), стадия 2 (перисинусоидальный и перипортальный фиброз), стадия 3 (мостовидный фиброз) и стадия 4 (цирроз). Эти гистологические диагнозы использовали для определения следующих фенотипов: 1) нормальный: нет признаков стеатоза, NASH или фиброза; 2) простой стеатоз: стеатоз (независимо от степени тяжести) без признаков NASH или фиброза; 3) NASH: любое присутствие лобулярного воспаления или вспучивания клеток печени (независимо от степени тяжести) или любое наличие фиброза (независимо от стадии); 4) фиброз: любое наличие фиброза (независимо от стадии).
Подготовка образцов, секвенирование и генотипирование
[00455] Подготовку образцов ДНК и секвенирование всего экзома у участников исследования DiscovEHR, кардиологического исследования в Далласе и медицинского биобанка Penn Medicine Biobank проводили в компании Regeneron Genetics (Dewey et al., Science In Press, 2016, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). В исследовании печени в Далласе и исследовании печени у детей в Далласе rs72613567 HSD17B13 генотипировали с помощью анализа Taqman (и проверяли с помощью секвенирования по Сэнджеру у 5 индивидуумов каждого генотипа).
[00456] В частности, захват экзома проводили с помощью зондов NimbleGen в соответствии с рекомендованным производителем протоколом (Roche NimbleGen). Захваченную ДНК амплифицировали с помощью ПЦР и количественно оценивали с помощью qRT-PCR (Кара Biosystems). Мультиплексированные образцы секвенировали с помощью секвенирования спаренных концов размером 75 п. о. на Illumina v4 HiSeq 2500 до глубины охвата, достаточной для обеспечения более чем 20-кратной глубины гаплоидного считывания более 85% целевых оснований у 96% образцов (примерно 80-кратная средняя глубина гаплоидного считывания целевых оснований). Необработанные данные по последовательностям из каждого цикла Illumina Hiseq 2500 загружали на платформу DNAnexus (Reid et al., BMC Bioinformatics, 2014, 15, 30, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) для выравнивания результатов считывания последовательностей и выявления вариантов. Вкратце: необработанные данные о последовательностях преобразованы из файлов BCL в специфичные по образцу FASTQ-файлы, для которых проводили выравнивание с человеческой эталонной сборкой GRCh37.p13 с BWA-mem (Li et al., Bioinformatics, 2009, 25, 1754-60, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Однонуклеотидные варианты (SNV) и варианты последовательностей со вставками/делециями (indel) выявляли с помощью инструментария Genome Analysis Toolkit (McKenna et al., Genome Res., 2010, 20, 1297-303, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей).
Анализ полноэкзомных ассоциаций ферментов печени и фенотипов хронических заболеваний печени
[00457] Использовали линейные смешанные модели для тестирования 502219 двухаллельных вариантов, которые имели степень потерянной информации <1%, р-значение закона Харди-Вайнберга >1,0×10-6 и частоту минорного аллеля >0,1%, с целью определения ассоциации с уровнями трансаминаз. В случае вариантов с полногеномными значимыми ассоциациями с трансамин азами (p<1×10-7) в группе исследования GHS проводили анализы ассоциаций и метаанализ в описанных выше репликационных исследованиях на лицах европейского происхождения. Для определения реплицированных ассоциаций использовали порог значимости Бонферрони, определяемый количеством протестированных вариантов. Также проводили метаанализ результатов поисковых и репликационных исследований. Все раскрытые в тексте р-значения соответствовали аллельной модели.
[00458] Затем тестировали ассоциированные с транс аминазами однонуклеотидные варианты в отношении ассоциаций с фенотипами хронических заболеваний печени. Для определения значимости ассоциаций использовали порог значимости Бонферрони, определяемый количеством вариантов и широкими категориями хронических заболеваний печени. Дополнительно тестировали реплицируемые новые варианты в отношении ассоциации с определенными с точки зрения гистопатологии фенотипами печени из группы бариатрической хирургии GHS. Также проводили полнофеномный поиск ассоциаций реплицируемых новых вариантов с 405 количественными клиническими показателями и 3168 клиническими диагнозами.
[00459] В частности, было протестировано 502219 двухаллельных вариантов со степенью потерянной информации <1%, P-значением закона Харди-Вайнберга >1,0×10-6 и частотой минорного аллеля >0,1% для ассоциации с уровнями трансаминаз. Log10-преобразованные медианные ALT и AST корректировали по возрасту, возрасту2, полу, BMI и первым четырем основным компонентам происхождения. Для учета родства среди участников исследования также использовали матрицу генетического родства в качестве ковариаты случайных эффектов. Как основные компоненты, так и матрица генетического родства были сконструированы из 39858 не относящихся к МНС маркеров в примерном равновесии связей и с частотой минорного аллеля >0,1%. Использовали линейные смешанные модели, реализованные в пакете GCTA (Yang et al., Am. J. Hum. Genet., 2011, 88, 76-82, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) для тестирования связи между невязками признаков и однонуклеотидными вариантами. Все раскрытые в тексте р-значения соответствовали аллельной модели.
[00460] Была предпринята попытка реплицировать ассоциации в группе исследования GHS, в трех отдельных группах с европейским происхождением: в группе бариатрической хирургии GHS, кардиологического исследования в Далласе и медицинского биобанка Penn Medicine Biobank (описано выше). Результаты измерений ALT и AST из группы бариатрической хирургии GHS и из медицинского биобанка Penn Medicine Biobank преобразовывали в log10 и корректировали по возрасту, возрасту2, полу, BMI и первым четырем основным компонентам происхождения. Включали матрицы генетического родства как ковариаты случайных эффектов и проводили анализ с использованием линейных смешанных моделей в GCTA. В кардиологическом исследовании в Далласе, преобразованные в log10 показатели ALT и AST корректировали по возрасту, возрасту2, полу, BMI и первым десяти основным компонентам происхождения и проводили анализ с использованием линейной регрессии, реализованной в PLINK. Сводная статистика для трех репликационных групп была подвергнута метаанализу с помощью METAL (Willer et al., Bioinformatics, 2010, 26, 2190-1, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей) (репликационного метаанализа). Аналогичным образом была подвергнута метаанализу сводная статистика для исследуемой группы и трех репликационных групп (совместный метаанализ).
Анализ ассоциаций с фенотипами хронического заболевания печени
[00461] Анализировали тринадцать значимых и реплицированных однонуклеотидных вариантов из ExWAS ферментов печени в отношении ассоциаций с фенотипами хронического заболевания печени, определенными из исследуемой группы GHS, как описано выше. Использовали порог значимости Бонферрони Р<0,05/26 (Р<1,92×10-3) для учета протестированных тринадцати вариантов и двух обширных категории хронических заболеваний печени (алкогольных и неалкогольных). Вариант rs72613567 HSD17B13 дополнительно тестировали в отношении ассоциации с определенными с точки зрения гистопатологии фенотипами печени из группы бариатрической хирургии GHS, как описано выше. Отношения шансов оценивали с помощью вероятностного способа логистической регрессии с поправкой Фирта после коррекции по возрасту, возрасту2, полу, BMI и первых четырех основных компонентов происхождения. Генотипические отношения шансов оценивали для rs72613567 HSD17B13, используя те же ковариаты.
[00462] Отношения шансов для заболевания печени в DLS оценивали с помощью логистической регрессии с коррекцией по возрасту, возрасту2, полу, индексу массы тела и сообщенной участником этнической принадлежности. Участников кардиологического исследования в Далласе с имеющимися генотипами rs72613567 использовали в качестве нормальных контролей (n=4279). Отношения шансов в DPLS оценивали с помощью логистической регрессии.
Полнофеномный поиск ассоциаций rs72613567 HSD17B13
[00463] Проводили полнофеномный поиск ассоциаций rs72613567 HSD17B13 с 405 количественными, полученными из EHR антропометрическими показателями, показателями жизненно важных функций, лабораторными, электрокардиографическими и эхокардиографическими показателями и показателями, полученными в результате денситометрии кости, а также с 3168 полученными из EHR клиническими диагнозами. Медианные лабораторные значения для индивидуумов с серийными амбулаторными показателями рассчитывали после удаления вероятных ложных значений, которые были > 3 стандартных отклонений от внутрииндивидуального медианного значения; также рассчитывали максимальные и минимальные значения. Затем рассчитывали невязки признаков для всех лабораторных признаков после корректировки по возрасту, возрасту2, полу и первым десяти основным компонентам происхождения и применяли соответствующие преобразования перед проведением анализа ассоциаций. Диагностические коды на основе ICD-9 объединяли в иерархические группы клинических заболеваний и соответствующие контроли с помощью модифицированной версии методики объединения в группы, предложенной Denny et al (Denny et al., Nature Biotechnology, 2013, 31, 1102-10 и Denny et al., Bioinformatics, 2010, 26, 1205-10, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Для постановки диагноза на основе ICD-9 было необходимо одно или несколько из следующих: запись диагностического кода в перечне проблем или эпизодический диагностический код, зафиксированный по двум отдельным клиническим эпизодам в разные календарные дни.
[00464] Анализы ассоциаций с преобразованными невязками количественных клинических показателей проводили с использованием линейной регрессии, а анализы ассоциаций с клиническими диагнозами проводили с использованием логистической регрессии с коррекцией по возрасту, возрасту2, полу и первым четырем основным компонентам. Аллелям присваивали коды с использованием аддитивных (0 для эталонных аллельных гомозигот, 1 для гетерозигот и 2 для альтернативных аллельных гомозигот) и рецессивных (0 для эталонных аллельных гомозигот и гетерозигот, 1 для альтернативных аллельных гомозигот).
Программное обеспечение
[00465] Анализ генетических ассоциаций проводили с использованием программного обеспечения GCTA, версии 1.25.07, и PLINK, версии 1.9.0. Квантиль-квантиль график и график Манхэттена строили с использованием программного обеспечения R, версии 3.2.1 (R Project for Statistical Computing). Графики региональных ассоциаций строили с помощью LocusZoom (Pruim et al., Bioinformatics, 2010, 26, 2336-7, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей).
Исследования по секвенированию РНК
[00466] Качество и концентрацию РНК оценивали путем прогона общей РНК на чипе Agilent RNA Nano Bioanalyzer; все образцы имели число целостности РНК (RIN), превышавшее 8. Полиаденилированные РНК-транскрипты выделяли с использованием двух циклов обогащения микроносителями с олиго(dT)25 (Thermo Fisher Scientific). Образцы очищали и концентрировали с микроносителями RNAclean XP (Beckman Coulter) и подвергали тепловой фрагментации примерно до 140 пар оснований. Синтез первой цепи осуществляли с использованием обратной транскриптазы SuperScript III (Thermo Fisher Scientific) с применением случайных гексамеров; во время синтеза второй цепи dTTP заменяли на dUTP. Образцы обрабатывали в соответствии с нашим стандартным способом получения библиотеки ДНК, упомянутой выше для экзомов, с добавлением стадии с использованием урацил-ДНК-гликозилазы для получения библиотек для специфичного по цепи секвенирования.
Выявление и проверка новых транскриптов HSD17B13
[00467] Результаты считывания наносили на карту относительно Human.B38 с помощью программного обеспечения ArrayStudio® (OmicSoft®, Кэри, Северная Каролина), допуская два несоответствия. Для выявления новых транскриптов HSD17B13 использовали два подхода. Новые соединения экзонов искали относительно Gencode v24 с помощью ArrayStudio. De novo сборку транскриптов проводили с помощью Trinity (v2.2.0) с настройками по умолчанию. Строили индивидуальные генные модели с включением новых транскриптов HSD17B13 и производили количественную оценку транскриптов путем выравнивания результатов считывания с индивидуальной генной моделью. Результаты выравнивания белковых последовательностей для всех выявленных изоформ HSD17B13 показаны на фиг. 7А и 7В. RT-PCR на общей РНК из образцов печени человека проводили с использованием системы SuperScriptTM One-Step RT-PCR System с ДНК-полимеразой Taq DNA Polymerase PlatinumTM (ThermoFisher). Каждые 50 мкл реакционной смеси для RT-PCR содержали 1× реакционного микса, 500 нМ каждого из прямого и обратного праймеров (PST516: ATGAACATCATCCTAGAAATCCTTC (SEQ ID NO: 251) и PST517: ATCATGCATACATCTCTGGCTGGAG (SEQ ID NO: 252)), 1 мкл RT/Platinum Taq и 75 нг РНК. Условия циклирования: один цикл при 45°С в течение 30 минут; один цикл при 94°С в течение 2 минут; 40 циклов при 94°С в течение 20 секунд, 53°С в течение 30 секунд и 72°С в течение 90 секунд; один цикл при 72°С в течение 5 минут; затем удержание на уровне 10°С. Продукты очищали с помощью набора для очистки ПЦР QIAquick PCR Purification Kit (Qiagen) и подвергали прямому секвенированию по Сэнджеру с применением праймера DE002 (ATCAGAACTTCAGGCCTTGG (SEQ ID NO: 253)). Для выявления транскриптов В и С продукты RT-PCR прогоняли на 2% агарозном геле с окрашиванием посредством красителя SYBR GoldSYBR® Gold Nucleic Acid Gel Stain (ThermoFisher) и бэнды с ожидаемой молекулярной массой вырезали и очищали с использованием набора для экстракции из геля QIAquick Gel Extraction Kit (Qiagen), затем подвергали клонированию с использованием набора для клонирования ТТОРО® ТА Cloning Kit (ThermoFisher). Секвенирование клонов ТОРО проводили с применением праймеров для секвенирования M13F и M13R. Анализ последовательностей проводили с использованием программного обеспечения для анализа ДНК Sequencher (Gene Codes Corporation).
Полноразмерные транскрипты HSD17B13 амплифицировали непосредственно с 50 нг общей РНК с помощью системы SuperScript III One-step RT-PCR System с Platinum Taq High Fidelity (ThermoFisher Scientific) с применением геноспецифических праймеров в первом (GCAAAGCCATGAACATCATCC (SEQ ID NO: 254)) и последнем экзонах (TCTTGATGTAGTGGGAGTCGGATT (SEQ ID NO: 255)) для получения ампликона размером приблизительно 2,2 т.о. (транскрипта с максимальным прогнозируемым размером). Ампликоны проверяли на биоанализаторе Agilent Bioanalyzer. К ампликонам пришивали PacBio-совместимые баркодированные адаптеры и очищали с микроносителями PacBio РВ (Pacific Biosciences). Библиотеки объединяли в пул в равных количествах и секвенировали на одной ячейке для SMRT в течение 180 минут на платформе PacBio RSII. Данные подвергали демультиплексированию с использованием программного обеспечения PacBio software smrtanalysis v2.3 tool labelzmw, а затем анализировали с помощью ConsensusTools AmpliconAnalysis. Полученные в результате ампликоны сравнивали с генами HSD17B13 из базы данных RefSeq для определения статуса изоформы генотипа.
Субклеточная локализация изоформ HSD17B13
[00468] Клетки HepG2 культивировали в минимальной основной среде Игла, дополненной 10% эмбриональной бычьей сыворотки. Транскрипты А и D HSD17B13 субклонировали в остовные лентивирусные конструкции Myc-DDK и получали лентивирус. Клетки HepG2 инфицировали лентивирусом, несущим транскрипты HSD17B13. Стабильные клеточные линии, экспрессирующие каждый транскрипт HSD17B13, подвергали отбору с помощью 1-3 мг/мл сульфата генетицина G-418 в полной культуральной среде в течение двух недель. После фиксации изоформы HSD17B13 детектировали с помощью мышиного антитела к Мус. Липидные капли метили красителем BODIPY FL (Sigma). Вторичными антителами для иммунофлуоресценции были конъюгированные с Alexa Fluor 488 антитела осла к IgG кролика и конъюгированные с Alexa Fluor 594 антитела осла к IgG мыши (Jackson ImmunoResearch).
Количественная оценка экспрессии белка HSD171B3 в ткани биоптата печени человека и стабильных клеточных линиях
[00469] Образцы человеческой печени и клеточной массы гомогенизировали в ледяном 1× буфере для лизиса RIPA (EMD Millipore) в присутствии смесей протеаз и ингибиторов фосфатаз (ThermoFisher). Собирали надосадочную жидкость и использовали ее для определения концентрации белка с помощью анализа белка ВСА (ThermoFisher). Человеческие ткани и клеточные лизаты загружали и разделяли на SDS/PAGE-гелях (Bio-Rad) и переносили на PVDF-мембраны (Bio-Rad). Мембраны блокировали в течение 1 часа посредством 5% (масса/объем) молока в 1× TBS с добавлением 0,1% твина 20 (Bio-Rad). Мембраны инкубировали с антителом при 4°С в течение ночи против HSD17B13 (1:200, Thermo-Fisher) и В-актина (1:500, Cell Signaling Technology). Связанное антитело детектировали с использованием конъюгированного с HRP антитела к антителам кролика (1:10000, Jackson ImmunoResearch) и проявляли с помощью хемилюминесцентного реагента (ThermoFisher). Интенсивность бэндов количественно оценивали с помощью программного обеспечения Image J.
Полуколичественная ПЦР в режиме реального времени
[00470] РНК выделяли из клетки с использованием TRIzol® (Invitrogen, Карлсбад, Калифорния). Синтезировали кДНК первой цепи с помощью Superscript III RT (Invitrogen) и использовали для полуколичественной ПЦР на основе интрон-охватывающих праймеров. Для измерения уровня экспрессии транскриптов использовали систему для ПЦР в режиме реального времени QuantStudio 6 Flex Real-Time PCR System. Праймеры HSD17B13 и ТВР заказывали у IDT (Integrated DNA Technologies). Относительную экспрессию гена анализировали способом ΔΔCt с получением кратного изменения экспрессии, нормализованного по отношению к конститутивному гену ТВР (ΔCt).
Выделение и характеристика липидных капель с помощью вестерн-блоттинга
[00471] Липидные капли получали из клеток HepG2, стабильно экспрессирующих транскрипт А HSD17B13 (IsoA) или транскрипт D (IsoD), как сообщалось ранее (Brasaemle DL, Wolins NE. Isolation of lipid droplets from cells by density gradient centrifugation, Current protocols in cell biology 2006; Chapter 3:Unit 3 15 и Ding et al., Nature Protocols, 2013, 8, 43-51, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Вкратце: клетки HepG2, стабильно экспрессирующие IsoA, IsoD HSD17B13, или исходную линию инкубировали в течение ночи с 1 мМ олеиновой кислоты. После увеличения содержания липидов клетки собирали и ресуспендировали в буфере для гипотонического лизирования (20 мМ трис, рН 7,5, 1 мМ ЭДТА) с добавлением 1× ингибиторов протеаз/фосфатаз Halt™ (Thermo) и лизировали путем кавитации при 50 бар в течение 8 минут. Лизаты центрифугировали при 1000 g/4°C в течение 10 минут, и образовавшуюся после разрушения ядер надосадочную жидкость (PNS) смешивали с сахарозой до конечного объема 2 мл и концентрации 20% в пробирках для ультрацентрифугирования. Затем поверх лизата наслаивали 1,5 мл 5% сахарозы и еще 1,5 мл буфера для гипотонического лизиса. Пробирки центрифугировали при 182000 g/4°C в течение 40 минут и слои липидных капель (LD) переносили в новые пробирки. Отсасывали оставшийся объем в пробирке и осадок (общая мембрана, ТМ) ресуспендировали в 0,5 мл буфера для гипотонического лизиса. Фракции PNS, LD и ТМ смешивали с 1× буфером для радиоиммунопреципитации (RIPA) (EMD) + буфером для разведения образцов NuPAGETM LDS Sample Buffer (Thermo) и β-меркаптоэтанолом и обрабатывали ультразвуком в течение 3 часов при 37°С. Лизат ТМ разводили в 2,5 раза для нормализации к PNS. Лизаты гнали на 4-20% SDS-PAGE-гелях (Biorad), переносили с помощью Trans-Blot (Biorad) на PVDF-мембраны для слабой флуоресценции и блокировали в течение 1 часа в блокирующем буфере Odyssey TBS Blocking Buffer. Мембраны инкубировали в течение ночи со следующими антителами: α-HSD17B13 (Abgent, кат. № АР5729а 1:500); маркером LD: α-ADRP (Proteintech, 152-94-1-АР, 1:2500); маркером LD: α-TIP47 (Proteintech, 10694 1:2000); маркером лизосом: α-LAMPI (Novus, NBP2-25183, 1:1000); цитозольным маркером: α-GAPDH (Proteintech, 60004-1-Ig, 1:2000); маркером эндоплазматического ретикулума: α-кальретикулином (Abcam, ab92516, 1:1000); митохондриальным маркером: α-СОХ IV (Abeam, ab33985, 1:500); маркером цитоскелета: α-актином (Sigma, А5441, 1:4000). На следующий день мембраны промывали 4 раза забуференным при помощи Трис солевым раствором + 0,1% твина, затем инкубировали в течение 1 часа при комнатной температуре с блокирующим буфером, содержащим вторичные α-кроличьи (800CW) и α-мышиные (680RD) (Li-Cor) антитела IRDye® в разведениях 1:5000 и 1:10000 соответственно. Гели снова промывали посредством TBST и визуализировали с помощью Odyssey.
Количественная оценка внутриклеточного содержания триглицерида
[00472] Содержание триглицеридов (TG) из стабильных клеток определяли с помощью набора для количественного определения TG (Abeam). В данном анализе ТГ превращали в свободные жирные кислоты и глицерин. Затем глицерин окисляли с получением продукта, который количественно определяли (спектрофотометрией с λ=570 нм).
Субстратный скрининг библиотек стероидов и биоактивных липидов относительно очищенного рекомбинантного HSD17B13
[00473] Реакции проводили в конечном объеме 40 мкл аналитического буфера (0,2 М трис-HCl, рН 7,5), который содержал 500 мкМ NAD+, 5 мкМ биоактивного липида или 50 мкМ стероида (все в конечной концентрации 5% DMSO) и 100 нг рекомбинантного HSD17B13 человека. Реакционные смеси инкубировали в течение 3 часов, при 23°С, после чего добавляли равный объем реагента для детекции NADH-Glo Detection Reagent (Promega). После 1-часовой инкубации при 23°С измеряли относительные световые единицы (RLU) на планшет-ридере Envision Plate Reader (Perkin Elmer). Необработанные значения RLU нормализовали как процент контроля (50 мкМ эстрадиола) после вычитания отрицательного контроля (5% DMSO) согласно следующей формуле: процент контроля (РОС) = 100 × (образец (RLU) - среднее отрицательного контроля) / (среднее положительного контроля среднее отрицательного контроля).
In vitro и клеточная характеристика ферментативной активности HSD17B13
[00474] Рекомбинантный белок HSD17B13 человека очищали из Е. coli (Genscript), трансформированной плазмидной ДНК, несущей транскрипт А или транскрипт D HSD17B13. Варианты HSD17B13 содержали 10×His маркер на С-конце, и их очищали из растворимой фракции с использованием аффинной очистки на Ni2+. Ферментативную активность определяли путем измерения продуцирования NADH с помощью системы для детекции NAD(P)H-Glo Detection System (Promega). Реакции проводили в течение 3 часов при 25°С в 0,2 М трис-HCl, рН 7,5, 0,5 мМ NAD+, 75 мкМ субстрата (Sigma) и 500 нг очищенного фермента в конечном объеме 100 мкл. После инкубации 20 мкл реакционной смеси объединяли с 20 мкл люциферазного реагента (Promega), инкубировали при комнатной температуре в течение 1 часа и считывали на планшет-ридере Envision Plate Reader (Perkin Elmer).
[00475] Клетки HEK293, сверхэкспрессирующие транскрипт А, транскрипт D HSD17B13 или зеленый флуоресцентный белок (GFP, контроль), использовали для исследования активности HSD17B13 в отношении эстрадиола в клеточном анализе. Эстрадиол (1 мкМ) вносили в клетки каждого типа. Спустя 48 часов собирали среду и определяли концентрацию эстрадиола и продукта его превращения под названием эстрон и количественно оценивали с помощью ЖХ-МС.
Ассоциация экзонных вариантов с аспартат- и аланинаминотрансферазами
[00476] Тестировали 502219 двухаллельных одиночных генетических вариантов в отношении ассоциации с сывороточными уровнями ALT или AST у 46544 индивидуумов европейского происхождения из исследования DiscovEHR («группы исследования GHS»; основные демографические данные представлены в таблице 6). Всего было обнаружено 35 вариантов в 19 генах, которые связаны с ALT или AST при Р<1,0×10-7 (фиг. 1А и 1В, а также таблица 7). Проводили репликационные исследования на трех группах индивидуумов европейского происхождения: 1) пациентах с бариатрической хирургией (n=2644) из DiscovEHR («группа бариатрической хирургии GHS»), 2) 1357 индивидуумах из кардиологического исследования в Далласе и 3) 8526 индивидуумах из медицинского биобанка Penn Medicine Biobank. В метаанализе репликационных групп с сывороточными уровнями ALT или AST значимо были ассоциированы тринадцать вариантов в девяти генах (порог значимости по Бонферрони Р<1,43×10-3 для 35 протестированных вариантов, таблица 8). В их число входили варианты, для которых ранее было раскрыто, что они ассоциированы с повышенными уровнями трансаминаз, такие как PNPLA37, TM6SF211, SERPINA122, SAMM5023 и ERLIN124. SERPINA1 кодирует альфа-1-антитрипсин, функциональный дефицит которого вызывает наследственное заболевание печени; ассоциация с SAMM50 опосредована неравновесной связью с варьированием PNPLA3, a ERLIN1 участвует в отложении жира в печени. Также выявляли варианты, которые ранее не были раскрыты как ассоциированные с заболеванием печени. В их число входило несколько вариантов у GPT и GOT1, генов, кодирующих ALT и AST соответственно, и SLC39A12, который кодирует представителя 12 семейства транспортеров растворенных веществ 39.
[00477] Также выявляли воспроизводимую ассоциацию между вариантом HSD17B13, гена, кодирующего гидроксистероид-17-бета-дегидрогеназу 13, неохарактеризованного представителя семейства 17-бета-гидроксистероиддегидрогеназ, и сниженными уровнями ALT (Р обнаружения = 4,2×10-12, репликационный P=1.7×10-4) и AST (Р обнаружения = 6,2×10-10, репликационный P=1.7×10-4, таблица 8). Ассоциированный вариант rs72613567 представлял собой вставку аденина рядом с донорным сайтом сплайсинга шестого экзона (аллель ТА) и имел частоту аллеля 26,0% в группе исследования GHS. Ранее у Chambers et al. был выявлен близлежащий локус в 4q22 (rs6834314), ассоциированный с уровнями ALT (Chambers et al., Nat. Genet., 2011, 43, 1131-1138, doi:10.1038/ng.970, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей); rs72613567 до настоящего момента не был раскрыт как ассоциированный с уровнями трансаминаз. HSD17B13 находится на 30 т.о. выше HSD17B11, другого представителя того же семейства генов. Не наблюдали полноэкзомных значимых ассоциаций между кодирующими или сплайс-вариантами у HSD17B11 и уровнями трансаминаз в группе исследования (фиг. 5А и 5В) или в совместном метаанализе группы исследования и трех репликационных групп. Кроме того, дисбаланс связей rs72613567 с вариантами HSD17B11 было умеренным во всех группах этнического происхождения (r2<0,4 со всеми установленными вариантами в HSD17B11 во всех группах этнического происхождения). В совокупности эти результаты свидетельствовали о HSD17B13 в качестве гена в геномном участке, который наиболее вероятно функционально связан с уровнями трансаминаз.
[00478]
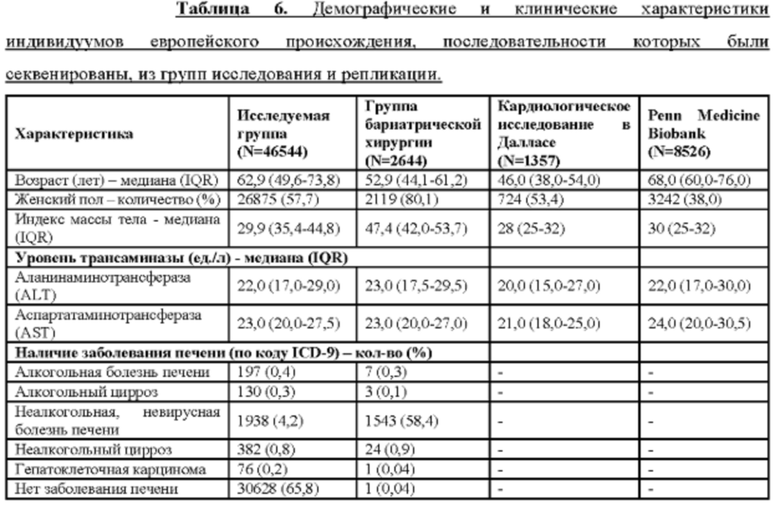
[00479] Таблица 7. Однонуклеотидные варианты, связанные с уровнями трансаминаз в сыворотке при P<1,0×10-7 в группе исследования.
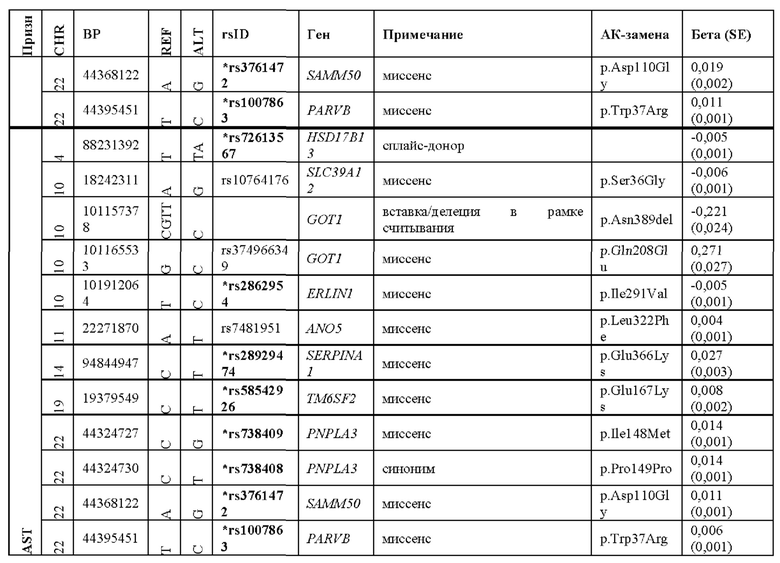
[00480]

[00481]
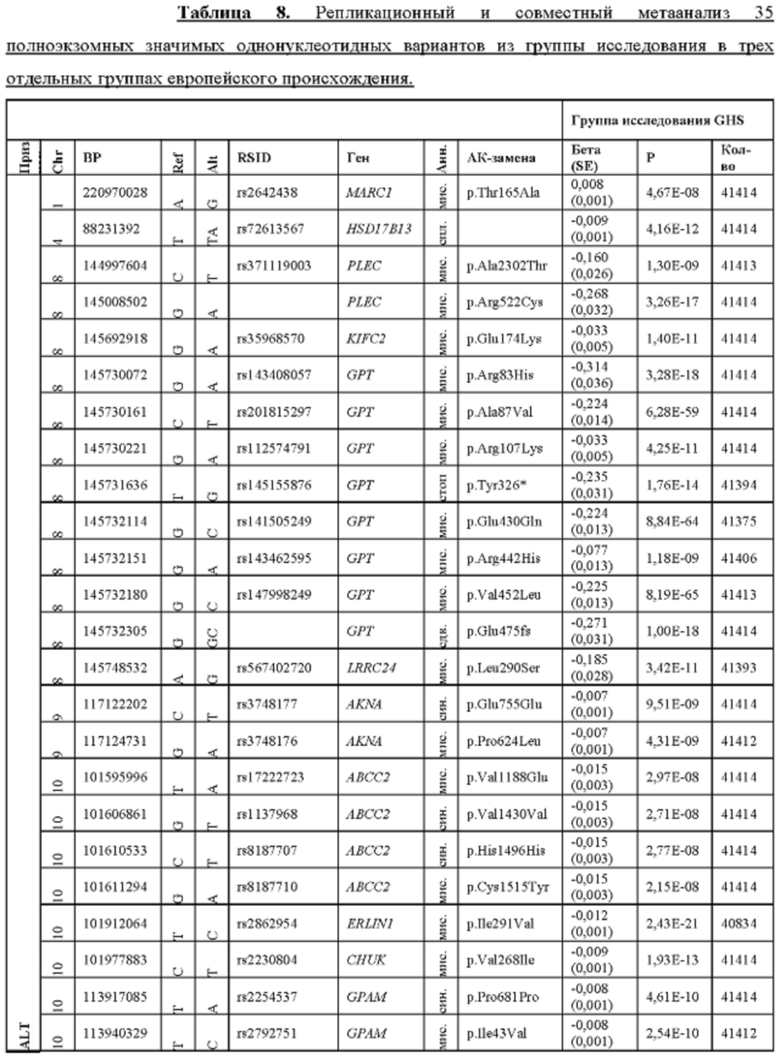
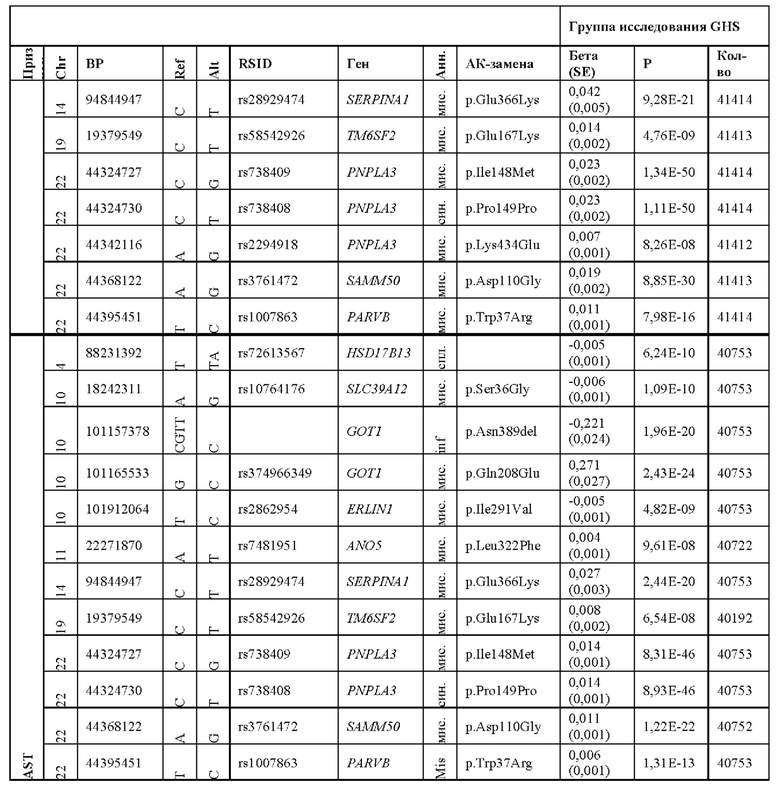


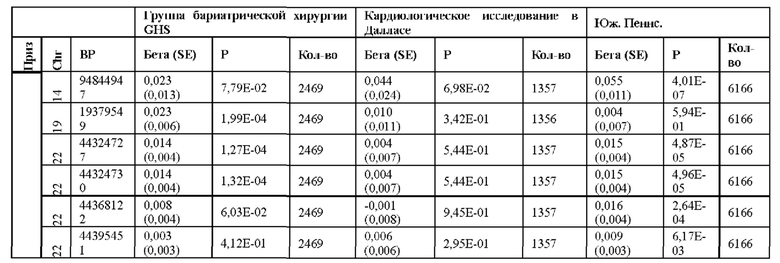
[00483]
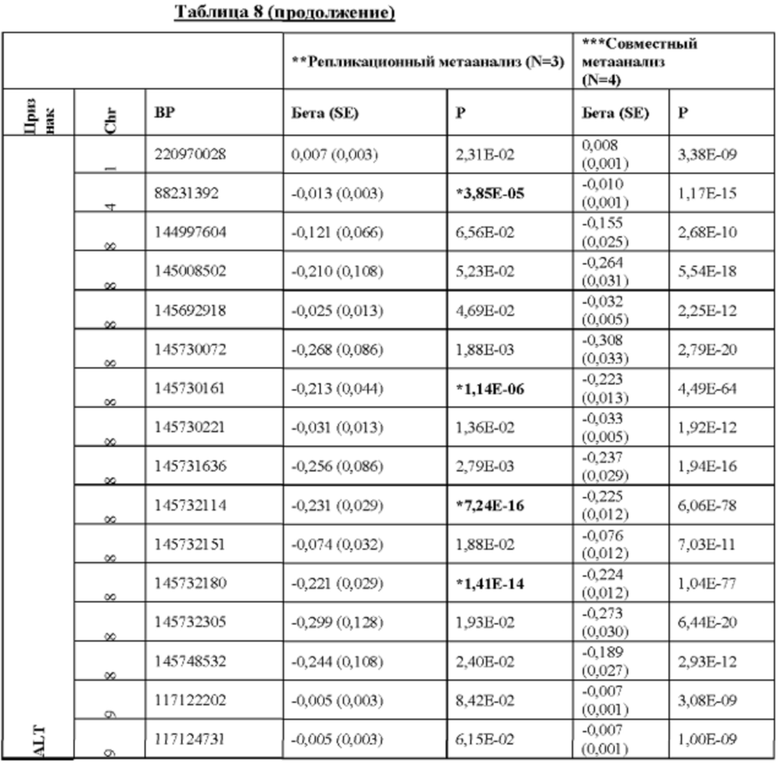
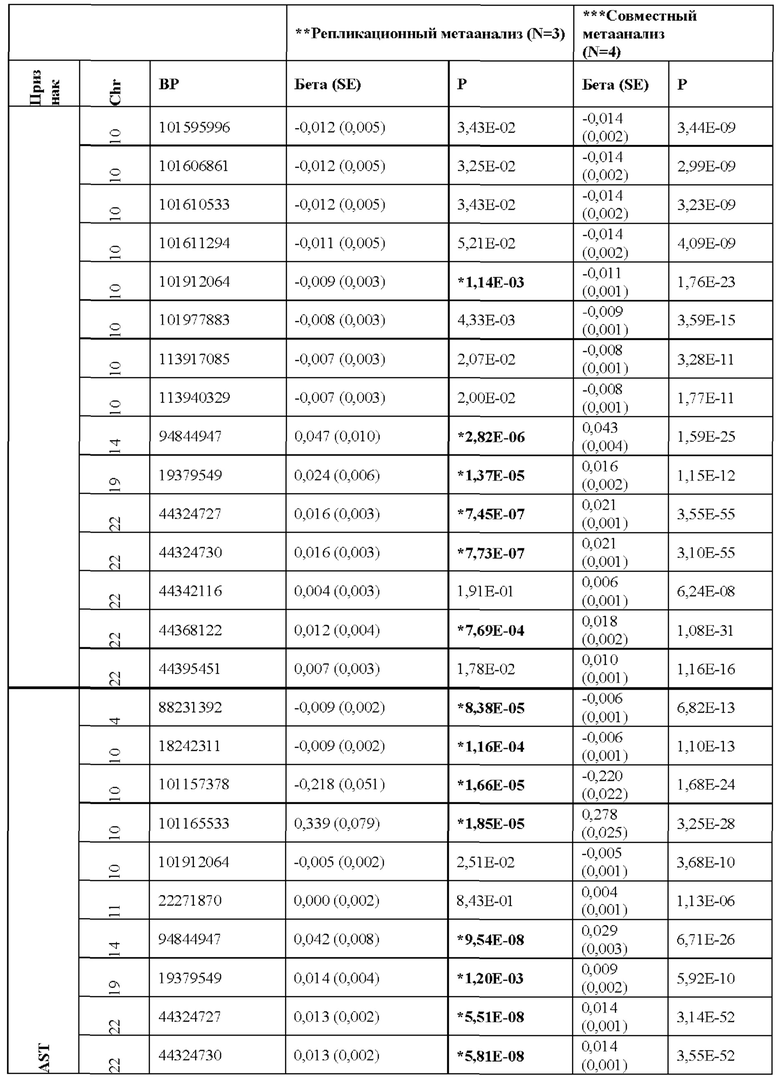
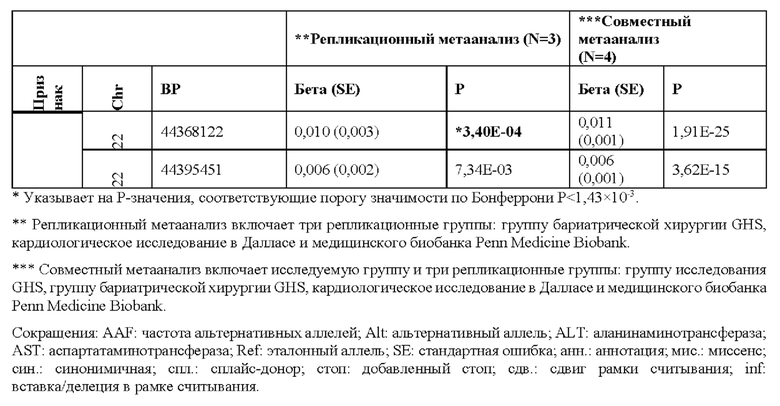
Ассоциация экэонных вариантов с клиническими диагнозами хронического заболевания печени
[00484] Затем анализировали взаимосвязь между тринадцатью ассоциированными с уровнями трансаминаз вариантами девяти генов, обнаруженных в группе исследования и репликационных группах, и хроническим заболеванием печени, в том числе алкогольной и неалкогольной (невирусной) болезнью печени, а также наиболее запущенными формами хронического заболевания печени: алкогольным циррозом, неалкогольным циррозом и гепатоклеточной карциномой (НСС). С помощью порога значимости по Бонферрони Р<1,92×10-3 для тринадцати протестированных вариантов были обнаружены значимые ассоциации между шестью вариантами пяти генов (HSD17B13, SERPINA1, TM6SF2, PNPLA3 и SAMM50) и фенотипами хронических заболеваний печени (таблица 9). Ассоциации SERPINA1, TM6SF2, PNPLA3 и SAMM50 подтверждали раскрытые ранее ассоциации. В группе исследования rs72613567:TA HSD17B13 был ассоциирован с более низкими шансами развития всех полученных из EHR категорий как алкогольной, так и неалкогольной болезни печени зависимым от дозы алелля образом (фиг. 2А): все категории алкогольной болезни печени, отношение шансов для гетерозигот (ORhet) (95% доверительный интервал) 0,58 (0,42-0,80), OR для гомозигот (ORhom) 0,47 (0,23-0,97), OR для аллельных вариантов (ORallelic) 0,62 (0,48-0,81), Р=1,8×10-4; все категории неалкогольной болезни печени: ORhet 0,83 (0,75-0,92), ORhom 0,70 (0,57-0,87), ORallelic 0,84 (0,78-0,91), Р=1,3×10-5. rs72613567:TA HSD17B13 также был ассоциирован с более низкими шансами развития алкогольного и неалкогольного цирроза, с 42% и 73% снижением шансов развития алкогольного цирроза в случае соответственно гетерозигот и гомозигот (ORhet 0,58 (0,39-0,86), ORhom 0,27 (0,09-0,85), ORallelic 0,56 (0,41-0,78), Р=3,4×10-4), и 26% и 49% снижением шансов развития неалкогольного цирроза в случае соответственно гетерозигот и гомозигот (ORhet 0,74 (0,60-0,93), ORhom 0,51 (0,31-0,85), ORallelic 0,74 (0,62-0,88), P=4,5×10-4). rs72613567:TA HSD17B13 также был номинально ассоциирован с более низкими шансами развития ННС.
[00485] Была предпринята попытка подтвердить и расширить эти результаты в многоэтническом исследовании печени в Далласе (DLS) и исследовании печени у детей в Далласе (DPLS) (таблица 10). В DLS аллель ТА был ассоциирован со снижением шансов развития какого-либо заболевания печени зависимым от дозы аллеля образом (ORhet 0,74 (0,57-0,97), ORhom 0,41 (0,21-0,83), ORallelic 0,70 (0,5-0,88), Р=1,8×10-3, фиг. 8). Схожие эффекты наблюдали для всех подтипов заболеваний печени, полученных из EHR, в том числе защитные ассоциации с распространенными циррозными формами алкогольной (ORallelic 0,72 (0,53-0,99), Р=4,4×10-2) и неалкогольной (ORallelic 0,65 (0,40-1,07), Р=9,0×10-2) болезни печени. В подгруппах анализов индивидуумов, сгруппированных по этнической принадлежности, о которой они сами сообщали, ассоциация с заболеванием печени была значимой у латиноамериканцов (n=326 случаев и 722 контроля, ORallelic 0,51 (0,35-0,74), Р=4,0×10-4); аналогичные числовые тенденции, которые не достигали статистической значимости, были также отмечены в подгруппах афроамериканцев (n=33 случая и 2291 контроль, ORallelic 0,74 (0,25-2,47), Р=0,67) и американцев европейского происхождения (n=158 случаев и 1266 контролей, ORallelic 0,87 (0,65-1,15), Р=0,32) из DLS. В DPLS, отдельном исследовании детей-пациентов латиноамериканцев с заболеванием печени и страдающих избыточным весом контролей, аллель ТА также был ассоциирован со снижением шансов развития заболевания печени (ORallelic 0,61 (0,37-0,99), Р=4,6×10-2). Так, rs72613567:TA HSD17B13 был ассоциирован со сниженными шансами развития множественных форм хронического заболевания печени, в том числе цирроза, у взрослых и детей в трех независимых контингентах.
[00486]
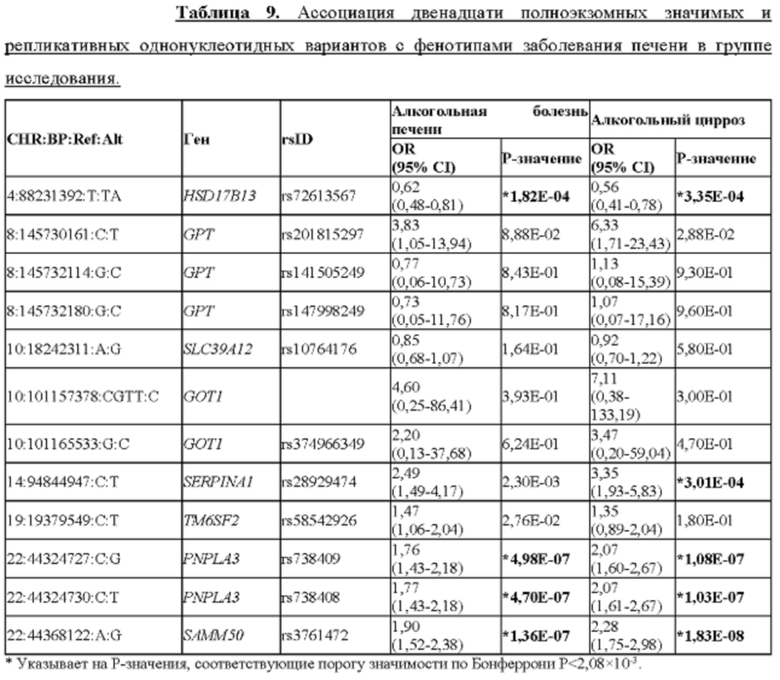
[00487]
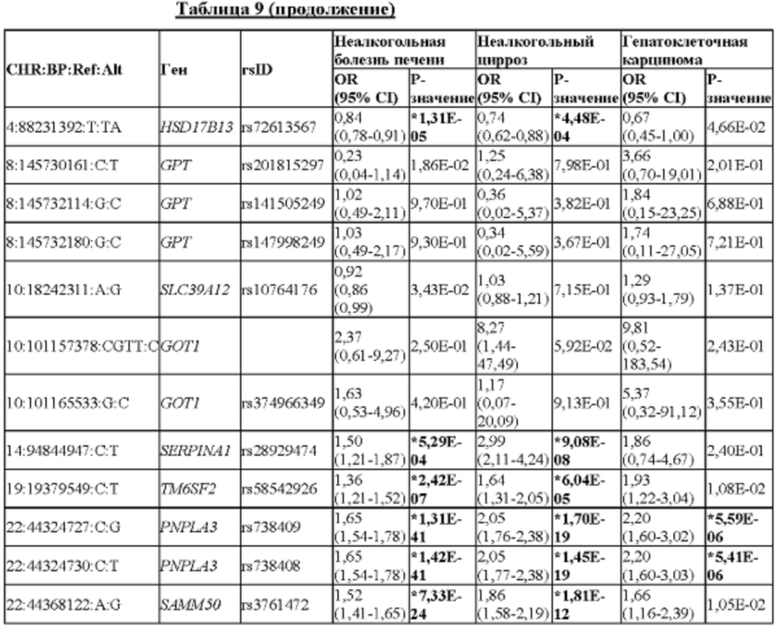
[00488]
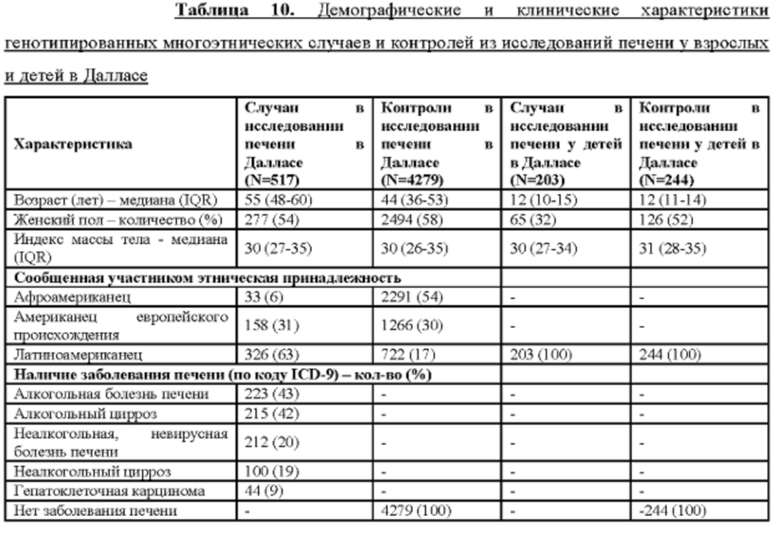
Ассоциация rs72613567:TA HSD17B13 с патологией печени
[00489] Под NAFLD описывается спектр заболеваний, варьирующий от накопления жира в печени без признаков значительного воспаления (простой стеатоз) до более клинически значимого NASH. Для подтверждения ассоциации между полученными из EHR диагностическими кодами заболеваний печени и rs72613567:TA HSD17B13 и дополнительного понимания его ассоциации с гистопатологическим прогрессированием стеатоза в NASH проводили тесты по выявлению ассоциации в группе бариатрической хирургии GHS. В данной группе из 2391 индивидуумов с полностью секвенированным экзомом, которых оценивали по результатам биопсии печени во время бариатрической хирургической операции, у 555 (23%) индивидуумов не было признаков стеатоза, стеатогепатита или фиброза («нормальные»), 830 (35%) имели простой стеатоз, а 1006 (42%) имели NASH. При сравнении доли случаев с нормальной печенью, простым стеатозом и NASH по генотипу наблюдали, что доля случаев с нормальной печенью, по-видимому, не отличалась по генотипу (23%, 24% и 23% соответственно для Т/Т, Т/ТА и ТА/ТА носителей, Р=0,5 по критерию хи-квадрат для тенденции в пропорциях), но что снижалась доля случаев с NASH (45%, 40% и 31% соответственно для Т/Т, Т/ТА и ТА/ТА носителей, Р=1,6×10-4) и что увеличивалась доля случаев с простым стеатозом (33%, 35% и 47% соответственно для Т/Т, Т/ТА и ТА/ТА носителей, P=1,1×10-3) с каждым аллелем ТА (фиг. 9). Среди индивидуумов со стеатозом аллель ТА был ассоциирован со статистически значимо более низкими шансами как NASH, так и фиброза, по сравнению с простым стеатозом (ORallelic 0,77 (0,66-0,90), Р=6,5×10-4 в случае NASH; ORallelic 0,74 (0,62-0,88), Р=4,15×10-4 в случае; фиг. 2В), зависимым от дозы аллеля образом. В целом, эти данные свидетельствовали о роли HSD17B13 в опосредовании прогрессирования NAFLD от простого стеатоза до более запущенных стадий NASH и фиброза.
Ассоциация rs72613567:TA HSD17B13 с клиническими количественными признаками и диагнозами
[00490] Для более исчерпывающего изучения клинических последствий сплайс-варианта HSD17B13 проводили полнофеномный поиск ассоциаций rs72613567:TA HSD17B13 с 405 количественными, полученными из EHR антропометрическими показателями, показателями жизненно важных функций, лабораторными, электрокардиографическими и эхокардиографическими показателями и показателями, полученными в результате денситометрии кости, а также с 3168 полученными из EHR клиническими диагнозами. С помощью значений порога значимости по Бонферрони 1,23×10-4 и 1,58×10-5 для ассоциаций с количественными клиническими показателями и клиническими диагнозами соответственно были выявлены статистически значимые ассоциации аллеля rs72613567:TA HSD17B13 с более высоким количеством тромбоцитов, помимо ассоциаций с трансаминазами печени (таблица 11). Статистически значимые ассоциации с клиническими диагнозами, отличными от хронического заболевания печени, выявлено не было (OR (95% CI) = 0,88 (0,84-0,93); Р=9,14×10-6; AAF = 0,263; всего случаев = 4031, Т/Т = 2331, Т/ТА = 1449, ТА/ТА = 251; всего контролей = 35701, Т/Т = 19238, Т/ТА = 13984, ТА/ТА = 2479).
[00491]

Влияние rs72613567:TA HSD17B13 на экспрессию мРНК HSD17B13 и белка HSD17B13
[00492] Затем изучали влияние аллеля rs72613567:TA HSD17B13 на экспрессию известных и новых транскриптов гена. Использовали секвенирование РНК для оценки экспрессии мРНК HSD17B13 в нормальных с точки зрения гистологии образцах печени, полученных от 22 Т/Т гомозиготных, 30 Т/ТА гетерозиготных и 17 ТА/ТА гомозиготных носителей сплайс-варианта rs72613567 HSD17B13. Помимо двух известных транскриптов HSD17B13, А и В, были выявлены два новых транскрипта: транскрипт С, у которого отсутствовал экзон 6, и транскрипт D, который содержал вставку гуанинового нуклеотида на 3'-конце экзона 6, что, согласно прогнозам, приводило бы к преждевременному укорочению белка. Четыре дополнительных транскрипта (Е-Н) характеризовались очень низкими уровнями экспрессии (фиг. 3A-3D и 6A-6D). Транскрипты проверяли с помощью RT-PCR и секвенирования по Сэнджеру. Также транскрипт D проверяли с помощью секвенирования кДНК с длинными участками считывания. Результаты выравнивания белковых последовательностей для всех выявленных изоформ (А-Н) HSD17B13 показаны на фиг. 7А и 7В. Уровни экспрессии этих транскриптов варьировали в соответствии с генотипом rs72613567 HSD17B13; уровни транскриптов А и В снижались, тогда как уровни транскриптов С и D возрастали зависимым от дозы аллеля образом с каждым аллелем ТА (фиг. 3A-3D). Транскрипт А, кодирующий полноразмерный белок из 300 аминокислот, был преобладающим транскриптом у Т/Т гомозигот, тогда как транскрипт D, кодирующий преждевременно укороченный белок, был преобладающим транскриптом у ТА/ТА гомозигот. В ткани биоптата печени человека укороченный белок изоформы D минимально присутствовал у гетерозигот и ТА/ТА гомозигот, а содержание белка изоформы А снижалось зависимым от дозы аллеля образом (фиг. 10В и 10С). Гетерологичная экспрессия изоформ А и D в клетках HEK 293 указывала на пониженное содержание изоформы D относительно экспрессии мРНК, что свидетельствовало о нестабильности изоформы D по сравнению с изоформой А (фиг. 11А-11С). Эти данные указывали на rs72613567 HSD17B13, который изменял сплайсинг мРНК, что приводило к синтезу укороченной формы белка со значительно сниженной экспрессией в печени человека.
Экспрессия HSD17B13 в клетках печени человека
[00493] HSD17B13 экспрессируется преимущественно в печени (Liu et al., Acta Biochim. Pol. 2007, 54, 213-218, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), где он локализуется в липидных каплях (Su et al., Proc. Natl. Acad. Sci. USA, 2014, 111, 11437-11442, doi:10.1073/pnas.1410741111, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), что согласуется с ролью в патогенезе жировой болезни печени. Экспрессию HSD17B13 и его локализацию оценивали в иммортализированной линии клеток печени человека, стабильно трансдуцированной лентивирусом, экспрессирующим транскрипты А или D HSD17B13. Изоформу А HSD17B13 преимущественно детектировали на мембранах, окружающих меченные посредством BODIPY липидные капли (данные не показаны). Схожую субклеточную локализацию наблюдали для изоформы D HSD17B13 на поверхности липидных капель (данные не показаны и фиг. 12). Не наблюдали никаких различий во внутриклеточном содержании триглицеридов при обработке олеиновой кислотой клеточных линий со сверхэкспрессией GFP-контроля или изоформ А или D HSD17B13 (фиг. 13A-13D).
Влияние rs72613567:TA на активность HSD17B13 in vitro и в клеточных моделях
[00494] Чтобы понять функциональные последствия преждевременного укорочения белка HSD17B13, вызванной rs72613567:TA, оценивали ферментативную активность изоформ А и D in vitro с помощью рекомбинантного белка и никотинамидаденозинового динуклеотида в качестве кофактора. Были протестированы 265 уникальных предполагаемых субстратов и выявлены стероидные субстраты и биоактивные липиды (например, лейкотриен В4) в качестве ферментативных субстратов HS17B13. При последующем анализе характеристик ферментативной активности HSD17B13 делали фокус на ферментативном превращении эстрадиола (значения Vmax и Km на фиг. 14), которое приводило к окислению гидроксильной группы до кетонной группы. В случае изоформы D HSD17B13 наблюдали значительно сниженную активность в отношении эстрадиола in vitro (фиг. 10D) и в клеточных анализах ферментативного превращения (фиг. 10Е) по сравнению с изоформой А HSD17B13.
[00495] Путем связывания результатов крупномасштабного секвенирования экзома с полученными из EHR клиническими фенотипами была выявлена новая ассоциация между сплайс-вариантом HSD17B13 и сниженными уровнями сывороточной трансами назы, а также сниженным риском неалкогольных и алкогольных форм болезни печени. Эти ассоциации одинаково наблюдали в четырех независимых группах и у нескольких различных категорий заболеваний печени, включая запущенные циррозные формы заболеваний печени и НСС. Аллель rs72613567:TA HSD17B13 не был ассоциирован с простым стеатозом, но был ассоциирован со сниженным риском развития NASH и фиброза, что позволяло предположить, что данный вариант аллеля защищает от прогрессирования до более клинически запущенных стадий хронического заболевания печени. В полнофеномном поиске ассоциаций rs72613567:TA HSD17B13 не был значимо ассоциирован с клиническими диагнозами или показателями, отличными от хронического заболевания печени и ассоциированных клинических показателей (печеночных трансаминаз и количества тромбоцитов), что свидетельствовало, что клинические эффекты варианта аллеля могут быть специфическими для хронического заболевания печени.
[00496] Другие представители семейства гидроксистероид-17-бета-дегидрогеназ вовлечены в метаболизм половых стероидов и жирных кислот (Moeller, Mol. Cell. Endocrinol., 2009, 301, 7-19, doi:10.1016/j.mce.2008.10.040, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей), но о функции HSD17B13 мало известно. Ранее было показано, что сверхэкспрессия HSD17B13 увеличивает липогенез в печени мыши и увеличивает количество и размер липидных капель в культивируемых гепатоцитах (Su et al., Proc. Natl. Acad. Sci. USA, 2014, 111, 11437-11442, doi:10.1073/pnas.1410741111, включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Из результатов двух ранее проведенных исследования также видно, что экспрессия в печени белка HSD17B13 увеличивалась у пациентов с жировым метаморфозом печени (Su et al., Proc. Natl. Acad. Sci. USA, 2014, 111, 11437-11442, doi:10.1073/pnas.l410741111 и Kampf et al., FASEB J., 2014, 28, 2901-2914, doi:10.1096/fj.14-250555, каждая из которых включена в настоящий документ посредством ссылки в полном своем объеме для всех целей). Наши данные позволяют предположить, что обе изоформы HSD17B13 экспрессируются на мембране липидной капли, но, по-видимому, не модулируют внутриклеточное содержание нейтрального жира, что отражается в отсутствии ассоциации между rs72613567:TA HSD17B13 и простым стеатозом у людей. Хотя физиологические субстраты HSD17B13 не известны, из результатов ферментативных исследований видно, что изоформа HSD17B13, кодируемая аллелем rs72613567:TA HSD17B13, каталитически дефектна в отношении эстрадиола. Хотя в настоящее время остается неясным, являются ли какие-либо из протестированных субстратов критическими для заболевания печени, интригует тот факт, что HSD17B13 обладает ферментативной активностью в отношении нескольких биологически активных форм липидов (например, лейкотриена В4), которые, как было установлено ранее, вовлечены в опосредованное липидами воспаление (Li et al., Nature Medicine, 2015, 21, 239-247, doi:10.1038/nm.3800, полностью включена в настоящий документ посредством ссылки для всех целей).
[00497] Вариант HSD17B13 может стать подходом для создания новых терапевтических стратегий для целенаправленного лечения хронического заболевания печени, подобно генетическим вариантам, которые привели к разработке новых терапевтических средств в других областях. Наши данные свидетельствуют о том, что HSD17B13 изменяет прогрессирование заболевания печени от стеатоза до более запущенных стадий NASH, фиброза и цирроза печени, которые ассоциированы со значительной заболеваемостью и смертностью и для которых в настоящее время нет эффективных средств лечения.
Пример 4 Модификация мышиного локуса Hsd17b13 с помощью CRISPR/Cas9 ex vivo и in vivo
[00498] В качестве доказательства идеи целенаправленного воздействия на Hsd17b13 с помощью системы CRISPR/Cas9 тестировали мышиные гидовые РНК Hsd17b13, нацеленные либо на участок экзона 1, либо на участок экзона 6/7 мышиного локуса Hsd17b13. Целевые для гидовой РНК последовательности приведены в таблице 12. ДНК-нацеливающие сегменты гидовой РНК, соответствующие SEQ ID NO: 259-268, представлены соответственно под SEQ ID NO: 1643-1652, которые идентичны SEQ ID NO: 259-268 за исключением урацилов вместо тиминов. Идентификатором гена в NCBI для мышиной Hsd17b13 (гидроксистероид-(17-бета)-дегидрогеназы 13) является 243168 (SEQ ID NO: 269). Мышиный геномный локус расположен на хромосоме 5, NC_000071.6 (103955442..103977388, комплементарная цепь).
[00499]
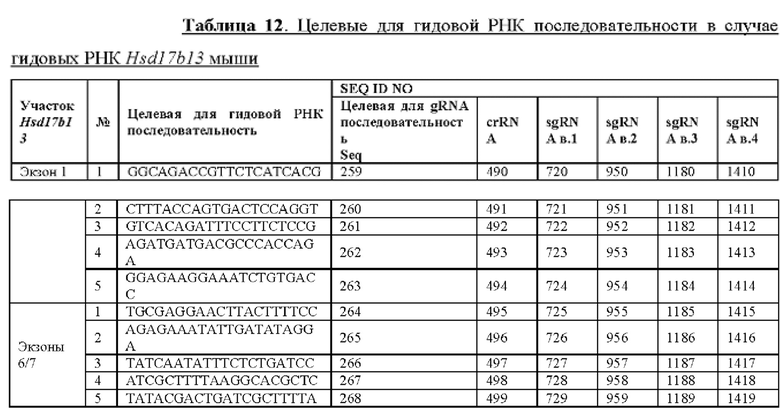
[00500] Гидовые РНК сначала тестировали ex viva на первичных гепатоцитах мыши, выделенных из гибридных мышей дикого типа (75% C57BL/6NTac 25% 129S6/SvEvTac). Печени от мышей перфузировали 50 мл среды для перфузии печени, содержащей 1 × пенициллин/стрептомицин, с последующим добавлением 50 мл среды для переваривания печени (HBSS, 100 мМ CaCl2, 500 мМ HEPES, коллагеназа). После расщепления печени ее помещали в промывочную среду, содержащую 1 × пенициллин/стрептомицин и L-глутамин. Печень разрывали для высвобождения гепатоцитов из печени путем легкого встряхивания. После высвобождения клеток их пропускали через фильтр с размером ячейки 70 мкм и центрифугировали при 50 g в течение 4 минут при 4°С. Осадки 2× промывали промывочным буфером. Затем осадки ресуспендировали в 20 мл 38-40% перколла и крутили при 200 g × 10 минут при 4°С. Осадок 2× промывали и ресуспендировали в среде для посева (среда Уильямов Е, 1 × пенициллин/стрептомицин, 1 × L-глутамин, 5% FBS). Клетки высевали в количестве 300000 клеток на лунку в 24-луночные покрытые коллагеном планшеты для культивирования тканей. После того как клеткам давали прикрепиться в течение 6-18 часов, среду для посева заменяли средой без FBS. Используемые реагенты приведены в таблице 13.
[00501]
[00502] Рибонуклеопротеиновые комплексы (RNP), содержащие Cas9 и мышиную gRNA Hsd17b13, добавляли к свежевыделенным первичным гепатоцитам мышей. В случае экспериментов ex vivo на первичных гепатоцитах мышей использовали модульные гидовые РНК с раздельными crRNA и tracrRNA. SEQ ID NO crRNA приведены в таблице 12, а последовательность tracrRNA приведена под SEQ ID NO: 1422. Каждый RNP-комплекс Cas9/gRNA трансфицировали в конечной концентрации 2 нМ с использованием CRISPRMAX™. Спустя 48 часов из клеток готовили ДНК-лизаты и проводили секвенирование следующего поколения для каждой тестируемой гидовой РНК с целью определения частоты вставок/делеций (indel) по прогнозируемым сайтам разреза.
[00503] На фиг. 15 показаны уровни редактирования (% считывания со вставками/делециями) в мышином гене Hsd17b13 с каждой из гидовых РНК в первичных гепатоцитах мыши, включая каждую из пяти гидовых РНК, нацеленных на участок экзон 1, и каждой из пяти гидовых РНК, нацеленных на участок экзон 6/7. Эффективность редактирования относится к общему количеству вставок или делеций, наблюдаемых по общему количеству последовательностей, считанных в ПЦР-реакции из пула лизированных клеток, при определении с помощью секвенирования следующего поколения. Почти у всех гидовых РНК наблюдали эффективность редактирования по меньшей мере 20%.
[00504] Затем пять гидовых РНК к мышиному Hsd17b13 тестировали in vivo на мышах со встроенным в геном геном Cas9 (Cas9-готовых мышах). В случае экспериментов in vivo на мышах использовали химерные одиночные гидовые РНК. Нацеленная на ДНК последовательность для каждой гидовой РНК была эквивалентна целевой последовательности для гидовой РНК, приведенной в таблице 12, с урациалми вместо тиминов. Каждая отдельная гидовая РНК включала нацеленную на ДНК последовательность выше (5') остовной gRNA, приведенной под SEQ ID NO: 1420. SEQ ID NO sgRNA приведены в таблице 12 (столбец sgRNA вар. 1). В таблицу 12 включены и другие варианты sgRNA с использованием другого остова гидовой РНК, но с ними не проводили тестирование. На каждую группу трем самцам Cas9-готовых мышей вводили каждую гидовую РНК. Гидовые РНК вводили с помощью аденоассоциированного вируса (AAV8), несущего кассету экспрессии sgRNA, посредством инъекции в хвостовую вену (1×1011 на мышь в 100 мкл PBS). Мышам дикого типа, которые не экспрессировали Cas9, вводили дозу всех пяти гидовых РНК в качестве отрицательного контроля. Через три недели после инъекции животных умерщвляли и собирали сыворотку крови вместе с печенью и другими тканями. Ткани перерабатывали в ДНК-лизаты, которые затем анализировали с помощью NGS-секвенирования.
[00505] Как показано на фиг. 16, из результатов NGS-секвенирования было видно значимое редактирование в печения для всех пяти гидовых РНК (процент редактирования составлял по меньшей мере 20% для каждой из них). Эффективность редактирования относится к общему количеству вставок или делеций, наблюдаемых по общему количеству последовательностей, считанных в ПЦР-реакции из пула лизированных клеток. В других тканях наблюдали минимальные или статистически не значимые уровни редактирования генов (данные не показаны).
[00506] Из результатов химического анализа сыворотки в отношении ферментов печени ALT, AST, триглицеридов, общего холестерина, HDL, LDL, неэтерифицированных жирных кислот (NEFA) и альбумина была видна небольшая разница между различными группами обработки (данные не показаны).
[00507] Экспрессию Hsd17b13 оценивали путем оценки равных массовых количеств РНК из печени с помощью RT-qPCR. Геномную ДНК расщепляли так, чтобы она не учитывалась в qPCR-реакции. РНК подвергали обратной транскрипции, а затем использовали Cas9-специфичную пробу для детекции транскриптов Cas9. Для каждой индивидуальной гидовой РНК к Hsd17b13 наблюдали по меньшей мере 50% нарушение экспрессии мРНК Hsd17b13. См. фиг. 17А. Напротив, не наблюдали значимого снижения экспрессии нецелевого представителя семейства HSD. См. фиг. 17В.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Регенерон Фармасьютикалс, Инк.
<120> Варианты HSD17B13 и их применения
<130> 057766/507242
<150> US 62/449335
<151> 2017-01-23
<150> US 62/472972
<151> 2017-03-17
<150> US 62/581918
<151> 2017-11-06
<160> 1652
<170> PatentIn version 3.5
<210> 1
<211> 19118
<212> DNA
<213> Homo sapiens
<400> 1
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataaggtaat gtatacatct tccaactttt 300
taaagtcaca gagtaagata tgtattttaa gaattatttg acttaccatc tacttatctt 360
tgtatttttg tttttcaaag tttgataaat tccctggtcc cttagtctgt atatgtgtca 420
ggttagttag atgaagggaa tgtaattaag aactaagcag cgatttttat gacatggtgt 480
gcaggttgat agaaagactc aggagccagt ctccttccaa gctgctaaat gaggcaagtc 540
acatattatc tctcagcctg ttttcttggc tctgaagtgg ggataataac ttaggggatg 600
ggcaagaacg ggatctgaaa attacagcta caaacaaaag tcaaacgaag aacttgcaac 660
agaaaccttt agtgcctccc ctcatgcaca agcaacacag ttctaaaata tttactgtct 720
gaccctttac agaaaatgtt tgccagtccg tagtcaaaag gattaaataa gtaatatttt 780
cagcacttag catatgataa acgatacgtg gcacatgata aacaataact gtgttaaata 840
aaatatgtgc gcagtgagtc aggcttttcc ttggacatta gtatttttcc tgtgttctta 900
cttgtaaaca ctacattaac aaccccaaat aaaactgaag gaactgaaat cttgtatcat 960
tttctctaaa cttgtaaatt ctggtaaggc catgaaaata tatgcagaga agtgtttaca 1020
ggattttagg attggaaaaa ttgtgaagta ctccttgaga atcacatttt ctgcaaatta 1080
cagtggtttt aattaccatt atattattac tttctcatgt tctttgctgt catgtttagt 1140
tgaaacctaa aatgtctctt acacttagag aactaattct tttctgtttt ttttctgaat 1200
agtgaagaat actatacaaa aaagctacta catttttatt taacagatat gagcatttat 1260
ataatagagg agttgatgta tataaaaatg atttgccatc tttttggtct ttgaagaaat 1320
tcgaatgaac tttctggaag atagcaagaa tttacaaata gagaaaattg ttgcctgctg 1380
ttctcaggca tttgtccaaa aatataaata agtataaatc tatgaaaagg gcttgatgaa 1440
atctaacctt caaatctctt tccagatgtg tatttttggg gaaagggcta tatttattaa 1500
gtttttttta aattttaaaa tttccagaga caagagaaaa gtaaattaga aggaagtcgt 1560
attaaaaatg acttaagggc gggtgcagtg gctcacacct gtaatcccag cactttggga 1620
gacggaggtg ggcagattgc tggagcccag gagttcaaga ccagcctggg cagcacagca 1680
aaacccccaa ctctacaaaa aatacaaaaa ttagctgggt gcgggggtgc acacccgtag 1740
tcccagctac tcgggaggct gaggtgggag gatcgtttca gttcaggaag ccaaggctgc 1800
aatgagctat gatggcatca ttgcactcca agctgggcaa tagagccagg ctctgtctca 1860
aaaaaaataa aaaaagactt aagaaaaata ggtaacccaa cctcaaaaat tctctttgaa 1920
tcattaaatt tcatggttaa acatttaagc tactgaatga ttcactctaa ggctgtaatg 1980
taactcagat ctcctttagg cgaggaagat gctggctgag ttttcatcat aactggctcc 2040
ttttgccctg tgagatgaga gacacagtag cagtttggct cttatgcaat ctaaactgtt 2100
gcgttgggaa tacggttcaa aaaacacatt ggagtttaag ctaaagcaag tgttttgcta 2160
acaaaaagac aaggcatcac attttgcaat tgtctagctc agttataaaa cagaagaata 2220
ggccggacgc ggtggctcac gcctgtaatc ccagcacttt gggaggccga gacgggcgga 2280
tcacgaggtc aggagatcga gaccatcctg gataacacag tgaaaccccg tctctactaa 2340
aaatacaaaa aaattagcca ggcgtagtgg cgggcgcctg tagtcccagc tactcgggag 2400
gctgaggcag gagaatggtg tgaacccggg aggcggagct tgcagtgagc cgagatgacg 2460
ccactgcact ccagcctggg cgacagagcg agactccgtc tcaaaaaaaa aaaaaaaaaa 2520
aaaactgaag aataattaat tcttcaatca aaacatctga tgaatgctct ggtaacttat 2580
gctctctact gacctagaaa caaatgagag agtatggtgt ggtttgtgca atctggcagt 2640
gagcaagcta ccaactaaat cagtgaaaga ctctcctatt ctttttttac tcttctgcaa 2700
tcccacaaaa ggctatttga ggggatactg actttgagac tgggtcctaa catccatgtt 2760
tggggagttc aggctgctgc tccagggttt agcctacagt agcgaaatac aaaggaccca 2820
gagaccactc attcaaggtt tgccctaaat agcagcaaca ccactgtcat ctcaatacac 2880
gaagaatagg gcttttcagg tatccttgcc tctttgtcac agagaagagt ttacagattg 2940
tgagacggaa aagtataatt tttaaaacct tataatattt tctataaaag tcacctgagg 3000
tgaaaacttg aaaagaatta taattttcca gaatgtgagt caagaaacat tagagcaatt 3060
ttatcttagg aaagaggtct ttgaatttag gctgaaagta aattgctctg tctccatgtc 3120
ctatggttat gggcaagttt ggtacataaa tgagaaatcc atccagtggc cttgcccatc 3180
tcactcccaa acacctgaag aatgtaatgt tatatctcct agagtagcag catggtctcc 3240
ctatgaaagt ccttcttctt taaggagact tctttccctt ccctcctagg aggatgagtc 3300
agaatcatca agaaaaatat gatgggcaga ggcatacagt ttaccattac cactagttta 3360
gaattactac ttagcacttt actgcctatt acatagttgg tgctcaacaa atgtatgata 3420
aattaatggt tgagtttttc tttcttctcc atattcatct tccatgacac cacgaagagc 3480
aatgtttttc aagaatgttc ttcaaggttt gaaagtagcc tgctttagag aaactgccta 3540
ctgtacagcc tccaaccaag aggaaaagct gaaaaaagca tgaagggatt ttgttttgtt 3600
ttgtttgttt tggttttaat atgagcattc cctggcagaa aagccagggg taatctcatt 3660
gcaactaggc aatcactctc aagaaatttt ctaacaaata aggaggccaa tttttatttt 3720
attttgagac gaagtcccac tctgtcaccc aggttggagt gcaatggaat gatttcagct 3780
cactgcaacc tccgcctccc gggttcaagt gattctcctg tctaaacttc ccgagtagct 3840
gggattacag gctcccacca ccacgcccag ctaatttttt gtatttttag tagagatggg 3900
gtttcaccat tttggccaga ctggtctcaa actcctgacc tcaagtgatc caccctcctc 3960
ggcctcctaa agtgctggga ttacaggcgt gagccaccac acctgaccca ggaggccaat 4020
ttttaaaagg ttaactaatc ttcatgtcca aaatgaatgt taattgttca ttttggacat 4080
gaatgttaat tttttttttt tttttttttg agacagagtc tcactctgtt gcccaggctg 4140
gagtccagtg gcactatctc cactcactgc aacttcctcc tcccaggttc aagcaattat 4200
cctgcctcag cctcccaagt agctgggatt acaggcccac accatcaggc ctggctaatt 4260
tttgtatttt tagtagagac ggggtttcac catgttggcc aggctggtct tgaactcctg 4320
acctcgtgat ccgccctcct cggccaacca aagtgctggg attacaggcg tgagccaccg 4380
cgcctagccg aatgttaatt gtctaaaaat ttttcttctc caatgtcttc tcctccactt 4440
ttttcggaat ttgtttcttc ctaattacag cgcggtgtgg aggaaactgc agctgagtgc 4500
cgaaaactag gcgtcactgc gcatgcgtat gtggtagact gcagcaacag agaagagatc 4560
tatcgctctc taaatcaggt gagactgcag gttcacaaat ttcttcagat tattttgttt 4620
cctaggacgc tgacgtggaa aatgagaaag gtctttatga ctgcctgatt taaattggat 4680
tttagctgct aactgaagta gttatgtcac caaggaagga tatatacttt ttttcttgta 4740
tgtaatccac tcagctctgc ccattattat tgttcatatt attaatcaat ttcattctga 4800
tcagaagtgt gagcagtggc acagagtgac tgacaaaaga tttatcatca gggaatatgg 4860
atcacttcct agttttgttt tagtcctatt aactttgcag taattccagc ttctctttaa 4920
ttatttccct tgtgagattt tattttggtg ttaatgtagt cttctgtaga aaatgtaata 4980
ttaataatta ttatcacaat tattttaaaa gagtaaatac caaataatca caatgaacta 5040
agcactctaa caaactttac attttttaat tcaatcccta caataactct gtaaacttca 5100
ttttacagat aagcaaatta tgactcagag aggttaagcc agacccaggt catgtagtta 5160
ttaggttatg aaaccaggat ttctcaacca gcactttaga ccaggtgcgg tggttcacac 5220
atgtaatccc agcactttgt gaggccaagg tggaaggatc acatgagacc aagagttcaa 5280
gaccagccca ggcaacatag tgagacccta tctctaaaaa aaaaaaaaaa aaaaaaaaaa 5340
aaagtttaaa gaaaaacaca tttttaaaaa atgaacactt taaaaatatt tggtcagaat 5400
ttatatagga atttatcaac ataaatgtta atttcacttt actgataaac ttgcaaaaca 5460
tgatgtgctg ggtactgaaa tttagatgtt aaaagaacag tttatcccac ctttatgaca 5520
gtgttccctt ggcctccacg atttgagctc aacagtctgt cttgcctgaa ctctgagaga 5580
cctcatacaa tagaagaaag actctcatct ttggattata ttggtcccaa aactttgagt 5640
ttgaataata cacccagtga aagtgttctt tcaatttcaa aaggtgaaga aagaagtggg 5700
tgatgtaaca atcgtggtga ataatgctgg gacagtatat ccagccgatc ttctcagcac 5760
caaggatgaa gagattacca agacatttga ggtcaacatc ctaggacatt tttgggtgag 5820
tgtgagtcag aaacatttct gatttgtgca ccttctctta agatacatga aacttataac 5880
ggagttcaca tacttctgga caggaaactg gccagatctt tgccttaatc aagaatcatt 5940
aaatttgttt gagtagaaga gccacagagt ctctgacaca aggacacaga attcaagtgg 6000
acacaacaca ccagaatgta agctacttgg tctgtcttgt ccaccagtat ctgacacaaa 6060
gcttggcatg taccaggagc tcaacaaatg tttgtggagg tttgttaagg gttgtcagtg 6120
tacatctttt caatgctgtc acttgtgact tcattttttt ccctccacac catgattttg 6180
taatgtgtcc tcattttgtg gaattttaga atggaaagga catcagaagt aattacttgg 6240
atgtatatag gatcgaggac acttttggac gagactctga ggcaagtgtt ctagatccat 6300
ggggtgctgg aactgagaaa tgcagctata cagacctcat ataattggtt agttttgtgg 6360
gagatggaaa tatcaacttc aactgccttt gtatagaaat ttttatgatt aatcttccag 6420
tgcctcaata ttagtgtaga atctagggca gatctggatt ctagaagaaa gaagaaaaaa 6480
aagagatgtg tcccccttac ctttaccagc tcttcacata tgtgaattgg ctcccatgcc 6540
caccaaacta cacggagacc tcatacatta gctacctata gctgcataac aaattataca 6600
aaacttagtg gtttaaagca acaatgtatg ttcactatcc tctcacagtt tctatgggtt 6660
gggaatttgg aggtagcttg ggttgggagt tctagttcta tgaatttgca taggatttat 6720
taaattctta taaaatttta ttgatgtttc tcacaaaaga ggtttttgga aaaaaagaaa 6780
gacttgtttt ctgtaacatc aacatataat atacaatatt acaaataggg agatagtgaa 6840
ttcaatcatg attcattagt gtggtgtaga actctcagct tacactactc aactgtctta 6900
atacagttac acaagatttc actcttttaa ttagaatgat aaagccccaa accaaaaaat 6960
tatatgacac caaattatca taaggaataa ttttagttct gaaaactctg aatttttccc 7020
ttaatattgt ttagatgaca tatccaaaaa aggatctatt tgattccttc tgaagggaag 7080
gagggggagt actgagatta gtgttggcat ggggcttacc ataccaataa atttgtatct 7140
ttatttctat catttgtaaa gaattaatca tggaatgctt ggaagtattt tatttcattg 7200
tataagttct ctcaaatgcc tttctgtctt aacaaaaata aaactacctg atttggaaac 7260
ctaacgtcta tgtcattgtc tttcttcttt ctgcaatgat ccttaagatc acaaaagcac 7320
ttcttccatc gatgatggag agaaatcatg gccacatcgt cacagtggct tcagtgtgcg 7380
gccacgaagg gattccttac ctcatcccat attggtaagt atcacatgcc agccatgtta 7440
tatattttta tactttgaag ggagcattac acttcaaatt gttaccactg gagagtcctg 7500
gttcttggca tcttgaacaa agaattggac aaaactcacc aacaaagcca ggaaagaatg 7560
aagcaacaaa agcagagatt tattgaaaat gaaagtacgc tttacagggt gggagtgggc 7620
ccaagcacag gggctcaaga gccaattaca gaattttctg gggtttaaat accccctaga 7680
ggtttccact ggttacttgg tgtacgccct atgtaaatga agaggatgaa ttaaagttac 7740
agagtcgttt actcagtgta caccatatgt aaatggagag gatatttcct gtcatagctg 7800
gagtgtttcc atttgattta gttctaggaa gtcagcatga atcggcctta tgttccctgc 7860
ctccagaccc tgttctcctg cctcaagatt acaatgctga gagcagagtg atttggattt 7920
acagaattta aatttatagt agtttagaat gattttttaa atgacttttt ctaaaacaat 7980
gaaaccaggt tgtaattata tttaagatat ttttagattt ctgcaaactc ctctgtagaa 8040
caatgagaga aaacagtaat gccaagcatg tttccattgt ttcctggaat aagaaacaga 8100
aaccccacag actgagaagc aaaacctaca gaagctaaaa tgaacacatg tctatgtcat 8160
ggccttggtg cccaagataa gacaatcaga gtggtccctg gatcaaaaca ttttacagtg 8220
tgcttgtgcc atgaaagtgt gtgtgtgtgt gtgtgtgtgt gtgtgagaga gagagagaga 8280
gagaaaacga ctctacctga ctaaaagttg cagataccac actccatgca ccaccaaaga 8340
cataaaggga aggaggtgag aggcgttaag gatgtactgc tgtatttgcc aaatatcctt 8400
tcctgtaaac tcttctccag atcctcataa taaaattaag aggccaaagt ggcaaccatt 8460
gtcaagagaa aaactatcaa ccattgtcaa gagaataact cagttattga gagagagagg 8520
agaaatgagc agagtcctac agaagtctgt caacacagat accagttttg tagaatttct 8580
aaatgtattt ttcctgattc atatttttca aaataaaagc agcaataaaa actgattaga 8640
aaacagtttg aagattcaat ggaaaaacct tacatgtagg atggaaaact gaacattaag 8700
ccaatcaata gagttatttt tgttcttttg ttatcattgt tgtttaagaa atgagatacg 8760
ttcacaattc tgcttaatca tgtaagaaaa tgaaaatgaa ttgccattta tactctcaga 8820
aaaatcacaa gtggctgatt tttggcttcc acttgttctt aagccaaatg ataccgcctt 8880
ctcacagaaa gctgaggatt ggtttcactc tcccttagct aacaatgctt aataattctc 8940
ttacagttcc agcaaatttg ccgctgttgg ctttcacaga ggtctgacat cagaacttca 9000
ggccttggga aaaactggta tcaaaacctc atgtctctgc ccagtttttg tgaatactgg 9060
gttcaccaaa aatccaagca caaggtaagg tcaaaatcaa gttagaatgg gtatgtggta 9120
tgataaattg atatgaaaac taatgagaaa tgtttaggca ggccaactaa tagaagaaaa 9180
tgaagaagga aaaataattt ttcttattat tattattatc ttgaaattaa aggaataaag 9240
ggggaaaaca cattagggac tagcaggaat gatcagccac cgatgaactg ggatatttat 9300
ttgtgtccgg gagaaagcac atacatttga tcaccgttac caccctgtct ttaaaatgca 9360
aatgttccaa ggaccagcaa ataaattgag tatctagctc cttagtcaag gtgaatttct 9420
gcaagaactc ttgtctctgg tgagacagga tttgagacca caagagaaga aaaattagtc 9480
ctgaaaggag aagaaaaaag caggaaggtg tggataagaa cccgaaaatt aagccatctg 9540
cttaacaaat ttttctaatc ctagtatata ttctgctgca ggttaacaaa atatactaag 9600
cttaatgatt cgaaaccaat tttttactgg aagggaatta atcctaaata tattcattca 9660
aaagaactaa acaattctct gttgagtgcc gcctcatttg aggatactga ctcttacagc 9720
ctgagttagc tatgtggtct ctgcagctgg aatcactccc tgccactgga gtccttcatg 9780
gtgttagacc ataggtactg ttgactaaag aaaaaaaaaa gtttttgttt ttatttttgt 9840
tttttttgag acagagtctc actctgtcac ccaggctgga gtacagtggc gcgatctcag 9900
ctcaccgcaa cctccgcctt tctgggttca agcaattctc cttcctcagc ctcctgagta 9960
tttggattac aggcgcccac caccacgcct ggctaatttt tgtattttta gtagagacgg 10020
ggtttcacca tgttggccag gctggtctca aactcctgac ctcaggtgtc ctacctgcct 10080
tggcctccta aaatgctggg attacaggag tgagccacca tgcccggcca aaaaaataag 10140
tttttaaaga attaaaggtc atcctggcta acacagtgaa accccgtctc tactaaaaaa 10200
cacaaaaaaa ttagccgggc gtggtggcgg gcgcctgtag tcccagctgc gcgggaggct 10260
gaggcaggag aatggcgtga acccgggagg cggagcttgc agtgagccga gatcgcgcca 10320
ctgcactcca gcctgggcga cagagcgaga ctccgtctca aaaaaaaaaa aaaaaaaaaa 10380
aaaaaaaaga attaaaggtg ttaattttat ttagaagcct tactgaagac tacagtcgga 10440
ggcctatagc ctgagagcag ccctttagag aggttcagtt gaactgttct gatagtgggg 10500
gccatgtgct ctatcctgta ttgtcttcaa agcatctttc cagagagctg cacattgtca 10560
cagagtcagg gactttgtga aattatgctg acaaccagaa gtgagtaaac gtggcttctt 10620
acatttgcta cgttgtctca cagtacttaa taagtatgca atatgtaagt aaatactata 10680
gtactattgc aactcctgat tgttttctta gacaaggaat tgggcccaat aaaaaccctc 10740
ttggtaggca ttcaggcttc gtgtaccatg agctttccta agggtatcct gccactcttg 10800
gggaaggcat gatagatgag gggagtaagg ataatggaac tctgggtaca gggttcctgg 10860
gggctaactt agaggtagac acaggcaatg ctaaatattt gggattgatt ttatagaggt 10920
tgctagattg tgaatttcct tagtaagggc taaggcattg atatgtaatg tcacacttgg 10980
ctccgaggct gggttgttgg atccatgtag atgaaatcag ggagagaaag ggcagaacgg 11040
agtaatttag aaatgtattg atttgtatta ctctctgttg gcttgctatt caaggcagtg 11100
gagaactcaa tcacataata atctgcagca aaccacagat catcccaggg aatgaagttt 11160
taacattcgc tggctcccta actcctcacc cagcctttac attcactggc tgttcagtcc 11220
atgcctggac atcttaattt gaatacaaca ttttaaatcc atttttctgt catcatcttg 11280
cactaacaga caattctaca ctaagcctat gtttatgaat atttctcaag agtacatgta 11340
cacagccttc agtataagga aaactggaag tatgacatac ctccagttgt catactcctt 11400
gggcccctct taaattctca ttaaactgca ggataggcaa gtcagaggtg aatctcaaat 11460
acgaaattct taccggaaag gggttccaat ccagacccca agagagggtt cttagatttc 11520
tcgcaagaaa taattcgggg caaggccaca gtgcaaagca aaagcaagtt tattaggaaa 11580
gtaaaggagt agagaacagc tactccatgg agaagaatgg cttgagctgc tccaccaagg 11640
gtatttagag ttacttcttg attatatgct aaacaagggg tggattattc atgagttttc 11700
cgggaaaagg gtgagcaatt cccagaactg agatttcctc ccctttttag gccatatagg 11760
gtaacttcct gccattgcca tggtatttgt aaactgtcat agtgctggtg gaagtgtctc 11820
ttagcttgct aatgtattat agttagctta taatgagcag tgaggacaac cagaggtcac 11880
tttcatcacc atcttggttt tggtgggttt tggccggctt ctttactgca ccctatttta 11940
tcaacaaggt ctttatgacc tgaatcttgt gccaacctcc tatctcatcc tgtgacaaag 12000
aatgccttaa cttcctggga atgcagccca gtaggtgtca gccttatttt acccagaccc 12060
tattcaagat ggagttgctc tgatttaaac gcctctgaca aaatgacgac ctcaaaacaa 12120
tccagcttta tggaatacct ccacaagaaa gaaagtatac ttagctatag aattttctcc 12180
ttgcatccaa caggctttga gatgtcagat gtttccttcc tgtcccatga ttaatcctag 12240
ccattcctct ttcttgtctg gctccactac tccttaccat ctaatgcctc gccaccattt 12300
tgatattttg actaagtgag ctatgaaaca cacctactgg atatgaaagt ataagtttct 12360
gataacaaaa catcaacatg ggatgtggag gaagtgggta gggtggcatt aatgcagcaa 12420
atcctggaat attttaaatc ttcattctaa atttagtaaa aatataggat aattttcctg 12480
ccatcattta cttataaaat taaaatttta gaaaataaaa ataatatttt cctcttttta 12540
atcacagatt atggcctgta ttggagacag atgaagtcgt aagaagtctg atagatggaa 12600
tacttaccaa taagaaaatg atttttgttc catcgtatat caatatcttt ctgagactac 12660
agaagtaagt acagcacaga acacccaaat actaaaacac caatagagct tttttttttg 12720
cttttttttt ttttagacag agtctcactc tgtcaccctg gctggattgc ggtggttgca 12780
gtggcatgat cttggctcac tgcaacctcc gcctcctggg ttcaagcaat tctcatgcct 12840
cagaccccca agtaactggg attataggtg tgtgctgcca cactacaccc agctaatttt 12900
tgtatttttt gatagagaca ggtttcccca tgttggccag gctggactcg aactcctgac 12960
ctcaagttat cctcctgtct cggcctccca aagtgctggg attacagtca tgagccacca 13020
tgcctggccc aatagagcta ttattatgga gcatctttca gttgtgaaaa ttggcatgga 13080
aactctccat ccctggggag aacagttatt tcctctgtta ttttcctacc cagtctataa 13140
aaagagagtg attcattttc tctaccaaat ctactgtctc tgcccaaact ttgctgaaga 13200
ctattctaac taaaggaaac acagtttaaa aagaatgcaa tatagtgaag tagttaataa 13260
taaagactcc atttttaaaa gtctgctgga agtttggttg ggattgcact gaatctatag 13320
agcaattggg gagtattgac atatcaacaa tattgagttt tctaatccaa gaacataata 13380
tctattttta aaatcttctt caaaatcttt aaatctttaa attgtatttt gtagtttttg 13440
gtgtttaagt cttgcacata ttttgtcaga tttattccaa agtatttcac gggttctttt 13500
tttttttttt tttttttttt ttgagacaga gtttcaccct tgttgcccag gctggagtgc 13560
agtggcgtga tcttggctca ctgcagcttc tgcctcctgg cttcaagtga ttctcctgcc 13620
tcagcctccc aagtagctgg gattacaggc acctgccccc tcgcccaact aactttttgt 13680
gtttgtagta gagacagggt ttcaccatgt tggccaggct ggtctcgaac tcctgacctc 13740
atgtgatcca cctgcctcag cctcccaaag tgctgggatt acaggcatga gccatcatgc 13800
ccagccctat ttgacggttt ttgacgctaa tgcaagtggc attttaaaaa attttatatt 13860
tcccattgtt tgttgtcagt atatattgga tttttgtaat ttgatctcat attttgcagt 13920
cttgctaaat tgctaaacct ctttttgcta aactcgataa gctttttttt ttttggtaga 13980
ttcctgggcc tctaattttc tttatgggaa agtttttaat tacaaattta atttctttaa 14040
tagctacatg gctattcaat ttacttatta attcttggta atgtgtgtct ttcaaggaat 14100
ttgtccattt catctaagtt gtagaatttc tttggcataa atttgtacat aacattccct 14160
tattatcctt ttaatgtctt tagaatgtct tatttattta tttatttatt tttattatat 14220
ttttttgaga cagagtctcg ctctgttgcc caggctggag tgcagtggca caatcttggc 14280
tcactgcaag ctccgccttc tgggttcatg ccattctcct gcctcagcct ccctagttgc 14340
tgggactaca ggcgcctgca accatgccca gcttattttt tttttttttt tttttttttt 14400
tttttttttt tttttttttt tagtagagac ggggtttcac cctgttagcc aggatggtct 14460
cgatctcctg acctggtgat ccgcccgcct cagcctccca aagtgctggg attacaggcg 14520
tgagccacca agcccagcct atttatttat ttagtagaga cagtctcact ttgctgccca 14580
ggcaacaaag gttttgaatg cctggcctca agcagtcctc ctgccttggc ctcccaaagt 14640
gctgggatta caggcatgag ccactgcacc tggccaaatg aatatgctga taatatcttc 14700
tttataagga tgacataaga ataaaataat gtaatacaaa caaagcccct gtcactgaaa 14760
atgtatagac ttcaaatgtt aaagtcttag agaacagaat ttatatgaaa tagcaacagc 14820
aacaatttcc cagaggaaat actctctcag ctttcttctg aggagcagtt tctaaattga 14880
aattgtatca gtgagaagat aactatacta acttcataag ccttgggcct ttttgaaaca 14940
aatccatata aactatgaac aaacttgaaa tagaacaatt tgagaacagg gtacaaactg 15000
cattggtgta tcaatttcag tatttggttt tagcttaaat agactgactt gagataacat 15060
aaggagaacc ttgaccccca agcaacatca tctcgcgagt tgactaggcc gggtgtggtg 15120
tctcacgcct gtaattccag cactttggga ggccacagca ggcagatcac ttgaggtcag 15180
gcattcgaga ccagcctggc caacatggtg aaacctcagc tctactaaag atacgaaaat 15240
tagcaggcat agtggcctgc acctgtaata ccaggcactc gcaggagaat cccttgaacc 15300
cggaaggcgg agattgcagt aaaccatgat tgtgccactg cactccagcc tgggcaacag 15360
gagactctgt ctcggaaaaa taaatttttt aaaaaaatga aaaaaaataa aagttgacta 15420
aattagtgtc ttggtactaa gcactgtagg aagtgagttt catggaaccc caactctctt 15480
ggggcccaaa gcaagtcata ttaatattga aaattacatg catatacatg catatgacca 15540
aggtgataaa aacaattatt ctgcctgagt tggagaatag tatcccagta aaataaacaa 15600
gagtctcaaa gtcttttgta tcctttgaag ctgtcatggt ggtttgtaac taggcaacag 15660
gtatatattg ttaatcttct ttgcatttaa ttccttttat agagagacac aattttacga 15720
gcagatgcaa ttactagcat gaaggtttct ttgtgagggt agttaaaagg cccacatgag 15780
ctctcttctt atccttgtcc ttctttcagc cagatcttcc ctgccccttt gctcattcca 15840
tctttcaccc acctaccccc aaaacaagga agtaaatctt gcattagtca acaataccaa 15900
agtgattttc aatatgactt tctctgcaga atgttattat ttctgcctct ttacattcac 15960
atactgtctt cctttttttt tttttttttt tttttttttt tagattgggt ctcactctgt 16020
tgcccaggct ggagtgcagt ggcttgatct cagctcactg taacctccac ctcctgagtt 16080
caagcaattc tcctgcctca gcctcctgag tagctgggat tacaggcatg tgccaccaca 16140
cctggctagt ttttttgtat ttttagtaga gacagggttt caccatgttg gtcaagctgg 16200
tctcgaactc ctgacctcat gatctgacca cctgtgcctc tcaaagtgct gggattacag 16260
gcgtgagcca ccgggccagc cactctcttc ctttcagttg cctactcatc tcttatgcat 16320
tcctggacat cagttgtcct tttgaagctt tcctccacta tcccagccca tgtgaatcct 16380
ccttccagtt atagccctta attctagatg gctgatattt ttcaataatt gttttaagat 16440
gaccatttta gcctatcagc taaacaatat caaagacaat agctattttt caagtacttt 16500
agtttacctt attatagagt gcataataga tattcagtaa atagtaaagg agaggtgaag 16560
gcttgcatag aatggattct ggtggtgtct cttggtgagc ttttagcatc aagattaatc 16620
agcagtttca gcaatgagct cagaccttca gttttagatc tttactcata tcagataaga 16680
gagtgagaag agtggtatgt atcagtgctt tatttatatt tgcatccaat ttgaactatg 16740
aatattacaa aggtgcacac ataggttcag acagattgat ttaaaatgac caaagatgac 16800
ctgtcgtaag caacctgggt atcttaagat gcactccttg gagagggaat gttcctaaaa 16860
acattttcag agggacgaac tgtatgaaat tcagtaaaac ataaatcatg aggaaaactg 16920
attactctct ttttgacatg aaatgagagt tttaatgcat ggttacgatt attaacgtac 16980
tccgctgcaa gacgttaata aagttactgt tttgcaggct agaatgtctt gatgctgtaa 17040
tcagaacaca ctttttcccc tttcttccag cttcaaatgc agattcataa ttgggctgac 17100
ttctaataac tgcaatgttt tctgccttgg gcttgcagca gaagcctgac aaaatagtgt 17160
ttgtttaggc aataatttat ttatttattt attgagatgg agtttcattc ttgtcgccca 17220
ggctggagtg caatggcgtg atctcggctc actgcaacct ctgtgttcag gcaataattt 17280
agactttacc ttacttgtga ttactatagc aattactata gccacaaggc ataattttac 17340
tgtctcattt caattttatg aatttgaatg tttttacact tttcctaatg aagtccacta 17400
tgaagttatg tcaaaaaaaa aaaagaaaaa gaaagatgca cacgtaaaag agaggtggtt 17460
gcaagagaag aaaagaacgg aggaaagtta aacgcaaacc agataactct cagcgtattc 17520
taaatgacca aaaacagaac tctgttgtca aagattttaa atggaaaatt tttcaatttt 17580
tttttctttt ttgtacaggt ttcttcctga acgcgcctca gcgattttaa atcgtatgca 17640
gaatattcaa tttgaagcag tggttggcca caaaatcaaa atgaaatgaa taaataagct 17700
ccagccagag atgtatgcat gataatgata tgaatagttt cgaatcaatg ctgcaaagct 17760
ttatttcaca ttttttcagt cctgataata ttaaaaacat tggtttggca ctagcagcag 17820
tcaaacgaac aagattaatt acctgtcttc ctgtttctca agaatattta cgtagttttt 17880
cataggtctg tttttccttt catgcctctt aaaaacttct gtgcttacat aaacatactt 17940
aaaaggtttt ctttaagata ttttattttt ccatttaaag gtggacaaaa gctacctccc 18000
taaaagtaaa tacaaagaga acttatttac acagggaagg tttaagactg ttcaagtagc 18060
attccaatct gtagccatgc cacagaatat caacaagaac acagaatgag tgcacagcta 18120
agagatcaag tttcagcagg cagctttatc tcaacctgga catattttaa gattcagcat 18180
ttgaaagatt tccctagcct cttccttttt cattagccca aaacggtgca actctattct 18240
ggactttatt acttgattct gtcttctgta taactctgaa gtccaccaaa agtggaccct 18300
ctatatttcc tcccttttta tagtcttata agatacatta tgaaaggtga ccgactctat 18360
tttaaatctc agaattttaa gttctagccc catgataacc tttttctttg taatttatgc 18420
tttcatatat ccttggtccc agagatgttt agacaatttt aggctcaaaa attaaagcta 18480
acacaggaaa aggaactgta ctggctatta cataagaaac aatggaccca agagaagaaa 18540
aggaagaaag aaaggttttt tggtttttgt tttgttttgt tttgtttttt gtttttttga 18600
gatggagtct cactctttcg cccaggctgg agtgcagtgg tatgatctca gctcactgca 18660
agctccacct cccgggttca cgccattctc ctgcctcagc ctcctgagta gctgggacta 18720
caggcgcccg ccaccacacc cggctaattt tttgtatttt ttgtagagac ggggtttcac 18780
catgttagcc aagatggtct cgatctcctg acctcgtgat ccacctgcct cggcctccca 18840
aagtgctggg attacgggtg tgagccaccg tgcccagcct tttttttttt aatagaaaaa 18900
ataatccgac tcccactaca tcaagactaa tcttgttttg tgtgtttttc acatgtatta 18960
tagaatgctt ttgcatggac tatcctcttg tttttattaa aaacaaatga tttttttaaa 19020
agtcacaaaa acaattcact aaaaataaat atgtcattgt gctttaaaaa aataacctct 19080
tgtagttata aaataaaacg tttgacttct aaactctg 19118
<210> 2
<211> 19119
<212> DNA
<213> Homo sapiens
<400> 2
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataaggtaat gtatacatct tccaactttt 300
taaagtcaca gagtaagata tgtattttaa gaattatttg acttaccatc tacttatctt 360
tgtatttttg tttttcaaag tttgataaat tccctggtcc cttagtctgt atatgtgtca 420
ggttagttag atgaagggaa tgtaattaag aactaagcag cgatttttat gacatggtgt 480
gcaggttgat agaaagactc aggagccagt ctccttccaa gctgctaaat gaggcaagtc 540
acatattatc tctcagcctg ttttcttggc tctgaagtgg ggataataac ttaggggatg 600
ggcaagaacg ggatctgaaa attacagcta caaacaaaag tcaaacgaag aacttgcaac 660
agaaaccttt agtgcctccc ctcatgcaca agcaacacag ttctaaaata tttactgtct 720
gaccctttac agaaaatgtt tgccagtccg tagtcaaaag gattaaataa gtaatatttt 780
cagcacttag catatgataa acgatacgtg gcacatgata aacaataact gtgttaaata 840
aaatatgtgc gcagtgagtc aggcttttcc ttggacatta gtatttttcc tgtgttctta 900
cttgtaaaca ctacattaac aaccccaaat aaaactgaag gaactgaaat cttgtatcat 960
tttctctaaa cttgtaaatt ctggtaaggc catgaaaata tatgcagaga agtgtttaca 1020
ggattttagg attggaaaaa ttgtgaagta ctccttgaga atcacatttt ctgcaaatta 1080
cagtggtttt aattaccatt atattattac tttctcatgt tctttgctgt catgtttagt 1140
tgaaacctaa aatgtctctt acacttagag aactaattct tttctgtttt ttttctgaat 1200
agtgaagaat actatacaaa aaagctacta catttttatt taacagatat gagcatttat 1260
ataatagagg agttgatgta tataaaaatg atttgccatc tttttggtct ttgaagaaat 1320
tcgaatgaac tttctggaag atagcaagaa tttacaaata gagaaaattg ttgcctgctg 1380
ttctcaggca tttgtccaaa aatataaata agtataaatc tatgaaaagg gcttgatgaa 1440
atctaacctt caaatctctt tccagatgtg tatttttggg gaaagggcta tatttattaa 1500
gtttttttta aattttaaaa tttccagaga caagagaaaa gtaaattaga aggaagtcgt 1560
attaaaaatg acttaagggc gggtgcagtg gctcacacct gtaatcccag cactttggga 1620
gacggaggtg ggcagattgc tggagcccag gagttcaaga ccagcctggg cagcacagca 1680
aaacccccaa ctctacaaaa aatacaaaaa ttagctgggt gcgggggtgc acacccgtag 1740
tcccagctac tcgggaggct gaggtgggag gatcgtttca gttcaggaag ccaaggctgc 1800
aatgagctat gatggcatca ttgcactcca agctgggcaa tagagccagg ctctgtctca 1860
aaaaaaataa aaaaagactt aagaaaaata ggtaacccaa cctcaaaaat tctctttgaa 1920
tcattaaatt tcatggttaa acatttaagc tactgaatga ttcactctaa ggctgtaatg 1980
taactcagat ctcctttagg cgaggaagat gctggctgag ttttcatcat aactggctcc 2040
ttttgccctg tgagatgaga gacacagtag cagtttggct cttatgcaat ctaaactgtt 2100
gcgttgggaa tacggttcaa aaaacacatt ggagtttaag ctaaagcaag tgttttgcta 2160
acaaaaagac aaggcatcac attttgcaat tgtctagctc agttataaaa cagaagaata 2220
ggccggacgc ggtggctcac gcctgtaatc ccagcacttt gggaggccga gacgggcgga 2280
tcacgaggtc aggagatcga gaccatcctg gataacacag tgaaaccccg tctctactaa 2340
aaatacaaaa aaattagcca ggcgtagtgg cgggcgcctg tagtcccagc tactcgggag 2400
gctgaggcag gagaatggtg tgaacccggg aggcggagct tgcagtgagc cgagatgacg 2460
ccactgcact ccagcctggg cgacagagcg agactccgtc tcaaaaaaaa aaaaaaaaaa 2520
aaaactgaag aataattaat tcttcaatca aaacatctga tgaatgctct ggtaacttat 2580
gctctctact gacctagaaa caaatgagag agtatggtgt ggtttgtgca atctggcagt 2640
gagcaagcta ccaactaaat cagtgaaaga ctctcctatt ctttttttac tcttctgcaa 2700
tcccacaaaa ggctatttga ggggatactg actttgagac tgggtcctaa catccatgtt 2760
tggggagttc aggctgctgc tccagggttt agcctacagt agcgaaatac aaaggaccca 2820
gagaccactc attcaaggtt tgccctaaat agcagcaaca ccactgtcat ctcaatacac 2880
gaagaatagg gcttttcagg tatccttgcc tctttgtcac agagaagagt ttacagattg 2940
tgagacggaa aagtataatt tttaaaacct tataatattt tctataaaag tcacctgagg 3000
tgaaaacttg aaaagaatta taattttcca gaatgtgagt caagaaacat tagagcaatt 3060
ttatcttagg aaagaggtct ttgaatttag gctgaaagta aattgctctg tctccatgtc 3120
ctatggttat gggcaagttt ggtacataaa tgagaaatcc atccagtggc cttgcccatc 3180
tcactcccaa acacctgaag aatgtaatgt tatatctcct agagtagcag catggtctcc 3240
ctatgaaagt ccttcttctt taaggagact tctttccctt ccctcctagg aggatgagtc 3300
agaatcatca agaaaaatat gatgggcaga ggcatacagt ttaccattac cactagttta 3360
gaattactac ttagcacttt actgcctatt acatagttgg tgctcaacaa atgtatgata 3420
aattaatggt tgagtttttc tttcttctcc atattcatct tccatgacac cacgaagagc 3480
aatgtttttc aagaatgttc ttcaaggttt gaaagtagcc tgctttagag aaactgccta 3540
ctgtacagcc tccaaccaag aggaaaagct gaaaaaagca tgaagggatt ttgttttgtt 3600
ttgtttgttt tggttttaat atgagcattc cctggcagaa aagccagggg taatctcatt 3660
gcaactaggc aatcactctc aagaaatttt ctaacaaata aggaggccaa tttttatttt 3720
attttgagac gaagtcccac tctgtcaccc aggttggagt gcaatggaat gatttcagct 3780
cactgcaacc tccgcctccc gggttcaagt gattctcctg tctaaacttc ccgagtagct 3840
gggattacag gctcccacca ccacgcccag ctaatttttt gtatttttag tagagatggg 3900
gtttcaccat tttggccaga ctggtctcaa actcctgacc tcaagtgatc caccctcctc 3960
ggcctcctaa agtgctggga ttacaggcgt gagccaccac acctgaccca ggaggccaat 4020
ttttaaaagg ttaactaatc ttcatgtcca aaatgaatgt taattgttca ttttggacat 4080
gaatgttaat tttttttttt tttttttttg agacagagtc tcactctgtt gcccaggctg 4140
gagtccagtg gcactatctc cactcactgc aacttcctcc tcccaggttc aagcaattat 4200
cctgcctcag cctcccaagt agctgggatt acaggcccac accatcaggc ctggctaatt 4260
tttgtatttt tagtagagac ggggtttcac catgttggcc aggctggtct tgaactcctg 4320
acctcgtgat ccgccctcct cggccaacca aagtgctggg attacaggcg tgagccaccg 4380
cgcctagccg aatgttaatt gtctaaaaat ttttcttctc caatgtcttc tcctccactt 4440
ttttcggaat ttgtttcttc ctaattacag cgcggtgtgg aggaaactgc agctgagtgc 4500
cgaaaactag gcgtcactgc gcatgcgtat gtggtagact gcagcaacag agaagagatc 4560
tatcgctctc taaatcaggt gagactgcag gttcacaaat ttcttcagat tattttgttt 4620
cctaggacgc tgacgtggaa aatgagaaag gtctttatga ctgcctgatt taaattggat 4680
tttagctgct aactgaagta gttatgtcac caaggaagga tatatacttt ttttcttgta 4740
tgtaatccac tcagctctgc ccattattat tgttcatatt attaatcaat ttcattctga 4800
tcagaagtgt gagcagtggc acagagtgac tgacaaaaga tttatcatca gggaatatgg 4860
atcacttcct agttttgttt tagtcctatt aactttgcag taattccagc ttctctttaa 4920
ttatttccct tgtgagattt tattttggtg ttaatgtagt cttctgtaga aaatgtaata 4980
ttaataatta ttatcacaat tattttaaaa gagtaaatac caaataatca caatgaacta 5040
agcactctaa caaactttac attttttaat tcaatcccta caataactct gtaaacttca 5100
ttttacagat aagcaaatta tgactcagag aggttaagcc agacccaggt catgtagtta 5160
ttaggttatg aaaccaggat ttctcaacca gcactttaga ccaggtgcgg tggttcacac 5220
atgtaatccc agcactttgt gaggccaagg tggaaggatc acatgagacc aagagttcaa 5280
gaccagccca ggcaacatag tgagacccta tctctaaaaa aaaaaaaaaa aaaaaaaaaa 5340
aaagtttaaa gaaaaacaca tttttaaaaa atgaacactt taaaaatatt tggtcagaat 5400
ttatatagga atttatcaac ataaatgtta atttcacttt actgataaac ttgcaaaaca 5460
tgatgtgctg ggtactgaaa tttagatgtt aaaagaacag tttatcccac ctttatgaca 5520
gtgttccctt ggcctccacg atttgagctc aacagtctgt cttgcctgaa ctctgagaga 5580
cctcatacaa tagaagaaag actctcatct ttggattata ttggtcccaa aactttgagt 5640
ttgaataata cacccagtga aagtgttctt tcaatttcaa aaggtgaaga aagaagtggg 5700
tgatgtaaca atcgtggtga ataatgctgg gacagtatat ccagccgatc ttctcagcac 5760
caaggatgaa gagattacca agacatttga ggtcaacatc ctaggacatt tttgggtgag 5820
tgtgagtcag aaacatttct gatttgtgca ccttctctta agatacatga aacttataac 5880
ggagttcaca tacttctgga caggaaactg gccagatctt tgccttaatc aagaatcatt 5940
aaatttgttt gagtagaaga gccacagagt ctctgacaca aggacacaga attcaagtgg 6000
acacaacaca ccagaatgta agctacttgg tctgtcttgt ccaccagtat ctgacacaaa 6060
gcttggcatg taccaggagc tcaacaaatg tttgtggagg tttgttaagg gttgtcagtg 6120
tacatctttt caatgctgtc acttgtgact tcattttttt ccctccacac catgattttg 6180
taatgtgtcc tcattttgtg gaattttaga atggaaagga catcagaagt aattacttgg 6240
atgtatatag gatcgaggac acttttggac gagactctga ggcaagtgtt ctagatccat 6300
ggggtgctgg aactgagaaa tgcagctata cagacctcat ataattggtt agttttgtgg 6360
gagatggaaa tatcaacttc aactgccttt gtatagaaat ttttatgatt aatcttccag 6420
tgcctcaata ttagtgtaga atctagggca gatctggatt ctagaagaaa gaagaaaaaa 6480
aagagatgtg tcccccttac ctttaccagc tcttcacata tgtgaattgg ctcccatgcc 6540
caccaaacta cacggagacc tcatacatta gctacctata gctgcataac aaattataca 6600
aaacttagtg gtttaaagca acaatgtatg ttcactatcc tctcacagtt tctatgggtt 6660
gggaatttgg aggtagcttg ggttgggagt tctagttcta tgaatttgca taggatttat 6720
taaattctta taaaatttta ttgatgtttc tcacaaaaga ggtttttgga aaaaaagaaa 6780
gacttgtttt ctgtaacatc aacatataat atacaatatt acaaataggg agatagtgaa 6840
ttcaatcatg attcattagt gtggtgtaga actctcagct tacactactc aactgtctta 6900
atacagttac acaagatttc actcttttaa ttagaatgat aaagccccaa accaaaaaat 6960
tatatgacac caaattatca taaggaataa ttttagttct gaaaactctg aatttttccc 7020
ttaatattgt ttagatgaca tatccaaaaa aggatctatt tgattccttc tgaagggaag 7080
gagggggagt actgagatta gtgttggcat ggggcttacc ataccaataa atttgtatct 7140
ttatttctat catttgtaaa gaattaatca tggaatgctt ggaagtattt tatttcattg 7200
tataagttct ctcaaatgcc tttctgtctt aacaaaaata aaactacctg atttggaaac 7260
ctaacgtcta tgtcattgtc tttcttcttt ctgcaatgat ccttaagatc acaaaagcac 7320
ttcttccatc gatgatggag agaaatcatg gccacatcgt cacagtggct tcagtgtgcg 7380
gccacgaagg gattccttac ctcatcccat attggtaagt atcacatgcc agccatgtta 7440
tatattttta tactttgaag ggagcattac acttcaaatt gttaccactg gagagtcctg 7500
gttcttggca tcttgaacaa agaattggac aaaactcacc aacaaagcca ggaaagaatg 7560
aagcaacaaa agcagagatt tattgaaaat gaaagtacgc tttacagggt gggagtgggc 7620
ccaagcacag gggctcaaga gccaattaca gaattttctg gggtttaaat accccctaga 7680
ggtttccact ggttacttgg tgtacgccct atgtaaatga agaggatgaa ttaaagttac 7740
agagtcgttt actcagtgta caccatatgt aaatggagag gatatttcct gtcatagctg 7800
gagtgtttcc atttgattta gttctaggaa gtcagcatga atcggcctta tgttccctgc 7860
ctccagaccc tgttctcctg cctcaagatt acaatgctga gagcagagtg atttggattt 7920
acagaattta aatttatagt agtttagaat gattttttaa atgacttttt ctaaaacaat 7980
gaaaccaggt tgtaattata tttaagatat ttttagattt ctgcaaactc ctctgtagaa 8040
caatgagaga aaacagtaat gccaagcatg tttccattgt ttcctggaat aagaaacaga 8100
aaccccacag actgagaagc aaaacctaca gaagctaaaa tgaacacatg tctatgtcat 8160
ggccttggtg cccaagataa gacaatcaga gtggtccctg gatcaaaaca ttttacagtg 8220
tgcttgtgcc atgaaagtgt gtgtgtgtgt gtgtgtgtgt gtgtgagaga gagagagaga 8280
gagaaaacga ctctacctga ctaaaagttg cagataccac actccatgca ccaccaaaga 8340
cataaaggga aggaggtgag aggcgttaag gatgtactgc tgtatttgcc aaatatcctt 8400
tcctgtaaac tcttctccag atcctcataa taaaattaag aggccaaagt ggcaaccatt 8460
gtcaagagaa aaactatcaa ccattgtcaa gagaataact cagttattga gagagagagg 8520
agaaatgagc agagtcctac agaagtctgt caacacagat accagttttg tagaatttct 8580
aaatgtattt ttcctgattc atatttttca aaataaaagc agcaataaaa actgattaga 8640
aaacagtttg aagattcaat ggaaaaacct tacatgtagg atggaaaact gaacattaag 8700
ccaatcaata gagttatttt tgttcttttg ttatcattgt tgtttaagaa atgagatacg 8760
ttcacaattc tgcttaatca tgtaagaaaa tgaaaatgaa ttgccattta tactctcaga 8820
aaaatcacaa gtggctgatt tttggcttcc acttgttctt aagccaaatg ataccgcctt 8880
ctcacagaaa gctgaggatt ggtttcactc tcccttagct aacaatgctt aataattctc 8940
ttacagttcc agcaaatttg ccgctgttgg ctttcacaga ggtctgacat cagaacttca 9000
ggccttggga aaaactggta tcaaaacctc atgtctctgc ccagtttttg tgaatactgg 9060
gttcaccaaa aatccaagca caaggtaagg tcaaaatcaa gttagaatgg gtatgtggta 9120
tgataaattg atatgaaaac taatgagaaa tgtttaggca ggccaactaa tagaagaaaa 9180
tgaagaagga aaaataattt ttcttattat tattattatc ttgaaattaa aggaataaag 9240
ggggaaaaca cattagggac tagcaggaat gatcagccac cgatgaactg ggatatttat 9300
ttgtgtccgg gagaaagcac atacatttga tcaccgttac caccctgtct ttaaaatgca 9360
aatgttccaa ggaccagcaa ataaattgag tatctagctc cttagtcaag gtgaatttct 9420
gcaagaactc ttgtctctgg tgagacagga tttgagacca caagagaaga aaaattagtc 9480
ctgaaaggag aagaaaaaag caggaaggtg tggataagaa cccgaaaatt aagccatctg 9540
cttaacaaat ttttctaatc ctagtatata ttctgctgca ggttaacaaa atatactaag 9600
cttaatgatt cgaaaccaat tttttactgg aagggaatta atcctaaata tattcattca 9660
aaagaactaa acaattctct gttgagtgcc gcctcatttg aggatactga ctcttacagc 9720
ctgagttagc tatgtggtct ctgcagctgg aatcactccc tgccactgga gtccttcatg 9780
gtgttagacc ataggtactg ttgactaaag aaaaaaaaaa gtttttgttt ttatttttgt 9840
tttttttgag acagagtctc actctgtcac ccaggctgga gtacagtggc gcgatctcag 9900
ctcaccgcaa cctccgcctt tctgggttca agcaattctc cttcctcagc ctcctgagta 9960
tttggattac aggcgcccac caccacgcct ggctaatttt tgtattttta gtagagacgg 10020
ggtttcacca tgttggccag gctggtctca aactcctgac ctcaggtgtc ctacctgcct 10080
tggcctccta aaatgctggg attacaggag tgagccacca tgcccggcca aaaaaataag 10140
tttttaaaga attaaaggtc atcctggcta acacagtgaa accccgtctc tactaaaaaa 10200
cacaaaaaaa ttagccgggc gtggtggcgg gcgcctgtag tcccagctgc gcgggaggct 10260
gaggcaggag aatggcgtga acccgggagg cggagcttgc agtgagccga gatcgcgcca 10320
ctgcactcca gcctgggcga cagagcgaga ctccgtctca aaaaaaaaaa aaaaaaaaaa 10380
aaaaaaaaga attaaaggtg ttaattttat ttagaagcct tactgaagac tacagtcgga 10440
ggcctatagc ctgagagcag ccctttagag aggttcagtt gaactgttct gatagtgggg 10500
gccatgtgct ctatcctgta ttgtcttcaa agcatctttc cagagagctg cacattgtca 10560
cagagtcagg gactttgtga aattatgctg acaaccagaa gtgagtaaac gtggcttctt 10620
acatttgcta cgttgtctca cagtacttaa taagtatgca atatgtaagt aaatactata 10680
gtactattgc aactcctgat tgttttctta gacaaggaat tgggcccaat aaaaaccctc 10740
ttggtaggca ttcaggcttc gtgtaccatg agctttccta agggtatcct gccactcttg 10800
gggaaggcat gatagatgag gggagtaagg ataatggaac tctgggtaca gggttcctgg 10860
gggctaactt agaggtagac acaggcaatg ctaaatattt gggattgatt ttatagaggt 10920
tgctagattg tgaatttcct tagtaagggc taaggcattg atatgtaatg tcacacttgg 10980
ctccgaggct gggttgttgg atccatgtag atgaaatcag ggagagaaag ggcagaacgg 11040
agtaatttag aaatgtattg atttgtatta ctctctgttg gcttgctatt caaggcagtg 11100
gagaactcaa tcacataata atctgcagca aaccacagat catcccaggg aatgaagttt 11160
taacattcgc tggctcccta actcctcacc cagcctttac attcactggc tgttcagtcc 11220
atgcctggac atcttaattt gaatacaaca ttttaaatcc atttttctgt catcatcttg 11280
cactaacaga caattctaca ctaagcctat gtttatgaat atttctcaag agtacatgta 11340
cacagccttc agtataagga aaactggaag tatgacatac ctccagttgt catactcctt 11400
gggcccctct taaattctca ttaaactgca ggataggcaa gtcagaggtg aatctcaaat 11460
acgaaattct taccggaaag gggttccaat ccagacccca agagagggtt cttagatttc 11520
tcgcaagaaa taattcgggg caaggccaca gtgcaaagca aaagcaagtt tattaggaaa 11580
gtaaaggagt agagaacagc tactccatgg agaagaatgg cttgagctgc tccaccaagg 11640
gtatttagag ttacttcttg attatatgct aaacaagggg tggattattc atgagttttc 11700
cgggaaaagg gtgagcaatt cccagaactg agatttcctc ccctttttag gccatatagg 11760
gtaacttcct gccattgcca tggtatttgt aaactgtcat agtgctggtg gaagtgtctc 11820
ttagcttgct aatgtattat agttagctta taatgagcag tgaggacaac cagaggtcac 11880
tttcatcacc atcttggttt tggtgggttt tggccggctt ctttactgca ccctatttta 11940
tcaacaaggt ctttatgacc tgaatcttgt gccaacctcc tatctcatcc tgtgacaaag 12000
aatgccttaa cttcctggga atgcagccca gtaggtgtca gccttatttt acccagaccc 12060
tattcaagat ggagttgctc tgatttaaac gcctctgaca aaatgacgac ctcaaaacaa 12120
tccagcttta tggaatacct ccacaagaaa gaaagtatac ttagctatag aattttctcc 12180
ttgcatccaa caggctttga gatgtcagat gtttccttcc tgtcccatga ttaatcctag 12240
ccattcctct ttcttgtctg gctccactac tccttaccat ctaatgcctc gccaccattt 12300
tgatattttg actaagtgag ctatgaaaca cacctactgg atatgaaagt ataagtttct 12360
gataacaaaa catcaacatg ggatgtggag gaagtgggta gggtggcatt aatgcagcaa 12420
atcctggaat attttaaatc ttcattctaa atttagtaaa aatataggat aattttcctg 12480
ccatcattta cttataaaat taaaatttta gaaaataaaa ataatatttt cctcttttta 12540
atcacagatt atggcctgta ttggagacag atgaagtcgt aagaagtctg atagatggaa 12600
tacttaccaa taagaaaatg atttttgttc catcgtatat caatatcttt ctgagactac 12660
agaagttaag tacagcacag aacacccaaa tactaaaaca ccaatagagc tttttttttt 12720
gctttttttt tttttagaca gagtctcact ctgtcaccct ggctggattg cggtggttgc 12780
agtggcatga tcttggctca ctgcaacctc cgcctcctgg gttcaagcaa ttctcatgcc 12840
tcagaccccc aagtaactgg gattataggt gtgtgctgcc acactacacc cagctaattt 12900
ttgtattttt tgatagagac aggtttcccc atgttggcca ggctggactc gaactcctga 12960
cctcaagtta tcctcctgtc tcggcctccc aaagtgctgg gattacagtc atgagccacc 13020
atgcctggcc caatagagct attattatgg agcatctttc agttgtgaaa attggcatgg 13080
aaactctcca tccctgggga gaacagttat ttcctctgtt attttcctac ccagtctata 13140
aaaagagagt gattcatttt ctctaccaaa tctactgtct ctgcccaaac tttgctgaag 13200
actattctaa ctaaaggaaa cacagtttaa aaagaatgca atatagtgaa gtagttaata 13260
ataaagactc catttttaaa agtctgctgg aagtttggtt gggattgcac tgaatctata 13320
gagcaattgg ggagtattga catatcaaca atattgagtt ttctaatcca agaacataat 13380
atctattttt aaaatcttct tcaaaatctt taaatcttta aattgtattt tgtagttttt 13440
ggtgtttaag tcttgcacat attttgtcag atttattcca aagtatttca cgggttcttt 13500
tttttttttt tttttttttt tttgagacag agtttcaccc ttgttgccca ggctggagtg 13560
cagtggcgtg atcttggctc actgcagctt ctgcctcctg gcttcaagtg attctcctgc 13620
ctcagcctcc caagtagctg ggattacagg cacctgcccc ctcgcccaac taactttttg 13680
tgtttgtagt agagacaggg tttcaccatg ttggccaggc tggtctcgaa ctcctgacct 13740
catgtgatcc acctgcctca gcctcccaaa gtgctgggat tacaggcatg agccatcatg 13800
cccagcccta tttgacggtt tttgacgcta atgcaagtgg cattttaaaa aattttatat 13860
ttcccattgt ttgttgtcag tatatattgg atttttgtaa tttgatctca tattttgcag 13920
tcttgctaaa ttgctaaacc tctttttgct aaactcgata agcttttttt tttttggtag 13980
attcctgggc ctctaatttt ctttatggga aagtttttaa ttacaaattt aatttcttta 14040
atagctacat ggctattcaa tttacttatt aattcttggt aatgtgtgtc tttcaaggaa 14100
tttgtccatt tcatctaagt tgtagaattt ctttggcata aatttgtaca taacattccc 14160
ttattatcct tttaatgtct ttagaatgtc ttatttattt atttatttat ttttattata 14220
tttttttgag acagagtctc gctctgttgc ccaggctgga gtgcagtggc acaatcttgg 14280
ctcactgcaa gctccgcctt ctgggttcat gccattctcc tgcctcagcc tccctagttg 14340
ctgggactac aggcgcctgc aaccatgccc agcttatttt tttttttttt tttttttttt 14400
tttttttttt tttttttttt ttagtagaga cggggtttca ccctgttagc caggatggtc 14460
tcgatctcct gacctggtga tccgcccgcc tcagcctccc aaagtgctgg gattacaggc 14520
gtgagccacc aagcccagcc tatttattta tttagtagag acagtctcac tttgctgccc 14580
aggcaacaaa ggttttgaat gcctggcctc aagcagtcct cctgccttgg cctcccaaag 14640
tgctgggatt acaggcatga gccactgcac ctggccaaat gaatatgctg ataatatctt 14700
ctttataagg atgacataag aataaaataa tgtaatacaa acaaagcccc tgtcactgaa 14760
aatgtataga cttcaaatgt taaagtctta gagaacagaa tttatatgaa atagcaacag 14820
caacaatttc ccagaggaaa tactctctca gctttcttct gaggagcagt ttctaaattg 14880
aaattgtatc agtgagaaga taactatact aacttcataa gccttgggcc tttttgaaac 14940
aaatccatat aaactatgaa caaacttgaa atagaacaat ttgagaacag ggtacaaact 15000
gcattggtgt atcaatttca gtatttggtt ttagcttaaa tagactgact tgagataaca 15060
taaggagaac cttgaccccc aagcaacatc atctcgcgag ttgactaggc cgggtgtggt 15120
gtctcacgcc tgtaattcca gcactttggg aggccacagc aggcagatca cttgaggtca 15180
ggcattcgag accagcctgg ccaacatggt gaaacctcag ctctactaaa gatacgaaaa 15240
ttagcaggca tagtggcctg cacctgtaat accaggcact cgcaggagaa tcccttgaac 15300
ccggaaggcg gagattgcag taaaccatga ttgtgccact gcactccagc ctgggcaaca 15360
ggagactctg tctcggaaaa ataaattttt taaaaaaatg aaaaaaaata aaagttgact 15420
aaattagtgt cttggtacta agcactgtag gaagtgagtt tcatggaacc ccaactctct 15480
tggggcccaa agcaagtcat attaatattg aaaattacat gcatatacat gcatatgacc 15540
aaggtgataa aaacaattat tctgcctgag ttggagaata gtatcccagt aaaataaaca 15600
agagtctcaa agtcttttgt atcctttgaa gctgtcatgg tggtttgtaa ctaggcaaca 15660
ggtatatatt gttaatcttc tttgcattta attcctttta tagagagaca caattttacg 15720
agcagatgca attactagca tgaaggtttc tttgtgaggg tagttaaaag gcccacatga 15780
gctctcttct tatccttgtc cttctttcag ccagatcttc cctgcccctt tgctcattcc 15840
atctttcacc cacctacccc caaaacaagg aagtaaatct tgcattagtc aacaatacca 15900
aagtgatttt caatatgact ttctctgcag aatgttatta tttctgcctc tttacattca 15960
catactgtct tccttttttt tttttttttt tttttttttt ttagattggg tctcactctg 16020
ttgcccaggc tggagtgcag tggcttgatc tcagctcact gtaacctcca cctcctgagt 16080
tcaagcaatt ctcctgcctc agcctcctga gtagctggga ttacaggcat gtgccaccac 16140
acctggctag tttttttgta tttttagtag agacagggtt tcaccatgtt ggtcaagctg 16200
gtctcgaact cctgacctca tgatctgacc acctgtgcct ctcaaagtgc tgggattaca 16260
ggcgtgagcc accgggccag ccactctctt cctttcagtt gcctactcat ctcttatgca 16320
ttcctggaca tcagttgtcc ttttgaagct ttcctccact atcccagccc atgtgaatcc 16380
tccttccagt tatagccctt aattctagat ggctgatatt tttcaataat tgttttaaga 16440
tgaccatttt agcctatcag ctaaacaata tcaaagacaa tagctatttt tcaagtactt 16500
tagtttacct tattatagag tgcataatag atattcagta aatagtaaag gagaggtgaa 16560
ggcttgcata gaatggattc tggtggtgtc tcttggtgag cttttagcat caagattaat 16620
cagcagtttc agcaatgagc tcagaccttc agttttagat ctttactcat atcagataag 16680
agagtgagaa gagtggtatg tatcagtgct ttatttatat ttgcatccaa tttgaactat 16740
gaatattaca aaggtgcaca cataggttca gacagattga tttaaaatga ccaaagatga 16800
cctgtcgtaa gcaacctggg tatcttaaga tgcactcctt ggagagggaa tgttcctaaa 16860
aacattttca gagggacgaa ctgtatgaaa ttcagtaaaa cataaatcat gaggaaaact 16920
gattactctc tttttgacat gaaatgagag ttttaatgca tggttacgat tattaacgta 16980
ctccgctgca agacgttaat aaagttactg ttttgcaggc tagaatgtct tgatgctgta 17040
atcagaacac actttttccc ctttcttcca gcttcaaatg cagattcata attgggctga 17100
cttctaataa ctgcaatgtt ttctgccttg ggcttgcagc agaagcctga caaaatagtg 17160
tttgtttagg caataattta tttatttatt tattgagatg gagtttcatt cttgtcgccc 17220
aggctggagt gcaatggcgt gatctcggct cactgcaacc tctgtgttca ggcaataatt 17280
tagactttac cttacttgtg attactatag caattactat agccacaagg cataatttta 17340
ctgtctcatt tcaattttat gaatttgaat gtttttacac ttttcctaat gaagtccact 17400
atgaagttat gtcaaaaaaa aaaaagaaaa agaaagatgc acacgtaaaa gagaggtggt 17460
tgcaagagaa gaaaagaacg gaggaaagtt aaacgcaaac cagataactc tcagcgtatt 17520
ctaaatgacc aaaaacagaa ctctgttgtc aaagatttta aatggaaaat ttttcaattt 17580
ttttttcttt tttgtacagg tttcttcctg aacgcgcctc agcgatttta aatcgtatgc 17640
agaatattca atttgaagca gtggttggcc acaaaatcaa aatgaaatga ataaataagc 17700
tccagccaga gatgtatgca tgataatgat atgaatagtt tcgaatcaat gctgcaaagc 17760
tttatttcac attttttcag tcctgataat attaaaaaca ttggtttggc actagcagca 17820
gtcaaacgaa caagattaat tacctgtctt cctgtttctc aagaatattt acgtagtttt 17880
tcataggtct gtttttcctt tcatgcctct taaaaacttc tgtgcttaca taaacatact 17940
taaaaggttt tctttaagat attttatttt tccatttaaa ggtggacaaa agctacctcc 18000
ctaaaagtaa atacaaagag aacttattta cacagggaag gtttaagact gttcaagtag 18060
cattccaatc tgtagccatg ccacagaata tcaacaagaa cacagaatga gtgcacagct 18120
aagagatcaa gtttcagcag gcagctttat ctcaacctgg acatatttta agattcagca 18180
tttgaaagat ttccctagcc tcttcctttt tcattagccc aaaacggtgc aactctattc 18240
tggactttat tacttgattc tgtcttctgt ataactctga agtccaccaa aagtggaccc 18300
tctatatttc ctcccttttt atagtcttat aagatacatt atgaaaggtg accgactcta 18360
ttttaaatct cagaatttta agttctagcc ccatgataac ctttttcttt gtaatttatg 18420
ctttcatata tccttggtcc cagagatgtt tagacaattt taggctcaaa aattaaagct 18480
aacacaggaa aaggaactgt actggctatt acataagaaa caatggaccc aagagaagaa 18540
aaggaagaaa gaaaggtttt ttggtttttg ttttgttttg ttttgttttt tgtttttttg 18600
agatggagtc tcactctttc gcccaggctg gagtgcagtg gtatgatctc agctcactgc 18660
aagctccacc tcccgggttc acgccattct cctgcctcag cctcctgagt agctgggact 18720
acaggcgccc gccaccacac ccggctaatt ttttgtattt tttgtagaga cggggtttca 18780
ccatgttagc caagatggtc tcgatctcct gacctcgtga tccacctgcc tcggcctccc 18840
aaagtgctgg gattacgggt gtgagccacc gtgcccagcc tttttttttt taatagaaaa 18900
aataatccga ctcccactac atcaagacta atcttgtttt gtgtgttttt cacatgtatt 18960
atagaatgct tttgcatgga ctatcctctt gtttttatta aaaacaaatg atttttttaa 19020
aagtcacaaa aacaattcac taaaaataaa tatgtcattg tgctttaaaa aaataacctc 19080
ttgtagttat aaaataaaac gtttgacttc taaactctg 19119
<210> 3
<211> 600
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(499)
<223> Выше TSS
<400> 3
cactgcacca cgaactcatg gactgaagca atcctcctgc ctcagcctcc tgggtagctg 60
ggactacaga cacatgccac catatccagc taattttttt ctatagtttt tttttttttt 120
tttgagacag ggtcttacta tgttgcccag actggtctcg aactcctggg ctcaagcaat 180
cctctgcctc agcctcccaa agtgctggga ttacagatgt gagccactgc acctggcccc 240
tagaattgtt tctagaggtg aaacttcaag gtgaaatata gtacataact gcttttcaga 300
taaacaagtc cagagagcac actctcttgt gctcttggca tcacttggca tcacttcata 360
tttgaggtgt ttcaaaccca ttagaacacg tgaacaaggc ctgcttccaa agctggcttc 420
catctggtag tcccattaac aactgggcac accccttccc tagagctctg tgtagacagt 480
acctcctccc taggactaca caaggactga accagaagga agaggacaga gcaaagccat 540
gaacatcatc ctagaaatcc ttctgcttct gatcaccatc atctactcct acttggagtc 600
<210> 4
<211> 2397
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(383)
<223> Экзон 2
<220>
<221> misc_feature
<222> (384)..(515)
<223> Экзон 3
<220>
<221> misc_feature
<222> (516)..(622)
<223> Экзон 4
<220>
<221> misc_feature
<222> (623)..(760)
<223> Экзон 5
<220>
<221> misc_feature
<222> (761)..(877)
<223> Экзон 6v1
<220>
<221> misc_feature
<222> (878)..(2397)
<223> Экзон 7
<400> 4
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataagcgcgg tgtggaggaa actgcagctg 300
agtgccgaaa actaggcgtc actgcgcatg cgtatgtggt agactgcagc aacagagaag 360
agatctatcg ctctctaaat caggtgaaga aagaagtggg tgatgtaaca atcgtggtga 420
ataatgctgg gacagtatat ccagccgatc ttctcagcac caaggatgaa gagattacca 480
agacatttga ggtcaacatc ctaggacatt tttggatcac aaaagcactt cttccatcga 540
tgatggagag aaatcatggc cacatcgtca cagtggcttc agtgtgcggc cacgaaggga 600
ttccttacct catcccatat tgttccagca aatttgccgc tgttggcttt cacagaggtc 660
tgacatcaga acttcaggcc ttgggaaaaa ctggtatcaa aacctcatgt ctctgcccag 720
tttttgtgaa tactgggttc accaaaaatc caagcacaag attatggcct gtattggaga 780
cagatgaagt cgtaagaagt ctgatagatg gaatacttac caataagaaa atgatttttg 840
ttccatcgta tatcaatatc tttctgagac tacagaagtt tcttcctgaa cgcgcctcag 900
cgattttaaa tcgtatgcag aatattcaat ttgaagcagt ggttggccac aaaatcaaaa 960
tgaaatgaat aaataagctc cagccagaga tgtatgcatg ataatgatat gaatagtttc 1020
gaatcaatgc tgcaaagctt tatttcacat tttttcagtc ctgataatat taaaaacatt 1080
ggtttggcac tagcagcagt caaacgaaca agattaatta cctgtcttcc tgtttctcaa 1140
gaatatttac gtagtttttc ataggtctgt ttttcctttc atgcctctta aaaacttctg 1200
tgcttacata aacatactta aaaggttttc tttaagatat tttatttttc catttaaagg 1260
tggacaaaag ctacctccct aaaagtaaat acaaagagaa cttatttaca cagggaaggt 1320
ttaagactgt tcaagtagca ttccaatctg tagccatgcc acagaatatc aacaagaaca 1380
cagaatgagt gcacagctaa gagatcaagt ttcagcaggc agctttatct caacctggac 1440
atattttaag attcagcatt tgaaagattt ccctagcctc ttcctttttc attagcccaa 1500
aacggtgcaa ctctattctg gactttatta cttgattctg tcttctgtat aactctgaag 1560
tccaccaaaa gtggaccctc tatatttcct ccctttttat agtcttataa gatacattat 1620
gaaaggtgac cgactctatt ttaaatctca gaattttaag ttctagcccc atgataacct 1680
ttttctttgt aatttatgct ttcatatatc cttggtccca gagatgttta gacaatttta 1740
ggctcaaaaa ttaaagctaa cacaggaaaa ggaactgtac tggctattac ataagaaaca 1800
atggacccaa gagaagaaaa ggaagaaaga aaggtttttt ggtttttgtt ttgttttgtt 1860
ttgttttttg tttttttgag atggagtctc actctttcgc ccaggctgga gtgcagtggt 1920
atgatctcag ctcactgcaa gctccacctc ccgggttcac gccattctcc tgcctcagcc 1980
tcctgagtag ctgggactac aggcgcccgc caccacaccc ggctaatttt ttgtattttt 2040
tgtagagacg gggtttcacc atgttagcca agatggtctc gatctcctga cctcgtgatc 2100
cacctgcctc ggcctcccaa agtgctggga ttacgggtgt gagccaccgt gcccagcctt 2160
ttttttttta atagaaaaaa taatccgact cccactacat caagactaat cttgttttgt 2220
gtgtttttca catgtattat agaatgcttt tgcatggact atcctcttgt ttttattaaa 2280
aacaaatgat ttttttaaaa gtcacaaaaa caattcacta aaaataaata tgtcattgtg 2340
ctttaaaaaa ataacctctt gtagttataa aataaaacgt ttgacttcta aactctg 2397
<210> 5
<211> 2289
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(407)
<223> Экзон 3
<220>
<221> misc_feature
<222> (408)..(514)
<223> Экзон 4
<220>
<221> misc_feature
<222> (515)..(652)
<223> Экзон 5
<220>
<221> misc_feature
<222> (653)..(769)
<223> Экзон 6v1
<220>
<221> misc_feature
<222> (770)..(2289)
<223> Экзон 7
<400> 5
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataaggtgaa gaaagaagtg ggtgatgtaa 300
caatcgtggt gaataatgct gggacagtat atccagccga tcttctcagc accaaggatg 360
aagagattac caagacattt gaggtcaaca tcctaggaca tttttggatc acaaaagcac 420
ttcttccatc gatgatggag agaaatcatg gccacatcgt cacagtggct tcagtgtgcg 480
gccacgaagg gattccttac ctcatcccat attgttccag caaatttgcc gctgttggct 540
ttcacagagg tctgacatca gaacttcagg ccttgggaaa aactggtatc aaaacctcat 600
gtctctgccc agtttttgtg aatactgggt tcaccaaaaa tccaagcaca agattatggc 660
ctgtattgga gacagatgaa gtcgtaagaa gtctgataga tggaatactt accaataaga 720
aaatgatttt tgttccatcg tatatcaata tctttctgag actacagaag tttcttcctg 780
aacgcgcctc agcgatttta aatcgtatgc agaatattca atttgaagca gtggttggcc 840
acaaaatcaa aatgaaatga ataaataagc tccagccaga gatgtatgca tgataatgat 900
atgaatagtt tcgaatcaat gctgcaaagc tttatttcac attttttcag tcctgataat 960
attaaaaaca ttggtttggc actagcagca gtcaaacgaa caagattaat tacctgtctt 1020
cctgtttctc aagaatattt acgtagtttt tcataggtct gtttttcctt tcatgcctct 1080
taaaaacttc tgtgcttaca taaacatact taaaaggttt tctttaagat attttatttt 1140
tccatttaaa ggtggacaaa agctacctcc ctaaaagtaa atacaaagag aacttattta 1200
cacagggaag gtttaagact gttcaagtag cattccaatc tgtagccatg ccacagaata 1260
tcaacaagaa cacagaatga gtgcacagct aagagatcaa gtttcagcag gcagctttat 1320
ctcaacctgg acatatttta agattcagca tttgaaagat ttccctagcc tcttcctttt 1380
tcattagccc aaaacggtgc aactctattc tggactttat tacttgattc tgtcttctgt 1440
ataactctga agtccaccaa aagtggaccc tctatatttc ctcccttttt atagtcttat 1500
aagatacatt atgaaaggtg accgactcta ttttaaatct cagaatttta agttctagcc 1560
ccatgataac ctttttcttt gtaatttatg ctttcatata tccttggtcc cagagatgtt 1620
tagacaattt taggctcaaa aattaaagct aacacaggaa aaggaactgt actggctatt 1680
acataagaaa caatggaccc aagagaagaa aaggaagaaa gaaaggtttt ttggtttttg 1740
ttttgttttg ttttgttttt tgtttttttg agatggagtc tcactctttc gcccaggctg 1800
gagtgcagtg gtatgatctc agctcactgc aagctccacc tcccgggttc acgccattct 1860
cctgcctcag cctcctgagt agctgggact acaggcgccc gccaccacac ccggctaatt 1920
ttttgtattt tttgtagaga cggggtttca ccatgttagc caagatggtc tcgatctcct 1980
gacctcgtga tccacctgcc tcggcctccc aaagtgctgg gattacgggt gtgagccacc 2040
gtgcccagcc tttttttttt taatagaaaa aataatccga ctcccactac atcaagacta 2100
atcttgtttt gtgtgttttt cacatgtatt atagaatgct tttgcatgga ctatcctctt 2160
gtttttatta aaaacaaatg atttttttaa aagtcacaaa aacaattcac taaaaataaa 2220
tatgtcattg tgctttaaaa aaataacctc ttgtagttat aaaataaaac gtttgacttc 2280
taaactctg 2289
<210> 6
<211> 2280
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(383)
<223> Экзон 2
<220>
<221> misc_feature
<222> (384)..(515)
<223> Экзон 3
<220>
<221> misc_feature
<222> (516)..(622)
<223> Экзон 4
<220>
<221> misc_feature
<222> (623)..(760)
<223> Экзон 5
<220>
<221> misc_feature
<222> (761)..(2280)
<223> Экзон 7
<400> 6
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataagcgcgg tgtggaggaa actgcagctg 300
agtgccgaaa actaggcgtc actgcgcatg cgtatgtggt agactgcagc aacagagaag 360
agatctatcg ctctctaaat caggtgaaga aagaagtggg tgatgtaaca atcgtggtga 420
ataatgctgg gacagtatat ccagccgatc ttctcagcac caaggatgaa gagattacca 480
agacatttga ggtcaacatc ctaggacatt tttggatcac aaaagcactt cttccatcga 540
tgatggagag aaatcatggc cacatcgtca cagtggcttc agtgtgcggc cacgaaggga 600
ttccttacct catcccatat tgttccagca aatttgccgc tgttggcttt cacagaggtc 660
tgacatcaga acttcaggcc ttgggaaaaa ctggtatcaa aacctcatgt ctctgcccag 720
tttttgtgaa tactgggttc accaaaaatc caagcacaag gtttcttcct gaacgcgcct 780
cagcgatttt aaatcgtatg cagaatattc aatttgaagc agtggttggc cacaaaatca 840
aaatgaaatg aataaataag ctccagccag agatgtatgc atgataatga tatgaatagt 900
ttcgaatcaa tgctgcaaag ctttatttca cattttttca gtcctgataa tattaaaaac 960
attggtttgg cactagcagc agtcaaacga acaagattaa ttacctgtct tcctgtttct 1020
caagaatatt tacgtagttt ttcataggtc tgtttttcct ttcatgcctc ttaaaaactt 1080
ctgtgcttac ataaacatac ttaaaaggtt ttctttaaga tattttattt ttccatttaa 1140
aggtggacaa aagctacctc cctaaaagta aatacaaaga gaacttattt acacagggaa 1200
ggtttaagac tgttcaagta gcattccaat ctgtagccat gccacagaat atcaacaaga 1260
acacagaatg agtgcacagc taagagatca agtttcagca ggcagcttta tctcaacctg 1320
gacatatttt aagattcagc atttgaaaga tttccctagc ctcttccttt ttcattagcc 1380
caaaacggtg caactctatt ctggacttta ttacttgatt ctgtcttctg tataactctg 1440
aagtccacca aaagtggacc ctctatattt cctccctttt tatagtctta taagatacat 1500
tatgaaaggt gaccgactct attttaaatc tcagaatttt aagttctagc cccatgataa 1560
cctttttctt tgtaatttat gctttcatat atccttggtc ccagagatgt ttagacaatt 1620
ttaggctcaa aaattaaagc taacacagga aaaggaactg tactggctat tacataagaa 1680
acaatggacc caagagaaga aaaggaagaa agaaaggttt tttggttttt gttttgtttt 1740
gttttgtttt ttgttttttt gagatggagt ctcactcttt cgcccaggct ggagtgcagt 1800
ggtatgatct cagctcactg caagctccac ctcccgggtt cacgccattc tcctgcctca 1860
gcctcctgag tagctgggac tacaggcgcc cgccaccaca cccggctaat tttttgtatt 1920
ttttgtagag acggggtttc accatgttag ccaagatggt ctcgatctcc tgacctcgtg 1980
atccacctgc ctcggcctcc caaagtgctg ggattacggg tgtgagccac cgtgcccagc 2040
cttttttttt ttaatagaaa aaataatccg actcccacta catcaagact aatcttgttt 2100
tgtgtgtttt tcacatgtat tatagaatgc ttttgcatgg actatcctct tgtttttatt 2160
aaaaacaaat gattttttta aaagtcacaa aaacaattca ctaaaaataa atatgtcatt 2220
gtgctttaaa aaaataacct cttgtagtta taaaataaaa cgtttgactt ctaaactctg 2280
<210> 7
<211> 2398
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(383)
<223> Экзон 2
<220>
<221> misc_feature
<222> (384)..(515)
<223> Экзон 3
<220>
<221> misc_feature
<222> (516)..(622)
<223> Экзон 4
<220>
<221> misc_feature
<222> (623)..(760)
<223> Экзон 5
<220>
<221> misc_feature
<222> (761)..(878)
<223> Экзон 6v2 - включает дополнительный остаток 878 на 3’-конце
<220>
<221> misc_feature
<222> (879)..(2398)
<223> Экзон 7
<400> 7
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataagcgcgg tgtggaggaa actgcagctg 300
agtgccgaaa actaggcgtc actgcgcatg cgtatgtggt agactgcagc aacagagaag 360
agatctatcg ctctctaaat caggtgaaga aagaagtggg tgatgtaaca atcgtggtga 420
ataatgctgg gacagtatat ccagccgatc ttctcagcac caaggatgaa gagattacca 480
agacatttga ggtcaacatc ctaggacatt tttggatcac aaaagcactt cttccatcga 540
tgatggagag aaatcatggc cacatcgtca cagtggcttc agtgtgcggc cacgaaggga 600
ttccttacct catcccatat tgttccagca aatttgccgc tgttggcttt cacagaggtc 660
tgacatcaga acttcaggcc ttgggaaaaa ctggtatcaa aacctcatgt ctctgcccag 720
tttttgtgaa tactgggttc accaaaaatc caagcacaag attatggcct gtattggaga 780
cagatgaagt cgtaagaagt ctgatagatg gaatacttac caataagaaa atgatttttg 840
ttccatcgta tatcaatatc tttctgagac tacagaaggt ttcttcctga acgcgcctca 900
gcgattttaa atcgtatgca gaatattcaa tttgaagcag tggttggcca caaaatcaaa 960
atgaaatgaa taaataagct ccagccagag atgtatgcat gataatgata tgaatagttt 1020
cgaatcaatg ctgcaaagct ttatttcaca ttttttcagt cctgataata ttaaaaacat 1080
tggtttggca ctagcagcag tcaaacgaac aagattaatt acctgtcttc ctgtttctca 1140
agaatattta cgtagttttt cataggtctg tttttccttt catgcctctt aaaaacttct 1200
gtgcttacat aaacatactt aaaaggtttt ctttaagata ttttattttt ccatttaaag 1260
gtggacaaaa gctacctccc taaaagtaaa tacaaagaga acttatttac acagggaagg 1320
tttaagactg ttcaagtagc attccaatct gtagccatgc cacagaatat caacaagaac 1380
acagaatgag tgcacagcta agagatcaag tttcagcagg cagctttatc tcaacctgga 1440
catattttaa gattcagcat ttgaaagatt tccctagcct cttccttttt cattagccca 1500
aaacggtgca actctattct ggactttatt acttgattct gtcttctgta taactctgaa 1560
gtccaccaaa agtggaccct ctatatttcc tcccttttta tagtcttata agatacatta 1620
tgaaaggtga ccgactctat tttaaatctc agaattttaa gttctagccc catgataacc 1680
tttttctttg taatttatgc tttcatatat ccttggtccc agagatgttt agacaatttt 1740
aggctcaaaa attaaagcta acacaggaaa aggaactgta ctggctatta cataagaaac 1800
aatggaccca agagaagaaa aggaagaaag aaaggttttt tggtttttgt tttgttttgt 1860
tttgtttttt gtttttttga gatggagtct cactctttcg cccaggctgg agtgcagtgg 1920
tatgatctca gctcactgca agctccacct cccgggttca cgccattctc ctgcctcagc 1980
ctcctgagta gctgggacta caggcgcccg ccaccacacc cggctaattt tttgtatttt 2040
ttgtagagac ggggtttcac catgttagcc aagatggtct cgatctcctg acctcgtgat 2100
ccacctgcct cggcctccca aagtgctggg attacgggtg tgagccaccg tgcccagcct 2160
tttttttttt aatagaaaaa ataatccgac tcccactaca tcaagactaa tcttgttttg 2220
tgtgtttttc acatgtatta tagaatgctt ttgcatggac tatcctcttg tttttattaa 2280
aaacaaatga tttttttaaa agtcacaaaa acaattcact aaaaataaat atgtcattgt 2340
gctttaaaaa aataacctct tgtagttata aaataaaacg tttgacttct aaactctg 2398
<210> 8
<211> 2469
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(383)
<223> Экзон 2
<220>
<221> misc_feature
<222> (384)..(515)
<223> Экзон 3
<220>
<221> misc_feature
<222> (516)..(587)
<223> Экзон 3'
<220>
<221> misc_feature
<222> (588)..(694)
<223> Экзон 4
<220>
<221> misc_feature
<222> (695)..(832)
<223> Экзон 5
<220>
<221> misc_feature
<222> (833)..(949)
<223> Экзон 6v1
<220>
<221> misc_feature
<222> (950)..(2469)
<223> Экзон 7
<400> 8
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataagcgcgg tgtggaggaa actgcagctg 300
agtgccgaaa actaggcgtc actgcgcatg cgtatgtggt agactgcagc aacagagaag 360
agatctatcg ctctctaaat caggtgaaga aagaagtggg tgatgtaaca atcgtggtga 420
ataatgctgg gacagtatat ccagccgatc ttctcagcac caaggatgaa gagattacca 480
agacatttga ggtcaacatc ctaggacatt tttggaatgg aaaggacatc agaagtaatt 540
acttggatgt atataggatc gaggacactt ttggacgaga ctctgagatc acaaaagcac 600
ttcttccatc gatgatggag agaaatcatg gccacatcgt cacagtggct tcagtgtgcg 660
gccacgaagg gattccttac ctcatcccat attgttccag caaatttgcc gctgttggct 720
ttcacagagg tctgacatca gaacttcagg ccttgggaaa aactggtatc aaaacctcat 780
gtctctgccc agtttttgtg aatactgggt tcaccaaaaa tccaagcaca agattatggc 840
ctgtattgga gacagatgaa gtcgtaagaa gtctgataga tggaatactt accaataaga 900
aaatgatttt tgttccatcg tatatcaata tctttctgag actacagaag tttcttcctg 960
aacgcgcctc agcgatttta aatcgtatgc agaatattca atttgaagca gtggttggcc 1020
acaaaatcaa aatgaaatga ataaataagc tccagccaga gatgtatgca tgataatgat 1080
atgaatagtt tcgaatcaat gctgcaaagc tttatttcac attttttcag tcctgataat 1140
attaaaaaca ttggtttggc actagcagca gtcaaacgaa caagattaat tacctgtctt 1200
cctgtttctc aagaatattt acgtagtttt tcataggtct gtttttcctt tcatgcctct 1260
taaaaacttc tgtgcttaca taaacatact taaaaggttt tctttaagat attttatttt 1320
tccatttaaa ggtggacaaa agctacctcc ctaaaagtaa atacaaagag aacttattta 1380
cacagggaag gtttaagact gttcaagtag cattccaatc tgtagccatg ccacagaata 1440
tcaacaagaa cacagaatga gtgcacagct aagagatcaa gtttcagcag gcagctttat 1500
ctcaacctgg acatatttta agattcagca tttgaaagat ttccctagcc tcttcctttt 1560
tcattagccc aaaacggtgc aactctattc tggactttat tacttgattc tgtcttctgt 1620
ataactctga agtccaccaa aagtggaccc tctatatttc ctcccttttt atagtcttat 1680
aagatacatt atgaaaggtg accgactcta ttttaaatct cagaatttta agttctagcc 1740
ccatgataac ctttttcttt gtaatttatg ctttcatata tccttggtcc cagagatgtt 1800
tagacaattt taggctcaaa aattaaagct aacacaggaa aaggaactgt actggctatt 1860
acataagaaa caatggaccc aagagaagaa aaggaagaaa gaaaggtttt ttggtttttg 1920
ttttgttttg ttttgttttt tgtttttttg agatggagtc tcactctttc gcccaggctg 1980
gagtgcagtg gtatgatctc agctcactgc aagctccacc tcccgggttc acgccattct 2040
cctgcctcag cctcctgagt agctgggact acaggcgccc gccaccacac ccggctaatt 2100
ttttgtattt tttgtagaga cggggtttca ccatgttagc caagatggtc tcgatctcct 2160
gacctcgtga tccacctgcc tcggcctccc aaagtgctgg gattacgggt gtgagccacc 2220
gtgcccagcc tttttttttt taatagaaaa aataatccga ctcccactac atcaagacta 2280
atcttgtttt gtgtgttttt cacatgtatt atagaatgct tttgcatgga ctatcctctt 2340
gtttttatta aaaacaaatg atttttttaa aagtcacaaa aacaattcac taaaaataaa 2400
tatgtcattg tgctttaaaa aaataacctc ttgtagttat aaaataaaac gtttgacttc 2460
taaactctg 2469
<210> 9
<211> 1715
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(383)
<223> Экзон 2
<220>
<221> misc_feature
<222> (384)..(515)
<223> Экзон 3
<220>
<221> misc_feature
<222> (516)..(622)
<223> Экзон 4
<220>
<221> misc_feature
<222> (623)..(760)
<223> Экзон 5
<220>
<221> misc_feature
<222> (761)..(1715)
<223> Экзон 6v3
<220>
<221> misc_feature
<222> (878)..(1715)
<223> Участок считывания из экзона 6 в интрон 6
<220>
<221> misc_feature
<222> (1716)..(1715)
<223> Участок считывания из экзона 6 в интрон 6
<400> 9
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataagcgcgg tgtggaggaa actgcagctg 300
agtgccgaaa actaggcgtc actgcgcatg cgtatgtggt agactgcagc aacagagaag 360
agatctatcg ctctctaaat caggtgaaga aagaagtggg tgatgtaaca atcgtggtga 420
ataatgctgg gacagtatat ccagccgatc ttctcagcac caaggatgaa gagattacca 480
agacatttga ggtcaacatc ctaggacatt tttggatcac aaaagcactt cttccatcga 540
tgatggagag aaatcatggc cacatcgtca cagtggcttc agtgtgcggc cacgaaggga 600
ttccttacct catcccatat tgttccagca aatttgccgc tgttggcttt cacagaggtc 660
tgacatcaga acttcaggcc ttgggaaaaa ctggtatcaa aacctcatgt ctctgcccag 720
tttttgtgaa tactgggttc accaaaaatc caagcacaag attatggcct gtattggaga 780
cagatgaagt cgtaagaagt ctgatagatg gaatacttac caataagaaa atgatttttg 840
ttccatcgta tatcaatatc tttctgagac tacagaagtt aagtacagca cagaacaccc 900
aaatactaaa acaccaatag agcttttttt tttgcttttt ttttttttag acagagtctc 960
actctgtcac cctggctgga ttgcggtggt tgcagtggca tgatcttggc tcactgcaac 1020
ctccgcctcc tgggttcaag caattctcat gcctcagacc cccaagtaac tgggattata 1080
ggtgtgtgct gccacactac acccagctaa tttttgtatt ttttgataga gacaggtttc 1140
cccatgttgg ccaggctgga ctcgaactcc tgacctcaag ttatcctcct gtctcggcct 1200
cccaaagtgc tgggattaca gtcatgagcc accatgcctg gcccaataga gctattatta 1260
tggagcatct ttcagttgtg aaaattggca tggaaactct ccatccctgg ggagaacagt 1320
tatttcctct gttattttcc tacccagtct ataaaaagag agtgattcat tttctctacc 1380
aaatctactg tctctgccca aactttgctg aagactattc taactaaagg aaacacagtt 1440
taaaaagaat gcaatatagt gaagtagtta ataataaaga ctccattttt aaaagtctgc 1500
tggaagtttg gttgggattg cactgaatct atagagcaat tggggagtat tgacatatca 1560
acaatattga gttttctaat ccaagaacat aatatctatt tttaaaatct tcttcaaaat 1620
ctttaaatct ttaaattgta ttttgtagtt tttggtgttt aagtcttgca catattttgt 1680
cagatttatt ccaaagtatt tcacgggttc ttttt 1715
<210> 10
<211> 2290
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(407)
<223> Экзон 3
<220>
<221> misc_feature
<222> (408)..(514)
<223> Экзон 4
<220>
<221> misc_feature
<222> (515)..(652)
<223> Экзон 5
<220>
<221> misc_feature
<222> (653)..(770)
<223> Экзон 6v2 - включает дополнительный остаток 770 на 3’-конце
<220>
<221> misc_feature
<222> (771)..(2290)
<223> Экзон 7
<400> 10
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataaggtgaa gaaagaagtg ggtgatgtaa 300
caatcgtggt gaataatgct gggacagtat atccagccga tcttctcagc accaaggatg 360
aagagattac caagacattt gaggtcaaca tcctaggaca tttttggatc acaaaagcac 420
ttcttccatc gatgatggag agaaatcatg gccacatcgt cacagtggct tcagtgtgcg 480
gccacgaagg gattccttac ctcatcccat attgttccag caaatttgcc gctgttggct 540
ttcacagagg tctgacatca gaacttcagg ccttgggaaa aactggtatc aaaacctcat 600
gtctctgccc agtttttgtg aatactgggt tcaccaaaaa tccaagcaca agattatggc 660
ctgtattgga gacagatgaa gtcgtaagaa gtctgataga tggaatactt accaataaga 720
aaatgatttt tgttccatcg tatatcaata tctttctgag actacagaag gtttcttcct 780
gaacgcgcct cagcgatttt aaatcgtatg cagaatattc aatttgaagc agtggttggc 840
cacaaaatca aaatgaaatg aataaataag ctccagccag agatgtatgc atgataatga 900
tatgaatagt ttcgaatcaa tgctgcaaag ctttatttca cattttttca gtcctgataa 960
tattaaaaac attggtttgg cactagcagc agtcaaacga acaagattaa ttacctgtct 1020
tcctgtttct caagaatatt tacgtagttt ttcataggtc tgtttttcct ttcatgcctc 1080
ttaaaaactt ctgtgcttac ataaacatac ttaaaaggtt ttctttaaga tattttattt 1140
ttccatttaa aggtggacaa aagctacctc cctaaaagta aatacaaaga gaacttattt 1200
acacagggaa ggtttaagac tgttcaagta gcattccaat ctgtagccat gccacagaat 1260
atcaacaaga acacagaatg agtgcacagc taagagatca agtttcagca ggcagcttta 1320
tctcaacctg gacatatttt aagattcagc atttgaaaga tttccctagc ctcttccttt 1380
ttcattagcc caaaacggtg caactctatt ctggacttta ttacttgatt ctgtcttctg 1440
tataactctg aagtccacca aaagtggacc ctctatattt cctccctttt tatagtctta 1500
taagatacat tatgaaaggt gaccgactct attttaaatc tcagaatttt aagttctagc 1560
cccatgataa cctttttctt tgtaatttat gctttcatat atccttggtc ccagagatgt 1620
ttagacaatt ttaggctcaa aaattaaagc taacacagga aaaggaactg tactggctat 1680
tacataagaa acaatggacc caagagaaga aaaggaagaa agaaaggttt tttggttttt 1740
gttttgtttt gttttgtttt ttgttttttt gagatggagt ctcactcttt cgcccaggct 1800
ggagtgcagt ggtatgatct cagctcactg caagctccac ctcccgggtt cacgccattc 1860
tcctgcctca gcctcctgag tagctgggac tacaggcgcc cgccaccaca cccggctaat 1920
tttttgtatt ttttgtagag acggggtttc accatgttag ccaagatggt ctcgatctcc 1980
tgacctcgtg atccacctgc ctcggcctcc caaagtgctg ggattacggg tgtgagccac 2040
cgtgcccagc cttttttttt ttaatagaaa aaataatccg actcccacta catcaagact 2100
aatcttgttt tgtgtgtttt tcacatgtat tatagaatgc ttttgcatgg actatcctct 2160
tgtttttatt aaaaacaaat gattttttta aaagtcacaa aaacaattca ctaaaaataa 2220
atatgtcatt gtgctttaaa aaaataacct cttgtagtta taaaataaaa cgtttgactt 2280
ctaaactctg 2290
<210> 11
<211> 2470
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(383)
<223> Экзон 2
<220>
<221> misc_feature
<222> (384)..(515)
<223> Экзон 3
<220>
<221> misc_feature
<222> (516)..(587)
<223> Экзон 3'
<220>
<221> misc_feature
<222> (588)..(694)
<223> Экзон 4
<220>
<221> misc_feature
<222> (695)..(832)
<223> Экзон 5
<220>
<221> misc_feature
<222> (833)..(950)
<223> Экзон 6v2 - включает дополнительный остаток 950 на 3’-конце
<220>
<221> misc_feature
<222> (951)..(2470)
<223> Экзон 7
<400> 11
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataagcgcgg tgtggaggaa actgcagctg 300
agtgccgaaa actaggcgtc actgcgcatg cgtatgtggt agactgcagc aacagagaag 360
agatctatcg ctctctaaat caggtgaaga aagaagtggg tgatgtaaca atcgtggtga 420
ataatgctgg gacagtatat ccagccgatc ttctcagcac caaggatgaa gagattacca 480
agacatttga ggtcaacatc ctaggacatt tttggaatgg aaaggacatc agaagtaatt 540
acttggatgt atataggatc gaggacactt ttggacgaga ctctgagatc acaaaagcac 600
ttcttccatc gatgatggag agaaatcatg gccacatcgt cacagtggct tcagtgtgcg 660
gccacgaagg gattccttac ctcatcccat attgttccag caaatttgcc gctgttggct 720
ttcacagagg tctgacatca gaacttcagg ccttgggaaa aactggtatc aaaacctcat 780
gtctctgccc agtttttgtg aatactgggt tcaccaaaaa tccaagcaca agattatggc 840
ctgtattgga gacagatgaa gtcgtaagaa gtctgataga tggaatactt accaataaga 900
aaatgatttt tgttccatcg tatatcaata tctttctgag actacagaag gtttcttcct 960
gaacgcgcct cagcgatttt aaatcgtatg cagaatattc aatttgaagc agtggttggc 1020
cacaaaatca aaatgaaatg aataaataag ctccagccag agatgtatgc atgataatga 1080
tatgaatagt ttcgaatcaa tgctgcaaag ctttatttca cattttttca gtcctgataa 1140
tattaaaaac attggtttgg cactagcagc agtcaaacga acaagattaa ttacctgtct 1200
tcctgtttct caagaatatt tacgtagttt ttcataggtc tgtttttcct ttcatgcctc 1260
ttaaaaactt ctgtgcttac ataaacatac ttaaaaggtt ttctttaaga tattttattt 1320
ttccatttaa aggtggacaa aagctacctc cctaaaagta aatacaaaga gaacttattt 1380
acacagggaa ggtttaagac tgttcaagta gcattccaat ctgtagccat gccacagaat 1440
atcaacaaga acacagaatg agtgcacagc taagagatca agtttcagca ggcagcttta 1500
tctcaacctg gacatatttt aagattcagc atttgaaaga tttccctagc ctcttccttt 1560
ttcattagcc caaaacggtg caactctatt ctggacttta ttacttgatt ctgtcttctg 1620
tataactctg aagtccacca aaagtggacc ctctatattt cctccctttt tatagtctta 1680
taagatacat tatgaaaggt gaccgactct attttaaatc tcagaatttt aagttctagc 1740
cccatgataa cctttttctt tgtaatttat gctttcatat atccttggtc ccagagatgt 1800
ttagacaatt ttaggctcaa aaattaaagc taacacagga aaaggaactg tactggctat 1860
tacataagaa acaatggacc caagagaaga aaaggaagaa agaaaggttt tttggttttt 1920
gttttgtttt gttttgtttt ttgttttttt gagatggagt ctcactcttt cgcccaggct 1980
ggagtgcagt ggtatgatct cagctcactg caagctccac ctcccgggtt cacgccattc 2040
tcctgcctca gcctcctgag tagctgggac tacaggcgcc cgccaccaca cccggctaat 2100
tttttgtatt ttttgtagag acggggtttc accatgttag ccaagatggt ctcgatctcc 2160
tgacctcgtg atccacctgc ctcggcctcc caaagtgctg ggattacggg tgtgagccac 2220
cgtgcccagc cttttttttt ttaatagaaa aaataatccg actcccacta catcaagact 2280
aatcttgttt tgtgtgtttt tcacatgtat tatagaatgc ttttgcatgg actatcctct 2340
tgtttttatt aaaaacaaat gattttttta aaagtcacaa aaacaattca ctaaaaataa 2400
atatgtcatt gtgctttaaa aaaataacct cttgtagtta taaaataaaa cgtttgactt 2460
ctaaactctg 2470
<210> 12
<211> 300
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(106)
<223> Участок, кодируемый экзоном 2
<220>
<221> MISC_FEATURE
<222> (107)..(150)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (151)..(185)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (186)..(232)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (233)..(271)
<223> Участок, кодируемый экзоном 6v1
<220>
<221> MISC_FEATURE
<222> (272)..(300)
<223> Участок, кодируемый экзоном 7
<400> 12
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met
145 150 155 160
Glu Arg Asn His Gly His Ile Val Thr Val Ala Ser Val Cys Gly His
165 170 175
Glu Gly Ile Pro Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala
180 185 190
Val Gly Phe His Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys
195 200 205
Thr Gly Ile Lys Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly
210 215 220
Phe Thr Lys Asn Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp
225 230 235 240
Glu Val Val Arg Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met
245 250 255
Ile Phe Val Pro Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Phe
260 265 270
Leu Pro Glu Arg Ala Ser Ala Ile Leu Asn Arg Met Gln Asn Ile Gln
275 280 285
Phe Glu Ala Val Val Gly His Lys Ile Lys Met Lys
290 295 300
<210> 13
<211> 264
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(114)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (115)..(149)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (150)..(196)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (197)..(235)
<223> Участок, кодируемый экзоном 6v1
<220>
<221> MISC_FEATURE
<222> (236)..(264)
<223> Участок, кодируемый экзоном 7
<400> 13
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Val Lys Lys Glu Val Gly Asp Val Thr Ile
65 70 75 80
Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp Leu Leu Ser Thr
85 90 95
Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn Ile Leu Gly His
100 105 110
Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met Glu Arg Asn His
115 120 125
Gly His Ile Val Thr Val Ala Ser Val Cys Gly His Glu Gly Ile Pro
130 135 140
Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala Val Gly Phe His
145 150 155 160
Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys Thr Gly Ile Lys
165 170 175
Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly Phe Thr Lys Asn
180 185 190
Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp Glu Val Val Arg
195 200 205
Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met Ile Phe Val Pro
210 215 220
Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Phe Leu Pro Glu Arg
225 230 235 240
Ala Ser Ala Ile Leu Asn Arg Met Gln Asn Ile Gln Phe Glu Ala Val
245 250 255
Val Gly His Lys Ile Lys Met Lys
260
<210> 14
<211> 261
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(106)
<223> Участок, кодируемый экзоном 2
<220>
<221> MISC_FEATURE
<222> (107)..(150)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (151)..(185)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (186)..(232)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (233)..(261)
<223> Участок, кодируемый экзоном 7
<400> 14
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met
145 150 155 160
Glu Arg Asn His Gly His Ile Val Thr Val Ala Ser Val Cys Gly His
165 170 175
Glu Gly Ile Pro Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala
180 185 190
Val Gly Phe His Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys
195 200 205
Thr Gly Ile Lys Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly
210 215 220
Phe Thr Lys Asn Pro Ser Thr Arg Phe Leu Pro Glu Arg Ala Ser Ala
225 230 235 240
Ile Leu Asn Arg Met Gln Asn Ile Gln Phe Glu Ala Val Val Gly His
245 250 255
Lys Ile Lys Met Lys
260
<210> 15
<211> 274
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(106)
<223> Участок, кодируемый экзоном 2
<220>
<221> MISC_FEATURE
<222> (107)..(150)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (151)..(185)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (186)..(232)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (233)..(271)
<223> Участок, кодируемый экзоном 6v2
<220>
<221> MISC_FEATURE
<222> (272)..(274)
<223> Участок, кодируемый экзоном 7
<400> 15
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met
145 150 155 160
Glu Arg Asn His Gly His Ile Val Thr Val Ala Ser Val Cys Gly His
165 170 175
Glu Gly Ile Pro Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala
180 185 190
Val Gly Phe His Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys
195 200 205
Thr Gly Ile Lys Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly
210 215 220
Phe Thr Lys Asn Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp
225 230 235 240
Glu Val Val Arg Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met
245 250 255
Ile Phe Val Pro Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Val
260 265 270
Ser Ser
<210> 16
<211> 324
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(106)
<223> Участок, кодируемый экзоном 2
<220>
<221> MISC_FEATURE
<222> (107)..(150)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (151)..(174)
<223> Участок, кодируемый экзоном 3'
<220>
<221> MISC_FEATURE
<222> (175)..(209)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (210)..(256)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (257)..(295)
<223> Участок, кодируемый экзоном 6v1
<220>
<221> MISC_FEATURE
<222> (296)..(324)
<223> Участок, кодируемый экзоном 7
<400> 16
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Asn Gly Lys Asp Ile Arg Ser Asn Tyr Leu
145 150 155 160
Asp Val Tyr Arg Ile Glu Asp Thr Phe Gly Arg Asp Ser Glu Ile Thr
165 170 175
Lys Ala Leu Leu Pro Ser Met Met Glu Arg Asn His Gly His Ile Val
180 185 190
Thr Val Ala Ser Val Cys Gly His Glu Gly Ile Pro Tyr Leu Ile Pro
195 200 205
Tyr Cys Ser Ser Lys Phe Ala Ala Val Gly Phe His Arg Gly Leu Thr
210 215 220
Ser Glu Leu Gln Ala Leu Gly Lys Thr Gly Ile Lys Thr Ser Cys Leu
225 230 235 240
Cys Pro Val Phe Val Asn Thr Gly Phe Thr Lys Asn Pro Ser Thr Arg
245 250 255
Leu Trp Pro Val Leu Glu Thr Asp Glu Val Val Arg Ser Leu Ile Asp
260 265 270
Gly Ile Leu Thr Asn Lys Lys Met Ile Phe Val Pro Ser Tyr Ile Asn
275 280 285
Ile Phe Leu Arg Leu Gln Lys Phe Leu Pro Glu Arg Ala Ser Ala Ile
290 295 300
Leu Asn Arg Met Gln Asn Ile Gln Phe Glu Ala Val Val Gly His Lys
305 310 315 320
Ile Lys Met Lys
<210> 17
<211> 284
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(106)
<223> Участок, кодируемый экзоном 2
<220>
<221> MISC_FEATURE
<222> (107)..(150)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (151)..(185)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (186)..(232)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (233)..(284)
<223> Участок, кодируемый экзоном 6v3
<220>
<221> MISC_FEATURE
<222> (272)..(284)
<223> Участок, кодируемый участком считывания в интрон 6
<400> 17
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met
145 150 155 160
Glu Arg Asn His Gly His Ile Val Thr Val Ala Ser Val Cys Gly His
165 170 175
Glu Gly Ile Pro Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala
180 185 190
Val Gly Phe His Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys
195 200 205
Thr Gly Ile Lys Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly
210 215 220
Phe Thr Lys Asn Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp
225 230 235 240
Glu Val Val Arg Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met
245 250 255
Ile Phe Val Pro Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Leu
260 265 270
Ser Thr Ala Gln Asn Thr Gln Ile Leu Lys His Gln
275 280
<210> 18
<211> 238
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(114)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (115)..(149)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (150)..(196)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (197)..(235)
<223> Участок, кодируемый экзоном 6v2
<220>
<221> MISC_FEATURE
<222> (236)..(238)
<223> Участок, кодируемый экзоном 7
<400> 18
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Val Lys Lys Glu Val Gly Asp Val Thr Ile
65 70 75 80
Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp Leu Leu Ser Thr
85 90 95
Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn Ile Leu Gly His
100 105 110
Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met Glu Arg Asn His
115 120 125
Gly His Ile Val Thr Val Ala Ser Val Cys Gly His Glu Gly Ile Pro
130 135 140
Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala Val Gly Phe His
145 150 155 160
Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys Thr Gly Ile Lys
165 170 175
Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly Phe Thr Lys Asn
180 185 190
Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp Glu Val Val Arg
195 200 205
Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met Ile Phe Val Pro
210 215 220
Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Val Ser Ser
225 230 235
<210> 19
<211> 298
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(106)
<223> Участок, кодируемый экзоном 2
<220>
<221> MISC_FEATURE
<222> (107)..(150)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (151)..(174)
<223> Участок, кодируемый экзоном 3'
<220>
<221> MISC_FEATURE
<222> (175)..(209)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (210)..(256)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (257)..(295)
<223> Участок, кодируемый экзоном 6v2
<220>
<221> MISC_FEATURE
<222> (296)..(298)
<223> Участок, кодируемый экзоном 7
<400> 19
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Asn Gly Lys Asp Ile Arg Ser Asn Tyr Leu
145 150 155 160
Asp Val Tyr Arg Ile Glu Asp Thr Phe Gly Arg Asp Ser Glu Ile Thr
165 170 175
Lys Ala Leu Leu Pro Ser Met Met Glu Arg Asn His Gly His Ile Val
180 185 190
Thr Val Ala Ser Val Cys Gly His Glu Gly Ile Pro Tyr Leu Ile Pro
195 200 205
Tyr Cys Ser Ser Lys Phe Ala Ala Val Gly Phe His Arg Gly Leu Thr
210 215 220
Ser Glu Leu Gln Ala Leu Gly Lys Thr Gly Ile Lys Thr Ser Cys Leu
225 230 235 240
Cys Pro Val Phe Val Asn Thr Gly Phe Thr Lys Asn Pro Ser Thr Arg
245 250 255
Leu Trp Pro Val Leu Glu Thr Asp Glu Val Val Arg Ser Leu Ile Asp
260 265 270
Gly Ile Leu Thr Asn Lys Lys Met Ile Phe Val Pro Ser Tyr Ile Asn
275 280 285
Ile Phe Leu Arg Leu Gln Lys Val Ser Ser
290 295
<210> 20
<211> 20
<212> DNA
<213> Homo sapiens
<400> 20
tgaagttttt cattcctcag 20
<210> 21
<211> 20
<212> DNA
<213> Homo sapiens
<400> 21
cttcaccaac gactccaagt 20
<210> 22
<211> 20
<212> DNA
<213> Homo sapiens
<400> 22
ctactcctac ttggagtcgt 20
<210> 23
<211> 20
<212> DNA
<213> Homo sapiens
<400> 23
ctccaagtag gagtagatga 20
<210> 24
<211> 20
<212> DNA
<213> Homo sapiens
<400> 24
caccatcatc tactcctact 20
<210> 25
<211> 20
<212> DNA
<213> Homo sapiens
<400> 25
tgatggtgat cagaagcaga 20
<210> 26
<211> 20
<212> DNA
<213> Homo sapiens
<400> 26
tcagaagcag aaggatttct 20
<210> 27
<211> 20
<212> DNA
<213> Homo sapiens
<400> 27
gatttctagg atgatgttca 20
<210> 28
<211> 20
<212> DNA
<213> Homo sapiens
<400> 28
ttgctctgtc ctcttccttc 20
<210> 29
<211> 20
<212> DNA
<213> Homo sapiens
<400> 29
aggactgaac cagaaggaag 20
<210> 30
<211> 20
<212> DNA
<213> Homo sapiens
<400> 30
tacacaagga ctgaaccaga 20
<210> 31
<211> 20
<212> DNA
<213> Homo sapiens
<400> 31
ttcagtcctt gtgtagtcct 20
<210> 32
<211> 20
<212> DNA
<213> Homo sapiens
<400> 32
tcagtccttg tgtagtccta 20
<210> 33
<211> 20
<212> DNA
<213> Homo sapiens
<400> 33
gtccttgtgt agtcctaggg 20
<210> 34
<211> 20
<212> DNA
<213> Homo sapiens
<400> 34
cttgtgtagt cctagggagg 20
<210> 35
<211> 20
<212> DNA
<213> Homo sapiens
<400> 35
ctcctcccta ggactacaca 20
<210> 36
<211> 20
<212> DNA
<213> Homo sapiens
<400> 36
gtagacagta cctcctccct 20
<210> 37
<211> 20
<212> DNA
<213> Homo sapiens
<400> 37
tactgtctac acagagctct 20
<210> 38
<211> 20
<212> DNA
<213> Homo sapiens
<400> 38
actgtctaca cagagctcta 20
<210> 39
<211> 20
<212> DNA
<213> Homo sapiens
<400> 39
tctacacaga gctctaggga 20
<210> 40
<211> 20
<212> DNA
<213> Homo sapiens
<400> 40
ctacacagag ctctagggaa 20
<210> 41
<211> 20
<212> DNA
<213> Homo sapiens
<400> 41
tacacagagc tctagggaag 20
<210> 42
<211> 20
<212> DNA
<213> Homo sapiens
<400> 42
tgtcaggtta gttagatgaa 20
<210> 43
<211> 20
<212> DNA
<213> Homo sapiens
<400> 43
gtgtcaggtt agttagatga 20
<210> 44
<211> 20
<212> DNA
<213> Homo sapiens
<400> 44
cctgacacat atacagacta 20
<210> 45
<211> 20
<212> DNA
<213> Homo sapiens
<400> 45
ctgacacata tacagactaa 20
<210> 46
<211> 20
<212> DNA
<213> Homo sapiens
<400> 46
ccttagtctg tatatgtgtc 20
<210> 47
<211> 20
<212> DNA
<213> Homo sapiens
<400> 47
catatacaga ctaagggacc 20
<210> 48
<211> 20
<212> DNA
<213> Homo sapiens
<400> 48
atatacagac taagggacca 20
<210> 49
<211> 20
<212> DNA
<213> Homo sapiens
<400> 49
tcaaagtttg ataaattccc 20
<210> 50
<211> 20
<212> DNA
<213> Homo sapiens
<400> 50
aaaatacaaa gataagtaga 20
<210> 51
<211> 20
<212> DNA
<213> Homo sapiens
<400> 51
actctgtgac tttaaaaagt 20
<210> 52
<211> 20
<212> DNA
<213> Homo sapiens
<400> 52
ggttctgtgg gatattaata 20
<210> 53
<211> 20
<212> DNA
<213> Homo sapiens
<400> 53
acagagcata ttggttctgt 20
<210> 54
<211> 20
<212> DNA
<213> Homo sapiens
<400> 54
gacagagcat attggttctg 20
<210> 55
<211> 20
<212> DNA
<213> Homo sapiens
<400> 55
tgcaaaacga cagagcatat 20
<210> 56
<211> 20
<212> DNA
<213> Homo sapiens
<400> 56
gagctgggca tggaataggc 20
<210> 57
<211> 20
<212> DNA
<213> Homo sapiens
<400> 57
actggagctg ggcatggaat 20
<210> 58
<211> 20
<212> DNA
<213> Homo sapiens
<400> 58
ctcattactg gagctgggca 20
<210> 59
<211> 20
<212> DNA
<213> Homo sapiens
<400> 59
ttgttctcat tactggagct 20
<210> 60
<211> 20
<212> DNA
<213> Homo sapiens
<400> 60
attgttctca ttactggagc 20
<210> 61
<211> 20
<212> DNA
<213> Homo sapiens
<400> 61
ggggagattg ttctcattac 20
<210> 62
<211> 20
<212> DNA
<213> Homo sapiens
<400> 62
gaggagaaaa tctgtggctg 20
<210> 63
<211> 20
<212> DNA
<213> Homo sapiens
<400> 63
agaggagaaa atctgtggct 20
<210> 64
<211> 20
<212> DNA
<213> Homo sapiens
<400> 64
cagaggagaa aatctgtggc 20
<210> 65
<211> 20
<212> DNA
<213> Homo sapiens
<400> 65
tcctcagagg agaaaatctg 20
<210> 66
<211> 20
<212> DNA
<213> Homo sapiens
<400> 66
ggggtgtgcc cagttgttaa 20
<210> 67
<211> 20
<212> DNA
<213> Homo sapiens
<400> 67
gggtgtgccc agttgttaat 20
<210> 68
<211> 20
<212> DNA
<213> Homo sapiens
<400> 68
tggtagtccc attaacaact 20
<210> 69
<211> 20
<212> DNA
<213> Homo sapiens
<400> 69
ctggtagtcc cattaacaac 20
<210> 70
<211> 20
<212> DNA
<213> Homo sapiens
<400> 70
ttgttaatgg gactaccaga 20
<210> 71
<211> 20
<212> DNA
<213> Homo sapiens
<400> 71
taccagatgg aagccagctt 20
<210> 72
<211> 20
<212> DNA
<213> Homo sapiens
<400> 72
ttccaaagct ggcttccatc 20
<210> 73
<211> 20
<212> DNA
<213> Homo sapiens
<400> 73
tggaagccag ctttggaagc 20
<210> 74
<211> 20
<212> DNA
<213> Homo sapiens
<400> 74
acaaggcctg cttccaaagc 20
<210> 75
<211> 20
<212> DNA
<213> Homo sapiens
<400> 75
gccttgttca cgtgttctaa 20
<210> 76
<211> 20
<212> DNA
<213> Homo sapiens
<400> 76
ccttgttcac gtgttctaat 20
<210> 77
<211> 20
<212> DNA
<213> Homo sapiens
<400> 77
cccattagaa cacgtgaaca 20
<210> 78
<211> 20
<212> DNA
<213> Homo sapiens
<400> 78
ttggcatcac ttcatatttg 20
<210> 79
<211> 20
<212> DNA
<213> Homo sapiens
<400> 79
cttgtgctct tggcatcact 20
<210> 80
<211> 20
<212> DNA
<213> Homo sapiens
<400> 80
agcacactct cttgtgctct 20
<210> 81
<211> 20
<212> DNA
<213> Homo sapiens
<400> 81
gcacaagaga gtgtgctctc 20
<210> 82
<211> 20
<212> DNA
<213> Homo sapiens
<400> 82
gcttaatctc acacatagaa 20
<210> 83
<211> 20
<212> DNA
<213> Homo sapiens
<400> 83
cttaatctca cacatagaaa 20
<210> 84
<211> 20
<212> DNA
<213> Homo sapiens
<400> 84
ttaatctcac acatagaaag 20
<210> 85
<211> 20
<212> DNA
<213> Homo sapiens
<400> 85
aggagtgctg gtttatcaac 20
<210> 86
<211> 20
<212> DNA
<213> Homo sapiens
<400> 86
ttctttgaca gcaggagtgc 20
<210> 87
<211> 20
<212> DNA
<213> Homo sapiens
<400> 87
actctggttt ctttgacagc 20
<210> 88
<211> 20
<212> DNA
<213> Homo sapiens
<400> 88
accagagttg agaaaacccc 20
<210> 89
<211> 20
<212> DNA
<213> Homo sapiens
<400> 89
tccaggggtt ttctcaactc 20
<210> 90
<211> 20
<212> DNA
<213> Homo sapiens
<400> 90
cagttattaa atgaatccag 20
<210> 91
<211> 20
<212> DNA
<213> Homo sapiens
<400> 91
gcagttatta aatgaatcca 20
<210> 92
<211> 20
<212> DNA
<213> Homo sapiens
<400> 92
ggcagttatt aaatgaatcc 20
<210> 93
<211> 20
<212> DNA
<213> Homo sapiens
<400> 93
tggatggtaa cagctacatc 20
<210> 94
<211> 20
<212> DNA
<213> Homo sapiens
<400> 94
gctgttacca tccacatcct 20
<210> 95
<211> 20
<212> DNA
<213> Homo sapiens
<400> 95
tcaagaacca aggatgtgga 20
<210> 96
<211> 20
<212> DNA
<213> Homo sapiens
<400> 96
tccttcaaga accaaggatg 20
<210> 97
<211> 20
<212> DNA
<213> Homo sapiens
<400> 97
tgagtgtcct tcaagaacca 20
<210> 98
<211> 20
<212> DNA
<213> Homo sapiens
<400> 98
ttttatttta taactacaag 20
<210> 99
<211> 20
<212> DNA
<213> Homo sapiens
<400> 99
ttgtttttaa taaaaacaag 20
<210> 100
<211> 20
<212> DNA
<213> Homo sapiens
<400> 100
tattatagaa tgcttttgca 20
<210> 101
<211> 20
<212> DNA
<213> Homo sapiens
<400> 101
caagattagt cttgatgtag 20
<210> 102
<211> 20
<212> DNA
<213> Homo sapiens
<400> 102
aagattagtc ttgatgtagt 20
<210> 103
<211> 20
<212> DNA
<213> Homo sapiens
<400> 103
agtcttgatg tagtgggagt 20
<210> 104
<211> 20
<212> DNA
<213> Homo sapiens
<400> 104
tttttctatt aaaaaaaaaa 20
<210> 105
<211> 20
<212> DNA
<213> Homo sapiens
<400> 105
tctattaaaa aaaaaaaggc 20
<210> 106
<211> 20
<212> DNA
<213> Homo sapiens
<400> 106
ctattaaaaa aaaaaaggct 20
<210> 107
<211> 20
<212> DNA
<213> Homo sapiens
<400> 107
aaaaaaaaaa aggctgggca 20
<210> 108
<211> 20
<212> DNA
<213> Homo sapiens
<400> 108
aaaaaaaagg ctgggcacgg 20
<210> 109
<211> 20
<212> DNA
<213> Homo sapiens
<400> 109
cacccgtaat cccagcactt 20
<210> 110
<211> 20
<212> DNA
<213> Homo sapiens
<400> 110
acccgtaatc ccagcacttt 20
<210> 111
<211> 20
<212> DNA
<213> Homo sapiens
<400> 111
cgtaatccca gcactttggg 20
<210> 112
<211> 20
<212> DNA
<213> Homo sapiens
<400> 112
tcccaaagtg ctgggattac 20
<210> 113
<211> 20
<212> DNA
<213> Homo sapiens
<400> 113
ctcccaaagt gctgggatta 20
<210> 114
<211> 20
<212> DNA
<213> Homo sapiens
<400> 114
cccagcactt tgggaggccg 20
<210> 115
<211> 20
<212> DNA
<213> Homo sapiens
<400> 115
cctcggcctc ccaaagtgct 20
<210> 116
<211> 20
<212> DNA
<213> Homo sapiens
<400> 116
gcactttggg aggccgaggc 20
<210> 117
<211> 20
<212> DNA
<213> Homo sapiens
<400> 117
ctttgggagg ccgaggcagg 20
<210> 118
<211> 20
<212> DNA
<213> Homo sapiens
<400> 118
gccgaggcag gtggatcacg 20
<210> 119
<211> 20
<212> DNA
<213> Homo sapiens
<400> 119
acctcgtgat ccacctgcct 20
<210> 120
<211> 20
<212> DNA
<213> Homo sapiens
<400> 120
ggcaggtgga tcacgaggtc 20
<210> 121
<211> 20
<212> DNA
<213> Homo sapiens
<400> 121
tcaggagatc gagaccatct 20
<210> 122
<211> 20
<212> DNA
<213> Homo sapiens
<400> 122
cgagaccatc ttggctaaca 20
<210> 123
<211> 20
<212> DNA
<213> Homo sapiens
<400> 123
tttcaccatg ttagccaaga 20
<210> 124
<211> 20
<212> DNA
<213> Homo sapiens
<400> 124
ttgtattttt tgtagagacg 20
<210> 125
<211> 20
<212> DNA
<213> Homo sapiens
<400> 125
tttgtatttt ttgtagagac 20
<210> 126
<211> 20
<212> DNA
<213> Homo sapiens
<400> 126
ttttgtattt tttgtagaga 20
<210> 127
<211> 20
<212> DNA
<213> Homo sapiens
<400> 127
aaaaaataca aaaaattagc 20
<210> 128
<211> 20
<212> DNA
<213> Homo sapiens
<400> 128
aaaaatacaa aaaattagcc 20
<210> 129
<211> 20
<212> DNA
<213> Homo sapiens
<400> 129
tacaaaaaat tagccgggtg 20
<210> 130
<211> 20
<212> DNA
<213> Homo sapiens
<400> 130
aaaaaattag ccgggtgtgg 20
<210> 131
<211> 20
<212> DNA
<213> Homo sapiens
<400> 131
aaattagccg ggtgtggtgg 20
<210> 132
<211> 20
<212> DNA
<213> Homo sapiens
<400> 132
aattagccgg gtgtggtggc 20
<210> 133
<211> 20
<212> DNA
<213> Homo sapiens
<400> 133
caggcgcccg ccaccacacc 20
<210> 134
<211> 20
<212> DNA
<213> Homo sapiens
<400> 134
gcctgtagtc ccagctactc 20
<210> 135
<211> 20
<212> DNA
<213> Homo sapiens
<400> 135
tgtagtccca gctactcagg 20
<210> 136
<211> 20
<212> DNA
<213> Homo sapiens
<400> 136
tcctgagtag ctgggactac 20
<210> 137
<211> 20
<212> DNA
<213> Homo sapiens
<400> 137
cccagctact caggaggctg 20
<210> 138
<211> 20
<212> DNA
<213> Homo sapiens
<400> 138
cctcagcctc ctgagtagct 20
<210> 139
<211> 20
<212> DNA
<213> Homo sapiens
<400> 139
gcctcagcct cctgagtagc 20
<210> 140
<211> 20
<212> DNA
<213> Homo sapiens
<400> 140
aggaggctga ggcaggagaa 20
<210> 141
<211> 20
<212> DNA
<213> Homo sapiens
<400> 141
gcaggagaat ggcgtgaacc 20
<210> 142
<211> 20
<212> DNA
<213> Homo sapiens
<400> 142
caggagaatg gcgtgaaccc 20
<210> 143
<211> 20
<212> DNA
<213> Homo sapiens
<400> 143
gagaatggcg tgaacccggg 20
<210> 144
<211> 20
<212> DNA
<213> Homo sapiens
<400> 144
aatggcgtga acccgggagg 20
<210> 145
<211> 20
<212> DNA
<213> Homo sapiens
<400> 145
cactgcaagc tccacctccc 20
<210> 146
<211> 20
<212> DNA
<213> Homo sapiens
<400> 146
tcactgcaag ctccacctcc 20
<210> 147
<211> 20
<212> DNA
<213> Homo sapiens
<400> 147
cataccactg cactccagcc 20
<210> 148
<211> 20
<212> DNA
<213> Homo sapiens
<400> 148
ataccactgc actccagcct 20
<210> 149
<211> 20
<212> DNA
<213> Homo sapiens
<400> 149
tcgcccaggc tggagtgcag 20
<210> 150
<211> 20
<212> DNA
<213> Homo sapiens
<400> 150
tctcactctt tcgcccaggc 20
<210> 151
<211> 20
<212> DNA
<213> Homo sapiens
<400> 151
ggagtctcac tctttcgccc 20
<210> 152
<211> 20
<212> DNA
<213> Homo sapiens
<400> 152
tgttttttgt ttttttgaga 20
<210> 153
<211> 20
<212> DNA
<213> Homo sapiens
<400> 153
aggaagaaag aaaggttttt 20
<210> 154
<211> 20
<212> DNA
<213> Homo sapiens
<400> 154
agaagaaaag gaagaaagaa 20
<210> 155
<211> 20
<212> DNA
<213> Homo sapiens
<400> 155
ctttcttcct tttcttctct 20
<210> 156
<211> 20
<212> DNA
<213> Homo sapiens
<400> 156
tttcttcctt ttcttctctt 20
<210> 157
<211> 20
<212> DNA
<213> Homo sapiens
<400> 157
aatggaccca agagaagaaa 20
<210> 158
<211> 20
<212> DNA
<213> Homo sapiens
<400> 158
ggctattaca taagaaacaa 20
<210> 159
<211> 20
<212> DNA
<213> Homo sapiens
<400> 159
cacaggaaaa ggaactgtac 20
<210> 160
<211> 20
<212> DNA
<213> Homo sapiens
<400> 160
attaaagcta acacaggaaa 20
<210> 161
<211> 20
<212> DNA
<213> Homo sapiens
<400> 161
tcaaaaatta aagctaacac 20
<210> 162
<211> 20
<212> DNA
<213> Homo sapiens
<400> 162
taaaattgtc taaacatctc 20
<210> 163
<211> 20
<212> DNA
<213> Homo sapiens
<400> 163
agagatgttt agacaatttt 20
<210> 164
<211> 20
<212> DNA
<213> Homo sapiens
<400> 164
tctaaacatc tctgggacca 20
<210> 165
<211> 20
<212> DNA
<213> Homo sapiens
<400> 165
tttatgcttt catatatcct 20
<210> 166
<211> 20
<212> DNA
<213> Homo sapiens
<400> 166
agcataaatt acaaagaaaa 20
<210> 167
<211> 20
<212> DNA
<213> Homo sapiens
<400> 167
tacaaagaaa aaggttatca 20
<210> 168
<211> 20
<212> DNA
<213> Homo sapiens
<400> 168
acaaagaaaa aggttatcat 20
<210> 169
<211> 20
<212> DNA
<213> Homo sapiens
<400> 169
caaagaaaaa ggttatcatg 20
<210> 170
<211> 20
<212> DNA
<213> Homo sapiens
<400> 170
tctgagattt aaaatagagt 20
<210> 171
<211> 20
<212> DNA
<213> Homo sapiens
<400> 171
cttataagat acattatgaa 20
<210> 172
<211> 20
<212> DNA
<213> Homo sapiens
<400> 172
tatcttataa gactataaaa 20
<210> 173
<211> 20
<212> DNA
<213> Homo sapiens
<400> 173
atcttataag actataaaaa 20
<210> 174
<211> 20
<212> DNA
<213> Homo sapiens
<400> 174
ttataagact ataaaaaggg 20
<210> 175
<211> 20
<212> DNA
<213> Homo sapiens
<400> 175
taaaaaggga ggaaatatag 20
<210> 176
<211> 20
<212> DNA
<213> Homo sapiens
<400> 176
aaaaagggag gaaatataga 20
<210> 177
<211> 20
<212> DNA
<213> Homo sapiens
<400> 177
aaatatagag ggtccacttt 20
<210> 178
<211> 20
<212> DNA
<213> Homo sapiens
<400> 178
tatagagggt ccacttttgg 20
<210> 179
<211> 20
<212> DNA
<213> Homo sapiens
<400> 179
actctgaagt ccaccaaaag 20
<210> 180
<211> 20
<212> DNA
<213> Homo sapiens
<400> 180
agaatagagt tgcaccgttt 20
<210> 181
<211> 20
<212> DNA
<213> Homo sapiens
<400> 181
aaaacggtgc aactctattc 20
<210> 182
<211> 20
<212> DNA
<213> Homo sapiens
<400> 182
ccgttttggg ctaatgaaaa 20
<210> 183
<211> 20
<212> DNA
<213> Homo sapiens
<400> 183
cctttttcat tagcccaaaa 20
<210> 184
<211> 20
<212> DNA
<213> Homo sapiens
<400> 184
tgggctaatg aaaaaggaag 20
<210> 185
<211> 20
<212> DNA
<213> Homo sapiens
<400> 185
taatgaaaaa ggaagaggct 20
<210> 186
<211> 20
<212> DNA
<213> Homo sapiens
<400> 186
aatgaaaaag gaagaggcta 20
<210> 187
<211> 20
<212> DNA
<213> Homo sapiens
<400> 187
ctgaatctta aaatatgtcc 20
<210> 188
<211> 20
<212> DNA
<213> Homo sapiens
<400> 188
caggcagctt tatctcaacc 20
<210> 189
<211> 20
<212> DNA
<213> Homo sapiens
<400> 189
ctaagagatc aagtttcagc 20
<210> 190
<211> 20
<212> DNA
<213> Homo sapiens
<400> 190
gtgttcttgt tgatattctg 20
<210> 191
<211> 20
<212> DNA
<213> Homo sapiens
<400> 191
cttgttgata ttctgtggca 20
<210> 192
<211> 20
<212> DNA
<213> Homo sapiens
<400> 192
tctgtggcat ggctacagat 20
<210> 193
<211> 20
<212> DNA
<213> Homo sapiens
<400> 193
agaacttatt tacacaggga 20
<210> 194
<211> 20
<212> DNA
<213> Homo sapiens
<400> 194
aaagagaact tatttacaca 20
<210> 195
<211> 20
<212> DNA
<213> Homo sapiens
<400> 195
caaagagaac ttatttacac 20
<210> 196
<211> 20
<212> DNA
<213> Homo sapiens
<400> 196
ttctctttgt atttactttt 20
<210> 197
<211> 20
<212> DNA
<213> Homo sapiens
<400> 197
tctctttgta tttactttta 20
<210> 198
<211> 20
<212> DNA
<213> Homo sapiens
<400> 198
ctttgtattt acttttaggg 20
<210> 199
<211> 20
<212> DNA
<213> Homo sapiens
<400> 199
agcttttgtc cacctttaaa 20
<210> 200
<211> 20
<212> DNA
<213> Homo sapiens
<400> 200
tttatttttc catttaaagg 20
<210> 201
<211> 20
<212> DNA
<213> Homo sapiens
<400> 201
tattttattt ttccatttaa 20
<210> 202
<211> 20
<212> DNA
<213> Homo sapiens
<400> 202
cttacataaa catacttaaa 20
<210> 203
<211> 20
<212> DNA
<213> Homo sapiens
<400> 203
taagcacaga agtttttaag 20
<210> 204
<211> 20
<212> DNA
<213> Homo sapiens
<400> 204
aagtttttaa gaggcatgaa 20
<210> 205
<211> 20
<212> DNA
<213> Homo sapiens
<400> 205
atatttacgt agtttttcat 20
<210> 206
<211> 20
<212> DNA
<213> Homo sapiens
<400> 206
cgtaaatatt cttgagaaac 20
<210> 207
<211> 20
<212> DNA
<213> Homo sapiens
<400> 207
ttcttgagaa acaggaagac 20
<210> 208
<211> 20
<212> DNA
<213> Homo sapiens
<400> 208
taatattaaa aacattggtt 20
<210> 209
<211> 20
<212> DNA
<213> Homo sapiens
<400> 209
ccaatgtttt taatattatc 20
<210> 210
<211> 20
<212> DNA
<213> Homo sapiens
<400> 210
cctgataata ttaaaaacat 20
<210> 211
<211> 20
<212> DNA
<213> Homo sapiens
<400> 211
cattatcatg catacatctc 20
<210> 212
<211> 20
<212> DNA
<213> Homo sapiens
<400> 212
atcatgcata catctctggc 20
<210> 213
<211> 20
<212> DNA
<213> Homo sapiens
<400> 213
ttcatttcat tttgattttg 20
<210> 214
<211> 20
<212> DNA
<213> Homo sapiens
<400> 214
attcaatttg aagcagtggt 20
<210> 215
<211> 20
<212> DNA
<213> Homo sapiens
<400> 215
gaatattcaa tttgaagcag 20
<210> 216
<211> 20
<212> DNA
<213> Homo sapiens
<400> 216
catacgattt aaaatcgctg 20
<210> 217
<211> 20
<212> DNA
<213> Homo sapiens
<400> 217
aaaatcgctg aggcgcgttc 20
<210> 218
<211> 20
<212> DNA
<213> Homo sapiens
<400> 218
tttttttttc ttttttgtac 20
<210> 219
<211> 20
<212> DNA
<213> Homo sapiens
<400> 219
ctgttgtcaa agattttaaa 20
<210> 220
<211> 20
<212> DNA
<213> Homo sapiens
<400> 220
tgacaacaga gttctgtttt 20
<210> 221
<211> 20
<212> DNA
<213> Homo sapiens
<400> 221
agaatacgct gagagttatc 20
<210> 222
<211> 20
<212> DNA
<213> Homo sapiens
<400> 222
gcaagagaag aaaagaacgg 20
<210> 223
<211> 20
<212> DNA
<213> Homo sapiens
<400> 223
gttgcaagag aagaaaagaa 20
<210> 224
<211> 20
<212> DNA
<213> Homo sapiens
<400> 224
atgcacacgt aaaagagagg 20
<210> 225
<211> 20
<212> DNA
<213> Homo sapiens
<400> 225
aagatgcaca cgtaaaagag 20
<210> 226
<211> 20
<212> DNA
<213> Homo sapiens
<400> 226
atcatgcata catctctggc 20
<210> 227
<211> 20
<212> DNA
<213> Homo sapiens
<400> 227
ttcatttcat tttgattttg 20
<210> 228
<211> 20
<212> DNA
<213> Homo sapiens
<400> 228
attcaatttg aagcagtggt 20
<210> 229
<211> 20
<212> DNA
<213> Homo sapiens
<400> 229
gaatattcaa tttgaagcag 20
<210> 230
<211> 20
<212> DNA
<213> Homo sapiens
<400> 230
catacgattt aaaatcgctg 20
<210> 231
<211> 20
<212> DNA
<213> Homo sapiens
<400> 231
aaaatcgctg aggcgcgttc 20
<210> 232
<211> 20
<212> DNA
<213> Homo sapiens
<400> 232
tttttttttc ttttttgtac 20
<210> 233
<211> 20
<212> DNA
<213> Homo sapiens
<400> 233
ctgttgtcaa agattttaaa 20
<210> 234
<211> 20
<212> DNA
<213> Homo sapiens
<400> 234
tgacaacaga gttctgtttt 20
<210> 235
<211> 20
<212> DNA
<213> Homo sapiens
<400> 235
agaatacgct gagagttatc 20
<210> 236
<211> 20
<212> DNA
<213> Homo sapiens
<400> 236
gcaagagaag aaaagaacgg 20
<210> 237
<211> 20
<212> DNA
<213> Homo sapiens
<400> 237
gttgcaagag aagaaaagaa 20
<210> 238
<211> 20
<212> DNA
<213> Homo sapiens
<400> 238
atgcacacgt aaaagagagg 20
<210> 239
<211> 20
<212> DNA
<213> Homo sapiens
<400> 239
aagatgcaca cgtaaaagag 20
<210> 240
<211> 300
<212> PRT
<213> Homo sapiens
<400> 240
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met
145 150 155 160
Glu Arg Asn His Gly His Ile Val Thr Val Ala Ser Val Cys Gly His
165 170 175
Glu Gly Ile Pro Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala
180 185 190
Val Gly Phe His Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys
195 200 205
Thr Gly Ile Lys Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly
210 215 220
Phe Thr Lys Asn Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp
225 230 235 240
Glu Val Val Arg Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met
245 250 255
Ile Phe Val Pro Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Phe
260 265 270
Leu Pro Glu Arg Ala Ser Ala Ile Leu Asn Arg Met Gln Asn Ile Gln
275 280 285
Phe Glu Ala Val Val Gly His Lys Ile Lys Met Lys
290 295 300
<210> 241
<211> 264
<212> PRT
<213> Homo sapiens
<400> 241
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Val Lys Lys Glu Val Gly Asp Val Thr Ile
65 70 75 80
Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp Leu Leu Ser Thr
85 90 95
Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn Ile Leu Gly His
100 105 110
Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met Glu Arg Asn His
115 120 125
Gly His Ile Val Thr Val Ala Ser Val Cys Gly His Glu Gly Ile Pro
130 135 140
Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala Val Gly Phe His
145 150 155 160
Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys Thr Gly Ile Lys
165 170 175
Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly Phe Thr Lys Asn
180 185 190
Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp Glu Val Val Arg
195 200 205
Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met Ile Phe Val Pro
210 215 220
Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Phe Leu Pro Glu Arg
225 230 235 240
Ala Ser Ala Ile Leu Asn Arg Met Gln Asn Ile Gln Phe Glu Ala Val
245 250 255
Val Gly His Lys Ile Lys Met Lys
260
<210> 242
<211> 300
<212> PRT
<213> Homo sapiens
<400> 242
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met
145 150 155 160
Glu Arg Asn His Gly His Ile Val Thr Val Ala Ser Val Cys Gly His
165 170 175
Glu Gly Ile Pro Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala
180 185 190
Val Gly Phe His Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys
195 200 205
Thr Gly Ile Lys Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly
210 215 220
Phe Thr Lys Asn Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp
225 230 235 240
Glu Val Val Arg Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met
245 250 255
Ile Phe Val Pro Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Phe
260 265 270
Leu Pro Glu Arg Ala Ser Ala Ile Leu Asn Arg Met Gln Asn Ile Gln
275 280 285
Phe Glu Ala Val Val Gly His Lys Ile Lys Met Lys
290 295 300
<210> 243
<211> 264
<212> PRT
<213> Homo sapiens
<400> 243
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Val Lys Lys Glu Val Gly Asp Val Thr Ile
65 70 75 80
Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp Leu Leu Ser Thr
85 90 95
Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn Ile Leu Gly His
100 105 110
Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met Glu Arg Asn His
115 120 125
Gly His Ile Val Thr Val Ala Ser Val Cys Gly His Glu Gly Ile Pro
130 135 140
Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala Val Gly Phe His
145 150 155 160
Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys Thr Gly Ile Lys
165 170 175
Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly Phe Thr Lys Asn
180 185 190
Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp Glu Val Val Arg
195 200 205
Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met Ile Phe Val Pro
210 215 220
Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys Phe Leu Pro Glu Arg
225 230 235 240
Ala Ser Ala Ile Leu Asn Arg Met Gln Asn Ile Gln Phe Glu Ala Val
245 250 255
Val Gly His Lys Ile Lys Met Lys
260
<210> 244
<211> 2397
<212> DNA
<213> Homo sapiens
<400> 244
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataagcgcgg tgtggaggaa actgcagctg 300
agtgccgaaa actaggcgtc actgcgcatg cgtatgtggt agactgcagc aacagagaag 360
agatctatcg ctctctaaat caggtgaaga aagaagtggg tgatgtaaca atcgtggtga 420
ataatgctgg gacagtatat ccagccgatc ttctcagcac caaggatgaa gagattacca 480
agacatttga ggtcaacatc ctaggacatt tttggatcac aaaagcactt cttccatcga 540
tgatggagag aaatcatggc cacatcgtca cagtggcttc agtgtgcggc cacgaaggga 600
ttccttacct catcccatat tgttccagca aatttgccgc tgttggcttt cacagaggtc 660
tgacatcaga acttcaggcc ttgggaaaaa ctggtatcaa aacctcatgt ctctgcccag 720
tttttgtgaa tactgggttc accaaaaatc caagcacaag attatggcct gtattggaga 780
cagatgaagt cgtaagaagt ctgatagatg gaatacttac caataagaaa atgatttttg 840
ttccatcgta tatcaatatc tttctgagac tacagaagtt tcttcctgaa cgcgcctcag 900
cgattttaaa tcgtatgcag aatattcaat ttgaagcagt ggttggccac aaaatcaaaa 960
tgaaatgaat aaataagctc cagccagaga tgtatgcatg ataatgatat gaatagtttc 1020
gaatcaatgc tgcaaagctt tatttcacat tttttcagtc ctgataatat taaaaacatt 1080
ggtttggcac tagcagcagt caaacgaaca agattaatta cctgtcttcc tgtttctcaa 1140
gaatatttac gtagtttttc ataggtctgt ttttcctttc atgcctctta aaaacttctg 1200
tgcttacata aacatactta aaaggttttc tttaagatat tttatttttc catttaaagg 1260
tggacaaaag ctacctccct aaaagtaaat acaaagagaa cttatttaca cagggaaggt 1320
ttaagactgt tcaagtagca ttccaatctg tagccatgcc acagaatatc aacaagaaca 1380
cagaatgagt gcacagctaa gagatcaagt ttcagcaggc agctttatct caacctggac 1440
atattttaag attcagcatt tgaaagattt ccctagcctc ttcctttttc attagcccaa 1500
aacggtgcaa ctctattctg gactttatta cttgattctg tcttctgtat aactctgaag 1560
tccaccaaaa gtggaccctc tatatttcct ccctttttat agtcttataa gatacattat 1620
gaaaggtgac cgactctatt ttaaatctca gaattttaag ttctagcccc atgataacct 1680
ttttctttgt aatttatgct ttcatatatc cttggtccca gagatgttta gacaatttta 1740
ggctcaaaaa ttaaagctaa cacaggaaaa ggaactgtac tggctattac ataagaaaca 1800
atggacccaa gagaagaaaa ggaagaaaga aaggtttttt ggtttttgtt ttgttttgtt 1860
ttgttttttg tttttttgag atggagtctc actctttcgc ccaggctgga gtgcagtggt 1920
atgatctcag ctcactgcaa gctccacctc ccgggttcac gccattctcc tgcctcagcc 1980
tcctgagtag ctgggactac aggcgcccgc caccacaccc ggctaatttt ttgtattttt 2040
tgtagagacg gggtttcacc atgttagcca agatggtctc gatctcctga cctcgtgatc 2100
cacctgcctc ggcctcccaa agtgctggga ttacgggtgt gagccaccgt gcccagcctt 2160
ttttttttta atagaaaaaa taatccgact cccactacat caagactaat cttgttttgt 2220
gtgtttttca catgtattat agaatgcttt tgcatggact atcctcttgt ttttattaaa 2280
aacaaatgat ttttttaaaa gtcacaaaaa caattcacta aaaataaata tgtcattgtg 2340
ctttaaaaaa ataacctctt gtagttataa aataaaacgt ttgacttcta aactctg 2397
<210> 245
<211> 2289
<212> DNA
<213> Homo sapiens
<400> 245
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataaggtgaa gaaagaagtg ggtgatgtaa 300
caatcgtggt gaataatgct gggacagtat atccagccga tcttctcagc accaaggatg 360
aagagattac caagacattt gaggtcaaca tcctaggaca tttttggatc acaaaagcac 420
ttcttccatc gatgatggag agaaatcatg gccacatcgt cacagtggct tcagtgtgcg 480
gccacgaagg gattccttac ctcatcccat attgttccag caaatttgcc gctgttggct 540
ttcacagagg tctgacatca gaacttcagg ccttgggaaa aactggtatc aaaacctcat 600
gtctctgccc agtttttgtg aatactgggt tcaccaaaaa tccaagcaca agattatggc 660
ctgtattgga gacagatgaa gtcgtaagaa gtctgataga tggaatactt accaataaga 720
aaatgatttt tgttccatcg tatatcaata tctttctgag actacagaag tttcttcctg 780
aacgcgcctc agcgatttta aatcgtatgc agaatattca atttgaagca gtggttggcc 840
acaaaatcaa aatgaaatga ataaataagc tccagccaga gatgtatgca tgataatgat 900
atgaatagtt tcgaatcaat gctgcaaagc tttatttcac attttttcag tcctgataat 960
attaaaaaca ttggtttggc actagcagca gtcaaacgaa caagattaat tacctgtctt 1020
cctgtttctc aagaatattt acgtagtttt tcataggtct gtttttcctt tcatgcctct 1080
taaaaacttc tgtgcttaca taaacatact taaaaggttt tctttaagat attttatttt 1140
tccatttaaa ggtggacaaa agctacctcc ctaaaagtaa atacaaagag aacttattta 1200
cacagggaag gtttaagact gttcaagtag cattccaatc tgtagccatg ccacagaata 1260
tcaacaagaa cacagaatga gtgcacagct aagagatcaa gtttcagcag gcagctttat 1320
ctcaacctgg acatatttta agattcagca tttgaaagat ttccctagcc tcttcctttt 1380
tcattagccc aaaacggtgc aactctattc tggactttat tacttgattc tgtcttctgt 1440
ataactctga agtccaccaa aagtggaccc tctatatttc ctcccttttt atagtcttat 1500
aagatacatt atgaaaggtg accgactcta ttttaaatct cagaatttta agttctagcc 1560
ccatgataac ctttttcttt gtaatttatg ctttcatata tccttggtcc cagagatgtt 1620
tagacaattt taggctcaaa aattaaagct aacacaggaa aaggaactgt actggctatt 1680
acataagaaa caatggaccc aagagaagaa aaggaagaaa gaaaggtttt ttggtttttg 1740
ttttgttttg ttttgttttt tgtttttttg agatggagtc tcactctttc gcccaggctg 1800
gagtgcagtg gtatgatctc agctcactgc aagctccacc tcccgggttc acgccattct 1860
cctgcctcag cctcctgagt agctgggact acaggcgccc gccaccacac ccggctaatt 1920
ttttgtattt tttgtagaga cggggtttca ccatgttagc caagatggtc tcgatctcct 1980
gacctcgtga tccacctgcc tcggcctccc aaagtgctgg gattacgggt gtgagccacc 2040
gtgcccagcc tttttttttt taatagaaaa aataatccga ctcccactac atcaagacta 2100
atcttgtttt gtgtgttttt cacatgtatt atagaatgct tttgcatgga ctatcctctt 2160
gtttttatta aaaacaaatg atttttttaa aagtcacaaa aacaattcac taaaaataaa 2220
tatgtcattg tgctttaaaa aaataacctc ttgtagttat aaaataaaac gtttgacttc 2280
taaactctg 2289
<210> 246
<211> 1714
<212> DNA
<213> Homo sapiens
<220>
<221> misc_feature
<222> (1)..(275)
<223> Экзон 1
<220>
<221> misc_feature
<222> (276)..(383)
<223> Экзон 2
<220>
<221> misc_feature
<222> (384)..(515)
<223> Экзон 3
<220>
<221> misc_feature
<222> (516)..(622)
<223> Экзон 4
<220>
<221> misc_feature
<222> (623)..(760)
<223> Экзон 5
<220>
<221> misc_feature
<222> (761)..(1714)
<223> Экзон 6v4
<220>
<221> misc_feature
<222> (878)..(1714)
<223> Участок считывания из экзона 6 в интрон 6
<400> 246
agacagtacc tcctccctag gactacacaa ggactgaacc agaaggaaga ggacagagca 60
aagccatgaa catcatccta gaaatccttc tgcttctgat caccatcatc tactcctact 120
tggagtcgtt ggtgaagttt ttcattcctc agaggagaaa atctgtggct ggggagattg 180
ttctcattac tggagctggg catggaatag gcaggcagac tacttatgaa tttgcaaaac 240
gacagagcat attggttctg tgggatatta ataagcgcgg tgtggaggaa actgcagctg 300
agtgccgaaa actaggcgtc actgcgcatg cgtatgtggt agactgcagc aacagagaag 360
agatctatcg ctctctaaat caggtgaaga aagaagtggg tgatgtaaca atcgtggtga 420
ataatgctgg gacagtatat ccagccgatc ttctcagcac caaggatgaa gagattacca 480
agacatttga ggtcaacatc ctaggacatt tttggatcac aaaagcactt cttccatcga 540
tgatggagag aaatcatggc cacatcgtca cagtggcttc agtgtgcggc cacgaaggga 600
ttccttacct catcccatat tgttccagca aatttgccgc tgttggcttt cacagaggtc 660
tgacatcaga acttcaggcc ttgggaaaaa ctggtatcaa aacctcatgt ctctgcccag 720
tttttgtgaa tactgggttc accaaaaatc caagcacaag attatggcct gtattggaga 780
cagatgaagt cgtaagaagt ctgatagatg gaatacttac caataagaaa atgatttttg 840
ttccatcgta tatcaatatc tttctgagac tacagaagta agtacagcac agaacaccca 900
aatactaaaa caccaataga gctttttttt ttgctttttt tttttttaga cagagtctca 960
ctctgtcacc ctggctggat tgcggtggtt gcagtggcat gatcttggct cactgcaacc 1020
tccgcctcct gggttcaagc aattctcatg cctcagaccc ccaagtaact gggattatag 1080
gtgtgtgctg ccacactaca cccagctaat ttttgtattt tttgatagag acaggtttcc 1140
ccatgttggc caggctggac tcgaactcct gacctcaagt tatcctcctg tctcggcctc 1200
ccaaagtgct gggattacag tcatgagcca ccatgcctgg cccaatagag ctattattat 1260
ggagcatctt tcagttgtga aaattggcat ggaaactctc catccctggg gagaacagtt 1320
atttcctctg ttattttcct acccagtcta taaaaagaga gtgattcatt ttctctacca 1380
aatctactgt ctctgcccaa actttgctga agactattct aactaaagga aacacagttt 1440
aaaaagaatg caatatagtg aagtagttaa taataaagac tccattttta aaagtctgct 1500
ggaagtttgg ttgggattgc actgaatcta tagagcaatt ggggagtatt gacatatcaa 1560
caatattgag ttttctaatc caagaacata atatctattt ttaaaatctt cttcaaaatc 1620
tttaaatctt taaattgtat tttgtagttt ttggtgttta agtcttgcac atattttgtc 1680
agatttattc caaagtattt cacgggttct tttt 1714
<210> 247
<211> 271
<212> PRT
<213> Homo sapiens
<220>
<221> MISC_FEATURE
<222> (1)..(70)
<223> Участок, кодируемый экзоном 1
<220>
<221> MISC_FEATURE
<222> (71)..(106)
<223> Участок, кодируемый экзоном 2
<220>
<221> MISC_FEATURE
<222> (107)..(150)
<223> Участок, кодируемый экзоном 3
<220>
<221> MISC_FEATURE
<222> (151)..(185)
<223> Участок, кодируемый экзоном 4
<220>
<221> MISC_FEATURE
<222> (186)..(232)
<223> Участок, кодируемый экзоном 5
<220>
<221> MISC_FEATURE
<222> (233)..(271)
<223> Участок, кодируемый экзоном 6v4
<400> 247
Met Asn Ile Ile Leu Glu Ile Leu Leu Leu Leu Ile Thr Ile Ile Tyr
1 5 10 15
Ser Tyr Leu Glu Ser Leu Val Lys Phe Phe Ile Pro Gln Arg Arg Lys
20 25 30
Ser Val Ala Gly Glu Ile Val Leu Ile Thr Gly Ala Gly His Gly Ile
35 40 45
Gly Arg Gln Thr Thr Tyr Glu Phe Ala Lys Arg Gln Ser Ile Leu Val
50 55 60
Leu Trp Asp Ile Asn Lys Arg Gly Val Glu Glu Thr Ala Ala Glu Cys
65 70 75 80
Arg Lys Leu Gly Val Thr Ala His Ala Tyr Val Val Asp Cys Ser Asn
85 90 95
Arg Glu Glu Ile Tyr Arg Ser Leu Asn Gln Val Lys Lys Glu Val Gly
100 105 110
Asp Val Thr Ile Val Val Asn Asn Ala Gly Thr Val Tyr Pro Ala Asp
115 120 125
Leu Leu Ser Thr Lys Asp Glu Glu Ile Thr Lys Thr Phe Glu Val Asn
130 135 140
Ile Leu Gly His Phe Trp Ile Thr Lys Ala Leu Leu Pro Ser Met Met
145 150 155 160
Glu Arg Asn His Gly His Ile Val Thr Val Ala Ser Val Cys Gly His
165 170 175
Glu Gly Ile Pro Tyr Leu Ile Pro Tyr Cys Ser Ser Lys Phe Ala Ala
180 185 190
Val Gly Phe His Arg Gly Leu Thr Ser Glu Leu Gln Ala Leu Gly Lys
195 200 205
Thr Gly Ile Lys Thr Ser Cys Leu Cys Pro Val Phe Val Asn Thr Gly
210 215 220
Phe Thr Lys Asn Pro Ser Thr Arg Leu Trp Pro Val Leu Glu Thr Asp
225 230 235 240
Glu Val Val Arg Ser Leu Ile Asp Gly Ile Leu Thr Asn Lys Lys Met
245 250 255
Ile Phe Val Pro Ser Tyr Ile Asn Ile Phe Leu Arg Leu Gln Lys
260 265 270
<210> 248
<211> 23
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> misc_feature
<222> (2)..(21)
<223> n = A, T, C или G
<400> 248
gnnnnnnnnn nnnnnnnnnn ngg 23
<210> 249
<211> 23
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> misc_feature
<222> (1)..(21)
<223> n = A, T, C или G
<400> 249
nnnnnnnnnn nnnnnnnnnn ngg 23
<210> 250
<211> 25
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<220>
<221> misc_feature
<222> (3)..(23)
<223> n = A, T, C или G
<400> 250
ggnnnnnnnn nnnnnnnnnn nnngg 25
<210> 251
<211> 25
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 251
atgaacatca tcctagaaat ccttc 25
<210> 252
<211> 25
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 252
atcatgcata catctctggc tggag 25
<210> 253
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 253
atcagaactt caggccttgg 20
<210> 254
<211> 21
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 254
gcaaagccat gaacatcatc c 21
<210> 255
<211> 24
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 255
tcttgatgta gtgggagtcg gatt 24
<210> 256
<211> 82
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 256
guuggaacca uucaaaacag cauagcaagu uaaaauaagg cuaguccguu aucaacuuga 60
aaaaguggca ccgagucggu gc 82
<210> 257
<211> 76
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 257
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu 60
ggcaccgagu cggugc 76
<210> 258
<211> 86
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 258
guuuaagagc uaugcuggaa acagcauagc aaguuuaaau aaggcuaguc cguuaucaac 60
uugaaaaagu ggcaccgagu cggugc 86
<210> 259
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 259
ggcagaccgt tctcatcacg 20
<210> 260
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 260
ctttaccagt gactccaggt 20
<210> 261
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 261
gtcacagatt tccttctccg 20
<210> 262
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 262
agatgatgac gcccaccaga 20
<210> 263
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 263
ggagaaggaa atctgtgacc 20
<210> 264
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 264
tgcgaggaac ttacttttcc 20
<210> 265
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 265
agagaaatat tgatatagga 20
<210> 266
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 266
tatcaatatt tctctgatcc 20
<210> 267
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 267
atcgctttta aggcacgctc 20
<210> 268
<211> 20
<212> DNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 268
tatacgactg atcgctttta 20
<210> 269
<211> 21947
<212> DNA
<213> Mus musculus
<400> 269
gcagcagccc ataatacctg acagggctct ctgtggagct ctgagcagag gtgtctgttg 60
tgagaacaga gccatgaacc tcatcctgga atttctcctt ctggtgggcg tcatcatcta 120
ctcctacctg gagtcactgg taaagttctt cattccccgg agaaggaaat ctgtgaccgg 180
gcagaccgtt ctcatcacgg gggccggaca cggaataggc aggctgactg catatgaatt 240
tgcaaagcag aaaagcagac tggttctatg ggatatcaat aaggtaataa taggtgtttc 300
taacaatctt ttaaagtcac aaaataacaa ggcatatgtt tctctagaga cgtaacttag 360
tgctggcttt tctctgtgtt ttcctttttc aaagttttaa taaactcctg ggtcctttct 420
ggatccctgc gacaggtgaa caagatggaa ccaacgagct ggagaactaa gcctgggggg 480
tttcattttg tttgactggt tggttggttg gttggttggt tctactgtgg tgtacaactt 540
gataacaaga gacgcaggag ccagagagct tcccagctgc tgaaattagg caagccacat 600
aacttacctc aacttcaaat ttaaaaaaca aaaaacaaac aaaaaactct accattggga 660
taaaatctca gagggagaca aaatctacag gtaatgattg ccagcaacca aattggccaa 720
tatgcagttt attacagcta ggggtgactt ttatactttt agattttcat ttattctgtt 780
attcatttgt tcctttctgt gatagactga agctgggtct tagtacctac cactgagcca 840
cagtacctta gcacgtgcca ggcaagcact ctatcaccga gcaaccgctc aggtttttac 900
acatttgagg gattgattct caaaacaaac aaacaaaacc acatgaagaa ctaacagcag 960
aaacccatgg tacttccact cacactccag caatcctaaa atacttacat ctgacctttt 1020
gcagaagaag aaaaaatact tgccgggtcc tgagcaagaa gggtaaagaa taatacttaa 1080
gtgcttagca tggcctggtg cacgcagcat cacatatgga aatgtctaca ccagtgcctg 1140
gctttcacta gtacatttaa gaggttgttg atctctgttt atttataaat gctcttaact 1200
accttaagtc tgaaagtgtt gacgtcctgt gtcactttct cctaaacatg cacattccca 1260
aaaggtcgtg aaaaagatgt gccacaacaa actgtctctg ggattgtaat ctttgaagaa 1320
ctggactggg gatgtagccc agcggtacaa caattatcta gaatatacaa actagatatt 1380
agatccagtt gtggctgggt gtgagcctca gcactacaag gaaaccacaa accgctgctg 1440
tcatgtaccg gatacagtgg ctcagactag agagactggg gttttggtgt ttgtttgttt 1500
gtttgtttgt ttgtttgttg agaagcagag tttccaaagc tttccttggg gttttgctcg 1560
gcttcaggta accctctgtt acgtttttgc catctgcagt taacaccact tttcccagcc 1620
ttcagaacgc tcttgaacat aaggagctaa atctgctttg gttccttttg agcagtgaag 1680
aatcccgaga ggcaccatgc atctttgctt agcagataca ggcgttggtg ggcaagatga 1740
gctaacacgg tacttggaga tgattctgag attatcaaat cctgcacagg tcccaggcag 1800
tcttcttgga gctgagaaaa ctgagtcagc tgttctcagt ccctcgtaaa aaatataaac 1860
gagaatgaga atacatcatg aagatggtcc acgggtcttc agatcacctc ccaagtgtgc 1920
atggtgggaa gacatcaata cttagtagac tacctttaat ttgaaaattt tcagacacag 1980
aaagagtaaa actagaaaga ggtcacatta aagatgactt ccactttttt tttcctgtgc 2040
ctgaaaactg agtaactcaa ccttgtaaat ttctctttga attcttgaac ttcatgatga 2100
aacatttaag acatcaaatg attgactgaa aagctgcaag tgtaactcca gtttccctgt 2160
tcactgagag gccagctagc tttccatcaa agaaatatgg ctccttttcc ttaaaaggca 2220
agaagcaagg ccacagagaa gcctcctcca cccccagctt taaaaacagt ggcacagaga 2280
tgtgaaacac aactgttagg aagggaattc agcagtgggg agggaggggc tgcaggaagg 2340
taacagggga taaaataata agagcataca tgtgtatgca aggaatgaaa agcaaattta 2400
agaagaacgc aatgacacaa tagcaaaact gtggacccag ccaaggtgcc cacccagaga 2460
catgggttgg aaaatgtggt ttcacacaca ggacatcgtt cagtcttttg gaagaatgac 2520
atcattctgt ttgctggaca gtggaaaaaa ctggcagtca caactttagg tgaaaaggga 2580
gccagactca caaagggaag gatcagtggg gggcgggggg ggggggaggt cactctgctc 2640
tggtcccagc attggaccag gaaccaaggc aagagggttg ctgcaagtta gaacccagcc 2700
ctggctacag atgaaattat agcaagagac aaaagagggt aggggaaggc agcaagcatg 2760
ggctctgtca tgcctcgtgt gtgggaatgt catcatggaa catactagtt tgtacgttaa 2820
taaaaaggac acagggatta gcaatgggca gaggggttga caccccagaa ggtgtttctt 2880
gtgcaaaagt gacgaagaga tacattcttc catgctccag attaattata aggctggata 2940
gttcgcaaat tattagcatc tatcactcat acatgatgct aggtttcacc gtaagctttc 3000
atagatgtgt atattctggt catattcact gttcacccat cattatctct ggtcccctcc 3060
cactcttgct gacccccttt ctaagtgccc ccttgtgctt tcatgtgtgt gtgctaatga 3120
ctttagatag gattgctaac ttgagcctag acgagggctt gttacaggag tagggatggc 3180
ttgctggtgg cgaagaaagt aaccctgata cactgtttct tcagtcacat cttttgctta 3240
tgctactgaa gaaagtaaca ctgataaatt gtttcttctt cagtcaaatc tttgcttgag 3300
gctctggtta ctgcctctct ctacagctct gacgtctgta tgaggctagt gaggagggca 3360
gctgggccat ctgacagtaa gcagccttca ttagtggaaa taaagccaac attagtggaa 3420
aactctccta cccagaccac agctctattc cacagcccca cgaggaccta tgctctatgc 3480
ggcagtccat cccaagtcta tgtgaagact atgttggttt cgtaagtgac accttaaccc 3540
tccttaaagt ctgttcttta agacttctat tgttgctttg tggtttagcc aagataagga 3600
aatgcaaagg gtcggagggg aggcaagcag tgtaagccaa acataccatc cactcaggca 3660
aggcccaaat gatggctaca ctattgccat cccagcttgc acaagaaaga gcttcctggg 3720
tgtcctcaca gagaagtggg gacaaccatc agaggaaaaa gtgtgatttt taaaaagaat 3780
ttgaacccca taaaatcaat aggtgtcaaa acttggaaag agtccaaatt ttcctgaaat 3840
gtaggttaaa aaaaaaaaaa aagccaaggg tagtgactca cacctgtaag tgcttgagga 3900
ggttgaggca ggaggattgg cctgagttga agacccgctt agggttcaca tagtaagcaa 3960
gggatagaga atgacacctg gtctaaaaca ccaagagaga acacacagac acacagacac 4020
acacacacac accacattag gacaatctta ggtaagaggc ttctggattt aggctaaaaa 4080
ttagctgtct ccatgtcttg gaggacttgt gtgcaattct cactttcgtg agaattcagc 4140
tgttggctct tggccttttt ccctccagag cttcagaaga ataaatactc agttaagact 4200
gtcacgtgta gtaagttaca gtttcctcgt gcaagctttt cctggataag attttctcct 4260
cgcatccgcg ggggtgaatc cacgttgtga taagccatct agtgtcactc tagagggctg 4320
cttagcactc tactgccaac aatgggtgat gcccggcaac tgtgtggact agtcttcctt 4380
ctgtcctccg tgctcagaca tcttccctga cactgtgagg agcagcatct tcgataactt 4440
ataggaagcc cttcatgcta ccacagcatg acgctagaca aatcacctac aggcactccc 4500
aggacaagat gcgaagctgc aaaagcctta acccgaaggg cttgtttctc tttgattccg 4560
ctgctgcttt ggccagctaa ttagttaatg tagttagatg agttcacata acgaggctta 4620
ttggacaagg atccgaaagt attttacaga gcctgatctt ttctcaaatt tgcctccccc 4680
ctccttcacc cctcccgccc cccaccccgc ccagcgtggt gttgaggaaa ccgcggacaa 4740
atgcaggaaa ctgggggccg tcgtgcacgt gtttgtggtg gactgcagca accgggccga 4800
gatttacaac tctgtggatc aggtgagaac ccgggtgcag gtgtcgcatc agctgacact 4860
gagctgggaa acgaaaaggt gggctaggga aggttgactt ccggtagacg gcagccgccc 4920
tgtcaagttg ctgtgtgacc gaaggagtga atattaatat ccactcagct ctggcctctg 4980
tcactgtttc atattctgag tcaaatctgc tgtgtctaaa aagtttggcc cagaggaaac 5040
gaaagatgat ttatcaagag aaaatttgat ctcttttcta acttttttaa aattagactt 5100
tgttctgctt tttctttaac ttattccctc tgagatttta ttggtttgcg tataatctag 5160
tcttttatga aaaacagagt taatgacttt ttaaacattg tatttgtttt atttactgca 5220
tcgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtgcgcgc gcgcatggtg 5280
ggggtgtatg tagaatttag atgacaaatt ttgaaagctg gtctctcctt ccatgatggg 5340
gattctgggg gttgttaggt atggcaaaaa cagaagagga ggaggaggag gaggaggagg 5400
aggaggagga ggaggtggtg gtggtggtgg tggtggtgaa gaagaagaag gagcaggagg 5460
aggggaggag gggaggagta agaggagaaa gaggaaaggg aaggggaggg gggaggggga 5520
gaagaaaatg cctttaccca ctgagccgtc tccccacccc acaataccta ctctttcaaa 5580
aagtgaacat caagagctta ccaaaaaaaa aaaaacaaaa aacaaaaaac aaaaaaacaa 5640
aaaacaaacc ctccgctaag ctactgcctt ctttaatctg atttaatttt taaaaatccc 5700
attagcctca ctatagataa gcaaatagaa gcttggaagg tttggcccag acaaagacca 5760
cgtggccagt gggtagcaaa accaagatta attacttaac actttaaaaa tatcttaaca 5820
gaacttaagt attatttacc agcacaaaca ttaattcttg tttatttaag ttcataagtt 5880
ttcaccctac ctatcacata ctgagcacag aatttagggt tttaaccaaa acaggttagc 5940
cctacctcat gacaggtttc ccttcctgtc actgcttggg ctggttcttt cacgcctgaa 6000
ctctgagacc tcgtgcactg gaataacaaa agcctagatg gcatagtgac caagctagct 6060
gtcaccaaag ccagtgagcc cagacagaaa tgcatcgtaa agctattctt tattcttgga 6120
agtcagatcg atatcttgga tatctgtctt ccatgtcacg ctgtgatgtc tgtgtgtctc 6180
tcattatcat cccttggaca acttcctcct tttgtgttat tgggctcagg catttcttgg 6240
agttgagttg gcctagttta tgctggtatc aatcttagac tcttgggatg gctaggggta 6300
gggtgtggag tggttagctc agaacacatg gtgtggagtt gctgagctat aaatattgtg 6360
tatgctctgg tttcttttta catggtggag tatatatatc cttataatat agaacataaa 6420
aatgcctcta cccatctgtc tcctatcaac atgagctttg tgagaatgac cggtcctgtg 6480
tgtgcatttt aaaaggtaaa gagagaagta ggtgatgtcg agatcgtggt aaacaacgcc 6540
ggggcgatat atccagcaga ccttcttagt gccaaggacg aggagatcac caagaccttt 6600
gaggtcaata tcctcggaca tttttgggta agtcagaaac atttctgggt gtgtgtgtct 6660
gacatctctt cggtacagga aactcattga ggtcacaata tttgtggagg gaggtgtttt 6720
tggttttgtt tcttaagatt tatttattta tttattttat gtatgtgagt acactgtagc 6780
tgtcttcaga cacaccagaa gagggcatcc gatcccgtta cagatggttg tgagccacca 6840
tgaggttgtt gggaattgaa ctcagtacct ctgggagtgc tattaaccac tgagccatct 6900
ctccagcctt gttttttgtt ttgcaagtca ggaatcttat aatgtgtttg aatagaagag 6960
ccacaggtct ctggcaacag ggttcagagt tcaaggagac acaggacacc caaaagagag 7020
agccatttgg tttattttgt caaacaaaat ctaacatagg gcctgatgaa taccagatct 7080
acaaaaagta aattaataat aaaaaattta ttaattaaaa atatttgtaa aggtttactg 7140
atatttcctc tgtacatctt ttcactattt tagaggaaat gtatgctgca tacattggtc 7200
tgcatcttcc tttcataggt gtttagagct gaaaggacat agaatactta gatttgtgca 7260
gaagttgggg acacttagag ttaggctctg agataagtag acattttgac ataacattat 7320
tatctacaaa gttcctgcct gagaacttca caattgagtt accaataggg gggggaaaca 7380
atctcaaaat gttttactgt agctcacaaa tttgcattga tctatattcc cagctaatct 7440
cagtcataag cagcctatgg gcggtgggat gagcatgcct attagttctg gagtgtagag 7500
agaatgaatc caattcctta ctattgggag tttgtgagaa actgaagtac ccttctcagc 7560
cctttggagg gagcatttgt atgaattaat accgtgggac ctcaaaatta ctatagcacc 7620
tggagcaaac ctggatttga agaacatcct tcatcctcat gacccagcta acatgacagt 7680
cacacaacac agggacccta tatactagtt atctaataat taataggaaa ttataactat 7740
gtcacaaaat atatcaaaag gtagaggttt agagaaccag gagccctcat tacctccata 7800
cagatctgca ggcagggctg atagggggat tatcttggca acctgggctc taggaattta 7860
cacagaattt gaagagtggg tctgtaaaat tctcataaaa tgttctgata tttcccactg 7920
tacagtgagg cttttgtttc ccataatgta tcctatactg tatactgtta caagagagag 7980
atttgggtct gctccctgtg cacggtggtt gtgggccagg atagagcttc caccgctttg 8040
acgtgagtaa agatccccat tcttcaaatt ggagccaaga acttgtaaaa cggtaaacca 8100
tcctccagtt cccgcactgg tggatctctc ccttgacaat attgcttagg gctcagcagc 8160
accaggcttg cctggcatgc acaaagtcct ctggttaatt cctagcacgg cggaaaagta 8220
acaatgttgc ttctgtggca aatcccggaa ggagctactt aattcttcct ggagggatga 8280
aggggcatgc tcgcccgcta gcactggtcc tcagcacaat ctaggggact cagctctgag 8340
tgctgtatgc acgcttgcac aggtcctctg catgatctaa gggacactct gagtactgcc 8400
tggaaccatc acagttactc tctctttctg tcactgtgtg gaaggaacct cagggtctta 8460
caagtgtgta atttccaatg cctcctcctc cgctatggtg gaggcattgg aaatgaaaga 8520
gcctggcctg gaatccacct aataaccgtg tcattgtttc tgttcttttt acgtggacct 8580
taagatcata aaagcactcc ttccatcgat gctgagaaga aactctggcc acattgtcac 8640
agtggcttcg gtgtgcggcc atggagtgat tccttatctc atcccttatt ggtaggtgtc 8700
tcttgctcac agcgctgtgc atttctgctt attagtttgg ggattattta tagggattat 8760
tataattagt atatttatgt gttttataaa tattatatac tataatttta taaatgtatt 8820
catatattat ataactaaat aattataggg attaattata ggaaattatt agggattatt 8880
agttttcagg cctcaatgcc cacagcctag taaagtgccc agaagctaac acaagcacag 8940
aggtatagcg tggtctatgt gtgtgcatgt gtgagagaag agaacggagc agccctgcct 9000
tcccaggaga tgcacacact gcacaccatg tcccagggac ccagacacac tcgcagtctg 9060
cagcatatcc ctgagaagtc tgctattgcc atcttgtttc cttactgcta tacgatgatc 9120
tcagaaacac ttttaccaag atctgtggat gtagcagatg caggtgttat ttctttatag 9180
gcatattcaa agctaaactt gaccatggac taagcaatct ctcacactat acatggtcac 9240
agatgagtta aatttcagtg tagttcaggt ttccattaca cgcacacact cacacacata 9300
cacatacaca cacaatacac acacatgcac acacatacac cacacacacc atacacaaca 9360
catacacaca caacacacat tcacatatgc acacacatat acacacacac acatcacaca 9420
tgcacacagg agaggaagac agagacagca ataacttcta acactgatat tttttgtcac 9480
ctggacaaaa actacaatta aaacttatat agctccatat atgcatatgc taatttattg 9540
aattctccaa gacagcctta tattttgtta tctatatttt ataatcagaa aaatgaaaga 9600
acaaaaggtt aaaggttgca catgcaaaca caaccctacc aatgccatct ggagcccctt 9660
tgctccaaat actcgttgtg acattaggac acacatgaga ctgccagtgt caagggagaa 9720
aacatcagct ctttaatcaa gagaagagaa atgagctcag acccacaagt ttgtcgatgt 9780
tggtttcaca gtgttttaag tgtgtttttc tgactcacat ttttggagat aaaaactaat 9840
aaaacctgat taagaagcac ttggaaggtt gcctggacca tagagaaggg tcacacatac 9900
agtgagaaac attaagccta tctgtaaagt ggtccttttg ctatctttgc tagttagaag 9960
ttagaaacag gggctttggg gttagcaatg gttgaagaca tgcatgtgtc aatccctgag 10020
ttcaatcccc agcacctaaa agaaaaaagg aaaaagaaac aggtattttg catgctcaga 10080
attctgcttc ctacataaga aaataaactc gagcggcagg tgacaggtct ggacacatga 10140
agctgccttc tctcaggcag ctgagactgt ccccaccacc accaccacca ccaccaccac 10200
cacacacaca cacacacaca cacacacaca cacacacaca cacacacaca cacctttgct 10260
gatggctgat tttctcacag ctccagcaag tttgctgctg tgggcttcca ccgagcactg 10320
accgcagaac tggacacctt ggggaaaacc ggtatccaaa cctcgtgtct ctgccctgtg 10380
ttcgtgaata ctggcttcac caaaaacccg agtacaaggt gagatggaag acctttaagc 10440
gggagccccc ccctccaggc aatggagtct tgagtaggtc ataggtactt aacaagcatg 10500
tcacgatcat ttaaataagt ccagtagcat gatctcaacc cctaggctga cactgaggtg 10560
cccatgtcag atgtggctct aaggctggat tgaagactcc attgcaaaat taggaagggg 10620
agagacagag gcaggataat ctaaaaagaa ttgatttgtg ctgctgtgtg atggtttgct 10680
agtccacatc acaggcatca caatcgcacg ggagtctgcg gaaaccacag actatctcat 10740
gggattcagt tccaacaccc acacagcctt gcctcggcat gctaaccttg gtgtgacatc 10800
taaaatccat ttttatgctt tgatttttcc ctcctcttca ttaatgggca aggctccaac 10860
actggacatg ggttatacgc atttctcagt tatacacggg tgcaactttt gaatcacgaa 10920
acgccccagt gctgtaacag aacactcctt gtacaaatct taaattccca taaaattgca 10980
ggagaagcaa atcttagtac gagatgcctc agaacaactc agctctgtgg atacccacac 11040
acaacgggaa gtgtactcag acatagaata ttctttttac atacagcagg cattggagat 11100
ccggtgtttc cttcctggcc attttgtccc ctggctcctc agtccctccc catcttgtac 11160
cttgttgtga tttaattatt ttgacctggt gaggtaacag acagacctac tggacccaag 11220
agtacaagaa catgataaca agttatcagt gtgggatgtg ggaaggggag agggtgatgt 11280
tcactcaggg aagcctggaa cattgtaaat cctaattcta aatttggtga agagacagga 11340
taatttcctt catttgttaa ttaaaaacaa tataaatcaa gaggctgggg ttttacctca 11400
acatcagggt atgtgtgaaa gttctggatt taattccagc atccaaagga agaaataaaa 11460
tataaacgga gataaactct ttcccccttt ccttcaggtt atggcctgta ttagagccgg 11520
aagaagttgc aaggagtctg atcaatggaa tacttaccaa caagaaaatg atcttcgttc 11580
catcctatat caatatttct ctgatcctgg aaaagtaagt tcctcgcaga accctgaaac 11640
actcaaacac aaatgttaga aatgttgcta tggaaactcc ccatggctgc agtgaaagtc 11700
acgttttcca tgttgcctta cccatgagtc tttgattccc tgataccctt tctggaatac 11760
atctaccatc ttggcccaaa ttttgttcaa gtctatttaa cagaaagaaa gaaaaactac 11820
agggtatggg cacaccataa aggctctgtg tgtgtgttca tgtggtgtat gtgggggata 11880
gagatgtggt acttgtggag acagaggcaa agctctcagg aaccatccac ctaggtttct 11940
gatttgggtt tatttctctt ctctccttcc cctcccctcc cctcccctcc cttcccctcc 12000
cttcccctcc cctcccctcc tctcttcctt ttcctttcct ttcctttcct tttcttttct 12060
tttcttctct tctcttctct tctcttctct tctcttctct tctcttctct tctcttcctc 12120
ttccttccct ccctccctcc ctctctttcc tctctctctt tttttctgat acaatctctt 12180
attgagacct ggactcacca gcttggctag gatagatatg catcaccagg gatatcccag 12240
gccctctatt cccacagtac tggggttaca agtgctgaca gctatgccca gctgtgtgtg 12300
tgttctgagg attgaactca ggccctcctg ttgtcgtggc aatctcccta aaccctcggg 12360
tgctcagttt tgcagttctt tgtgacagtg tgcatctcga atgtctcata acatgctttc 12420
acatgtttct atttcacttc atttataacc caagagcctt atgatagaat cacttctttt 12480
catctcaggc ttgtgagccc ccaaagaggg caaaatccca caaattgaat ttcctcaaca 12540
cagctcaaaa caggtctcat taaaagttga aaaaaaattc caaaattggc atgtatatga 12600
ccaaagtgca aaaatggttg actgctctgc ctgggatagg accagttacc tgtctcaaag 12660
cccttgccac actgtgggtg atttgttacg ctggcaacag ggatttttgt tgctccttgc 12720
atttaactcc ttttggagag agacagttct gtgatcagaa gcaatcacta gtctctgggc 12780
ttgttgttca atggcctgag caagctctct ccttaccacc tcacagcagg gtccccacct 12840
cccagtctct tctcccacct tctgccctct atccagggaa gtaaatgttg ctttagtaaa 12900
caataccaat gtgatctgaa atttggcttt ctctgtagaa atcgcacctc ctgcctcttt 12960
cacttcactc acacccttcc ctattctgtc aactcagacc taaccctccg acatgctgag 13020
aggtattcag cccctgtaaa cccttccttc cacatcttgt tatagtcagc tgatggtttt 13080
ctgtattcat ttccagttgg ccatgtttaa ccgctgggca gcgcaataca ctaagaggca 13140
gtagcttttg aaaaggctct ttagtttttc tttctctaaa ataataggtc tggacacagg 13200
cagagggggg aagggggtgt atgtggtgga ctctcagttc ttggccccag aatacactgc 13260
cgttttttgg aagtgagctt ggtgtcggtt ttaggttgtc attggtagaa gacgtcatcc 13320
aattttgctg ttctgagaga acaacgctat attatatata gataactgta gatggggtgg 13380
gggaggggag actaaacaat aagcaaagag tagaggaaaa ctggacttga gtaagtatgc 13440
tcttggcgct ttttaaattg acaacaaaaa ggaacactgg catcaaaact tcaaactgaa 13500
atttctcatt ttatttttaa tgtaggtttc ttcctgagcg tgccttaaaa gcgatcagtc 13560
gtatacagaa cattcaattt gaagcaattg tgggccacaa aaccaagatg aagtagtgca 13620
tgcagagacg tgtggacacc aatgatgtga agccaagttt agaggggaca cacagctttc 13680
tttcacatgt tttaagtgtt ccacatgttt aaaatgtagg cttgacccta gcagccatcg 13740
agtgcataag cgtggtcaac tgtccttcta gtttcctata cttatagatg tctcagctcc 13800
tggtagtggg ttctggcaat ggatatgtaa aggagggaaa aggcaatcca tgtgttttta 13860
taaaataaat tactaaatag aaatgtgggc aaaagggcaa gataataaag tcttgggcaa 13920
atgttggcac gctgtagtga tttctctttc gatggagaga ttggtggtgt tttaaatttg 13980
ctctttgtgg acaggatgaa aactctacct ggtttccttg acatatgaat taggaaggag 14040
tatttttgtt ccacattttt tttctcagtg tgaggttttc ttcacatcat gtgaatggac 14100
aatgaggtca ttttatttac ctaacttggg gaagcaaata ttgacacaga tttgcaactg 14160
tgaaatctca acaccttgtt ctttgcttta gaaagacctt ggacacattt gtttttcttt 14220
ttattctatt aggcaataac tgaaaatacc caaccgtata catagtaaca gtgacatgac 14280
ctcccacaca gatcccatta tgaagcagga aaccttcctg ggttttcctt tggcttctgt 14340
attttgctgc aggggactga tcccacagac taagacaggt tacataggca gtctgtgttg 14400
acctcctgtc tatcaacttg cagagttact tccacatttt gctttgatac ctttaaagtg 14460
aagctcagag ggatgcctca gtatgtgaca atgatctctt ttgcccacgt ctgcatattt 14520
tgagtgtaaa tatcttccct acaaggtcca gctccatttc taaatatttt tctatgataa 14580
tcttcacagt cacagttctc actcctgcta accattgcag ccgcttaggc taccccagga 14640
agcacagatc cctaagtaag accccatcta cacaaatcat ttagttagag acacatcata 14700
gaattcctag gtccctgcga gcacggggtc atgatttatt gatgagtatt tcagaatggt 14760
ggaagtattt gtttattgtc tactcagttc atggaaacaa cctttttggt aagcagacag 14820
tatctcctct tttttttaat ggtagaatat gatattgcat atacataagc acattttgaa 14880
catagctgaa tataagtatc actgtttcat ccttcttctt cttcttcttc ttcttcttct 14940
tcttcttctt cttcttcttc ttcttccgga gcatataatt ttattgctct tcttttttaa 15000
agaatagaag aatatctttt atactaggtc catgggctat ctagtgtctg gtttttgttc 15060
acccaagcag tgctagatac aagctccatc ttgtgtagta ggctttaagt caaaacagat 15120
ttggttggtt actctcacaa cattgggcta ccattttcct accatatctt ttaggcacaa 15180
caccaatgtt gatccatggg tgtgtggctt tgtttgttgt tcttttccta ggagcatgca 15240
gaatgacttc ctacacctaa gatactagta gataggagtg aagagtctat gtagtcacca 15300
gattgacttc tagatgttca gtgagttgag ttgcctttag caataggacc ttgttgacag 15360
gtggtgtaga gaaacatatg gccatggtaa cagcttgagt tctttgcaga ttcccatgta 15420
aacatttgga caacatctca gtaaaatgta attgagtttg aatactggaa gcttcttttg 15480
gtgaggaaag ataccctgtt agaaacaagt ctcctttgtt atagggtgat ttcatttata 15540
tgtgtaattg cctttgtatg tgtatatttt tctatcaatc tgtctatgta tcatctatct 15600
aatctctctg tcatctaata tatgtatcat ctgtcagtta tccacctatc acacatatat 15660
agctattgtc tatcaatcac cttttaatct accttttcat tctatcaaac tattaatata 15720
tcaatcactt atgtatttgt gattgatcta tcttttaacc atctaattat cacatatctt 15780
tcttctattg atgatctatt ttcatctgtt tctctgtctg cctctcattt ccttttgaat 15840
agctctagct atcatatgca aagggaagac attttcacaa aaataagcag tcttgagggt 15900
cctcatgatg ttttcagtcc ctcagtgtct cattccaaaa gcccaaagac accatagaat 15960
tcagacagaa tgccatgtaa caaagcccca cactcaatac ccagttacaa ccaccttagc 16020
agaacactaa agagattaaa ctatccttgg ctgccactaa aatcaggtag tcttcatggt 16080
aagtgtggat ctagcttttg gggaagtcct gtattctttg ctaacccact ttcatttcaa 16140
gattgaacaa aacacaggaa gatggtgccc tgtgaggcct tgacactcac atagcctcag 16200
tccagttagc tccctgtcca tgtgttcaca acagatgctt gtgataaaga aagccccaca 16260
ggatgggtga ggtctgagtt tctgaccaca gatccttcct gttatttttg atgttatttt 16320
tatgtttgtg tggttatctt cttttgggtt tgttgaaaga ggatttcttt ttctttttct 16380
aaggtgtagt ttcccttctt gtgttggcat tttccatcta ttatccttta tagggctgga 16440
tttgtggaaa gatattgtgt aaatttggtt tgtcatggaa tatcttggtc tctctgtcta 16500
tggtaattga gagttttgct gggtatagta gcctaggctg gcatttgtgt tctcttagga 16560
tctgtatgac atctgcctag gatcttctag ctttcatagt ctctggtaag aagtctggtg 16620
gaattctgat aggtctgcct tttacttgac cttttcccct tactgctttt aatattcttt 16680
ctttgttttg tgcatttgat gttttgatta ttatgtgaca ggaggaattt cttttctggt 16740
caagtctctt tggagttctg taggcttctt gtatgtttat gggcatctct ttctttagat 16800
tagggaagtt ttcttctata actttgttaa agatatttac tggcccttta agttggggat 16860
cttcactctc ttctatacct attatcctta ggtttggtct tctcattgtg tcctggattt 16920
ccaggatgtt ttgggttagg agctttttgc tttttgcatt ttctttgact gttgtgtcaa 16980
tgttttctat ggtatcttct gcacctgaga ttctctcttc tacctcttgt atactgctgg 17040
tgatgcttgc atctataact actgatctct ttcctaggtt ttctatctcc agggttgtcc 17100
ctttgtgatt tctttattgt ttctatttcc attttcagat cctggatagt tttgttcaac 17160
tccttcacct atttggttgt gttttcctgt aattctttaa gggagttttg tgtttcctct 17220
ttaagggttt ctagacattt acctgtgttt ctttaaagga gttatttatg tccttaaagt 17280
cctctatcat catcatgaga tgtgatttta actcagggtc ttgcttttct ggtgtgttgg 17340
gatatccagg gctcactgtg ttgggagaac tgggttctga tgatgccaag tagccttggt 17400
ttctgttgct tatgttcttg ctcttacctc ttgccacctg gttatctctg gtgttaactg 17460
gtctggctgt ctctgactgt ggcttgtccc ttctgcaagt ctgcattgtt agtacttctg 17520
ggagaccagt tctctcagga ggaatttggg tatggagatc tgtggcacag ggtcagctct 17580
ggagtgtaga cagaaaccag aaggatcctg tgcctggttg ttccttgatt cctgtgtcct 17640
gatggctctg ggaaggtttc gcttgggcca cgagtttgaa cagaagtggt gctcttacct 17700
gtgctcgggg gtgtgtcagc actcctggca aaccagctct ctcctggccg tatttgggta 17760
tgtagcggtg tggcacagga tcagctccgg gcacagacgc aaaccagaaa gcttctgtcc 17820
caggccgctc ctggttcctg tgtcctgaga gttccaggag ggtccttctg agcagcagtg 17880
gtggttttac ctgagttcaa aggcttgtcc acactcctgg gaggcaagct ctctcccagc 17940
actatttggg tatggagccc tgtggcagag gatcagctct gggccaaaca taaaccggaa 18000
ggctgacatc tttcaaaagg agtctttttt tctttctttt tttttggggg ggggggggtt 18060
gatttttttt tttttcgaga cagggtgtct ctgtatagcc ctggctgtcc tggaactcac 18120
tctgtagacc aggctggtct cgaactcaga aatctgcctg cctctgcctc ccgcatgctg 18180
ggattaaagg tgtgcgccac cacgacccgc tcaaaaggac ttaatgttct ttatttaaca 18240
atgttatata ttttctagca acctatcttc tctccctctc tccctttctg cttctctctt 18300
tcttcctctt tctttttctg cacctttctc tctctgcctc tctctcttcc tttctctctg 18360
tctctgtctc tctctctctc tctttctctc tctctctctc tctctctctc tctctctcgt 18420
gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtgtgtgt gtgtattaaa ggctgcatta 18480
caccaggcaa aggctgtgtt acaccaggta aaggctacat tacacaggta aaggttgtgt 18540
tatgccaagc aaaggagttt ctttgaactt taatctcatt gcacccacaa gagagtaagc 18600
atacctcagg ctaggtgtgt tcactggaat acgtgaagta aaggttgaaa cttctgttta 18660
tgactaattc tttatcttaa aactttgttt tttacagtat atacgatatg tgattatatt 18720
atgacacgat atatactaag taaatattag gtacacactg aagtttagct gcaatgctca 18780
gtcagaccca cagtcctttg cccactcctg ttctctctct ctctctctcc ctctctgtct 18840
ctctctctct ctcgctctgt actctatagc tgtctttgag caaagatcac aggaaccaaa 18900
aaactgagag gtccagagca tgagcctagt gcacatttta gtattcctac atggttatag 18960
gtgcactgtc tcctgtccat ggtgtctacc accctaggaa aagctgcagg gtctagagat 19020
cttagaaata gaataagcca ggctaaatac acagaccact gggtaaatca gtgcagtggt 19080
cactgttgtg ggctagtcac cagagaagac tgttcaagcc aatggaaagt cattactagc 19140
tgactggtga ctacactgga tgttcaggat cccagtgtag cactgagcct tcctcaggtt 19200
gagcttttaa gcacaaaaac catgttcttg gttgacgtac ttcagttaac aagaacagtt 19260
agctggaaga ggaactacag aagctaaaag gcaacattag tacattatga gactttacca 19320
aaacaatgaa ccctaatgaa ttatgccttt tagttttgac aggtagggct gtctaagtgc 19380
tgagttttac agcctgaaca gtacttccat catggagtca gatatgctaa gatctgggac 19440
cctgttacat tccacactgt tcttagtgaa tgagtcaaat catggattca tcctgtttta 19500
gtttaagatg aagaatgaga attaaagtgc tgacacaaaa tcattagcca gggagaccac 19560
gagaatggag actggtactg actacttcct caaccatgtc tatattttct ctaattattc 19620
cttaccaatt agtatagaaa caataggttt ctttcagagc catgtatagt ttcctttatg 19680
tcatttgaag tctaggcctc tccaattttg tagggagaat ctacctgcaa gggaatctaa 19740
ctgttgtttt aattcagaaa ctgaaacttc aaatacctgt aggtcctgat caacctatcc 19800
tcttagtttg gaaagcctca gaagaggcat atttagtcaa accattaata tatgctcacc 19860
aagtagtctc agaggtttgg taatattgct gttgtccaga tgaattaaac catcgggcag 19920
aataacacac cttggaatgt ggggaagtgc gtaggttaaa aattttggat attagacagg 19980
ttttagcccc ttctaagtcc tctagcatta atctgggctg cccccagtca cagtgagttt 20040
tgtcagtcaa tagattggtt agtccccatt tctctgtcct acagcagatg ggtgtctaag 20100
cagtttcatc aaccctggat ggttttgact gggcatgatt acctctgcct cccagggact 20160
ctttctgtgg catagagtct gatgctccag gcctttttgg ttttacaaag accagaatct 20220
tttaaagagt tttgggggcc tagtgagatg gttaagaaca ctgactgttc ttccaaaggt 20280
cctgagttca attcccagcg accacatggt gggtcacaac catccgtaat gagatctgac 20340
accctcttct ggtgtgtctg aagacagcta cagtgtagtg tacttagata taataataaa 20400
taaatctaaa agaattttct gtaggaagct tcggttttaa gttccaattt ttggaatgga 20460
aactcctctg ttttaagctg tggtacaggg tcagctctgg ggttcagaca gaaaccagaa 20520
ggatcctgtc cccaactgtt ctttggttcc agtgtcctga tggctctggg catgtccctc 20580
ttgggccagg aatttgagca gaaatggtgg tcttacctgt ccacactcac aggcatgtcc 20640
acactcctgg gagaccagtt ctctcctggt ggtatttggg tatggagcgc tgtggcacag 20700
gatcaagctc tgggtacagg cggccagtat ctcctctcgt ttgatcctga attgttaaat 20760
tctttattat caaataaacc attcttggcc aatcttgtgg ccacactgaa tgccagtagt 20820
aaggagccta tgaacatctg acatcactgt gcaacaataa aagacggcac catgttcaga 20880
cataaccaat ggaattggat tcatagatgt acacatgtgg agtgaagatc aatagtccag 20940
agccagaaga ggaccagagg ccccagcaac attgctccca gacctgctaa cactttggtt 21000
tcctccatac actgctgaac aagagagtac ttactccaga aagctatgag ctgggtgatg 21060
aggagagggc tcaggtttta aaagcacttc tctttctttc agaggacctg gattcagttc 21120
caagcaccca catggtggtt ctcaacagcc tgttactcca atcccagggg atttgacacc 21180
ctcttcagac tttctcaaac actaggcact cgtggggtac gcatacgtca ttcaggcaag 21240
ccctcataca cataaaatag aataagcaca tattttaaat gtgctactat tactgatgtt 21300
ttaagccagt taaattatgt gattttctta acagcaataa aatattgtag ataatgtcat 21360
catgctagct gcatcactct gactgtataa acccaagaga atttcactcc atctccttct 21420
gccaaaatat ttacaacact taagaaataa gggctgattt ttattttttt taacataaaa 21480
tatatgagac agttgtatcc tgtcagctaa aaatgttttc tctggcctgt gagttggctc 21540
agtagatgaa gacacttgct gccaagccta acaacctgaa ttcaactccc agaacccatg 21600
taatgggaag acagatggga ttcagagaaa ttgtcctctg atcattgtac ccatgccttg 21660
gtatatgagt atctgtgatg gtttgtatat gctcagtcca gggagtggca ctactagaag 21720
gtgtggtcct gttggagtag atgtatcact gtgggtgtgg gctttaataa cctcatccta 21780
gctgcctgga agccagtatt ctgctagcag ccttcatatg aagatacaga actctcagct 21840
gtgtttgcac catgcctgca tgaacgctgc catgctccca ccttgatgat aatggactga 21900
acctctgaac ctgtaagcca gccccgatta aatgttgtca cttataa 21947
<210> 270
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 270
ugucagguua guuagaugaa guuuuagagc uaugcu 36
<210> 271
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 271
gugucagguu aguuagauga guuuuagagc uaugcu 36
<210> 272
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 272
ccugacacau auacagacua guuuuagagc uaugcu 36
<210> 273
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 273
cugacacaua uacagacuaa guuuuagagc uaugcu 36
<210> 274
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 274
ccuuagucug uauauguguc guuuuagagc uaugcu 36
<210> 275
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 275
cauauacaga cuaagggacc guuuuagagc uaugcu 36
<210> 276
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 276
auauacagac uaagggacca guuuuagagc uaugcu 36
<210> 277
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 277
ucaaaguuug auaaauuccc guuuuagagc uaugcu 36
<210> 278
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 278
aaaauacaaa gauaaguaga guuuuagagc uaugcu 36
<210> 279
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 279
acucugugac uuuaaaaagu guuuuagagc uaugcu 36
<210> 280
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 280
gguucugugg gauauuaaua guuuuagagc uaugcu 36
<210> 281
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 281
acagagcaua uugguucugu guuuuagagc uaugcu 36
<210> 282
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 282
gacagagcau auugguucug guuuuagagc uaugcu 36
<210> 283
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 283
ugcaaaacga cagagcauau guuuuagagc uaugcu 36
<210> 284
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 284
gagcugggca uggaauaggc guuuuagagc uaugcu 36
<210> 285
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 285
acuggagcug ggcauggaau guuuuagagc uaugcu 36
<210> 286
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 286
cucauuacug gagcugggca guuuuagagc uaugcu 36
<210> 287
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 287
uuguucucau uacuggagcu guuuuagagc uaugcu 36
<210> 288
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 288
auuguucuca uuacuggagc guuuuagagc uaugcu 36
<210> 289
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 289
ggggagauug uucucauuac guuuuagagc uaugcu 36
<210> 290
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 290
gaggagaaaa ucuguggcug guuuuagagc uaugcu 36
<210> 291
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 291
agaggagaaa aucuguggcu guuuuagagc uaugcu 36
<210> 292
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 292
cagaggagaa aaucuguggc guuuuagagc uaugcu 36
<210> 293
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 293
uccucagagg agaaaaucug guuuuagagc uaugcu 36
<210> 294
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 294
ugaaguuuuu cauuccucag guuuuagagc uaugcu 36
<210> 295
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 295
cuucaccaac gacuccaagu guuuuagagc uaugcu 36
<210> 296
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 296
cuacuccuac uuggagucgu guuuuagagc uaugcu 36
<210> 297
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 297
cuccaaguag gaguagauga guuuuagagc uaugcu 36
<210> 298
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 298
caccaucauc uacuccuacu guuuuagagc uaugcu 36
<210> 299
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 299
ugauggugau cagaagcaga guuuuagagc uaugcu 36
<210> 300
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 300
ucagaagcag aaggauuucu guuuuagagc uaugcu 36
<210> 301
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 301
gauuucuagg augauguuca guuuuagagc uaugcu 36
<210> 302
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 302
uugcucuguc cucuuccuuc guuuuagagc uaugcu 36
<210> 303
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 303
aggacugaac cagaaggaag guuuuagagc uaugcu 36
<210> 304
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 304
uacacaagga cugaaccaga guuuuagagc uaugcu 36
<210> 305
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 305
uucaguccuu guguaguccu guuuuagagc uaugcu 36
<210> 306
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 306
ucaguccuug uguaguccua guuuuagagc uaugcu 36
<210> 307
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 307
guccuugugu aguccuaggg guuuuagagc uaugcu 36
<210> 308
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 308
cuuguguagu ccuagggagg guuuuagagc uaugcu 36
<210> 309
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 309
cuccucccua ggacuacaca guuuuagagc uaugcu 36
<210> 310
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 310
guagacagua ccuccucccu guuuuagagc uaugcu 36
<210> 311
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 311
uacugucuac acagagcucu guuuuagagc uaugcu 36
<210> 312
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 312
acugucuaca cagagcucua guuuuagagc uaugcu 36
<210> 313
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 313
ucuacacaga gcucuaggga guuuuagagc uaugcu 36
<210> 314
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 314
cuacacagag cucuagggaa guuuuagagc uaugcu 36
<210> 315
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 315
uacacagagc ucuagggaag guuuuagagc uaugcu 36
<210> 316
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 316
ggggugugcc caguuguuaa guuuuagagc uaugcu 36
<210> 317
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 317
gggugugccc aguuguuaau guuuuagagc uaugcu 36
<210> 318
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 318
ugguaguccc auuaacaacu guuuuagagc uaugcu 36
<210> 319
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 319
cugguagucc cauuaacaac guuuuagagc uaugcu 36
<210> 320
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 320
uuguuaaugg gacuaccaga guuuuagagc uaugcu 36
<210> 321
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 321
uaccagaugg aagccagcuu guuuuagagc uaugcu 36
<210> 322
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 322
uuccaaagcu ggcuuccauc guuuuagagc uaugcu 36
<210> 323
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 323
uggaagccag cuuuggaagc guuuuagagc uaugcu 36
<210> 324
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 324
acaaggccug cuuccaaagc guuuuagagc uaugcu 36
<210> 325
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 325
gccuuguuca cguguucuaa guuuuagagc uaugcu 36
<210> 326
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 326
ccuuguucac guguucuaau guuuuagagc uaugcu 36
<210> 327
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 327
cccauuagaa cacgugaaca guuuuagagc uaugcu 36
<210> 328
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 328
uuggcaucac uucauauuug guuuuagagc uaugcu 36
<210> 329
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 329
cuugugcucu uggcaucacu guuuuagagc uaugcu 36
<210> 330
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 330
agcacacucu cuugugcucu guuuuagagc uaugcu 36
<210> 331
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 331
gcacaagaga gugugcucuc guuuuagagc uaugcu 36
<210> 332
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 332
gcuuaaucuc acacauagaa guuuuagagc uaugcu 36
<210> 333
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 333
cuuaaucuca cacauagaaa guuuuagagc uaugcu 36
<210> 334
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 334
uuaaucucac acauagaaag guuuuagagc uaugcu 36
<210> 335
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 335
aggagugcug guuuaucaac guuuuagagc uaugcu 36
<210> 336
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 336
uucuuugaca gcaggagugc guuuuagagc uaugcu 36
<210> 337
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 337
acucugguuu cuuugacagc guuuuagagc uaugcu 36
<210> 338
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 338
accagaguug agaaaacccc guuuuagagc uaugcu 36
<210> 339
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 339
uccagggguu uucucaacuc guuuuagagc uaugcu 36
<210> 340
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 340
caguuauuaa augaauccag guuuuagagc uaugcu 36
<210> 341
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 341
gcaguuauua aaugaaucca guuuuagagc uaugcu 36
<210> 342
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 342
ggcaguuauu aaaugaaucc guuuuagagc uaugcu 36
<210> 343
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 343
uggaugguaa cagcuacauc guuuuagagc uaugcu 36
<210> 344
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 344
gcuguuacca uccacauccu guuuuagagc uaugcu 36
<210> 345
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 345
ucaagaacca aggaugugga guuuuagagc uaugcu 36
<210> 346
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 346
uccuucaaga accaaggaug guuuuagagc uaugcu 36
<210> 347
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 347
ugaguguccu ucaagaacca guuuuagagc uaugcu 36
<210> 348
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 348
uuuuauuuua uaacuacaag guuuuagagc uaugcu 36
<210> 349
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 349
uuguuuuuaa uaaaaacaag guuuuagagc uaugcu 36
<210> 350
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 350
uauuauagaa ugcuuuugca guuuuagagc uaugcu 36
<210> 351
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 351
caagauuagu cuugauguag guuuuagagc uaugcu 36
<210> 352
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 352
aagauuaguc uugauguagu guuuuagagc uaugcu 36
<210> 353
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 353
agucuugaug uagugggagu guuuuagagc uaugcu 36
<210> 354
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 354
uuuuucuauu aaaaaaaaaa guuuuagagc uaugcu 36
<210> 355
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 355
ucuauuaaaa aaaaaaaggc guuuuagagc uaugcu 36
<210> 356
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 356
cuauuaaaaa aaaaaaggcu guuuuagagc uaugcu 36
<210> 357
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 357
aaaaaaaaaa aggcugggca guuuuagagc uaugcu 36
<210> 358
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 358
aaaaaaaagg cugggcacgg guuuuagagc uaugcu 36
<210> 359
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 359
cacccguaau cccagcacuu guuuuagagc uaugcu 36
<210> 360
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 360
acccguaauc ccagcacuuu guuuuagagc uaugcu 36
<210> 361
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 361
cguaauccca gcacuuuggg guuuuagagc uaugcu 36
<210> 362
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 362
ucccaaagug cugggauuac guuuuagagc uaugcu 36
<210> 363
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 363
cucccaaagu gcugggauua guuuuagagc uaugcu 36
<210> 364
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 364
cccagcacuu ugggaggccg guuuuagagc uaugcu 36
<210> 365
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 365
ccucggccuc ccaaagugcu guuuuagagc uaugcu 36
<210> 366
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 366
gcacuuuggg aggccgaggc guuuuagagc uaugcu 36
<210> 367
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 367
cuuugggagg ccgaggcagg guuuuagagc uaugcu 36
<210> 368
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 368
gccgaggcag guggaucacg guuuuagagc uaugcu 36
<210> 369
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 369
accucgugau ccaccugccu guuuuagagc uaugcu 36
<210> 370
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 370
ggcaggugga ucacgagguc guuuuagagc uaugcu 36
<210> 371
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 371
ucaggagauc gagaccaucu guuuuagagc uaugcu 36
<210> 372
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 372
cgagaccauc uuggcuaaca guuuuagagc uaugcu 36
<210> 373
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 373
uuucaccaug uuagccaaga guuuuagagc uaugcu 36
<210> 374
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 374
uuguauuuuu uguagagacg guuuuagagc uaugcu 36
<210> 375
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 375
uuuguauuuu uuguagagac guuuuagagc uaugcu 36
<210> 376
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 376
uuuuguauuu uuuguagaga guuuuagagc uaugcu 36
<210> 377
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 377
aaaaaauaca aaaaauuagc guuuuagagc uaugcu 36
<210> 378
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 378
aaaaauacaa aaaauuagcc guuuuagagc uaugcu 36
<210> 379
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 379
uacaaaaaau uagccgggug guuuuagagc uaugcu 36
<210> 380
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 380
aaaaaauuag ccgggugugg guuuuagagc uaugcu 36
<210> 381
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 381
aaauuagccg gguguggugg guuuuagagc uaugcu 36
<210> 382
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 382
aauuagccgg gugugguggc guuuuagagc uaugcu 36
<210> 383
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 383
caggcgcccg ccaccacacc guuuuagagc uaugcu 36
<210> 384
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 384
gccuguaguc ccagcuacuc guuuuagagc uaugcu 36
<210> 385
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 385
uguaguccca gcuacucagg guuuuagagc uaugcu 36
<210> 386
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 386
uccugaguag cugggacuac guuuuagagc uaugcu 36
<210> 387
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 387
cccagcuacu caggaggcug guuuuagagc uaugcu 36
<210> 388
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 388
ccucagccuc cugaguagcu guuuuagagc uaugcu 36
<210> 389
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 389
gccucagccu ccugaguagc guuuuagagc uaugcu 36
<210> 390
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 390
aggaggcuga ggcaggagaa guuuuagagc uaugcu 36
<210> 391
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 391
gcaggagaau ggcgugaacc guuuuagagc uaugcu 36
<210> 392
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 392
caggagaaug gcgugaaccc guuuuagagc uaugcu 36
<210> 393
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 393
gagaauggcg ugaacccggg guuuuagagc uaugcu 36
<210> 394
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 394
aauggcguga acccgggagg guuuuagagc uaugcu 36
<210> 395
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 395
cacugcaagc uccaccuccc guuuuagagc uaugcu 36
<210> 396
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 396
ucacugcaag cuccaccucc guuuuagagc uaugcu 36
<210> 397
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 397
cauaccacug cacuccagcc guuuuagagc uaugcu 36
<210> 398
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 398
auaccacugc acuccagccu guuuuagagc uaugcu 36
<210> 399
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 399
ucgcccaggc uggagugcag guuuuagagc uaugcu 36
<210> 400
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 400
ucucacucuu ucgcccaggc guuuuagagc uaugcu 36
<210> 401
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 401
ggagucucac ucuuucgccc guuuuagagc uaugcu 36
<210> 402
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 402
uguuuuuugu uuuuuugaga guuuuagagc uaugcu 36
<210> 403
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 403
aggaagaaag aaagguuuuu guuuuagagc uaugcu 36
<210> 404
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 404
agaagaaaag gaagaaagaa guuuuagagc uaugcu 36
<210> 405
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 405
cuuucuuccu uuucuucucu guuuuagagc uaugcu 36
<210> 406
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 406
uuucuuccuu uucuucucuu guuuuagagc uaugcu 36
<210> 407
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 407
aauggaccca agagaagaaa guuuuagagc uaugcu 36
<210> 408
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 408
ggcuauuaca uaagaaacaa guuuuagagc uaugcu 36
<210> 409
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 409
cacaggaaaa ggaacuguac guuuuagagc uaugcu 36
<210> 410
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 410
auuaaagcua acacaggaaa guuuuagagc uaugcu 36
<210> 411
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 411
ucaaaaauua aagcuaacac guuuuagagc uaugcu 36
<210> 412
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 412
uaaaauuguc uaaacaucuc guuuuagagc uaugcu 36
<210> 413
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 413
agagauguuu agacaauuuu guuuuagagc uaugcu 36
<210> 414
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 414
ucuaaacauc ucugggacca guuuuagagc uaugcu 36
<210> 415
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 415
uuuaugcuuu cauauauccu guuuuagagc uaugcu 36
<210> 416
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 416
agcauaaauu acaaagaaaa guuuuagagc uaugcu 36
<210> 417
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 417
uacaaagaaa aagguuauca guuuuagagc uaugcu 36
<210> 418
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 418
acaaagaaaa agguuaucau guuuuagagc uaugcu 36
<210> 419
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 419
caaagaaaaa gguuaucaug guuuuagagc uaugcu 36
<210> 420
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 420
ucugagauuu aaaauagagu guuuuagagc uaugcu 36
<210> 421
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 421
cuuauaagau acauuaugaa guuuuagagc uaugcu 36
<210> 422
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 422
uaucuuauaa gacuauaaaa guuuuagagc uaugcu 36
<210> 423
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 423
aucuuauaag acuauaaaaa guuuuagagc uaugcu 36
<210> 424
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 424
uuauaagacu auaaaaaggg guuuuagagc uaugcu 36
<210> 425
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 425
uaaaaaggga ggaaauauag guuuuagagc uaugcu 36
<210> 426
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 426
aaaaagggag gaaauauaga guuuuagagc uaugcu 36
<210> 427
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 427
aaauauagag gguccacuuu guuuuagagc uaugcu 36
<210> 428
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 428
uauagagggu ccacuuuugg guuuuagagc uaugcu 36
<210> 429
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 429
acucugaagu ccaccaaaag guuuuagagc uaugcu 36
<210> 430
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 430
agaauagagu ugcaccguuu guuuuagagc uaugcu 36
<210> 431
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 431
aaaacggugc aacucuauuc guuuuagagc uaugcu 36
<210> 432
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 432
ccguuuuggg cuaaugaaaa guuuuagagc uaugcu 36
<210> 433
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 433
ccuuuuucau uagcccaaaa guuuuagagc uaugcu 36
<210> 434
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 434
ugggcuaaug aaaaaggaag guuuuagagc uaugcu 36
<210> 435
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 435
uaaugaaaaa ggaagaggcu guuuuagagc uaugcu 36
<210> 436
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 436
aaugaaaaag gaagaggcua guuuuagagc uaugcu 36
<210> 437
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 437
cugaaucuua aaauaugucc guuuuagagc uaugcu 36
<210> 438
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 438
caggcagcuu uaucucaacc guuuuagagc uaugcu 36
<210> 439
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 439
cuaagagauc aaguuucagc guuuuagagc uaugcu 36
<210> 440
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 440
guguucuugu ugauauucug guuuuagagc uaugcu 36
<210> 441
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 441
cuuguugaua uucuguggca guuuuagagc uaugcu 36
<210> 442
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 442
ucuguggcau ggcuacagau guuuuagagc uaugcu 36
<210> 443
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 443
agaacuuauu uacacaggga guuuuagagc uaugcu 36
<210> 444
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 444
aaagagaacu uauuuacaca guuuuagagc uaugcu 36
<210> 445
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 445
caaagagaac uuauuuacac guuuuagagc uaugcu 36
<210> 446
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 446
uucucuuugu auuuacuuuu guuuuagagc uaugcu 36
<210> 447
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 447
ucucuuugua uuuacuuuua guuuuagagc uaugcu 36
<210> 448
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 448
cuuuguauuu acuuuuaggg guuuuagagc uaugcu 36
<210> 449
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 449
agcuuuuguc caccuuuaaa guuuuagagc uaugcu 36
<210> 450
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 450
uuuauuuuuc cauuuaaagg guuuuagagc uaugcu 36
<210> 451
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 451
uauuuuauuu uuccauuuaa guuuuagagc uaugcu 36
<210> 452
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 452
cuuacauaaa cauacuuaaa guuuuagagc uaugcu 36
<210> 453
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 453
uaagcacaga aguuuuuaag guuuuagagc uaugcu 36
<210> 454
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 454
aaguuuuuaa gaggcaugaa guuuuagagc uaugcu 36
<210> 455
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 455
auauuuacgu aguuuuucau guuuuagagc uaugcu 36
<210> 456
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 456
cguaaauauu cuugagaaac guuuuagagc uaugcu 36
<210> 457
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 457
uucuugagaa acaggaagac guuuuagagc uaugcu 36
<210> 458
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 458
uaauauuaaa aacauugguu guuuuagagc uaugcu 36
<210> 459
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 459
ccaauguuuu uaauauuauc guuuuagagc uaugcu 36
<210> 460
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 460
ccugauaaua uuaaaaacau guuuuagagc uaugcu 36
<210> 461
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 461
cauuaucaug cauacaucuc guuuuagagc uaugcu 36
<210> 462
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 462
aucaugcaua caucucuggc guuuuagagc uaugcu 36
<210> 463
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 463
uucauuucau uuugauuuug guuuuagagc uaugcu 36
<210> 464
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 464
auucaauuug aagcaguggu guuuuagagc uaugcu 36
<210> 465
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 465
gaauauucaa uuugaagcag guuuuagagc uaugcu 36
<210> 466
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 466
cauacgauuu aaaaucgcug guuuuagagc uaugcu 36
<210> 467
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 467
aaaaucgcug aggcgcguuc guuuuagagc uaugcu 36
<210> 468
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 468
uuuuuuuuuc uuuuuuguac guuuuagagc uaugcu 36
<210> 469
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 469
cuguugucaa agauuuuaaa guuuuagagc uaugcu 36
<210> 470
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 470
ugacaacaga guucuguuuu guuuuagagc uaugcu 36
<210> 471
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 471
agaauacgcu gagaguuauc guuuuagagc uaugcu 36
<210> 472
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 472
gcaagagaag aaaagaacgg guuuuagagc uaugcu 36
<210> 473
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 473
guugcaagag aagaaaagaa guuuuagagc uaugcu 36
<210> 474
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 474
augcacacgu aaaagagagg guuuuagagc uaugcu 36
<210> 475
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 475
aagaugcaca cguaaaagag guuuuagagc uaugcu 36
<210> 476
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 476
aucaugcaua caucucuggc guuuuagagc uaugcu 36
<210> 477
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 477
uucauuucau uuugauuuug guuuuagagc uaugcu 36
<210> 478
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 478
auucaauuug aagcaguggu guuuuagagc uaugcu 36
<210> 479
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 479
gaauauucaa uuugaagcag guuuuagagc uaugcu 36
<210> 480
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 480
cauacgauuu aaaaucgcug guuuuagagc uaugcu 36
<210> 481
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 481
aaaaucgcug aggcgcguuc guuuuagagc uaugcu 36
<210> 482
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 482
uuuuuuuuuc uuuuuuguac guuuuagagc uaugcu 36
<210> 483
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 483
cuguugucaa agauuuuaaa guuuuagagc uaugcu 36
<210> 484
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 484
ugacaacaga guucuguuuu guuuuagagc uaugcu 36
<210> 485
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 485
agaauacgcu gagaguuauc guuuuagagc uaugcu 36
<210> 486
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 486
gcaagagaag aaaagaacgg guuuuagagc uaugcu 36
<210> 487
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 487
guugcaagag aagaaaagaa guuuuagagc uaugcu 36
<210> 488
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 488
augcacacgu aaaagagagg guuuuagagc uaugcu 36
<210> 489
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 489
aagaugcaca cguaaaagag guuuuagagc uaugcu 36
<210> 490
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 490
ggcagaccgu ucucaucacg guuuuagagc uaugcu 36
<210> 491
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 491
cuuuaccagu gacuccaggu guuuuagagc uaugcu 36
<210> 492
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 492
gucacagauu uccuucuccg guuuuagagc uaugcu 36
<210> 493
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 493
agaugaugac gcccaccaga guuuuagagc uaugcu 36
<210> 494
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 494
ggagaaggaa aucugugacc guuuuagagc uaugcu 36
<210> 495
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 495
ugcgaggaac uuacuuuucc guuuuagagc uaugcu 36
<210> 496
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 496
agagaaauau ugauauagga guuuuagagc uaugcu 36
<210> 497
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 497
uaucaauauu ucucugaucc guuuuagagc uaugcu 36
<210> 498
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 498
aucgcuuuua aggcacgcuc guuuuagagc uaugcu 36
<210> 499
<211> 36
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 499
uauacgacug aucgcuuuua guuuuagagc uaugcu 36
<210> 500
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 500
ugucagguua guuagaugaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 501
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 501
gugucagguu aguuagauga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 502
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 502
ccugacacau auacagacua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 503
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 503
cugacacaua uacagacuaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 504
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 504
ccuuagucug uauauguguc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 505
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 505
cauauacaga cuaagggacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 506
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 506
auauacagac uaagggacca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 507
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 507
ucaaaguuug auaaauuccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 508
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 508
aaaauacaaa gauaaguaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 509
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 509
acucugugac uuuaaaaagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 510
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 510
gguucugugg gauauuaaua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 511
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 511
acagagcaua uugguucugu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 512
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 512
gacagagcau auugguucug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 513
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 513
ugcaaaacga cagagcauau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 514
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 514
gagcugggca uggaauaggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 515
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 515
acuggagcug ggcauggaau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 516
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 516
cucauuacug gagcugggca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 517
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 517
uuguucucau uacuggagcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 518
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 518
auuguucuca uuacuggagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 519
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 519
ggggagauug uucucauuac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 520
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 520
gaggagaaaa ucuguggcug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 521
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 521
agaggagaaa aucuguggcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 522
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 522
cagaggagaa aaucuguggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 523
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 523
uccucagagg agaaaaucug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 524
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 524
ugaaguuuuu cauuccucag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 525
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 525
cuucaccaac gacuccaagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 526
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 526
cuacuccuac uuggagucgu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 527
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 527
cuccaaguag gaguagauga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 528
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 528
caccaucauc uacuccuacu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 529
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 529
ugauggugau cagaagcaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 530
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 530
ucagaagcag aaggauuucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 531
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 531
gauuucuagg augauguuca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 532
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 532
uugcucuguc cucuuccuuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 533
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 533
aggacugaac cagaaggaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 534
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 534
uacacaagga cugaaccaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 535
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 535
uucaguccuu guguaguccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 536
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 536
ucaguccuug uguaguccua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 537
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 537
guccuugugu aguccuaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 538
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 538
cuuguguagu ccuagggagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 539
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 539
cuccucccua ggacuacaca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 540
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 540
guagacagua ccuccucccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 541
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 541
uacugucuac acagagcucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 542
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 542
acugucuaca cagagcucua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 543
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 543
ucuacacaga gcucuaggga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 544
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 544
cuacacagag cucuagggaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 545
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 545
uacacagagc ucuagggaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 546
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 546
ggggugugcc caguuguuaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 547
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 547
gggugugccc aguuguuaau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 548
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 548
ugguaguccc auuaacaacu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 549
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 549
cugguagucc cauuaacaac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 550
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 550
uuguuaaugg gacuaccaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 551
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 551
uaccagaugg aagccagcuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 552
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 552
uuccaaagcu ggcuuccauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 553
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 553
uggaagccag cuuuggaagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 554
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 554
acaaggccug cuuccaaagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 555
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 555
gccuuguuca cguguucuaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 556
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 556
ccuuguucac guguucuaau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 557
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 557
cccauuagaa cacgugaaca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 558
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 558
uuggcaucac uucauauuug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 559
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 559
cuugugcucu uggcaucacu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 560
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 560
agcacacucu cuugugcucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 561
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 561
gcacaagaga gugugcucuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 562
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 562
gcuuaaucuc acacauagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 563
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 563
cuuaaucuca cacauagaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 564
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 564
uuaaucucac acauagaaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 565
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 565
aggagugcug guuuaucaac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 566
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 566
uucuuugaca gcaggagugc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 567
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 567
acucugguuu cuuugacagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 568
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 568
accagaguug agaaaacccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 569
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 569
uccagggguu uucucaacuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 570
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 570
caguuauuaa augaauccag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 571
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 571
gcaguuauua aaugaaucca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 572
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 572
ggcaguuauu aaaugaaucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 573
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 573
uggaugguaa cagcuacauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 574
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 574
gcuguuacca uccacauccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 575
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 575
ucaagaacca aggaugugga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 576
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 576
uccuucaaga accaaggaug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 577
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 577
ugaguguccu ucaagaacca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 578
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 578
uuuuauuuua uaacuacaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 579
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 579
uuguuuuuaa uaaaaacaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 580
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 580
uauuauagaa ugcuuuugca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 581
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 581
caagauuagu cuugauguag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 582
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 582
aagauuaguc uugauguagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 583
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 583
agucuugaug uagugggagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 584
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 584
uuuuucuauu aaaaaaaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 585
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 585
ucuauuaaaa aaaaaaaggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 586
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 586
cuauuaaaaa aaaaaaggcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 587
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 587
aaaaaaaaaa aggcugggca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 588
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 588
aaaaaaaagg cugggcacgg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 589
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 589
cacccguaau cccagcacuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 590
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 590
acccguaauc ccagcacuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 591
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 591
cguaauccca gcacuuuggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 592
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 592
ucccaaagug cugggauuac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 593
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 593
cucccaaagu gcugggauua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 594
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 594
cccagcacuu ugggaggccg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 595
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 595
ccucggccuc ccaaagugcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 596
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 596
gcacuuuggg aggccgaggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 597
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 597
cuuugggagg ccgaggcagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 598
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 598
gccgaggcag guggaucacg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 599
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 599
accucgugau ccaccugccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 600
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 600
ggcaggugga ucacgagguc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 601
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 601
ucaggagauc gagaccaucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 602
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 602
cgagaccauc uuggcuaaca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 603
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 603
uuucaccaug uuagccaaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 604
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 604
uuguauuuuu uguagagacg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 605
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 605
uuuguauuuu uuguagagac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 606
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 606
uuuuguauuu uuuguagaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 607
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 607
aaaaaauaca aaaaauuagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 608
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 608
aaaaauacaa aaaauuagcc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 609
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 609
uacaaaaaau uagccgggug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 610
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 610
aaaaaauuag ccgggugugg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 611
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 611
aaauuagccg gguguggugg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 612
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 612
aauuagccgg gugugguggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 613
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 613
caggcgcccg ccaccacacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 614
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 614
gccuguaguc ccagcuacuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 615
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 615
uguaguccca gcuacucagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 616
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 616
uccugaguag cugggacuac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 617
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 617
cccagcuacu caggaggcug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 618
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 618
ccucagccuc cugaguagcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 619
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 619
gccucagccu ccugaguagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 620
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 620
aggaggcuga ggcaggagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 621
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 621
gcaggagaau ggcgugaacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 622
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 622
caggagaaug gcgugaaccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 623
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 623
gagaauggcg ugaacccggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 624
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 624
aauggcguga acccgggagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 625
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 625
cacugcaagc uccaccuccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 626
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 626
ucacugcaag cuccaccucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 627
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 627
cauaccacug cacuccagcc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 628
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 628
auaccacugc acuccagccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 629
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 629
ucgcccaggc uggagugcag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 630
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 630
ucucacucuu ucgcccaggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 631
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 631
ggagucucac ucuuucgccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 632
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 632
uguuuuuugu uuuuuugaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 633
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 633
aggaagaaag aaagguuuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 634
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 634
agaagaaaag gaagaaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 635
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 635
cuuucuuccu uuucuucucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 636
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 636
uuucuuccuu uucuucucuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 637
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 637
aauggaccca agagaagaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 638
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 638
ggcuauuaca uaagaaacaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 639
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 639
cacaggaaaa ggaacuguac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 640
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 640
auuaaagcua acacaggaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 641
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 641
ucaaaaauua aagcuaacac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 642
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 642
uaaaauuguc uaaacaucuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 643
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 643
agagauguuu agacaauuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 644
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 644
ucuaaacauc ucugggacca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 645
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 645
uuuaugcuuu cauauauccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 646
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 646
agcauaaauu acaaagaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 647
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 647
uacaaagaaa aagguuauca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 648
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 648
acaaagaaaa agguuaucau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 649
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 649
caaagaaaaa gguuaucaug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 650
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 650
ucugagauuu aaaauagagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 651
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 651
cuuauaagau acauuaugaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 652
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 652
uaucuuauaa gacuauaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 653
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 653
aucuuauaag acuauaaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 654
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 654
uuauaagacu auaaaaaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 655
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 655
uaaaaaggga ggaaauauag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 656
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 656
aaaaagggag gaaauauaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 657
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 657
aaauauagag gguccacuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 658
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 658
uauagagggu ccacuuuugg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 659
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 659
acucugaagu ccaccaaaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 660
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 660
agaauagagu ugcaccguuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 661
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 661
aaaacggugc aacucuauuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 662
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 662
ccguuuuggg cuaaugaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 663
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 663
ccuuuuucau uagcccaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 664
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 664
ugggcuaaug aaaaaggaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 665
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 665
uaaugaaaaa ggaagaggcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 666
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 666
aaugaaaaag gaagaggcua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 667
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 667
cugaaucuua aaauaugucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 668
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 668
caggcagcuu uaucucaacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 669
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 669
cuaagagauc aaguuucagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 670
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 670
guguucuugu ugauauucug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 671
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 671
cuuguugaua uucuguggca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 672
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 672
ucuguggcau ggcuacagau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 673
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 673
agaacuuauu uacacaggga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 674
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 674
aaagagaacu uauuuacaca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 675
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 675
caaagagaac uuauuuacac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 676
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 676
uucucuuugu auuuacuuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 677
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 677
ucucuuugua uuuacuuuua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 678
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 678
cuuuguauuu acuuuuaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 679
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 679
agcuuuuguc caccuuuaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 680
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 680
uuuauuuuuc cauuuaaagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 681
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 681
uauuuuauuu uuccauuuaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 682
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 682
cuuacauaaa cauacuuaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 683
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 683
uaagcacaga aguuuuuaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 684
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 684
aaguuuuuaa gaggcaugaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 685
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 685
auauuuacgu aguuuuucau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 686
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 686
cguaaauauu cuugagaaac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 687
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 687
uucuugagaa acaggaagac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 688
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 688
uaauauuaaa aacauugguu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 689
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 689
ccaauguuuu uaauauuauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 690
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 690
ccugauaaua uuaaaaacau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 691
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 691
cauuaucaug cauacaucuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 692
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 692
aucaugcaua caucucuggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 693
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 693
uucauuucau uuugauuuug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 694
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 694
auucaauuug aagcaguggu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 695
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 695
gaauauucaa uuugaagcag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 696
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 696
cauacgauuu aaaaucgcug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 697
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 697
aaaaucgcug aggcgcguuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 698
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 698
uuuuuuuuuc uuuuuuguac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 699
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 699
cuguugucaa agauuuuaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 700
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 700
ugacaacaga guucuguuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 701
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 701
agaauacgcu gagaguuauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 702
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 702
gcaagagaag aaaagaacgg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 703
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 703
guugcaagag aagaaaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 704
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 704
augcacacgu aaaagagagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 705
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 705
aagaugcaca cguaaaagag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 706
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 706
aucaugcaua caucucuggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 707
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 707
uucauuucau uuugauuuug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 708
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 708
auucaauuug aagcaguggu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 709
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 709
gaauauucaa uuugaagcag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 710
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 710
cauacgauuu aaaaucgcug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 711
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 711
aaaaucgcug aggcgcguuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 712
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 712
uuuuuuuuuc uuuuuuguac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 713
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 713
cuguugucaa agauuuuaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 714
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 714
ugacaacaga guucuguuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 715
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 715
agaauacgcu gagaguuauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 716
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 716
gcaagagaag aaaagaacgg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 717
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 717
guugcaagag aagaaaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 718
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 718
augcacacgu aaaagagagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 719
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 719
aagaugcaca cguaaaagag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 720
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 720
ggcagaccgu ucucaucacg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 721
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 721
cuuuaccagu gacuccaggu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 722
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 722
gucacagauu uccuucuccg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 723
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 723
agaugaugac gcccaccaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 724
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 724
ggagaaggaa aucugugacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 725
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 725
ugcgaggaac uuacuuuucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 726
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 726
agagaaauau ugauauagga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 727
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 727
uaucaauauu ucucugaucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 728
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 728
aucgcuuuua aggcacgcuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 729
<211> 97
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 729
uauacgacug aucgcuuuua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugcu 97
<210> 730
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 730
ugucagguua guuagaugaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 731
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 731
gugucagguu aguuagauga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 732
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 732
ccugacacau auacagacua guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 733
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 733
cugacacaua uacagacuaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 734
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 734
ccuuagucug uauauguguc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 735
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 735
cauauacaga cuaagggacc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 736
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 736
auauacagac uaagggacca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 737
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 737
ucaaaguuug auaaauuccc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 738
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 738
aaaauacaaa gauaaguaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 739
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 739
acucugugac uuuaaaaagu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 740
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 740
gguucugugg gauauuaaua guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 741
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 741
acagagcaua uugguucugu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 742
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 742
gacagagcau auugguucug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 743
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 743
ugcaaaacga cagagcauau guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 744
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 744
gagcugggca uggaauaggc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 745
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 745
acuggagcug ggcauggaau guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 746
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 746
cucauuacug gagcugggca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 747
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 747
uuguucucau uacuggagcu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 748
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 748
auuguucuca uuacuggagc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 749
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 749
ggggagauug uucucauuac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 750
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 750
gaggagaaaa ucuguggcug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 751
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 751
agaggagaaa aucuguggcu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 752
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 752
cagaggagaa aaucuguggc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 753
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 753
uccucagagg agaaaaucug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 754
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 754
ugaaguuuuu cauuccucag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 755
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 755
cuucaccaac gacuccaagu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 756
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 756
cuacuccuac uuggagucgu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 757
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 757
cuccaaguag gaguagauga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 758
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 758
caccaucauc uacuccuacu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 759
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 759
ugauggugau cagaagcaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 760
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 760
ucagaagcag aaggauuucu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 761
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 761
gauuucuagg augauguuca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 762
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 762
uugcucuguc cucuuccuuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 763
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 763
aggacugaac cagaaggaag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 764
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 764
uacacaagga cugaaccaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 765
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 765
uucaguccuu guguaguccu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 766
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 766
ucaguccuug uguaguccua guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 767
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 767
guccuugugu aguccuaggg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 768
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 768
cuuguguagu ccuagggagg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 769
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 769
cuccucccua ggacuacaca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 770
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 770
guagacagua ccuccucccu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 771
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 771
uacugucuac acagagcucu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 772
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 772
acugucuaca cagagcucua guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 773
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 773
ucuacacaga gcucuaggga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 774
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 774
cuacacagag cucuagggaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 775
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 775
uacacagagc ucuagggaag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 776
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 776
ggggugugcc caguuguuaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 777
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 777
gggugugccc aguuguuaau guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 778
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 778
ugguaguccc auuaacaacu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 779
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 779
cugguagucc cauuaacaac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 780
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 780
uuguuaaugg gacuaccaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 781
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 781
uaccagaugg aagccagcuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 782
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 782
uuccaaagcu ggcuuccauc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 783
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 783
uggaagccag cuuuggaagc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 784
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 784
acaaggccug cuuccaaagc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 785
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 785
gccuuguuca cguguucuaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 786
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 786
ccuuguucac guguucuaau guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 787
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 787
cccauuagaa cacgugaaca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 788
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 788
uuggcaucac uucauauuug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 789
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 789
cuugugcucu uggcaucacu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 790
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 790
agcacacucu cuugugcucu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 791
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 791
gcacaagaga gugugcucuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 792
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 792
gcuuaaucuc acacauagaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 793
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 793
cuuaaucuca cacauagaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 794
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 794
uuaaucucac acauagaaag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 795
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 795
aggagugcug guuuaucaac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 796
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 796
uucuuugaca gcaggagugc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 797
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 797
acucugguuu cuuugacagc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 798
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 798
accagaguug agaaaacccc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 799
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 799
uccagggguu uucucaacuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 800
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 800
caguuauuaa augaauccag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 801
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 801
gcaguuauua aaugaaucca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 802
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 802
ggcaguuauu aaaugaaucc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 803
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 803
uggaugguaa cagcuacauc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 804
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 804
gcuguuacca uccacauccu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 805
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 805
ucaagaacca aggaugugga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 806
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 806
uccuucaaga accaaggaug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 807
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 807
ugaguguccu ucaagaacca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 808
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 808
uuuuauuuua uaacuacaag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 809
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 809
uuguuuuuaa uaaaaacaag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 810
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 810
uauuauagaa ugcuuuugca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 811
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 811
caagauuagu cuugauguag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 812
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 812
aagauuaguc uugauguagu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 813
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 813
agucuugaug uagugggagu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 814
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 814
uuuuucuauu aaaaaaaaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 815
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 815
ucuauuaaaa aaaaaaaggc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 816
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 816
cuauuaaaaa aaaaaaggcu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 817
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 817
aaaaaaaaaa aggcugggca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 818
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 818
aaaaaaaagg cugggcacgg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 819
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 819
cacccguaau cccagcacuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 820
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 820
acccguaauc ccagcacuuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 821
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 821
cguaauccca gcacuuuggg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 822
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 822
ucccaaagug cugggauuac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 823
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 823
cucccaaagu gcugggauua guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 824
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 824
cccagcacuu ugggaggccg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 825
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 825
ccucggccuc ccaaagugcu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 826
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 826
gcacuuuggg aggccgaggc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 827
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 827
cuuugggagg ccgaggcagg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 828
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 828
gccgaggcag guggaucacg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 829
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 829
accucgugau ccaccugccu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 830
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 830
ggcaggugga ucacgagguc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 831
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 831
ucaggagauc gagaccaucu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 832
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 832
cgagaccauc uuggcuaaca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 833
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 833
uuucaccaug uuagccaaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 834
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 834
uuguauuuuu uguagagacg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 835
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 835
uuuguauuuu uuguagagac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 836
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 836
uuuuguauuu uuuguagaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 837
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 837
aaaaaauaca aaaaauuagc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 838
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 838
aaaaauacaa aaaauuagcc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 839
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 839
uacaaaaaau uagccgggug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 840
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 840
aaaaaauuag ccgggugugg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 841
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 841
aaauuagccg gguguggugg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 842
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 842
aauuagccgg gugugguggc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 843
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 843
caggcgcccg ccaccacacc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 844
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 844
gccuguaguc ccagcuacuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 845
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 845
uguaguccca gcuacucagg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 846
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 846
uccugaguag cugggacuac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 847
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 847
cccagcuacu caggaggcug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 848
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 848
ccucagccuc cugaguagcu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 849
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 849
gccucagccu ccugaguagc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 850
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 850
aggaggcuga ggcaggagaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 851
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 851
gcaggagaau ggcgugaacc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 852
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 852
caggagaaug gcgugaaccc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 853
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 853
gagaauggcg ugaacccggg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 854
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 854
aauggcguga acccgggagg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 855
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 855
cacugcaagc uccaccuccc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 856
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 856
ucacugcaag cuccaccucc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 857
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 857
cauaccacug cacuccagcc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 858
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 858
auaccacugc acuccagccu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 859
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 859
ucgcccaggc uggagugcag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 860
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 860
ucucacucuu ucgcccaggc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 861
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 861
ggagucucac ucuuucgccc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 862
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 862
uguuuuuugu uuuuuugaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 863
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 863
aggaagaaag aaagguuuuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 864
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 864
agaagaaaag gaagaaagaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 865
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 865
cuuucuuccu uuucuucucu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 866
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 866
uuucuuccuu uucuucucuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 867
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 867
aauggaccca agagaagaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 868
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 868
ggcuauuaca uaagaaacaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 869
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 869
cacaggaaaa ggaacuguac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 870
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 870
auuaaagcua acacaggaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 871
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 871
ucaaaaauua aagcuaacac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 872
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 872
uaaaauuguc uaaacaucuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 873
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 873
agagauguuu agacaauuuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 874
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 874
ucuaaacauc ucugggacca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 875
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 875
uuuaugcuuu cauauauccu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 876
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 876
agcauaaauu acaaagaaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 877
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 877
uacaaagaaa aagguuauca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 878
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 878
acaaagaaaa agguuaucau guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 879
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 879
caaagaaaaa gguuaucaug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 880
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 880
ucugagauuu aaaauagagu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 881
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 881
cuuauaagau acauuaugaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 882
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 882
uaucuuauaa gacuauaaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 883
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 883
aucuuauaag acuauaaaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 884
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 884
uuauaagacu auaaaaaggg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 885
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 885
uaaaaaggga ggaaauauag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 886
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 886
aaaaagggag gaaauauaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 887
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 887
aaauauagag gguccacuuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 888
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 888
uauagagggu ccacuuuugg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 889
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 889
acucugaagu ccaccaaaag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 890
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 890
agaauagagu ugcaccguuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 891
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 891
aaaacggugc aacucuauuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 892
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 892
ccguuuuggg cuaaugaaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 893
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 893
ccuuuuucau uagcccaaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 894
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 894
ugggcuaaug aaaaaggaag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 895
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 895
uaaugaaaaa ggaagaggcu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 896
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 896
aaugaaaaag gaagaggcua guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 897
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 897
cugaaucuua aaauaugucc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 898
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 898
caggcagcuu uaucucaacc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 899
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 899
cuaagagauc aaguuucagc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 900
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 900
guguucuugu ugauauucug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 901
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 901
cuuguugaua uucuguggca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 902
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 902
ucuguggcau ggcuacagau guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 903
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 903
agaacuuauu uacacaggga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 904
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 904
aaagagaacu uauuuacaca guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 905
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 905
caaagagaac uuauuuacac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 906
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 906
uucucuuugu auuuacuuuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 907
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 907
ucucuuugua uuuacuuuua guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 908
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 908
cuuuguauuu acuuuuaggg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 909
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 909
agcuuuuguc caccuuuaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 910
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 910
uuuauuuuuc cauuuaaagg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 911
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 911
uauuuuauuu uuccauuuaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 912
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 912
cuuacauaaa cauacuuaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 913
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 913
uaagcacaga aguuuuuaag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 914
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 914
aaguuuuuaa gaggcaugaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 915
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 915
auauuuacgu aguuuuucau guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 916
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 916
cguaaauauu cuugagaaac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 917
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 917
uucuugagaa acaggaagac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 918
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 918
uaauauuaaa aacauugguu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 919
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 919
ccaauguuuu uaauauuauc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 920
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 920
ccugauaaua uuaaaaacau guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 921
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 921
cauuaucaug cauacaucuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 922
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 922
aucaugcaua caucucuggc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 923
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 923
uucauuucau uuugauuuug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 924
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 924
auucaauuug aagcaguggu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 925
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 925
gaauauucaa uuugaagcag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 926
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 926
cauacgauuu aaaaucgcug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 927
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 927
aaaaucgcug aggcgcguuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 928
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 928
uuuuuuuuuc uuuuuuguac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 929
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 929
cuguugucaa agauuuuaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 930
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 930
ugacaacaga guucuguuuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 931
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 931
agaauacgcu gagaguuauc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 932
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 932
gcaagagaag aaaagaacgg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 933
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 933
guugcaagag aagaaaagaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 934
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 934
augcacacgu aaaagagagg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 935
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 935
aagaugcaca cguaaaagag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 936
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 936
aucaugcaua caucucuggc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 937
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 937
uucauuucau uuugauuuug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 938
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 938
auucaauuug aagcaguggu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 939
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 939
gaauauucaa uuugaagcag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 940
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 940
cauacgauuu aaaaucgcug guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 941
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 941
aaaaucgcug aggcgcguuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 942
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 942
uuuuuuuuuc uuuuuuguac guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 943
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 943
cuguugucaa agauuuuaaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 944
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 944
ugacaacaga guucuguuuu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 945
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 945
agaauacgcu gagaguuauc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 946
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 946
gcaagagaag aaaagaacgg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 947
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 947
guugcaagag aagaaaagaa guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 948
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 948
augcacacgu aaaagagagg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 949
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 949
aagaugcaca cguaaaagag guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 950
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 950
ggcagaccgu ucucaucacg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 951
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 951
cuuuaccagu gacuccaggu guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 952
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 952
gucacagauu uccuucuccg guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 953
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 953
agaugaugac gcccaccaga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 954
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 954
ggagaaggaa aucugugacc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 955
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 955
ugcgaggaac uuacuuuucc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 956
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 956
agagaaauau ugauauagga guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 957
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 957
uaucaauauu ucucugaucc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 958
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 958
aucgcuuuua aggcacgcuc guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 959
<211> 102
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 959
uauacgacug aucgcuuuua guuggaacca uucaaaacag cauagcaagu uaaaauaagg 60
cuaguccguu aucaacuuga aaaaguggca ccgagucggu gc 102
<210> 960
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 960
ugucagguua guuagaugaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 961
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 961
gugucagguu aguuagauga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 962
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 962
ccugacacau auacagacua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 963
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 963
cugacacaua uacagacuaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 964
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 964
ccuuagucug uauauguguc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 965
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 965
cauauacaga cuaagggacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 966
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 966
auauacagac uaagggacca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 967
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 967
ucaaaguuug auaaauuccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 968
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 968
aaaauacaaa gauaaguaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 969
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 969
acucugugac uuuaaaaagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 970
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 970
gguucugugg gauauuaaua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 971
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 971
acagagcaua uugguucugu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 972
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 972
gacagagcau auugguucug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 973
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 973
ugcaaaacga cagagcauau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 974
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 974
gagcugggca uggaauaggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 975
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 975
acuggagcug ggcauggaau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 976
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 976
cucauuacug gagcugggca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 977
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 977
uuguucucau uacuggagcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 978
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 978
auuguucuca uuacuggagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 979
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 979
ggggagauug uucucauuac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 980
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 980
gaggagaaaa ucuguggcug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 981
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 981
agaggagaaa aucuguggcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 982
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 982
cagaggagaa aaucuguggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 983
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 983
uccucagagg agaaaaucug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 984
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 984
ugaaguuuuu cauuccucag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 985
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 985
cuucaccaac gacuccaagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 986
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 986
cuacuccuac uuggagucgu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 987
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 987
cuccaaguag gaguagauga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 988
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 988
caccaucauc uacuccuacu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 989
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 989
ugauggugau cagaagcaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 990
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 990
ucagaagcag aaggauuucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 991
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 991
gauuucuagg augauguuca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 992
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 992
uugcucuguc cucuuccuuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 993
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 993
aggacugaac cagaaggaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 994
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 994
uacacaagga cugaaccaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 995
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 995
uucaguccuu guguaguccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 996
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 996
ucaguccuug uguaguccua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 997
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 997
guccuugugu aguccuaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 998
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 998
cuuguguagu ccuagggagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 999
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 999
cuccucccua ggacuacaca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1000
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1000
guagacagua ccuccucccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1001
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1001
uacugucuac acagagcucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1002
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1002
acugucuaca cagagcucua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1003
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1003
ucuacacaga gcucuaggga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1004
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1004
cuacacagag cucuagggaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1005
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1005
uacacagagc ucuagggaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1006
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1006
ggggugugcc caguuguuaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1007
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1007
gggugugccc aguuguuaau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1008
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1008
ugguaguccc auuaacaacu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1009
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1009
cugguagucc cauuaacaac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1010
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1010
uuguuaaugg gacuaccaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1011
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1011
uaccagaugg aagccagcuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1012
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1012
uuccaaagcu ggcuuccauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1013
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1013
uggaagccag cuuuggaagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1014
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1014
acaaggccug cuuccaaagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1015
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1015
gccuuguuca cguguucuaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1016
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1016
ccuuguucac guguucuaau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1017
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1017
cccauuagaa cacgugaaca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1018
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1018
uuggcaucac uucauauuug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1019
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1019
cuugugcucu uggcaucacu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1020
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1020
agcacacucu cuugugcucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1021
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1021
gcacaagaga gugugcucuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1022
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1022
gcuuaaucuc acacauagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1023
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1023
cuuaaucuca cacauagaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1024
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1024
uuaaucucac acauagaaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1025
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1025
aggagugcug guuuaucaac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1026
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1026
uucuuugaca gcaggagugc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1027
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1027
acucugguuu cuuugacagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1028
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1028
accagaguug agaaaacccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1029
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1029
uccagggguu uucucaacuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1030
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1030
caguuauuaa augaauccag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1031
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1031
gcaguuauua aaugaaucca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1032
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1032
ggcaguuauu aaaugaaucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1033
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1033
uggaugguaa cagcuacauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1034
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1034
gcuguuacca uccacauccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1035
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1035
ucaagaacca aggaugugga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1036
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1036
uccuucaaga accaaggaug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1037
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1037
ugaguguccu ucaagaacca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1038
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1038
uuuuauuuua uaacuacaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1039
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1039
uuguuuuuaa uaaaaacaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1040
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1040
uauuauagaa ugcuuuugca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1041
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1041
caagauuagu cuugauguag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1042
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1042
aagauuaguc uugauguagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1043
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1043
agucuugaug uagugggagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1044
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1044
uuuuucuauu aaaaaaaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1045
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1045
ucuauuaaaa aaaaaaaggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1046
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1046
cuauuaaaaa aaaaaaggcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1047
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1047
aaaaaaaaaa aggcugggca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1048
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1048
aaaaaaaagg cugggcacgg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1049
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1049
cacccguaau cccagcacuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1050
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1050
acccguaauc ccagcacuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1051
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1051
cguaauccca gcacuuuggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1052
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1052
ucccaaagug cugggauuac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1053
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1053
cucccaaagu gcugggauua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1054
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1054
cccagcacuu ugggaggccg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1055
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1055
ccucggccuc ccaaagugcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1056
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1056
gcacuuuggg aggccgaggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1057
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1057
cuuugggagg ccgaggcagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1058
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1058
gccgaggcag guggaucacg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1059
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1059
accucgugau ccaccugccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1060
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1060
ggcaggugga ucacgagguc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1061
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1061
ucaggagauc gagaccaucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1062
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1062
cgagaccauc uuggcuaaca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1063
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1063
uuucaccaug uuagccaaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1064
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1064
uuguauuuuu uguagagacg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1065
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1065
uuuguauuuu uuguagagac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1066
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1066
uuuuguauuu uuuguagaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1067
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1067
aaaaaauaca aaaaauuagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1068
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1068
aaaaauacaa aaaauuagcc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1069
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1069
uacaaaaaau uagccgggug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1070
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1070
aaaaaauuag ccgggugugg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1071
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1071
aaauuagccg gguguggugg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1072
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1072
aauuagccgg gugugguggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1073
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1073
caggcgcccg ccaccacacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1074
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1074
gccuguaguc ccagcuacuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1075
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1075
uguaguccca gcuacucagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1076
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1076
uccugaguag cugggacuac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1077
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1077
cccagcuacu caggaggcug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1078
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1078
ccucagccuc cugaguagcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1079
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1079
gccucagccu ccugaguagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1080
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1080
aggaggcuga ggcaggagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1081
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1081
gcaggagaau ggcgugaacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1082
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1082
caggagaaug gcgugaaccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1083
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1083
gagaauggcg ugaacccggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1084
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1084
aauggcguga acccgggagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1085
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1085
cacugcaagc uccaccuccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1086
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1086
ucacugcaag cuccaccucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1087
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1087
cauaccacug cacuccagcc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1088
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1088
auaccacugc acuccagccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1089
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1089
ucgcccaggc uggagugcag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1090
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1090
ucucacucuu ucgcccaggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1091
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1091
ggagucucac ucuuucgccc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1092
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1092
uguuuuuugu uuuuuugaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1093
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1093
aggaagaaag aaagguuuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1094
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1094
agaagaaaag gaagaaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1095
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1095
cuuucuuccu uuucuucucu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1096
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1096
uuucuuccuu uucuucucuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1097
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1097
aauggaccca agagaagaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1098
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1098
ggcuauuaca uaagaaacaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1099
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1099
cacaggaaaa ggaacuguac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1100
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1100
auuaaagcua acacaggaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1101
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1101
ucaaaaauua aagcuaacac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1102
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1102
uaaaauuguc uaaacaucuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1103
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1103
agagauguuu agacaauuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1104
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1104
ucuaaacauc ucugggacca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1105
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1105
uuuaugcuuu cauauauccu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1106
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1106
agcauaaauu acaaagaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1107
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1107
uacaaagaaa aagguuauca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1108
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1108
acaaagaaaa agguuaucau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1109
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1109
caaagaaaaa gguuaucaug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1110
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1110
ucugagauuu aaaauagagu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1111
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1111
cuuauaagau acauuaugaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1112
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1112
uaucuuauaa gacuauaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1113
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1113
aucuuauaag acuauaaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1114
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1114
uuauaagacu auaaaaaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1115
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1115
uaaaaaggga ggaaauauag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1116
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1116
aaaaagggag gaaauauaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1117
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1117
aaauauagag gguccacuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1118
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1118
uauagagggu ccacuuuugg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1119
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1119
acucugaagu ccaccaaaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1120
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1120
agaauagagu ugcaccguuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1121
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1121
aaaacggugc aacucuauuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1122
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1122
ccguuuuggg cuaaugaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1123
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1123
ccuuuuucau uagcccaaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1124
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1124
ugggcuaaug aaaaaggaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1125
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1125
uaaugaaaaa ggaagaggcu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1126
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1126
aaugaaaaag gaagaggcua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1127
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1127
cugaaucuua aaauaugucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1128
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1128
caggcagcuu uaucucaacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1129
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1129
cuaagagauc aaguuucagc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1130
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1130
guguucuugu ugauauucug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1131
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1131
cuuguugaua uucuguggca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1132
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1132
ucuguggcau ggcuacagau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1133
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1133
agaacuuauu uacacaggga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1134
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1134
aaagagaacu uauuuacaca guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1135
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1135
caaagagaac uuauuuacac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1136
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1136
uucucuuugu auuuacuuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1137
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1137
ucucuuugua uuuacuuuua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1138
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1138
cuuuguauuu acuuuuaggg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1139
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1139
agcuuuuguc caccuuuaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1140
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1140
uuuauuuuuc cauuuaaagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1141
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1141
uauuuuauuu uuccauuuaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1142
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1142
cuuacauaaa cauacuuaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1143
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1143
uaagcacaga aguuuuuaag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1144
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1144
aaguuuuuaa gaggcaugaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1145
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1145
auauuuacgu aguuuuucau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1146
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1146
cguaaauauu cuugagaaac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1147
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1147
uucuugagaa acaggaagac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1148
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1148
uaauauuaaa aacauugguu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1149
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1149
ccaauguuuu uaauauuauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1150
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1150
ccugauaaua uuaaaaacau guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1151
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1151
cauuaucaug cauacaucuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1152
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1152
aucaugcaua caucucuggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1153
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1153
uucauuucau uuugauuuug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1154
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1154
auucaauuug aagcaguggu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1155
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1155
gaauauucaa uuugaagcag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1156
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1156
cauacgauuu aaaaucgcug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1157
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1157
aaaaucgcug aggcgcguuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1158
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1158
uuuuuuuuuc uuuuuuguac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1159
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1159
cuguugucaa agauuuuaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1160
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1160
ugacaacaga guucuguuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1161
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1161
agaauacgcu gagaguuauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1162
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1162
gcaagagaag aaaagaacgg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1163
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1163
guugcaagag aagaaaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1164
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1164
augcacacgu aaaagagagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1165
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1165
aagaugcaca cguaaaagag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1166
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1166
aucaugcaua caucucuggc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1167
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1167
uucauuucau uuugauuuug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1168
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1168
auucaauuug aagcaguggu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1169
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1169
gaauauucaa uuugaagcag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1170
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1170
cauacgauuu aaaaucgcug guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1171
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1171
aaaaucgcug aggcgcguuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1172
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1172
uuuuuuuuuc uuuuuuguac guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1173
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1173
cuguugucaa agauuuuaaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1174
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1174
ugacaacaga guucuguuuu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1175
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1175
agaauacgcu gagaguuauc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1176
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1176
gcaagagaag aaaagaacgg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1177
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1177
guugcaagag aagaaaagaa guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1178
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1178
augcacacgu aaaagagagg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1179
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1179
aagaugcaca cguaaaagag guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1180
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1180
ggcagaccgu ucucaucacg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1181
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1181
cuuuaccagu gacuccaggu guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1182
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1182
gucacagauu uccuucuccg guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1183
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1183
agaugaugac gcccaccaga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1184
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1184
ggagaaggaa aucugugacc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1185
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1185
ugcgaggaac uuacuuuucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1186
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1186
agagaaauau ugauauagga guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1187
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1187
uaucaauauu ucucugaucc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1188
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1188
aucgcuuuua aggcacgcuc guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1189
<211> 96
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1189
uauacgacug aucgcuuuua guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc 60
cguuaucaac uugaaaaagu ggcaccgagu cggugc 96
<210> 1190
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1190
ugucagguua guuagaugaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1191
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1191
gugucagguu aguuagauga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1192
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1192
ccugacacau auacagacua guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1193
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1193
cugacacaua uacagacuaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1194
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1194
ccuuagucug uauauguguc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1195
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1195
cauauacaga cuaagggacc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1196
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1196
auauacagac uaagggacca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1197
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1197
ucaaaguuug auaaauuccc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1198
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1198
aaaauacaaa gauaaguaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1199
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1199
acucugugac uuuaaaaagu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1200
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1200
gguucugugg gauauuaaua guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1201
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1201
acagagcaua uugguucugu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1202
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1202
gacagagcau auugguucug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1203
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1203
ugcaaaacga cagagcauau guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1204
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1204
gagcugggca uggaauaggc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1205
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1205
acuggagcug ggcauggaau guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1206
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1206
cucauuacug gagcugggca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1207
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1207
uuguucucau uacuggagcu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1208
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1208
auuguucuca uuacuggagc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1209
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1209
ggggagauug uucucauuac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1210
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1210
gaggagaaaa ucuguggcug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1211
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1211
agaggagaaa aucuguggcu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1212
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1212
cagaggagaa aaucuguggc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1213
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1213
uccucagagg agaaaaucug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1214
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1214
ugaaguuuuu cauuccucag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1215
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1215
cuucaccaac gacuccaagu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1216
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1216
cuacuccuac uuggagucgu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1217
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1217
cuccaaguag gaguagauga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1218
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1218
caccaucauc uacuccuacu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1219
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1219
ugauggugau cagaagcaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1220
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1220
ucagaagcag aaggauuucu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1221
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1221
gauuucuagg augauguuca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1222
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1222
uugcucuguc cucuuccuuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1223
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1223
aggacugaac cagaaggaag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1224
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1224
uacacaagga cugaaccaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1225
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1225
uucaguccuu guguaguccu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1226
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1226
ucaguccuug uguaguccua guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1227
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1227
guccuugugu aguccuaggg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1228
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1228
cuuguguagu ccuagggagg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1229
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1229
cuccucccua ggacuacaca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1230
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1230
guagacagua ccuccucccu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1231
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1231
uacugucuac acagagcucu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1232
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1232
acugucuaca cagagcucua guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1233
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1233
ucuacacaga gcucuaggga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1234
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1234
cuacacagag cucuagggaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1235
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1235
uacacagagc ucuagggaag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1236
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1236
ggggugugcc caguuguuaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1237
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1237
gggugugccc aguuguuaau guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1238
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1238
ugguaguccc auuaacaacu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1239
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1239
cugguagucc cauuaacaac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1240
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1240
uuguuaaugg gacuaccaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1241
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1241
uaccagaugg aagccagcuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1242
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1242
uuccaaagcu ggcuuccauc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1243
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1243
uggaagccag cuuuggaagc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1244
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1244
acaaggccug cuuccaaagc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1245
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1245
gccuuguuca cguguucuaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1246
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1246
ccuuguucac guguucuaau guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1247
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1247
cccauuagaa cacgugaaca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1248
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1248
uuggcaucac uucauauuug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1249
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1249
cuugugcucu uggcaucacu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1250
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1250
agcacacucu cuugugcucu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1251
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1251
gcacaagaga gugugcucuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1252
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1252
gcuuaaucuc acacauagaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1253
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1253
cuuaaucuca cacauagaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1254
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1254
uuaaucucac acauagaaag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1255
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1255
aggagugcug guuuaucaac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1256
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1256
uucuuugaca gcaggagugc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1257
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1257
acucugguuu cuuugacagc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1258
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1258
accagaguug agaaaacccc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1259
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1259
uccagggguu uucucaacuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1260
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1260
caguuauuaa augaauccag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1261
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1261
gcaguuauua aaugaaucca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1262
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1262
ggcaguuauu aaaugaaucc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1263
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1263
uggaugguaa cagcuacauc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1264
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1264
gcuguuacca uccacauccu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1265
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1265
ucaagaacca aggaugugga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1266
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1266
uccuucaaga accaaggaug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1267
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1267
ugaguguccu ucaagaacca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1268
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1268
uuuuauuuua uaacuacaag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1269
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1269
uuguuuuuaa uaaaaacaag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1270
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1270
uauuauagaa ugcuuuugca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1271
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1271
caagauuagu cuugauguag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1272
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1272
aagauuaguc uugauguagu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1273
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1273
agucuugaug uagugggagu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1274
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1274
uuuuucuauu aaaaaaaaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1275
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1275
ucuauuaaaa aaaaaaaggc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1276
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1276
cuauuaaaaa aaaaaaggcu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1277
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1277
aaaaaaaaaa aggcugggca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1278
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1278
aaaaaaaagg cugggcacgg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1279
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1279
cacccguaau cccagcacuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1280
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1280
acccguaauc ccagcacuuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1281
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1281
cguaauccca gcacuuuggg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1282
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1282
ucccaaagug cugggauuac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1283
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1283
cucccaaagu gcugggauua guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1284
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1284
cccagcacuu ugggaggccg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1285
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1285
ccucggccuc ccaaagugcu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1286
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1286
gcacuuuggg aggccgaggc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1287
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1287
cuuugggagg ccgaggcagg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1288
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1288
gccgaggcag guggaucacg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1289
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1289
accucgugau ccaccugccu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1290
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1290
ggcaggugga ucacgagguc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1291
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1291
ucaggagauc gagaccaucu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1292
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1292
cgagaccauc uuggcuaaca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1293
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1293
uuucaccaug uuagccaaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1294
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1294
uuguauuuuu uguagagacg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1295
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1295
uuuguauuuu uuguagagac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1296
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1296
uuuuguauuu uuuguagaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1297
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1297
aaaaaauaca aaaaauuagc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1298
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1298
aaaaauacaa aaaauuagcc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1299
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1299
uacaaaaaau uagccgggug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1300
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1300
aaaaaauuag ccgggugugg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1301
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1301
aaauuagccg gguguggugg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1302
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1302
aauuagccgg gugugguggc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1303
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1303
caggcgcccg ccaccacacc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1304
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1304
gccuguaguc ccagcuacuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1305
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1305
uguaguccca gcuacucagg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1306
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1306
uccugaguag cugggacuac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1307
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1307
cccagcuacu caggaggcug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1308
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1308
ccucagccuc cugaguagcu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1309
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1309
gccucagccu ccugaguagc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1310
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1310
aggaggcuga ggcaggagaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1311
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1311
gcaggagaau ggcgugaacc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1312
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1312
caggagaaug gcgugaaccc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1313
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1313
gagaauggcg ugaacccggg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1314
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1314
aauggcguga acccgggagg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1315
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1315
cacugcaagc uccaccuccc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1316
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1316
ucacugcaag cuccaccucc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1317
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1317
cauaccacug cacuccagcc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1318
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1318
auaccacugc acuccagccu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1319
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1319
ucgcccaggc uggagugcag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1320
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1320
ucucacucuu ucgcccaggc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1321
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1321
ggagucucac ucuuucgccc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1322
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1322
uguuuuuugu uuuuuugaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1323
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1323
aggaagaaag aaagguuuuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1324
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1324
agaagaaaag gaagaaagaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1325
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1325
cuuucuuccu uuucuucucu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1326
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1326
uuucuuccuu uucuucucuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1327
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1327
aauggaccca agagaagaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1328
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1328
ggcuauuaca uaagaaacaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1329
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1329
cacaggaaaa ggaacuguac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1330
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1330
auuaaagcua acacaggaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1331
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1331
ucaaaaauua aagcuaacac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1332
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1332
uaaaauuguc uaaacaucuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1333
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1333
agagauguuu agacaauuuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1334
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1334
ucuaaacauc ucugggacca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1335
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1335
uuuaugcuuu cauauauccu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1336
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1336
agcauaaauu acaaagaaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1337
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1337
uacaaagaaa aagguuauca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1338
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1338
acaaagaaaa agguuaucau guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1339
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1339
caaagaaaaa gguuaucaug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1340
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1340
ucugagauuu aaaauagagu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1341
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1341
cuuauaagau acauuaugaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1342
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1342
uaucuuauaa gacuauaaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1343
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1343
aucuuauaag acuauaaaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1344
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1344
uuauaagacu auaaaaaggg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1345
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1345
uaaaaaggga ggaaauauag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1346
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1346
aaaaagggag gaaauauaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1347
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1347
aaauauagag gguccacuuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1348
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1348
uauagagggu ccacuuuugg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1349
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1349
acucugaagu ccaccaaaag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1350
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1350
agaauagagu ugcaccguuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1351
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1351
aaaacggugc aacucuauuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1352
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1352
ccguuuuggg cuaaugaaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1353
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1353
ccuuuuucau uagcccaaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1354
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1354
ugggcuaaug aaaaaggaag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1355
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1355
uaaugaaaaa ggaagaggcu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1356
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1356
aaugaaaaag gaagaggcua guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1357
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1357
cugaaucuua aaauaugucc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1358
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1358
caggcagcuu uaucucaacc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1359
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1359
cuaagagauc aaguuucagc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1360
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1360
guguucuugu ugauauucug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1361
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1361
cuuguugaua uucuguggca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1362
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1362
ucuguggcau ggcuacagau guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1363
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1363
agaacuuauu uacacaggga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1364
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1364
aaagagaacu uauuuacaca guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1365
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1365
caaagagaac uuauuuacac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1366
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1366
uucucuuugu auuuacuuuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1367
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1367
ucucuuugua uuuacuuuua guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1368
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1368
cuuuguauuu acuuuuaggg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1369
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1369
agcuuuuguc caccuuuaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1370
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1370
uuuauuuuuc cauuuaaagg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1371
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1371
uauuuuauuu uuccauuuaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1372
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1372
cuuacauaaa cauacuuaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1373
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1373
uaagcacaga aguuuuuaag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1374
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1374
aaguuuuuaa gaggcaugaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1375
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1375
auauuuacgu aguuuuucau guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1376
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1376
cguaaauauu cuugagaaac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1377
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1377
uucuugagaa acaggaagac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1378
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1378
uaauauuaaa aacauugguu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1379
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1379
ccaauguuuu uaauauuauc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1380
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1380
ccugauaaua uuaaaaacau guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1381
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1381
cauuaucaug cauacaucuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1382
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1382
aucaugcaua caucucuggc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1383
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1383
uucauuucau uuugauuuug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1384
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1384
auucaauuug aagcaguggu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1385
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1385
gaauauucaa uuugaagcag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1386
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1386
cauacgauuu aaaaucgcug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1387
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1387
aaaaucgcug aggcgcguuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1388
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1388
uuuuuuuuuc uuuuuuguac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1389
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1389
cuguugucaa agauuuuaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1390
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1390
ugacaacaga guucuguuuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1391
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1391
agaauacgcu gagaguuauc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1392
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1392
gcaagagaag aaaagaacgg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1393
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1393
guugcaagag aagaaaagaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1394
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1394
augcacacgu aaaagagagg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1395
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1395
aagaugcaca cguaaaagag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1396
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1396
aucaugcaua caucucuggc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1397
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1397
uucauuucau uuugauuuug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1398
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1398
auucaauuug aagcaguggu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1399
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1399
gaauauucaa uuugaagcag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1400
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1400
cauacgauuu aaaaucgcug guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1401
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1401
aaaaucgcug aggcgcguuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1402
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1402
uuuuuuuuuc uuuuuuguac guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1403
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1403
cuguugucaa agauuuuaaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1404
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1404
ugacaacaga guucuguuuu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1405
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1405
agaauacgcu gagaguuauc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1406
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1406
gcaagagaag aaaagaacgg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1407
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1407
guugcaagag aagaaaagaa guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1408
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1408
augcacacgu aaaagagagg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1409
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1409
aagaugcaca cguaaaagag guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1410
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1410
ggcagaccgu ucucaucacg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1411
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1411
cuuuaccagu gacuccaggu guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1412
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1412
gucacagauu uccuucuccg guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1413
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1413
agaugaugac gcccaccaga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1414
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1414
ggagaaggaa aucugugacc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1415
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1415
ugcgaggaac uuacuuuucc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1416
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1416
agagaaauau ugauauagga guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1417
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1417
uaucaauauu ucucugaucc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1418
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1418
aucgcuuuua aggcacgcuc guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1419
<211> 106
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1419
uauacgacug aucgcuuuua guuuaagagc uaugcuggaa acagcauagc aaguuuaaau 60
aaggcuaguc cguuaucaac uugaaaaagu ggcaccgagu cggugc 106
<210> 1420
<211> 77
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1420
guuuuagagc uagaaauagc aaguuaaaau aaggcuaguc cguuaucaac uugaaaaagu 60
ggcaccgagu cggugcu 77
<210> 1421
<211> 16
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1421
guuuuagagc uaugcu 16
<210> 1422
<211> 67
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1422
agcauagcaa guuaaaauaa ggcuaguccg uuaucaacuu gaaaaagugg caccgagucg 60
gugcuuu 67
<210> 1423
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1423
ugucagguua guuagaugaa 20
<210> 1424
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1424
gugucagguu aguuagauga 20
<210> 1425
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1425
ccugacacau auacagacua 20
<210> 1426
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1426
cugacacaua uacagacuaa 20
<210> 1427
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1427
ccuuagucug uauauguguc 20
<210> 1428
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1428
cauauacaga cuaagggacc 20
<210> 1429
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1429
auauacagac uaagggacca 20
<210> 1430
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1430
ucaaaguuug auaaauuccc 20
<210> 1431
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1431
aaaauacaaa gauaaguaga 20
<210> 1432
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1432
acucugugac uuuaaaaagu 20
<210> 1433
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1433
gguucugugg gauauuaaua 20
<210> 1434
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1434
acagagcaua uugguucugu 20
<210> 1435
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1435
gacagagcau auugguucug 20
<210> 1436
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1436
ugcaaaacga cagagcauau 20
<210> 1437
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1437
gagcugggca uggaauaggc 20
<210> 1438
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1438
acuggagcug ggcauggaau 20
<210> 1439
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1439
cucauuacug gagcugggca 20
<210> 1440
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1440
uuguucucau uacuggagcu 20
<210> 1441
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1441
auuguucuca uuacuggagc 20
<210> 1442
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1442
ggggagauug uucucauuac 20
<210> 1443
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1443
gaggagaaaa ucuguggcug 20
<210> 1444
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1444
agaggagaaa aucuguggcu 20
<210> 1445
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1445
cagaggagaa aaucuguggc 20
<210> 1446
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1446
uccucagagg agaaaaucug 20
<210> 1447
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1447
ugaaguuuuu cauuccucag 20
<210> 1448
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1448
cuucaccaac gacuccaagu 20
<210> 1449
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1449
cuacuccuac uuggagucgu 20
<210> 1450
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1450
cuccaaguag gaguagauga 20
<210> 1451
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1451
caccaucauc uacuccuacu 20
<210> 1452
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1452
ugauggugau cagaagcaga 20
<210> 1453
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1453
ucagaagcag aaggauuucu 20
<210> 1454
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1454
gauuucuagg augauguuca 20
<210> 1455
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1455
uugcucuguc cucuuccuuc 20
<210> 1456
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1456
aggacugaac cagaaggaag 20
<210> 1457
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1457
uacacaagga cugaaccaga 20
<210> 1458
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1458
uucaguccuu guguaguccu 20
<210> 1459
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1459
ucaguccuug uguaguccua 20
<210> 1460
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1460
guccuugugu aguccuaggg 20
<210> 1461
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1461
cuuguguagu ccuagggagg 20
<210> 1462
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1462
cuccucccua ggacuacaca 20
<210> 1463
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1463
guagacagua ccuccucccu 20
<210> 1464
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1464
uacugucuac acagagcucu 20
<210> 1465
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1465
acugucuaca cagagcucua 20
<210> 1466
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1466
ucuacacaga gcucuaggga 20
<210> 1467
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1467
cuacacagag cucuagggaa 20
<210> 1468
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1468
uacacagagc ucuagggaag 20
<210> 1469
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1469
ggggugugcc caguuguuaa 20
<210> 1470
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1470
gggugugccc aguuguuaau 20
<210> 1471
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1471
ugguaguccc auuaacaacu 20
<210> 1472
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1472
cugguagucc cauuaacaac 20
<210> 1473
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1473
uuguuaaugg gacuaccaga 20
<210> 1474
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1474
uaccagaugg aagccagcuu 20
<210> 1475
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1475
uuccaaagcu ggcuuccauc 20
<210> 1476
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1476
uggaagccag cuuuggaagc 20
<210> 1477
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1477
acaaggccug cuuccaaagc 20
<210> 1478
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1478
gccuuguuca cguguucuaa 20
<210> 1479
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1479
ccuuguucac guguucuaau 20
<210> 1480
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1480
cccauuagaa cacgugaaca 20
<210> 1481
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1481
uuggcaucac uucauauuug 20
<210> 1482
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1482
cuugugcucu uggcaucacu 20
<210> 1483
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1483
agcacacucu cuugugcucu 20
<210> 1484
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1484
gcacaagaga gugugcucuc 20
<210> 1485
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1485
gcuuaaucuc acacauagaa 20
<210> 1486
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1486
cuuaaucuca cacauagaaa 20
<210> 1487
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1487
uuaaucucac acauagaaag 20
<210> 1488
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1488
aggagugcug guuuaucaac 20
<210> 1489
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1489
uucuuugaca gcaggagugc 20
<210> 1490
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1490
acucugguuu cuuugacagc 20
<210> 1491
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1491
accagaguug agaaaacccc 20
<210> 1492
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1492
uccagggguu uucucaacuc 20
<210> 1493
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1493
caguuauuaa augaauccag 20
<210> 1494
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1494
gcaguuauua aaugaaucca 20
<210> 1495
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1495
ggcaguuauu aaaugaaucc 20
<210> 1496
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1496
uggaugguaa cagcuacauc 20
<210> 1497
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1497
gcuguuacca uccacauccu 20
<210> 1498
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1498
ucaagaacca aggaugugga 20
<210> 1499
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1499
uccuucaaga accaaggaug 20
<210> 1500
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1500
ugaguguccu ucaagaacca 20
<210> 1501
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1501
uuuuauuuua uaacuacaag 20
<210> 1502
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1502
uuguuuuuaa uaaaaacaag 20
<210> 1503
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1503
uauuauagaa ugcuuuugca 20
<210> 1504
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1504
caagauuagu cuugauguag 20
<210> 1505
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1505
aagauuaguc uugauguagu 20
<210> 1506
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1506
agucuugaug uagugggagu 20
<210> 1507
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1507
uuuuucuauu aaaaaaaaaa 20
<210> 1508
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1508
ucuauuaaaa aaaaaaaggc 20
<210> 1509
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1509
cuauuaaaaa aaaaaaggcu 20
<210> 1510
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1510
aaaaaaaaaa aggcugggca 20
<210> 1511
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1511
aaaaaaaagg cugggcacgg 20
<210> 1512
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1512
cacccguaau cccagcacuu 20
<210> 1513
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1513
acccguaauc ccagcacuuu 20
<210> 1514
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1514
cguaauccca gcacuuuggg 20
<210> 1515
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1515
ucccaaagug cugggauuac 20
<210> 1516
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1516
cucccaaagu gcugggauua 20
<210> 1517
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1517
cccagcacuu ugggaggccg 20
<210> 1518
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1518
ccucggccuc ccaaagugcu 20
<210> 1519
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1519
gcacuuuggg aggccgaggc 20
<210> 1520
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1520
cuuugggagg ccgaggcagg 20
<210> 1521
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1521
gccgaggcag guggaucacg 20
<210> 1522
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1522
accucgugau ccaccugccu 20
<210> 1523
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1523
ggcaggugga ucacgagguc 20
<210> 1524
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1524
ucaggagauc gagaccaucu 20
<210> 1525
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1525
cgagaccauc uuggcuaaca 20
<210> 1526
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1526
uuucaccaug uuagccaaga 20
<210> 1527
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1527
uuguauuuuu uguagagacg 20
<210> 1528
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1528
uuuguauuuu uuguagagac 20
<210> 1529
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1529
uuuuguauuu uuuguagaga 20
<210> 1530
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1530
aaaaaauaca aaaaauuagc 20
<210> 1531
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1531
aaaaauacaa aaaauuagcc 20
<210> 1532
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1532
uacaaaaaau uagccgggug 20
<210> 1533
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1533
aaaaaauuag ccgggugugg 20
<210> 1534
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1534
aaauuagccg gguguggugg 20
<210> 1535
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1535
aauuagccgg gugugguggc 20
<210> 1536
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1536
caggcgcccg ccaccacacc 20
<210> 1537
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1537
gccuguaguc ccagcuacuc 20
<210> 1538
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1538
uguaguccca gcuacucagg 20
<210> 1539
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1539
uccugaguag cugggacuac 20
<210> 1540
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1540
cccagcuacu caggaggcug 20
<210> 1541
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1541
ccucagccuc cugaguagcu 20
<210> 1542
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1542
gccucagccu ccugaguagc 20
<210> 1543
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1543
aggaggcuga ggcaggagaa 20
<210> 1544
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1544
gcaggagaau ggcgugaacc 20
<210> 1545
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1545
caggagaaug gcgugaaccc 20
<210> 1546
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1546
gagaauggcg ugaacccggg 20
<210> 1547
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1547
aauggcguga acccgggagg 20
<210> 1548
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1548
cacugcaagc uccaccuccc 20
<210> 1549
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1549
ucacugcaag cuccaccucc 20
<210> 1550
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1550
cauaccacug cacuccagcc 20
<210> 1551
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1551
auaccacugc acuccagccu 20
<210> 1552
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1552
ucgcccaggc uggagugcag 20
<210> 1553
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1553
ucucacucuu ucgcccaggc 20
<210> 1554
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1554
ggagucucac ucuuucgccc 20
<210> 1555
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1555
uguuuuuugu uuuuuugaga 20
<210> 1556
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1556
aggaagaaag aaagguuuuu 20
<210> 1557
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1557
agaagaaaag gaagaaagaa 20
<210> 1558
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1558
cuuucuuccu uuucuucucu 20
<210> 1559
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1559
uuucuuccuu uucuucucuu 20
<210> 1560
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1560
aauggaccca agagaagaaa 20
<210> 1561
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1561
ggcuauuaca uaagaaacaa 20
<210> 1562
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1562
cacaggaaaa ggaacuguac 20
<210> 1563
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1563
auuaaagcua acacaggaaa 20
<210> 1564
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1564
ucaaaaauua aagcuaacac 20
<210> 1565
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1565
uaaaauuguc uaaacaucuc 20
<210> 1566
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1566
agagauguuu agacaauuuu 20
<210> 1567
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1567
ucuaaacauc ucugggacca 20
<210> 1568
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1568
uuuaugcuuu cauauauccu 20
<210> 1569
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1569
agcauaaauu acaaagaaaa 20
<210> 1570
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1570
uacaaagaaa aagguuauca 20
<210> 1571
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1571
acaaagaaaa agguuaucau 20
<210> 1572
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1572
caaagaaaaa gguuaucaug 20
<210> 1573
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1573
ucugagauuu aaaauagagu 20
<210> 1574
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1574
cuuauaagau acauuaugaa 20
<210> 1575
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1575
uaucuuauaa gacuauaaaa 20
<210> 1576
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1576
aucuuauaag acuauaaaaa 20
<210> 1577
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1577
uuauaagacu auaaaaaggg 20
<210> 1578
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1578
uaaaaaggga ggaaauauag 20
<210> 1579
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1579
aaaaagggag gaaauauaga 20
<210> 1580
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1580
aaauauagag gguccacuuu 20
<210> 1581
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1581
uauagagggu ccacuuuugg 20
<210> 1582
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1582
acucugaagu ccaccaaaag 20
<210> 1583
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1583
agaauagagu ugcaccguuu 20
<210> 1584
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1584
aaaacggugc aacucuauuc 20
<210> 1585
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1585
ccguuuuggg cuaaugaaaa 20
<210> 1586
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1586
ccuuuuucau uagcccaaaa 20
<210> 1587
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1587
ugggcuaaug aaaaaggaag 20
<210> 1588
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1588
uaaugaaaaa ggaagaggcu 20
<210> 1589
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1589
aaugaaaaag gaagaggcua 20
<210> 1590
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1590
cugaaucuua aaauaugucc 20
<210> 1591
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1591
caggcagcuu uaucucaacc 20
<210> 1592
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1592
cuaagagauc aaguuucagc 20
<210> 1593
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1593
guguucuugu ugauauucug 20
<210> 1594
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1594
cuuguugaua uucuguggca 20
<210> 1595
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1595
ucuguggcau ggcuacagau 20
<210> 1596
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1596
agaacuuauu uacacaggga 20
<210> 1597
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1597
aaagagaacu uauuuacaca 20
<210> 1598
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1598
caaagagaac uuauuuacac 20
<210> 1599
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1599
uucucuuugu auuuacuuuu 20
<210> 1600
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1600
ucucuuugua uuuacuuuua 20
<210> 1601
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1601
cuuuguauuu acuuuuaggg 20
<210> 1602
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1602
agcuuuuguc caccuuuaaa 20
<210> 1603
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1603
uuuauuuuuc cauuuaaagg 20
<210> 1604
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1604
uauuuuauuu uuccauuuaa 20
<210> 1605
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1605
cuuacauaaa cauacuuaaa 20
<210> 1606
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1606
uaagcacaga aguuuuuaag 20
<210> 1607
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1607
aaguuuuuaa gaggcaugaa 20
<210> 1608
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1608
auauuuacgu aguuuuucau 20
<210> 1609
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1609
cguaaauauu cuugagaaac 20
<210> 1610
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1610
uucuugagaa acaggaagac 20
<210> 1611
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1611
uaauauuaaa aacauugguu 20
<210> 1612
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1612
ccaauguuuu uaauauuauc 20
<210> 1613
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1613
ccugauaaua uuaaaaacau 20
<210> 1614
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1614
cauuaucaug cauacaucuc 20
<210> 1615
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1615
aucaugcaua caucucuggc 20
<210> 1616
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1616
uucauuucau uuugauuuug 20
<210> 1617
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1617
auucaauuug aagcaguggu 20
<210> 1618
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1618
gaauauucaa uuugaagcag 20
<210> 1619
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1619
cauacgauuu aaaaucgcug 20
<210> 1620
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1620
aaaaucgcug aggcgcguuc 20
<210> 1621
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1621
uuuuuuuuuc uuuuuuguac 20
<210> 1622
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1622
cuguugucaa agauuuuaaa 20
<210> 1623
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1623
ugacaacaga guucuguuuu 20
<210> 1624
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1624
agaauacgcu gagaguuauc 20
<210> 1625
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1625
gcaagagaag aaaagaacgg 20
<210> 1626
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1626
guugcaagag aagaaaagaa 20
<210> 1627
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1627
augcacacgu aaaagagagg 20
<210> 1628
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1628
aagaugcaca cguaaaagag 20
<210> 1629
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1629
aucaugcaua caucucuggc 20
<210> 1630
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1630
uucauuucau uuugauuuug 20
<210> 1631
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1631
auucaauuug aagcaguggu 20
<210> 1632
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1632
gaauauucaa uuugaagcag 20
<210> 1633
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1633
cauacgauuu aaaaucgcug 20
<210> 1634
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1634
aaaaucgcug aggcgcguuc 20
<210> 1635
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1635
uuuuuuuuuc uuuuuuguac 20
<210> 1636
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1636
cuguugucaa agauuuuaaa 20
<210> 1637
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1637
ugacaacaga guucuguuuu 20
<210> 1638
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1638
agaauacgcu gagaguuauc 20
<210> 1639
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1639
gcaagagaag aaaagaacgg 20
<210> 1640
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1640
guugcaagag aagaaaagaa 20
<210> 1641
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1641
augcacacgu aaaagagagg 20
<210> 1642
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1642
aagaugcaca cguaaaagag 20
<210> 1643
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1643
ggcagaccgu ucucaucacg 20
<210> 1644
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1644
cuuuaccagu gacuccaggu 20
<210> 1645
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1645
gucacagauu uccuucuccg 20
<210> 1646
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1646
agaugaugac gcccaccaga 20
<210> 1647
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1647
ggagaaggaa aucugugacc 20
<210> 1648
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1648
ugcgaggaac uuacuuuucc 20
<210> 1649
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1649
agagaaauau ugauauagga 20
<210> 1650
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1650
uaucaauauu ucucugaucc 20
<210> 1651
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1651
aucgcuuuua aggcacgcuc 20
<210> 1652
<211> 20
<212> RNA
<213> Искусственная последовательность
<220>
<223> Синтетическая
<400> 1652
uauacgacug aucgcuuuua 20
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| КОМПОЗИЦИИ И СПОСОБЫ, ПРЕДНАЗНАЧЕННЫЕ ДЛЯ ЛЕЧЕНИЯ ГЕМОГЛОБИНОПАТИЙ | 2018 |
|
RU2834231C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ЛЕЧЕНИЯ ГЕМОГЛОБИНОПАТИЙ | 2016 |
|
RU2812491C2 |
| РНК-НАПРАВЛЯЕМАЯ ИНЖЕНЕРИЯ ГЕНОМА ЧЕЛОВЕКА | 2013 |
|
RU2766685C2 |
| КОНСТРУИРОВАНИЕ СИСТЕМ, СПОСОБЫ И ОПТИМИЗИРОВАННЫЕ НАПРАВЛЯЮЩИЕ КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796017C2 |
| Т-КЛЕТКИ С ХИМЕРНЫМИ АНТИГЕННЫМИ РЕЦЕПТОРАМИ (CAR-T) ДЛЯ ЛЕЧЕНИЯ РАКА | 2019 |
|
RU2811466C2 |
| СТРАТЕГИИ CRISPR И AAV ДЛЯ ТЕРАПИИ Х-СЦЕПЛЕННОГО ЮВЕНИЛЬНОГО РЕТИНОШИЗИСА | 2020 |
|
RU2833486C1 |
| ПЛАТФОРМА CRISPR/CAS ДЛЯ ИСКЛЮЧАЮЩЕГО СКРИНИНГА ДЛЯ ВЫЯВЛЕНИЯ ГЕНЕТИЧЕСКИХ УЯЗВИМОСТЕЙ, СВЯЗАННЫХ С АГРЕГАЦИЕЙ ТАУ-БЕЛКА | 2020 |
|
RU2808829C2 |
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ ИММУНООНКОЛОГИИ | 2016 |
|
RU2771624C2 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796549C2 |
| ОЛИГОНУКЛЕОТИД ДЛЯ ЛЕЧЕНИЯ ПАЦИЕНТОВ С МЫШЕЧНОЙ ДИСТРОФИЕЙ | 2013 |
|
RU2789279C2 |
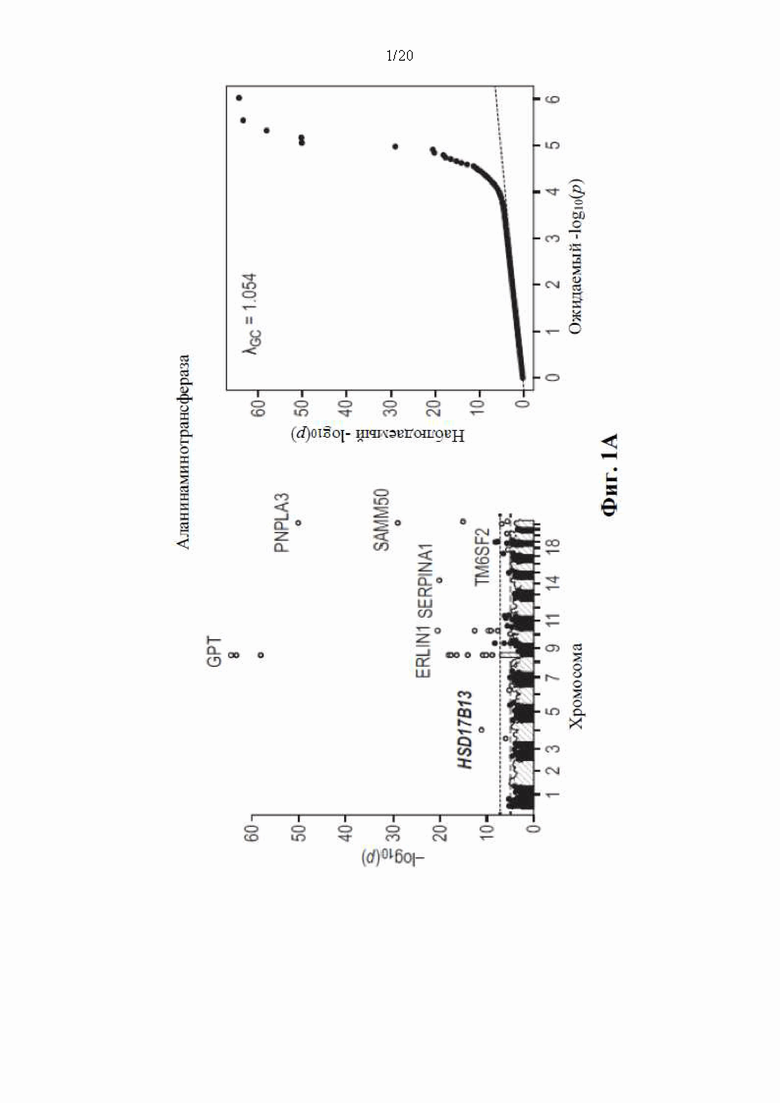
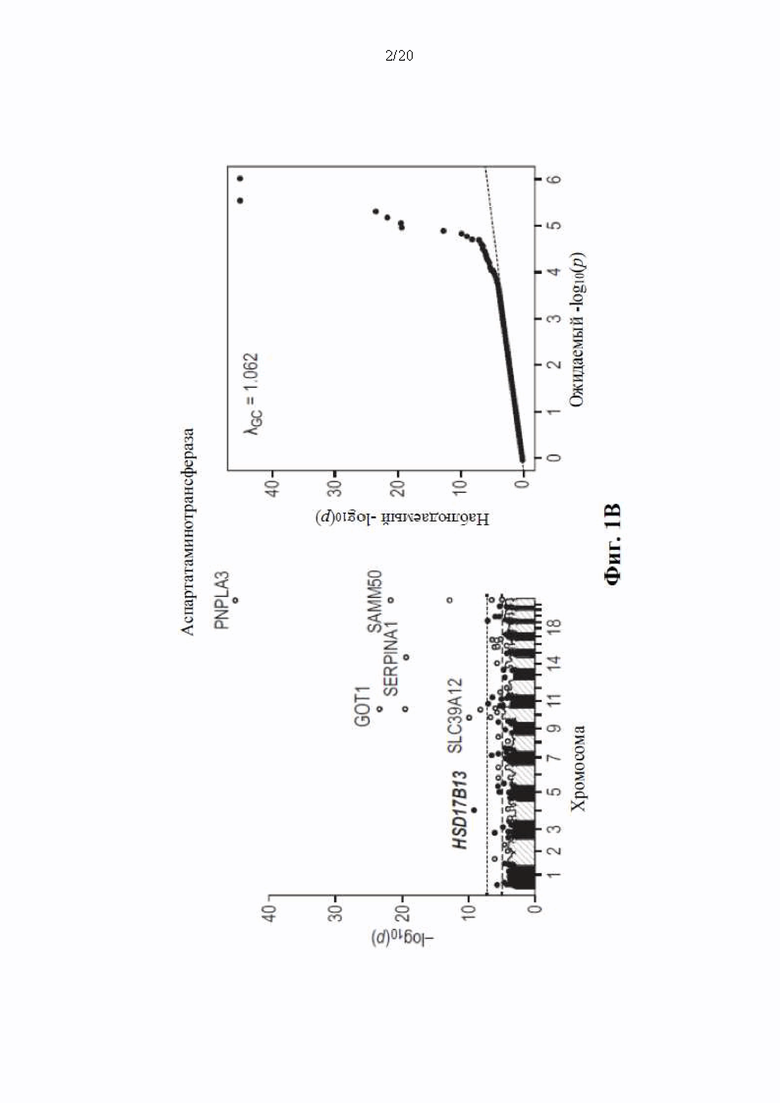
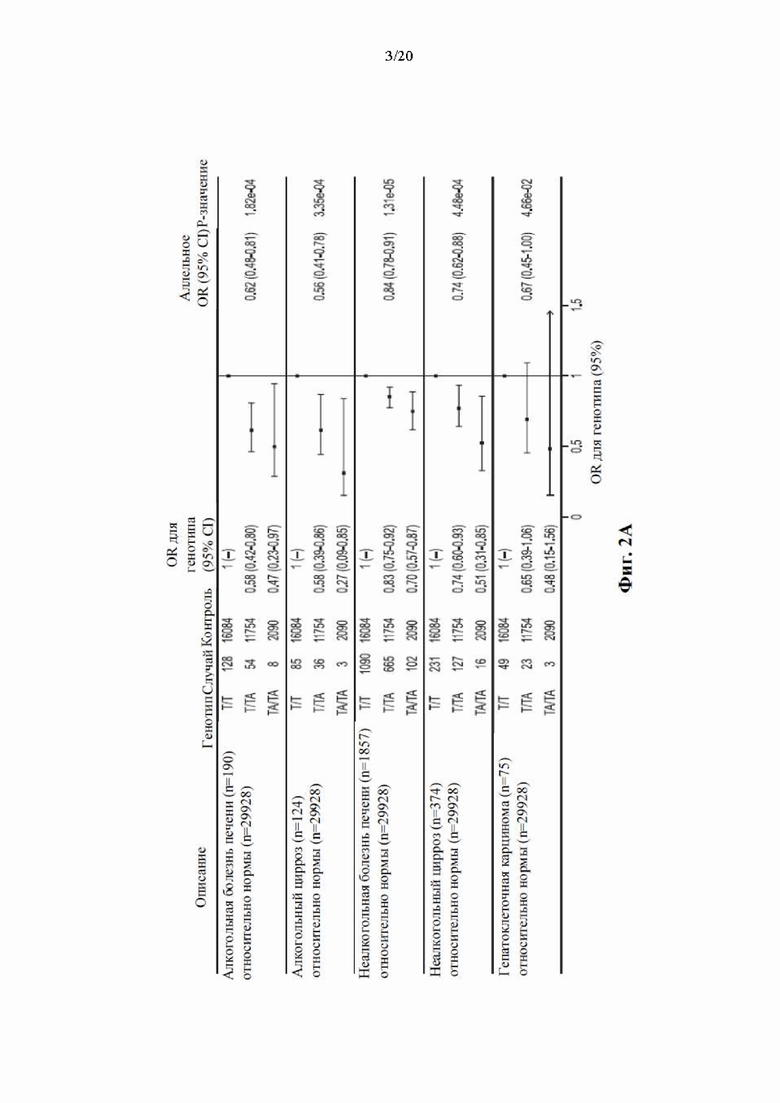
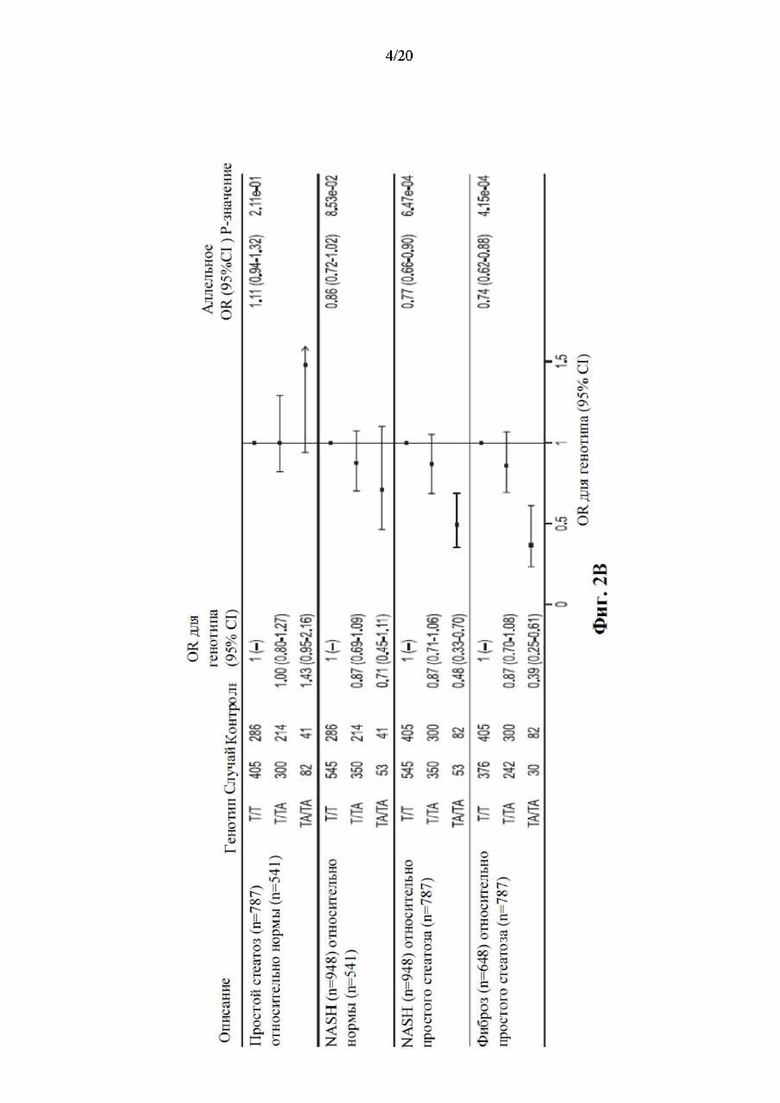
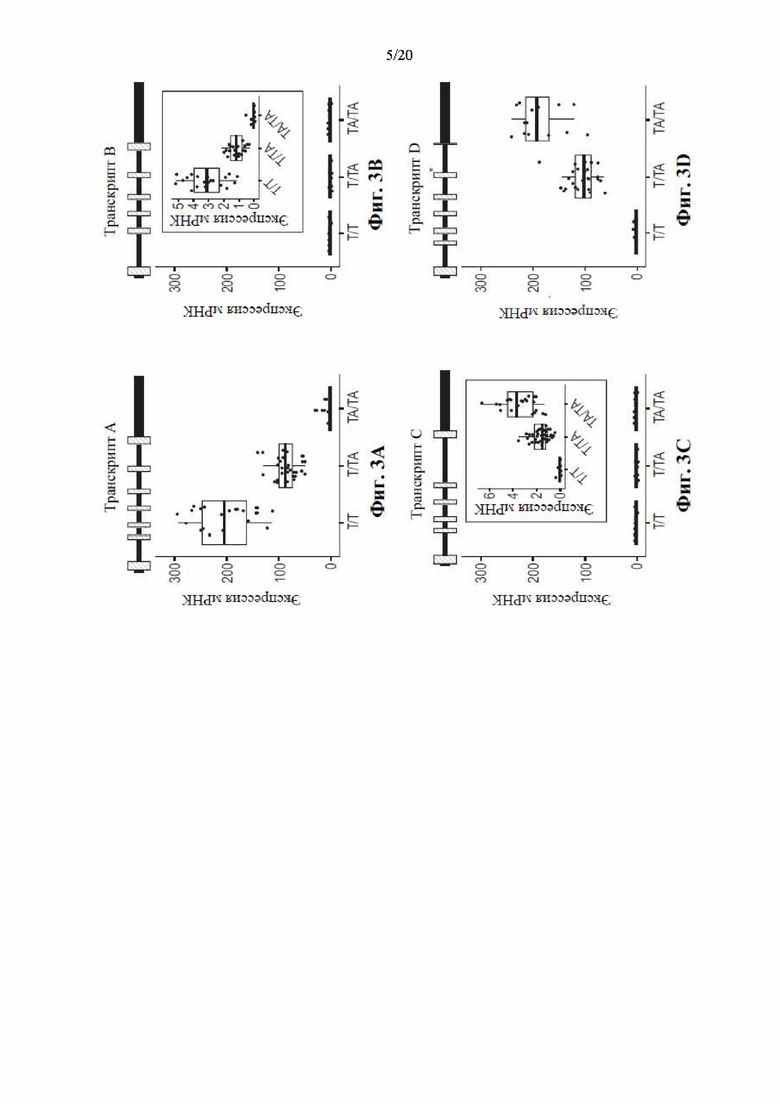
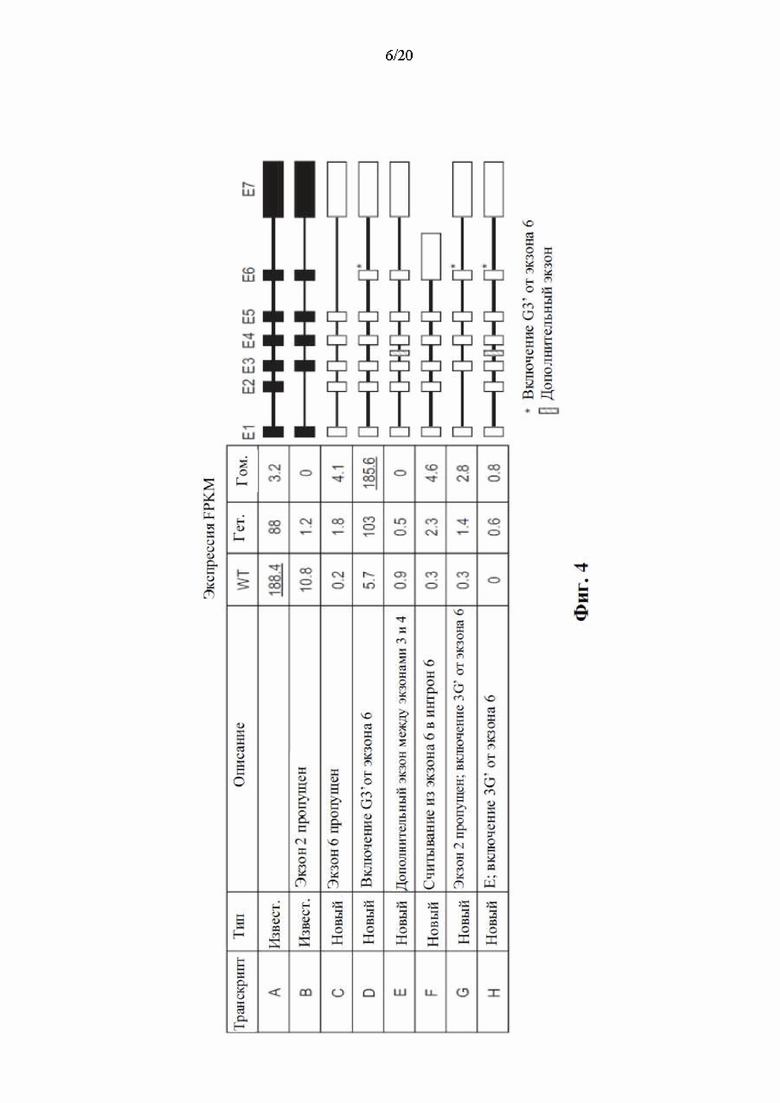
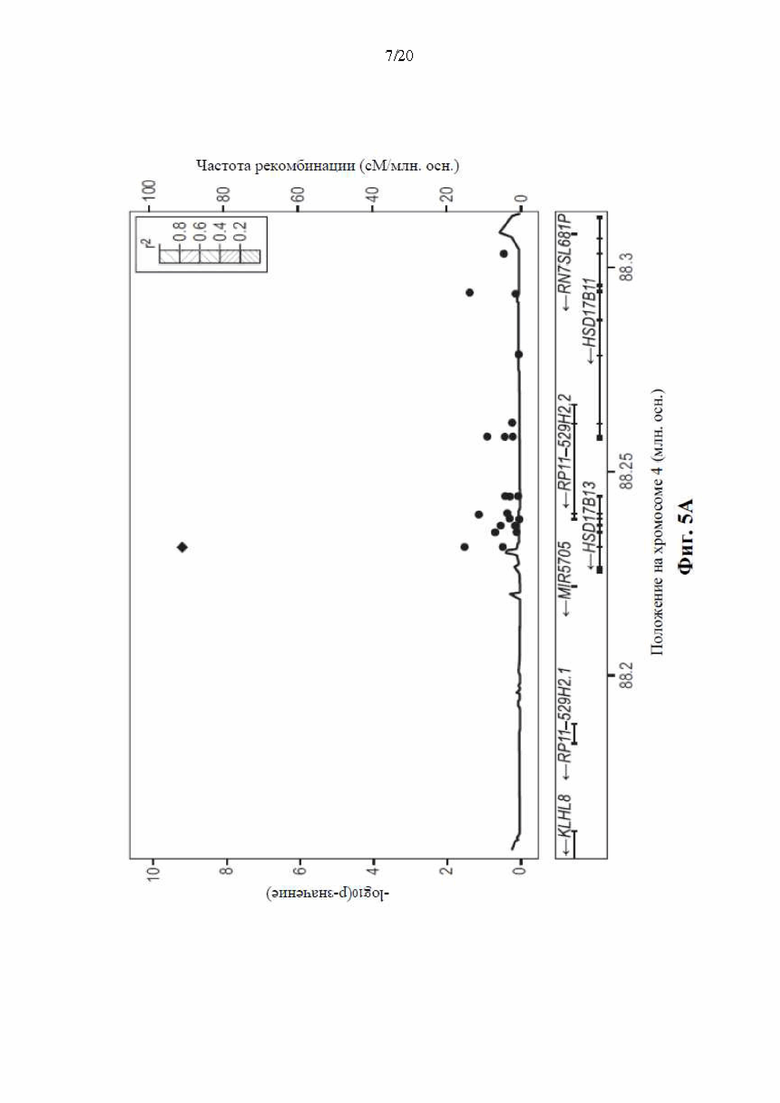
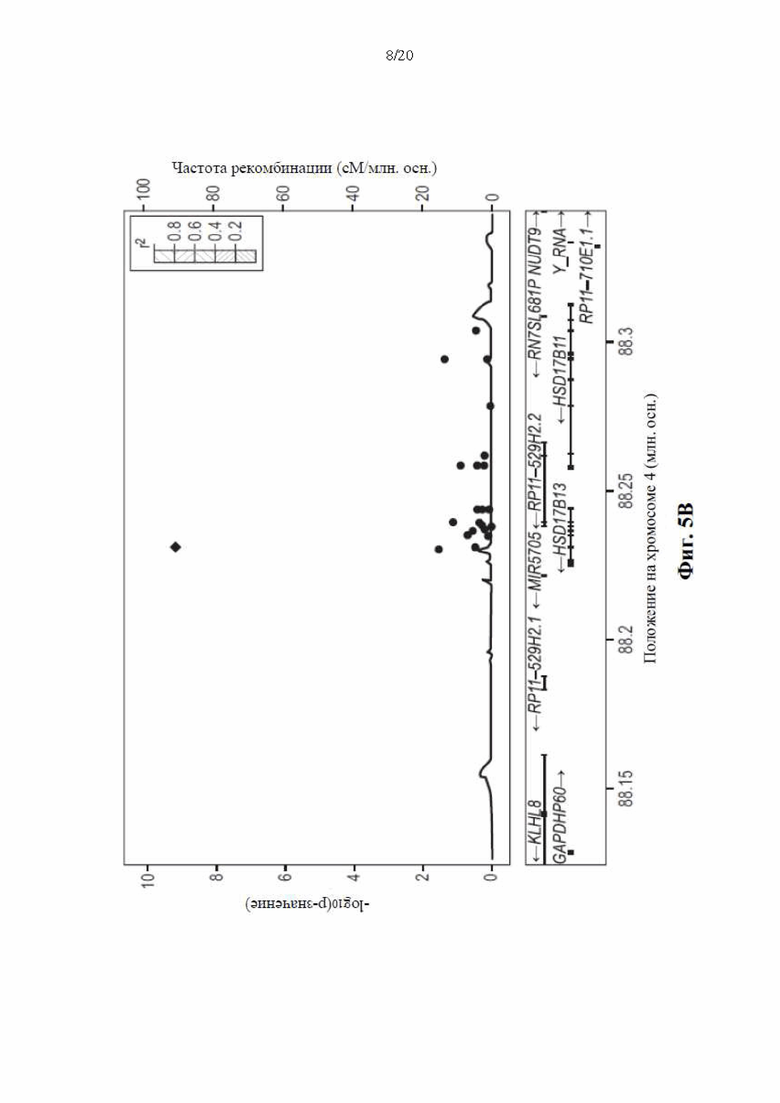
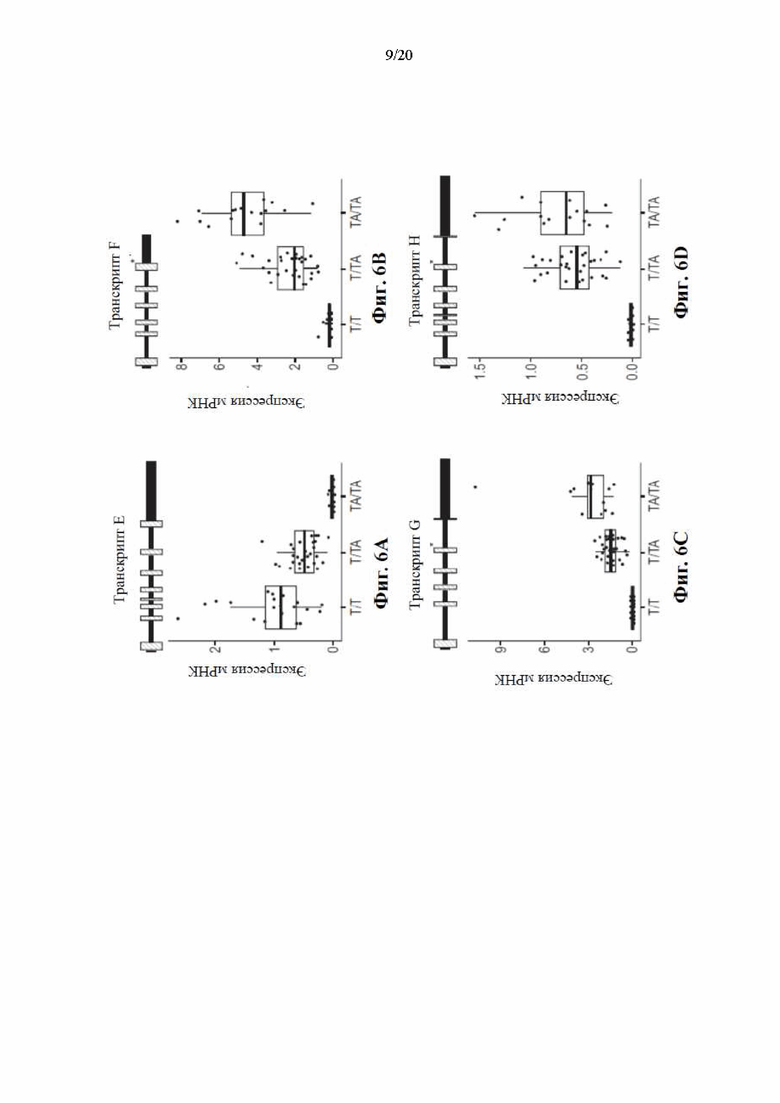
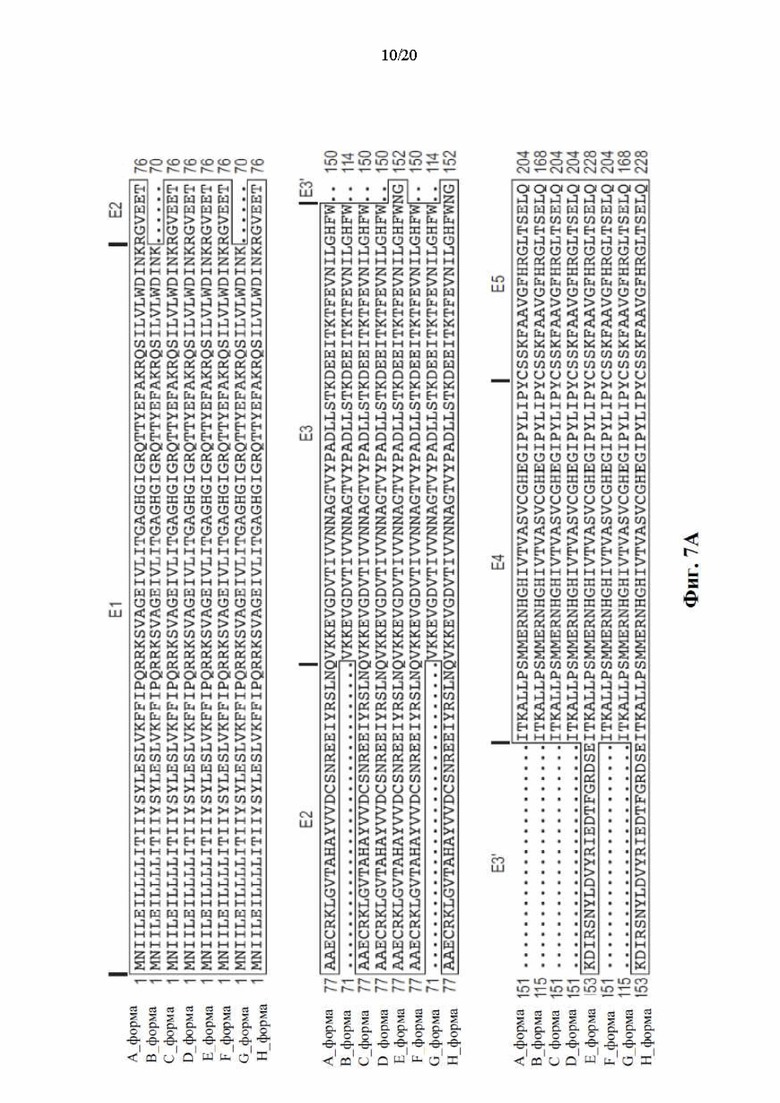
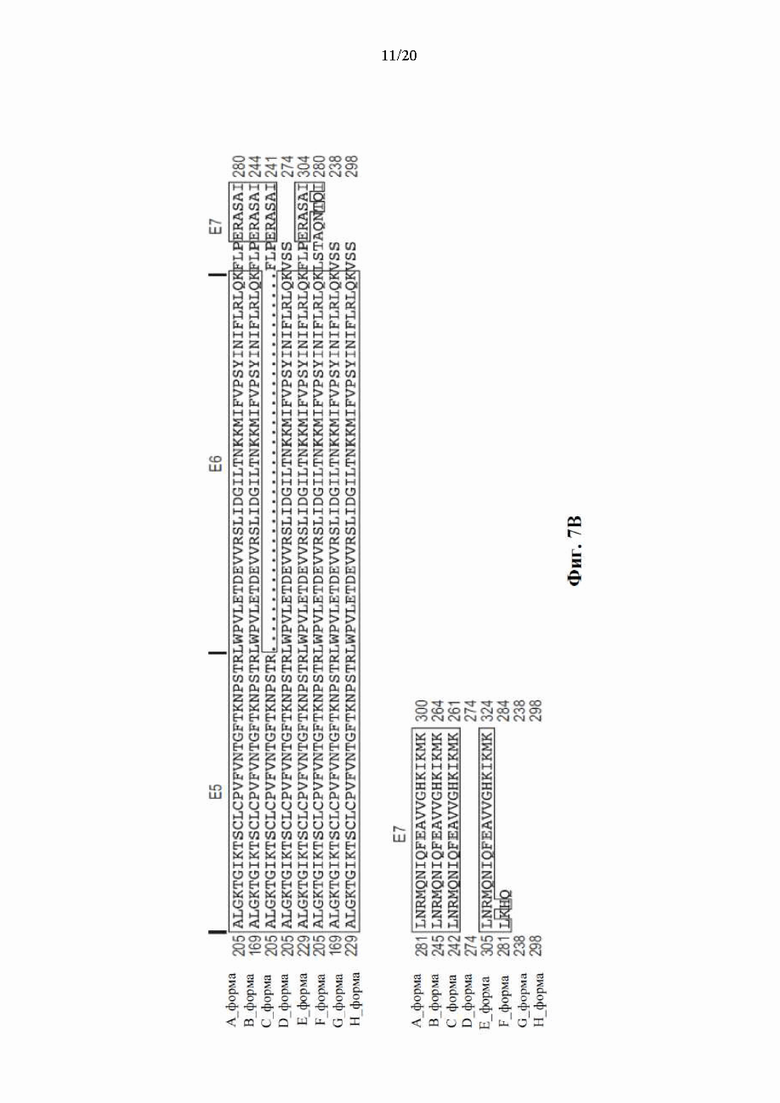
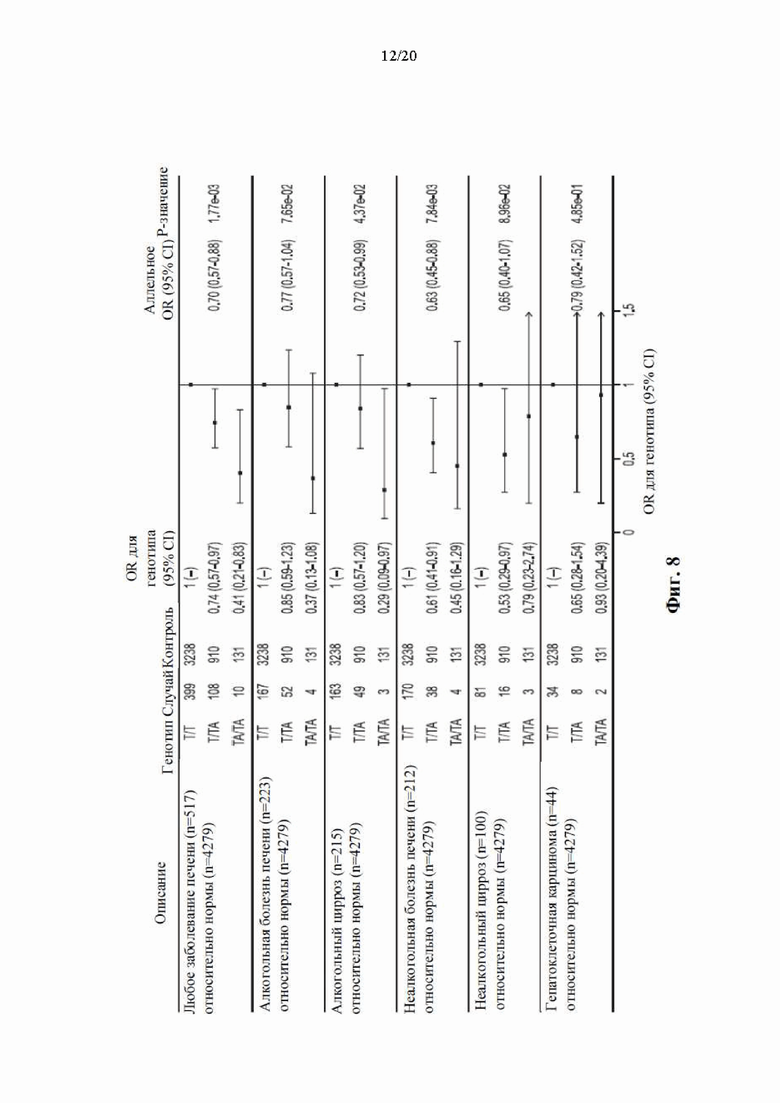
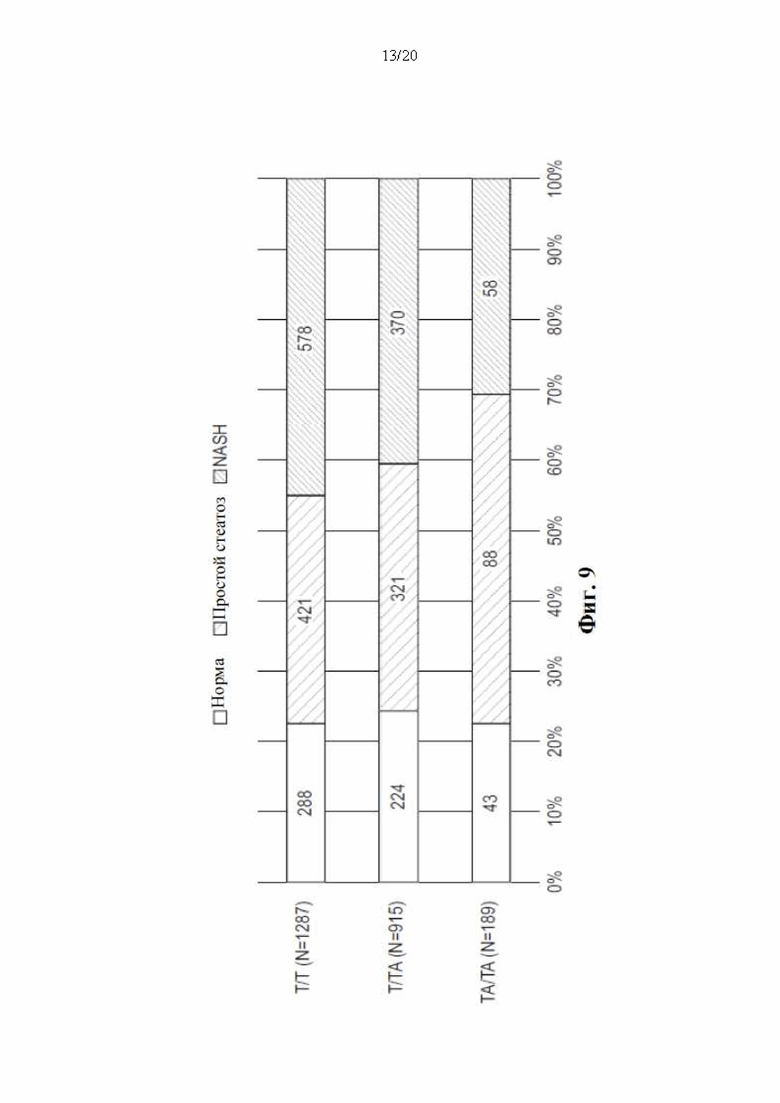
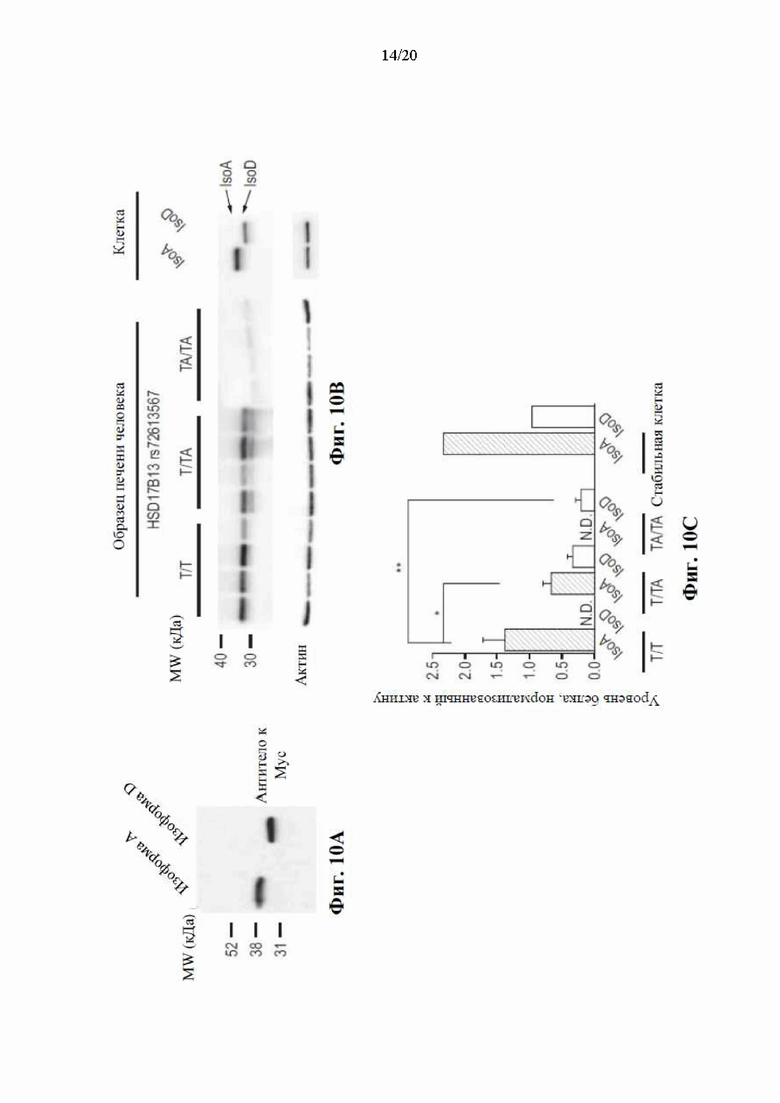
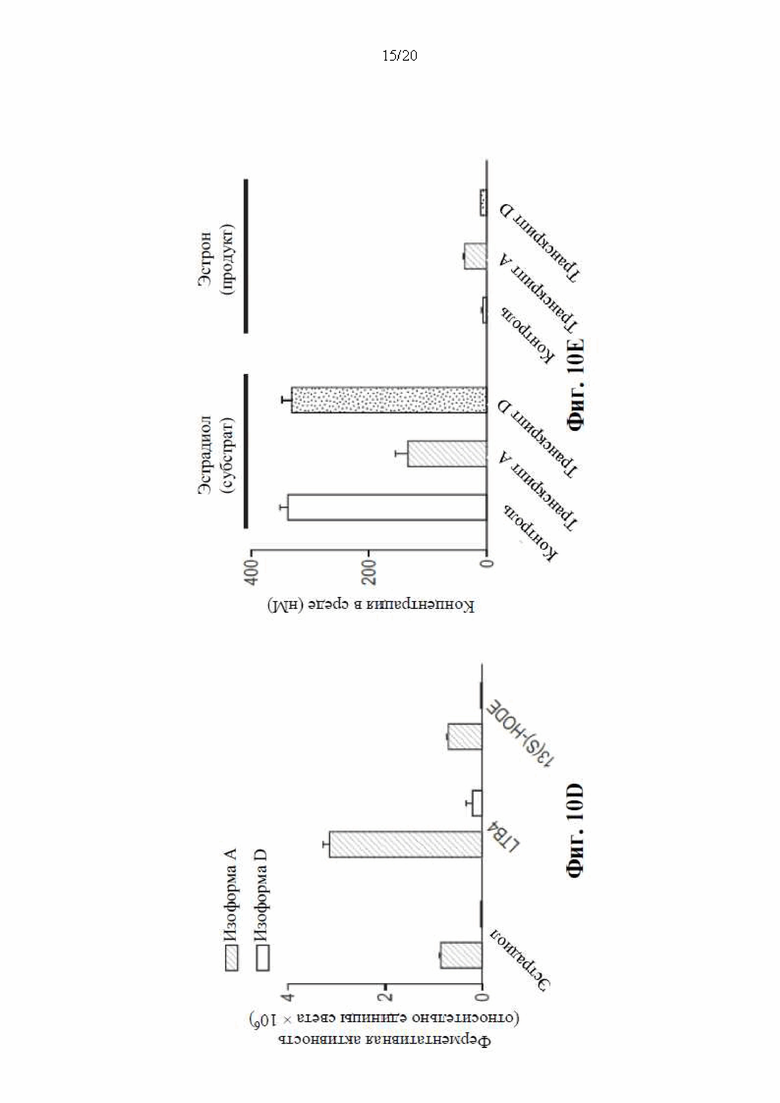
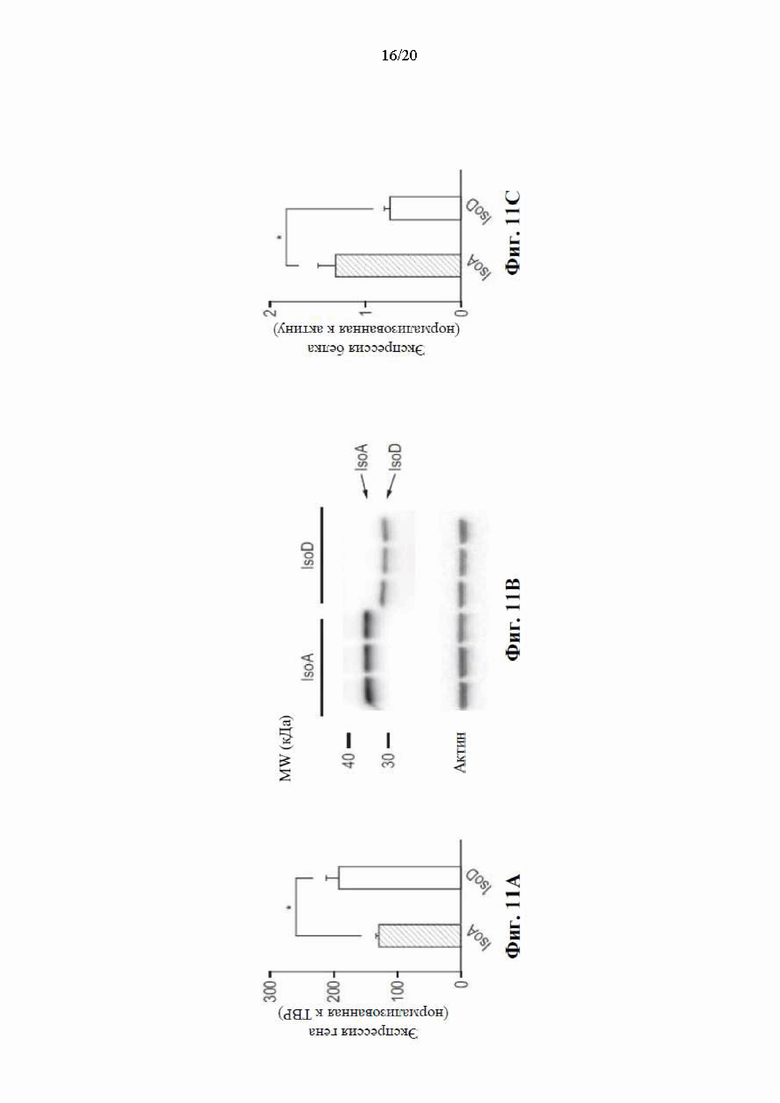

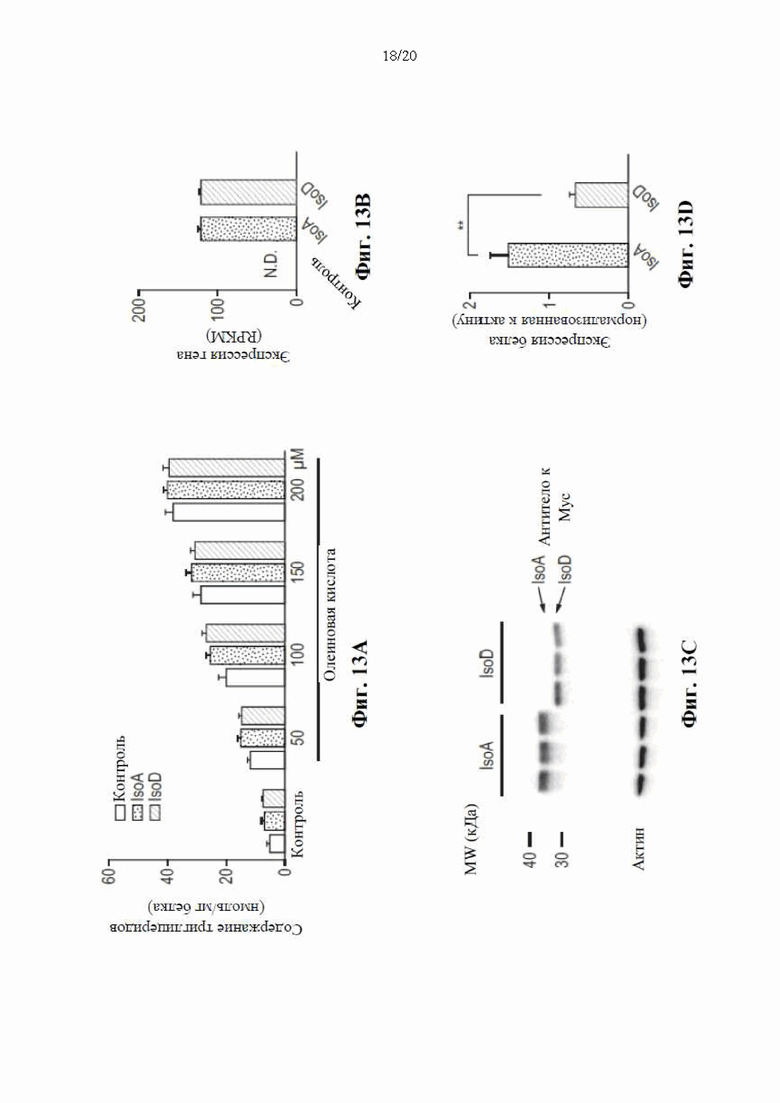
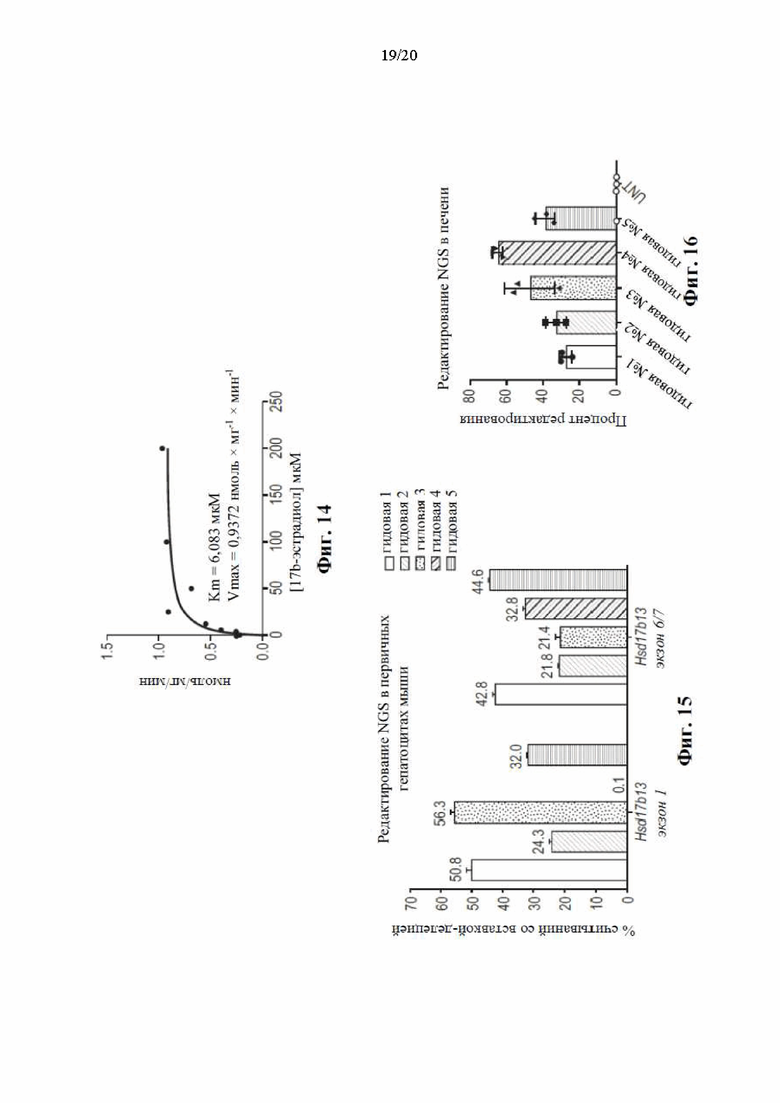
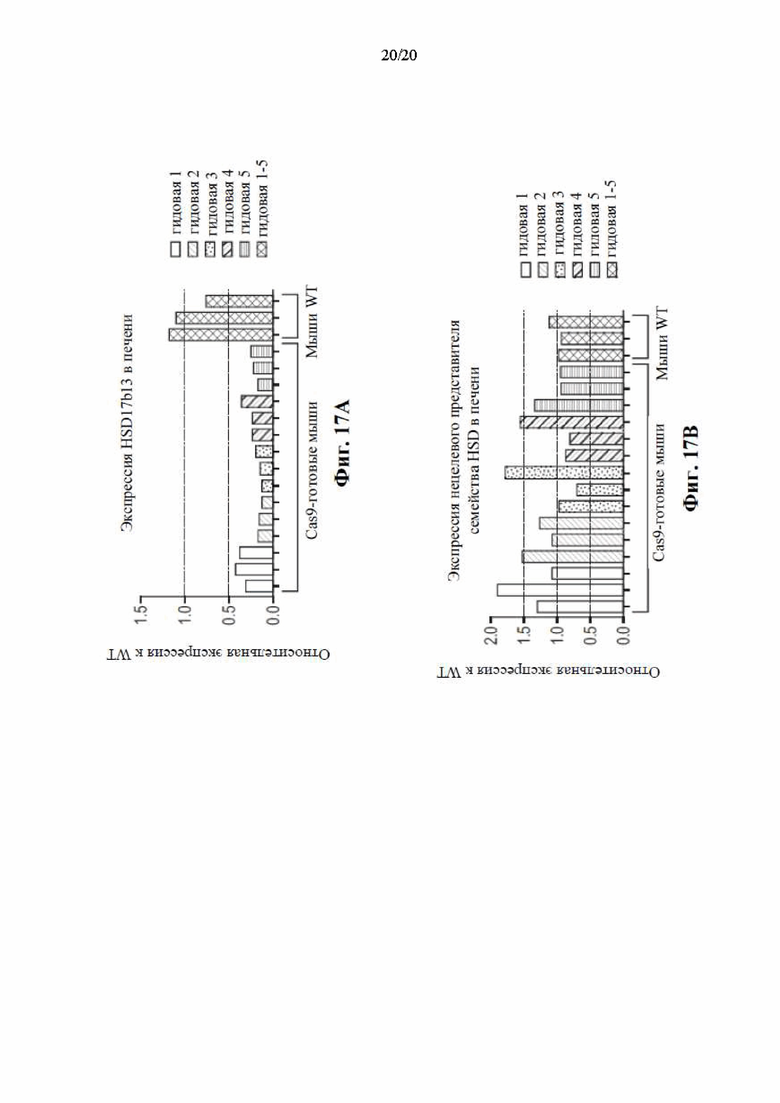
Изобретение относится к области биотехнологии, в частности к способу модификации гена HSD17B13 в клетке, предусматривающему введение в клетку белка Cas9 или нуклеиновой кислоты, кодирующей белок Cas9, и первой гидовой РНК или ДНК, кодирующей первую гидовую РНК, которая образует комплекс с белком Cas9 и нацелена на первую целевую для гидовой РНК последовательность в гене HSD17B13. Также раскрыт способ изменения экспрессии гена HSD17B13 в клетке, предусматривающий введение в клетку белка Cas9 или нуклеиновой кислоты, кодирующей белок Cas9, причем белок Cas9 является не обладающим нуклеазной активностью белком Cas9, слитым с доменом-репрессором транскрипции или с доменом-активатором транскрипции, и первой гидовой РНК или ДНК, кодирующей первую гидовую РНК, нацеленной на первую целевую для гидовой РНК последовательность в гене HSD17B13. Изобретение эффективно для терапии или профилактики заболеваний печени. 2 н. и 79 з.п. ф-лы, 17 ил., 13 табл., 4 пр.
1. Способ модификации гена HSD17B13 в клетке, предусматривающий введение в клетку:
(a) белка Cas9 или нуклеиновой кислоты, кодирующей белок Cas9; и
(b) первой гидовой РНК или ДНК, кодирующей первую гидовую РНК, причем первая гидовая РНК содержит первую часть РНК CRISPR (crRNA) и первую часть транс-активирующей РНК CRISPR (tracrRNA), причем первая гидовая РНК образует комплекс с белком Cas9 и нацелена на первую целевую для гидовой РНК последовательность в гене HSD17B13, причем первая целевая для гидовой РНК последовательность включает старт-кодон гена HSD17B13 или находится в непосредственной близости от него, и
причем белок Cas9 расщепляет первую целевую для гидовой РНК последовательность с образованием целенаправленной генетической модификации в гене HSD17B13.
2. Способ по п. 1, предусматривающий, что
белок Cas9 является обладающим нуклеазной активностью белком Cas9, а ген HSD17B13 не имеет тимина, вставленного между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1.
3. Способ по п. 1 или 2, предусматривающий, что:
(a) первая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 20-81 и 259-263; и/или
(b) первая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1423-1484 и 1643-1647; и/или
(c) первая гидовая РНК содержит любую из SEQ ID NO: 500-561, 730-791, 960-1021, 1190-1251, 720-724, 950-954, 1180-1184 и 1410-1414.
4. Способ по любому из пп. 1-3, предусматривающий, что первая целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 1 из SEQ ID NO: 1 или 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1 или 2.
5. Способ по любому из пп. 1-4, предусматривающий, что первая целевая для гидовой РНК последовательность находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона для гена HSD17B13 или содержит старт-кодон для гена HSD17B13.
6. Способ по любому из пп. 1-5, дополнительно предусматривающий введение в клетку второй гидовой РНК или ДНК, кодирующей вторую гидовую РНК,
причем вторая гидовая РНК содержит вторую часть РНК CRISPR (crRNA) и вторую часть транс-активирующей РНК CRISPR (tracrRNA),
причем вторая гидовая РНК образует комплекс с белком Cas9 и нацелена на вторую целевую для гидовой РНК последовательность в гене HSD17B13, и
причем вторая целевая для гидовой РНК последовательность включает старт-кодон гена HSD17B13 или находится в непосредственной близости от него.
7. Способ по п. 6, предусматривающий, что вторая целевая для гидовой РНК последовательность находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона для гена HSD17B13 или содержит старт-кодон для гена HSD17B13.
8. Способ по любому из пп. 1-7, предусматривающий, что способ приводит к разрушению старт-кодона гена HSD17B13.
9. Способ по любому из пп. 1-5, дополнительно предусматривающий введение в клетку второй гидовой РНК или ДНК, кодирующей вторую гидовую РНК,
причем вторая гидовая РНК содержит вторую часть РНК CRISPR (crRNA) и вторую часть транс-активирующей РНК CRISPR (tracrRNA),
причем вторая гидовая РНК образует комплекс с белком Cas9 и нацелена на вторую целевую для гидовой РНК последовательность в гене HSD17B13,
причем вторая целевая для гидовой РНК последовательность включает стоп-кодон гена HSD17B13 или находится в непосредственной близости от него, и
причем клетку модифицируют так, чтобы у нее была делеция кодирующего участка HSD17B13 между первой целевой для гидовой РНК последовательностью и второй целевой для гидовой РНК последовательностью.
10. Способ по п. 9, предусматривающий, что:
(a) вторая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 82-225; и/или
(b) вторая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1485-1628; и/или
(c) вторая гидовая РНК содержит любую из SEQ ID NO: 562-705, 792-935, 1022-1165 и 1252-1395.
11. Способ по п. 9 или 10, предусматривающий, что вторая целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 7 из SEQ ID NO: 1 или 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1 или 2.
12. Способ по любому из пп. 9-11, предусматривающий, что вторая целевая для гидовой РНК последовательность находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от стоп-кодона для гена HSD17B13 или содержит стоп-кодон для гена HSD17B13.
13. Способ по любому из пп. 9-12, предусматривающий, что делеции подвергают кодирующую последовательность гена HSD17B13.
14. Способ по любому из пп. 1-13, предусматривающий, что целенаправленную генетическую модификацию получают путем репарации отщепленной первой целевой для гидовой РНК последовательности посредством негомологичного соединения концов.
15. Способ по любому из пп. 1-14, предусматривающий, что способ приводит к потере функции гена HSD17B13.
16. Способ по любому из пп. 1-15, дополнительно предусматривающий введение вектора экспрессии в клетку, причем вектор экспрессии содержит рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1.
17. Способ по п. 16, предусматривающий, что рекомбинантный ген HSD17B13 является геном человека.
18. Способ по п. 16 или 17, предусматривающий, что рекомбинантный ген HSD17B13 представляет собой миниген HSD17B13, в котором были делетированы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа, причем делетированные сегменты содержат одну или несколько интронных последовательностей, и причем миниген HSD17B13 содержит интрон, соответствующий интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2.
19. Способ по любому из пп. 1-15, дополнительно предусматривающий введение вектора экспрессии в клетку, причем вектор экспрессии содержит нуклеиновую кислоту, кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15, и необязательно причем нуклеиновая кислота, кодирующая белок HSD17B13, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична SEQ ID NO: 7 при оптимальном выравнивании с SEQ ID NO: 7.
20. Способ по любому из пп. 1-15, дополнительно предусматривающий введение белка HSD17B13 или его фрагмента в клетку, причем белок HSD17B13 или его фрагмент по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15.
21. Способ по любому из пп. 1-15, дополнительно предусматривающий введение экзогенной донорной последовательности в клетку, причем экзогенная донорная последовательность рекомбинирует с целевым геномным локусом в гене HSD17B13 с получением целенаправленной генетической модификации.
22. Способ по п. 21, предусматривающий, что репарация гена HSD17B13 с помощью экзогенной донорной последовательности происходит посредством вставки, опосредованной негомологичным соединением концов.
23. Способ по п. 21, предусматривающий, что репарация гена HSD17B13 с помощью экзогенной донорной последовательности происходит посредством гомологичной репарации.
24. Способ по п. 23, предусматривающий, что экзогенная донорная последовательность содержит 5' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 5' от положения, соответствующего положению 12666 из SEQ ID NO: 2, и 3' гомологичное плечо, которое гибридизируется с целевой последовательностью, находящейся 3' от положения, соответствующего положению 12666 из SEQ ID NO: 2, причем экзогенная донорная последовательность рекомбинирует с геном HSD17B13.
25. Способ по п. 24, предусматривающий, что экзогенная донорная последовательность дополнительно содержит вставку нуклеиновой кислоты, фланкированную 5' гомологичным плечом и 3' гомологичным плечом.
26. Способ по п. 25, предусматривающий, что вставка нуклеиновой кислоты содержит тимин, и причем при рекомбинации экзогенной донорной последовательности с геном HSD17B13 тимин вставляется между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1.
27. Способ по любому из пп. 21-26, предусматривающий, что экзогенная донорная последовательность составляет от приблизительно 50 нуклеотидов до приблизительно 1 т.о. в длину.
28. Способ по п. 27, предусматривающий, что экзогенная донорная последовательность составляет от приблизительно 80 нуклеотидов до приблизительно 200 нуклеотидов в длину.
29. Способ по любому из пп. 21-28, предусматривающий, что экзогенная донорная последовательность является одноцепочечным олигодезоксинуклеотидом.
30. Способ по любому из пп. 1-28, предусматривающий, что первая гидовая РНК представляет собой одномолекулярную гидовую РНК, в которой первая часть crRNA связана с первой частью tracrRNA.
31. Способ по п. 30, предусматривающий, что первая гидовая РНК содержит последовательность, представленную под SEQ ID NO: 1420, 256, 257 или 258.
32. Способ по любому из пп. 1-28, предусматривающий, что первая часть crRNA и первая часть tracrRNA являются отдельными молекулами РНК.
33. Способ по п. 32, предусматривающий, что первая часть crRNA содержит последовательность, представленную под SEQ ID NO: 1421, и/или первая часть tracrRNA содержит последовательность, представленную под SEQ ID NO: 1422.
34. Способ по любому из пп. 1-33, предусматривающий, что первая гидовая РНК содержит модификацию, обеспечивающую измененную или регулируемую стабильность.
35. Способ по любому из пп. 1-34, причем способ предусматривает введение в клетку нуклеиновой кислоты, кодирующей белок Cas9, причем нуклеиновая кислота, кодирующая белок Cas9, представляет собой ДНК.
36. Способ по любому из пп. 1-34, причем способ предусматривает введение в клетку нуклеиновой кислоты, кодирующей белок Cas9, причем нуклеиновая кислота, кодирующая белок Cas9, представляет собой РНК.
37. Способ по любому из пп. 1-36, причем способ предусматривает введение в клетку первой гидовой РНК в форме РНК.
38. Способ по любому из пп. 1-36, причем способ предусматривает введение в клетку ДНК, кодирующей первую гидовую РНК.
39. Способ по любому из пп. 1-38, предусматривающий, что белок Cas9 или нуклеиновую кислоту, кодирующую белок Cas9, и/или первую гидовую РНК или ДНК, кодирующую первую гидовую РНК, вводят в клетку посредством доставки, опосредованной липидными наночастицами.
40. Способ по любому из пп. 1-39, предусматривающий, что белок Cas9 или нуклеиновую кислоту, кодирующую белок Cas9, и/или первую гидовую РНК или ДНК, кодирующую первую гидовую РНК, вводят в клетку посредством аденоассоциированного вируса.
41. Способ по любому из пп. 1-40, предусматривающий, что клетка представляет собой клетку мыши, клетку крысы или клетку человека.
42. Способ по п. 41, предусматривающий, что клетка представляет собой клетку печени человека, клетку печени мыши, клетку печени крысы, плюрипотентную клетку мыши или плюрипотентную клетку крысы.
43. Способ по п. 42, предусматривающий, что клетка представляет собой клетку печени человека.
44. Способ по любому из пп. 1-43, предусматривающий, что клетка представляет собой клетку человека, и причем:
(a) первая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 20-81; и/или
(b) первая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1423-1484; и/или
(c) первая гидовая РНК содержит любую из SEQ ID NO: 500-561, 730-791, 960-1021 и 1190-1251,
необязательно причем:
(a) первая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 20-41, любую из SEQ ID NO: 21-23, 33 и 35 или любую из SEQ ID NO: 33 и 35; и/или
(b) первая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1447-1468, любую из SEQ ID NO: 1448-1450, 1460 и 1462 или любую из SEQ ID NO: 1460 и 1462; и/или
(c) первая гидовая РНК содержит любую из SEQ ID NO: 524-545, 754-775, 984-1005 и 1214-1235, или любую из SEQ ID NO: 295-297, 525-527, 755-757, 985-987, 1215-1217, 307, 309, 537, 539, 767, 769, 997, 999, 1227 и 1229, или любую из SEQ ID NO: 307, 309, 537, 539, 767, 769, 997, 999, 1227 и 1229.
45. Способ по любому из пп. 1-42, предусматривающий, что клетка представляет собой клетку мыши, и причем:
(а) первая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 259-263; и/или
(b) первая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1643-1647; и/или
(c) первая гидовая РНК содержит любую из SEQ ID NO: 720-724, 950-954, 1180-1184 и 1410-1414.
46. Способ по любому из пп. 1-45, предусматривающий, что клетка находится в условиях ex vivo или in vivo.
47. Способ по п. 46, предусматривающий, что клетка находится в условиях in vivo.
48. Способ по п. 47, предусматривающий, что клетка представляет собой клетку печени, и причем белок Cas9 или нуклеиновую кислоту, кодирующую белок Cas9, и первую гидовую РНК или ДНК, кодирующую первую гидовую РНК, вводят в печень в условиях in vivo.
49. Способ по любому из пп. 1-48, предусматривающий, что клетка представляет собой клетку печени у субъекта, имеющего хроническое заболевание печени или подверженного риску его развития, необязательно причем хроническое заболевание печени представляет собой неалкогольную жировую болезнь печени, алкогольную жировую болезнь печени, цирроз или гепатоклеточную карциному, и необязательно причем субъект имеет стеатогепатит, фиброз, цирроз и/или гепатоклеточную карциному или подвержен риску их развития.
50. Способ изменения экспрессии гена HSD17B13 в клетке, предусматривающий введение в клетку:
(a) белка Cas9 или нуклеиновой кислоты, кодирующей белок Cas9, причем белок Cas9 является не обладающим нуклеазной активностью белком Cas9, слитым с доменом-репрессором транскрипции или с доменом-активатором транскрипции; и
(b) первой гидовой РНК или ДНК, кодирующей первую гидовую РНК, причем первая гидовая РНК содержит первую часть РНК CRISPR (crRNA) и первую часть транс-активирующей РНК CRISPR (tracrRNA), причем первая гидовая РНК образует комплекс с белком Cas9 и нацелена на первую целевую для гидовой РНК последовательность в гене HSD17B13, причем первая целевая для гидовой РНК последовательность включает старт-кодон гена HSD17B13 или находится в непосредственной близости от него, и
причем белок Cas9 связывает первую целевую для гидовой РНК последовательность и изменяет экспрессию гена HSD17B13.
51. Способ по п. 50, предусматривающий, что:
(а) белок Cas9 является не обладающим нуклеазной активностью белком Cas9, слитым с доменом-репрессором транскрипции, а ген HSD17B13 не имеет тимина, вставленного между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальной выравнивании гена HSD17B13 с SEQ ID NO: 1; или
(b) белок Cas9 является не обладающим нуклеазной активностью белком Cas9, слитым с доменом-активатором транскрипции, и причем ген HSD17B13 содержит тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальной выравнивании гена HSD17B13 с SEQ ID NO: 1.
52. Способ по п. 50 или 51, предусматривающий, что:
(a) первая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 20-81 и 259-263; и/или
(b) первая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1423-1484 и 1643-1647; и/или
(c) первая гидовая РНК содержит любую из SEQ ID NO: 500-561, 730-791, 960-1021, 1190-1251, 720-724, 950-954, 1180-1184 и 1410-1414.
53 Способ по любому из пп. 50-52, предусматривающий, что первая целевая для гидовой РНК последовательность находится в пределах участка, соответствующего экзону 1 из SEQ ID NO: 1 или 2 при оптимальном выравнивании гена HSD17B13 с SEQ ID NO: 1 или 2.
54. Способ по любому из пп. 50-53, предусматривающий, что первая целевая для гидовой РНК последовательность находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона для гена HSD17B13 или содержит старт-кодон для гена HSD17B13.
55. Способ по любому из пп. 50-54, дополнительно предусматривающий введение в клетку второй гидовой РНК или ДНК, кодирующей вторую гидовую РНК,
причем вторая гидовая РНК содержит вторую часть РНК CRISPR (crRNA) и вторую часть транс-активирующей РНК CRISPR (tracrRNA),
причем вторая гидовая РНК образует комплекс с белком Cas9 и нацелена на вторую целевую для гидовой РНК последовательность в гене HSD17B13, и
причем вторая целевая для гидовой РНК последовательность включает старт-кодон гена HSD17B13 или находится в непосредственной близости от него.
56. Способ по п. 55, предусматривающий, что вторая целевая для гидовой РНК последовательность находится в пределах приблизительно 10, 20, 30, 40, 50, 100, 200, 300, 400, 500 или 1000 нуклеотидов от старт-кодона для гена HSD17B13 или содержит старт-кодон для гена HSD17B13.
57. Способ по любому из пп. 50-56, дополнительно предусматривающий введение вектора экспрессии в клетку, причем вектор экспрессии содержит рекомбинантный ген HSD17B13, содержащий тимин, вставленный между нуклеотидами, соответствующими положениям 12665 и 12666 из SEQ ID NO: 1 при оптимальном выравнивании рекомбинантного гена HSD17B13 с SEQ ID NO: 1.
58. Способ по п. 57, предусматривающий, что рекомбинантный ген HSD17B13 является геном человека.
59. Способ по п. 57 или 58, предусматривающий, что рекомбинантный ген HSD17B13 представляет собой миниген HSD17B13, в котором были делетированы один или несколько несущественных сегментов гена относительно соответствующего гена HSD17B13 дикого типа, причем делетированные сегменты содержат одну или несколько интронных последовательностей, и причем миниген HSD17B13 содержит интрон, соответствующий интрону 6 из SEQ ID NO: 2 при оптимальном выравнивании с SEQ ID NO: 2.
60. Способ по любому из пп. 50-56, дополнительно предусматривающий введение вектора экспрессии в клетку, причем вектор экспрессии содержит нуклеиновую кислоту, кодирующую белок HSD17B13, который по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15, и необязательно причем нуклеиновая кислота, кодирующая белок HSD17B13, по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентична SEQ ID NO: 7 при оптимальном выравнивании с SEQ ID NO: 7.
61. Способ по любому из пп. 50-56, дополнительно предусматривающий введение белка HSD17B13 или его фрагмента в клетку, причем белок HSD17B13 или его фрагмент по меньшей мере на 90%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% или на 100% идентичен SEQ ID NO: 15.
62. Способ по любому из пп. 50-61, предусматривающий, что первая гидовая РНК представляет собой одномолекулярную гидовую РНК, в которой первая часть crRNA связана с первой частью tracrRNA.
63. Способ по п. 62, предусматривающий, что первая гидовая РНК содержит последовательность, представленную под SEQ ID NO: 1420, 256, 257 или 258.
64. Способ по любому из пп. 50-61, предусматривающий, что первая часть crRNA и первая часть tracrRNA являются отдельными молекулами РНК.
65. Способ по п. 64, предусматривающий, что первая часть crRNA содержит последовательность, представленную под SEQ ID NO: 1421, и/или первая часть tracrRNA содержит последовательность, представленную под SEQ ID NO: 1422.
66. Способ по любому из пп. 50-65, предусматривающий, что первая гидовая РНК содержит модификацию, обеспечивающую измененную или регулируемую стабильность.
67. Способ по любому из пп. 50-66, причем способ предусматривает введение в клетку нуклеиновой кислоты, кодирующей белок Cas9, причем нуклеиновая кислота, кодирующая белок Cas9, представляет собой ДНК.
68. Способ по любому из пп. 50-66, причем способ предусматривает введение в клетку нуклеиновой кислоты, кодирующей белок Cas9, причем нуклеиновая кислота, кодирующая белок Cas9, представляет собой РНК.
69. Способ по любому из пп. 50-68, причем способ предусматривает введение в клетку первой гидовой РНК в форме РНК.
70. Способ по любому из пп. 50-68, причем способ предусматривает введение в клетку ДНК, кодирующей первую гидовую РНК.
71. Способ по любому из пп. 50-70, предусматривающий, что белок Cas9 или нуклеиновую кислоту, кодирующую белок Cas9, и/или первую гидовую РНК или ДНК, кодирующую первую гидовую РНК, вводят в клетку посредством доставки, опосредованной липидными наночастицами.
72. Способ по любому из пп. 50-71, предусматривающий, что белок Cas9 или нуклеиновую кислоту, кодирующую белок Cas9, и/или первую гидовую РНК или ДНК, кодирующую первую гидовую РНК, вводят в клетку посредством аденоассоциированного вируса.
73. Способ по любому из пп. 50-72, предусматривающий, что клетка представляет собой клетку мыши, клетку крысы или клетку человека.
74. Способ по п. 73, предусматривающий, что клетка представляет собой клетку печени человека, клетку печени мыши, клетку печени крысы, плюрипотентную клетку мыши или плюрипотентную клетку крысы.
75. Способ по п. 74, предусматривающий, что клетка представляет собой клетку печени человека.
76. Способ по любому из пп. 50-75, предусматривающий, что клетка представляет собой клетку человека, и причем:
(а) первая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 20-81; и/или
(b) первая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1423-1484; и/или
(c) первая гидовая РНК содержит любую из SEQ ID NO: 500-561, 730-791, 960-1021 и 1190-1251,
необязательно причем:
(a) первая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 20-41, любую из SEQ ID NO: 21-23, 33 и 35 или любую из SEQ ID NO: 33 и 35; и/или
(b) первая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1447-1468, любую из SEQ ID NO: 1448-1450, 1460 и 1462 или любую из SEQ ID NO: 1460 и 1462; и/или
(c) первая гидовая РНК содержит любую из SEQ ID NO: 524-545, 754-775, 984-1005 и 1214-1235, или любую из SEQ ID NO: 295-297, 525-527, 755-757, 985-987, 1215-1217, 307, 309, 537, 539, 767, 769, 997, 999, 1227 и 1229, или любую из SEQ ID NO: 307, 309, 537, 539, 767, 769, 997, 999, 1227 и 1229.
77. Способ по любому из пп. 50-74, предусматривающий, что клетка представляет собой клетку мыши, и причем:
(a) первая целевая для гидовой РНК последовательность содержит любую из SEQ ID NO: 259-263; и/или
(b) первая гидовая РНК содержит нацеливающий на ДНК сегмент, который содержит любую из SEQ ID NO: 1643-1647; и/или
(c) первая гидовая РНК содержит любую из SEQ ID NO: 720-724, 950-954, 1180-1184 и 1410-1414.
78. Способ по любому из пп. 50-77, предусматривающий, что клетка находится в условиях ex vivo или in vivo.
79. Способ по п. 78, предусматривающий, что клетка находится в условиях in vivo.
80. Способ по п. 79, предусматривающий, что клетка представляет собой клетку печени, и причем белок Cas9 или нуклеиновую кислоту, кодирующую белок Cas9, и первую гидовую РНК или ДНК, кодирующую первую гидовую РНК, вводят в печень в условиях in vivo.
81. Способ по любому из пп. 50-80, предусматривающий, что клетка представляет собой клетку печени у субъекта, имеющего хроническое заболевание печени или подверженного риску его развития, необязательно причем хроническое заболевание печени представляет собой неалкогольную жировую болезнь печени, алкогольную жировую болезнь печени, цирроз или гепатоклеточную карциному, и необязательно причем субъект имеет стеатогепатит, фиброз, цирроз и/или гепатоклеточную карциному или подвержен риску их развития.
| WEN SU et al | |||
| Печь для сжигания твердых и жидких нечистот | 1920 |
|
SU17A1 |
| Говорящий кинематограф | 1920 |
|
SU111A1 |
| Способ очистки нефти и нефтяных продуктов и уничтожения их флюоресценции | 1921 |
|
SU31A1 |
| Устройство для преобразования электрического переменного тока в постоянный | 1928 |
|
SU11437A1 |
| LINLIN SUN et al | |||
| Разборный с внутренней печью кипятильник | 1922 |
|
SU9A1 |
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| KAREEM HASSAN et al | |||
Авторы
Даты
2021-11-30—Публикация
2018-01-19—Подача