ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к новым антителам, распознающим белок запрограммированной гибели клеток 1 (PD-1), новым антителам, распознающим белок гена 3 активации лимфоцитов (LAG-3), и биспецифическим PD-1/LAG-3-связывающим белкам, полученным на основе этих антител, таким как связывающие FIT-Ig белки. Антитела и биспецифические связывающие белки полезны для лечения иммунологических заболеваний и гематологических злокачественных новообразований.
УРОВЕНЬ ТЕХНИКИКИ
Белок запрограммированной гибели клеток 1 (PD-1)
Белок запрограммированной гибели клеток 1 (PD-1, CD279) является членом семейства рецепторов CD28, которое включает CD28, CTLA-4, ICOS, PD-1 и BTLA. Экспрессия PD-1 часто обнаруживается в иммунных клетках, таких как Т-клетки, В-клетки, моноциты и естественные киллерные (NK) клетки. PD-1 и аналогичные члены семейства представляют собой трансмембранные гликопротеины типа I, содержащие иммуноглобулин-подобный домен, напоминающий вариабельный домен Ig, который отвечает за связывание лиганда, и цитоплазматический хвост, который отвечает за связывание сигнальных молекул. Цитоплазматический хвост PD-1 содержит два сигнальных мотива на основе тирозина, ITIM (иммунорецепторный ингибирующий мотив на основе тирозина) и ITSM (иммунорецепторный мотив переключения на основе тирозина). Vivier et al., Immunol. Today, 18:286-291 (1997) and Chemnitz et al., J. Immunol., 173:945-954 (2004).
Идентифицированы два гликопротеиновых лиганда клеточной поверхности белка PD-1: лиганд запрограммированной смерти 1 (PD-L1, CD274, B7-H1) и PD-L2 (CD273, B7-DC), и показано, что они индуцируют внутриклеточную передачу сигнала, которая подавляет CD3- и CD28-опосредованную активацию Т-клеток. Riley, Immunol. Rev., 229:114-125 (2009). Это подавление активации Т-клеток, в свою очередь, приводит к снижению пролиферации Т-клеток, секреции IL-2, секреции IFN-γ и секреции других факторов роста и цитокинов. Freeman et al., J. Exp. Med., 192:1027-1034 (2000); Latchman et al., Nat. Immunol., 2:261-8 (2001); Carter et al., Eur. J. Immunol., 32:634-43 (2002); Ohigashi et al., Clin. Cancer Res., 11:2947-53 (2005). Предполагается, что передача сигналов через взаимодействие PD-1/PD-L1 осуществляет важные, незаменимые функции в иммунной системе, обеспечивая отрицательную регуляцию Т-клеточных ответов. Эта регуляция связана с развитием Т-клеток в тимусе, регуляцией хронических воспалительных реакций и поддержанием как периферической толерантности, так и иммунных привилегий. Ключевая роль этих функций проиллюстрирована на примере мышей с дефицитом PD-1 с проявленным аутоиммунным фенотипом. Дефицит PD-1 у мышей C57BL/6 приводит к развитию хронического прогрессирующего волчаночноподобного гломерулонефрита и артрита. У мышей Balb/c дефицит PD-1 приводит к развитию тяжелой кардиомиопатии из-за присутствия аутореактивных антител, специфичных к сердечной ткани.
После стимуляции Т-клеток PD-1 рекрутирует тирозинфосфатазу SHP-2 к мотиву ITSM, расположенному в цитоплазматическом хвосте, что приводит к дефосфорилированию эффекторных молекул, таких как CD3-ζ, PKC-θ и ZAP70, участвующих в CD3 T-клеточном сигнальном каскаде. Механизм, с помощью которого PD-1 подавляет Т-клеточные ответы, аналогичен механизму CTLA-4, но отличается от него, поскольку обе молекулы регулируют перекрывающийся набор сигнальных белков. Parry et al., Mol. Cell Biol., 25:9543-9553 (2005). Как правило, подавляющий сигнал, опосредованный PD-1, играет важную роль в иммунной толерантности. Bour-Jordan et al., Immunol. Rev., 241:180-205 (2011).
Увеличенная экспрессия PD-1 обнаруживается в опухоль-инфильтрирующих лимфоцитах (TIL), и, согласно опубликованным сообщениям, экспрессия лигандов PD-1 в опухолевых клетках наблюдается при различных раковых заболеваниях различных тканей, включая легкие, печень, желудок, почки, грудную железу, яичники, поджелудочную железу, меланоциты и пищевод. В целом, для многих типов опухолей экспрессия лиганда PD-1 на опухолевых клетках коррелирует с плохим прогнозом для онкологических пациентов. Okazaki and Honjo, Int. Immunol., 19:813-824 (2007).
Блокада взаимодействия PD-1/PD-L1 может привести к усилению опухолеспецифического Т-клеточного иммунитета и, следовательно, быть полезной для уничтожения опухолевых клеток иммунной системой. На мышиной модели агрессивного рака поджелудочной железы T. Nomi с сотрудниками продемонстрировали терапевтическую эффективность блокады PD-1/PD-L1, показав, что введение анти-PD-1 или анти-PD-L1 антитела приводит к существенному подавлению роста опухоли. Nomi et al., Clin. Cancer Res., 13:2151-2157 (2007). Блокада антителами оказалась эффективной в отношении инфильтрации в опухоль опухолевых CD8+ Т-клеток, приводя к усилению активности противоопухолевых эффекторов, включая IFN-γ, гранзим B и перфорин. В другом исследовании на мышиной модели плоскоклеточного рака блокада анти-PD-1 или анти-PD-L1 антителами существенно подавляла рост опухоли. Tsushima et al., Oral Oncol., 42:268-274 (2006).
Недавно было показано, что PD-1 имеет высокий уровень экспрессии в Т-клетках ВИЧ-инфицированных людей, и что экспрессия этого рецептора коррелирует с нарушением функции Т-клеток и прогрессированием заболевания. Day et al., Nature, 443:350-354 (2006); Trautmann et al., Nat. Med., 12:1198-1202 (2006). В обоих исследованиях блокада лиганда PD-L1 значительно увеличивала размножение ВИЧ-специфических, IFN-γ-продуцирующих клеток in vitro.
Соответственно, терапевтическая модуляция передачи сигналов PD-1 молекулами-антагонистами может изменить направление развития иммунных клеток от толерантности и реактивировать их на уничтожение рака и устранение хронических вирусных инфекций.
Ген 3 активации лимфоцитов (LAG-3)
Белок гена 3 активации лимфоцитов (LAG-3, CD223) является негативно регулируемым костимулирующим рецептором, который модулирует гомеостаз, пролиферацию и активацию Т-клеток. Sierro et al., Expert Opin. Ther. Targets, 15:91-101 (2010). Член суперсемейства иммуноглобулинов, LAG-3 представляет собой CD4-подобный белок, который, как и CD4, связывается с молекулами MHC класса II, но с двукратно превышающей аффинностью и в участке, отличном от участка для CD4. Huard et al., Proc. Natl Acad. Sci. USA, 94:5744-9 (1997). LAG-3 экспрессируется на активированных CD8+ T-клетках, γδ T-клетках, естественных киллерах, B-клетках, плазматических дендритных клетках и регуляторных T-клетках (Treg). Роль LAG-3 в качестве негативного регулятора Т-клеточных ответов определена на основе исследований на мышах, нокаутных по гену LAG-3, и использовании блокирующих анти-LAG-3 антител в модельных системах in vitro и in vivo. Sierro et al. (2010), op. cit.; Hannier et al., J. Immunol., 161:4058-65 (1998); Macon-Lemaitre et al., Immunology, 115:170-8 (2005); Workman et al., Eur. J. Immunol., 33:970-9 (2003). Как естественные, так и индуцированные Treg экспрессируют повышенные уровни LAG-3, которые необходимы для максимального проявления их подавляющей функции. Camisaschi et al., J. Immunol., 184:6545-6551 (2010); Huang, et al., Immunity, 21:503-513 (2004). Кроме того, эктопическая экспрессия LAG-3 на эффекторных CD4+ T-клетках снижает их пролиферативную способность и наделяет их регуляторной способностью, направленной против сторонних T-клеток. Huang, см. там же. Недавние исследования также показали, что высокий уровень экспрессии LAG-3 на истощенных Т-лимфоцитах, специфичных к вирусу лимфоцитарного хориоменингита (LCMV), способствует их переходу в невосприимчивое состояние и ограничивает противоопухолевые ответы CD8+ Т-клеток. Blackburn et al., Nat. Immunol., 10:29-37 (2009) и Grosso et al., J. Clin. Invest., 117:3383-3392 (2007).
Важная роль LAG-3 в противоопухолевом иммунном ответе и иммунном ответе на инфекцию делает его мишенью для иммунотерапии. Для лечения некоторых видов рака и хронических вирусных инфекций было предложено блокировать LAG-3 антагонистами, включая моноклональные антитела. Turnis et al., Eur. J. Immunol., 45:1892-1905 (2015).
По мере прояснения важности опосредованной PD-1 и LAG-3 передачи сигналов потребность в новых ингибирующих анти-PD-1 и анти-LAG-3 антителах, которые могли бы эффективно изменять функциональность Т-клеток или увеличивать реактивность опухолевых клеток к иммунным эффекторным клеткам, становится все более актуальной. Более того, разработка биспецифических молекул, сочетающих эффекты ингибирования PD-1 и LAG-3, могла бы привести к желаемому улучшению терапевтических подходов к лечению рака также.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к новым антителам, которые связываются с PD-1 с высокой аффинностью (сродством), и новым антителам, которые связываются с LAG-3 с высокой аффинностью. Изобретение также относится к PD-1/LAG-3 биспецифическим иммуноглобулинам, полученным на базе платформы Fabs-in-Tandem, (FIT-Ig), которые реагируют как с PD-1, так и с LAG-3. Антитела и биспецифические связывающие белки по настоящему изобретению могут блокировать LAG-3 на TIL для уменьшения популяции опухоль-инфильтрированных Treg-клеток или для восстановления TIL до цитотоксического фенотипа. Кроме того, антитела и биспецифические связывающие белки по изобретению можно использовать для подавления передачи сигналов PD-1/PD-L1, с целью реактивации опухоль-инфильтрованных цитотоксических Т-клеток. Биспецифические поливалентные связывающие белки, раскрытые в настоящей заявке, будут полезны в качестве биспецифических ингибиторов PD-1/LAG-3 для обеспечения синергетического комбинированного эффекта, позволяющего преодолеть подавление противоопухолевого иммунного ответа и, таким образом, улучшить результаты даже у тех пациентов, которые не реагируют или перестали реагировать либо на анти-PD-1 терапию, либо на анти-LAG-3 терапию, предоставляемые по отдельности.
Изобретение также относится к способам получения и применения анти-PD-1 и анти-LAG-3 антител и биспецифических PD-1/LAG-3-связывающих белков по настоящему изобретению, а также различных композиций, которые можно использовать в способах обнаружения PD-1 и/или LAG-3 в образце или в способах лечения или профилактики у индивидуума расстройства, ассоциированнго с активностью PD-1 и/или LAG-3.
В дополнительном варианте осуществления изобретение относится к биспецифическому связывающему Fabs-in-Tandem иммуноглобулиновому (FIT-Ig) белку, содержащему первую, вторую и третью полипептидные цепи,
причем указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, (i) VLA-CL-VHB-CH1-Fc, где CL слит непосредственно с VHB, или (ii) VHB-CH1-VLA-CL-Fc, где CH1 слит непосредственно с VLA;
причем указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHA-CH1; и
причем указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLB-CL;
причем VL представляет собой вариабельный домен легкой цепи, CL представляет собой константный домен легкой цепи, VH представляет собой вариабельный домен тяжелой цепи, CH1 представляет собой константный домен тяжелой цепи, Fc представляет собой Fc-область иммуноглобулина, A представляет собой эпитоп PD-1 или LAG-3, и B представляет собой эпитоп PD-1 или LAG-3, при условии, что A и B разные. В соответствии с настоящим изобретением такие связывающие FIT-Ig белки связываются как с PD-1, так и с LAG-3.
В предпочтительных вариантах осуществления Fab-фрагменты таких связывающих FIT-Ig белков включают домены VLA-CL и VHA-CH1 родительского антитела, связывающиеся с одним из целевых антигенов PD-1 или LAG-3, и включают домены VLB-CL и VHB-CH1 другого родительского антитела, связывающиеся с другим целевым антигеном PD-1 и LAG-3. Таким образом, спаривание VH-CH1/VL-CL приводит к образованию тандемных Fab-фрагментов, распознающих PD-1 и LAG-3.
В соответствии с настоящим изобретением PD-1/LAG-3-связывающий FIT-Ig белок преимущественно может содержать первую, вторую и третью полипептидные цепи, причем указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLPD-1-CL-VHLAG-3-CH1-Fc, где CL слит непосредственно с VHLAG-3, причем указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHPD-1-CH1; и причем указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLLAG-3-CL; причем VLPD-1 представляет собой вариабельный домен легкой цепи анти-PD-1 антитела, CL представляет собой константный домен легкой цепи, VHPD-1 представляет собой вариабельный домен тяжелой цепи анти-PD-1 антитела, CH1 представляет собой константный домен тяжелой цепи, VLLAG-3 представляет собой вариабельный домен легкой цепи анти-LAG-3 антитела, VHLAG-3 представляет собой вариабельный домен тяжелой цепи анти-LAG-3 антитела, и Fc представляет собой Fc-область иммуноглобулина. Преимущественно, в первой полипептидной цепи домены VLPD-1-CL являются такими же, как легкая цепь родительского анти-PD-1 антитела, домены VHPD-1-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-PD-1 антитела, домены VLLAG-3-CL являются такими же, как легкая цепь родительского анти-LAG-3 антитела, и домены VHLAG-3-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-LAG-3 антитела.
В альтернативных вариантах осуществления PD-1/LAG-3-связывающий FIT-Ig белок может преимущественно содержать первую, вторую и третью полипептидные цепи, причем указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLLAG-3-CL-VHPD-1-CH1-Fc, где CL слит непосредственно с VHPD-1, причем указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHLAG-3-CH1; и причем указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLPD-1-CL; причем VLPD-1 представляет собой вариабельный домен легкой цепи анти-PD-1 антитела, CL представляет собой константный домен легкой цепи, VHPD-1 представляет собой вариабельный домен тяжелой цепи анти-PD-1 антитела, CH1 представляет собой константный домен тяжелой цепи, VLLAG-3 представляет собой вариабельный домен легкой цепи анти-LAG-3 антитела, VHLAG-3 представляет собой вариабельный домен тяжелой цепи анти-LAG-3 антитела, и Fc представляет собой Fc-область иммуноглобулина. Преимущественно, в первой полипептидной цепи домены VLLAG-3-CL являются такими же, как легкая цепь родительского анти-LAG-3 антитела, домены VHLAG-3-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-LAG-3 антитела, домены VLPD-1-CL являются такими же, как легкая цепь родительского анти-PD-1 антитела, и домены VHPD-1-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-PD-1 антитела.
В альтернативных вариантах осуществления PD-1/LAG-3-связывающий FIT-Ig белок может преимущественно содержать первую, вторую и третью полипептидные цепи, причем указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHLAG-3-CH1-VLPD-1-CL-Fc, где CH1 слит непосредственно с VLPD-1, причем указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLLAG-3-CL; и в котором указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHPD-1-CH1; причем VLPD-1 представляет собой вариабельный домен легкой цепи анти-PD-1 антитела, CL представляет собой константный домен легкой цепи, VHPD-1 представляет собой вариабельный домен тяжелой цепи анти-PD-1 антитела, CH1 представляет собой константный домен тяжелой цепи, VLLAG-3 представляет собой вариабельный домен легкой цепи анти-LAG-3 антитела, VHLAG-3 представляет собой вариабельный домен тяжелой цепи анти-LAG-3 антитела, и Fc представляет собой Fc-область иммуноглобулина. Преимущественно, в первой полипептидной цепи домены VLLAG-3-CL являются такими же, как легкая цепь родительского анти-LAG-3 антитела, домены VHLAG-3-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-LAG-3 антитела, домены VLPD-1-CL являются такими же, как легкая цепь родительского анти-PD-1 антитела, и домены VHPD-1-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-PD-1 антитела.
В альтернативных вариантах осуществления PD-1/LAG-3-связывающий FIT-Ig белок преимущественно может содержать первую, вторую и третью полипептидные цепи, причем указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHPD-1-CH1-VLLAG-3-CL-Fc, где CH1 слит непосредственно с VLLAG-3, причем указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLPD-1-CL; и причем указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHLAG-3-CH1; причем VLPD-1 представляет собой вариабельный домен легкой цепи анти-PD-1 антитела, CL представляет собой константный домен легкой цепи, VHPD-1 представляет собой вариабельный домен тяжелой цепи анти-PD-1 антитела, CH1 представляет собой константный домен тяжелой цепи, VLLAG-3 представляет собой вариабельный домен легкой цепи анти-LAG-3 антитела, VHLAG-3 представляет собой вариабельный домен тяжелой цепи анти-LAG-3 антитела, и Fc представляет собой Fc-область иммуноглобулина. Преимущественно, в первой полипептидной цепи домены VLLAG-3-CL являются такими же, как легкая цепь родительского анти-LAG-3 антитела, домены VHLAG-3-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-LAG-3 антитела, домены VLPD-1-CL являются такими же, как легкая цепь родительского анти-PD-1 антитела, и домены VHPD-1-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-PD-1 антитела.
В приведенных выше формулах первой полипептидной цепи связывающего FIT-Ig белка Fc-область может быть природной или измененной Fc-областью. В конкретных вариантах осуществления Fc-область представляет собой человеческую Fc-область из IgG1, IgG2, IgG3, IgG4, IgA, IgM, IgE или IgD. В конкретных вариантах осуществления Fc представляет собой человеческий Fc из IgG1 или модифицированный человеческий Fc, такой как показано в таблице 6 ниже (SEQ ID NO: 28).
В одном из вариантов осуществления изобретения связывающие FIT-Ig белки по настоящему изобретению сохраняют одно или более свойств родительских антител, из которых получены последовательности Fab-фрагментов и которые включены в структуру FIT-Ig. В предпочтительных вариантах осуществления FIT-Ig сохраняет аффинность связывания с целевыми антигенами (например, LAG-3 и PD-1), сопоставимую с аффинность связывания родительских антител, что означает, что аффинность связывания связывающего FIT-Ig белка с целевыми антигенами PD-1 и LAG-3 не отличается более чем в 10 раз от аффинности связывания родительских антител с соответствующими целевыми антигенами, согласно результатам измерения методом поверхностного плазмонного резонанса или биослойной интерферометрии.
В одном из варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-679 SEQ ID NO: 78; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 20-240 SEQ ID NO: 83; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-236 SEQ ID NO: 86 (см. таблицу 27).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-684 SEQ ID NO: 88; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 20-235 SEQ ID NO: 91; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-236 SEQ ID NO: 93 (см. таблицу 28).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-679 SEQ ID NO: 95; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 20-242 SEQ ID NO: 98; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-236 SEQ ID NO: 100 (см. таблицу 29).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-684 SEQ ID NO: 102; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 20-235 SEQ ID NO: 105; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-236 SEQ ID NO: 107 (см. таблицу 30).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-679 SEQ ID NO: 140; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 144; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 146 (см. FIT107-1-6a-1; таблица 41).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-684 SEQ ID NO: 147; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 151; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 153 (см. FIT-107-1-6b-1; таблица 42).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-679 SEQ ID NO: 154; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 158; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 160 (см. FIT-107-1-6a-2; таблица 43).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-684 SEQ ID NO: 161; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 165; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 167 (см. FIT-107-1-6b-2; таблица 44).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-679 SEQ ID NO: 168; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 172; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 174 (см. FIT-107-1-6a-3; таблица 45).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-684 SEQ ID NO: 175; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 179; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 181 (см. FIT-107-1-6b-3; таблица 46).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-679 SEQ ID NO: 182; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 186; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 188 (см. FIT-107-1-7a-1; таблица 47).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-687 SEQ ID NO: 189; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 193; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 195 (см. FIT-107-1-7b-1; таблица 48).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-679 SEQ ID NO: 196; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 200; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 202 (см. FIT-107-1-7a-2; таблица 49).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-687 SEQ ID NO: 203; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 207; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 209 (см. FIT-107-1-7b-2; таблица 50).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-679 SEQ ID NO: 210; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 214; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 216 (см. FIT-107-1-7a-3; таблица 51).
В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-687 SEQ ID NO: 217; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 221; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из аминокислотной последовательности SEQ ID NO: 223 (см. FIT-107-1-7b-3; таблица 52).
Изобретение также относится к новым антителам, способным связывать человеческий PD-1, в которых антигенсвязывающий домен указанных антител содержит набор из шести CDR, т.е. CDR-H1, CDR-H2, CDR-H3, CDR-L1, CDR-L2 и CDR-L3, выбранных из группы наборов CDR, определенных ниже:
Аминокислотная последовательность
Изобретение также относится к новым антителам, способным связывать человеческий LAG-3, в которых антигенсвязывающий домен указанных антител содержит набор из шести CDR, т.е. CDR-H1, CDR-H2, CDR-H3, CDR-L1, CDR-L2 и CDR-L3, выбранных из группы наборов CDR, определенных ниже:
Аминокислотная последовательность
В одном из вариантов осуществления связывающий белок по изобретению представляет собой биспецифический, поливалентный иммуноглобулиновый связывающий белок, содержащий два или более антигенсвязывающих участков, причем по меньшей мере один антигенсвязывающий участок содержит набор CDR, выбранный из указанных выше наборов CDR 1, 2, 3 и 4, и по меньшей мере один антигенсвязывающий участок содержит приведенный выше набор 5 CDR.
В одном из вариантов осуществления анти-PD-1 антитело по изобретению содержит домены VH и VL, причем два вариабельных домена содержат аминокислотные последовательности, выбранные из группы, состоящей из:
В дополнительном варианте осуществления анти-LAG-3 антитело по изобретению содержит домены VH и VL, причем два вариабельных домена содержат аминокислотные последовательности, выбранные из группы, состоящей из:
* где SEQ ID NO: 226 является такой же, как SEQ ID NO: 135, за исключением аминокислоты в положении 56, которая представляет собой Ala (A) вместо Gly (G) (замена G55A по нумерации Кабат); и SEQ ID NO: 227 является такой же, как SEQ ID NO: 136, за исключением аминокислоты в положении 56, которая представляет собой Ala (A) вместо Gly (G) (замена G55A по нумерации Кабат).
В другом варианте осуществления анти-PD-1 антитело или анти-LAG-3 антитело можно использовать для получения производных связывающих белков, распознающих тот же самый целевой антиген, с помощью методов, хорошо известных в данной области. Такое производное может представлять собой, например, одноцепочечное антитело (scFv), Fab-фрагмент (Fab), Fab'-фрагмент, F(ab')2, Fv и связанный дисульфидной связью Fv.
В другом аспекте изобретения антитело или биспецифический связывающий белок по настоящему изобретению способны модулировать биологическую функцию PD-1, LAG-3 или обоих. В другом аспекте раскрытое в настоящем описании пнти-PD-1 антитело способно подавлять передачу сигнала PD-1/PD-L1. Подавление сигнала можно измерить с помощью анализа реакции смешанной культуры лимфоцитов, например описанного ниже в рабочих примерах. В другом аспекте раскрытое в настоящем описании анти-LAG-3 антитело способно подавлять взаимодействие MHC класса II/LAG-3. Такое подавление можно измерить с помощью анализа активации PBMC с помощью SEB, например описанного ниже в рабочих примерах. В другом аспекте раскрытый в настоящем описании PD-1/LAG-3-биспецифический связывающий FIT-Ig белок способен подавлять как передачу сигналов PD-1/PD-L1, так и взаимодействие MHC класса II/LAG-3.
В одном из вариантов осуществления анти-PD-1 антитело, раскрытое в настоящем описании, или его антигенсвязывающий фрагмент имеет константу скорости ассоциации (kon) с человеческим PD-1, составляющую по меньшей мере 1×104 M-1с-1, по меньшей мере 3×104 M-1с-1, по меньшей мере 5×104 M-1с-1, по меньшей мере 7×104 M-1с-1, по меньшей мере 9×104 M-1с-1, по меньшей мере 1×105 M-1с-1, по меньшей мере 1,1×105 M-1с-1, по меньшей мере 1×105 M-1с-1, по меньшей мере 1,25×105 M-1с-1, по меньшей мере 1,4×105 M-1с-1, по меньшей мере 1,5×105 M-1с-1, по меньшей мере, 3×105 M-1с-1 или более, измеренную методом поверхностного плазмонного резонанса или биослойной интерферометрии.
В другом варианте осуществления раскрытое в настоящем описании анти-PD-1 антитело или его антигенсвязывающий фрагмент имеет константу скорости диссоциации (koff) с человеческим PD-1 менее 1×10-3 с-1, менее 5×10-4 с-1, менее 3×10-4 с-1, менее 1×10-4 с-1, менее 8×10-5 с-1, менее 6×10-5 с-1, менее 4×10-5 с-1, менее 3×10-5 с-1 или менее 1×10-5 с-1, измеренную методом поверхностного плазмонного резонанса или биослойной интерферометрии.
В другом варианте осуществления раскрытое в настоящем описании анти-PD-1 антитело или его антигенсвязывающий фрагмент имеет константу диссоциации (KD) с человеческим PD-1 менее 1×10-8 М, менее 5×10-9 М, менее 3×10-9 М, менее 1×10-9 М, менее 8×10-10 М, менее 6×10-10 М, менее 4×10-10 М, менее 2×10-10 М или менее 1×10-10 М.
В одном из вариантов осуществления анти-LAG-3 антитело, раскрытое в настоящем описании, или его антигенсвязывающий фрагмент имеет константу скорости ассоциации (kon) с человеческим LAG-3, составляющую по меньшей мере 5×103 M-1с-1, по меньшей мере 7×103 M-1с-1, по меньшей мере 1×104 M-1с-1, по меньшей мере 3×104 M-1с-1, по меньшей мере 5×104 M-1с-1, по меньшей мере 7×104 M-1с-1, по меньшей мере 1×105 M-1с-1 или по меньшей мере 2×105 M-1с-1 или более, измеренную методом поверхностного плазмонного резонанса или биослойной интерферометрии.
В другом варианте осуществления раскрытое в настоящем описании анти-LAG-3 антитело или его антигенсвязывающий фрагмент имеет константу скорости диссоциации (koff) с человеческим LAG-3 менее 1,5×10-3 с-1, менее 1×10-3 с-1, менее 8×10-4 с-1, менее 6×10-4 с-1, менее 4×10-4 с-1, менее 2×10-4 с-1, менее 1×10-4 с-1, менее 9×10-5 с-1, менее 8×10-5 с-1, менее 7×10-5 с-1, менее 5×10-5 с-1, менее 4×10-5 с-1, менее 2×10-5 с-1 или менее 1×10-5 с-1, измеренную методом поверхностного плазмонного резонанса или биослойной интерферометрии.
В другом варианте осуществления раскрытое в настоящем описании анти-LAG-3 антитело или его антигенсвязывающий фрагмент имеет константу диссоциации (KD) с LAG-3 менее 5×10-7 M, менее 2×10-7 M, менее 1×10-7 M, менее 8×10-8 M, менее 6×10-8 M, менее 4×10-8 M; менее 2×10-8 М; менее 1×10-8 М; менее 8×10-9 М; менее 6×10-9 М, менее 4×10-9 М; менее 2×10-9 М; или менее 1×10-9 М.
В одном из вариантов осуществления биспецифический связывающий FIT-Ig белок по изобретению, способный связывать PD-1 и LAG-3, имеет константу скорости ассоциации (kon) с человеческим PD-1, составляющую по меньшей мере 5×103 M-1с-1, по меньшей мере 1×104 M-1с-1, по меньшей мере 5×104 M-1с-1, по меньшей мере 1×105 M-1с-1, по меньшей мере 3×105 M-1с-1 или по меньшей мере 5×105 M-1с-1 или более, и тот же связывающий белок имеет константу скорости ассоциации (kon) с человеческим LAG-3, составляющую по меньшей мере 5×103 M-1с-1, по меньшей мере 1×104 M-1с-1, по меньшей мере 5×104 M-1с-1, по меньшей мере 1×105 M-1с-1, по меньшей мере 3×105 M-1с-1 или по меньшей мере 5×105 M-1с-1 или более, измеренные методом поверхностного плазмонного резонанса или биослойной интерферометрии. В дополнительных вариантах осуществления биспецифический связывающий FIT-Ig белок по изобретению, способный связывать PD-1 и LAG-3, имеет константы скорости ассоциации (kon) с человеческими PD-1 и LAG-3, которые не более чем в 10 раз меньше значений kon для PD-1 и LAG-3 родительского анти-PD-1 антитела и родительского анти-LAG-3 антитела, соответственно, из которых были получены соответствующие анти-PD-1 и анти-LAG-3 специфичности связывающего FIT-Ig белка. Другими словами, сохраненная константа скорости ассоциации связывающего FIT-Ig белка с каждым антигеном (PD-1 или LAG-3) выше, равна или не более чем на один порядок меньше константы скорости ассоциации (kon), наблюдаемой у родительских антител, реагирующих с соответствующими PD-1 или LAG-3 антигенами. В контексте настоящего описания, PD-1/LAG-3-связывающий FIT-Ig белок может иметь улучшенную kon по отношению к одному или обоим антигенам по сравнению с kon для соответствующих антигенов, наблюдаемых у родительских антител, или значение kon для одного или обоих антигенов может оставаться по существу таким же, как у родительских антител, соответственно, или, в случае уменьшения величины kon для одного или обоих антигенов, наблюдаемого у связывающего FIT-Ig белка относительно значений kon родительских антител, такое уменьшение является не более чем 10-кратным. Предпочтительно уменьшение величины kon у FIT-Ig для конкретного антигена по сравнению с kon для этого антигена у родительского антитела составляет менее 50%, более предпочтительно менее 25%. Такие высокие значения kon биспецифического FIT-Ig по сравнению с kon родительских антител являются неожиданным достижением в данной области техники.
В одном из вариантов осуществления биспецифический связывающий FIT-Ig белок по изобретению, способный связывать PD-1 и LAG-3, имеет константу скорости диссоциации (koff) с человеческим PD-1 менее 2×0-4 с-1, менее 1×10-4 с-1, менее 5×10-5 с-1, менее 3×10-5 с-1, менее 2×10-5 с-1, менее 1×10-5 с-1 или менее 8×10-6 с-1, и тот же связывающий белок имеет константу скорости диссоциации (koff) с человеческим LAG-3 менее 2×10-4 с-1, менее 1×10-4 с-1, менее 5×10-5 с-1, менее 3×10-5 с-1, менее 2×10-5 с-1, менее 1×10-5 с-1 или менее 8×10-6 с-1, измеренную методом поверхностного плазмонного резонанса или биослойной интерферометрии.
В другом варианте осуществления биспецифический FIT-Ig связывающий белок по изобретению, способный связывать PD-1 и LAG-3, имеет константу диссоциации (KD) с PD-1 менее 2×10-8 М, менее 1×10-8 M, менее 5×10-9 M, менее 1×10-9 M, менее 6×10-10 M, менее 5×10-10 M, менее 3×10-10 M, менее 2×10-10 M, менее 1×10-10 M, менее 8×10-11 M, менее 6×10-11 M, менее 4×10-11 M или менее 1×10-11 M, и тот же связывающий белок имеет константу диссоциации (KD) с человеческим LAG-3 менее 2×10-8 M, менее 1×10-8 M, менее 5×10-9 M, менее 1×10-9 M, менее 6×10-10 M, менее 5×10-10 M, менее 3×10-10 M, менее 2×10-10 M, менее 1×10-10 M, менее 8×10-11 M, менее 6×10-11 M, менее 4×10-11 M или менее 1×10-11 M. В дополнительных вариантах осуществления биспецифический связывающий FIT-Ig белок по изобретению, способный связывать PD-1 и LAG-3, имеет константы диссоциации (KD) для человеческих PD-1 и LAG-3, которые не более чем в 10 раз отличаются от величин KD для PD-1 и для LAG-3 родительского анти-PD-1 антитела и родительского анти-LAG-3 антитела, соответственно, из которых были получены соответствующие анти-PD-1 и анти-LAG-3 специфичности связывающего FIT-Ig белка. Другими словами, у связывающего FIT-Ig белка сохранена аффинность связывания родительских антител к каждому антигену (PD-1 или LAG-3), на что указывает константа диссоциации (KD), которая находится в пределах одного порядка величины с KD, наблюдаемой у родительских антител, реагирующих с PD-1 или LAG-3 антигенами, соответственно. В контексте настоящего описания, PD-1/LAG-3-связывающий FIT-Ig белок может иметь улучшенную KD (т.е. может иметь более низкое значение KD; т.е. более сильное связывание) для одного или обоих антигенов по сравнению с KD для соответствующего антигена, наблюдаемой у родительских антител, или может иметь по существу такую же KD для одного или обоих антигенов, что и у родительских антител, соответственно, или наблюдаемая у связывающего FIT-Ig белка KD для одного или обоих антигенов может быть снижена (т.е. иметь более высокое значение KD, т.е. более сильное связывание) относительно KD родительского антитела, но в случае различия величин KD между связывающим FIT-Ig белом и родительским антителом, такое различие является не более чем 10-кратным. Предпочтительно PD-1/LAG-3-связывающий FIT-Ig белок имеет более низкое значение KD (более прочно связывается) для одного или обоих антигенов по сравнению со значениями KD для соответствующих антигенов, наблюдаемых у одного или обоих родительских антител. Сохранение аффинности связывания родительских анти-PD-1 и анти-LAG-3 антител в пределах ± 10-кратного изменения KD является неожиданным достижением в данной области техники.
Изобретение также относится к фармацевтическим композициям, содержащим по меньшей мере одно анти-PD-1 антитело или его антигенсвязывающий фрагмент, раскрытое в настоящем описании, и фармацевтически приемлемый носитель. Изобретение также относится к фармацевтическим композициям, содержащим по меньшей мере одно анти-LAG-3 антитело или его антигенсвязывающие фрагменты и фармацевтически приемлемый носитель. Изобретение также относится к фармацевтическим композициям, содержащим комбинацию анти-PD-1 и анти-LAG-3 антител, раскрытых в настоящем описании, или их антигенсвязывающих фрагментов, и фармацевтически приемлемый носитель. Изобретение также относится к биспецифическим, поливалентным связывающим иммуноглобулиновым белкам, реагирующим как с PD-1, так и с LAG-3, причем эти связывающие белки включают связывающие участки VH/VL, полученные из анти-PD-1 и анти-LAG-3 антител, раскрытых в настоящем описании. В частности, изобретение относится к фармацевтическим композициям, содержащим по меньшей мере один связывающий FIT-Ig белок, способный связывать PD-1 и LAG-3, и фармацевтически приемлемый носитель. Фармацевтические композиции по изобретению могут дополнительно содержать по меньшей мере один дополнительный активный ингредиент. В одном из вариантов осуществления такой дополнительный ингредиент включает, без ограничения, терапевтический агент, агент визуализации, цитотоксический агент, ингибитор ангиогенеза, ингибитор киназы, блокатор костимулирующей молекулы, блокатор молекулы адгезии, антитело с другой специфичностью или его функциональный фрагмент, детектируемую метку или репортер; агонист или антагонист определенного цитокина(ов), наркотик, нестероидное противовоспалительное средство (НПВП), анальгетик, анестетик, седативный препарат, местный анестетик, нейромышечный блокатор, противомикробный агент, кортикостероид, анаболический стероид, эритропоэтин, иммуноген, иммуносупрессивное средство, гормон роста, лекарственное вещество-заместитель гормона, радиофармацевтическое средство, антидепрессант, нейролептик, стимулятор (например, амфетамин, кофеин и т.д.), бета-агонист, ингаляционный стероид, адреналин или аналог, цитокин.
В другом варианте осуществления фармацевтическая композиция дополнительно содержит по меньшей мере один дополнительный терапевтический агент для лечения расстройства, при котором PD-1 и/или LAG-3-опосредованная сигнальная активность является вредной.
В следующем варианте осуществления изобретение относится к выделенным нуклеиновым кислотам, кодирующим одну или более аминокислотных последовательностей анти-PD-1 антитела по изобретению или его антигенсвязывающего фрагмента; выделенным нуклеиновым кислотам, кодирующим одну или более аминокислотных последовательностей анти-LAG-3 антитела по изобретению или его антигенсвязывающего фрагмента; и выделенным нуклеиновым кислотам, кодирующим одну или более аминокислотных последовательностей биспецифического связывающего Fabs-in-Tandem иммуноглобулинового (FIT-Ig) белка, способного связываться как с PD-1, так и LAG-3. Такие нуклеиновые кислоты могут быть вставлены в вектор для выполнения различных генетических анализов или для экспрессии, определения характерных признаков или улучшения одного или более свойств антитела или связывающего белка, раскрытых в настоящем описании. Вектор может содержать одну или более молекул нуклеиновых кислот, кодирующих одну или более аминокислотных последовательностей антитела или связывающего белка, раскрытых в настоящем описании, причем одна или более молекул нуклеиновых кислот функционально связаны с соответствующими транскрипционными и/или трансляционными последовательностями, которые обеспечивают экспрессию антитела или связывающего белка в конкретной клетке-хозяине, несущей вектор. Примеры векторов клонирования или экспрессии нуклеиновых кислот, кодирующих аминокислотные последовательности связывающих белков, раскрытых в настоящем описании, включают, без ограничения, pcDNA, pTT, pTT3, pEFBOS, pBV, pJV и pBJ и их производные.
Изобретение также относится к клетке-хозяину, содержащей вектор, содержащий нуклеиновую кислоту, кодирующую одну или более аминокислотных последовательностей антитела или связывающего белка, раскрытых в настоящем описании. Клетки-хозяева, используемые по изобретению, могут быть прокариотическими или эукариотическими. Примером прокариотической клетки-хозяина является Escherichia coli. Эукариотические клетки, используемые в качестве клеток-хозяев по изобретению, включают клетки протистов, клетки животных, клетки растений и клетки грибов. Примером клетки грибов является дрожжевая клетка, включая Saccharomyces cerevisiae. Типичная животная клетка, используемая в качестве клетки-хозяина по изобретению, включает, без ограничения, клетку млекопитающих, клетку птиц и клетку насекомых. Предпочтительные клетки млекопитающих включают, без ограничения, клетки CHO, клетки HEK и клетки COS. Клетка насекомых, используемая в качестве клетки-хозяина по изобретению, представляет собой клетку насекомых Sf9.
В другом аспекте изобретение относится к способу получения анти-PD-1 антитела или его функционального фрагмента, включающему культивирование клетки-хозяина, содержащей вектор экспрессии, кодирующий антитело или функциональный фрагмент, в культуральной среде в условиях, достаточных для того, чтобы вызвать экспрессию клеткой-хозяином антитела или фрагмента, способного связывать PD-1. В другом аспекте изобретение относится к способу получения анти-LAG-3 антитела или его функционального фрагмента, включающему культивирование клетки-хозяина, содержащей вектор экспрессии, кодирующий антитело или функциональный фрагмент, в культуральной среде в условиях, достаточных для того, чтобы вызвать экспрессию клеткой-хозяином антитела или фрагмента, способного связывать LAG-3. В другом аспекте изобретение относится к способу получения биспецифического, поливалентного связывающего белка, способного связывать PD-1 и LAG-3, в частности, связывающего FIT-Ig белка, который связывается с PD-1 и LAG-3, включающему культивирование клетки-хозяина, содержащей вектор экспрессии, кодирующий FIT-Ig связывающий белок, в культуральной среде в условиях, достаточных для того, чтобы вызвать экспрессию клеткой-хозяином связывающего белка, способного связывать PD-1 и LAG-3. Полученные таким образом белки можно выделить и использовать в различных композициях и способах, раскрытых в настоящем описании.
В одном из вариантов осуществления настоящее изобретение относится к способу лечения рака у нуждающегося в этом субъекта, включающему введение субъекту анти-PD-1 антитела или его PD-1-связывающего фрагмента, раскрытого в настоящем описании, причем указанное антитело или связывающий фрагмент способны связывать PD-1 и ингибировать PD-1/PD-L1 или PD-1/PD-L2-опосредованную передачу сигналов в клетке, экспрессирующей PD-1. В другом варианте осуществления настоящее изобретение относится к способу лечения рака у нуждающегося в этом субъекта, включающему введение субъекту анти-LAG-3 антитела или его LAG-3-связывающего фрагмента, раскрытого в настоящем описании, где указанное антитело или связывающий фрагмент способны связывать LAG-3 и ингибировать передачу сигналов, опосредованную MHC класса II/LAG-3, в клетке, экспрессирующей LAG-3. В другом варианте осуществления настоящее изобретение относится к способу лечения рака у нуждающегося в этом субъекта, включающему введение субъекту раскрытого в настоящем описании биспецифического связывающего FIT-Ig белка, способного связывать LAG-3 и PD-1, причем связывающий белок способен связывать LAG-3 и PD-1 и ингибировать передачу сигналов, опосредованную MHC класса II/LAG-3, в клетке, экспрессирующей LAG-3, и ингибировать PD-1/PD-L1 или PD-1/PD-L2-опосредованную передачу сигналов в клетке, экспрессирующей PD-1. В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-684 SEQ ID NO: 102; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 20-235 SEQ ID NO: 105; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-236 SEQ ID NO: 107 (см. таблицу 30). В следующем варианте осуществления связывающий FIT-Ig белок по настоящему изобретению связывает PD-1 и LAG-3 и состоит из первой полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-687 SEQ ID NO: 189; второй полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 20-235 SEQ ID NO: 192; и третей полипептидной цепи, содержащей, по существу состоящей или состоящей из последовательности аминокислот 23-236 SEQ ID NO: 194 (см. таблицу 48).
В другом варианте осуществления настоящее изобретение относится к способу лечения аутоиммунного заболевания или рака у нуждающегося в этом субъекта, причем связывающий белок способен связывать LAG-3 и PD-1, и причем аутоиммунное заболевание или рак представляет собой аутоиммунное заболевание или рак, обычно реагирующий на иммунотерапию. В другом варианте осуществления рак представляет собой рак, не связанный с иммунотерапией. В другом варианте осуществления рак представляет собой рак, который является рефрактерным или рецидивирующим злокачественным новообразованием. В другом варианте связывающий белок подавляет рост или выживание опухолевых клеток. В другом варианте осуществления рак выбирают из группы, состоящей из меланомы (например, метастатической злокачественной меланомы), рака почек (например, светлоклеточной карциномы), рака простаты (например, гормонорезистентной аденокарциномы простаты), аденокарциномы поджелудочной железы, рака груди, рака толстой кишки, рака легких (например, немелкоклеточного рака легких), рака пищевода, плоскоклеточного рака головы и шеи, рака печени, рака яичников, рака шейки матки, рака щитовидной железы, глиобластомы, глиомы, лейкоза, лимфомы и других злокачественных новообразований.
Способы лечения, раскрытые в настоящем описании, могут дополнительно включать введение нуждающемуся в этом субъекту, иммуностимулирующего адъюванта, такого как олигодезоксинуклеотид CpG (CpG ODN), содержащий полный или часть фосфодиэфирного или фосфоротиоатного остова. Например, в способе лечения по настоящему изобретению иммуностимулирующий адъювант может быть включен в композицию, содержащую антитело или связывающий FIT-Ig белок по настоящему изобретению, причем композицию вводят нуждающемуся в лечении субъекту. В другом варианте осуществления способ лечения по настоящему изобретению может включать этап введения нуждающемуся в лечении субъекту антитела или связывающего FIT-Ig белка по настоящему изобретению и отдельный этап введения субъекту иммуностимулирующего адъюванта до, одновременно или после этапа введения субъекту антитела или связывающего FIT-Ig белка по изобретению.
КРАТКОЕ ОПИСАНИЕ РИСУНКОВ
На фиг. 1A и 1B представлена гистограмма, показывающая уровни продуцирования IL-2 в реакции смешанной культуры лимфоцитов для оценки действия различных анти-PD-1 антител, раскрытых в настоящем описании, по сравнению с двумя рекомбинантными анти-PD-1 антителами, полученными из опубликованных последовательностей (ниволумаб и пембролизумаб) и контрольных человеческих и мышиных антител, направленных против нерелевантных антигенов («hIgG4» и «mIgG»). На фиг. 1A и 1B показаны отдельные тесты MLR с использованием реактивных лимфоцитов, полученных от разных доноров.
На фиг. 2A и 2B представлены гистограммы, показывающие уровни продуцирования гамма-интерферона (IFN-гамма) в реакции смешанной культуры лимфоцитов для оценки действия различных анти-PD-1 антител, раскрытых в настоящем описании, по сравнению с двумя рекомбинантными анти-PD-1 антителами, полученными из опубликованных последовательностей (ниволумаб и пембролизумаб) и контрольных человеческих и мышиных антител, направленных против нерелевантных антигенов («hIgG4» и «mIgG»). На фиг. 2A и 2B показаны отдельные тесты MLR с использованием реактивных лимфоцитов, полученных от разных доноров.
На фиг. 3 показаны гистограммы уровней продуцирования IL-2 в реакции смешанной культуры лимфоцитов для оценки действия различных гуманизированных анти-PD-1 антител, раскрытых в настоящем описании, по сравнению с рекомбинантным терапевтическим анти-PD-1 антителом, полученным из опубликованных последовательностей (ниволумаб), и контрольным человеческим антителом, направленным против нерелевантного антигена («HuF0323-1»).
На фиг. 4 представлены гистограммы уровней продуцирования IL-2 в реакции смешанной культуры лимфоцитов для оценки действия различных гуманизированных анти-PD-1 антител, раскрытых в настоящем описании, по сравнению с исходным мышиным mAb709, рекомбинантным терапевтическим анти-PD-1 антителом, полученным из опубликованной последовательности (ниволумаб), и контрольным человеческим антителом, направленным против нерелевантного антигена («HuF0323-1»).
На фиг. 5 представлены гистограммы уровней продуцирования IL-2 в реакции смешанной культуры лимфоцитов для оценки действия различных гуманизированных анти-PD-1 антител, раскрытых в настоящем описании, по сравнению с химерой с родительскими вариабельными доменами мышиного mAb713 (mAb713c), рекомбинантным терапевтическим анти-PD-1 антителом, полученным из опубликованных последовательностей (ниволумаб), и контрольным человеческим антителом, направленным против нерелевантного антигена («HuF0323-1»). На фиг. 3, 4 и 5 показаны отдельные тесты MLR с использованием реактивных лимфоцитов, полученных от разных доноров.
На фиг. 6 представлена гистограмма уровней продуцирования IL-2 в анализе SEB-опосредованной активации Т-клеток, в котором выполняют сравнение обратимости подавляющего эффекта Т-клеток при различных концентрациях двух раскрытых в настоящем описании мышиных анти-LAG-3 антител. Функциональность анти-LAG-3 антител по изобретению («3502-mAb746» и 3502-mAb747») сравнивают с рекомбинантным анти-LAG-3 mAb, полученным из опубликованной последовательности («BMS-986016»), рекомбинантным мышиным анти-LAG-3 антителом, полученным из опубликованной последовательности («BAP050»), и контрольными человеческими и мышиными антителами, направленными против нерелевантных антигенов («hIgG4» и «mIgG»).
На фиг. 7 представлена гистограмма, показывающая продуцирование IL-2 в анализе SEB-опосредованной активации Т-клеток, в котором выполняют сравнение обратимости подавляющего эффекта Т-клеток при различных концентрациях нескольких, раскрытых в настоящем описании биспецифических связывающих FIT-Ig белков, FIT107-1-2a. Функциональность FIT107-1-2a сравнивают с комбинацией рекомбинантного анти-LAG-3 mAb с известной последовательностью (BMS-986016) и рекомбинантного анти-PD-1 mAb с известной последовательностью (ниволумаб), и только с одним анти-PD-1 mAb (раскрытым в настоящем описании «PD-1», mAb709).
На фиг. 8 представлены кривые, показывающие относительные уровни продуцирования гамма-интерферона (IFN-g) в реакции смешанной культуры лимфоцитов для оценки действия биспецифического связывающего FIT-Ig белка FIT107-1-2a в различных концентрациях по сравнению с комбинацией рекомбинантного анти-LAG-3 mAb с известной последовательностью (BMS-986016) и рекомбинантного анти-PD-1 mAb с известной последовательностью (ниволумаб), и раскрытого в настоящем описании гуманизированного анти-PD-1 антитела, HumAb709-8.
На фиг.9 представлена гистограмма, показывающая продуцирование IL-2 в анализе SEB-опосредованной активации Т-клеток, в котором выполняют сравнение обратимости подавляющего эффекта Т-клеток при различных концентрациях химерного анти-LAG-3 антитела mAb747c и гуманизированного анти-LAG-3 антитела HumAb747-60. См. пример 13. Функциональность гуманизированного анти-LAG-3 антитела по изобретению (HumAb747-60) сравнивают с химерным анти-LAG-3 mAb, полученным с использованием мышиных вариабельных доменов, раскрытых в настоящем описании, и человеческим антителом, направленным против нерелевантного антигена («hIgG4», контроль).
На фиг.10 представлена гистограмма, показывающая продуцирование IL-2 в анализе SEB-опосредованной активации Т-клеток, в котором выполняют сравнение обратимости подавляющего эффекта Т-клеток при различных концентрациях химерного анти-LAG-3 антитела mAb747c и высокоаффинных вариантов гуманизированного анти-LAG-3 антитела HumAb747-60, включающих мутации, указанные после экспериментов по созреванию аффинности. См. пример 14. Функциональность измененных анти-LAG-3 антител по изобретению (HumAb747V-66 к HumAb747V-73) сравнивают с химерным анти-LAG-3 mAb, полученным с использованием мышиных вариабельных доменов, раскрытых в настоящем описании, и человеческим антителом, направленным против нерелевантного антигена («hIgG4», контроль).
На фиг.11 представлена гистограмма, показывающая продуцирование IL-2 в анализе SEB-опосредованной активации Т-клеток, в котором выполняют сравнение обратимости подавляющего эффекта Т-клеток при различных концентрациях связывающего FIT-Ig белка, специфичного как для целевого LAG-3, так и для целевого PD-1. См. пример 16.2. Функциональность PD-1/LAG-3-биспецифических FIT-Ig антител по изобретению сравнивают с комбинацией рекомбинантных моноклональных анти-PD-1 и анти-LAG-3 антител, полученных из опубликованных последовательностей («ниволумаб+BMS 986016»), и человеческого антитела, направленного против нерелевантного антигена («hIgG», контроль).
На фиг.12 представлена гистограмма, показывающая результаты анализа блокирования рецепторов, показывающая способность анти-LAG-3 антитела по изобретению (HumAb747V-67) и PD-1/LAG-3-связывающего FIT-Ig белка по изобретению (FIT107-1-7b-1) блокировать взаимодействие между человеческим LAG-3 и фибриноген-подобным белком 1 (FGL1). См. пример 16.5.
На фиг.13 представлена серия графиков, полученных по результатам оценки связывания клеточной поверхности с PD-1 и LAG-3, экспрессируемыми на Т-клетках. Результаты показывают, что биспецифический FIT-Ig белок FIT107-1-7b-1 распознает на Т-клетках как PD-1, так и LAG-3 поверхностные белки.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к новым анти-PD-1 антителам, новым анти-LAG-3 антителам, их антигенсвязывающим частям и поливалентным биспецифическим связывающим белкам, таким как Fabs-in-Tandem иммуноглобулины (FIT-Ig), которые связываются как с PD-1, так и LAG-3. Различные аспекты изобретения относятся к анти-PD-1 и анти-LAG-3 антителам и фрагментам антител, связывающим FIT-Ig белкам, которые связываются с человеческим PD-1 и человеческим LAG-3, и их фармацевтическим композициям, а также к нуклеиновым кислотам, рекомбинантным векторам экспрессии и клеткам-хозяевам для получения таких антител, функциональных фрагментов антител и связывающих белков. В объем настоящего изобретения также входят способы использования антител, функциональных фрагментов антител и биспецифических связывающих белков по изобретению для обнаружения человеческого PD-1, человеческого LAG-3 или обоих; для ингибирования активности человеческого PD-1 и/или человеческого LAG-3 либо in vitro, либо in vivo; и для лечения заболеваний, особенно рака, которые опосредованы связыванием PD-1 и/или LAG-3 с соответствующими лигандами, т.е. PD-1 и MHC класса II.
Если не указано иное, научные и технические термины, используемые в контексте настоящего изобретения, должны иметь значения, которые обычно понимают специалисты в данной области техники. Значение и объем терминов должны быть ясны, однако в случае любой скрытой неоднозначности приведенные в настоящем описании определения имеют приоритет над любым словарным определением или определением, найденным вне настоящего документа. Кроме того, если из контекста не следует иное, термины в единственном числе включают множественное число, а термины во множественном числе включают единственное число. В настоящей заявке использование союза «или» означает «и/или», если не указано иное. Кроме того, использование термина «включающий», а также других форм, таких как «включает» и «включенный», не является ограничивающим. Аналогично, такие термины, как «элемент» или «компонент», охватывают как элементы, так и компоненты, содержащие одну единицу, и элементы и компоненты, которые содержат более одной субъединицы, если специально не указано иное.
Как правило, представленные в настоящем описании обозначения и методы культивирования клеток и тканей, молекулярной биологии, иммунологии, микробиологии, генетики и химии белков и нуклеиновых кислот, а также методы гибридизации хорошо известны и широко используются в данной области. Способы и методы по настоящему изобретению обычно выполняют в соответствии с обычными способами, хорошо известными в данной области техники и описанными в общем виде и более конкретно в различных документах, которые цитируются и обсуждаются в настоящем описании, если не указано иное. Ферментативные реакции и методы очистки выполняют в соответствии со спецификациями производителя, как это обычно осуществляется в данной области техники или как раскрыто в настоящем описании. Обозначения, используемые в связи с лабораторными процедурами и методами аналитической химии, синтетической органической химии, медицинской и фармацевтической химии, используемые в настоящем описании, хорошо известны и широко используются в данной области. Для химического синтеза, химического анализа, приготовления фармацевтического препарата, составления лекарственной формы и доставки, а также лечения пациентов используются стандартные методы.
Для более полного понимания настоящего изобретения ниже приведено определение некоторых терминов.
Термин «полипептид» относится к любой полимерной аминокислотной цепи. Термины «пептид» и «белок» используются взаимозаменяемо с термином «полипептид», а также относятся к полимерной аминокислотной цепи. Термин «полипептид» охватывает природные или искусственные белки, фрагменты белка и полипептидные аналоги аминокислотной последовательности белка. Термин «полипептид» охватывает его фрагменты и варианты (включая фрагменты вариантов), если из контекста не следует иное. Для антигенного полипептида фрагмент полипептида необязательно содержит по меньшей мере один непрерывный или нелинейный эпитоп полипептида. Точные границы по меньшей мере одного фрагмента эпитопа могут быть подтверждены обычным специалистом в данной области. Фрагмент содержит по меньшей мере примерно 5 смежных аминокислот, например, по меньшей мере примерно 10 смежных аминокислот, по меньшей мере примерно 15 смежных аминокислот или по меньшей мере примерно 20 смежных аминокислот. Вариант полипептида является таким, как он описан в настоящей заявке.
Термин «выделенный белок» или «выделенный полипептид» представляет белок или полипептид, который в силу своего происхождения или источника происхождения не связан с естественным образом ассоциированными компонентами, которые сопровождают его в нативном состоянии; практически не содержит других белков того же вида; экспрессируется клеткой другого вида; или не встречается в природе. Таким образом, полипептид, который синтезирован химическим способом или синтезирован в клеточной системе, отличной от клетки, из которой он происходит в естественных условиях, будет «выделен» из компонентов, связанных с ним в естественных условиях. Белок также можно сделать практически свободным от компонентов, связанных с ним в естественных условиях, с помощью методов очистки белка, хорошо известных в данной области.
Термин «извлечение» относится к процессу освобождения химического вещества, такого как полипептид, от связанных с ним в естественных условиях компонентов путем выделения, например, с помощью методов очистки белков, хорошо известных в данной области.
Термин «биологическая активность» относится ко всем биологическим свойствам раскрытых в настоящем описании анти-PD-1 или анти-LAG-3 антител. Биологические свойства анти-PD-1 антител включают, без ограничения, связывание с белком PD-1; биологические свойства анти-LAG-3 антител включают, без ограничения, связывание с белками MHC класса II.
Термин «специфическое связывание» или «специфически связывающийся» применительно к взаимодействию антитела, связывающего белка или пептида со вторым химическим веществом означает, что взаимодействие зависит от наличия у второго химического вещества конкретной структуры (например, антигенной детерминанты или эпитопа). Например, антитело распознает и связывается с определенной структурой белка, и не с белками в целом. Если антитело является специфическим к эпитопу «А», наличие молекулы, содержащей эпитоп А (или свободный немеченый А), в реакции, содержащей меченный «А» и антитело, уменьшает количество меченного А, связанного с антителом.
Термин «антитело» в широком смысле относится к любой молекуле иммуноглобулина (Ig), состоящей из четырех полипептидных цепей, двух тяжелых (H) цепей и двух легких (L) цепей, или любого их функционального фрагмента, мутанта, варианта или производного, которое сохраняет важные особенности связывания эпитопа, свойственные молекуле Ig. Такие форматы мутантных, вариантных или производных антител известны в данной области. Их неограничивающие варианты осуществления обсуждаются ниже.
В полноразмерном антителе каждая тяжелая цепь состоит из вариабельной области тяжелой цепи (сокращенно обозначенной в настоящем описании VH) и константной области тяжелой цепи. Константная область тяжелой цепи содержит три домена: СН1, СН2 и СН3. Каждая легкая цепь состоит из вариабельной области легкой цепи (сокращенно обозначенной в настоящем описании VL) и константной области легкой цепи. Константная область легкой цепи состоит из одного домена CL. Области VH и VL могут быть дополнительно подразделены на области гипервариабельности, называемые областями, определяющими комплементарность (CDR), которые чередуются с более консервативными областями, называемыми каркасными областями (FR). Каждая из VH и VL содержит три CDR и четыре FR, расположенные в направлении от аминоконца к карбоксиконцу в следующем порядке: FR1, CDR1, FR2, CDR2, FR3, CDR3, FR4. Первую, вторую и третью CDR домена VH обычно обозначают как CDR-H1, CDR-H2 и CDR-H3; аналогично, первую, вторую и третью CDR домена VL обычно обозначают как CDR-L1, CDR-L2 и CDR-L3. Молекулы иммуноглобулинов могут быть любого типа (например, IgG, IgE, IgM, IgD, IgA и IgY), класса (например, IgG1, IgG2, IgG3, IgG4, IgA1 и IgA2) или подкласса.
Термин «Fc-область» используется для определения С-концевой области тяжелой цепи иммуноглобулина, которая может быть образована при расщеплении интактного антитела папаином. Fc-область может быть Fc-областью с нативной последовательностью или измененной Fc-областью. Fc-область иммуноглобулина обычно включает два константных домена, т.е. домен CH2 и домен CH3, и необязательно включает домен CH4, например, как в случае Fc-областей антител IgM и IgE. Fc-область антител IgG, IgA и IgD включает шарнирную область, домен CH2 и домен CH3. Напротив, Fc-область антител IgM и IgE не имеет шарнирной области, но содержит домен CH2, домен CH3 и домен CH4. Измененные Fc-области, имеющие замены аминокислотных остатков в Fc-части для изменения эффекторной функции антитела, известны в данной области (см., например, Winter et al., патенты США №№ 5648260 и 5624821). Fc-часть антитела опосредует несколько важных эффекторных функций, например, индукцию цитокинов, ADCC, фагоцитоз, комплементзависимую цитотоксичность (CDC) и период полужизни/выведения антитела и образование комплексов антиген-антитело. В зависимости от терапевтических целей, в некоторых случаях эти эффекторные функции являются желательными для терапевтического антитела, но в других случаях могут быть ненужными или даже вредными. Определенные изотипы человеческого IgG, в частности IgG1 и IgG3, опосредуют ADCC и CDC посредством связывания с FcgR и комплементом C1q, соответственно. В еще одном варианте осуществления в константной области антитела, например в Fc-области антитела, заменен по меньшей мере один аминокислотный остаток с изменением эффекторных функций антитела. Димеризация двух идентичных тяжелых цепей иммуноглобулина опосредуется димеризацией доменов CH3 и стабилизируется дисульфидными связями в шарнирной области, которая соединяет константные домены CH1 с константными доменами Fc (например, CH2 и CH3). Противовоспалительная активность IgG полностью зависит от сиалирования N-связанного гликана Fc-фрагмента IgG. Определены точные требования в отношении гликанов для обеспечения противовоспалительной активности, позволяющие создавать соответствующий фрагмент Fc IgG1, что, в свою очередь, позволяет создавать полностью рекомбинантный сиалированный Fc IgG1 со существенно повышенной эффективностью (см. Anthony et al., Science, 320:373-376 (2008)).
Термины «антигенсвязывающая часть» и «антигенсвязывающий фрагмент» или «функциональный фрагмент» антитела используются взаимозаменяемо и относятся к одному или более фрагментам антитела, которые сохраняют способность специфически связываться с антигеном, т.е. тем же самым антигеном (например, PD-1, LAG-3), с которым связывается полноразмерное антитело, от которого происходит указанная часть или фрагмент. Было показано, что антигенсвязывающая функция антитела может выполняться фрагментами полноразмерного антитела. Такие варианты осуществления антител также могут иметь биспецифический, двойной специфический или полиспецифический формат; специфическое связывание с двумя или более разными антигенами (например, PD-1 и другим антигеном, таким как LAG-3). Примеры связывающих фрагментов, охватываемых термином «антигенсвязывающая часть» антитела, включают (i) фрагмент Fab, моновалентный фрагмент, состоящий из доменов VL, VH, CL и CH1; (ii) F(ab')2 фрагмент, двухвалентный фрагмент, содержащий два фрагмента Fab, связанных дисульфидным мостиком в шарнирной области; (iii) Fd фрагмент, состоящий из доменов VH и CH1; (iv) фрагмент Fv, состоящий из доменов VL и VH одного плеча антитела, (v) фрагмент dAb (Ward et al., Nature, 341:544-546 (1989); публикация РСТ № WO 90/05144), который содержит единственный вариабельный домен; и (vi) выделенную определяющую комплементарность область (CDR). Кроме того, хотя два домена Fv фрагмента, VL и VH, кодируются отдельными генами, они могут быть объединены с помощью рекомбинантных методов посредством синтетического линкера, обеспечивающего образование одной белковой цепи, в которой VL и VH области спарены с образованием одновалентных молекул (известных как одноцепочечные Fv (scFv); см., например, Bird et al., Science 242:423-426, 1988 и Huston et al., Proc. Natl. Acad. Sci. USA 85:5879-5883 (1988)). Такие одноцепочечные антитела также охвачены термином «антигенсвязывающая часть» антитела и эквивалентными терминами, приведенными выше. Также включены другие формы одноцепочечных антител, такие как диатела. Диатела представляют собой двухвалентные биспецифические антитела, в которых домены VH и VL экспрессируются в одной полипептидной цепи, но с использованием линкера, который является слишком коротким, чтобы обеспечить спаривание между двумя доменами одной цепи, тем самым приводя к спариванию доменов с комплементарными доменами другой цепи и образованию двух сайтов связывания антигена (см., например, Holliger et al., Proc. Natl. Acad. Sci. USA, 90:6444-6448 (1993)). Такие участки связывания антитела известны в данной области (Kontermann и Dübel eds., Antibody Engineering (Springer-Verlag, New York, 2001), p. 790 (ISBN 3-540-41354-5)). Кроме того, одноцепочечные антитела также включают «линейные антитела», содержащие пару тандемных Fv-сегментов (VH-CH1-VH-CH1), которые вместе с комплементарными полипептидами легкой цепи образуют пару антигенсвязывающих областей (Zapata et al., Protein Eng., 8(10):1057-1062 (1995); и патент США № 5641870).
Константный (C) домен иммуноглобулина относится к константному домену тяжелой (CH) или легкой (CL) цепи. Аминокислотные последовательности константных доменов тяжелой цепи и легкой цепи IgG мыши и человека известны в данной области.
Термин «моноклональное антитело» или «mAb» относится к антителу, полученному из популяции по существу гомогенных антител, т.е. отдельные антитела, составляющие популяцию, являются идентичными, за исключением возможных встречающихся в природе мутаций, которые могут присутствовать в незначительных количествах. Моноклональные антитела являются высокоспецифичными и направлены против одной антигенной детерминанты (эпитопа). Более того, в отличие от препаратов поликлональных антител, которые обычно включают разные антитела, направленные против разных детерминант (эпитопов), каждое mAb направлено против одного детерминанта на антигене. Модификатор «моноклональный» не следует толковать как требующий получения антитела каким-либо конкретным способом.
Термин «человеческое антитело» включает антитела, имеющие вариабельные и константные области, полученные из последовательностей иммуноглобулинов человеческой зародышевой линии. Человеческие антитела по изобретению могут включать аминокислотные остатки, не кодируемые последовательностями иммуноглобулинов человеческой зародышевой линии (например, мутации, введенные случайным или сайт-специфическим мутагенезом in vitro, или соматические мутации in vivo), например, в CDR и, в частности, в CDR3. Однако термин «человеческое антитело» не включает антитела, в которых к человеческим каркасным последовательностям привиты последовательности CDR, полученные из зародышевой линии другого вида млекопитающих, такого как мышь.
Термин «рекомбинантное человеческое антитело» включает все человеческие антитела, которые получают, экспрессируют, создают или выделяют рекомбинантными способами, такие как антитела, экспрессируемые с использованием рекомбинантного вектора экспрессии, трансфицированного в клетку-хозяина, антитела, выделенные из рекомбинантной комбинаторной библиотеки человеческих антител (Hoogenboom, HR, Trends Biotechnol., 15:62-70 (1997); Azzazy and Highsmith, Clin. Biochem., 35:425-445 (2002); Gavilondo and Larrick, BioTechniques, 29:128-145 (2000); Hoogenboom and Chames, Immunol. Today, 21:371-378 (2000)), антитела, выделенные из животного (например, мыши), которое является трансгенным для человеческих генов иммуноглобулина (см., например, Taylor et al., Nucl. Acids Res., 20:6287-6295 (1992); Kellermann and Green, Curr. Opin. Biotechnol., 13:593-597 (2002); Little et al., Immunol. Today, 21:364-370 (2000)); или антитела, полученные, экспрессированные, созданные или выделенные любыми другими способами, которые включают сплайсинг последовательностей человеческого гена иммуноглобулина с другими ДНК-последовательностями. Такие рекомбинантные человеческие антитела имеют вариабельные и константные области, происходящие из последовательностей иммуноглобулинов человеческой зародышевой линии. Однако в некоторых вариантах осуществления такие рекомбинантные человеческие антитела подвергаются мутагенезу in vitro (или соматическому мутагенезу in vivo, когда используется животное, трансгенное в отношении последовательностей человеческого Ig) и, таким образом, аминокислотные последовательности областей VH и VL рекомбинантных антител представляют собой последовательности, которые, хотя и происходят из последовательностей VH и VL человеческой зародышевой линии и связаны с ними, могут не существовать в природе в репертуаре человеческих антител зародышевой линии in vivo.
Термин «химерное антитело» относится к антителам, которые содержат последовательности вариабельной области тяжелой и легкой цепей от одного вида и последовательности константной области от другого вида, такие как антитела, имеющие мышиные вариабельные области тяжелой и легкой цепей, связанные с человеческими константными областями.
Термин «CDR-привитое антитело» относится к антителам, которые содержат последовательности вариабельных областей тяжелой и легкой цепей одного вида, но в которых последовательности одной или более областей CDR VH и/или VL заменены последовательностями CDR другого вида, например, антитела, имеющие вариабельные области тяжелой и легкой цепей человека, в которых одна или более человеческих CDR заменены мышиными последовательностями CDR.
Термин «гуманизированное антитело» относится к антителам, которые содержат последовательности вариабельной области тяжелой и легкой цепей из не относящихся к человеку видов (например, мыши), но в которых, по меньшей мере, часть последовательности VH и/или VL была изменена «человекоподобной», т.е. более похожей на последовательности вариабельных областей человеческой зародышевой линии. Один из типов гуманизированного антитела представляет собой CDR-привитое антитело, в котором последовательности CDR из не относящихся к человеку видов (например, мыши) введены в человеческие каркасные последовательности VH и VL. Гуманизированное антитело представляет собой антитело или его вариант, производное, аналог или фрагмент, которое иммуноспецифично связывается с представляющим интерес антигеном и которое содержит каркасные области и константные области, имеющие по существу аминокислотную последовательность человеческого антитела, и при этом содержит определяющие комплементарность области (CDR), имеющие по существу аминокислотную последовательность не относящегося к человеческому антитела. В контексте настоящего описания термин «по существу» применительно к CDR относится к CDR, имеющей аминокислотную последовательность, которая на по меньшей мере 80%, по меньшей мере 85%, по меньшей мере, 90%, по меньшей мере 95%, по меньшей мере 98% или по меньшей мере 99% идентична аминокислотной последовательности CDR не относящегося к человеческому антитела. Гуманизированное антитело включает практически все из по меньшей мере одного, а обычно двух вариабельных доменов (Fab, Fab', F(ab')2, Fv), в которых все или по существу все области CDR соответствуют CDR не относящегося к человеческому иммуноглобулина (т.е. донорского антитела), и все или по существу все каркасные области являются областями из консенсусной последовательности человеческого иммуноглобулина. В одном из вариантов осуществления гуманизированное антитело также содержит по меньшей мере часть константной области (Fc) иммуноглобулина, как правило человеческого иммуноглобулина. В некоторых вариантах осуществления гуманизированное антитело содержит как легкую цепь, так и по меньшей мере вариабельный домен тяжелой цепи. Антитело также может включать участки СН1, шарнир, СН2, СН3 и СН4 тяжелой цепи. В некоторых вариантах осуществления гуманизированное антитело содержит только гуманизированную легкую цепь. В некоторых вариантах осуществления гуманизированное антитело содержит только гуманизированную тяжелую цепь. В конкретных вариантах осуществления гуманизированное антитело содержит только гуманизированный вариабельный домен легкой цепи и/или гуманизированную тяжелую цепь.
Гуманизированное антитело может быть выбрано из любого класса иммуноглобулинов, включая IgM, IgG, IgD, IgA и IgE, и любого изотипа, включая, без ограничения, IgG1, IgG2, IgG3 и IgG4. Гуманизированное антитело может содержать последовательности из более чем одного класса или изотипа, и для оптимизации желаемых эффекторных функций могут быть выбраны конкретные константные домены с помощью методов, хорошо известных в данной области.
Каркасная область и области CDR гуманизированного антитела не обязательно должны точно соответствовать родительским последовательностям, например CDR донорного антитела, или акцепторная каркасная область может быть модифицирована путем замены, вставки и/или удаления по меньшей мере одного аминокислотного остатка так, что CDR или остаток каркаса в этом участке не будет соответствовать ни донорному антителу, ни консенсусному каркасу. Однако в типичном варианте осуществления такие мутации не будут обширными. Обычно по меньшей мере 80%, предпочтительно по меньшей мере 85%, более предпочтительно по меньшей мере 90% и наиболее предпочтительно по меньшей мере 95% остатков гуманизированных антител будут соответствовать остаткам исходных последовательностей FR и CDR. Для сохранения конкретной петлевой структуры или для правильной ориентации последовательностей CDR, необходимой для контакта с целевым антигеном, часто используется обратная мутация в конкретном положении каркаса для восстановления той же аминокислоты, которая содержится в этом положении в донорском антителе.
Термин «CDR» относится к определяющим комплементарность участкам в последовательностях вариабельных доменов антитела. В каждой из вариабельных областей тяжелой цепи и легкой цепи содержатся три CDR, которые обозначены как CDR-H1, CDR-H2, CDR-H3, CDR-L1, CDR-L2 и CDR-L3. В контексте настоящего описания, термин «набор CDR» относится к группе из трех CDR, которые встречаются в одной вариабельной области, способной связывать антиген. Точные границы этих CDR различаются в зависимости от разных систем определения. Система, описанная Кабатом (Kabat et al., Sequences of Proteins of Immunological Interest (National Institutes of Health, Bethesda, Maryland (1987) и (1991)), не только обеспечивает однозначную систему нумерации остатков, применимую к любой вариабельной области антитела, но также позволяет определить точные границы остатков, определяющих три CDR.
Термин «нумерация по Кабат», которая известна в данной области, относится к системе нумерации аминокислотных остатков, которые более вариабельны (т.е. гипервариабельны), чем другие аминокислотные остатки в вариабельных областях тяжелой и легкой цепей антитела или его антигенсвязывающей части. См. Kabat et al., Ann. NY Acad. Sci., 190: 382-391 (1971); и Kabat et al., Sequences of Proteins of Immunological Interest, Fifth Edition, U.S. Department of Health and Human Services, NIH Publication No. 91-3242 (1991).
Рост и анализ обширных общедоступных баз данных аминокислотных последовательностей вариабельных областей тяжелых и легких цепей за последние двадцать лет привели к пониманию типичных границ между каркасными областями (FR) и последовательностями CDR в последовательностях вариабельных областей и позволили специалистам в данной области техники более точно определять CDR в соответствии с нумерацией Кабат, нумерацией Чотиа или других систем. См., например, Martin, «Protein Sequence and Structure Analysis of Antibody Variable Domains», In Kontermann and Dübel, eds., Antibody Engineering (Springer-Verlag, Berlin, 2001), глава 31, стр. 432-433.
Термин «поливалентный связывающий белок» означает связывающий белок, содержащий два или более антигенсвязывающих участка. Поливалентный связывающий белок предпочтительно конструируют таким образом, чтобы он имел три или более антигенсвязывающих участка и, как правило, не относился к встречающемуся в природе антителу. Термин «биспецифический связывающий белок» относится к связывающему белку, способному связывать две мишени с разной специфичностью. Связывающие «Fabs-in-Tandem иммуноглобулиновые» (FIT-Ig) белки по изобретению содержат два или более антигенсвязывающих участка и обычно представляют собой четырехвалентные связывающие белки. FIT-Ig может быть моноспецифическим, т.е. способным связывать один антиген, или полиспецифическим, т.е. способным связывать два или более антигенов. Предпочтительный FIT-Ig по настоящему изобретению связывает как PD-1, так и LAG-3 и, следовательно, является биспецифическим. Связывающий FIT-Ig белок, содержащий два полипептида с длинной (тяжелой) цепью V-C-V-C-Fc и четыре полипептида с короткой (легкой) цепью V-C, образует гексамер, демонстрирующий четыре антигенсвязывающих участка Fab (VH-CH1, спаренный с VL-CL, иногда обозначаемый как VH-CH1::VL-CL). Каждая половина FIT-Ig содержит полипептид тяжелой цепи и два полипептида легкой цепи, и спаривание комплементарных иммуноглобулинов, элементов VH-CH1 и VL-CL трех цепей, приводит к образованию двух Fab-структурированных антигенсвязывающих участков, расположенных в тандеме. В настоящем изобретении предпочтительно, чтобы домены иммуноглобулина, содержащие Fab-элементы, были непосредственно слиты с полипептидом тяжелой цепи без использования междоменных линкеров. Иными словами, N-концевой элемент V-C длинных (тяжелых) полипептидных цепей непосредственно слит своим С-концом с N-концом другого элемента V-C, который, в свою очередь, связан с C-концевой Fc-областью. В биспецифических связывающих FIT-Ig белках тандемно расположенные Fab-элементы будут реагировать с различными антигенами. Каждый связывающий участок антигена Fab включает вариабельный домен тяжелой цепи и вариабельный домен легкой цепи, всего шесть CDR в антигенсвязывающем участке.
Описание конструкции, экспрессии и характеристики молекул FIT-Ig представлено в публикации РСТ WO 2015/103072. Предпочтительный пример таких FIT-Ig молекул включает тяжелую цепь и две разные легкие цепи. Тяжелая цепь имеет структурную формулу VLA-CL-VHB-CH1-Fc, где CL слит непосредственно с VHB, или VHB-CH1-VLA-CL-Fc, где CH1 слит непосредственно с VLA, где VLA представляет собой вариабельный домен легкой цепи из родительского антитела, которое связывает антиген A, VHB представляет собой вариабельный домен тяжелой цепи из родительского антитела, которое связывает антиген B, CL представляет собой константный домен легкой цепи, CH1 представляет собой константный домен тяжелой цепи, и Fc представляет собой Fc-область иммуноглобулина (например, часть C -концевой шарнир-CH2-CH3 тяжелой цепи антитела IgG1). Две легкие полипептидные цепи FIT-Ig имеют формулы VHA-CH1 и VLB-CL, соответственно. В вариантах осуществления биспецифического FIT-Ig антиген A и антиген B представляют собой разные антигены или разные эпитопы одного и того же антигена. В настоящем изобретении один из A и B представляет собой PD-1, а другой - LAG-3.
Термин «активность» включает такие свойства, как способность специфически связывать целевой антиген, аффинность (сродство) антитела к антигену, способность нейтрализовать биологическую активность целевого антигена, способность ингибировать взаимодействие целевого антигена с его естественным рецептором(ами) и т.п. Предпочтительные антитела и связывающие белки по настоящему изобретению обладают способностью ингибировать связывание PD-1 с лигандом PD-L1, способностью ингибировать связывание LAG-3 с лигандом MHC класса II или и тем, и другим в случае раскрытых в настоящем описании биспецифических связывающих белков.
Термин «kon» (также «Kon», «kon») в контексте настоящего описания обозначает константы скорости ассоциации связывающего белка (например, антитела) с антигеном с образованием ассоциативного комплекса, например комплекса антитело/антиген, известного в данной области. «Kon» также известна под термином «константа скорости ассоциации» или «ka», которая используются в настоящем описании взаимозаменяемо. Это значение указывает скорость связывания антитела с его целевым антигеном или скорость образования комплекса антитело-антиген, как показано приведенным ниже уравнением:
Антитело («Ab») + Антиген («Ag») → AAb-Ag.
Термин «koff» (также «Koff», «koff»), используемый в настоящем описании, обозначает константы скорости диссоциации или «константы скорости диссоциации» связывающего белка (например, антитела) из ассоциативного комплекса (например, комплекса антитело/антиген), известного в данной области. Это значение указывает на скорость диссоциации антитела и его целевого антигена или разделения комплекса Ab-Ag с течением времени на свободное антитело и антиген, как показано приведенным ниже уравнением:
Ab+Ag ← Ab-Ag.
Термин «KD» (также «Kd»), используемый в настоящем описании, обозначает «константы равновесной диссоциации» и относится к значению, полученному при измерении титрования в равновесном состоянии или путем деления константы скорости диссоциации (koff) на константу скорости ассоциации (kon). Константа скорости ассоциации (kon), константа скорости диссоциации (koff) и константа равновесной диссоциации (KD) используются для представления аффинности связывания антитела с антигеном. Способы определения констант скорости ассоциации и диссоциации хорошо известны в данной области. Использование методов, основанных на флуоресценции, обеспечивает высокую чувствительность и возможность исследовать образцы в физиологических буферах в состоянии равновесия. Могут быть использованы другие экспериментальные подходы и инструменты, такие как анализ BIAcore® (анализ биомолекулярного взаимодействия) (например, с помощью инструмента, доступного от BIAcore International AB, компании GE Healthcare, Uppsala, Sweden). Биослойная интерферометрия (BLI) с использованием, например, системы Octet® RED96 (Pall FortéBio LLC) является еще одним методом анализа аффинности. Кроме того, также можно использовать анализ KinExA® (анализ кинетического исключения), доступный от Sapidyne Instruments (Boise, Idaho).
Термин «выделенная нуклеиновая кислота» означает полинуклеотид (например, геномную, кДНК, или синтетического происхождения или некоторую их комбинацию), который в результате вмешательства со стороны человека не связан со всеми или частью полинуклеотидов, с которыми он существует в природе; функционально связан с полинуклеотидом, с которым он не связан в природе; или не встречается в природе как часть более крупной последовательности.
Термин «вектор» в контексте настоящего описания относится к молекуле нуклеиновой кислоты, способной к переносу другой нуклеиновой кислоты, с которой она связана. Одним из типов векторов является «плазмида», которая относится к кольцевой двухцепочечной петле ДНК, в которую могут быть лигированы дополнительные сегменты ДНК. Другой тип вектора представляет собой вирусный вектор, в котором в вирусный геном могут быть лигированы дополнительные сегменты ДНК. Некоторые векторы способны к автономной репликации в клетке-хозяине, в которую они введены (например, бактериальные векторы, имеющие бактериальный источник репликации, и эписомальные векторы млекопитающих). При введении в клетку-хозяина в ее геном могут быть интегрированы другие векторы (например, неэписомальные векторы млекопитающих) и, таким образом, реплицироваться вместе с геномом-хозяином. Более того, некоторые векторы способны направлять экспрессию генов, с которыми они функционально связаны. Такие векторы называются в настоящем описании «рекомбинантными векторами экспрессии» (или просто «векторами экспрессии»). Как правило, векторы экспрессии, используемые в методах рекомбинантной ДНК, часто находятся в форме плазмид. В настоящем описании «плазмида» и «вектор» могут использоваться взаимозаменяемо, поскольку плазмида является наиболее часто используемой формой вектора. Однако изобретение предназначено для включения других форм векторов экспрессии, таких как вирусные векторы (например, ретровирусы с дефектом репликации, аденовирусы и аденоассоциированные вирусы), которые выполняют эквивалентные функции.
Термин «функционально связанный» относится к размещению рядом или смежному положению, при котором описанные таким образом компоненты находятся во взаимосвязи, позволяющей им функционировать по назначению. Контрольную последовательность, «функционально связанную» с кодирующей последовательностью, лигируют таким образом, чтобы экспрессия кодирующей последовательности происходила в условиях, совместимых для контрольной последовательности. «Функционально связанные» последовательности включают как контрольные экспрессирующие последовательности, которые примыкают к представляющему интерес гену, так и контрольные экспрессирующие последовательности, которые обеспечивают транс-воздействие или действуют на расстоянии, обеспечивая контроль представляющего интерес гена. Используемый в настоящем описании термин «контрольная экспрессирующая последовательность» относится к полинуклеотидным последовательностям, которые необходимы для осуществления экспрессии и процессинга кодирующих последовательностей, с которыми они лигированы. Контрольные экспрессирующие последовательности включают соответствующие последовательности инициации, терминации транскрипции, промоторные и энхансерные последовательности; эффективные сигналы обработки РНК, такие как сигналы сплайсинга и полиаденилирования; последовательности, стабилизирующие цитоплазматическую мРНК; последовательности, которые повышают эффективность трансляции (т.е. консенсусную последовательность Козак); последовательности, повышающие стабильность белка; и, если необходимо, последовательности, которые усиливают секрецию белка. Природа таких контрольных последовательностей различается в зависимости от организма-хозяина; в прокариотах такие контрольные последовательности обычно включают промотор, сайт связывания рибосомы и последовательность терминации транскрипции; у эукариот такие контрольные последовательности обычно включают промоторы и последовательность терминации транскрипции. Термин «контрольные последовательности» предназначен для включения компонентов, присутствие которых необходимо для экспрессии и процессинга, а также может включать дополнительные компоненты, присутствие которых является благоприятным, например лидерные или сигнальные последовательности и последовательности партнеров слияния.
«Трансформация» в контексте настоящего описания относится к любому процессу, посредством которого экзогенная ДНК проникает в клетку-хозяин. Трансформация может происходить в естественных или искусственных условиях с помощью различных методов, хорошо известных в данной области. Трансформация может основываться на любом известном способе встраивания последовательностей чужеродных нуклеиновых кислот в прокариотическую или эукариотическую клетку-хозяин. Метод выбирают в зависимости от трансформируемой клетки-хозяина, который может включать, помимо прочего, трансфекцию, вирусную инфекцию, электропорацию, липофекцию и бомбардировку частицами. Такие «трансформированные» клетки включают стабильно трансформированные клетки, в которых встроенная ДНК способна к репликации либо в виде автономно реплицирующейся плазмиды, либо в виде части хромосомы хозяина. Они также включают клетки, которые временно экспрессируют встроенную ДНК или РНК в течение ограниченных периодов времени.
Термин «рекомбинантная клетка-хозяин» (или просто «клетка-хозяин») предназначен для обозначения клетки, в которую была введена экзогенная ДНК. В одном из вариантов осуществления клетка-хозяин содержит две или более (например, несколько) нуклеиновых кислот, кодирующих антитела, такие как клетки-хозяева, описанные, например, в патенте США № 7,262,028. Такие термины предназначены для обозначения не только конкретной рассматриваемой клетки, но также и потомства такой клетки. Поскольку из-за мутаций или влияний окружающей среды в последующих поколениях могут происходить определенные модификации, такое потомство может фактически не быть идентичным родительской клетке, но оно все же включено в объем термина «клетка-хозяин», который используется настоящем описании. В одном из вариантов осуществления клетки-хозяева включают прокариотические и эукариотические клетки, выбранные из любого из Царств природы. В другом варианте осуществления эукариотические клетки включают клетки простейших, грибов, растений и животных. В другом варианте осуществления клетки-хозяева включают, без ограничения, линию прокариотических клеток Escherichia coli; линии клеток млекопитающих СНО, НЕК 293, COS, NS0, SP2 и PER.C6; линию клеток насекомых Sf9; и клетки гриба Saccharomyces cerevisiae.
Для рекомбинантной ДНК, синтеза олигонуклеотидов, а также для культивирования и трансформации тканей (например, электропорации, липофекции) могут использоваться стандартные методы. Ферментативные реакции и методы очистки могут быть выполнены в соответствии с техническими условиями производителя или в соответствии с обычными методами, используемыми в данной области техники, или представленными в настоящем описании. Вышеупомянутые методы и процедуры как правило могут выполняться в соответствии с обычными способами, хорошо известными в данной области техники и описанными в общем виде и более конкретно в ссылках, которые цитируются и обсуждаются в настоящем описании. См., например, Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd ed. (Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 1989).
Термин «агонист», используемый в настоящем описании, относится к модулятору, который при контакте с представляющей интерес молекулой вызывает увеличение определенной активности или функции молекулы по сравнению с величиной активности или функции, наблюдаемой в отсутствие агониста. Термины «антагонист» и «ингибитор» в контексте настоящего описания относятся к модуляторам, которые при контакте с представляющей интерес молекулой вызывают уменьшение определенной активности или функции молекулы по сравнению с величиной активности или функции, наблюдаемой в отсутствие антагониста. Конкретные представляющие интерес антагонисты включают антагонисты, которые блокируют или снижают биологическую или иммунологическую активность человеческого PD-1 и человеческого LAG-3.
Используемый в настоящем описании термин «эффективное количество» относится к количеству лечебного средства, которое достаточно для уменьшения или облегчения тяжести и/или продолжительности расстройства или одного или более его симптомов; предотвращения развития расстройства; вызова регрессии расстройства; предотвращения рецидива, развития или прогрессирования одного или более симптомов, связанных с расстройством; обнаружения расстройства; или усиления или улучшения профилактического или терапевтического эффекта(ов), обеспечиваемых другими терапевтическими средствами (например, профилактическими или терапевтическими агентами).
Продуцирование анти-PD-1 и анти-LAG-3 антител
Анти-PD-1 и анти-LAG-3 антитела по настоящему изобретению могут быть получены любым из нескольких способов, известных в данной области. Например, экспрессия из клеток-хозяев, причем вектор(ы) экспрессии, кодирующие тяжелую и легкую цепи, трансфицируют в клетку-хозяин стандартными методами. Подразумевается, что различные формы термина «трансфекция» охватывают широкий спектр методов, обычно используемых для введения экзогенной ДНК в прокариотическую или эукариотическую клетку-хозяина, например электропорацию, преципитацию фосфатом кальция, трансфекцию DEAE-декстраном и т.п. Хотя экспрессия антитела по настоящему изобретению возможна в прокариотических или эукариотических клетках-хозяевах, экспрессия антител в эукариотических клетках является предпочтительной и наиболее предпочтительной в клетках-хозяевах млекопитающих, поскольку такие эукариотические клетки (и, в частности, клетки млекопитающих) являются более подходящими, чем прокариотические клетки, для сборки и секретирования правильно свернутых и иммунологически активных антител.
Предпочтительные клетки-хозяева млекопитающих для экспрессии рекомбинантных антител по изобретению включают клетки яичника китайского хомячка (клетки CHO) (включая клетки dhfr-CHO, описанные в Urlaub and Chasin, Proc. Natl. Acad. Sci. USA, 77:4216-4220 (1980)), используемые с селективным маркером DHFR, например, как описано у Kaufman and Sharp, J. Mol. Biol., 159:601-621 (1982)), клетки миеломы NS0, клетки COS и клетки SP2. Когда рекомбинантные векторы экспрессии, кодирующие гены антител, вводят в клетки-хозяева млекопитающих, антитела получают путем культивирования клеток-хозяев в течение периода времени, достаточного для обеспечения экспрессии антитела в клетках-хозяевах или, более предпочтительно, секреции антитела в культуральную среду, в которой выращивают клетки-хозяева. Антитела можно выделить из культуральной среды с помощью стандартных методов очистки белков.
Клетки-хозяева также можно использовать для получения функциональных фрагментов антител, таких как фрагменты Fab или молекулы scFv. Следует понимать, что изменения вышеупомянутой процедуры находятся в пределах объема настоящего изобретения. Например, может быть желательна трансфекция клетки-хозяина ДНК, кодирующей функциональные фрагменты легкой цепи и/или тяжелой цепи антитела по настоящему изобретению. Технология рекомбинантной ДНК также может быть использована для удаления части или всей ДНК, кодирующей одну или обе легкие и тяжелые цепи, которая не является необходимой для связывания с представляющими интерес антигенами. Молекулы, экспрессируемые из таких усеченных молекул ДНК, также охвачены антителами по изобретению. Кроме того, путем перекрестного связывания антитела по настоящему изобретению со вторым антителом стандартными методами химической сшивки могут быть получены бифункциональные антитела, в которых одна тяжелая и одна легкая цепь представляют собой антитело по изобретению, а другая тяжелая и легкая цепь являются специфичными к антигену, который отличается от представляющих интерес антигенов.
В иллюстративной системе для рекомбинантной экспрессии антитела или его антигенсвязывающей части по изобретению рекомбинантный вектор экспрессии, кодирующий как тяжелую цепь антитела, так и легкую цепь антитела, вводят в клетки dhfr-CHO посредством трансфекции, опосредованной фосфатом кальция. В рекомбинантном векторе экспрессии каждый из генов тяжелой и легкой цепей антитела функционально связан с регуляторными элементами энхансера CMV/промотора AdMLP для управления высокими уровнями транскрипции генов. Рекомбинантный вектор экспрессии также несет ген DHFR, который позволяет отбирать клетки СНО, трансфицированные вектором, путем селекции/индуцированной метотрексатом амплификации. Отобранные трансфицированные клетки-хозяева культивируют, обеспечивая экспрессию тяжелой и легкой цепей антитела, и интактное антитело выделяют из культуральной среды. Получение рекомбинантного вектора экспрессии, трансфекцию клеток-хозяев, отбор трансфектантов, культивирование клеток-хозяев и извлечение антитела из культуральной среды осуществляют с помощью стандартных методов молекулярной биологии. Кроме того, изобретение относится к способу получения рекомбинантного анти-PD-1 или анти-LAG-3 антитела по настоящему изобретению путем культивирования трансфицированной клетки-хозяина по настоящему изобретению в подходящей культуральной среде до тех пор, пока продуцируется рекомбинантное антитело по настоящему изобретению. Способ может дополнительно включать выделение рекомбинантного антитела из культуральной среды.
Получение биспецифических FIT-Ig, связывающих PD-1 и LAG-3
Клинические исследования с использованием ингибиторов иммунных контрольных точек, таких как антитела, нацеленные на PD-1, PD-L1 и CTLA-4, дали многообещающие результаты, однако было замечено, что только часть пациентов изначально реагирует на эти ингибиторы, и растущие клинические данные указывают на то, что у значительной части пациентов, изначально отвечающих на лечение, в конечном итоге через несколько месяцев или лет наблюдается рецидив с летальным исходом, развитием устойчивости заболевания к лекарственному средству. Syn et al., Lancet Oncology, 18(12):e731-e741 (2017). Как LAG-3, так и PD-1 коэкспрессируются на толеризованных лимфоцитах, инфильтрированных опухолью (TILS), что способствует подавлению иммунитета в опухолях; и в качестве средства восстановления противоопухолевой функции CD8+ Т-клеток была предложена двойная блокада LAG-3 и PD-1. Matsuzaki et al., Proc. Natl. Акад. Sci. США, 107(17):7875-7880 (2010). Соответственно, разработка биспецифических LAG-3/PD-1-связывающих белков, которые могут блокировать одновременно обе мишени на иммуносупрессивных Т-клетках, может обеспечить прогресс в этой терапевтической области.
Настоящее изобретение относится к связывающим Fabs-in-Tandem иммуноглобулиновым (FIT-Ig) белкам, которые связываются как с PD-1, так и с LAG-3. Примерный вариант осуществления таких FIT-Ig молекул включает (1) тяжелую полипептидную цепь, которая содержит либо структурную формулу (i) VLA-CL-VHB-CH1-Fc, где CL слит непосредственно с VHB, либо структурную формулу (ii) VHB-CH1-VLA-CL-Fc, где CH1 слит непосредственно с VLA; (2) легкую полипептидную цепь формулы VHA-CH1; и (3) другую легкую полипептидную цепь формулы VLB-CL,
где VL представляет собой вариабельный домен легкой цепи, CL представляет собой константный домен легкой цепи, VH представляет собой вариабельный домен тяжелой цепи, CH1 представляет собой константный домен тяжелой цепи, Fc представляет собой Fc-область иммуноглобулина, A представляет собой эпитоп PD-1 или LAG-3, и B представляет собой эпитоп PD-1 или LAG-3, при условии, что A и B разные. В соответствии с настоящим изобретением такие связывающие FIT-Ig белки связываются как с PD-1, так и с LAG-3.
FIT-Ig может содержать две такие тяжелые цепи (1), две такие легкие цепи (2) и две такие легкие цепи (3), образуя мономер связывающего белка с шестью цепями, имеющий четыре функциональных антигенсвязывающих Fab-участка. Такой связывающий FIT-Ig белок содержит две идентичные субъединицы, причем каждая субъединица включает одну тяжелую цепь (1), одну легкую цепь (2) и одну легкую цепь (3), которые вместе образуют пару связывающих Fab-участков, расположенных в тандеме. Спаривание Fc-областей двух таких субъединиц дает шестицепочечный биспецифический связывающий FIT-Ig белок по изобретению, имеющий в общей сложности четыре функциональных связывающих звена Fab.
Можно использовать пептидный линкер на тяжелой цепи для разделения тандемно связанных Fab-фрагментов, однако для биспецифических FIT-Ig по изобретению предпочтительно исключение таких линкерных последовательностей. При этом, в данной области техники считается, что в иммуноглобулинах, сконструированных в поливалентных форматах, имеющих тандемно расположенные связывающие участки, соседние связывающие участки будут мешать друг другу, если не использовать гибкий линкер для пространственного разделения связывающих участков. Однако в отношении PD-1/LAG-3 FIT-Ig по настоящему изобретению было обнаружено, что расположение иммуноглобулиновых доменов в соответствии с приведенными выше формулами цепи обеспечивает полипептидные цепи, которые хорошо экспрессируются в трансфицированных клетках млекопитающих, собираются соответствующим образом и секретируются в виде биспецифических, поливалентных иммуноглобулин-подобных связывающих белков, которые связывают целевые антигены PD-1 и LAG-3. См. пример 10 ниже. Несмотря на отсутствие каких-либо линкерных последовательностей между связывающими участками Fab, PD-1/LAG-3 FIT-Ig по настоящему изобретению сохраняют аффинность связывания с целевыми антигенами, демонстрируя аффинность связывания, сопоставимую с таковой у родительских mAb. Кроме того, исключение синтетических линкерных последовательностей из связывающих белков позволяет избежать создания антигенных участков, распознаваемых иммунной системой млекопитающих, и, таким образом, исключение линкеров снижает возможную иммуногенность FIT-Ig и обеспечивает период полужизни в кровотоке, который аналогичен периоду полужизни естественного антитела, т.е. FIT-Ig не выводится быстро через иммунную опсонизацию и захват в печени.
Каждый вариабельный домен (VH или VL) в FIT-Ig может быть получен из одного или более «родительских» моноклональных антител, которые связывают один из целевых антигенов, т.е. PD-1 или LAG-3. Связывающие белки FIT-Ig преимущественно получают, используя последовательности вариабельных доменов моноклональных анти-PD-1 и анти-LAG-3 антител, раскрытых в настоящем описании. Предпочтительно родительские антитела представляют собой гуманизированные антитела.
В одном из аспектов настоящее изобретение относится к выбору родительских антител по меньшей мере с одним или более свойствами, необходимыми для молекулы FIT-Ig. В одном из вариантов осуществления свойства антитела выбирают из группы, состоящей из антигенной специфичности, аффинности к антигену, активности, биологической функции, узнавания эпитопа, стабильности, растворимости, эффективности продуцирования, отсутствия иммуногенности, фармакокинетики, биодоступности, перекрестной реактивности тканей и связывания ортологичных антигенов. PD-1 и LAG-3 являются белками клеточной поверхности, и взаимодействие с соответствующими лигандами PD-L1 (рецептор клеточной поверхности) и MHC класса II (поверхностные белки на антигенпрезентирующих клетках) приводит к внутриклеточной передаче сигналов, связанной с подавлением Т-клеток и иммунным ответом. Соответственно, способность анти-PD-1 антител, анти-LAG-3 антител и FIT-Ig белков, связывающих PD-1/LAG-3, по изобретению ингибировать взаимодействие PD-1/PD-L1 и/или MHC класса II/LAG-3 делает их мощными регуляторами активации иммунных клеток и активности иммунных эффекторных клеток.
Антитела, их функциональные фрагменты и связывающие белки по изобретению могут быть очищены (для предполагаемого использования) с помощью одного или нескольких из множества способов и материалов, доступных в данной области для очистки антител и связывающих белков. Такие методы и материалы включают, без ограничения, аффинную хроматографию (например, с использованием смол, частиц или мембран, конъюгированных с белком A, белком G, белком L или специфическим лигандом антитела, его функциональным фрагментом или связывающим белком), ионообменную хроматографию (например, с использованием ионообменных частиц или мембран), хроматографию гидрофобного взаимодействия («HIC»; например, с использованием гидрофобных частиц или мембран), ультрафильтрацию, нанофильтрацию, диафильтрацию, эксклюзионную хроматографию («SEC»), обработку с низким pH (для инактивации контаминирующих вирусов) и их комбинации для получения приемлемой чистоты для предполагаемого использования. Неограничивающий пример обработки с низким pH для инактивации контаминирующих вирусов включает снижение pH раствора или суспензии, содержащей антитело, его функциональный фрагмент или связывающий белок по изобретению, до pH 3,5 с помощью 0,5 M фосфорной кислоты при 18°C-25°C в течение 60-70 минут.
Применение антител и связывающих белков по изобретению
Учитывая способность связываться с человеческим PD-1 и/или LAG-3, раскрытые в настоящем описании антитела, их функциональные фрагменты и биспецифические поливалентные связывающие белки могут быть использованы для обнаружения PD-1 или LAG-3, или обоих, например в биологическом образце, содержащем клетки, которые экспрессируют один или оба этих целевых антигена. Антитела, функциональные фрагменты и связывающие белки по изобретению можно использовать в обычном иммуноанализе, таком как иммуноферментный анализ (ELISA), радиоиммуноанализ (RIA) или тканевый иммуногистохимический анализ. Изобретение относится к способу обнаружения PD-1 или LAG-3 в биологическом образце, включающему приведение биологического образца в контакт с антителом, его антигенсвязывающей частью или связывающим белком по изобретению и определение того, происходит ли связывание с целевым антигеном, тем самым выполняя обнаружение наличия или отсутствия мишени в биологическом образце. Обнаружение связанного или несвязанного антитела/фрагмента/связывающего белка может быть выполнено с помощью непосредственного или опосредованного мечения антитела, функционального фрагмента или связывающего белка детектируемым веществом. Подходящие детектируемые вещества включают различные ферменты, простетические группы, флуоресцентные материалы, люминесцентные материалы и радиоактивные материалы. Примеры подходящих ферментов включают пероксидазу хрена, щелочную фосфатазу, β-галактозидазу или ацетилхолинэстеразу. Примеры подходящих комплексов простетических групп включают стрептавидин/биотин и авидин/биотин; примеры подходящих флуоресцентных материалов включают умбеллиферон, флуоресцеин, флуоресцеинизотиоцианат, родамин, дихлортриазиниламин, флуоресцеин, дансилхлорид или фикоэритрин; пример люминесцентного материала включает люминол; и примеры подходящего радиоактивного материала включают 3H, 14C, 35S, 90Y, 99Tc, 111In, 125I, 131I, 177Lu, 166Ho или 153Sm.
Антитела, их функциональные фрагменты и связывающие белки по изобретению предпочтительно способны нейтрализовать активность человеческого PD-1 и/или человеческого LAG-3 как in vitro, так и in vivo. Соответственно, антитела, их функциональные фрагменты и связывающие белки по изобретению могут быть использованы для ингибирования активности человеческого PD-1 и/или человеческого LAG-3, например, ингибирования передачи клеточных сигналов, опосредованной PD-1/PD-L1 (или PD -1/PD-L2), и/или взаимодействия MHC класса II/LAG-3 в культуре клеток, содержащей клетки, экспрессирующие PD-1 и/или LAG-3, у субъектов-людей или у других субъектов-млекопитающих, имеющих PD-1 или LAG-3, с которым перекрестно реагирует антитело, его функциональный фрагмент или связывающий белок по изобретению. В одном из вариантов осуществления изобретение относится к способу восстановления активности активированных Т-клеток (реверсия супрессии), включающему контактирование клеток, экспрессирующих человеческий PD-1, с анти-PD-1 антителом или PD-1-связывающим белком по настоящему изобретению, таким образом подавляя активность PD-1. В другом варианте осуществления изобретение относится к способу восстановления активности активированных Т-клеток (реверсия супрессии), включающему контактирование клеток, экспрессирующих человеческий LAG-3, с анти-LAG-3 антителом или LAG-3-связывающим белком по изобретению, таким образом подавляя активность LAG-3.
В другом варианте осуществления изобретение относится к способу лечения субъекта, страдающего заболеванием или нарушением, при котором активность PD-1 и/или LAG-3 является вредной, такой способ включает введение субъекту антитела или связывающего белка по настоящему изобретению в эффективном количестве, таким образом снижая у субъекта активность, опосредованную связыванием PD-1/PD-L1 или PD-1/PD-L2 и/или связыванием MHC класс II/LAG-3.
В контексте настоящего описания термин «расстройство, при котором активность PD-1 и/или LAG-3 является вредной» включает заболевания и другие расстройства, при которых взаимодействие PD-1 с одним или обоими его лигандами (PD-L1, PD-L2) или взаимодействие LAG-3 с его лигандом (MHC класс II) у страдающего расстройством субъекта либо отвечает за патофизиологию расстройства, либо является фактором, который способствует ухудшению расстройства. Соответственно, расстройство, при котором активность PD-1 и/или LAG-3 является вредной, представляет собой расстройство, при котором ожидается, что подавление активности PD-1 и/или LAG-3 облегчит симптомы и/или прогрессирование расстройства.
В другом варианте осуществления настоящее изобретение относится к способу лечения аутоиммунного заболевания или рака у нуждающегося в этом субъекта, включающему введение субъекту антитела, его функционального фрагмента или связывающего белка, раскрытого в настоящем описании, который способен связывать LAG-3, PD-1 или оба LAG-3 и PD-1, причем аутоиммунное заболевание или рак представляет собой заболевание, которое реагирует на иммунотерапию. В другом варианте осуществления способ по настоящему изобретению используется для лечения аутоиммунного заболевания или рака, которые не были связаны с иммунотерапией. В другом варианте осуществления способ по изобретению используется для лечения рака, который является рефрактерным или рецидивирующим злокачественным новообразованием. В другом варианте осуществления анти-LAG-3 или анти-PD-1 антитело, его функциональный фрагмент или биспецифический LAG-3/PD-1-связывающий белок по изобретению используется в способе, который ингибирует рост или выживание опухолевых клеток.
В другом варианте осуществления изобретение относится к способу лечения рака у субъекта, включающему этап введения субъекту анти-PD-1 или анти-LAG-3 антитела по настоящему изобретению, его функционального фрагмента или LAG-3/PD-1-биспецифического связывающего белка по настоящему изобретению, например, такого как связывающий Fabs-in-tandem иммуноглобулиновый (FIT-Ig) белок, при этом рак выбирают из группы, состоящей из: меланомы (например, метастатической злокачественной меланомы), почечного рака (например, светлоклеточной карциномы), рака простаты (например, гормонорезистентной аденокарциномы простаты), аденокарциномы поджелудочной железы, рака груди, рака толстой кишки, рака легких (например, немелкоклеточного рака легких), рака пищевода, плоскоклеточного рака головы и шеи, рака печени, рака яичников, рака шейки матки, рака щитовидной железы, глиобластомы, глиомы, лейкемии, лимфомы, первичного рака кости (например, остеосаркомы, саркомы Юинга, злокачественной фиброзной гистиоцитомы и хондросаркомы), метастатического рака и других злокачественных новообразований.
Изобретение также относится к фармацевтическим композициям, содержащим антитело или его антигенсвязывающую часть, или биспецифический поливалентный связывающий белок по изобретению (т.е. основной активный ингредиент) и фармацевтически приемлемый носитель. Фармацевтические композиции, содержащие белки по изобретению, предназначены для применения, без ограничения, для диагностики, обнаружения или мониторинга нарушения; лечения, контроля или облегчения расстройства или одного или более его симптомов; и/или исследования. В конкретном варианте осуществления композиция содержит одно или более антител или связывающих белков по изобретению. В другом варианте осуществления фармацевтическая композиция содержит одно или более антител или связывающих белков по изобретению и одно или более профилактических или терапевтических средств, отличных от антител или связывающих белков по изобретению, для лечения расстройства, при котором вредна активность PD-1 и/или LAG-3. В одном из вариантов осуществления профилактические или терапевтические агенты, как известно, могут быть полезны или применялись, или в настоящее время применяются для профилактики, лечения, контроля или облегчения расстройства или одного или более симптомов. В соответствии с этими вариантами осуществления композиция может дополнительно содержать носитель, разбавитель или вспомогательное вещество. Наполнитель обычно представляет собой любое соединение или комбинацию соединений, которые придают композиции желаемую характеристику, отличную от свойства основного активного ингредиента (т.е. отличную от антитела, его функциональной части или связывающего белка по изобретению).
Антитела (включая их функциональные фрагменты) и связывающие белки по изобретению могут быть включены в фармацевтические композиции, подходящие для введения субъекту. Обычно фармацевтическая композиция содержит антитело или связывающий белок по изобретению и фармацевтически приемлемый носитель. Используемый в настоящей заявке термин «фармацевтически приемлемый носитель» включает любые растворители, дисперсионные среды, покрытия, антибактериальные и противогрибковые агенты, регулирующие изотоничность агенты и задерживающие абсорбцию агенты и т.п., которые являются физиологически совместимыми. Примеры фармацевтически приемлемых носителей включают один или более из следующих компонентов: воду, физиологический раствор, забуференный фосфатом физиологический раствор, декстрозу, глицерин, этанол и т.п., а также их комбинации. Во многих случаях предпочтительно включение в композицию агентов, регулирующих изотоничность, например сахаров, полиспиртов (таких как маннит или сорбит) или хлорида натрия. Фармацевтически приемлемые носители могут дополнительно содержать незначительные количества вспомогательных веществ, таких как смачивающие или эмульгирующие агенты, консерванты или буферы, которые увеличивают срок хранения или эффективность антитела или связывающего белка, присутствующего в композиции.
Состав фармацевтической композиции по изобретению подбирают таким образом, чтобы он был совместим с предполагаемым путем введения. Примеры способов введения включают, без ограничения, парентеральное (например, внутривенное, внутрикожное, подкожное, внутримышечное), пероральное, интраназальное (например, ингаляционное), трансдермальное (например, местное), внутриопухолевое, трансмукозальное и ректальное введение. В конкретном варианте осуществления композицию готовят в соответствии с обычными процедурами в виде фармацевтической композиции, адаптированной для внутривенного, подкожного, внутримышечного, перорального, интраназального или местного введения человеку. Обычно композиции для внутривенного введения представляют собой растворы в стерильном изотоническом водном буфере. При необходимости композиция также может включать солюбилизирующий агент и местный анестетик для облегчения боли в месте инъекции, такой как лидокаин (ксилокаин, лигнокаин).
Способ по настоящему изобретению может включать введение композиции, составленной для парентерального введения, путем инъекции (например, путем болюсной инъекции или непрерывной инфузии). Составы для инъекций могут быть представлены в виде стандартной лекарственной формы (например, в ампулах или в многодозовых контейнерах) с добавленным консервантом. Композиции могут принимать такие формы, как суспензии, растворы или эмульсии в масляных или водных носителях, и могут содержать агенты для составления рецептур, такие как суспендирующие, стабилизирующие и/или диспергирующие агенты. Альтернативно, первичный активный ингредиент может быть в форме порошка, предназначенный для смешивания перед использованием с подходящим носителем (например, стерильной апирогенной водой).
Способы по изобретению могут дополнительно включать введение композиций, приготовленных в виде препаратов-депо. Такие составы длительного действия можно вводить путем имплантации (например, подкожно или внутримышечно) или путем внутримышечной инъекции. Таким образом, композиции, например, могут быть составлены с подходящими полимерными или гидрофобными материалами (например, в виде эмульсии в приемлемом масле) или ионообменными смолами, или в виде труднорастворимых производных (например, в виде умеренно растворимой соли).
Антитело, его функциональный фрагмент или связывающий белок по изобретению также можно вводить с одним или более дополнительными терапевтическими агентами, пригодными для лечения различных заболеваний. Антитела, их функциональные фрагменты и связывающие белки, раскрытые в настоящем описании, можно использовать отдельно или в комбинации с дополнительным агентом, например терапевтическим агентом, причем указанный дополнительный агент выбирается квалифицированным специалистом в зависимости от предполагаемого назначения. Например, дополнительный агент может быть терапевтическим агентом, который в данной области техники считается полезным для лечения заболевания или состояния, которое подлежит лечению с помощью антитела или связывающего белка по настоящему изобретению. Дополнительный агент также может быть агентом, который придает полезные свойства терапевтической композиции, например агентом, который влияет на вязкость композиции.
Настоящее изобретение будет более понятным после изучения подробного описания со ссылкой на приведенные ниже примеры, которые включены только с целью иллюстрации и не предназначены для ограничения изобретения.
Примеры
Пример 1: Создание моноклональных антител к человеческому PD-1.
Моноклональные антитела к человеческому PD-1 получали следующим образом:
Пример 1.1: Иммунизация человеческим антигеном PD-1
50 мкг рекомбинантного очищенного полипептида внеклеточного домена (ECD) человеческого PD-1, смешанного с полным адъювантом Фрейнда, вводили внутрибрюшинно пяти 6-8-недельным мышам Balb/C и пяти мышам SJL в день 1. В дни 16 и 26 тем же мышам вводили внутрибрюшинно 25 мкг рекомбинантного очищенного человеческого иммуногена PD-1 ECD, смешанного с неполным адъювантом Фрейнда. Заключительную бустерную иммунизацию 25 мкг иммуногена проводили за 3-4 дня до слияния.
Пример 1.2: Создание гибридом
Для создания гибридом спленоциты, полученные от иммунизированных мышей, описанных в примере 1.1, сливали с клетками SP2/0-Ag-14 в соотношении 5:1 в соответствии с установленным методом, описанным в Kohler and Milstein, Nature, 256:495-497 (1975). Продукты слияния помещали в 96-луночные планшеты, в среду для селекции, содержащую гипоксантин-аминоптерин-тимидин (HAT) с плотностью 1×105 клеток селезенки на лунку. Через семь-десять дней после слияния наблюдали макроскопические колонии гибридомы.
Пример 1.3: Оценка активности связывания PD-1 методами ELISA и FACS
Присутствие PD-1-специфических антител определяли с помощью иммуноферментного анализа (ELISA) следующим образом:
Во-первых, изготавливали синтетические мишени к человеческому PD-1, PD-1 яванского макака и мышиному PD-1 по заказу Synbio Technologies (Suzhou, China). Каждая мишень состояла из полипептидного сегмента внеклеточного домена (ECD) белка PD-1 человека, яванского макака или мыши, слитого с Fc-областью человеческого IgG. Синтетические гены, кодирующие каждый слитый белок ECD-Fc, субклонировали в вектор экспрессии pCP (Chempartner, Shanghai, China), и плазмиды экспрессии временно трансфицировали в клетки HEK 293E в 1-3 литрах среды и культивировали в течение семи дней в шейкере с CO2. Последовательности ECD, используемые для каждого слитого белка, представлены в таблице 1 ниже. Часть ECD PD-1 каждого слитого белка подчеркнута.
Таблица 1: Аминокислотные последовательности для целевых слитых белков PD-1 ECD-Fc
1234567890123456789012345678901234567890
Супернатанты трансфектантов HEK 293E, содержащие рекомбинантные слитые белки ECD/Fc, собирали центрифугированием при 4000 ×g в течение 30 минут с последующей очисткой с белком A с использованием аффинной смолы MabSelect SuRe™ (GE Healthcare). Продукты слияния диализовали против фосфатно-солевого буфера (PBS) при pH 7,4 и хранили при -80°C.
Планшеты для ELISA инкубировали в течение ночи при 4°C с 50 мкл синтетических мишеней описанного выше слитого белка PD-1 ECD/Fc, разведенного в буфере PBS, pH 7,4, с концентрацией 1 мкг/мл. Планшеты промывали четыре раза в промывочном буфере (PBS, содержащем 0,05% Твин 20) и блокировали в течение 1 часа при 37°C, используя 200 мкл на лунку блокирующего буфера (1% BSA в PBS, содержащем 0,05% Твин 20). После удаления блокирующего буфера в лунки добавляли супернатант гибридомы (или позже разведенные очищенные mAb) в количестве 100 мкл на лунку и инкубировали при 37°C в течение 1 часа. Лунки промывали четыре раза промывочным буфером и антимышиную HRP (Sigma) для характеристики мышиного антитела к человеческому PD-1 разбавляли 1:5000 и добавляли в лунки по 100 мкл на лунку. Планшеты инкубировали в течение 1 часа при 37°C и четыре раза промывали промывочным буфером. В лунку добавляли 100 мкл хромогенного раствора тетраметилбензидина (TMB). После появления окраски реакцию останавливали 1 нормальным раствором HCl и измеряли оптическую плотность при 450 нм на планшетном ридере SpectraMax® M5e (Molecular Devices; San Jose, California, US).
Пример 1.4: Получение линий клеток, экспрессирующих PD-1, и анализ FACS
Стабильные клеточные линии, сверхэкспрессирующие человеческий PD-1 или PD-1 яванского макака, получали путем трансфекции клеток CHO-K1 (полученных из ATCC) лентивирусными плазмидными векторами pLvx (Clontech), содержащими встроенные гены, кодирующие человеческий PD-1 или PD-1 яванского макака. Единичные клоны выделяли методом предельного разведения. Клоны подвергали скринингу на уровень экспрессии с помощью анализа FACS, используя анти-PD-1 антитела, полученные рекомбинантно из известных последовательностей антител (Chempartner), и для использования в FACS-анализах связывания и функциональных анализах отбирали клоны, имеющие самый высокий уровень экспрессии PD-1, как описано ниже.
Анализ связывания для мишеней на клеточной поверхности: Способность очищенных антител связываться с человеческим PD-1 клеточной мембраны или PD-1 яванского макака определяли с помощью FACS-анализа. Клетки CHO-K1, стабильно экспрессирующие человеческий PD-1 (клетки CHO-K1-hPD-1) или PD-1 яванского макака (CHO-K1-cynoPD-1), ресуспендировали в PBS, содержащем 2% FBS (буфер FACS), и высевали с плотностью 2×104 клеток/лунка в 96-луночные круглодонные планшеты (Corning; кат. № 3799). В лунки добавляли супернатанты гибридом, продуцирующих анти-PD-1 антитела, и детектировали с помощью вторичных антител осла к мышиным IgG (H+L) AlexaFluor® 488 с высокой перекрестной адсорбцией (Invitrogen; кат. № A-21202), и затем аналитический планшет считывали на проточном цитометре. Гибридомы, генерирующие супернатанты с положительным сигналом к мишеням, экспрессирующим человеческий PD-1, дополнительно характеризовали с помощью клеток CHO-K1/cynoPD-1 для определения перекрестной реактивности антител к PD-1 яванского макака.
Пример 1.5: Анализ блокирования рецепторов (RBA)
Супернатанты, демонстрирующие PD-1-специфическую активность, тестировали на способность блокировать связывание PD-1 с лигандами PD-L1 и PD-L2, используя в качестве мишени иммобилизованный белок человеческого PD-1 ECD/Fc и слитые белки PD-L1/Fc, и PD-L2/Fc, полученными таким же образом, как и PD-1 ECD/Fc-связывающие белки, описанные в примере 1.3 выше. Для определения относительной эффективности супернатантов, содержащих антитела, оценивали их способность ингибировать связывание лиганда человеческого PD-1 (PD-L1 или PD-L2) с человеческим белком PD-1. Планшеты ELISA покрывали 100 мкл 50 нг/мл huPD-1/Fc (т.е. внеклеточного домена PD-1, привитого на N-конец человеческой Fc-области, выделенной в виде гомодимера) в PBS и инкубировали в течение ночи при 4°C. Планшеты промывали четыре раза в буфере для промывки (PBS, содержащий 0,05% Твин 20) и блокировали в течение 1 часа при 37°C, используя 200 мкл на лунку блокирующего буфера (1% BSA в PBS, содержащем 0,05% Твин 20). После удаления блокирующего буфера в лунки добавляли супернатант гибридомы (50 мкл), смешанный либо с 50 мкл биотинилированного человеческого PD-L1/Fc (конечная концентрация 1,0 мг/мл) в блокирующем буфере, либо с 50 мкл биотинилированного человеческого PD-L2/Fc (конечная концентрация 50 мкг/мл) в блокирующем буфере, затем инкубировали при 37°C в течение 1 часа. Сигнал проявляли путем добавления стрептавидин-HRP (Sigma, кат. № S2468) (100 мкл/лунка стрептавидин-HRP при разведении 1:5000) и инкубации в течение 40 минут при 37°C и четырехкратной промывки в промывочном буфере. Добавляли 100 мкл раствора TMB на лунку. После появления цвета реакцию останавливали 1 нормальным раствором HCl и измеряли оптическую плотность при 450 нм.
Пример 1.6: Экспрессия и очистка моноклональных анти-PD-1 антител
Клетки гибридомы, продуцирующие мышиные моноклональные антитела, культивировали в среде для экспрессии FreeStyle™ 293 (Gibco/Life Technologies) в шейкере с CO2 при 37°C в течение 5-7 дней. Кондиционированную среду собирали центрифугированием при 4000 ×g в течение 30 минут для удаления всех клеток и клеточного дебриса, затем перед очисткой фильтровали через 0,22 мкм мембрану. Мышиные антитела наносили и связывали, пропуская через колонку, заполненную смолой MabSelect™ SuRe (GE Healthcare) с белком A, в соответствии с инструкциями производителя, промывали PBS, элюировали буфером, содержащим 20 мМ цитрат, 150 мМ NaCl, pH 3,5. Элюированные материалы сразу нейтрализовали 1М Трис при pH 8,0 и диализовали против PBS. Одностадийно очищенные антитела обычно имели чистоту выше 90%, определенную с помощью SEC-ВЭЖХ. Концентрации белка определяли путем измерения оптической плотности при 280 нм или с помощью микрообъемного спектрофотометра NanoDrop™ (Thermo Scientific). Очищенные антитела хранили в виде аликвот в морозильной камере при -80°C.
Пример 2: Связывающая активность очищенных анти-PD-1 антител
Примеры 2.1: Характеристика с помощью ELISA
Связывающий ELISA выполняли таким же образом, как описано в примере 1.3 выше. Каждое очищенное антитело подвергали 10-кратному серийному разведению и дублировали. После блокирования 96-луночного аналитического планшета с лунками, содержащими иммобилизованные мишени слитого белка PD-1 ECD/Fc, серийные разведения очищенных образцов антител добавляли в лунки аналитических планшетов. Связанное с HRP антитело к мышиному IgG (A0168, Sigma) и реагент TMB использовали для обнаружения и проявления сигнала ELISA, который считывали на планшетном ридере SpectraMax® M5e при длине волны 450 нм. Подгонку кривых выполняли с помощью программного обеспечения GraphPad, и вычисляли ЕС50. Анализ блокирования рецепторов (RBA) выполняли аналогичным образом, как описано в Примере 1.5, с титрованными очищенными антителами, и определяли верхние проценты блокирования и значения IC50.
Пример 2.2: Характеристика с помощью FACS
Описанные выше клетки CHO-K1/huPD-1 или CHO-K1/cynoPD-1 вносили в 96-луночные круглодонные аналитические планшеты для анализа (кат. № 3799; Corning) в количестве 2×104 клеток на лунку и окрашивали очищенными анти-PD-1 антителами. Анти-PD-1 антитела детектировали с помощью вторичных антител осла к мышиному IgG (H+L) AlexaFluor® с высокой перекрестной адсорбцией (кат. № A21202; Invitrogen), и флуоресценцию клеток контролировали с помощью проточного цитометра. Данные обрабатывали с помощью программного обеспечения GraphPad и вычисляли значения EC50.
Результаты этих анализов по характеристике связывания приведены в таблице 2 ниже.
Таблица 2: Связывающая активность очищенных анти-PD-1 антител
(huPD-L1)
(huPD-L2)
(нМ)
TOP (%)
Пример 2.3: Измерение аффинности методом поверхностного плазмонного резонанса (SPR)
Кинетику связывания очищенных антител измеряли методом поверхностного плазмонного резонанса с помощью прибора Biacore T200 (GE Healthcare), используя стандартные процедуры. Вкратце, козьи поликлональные антитела к Fc мышиного IgG (Genway) иммобилизовали непосредственно на биосенсорном чипе, и образцы антител вводили в реакционные матрицы со скоростью потока 5 мкл/мин. Константы скорости ассоциации и диссоциации, kon (M-1с-1) и koff (с-1), соответственно, определяли при непрерывной скорости потока 30 мкл/мин. Константы скоростей получали путем измерения кинетического связывания при пяти различных концентрациях слитого белка человеческого PD-1/Fc. Затем из констант кинетических скоростей вычисляли константу равновесной диссоциации KD (M) реакции между антителами и родственными целевыми белками по формуле: KD=koff/kon. Измеряли аффинность к одиннадцати мышиным анти-PD-1 антителам, как показано в таблице 3 ниже.
Таблица 3: Измерение аффинности у 11 моноклональных анти-PD-1 антител
Пример 3: Функциональная активность анти-PD-1 антител
Анализ реакции смешанной культуры лимфоцитов (MLR) выполняли с использованием дендритных клеток, полученных из моноцитов от одного донора и аллогенных CD4+ Т-клеток от другого донора. Образцы цельной крови собирали у здоровых доноров, и PBMC выделяли из цельной крови путем центрифугирования в градиенте Ficoll-Pague. В день 1 получали PBMC от одного донора, которые разбавляли бессывороточной средой RPMI 1640 до концентрации 1×106 клеток/мл. Разбавленные РВМС высевали в 6-луночный планшет для тканевых культур из расчета 3 мл/лунку и инкубировали в течение 3 часов. Супернатант удаляли и неиммобилизованные клетки смывали. Иммобилизованные моноциты поляризовали в дендритные клетки с помощью 250 ед./Мл IL-4 и 500 ед./Мл GM-CSF в RPMI 1640 с 10% FBS. На четвертый день среду заменяли свежей средой, содержащей IL-4 и GM-CSF. На седьмой день незрелые дендритные клетки собирали и обрабатывали 1 мкг/мл бактериального липополисахарида (LPS) (Sigma) в RPMI 1640 с 10% FBS в течение дополнительных 24 часов до созревания. В день 8 выделяли CD4+ Т-клетки из PBMC, полученных от другого донора, путем отрицательного отбора и доводили до конечной концентрации 2×106 клеток/мл. Зрелые дендритные клетки обрабатывали митомицином C при 37°C в течение 1,5 часов, затем дендритные клетки промывали PBS и доводили до конечной концентрации 1×106 клеток/мл. CD4+ Т-клетки (клетки-респондеры) добавляли в 96-луночные планшеты в количестве 100 мкл/лунку и предварительно обрабатывали тестируемым антителом в разбавленной концентрации в течение 30 минут. Зрелые дендритные клетки (клетки-стимуляторы) добавляли в лунки в количестве 100 мкл/лунку. Конечный объем в каждой лунке составляет 200 мкл. Смешанную культуру лимфоцитов инкубировали при 37°C. Продуцирование IL-2 измеряли через 72 часа (см. фиг. 1A и 1B); IFN-γ измеряли через 120 часов (см. фиг. 2A и 2B).
Пример 4: Клонирование и анализ последовательности анти-PD-1 mAb
Общую РНК каждого клона гибридомы выделяли из >5×106 клеток с помощью реагента TRIzol (Кат. № 15596; Invitrogen). кДНК синтезировали с помощью набора SuperMix для синтеза первой цепи SuperScript™ III (Кат. № 18080; Invitrogen) и применяли в качестве ПЦР-матрицы для набора праймеров мышиного Ig (Кат. № 69831-3; Novagen). Продукты ПЦР анализировали с помощью электрофореза на 1,2% агарозном геле с окрашиванием геля красителем для ДНК SYBR™ Safe (Invitrogen). Фрагменты ДНК правильного размера очищали с помощью набора NucleoSpin® Gel и PCR Clean-up (Кат. № 740609; Macherey-Nagel GmbH) в соответствии с инструкциями производителя и по отдельности субклонировали в вектор pMD18-T (Sino Biological Inc.). Из каждой трансформации отбирали пятнадцать колоний, и последовательности инсерционных фрагментов анализировали секвенированием ДНК. Последовательности подтверждали при наличии не менее 8 совпадений с консенсусными последовательностями для VH и VL. Белковые последовательности вариабельных областей мышиных анти-PD-1 mAb анализировали путем выравнивания с гомологичными последовательностями, которые приведены в таблице 4. Определяющие комплементарность области (CDR) идентифицировали согласно нумерации Кабат, которые представлены в таблице 4 путем подчеркивания.
Таблица 4: Аминокислотные последовательности 8 мышиных моноклональных анти-PD-1 антител
1234567890123456789012345678901234567890
Пример 5: Гуманизация мышиных анти-PD-1 антител
На основании активности связывания человеческого PD-1, перекрестной реактивности с PD-1 яванского макака, аналогичной человеческой, почти 100% блокирующей активности в анализе RBA, функциональной активности в MLR и аффинности по меньшей мере в наномольном диапазоне, измеренной с помощью Biacore, для гуманизации были отобраны четыре анти-PD-1 антитела, mAb709, mAb713, mAb703 и mAb719.
Пример 5.1: Гуманизация мышиного антитела mAb709
Гены вариабельной области mAb709 использовали для создания гуманизированного антитела. На первом этапе этого процесса аминокислотные последовательности VH и VL mAb709 сравнивали с доступной базой данных последовательностей V-генов человеческого Ig, чтобы найти в целом наиболее подходящие последовательности V-генов человеческого Ig зародышевой линии. Кроме того, сегмент 4 каркасной области VH или VL сравнивали с базой данных J-областей, чтобы найти человеческую каркасную область, имеющую наивысшую гомологию с мышиными областями VH и VL, соответственно. Наиболее близкое соответствие с человеческим V-геном легкой цепи показал ген O12; и наиболее близкое соответствие с человеческим геном тяжелой цепи показал ген VH3-7. Затем конструировали гуманизированные последовательности вариабельных доменов, где CDR-L1, CDR-L2 и CDR-L3 легкой цепи mAb709 прививали на каркасную последовательность гена O12 с последовательностью каркасной области 4 JK4 после CDR-L3; и CDR-H1, CDR-H2 и CDR-H3 тяжелой цепи mAb709 прививали на последовательности каркасной области VH3-7 с последовательностью каркасной области 4 JH1 после CDR-H3. Затем создавали трехмерную Fv-модель mAb709, чтобы определить, существуют ли какие-либо положения в каркасе, в которых мышиные аминокислоты являются критическими для поддержания петлевых структур или интерфейса VH/VL. Эти остатки в гуманизированных последовательностях следует подвергнуть обратной мутации на мышиные остатки в том же положении с целью сохранения аффинности/активности. В случае легкой цепи в качестве желательных обратных мутаций идентифицировали обратную мутацию Phe в Tyr в положении 71 (F71Y, нумерация по Кабат), обратную мутацию Tyr в Ser в положении 49 (Y49S, нумерация по Кабат), обратную мутацию Gln в Val в положении 3 (Q3V, нумерация Кабат), обратную мутацию Leu в Val в положении 46 (L46V, нумерация Кабат), Ser в Thr в положении 63 (S63T нумерация по Кабат), обратную мутацию Ala в Ser в положении 43 (A43S нумерация по Кабат) и обратную мутацию Pro в Leu в положении 44 (P44L нумерация по Кабат). В случае тяжелой цепи в качестве желательных обратных мутаций идентифицировали мутацию Arg в Gly в положении 98 (R94G нумерация по Кабат) и мутацию Gly в Arg в положении 44 (G44R нумерация по Кабат). Конструировали мутированные вариабельные домены, содержащие одну или более из этих обратных мутаций. См. таблицу 5 ниже. (Обратно мутированные аминокислотные остатки в каркасной области обозначены двойным подчеркиванием; мышиные CDR исходного родительского антитела просто подчеркнуты.)
Таблица 5: Дизайн гуманизированных VH/VL для mAb709 с w/обратными мутациями на мышиные остатки
1234567890123456789012345678901234567890
Гены гуманизированных VH и VK получали синтетически и затем соответственно клонировали в векторы, содержащие константные домены человеческого IgG1 и человеческой каппа-цепи (см. таблицу 6 ниже.)
Таблица 6: Последовательность человеческой константной области, используемая для гуманизации антител
1234567890123456789012345678901234567890
Спаривание цепей гуманизированной VH и гуманизированной VK привело к созданию 15 гуманизированных антител, названных HumAb709-1 - HumAb709-15 (таблица 7). В качестве положительного контроля для сравнения аффинности также получали химерное антитело с последовательностями родительских мышиных VH/VL и человеческой константной области (mAb709c). Все рекомбинантные mAb экспрессировали и очищали.
Таблица 7: Список полученных гуманизированных анти-PD-1 антител mAb709
Характеристики 15 гуманизированных антител и химерного антитела (mAb709c) получали с помощью методов связывающего ELISA и клеточного RBA. Для клеточного RBA в предварительно заблокированный 96-луночный круглодонный планшет добавляли клетки CHO-K1-huPD1 в количестве 2×105 клеток/лунку и после промывки в каждую лунку добавляли 50 мкл разбавленных антител в диапазоне от 0,064 нМ до 200 нМ. Затем добавляли 50 мкл биотинилированного белка PD-L1/Fc или биотинилированного белка PD-L2/Fc в концентрации 60 мкг/мл. После осторожного перемешивания и инкубации при 4°C клетки промывали и окрашивали раствором стрептавидина Alexa Fluor™ 488 (1:1000, ThermoFisher Scientific; кат. № S32354). Сигналы считывали с помощью FACS, а кривые строили с помощью программного обеспечения GraphPad. Расчетные значения IC50 показаны в Таблице 8 ниже. Антитела, имеющие положительные (низкие) значения IC50 (т.е. ниже примерно 1,0 нМ для по меньшей мере одного лиганда PD-1), дополнительно анализировали на аффинность связывания методом поверхностного плазмонного резонанса с помощью прибора Biacore T200. Вкратце, козье поликлональное антитело к Fc человеческого IgG иммобилизовали непосредственно на чипе биосенсора, и образцы гуманизированного анти-PD-1 антитела или химерного антитела вводили в реакционные матрицы со скоростью потока 5 мкл/мин. Константы скорости ассоциации и диссоциации, kon (M-1с-1) и koff (с-1), соответственно, определяли выполняя измерение кинетического связывания при пяти различных концентрациях человеческого белка PD-1-His при непрерывной скорости потока 30 мкл/мин. Константу равновесной диссоциации KD (M) реакции между антителами и родственными белками-мишенями рассчитывали из констант кинетических скоростей по формуле: KD=koff/kon. Аффинность к пяти из производных гуманизированных анти-PD-1 mAb709 показана в таблице 8. HumAb709-8 имело минимальное количество обратных мутаций и максимально сохраненную аффинность родительских вариабельных доменов химерного mAb709c.
Таблица 8: Значения RBA и аффинности связывания гуманизированных анти-PD-1 антител mAb709
IC50 (нМ)
IC50 (нМ)
Пример 5.2: Гуманизация мышиного антитела mAb713
Гены вариабельной области анти-PD-1 mAb713 использовали для создания гуманизированного антитела. Аминокислотные последовательности VH и VK mAb713 сравнивали с доступной базой данных последовательностей V-генов человеческого Ig, чтобы найти в целом наиболее подходящие последовательности V-генов человеческого Ig зародышевой линии. Кроме того, сегмент 4 каркасной области VH или VL сравнивали с базой данных J-областей, чтобы найти каркасную область, имеющую наивысшую гомологию с мышиными областями VH и VL, соответственно. Наиболее близкое соответствие с человеческим V-геном легкой цепи показал ген O18; и наиболее близкое соответствие с человеческим геном тяжелой цепи показал ген VH3-48. Затем были конструировали гуманизированные последовательности вариабельных доменов, где CDR-L1, CDR-L2 и CDR-L3 легкой цепи mAb713 прививали на каркасные последовательности гена O18 с последовательностью каркасной области 4 JK4 после CDR-L3; и CDR-H1, CDR-H2 и CDR-H3 тяжелой цепи mAb713 прививали на каркасные последовательности VH3-48 с последовательностью каркасной области 4 JH6 после CDR-H3. Затем создавали трехмерную Fv-модель mAb709, чтобы определить, существуют ли какие-либо положения в каркасе, в которых мышиные аминокислоты являются критическими для сохранения петлевых структур или интерфейса VH/VL. Эти остатки в гуманизированных последовательностях следует подвергнуть обратной мутации на мышиные остатки в том же положении с целью сохранения аффинности/активности. В случае легкой цепи в качестве желательных обратных мутаций идентифировали обратную мутацию Phe в Tyr в положении 71 (F71Y, нумерация Кабат), обратную мутацию Tyr в Ser в положении 49 (Y49S, нумерация Кабат) и обратную мутацию Thr в Lys в положении 69 (T69K, нумерация Кабат). В случае тяжелой цепи в качестве желательных обратных мутаций идентифировали мутацию Arg в Lys в положении 98 (R94K, нумерация Кабат), обратную мутацию Phe в Ser в положении 29 (F29S, нумерация Кабат) и обратную мутацию Ser в Ala в положении 49 (S49A, нумерация Кабат). Конструировали мутированные вариабельные домены, содержащие одну или более из этих обратных мутаций. См. таблицу 9 ниже. (Обратно мутированные аминокислотные остатки в каркасной области обозначены двойным подчеркиванием; мышиные CDR исходного родительского антитела просто подчеркнуты.)
Таблица 9: Варианты последовательностей вариабельных доменов VH и VL mAb713
1234567890123456789012345678901234567890
Гуманизированные гены VH и VK получали синтетически, и затем отдельно клонировали в векторы, содержащие константные домены человеческого IgG1 и человеческой каппа-цепи (см. таблицу 6 выше). Спаривание вариантов человеческой VH и вариантов человеческой VK дало 16 гуманизированных антител, названных HumAb713-1 - HumAb713-16 (таблица 10). в качестве положительного контроля для сравнения аффинности также получали химерное антитело (mAb713c) с родительскими мышиными VH/VL и человеческими константными последовательностями.
Таблица 10: Список полученных гуманизированных анти-PD-1 антител mAb713
Характеристику всех 16 гуманизированных антител и химерного антитела (mAb713c) получали с помощью методов связывающего ELISA, клеточного RBA и тестирования аффинности с помощью Biacore. Полученные результаты обобщены в таблице 11.
Таблица 11: Значения RBA и аффинности связывания гуманизированных анти-PD-1 антител mAb713
IC50 (нМ)
IC50 (нМ)
HumAb713-7 имел минимальное количество обратных мутаций с сохранением характеристик аффинности родительских вариабельных доменов химерного антитела mAb713c. Функциональную активность гуманизированных антител mAb713 подтверждали в анализе MLR, как описано в примере 3. Как видно на фиг. 5, HumAb713-7 проявлял активность, сопоставимую с активностью химерного антитела mAb713c в MLR, что согласуется с его сохраненными связывающими свойствами.
Пример 5.3: Гуманизация мышиного антитела mAb703
Следуя той же процедуре, что и в Примерах 5.1 и 5.2, было выбрано мышиное анти-PD-1 антитело mAb703, которое гуманизировали. Конструировали гуманизированные вариабельные домены, причем некоторые из них содержали одну или более обратных мутаций, аминокислотные последовательности которых представлены в таблице 12 ниже. (Обратно мутированные аминокислотные остатки в каркасной области обозначены двойным подчеркиванием; мышиные CDR исходного родительского антитела просто подчеркнуты.)
Таблица 12: Варианты последовательности вариабельных доменов VH и VL mAb703
1234567890123456789012345678901234567890
Гуманизированные гены VH и VK получали синтетически, а затем отдельно клонировали в векторы, содержащие константные домены человеческого IgG1 и человеческой каппа-цепи (см. таблицу 6 выше). Спаривание вариантов человеческой VH и вариантов человеческой VK дало 25 гуманизированных антител, названных HumAb703-1 - HumAb703-25 (таблица 13). В качестве положительного контроля для сравнения аффинности также получали химерное антитело с родительскими мышиными VH/VL и человеческими константными последовательностями.
Таблица 13: Список полученных гуманизированных анти-PD-1 антител mAb703
Характеристики всех 25 гуманизированных антител и химерного антитела (mAb703c) получали с помощью методов связывающего ELISA и тестирования аффинности с помощью Biacore. Результаты оценки аффинности, наблюдаемой у положительных связующих агентов суммированы в таблице 14.
Таблица 14: Аффинность связывания выбранных гуманизированных анти-PD-1 антител mAb703
Функциональную активность гуманизированных антител mAb703 подтверждали в анализах MLR, которые выполняли согласно тому, как описано в примере 3, как показано на фиг. 2A и 3.
Пример 5.4: Гуманизация мышиного антитела mAb719
Следуя той же процедуре, что и в примерах 5.1 и 5.2, было выбрано мышиное анти-PD-1 антитело mAb719, которое гуманизировали. Конструировали гуманизированные вариабельные домены, причем некоторые из них содержали одну или более обратных мутаций; аминокислотные последовательности представлены в таблице 15 ниже. (Обратной мутированные аминокислотные остатки в каркасной области обозначены двойным подчеркиванием; мышиные CDR исходного родительского антитела просто подчеркнуты.) Кроме того, выполняли замену Asp → Ala в CDR-H2 и Ser → Ala в CDR-H3, чтобы избежать возможности изомеризации Asp, часто наблюдаемой в рекомбинантных антителах. (См. последовательности mAb719 VH.1E и mAb719 VH.1F в таблице 15.)
Таблица 15: Варианты последовательности вариабельных доменов VH и VL mAb719
1234567890123456789012345678901234567890
Гуманизированные гены VH и VK получали синтетически, и затем отдельно клонировали в векторы, содержащие константные домены человеческого IgG1 и человеческой каппа-цепи (см. таблицу 6 выше). Спаривание вариантов человеческой VH и вариантов человеческой VK привело к образованию 21 гуманизированного антитела, названного HumAb719-1 - HumAb719-21 (таблица 16). В качестве положительного контроля для сравнения аффинности также получали химерное антитело с родительскими мышиными VH/VL и человеческими константными последовательностями. Все рекомбинантные mAb экспрессировали и очищали.
Таблица 16: Список полученных гуманизированных анти-PD-1 антител mAb719
Характеристику всех 21 гуманизированного антитела и химерного антитела (mAb719c) получали с помощью связывающего ELISA и определения аффинности с помощью системы биослойной интерферометрии на устройстве Octet® RED96 (Pall FortéBio LLC) с использованием биосенсора с иммобилизованным слитым белком человеческого PD-1/Fc в качестве целевого антитела. Константы скоростей получали путем измерения кинетического связывания при пяти различных концентрациях антител. Аффинность была выше, чем в предыдущем тестировании с помощью Biacore, что явилось результатом использования мишени с двухвалентным связыванием. Результаты по оценке аффинности, наблюдаемой у положительных связующих суммированы в таблице 17.
Таблица 17: Аффинность связывания выбранных гуманизированных анти-PD-1 антител mAb719
Функциональную активность гуманизированных антител mAb719 подтверждали в анализах MLR, как показано на фиг. 2B и 3.
Пример 6: Фармакокинетические свойства ведущих анти-PD-1 антител.
Фармакокинетические свойства HumAb709-8 и HumAb713-7 оценивали, используя самцов крыс Sprague-Dawley (SD). Антитела вводили крысам-самцам SD в виде однократной внутривенной дозы 5 мг/кг. Образцы сыворотки собирали в разные моменты времени в течение 28 дней: через 0, 5, 15 и 30 минут; 1, 2, 4, 8 и 24 часа; и 2, 4, 7, 10, 14, 21 и 28 дней путем серийного забора крови через хвостовую вену, и анализировали с помощью общего ELISA. Вкратце, планшеты для ELISA покрывали 125 нг/лунку козьего антитела к Fc человеческого IgG (Rockland, кат. № 609-101-017) при 4°C в течение ночи, блокировали 1X PBS/1% BSA/0,05% Твин-20/0,05% ProClin™ 300. Все образцы сыворотки сначала 20-кратно разбавляли блокирующим буфером. Дополнительное разведение выполняли, используя 5% объединенную крысиную сыворотку, и инкубировали на планшете в течение 60 минут при 37°C. Детектирование выполняли с помощью конъюгированных с пероксидазой антител к человеческому IgG (фрагмент Fab) (Sigma; Кат. № A0293), и концентрации определяли с помощью стандартных кривых путем подгонки четырехпараметрической логистической кривой. Значения фармакокинетических параметров определяли с помощью некомпартментной модели, используя программное обеспечение WinNonlin (Pharsight Corporation, Mountain View, CA). Согласно полученным результатам (Таблица 18), HumAb09-8 и HumAb13-7 демонстрируют стабильные свойства.
Таблица 18: Фармакокинетические свойства HumAb709-8 и HumAb713-7
Пример 7: Создание моноклональных анти-LAG-3 антител
Моноклональные анти-LAG-3 антитела (mAb) получали слиянием гибридом.
Пример 7.1: Иммунизация, слияние гибридом и клонирование.
Иммунизацию мышей Balb/C выполняли таким же образом, как описано выше для получения анти-PD-1 антител (Пример 1), за исключением использования в качестве иммуногена гомодимера: D1-D2 человеческого LAG-3/мышиного Fc. Иммунизированных животных подвергали повторной вакцинации 2-4 раза с интервалом в 2-3 недели. Через три дня после окончательной иммунизации из иммунизированных мышей выделяли спленоциты, которые сливали с линией клеток мышиной миеломы SP2/0 с помощью стандартных методов.
Пример 7.2: Идентификация и характеристика анти-LAG-3 антител
Изготавливали синтетические мишени к человеческому LAG-3 и LAG-3 яванского макака по заказу Synbio Technologies (Suzhou, China). Каждая мишень состояла из полипептидного сегмента внеклеточного домена белка LAG-3 человека или яванского макака, слитого с Fc-областью человеческого IgG. Синтетические гены, кодирующие каждый слитый белок LAG-3 ECD/Fc, субклонировали в вектор экспрессии pCP (Chempartner, Shanghai, CN), и плазмиды экспрессии временно трансфицировали в клетки HEK 293E в 1-3 литрах среды и культивировали в течение семи дней в шейкере с CO2. Последовательности ECD, используемые для каждого слияния, представлены в таблице 19 ниже. ECD-часть LAG-3 каждого слитого белка подчеркнута.
Таблица 19: Аминокислотные последовательности целевых слитых белков LAG-3 ECD/Fc
1234567890123456789012345678901234567890
Супернатанты клонов гибридом в первую очередь проверяли с помощью ELISA. Вкратце, 1 мкг/мл человеческого LAG-3 ECD/Fc в NaHCO3 наносили на каждую лунку 96-луночного планшета в количестве 50 мкл/лунку оставляли на ночь. Планшеты промывали 3 раза 1X PBST по 300 мкл на лунку. После блокирования 1% BSA в PBST в количестве 250 мкл на лунку и инкубации при комнатной температуре в течение 1 часа добавляли супернатанты гибридом в количестве 50 мкл на лунку и инкубировали при 37°C в течение 1 часа. После промывки добавляли конъюгированное с HRP вторичное козье антитело к Fc мышиного IgG (кат. № A0168, Sigma) в количестве 100 мкл/лунку, и планшеты инкубировали при комнатной температуре в течение 1 часа. Реагент TMB (InnoReagents) использовали для обнаружения и проявления сигнала ELISA в количестве 100 мкл/лунку в течение 15 минут, реакцию останавливали 1 нормальным HCl. Планшеты считывали с помощью планшетного ридера (SpectraMax® M5e, Molecular Devices, USA) при длине волны 450 нм. ELISA-положительные клоны-продуценты антител дополнительно проверяли с помощью анализа FACS, используя методы, аналогичные описанным выше в примере 1.4, за исключением использования стабильных клеточных линий HEK 293F, экспрессирующих LAG-3 человека или LAG-3 яванского макака. Отбирали гибридомы, демонстрирующие активность связывания LAG-3, которые дополнительно характеризовали в анализе блокирования рецепторов (RBA).
Пример 7.3: Анализ блокирования рецепторов (RBA)
Супернатанты, демонстрирующие LAG-3-специфическую активность, тестировали на способность блокировать связывание рецептора LAG-3 с MHC класса II. В-клеточные лимфобласты человека Raji экспрессируют высокие уровни MCH класса II, и их использовали в качестве связывающих мишеней для белков LAG-3 ECD/Fc, описанных выше. Вкратце, клетки Raji собирали, ресуспендировали в буфере для FACS и высевали в 96-луночные планшеты (2×105 клеток/лунку). Супернатанты анти-LAG-3 гибридомы смешивали с растворимым LAG-3 ECD/Fc, и смесь добавляли в лунки до конечного объема 100 мкл/лунка. После добавления смеси к клеткам планшеты инкубировали при комнатной температуре в течение 30 минут. После двукратной промывки PBS клетки инкубировали со вторичным антителом к человеческому IgG Alexa Fluor® 488 при 4°C в течение 1 часа, дважды промывали PBS, и затем измеряли флуоресценцию на проточном цитометре.
Пример 7.4: Экспрессия и очистка анти-LAG-3 моноклональных антител
Клетки гибридомы, продуцирующие мышиные моноклональные антитела, культивировали в среде для экспрессии FreeStyle™ 293 (Gibco/Life Technologies) в шейкере с CO2 при 37°C в течение 5-7 дней. Кондиционированную среду собирали центрифугированием при 4000 ×g в течение 30 минут для удаления всех клеток и клеточного дебриса, затем перед очисткой фильтровали через 0,22 мкм мембрану. Наносили мышиные антитела и связывали, пропуская через колонку, заполненную смолой MabSelect™ SuRe (GE Healthcare) с белком A, в соответствии с инструкциями производителя, промывали PBS, элюировали буфером, содержащим 20 мМ цитрат, 150 мМ NaCl, pH 3,5. Элюированные материалы сразу нейтрализовали 1М Трис при pH 8,0 и диализовали против PBS. Одностадийно очищенные антитела обычно имели чистоту выше 90%, определенную с помощью SEC-ВЭЖХ. Концентрации белка определяли путем измерения оптической плотности при 280 нм или с помощью микрообъемного спектрофотометра NanoDrop™ (Thermo Scientific). Очищенные антитела хранили в виде аликвот в морозильной камере при -80°C.
Пример 7.5: Связывающая активность очищенных анти-LAG-3 антител
Характеристика с помощью ELISA
Связывающий ELISA выполняли таким же образом, как описано в примере 7.2 выше. Каждое очищенное антитело подвергали 10-кратному серийному разведению. После блокирования 96-луночного аналитического планшета с лунками, содержащими иммобилизованные мишени слитого белка LAG-3 ECD/Fc, в лунки аналитических планшетов добавляли очищенные разведенные образцы антител. Связанное с HRP антитело к мышиному IgG (A0168, Sigma) и реагент TMB использовали для обнаружения и проявления сигнала ELISA, который считывали на планшетном ридере SpectraMax® M5e при длине волны 450 нм. Кривые подгоняли с помощью программного обеспечения GraphPad, и вычисляли значения ЕС50. Аналогичным образом выполняли RBA, как описано в Примере 7.3, используя титры очищенных антител, и определяли верхние проценты блокирования и значения IC50.
Характеристика с помощью FACS
FACS-анализ выполняли, используя методы, аналогичные описанным в примере 1.4 выше, за исключением использования стабильных клеточных линий HEK 293F, экспрессирующих либо LAG-3 человека, либо LAG-3 яванского макака. LAG-3-экспрессирующие клетки загружали в количестве 2×104 клеток на лунку в 96-луночные круглодонные аналитические планшеты для анализа (Кат. № 3799; Corning) и окрашивали очищенными анти-LAG-3 антителами. Анти-LAG-3 антитела детектировали с помощью вторичных антител осла к мышиному IgG (H+L) AlexaFluor® с высокой перекрестной адсорбцией (кат. № A21202; Invitrogen), и флуоресценцию клеток контролировали с помощью проточного цитометра. Данные обрабатывали с помощью программного обеспечения GraphPad и вычисляли значения EC50.
Результаты этих анализов для характеристики связывания приведены в таблице 20 ниже.
Таблица 20: Связывающая активность очищенных мышиных анти-LAG-3 антител
В этой таблице «N/A» означает не поддающуюся измерению активность связывания.
Пример 7.6: Характеристика с помощью метода RBA и анализа антиген-зависимой активации
Очищенные анти-LAG-3 антитела также тестировали методом RBA аналогично тому, как описано в примере 7.3. Антитела также тестировали в анализе активации антиген-специфических Т-клеток, как показано ниже: линию мышиных Т-гибридомных клеток, экспрессирующую huLAG-3, получали согласно ChemPartner (Shanghai, CN). В качестве эффекторных клеток использовали спленоциты мышей той же линии. Гибридома, экспрессирующая рецепторный белок huLAG-3, способна связываться с мышиными спленоцитами, положительными по MHC-класса II, с ингибирующим эффектом, обусловленным взаимодействием с классом II. Аналитические тесты для опосредованной антителами LAG-3 обратимости ингибирующего эффекта измеряли по увеличению уровня продуцирования IL-2. Мышиные спленоциты собирали у 6-8 недельных самок мышей C57BL/6, эритроциты лизировали с помощью буфера для лизиса красных кровяных клеток (Sigma-Aldrich; R7757) в соответствии с инструкциями производителя. Затем 50 мкл клеток Т-клеточной гибридомы-huLAG-3 (2×106 клеток/мл) высевали в каждую лунку 96-луночного культурального планшета, и затем добавляли серийные разведения моноклональных анти-LAG-3 антител в концентрации 50 мкл/лунка и инкубировали при 37°С в течение 30 мин. Мышиные спленоциты (4×106 клеток/мл) и антиген (20 мкг/мл) смешивали и инкубировали при 37°C в течение 30 минут. Смесь (100 мкл/лунку) добавляли в каждую лунку, которая уже была засеяна клетками Т-клеточной гибридомы-huLAG-3 и анти-LAG-3 mAb. Смесь антител, клетки Т-клеточной гибридомы-huLAG-3, мышиные спленоциты и антиген культивировали в течение 3 дней. Через 72 часа 100 мкл супернатанта клеточной культуры аспирировали и разбавляли до подходящих концентраций для проведения количественного ELISA для определения мышиного IL-2 с помощью набора ELISA (R&D Systems) в соответствии с протоколом производителя. Планшет для ELISA считывали на планшетном ридере SpectraMax M5 (Molecular Devices) с использованием протокола ELISA-Endpoint-TMB и HRP. Результаты анализа RBA и антиген-зависимой активации показаны в таблице 21.
Таблица 21: Характеристика мышиных анти-LAG-3 антител
Пример 7.7: Определение аффинности связывания с помощью Biacore
Для антител с высокой аффинностью связывания в анализах ELISA и FACS, а также с высокой функциональной активностью, определяли аффинность связывания на основании измерения кинетических констант связывания в реакциях связывания в реальном времени методом поверхностного плазмонного резонанса с помощью Biacore. Вкратце, анализ связывания антитела с антигеном выполняли с помощью системы Biacore T200, используя метод захвата антител. Антитело к Fc мышиного IgG иммобилизовали на сенсорном чипе CM5 в соответствии с инструкциями производителя. Вводили тестируемое мышиное моноклональное анти-LAG-3 антитело и захватывали иммобилизованным антителом к Fc мышиного IgG. Затем отдельно вводили серийные разведения антигена LAG-3 и записывали профиль связывания для каждого разведения анализируемого антигена. Температура анализа составляла 25°C, а времена ассоциации и диссоциации составляли 180 и 1200 секунд, соответственно. Данные Biacore подгоняли с помощью программного обеспечения 1.0 для оценки Biacore T200 в соответствии с моделью связывания 1:1 для расчета констант скоростей ассоциации (kon) и диссоциации (koff), и из этих расчетов определяли константу равновесной диссоциации (KD). Значения аффинности (KD) у четырех выбранных анти-LAG-3 антител приведены в таблице 22 ниже.
Таблица 22: Аффинность связывания выбранных анти-LAG-3 антител
Пример 7.8: Сравнение функции анти-LAG-3 антител в анализе PBMC
Для дополнительной проверки функции анти-LAG-3 антител в человеческих PBMC выполняли анализ стимуляции с помощью бактериального токсина, суперантигенного энтеротоксина B Staphylococcus aureus (SEB). SEB является известным суперантигеном для активации иммунной системы путем стимуляции человеческих Т-клеток, что, в свою очередь, вызывает избыточное продуцирование нескольких цитокинов. PBMC выделяли из образца крови здорового донора-человека. PBMC высевали в 96-луночный аналитический планшет в количестве 2×105 клеток/лунку, затем в планшеты добавляли различные тестируемые анти-LAG-3 антитела и инкубировали с PBMC при 37°C в течение 30 мин. Добавляли раствор SEB до конечной концентрации 10 нг/мл. Затем планшеты инкубировали в течение 96 часов. В конце этой инкубации собирали 100 мкл супернатанта клеточной культуры и измеряли уровень продуцирования IL-2 с помощью набора ELISA для определения IL-2 (R&D Systems; Кат. № DY202). Результаты показаны на фиг. 6.
Пример 8: Секвенирование вариабельных областей мышиных анти-LAG-3 антител
Для амплификации вариабельных областей тяжелой и легкой цепей выделяли общую РНК из >5×106 клеток выбранных клонов гибридомы с помощью реагента для выделения РНК TRIzol® (Invitrogen; кат. № 15596). кДНК синтезировали с помощью набора SuperMix для синтеза первичной цепи SuperScript™ III (Invitrogen; кат. № 18080) и применяли в качестве ПЦР-матрицы для набора праймеров мышиного Ig (Novagen; кат. № 69831-3). Продукты ПЦР анализировали с помощью электрофореза на 1,2% агарозном геле с окрашиванием геля красителем для ДНК SYBR™ Safe (Invitrogen). Фрагменты ДНК правильного размера очищали с помощью наборов NucleoSpin® Gel и PCR Clean-up (Macherey-Nagel GmbH; Кат. № 740609) в соответствии с инструкциями производителя и субклонировали отдельно в клонирующие векторы pMD18-T. Отбирали пятнадцать колоний из каждой трансформации, и последовательности инсерционных фрагментов анализировали путем секвенирования ДНК. Последовательности подтверждали, если было найдено не менее 8 совпадений с консенсусными последовательностями для VH и VL. Последовательности вариабельной области семи мышиных mAb, проанализированные путем выравнивания гомологичных последовательностей, приведены в таблице 23. Определяющие комплементарность области (CDR) идентифицировали согласно нумерации Кабат.
Таблица 23: Аминокислотные последовательности VH/VL 7 мышиных анти-LAG-3 антител
1234567890123456789012345678901234567890
Пример 9: Гуманизация мышиного анти-LAG-3 антитела mAb747
Для гуманизации выбирали mAb747, исходя из антигенсвязывающей активности, перекрестной реактивности с белком LAG-3 яванского макака, функциональной активности и аффинности.
Пример 9.1: Гуманизация mAb747
Для создания гуманизированного антитела использовали гены вариабельной области анти-LAG-3 mAb747. На первом этапе этого процесса аминокислотные последовательности VH и VL mAb747 (SEQ ID NO:60 и SEQ ID NO:62) сравнивали с доступной базой данных последовательностей V-генов человеческого Ig, чтобы найти в целом наиболее подходящие последовательности V-генов человеческого Ig зародышевой линии. Кроме того, последовательность каркасной области 4 VH или VL сравнивали с базой данных J-областей, чтобы найти человеческую каркасную область, имеющую наивысшую гомологию с мышиными областями VH и VL, соответственно. Наиболее близкое соответствие с человеческим V-геном легкой цепи показал ген А1; и наиболее близкое соответствие с человеческой тяжелой цепью показал ген VH1-f. Затем конструировали гуманизированные последовательности вариабельных доменов, в которых CDR-L1, CDR-L2 и CDR-L3 легкой цепи mAb747 прививали на каркасные последовательности гена А1 с последовательностью каркасной области 4 JK4 после CDR-L3; и CDR-H1, CDR-H2 и CDR-H3 тяжелой цепи mAb747 прививали на последовательности каркасной области VH1-f с последовательностью каркасной области 4 JH4 после CDR-H3. Затем создавали трехмерную Fv-модель mAb747, чтобы определить, существуют ли какие-либо положения в каркасной области, в которых мышиные аминокислоты являются критическими для сохранения петлевых структур или интерфейса VH/VL. Такие остатки в гуманизированных последовательностях следует подвергнуть обратной мутации на мышиные остатки в том же положении для сохранения аффинности/активности. Было показано несколько желательных обратных мутаций для VH и VL mAb747, и были созданы три альтернативные конструкции VH и VL, показанные в таблице 24 ниже. (Обратно мутированные аминокислотные остатки в каркасной области обозначены двойным подчеркиванием; мышиные CDR исходного родительского антитела просто подчеркнуты.)
Таблица 24: Дизайн гуманизированных VH/VL mAb747 - обратные мутации на мышиные остатки
1234567890123456789012345678901234567890
Гуманизированные гены VH и VK получали синтетически и затем клонировали в векторы, содержащие константные домены человеческой каппа-цепи, соответственно. Спаривание человеческой VH и человеческой VK привело к образованию 9 гуманизированных анти-LAG-3 антител, названных HumAb747-34-42 (таблица 25). В качестве положительного контроля для сравнения аффинности также получали (mAb747c) химерное антитело с родительскими мышиными VH/VL и человеческими константными последовательностями.
Таблица 25: Список полученных гуманизированных анти-LAG-3 антител mAb747
Все 9 гуманизированных антител (таблица 25) и химерное антитело, имеющее родительские мышиные домены VH и VL (mAb747c), ранжировали по константе скорости диссоциации (koff). Вкратце, антитела были охарактеризованы по аффинности и кинетике связывания методом биослойной интерферометрии, выполненным на устройстве Octet® RED96 (Pall FortéBio LLC). Антитела захватывали биосенсорами Anti-HIgG Fc Capture (AHC) (Pall) в концентрации 100 нМ в течение 30 секунд. Затем сенсоры погружали в рабочий буфер (1X pH 7,2 PBS, 0,05% Твин 20, 0,1% BSA) на 60 секунд для проверки исходного уровня. Связывание измеряли путем погружения сенсоров в одну концентрацию рекомбинантного человеческого белка LAG-3-his (новопротеин). Диссоциацию осуществляли путем погружения сенсоров в рабочий буфер на 1200 секунд. Кривые ассоциации и диссоциации подгоняли к модели связывания Ленгмюра 1:1 с помощью программного обеспечения FortéBio Data Analysis (Pall). Результаты приведены в таблице 26.
Таблица 26: ранжирование гуманизированных анти-LAG-3 антител по скорости диссоциации
HumAb747-42 имело константу скорости диссоциации, которая превышала константу скорости диссоциации контрольного химерного антитела с родительскими вариабельными доменами, только в 2,2 раза.
Пример 10: Получение PD-1/LAG-3 поливалентных FIT (Fabs-in-tandem) иммуноглобулинов (FIT-Ig)
Конструировали биспецифические связывающие Fabs-in-Tandem иммуноглобулиновые белки, распознающие как человеческий PD-1, так и человеческий LAG-3.
Для каждой из конструкций FIT-Ig, описанных в приведенных ниже таблицах, показана сигнальная последовательность, используемая в векторе экспрессии для каждой из трехкомпонентных полипептидных цепей. При получении описанных ниже FIT-Ig белков использовали либо MDMRVPAQLLGLLLLWFPGSRC (SEQ ID NO: 79), либо MEFGLSWLFLVAILKGVQC (SEQ ID NO: 84), хотя специалистам в данной области как правило известны многие альтернативные сигнальные пептиды, которые также могут быть использованы. Будет понятно, что такие сигнальные последовательности расщепляются во время секреции полипептидов экспрессирующей клеткой-хозяином, и, таким образом, сигнальные последовательности не являются частью конечных связывающих FIT-Ig белков.
Пример 10.1: FIT107-1-2a
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-2a, конструировали, используя кодирующие последовательности для доменов иммуноглобулина, полученные из родительских антител mAb709 (мышиных анти-PD-1, см. таблицу 4 выше) и mAb746 (мышиных анти-LAG-3, см. таблицу 23 выше). FIT-Ig FIT107-1-2a является гексамером, состоящим из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL mAb709, слитого непосредственно с VH-CH1 mAb709, слитого непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1 (см. таблицу 6 выше);
Полипептидная цепь #2 имеет формулу домена: VH-CH1 mAb709; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) mAb709.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-2a показаны в Таблице 27 ниже.
Таблица 27: Аминокислотные последовательности цепей компонентов FIT107-1-2a
1234567890123456789012345678901234567890
мышиное mAb709
(VL подчеркнута)
мышиное mAb746
(VH подчеркнута)
человеческого IgG1
FIT107-1-2a
мышиного mAb709
(VH подчеркнута)
мышиного mAb746
(VL подчеркнута)
Пример 10.2: FIT107-1-2b
Конструировали другой биспецифический Fabs-in-Tandem иммуноглобулин, распознающий как человеческий PD-1, так и человеческий LAG-3. Этот PD-1/LAG-3 FIT-Ig обозначен как FIT107-1-2b. При создании связывающего белка FIT107-1-2b использовали кодирующие последовательности доменов иммуноглобулина, полученные из родительских мышиных антител mAb709 и mAb746, но в этой конструкции LAG-3-связывающий домен FIT-Ig находился в N-терминальном (внешнем) положении, и PD-1-связывающий домен находился во внутреннем положении, слитый на С-конце с доменами VL-CL LAG-3-связывающей области. FIT-Ig FIT107-1-2b представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL mAb746, слитого непосредственно с VH-CH1 mAb746, слитого непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1 (см. таблицу 6 выше);
Полипептидная цепь #2 имеет формулу домена: VH-CH1 mAb746; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) mAb709.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-2b показаны в Таблице 28 ниже:
Таблица 28: Аминокислотные последовательности цепей компонентов FIT107-1-2b
1234567890123456789012345678901234567890
мышиного mAb746
(VL подчеркнута)
мышиного mAb709
(VH подчеркнута)
человеческого IgG1
FIT107-1-2b
(VH подчеркнута)
мышиного mAb709
(VL подчеркнута)
Пример 10.3: FIT107-1-5a
FIT-Ig PD-1/LAG-3, обозначенный FIT107-1-5a, сконструировали, используя кодирующие последовательности доменов иммуноглобулинов, полученных из родительских гуманизированных антител HumAb709-8 (анти-PD-1, SEQ ID NO: 21 и SEQ ID NO: 25) и HumAb747-42 (SEQ ID NO: 73 и SEQ ID NO: 77). FIT-Ig FIT107-1-5a является гексамером, состоящим из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb709-8, слитые непосредственно с VH-CH1 HumAb747-42, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1 (см. таблицу 6 выше);
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb709-8; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb747-42.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-5a показаны в таблице 29 ниже:
Таблица 29: Аминокислотные последовательности цепей компонентов FIT107-1-5a
1234567890123456789012345678901234567890
HumAb709-8
(VL подчеркнута)
HumAb747-42
(VH подчеркнута)
человеческого IgG1
HumAb709-8
(VH подчеркнута)
HumAb747-42
(VL подчеркнута)
Пример 10.4: FIT107-1-5b
Конструировали другой биспецифический Fabs-in-Tandem иммуноглобулин, распознающий как человеческий PD-1, так и человеческий LAG-3. Этот PD-1/LAG-3 FIT-Ig обозначен как FIT107-1-5b. При создании связывающего белка FIT107-1-5b использовали кодирующие последовательности доменов иммуноглобулинов, полученных из родительских гуманизированных антител HumAb709-8 и HumAb747-42, но в этой FIT-Ig конструкции LAG-3-связывающий домен находится в N-терминальном (внешнем) положении, и PD-1-связывающий домен находится во внутреннем положении, слитый на C-конце с доменами VL-CL N-концевого LAG-3-связывающего участка. FIT-Ig FIT107-1-5b представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb747-42, слитые непосредственно с VH-CH1 HumAb709-8, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1 (см. таблицу 6 выше);
Полипептидная цепь #2 имеет формулу домена: VH-CH1 mAb747-42; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb709-8.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-5b показаны в Таблице 30 ниже:
Таблица 30: Аминокислотные последовательности цепей компонентов FIT107-1-5b
1234567890123456789012345678901234567890
HumAb747-42
(VL подчеркнута)
HumAb709-8
(VH подчеркнута)
человеческого IgG1
HumAb747-42
(VH подчеркнута)
HuMAb709-8
(VL подчеркнута)
Пример 10.5: Экспрессия и очистка FIT-Ig
Четыре PD-1/LAG-3 FIT-Ig конструкции: FIT107-1-2a, FIT107-1-2b, FIT107-1-5a и FIT107-1-5b, представляют собой тип биспецифического поливалентного связывающего белка, известного как Fabs-In-Tandem иммуноглобулин (или FIT-Ig), описанный в общем в WO 2015/103072 и WO 2017/136820. Связывающие белки получали коэкспрессией трехкомпонентных полипептидных цепей в клетке-хозяине млекопитающего, трансфицированной векторами экспрессии для всех трех цепей. Согласно конструкции связывающего белка требуется, чтобы длинная полипептидная цепь (Цепь #1) была спарена с обеими короткими полипептидными цепями (Цепи #2 и #3) для образования функциональных тандемных Fab-фрагментов, к тому же длинная цепь предназначена для димеризации через Fc-область (шарнир-CH2-CH3), так что образуется шестицепочечный связывающий белок, демонстрирующий четыре интактных Fab-связывающих участка. В связывающих белках FIT107-1-2a и FIT107-1-5a N-терминальные или «внешние» Fab-связывающие участки связываются с PD-1, а соседние «внутренние» Fab-связывающие участки связываются с LAG-3. Внешний Fab-фрагмент (анти-PD-1) FIT107-1-2a и FIT107-1-5a присоединен к внутреннему Fab-фрагменту (анти-LAG-3) только через длинную цепь (Цепь #1) путем прямого слияния VL-CLmAb709 или VL-CLHumAb709-8, поскольку в этом случае может находиться на его С-конце с N-концом VH-CH1mAb746 или VH-CH1HumAb747-42, соответственно, без использования линкеров, соединяющих иммуноглобулиновые домены. Аналогично, внешний Fab-фрагмент (анти-LAG-3) FIT107-1-2b и FIT107-1-5b присоединен к внутреннему Fab-фрагменту (анти-PD-1) только через длинную цепь (Цепь #1) путем прямого слияния VL-CLmAb746 или VL-CLmAb747-42, поскольку в этом случае может находиться на его C-конце с N-концом VH-CH1mAb709 или VH-CH1mAb709-8, соответственно, без использования линкеров, соединяющих иммуноглобулиновые домены.
Векторы экспрессии, кодирующие полипептидные цепи #1, #2 и #3 каждого из FIT-Ig (FIT107-1-2a, FIT107-1-2b, FIT107-1-5a и FIT107-1-5b), временно ко-экспрессировали, используя полиэтиленимин (PEI) в качестве агента трансфекции в клетках 293E эмбриональной почки человека. Вкратце, ДНК в экспрессионной среде FreeStyle™ 293 смешивали с PEI с конечной концентрацией ДНК к PEI в соотношении 1:2, инкубировали в течение 15-20 минут при комнатной температуре, а затем добавляли к клеткам HEK293E (1,0-1,2×106/мл, жизнеспособность клеток >95%) при 60 мкг ДНК/120 мл культуры. Через 6-24 часа культивирования в шейкере к трансфицированным клеткам добавляли пептон до конечной концентрации 5% при встряхивании со скоростью 125 об/мин при 37°C и ±8% CO2. На 6-7 день супернатант собирали центрифугированием и фильтрованием, и FIT-Ig белок очищали с помощью хроматографии с белком А (Pierce, Rockford, IL) в соответствии с инструкциями производителя.
Для экспрессии ДНК FIT107-1-2a, FIT107-1-2b, FIT107-1-5a и FIT107-1-5b, кодирующей для экспрессирования цепей #1, #2 и #3, трансфицировали, используя молярное соотношение Цепь #1:Цепь #2:Цепь #3, равное 1:3:3. Это было разработано для пропорционального увеличения экспрессии коротких цепей #2 и #3 по сравнению с длинной цепью (Цепь #1), что, в свою очередь, уменьшило бы появление сегментов VL-CL и VH-CH1 в длинной цепи (Цепь #1), которые не были спарены с соответствующими легкими цепями и, таким образом, не смогли бы образовать функциональный Fab-фрагмент. Продукты экспрессии FIT-Ig белка очищали c gjvjom. хроматографии с белком А. Состав и чистоту очищенных FIT-Ig анализировали с помощью эксклюзионной хроматографии (SEC). Очищенный FIT-Ig в PBS наносили на колонку TSKgel SuperSW3000, 300×4,6 мм (TOSOH). Прибор для ВЭЖХ модели U3000 (DIONEX) использовали для SEC с использованием УФ-детекции при 280 нм и 214 нм. См. таблицу 31 ниже.
Таблица 31: Анализ экспрессии и SEC PD-1/LAG-3-связывающих FIT-Ig белков
Более низкое содержание мономерной фракции для FIT107-1-2b, -5a и -5b указывает на некоторую возможную агрегацию.
Пример 11: Аффинность связывания FIT-Ig с целевыми антигенами
Кинетику связывания FIT-Ig с мишенями PD-1 и LAG-3 определяли с помощью измерений SPR Biacore. Значения аффинности связывания FIT107-1-2a и FIT107-1-2b с обоими целевыми антигенами PDL-1 и LAG-3 показаны в Таблице 32 ниже.
Таблица 32: Аффинность связывания FIT107-1-2a и FIT107-1-2b
HuLAG-3 ECD/Fc
внутренний: LAG-3
внутренний: PD-1
Пример 12: Определение специфичности и функции FIT-Ig
Анти-PD1 и анти-LAG-3 биспецифичность и биологическая активность связывающего белка FIT107-1-2a тестировали в анализе активации PBMC с использованием стафилококкового энтеротоксина B (SEB) в качестве суперантигена (см. пример 7.8). Вкратце, PBMC выделяли из здорового донора, затем высевали в 96-луночный планшет по 50 мкл/лунка с плотностью 2×105 клеток/лунка. Тестируемые связывающие белки (например, FIT107-1-2a, комбинация коммерчески доступных моноклональных анти-PD-1 и анти-LAG-3 антител, или моноклональное анти-PD-1 антитело) добавляли в планшеты и инкубировали с PBMC при 37°C в течение 30 мин. Раствор SEB добавляли до конечной концентрации 10 нг/мл. Планшеты инкубировали в течение 96 часов, затем собирали 100 мкл супернатанта клеточной культуры и измеряли уровень продуцирования IL-2 с помощью набора для определения IL-2 для ELISA (R&D Systems; Кат. № DY202). Результаты показаны на фиг. 7. Биспецифический FIT-Ig белок FIT107-1-2a способен усиливать активацию Т-клеток, на что указывает уровень продуцирования IL-2, по сравнению с одним анти-PD-1 антителом или смесью анти-LAG-3 и анти-PD-1 антител.
Кроме того, анализ реакции смешанной культуры лимфоцитов (MLR) выполняли аналогично тому, как описано в Примере 3, для дополнительной проверки анти-PD-1 и анти-LAG-3 функции FIT107-1-2a. Реакция смешанной культуры лимфоцитов - это клеточный иммунный ответ ex vivo, который возникает между двумя популяциями аллогенных лимфоцитов при смешивании друг с другом. Аллогенные лимфоциты претерпевают бластную трансформацию, синтез ДНК и клеточную пролиферацию в ответ на различия основных антигенов гистосовместимости (MHC Class I и II) между двумя популяциями клеток, которые обозначены как клетки-респондеры и клетки-стимуляторы. При MLR для тестирования FIT107-1-2a в день 1 очищали PBMC, полученные от здоровых доноров и выделяли моноциты CD14+. Моноциты высевали в 6-луночные планшеты и обрабатывали 35 нг/мл IL-4 (R&D Systems) и 50 нг/мл GM-CSF (R&D Systems) в среде RPMI 1640 плюс 10% FBS. Среду заменяли через 4 дня. На седьмой день моноциты, дифференцированные в незрелые дендритные клетки, собирали и дополнительно обрабатывали в течение двух дней в среде для созревания с 20 нг/мл TNF-α (R&D Systems), 50 мкг/мл Poly I:C (Sigma), 35 н/мл IL-4 и 50 нг/мл GM-CSF. Для анализов MLR при совместном культивировании использовали бессывороточную среду X-VIVO™ 15, чтобы избежать влияния сыворотки на эффективность антител. 96-луночные планшеты с U-образным дном засевали аллогенными CD4+ Т-клетками (клетками-респондерами) из расчета 1×105 клеток/лунку и предварительно обрабатывали тестируемым связывающим белком (т.е. FIT107-1-2a, комбинацией коммерчески доступных моноклональных анти-PD-1 и анти-LAG-3 антител, или моноклональных анти-PD-1 антител) в течение 30 мин. Затем зрелые дендритные клетки (клетки-стимуляторы) высевали в лунки с плотностью 1×104 клеток/лунку и культивировали совместно с клетками-респондерами в течение пяти дней, за это время собирали 100 мкл супернатанта и измеряли уровень продуцирования IFN-γ с помощью ELISA. Результаты показаны на Фиг. 8. Биспецифический связывающий FIT-Ig белок показал ЕС50, равный 0,5084 нМ, по сравнению со значением ЕС50, составляющим 16,84 нМ для комбинации анти-PD-1 и анти-LAG-3 антител или только анти-PD-1 антитела. Таким образом, связывающий FIT-Ig белок усиливал продуцирование IFN-γ (гамма-интерферона) в MLR в концентрации, которая более чем в 30 раз ниже, чем концентрация одного антитела или комбинации антител.
Пример 13: Гуманизация mAb747 из новой партии
Гены вариабельной области анти-LAG-3 mAb747 использовали для создания гуманизированного антитела. На первом этапе этого процесса аминокислотные последовательности VH и VL mAb747 (SEQ ID NO:60 и SEQ ID NO:62) сравнивали с доступной базой данных последовательностей V-генов человеческого Ig, чтобы найти в целом наиболее подходящие последовательности V-генов человеческого Ig зародышевой линии. Кроме того, последовательность каркасной области 4 (FW4) VH или VL сравнивали с базой данных J-областей, чтобы найти человеческую каркасную область, имеющую наивысшую гомологию с мышиными областями VH и VL, соответственно. Наиболее близкое соответствие человеческого V-гена легкой цепи показал ген А30; и наиболее близкое соответствие человеческой тяжелой цепи показал ген VH1-69-2. Затем конструировали гуманизированные последовательности вариабельных доменов, в которых CDR-L1, CDR-L2 и CDR-L3 легкой цепи mAb747 прививали на каркасные последовательности гена А30 с последовательностью каркасной области 4 JK4 после CDR-L3; и CDR-H1, CDR-H2 и CDR-H3 тяжелой цепи mAb747 прививали на каркасные последовательности VH1-69-2 с последовательностью каркасной области 4 JH6 после CDR-H3. Затем создавали трехмерную Fv-модель mAb747, чтобы определить, существуют ли какие-либо положения каркаса, в которых мышиные аминокислоты являются критическими для сохранения петлевых структур или интерфейса VH/VL. Такие остатки в гуманизированных последовательностях следует подвергнуть обратной мутации на мышиные остатки в том же положении для сохранения аффинности/активности. Было идентифицировано несколько желательных обратных мутаций для VH и VL mAb747, и были созданы три альтернативные конструкции VH и VL, показанные в таблице 33 ниже. (Обратно мутированные аминокислотные остатки каркасной области обозначены двойным подчеркиванием; CDR подчеркнуты в соответствии с системой нумерации Кабат, за исключением VH CDR1, определенной с помощью системы нумерации ABM.)
Таблица 33: Дизайн гуманизированных VH/VL для mAb747 - обратные мутации на мышиные остатки
1234567890123456789012345678901234567890
Гуманизированные гены VH и VK получали синтетически и затем клонировали в векторы, содержащие константные домены человеческой каппа-цепи, соответственно. Спаривание человеческой VH и человеческой VK привело к образованию 9 гуманизированных анти-LAG-3 антител, названы HumAb747-43 - HumAb747-60 (таблица 60). Для сравнения аффинности также использовали (mAb747c) в качестве положительного контроля химерное антитело с родительскими мышиными VH/VL и человеческими константными последовательностями, описанными выше.
Таблица 34: Список полученных гуманизированных анти-LAG-3 антител mAb747
Все 18 гуманизированных антител (таблица 34) и химерное антитело, имеющее родительские мышиные домены VH и VL (mAb747c), ранжировали по константе скорости диссоциации (koff). Вкратце, антитела характеризовали по аффинности и кинетике связывания методом биослойной интерферометрии, выполненным на устройстве Octet® RED96 (Pall FortéBio LLC). Антитела захватывали биосенсорами Anti-HIgG Fc Capture (AHC) (Pall) с концентрацией 100 нМ в течение 30 секунд. Затем сенсоры погружали в рабочий буфер (1X pH 7,2 PBS, 0,05% Твин 20, 0,1% BSA) на 60 секунд для проверки исходного уровня. Связывание измеряли путем погружения сенсоров в одну концентрацию рекомбинантного человеческого белка LAG-3-his (новопротеин). Диссоциацию получали путем погружения сенсоров в рабочий буфер на 1200 секунд. Кривые ассоциации и диссоциации подгоняли к модели связывания Ленгмюра 1:1 с помощью программного обеспечения FortéBio Data Analysis (Pall). Результаты приведены в таблице 35. В каждой тестовой группе скорости диссоциации антител можно было сравнить со скоростью диссоциации mAb747c. Отношения скоростей диссоциации вычисляли путем отношения скорости диссоциации антитела к скорости диссоциации mAb747c в группе и сравнивали все значения. Чем ниже отношение, тем выше аффинность антитела.
Таблица 35: Ранжирование гуманизированных анти-LAG-3 антител по скорости диссоциации
(1/с)
HumAb747-60 показал константу скорости диссоциации, которая превышала скорость диссоциации контрольного химерного антитела с родительскими вариабельными доменами, только 1,5 раза.
Для дополнительной проверки функции анти-LAG-3 антител в человеческих PBMC выполняли анализ стимуляции бактериального токсина с использованием суперантигенного энтеротоксина B Staphylococcus aureus (SEB). Продуцирование IL-2 измеряли с помощью набора для определения IL-2 PerkinElmer (PerkinElmer; кат. № TRF1221M). HumAb747-60 был способен усиливать секрецию IL-2 PBMC, стимулированных SEB, путем блокирования сигнального пути LAG-3. Результаты показаны на фиг. 9. Таким образом, HumAb747-60 оказался функциональным и был выбран для дальнейшей разработки.
Пример 14: Созревание аффинности HumAb747-60
Пример 14.1: Создание и скрининг библиотеки созревания аффинности
Хотя HumAb747-60 показало константу скорости диссоциации, которая превышала скорость диссоциации химерного контрольного mAb747c, только в 1,5 раза, результаты анализа PBMC-SEB показали, что HumAb747-60 имеет несколько более слабую функциональную активность. Для дальнейшего улучшения аффинности остатки CDR (по системе нумерации ABM) оптимизировали путем созревания аффинности на основе HumAb747-39 (см. таблицы 24, 25). Были спроектированы и созданы две фаговые библиотеки. Одна была разработана для получения мутированных CDR-L1, CDR-L3 и CDR-H3 (нумерация ABM), каждая из которых имела один случайно мутированный остаток. Другая была разработана для получения мутированных CDR-L2, CDR-H1 и CDR-H2 (нумерация ABM), каждая из которых имела один случайно мутированный остаток.
Библиотеки фагового дисплея создавали с помощью метода, описанного в Journal of Immunological Methods, 201:35-55 (1997). Вкратце, VH-CH1 и VL-CL амплифицировали с вырожденными праймерами, которые вводят мутации, а затем последовательно клонировали в два сайта множественного клонирования (MCS) фагемидного вектора. Затем фагемидные векторы электротрансформировали в TG1 (кат. номер 60502-1, Lucigen), в результате чего получали библиотеки соответственно из приблизительно 1,2×108 клонов, демонстрирующие высокое разнообразие последовательностей. Библиотеки спасали с помощью фага-хелпера M13K07 (Кат. № N0315S) в соотношении приблизительно 1:20 (клетка на фаг). Отбор библиотеки фагового дисплея выполняли с помощью рекомбинантного человеческого белка LAG-3 с последующими стадиями промывки. Fab-фрагмент HumAb747-39 также конструировали с помощью фагемидного вектора и использовали в качестве положительного контроля. Выбранные фаги использовали для заражения клеток-хозяев. Связывающую способность супернатантов Fab из отдельных клонов скринировали с помощью ELISA. Вкратце, супернатанты Fab отдельных клонов получали путем культивирования в течение ночи при 30°C с 1 мМ IPTG. 2 мкг/мл человеческого белка LAG-3 в 100 мкл забуференного фосфатом физиологического раствора (PBS) наносили непосредственно на каждую лунку 96-луночных планшетов. Связанное с HRP анти-c-myc антитело (кат. номер Ab1261, Abcam) и реагент TMB использовали для обнаружения и проявки сигнала ELISA, который считывали с помощью планшетного ридера (планшетный ридер SpectraMax® Plus 384, Molecular Devices) при длине волны 450 нм.
Положительные клоны в скрининговом ELISA отбирали для секвенирования. Учитывая избыточность последовательностей и силу сигнала в скрининговом ELISA, для дальнейшей оценки были отобраны приведенные ниже 7 клонов. Последовательности VH/VL показаны в Таблице 36. (Мутировавшие аминокислотные остатки, идентифицированные по созреванию аффинности, обозначены двойным подчеркиванием; CDR подчеркнуты в соответствии с системой нумерации Кабат).
Таблица 36: Аминокислотные последовательности VH/VL 7-ми содержащими мутации антител с созревшей аффинностью
1234567890123456789012345678901234567890
Пример 14.2: Преобразование IgG и характеристика положительных клонов
Семь клонов Fab были преобразованы в полные белки IgG. Вкратце, гены VH и VL получали синтетически, а затем клонировали в векторы, содержащие кодирующие последовательности для константных доменов человеческого IgG1 и человеческой каппа-цепи, соответственно. Плазмиды тяжелой цепи и родственной легкой цепи котрансфицировали в клетки HEK 293E по отдельности. Примерно через шесть дней культивирования клеток после трансфекции супернатанты собирали и подвергали аффинной хроматографии с белком А. Очищенные антитела ранжировали по значениям аффинности, используя метод биослойной интерферометрии на устройстве Octet® RED96 (см. пример 9.1 выше). Результаты приведены в таблице 37.
Таблица 37: Ранжирование скорости диссоциации анти-LAG-3 антител после созревания аффинности
D1-70-IgG и B2-53-IgG показали минимально увеличенную константу скорости диссоциации по сравнению с таковой антитела HumAb747-39, что является отражением наибольшего увеличения аффинности после мутаций. Следовательно, D1-70-IgG и B2-53-IgG, содержащие мутации, введенные в последовательность HumAb747-60, оказались наилучшими кандидатами после гуманизации.
Пример 14.3: Создание и характеристика антител, сконструированных дополнительно
Мутации в D1-70, идентифицированные в процессе созревания аффинности, представляли собой D26G, E53R и E58M в домене VH и S56L в домене VL (положение остатка определено по системе нумерации Кабат). Мутации в B2-53, идентифицированные в процессе созревания аффинности, представляли собой D26G и E58V в домене VH и T53A в домене VL (положение остатка определено по системе нумерации Кабат). Эти мутации включали в последовательности VH/VL HumAb747-60, отдельно или в комбинации.
В CDR-H2 HumAb747-60 имелся NG-паттерн, который мог привести к гетерогенности во время создания антител в результате реакций дезаминирования, поэтому также оценивали мутацию NG в NA. Согласно рассчетам, мутация G55A в домене VH не приводила бы к нарушению активности HumAb747-60 при нарушении паттерна NG. Аминокислотные последовательности для вариантов антител, включая мутации, обсуждаемые выше, приведены в таблице 38. (CDR определены согласно нумерации Кабат.)
Таблица 38: Разработанная конструкция VH/VL для HumAb747-60
1234567890123456789012345678901234567890
Сконструированные гены VH и VK получали синтетически и затем соответственно клонировали в векторы, содержащие константные домены человеческого IgG1 и человеческой каппа-цепи. Спаривание человеческой VH и человеческой VK привело к образованию 13 сконструированных анти-LAG-3 антител, названных HumAb747V-61 - HumAb747V-73 (таблица 25). В качестве положительного контроля для сравнения аффинности также получали химерное антитело с родительскими мышиными VH/VL и человеческими константными последовательностями (mAb747c).
Таблица 39: Список полученных сконструированных анти-LAG-3 антител
Все 13 гуманизированных антител (таблица 39) и химерное анти-LAG-3 антитело mAb747c, имеющее родительские мышиные домены VH и VL, ранжировали по константе скорости диссоциации (koff) таким же образом, как описано в примере 9.1 выше. Результаты приведены в таблице 40. В каждой тестовой группе скорости диссоциации антител можно было сравнить со скоростью диссоциации mAb747c. Отношения скоростей диссоциации вычисляли путем отношения скорости диссоциации антитела к скорости диссоциации mAb747c в группе, и сравнивали все полученные значения. Чем ниже отношение, тем выше аффинность антитела.
Таблица 40: Ранжирование анти-LAG-3 антител с дальнейшей разработкой
Большинство антител на основе HumAb747-60, содержащие мутации в CDR, принятые после созревания аффинности, показали улучшенную скорость диссоциации по сравнению со скоростью диссоциации mAb747c, что предсказывает улучшение аффинности. HumAb747V-61 показало скорость диссоциации, аналогичную скорости диссоциации mAb747, что указывает на то, что аминокислотная замена VH G55A не ухудшает аффинность. Антитела с отношениями скоростей диссоциации ниже 60% относительно скорости диссоциации mAb747c в пределах тестовой группы, дополнительно оценивали с помощью клеточного функционального анализа.
Анти-LAG-3 активность тестировали в анализе активации PBMC с использованием стафилококкового энтеротоксина B (SEB) в качестве суперантигена. Вкратце, PBMC высевали в 96-луночный планшет в количестве 2×105 клеток/лунку. Тестовые белки (моноклональные анти-LAG-3 антитела) добавляли в планшеты и инкубировали с PBMC при 37°C в течение 30 мин. Раствор SEB добавляли с конечной концентрацией 10 нг/мл. Планшеты инкубировали в течение 96 часов, затем собирали 100 мкл супернатанта клеточной культуры и измеряли уровень продуцирования IL-2 с помощью набора для определения IL-2 PerkinElmer (PerkinElmer; кат. номер TRF1221M). Результаты показаны на фиг. 10. Результаты показывают, что сконструированные варианты антител могут усиливать продуцирование IL-2 из стимулированных SEB PBMC путем блокирования передачи сигналов, опосредованной LAG-3.
Судя по результатам анализа PBMC-SEB, HumAb747V-67, HumAb747V-72 и HumAb747V-73 продемонстрировали превосходную активность по блокированию LAG-3; поэтому эти три антитела использовали для создания связывающих FIT-Ig белков, нацеленных на LAG-3, а также на PD-1.
Пример 15: Создание PD-1/LAG-3 FIT-Ig с использованием новых последовательностей анти-LAG-3 антител
Анти-LAG-3 антитела HumAb747V-67, HumAb747V-72 и HumAb747V-73, полученные, как описано выше, и два анти-PD-1 антитела, HumAb709-8 (см. Таблицы 5 и 7 выше) и HumAb713-7 (см. Таблицы 9 и 10 выше), использовали для создания связывающих FIT-Ig белков, следуя процедурам, описанным выше в примере 10. Мутацию G55A включали в структуру последовательностей всех доменов VH ани-LAG-3 Fab-фрагментов.
Пример 15.1: Создание PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-6a-1
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-6a-1, конструировали, используя кодирующие последовательности для доменов иммуноглобулина из родительских антител mAb709-8 (гуманизированное анти-PD-1, см. таблицы 5 и 6 выше) и HumAb747V-67 (гуманизированное анти-LAG-3, см. таблицы 38 и 39 выше). FIT-Ig FIT107-1-6a-1 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb709-8, слитые непосредственно с VH-CH1 HumAb747V-67, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1 (см. таблицу 6 выше);
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb709-8; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb747V-676.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-6a-1 показаны в Таблице 41 ниже.
Таблица 41: Аминокислотные последовательности цепей компонентов FIT107-1-6a
1234567890123456789012345678901234567890
HumAb709-8
(VL подчеркнута)
HumAb747V-67
(VH подчеркнута)
человеческого IgG1
PDSVKGRFTISRDNAKNSLYLQMNSLRAEDTAVYYCAGQGGNYLFAYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb709-8
(VH подчеркнута)
HumAb747V-67
(VL подчеркнута)
Пример 15.2: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-6b-1
Конструировали другой биспецифический Fabs-in-Tandem иммуноглобулин, распознающий как человеческий PD-1, так и человеческий LAG-3. Этот PD-1/LAG-3 FIT-Ig обозначен как FIT107-1-6b-1. При конструировании связывающего белка FIT107-1-6b-1 использовали кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb747V-67 (анти-LAG-3) и HumAb709-8 (анти-PD-1). Эта конструкция FIT-Ig имела LAG-3-связывающий домен в N-концевом (внешнем) положении и PD-1-связывающий домен во внутреннем положении, слитом на C-конце с доменами VL-CL LAG-3-связывающей области. FIT-Ig FIT107-1-6b-1 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb747-67, слитые непосредственно с VH-CH1 HumAb709-8, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1 (см. таблицу 6 выше);
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb747V-67; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb709-8.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-6b-1 показаны в Таблице 42 ниже:
Таблица 42: Аминокислотные последовательности цепей компонентов FIT107-1-6b-1
1234567890123456789012345678901234567890
FIT107-1-6b-1
HumAb747V-67 (VL подчеркнута)
HumAb709-8
(VH подчеркнута)
человеческого IgG1
FIT107-1-6b-1
ASKFQGKATITADTSTNTAYLELSSLRSEDTAVYYCTVYGDYWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb747V-67
(VH подчеркнута)
FIT107-1-6b-1
HumAb709-8
(VL подчеркнута)
Пример 15.3: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-6a-2
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-6a-2, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител mAb709-8 (гуманизированное анти-PD-1) и HumAb747V-72 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-6a-2 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb709-8, слитые непосредственно с VH-CH1 HumAb747V-72, слитые непосредственно с шарнир-CH2-CH3 мутантной константной области человеческого IgG1 (см. таблицу 6 выше);
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb709-8; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb747V-72.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-6a-2 показаны в Таблице 43 ниже.
Таблица 43: Аминокислотные последовательности цепей компонентов FIT107-1-6a
1234567890123456789012345678901234567890
FIT107-1-6a-2
HumAb709-8
(VL подчеркнута)
HumAb747V-72
(VH подчеркнута)
человеческого IgG1
FIT107-1-6a-2
PDSVKGRFTISRDNAKNSLYLQMNSLRAEDTAVYYCAGQGGNYLFAYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb709-8
(VH подчеркнута)
FIT107-1-6a-2
HumAb747V-72 (VL подчеркнута)
Пример 15.4: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-6b-2
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-6b-2, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb709-8 (гуманизированное анти-PD-1) и HumAb747V-72 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-6b-2 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb747V-72, слитые непосредственно с VH-CH1 HumAb709-8, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1;
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb747V-72; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb709-8.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-6b-2 показаны в Таблице 44 ниже.
Таблица 44: Аминокислотные последовательности цепей компонентов FIT107-1-6b-2
1234567890123456789012345678901234567890
FIT107-1-6b-2
HumAb747V-72 (VL подчеркнута)
HumAb709-8 (VH подчеркнута)
человеческого IgG1
FIT107-1-6b-2
ASKFQGKATITADTSTNTAYLELSSLRSEDTAVYYCTVYGDYWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb747V-72
(VH подчеркнута)
FIT107-1-6b-2
HumAb709-8
(VL подчеркнута)
Пример 15.5: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-6a-3
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-6a-3, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител mAb709-8 (гуманизированное анти-PD-1) и HumAb747V-73 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-6a-3 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb709-8, слитые непосредственно с VH-CH1 HumAb747V-73, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1;
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb709-8; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb747V-73.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-6a-3 показаны в Таблице 45 ниже.
Таблица 45: Аминокислотные последовательности цепей компонентов FIT107-1-6a
1234567890123456789012345678901234567890
FIT107-1-6a-3
HumAb709-8
(VL подчеркнута)
HumAb747V-73
(VH подчеркнута)
человеческого IgG1
FIT107-1-6a-3
PDSVKGRFTISRDNAKNSLYLQMNSLRAEDTAVYYCAGQGGNYLFAYWGQGTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb709-8
(VH подчеркнута)
FIT107-1-6a-3
HumAb747V-73
(VL подчеркнута)
Пример 15.6: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-6b-3
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-6b-3, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb709-8 (гуманизированное анти-PD-1) и HumAb747V-73 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-6b-3 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb747V-73, слитые непосредственно с VH-CH1 HumAb709-8, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1;
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb747V-73; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb709-8.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-6b-3 показаны в Таблице 46 ниже.
Таблица 46: Аминокислотные последовательности цепей компонентов FIT107-1-6b-3
1234567890123456789012345678901234567890
FIT107-1-6b-3
HumAb747V-73 (VL подчеркнута)
HumAb709-8 (VH подчеркнута)
человеческого IgG1
FIT107-1-6b-3
ASKFQGKATITADTSTNTAYLELSSLRSEDTAVYYCTVYGDYWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb747V-73
(VH подчеркнута)
FIT107-1-6b-3
HumAb709-8
(VL подчеркнута)
Пример 15.7: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-7a-1
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-7a-1, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb713-7 (гуманизированное анти-PD-1, см. таблицы 9 и 10 выше) и HumAb747V-67 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-7a-1 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb713-7, слитые непосредственно с VH-CH1 HumAb747V-67, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1;
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb713-7; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb747V-67.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-7a-1 показаны в Таблице 47 ниже.
Таблица 47: Аминокислотные последовательности цепей компонентов FIT107-1-7a
1234567890123456789012345678901234567890
FIT107-1-7a-1
HumAb713-7
(VL подчеркнута)
HumAb747V-67
(VH подчеркнута)
человеческого IgG1
FIT107-1-7a-1
ADTVKGRFTISRDNAKNSLYLQMNSLRDEDTAVYYCAKRGGSSHVNVMDYWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb713-7
(VH подчеркнута)
FIT107-1-7a-1
HumAb747V-67
(VL подчеркнута)
Пример 15.8: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-7b-1
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-7b-1, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb713-7 (гуманизированное анти-PD-1) и HumAb747V-67 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-7b-1 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb747V-67, слитые непосредственно с VH-CH1 HumAb713-7, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1;
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb747V-67; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb713-7.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-7b-1 показаны в Таблице 48 ниже.
Таблица 48: Аминокислотные последовательности цепей компонентов FIT107-1-7b-1
1234567890123456789012345678901234567890
FIT107-1-7b-1
HumAb747V-67 (VL подчеркнута)
HumAb713-7 (VH подчеркнута)
человеческого IgG1
FIT107-1-7b-1
ASKFQGKATITADTSTNTAYLELSSLRSEDTAVYYCTVYGDYWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb747V-67
(VH подчеркнута)
FIT107-1-7b-1
HumAb713-7
(VL подчеркнута)
Пример 15.9: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-7a-2
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-7a-2, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb713-7 (гуманизированное анти-PD-1) и HumAb747V-72 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-7a-2 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb713-7, слитые непосредственно с VH-CH1 HumAb747V-72, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1 (см. таблицу 6 выше);
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb713-7; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb747V-72.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-7a-2 показаны в Таблице 49 ниже.
Таблица 49: Аминокислотные последовательности цепей компонентов FIT107-1-7a
1234567890123456789012345678901234567890
FIT107-1-7a-2
HumAb713-7
(VL подчеркнута)
HumAb747V-72
(VH подчеркнута)
человеческого IgG1
FIT107-1-7a-2
HumAb713-7
(VH подчеркнута)
FIT107-1-7a-2
HumAb747V-72
(VL подчеркнута)
Пример 15.10: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-7b-2
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-7b-2, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb713-7 (гуманизированное анти-PD-1) и HumAb747V-72 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-7b-2 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb747V-72, слитые непосредственно с VH-CH1 HumAb713-7, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1;
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb747V-72; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb713-7.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-7b-2 показаны в Таблице 50 ниже.
Таблица 50: Аминокислотные последовательности цепей компонентов FIT107-1-7b-2
1234567890123456789012345678901234567890
FIT107-1-7b-2
HumAb747V-72 (VL подчеркнута)
HumAb713-7
(VH подчеркнута)
человеческого IgG1
FIT107-1-7b-2
ASKFQGKATITADTSTNTAYLELSSLRSEDTAVYYCTVYGDYWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb747V-72
(VH подчеркнута)
FIT107-1-7b-2
HumAb713-7
(VL подчеркнута)
Пример 15.11: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-7a-3
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-7a-3, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb713-7 (гуманизированное анти-PD-1) и HumAb747V-73 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-7a-3 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb713-7, слитые непосредственно с VH-CH1 HumAb747V-73, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1;
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb713-7; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb747V-73.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-7a-3 показаны в Таблице 51 ниже.
Таблица 51: Аминокислотные последовательности цепей компонентов FIT107-1-7a-3
1234567890123456789012345678901234567890
FIT107-1-7a-3
HumAb713-7
(VL подчеркнута)
HumAb747V-73
(VH подчеркнута)
человеческого IgG1
FIT107-1-7a-3
ADTVKGRFTISRDNAKNSLYLQMNSLRDEDTAVYYCAKRGGSSHVNVMDYWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb713-7
(VH подчеркнута)
FIT107-1-7a-3
HumAb747V-73
(VL подчеркнута)
Пример 15.12: Получение PD-1/LAG-3-связывающего FIT-Ig белка FIT107-1-7b-3
PD-1/LAG-3 FIT-Ig, обозначенный FIT107-1-7b-3, конструировали, используя кодирующие последовательности доменов иммуноглобулина из родительских антител HumAb713-7 (гуманизированное анти-PD-1) и HumAb747V-73 (гуманизированное анти-LAG-3). FIT-Ig FIT107-1-7b-3 представляет собой гексамер, состоящий из трехкомпонентных полипептидных цепей:
Полипептидная цепь #1 имеет формулу домена: VL-CL HumAb747V-73, слитые непосредственно с VH-CH1 HumAb713-7, слитые непосредственно с участком шарнир-CH2-CH3 мутантной константной области человеческого IgG1;
Полипептидная цепь #2 имеет формулу домена: VH-CH1 HumAb747V-73; и
Полипептидная цепь #3 имеет формулу домена: легкая цепь (VL-CL) HumAb713-7.
Аминокислотные последовательности трех экспрессируемых полипептидных цепей FIT107-1-7b-3 показаны в Таблице 52 ниже.
Таблица 52: Аминокислотные последовательности цепей компонентов FIT107-1-7b-3
1234567890123456789012345678901234567890
FIT107-1-7b-3
HumAb747V-73 (VL подчеркнута)
HumAb713-7 (VH подчеркнута)
человеческого IgG1
FIT107-1-7b-3
ASKFQGKATITADTSTNTAYLELSSLRSEDTAVYYCTVYGDYWGQGTTVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSC
HumAb747V-73
(VH подчеркнута)
FIT107-1-7b-3
HumAb713-7
(VL подчеркнута)
Пример 16: Характеристика новых FIT-Ig белков
Пример 16,1: Экспрессия и анализ SEC
12 связывающих FIT-Ig белков, описанных выше (таблицы 41-52), экспрессировали таким же образом, как описано выше в примере 10.5, и очищали с помощью хроматографии с белком А. Состав и чистоту очищенных FIT-Ig анализировали с помощью эксклюзионной хроматографии (SEC). Очищенный FIT-Ig в PBS наносили на колонку TSKgel SuperSW3000, 300×4,6 мм (TOSOH). Прибор для ВЭЖХ DIONEX™ UltiMate 3000 (Thermo Scientific) использовали для выполнения SEC, используя УФ-детектирование при 280 нм и 214 нм. См. таблицу 53 ниже.
Таблица 53: Экспрессия и SEC анализ связывающих PD-1/LAG-3 FIT-Ig белков
FIT-Ig белки, которые имели более низкое содержание мономерной фракции (<80%), исключали из дальнейших исследований по определению характеристик белков.
Пример 16.2: Функциональные анализы
Активность PD-1/LAG-3 FIT-Ig тестировали в анализе активации PBMC с использованием стафилококкового энтеротоксина B (SEB) в качестве суперантигена, как описано в примере 12. Результаты показаны на фиг. 11. Результаты показали, что все протестированные варианты FIT-Ig могут усиливать секрецию IL-2 из PBMC, стимулированных SEB. Это увеличение было по каким-то причинам инвертировано при использовании самых высоких доз FIT107-1-7b-2 и FIT107-1-7b-3, поэтому этим двум FIT-Ig белкам не было отдано предпочтение в качестве лидирующих молекул.
Пример 16.3: Связывающая активность
Кинетики связывания FIT-Ig с мишенями PD-1 и LAG-3 определяли методом биослойной интерферометрии с помощью системы Octet® RED96 (Pall FortéBio LLC). Аффинности связывания к обоим целевым антигенам PD-1 и LAG-3 показаны в таблице 54 ниже. Все FIT-Ig белки сохраняли аффинность как к huPD-1, так и к huLAG-3. Все FIT-Ig белки, которые были на активность к антигенам яванского макака, также показали перекрестную реактивность с антигенами яванского макака.
Таблица 54: Аффинность связывания PD-1/LAG-3-связывающих FIT-Ig белков
Пример 16.4: Фармакокинетические данные, полученные на крысах
Исходя из чистоты после одноэтапной очистки, титра экспрессии при временной трансфекции, сохраненной аффинности связывания, а также функциональной активности в анализе PBMC-SEB, в качестве ведущей молекулы было отобрано FIT107-1-7b-1. Фармакокинетические свойства FIT107-1-7b-1 оценивали на самцах крыс Sprague-Dawley (SD). FIT-Ig белок вводили самцам крыс SD однократно внутривенно в дозе 5 мг/кг. Образцы сыворотки собирали в разные моменты времени в течение 28 дней: через 0, 5, 15 и 30 минут; 1, 2, 4, 8 и 24 часа; и 2, 4, 7, 10, 14, 21 и 28 дней серийного забора образцов крови через хвостовую вену, и анализировали с помощью общего ELISA. Вкратце, планшеты для ELISA покрывали козьим антителом к Fc человеческого IgG (Rockland, кат. № 609-101-017) в количестве 125 нг/лунку при 4°C в течение ночи, блокировали 1X PBS/1% BSA/0,05% Твин-20/0,05% ProClinТМ 300. Все образцы сыворотки сначала 20-кратно разбавляли блокирующим буфером. Дополнительное разведение выполняли, используя 5% объединенную крысиную сыворотку, и инкубировано на планшете в течение 60 минут при 37°C. Детектирование выполняли с помощью конъюгированного с пероксидазой козьего антитела к Fab человеческого IgG (Sigma-Aldrich; Кат. № A0293), и концентрации определяли с помощью стандартных кривых путем подгонки, используя четырех-параметрическую логистическую модель. Значения фармакокинетических параметров определяли с помощью некомпартментной модели с помощью программного обеспечения WinNonlin (Pharsight Corporation, Mountain View, CA). Согласно результатам, приведенным в таблице 55, FIT107-1-7b-1 продемонстрировало стабильные свойства in vivo.
Таблица 55: Фармакокинетические свойства FIT107-1-7b-1
Пример 16.5: Анализ блокирования рецептора FGL1 (RBA)
Согласно недавно опубликованным сообщениям, фибриноген-подобный белок 1 (FGL1) является основным функциональным лигандом LAG-3, независимым от MHC Class II (Wang J. et al., Cell, 176 (1): 334-47 (2019)). Блокада взаимодействия FGL1/LAG-3 антителами стимулирует опухолевый иммунитет. Для оценки блокирующей активности анти-LAG-3 антитела или FIT-Ig, FGL1 (Wuhan USCN, кат. № RPD022Hu01) разбавляли до 5 мкг/мл физиологическим раствором, используя фосфатный буфер Дульбекко, и добавляли по 100 мкл в 96-луночный планшет и инкубировали при 4°C в течение ночи. Планшет трижды промывали 300 мкл/лунку PBS+Твин 20 (PBST). Добавляли HumAb747V-67, FIT107-1-7b-1, hIgG (рабочая концентрация: 100 нМ, 10 нМ, 1 нМ и 0,1 нМ) и 1 мкг/мл биотинилированного LAG-3 (AcroBiosystem, кат. № H82E5) и инкубировали при комнатной температуре в течение 2 часов. Планшет трижды промывали 300 мкл/лунку PBST, затем считывали с помощью считывающего устройства для микропланшетов VARIOSKANТМ LUX (Thermo Scientific), используя протокол ELISA-Endpoint-TMB/HRP. Результаты показаны на фиг. 12. Результаты показали, что как FIT-Ig FIT107-1-7b-1, так и его исходное анти-LAG-3 антитело HumAb747V-67 могут блокировать связывание человеческого LAG-3 с белком FGL1.
Пример 16.6: Связывающая активность клеточного белка FIT107-1-7b-1 в первичных клетках.
Вышеупомянутые анализы продемонстрировали, что PD-1/LAG-3 FIT-Ig белки могут связывать рекомбинантные антигенные белки. Для дальнейшей оценки связывающей способности FIT107-1-7b-1 на поверхности клеток, родительские антитела HumAb713-7, HumAb747V-67 и биспецифический FIT-Ig FIT107-1-7b-1 биотинилировали, используя биотин в качестве реагента (Sigma, Cat № S3259). Нестимулированные РВМС ресуспендировали в количестве 5×106 клеток/мл. Для тестирования анти-PD-1 антитела (HumAb713-7) или анти-LAG-3 антитела (HumAb747V-67) добавляли 100 мкг/мл антитела, и реакционную смесь инкубировали при 37°C в течение 40 минут отдельно с последующими двумя промывками буфером FACS. Затем в лунки 96-луночного планшета добавляли 100 мкл, содержащие 5×105 РВМС/лунка (необработанная группа, группа, обработанная анти-PD-1 антителом, и группа, обработанная анти-LAG-3 антителом). Добавляли биотинилированные HumAb713-7, HumAb747V-67 и FIT107-1-7b-1 и инкубировали при 37°C в течение 40 минут (с конечной рабочей концентрацией, начиная со 100 нМ с 3-кратными серийными разведениями) с последующей двукратной промывкой буфером FACS. Добавляли FITC-стрептавидин и BV421-антитело к человеческому CD3, и аналитический планшет инкубировали при 4°C в течение 30 минут с последующей промывкой дважды буфером FACS. Планшет анализировали с помощью проточного цитометра Beckman Coulter CytoFlex. Группы со стимулированными PBMC стимулировали, используя анти-CD3 плюс анти-CD28 антитела в течение 72 часов для индукции экспрессии PD-1 и LAG-3 на Т-клетках. Связывание HumAb713-7, HumAb747V-67 и FIT107-1-7b-1 тестировали на стимулированных PBMC, используя стратегию группировки (необработанные, обработанные анти-PD-1 антителом и обработанные анти-LAG-3 антителом), аналогичную использованной в эксперименте с нестимулированными PBMC. Связывание тестируемых антител исследовали на субпопуляции CD3-T клеток методом FACS. Результаты показаны на Фиг. 13. Результаты показали, что FIT107-7b-1 проявляет уникальный паттерн связывания, указывающий на связывание как с мишенью PD-1, так и с мишенью LAG-3 на Т-клетках.
Содержание всех ссылок (включая литературные источники, патенты, заявки на патенты и веб-сайты), которые приведены в настоящей заявке, включены в настоящее описание в явном виде во всей своей полноте в качестве ссылки. Для практического осуществления изобретения используются, если не указано иное, обычные методы иммунологии, молекулярной биологии и клеточной биологии, которые хорошо известны в данной области.
Изобретение может быть реализовано в других конкретных формах без отступления от основных характеристик изобретения, описанных выше. Следовательно, вышеизложенные варианты осуществления следует рассматривать, как иллюстративные, а не ограничивающие раскрытое в настоящем описании изобретение. Объем изобретения определен прилагаемой формулой изобретения.
--->
СПИСКИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Shanghai EpimAb Biotherapeutics Co., Ltd.
XU, Xuan
GONG, Shiyong
WU, Chengbin
<120> ВЫСОКОАФФИННЫЕ АНТИ-PD-1 И АНТИ-LAG-3 АНТИТЕЛА И ПОЛУЧЕННЫЕ ИЗ
НИХ БИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ БЕЛКИ
<130> EPM-107.1PCT (PF190265PCT)
<140> PCT/CN2019/085164
<141> 2019-04-30
<150> PCT/CN2018/085468
<151> 2018-05-03
<160> 223
<170> PatentIn version 3.5
<210> 1
<211> 380
<212> Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок человеческого PD-1/Fc
<400> 1
Leu Asp Ser Pro Asp Arg Pro Trp Asn Pro Pro Thr Phe Ser Pro Ala
1 5 10 15
Leu Leu Val Val Thr Glu Gly Asp Asn Ala Thr Phe Thr Cys Ser Phe
20 25 30
Ser Asn Thr Ser Glu Ser Phe Val Leu Asn Trp Tyr Arg Met Ser Pro
35 40 45
Ser Asn Gln Thr Asp Lys Leu Ala Ala Phe Pro Glu Asp Arg Ser Gln
50 55 60
Pro Gly Gln Asp Cys Arg Phe Arg Val Thr Gln Leu Pro Asn Gly Arg
65 70 75 80
Asp Phe His Met Ser Val Val Arg Ala Arg Arg Asn Asp Ser Gly Thr
85 90 95
Tyr Leu Cys Gly Ala Ile Ser Leu Ala Pro Lys Ala Gln Ile Lys Glu
100 105 110
Ser Leu Arg Ala Glu Leu Arg Val Thr Glu Arg Arg Ala Glu Val Pro
115 120 125
Thr Ala His Pro Ser Pro Ser Pro Arg Pro Ala Gly Gln Phe Gln Ile
130 135 140
Glu Gly Arg Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
145 150 155 160
Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe
165 170 175
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
180 185 190
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
195 200 205
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
210 215 220
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
225 230 235 240
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
245 250 255
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
260 265 270
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
275 280 285
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
290 295 300
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
305 310 315 320
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
325 330 335
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
340 345 350
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
355 360 365
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375 380
<210> 2
<211> 380
<212> Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок PD-1 яванского макака/Fc
<400> 2
Leu Glu Ser Pro Asp Arg Pro Trp Asn Ala Pro Thr Phe Ser Pro Ala
1 5 10 15
Leu Leu Leu Val Thr Glu Gly Asp Asn Ala Thr Phe Thr Cys Ser Phe
20 25 30
Ser Asn Ala Ser Glu Ser Phe Val Leu Asn Trp Tyr Arg Met Ser Pro
35 40 45
Ser Asn Gln Thr Asp Lys Leu Ala Ala Phe Pro Glu Asp Arg Ser Gln
50 55 60
Pro Gly Gln Asp Cys Arg Phe Arg Val Thr Arg Leu Pro Asn Gly Arg
65 70 75 80
Asp Phe His Met Ser Val Val Arg Ala Arg Arg Asn Asp Ser Gly Thr
85 90 95
Tyr Leu Cys Gly Ala Ile Ser Leu Ala Pro Lys Ala Gln Ile Lys Glu
100 105 110
Ser Leu Arg Ala Glu Leu Arg Val Thr Glu Arg Arg Ala Glu Val Pro
115 120 125
Thr Ala His Pro Ser Pro Ser Pro Arg Pro Ala Gly Gln Phe Gln Ile
130 135 140
Glu Gly Arg Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
145 150 155 160
Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe
165 170 175
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
180 185 190
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
195 200 205
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
210 215 220
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
225 230 235 240
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
245 250 255
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
260 265 270
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
275 280 285
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
290 295 300
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
305 310 315 320
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
325 330 335
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
340 345 350
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
355 360 365
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375 380
<210> 3
<211> 380
<212> Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок мышиного PD-1 /Fc
<400> 3
Leu Glu Val Pro Asn Gly Pro Trp Arg Ser Leu Thr Phe Tyr Pro Ala
1 5 10 15
Trp Leu Thr Val Ser Glu Gly Ala Asn Ala Thr Phe Thr Cys Ser Leu
20 25 30
Ser Asn Trp Ser Glu Asp Leu Met Leu Asn Trp Asn Arg Leu Ser Pro
35 40 45
Ser Asn Gln Thr Glu Lys Gln Ala Ala Phe Cys Asn Gly Leu Ser Gln
50 55 60
Pro Val Gln Asp Ala Arg Phe Gln Ile Ile Gln Leu Pro Asn Arg His
65 70 75 80
Asp Phe His Met Asn Ile Leu Asp Thr Arg Arg Asn Asp Ser Gly Ile
85 90 95
Tyr Leu Cys Gly Ala Ile Ser Leu His Pro Lys Ala Lys Ile Glu Glu
100 105 110
Ser Pro Gly Ala Glu Leu Val Val Thr Glu Arg Ile Leu Glu Thr Ser
115 120 125
Thr Arg Tyr Pro Ser Pro Ser Pro Lys Pro Glu Gly Arg Phe Gln Ile
130 135 140
Glu Gly Arg Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
145 150 155 160
Pro Cys Pro Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe
165 170 175
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
180 185 190
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
195 200 205
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
210 215 220
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
225 230 235 240
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
245 250 255
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
260 265 270
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
275 280 285
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
290 295 300
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
305 310 315 320
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
325 330 335
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
340 345 350
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
355 360 365
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
370 375 380
<210> 4
<211> 117
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb701
<400> 4
Glu Val Leu Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Met Met Ser Trp Ile Arg Gln Thr Pro Glu Arg Arg Leu Glu Trp Val
35 40 45
Ala Ser Met Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Ser Ser Leu Arg Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg Arg Gly Thr Tyr Ala Met Asp Tyr Trp Gly Gln Gly Thr Ser
100 105 110
Val Thr Val Ser Ser
115
<210> 5
<211> 107
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb701
<400> 5
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Gln Ser Ala Ser Leu Gly
1 5 10 15
Glu Ser Val Thr Ile Thr Cys Leu Ala Ser Gln Thr Ile Gly Thr Trp
20 25 30
Leu Thr Trp Tyr Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Ala Ala Thr Ser Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Lys Phe Ser Phe Lys Ile Ser Ser Leu Gln Ala
65 70 75 80
Glu Asp Phe Val Ser Tyr Tyr Cys Gln Gln Leu Tyr Ser Thr Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 6
<211> 121
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb703
<400> 6
Asp Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Gln
1 5 10 15
Ser Leu Ser Leu Thr Cys Ser Val Thr Gly Tyr Ser Ile Thr Thr Gly
20 25 30
Tyr Tyr Trp Asn Trp Ile Arg Gln Phe Pro Gly Asn Lys Leu Glu Trp
35 40 45
Met Gly Tyr Met Ser Tyr Asp Gly Asn Asn Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Asn Arg Ile Ser Ile Thr Arg Asp Thr Ser Lys Asn Gln Phe Leu
65 70 75 80
Leu Arg Leu Asn Ser Val Thr Thr Glu Asp Thr Ala Thr Tyr Phe Cys
85 90 95
Ala Arg Asp Arg Gly Thr Thr Ile Leu Gly Gly Thr Met Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Ser Val Thr Val Ser
115 120
<210> 7
<211> 107
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb703
<400> 7
Ser Ile Val Met Thr Gln Thr Pro Lys Phe Leu Phe Val Ser Ala Gly
1 5 10 15
Asp Arg Val Thr Ile Ala Cys Lys Ala Ser Gln Ser Val Ser Asn Asp
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Ala Phe Tyr Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Tyr Gly Thr Asp Phe Thr Phe Thr Ile Ser Thr Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Val Tyr Phe Cys Gln Gln Asp Tyr Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 8
<211> 118
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb709
<400> 8
Glu Val Lys Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Thr Pro Glu Lys Arg Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu His Met Ser Ser Leu Arg Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ala
115
<210> 9
<211> 107
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb709
<400> 9
Asp Ile Val Met Thr Gln Ser His Lys Phe Met Ser Thr Ser Val Gly
1 5 10 15
Asp Ser Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Leu Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ala Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Leu Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Gln Leu Glu Ile Lys
100 105
<210> 10
<211> 121
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb713
<400> 10
Glu Val Lys Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Glu Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Glu Lys Gly Leu Glu Trp Val
35 40 45
Ala Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Met Thr Ser Leu Arg Ser Glu Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Ser Val Thr Val Ser Ser
115 120
<210> 11
<211> 107
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb713
<400> 11
Asp Ile Gln Met Thr Gln Ser Ser Ser Tyr Leu Ser Val Ser Leu Gly
1 5 10 15
Gly Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Asn Ala Pro Arg Leu Leu Ile
35 40 45
Ser Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Lys Asp Tyr Thr Leu Ser Ile Thr Ser Leu Gln Thr
65 70 75 80
Glu Asp Val Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 12
<211> 117
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb714
<400> 12
Glu Val His Leu Gln Gln Ser Gly Pro Glu Leu Val Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Ile Phe Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Asn
20 25 30
Asn Val Glu Trp Val Lys Gln Ser His Gly Lys Ser Leu Glu Trp Ile
35 40 45
Gly Asp Ile Asn Pro Asn Asn Gly Asp Thr Leu Tyr Ser Gln Tyr Phe
50 55 60
Lys Asp Lys Ala Thr Leu Thr Val Asp Lys Ser Ser Thr Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Ser Glu Asp Thr Gly Leu Tyr Tyr Cys
85 90 95
Ala Arg Gly Lys Ser Asp Gln Phe Asp Tyr Trp Gly Gln Gly Thr Thr
100 105 110
Leu Thr Val Ser Ser
115
<210> 13
<211> 107
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb714
<400> 13
Asp Ile Gln Met Thr Gln Ser Pro Ala Ser Gln Ser Ala Ser Leu Gly
1 5 10 15
Glu Ser Val Thr Ile Thr Cys Leu Ala Ser Gln Thr Ile Gly Thr Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ser Pro Gln Leu Leu Ile
35 40 45
Tyr Ala Ala Thr Ser Leu Ala Asp Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Lys Phe Ser Phe Lys Ile Ser Ser Leu Gln Gly
65 70 75 80
Glu Asp Phe Val Ser Tyr Tyr Cys Gln Gln Leu Tyr Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 14
<211> 118
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb715
<400> 14
Glu Val Met Leu Val Glu Ser Gly Gly Gly Leu Leu Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Thr Pro Glu Lys Arg Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Thr Ser Leu Arg Ser Glu Asp Thr Ala Phe Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Thr Tyr Leu Phe Ala Ser Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ala
115
<210> 15
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL mAb715
<400> 15
Asp Ile Val Met Thr Gln Ser His Lys Phe Met Ser Thr Ser Val Gly
1 5 10 15
Asp Ser Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Ala
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Pro Pro Lys Val Leu Ile
35 40 45
Tyr Trp Ala Ser Thr Arg His Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Leu Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 16
<211> 120
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb718
<400> 16
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Thr Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Glu Met His Trp Ala Lys Gln Thr Pro Val His Gly Leu Glu Trp Ile
35 40 45
Gly Val Ile Glu Pro Glu Ser Gly Gly Thr Val Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Lys Leu Thr Ala Asp Lys Ser Ser Arg Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Glu Gly Phe Asn Ser Asp His Tyr Phe Asp Tyr Trp Gly Gln
100 105 110
Gly Thr Thr Leu Thr Val Ser Ser
115 120
<210> 17
<211> 112
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb718
<400> 17
Asp Val Leu Met Thr Gln Thr Pro Leu Ser Leu Pro Val Ser Leu Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Arg Ser Ser Gln Asn Ile Val His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu Glu Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Lys Leu Leu Ile Tyr Lys Val Phe Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Phe Gln Gly
85 90 95
Ser His Val Pro Tyr Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 18
<211> 116
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb719
<400> 18
Glu Val Lys Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Thr Ala Ser Gly Phe Ser Phe Ser Ser His
20 25 30
Leu Met Ser Trp Val Arg Gln Thr Pro Glu Lys Arg Leu Glu Trp Val
35 40 45
Ala Ala Ile Ser Gly Gly Gly Ala Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Arg Ser Leu Arg Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Thr Arg Gln Ile Leu Ala Phe Asp Ser Trp Gly Gln Gly Thr Thr Leu
100 105 110
Thr Val Ser Ser
115
<210> 19
<211> 107
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb719
<400> 19
Asp Ile Gln Met Asn Gln Ser Pro Ser Ser Leu Ser Val Ser Leu Gly
1 5 10 15
Asp Thr Ile Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Asn Ile Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Gly Gly Ser Gly Thr Gly Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Ser Tyr Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 20
<211> 118
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb709 VH.1
<400> 20
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 21
<211> 118
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb709 VH.1A
<400> 21
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 22
<211> 118
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb709 VH.1B
<400> 22
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Arg Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser
115
<210> 23
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb709 VK.1A
<400> 23
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 24
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb709 VK.1B
<400> 24
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 25
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb709 VK.1C
<400> 25
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 26
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb709 VK.1D
<400> 26
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 27
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb709 VK.1E
<400> 27
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ser Leu Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 28
<211> 330
<212> Белок
<213> Homo sapiens
<400> 28
Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys
1 5 10 15
Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr
20 25 30
Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser
35 40 45
Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser
50 55 60
Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr
65 70 75 80
Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys
85 90 95
Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys
100 105 110
Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro
115 120 125
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
130 135 140
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
145 150 155 160
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
165 170 175
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
180 185 190
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn
195 200 205
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
210 215 220
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu
225 230 235 240
Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
245 250 255
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
260 265 270
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
275 280 285
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
290 295 300
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
305 310 315 320
Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
325 330
<210> 29
<211> 107
<212> Белок
<213> Homo sapiens
<400> 29
Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu
1 5 10 15
Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe
20 25 30
Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln
35 40 45
Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser
50 55 60
Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu
65 70 75 80
Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser
85 90 95
Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
100 105
<210> 30
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb713 VH.1
<400> 30
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 31
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb713 VH.1A
<400> 31
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 32
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb713 VH.1B
<400> 32
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 33
<211> 121
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb713 VH.1C
<400> 33
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 34
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb713 VK.1
<400> 34
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 35
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb713 VK.1A
<400> 35
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 36
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb713 VK.1B
<400> 36
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 37
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb713 VK.1C
<400> 37
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Ser Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Lys Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 38
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb703 VH.1A
<400> 38
Glu Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Tyr Ser Ile Ser Thr Gly
20 25 30
Tyr Tyr Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Tyr Met Ser Tyr Asp Gly Asn Asn Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Asn Arg Val Thr Ile Ser Arg Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Gly Thr Thr Ile Leu Gly Gly Thr Met Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 39
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb703 VH.1B
<400> 39
Glu Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Tyr Ser Ile Ser Thr Gly
20 25 30
Tyr Tyr Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Ile Gly Tyr Met Ser Tyr Asp Gly Asn Asn Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Asn Arg Ile Thr Ile Ser Arg Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Gly Thr Thr Ile Leu Gly Gly Thr Met Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 40
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb703 VH.1C
<400> 40
Glu Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Tyr Ser Ile Ser Thr Gly
20 25 30
Tyr Tyr Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Met Gly Tyr Met Ser Tyr Asp Gly Asn Asn Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Asn Arg Ile Thr Ile Ser Arg Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Gly Thr Thr Ile Leu Gly Gly Thr Met Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 41
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb703 VH.1D
<400> 41
Glu Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Tyr Ser Ile Thr Thr Gly
20 25 30
Tyr Tyr Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu Trp
35 40 45
Met Gly Tyr Met Ser Tyr Asp Gly Asn Asn Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Asn Arg Ile Thr Ile Ser Arg Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Asp Arg Gly Thr Thr Ile Leu Gly Gly Thr Met Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 42
<211> 122
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb703 VH.1E
<400> 42
Glu Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Ala Val Ser Gly Tyr Ser Ile Thr Thr Gly
20 25 30
Tyr Tyr Trp Asn Trp Ile Arg Gln Pro Pro Gly Lys Lys Leu Glu Trp
35 40 45
Met Gly Tyr Met Ser Tyr Asp Gly Asn Asn Asn Tyr Asn Pro Ser Leu
50 55 60
Lys Asn Arg Ile Thr Ile Ser Arg Asp Thr Ser Lys Asn Gln Phe Ser
65 70 75 80
Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ala Arg Asp Arg Gly Thr Thr Ile Leu Gly Gly Thr Met Asp Tyr Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 43
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb703 VK.1
<400> 43
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Ser Asn Asp
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Ala Phe Tyr Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Asp Tyr Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 44
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb703 VK.1A
<400> 44
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Ser Asn Asp
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Ala Phe Tyr Arg Tyr Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Tyr Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Asp Tyr Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 45
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb703 VK.1B
<400> 45
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Ser Asn Asp
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Ala Phe Tyr Arg Tyr Thr Gly Val Pro Asp Arg Phe Ser Gly
50 55 60
Ser Gly Tyr Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Asp Tyr Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 46
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb703 VK.1C
<400> 46
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Ser Asn Asp
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Ala Phe Tyr Arg Tyr Thr Gly Val Pro Asp Arg Phe Ser Gly
50 55 60
Ser Gly Tyr Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Asp Tyr Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 47
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb703 VK.1D
<400> 47
Ser Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Ser Val Ser Asn Asp
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ser Pro Lys Leu Leu Ile
35 40 45
Tyr Tyr Ala Phe Tyr Arg Tyr Thr Gly Val Pro Asp Arg Phe Thr Gly
50 55 60
Ser Gly Tyr Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Phe Cys Gln Gln Asp Tyr Ser Ser Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 48
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb719 VH.1
<400> 48
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Leu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Gly Gly Ala Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gln Ile Leu Ala Phe Asp Ser Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 49
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb719 VH.1A
<400> 49
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser His
20 25 30
Leu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Gly Gly Ala Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Gln Ile Leu Ala Phe Asp Ser Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 50
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb719 VH.1B
<400> 50
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser His
20 25 30
Leu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Gly Gly Ala Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Gln Ile Leu Ala Phe Asp Ser Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 51
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb719 VH.1C
<400> 51
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser His
20 25 30
Leu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ala Ile Ser Gly Gly Gly Ala Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Gln Ile Leu Ala Phe Asp Ser Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 52
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb719 VH.1D
<400> 52
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser His
20 25 30
Leu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Arg Leu Glu Trp Val
35 40 45
Ala Ala Ile Ser Gly Gly Gly Ala Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Gln Ile Leu Ala Phe Asp Ser Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 53
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb719 VH.1E
<400> 53
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser His
20 25 30
Leu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Ala Ile Ser Gly Gly Gly Ala Asp Thr Tyr Tyr Pro Ala Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Gln Ile Leu Ala Phe Asp Ala Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 54
<211> 116
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb719 VH.1F
<400> 54
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Ser Phe Ser Ser His
20 25 30
Leu Met Ser Trp Val Arg Gln Ala Pro Gly Lys Arg Leu Glu Trp Val
35 40 45
Ala Ala Ile Ser Gly Gly Gly Ala Asp Thr Tyr Tyr Pro Ala Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Arg Gln Ile Leu Ala Phe Asp Ala Trp Gly Gln Gly Thr Thr Val
100 105 110
Thr Val Ser Ser
115
<210> 55
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb719 VK.1
<400> 55
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Ser Tyr Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 56
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb719 VK.1A
<400> 56
Asp Ile Gln Met Asn Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Ser Tyr Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 57
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb719 VK.1B
<400> 57
Asp Ile Gln Met Asn Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys His Ala Ser Gln Asn Ile Tyr Val Trp
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ile Pro Lys Leu Leu Ile
35 40 45
Tyr Lys Ala Ser Asn Leu His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Gly Gln Ser Tyr Pro Trp
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 58
<211> 665
<212> Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок человеческого LAG-3 /Fc
<400> 58
Leu Gln Pro Gly Ala Glu Val Pro Val Val Trp Ala Gln Glu Gly Ala
1 5 10 15
Pro Ala Gln Leu Pro Cys Ser Pro Thr Ile Pro Leu Gln Asp Leu Ser
20 25 30
Leu Leu Arg Arg Ala Gly Val Thr Trp Gln His Gln Pro Asp Ser Gly
35 40 45
Pro Pro Ala Ala Ala Pro Gly His Pro Leu Ala Pro Gly Pro His Pro
50 55 60
Ala Ala Pro Ser Ser Trp Gly Pro Arg Pro Arg Arg Tyr Thr Val Leu
65 70 75 80
Ser Val Gly Pro Gly Gly Leu Arg Ser Gly Arg Leu Pro Leu Gln Pro
85 90 95
Arg Val Gln Leu Asp Glu Arg Gly Arg Gln Arg Gly Asp Phe Ser Leu
100 105 110
Trp Leu Arg Pro Ala Arg Arg Ala Asp Ala Gly Glu Tyr Arg Ala Ala
115 120 125
Val His Leu Arg Asp Arg Ala Leu Ser Cys Arg Leu Arg Leu Arg Leu
130 135 140
Gly Gln Ala Ser Met Thr Ala Ser Pro Pro Gly Ser Leu Arg Ala Ser
145 150 155 160
Asp Trp Val Ile Leu Asn Cys Ser Phe Ser Arg Pro Asp Arg Pro Ala
165 170 175
Ser Val His Trp Phe Arg Asn Arg Gly Gln Gly Arg Val Pro Val Arg
180 185 190
Glu Ser Pro His His His Leu Ala Glu Ser Phe Leu Phe Leu Pro Gln
195 200 205
Val Ser Pro Met Asp Ser Gly Pro Trp Gly Cys Ile Leu Thr Tyr Arg
210 215 220
Asp Gly Phe Asn Val Ser Ile Met Tyr Asn Leu Thr Val Leu Gly Leu
225 230 235 240
Glu Pro Pro Thr Pro Leu Thr Val Tyr Ala Gly Ala Gly Ser Arg Val
245 250 255
Gly Leu Pro Cys Arg Leu Pro Ala Gly Val Gly Thr Arg Ser Phe Leu
260 265 270
Thr Ala Lys Trp Thr Pro Pro Gly Gly Gly Pro Asp Leu Leu Val Thr
275 280 285
Gly Asp Asn Gly Asp Phe Thr Leu Arg Leu Glu Asp Val Ser Gln Ala
290 295 300
Gln Ala Gly Thr Tyr Thr Cys His Ile His Leu Gln Glu Gln Gln Leu
305 310 315 320
Asn Ala Thr Val Thr Leu Ala Ile Ile Thr Val Thr Pro Lys Ser Phe
325 330 335
Gly Ser Pro Gly Ser Leu Gly Lys Leu Leu Cys Glu Val Thr Pro Val
340 345 350
Ser Gly Gln Glu Arg Phe Val Trp Ser Ser Leu Asp Thr Pro Ser Gln
355 360 365
Arg Ser Phe Ser Gly Pro Trp Leu Glu Ala Gln Glu Ala Gln Leu Leu
370 375 380
Ser Gln Pro Trp Gln Cys Gln Leu Tyr Gln Gly Glu Arg Leu Leu Gly
385 390 395 400
Ala Ala Val Tyr Phe Thr Glu Leu Ser Ser Pro Gly Ala Gln Arg Ser
405 410 415
Gly Arg Ala Pro Gly Ala Leu Pro Ala Gly His Leu Ile Glu Gly Arg
420 425 430
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
435 440 445
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
450 455 460
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
465 470 475 480
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
485 490 495
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
500 505 510
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
515 520 525
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
530 535 540
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
545 550 555 560
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
565 570 575
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
580 585 590
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
595 600 605
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
610 615 620
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
625 630 635 640
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
645 650 655
Lys Ser Leu Ser Leu Ser Pro Gly Lys
660 665
<210> 59
<211> 665
<212> Белок
<213> Искусственная последовательность
<220>
<223> Слитый белок LAG-3 яванского макака/Fc
<220>
<221> misc_feature
<222> (52)..(52)
<223> Xaa может представлять собой любую встречающуюся в природе
аминокислоту
<400> 59
Pro Gln Pro Gly Ala Glu Ile Ser Val Val Trp Ala Gln Glu Gly Ala
1 5 10 15
Pro Ala Gln Leu Pro Cys Ser Pro Thr Ile Pro Leu Gln Asp Leu Ser
20 25 30
Leu Leu Arg Arg Ala Gly Val Thr Trp Gln His Gln Pro Asp Ser Gly
35 40 45
Pro Pro Ala Xaa Ala Pro Gly His Pro Pro Val Pro Gly His Arg Pro
50 55 60
Ala Ala Pro Tyr Ser Trp Gly Pro Arg Pro Arg Arg Tyr Thr Val Leu
65 70 75 80
Ser Val Gly Pro Gly Gly Leu Arg Ser Gly Arg Leu Pro Leu Gln Pro
85 90 95
Arg Val Gln Leu Asp Glu Arg Gly Arg Gln Arg Gly Asp Phe Ser Leu
100 105 110
Trp Leu Arg Pro Ala Arg Arg Ala Asp Ala Gly Glu Tyr Arg Ala Thr
115 120 125
Val His Leu Arg Asp Arg Ala Leu Ser Cys Arg Leu Arg Leu Arg Val
130 135 140
Gly Gln Ala Ser Met Thr Ala Ser Pro Pro Gly Ser Leu Arg Thr Ser
145 150 155 160
Asp Trp Val Ile Leu Asn Cys Ser Phe Ser Arg Pro Asp Arg Pro Ala
165 170 175
Ser Val His Trp Phe Arg Ser Arg Gly Gln Gly Arg Val Pro Val Gln
180 185 190
Gly Ser Pro His His His Leu Ala Glu Ser Phe Leu Phe Leu Pro His
195 200 205
Val Gly Pro Met Asp Ser Gly Leu Trp Gly Cys Ile Leu Thr Tyr Arg
210 215 220
Asp Gly Phe Asn Val Ser Ile Met Tyr Asn Leu Thr Val Leu Gly Leu
225 230 235 240
Glu Pro Ala Thr Pro Leu Thr Val Tyr Ala Gly Ala Gly Ser Arg Val
245 250 255
Glu Leu Pro Cys Arg Leu Pro Pro Ala Val Gly Thr Gln Ser Phe Leu
260 265 270
Thr Ala Lys Trp Ala Pro Pro Gly Gly Gly Pro Asp Leu Leu Val Ala
275 280 285
Gly Asp Asn Gly Asp Phe Thr Leu Arg Leu Glu Asp Val Ser Gln Ala
290 295 300
Gln Ala Gly Thr Tyr Ile Cys His Ile Arg Leu Gln Gly Gln Gln Leu
305 310 315 320
Asn Ala Thr Val Thr Leu Ala Ile Ile Thr Val Thr Pro Lys Ser Phe
325 330 335
Gly Ser Pro Gly Ser Leu Gly Lys Leu Leu Cys Glu Val Thr Pro Ala
340 345 350
Ser Gly Gln Glu His Phe Val Trp Ser Pro Leu Asn Thr Pro Ser Gln
355 360 365
Arg Ser Phe Ser Gly Pro Trp Leu Glu Ala Gln Glu Ala Gln Leu Leu
370 375 380
Ser Gln Pro Trp Gln Cys Gln Leu His Gln Gly Glu Arg Leu Leu Gly
385 390 395 400
Ala Ala Val Tyr Phe Thr Glu Leu Ser Ser Pro Gly Ala Gln Arg Ser
405 410 415
Gly Arg Ala Pro Gly Ala Leu Arg Ala Gly His Leu Ile Glu Gly Arg
420 425 430
Met Asp Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro
435 440 445
Ala Pro Glu Leu Leu Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
450 455 460
Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
465 470 475 480
Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr
485 490 495
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
500 505 510
Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
515 520 525
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
530 535 540
Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
545 550 555 560
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met
565 570 575
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
580 585 590
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
595 600 605
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
610 615 620
Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val
625 630 635 640
Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln
645 650 655
Lys Ser Leu Ser Leu Ser Pro Gly Lys
660 665
<210> 60
<211> 113
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb746 и VH mAb747
<400> 60
Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser
100 105 110
Ser
<210> 61
<211> 107
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb746
<400> 61
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Ser Leu Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Ser Asp Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 62
<211> 107
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb747
<400> 62
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Ser Leu Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Asp Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Ala Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 63
<211> 116
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb742
<400> 63
Gln Gly Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Thr Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Asn Asp Tyr
20 25 30
Glu Met His Trp Val Lys Gln Thr Pro Val His Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Asp Pro Glu Thr Gly Gly Thr Ala Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Ile Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Glu Leu Arg Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ile Arg Trp Gly Ser Thr Val Phe Pro Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser
115
<210> 64
<211> 112
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb742
<400> 64
Asp Gly Val Leu Thr Gln Thr Pro Leu Ser Leu Pro Val Asn Ile Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Lys Ser Thr Lys Ser Leu Leu Asn Ser
20 25 30
Asp Gly Phe Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Phe Gln Ser
85 90 95
Asn Tyr Leu Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 65
<211> 116
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb744
<400> 65
Gln Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Thr
1 5 10 15
Ser Val Thr Leu Ser Cys Lys Ala Ser Gly Tyr Thr Phe Thr Asp Tyr
20 25 30
Glu Met His Trp Met Lys Gln Thr Pro Val His Gly Leu Glu Trp Ile
35 40 45
Gly Ala Ile Asp Pro Ala Thr Gly Gly Thr Ala Tyr Asn Gln Lys Phe
50 55 60
Lys Gly Lys Ala Ile Leu Thr Ala Asp Lys Ser Ser Ser Thr Ala Tyr
65 70 75 80
Met Asp Phe Arg Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Ile Arg Trp Gly Thr Thr Val Phe Pro Tyr Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser
115
<210> 66
<211> 112
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb744
<400> 66
Asp Val Val Leu Thr Gln Thr Pro Leu Ser Leu Pro Val Asn Ile Gly
1 5 10 15
Asp Gln Ala Ser Ile Ser Cys Lys Ser Thr Lys Ser Leu Leu Asn Ser
20 25 30
Asp Gly Phe Thr Tyr Leu Asp Trp Tyr Leu Gln Lys Pro Gly Gln Ser
35 40 45
Pro Gln Leu Leu Ile Tyr Leu Val Ser Asn Arg Phe Ser Gly Val Pro
50 55 60
Asp Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Phe Gln Ser
85 90 95
Asn Tyr Leu Pro Trp Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 67
<211> 112
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb748
<400> 67
Glu Val Gln Met Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Thr Val Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Asp Pro Glu Asn Gly Asp Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Asn Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Tyr Phe Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser
100 105 110
<210> 68
<211> 112
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb748 и VL mAb750
<400> 68
Asp Val Val Met Thr Gln Thr Pro Leu Thr Leu Ser Val Thr Ile Gly
1 5 10 15
Gln Pro Ala Ser Ile Ser Cys Lys Ser Ser Gln Ser Leu Leu Asp Ser
20 25 30
Asp Gly Lys Thr Tyr Leu Asn Trp Leu Leu Gln Arg Pro Gly Gln Ser
35 40 45
Pro Lys Arg Leu Ile Tyr Leu Val Ser Lys Leu Asp Ser Gly Val Pro
50 55 60
Asp Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Lys Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Leu Gly Val Tyr Tyr Cys Trp Gln Gly
85 90 95
Ser His Phe Pro Gln Thr Phe Gly Gly Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 69
<211> 116
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb749
<400> 69
Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Val His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Asp Pro Glu Asn Gly Asp Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Phe Cys
85 90 95
Ser Thr Trp Asp Ala Glu Glu Asn Tyr Trp Gly Gln Gly Thr Thr Leu
100 105 110
Ser Val Ser Ser
115
<210> 70
<211> 112
<212> Белок
<213> Mus musculus
<220>
<223> VL mAb749
<400> 70
Asp Ile Val Leu Thr Gln Ala Ala Pro Ser Val Pro Val Thr Pro Gly
1 5 10 15
Glu Ser Val Ser Ile Ser Cys Arg Ser Ser Lys Ser Leu Leu His Ser
20 25 30
Asn Gly Asn Thr Tyr Leu Tyr Trp Phe Leu Gln Arg Pro Gly Gln Ser
35 40 45
Pro Gln Val Leu Ile Tyr Arg Met Ser Asn Leu Ala Ser Gly Val Pro
50 55 60
Val Arg Phe Ser Gly Ser Gly Ser Gly Thr Ala Phe Thr Leu Arg Ile
65 70 75 80
Ser Arg Val Glu Ala Glu Asp Val Gly Val Tyr Tyr Cys Met Gln His
85 90 95
Leu Glu Tyr Pro Phe Thr Phe Gly Ser Gly Thr Lys Leu Glu Ile Lys
100 105 110
<210> 71
<211> 116
<212> Белок
<213> Mus musculus
<220>
<223> VH mAb750
<400> 71
Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Thr Pro Ser Gly Leu Asn Ile Lys Asp Asp
20 25 30
Tyr Ile His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Asp Pro Glu Asn Gly Asp Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Ser Ala Val Tyr Tyr Cys
85 90 95
Cys Thr Ala Asp Tyr Arg Asn Trp Tyr Trp Gly Gln Gly Thr Thr Leu
100 105 110
Thr Val Ser Ser
115
<210> 72
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb747 VH.2A
<400> 72
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 73
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb747 VH.2B
<400> 73
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 74
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH mAb747 VH.1G
<400> 74
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Gln Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 75
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb747 VK.1E
<400> 75
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 76
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VK mAb747 VK.2A
<400> 76
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Lys Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 77
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> Vk mAb747 VK.2B
<400> 77
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 78
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-2a
Полипептидная цепь #1 FIT-Ig
<400> 78
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser His Lys Phe
20 25 30
Met Ser Thr Ser Val Gly Asp Ser Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln
50 55 60
Ser Leu Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ala Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Leu Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Gln Leu Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala Ser Val Lys Leu
245 250 255
Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp Tyr Met His Trp
260 265 270
Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile Gly Trp Ile Val
275 280 285
Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe Gln Gly Lys Ala
290 295 300
Thr Ile Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr Leu Gln Leu Ser
305 310 315 320
Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys Thr Val Tyr Gly
325 330 335
Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser Ser Ala Ser Thr
340 345 350
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
355 360 365
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
370 375 380
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
385 390 395 400
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
405 410 415
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
420 425 430
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
435 440 445
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
450 455 460
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
465 470 475 480
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
485 490 495
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
500 505 510
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
515 520 525
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
530 535 540
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
545 550 555 560
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
565 570 575
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
580 585 590
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
595 600 605
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
610 615 620
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
625 630 635 640
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
645 650 655
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
660 665 670
Leu Ser Leu Ser Pro Gly Lys
675
<210> 79
<211> 22
<212> Белок
<213> Искусственная последовательность
<220>
<223> сигнальная последовательность
<400> 79
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys
20
<210> 80
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL mAb709
<400> 80
Asp Ile Val Met Thr Gln Ser His Lys Phe Met Ser Thr Ser Val Gly
1 5 10 15
Asp Ser Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Leu Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ala Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Leu Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Gln Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 81
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 mAb746
<400> 81
Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 82
<211> 227
<212> Белок
<213> Искусственная последовательность
<220>
<223> шарнир-CH2-CH3 человеческого IgG1
<400> 82
Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly
1 5 10 15
Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met
20 25 30
Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser His
35 40 45
Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val
50 55 60
His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr
65 70 75 80
Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly
85 90 95
Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile
100 105 110
Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val
115 120 125
Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser
130 135 140
Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu
145 150 155 160
Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro
165 170 175
Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val
180 185 190
Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met
195 200 205
His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser
210 215 220
Pro Gly Lys
225
<210> 83
<211> 240
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-2a
Полипептидная цепь #2 FIT-Ig
<400> 83
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Lys Leu Val Glu Ser Gly Gly Gly Leu Val Lys
20 25 30
Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Phe Tyr Thr Met Ser Trp Val Arg Gln Thr Pro Glu Lys Arg Leu
50 55 60
Glu Trp Val Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Thr Leu Tyr Leu His Met Ser Ser Leu Arg Ser Glu Asp Thr Ala Leu
100 105 110
Tyr Tyr Cys Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly
115 120 125
Gln Gly Thr Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235 240
<210> 84
<211> 19
<212> Белок
<213> Искусственная последовательность
<220>
<223> сигнальная последовательность
<400> 84
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys
<210> 85
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 мышиного mAb709
<400> 85
Glu Val Lys Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Thr Pro Glu Lys Arg Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu His Met Ser Ser Leu Arg Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 86
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-2a
Полипептидная цепь #3 FIT-Ig
<400> 86
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Leu Gly Glu Arg Val Ser Leu Asn Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Ser Asp Gly
50 55 60
Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val
65 70 75 80
Pro Lys Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Ser Leu Thr
85 90 95
Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 87
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL мышиного mAb746
<400> 87
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Ser Leu Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Ser Asp Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 88
<211> 684
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-2b
Полипептидная цепь #1 FIT-Ig
<400> 88
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Leu Gly Glu Arg Val Ser Leu Asn Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Ser Asp Gly
50 55 60
Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val
65 70 75 80
Pro Lys Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Ser Leu Thr
85 90 95
Ile Ser Ser Leu Glu Ser Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Lys Leu
225 230 235 240
Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly Ser Leu Lys Leu
245 250 255
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr Thr Met Ser Trp
260 265 270
Val Arg Gln Thr Pro Glu Lys Arg Leu Glu Trp Val Ala Thr Ile Ser
275 280 285
Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val Lys Gly Arg Phe
290 295 300
Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr Leu His Met Ser
305 310 315 320
Ser Leu Arg Ser Glu Asp Thr Ala Leu Tyr Tyr Cys Ala Gly Gln Gly
325 330 335
Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
340 345 350
Ser Ala Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
580 585 590
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
675 680
<210> 89
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL mAb746
<400> 89
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Leu Gly
1 5 10 15
Glu Arg Val Ser Leu Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Ser Asp Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Lys Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Ser Leu Thr Ile Ser Ser Leu Glu Ser
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Ala Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 90
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 mAb709
<400> 90
Glu Val Lys Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Thr Pro Glu Lys Arg Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Tyr
65 70 75 80
Leu His Met Ser Ser Leu Arg Ser Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ala Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 91
<211> 235
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-2b
Полипептидная цепь #2 FIT-Ig
<400> 91
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg
20 25 30
Pro Gly Ala Ser Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile
35 40 45
Lys Asp Asp Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu
50 55 60
Asp Trp Ile Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala
65 70 75 80
Ser Lys Phe Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ser Asn
85 90 95
Thr Ala Tyr Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Leu
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235
<210> 92
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 mAb746
<400> 92
Glu Val Gln Leu Gln Gln Ser Gly Ala Glu Leu Val Arg Pro Gly Ala
1 5 10 15
Ser Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ser Asn Thr Ala Tyr
65 70 75 80
Leu Gln Leu Ser Ser Leu Thr Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Leu Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 93
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-2b
Полипептидная цепь #3 FIT-Ig
<400> 93
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser His Lys Phe
20 25 30
Met Ser Thr Ser Val Gly Asp Ser Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln
50 55 60
Ser Leu Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ala Arg Phe Thr Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Val Gln Ala Glu Asp Leu Ala Leu Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Gln Leu Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 94
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL mAb709
<400> 94
Asp Ile Val Met Thr Gln Ser His Lys Phe Met Ser Thr Ser Val Gly
1 5 10 15
Asp Ser Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Gln Ser Leu Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ala Arg Phe Thr Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Val Gln Ala
65 70 75 80
Glu Asp Leu Ala Leu Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Gln Leu Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 95
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-5a
Полипептидная цепь #1 FIT-Ig
<400> 95
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Ser Val Lys Val
245 250 255
Ser Cys Lys Ala Ser Asp Phe Asn Ile Lys Asp Asp Tyr Met His Trp
260 265 270
Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile Gly Trp Ile Val
275 280 285
Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe Gln Gly Lys Ala
290 295 300
Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr Met Glu Leu Ser
305 310 315 320
Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys Thr Val Tyr Gly
325 330 335
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
340 345 350
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
355 360 365
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
370 375 380
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
385 390 395 400
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
405 410 415
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
420 425 430
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
435 440 445
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
450 455 460
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
465 470 475 480
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
485 490 495
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
500 505 510
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
515 520 525
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
530 535 540
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
545 550 555 560
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
565 570 575
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
580 585 590
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
595 600 605
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
610 615 620
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
625 630 635 640
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
645 650 655
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
660 665 670
Leu Ser Leu Ser Pro Gly Lys
675
<210> 96
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb709-8
<400> 96
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 97
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 mAb
HumAb747-42
<400> 97
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 98
<211> 240
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-5a
Полипептидная цепь #2 FIT-Ig
<400> 98
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Phe Tyr Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly
115 120 125
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235 240
<210> 99
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb709-8
<400> 99
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 100
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-5a
Полипептидная цепь #3 FIT-Ig
<400> 100
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Asn Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly
50 55 60
Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 101
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747-42
<400> 101
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 102
<211> 684
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-5b
Полипептидная цепь #1 FIT-Ig
<400> 102
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Asn Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly
50 55 60
Thr Ile Lys Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
245 250 255
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr Thr Met Ser Trp
260 265 270
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Thr Ile Ser
275 280 285
Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val Lys Gly Arg Phe
290 295 300
Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn
305 310 315 320
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Gly Gln Gly
325 330 335
Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
340 345 350
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
580 585 590
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
675 680
<210> 103
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747-42
<400> 103
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 104
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb709-8
<400> 104
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 105
<211> 235
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-5b
Полипептидная цепь #2 FIT-Ig
<400> 105
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Ser Val Lys Val Ser Cys Lys Ala Ser Asp Phe Asn Ile
35 40 45
Lys Asp Asp Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu
50 55 60
Glu Trp Ile Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala
65 70 75 80
Ser Lys Phe Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ile Asn
85 90 95
Thr Ala Tyr Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235
<210> 106
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747-42
<400> 106
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 107
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-5b
Полипептидная цепь #3 FIT-Ig
<400> 107
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 108
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL mAb HuMAb709-8
<400> 108
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 109
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv1
<400> 109
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Ile Ser Cys Lys Val Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Gln Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 110
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv2
<400> 110
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Lys Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Gln Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 111
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv3
<400> 111
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Gln Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Thr Asp Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 112
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv4
<400> 112
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Ala Pro Gly Lys Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 113
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv5
<400> 113
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 114
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv6
<400> 114
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 115
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для huEpi001 VLv1
<400> 115
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ala Ile Lys Ser Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 116
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для huEpi001 VLv2
<400> 116
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Lys Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 117
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для huEpi001 VLv3
<400> 117
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 118
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для huEpi001 VLv4
<400> 118
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 119
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для антитела B2-53
<400> 119
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 120
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для антитела B2-53
<400> 120
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 121
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для антитела B3-21
<400> 121
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 122
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для антитела B3-21
<400> 122
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Met Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Tyr Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 123
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для антитела B3-43
<400> 123
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Cys Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 124
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для антитела B3-43
<400> 124
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser His Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 125
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для антитела B3-46
<400> 125
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Leu Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 126
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для антитела B3-46
<400> 126
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Thr Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 127
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для антитела B3-48
<400> 127
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Asp Phe Ser Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Lys Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 128
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для антитела B3-48
<400> 128
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Met Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 129
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для антитела B3-69
<400> 129
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr His Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 130
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для антитела B3-69
<400> 130
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Glu Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 131
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для антитела D1-70
<400> 131
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Ser Val Lys Val Ser Cys Lys Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Arg Gln Ala Pro Gly Gln Gly Leu Glu Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Gly Asn Thr Met Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Arg Val Thr Ile Thr Ala Asp Thr Ser Ile Asn Thr Ala Tyr
65 70 75 80
Met Glu Leu Ser Arg Leu Arg Ser Asp Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 132
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для антитела D1-70
<400> 132
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Asn Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Glu Gly Thr Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Leu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys
100 105
<210> 133
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv6(G55A)
<400> 133
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Asp Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Ala Asn Thr Glu Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 134
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv6.1
<400> 134
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Gly Asn Thr Met Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 135
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv6.2
<400> 135
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Gly Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 136
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH для huEpi001-VHv6.3
<400> 136
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Gly Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 137
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для huEpi001-VLv3.4
<400> 137
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Leu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 138
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для huEpi001-VLv3.5
<400> 138
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 139
<211> 107
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL для huEpi001-VLv3.6
<400> 139
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys
100 105
<210> 140
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-1
Полипептидная цепь #1 FIT-Ig
<400> 140
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Thr Val Lys Leu
245 250 255
Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp Tyr Met His Trp
260 265 270
Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile Gly Trp Ile Val
275 280 285
Pro Glu Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe Gln Gly Lys Ala
290 295 300
Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr Leu Glu Leu Ser
305 310 315 320
Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys Thr Val Tyr Gly
325 330 335
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
340 345 350
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
355 360 365
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
370 375 380
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
385 390 395 400
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
405 410 415
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
420 425 430
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
435 440 445
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
450 455 460
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
465 470 475 480
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
485 490 495
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
500 505 510
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
515 520 525
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
530 535 540
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
545 550 555 560
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
565 570 575
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
580 585 590
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
595 600 605
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
610 615 620
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
625 630 635 640
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
645 650 655
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
660 665 670
Leu Ser Leu Ser Pro Gly Lys
675
<210> 141
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb709-8
<400> 141
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 142
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-67
<400> 142
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 143
<211> 240
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-1
Полипептидная цепь #2 FIT-Ig
<400> 143
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Phe Tyr Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly
115 120 125
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235 240
<210> 144
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb709-8
<400> 144
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 145
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-1
Полипептидная цепь #3 FIT-Ig
<400> 145
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 146
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-67
<400> 146
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 147
<211> 684
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-1
Полипептидная цепь #1 FIT-Ig
<400> 147
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
245 250 255
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr Thr Met Ser Trp
260 265 270
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Thr Ile Ser
275 280 285
Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val Lys Gly Arg Phe
290 295 300
Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn
305 310 315 320
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Gly Gln Gly
325 330 335
Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
340 345 350
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
580 585 590
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
675 680
<210> 148
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-67
<400> 148
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 149
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb709-8
<400> 149
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 150
<211> 235
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-1
Полипептидная цепь #2 FIT-Ig
<400> 150
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile
35 40 45
Lys Asp Asp Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu
50 55 60
Asp Trp Ile Gly Trp Ile Val Pro Glu Asn Ala Asn Thr Val Tyr Ala
65 70 75 80
Ser Lys Phe Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn
85 90 95
Thr Ala Tyr Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235
<210> 151
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-67
<400> 151
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 152
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-1
Полипептидная цепь #3 FIT-Ig
<400> 152
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 153
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb709-8
<400> 153
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 154
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-2
Полипептидная цепь #1 FIT-Ig
<400> 154
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Thr Val Lys Leu
245 250 255
Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp Tyr Met His Trp
260 265 270
Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile Gly Trp Ile Val
275 280 285
Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe Gln Gly Lys Ala
290 295 300
Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr Leu Glu Leu Ser
305 310 315 320
Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys Thr Val Tyr Gly
325 330 335
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
340 345 350
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
355 360 365
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
370 375 380
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
385 390 395 400
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
405 410 415
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
420 425 430
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
435 440 445
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
450 455 460
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
465 470 475 480
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
485 490 495
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
500 505 510
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
515 520 525
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
530 535 540
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
545 550 555 560
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
565 570 575
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
580 585 590
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
595 600 605
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
610 615 620
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
625 630 635 640
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
645 650 655
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
660 665 670
Leu Ser Leu Ser Pro Gly Lys
675
<210> 155
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb709-8
<400> 155
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 156
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-72
<400> 156
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 157
<211> 240
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-2
Полипептидная цепь #2 FIT-Ig
<400> 157
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Phe Tyr Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly
115 120 125
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235 240
<210> 158
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb709-8
<400> 158
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 159
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-2
Полипептидная цепь #3 FIT-Ig
<400> 159
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 160
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-72
<400> 160
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 161
<211> 684
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-2
Полипептидная цепь #1 FIT-Ig
<400> 161
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
245 250 255
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr Thr Met Ser Trp
260 265 270
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Thr Ile Ser
275 280 285
Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val Lys Gly Arg Phe
290 295 300
Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn
305 310 315 320
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Gly Gln Gly
325 330 335
Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
340 345 350
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
580 585 590
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
675 680
<210> 162
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-72
<400> 162
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 163
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb709-8
<400> 163
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 164
<211> 235
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-2
Полипептидная цепь #2 FIT-Ig
<400> 164
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile
35 40 45
Lys Asp Asp Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu
50 55 60
Asp Trp Ile Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala
65 70 75 80
Ser Lys Phe Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn
85 90 95
Thr Ala Tyr Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235
<210> 165
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-72
<400> 165
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 166
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-2
Полипептидная цепь #3 FIT-Ig
<400> 166
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 167
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb709-8
<400> 167
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 168
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-3
Полипептидная цепь #1 FIT-Ig
<400> 168
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Thr Val Lys Leu
245 250 255
Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp Tyr Met His Trp
260 265 270
Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile Gly Trp Ile Val
275 280 285
Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe Gln Gly Lys Ala
290 295 300
Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr Leu Glu Leu Ser
305 310 315 320
Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys Thr Val Tyr Gly
325 330 335
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
340 345 350
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
355 360 365
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
370 375 380
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
385 390 395 400
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
405 410 415
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
420 425 430
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
435 440 445
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
450 455 460
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
465 470 475 480
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
485 490 495
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
500 505 510
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
515 520 525
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
530 535 540
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
545 550 555 560
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
565 570 575
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
580 585 590
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
595 600 605
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
610 615 620
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
625 630 635 640
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
645 650 655
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
660 665 670
Leu Ser Leu Ser Pro Gly Lys
675
<210> 169
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb709-8
<400> 169
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 170
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-73
<400> 170
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 171
<211> 240
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-3
Полипептидная цепь #2 FIT-Ig
<400> 171
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe
35 40 45
Ser Phe Tyr Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro
65 70 75 80
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly
115 120 125
Gln Gly Thr Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
130 135 140
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
145 150 155 160
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
165 170 175
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
180 185 190
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
195 200 205
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
210 215 220
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235 240
<210> 172
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb709-8
<400> 172
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 173
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6a-3
Полипептидная цепь #3 FIT-Ig
<400> 173
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 174
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-73
<400> 174
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 175
<211> 684
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-3
Полипептидная цепь #1 FIT-Ig
<400> 175
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
245 250 255
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr Thr Met Ser Trp
260 265 270
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Thr Ile Ser
275 280 285
Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val Lys Gly Arg Phe
290 295 300
Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn
305 310 315 320
Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala Gly Gln Gly
325 330 335
Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
340 345 350
Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser
355 360 365
Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys
370 375 380
Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu
385 390 395 400
Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu
405 410 415
Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr
420 425 430
Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val
435 440 445
Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro
450 455 460
Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe
465 470 475 480
Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val
485 490 495
Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe
500 505 510
Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro
515 520 525
Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr
530 535 540
Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val
545 550 555 560
Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala
565 570 575
Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg
580 585 590
Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly
595 600 605
Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro
610 615 620
Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser
625 630 635 640
Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln
645 650 655
Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His
660 665 670
Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
675 680
<210> 176
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-73
<400> 176
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 177
<211> 221
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb709-8
<400> 177
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Phe Tyr
20 25 30
Thr Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Thr Ile Ser Gly Gly Gly Arg Asp Thr Tyr Tyr Pro Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Gly Gln Gly Gly Asn Tyr Leu Phe Ala Tyr Trp Gly Gln Gly Thr
100 105 110
Leu Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro
115 120 125
Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly
130 135 140
Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn
145 150 155 160
Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln
165 170 175
Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser
180 185 190
Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser
195 200 205
Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 178
<211> 235
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-3
Полипептидная цепь #2 FIT-Ig
<400> 178
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile
35 40 45
Lys Asp Asp Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu
50 55 60
Asp Trp Ile Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala
65 70 75 80
Ser Lys Phe Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn
85 90 95
Thr Ala Tyr Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235
<210> 179
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-73
<400> 179
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 180
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-6b-3
Полипептидная цепь #3 FIT-Ig
<400> 180
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Val Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Gln Asp Val Asn Thr Val Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Val Leu Ile Ser Trp Ala Ser Thr Arg His Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
His Tyr Thr Thr Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 181
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb709-8
<400> 181
Asp Ile Val Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Gln Asp Val Asn Thr Val
20 25 30
Val Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Val Leu Ile
35 40 45
Ser Trp Ala Ser Thr Arg His Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln His Tyr Thr Thr Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 182
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-1
Полипептидная цепь #1 FIT-Ig
<400> 182
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Asp His Ile Asn Asn Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Leu Leu Ile Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
Tyr Trp Ser Pro Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Thr Val Lys Leu
245 250 255
Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp Tyr Met His Trp
260 265 270
Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile Gly Trp Ile Val
275 280 285
Pro Glu Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe Gln Gly Lys Ala
290 295 300
Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr Leu Glu Leu Ser
305 310 315 320
Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys Thr Val Tyr Gly
325 330 335
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
340 345 350
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
355 360 365
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
370 375 380
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
385 390 395 400
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
405 410 415
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
420 425 430
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
435 440 445
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
450 455 460
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
465 470 475 480
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
485 490 495
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
500 505 510
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
515 520 525
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
530 535 540
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
545 550 555 560
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
565 570 575
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
580 585 590
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
595 600 605
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
610 615 620
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
625 630 635 640
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
645 650 655
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
660 665 670
Leu Ser Leu Ser Pro Gly Lys
675
<210> 183
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL mAb HumAb713-7
<400> 183
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 184
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-67
<400> 184
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 185
<211> 243
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-1
Полипептидная цепь #2 FIT-Ig
<400> 185
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser
35 40 45
Ser Asp Tyr Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
225 230 235 240
Lys Ser Cys
<210> 186
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb713-7
<400> 186
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 187
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-1
Полипептидная цепь #3 FIT-Ig
<400> 187
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 188
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-67
<400> 188
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 189
<211> 687
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-1
Полипептидная цепь #1 FIT-Ig
<400> 189
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
245 250 255
Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr Gly Met His Trp
260 265 270
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Tyr Ile Ser
275 280 285
Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val Lys Gly Arg Phe
290 295 300
Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn
305 310 315 320
Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys Ala Lys Arg Gly
325 330 335
Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly Gln Gly Thr Thr
340 345 350
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
355 360 365
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
370 375 380
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
385 390 395 400
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
405 410 415
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
420 425 430
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
435 440 445
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
450 455 460
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
465 470 475 480
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
485 490 495
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
500 505 510
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
515 520 525
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
530 535 540
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
545 550 555 560
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
565 570 575
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
580 585 590
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
595 600 605
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
610 615 620
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
625 630 635 640
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
645 650 655
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
660 665 670
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
675 680 685
<210> 190
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-67
<400> 190
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 191
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb713-7
<400> 191
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 192
<211> 235
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-1
Полипептидная цепь #2 FIT-Ig
<400> 192
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile
35 40 45
Lys Asp Asp Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu
50 55 60
Asp Trp Ile Gly Trp Ile Val Pro Glu Asn Ala Asn Thr Val Tyr Ala
65 70 75 80
Ser Lys Phe Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn
85 90 95
Thr Ala Tyr Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235
<210> 193
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-67
<400> 193
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 194
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-1
Полипептидная цепь #3 FIT-Ig
<400> 194
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Asp His Ile Asn Asn Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Leu Leu Ile Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
Tyr Trp Ser Pro Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 195
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb713-7
<400> 195
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 196
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-2
Полипептидная цепь #1 FIT-Ig
<400> 196
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Asp His Ile Asn Asn Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Leu Leu Ile Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
Tyr Trp Ser Pro Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Thr Val Lys Leu
245 250 255
Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp Tyr Met His Trp
260 265 270
Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile Gly Trp Ile Val
275 280 285
Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe Gln Gly Lys Ala
290 295 300
Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr Leu Glu Leu Ser
305 310 315 320
Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys Thr Val Tyr Gly
325 330 335
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
340 345 350
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
355 360 365
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
370 375 380
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
385 390 395 400
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
405 410 415
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
420 425 430
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
435 440 445
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
450 455 460
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
465 470 475 480
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
485 490 495
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
500 505 510
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
515 520 525
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
530 535 540
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
545 550 555 560
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
565 570 575
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
580 585 590
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
595 600 605
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
610 615 620
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
625 630 635 640
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
645 650 655
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
660 665 670
Leu Ser Leu Ser Pro Gly Lys
675
<210> 197
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb713-7
<400> 197
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 198
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-72
<400> 198
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 199
<211> 243
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-2
Полипептидная цепь #2 FIT-Ig
<400> 199
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser
35 40 45
Ser Asp Tyr Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
225 230 235 240
Lys Ser Cys
<210> 200
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb713-7
<400> 200
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 201
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-2
Полипептидная цепь #3 FIT-Ig
<400> 201
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 202
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-72
<400> 202
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 203
<211> 687
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-2
Полипептидная цепь #1 FIT-Ig
<400> 203
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
245 250 255
Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr Gly Met His Trp
260 265 270
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Tyr Ile Ser
275 280 285
Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val Lys Gly Arg Phe
290 295 300
Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn
305 310 315 320
Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys Ala Lys Arg Gly
325 330 335
Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly Gln Gly Thr Thr
340 345 350
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
355 360 365
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
370 375 380
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
385 390 395 400
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
405 410 415
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
420 425 430
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
435 440 445
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
450 455 460
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
465 470 475 480
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
485 490 495
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
500 505 510
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
515 520 525
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
530 535 540
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
545 550 555 560
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
565 570 575
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
580 585 590
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
595 600 605
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
610 615 620
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
625 630 635 640
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
645 650 655
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
660 665 670
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
675 680 685
<210> 204
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL mAb HumAb747V-72
<400> 204
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Ala Leu Asp Leu Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 205
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb713-7
<400> 205
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 206
<211> 235
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-2
Полипептидная цепь #2 FIT-Ig
<400> 206
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile
35 40 45
Lys Asp Asp Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu
50 55 60
Asp Trp Ile Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala
65 70 75 80
Ser Lys Phe Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn
85 90 95
Thr Ala Tyr Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235
<210> 207
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-72
<400> 207
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 208
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-2
Полипептидная цепь #3 FIT-Ig
<400> 208
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Asp His Ile Asn Asn Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Leu Leu Ile Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
Tyr Trp Ser Pro Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 209
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb713-7
<400> 209
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 210
<211> 679
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-3
Полипептидная цепь #1 FIT-Ig
<400> 210
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Asp His Ile Asn Asn Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Leu Leu Ile Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
Tyr Trp Ser Pro Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala Thr Val Lys Leu
245 250 255
Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp Tyr Met His Trp
260 265 270
Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile Gly Trp Ile Val
275 280 285
Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe Gln Gly Lys Ala
290 295 300
Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr Leu Glu Leu Ser
305 310 315 320
Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys Thr Val Tyr Gly
325 330 335
Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr
340 345 350
Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser
355 360 365
Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu
370 375 380
Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His
385 390 395 400
Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser
405 410 415
Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys
420 425 430
Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu
435 440 445
Pro Lys Ser Cys Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
450 455 460
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
465 470 475 480
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
485 490 495
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
500 505 510
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
515 520 525
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
530 535 540
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Ala Leu
545 550 555 560
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
565 570 575
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Glu Glu Met Thr Lys
580 585 590
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
595 600 605
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
610 615 620
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
625 630 635 640
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
645 650 655
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
660 665 670
Leu Ser Leu Ser Pro Gly Lys
675
<210> 211
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb713-7
<400> 211
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 212
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-73
<400> 212
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 213
<211> 243
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-3
Полипептидная цепь #2 FIT-Ig
<400> 213
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln
20 25 30
Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser
35 40 45
Ser Asp Tyr Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
50 55 60
Glu Trp Val Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala
65 70 75 80
Asp Thr Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
85 90 95
Ser Leu Tyr Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp
115 120 125
Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys
130 135 140
Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly
145 150 155 160
Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro
165 170 175
Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr
180 185 190
Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val
195 200 205
Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn
210 215 220
Val Asn His Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro
225 230 235 240
Lys Ser Cys
<210> 214
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb713-7
<400> 214
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 215
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7a-3
Полипептидная цепь #3 FIT-Ig
<400> 215
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 216
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-73
<400> 216
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 217
<211> 687
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-3
Полипептидная цепь #1 FIT-Ig
<400> 217
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser
35 40 45
Gln Glu Ile Ser Gly Tyr Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly
50 55 60
Ala Ile Lys Arg Leu Ile Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln
100 105 110
Tyr Ala Ser Tyr Pro Leu Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys Glu Val Gln Leu
225 230 235 240
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu
245 250 255
Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr Gly Met His Trp
260 265 270
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Tyr Ile Ser
275 280 285
Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val Lys Gly Arg Phe
290 295 300
Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn
305 310 315 320
Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys Ala Lys Arg Gly
325 330 335
Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly Gln Gly Thr Thr
340 345 350
Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu
355 360 365
Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys
370 375 380
Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser
385 390 395 400
Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser
405 410 415
Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser
420 425 430
Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn
435 440 445
Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys Asp Lys Thr His
450 455 460
Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
465 470 475 480
Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr
485 490 495
Pro Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu
500 505 510
Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
515 520 525
Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser
530 535 540
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
545 550 555 560
Cys Lys Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile
565 570 575
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
580 585 590
Pro Ser Arg Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
595 600 605
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
610 615 620
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
625 630 635 640
Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg
645 650 655
Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu
660 665 670
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Lys
675 680 685
<210> 218
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb747V-73
<400> 218
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Glu Ile Ser Gly Tyr
20 25 30
Leu Ser Trp Leu Gln Gln Lys Pro Gly Gly Ala Ile Lys Arg Leu Ile
35 40 45
Tyr Ala Ala Ser Thr Leu Asp Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Arg Ser Gly Ser Asp Tyr Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Asp Tyr Tyr Cys Leu Gln Tyr Ala Ser Tyr Pro Leu
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Leu Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 219
<211> 224
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb713-7
<400> 219
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Ser Ser Asp Tyr
20 25 30
Gly Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Gly Ser Tyr Thr Ile Tyr Tyr Ala Asp Thr Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Asp Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Arg Gly Gly Ser Ser His Val Asn Val Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Thr Val Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser
115 120 125
Val Phe Pro Leu Ala Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala
130 135 140
Ala Leu Gly Cys Leu Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val
145 150 155 160
Ser Trp Asn Ser Gly Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala
165 170 175
Val Leu Gln Ser Ser Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val
180 185 190
Pro Ser Ser Ser Leu Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His
195 200 205
Lys Pro Ser Asn Thr Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
210 215 220
<210> 220
<211> 235
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-3
Полипептидная цепь #2 FIT-Ig
<400> 220
Met Glu Phe Gly Leu Ser Trp Leu Phe Leu Val Ala Ile Leu Lys Gly
1 5 10 15
Val Gln Cys Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys
20 25 30
Pro Gly Ala Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile
35 40 45
Lys Asp Asp Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu
50 55 60
Asp Trp Ile Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala
65 70 75 80
Ser Lys Phe Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn
85 90 95
Thr Ala Tyr Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val
100 105 110
Tyr Tyr Cys Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val
115 120 125
Thr Val Ser Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala
130 135 140
Pro Ser Ser Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu
145 150 155 160
Val Lys Asp Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly
165 170 175
Ala Leu Thr Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser
180 185 190
Gly Leu Tyr Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu
195 200 205
Gly Thr Gln Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr
210 215 220
Lys Val Asp Lys Lys Val Glu Pro Lys Ser Cys
225 230 235
<210> 221
<211> 216
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH-CH1 HumAb747V-73
<400> 221
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser Ala Ser Thr Lys Gly Pro Ser Val Phe Pro Leu Ala Pro Ser Ser
115 120 125
Lys Ser Thr Ser Gly Gly Thr Ala Ala Leu Gly Cys Leu Val Lys Asp
130 135 140
Tyr Phe Pro Glu Pro Val Thr Val Ser Trp Asn Ser Gly Ala Leu Thr
145 150 155 160
Ser Gly Val His Thr Phe Pro Ala Val Leu Gln Ser Ser Gly Leu Tyr
165 170 175
Ser Leu Ser Ser Val Val Thr Val Pro Ser Ser Ser Leu Gly Thr Gln
180 185 190
Thr Tyr Ile Cys Asn Val Asn His Lys Pro Ser Asn Thr Lys Val Asp
195 200 205
Lys Lys Val Glu Pro Lys Ser Cys
210 215
<210> 222
<211> 236
<212> Белок
<213> Искусственная последовательность
<220>
<223> FIT107-1-7b-3
Полипептидная цепь #3 FIT-Ig
<400> 222
Met Asp Met Arg Val Pro Ala Gln Leu Leu Gly Leu Leu Leu Leu Trp
1 5 10 15
Phe Pro Gly Ser Arg Cys Asp Ile Gln Met Thr Gln Ser Pro Ser Ser
20 25 30
Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Lys Ala Ser
35 40 45
Asp His Ile Asn Asn Trp Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys
50 55 60
Ala Pro Lys Leu Leu Ile Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val
65 70 75 80
Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr
85 90 95
Ile Ser Ser Leu Gln Pro Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln
100 105 110
Tyr Trp Ser Pro Pro Tyr Thr Phe Gly Gly Gly Thr Lys Val Glu Ile
115 120 125
Lys Arg Thr Val Ala Ala Pro Ser Val Phe Ile Phe Pro Pro Ser Asp
130 135 140
Glu Gln Leu Lys Ser Gly Thr Ala Ser Val Val Cys Leu Leu Asn Asn
145 150 155 160
Phe Tyr Pro Arg Glu Ala Lys Val Gln Trp Lys Val Asp Asn Ala Leu
165 170 175
Gln Ser Gly Asn Ser Gln Glu Ser Val Thr Glu Gln Asp Ser Lys Asp
180 185 190
Ser Thr Tyr Ser Leu Ser Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr
195 200 205
Glu Lys His Lys Val Tyr Ala Cys Glu Val Thr His Gln Gly Leu Ser
210 215 220
Ser Pro Val Thr Lys Ser Phe Asn Arg Gly Glu Cys
225 230 235
<210> 223
<211> 214
<212> Белок
<213> Искусственная последовательность
<220>
<223> VL-CL HumAb713-7
<400> 223
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Lys Ala Ser Asp His Ile Asn Asn Trp
20 25 30
Leu Ala Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Gly Ala Thr Ser Leu Glu Thr Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ile Ala Thr Tyr Tyr Cys Gln Gln Tyr Trp Ser Pro Pro Tyr
85 90 95
Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg Thr Val Ala Ala
100 105 110
Pro Ser Val Phe Ile Phe Pro Pro Ser Asp Glu Gln Leu Lys Ser Gly
115 120 125
Thr Ala Ser Val Val Cys Leu Leu Asn Asn Phe Tyr Pro Arg Glu Ala
130 135 140
Lys Val Gln Trp Lys Val Asp Asn Ala Leu Gln Ser Gly Asn Ser Gln
145 150 155 160
Glu Ser Val Thr Glu Gln Asp Ser Lys Asp Ser Thr Tyr Ser Leu Ser
165 170 175
Ser Thr Leu Thr Leu Ser Lys Ala Asp Tyr Glu Lys His Lys Val Tyr
180 185 190
Ala Cys Glu Val Thr His Gln Gly Leu Ser Ser Pro Val Thr Lys Ser
195 200 205
Phe Asn Arg Gly Glu Cys
210
<210> 224
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR-H2 из анти-LAG-3 антитела HumAb747V-67 с заменой G55A по
нумерации Кабат
<400> 224
Trp Ile Val Pro Glu Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe Gln
1 5 10 15
Gly
<210> 225
<211> 17
<212> Белок
<213> Искусственная последовательность
<220>
<223> CDR-H2 из анти-LAG-3 антитела HumAb747V-72 или HumAb747V-73
с заменой G55A по нумерации Кабат
<400> 225
Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe Gln
1 5 10 15
Gly
<210> 226
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH с заменой G55A по нумерации Кабат
<400> 226
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Glu Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<210> 227
<211> 113
<212> Белок
<213> Искусственная последовательность
<220>
<223> VH с заменой G55A по нумерации Кабат
<400> 227
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Ala
1 5 10 15
Thr Val Lys Leu Ser Cys Thr Ala Ser Gly Phe Asn Ile Lys Asp Asp
20 25 30
Tyr Met His Trp Val Lys Gln Arg Pro Glu Gln Gly Leu Asp Trp Ile
35 40 45
Gly Trp Ile Val Pro Arg Asn Ala Asn Thr Val Tyr Ala Ser Lys Phe
50 55 60
Gln Gly Lys Ala Thr Ile Thr Ala Asp Thr Ser Thr Asn Thr Ala Tyr
65 70 75 80
Leu Glu Leu Ser Ser Leu Arg Ser Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Val Tyr Gly Asp Tyr Trp Gly Gln Gly Thr Thr Val Thr Val Ser
100 105 110
Ser
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| РЕЖИМЫ ДОЗИРОВАНИЯ АНТИТЕЛ АНТИ-LAG-3 И ИХ ПРИМЕНЕНИЕ | 2018 |
|
RU2801208C2 |
| АНТИ-CD39 АНТИТЕЛА, КОМПОЗИЦИИ, СОДЕРЖАЩИЕ АНТИ-CD39 АНТИТЕЛА, И СПОСОБЫ ПРИМЕНЕНИЯ АНТИ-CD39 АНТИТЕЛ | 2018 |
|
RU2795348C2 |
| АНТИ-ROR1/АНТИ-CD3 БИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ | 2020 |
|
RU2831608C2 |
| Анти-PD-L1 и IL-2 цитокины | 2017 |
|
RU2769282C2 |
| ФАРМАЦЕВТИЧЕСКАЯ КОМПОЗИЦИЯ, СОДЕРЖАЩАЯ АНТИТЕЛО К LAG-3, И ЕЕ ПРИМЕНЕНИЕ | 2018 |
|
RU2771384C2 |
| АНТИ-LAG-3 АНТИТЕЛА И ИХ КОМПОЗИЦИИ | 2017 |
|
RU2755503C2 |
| Антитело против FXI/FXIA, его антигенсвязывающий фрагмент и фармацевтическое применение | 2021 |
|
RU2838408C1 |
| СЕЛЕКТИВНЫЕ В ОТНОШЕНИИ ИЗОФОРМЫ АНТИТЕЛА К TGF-БЕТА И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2021 |
|
RU2839151C1 |
| МОЛЕКУЛЫ АНТИ-SIGLEC-9 АНТИТЕЛА | 2020 |
|
RU2833330C1 |
| АНТИ-PD-1 АНТИТЕЛА | 2019 |
|
RU2788095C2 |
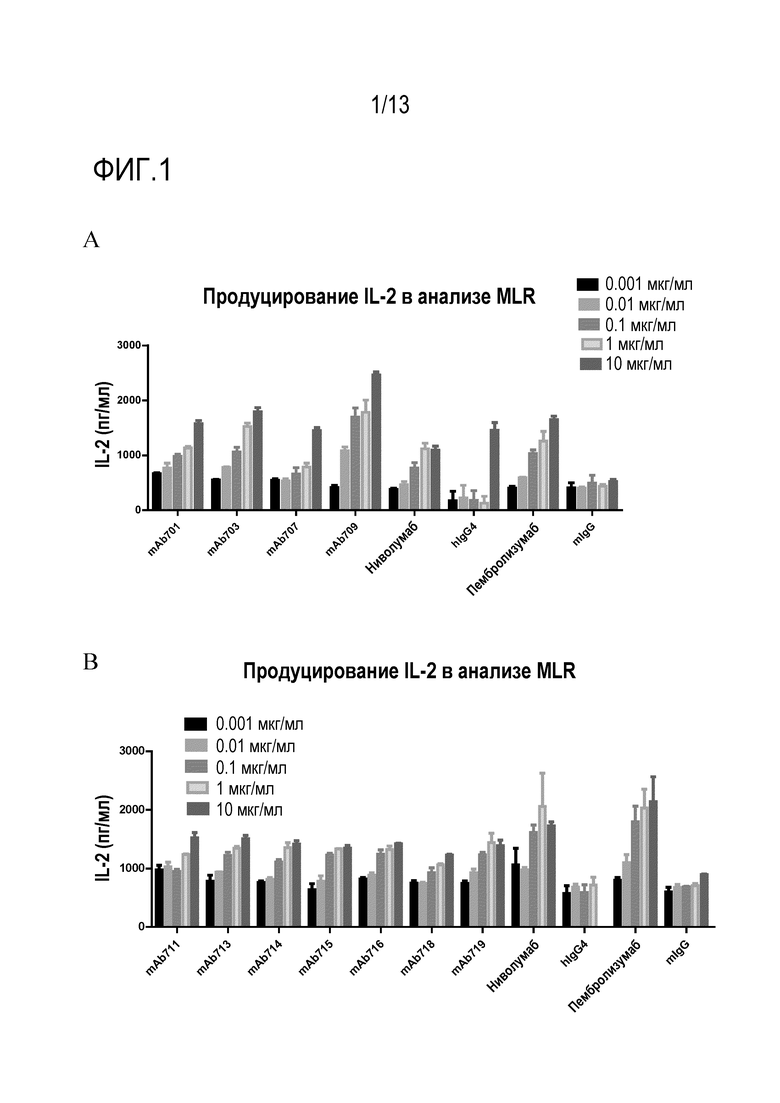
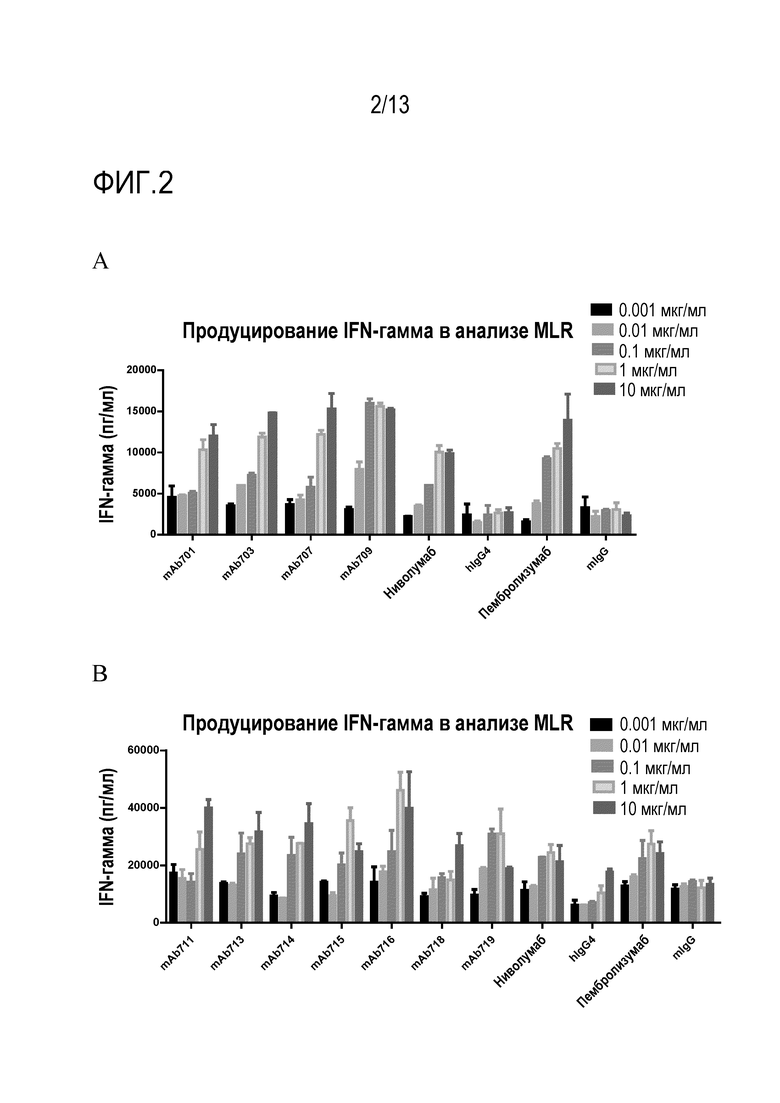
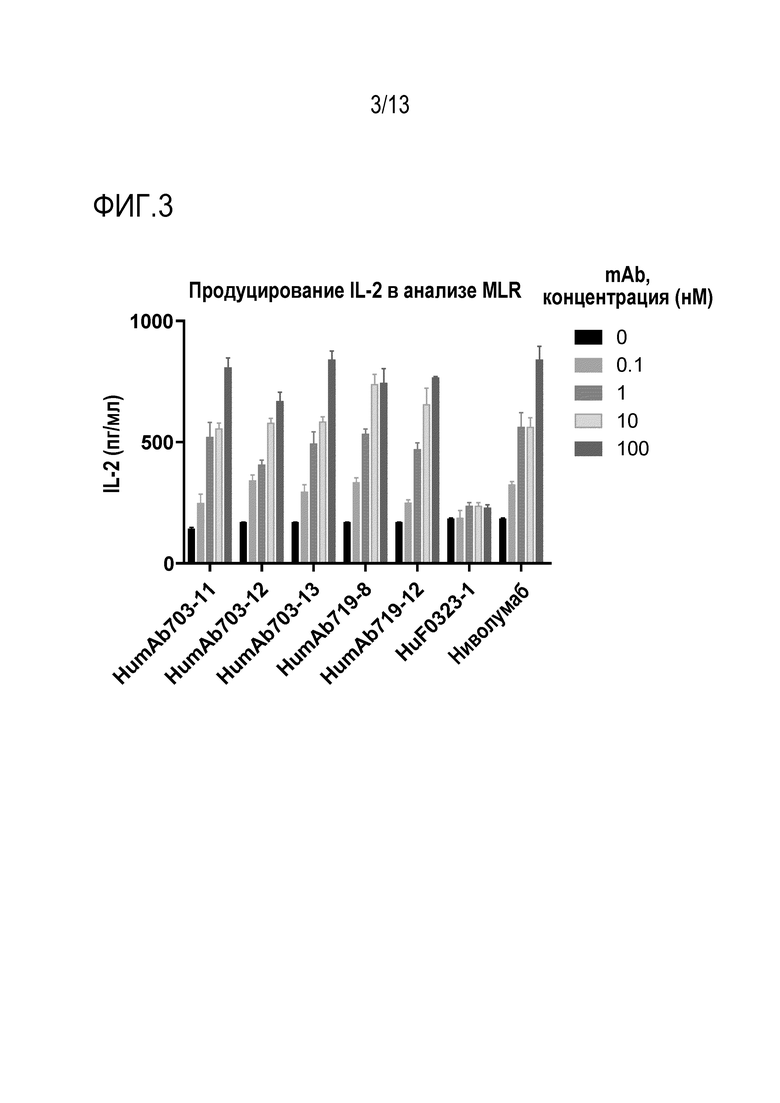
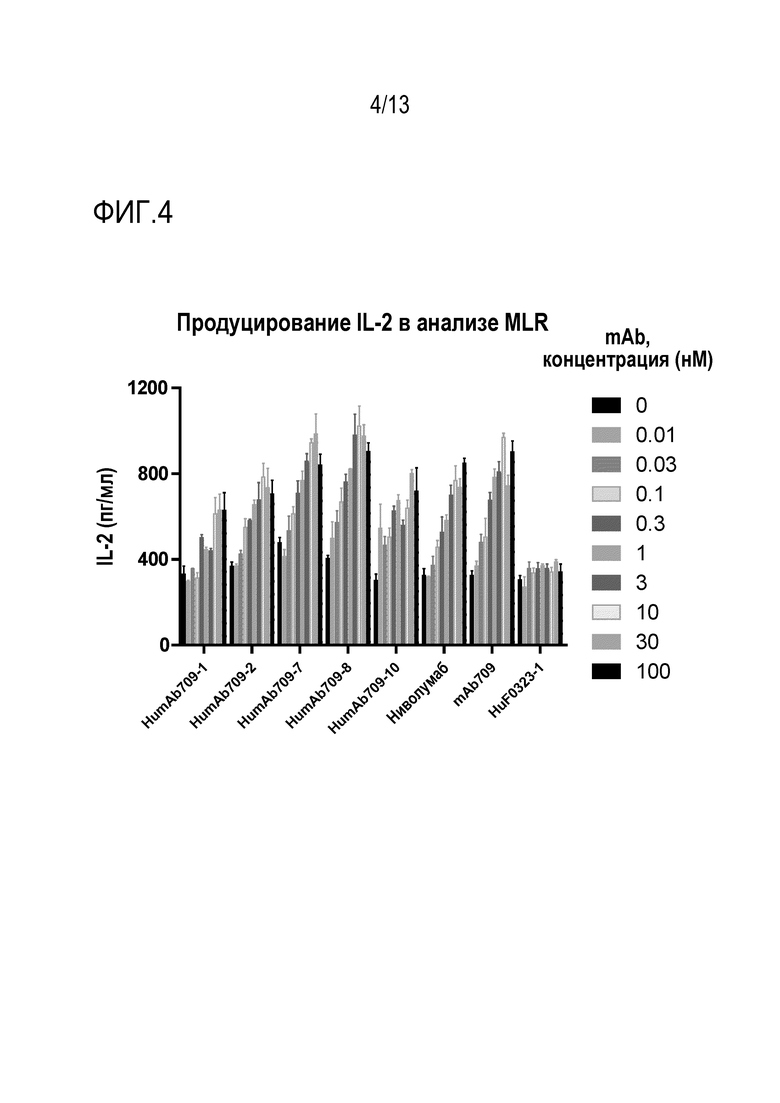
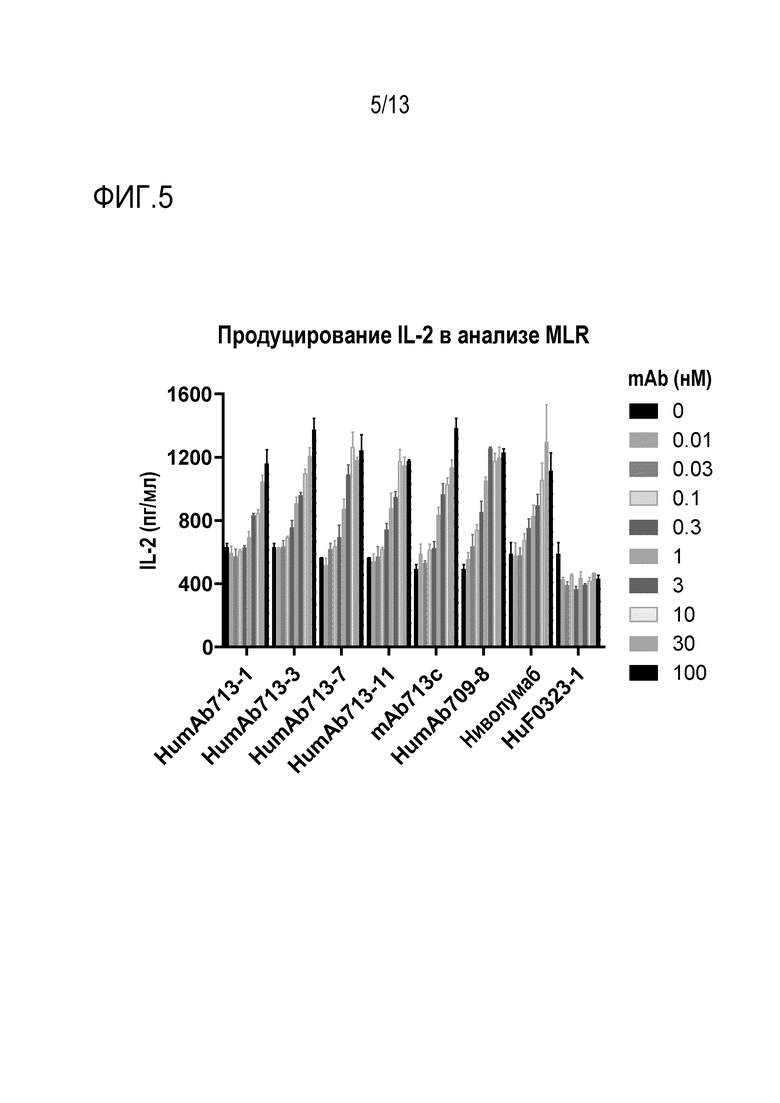
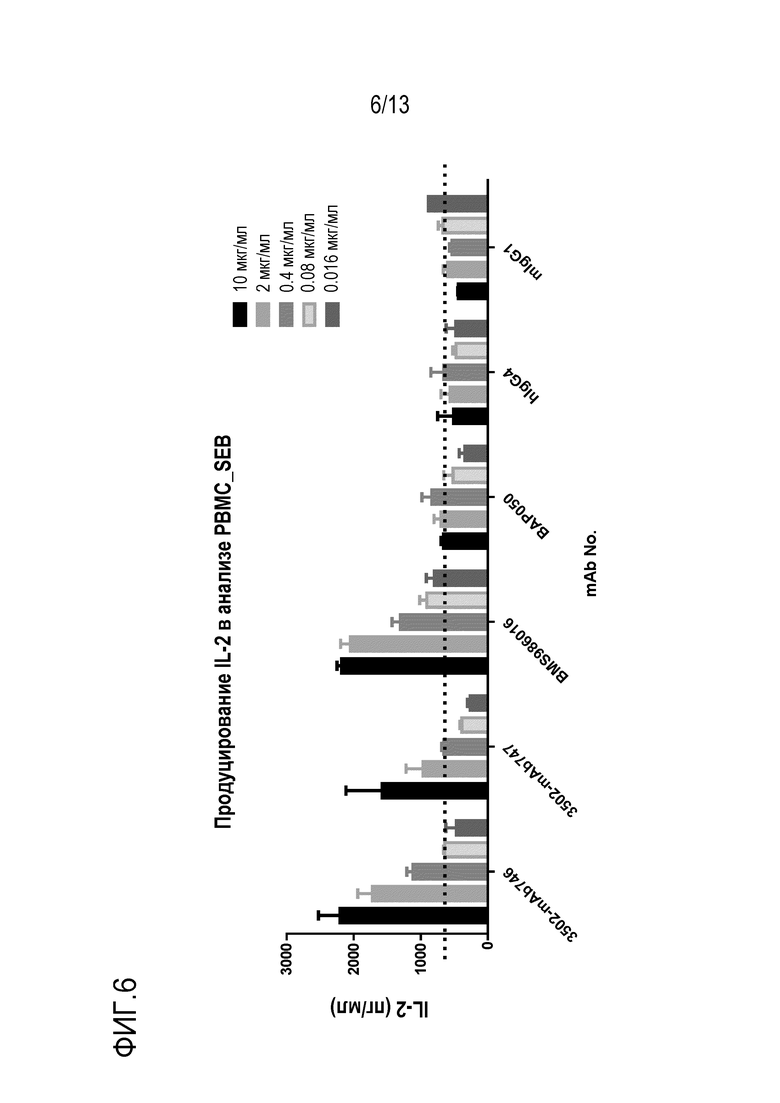
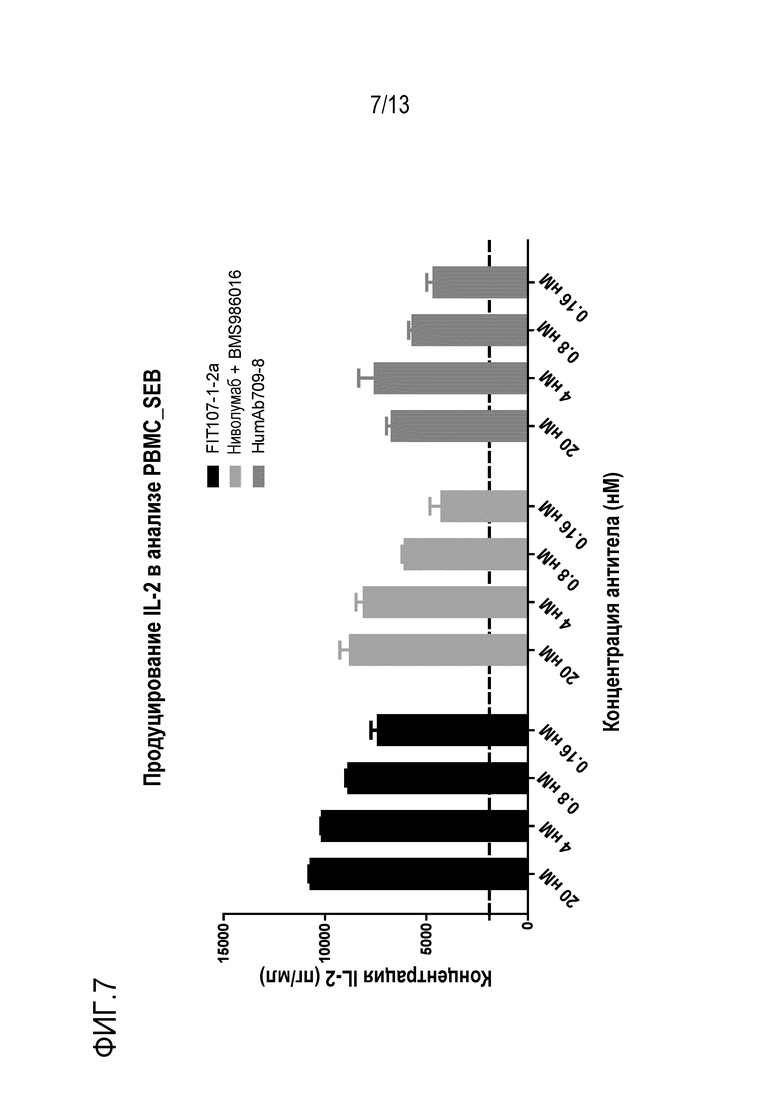
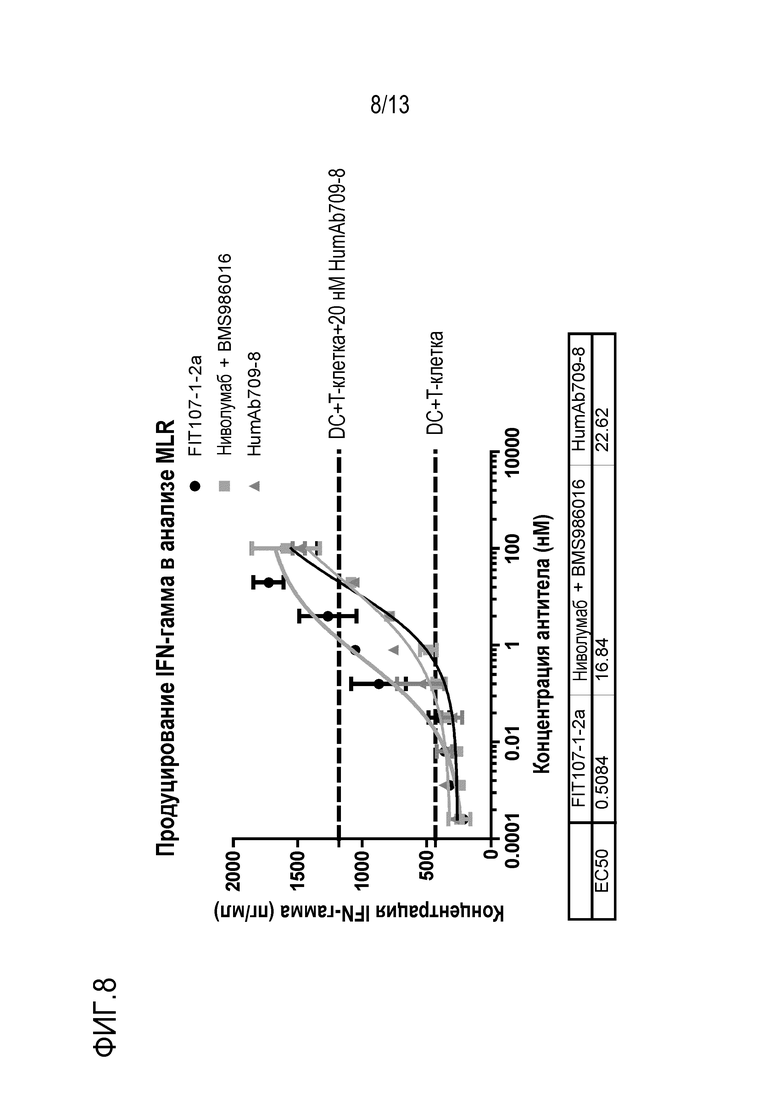
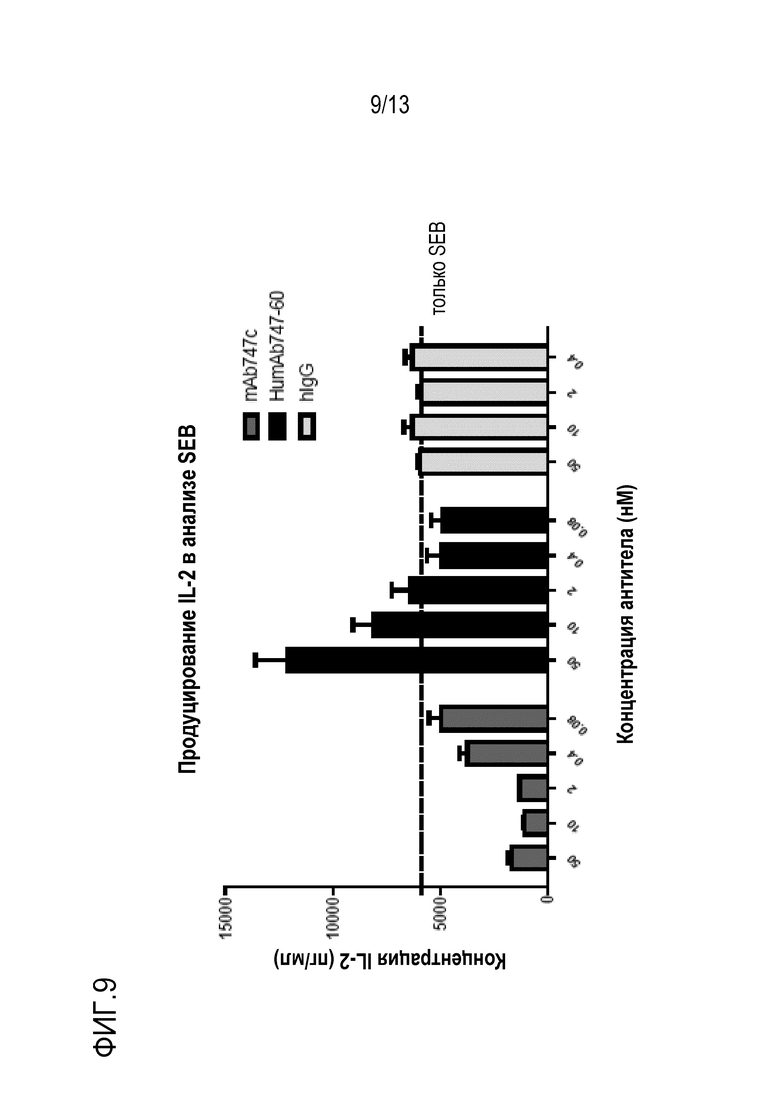
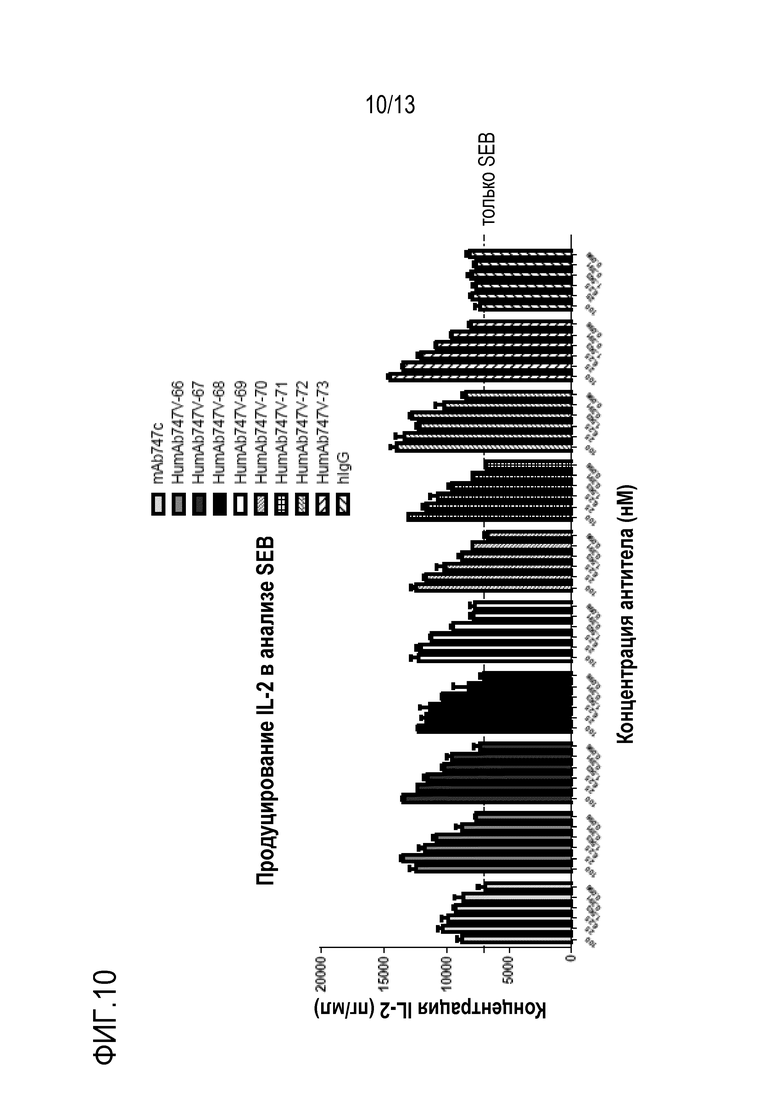
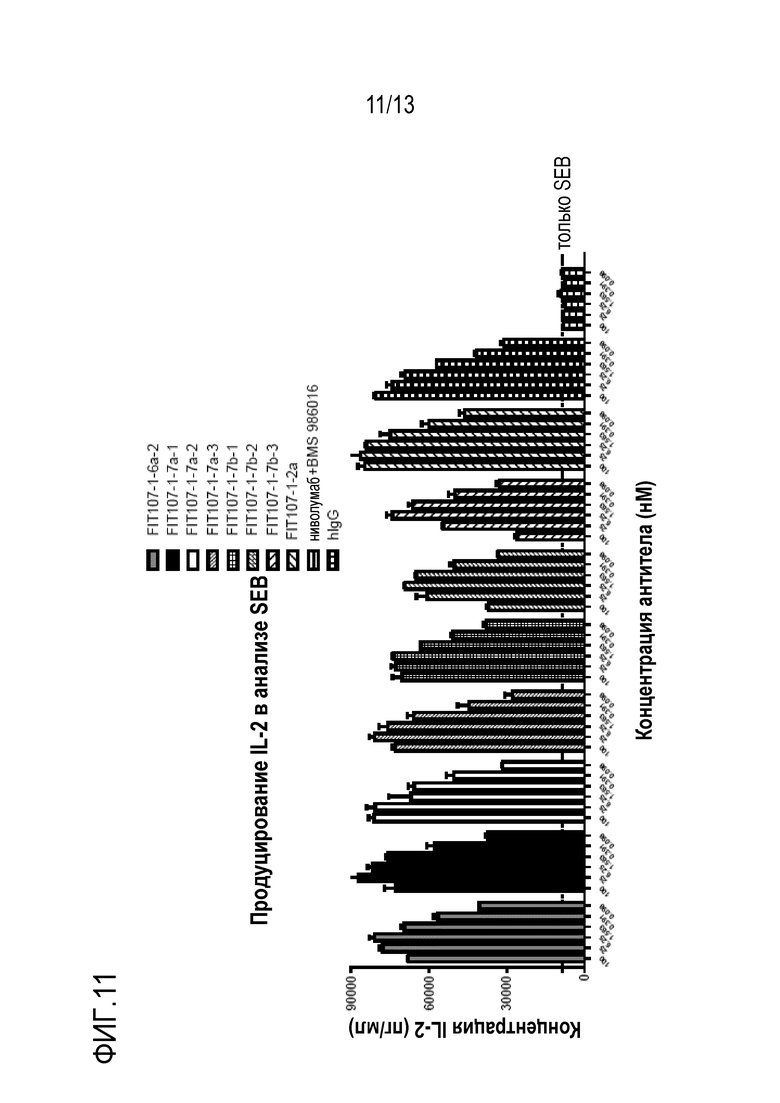
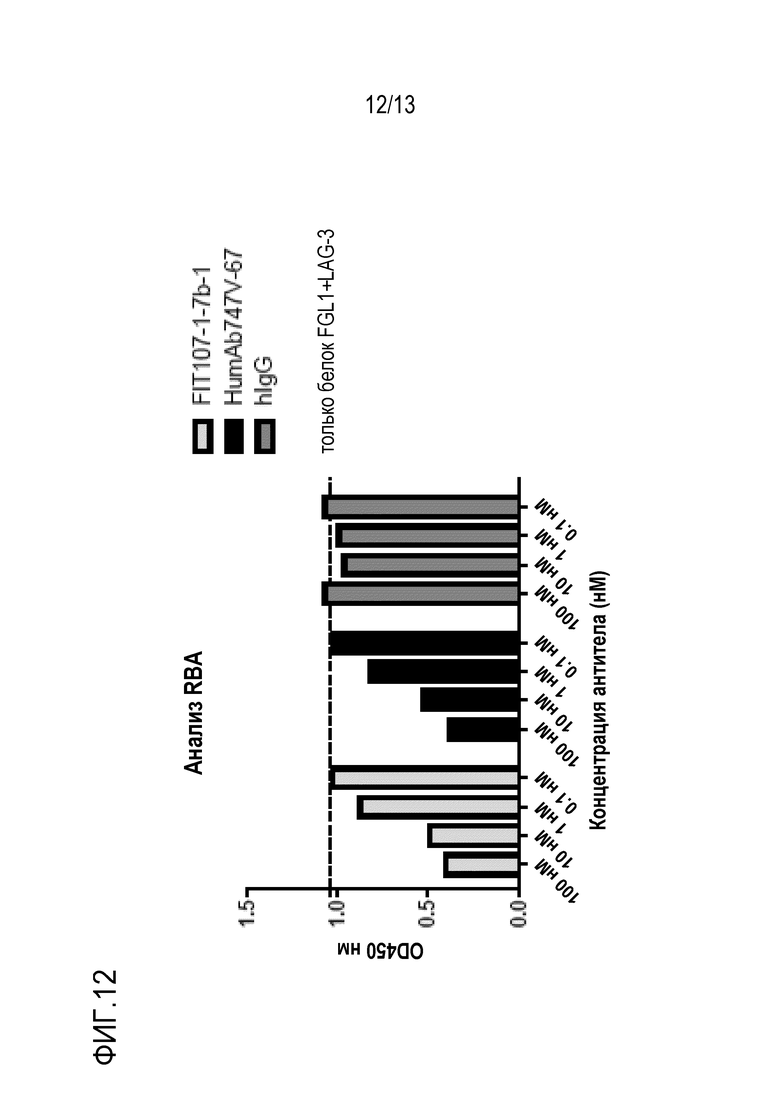
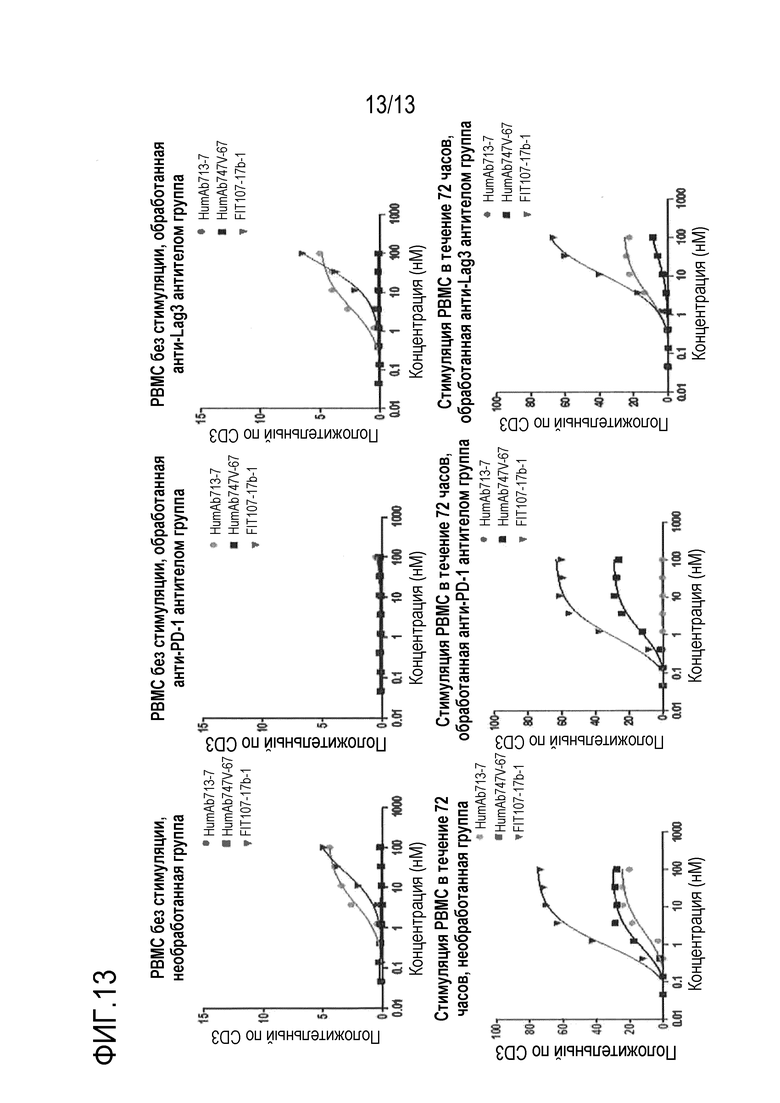
Изобретение относится к области биотехнологии, в частности к биспецифическому поливалентному связывающему белку, способному связывать PD-1 и LAG-3, а также к анти-LAG-3 антителу или его антигенсвязывающей части. Также раскрыта композиция, содержащая вышеуказанный белок или антитело, а также способ изготовления лекарственного средства. Изобретение эффективно для лечения расстройства, при котором активность, опосредованная PD-1 и/или опосредованная LAG-3, является вредной. 9 н. и 22 з.п. ф-лы, 13 ил., 55 табл., 16 пр.
1. Биспецифический поливалентный связывающий белок, содержащий первую, вторую и третью полипептидные цепи, причем
указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, (i) VLA-CL-VHB-CH1-Fc, где CL слит непосредственно с VHB, или (ii) VHB-CH1-VLA-CL-Fc, где CH1 слит непосредственно с VLA;
указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHA-CH1; и
указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLB-CL;
причем VL представляет собой вариабельный домен легкой цепи, CL представляет собой константный домен легкой цепи, VH представляет собой вариабельный домен тяжелой цепи, CH1 представляет собой константный домен тяжелой цепи, Fc представляет собой Fc-область иммуноглобулина, A представляет собой эпитоп PD-1 или LAG-3 и B представляет собой эпитоп PD-1 или LAG-3 при условии, что A и B разные, причем указанный связывающий белок способен связывать как PD-1, так и LAG-3; где
VHA и VLA соответственно представляют VHLAG-3 и VLLAG-3 и VLB и VHB соответственно представляют VLPD-1 и VHPD-1, если A представляет собой эпитоп LAG-3 и B представляет собой эпитоп PD-1, или
VLB и VHB соответственно представляют собой VLLAG-3 и VHLAG-3 и VLA и VHA соответственно представляют собой VLPD-1 и VHPD-1, когда B представляет собой эпитоп LAG-3 и A представляет собой эпитоп PD-1, и
указанные VHLAG-3 и VLLAG-3 соответственно содержат CDR, а именно CDR-H1, CDR-H2, CDR-H3 и CDR-L1, CDR-L2, CDR-L3, выбранные из группы, состоящей из следующих наборов CDR:
CDR-H1, содержащая DDYMH (остатки 31-35 SEQ ID NO: 135), CDR-H2, содержащая WIVPENANTVYASKFQG (SEQ ID NO: 224), CDR-H3, содержащая YGDY (остатки 99-102 SEQ ID NO: 135), CDR-L1, содержащая RASQEISGYLS (остатки 24-34 SEQ ID NO: 138), CDR-L2, содержащая AASALDS (остатки 50-56 SEQ ID NO: 138), и CDR-L3, содержащая LQYASYPLT (остатки 89-97 SEQ ID NO: 138);
CDR-H1, содержащая DDYMH (остатки 31-35 SEQ ID NO: 136), CDR-H2, содержащая WIVPRNANTVYASKFQG (SEQ ID NO: 225), CDR-H3, содержащая YGDY (остатки 99-102 SEQ ID NO: 136), CDR-L1, содержащая RASQEISGYLS (остатки 24-34 SEQ ID NO: 139), CDR-L2, содержащая AASALDL (остатки 50-56 SEQ ID NO: 139), и CDR-L3 содержащая LQYASYPLT (остатки 89-97 SEQ ID NO: 139); или
CDR-H1, содержащая DDYMH (остатки 31-35 SEQ ID NO: 136), CDR-H2, содержащая WIVPRNANTVYASKFQG (SEQ ID NO: 225), CDR-H3, содержащая YGDY (остатки 99-102 SEQ ID NO: 136), CDR-L1, содержащая RASQEISGYLS (остатки 24-34 SEQ ID NO: 117), CDR-L2, содержащая AASTLDS (остатки 50-56 SEQ ID NO: 117), и CDR-L3, содержащая LQYASYPLT (остатки 89-97 SEQ ID NO: 117); и
где указанные VHPD-1 и VLPD-1 соответственно содержат CDR, а именно CDR-H1, CDR-H2, CDR-H3 и CDR-L1, CDR-L2, CDR-L3, которые соответственно представляют собой следующие:
CDR-H1, содержащая DYGMH (остатки 31-35 SEQ ID NO: 10), CDR-H2, содержащая YISSGSYTIYYADTVKG (остатки 50-66 SEQ ID NO: 10), CDR-H3, содержащая RGGSSHVNVMDY (остатки 99-110 SEQ ID NO: 10), CDR-L1, содержащая KASDHINNWLA (остатки 24-34 SEQ ID NO: 11), CDR-L2, содержащая GATSLET (остатки 50-56 SEQ ID NO: 11), и CDR-L3, содержащая QQYWSPPYT (остатки 89-97 SEQ ID NO: 11).
2. Биспецифический поливалентный связывающий белок, содержащий первую, вторую и третью полипептидные цепи, причем
указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, (i) VLA-CL-VHB-CH1-Fc, где CL слит непосредственно с VHB, или (ii) VHB-CH1-VLA-CL-Fc, где CH1 слит непосредственно с VLA;
указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHA-CH1; и
указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLB-CL;
причем VL представляет собой вариабельный домен легкой цепи, CL представляет собой константный домен легкой цепи, VH представляет собой вариабельный домен тяжелой цепи, CH1 представляет собой константный домен тяжелой цепи, Fc представляет собой Fc-область иммуноглобулина, A представляет собой эпитоп PD-1 или LAG-3 и B представляет собой эпитоп PD-1 или LAG-3 при условии, что A и B разные, причем указанный связывающий белок способен связывать как PD-1, так и LAG-3; где
VHA и VLA соответственно представляют VHLAG-3 и VLLAG-3 и VLB и VHB соответственно представляют VLPD-1 и VHPD-1, если A представляет собой эпитоп LAG-3 и B представляет собой эпитоп PD-1, или
VLB и VHB соответственно представляют собой VLLAG-3 и VHLAG-3 и VLA и VHA соответственно представляют собой VLPD-1 и VHPD-1, когда B представляет собой эпитоп LAG-3 и A представляет собой эпитоп PD-1, и
где указанные VHLAG-3 и VLLAG-3, соответственно, содержат аминокислотные последовательности, выбранные из группы, состоящей из:
SEQ ID NO: 135 и SEQ ID NO: 138,
SEQ ID NO: 136 и SEQ ID NO: 139,
SEQ ID NO: 136 и SEQ ID NO: 117,
SEQ ID NO: 226 и SEQ ID NO: 138,
SEQ ID NO: 227 и SEQ ID NO: 139 и
SEQ ID NO: 227 и SEQ ID NO: 117;
и где указанные VHPD-1 и VLPD-1 соответственно содержат аминокислотные последовательности, как показано в SEQ ID NO: 32 и SEQ ID NO: 35.
3. Связывающий белок по п.1, в котором домены VLA-CL и VHA-CH1 происходят от родительского антитела, связывающегося с одним из целевых антигенов PD-1 или LAG-3, и домены VLB-CL и VHB-CH1 происходят от другого родительского антитела, связывающегося с другим целевым антигеном PD-1 или LAG-3.
4. Связывающий белок по п.3, содержащий первую, вторую и третью полипептидные цепи, причем указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLPD-1-CL-VHLAG-3-CH1-Fc, где CL слит непосредственно с VHLAG-3,
причем указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHPD-1-CH1; и
причем указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLLAG-3-CL;
где VLPD-1 представляет собой вариабельный домен легкой цепи анти-PD-1 антитела, CL представляет собой константный домен легкой цепи, VHPD-1 представляет собой вариабельный домен тяжелой цепи анти-PD-1 антитела, CH1 представляет собой константный домен тяжелой цепи, VLLAG-3 представляет собой вариабельный домен легкой цепи анти-LAG-3 антитела, VHLAG-3 представляет собой вариабельный домен тяжелой цепи анти-LAG-3 антитела и Fc представляет собой Fc-область иммуноглобулина.
5. Связывающий белок по п.4, в котором в первой полипептидной цепи домены VLPD-1-CL являются такими же, как легкая цепь родительского анти-PD-1 антитела, домены VHPD-1-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-PD-1 антитела, домены VLLAG-3-CL являются такими же, как легкая цепь родительского анти-LAG-3 антитела, и домены VHLAG-3-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-LAG-3 антитела.
6. Связывающий белок по п.3, содержащий первую, вторую и третью полипептидные цепи, причем указанная первая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLLAG-3-CL-VHPD-1-CH1-Fc, где CL слит непосредственно с VHPD-1,
причем указанная вторая полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VHLAG-3-CH1; и
причем указанная третья полипептидная цепь содержит, в направлении от аминоконца к карбоксильному концу, VLPD-1-CL; где VLPD-1 представляет собой вариабельный домен легкой цепи анти-PD-1 антитела, CL представляет собой константный домен легкой цепи, VHPD-1 представляет собой вариабельный домен тяжелой цепи анти-PD-1 антитела, CH1 представляет собой константный домен тяжелой цепи, VLLAG-3 представляет собой вариабельный домен легкой цепи анти-LAG-3 антитела, VHLAG-3 представляет собой вариабельный домен тяжелой цепи анти-LAG-3 антитела и Fc представляет собой Fc-область иммуноглобулина.
7. Связывающий белок по п.6, в котором в первой полипептидной цепи домены VLLAG-3-CL являются такими же, как легкая цепь родительского анти-LAG-3 антитела, домены VHLAG-3-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-LAG-3 антитела, домены VLPD-1-CL являются такими же, как легкая цепь родительского анти-PD-1 антитела, и домены VHPD-1-CH1 являются такими же, как вариабельный домен тяжелой цепи и константный домен тяжелой цепи родительского анти-PD-1 антитела.
8. Связывающий белок по п.1, в котором указанная первая полипептидная цепь содержит последовательность аминокислот 23-679 SEQ ID NO: 182, указанная вторая полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 186 и указанная третья полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 188.
9. Связывающий белок по п.1, в котором указанная первая полипептидная цепь содержит последовательность аминокислот 23-687 SEQ ID NO: 189, указанная вторая полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 193 и указанная третья полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 195.
10. Связывающий белок по п.1, в котором указанная первая полипептидная цепь содержит последовательность аминокислот 23-679 SEQ ID NO: 196, указанная вторая полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 200 и указанная третья полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 202.
11. Связывающий белок по п.1, в котором указанная первая полипептидная цепь содержит последовательность аминокислот 23-687 SEQ ID NO: 203, указанная вторая полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 207 и указанная третья полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 209.
12. Связывающий белок по п.1, в котором указанная первая полипептидная цепь содержит последовательность аминокислот 23-679 SEQ ID NO: 210, указанная вторая полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 214 и указанная третья полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 216.
13. Связывающий белок по п.1, в котором указанная первая полипептидная цепь содержит последовательность аминокислот 23-687 SEQ ID NO: 217, указанная вторая полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 221 и указанная третья полипептидная цепь содержит аминокислотную последовательность SEQ ID NO: 223.
14. Анти-LAG-3 антитело или его антигенсвязывающая часть, способные связывать LAG-3 человека, где антитело или антигенсвязывающая часть антитела содержат набор из шести CDR, CDR-H1, CDR-H2, CDR-H3, CDR-L1, CDR-L2 и CDR-L3, выбранных из группы, состоящей из следующих наборов CDR:
CDR-H1, состоящая из DDYMH (остатки 31-35 SEQ ID NO: 135), CDR-H2, состоящая из WIVPENANTVYASKFQG (SEQ ID NO: 224), CDR-H3, состоящая из YGDY (остатки 99-102 SEQ ID NO: 135), CDR-L1, состоящая из RASQEISGYLS (остатки 24-34 SEQ ID NO: 138), CDR-L2, состоящая из AASALDS (остатки 50-56 SEQ ID NO: 138), и CDR-L3, состоящая из LQYASYPLT (остатки 89-97 SEQ ID NO: 138);
CDR-H1, состоящая из DDYMH (остатки 31-35 SEQ ID NO: 136), CDR-H2, содержащая WIVPRNANTVYASKFQG (SEQ ID NO: 225), CDR-H3, состоящая из YGDY (остатки 99-102 SEQ ID NO: 136), CDR-L1, состоящая из RASQEISGYLS (остатки 24-34 SEQ ID NO: 139), CDR-L2, состоящая из AASALDL (остатки 50-56 SEQ ID NO: 139), и CDR-L3, состоящая из LQYASYPLT (остатки 89-97 SEQ ID NO: 139); или
CDR-H1, состоящая из DDYMH (остатки 31-35 SEQ ID NO: 136), CDR-H2, состоящая из WIVPRNANTVYASKFQG (SEQ ID NO: 225), CDR-H3, состоящая из YGDY (остатки 99-102 SEQ ID NO: 136), CDR-L1, состоящая из RASQEISGYLS (остатки 24-34 SEQ ID NO: 117), CDR-L2, состоящая из AASTLDS (остатки 50-56 SEQ ID NO: 117), и CDR-L3, состоящая из LQYASYPLT (остатки 89-97 SEQ ID NO: 117).
15. Анти-LAG-3 антитело или его антигенсвязывающая часть, способные связывать LAG-3 человека, где антитело или антигенсвязывающая часть антитела содержат пару доменов VH и VL, где пара доменов VH и VL содержит аминокислотные последовательности, выбранные из группы, состоящей из:
SEQ ID NO: 135 и SEQ ID NO: 138,
SEQ ID NO: 136 и SEQ ID NO: 139,
SEQ ID NO: 136 и SEQ ID NO: 117,
SEQ ID NO: 226 и SEQ ID NO: 138,
SEQ ID NO: 227 и SEQ ID NO: 139 и
SEQ ID NO: 227 и SEQ ID NO: 117.
16. Антитело по любому из пп. 14, 15 или связывающий белок по любому из пп. 1-13, содержащие Fc-область, содержащую SEQ ID NO: 28.
17. Фармацевтическая композиция, содержащая по меньшей мере одно анти-LAG-3 антитело или его антигенсвязывающий фрагмент по п.14 в эффективном количестве и фармацевтически приемлемый носитель, для лечения заболевания или расстройства, при котором активность, опосредованная LAG-3, является вредной.
18. Фармацевтическая композиция, содержащая по меньшей мере один биспецифический связывающий белок по п.1 в эффективном количестве и фармацевтически приемлемый носитель, для лечения заболевания или расстройства, при котором активность, опосредованная PD-1, и/или активность, опосредованная LAG-3, является вредной.
19. Применение биспецифического связывающего белка по п.1 для лечения заболевания или расстройства, при котором активность, опосредованная PD-1, и/или активность, опосредованная LAG-3, является вредной.
20. Применение по п.19, где указанное заболевание представляет собой рак.
21. Применение по п.20, где рак представляет собой меланому, почечный рак, рак простаты, аденокарциному поджелудочной железы, рак груди, рак толстой кишки, рак легких, рак пищевода, плоскоклеточный рак головы и шеи, рак печени, рак яичников, рак шейки матки, рак щитовидной железы, глиобластому, глиому, лейкемию, лимфому или первичный рак кости.
22. Применение по п. 21, где меланома представляет собой метастатическую злокачественную меланому, почечный рак представляет собой светлоклеточную карциному, рак простаты представляет собой гормонорезистентную аденокарциному простаты, рак легких представляет собой немелкоклеточный рак легких или первичный рак кости представляет собой остеосаркому, саркому Юинга, злокачественную фиброзную гистиоцитому и хондросаркому.
23. Способ изготовления лекарственного средства для лечения заболевания или расстройства, при котором активность, опосредованная PD-1, и/или активность, опосредованная LAG-3, является вредной, включающий приготовление по меньшей мере одного биспецифического связывающего белка по п.1 с фармацевтически приемлемым носителем.
24. Способ по п.23, где указанное заболевание представляет собой рак.
25. Способ по п.24, где рак представляет собой меланому, почечный рак, рак простаты, аденокарциному поджелудочной железы, рак груди, рак толстой кишки, рак легких, рак пищевода, плоскоклеточный рак головы и шеи, рак печени, рак яичников, рак шейки матки, рак щитовидной железы, глиобластому, глиому, лейкемию, лимфому или первичный рак кости.
26. Способ по п.25, где меланома представляет собой метастатическую злокачественную меланому, почечный рак представляет собой светлоклеточную карциному, рак простаты представляет собой гормонорезистентную аденокарциному простаты, рак легких представляет собой немелкоклеточный рак легких или первичный рак кости педставляет собой остеосаркому, саркому Юинга, злокачественную фиброзную гистиоцитому и хондросаркому.
27. Способ лечения расстройства, при котором активность, опосредованная PD-1 и/или опосредованная LAG-3, является вредной, включающий введение нуждающемуся в этом субъекту эффективного количества фармацевтической композиции по п.17 или 18 или их комбинации.
28. Способ по п.27, где указанное заболевание представляет собой рак.
29. Способ по п.28, где рак представляет собой меланому, почечный рак, рак простаты, аденокарциному поджелудочной железы, рак груди, рак толстой кишки, рак легких, рак пищевода, плоскоклеточный рак головы и шеи, рак печени, рак яичников, рак шейки матки, рак щитовидной железы, глиобластому, глиому, лейкемию, лимфому или первичный рак кости.
30. Способ по п.29, где меланома представляет собой метастатическую злокачественную меланому, почечный рак представляет собой светлоклеточную карциному, рак простаты представляет собой гормонорезистентную аденокарциному простаты, рак легких представляет собой немелкоклеточный рак легких или первичный рак кости педставляет собой остеосаркому, саркому Юинга, злокачественную фиброзную гистиоцитому и хондросаркому.
31. Способ по любому из пп.27-30, где субъект представляет собой человека.
| US 2017247456 A1, 31.08.2017 | |||
| EVA DAHLEN et al., Bispecific antibodies in cancer immunotherapy, Therapeutic Advances in Vaccines and Immunotherapy, 7 February 2018, 6(1) | |||
| MERCK KGAA, F-Star shake on immune-oncology bispecifics, NATURE BIOTECHNOLOGY, JULY 2017, VOLUME 35 NUMBER 7 | |||
| МОНОКЛОНАЛЬНЫЕ АНТИТЕЛА ЧЕЛОВЕКА К БЕЛКУ ПРОГРАММИРУЕМОЙ СМЕРТИ 1 (PD-1) И СПОСОБЫ ЛЕЧЕНИЯ РАКА С ИСПОЛЬЗОВАНИЕМ АНТИ-PD-1-АНТИТЕЛ САМОСТОЯТЕЛЬНО ИЛИ В КОМБИНАЦИИ С ДРУГИМИ ИММУНОТЕРАПЕВТИЧЕСКИМИ СРЕДСТВАМИ | 2006 |
|
RU2494107C2 |
Авторы
Даты
2022-10-26—Публикация
2019-04-30—Подача