Уровень техники
Несмотря на то, что биологические препараты пригодны для лечения многих заболеваний, в том числе различных видов рака, короткий период полужизни таких молекул в кровотоке представляет собой основное ограничение.
Биологические препараты пригодны для лечения рака различными способами. Лекарственные препараты на основе цитокинов могут воздействовать непосредственно на раковые клетки в результате влияния на рост и размножение таких клеток. Цитокины могут также стимулировать иммунную систему, стимулируя рост киллерных Т-клеток и других клеток, которые поражают раковые клетки. Кроме того, цитокины могут стимулировать передачу раковыми клетками химических веществ, которые привлекают клетки иммунной системы. См., например, Dranoff, Nature Reviews Cancer 4: 11-22 (2004); и Zhang et al., Proc Natl Acad Sci USA 106 (18): 7513-7518 (2009). Антитела являются желательными в качестве терапевтических средств благодаря их способности распознавать мишени как со специфичностью, так и с высокой аффинностью. Лекарственные препараты на основе моноклональных антител, включая те, которые целенаправленно воздействуют на опухолевые поверхностные антигены и ингибирующие сигналы, которые ограничивают активацию Т-клеток, уже более 20 лет представляли собой стандартный компонент терапевтических средств для лечения рака. См., например, Weiner, Nat Rev Cancer 15 (6): 361-370 (2015).
Короткий период полужизни в кровотоке представляет собой основное ограничение для многих биологических препаратов. См., например, Perdreau et al., European Cytokine Network 21: 297-307 (2010). Такие терапевтические средства короткого действия требуют профилей частого дозирования, которые могут снизить применимость в клинике, особенно в случае хронических состояний. Длительный период полужизни в сыворотке крови является желательным, поскольку он привел бы к уменьшению необходимости повторных инъекций молекулы для достижения терапевтически значимой концентрации в сыворотке крови. Способы продления периода полужизни терапевтических белков включают в себя пегилирование, слияние с человеческим сывороточным альбумином (HSA), слияние с константным фрагментом (Fc) иммуноглобулина IgG человека и слияние с неструктурированными полипептидами, такими как XTEN. См., например, Stohl, BioDrugs 29 (4): 215-239 (2015). Технологии продления периода полужизни создают возможности для новых и усовершенствованных биологических препаратов, которые приводят к снижению стоимости и неудобства частого дозирования. Таким образом, сохраняется постоянная потребность в новых реагентах и способах, пригодных для продления периодов полужизни терапевтических средств на основе белков и пептидов.
Краткое описание сущности изобретения
В данном документе предложены композиции, которые содержат альбумин-связывающий домен (ABD). Описанные в данном документе биологические препараты, которые содержат рассматриваемые альбумин-связывающие домены (т.е. слитые белки на основе альбумин-связывающего домена), предпочтительно демонстрируют увеличенные периоды полужизни и более эффективную фармакокинетику in vivo по сравнению с биологическими препаратами без ABD.
В одном аспекте в данном документе предложена композиция, которая содержит альбумин-связывающий домен (ABD). ABD содержит: а) вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2; и b) вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 любой из вариабельных областей легких цепей, изображенных на фиг. 2.
В некоторых вариантах осуществления vhCDR1 содержит последовательность vhCDR1, vhCDR2 содержит последовательность vhCDR2 и vhCDR3 содержит последовательность vhCDR3 в соответствии с любой из последовательностей vhCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В одном варианте осуществления вариабельная область тяжелой цепи содержит последовательность любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2. В некоторых вариантах осуществления vlCDR1 содержит последовательность vlCDR1, vlCDR2 содержит последовательность vlCDR2 и vlCDR3 содержит последовательность vlCDR3 в соответствии с любой из последовательностей vlCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В одном варианте осуществления вариабельная область легкой цепи содержит последовательность любой из вариабельных областей легких цепей, продемонстрированных на фиг. 2. В иллюстративном варианте осуществления альбумин-связывающий домен содержит вариабельную область тяжелой цепи и вариабельную область легкой цепи A10m3 (фиг. 2D).
В другом аспекте в данном документе предложена композиция, которая содержит вариантный IL-15. Вариантный IL-15 содержит одну или несколько аминокислотных замен, выбранных из группы, состоящей из K86A, K86R, N112A, N112S, N112Q, K86A/N112A, K86R/N112A, K86A/N112S, K86R/N112S, K86A/N112Q, K86R/N112Q, K86A/N112A/N79A, K86R/N112A/N79A, K86A/N112A/N79D, K86R/N112A/N79D, K86A/N112A/N79Q, K86R/N112A/N79Q, K86A/N112A/N71D, K86R/N112A/N71D, K86A/N112A/N71Q, K86R/N112A/N71Q, K86A/N112A/N71D/N79A, K86A/N112A/N71D/N79D, K86A/N112A/N71Q/N79A, K86A/N112A/N71Q/N79D, K86R/N112A/N71D/N79A, K86R/N112A/N71D/N79D, K86R/N112A/N71D/N79Q, K86R/N112A/N71Q/N79A, K86R/N112A/N71Q/N79D и K86R/N112A/N71Q/N79Q, по сравнению с исходным IL-15.
В некоторых вариантах осуществления вариантный IL-15 содержит аминокислотную последовательность любого из вариантных IL-15, продемонстрированных на фиг. 3. В определенных вариантах осуществления вариантный IL-15 дополнительно содержит альфа-рецептор IL-15 (IL-15Rα), присоединенный к указанному IL-15.
В одном аспекте в данном документе предложен слитый белок на основе альбумин-связывающего домена (ABD), который содержит ABD, присоединенный к партнеру по слиянию. ABD содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 любой из вариабельных областей легких цепей, продемонстрированных на фиг. 2.
В некоторых вариантах осуществления vhCDR1 содержит последовательность vhCDR1, vhCDR2 содержит последовательность vhCDR2 и vhCDR3 содержит последовательность vhCDR3 в соответствии с любой из последовательностей vhCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В некоторых вариантах осуществления вариабельная область тяжелой цепи содержит последовательность любой из последовательностей вариабельной области тяжелых цепей, продемонстрированных на фиг. 2.
В определенных вариантах осуществления vlCDR1 содержит последовательность vlCDR1, vlCDR2 содержит последовательность vlCDR2 и vlCDR3 содержит последовательность vlCDR3 в соответствии с любой из последовательностей vlCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В определенных вариантах осуществления вариабельная область легкой цепи содержит последовательность любой из последовательностей вариабельной области легкой цепи, продемонстрированных на фиг. 2.
В иллюстративном варианте осуществления вариабельная область тяжелой цепи и вариабельная область легкой цепи содержат вариабельную область тяжелой цепи и вариабельную область легкой цепи A10m3 соответственно (фиг. 2D).
В некоторых вариантах осуществления партнер по слиянию представляет собой цитокин. В определенных вариантах осуществления цитокин выбирают из IL-2, IL-7, IL-12, IL-15, IL-18, IL-21, GM-CSF и IFN-α.
В определенных вариантах осуществления партнер по слиянию представляет собой связывающий фрагмент. В некоторых вариантах осуществления связывающий фрагмент представляет собой scFv, который содержит вариабельную область тяжелой цепи scFv и вариабельную область легкой цепи scFv. В некоторых вариантах осуществления scFv выбирают из анти-scFv TGFβ, анти-scFv PD-L1 и анти-scFv TNF. В некоторых вариантах осуществления scFv представляет собой анти-scFv интерлейкина. В иллюстративных вариантах осуществления scFv представляет собой анти-scFv IL-1, анти-scFv IL-6, анти-scFv IL-8, анти-scFv IL-17(A-F) или анти-scFv IL-23.
В некоторых вариантах осуществления ABD присоединяется к указанному партнеру по слиянию с помощью линкера. В иллюстративном варианте осуществления линкер представляет собой (GGGGS)x, где x представляет собой целое число от 1 до 10.
В другом аспекте в данном документе предложен слитый белок на основе IL15-альбумин-связывающего домена (ABD) в соответствии с формулой (IL-15)-L-(ABD). ABD содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 любой из вариабельных областей легких цепей, продемонстрированных на фиг. 2, а L представляет собой линкер.
В некоторых вариантах осуществления vhCDR1 содержит последовательность vhCDR1, vhCDR2 содержит последовательность vhCDR2 и vhCDR3 содержит последовательность vhCDR3 в соответствии с любой из последовательностей vhCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В некоторых вариантах осуществления вариабельная область тяжелой цепи содержит последовательность любой из последовательностей вариабельной области тяжелых цепей, продемонстрированных на фиг. 2.
В определенных вариантах осуществления vlCDR1 содержит последовательность vlCDR1, vlCDR2 содержит последовательность vlCDR2 и vlCDR3 содержит последовательность vlCDR3 в соответствии с любой из последовательностей vlCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В определенных вариантах осуществления вариабельная область легкой цепи содержит последовательность любой из последовательностей вариабельной области легкой цепи, продемонстрированных на фиг. 2.
В иллюстративном варианте осуществления вариабельная область тяжелой цепи и вариабельная область легкой цепи содержат вариабельную область тяжелой цепи и вариабельную область легкой цепи A10m3 соответственно (фиг. 2D).
В одном варианте осуществления IL-15 представляет собой вариантный IL-15, содержащий одну или несколько аминокислотных замен, выбранных из группы, состоящей из K86A, K86R, N112A, N112S, N112Q, K86A/N112A, K86R/N112A, K86A/N112S, K86R/N112S, K86A/N112Q, K86R/N112Q, K86A/N112A/N79A, K86R/N112A/N79A, K86A/N112A/N79D, K86R/N112A/N79D, K86A/N112A/N79Q, K86R/N112A/N79Q, K86A/N112A/N71D, K86R/N112A/N71D, K86A/N112A/N71Q, K86R/N112A/N71Q, K86A/N112A/N71D/N79A, K86A/N112A/N71D/N79D, K86A/N112A/N71Q/N79A, K86A/N112A/N71Q/N79D, K86R/N112A/N71D/N79A, K86R/N112A/N71D/N79D, K86R/N112A/N71D/N79Q, K86R/N112A/N71Q/N79A, K86R/N112A/N71Q/N79D и K86R/N112A/N71Q/N79Q, по сравнению с исходным IL-15. В некоторых вариантах осуществления вариантный IL-15 содержит аминокислотную последовательность любого из вариантных IL-15, продемонстрированных на фиг. 3. В иллюстративном варианте осуществления вариантный IL-15 содержит аминокислотную последовательность IL15 K86R/N112A.
В определенных вариантах осуществления IL-15 представляет собой IL-15 дикого типа. В некоторых вариантах осуществления IL-15 представляет собой IL-15 дикого типа, присоединенный к альфа-рецептору IL-15 (IL-15Rα).
В определенных вариантах осуществления линкер выбирают из любого из линкеров, продемонстрированных на фиг. 48. В некоторых вариантах осуществления линкер представляет собой (GGGGS)x, где x представляет собой целое число от 1 до 10.
В одном варианте осуществления слитый белок на основе IL15-ABD имеет аминокислотную последовательность в соответствии с любой из аминокислотных последовательностей, продемонстрированных на фиг. 4.
В другом аспекте в данном документе представлен слитый белок на основе IL12-альбумин-связывающего домена (ABD) в соответствии с формулой (IL-12)-L-(ABD). ABD содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 любой из вариабельных областей легких цепей, продемонстрированных на фиг. 2, а L представляет собой линкер.
В некоторых вариантах осуществления vhCDR1 содержит последовательность vhCDR1, vhCDR2 содержит последовательность vhCDR2 и vhCDR3 содержит последовательность vhCDR3 в соответствии с любой из последовательностей vhCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В некоторых вариантах осуществления вариабельная область тяжелой цепи содержит последовательность любой из последовательностей вариабельной области тяжелых цепей, продемонстрированных на фиг. 2.
В определенных вариантах осуществления vlCDR1 содержит последовательность vlCDR1, vlCDR2 содержит последовательность vlCDR2 и vlCDR3 содержит последовательность vlCDR3 в соответствии с любой из последовательностей vlCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В определенных вариантах осуществления вариабельная область легкой цепи содержит последовательность любой из последовательностей вариабельной области легкой цепи, продемонстрированных на фиг. 2.
В иллюстративном варианте осуществления вариабельная область тяжелой цепи и вариабельная область легкой цепи содержат вариабельную область тяжелой цепи и вариабельную область легкой цепи A10m3 соответственно (фиг. 2D).
В одном варианте осуществления IL-12 представляет собой одноцепочечный IL-12, который содержит субъединицу p35, субъединицу p40 и линкер IL-12, и линкер IL-12 ковалентно присоединяет субъединицу p35 к субъединице p40. В определенных вариантах осуществления линкер выбирают из любого из линкеров, продемонстрированных на фиг. 48. В иллюстративном варианте осуществления линкер представляет собой (GGGGS)x, где x представляет собой целое число от 1 до 10.
В иллюстративном варианте осуществления слитый белок на основе IL12-ABD содержит аминокислотную последовательность в соответствии с любой из аминокислотных последовательностей фиг. 20.
В другом аспекте в данном документе предложен слитый белок на основе альбумин-связывающего домена (ABD), имеющий формулу от N-конца к C-концу, выбранную из: a) (IL-12)-L1-(ABD)-L2-(IL-15); и b) (IL-15)-L1-(ABD)-L2-(IL-12). ABD содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 любой из вариабельных областей легких цепей, продемонстрированных на фиг. 2, а L1 и L2 представляют собой первый и второй линкер соответственно.
В некоторых вариантах осуществления vhCDR1 содержит последовательность vhCDR1, vhCDR2 содержит последовательность vhCDR2 и vhCDR3 содержит последовательность vhCDR3 в соответствии с любой из последовательностей vhCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В некоторых вариантах осуществления вариабельная область тяжелой цепи содержит последовательность любой из последовательностей вариабельной области тяжелых цепей, продемонстрированных на фиг. 2.
В определенных вариантах осуществления vlCDR1 содержит последовательность vlCDR1, vlCDR2 содержит последовательность vlCDR2 и vlCDR3 содержит последовательность vlCDR3 в соответствии с любой из последовательностей vlCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В определенных вариантах осуществления вариабельная область легкой цепи содержит последовательность любой из последовательностей вариабельной области легкой цепи, продемонстрированных на фиг. 2.
В иллюстративном варианте осуществления вариабельная область тяжелой цепи и вариабельная область легкой цепи содержат вариабельную область тяжелой цепи и вариабельную область легкой цепи A10m3 соответственно (фиг. 2D).
В некоторых вариантах осуществления IL-15 содержит полипептид IL-15 дикого типа. В некоторых вариантах осуществления IL-15 дикого типа присоединен к альфа-рецептору IL-15 (IL-15Rα).
В одном варианте осуществления IL-15 представляет собой вариантный IL-15, который содержит одну или несколько аминокислотных замен, выбранных из K86A, K86R, N112A, N112S, N112Q, K86A/N112A, K86R/N112A, K86A/N112S, K86R/N112S, K86A/N112Q, K86R/N112Q, K86A/N112A/N79A, K86R/N112A/N79A, K86A/N112A/N79D, K86R/N112A/N79D, K86A/N112A/N79Q, K86R/N112A/N79Q, K86A/N112A/N71D, K86R/N112A/N71D, K86A/N112A/N71Q, K86R/N112A/N71Q, K86A/N112A/N71D/N79A, K86A/N112A/N71D/N79D, K86A/N112A/N71Q/N79A, K86A/N112A/N71Q/N79D, K86R/N112A/N71D/N79A, K86R/N112A/N71D/N79D, K86R/N112A/N71D/N79Q, K86R/N112A/N71Q/N79A, K86R/N112A/N71Q/N79D и K86R/N112A/N71Q/N79Q, по сравнению с исходным IL-15. В одном варианте осуществления IL15 содержит аминокислотную последовательность в соответствии с любой из аминокислотных последовательностей, продемонстрированных на фиг. 3.
В определенных вариантах осуществления IL-12 представляет собой одноцепочечный IL-12, который содержит субъединицу p35, субъединицу p40 и линкер IL-12, где линкер IL-12 присоединяет субъединицу p35 к субъединице p40.
В некоторых вариантах осуществления каждый из первого линкера и второго линкера независимо выбирают из любого из линкеров, продемонстрированных на фиг. 48. В иллюстративном варианте осуществления линкер представляет собой (GGGGS)x, где x представляет собой целое число от 1 до 10.
В другом аспекте в данном документе предложен слитый белок с ABD, который содержит альбумин-связывающий домен (ABD), цитокин и линкер (L) в соответствии с формулой (цитокин)-L-(ABD). ABD содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 любой из вариабельных областей легких цепей, продемонстрированных на фиг. 2, а L представляет собой линкер. Цитокин выбирают из IL-2, IL-7, IL-12, IL-15, IL-18, IL-21, GM-CSF и IFN-α.
В некоторых вариантах осуществления vhCDR1 содержит последовательность vhCDR1, vhCDR2 содержит последовательность vhCDR2 и vhCDR3 содержит последовательность vhCDR3 в соответствии с любой из последовательностей vhCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В некоторых вариантах осуществления вариабельная область тяжелой цепи содержит последовательность любой из последовательностей вариабельной области тяжелых цепей, продемонстрированных на фиг. 2.
В определенных вариантах осуществления vlCDR1 содержит последовательность vlCDR1, vlCDR2 содержит последовательность vlCDR2 и vlCDR3 содержит последовательность vlCDR3 в соответствии с любой из последовательностей vlCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В определенных вариантах осуществления вариабельная область легкой цепи содержит последовательность любой из последовательностей вариабельной области легкой цепи, продемонстрированных на фиг. 2.
В иллюстративном варианте осуществления вариабельная область тяжелой цепи и вариабельная область легкой цепи содержат вариабельную область тяжелой цепи и вариабельную область легкой цепи A10m3 соответственно (фиг. 2D).
В некоторых вариантах осуществления линкер выбирают из любого из линкеров, продемонстрированных на фиг. 48. В иллюстративном варианте осуществления линкер представляет собой (GGGGS)x, где x представляет собой целое число от 1 до 10.
В другом аспекте в данном документе предложен слитый белок с ABD в соответствии с формулой: (FP1)-L1-(ABD)-L2-(FP2), где ABD представляет собой альбумин-связывающий домен, который включает вариабельную тяжелую цепь и вариабельную легкую цепь; где FP1 и FP2 представляют собой первый слитый белок и второй слитый белок соответственно; и где L1 и L2 представляют собой первый и второй линкер, соответственно. ABD содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 любой из вариабельных областей легких цепей, продемонстрированных на фиг. 2.
В некоторых вариантах осуществления vhCDR1 содержит последовательность vhCDR1, vhCDR2 содержит последовательность vhCDR2 и vhCDR3 содержит последовательность vhCDR3 в соответствии с любой из последовательностей vhCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В некоторых вариантах осуществления вариабельная область тяжелой цепи содержит последовательность любой из последовательностей вариабельной области тяжелых цепей, продемонстрированных на фиг. 2.
В определенных вариантах осуществления vlCDR1 содержит последовательность vlCDR1, vlCDR2 содержит последовательность vlCDR2 и vlCDR3 содержит последовательность vlCDR3 в соответствии с любой из последовательностей vlCDR1, vhCDR2 и vhCDR3, продемонстрированных на фиг. 2. В определенных вариантах осуществления вариабельная область легкой цепи содержит последовательность любой из последовательностей вариабельной области легкой цепи, продемонстрированных на фиг. 2.
В иллюстративном варианте осуществления вариабельная область тяжелой цепи и вариабельная область легкой цепи содержат вариабельную область тяжелой цепи и вариабельную область легкой цепи A10m3 соответственно (фиг. 2D).
В некоторых вариантах осуществления FP1 и FP2 представляют собой первый цитокин и второй цитокин соответственно. В иллюстративном варианте осуществления первый цитокин и указанный второй цитокин выбирают из IL-2 и IL-12; IL-7 и IL-15; IL-15 и IL-12; IL-18 и GM-CSF; IL-21 и IL-15; GM-CSF и IL-12; GM-CSF и IL-21; и IFN-α и IL-15.
В некоторых вариантах осуществления первого и второго партнеров по слиянию выбирают из анти-scFv PD-L1 и IL-12; анти-scFv PD-L1 и IL-15; анти-scFv PD-L1 и анти-scFv TGFβ; первого анти-scFv PD-L1 и второго scFv PD-L1; анти-scFv TGFβ и IL-12; анти-scFv TGFβ и IL-15; анти-scFv TGFβ и scFv PD-L1; и первого анти-scFv TGFβ и второго анти-scFv TGFβ.
В одном варианте осуществления FP1 и FP2 представляют собой первый связывающий фрагмент и второй связывающий фрагмент соответственно. В определенных вариантах осуществления каждый из первого связывающего фрагмента и второго связывающего фрагмента представляют собой scFv. В иллюстративном варианте осуществления первый связывающий фрагмент и второй связывающий фрагмент выбирают из scFv TNF и scFv IL-1; scFv TNF и scFv IL-6; scFv TNF и scFv IL-8; scFv TNF и scFv IL-17 (изоформы A-F); ScFv TNF и scFv IL-23 и первого scFv TNF и второго scFv TNF.
В некоторых вариантах осуществления каждый из первого линкера и второго линкера независимо выбирают из любого из линкеров, продемонстрированных на фиг. 48. В одном варианте осуществления каждый из первого линкера и второго линкера независимо друг от друга представляет собой (GGGGS)x, где x представляет собой целое число от 1 до 10.
В другом аспекте в данном документе представлен слитый белок на основе альбумин-связывающего домена (ABD), который содержит TGFβ-связывающий домен и альбумин-связывающий домен. Альбумин-связывающий домен содержит вариабельную область тяжелой цепи ABD и вариабельную область легкой цепи ABD, имеющие аминокислотные последовательности любой из вариабельных областей тяжелых цепей и вариабельных областей легких цепей на фиг. 2.
В иллюстративном варианте осуществления вариабельная область тяжелой цепи ABD и указанная вариабельная область легкой цепи ABD содержат аминокислотную последовательность вариабельной области тяжелой цепи и вариабельной области легкой цепи A10m3.
В одном варианте осуществления TGF-связывающий домен представляет собой scFv, который содержит вариабельную область тяжелой цепи и вариабельную область легкой цепи 4D9 (фиг. 40В). В некоторых вариантах осуществления ABD дополнительно содержит IL-12-, IL-15- или PD-L1-связывающий домен или второй TGFβ-связывающий домен.
В другом аспекте в данном документе предложен слитый белок на основе альбумин-связывающего домена (ABD), который содержит PD-L1-связывающий домен и альбумин-связывающий домен. Альбумин-связывающий домен содержит вариабельную область тяжелой цепи ABD и вариабельную область легкой цепи ABD, имеющие аминокислотные последовательности любой из вариабельных областей тяжелых цепей и вариабельных областей легких цепей на фиг. 2.
В некоторых вариантах осуществления вариабельная область тяжелой цепи ABD и указанная вариабельная область легкой цепи ABD содержат аминокислотную последовательность вариабельной области тяжелой цепи и вариабельной области легкой цепи A10m3 (фиг. 2D). В определенных вариантах осуществления PD-L1-связывающий домен представляет собой scFv, содержащий вариабельную область тяжелой цепи и вариабельную область легкой цепи 10D12 (фиг. 50).
В некоторых вариантах осуществления ABD дополнительно содержит IL-12-, IL-15- или TGFβ-связывающий домен или второй PD-L1-связывающий домен.
В другом аспекте в данном документе предложены нуклеиновая кислота, которая кодирует любой из альбумин-связывающих доменов, вариантных IL-15 или слитых белков на основе ABD, описанных в данном документе, клетки-хозяева, которые содержат любые такие нуклеиновые кислоты, и способы получения таких ABD, вариантных IL-15 или слитых белков на основе ABD.
В еще одном аспекте в данном документе предложен способ ингибирования или уменьшения опухоли у субъекта, нуждающегося в этом, при этом способ включает введение субъекту слитого белка на основе ABD.
Краткое описание графических материалов
На фиг. 1A-C продемонстрировано несколько иллюстративных слитых белков на основе альбумин-связывающего домена, раскрытых в данном документе, в том числе слитых белков, в которых альбумин-связывающий домен присоединен к цитокину (например, IL-12 или IL-15), и слитых белков, в которых альбумин-связывающий домен присоединен к 1) двум цитокинам; 2) двум связывающим фрагментам (например, scFvs); 3) связывающему фрагменту и цитокину.
На фиг. 2А-2Z продемонстрированы последовательности иллюстративных альбумин-связывающих доменов, включенных в определенные варианты осуществления рассматриваемых слитых белков на основе альбумин-связывающего домена, описанных в данном документе. В эти фигуры включены последовательности вариабельного домена тяжелой цепи и вариабельного домена легкой цепи, а также определенные последовательности vhCDR1-3 и vlCDR1-3.
На фиг. 3 продемонстрированы последовательности иллюстративных вариантных IL-15, описанных в данном документе.
На фиг. 4 продемонстрированы иллюстративные варианты осуществления слитых белков на основе IL-15-ABD, описанных в данном документе. Слитые белки на основе IL-15-ABD, продемонстрированные на фиг. 4, включают в себя ABD A10m3.
На фиг. 5 продемонстрированы результаты исследований, показывающих, что транскрипция не является причиной низкой экспрессии IL-15-A10m3 в клетках HEK293T. Проверка мРНК IL15-A10m3 в трансфицированных клетках. А) Экспрессия IL15-A10m3 в HEK293 не может быть выявлена с помощью вестерн-блоттинга с использованием анти-His-тэг антитела (слева), или с помощью функционального связывания с использованием ИФА с MSA (справа). М: маркер, CK: среда для культивирования нетрансфицированных клеток в качестве контроля, 1-3: среда из 3 независимо трансфицированных клеточных культур (100, 200 и 250 = 100, 200, 250 мкг/мл зеоцина соответственно). 10 мкг/мл продуцированного E. coli IL15-A10m3 выступали в качестве положительного контроля. B) мРНК готовили из 4 независимо трансфицированных клеток IL15-A10m3 и проводили RT-PCR для количественного определения уровня мРНК из мРНК IL15-A10m3 по сравнению с уровнем гена домашнего хозяйства, GAPDH. Полоса 1) контроль с использованием нетрансфицированных клеток; 2-5) транскрипция мРНК IL15-A10m3, по-видимому, является обычной во всех трансфицированных клетках; 6) положительный контроль мРНК GAPDH.
На фиг. 6 продемонстрирована идентификация потенциальных сайтов убиквитинирования IL-15, которые предположительно защищены альфа-рецептором IL-15 (IL-15Rα), связывающимся с IL-15. (A) K86 в красном цвете представляет собой предполагаемый сайт убиквитинирования, который находится рядом с сайтами связывания IL-15Rα (отмечены звездочками); (B) K86 представляет собой мишень для убиквитинирования от UbPred, онлайн базы данных сайтов убиквитинирования (www.ubpred.org).
На фиг. 7 продемонстрированы схематические иллюстрации различных слитых белков на основе IL-15-ABD с повышенной стабильностью, описанных в данном документе, в том числе IL-15-ABD с партнером по слиянию IL-15Ra/IL15 «домен sushi» (A) и слитые белки на основе IL-15-ABD, которые включают в себя партнеров по слиянию вариантного IL-15, имеющих аминокислотные замены в предполагаемом сайте убиквитинирования K86 (B).
На фиг. 8 продемонстрированы исследования, оценивающие уровень экспрессии (фиг. 8А) и способность продуцируемых клетками HEK293 вариантов IL-15-ABD K86R и K86A и IL-15Rα/IL-15-ABD связываться с мышиным сывороточным альбумином (фиг. 8A) и IL-15Rα (фиг. 8В). A) Показания ИФА в случае связывания IL15-ABD с мышиным сывороточным альбумином (MSA) 12 клонов IL-1510-A3m3 ДТ (ДТ), 12 клонов IL-15Rα/IL-15-A10m3 (IL15Ra), 6 мутантных клонов IL-15 K86A-A10m3 (K86A) и 6 мутантных клонов IL-15 K86R-A10m3 (K86R). Среду для культивирования из каждой лунки образца из 24-луночных планшетов добавляли в планшеты для ИФА, покрытые MSA. B) ИФА в случае связывания IL-15 с альфа-рецептором IL-15 (IL-15Rα), который был нанесен на планшет, использовали для подтверждения того, что мутации K86A (клон A3, звездочка желтого цвета) и K86R (клон R6, звездочка зеленого цвета) не оказывали влияния на активность связывания IL-15 с IL-15Rα. Внутренний IL-15Rα в IL-15Rα-IL-15-A10m3 (клон alpha1, звездочка красного цвета) мог связываться с внутренним IL15 и, таким образом, блокировать его связывание с IL-15Rα, нанесенным на планшет. 10 мкг/мл продуцированного E. coli IL-15-A10m3 ДТ использовали в качестве положительного контроля.
На фиг. 9 продемонстрировано расширенное производство клона №6 IL15K86R-A10m3. А) Хроматограмма IL15K86R-A10m3 с использованием колонки для эксклюзионной хроматографии; В) анализ SDS-PAGE фракций SEC от 14 до 42; C) конечные продукты (1: IL15K86R-A10m3, 2: IL15Ra-IL15-A10m3) были окончательно подтверждены с помощью SDS-PAGE (слева) и вестерн-блоттинга с анти-His-тэг антитела (справа).
На фиг. 10 продемонстрированы результаты анализов связывания in vitro, подтверждающие способность IL-15 K86R-A10m3 связывать мышиный сывороточный альбумин (MSA).
На фиг. 11 продемонстрированы результаты анализов пролиферации CTLL2, показывающие, что полипептиды IL-15 K86R-A10m3, продуцируемые из клеток HEK293T, имеют пониженную биологическую активность по сравнению с IL-15 дикого типа и IL-15-A10m3, продуцируемыми из E. coli.
На фиг. 12 продемонстрированы результаты исследований, показывающие, что пониженная биологическая активность продуцируемого клетками HEK IL-15 K86R-A10m3, по меньшей мере, частично обусловлена его гликозилированием.
На фиг. 13 продемонстрированы результаты исследований, показывающие, что мутация N112A, введенная в IL15 K86R-A10m3, может приводить к восстановлению биологической активности IL-15, сопоставимой с таковой дегликозилированного IL-15R-A10m3 в анализах пролиферации CTLL2 (A). (B) Дополнительно показано, что слитые белки на основе IL-15R-A10m3 с аминокислотными заменами IL-15 N112A, N112Q и N112S проявляли повышенную биологическую активность, обратно пропорциональную размеру замещенной боковой цепи.
На фиг. 14 продемонстрирована экспериментальная схема исследования для оценки in vivo эффектов IL-15 и IL-15-ABD на опухолевый рост в модели меланомы мыши B16-F10.
На фиг. 15 продемонстрирован график, показывающий величину ингибирования опухолевого роста B16-F10 у мышей C57BL/6, обработанных слитым белком на основе IL-15-ABD, через 11 дней после обработки.
На фиг. 16 продемонстрированы анализы FACS, изображающие влияние обработки IL-15-ABD на популяции лимфоцитов, инфильтрующих опухоль.
На фиг. 17 и 18 продемонстрирован обобщенный результат обработки IL-15-ABD на популяции лимфоцитов в селезенке и опухолях.
На фиг. 19A и B продемонстрированы результаты исследований, показывающие стабильность рассматриваемого IL-15-ABD в мышиной модели (A) и в сыворотке крови человека (B) по сравнению с контрольным IL-15 ДТ.
На фиг. 20 продемонстрированы последовательности иллюстративных IL-12-ABD, описанных в данном документе: mIL-12sc-A10m3 и IL-12sc-A10m3 человека.
На фиг. 21 и 22 продемонстрированы исследования, показывающие, что рассматриваемый IL-12-ABD, продуцируемый из клеток HEK293T, является биологически активным в анализах in vitro и клеточных анализах.
На фиг. 23 продемонстрирована экспериментальная схема исследования для оценки in vivo эффектов IL-12 и IL-12-ABD на опухолевый рост в модели меланомы мыши B16-F10. Аналогичные молярные концентрации IL-12 и IL-12 были использованы в трех различных концентрациях. Например, 3 мкг IL-12 представляет собой такую же самую молярную концентрацию, что и 4,5 мкг IL-12-ABD, 10 мкг IL-12 представляет собой такую самую же молярную концентрацию, что и 15 мкг IL-12-ABD, а 20 мкг IL-12 представляет собой такую же самую молярную концентрацию, что и 30 мкг IL-12-ABD. Молекулярная масса IL-12 составляет 70 кДа, а молекулярная масса IL-12-ABD составляет 107 кДа.
На фиг. 24 продемонстрированы графики, изображающие кинетику роста опухолей различных групп в исследованиях IL-12-ABD/IL-12 in vivo. На фиг. 24A продемонстрированы результаты оценок размера опухоли в группах IL-12 и IL-12-ABD, на фиг. 24B продемонстрированы группы обработки IL-12 отдельно, а на фиг. 24C продемонстрированы группы обработки IL-12-ABD отдельно.
На фиг. 25 продемонстрированы графики, изображающие кинетику опухолевого роста у отдельных животных в каждой из различных групп исследований IL-12-ABD/IL-12 in vivo.
На фиг. 26 продемонстрированы графики, показывающие объемы опухоли различных групп в исследованиях IL-12-ABD/IL-12 in vivo, через 10 дней после обработки.
На фиг. 27 продемонстрированы измерения продольной массы тела (слева) и % массы тела (справа) у мышей, несущих опухоль B16-F10, в различные временные точки после обработки IL-12 или IL-12-ABD.
На фиг. 28 продемонстрированы кривые псевдо-выживания Каплана-Мейера для различных групп в исследованиях IL-12-ABD/IL-12 in vivo.
На фиг. 29 продемонстрирован обобщенный результат исследований, сравнивающих фармакодинамические эффекты однократной дозы IL-12-ABD и IL-12 в отношении мышей, несущих опухоль B16-F10, через пять дней. Продемонстрированы сравнения массы опухоли, массы селезенки, сывороточного IFN-γ и массы тела.
На фиг. 30 продемонстрирован результат исследования, сравнивающего объем опухоли у мышей, несущих опухоль B16-F10, через 10 дней, которым вводили IL-12-ABD (1,3 мкг), IL-12 (30 мкг) или плацебо. В указанном исследовании IL-12-ABD вводят в ~ 30 раз более низкой молярной дозе, чем IL-12.
На фиг. 31 дополнительно продемонстрированы гематопоэтические эффекты IL-12-ABD и IL-12 у мышей из исследования, продемонстрированного на фиг. 32, через 3 и 7 дней.
На фиг. 32 продемонстрировано исследование, показывающее влияние комбинированных видов терапии однократными дозами с использованием IL-12-ABD или IL-12 с анти-PD-1 антителами на опухолевый рост у мышей, несущих опухоль B16-F10.
На фиг. 33 продемонстрированы результаты анализа сыворотки, показывающие увеличение PK в случае IL12-ABD по сравнению с IL-12 ДТ.
На фиг. 34 продемонстрированы последовательности иллюстративного рассматриваемого IL-15-ABD-IL-12, описанного в данном документе: 1) hIL-15 (K86R/N112A)-A10m3-mIL-12sc; 2) mIL-12sc-A10m3-hIL-15 (K86R); и 3) mIL-12sc-A10m3-hIL-15 (K86R/N112A).
На фиг. 35 продемонстрированы активности связывания как hIL -15 (K86R)-A10m3-mIL-12sc, так и mIL-12sc -A10m3-hIL-15 (K86R) с MSA (A), альфа-рецептором IL-15 и бета2-рецептором IL-12 (B), подтвержденные ИФА.
На фиг. 36 продемонстрированы последовательности дополнительного иллюстративного рассматриваемого IL-15-ABD-IL-12, описанные в данном документе: 1) hIL-15 (K86R) -A10m3-hIL-12sc; 2) hIL-12sc-A10m3-hIL-15 (K86R); и 3) hIL-12sc-A10m3-hIL-15 (K86R/N112A).
На фиг. 37 и 38 продемонстрированы результаты экспериментов, показывающих, что IL-15-ABD-IL-12, описанный в данном документе, проявляет как активность IL-12 (фиг. 39), так и активность IL-15 (фиг. 40).
На фиг. 39 продемонстрированы результаты исследования, показывающие, что IL-15-ABD-IL-12, описанный в данном документе, проявлял более высокую активность по сравнению с IL-12-ABD в отдельности.
На фиг. 40А и В продемонстрированы последовательности двух иллюстративных TGFβ-связывающих доменов, описанных в данном документе, 4H7 и 4D9, в том числе последовательности вариабельной области тяжелой цепи, вариабельной области легкой цепи и форма scFv.
На фиг. 41 продемонстрированы результаты исследования, показывающие, что scFv TFFβ, описанные в данном документе, способны блокировать индуцированную TGFβ1 экспансию Treg (CD4+Foxp3+).
На фиг. 42 и 43 продемонстрировано, что блокада TGF-β обращает индуцированный TGF-β1 эпителиально-мезенхимальный переход (фиг. 44) и миграцию (фиг. 45). (фиг. 42А). Схематическое изображение потери E-кадгерина и индуцированной экспрессии виментина во время EMT. (фиг. 44B). Клетки мыши 4T1 культивировали в средах для роста с добавлением только TGF-β1 или совместно с 1D11 (панель 3) или анти-scFv TGF-β1 (панель 4), затем фиксировали и окрашивали антителом к E-кадгерину (зеленый цвет) и антителом к виментину (фиолетовый цвет). Ядра были докрашены DAPI (синий цвет). Обработка TGF-β1 индуцировала потерю E-кадгерина из межклеточных контактов и приводила к повышению экспрессии виментина. Указанный эффект обращается в результате добавления 1D11 или рассматриваемого анти-scFv TGF-β1, описанного в данном документе. На фиг. 43 дополнительно продемонстрировано, что рассматриваемые анти-scFv TGF-β1, описанные в данном документе, блокируют опосредованную TGF-β1 миграцию клеток карциномы.
На фиг. 44 представлен обобщенный результат исследований, показывающих, что рассматриваемые анти-scFv TGF-β1, описанные в данном документе, способны ингибировать опосредованное TGF-β фосфорилирование Smad2 в клетках человека (A) и мыши (B). В случае А рассматриваемые анти-scFv TGF-β1 были способны ингибировать опосредованное TGF-β1 фосфорилирование Smad2 в клетках человека дозозависимым образом. В случае B рассматриваемые анти-scFv TGF-β1 были способны ингибировать опосредованное TGF-β1, -β2 и -β3 мыши фосфорилирование Smad2 в клетках мыши.
На фиг. 45A продемонстрированы последовательности иллюстративных конструкций scFv TGF-β1-ABD (4D9M-A6m и 4H7-A6m).
На фиг. 45B продемонстрирован обобщенный результат исследования, показывающий, что анти-TGFβ-1-ABD расширяет анти-scFv TGFβ-1.
На фиг. 46 продемонстрирован обобщенный результат исследования, показывающий, что различные анти-scFv TGF-β1-ABD (бивалент в случае TGF-β1), продуцируемые в клетках E. coli или млекопитающих (HEK), способны связываться с мышиным сывороточным альбумином.
На фиг. 47 продемонстрирован обобщенный результат исследования, показывающий, что опосредованное TGFβ-1 ингибирование пролиферации Т-клеток было обращено (т.е. повышение пролиферации Т-клеток) с помощью иллюстративных конструкций scFv TGF-β1-ABD (4D9M-A6m и 4H7-A6m).
На фиг. 48 продемонстрированы иллюстративные линкеры, которые могут быть использованы с рассматриваемыми слитыми белками. Такие линкеры могут быть использованы в качестве линкеров scFv для вариабельных областей тяжелых цепей и вариабельных областей легких цепей ABD. Такие линкеры также могут быть использованы для присоединения альбумин-связывающих доменов, описанных в данном документе, к партнерам по слиянию IL-12 и IL-15 или для соединения компонентов партнеров по слиянию IL-12 (p35 и p40) или партнеров по слиянию IL-15 (IL-15 и IL-15Rα) друг с другом.
На фиг. 49A-G продемонстрирована аминокислотная последовательность иллюстративных цитокинов, которые могут быть включены в слитые белки на основе ABD, описанные в данном документе. На фиг. 49A-G также продемонстрированы иллюстративные слитые белки цитокин-ABD, при этом ABD представляет собой A10m3. Несмотря на то, что такие слитые белки на основе цитокина и ABD изображены с A10m3 ABD, любой ABD, в том числе ABD, описанный в данном документе, может быть включен в слитые белки цитокин-ABD.
На фиг. 50 продемонстрирована аминокислотная последовательность иллюстративного анти-PD-L1-связывающего домена, 10D12, который может быть использован с описанными в данном документе слитыми белками на основе ABD. 10D12 связывается с hPD-L1 при низком значении pH и перекрестно реагирует с mPD-L1. 10D12 не связывается с hPD-L2 или mPD-L2. Кроме того, 10D12 блокирует взаимодействие PD-1/PD-L1, а также взаимодействие B71/PD-L1.
На фиг. 51A и B продемонстрировано несколько пригодных комбинаций цитокин-ABD-цитокин (A) и связывающий фрагмент-ABD-цитокин/связывающий фрагмент (B). «A» и «B» в каждой комбинации могут переключаться к противоположной ориентации. На этих фигурах также продемонстрирован остов линкер-A10m3-линкер, который может быть включен в комбинации цитокин-ABD-цитокин и связывающий фрагмент-ABD-цитокин/связывающий фрагмент. Несмотря на то, что продемонстрирован A10m3 ABD, любой ABD, в том числе любой из продемонстрированных в данном документе (например, фиг. 2), может быть использован в таких конструкциях.
Подробное описание сущности изобретения
A. Обзор
Биологические препараты, в том числе цитокины и терапевтические средства на основе антител, пригодны для лечения видов рака.
Современные и потенциальные терапевтические средства на основе цитокинов включают средства, использующие IL-2, IL-7, IL-12, IL-15, IL-18, IL-21 GM-CSF и IFN-α.
IL-12 способен опосредовать иммунные эффекторные функции способом, совместимым с усилением провоспалительного эндогенного противоопухолевого иммунного ответа. (См., например, Boggio et al., J Exp Med 188:589-96 (1998); Cavallo et al., Cancer Res 59:414-21 (1999); Yu et al. Int Immunol 8:855-65 (1996); Nastala et al., J Immunol 153:1697:706 (1994); Brunda et al., J Exp Med 178: 1223-30 (1993). Известно, что IL-12 индуцирует воспалительный ответ Th1 CD4+ T-клеток, а также усиливает цитотоксичность CD8+ T-клеток. Исследования также показали, что секреция IFNγ Т-клетками, опосредованная IL-12, может обратить анергию Т-клеток и придать резистентность эффекторным Т-клетками по отношению к иммуносупрессорным регуляторным Т-клеткам. Способность IL-12 не только активировать адаптивную и врожденную иммунную системы, но и дополнительно модулировать в ином смысле враждебное в отношении иммунной системы микроокружение опухоли делает IL-12 идеальным кандидатом для иммунотерапии опухолей.
IL-15 способен стимулировать пролиферацию Т-клеток внутри опухолей (см., например, Miecnik et al., Sci Transl Med 6 (228): 228ra37 (2014)), а также продлевать выживаемость эффекторных CD8+ T-клеток памяти и является определяющим для развития NK-клеток. Таким образом, считается, что IL-15 может усиливать действие ингибиторов контрольных точек и других иммунотерапевтических средств, которые используют Т-клетки для атаки раковых клеток. В то же время мономер IL-15 имеет короткий период полужизни, составляющий менее 40 минут in vivo. Модификации мономера IL-15 могут улучшить его фармакокинетику in vivo при лечении видов рака. Эти модификации в основном были направлены на улучшение транс-презентации IL-15 альфа-субъединицей рецептора IL-15, IL-15Rα. Такие модификации включают в себя: 1) предассоциацию слияния IL-15 и слияния субъединицы его растворимого альфа-рецептора с Fc с образованием комплекса IL-15:IL-15Rα-Fc (см., например, Rubinstein et al., Proc Natl Acad Sci USA 103:9166-71 (2006)); 2) экспрессию гиперагонистов белка IL-15-sIL-15Rα-sushi (см., например, Bessard et al., Molecular Cancer Therapeutics 8: 2736-45 (2009)); и 3) предассоциацию мутантной формы IL-15 человека IL-15N72D с комплексом слияния IL-15Rα-Fc sushi-Fc (см., например, Zhu et al., Journal of Immunology 183:3598-6007 (2009)).
Лекарственные препараты на основе моноклональных антител, включая те, которые целенаправленно воздействуют на опухолевые поверхностные антигены и ингибирующие сигналы, которые ограничивают активацию Т-клеток, уже более 20 лет представляли собой стандартный компонент терапевтических средств для лечения рака. См., например, Weiner, Nat Rev Cancer 15 (6): 361-370 (2015).
Короткий период полужизни в кровотоке представляет собой серьезное препятствие для многих биологических препаратов, в том числе лекарственных препаратов на основе цитокинов и антител. См., например, Herrington-Symes et al., Advances in Bioscience and Biotechnology 4: 689-698 (2013) и Perdreau et al., European Cytokine Network 21: 297-307 (2010). Такие терапевтические средства короткого действия требуют профилей частого дозирования, которые могут снизить применимость в клинике, особенно в случае хронических состояний. Длительный период полужизни в сыворотке крови является желательным, поскольку он привел бы к уменьшению необходимости повторных инъекций молекулы для достижения терапевтически значимой концентрации в сыворотке крови. Способы продления периода полужизни терапевтических белков включают в себя пегилирование, слияние с человеческим сывороточным альбумином (HSA), слияние с константным фрагментом (Fc) иммуноглобулина IgG человека и слияние с неструктурированными слитыми белками, такими как XTEN. См., например, Stohl, BioDrugs 29 (4): 215-239 (2015). Технологии продления периода полужизни создают возможности для усовершенствованных или новых биологических препаратов, которые приводят к снижению стоимости и неудобства частого дозирования. Таким образом, сохраняется постоянная потребность в новых реагентах и способах, которые могут приводить к продлению периодов полужизни терапевтических средств на основе белков и пептидов.
В данном документе предложены новые слитые белки на основе альбумин-связывающего домена (ABD), пригодные для продления периодов полужизни биологических препаратов (например, интерлейкинов и антител). Сывороточный альбумин имеет длительный период полужизни в диапазоне 2-4 недель вследствие рециркуляции посредством неонатального Fc-рецептора (FcRn). Альбумин поглощается эндотелиальными клетками в результате макропиноцитоза и связывается с FcRn pH-зависимым образом в кислой среде ранней эндосомы. Связывание альбумина и FcRn способствует отклонению молекул альбумина от разрушения в лизосомальном компартменте и перенаправляет молекулы альбумина к плазматической мембране, где они высвобождаются обратно в плазму крови благодаря нейтральному значению pH.
Альбумин-связывающие домены (ABD), описанные в данном документе, не конкурируют с FcRN за связывание альбумина и связывают альбумин в диапазоне значений рН, который позволяет ABD также подвергаться рециркуляции эндосомального альбумина под действием FcRn при связывании с альбумином. Таким образом, биологические препараты, которые содержит рассматриваемый альбумин-связывающий домен (ABD), способны избегать лизосомального разрушения в результате пути с участием альбумина и FcRn и, следовательно, проявляют более длительные периоды полужизни в сыворотке, чем аналоги, не имеющие ABD.
Кроме того, такие ABD-содержащие терапевтические средства предпочтительно локализуются в опухолях, которые, как известно, содержат высокие уровни сывороточного альбумина. Таким образом, такие ABD-содержащие терапевтические средства особенно пригодны для лечения видов рака.
B. Альбумин-связывающие домены
В одном аспекте в данном документе предложены композиции, которые содержат альбумин-связывающий домен. Используемый в данном документе термин «сывороточный альбумин» относится к члену семейства глобулярных белков, продуцируемых печенью, который функционирует главным образом в качестве белка-переносчика стероидов, жирных кислот и гормонов щитовидной железы в крови. Сывороточный альбумин также играет основную роль в стабилизации объема внеклеточной жидкости, в результате участия в поддержании онкотического давления плазмы крови, и включает, но не ограничиваясь ими, человеческий сывороточный альбумин (HSA, номера доступа в Genbank: NM_000477 и NP_000468) и мышиный сывороточный альбумин (MSA, номера доступа в Genbank: NM_009654 и NP_0033784). Структура альбумина характеризуется несколькими длинными α-спиралями и содержит одиннадцать различных связывающих доменов для гидрофобных соединений. У людей сывороточный альбумин кодируется геном ALB.
Слитые белки, которые включают в себя рассматриваемый альбумин-связывающий домен (ABD), могут связывать сывороточный альбумин (SA), что способствует поглощению слитого белка клетками в результате макропиноцитоза. В определенных вариантах осуществления ABD, описанные в данном документе, связываются в диапазоне значений рН от приблизительно 5,5 до приблизительно 7,2. В ранней эндосоме такие связанные SA слитые белки на основе ABD связываются с FcRn с помощью SA при кислом значении pH (например, pH 5,5), что, в свою очередь, отклоняет связанный SA слитый белок с ABD от лизосомного компартмента клетки и направляет обратно к плазматической мембране. В области плазматической мембраны SA диссоциирует от FcRn вследствие нейтрального значения pH (например, значения pH 7,1-7,5) и SA и слитый белок с ABD высвобождаются обратно в кровоток. Поскольку терапевтические средства, которые содержат рассматриваемый ABD, способны связываться с альбумином в диапазоне значений рН от приблизительно 5,5 до приблизительно 7,2, такие терапевтические средства также предпочтительно подвергаются рециркуляции эндосомального альбумина под действием FcRn и, таким образом, избегают лизосомального расщепления. Соответственно, терапевтические средства, которые содержат такие ABD, предпочтительно демонстрируют более длительные периоды полужизни в сыворотке крови, чем аналоги, не имеющие ABD. Такие терапевтические средства особенно пригодны для лечения видов рака, которые, как известно, содержат высокие уровни сывороточного альбумина.
В некоторых вариантах осуществления альбумин-связывающий домен связывает альбумин в сайте, который не препятствует связыванию SA с неонатальным Fc-рецептором (FcRn). Используемый в данном документе термин «FcRn» или «неонатальный Fc-рецептор» означает белок, который связывает Fc-область антитела IgG и кодируется, по меньшей мере, отчасти, геном FcRn. FcRn может происходить из любого организма, в том числе, но не ограничиваясь ими, людей, мышей, крыс, кроликов и обезьян. Как известно в данной области техники, функциональный белок FcRn включает в себя два слитых белка, часто обозначаемых тяжелой цепью и легкой цепью. Легкая цепь представляет собой бета-2-микроглобулин, а тяжелая цепь кодируется геном FcRn. Если в данном документе не указано иное, FcRn или белок FcRn относится к комплексу тяжелой цепи FcRn с бета-2-микроглобулином.
В некоторых вариантах осуществления альбумин-связывающий домен, описанный или приведенный в качестве примера в данном документе, предпочтительно специфически связывается с сывороточным альбумином (например, HSA) в эпитопе в молекуле сывороточного альбумина, который не участвует во взаимодействии молекулы сывороточного альбумина с FcRn. Связывание SA-связывающего фрагмента с молекулой сывороточного альбумина, таким образом, предпочтительно по сути не нарушает, не ингибирует, не предупреждает или иным образом не снижает связывание молекулы сывороточного альбумина (например, HSA) с FcRn. Предпочтительно альбумин-связывающий домен не конкурирует с FcRn за связывание с молекулой сывороточного альбумина. Предпочтительно альбумин-связывающий домен стерически не ингибирует связывание сывороточного альбумина с FcRn. Предпочтительно, SA-связывающий фрагмент не изменяет конформацию молекулы сывороточного альбумина таким образом, что альбумин не может взаимодействовать с FcRn.
В некоторых вариантах осуществления альбумин-связывающий домен связывает SA (например, HSA) при значении pH 5,0 ± 0,1, 0,2, 0,3, 0,4, 0,5, 0,6, 0,7, 0,8, 0,9, 1,0, 1,1, 1,2, 1,3, 1,4, 1,5, 1,6, 1,7, 1,8, 1,9, 2,0, 2,1, 2,2, 2,3, 2,4, 2,5, 2,6, 2,7, 2,8, 2,9 или 3,0. В некоторых вариантах осуществления альбумин-связывающий домен связывает SA в диапазоне значений рН от приблизительно 5,5 до приблизительно 7,2. В некоторых вариантах осуществления SA-связывающий фрагмент связывает SA при значении pH 5,5.
В некоторых вариантах осуществления альбумин-связывающий домен представляет собой домен, связывающий человеческий сывороточный альбумин (HSA). HSA-связывающие домены включают в себя, но не ограничиваясь ими, альбумин-связывающие домены, которые могут связываться с молекулой HSA, такой как целая молекула HSA или фрагмент HSA. В некоторых вариантах осуществления HSA-связывающий домен также связывает мышиный сывороточный альбумин. В некоторых вариантах осуществления HSA-связывающий домен также связывает альбумин яванского макака. В некоторых вариантах осуществления HSA-связывающий домен не связывается с бычьим сывороточным альбумином (BSA).
Альбумин-связывающие домены, предложенные в данном документе, могут содержать только вариабельную область тяжелой цепи или вариабельную область тяжелой цепи в сочетании с вариабельной областью легкой цепи. В некоторых вариантах осуществления альбумин-связывающий домен содержит вариабельную область тяжелой цепи. В некоторых вариантах осуществления вариабельная область тяжелой цепи содержит vhCDR1, vhCDR2 и vhCDR3 (области, определяющие комплементарность, вариабельной области тяжелой цепи 1-3). В определенных вариантах осуществления антигенсвязывающий домен также содержит вариабельную область легкой цепи. В некоторых вариантах осуществления вариабельная область легкой цепи содержит vlCDR1, vlCDR2 и vlCDR3 (области, определяющие комплементарность, вариабельной области легкой цепи 1-3).
В некоторых вариантах осуществления альбумин-связывающий домен содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей, продемонстрированных на фиг. 2. В некоторых вариантах осуществления альбумин-связывающий домен содержит vhCDR1, vhCDR2 и vhCDR3 вариабельной области тяжелой цепи A10m3, как продемонстрировано на фиг. 2D. В некоторых вариантах осуществления альбумин-связывающий домен содержит vhCDR1, vhCDR2 и vhCDR3 A10m3, как продемонстрировано на фиг. 2D.
В некоторых вариантах осуществления альбумин-связывающий домен также содержит вариабельную область легкой цепи. В иллюстративном варианте осуществления альбумин-связывающий домен содержит вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 любой из вариабельных областей легких цепей, как продемонстрировано на фиг. 2. В некоторых вариантах осуществления альбумин-связывающий домен содержит vlCDR1, vlCDR2 и vlCDR3 вариабельной области легкой цепи A10m3, как продемонстрировано на фиг. 2D. В некоторых вариантах осуществления альбумин-связывающий домен содержит vlCDR1, vlCDR2 и vlCDR3 A10m3, как продемонстрировано на фиг. 2D.
В некоторых вариантах осуществления альбумин-связывающий домен (например, HSA-связывающий домен) представляет собой антитело или фрагмент антитела. В некоторых вариантах осуществления альбумин-связывающий домен (например, HSA-связывающий домен) представляет собой scFv.
В некоторых вариантах осуществления, в которых ABD содержит как вариабельную область тяжелой цепи, так и вариабельную область легкой цепи, вариабельная область тяжелой цепи и вариабельная область легкой цепи присоединяются друг к другу с помощью линкера (например, линкера scFv). В некоторых вариантах осуществления линкер присоединен к вариабельной области тяжелой цепи на своем С-конце и к вариабельной области легкой цепи на своем N-конце. Подходящие линкеры описаны в данном документе и на фиг. 48. В некоторых вариантах осуществления линкер представляет собой линкер (Gly4Ser)x, где x представляет собой 1, 2, 3, 4, 5, 6, 7 или 8. В некоторых вариантах осуществления линкер представляет собой линкер (Gly4Ser)5.
В некоторых вариантах осуществления альбумин-связывающий домен также содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 A10m3, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vlCDR3 A10m3 (фиг. 2D). В одном варианте осуществления альбумин-связывающий домен содержит последовательность вариабельной области тяжелой цепи и последовательность вариабельной области легкой цепи ABD A10m3, продемонстрированные на фиг. 2D.
C. Варианты интерлейкина-15
В другом аспекте в данном документе предложены композиции, которые содержит вариантные IL-15 с повышенной стабильностью и/или биологической активностью in vivo по сравнению с IL-15 дикого типа.
Все используемые в данном документе термины «интерлейкин 15», «IL-15» и «IL15» относятся к интерлейкину, который связывается и передает сигналы посредством комплекса, состоящего из альфа-цепи специфического в отношении IL-15 рецептора, бета-цепи рецептора IL-2/IL-15 (CD122) и общей гамма-цепи (гамма-C, CD132) (номера доступа в Genbank: NM_00000585 и NP_000576 (человек); и NM_001254747 и NP_001241676 (мышь)).
Было показано, что IL-15 стимулирует пролиферацию Т-клеток внутри опухолей (см., например, Miecnik et al., Sci Transl Med 6 (228):228ra37 (2014)). IL-15 также способен приводить к продлению выживаемости эффекторных CD8+ T-клеток памяти и имеет определяющее значение для развития NK-клеток. Считается, что IL-15 может усиливать действие ингибиторов контрольных точек и других иммунотерапевтических средств, которые используют Т-клетки для атаки раковых клеток. Таким образом, не связываясь каким-либо конкретным принципом действия, считается, что IL-15, описанные в данном документе, пригодны для лечения видов рака.
В то же время мономер IL-15 имеет короткий период полужизни, составляющий менее 40 минут in vivo. Модификации мономера IL-15 могут приводить к улучшению его фармакокинетики in vivo при лечении видов рака. Эти модификации в основном были направлены на улучшение транс-презентации IL-15 альфа-субъединицей рецептора IL-15, IL-15Rα. Такие модификации включают в себя: 1) предассоциацию слияния IL-15 и слияния субъединицы его растворимого альфа-рецептора с Fc с образованием комплекса IL-15:IL-15Rα-Fc (см., например, Rubinstein et al., Proc Natl Acad Sci USA 103:9166–71 (2006)); 2) экспрессию гиперагонистов белка IL-15-sIL-15Rα-sushi (см., например, Bessard et al., Molecular Cancer Therapeutics 8: 2736-45 (2009)); и 3) предассоциацию мутантной формы IL-15 человека IL-15N72D с комплексом слияния IL-15Rα-Fc sushi-Fc (см., например, Zhu et al., Journal of Immunology 183:3598-6007 (2009)).
В некоторых вариантах осуществления IL-15 представляет собой вариант исходного IL-15 с повышенной стабильностью по сравнению с IL-15 дикого типа. В конкретных вариантах осуществления вариантный IL-15 представляет собой вариант IL-15 человека дикого типа. В иллюстративном варианте осуществления вариантный IL-15 содержит аминокислотную замену в положении K86 исходного IL-15, продемонстрированную на фиг. 3. Описанный в данном документе K86 представляет собой предполагаемый сайт убиквитин-зависимого расщепления (см. Пример 2) при использовании с определенными типами клеток (например, Т-клеток HEK293). Таким образом, не связываясь каким-либо конкретным принципом действия, считается, что удаление сайта убиквитинирования K86 в результате аминокислотного замещения повышает стабильность IL-15 (см. Примеры 2 и 3).
В определенных вариантах осуществления IL-15 представляет собой вариантный IL-15, имеющий аминокислотную замену в положении N112. Положение аминокислоты N112 представляет собой основной сайт биологической активности IL-15, поскольку оно является определяющим для соответствующего взаимодействия гамма-рецептора IL-15/IL-15, особенно когда IL-15 присоединен к ABD. Таким образом, не связываясь каким-либо конкретным принципом действия, считается, что мутации в положении N112 могут приводить к усилению одной или нескольких функций IL-15, в том числе, но не ограничиваясь ими, стимулирования пролиферации Т-клеток в опухолевых микроокружениях, повышения выживаемости CD8+ T клеток и активации развития NK-клеток.
Определенные аминокислотные замены которые приводят к повышению стабильности и/или биологической активности IL-15 in vivo, включают в себя, но не ограничиваясь ими: K86A, K86R, N112A, N112S, N112Q, K86A/N112A, K86R/N112A, K86A/N112S, K86R/N112S, K86A/N112Q, K86R/N112Q, K86A/N112A/N79A, K86R/N112A/N79A, K86A/N112A/N79D, K86R/N112A/N79D, K86A/N112A/N79Q, K86R/N112A/N79Q, K86A/N112A/N71D, K86R/N112A/N71D, K86A/N112A/N71Q, K86R/N112A/N71Q, K86A/N112A/N71D/N79A, K86A/N112A/N71D/N79D, K86A/N112A/N71Q/N79A, K86A/N112A/N71Q/N79D, K86R/N112A/N71D/N79A, K86R/N112A/N71D/N79D, K86R/N112A/N71D/N79Q, K86R/N112A/N71Q/N79A, K86R/N112A/N71Q/N79D и K86R/N112A/N71Q/N79Q. Иллюстративные вариантные IL-15, которые содержит одну или несколько таких аминокислотных замен, продемонстрированы на фиг. 3. В иллюстративном варианте осуществления вариантный IL-15 содержит аминокислотные замены K86A и N112A.
В одном варианте осуществления IL-15, описанный в данном документе (IL-15 дикого типа и вариантные IL-15), присоединен к альфа-IL-15R. Было показано, что такой IL-15, представленный в транс-положении с его рецептором, имеет длительный период полужизни и более высокую активность по сравнению с нативным Il-15 в отдельности. См., например, Wu, J Mol Genet Med 7, 85 (2013).
D. IL-12
В другом аспекте в данном документе предложены композиции, которые содержат IL-12. Все используемые в данном документе термины «интерлейкин 12», «IL-12» и «IL12» относятся к интерлейкину, который представляет собой гетеродимерный цитокин, кодируемый генами IL-12A и IL-12B (номера доступа в Genbank: NM_000882 (IL-12A) и NM_002187 (IL-12B)). IL-12 состоит из пучка четырех альфа-спиралей и участвует в дифференцировке нативных Т-клеток в TH1-клетки. IL-12 связывается с рецептором IL-12, который является гетеродимерным рецептором, образованным IL-12R-β1 и IL-12R-β2. IL-12 известен в качестве фактора, стимулирующего Т-клетки, который может стимулировать рост и функцию Т-клеток. В частности, IL-12 может стимулировать продуцирование интерферона гамма (IFN-γ) и фактора некроза опухоли-альфа (TNF-α) из Т-клеток и естественных клеток-киллеров (NK) и приводить к уменьшению опосредуемой IL-4 супрессии IFN-γ. IL-12 может дополнительно опосредовать усиление цитотоксической активности NK-клеток и CD8+ цитотоксических Т-лимфоцитов. Кроме того, IL-12 также может иметь антиангиогенную активность за счет повышения продуцирования гамма-интерферона, что, в свою очередь, приводит к повышению продуцирования белка, индуцируемого хемокином-10 (IP-10 или CXCL10). Затем IP-10 опосредует этот антиангиогенный эффект. Не связываясь каким-либо конкретным принципом действия, считают, что IL-12 в связи с его способностью индуцировать иммунные ответы и его антиангиогенной активностью может быть использован для лечения видов рака.
В некоторых вариантах осуществления IL-12 представляет собой IL-12 мыши. В других вариантах осуществления IL-12 представляет собой IL-12 человека.
В некоторых вариантах осуществления IL-12 представляет собой одноцепочечный полипептид IL-12, содержащий субъединицу p35 IL-12, присоединенную к субъединице p40 IL-12. Такие одноцепочечные полипептиды IL-12 предпочтительно сохраняют одну или несколько из биологических активностей IL-12 дикого типа. В некоторых вариантах осуществления одноцепочечный полипептид IL-12, описанный в данном документе, соответствует формуле, от N-конца к C-концу, (p40)-(L)-(p35), где «p40» представляет собой субъединицу р40 IL-12, «p35» представляет собой субъединицу p35 IL-12, а L представляет собой линкер. В других вариантах осуществления одноцепочечный IL-12 соответствует формуле, от N-конца к C-концу, (p35)-(L)-(p40). Любой подходящий линкер может быть использован в одноцепочечном полипептиде IL-12, в том числе линкеры, которые описаны в данном документе и раскрыты на фиг. 49C. Подходящие линкеры могут включать в себя, например, линкеры, имеющие аминокислотную последовательность (GGGGS)x, где x представляет собой целое число от 1 до 10. Другие подходящие линкеры содержат, например, аминокислотную последовательность GGGGGGS. Иллюстративные одноцепочечные линкеры IL-12, которые могут быть использованы с рассматриваемыми одноцепочечными полипептидами IL-12, также описаны в Lieschke et al., Nature Biotechnology 15:35-40 (1997), которая полностью включена в данный документ посредством ссылки и особенно в связи с описанием полипептидных линкеров IL-12.
В одном иллюстративном варианте осуществления одноцепочечный полипептид IL-12 представляет собой одноцепочечный полипептид IL-12 человека (т.е. он содержит субъединицу p35 и p40 IL-12 человека). В определенных вариантах осуществления одноцепочечный полипептид IL-12 представляет собой одноцепочечный полипептид IL-12 мыши. Иллюстративные одноцепочечные IL-12 мыши и человека продемонстрированы на фиг. 20 (показаны в виде слитого пептида с ABD) и 49C.
Е. Слитые белки на основе ABD
В одном аспекте в данном документе предложены композиции на основе ABD, которые содержат альбумин-связывающий домен, присоединенный к одному или нескольким партнерам по слиянию (например, первому партнеру по слиянию, второму партнеру по слиянию и т.д.) с помощью линкера. Как описано в данном документе, слитые белки на основе ABD могут подвергаться эндосомной рециркуляции, опосредованной FcRn, и, таким образом, предпочтительно демонстрируют увеличенный период полужизни по сравнению с аналогами, которые не содержат такие ABD.
ABD, пригодные для таких слитых белков на основе ABD, включают в себя, но не ограничиваясь ими, таковые, описанные в данном документе. Аминокислотные последовательности таких ABD, в том числе vhCDR1-3, vlCDR1-3, последовательности вариабельных областей тяжелых цепей и вариабельных областей легких цепей, раскрыты, например, на фиг. 2. В некоторых вариантах осуществления слитый белок с ABD содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 любой из вариабельных областей тяжелых цепей ABD на фиг. 2, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vCDR3 любой из вариабельных областей легких цепей ABD на фиг. 2. В некоторых вариантах осуществления ABD содержит вариабельную область тяжелой цепи, имеющую vhCDR1, vhCDR2 и vhCDR3 ABD, раскрытые на фиг. 2, и вариабельную область легкой цепи, имеющую vhCDR1, vhCDR2 и vhCDR3 ABD, раскрытые на фиг. 2. В некоторых вариантах осуществления слитый белок с ABD содержит вариабельную область тяжелой цепи и вариабельную область легкой цепи ABD, раскрытые на фиг. 2.
В одном иллюстративном варианте осуществления слитый белок с ABD содержит вариабельную область тяжелой цепи, которая содержит vhCDR1, vhCDR2 и vhCDR3 вариабельной области тяжелой цепи A10m3, и вариабельную область легкой цепи, которая содержит vlCDR1, vlCDR2 и vCDR3 вариабельной области легкой цепи A10m3 (фиг. 2D). В некоторых вариантах осуществления ABD содержит вариабельную область тяжелой цепи, имеющую vhCDR1, vhCDR2 и vhCDR3 A10m3, и вариабельную область легкой цепи, имеющую vhCDR1, vhCDR2 и vhCDR3 A10m3 (фиг. 2D). В некоторых вариантах осуществления слитый белок с ABD содержит вариабельную область тяжелой цепи и вариабельную область легкой цепи A10m3 (фиг. 2D).
Слитые белки на основе ABD, описанные в данном документе, содержат партнера по слиянию. В некоторых вариантах осуществления партнер по слиянию включает в себя двух партнеров по слиянию (первого партнера по слиянию (FP1) и второго партнера по слиянию (FP2). В вариантах осуществления, которые включают в себя двух партнеров по слиянию, партнеры по слиянию могут быть присоединены к ABD в нескольких ориентациях. В некоторых вариантах осуществления слитый белок с ABD соответствует формуле, от N-конца к C-концу: FP1-ABD-FP2, FP1-PF2-ABD или ABD-FP1-FP2, где FP1 представляет собой первого партнера по слиянию, а FP2 представляет собой второго партнера по слиянию.
Любой подходящий партнер по слиянию, для которого желательно продление периода полужизни партнера по слиянию, может быть включен в рассматриваемые слитые белки на основе ABD. Партнеры слияния могут включать в себя, например, цитокины (например, интерфероны и интерлейкины), факторы роста, полипептиды, белки и гормоны (например, гормоны роста, гормон паращитовидной железы).
В определенных вариантах осуществления партнер по слиянию представляет собой связывающий фрагмент на основе антитела, который содержит вариабельную область тяжелой цепи и вариабельную область легкой цепи. Такие связывающие фрагменты могут связываться с любой мишенью, представляющей интерес, в том числе, например, специфичными для опухоли мишенями или цитокинами. В иллюстративном варианте осуществления партнер слияния представляет собой одноцепочечный вариабельный фрагмент (scFv). Партнеры по слиянию на основе антител также включают в себя, но не ограничиваясь ими, ds-scFv, однодоменные антитела (sdAb), диатела, dsFv, ds-scFv, Fab и полноразмерные антитела. Партнеры по слиянию на основе антител также включают в себя мультиспецифические (например, биспецифические) антитела и фрагменты.
Рассматриваемые слитые белки на основе ABD, описанные в данном документе, используют линкеры между компонентами (ABD и партнерами по слиянию) и внутри компонентов. Например, партнеры по слиянию scFv используют стандартные пептидные линкеры, как правило, на основе глицина и серина, для присоединения вариабельных областей тяжелых и легких цепей с образованием scFv. Кроме того, стандартные пептидные линкеры используются для присоединения ABD к партнерам по слиянию (например, цитокиновым партнерам по слиянию). Кроме того, линкеры используются для присоединения компонентов определенных фрагментов, например, субъединицы p35 и p40 IL-12 и IL-15 с IL-15Rα. В некоторых вариантах осуществления линкер представляет собой линкер (Gly4Ser)x, где x представляет собой 1, 2, 3, 4, 6, 7 или 8. В конкретных вариантах осуществления ABD соединяется с партнерами по слиянию с помощью линкеров (Gly4Ser)5.
Как показано в данном документе, существует ряд подходящих линкеров, которые могут быть использованы, в том числе традиционные пептидные связи, полученные с помощью рекомбинантных методик. Линкерный пептид может преимущественно содержать следующие аминокислотные остатки: Gly, Ser, Ala или Thr. Линкерный пептид должен иметь длину, которая является достаточной для связывания двух молекул таким образом, чтобы они принимали правильную конформацию относительно друг друга, с целью того, чтобы они сохраняли необходимую активность. В одном варианте осуществления линкер состоит от приблизительно 1 до 50 аминокислот в длину, предпочтительно от приблизительно 1 до 30 аминокислот в длину. В одном варианте осуществления могут быть использованы линкеры от 1 до 20 аминокислот в длину, при этом в некоторых вариантах осуществления находят применение линкеры от приблизительно 5 до приблизительно 10 аминокислот. Пригодные линкеры включают в себя глицин-сериновые полимеры, в том числе, например, (GS)n, (GSGGS)n, (GGGGS)n и (GGGS)n, где n представляет собой целое число, из, по меньшей мере, одного (и, как правило, от 3 до 4), глицин-аланиновые полимеры, аланин-сериновые полимеры и другие гибкие линкеры. В качестве альтернативы, различные непротеиновые полимеры, в том числе, но не ограничиваясь ими, полиэтиленгликоль (ПЭГ), полипропиленгликоль, полиоксиалкилены или сополимеры полиэтиленгликоля и полипропиленгликоля, могут найти применение в качестве линкеров, т.е. могут найти применение в качестве линкеров.
Другие линкерные последовательности могут содержать любую последовательность любой длины домена CL/CH1, однако не все остатки домена CL/CH1; например, первые 5-12 аминокислотных остатков доменов CL/CH1. Линкеры могут быть получены из легкой цепи иммуноглобулинов, например Cκ или Cλ. Линкеры могут быть получены из тяжелых цепей иммуноглобулинов любого изотипа, в том числе, например, Cγ1, Cγ2, Cγ3, Cγ4, Cα1, Cα2, Cσ, Cε и Cμ. Линкерные последовательности также могут быть получены из других белков, таких как Ig-подобные белки (например, TCR, FcR, KIR), последовательности, происходящие из шарнирных областей, и другие природные последовательности из других белков.
Часто линкер является по сути невосприимчивым к внеклеточной среде. Используемый в данном документе термин «по сути невосприимчивый к внеклеточной среде» в контексте линкера означает, что не более чем приблизительно 20%, 15%, 10%, 5%, 3% или не более чем приблизительно 1% из линкеров в образце соединения конъюгата антитело-лекарственное средство расщепляются, когда соединение конъюгата антитело-лекарственное средство присутствует во внеклеточной среде (например, в плазме крови).
Является ли линкер по сути невосприимчивым к внеклеточной среде, можно определить, например, с помощью инкубации с плазмой крови соединения конъюгата антитело-лекарственное средство в течение предварительно определенного периода времени (например, 2, 4, 8, 16 или 24 часов) и затем количественного определения количества свободного лекарственного средства, присутствующего в плазме крови.
В других, не являющихся взаимоисключающими, вариантах осуществления линкер способствует клеточной интернализации. В некоторых вариантах осуществления линкер стимулирует клеточную интернализацию при конъюгировании с терапевтическим агентом (т.е. в среде фрагмента линкер-терапевтический агент соединения конъюгата антитело-лекарственное средство, как описано в данном документе). В еще одним вариантах осуществления линкер способствует клеточной интернализации при конъюгировании как с соединением ауристатина, так и слитыми белками на основе ABD по данному изобретению.
Различные иллюстративные линкеры, которые могут быть использованы с композициями и способами данного изобретения, описаны в WO 2004-010957, публикации США № 2006/0074008, публикации США № 20050238649 и публикации США № 2006/0024317 (каждое из которых включено в данный документ посредством ссылки во полном объеме и для всех целей).
Иллюстративные линкеры, которые могут быть использованы с рассматриваемыми ABD в качестве линкеров доменов, линкеров scFv, а также компонентов присоединения определенных партнеров по слиянию, дополнительно продемонстрированы на фиг. 48.
В некоторых вариантах осуществления линкер представляет собой «линкер домена», используемый для связывания любых двух доменов, как указано в данном документе, вместе (например, цитокинового партнера по слиянию (например, интерлейкина) и ABD). Несмотря на то, что может быть использован любой подходящий линкер, во многих вариантах осуществления используется глицин-сериновый полимер, в том числе, например, (GS)n, (GSGGS)n, (GGGGS)n и (GGGS)n, где n представляет собой целое число, из, по меньшей мере, одного (и, как правило, от 3 до 4 и до 5), а также любая пептидная последовательность, которая обеспечивает рекомбинантное присоединение двух доменов с достаточной длиной и гибкостью с тем, чтобы каждый домен сохранял свою биологическую функцию.
В некоторых вариантах осуществления партнер по слиянию представляет собой scFv, который содержит вариабельную область тяжелой цепи и вариабельную область легкой цепи. В таких вариантах осуществления вариабельная область тяжелой цепи ABD присоединяется к вариабельной области легкой цепи с помощью линкера scFv.
Иллюстративные слитые белки на основе ABD дополнительно описаны ниже.
1. Слитые белки цитокин-ABD
В некоторых вариантах осуществления слитый белок с ABD содержит цитокинового партнера по слиянию, т.е. относится к слитым белкам на основе цитокин-альбумин-связывающего домена (цитокин-ABD) (фиг. 1). В некоторых вариантах осуществления слитый белок цитокин-ABD содержит IL-2, IL-7, IL-12, IL-15, IL-18, IL-21, IL-27, GM-CSF или IFN-α. Рассматриваемые слитые белки цитокин-ABD пригодны для иммуномодулирующих эффектов у субъекта, в случае, если необходимы такие иммуномодулирующие эффекты (например, лечение рака или аутоиммунного заболевания). Кроме того, рассматриваемые цитокин-ABD проявляют более длительный период полужизни и улучшенные фармакокинетические свойства по сравнению с терапевтическими средствами на основе интерлейкинов в отдельности.
Любой ABD может быть использован с слитыми белками цитокин-ABD, описанными в данном документе. В некоторых вариантах осуществления ABD содержит вариабельную область тяжелой цепи ABD. В определенных вариантах осуществления ABD содержит вариабельную область легкой цепи ABD. В иллюстративных вариантах осуществления ABD представляет собой scFv, который содержит вариабельную область тяжелой цепи, присоединенную к вариабельной области легкой цепи с помощью линкера (например, любого из линкеров, раскрытых в данном документе и на фиг. 48).
В определенных вариантах осуществления вариабельная область тяжелой цепи содержит vhCDR1-3 любой из вариабельных областей тяжелых цепей ABD, описанных в данном документе, в том числе такие вариабельные области тяжелых цепей ABD, продемонстрированные на фиг. 2. В определенных вариантах осуществления вариабельная область тяжелой цепи ABD содержит vhCDR1-3 вариабельной области тяжелой цепи A10m3 (фиг. 2D). В некоторых вариантах осуществления вариабельная область тяжелой цепи ABD содержит vhCDR1-3 A10m3, как продемонстрировано на фиг. 2D. В иллюстративных вариантах осуществления вариабельная область тяжелой цепи ABD имеет аминокислотную последовательность вариабельной области тяжелой цепи A10m3.
В определенных вариантах осуществления ABD содержит вариабельную область легкой цепи ABD. В некоторых вариантах осуществления вариабельная область легкой цепи ABD содержит vlCDR1-3 любой из вариабельных областей легких цепей ABD, описанных в данном документе, в том числе такие вариабельные области легких цепей ABD, продемонстрированные на фиг. 2. В определенных вариантах осуществления вариабельная область легкой цепи ABD содержит vlCDR1-3 вариабельной области легкой цепи A10m3 (фиг. 2D). В некоторых вариантах осуществления вариабельная область легкой цепи ABD содержит vlCDR1-3 A10m3, как продемонстрировано на фиг. 2D. В иллюстративных вариантах осуществления вариабельная область легкой цепи ABD имеет последовательность вариабельной области легкой цепи A10m3.
Аминокислотные последовательности иллюстративных цитокинов, которые могут быть использованы в рассматриваемых слияниях цитокин-ABD, а также иллюстративных слитых белках цитокин-ABD, где ABD представляет собой A10m3, продемонстрированы на фиг. 49A-G.
В некоторых вариантах осуществления IL-ABD соответствует формуле, от N-конца к C-концу, цитокин-L-ABD или ABD-L-цитокин, где L представляет собой линкер, который присоединяет цитокин к ABD (например, пептидный линкер). В некоторых вариантах осуществления цитокин-ABD содержит вариабельную область тяжелой цепи, и цитокин присоединен к N-концу вариабельной области тяжелой цепи ABD. В некоторых вариантах осуществления этот цитокин присоединен к C-концу вариабельной области тяжелой цепи ABD. В иллюстративном варианте осуществления интерлейкин-ABD содержит ABD, который также содержит вариабельную область легкой цепи (например, scFv ABD). В некоторых вариантах осуществления этот цитокин присоединен к С-концу вариабельной области легкой цепи. В определенном варианте осуществления N-конец цитокина присоединен к ABD. В других вариантах осуществления C-конец цитокина присоединен к ABD.
В определенных вариантах осуществления цитокин представляет собой цитокин, выбранный из IL-2, IL-7, IL-12, IL-15, IL-18, IL-21, IL-27, GM-CSF и IFN-α. Молекулы цитокинов включают в себя, например, полноразмерные цитокины и фрагменты цитокинов, например, часть, которая важна для функции определенного цитокина (например, часть интерлейкина, которая связывается с его рецептором).
В некоторых вариантах осуществления цитокин-ABD содержит молекулу IL-2 или ее фрагмент. Все используемые в данном документе термины «интерлейкин 2», «IL-2» и «IL2» относятся к члену семейства цитокинов, имеющему четыре альфа-спиральных пучка, и передающему сигналы посредством рецептора IL-2 (номера доступа в Genbank: NM_000586 и NP_000577 (человек) и NM_008366 и NP_032392 (мышь)). IL-2 играет определяющие роли в функционировании иммунной системы, переносимости и иммунитете, главным образом в результате его непосредственных эффектов на Т-клетки. В тимусе IL-2 предупреждает аутоиммунные заболевания, способствуя дифференцировке определенных незрелых Т-клеток в регуляторные Т-клетки, которые уничтожают другие Т-клетки, которые примированы для атаки нормальных здоровых клеток в организме. IL-2 использовали для лечения видов рака (злокачественной меланомы, почечно-клеточного рака) в больших дозах с интервальным введением и широко использовали в дозах с непрерывным введением. Иллюстративные IL-2-ABD продемонстрированы на фиг. 49A.
В некоторых вариантах осуществления слитый белок цитокин-ABD содержит молекулу IL-7 или ее фрагмент. Все используемые в данном документе термины «интерлейкин 7», «IL-7» и «IL7» (номера доступа в Genbank: NM_000880 и NP_000871 (человек); и NM_008371 и NP_032397 (мышь)) относятся к члену семейства цитокинов, который представляет собой гематопоэтический фактор роста, секретируемый стромальными клетками в костном мозге и тимусе, и связывается с рецептором IL-7. Интерлейкин-7 (IL-7) представляет собой цитокин, происходящий из не относящихся к гематопоэтическим клеток, при этом имеет центральную роль в адаптивной иммунной системе. Он способствует развитию лимфоцитов в тимусе, поддерживает гомеостаз вследствие выживаемости наивных Т-клеток и Т-клеток памяти на периферии. Кроме того, он играет важную роль для органогенеза лимфатических узлов (LN) и для поддержания активированных Т-клеток, подвергшихся рекрутингу во вторичные лимфоидные органы (SLO). IL-7 представляет собой идеальное решение для восстановления пациентов с ослабленной иммунной системой, имеющих рак, в результате экспансии периферических Т-клеток. На животных моделях было доказано, что IL-7 способствует продлению выживаемости опухоль-несущих хозяев. Иллюстративные IL-7-ABD продемонстрированы на фиг. 49B.
В определенных вариантах осуществления цитокин-ABD содержит молекулу IL-12 или ее фрагмент. В некоторых вариантах осуществления IL-12 представляет собой одноцепочечный IL-12, описанный в данном документе, и продемонстрированный на фиг. 20 (показано в виде слитого пептида с ABD) и 49C.
В определенных вариантах осуществления цитокин-ABD содержит молекулу IL-15 или ее фрагмент. В некоторых вариантах осуществления IL-15-ABD содержит вариантный IL-15, как описано в данном документе и на фиг. 3 и 4. В некоторых вариантах осуществления IL-15 представляет собой IL-15 дикого типа, как продемонстрировано на фиг. 3 (например, «исходный IL-15»). В некоторых вариантах осуществления IL-15 дикого типа присоединен к альфа-рецептору IL-15 (IL-15Rα).
В определенных вариантах осуществления цитокин-ABD содержит молекулу IL-21 или ее фрагмент. Все используемые в данном документе термины «интерлейкин 21», «IL-21» и «IL21» (номера доступа в Genbank: NM_001207006 и NP_001193935 (человек) и NM_0001291041 и NP_001277970 (мышь)) относятся к члену семейства цитокинов, который связывается с рецептором IL-21 и оказывает сильные регуляторные эффекты на клетки иммунной системы, в том числе естественные клетки-киллеры (NK) и цитотоксические клетки, и связывается с рецептором IL-21, который может разрушать инфицированные вирусом или раковые клетки. Таким образом, не связываясь каким-либо конкретным принципом действия, считается, что рассматриваемые цитокин-ABD, имеющие IL-21, пригодны для лечения различных видов рака. Иллюстративные IL-21-ABD продемонстрированы на фиг. 49E.
Другие пригодные цитокины, которые могут быть включены в рассматриваемые цитокин-ABD, включают в себя, но не ограничиваясь ими, IL-27, IFN-α и GM-CSF. В определенных вариантах осуществления полипептид интерлейкин-ABD также содержит молекулу интерферона-альфа, интерферона-бета или GM-CSF. Такие молекулы являются пригодными для доставки в опухоли и снижения токсичности.
В некоторых вариантах осуществления цитокин-ABD содержит два цитокина, присоединенных к ABD, выбранные из следующего: IL-2 и IL-12; IL-7 и IL-15; IL-15 и IL-12; IL-18 и GMC SF; IL-21 и IL-15; GMC-SF и IL-12; GMC-SF и IL-21; и IFN-α и IL-15. На фиг. 1 продемонстрированы несколько иллюстративных ориентаций, в которых два цитокина могут быть присоединены к ABD. Любой ABD может быть использован в таком слитом белке, в том числе те, которые содержат любые вариабельные домены тяжелый цепей ABD и вариабельные домены легких цепей, продемонстрированные на фиг. 2. В некоторых вариантах осуществления ABD представляет собой A10m3.
Любой линкер может быть использован для присоединения каждого цитокина к ABD, в том числе любой линкер, продемонстрированный на фиг. 48. Иллюстративная последовательность остова линкер-A10m3-линкер для использования в таких слитых белках продемонстрирована на фиг. 50. Иллюстративные линкеры, которые могут быть использованы, включают в себя линкеры (GGGGS)х, где Х представляет собой от 1 до 10. В определенных вариантах осуществления (GGGGS)5 используется для присоединения цитокина к ABD.
В некоторых вариантах осуществления цитокин-ABD соответствует формуле, выбранной из следующего (от N-конца к C-концу): цитокин 1-L1-ABD-L2-цитокин 2; цитокин 1-L1-цитокин 2-L2-ABD; и ABD-L1-цитокин 1-L2-цитокин 2, где L1 и L2 представляют собой линкеры, которые связывают цитокины и компоненты ABD, а цитокин 1 и цитокин 2 выбирают из следующих пар цитокинов: IL-2 и IL-12; IL-7 и IL-15;IL-15 и IL-12; IL-18 и GM-CSF; IL-21 и IL-15; GM-CSF и IL-12; GM-CSF и IL-21; и IFN-α и IL-15. В некоторых вариантах осуществления ABD представляет собой A10m3 (фиг. 2D).
В таких цитокин-ABD любой цитокин может представлять собой «цитокин 1», а другой цитокин может представлять собой «цитокин 2». Например, в одном варианте осуществления пары цитокинов IL-2 и IL-12 IL-2 представляет собой «цитокин 1», а IL-12 представляет собой «цитокин 2». В другом варианте осуществления пары цитокинов IL-2 и IL-12. IL-12 представляет собой «цитокин 1», а IL-2 представляет собой «цитокин 2».
В некоторых вариантах осуществления цитокин-ABD содержит два одинаковых интерлейкина. В других вариантах осуществления полипептид интерлейкин-ABD содержит два разных интерлейкина (например, IL-12 и IL-15), описанные выше. Слитые белки на основе ABD, которые включают в себя определенные комбинации цитокинов, раскрыты на фиг. 51. Иллюстративные слитые белки цитокин-ABD, которые включают в себя комбинацию различных цитокинов, дополнительно описаны подробно ниже.
А. IL-12 и IL-15
В определенных вариантах осуществления слитый белок цитокин-ABD содержит IL-12 и IL-15. Иллюстративные последовательности IL-12 и IL-15, которые могут быть включены в такие варианты осуществления, продемонстрированы на фиг. 3 и 49C. Считается, что цитокин-ABD, которые включают как партнера по слиянию IL-12, так и партнера по слиянию IL-15, проявляют противоопухолевые эффекты обоих интерлейкинов. Было показано, что комбинация IL-12 и IL-15 индуцирует повышенную противоопухолевую активность по сравнению с каким-либо цитокином в отдельности. Такое усиление противоопухолевой активности коррелировало с обратной активацией каждого цитокинового рецептора посредством синергетической индукции IFN-γ. Кроме того, было показано, что IL-12 в сочетании с IL-15 стимулирует противоопухолевую активность в перитонеальных макрофагах посредством синтеза оксида азота. Не связываясь каким-либо конкретным принципом действия, считают, что полипептиды, имеющие как партнера по слиянию IL-12, так и партнера по слиянию IL-15, способны быстро активировать врожденный ответ (IL-12), а также эффективно стимулировать пролиферацию T-клеток и поддерживают CD8+ Т-клетки памяти (IL-15). Исследования на животных показывают, что последовательная доставка клеток, экспрессирующих IL-12 и IL-15, также приводила к излечению мышей в терапевтических условиях развившейся опухоли. Истощение CD8+ клеток приводило к устранению защиты указанной терапии, что предполагает клональную экспансию CTL опухоли. См., например, Croce et al., Clin Cancer Res 11(2 Pt 1):735-42 (2005).
В некоторых вариантах осуществления цитокин-ABD соответствует формуле, от N-конца к C-концу:
а) (IL-12)-L1-(ABD)-L2-(IL-15); или же
b) (IL-15)-L1-(ABD)-L2-(IL-12).
Может быть использован любой подходящий ABD, в том числе, например, ABD, имеющий вариабельную область тяжелой цепь ABD, которая содержит vhCDR1-3 любой из вариабельных областей тяжелых цепей ABD, приведенных на фиг. 2. В иллюстративном варианте осуществления вариабельная область тяжелой цепи содержит vhCDR1-3 вариабельной области тяжелой цепи A10m3 (фиг. 2D). В определенных вариантах осуществления ABD содержит вариабельную область легкой цепи ABD, которая содержит vlCDR1-3 вариабельной области легкой цепи A10m3. В иллюстративном варианте осуществления ABD представляет собой scFv A10m3.
Могут быть использованы любые подходящие IL-12 и IL-15. В некоторых вариантах осуществления IL-15 представляет собой вариантный IL-15, описанный в данном документе (см., например, фиг. 3). IL-15 также может представлять собой IL-15 дикого типа или IL-15 дикого типа, который присоединен к IL-15R. В одном варианте осуществления IL-15 представляет собой вариантный IL-15, выбранный из тех, которые продемонстрированы на фиг. 3. В иллюстративном варианте осуществления Il-15 представляет собой вариантный IL-15, имеющий аминокислотные замены K86R и N112A.
IL-12, которые могут быть использованы, включают в себя те IL-12, которые имеют домен p35 и p40. В некоторых вариантах осуществления IL-12 представляет собой одноцепочечный IL-12, описанный в данном документе (см., например, фиг. 20, как показано в виде части слитого полипептида на основе IL-12-ABD, и фиг. 49C).
В таких вариантах осуществления L1 и L2 представляют собой первый и второй линкер соответственно, L1 и L2 могут представлять собой любой линкер, который является подходящим для присоединения доменов IL-15 и IL-12 к домену ABD (например, линкеры, приведенные на фиг. 48). Иллюстративные линкеры, которые могут быть использованы, включают в себя линкеры (GGGGS)х, где Х представляет собой от 1 до 10. В определенных вариантах осуществления каждый из L1 и L2 представляет собой (GGGGS)5.
Последовательности иллюстративных полипептидов ABD, которые включают в себя IL-12 и IL-15, продемонстрированы на фиг. 34 и 36.
В. IL-2 и IL-12
В определенных вариантах осуществления цитокин-ABD содержит IL-2 и IL-12.
IL-2 и IL-12 взаимно активируют рецепторы друг друга и используют отдельные сигнальные пути для индукции различных, но комплементарных биологических эффектов. Как IL-2, так и IL-12 могут стимулировать митоген- или CD3-активированные Т-клетки с целью пролиферации и продуцирования IFN-γ. Кроме того, исследования показывают, что доставка генов как IL-2, так и IL-12 мышам, несущим меланому B16, вызывала значительное снижение опухолевой нагрузки и повышение общей выживаемости (см., например, Dietrich et al., Arch Surg 387(34):177-82 (2002)). Таким образом, считается, что слитые белки цитокин-ABD, имеющие как IL-2, так и IL-12, являются пригодными для уменьшения опухолей и лечения видов рака.
С. IL-2 и IL-15
В определенных вариантах осуществления слитый белок цитокин-ABD содержит IL-2 и IL-15. Как IL-2, так и IL-15 способны стимулировать пролиферацию NK-клеток и активированных Т-клеток, а также поддерживать экспансию эффекторных Т-клеток. Считается, что слитые белки цитокин-ABD, которые содержат как IL-2, так и IL-15, являются пригодными для уменьшения опухолей и лечения видов рака.
D. IL-7 и IL-12
В определенных вариантах осуществления слитый белок цитокин-ABD содержит партнера по слиянию IL-7 и партнера по слиянию IL-12.
Интерлейкин-7 (IL-7) представляет собой цитокин, происходящий из не относящихся к гематопоэтическим клеток, при этом имеет центральную роль в адаптивной иммунной системе. Он способствует развитию лимфоцитов в тимусе, поддерживает гомеостаз вследствие выживаемости наивных Т-клеток и Т-клеток памяти на периферии. Кроме того, он играет важную роль для органогенеза лимфатических узлов (LN) и для поддержания активированных Т-клеток, подвергшихся рекрутингу во вторичные лимфоидные органы (SLO). Иммунная активность пациентов, больных раком, подавлена и характеризуется меньшим количеством Т-клеток, меньшей инфильтрацией эффекторных иммунных клеток, более высокими уровнями истощенных эффекторных клеток и более высокими уровнями иммуносупрессорных цитокинов, таких как трансформирующий фактор роста β (TGF-β). IL-7 представляет собой идеальное решение для восстановления пациентов с ослабленной иммунной системой, имеющих рак, в результате экспансии периферических Т-клеток. На животных моделях было доказано, что IL-7 способствует продлению выживаемости опухоль-несущих хозяев. См. Gao et al., Int. J. Mol. Sci. 16: 10267-10280 (2015).
IL-12 действует непосредственно на CD8+ T-клетки с целью усиления их пролиферации, опосредованной IL-7. Считают, что слитые белки цитокин-ABD, которые содержат IL-7- и IL-12-связывающий домен, предпочтительно способствуют пролиферации CD8+ T-клеток и усиливают цитолитическую активность против опухолей.
IL-7 и IL-15
В определенных вариантах осуществления слитый белок цитокин-ABD содержит IL-7 и IL-15. Как упомянуто выше, интерлейкины 7 и 15 считаются сильными провоспалительными цитокинами, которые способны уменьшать опухолевый процесс. Считают, что слитые белки цитокин-ABD, которые содержат себя как IL-7, так и IL-15, являются пригодными для уменьшения опухолей и лечения видов рака.
E. IL-12 и IL-21
В определенных вариантах осуществления цитокин-ABD содержит IL-12 и IL-21.
Как упомянуто выше, IL-12 способен стимулировать пролиферацию NK-клеток и активированных Т-клеток и поддерживать экспансию эффекторных Т-клеток. IL-21 представляет собой регулятор функции NK- и T-клеток, который связывает врожденную и адаптивную иммунные системы. IL-21 способствует созреванию NK-клеток из предшественников костного мозга, активирует периферические NK-клетки человека, способствует экспансии и созреванию NK и усиливает опосредуемые CD8+ T-клетками эффекторные функции.
Считается, что слитые белки цитокин-ABD, которые содержат как IL-12, так и IL-21, являются пригодными для уменьшения опухолей и лечения видов рака.
F. IL-12 и IL-18
В определенных вариантах осуществления цитокин-ABD содержит IL-12 и IL-18.
Известно, что IL-18 индуцирует продуцирование IFN-γ, способствует развитию Th1-клеток и активации NK. Известно, что IL-12 индуцирует активацию рецептора IL-18 зависимым от IFN-γ образом. Введение клеток карциномы молочной железы мышей SCK, коэкспрессирующих IL-18 и IL-12, мышам способствовало уменьшению опухолевой нагрузки и ингибированию ангиогенеза (см., например, Coughlin et al., J Clin Invest 101 (6):1441-1452 (1998)). Введение IL-18 в комбинации с IL-12 мышам, несущим опухоль, синергически индуцировало продление уровня IFN-γ в сыворотке крови, тогда как мыши, несущие опухоль, обработанные IL-18 или IL-12 в отдельности, индуцировали минимальный уровень IFN-γ в сыворотке крови, который быстро снижался (см., например, Subleski et al., Cancer Res 66 (22):11005-1012 (2006)). Иллюстративные IL-18-ABD продемонстрированы на фиг. 37.
Считается, что слитые белки цитокин-ABD, которые содержат как партнера по слиянию IL-12, так и партнера по слиянию IL-21, являются пригодными для уменьшения опухолей и лечения видов рака.
G. GM-CSF и IL-12
В определенных вариантах осуществления цитокин-ABD содержит GM-CSF и IL-18. GM-CSF регулирует дифференцировку и пролиферацию гемопоэтических клеток-предшественников. GM-CSF также приводит к усилению способности APC к процессингу и презентации антигена, что, в свою очередь, приводит к активации цитотоксических Т-клеток, увеличению продуцирования IFN-γ и, в конечном итоге, к регрессии опухоли. Как GM-CSF, так и IL-12 способны вызывать значительные противоопухолевые ответы в нескольких различных доклинических моделях опухолей, в том числе модели опухоли печени и модели опухоли легкого. (См., например, Kilinc et al., J. Immunol. 177(10):6962-973 (2006)).
Считается, что слитые белки цитокин-ABD, которые содержат как партнера по слиянию GM-CSF, так и партнера по слиянию IL-12, являются пригодными для уменьшения опухолей и лечения видов рака.
H. IFN-альфа и IL-12
Совместная природа этих двух цитокинов выходит за рамки простого сходных биологических эффектов. Например, IL-12, который, как хорошо известно, индуцирует продуцирование IFN-γ, может приводить к продуцированию дополнительных растворимых факторов, которые усиливают передачу сигнала с участием IFN-α. Известно, что интерфероны, в том числе интерферон альфа, вызывают апоптоз в злокачественных клетках. См., например, Thyrell, L. et al., Oncogene 21, 1251-262 (2002).
Считается, что слитые белки цитокин-ABD, которые содержат как IFN альфа, так и IL-12, являются пригодными для уменьшения опухолей и лечения видов рака.
Дополнительные комбинации цитокин-цитокин, которые могут быть включены в рассматриваемые слитые белки цитокин-ABD, продемонстрированы на фиг. 51А.
2. Слитые белки связывающий фрагмент-ABD
В некоторых вариантах осуществления слитые белки на основе ABD содержат связывающий фрагмент (например, scFv), т.е. слитый белок BM-ABD. В некоторых вариантах осуществления BM-ABD содержит один связывающий фрагмент. В некоторых вариантах осуществления BM-ABD содержит два связывающих фрагмента (например, scFv). В других вариантах осуществления BM-ABD содержит цитокин и связывающий фрагмент (фиг. 1B). На фиг. 1 продемонстрировано несколько иллюстративных ориентаций, в которых комбинации связывающего фрагмента или связывающий фрагмент-цитокин/связывающий фрагмент-связывающий фрагмент могут быть присоединены к ABD.
Связывающие фрагменты, которые являются пригодными для практической реализации с рассматриваемыми BM-ABD-связывающими фрагментами, основаны на вариабельном домене тяжелой цепи и вариабельном домене легкой цепи антител. В некоторых вариантах осуществления связывающий фрагмент содержит вариабельный домен тяжелой цепи и вариабельный домен легкой цепи. В некоторых вариантах осуществления связывающий фрагмент представляет собой одноцепочечный вариабельный фрагмент (scFv).
Любой ABD может быть использован в таком слитом белке, в том числе те, которые содержат любые вариабельные домены тяжелый цепей ABD и вариабельные домены легких цепей, продемонстрированные на фиг. 2. В определенных вариантах осуществления ABD содержит вариабельные домены тяжелых и легких цепей A10m3. В некоторых вариантах осуществления ABD представляет собой scFv A10m3.
Любой линкер может быть использован для присоединения цитокина и/или связывающего фрагмента к ABD, в том числе любой линкер, продемонстрированный на фиг. 48. В иллюстративном варианте осуществления линкер представляет собой (GGGGS)5. Иллюстративная последовательность остова линкер-A10m3-линкер для использования в таких слитых белках продемонстрирована на фиг. 48.
Иллюстративные комбинации двух связывающих фрагментов (например, scFv) или цитокина и связывающего фрагмента, которые могут быть представлять собой такие слитые белки на основе ABD, продемонстрированы на фиг. 51B.
В некоторых вариантах осуществления BM-ABD содержит анти-TGFβ связывающий домен (например, анти-scFv TGFβ). Иллюстративные последовательности анти-scFv TGFβ продемонстрированы на фиг. 45. В некоторых вариантах осуществления анти-scFv TGFβ содержит вариабельный домен тяжелой цепи и вариабельный домен легкой цепи анти-scFv TGFβ 4G9. В некоторых вариантах осуществления слитый белок с ABD содержит анти-scFv TGFβ и цитокин или дополнительный связывающий фрагмент, при этом цитокин или дополнительный связывающий фрагмент представляет собой второй анти-scFv TGFβ-, IL-15-, IL-12- или анти-PD-L1-связывающий домен (10D12). См. фиг. 51B.
В некоторых вариантах осуществления BM-ABD содержит анти-PD-L1-связывающий домен (например, анти-PD-L1 scFv). В некоторых вариантах осуществления анти-PD-L1 scFv содержит вариабельный домен тяжелой цепи и вариабельный домен легкой цепи анти-PD-L1 10D12 scFv 4D9. В некоторых вариантах осуществления слитый белок с ABD содержит анти-PD-L1 scFv и цитокин или дополнительный связывающий фрагмент, при этом цитокин или дополнительный связывающий фрагмент представляет собой второй анти-PD-L1 scFv, IL-15-, IL-12- или анти-TGFβ-связывающий домен (4D9). См. фиг. 51B.
A. TGF-β-связывающие фрагменты
В определенных вариантах осуществления BM-ABD, предложенный в данном документе, содержит TGF-β-связывающий фрагмент. Все используемые в данном документе термины «TGF-β», «TGFβ», «TGFb» и «трансформирующий фактор роста бета» относятся к члену семейства цитокинов, которые участвуют в контроле пролиферации, клеточной дифференцировки и других функциях в большинстве клеток и существуют по крайней мере в трех изоформах: TGFβ1 (номера доступа в Genbank: NM_000660 и NP_000651 (человек) и NM_011577 и NP_035707 (мышь)), TGFβ2 (номера доступа в Genbank NM_001135599 и NP_001129071 (человек)) и NM_009363 и NP_33) и TGFβ3 (номер доступа в Genbank: NM_003239). Члены семейства TGFβ имеют N-концевой сигнальный пептид из 20-30 аминокислот, который необходим для секреции из клеток, про-область и С-концевую область из 112-113 аминокислот, которая становится зрелой молекулой TGFβ после своего высвобождения в форме про-области в результате протеолитического расщепления. В некоторых вариантах осуществления зрелый белок TGFβ может димеризоваться с образованием активной молекулы с молекулярной массой 25 кДа со многими консервативными структурными мотивами, в том числе девятью остатками цистеина, восемь которых образуют дисульфидные связи в молекуле TGFβ с образованием структуры цистеинового узла. Девятый консервативный цистеин образует связь с девятым цистеином другого TGFβ с образованием димера.
Не связываясь каким-либо конкретным принципом действия, считают, что рассматриваемый BM-ABD, который связывает TGFβ, может быть использован для лечения субъектов, имеющих рак (например, рак поздней стадии). В определенных вариантах осуществления BM-ABD содержит TGFβ1-связывающий фрагмент. В определенных вариантах осуществления BM-ABD содержит TGFβ2-связывающий фрагмент. В определенных вариантах осуществления BM-ABD содержит TGFβ3-связывающий фрагмент. В некоторых вариантах осуществления мультивалентный связывающий полипептид содержит связывающий фрагмент, который может связывать TGFβ1, TGFβ2 и/или TGFβ3 или любую их комбинацию (например, связывает TGFβ1 и TGFβ2; связывает TGFβ2 и TGFβ3; связывает TGFβ1 и TGFβ3; или связывает TGFβ1, TGFβ2 и TGFβ3). В некоторых вариантах осуществления TGFβ-связывающий фрагмент связывает TGFβ1, TGFβ2 и TGFβ3. В некоторых вариантах осуществления TGFβ-связывающий фрагмент содержит вариабельный домен тяжелой цепи и вариабельный домен легкой цепи TGFβ-связывающего фрагмента на фиг. 40. В одном варианте осуществления TGFβ-связывающий фрагмент содержит вариабельный домен тяжелой цепи и вариабельный домен легкой цепи TGFβ-связывающего фрагмента 4D9 (фиг. 40B). В конкретном варианте осуществления TGFβ-связывающий фрагмент представляет собой scFv 4D9. Было показано, что 4D9 предупреждает экспансию регуляторных CD4+FoxP3+ Т-клеток, предупреждает активацию Smad (например, фосфорилирование Smad2), а также предупреждает клеточный эпителиально-мезенхимальный переход и/или миграцию раковых клеток.
В некоторых вариантах осуществления слитый белок TGFβ-связывающий фрагмент-ABD дополнительно присоединяется к другому связывающему фрагменту или цитокину. В определенных вариантах осуществления другой связывающий фрагмент представляет собой PD-L1-связывающий фрагмент или другой TGFβ-связывающий фрагмент. В некоторых вариантах осуществления этот цитокин представляет собой IL-15 или IL-12 (см. фиг. 51B).
B. PD-L1-связывающие фрагменты
В определенных вариантах осуществления BM-ABD, предложенный в данном документе, содержит связывающий фрагмент лиганда 1 белка запрограммированной клеточной смерти 1 (PD-L1). Все используемые в данном документе термины «лиганд 1 белка запрограммированной клеточной смерти», «лиганд 1 белка запрограммированной смерти», «PDL1» и «PD-L1» (номера доступа в Genbank NM_001267706 и NP_001254635 (человек) и NM_021893 и NP_068693 (мышь)) относятся к члену семейства трансмембранных белков 1 типа с молекулярной массой 40 кДа, который связывается с рецептором PD1, встречающимся на активированных Т-клетках, В-клетках и миелоидных клетках, с целью модуляции активации или ингибирования. Повышенная регуляция PD-L1 способствует тому, что виды рака ускользают от иммунной системы. См., например, Hamanishi et al., Proc Natl Acad Sci USA 104(9): 3360-5 (2007). Таким образом, считается, что рассматриваемые BM-ABD, которые содержат PD-L1-связывающий фрагмент, могут быть пригодными при лечении видов рака.
В одном варианте осуществления PD-L1-связывающий фрагмент содержит вариабельный домен тяжелой цепи и вариабельный домен легкой цепи PD-L1-связывающего фрагмента 10D12 (фиг. 50). В конкретном варианте осуществления PD-L1-связывающий фрагмент представляет собой scFv 10D12 (фиг. 50). 10D12 связывается с hPD-L1 при низком значении pH и перекрестно реагирует с mPD-L1. 10D12 не связывается с hPD-L2 или mPD-L2. Кроме того, 10D12 блокирует взаимодействие PD-1/PD-L1, а также взаимодействие B71/PD-L1.
В некоторых вариантах осуществления слитый белок PD-L1-связывающий фрагмент-ABD дополнительно присоединяется к другому связывающему фрагменту или цитокину. В определенных вариантах осуществления другой связывающий фрагмент представляет собой TGFβ-связывающий фрагмент или другой PD-L1-связывающий фрагмент. В некоторых вариантах осуществления этот цитокин представляет собой IL-15 или IL-12 (см. фиг. 51B).
C. TNF и другие связывающие фрагменты
В одном варианте осуществления слитый белок с ABD, предложенный в данном документе, содержит связывающий фрагмент фактора некроза опухоли (TNF). Не связываясь каким-либо конкретным принципом действия, считается, что такие слитые белки на основе ABD являются пригодными в качестве противовоспалительных и/или противоопухолевых средств. В некоторых вариантах осуществления TNF-связывающий фрагмент представляет собой scFv. В конкретных вариантах осуществления слитый белок TNF-связывающий фрагмент-ABD дополнительно присоединяется к другому партнеру по слиянию, который представляет собой связывающий фрагмент или ингибирующий пептид. В определенных вариантах осуществления второй связывающий фрагмент представляет собой второй TNF-связывающий фрагмент, IL-1-связывающий фрагмент, IL-6-связывающий фрагмент, IL-8-связывающий фрагмент, IL-17- (изоформы A-F) связывающий фрагмент или IL-IL-23-связывающий фрагмент.
В другом варианте осуществления слитый белок с ABD содержит связывающий фрагмент, выбранный из IL-1-, IL-6-, IL-8-, IL-17- (A-F) и IL-23-связывающего фрагмента.
В некоторых вариантах осуществления такие TNF- и интерлейкин-связывающие фрагменты-ABD являются пригодными для лечения заболеваний, таких как ревматоидный артрит, болезнь Крона, псориатический артрит, анкилозирующий спондилит, язвенный колит, хронический бляшечный псориаз и заболевания на основе TNF.
F. Диагностические пути применения
В другом аспекте в данном документе предложены способы визуализации и/или выявления опухолей. В некоторых аспектах способы включают приведение в контакт опухолевой клетки, культуры опухолевых клеток, клетки сосудистой сети опухоли, культуры сосудистой сети опухоли, опухолевой ткани и других тканей и клеток с рассматриваемым слитым белком на основе ABD по настоящему изобретению, который является меченым.
Слитый белок с ABD также находит применение в визуализации in vitro или in vivo опухолей или аутоиммунных заболеваний, ассоциированных с антигенсвязывающими партнерами слитого белка на основе ABD в данном документе. В некоторых вариантах осуществления слитый белок, описанный в данном документе, используется как для диагностики, так и для лечения, или только для диагностики. В некоторых вариантах осуществления рассматриваемый слитый белок с ABD является меченым.
Диагностика может быть выполнена либо in vivo, с помощью введения диагностического белка, который позволяет получать визуализацию всего организма, как описано ниже, либо in vitro на образцах, извлеченных от пациента. «Образец» в данном контексте включает в себя любое количество объектов, в том числе, но не ограничиваясь ими, биологические жидкости (в том числе, но не ограничиваясь ими, кровь, мочу, сыворотку крови, лимфу, слюну, анальные и вагинальные выделения, пот и сперму), а также образцы тканей, например, полученные в результате биопсии соответствующих тканей.
Под «меченным» в данном документе подразумевается, что слитый белок с ABD, раскрытый в данном документе, имеет один или несколько элементов, изотопов или химических соединений, присоединенных для обеспечения возможности выявления в ходе скрининга или диагностической процедуры. Как правило, метки делятся на несколько классов: а) иммунные метки, которые могут представлять собой эпитоп, включенные в качестве партнера по слиянию, который распознается антителом, б) изотопные метки, которые могут представлять собой радиоактивные или тяжелые изотопы, в) метки на основе малых молекул, которые могут включать в себя флуоресцентные и колориметрические красители или молекулы, такие как биотин, которые позволяют другие способы маркировки, и d) метки, такие как частицы (в том числе пузырьки для ультразвуковой маркировки) или парамагнитные метки, которые позволяют визуализировать организм Метки могут быть включены в белки в любом положении (например, с помощью одного или нескольких линкеров, описанных в данном документе) и могут быть включены in vitro или in vivo во время экспрессии белка, как известно в данной области техники. Конкретные метки включают в себя оптические красители, в том числе, но не ограничиваясь ими, хромофоры, люминофоры и флуорофоры, причем последние являются во многих случаях специфичными. Флуорофоры могут представлять собой как флуоры на основе «малых молекул», так и белковые флуоры.
Под «флуоресцентной меткой» подразумевается любая молекула, которая может быть выявлена с помощью присущих ей флуоресцентных свойств. Подходящие флуоресцентные метки включают в себя, но не ограничиваясь ими, флуоресцеин, родамин, тетраметилродамин, эозин, эритрозин, кумарин, метил-кумарины, пирен, малацитовый зеленый, стильбен, люцифер желтый, Cascade BlueJ, Texas Red, IAEDANS, EDANS, BODIPY FL, LC Red 640, Cy 5, Cy 5.5, LC Red 705, зеленый Орегон, красители Alexa-Fluor (Alexa Fluor 350, Alexa Fluor 430, Alexa Fluor 488, Alexa Fluor 546, Alexa Fluor 568, Alexa Fluor 594, Alexa Fluor 633 , Alexa Fluor 660, Alexa Fluor 680), Cascade Blue, Cascade Yellow и R-фикоэритрин (PE) (Molecular Probes, Юджин, Орегон), FITC, родамин и Texas Red (Pierce, Рокфорд, Иллинойс), Cy5, Cy5.5, Cy7 (Amersham Life Science, Питтсбург, Пенсильвания). Подходящие оптические красители, в том числе флуорофоры, описаны в Molecular Probes Handbook by Rihard P. Haugland, полностью включенной в качестве ссылки.
Подходящие белковые флуоресцентные метки также включают в себя, но не ограничиваясь ими, зеленый флуоресцентный белок, в том числе GFP видов Renilla, Ptilosarcus или Aequorea (Chalfie et al., Science 263:802-805 (1994)), EGFP (Clontech Laboratories, Inc., Genbank, номер доступа U55762), синий флуоресцентный белок (BFP, Quantum Biotechnologies, Inc. 1801 de Maisonneuve Blvd. West, 8th Floor, Montreal, Quebec, Canada H3H 1J9; Stauber, 1998, Biotechniques 24:462-471; Heim et al., 1996, Curr. Biol. 6:178-182), усиленный желтый флуоресцентный белок (EYFP, Clontech Laboratories, Inc.), люциферазу (Ichiki et al., J. Immunol. 150:5408-5417 (1993)), β-галактозидазу (Nolan et al., Proc. Natl. Acad. Sci. USA, 85:2603-2607 (1998)) и Renilla (WO92/15673, WO95/07463, WO98/14605, WO98/26277, WO99/49019, патенты США №№ 5292658, 5418155, 5683888, 5741668, 5777079, 5804387, 5874304, 5876995, 5925558). Все вышеприведенные ссылки в этом параграфе явно включены в данный документе посредством ссылки.
G. Получение альбумин-связывающих доменов и слитых белков
Как будет понятно специалистам в данной области техники, стандартные протоколы используются для получения рассматриваемых ABD. Общие способы молекулярной биологии, экспрессии, очистки и скрининга антител описаны в Antibody Engineering, edited by Kontermann & Dubel, Springer, Heidelberg, 2001; and Hayhurst & Georgiou, Curr Opin Chem Biol 5:683-689 (2001); Maynard & Georgiou, Annu Rev Biomed Eng 2:339-76 (2000).
В одном варианте осуществления, раскрытом в данном документе, создают нуклеиновые кислоты, которые кодируют слитый белок с ABD и которые затем могут быть клонированы в клетки-хозяева, экспрессированы и проанализированы, при необходимости. Таким образом, могут быть получены нуклеиновые кислоты и, в частности, ДНК, которые кодируют каждую последовательность белка. Эти практические варианты реализации выполняют с использованием хорошо известных процедур. Например, различные способы, которые могут найти применение при создании слитых белков на основе ABD, аналогичные получению антител, раскрыты в данном документе, описаны в Molecular Cloning - A Laboratory Manual, 3rd Ed. (Maniatis, Cold Spring Harbor Laboratory Press, New York, 2001) и Current Protocols in Molecular Biology (John Wiley & Sons), оба полностью включены посредством ссылки. Существует множество методик, которые могут быть использованы для эффективного получения ДНК, кодирующей ABD, раскрытые в данном документе. Такие способы включают, но не ограничиваясь ими, способы сборки генов, способ на основе ПЦР и способы, в которых используют вариации ПЦР, способы, основанные на цепной реакции с использованием лигазы, способы с использованием объединенных олигонуклеотидов, такие как способы, используемые в синтетическом шаффлинге, способы амплификации сниженной точности и способы, в которых используют олигонуклеотиды со случайными мутациями, классические способы сайт-направленного мутагенеза, кассетный мутагенез и другие способы амплификации и синтеза генов. Как известно в данной области техники, существует множество коммерчески доступных наборов и способов для сборки генов, мутагенеза, субклонирования векторов и т.п., и такие коммерческие продукты находят применение для создания нуклеиновых кислот, которые кодируют слитые белки на основе ABD.
ABD, раскрытые в данном документе, могут быть получены путем культивирования клетки-хозяина, трансформированной нуклеиновой кислотой, например, вектором экспрессии, содержащим нуклеиновую кислоту, кодирующую слитые белки на основе ABD, в соответствующих условиях с тем, чтобы индуцировать или вызывать экспрессию белка. Условия, подходящие для экспрессии, будут варьироваться в зависимости от выбора вектора экспрессии и клетки-хозяина и будут легко установлены специалистом в данной области техники с помощью стандартного экспериментирования. Может быть использован широкий спектр подходящих клеток-хозяев, в том числе, но не ограничиваясь ими, клеток млекопитающих, бактерий, клеток насекомых, дрожжевых грибов и растительных клеток. Например, различные клеточные линии, которые могут найти применение при получении слитых белков на основе ABD, раскрытых в данном документе, описаны в каталоге клеточных линий ATCC®, доступном из Американской коллекции типовых культур.
В одном варианте осуществления ABD экспрессируются в системах экспрессии млекопитающих, в том числе системах, в которых экспрессионные конструкции вводят в клетки млекопитающих с использованием вируса, такого как ретровирус или аденовирус. Могут быть использованы любые клетки млекопитающих, например, клетки человека, мыши, крысы, хомяка и приматов. Подходящие клетки также включают в себя известные клетки для научных исследований, в том числе, но не ограничиваясь ими, клетки Jurkat T, клетки NIH3T3, CHO, BHK, COS, HEK293, PER C.6, HeLa, Sp2/0, NS0 и их варианты. В альтернативном варианте осуществления библиотечные белки экспрессируются в бактериальных клетках. Бактериальные экспрессионные системы хорошо известны в данной области техники и включают в себя Escherichia coli (E. coli), Bacillus subtilis, Streptococcus cremoris и Streptococcus lividans. В альтернативных вариантах осуществления слитые белки на основе ABD продуцируются в клетках насекомых (например, Sf21/Sf9, Trichoplusia ni Bti-Tn5b1-4) или клетках дрожжей (например, S. cerevisiae, Pichia и т.д.). В альтернативном варианте осуществления полипептиды на основе ABD экспрессируются in vitro с использованием бесклеточных систем трансляции. Доступны системы трансляции in vitro, полученные как из прокариотических (например, E. coli), так и из эукариотических (например, зародышей пшеницы, ретикулоцитов кролика) клеток, и они могут быть выбраны на основании уровней экспрессии и функциональных свойств белка, представляющего интерес. Например, как понятно специалистам в данной области техники, трансляция in vitro требуется для некоторых технологий дисплея, например рибосомного дисплея. Кроме того, слитые белки на основе ABD могут быть получены с помощью способов химического синтеза. Также трансгенные экспрессионные системы могут быть как животными (например, коровье, овечье или козье молоко, развивающиеся куриные эмбрионы, целые личинки насекомых и т.д.), так и растительными (например, кукуруза, табак, ряска и т.д.).
Нуклеиновые кислоты, которые кодируют слитые белки на основе ABD, раскрытые в данном документе, могут быть включены в экспрессионный вектор для экспрессии белка. Разнообразные экспрессионные векторы могут быть использованы для экспрессии белка. Экспрессионные векторы могут содержать самореплицирующиеся внехромосомные векторы или векторы, которые интегрируются в геном хозяина. Векторы экспрессии конструируются таким образом, чтобы они были совместимыми с типом клетки-хозяина. Таким образом, экспрессионные векторы, которые находят применение для получения антител, раскрытых в данном документе, включают, но не ограничиваясь ими, векторы, которые обеспечивают экспрессию белка в клетках млекопитающих, бактериях, клетках насекомых, дрожжевых грибах и в системах in vitro. Как известно в данной области техники, доступно множество векторов экспрессии, коммерчески или иным образом, которые могут найти применение для экспрессии антител, раскрытых в данном документе.
Раскрытые слитые белки на основе ABD могут кодироваться несколькими молекулами нуклеиновых кислот. Например, вариабельные области тяжелых и легких цепей могут быть введены в клетку-хозяина независимо друг от друга. Несмотря на то, что они присутствуют в отдельных нуклеиновых кислотах, их экспрессия приводит к образованию одного полипептида.
Экспрессионные векторы в типичном случае содержат белок, функционально связанный с контрольными или регуляторными последовательностями, селектируемыми маркерами, любыми партнерами по слиянию и/или дополнительными элементами. Под «функционально связанным» в данном документе подразумевается, что нуклеиновая кислота находится в функциональном отношении с другой последовательностью нуклеиновой кислоты. Как правило, эти экспрессионные векторы содержат нуклеиновую кислоту для регуляции транскрипции и трансляции, функционально связанную с нуклеиновой кислотой, кодирующей мультивалентные слитые белки на основе ABD, и в типичном случае они являются подходящими для клетки-хозяина, используемой для экспрессии белка. Как правило, последовательности регуляции транскрипции и трансляции могут содержать промоторные последовательности, сайты связывания рибосом, последовательности инициации и терминации транскрипции, последовательности инициации и терминации трансляции и энхансерные или активаторные последовательности. Как также известно в данной области техники, экспрессионные векторы в типичном случае содержат селектируемый ген или маркер, позволяющий осуществлять селекцию трансформированных клеток-хозяев, содержащих экспрессионный вектор. Селектируемые гены хорошо известны в данной области техники и будут варьироваться в зависимости от используемой клетки-хозяина.
В одном варианте осуществления ABD очищают или выделяют после экспрессии. ABD и слитые белки на основе ABD могут быть выделены или очищены различными способами, известными специалистам в данной области техники. Очистка может быть особенно пригодной для отделения молекул гетеродимерных тяжелых цепей от молекул гомодимерных тяжелых цепей, описанных в данном документе. Стандартные способы очистки включают в себя хроматографические методики, в том числе ионообменные, гидрофобные, аффинные, эксклюзионные или гель-фильтрационные и обращенно-фазовые, осуществляемую при атмосферном давлении или при высоком давлении с использованием таких систем, как FPLC и ВЭЖХ. Способы очистки также включают в себя электрофоретические методики, методики изоэлектрической фокусировки, иммунологические, преципитационные, диалитические способы и способы хроматофокусировки. Методики ультрафильтрации и диафильтрации в сочетании с концентрацией белка также являются пригодными. Используется слияние, Ni+2 аффинная хроматография, если используется His-тэг, или иммобилизованное анти-flag антитело, если используется flag-тэг. Общее руководство по подходящим методикам очистки см., например, включенное полностью посредством ссылки Protein Purification: Principles and Practice, 3rd Ed., Scopes, Springer-Verlag, NY, 1994, полностью включенная посредством ссылки. Степень необходимой очистки будет варьироваться в зависимости от скрининга или применения антител. В некоторых случаях очистка не требуется.
H. Терапевтические пути применения альбумин-связывающих доменов и слитых белков на основе ABD
Рассматриваемые ABD и слитые белки на основе ABD находят применение при различных терапевтических путях применениях, как описано в данном документе.
В одном аспекте в данном документе предложен способ ингибирования опухолевого роста у субъекта, нуждающегося в этом, с помощью рассматриваемых слитых белков на основе альбумин-связывающего домена (ABD), описанных в данном документе. Пригодные слитые белки на основе ABD включают в себя, но не ограничиваясь ими, белки, раскрытые на фиг. 4, 20, 34, 36, 40, 45, 50-51.
В некоторых вариантах осуществления слитый белок с ABD содержит IL-12 или IL-15 (например, слитый белок IL-12-ABD или IL-15-ABD). Описанные в данном документе слитые белки IL-15-ABD способны ингибировать опухолевый рост дозозависимым образом. Такое опосредованное IL-15 ингибирование опухолевого роста сопровождается повышением количества лимфоцитов, инфильтрирующих опухоль, в том числе цитотоксических Т-лимфоцитов (CTL) и активированнх естественных клеток-киллеров (NK). В определенных вариантах осуществления слитый белок с ABD содержит молекулу IL-12. В некоторых вариантах осуществления слитый белок с ABD содержит IL-15. В еще одних вариантах осуществления цитокин-ABD содержит IL-12 и IL-15. В некоторых вариантах осуществления IL-15-ABD содержит вариантный IL-15, описанный в данном документе (см., например, фиг. 3).
В данном документе также предложен способ лечения субъекта, имеющего рак, с помощью введения субъекту слитого белка на основе альбумин-связывающего домена (ABD). В некоторых вариантах осуществления слитый белок с ABD содержит IL-12 или IL-15 (например, слитый белок IL-12-ABD или IL-15-ABD). IL-12 и IL-15 представляют собой цитокины для иммуномодуляции микроокружения опухоли благодаря их способности пролиферировать и продлевать выживаемость CD8+ T-клеток. Другие пригодные слитые белки на основе ABD включают в себя, но не ограничиваясь ими, белки, раскрытые на фиг. 4, 20, 34, 36, 40, 45, 50-51.
Примеры рака, подлежащего лечению, в данном документе включают в себя, но не ограничиваясь ими, карциному, бластому, саркому, определенные виды лейкоза или лимфоидные злокачественные новообразования. Более конкретные примеры таких видов рака включают в себя плоскоклеточный рак (например, рак клеток плоского эпителия), рак легкого, в том числе мелкоклеточный рак легкого, немелкоклеточный рак легкого, аденокарциному легкого и плоскоклеточный рак легкого, рак брюшины, гепатоцеллюлярный рак, рак желудка, в том числе рак желудочно-кишечного тракта, рак поджелудочной железы, глиобластому, рак шейки матки, рак яичников, рак печени, рак мочевого пузыря, гепатому, рак молочной железы, рак толстой кишки, рак прямой кишки, колоректальный рак, рак эндометрия или матки, карциному слюнных желез, рак почки, рак предстательной железы, рак влагалища, рак щитовидной железы, рак печени, рак анального канала, рак полового члена, рак кожи/меланому, а также рак головы и шеи и метастазы, ассоциированные с любой из первичных опухолей.
В другом аспекте в данном документе предложен способ повышения пролиферации и/или выживаемости CD8+ T-клетки. В некоторых вариантах осуществления способ включает приведение в контакт клетки со слитым белком на основе альбумин-связывающего домена, который содержит IL-12 и/или IL-15 (например, слитый белок IL-12-ABD или IL-15-ABD). В определенных вариантах осуществления слитый белок с ABD содержит молекулу IL-12. В некоторых вариантах осуществления слитый белок с ABD содержит молекулу IL-15. В еще одних вариантах осуществления слитый белок с ABD содержит IL-12 и IL-15.
I. Фармацевтические составы, введение и дозировка
В другом аспекте в данном документе предложена терапевтическая композиция, содержащая любой рассматриваемый полипептид на основе альбумин-связывающего домена (ABD) и носитель. Рассматриваемые терапевтические композиции, используемые при практическом осуществлении вышеуказанных способов, могут быть составлены в фармацевтические композиции, содержащие носитель, подходящий для необходимого способа доставки. Подходящие носители включают в себя любое вещество, которое в сочетании с терапевтической композицией сохраняет противоопухолевую функцию терапевтической композиции и, как правило, не реагирует с иммунной системой пациента. Примеры включают в себя, но не ограничиваясь ими, любой из ряда стандартных фармацевтических носителей, такие как стерильные фосфатно-солевые буферные растворы, бактериостатическая вода и т.п. (см., в целом, Remington's Pharmaceutical Sciences 16th Edition, A. Osal., Ed., 1980).
1. Композиции для введения in vivo
Составы слитого белка на основе альбумин-связывающего домена (ABD), используемые в соответствии с данным изобретением, готовят для хранения с помощью смешивания слитого белка на основе ABD, имеющего необходимую степень чистоты, с необязательными фармацевтически приемлемыми носителями, наполнителями или стабилизаторами (Remington's Pharmaceutical Sciences 16th edition, Osol, A. Ed. [1980]), в форме лиофилизированных составов или водных растворов. Приемлемые носители, наполнители или стабилизаторы являются нетоксичными для реципиентов в используемых дозировках и концентрациях и включают в себя буферы, такие как фосфатный, цитратный и на основе других органических кислот; антиоксиданты, в том числе аскорбиновую кислоту и метионин; консерванты (такие как октадецилдиметилбензиламмонийхлорид; хлорид гексаметония; хлорид бензалкония, хлорид бензетония; феноловый, бутиловый или бензиловый спирт; алкилпарабены, такие как метил- или пропилпарабен; катехол; резорцинол; циклогексанол; 3-пентанол; и м-крезол); низкомолекулярные (менее чем приблизительно 10 остатков) слитые белки; белки, такие как сывороточный альбумин, желатин или иммуноглобулины; гидрофильные полимеры, такие как поливинилпирролидон; аминокислоты, такие как глицин, глутамин, аспарагин, гистидин, аргинин или лизин; моносахариды, дисахариды и другие углеводы, в том числе глюкоза, манноза или декстрины; хелатообразующие агенты, такие как ЭДТА или DPTA; сахара, такие как сахароза, маннит, трегалоза или сорбит; солеобразующие противоионы, такие как натрий; комплексы металлов (например, комплексы Zn-белок); и/или неионные поверхностно-активные вещества, такие как TWEEN™, PLURONICS™ или полиэтиленгликоль (ПЭГ) с различными молекулярными массами.
Композиция в данном документе также может содержать более одного активного соединения, которое необходимо для конкретного показания, подлежащего лечению, предпочтительно, с комплементарными активностями, которые не оказывают отрицательного воздействия друг на друга. Например, может быть желательным придание слитым белкам на основе ABD других специфических особенностей. В качестве альтернативы или в качестве дополнения, композиция может содержать цитотоксический агент, цитокин, ингибитор роста и/или антагонист на основе малой молекулы. Такие молекулы подходящим образом присутствуют в комбинации в количествах, которые являются эффективными для предполагаемой цели.
Активные ингредиенты также могут быть захвачены в микрокапсулы, приготовленные, например, с помощью методик коацервации или с помощью полимеризации на границе раздела фаз, например, в микрокапсулы на основе гидроксиметилцеллюлозы или желатина и микрокапсулы на основе поли-(метилметацилата) соответственно, в системы доставки коллоидных лекарственных средств (например, липосомы, альбуминовые микросферы, микроэмульсии, наночастицы и нанокапсулы) или в макроэмульсии. Такие методики описаны в Remington's Pharmaceutical Sciences 16th edition, Osol, A. Ed. (1980).
Составы, подлежащие использованию для введения in vivo, должны быть стерильными или почти стерильными. Это легко достигается с помощью фильтрации с использованием стерильных фильтрующих мембран.
Могут быть получены препараты с замедленным высвобождением. Подходящие примеры препаратов с замедленным высвобождением включают в себя полупроницаемые матрицы из твердых гидрофобных полимеров, содержащих антитело, при этом эти матрицы находятся в форме изделий определенной формы, например, пленок или микрокапсул. Примеры матриц с замедленным высвобождением включают в себя сложные полиэфиры, гидрогели (например, поли(2-гидроксиэтилметакрилат) или поли(виниловый спирт)), полилактиды (патент США № 3773919), сополимеры L-глутаминовой кислоты и гамма-этил-L-глутамата, неразлагаемый этилен-винилацетат, разлагаемые сополимеры молочной кислоты и гликолевой кислоты, такие как LUPRON DEPOT™ (инъекционные микросферы, состоящие из сополимера молочной кислоты и гликолевой кислоты и ацетата лейпролида) и поли-D-(-)-3-гидроксимасляную кислоту. Несмотря на то, что такие полимеры, как этилен-винилацетат и молочная кислота и гликолевая кислота, позволяют высвобождать молекулы в течение более 100 дней, определенные гидрогели высвобождают белки в течение более коротких периодов времени.
Когда инкапсулированные слитые белки на основе альбумин-связывающего домена остаются в организме в течение длительного времени, они могут денатурировать или агрегировать в результате воздействия влаги при 37°C, что приводит к потере биологической активности и возможным изменениям иммуногенности. Для стабилизации могут быть разработаны рациональные стратегии в зависимости от используемого механизма. Например, если обнаружено, что механизм агрегации заключается в образовании межмолекулярных S-S-связей посредством тиодисульфидного обмена, стабилизация может быть достигнута с помощью модификации сульфгидрильных остатков, лиофилизации из кислых растворов, контроля содержания влаги, использования соответствующих добавок и разработки специфических полимерных матричных композиций.
2. Способы введения
Рассматриваемые слитые белки и терапевтические агенты на основе альбумин-связывающего домена вводят субъекту в соответствии с известными способами, такими как внутривенное введение в виде болюса или путем непрерывной инфузии в течение некоторого периода времени, с помощью внутримышечных, внутрибрюшинных, внутрицереброспинальных, подкожных, внутрикожных, внутрисуставных, интрасиновиальных, интратекальных, пероральных, местных или ингаляционных путей. Внутривенное или подкожное введение антитела является предпочтительным.
3. Способы лечения
В способах, предложенных в данном документе, терапия используется для обеспечения положительного терапевтического ответа в отношении заболевания или патологического состояния. Под «положительным терапевтическим ответом» подразумевается нормализация заболевания или состояния и/или нормализация симптомов, ассоциированных с заболеванием или патологическим состоянием. Например, положительный терапевтический ответ может относиться к одному или нескольким из следующих видов нормализации заболевания: (1) уменьшению количества опухолевых клеток; (2) увеличению гибели опухолевых клеток; (3) ингибированию выживания опухолевых клеток; (5) ингибированию (т.е. замедлению до некоторой степени, предпочтительно остановке) опухолевого роста; (6) увеличению выживаемости пациентов; и (7) некоторому облегчению от одного или нескольких симптомов, ассоциированных с заболеванием или патологическим состоянием.
Положительные терапевтические ответы при любом определенном заболевании или патологическом состоянии могут быть определены с помощью стандартизированных критериев ответа, специфичных в отношении этого заболевания или патологического состояния. Реакцию опухоли можно оценивать в отношении изменений морфологии опухоли (т.е. общей опухолевой нагрузки, размера опухоли и т.п.) с использованием методик скрининга, таких как магнитно-резонансная томография (МРТ), рентгенография, компьютерная томография (КТ), сканирование костей скелета, эндоскопия и биопсия опухоли, в том числе аспирация костного мозга (BMA) и подсчет опухолевых клеток в кровотоке.
В дополнение к этим положительным терапевтическим ответам, субъект, подвергающийся терапии, может испытывать положительный эффект в виде нормализации симптомов, ассоциированных с заболеванием.
Таким образом, например, в случае В-клеточных опухолей субъект может испытывать уменьшение так называемых В-симптомов, т.е. ночных приливов, лихорадки, потери массы тела и/или крапивницы. В случае предзлокачественных патологических состояний терапия мультивалентным терапевтическим агентом может блокировать и/или продлевать время до развития связанного злокачественного состояния, например, развития множественной миеломы у субъектов, страдающих моноклональной гаммопатией неясного генеза (MGUS).
Нормализацию заболевания можно охарактеризовать в виде полного ответа. Под «полным ответом» подразумевается отсутствие клинически выявляемого заболевания с нормализацией любых ранее аномальных рентгенологических исследований, костного мозга и спинномозговой жидкости (CSF) или аномального моноклонального белка в случае миеломы.
Такой ответ может сохраняться в течение, по меньшей мере, 4-8 недель или иногда 6-8 недель после лечения в соответствии с рассматриваемыми способами. В качестве альтернативы, нормализация заболевания может быть классифицирована в виде частичного ответа. Под «частичным ответом» подразумевается, по меньшей мере, приблизительно 50% снижение всей измеряемой опухолевой нагрузки (т.е. количества злокачественных клеток, присутствующих у субъекта, или измеренной массы опухолевых масс или количества аномального моноклонального белка) в отсутствие новых поражений, которые могут сохраняться в течение 4-8 недель или 6-8 недель.
Лечение включает в себя «терапевтически эффективное количество» используемых лекарственных препаратов. «Терапевтически эффективное количество» относится к количеству, эффективному в дозировках и в течение периодов времени, необходимых для достижения необходимого терапевтического результата.
Терапевтически эффективное количество может варьироваться в зависимости от таких факторов, как состояние заболевания, возраст, пол и масса тела индивидуума, а также способность лекарственных препаратов вызывать необходимый ответ у индивидуума. Терапевтически эффективное количество также представляет собой количество, при котором любые токсические или вредные эффекты антитела или части антитела компенсируются терапевтически полезными эффектами.
«Терапевтически эффективное количество» в случае терапии опухолей также может быть измерено по его способности стабилизировать прогрессирование заболевания. Способность соединения ингибировать рак может быть оценена в животной модели, прогнозирующей эффективность при опухолях человека.
В качестве альтернативы, это свойство композиции может быть оценено путем исследования способности соединения ингибировать клеточный рост или индуцировать апоптоз с помощью анализов in vitro, известных специалисту в данной области техники. Терапевтически эффективное количество терапевтического соединения может приводить к уменьшению размера опухоли или иным образом ослаблять симптомы у субъекта. Специалист в данной области техники сможет определить такие количества на основе таких факторов, как размер субъекта, тяжесть симптомов субъекта и конкретная композиция или выбранный способ введения.
Примеры
Пример 1. Скрининг и идентификация связывающих доменов сывороточного альбумина человека
Способы твердофазного пэннинга и пэннинга в растворе осуществляли для идентификации фаговых альбумин-связывающих доменов scFv (ABD). Альбумин-связывающие домены, которые были выбирали в результате первичного скрининга связывающих доменов человеческого сывороточного альбумина, впоследствии подвергали скринингу в отношении перекрестной реактивности с мышиным сывороточным альбумином с использованием стандартных методик ИФА. Первичные кандидаты альбумин-связывающих доменов, полученные с использованием способов скрининга, секвенировали и затем анализировали в отношении зависимого от концентрации связывания с мишенью, стабильности рН, интерференции связывания FcRn и кинетики связывания. В частности, кандидатные ABD выбирали в отношении их способности связывать человеческий сывороточный альбумин (кД ~ 20-60 нМ), мышиный сывороточный альбумин (кД ~ 10-30 нМ) и сывороточный альбумин яванского макака (кД ~ 20-60 нМ) при низком значении рН (рН 5,5) и нейтральном значении рН (рН 7,7). Кандадатные ABD анализировали с тем, чтобы убедиться, что они не конкурируют с FcRn, связывающимися с сывороточным альбумином. Как описано в данном документе, ABD, которые связываются при таком значении pH и не конкурируют за связывание с FcRn, способны подвергаться опосредованной FcRn эндосомной рециркуляции. Таким образом, биологические препараты (например, цитокины и биологические препараты на основе антител), которые содержит такие ABD, также способны подвергаться такому опосредованному FcRn рециклингу и, таким образом, проявляют более длительный период полужизни по сравнению с аналогами, которые не содержат такие ABD.
Пять альбумин-связывающих клонов отбирали на основе этих критериев: A9, A10, A6, 2B4, 2H10. Из этих пяти клонов A10 отбирали на основе высокого уровня экспрессии и наилучшего профиля активности. Затем A10 мутировали для исключения областей, которые предположительно могли вызвать иммуногенность. Из этих вариантов A10 A10m3 отбирали в качестве примера из-за его высокого сродства к сывороточному альбумину.
Последовательности иллюстративных связывающих доменов человеческого сывороточного альбумина продемонстрированы на фиг. 2, в том числе A10m3 (фиг. 2D).
Пример 2. Вариантный IL-15 и IL-15-ABD
Экспрессия белка IL15-A10m3 клетками HEK293T является чрезвычайно низкой, что нельзя объяснить транскрипцией
Конструкции IL-15-ABD (IL-15-A10m3) получали в клетках HEK293 в трех независимо трансфицированных культурах клеток и оценивали с помощью вестерн-блоттинга с использованием либо анти-His-тэг антитела (фиг. 5А, слева), либо с помощью функционального связывания ИФА с мышиным сывороточным альбумином (фиг. 5А, справа). Как продемонстрировано на фиг. 5А, экспрессию IL-15-A10m3 не могли оценить ни одним из этих способов. Чтобы оценить, была ли экспрессия IL-15-A10m3 недостаточной в клетках HEK293 вследствие низких уровней транскрипции, мРНК получали из четырех независимо трансфицированных клеток IL-15-A10m3 и проводили RT-PCR для количественного определения уровня мРНК IL15-A10m3 (фиг. 5В, полосы 2-5) по сравнению с мРНК гена домашнего хозяйства, GAPDH (фиг. 5В, полоска 6). Как продемонстрировано на фиг. 5В, мРНК IL15-A10m3 обнаруживали в значительных количествах в трансфицированных клетках по отношению к этому контрольному GAPDH, что позволяет предположить, что низкие уровни экспрессии IL-15-A10m3, продуцируемого в клетках HEK293, обусловлены не транскрипцией, а, скорее всего, связаны с трансляцией или посттрансляционным процессом.
Идентификация предполагаемого сайта убиквитинирования в IL15, который прилегает к сайту связывания альфа-рецептора IL15
Исследования показали, что белок IL15 экспрессируется в клетках, однако является очень нестабильным при коротком периоде полужизни. Коэкспрессия IL15Rα с IL15 в одной и той же клетке значительно увеличило количество IL15R клеточной поверхности α, а также IL15. Дальнейшие исследования подтверждали, что IL15Rα выступает в качестве шаперона IL15 и связывает IL-15 внутриклеточно для защиты и стабилизации IL-15 перед секрецией. Эти результаты предполагают, что трансляция не может объяснить низкое продуцирование IL15. Скорее всего, пост-трансляционная модификация (PTM) может играть определенную роль во внутриклеточной нестабильности IL15, и внутриклеточная нестабильность IL15 можно преодолеть с помощью блокирования IL15Rα специфический, но пока еще неизвестной пост-трансляционной модификации. Убиквитинирование является хорошо описанным механизмом, который позволяет клеткам маркировать внутриклеточные белки для расщепления.
Учитывая, что IL15 является очень мощным провоспалительным цитокином, экспрессия которого строго контролируется клетками, возможно, что клетки используют убиквитинирование для активного контроля уровней белка IL15. Идентифицировали потенциальные сайты убиквитинирования на IL-15, которые предположительно защищены при связывании с IL-15Rα (фиг. 6). В частности, аминокислота K86 является предполагаемым сайтом убиквитинирования, который находится рядом с сайтом связывания IL-15/IL-15Rα (фиг. 6A), подтверждая возможность того, что связывание IL15Rα с IL15 блокирует доступность убиквитинлигазы (например, E3) в отношении K86 в белке IL15. K86 был далее подтверждали в качестве сайта убиквитинирования с использованием UbPred, онлайн базы данных сайтов убиквитинирования (www.ubpred.org), фиг. 6B.
Мутация K86 в IL15 восстанавливает экспрессию белка IL15-A10m3 клетками HEK293T
Чтобы оценить, влияет ли убиквитинирование в K86 на внутриклеточную стабильность IL-15, получали варианты IL-15, содержащие аминокислотные замены в K86, в том числе K86A и K86R. Последовательности нескольких из этих вариантов IL-15 продемонстрированы на фиг. 3. Не связываясь каким-либо конкретным принципом действия, считается, что аминокислотные замены в этих конкретных сайтах убиквитинирования приводят к образованию устойчивого к убиквитинированию IL-15, который проявляет повышенную стабильность по сравнению с IL-15 дикого типа. Такие варианты IL-15 присоединяли к ABD (A10m3) для дальнейшего продления периода полужизни (фиг. 4). На фиг. 7 предложены схематические иллюстрации таких слитых белков IL-15-ABD, в том числе IL-15-ABD с «доменом sushi» (A) IL-15Rα/IL15, и слитых белков IL-15-ABD, которые содержат варианты IL-15, имеющие аминокислотные замены в месте убиквитинирования K86 (B).
Продуцированные в клетках HEK293 варианты IL-15-ABD K86R и K86A и IL-15Rα/IL15-ABD оценивали в отношении их способности связывать мышиный сывороточный альбумин (MSA) и IL-15Rα. Как продемонстрировано на фиг. 8А, продуцированные HEK293 варианты IL-15-ABD K86R и K86A (12 клонов) и IL-15Rα/IL15-ABD (12 клонов) демонстрируют более высокую экспрессию по сравнению с продуцируемым HEK-293 IL-15-ABD дикого типа (12 клонов). Такие конструкции также были способны связываться с MSA. Более того, как продемонстрировано на фиг. 8В, замены K86R (клон R6, звездочка зеленого цвета) и K86A (клон A3, звездочка желтого цвета) не влияли на способность вариантов связываться с IL-15Rα. Интересно отметить, что слитый с IL15R α IL15-A10m3 не мог проявлять какое-либо связывание с IL15R, α, свидетельствуя о том, что внутренний домен sushi IL15R, α связывался с IL15 внутримолекулярно и, таким образом, блокировал связывание с внешним IL15R, α нанесенным на планшет. Это согласуется с нашим открытием и открытием других авторов в отношении того, что связывание IL15 с доменом sushi IL15R α повышает экспрессию IL15 (фиг. 7В).
Осуществляли продуцирование в увеличенном масштабе одного конкретного варианта IL-15-ABD, продуцируемого клеткой HEK293, содержащего аминокислотную замену K86R (IL-15 K86R-A10m3), и оценивали конструкцию IL-15 K86R-A10m3 в отношении связывания сывороточного альбумина in vitro. Как продемонстрировано на фиг. 9, IL-15-ABD, имеющий мутацию K86 (IL-15 K86R-A10m3) и IL-15Rα/IL15-ABD (IL-15Rα/IL15-A10m3), были способны продуцироваться в клетках HEK 293 в увеличенных количествах, что подтверждается SDS-PAGE (фиг. 9C, слева) и вестерн-блоттингом с анти-His-тег антителом (справа). Кроме того, как продемонстрировано на фиг. 10, K86R-A10m3 демонстрирует связывание с мышиным сывороточным альбумином (MSA).
Биологическая активность IL15 K86R-A10m3, продуцируемого из клеток HEK293T, нарушается, что может быть устранено путем дегликозилирования.
Биологическую активность различных IL-15-ABD исследовали с использованием анализов пролиферации CTLL2. Исследуемые IL-15-ABD включают IL-15-A10m3 дикого типа и три различных IL-15 K86R-A10m3, продуцируемых в клетках HEK293T. В качестве контролей использовали коммерчески доступный IL-15 и IL-15-ABD собственного производства, при этом оба получали с использованием E. coli. Как продемонстрировано на фиг. 11, IL-15 K86R-A10m3, продуцируемый из HEK293T, продемонстрировал значительно сниженную способность стимулировать пролиферацию CTLL2 по сравнению с контролями, продуцированными в E. coli, включая коммерческие IL-15 дикого типа (R&D) и IL-15-A10m3, продуцируемый собственным путем. Учитывая разницу в анализе пролиферации CTLL2 между IL15 K86R-A10m3 из HEK293T и IL15-A10m3 из E. coli, было предположено, что, поскольку IL15-A10m3, продуцируемый из E. coli, не подвергается классическому N-гликозилированию, как это происходит в клетках млекопитающих, возможно, что гликозилирование IL15 K86R -A10m3 в T-клетках HEK293 может нарушать взаимодействие IL15 K86R -A10m3 с его рецепторами.
Чтобы оценить, вызвана ли сниженная биологическая активность продуцируемого клетками HEK IL-15 K86R-A10m3 гликозилированием, IL-15 K86R-A10m3 подвергли дегликозилированию с использованием PNGазы и анализов пролиферации CTLL2 для оценки биологической активности дегликозилированного IL15 K86R-A10m3. После обработки смесью PNGазы IL-15 K86R-A10m3 в нативных условиях гликан полностью удаляли и визуализировали с помощью SDS-PAGE с последующим окрашиванием гликаном (фиг. 12A, слева) и окрашиванием кумасси синим (фиг. 12A, справа). 1) смесь IL-15R-A10m3 + 5 мкл PNGазы; 2) смесь белка + 10 мкл PNGазы; 3) контроль ферментом отсутствует. Как продемонстрировано на фиг. 12B, дегликозилирование IL-15 K86R-A10m3 (синий цвет) почти полностью восстанавливало активность в анализах пролиферации CTLL2 по сравнению с необработанными образцами (фиолетовый цвет, желтый цвет). В качестве положительных контролей использовали IL-15 ДТ от R&D systems (красный цвет) и продуцируемый E. coli IL-15-A10m3 собственного производства (черный цвет).
N112 IL-15 K86R-A10m3 является определяющим для его биологической активности для активации пролиферации CTLL2
Положение аминокислоты N112 IL-15 K86R-A10m3 является основным сайтом для биологической активности IL-15, особенно в контексте IL-15-ABD, так как оно является определяющим для соответствующего установления взаимодействия между IL-15 и гамма-рецептором IL15. Вариант IL-15 K86R дополнительно мутировали в N112A, чтобы определить, могут ли мутации в этом сайте восстанавливать биологическую активность IL-15, аналогично дегликозилированному IL-15. В частности, IL15 K86R-A10m3 мутировали для дополнительного включения аминокислотной замены N112Q, N112A или N112S IL-15 и выполняли анализы пролиферации CTTLL2 для проверки биологической активности. Как продемонстрировано на фиг. 13А, введение аминокислотной замены N112A в IL-15 K86R-A10m3 (синий цвет) восстановило его биологическую активность, сопоставимую с таковой дегликозилированного IL-15 K86R-A10m3 (зеленый цвет) в анализах пролиферации CTLL2, в то время как мутация N112Q (красный цвет) не оказывала влияния на биологическую активность по отношению к исходному IL-15R-A10m3 без дегликозилирования (желтый цвет). IL-15 ДТ (черный цвет) от R&D systems выступал в качестве положительного контроля.
Мутации с различными боковыми цепями в N112 исследовали, чтобы дополнительно продемонстрировать влияние размера на биологическую активность. Как продемонстрировано на фиг. 13B, N112Q (большой размер, красный цвет), N112S (средний размер, зеленый цвет) и N112A (малый размер, синий цвет) проявляли повышенную биологическую активность, обратно пропорциональную размеру боковых цепей. Более того, предполагаемая водородная связь, образованная N112 IL-15 и Y103 гамма-рецептора IL15, по-видимому, не была важна для этой активности, поскольку N112A не может образовывать такую связь. В качестве положительного контроля использовали IL-15 ДТ от R&D systems (черный цвет) и IL-15-A10m3 E. coli собственного производства (фиолетовый цвет); в качестве отрицательного контроля использовали исходный IL15 K86R-A10m3 без дегликозилирования (желтый цвет).
Пример 3. Активность in vivo вариантного IL-15 и IL-15-ABD
Способность IL-15 и IL-15-ABD ингибировать опухолевый рост оценивали с использованием модели меланомы мыши B16-F10. Как продемонстрировано на фиг. 14, мышей обрабатывали IL-15, плацебо PBS или различными дозами IL-15-ABD с помощью внутривенной инъекции в четыре различные временные точки, с интервалом 48 часов. Как продемонстрировано на фиг. 16, IL-15-ABD ингибирует опухолевый рост дозозависимым образом.
Анализы FACS проводили для дальнейшей оценки профиля популяций лимфоцитов, инфильтрующих опухоль, у мышей, обработанных IL-15-ABD, на основании этих исследований. Как продемонстрировано на фиг. 16, опухоли у мышей, обработанных IL-15-ABD, показали увеличение популяций NK-клеток. Эти данные в сочетании с наблюдениями повышенного скопления и сохранения опухолей у мышей, обработанных IL-15-ABD, как описано выше, позволяют предположить, что ABD усиливает провоспалительный эффект IL-15 в опухоли. Эффекты обработки IL-15-ABD на популяции лимфоцитов в селезенке и опухолях представлены на фиг. 17 и 18. Как продемонстрировано на фиг. 17 и 18, анализ FACS популяций лимфоцитов показывает 3-6-кратное увеличение популяций CTL, инфильтрирующих опухоли, и NK-клеток в опухолях мышей, обработанных IL-15 ABD. Никаких значимых различий в селезенке не наблюдали. В целом, результаты этих исследований показывают иммуномодулирующую способность IL-15-ABD в отношении опухоли in vivo.
Чтобы оценить способность слитых белков на основе ABD увеличивать период полужизни IL-15, мышам C57B внутривенно вводили 5 мкг IL-15-ABD или IL-15 в отдельности и затем оценивали концентрации IL-15-ABD и IL- 15 в сыворотке крови. Как продемонстрировано на фиг. 20А, IL-15-ABD проявлял более высокую PK по сравнению с IL-15 ДТ. T ½β IL-15 = 0,6 часа, аналогично таковым, о которых сообщалось в открытом доступе (~ 0,5 часа). Результаты исследования показывают, что ABD увеличивает T ½β IL-15 до ~ 7,0 часов, что составляет в ~ 10 раз больше. IL-15-ABD также анализировали в отношении стабильности в сыворотке крови человека с использованием клеточного анализа. Как продемонстрировано на фиг. 19B, IL-15-ABD был более стабильным в сыворотке крови человека по сравнению с коммерческим контролем IL-15 без ABD.
Пример 4. IL-12-ABD
Конструкции одноцепочечных IL-12 мыши-ABD получали в клетках HEK293T и очищали с помощью эксклюзионной хроматографии. IL-12-A10m3, продуцируемый из клеток HEK293T, был полностью активен как в анализах in vitro, так и в клеточных анализах. Как продемонстрировано на фиг. 21А, IL12-A10m3 способен связываться с мышиным сывороточным альбумином с равновесной константой диссоциации (KD), составляющей 2,1 нМ. IL12-A10m3, продуцируемый из HEK293T, также был способен стимулировать пролиферацию PBMC человека, сопоставимую с пролиферацией IL12 мыши собственного производства и коммерчески доступного мышиного IL-12 (R&D) (фиг. 21B). Кроме того, IL12-A10m3, продуцируемый из HEK293T, стимулировал секрецию гамма-интерферона из PBMC человека, сопоставимую с таковой с продуцируемым IL-12 мыши собственного производства и коммерчески доступного IL-12 мыши (R&D) (фиг. 22).
Обработка IL-12-ABD приводит к уменьшению объема опухоли in vivo.
Способность IL-12 и IL-12-ABD ингибировать опухолевый рост оценивали с использованием модели меланомы мыши B16-F10. Как продемонстрировано на фиг. 23, мышей обрабатывали либо IL-12-ABD, либо IL-12 в трех аналогичных дозах с помощью внутривенной инъекции на 7 день после инокуляции опухоли (день 0), когда объем опухоли достигал 100 мм3. Опухолевый рост контролировали каждые 2 дня после обработки в течение 10 дней. Плацебо PBS выступал в качестве контроля. Как продемонстрировано на фиг. 24-26 и 28, как IL-12, так и IL-12-ABD были способны уменьшать опухолевый рост дозозависимым образом. Более того, IL-12-ABD был способен более эффективно уменьшать объем опухоли по сравнению с IL-12 в отдельности при аналогичных концентрациях (см., например, фиг. 26 через 10 дней и фиг. 28, медианы дней для того, чтобы 50% опухолей достигли 2000 мм3). Измерения продольной массы тела мышей из этих исследований показывают минимальные изменения массы тела во всех группах обработки IL-12-ABD (фиг. 27). Отсутствие наблюдаемых значимых изменений массы указывает на отсутствие токсичности IL-12-ABD в группах обработки в течение 12-дневного последующего курса обработки.
Дальнейшая характеристика фармакодинамических эффектов однократной дозы IL-12-ABD (4,5 мкг IL-12-ABD, такая же молярная доза, что и контроль 3 мкг IL-12) у мышей, несущих опухоли B16-F10, через 5 дней продемонстрировала, что IL -12-ABD проявлял аналогичное более высокое подавление опухолевого роста по сравнению с аналогичной молярной дозой контроля IL-12. У мышей, обработанных IL-12-ABD, также наблюдалось соответствующее увеличение иммунной активации, о чем свидетельствовало увеличение массы селезенки и IFN-γ, при этом влияние на массу тела мыши по сравнению с контролем отсутствовало (фиг. 29).
На фиг. 30 дополнительно продемонстрирован результат исследования, сравнивающего объем опухоли у мышей, несущих опухоль B16-F10, через 10 дней, которым вводили IL-12-ABD (1,3 мкг), IL-12 (30 мкг) или плацебо. Несмотря на то, что IL-12 (1 мкг) и IL-12-ABD (1,3 мкг) являются молярно эквивалентными и имеют одинаковую биологическую активность in vitro, IL-12-ABD является в ~ 30 раз более эффективным, чем IL-12, in vivo (ср. результаты в день 10 на фиг. 30, 1,3 мкг IL-12-ABD > 30 мкг IL-12). На фиг. 31 дополнительно продемонстрированы гематопоэтические эффекты IL-12-ABD и IL-12 у мышей из исследования, продемонстрированного на фиг. 30, через 3 и 7 дней. Как продемонстрировано на фиг. 31, мыши, обработанные IL-12-ABD, на день 3 проявляли временное снижение уровня WBC, нейтрофилов и лимфоцитов по сравнению с мышами, обработанными IL-12 и контролем плацебо. В то же время такие клеточные популяции возвращались к нормальному состоянию на день 7. Кроме того, уровни IFN-γ у мышей, обработанных IL-12-ABD, были выше в дни 3 и 7 по сравнению с мышами, обработанными IL-12 и контролем.
Оценка противоопухолевых эффектов IL-12-ABD или IL-12 в комбинации с анти-PD-1 антителом in vivo.
Эффект комбинированных видов терапии с использованием однократных доз с использованием IL-12-ABD или IL-12 с анти-PD-1 антителами оценивали у мышей, несущих опухоли B16-F10, через 8 дней (фиг. 32).
Животных (в возрасте 7-10 недель) разделяли на 8 групп (по 8 животных в группе) через 10 дней после инокуляции опухолевых клеток B16-F10 (2×104 клеток/мышь). Животных распределяли на основании объема опухоли. На момент распределения средний объем опухоли на группу составлял 100 мм3. В день 0 (когда опухоли достигли 100 мм3) каждой группе вводили внутривенную однократную дозу либо PBS (плацебо), либо IL12-ABD (1,5 мкг, 5 мкг, 15 мкг), либо IL15-ABD-IL12 (1,7 мкг, 6 17, 17)
Группы исследовали в отношении массы тела, объема опухоли и псевдо-выживаемости. Массы тела измеряли до инокуляции опухоли и во время измерений опухоли. Размер опухоли измеряли каждые 2 дня в двух измерениях, с помощью штангенциркуля, а объем выражали в мм3 с помощью формулы: V = 0,5 x a x b2, где a и b представляют собой длинный и короткий диаметры опухоли. Исследование проводили в виде псевдо-выживания; каждую мышь подвергали эвтаназии, когда ее опухоль достигала 2000 мм3 или когда определяли, что она умирает.
Как продемонстрировано на фиг. 33, IL-12-ABD был более эффективным, чем обработка либо анти-PD-1, либо молярно эквивалентной дозой рекомбинантного IL-12. Более того, IL-12-ABD был так же эффективен, как и комбинация обработки рекомбинантным IL-12 и анти-PD-1. Интересно отметить, что добавление анти-PD-1 Ат к рекомбинантному IL-12 повышало эффективность любой обработки в отдельности, тогда как обработка анти-PD-1 не давала дополнительной пользы для IL-12-ABD.
Чтобы оценить способность слитых белков на основе ABD увеличивать период полужизни IL-12, мышам C57B внутривенно вводили 5 мкг IL-12-ABD или только IL-12 и оценивали концентрации IL-12-ABD и IL-12 в сыворотке крови. Как продемонстрировано на фиг. 33, IL-12-ABD проявлял более высокую PK, чем IL-12 ДТ. T ½β IL-12 = 2,5 часа, аналогично таковым, о которых сообщалось в открытом доступе (~ 3,5 часа). Результаты исследования показывают, что ABD продлевало T ½β IL-12 до 9,5 часов, что составляло увеличение в ~ 4 раза.
Пример 5. Биспецифический IL-15-ABD-IL-12
Конструкции IL-15-ABD-IL-12, hIL15 (K86R/N112A)-A10m3-mIL-12sc и mIL-12sc-A10m3-hIL15 (K86R/N112A) получали в клетках HEK293T и очищали с помощью эксклюзионной хроматографии. Последовательности этих конструкций продемонстрированы на фиг. 34. Способность hIL15 (K86R/N112A)-A10m3-mIL-12sc и mIL-12sc-A10m3-hIL15 (K86R/N112A) связывать MSA, бета 2-рецептор IL12 и альфа-рецептор IL-15 оценивали с помощью ИФА. Как продемонстрировано на фиг. 35, обе конструкции IL-15-ABD-IL-12 были способны связываться с MSA дозозависимым образом в среде для культивирования клеток. Кроме того, обе биспецифичные конструкции были способны связываться с бета 2-рецептором IL-12 и альфа-рецептором IL-15 дозозависимым образом в среде для культивирования клеток. Как продемонстрировано на фиг. 35, IL-12/IL-15-ABD, имеющие ориентацию, от N-конца к C-концу, IL-15-ABD-IL-12, проявляли более высокое связывание антигена по сравнению с IL-12-ABD-IL-15. Дополнительные биспецифические соединения, которые содержат IL-15 и IL-12, раскрыты на фиг. 36.
Оценка IL-15-ABD-IL-12 в отношении активности IL-12 и IL-15
IL-15-ABD-IL-12 дополнительно оценивали в отношении активности IL-12 и IL-15 (фиг. 37 и 38).
Для оценки активности IL-12 лимфоциты из PBMC подвергали превращению в бласты с помощью обработки PHA-P в течение четырех дней и rhIL-2 на третий день. Затем лимфобласты обрабатывали либо IL-15-ABD-IL-12, либо контролем IL-12 в течение двух дней, и оценивали активность IL-12 на основе пролиферации лимфобластов и секреции IFN-γ (фиг. 37A). Активность IL-15 оценивали с использованием анализа пролиферации цитотоксических Т-лимфоцитов CTLL-2 (фиг. 38A).
Как продемонстрировано на фиг. 37, IL-15-ABD-IL-12 проявлял активность IL-12, как оценивали по пролиферации лимфобластов (фиг. 37B) и секреции IFN-γ (фиг. 37C). Кроме того, IL-15-ABD-IL-12 проявлял IL-15 в анализе пролиферации CTLL-2 (фиг. 38B). Таким образом, рассматриваемый IL-15-ABD-IL-12 проявлял биологическую активность как IL-12, так и IL-15.
Противоопухолевые эффекты IL-15-ABD-IL-12 в модели меланомы мыши B16-F10
Не связываясь каким-либо конкретным принципом действия, считают, что ABD IL-15/IL-12 обеспечивают синергетическую биологическую активность. В частности, IL-12 увеличивает активность альфа-рецептора IL-15, IFN-γ NK/T-клеток и TH1-иммунитет, при этом снижается активность Treg-клеток. IL-15 увеличивает активность бета 1-рецептора IL-12 и NK-клеток, при этом уменьшается снижается потеря клеток CD8 клеток памяти.
Противоопухолевые эффекты и псевдо-выживаемость IL-12-ABD по отношению к IL15-ABD-IL12 оценивали с использованием модели меланомы мыши B16-F10 (фиг. 39).
Животных (в возрасте 7-10 недель) разделяли на 8 групп (по 8 животных в группе) через 10 дней после инокуляции опухолевых клеток B16-F10 (2×104 клеток/мышь). Животных распределяли на основании объема опухоли. На момент распределения средний объем опухоли на группу составлял 100 мм3. В день 0 (когда опухоли достигли 100 мм3) каждой группе вводили внутривенную однократную дозу либо PBS (плацебо), либо молярно эквивалентные дозы IL12-ABD (1,5 мкг, 5 мкг, 15 мкг), либо IL15-ABD-IL12 (1,7 мкг, 6 мкг, 17 мкг).
Как продемонстрировано на фиг. 39, IL-15-ABD-IL-12 был более эффективным в отношении противоопухолевой активности по сравнению с IL-12-ABD в мышиной модели B16-F10 при одинаковой молярной концентрации. Другие аналогичные исследования in vivo показывают, что свободный IL-12 (5 мкг) в сочетании с IL-15 (1 мкг) является менее чем на 50% эффективнее, чем IL-15-ABD-IL-12 (6 мкг) (данные не показаны).
Пример 6. Анти-TGFβ-ABD
После биопэннинга и скрининга, проведенного с использованием технологии поверхностного плазмонного резонанса, идентифицировали анти-hTGFβ1-связывающие домены: 1A10, 1F11, 2H6, 4B9, 4C10, 4D9, 4G3, 4G6, 4H4, 4H7 и 6H11. Эти клоны проявляли перекрестную реактивность в отношении к hTGFβ2, 3 и mTGFβ1 и потенциальное ингибирование связывания hTGFβ1 с его рецептором II. Затем клоны выбирали для очистки в виде scFv и дальнейшей характеристики.
ScFv подвергали скринингу в отношении перекрестной реактивности в отношении к TGF-1 мыши и человека с использованием стандартных методик ИФА. Связывание при ИФА показывает, что 2H6, 4G3, 4H7, 4B9, 4D9 и 6H11 имеют высокую перекрестную реактивность по отношению как к mTGFβ-1, так и к hTGFβ-1.
Связывание и блокирование при ИФА проводили для определения того, способны ли анти-scFv TGFβ-1 связывать TGFβ-1 и блокировать его взаимодействие с TGFβR-II. Все из 2H6, 4G3, 4H7, 4B9 и 4D9 демонстрируют высокую эффективность блокирования и ингибирование взаимодействия mTGFβ и mTGFβR-II.
Некоторые из этих анти-scFv TGFβ-1 исследовали в отношении их способности нарушать биологическую активность TGFβ-1. Последовательности анти-scFv TGFβ 14H7 и 4D9 продемонстрированы на фиг. 40A и B.
Блокада TGFβ1-индуцированной экспансии CD4+Foxp3+ регуляторных Т-клеток
Регуляторные Т-клетки (Treg) способны влиять на гомеостаз иммунной системы. Такие Treg необходимы для поддержания аутотолерантности, поскольку дефекты могут приводить к тяжелым аутоиммунным заболеваниям. При раке опухолевые клетки способны секретировать цитокины, которые влияют на гомеостаз иммунной системы. В частности, опухолевые клетки могут секретировать TGFβ, который затем может влиять на количество циркулирующих Treg. Ранее было продемонстрировано, что воздействие TGFβ1 приводит к экспансии подсовокупностей CD4+Foxp3+ Treg из CD4+Foxp3- T-клеток. Эти индуцированные Treg затем могут способствовать индукции анергического ответа Т-клеток путем ингибирования активации опухолевых антигенспецифических цитотоксических CD8+ Т-клеток.
Как продемонстрировано на фиг. 41, рекомбинантный TGFβ1 способен стимулировать экспансию CD4+Foxp3+ Treg из смешанной популяции Т-клеток, выделенных из РВМС здоровых доноров-людей. В то же время блокада TGFβ всеми из анти-TGFβ 1D11 антителом или scFv TGF-β1 2H6, 4H7 и 4D9 значительно ингибирует индуцированную TGFβ экспансию CD4+Foxp3+ Treg также дозозависимым образом. Таким образом, такие TGFβ1scFv являются пригодными для уменьшения экспансии TReg при видах рака.
Блокада индуцированного TGFβ1 эпителиально-мезенхимального перехода (EMT)
Известно, что воздействие TGFβ индуцирует эпителиально-мезенхимальный переход. Во время этого процесса эпителиальные клетки трансдифференцируются из организованного, поляризованного и тесно связанного эпителиального слоя клеток с галькообразной морфологией в неорганизованные и подвижные клетки, которые по морфологии кажутся мезенхимальными. Во время EMT, инвазивная способность клеток активируется и, таким образом, усиливается онкогенная способность клеток. Е-кадгерин является широко используемым маркером эпителиальных клеток и локализуется в адгезионных контактах между эпителиальными клетками. Потеря E-cadherin является выраженным маркером EMT, который указывает на процесс трансдифференцировки. (фиг. 42А). Кроме того, виментин ассоциирован с высокоподвижными клетками. Таким образом, индукция экспрессии виментина в клетках также свидетельствует о повышенной подвижности и повышенной локализованной инвазии in vivo.
Как продемонстрировано на фиг. 42 и 43, блокада TGF-β анти-TGF-β 1D11 антителом или scFv TGF-β1 обращает индуцированный TGF-β1 эпителиально-мезенхимальный переход (фиг. 42) и миграцию (фиг. 43). Клетки мыши 4T1 культивировали в среде для роста с добавкой TGF-β1 (панель 2); TGF-β1 и 1D11 (панель 3); или TGF-β1 и анти-scFv TGF-β1 (панель 4), затем фиксировали и окрашивали антителом к E-кадгериун (зеленый цвет) и антителом к виментину (фиолетовый цвет). Ядра докрашивали DAPI (синий цвет). Обработка TGF-β1 индуцировала потерю E-кадгерина из межклеточных контактов и приводила к повышению экспрессии виментина. Указанный эффект обращается в результате добавления 1D11 или рассматриваемого анти-scFv TGF-β1, описанного в данном документе (фиг. 42, панели 3 и 4). Кроме того, анти-scFv TGF-β1, описанное в данном документе, способно блокировать опосредованную TGF-β1 миграцию клеток карциномы (фиг. 43).
Нейтрализация индуцированной ТGFβ1 активации Smad
Суперсемейство TGFβ состоит из плейотропных цитокинов, которые регулируют различные биологические процессы, в том числе пролиферацию, дифференцировку, миграцию, выживание клеток, ангиогенез, заживление ран и иммунный надзор. У людей преобладающей изоформой является TGFβ1, которая экспрессируется в различных типах тканей. TGFβ ингибирует пролиферацию большинства нормальных эпителиальных клеток. Кроме того, на ранних стадиях видов рака эпителиального происхождения TGFβ выступает в качестве ингибитора клеточного роста. Таким образом, во время начала рака TGFβ выступает в качестве супрессора опухоли. В то же время на более поздних стадиях прогрессирования рака опухолевые клетки становятся устойчивыми к эффектам ингибирующего роста TGFβ, и TGFβ выполняет роль промотора опухоли. Действительно, было показано, что TGFβ1 сверхэкспрессируется в различных опухолях. Активация пути с участием TGFβ происходит посредством связывания лиганда TGFβ с рецептором TGFβ II типа (TβRII), который затем индуцирует ассоциацию и олигомеризацию между TβRII и TβRI. Когда этот олигомер образуется, Smad2 и Smad3 подвергаются рекрутингу и фосфорилируются TβRI. Затем фосфорилированные Smad2 или Smad3 связываются с Smad4 в цитоплазме, и этот комплекс транслоцируется в ядро, где он взаимодействует с промоторной областью, и активирует транскрипцию генов-мишеней. Таким образом, активация пути с участием TGFβ измерима с помощью быстрого фосфорилирования Smad2 после добавления TGFβ к клеткам, лишенным сыворотки крови. В то же время эффективная блокада TGFβ будет ингибировать фосфорилирование Smad2. В данном документе отсутствие фосфорилирования Smad2 может быть использовано в качестве меры эффективной блокады TGFβ рассматриваемыми анти-scFv TGFβ конструкциями.
С помощью лишенных сыворотки крови клеток человека (фиг. 44А) или мыши (фиг. 44В) определяли, что добавление рекомбинантного TGFβ1 человека (фиг. 44А) или TGF-β1, -β2 и -β3 мыши (фиг. 44В) индуцировало фосфорилирование Smad2. Такое фосфорилирование снижается дозозависимым образом, когда TGFβ предварительно инкубируют с контрольным анти-TGFβ 1D11 антителом или конструкциями scFv TGF-β1 2H6, 4H7 и 4D9. Эти данные свидетельствуют о том, что конструкции scFv 2H6, 4H7 и 4D9 способны секвестрировать TGFβ1 и ингибировать его взаимодействие с TβRII/TβRI, тем самым, ингибируя каскад активации TGFβ во время поздней стадии рака.
Анти-TGFβ-1-ABD 4D9
Последовательности иллюстративных конструкций TGF-β1 scGv-ABD (4D9M-A6m и 4H7-A6m) показаны на фиг. 45A. Как продемонстрировано на фиг. 45B, анти-TGFβ-1-ABD продлевал T ½β анти-scFv TGFβ-1 со 106 минут до 10,6 часов.
Анти-scFv TGF-β1-ABD (бивалентные в случае TGF-β1), продуцируемый в клетках E. coli и HEK, оценивали в отношении связывания с мышиным сывороточным альбумином (фиг. 47). Оценивали три различные ориентации конструкций: две анти-scFv TGF-β1, присоединенные к N-концу ABD («Bi N Term»); две анти-scFv TGF-β1, присоединенные к C-концу ABD («Bi C Term»); или один анти-TGF-β1 scFv, присоединенный к каждому из N-конца и C-конца ABD («Bi Mid»). Как продемонстрировано на фиг. 46, все конструкции проявляли связывание с мышиным сывороточным альбумином. В отношении конструкций, продуцируемых в E. coli, Bi Mid проявляла наиболее эффективное связывание с MSA, чем ориентация N Term. В отношении конструкций, продуцируемых в клетках HEK, ориентация Bi N Term проявляла более эффективное связывание с MSA, чем ориентация Bi Mid или Bi C term.
Как продемонстрировано на фиг. 47, опосредованное TGFβ-1 ингибирование пролиферации Т-клеток обращали (т.е. повышение пролиферации Т-клеток) с помощью таких конструкций (фиг. 46B). Кроме того, было показано, что 4D9M-ABD блокирует связывание TGFβ-1 человека и TGFβ-3 человека с когнатными рецепторами (данные не показаны).
Все цитируемые ссылки явно включены в данный документ посредством ссылки в их полном объеме.
Принимая во внимание, что конкретные варианты осуществления изобретения были описаны выше в иллюстративных целях, специалистам в данной области техники будет понятно, что могут быть выполнены многочисленные вариации деталей, не отклоняясь от изобретения, описанного в прилагаемой формуле изобретения.
--->
SEQUENCE LISTING
<110> Sonnet Bio Therapeutics
<120> Albumin Binding Domain Fusion Proteins
<130> 116076-5007-WO
<140> PCT/US2018/000085
<141> 2018-02-20
<160> 310
<170> PatentIn version 3.5
<210> 1
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 1
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Phe
65 70 75 80
Leu Gln Met Asn Arg Leu Arg Pro Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 2
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 2
Gly Ile Thr Phe Asp Asp Tyr Val
1 5
<210> 3
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 3
Ile Ser Ser Asn Ser Gly Tyr Ile
1 5
<210> 4
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 4
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile
1 5 10 15
<210> 5
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 5
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 6
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 6
Asn Ile Gly Thr Lys Ser
1 5
<210> 7
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 7
Ala Asp Ser
1
<210> 8
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 8
Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val
1 5 10
<210> 9
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 9
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp His
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 10
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 10
Gly Ile Thr Phe Asp Asp His Val
1 5
<210> 11
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 11
Ile Ser Ser Asn Ser Gly Tyr Ile
1 5
<210> 12
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 12
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile
1 5 10 15
<210> 13
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 13
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 14
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 14
Asn Ile Gly Thr Lys Ser
1 5
<210> 15
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 15
Ala Asp Ser
1
<210> 16
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 16
Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val
1 5 10
<210> 17
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 17
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 18
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 18
Gly Ile Thr Phe Asp Asp Tyr Ala
1 5
<210> 19
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 19
Ile Ser Ser Asn Ser Gly Tyr Ile
1 5
<210> 20
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 20
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile
1 5 10 15
<210> 21
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 21
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 22
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 22
Asn Ile Gly Thr Lys Ser
1 5
<210> 23
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 23
Ala Asp Ser
1
<210> 24
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 24
Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val
1 5 10
<210> 25
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 25
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser
115 120
<210> 26
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 26
Gly Ile Thr Phe Asp Asp Ala Val
1 5
<210> 27
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 27
Ile Ser Ser Asn Ser Gly Tyr Ile
1 5
<210> 28
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 28
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile
1 5 10 15
<210> 29
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 29
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
1 5 10 15
Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val
20 25 30
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
35 40 45
Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His
85 90 95
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 30
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 30
Asn Ile Gly Thr Lys Ser
1 5
<210> 31
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 31
Ala Asp Ser
1
<210> 32
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 32
Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val
1 5 10
<210> 33
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 33
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Asn Ser Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Ala Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Phe Ser Val Asp Lys Ser Ile Asn Thr Ala Tyr
65 70 75 80
Leu Val Trp Thr Ser Leu Lys Ala Ser Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Gln Arg Trp Gly Ser Ser Ser Phe Asp Leu Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 34
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 34
Gly Tyr Ser Phe Asn Ser Tyr Trp
1 5
<210> 35
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 35
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 36
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 36
Ala Arg Gln Arg Trp Gly Ser Ser Ser Phe Asp Leu
1 5 10
<210> 37
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 37
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Glu Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Gly Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 38
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 38
Ser Ser Asn Ile Gly Asn Asn Tyr
1 5
<210> 39
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 39
Glu Asn Asn
1
<210> 40
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 40
Gly Thr Trp Asp Ser Ser Leu Ser Ala Gly Val
1 5 10
<210> 41
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 41
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Asn Ser Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Phe Ser Val Asp Lys Ser Ile Asn Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Ala Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Gln Arg Trp Gly Ser Ser Ser Phe Asp Ala Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 42
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 42
Gly Tyr Ser Phe Asn Ser Tyr Trp
1 5
<210> 43
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 43
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 44
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 44
Ala Arg Gln Arg Trp Gly Ser Ser Ser Phe Asp Ala
1 5 10
<210> 45
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 45
Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala Ala Pro Gly Gln
1 5 10 15
Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn
20 25 30
Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu
35 40 45
Ile Tyr Glu Asn Asn Lys Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser
50 55 60
Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln
65 70 75 80
Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu
85 90 95
Ser Ala Gly Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 46
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 46
Ser Ser Asn Ile Gly Asn Asn Tyr
1 5
<210> 47
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 47
Glu Asn Asn
1
<210> 48
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 48
Gly Thr Trp Asp Ser Ser Leu Ser Ala Gly Val
1 5 10
<210> 49
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 49
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Ser Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Glu Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 50
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 50
Gly Tyr Ser Phe Thr Ser Tyr Trp
1 5
<210> 51
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 51
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 52
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 52
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr
1 5 10
<210> 53
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 53
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Ile Ser Ser Leu Ala Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Glu Tyr Ser Cys Ser Ser Tyr Ala Gly Arg
85 90 95
Asn Ala Val Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 54
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 54
Ser Ser Leu Ala Gly Ser Tyr Asn Leu
1 5
<210> 55
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 55
Glu Val Thr
1
<210> 56
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 56
Ser Ser Tyr Ala Gly Arg Asn Ala Val Ala Val
1 5 10
<210> 57
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 57
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Ser Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Glu Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Ala Tyr Tyr Cys
85 90 95
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 58
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 58
Gly Tyr Ser Phe Thr Ser Tyr Trp
1 5
<210> 59
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 59
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 60
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 60
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr
1 5 10
<210> 61
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 61
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Ile Ser Ser Leu Ala Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Glu Tyr Ser Cys Ser Ser Tyr Ala Gly Arg
85 90 95
Asn Ala Val Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 62
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 62
Ser Ser Leu Ala Gly Ser Tyr Asn Leu
1 5
<210> 63
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 63
Glu Val Thr
1
<210> 64
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 64
Ser Ser Tyr Ala Gly Arg Asn Ala Val Ala Val
1 5 10
<210> 65
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 65
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Val Val Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Ser
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Ser Ser Gly Trp Tyr Pro Tyr Tyr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 66
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 66
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 67
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 67
Ile Ser Gly Ser Gly Gly Ser Thr
1 5
<210> 68
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 68
Ala Lys Ser Ser Gly Trp Tyr Pro Tyr Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 69
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 69
Ser Tyr Glu Leu Met Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asn Asn Leu Gly Asp Lys Tyr Thr
20 25 30
Ser Trp Tyr Gln His Lys Pro Gly Gln Ser Pro Val Met Val Ile Tyr
35 40 45
Gln Asp Thr Lys Arg Pro Ser Gly Ile Ala Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Leu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Thr Gly Thr Ala Val
85 90 95
Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105
<210> 70
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 70
Asn Leu Gly Asp Lys Tyr
1 5
<210> 71
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 71
Gln Asp Thr
1
<210> 72
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 72
Gln Ala Trp Asp Thr Gly Thr Ala Val
1 5
<210> 73
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 73
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Val Val Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Ser
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ala Tyr Tyr Cys
85 90 95
Ala Lys Ser Ser Gly Trp Tyr Pro Tyr Tyr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 74
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 74
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 75
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 75
Ile Ser Gly Ser Gly Gly Ser Thr
1 5
<210> 76
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 76
Ala Lys Ser Ser Gly Trp Tyr Pro Tyr Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 77
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 77
Ser Tyr Glu Leu Met Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asn Asn Leu Gly Asp Lys Tyr Thr
20 25 30
Ser Trp Tyr Gln His Lys Pro Gly Gln Ser Pro Val Met Val Ile Tyr
35 40 45
Gln Asp Thr Lys Arg Pro Ser Gly Ile Ala Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Leu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Thr Gly Thr Ala Val
85 90 95
Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105
<210> 78
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 78
Asn Leu Gly Asp Lys Tyr
1 5
<210> 79
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 79
Gln Asp Thr
1
<210> 80
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 80
Gln Ala Trp Asp Thr Gly Thr Ala Val
1 5
<210> 81
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 81
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Gln Ala Phe Gly Tyr Asn Phe Asn Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Gln Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Arg Ser Thr Val Thr Lys Ser Phe Asp Ile Trp Gly Gln Gly
100 105 110
Thr Met Val Thr Val Ser Ser
115
<210> 82
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 82
Gly Tyr Asn Phe Asn Asn Tyr Trp
1 5
<210> 83
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 83
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 84
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 84
Ala Arg Arg Ser Thr Val Thr Lys Ser Phe Asp Ile
1 5 10
<210> 85
<211> 111
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 85
Gln Ala Gly Leu Thr Gln Pro Pro Ser Val Ser Lys Gly Leu Arg Gln
1 5 10 15
Thr Ala Thr Leu Thr Cys Thr Gly Asn Ser Asn Asn Val Gly Lys Gln
20 25 30
Gly Ala Ala Trp Leu Gln Gln His Gln Gly His Pro Pro Lys Leu Leu
35 40 45
Ser Tyr Ser Ser Ile Asn Arg Pro Val Gly Ile Ser Glu Arg Phe Ser
50 55 60
Ala Ser Arg Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Leu Gln
65 70 75 80
Pro Glu Asp Glu Ala Asp Tyr Tyr Cys Ser Ala Trp Asp Ser Ser Leu
85 90 95
Ser Ala Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
<210> 86
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 86
Ser Asn Asn Val Gly Lys Gln Gly
1 5
<210> 87
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 87
Ser Ser Ile
1
<210> 88
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 88
Ser Ala Trp Asp Ser Ser Leu Ser Ala Trp Val
1 5 10
<210> 89
<211> 130
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 89
Gln Val Gln Leu Gln Glu Ser Gly Pro Gly Leu Val Lys Pro Ser Glu
1 5 10 15
Thr Leu Ser Leu Thr Cys Thr Val Ser Gly Gly Ser Ile Ser Ser Ser
20 25 30
Ser Tyr Tyr Trp Gly Trp Ile Arg Gln Pro Pro Gly Lys Gly Leu Glu
35 40 45
Trp Ile Gly Ser Ile Tyr Tyr Ser Gly Ser Thr Tyr Tyr Asn Pro Ser
50 55 60
Leu Lys Ser Arg Val Thr Ile Ser Val Asp Thr Ser Lys Asn Gln Phe
65 70 75 80
Ser Leu Lys Leu Ser Ser Val Thr Ala Ala Asp Thr Ala Val Tyr Tyr
85 90 95
Cys Ala Arg Arg Ser Ala Trp Ser Ser Gly Trp Ser Thr Leu Arg Tyr
100 105 110
Tyr Tyr Tyr Gly Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val
115 120 125
Ser Ser
130
<210> 90
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 90
Gly Gly Ser Ile Ser Ser Ser Ser Tyr Tyr
1 5 10
<210> 91
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 91
Ile Tyr Tyr Ser Gly Ser Thr
1 5
<210> 92
<211> 22
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 92
Ala Arg Arg Ser Ala Trp Ser Ser Gly Trp Ser Thr Leu Arg Tyr Tyr
1 5 10 15
Tyr Tyr Gly Met Asp Val
20
<210> 93
<211> 109
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 93
Glu Ile Val Leu Thr Gln Ser Pro Ala Thr Leu Ser Leu Ser Pro Gly
1 5 10 15
Glu Arg Ala Thr Leu Ser Cys Arg Ala Ser Gln Ser Ile Ala Thr Tyr
20 25 30
Leu Ala Trp Tyr Gln Gln Asn Pro Gly Gln Ala Pro Arg Leu Leu Ile
35 40 45
Tyr Asp Ala Ser Thr Arg Ala Thr Gly Ile Pro Ala Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Glu Pro
65 70 75 80
Glu Asp Phe Ala Val Tyr Tyr Cys Gln Glu Arg Ser Asn Trp Ser Arg
85 90 95
Leu Thr Phe Gly Gly Gly Thr Lys Val Glu Ile Lys Arg
100 105
<210> 94
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 94
Gln Ser Ile Ala Thr Tyr
1 5
<210> 95
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 95
Asp Ala Ser
1
<210> 96
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 96
Gln Glu Arg Ser Asn Trp Ser Arg Leu Thr
1 5 10
<210> 97
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 97
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Val Pro Gly Arg Gly Leu Glu Trp Leu
35 40 45
Ala Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Glu Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 98
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 98
Gly Tyr Ser Phe Thr Asn Tyr Trp
1 5
<210> 99
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 99
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 100
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 100
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr
1 5 10
<210> 101
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 101
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Ile Ser Ser Leu Ala Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Glu Tyr Ser Cys Ser Ser Tyr Ala Gly Arg
85 90 95
Asn Ala Val Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 102
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 102
Ser Ser Leu Ala Gly Ser Tyr Asn Leu
1 5
<210> 103
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 103
Glu Val Thr
1
<210> 104
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 104
Ser Ser Tyr Ala Gly Arg Asn Ala Val Ala Val
1 5 10
<210> 105
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 105
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Glu Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Glu Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 106
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 106
Gly Tyr Ser Phe Thr Asn Tyr Trp
1 5
<210> 107
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 107
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 108
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 108
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr
1 5 10
<210> 109
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 109
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Ile Ser Ser Leu Ala Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Glu Tyr Ser Cys Ser Ser Tyr Ala Gly Arg
85 90 95
Asn Ala Val Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 110
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 110
Ser Ser Leu Ala Gly Ser Tyr Asn Leu
1 5
<210> 111
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 111
Glu Val Thr
1
<210> 112
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 112
Ser Ser Tyr Ala Gly Arg Asn Ala Val Ala Val
1 5 10
<210> 113
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 113
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Glu Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Glu Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Asn Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 114
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 114
Gly Tyr Ser Phe Thr Asn Tyr Trp
1 5
<210> 115
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 115
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 116
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 116
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr
1 5 10
<210> 117
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 117
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Ile Ser Ser Leu Ala Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Glu Tyr Ser Cys Ser Ser Tyr Ala Gly Arg
85 90 95
Asn Ala Val Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 118
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 118
Ser Ser Leu Ala Gly Ser Tyr Asn Leu
1 5
<210> 119
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 119
Glu Val Thr
1
<210> 120
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 120
Ser Ser Tyr Ala Gly Arg Asn Ala Val Ala Val
1 5 10
<210> 121
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 121
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Val Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Gly Ser Ser Asn Ser Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ala Arg Asp Asn Ala Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Val Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His His Gly Arg Leu Tyr Tyr Tyr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 122
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 122
Gly Phe Thr Phe Ser Asn Tyr Ala
1 5
<210> 123
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 123
Ile Ser Gly Ser Ser Asn Ser Ile
1 5
<210> 124
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 124
Ala Arg His His Gly Arg Leu Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 125
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 125
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Lys Leu Gly Glu Lys Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Met Val Ile Tyr
35 40 45
Gln Asp Arg Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Met
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Thr Thr Thr Pro Gly
85 90 95
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly
100 105
<210> 126
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 126
Lys Leu Gly Glu Lys Tyr
1 5
<210> 127
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 127
Gln Asp Arg
1
<210> 128
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 128
Gln Ala Trp Asp Thr Thr Thr Pro Gly
1 5
<210> 129
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 129
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Tyr
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val
50 55 60
Arg Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Val Asn Arg Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg His His Gly Arg Leu Tyr Tyr Tyr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 130
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 130
Gly Ile Thr Phe Asp Asp Tyr Val
1 5
<210> 131
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 131
Ile Ser Ser Asn Ser Gly Tyr Ile
1 5
<210> 132
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 132
Ala Arg His His Gly Arg Leu Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 133
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 133
Ser Tyr Glu Leu Thr Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Ser Ile Thr Cys Ser Gly Asp Lys Leu Gly Glu Lys Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ser Pro Val Met Val Ile Tyr
35 40 45
Gln Asp Arg Lys Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Met
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Thr Thr Thr Pro Gly
85 90 95
Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly
100 105
<210> 134
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 134
Lys Leu Gly Glu Lys Tyr
1 5
<210> 135
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 135
Gln Asp Arg
1
<210> 136
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 136
Gln Ala Trp Asp Thr Thr Thr Pro Gly
1 5
<210> 137
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 137
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Ser Ser Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Glu Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 138
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 138
Gly Tyr Ser Phe Ser Ser Tyr Trp
1 5
<210> 139
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 139
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 140
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 140
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr
1 5 10
<210> 141
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 141
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Ile Ser Ser Leu Ala Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Glu Tyr Ser Cys Ser Ser Tyr Ala Gly Arg
85 90 95
Asn Ala Val Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 142
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 142
Ser Ser Leu Ala Gly Ser Tyr Asn Leu
1 5
<210> 143
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 143
Glu Val Thr
1
<210> 144
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 144
Ser Ser Tyr Ala Gly Arg Asn Ala Val Ala Val
1 5 10
<210> 145
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 145
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Asn Phe Asn Ser Tyr
20 25 30
Trp Ile Ala Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Gly Pro Pro Phe
50 55 60
Lys Gly Gln Ala Thr Ile Ser Ala Asp Lys Ser Ile Asn Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Arg Pro Ser Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Leu Val Gly Glu Gly Arg Ser Ile Gln Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 146
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 146
Gly Tyr Asn Phe Asn Ser Tyr Trp
1 5
<210> 147
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 147
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 148
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 148
Ala Arg Leu Val Gly Glu Gly Arg Ser Ile Gln Tyr
1 5 10
<210> 149
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 149
Gln Pro Val Leu Thr Gln Pro Pro Ser Ala Ser Ala Ser Leu Gly Ala
1 5 10 15
Ser Val Thr Leu Thr Cys Thr Leu Thr Ser Gly Tyr Arg Asn Tyr Lys
20 25 30
Val Asp Trp Tyr Gln Gln Arg Pro Gly Lys Gly Pro Arg Phe Val Met
35 40 45
Arg Val Gly Thr Gly Gly Ile Val Gly Ser Lys Gly Asp Gly Ile Pro
50 55 60
Asp Arg Phe Ser Ala Leu Gly Ser Gly Leu Asn Arg Tyr Leu Thr Ile
65 70 75 80
Lys Asp Ile Gln Glu Glu Asp Glu Ser Asp Tyr Tyr Cys Gly Ala Asp
85 90 95
Tyr Gly Ser Gly Ser Asn Phe Leu Val Val Phe Gly Gly Gly Thr Lys
100 105 110
Leu Thr Val Leu Gly
115
<210> 150
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 150
Ser Gly Tyr Arg Asn Tyr Lys
1 5
<210> 151
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 151
Val Gly Thr Gly Gly Ile Val Gly
1 5
<210> 152
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 152
Gly Ala Asp Tyr Gly Ser Gly Ser Asn Phe Leu Val Val
1 5 10
<210> 153
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 153
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Ala Ser Gly Tyr Asp Phe Thr Arg Tyr
20 25 30
Trp Ile Val Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Tyr Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Gly Pro Pro Phe
50 55 60
Lys Gly Gln Ala Thr Ile Ser Ala Asp Lys Ser Ile Asn Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Arg Pro Ser Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Arg Leu Val Gly Glu Gly Arg Ser Ile Gln Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 154
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 154
Gly Tyr Asp Phe Thr Arg Tyr Trp
1 5
<210> 155
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 155
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 156
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 156
Ala Arg Leu Val Gly Glu Gly Arg Ser Ile Gln Tyr
1 5 10
<210> 157
<211> 117
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 157
Gln Pro Val Leu Thr Gln Pro Pro Ser Ala Ser Ala Ser Leu Gly Ala
1 5 10 15
Ser Val Thr Leu Thr Cys Thr Leu Thr Ser Gly Tyr Arg Asn Tyr Lys
20 25 30
Val Asp Trp Tyr Gln Gln Arg Pro Gly Lys Gly Pro Arg Phe Val Met
35 40 45
Arg Val Gly Thr Gly Gly Ile Val Gly Ser Lys Gly Asp Gly Ile Pro
50 55 60
Asp Arg Phe Ser Ala Leu Gly Ser Gly Leu Asn Arg Tyr Leu Thr Ile
65 70 75 80
Lys Asp Ile Gln Glu Glu Asp Glu Ser Asp Tyr Tyr Cys Gly Ala Asp
85 90 95
Tyr Gly Ser Gly Ser Asn Phe Leu Val Val Phe Gly Gly Gly Thr Lys
100 105 110
Val Thr Val Leu Gly
115
<210> 158
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 158
Ser Gly Tyr Arg Asn Tyr Lys
1 5
<210> 159
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 159
Val Gly Thr Gly Gly Ile Val Gly
1 5
<210> 160
<211> 13
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 160
Gly Ala Asp Tyr Gly Ser Gly Ser Asn Phe Leu Val Val
1 5 10
<210> 161
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 161
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Leu Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Arg Ser
20 25 30
Trp Ile Ala Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Glu Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 162
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 162
Gly Tyr Ser Phe Thr Arg Ser Trp
1 5
<210> 163
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 163
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 164
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 164
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr
1 5 10
<210> 165
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 165
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Ile Ser Ser Leu Ala Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Glu Tyr Ser Cys Ser Ser Tyr Ala Gly Arg
85 90 95
Asn Ala Val Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 166
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 166
Ser Ser Leu Ala Gly Ser Tyr Asn Leu
1 5
<210> 167
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 167
Glu Val Thr
1
<210> 168
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 168
Ser Ser Tyr Ala Gly Arg Asn Ala Val Ala Val
1 5 10
<210> 169
<211> 119
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 169
Glu Val Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu
1 5 10 15
Ser Pro Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Thr Asn Tyr
20 25 30
Trp Ile Gly Trp Val Arg Gln Met Pro Gly Glu Gly Leu Glu Trp Met
35 40 45
Gly Ile Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe
50 55 60
Glu Gly Gln Val Thr Ile Ser Ala Asp Lys Ser Ile Ser Thr Ala Tyr
65 70 75 80
Leu Gln Trp Ser Ser Leu Lys Ala Ser Asp Thr Ala Met Tyr Tyr Cys
85 90 95
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr Trp Gly Gln Gly
100 105 110
Thr Leu Val Thr Val Ser Ser
115
<210> 170
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 170
Gly Tyr Ser Phe Thr Asn Tyr Trp
1 5
<210> 171
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 171
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 172
<211> 12
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 172
Ala Arg Leu His Gly Val Val Ser Arg Gly Asp Tyr
1 5 10
<210> 173
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 173
Gln Ser Ala Leu Thr Gln Pro Ala Ser Val Ser Gly Ser Pro Gly Gln
1 5 10 15
Ser Ile Thr Ile Ser Cys Ser Gly Ile Ser Ser Leu Ala Gly Ser Tyr
20 25 30
Asn Leu Val Ser Trp Tyr Gln Gln Tyr Pro Gly Lys Ala Pro Lys Leu
35 40 45
Ile Ile Tyr Glu Val Thr Lys Arg Pro Ser Gly Val Ser Asn Arg Phe
50 55 60
Ser Gly Ser Lys Ser Gly Asn Thr Ala Ser Leu Thr Ile Ser Gly Leu
65 70 75 80
Gln Ala Asp Asp Glu Ala Glu Tyr Ser Cys Ser Ser Tyr Ala Gly Arg
85 90 95
Asn Ala Val Ala Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105 110
<210> 174
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 174
Ser Ser Leu Ala Gly Ser Tyr Asn Leu
1 5
<210> 175
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 175
Glu Val Thr
1
<210> 176
<211> 11
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 176
Ser Ser Tyr Ala Gly Arg Asn Ala Val Ala Val
1 5 10
<210> 177
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 177
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Val Val Arg Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Asp Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Ser
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Ser Ser Gly Trp Tyr Pro Tyr Tyr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 178
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 178
Gly Phe Thr Phe Asp Asp Tyr Ala
1 5
<210> 179
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 179
Ile Ser Gly Ser Gly Gly Ser Thr
1 5
<210> 180
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 180
Ala Lys Ser Ser Gly Trp Tyr Pro Tyr Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 181
<211> 107
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 181
Ser Tyr Glu Leu Met Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asn Asn Leu Gly Asp Lys Tyr Thr
20 25 30
Ser Trp Tyr Gln His Lys Pro Gly Gln Ser Pro Val Met Val Ile Tyr
35 40 45
Gln Asp Thr Lys Arg Pro Ser Gly Ile Ala Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Leu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Thr Gly Thr Ala Val
85 90 95
Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly
100 105
<210> 182
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 182
Asn Leu Gly Asp Lys Tyr
1 5
<210> 183
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 183
Gln Asp Thr
1
<210> 184
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 184
Gln Ala Trp Asp Thr Gly Thr Ala Val
1 5
<210> 185
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 185
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Val Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asn Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Gly Ile Ser Gly Ser Ser Gly Ser Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ala Arg Asp Asn Ala Lys Asn Thr Leu Phe
65 70 75 80
Leu Gln Val Asn Ser Leu Arg Ala Glu Asp Thr Ala Leu Tyr Tyr Cys
85 90 95
Ala Arg His His Gly Arg Leu Tyr Tyr Tyr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 186
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 186
Gly Phe Thr Phe Ser Asn Tyr Ala
1 5
<210> 187
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 187
Ile Ser Gly Ser Ser Gly Ser Ile
1 5
<210> 188
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 188
Ala Arg His His Gly Arg Leu Tyr Tyr Tyr Tyr Gly Met Asp Val
1 5 10 15
<210> 189
<211> 108
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 189
Ser Tyr Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala Leu Gly Gln
1 5 10 15
Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu Arg Ser Tyr Tyr Ala
20 25 30
Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Ile Tyr
35 40 45
Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser
50 55 60
Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Ala Gln Ala Glu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Asp Ser Ser Gly Thr Val
85 90 95
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105
<210> 190
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 190
Ser Leu Arg Ser Tyr Tyr
1 5
<210> 191
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 191
Gly Lys Asn
1
<210> 192
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 192
Asn Ser Arg Asp Ser Ser Gly Thr Val Val
1 5 10
<210> 193
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 193
Glu Val Gln Leu Val Glu Thr Gly Gly Gly Val Val Arg Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Ser
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Ser Ser Gly Trp Tyr Pro Tyr Tyr Tyr Gly Met Asp Val Trp
100 105 110
Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120
<210> 194
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 194
Gly Tyr Ser Phe Thr Ser Tyr Trp
1 5
<210> 195
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 195
Ile Tyr Pro Gly Asp Ser Asp Thr
1 5
<210> 196
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 196
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile
1 5 10 15
<210> 197
<211> 110
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 197
Ser Tyr Glu Leu Met Gln Pro Pro Ser Val Ser Val Ser Pro Gly Gln
1 5 10 15
Thr Ala Arg Ile Thr Cys Ser Gly Asn Asn Leu Gly Asp Lys Tyr Thr
20 25 30
Ser Trp Tyr Gln His Lys Pro Gly Gln Ser Pro Val Met Val Ile Tyr
35 40 45
Gln Asp Thr Lys Arg Pro Ser Gly Ile Ala Glu Arg Phe Ser Gly Ser
50 55 60
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Gly Thr Gln Ala Leu
65 70 75 80
Asp Glu Ala Asp Tyr Tyr Cys Gln Ala Trp Asp Thr Gly Thr Ala Val
85 90 95
Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly Leu Gly Gly
100 105 110
<210> 198
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 198
Asn Leu Gly Asp Lys Tyr
1 5
<210> 199
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 199
Gln Asp Thr
1
<210> 200
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 200
Gln Ala Trp Asp Thr Gly Thr Ala Val
1 5
<210> 201
<211> 253
<212> PRT
<213> Artificial Sequence
<220>
<223> A10m3 amino acid sequence
<400> 201
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser
130 135 140
Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr
145 150 155 160
Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His
165 170 175
Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala
180 185 190
Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn
195 200 205
Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp
210 215 220
Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu
225 230 235 240
Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
245 250
<210> 202
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy chain
<400> 202
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala
20 25 30
Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr
115 120 125
<210> 203
<211> 113
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light chain
<400> 203
Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala
1 5 10 15
Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr
20 25 30
Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu
35 40 45
Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val
50 55 60
Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val
65 70 75 80
Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg
85 90 95
Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
100 105 110
Gly
<210> 204
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Parental IL15
<400> 204
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Asn
100 105 110
Thr Ser
<210> 205
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15 K86A
<400> 205
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Ala Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Asn
100 105 110
Thr Ser
<210> 206
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15 K86R
<400> 206
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Arg Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Asn
100 105 110
Thr Ser
<210> 207
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15 N112A
<400> 207
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Ala
100 105 110
Thr Ser
<210> 208
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15 N112S
<400> 208
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Ser
100 105 110
Thr Ser
<210> 209
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15 N112Q
<400> 209
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Gln
100 105 110
Thr Ser
<210> 210
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15 K86R/N112A
<400> 210
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Arg Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Ala
100 105 110
Thr Ser
<210> 211
<211> 392
<212> PRT
<213> Artificial Sequence
<220>
<223> Parental IL15-A10m3
<400> 211
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Asn
100 105 110
Thr Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val
130 135 140
Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser
145 150 155 160
Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val
165 170 175
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser
180 185 190
Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
195 200 205
Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg
210 215 220
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr
225 230 235 240
Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met
245 250 255
Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly
260 265 270
Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln
275 280 285
Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys
290 295 300
Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys
305 310 315 320
Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro
325 330 335
Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala
340 345 350
Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr
355 360 365
Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly
370 375 380
Gly Thr Lys Leu Thr Val Leu Gly
385 390
<210> 212
<211> 392
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15 K86A -A10m3 K86A
<400> 212
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Ala Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Asn
100 105 110
Thr Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val
130 135 140
Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser
145 150 155 160
Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val
165 170 175
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser
180 185 190
Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
195 200 205
Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg
210 215 220
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr
225 230 235 240
Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met
245 250 255
Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly
260 265 270
Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln
275 280 285
Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys
290 295 300
Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys
305 310 315 320
Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro
325 330 335
Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala
340 345 350
Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr
355 360 365
Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly
370 375 380
Gly Thr Lys Leu Thr Val Leu Gly
385 390
<210> 213
<211> 392
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15 K86R -A10m3
<400> 213
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Arg Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Asn
100 105 110
Thr Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val
130 135 140
Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser
145 150 155 160
Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val
165 170 175
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser
180 185 190
Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
195 200 205
Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg
210 215 220
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr
225 230 235 240
Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met
245 250 255
Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly
260 265 270
Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln
275 280 285
Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys
290 295 300
Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys
305 310 315 320
Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro
325 330 335
Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala
340 345 350
Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr
355 360 365
Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly
370 375 380
Gly Thr Lys Leu Thr Val Leu Gly
385 390
<210> 214
<211> 392
<212> PRT
<213> Artificial Sequence
<220>
<223> IL15R K86R/ N112A -A10m3
<400> 214
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Arg Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Ala
100 105 110
Thr Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val
130 135 140
Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser
145 150 155 160
Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val
165 170 175
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser
180 185 190
Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
195 200 205
Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg
210 215 220
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr
225 230 235 240
Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met
245 250 255
Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly
260 265 270
Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln
275 280 285
Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys
290 295 300
Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys
305 310 315 320
Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro
325 330 335
Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala
340 345 350
Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr
355 360 365
Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly
370 375 380
Gly Thr Lys Leu Thr Val Leu Gly
385 390
<210> 215
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> IL-15 ubiquitination sites
<400> 215
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr
35
<210> 216
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> IL-15 ubiquintination sites
<400> 216
Ala Met Lys Cys Phe Leu Leu Glu Leu Gln Val Ile Ser Leu Glu Ser
1 5 10 15
Gly Asp Ala Ser Ile His Asp Thr Val Glu Asn Leu Ile Ile Leu Ala
20 25 30
Asn Asn Ser Leu Ser Ser
35
<210> 217
<211> 38
<212> PRT
<213> Artificial Sequence
<220>
<223> IL-15 ubiquitination sites
<400> 217
Asn Gly Asn Val Thr Glu Ser Gly Cys Lys Glu Cys Glu Glu Leu Glu
1 5 10 15
Glu Lys Asn Ile Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln
20 25 30
Met Phe Ile Asn Thr Ser
35
<210> 218
<211> 799
<212> PRT
<213> Artificial Sequence
<220>
<223> mIL12sc-A10m3
<400> 218
Met Trp Glu Leu Glu Lys Asp Val Tyr Val Val Glu Val Asp Trp Thr
1 5 10 15
Pro Asp Ala Pro Gly Glu Thr Val Asn Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Asp Ile Thr Trp Thr Ser Asp Gln Arg His Gly Val Ile Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Thr Val Lys Glu Phe Leu Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Thr Leu Ser His Ser His Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asn Gly Ile Trp Ser Thr Glu Ile Leu Lys
85 90 95
Asn Phe Lys Asn Lys Thr Phe Leu Lys Cys Glu Ala Pro Asn Tyr Ser
100 105 110
Gly Arg Phe Thr Cys Ser Trp Leu Val Gln Arg Asn Met Asp Leu Lys
115 120 125
Phe Asn Ile Lys Ser Ser Ser Ser Ser Pro Asp Ser Arg Ala Val Thr
130 135 140
Cys Gly Met Ala Ser Leu Ser Ala Glu Lys Val Thr Leu Asp Gln Arg
145 150 155 160
Asp Tyr Glu Lys Tyr Ser Val Ser Cys Gln Glu Asp Val Thr Cys Pro
165 170 175
Thr Ala Glu Glu Thr Leu Pro Ile Glu Leu Ala Leu Glu Ala Arg Gln
180 185 190
Gln Asn Lys Tyr Glu Asn Tyr Ser Thr Ser Phe Phe Ile Arg Asp Ile
195 200 205
Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Met Lys Pro Leu Lys Asn
210 215 220
Ser Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Ser Trp Ser Thr Pro
225 230 235 240
His Ser Tyr Phe Ser Leu Lys Phe Phe Val Arg Ile Gln Arg Lys Lys
245 250 255
Glu Lys Met Lys Glu Thr Glu Glu Gly Cys Asn Gln Lys Gly Ala Phe
260 265 270
Leu Val Glu Lys Thr Ser Thr Glu Val Gln Cys Lys Gly Gly Asn Val
275 280 285
Cys Val Gln Ala Gln Asp Arg Tyr Tyr Asn Ser Ser Cys Ser Lys Trp
290 295 300
Ala Cys Val Pro Cys Arg Val Arg Ser Gly Gly Gly Gly Ser Gly Gly
305 310 315 320
Gly Gly Ser Gly Gly Gly Gly Ser Arg Val Ile Pro Val Ser Gly Pro
325 330 335
Ala Arg Cys Leu Ser Gln Ser Arg Asn Leu Leu Lys Thr Thr Asp Asp
340 345 350
Met Val Lys Thr Ala Arg Glu Lys Leu Lys His Tyr Ser Cys Thr Ala
355 360 365
Glu Asp Ile Asp His Glu Asp Ile Thr Arg Asp Gln Thr Ser Thr Leu
370 375 380
Lys Thr Cys Leu Pro Leu Glu Leu His Lys Asn Glu Ser Cys Leu Ala
385 390 395 400
Thr Arg Glu Thr Ser Ser Thr Thr Arg Gly Ser Cys Leu Pro Pro Gln
405 410 415
Lys Thr Ser Leu Met Met Thr Leu Cys Leu Gly Ser Ile Tyr Glu Asp
420 425 430
Leu Lys Met Tyr Gln Thr Glu Phe Gln Ala Ile Asn Ala Ala Leu Gln
435 440 445
Asn His Asn His Gln Gln Ile Ile Leu Asp Lys Gly Met Leu Val Ala
450 455 460
Ile Asp Glu Leu Met Gln Ser Leu Asn His Asn Gly Glu Thr Leu Arg
465 470 475 480
Gln Lys Pro Pro Val Gly Glu Ala Asp Pro Tyr Arg Val Lys Met Lys
485 490 495
Leu Cys Ile Leu Leu His Ala Phe Ser Thr Arg Val Val Thr Ile Asn
500 505 510
Arg Val Met Gly Tyr Leu Ser Ser Ala Gly Gly Gly Gly Ser Gly Gly
515 520 525
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
530 535 540
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro
545 550 555 560
Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp
565 570 575
Asp Ala Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
580 585 590
Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp
595 600 605
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
610 615 620
Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr
625 630 635 640
Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp
645 650 655
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly
660 665 670
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His
675 680 685
Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly
690 695 700
Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser
705 710 715 720
Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val
725 730 735
Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly
740 745 750
Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala
755 760 765
Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp
770 775 780
His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
785 790 795
<210> 219
<211> 788
<212> PRT
<213> Artificial Sequence
<220>
<223> Human IL-12sc-A10m3
<400> 219
Ile Trp Glu Leu Lys Lys Asp Val Tyr Val Val Glu Leu Asp Trp Tyr
1 5 10 15
Pro Asp Ala Pro Gly Glu Met Val Val Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln Ser Ser Glu Val Leu Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Gln Val Lys Glu Phe Gly Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Val Leu Ser His Ser Leu Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asp Gly Ile Trp Ser Thr Asp Ile Leu Lys
85 90 95
Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe Leu Arg Cys Glu Ala Lys
100 105 110
Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp Leu Thr Thr Ile Ser Thr
115 120 125
Asp Leu Thr Phe Ser Val Lys Ser Ser Arg Gly Ser Ser Asp Pro Gln
130 135 140
Gly Val Thr Cys Gly Ala Ala Thr Leu Ser Ala Glu Arg Val Arg Gly
145 150 155 160
Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu Cys Gln Glu Asp Ser Ala
165 170 175
Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile Glu Val Met Val Asp Ala
180 185 190
Val His Lys Leu Lys Tyr Glu Asn Tyr Thr Ser Ser Phe Phe Ile Arg
195 200 205
Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Leu Lys Pro Leu
210 215 220
Lys Asn Ser Arg Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Thr Trp
225 230 235 240
Ser Thr Pro His Ser Tyr Phe Ser Leu Thr Phe Cys Val Gln Val Gln
245 250 255
Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg Val Phe Thr Asp Lys Thr
260 265 270
Ser Ala Thr Val Ile Cys Arg Lys Asn Ala Ser Ile Ser Val Arg Ala
275 280 285
Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser Glu Trp Ala Ser Val Pro
290 295 300
Cys Ser Gly Gly Gly Gly Gly Gly Ser Arg Asn Leu Pro Val Ala Thr
305 310 315 320
Pro Asp Pro Gly Met Phe Pro Cys Leu His His Ser Gln Asn Leu Leu
325 330 335
Arg Ala Val Ser Asn Met Leu Gln Lys Ala Arg Gln Thr Leu Glu Phe
340 345 350
Tyr Pro Cys Thr Ser Glu Glu Ile Asp His Glu Asp Ile Thr Lys Asp
355 360 365
Lys Thr Ser Thr Val Glu Ala Cys Leu Pro Leu Glu Leu Thr Lys Asn
370 375 380
Glu Ser Cys Leu Asn Ser Arg Glu Thr Ser Phe Ile Thr Asn Gly Ser
385 390 395 400
Cys Leu Ala Ser Arg Lys Thr Ser Phe Met Met Ala Leu Cys Leu Ser
405 410 415
Ser Ile Tyr Glu Asp Leu Lys Met Tyr Gln Val Glu Phe Lys Thr Met
420 425 430
Asn Ala Lys Leu Leu Met Asp Pro Lys Arg Gln Ile Phe Leu Asp Gln
435 440 445
Asn Met Leu Ala Val Ile Asp Glu Leu Met Gln Ala Leu Asn Phe Asn
450 455 460
Ser Glu Thr Val Pro Gln Lys Ser Ser Leu Glu Glu Pro Asp Phe Tyr
465 470 475 480
Lys Thr Lys Ile Lys Leu Cys Ile Leu Leu His Ala Phe Arg Ile Arg
485 490 495
Ala Val Thr Ile Asp Arg Val Thr Ser Tyr Leu Asn Ala Ser Gly Gly
500 505 510
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
515 520 525
Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
530 535 540
Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser
545 550 555 560
Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln Ala Pro
565 570 575
Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr
580 585 590
Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
595 600 605
Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu
610 615 620
Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg
625 630 635 640
Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser
645 650 655
Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
660 665 670
Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val
675 680 685
Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn
690 695 700
Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
705 710 715 720
Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro
725 730 735
Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile
740 745 750
Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp
755 760 765
Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu
770 775 780
Thr Val Leu Gly
785
<210> 220
<211> 938
<212> PRT
<213> Artificial Sequence
<220>
<223> hIL15RA-A10m3-mIL-12sc
<400> 220
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Arg Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Ala
100 105 110
Thr Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val
130 135 140
Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser
145 150 155 160
Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val
165 170 175
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser
180 185 190
Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
195 200 205
Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg
210 215 220
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr
225 230 235 240
Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met
245 250 255
Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly
260 265 270
Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln
275 280 285
Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys
290 295 300
Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys
305 310 315 320
Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro
325 330 335
Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala
340 345 350
Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr
355 360 365
Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly
370 375 380
Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Gly Ser Gly Gly Gly
385 390 395 400
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
405 410 415
Ser Met Trp Glu Leu Glu Lys Asp Val Tyr Val Val Glu Val Asp Trp
420 425 430
Thr Pro Asp Ala Pro Gly Glu Thr Val Asn Leu Thr Cys Asp Thr Pro
435 440 445
Glu Glu Asp Asp Ile Thr Trp Thr Ser Asp Gln Arg His Gly Val Ile
450 455 460
Gly Ser Gly Lys Thr Leu Thr Ile Thr Val Lys Glu Phe Leu Asp Ala
465 470 475 480
Gly Gln Tyr Thr Cys His Lys Gly Gly Glu Thr Leu Ser His Ser His
485 490 495
Leu Leu Leu His Lys Lys Glu Asn Gly Ile Trp Ser Thr Glu Ile Leu
500 505 510
Lys Asn Phe Lys Asn Lys Thr Phe Leu Lys Cys Glu Ala Pro Asn Tyr
515 520 525
Ser Gly Arg Phe Thr Cys Ser Trp Leu Val Gln Arg Asn Met Asp Leu
530 535 540
Lys Phe Asn Ile Lys Ser Ser Ser Ser Ser Pro Asp Ser Arg Ala Val
545 550 555 560
Thr Cys Gly Met Ala Ser Leu Ser Ala Glu Lys Val Thr Leu Asp Gln
565 570 575
Arg Asp Tyr Glu Lys Tyr Ser Val Ser Cys Gln Glu Asp Val Thr Cys
580 585 590
Pro Thr Ala Glu Glu Thr Leu Pro Ile Glu Leu Ala Leu Glu Ala Arg
595 600 605
Gln Gln Asn Lys Tyr Glu Asn Tyr Ser Thr Ser Phe Phe Ile Arg Asp
610 615 620
Ile Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Met Lys Pro Leu Lys
625 630 635 640
Asn Ser Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Ser Trp Ser Thr
645 650 655
Pro His Ser Tyr Phe Ser Leu Lys Phe Phe Val Arg Ile Gln Arg Lys
660 665 670
Lys Glu Lys Met Lys Glu Thr Glu Glu Gly Cys Asn Gln Lys Gly Ala
675 680 685
Phe Leu Val Glu Lys Thr Ser Thr Glu Val Gln Cys Lys Gly Gly Asn
690 695 700
Val Cys Val Gln Ala Gln Asp Arg Tyr Tyr Asn Ser Ser Cys Ser Lys
705 710 715 720
Trp Ala Cys Val Pro Cys Arg Val Arg Ser Gly Gly Gly Gly Ser Gly
725 730 735
Gly Gly Gly Ser Gly Gly Gly Gly Ser Arg Val Ile Pro Val Ser Gly
740 745 750
Pro Ala Arg Cys Leu Ser Gln Ser Arg Asn Leu Leu Lys Thr Thr Asp
755 760 765
Asp Met Val Lys Thr Ala Arg Glu Lys Leu Lys His Tyr Ser Cys Thr
770 775 780
Ala Glu Asp Ile Asp His Glu Asp Ile Thr Arg Asp Gln Thr Ser Thr
785 790 795 800
Leu Lys Thr Cys Leu Pro Leu Glu Leu His Lys Asn Glu Ser Cys Leu
805 810 815
Ala Thr Arg Glu Thr Ser Ser Thr Thr Arg Gly Ser Cys Leu Pro Pro
820 825 830
Gln Lys Thr Ser Leu Met Met Thr Leu Cys Leu Gly Ser Ile Tyr Glu
835 840 845
Asp Leu Lys Met Tyr Gln Thr Glu Phe Gln Ala Ile Asn Ala Ala Leu
850 855 860
Gln Asn His Asn His Gln Gln Ile Ile Leu Asp Lys Gly Met Leu Val
865 870 875 880
Ala Ile Asp Glu Leu Met Gln Ser Leu Asn His Asn Gly Glu Thr Leu
885 890 895
Arg Gln Lys Pro Pro Val Gly Glu Ala Asp Pro Tyr Arg Val Lys Met
900 905 910
Lys Leu Cys Ile Leu Leu His Ala Phe Ser Thr Arg Val Val Thr Ile
915 920 925
Asn Arg Val Met Gly Tyr Leu Ser Ser Ala
930 935
<210> 221
<211> 938
<212> PRT
<213> Artificial Sequence
<220>
<223> mIL-12sc-A10m3-hIL15R
<400> 221
Met Trp Glu Leu Glu Lys Asp Val Tyr Val Val Glu Val Asp Trp Thr
1 5 10 15
Pro Asp Ala Pro Gly Glu Thr Val Asn Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Asp Ile Thr Trp Thr Ser Asp Gln Arg His Gly Val Ile Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Thr Val Lys Glu Phe Leu Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Thr Leu Ser His Ser His Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asn Gly Ile Trp Ser Thr Glu Ile Leu Lys
85 90 95
Asn Phe Lys Asn Lys Thr Phe Leu Lys Cys Glu Ala Pro Asn Tyr Ser
100 105 110
Gly Arg Phe Thr Cys Ser Trp Leu Val Gln Arg Asn Met Asp Leu Lys
115 120 125
Phe Asn Ile Lys Ser Ser Ser Ser Ser Pro Asp Ser Arg Ala Val Thr
130 135 140
Cys Gly Met Ala Ser Leu Ser Ala Glu Lys Val Thr Leu Asp Gln Arg
145 150 155 160
Asp Tyr Glu Lys Tyr Ser Val Ser Cys Gln Glu Asp Val Thr Cys Pro
165 170 175
Thr Ala Glu Glu Thr Leu Pro Ile Glu Leu Ala Leu Glu Ala Arg Gln
180 185 190
Gln Asn Lys Tyr Glu Asn Tyr Ser Thr Ser Phe Phe Ile Arg Asp Ile
195 200 205
Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Met Lys Pro Leu Lys Asn
210 215 220
Ser Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Ser Trp Ser Thr Pro
225 230 235 240
His Ser Tyr Phe Ser Leu Lys Phe Phe Val Arg Ile Gln Arg Lys Lys
245 250 255
Glu Lys Met Lys Glu Thr Glu Glu Gly Cys Asn Gln Lys Gly Ala Phe
260 265 270
Leu Val Glu Lys Thr Ser Thr Glu Val Gln Cys Lys Gly Gly Asn Val
275 280 285
Cys Val Gln Ala Gln Asp Arg Tyr Tyr Asn Ser Ser Cys Ser Lys Trp
290 295 300
Ala Cys Val Pro Cys Arg Val Arg Ser Gly Gly Gly Gly Ser Gly Gly
305 310 315 320
Gly Gly Ser Gly Gly Gly Gly Ser Arg Val Ile Pro Val Ser Gly Pro
325 330 335
Ala Arg Cys Leu Ser Gln Ser Arg Asn Leu Leu Lys Thr Thr Asp Asp
340 345 350
Met Val Lys Thr Ala Arg Glu Lys Leu Lys His Tyr Ser Cys Thr Ala
355 360 365
Glu Asp Ile Asp His Glu Asp Ile Thr Arg Asp Gln Thr Ser Thr Leu
370 375 380
Lys Thr Cys Leu Pro Leu Glu Leu His Lys Asn Glu Ser Cys Leu Ala
385 390 395 400
Thr Arg Glu Thr Ser Ser Thr Thr Arg Gly Ser Cys Leu Pro Pro Gln
405 410 415
Lys Thr Ser Leu Met Met Thr Leu Cys Leu Gly Ser Ile Tyr Glu Asp
420 425 430
Leu Lys Met Tyr Gln Thr Glu Phe Gln Ala Ile Asn Ala Ala Leu Gln
435 440 445
Asn His Asn His Gln Gln Ile Ile Leu Asp Lys Gly Met Leu Val Ala
450 455 460
Ile Asp Glu Leu Met Gln Ser Leu Asn His Asn Gly Glu Thr Leu Arg
465 470 475 480
Gln Lys Pro Pro Val Gly Glu Ala Asp Pro Tyr Arg Val Lys Met Lys
485 490 495
Leu Cys Ile Leu Leu His Ala Phe Ser Thr Arg Val Val Thr Ile Asn
500 505 510
Arg Val Met Gly Tyr Leu Ser Ser Ala Gly Gly Gly Gly Ser Gly Gly
515 520 525
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
530 535 540
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro
545 550 555 560
Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp
565 570 575
Asp Ala Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
580 585 590
Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp
595 600 605
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
610 615 620
Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr
625 630 635 640
Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp
645 650 655
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly
660 665 670
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His
675 680 685
Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly
690 695 700
Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser
705 710 715 720
Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val
725 730 735
Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly
740 745 750
Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala
755 760 765
Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp
770 775 780
His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly
785 790 795 800
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
805 810 815
Gly Gly Ser Gly Gly Gly Gly Ser Asn Trp Val Asn Val Ile Ser Asp
820 825 830
Leu Lys Lys Ile Glu Asp Leu Ile Gln Ser Met His Ile Asp Ala Thr
835 840 845
Leu Tyr Thr Glu Ser Asp Val His Pro Ser Cys Lys Val Thr Ala Met
850 855 860
Lys Cys Phe Leu Leu Glu Leu Gln Val Ile Ser Leu Glu Ser Gly Asp
865 870 875 880
Ala Ser Ile His Asp Thr Val Glu Asn Leu Ile Ile Leu Ala Asn Asn
885 890 895
Ser Leu Ser Ser Asn Gly Asn Val Thr Glu Ser Gly Cys Arg Glu Cys
900 905 910
Glu Glu Leu Glu Glu Lys Asn Ile Lys Glu Phe Leu Gln Ser Phe Val
915 920 925
His Ile Val Gln Met Phe Ile Asn Thr Ser
930 935
<210> 222
<211> 938
<212> PRT
<213> Artificial Sequence
<220>
<223> mIL-12sc-A10m3-hIL15RA
<400> 222
Met Trp Glu Leu Glu Lys Asp Val Tyr Val Val Glu Val Asp Trp Thr
1 5 10 15
Pro Asp Ala Pro Gly Glu Thr Val Asn Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Asp Ile Thr Trp Thr Ser Asp Gln Arg His Gly Val Ile Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Thr Val Lys Glu Phe Leu Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Thr Leu Ser His Ser His Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asn Gly Ile Trp Ser Thr Glu Ile Leu Lys
85 90 95
Asn Phe Lys Asn Lys Thr Phe Leu Lys Cys Glu Ala Pro Asn Tyr Ser
100 105 110
Gly Arg Phe Thr Cys Ser Trp Leu Val Gln Arg Asn Met Asp Leu Lys
115 120 125
Phe Asn Ile Lys Ser Ser Ser Ser Ser Pro Asp Ser Arg Ala Val Thr
130 135 140
Cys Gly Met Ala Ser Leu Ser Ala Glu Lys Val Thr Leu Asp Gln Arg
145 150 155 160
Asp Tyr Glu Lys Tyr Ser Val Ser Cys Gln Glu Asp Val Thr Cys Pro
165 170 175
Thr Ala Glu Glu Thr Leu Pro Ile Glu Leu Ala Leu Glu Ala Arg Gln
180 185 190
Gln Asn Lys Tyr Glu Asn Tyr Ser Thr Ser Phe Phe Ile Arg Asp Ile
195 200 205
Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Met Lys Pro Leu Lys Asn
210 215 220
Ser Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Ser Trp Ser Thr Pro
225 230 235 240
His Ser Tyr Phe Ser Leu Lys Phe Phe Val Arg Ile Gln Arg Lys Lys
245 250 255
Glu Lys Met Lys Glu Thr Glu Glu Gly Cys Asn Gln Lys Gly Ala Phe
260 265 270
Leu Val Glu Lys Thr Ser Thr Glu Val Gln Cys Lys Gly Gly Asn Val
275 280 285
Cys Val Gln Ala Gln Asp Arg Tyr Tyr Asn Ser Ser Cys Ser Lys Trp
290 295 300
Ala Cys Val Pro Cys Arg Val Arg Ser Gly Gly Gly Gly Ser Gly Gly
305 310 315 320
Gly Gly Ser Gly Gly Gly Gly Ser Arg Val Ile Pro Val Ser Gly Pro
325 330 335
Ala Arg Cys Leu Ser Gln Ser Arg Asn Leu Leu Lys Thr Thr Asp Asp
340 345 350
Met Val Lys Thr Ala Arg Glu Lys Leu Lys His Tyr Ser Cys Thr Ala
355 360 365
Glu Asp Ile Asp His Glu Asp Ile Thr Arg Asp Gln Thr Ser Thr Leu
370 375 380
Lys Thr Cys Leu Pro Leu Glu Leu His Lys Asn Glu Ser Cys Leu Ala
385 390 395 400
Thr Arg Glu Thr Ser Ser Thr Thr Arg Gly Ser Cys Leu Pro Pro Gln
405 410 415
Lys Thr Ser Leu Met Met Thr Leu Cys Leu Gly Ser Ile Tyr Glu Asp
420 425 430
Leu Lys Met Tyr Gln Thr Glu Phe Gln Ala Ile Asn Ala Ala Leu Gln
435 440 445
Asn His Asn His Gln Gln Ile Ile Leu Asp Lys Gly Met Leu Val Ala
450 455 460
Ile Asp Glu Leu Met Gln Ser Leu Asn His Asn Gly Glu Thr Leu Arg
465 470 475 480
Gln Lys Pro Pro Val Gly Glu Ala Asp Pro Tyr Arg Val Lys Met Lys
485 490 495
Leu Cys Ile Leu Leu His Ala Phe Ser Thr Arg Val Val Thr Ile Asn
500 505 510
Arg Val Met Gly Tyr Leu Ser Ser Ala Gly Gly Gly Gly Ser Gly Gly
515 520 525
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
530 535 540
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro
545 550 555 560
Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp
565 570 575
Asp Ala Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
580 585 590
Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp
595 600 605
Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser
610 615 620
Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr
625 630 635 640
Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp
645 650 655
Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly
660 665 670
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His
675 680 685
Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly
690 695 700
Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser
705 710 715 720
Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val
725 730 735
Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly
740 745 750
Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala
755 760 765
Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp
770 775 780
His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly
785 790 795 800
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
805 810 815
Gly Gly Ser Gly Gly Gly Gly Ser Asn Trp Val Asn Val Ile Ser Asp
820 825 830
Leu Lys Lys Ile Glu Asp Leu Ile Gln Ser Met His Ile Asp Ala Thr
835 840 845
Leu Tyr Thr Glu Ser Asp Val His Pro Ser Cys Lys Val Thr Ala Met
850 855 860
Lys Cys Phe Leu Leu Glu Leu Gln Val Ile Ser Leu Glu Ser Gly Asp
865 870 875 880
Ala Ser Ile His Asp Thr Val Glu Asn Leu Ile Ile Leu Ala Asn Asn
885 890 895
Ser Leu Ser Ser Asn Gly Asn Val Thr Glu Ser Gly Cys Arg Glu Cys
900 905 910
Glu Glu Leu Glu Glu Lys Asn Ile Lys Glu Phe Leu Gln Ser Phe Val
915 920 925
His Ile Val Gln Met Phe Ile Ala Thr Ser
930 935
<210> 223
<211> 927
<212> PRT
<213> Artificial Sequence
<220>
<223> hIL15RA-A10m3-hIL-12sc
<400> 223
Asn Trp Val Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile
1 5 10 15
Gln Ser Met His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His
20 25 30
Pro Ser Cys Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln
35 40 45
Val Ile Ser Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu
50 55 60
Asn Leu Ile Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val
65 70 75 80
Thr Glu Ser Gly Cys Arg Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile
85 90 95
Lys Glu Phe Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Ala
100 105 110
Thr Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val
130 135 140
Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser
145 150 155 160
Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val
165 170 175
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser
180 185 190
Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr
195 200 205
Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg
210 215 220
Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr
225 230 235 240
Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met
245 250 255
Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly
260 265 270
Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln
275 280 285
Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys
290 295 300
Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys
305 310 315 320
Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro
325 330 335
Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala
340 345 350
Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr
355 360 365
Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly
370 375 380
Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Gly Ser Gly Gly Gly
385 390 395 400
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
405 410 415
Ser Ile Trp Glu Leu Lys Lys Asp Val Tyr Val Val Glu Leu Asp Trp
420 425 430
Tyr Pro Asp Ala Pro Gly Glu Met Val Val Leu Thr Cys Asp Thr Pro
435 440 445
Glu Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln Ser Ser Glu Val Leu
450 455 460
Gly Ser Gly Lys Thr Leu Thr Ile Gln Val Lys Glu Phe Gly Asp Ala
465 470 475 480
Gly Gln Tyr Thr Cys His Lys Gly Gly Glu Val Leu Ser His Ser Leu
485 490 495
Leu Leu Leu His Lys Lys Glu Asp Gly Ile Trp Ser Thr Asp Ile Leu
500 505 510
Lys Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe Leu Arg Cys Glu Ala
515 520 525
Lys Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp Leu Thr Thr Ile Ser
530 535 540
Thr Asp Leu Thr Phe Ser Val Lys Ser Ser Arg Gly Ser Ser Asp Pro
545 550 555 560
Gln Gly Val Thr Cys Gly Ala Ala Thr Leu Ser Ala Glu Arg Val Arg
565 570 575
Gly Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu Cys Gln Glu Asp Ser
580 585 590
Ala Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile Glu Val Met Val Asp
595 600 605
Ala Val His Lys Leu Lys Tyr Glu Asn Tyr Thr Ser Ser Phe Phe Ile
610 615 620
Arg Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Leu Lys Pro
625 630 635 640
Leu Lys Asn Ser Arg Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Thr
645 650 655
Trp Ser Thr Pro His Ser Tyr Phe Ser Leu Thr Phe Cys Val Gln Val
660 665 670
Gln Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg Val Phe Thr Asp Lys
675 680 685
Thr Ser Ala Thr Val Ile Cys Arg Lys Asn Ala Ser Ile Ser Val Arg
690 695 700
Ala Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser Glu Trp Ala Ser Val
705 710 715 720
Pro Cys Ser Gly Gly Gly Gly Gly Gly Ser Arg Asn Leu Pro Val Ala
725 730 735
Thr Pro Asp Pro Gly Met Phe Pro Cys Leu His His Ser Gln Asn Leu
740 745 750
Leu Arg Ala Val Ser Asn Met Leu Gln Lys Ala Arg Gln Thr Leu Glu
755 760 765
Phe Tyr Pro Cys Thr Ser Glu Glu Ile Asp His Glu Asp Ile Thr Lys
770 775 780
Asp Lys Thr Ser Thr Val Glu Ala Cys Leu Pro Leu Glu Leu Thr Lys
785 790 795 800
Asn Glu Ser Cys Leu Asn Ser Arg Glu Thr Ser Phe Ile Thr Asn Gly
805 810 815
Ser Cys Leu Ala Ser Arg Lys Thr Ser Phe Met Met Ala Leu Cys Leu
820 825 830
Ser Ser Ile Tyr Glu Asp Leu Lys Met Tyr Gln Val Glu Phe Lys Thr
835 840 845
Met Asn Ala Lys Leu Leu Met Asp Pro Lys Arg Gln Ile Phe Leu Asp
850 855 860
Gln Asn Met Leu Ala Val Ile Asp Glu Leu Met Gln Ala Leu Asn Phe
865 870 875 880
Asn Ser Glu Thr Val Pro Gln Lys Ser Ser Leu Glu Glu Pro Asp Phe
885 890 895
Tyr Lys Thr Lys Ile Lys Leu Cys Ile Leu Leu His Ala Phe Arg Ile
900 905 910
Arg Ala Val Thr Ile Asp Arg Val Thr Ser Tyr Leu Asn Ala Ser
915 920 925
<210> 224
<211> 927
<212> PRT
<213> Artificial Sequence
<220>
<223> Human IL-12sc-A10m3-hIL15R
<400> 224
Ile Trp Glu Leu Lys Lys Asp Val Tyr Val Val Glu Leu Asp Trp Tyr
1 5 10 15
Pro Asp Ala Pro Gly Glu Met Val Val Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln Ser Ser Glu Val Leu Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Gln Val Lys Glu Phe Gly Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Val Leu Ser His Ser Leu Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asp Gly Ile Trp Ser Thr Asp Ile Leu Lys
85 90 95
Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe Leu Arg Cys Glu Ala Lys
100 105 110
Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp Leu Thr Thr Ile Ser Thr
115 120 125
Asp Leu Thr Phe Ser Val Lys Ser Ser Arg Gly Ser Ser Asp Pro Gln
130 135 140
Gly Val Thr Cys Gly Ala Ala Thr Leu Ser Ala Glu Arg Val Arg Gly
145 150 155 160
Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu Cys Gln Glu Asp Ser Ala
165 170 175
Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile Glu Val Met Val Asp Ala
180 185 190
Val His Lys Leu Lys Tyr Glu Asn Tyr Thr Ser Ser Phe Phe Ile Arg
195 200 205
Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Leu Lys Pro Leu
210 215 220
Lys Asn Ser Arg Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Thr Trp
225 230 235 240
Ser Thr Pro His Ser Tyr Phe Ser Leu Thr Phe Cys Val Gln Val Gln
245 250 255
Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg Val Phe Thr Asp Lys Thr
260 265 270
Ser Ala Thr Val Ile Cys Arg Lys Asn Ala Ser Ile Ser Val Arg Ala
275 280 285
Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser Glu Trp Ala Ser Val Pro
290 295 300
Cys Ser Gly Gly Gly Gly Gly Gly Ser Arg Asn Leu Pro Val Ala Thr
305 310 315 320
Pro Asp Pro Gly Met Phe Pro Cys Leu His His Ser Gln Asn Leu Leu
325 330 335
Arg Ala Val Ser Asn Met Leu Gln Lys Ala Arg Gln Thr Leu Glu Phe
340 345 350
Tyr Pro Cys Thr Ser Glu Glu Ile Asp His Glu Asp Ile Thr Lys Asp
355 360 365
Lys Thr Ser Thr Val Glu Ala Cys Leu Pro Leu Glu Leu Thr Lys Asn
370 375 380
Glu Ser Cys Leu Asn Ser Arg Glu Thr Ser Phe Ile Thr Asn Gly Ser
385 390 395 400
Cys Leu Ala Ser Arg Lys Thr Ser Phe Met Met Ala Leu Cys Leu Ser
405 410 415
Ser Ile Tyr Glu Asp Leu Lys Met Tyr Gln Val Glu Phe Lys Thr Met
420 425 430
Asn Ala Lys Leu Leu Met Asp Pro Lys Arg Gln Ile Phe Leu Asp Gln
435 440 445
Asn Met Leu Ala Val Ile Asp Glu Leu Met Gln Ala Leu Asn Phe Asn
450 455 460
Ser Glu Thr Val Pro Gln Lys Ser Ser Leu Glu Glu Pro Asp Phe Tyr
465 470 475 480
Lys Thr Lys Ile Lys Leu Cys Ile Leu Leu His Ala Phe Arg Ile Arg
485 490 495
Ala Val Thr Ile Asp Arg Val Thr Ser Tyr Leu Asn Ala Ser Gly Gly
500 505 510
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
515 520 525
Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
530 535 540
Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser
545 550 555 560
Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln Ala Pro
565 570 575
Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr
580 585 590
Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
595 600 605
Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu
610 615 620
Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg
625 630 635 640
Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser
645 650 655
Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
660 665 670
Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val
675 680 685
Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn
690 695 700
Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
705 710 715 720
Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro
725 730 735
Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile
740 745 750
Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp
755 760 765
Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu
770 775 780
Thr Val Leu Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
785 790 795 800
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Trp Val
805 810 815
Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile Gln Ser Met
820 825 830
His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His Pro Ser Cys
835 840 845
Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln Val Ile Ser
850 855 860
Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu Asn Leu Ile
865 870 875 880
Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val Thr Glu Ser
885 890 895
Gly Cys Arg Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile Lys Glu Phe
900 905 910
Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Asn Thr Ser
915 920 925
<210> 225
<211> 927
<212> PRT
<213> Artificial Sequence
<220>
<223> Human IL-12sc-A10m3-hIL15RA
<400> 225
Ile Trp Glu Leu Lys Lys Asp Val Tyr Val Val Glu Leu Asp Trp Tyr
1 5 10 15
Pro Asp Ala Pro Gly Glu Met Val Val Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln Ser Ser Glu Val Leu Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Gln Val Lys Glu Phe Gly Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Val Leu Ser His Ser Leu Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asp Gly Ile Trp Ser Thr Asp Ile Leu Lys
85 90 95
Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe Leu Arg Cys Glu Ala Lys
100 105 110
Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp Leu Thr Thr Ile Ser Thr
115 120 125
Asp Leu Thr Phe Ser Val Lys Ser Ser Arg Gly Ser Ser Asp Pro Gln
130 135 140
Gly Val Thr Cys Gly Ala Ala Thr Leu Ser Ala Glu Arg Val Arg Gly
145 150 155 160
Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu Cys Gln Glu Asp Ser Ala
165 170 175
Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile Glu Val Met Val Asp Ala
180 185 190
Val His Lys Leu Lys Tyr Glu Asn Tyr Thr Ser Ser Phe Phe Ile Arg
195 200 205
Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Leu Lys Pro Leu
210 215 220
Lys Asn Ser Arg Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Thr Trp
225 230 235 240
Ser Thr Pro His Ser Tyr Phe Ser Leu Thr Phe Cys Val Gln Val Gln
245 250 255
Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg Val Phe Thr Asp Lys Thr
260 265 270
Ser Ala Thr Val Ile Cys Arg Lys Asn Ala Ser Ile Ser Val Arg Ala
275 280 285
Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser Glu Trp Ala Ser Val Pro
290 295 300
Cys Ser Gly Gly Gly Gly Gly Gly Ser Arg Asn Leu Pro Val Ala Thr
305 310 315 320
Pro Asp Pro Gly Met Phe Pro Cys Leu His His Ser Gln Asn Leu Leu
325 330 335
Arg Ala Val Ser Asn Met Leu Gln Lys Ala Arg Gln Thr Leu Glu Phe
340 345 350
Tyr Pro Cys Thr Ser Glu Glu Ile Asp His Glu Asp Ile Thr Lys Asp
355 360 365
Lys Thr Ser Thr Val Glu Ala Cys Leu Pro Leu Glu Leu Thr Lys Asn
370 375 380
Glu Ser Cys Leu Asn Ser Arg Glu Thr Ser Phe Ile Thr Asn Gly Ser
385 390 395 400
Cys Leu Ala Ser Arg Lys Thr Ser Phe Met Met Ala Leu Cys Leu Ser
405 410 415
Ser Ile Tyr Glu Asp Leu Lys Met Tyr Gln Val Glu Phe Lys Thr Met
420 425 430
Asn Ala Lys Leu Leu Met Asp Pro Lys Arg Gln Ile Phe Leu Asp Gln
435 440 445
Asn Met Leu Ala Val Ile Asp Glu Leu Met Gln Ala Leu Asn Phe Asn
450 455 460
Ser Glu Thr Val Pro Gln Lys Ser Ser Leu Glu Glu Pro Asp Phe Tyr
465 470 475 480
Lys Thr Lys Ile Lys Leu Cys Ile Leu Leu His Ala Phe Arg Ile Arg
485 490 495
Ala Val Thr Ile Asp Arg Val Thr Ser Tyr Leu Asn Ala Ser Gly Gly
500 505 510
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
515 520 525
Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
530 535 540
Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser
545 550 555 560
Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln Ala Pro
565 570 575
Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr
580 585 590
Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
595 600 605
Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu
610 615 620
Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg
625 630 635 640
Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser
645 650 655
Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
660 665 670
Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val
675 680 685
Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn
690 695 700
Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
705 710 715 720
Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro
725 730 735
Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile
740 745 750
Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp
755 760 765
Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu
770 775 780
Thr Val Leu Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
785 790 795 800
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asn Trp Val
805 810 815
Asn Val Ile Ser Asp Leu Lys Lys Ile Glu Asp Leu Ile Gln Ser Met
820 825 830
His Ile Asp Ala Thr Leu Tyr Thr Glu Ser Asp Val His Pro Ser Cys
835 840 845
Lys Val Thr Ala Met Lys Cys Phe Leu Leu Glu Leu Gln Val Ile Ser
850 855 860
Leu Glu Ser Gly Asp Ala Ser Ile His Asp Thr Val Glu Asn Leu Ile
865 870 875 880
Ile Leu Ala Asn Asn Ser Leu Ser Ser Asn Gly Asn Val Thr Glu Ser
885 890 895
Gly Cys Arg Glu Cys Glu Glu Leu Glu Glu Lys Asn Ile Lys Glu Phe
900 905 910
Leu Gln Ser Phe Val His Ile Val Gln Met Phe Ile Ala Thr Ser
915 920 925
<210> 226
<211> 131
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 226
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Met Arg Tyr Cys Ser Gly Gly Ser Cys Tyr Pro Tyr Ala
100 105 110
Tyr Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr
130
<210> 227
<211> 115
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 227
Val His Ser Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser Val Ala
1 5 10 15
Leu Gly Gln Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu Arg Ser
20 25 30
Tyr Tyr Ala Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu
35 40 45
Val Ile Tyr Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp Arg Phe
50 55 60
Ser Gly Ser Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr Gly Ala
65 70 75 80
Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Asp Ser Ser
85 90 95
Gly Asn Leu Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
100 105 110
Leu Gly Gly
115
<210> 228
<211> 261
<212> PRT
<213> Artificial Sequence
<220>
<223> scFv
<400> 228
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Met Arg Tyr Cys Ser Gly Gly Ser Cys Tyr Pro Tyr Ala
100 105 110
Tyr Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Val His Ser Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser
145 150 155 160
Val Ala Leu Gly Gln Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu
165 170 175
Arg Ser Tyr Tyr Ala Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
180 185 190
Val Leu Val Ile Tyr Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp
195 200 205
Arg Phe Ser Gly Ser Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr
210 215 220
Gly Ala Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Asp
225 230 235 240
Ser Ser Gly Asn Leu Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val
245 250 255
Leu Gly Leu Gly Gly
260
<210> 229
<211> 125
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable heavy (vh) chain
<400> 229
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
1 5 10 15
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala
20 25 30
Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
35 40 45
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ala Tyr Tyr Cys Ala
85 90 95
Lys Glu Ile Ser Gly Ser Tyr Leu Gly Leu Val Ala Phe Asp Ile Trp
100 105 110
Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr
115 120 125
<210> 230
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable light (vl) chain
<400> 230
Val His Ser Ser Tyr Val Leu Thr Gln Pro Leu Ser Val Ser Val Ala
1 5 10 15
Pro Gly Gln Thr Ala Arg Met Thr Cys Gly Gly Asp Asn Val Gly Ala
20 25 30
Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu
35 40 45
Leu Ile Tyr Tyr Asp His Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe
50 55 60
Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Thr Arg Val
65 70 75 80
Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Pro Ser
85 90 95
Ser Asp Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly Leu
100 105 110
Gly Gly
<210> 231
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> scFv
<400> 231
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ala Tyr Tyr Cys
85 90 95
Ala Lys Glu Ile Ser Gly Ser Tyr Leu Gly Leu Val Ala Phe Asp Ile
100 105 110
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser
130 135 140
Ser Tyr Val Leu Thr Gln Pro Leu Ser Val Ser Val Ala Pro Gly Gln
145 150 155 160
Thr Ala Arg Met Thr Cys Gly Gly Asp Asn Val Gly Ala Lys Ser Val
165 170 175
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Leu Ile Tyr
180 185 190
Tyr Asp His Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
195 200 205
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Thr Arg Val Glu Ala Gly
210 215 220
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Pro Ser Ser Asp Val
225 230 235 240
Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly Leu Gly Gly
245 250 255
<210> 232
<211> 261
<212> PRT
<213> Artificial Sequence
<220>
<223> 4H7m scFv
<400> 232
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Met Arg Tyr Cys Ser Gly Gly Ser Cys Tyr Pro Tyr Ala
100 105 110
Tyr Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Val His Ser Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser
145 150 155 160
Val Ala Leu Gly Gln Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu
165 170 175
Arg Ser Tyr Tyr Ala Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
180 185 190
Val Leu Val Ile Tyr Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp
195 200 205
Arg Phe Ser Gly Ser Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr
210 215 220
Gly Ala Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Asp
225 230 235 240
Ser Ser Gly Asn Leu Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val
245 250 255
Leu Gly Leu Gly Gly
260
<210> 233
<211> 537
<212> PRT
<213> Artificial Sequence
<220>
<223> 4H7m scFV-A6m
<400> 233
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Arg
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Val Ile Ser Tyr Asp Gly Ser Asn Lys Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Glu Met Arg Tyr Cys Ser Gly Gly Ser Cys Tyr Pro Tyr Ala
100 105 110
Tyr Tyr Met Asp Val Trp Gly Gln Gly Thr Thr Val Thr Val Ser Ser
115 120 125
Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Val His Ser Ser Ser Glu Leu Thr Gln Asp Pro Ala Val Ser
145 150 155 160
Val Ala Leu Gly Gln Thr Val Arg Ile Thr Cys Gln Gly Asp Ser Leu
165 170 175
Arg Ser Tyr Tyr Ala Ser Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
180 185 190
Val Leu Val Ile Tyr Gly Lys Asn Asn Arg Pro Ser Gly Ile Pro Asp
195 200 205
Arg Phe Ser Gly Ser Ser Ser Gly Asn Thr Ala Ser Leu Thr Ile Thr
210 215 220
Gly Ala Gln Ala Glu Asp Glu Ala Asp Tyr Tyr Cys Asn Ser Arg Asp
225 230 235 240
Ser Ser Gly Asn Leu Val Val Phe Gly Gly Gly Thr Lys Leu Thr Val
245 250 255
Leu Gly Leu Gly Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
260 265 270
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
275 280 285
Gln Leu Val Gln Ser Gly Ala Glu Val Lys Lys Pro Gly Glu Ser Leu
290 295 300
Lys Ile Ser Cys Lys Gly Ser Gly Tyr Ser Phe Asn Ser Tyr Trp Ile
305 310 315 320
Gly Trp Val Arg Gln Met Pro Gly Lys Gly Leu Glu Trp Met Gly Ile
325 330 335
Ile Tyr Pro Gly Asp Ser Asp Thr Arg Tyr Ser Pro Ser Phe Gln Gly
340 345 350
Gln Val Thr Phe Ser Val Asp Lys Ser Ile Asn Thr Ala Tyr Leu Gln
355 360 365
Trp Ser Ser Leu Lys Ala Ser Asp Ala Ala Met Tyr Tyr Cys Ala Arg
370 375 380
Gln Arg Trp Gly Ser Ser Ser Phe Asp Ala Trp Gly Gln Gly Thr Leu
385 390 395 400
Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly
405 410 415
Gly Ser Gly Gly Gly Gly Ser Val His Ser Gln Ser Val Leu Thr Gln
420 425 430
Pro Pro Ser Val Ser Ala Ala Pro Gly Gln Lys Val Thr Ile Ser Cys
435 440 445
Ser Gly Ser Ser Ser Asn Ile Gly Asn Asn Tyr Val Ser Trp Tyr Gln
450 455 460
Gln Leu Pro Gly Thr Ala Pro Lys Leu Leu Ile Tyr Glu Asn Asn Lys
465 470 475 480
Arg Pro Ser Gly Ile Pro Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr
485 490 495
Ser Ala Thr Leu Gly Ile Thr Gly Leu Gln Thr Gly Asp Glu Ala Asp
500 505 510
Tyr Tyr Cys Gly Thr Trp Asp Ser Ser Leu Ser Ala Gly Val Phe Gly
515 520 525
Gly Gly Thr Gln Leu Thr Val Leu Gly
530 535
<210> 234
<211> 255
<212> PRT
<213> Artificial Sequence
<220>
<223> 4D9m scFV
<400> 234
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ala Tyr Tyr Cys
85 90 95
Ala Lys Glu Ile Ser Gly Ser Tyr Leu Gly Leu Val Ala Phe Asp Ile
100 105 110
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser
130 135 140
Ser Tyr Val Leu Thr Gln Pro Leu Ser Val Ser Val Ala Pro Gly Gln
145 150 155 160
Thr Ala Arg Met Thr Cys Gly Gly Asp Asn Val Gly Ala Lys Ser Val
165 170 175
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Leu Ile Tyr
180 185 190
Tyr Asp His Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
195 200 205
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Thr Arg Val Glu Ala Gly
210 215 220
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Pro Ser Ser Asp Val
225 230 235 240
Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly Leu Gly Gly
245 250 255
<210> 235
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> VH
<400> 235
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ala Tyr Tyr Cys
85 90 95
Ala Lys Glu Ile Ser Gly Ser Tyr Leu Gly Leu Val Ala Phe Asp Ile
100 105 110
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr
115 120 125
<210> 236
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 236
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 237
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 237
Ser Gly Ser Gly Gly Ser Thr
1 5
<210> 238
<211> 24
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 238
Ala Glu Asp Thr Ala Ala Tyr Tyr Cys Ala Lys Glu Ile Ser Gly Ser
1 5 10 15
Tyr Leu Gly Leu Val Ala Phe Asp
20
<210> 239
<211> 114
<212> PRT
<213> Artificial Sequence
<220>
<223> VL
<400> 239
Val His Ser Ser Tyr Val Leu Thr Gln Pro Leu Ser Val Ser Val Ala
1 5 10 15
Pro Gly Gln Thr Ala Arg Met Thr Cys Gly Gly Asp Asn Val Gly Ala
20 25 30
Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu
35 40 45
Leu Ile Tyr Tyr Asp His Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe
50 55 60
Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Thr Arg Val
65 70 75 80
Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Pro Ser
85 90 95
Ser Asp Val Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly Leu
100 105 110
Gly Gly
<210> 240
<211> 6
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 240
Asn Val Gly Ala Lys Ser
1 5
<210> 241
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 241
Tyr Asp His
1
<210> 242
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 242
Gln Val Trp Asp Pro Ser Ser Asp Val Val
1 5 10
<210> 243
<211> 531
<212> PRT
<213> Artificial Sequence
<220>
<223> 4D9m scFV-A6m
<400> 243
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Ile Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Ala Tyr Tyr Cys
85 90 95
Ala Lys Glu Ile Ser Gly Ser Tyr Leu Gly Leu Val Ala Phe Asp Ile
100 105 110
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser
130 135 140
Ser Tyr Val Leu Thr Gln Pro Leu Ser Val Ser Val Ala Pro Gly Gln
145 150 155 160
Thr Ala Arg Met Thr Cys Gly Gly Asp Asn Val Gly Ala Lys Ser Val
165 170 175
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Leu Ile Tyr
180 185 190
Tyr Asp His Asp Arg Pro Ser Gly Ile Pro Glu Arg Phe Ser Gly Ser
195 200 205
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Thr Arg Val Glu Ala Gly
210 215 220
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Pro Ser Ser Asp Val
225 230 235 240
Val Phe Gly Gly Gly Thr Gln Leu Thr Val Leu Gly Leu Gly Gly Gly
245 250 255
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
260 265 270
Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Gln Ser Gly
275 280 285
Ala Glu Val Lys Lys Pro Gly Glu Ser Leu Lys Ile Ser Cys Lys Gly
290 295 300
Ser Gly Tyr Ser Phe Asn Ser Tyr Trp Ile Gly Trp Val Arg Gln Met
305 310 315 320
Pro Gly Lys Gly Leu Glu Trp Met Gly Ile Ile Tyr Pro Gly Asp Ser
325 330 335
Asp Thr Arg Tyr Ser Pro Ser Phe Gln Gly Gln Val Thr Phe Ser Val
340 345 350
Asp Lys Ser Ile Asn Thr Ala Tyr Leu Gln Trp Ser Ser Leu Lys Ala
355 360 365
Ser Asp Ala Ala Met Tyr Tyr Cys Ala Arg Gln Arg Trp Gly Ser Ser
370 375 380
Ser Phe Asp Ala Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Ala
385 390 395 400
Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
405 410 415
Ser Val His Ser Gln Ser Val Leu Thr Gln Pro Pro Ser Val Ser Ala
420 425 430
Ala Pro Gly Gln Lys Val Thr Ile Ser Cys Ser Gly Ser Ser Ser Asn
435 440 445
Ile Gly Asn Asn Tyr Val Ser Trp Tyr Gln Gln Leu Pro Gly Thr Ala
450 455 460
Pro Lys Leu Leu Ile Tyr Glu Asn Asn Lys Arg Pro Ser Gly Ile Pro
465 470 475 480
Asp Arg Phe Ser Gly Ser Lys Ser Gly Thr Ser Ala Thr Leu Gly Ile
485 490 495
Thr Gly Leu Gln Thr Gly Asp Glu Ala Asp Tyr Tyr Cys Gly Thr Trp
500 505 510
Asp Ser Ser Leu Ser Ala Gly Val Phe Gly Gly Gly Thr Gln Leu Thr
515 520 525
Val Leu Gly
530
<210> 244
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Gly-Ser 15
<400> 244
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 245
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Whitlow linker
<400> 245
Gly Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr
1 5 10 15
Lys Gly
<210> 246
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> 6paxA_1(+A)
<400> 246
Ile Arg Pro Arg Ala Ile Gly Gly Ser Lys Pro Arg Val Ala
1 5 10
<210> 247
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> +B
<400> 247
Gly Lys Gly Gly Ser Gly Lys Gly Gly Ser Gly Lys Gly Gly Ser
1 5 10 15
<210> 248
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> +C
<400> 248
Gly Gly Lys Gly Ser Gly Gly Lys Gly Ser Gly Gly Lys Gly Ser
1 5 10 15
<210> 249
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> +D
<400> 249
Gly Gly Gly Lys Ser Gly Gly Gly Lys Ser Gly Gly Gly Lys Ser
1 5 10 15
<210> 250
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> +E
<400> 250
Gly Lys Gly Lys Ser Gly Lys Gly Lys Ser Gly Lys Gly Lys Ser
1 5 10 15
<210> 251
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> +F
<400> 251
Gly Gly Gly Lys Ser Gly Gly Lys Gly Ser Gly Lys Gly Gly Ser
1 5 10 15
<210> 252
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> +G
<400> 252
Gly Lys Pro Gly Ser Gly Lys Pro Gly Ser Gly Lys Pro Gly Ser
1 5 10 15
<210> 253
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> +H
<400> 253
Gly Lys Pro Gly Ser Gly Lys Pro Gly Ser Gly Lys Pro Gly Ser Gly
1 5 10 15
Lys Pro Gly Ser
20
<210> 254
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> +I
<400> 254
Gly Lys Gly Lys Ser Gly Lys Gly Lys Ser Gly Lys Gly Lys Ser Gly
1 5 10 15
Lys Gly Lys Ser
20
<210> 255
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Gly-Ser 15
<400> 255
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 256
<211> 14
<212> PRT
<213> Artificial Sequence
<220>
<223> 3hsc_2(-A)
<400> 256
Ser Thr Ala Gly Asp Thr His Leu Gly Gly Glu Asp Phe Asp
1 5 10
<210> 257
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> -B
<400> 257
Gly Glu Gly Gly Ser Gly Glu Gly Gly Ser Gly Glu Gly Gly Ser
1 5 10 15
<210> 258
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> -C
<400> 258
Gly Gly Glu Gly Ser Gly Gly Glu Gly Ser Gly Gly Glu Gly Ser
1 5 10 15
<210> 259
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> -D
<400> 259
Gly Gly Gly Glu Ser Gly Gly Gly Glu Ser Gly Gly Gly Glu Ser
1 5 10 15
<210> 260
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> -E
<400> 260
Gly Glu Gly Glu Ser Gly Glu Gly Glu Ser Gly Glu Gly Glu Ser
1 5 10 15
<210> 261
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> -F
<400> 261
Gly Gly Gly Glu Ser Gly Gly Glu Gly Ser Gly Glu Gly Gly Ser
1 5 10 15
<210> 262
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> -G
<400> 262
Gly Glu Gly Glu Ser Gly Glu Gly Glu Ser Gly Glu Gly Glu Ser Gly
1 5 10 15
Glu Gly Glu Ser
20
<210> 263
<211> 15
<212> PRT
<213> Artificial Sequence
<220>
<223> Additional linkers
<400> 263
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 264
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Additional linker
<400> 264
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 265
<211> 18
<212> PRT
<213> Artificial Sequence
<220>
<223> Additional linker
<400> 265
Gly Ser Thr Ser Gly Ser Gly Lys Pro Gly Ser Gly Glu Gly Ser Thr
1 5 10 15
Lys Gly
<210> 266
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Additional linker
<400> 266
Pro Arg Gly Ala Ser Lys Ser Gly Ser Ala Ser Gln Thr Gly Ser Ala
1 5 10 15
Pro Gly Ser
<210> 267
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Additional linker
<400> 267
Gly Thr Ala Ala Ala Gly Ala Gly Ala Ala Gly Gly Ala Ala Ala Gly
1 5 10 15
Ala Ala Gly
<210> 268
<211> 19
<212> PRT
<213> Artificial Sequence
<220>
<223> Artificial linker
<400> 268
Gly Thr Ser Gly Ser Ser Gly Ser Gly Ser Gly Gly Ser Gly Ser Gly
1 5 10 15
Gly Gly Gly
<210> 269
<211> 20
<212> PRT
<213> Artificial Sequence
<220>
<223> Additional linker
<400> 269
Gly Lys Pro Gly Ser Gly Lys Pro Gly Ser Gly Lys Pro Gly Ser Gly
1 5 10 15
Lys Pro Gly Ser
20
<210> 270
<211> 7
<212> PRT
<213> Artificial Sequence
<220>
<223> Additional linker
<400> 270
Gly Gly Gly Gly Gly Gly Ser
1 5
<210> 271
<211> 133
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-2
<400> 271
Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His
1 5 10 15
Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys
20 25 30
Asn Pro Lys Leu Thr Arg Met Leu Thr Phe Lys Phe Tyr Met Pro Lys
35 40 45
Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys
50 55 60
Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu
65 70 75 80
Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu
85 90 95
Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala
100 105 110
Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile
115 120 125
Ile Ser Thr Leu Thr
130
<210> 272
<211> 411
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-2-A10m3
<400> 272
Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His
1 5 10 15
Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys
20 25 30
Asn Pro Lys Leu Thr Arg Met Leu Thr Phe Lys Phe Tyr Met Pro Lys
35 40 45
Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys
50 55 60
Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu
65 70 75 80
Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu
85 90 95
Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala
100 105 110
Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile
115 120 125
Ile Ser Thr Leu Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
145 150 155 160
Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu
165 170 175
Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met
180 185 190
His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly
195 200 205
Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly
210 215 220
Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln
225 230 235 240
Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys
245 250 255
Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln
260 265 270
Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser
275 280 285
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val
290 295 300
Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr
305 310 315 320
Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr
325 330 335
Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser
340 345 350
Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly
355 360 365
Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala
370 375 380
Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val
385 390 395 400
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
405 410
<210> 273
<211> 149
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL-2
<400> 273
Ala Pro Thr Ser Ser Ser Thr Ser Ser Ser Thr Ala Glu Ala Gln Gln
1 5 10 15
Gln Gln Gln Gln Gln Gln Gln Gln Gln Gln His Leu Glu Gln Leu Leu
20 25 30
Met Asp Leu Gln Glu Leu Leu Ser Arg Met Glu Asn Tyr Arg Asn Leu
35 40 45
Lys Leu Pro Arg Met Leu Thr Phe Lys Phe Tyr Leu Pro Lys Gln Ala
50 55 60
Thr Glu Leu Lys Asp Leu Gln Cys Leu Glu Asp Glu Leu Gly Pro Leu
65 70 75 80
Arg His Val Leu Asp Leu Thr Gln Ser Lys Ser Phe Gln Leu Glu Asp
85 90 95
Ala Glu Asn Phe Ile Ser Asn Ile Arg Val Thr Val Val Lys Leu Lys
100 105 110
Gly Ser Asp Asn Thr Phe Glu Cys Gln Phe Asp Asp Glu Ser Ala Thr
115 120 125
Val Val Asp Phe Leu Arg Arg Trp Ile Ala Phe Cys Gln Ser Ile Ile
130 135 140
Ser Thr Ser Pro Gln
145
<210> 274
<211> 427
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL2-A10m3
<400> 274
Ala Pro Thr Ser Ser Ser Thr Ser Ser Ser Thr Ala Glu Ala Gln Gln
1 5 10 15
Gln Gln Gln Gln Gln Gln Gln Gln Gln Gln His Leu Glu Gln Leu Leu
20 25 30
Met Asp Leu Gln Glu Leu Leu Ser Arg Met Glu Asn Tyr Arg Asn Leu
35 40 45
Lys Leu Pro Arg Met Leu Thr Phe Lys Phe Tyr Leu Pro Lys Gln Ala
50 55 60
Thr Glu Leu Lys Asp Leu Gln Cys Leu Glu Asp Glu Leu Gly Pro Leu
65 70 75 80
Arg His Val Leu Asp Leu Thr Gln Ser Lys Ser Phe Gln Leu Glu Asp
85 90 95
Ala Glu Asn Phe Ile Ser Asn Ile Arg Val Thr Val Val Lys Leu Lys
100 105 110
Gly Ser Asp Asn Thr Phe Glu Cys Gln Phe Asp Asp Glu Ser Ala Thr
115 120 125
Val Val Asp Phe Leu Arg Arg Trp Ile Ala Phe Cys Gln Ser Ile Ile
130 135 140
Ser Thr Ser Pro Gln Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
145 150 155 160
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
165 170 175
Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu
180 185 190
Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met
195 200 205
His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly
210 215 220
Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly
225 230 235 240
Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln
245 250 255
Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys
260 265 270
Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln
275 280 285
Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser
290 295 300
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val
305 310 315 320
Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr
325 330 335
Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr
340 345 350
Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser
355 360 365
Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly
370 375 380
Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala
385 390 395 400
Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val
405 410 415
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
420 425
<210> 275
<211> 152
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-7
<400> 275
Asp Cys Asp Ile Glu Gly Lys Asp Gly Lys Gln Tyr Glu Ser Val Leu
1 5 10 15
Met Val Ser Ile Asp Gln Leu Leu Asp Ser Met Lys Glu Ile Gly Ser
20 25 30
Asn Cys Leu Asn Asn Glu Phe Asn Phe Phe Lys Arg His Ile Cys Asp
35 40 45
Ala Asn Lys Glu Gly Met Phe Leu Phe Arg Ala Ala Arg Lys Leu Arg
50 55 60
Gln Phe Leu Lys Met Asn Ser Thr Gly Asp Phe Asp Leu His Leu Leu
65 70 75 80
Lys Val Ser Glu Gly Thr Thr Ile Leu Leu Asn Cys Thr Gly Gln Val
85 90 95
Lys Gly Arg Lys Pro Ala Ala Leu Gly Glu Ala Gln Pro Thr Lys Ser
100 105 110
Leu Glu Glu Asn Lys Ser Leu Lys Glu Gln Lys Lys Leu Asn Asp Leu
115 120 125
Cys Phe Leu Lys Arg Leu Leu Gln Glu Ile Lys Thr Cys Trp Asn Lys
130 135 140
Ile Leu Met Gly Thr Lys Glu His
145 150
<210> 276
<211> 430
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-7-A10m3
<400> 276
Asp Cys Asp Ile Glu Gly Lys Asp Gly Lys Gln Tyr Glu Ser Val Leu
1 5 10 15
Met Val Ser Ile Asp Gln Leu Leu Asp Ser Met Lys Glu Ile Gly Ser
20 25 30
Asn Cys Leu Asn Asn Glu Phe Asn Phe Phe Lys Arg His Ile Cys Asp
35 40 45
Ala Asn Lys Glu Gly Met Phe Leu Phe Arg Ala Ala Arg Lys Leu Arg
50 55 60
Gln Phe Leu Lys Met Asn Ser Thr Gly Asp Phe Asp Leu His Leu Leu
65 70 75 80
Lys Val Ser Glu Gly Thr Thr Ile Leu Leu Asn Cys Thr Gly Gln Val
85 90 95
Lys Gly Arg Lys Pro Ala Ala Leu Gly Glu Ala Gln Pro Thr Lys Ser
100 105 110
Leu Glu Glu Asn Lys Ser Leu Lys Glu Gln Lys Lys Leu Asn Asp Leu
115 120 125
Cys Phe Leu Lys Arg Leu Leu Gln Glu Ile Lys Thr Cys Trp Asn Lys
130 135 140
Ile Leu Met Gly Thr Lys Glu His Gly Gly Gly Gly Ser Gly Gly Gly
145 150 155 160
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
165 170 175
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly
180 185 190
Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp
195 200 205
Ala Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
210 215 220
Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser
225 230 235 240
Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu
245 250 255
Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr
260 265 270
Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile
275 280 285
Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly
290 295 300
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser
305 310 315 320
Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln
325 330 335
Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val
340 345 350
His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr
355 360 365
Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser
370 375 380
Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly
385 390 395 400
Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His
405 410 415
Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
420 425 430
<210> 277
<211> 129
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL-7
<400> 277
Glu Cys His Ile Lys Asp Lys Glu Gly Lys Ala Tyr Glu Ser Val Leu
1 5 10 15
Met Ile Ser Ile Asp Glu Leu Asp Lys Met Thr Gly Thr Asp Ser Asn
20 25 30
Cys Pro Asn Asn Glu Pro Asn Phe Phe Arg Lys His Val Cys Asp Asp
35 40 45
Thr Lys Glu Ala Ala Phe Leu Asn Arg Ala Ala Arg Lys Leu Lys Gln
50 55 60
Phe Leu Lys Met Asn Ile Ser Glu Glu Phe Asn Val His Leu Leu Thr
65 70 75 80
Val Ser Gln Gly Thr Gln Thr Leu Val Asn Cys Thr Ser Lys Glu Glu
85 90 95
Lys Asn Val Lys Glu Gln Lys Lys Asn Asp Ala Cys Phe Leu Lys Arg
100 105 110
Leu Leu Arg Glu Ile Lys Thr Cys Trp Asn Lys Ile Leu Lys Gly Ser
115 120 125
Ile
<210> 278
<211> 407
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL-7-A10m3
<400> 278
Glu Cys His Ile Lys Asp Lys Glu Gly Lys Ala Tyr Glu Ser Val Leu
1 5 10 15
Met Ile Ser Ile Asp Glu Leu Asp Lys Met Thr Gly Thr Asp Ser Asn
20 25 30
Cys Pro Asn Asn Glu Pro Asn Phe Phe Arg Lys His Val Cys Asp Asp
35 40 45
Thr Lys Glu Ala Ala Phe Leu Asn Arg Ala Ala Arg Lys Leu Lys Gln
50 55 60
Phe Leu Lys Met Asn Ile Ser Glu Glu Phe Asn Val His Leu Leu Thr
65 70 75 80
Val Ser Gln Gly Thr Gln Thr Leu Val Asn Cys Thr Ser Lys Glu Glu
85 90 95
Lys Asn Val Lys Glu Gln Lys Lys Asn Asp Ala Cys Phe Leu Lys Arg
100 105 110
Leu Leu Arg Glu Ile Lys Thr Cys Trp Asn Lys Ile Leu Lys Gly Ser
115 120 125
Ile Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu
145 150 155 160
Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys
165 170 175
Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg
180 185 190
Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn
195 200 205
Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile
210 215 220
Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu
225 230 235 240
Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser
245 250 255
Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val
260 265 270
Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly
275 280 285
Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro
290 295 300
Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly
305 310 315 320
Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro
325 330 335
Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser
340 345 350
Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr
355 360 365
Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys
370 375 380
Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly
385 390 395 400
Thr Lys Leu Thr Val Leu Gly
405
<210> 279
<211> 510
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-12sc
<400> 279
Ile Trp Glu Leu Lys Lys Asp Val Tyr Val Val Glu Leu Asp Trp Tyr
1 5 10 15
Pro Asp Ala Pro Gly Glu Met Val Val Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln Ser Ser Glu Val Leu Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Gln Val Lys Glu Phe Gly Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Val Leu Ser His Ser Leu Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asp Gly Ile Trp Ser Thr Asp Ile Leu Lys
85 90 95
Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe Leu Arg Cys Glu Ala Lys
100 105 110
Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp Leu Thr Thr Ile Ser Thr
115 120 125
Asp Leu Thr Phe Ser Val Lys Ser Ser Arg Gly Ser Ser Asp Pro Gln
130 135 140
Gly Val Thr Cys Gly Ala Ala Thr Leu Ser Ala Glu Arg Val Arg Gly
145 150 155 160
Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu Cys Gln Glu Asp Ser Ala
165 170 175
Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile Glu Val Met Val Asp Ala
180 185 190
Val His Lys Leu Lys Tyr Glu Asn Tyr Thr Ser Ser Phe Phe Ile Arg
195 200 205
Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Leu Lys Pro Leu
210 215 220
Lys Asn Ser Arg Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Thr Trp
225 230 235 240
Ser Thr Pro His Ser Tyr Phe Ser Leu Thr Phe Cys Val Gln Val Gln
245 250 255
Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg Val Phe Thr Asp Lys Thr
260 265 270
Ser Ala Thr Val Ile Cys Arg Lys Asn Ala Ser Ile Ser Val Arg Ala
275 280 285
Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser Glu Trp Ala Ser Val Pro
290 295 300
Cys Ser Gly Gly Gly Gly Gly Gly Ser Arg Asn Leu Pro Val Ala Thr
305 310 315 320
Pro Asp Pro Gly Met Phe Pro Cys Leu His His Ser Gln Asn Leu Leu
325 330 335
Arg Ala Val Ser Asn Met Leu Gln Lys Ala Arg Gln Thr Leu Glu Phe
340 345 350
Tyr Pro Cys Thr Ser Glu Glu Ile Asp His Glu Asp Ile Thr Lys Asp
355 360 365
Lys Thr Ser Thr Val Glu Ala Cys Leu Pro Leu Glu Leu Thr Lys Asn
370 375 380
Glu Ser Cys Leu Asn Ser Arg Glu Thr Ser Phe Ile Thr Asn Gly Ser
385 390 395 400
Cys Leu Ala Ser Arg Lys Thr Ser Phe Met Met Ala Leu Cys Leu Ser
405 410 415
Ser Ile Tyr Glu Asp Leu Lys Met Tyr Gln Val Glu Phe Lys Thr Met
420 425 430
Asn Ala Lys Leu Leu Met Asp Pro Lys Arg Gln Ile Phe Leu Asp Gln
435 440 445
Asn Met Leu Ala Val Ile Asp Glu Leu Met Gln Ala Leu Asn Phe Asn
450 455 460
Ser Glu Thr Val Pro Gln Lys Ser Ser Leu Glu Glu Pro Asp Phe Tyr
465 470 475 480
Lys Thr Lys Ile Lys Leu Cys Ile Leu Leu His Ala Phe Arg Ile Arg
485 490 495
Ala Val Thr Ile Asp Arg Val Thr Ser Tyr Leu Asn Ala Ser
500 505 510
<210> 280
<211> 788
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-12sc-A10m3
<400> 280
Ile Trp Glu Leu Lys Lys Asp Val Tyr Val Val Glu Leu Asp Trp Tyr
1 5 10 15
Pro Asp Ala Pro Gly Glu Met Val Val Leu Thr Cys Asp Thr Pro Glu
20 25 30
Glu Asp Gly Ile Thr Trp Thr Leu Asp Gln Ser Ser Glu Val Leu Gly
35 40 45
Ser Gly Lys Thr Leu Thr Ile Gln Val Lys Glu Phe Gly Asp Ala Gly
50 55 60
Gln Tyr Thr Cys His Lys Gly Gly Glu Val Leu Ser His Ser Leu Leu
65 70 75 80
Leu Leu His Lys Lys Glu Asp Gly Ile Trp Ser Thr Asp Ile Leu Lys
85 90 95
Asp Gln Lys Glu Pro Lys Asn Lys Thr Phe Leu Arg Cys Glu Ala Lys
100 105 110
Asn Tyr Ser Gly Arg Phe Thr Cys Trp Trp Leu Thr Thr Ile Ser Thr
115 120 125
Asp Leu Thr Phe Ser Val Lys Ser Ser Arg Gly Ser Ser Asp Pro Gln
130 135 140
Gly Val Thr Cys Gly Ala Ala Thr Leu Ser Ala Glu Arg Val Arg Gly
145 150 155 160
Asp Asn Lys Glu Tyr Glu Tyr Ser Val Glu Cys Gln Glu Asp Ser Ala
165 170 175
Cys Pro Ala Ala Glu Glu Ser Leu Pro Ile Glu Val Met Val Asp Ala
180 185 190
Val His Lys Leu Lys Tyr Glu Asn Tyr Thr Ser Ser Phe Phe Ile Arg
195 200 205
Asp Ile Ile Lys Pro Asp Pro Pro Lys Asn Leu Gln Leu Lys Pro Leu
210 215 220
Lys Asn Ser Arg Gln Val Glu Val Ser Trp Glu Tyr Pro Asp Thr Trp
225 230 235 240
Ser Thr Pro His Ser Tyr Phe Ser Leu Thr Phe Cys Val Gln Val Gln
245 250 255
Gly Lys Ser Lys Arg Glu Lys Lys Asp Arg Val Phe Thr Asp Lys Thr
260 265 270
Ser Ala Thr Val Ile Cys Arg Lys Asn Ala Ser Ile Ser Val Arg Ala
275 280 285
Gln Asp Arg Tyr Tyr Ser Ser Ser Trp Ser Glu Trp Ala Ser Val Pro
290 295 300
Cys Ser Gly Gly Gly Gly Gly Gly Ser Arg Asn Leu Pro Val Ala Thr
305 310 315 320
Pro Asp Pro Gly Met Phe Pro Cys Leu His His Ser Gln Asn Leu Leu
325 330 335
Arg Ala Val Ser Asn Met Leu Gln Lys Ala Arg Gln Thr Leu Glu Phe
340 345 350
Tyr Pro Cys Thr Ser Glu Glu Ile Asp His Glu Asp Ile Thr Lys Asp
355 360 365
Lys Thr Ser Thr Val Glu Ala Cys Leu Pro Leu Glu Leu Thr Lys Asn
370 375 380
Glu Ser Cys Leu Asn Ser Arg Glu Thr Ser Phe Ile Thr Asn Gly Ser
385 390 395 400
Cys Leu Ala Ser Arg Lys Thr Ser Phe Met Met Ala Leu Cys Leu Ser
405 410 415
Ser Ile Tyr Glu Asp Leu Lys Met Tyr Gln Val Glu Phe Lys Thr Met
420 425 430
Asn Ala Lys Leu Leu Met Asp Pro Lys Arg Gln Ile Phe Leu Asp Gln
435 440 445
Asn Met Leu Ala Val Ile Asp Glu Leu Met Gln Ala Leu Asn Phe Asn
450 455 460
Ser Glu Thr Val Pro Gln Lys Ser Ser Leu Glu Glu Pro Asp Phe Tyr
465 470 475 480
Lys Thr Lys Ile Lys Leu Cys Ile Leu Leu His Ala Phe Arg Ile Arg
485 490 495
Ala Val Thr Ile Asp Arg Val Thr Ser Tyr Leu Asn Ala Ser Gly Gly
500 505 510
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
515 520 525
Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
530 535 540
Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser
545 550 555 560
Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln Ala Pro
565 570 575
Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr
580 585 590
Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
595 600 605
Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu
610 615 620
Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg
625 630 635 640
Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser
645 650 655
Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
660 665 670
Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val
675 680 685
Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn
690 695 700
Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala
705 710 715 720
Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro
725 730 735
Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile
740 745 750
Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp
755 760 765
Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu
770 775 780
Thr Val Leu Gly
785
<210> 281
<211> 157
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-18-A10m3
<400> 281
Tyr Phe Gly Lys Leu Glu Ser Lys Leu Ser Val Ile Arg Asn Leu Asn
1 5 10 15
Asp Gln Val Leu Phe Ile Asp Gln Gly Asn Arg Pro Leu Phe Glu Asp
20 25 30
Met Thr Asp Ser Asp Cys Arg Asp Asn Ala Pro Arg Thr Ile Phe Ile
35 40 45
Ile Ser Met Tyr Lys Asp Ser Gln Pro Arg Gly Met Ala Val Thr Ile
50 55 60
Ser Val Lys Cys Glu Lys Ile Ser Thr Leu Ser Cys Glu Asn Lys Ile
65 70 75 80
Ile Ser Phe Lys Glu Met Asn Pro Pro Asp Asn Ile Lys Asp Thr Lys
85 90 95
Ser Asp Ile Ile Phe Phe Gln Arg Ser Val Pro Gly His Asp Asn Lys
100 105 110
Met Gln Phe Glu Ser Ser Ser Tyr Glu Gly Tyr Phe Leu Ala Cys Glu
115 120 125
Lys Glu Arg Asp Leu Phe Lys Leu Ile Leu Lys Lys Glu Asp Glu Leu
130 135 140
Gly Asp Arg Ser Ile Met Phe Thr Val Gln Asn Glu Asp
145 150 155
<210> 282
<211> 435
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-18-A10m3
<400> 282
Tyr Phe Gly Lys Leu Glu Ser Lys Leu Ser Val Ile Arg Asn Leu Asn
1 5 10 15
Asp Gln Val Leu Phe Ile Asp Gln Gly Asn Arg Pro Leu Phe Glu Asp
20 25 30
Met Thr Asp Ser Asp Cys Arg Asp Asn Ala Pro Arg Thr Ile Phe Ile
35 40 45
Ile Ser Met Tyr Lys Asp Ser Gln Pro Arg Gly Met Ala Val Thr Ile
50 55 60
Ser Val Lys Cys Glu Lys Ile Ser Thr Leu Ser Cys Glu Asn Lys Ile
65 70 75 80
Ile Ser Phe Lys Glu Met Asn Pro Pro Asp Asn Ile Lys Asp Thr Lys
85 90 95
Ser Asp Ile Ile Phe Phe Gln Arg Ser Val Pro Gly His Asp Asn Lys
100 105 110
Met Gln Phe Glu Ser Ser Ser Tyr Glu Gly Tyr Phe Leu Ala Cys Glu
115 120 125
Lys Glu Arg Asp Leu Phe Lys Leu Ile Leu Lys Lys Glu Asp Glu Leu
130 135 140
Gly Asp Arg Ser Ile Met Phe Thr Val Gln Asn Glu Asp Gly Gly Gly
145 150 155 160
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
165 170 175
Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly
180 185 190
Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
195 200 205
Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln Ala Pro Gly
210 215 220
Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile
225 230 235 240
Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
245 250 255
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu Asp
260 265 270
Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly
275 280 285
Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
290 295 300
Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
305 310 315 320
Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser
325 330 335
Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile
340 345 350
Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
355 360 365
Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu
370 375 380
Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser
385 390 395 400
Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp
405 410 415
Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
420 425 430
Val Leu Gly
435
<210> 283
<211> 435
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL-18-A10m3
<400> 283
Asn Phe Gly Arg Leu His Cys Thr Thr Ala Val Ile Arg Asn Ile Asn
1 5 10 15
Asp Gln Val Leu Phe Val Asp Lys Arg Gln Pro Val Phe Glu Asp Met
20 25 30
Thr Asp Ile Asp Gln Ser Ala Ser Glu Pro Gln Thr Arg Leu Ile Ile
35 40 45
Tyr Met Tyr Lys Asp Ser Glu Val Arg Gly Leu Ala Val Thr Leu Ser
50 55 60
Val Lys Asp Ser Lys Met Ser Thr Leu Ser Cys Lys Asn Lys Ile Ile
65 70 75 80
Ser Phe Glu Glu Met Asp Pro Pro Glu Asn Ile Asp Asp Ile Gln Ser
85 90 95
Asp Leu Ile Phe Phe Gln Lys Arg Val Pro Gly His Asn Lys Met Glu
100 105 110
Phe Glu Ser Ser Leu Tyr Glu Gly His Phe Leu Ala Cys Gln Lys Glu
115 120 125
Asp Asp Ala Phe Lys Leu Ile Leu Lys Lys Lys Asp Glu Asn Gly Asp
130 135 140
Lys Ser Val Met Phe Thr Leu Thr Asn Leu His Gln Ser Gly Gly Gly
145 150 155 160
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
165 170 175
Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly
180 185 190
Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly
195 200 205
Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln Ala Pro Gly
210 215 220
Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile
225 230 235 240
Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
245 250 255
Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu Asp
260 265 270
Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly
275 280 285
Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser
290 295 300
Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
305 310 315 320
Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser
325 330 335
Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile
340 345 350
Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro
355 360 365
Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu
370 375 380
Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser
385 390 395 400
Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp
405 410 415
Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
420 425 430
Val Leu Gly
435
<210> 284
<211> 133
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-21
<400> 284
Gln Gly Gln Asp Arg His Met Ile Arg Met Arg Gln Leu Ile Asp Ile
1 5 10 15
Val Asp Gln Leu Lys Asn Tyr Val Asn Asp Leu Val Pro Glu Phe Leu
20 25 30
Pro Ala Pro Glu Asp Val Glu Thr Asn Cys Glu Trp Ser Ala Phe Ser
35 40 45
Cys Phe Gln Lys Ala Gln Leu Lys Ser Ala Asn Thr Gly Asn Asn Glu
50 55 60
Arg Ile Ile Asn Val Ser Ile Lys Lys Leu Lys Arg Lys Pro Pro Ser
65 70 75 80
Thr Asn Ala Gly Arg Arg Gln Lys His Arg Leu Thr Cys Pro Ser Cys
85 90 95
Asp Ser Tyr Glu Lys Lys Pro Pro Lys Glu Phe Leu Glu Arg Phe Lys
100 105 110
Ser Leu Leu Gln Lys Met Ile His Gln His Leu Ser Ser Arg Thr His
115 120 125
Gly Ser Glu Asp Ser
130
<210> 285
<211> 411
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-21-A10m3
<400> 285
Gln Gly Gln Asp Arg His Met Ile Arg Met Arg Gln Leu Ile Asp Ile
1 5 10 15
Val Asp Gln Leu Lys Asn Tyr Val Asn Asp Leu Val Pro Glu Phe Leu
20 25 30
Pro Ala Pro Glu Asp Val Glu Thr Asn Cys Glu Trp Ser Ala Phe Ser
35 40 45
Cys Phe Gln Lys Ala Gln Leu Lys Ser Ala Asn Thr Gly Asn Asn Glu
50 55 60
Arg Ile Ile Asn Val Ser Ile Lys Lys Leu Lys Arg Lys Pro Pro Ser
65 70 75 80
Thr Asn Ala Gly Arg Arg Gln Lys His Arg Leu Thr Cys Pro Ser Cys
85 90 95
Asp Ser Tyr Glu Lys Lys Pro Pro Lys Glu Phe Leu Glu Arg Phe Lys
100 105 110
Ser Leu Leu Gln Lys Met Ile His Gln His Leu Ser Ser Arg Thr His
115 120 125
Gly Ser Glu Asp Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val
145 150 155 160
Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu
165 170 175
Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met
180 185 190
His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly
195 200 205
Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly
210 215 220
Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln
225 230 235 240
Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys
245 250 255
Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln
260 265 270
Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser
275 280 285
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val
290 295 300
Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr
305 310 315 320
Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr
325 330 335
Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser
340 345 350
Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly
355 360 365
Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala
370 375 380
Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp Val
385 390 395 400
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
405 410
<210> 286
<211> 122
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL-21
<400> 286
Pro Asp Arg Leu Leu Ile Arg Leu Arg His Leu Ile Asp Ile Val Glu
1 5 10 15
Gln Leu Lys Ile Tyr Glu Asn Asp Leu Asp Pro Glu Leu Leu Ser Ala
20 25 30
Pro Gln Asp Val Lys Gly His Cys Glu His Ala Ala Phe Ala Cys Phe
35 40 45
Gln Lys Ala Lys Leu Lys Pro Ser Asn Pro Gly Asn Asn Lys Thr Phe
50 55 60
Ile Ile Asp Leu Val Ala Gln Leu Arg Arg Arg Leu Pro Ala Arg Arg
65 70 75 80
Gly Gly Lys Lys Gln Lys His Ile Ala Lys Cys Pro Ser Cys Asp Ser
85 90 95
Tyr Glu Lys Arg Thr Pro Lys Glu Phe Leu Glu Arg Leu Lys Trp Leu
100 105 110
Leu Gln Lys Met Ile His Gln His Leu Ser
115 120
<210> 287
<211> 400
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL-21-A10m3
<400> 287
Pro Asp Arg Leu Leu Ile Arg Leu Arg His Leu Ile Asp Ile Val Glu
1 5 10 15
Gln Leu Lys Ile Tyr Glu Asn Asp Leu Asp Pro Glu Leu Leu Ser Ala
20 25 30
Pro Gln Asp Val Lys Gly His Cys Glu His Ala Ala Phe Ala Cys Phe
35 40 45
Gln Lys Ala Lys Leu Lys Pro Ser Asn Pro Gly Asn Asn Lys Thr Phe
50 55 60
Ile Ile Asp Leu Val Ala Gln Leu Arg Arg Arg Leu Pro Ala Arg Arg
65 70 75 80
Gly Gly Lys Lys Gln Lys His Ile Ala Lys Cys Pro Ser Cys Asp Ser
85 90 95
Tyr Glu Lys Arg Thr Pro Lys Glu Phe Leu Glu Arg Leu Lys Trp Leu
100 105 110
Leu Gln Lys Met Ile His Gln His Leu Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
130 135 140
Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln
145 150 155 160
Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe
165 170 175
Asp Asp Ala Val Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu
180 185 190
Glu Trp Val Ala Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala
195 200 205
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
210 215 220
Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val
225 230 235 240
Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe
245 250 255
Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr
260 265 270
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val
275 280 285
His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro
290 295 300
Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys
305 310 315 320
Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val
325 330 335
Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser
340 345 350
Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu
355 360 365
Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser
370 375 380
Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
385 390 395 400
<210> 288
<211> 209
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-27sc
<400> 288
Arg Lys Gly Pro Pro Ala Ala Leu Thr Leu Pro Arg Val Gln Cys Arg
1 5 10 15
Ala Ser Arg Tyr Pro Ile Ala Val Asp Cys Ser Trp Thr Leu Pro Pro
20 25 30
Ala Pro Asn Ser Thr Ser Pro Val Ser Phe Ile Ala Thr Tyr Arg Leu
35 40 45
Gly Met Ala Ala Arg Gly His Ser Trp Pro Cys Leu Gln Gln Thr Pro
50 55 60
Thr Ser Thr Ser Cys Thr Ile Thr Asp Val Gln Leu Phe Ser Met Ala
65 70 75 80
Pro Tyr Val Leu Asn Val Thr Ala Val His Pro Trp Gly Ser Ser Ser
85 90 95
Ser Phe Val Pro Phe Ile Thr Glu His Ile Ile Lys Pro Asp Pro Pro
100 105 110
Glu Gly Val Arg Leu Ser Pro Leu Ala Glu Arg Gln Leu Gln Val Gln
115 120 125
Trp Glu Pro Pro Gly Ser Trp Pro Phe Pro Glu Ile Phe Ser Leu Lys
130 135 140
Tyr Trp Ile Arg Tyr Lys Arg Gln Gly Ala Ala Arg Phe His Arg Val
145 150 155 160
Gly Pro Ile Glu Ala Thr Ser Phe Ile Leu Arg Ala Val Arg Pro Arg
165 170 175
Ala Arg Tyr Tyr Val Gln Val Ala Ala Gln Asp Leu Thr Asp Tyr Gly
180 185 190
Glu Leu Ser Asp Trp Ser Leu Pro Ala Thr Ala Thr Met Ser Leu Gly
195 200 205
Lys
<210> 289
<211> 716
<212> PRT
<213> Artificial Sequence
<220>
<223> Human hIL-27sc-A10m3
<400> 289
Lys Gly Pro Pro Ala Ala Leu Thr Leu Pro Arg Val Gln Cys Arg Ala
1 5 10 15
Ser Arg Tyr Pro Ile Ala Val Asp Cys Ser Trp Thr Leu Pro Pro Ala
20 25 30
Pro Asn Ser Thr Ser Pro Val Ser Phe Ile Ala Thr Tyr Arg Leu Gly
35 40 45
Met Ala Ala Arg Gly His Ser Trp Pro Cys Leu Gln Gln Thr Pro Thr
50 55 60
Ser Thr Ser Cys Thr Ile Thr Asp Val Gln Leu Phe Ser Met Ala Pro
65 70 75 80
Tyr Val Leu Asn Val Thr Ala Val His Pro Trp Gly Ser Ser Ser Ser
85 90 95
Phe Val Pro Phe Ile Thr Glu His Ile Ile Lys Pro Asp Pro Pro Glu
100 105 110
Gly Val Arg Leu Ser Pro Leu Ala Glu Arg Gln Leu Gln Val Gln Trp
115 120 125
Glu Pro Pro Gly Ser Trp Pro Phe Pro Glu Ile Phe Ser Leu Lys Tyr
130 135 140
Trp Ile Arg Tyr Lys Arg Gln Gly Ala Ala Arg Phe His Arg Val Gly
145 150 155 160
Pro Ile Glu Ala Thr Ser Phe Ile Leu Arg Ala Val Arg Pro Arg Ala
165 170 175
Arg Tyr Tyr Val Gln Val Ala Ala Gln Asp Leu Thr Asp Tyr Gly Glu
180 185 190
Leu Ser Asp Trp Ser Leu Pro Ala Thr Ala Thr Met Ser Leu Gly Lys
195 200 205
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Phe
210 215 220
Pro Arg Pro Pro Gly Arg Pro Gln Leu Ser Leu Gln Glu Leu Arg Arg
225 230 235 240
Glu Phe Thr Val Ser Leu His Leu Ala Arg Lys Leu Leu Ser Glu Val
245 250 255
Arg Gly Gln Ala His Arg Phe Ala Glu Ser His Leu Pro Gly Val Asn
260 265 270
Leu Tyr Leu Leu Pro Leu Gly Glu Gln Leu Pro Asp Val Ser Leu Thr
275 280 285
Phe Gln Ala Trp Arg Arg Leu Ser Asp Pro Glu Arg Leu Cys Phe Ile
290 295 300
Ser Thr Thr Leu Gln Pro Phe His Ala Pro Leu Gly Gly Leu Gly Thr
305 310 315 320
Gln Gly Arg Trp Thr Asn Met Glu Arg Met Gln Leu Trp Ala Met Arg
325 330 335
Leu Asp Leu Arg Asp Leu Gln Arg His Leu Arg Phe Gln Val Leu Ala
340 345 350
Ala Gly Phe Asn Leu Pro Glu Glu Glu Glu Glu Glu Glu Glu Glu Glu
355 360 365
Glu Glu Glu Arg Lys Gly Leu Leu Pro Gly Ala Leu Gly Ser Ala Leu
370 375 380
Gln Gly Pro Ala Gln Val Ser Trp Pro Gln Leu Leu Ser Thr Tyr Arg
385 390 395 400
Leu Leu His Ser Leu Glu Leu Val Leu Ser Arg Ala Val Arg Glu Leu
405 410 415
Leu Leu Leu Ser Lys Ala Gly His Ser Val Trp Pro Leu Gly Phe Pro
420 425 430
Thr Leu Ser Pro Gln Pro Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
435 440 445
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
450 455 460
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser
465 470 475 480
Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val
485 490 495
Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
500 505 510
Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys
515 520 525
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu
530 535 540
Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val
545 550 555 560
Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly
565 570 575
Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly
580 585 590
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr
595 600 605
Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala
610 615 620
Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp
625 630 635 640
Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp
645 650 655
Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser
660 665 670
Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu
675 680 685
Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp
690 695 700
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
705 710 715
<210> 290
<211> 210
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL-27sc
<400> 290
Tyr Thr Glu Thr Ala Leu Val Ala Leu Ser Gln Pro Arg Val Gln Cys
1 5 10 15
His Ala Ser Arg Tyr Pro Val Ala Val Asp Cys Ser Trp Thr Pro Leu
20 25 30
Gln Ala Pro Asn Ser Thr Arg Ser Thr Ser Phe Ile Ala Thr Tyr Arg
35 40 45
Leu Gly Val Ala Thr Gln Gln Gln Ser Gln Pro Cys Leu Gln Arg Ser
50 55 60
Pro Gln Ala Ser Arg Cys Thr Ile Pro Asp Val His Leu Phe Ser Thr
65 70 75 80
Val Pro Tyr Met Leu Asn Val Thr Ala Val His Pro Gly Gly Ala Ser
85 90 95
Ser Ser Leu Leu Ala Phe Val Ala Glu Arg Ile Ile Lys Pro Asp Pro
100 105 110
Pro Glu Gly Val Arg Leu Arg Thr Ala Gly Gln Arg Leu Gln Val Leu
115 120 125
Trp His Pro Pro Ala Ser Trp Pro Phe Pro Asp Ile Phe Ser Leu Lys
130 135 140
Tyr Arg Leu Arg Tyr Arg Arg Arg Gly Ala Ser His Phe Arg Gln Val
145 150 155 160
Gly Pro Ile Glu Ala Thr Thr Phe Thr Leu Arg Asn Ser Lys Pro His
165 170 175
Ala Lys Tyr Cys Ile Gln Val Ser Ala Gln Asp Leu Thr Asp Tyr Gly
180 185 190
Lys Pro Ser Asp Trp Ser Leu Pro Gly Gln Val Glu Ser Ala Pro His
195 200 205
Lys Pro
210
<210> 291
<211> 709
<212> PRT
<213> Artificial Sequence
<220>
<223> Mouse mIL-27sc-A10m3
<400> 291
Tyr Thr Glu Thr Ala Leu Val Ala Leu Ser Gln Pro Arg Val Gln Cys
1 5 10 15
His Ala Ser Arg Tyr Pro Val Ala Val Asp Cys Ser Trp Thr Pro Leu
20 25 30
Gln Ala Pro Asn Ser Thr Arg Ser Thr Ser Phe Ile Ala Thr Tyr Arg
35 40 45
Leu Gly Val Ala Thr Gln Gln Gln Ser Gln Pro Cys Leu Gln Arg Ser
50 55 60
Pro Gln Ala Ser Arg Cys Thr Ile Pro Asp Val His Leu Phe Ser Thr
65 70 75 80
Val Pro Tyr Met Leu Asn Val Thr Ala Val His Pro Gly Gly Ala Ser
85 90 95
Ser Ser Leu Leu Ala Phe Val Ala Glu Arg Ile Ile Lys Pro Asp Pro
100 105 110
Pro Glu Gly Val Arg Leu Arg Thr Ala Gly Gln Arg Leu Gln Val Leu
115 120 125
Trp His Pro Pro Ala Ser Trp Pro Phe Pro Asp Ile Phe Ser Leu Lys
130 135 140
Tyr Arg Leu Arg Tyr Arg Arg Arg Gly Ala Ser His Phe Arg Gln Val
145 150 155 160
Gly Pro Ile Glu Ala Thr Thr Phe Thr Leu Arg Asn Ser Lys Pro His
165 170 175
Ala Lys Tyr Cys Ile Gln Val Ser Ala Gln Asp Leu Thr Asp Tyr Gly
180 185 190
Lys Pro Ser Asp Trp Ser Leu Pro Gly Gln Val Glu Ser Ala Pro His
195 200 205
Lys Pro Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Phe Pro Thr Asp Pro Leu Ser Leu Gln Glu Leu Arg Arg Glu Phe
225 230 235 240
Thr Val Ser Leu Tyr Leu Ala Arg Lys Leu Leu Ser Glu Val Gln Gly
245 250 255
Tyr Val His Ser Phe Ala Glu Ser Arg Leu Pro Gly Val Asn Leu Asp
260 265 270
Leu Leu Pro Leu Gly Tyr His Leu Pro Asn Val Ser Leu Thr Phe Gln
275 280 285
Ala Trp His His Leu Ser Asp Ser Glu Arg Leu Cys Phe Leu Ala Thr
290 295 300
Thr Leu Arg Pro Phe Pro Ala Met Leu Gly Gly Leu Gly Thr Gln Gly
305 310 315 320
Thr Trp Thr Ser Ser Glu Arg Glu Gln Leu Trp Ala Met Arg Leu Asp
325 330 335
Leu Arg Asp Leu His Arg His Leu Arg Phe Gln Val Leu Ala Ala Gly
340 345 350
Phe Lys Cys Ser Lys Glu Glu Glu Asp Lys Glu Glu Glu Glu Glu Glu
355 360 365
Glu Glu Glu Glu Lys Lys Leu Pro Leu Gly Ala Leu Gly Gly Pro Asn
370 375 380
Gln Val Ser Ser Gln Val Ser Trp Pro Gln Leu Leu Tyr Thr Tyr Gln
385 390 395 400
Leu Leu His Ser Leu Glu Leu Val Leu Ser Arg Ala Val Arg Asp Leu
405 410 415
Leu Leu Leu Ser Leu Pro Arg Arg Pro Gly Ser Ala Trp Asp Ser Gly
420 425 430
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
435 440 445
Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
450 455 460
Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala Ala
465 470 475 480
Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln Ala
485 490 495
Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser Gly
500 505 510
Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
515 520 525
Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg Ala
530 535 540
Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn Pro
545 550 555 560
Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr Val
565 570 575
Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
580 585 590
Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro Ser
595 600 605
Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly Asn
610 615 620
Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly Gln
625 630 635 640
Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly Ile
645 650 655
Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu Thr
660 665 670
Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln Val
675 680 685
Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr Lys
690 695 700
Leu Thr Val Leu Gly
705
<210> 292
<211> 144
<212> PRT
<213> Artificial Sequence
<220>
<223> Human GM-CSF
<400> 292
Met Trp Leu Gln Ser Leu Leu Leu Leu Gly Thr Val Ala Cys Ser Ile
1 5 10 15
Ser Ala Pro Ala Arg Ser Pro Ser Pro Ser Thr Gln Pro Trp Glu His
20 25 30
Val Asn Ala Ile Gln Glu Ala Arg Arg Leu Leu Asn Leu Ser Arg Asp
35 40 45
Thr Ala Ala Glu Met Asn Glu Thr Val Glu Val Ile Ser Glu Met Phe
50 55 60
Asp Leu Gln Glu Pro Thr Cys Leu Gln Thr Arg Leu Glu Leu Tyr Lys
65 70 75 80
Gln Gly Leu Arg Gly Ser Leu Thr Lys Leu Lys Gly Pro Leu Thr Met
85 90 95
Met Ala Ser His Tyr Lys Gln His Cys Pro Pro Thr Pro Glu Thr Ser
100 105 110
Cys Ala Thr Gln Ile Ile Thr Phe Glu Ser Phe Lys Glu Asn Leu Lys
115 120 125
Asp Phe Leu Leu Val Ile Pro Phe Asp Cys Trp Glu Pro Val Gln Glu
130 135 140
<210> 293
<211> 422
<212> PRT
<213> Artificial Sequence
<220>
<223> Human GM-CSF-A10m3
<400> 293
Met Trp Leu Gln Ser Leu Leu Leu Leu Gly Thr Val Ala Cys Ser Ile
1 5 10 15
Ser Ala Pro Ala Arg Ser Pro Ser Pro Ser Thr Gln Pro Trp Glu His
20 25 30
Val Asn Ala Ile Gln Glu Ala Arg Arg Leu Leu Asn Leu Ser Arg Asp
35 40 45
Thr Ala Ala Glu Met Asn Glu Thr Val Glu Val Ile Ser Glu Met Phe
50 55 60
Asp Leu Gln Glu Pro Thr Cys Leu Gln Thr Arg Leu Glu Leu Tyr Lys
65 70 75 80
Gln Gly Leu Arg Gly Ser Leu Thr Lys Leu Lys Gly Pro Leu Thr Met
85 90 95
Met Ala Ser His Tyr Lys Gln His Cys Pro Pro Thr Pro Glu Thr Ser
100 105 110
Cys Ala Thr Gln Ile Ile Thr Phe Glu Ser Phe Lys Glu Asn Leu Lys
115 120 125
Asp Phe Leu Leu Val Ile Pro Phe Asp Cys Trp Glu Pro Val Gln Glu
130 135 140
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
145 150 155 160
Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser
165 170 175
Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala
180 185 190
Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln
195 200 205
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser
210 215 220
Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
225 230 235 240
Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg
245 250 255
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn
260 265 270
Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr
275 280 285
Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
290 295 300
Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro
305 310 315 320
Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly
325 330 335
Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly
340 345 350
Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly
355 360 365
Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu
370 375 380
Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln
385 390 395 400
Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr
405 410 415
Lys Leu Thr Val Leu Gly
420
<210> 294
<211> 166
<212> PRT
<213> Artificial Sequence
<220>
<223> Human IFN-?
<400> 294
Cys Asp Leu Pro Glu Thr His Ser Leu Asp Asn Arg Arg Thr Leu Met
1 5 10 15
Leu Leu Ala Gln Met Ser Arg Ile Ser Pro Ser Ser Cys Leu Met Asp
20 25 30
Arg His Asp Phe Gly Phe Pro Gln Glu Glu Phe Asp Gly Asn Gln Phe
35 40 45
Gln Lys Ala Pro Ala Ile Ser Val Leu His Glu Leu Ile Gln Gln Ile
50 55 60
Phe Asn Leu Phe Thr Thr Lys Asp Ser Ser Ala Ala Trp Asp Glu Asp
65 70 75 80
Leu Leu Asp Lys Phe Cys Thr Glu Leu Tyr Gln Gln Leu Asn Asp Leu
85 90 95
Glu Ala Cys Val Met Gln Glu Glu Arg Val Gly Glu Thr Pro Leu Met
100 105 110
Asn Ala Asp Ser Ile Leu Ala Val Lys Lys Tyr Phe Arg Arg Ile Thr
115 120 125
Leu Tyr Leu Thr Glu Lys Lys Tyr Ser Pro Cys Ala Trp Glu Val Val
130 135 140
Arg Ala Glu Ile Met Arg Ser Leu Ser Leu Ser Thr Asn Leu Gln Glu
145 150 155 160
Arg Leu Arg Arg Lys Glu
165
<210> 295
<211> 444
<212> PRT
<213> Artificial Sequence
<220>
<223> Human IFN-?-A10m3
<400> 295
Cys Asp Leu Pro Glu Thr His Ser Leu Asp Asn Arg Arg Thr Leu Met
1 5 10 15
Leu Leu Ala Gln Met Ser Arg Ile Ser Pro Ser Ser Cys Leu Met Asp
20 25 30
Arg His Asp Phe Gly Phe Pro Gln Glu Glu Phe Asp Gly Asn Gln Phe
35 40 45
Gln Lys Ala Pro Ala Ile Ser Val Leu His Glu Leu Ile Gln Gln Ile
50 55 60
Phe Asn Leu Phe Thr Thr Lys Asp Ser Ser Ala Ala Trp Asp Glu Asp
65 70 75 80
Leu Leu Asp Lys Phe Cys Thr Glu Leu Tyr Gln Gln Leu Asn Asp Leu
85 90 95
Glu Ala Cys Val Met Gln Glu Glu Arg Val Gly Glu Thr Pro Leu Met
100 105 110
Asn Ala Asp Ser Ile Leu Ala Val Lys Lys Tyr Phe Arg Arg Ile Thr
115 120 125
Leu Tyr Leu Thr Glu Lys Lys Tyr Ser Pro Cys Ala Trp Glu Val Val
130 135 140
Arg Ala Glu Ile Met Arg Ser Leu Ser Leu Ser Thr Asn Leu Gln Glu
145 150 155 160
Arg Leu Arg Arg Lys Glu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
165 170 175
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
180 185 190
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser
195 200 205
Leu Arg Leu Ser Cys Ala Ala Ser Gly Ile Thr Phe Asp Asp Ala Val
210 215 220
Met His Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala
225 230 235 240
Gly Ile Ser Ser Asn Ser Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys
245 250 255
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu
260 265 270
Gln Met Asn Arg Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val
275 280 285
Lys Gly Leu Tyr Ser Asn Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly
290 295 300
Gln Gly Thr Met Val Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly
305 310 315 320
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Val His Ser Ser Tyr
325 330 335
Val Leu Thr Gln Pro Pro Ser Val Ser Val Ala Pro Gly Gln Thr Ala
340 345 350
Thr Ile Thr Cys Gly Gly Asn Asn Ile Gly Thr Lys Ser Val His Trp
355 360 365
Tyr Gln Gln Lys Pro Gly Gln Ala Pro Val Leu Val Val Tyr Ala Asp
370 375 380
Ser Asp Arg Pro Ser Gly Ile Pro Glu Arg Val Ser Gly Ser Asn Ser
385 390 395 400
Gly Asn Thr Ala Thr Leu Thr Ile Ser Arg Val Glu Ala Gly Asp Glu
405 410 415
Ala Asp Tyr Tyr Cys Gln Val Trp Asp Ser Arg Ser Asp His Leu Trp
420 425 430
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly
435 440
<210> 296
<211> 118
<212> PRT
<213> Artificial Sequence
<220>
<223> Anti-PD-L1 10D12, Variable Heavy Domain
<400> 296
Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly Ser
1 5 10 15
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr Tyr
20 25 30
Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
35 40 45
Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val Lys
50 55 60
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu
65 70 75 80
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
85 90 95
Ser Arg Ser Ser Met Ala Gln His Trp Gly Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Ala Ser Thr
115
<210> 297
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR1
<400> 297
Gly Phe Thr Phe Ser Asp Tyr Tyr
1 5
<210> 298
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR2
<400> 298
Ile Ser Ser Ser Gly Ser Thr Ile
1 5
<210> 299
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vhCDR3
<400> 299
Ala Ser Arg Ser Ser Met Ala Gln His
1 5
<210> 300
<211> 112
<212> PRT
<213> Artificial Sequence
<220>
<223> Variable Light Domain
<400> 300
Ser Asn Phe Met Leu Thr Gln Pro His Ser Val Ser Glu Ser Pro Gly
1 5 10 15
Lys Thr Val Thr Ile Ser Cys Thr Arg Ser Ser Gly Ser Ile Ala Ser
20 25 30
Tyr Tyr Val Gln Trp Tyr Gln Gln Arg Pro Gly Ser Ala Pro Thr Thr
35 40 45
Val Ile Tyr Glu Asp Asn Gln Arg Pro Ser Gly Val Pro Asp Arg Phe
50 55 60
Ser Gly Ser Ile Asp Ser Ser Ser Asn Ser Ala Ser Leu Thr Ile Ser
65 70 75 80
Gly Leu Lys Thr Glu Asp Glu Ala Asp Tyr Tyr Cys Gln Ser Tyr Asp
85 90 95
Ser Asn Asn Gln Val Phe Gly Gly Gly Thr Lys Val Thr Val Leu Gly
100 105 110
<210> 301
<211> 8
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR1
<400> 301
Ser Gly Ser Ile Ala Ser Tyr Tyr
1 5
<210> 302
<211> 3
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR2
<400> 302
Glu Asp Asn
1
<210> 303
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> vlCDR3
<400> 303
Gln Ser Tyr Asp Ser Asn Asn Gln Val
1 5
<210> 304
<211> 245
<212> PRT
<213> Artificial Sequence
<220>
<223> scFv
<400> 304
Gln Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Lys Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Asp Tyr
20 25 30
Tyr Met Ser Trp Ile Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Tyr Ile Ser Ser Ser Gly Ser Thr Ile Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Ser Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ser Arg Ser Ser Met Ala Gln His Trp Gly Gln Gly Thr Leu Val
100 105 110
Thr Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Gly Ser Asn Phe Met Leu Thr Gln Pro His Ser Val
130 135 140
Ser Glu Ser Pro Gly Lys Thr Val Thr Ile Ser Cys Thr Arg Ser Ser
145 150 155 160
Gly Ser Ile Ala Ser Tyr Tyr Val Gln Trp Tyr Gln Gln Arg Pro Gly
165 170 175
Ser Ala Pro Thr Thr Val Ile Tyr Glu Asp Asn Gln Arg Pro Ser Gly
180 185 190
Val Pro Asp Arg Phe Ser Gly Ser Ile Asp Ser Ser Ser Asn Ser Ala
195 200 205
Ser Leu Thr Ile Ser Gly Leu Lys Thr Glu Asp Glu Ala Asp Tyr Tyr
210 215 220
Cys Gln Ser Tyr Asp Ser Asn Asn Gln Val Phe Gly Gly Gly Thr Lys
225 230 235 240
Val Thr Val Leu Gly
245
<210> 305
<211> 303
<212> PRT
<213> Artificial Sequence
<220>
<223> linker-ABD-linker
<400> 305
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser
20 25 30
Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala
35 40 45
Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln
50 55 60
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser
65 70 75 80
Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
85 90 95
Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg
100 105 110
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn
115 120 125
Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr
130 135 140
Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro
165 170 175
Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly
180 185 190
Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly
195 200 205
Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly
210 215 220
Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu
225 230 235 240
Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln
245 250 255
Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr
260 265 270
Lys Leu Thr Val Leu Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
275 280 285
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
290 295 300
<210> 306
<211> 303
<212> PRT
<213> Artificial Sequence
<220>
<223> linker-ABD-linker
<400> 306
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser
20 25 30
Gly Gly Gly Leu Ile Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Ala
35 40 45
Ala Ser Gly Ile Thr Phe Asp Asp Ala Val Met His Trp Val Arg Gln
50 55 60
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Gly Ile Ser Ser Asn Ser
65 70 75 80
Gly Tyr Ile Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser
85 90 95
Arg Asp Asn Ala Lys Asn Ser Leu Tyr Leu Gln Met Asn Arg Leu Arg
100 105 110
Ala Glu Asp Thr Ala Val Tyr Tyr Cys Val Lys Gly Leu Tyr Ser Asn
115 120 125
Pro Arg Gly Gly Ala Phe Asp Ile Trp Gly Gln Gly Thr Met Val Thr
130 135 140
Val Ser Ser Ala Ser Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
145 150 155 160
Gly Gly Gly Gly Ser Val His Ser Ser Tyr Val Leu Thr Gln Pro Pro
165 170 175
Ser Val Ser Val Ala Pro Gly Gln Thr Ala Thr Ile Thr Cys Gly Gly
180 185 190
Asn Asn Ile Gly Thr Lys Ser Val His Trp Tyr Gln Gln Lys Pro Gly
195 200 205
Gln Ala Pro Val Leu Val Val Tyr Ala Asp Ser Asp Arg Pro Ser Gly
210 215 220
Ile Pro Glu Arg Val Ser Gly Ser Asn Ser Gly Asn Thr Ala Thr Leu
225 230 235 240
Thr Ile Ser Arg Val Glu Ala Gly Asp Glu Ala Asp Tyr Tyr Cys Gln
245 250 255
Val Trp Asp Ser Arg Ser Asp His Leu Trp Val Phe Gly Gly Gly Thr
260 265 270
Lys Leu Thr Val Leu Gly Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
275 280 285
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
290 295 300
<210> 307
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> ABD linker
<400> 307
Gly Gly Gly Gly Ser
1 5
<210> 308
<211> 2
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker
<400> 308
Gly Ser
1
<210> 309
<211> 5
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker
<400> 309
Gly Ser Gly Gly Ser
1 5
<210> 310
<211> 4
<212> PRT
<213> Artificial Sequence
<220>
<223> Linker
<400> 310
Gly Gly Gly Ser
1
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| IL-17A-связывающие полипептиды | 2016 |
|
RU2749712C2 |
| IL-12 ГЕТЕРОДИМЕРНЫЕ СЛИТЫЕ БЕЛКИ FC | 2019 |
|
RU2819097C2 |
| ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2018 |
|
RU2815452C2 |
| АНТИ-PVRIG АНТИТЕЛА И СПОСОБЫ ПРИМЕНЕНИЯ | 2016 |
|
RU2732042C2 |
| АНТИ-PVRIG АНТИТЕЛА И СПОСОБЫ ПРИМЕНЕНИЯ | 2016 |
|
RU2827926C2 |
| РЕКРУТИРУЮЩИЕ Т-КЛЕТКИ ПОЛИПЕПТИДЫ, СПОСОБНЫЕ СВЯЗЫВАТЬ CD123 И TCR АЛЬФА/БЕТА | 2017 |
|
RU2775063C2 |
| ЛИГАНДСВЯЗЫВАЮЩАЯ МОЛЕКУЛА С РЕГУЛИРУЕМОЙ ЛИГАНДСВЯЗЫВАЮЩЕЙ АКТИВНОСТЬЮ | 2017 |
|
RU2803067C2 |
| ПОЛИПЕПТИД, СОДЕРЖАЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2017 |
|
RU2827545C2 |
| АНТИТЕЛО ПРОТИВ IL-17A И ЕГО ПРИМЕНЕНИЕ | 2019 |
|
RU2816204C2 |
| АНТИГЕНСВЯЗЫВАЮЩАЯ МОЛЕКУЛА, СОДЕРЖАЩАЯ ДВА АНТИГЕНСВЯЗЫВАЮЩИХ ДОМЕНА, КОТОРЫЕ СВЯЗАНЫ ДРУГ С ДРУГОМ | 2019 |
|
RU2838581C2 |
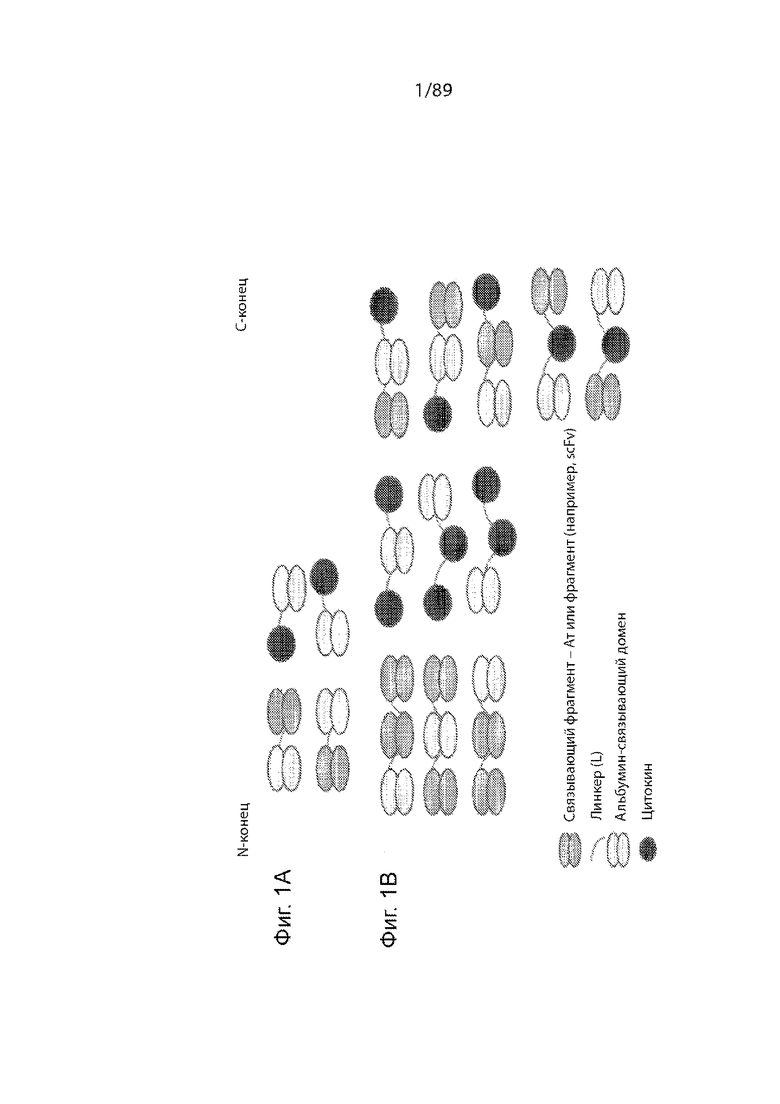





























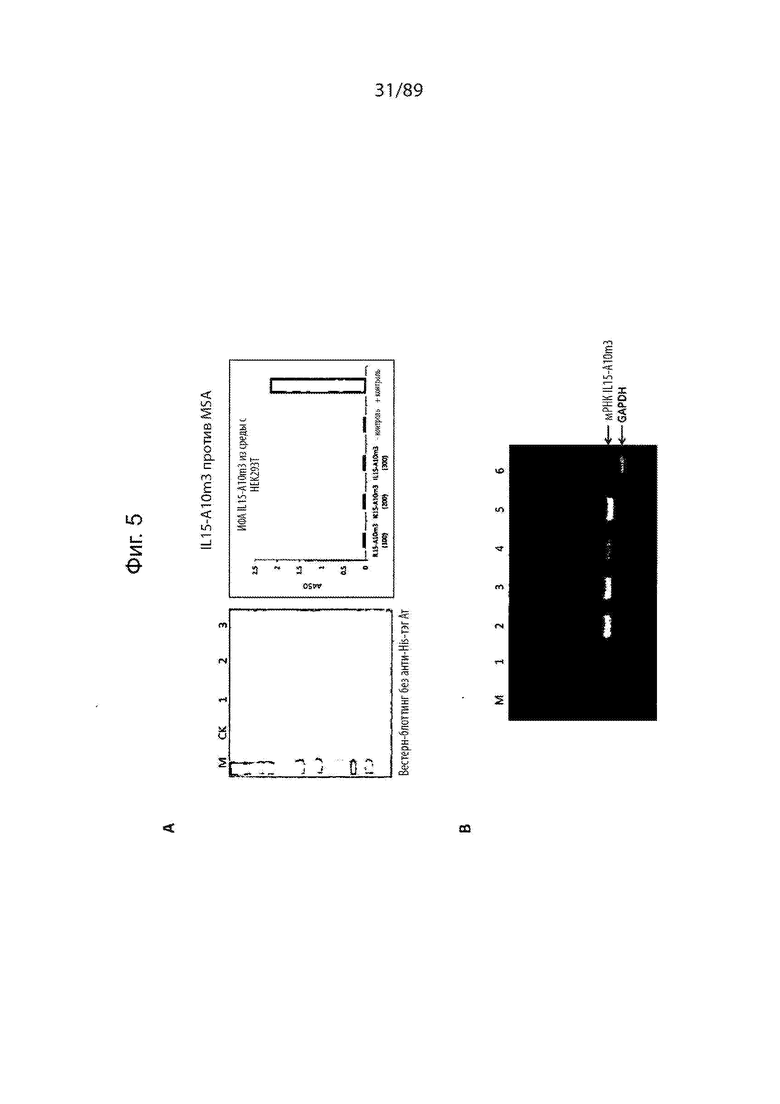

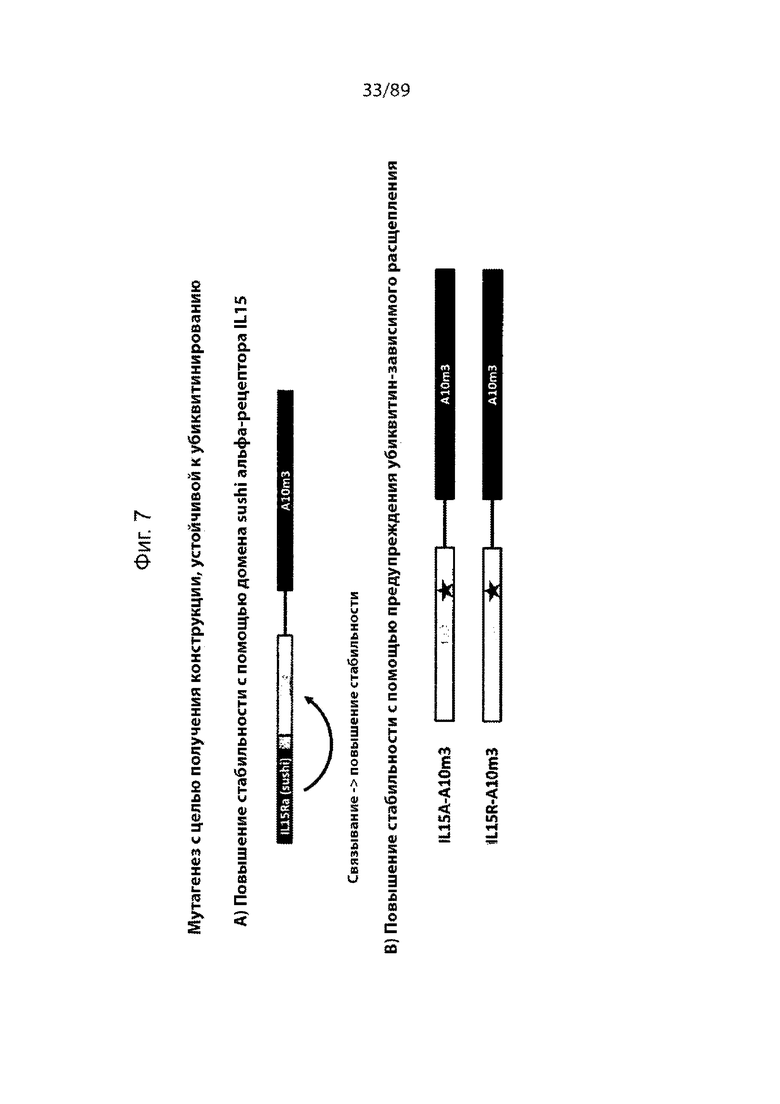
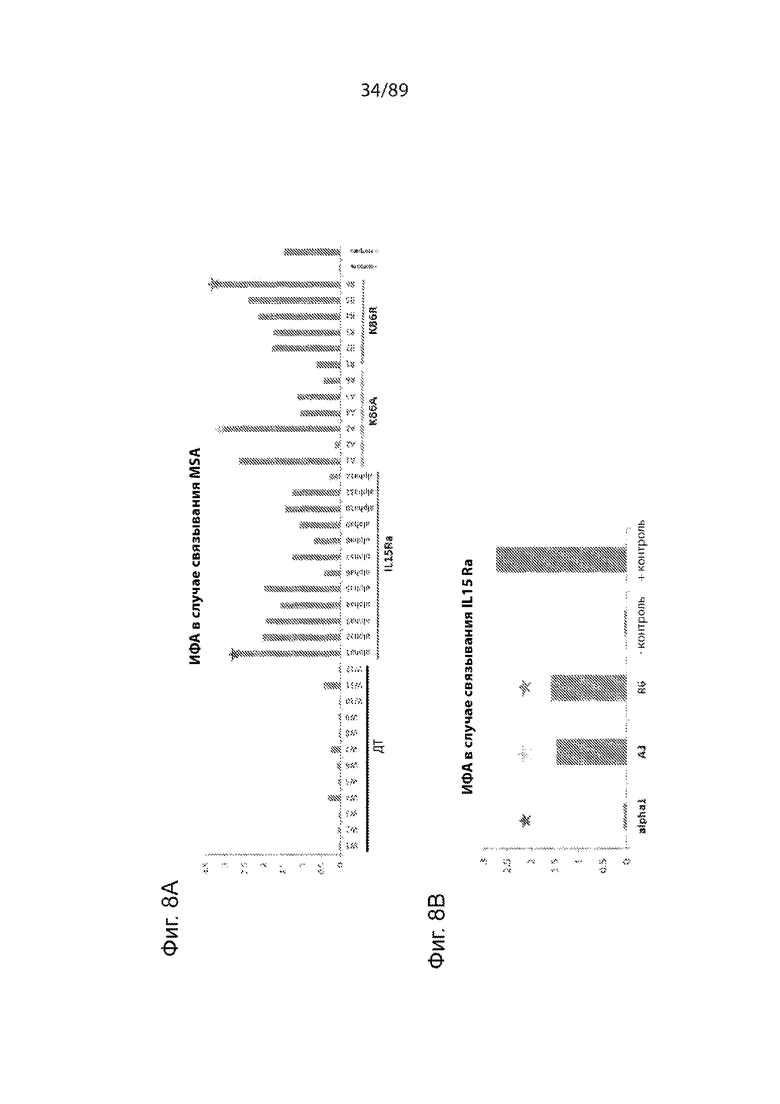

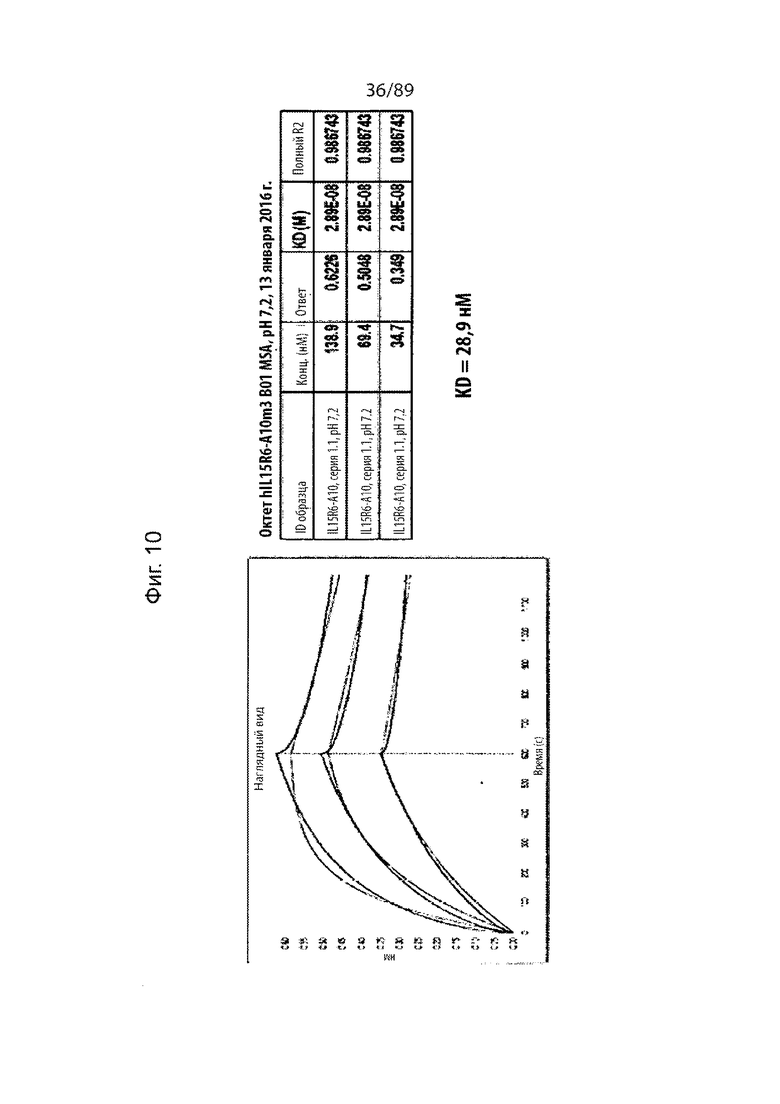
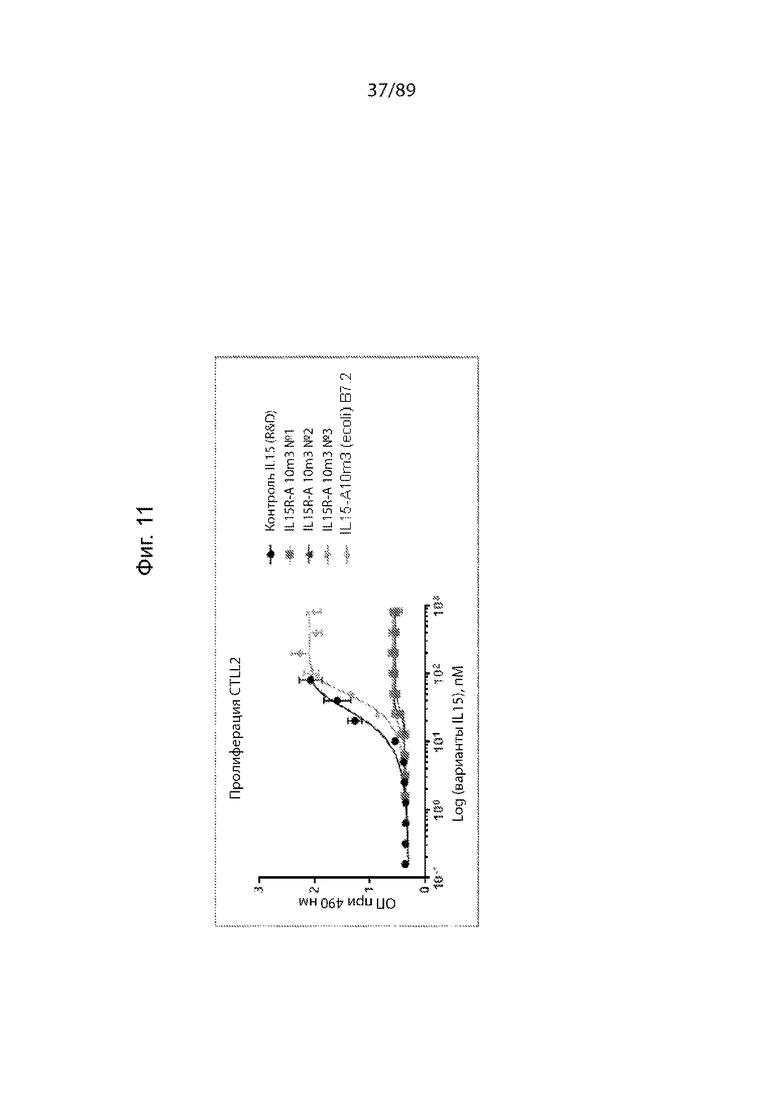
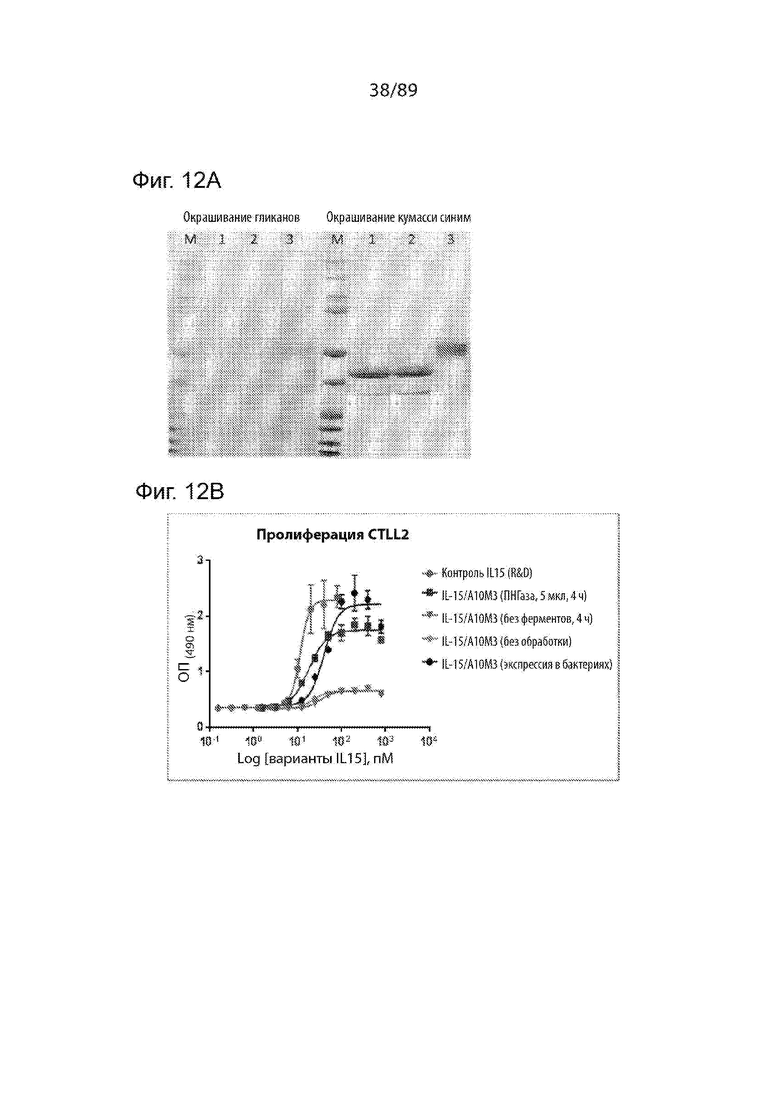
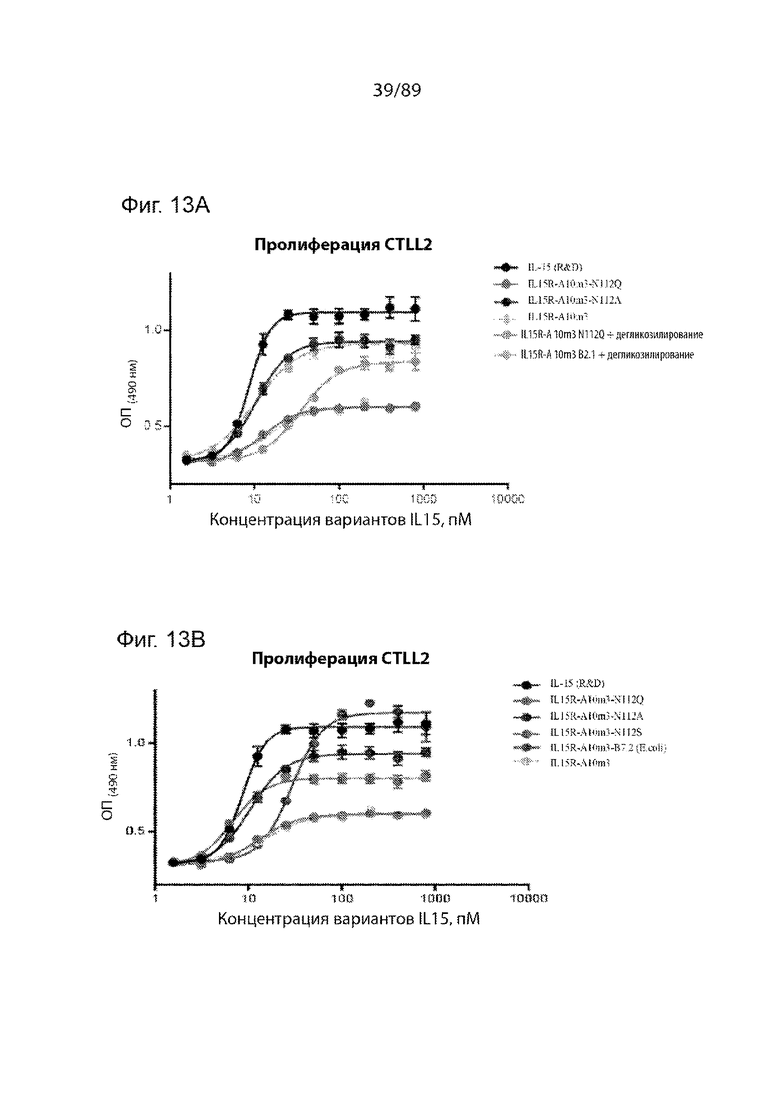
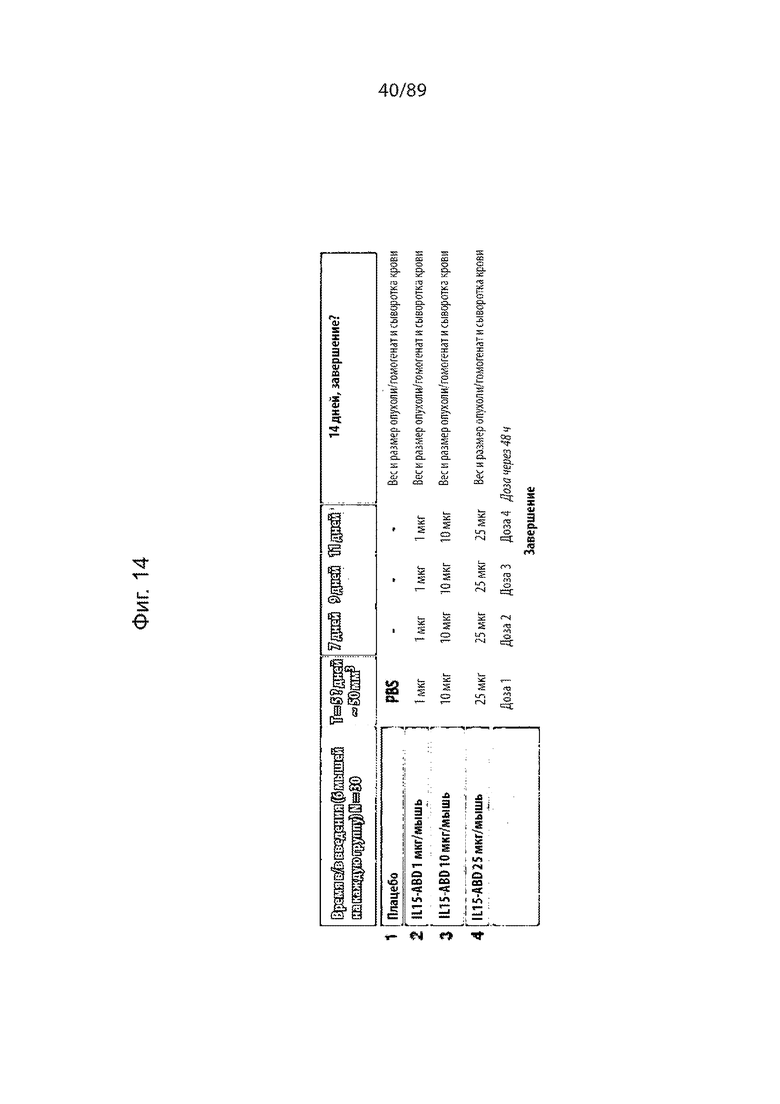
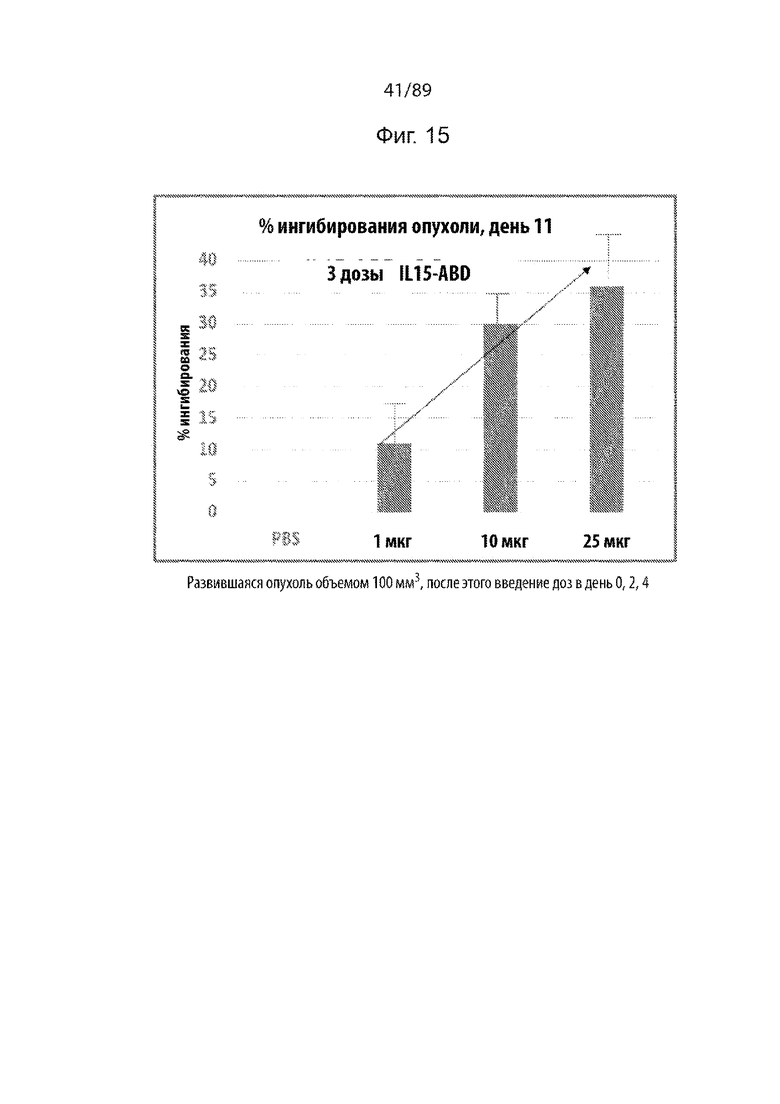
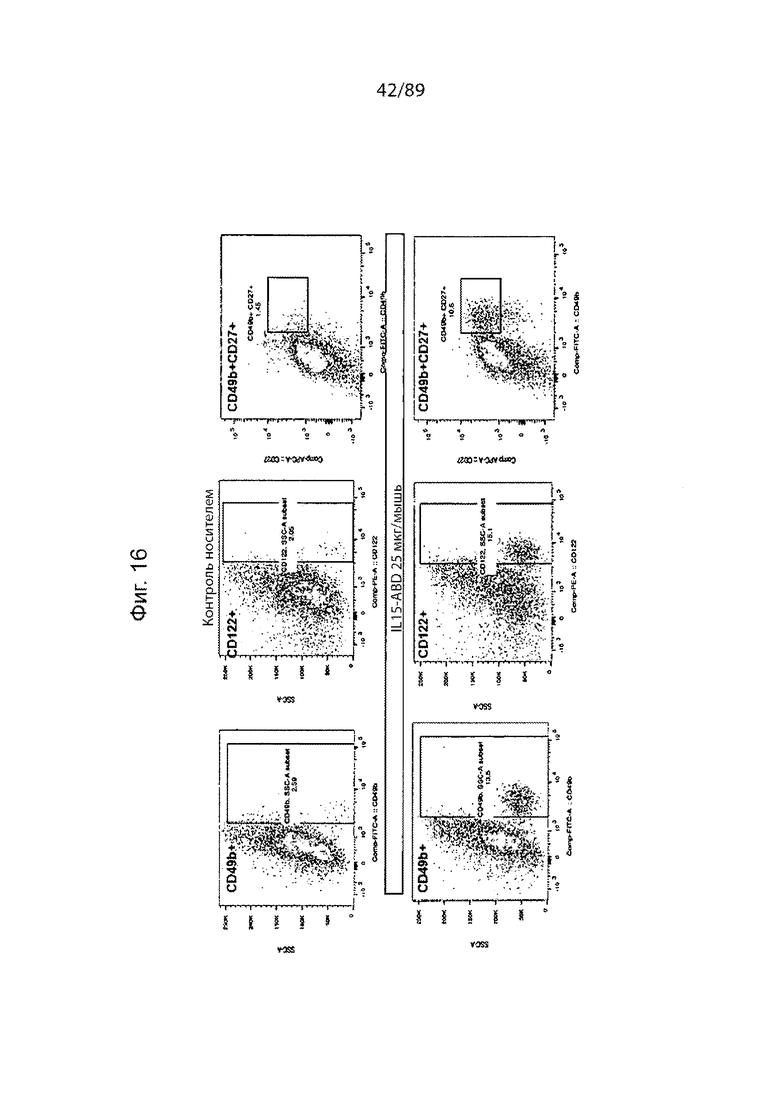
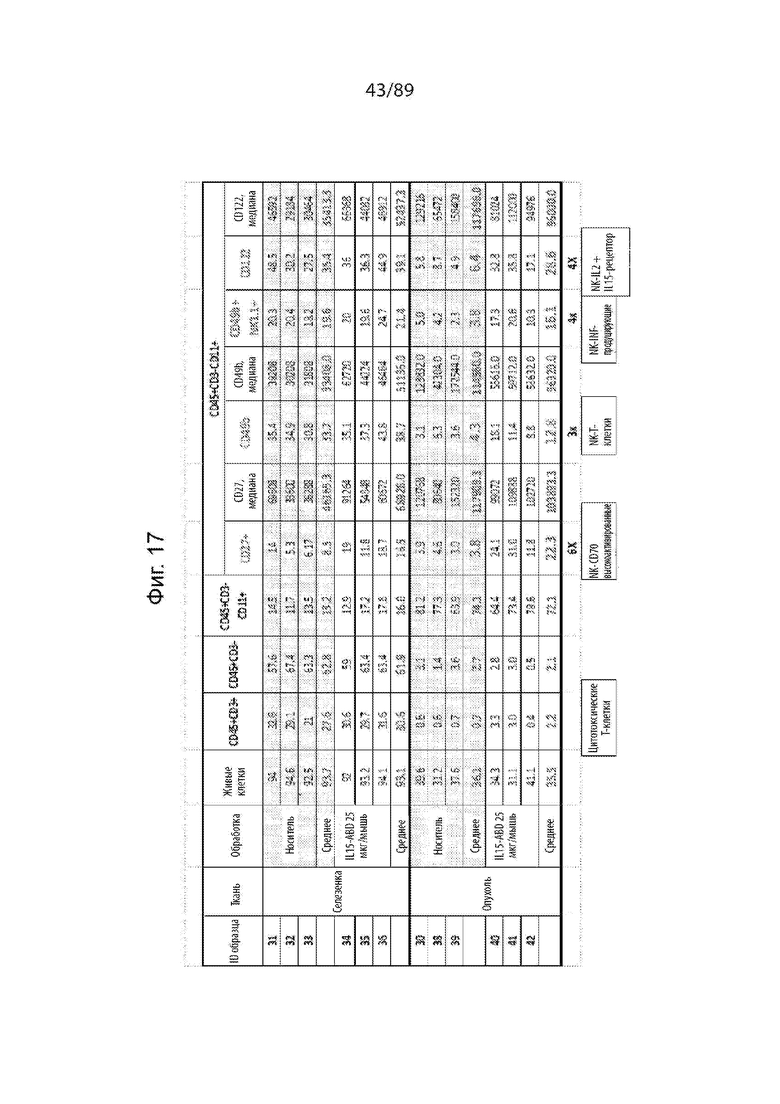
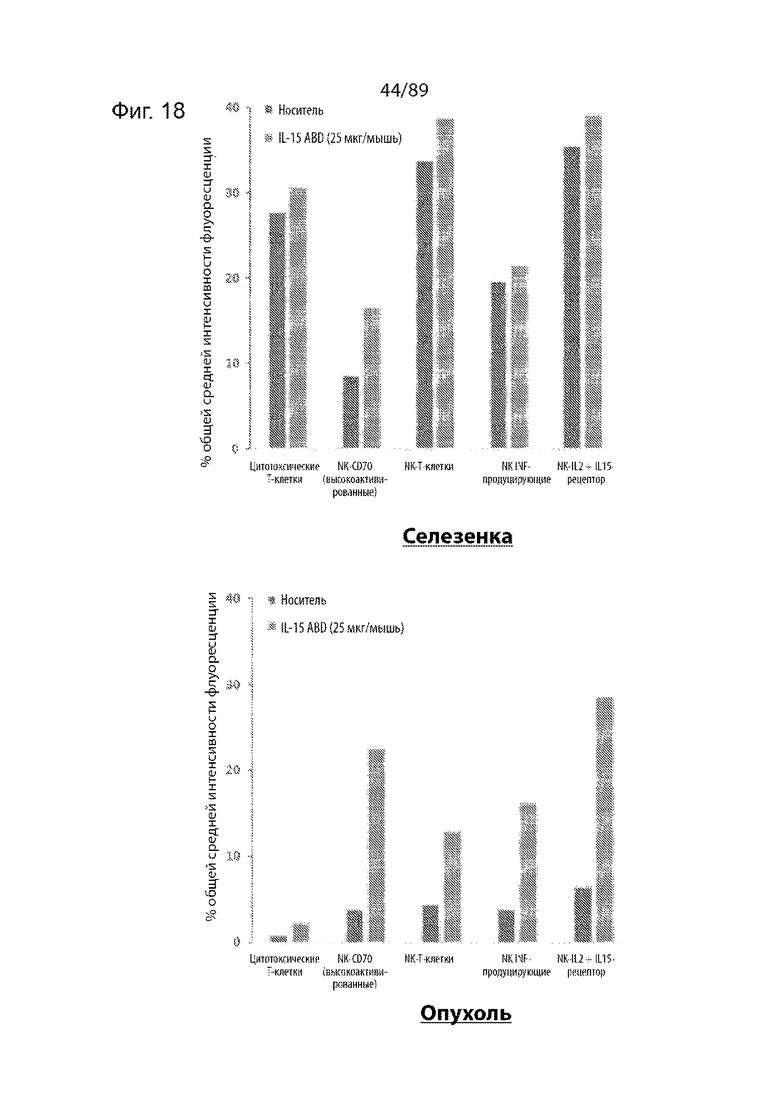
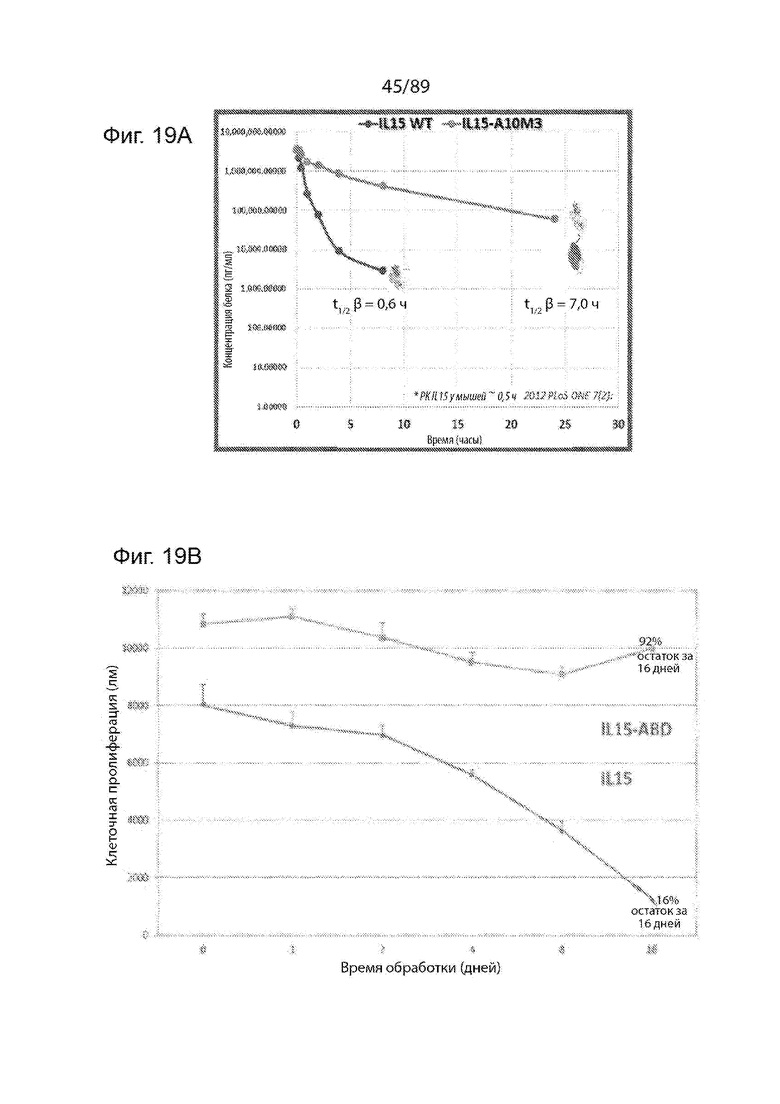

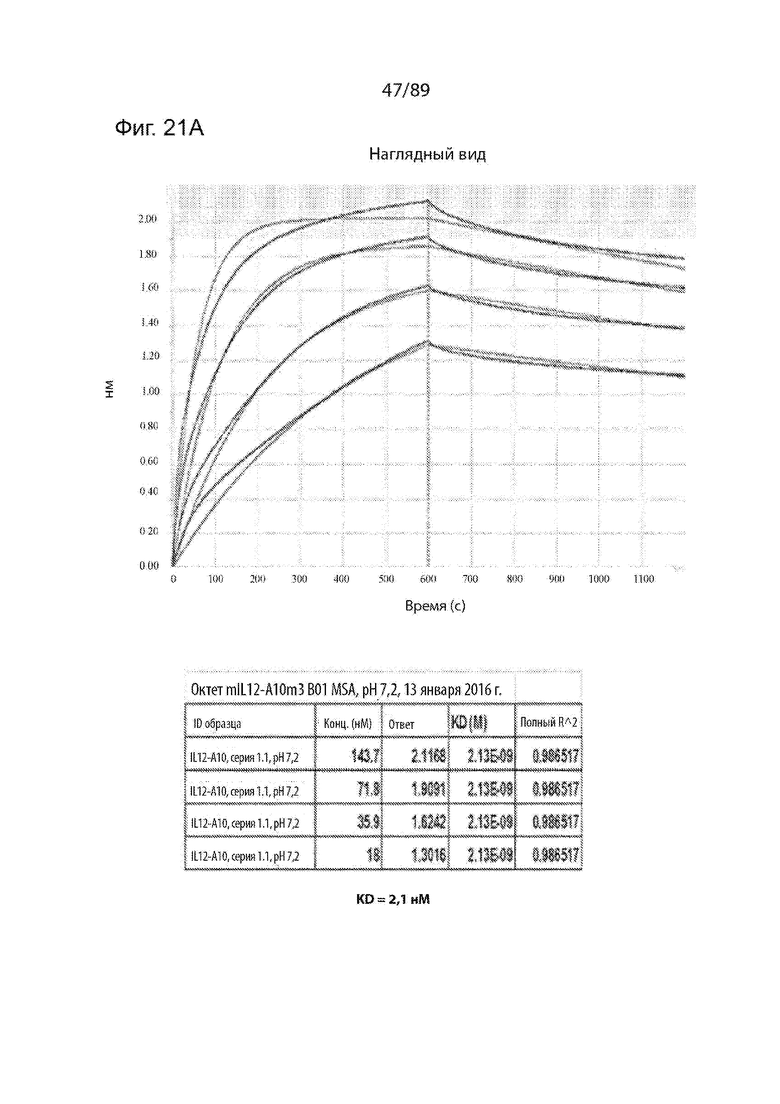
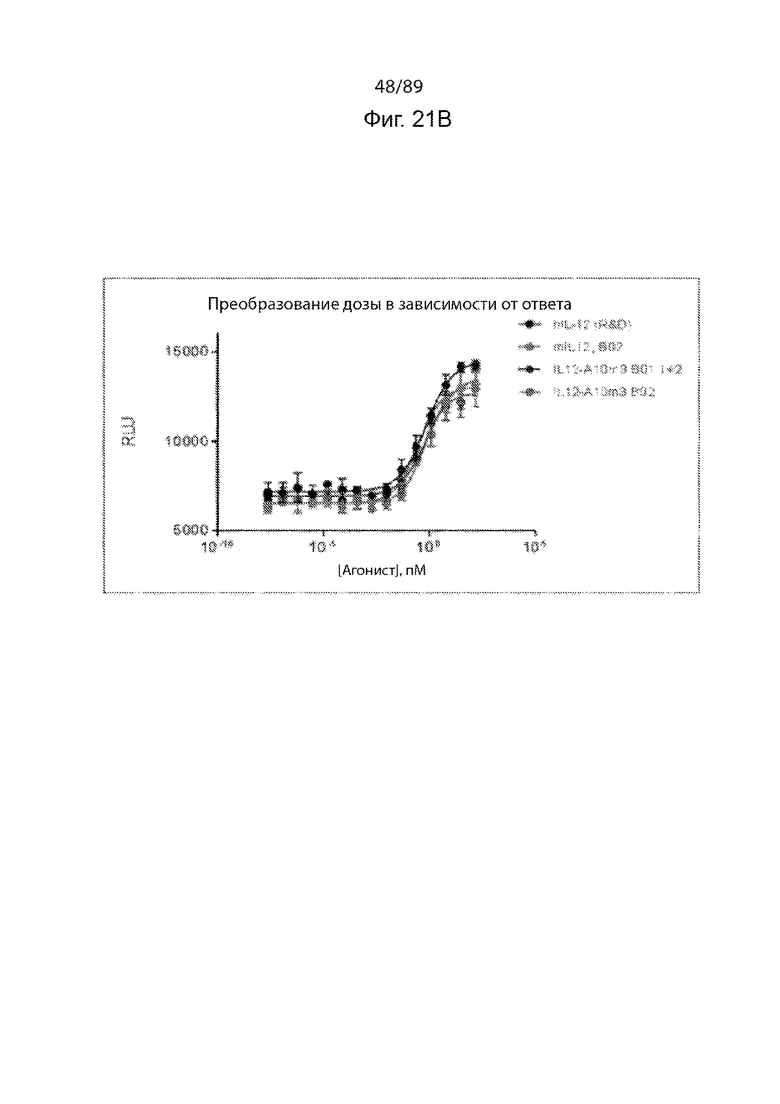
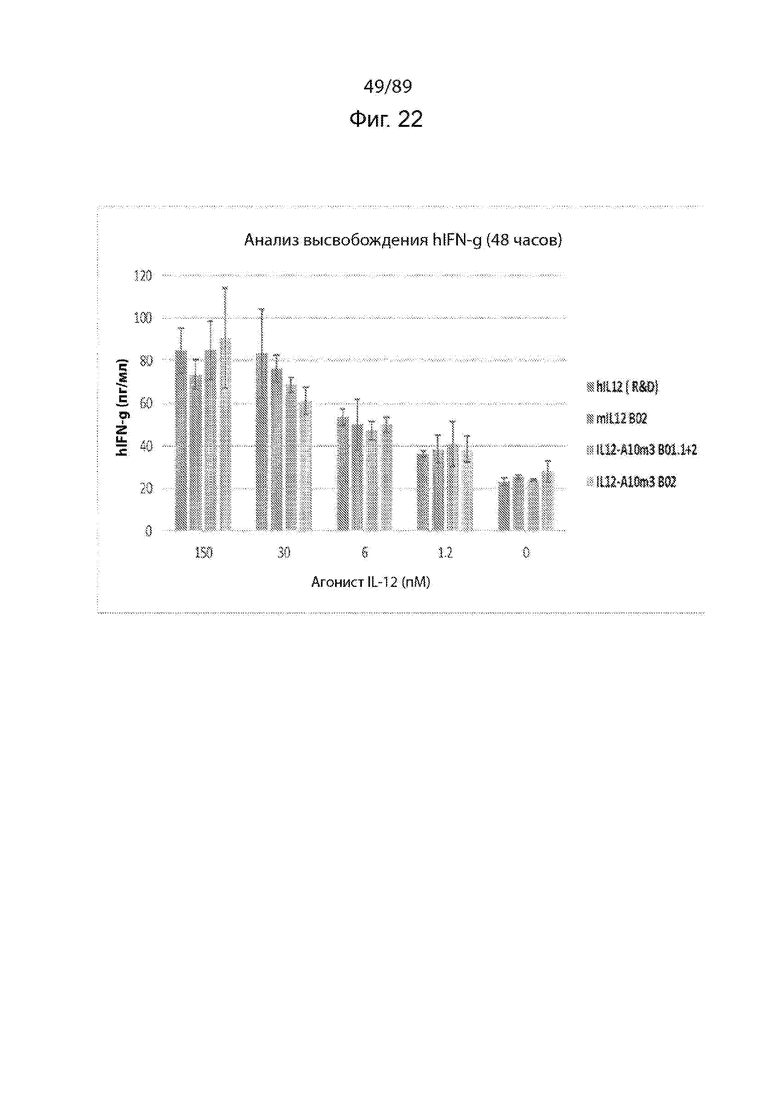
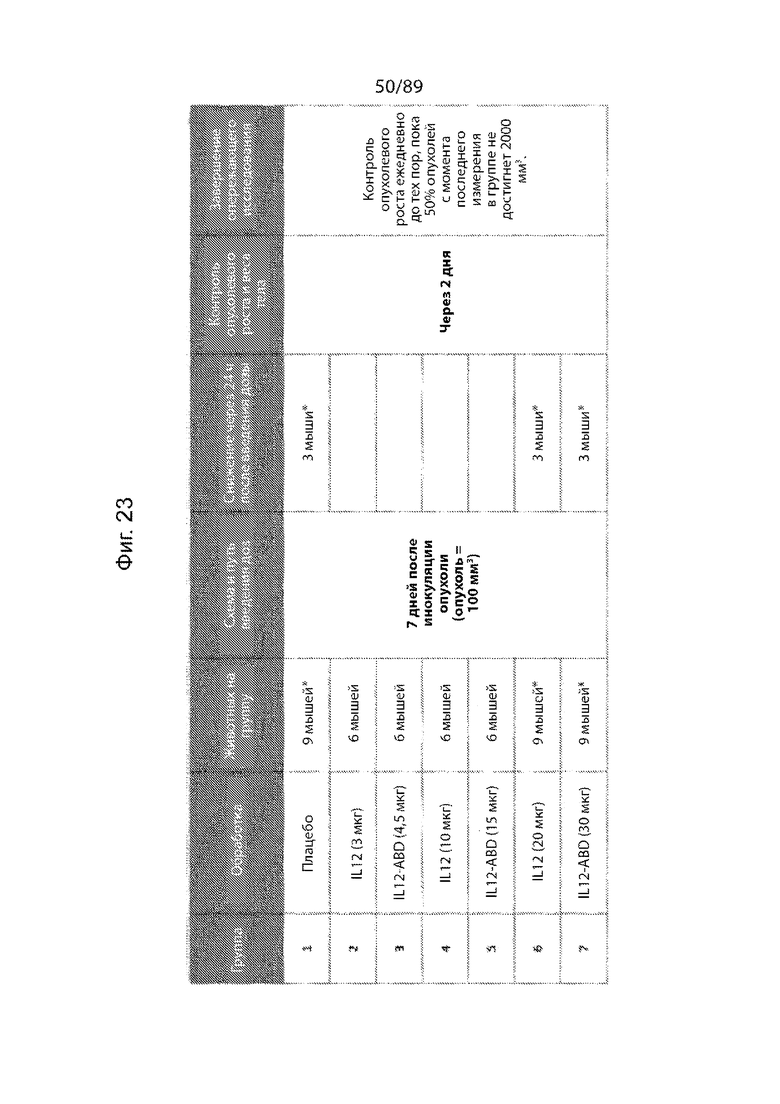
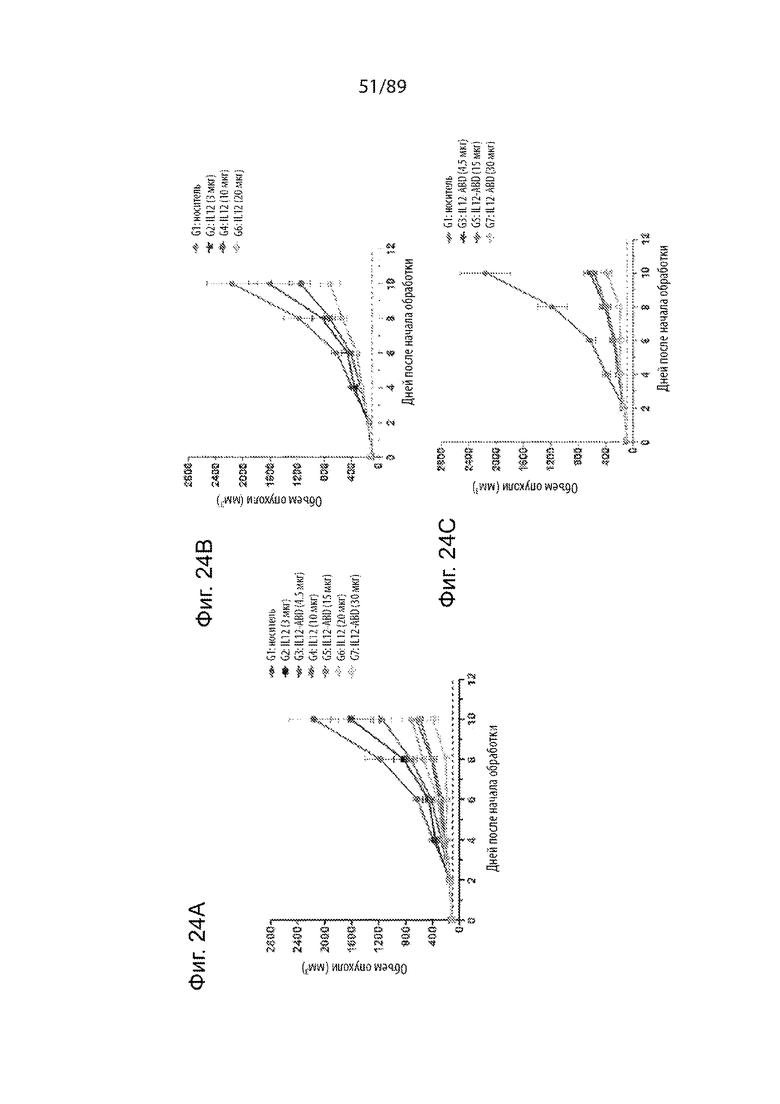
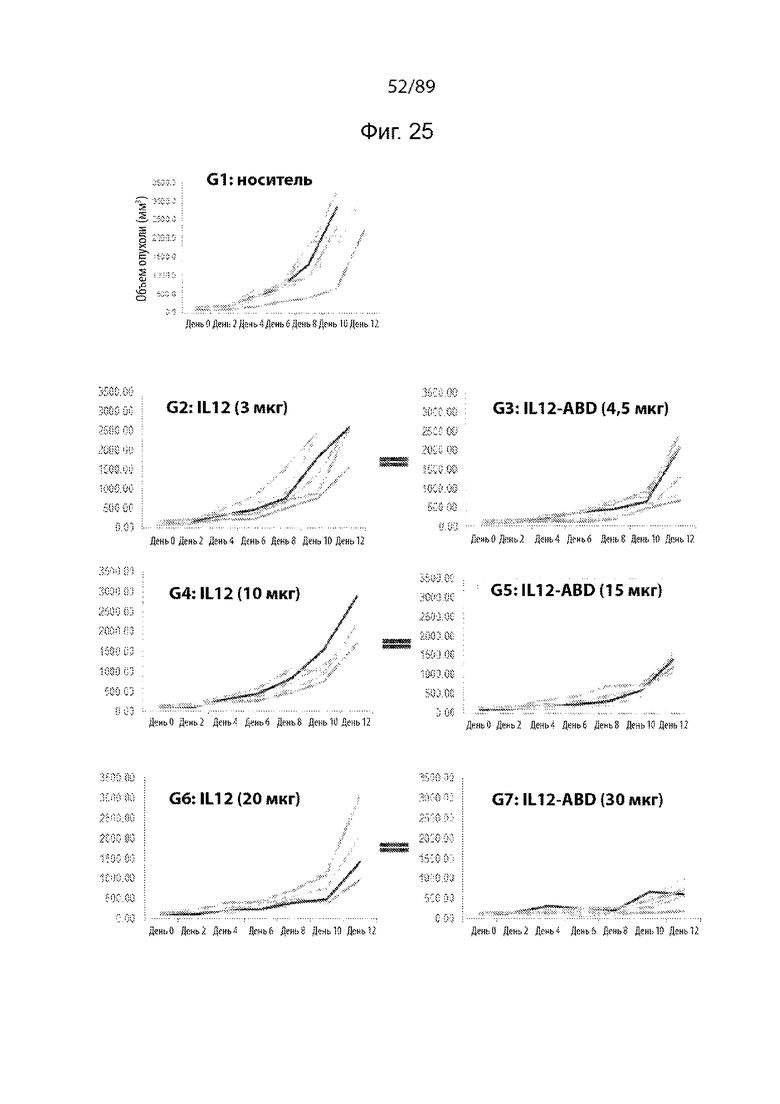
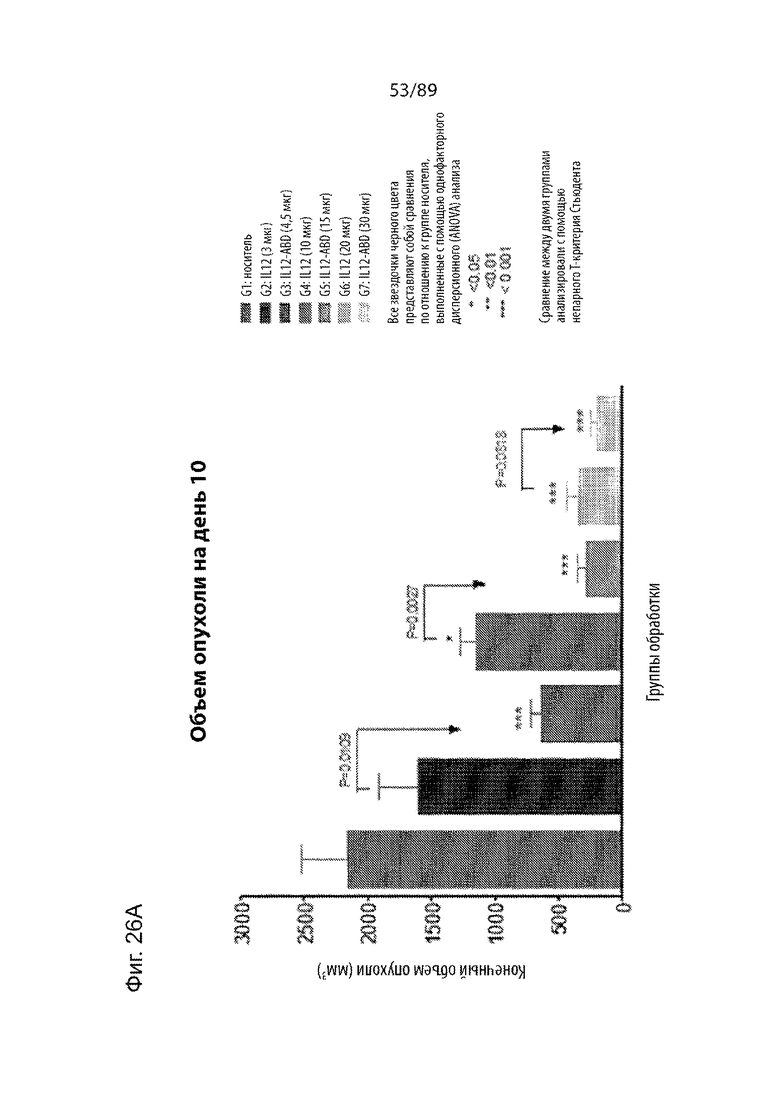
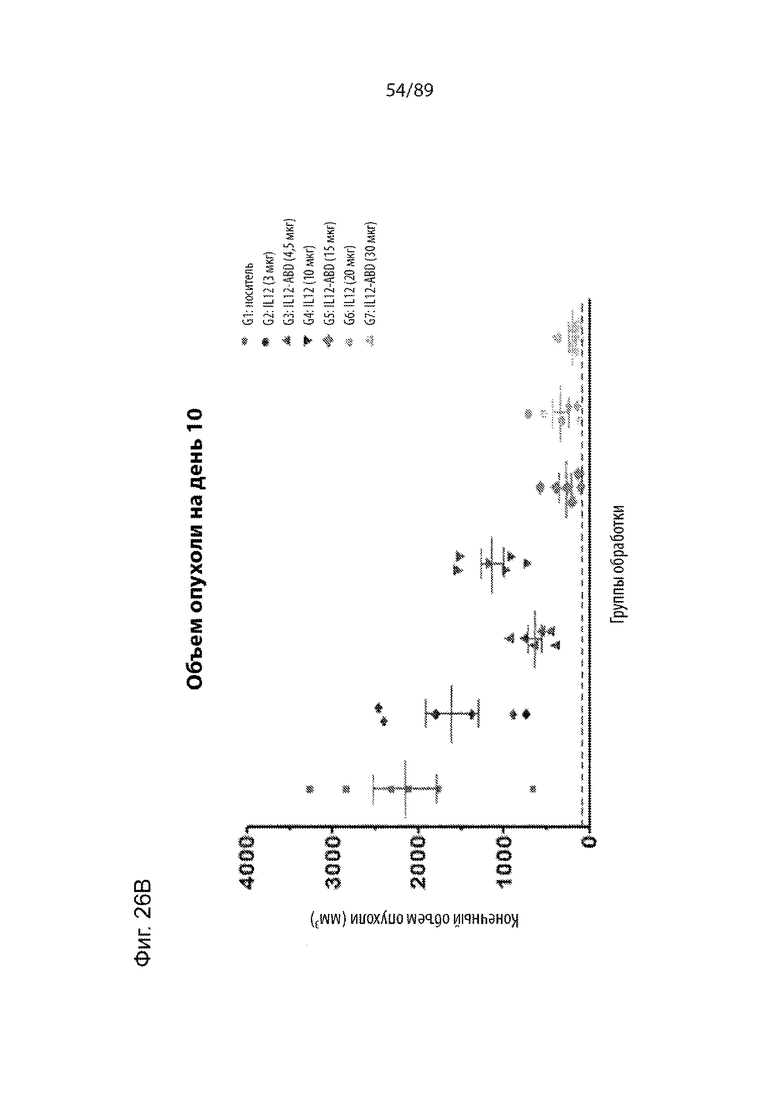
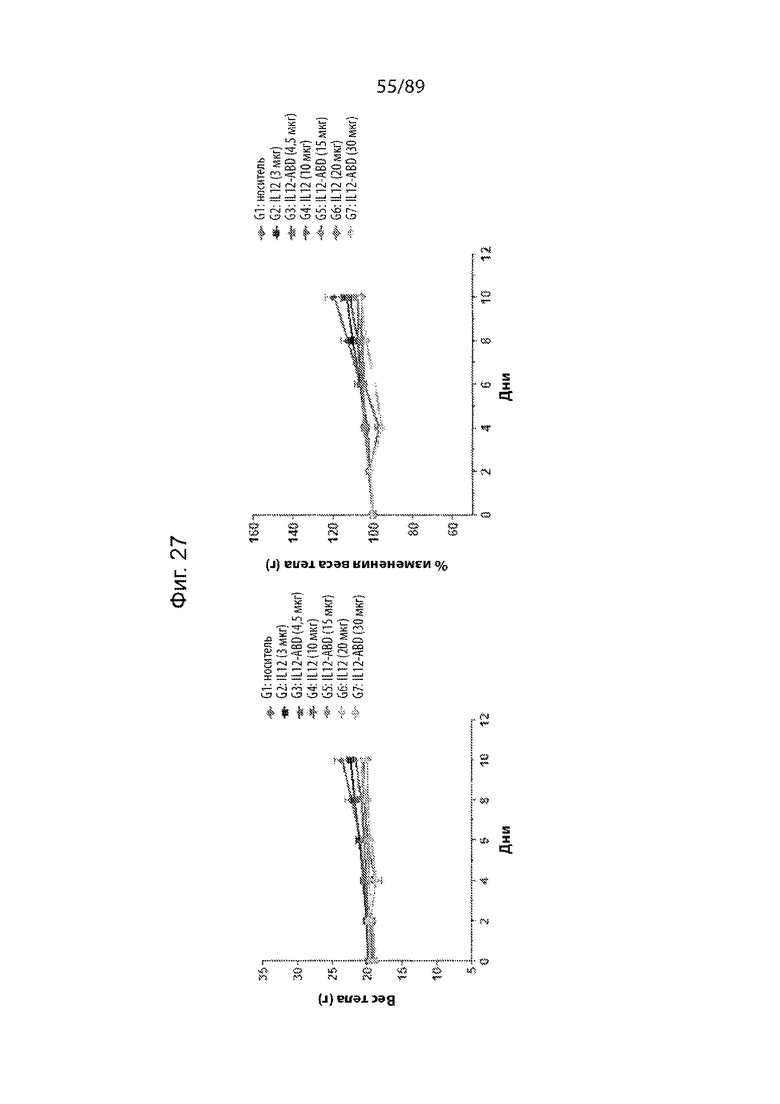



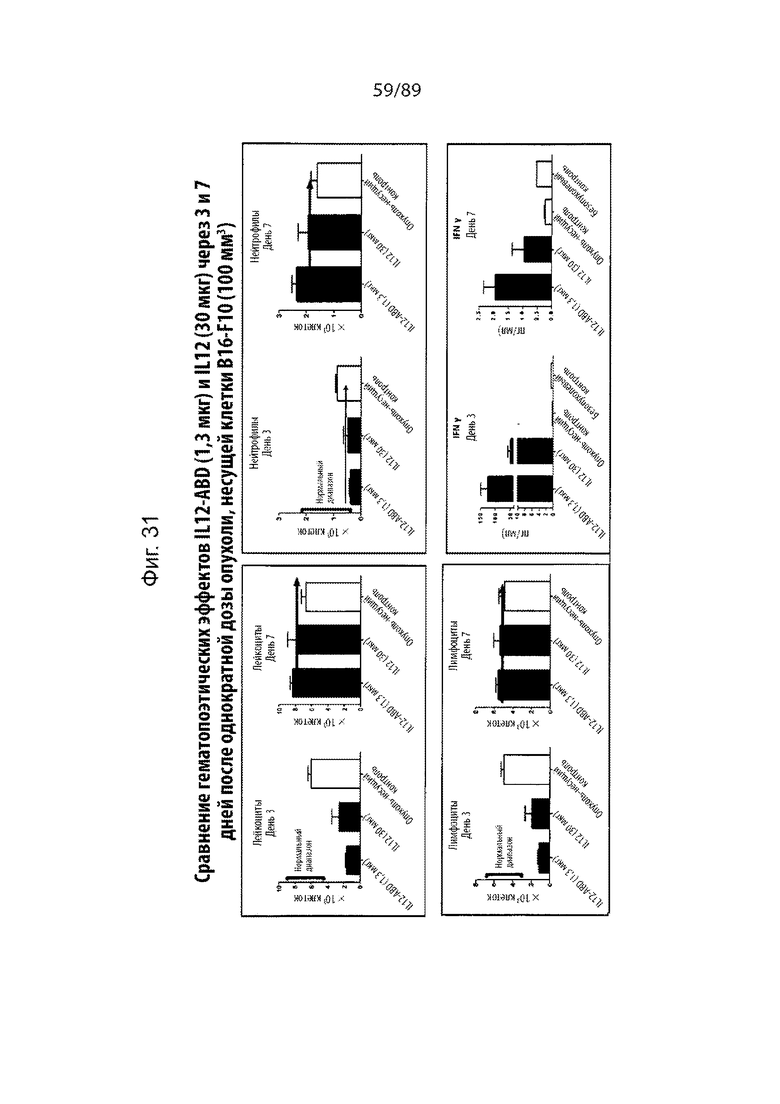

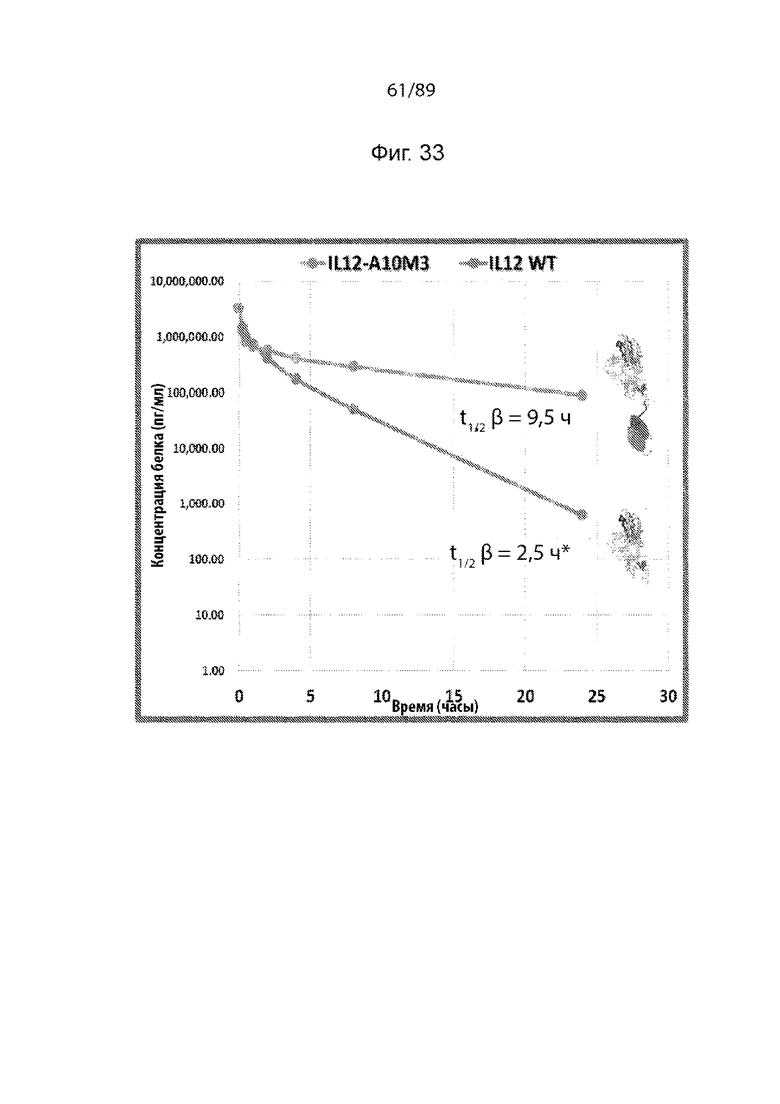

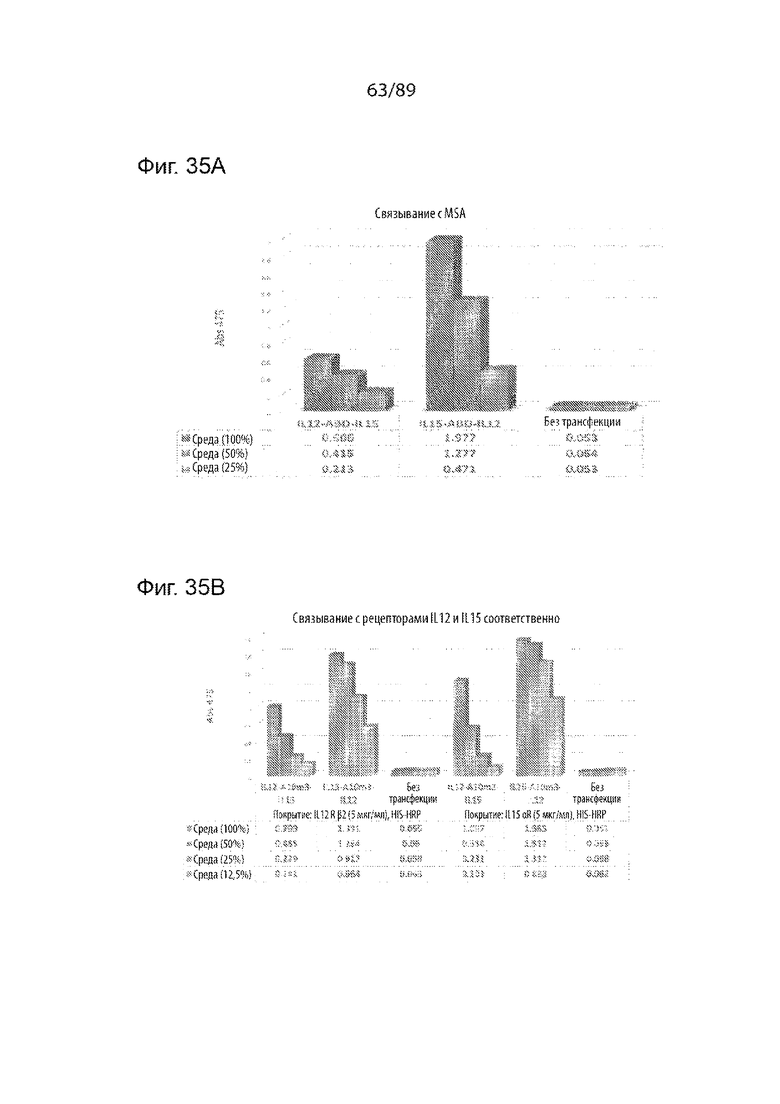

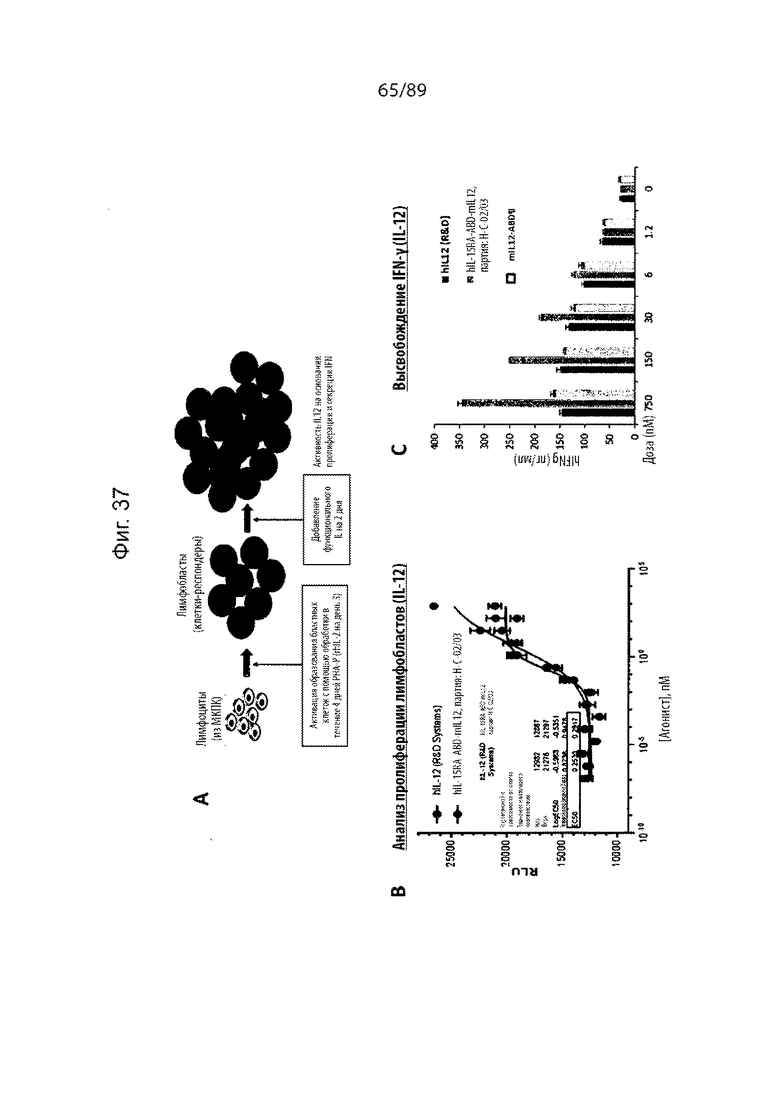
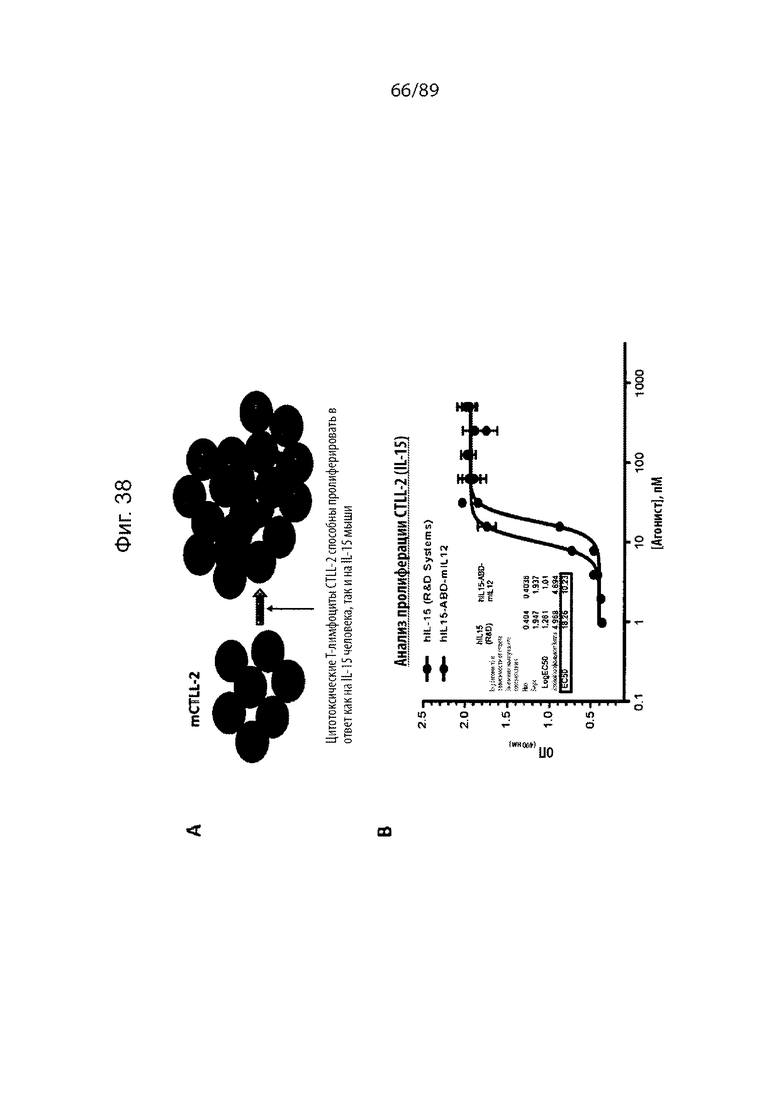
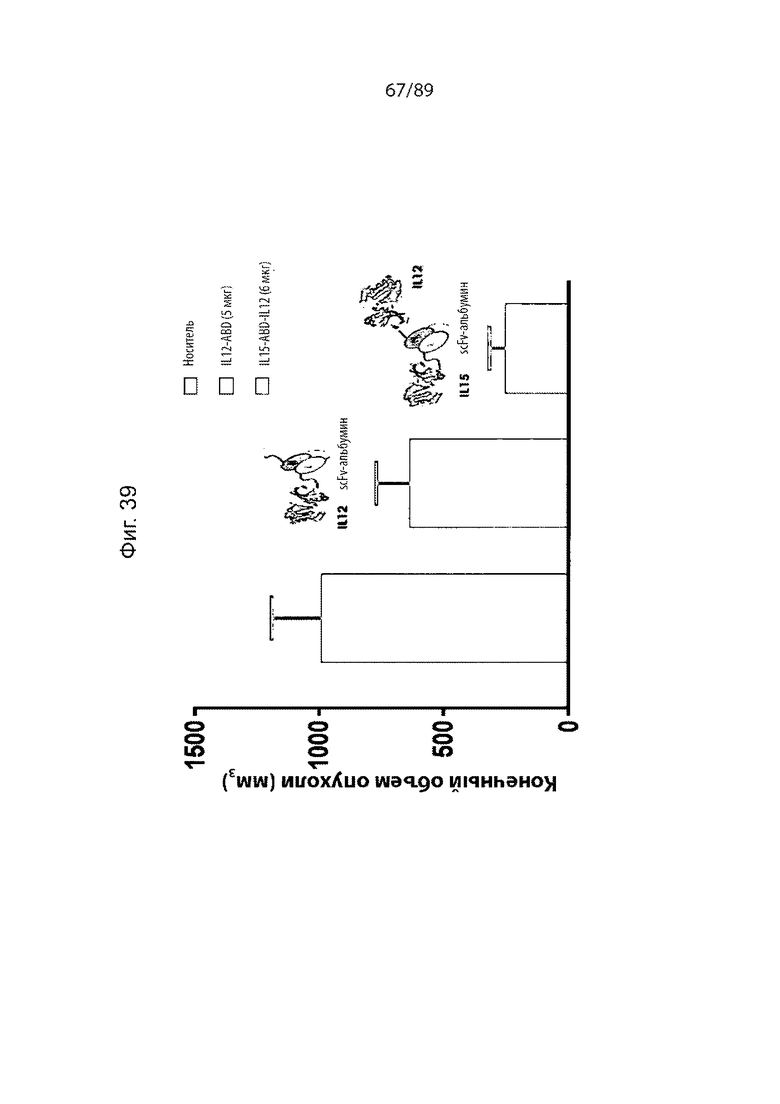


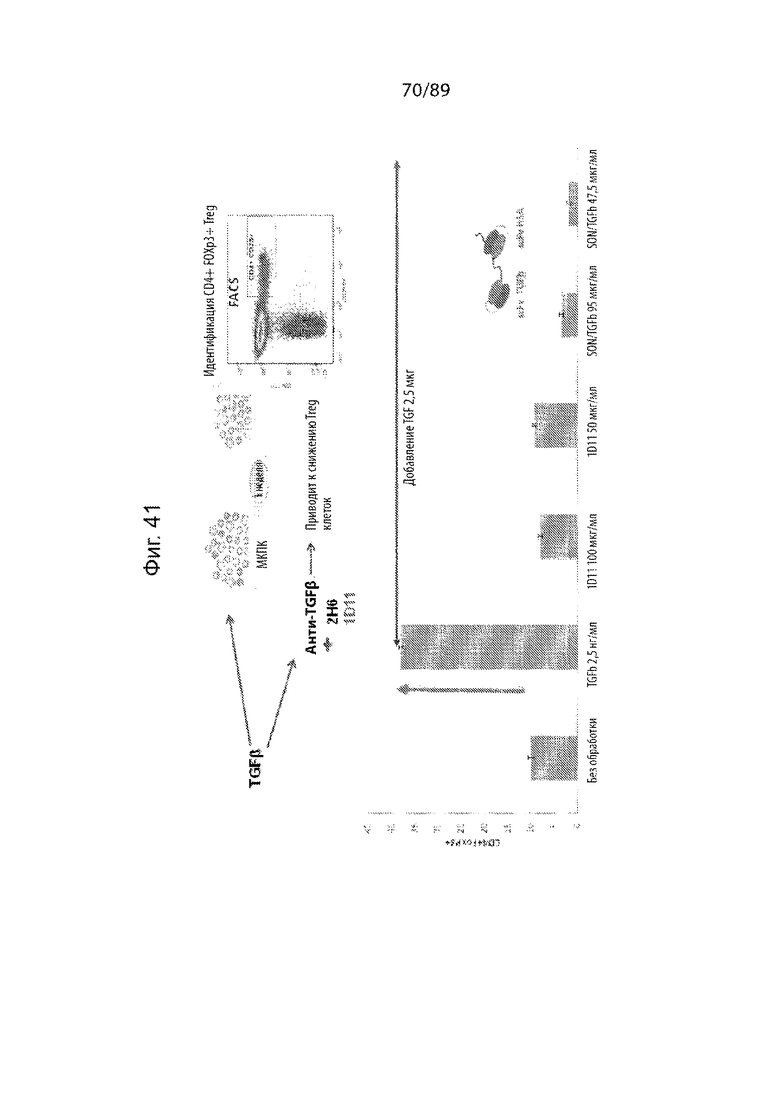
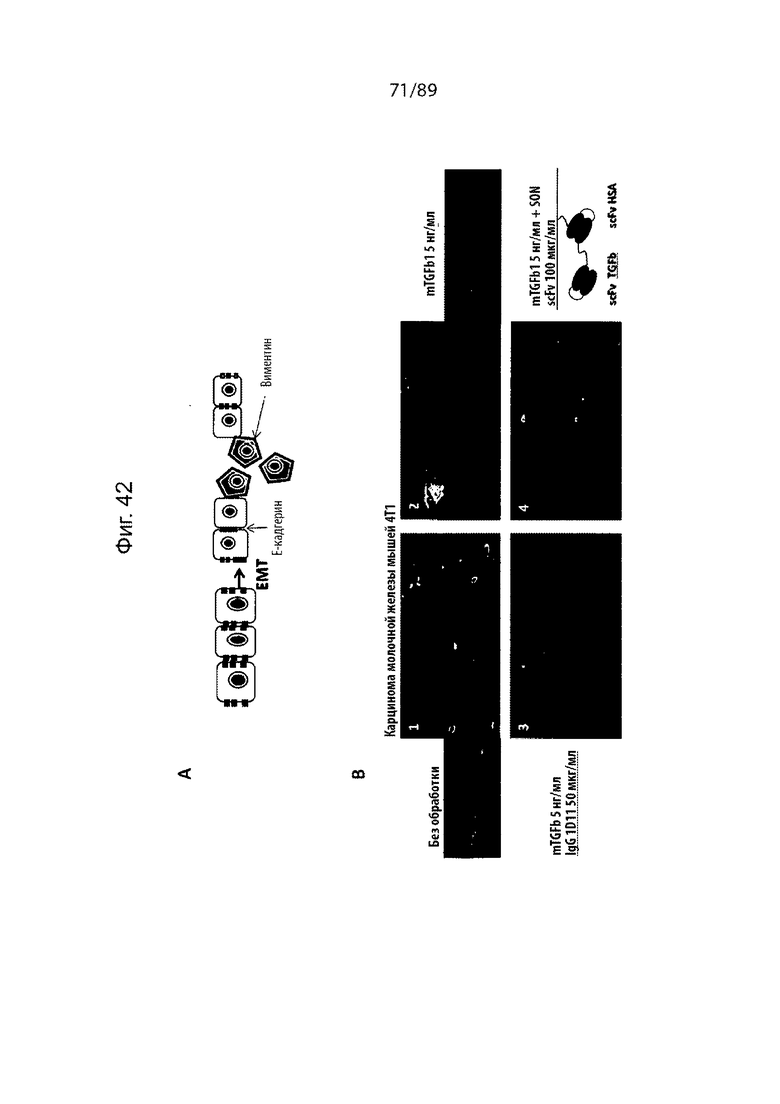
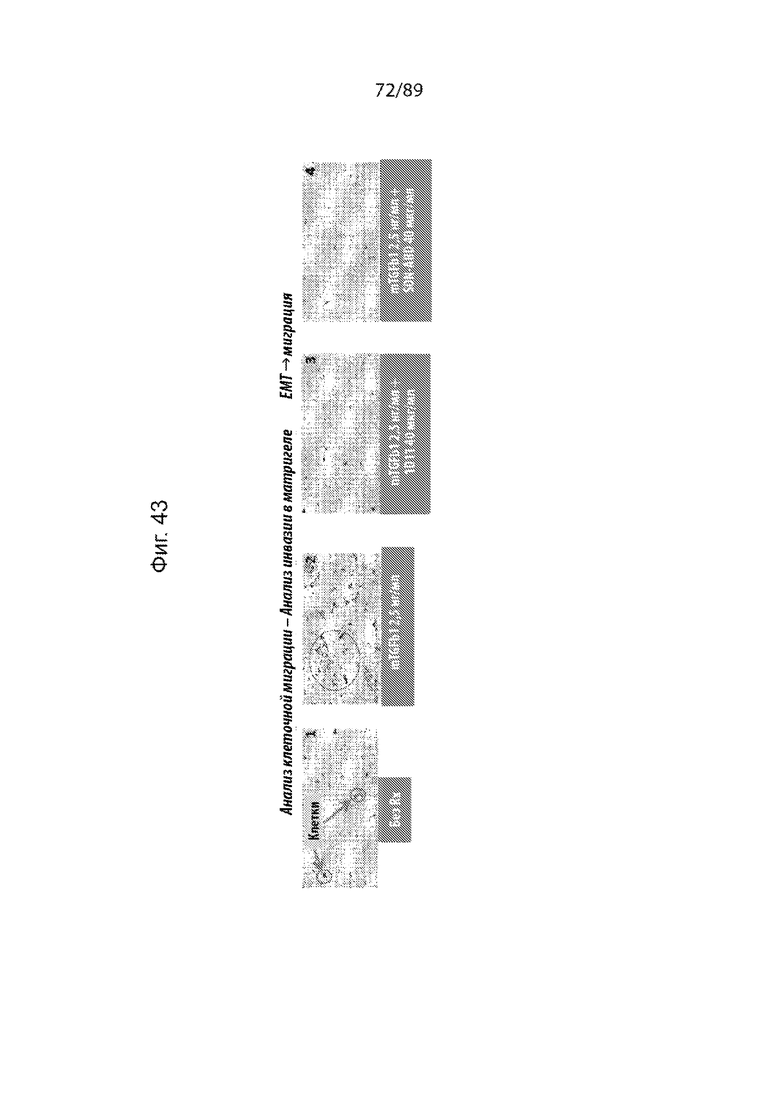
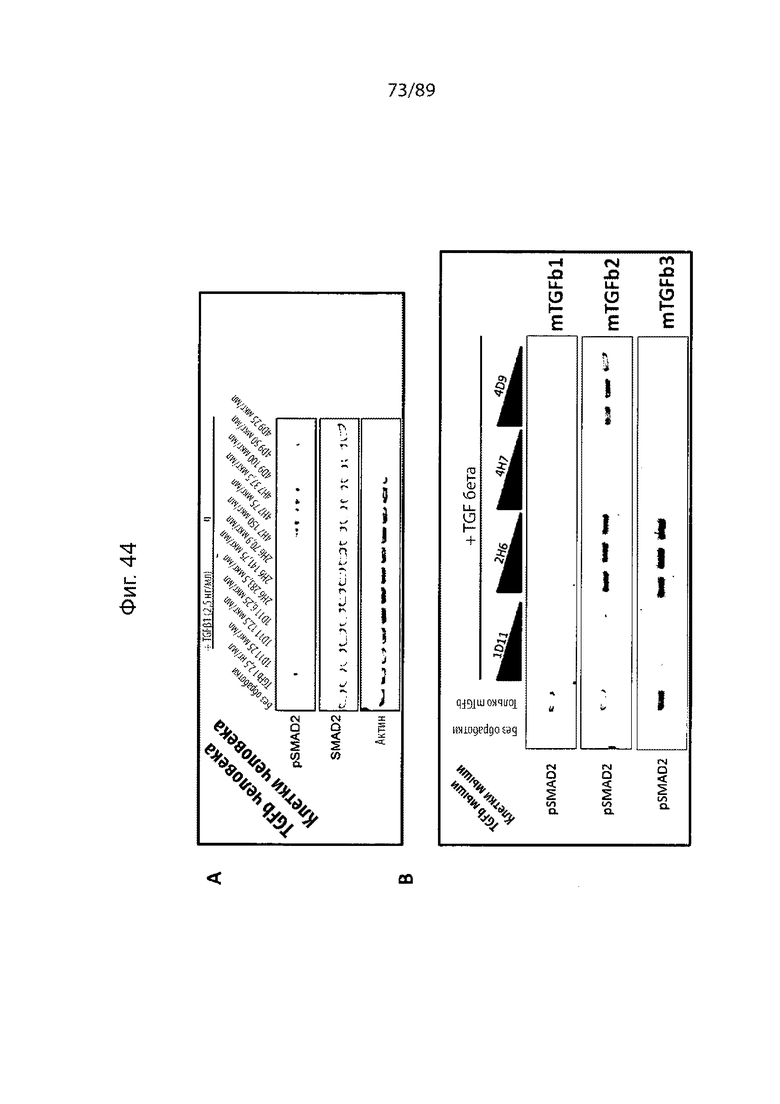



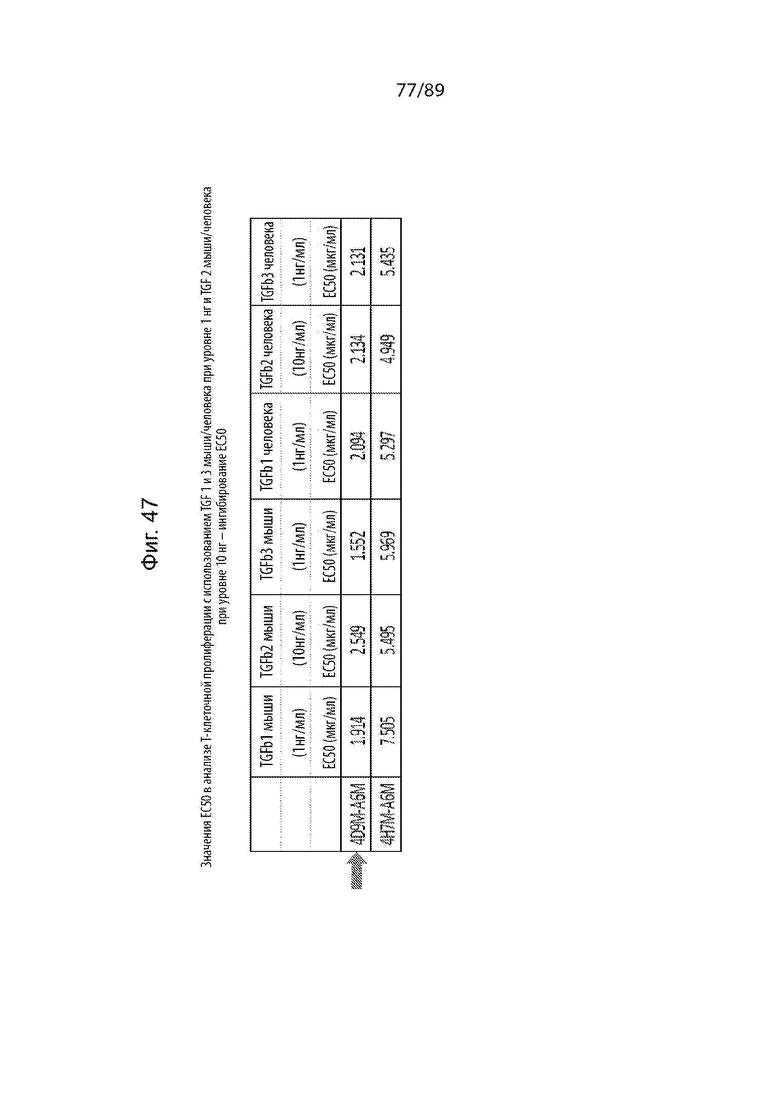












Изобретение относится к области биотехнологии, конкретно к слитым белкам с альбумин-связывающим доменом (ABD), и может быть использовано для рекомбинантного получения слитого белка с АВD. Рекомбинантным путем получают слитый белок с ABD, в котором ABD присоединен к одному или нескольким партнерам по слиянию и содержит vhCDR1, vhCDR2, vhCDR3 тяжелой цепи из SEQ ID NO: 25 и vlCDR1, vlCDR2, vlCDR3 легкой цепи из SEQ ID NO: 29. Изобретение обеспечивает получение слитого белка, который демонстрирует увеличенный период полужизни, улучшенную фармакокинетику in vivo по сравнению с биологическими препаратами без ABD и увеличенную локализацию в опухоли. 4 н. и 18 з. п. ф-лы, 51 ил., 6 пр.
1. Слитый белок с альбумин-связывающим доменом (ABD) с улучшенной фармакокинетикой in vivo, содержащий ABD, присоединенный к одному или нескольким партнерам по слиянию, при этом указанный ABD содержит вариабельную область тяжелой цепи, содержащую vhCDR1, vhCDR2 и vhCDR3 из вариабельной области тяжелой цепи, имеющей аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи, содержащую vlCDR1, vlCDR2 и vlCDR3 из вариабельной области легкой цепи, имеющей аминокислотную последовательность SEQ ID NO: 29.
2. Слитый белок с ABD по п. 1, отличающийся тем, что указанная вариабельная область тяжелой цепи имеет аминокислотную последовательность SEQ ID NO: 25 и указанная вариабельная область легкой цепи имеет аминокислотную последовательность SEQ ID NO: 29.
3. Слитый белок с ABD по п. 1 или 2, отличающийся тем, что указанный один или несколько партнеров по слиянию выбирают из группы, состоящей из: IL-2, IL-7, IL-12, IL-15, IL-18, IL-21, GM-CSF, IFN-α, анти-TGFβ scFv, анти-PD-L1 scFv, анти-TNF scFv, анти-IL-1 scFv, анти-IL-6 scFv, анти-IL-8 scFv, анти-IL-17 (AF) scFv и анти-IL-23 scFv и их комбинаций.
4. Слитый белок с ABD по любому из пп. 1-3, отличающийся тем, что указанный один или несколько партнеров по слиянию выбирают из группы, состоящей из: IL-12 и IL-15, IL-18 и IL-12, IL-18 и GM-CSF, GM-CSF и IL-12, IL12-ABD-IL12 и IL7-ABD-IL15.
5. Слитый белок с ABD по любому из пп. 1-4, отличающийся тем, что указанные один или несколько партнеров по слиянию ковалентно присоединены к указанному ABD с помощью линкера.
6. Слитый белок с ABD по п. 4, отличающийся тем, что указанный один или несколько партнеров по слиянию включают IL-18 и IL-12.
7. Слитый белок с ABD по п. 6, отличающийся тем, что указанный слитый белок имеет формулу с N-конца к C-концу, представленную (IL-18)-L1-(ABD)-L2-(IL-12), где L1 и L2 являются первым и вторым линкером соответственно.
8. Слитый белок с ABD по п. 4, отличающийся тем, что указанный один или несколько партнеров по слиянию включают IL-18 и GM-CSF.
9. Слитый белок с ABD по п. 8, отличающийся тем, что указанный слитый белок имеет формулу с N-конца к C-концу, представленную (IL-18)-L1-(ABD)-L2-(GM-CSF), где L1 и L2 являются первым и вторым линкером соответственно.
10. Слитый белок с ABD по п. 4, отличающийся тем, что указанный один или несколько партнеров по слиянию включают GM-CSF и IL-12.
11. Слитый белок с ABD по п. 10, отличающийся тем, что указанный слитый белок имеет формулу с N-конца к C-концу, представленную (GM-CSF)-L1-(ABD)-L2-(IL-12), где L1 и L2 являются первым и вторым линкером соответственно.
12. Слитый белок с ABD по п. 4, отличающийся тем, что указанный один или несколько партнеров по слиянию включают IL-12 и IL-15.
13. Слитый белок с ABD по п. 12, отличающийся тем, что указанный слитый белок имеет формулу с N-конца к C-концу, представленную (IL-12)-L1-(ABD)-L2-(IL-15), где L1 и L2 являются первым и вторым линкером соответственно.
14. Слитый белок с ABD по п. 4, отличающийся тем, что указанный один или несколько партнеров по слиянию включают IL-12.
15. Слитый белок с ABD по п. 14, отличающийся тем, что указанный слитый белок имеет формулу с N-конца к C-концу, представленную (IL-12)-L1-(ABD)-L2-(IL-12), где L1 и L2 являются первым и вторым линкером соответственно.
16. Слитый белок с ABD по п. 4, отличающийся тем, что указанный один или несколько партнеров по слиянию включают IL-7 и IL-15.
17. Слитый белок с ABD по п. 16, отличающийся тем, что указанный слитый белок имеет формулу с N-конца к C-концу, представленную (IL-7)-L1-(ABD)-L2-(IL-15), где L1 и L2 являются первым и вторым линкером соответственно.
18. Слитый белок с ABD по п. 3 или 4, где IL-15 является вариантом IL-15, содержащим одну или несколько аминокислотных замен, выбранных из группы, состоящей из K86A, K86R, N112A, N112S, N112Q, K86A/N112A, K86R/N112A, K86A/N112S, K86R/N112S, K86A/N112Q, K86R/N112Q, K86A/N112A/N79A, K86R/N112A/N79A, K86A/N112A/N79D, K86R/N112A/N79D, K86A/N112A/N79Q, K86R/N112A/N79Q, K86A/N112A/N71D, K86R/N112A/N71D, K86A/N112A/N71Q, K86R/N112A/N71Q, K86A/N112A/N71D/N79A, K86A/N112A/N71D/N79D, K86A/N112A/N71Q/N79A, K86A/N112A/N71Q/N79D, K86R/N112A/N71D/N79A, K86R/N112A/N71D/N79D, K86R/N112A/N71D/N79Q, K86R/N112A/N71Q/N79A, K86R/N112A/N71Q/N79D и K86R/N112A/N71Q/N79Q, по сравнению с родительским IL-15.
19. Слитый белок с ABD по п. 3 или 4, отличающийся тем, что IL-15 представляет собой IL-15 дикого типа, присоединенный к альфа-рецептору IL-15 (IL-15Rα).
20. Нуклеиновая кислота, кодирующая слитый белок с ABD по любому из пп. 1-19, содержащий ABD, присоединенный одному или нескольким партнерам по слиянию, при этом указанный ABD содержит вариабельную область тяжелой цепи, содержащую vhCDR1, vhCDR2 и vhCDR3 из вариабельной области тяжелой цепи, имеющей аминокислотную последовательность SEQ ID NO: 25, и вариабельную область легкой цепи, содержащую vlCDR1, vlCDR2 и vlCDR3 из вариабельной области легкой цепи, имеющей аминокислотную последовательность SEQ ID NO: 29.
21. Клетка-хозяин для получения слитого белка с ABD по любому из пп. 1-19, содержащая указанную нуклеиновую кислоту по п. 20.
22. Способ получения слитого белка с ABD по любому из пп. 1-19, включающий культивирование указанной клетки-хозяина по п. 21 в условиях, в которых слитый белок с ABD продуцируется, и извлечение указанного слитого белка на основе ABD.
| WO 2013068571 A1, 16.05.2013 | |||
| COLMAN P | |||
| M., Effects of amino acid sequence changes on antibody-antigen interactions, Research in Immunology, 1994, V | |||
| Заслонка для русской печи | 1919 |
|
SU145A1 |
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| SAFDARI Y | |||
| et al., Antibody humanization methods-a review and update, Biotechnology and Genetic Engineering Reviews, 2013, V | |||
| Солесос | 1922 |
|
SU29A1 |
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| SHEN J | |||
| et al., Single variable | |||
Авторы
Даты
2022-12-21—Публикация
2018-02-20—Подача