Изобретение относится к области биотехнологии, биоинженерии, биофармакологии и может быть использовано в качестве препарата, нейтрализующего рициновый токсин и использоваться для специфической терапии при отравлениях рициновым токсином.
Рицин (РТ) является фитотоксином, вырабатываемый растением, клещевина Ricinus communis [Endo Y, Tsurugi K. // J. Biol. Chem. - 1987. - Vol 262. - №. 17. - Р. 8128-30]. Токсин вырабатывается в клетках эндосперма созревающих семян клещевины, где он накапливается в вакуолях до 5% от общего количества белка [Lord J.M, Roberts L.M, Robertus J.D. // FASEB J. - 1994. - Vol. 8. - №. 2. - P. 201-208]. Рицин относится к AB семейству токсинов и второму типу рибосом-инактивируемых белков [Peumans W.J., Hao Q., van Damme E.J. // FASEB J. - 2001. - Vol. 15. - P. 1493-1506]. Рицин представляет собой гликопротеин с молекулярной массой 60-65 kDa, состоящий из двух полипептидных цепей А и В, связанных дисульфидной связью [Olsnes S., Pihl A. // Pharmacol. & Ther. - 1981. - Vol. 15 - №. 3. - P. 355-381]. Цепь А (РТА) ~32 kDa, РНК специфическая N-гликозидаза, которая удаляет аденин в положении 4324 (A4324) в 28S рРНК 60S субъединицы рибосомы печени крысы [Endo Y., Mitsui K., Motizuki M., Tsurugi K. // J. Biol. Chem. - 1987. - Vol. 262. - P. 5908-12; Barbieri L., Battelli M.G., Stirpe F. // Biochim Biophys Acta. - 1993. - Vol. 1154. - P. 237-282]. Это специфическое пуриновое основание A4324 расположено внутри универсальной консервативной сарцин/рициновой петли (Sarcin/Ricin Loop) [May K.L., Yan Q., Tumer N.E. // Toxicon. - 2013. - Vol. 69. - P. 143-151], отщепление аденина приводит к ингибированию синтеза белка и гибели клетки. Цепь B (РТB) ~34 kDa, является специфическим лектином [Sandvig K., Olsnes S., Pihl A. // J. Biol. Chem. - 1976. - Vol. 251. - P. 3977-3984], связывается с галактозными остатками гликопротеинов и гликолипидов на поверхности эукариотических клеток, и способствует обратному транспорту РTA в цитозоль [Sandvig K., van Deurs B. // FEBS Lett. - 2002. - Vol. 529. P. 49-53].
Симптомы перорального отравления РТ неспецифичны появляются в течение 12 часов и могут включать тошноту, рвоту, диарею и боль в животе. могут прогрессировать до артериальной гипотензии, печеночной недостаточности, нарушения функции почек и смерти из-за полиорганной недостаточности или сердечно-сосудистого коллапса. РТ запускает апоптоз у 50% клеток при концентрации ниже 1 нг/мл [Tesh V.L. // Curr. Top Microbiol. Immunol - 2012. - Vol. 357. - P. 137-178], [Spooner R.A., Lord J.M. // Curr. Top Microbiol. Immunol. - Vol. 357. - P. 19-40]. Предполагается, что при вдыхании РТ появление симптомов в течении 8 часов в виде кашля, одышки, артралгии и лихорадки и может привести к дыхательной недостаточности и смерти. У человека предполагаемая смертельная доза РТ при оральном отравлении составляет от 1 до 20 мг/кг [Audi J., Belson M., Patel M., Schier J., Osterloh J. // JAMA - 2005. - Vol. 294. - № 18. - P. 2342-2351], [Franz D.R., Jahrling P.B., Friedlander A.M., McClain D.J., Hoover D.L., et al. // JAMA - 1997. - Vol. 278. - P. 399-411]. При аэрозольном отравлении смертельная доза составляет приблизительно 5 мкг/кг токсина [Brey R.N., Mantis N.J., Pincus S.H., Vitetta E.S., Smith L.A., et al. // Hum. Vaccin. Immunother. - 2016. - Vol. 12. - P. 1196-1201].
В научной литературе, имеются статьи с описаниями получения вакцин против РТ. В качестве компонента вакцины использовали рекомбинантный РТА (рРТА) [Olson M.A., Carra J.H., Roxas-Duncan V., Wannemacher R.W., Smith L.A., et al. // Protein Eng. Des. Sel. - 2004. - Vol. 17. - P. 391-397], вакцина RiVax на основе рРТА [Smallshaw J.E., Richardson J.A., Vitetta E.S. // Vaccine - 2007. - Vol. 25. - P. 7459-7469], [Vitetta E.S., Smallshaw J.E., Coleman E., Jafri H., Foster C., et al. // Proc. Natl. Acad. Sci. USA - 2006. - Vol. 103. - P. 2268-2273], вакцина RVEc была разработана для снижения или устранения активности N-гликозидазы РТА путем изменения риботоксического сайта [McLain D. E., Horn T. L., Detrisac C. J., Lindsey C. Y., Smith L. A. // International Journal of Toxicology - 2011. - Vol. 30. - P. 143-152].
Пассивная иммунизация остается одним из наиболее эффективных методов профилактики и лечения токсических отравлений [Binder P., Attre O., Boutin J.P., Cavallo J.D., Debord T., et al. // Comp. Immunol. Microbiol. Infect. Dis. - 2003. - Vol. 26. P. 401-421].
Уже доказана эффективность применения моноклональных антител (МКАт) способных нейтрализовать РТ. GD12 - мышиное МКАт подкласса IgG1, имеющее эпитоп в 163-174 участке (TLARSFIICIQM) в РTA [Neal L.M., O'Hara J, Brey R.N, Mantis N. J. // American Society for Microbiology Infection and Immunity - 2010. - Vol. 78. - P. 552-561], UNIVAX 70 и 138 мышиные МКАт, подкласса IgG1, специфичны к РТА [Lemley P.V., Amanatides P., Wright D. C. // Hybridoma - 1994. - Vol. 13. P. 417-421], CETUS 75/3B12 мышиные МКАт против РTВ [Colombatti M., Johnson V.G., Skopicki H.A., Fendley B., Lewis M.S., Youle R.J // J. Immunol. - 1987. - Vol. 138. - P. 3339-3344], мышиные МКАт против цепей РTA (RА37, RA36) и РTB (RB34) [Prigent J., Panigai L., Lamourette P., Sauvaire D., Devilliers K., Plaisance M., Volland H., Creminon C., Simon S. // PLOS ONE - 2011. - Vol. 6. - P. 1-10], 3D74 и 4C13 мышиные МКАт имеющие эпитопы на обоих цепях РT [Guo J., Shen B., Sun Y. et al // Hybridoma - 2006. - Vol. 25. P. 225-229]. Химерное антитело c4C13 против РТ [Wang Y., Guo L., Zhao K., Chen J., Feng J., Sun Y., Li Y., Shen // Biotechnology Letters -2007. - Vol. 29. P. 1811-1816]. Рекомбинантное гуманизированное МКАт MIL50 против РТ [GAO Y., ZHU X., MENG Z., GAN H., GU R., WU Z., SUN W., DOU G. // Acta Pharmaceutica Sinica - 2022. - Vol. 57. - P. 480-483] и гуманизированное МКАт D9 [Hu W., Yin J., Chau D., Negrych L. M., Cherwonogrodzky J. W. // PLOS ONE - 2012. - Vol. 7. - Is. 9. - P. 1-9]. Приведённые выше работы демонстрируют потенциальную эффективность использования МКАт в терапии и на ранних стадиях токсического отравления РТ.
Национальный институт аллергии и инфекционных заболеваний (NIAID) и центры по контролю и профилактике заболеваний (CDC) США классифицируют РТ как приоритетный патоген категории В [NIAID Emerging Infectious Diseases/Pathogens // National institute of Allergy and Infectious diseases, - 2016. - National Institute of Health USA]. РТ является одиним из сильных и смертельных токсинов. Легкость, с которой можно получить токсин, делает РТ привлекательным оружием [Romano J.A., Lukey B.J., Salem H. // Pharmacology, Toxicology, and Therapeutics - 2007. - 2 изд., P. 752], [Balali-Mood M., Moshiri M., Etemad L. // Toxicon - 2013. - Vol. 69. - P. 131-142]. В настоящее время не существует противоядия, эффективных вакцин или других специфических терапий для лечения или профилактики отравления РТ.
Технический результат заключается в получении двух МКАт с одинаковыми вариабельными доменами: мышиного и рекомбинантного, в частности химерного (мышь-человек), которые способны к высокоаффинному и высокоспецифичному связыванию с цепью А рицинового токсина Ricinus communis и обладают нейтрализующим действием в отношении РТ.
Технический результат достигается тем, что предложен штамм мышиной гибридной клеточной линии Mus musculus 5B9 - продуцент моноклонального антитела 5B9, полученной в результате гибридизации мышиной миеломной линии X63-Ag8.653 со спленоцитами мыши линии BALB/c, гипериммунной в отношении рРTA рицинового токсина Ricinus communis. Штамм гибридных культивируемых клеток животных Mus musculus (гибридомы), являющийся продуцентом МКат 5B9, депонирован в «ГКПМ-Оболенск» ФБУН ГНЦ «Государственный научный центр прикладной микробиологии и биотехнологии» 30.03.2022 г. под № Н-97.
Технический результат также достигается тем, что предложено мышиное МКАт 5B9 специфичное к цепи А рицинового токсина Ricinus communis и нейтрализующее рициновый токсин Ricinus communis, представленное двумя идентичными тяжелыми и двумя идентичными легкими цепями, с известными аминокислотными последовательностями легкой цепи SEQ ID NO: 1 и Fd фрагмента тяжелой цепи SEQ ID NO: 2.
Технический результат также достигается тем, что предложено рекомбинантное химерное МКАт xi5B9 специфичное к цепи А рицинового токсина Ricinus communis, нейтрализующее рициновый токсин Ricinus communis, синтезируемое в эукариотической клеточной линии ExpiCHO-S, представленное двумя идентичными тяжелыми и двумя идентичными легкими цепями, с аминокислотной последовательностью легкой цепи SEQ ID NO:3 и с аминокислотной последовательностью тяжелой цепи SEQ ID NO:4, полученное путем замены константных доменов легкой цепи и тяжелой цепи мышиного антитела 5B9 на аналогичные области иммуноглобулина G человека.
Изобретение иллюстрируют следующие графические материалы:
Фиг. 1. Графическое представление результата выделения и очистки МКАт 5B9 и xi5B9 электрофоретического разделения в 10%-ом ПААГ. Окраска геля кумасси бриллиантовым синим R-250.
Дорожка 1 - маркер молекулярной массы белков PageRuler Plus Prestained Protein Ladder (Fermentas). Дорожка 2 - образец очищенного МКАт 5B9 (нередуцирующие условия). Дорожка 3 - образец очищенного МКАт 5B9 с добавлением 2-меркаптоэтанола (редуцирующие условия). Дорожка 4 - образец очищенного МКАт xi5B9 (нередуцирующие условия). Дорожка 5 - образец очищенного МКАт xi5B9 с добавлением 2-меркаптоэтанола (редуцирующие условия).
Фиг. 2. Результат электрофоретического разделения РТ и рекомбинантного белка рРТА, связывающимся с МКАт 5B9 и xi5B9. Окраска геля кумасси бриллиантовым синим R-250.
Дорожка 1 и 4 - маркер молекулярной массы белков PageRuler Prestained Protein Ladder (Thermo Fisher). Дорожка 2 - рицин (нередуцирующие условия). Дорожка 3 - рицин с добавлением 2-меркаптоэтанола (редуцирующие условия). Дорожка 5 - рекомбинантный белок рРТА.
Фиг. 3. Данные иммуноблота по связыванию МКАт 5B9 с рицином и рекомбинантным белком рРТА.
Дорожка 1 - маркер молекулярной массы белков PageRuler Prestained Protein Ladder (Thermo Fisher). Дорожка 2 -связывание МКАт 5B9 с рицином. Дорожка 3 -связывание МКАт 5B9 с рекомбинантным рРТА.
Фиг. 4. Данные иммуноблота по связыванию МКАт xi5B9 с рицином и рекомбинантным белком рРТА.
Дорожка 1 - маркер молекулярной массы белков PageRuler Plus Prestained Protein Ladder (Fermentas). Дорожка 2 -связывание МКАт xi5B9 с рицином. Дорожка 3 -связывание МКАт xi5B9 с рицином (редуцирующие условия). Дорожка 4 -связывание МКАт xi5B9 с рекомбинантным рРТА.
Фиг. 5. Результат определения подкласса и изотипа легкой цепи очищенных МКАт 5B9 с помощью иммунохроматографического экспресс-теста IsoQuick™ Strips for Mouse Monoclonal Isotyping (Envirologix, США).
Фиг. 6. Праймеры для сборки химерной легкой и тяжелой цепи МКАт (5B9) с кратким описанием.
Фиг. 7. Кривые зависимости абсорбции при длине волны 492 нм от концентрации антител 5B9 и xi5B9 в ИФА.
Фиг. 8. График расчета равновесной константы диссоциации МКАт xi5B9 с рекомбинантным белком рРТА.
Фиг. 9. Графическое представление результата амплификации ДНК, кодирующих различные участки МКАт после электрофоретического разделения в 1,5% агарозном геле.
Дорожка 1 - Маркер молекулярной массы ДНК GeneRuler 100 bp Plus DNA Ladder (#SM0321, Thermo Fisher). Дорожка 2 - легкая цепь 5B9. Дорожка 3 - тяжелая цепь 5B9 (Fd фрагмент). Дорожка 4 - VL вариабельный домен легкой цепи 5B9. Дорожка 5 - VH вариабельный домен тяжелой цепи 5B9. Дорожка 6 - легкая цепь xi5B9. Дорожка 7 - тяжелая цепь xi5B9. Дорожка 8 - ампликон тяжелая цепь+ легкая цепь xi5B9
Фиг. 10. Данные иммуноблота, отражающего взаимодействие МКат xi5B9 с антителами против его константных областей.
Дорожка 1, 3 - маркер молекулярной массы белков PageRuler Plus Prestained Protein Ladder (Fermentas). Дорожка 2 - образец очищенного МКАт xi5B9 после взаимодействия с антителами против к-цепи человеческого иммуноглобулина. Дорожка 4 - образец очищенного МКАт xi5B9 после взаимодействия с антителами против Fc фрагмента человеческого иммуноглобулина G.
Фиг 11. Графическое представление результата выделения и очистки МКАт xi5B9 из культуральной жидкости после электрофоретического разделения в 7,5%-ом ПААГ.
Окраска геля кумасси бриллиантовым синим R-250. Дорожка 1 - маркер молекулярной массы белков PageRuler Plus Prestained Protein Ladder (Fermentas). Дорожка 2 - образец очищенного МКАт xi5B9 (нередуцирующие условия).
Фиг 12. Определение токсиннейтрализующей активности МКАт 5B9 и xi5B9 in vitro в тесте МТТ.
Изобретение осуществляют следующим образом.
Для иммунизации использовали мышей линии BALB/c. Мышей иммунизировали двукратно, с четырнадцатидневной экспозицией, методом подкожного введения рРТА (Seq ID NO: 5) в дозе 100 мкг/мышь эмульгированного с полным (первая иммунизация) и неполным (вторая иммунизация) адъювантом Фрейнда (Sigma). Бустер-дозы препарата 50 мкг/мышь вводили внутривенно в физиологическом растворе. На третьи сутки после последней иммунизации проводится гибридизация спленоцитов иммунных мышей с партнером для слияния - клетками мышиной миеломной линии Х63-Ag8.653. Гибридизацию производят по методу Келера и Мильштейна [Köhler G., Milstein С. // Nature. - 1975. - Vol. 256. - №. 5517. - P. 495-497.] с модификациями. После гибридизации проводят селекцию, скрининг, клонирование и криоконсервацию гибридных клеток. Полученный моноклональный штамм гибридомы депонируют в коллекцию «ГКПМ-Оболенск» при ФБУН ГНЦ «Государственный научный центр прикладной микробиологии и биотехнологии» под названием «штамм гибридных культивируемых клеток животных Mus musculus 5B9». Для получения препаративных количеств МКАт 5B9 гибридомные клетки штамма 5B9 культивируют в культуральной среде RPMI medium 1640 с добавлением 20% фетальной бычьей сыворотки с последующим выделением из нее иммуноглобулиновой фракции посредством аффинной хроматографии. Готовый белковый продукт валидируют электрофорезом по Лэммли по типовому распределению иммуноглобулина G в полиакриламидном геле (ПААГ) до и после воздействия редуцирующего агента в сравнении с маркером молекулярной массы: без восстановления дисульфидных связей молекулярной вес белка представлен монопродуктом с массой 150 кДа, а редуцированная молекула отображается в виде двух полос с массами 25 и 50 кДа (Фиг. 1 дорожки 2 и 3).
Подкласс антитела и изотип легкой цепи определяют в иммунохроматографическом тесте (Фиг. 5). Для создания рекомбинантного химерного антитела, именуемого xi5B9, (константная часть от иммуноглобулина IgG1 человека, вариабельная часть от мышинокого МКАт 5B9) выделяют фракцию тотальной РНК штамма гибридных клеток 5B9, проводят реакцию обратной транскрипции, затем амплифицируют методом 5'-РАСЕ кДНК, кодирующие Fab-фрагмент, по методике (с модификациями), описанной в [Bradbury A. // Antibody Engineering. - Springer, Berlin, Heidelberg, 2010. - P. 15-20.]. Продукты ПЦР реакции (Фиг. 9, дорожки 2 и 3) клонируют в вектор pUC19 и секвенируют. По результатам секвенирования определяют границы вариабельной части и осуществляют «химеризацию» антитела путем амплификации участков вариабельной легкой цепи (VL) и вариабельной тяжелой цепи (VH) (Фиг. 9, дорожки 4 и 5) с последующим клонированием ПЦР продуктов в коммерческие вектора с константной легкой цепью (CL) и константной тяжелой цепью (CH) доменами человеческого IgG1. Получив таким образом гены полноразмерных цепей (Фиг. 9, дорожки 6 и 7) в двух отдельных плазмидах, далее собирают рекомбинантную ДНК, кодирующую молекулу химерного иммуноглобулина путем соединения генов двух цепей с помощью OE-PCR. В итоге получают молекулу ДНК (Фиг. 9, дорожка 8; Seq ID NO: 30) с кодонами следующих полипептидных участков (сверху вниз по ходу транскрипции): тяжелая цепь МКАт xi5B9, сайт узнавания протеазой фурином RAKR, глицин-сериновый линкер GSG, Т2А пептид, легкая цепь МКАт xi5B9. Полученный ПЦР продукт клонируют в плазмидный вектор pcDNA 3.4 для экспрессии в эукариотических клетках и именуют его pcDNA3.4_xi5B9. Данный вектор трансфецируют в клетки яичника китайского хомячка (СНО) и получают продуцента МКАт xi5B9. Клетки-продуценты культивируют в бессывороточной среде в колбах Эрленмейера, затем выделяют МКАт из культуральной жидкости с применением концентрирования и аффинной хроматографии. Белковый продукт валидируют электрофоретически подобно антителу 5B9 (Фиг. 1, дорожка 4 и 5), а также показывают взаимодействие белка с антителами к константным человеческим регионам в иммуноблоте (Фиг. 10), что отражает химерное строение рекомбинантного иммуноглобулина. С очищенными МКАт 5B9 и xi5B9 проводят ряд анализов: оценку специфичности связывания с антигеном в иммуноблоте (Фиг. 3 и 4), оценку количественного взаимодействия с антигеном в иммуноферментном анализе (ИФА) (Фиг. 7), определение равновесной константы диссоциации (Kd) методом поверхностного плазмонного резонанса (Фиг. 8), определение токсиннейтрализующей активности in vitro на клеточной линии Vero (CCL-81™) в микротесте МТТ (Фиг. 12) используя РТ Ricinus communis.
Устанавливают, что предложенные антитела в равной степени высокоаффинно связываются с антигеном рPTA и обладает токсиннейтрализующей активностью против РТ Ricinus Communis. Другие характерные признаки антител, установленные в процессе их создания, кратко перечислены ниже.
Мышиное МКАт 5B9:
A) Предоставлено иммуноглобулином IgG1 с легкой цепью κ;
Б) Эпитоп локализован в цепи А рицинового токсина Ricinus communis;
B) Молекулярный вес = 145 кДа;
Г) Сигнальная последовательность и последовательность легкой цепи Seq ID NO: 1;
Д) Сигнальная последовательность и последовательность Fd-фрагмента тяжелой цепи (вариабельная область и СН1 домен) Seq ID NO: 2;
Е) Последовательность вариабельной области легкой цепи Seq ID NO: 7;
Ж) Последовательность вариабельной области тяжелой цепи Seq ID NO: 9.
Химерное (мыши-человек) МКАт xi5B9:
A) Предоставлено иммуноглобулином IgG1 с легкой цепью κ;
Б) Эпитоп локализован в цепи А рицинового токсина Ricinus communis;
B) Kd = 2,63×10-8 М,
Г) Молекулярный вес = 147 кДа;
Д) Сигнальная последовательность и последовательность легкой цепи Seq ID NO: 3;
Е) Сигнальная последовательность и последовательность тяжелой цепи Seq ID NO: 4;
Для лучшего понимания сущности изобретения ниже следуют примеры его конкретного выполнения.
Пример 1. Получение и культивирование гибридной клеточной линии. Иммунизация лабораторных животных. Мышей линии BALB/c весом 18-20 г иммунизируют рекомбинантным белком рРТА по следующей схеме:
1) Вводят рекомбинантный белок рRTA с концентрацией 1 мг/мл в дозе 100 мкг/мышь с полным адъювантом Фрейнда в объемном соотношении 1:1 подкожно.
2) Через 2 недели вводят рекомбинантный белок рРТА с концентрацией 1 мг/мл в дозе 100 мкг/мышь с неполным адъювантом Фрейнда в объемном соотношении 1:1 подкожно.
3) Через 2 недели вводят рекомбинантный белок рРТА в дозе 50 мкг/мышь внутривенно.
Кровь иммунизированных мышей отбирают на третий день после финальной инъекции. Титры специфических антител в полученных сыворотках крови определяют с помощью непрямого твердофазного ИФА.
Гибридизация. Через 3 суток после финальной иммунизации мышей, чьи сыворотки крови показали наилучшие результаты при тестировании в ИФА, умерщвляют методом дислокации шейных позвонков, извлекают селезенки, и проводят процедуру слияния (гибридизации) 1×108 спленоцитов гипериммунной мыши с 1×107 клеток миеломной мышиной линии Х63-Ag8.653 (АТСС® CRL-1581™). В качестве агента для слияния применяют раствор полиэтиленгликоля в диметилсульфоксиде (Sigma Р7306, США).
Слившиеся клетки рассевают в лунки культуральных планшетов с заранее подготовленным фидерным слоем, состоящим из мышиных перитонеальных макрофагов в количестве 1×104 клеток на лунку. Культивирование полученных гибридных клеток осуществляют в СО2-инкубаторе Forma Serias II (Thermo Fisher, США) при температуре 37°С во влажной атмосфере, содержащей 5% СО2, в среде RPMI medium 1640 с 20% эмбриональной телячьей сывороткой, 2 мМ L-глутамина, 100 мкг/мл гентамицина, однократным раствором гипоксантина-аминоптерина-тимидина (HAT) (Gibco 21060-017, Thermo Fisher, США).
Селекция. Селекцию полученных гибридных клеток проводят с использованием однократного раствора HAT в течение 3 недель.
Скрининг гибридных клеток. Отбор положительных гибридных клеток, продуцирующих специфичные антитела, осуществляют методом непрямого твердофазного ИФА. Для проведения непрямого варианта ИФА в лунки 96-луночного полистиролового планшета для иммуноферментного анализа вносят по 1 мкг/лунку антигена рРТА в фосфатно-солевом буфере (ФСБ) (137 мМ NaCl, 2,7 мМ KCl, 10 мМ Na2HPO4, 1,76 мМ KH2PO4, рН 7.4) и инкубируют при температуре 37°С в течение 1 часа на планшетном орбитальной шейкере (Elmi, Латвия) при скорости вращения платформы 300 об./мин. Трижды отмывают ФСБ, содержащим 0,5% детергента Tween® 20 (ФСБ-Тв) по 200 мкл. Затем свободные валентности пластика блокируют молоком с массовой долей жира не более 0,5%, внося по 200 мкл на лунку, и инкубируют при температуре 37°С в течение 1 часа при тех же условиях. Трижды отмывают ФСБ-Тв. Подготовленные таким образом планшеты также используют в примерах: 2 и 8. Далее вносят в лунки супернатант культуральной жидкости, в которой выращивали гибридные клетки, в объеме 100 мкл, инкубируют при температуре 37°С в течение 1 часа на шейкере. После этого лунки планшета трижды отмывают ФСБ-Тв, и добавляют в лунки раствор кроличьих антител к целой молекуле IgG мыши, конъюгированных с пероксидазой хрена (Sigma-Aldrich, США), в рабочем разведении 1:10000. Планшет инкубируют при температуре 37°С в течение 1 часа на шейкере, затем 6 раз отмывают буфером ФСБ-Тв. После этого в лунки вносят по 100 мкл субстрат-индикаторного раствора (10 мкл 30% Н2О2 и 8 мг ортофенилендиамина на 10 мл фосфатно-цитратного буфера рН 5.0). Положительную реакцию оценивают по появлению желто-коричневого окрашивания раствора. Реакцию останавливают добавлением в лунки по 50 мкл 4N серной кислоты. Детекцию проводят на планшетном спектрофотометре (Bio-Rad xMark, США) при длине волны 492 нм.
Клонирование. Клонирование гибридных клеток проводят методом лимитирующих разведений в 96-луночных планшетах на фидерном слое, состоящем из перитонеальных макрофагов в количестве 1×104 клеток на лунку. Скрининг клонов проводят методом непрямого твердофазного ИФА, как было описано выше. Скринируют только те лунки, которые содержат моноклональную популяцию клеток. Клоны, показавшие наилучшие результаты в ИФА, тестируют не менее трех раз с интервалом 3-4 дня. Наиболее стабильные клоны гибридных клеток отбирают для дальнейшей работы. Наилучшие и стабильные результаты в ИФА показал клон гибридной клеточной линии 5B9.
Культивирование. Культивирование клона гибридной клеточной линии 5B9 ведут в культуральных флаконах Corning® Т-25 и Т-75 при температуре 37°С в атмосфере, содержащей 5% CO2. Средой культивирования является RPMI medium 1640, содержащая 20% эмбриональной телячьей сыворотки, 2 мМ L-глутамина, 100 мкг/мл гентамицина.
Количество клеток при субкультивировании поддерживают в диапазоне между 1×105-1×106 на 1 мл культуральной среды. Обновление среды путем частичной или полной замены производят каждые 2-3 дня.
Выявление контаминации гибридной клеточной линии клона 5В9. Контаминацию культуры гибридных клеток 5В9 оценивают визуально и с помощью набора флюоресцентных красителей для определения бактериальной и грибковой контаминации клеточных культур Cell Culture Contamination Detection Kit C-7028 (Thermo Fisher, США) и флюоресцентного красителя для ДНК, подходящего для определения контаминации микоплазмами Hoechst 33258 (Sigma-Aldrich, США). Всю пробоподготовку клеток гибридной линии и культуральной жидкости проводят согласно рекомендациям производителей. Подготовленные образцы рассматривают с помощью инвертированного флуоресцентного микроскопа CKX41 с ртутным осветителем URFLT50 (Olympus, Япония).
Криоконсервация. Замораживание клонов гибридных клеток 5В9 производят в специальных криопробирках (виалах) объемом 2 мл. В качестве среды для криоконсервации используем фетальную телячью сыворотку с добавлением 10% диметилсульфоксида. Снятие клеток с поверхностей культуральных флаконов осуществляют потряхиванием и пипетированием. Суспензию клеток центрифугируют при 125×g в течение 5 минут, удаляем супернатант и ресуспендируем в 1 мл среды для криоконсервации. Криопробирки охлаждаем до -80°С со скоростью заморозки 1°С/мин. Хранение замороженных культур осуществляем в жидком азоте. При разморозке виал нагревают на водяной бане при 37°С до растапливания льда, содержимое переносят в пробирку со средой RPMI medium 1640, центрифугируют 5 минут при 200×g. После чего супернатант удаляют, а клеточный осадок высевают и продолжают культивирование.
Культивирование в организме животного. Мышам линии BALB/c в возрасте 18-20 недель внутрибрюшинно вводят по 0,5 мл пристана (Sigma, США), а через 2-3 недели вводят внутрибрюшинно гибридные клетки 5B9 в пределах от 5×105 до 2×106 в среде DMEM без фетальной телячьей сыворотки. Иммуноасциты появляются на 7-15 сутки, а отбор асцитической жидкости производят на 10-21 сутки в соответствии со скоростью созревания каждого отдельного асцита. Клеточные компоненты асцитической жидкости удаляют центрифугированием (1000×g 15 мин), преципитируют белковую фракцию асцитической жидкости добавлением насыщенного раствора (NH4)2SO4 в соотношении 1:1, выдерживают при температуре 4°С не менее 12 часов.
Пример 2. Очистка и валидация МКАт 5B9
Выделение МКАт 5B9 из асцитической жидкости проводят методом аффинной хроматографии на колонке с сорбентом Protein G сефарозой (Protein G Sepharose 4 FastFlow, GE Healthcare, Великобритания). Для очисти МКАт 5B9 используют хроматографическую систему ÄCTA Pure (GE Healthcare, Швеция).
Очистка. Преципитат антител сульфатом аммония осаждают центрифугированием при 10000×g в течение 20 минут, удаляют супернатант. Осадок растворяют в 50 мл буфера нанесения 50 мМ Tris, 100 мМ NaCl, 0,02% NaN3, рН 8,0. Колонку емкостью 3 мл, заполненную Protein G сефарозой, уравновешивают буфером нанесения. Производят нанесение образца со скоростью 2 мл/мин на льду. Далее колонку отмывают от не связавшихся компонентов буфером нанесения, и проводят элюцию иммуноглобулинов буфером состава: 100 мМ глицина, 100 мМ NaCl, рН 2.4. Элюат собирают после начала экспоненциального роста величины абсорбции при 280 нм (А280) и заканчивают, когда А280 начинает снижаться по логарифмическому закону (выходить на плато). Уровень рН элюата доводят до 7,5 - 8,0 добавлением 1 М раствора Tris основания. Впоследствии элюат иммуноглобулиновой фракции вносят в коммерчески доступную колонку HiPrep™ 26/10 Desalting (GE Healthcare) емкостью 53 мл, уравновешенную ФСБ на скорости потока 10 мл/мин. Иммуноглобулиновую фракцию собирают через некоторое время, идентифицируя ее по изменению А280. Для измерения концентрации МКАт проводят спектрофотометрию при А280, затем для расчета концентрации [мг/мл] полученное значение разделяют на коэффициент массовой экстинкции для IgG, равный 1,37. Чистоту полученной иммуноглобулиновой фракции оценивают методом SDS-электрофореза по Лэммли в полиакриламидном геле (ПААГ) в редуцирующих и нередуцирующих условиях (Фиг. 1 дорожка 2, 3).
Оценку иммунологической специфичности очищенного МКАт 5B9 проводят методом иммуноблота (Вестерн-блоттинга) с РТ Ricinus communis и рекомбинантным рРТА (Seq ID NO: 5). Электрофоретическое разделение антигенов изображено на Фиг. 2. Белки подвергают электрофорезу в 10% ПААГ, после чего осуществляют горизонтальный перенос белков из геля на нитроцеллюлозную мембрану Hybond-C Extra (GE Healthcare, Великобритания) с помощью оборудования Trans-Blot® Turbo™ Tr ansfer System (Bio-Rad, США). После завершения переноса мембрану блокируют погружением в обезжиренное молоко с массовой долей жира не более 0,5% и инкубируют в течение 1 часа на орбитальном термостатируемом шейкере при 300 об./мин и температуре 37°С. Далее, после трехкратной промывки буфером ФСБ-Тв, мембрану инкубируют с МКАт 5B9, разведенным до концентрации 0,1-1 мкг/мл в ФСБ. После трехкратной промывки буфером ФСБ-Тв мембрану инкубируют с антителами к суммарной фракции IgG мыши, конъюгированными с пероксидазой хрена (А9044, Sigma, США) в разведении 1:10000 в буфере ФСБ-Тв в течение 30 минут. Затем мембрану промывают 6 раз в ФСБ-Тв. Визуализацию реакции проводят раствором субстратной смеси на основе диаминобензидина (0,05% диаминобензидина (Sigma, США), 0,015% H2O2 в ФСБ рН 7,4). Реакцию останавливают после отчетливо различимого окрашивания образцов путем погружения мембраны в дистиллированную воду. Как показывает проведенный анализ, результаты которого отражены на Фиг. 3 (дорожки 1 и 2), МКАт 5B9 специфически взаимодействует с РТ и рРТА.
Для оценки количественного взаимодействия очищенного МКАт 5B9 с рРTA, в лунки 96-луночного планшета с иммобилизированным антигеном, который подготовлен по методике из Примера 1, вносят методом двукратных разведений антитело в концентрациях 10000, 5000, 2500, 1250, 625, 312.5, 156.3, 78.1, 39.1, 19.5, 9.8, 4.9, 2.5, 1.2 нг/лунку. Инкубируют на термостатируемом шейкере при температуре 37°С в течение 1 часа 300 об/мин. После этого лунки планшета трижды отмывают ФСБ-Тв и добавляют в лунки раствор кроличьих антител к целой молекуле IgG мыши, конъюгированных с пероксидазой хрена в рабочем разведении 1:10000. Планшет инкубируют при температуре 37°С в течение 1 часа на шейкере, затем 6 раз отмывают буфером ФСБ-Тв. После этого в лунки вносят по 100 мкл субстрат-индикаторного раствора на основе ортофенилендиамина. Положительную реакцию оценивают по появлению желто-коричневого окрашивания раствора. Реакцию останавливают добавлением в лунки по 50 мкл 4N серной кислоты. Детекцию проводят на планшетном спектрофотометре (Bio-Rad xMark, США) при длине волны 492 нм. По результатам данного анализа, отраженного на Фиг. 7, минимально детектируемая концентрация МКАт 5B9 составила 25 нг/мл, кривая А = f(C) соответствует типовой кривой для высокоаффинных антител в непрямом твердофазном ИФА.
Пример 3. Определение подкласса очищенных МКАт 5B9.
Для определения используют иммунохроматографический экспресс-тест IsoQuick™ Strips for Mouse Monoclonal Isotyping (Envirologix, США). Перед использованием экспресс-теста очищенные МКАт разводят до концентрации 0,1-1 мкг/мл в 200 мкл буфера ФСБ в микроцентрифужной пробирке объемом 1,5 мл. После этого в пробирку погружают тест-полоску и оставляют до проявки контрольной полосы. По результатам иммунохроматографического изотипирования МКАт 5B9 относятся к мышиным иммуноглобулинам G, подкласс IgG1, изотип легкой цепи κ. Результаты отображены на Фиг. 5.
Пример 4. Установление первичной последовательности Fab фрагмента МКАт 5B9.
Для установления генетической и первичной аминокислотной последовательности участков легкой и тяжелой цепи (LC и Fd фрагмента) МКАт 5B9 используют методику 5'RACE-метода амплификации кДНК. На первом этапе выделяют тотальную РНК из 2×106-4×106 клеток гибридомы 5B9 с помощью набора QIAamp RNA Blood Mini Kit (QIAGEN) согласно методике производителя для суспензионной культуры клеток, с включением в протокол этапа расщепления остаточной ДНК непосредственно на колонке с использованием набора RNase-Free DNase Set (QIAGEN). Далее проводят две независимые реакции обратной транскрипции, в каждой реакции задействуют по 1 мкг полученной РНК и реактивы набора RevertAid First Strand cDNA Synthesis Kit (Thermo Fisher). Для синтеза первой цепи кДНК LC иммуноглобулина 5B9 используют праймер Oligo(dT)18 (Seq ID NO: 14), гибридизующийся с поли(А)-хвостом мРНК. Синтез первой цепи кДНК Fd фрагмента антитела 5B9 осуществляют с помощью ген-специфического праймера RACEMOG1 (ID NO: 15), комплементарного области мРНК, кодирующей «шарнирный» участок молекулы иммуноглобулина.
Следующим этапом к 3'-концу полученных кДНК присоединяют поли(А)-хвост ферментом концевой дезоксинуклеотидилтрансферазой (TdT). На одну реакцию объемом 10 мкл используют 1 мкл продукта реакции обратной транскрипции, 2 мМ дАТФ и 5 ед. TdT (Thermo Fisher). Смесь инкубируют в течение 5 минут, после чего фермент подвергают термической инактивации: 10 минут при 70°С.
Первую цепь кДНК LC амплифицируют с парой олигонуклеотидных праймеров CKMOsp (ID NO: 16) и XSCTnTag (ID NO: 17). Реакционная смесь (25 мкл) помимо воды, свободной от нуклеиновых кислот и нуклеаз, по 5 пкМ каждого праймера, 0,5 мкл продукта реакции полиаденилирования кДНК и смесь Phusion High-Fidelity PCR Master Mix (Thermo Fisher). Амплификацию проводят, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 40 циклов: 98°С 15 с, 40°С 15 с, 72°С 30 с, затем финальная элонгация при 72°С в течение 5 минут.
Первую цепь кДНК Fd амплифицируют с парой праймеров MOCG12FOR (ID NO: 18) и XSCTnTag. Для приготовления ПЦР смеси (25 мкл) используют воду, свободную от нуклеиновых кислот и нуклеаз, по 5 пкМ каждого праймера, 0,5 мкл продукта реакции полиаденилирования кДНК и смесь Phusion High-Fidelity PCR Master Mix (Thermo Fisher). Амплификацию проводят, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 40 циклов: 98°С 15 с, 40°С 15 с, 72°С 30 с, затем финальная элонгация при 72°С в течение 5 минут.
ПЦР продукты LC и Fd (Фиг. 9, дорожки 2 и 3) очищают с помощью электрофореза в агарозном геле при помощи QIAquick Gel Extraction Kit (QIAGEN). Очищенные ампликоны в количестве 100 нг подвергают обработке Т4 полинуклеотидкиназой и одновременно гидролизуют эндонуклеазой рестрикции Sall FastDigest™ в буфере FD с добавлением 0,5 мМ АТФ. В результате получают фрагменты ДНК с «липким» и «тупым» концами, готовые к встраиванию в вектор для клонирования pUC19. Плазмиду pUC19 в количестве 100 нг разрезают эндонуклеазами рестрикции Sall и Smal FastDigest™ и обрабатывают щелочной фосфатазой FastAP. Спустя час инкубации при 37°С ферменты в реакциях с плазмидой и вставками инактивируют температурой 75°С в течение 5 минут, затем соединяют по 50 нг плазмиды и вставки, добавляют АТФ до 0,5 мМ и 5 ед. Т4 ДНК-лигазы (все реактивы Thermo Fisher).
Выдерживают 2-3 часа при комнатной температуре, после чего подвергают фенол-хлороформной экстракции и спиртовой преципитации. Осадки ДНК растворяют в 1 мкл деионизованной воды и используют для электротрансформации компетентных клеток E.coli DH12S. Из позитивных трансформантов, выросших в виде единичных колоний на агаризованной среде LB с добавлением изопропил-β-D-1-тиогалактопиранозид (ИПТГ) и X-gal с ампициллином, выделяют плазмидную ДНК, которую впоследствии секвенируют по Сэнгеру с праймеров M13/pUC For (Seq ID NO: 19) и M13 Rev (Seq ID NO: 20). В результате определяют первичную аминокислотную последовательность легкой цепи (включая сигнальную последовательность и исключая аминокислотный остаток цистеина на с-конце), обозначенную в Seq ID NO: 1; определяют первичную аминокислотную последовательность Fd-фрагмента тяжелой цепи (включая сигнальную последовательность и исключая часть с-концевых аминокислот в домене CH1), обозначенную в Seq ID NO: 2. Устанавливают следующие границы вариабельных доменов: для легкой цепи Seq ID NO: 6, для тяжелой цепи Seq ID NO: 7.
Последовательности участков, определяющих комплементарность антитела, определяют, аннотируя аминокислотные последовательности VL и VH с использованием системы нумерации Кэбота: CDRL-1 - Seq ID NO: 8, CDRL-2 - Seq ID NO: 9, CDRL-3 -Seq ID NO: 10, CDRH-1 - Seq ID NO: 11, CDRH-2 - Seq ID NO: 12, CDRH-3 - Seq ID NO:13.
Пример 5. Сборка ДНК, кодирующей МКАт xi5B9
Создают плазмидные вектора pSF-CMV-xi5B9_LC и pSF-CMV-xi5B9_HC, содержащие генные конструкции полноразмерных цепей химерного иммуноглобулина xi5B9, на основе векторов pSF-CMV-HuKappa_LC (OG528) и pSF-CMV-HuIgG1_HC (OG527) из набора векторов для инженерии химерных или человеческих иммуноглобулинов Human IgG Vector Set (PP2409, Oxford Genetics). Осуществляют дизайн и синтез олигонуклеотидов 5B9VL SSL Nco_For (Seq ID NO: 21), 5B9VL BseRI Rev (Seq ID NO: 22), 5B9VH SSH Nco For (Seq ID NO: 23), 5B9 VH BseRI Rev (Seq ID NO: 24) для амплификации VL- и VH-кодирующих участков МКАт 5B9, которые устанавливают по методике примера 4. В состав прямых праймеров вводят сайт рестрикции Ncol и кодоны, определяющие синтез сигнальной последовательности, а обратных - сайт BseRI в определенном положении для «бесшовного соединения» VL-CL и VH-CH доменов. Описание олигонуклеотидов дано в таблице на Фиг. 6. Осуществляют дизайн и синтез олигонуклеотидов HC F1/2 (Seq ID NO: 28), HC R1 (Seq ID NO: 29), LC F1 (Seq ID NO: 26), LC R1/2 (Seq ID NO: 27) для амплификации полноразмерных цепей химерного МКАт xi5B9 и соединения двух цепей в единый ДНК-конструкт.
Получение химерной легкой цепи путем создания плазмидного вектора pSF-CMVxi5B9_LC. Составляют амлификационную смесь объемом 25 мкл из Phusion High-Fidelity PCR Master Mix, праймеров 5B9VL_SSL_Nco_F и 5B9VL_BseRI_R (no 5 пкМ каждого), воды для ПЦР. В качестве матрицы используют 10-20 нг плазмидной ДНК, полученной после лигирования pUC19 с кДНК легкой цепи МКАт 5B9 по примеру 4.
Амплификацию проводят, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 25 циклов: 98°С 15 с, 58°С 15 с, 72°С 30 с, затем финальная элонгация при 72°С в течение 5 минут. Качественный результат реакции оценивают электрофоретически (Фиг. 9, дорожка 4). После проведения ПЦР ДНК подвергают фенол-хлороформной экстракции и спиртовой преципитации. Далее 100 нг очищенного ПЦР-продукта гидролизуют эндонуклеазами рестрикции Ncol FastDigest и BseRI (NEB) в буфере FD. Плазмидный вектор OG528 в количестве 100 нг гидролизуют рестриктазами Ncol FastDigest и BseRI в буфере FD с добавлением щелочной фосфатазы FastAP. Спустя час инкубации при 37°С ДНК в обеих реакциях подвергают фенол-хлороформной экстракции и спиртовой преципитации. Плазмиду и вставку соединяют в одной реакционной смеси для лигирования с помощью Т4 ДНК-лигазы (Thermo Fisher). Пробирку выдерживают 2-3 часа при комнатной температуре, после чего подвергают фенол-хлороформной экстракции и спиртовой преципитации. Осадки ДНК растворяют в 1 мкл деионизованной воды и используют для электротрансформации компетентных клеток E. coli DH12S. Из позитивных трансформантов, выросших в виде единичных колоний на агаризованной ср еде LB с добавлением канамицина, выделяют плазмидную ДНК, которую впоследствии секвенируют по Сэнгеру с праймера CMV For (Seq ID NO: 25).
Получение химерной тяжелой цепи путем создания плазмидного вектора pSF-CMVxi5B9_HC. Составляют амлификационную смесь объемом 25 мкл из Phusion High-Fidelity PCR Master Mix, праймеров 5B9VH_SSH_Nco_F и 5B9VH_BseRI_R (пo 5 пкМ каждого), воды для ПЦР. В качестве матрицы используют 10-20 нг плазмидной ДНК, полученной после лигирования pUC19 с кДНК тяжелой цепи МКАт 5B9 по примеру 4.
Амплификацию проводят, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 25 циклов: 98°С 15 с, 58°С 15 с, 72°С 30 с, затем финальная элонгация при 72°С в течение 5 минут. Качественный результат реакции оценивают электрофоретически (Фиг. 9, дорожка 5). Далее методика получения вектора pSF-CMV-xi5B9_HC не отличается от методики получения pSF-CMV-xi5B9_LC, описанной выше, за исключением использования плазмиды OG527 для клонирования ПЦР-продукта.
Следующим этапом производят сборку рекомбинантной ДНК с открытой рамкой считывания, которая кодирует химерный полипептид (Seq ID NO: 30): тяжелая цепь xi5B9, аминокислотная последовательность RAKRGSGEGRGSLLTCGDVEENPGP, легкая цепь xi5B9. Сначала проводят две отдельные ПЦР с использованием Phusion High-Fidelity PCR Master Mix. Первую реакцию подготавливают с праймерами HC F1/2 и HC R1 (по 2 пкМ каждого), а также плазмидой pSF-CMV-xi5B9_HC в качестве матрицы (10-20 нг). Вторую - с праймерами LC F1 и LC R1/2 (по 2 пкМ каждого) и плазмидой pSF-CMV-xi5B9_LC в качестве матрицы (10-20 нг). Проводят амплификацию, используя следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 18 циклов: 98°С 15 с, 56°С 15 с, 72°С 45 с, затем финальная элонгация при 72°С в течение 5 минут. Качественный результат реакции оценивают электрофоретически. Изображение ампликона полноразмерной легкой цепи xi5B9 (Фиг. 9 в дорожке 6). Изображение ампликона полноразмерной тяжелой цепи xi5B9 (Фиг. 9 в дорожке 7). Далее подготавливают реакционную смесь с Phusion High-Fidelity PCR Master Mix в качестве основы, праймерами HC F1/2 и LC R1/2 (по 5 пкМ каждого) и по 0,1 мкл продукта из каждой проведенной ранее ПЦР. Для ПЦР используют следующие параметры: предварительная денатурация 98°С в течение 1 мин, далее 25 циклов: 98°С 15 с, 56°С 15 с, 72°С 1,5 мин, затем финальная элонгация при 72°С в течение 5 минут. Качественный результат реакции оценивают электрофоретически (Фиг. 9, дорожка 8). После проведения ПЦР ДНК подвергают фенол-хлороформной экстракции и спиртовой преципитации.
Пример 6. Получение плазмидного вектора для экспрессии МКАт xi5B9
ДНК, получаемую по примеру 5, в количестве 1 мкг, вносят в реакционную смесь объемом 50 мкл, состоящую из 1х Taq-буфера с 1,5 мМ MgCl2, 0,2 мМ dATP и 1 ед Taq-полимеразы. Реакцию инкубируют 20 мин при температуре 72°С.Затем производят очистку от фермента на колонке QIAquick PCR Purification Kit (Qiagen) согласно инструкции. Очищенную ДНК клонируют в вектор pcDNA3.4 из набора pcDNA 3.4-ТОРО ТА Cloning Kit (Thermo Fisher) по методу, описанному в инструкции производителя, и смешивают продукт реакции с хемокомпетентными клетками Е. coli One Shot ТОР10 (Thermo Fisher). Из позитивных трансформантов, выросших в виде единичных колоний на агаризованной среде LB с добавлением канамицина, выделяют плазмидную ДНК набором PureLink HiPure Plasmid Maxiprep Kit (Invitrogen). Полученный плазмидный вектор именуют pcDNA3.4_xi5B9.
Пример 7. Экспрессия МКАт xi5B9 в клетках СНО.
Клетки ExpiCHO-S, являющиеся частью набора ExpiCHO Expression System Kit (Gibco, Thermo Fisher), выращивают в среде ExpiCHO Expression Medium в одноразовых колбах Эрленмейера объемом 500 мл с вентилируемой крышкой (Corning) на шейкере Minitron (Infors НТ), режим 125 мин -1 в атмосфере 8% СО2 при 37°С. По достижении плотности 6×106 кл/мл и количества жизнеспособных клеток не менее 95% (оценивают по окрашиванию трипановым синим), проводят трансфекцию клеток плазмидой pcDNA3.4_xi5B9, полученной по примеру 6. В день трансфекции частично или полностью производят замену среды на аналогичную новую. Трансфекцию проводят с использованием реактивов и методики набора ExpiCHO Expression System Kit, а затем клетки выращивают согласно протоколу Мах Titer Protocols. Кратко: в каждую колбу объемом 500 мл, в которой находится 100 мл культуры ExpiCHO, добавляют смесь из 80 мкл плазмидной ДНК pcDNA3.4_xi5B9 с концентрацией 1 мкг/мл, 7,7 мл среды OptiPRO SFM и 320 мкл ExpiFectamine СНО Reagent. Культуру растят на шейкере при 32°С в атмосфере 5% СО2. Через 24 часа после трансфекции в колбу добавляют 600 мкл энхансера ExpiCHO Enhancer и 16 мл подкормки ExpiCHO Feed. На 5 сутки после трансфекции добавляют еще 16 мл ExpiCHO Feed. На 14 день культуру центрифугируют 1000 g в течение 10 минут. Супернатант, содержащий антитела xi5B9, дополнительно фильтруют через мембрану с диаметром пор 0,2 мкм, затем концентрируют десятикратно в ячейке Amicon Stirred Cell (Merck Millipore). При необходимости замораживают -20°С или сразу переходят к очистке по примеру 8.
Пример 8. Очистка и валидация МКАт xi5B9
Выделение МКАт xi5B9 из концентрированной культуральной жидкости проводят методом аффинной хроматографии на колонке с сорбентом Protein G сефарозой с использованием хроматографической системы Pure. Колонку с полезной емкостью 3 мл, заполненную Protein G сефарозой (Cytiva™), уравновешивают буфером нанесения (50 мМ Tris, 100 мМ NaCl, 0,02% NaN3, рН 8,0). Производят нанесение образца со скоростью 2 мл/мин на льду. Далее колонку отмывают от не связавшихся компонентов буфером нанесения, и проводят элюцию иммуноглобулинов буфером состава: 100 мМ глицина, 100 мМ NaCl, рН 2.4. Уровень рН элюата доводят до 7,5 - 8,0 добавлением 1 М раствора Tris основания. Впоследствии элюат иммуноглобулиновой фракции вносят в коммерчески доступную колонку HiTrap Desalting (GE Healthcare) емкостью 5 мл, уравновешенную фосфатно-солевым буфером ФСБ на скорости потока 5 мл/мин.
Иммуноглобулиновую фракцию собирают через некоторое время, идентифицируя ее по изменению А280. Для измерения концентрации МКАт проводят спектрофотометрию при А280, затем для расчета концентрации [мг/мл] полученное значение разделяют на коэффициент массовой экстинкции для IgG, равный 1,37. Чистоту полученной иммуноглобулиновой фракции оценивают методом SDS-электрофореза по Лэммли в 10% ПААГ в редуцирующих и нередуцирующих условиях. (Фиг. 1 дорожка 4, 5). Как правило, выход составляет 20-25 мг белка с 1 литра культуры.
Наличие человеческого константного региона в МКАт xi5B9 подтверждают методом иммуноблота. Антитело подвергают электрофорезу в 10% ПААГ в нередуцирующих условиях, после чего осуществляют горизонтальный перенос белка из геля на нитроцеллюлозную мембрану Hybond-C Extra. После завершения переноса мембрану блокируют погружением в обезжиренное молоко с массовой долей жира не более 0,5% и инкубируют в течение 1 ч на орбитальном термостатируемом шейкере при 300 об./мин и температуре 37°С. Далее, после трехкратной промывки буфером ФСБ-Тв, мембрану разрезают и инкубируют в ФСБ с разведенными (согласно паспортным рекомендациям для вестерн-блоттинга) HRP-конъюгированными поликлональными антителами козы: одну дорожку с антителами против κ цепи человеческого IgG (Sigma-Aldrich А7164), другую с антителами против Fc фрагмента человеческого IgG (Sigma-Aldrich АО 170). Спустя 1 час инкубации при 37°С 300 об/мин мембраны промывают 6 раз в ФСБ-Тв. Визуализацию реакции проводят раствором субстратной смеси на основе диаминобензидина. В результате проведения данного анализа, появление специфического окрашивания, отраженного на Фиг. 10, рекомбинантное МКАт xi5B9 в действительности является химерным и содержит человеческую константную часть.
Оценку иммунологической специфичности очищенного МКАт xi5B9 проводят методом иммуноблота с рРТА и РТ Ricinus communis. Белки подвергают электрофорезу в 12% ПААГ, после чего осуществляют горизонтальный перенос белков из геля на нитроцеллюлозную мембрану Hybond-C Extra. После завершения переноса мембрану блокируют погружением в обезжиренное молоко с массовой долей жира не более 0,5% и инкубируют в течение 1 ч на орбитальном термостатируемом шейкере при 300 об./мин и температуре 37°С. Далее, после трехкратной промывки буфером ФСБ-Тв, мембрану инкубируют с МКАт xi5B9, разведенным до концентрации 0,1-1 мкг/мл в ФСБ. После трехкратной промывки буфером ФСБ-Тв мембрану инкубируют с антителами к Fc фрагменту человеческого IgG (Sigma-Aldrich А0170) в разведении 1:10000 в буфере ФСБ-Тв в течение 30 минут. Затем мембрану промывают 6 раз в ФСБ-Тв. Визуализацию реакции проводят раствором субстратной смеси на основе диаминобензидина (0,05% диаминобензидина (Sigma, США), 0,015% Н2О2 в ФСБ рН 7,4). Реакцию останавливают после отчетливо различимого окрашивания образцов путем погружения мембраны в дистиллированную воду. Как показывает проведенный анализ, результаты которого отражены на Фиг. 4 в дорожках, МКАт xi5B9 специфически взаимодействует с цепью А РТ и рРТА.
Для оценки количественного взаимодействия очищенного МКАт xi5B9 с белком рРТА, в лунки 96-луночного планшета с иммобилизированым антигеном, который подготовлен по методике из Примера 1, вносят методом двукратных разведений антитело в концентрациях 10000, 5000, 2500, 1250, 625, 312.5, 156.3, 78.1, 39.1, 19.5, 9.8, 4.9, 2.5, 1.2 нг/лунку. Инкубируют на термостатируемом шейкере при температуре 37°С в течение 1 часа 300 об./мин. После этого лунки планшета трижды отмывают ФСБ-Тв и добавляют в лунки раствор антител к Fc фрагменту человеческого IgG (Sigma-Aldrich А0170), конъюгированных с пероксидазой хрена в рабочем разведении 1:10000. Планшет инкубируют при температуре 37°С в течение 1 часа на шейкере, затем 6 раз отмывают буфером ФСБ-Тв. После этого в лунки вносят по 100 мкл субстрат-индикаторного раствора на основе ортофенилендиамина. Положительную реакцию оценивают по появлению желто-коричневого окрашивания раствора. Реакцию останавливают добавлением в лунки по 50 мкл 4N серной кислоты. Детекцию проводят на планшетном спектрофотометре (Bio-Rad xMark, США) при длине волны 492 нм. По результатам данного анализа минимально детектируемая концентрация МКАт xi5B9 составила 25 нг/мл, кривая А = f(C) соответствует типовой кривой для высокоаффинных антител в непрямом тведофазном ИФА.
Пример 9. Определение равновесной константы диссоциации МКАт xi5B9
Определение равновесной константы диссоциации МКАт xi5B9 с целевым антигеном рРТА определяют методом поверхностного плазмонного резонанса (PPR) на приборе BIAcore 2000 (Biacore, Швеция) с использованием сенсорного чипа CM5. Предварительно на чип СМ5 иммобилизуют анти-гистидиновые антитела при помощи набора His Capture Kit type 2 (Cytiva) в соответствии с инструкцией производителя. В качестве лиганда используют рРТА, несущий His- тэг в своем составе. Лиганд наносят на чип СМ5 в концентрации 10 мкг/мл и скоростью 30 мкл/мин в течение 3 минут. Против лиганда наносят с той же скоростью в течение 2 минут раствор антител (МКАт 5B9 и xi5B9), титруют с двукратным шагом в диапазоне концентраций от 125 нМ до 6.25 нМ и. Все разведения белков проводят в буфере HBS-EP (10 мМ Hepes, 150 мМ NaCl, 3 мМ EDTA и 0,005% P20 (поверхностно-активного вещества), pH 7.4) при температуре 25°. Регенерацию чипа осуществляют 10 мМ глицином pH 1,7 в течение 30 с при скорости потока 50 мкл/мин. Полученные сенсограммы нормализуют путем вычитания базовых значений RU из эталонной проточной кюветы (без захвата МКАт) и анализируют с использованием модели связывания Ленгмюра 1:1 с помощью программного обеспечения Biacore T200 Evaluation Software. Измеренные равновесные константы диссоциации (KD) для МКАт KD xi5B9 = 2.63×10-8 М (Фиг. 8). с помощью программного обеспечения Biacore T200 Evaluation Software.
Пример 10. Определение токсиннейтрализующей активности МКАт in vitro.
Клеточная линия Vero была получена из Государственной коллекции патогенных микроорганизмов и клеточных культур (ГКПМ-Оболенск). В качестве среды для культивирования используют среду Игла в модификации Дульбекко (DMEM) с добавлением 2мМ L-глутамина, раствора антибиотика-антимикотика однократной концентрации и 10% инактивированной фетальной бычьей сыворотки (Gibco, Thermo Fisher, США). Клетки выращивают в 75 см2 культуральных флаконах (Corning, США) при атмосфере 5% CO2 при 37°C, не превышая 70%-80% конфлюэнции. За день до эксперимента предварительно выращенные адгезивные клетки собирают при помощи 0,05% раствора трипсина-ЭДТА (Gibco, Thermo Fisher, США) и готовят суспензию в полной ростовой среде. Для определения концентрации клеток используют автоматический счетчик клеток TC20™ (Bio-Rad, США). Жизнеспособность клеток определяют с помощью 0,4% раствора трипанового синего (Gibco, Thermo Fisher, США). В микрослайд счетчика клеток вносят смесь клеточной суспензии и раствора трипанового синего в соотношении 1:1. Далее подготавливают 96-луночные планшеты (Corning, США), заполняют лунки клеточной суспензией по 100 мкл. Концентрацию суспензии клеток рассчитывают таким образом, чтобы в каждой лунке оказалось 1×104 жизнеспособных клеток в объеме среды 100 мкл. Планшеты оставляли в CO2-инкубаторе (Panasonic, Япония) на 24 часа для прикрепления клеток ко дну лунок.
Исследования цитотоксичности РТ Ricinus communis и нейтрализующего действия полученных МКАт 5В9 и xi5B9. Для определения IC50 РТ, в культуральные планшеты с клеточной линеей Vero, в концентрации 1×104 клеток/лунку, добавляют РТ в различных концентрациях: 400, 200, 100, 50, 25, 12,5, 6,25, 3,75 пг/мл. В качестве контроля используют лунки без добавления РТ (достигая 100% жизнеспособных клеток, контроль сравнения К1) и лунки с добавлением мертиолята натрия (Oskar Tropitzsch, Германия) до конечной концентрации 0,024% (достигая 100% нежизнеспособных клеток, контроль сравнения К2). Все исследуемые точки дублировали в 6 повторностях. Подготовленные культуральные планшеты оставляют на 72 часа в CO2-инкубаторе. Раствор МТТ подготавливают разбавлением готовой навески из набора Vybrant МТТ Cell Proliferation Assay Kit (Thermo Fisher) в 1 мл ФСБ. Через 72 часа инкубации в лунки добавляют по 10 мкл MTT и инкубируют еще 4 часа. Затем аккуратно убирают культуральную среду с MTT и лизируем клеточный слой добавлением 200 мкл диметилсульфокси да (ДМСО, Sigma-Aldrich, США). Для полного лизиса клеток и равномерного растворения кристаллов формазана планшеты помещают на платформу шейкера TS-100C (BioSan, Латвия) и качали 10 минут 500 об/мин при комнатной температуре. Оптическую плотность клеточного лизата измеряют при длине волны 540 нм (ОП540) на планшетном спектрофотометре xMark (Bio-Rad, США). Процент выживших клеток после добавления РТ определяют по формуле:

Для определения способности МКАт нейтрализовать РТ Ricinus communis берем в различных молярных соотношениях по отношению к IC50 РТ (составила 25 пкг\мл). Соотношение РТ:МКАт - 1:15, 1:12, 1:9, 1:6, 1:3, 1:2, 1:1, 1:0.5. Данную смесь инкубируют при 37° 1 час, затем добавляли в подготовленные культуральные планшеты, с клеточной линией Vero, в концентрации 1×104 клеток/лунку. В качестве контролей 100% жизнеспособных клеток и 100% нежизнеспособных клеток используют те же контроли, что и при определении IC50 РТ (К1 и К2). Дальнейшая постановка микротеста МТТ не отличалась от постановки по определению IC50 РТ в отношении клеточной линии Vero.
По кривым зависимости жизнеспособности клеток от концентрации МКАт (Фиг. 12) делают вывод о том, что и МКАт 5B9, и МКАт xi5B9 в МТТ-тесте нейтрализуют действие РТ Ricinus communis в соотношениях 1:6 и выше, однако при избытке антигена нейтрализующая способность химерного xi5B9 немного уступает по сравнению с таковой у мышиного 5B9.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Рогозин проект
xi5B9 230523.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-05-23">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022128601/10(062729)</ApplicationNumberText>
<FilingDate>2022-11-03</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Рогозин Метхун
Мадибронович</ApplicantName>
<ApplicantNameLatin>Rogozin Methun
Madibronovich</ApplicantNameLatin>
<InventionTitle languageCode="ru">Штамм гибридных культивируемых
клеток животных Mus musculus 5B9-продуцент мышиного моноклонального
антитела 5B9, антитело моноклональное мышиное 5В9 и антитело
рекомбинантное химерное (мышь-человек) xi5В9, нейтрализующие рицин
Ricinus communis</InventionTitle>
<SequenceTotalQuantity>30</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>266</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SIGNAL</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..266</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MESQTQVFVYMLLWLSGVDGDIVMTQSQKFMSTSVGDRVSVTCKASQIV
GTSVAWYQQRPGQPPKPLIYSASFRYSGVPDRFTGSGSGTDFTLTISNVQSEDLAEYFCHHYYSYPFTFG
SGTKLEINRADAAPTVSIFPPSSEQLTSGGASVVCFLNNFYPKDINVKWKIDGSERQNGVLNSWTDQDSK
DSTCSMSSTLTLTKDEYERHNSYTCEATHKTSTSPVVKSFNRNEGYRARIHWPSFYNVVTGKTLALPNLI
ALQHIPL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>273</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SIGNAL</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..273</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MDMITPSLHACRSTSIFIFFFLNIWTGSSPQTLNTLTLTMGWSWIFLFL
LSETAGVLSEVQLQQSGPELVKPGASVKISCKPSGYSFSDYFIHWVKQSRVKSLEWIGRISPYNAATNYN
RNFKDKAILTVDRSSSTAYMELHSLTSEDSAVYYCARLRRSDGDENWGQGTSVTVSSAKTTPPSVYPLAP
GSAAQTNSMVTLGCLVKGYFPEPVTVTWNSGSLSSGVHTFPAVLQSDLYTLSSSVTVPSSTWPSETVTCN
VAHPASSTKVDKKI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>236</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SIGNAL</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..236</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MDMRVPAQLLGLLLLWLSGARCDIVMTQSQKFMSTSVGDRVSVTCKASQ
IVGTSVAWYQQRPGQPPKPLIYSASFRYSGVPDRFTGSGSGTDFTLTISNVQSEDLAEYFCHHYYSYPFT
FGSGTKLEINRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTEQD
SKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>277</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SIGNAL</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..277</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEFGLSWVFLVALFRGVQCEVQLQQSGPELVKPGASVKISCKPSGYSFS
DYFIHWVKQSRVKSLEWIGRISPYNAATNYNRNFKDKAILTVDRSSSTAYMELHSLTSEDSAVYYCARLR
RSDGDENWGQGTSVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHT
FPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPS
VFLFPPQAQRHTDDLQDP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>299</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SIGNAL</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..299</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Ricinus communis</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MKYLLPTAAAGLLLLAAQPAMAMDIFPKQYPIINFTTAGATVQSYTNFI
RAVRGRLTTGADVRHEIPVLPNRVGLPINQRFILVELSNHAELSVTLALDVTNAYVVGYRAGNSAYFFHP
DNQEDAEAITHLFTDVQNRYTFAFGGNYDRLEQLAGNLRENIELGNGPLEEAISALYYYSTGGTQLPTLA
RSFIICIQMISEAARFQYIEGEMRTRIRYNRRSAPDPSVITLENSWGRLSTAIQESNQGAFASPIQLQRR
NGSKFSVYDVSILIPIIALMVYRCAPPPSSQFLEHHHHHH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>110</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..110</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DIVMTQSQKFMSTSVGDRVSVTCKASQIVGTSVAWYQQRPGQPPKPLIY
SASFRYSGVPDRFTGSGSGTDFTLTISNVQSEDLAEYFCHHYYSYPFTFGSGTKLEINRAD</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>118</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..118</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EVQLQQSGPELVKPGASVKISCKPSGYSFSDYFIHWVKQSRVKSLEWIG
RISPYNAATNYNRNFKDKAILTVDRSSSTAYMELHSLTSEDSAVYYCARLRRSDGDENWGQGTSVTVSS<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q8">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CDR L-1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KASQIVGTSVA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q10">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CDR L-2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SASFRYS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>HHYYSYPFT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CDR H-1</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DYFIH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CDR H-2</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RISPYNAATNYNRNFKD</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CDR H-3</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mus musculus</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LRRSDGDEN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttttttttttttttttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatgcaaggcttacaaccaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q23">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcattcctgttgaagctcttgacaatggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactcgagtcgacatcgattttttttttttttttt</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcaattttcttgtccaccttggtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtaaaacgacggccagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacagctatgaccatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>91</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..91</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..91</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataccatggacatgagagtgccggcccagctgctgggactcctgctcct
gtggctgagcggcgcaaggtgcgacattgtgatgacccagtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatgaggaggcatgcatctgtttatttccaactttgtccc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>82</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..82</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..82</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ataccatggagtttgggctgagctgggttttcctcgttgctctttttag
gggcgtccagtgtgaggtccagctgcaacagtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>38</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tatgaggaggcatgcatgctgaggagacggtgactgag</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggcgtgtacggtgggag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>70</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..70</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..70</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagaggcagcctgctgacctgcggcgacgtggaggagaaccccggcccc
atggacatgagagtgccggcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q45">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctagcactcgcccctgttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q47">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accatggactggacgtggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>69</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..69</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q49">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..69</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgcaggtcagcaggctgcctctgccctcgccgctgcctctcttggctc
tcttgccagggctcagggac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>728</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..728</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q51">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value> </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..728</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEFGLSWVFLVALFRGVQCEVQLQQSGPELVKPGASVKISCKPSGYSFS
DYFIHWVKQSRVKSLEWIGRISPYNAATNYNRNFKDKAILTVDRSSSTAYMELHSLTSEDSAVYYCARLR
RSDGDENWGQGTSVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHT
FPAVLQSSGLYSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPS
VFLFPPKPKDTLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVL
HQDWLNGKEYKCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAV
EWESNGQPENNYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGKRA
KRGSGEGRGSLLTCGDVEENPGPMDMRVPAQLLGLLLLWLSGARCDIVMTQSQKFMSTSVGDRVSVTCKA
SQIVGTSVAWYQQRPGQPPKPLIYSASFRYSGVPDRFTGSGSGTDFTLTISNVQSEDLAEYFCHHYYSYP
FTFGSGTKLEINRTVAAPSVFIFPPSDEQLKSGTASVVCLLNNFYPREAKVQWKVDNALQSGNSQESVTE
QDSKDSTYSLSSTLTLSKADYEKHKVYACEVTHQGLSSPVTKSFNRGEC</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА И ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2821577C2 |
| АНТИТЕЛА ПРОТИВ ПРОПЕРДИНА И ИХ ПОЛУЧЕНИЕ | 2021 |
|
RU2830308C1 |
| АНТИТЕЛА ПРОТИВ IDE И ИХ ПРИМЕНЕНИЕ | 2020 |
|
RU2839026C1 |
| ХИМЕРНЫЕ ЦИТОКИНОВЫЕ РЕЦЕПТОРЫ, НЕСУЩИЕ ЭКТОДОМЕН PD-1 | 2020 |
|
RU2828214C2 |
| Моноклональные антитела, специфичные к белку рустицианин, и использование антител для диагностики и лечения злокачественных новообразований | 2024 |
|
RU2823344C1 |
| МОНОКЛОНАЛЬНЫЕ АНТИТЕЛА ПРОТИВ РЕЦЕПТОР-СВЯЗЫВАЮЩЕГО ДОМЕНА SPIKE-БЕЛКА ВИРУСА SARS-COV-2 И ИХ АНТИГЕНСВЯЗЫВАЮЩИЕ ФРАГМЕНТЫ, КОДИРУЮЩИЕ ИХ НУКЛЕИНОВЫЕ КИСЛОТЫ, А ТАКЖЕ СПОСОБЫ ПРИМЕНЕНИЯ | 2021 |
|
RU2800649C2 |
| ПЕПТИД, ПРИМЕНЯЕМЫЙ ДЛЯ ИММУНОТЕРАПЕВТИЧЕСКИХ СРЕДСТВ | 2021 |
|
RU2836172C1 |
| Моноклональное антитело iC2 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2023 |
|
RU2817697C1 |
| ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2018 |
|
RU2841528C2 |
| Плазмида для экспрессии рекомбинантного хорионического гонадотропина человека (ХГЧ), плазмида для экспрессии рекомбинантных альфа- и бета-субъединиц ХГЧ, моноклональная линия клеток млекопитающих - продуцент ХГЧ, способ получения рекомбинантного ХГЧ | 2024 |
|
RU2834784C1 |
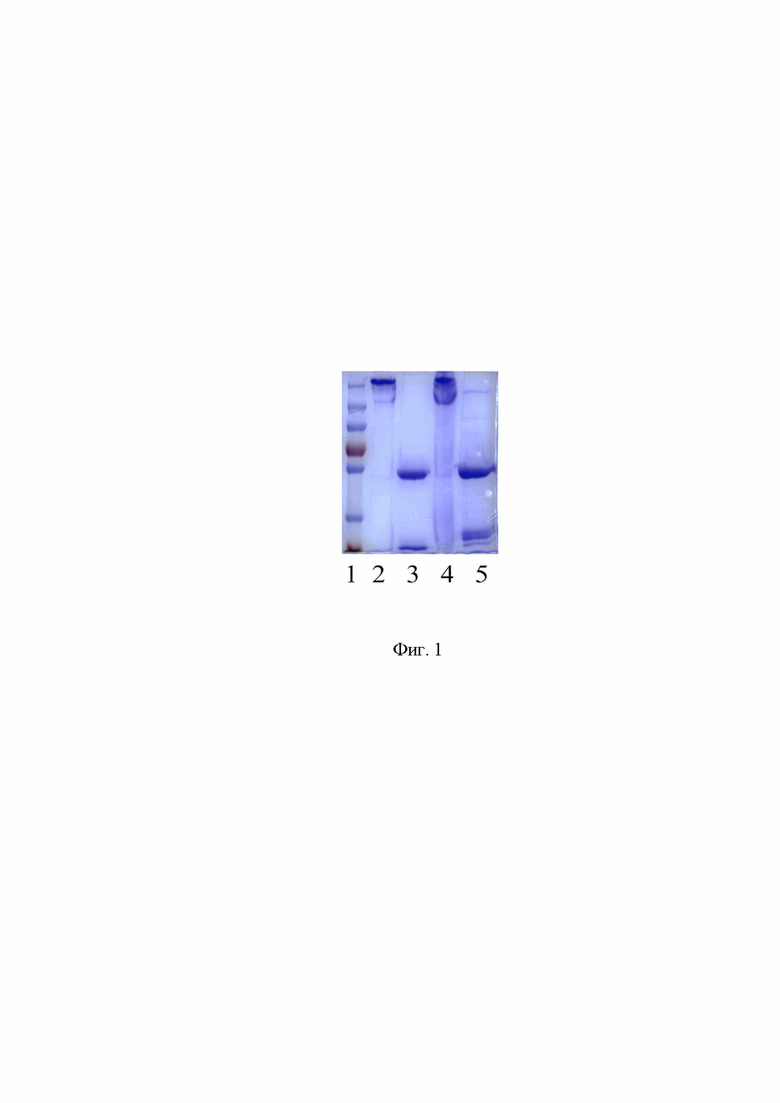
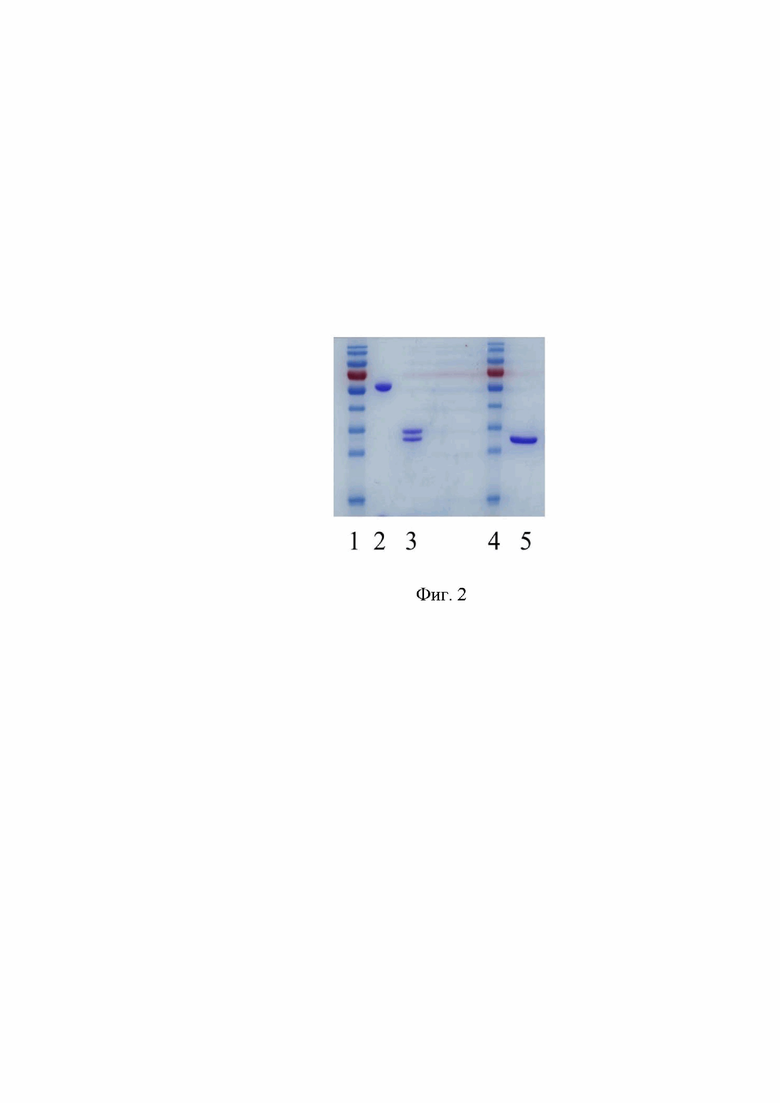



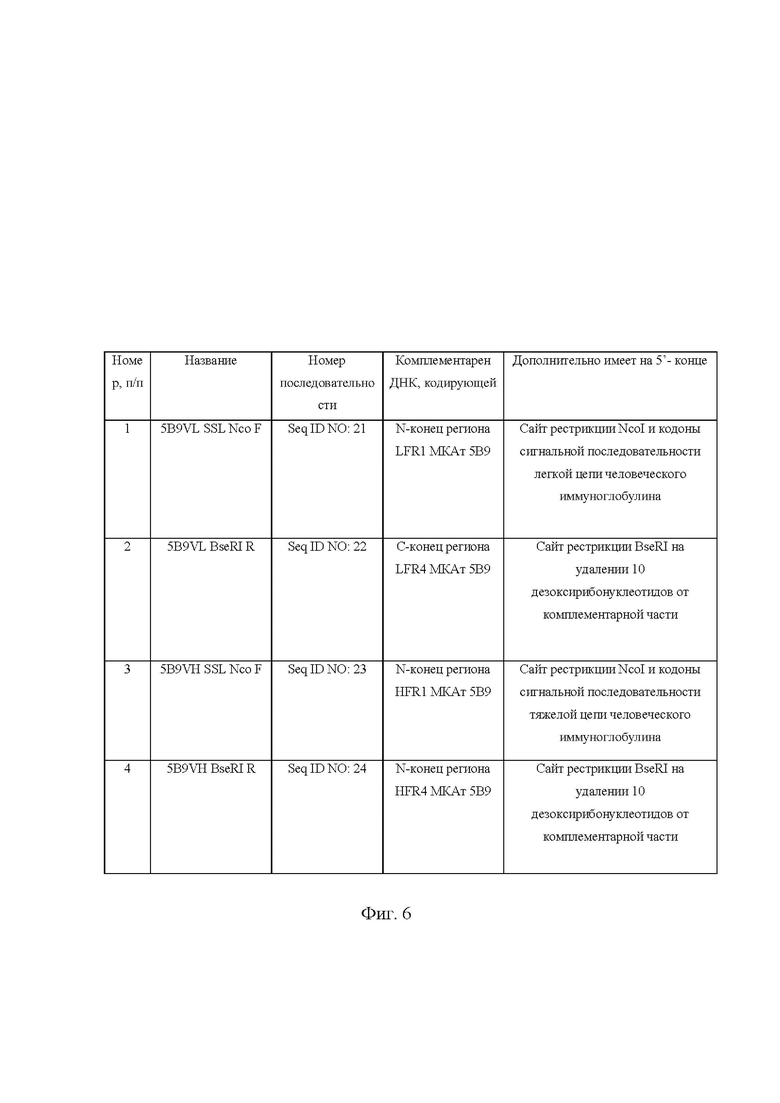
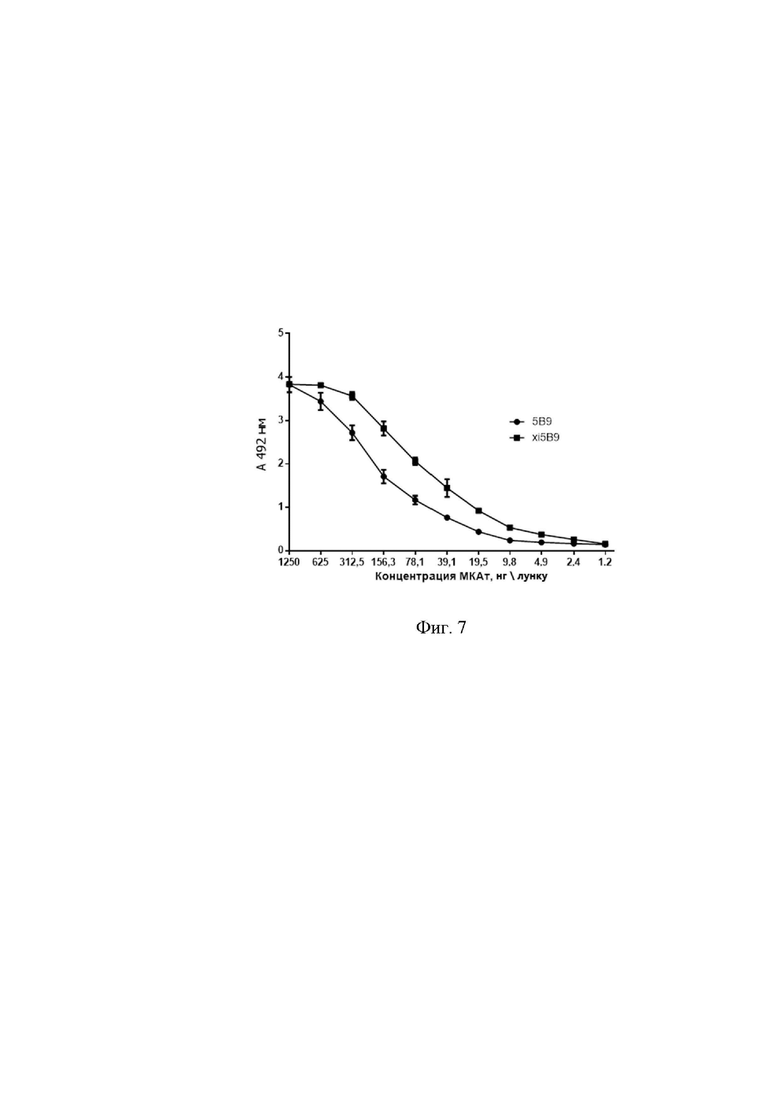
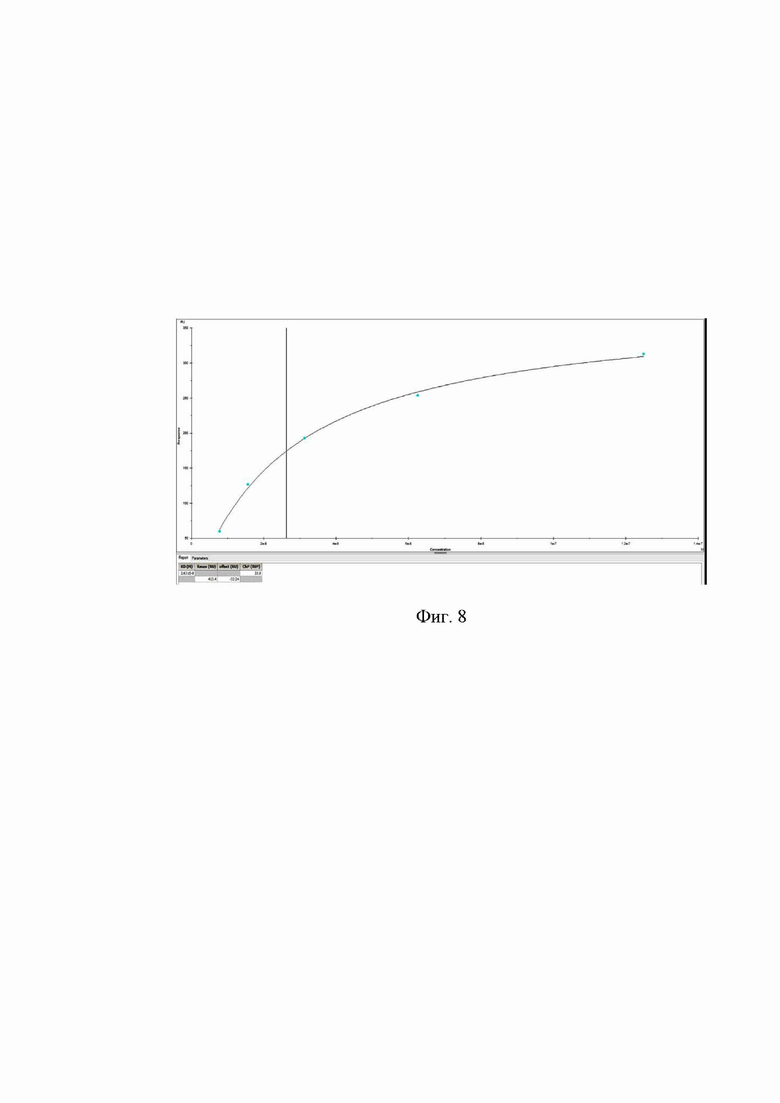

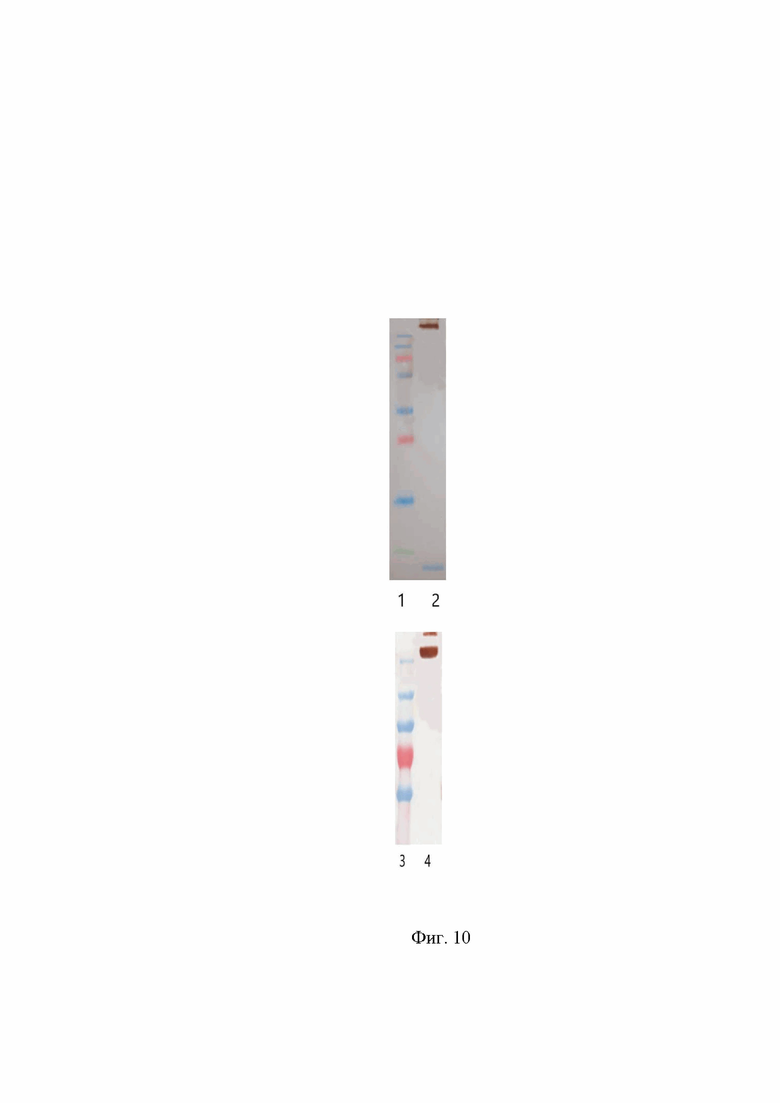

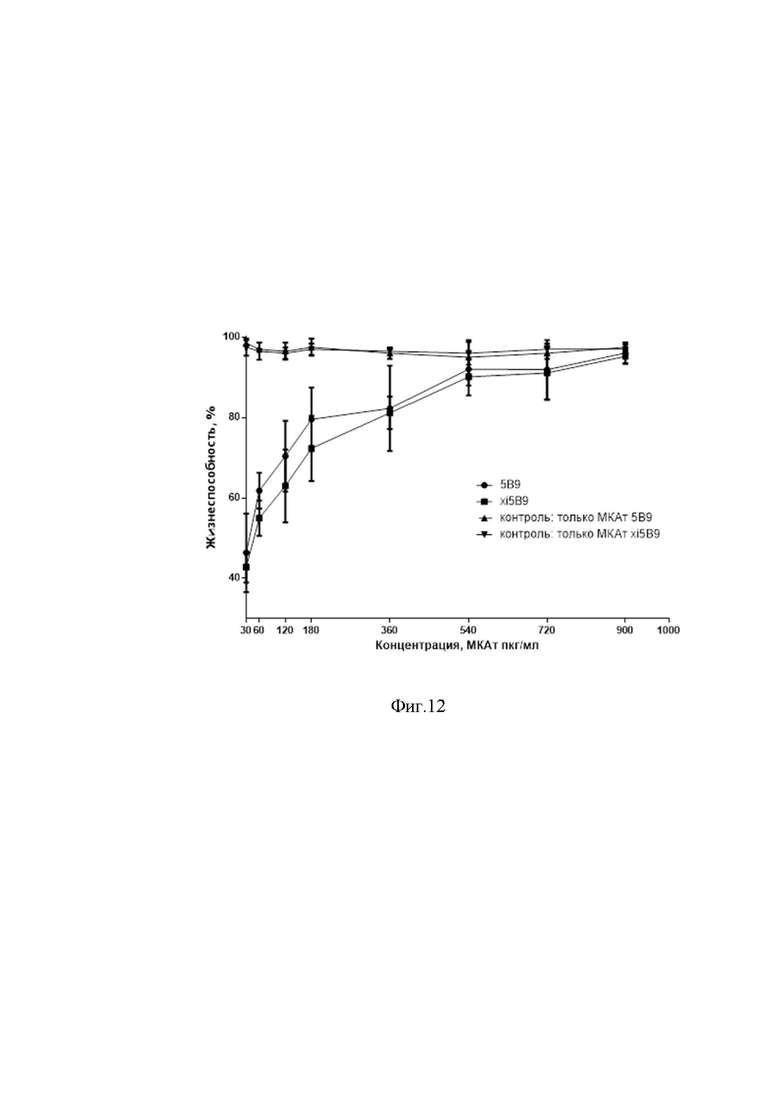
Изобретение относится к биотехнологии. Предложено мышиное моноклональное антитело 5B9, нейтрализующее рицин Ricinus communis, специфически связывающееся с рициновым токсином Ricinus communis в области А цепи и представленное двумя идентичными легкими и двумя идентичными цепями тяжелыми, образующими молекулу IgG1, с аминокислотными последовательностями легкой цепи SEQ ID NO: 1 и Fd фрагмента тяжелой цепи SEQ ID NO: 2. Также предложен штамм гибридных культивируемых клеток животных Mus musculus, депонированный в государственной коллекции патогенных микроорганизмов и клеточных культур «ГКПМ-Оболенск» № Н-97, продуцирующий вышеуказанное антитело 5B9. Также предложено химерное рекомбинантное моноклональное антитело мышь-человек xi5B9, полученное путем замены константных доменов легкой и тяжелой цепи вышеуказанного антитела 5B9. Антитело xi5B9 нейтрализует рицин Ricinus communis, специфически связывается с рициновым токсином Ricinus communis в области А цепи, экспрессируется в эукариотических клетках линии ExpiCHO-S, характеризуется константой равновесной диссоциации 2,63×10-8. Антитело xi5B9 представлено двумя идентичными легкими цепями и двумя идентичными тяжелыми цепями, несущими в своем составе константные домены иммуноглобулина G1 человека, образующими молекулу IgG1 с аминокислотными последовательностями легкой цепи SEQ ID NO: 3 и тяжелой цепи SEQ ID NO: 4. Изобретение обеспечивает получение антител с высокоаффинным и высокоспецифичным связыванием с цепью А рицинового токсина Ricinus communis, обладающих нейтрализующим действием в отношении рицинового токсина Ricinus communis. 3 н.п. ф-лы, 12 ил., 10 пр.
1. Мышиное моноклональное антитело 5B9, представленное двумя идентичными легкими и двумя идентичными цепями тяжелыми, образующими молекулу IgG1, с аминокислотными последовательностями легкой цепи SEQ ID NO: 1 и Fd фрагмента тяжелой цепи SEQ ID NO: 2, нейтрализующее рицин Ricinus communis, специфически связывающееся в области А цепи.
2. Химерное рекомбинантное моноклональное антитело мышь-человек xi5B9, полученное путем замены константных доменов легкой и тяжелой цепи антитела 5B9 по п. 1, представленное двумя идентичными легкими цепями и двумя идентичными тяжелыми цепями, несущими в своем составе константные домены иммуноглобулина G1 человека, образующими молекулу IgG1 с аминокислотными последовательностями легкой цепи SEQ ID NO: 3 и тяжелой цепи SEQ ID NO: 4, нейтрализующее рицин Ricinus communis, специфически связывающееся в области А цепи, экспрессируемое в эукариотических клетках линии ExpiCHO-S, характеризующееся константой равновесной диссоциации 2,63×10-8 М.
3. Штамм-продуцент мышиного моноклонального антитела 5B9 по п.1, представлен гибридными культивируемыми клетками животных Mus musculus, депонированный в государственной коллекции патогенных микроорганизмов и клеточных культур «ГКПМ-Оболенск» № Н-97.
| US 5626844 A1, 06.05.1997 | |||
| US 8344109 B2, 01.01.2013 | |||
| US 20140050722 A1, 20.02.2014 | |||
| PRIGENT J | |||
| ET AL | |||
| Neutralising antibodies against ricin toxin | |||
| PLoS One | |||
| Способ приготовления лака | 1924 |
|
SU2011A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Найдено онлайн: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3102095/ Дата обращения 27.04.2023 | |||
| ПОПОВА Е | |||
| Н | |||
| Взаимодействие рицина с | |||
Авторы
Даты
2023-08-28—Публикация
2022-11-03—Подача