Область техники, к которой относится изобретение
Настоящее изобретение относится к моноклональным антителам и их антигенсвязывающим фрагментам, которые связываются с рецептор-связывающим доменом (RBD) Spike-белка вируса SARS-CoV-2.
Уровень техники
За два года пандемии, вызванной бетакоронавирусом SARS-CoV-2, была зарегистрирована гибель порядка 5 млн человек. Использование ряда средств профилактики и терапии коронавирусной инфекции, вызванной SARS-CoV-2 (также называемой в данном документе ковид-19), позволяет снизить вероятность заражения, госпитализации и летального исхода. К таким средствам относятся вакцинопрофилактика, использование вирус-специфических малых молекул, препаратов плазмы реконвалесцентов, рекомбинантных Spike-специфичных белков (в том числе антител) и их производных. Несмотря на повсеместное проведение кампаний по иммунизации населения и идентификации нейтрализующих моноклональных антител против Spike-белка вируса SARS-CoV-2, существует значительная потребность в получении новых вариантов таких антител для расширения арсенала диагностических, профилактических и терапевтических средств особенно с учетом появления новых вариантов вируса. Кроме того, существуют группы населения, которым вакцинация либо не показана по медицинским соображениям, либо у которых не может быть выработан протективный иммунный ответ. Таким образом, проведение пассивной иммунизации – введение препаратов моноклональных антител в качестве пред- или пост-экспозиционной профилактики или терапии позволяет защитить таких людей, обеспечить менее длительное и тяжелое течение болезни, в том числе предотвратить развитие пост-ковидных осложнений, и снизить вероятность дальнейшей передачи вируса.
Известно, что из трех поверхностных белков SARS-CoV-2 лишь один, Spike, вызывает образование вируснейтрализующих антител. Также было показано, что подавляющее число полученных к настоящему моменту Spike-специфических моноклональных антител, обладающих способностью нейтрализовать вирус, направлены против RBD-домена Spike, ответственного за связывание с основным входным рецептором вируса, белком ACE2.
Идентификация SARS-CoV-2-специфических нейтрализующих моноклональных антител в основном осуществляется одним из нескольких способов, таких как секвенирование генов иммуноглобулинов из единичных В-клеток людей, перенесших ковид-19, или иммунизированных животных (в. т.ч. гуманизированных) (Brouwer, P. J. M. et al. Potent neutralizing antibodies from COVID-19 patients define multiple targets of vulnerability. Science 369, 643–650 (2020); Wu, Y. et al. A noncompeting pair of human neutralizing antibodies block COVID-19 virus binding to its receptor ACE2. Science 368, 1274–1278 (2020); Jones, B. E. et al. The neutralizing antibody, LY-CoV555, protects against SARS-CoV-2 infection in nonhuman primates. Sci. Transl. Med. 13, eabf1906 (2021); Hansen, J. et al. Studies in humanized mice and convalescent humans yield a SARS-CoV-2 antibody cocktail. Science 369, 1010–1014 (2020); Wang, C. et al. A human monoclonal antibody blocking SARS-CoV-2 infection. Nat. Commun. 11, 2251 (2020); Seydoux, E. et al. Analysis of a SARS-CoV-2-infected individual reveals development of potent neutralizing antibodies with limited somatic mutation. Immunity 53, 98–105.e5 (2020); Zost, S. J. et al. Rapid isolation and profiling of a diverse panel of human monoclonal antibodies targeting the SARS-CoV-2 spike protein. Nat. Med. 26, 1422–1427 (2020); Rogers, T. F. et al. Isolation of potent SARS-CoV-2 neutralizing antibodies and protection from disease in a small animal model. Science 369, 956–963 (2020); Shi, R. et al. A human neutralizing antibody targets the receptor-binding site of SARS-CoV-2. Nature 584, 120–124 (2020); Ju, B. et al. Human neutralizing antibodies elicited by SARS-CoV-2 infection. Nature 584, 115–119 (2020); Robbiani, D. F. et al. Convergent antibody responses to SARS-CoV-2 in convalescent individuals. Nature 584, 437–442 (2020); Liu, L. et al. Potent neutralizing antibodies against multiple epitopes on SARS-CoV-2 spike. Nature 584, 450–456 (2020); Chi, X. et al. A neutralizing human antibody binds to the N-terminal domain of the Spike protein of SARS-CoV-2. Science 369, 650–655 (2020); Wan, J. et al. Human-IgG-neutralizing monoclonal antibodies block the SARS-CoV-2 infection. Cell Rep. 32, 107918 (2020); Kreer, C. et al. Longitudinal isolation of potent near-germline SARS-CoV-2-neutralizing antibodies from COVID-19 patients. Cell 182, 843–854.e12 (2020); Kreye, J. et al. A therapeutic non-self-reactive SARS-CoV-2 antibody protects from lung pathology in a COVID-19 hamster model. Cell 183, 1058–1069.e19 (2020); McCallum, M. et al. N-terminal domain antigenic mapping reveals a site of vulnerability for SARS-CoV-2. Cell 184, 2332–2347.e16 (2021); Chen, X. et al. Human monoclonal antibodies block the binding of SARS-CoV-2 spike protein to angiotensin converting enzyme 2 receptor. Cell. Mol. Immunol. 17, 647–649 (2020); Cao, Y. et al. Potent neutralizing antibodies against SARS-CoV-2 identified by high-throughput single-cell sequencing of convalescent patients’ B cells. Cell 182, 73–84.e16 (2020); Andreano, E. et al. Extremely potent human monoclonal antibodies from COVID-19 convalescent patients. Cell. 184(7), 1821–1835.e16 (2021)), людей, перенесших инфекцию SARS-CoV (Wec, A. Z. et al. Broad sarbecovirus neutralizing antibodies define a key site of vulnerability on the SARS-CoV-2 spike protein. bioRxiv https://doi.org/10.1101/2020.05.15.096511 (2020); Tian, X. et al. Potent binding of 2019 novel coronavirus spike protein by a SARS coronavirus-specific human monoclonal antibody. Emerg. Microbes Infect. 9, 382–385 (2020); Pinto, D. et al. Cross-neutralization of SARS-CoV-2 by a human monoclonal SARS-CoV antibody. Nature 583, 290–295 (2020)), скрининг библиотек антител, scFv-, Fab- или VH- фрагментов (Li, W. et al. Rapid identification of a human antibody with high prophylactic and therapeutic efficacy in three animal models of SARS-CoV-2 infection. Proc. Natl. Acad. Sci. USA 117, 29832–29838 (2020); Parray, H. A. et al. Identification of an anti-SARS-CoV-2 receptor-binding domain-directed human monoclonal antibody from a naïve semisynthetic library. J. Biol. Chem. 295, 12814–12821 (2020); Wang, B. et al. Bivalent binding of a fully human IgG to the SARS-CoV-2 spike proteins reveals mechanisms of potent neutralization. bioRxiv https://doi.org/10.1101/2020.07.14.203414 (2020); Wu, Y. et al. Identification of Human Single-Domain Antibodies against SARS-CoV-2. Cell Host Microbe 27, 891–898.e5 (2020); Ku, Z. et al. Molecular determinants and mechanism for antibody cocktail preventing SARS-CoV-2 escape. Nat. Commun. 12, 469 (2021); Fagre, A. C. et al. A potent SARS-CoV-2 neutralizing human monoclonal antibody that reduces viral burden and disease severity in Syrian hamsters. Front. Immunol. 11, 614256 (2020); Noy-Porat, T. et al. A panel of human neutralizing mAbs targeting SARS-CoV-2 spike at multiple epitopes. Nat. Commun. 11, 4303 (2020); Yao, H. et al. Rational development of a human antibody cocktail that deploys multiple functions to confer Pan-SARS-CoVs protection. Cell Res. 31, 25–36 (2021); Kim, C. et al. A therapeutic neutralizing antibody targeting receptor binding domain of SARS-CoV-2 spike protein. Nat. Commun. 12, 288 (2021)).
В настоящий момент уже получены сотни различных SARS-CoV-2-специфических, в том числе и вируснейтрализующих, антител, обладающих различными профилями потентности и широты нейтрализации. Однако разработанные моноклональные антитела могут быть неактивными в отношении появившихся новых вариантов SARS-CoV-2. Так, например, моноклональное антитело бамланивимаб не способно нейтрализовать дельта-вариант SARS-CoV-2 (Planas, D. et al. Reduced sensitivity of SARS-CoV-2 variant Delta to antibody neutralization. Nature 596, 276–280 (2021)). В отношении бета-варианта бамланивимаб и этесивимаб полностью неактивны, а активность касиривимаба значительно снижена (Wang, P. et al. Antibody resistance of SARS-CoV-2 variants B.1.351 and B.1.1.7. Nature 593, 130–135 (2021)). Таким образом, идентификация и всесторонняя характеризация новых SARS-CoV-2-специфических антител является актуальной задачей и позволяет создавать композиции антител, против новых вирусных вариантов SARS-CoV-2 и потенциально против других, в том числе неописанных, коронавирусов.
Известно моноклональное антитело мыши IgM изотипа к RBD фрагменту в составе S-белка вируса SARS-CoV-2 (патент RU 2744274). Данное антитело способно детектировать до 1 нг рекомбинантного RBD фрагмента S-белка вируса SARS-CoV-2 в иммуноферментном анализе и обладает вируснейтрализующей активностью in vitro. Недостатками данного мышиного антитела для целей терапии и профилактики коронавирусной инфекции, вызванной SARS-CoV-2, являются его чужеродность для организма человека и короткое время полужизни, вследствие чего введение такого антитела пациентам может быть иммуногенным и сопровождаться нежелательными побочными реакциями. Кроме того, неизвестны уровень и механизм постулированной авторами нейтрализующей активности данного антитела в отношении SARS-CoV-2, детали эпитопной специфичности (а следовательно, и узнавание им вирусных вариантов, отличных от исходного штамма Wuhan-Hu-1, более не представленного среди циркулирующих изолятов) и возможность практического использования в системах, основанных на проточной цитометрии. Также не известен уровень (ауто)реактивности данного антитела по отношению к клеткам человека, поскольку оно было получено в иммунизированных мышах и в процессе созревания В-клетки проходило негативную селекцию против антигенов мыши, а не человека. Наконец, известно, что производство антител IgM изотипа является крайне трудоемким и сопровождается низким выходом, а сохранение всех заявленных свойств данного антитела при его возможной конверсии в IgG1-формат для преодоления данного ограничения является сомнительным, поскольку будет связано как минимум со снижением его авидности.
Существует множество публикаций, описывающих получение и характеризацию RBD-специфических моноклональных антител человека, в том числе и обладающих вируснейтрализующей активностью. Например, известно моноклональное антитело человека P4A1 (Guo, Y. et al., Nat Commun.12(1), 2623 (2021)). Однако в настоящий момент нет информации о его последовательности, а кроме того нет данных о сохранении им вируснейтрализующей активности в отношении циркулирующих в настоящее время вариантов SARS-CoV-2, а следовательно и о сохранении им терапевтической активности в отношении таких вирусов. Еще одним примером являются упомянутые выше человеческие RBD-специфические нейтрализующие антитела бамланивимаб (LY-CoV555) (Jones, B. et al. The neutralizing antibody, LY-CoV555, protects against SARS-CoV-2 infection in nonhuman primates. Sci. Transl. Med. 13(593), 1-17 (2021) и этесивимаб (LY-CoV016/JS016/CB6) (Shi, R. et al. A human neutralizing antibody targets the receptor-binding site of SARS-CoV-2. Nature 584, 120–124 (2020)), последовательности которых известны. Однако существенным недостатком данных антител является их неспособность нейтрализовать ряд циркулирующих вирусных вариантов SARS-CoV-2 (https://covdb.stanford.edu/page/susceptibility-data/), что ограничивает их профилактическое и терапевтическое использование.
Известна общедоступная база данных CoV-AbDab (http://opig.stats.ox.ac.uk/webapps/covabdab/), содержащая информацию о последовательностях и свойствах сотен RBD-специфических моноклональных антител различного происхождения (в том числе и человеческих), многие из которых обладают вируснейтрализующей активностью в отношении SARS-CoV-2. Недостатками большинства таких антител является их неполная характеризация в плане устойчивости к циркулирующим вариантам SARS-CoV-2, поскольку такие варианты появились позже, чем были получены антитела, а также в плане эпитопной специфичности, широты нейтрализации, наличия или отсутствия ауто- и полиреактивности и т.д.
Наиболее близким аналогом настоящего изобретения является разработка, описанная в заявке WO2021211775A1, где раскрыты несколько моноклональных антител человека, способных связываться с RBD фрагментом Spike-белка SARS-CoV-2 и ряда других сарбековирусов, а также обладающих широкой вируснейтрализующей активностью по отношению к различным циркулирующим вариантам SARS-CoV-2 и другим сарбековирусам. Поскольку пандемия, вызванная SARS-CoV-2, продолжается, и согласно молекулярно-эпидемиологическим наблюдениям наблюдается активное формирование все новых и новых вирусных вариантов, устойчивых к уже имеющимся моноклональным антителам и их сочетаниям, в будущем не исключено возникновение вариантов, устойчивых и к антителам, описанным в заявке WO2021211775A1. Таким образом, получение новых детально охарактеризованных RBD-специфических моноклональных антител как нейтрализующих, так и ненейтрализующих, является крайне актуальной задачей, поскольку может обеспечить рациональный дизайн композиций антител, предотвращающих мутационное ускользание вируса, а также более точную диагностику, эффективную терапию и профилактику коронавирусной инфекции.
Технической проблемой, решаемой настоящим изобретением, является получение новых моноклональных антител, предпочтительно человеческих моноклональных антител, которые способны к связыванию RBD фрагмента Spike-белка вируса SARS-CoV-2 и с высокой аффинностью распознают различные варианты вируса SARS-CoV-2.
Раскрытие сущности изобретения
Новые варианты вируса SARS-CoV-2 все чаще проявляют устойчивость к нейтрализации одобренными к применению моноклональными антителами. Поэтому расширение репертуара эффективных антител против SARS-CoV-2 продолжает оставаться важнейшей целью в условиях продолжающейся пандемии.
Таким образом задачей настоящего изобретения является получение новых моноклональных антител, способных к связыванию с RBD-районом Spike-белка SARS-CoV-2, предпочтительно обладающих вируснейтрализующей активностью, а также антиген-связывающих фрагментов таких антител.
Для ее решения авторами настоящего изобретения была получена панель антител человека, специфичных к RBD Spike-белка SARS-CoV-2 и охарактеризована их активность in vitro и in vivo.
Техническим результатом настоящего изобретения является получение новых моноклональных антител, способных к связыванию с RBD Spike-белка SARS-CoV-2. Высокая аффинность заявляемых антител позволит снизить их дозировку при диагностическом, профилактическом и терапевтическом применении.
Дополнительным техническим результатом для ряда антител является их высокая вируснейтрализующая активность, что обеспечивает возможность их использования в качестве средств профилактики (до- и пост-контактной) и терапии коронавирусной инфекции, вызванной SARS-CoV-2 и близкородственными ему вирусами.
Также полученные авторами настоящего изобретения данные говорят о том, что возможен рациональный дизайн широкого репертуара RBD-специфичных коктейлей антител, содержащих неконкурирующие нейтрализующие антитела, которые будут активными как в отношении циркулирующих в настоящее время вариантов вирусов, так и возможных будущих вариантов.
Поэтому дополнительным техническим результатом настоящего изобретения является создание коктейлей антител, содержащих комбинацию антител изобретения, которые обладают широкой вируснейтрализующей активностью по отношению к различным вариантам SARS-CoV-2.
Антитела и антигенсвязывающие фрагменты
Поставленная задача была решена получением моноклональных антител человека, селективно связывающих RBD фрагмент Spike-белка вируса SARS-CoV-2.
Используемый в настоящей заявке термин «антитело» предназначен для обозначения молекулы иммуноглобулина, составленной из четырех полипептидных цепей, при этом две тяжелые (Н) цепи и две легкие (L) цепи связываются друг с другом дисульфидными связями. Каждая тяжелая цепь содержит вариабельную область тяжелой цепи (сокращенно называемую в настоящем документе VH) и константную область тяжелой цепи. Константная область тяжелой цепи состоит из трех доменов: CH1, СН2 и СН3. Каждая легкая цепь содержит вариабельную область легкой цепи (сокращенно называемую в настоящем документе VL) и константную область легкой цепи. Константная область легкой цепи состоит из одного домена: CL.
Области VH и VL могут далее подразделяться на гипервариабельные районы, которые называются определяющими комплементарность областями (CDR), перемежаемые областями с более высоким уровнем консервативности, называемыми каркасными областями (FR). Каждая область VH и VL образована тремя CDR и четырьмя FR, расположенными от амино-терминального конца к карбокси-терминальному концу в следующем порядке: FR1, CDR1, FR2, CDR2, FR3, CDR3, FR4.
Четыре каркасных участка формируют конформацию типа бета-складчатого слоя. Участки CDR расположены в близком соседстве друг с другом благодаря каркасным участкам и вносят вклад в образование антигенсвязывающего участка. Участки CDR и каркасные участки антител могут быть определены путем ссылки на нумерационную систему Кабата [Kabat numbering system, Kabat et al., 1987, "Sequences of Proteins of Immunological Interest", US Dept. of Health and Human Services, US Government Printing Office] в сочетании с данными рентгеноструктурного анализа, как указано в заявке WO 91/09967. Участки CDR и каркасные участки антител могут также быть определены по номенклатуре Международной информационной системы по иммуногенетике (International Immunogenetics Information System, www.imgt.org).
Если не указано иное, любая представленная в описании полипептидная цепь имеет аминокислотную последовательность, которая начинается на N-конце и заканчивается на С-конце. Если антигенсвязывающий центр содержит и VH-, и VL-области, то они могут быть расположены на одной и той же полипептидной молекуле или предпочтительно каждая область может располагаться на разных цепях, VH-область может быть частью тяжелой цепи иммуноглобулина или ее фрагмента, а VL-область - частью легкой цепи иммуноглобулина или ее фрагмента.
Как хорошо известно, минорные изменения аминокислотной последовательности, такие как делеция, добавление или замена одной, небольшого количества или даже нескольких аминокислот, могут приводить к получению аллельной формы исходного белка, которая обладает практически идентичными свойствами. Более того, известен способ «перевивки» CDR-петель одного антитела в контекст каркасных последовательностей другого антитела, также с сохранением исходных свойств, что в частности позволяет производить гуманизацию антител, полученных в других млекопитающих. Таким образом, антитело изобретения включает те варианты, аминокислотная последовательность которых вне CDR петель отличается от приведенных в настоящей заявке последовательностей, но которые сохранили ту же антигенсвязывающую активность.
Антитело настоящего изобретения может быть человеческим антителом, гуманизованным антителом, мышиным антителом, химерным антителом и другими генно-инженерными антителами.
Также антитело изобретение может представлять собой аффинно-зрелое антитело.
Используемый в настоящем документе термин «антиген-связывающий фрагмент» антитела обозначает фрагмент антитела, который сохраняет способность к специфическому связыванию с антигеном. К примерам таких фрагментов, включенных в термин «антиген-связывающий фрагмент» антитела, относятся (i) фрагмент Fab, моновалентный фрагмент, состоящий из доменов VL, VH, CL и CH1; (ii) фрагмент F(ab')2, бивалентный фрагмент, состоящий из двух фрагментов Fab, связанных дисульфидным мостиком в шарнирной области; (iii) фрагмент Fd, состоящий из доменов VH и CH1; (iv) фрагмент Fv, состоящий из доменов VL и VH отдельной области антитела; (v) фрагмент dAb (Ward, E.S. et al. Nature. 241, 544-546, (1989)), состоящий из домена VH; и (vi) отдельная определяющая комплементарность область (CDR). Более того, несмотря на то, что два домена во фрагменте Fv, VL и VH кодируются различными генами, их можно объединить, используя рекомбинантные технологии, посредством синтетического линкера, который позволяет построить из них единую белковую цепочку, в которой области VL и VH связываются с образованием моновалентных молекул (известных как одноцепочечные антитела или scFv; см., например, Bird, R.E. et al. Science. 24, 423-426 (1988); Huston, J.S. et al. Proc. Natl. Acad. Sci. USA 85:5879-5883 (1988)). Такие одноцепочечные антитела также включаются в сферу охвата термина «антиген-связывающий фрагмент» антитела.
Моноклональные антитела человека являются минимально иммуногенными молекулами, поэтому многие современные методы направлены на их получение. Для этого в частности могут использоваться В лимфоциты, выделенные из периферической крови переболевших или вакцинированных доноров.
Специфические в отношении RBD Spike-белка SARS-CoV-2 моноклональные антитела настоящего изобретения были получены путем секвенирования и клонирования кДНК генов иммуноглобулинов в отдельных RBD-специфических В-лимфоцитах доноров, перенесших коронавирусную инфекцию ковид-19 в тяжелой форме и сформировавших высокие титры вируснейтрализующих антител в плазме крови.
Подходящий способ для выделения антител, эффективных для использования в рамках настоящего изобретения, включает (i) выделение В-лимфоцитов периферической крови доноров, перенесших коронавирусную инфекцию ковид-19 и сформировавших высокие нейтрализующие титры антител в сыворотке крови, (ii) окрашивание В-лимфоцитов флуоресцентно меченными препаратами RBD SARS-CoV-2, (iii) сортировка единичных окрашенных RBD+ В-лимфоцитов, (iv) проведение обратной транскрипции и ПЦР с наборами вырожденных праймеров, комплементарных лидерным и константным последовательностям генов иммуноглобулинов, (v) секвенирование полученных ПЦР продуктов с целью выяснения нуклеотидной и аминокислотной последовательностей экспрессируемых такими В-клетками антител, (vi) клонирование нуклеотидных последовательностей таких антител в экспрессионные векторы для наработки в культуре клеток млекопитающих, (vii) очистка, анализ специфичности и активности рекомбинантных моноклональных антител человека. В последующем также предусмотрено определение аминокислотной последовательности вариабельных доменов таких антител, измерение аффинности и специфичности, нейтрализующих свойств in vitro и in vivo.
В результате были получены нуклеотидные последовательности, на основании которых были определены аминокислотные последовательности вариабельных доменов тяжелых и легких цепей антител настоящего изобретения. Участки CDRs были определены по номенклатуре Kabat [Kabat numbering system, Kabat et al., 1987 "Sequences of Proteins of Immunological Interest", US Dept. of Health and Human Services, US Government Printing Office].
Полученное антитело включает вариабельную область тяжелой цепи (VH), которая содержит последовательности гипервариабельных районов: CDRH1, имеющего аминокислотную последовательность, выбранную из SEQ ID NO: 1-23, CDRH2, имеющего аминокислотную последовательность, выбранную из SEQ ID NO: 24-46, и CDRH3, имеющего аминокислотную последовательность, выбранную из SEQ ID NO: 47-69, и вариабельную область легкой цепи (VL), которая содержит последовательности гипервариабельных районов: CDRL1, имеющего аминокислотную последовательность, выбранную из SEQ ID NO: 70-92, CDRL2, имеющего аминокислотную последовательность, выбранную из AAS (SEQ ID NO:93), SNN (SEQ ID NO:94), WAT (SEQ ID NO:95), EVS (SEQ ID NO:96), DAS (SEQ ID NO:97), EVT (SEQ ID NO:98), KAS (SEQ ID NO:99), GAS (SEQ ID NO:100), NNN (SEQ ID NO:101), AAS (SEQ ID NO:102), DAS (SEQ ID NO:103), DVS (SEQ ID NO:104), GAS (SEQ ID NO:105), EVS (SEQ ID NO:106), KAS (SEQ ID NO:107), DAS (SEQ ID NO:108), GTS (SEQ ID NO:109), GAS (SEQ ID NO:110), GAS (SEQ ID NO:111), WAS (SEQ ID NO:112), DAS (SEQ ID NO:113), AAS (SEQ ID NO:114), или AAS (SEQ ID NO:115) и CDRL3, имеющего аминокислотную последовательность, выбранную из SEQ ID NO: 116-138. Также настоящим изобретением предусмотрены антиген-связывающие фрагменты данных моноклональных антител.
В следующем аспекте изобретения предусмотрено моноклональное антитело или его антигенсвязывающий фрагмент, содержащие вариабельную область тяжелой цепи (VH) с последовательностью аминокислот не менее чем на 90% гомологичную одной из последовательностей SEQ ID NO: 139-161, и вариабельную область легкой цепи (VL) с последовательностью аминокислот не менее чем на 90% гомологичной одной из последовательностей SEQ ID NO: 162-184.
В одном из воплощений моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельный участок тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности GFTFSSYA (SEQ ID NO: 1);
(ii) CDRH2 последовательности ISYDGSNK (SEQ ID NO: 24);
(iii) CDRH3 последовательности ARARPWRYSYGLYYYYGMDV (SEQ ID NO: 47), и
вариабельный участок легкой цепи (VL) содержит:
(i) CDRL1 последовательности QSISSY (SEQ ID NO: 70);
(ii) CDRL2 последовательности AAS (SEQ ID NO: 93);
(iii) CDRL3 последовательности QQSYSTPGS (SEQ ID NO: 116) [антитело iB0].
В другом воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 2;
(ii) CDRH2 последовательности SEQ ID NO: 25;
(iii) CDRH3 последовательности SEQ ID NO: 48, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 71;
(ii) CDRL2 последовательности SNN (SEQ ID NO:94);
(iii) CDRL3 последовательности SEQ ID NO: 117 [антитело iB1].
В следующем воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 3;
(ii) CDRH2 последовательности SEQ ID NO: 26;
(iii) CDRH3 последовательности SEQ ID NO: 49, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 72;
(ii) CDRL2 последовательности WAT (SEQ ID NO:95);
(iii) CDRL3 последовательности SEQ ID NO: 118 [антитело iB2].
В другом воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 4;
(ii) CDRH2 последовательности SEQ ID NO: 27;
(iii) CDRH3 последовательности SEQ ID NO: 50, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 73;
(ii) CDRL2 последовательности EVS (SEQ ID NO:96);
(iii) CDRL3 последовательности SEQ ID NO: 119 [антитело iB3].
В еще одном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH)содержит:
(i) CDRH1 последовательности SEQ ID NO: 5;
(ii) CDRH2 последовательности SEQ ID NO: 28;
(iii) CDRH3 последовательности SEQ ID NO: 51, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 74;
(ii) CDRL2 последовательности DAS (SEQ ID NO:97);
(iii) CDRL3 последовательности SEQ ID NO: 120 [антитело iB4].
В дополнительном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 6;
(ii) CDRH2 последовательности SEQ ID NO: 29;
(iii) CDRH3 последовательности SEQ ID NO: 52, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 75;
(ii) CDRL2 последовательности EVT (SEQ ID NO:98);
(iii) CDRL3 последовательности SEQ ID NO: 121 [антитело iB5]
В следующем воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 7;
(ii) CDRH2 последовательности SEQ ID NO: 30;
(iii) CDRH3 последовательности SEQ ID NO: 53, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 76;
(ii) CDRL2 последовательности KAS (SEQ ID NO:99);
(iii) CDRL3 последовательности SEQ ID NO: 122 [антитело iB6].
В другом воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 8;
(ii) CDRH2 последовательности SEQ ID NO: 31;
(iii) CDRH3 последовательности SEQ ID NO: 54, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 77;
(ii) CDRL2 последовательности GAS (SEQ ID NO:100);
(iii) CDRL3 последовательности SEQ ID NO: 123 [антитело iB9].
В еще одном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 9;
(ii) CDRH2 последовательности SEQ ID NO: 32;
(iii) CDRH3 последовательности SEQ ID NO: 55, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 78;
(ii) CDRL2 последовательности NNN (SEQ ID NO:101);
(iii) CDRL3 последовательности SEQ ID NO: 124 [антитело iB10].
В альтернативном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 10;
(ii) CDRH2 последовательности SEQ ID NO: 33;
(iii) CDRH3 последовательности SEQ ID NO: 56, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 79;
(ii) CDRL2 последовательности AAS (SEQ ID NO:102);
(iii) CDRL3 последовательности SEQ ID NO: 125 [антитело iB11].
В другом воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 11;
(ii) CDRH2 последовательности SEQ ID NO: 34;
(iii) CDRH3 последовательности SEQ ID NO: 57, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 80;
(ii) CDRL2 последовательности DAS (SEQ ID NO:103);
(iii) CDRL3 последовательности SEQ ID NO: 126 [антитело iB12]
В следующем воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 12;
(ii) CDRH2 последовательности SEQ ID NO: 35;
(iii) CDRH3 последовательности SEQ ID NO: 58, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 81;
(ii) CDRL2 последовательности DVS (SEQ ID NO:104);
(iii) CDRL3 последовательности SEQ ID NO: 127 [антитело iB13]
В ином воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 13;
(ii) CDRH2 последовательности SEQ ID NO: 36;
(iii) CDRH3 последовательности SEQ ID NO: 59, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 82;
(ii) CDRL2 последовательности GAS (SEQ ID NO:105);
(iii) CDRL3 последовательности SEQ ID NO: 128 [антитело iB14]
В дополнительном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 14;
(ii) CDRH2 последовательности SEQ ID NO: 37;
(iii) CDRH3 последовательности SEQ ID NO: 60, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 83;
(ii) CDRL2 последовательности EVS (SEQ ID NO:106);
(iii) CDRL3 последовательности SEQ ID NO: 129 [антитело iB15].
В альтернативном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 15;
(ii) CDRH2 последовательности SEQ ID NO: 38;
(iii) CDRH3 последовательности SEQ ID NO: 61, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 84;
(ii) CDRL2 последовательности KAS (SEQ ID NO:107);
(iii) CDRL3 последовательности SEQ ID NO: 130 [антитело iB16].
В ином воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 16;
(ii) CDRH2 последовательности SEQ ID NO: 39;
(iii) CDRH3 последовательности SEQ ID NO: 62, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 85;
(ii) CDRL2 последовательности DAS (SEQ ID NO:108);
(iii) CDRL3 последовательности SEQ ID NO: 131 [антитело iB17].
В еще одном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 17;
(ii) CDRH2 последовательности SEQ ID NO: 40;
(iii) CDRH3 последовательности SEQ ID NO: 63, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 86;
(ii) CDRL2 последовательности GTS (SEQ ID NO:109);
(iii) CDRL3 последовательности SEQ ID NO: 132 [антитело iB18].
В другом воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 18;
(ii) CDRH2 последовательности SEQ ID NO: 41;
(iii) CDRH3 последовательности SEQ ID NO: 64, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 87;
(ii) CDRL2 последовательности GAS (SEQ ID NO:110);
(iii) CDRL3 последовательности SEQ ID NO: 133 [антитело iB19].
В дополнительном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 19;
(ii) CDRH2 последовательности SEQ ID NO: 42;
(iii) CDRH3 последовательности SEQ ID NO: 65, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 88;
(ii) CDRL2 последовательности GAS (SEQ ID NO:111);
(iii) CDRL3 последовательности SEQ ID NO: 134 [антитело iB20].
В еще одном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 20;
(ii) CDRH2 последовательности SEQ ID NO: 43;
(iii) CDRH3 последовательности SEQ ID NO: 66, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 89;
(ii) CDRL2 последовательности WAS (SEQ ID NO:112);
(iii) CDRL3 последовательности SEQ ID NO: 135 [антитело iB21].
В альтернативном воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 21;
(ii) CDRH2 последовательности SEQ ID NO: 44;
(iii) CDRH3 последовательности SEQ ID NO: 67, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 90;
(ii) CDRL2 последовательности DAS (SEQ ID NO:113);
(iii) CDRL3 последовательности SEQ ID NO: 136 [антитело iB24].
В следующем воплощений моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что содержит:
(i) CDRH1 последовательности SEQ ID NO: 22;
(ii) CDRH2 последовательности SEQ ID NO: 45;
(iii) CDRH3 последовательности SEQ ID NO: 68, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 91;
(ii) CDRL2 последовательности AAS (SEQ ID NO:114);
(iii) CDRL3 последовательности SEQ ID NO: 137 [антитело iB25].
В ином воплощении моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающие RBD Spike-белка SARS-CoV-2, характеризуются тем, что вариабельная область тяжелой цепи (VH) содержит:
(i) CDRH1 последовательности SEQ ID NO: 23;
(ii) CDRH2 последовательности SEQ ID NO: 46;
(iii) CDRH3 последовательности SEQ ID NO: 69, и
вариабельная область легкой цепи (VL) содержит:
(i) CDRL1 последовательности SEQ ID NO: 92;
(ii) CDRL2 последовательности AAS (SEQ ID NO:115);
(iii) CDRL3 последовательности SEQ ID NO: 138 [антитело iB26]
В следующем воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, выбранной из SEQ ID NO: 139-161; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, выбранной из SEQ ID NO: 162-184.
В частности моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 139; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 162 [антитело iB0].
В следующем воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 140; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 163 [антитело iB1].
В ином воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 141; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 164 [антитело iB2].
В другом воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 142; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 165 [антитело iB3].
В альтернативном воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 143; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 166 [антитело iB4].
В еще одном воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 144; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 167 [антитело iB5].
В дополнительном воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 145; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 168 [антитело iB6].
В альтернативном моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 146; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 169 [антитело iB9].
В еще одном моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 147; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 170 [антитело iB10].
В следующем воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 148; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 171 [антитело iB11].
В альтернативном моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 149; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 172 [антитело iB12].
В ином воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую ий последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 150; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 173 [антитело iB13].
В дополнительном воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 151; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 174 [антитело iB14].
В другом воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 152; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 175 [антитело iB15].
В еще одном воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 153; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 176 [антитело iB16].
В следующем воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 154; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 177 [антитело iB17].
В дополнительном моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 155; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 178 [антитело iB18].
В еще одном моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 156; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 179 [антитело iB19].
В альтернативном воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 157; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 180 [антитело iB20].
В дополнительном воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 158; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 181 [антитело iB21].
В другом воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 159; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 182 [антитело iB24].
В следующем воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 160; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 183 [антитело iB25].
В дополнительном воплощении моноклональное антитело, селективно связывающее RBD Spike-белка SARS-CoV-2, включает вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 161; и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот не менее чем на 90% гомологичную последовательности, SEQ ID NO: 184 [антитело iB26].
В частности, антитела согласно настоящему изобретению обладают способностью к нейтрализации вируса SARS-CoV-2.
Новые антитела человека, селективно связываются с RBD Spike-белка SARS-CoV-2, содержат приведенные выше последовательности вариабельных областей и могут содержать консервативные участки обеих цепей, необходимые для функционирования указанных антител. Консервативные участки тяжелых цепей иммуноглобулинов классов IgG, IgM, IgA, IgD или IgE человека и других млекопитающих могут быть использованы в качестве консервативных участков для тяжелых цепей согласно настоящему изобретению. Например, антитело или его антигенсвязывающий фрагмент, согласно изобретению, могут содержать консервативные участки тяжелых цепей, принадлежащие к изотипу IgG1, IgG2, IgG3 или IgG4. Консервативные участки легкой цепи иммуноглобулинов изотипов каппа или лямбда человека и других млекопитающих могут быть использованы в качестве консервативных участков для легких цепей антител согласно настоящему изобретению.
В настоящем изобретении фраза «нейтрализующее моноклональное антитело» означает молекулу, которая связывается с SARS-CoV-2, образует стабильный комплекс и препятствует прикреплению и/или проникновению вируса SARS-CoV-2 в клетку. Способность антитела к связыванию с антигеном может быть определена специалистом в данной области с использованием методов, включающих, но не ограничивающихся методом иммуноферментного анализа (ИФА), равновесным диализом, с использованием плазмон-поверхностного резонанса и биослойной интерферометрии (BLI). Методы определения аффинности хорошо известны специалисту в данной области техники, подробно описаны Janeway и соавт. (Immunobiology: The Immune System in Health and Disease (Garland Publishing Company, 1996)).
Существующий уровень техники является таким, что позволяет специалисту в данной области синтезировать молекулы ДНК, предлагаемые в изобретении на основе представленной в настоящем описании информации, т.е. аминокислотных последовательностей гипервариабельных участков.
Нуклеиновые кислоты
Для решения поставленной задачи также были получены нуклеиновые кислоты, кодирующие VH указанных антител, в частности имеющие нуклеотидные последовательности SEQ ID NO: 185-207, и VL указанных антител, в частности имеющие нуклеотидные последовательности SEQ ID NO: 208-230.
Используемый в настоящем описании термин нуклеиновая кислота относится к фрагменту ДНК или РНК. Предпочтительно нуклеиновой кислотой, кодирующей антитело согласно настоящему изобретению, является фрагмент ДНК, кодирующий вариабельные тяжелую или легкую цепи моноклонального антитела, селективно связывающего RBD Spike-белка SARS-CoV-2, где данное антитело содержит последовательность аминокислот не менее чем на 90% гомологичную SEQ ID NO: 139-161, и последовательность аминокислот не менее чем на 90% гомологичную SEQ ID NO: 162-184.
Специалистам в данной области техники известно, что ввиду вырожденности генетического кода могут быть различия в последовательности ДНК кодирующей одну и ту же аминокислотную последовательность [Льюин Б. Гены. Москва.: Мир, 1987.]. Поэтому последовательности нуклеиновых кислот согласно настоящему изобретению могут отличаться, при условии, что они кодируют участки цепей антитела, содержащего последовательность аминокислот не менее чем на 90% гомологичную SEQ ID NO: 139-161, и последовательность аминокислот не менее чем на 90% гомологичную SEQ ID NO: 162-184.
Нуклеиновые кислоты изобретения также включают нуклеиновые кислоты, кодирующие тяжелую и легкую цепи моноклональных антител, селективно связывающих RBD Spike-белка SARS-CoV-2, содержащих последовательности аминокислот SEQ ID NO: 231-253 и последовательности аминокислот SEQ ID NO: 254-276. В частности, этими нуклеиновыми кислотами могут быть ДНК последовательности, которые приведены в перечне последовательностей под номерами SEQ ID NO: 277-299 и SEQ ID NO: 300-322.
Экспрессионный вектор
В одном воплощении изобретения предусмотрены рекомбинантные экспрессионные векторы, содержащие молекулу нуклеиновой кислоты по изобретению, причем вектор содержит контролирующую экспрессию последовательность, которая способствует экспрессии кодируемого полипептида в прокариотических или эукариотических клетках-хозяевах и функционально связана с данной молекулой нуклеиновой кислоты.
Рекомбинантный вектор также может включать последовательности нуклеотидов, которые способствуют, контролируют или регулируют экспрессию и транскрипцию полинуклеотида по изобретению, а также трансляцию полипептида по изобретению, причем эти последовательности выбирают в соответствии с используемыми клетками-хозяевами.
Клетки-хозяева, производящие антитела
При использовании в данном документе термин «клетка-хозяин» относится к клетке, в которую была введена экзогенная нуклеиновая кислота, такая как ДНК, например, в составе экспрессионного вектора, и которая способна экспрессировать антитело или антигенсвязывающий фрагмент изобретения. Специалистам в данной области понятно, что такой термин относятся не только к конкретной клетке, в которую была введена экзогенная нуклеиновая кислота, но также к потомству такой клетки. Поскольку определенные изменения могут произойти в последующих поколениях из-за мутаций или воздействий окружающей среды, такое потомство может быть не идентично исходной клетке, но все еще включено в объем термина «клетка-хозяин», используемого в данном документе. В некоторых воплощениях клетки-хозяева включают прокариотические и эукариотические клетки, которые пригодны для экспрессии экзогенной ДНК (например, рекомбинантной последовательности нуклеиновой кислоты). Типичные клетки включают клетки прокариот и эукариот (одноклеточных или многоклеточных), такие как бактериальные клетки, клетки грибов, дрожжевые клетки, клетки растений, клетки насекомых, клетки животных, отличных от человека, и клетки человека. В некоторых воплощениях клетка-хозяин представляет собой клетку HEK293T.
Способы получения антитела
Антитела изобретения могут быть получены рекомбинантным путем, при помощи временной доставки или постоянной интеграции кодирующих их нуклеиновых кислот, например в составе экспрессионного вектора, в клетки-хозяева, такие как клетки млекопитающих, насекомых, дрожжей или растений. После наработки в рекомбинантной системе антитела должны быть очищены до гомогенного состояния при помощи традиционных способов очистки антител, например одной или нескольких стадий хроматографического процесса. Например, антитело или его антигенсвязывающий фрагмент по изобретению может быть получен способом, включающим культивирование или поддержание клетки-хозяина в таких условиях, чтобы указанная клетка-хозяин продуцировала или экспрессировала антитело или его антигенсвязывающий фрагмент по любому изобретению, посредством экспрессии нуклеиновой кислоты, кодирующей тяжелую цепь антитела, и нуклеиновой кислоты, кодирующей легкую цепь антитела, например, в составе вектора, содержащего нуклеиновую кислоту, кодирующую тяжелую цепь антитела, и нуклеиновую кислоту, кодирующую легкую цепь антитела, или векторов, один из которых содержит нуклеиновую кислоту, кодирующую тяжелую цепь антитела, а второй, нуклеиновую кислоту, кодирующую легкую цепь антитела, и, при необходимости, дополнительно включающий выделение антитела или его антигенсвязывающего фрагмента, полученного таким способом.
Кроме того, известны способы получения антител посредством генетического переноса кодирующих их нуклеиновых кислот in situ или in vivo с использованием, например, внутримышечной инъекции, электропорации, вирусных векторов, наночастиц и липосом, позволяющие обеспечить синтез и продукцию антител и их комбинаций прямо в организме, в результате чего достигается искомый профилактический и/или терапевтический эффект.
Коктейли и комбинации антител
Для SARS-CoV-2 описано несколько основных механизмов прямой вируснейтрализующей активности антител. Первый основан на экранировании сайтов связывания S-белка с рецептором Ace2 за счет конкурентного узнавания антителом так называемого рецептор-связывающего мотива (RBM) в пределах RBD. Второй механизм также предотвращает связывание S-белка с Ace2, но за счет узнавания антителом эпитопов, отличных от RBM, при котором молекула антитела стерически мешает взаимодействию Ace2 с RBM. Третьим является дестабилизация антителом пред-фьюжн структуры Spike-белка и индукция его необратимого перехода в пост-фьюжн конформацию. Четвертый, наоборот, основан на стабилизации пред-фьюжн конформации Spike-белка.
Очевидно, что площадь контакта антитела с интерфейсом RBD-Ace2 прямо коррелирует с потенциальной устойчивостью антитела к аминокислотным заменам в Spike-белке. Так как максимальная площадь контакта антитела и антигена ограничена базовыми особенностями структуры вариабельных доменов тяжелой и легкой цепи, она не может быть существенно увеличена для каждого конкретного антитела. Однако, тот же эффект может достигаться за счет одновременного применения различных антител, связывающихся с независимыми или частично перекрывающимися эпитопами на поверхности Spike-белка, поскольку возникновение двух и более мутаций, инактивирующих сразу несколько вируснейтрализующих антител, без негативных последствий для приспособленности вируса крайне маловероятно. Таким образом, оправдано применение коктейлей антител, сформулированных на основе информации об эпитопах связывания со Spike-белком. При этом существует два формальных сценария использования антител в коктейле.
Первый из них рассчитан на одновременное связывание двух или более антител с вирусом (т.е. со Spike-белком) без взаимной конкуренции. Это значительно повышает эффективность вируснейтрализации, однако не является обязательным при использовании высокопотентных антител, каждое из которых по отдельности обладает высоким нейтрализационным потенциалом. Для такого варианта использования антитела не должны конкурировать, что можно установить при помощи специальных способов постановки иммуноферментного анализа, биослойной интерферометрии или структурного анализа комплексов антитело-антиген (криоЭМ, рентгеноструктурный анализ и т.д.).
Второй сценарий использования коктейлей предусматривает комбинирование антител вне зависимости от их сайтов связывания со Spike-белком, но с учетом их чувствительности к мутациям в геноме вируса, приводящим к заменам аминокислотных остатков в Spike-белке. При таком комбинировании антитела подбираются таким образом, чтобы возникающие мутации не инактивировали все антитела в коктейле. Для такого дизайна коктейлей не требуются данные о конкуренции антител, но нужна детальная картина мутационной чувствительности антител, что может быть определено в нейтрализационных тестах с панелью мутантных вариантов Spike-псевдотипированных вирусов или в условиях in vitro/in vivo эволюции системы вирус/антитело. При этом сценарии использования коктейлей разные антитела не взаимодействуют со Spike-белком одновременно, но гарантируют повышенную устойчивость препарата к вновь появляющимся мутациям.
Помимо информации о наличии или отсутствии взаимной конкуренции антител за связывание с целевым белком (например, c S-белком SARS-CoV-2), крайне важны данные о механизмах активности, аффинности и фармакодинамики. Например, формирование коктейля из двух антител, связывание одного из которых приводит к стерической реорганизации мишени и исчезновению эпитопа для другого антитела, является заведомо непродуктивным. Кроме того, известны примеры синергичной активности не только вируснейтрализующих антител, но и ненейтрализующих (слабонейтрализующих) антител друг с другом. Наконец, соотношение компонент антительного коктейля не обязательно должно быть эквимолярным, и тем самым оно может в значительной мере определять диапазон терапевтической и/ или профилактической активности.
Таким образом, полученная в данной работе информация о взаимной конкуренции антител друг с другом за связывание с RBD, а также данные о чувствительности антител к аминокислотным заменам в RBD, позволяют осуществлять рациональный дизайн коктейлей антител друг с другом и известными коммерческими нейтрализующими антителами. Например, антитела iB6 можно комбинировать не только друг с другом iB20 (как показано в соответствующих примерах), но и с REGN10987 и формулировать тем самым трехкомпонентный коктейль, целиком состоящий из неконкурирующих нейтрализующих антител против района взаимодействия ACE2 и RBD. Также возможны другие подобные комбинации, обеспечивающие большую устойчивость к возникающим заменам в структуре Spike-белка SARS-CoV-2, такие как iB14 + COVA2-15 или iB14 + COV2-2504 и т.д.
Применение антител в лечении и диагностике
Возможность использования моноклональных антител для целей диагностики является их ценным свойством. В частности распространены тест-системы, основанные на принципах иммуноферментного и иммунохроматографического (lateral flow) анализа, позволяющие специфично и селективно детектировать и измерять уровень интересующего аналита в сложных смесях и биологических жидкостях организма. Важными областями применения RBD-специфических моноклональных антител являются диагностика наличия коронавирусной инфекции у человека и других млекопитающих, оценка ее динамики, выяснение принадлежности обнаруженного изолята SARS-CoV-2 к той или иной кладе и, следовательно, прогноз его устойчивости к препаратам моноклональных антител, а также оценка уровня нейтрализующих антител в сыворотке крови и других биологических жидкостях млекопитающих.
Для целей терапии RBD-специфические моноклональные антитела и их комбинации могут быть использованы при введении человеку или другому млекопитающему в короткий срок после появления первых симптомов заболевания, вызванного SARS-CoV-2. Пути введения антител могут включать внутривенную и внутримышечную инъекцию, доставку аэрозолем в дыхательные пути или подкожное введение. Антитело или его антигенсвязывающий фрагмент изобретения могут быть введены системно, назально или путем прямой доставки кодирующих его нуклеиновых кислот в организм млекопитающих. Помимо терапевтического применения антитела могут быть введены заранее в качестве меры профилактики. В таком случае целесообразно использование антител для лиц, имеющих высокий риск заражения вирусом SARS-CoV-2, тяжелого течения заболевания и не подлежащих вакцинации по медицинским или иным причинам. При профилактическом использовании необходимо учитывать период полужизни антител в кровотоке, который в среднем для антител изотипа IgG1 составляет 21 день, но может быть значительно увеличен за счет модификации последовательности аминокислотных остатков в Fc-части.
Кроме того, необходимо учитывать то, что человек – не единственный вид млекопитающих, для которого показана возможность инфицирования SARS-CoV-2 и близкими ему вирусами. В частности, случаи заражения SARS-CoV-2 были показаны для многих представителей семейства кошачьих (кошка, лев, тигр, снежный барс, пума), куньих (норка, хорек), приматов (горилла, макаки), псовых (собака, енотовидная собака) рукокрылых, грызунов (хомячки), оленевых (белохвостый олень) и панголинов. Несмотря на то, что эффективный перенос вируса и его вариантов внутри этих видов животных и тем более обратного заражения от них человека происходит редко (например, известны случаи заражения людей от выращиваемых в зверохозяйствах норок), возможность формирования новых зоонозных очагов SARS-CoV-2 является эпидемиологической угрозой и требует развития надежного молекулярного инструментария, позволяющего детектировать, типировать, а при необходимости и нейтрализовать SARS-CoV-2, включая его варианты, также и в других видах млекопитающих.
Химерные антигенные рецепторы
В некоторых воплощениях настоящее изобретение относится к химерному антигенному рецептору, содержащему антигенсвязывающий сайт антитела настоящего изобретения. Используемый в данном документе термин «химерный антигенный рецептор» или «CAR» относится к рекомбинантному трансмембранному белку, экспрессируемому в клетке млекопитающего, который включает, по меньшей мере, внеклеточный домен, трансмембранный домен и внутриклеточный домен сигнализации. Необязательно, белок CAR включает «спейсер», который ковалентно связывает внеклеточный домен с трансмембранным доменом. Спейсер часто представляет собой полипептид. CAR обычно экспрессируется в лимфоцитах млекопитающих. В некоторых воплощениях CAR экспрессируется в клетке млекопитающих, такой как Т-клетка, NK-клетка, макрофаг или инфильтрирующий опухоль лимфоцит (TIL). CAR, экспрессируемый на Т-клетках, упоминается в данном документе как “CAR T-клетка” или “CAR-T.” В некоторых воплощениях CAR-T представляет собой хелперную Т-клетку, цитотоксическую Т-клетку, Т-клетку естественного киллера, Т-клетку памяти, регуляторную Т-клетку или гамма-дельта-Т-клетку. CAR-Т часто является аутологичной CAR-Т, хотя аллогенные CAR-Т включены в объем изобретения. Внеклеточный домен CAR включает антигенсвязывающую область, такую как антитело или его антигенсвязывающий фрагмент (например, scFv), который специфически связывается в физиологических условиях с антигеном, таким как S-белок вируса SARS-CoV-2. При специфическом связывании биохимическая цепь событий (т.е. сигнальная трансдукция) приводит к модуляции иммунологической активности CAR-T. Так, например, специфическое связывание антигенсвязывающей области CAR с его антигеном может приводить к изменениям иммунологической активности активности CAR Т-клеток, что отражается на изменениях цитотоксичности, пролиферации или продукции цитокинов. Передача сигнала при активации CAR достигается в некоторых воплощениях CD3-дзета цепью («CD3z»), которая участвует в трансдукции сигнала в нативных Т-клетках млекопитающего. CAR-T могут дополнительно содержать один или несколько костимулирующих доменов, заимствованных от таких белков как CD28, 41BB, OX40 или ICOS для дальнейшей модуляции иммуномодулирующего ответа Т-клетки. CD3z включает консервативный мотив, известный как мотив активации на основе тирозина на основе иммунорецептора (ITAM), который участвует в трансдукции сигнала Т-клеточного рецептора.
Краткое описание чертежей и таблиц, иллюстрирующих изобретение
На Фиг. 1 показаны результаты иммуноферментного анализа сывороток четырех доноров-реконвалесцентов (С33, С34, С35, С36) и контрольной сыворотки здорового донора (К-) против RBD SARS-CoV-2 (изолят Wuhan-Hu-1), иммобилизованного в лунках планшета.
На Фиг. 2 показана стратегия гейтирования и репрезентативные результаты FACS-окрашивания В-лимфоцитов донора-реконвалесцента С34.
На Фиг. 3 показаны результаты разделения очищенных рекомбинантных RBD-специфических антител в SDS-полиакриламидном геле. М – маркер молекулярного веса, LC – легкая цепь антитела, HC – тяжелая цепь антитела.
На Фиг. 4 показаны результаты BLI-анализа аффинности моноклональных антител к RBD SARS-CoV-2 (26,7 нМ). Представлены только данные для антител, обнаруживших заметное связывание с RBD. Данные для остальных антител аналогичны glVRC01 (негативный контроль, отсутствие связывания).
На Фиг. 5 показаны результаты BLI-анализа аффинности наиболее потентных моноклональных антител к RBD SARS-CoV-2.
На Фиг. 6 показаны результаты цитометрического анализа окрашивания клеток линии HEK293T с поверхностной экспрессией Spike-белка коронавирусов HCoV-229E, HCoV-NL63 и SARS-CoV-2 моноклональными антителами против RBD SARS-CoV-2.
На Фиг. 7 показаны результаты нейтрализационных тестов моноклональных антител iB-серии, а также известных референсных SARS-CoV-2-нейтрализующих антител (REGN10933, REGN10987, COVA2-15, COV2-2504) в псевдовирусной (А) и аутентичной (Б) вирусной системе.
На Фиг. 8 показаны данные анализа попарной конкуренции моноклональных антител за связывание с RBD SARS-CoV-2, полученные при помощи биослойной интерферометрии.
На Фиг. 9 представлены результаты BLI-анализа конкуренции вируснейтрализующих RBD-спрецифических моноклональных антител COVA2-15, iB12, iB15, REGN10933, iB4, iB6 с REGN10987, COVA2-15, iB12 и iB20 за связывание с RBD SARS-CoV-2.
На Фиг. 10 показаны данные BLI-анализа, свидетельствующие об отсутствии взаимной конкуренции вируснейтрализующих моноклональных антител iB20, REGN10987 и iB6 за связывание с RBD SARS-CoV-2 (A), и Венн-диаграмма перекрывания эпитопов представленных моноклональных антител.
На Фиг. 11 приведены данные анализа активности моноклональных антител iB12, iB14 и смеси iB6+iB20 терапевтическом и профилактическом введении на модели заражения SARS-CoV-2 сирийских хомячков. А – динамика изменения веса подопытных животных, Б - вирусная нагрузка (количество копий вирусной РНК на 1 мг ткани, данные для гена RdRp SARS-CoV-2) в легких хомяков на 5 день после заражения, В – степень легочной патологии.
На Фиг. 12 приведены данные гистопатологического анализа срезов легких хомяков на 5 день после инфицирования SARS-CoV-2 (окрашивание гематоксилин-эозином, представлены репрезентативные изображения для каждой группы).
На Фиг. 13 представлена диаграмма изменения нейтрализующих свойств моноклональных RBD-специфических антител против вариантов Spike-белка (одноаминокислотные замены (А) и комбинации замен (Б)) на системе нейтрализации Spike-псевдотипированных лентивирусных частиц. * - замены, отсутствующие среди циркулирующих изолятов SARS-CoV-2.
На Фиг. 14 показано сохранение (зеленый круг) или потеря (красный квадрат) нейтрализующей активности в отношении SKEN-псевдотипированных лентивирусных частиц различными моноклональными антителами и их комбинациями. Представлено изменение значений IC50 относительно предкового варианта Spike (изолят Wuhan-Hu-1), серым выделен диапазон 5-кратного изменения IC50, не являющийся существенным в плане потентности исследуемых антител.
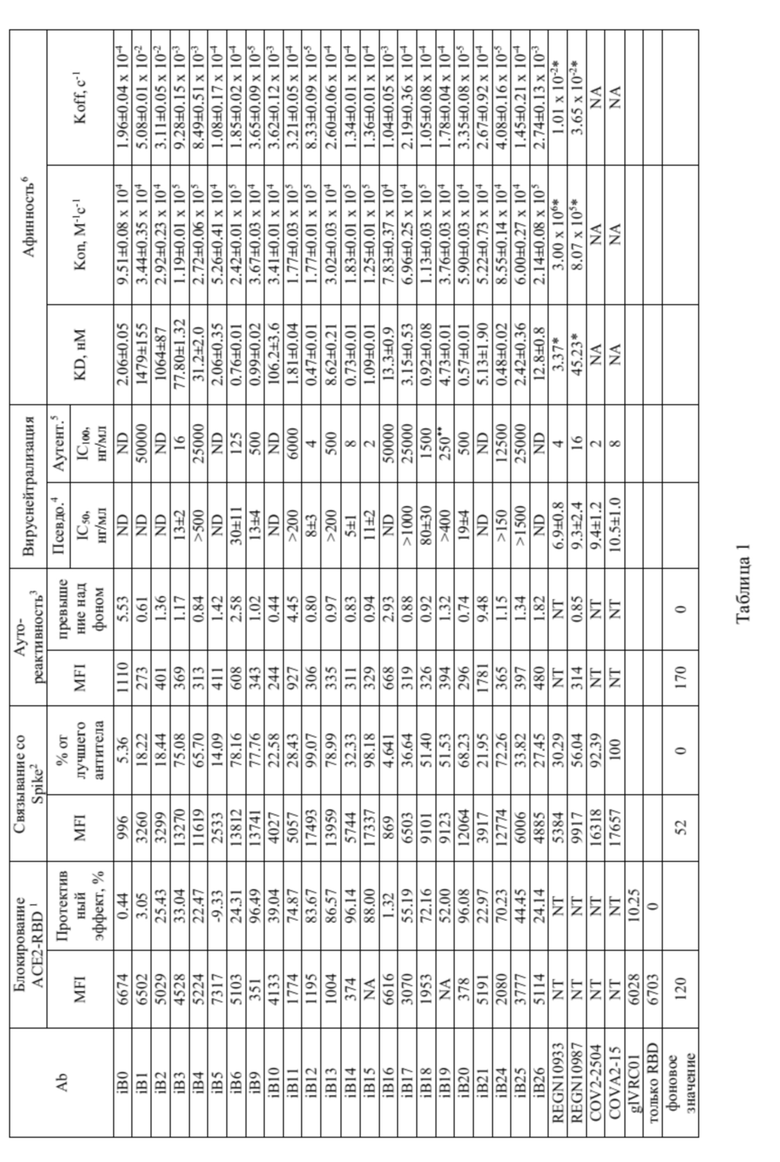
В Табл. 1 представлены характеристики полученных моноклональных антител (серия iB) и известных антител, специфичных к RBD SARS-CoV-2. Блокирование ACE2-RBD 1 было протестировано путем добавления смеси антитело /RBD к клеткам линии ACE2-HEK293T. Способность моноклонального антитела предотвращать связывание RBD с ACE2 была измерена при помощи проточной цитометрии. Протективный эффект был рассчитан по формуле (1 - (MFIантитело - MFIфон)/(MFIтолько RBD – MFIфон)) × 100%. Способность моноклональных антител связываться со Spike-белком на клеточной поверхности была исследована с использованием клеток линии HEK293T с поверхностной экспрессией Spike-белка SARS-CoV-2. Аутореактивность3 исследована при помощи окрашивания клеток линии HEP-2, параметры вируснейтрализации - в системе Spike-псевдотипированных лентивирусных частиц4 и аутентичного вируса SARS-CoV-25. Константы афинности6 были измерены при помощи биослойной интерферометрии или получены из литературных источников. MFI – средняя интенсивность флуоресценции, NT – не исследовано, ND – не детектировано, NA – не известно.
В Табл. 2 представлены результаты иммуноферментного анализа способности полученных моноклональных антител связываться с RBD SARS-CoV-2 дикого типа (изолят Wuhan-Hu-1), а также с RBD вариантов delta и beta. (+) – связывание, (-) – отсутствие связывания, (+/-) – слабое связывание.
В Табл. 3 представлены результаты Вестерн-блот гибридизации очищенного RBD SARS-CoV-2 и неочищенного рекомбинантного S-белка SARS-CoV-2 с полученными моноклональными антителами в присутствии и в отсутствие восстановителя. (+) – связывание, (-) – отсутствие связывания, (-/+) – слабое связывание.
Осуществление изобретения
Последующие примеры приведены исключительно для целей объяснения и не предназначены для ограничения каким-либо образом объема настоящего изобретения.
Пример 1. Выделение В-лимфоцитов человека, специфических к RBD Spike-белка вируса SARS-CoV-2.
Образцы инактивированной прогреванием в течение 30 минут при +56°С плазмы доноров, перенесших коронавирусную инфекцию ковид-19, были проанализированы при помощи набора SARS-СоV-2-IgG-ИФА (ООО «Хема-Медика»). Затем, у доноров, образцы плазмы которых обнаруживали титры RBD-специфических антител 1:5000 и выше (Фиг. 1), был произведен забор периферической венозной крови и выделение живых CD19+ IgG+ B-лимфоцитов (окрашивание DAPI, конъюгатами anti-human CD19-PE (ООО «Сорбент»), anti-human IgG-FITC (ООО «Сорбент»), 5 мкл/106 мононуклеарных клеток крови). B-лимфоциты были дополнительно окрашены биотинилированным рекомбинантным RBD SARS-CoV-2 (2 мкг/мл) с последующей детекцией при помощи конъюгата APC-стрептавидин (Thermo Fisher Scientific). Поверхностный фенотип целевой популяции В-лимфоцитов для сортировки единичных клеток в индивидуальные ПЦР-пробирки таким образом был RBDhighCD19+IgG+DAPI− (Фиг. 2). Окрашенные аналогичным образом В-лимфоциты здоровых доноров были использованы в качестве негативного контроля.
Пример 2. Получение моноклональных антитела человека против RBD Spike-белка вируса SARS-CoV-2.
Процедуры синтеза кДНК с единичных В-лимфоцитов и ПЦР с обратной транскрипцией проводили, как описано в работе Guselnikov и соавт (Guselnikov, S. V. et al. A simple way to increase recovery of the expressed VH and VL genes in single-sorted human B cells. BioTechniques 67, 184–187 (2019)), за исключением того, что для амплификации последовательностей, кодирующих γ- и λ-цепи антител, был использован протокол для μ-цепей. В частности для амплификации последовательностей генов VHγ, Vκ и Vλ были использован набор вырожденных праймеров из работы Tiller и соавт. (Tiller, T. et al. Efficient generation of monoclonal antibodies from single human B cells by single cell RT-PCR and expression vector cloning. J. Immunol. Methods 329, 112–124 (2008)). Парные образцы ПЦР-ампликонов (VHγ + Vκ или Vλ) секвенировали по Сэнгеру и клонировали в векторы для эукариотической экспрессии, кодирующие константные районы γ1, κ или λ-цепей иммуноглобулинов человека (Tiller, T. et al. Efficient generation of monoclonal antibodies from single human B cells by single cell RT-PCR and expression vector cloning. J. Immunol. Methods 329, 112–124 (2008)).
Таким образом были получены пары последовательностей VH- и VL-генов для 33 B-лимфоцитов, из которых 28 были представлены уникальными комбинациями последовательностей. Смесь плазмидных ДНК экспрессионных конструкций, кодирующих корректные пары легких и тяжелых цепей антител, была доставлена в клетки линии HEK293T, используя метод кальций-фосфатной трансфекции (Chenuet, S. et al. Calcium phosphate transfection generates mammalian recombinant cell lines with higher specific productivity than polyfection. Biotechnol. Bioeng. 101, 937–945 (2008)). Как правило, за 12 часов до проведения трансфекции 107 клеток рассаживали в культуральные флаконы площадью 150 см2 в среде IMDM (Gibco, Thermo Fisher Scientific) с добавлением 10% FBS (Gibco, Thermo Fisher Scientific) и смеси пенициллина/стрептомицина (Gibco, Thermo Fisher Scientific). Плазмидные ДНК смешивали в молярном соотношении 1:1, к полученной смеси добавляли 2 М CaCl2 до финальной концентрации 25 мМ и доводили суммарный объем смеси стерильной водой до 3 мл. Затем смесь медленно добавляли к 3 мл раствора 2× HBS (50 мМ HEPES, 1,5 мМ Na2HPO4, 280 мМ NaCl, 10 мМ KCl, 12 мМ сахароза, pH = 7.11) при постоянном интенсивном перемешивании. Полученный раствор добавляли по каплям к клеткам. Спустя 8 часов, культуральную среду заменяли на бессывороточную среду EX-CELL® 293 Serum-Free Medium (Sigma). Трансфицированные клетки росли в течение 6 дней в CO2-инкубаторе при 37°C, 5% CO2. Супернатантны, очищенные от клеточного дебриса при помощи центрифугирования (4000× g, 10 мин), фильтровали через 0.22 мкм PES-фильтр (TPP). Антитела очищали на колонке с Protein A агарозой (McLab, USA, #PPA-503) в соответствии с инструкциями производителя. Вкратце, после загрузки колонку промывали дважды PBS. Антитела элюировали раствором 0.1 M глицина, pH 2,7, 150 мМ NaCl в нейтрализующий буфер состава 1 M Tris, pH 8,0, в соотношении 1:10. Чистоту и выход препаратов антител контролировали посредством разделения в 15% SDS-полиакриламидном геле (Фиг. 3). Затем проводили диализ в течение ночи в PBS и концентрировали с использованием колонок Ultra-15 Ultracel-100K (Amicon) до финальной концентрации 2 мг/мл.
Для измерения кинетики взаимодействия антител с RBD была использована биослойная интерферометрия (прибор Octet (ForteBio), биосенсоры NTA). С этой целью была вначале проведена иммобилизация рекомбинантного белка RBD-His6 (30 мкг/мл) на поверхности биосенсора. Нерелевантный белок Fn3, также несущий His6-эпитоп и обладающий схожим молекулярным весом, был иммобилизован на контрольном сенсоре (30 мкг/мл), после чего производили необратимую иммобилизацию данных белков на сенсорах при помощи EDC/NHS, в соответствии с инструкциями производителя. Значения KD были получены с использованием концентрации RBD-His6, равной 26,7 нМ. Для обработки данных были использованы параметры полного локального фиттинга, бивалентная модель, R2 > 0,9, Χ2 < 3 (Фиг. 4). Затем для девяти наиболее аффинных моноклональных антител (iB3, iB6, iB9, iB12, iB13, iB14, iB15, iB19 и iB20) была проведена более точная характеризация связывания в диапазоне 6 концентраций RBD-His6 (6,25 нМ, 12,5 нМ, 25 нМ, 50 нМ, 100 нМ, 200 нМ). С этой целью были использованы AHC-биосенсоры и проведена иммобилизация антител. В качестве нерелевантного антитела было использовано моноклональное антитело человека glVRC01 (Georgiev I. S. et al. Antibodies VRC01 and 10E8 neutralize HIV-1 with high breadth and potency even with Ig-framework regions substantially reverted to germline. J Immunol. 192(3), 1100-1106 (2014)). Для обработки данных были использованы параметры полного глобального фиттинга, бивалентная модель, R2 > 0,9, Χ2 < 3 и последовательный переход между режимами: Baseline 30 с, Loading 300 с, Baseline2 60 с, Association 600 с, Dissociation 1500 с, Regeneration 3 × 5 с (Фиг. 5). Значения KD, Kon и Koff для 23 моноклональных антител приведены в Табл. 1.
Способность всех 23 моноклональных антител узнавать RBD в контексте Spike-белка SARS-CoV-2, а не только рекомбинантный RBD, была подтверждена с использованием проточной цитометрии клеток HEK293T, экспрессирующих Spike-белок SARS-CoV-2 на своей поверхности (Фиг. 6, Табл. 1). Более того, ни одно из полученных моноклональных антител не было способно узнавать Spike-белки двух родственных альфакоронавирусов, HCoV-229E и HCoV-NL63 (Фиг. 6), в полном соответствии со значительной степенью эволюционной дивергенции их последовательностей RBD и использованной стратегией отбора В-лимфоцитов именно против RBD Spike-белка SARS-CoV-2.
На основании полученных данных с учетом генетического кода были определены аминокислотные последовательности вариабельных доменов тяжелых (SEQ ID NO: 139-161) и легких (SEQ ID NO: 162-184) цепей 23 моноклональных антител, специфичных к RBD Spike-белка SARS-CoV-2. Участки CDRs VH (SEQ ID NO: 1-69) и VL (SEQ ID NO: 70-138) были определены по номенклатуре Kabat [Kabat numbering system, Kabat et al., 1987 "Sequences of Proteins of Immunological Interest", US Dept. of Health and Human Services, US Government Printing Office].
Таким образом, были получены аминокислотные последовательности SEQ ID NO: 139-161 и SEQ ID NO: 162-184, а также кодирующие их нуклеотидные последовательности SEQ ID NO: 185-207 и SEQ ID NO: 208-230.
Проведенный биоинформатический анализ последовательностей этих антител против известных последовательностей антител к SARS-CoV-2 не выявил антител, на 100% совпадающих по последовательностям легкой и тяжелой цепи с антителами изобретения.
Пример 3. Определение вируснейтрализующей активности моноклональных антител человека in vitro.
Для оценки уровня вируснейтрализующей активности моноклональных антител использовали два подхода: суррогатный тест с использованием псевдотипированных лентивирусных частиц и микронейтрализационный тест с использованием аутентичного вируса SARS-CoV-2.
Для получения лентивирусных частиц, псевдотипированных Spike-белком SARS-CoV-2 проводили кальций-фосфатную трансфекцию клеток линии HEK293T смесью плазмид psPAX2, pLV-EGFP и pCAGGS-SpikeΔ19 в молярном соотношении 4:6:3. Плазмида pCAGGS-SpikeΔ19 кодировала укороченный с С-конца на 19 аминокислотных остатков Spike-белок, соответствующий исходному штамму Wuhan-Hu-1 SARS-CoV-2 или мутантным вариантам Spike SARS-CoV-2 (SΔ19). Через восемь часов после трансфекции среду заменяли на Opti-Mem (Gibco, Thermo Fisher Scientific) с добавлением 2,5% FBS (Gibco, Thermo Fisher Scientific). Супернатанты собирали два дня спустя, центрифугировали при 4000 об/мин в течение 5 минут, после чего лентивирусные частицы концентрировали путем центрифугирования при 20000× g, 4°C в течение 90 минут. Для проведения нейтрализационного теста в качестве мишеней использовали клетки линии ACE2-HEK293T со стабильной поверхностной экспрессией белка ACE2, рассаженные с плотностью 30000 клеток/лунку в 96-луночном штативе в день проведения теста. Антитела серийно двукратно разводили в среде Opti-MEM с добавлением 2,5% FBS в диапазоне от 0,5 нг/мл до 1 мкг/мл и ко-инкубировали с 20000 лентивирусных частиц, псевдотипированных Spike-белком SARS-CoV-2 в течение 30 мин при 37°C в объеме 100 мкл. Полученную смесь антител и лентивирусных частиц добавляли к клеткам ACE2-HEK293T и возвращали в CO2-инкубатор. Спустя 72 часа после трансдукции измеряли процент трансдуцированных (GFP+) клеток, используя проточный цитометр. Значение концентрации полумаксимального ингибирования IC50 определяли, используя нелинейную регрессию, как концентрацию, обеспечивавшую нейтрализацию 50% псевдотипированных лентивирусных частиц (Фиг. 7а).
Микронейтрализационный тест с использованием аутентичного вируса SARS-CoV-2 проводили, как описано в работе Nurtop и соавторов (Nurtop, E. et al. Combination of ELISA screening and seroneutralisation tests to expedite Zika virus seroprevalence studies. Virol. J. 15, 192 (2018)) в 96-луночных планшетах (Costar), используя в качестве мишеней клетки линии VeroE6, культивируемые в среде DMEM с добавлением 5% FBS. Вирусный супернатант (100 TCID50) смешивали с равным объемом серийных разведений моноклональных антител и инкубировали при 37°C в течение 1 часа. Затем полученные смеси вирус/антитело (100 мкл) переносили в лунки планшета с монослоем клеток линии VeroE6. Спустя 5-7 дней лунки c клетками анализировали визуально при помощи инвертированного микроскопа и выясняли IC100 (наименьшая концентрация антитела, обеспечивавшая полную защиту от заражения 100 TCID50 вируса SARS-CoV-2 (Фиг. 7б). Значения вируснейтрализующей активности моноклональных антител в обоих тестах in vitro приведены в Табл. 1. Таким образом, установлено, что моноклональные антитела iВ12, iВ15, iB14, iB3 являются ультрапотентными нейтрализаторами SARS-CoV-2 (IC100<16 нг/мл), вируснейтрализующая активность iB9, iB20, iB6, iB13, iB19, iB18, iB24, iB11, iB4 значительно ниже и находится в диапазоне 125 нг/мл – 25 мкг/мл, а остальные моноклональные RBD-специфические антитела не обладают выраженной вируснейтрализующей активностью (Табл. 1).
Пример 4. Определение конкурентных взаимоотношений между моноклональными антителами за связывание с эпитопами RBD.
Мы обратились к анализу на основе биослойной интерферометрии для выяснения попарной конкуренции 22 RBD-специфических моноклональных антител за связывание с RBD. С этой целью рекомбинантный RBD-His6 (14 мкг/мл) был иммобилизован на NTA-биосенсор, после чего к нему было добавлено насыщающее количество первого антитела (Ab1, 75 мкг/мл (500 нМ). Сформированный таким образом комплекс RBD-Ab1 затем инкубировали со вторым антителом, Ab2. В целом была использована следующая 6-шаговая схема: Baseline 60 с, Loading 200 с, Baseline2 30 с, Saturation Ab1 600 с, Competing Ab2 600 с, Regeneration в глицин-HCl, pH 1,7, 5 × 3 с.
Большинство нейтрализующих моноклональных антител формировали два крупных кластера (Фиг. 8). В первый кластер входили высокопотентные антитела iB3, iB12, iB14 и iB15, а также умереннопотентное iB13 и слабопотентное iB11. Кластер II состоял из умеренно- и слабопотентных антител iB9, iB17, iB18, iB19, iB20, iB24 и iB25. Нейтрализующее моноклональное антитело iB6 не принадлежало ни одному из этих кластеров. Ненейтрализующие антитела также формировали свой отдельный кластер.
Данный анализ был расширен с целью выяснения возможности комбинирования выделенных нами 9 лидирующих нейтрализующих антител с рядом известных нейтрализующих антител, в частности REGN10933, REGN10987, COVA2-15 и COV2-2504 (Brouwer, P. J. M. et al. Potent neutralizing antibodies from COVID-19 patients define multiple targets of vulnerability. Science 369, 643–650 (2020); Hansen, J. et al. Studies in humanized mice and convalescent humans yield a SARS-CoV-2 antibody cocktail. Science 369, 1010-1014 (2020). Zost, S. J. et al. Rapid isolation and profiling of a diverse panel of human monoclonal antibodies targeting the SARS-CoV-2 spike protein. Nat. Med. 26, 1422–1427 (2020)). Было обнаружено, что моноклональные нейтрализующие антитела iB20, iB6, и REGN10987 не конкурируют друг с другом и могут одновременно связываться с RBD, т.е. их эпитопы не перекрываются. Эпитопы остальных моноклональных антител в разной степени перекрываются друг с другом (Фиг. 9, Фиг. 10). В частности, моноклональные нейтрализующие антитела iB3, iB12, iB13, iB14, iB15, REGN10933 и COV2-2504 конкурируют друг с другом за связывание с RBD, однако различаются между собой в плане конкуренции с СOVA2-15 и REGN10987. Последние два антитела конкурируют с iB3 и iB12, но не с iB13 и iB14. В то же время, REGN10933, iB15 и COV2-2504 не обнаруживают конкуренции с REGN10987, но конкурируют с COVA2-15. Антитела iB3, iB12, iB13, iB14, iB15, REGN10933, СOV2-2504 и COVA2-15, связанные с RBD, предотвращают связывание iB9, iB19 и iB20. Однако при обратной постановке эксперимента блокирование оказывается неполным. Таким образом, проведенный конкурентный анализ свидетельствует о том, что 13 нейтрализующих моноклональных антител образуют 8 эпитопных групп: iB13/iB14, REGN10933/COV2-2504/iB15, iB3/iB12, iB9, COVA2-15, iB19/iB20, iB6 и REGN10987 (Фиг. 10), что открывает значительный простор для создания различных композиций неконкурирующих друг с другом вируснейтрализующих антител. Например, такие композиции могут быть образованы сочетаниями iB14 + COVA2-15, iB14 + REGN10987, iB20 + iB6, iB15 + REGN10987, COV2-2504 + REGN10987, iB20 + REGN10987 или iB6 + iB20 + REGN10987.
Пример 5. Вируснейтрализующая активность моноклональных антител и их комбинаций in vivo.
Для оценки вируснейтрализующей активности моноклональных антител и их комбинаций in vivo была использована модель инфекции SARS-CoV-2 на сирийских хомячках. Вначале была исследована активность iB12 и смеси iB6/iB20 (1:1) в качестве профилактики и/или терапии инфекции SARS-CoV-2 и вызванной этим вирусом легочной патологии (Фиг. 11, верхняя панель). Дополнительно был проведен независимый эксперимент с антителами iB14, поскольку оно было более устойчиво к некоторым из замен в последовательности Spike-белка у циркулировавших в 2020-2021 годах вариантов SARS-CoV-2 (Фиг. 11, нижняя панель). В профилактической схеме было сформировано семь групп животных (n = 6), которым внутрибрюшинно вводили моноклональные антитела: 10 мг/кг iB12 (группа 1), 1 мг/кг iB12 (группа 2), 10 мг/кг 1:1 смесь iB6 и iВ20 (группа 3), 1 мг/кг смесь iB6 и iВ20 (группа 4), 10 мг/кг суммарный иммуноглобулин IgG человека (группа 5, негативный контроль) за 24 часа до инфекции (–1 dpi). Животные, не получившие ни антител, ни вирус (группа 6), были использованы в качестве дополнительного негативного контроля. На следующий день (0 dpi) животных интаназально зараражали SARS-CoV-2 (50 мкл/нозд.) в суммарной дозе 1,3 × 103 БОЕ. В терапевтической схеме животные получали iB12 (10 мг/кг, внутрибрюшинно) 6 часов после заражения (группа 7). Животных взвешивали ежедневно. В каждой группе троих животных усыпляли на 5 день после заражения (5 dpi), легкие выделяли для оценки вирусной нагрузки и патолого-морфологического исследования. Также производили забор крови для измерения уровня иммуноглобулинов человека IgG в сыворотке животных. В эксперименте по оценке протективных свойств моноклонального антитела iB14 была проверена дополнительная временная точка, 24 ч после инфекции (6-7 животных на группу).
Как профилактическое, так и терапевтическое введение антител обладало выраженным эффектом. В то время как контрольные животные прогрессивно теряли в весе (снижение на 15% к 5 дню после заражения), все животные из экспериментальных групп прибавляли в весе, начиная с 3 дня после заражения (Фиг. 11А). Кроме того, измерение уровней вирусной нагрузки в гомогенатах легких хомяков (ртПЦР) свидетельствовало о том, что животные, получившие моноклональные антитела профилактически имели уровни вирусной РНК, сниженные по сравнению с контролем, на 2–6 порядков. Терапевтическое применение моноклональных антител iB12 и iB14 в дозировке 1 мг на животное также обладало протективным эффектом, выражавшимся в снижении вирусной нагрузки в легких (Фиг. 11Б). В легких контрольных животных были обнаружены признаки интерстициальной пневмонии, включая множественные перибронхиальные и периваскулярные очаги лейкоцитарной инфильтрации в сочетании с отеком, множественными некротическими изменениями в интерстиции и выраженной консолидацией легочной ткани (более 50%). Профилактическое введение iB12 (1 мг) обладало наибольшим протективным эффектом в отношении легких: у животных из этой группы уровень легочной патологии был минимальным. Снижение дозы моноклонального антитела iB12 (0,1 мг), использование смеси iB6 + iB20 (0,1 и 1 мг), а также терапевтическое введение iB12 предотвращало развитие пневмонии, хотя иногда и наблюдалось утолщение альвеолярных перегородок с незначительной гиперплазией альвеолярных эпителиоцитов второго типа или десквамацией эпителия бронхиол, а также периваскулярные отеки (Фиг. 11В, Фиг. 12). Терапевтическое введение сниженной дозы моноклонального антитела iB14 или его отложенное введение через 24 часа после заражения не отражалось на уровне вирусной РНК в легких хомяков, однако потеря веса у таких животных была значительно меньше, чем в контроле (Фиг. 11А,Б). Измерение уровня вирусной РНК в гомогенатах легких хомяков проводили с использованием количественной обратной ПЦР в реальном времени. Для этой цели половину левой доли легкого взвешивали, помещали в раствор LIRA (Биолабмикс) и замораживали. Образцы тщательно гомогенизировали и использовали для выделения РНК в соответствии с инструкциями производителя. Используя набор RevertAid cDNA synthesis kit (Thermo Scientific) и случайные гексамерные олигонуклеотиды, была получена кДНК с 1 мг суммарной РНК. Для проведения обратной ПЦР в реальном времени были использованы два набора праймеров, специфически узнающих последовательности генов RdRp и E SARS-CoV-2 (Corman, V. M. et al. Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveill. 25, 2000045 (2020)), а также нормировочные праймеры, соответствующие транскриптам гена Rpl18 хомяка (Zivcec, M., Safronetz, D., Haddock, E., Feldmann, H. & Ebihara, H. Validation of assays to monitor immune responses in the Syrian golden hamster (Mesocricetus auratus). J. Immunol. Methods 368, 24–35 (2011)).
Пример 6. Изучение чувствительности нейтрализующих моноклональных антител к разнообразным заменам в Spike-белка SARS-CoV-2, в том числе присутствующим в циркулирующих вирусных вариантах.
С целью выяснения возможной чувствительности нейтрализующих моноклональных антител к заменам в RBD Spike-белка SARS-CoV-2 с использованием направленного мутагенеза была получена панель экспрессионных конструкций, кодирующих 9 одноаминокислотных вариантов Spike-белка (K417N, N439K, K444Q, N460T, A475V, Е484K, F486K, F490L и N501Y) SARS-CoV-2. Такие конструкции были использованы для получения псевдотипированных лентивирусных частиц и проведения суррогатного теста на вируснейтрализацию с моноклональными нейтрализующими антителами iB3, iB6, iB9, iB12, iB14, iB15 и iB20), а также четырьмя референсными нейтрализующими антителами REGN10987, REGN10933, COVA2-15 и COV2-2504. Замена N501Y присутствует в нескольких циркулирующих вариантах вируса SARS-CoV-2, а именно в 501Y.V1 (клада B.1.1.7), 501Y.V2 (клада B.1.351) и 501Y.V3 (клада P.1). В последних двух случаях, N501Y встречается в сочетании с заменами K417N и E484K. В свою очередь замена E484K встречается в кладе B.1.525 и обусловливает устойчивость таких вирусов к некоторым известным нейтрализующим моноклональным антителам (Jones, B.E. et al. The neutralizing antibody, LY-CoV555, protects against SARS-CoV-2 infection in nonhuman primates. Sci. Transl. Med. 13, eabf1906 (2021); Hansen, J. et al. Studies in humanized mice and convalescent humans yield a SARS-CoV-2 antibody cocktail. Science 369, 1010–1014 (2020); Barnes, C.O. et al. Structures of human antibodies bound to SARS-CoV-2 spike reveal common epitopes and recurrent features of antibodies. Cell 182, 828–842.e16 (2020); Weisblum, Y. et al. Escape from neutralizing antibodies by SARS-CoV-2 spike protein variants. elife 9, e61312 (2020); Starr, T.N. et al. Prospective mapping of viral mutations that escape antibodies used to treat COVID-19. Science 371, 850–854 (2021); Baum, A. et al. Antibody cocktail to SARS-CoV-2 spike protein prevents rapid mutational escape seen with individual antibodies. Science 369, 1014–1018 (2020); Greaney, A.J. et al. Complete mapping of mutations to the SARS-CoV-2 spike receptor-binding domain that escape antibody recognition. Cell Host Microbe 29, 44–57.e9 (2021)).
Полученные нами данные свидетельствуют о том, что из исследованных вирусных вариантов три - Е484K, F486K и F490L обладают наибольшим негативным эффектом на нейтрализующую активность протестированных моноклональных антител (Фиг. 13А). В частности, вариант E484K не нейтрализовался 7 из 11 антител. Три антитела, - iB14, iB20 и REGN10987 сохраняли активность в отношении этого варианта, в то время как нейтрализующая активность REGN10933 была снижена в ~11 раз. F490L полностью блокировал активность iB12 и iB15, снижал активность iB3 и COV2-2504 примерно в 20 раз, и незначительно снижал активность iB9, iB20 и COVA2-15. Антитело iB20 теряло нейтрализующую активность по отношению к вариантам N460T и A475V. В соответствии с опубликованными данными замена K417N приводила к снижению нейтрализующей активности REGN10933, а варианты N439K и K444Q негативно влияли на активность REGN10987 (Starr, T.N. et al. Prospective mapping of viral mutations that escape antibodies used to treat COVID-19. Science 371, 850–854 (2021); Baum, A. et al. Antibody cocktail to SARS-CoV-2 spike protein prevents rapid mutational escape seen with individual antibodies. Science 369, 1014–1018 (2020)). Важно отметить, что ни одна из последних трех замен не влияла на нейтрализующую активность полученных нами моноклональных нейтрализующих антител. Из исследованных моноклональных антител лишь одно, COVA2-15, проявляло умеренную чувствительность к варианту N501Y в соответствии с опубликованными данными (Shen, X. et al. SARS-CoV-2 variant B.1.1.7 is susceptible to neutralizing antibodies elicited by ancestral Spike vaccines. Cell Host Microbe. 29, 529-539.e3 (2021)).
Затем мы проверили, сохраняют ли моноклональные антитела iB14 и iB20 нейтрализующую активность против вариантов Spike-белка с тремя заменами - K417, E484 и N501 (KEN) – в пределах RBD, поскольку данная комбинация замен встречается в двух кладах циркулирующих вариантов SARS-CoV-2, 501Y.V2 и 501Y.V3. В полном соответствии с данными, что индивидуальные замены приводят к устойчивости таких вариантов Spike к iB14 и iB20, а также к REGN10987, наблюдалось сохранение полной нейтрализующей активности этих трех антител по отношению к KEN-варианту Spike. Антитела iB6 и COVA2-15 при этом полностью теряли такую активность (Фиг. 13Б). Соответственно, композиции антитела iB6 с iB20 или REGN10987, а также антитела COVA2-15 с iB14 также полностью нейтрализовали данный вариант (Фиг. 14).
Наконец, отдельно была исследована способность моноклональных нейтрализующих антител iB6, iB14 и iB20 нейтрализовать лентивирусные частицы, псевдотипированные вариантами Spike-белка с заменами в RBD, присутствующими в кладах SARS-CoV-2 delta (B.1.617.2 (L452R + T478K)), kappa (B.1.617.1 (L452R + E484Q)) и lambda (C.37 (L452Q + F490S)), которые появились в циркуляции в 2020-2021 годах. Антитела iB14 и iB20 сохраняют активность на полном уровне в отношении этих трех вариантов вируса (Фиг. 13Б), в отличие от iB6, которое оказалось чувствительно к заменам по остатку L452. Таким образом, полученные данные свидетельствуют о возможности применения моноклональных антител iB14 и iB20 в качестве профилактики заражения и терапии пациентов, зараженных данными вариантами SARS-CoV-2.
Пример 7. Оценка способности RBD-специфических моноклональных антител узнавать RBD SARS-CoV-2, а также RBD циркулирующих вариантов вируса, delta и beta, в формате иммуноферментного анализа.
С целью верификации возможности использования полученных RBD-специфических рекомбинантных моноклональных антител в диагностических целях был проведен иммуноферментный анализ их связывания с RBD Spike-белка SARS-CoV-2, соответствующих трем его вариантам (исходный изолят Wuhan-Hu-1, delta (замены L452R + T478K) и beta (замены K417N+E484K+N501Y, KEN). Антиген (RBD-His6) был разбавлен в 0,1 M NaHCO3 до концентрации 3 мкг/мл и перенесен в объеме 100 мкл в лунки 96-луночного планшета повышенной сорбции, Greiner Bio-One, Германия. После инкубирования в течение ночи при 4°C раствор, содержащий антиген, удаляли, промывали 5 раз отмывочным буфером, содержащим 0,1 M NaHCO3 и 0,1% Triton X-100, и проводили блокировку свободных центров связывания с использованием 150 мкл 2% (w/v) обезжиренного сухого молока (Santa Cruz, США) в 0,1 M NaHCO3 в течение 1 часа при комнатной температуре. Затем лунки 5 раз промывали отмывочным буфером и добавляли двукратные разведения антител в диапазоне от 1 мкг/мл до 7,5 нг/мл в отмывочном буфере (100 мкл на лунку). По завершении инкубирования при комнатной температуре в течение 1 часа лунки промывали 5 раз отмывочным буфером. Затем в каждую лунку добавляли по 100 мкл конъюгата мышиных моноклональных антител против IgG человека с пероксидазой (Вектор, Кольцово) (разведение 1:12000) в отмывочном буфере. Через 1час инкубирования планшета при комнатной температуре лунки промывали пять раз отмывочным буфером и добавляли 100 мкл субстратного раствора для пероксидазы: 0,5 M Na-цитратный буфер, pH 5,2, содержащий 5 мг/мл орто-фенилендиамина и 0,01% H2O2. После развития цветной реакции в течение 5-10 минут окрашивание останавливали внесением 0,5 М серной кислоты (100 мкл на лунку). Измерения оптической плотности сформированного в результате реакции продукта проводили при длине волны 490 нм (OD490) с использованием планшетного спектрофотометра Multiskan. Специфичность по отношению к RBD SARS-CoV-2 исходного изолята Wuhan-Hu-1 была подтверждена для всех приведенных в Таблице 2 антител. Вместе с тем необходимо отметить, что в соответствии с данными экспериментов по изучению чувствительности нейтрализующих моноклональных антител к разнообразным заменам в Spike-белке SARS-CoV-2, в том числе и присутствующим в циркулирующих вирусных вариантах (Пример 6), в формате иммуноферментного анализа обнаруживалась неспособность некоторых из моноклональных антител детектировать варианты RBD, соответствующие последовательностям Spike в кладах delta и beta. Другими словами, использование нечувствительных к указанным заменам антител (например, iB0, iB14, iB20) позволяет детектировать SARS-CoV-2 исходного типа, delta и beta, а использование комбинаций антител, напротив, теряющих сродство к RBD delta и/или beta, позволяет дифференцировать/типировать такие образцы и отличать от исходного, немутированного варианта, при проведении иммуноферментного анализа.
антитела
(delta)
Таблица 2
Пример 8. Оценка способности RBD-специфических моноклональных антител узнавать RBD и S-белок SARS-CoV-2 в условиях иммуноблоттинга.
Важным свойством любого моноклонального антитела является его способность узнавать целевой антиген в условиях иммуноблоттинга, при которых данный антиген может быть полностью или частично денатурирован/ренатурирован. Как правило, антитела, узнающие линейные эпитопы, обладают способностью эффективно детектировать целевые антигены в условиях иммуноблотинга независимо от условий денатурации, а именно присутствия или отсутствия в системе восстановителей дисульфидных связей, таких как бета-меркаптоэтанол, дитиотреитол и т.д. Полученные в данной заявке антитела были протестированы на способность узнавать RBD и S-белок SARS-CoV-2 в иммуноблоттинге при добавлении или без добавления бета-меркаптоэтанола. Данные, полученные в результате этих экспериментов, суммированы в Табл. 3, и свидетельствуют о том, что в отсутствие восстановителя большинство протестированных RBD-специфических моноклональных антител способно распознавать RBD и S-белок SARS-CoV-2, т.е. такие агенты обладают потенциалом для использования в качестве инструмента детекции. В то же время необходимо отметить, что в денатурирующих условиях ни одно из антител не узнает RBD или S-белок, что косвенно указывает на то, что сайты связывания полученных антител представлены сложными конформационными эпитопами.
Таблица 3
Пример 9. Оценка уровня аутореактивности RBD-специфических моноклональных антител.
Чтобы узнать, обладают ли полученные моноклональные RBD-специфические антитела аутореактивностью, т.е. способностью связываться с антигенами человека, как это было показано для некоторых SARS-CoV-2-специфических антител (Kreer, C. et al. Longitudinal isolation of potent near-germline SARS-CoV-2-neutralizing antibodies from COVID-19 patients. Cell 182, 843–854.e12 (2020); Kreye, J. et al. A therapeutic non-self-reactive SARS-CoV-2 antibody protects from lung pathology in a COVID-19 hamster model. Cell 183, 1058–1069.e19 (2020)), был использован протокол иммуноокрашивания клеток линии HEp-2 с последующей детекцией при помощи проточной цитометрии. Для этого клетки данной линии открепляли от поверхности культурального пластика используя раствор Версена, трижды промывали PBS и фиксировали в 2% параформальдегиде на льду в течение 10 минут. Затем для пермеабилизации клеток к ним добавляли сапонин до финальной концентрации 0,05%. В качестве позитивных контролей использовали первичные антитела против актина (ab3280, Abcam) и BIP (ab21685, Abcam). Для всех последующих шагов иммуноокрашивания использовали FACS-буфер с сапонином. Суспензии клеток инкубировали с антителами (5 мкг/мл) в течение получаса, трижды отмывали, после чего к клеткам добавляли APC-конъюгированные антитела donkey-anti-human IgG (1:600) (Jackson ImmunoResearch), goat-anti-rabbit-Alexa488 (1:600, Thermo Fisher Scientific), или goat-anti-mouse-Alexa488 (1:600, Thermo Fisher Scientific). Клетки без добавления первичных антител (но с добавлением конъюгатов) использовали для определения уровня фоновой флуоресценции. Уровень аутореактивности рассчитывали по формуле: MFI(антитело)–MFI(фон)/MFI(фон) (Табл. 1).
--->
Sequence Listing Information:
DTD Version: V1_3
File Name: R211040 sequence listing.xml
Software Name: WIPO Sequence
Software Version: 1.1.0
Production Date: 2021-12-28
General Information:
Current application / Applicant file reference: R21100410
Applicant name: Общество с ограниченной ответственностью "Имген+"
Applicant name / Language: ru
Applicant name / Name Latin: Obshchestvo s ogranichennoj otvetstvennostju
"Imgen+"
Invention title: Моноклональные антитела против рецептор-связывающего
домена SPIKE-белка вируса SARS-COV-2 и их антигенсвязывающие фрагменты,
кодирующие их нуклеиновые кислоты, а также способы применения (Ru)
Sequence Total Quantity: 322
Sequences:
Sequence Number (ID): 1
Length: 11
Molecule Type: AA
Residues:
GFTFSSYA 8
Sequence Number (ID): 2
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GFSFNTYS 8
Sequence Number (ID): 3
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GGSLSGYY 8
Sequence Number (ID): 4
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GYTFTGYY 8
Sequence Number (ID): 5
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GYTFTGYY 8
Sequence Number (ID): 6
Length: 10
Molecule Type: AA
Residues:
GGSISSGDYY 10
Sequence Number (ID): 7
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GGTFITYA 8
Sequence Number (ID): 8
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GGTFSNYA 8
Sequence Number (ID): 9
Length: 11
Molecule Type: AA
Residues:
GFTFSDYY 8
Sequence Number (ID): 10
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GYTFTSYY 8
Sequence Number (ID): 11
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GYLFTGYY 8
Sequence Number (ID): 12
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GFTFSSYA 8
Sequence Number (ID): 13
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GFTFPSSA 8
Sequence Number (ID): 14
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GYTFTNYD 8
Sequence Number (ID): 15
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GGSISSSSYY 10
Sequence Number (ID): 16
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GYTFTSYY 8
Sequence Number (ID): 17
Length: 11
Molecule Type: AA
Residues:
GFTVSSNY 8
Sequence Number (ID): 18
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
EFTVSSNY 8
Sequence Number (ID): 19
Length: 11
Molecule Type: AA
Residues:
GFTVSRNY 8
Sequence Number (ID): 20
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GFTFSSYA 8
Sequence Number (ID): 21
Length: 11
Molecule Type: AA
Residues:
GFTVSSNY 8
Sequence Number (ID): 22
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GLTVSSNY 8
Sequence Number (ID): 23
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH1
> ORGANISM, synthetic construct
Residues:
GFTFSSYG 8
Sequence Number (ID): 24
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
ISYDGSNK 8
Sequence Number (ID): 25
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
ISSSGSIL 8
Sequence Number (ID): 26
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
ITQSGGT 7
Sequence Number (ID): 27
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
INPNSGAT 8
Sequence Number (ID): 28
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
INPNSGGT 8
Sequence Number (ID): 29
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IFYSGST 7
Sequence Number (ID): 30
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IIPILGIT 8
Sequence Number (ID): 31
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IIPILGIA 8
Sequence Number (ID): 32
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
ISSSGSTI 8
Sequence Number (ID): 33
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
INPSGGST 8
Sequence Number (ID): 34
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
INPNSGGT 8
Sequence Number (ID): 35
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
VSGSGGST 8
Sequence Number (ID): 36
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IVVGSGNT 8
Sequence Number (ID): 37
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
MNPNSGNT 8
Sequence Number (ID): 38
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IYYSGST 7
Sequence Number (ID): 39
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
INPSGGST 8
Sequence Number (ID): 40
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IYSGGST 7
Sequence Number (ID): 41
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IYSGGST 7
Sequence Number (ID): 42
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IYSGGST 7
Sequence Number (ID): 43
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
ISGSGGST 8
Sequence Number (ID): 44
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IYSGGST 7
Sequence Number (ID): 45
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
IYSGGST 7
Sequence Number (ID): 46
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH2
> ORGANISM, synthetic construct
Residues:
ISYDGSNK 8
Sequence Number (ID): 47
Length: 20
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..20
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARARPWRYSY GLYYYYGMDV 20
Sequence Number (ID): 48
Length: 20
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..20
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARVGTRVGAS NFDYYYGMDV 20
Sequence Number (ID): 49
Length: 17
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..17
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARGGYDDNRG YYAYFDY 17
Sequence Number (ID): 50
Length: 28
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..28
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARVAQVVSNP LPYDVLTGLT YYYYGMDV 28
Sequence Number (ID): 51
Length: 14
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..14
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARDYYYDSSV NPDY 14
Sequence Number (ID): 52
Length: 15
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..15
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARRCRFVDTS MAFDY 15
Sequence Number (ID): 53
Length: 14
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..14
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARGGRGANPG WFDY 14
Sequence Number (ID): 54
Length: 16
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..16
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARTAYEYDSS GYFLDY 16
Sequence Number (ID): 55
Length: 13
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..13
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARHPMVRGVI GDY 13
Sequence Number (ID): 56
Length: 18
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..18
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARGHSGYDRL PQPPYFDY 18
Sequence Number (ID): 57
Length: 14
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..14
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARSGYYTSAV NFDY 14
Sequence Number (ID): 58
Length: 25
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..25
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
AKTVLAGRDD SSGYFTVYYY FGMDV 25
Sequence Number (ID): 59
Length: 16
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..16
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
AAPYCSGGSC YDGFDI 16
Sequence Number (ID): 60
Length: 24
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..24
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARAYQNRPIA VTDMGYYYYY GMDV 24
Sequence Number (ID): 61
Length: 16
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..16
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARGGSIYDYG DYEFDS 16
Sequence Number (ID): 62
Length: 14
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..14
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
AILGLHLGEL LFDP 14
Sequence Number (ID): 63
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARDFGEQWFD Y 11
Sequence Number (ID): 64
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARDFGDYYFD Y 11
Sequence Number (ID): 65
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARDLLEVGGS DY 12
Sequence Number (ID): 66
Length: 21
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..21
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
AKPLLGYDFW SGYYRGGWFD P 21
Sequence Number (ID): 67
Length: 13
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..13
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARDPYSYGRG GGY 13
Sequence Number (ID): 68
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
ARDGVYYGMD V 11
Sequence Number (ID): 69
Length: 17
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..17
> MOL_TYPE, protein
> NOTE, CDRH3
> ORGANISM, synthetic construct
Residues:
AKDYDFWSGY YPQPFDY 17
Sequence Number (ID): 70
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSISSY 6
Sequence Number (ID): 71
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
SSNIGRNT 8
Sequence Number (ID): 72
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSVLYSFRNK IC 12
Sequence Number (ID): 73
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
SSDVGSYNL 9
Sequence Number (ID): 74
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QDISNY 6
Sequence Number (ID): 75
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
SSDVGSYDL 9
Sequence Number (ID): 76
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSISSW 6
Sequence Number (ID): 77
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSVSSSY 7
Sequence Number (ID): 78
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
SSNIGSNT 8
Sequence Number (ID): 79
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSISKY 6
Sequence Number (ID): 80
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QDISNY 6
Sequence Number (ID): 81
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
SSDIGGYNY 9
Sequence Number (ID): 82
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSVSSSY 7
Sequence Number (ID): 83
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
SSDVGSYNL 9
Sequence Number (ID): 84
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSISSW 6
Sequence Number (ID): 85
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QDISNY 6
Sequence Number (ID): 86
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSVSSSY 7
Sequence Number (ID): 87
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSVSSSY 7
Sequence Number (ID): 88
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSISSSY 7
Sequence Number (ID): 89
Length: 15
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..15
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QSVLYSSNNK NY 12
Sequence Number (ID): 90
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QDISNY 6
Sequence Number (ID): 91
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QGISSY 6
Sequence Number (ID): 92
Length: 9
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..9
> MOL_TYPE, protein
> NOTE, CDRL1
> ORGANISM, synthetic construct
Residues:
QGIRND 6
Sequence Number (ID): 93
Residues:
000
Sequence Number (ID): 94
Residues:
000
Sequence Number (ID): 95
Residues:
000
Sequence Number (ID): 96
Residues:
000
Sequence Number (ID): 97
Residues:
000
Sequence Number (ID): 98
Residues:
000
Sequence Number (ID): 99
Residues:
000
Sequence Number (ID): 100
Residues:
000
Sequence Number (ID): 101
Residues:
000
Sequence Number (ID): 102
Residues:
000
Sequence Number (ID): 103
Residues:
000
Sequence Number (ID): 104
Residues:
000
Sequence Number (ID): 105
Residues:
000
Sequence Number (ID): 106
Residues:
000
Sequence Number (ID): 107
Residues:
000
Sequence Number (ID): 108
Residues:
000
Sequence Number (ID): 109
Residues:
000
Sequence Number (ID): 110
Residues:
000
Sequence Number (ID): 111
Residues:
000
Sequence Number (ID): 112
Residues:
000
Sequence Number (ID): 113
Residues:
000
Sequence Number (ID): 114
Residues:
000
Sequence Number (ID): 115
Residues:
000
Sequence Number (ID): 116
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQSYSTPGS 9
Sequence Number (ID): 117
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
AAWDDSLNGL V 11
Sequence Number (ID): 118
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQFYSTPWT 9
Sequence Number (ID): 119
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
CSYADSRTLV 10
Sequence Number (ID): 120
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYDNLPPRV T 11
Sequence Number (ID): 121
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
CSFAGSSPL 9
Sequence Number (ID): 122
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYNSYSPM 9
Sequence Number (ID): 123
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
HQYGSSPRT 9
Sequence Number (ID): 124
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
AAWDDSLNGY V 11
Sequence Number (ID): 125
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQSYSTPPT 9
Sequence Number (ID): 126
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYDNLPPRL T 11
Sequence Number (ID): 127
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
SSYTRSSTQV V 11
Sequence Number (ID): 128
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYGTSPWT 9
Sequence Number (ID): 129
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
CSYARISTPG FV 12
Sequence Number (ID): 130
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYNSYST 8
Sequence Number (ID): 131
Length: 13
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..13
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYDNLLGVT 10
Sequence Number (ID): 132
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYGSSPRT 9
Sequence Number (ID): 133
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYGSSPRT 9
Sequence Number (ID): 134
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYGSSPPIT 10
Sequence Number (ID): 135
Length: 10
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..10
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYYSTLLLT 10
Sequence Number (ID): 136
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QQYDNLPIT 9
Sequence Number (ID): 137
Length: 11
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..11
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
QHLNSYPSMY T 11
Sequence Number (ID): 138
Length: 12
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..12
> MOL_TYPE, protein
> NOTE, CDRL3
> ORGANISM, synthetic construct
Residues:
LQDYNYPRT 9
Sequence Number (ID): 139
Length: 127
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..127
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG VVQPGRSLRL SCAASGFTFS SYAMHWVRQA PGKGLEWVAV ISYDGSNKYY 60
ADSVKGRFTI SRDNSKNTLY LQMNSLRAED TAVYYCARAR PWRYSYGLYY YYGMDVWGQG 120
TTVTVSS 127
Sequence Number (ID): 140
Length: 127
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..127
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG LVQSGGSLRL SCAASGFSFN TYSMNWVRQA PGKGLEWVSY ISSSGSILHY 60
ADSVKGRLTI SRDNAKNSLY LQMNSLRAED TAVYYCARVG TRVGASNFDY YYGMDVWGQG 120
TTVTVSS 127
Sequence Number (ID): 141
Length: 123
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..123
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLRQWGSG LLKPSETLSL TCAVYGGSLS GYYWSYFRQP PGKGLEWIGE ITQSGGTNYN 60
PSLKSRITIS VDTSKNQFSL DLSSVTAADT AVYYCARGGY DDNRGYYAYF DYWGQGTLVT 120
VSS 123
Sequence Number (ID): 142
Length: 135
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..135
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT GYYIHWVRQA PGQGLEWMGW INPNSGATNY 60
AQKFQGRVTV TRDTSISTAY MELSRLRSDD TAVYYCARVA QVVSNPLPYD VLTGLTYYYY 120
GMDVWGQGTT VTVSS 135
Sequence Number (ID): 143
Length: 121
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..121
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT GYYMHWVRQA PGQGLEWMGW INPNSGGTNY 60
AQKFQGRVTM TRDTSITTAY MELSRLRSDD TAVYYCARDY YYDSSVNPDY WGQGTLVTVS 120
S 121
Sequence Number (ID): 144
Length: 123
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..123
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLQESGPG LVKPSQTLSL TCTVSGGSIS SGDYYWSWIR QHPGKGLEWI GYIFYSGSTY 60
YNPSLKSRVT ISVDTSKNQF SLKLSSVTAA DTAVYYCARR CRFVDTSMAF DYWGQGTLVT 120
VSS 123
Sequence Number (ID): 145
Length: 121
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..121
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGSSVKV SCKASGGTFI TYAFSWVRQA PGQGLEWMGR IIPILGITNY 60
AQKFQGRVTI TADKSTSTAY MELSSLRSED TAVYYCARGG RGANPGWFDY WGQGTLVTVS 120
S 121
Sequence Number (ID): 146
Length: 123
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..123
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGSSVKV SCKASGGTFS NYAISWVRQA PGQGLEWMGR IIPILGIANY 60
AQKFQGRLTI TADKSTSTAY MELSSLRSED TAVYYCARTA YEYDSSGYFL DYWGQGTLVT 120
VSS 123
Sequence Number (ID): 147
Length: 120
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..120
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLLESGGG LVKPGGSLRL SCAASGFTFS DYYMSWIRQA PGKGLEWVSY ISSSGSTIYY 60
ADSVKGRFTI SRDNAKNSLY LQMNSLRAED TAVYYCARHP MVRGVIGDYW GQGTLVTVSS 120
Sequence Number (ID): 148
Length: 125
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..125
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT SYYMHWVRQA PGQGLEWMGI INPSGGSTSY 60
VQKFQGRVTM TRDTSTSTVY MELSSLRFED TAVYYCARGH SGYDRLPQPP YFDYWGQGTL 120
VTVSS 125
Sequence Number (ID): 149
Length: 121
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..121
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYLFT GYYMHWVRQA PGQGLEWMGW INPNSGGTNY 60
AQKFQDRVTM TRDTSITTAY MELSRLRSDD TAVYYCARSG YYTSAVNFDY WGQGTLVTVS 120
S 121
Sequence Number (ID): 150
Length: 132
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..132
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLLESGGG LVQPGGSLRL SCAASGFTFS SYAMSWVRQA PGKGLEWVSA VSGSGGSTYY 60
ADSVKGRFTI SRDNSKNMLY LQMNSLRAED TAVYYCAKTV LAGRDDSSGY FTVYYYFGMD 120
VWGQGTTVTV SS 132
Sequence Number (ID): 151
Length: 123
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..123
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGPE VKKPGTSVKV SCKASGFTFP SSAVQWVRQA RGQRLEWIGW IVVGSGNTNY 60
AQKFQERVTI TRDMSTSTAY MELSSLRSED TAVYYCAAPY CSGGSCYDGF DIWGQGTMVT 120
VSS 123
Sequence Number (ID): 152
Length: 131
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..131
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT NYDINWVRQA TGQGLEWMGW MNPNSGNTGF 60
AQKFQGRVTM TRNTSIRTVY MELSSLRSED TAVYYCARAY QNRPIAVTDM GYYYYYGMDV 120
WGQGTTVTVS S 131
Sequence Number (ID): 153
Length: 124
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..124
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QLQLQESGPG LVKPSETLSL TCTVSGGSIS SSSYYWGWIR QPPGKGLEWI GSIYYSGSTY 60
YNPSLKSRVT ISVDTSKNQF SLKLSSVTAA DTALYYCARG GSIYDYGDYE FDSWGQGTLV 120
TVSS 124
Sequence Number (ID): 154
Length: 121
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..121
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT SYYMHWVRQA PGQGLEWMGI INPSGGSTSY 60
AQKFQGRVTM TRDTSTSTVY MELSSLRSED TAVYYCAILG LHLGELLFDP WGQGTLVTVS 120
S 121
Sequence Number (ID): 155
Length: 117
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..117
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG LIQPGGSLRL SCAASGFTVS SNYMSWVRQA PGKGLEWVSV IYSGGSTFYA 60
DSVKGRFTIS RDNSKNTLYL QMNSLRAEDT AVYYCARDFG EQWFDYWGQG TLVTVSS 117
Sequence Number (ID): 156
Length: 117
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..117
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG LVQPGGSLRL SCAASEFTVS SNYMSWVRQA PGKGLEWVSV IYSGGSTYYA 60
DSVKGRFTIS RDNSKNTLYL QMNSLRAEDT AVYYCARDFG DYYFDYWGQG TLVTVSS 117
Sequence Number (ID): 157
Length: 118
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..118
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLVETGGG LIQPGGSLRL SCAASGFTVS RNYMSWVRQA PGKGLEWVSL IYSGGSTFYA 60
DSVKGRFTIS RDDSKNTLYL QMNSLRAEDT AVYYCARDLL EVGGSDYWGQ GTLVTVSS 118
Sequence Number (ID): 158
Length: 128
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..128
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLLESGGG LVQPGGSLRL SCAASGFTFS SYAMSWVRQA PGKGLEWVSA ISGSGGSTYY 60
ADSVKGRFTI SRDNSKNTLY LQMNSLRAED TAVYYCAKPL LGYDFWSGYY RGGWFDPWGQ 120
GTLVTVSS 128
Sequence Number (ID): 159
Length: 119
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..119
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG LIQPGGSLRL SCAASGFTVS SNYMSWVRQA PGKGLEWVSV IYSGGSTYYA 60
DSVKGRFIIS RDNSKNTLYL QMNSLRAEDT AVYYCARDPY SYGRGGGYWG QGTLVTVSS 119
Sequence Number (ID): 160
Length: 117
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..117
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLVETGGG LIQPGGSLRL SCAASGLTVS SNYMNWVRQA PGKGLEWVSV IYSGGSTYYA 60
DSVKGRFTIS RDNSKNTLYL QMNSLRAEDT AVYYCARDGV YYGMDVWGQG TTVTVSS 117
Sequence Number (ID): 161
Length: 124
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..124
> MOL_TYPE, protein
> NOTE, VH
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG VVQPGRSLRL SCAASGFTFS SYGMHWVRQA PGKGLEWVAV ISYDGSNKYY 60
ADSVKGRFTI SRDNSKNTLY LQMNSLRAED TAVYYCAKDY DFWSGYYPQP FDYWGQGTLV 120
TVSS 124
Sequence Number (ID): 162
Length: 107
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..107
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCRASQSIS SYLNWYQQKP GKAPKLLIYA ASSLQSGVPS 60
RFSGSGSGTD FTLTISSLQP EDFATYYCQQ SYSTPGSFGQ GTKLEIK 107
Sequence Number (ID): 163
Length: 110
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..110
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
QSVLTQPPSA SGTPGQRVTI SCSGSSSNIG RNTVNWYQQL PGTAPKLVIY SNNQRPSGVP 60
DRFSGSKSGT SASLAISGLQ SEDEADYYCA AWDDSLNGLV FGGGTKLTVL 110
Sequence Number (ID): 164
Length: 113
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..113
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIVMTQSPDS LAVSLGERAT INCKSSQSVL YSFRNKICLA WYQQKPGQPP KLLIYWATTR 60
ESGVPDRFSG SGSGTDFTLT ISSLQAEDVA VYYCQQFYST PWTFGQGTKV EIK 113
Sequence Number (ID): 165
Length: 110
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..110
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
QSALTQSASV SGSPGQPITI SCTGTSSDVG SYNLVSWYQQ HPGKAPKLMI YEVSKRPSGV 60
SNRFSGSKSG NTASLTISGL QAEDEADYYC CSYADSRTLV FGGGTKLTVL 110
Sequence Number (ID): 166
Length: 109
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..109
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD ASNLETGVPS 60
RFSGSGSGTD FTFTISSLQP EDIATYYCQQ YDNLPPRVTF GQGTKVEIK 109
Sequence Number (ID): 167
Length: 109
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..109
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
QSALTQPASV SGSPGQSITI SCTGTSSDVG SYDLVSWYQQ HPGKAPKLMI YEVTKRPSGV 60
SNRFSGSKSG NTASLTISGL QAEDEADYYC CSFAGSSPLF GGGTKLTVL 109
Sequence Number (ID): 168
Length: 107
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..107
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPST LSASVGDRVT ITCRASQSIS SWLAWYQQKP GKAPKVLIYK ASSLESGVPS 60
RFSGSGSGTE FTLTISSLQP DDFATYYCQQ YNSYSPMFGQ GTKVEIK 107
Sequence Number (ID): 169
Length: 108
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..108
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQK PGQAPRLLIY GASSRATGIP 60
DRFSGSGSGT DFTLTISRLE PEDFAVYYCH QYGSSPRTFG QGTKVEIK 108
Sequence Number (ID): 170
Length: 110
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..110
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
QSVLTQPPSA SGTPGQRVTI SCSGSSSNIG SNTVNWYQQL PGTAPKLLIY NNNQRPSGVP 60
DRFSGSKSGT SASLAISGLQ SEDEADYYCA AWDDSLNGYV FGTGTKVTVL 110
Sequence Number (ID): 171
Length: 107
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..107
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSTSVGDRVT ITCRASQSIS KYLNWYQQKP GKAPKLLIYA ASSLQSGVPS 60
RFSGSGSGTD FTLTISSLQP EDFATYYCQQ SYSTPPTFGQ GTKVEIK 107
Sequence Number (ID): 172
Length: 109
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..109
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD ASNLETGVPS 60
RFSGSGSGTD FTFTISSLQP EDIATYYCQQ YDNLPPRLTF GGGTKVEIK 109
Sequence Number (ID): 173
Length: 111
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..111
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
QSALTQPASV SGSPGQSITI SCTGTSSDIG GYNYVSWYQQ HPGKAPKLMI YDVSNRPSGV 60
SNRFSGSKSG NTASLTISGL QAEDEAEYYC SSYTRSSTQV VFGGGTKLTV L 111
Sequence Number (ID): 174
Length: 108
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..108
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQK PGQAPRLLIY GASSRATGIP 60
DRFSGSGSGT DFTLTISRLE PEDFAVYHCQ QYGTSPWTFG QGTKVEIK 108
Sequence Number (ID): 175
Length: 112
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..112
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
QSALTQPASV SGSPGQSITI SCTGTSSDVG SYNLVSWYQQ HPGKAPKLMI YEVSKRPSGV 60
SNRFSGSKSG NTASLTISGL QAEDEADYYC CSYARISTPG FVFGTGTKVT VL 112
Sequence Number (ID): 176
Length: 106
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..106
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPST LSASVGDRVT ITCRASQSIS SWLAWYQQKP GKAPKLLIYK ASNLESGVPS 60
RFSGSGSGTE FTLTISSLQP DDFATYYCQQ YNSYSTFGQG TKVEIK 106
Sequence Number (ID): 177
Length: 108
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..108
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASLGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD ASNLQTGVPS 60
RFSGGGSGTD FTFTISSLQP EDIATYYCQQ YDNLLGVTFG PGTKVDIK 108
Sequence Number (ID): 178
Length: 108
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..108
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQS PGQAPRLLIY GTSSRATGIP 60
DRFSGSGSGT DFTLTISRLE PEDFAVYYCQ QYGSSPRTFG QGTKVEIK 108
Sequence Number (ID): 179
Length: 108
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..108
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQK PGQAPRLLIY GASSRATGIP 60
DRFSGSGSGT DFTLTISRLE PEDFAVYYCQ QYGSSPRTFG QGTKVEIK 108
Sequence Number (ID): 180
Length: 109
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..109
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSIS SSYLAWYQQK PGQAPRLLFY GASSRATGIP 60
DRFSGSGSGT DFTLTISKLE PEDFAVYYCQ QYGSSPPITF GQGTRLEIK 109
Sequence Number (ID): 181
Length: 114
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..114
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIVMTQSPDS LAVSLGERAT INCKSSQSVL YSSNNKNYLA WYQQKPGQPP KLLIYWASTR 60
ESGVPDRFSG SGSGTDFTLT ISSLQAEDVA VYYCQQYYST LLLTFGGGTK VEIK 114
Sequence Number (ID): 182
Length: 107
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..107
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD ASNLETGVPS 60
RFSGSGSGTD FTFTISSLQP EDIATYYCQQ YDNLPITFGQ GTRLEIK 107
Sequence Number (ID): 183
Length: 109
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..109
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQLTQSPSF LSASVGDRVT ITCRASQGIS SYLAWYQQKP GKAPKLLIFA ASTLQSGVPS 60
RFSASGSGTE FTLTISSLQP EDFATYYCQH LNSYPSMYTF GQGTKLEIK 109
Sequence Number (ID): 184
Length: 107
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..107
> MOL_TYPE, protein
> NOTE, VL
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCRASQGIR NDLGWYQQKP GKAPKLLIYA ASSLQSGVPS 60
RFSGSGSGTD FTLTISSLQP EDFATYYCLQ DYNYPRTFGQ GTKVEIK 107
Sequence Number (ID): 185
Length: 381
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..381
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatgcta tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcgtatg atggaagtaa taaatactac 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gagagcccgt 300
ccgtggagat acagctatgg cctctactac tactacggta tggacgtctg gggccaaggg 360
accacggtca ccgtctcctc a 381
Sequence Number (ID): 186
Length: 381
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..381
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tggtggagtc tgggggaggc ttggtacagt cgggggggtc cctgagactc 60
tcctgtgcag cctctggatt cagcttcaat acctatagca tgaactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtttcatac attagcagta gtggtagtat cttgcactac 180
gcagactctg tgaagggccg actcaccatt tccagagaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acggctgtgt attactgtgc gagagtgggg 300
acccgagtgg gagcttcaaa cttcgactac tactacggta tggacgtctg gggccaaggg 360
accacggtca ccgtctcctc a 381
Sequence Number (ID): 187
Length: 369
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..369
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tacgccagtg gggctcagga ctgttgaagc cttcggagac cctgtccctc 60
acctgcgctg tgtatggtgg gtctctaagt ggttactact ggagctactt ccgccagccc 120
ccagggaagg ggctggagtg gattggggaa atcactcaaa gtggcggcac caactacaac 180
ccgtccctca agagtcggat cacaatatca gtggacacgt ccaagaatca gttctctttg 240
gacctgagct ctgtgaccgc cgcggacacg gctgtgtatt actgtgcgag aggcggatac 300
gatgataata gaggttatta cgcgtatttt gactactggg gccagggaac cctggtcacc 360
gtctcctca 369
Sequence Number (ID): 188
Length: 405
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..405
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tacactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgc cacaaactat 180
gcacagaagt ttcagggcag ggtcaccgtg accagggaca cgtccatcag cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagttgcc 300
caagtcgtgt ccaacccact gccttacgat gttttgactg gtttaaccta ttactactac 360
ggtatggacg tctggggcca agggaccacg gtcaccgtct cctca 405
Sequence Number (ID): 189
Length: 363
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..363
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc ggctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcagggcag ggtcaccatg accagggaca cgtccatcac cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc gagagactac 300
tactatgata gtagtgtgaa ccctgactac tggggccagg gaaccctggt caccgtctcc 360
tca 363
Sequence Number (ID): 190
Length: 369
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..369
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tgcaggagtc gggcccagga ctggtgaagc cttcacagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagc agtggtgatt actactggag ctggatccgc 120
cagcacccag ggaagggcct ggagtggatt gggtacatct tttacagtgg gagcacctac 180
tacaacccgt ccctcaagag tcgggttacc atatcagtag acacgtctaa gaaccagttc 240
tccctgaagc tgagctctgt gactgccgcg gacacggccg tgtattactg tgcgaggagg 300
tgccgttttg tagatacatc tatggcattt gactactggg gccagggaac cctggtcacc 360
gtctcctca 369
Sequence Number (ID): 191
Length: 363
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..363
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcatc acctatgctt tcagctgggt acgacaggcc 120
cctggacaag ggcttgagtg gatgggaagg atcatcccta tccttggtat aacaaactac 180
gcacagaagt tccagggcag agtcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagaggggga 300
cgcggtgcta accccggttg gtttgactac tggggccagg gaaccctggt caccgtctcc 360
tca 363
Sequence Number (ID): 192
Length: 369
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..369
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctgggtcctc ggtgaaggtc 60
tcctgcaagg cttctggagg caccttcagc aactatgcta tcagctgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaagg atcatcccta tccttggtat agcaaactac 180
gcacagaagt tccagggcag actcacgatt accgcggaca aatccacgag cacagcctac 240
atggagctga gcagcctaag atctgaggac acggccgtgt attactgtgc gaggacggcg 300
tatgaatatg atagtagtgg ttatttcctt gactactggg gccagggaac cctggtcacc 360
gtctcctca 369
Sequence Number (ID): 193
Length: 360
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..360
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tgttggagtc tgggggaggc ttggtcaagc ctggagggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt gactactaca tgagctggat ccgccaggct 120
ccagggaagg ggctggagtg ggtttcatac attagtagta gtggtagtac catatactac 180
gcagactctg tgaagggccg attcaccatc tccagggaca acgccaagaa ctcactgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtgt attactgtgc gaggcatcct 300
atggttcggg gagttatcgg agactactgg ggccagggaa ccctggtcac cgtctcctca 360
Sequence Number (ID): 194
Length: 375
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..375
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg catctggata caccttcacc agctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaata atcaacccta gtggaggtag cacaagctac 180
gtacagaagt tccagggcag agtcaccatg accagggaca cgtccacgag cacagtctac 240
atggagctga gcagcctgag atttgaggac acggccgtgt attactgtgc gagaggtcat 300
agtggctacg atcggttacc tcaaccccct tactttgact actggggcca gggaaccctg 360
gtcaccgtct cctca 375
Sequence Number (ID): 195
Length: 363
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..363
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata cctcttcacc ggctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggatgg atcaacccta acagtggtgg cacaaactat 180
gcacagaagt ttcaggacag ggtcaccatg accagggaca cgtccatcac cacagcctac 240
atggagctga gcaggctgag atctgacgac acggccgtgt attactgtgc caggagtggt 300
tattatacga gtgctgtaaa ctttgactac tggggccagg gaaccctggt caccgtctcc 360
tca 363
Sequence Number (ID): 196
Length: 396
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..396
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagc agctatgcca tgagttgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct gttagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa catgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaaaccgtt 300
ttggctggtc gcgacgatag cagtggctac ttcacggttt actactactt cggtatggac 360
gtctggggcc aggggaccac ggtcaccgtc tcctca 396
Sequence Number (ID): 197
Length: 369
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..369
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tgggcctgag gtgaagaagc ctgggacctc agtgaaggtc 60
tcctgcaagg cttctggatt cacctttcct agctctgctg tgcagtgggt gcgacaggct 120
cgtggacaac gccttgagtg gataggatgg atcgtcgttg gcagtggtaa cacaaactac 180
gcacagaagt tccaggaaag agtcaccatt accagggaca tgtccacaag cacagcctac 240
atggagctga gcagcctgag atccgaggac acggccgtgt attactgtgc ggctccctat 300
tgtagtggtg gtagctgtta tgatggtttt gatatctggg gccaagggac aatggtcacc 360
gtctcttca 369
Sequence Number (ID): 198
Length: 393
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..393
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtc 60
tcctgcaagg cttctggata caccttcacc aattatgata tcaactgggt gcgacaggcc 120
actggacaag ggcttgagtg gatgggatgg atgaacccta acagtggtaa cacaggcttt 180
gcacagaagt tccagggcag agtcaccatg accaggaaca cctccataag aacagtctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gagagcctat 300
caaaatagac cgatagcagt aactgatatg ggttactact actactacgg tatggacgtc 360
tggggccaag ggaccacggt caccgtctcc tca 393
Sequence Number (ID): 199
Length: 372
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..372
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
cagctgcagc tgcaggagtc gggcccagga ctggtgaagc cttcggagac cctgtccctc 60
acctgcactg tctctggtgg ctccatcagc agtagtagtt actactgggg ctggatccgc 120
cagcccccag ggaaggggct ggagtggatt gggagtatct attatagtgg gagcacctac 180
tacaacccgt ccctcaagag tcgagtcacc atatcagtag acacgtccaa gaaccagttc 240
tccctgaagc tgagctctgt gaccgccgcg gacacggcct tgtattactg tgcgagaggg 300
gggtcgatct atgactacgg tgactacgaa tttgactcct ggggccaggg aaccctggtc 360
accgtctcct ca 372
Sequence Number (ID): 200
Length: 363
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..363
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
caggtgcagc tggtgcagtc tggggctgag gtgaagaagc ctggggcctc agtgaaggtt 60
tcctgcaagg catctggata caccttcacc agctactata tgcactgggt gcgacaggcc 120
cctggacaag ggcttgagtg gatgggaata atcaacccta gtggtggtag cacaagctac 180
gcacagaagt tccagggcag agtcaccatg accagggaca cgtccacgag cacagtctac 240
atggagctga gcagcctgag atctgaggac acggccgtgt attactgtgc gatattaggg 300
ttacatttgg gggagttatt attcgacccc tggggccagg gaaccctggt caccgtctcc 360
tca 363
Sequence Number (ID): 201
Length: 351
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..351
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tggtggagtc tggaggaggc ttgatccagc ctggggggtc cctgagactc 60
tcctgtgcag cctcggggtt caccgtcagt agcaactaca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagcg gtggtagcac attctacgca 180
gactccgtga agggccgatt caccatctcc agagacaatt ccaagaacac gctgtatctt 240
caaatgaaca gcctgagagc cgaggacacg gccgtgtatt actgtgcgag agatttcgga 300
gaacagtggt ttgactactg gggccaggga accctggtca ccgtctcctc a 351
Sequence Number (ID): 202
Length: 351
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..351
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tggtggagtc tgggggaggc ttggtccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctgaatt caccgtcagt agcaactaca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagcg gtggtagcac atactacgca 180
gactccgtga agggcagatt caccatctcc agagacaatt ccaagaacac gctgtatctt 240
caaatgaaca gcctgagagc cgaggacacg gctgtgtatt actgtgcgag agatttcggg 300
gactactact ttgactactg gggccaggga accctggtca ccgtctcctc a 351
Sequence Number (ID): 203
Length: 354
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..354
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tggtggagac tggaggaggc ttgatccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggctt caccgtcagt aggaactaca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcactt atttatagtg gtggtagcac attctacgca 180
gactccgtga agggccgatt caccatctcc agagacgatt ccaagaacac gttgtatctt 240
caaatgaaca gcctgagagc cgaggacacg gccgtgtatt actgtgcgag agatctcctc 300
gaggtgggag gcagcgacta ctggggccag ggaaccctgg tcaccgtctc ctca 354
Sequence Number (ID): 204
Length: 384
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..384
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tgttggagtc tgggggaggc ttggtacagc ctggggggtc cctgagactc 60
tcctgtgcag cctctggatt cacctttagc agctatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactac 180
gcagactccg tgaagggccg gttcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agccgaggac acggccgtat attactgtgc gaaaccactc 300
ttggggtacg atttttggag tggttattat cggggtggct ggttcgaccc ctggggccag 360
ggaaccctgg tcaccgtctc ctca 384
Sequence Number (ID): 205
Length: 357
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..357
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tggtggagtc tggaggaggc ttgatccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctgggtt caccgtcagt agcaactaca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagcg gtggtagcac atactacgca 180
gactccgtga agggccgatt catcatctcc agagacaatt ccaagaacac gctgtatctt 240
caaatgaaca gcctgagagc cgaggacacg gccgtgtatt actgtgcgag agatccatac 300
agctatggtc gaggtggcgg ctactggggc cagggaaccc tggtcaccgt ctcctca 357
Sequence Number (ID): 206
Length: 351
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..351
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tggtggagac tggaggaggc ttgatccagc ctggggggtc cctgagactc 60
tcctgtgcag cctctgggct caccgtcagt agcaactaca tgaactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagtt atttatagcg gtggtagcac atactacgca 180
gactccgtga agggccgatt caccatctcc agagacaatt ccaagaacac gctgtatctt 240
caaatgaaca gcctgagagc cgaggacacg gccgtgtatt actgtgcgag agatggggtc 300
tactacggta tggacgtctg gggccaaggg accacggtca ccgtctcctc a 351
Sequence Number (ID): 207
Length: 372
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..372
> mol_type, other DNA
> note, VH
> organism, synthetic construct
Residues:
gaggtgcagc tggtggagtc tgggggaggc gtggtccagc ctgggaggtc cctgagactc 60
tcctgtgcag cctctggatt caccttcagt agctatggca tgcactgggt ccgccaggct 120
ccaggcaagg ggctggagtg ggtggcagtt atatcatatg atggaagtaa taaatactac 180
gcagactccg tgaagggccg attcaccatc tccagagaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgag agctgaggac acggctgtgt attactgtgc gaaggattac 300
gatttttgga gtggttatta cccccagccc tttgactact ggggccaggg aaccctggtc 360
accgtctcct ca 372
Sequence Number (ID): 208
Length: 321
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..321
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttacagta cccctggcag ttttggccag 300
gggaccaagc tggagatcaa a 321
Sequence Number (ID): 209
Length: 330
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..330
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
cagtctgtgc tgactcagcc accctcagcg tctgggaccc ccgggcagag ggtcaccatc 60
tcttgttctg gaagtagctc caacatcgga agaaatactg taaactggta ccagcagctc 120
ccaggaacgg cccccaaact cgtcatctat agtaataatc agcggccctc aggggtccct 180
gaccgattct ctggctccaa gtctggcacc tcagcctccc tggccatcag tgggctccag 240
tctgaggatg aggctgatta ttactgtgca gcatgggatg acagcctgaa tggtctggta 300
ttcggcggag ggaccaagct gaccgtccta 330
Sequence Number (ID): 210
Length: 339
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..339
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagcttca ggaataagat ctgcttagct 120
tggtaccagc agaaaccagg acagcctcct aaactgctca tttactgggc aactacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggct gtttattact gtcagcaatt ttatagtact 300
ccgtggacgt tcggccaagg gaccaaggtg gaaatcaaa 339
Sequence Number (ID): 211
Length: 330
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..330
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
cagtctgccc tgactcagtc tgcctccgtg tctgggtctc ctggacagcc gatcaccatc 60
tcctgcactg gaaccagcag tgatgttggg agttataacc ttgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgaggtca gtaagcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgacaat ctctgggctc 240
caggctgagg acgaggctga ttattactgc tgctcatatg cagatagtag gactttggtg 300
ttcggcggag ggaccaagct gaccgtccta 330
Sequence Number (ID): 212
Length: 327
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..327
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tccctccgag ggtgacgttc 300
ggccaaggga ccaaggtgga aatcaaa 327
Sequence Number (ID): 213
Length: 327
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..327
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
cagtctgccc tgactcagcc tgcctccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgatgttggg agttatgacc ttgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgaggtca ctaagcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgacaat ctctgggctc 240
caggctgagg acgaggctga ttattactgc tgctcatttg caggtagtag ccccttgttc 300
ggcggaggga ccaagctgac cgtccta 327
Sequence Number (ID): 214
Length: 321
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..321
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaaggtcct gatctataag gcgtctagtt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attctccaat gttcggccaa 300
gggaccaagg tggaaatcaa a 321
Sequence Number (ID): 215
Length: 324
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..324
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcac cagtatggta gctcacctcg aacgttcggc 300
caagggacca aggtggagat caaa 324
Sequence Number (ID): 216
Length: 330
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..330
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
cagtctgtgc tgactcagcc accctcagcg tctgggaccc ccgggcagag ggtcaccatc 60
tcttgttctg gaagcagctc caacatcgga agtaatactg taaactggta ccagcagctc 120
ccaggaacgg cccccaaact cctcatctat aataataatc aacggccctc aggggtccct 180
gaccgattct ctggctccaa gtctggcacc tcagcctccc tggccatcag tgggctccag 240
tctgaggatg aggctgatta ttactgtgca gcatgggatg acagcctgaa tggttatgtc 300
ttcggaactg ggaccaaggt caccgtccta 330
Sequence Number (ID): 217
Length: 321
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..321
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccatcctcc ctgtctacat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gagcattagc aagtatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
aggttcagtg gcagtggatc tgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttacta ctgtcaacag agttatagta cccctccgac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
Sequence Number (ID): 218
Length: 327
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..327
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg ctacatatta ctgtcaacag tatgataatc tccctccgcg gctcactttc 300
ggcggaggga ccaaggtgga aatcaaa 327
Sequence Number (ID): 219
Length: 333
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..333
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
cagtctgccc tgactcagcc tgcctccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgacattggt ggttataact atgtctcctg gtaccaacaa 120
cacccaggca aagcccccaa actcatgatt tatgatgtca gtaatcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgaccat ctctgggctc 240
caggctgagg acgaggctga atattactgc agctcatata caagaagcag cactcaggtg 300
gtattcggcg gagggaccaa gctgaccgtc cta 333
Sequence Number (ID): 220
Length: 324
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..324
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcactatcag cagactggag 240
cctgaagatt ttgcagtgta tcactgtcag cagtatggta cctcaccgtg gacgttcggc 300
caagggacca aggtggaaat caaa 324
Sequence Number (ID): 221
Length: 336
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..336
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
cagtctgccc tgactcagcc tgcctccgtg tctgggtctc ctggacagtc gatcaccatc 60
tcctgcactg gaaccagcag tgatgttggg agttataacc ttgtctcctg gtaccaacag 120
cacccaggca aagcccccaa actcatgatt tatgaagtca gtaagcggcc ctcaggggtt 180
tctaatcgct tctctggctc caagtctggc aacacggcct ccctgacaat ctctgggctc 240
caggctgagg acgaggctga ttattactgc tgctcatatg cacgtattag cacccccgga 300
tttgtcttcg gaactgggac caaggtcacc gtccta 336
Sequence Number (ID): 222
Length: 318
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..318
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccttccacc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gagtattagt agctggttgg cctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctataag gcatctaatt tagaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tgggacagaa ttcactctca ccatcagcag cctgcagcct 240
gatgattttg caacttatta ctgccaacag tataatagtt attcgacgtt cggccaaggg 300
accaaggtgg agatcaaa 318
Sequence Number (ID): 223
Length: 324
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..324
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctctaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaggccc ctaagctcct gatctacgat gcatccaatt tgcaaacagg ggtcccatca 180
aggttcagtg gaggtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tcctgggagt cactttcggc 300
cctgggacca aagtggatat caaa 324
Sequence Number (ID): 224
Length: 324
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..324
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagagt 120
cctggccagg ctcccaggct cctcatctat ggtacatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcacctag aacgttcggc 300
caagggacca aggtggagat caaa 324
Sequence Number (ID): 225
Length: 324
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..324
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagtca gagtgttagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcatctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag cagactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcacctcg aacgttcggc 300
caagggacca aggtggagat caaa 324
Sequence Number (ID): 226
Length: 327
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..327
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gaaattgtgt tgacgcagtc tccaggcacc ctgtctttgt ctccagggga aagagccacc 60
ctctcctgca gggccagcca gagtattagc agcagctact tagcctggta ccagcagaaa 120
cctggccagg ctcccaggct cctcttctat ggtgcatcca gcagggccac tggcatccca 180
gacaggttca gtggcagtgg gtctgggaca gacttcactc tcaccatcag caaactggag 240
cctgaagatt ttgcagtgta ttactgtcag cagtatggta gctcacctcc gatcaccttc 300
ggccaaggga cacgactgga gattaaa 327
Sequence Number (ID): 227
Length: 342
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..342
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatcgtga tgacccagtc tccagactcc ctggctgtgt ctctgggcga gagggccacc 60
atcaactgca agtccagcca gagtgtttta tacagctcca acaataagaa ctacttagct 120
tggtaccagc agaaaccagg acagcctcct aagctgctca tttactgggc atctacccgg 180
gaatccgggg tccctgaccg attcagtggc agcgggtctg ggacagattt cactctcacc 240
atcagcagcc tgcaggctga agatgtggca gtttattact gtcagcaata ttatagtact 300
ctcttgctca ctttcggcgg agggaccaag gtggagatca aa 342
Sequence Number (ID): 228
Length: 321
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..321
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc aggcgagtca ggacattagc aactatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctacgat gcatccaatt tggaaacagg ggtcccatca 180
aggttcagtg gaagtggatc tgggacagat tttactttca ccatcagcag cctgcagcct 240
gaagatattg caacatatta ctgtcaacag tatgataatc tcccgatcac cttcggccaa 300
gggacacgac tggagattaa a 321
Sequence Number (ID): 229
Length: 327
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..327
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccagt tgacccagtc tccatccttc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggccagtca gggcattagc agttatttag cctggtatca gcaaaaacca 120
gggaaagccc ctaagctcct gatctttgct gcatccactt tgcagagtgg ggtcccatca 180
aggttcagcg ccagtggatc tgggacagaa ttcactctca caatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtcaacac cttaatagtt acccttctat gtacactttt 300
ggccagggga ccaagctgga gatcaaa 327
Sequence Number (ID): 230
Length: 321
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..321
> mol_type, other DNA
> note, VL
> organism, synthetic construct
Residues:
gacatccaga tgacccagtc tccatcctcc ctgtctgcat ctgtaggaga cagagtcacc 60
atcacttgcc gggcaagtca gggcattaga aatgatttag gctggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tacaaagtgg ggtcccatca 180
aggttcagcg gcagtggatc tggcacagat ttcactctca ccatcagcag cctgcagcct 240
gaagattttg caacttatta ctgtctacaa gattacaatt accctcggac gttcggccaa 300
gggaccaagg tggaaatcaa a 321
Sequence Number (ID): 231
Length: 457
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..457
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG VVQPGRSLRL SCAASGFTFS SYAMHWVRQA PGKGLEWVAV ISYDGSNKYY 60
ADSVKGRFTI SRDNSKNTLY LQMNSLRAED TAVYYCARAR PWRYSYGLYY YYGMDVWGQG 120
TTVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF 180
PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPKSC DKTHTCPPCP 240
APELLGGPSV FLFPPKPKDT LMISRTPEVT CVVVDVSHED PEVKFNWYVD GVEVHNAKTK 300
PREEQYNSTY RVVSVLTVLH QDWLNGKEYK CKVSNKALPA PIEKTISKAK GQPREPQVYT 360
LPPSREEMTK NQVSLTCLVK GFYPSDIAVE WESNGQPENN YKTTPPVLDS DGSFFLYSKL 420
TVDKSRWQQG NVFSCSVMHE ALHNHYTQKS LSLSPGK 457
Sequence Number (ID): 232
Length: 457
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..457
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG LVQSGGSLRL SCAASGFSFN TYSMNWVRQA PGKGLEWVSY ISSSGSILHY 60
ADSVKGRLTI SRDNAKNSLY LQMNSLRAED TAVYYCARVG TRVGASNFDY YYGMDVWGQG 120
TTVTVSSAST KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF 180
PAVLQSSGLY SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPKSC DKTHTCPPCP 240
APELLGGPSV FLFPPKPKDT LMISRTPEVT CVVVDVSHED PEVKFNWYVD GVEVHNAKTK 300
PREEQYNSTY RVVSVLTVLH QDWLNGKEYK CKVSNKALPA PIEKTISKAK GQPREPQVYT 360
LPPSREEMTK NQVSLTCLVK GFYPSDIAVE WESNGQPENN YKTTPPVLDS DGSFFLYSKL 420
TVDKSRWQQG NVFSCSVMHE ALHNHYTQKS LSLSPGK 457
Sequence Number (ID): 233
Length: 453
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..453
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLRQWGSG LLKPSETLSL TCAVYGGSLS GYYWSYFRQP PGKGLEWIGE ITQSGGTNYN 60
PSLKSRITIS VDTSKNQFSL DLSSVTAADT AVYYCARGGY DDNRGYYAYF DYWGQGTLVT 120
VSSASTKGPS VFPLAPSSKS TSGGTAALGC LVKDYFPEPV TVSWNSGALT SGVHTFPAVL 180
QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNTKVDKR VEPKSCDKTH TCPPCPAPEL 240
LGGPSVFLFP PKPKDTLMIS RTPEVTCVVV DVSHEDPEVK FNWYVDGVEV HNAKTKPREE 300
QYNSTYRVVS VLTVLHQDWL NGKEYKCKVS NKALPAPIEK TISKAKGQPR EPQVYTLPPS 360
REEMTKNQVS LTCLVKGFYP SDIAVEWESN GQPENNYKTT PPVLDSDGSF FLYSKLTVDK 420
SRWQQGNVFS CSVMHEALHN HYTQKSLSLS PGK 453
Sequence Number (ID): 234
Length: 465
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..465
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT GYYIHWVRQA PGQGLEWMGW INPNSGATNY 60
AQKFQGRVTV TRDTSISTAY MELSRLRSDD TAVYYCARVA QVVSNPLPYD VLTGLTYYYY 120
GMDVWGQGTT VTVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE PVTVSWNSGA 180
LTSGVHTFPA VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KRVEPKSCDK 240
THTCPPCPAP ELLGGPSVFL FPPKPKDTLM ISRTPEVTCV VVDVSHEDPE VKFNWYVDGV 300
EVHNAKTKPR EEQYNSTYRV VSVLTVLHQD WLNGKEYKCK VSNKALPAPI EKTISKAKGQ 360
PREPQVYTLP PSREEMTKNQ VSLTCLVKGF YPSDIAVEWE SNGQPENNYK TTPPVLDSDG 420
SFFLYSKLTV DKSRWQQGNV FSCSVMHEAL HNHYTQKSLS LSPGK 465
Sequence Number (ID): 235
Length: 451
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..451
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT GYYMHWVRQA PGQGLEWMGW INPNSGGTNY 60
AQKFQGRVTM TRDTSITTAY MELSRLRSDD TAVYYCARDY YYDSSVNPDY WGQGTLVTVS 120
SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV SWNSGALTSG VHTFPAVLQS 180
SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKRVE PKSCDKTHTC PPCPAPELLG 240
GPSVFLFPPK PKDTLMISRT PEVTCVVVDV SHEDPEVKFN WYVDGVEVHN AKTKPREEQY 300
NSTYRVVSVL TVLHQDWLNG KEYKCKVSNK ALPAPIEKTI SKAKGQPREP QVYTLPPSRE 360
EMTKNQVSLT CLVKGFYPSD IAVEWESNGQ PENNYKTTPP VLDSDGSFFL YSKLTVDKSR 420
WQQGNVFSCS VMHEALHNHY TQKSLSLSPG K 451
Sequence Number (ID): 236
Length: 453
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..453
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLQESGPG LVKPSQTLSL TCTVSGGSIS SGDYYWSWIR QHPGKGLEWI GYIFYSGSTY 60
YNPSLKSRVT ISVDTSKNQF SLKLSSVTAA DTAVYYCARR CRFVDTSMAF DYWGQGTLVT 120
VSSASTKGPS VFPLAPSSKS TSGGTAALGC LVKDYFPEPV TVSWNSGALT SGVHTFPAVL 180
QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNTKVDKR VEPKSCDKTH TCPPCPAPEL 240
LGGPSVFLFP PKPKDTLMIS RTPEVTCVVV DVSHEDPEVK FNWYVDGVEV HNAKTKPREE 300
QYNSTYRVVS VLTVLHQDWL NGKEYKCKVS NKALPAPIEK TISKAKGQPR EPQVYTLPPS 360
REEMTKNQVS LTCLVKGFYP SDIAVEWESN GQPENNYKTT PPVLDSDGSF FLYSKLTVDK 420
SRWQQGNVFS CSVMHEALHN HYTQKSLSLS PGK 453
Sequence Number (ID): 237
Length: 451
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..451
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGSSVKV SCKASGGTFI TYAFSWVRQA PGQGLEWMGR IIPILGITNY 60
AQKFQGRVTI TADKSTSTAY MELSSLRSED TAVYYCARGG RGANPGWFDY WGQGTLVTVS 120
SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV SWNSGALTSG VHTFPAVLQS 180
SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKRVE PKSCDKTHTC PPCPAPELLG 240
GPSVFLFPPK PKDTLMISRT PEVTCVVVDV SHEDPEVKFN WYVDGVEVHN AKTKPREEQY 300
NSTYRVVSVL TVLHQDWLNG KEYKCKVSNK ALPAPIEKTI SKAKGQPREP QVYTLPPSRE 360
EMTKNQVSLT CLVKGFYPSD IAVEWESNGQ PENNYKTTPP VLDSDGSFFL YSKLTVDKSR 420
WQQGNVFSCS VMHEALHNHY TQKSLSLSPG K 451
Sequence Number (ID): 238
Length: 453
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..453
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGSSVKV SCKASGGTFS NYAISWVRQA PGQGLEWMGR IIPILGIANY 60
AQKFQGRLTI TADKSTSTAY MELSSLRSED TAVYYCARTA YEYDSSGYFL DYWGQGTLVT 120
VSSASTKGPS VFPLAPSSKS TSGGTAALGC LVKDYFPEPV TVSWNSGALT SGVHTFPAVL 180
QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNTKVDKR VEPKSCDKTH TCPPCPAPEL 240
LGGPSVFLFP PKPKDTLMIS RTPEVTCVVV DVSHEDPEVK FNWYVDGVEV HNAKTKPREE 300
QYNSTYRVVS VLTVLHQDWL NGKEYKCKVS NKALPAPIEK TISKAKGQPR EPQVYTLPPS 360
REEMTKNQVS LTCLVKGFYP SDIAVEWESN GQPENNYKTT PPVLDSDGSF FLYSKLTVDK 420
SRWQQGNVFS CSVMHEALHN HYTQKSLSLS PGK 453
Sequence Number (ID): 239
Length: 450
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..450
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLLESGGG LVKPGGSLRL SCAASGFTFS DYYMSWIRQA PGKGLEWVSY ISSSGSTIYY 60
ADSVKGRFTI SRDNAKNSLY LQMNSLRAED TAVYYCARHP MVRGVIGDYW GQGTLVTVSS 120
ASTKGPSVFP LAPSSKSTSG GTAALGCLVK DYFPEPVTVS WNSGALTSGV HTFPAVLQSS 180
GLYSLSSVVT VPSSSLGTQT YICNVNHKPS NTKVDKRVEP KSCDKTHTCP PCPAPELLGG 240
PSVFLFPPKP KDTLMISRTP EVTCVVVDVS HEDPEVKFNW YVDGVEVHNA KTKPREEQYN 300
STYRVVSVLT VLHQDWLNGK EYKCKVSNKA LPAPIEKTIS KAKGQPREPQ VYTLPPSREE 360
MTKNQVSLTC LVKGFYPSDI AVEWESNGQP ENNYKTTPPV LDSDGSFFLY SKLTVDKSRW 420
QQGNVFSCSV MHEALHNHYT QKSLSLSPGK 450
Sequence Number (ID): 240
Length: 455
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..455
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT SYYMHWVRQA PGQGLEWMGI INPSGGSTSY 60
VQKFQGRVTM TRDTSTSTVY MELSSLRFED TAVYYCARGH SGYDRLPQPP YFDYWGQGTL 120
VTVSSASTKG PSVFPLAPSS KSTSGGTAAL GCLVKDYFPE PVTVSWNSGA LTSGVHTFPA 180
VLQSSGLYSL SSVVTVPSSS LGTQTYICNV NHKPSNTKVD KRVEPKSCDK THTCPPCPAP 240
ELLGGPSVFL FPPKPKDTLM ISRTPEVTCV VVDVSHEDPE VKFNWYVDGV EVHNAKTKPR 300
EEQYNSTYRV VSVLTVLHQD WLNGKEYKCK VSNKALPAPI EKTISKAKGQ PREPQVYTLP 360
PSREEMTKNQ VSLTCLVKGF YPSDIAVEWE SNGQPENNYK TTPPVLDSDG SFFLYSKLTV 420
DKSRWQQGNV FSCSVMHEAL HNHYTQKSLS LSPGK 455
Sequence Number (ID): 241
Length: 451
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..451
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYLFT GYYMHWVRQA PGQGLEWMGW INPNSGGTNY 60
AQKFQDRVTM TRDTSITTAY MELSRLRSDD TAVYYCARSG YYTSAVNFDY WGQGTLVTVS 120
SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV SWNSGALTSG VHTFPAVLQS 180
SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKRVE PKSCDKTHTC PPCPAPELLG 240
GPSVFLFPPK PKDTLMISRT PEVTCVVVDV SHEDPEVKFN WYVDGVEVHN AKTKPREEQY 300
NSTYRVVSVL TVLHQDWLNG KEYKCKVSNK ALPAPIEKTI SKAKGQPREP QVYTLPPSRE 360
EMTKNQVSLT CLVKGFYPSD IAVEWESNGQ PENNYKTTPP VLDSDGSFFL YSKLTVDKSR 420
WQQGNVFSCS VMHEALHNHY TQKSLSLSPG K 451
Sequence Number (ID): 242
Length: 462
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..462
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLLESGGG LVQPGGSLRL SCAASGFTFS SYAMSWVRQA PGKGLEWVSA VSGSGGSTYY 60
ADSVKGRFTI SRDNSKNMLY LQMNSLRAED TAVYYCAKTV LAGRDDSSGY FTVYYYFGMD 120
VWGQGTTVTV SSASTKGPSV FPLAPSSKST SGGTAALGCL VKDYFPEPVT VSWNSGALTS 180
GVHTFPAVLQ SSGLYSLSSV VTVPSSSLGT QTYICNVNHK PSNTKVDKRV EPKSCDKTHT 240
CPPCPAPELL GGPSVFLFPP KPKDTLMISR TPEVTCVVVD VSHEDPEVKF NWYVDGVEVH 300
NAKTKPREEQ YNSTYRVVSV LTVLHQDWLN GKEYKCKVSN KALPAPIEKT ISKAKGQPRE 360
PQVYTLPPSR EEMTKNQVSL TCLVKGFYPS DIAVEWESNG QPENNYKTTP PVLDSDGSFF 420
LYSKLTVDKS RWQQGNVFSC SVMHEALHNH YTQKSLSLSP GK 462
Sequence Number (ID): 243
Length: 453
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..453
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGPE VKKPGTSVKV SCKASGFTFP SSAVQWVRQA RGQRLEWIGW IVVGSGNTNY 60
AQKFQERVTI TRDMSTSTAY MELSSLRSED TAVYYCAAPY CSGGSCYDGF DIWGQGTMVT 120
VSSASTKGPS VFPLAPSSKS TSGGTAALGC LVKDYFPEPV TVSWNSGALT SGVHTFPAVL 180
QSSGLYSLSS VVTVPSSSLG TQTYICNVNH KPSNTKVDKR VEPKSCDKTH TCPPCPAPEL 240
LGGPSVFLFP PKPKDTLMIS RTPEVTCVVV DVSHEDPEVK FNWYVDGVEV HNAKTKPREE 300
QYNSTYRVVS VLTVLHQDWL NGKEYKCKVS NKALPAPIEK TISKAKGQPR EPQVYTLPPS 360
REEMTKNQVS LTCLVKGFYP SDIAVEWESN GQPENNYKTT PPVLDSDGSF FLYSKLTVDK 420
SRWQQGNVFS CSVMHEALHN HYTQKSLSLS PGK 453
Sequence Number (ID): 244
Length: 461
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..461
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT NYDINWVRQA TGQGLEWMGW MNPNSGNTGF 60
AQKFQGRVTM TRNTSIRTVY MELSSLRSED TAVYYCARAY QNRPIAVTDM GYYYYYGMDV 120
WGQGTTVTVS SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV SWNSGALTSG 180
VHTFPAVLQS SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKRVE PKSCDKTHTC 240
PPCPAPELLG GPSVFLFPPK PKDTLMISRT PEVTCVVVDV SHEDPEVKFN WYVDGVEVHN 300
AKTKPREEQY NSTYRVVSVL TVLHQDWLNG KEYKCKVSNK ALPAPIEKTI SKAKGQPREP 360
QVYTLPPSRE EMTKNQVSLT CLVKGFYPSD IAVEWESNGQ PENNYKTTPP VLDSDGSFFL 420
YSKLTVDKSR WQQGNVFSCS VMHEALHNHY TQKSLSLSPG K 461
Sequence Number (ID): 245
Length: 454
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..454
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QLQLQESGPG LVKPSETLSL TCTVSGGSIS SSSYYWGWIR QPPGKGLEWI GSIYYSGSTY 60
YNPSLKSRVT ISVDTSKNQF SLKLSSVTAA DTALYYCARG GSIYDYGDYE FDSWGQGTLV 120
TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP VTVSWNSGAL TSGVHTFPAV 180
LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK RVEPKSCDKT HTCPPCPAPE 240
LLGGPSVFLF PPKPKDTLMI SRTPEVTCVV VDVSHEDPEV KFNWYVDGVE VHNAKTKPRE 300
EQYNSTYRVV SVLTVLHQDW LNGKEYKCKV SNKALPAPIE KTISKAKGQP REPQVYTLPP 360
SREEMTKNQV SLTCLVKGFY PSDIAVEWES NGQPENNYKT TPPVLDSDGS FFLYSKLTVD 420
KSRWQQGNVF SCSVMHEALH NHYTQKSLSL SPGK 454
Sequence Number (ID): 246
Length: 451
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..451
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
QVQLVQSGAE VKKPGASVKV SCKASGYTFT SYYMHWVRQA PGQGLEWMGI INPSGGSTSY 60
AQKFQGRVTM TRDTSTSTVY MELSSLRSED TAVYYCAILG LHLGELLFDP WGQGTLVTVS 120
SASTKGPSVF PLAPSSKSTS GGTAALGCLV KDYFPEPVTV SWNSGALTSG VHTFPAVLQS 180
SGLYSLSSVV TVPSSSLGTQ TYICNVNHKP SNTKVDKRVE PKSCDKTHTC PPCPAPELLG 240
GPSVFLFPPK PKDTLMISRT PEVTCVVVDV SHEDPEVKFN WYVDGVEVHN AKTKPREEQY 300
NSTYRVVSVL TVLHQDWLNG KEYKCKVSNK ALPAPIEKTI SKAKGQPREP QVYTLPPSRE 360
EMTKNQVSLT CLVKGFYPSD IAVEWESNGQ PENNYKTTPP VLDSDGSFFL YSKLTVDKSR 420
WQQGNVFSCS VMHEALHNHY TQKSLSLSPG K 451
Sequence Number (ID): 247
Length: 447
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..447
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG LIQPGGSLRL SCAASGFTVS SNYMSWVRQA PGKGLEWVSV IYSGGSTFYA 60
DSVKGRFTIS RDNSKNTLYL QMNSLRAEDT AVYYCARDFG EQWFDYWGQG TLVTVSSAST 120
KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY 180
SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPKSC DKTHTCPPCP APELLGGPSV 240
FLFPPKPKDT LMISRTPEVT CVVVDVSHED PEVKFNWYVD GVEVHNAKTK PREEQYNSTY 300
RVVSVLTVLH QDWLNGKEYK CKVSNKALPA PIEKTISKAK GQPREPQVYT LPPSREEMTK 360
NQVSLTCLVK GFYPSDIAVE WESNGQPENN YKTTPPVLDS DGSFFLYSKL TVDKSRWQQG 420
NVFSCSVMHE ALHNHYTQKS LSLSPGK 447
Sequence Number (ID): 248
Length: 447
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..447
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG LVQPGGSLRL SCAASEFTVS SNYMSWVRQA PGKGLEWVSV IYSGGSTYYA 60
DSVKGRFTIS RDNSKNTLYL QMNSLRAEDT AVYYCARDFG DYYFDYWGQG TLVTVSSAST 120
KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY 180
SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPKSC DKTHTCPPCP APELLGGPSV 240
FLFPPKPKDT LMISRTPEVT CVVVDVSHED PEVKFNWYVD GVEVHNAKTK PREEQYNSTY 300
RVVSVLTVLH QDWLNGKEYK CKVSNKALPA PIEKTISKAK GQPREPQVYT LPPSREEMTK 360
NQVSLTCLVK GFYPSDIAVE WESNGQPENN YKTTPPVLDS DGSFFLYSKL TVDKSRWQQG 420
NVFSCSVMHE ALHNHYTQKS LSLSPGK 447
Sequence Number (ID): 249
Length: 448
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..448
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLVETGGG LIQPGGSLRL SCAASGFTVS RNYMSWVRQA PGKGLEWVSL IYSGGSTFYA 60
DSVKGRFTIS RDDSKNTLYL QMNSLRAEDT AVYYCARDLL EVGGSDYWGQ GTLVTVSSAS 120
TKGPSVFPLA PSSKSTSGGT AALGCLVKDY FPEPVTVSWN SGALTSGVHT FPAVLQSSGL 180
YSLSSVVTVP SSSLGTQTYI CNVNHKPSNT KVDKRVEPKS CDKTHTCPPC PAPELLGGPS 240
VFLFPPKPKD TLMISRTPEV TCVVVDVSHE DPEVKFNWYV DGVEVHNAKT KPREEQYNST 300
YRVVSVLTVL HQDWLNGKEY KCKVSNKALP APIEKTISKA KGQPREPQVY TLPPSREEMT 360
KNQVSLTCLV KGFYPSDIAV EWESNGQPEN NYKTTPPVLD SDGSFFLYSK LTVDKSRWQQ 420
GNVFSCSVMH EALHNHYTQK SLSLSPGK 448
Sequence Number (ID): 250
Length: 458
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..458
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLLESGGG LVQPGGSLRL SCAASGFTFS SYAMSWVRQA PGKGLEWVSA ISGSGGSTYY 60
ADSVKGRFTI SRDNSKNTLY LQMNSLRAED TAVYYCAKPL LGYDFWSGYY RGGWFDPWGQ 120
GTLVTVSSAS TKGPSVFPLA PSSKSTSGGT AALGCLVKDY FPEPVTVSWN SGALTSGVHT 180
FPAVLQSSGL YSLSSVVTVP SSSLGTQTYI CNVNHKPSNT KVDKRVEPKS CDKTHTCPPC 240
PAPELLGGPS VFLFPPKPKD TLMISRTPEV TCVVVDVSHE DPEVKFNWYV DGVEVHNAKT 300
KPREEQYNST YRVVSVLTVL HQDWLNGKEY KCKVSNKALP APIEKTISKA KGQPREPQVY 360
TLPPSREEMT KNQVSLTCLV KGFYPSDIAV EWESNGQPEN NYKTTPPVLD SDGSFFLYSK 420
LTVDKSRWQQ GNVFSCSVMH EALHNHYTQK SLSLSPGK 458
Sequence Number (ID): 251
Length: 449
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..449
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG LIQPGGSLRL SCAASGFTVS SNYMSWVRQA PGKGLEWVSV IYSGGSTYYA 60
DSVKGRFIIS RDNSKNTLYL QMNSLRAEDT AVYYCARDPY SYGRGGGYWG QGTLVTVSSA 120
STKGPSVFPL APSSKSTSGG TAALGCLVKD YFPEPVTVSW NSGALTSGVH TFPAVLQSSG 180
LYSLSSVVTV PSSSLGTQTY ICNVNHKPSN TKVDKRVEPK SCDKTHTCPP CPAPELLGGP 240
SVFLFPPKPK DTLMISRTPE VTCVVVDVSH EDPEVKFNWY VDGVEVHNAK TKPREEQYNS 300
TYRVVSVLTV LHQDWLNGKE YKCKVSNKAL PAPIEKTISK AKGQPREPQV YTLPPSREEM 360
TKNQVSLTCL VKGFYPSDIA VEWESNGQPE NNYKTTPPVL DSDGSFFLYS KLTVDKSRWQ 420
QGNVFSCSVM HEALHNHYTQ KSLSLSPGK 449
Sequence Number (ID): 252
Length: 447
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..447
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLVETGGG LIQPGGSLRL SCAASGLTVS SNYMNWVRQA PGKGLEWVSV IYSGGSTYYA 60
DSVKGRFTIS RDNSKNTLYL QMNSLRAEDT AVYYCARDGV YYGMDVWGQG TTVTVSSAST 120
KGPSVFPLAP SSKSTSGGTA ALGCLVKDYF PEPVTVSWNS GALTSGVHTF PAVLQSSGLY 180
SLSSVVTVPS SSLGTQTYIC NVNHKPSNTK VDKRVEPKSC DKTHTCPPCP APELLGGPSV 240
FLFPPKPKDT LMISRTPEVT CVVVDVSHED PEVKFNWYVD GVEVHNAKTK PREEQYNSTY 300
RVVSVLTVLH QDWLNGKEYK CKVSNKALPA PIEKTISKAK GQPREPQVYT LPPSREEMTK 360
NQVSLTCLVK GFYPSDIAVE WESNGQPENN YKTTPPVLDS DGSFFLYSKL TVDKSRWQQG 420
NVFSCSVMHE ALHNHYTQKS LSLSPGK 447
Sequence Number (ID): 253
Length: 454
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..454
> MOL_TYPE, protein
> NOTE, heavy chain
> ORGANISM, synthetic construct
Residues:
EVQLVESGGG VVQPGRSLRL SCAASGFTFS SYGMHWVRQA PGKGLEWVAV ISYDGSNKYY 60
ADSVKGRFTI SRDNSKNTLY LQMNSLRAED TAVYYCAKDY DFWSGYYPQP FDYWGQGTLV 120
TVSSASTKGP SVFPLAPSSK STSGGTAALG CLVKDYFPEP VTVSWNSGAL TSGVHTFPAV 180
LQSSGLYSLS SVVTVPSSSL GTQTYICNVN HKPSNTKVDK RVEPKSCDKT HTCPPCPAPE 240
LLGGPSVFLF PPKPKDTLMI SRTPEVTCVV VDVSHEDPEV KFNWYVDGVE VHNAKTKPRE 300
EQYNSTYRVV SVLTVLHQDW LNGKEYKCKV SNKALPAPIE KTISKAKGQP REPQVYTLPP 360
SREEMTKNQV SLTCLVKGFY PSDIAVEWES NGQPENNYKT TPPVLDSDGS FFLYSKLTVD 420
KSRWQQGNVF SCSVMHEALH NHYTQKSLSL SPGK 454
Sequence Number (ID): 254
Length: 214
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..214
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCRASQSIS SYLNWYQQKP GKAPKLLIYA ASSLQSGVPS 60
RFSGSGSGTD FTLTISSLQP EDFATYYCQQ SYSTPGSFGQ GTKLEIKRTV AAPSVFIFPP 120
SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ ESVTEQDSKD STYSLSSTLT 180
LSKADYEKHK VYACEVTHQG LSSPVTKSFN RGEC 214
Sequence Number (ID): 255
Length: 216
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..216
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
QSVLTQPPSA SGTPGQRVTI SCSGSSSNIG RNTVNWYQQL PGTAPKLVIY SNNQRPSGVP 60
DRFSGSKSGT SASLAISGLQ SEDEADYYCA AWDDSLNGLV FGGGTKLTVL GQPKAAPSVT 120
LFPPSSEELQ ANKATLVCLI SDFYPGAVTV AWKADSSPVK AGVETTTPSK QSNNKYAASS 180
YLSLTPEQWK SHRSYSCQVT HEGSTVEKTV APTECS 216
Sequence Number (ID): 256
Length: 220
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..220
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIVMTQSPDS LAVSLGERAT INCKSSQSVL YSFRNKICLA WYQQKPGQPP KLLIYWATTR 60
ESGVPDRFSG SGSGTDFTLT ISSLQAEDVA VYYCQQFYST PWTFGQGTKV EIKRTVAAPS 120
VFIFPPSDEQ LKSGTASVVC LLNNFYPREA KVQWKVDNAL QSGNSQESVT EQDSKDSTYS 180
LSSTLTLSKA DYEKHKVYAC EVTHQGLSSP VTKSFNRGEC 220
Sequence Number (ID): 257
Length: 216
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..216
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
QSALTQSASV SGSPGQPITI SCTGTSSDVG SYNLVSWYQQ HPGKAPKLMI YEVSKRPSGV 60
SNRFSGSKSG NTASLTISGL QAEDEADYYC CSYADSRTLV FGGGTKLTVL GQPKAAPSVT 120
LFPPSSEELQ ANKATLVCLI SDFYPGAVTV AWKADSSPVK AGVETTTPSK QSNNKYAASS 180
YLSLTPEQWK SHRSYSCQVT HEGSTVEKTV APTECS 216
Sequence Number (ID): 258
Length: 216
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..216
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD ASNLETGVPS 60
RFSGSGSGTD FTFTISSLQP EDIATYYCQQ YDNLPPRVTF GQGTKVEIKR TVAAPSVFIF 120
PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN SQESVTEQDS KDSTYSLSST 180
LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC 216
Sequence Number (ID): 259
Length: 215
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..215
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
QSALTQPASV SGSPGQSITI SCTGTSSDVG SYDLVSWYQQ HPGKAPKLMI YEVTKRPSGV 60
SNRFSGSKSG NTASLTISGL QAEDEADYYC CSFAGSSPLF GGGTKLTVLG QPKAAPSVTL 120
FPPSSEELQA NKATLVCLIS DFYPGAVTVA WKADSSPVKA GVETTTPSKQ SNNKYAASSY 180
LSLTPEQWKS HRSYSCQVTH EGSTVEKTVA PTECS 215
Sequence Number (ID): 260
Length: 214
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..214
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPST LSASVGDRVT ITCRASQSIS SWLAWYQQKP GKAPKVLIYK ASSLESGVPS 60
RFSGSGSGTE FTLTISSLQP DDFATYYCQQ YNSYSPMFGQ GTKVEIKRTV AAPSVFIFPP 120
SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ ESVTEQDSKD STYSLSSTLT 180
LSKADYEKHK VYACEVTHQG LSSPVTKSFN RGEC 214
Sequence Number (ID): 261
Length: 215
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..215
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQK PGQAPRLLIY GASSRATGIP 60
DRFSGSGSGT DFTLTISRLE PEDFAVYYCH QYGSSPRTFG QGTKVEIKRT VAAPSVFIFP 120
PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS QESVTEQDSK DSTYSLSSTL 180
TLSKADYEKH KVYACEVTHQ GLSSPVTKSF NRGEC 215
Sequence Number (ID): 262
Length: 216
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..216
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
QSVLTQPPSA SGTPGQRVTI SCSGSSSNIG SNTVNWYQQL PGTAPKLLIY NNNQRPSGVP 60
DRFSGSKSGT SASLAISGLQ SEDEADYYCA AWDDSLNGYV FGTGTKVTVL GQPKANPTVT 120
LFPPSSEELQ ANKATLVCLI SDFYPGAVTV AWKADSSPVK AGVETTTPSK QSNNKYAASS 180
YLSLTPEQWK SHRSYSCQVT HEGSTVEKTV APTECS 216
Sequence Number (ID): 263
Length: 214
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..214
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSTSVGDRVT ITCRASQSIS KYLNWYQQKP GKAPKLLIYA ASSLQSGVPS 60
RFSGSGSGTD FTLTISSLQP EDFATYYCQQ SYSTPPTFGQ GTKVEIKRTV AAPSVFIFPP 120
SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ ESVTEQDSKD STYSLSSTLT 180
LSKADYEKHK VYACEVTHQG LSSPVTKSFN RGEC 214
Sequence Number (ID): 264
Length: 216
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..216
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD ASNLETGVPS 60
RFSGSGSGTD FTFTISSLQP EDIATYYCQQ YDNLPPRLTF GGGTKVEIKR TVAAPSVFIF 120
PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN SQESVTEQDS KDSTYSLSST 180
LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC 216
Sequence Number (ID): 265
Length: 217
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..217
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
QSALTQPASV SGSPGQSITI SCTGTSSDIG GYNYVSWYQQ HPGKAPKLMI YDVSNRPSGV 60
SNRFSGSKSG NTASLTISGL QAEDEAEYYC SSYTRSSTQV VFGGGTKLTV LGQPKAAPSV 120
TLFPPSSEEL QANKATLVCL ISDFYPGAVT VAWKADSSPV KAGVETTTPS KQSNNKYAAS 180
SYLSLTPEQW KSHRSYSCQV THEGSTVEKT VAPTECS 217
Sequence Number (ID): 266
Length: 215
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..215
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQK PGQAPRLLIY GASSRATGIP 60
DRFSGSGSGT DFTLTISRLE PEDFAVYHCQ QYGTSPWTFG QGTKVEIKRT VAAPSVFIFP 120
PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS QESVTEQDSK DSTYSLSSTL 180
TLSKADYEKH KVYACEVTHQ GLSSPVTKSF NRGEC 215
Sequence Number (ID): 267
Length: 218
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..218
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
QSALTQPASV SGSPGQSITI SCTGTSSDVG SYNLVSWYQQ HPGKAPKLMI YEVSKRPSGV 60
SNRFSGSKSG NTASLTISGL QAEDEADYYC CSYARISTPG FVFGTGTKVT VLGQPKANPT 120
VTLFPPSSEE LQANKATLVC LISDFYPGAV TVAWKADSSP VKAGVETTTP SKQSNNKYAA 180
SSYLSLTPEQ WKSHRSYSCQ VTHEGSTVEK TVAPTECS 218
Sequence Number (ID): 268
Length: 213
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..213
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPST LSASVGDRVT ITCRASQSIS SWLAWYQQKP GKAPKLLIYK ASNLESGVPS 60
RFSGSGSGTE FTLTISSLQP DDFATYYCQQ YNSYSTFGQG TKVEIKRTVA APSVFIFPPS 120
DEQLKSGTAS VVCLLNNFYP REAKVQWKVD NALQSGNSQE SVTEQDSKDS TYSLSSTLTL 180
SKADYEKHKV YACEVTHQGL SSPVTKSFNR GEC 213
Sequence Number (ID): 269
Length: 215
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..215
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASLGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD ASNLQTGVPS 60
RFSGGGSGTD FTFTISSLQP EDIATYYCQQ YDNLLGVTFG PGTKVDIKRT VAAPSVFIFP 120
PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS QESVTEQDSK DSTYSLSSTL 180
TLSKADYEKH KVYACEVTHQ GLSSPVTKSF NRGEC 215
Sequence Number (ID): 270
Length: 215
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..215
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQS PGQAPRLLIY GTSSRATGIP 60
DRFSGSGSGT DFTLTISRLE PEDFAVYYCQ QYGSSPRTFG QGTKVEIKRT VAAPSVFIFP 120
PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS QESVTEQDSK DSTYSLSSTL 180
TLSKADYEKH KVYACEVTHQ GLSSPVTKSF NRGEC 215
Sequence Number (ID): 271
Length: 215
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..215
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSVS SSYLAWYQQK PGQAPRLLIY GASSRATGIP 60
DRFSGSGSGT DFTLTISRLE PEDFAVYYCQ QYGSSPRTFG QGTKVEIKRT VAAPSVFIFP 120
PSDEQLKSGT ASVVCLLNNF YPREAKVQWK VDNALQSGNS QESVTEQDSK DSTYSLSSTL 180
TLSKADYEKH KVYACEVTHQ GLSSPVTKSF NRGEC 215
Sequence Number (ID): 272
Length: 216
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..216
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
EIVLTQSPGT LSLSPGERAT LSCRASQSIS SSYLAWYQQK PGQAPRLLFY GASSRATGIP 60
DRFSGSGSGT DFTLTISKLE PEDFAVYYCQ QYGSSPPITF GQGTRLEIKR TVAAPSVFIF 120
PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN SQESVTEQDS KDSTYSLSST 180
LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC 216
Sequence Number (ID): 273
Length: 221
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..221
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIVMTQSPDS LAVSLGERAT INCKSSQSVL YSSNNKNYLA WYQQKPGQPP KLLIYWASTR 60
ESGVPDRFSG SGSGTDFTLT ISSLQAEDVA VYYCQQYYST LLLTFGGGTK VEIKRTVAAP 120
SVFIFPPSDE QLKSGTASVV CLLNNFYPRE AKVQWKVDNA LQSGNSQESV TEQDSKDSTY 180
SLSSTLTLSK ADYEKHKVYA CEVTHQGLSS PVTKSFNRGE C 221
Sequence Number (ID): 274
Length: 214
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..214
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCQASQDIS NYLNWYQQKP GKAPKLLIYD ASNLETGVPS 60
RFSGSGSGTD FTFTISSLQP EDIATYYCQQ YDNLPITFGQ GTRLEIKRTV AAPSVFIFPP 120
SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ ESVTEQDSKD STYSLSSTLT 180
LSKADYEKHK VYACEVTHQG LSSPVTKSFN RGEC 214
Sequence Number (ID): 275
Length: 216
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..216
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQLTQSPSF LSASVGDRVT ITCRASQGIS SYLAWYQQKP GKAPKLLIFA ASTLQSGVPS 60
RFSASGSGTE FTLTISSLQP EDFATYYCQH LNSYPSMYTF GQGTKLEIKR TVAAPSVFIF 120
PPSDEQLKSG TASVVCLLNN FYPREAKVQW KVDNALQSGN SQESVTEQDS KDSTYSLSST 180
LTLSKADYEK HKVYACEVTH QGLSSPVTKS FNRGEC 216
Sequence Number (ID): 276
Length: 214
Molecule Type: AA
Features Location/Qualifiers:
- SOURCE, 1..214
> MOL_TYPE, protein
> NOTE, light chain
> ORGANISM, synthetic construct
Residues:
DIQMTQSPSS LSASVGDRVT ITCRASQGIR NDLGWYQQKP GKAPKLLIYA ASSLQSGVPS 60
RFSGSGSGTD FTLTISSLQP EDFATYYCLQ DYNYPRTFGQ GTKVEIKRTV AAPSVFIFPP 120
SDEQLKSGTA SVVCLLNNFY PREAKVQWKV DNALQSGNSQ ESVTEQDSKD STYSLSSTLT 180
LSKADYEKHK VYACEVTHQG LSSPVTKSFN RGEC 214
Sequence Number (ID): 277
Length: 1428
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1428
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgg tggagtctgg gggaggcgtg gtccagcctg ggaggtccct gagactctcc 120
tgtgcagcct ctggattcac cttcagtagc tatgctatgc actgggtccg ccaggctcca 180
ggcaaggggc tggagtgggt ggcagttata tcgtatgatg gaagtaataa atactacgca 240
gactccgtga agggccgatt caccatctcc agagacaatt ccaagaacac gctgtatctg 300
caaatgaaca gcctgagagc tgaggacacg gctgtgtatt actgtgcgag agcccgtccg 360
tggagataca gctatggcct ctactactac tacggtatgg acgtctgggg ccaagggacc 420
acggtcaccg tctcctcagc gtcgaccaag ggcccatcgg tcttccccct ggcaccctcc 480
tccaagagca cctctggggg cacagcggcc ctgggctgcc tggtcaagga ctacttcccc 540
gaacctgtga cggtctcgtg gaactcaggc gccctgacca gcggcgtgca caccttcccg 600
gctgtcctac agtcctcagg actctactcc ctcagcagcg tggtgaccgt gccctccagc 660
agcttgggca cccagaccta catctgcaac gtgaatcaca agcccagcaa caccaaggtg 720
gacaagagag ttgagcccaa atcttgtgac aaaactcaca catgcccacc gtgcccagca 780
cctgaactcc tggggggacc gtcagtcttc ctcttccccc caaaacccaa ggacaccctc 840
atgatctccc ggacccctga ggtcacatgc gtggtggtgg acgtgagcca cgaagaccct 900
gaggtcaagt tcaactggta cgtggacggc gtggaggtgc ataatgccaa gacaaagccg 960
cgggaggagc agtacaacag cacgtaccgt gtggtcagcg tcctcaccgt cctgcaccag 1020
gactggctga atggcaagga gtacaagtgc aaggtctcca acaaagccct cccagccccc 1080
atcgagaaaa ccatctccaa agccaaaggg cagccccgag aaccacaggt gtacaccctg 1140
cccccatccc gggaggagat gaccaagaac caggtcagcc tgacctgcct ggtcaaaggc 1200
ttctatccca gcgacatcgc cgtggagtgg gagagcaatg ggcagccgga gaacaactac 1260
aagaccacgc ctcccgtgct ggactccgac ggctccttct tcctctatag caagctcacc 1320
gtggacaaga gcaggtggca gcaggggaac gtcttctcat gctccgtgat gcatgaggct 1380
ctgcacaacc actacacgca gaagagcctc tccctgtccc cgggtaaa 1428
Sequence Number (ID): 278
Length: 1428
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1428
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgg tggagtctgg gggaggcttg gtacagtcgg gggggtccct gagactctcc 120
tgtgcagcct ctggattcag cttcaatacc tatagcatga actgggtccg ccaggctcca 180
gggaaggggc tggagtgggt ttcatacatt agcagtagtg gtagtatctt gcactacgca 240
gactctgtga agggccgact caccatttcc agagacaacg ccaagaactc actgtatctg 300
caaatgaaca gcctgagagc cgaggacacg gctgtgtatt actgtgcgag agtggggacc 360
cgagtgggag cttcaaactt cgactactac tacggtatgg acgtctgggg ccaagggacc 420
acggtcaccg tctcctcagc gtcgaccaag ggcccatcgg tcttccccct ggcaccctcc 480
tccaagagca cctctggggg cacagcggcc ctgggctgcc tggtcaagga ctacttcccc 540
gaacctgtga cggtctcgtg gaactcaggc gccctgacca gcggcgtgca caccttcccg 600
gctgtcctac agtcctcagg actctactcc ctcagcagcg tggtgaccgt gccctccagc 660
agcttgggca cccagaccta catctgcaac gtgaatcaca agcccagcaa caccaaggtg 720
gacaagagag ttgagcccaa atcttgtgac aaaactcaca catgcccacc gtgcccagca 780
cctgaactcc tggggggacc gtcagtcttc ctcttccccc caaaacccaa ggacaccctc 840
atgatctccc ggacccctga ggtcacatgc gtggtggtgg acgtgagcca cgaagaccct 900
gaggtcaagt tcaactggta cgtggacggc gtggaggtgc ataatgccaa gacaaagccg 960
cgggaggagc agtacaacag cacgtaccgt gtggtcagcg tcctcaccgt cctgcaccag 1020
gactggctga atggcaagga gtacaagtgc aaggtctcca acaaagccct cccagccccc 1080
atcgagaaaa ccatctccaa agccaaaggg cagccccgag aaccacaggt gtacaccctg 1140
cccccatccc gggaggagat gaccaagaac caggtcagcc tgacctgcct ggtcaaaggc 1200
ttctatccca gcgacatcgc cgtggagtgg gagagcaatg ggcagccgga gaacaactac 1260
aagaccacgc ctcccgtgct ggactccgac ggctccttct tcctctatag caagctcacc 1320
gtggacaaga gcaggtggca gcaggggaac gtcttctcat gctccgtgat gcatgaggct 1380
ctgcacaacc actacacgca gaagagcctc tccctgtccc cgggtaaa 1428
Sequence Number (ID): 279
Length: 1416
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1416
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctac gccagtgggg ctcaggactg ttgaagcctt cggagaccct gtccctcacc 120
tgcgctgtgt atggtgggtc tctaagtggt tactactgga gctacttccg ccagccccca 180
gggaaggggc tggagtggat tggggaaatc actcaaagtg gcggcaccaa ctacaacccg 240
tccctcaaga gtcggatcac aatatcagtg gacacgtcca agaatcagtt ctctttggac 300
ctgagctctg tgaccgccgc ggacacggct gtgtattact gtgcgagagg cggatacgat 360
gataatagag gttattacgc gtattttgac tactggggcc agggaaccct ggtcaccgtc 420
tcctcagcgt cgaccaaggg cccatcggtc ttccccctgg caccctcctc caagagcacc 480
tctgggggca cagcggccct gggctgcctg gtcaaggact acttccccga acctgtgacg 540
gtctcgtgga actcaggcgc cctgaccagc ggcgtgcaca ccttcccggc tgtcctacag 600
tcctcaggac tctactccct cagcagcgtg gtgaccgtgc cctccagcag cttgggcacc 660
cagacctaca tctgcaacgt gaatcacaag cccagcaaca ccaaggtgga caagagagtt 720
gagcccaaat cttgtgacaa aactcacaca tgcccaccgt gcccagcacc tgaactcctg 780
gggggaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 840
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 900
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 960
tacaacagca cgtaccgtgt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 1020
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcc cagcccccat cgagaaaacc 1080
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 1140
gaggagatga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatcccagc 1200
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1260
cccgtgctgg actccgacgg ctccttcttc ctctatagca agctcaccgt ggacaagagc 1320
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1380
tacacgcaga agagcctctc cctgtccccg ggtaaa 1416
Sequence Number (ID): 280
Length: 1452
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1452
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg ggctgaggtg aagaagcctg gggcctcagt gaaggtctcc 120
tgcaaggctt ctggatacac cttcaccggc tactatatac actgggtgcg acaggcccct 180
ggacaagggc ttgagtggat gggatggatc aaccctaaca gtggtgccac aaactatgca 240
cagaagtttc agggcagggt caccgtgacc agggacacgt ccatcagcac agcctacatg 300
gagctgagca ggctgagatc tgacgacacg gccgtgtatt actgtgcgag agttgcccaa 360
gtcgtgtcca acccactgcc ttacgatgtt ttgactggtt taacctatta ctactacggt 420
atggacgtct ggggccaagg gaccacggtc accgtctcct cagcgtcgac caagggccca 480
tcggtcttcc ccctggcacc ctcctccaag agcacctctg ggggcacagc ggccctgggc 540
tgcctggtca aggactactt ccccgaacct gtgacggtct cgtggaactc aggcgccctg 600
accagcggcg tgcacacctt cccggctgtc ctacagtcct caggactcta ctccctcagc 660
agcgtggtga ccgtgccctc cagcagcttg ggcacccaga cctacatctg caacgtgaat 720
cacaagccca gcaacaccaa ggtggacaag agagttgagc ccaaatcttg tgacaaaact 780
cacacatgcc caccgtgccc agcacctgaa ctcctggggg gaccgtcagt cttcctcttc 840
cccccaaaac ccaaggacac cctcatgatc tcccggaccc ctgaggtcac atgcgtggtg 900
gtggacgtga gccacgaaga ccctgaggtc aagttcaact ggtacgtgga cggcgtggag 960
gtgcataatg ccaagacaaa gccgcgggag gagcagtaca acagcacgta ccgtgtggtc 1020
agcgtcctca ccgtcctgca ccaggactgg ctgaatggca aggagtacaa gtgcaaggtc 1080
tccaacaaag ccctcccagc ccccatcgag aaaaccatct ccaaagccaa agggcagccc 1140
cgagaaccac aggtgtacac cctgccccca tcccgggagg agatgaccaa gaaccaggtc 1200
agcctgacct gcctggtcaa aggcttctat cccagcgaca tcgccgtgga gtgggagagc 1260
aatgggcagc cggagaacaa ctacaagacc acgcctcccg tgctggactc cgacggctcc 1320
ttcttcctct atagcaagct caccgtggac aagagcaggt ggcagcaggg gaacgtcttc 1380
tcatgctccg tgatgcatga ggctctgcac aaccactaca cgcagaagag cctctccctg 1440
tccccgggta aa 1452
Sequence Number (ID): 281
Length: 1410
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1410
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg ggctgaggtg aagaagcctg gggcctcagt gaaggtctcc 120
tgcaaggctt ctggatacac cttcaccggc tactatatgc actgggtgcg acaggcccct 180
ggacaagggc ttgagtggat gggatggatc aaccctaaca gtggtggcac aaactatgca 240
cagaagtttc agggcagggt caccatgacc agggacacgt ccatcaccac agcctacatg 300
gagctgagca ggctgagatc tgacgacacg gccgtgtatt actgtgcgag agactactac 360
tatgatagta gtgtgaaccc tgactactgg ggccagggaa ccctggtcac cgtctcctca 420
gcgtcgacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 480
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaacctgt gacggtctcg 540
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 600
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 660
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 720
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 780
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 840
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 900
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 960
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 1020
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1080
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 1140
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1200
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1260
ctggactccg acggctcctt cttcctctat agcaagctca ccgtggacaa gagcaggtgg 1320
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1380
cagaagagcc tctccctgtc cccgggtaaa 1410
Sequence Number (ID): 282
Length: 1416
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1416
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgc aggagtcggg cccaggactg gtgaagcctt cacagaccct gtccctcacc 120
tgcactgtct ctggtggctc catcagcagt ggtgattact actggagctg gatccgccag 180
cacccaggga agggcctgga gtggattggg tacatctttt acagtgggag cacctactac 240
aacccgtccc tcaagagtcg ggttaccata tcagtagaca cgtctaagaa ccagttctcc 300
ctgaagctga gctctgtgac tgccgcggac acggccgtgt attactgtgc gaggaggtgc 360
cgttttgtag atacatctat ggcatttgac tactggggcc agggaaccct ggtcaccgtc 420
tcctcagcgt cgaccaaggg cccatcggtc ttccccctgg caccctcctc caagagcacc 480
tctgggggca cagcggccct gggctgcctg gtcaaggact acttccccga acctgtgacg 540
gtctcgtgga actcaggcgc cctgaccagc ggcgtgcaca ccttcccggc tgtcctacag 600
tcctcaggac tctactccct cagcagcgtg gtgaccgtgc cctccagcag cttgggcacc 660
cagacctaca tctgcaacgt gaatcacaag cccagcaaca ccaaggtgga caagagagtt 720
gagcccaaat cttgtgacaa aactcacaca tgcccaccgt gcccagcacc tgaactcctg 780
gggggaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 840
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 900
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 960
tacaacagca cgtaccgtgt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 1020
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcc cagcccccat cgagaaaacc 1080
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 1140
gaggagatga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatcccagc 1200
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1260
cccgtgctgg actccgacgg ctccttcttc ctctatagca agctcaccgt ggacaagagc 1320
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1380
tacacgcaga agagcctctc cctgtccccg ggtaaa 1416
Sequence Number (ID): 283
Length: 1410
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1410
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg ggctgaggtg aagaagcctg ggtcctcggt gaaggtctcc 120
tgcaaggctt ctggaggcac cttcatcacc tatgctttca gctgggtacg acaggcccct 180
ggacaagggc ttgagtggat gggaaggatc atccctatcc ttggtataac aaactacgca 240
cagaagttcc agggcagagt cacgattacc gcggacaaat ccacgagcac agcctacatg 300
gagctgagca gcctgagatc tgaggacacg gccgtgtatt actgtgcgag agggggacgc 360
ggtgctaacc ccggttggtt tgactactgg ggccagggaa ccctggtcac cgtctcctca 420
gcgtcgacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 480
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaacctgt gacggtctcg 540
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 600
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 660
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 720
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 780
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 840
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 900
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 960
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 1020
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1080
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 1140
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1200
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1260
ctggactccg acggctcctt cttcctctat agcaagctca ccgtggacaa gagcaggtgg 1320
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1380
cagaagagcc tctccctgtc cccgggtaaa 1410
Sequence Number (ID): 284
Length: 1416
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1416
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg ggctgaggtg aagaagcctg ggtcctcggt gaaggtctcc 120
tgcaaggctt ctggaggcac cttcagcaac tatgctatca gctgggtgcg acaggcccct 180
ggacaagggc ttgagtggat gggaaggatc atccctatcc ttggtatagc aaactacgca 240
cagaagttcc agggcagact cacgattacc gcggacaaat ccacgagcac agcctacatg 300
gagctgagca gcctaagatc tgaggacacg gccgtgtatt actgtgcgag gacggcgtat 360
gaatatgata gtagtggtta tttccttgac tactggggcc agggaaccct ggtcaccgtc 420
tcctcagcgt cgaccaaggg cccatcggtc ttccccctgg caccctcctc caagagcacc 480
tctgggggca cagcggccct gggctgcctg gtcaaggact acttccccga acctgtgacg 540
gtctcgtgga actcaggcgc cctgaccagc ggcgtgcaca ccttcccggc tgtcctacag 600
tcctcaggac tctactccct cagcagcgtg gtgaccgtgc cctccagcag cttgggcacc 660
cagacctaca tctgcaacgt gaatcacaag cccagcaaca ccaaggtgga caagagagtt 720
gagcccaaat cttgtgacaa aactcacaca tgcccaccgt gcccagcacc tgaactcctg 780
gggggaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 840
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 900
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 960
tacaacagca cgtaccgtgt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 1020
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcc cagcccccat cgagaaaacc 1080
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 1140
gaggagatga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatcccagc 1200
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1260
cccgtgctgg actccgacgg ctccttcttc ctctatagca agctcaccgt ggacaagagc 1320
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1380
tacacgcaga agagcctctc cctgtccccg ggtaaa 1416
Sequence Number (ID): 285
Length: 1407
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1407
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgt tggagtctgg gggaggcttg gtcaagcctg gagggtccct gagactctcc 120
tgtgcagcct ctggattcac cttcagtgac tactacatga gctggatccg ccaggctcca 180
gggaaggggc tggagtgggt ttcatacatt agtagtagtg gtagtaccat atactacgca 240
gactctgtga agggccgatt caccatctcc agggacaacg ccaagaactc actgtatctg 300
caaatgaaca gcctgagagc cgaggacacg gccgtgtatt actgtgcgag gcatcctatg 360
gttcggggag ttatcggaga ctactggggc cagggaaccc tggtcaccgt ctcctcagcg 420
tcgaccaagg gcccatcggt cttccccctg gcaccctcct ccaagagcac ctctgggggc 480
acagcggccc tgggctgcct ggtcaaggac tacttccccg aacctgtgac ggtctcgtgg 540
aactcaggcg ccctgaccag cggcgtgcac accttcccgg ctgtcctaca gtcctcagga 600
ctctactccc tcagcagcgt ggtgaccgtg ccctccagca gcttgggcac ccagacctac 660
atctgcaacg tgaatcacaa gcccagcaac accaaggtgg acaagagagt tgagcccaaa 720
tcttgtgaca aaactcacac atgcccaccg tgcccagcac ctgaactcct ggggggaccg 780
tcagtcttcc tcttcccccc aaaacccaag gacaccctca tgatctcccg gacccctgag 840
gtcacatgcg tggtggtgga cgtgagccac gaagaccctg aggtcaagtt caactggtac 900
gtggacggcg tggaggtgca taatgccaag acaaagccgc gggaggagca gtacaacagc 960
acgtaccgtg tggtcagcgt cctcaccgtc ctgcaccagg actggctgaa tggcaaggag 1020
tacaagtgca aggtctccaa caaagccctc ccagccccca tcgagaaaac catctccaaa 1080
gccaaagggc agccccgaga accacaggtg tacaccctgc ccccatcccg ggaggagatg 1140
accaagaacc aggtcagcct gacctgcctg gtcaaaggct tctatcccag cgacatcgcc 1200
gtggagtggg agagcaatgg gcagccggag aacaactaca agaccacgcc tcccgtgctg 1260
gactccgacg gctccttctt cctctatagc aagctcaccg tggacaagag caggtggcag 1320
caggggaacg tcttctcatg ctccgtgatg catgaggctc tgcacaacca ctacacgcag 1380
aagagcctct ccctgtcccc gggtaaa 1407
Sequence Number (ID): 286
Length: 1422
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1422
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg ggctgaggtg aagaagcctg gggcctcagt gaaggtttcc 120
tgcaaggcat ctggatacac cttcaccagc tactatatgc actgggtgcg acaggcccct 180
ggacaagggc ttgagtggat gggaataatc aaccctagtg gaggtagcac aagctacgta 240
cagaagttcc agggcagagt caccatgacc agggacacgt ccacgagcac agtctacatg 300
gagctgagca gcctgagatt tgaggacacg gccgtgtatt actgtgcgag aggtcatagt 360
ggctacgatc ggttacctca acccccttac tttgactact ggggccaggg aaccctggtc 420
accgtctcct cagcgtcgac caagggccca tcggtcttcc ccctggcacc ctcctccaag 480
agcacctctg ggggcacagc ggccctgggc tgcctggtca aggactactt ccccgaacct 540
gtgacggtct cgtggaactc aggcgccctg accagcggcg tgcacacctt cccggctgtc 600
ctacagtcct caggactcta ctccctcagc agcgtggtga ccgtgccctc cagcagcttg 660
ggcacccaga cctacatctg caacgtgaat cacaagccca gcaacaccaa ggtggacaag 720
agagttgagc ccaaatcttg tgacaaaact cacacatgcc caccgtgccc agcacctgaa 780
ctcctggggg gaccgtcagt cttcctcttc cccccaaaac ccaaggacac cctcatgatc 840
tcccggaccc ctgaggtcac atgcgtggtg gtggacgtga gccacgaaga ccctgaggtc 900
aagttcaact ggtacgtgga cggcgtggag gtgcataatg ccaagacaaa gccgcgggag 960
gagcagtaca acagcacgta ccgtgtggtc agcgtcctca ccgtcctgca ccaggactgg 1020
ctgaatggca aggagtacaa gtgcaaggtc tccaacaaag ccctcccagc ccccatcgag 1080
aaaaccatct ccaaagccaa agggcagccc cgagaaccac aggtgtacac cctgccccca 1140
tcccgggagg agatgaccaa gaaccaggtc agcctgacct gcctggtcaa aggcttctat 1200
cccagcgaca tcgccgtgga gtgggagagc aatgggcagc cggagaacaa ctacaagacc 1260
acgcctcccg tgctggactc cgacggctcc ttcttcctct atagcaagct caccgtggac 1320
aagagcaggt ggcagcaggg gaacgtcttc tcatgctccg tgatgcatga ggctctgcac 1380
aaccactaca cgcagaagag cctctccctg tccccgggta aa 1422
Sequence Number (ID): 287
Length: 1410
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1410
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg ggctgaggtg aagaagcctg gggcctcagt gaaggtctcc 120
tgcaaggctt ctggatacct cttcaccggc tactatatgc actgggtgcg acaggcccct 180
ggacaagggc ttgagtggat gggatggatc aaccctaaca gtggtggcac aaactatgca 240
cagaagtttc aggacagggt caccatgacc agggacacgt ccatcaccac agcctacatg 300
gagctgagca ggctgagatc tgacgacacg gccgtgtatt actgtgccag gagtggttat 360
tatacgagtg ctgtaaactt tgactactgg ggccagggaa ccctggtcac cgtctcctca 420
gcgtcgacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 480
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaacctgt gacggtctcg 540
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 600
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 660
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 720
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 780
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 840
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 900
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 960
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 1020
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1080
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 1140
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1200
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1260
ctggactccg acggctcctt cttcctctat agcaagctca ccgtggacaa gagcaggtgg 1320
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1380
cagaagagcc tctccctgtc cccgggtaaa 1410
Sequence Number (ID): 288
Length: 1443
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1443
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgt tggagtctgg gggaggcttg gtacagcctg gggggtccct gagactctcc 120
tgtgcagcct ctggattcac ctttagcagc tatgccatga gttgggtccg ccaggctcca 180
gggaaggggc tggagtgggt ctcagctgtt agtggtagtg gtggtagcac atactacgca 240
gactccgtga agggccggtt caccatctcc agagacaatt ccaagaacat gctgtatctg 300
caaatgaaca gcctgagagc cgaggacacg gccgtatatt actgtgcgaa aaccgttttg 360
gctggtcgcg acgatagcag tggctacttc acggtttact actacttcgg tatggacgtc 420
tggggccagg ggaccacggt caccgtctcc tcagcgtcga ccaagggccc atcggtcttc 480
cccctggcac cctcctccaa gagcacctct gggggcacag cggccctggg ctgcctggtc 540
aaggactact tccccgaacc tgtgacggtc tcgtggaact caggcgccct gaccagcggc 600
gtgcacacct tcccggctgt cctacagtcc tcaggactct actccctcag cagcgtggtg 660
accgtgccct ccagcagctt gggcacccag acctacatct gcaacgtgaa tcacaagccc 720
agcaacacca aggtggacaa gagagttgag cccaaatctt gtgacaaaac tcacacatgc 780
ccaccgtgcc cagcacctga actcctgggg ggaccgtcag tcttcctctt ccccccaaaa 840
cccaaggaca ccctcatgat ctcccggacc cctgaggtca catgcgtggt ggtggacgtg 900
agccacgaag accctgaggt caagttcaac tggtacgtgg acggcgtgga ggtgcataat 960
gccaagacaa agccgcggga ggagcagtac aacagcacgt accgtgtggt cagcgtcctc 1020
accgtcctgc accaggactg gctgaatggc aaggagtaca agtgcaaggt ctccaacaaa 1080
gccctcccag cccccatcga gaaaaccatc tccaaagcca aagggcagcc ccgagaacca 1140
caggtgtaca ccctgccccc atcccgggag gagatgacca agaaccaggt cagcctgacc 1200
tgcctggtca aaggcttcta tcccagcgac atcgccgtgg agtgggagag caatgggcag 1260
ccggagaaca actacaagac cacgcctccc gtgctggact ccgacggctc cttcttcctc 1320
tatagcaagc tcaccgtgga caagagcagg tggcagcagg ggaacgtctt ctcatgctcc 1380
gtgatgcatg aggctctgca caaccactac acgcagaaga gcctctccct gtccccgggt 1440
aaa 1443
Sequence Number (ID): 289
Length: 1416
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1416
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg gcctgaggtg aagaagcctg ggacctcagt gaaggtctcc 120
tgcaaggctt ctggattcac ctttcctagc tctgctgtgc agtgggtgcg acaggctcgt 180
ggacaacgcc ttgagtggat aggatggatc gtcgttggca gtggtaacac aaactacgca 240
cagaagttcc aggaaagagt caccattacc agggacatgt ccacaagcac agcctacatg 300
gagctgagca gcctgagatc cgaggacacg gccgtgtatt actgtgcggc tccctattgt 360
agtggtggta gctgttatga tggttttgat atctggggcc aagggacaat ggtcaccgtc 420
tcttcagcgt cgaccaaggg cccatcggtc ttccccctgg caccctcctc caagagcacc 480
tctgggggca cagcggccct gggctgcctg gtcaaggact acttccccga acctgtgacg 540
gtctcgtgga actcaggcgc cctgaccagc ggcgtgcaca ccttcccggc tgtcctacag 600
tcctcaggac tctactccct cagcagcgtg gtgaccgtgc cctccagcag cttgggcacc 660
cagacctaca tctgcaacgt gaatcacaag cccagcaaca ccaaggtgga caagagagtt 720
gagcccaaat cttgtgacaa aactcacaca tgcccaccgt gcccagcacc tgaactcctg 780
gggggaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 840
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 900
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 960
tacaacagca cgtaccgtgt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 1020
ggcaaggagt acaagtgcaa ggtctccaac aaagccctcc cagcccccat cgagaaaacc 1080
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 1140
gaggagatga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatcccagc 1200
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 1260
cccgtgctgg actccgacgg ctccttcttc ctctatagca agctcaccgt ggacaagagc 1320
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 1380
tacacgcaga agagcctctc cctgtccccg ggtaaa 1416
Sequence Number (ID): 290
Length: 1440
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1440
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg ggctgaggtg aagaagcctg gggcctcagt gaaggtctcc 120
tgcaaggctt ctggatacac cttcaccaat tatgatatca actgggtgcg acaggccact 180
ggacaagggc ttgagtggat gggatggatg aaccctaaca gtggtaacac aggctttgca 240
cagaagttcc agggcagagt caccatgacc aggaacacct ccataagaac agtctacatg 300
gagctgagca gcctgagatc tgaggacacg gccgtgtatt actgtgcgag agcctatcaa 360
aatagaccga tagcagtaac tgatatgggt tactactact actacggtat ggacgtctgg 420
ggccaaggga ccacggtcac cgtctcctca gcgtcgacca agggcccatc ggtcttcccc 480
ctggcaccct cctccaagag cacctctggg ggcacagcgg ccctgggctg cctggtcaag 540
gactacttcc ccgaacctgt gacggtctcg tggaactcag gcgccctgac cagcggcgtg 600
cacaccttcc cggctgtcct acagtcctca ggactctact ccctcagcag cgtggtgacc 660
gtgccctcca gcagcttggg cacccagacc tacatctgca acgtgaatca caagcccagc 720
aacaccaagg tggacaagag agttgagccc aaatcttgtg acaaaactca cacatgccca 780
ccgtgcccag cacctgaact cctgggggga ccgtcagtct tcctcttccc cccaaaaccc 840
aaggacaccc tcatgatctc ccggacccct gaggtcacat gcgtggtggt ggacgtgagc 900
cacgaagacc ctgaggtcaa gttcaactgg tacgtggacg gcgtggaggt gcataatgcc 960
aagacaaagc cgcgggagga gcagtacaac agcacgtacc gtgtggtcag cgtcctcacc 1020
gtcctgcacc aggactggct gaatggcaag gagtacaagt gcaaggtctc caacaaagcc 1080
ctcccagccc ccatcgagaa aaccatctcc aaagccaaag ggcagccccg agaaccacag 1140
gtgtacaccc tgcccccatc ccgggaggag atgaccaaga accaggtcag cctgacctgc 1200
ctggtcaaag gcttctatcc cagcgacatc gccgtggagt gggagagcaa tgggcagccg 1260
gagaacaact acaagaccac gcctcccgtg ctggactccg acggctcctt cttcctctat 1320
agcaagctca ccgtggacaa gagcaggtgg cagcagggga acgtcttctc atgctccgtg 1380
atgcatgagg ctctgcacaa ccactacacg cagaagagcc tctccctgtc cccgggtaaa 1440
Sequence Number (ID): 291
Length: 1419
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1419
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
ctgcagctgc aggagtcggg cccaggactg gtgaagcctt cggagaccct gtccctcacc 120
tgcactgtct ctggtggctc catcagcagt agtagttact actggggctg gatccgccag 180
cccccaggga aggggctgga gtggattggg agtatctatt atagtgggag cacctactac 240
aacccgtccc tcaagagtcg agtcaccata tcagtagaca cgtccaagaa ccagttctcc 300
ctgaagctga gctctgtgac cgccgcggac acggccttgt attactgtgc gagagggggg 360
tcgatctatg actacggtga ctacgaattt gactcctggg gccagggaac cctggtcacc 420
gtctcctcag cgtcgaccaa gggcccatcg gtcttccccc tggcaccctc ctccaagagc 480
acctctgggg gcacagcggc cctgggctgc ctggtcaagg actacttccc cgaacctgtg 540
acggtctcgt ggaactcagg cgccctgacc agcggcgtgc acaccttccc ggctgtccta 600
cagtcctcag gactctactc cctcagcagc gtggtgaccg tgccctccag cagcttgggc 660
acccagacct acatctgcaa cgtgaatcac aagcccagca acaccaaggt ggacaagaga 720
gttgagccca aatcttgtga caaaactcac acatgcccac cgtgcccagc acctgaactc 780
ctggggggac cgtcagtctt cctcttcccc ccaaaaccca aggacaccct catgatctcc 840
cggacccctg aggtcacatg cgtggtggtg gacgtgagcc acgaagaccc tgaggtcaag 900
ttcaactggt acgtggacgg cgtggaggtg cataatgcca agacaaagcc gcgggaggag 960
cagtacaaca gcacgtaccg tgtggtcagc gtcctcaccg tcctgcacca ggactggctg 1020
aatggcaagg agtacaagtg caaggtctcc aacaaagccc tcccagcccc catcgagaaa 1080
accatctcca aagccaaagg gcagccccga gaaccacagg tgtacaccct gcccccatcc 1140
cgggaggaga tgaccaagaa ccaggtcagc ctgacctgcc tggtcaaagg cttctatccc 1200
agcgacatcg ccgtggagtg ggagagcaat gggcagccgg agaacaacta caagaccacg 1260
cctcccgtgc tggactccga cggctccttc ttcctctata gcaagctcac cgtggacaag 1320
agcaggtggc agcaggggaa cgtcttctca tgctccgtga tgcatgaggc tctgcacaac 1380
cactacacgc agaagagcct ctccctgtcc ccgggtaaa 1419
Sequence Number (ID): 292
Length: 1410
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1410
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcccag 60
gtgcagctgg tgcagtctgg ggctgaggtg aagaagcctg gggcctcagt gaaggtttcc 120
tgcaaggcat ctggatacac cttcaccagc tactatatgc actgggtgcg acaggcccct 180
ggacaagggc ttgagtggat gggaataatc aaccctagtg gtggtagcac aagctacgca 240
cagaagttcc agggcagagt caccatgacc agggacacgt ccacgagcac agtctacatg 300
gagctgagca gcctgagatc tgaggacacg gccgtgtatt actgtgcgat attagggtta 360
catttggggg agttattatt cgacccctgg ggccagggaa ccctggtcac cgtctcctca 420
gcgtcgacca agggcccatc ggtcttcccc ctggcaccct cctccaagag cacctctggg 480
ggcacagcgg ccctgggctg cctggtcaag gactacttcc ccgaacctgt gacggtctcg 540
tggaactcag gcgccctgac cagcggcgtg cacaccttcc cggctgtcct acagtcctca 600
ggactctact ccctcagcag cgtggtgacc gtgccctcca gcagcttggg cacccagacc 660
tacatctgca acgtgaatca caagcccagc aacaccaagg tggacaagag agttgagccc 720
aaatcttgtg acaaaactca cacatgccca ccgtgcccag cacctgaact cctgggggga 780
ccgtcagtct tcctcttccc cccaaaaccc aaggacaccc tcatgatctc ccggacccct 840
gaggtcacat gcgtggtggt ggacgtgagc cacgaagacc ctgaggtcaa gttcaactgg 900
tacgtggacg gcgtggaggt gcataatgcc aagacaaagc cgcgggagga gcagtacaac 960
agcacgtacc gtgtggtcag cgtcctcacc gtcctgcacc aggactggct gaatggcaag 1020
gagtacaagt gcaaggtctc caacaaagcc ctcccagccc ccatcgagaa aaccatctcc 1080
aaagccaaag ggcagccccg agaaccacag gtgtacaccc tgcccccatc ccgggaggag 1140
atgaccaaga accaggtcag cctgacctgc ctggtcaaag gcttctatcc cagcgacatc 1200
gccgtggagt gggagagcaa tgggcagccg gagaacaact acaagaccac gcctcccgtg 1260
ctggactccg acggctcctt cttcctctat agcaagctca ccgtggacaa gagcaggtgg 1320
cagcagggga acgtcttctc atgctccgtg atgcatgagg ctctgcacaa ccactacacg 1380
cagaagagcc tctccctgtc cccgggtaaa 1410
Sequence Number (ID): 293
Length: 1398
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1398
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgg tggagtctgg aggaggcttg atccagcctg gggggtccct gagactctcc 120
tgtgcagcct cggggttcac cgtcagtagc aactacatga gctgggtccg ccaggctcca 180
gggaaggggc tggagtgggt ctcagttatt tatagcggtg gtagcacatt ctacgcagac 240
tccgtgaagg gccgattcac catctccaga gacaattcca agaacacgct gtatcttcaa 300
atgaacagcc tgagagccga ggacacggcc gtgtattact gtgcgagaga tttcggagaa 360
cagtggtttg actactgggg ccagggaacc ctggtcaccg tctcctcagc gtcgaccaag 420
ggcccatcgg tcttccccct ggcaccctcc tccaagagca cctctggggg cacagcggcc 480
ctgggctgcc tggtcaagga ctacttcccc gaacctgtga cggtctcgtg gaactcaggc 540
gccctgacca gcggcgtgca caccttcccg gctgtcctac agtcctcagg actctactcc 600
ctcagcagcg tggtgaccgt gccctccagc agcttgggca cccagaccta catctgcaac 660
gtgaatcaca agcccagcaa caccaaggtg gacaagagag ttgagcccaa atcttgtgac 720
aaaactcaca catgcccacc gtgcccagca cctgaactcc tggggggacc gtcagtcttc 780
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 840
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 900
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agtacaacag cacgtaccgt 960
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 1020
aaggtctcca acaaagccct cccagccccc atcgagaaaa ccatctccaa agccaaaggg 1080
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 1140
caggtcagcc tgacctgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1200
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1260
ggctccttct tcctctatag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1320
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1380
tccctgtccc cgggtaaa 1398
Sequence Number (ID): 294
Length: 1398
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1398
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgg tggagtctgg gggaggcttg gtccagcctg gggggtccct gagactctcc 120
tgtgcagcct ctgaattcac cgtcagtagc aactacatga gctgggtccg ccaggctcca 180
gggaaggggc tggagtgggt ctcagttatt tatagcggtg gtagcacata ctacgcagac 240
tccgtgaagg gcagattcac catctccaga gacaattcca agaacacgct gtatcttcaa 300
atgaacagcc tgagagccga ggacacggct gtgtattact gtgcgagaga tttcggggac 360
tactactttg actactgggg ccagggaacc ctggtcaccg tctcctcagc gtcgaccaag 420
ggcccatcgg tcttccccct ggcaccctcc tccaagagca cctctggggg cacagcggcc 480
ctgggctgcc tggtcaagga ctacttcccc gaacctgtga cggtctcgtg gaactcaggc 540
gccctgacca gcggcgtgca caccttcccg gctgtcctac agtcctcagg actctactcc 600
ctcagcagcg tggtgaccgt gccctccagc agcttgggca cccagaccta catctgcaac 660
gtgaatcaca agcccagcaa caccaaggtg gacaagagag ttgagcccaa atcttgtgac 720
aaaactcaca catgcccacc gtgcccagca cctgaactcc tggggggacc gtcagtcttc 780
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 840
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 900
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agtacaacag cacgtaccgt 960
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 1020
aaggtctcca acaaagccct cccagccccc atcgagaaaa ccatctccaa agccaaaggg 1080
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 1140
caggtcagcc tgacctgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1200
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1260
ggctccttct tcctctatag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1320
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1380
tccctgtccc cgggtaaa 1398
Sequence Number (ID): 295
Length: 1401
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1401
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgg tggagactgg aggaggcttg atccagcctg gggggtccct gagactctcc 120
tgtgcagcct ctggcttcac cgtcagtagg aactacatga gctgggtccg ccaggctcca 180
gggaaggggc tggagtgggt ctcacttatt tatagtggtg gtagcacatt ctacgcagac 240
tccgtgaagg gccgattcac catctccaga gacgattcca agaacacgtt gtatcttcaa 300
atgaacagcc tgagagccga ggacacggcc gtgtattact gtgcgagaga tctcctcgag 360
gtgggaggca gcgactactg gggccaggga accctggtca ccgtctcctc agcgtcgacc 420
aagggcccat cggtcttccc cctggcaccc tcctccaaga gcacctctgg gggcacagcg 480
gccctgggct gcctggtcaa ggactacttc cccgaacctg tgacggtctc gtggaactca 540
ggcgccctga ccagcggcgt gcacaccttc ccggctgtcc tacagtcctc aggactctac 600
tccctcagca gcgtggtgac cgtgccctcc agcagcttgg gcacccagac ctacatctgc 660
aacgtgaatc acaagcccag caacaccaag gtggacaaga gagttgagcc caaatcttgt 720
gacaaaactc acacatgccc accgtgccca gcacctgaac tcctgggggg accgtcagtc 780
ttcctcttcc ccccaaaacc caaggacacc ctcatgatct cccggacccc tgaggtcaca 840
tgcgtggtgg tggacgtgag ccacgaagac cctgaggtca agttcaactg gtacgtggac 900
ggcgtggagg tgcataatgc caagacaaag ccgcgggagg agcagtacaa cagcacgtac 960
cgtgtggtca gcgtcctcac cgtcctgcac caggactggc tgaatggcaa ggagtacaag 1020
tgcaaggtct ccaacaaagc cctcccagcc cccatcgaga aaaccatctc caaagccaaa 1080
gggcagcccc gagaaccaca ggtgtacacc ctgcccccat cccgggagga gatgaccaag 1140
aaccaggtca gcctgacctg cctggtcaaa ggcttctatc ccagcgacat cgccgtggag 1200
tgggagagca atgggcagcc ggagaacaac tacaagacca cgcctcccgt gctggactcc 1260
gacggctcct tcttcctcta tagcaagctc accgtggaca agagcaggtg gcagcagggg 1320
aacgtcttct catgctccgt gatgcatgag gctctgcaca accactacac gcagaagagc 1380
ctctccctgt ccccgggtaa a 1401
Sequence Number (ID): 296
Length: 1431
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1431
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgt tggagtctgg gggaggcttg gtacagcctg gggggtccct gagactctcc 120
tgtgcagcct ctggattcac ctttagcagc tatgccatga gctgggtccg ccaggctcca 180
gggaaggggc tggagtgggt ctcagctatt agtggtagtg gtggtagcac atactacgca 240
gactccgtga agggccggtt caccatctcc agagacaatt ccaagaacac gctgtatctg 300
caaatgaaca gcctgagagc cgaggacacg gccgtatatt actgtgcgaa accactcttg 360
gggtacgatt tttggagtgg ttattatcgg ggtggctggt tcgacccctg gggccaggga 420
accctggtca ccgtctcctc agcgtcgacc aagggcccat cggtcttccc cctggcaccc 480
tcctccaaga gcacctctgg gggcacagcg gccctgggct gcctggtcaa ggactacttc 540
cccgaacctg tgacggtctc gtggaactca ggcgccctga ccagcggcgt gcacaccttc 600
ccggctgtcc tacagtcctc aggactctac tccctcagca gcgtggtgac cgtgccctcc 660
agcagcttgg gcacccagac ctacatctgc aacgtgaatc acaagcccag caacaccaag 720
gtggacaaga gagttgagcc caaatcttgt gacaaaactc acacatgccc accgtgccca 780
gcacctgaac tcctgggggg accgtcagtc ttcctcttcc ccccaaaacc caaggacacc 840
ctcatgatct cccggacccc tgaggtcaca tgcgtggtgg tggacgtgag ccacgaagac 900
cctgaggtca agttcaactg gtacgtggac ggcgtggagg tgcataatgc caagacaaag 960
ccgcgggagg agcagtacaa cagcacgtac cgtgtggtca gcgtcctcac cgtcctgcac 1020
caggactggc tgaatggcaa ggagtacaag tgcaaggtct ccaacaaagc cctcccagcc 1080
cccatcgaga aaaccatctc caaagccaaa gggcagcccc gagaaccaca ggtgtacacc 1140
ctgcccccat cccgggagga gatgaccaag aaccaggtca gcctgacctg cctggtcaaa 1200
ggcttctatc ccagcgacat cgccgtggag tgggagagca atgggcagcc ggagaacaac 1260
tacaagacca cgcctcccgt gctggactcc gacggctcct tcttcctcta tagcaagctc 1320
accgtggaca agagcaggtg gcagcagggg aacgtcttct catgctccgt gatgcatgag 1380
gctctgcaca accactacac gcagaagagc ctctccctgt ccccgggtaa a 1431
Sequence Number (ID): 297
Length: 1404
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1404
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgg tggagtctgg aggaggcttg atccagcctg gggggtccct gagactctcc 120
tgtgcagcct ctgggttcac cgtcagtagc aactacatga gctgggtccg ccaggctcca 180
gggaaggggc tggagtgggt ctcagttatt tatagcggtg gtagcacata ctacgcagac 240
tccgtgaagg gccgattcat catctccaga gacaattcca agaacacgct gtatcttcaa 300
atgaacagcc tgagagccga ggacacggcc gtgtattact gtgcgagaga tccatacagc 360
tatggtcgag gtggcggcta ctggggccag ggaaccctgg tcaccgtctc ctcagcgtcg 420
accaagggcc catcggtctt ccccctggca ccctcctcca agagcacctc tgggggcaca 480
gcggccctgg gctgcctggt caaggactac ttccccgaac ctgtgacggt ctcgtggaac 540
tcaggcgccc tgaccagcgg cgtgcacacc ttcccggctg tcctacagtc ctcaggactc 600
tactccctca gcagcgtggt gaccgtgccc tccagcagct tgggcaccca gacctacatc 660
tgcaacgtga atcacaagcc cagcaacacc aaggtggaca agagagttga gcccaaatct 720
tgtgacaaaa ctcacacatg cccaccgtgc ccagcacctg aactcctggg gggaccgtca 780
gtcttcctct tccccccaaa acccaaggac accctcatga tctcccggac ccctgaggtc 840
acatgcgtgg tggtggacgt gagccacgaa gaccctgagg tcaagttcaa ctggtacgtg 900
gacggcgtgg aggtgcataa tgccaagaca aagccgcggg aggagcagta caacagcacg 960
taccgtgtgg tcagcgtcct caccgtcctg caccaggact ggctgaatgg caaggagtac 1020
aagtgcaagg tctccaacaa agccctccca gcccccatcg agaaaaccat ctccaaagcc 1080
aaagggcagc cccgagaacc acaggtgtac accctgcccc catcccggga ggagatgacc 1140
aagaaccagg tcagcctgac ctgcctggtc aaaggcttct atcccagcga catcgccgtg 1200
gagtgggaga gcaatgggca gccggagaac aactacaaga ccacgcctcc cgtgctggac 1260
tccgacggct ccttcttcct ctatagcaag ctcaccgtgg acaagagcag gtggcagcag 1320
gggaacgtct tctcatgctc cgtgatgcat gaggctctgc acaaccacta cacgcagaag 1380
agcctctccc tgtccccggg taaa 1404
Sequence Number (ID): 298
Length: 1398
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1398
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgg tggagactgg aggaggcttg atccagcctg gggggtccct gagactctcc 120
tgtgcagcct ctgggctcac cgtcagtagc aactacatga actgggtccg ccaggctcca 180
gggaaggggc tggagtgggt ctcagttatt tatagcggtg gtagcacata ctacgcagac 240
tccgtgaagg gccgattcac catctccaga gacaattcca agaacacgct gtatcttcaa 300
atgaacagcc tgagagccga ggacacggcc gtgtattact gtgcgagaga tggggtctac 360
tacggtatgg acgtctgggg ccaagggacc acggtcaccg tctcctcagc gtcgaccaag 420
ggcccatcgg tcttccccct ggcaccctcc tccaagagca cctctggggg cacagcggcc 480
ctgggctgcc tggtcaagga ctacttcccc gaacctgtga cggtctcgtg gaactcaggc 540
gccctgacca gcggcgtgca caccttcccg gctgtcctac agtcctcagg actctactcc 600
ctcagcagcg tggtgaccgt gccctccagc agcttgggca cccagaccta catctgcaac 660
gtgaatcaca agcccagcaa caccaaggtg gacaagagag ttgagcccaa atcttgtgac 720
aaaactcaca catgcccacc gtgcccagca cctgaactcc tggggggacc gtcagtcttc 780
ctcttccccc caaaacccaa ggacaccctc atgatctccc ggacccctga ggtcacatgc 840
gtggtggtgg acgtgagcca cgaagaccct gaggtcaagt tcaactggta cgtggacggc 900
gtggaggtgc ataatgccaa gacaaagccg cgggaggagc agtacaacag cacgtaccgt 960
gtggtcagcg tcctcaccgt cctgcaccag gactggctga atggcaagga gtacaagtgc 1020
aaggtctcca acaaagccct cccagccccc atcgagaaaa ccatctccaa agccaaaggg 1080
cagccccgag aaccacaggt gtacaccctg cccccatccc gggaggagat gaccaagaac 1140
caggtcagcc tgacctgcct ggtcaaaggc ttctatccca gcgacatcgc cgtggagtgg 1200
gagagcaatg ggcagccgga gaacaactac aagaccacgc ctcccgtgct ggactccgac 1260
ggctccttct tcctctatag caagctcacc gtggacaaga gcaggtggca gcaggggaac 1320
gtcttctcat gctccgtgat gcatgaggct ctgcacaacc actacacgca gaagagcctc 1380
tccctgtccc cgggtaaa 1398
Sequence Number (ID): 299
Length: 1419
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..1419
> mol_type, other DNA
> note, heavy chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgag 60
gtgcagctgg tggagtctgg gggaggcgtg gtccagcctg ggaggtccct gagactctcc 120
tgtgcagcct ctggattcac cttcagtagc tatggcatgc actgggtccg ccaggctcca 180
ggcaaggggc tggagtgggt ggcagttata tcatatgatg gaagtaataa atactacgca 240
gactccgtga agggccgatt caccatctcc agagacaatt ccaagaacac gctgtatctg 300
caaatgaaca gcctgagagc tgaggacacg gctgtgtatt actgtgcgaa ggattacgat 360
ttttggagtg gttattaccc ccagcccttt gactactggg gccagggaac cctggtcacc 420
gtctcctcag cgtcgaccaa gggcccatcg gtcttccccc tggcaccctc ctccaagagc 480
acctctgggg gcacagcggc cctgggctgc ctggtcaagg actacttccc cgaacctgtg 540
acggtctcgt ggaactcagg cgccctgacc agcggcgtgc acaccttccc ggctgtccta 600
cagtcctcag gactctactc cctcagcagc gtggtgaccg tgccctccag cagcttgggc 660
acccagacct acatctgcaa cgtgaatcac aagcccagca acaccaaggt ggacaagaga 720
gttgagccca aatcttgtga caaaactcac acatgcccac cgtgcccagc acctgaactc 780
ctggggggac cgtcagtctt cctcttcccc ccaaaaccca aggacaccct catgatctcc 840
cggacccctg aggtcacatg cgtggtggtg gacgtgagcc acgaagaccc tgaggtcaag 900
ttcaactggt acgtggacgg cgtggaggtg cataatgcca agacaaagcc gcgggaggag 960
cagtacaaca gcacgtaccg tgtggtcagc gtcctcaccg tcctgcacca ggactggctg 1020
aatggcaagg agtacaagtg caaggtctcc aacaaagccc tcccagcccc catcgagaaa 1080
accatctcca aagccaaagg gcagccccga gaaccacagg tgtacaccct gcccccatcc 1140
cgggaggaga tgaccaagaa ccaggtcagc ctgacctgcc tggtcaaagg cttctatccc 1200
agcgacatcg ccgtggagtg ggagagcaat gggcagccgg agaacaacta caagaccacg 1260
cctcccgtgc tggactccga cggctccttc ttcctctata gcaagctcac cgtggacaag 1320
agcaggtggc agcaggggaa cgtcttctca tgctccgtga tgcatgaggc tctgcacaac 1380
cactacacgc agaagagcct ctccctgtcc ccgggtaaa 1419
Sequence Number (ID): 300
Length: 699
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..699
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc atcctccctg tctgcatctg taggagacag agtcaccatc 120
acttgccggg caagtcagag cattagcagc tatttaaatt ggtatcagca gaaaccaggg 180
aaagccccta agctcctgat ctatgctgca tccagtttgc aaagtggggt cccatcaagg 240
ttcagtggca gtggatctgg gacagatttc actctcacca tcagcagtct gcaacctgaa 300
gattttgcaa cttactactg tcaacagagt tacagtaccc ctggcagttt tggccagggg 360
accaagctgg agatcaaacg tacggtggct gcaccatctg tcttcatctt cccgccatct 420
gatgagcagt tgaaatctgg aactgcctct gttgtgtgcc tgctgaataa cttctatccc 480
agagaggcca aagtacagtg gaaggtggat aacgccctcc aatcgggtaa ctcccaggag 540
agtgtcacag agcaggacag caaggacagc acctacagcc tcagcagcac cctgacgctg 600
agcaaagcag actacgagaa acacaaagtc tacgcctgcg aagtcaccca tcagggcctg 660
agctcgcccg tcacaaagag cttcaacagg ggagagtgt 699
Sequence Number (ID): 301
Length: 705
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..705
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggttc ctgggcccag 60
tctgtgctga ctcagccacc ctcagcgtct gggacccccg ggcagagggt caccatctct 120
tgttctggaa gtagctccaa catcggaaga aatactgtaa actggtacca gcagctccca 180
ggaacggccc ccaaactcgt catctatagt aataatcagc ggccctcagg ggtccctgac 240
cgattctctg gctccaagtc tggcacctca gcctccctgg ccatcagtgg gctccagtct 300
gaggatgagg ctgattatta ctgtgcagca tgggatgaca gcctgaatgg tctggtattc 360
ggcggaggga ccaagctgac cgtcctaggt cagcccaagg ctgccccctc ggtcactctg 420
ttcccaccct cgagtgagga gcttcaagcc aacaaggcca cactggtgtg tctcataagt 480
gacttctacc cgggagccgt gacagtggcc tggaaggcag atagcagccc cgtcaaggcg 540
ggagtggaga ccaccacacc ctccaaacaa agcaacaaca agtacgcggc cagcagctac 600
ctgagcctga cgcctgagca gtggaagtcc cacagaagct acagctgcca ggtcacgcat 660
gaagggagca ccgtggagaa gacagtcgcc ccaactgagt gttct 705
Sequence Number (ID): 302
Length: 717
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..717
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcggac 60
atcgtgatga cccagtctcc agactccctg gctgtgtctc tgggcgagag ggccaccatc 120
aactgcaagt ccagccagag tgttttatac agcttcagga ataagatctg cttagcttgg 180
taccagcaga aaccaggaca gcctcctaaa ctgctcattt actgggcaac tacccgggaa 240
tccggggtcc ctgaccgatt cagtggcagc gggtctggga cagatttcac tctcaccatc 300
agcagcctgc aggctgaaga tgtggctgtt tattactgtc agcaatttta tagtactccg 360
tggacgttcg gccaagggac caaggtggaa atcaaacgta cggtggctgc accatctgtc 420
ttcatcttcc cgccatctga tgagcagttg aaatctggaa ctgcctctgt tgtgtgcctg 480
ctgaataact tctatcccag agaggccaaa gtacagtgga aggtggataa cgccctccaa 540
tcgggtaact cccaggagag tgtcacagag caggacagca aggacagcac ctacagcctc 600
agcagcaccc tgacgctgag caaagcagac tacgagaaac acaaagtcta cgcctgcgaa 660
gtcacccatc agggcctgag ctcgcccgtc acaaagagct tcaacagggg agagtgt 717
Sequence Number (ID): 303
Length: 705
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..705
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggttc ctgggcccag 60
tctgccctga ctcagtctgc ctccgtgtct gggtctcctg gacagccgat caccatctcc 120
tgcactggaa ccagcagtga tgttgggagt tataaccttg tctcctggta ccaacagcac 180
ccaggcaaag cccccaaact catgatttat gaggtcagta agcggccctc aggggtttct 240
aatcgcttct ctggctccaa gtctggcaac acggcctccc tgacaatctc tgggctccag 300
gctgaggacg aggctgatta ttactgctgc tcatatgcag atagtaggac tttggtgttc 360
ggcggaggga ccaagctgac cgtcctaggt cagcccaagg ctgccccctc ggtcactctg 420
ttcccgccct cgagtgagga gcttcaagcc aacaaggcca cactggtgtg tctcataagt 480
gacttctacc cgggagccgt gacagtggcc tggaaggcag atagcagccc cgtcaaggcg 540
ggagtggaga ccaccacacc ctccaaacaa agcaacaaca agtacgcggc cagcagctac 600
ctgagcctga cgcctgagca gtggaagtcc cacagaagct acagctgcca ggtcacgcat 660
gaagggagca ccgtggagaa gacagtcgcc ccaactgagt gttct 705
Sequence Number (ID): 304
Length: 705
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..705
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc atcctccctg tctgcatctg taggagacag agtcaccatc 120
acttgccagg cgagtcagga cattagcaac tatttaaatt ggtatcagca gaaaccaggg 180
aaagccccta agctcctgat ctacgatgca tccaatttgg aaacaggggt cccatcaagg 240
ttcagtggaa gtggatctgg gacagatttt actttcacca tcagcagcct gcagcctgaa 300
gatattgcaa catattactg tcaacagtat gataatctcc ctccgagggt gacgttcggc 360
caagggacca aggtggaaat caaacgtacg gtggctgcac catctgtctt catcttcccg 420
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 480
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 540
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 600
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 660
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 705
Sequence Number (ID): 305
Length: 702
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..702
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggttc ctgggcccag 60
tctgccctga ctcagcctgc ctccgtgtct gggtctcctg gacagtcgat caccatctcc 120
tgcactggaa ccagcagtga tgttgggagt tatgaccttg tctcctggta ccaacagcac 180
ccaggcaaag cccccaaact catgatttat gaggtcacta agcggccctc aggggtttct 240
aatcgcttct ctggctccaa gtctggcaac acggcctccc tgacaatctc tgggctccag 300
gctgaggacg aggctgatta ttactgctgc tcatttgcag gtagtagccc cttgttcggc 360
ggagggacca agctgaccgt cctaggtcag cccaaggctg ccccctcggt cactctgttc 420
ccaccctcga gtgaggagct tcaagccaac aaggccacac tggtgtgtct cataagtgac 480
ttctacccgg gagccgtgac agtggcctgg aaggcagata gcagccccgt caaggcggga 540
gtggagacca ccacaccctc caaacaaagc aacaacaagt acgcggccag cagctacctg 600
agcctgacgc ctgagcagtg gaagtcccac agaagctaca gctgccaggt cacgcatgaa 660
gggagcaccg tggagaagac agtcgcccca actgagtgtt ct 702
Sequence Number (ID): 306
Length: 699
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..699
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc ttccaccctg tctgcatctg taggagacag agtcaccatc 120
acttgccggg ccagtcagag tattagtagc tggttggcct ggtatcagca gaaaccaggg 180
aaagccccta aggtcctgat ctataaggcg tctagtttag aaagtggggt cccatcaagg 240
ttcagcggca gtggatctgg gacagaattc actctcacca tcagcagcct gcagcctgat 300
gattttgcaa cttattactg ccaacagtat aatagttatt ctccaatgtt cggccaaggg 360
accaaggtgg aaatcaaacg tacggtggct gcaccatctg tcttcatctt cccgccatct 420
gatgagcagt tgaaatctgg aactgcctct gttgtgtgcc tgctgaataa cttctatccc 480
agagaggcca aagtacagtg gaaggtggat aacgccctcc aatcgggtaa ctcccaggag 540
agtgtcacag agcaggacag caaggacagc acctacagcc tcagcagcac cctgacgctg 600
agcaaagcag actacgagaa acacaaagtc tacgcctgcg aagtcaccca tcagggcctg 660
agctcgcccg tcacaaagag cttcaacagg ggagagtgt 699
Sequence Number (ID): 307
Length: 702
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..702
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcagaa 60
attgtgttga cgcagtctcc aggcaccctg tctttgtctc caggggaaag agccaccctc 120
tcctgcaggg ccagtcagag tgttagcagc agctacttag cctggtacca gcagaaacct 180
ggccaggctc ccaggctcct catctatggt gcatccagca gggccactgg catcccagac 240
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag actggagcct 300
gaagattttg cagtgtatta ctgtcaccag tatggtagct cacctcgaac gttcggccaa 360
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 420
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 480
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 540
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 600
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 660
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 702
Sequence Number (ID): 308
Length: 705
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..705
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggttc ctgggcccag 60
tctgtgctga ctcagccacc ctcagcgtct gggacccccg ggcagagggt caccatctct 120
tgttctggaa gcagctccaa catcggaagt aatactgtaa actggtacca gcagctccca 180
ggaacggccc ccaaactcct catctataat aataatcaac ggccctcagg ggtccctgac 240
cgattctctg gctccaagtc tggcacctca gcctccctgg ccatcagtgg gctccagtct 300
gaggatgagg ctgattatta ctgtgcagca tgggatgaca gcctgaatgg ttatgtcttc 360
ggaactggga ccaaggtcac cgtcctaggt cagcccaagg ccaaccccac tgtcactctg 420
ttcccaccct cgagtgagga gcttcaagcc aacaaggcca cactggtgtg tctcataagt 480
gacttctacc cgggagccgt gacagtggcc tggaaggcag atagcagccc cgtcaaggcg 540
ggagtggaga ccaccacacc ctccaaacaa agcaacaaca agtacgcggc cagcagctac 600
ctgagcctga cgcctgagca gtggaagtcc cacagaagct acagctgcca ggtcacgcat 660
gaagggagca ccgtggagaa gacagtcgcc ccaactgagt gttct 705
Sequence Number (ID): 309
Length: 699
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..699
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc atcctccctg tctacatctg taggagacag agtcaccatc 120
acttgccggg caagtcagag cattagcaag tatttaaatt ggtatcagca gaaaccaggg 180
aaagccccta agctcctgat ctatgctgca tccagtttgc aaagtggggt cccatcaagg 240
ttcagtggca gtggatctgg gacagatttc actctcacca tcagcagtct gcaacctgaa 300
gattttgcaa cttactactg tcaacagagt tatagtaccc ctccgacgtt cggccaaggg 360
accaaggtgg aaatcaaacg tacggtggct gcaccatctg tcttcatctt cccgccatct 420
gatgagcagt tgaaatctgg aactgcctct gttgtgtgcc tgctgaataa cttctatccc 480
agagaggcca aagtacagtg gaaggtggat aacgccctcc aatcgggtaa ctcccaggag 540
agtgtcacag agcaggacag caaggacagc acctacagcc tcagcagcac cctgacgctg 600
agcaaagcag actacgagaa acacaaagtc tacgcctgcg aagtcaccca tcagggcctg 660
agctcgcccg tcacaaagag cttcaacagg ggagagtgt 699
Sequence Number (ID): 310
Length: 705
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..705
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc atcctccctg tctgcatctg taggagacag agtcaccatc 120
acttgccagg cgagtcagga cattagcaac tatttaaatt ggtatcagca gaaaccaggg 180
aaagccccta agctcctgat ctacgatgca tccaatttgg aaacaggggt cccatcaagg 240
ttcagtggaa gtggatctgg gacagatttt actttcacca tcagcagcct gcagcctgaa 300
gatattgcta catattactg tcaacagtat gataatctcc ctccgcggct cactttcggc 360
ggagggacca aggtggaaat caaacgtacg gtggctgcac catctgtctt catcttcccg 420
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 480
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 540
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 600
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 660
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 705
Sequence Number (ID): 311
Length: 708
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..708
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggttc ctgggcccag 60
tctgccctga ctcagcctgc ctccgtgtct gggtctcctg gacagtcgat caccatctcc 120
tgcactggaa ccagcagtga cattggtggt tataactatg tctcctggta ccaacaacac 180
ccaggcaaag cccccaaact catgatttat gatgtcagta atcggccctc aggggtttct 240
aatcgcttct ctggctccaa gtctggcaac acggcctccc tgaccatctc tgggctccag 300
gctgaggacg aggctgaata ttactgcagc tcatatacaa gaagcagcac tcaggtggta 360
ttcggcggag ggaccaagct gaccgtccta ggtcagccca aggctgcccc ctcggtcact 420
ctgttcccac cctcgagtga ggagcttcaa gccaacaagg ccacactggt gtgtctcata 480
agtgacttct acccgggagc cgtgacagtg gcctggaagg cagatagcag ccccgtcaag 540
gcgggagtgg agaccaccac accctccaaa caaagcaaca acaagtacgc ggccagcagc 600
tacctgagcc tgacgcctga gcagtggaag tcccacagaa gctacagctg ccaggtcacg 660
catgaaggga gcaccgtgga gaagacagtc gccccaactg agtgttct 708
Sequence Number (ID): 312
Length: 702
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..702
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcagaa 60
attgtgttga cgcagtctcc aggcaccctg tctttgtctc caggggaaag agccaccctc 120
tcctgcaggg ccagtcagag tgttagcagc agctacttag cctggtacca gcagaaacct 180
ggccaggctc ccaggctcct catctatggt gcatccagca gggccactgg catcccagac 240
aggttcagtg gcagtgggtc tgggacagac ttcactctca ctatcagcag actggagcct 300
gaagattttg cagtgtatca ctgtcagcag tatggtacct caccgtggac gttcggccaa 360
gggaccaagg tggaaatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 420
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 480
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 540
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 600
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 660
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 702
Sequence Number (ID): 313
Length: 711
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..711
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggttc ctgggcccag 60
tctgccctga ctcagcctgc ctccgtgtct gggtctcctg gacagtcgat caccatctcc 120
tgcactggaa ccagcagtga tgttgggagt tataaccttg tctcctggta ccaacagcac 180
ccaggcaaag cccccaaact catgatttat gaagtcagta agcggccctc aggggtttct 240
aatcgcttct ctggctccaa gtctggcaac acggcctccc tgacaatctc tgggctccag 300
gctgaggacg aggctgatta ttactgctgc tcatatgcac gtattagcac ccccggattt 360
gtcttcggaa ctgggaccaa ggtcaccgtc ctaggtcagc ccaaggccaa ccccactgtc 420
actctgttcc cgccctcgag tgaggagctt caagccaaca aggccacact ggtgtgtctc 480
ataagtgact tctacccggg agccgtgaca gtggcctgga aggcagatag cagccccgtc 540
aaggcgggag tggagaccac cacaccctcc aaacaaagca acaacaagta cgcggccagc 600
agctacctga gcctgacgcc tgagcagtgg aagtcccaca gaagctacag ctgccaggtc 660
acgcatgaag ggagcaccgt ggagaagaca gtcgccccaa ctgagtgttc t 711
Sequence Number (ID): 314
Length: 696
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..696
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc ttccaccctg tctgcatctg taggagacag agtcaccatc 120
acttgccggg ccagtcagag tattagtagc tggttggcct ggtatcagca gaaaccaggg 180
aaagccccta agctcctgat ctataaggca tctaatttag aaagtggggt cccatcaagg 240
ttcagcggca gtggatctgg gacagaattc actctcacca tcagcagcct gcagcctgat 300
gattttgcaa cttattactg ccaacagtat aatagttatt cgacgttcgg ccaagggacc 360
aaggtggaga tcaaacgtac ggtggctgca ccatctgtct tcatcttccc gccatctgat 420
gagcagttga aatctggaac tgcctctgtt gtgtgcctgc tgaataactt ctatcccaga 480
gaggccaaag tacagtggaa ggtggataac gccctccaat cgggtaactc ccaggagagt 540
gtcacagagc aggacagcaa ggacagcacc tacagcctca gcagcaccct gacgctgagc 600
aaagcagact acgagaaaca caaagtctac gcctgcgaag tcacccatca gggcctgagc 660
tcgcccgtca caaagagctt caacagggga gagtgt 696
Sequence Number (ID): 315
Length: 702
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..702
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc atcctccctg tctgcatctc taggagacag agtcaccatc 120
acttgccagg cgagtcagga cattagcaac tatttaaatt ggtatcagca gaaaccaggg 180
aaggccccta agctcctgat ctacgatgca tccaatttgc aaacaggggt cccatcaagg 240
ttcagtggag gtggatctgg gacagatttt actttcacca tcagcagcct gcagcctgaa 300
gatattgcaa catattactg tcaacagtat gataatctcc tgggagtcac tttcggccct 360
gggaccaaag tggatatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 420
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 480
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 540
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 600
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 660
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 702
Sequence Number (ID): 316
Length: 702
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..702
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcagaa 60
attgtgttga cgcagtctcc aggcaccctg tctttgtctc caggggaaag agccaccctc 120
tcctgcaggg ccagtcagag tgttagcagc agctacttag cctggtacca gcagagtcct 180
ggccaggctc ccaggctcct catctatggt acatccagca gggccactgg catcccagac 240
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag actggagcct 300
gaagattttg cagtgtatta ctgtcagcag tatggtagct cacctagaac gttcggccaa 360
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 420
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 480
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 540
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 600
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 660
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 702
Sequence Number (ID): 317
Length: 702
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..702
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcagaa 60
attgtgttga cgcagtctcc aggcaccctg tctttgtctc caggggaaag agccaccctc 120
tcctgcaggg ccagtcagag tgttagcagc agctacttag cctggtacca gcagaaacct 180
ggccaggctc ccaggctcct catctatggt gcatccagca gggccactgg catcccagac 240
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcag actggagcct 300
gaagattttg cagtgtatta ctgtcagcag tatggtagct cacctcgaac gttcggccaa 360
gggaccaagg tggagatcaa acgtacggtg gctgcaccat ctgtcttcat cttcccgcca 420
tctgatgagc agttgaaatc tggaactgcc tctgttgtgt gcctgctgaa taacttctat 480
cccagagagg ccaaagtaca gtggaaggtg gataacgccc tccaatcggg taactcccag 540
gagagtgtca cagagcagga cagcaaggac agcacctaca gcctcagcag caccctgacg 600
ctgagcaaag cagactacga gaaacacaaa gtctacgcct gcgaagtcac ccatcagggc 660
ctgagctcgc ccgtcacaaa gagcttcaac aggggagagt gt 702
Sequence Number (ID): 318
Length: 705
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..705
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcagaa 60
attgtgttga cgcagtctcc aggcaccctg tctttgtctc caggggaaag agccaccctc 120
tcctgcaggg ccagccagag tattagcagc agctacttag cctggtacca gcagaaacct 180
ggccaggctc ccaggctcct cttctatggt gcatccagca gggccactgg catcccagac 240
aggttcagtg gcagtgggtc tgggacagac ttcactctca ccatcagcaa actggagcct 300
gaagattttg cagtgtatta ctgtcagcag tatggtagct cacctccgat caccttcggc 360
caagggacac gactggagat taaacgtacg gtggctgcac catctgtctt catcttcccg 420
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 480
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 540
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 600
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 660
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 705
Sequence Number (ID): 319
Length: 720
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..720
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcggac 60
atcgtgatga cccagtctcc agactccctg gctgtgtctc tgggcgagag ggccaccatc 120
aactgcaagt ccagccagag tgttttatac agctccaaca ataagaacta cttagcttgg 180
taccagcaga aaccaggaca gcctcctaag ctgctcattt actgggcatc tacccgggaa 240
tccggggtcc ctgaccgatt cagtggcagc gggtctggga cagatttcac tctcaccatc 300
agcagcctgc aggctgaaga tgtggcagtt tattactgtc agcaatatta tagtactctc 360
ttgctcactt tcggcggagg gaccaaggtg gagatcaaac gtacggtggc tgcaccatct 420
gtcttcatct tcccgccatc tgatgagcag ttgaaatctg gaactgcctc tgttgtgtgc 480
ctgctgaata acttctatcc cagagaggcc aaagtacagt ggaaggtgga taacgccctc 540
caatcgggta actcccagga gagtgtcaca gagcaggaca gcaaggacag cacctacagc 600
ctcagcagca ccctgacgct gagcaaagca gactacgaga aacacaaagt ctacgcctgc 660
gaagtcaccc atcagggcct gagctcgccc gtcacaaaga gcttcaacag gggagagtgt 720
Sequence Number (ID): 320
Length: 699
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..699
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc atcctccctg tctgcatctg taggagacag agtcaccatc 120
acttgccagg cgagtcagga cattagcaac tatttaaatt ggtatcagca gaaaccaggg 180
aaagccccta agctcctgat ctacgatgca tccaatttgg aaacaggggt cccatcaagg 240
ttcagtggaa gtggatctgg gacagatttt actttcacca tcagcagcct gcagcctgaa 300
gatattgcaa catattactg tcaacagtat gataatctcc cgatcacctt cggccaaggg 360
acacgactgg agattaaacg tacggtggct gcaccatctg tcttcatctt cccgccatct 420
gatgagcagt tgaaatctgg aactgcctct gttgtgtgcc tgctgaataa cttctatccc 480
agagaggcca aagtacagtg gaaggtggat aacgccctcc aatcgggtaa ctcccaggag 540
agtgtcacag agcaggacag caaggacagc acctacagcc tcagcagcac cctgacgctg 600
agcaaagcag actacgagaa acacaaagtc tacgcctgcg aagtcaccca tcagggcctg 660
agctcgcccg tcacaaagag cttcaacagg ggagagtgt 699
Sequence Number (ID): 321
Length: 705
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..705
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattcagac 60
atccagttga cccagtctcc atccttcctg tctgcatctg taggagacag agtcaccatc 120
acttgccggg ccagtcaggg cattagcagt tatttagcct ggtatcagca aaaaccaggg 180
aaagccccta agctcctgat ctttgctgca tccactttgc agagtggggt cccatcaagg 240
ttcagcgcca gtggatctgg gacagaattc actctcacaa tcagcagcct gcagcctgaa 300
gattttgcaa cttattactg tcaacacctt aatagttacc cttctatgta cacttttggc 360
caggggacca agctggagat caaacgtacg gtggctgcac catctgtctt catcttcccg 420
ccatctgatg agcagttgaa atctggaact gcctctgttg tgtgcctgct gaataacttc 480
tatcccagag aggccaaagt acagtggaag gtggataacg ccctccaatc gggtaactcc 540
caggagagtg tcacagagca ggacagcaag gacagcacct acagcctcag cagcaccctg 600
acgctgagca aagcagacta cgagaaacac aaagtctacg cctgcgaagt cacccatcag 660
ggcctgagct cgcccgtcac aaagagcttc aacaggggag agtgt 705
Sequence Number (ID): 322
Length: 699
Molecule Type: DNA
Features Location/Qualifiers:
- source, 1..699
> mol_type, other DNA
> note, light chain
> organism, synthetic construct
Residues:
atgggatggt catgtatcat cctttttcta gtagcaactg caaccggtgt acattctgac 60
atccagatga cccagtctcc atcctccctg tctgcatctg taggagacag agtcaccatc 120
acttgccggg caagtcaggg cattagaaat gatttaggct ggtatcagca gaaaccaggg 180
aaagccccta agctcctgat ctatgctgca tccagtttac aaagtggggt cccatcaagg 240
ttcagcggca gtggatctgg cacagatttc actctcacca tcagcagcct gcagcctgaa 300
gattttgcaa cttattactg tctacaagat tacaattacc ctcggacgtt cggccaaggg 360
accaaggtgg aaatcaaacg tacggtggct gcaccatctg tcttcatctt cccgccatct 420
gatgagcagt tgaaatctgg aactgcctct gttgtgtgcc tgctgaataa cttctatccc 480
agagaggcca aagtacagtg gaaggtggat aacgccctcc aatcgggtaa ctcccaggag 540
agtgtcacag agcaggacag caaggacagc acctacagcc tcagcagcac cctgacgctg 600
agcaaagcag actacgagaa acacaaagtc tacgcctgcg aagtcaccca tcagggcctg 660
agctcgcccg tcacaaagag cttcaacagg ggagagtgt 699
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Рекомбинантный белок, связывающийся с RBD S-белка SARS-CoV-2 | 2022 |
|
RU2778942C1 |
| ПОЛИПЕПТИД С АНТИ-SARS-CoV-2 АКТИВНОСТЬЮ И ПРОЛОНГИРОВАННЫМ ВРЕМЕНЕМ ЦИРКУЛЯЦИИ В КРОВОТОКЕ | 2023 |
|
RU2822355C1 |
| ИММУНОЛИПОСОМА, СВЯЗЫВАЮЩАЯСЯ С RBD S-БЕЛКА SARS-COV-2 | 2023 |
|
RU2827165C1 |
| Моноклональное антитело iC2 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2023 |
|
RU2817697C1 |
| Способ получения нокаута гена CD209 в эмбрионах Bos taurus путем трансдукции зигот адено-ассоциированными вирусами, кодирующими saCas9 и соответствующую гидовую РНК | 2022 |
|
RU2800917C1 |
| Моноклональное антитело iC1 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2023 |
|
RU2817696C1 |
| ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2018 |
|
RU2841528C2 |
| РАСТВОРИМЫЙ АПФ2, ЕГО СЛИТЫЙ БЕЛОК И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2021 |
|
RU2837537C1 |
| Однодоменное антитело ламы Н5 и его производное H5-Fc, специфически связывающие RBD-домен S-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2022 |
|
RU2793967C1 |
| ХИМЕРНЫЕ ЦИТОКИНОВЫЕ РЕЦЕПТОРЫ, НЕСУЩИЕ ЭКТОДОМЕН PD-1 | 2020 |
|
RU2828214C2 |
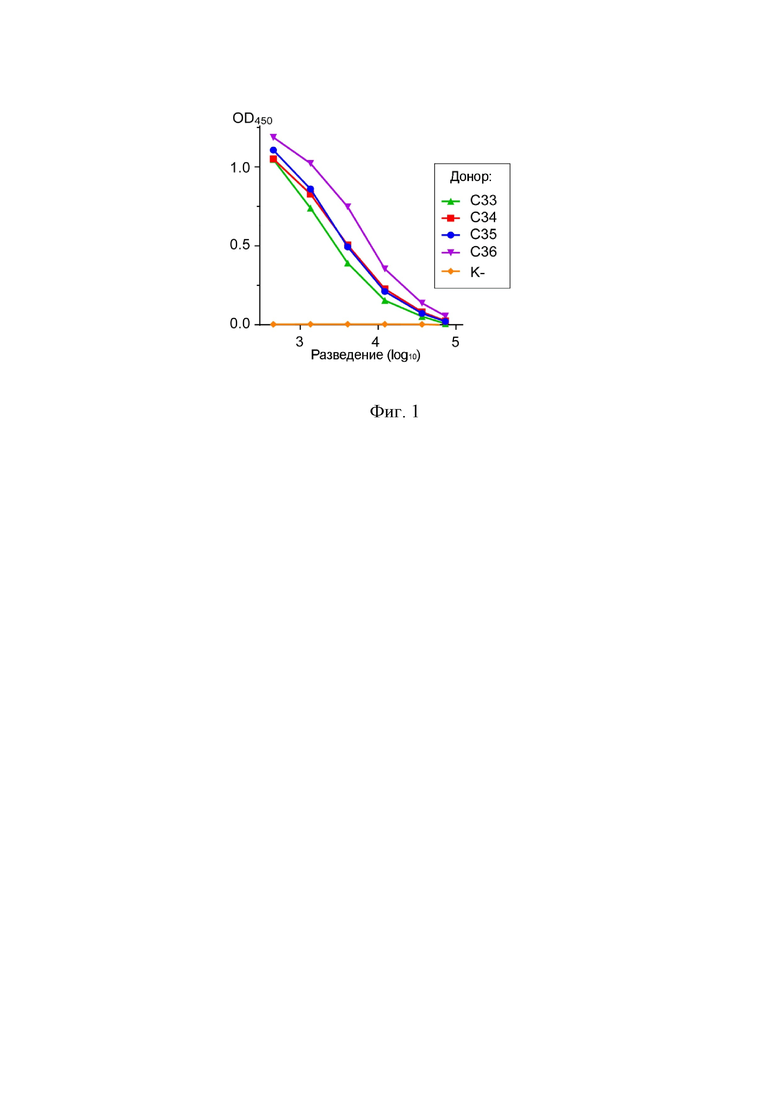
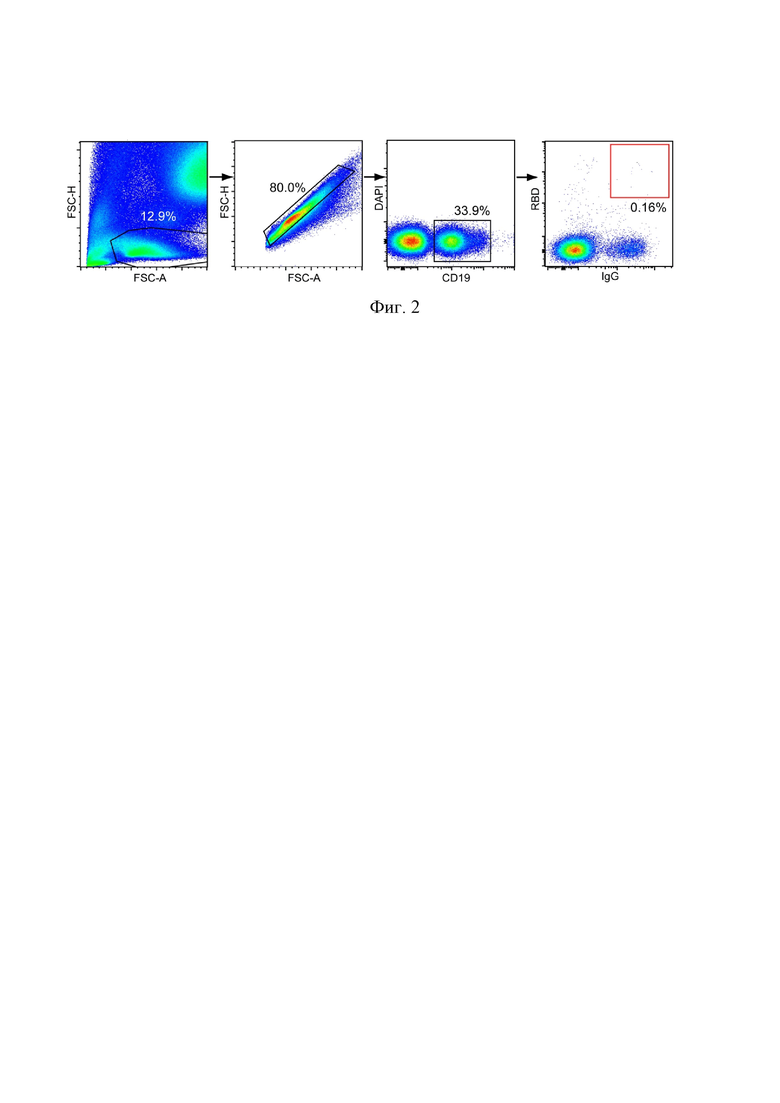
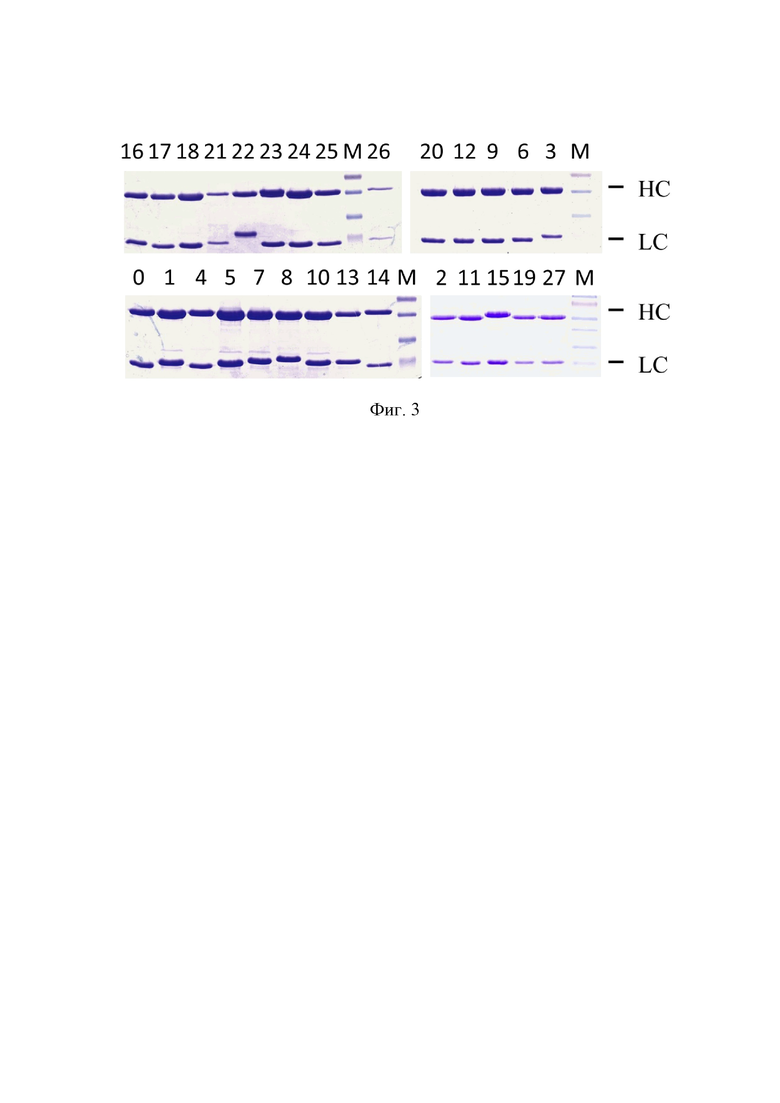

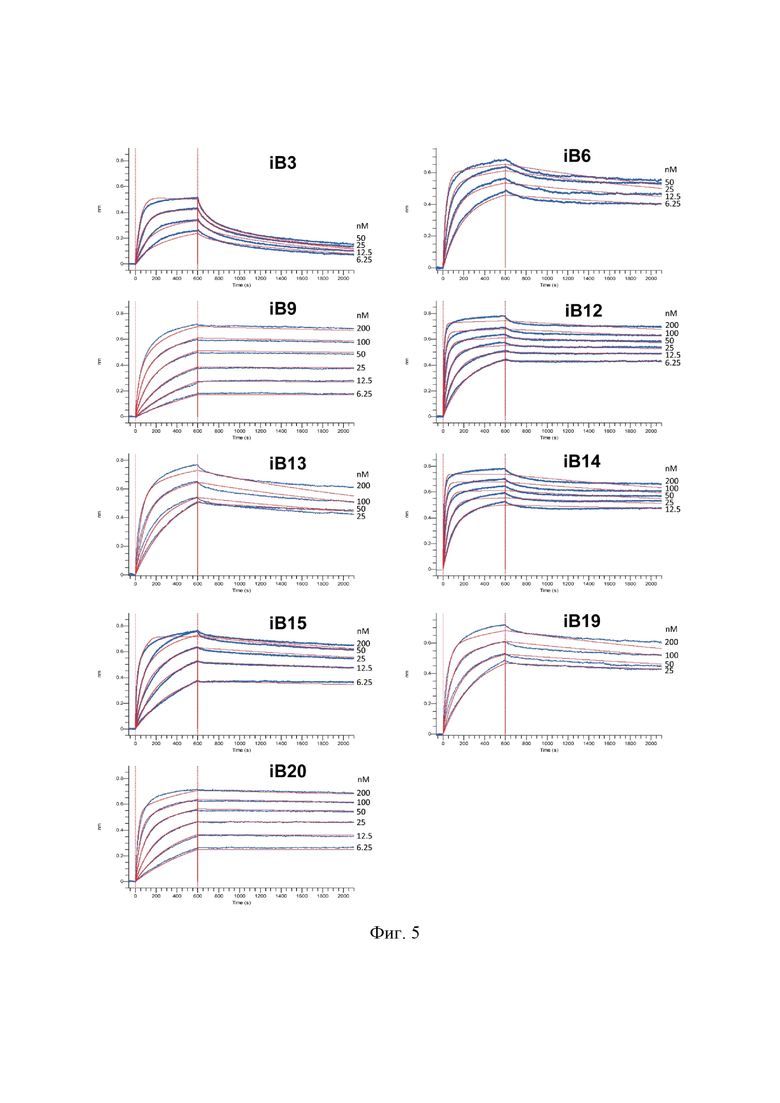

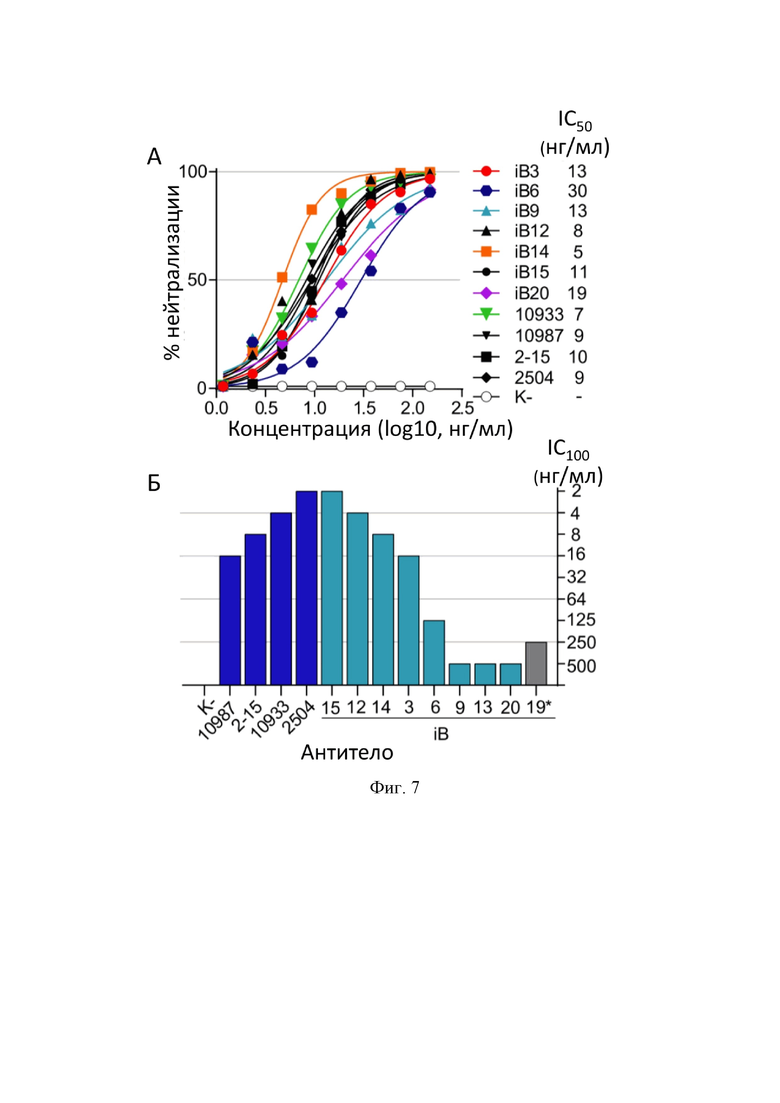


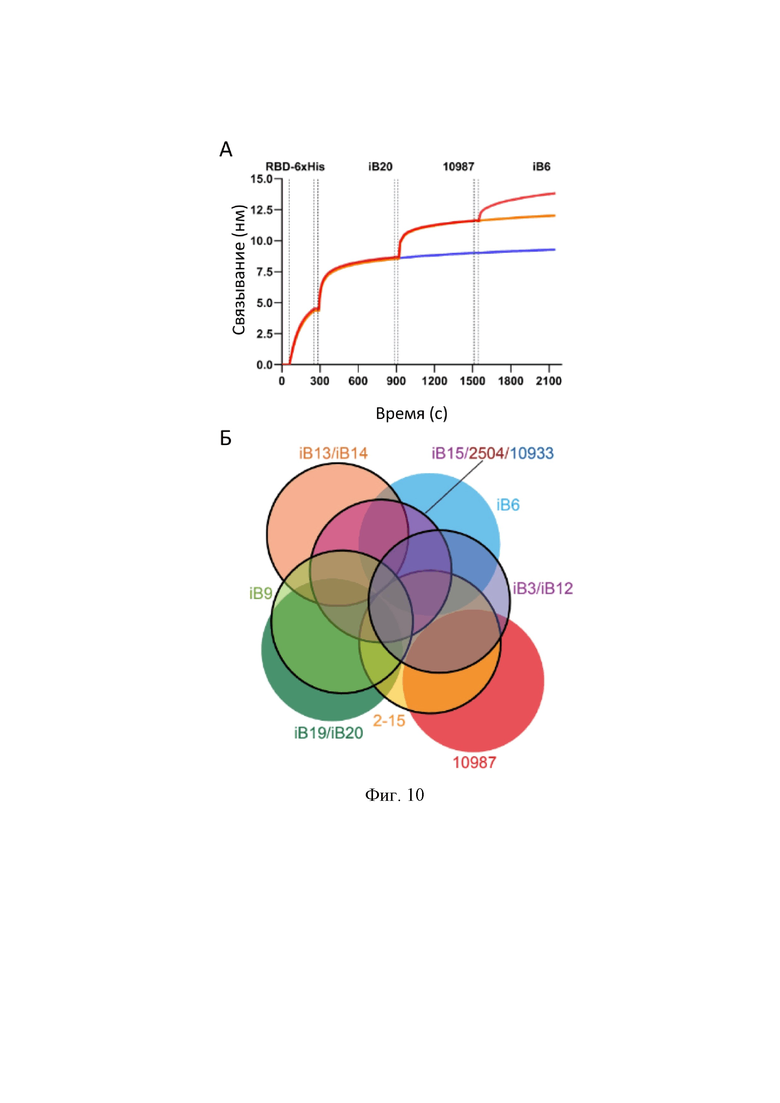
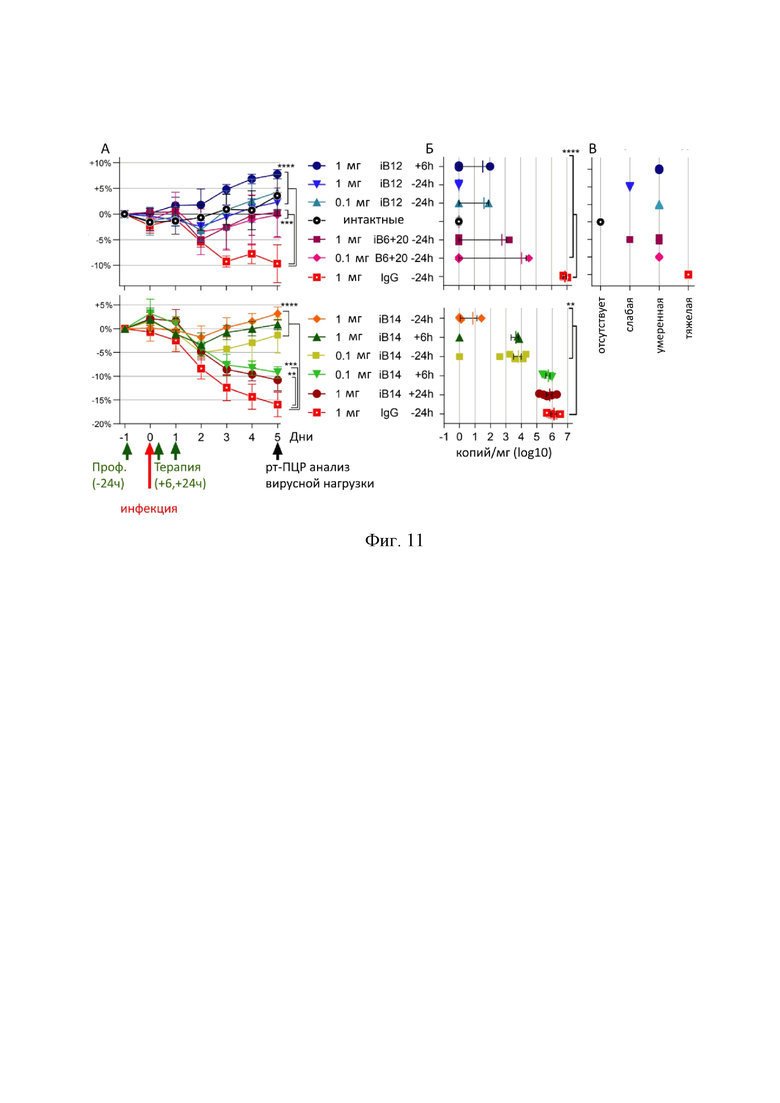
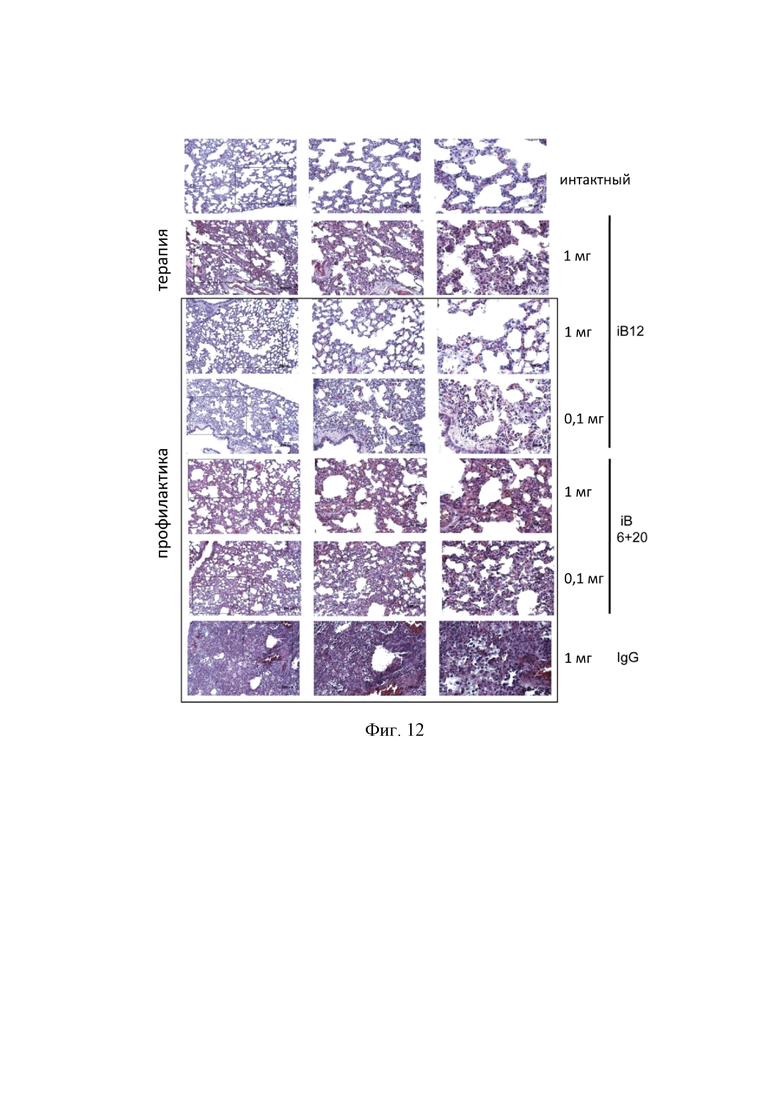
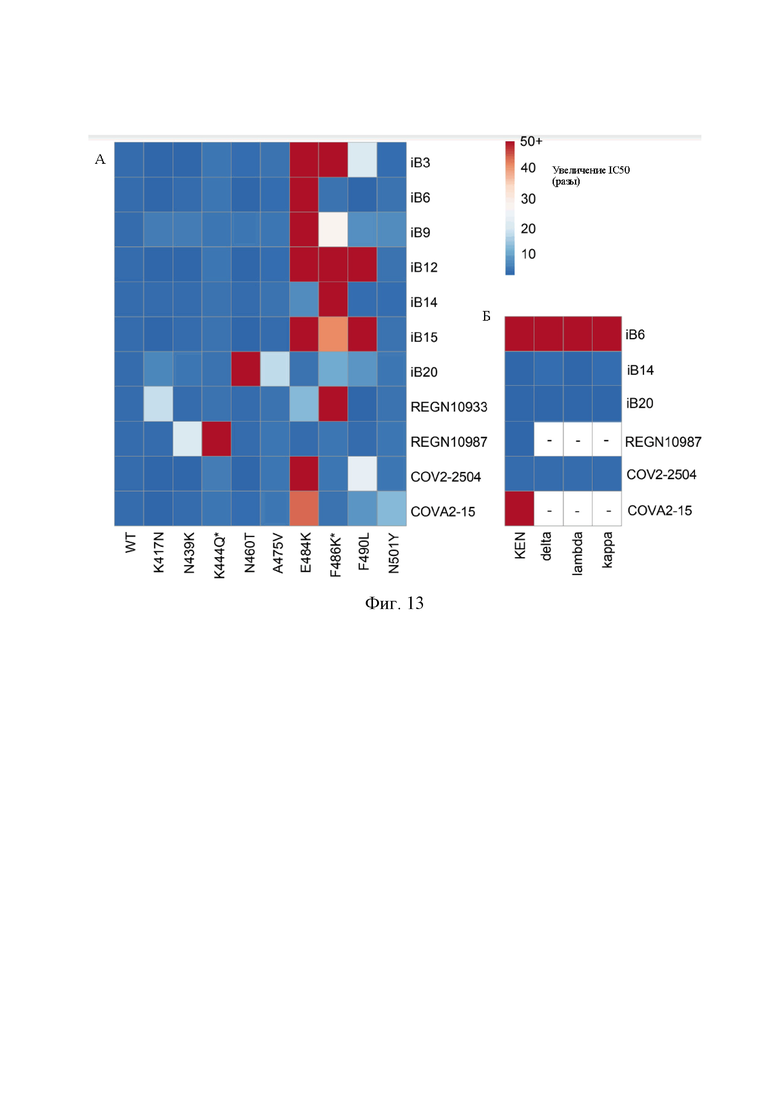
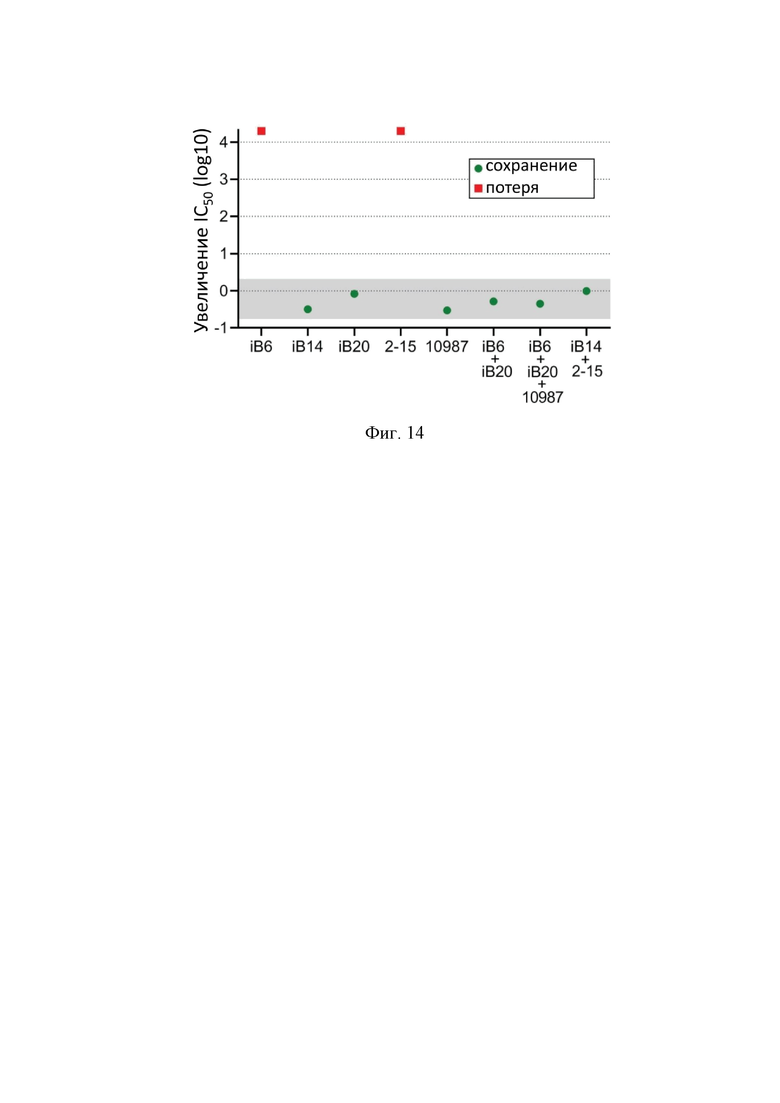
Изобретение относится к области биотехнологии, в частности к моноклональному антителу или его антигенсвязывающему фрагменту, селективно связывающему RBD фрагмент Spike-белка вируса SARS-CoV-2, способу его получения, а также к содержащей его комбинации. Также раскрыты нуклеиновая кислота, кодирующая вышеуказанное антитело, а также вектор, ее содержащий. Изобретение также относится к антигенному рецептору, который селективно связывает RBD фрагмент Spike-белка вируса SARS-CoV-2, содержащему антигенсвязывающую область, представленную вышеуказанным антителом или его антигенсвязывающим фрагментом. Изобретение эффективно для профилактики и терапии инфекции, вызванной SARS-CoV-2, а также для оценки наличия инфекции SARS-CoV-2 у млекопитающих. 9 н. и 15 з.п. ф-лы, 14 ил., 3 табл., 9 пр.
1. Моноклональное антитело или его антигенсвязывающий фрагмент, селективно связывающее RBD фрагмент Spike-белка вируса SARS-CoV-2, где антитело или антигенсвязывающий фрагмент выбраны из:
(a) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 1;
(ii) CDRH2 последовательности SEQ ID NO: 24;
(iii) CDRH3 последовательности SEQ ID NO: 47, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 70;
(ii) CDRL2 последовательности AAS (SEQ ID NO:93);
(iii) CDRL3 последовательности SEQ ID NO: 116;
(b) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 2;
(ii) CDRH2 последовательности SEQ ID NO: 25;
(iii) CDRH3 последовательности SEQ ID NO: 48, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 71;
(ii) CDRL2 последовательности SNN (SEQ ID NO:94);
(iii) CDRL3 последовательности SEQ ID NO: 117;
(с) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 3;
(ii) CDRH2 последовательности SEQ ID NO: 26;
(iii) CDRH3 последовательности SEQ ID NO: 49, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 72;
(ii) CDRL2 последовательности WAT (SEQ ID NO:95);
(iii) CDRL3 последовательности SEQ ID NO: 118;
(d) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 4;
(ii) CDRH2 последовательности SEQ ID NO: 27;
(iii) CDRH3 последовательности SEQ ID NO: 50, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 73;
(ii) CDRL2 последовательности EVS (SEQ ID NO:96);
(iii) CDRL3 последовательности SEQ ID NO: 119;
(e) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 5;
(ii) CDRH2 последовательности SEQ ID NO: 28;
(iii) CDRH3 последовательности SEQ ID NO: 51, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 74;
(ii) CDRL2 последовательности DAS (SEQ ID NO:97);
(iii) CDRL3 последовательности SEQ ID NO: 120;
(f) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 6;
(ii) CDRH2 последовательности SEQ ID NO: 29;
(iii) CDRH3 последовательности SEQ ID NO: 52, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 75;
(ii) CDRL2 последовательности EVT (SEQ ID NO:98);
(iii) CDRL3 последовательности SEQ ID NO: 121;
(g) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 7;
(ii) CDRH2 последовательности SEQ ID NO: 30;
(iii) CDRH3 последовательности SEQ ID NO: 53, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 76;
(ii) CDRL2 последовательности KAS (SEQ ID NO:99);
(iii) CDRL3 последовательности SEQ ID NO: 122;
(h) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 8;
(ii) CDRH2 последовательности SEQ ID NO: 31;
(iii) CDRH3 последовательности SEQ ID NO: 54, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 77;
(ii) CDRL2 последовательности GAS (SEQ ID NO:100);
(iii) CDRL3 последовательности SEQ ID NO: 123;
(i) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 9;
(ii) CDRH2 последовательности SEQ ID NO: 32;
(iii) CDRH3 последовательности SEQ ID NO: 55, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 78;
(ii) CDRL2 последовательности NNN (SEQ ID NO:101);
(iii) CDRL3 последовательности SEQ ID NO: 124;
(j) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 10;
(ii) CDRH2 последовательности SEQ ID NO: 33;
(iii) CDRH3 последовательности SEQ ID NO: 56, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 79;
(ii) CDRL2 последовательности AAS (SEQ ID NO:102);
(iii) CDRL3 последовательности SEQ ID NO: 125;
(k) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 11;
(ii) CDRH2 последовательности SEQ ID NO: 34;
(iii) CDRH3 последовательности SEQ ID NO: 57, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 80;
(ii) CDRL2 последовательности DAS (SEQ ID NO:103);
(iii) CDRL3 последовательности SEQ ID NO: 126;
(l) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 12;
(ii) CDRH2 последовательности SEQ ID NO: 35;
(iii) CDRH3 последовательности SEQ ID NO: 58, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 81;
(ii) CDRL2 последовательности DVS (SEQ ID NO:104);
(iii) CDRL3 последовательности SEQ ID NO: 127;
(m) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 13;
(ii) CDRH2 последовательности SEQ ID NO: 36;
(iii) CDRH3 последовательности SEQ ID NO: 59, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 82;
(ii) CDRL2 последовательности GAS (SEQ ID NO:105);
(iii) CDRL3 последовательности SEQ ID NO: 128;
(n) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 14;
(ii) CDRH2 последовательности SEQ ID NO: 37;
(iii) CDRH3 последовательности SEQ ID NO: 60, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 83;
(ii) CDRL2 последовательности EVS (SEQ ID NO:106);
(iii) CDRL3 последовательности SEQ ID NO: 129;
(o) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 15;
(ii) CDRH2 последовательности SEQ ID NO: 38;
(iii) CDRH3 последовательности SEQ ID NO: 61, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 84;
(ii) CDRL2 последовательности KAS (SEQ ID NO:107);
(iii) CDRL3 последовательности SEQ ID NO: 130;
(p) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 16;
(ii) CDRH2 последовательности SEQ ID NO: 39;
(iii) CDRH3 последовательности SEQ ID NO: 62, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 85;
(ii) CDRL2 последовательности DAS (SEQ ID NO:108);
(iii) CDRL3 последовательности SEQ ID NO: 131;
(q) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 17;
(ii) CDRH2 последовательности SEQ ID NO: 40;
(iii) CDRH3 последовательности SEQ ID NO: 63, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 86;
(ii) CDRL2 последовательности GTS (SEQ ID NO:109);
(iii) CDRL3 последовательности SEQ ID NO: 132;
(r) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 18;
(ii) CDRH2 последовательности SEQ ID NO: 41;
(iii) CDRH3 последовательности SEQ ID NO: 64, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 87;
(ii) CDRL2 последовательности GAS (SEQ ID NO:110);
(iii) CDRL3 последовательности SEQ ID NO: 133;
(s) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 19;
(ii) CDRH2 последовательности SEQ ID NO: 42;
(iii) CDRH3 последовательности SEQ ID NO: 65, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 88;
(ii) CDRL2 последовательности GAS (SEQ ID NO:111);
(iii) CDRL3 последовательности SEQ ID NO: 134;
(t) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 20;
(ii) CDRH2 последовательности SEQ ID NO: 43;
(iii) CDRH3 последовательности SEQ ID NO: 66, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 89;
(ii) CDRL2 последовательности WAS (SEQ ID NO:112);
(iii) CDRL3 последовательности SEQ ID NO: 135;
(u) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 21;
(ii) CDRH2 последовательности SEQ ID NO: 44;
(iii) CDRH3 последовательности SEQ ID NO: 67, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 90;
(ii) CDRL2 последовательности DAS (SEQ ID NO:113);
(iii) CDRL3 последовательности SEQ ID NO: 136;
(v) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 22;
(ii) CDRH2 последовательности SEQ ID NO: 45;
(iii) CDRH3 последовательности SEQ ID NO: 68, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 91;
(ii) CDRL2 последовательности AAS (SEQ ID NO:114);
(iii) CDRL3 последовательности SEQ ID NO: 137;
(w) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 23;
(ii) CDRH2 последовательности SEQ ID NO: 46;
(iii) CDRH3 последовательности SEQ ID NO: 69, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 92;
(ii) CDRL2 последовательности AAS (SEQ ID NO:115);
(iii) CDRL3 последовательности SEQ ID NO: 138.
2. Антитело или его антигенсвязывающий фрагмент по п.1, содержащее вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот, не менее чем на 90% гомологичную одной из SEQ ID NO: 139-161, и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот, не менее чем на 90% гомологичную одной из SEQ ID NO: 162-184.
3. Антитело или его антигенсвязывающий фрагмент по любому из пп.1-2, которое является антителом человека.
4. Антитело или его антигенсвязывающий фрагмент по любому из пп.1-2, где антитело представляет собой химерное антитело, мышиное антитело и/или антитело, прошедшее созревание аффинности.
5. Антитело или его антигенсвязывающий фрагмент по любому из пп.1-4, где антитело или антигенсвязывающий фрагмент обладают вируснейтрализующей активностью.
6. Антитело или его антигенсвязывающий фрагмент по любому из пп.1-5, где антитело или его антигенсвязывающий фрагмент содержат консервативные участки тяжелых цепей, относящиеся к классу IgG, IgM, IgA, IgD или IgE.
7. Антитело или его антигенсвязывающий фрагмент по любому из пп.1-6, где антитело или его антигенсвязывающий фрагмент содержат консервативные участки тяжелых цепей, принадлежащие к изотипу IgG1, IgG2, IgG3 или IgG4.
8. Антитело или его антигенсвязывающий фрагмент по любому из пп.1-5, где антигенсвязывающий фрагмент представляет собой Fv, scFv, Fab, F(ab′)2 или Fab′.
9. Нуклеиновая кислота, кодирующая антитело по любому из пп. 1-9.
10. Нуклеиновая кислота по п.9, которая представляет собой ДНК.
11. Нуклеиновая кислота по любому из пп.9-10, содержащая нуклеотидную последовательность, выбранную из SEQ ID NO: 185-207, и нуклеотидную последовательность, выбранную из SEQ ID NO: 208-230.
12. Нуклеиновая кислота по любому из пп.9-10 содержащая нуклеотидную последовательность, выбранную из SEQ ID NO: 277-299, и нуклеотидную последовательность, выбранную из SEQ ID NO: 300-322.
13. Экспрессионный вектор, содержащий нуклеиновую кислоту по любому из пп.9-12.
14. Клетка-хозяин для экспрессии антитела или его антигенсвязывающего фрагмента по любому из пп.1-8, содержащая нуклеиновую кислоту по любому из пп.9-12, или экспрессионный вектор по п.13.
15. Способ получения антитела или его антигенсвязывающего фрагмента по любому из пп.1-8, включающий культивирование или поддержание клетки-хозяина по п. 14 в таких условиях, чтобы указанная клетка-хозяин продуцировала или экспрессировала антитело или его антигенсвязывающий фрагмент по любому из пп.1-8, посредством экспрессии нуклеиновой кислоты по любому из пп. 9-12, например, в составе вектора по п.13, и, при необходимости, дополнительно включающий выделение антитела или его антигенсвязывающего фрагмента по любому из пп.1-8, полученного таким способом.
16. Комбинация антител для профилактики и терапии инфекции, вызванной SARS-CoV-2, у субъекта, содержащая по меньшей мере два антитела, где антитела направлены на эпитопы на поверхности Spike-белка, где по меньшей мере одно из этих антител представляет собой антитело по любому из пп.1-8, где данное антитело обладает нейтрализующей активностью и выбрано из:
(d) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 4;
(ii) CDRH2 последовательности SEQ ID NO: 27;
(iii) CDRH3 последовательности SEQ ID NO: 50, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 73;
(ii) CDRL2 последовательности EVS (SEQ ID NO:96);
(iii) CDRL3 последовательности SEQ ID NO: 119;
(e) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 5;
(ii) CDRH2 последовательности SEQ ID NO: 28;
(iii) CDRH3 последовательности SEQ ID NO: 51, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 74;
(ii) CDRL2 последовательности DAS (SEQ ID NO:97);
(iii) CDRL3 последовательности SEQ ID NO: 120;
(g) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 7;
(ii) CDRH2 последовательности SEQ ID NO: 30;
(iii) CDRH3 последовательности SEQ ID NO: 53, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 76;
(ii) CDRL2 последовательности KAS (SEQ ID NO:99);
(iii) CDRL3 последовательности SEQ ID NO: 122;
(h) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 8;
(ii) CDRH2 последовательности SEQ ID NO: 31;
(iii) CDRH3 последовательности SEQ ID NO: 54, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 77;
(ii) CDRL2 последовательности GAS (SEQ ID NO:100);
(iii) CDRL3 последовательности SEQ ID NO: 123;
(j) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 10;
(ii) CDRH2 последовательности SEQ ID NO: 33;
(iii) CDRH3 последовательности SEQ ID NO: 56, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 79;
(ii) CDRL2 последовательности AAS (SEQ ID NO:102);
(iii) CDRL3 последовательности SEQ ID NO: 125;
(k) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
i) CDRH1 последовательности SEQ ID NO: 11;
(ii) CDRH2 последовательности SEQ ID NO: 34;
(iii) CDRH3 последовательности SEQ ID NO: 57, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 80;
(ii) CDRL2 последовательности DAS (SEQ ID NO:103);
(iii) CDRL3 последовательности SEQ ID NO: 126;
(l) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 12;
(ii) CDRH2 последовательности SEQ ID NO: 35;
(iii) CDRH3 последовательности SEQ ID NO: 58, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 81;
(ii) CDRL2 последовательности DVS (SEQ ID NO:104);
(iii) CDRL3 последовательности SEQ ID NO: 127;
(m) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
i) CDRH1 последовательности SEQ ID NO: 13;
(ii) CDRH2 последовательности SEQ ID NO: 36;
(iii) CDRH3 последовательности SEQ ID NO: 59, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 82;
(ii) CDRL2 последовательности GAS (SEQ ID NO:105);
(iii) CDRL3 последовательности SEQ ID NO: 128;
(n) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 14;
(ii) CDRH2 последовательности SEQ ID NO: 37;
(iii) CDRH3 последовательности SEQ ID NO: 60, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 83;
(ii) CDRL2 последовательности EVS (SEQ ID NO:106);
(iii) CDRL3 последовательности SEQ ID NO: 129;
(r) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 18;
(ii) CDRH2 последовательности SEQ ID NO: 41;
(iii) CDRH3 последовательности SEQ ID NO: 64, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 87;
(ii) CDRL2 последовательности GAS (SEQ ID NO:110);
(iii) CDRL3 последовательности SEQ ID NO: 133;
(s) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 19;
(ii) CDRH2 последовательности SEQ ID NO: 42;
(iii) CDRH3 последовательности SEQ ID NO: 65, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 88;
(ii) CDRL2 последовательности GAS (SEQ ID NO:111);
(iii) CDRL3 последовательности SEQ ID NO: 134; или
(u) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 21;
(ii) CDRH2 последовательности SEQ ID NO: 44;
(iii) CDRH3 последовательности SEQ ID NO: 67, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 90;
(ii) CDRL2 последовательности DAS (SEQ ID NO:113);
(iii) CDRL3 последовательности SEQ ID NO: 136.
17. Комбинация антител по п.16, где субъектом является человек или другое млекопитающее.
18. Комбинация антител по любому из пп.16-17, которая нейтрализует различные варианты вируса.
19. Применение антитела или его антигенсвязывающего фрагмента по любому из пп.1-8, для лечения инфекции, вызванной вирусом SARS-CoV-2 у человека и других млекопитающих, где антитело выбрано из:
(d) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 4;
(ii) CDRH2 последовательности SEQ ID NO: 27;
(iii) CDRH3 последовательности SEQ ID NO: 50, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 73;
(ii) CDRL2 последовательности EVS (SEQ ID NO:96);
(iii) CDRL3 последовательности SEQ ID NO: 119;
(e) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 5;
(ii) CDRH2 последовательности SEQ ID NO: 28;
(iii) CDRH3 последовательности SEQ ID NO: 51, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 74;
(ii) CDRL2 последовательности DAS (SEQ ID NO:97);
(iii) CDRL3 последовательности SEQ ID NO: 120;
(g) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 7;
(ii) CDRH2 последовательности SEQ ID NO: 30;
(iii) CDRH3 последовательности SEQ ID NO: 53, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 76;
(ii) CDRL2 последовательности KAS (SEQ ID NO:99);
(iii) CDRL3 последовательности SEQ ID NO: 122;
(h) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 8;
(ii) CDRH2 последовательности SEQ ID NO: 31;
(iii) CDRH3 последовательности SEQ ID NO: 54, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 77;
(ii) CDRL2 последовательности GAS (SEQ ID NO:100);
(iii) CDRL3 последовательности SEQ ID NO: 123;
(j) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 10;
(ii) CDRH2 последовательности SEQ ID NO: 33;
(iii) CDRH3 последовательности SEQ ID NO: 56, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 79;
(ii) CDRL2 последовательности AAS (SEQ ID NO:102);
(iii) CDRL3 последовательности SEQ ID NO: 125;
(k) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
i) CDRH1 последовательности SEQ ID NO: 11;
(ii) CDRH2 последовательности SEQ ID NO: 34;
(iii) CDRH3 последовательности SEQ ID NO: 57, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 80;
(ii) CDRL2 последовательности DAS (SEQ ID NO:103);
(iii) CDRL3 последовательности SEQ ID NO: 126;
(l) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 12;
(ii) CDRH2 последовательности SEQ ID NO: 35;
(iii) CDRH3 последовательности SEQ ID NO: 58, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 81;
(ii) CDRL2 последовательности DVS (SEQ ID NO:104);
(iii) CDRL3 последовательности SEQ ID NO: 127;
(m) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
i) CDRH1 последовательности SEQ ID NO: 13;
(ii) CDRH2 последовательности SEQ ID NO: 36;
(iii) CDRH3 последовательности SEQ ID NO: 59, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 82;
(ii) CDRL2 последовательности GAS (SEQ ID NO:105);
(iii) CDRL3 последовательности SEQ ID NO: 128;
(n) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 14;
(ii) CDRH2 последовательности SEQ ID NO: 37;
(iii) CDRH3 последовательности SEQ ID NO: 60, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 83;
(ii) CDRL2 последовательности EVS (SEQ ID NO:106);
(iii) CDRL3 последовательности SEQ ID NO: 129;
(r) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 18;
(ii) CDRH2 последовательности SEQ ID NO: 41;
(iii) CDRH3 последовательности SEQ ID NO: 64, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 87;
(ii) CDRL2 последовательности GAS (SEQ ID NO:110);
(iii) CDRL3 последовательности SEQ ID NO: 133;
(s) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 19;
(ii) CDRH2 последовательности SEQ ID NO: 42;
(iii) CDRH3 последовательности SEQ ID NO: 65, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 88;
(ii) CDRL2 последовательности GAS (SEQ ID NO:111);
(iii) CDRL3 последовательности SEQ ID NO: 134; или
(u) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 21;
(ii) CDRH2 последовательности SEQ ID NO: 44;
(iii) CDRH3 последовательности SEQ ID NO: 67, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 90;
(ii) CDRL2 последовательности DAS (SEQ ID NO:113);
(iii) CDRL3 последовательности SEQ ID NO: 136.
20. Применение по п.19, где антитело или его антигенсвязывающий фрагмент применяют для лечения человека.
21. Применение по любому из пп.19-20, где антитело или его антигенсвязывающий фрагмент вводится системно, назально или путем прямой доставки кодирующих его нуклеиновых кислот в организм млекопитающих.
22. Применение антитела или его антигенсвязывающего фрагмента по любому из пп.1-8, где антитело выбрано из:
(a) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности GFTFSSYA (SEQ ID NO: 1);
(ii) CDRH2 последовательности ISYDGSNK (SEQ ID NO: 24);
(iii) CDRH3 последовательности ARARPWRYSYGLYYYYGMDV (SEQ ID NO: 47), и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности QSISSY (SEQ ID NO: 70);
(ii) CDRL2 последовательности AAS (SEQ ID NO: 93);
(iii) CDRL3 последовательности QQSYSTPGS (SEQ ID NO: 116); или
(m) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 13;
(ii) CDRH2 последовательности SEQ ID NO: 36;
(iii) CDRH3 последовательности SEQ ID NO: 59, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 82;
(ii) CDRL2 последовательности GAS (SEQ ID NO:105);
(iii) CDRL3 последовательности SEQ ID NO: 128; или
(s) антитела или его антигенсвязывающего фрагмента, содержащего вариабельную область тяжелой цепи (VH), содержащую:
(i) CDRH1 последовательности SEQ ID NO: 19;
(ii) CDRH2 последовательности SEQ ID NO: 42;
(iii) CDRH3 последовательности SEQ ID NO: 65, и
вариабельную область легкой цепи (VL), содержащую:
(i) CDRL1 последовательности SEQ ID NO: 88;
(ii) CDRL2 последовательности GAS (SEQ ID NO:111);
(iii) CDRL3 последовательности SEQ ID NO: 134,
для оценки наличия инфекции SARS-CoV-2 у млекопитающих.
23. Применение по п.22, где млекопитающее является человеком.
24. Химерный антигенный рецептор, который селективно связывает RBD фрагмент Spike-белка вируса SARS-CoV-2, где химерный антигенный рецептор содержит антигенсвязывающую область, представленную антителом или его антигенсвязывающим фрагментом по любому из пп. 1-8.
| WO2021211775 A1, 21.10.2021 | |||
| RUI SHI et al., A human neutralizing antibody targets the receptor-binding site of SARS-CoV-2, Nature, 2020, Vol | |||
| ПРИСПОСОБЛЕНИЕ ДЛЯ АВТОМАТИЧЕСКОЙ ПЕРЕСТАНОВКИ ЛЕНТЫ В УКАЗАТЕЛЯХ ОСТАНОВОК | 1914 |
|
SU584A1 |
| BRYAN E | |||
| JONES et al., The neutralizing antibody, LY-CoV555, protects against SARS-CoV-2 infection in nonhuman primates, Transl | |||
| Med., 12 May 2021, Vol | |||
| Насос | 1917 |
|
SU13A1 |
| Моноклональное антитело к RBD фрагменту в составе S белка вируса SARS-CoV-2 | 2020 |
|
RU2744274C1 |
Авторы
Даты
2023-07-25—Публикация
2021-12-29—Подача