Область техники
Настоящее изобретение относится к антителу или его антигенсвязывающей части, которое может связываться с пропердином (фактор Р). Антитело по настоящему изобретению далее используется в приготовлении лекарственного средства для лечения заболеваний посредством ингибирования альтернативного пути комплемента.
Уровень техники
Ученые десятилетиями стремились разработать лекарственные средства для лечения комплемент-опосредованных заболеваний. К концу 20-го века многие антикомплементарные агенты продемонстрировали многообещающие результаты in vitro и на животных моделях, но лишь немногие кандидаты в лекарственные средства были разработаны для людей, а те, которые были разработаны, не получили дальнейшего развития. Среди доклинических молекул, которые в то время тестировались, были антитела против компонентов комплемента, которые блокировали их функцию. Например, блокада функции С5 с использованием моноклональных антител против С5 мыши и против С5 человека была легко установлена с использованием моделей болезней животных как in vitro, так и in vivo. К началу 21 века гуманизированное моноклональное антитело против C5 человека, экулизумаб, проходило клиническую разработку, и в 2007 году оно было одобрено FDA для использования при редком, но разрушительном заболевании, пароксизмальной ночной гемоглобинурии (PNH). Это клиническое подтверждение антикомплементарной терапии стало важной вехой в открытии антикомплементарных лекарственных средств; этот прорыв в сочетании с новаторскими данными, полученными в результате полногеномных исследований (GWAS), которые продемонстрировали ключевую роль комплемента в широко распространенных заболеваниях, привел к возрождению открытия антикомплементарных лекарственных средств. Это привело нас к сегодняшнему дню со многими новыми лекарственными средствами в этой области, которые проходят поздние стадии клинической разработки, и многие другие находятся на стадии открытия или доклинических стадиях (1).
Однако высокие ожидания, связанные с лекарственным препаратом-кандидатом, разработанным для офтальмологических показаний (т.е. моноклональное антитело, нацеленное на фактор D (FD), ламполизумаб), не привели к значимым клиническим ответам в исследованиях фазы III, что указывает на то, что индивидуальные проблемы, связанные с биодоступностью или эффективностью препарата остаются не решенными. Недавнее одобрение FDA версии экулизумаба пролонгированного действия (ALXN1210/равулизумаб, Ultomiris, Alexion), которая характеризуется длительным пребыванием в плазме крови и требует введения только каждые 8 недель, а не раз в две недели, является важным шагом в улучшении ведения пациентов, но не может считаться полноценным появлением нового лекарственного средства в этой области. При текущей клинической доступности только одного комплемент-специфического лекарственного средства, которое, очевидно, не может удовлетворить все клинические показания, связанные с комплементом, потребность в терапевтических подходах, адаптированных к заболеванию, становится все более актуальной (2).
Однако следует отметить, что разработка лекарственных средств, нацеленных на путь комплемента, связана с рядом проблем, включая огромное количество белков, из которых нужно выбирать, количество циркулирующих или связанных с мембраной белков каждой такой мишени, естественная центральная роль комплемента в борьбе с инфекцией и влияние слишком сильной регуляции на безопасность, а также идентификация соответствующего заболевания или показания к применению лекарственного средства. При ряде заболеваний комплемент играет ведущую роль в патогенезе, тогда как при других комплемент является «усугубителем» заболевания, вызывая усиление патологии, инициированное другим триггером заболевания, тем самым вызывая воспаление и повреждение тканей.
Тем не менее, количество потенциальных лекарственных средств в системе комплемента увеличивается, поскольку роль комплемента помимо лизиса и в перекрестных взаимодействиях с другими биологическими системами, такими как коагуляция, становится все более ясной. В разработке находятся лекарственные средства, нацеленные на каждый из трех путей в системе комплемента, и эти лекарственные средства включают низкомолекулярные соединения, пептиды, биологические препараты, антитела и терапевтические средства на основе ДНК.
Из-за каскадной природы пути комплемента и большого количества белков, как растворимых, так и связанных с мембраной, из которых он состоит, выбор отдельных точек вмешательства может привести к различным терапевтическим эффектам. В большинстве случаев триггерный путь активации комплемента активируется чужеродными или измененными поверхностями. При классическом пути распознавание иммунных комплексов (и других неиммуноглобулиновых фрагментов) с помощью C1q активирует связанные сериновые протеазы, C1r и C1s, которые расщепляют белки плазмы C2 и C4 с образованием конвертазного комплекса C3 (C4b2a) на активирующей поверхности. Тот же результат достигается, когда паттерн-распознающие белки лектинового пути (связывающие маннозу лектины [MBL], фиколины, коллектины) связываются с углеводными паттернами на поверхности патогена и индуцируют MBL-ассоциированные сериновые протеазы для расщепления C2/C4. Альтернативный путь, посредством взаимодействия с протеазами, факторами B и D (FB, FD) и связанным с поверхностью C3b, формирует другой тип конвертазы C3. Во всех трех путях комплемента точкой схождения является генерация той или иной С3-конвертазы, которая активирует С3, что приводит как минимум к двум значимым событиям на поверхности пораженной клетки. Одним из них является депонирование C3b на поверхности-мишени (что делает его кандидатом на опсонизацию макрофагами) и одновременное высвобождение анафилатоксина C3a. Другой - образование C3bBb (конвертазы C3) и ее стабилизация пропердином для сборки конвертаз C5, которые расщепляют C5. Альтернативный путь является единственным путем, который остается конститутивно активным, обеспечивая постоянную минимальную фоновую активность, готовую к усилению при наличии правильного триггера. В то время как высвобожденный анафилатоксин C5a является одним из самых сильных хемоаттрактантов и провоспалительных модуляторов, работая в основном за счет связывания с рецептором 1 C5a (C5aR1), фрагмент C5b может индуцировать сборку мембраноатакующего комплекса (MAC), который повреждает или вызывает лизис атакуемых клеток (1).
Более 20 терапевтических агентов, нацеленных на отдельные компоненты и эффекторные пути каскада комплемента, в настоящее время находятся в стадии клинической разработки для различных показаний (2).
Петля амплификации альтернативного пути является основной движущей силой активации комплемента и часто определяет начало образования конвертазы С5, тем самым инициируя последующее расщепление С5 и активацию терминального пути. Даже при заболеваниях, при которых патологические механизмы опосредованы лектиновым или классическим путями, блокада альтернативного пути может обеспечить терапевтический эффект за счет уменьшения эффекта, генерируемого через петлю амплификации. Следовательно, разработанное специфическое вмешательство только в петлю амплификации считается привлекательным предложением при разработке лекарственных средств, поскольку это позволило бы лектиновым и классическим путям оставаться, по меньшей мере, частично функциональными для борьбы с инфекциями; в то время как блокирование альтернативного пути контролировало бы побочное повреждение нормальной ткани.
В цикле амплификации есть несколько мишеней, поддающихся воздействию лекарственных средств, например, C3 (или C3b), FB, FD и FP (пропердин), и все они были мишенями при разработке лекарственных средств (1).
Несмотря на то, что развитие лекарственных средств доходит до поздней стадии клинической разработки, даже спустя более десяти лет после запуска экулизумаба, на рынке антикомплементарных лекарственных средств по-прежнему не хватает каких-либо новых одобренных терапевтических средств, направленных против альтернативных мишеней в каскаде. Новые случаи недостаточного ответа на анти-С5 терапию и понимание того, что множественные триггеры и сложные генетические признаки могут исказить базальную активность комплемента у пациента, но плохо определяемым образом, указывают на необходимость более всесторонней стратификации пациентов и надежного мониторинга во время антикомплементарной терапии заболеваний, которые в настоящее время лечат экулизумабом.
Хотя одобрение экулизумаба резко изменило картину PNH, предоставив первую этиологическую терапию для этих пациентов, возникли неудовлетворенные клинические потребности, в том числе генетически обусловленный рефрактерный фенотип к анти-С5 у некоторых пациентов, остаточный гемолиз С3-опсонизированных клеток PNH во внесосудистых отделах, а также фармакокинетический/фармакодинамический (ФК/ФД) прорывной гемолиз, наблюдаемый в некоторых случаях, который вызывает сильную активацию комплемента (то есть острые инфекции), независимо от уровня дозировки препарата (2).
Важно отметить, что анти-С5 терапия связана с риском опасных для жизни и фатальных менингококковых инфекций, которые могут быстро стать опасными для жизни или смертельными, если их не распознать и не лечить на ранней стадии (3).
Таким образом, вполне возможно, что в большинстве ситуаций заболеваний, опосредованных комплементом, полное ингибирование образования MAC, происходящего из всех путей комплемента, не является существенным, и подавление, а не выключение комплемента, вероятно, будет достаточным для обеспечения терапевтического эффекта, позволяя достаточной части активности комплемента оставаться для защиты от критических инфекций. Целью такого лечения было бы обратить вспять дисрегуляцию и восстановить гомеостаз. В этом контексте петля амплификации представляет собой превосходную мишень. Путем уменьшения количества циклов посредством петли усиления за счет увеличения регуляции петли или путем снижения доступности конвертазы можно позволить точно настроить терапию для улучшения патологии при сохранении защитной роли комплемента в иммунной защите. Это будет значительным преимуществом для лечения пожилых и уязвимых к инфекции людей в сообществе. Несколько AP-специфических препаратов находятся на фазе 2 разработки, включая ингибиторы FB и FD. Другие препараты, обладающие модулирующими свойствами, уже не за горами; доставка функциональных доменов регуляторов, таких как FH, может напрямую модулировать ферменты конвертазы, как в доклинической молекуле AMY201 (Amyndas), укороченной, рекомбинантной форме FH, сконструированной для связывания с превосходной эффективностью с поверхностями-мишенями. Наконец, возможно блокировать естественные модификаторы комплемента; пропердин стабилизирует ферменты AP-конвертазы, а MASP3 активирует FD, вмешательство на этих уровнях с использованием таких препаратов, как CLG561 (антипропердин, Novartis) или OMS906 (доклинический анти-MASP3, Omeros), может подтолкнуть систему комплемента к восстановлению гомеостаза (4).
Настоящее изобретение относится к антителам, нацеленным на пропердин (фактор P) для ингибирования альтернативного пути.
Моноклональные антитела к пропердину известны в данной области и описаны, например, в WO 2006131874, WO 2009110918, WO 201109494, WO 2013006449 и WO 2018140956. Были разработаны различные антитела, которые связывают активированный пропердин, но, насколько нам известно, ни одно из них еще не одобрено. Существует одно антитело CLG561, нацеленное на пропердин, которое находится на ранней клинической стадии, когда его оценивали в комбинации с антителом против C5 (5).
Таким образом, все еще существует неудовлетворенная потребность в эффективной регуляции активности пропердина и, таким образом, в контроле альтернативного пути комплемента. Таким образом, настоящее изобретение относится к антителу против пропердина, которое может быть разработано для лечения родственных заболеваний.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
Настоящее изобретение относится к антителу или его антигенсвязывающей части, которое может связываться с пропердином (фактор P). Антитело по настоящему изобретению приводит к селективному ингибированию альтернативного пути комплемента, в то же время позволяя продолжаться классическому и лектиновому путям. Кроме того, антитело по настоящему изобретению может модифицировать или уменьшать связывание с FcγR для минимизации его ADCC-активности. Настоящее изобретение относится к антителу, которое содержит аминокислотную последовательность для минимизации его CDC-активности. Антитело по настоящему изобретению имеет более высокую аффинность связывания с FcRn, и, следовательно, антитело по настоящему изобретению может иметь длительный период полужизни в кровотоке организма пациента, и его можно вводить с уменьшенной частотой дозирования. Антитело по настоящему изобретению может дополнительно применяться для приготовления лекарственного средства для лечения заболеваний посредством ингибирования альтернативного пути комплемента.
Краткое описание чертежей
Фигура 1 демонстрирует дозозависимое связывание связывающих агентов против пропердина с нативным очищенным пропердином человека. Протестированные mAb против пропердина человека по настоящему изобретению показали значительное связывание с нативным антигеном.
Фигуры 2a и 2b демонстрируют, что mAb против пропердина человека по настоящему изобретению ингибируют опосредованный альтернативным комплементом человека лизис эритроцитов кролика (RBC). Этот лизис почти полностью ингибировался каждым из протестированных mAb против пропердина. Изотипический контроль mAb использовали в качестве отрицательного контроля, и он показал лишь незначительное ингибирование лизиса.
Фигура 3а демонстрирует перекрестную реактивность mAb против пропердина с пропердином обезьяны в ее сыворотке. Все протестированные гуманизированные mAb против пропердина человека по настоящему изобретению были способны ингибировать лизис эритроцитов кролика, опосредованный обезьяньей альтернативной системой комплемента, из сыворотки обезьяны.
Фигура 3b демонстрирует ингибирующую гемолиз активность mAb против пропердина P15 (GLS) у шести разных обезьян. P15 (GLS) было способно ингибировать на 85-95% лизис эритроцитов кролика, опосредованный обезьяньей сывороткой, у разных обезьян.
Фигура 4 демонстрирует дозозависимое ингибирование LPS-индуцированной активации комплемента всеми испытанными mAb по настоящему изобретению. Все протестированные mAb эффективно ингибировали активацию комплемента, опосредованную нормальной человеческой сывороткой (NHS), при обеих испытанных концентрациях. Все протестированные mAb продемонстрировали значительное ингибирование образования MAC.
Фигура 5 демонстрирует ингибирование индуцированного сывороткой человека гемолиза собственных эритроцитов, вызванного дисфункцией CD55 и CD59, с использованием mAb против пропердина по настоящему изобретению. Человеческие эритроциты обычно не лизируются в присутствии аутологичной человеческой сыворотки. Однако в присутствии антител против CD55 и против CD59 регуляция пути комплемента нарушается, что приводит к гемолизу собственных эритроцитов аутологичной сывороткой. Указанный лизис ингибировался каждым из протестированных mAb против пропердина.
Фигура 6 демонстрирует ингибирование индуцированного сывороткой человека гемолиза собственных эритроцитов, обработанных проназой, всеми протестированными mAb против пропердина по настоящему изобретению.
Фигура 7 демонстрирует снижение продукции TNF-α, индуцированной интратрахеальной инстилляцией LPS, у мышей с помощью кроличьего mAb против мышиного пропердина (суррогатное антитело антитела против пропердина по настоящему изобретению).
Фигура 8 демонстрирует снижение продукции IL-6, индуцированной интратрахеальной инстилляцией LPS, у мышей с помощью кроличьего mAb против мышиного пропердина (суррогатное антитело антитела против пропердина по настоящему изобретению).
Определения
Термин «антитело», как упоминается в настоящем описании, включает цельные антитела и любые антигенсвязывающие фрагменты (т.е. «антигенсвязывающая часть») или их отдельные цепи. «Антитело» относится к гликопротеину, содержащему по меньшей мере две тяжелые (Н) цепи и две легкие (L) цепи, соединенные между собой дисульфидными связями, или его антигенсвязывающую часть. Каждая тяжелая цепь состоит из вариабельной области тяжелой цепи (обозначенной в настоящем документе как VH) и константной области тяжелой цепи. Константная область тяжелой цепи состоит из трех доменов, CH1, CH2 и CH3. Каждая легкая цепь содержит вариабельную область легкой цепи (сокращенно обозначенную в настоящем описании как VL) и константную область легкой цепи. Константная область легкой цепи содержит один домен, CL. Области VH и VL могут быть дополнительно подразделены на области гипервариабельности, называемые областями, определяющими комплементарность (CDR), перемежающиеся с более консервативными областями, называемыми каркасными областями (FR). Каждая VH и VL состоит из трех CDR и четырех FR, расположенных от амино-конца к карбокси-концу в следующем порядке: FR1, CDR1, FR2, CDR2, FR3, CDR3, FR4. Вариабельные области тяжелой и легкой цепей содержат домен связывания, который взаимодействует с антигеном. Константные области антител могут опосредовать связывание иммуноглобулина с тканями или факторами хозяина, включая различные клетки иммунной системы (например, эффекторные клетки, такие как NK-клетки, Т-клетки, макрофаги и дендритные клетки и т.д.) и первые компонент (C1q) классической системы комплемента.
Термин «функционально связанный» предназначен для обозначения того, что ген антитела лигируют в вектор таким образом, что последовательности контроля транскрипции и трансляции внутри вектора выполняют свою предполагаемую функцию регуляции транскрипции и трансляции гена антитела.
Термин «Ka» представляет собой скорость ассоциации конкретного взаимодействия антитело-антиген, тогда как термин «Kd» представляет собой скорость диссоциации конкретного взаимодействия антитело-антиген. Термин «KD» представляет собой константу аффинности, которую получают из отношения Kd к Ka. Ее можно измерить, используя метод поверхностного плазмонного резонанса, который хорошо известен в данной области техники.
Термины «моноклональное антитело» или «композиция моноклонального антитела», используемые в настоящем документе, относятся к препарату молекул антител в виде одномолекулярной композиции. Композиция моноклонального антитела демонстрирует уникальную специфичность связывания и аффинность к конкретному эпитопу.
Термин «биспецифическое антитело» относится к гомогенной популяции антител, участвующих в высокоспецифическом распознавании и связывании двух разных антигенных детерминант или эпитопов.
Термин «рекомбинантное антитело», используемый в настоящем описании, включает все антитела, которые получают, экспрессируют, создают или выделяют рекомбинантными способами. Однако в некоторых вариантах осуществления такие рекомбинантные антитела могут быть получены путем мутагенеза in vitro, и, таким образом, аминокислотные последовательности областей VH и VL рекомбинантных антител, как описано в настоящем документе, представляют собой последовательности, которые могут не существовать в естественной среде в репертуаре зародышевой линии антител человека in vivo.
Термин «человеческое антитело» включает антитела, имеющие вариабельные и константные области (если они присутствуют), полученные из последовательностей иммуноглобулинов человека, предпочтительно последовательностей зародышевой линии человека.
Термин «химерное антитело и его антигенсвязывающие фрагменты», используемый в настоящем документе, включает части двух или более разных видов (например, мыши и человека). Химерные антитела могут быть получены с мышиными вариабельными областями желаемой специфичности, слитыми с человеческими константными доменами (например, как описано в патенте США No. 4816567). Таким образом, нечеловеческие антитела можно модифицировать, чтобы сделать их более подходящими для клинического применения у человека (например, способы лечения или профилактики комплемент-опосредованного расстройства у пациента).
Используемый в настоящем описании термин «гуманизированных» форм нечеловеческих (например, мышиных) антител представляет собой химерные антитела, которые содержат минимальную последовательность, полученную из нечеловеческого иммуноглобулина. По большей части гуманизированные антитела представляют собой иммуноглобулины человека (антитело-реципиент), в которых остатки из гипервариабельной области реципиента заменены остатками из гипервариабельной области вида, отличного от человека (донорное антитело), такого как мышь, крыса, кролик или низший примат, обладающими желаемой специфичностью, аффинностью и эффективностью. В некоторых случаях остатки каркасной области Fv (FR) человеческого иммуноглобулина заменены соответствующими нечеловеческими остатками. Более того гуманизированное антитело может содержать остатки, которые не обнаружены в рецепиентном антителе или в донорном антителе. Эти модификации сделаны для дальнейшего улучшения характеристик антител. В общем, гуманизированное антитело будет содержать практически весь, по меньшей мере один, и, как правило, два вариабельных домена, в которые все или почти все гипервариабельные петли соответствуют областям нечеловеческого иммуноглобулина, а все или практически все FR-области являются областями последовательности человеческого иммуноглобулина. Гуманизированное антитело необязательно также может содержать по меньшей мере часть иммуноглобулиновой константной области (Fc), как правило, из человеческого иммуноглобулина.
Термин «фармацевтический состав» относится к препаратам, которые находятся в такой форме, которая обеспечивает несомненную эффективность биологической активности активных ингредиентов. Термины «фармацевтический состав» или «фармацевтическая композиция» или «композиция» могут использоваться здесь взаимозаменяемо.
Термин «вспомогательное вещество» относится к агенту, который может быть добавлен к составу для стабилизации активного лекарственного вещества в препаративной форме для регулирования и поддержания осмоляльности и рН фармацевтических препаратов. Примеры обычно используемых вспомогательных веществ включают, но не ограничиваются ими, сахара, полиолы, аминокислоты, поверхностно-активные вещества и полимеры. «Фармацевтически приемлемые» вспомогательные вещества представляют собой такие, которые целесообразно вводить рассматриваемому млекопитающему для получения эффективной дозы используемого активного ингредиента.
Используемый в настоящем описании термин «лечение» или «терапевтические средства» относится к любому лечению заболевания у млекопитающего, особенно у человека. Он включает: (а) предотвращение возникновения заболевания у пациента, который может быть предрасположен к заболеванию или подвергаться риску заражения заболеванием, но еще не диагностирован как имеющий его; b) ингибирование заболевания, т.е. прекращение его развития; и (c) облегчение заболевания, т.е. вызывание регрессии заболевания.
Термины «пациент» и «субъект» используются взаимозаменяемо и используются в их обычном смысле для обозначения живого организма, страдающего или склонного к состоянию, которое можно предотвратить или лечить путем введения композиции по настоящему изобретению, и включает как человека, так и животных, отличных от человека. Примеры субъектов включают, но не ограничиваются ими, человека, шимпанзе и других обезьян и виды обезьян; сельскохозяйственных животных, таких как крупный рогатый скот, овцы, свиньи, козы и лошади; домашних млекопитающих, такие как собаки и кошки; лабораторных животных, включая грызунов, таких как мыши, крысы и морские свинки; птиц, включая домашних, диких и промысловых птиц, таких как куры, индейки и другие куриные, утки, гуси и т.п. Термин не обозначает определенный возраст. Таким образом, интерес представляют взрослые, молодые и новорожденные особи.
(3-буквенное)
(1-буквенное)
Другие сокращения, используемые в настоящей патентной заявке:
ADCC: антителозависимая клеточная цитотоксичность
аГУС: атипичный гемолитико-уремический синдром
CDC: Комплементзависимая цитотоксичность
CDR: область, определяющая комплементарность
CH: константная область тяжелой цепи
CL: константная область легкой цепи
ДЭПК: диэтилпирокарбонат
ЭДТА: этилендиаминтетрауксусная кислота.
EGTA: этиленгликоль-бис(β-аминоэтиловый эфир)-N,N,N',N'-тетрауксусная кислота
FcRn: неонатальный Fc-рецептор
FR: каркасная область
HCVR: вариабельная область тяжелой цепи
HC: тяжелая цепь
IPTG: изопропил β-D-1-тиогалактопиранозид
i.v.: внутривенно
Ka: константа ассоциации
Kd: константа диссоциации
KD: равновесная константа диссоциации
LCVR: вариабельная область легкой цепи
LC: легкая цепь
LPS: липополисахарид
mAb: моноклональное антитело
MAC: Мембранный атакующий комплекс
NHS: нормальная человеческая сыворотка
OD: оптическая плотность
P20: Полисорбат 20
PBS: фосфатно-солевой буфер.
Буфер PF: буфер периплазматической фракции
Pfx: корректирующая ДНК-полимераза, PfxTM от Invitrogen
PNH: пароксизмальная ночная гемоглобинурия
RBC: эритроцит
RPM: обороты в минуту
RPMI: Мемориальный институт Розуэлл-парка
sc: подкожно
scFv: одноцепочечный вариабельный фрагмент
SEQ/seq: последовательность
SPR: поверхностный плазмонный резонанс
ТМА: Тромботическая микроангиопатия
ТМБ: 3, 3', 5, 5'-тетраметилбензидин
VH: вариабельная область тяжелой цепи
VL: вариабельная область легкой цепи
Варианты осуществления изобретения
Описание настоящего изобретения относится к новым антителам против пропердина, которые можно использовать в терапевтических целях.
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению связывается с высокой аффинностью с пропердином человека.
В одном варианте осуществления настоящее изобретение относится к антителу против пропердина или его антигенсвязывающей части, содержащему:
(a) CDRH1 общей формулы (I): G-Y-X1a-X2a-X3a-X4a-X5a-X6a-X7a;
(b) CDRH2 общей формулы (II): X1b-I-X2b-X3b-X4b-X5b-X6b-X7b;
(c) CDRH3 общей формулы (III): X1c-X2c-X3c-X4c-X5c-X6c-X7c-X8c-X9c-X10c-X11c-X12c-X13c-X14c;
(d) CDRL1 общей формулы (IV): X1d-X2d-X3d-X4d-X5d-X6d-X7d-X8d-X9d-X10d-X11d-X12d-X13d-X14d-X15d-X16d-X17d;
(e) CDRL2 общей формулы (V): X1e-X2e-X3e-X4e-X5e-X6e-X7e и
(f) CDRL3 общей формулы (VI): X1f-X2f-X3f-X4f-X5f-X6f-X7f-X8f-X9f-X10f-X11f, где,
X1a представляет собой аминокислоту, выбранную из серина и треонина;
X2a представляет собой аминокислоту, выбранную из фенилаланина и изолейцина;
X3a представляет собой аминокислоту, выбранную из треонина и аланина;
X4a представляет собой аминокислоту, выбранную из аспарагиновой кислоты, серина и гистидина;
X5a представляет собой аминокислоту, выбранную из тирозина, аспарагина, глицина и треонина;
Каждый из X6a и X7a может присутствовать или отсутствовать, и, если он присутствует, то он представляет собой аминокислоту тирозин;
X1b представляет собой аминокислоту, выбранную из валина, лейцина, тирозина и глутаминовой кислоты;
X2b представляет собой аминокислоту, выбранную из серина, аспарагина и аспарагиновой кислоты;
X3b представляет собой аминокислоту, выбранную из треонина, пролина и тирозина;
X4b представляет собой аминокислоту, выбранную из тирозина, глицина, аспарагиновой кислоты и серина;
X5b представляет собой аминокислоту, выбранную из тирозина, треонина, глицина и аланина;
X6b представляет собой аминокислоту, выбранную из глицина, аспарагиновой кислоты и треонина;
X7b представляет собой аминокислоту, выбранную из аспарагиновой кислоты, тирозина и аспарагина;
X1c представляет собой аминокислоту, выбранную из аспарагиновой кислоты, глутаминовой кислоты, аланина и аргинина;
X2c представляет собой аминокислоту, выбранную из лейцина, аспарагиновой кислоты, лизина и глицина;
X3c представляет собой аминокислоту, выбранную из аспарагиновой кислоты, тирозина, серина и лейцина;
X4c представляет собой аминокислоту, выбранную из глицина, аспарагиновой кислоты, аргинина, тирозина, лейцина, серина и лизина;
X5c представляет собой аминокислоту, выбранную из тирозина, аргинина, аспарагиновой кислоты и глицина;
X6c представляет собой аминокислоту, выбранную из глутаминовой кислоты, серина, фенилаланина, тирозина и аспарагина;
X7c представляет собой аминокислоту, выбранную из серина, пролина, аспарагиновой кислоты и фенилаланина, или может отсутствовать;
X8c представляет собой аминокислоту, выбранную из метионина, триптофана, фенилаланина и валина, или может отсутствовать;
X9c представляет собой аминокислоту, выбранную из аспарагиновой кислоты и фенилаланина, или может отсутствовать;
Каждый из X10c и X11c может присутствовать или отсутствовать, и когда он присутствует, он представляет собой аминокислоту, независимо выбранную из тирозина и аланина;
Каждый из X12c, X13c и X14c может присутствовать или отсутствовать, и когда он присутствует, он представляет собой аминокислоту, независимо выбранную из метионина, аспарагиновой кислоты и тирозина;
X1d представляет собой аминокислоту, выбранную из аргинина, лизина, лейцина, серина, тирозина и глутаминовой кислоты;
X2d представляет собой аминокислоту, выбранную из пролина, серина, аланина, лейцина, глицина и глутамима;
X3d представляет собой аминокислоту, выбранную из серина, аспарагиновой кислоты, аргинина и триптофана;
X4d представляет собой аминокислоту, выбранную из глутамина, глицина, серина и лейцина;
X5d представляет собой аминокислоту, выбранную из аспарагиновой кислоты, серина, треонина, пролина и лейцина;
X6d представляет собой аминокислоту, выбранную из изолейцина, валина, лейцина, серина, аспарагина, фенилаланина и глицина;
X7d представляет собой аминокислоту, выбранную из аспарагина, лейцина, глицина, пролина, изолейцина, лизина и гистидина;
X8d представляет собой аминокислоту, выбранную из аспарагина, аспарагиновой кислоты, треонина, глицина, аргинина, глутамина и тирозина;
X9d представляет собой аминокислоту, выбранную из тирозина, изолейцина, триптофана, аспарагина, глицина, валина, гистидина и серина;
X10d представляет собой аминокислоту, выбранную из лейцина, аспарагина, треонина, лизина, аргинина и серина;
X11d представляет собой аминокислоту, выбранную из серина, глицина, аланина, тирозина, пролина, аспарагиновой кислоты и аспарагина;
X12d может отсутствовать;
X13d представляет собой аминокислоту, выбранную из треонина, серина, аспарагина и лизина, или может отсутствовать;
Каждый из X14d, X15d, X16d и X17d может присутствовать или отсутствовать, и когда он присутствует, он представляет собой аминокислоту, независимо выбранную из тирозина, лейцина, аспарагина и аланина;
X1e представляет собой аминокислоту, выбранную из аспарагиновой кислоты, триптофана, тирозина, лейцина и аланина;
X2e представляет собой аминокислоту, выбранную из аспарагина, аланина, треонина и валина;
X3e представляет собой аминокислоту, выбранную из аспарагина, серина и треонина;
X4e представляет собой аминокислоту, выбранную из лизина, треонина, аргинина и серина;
X5e представляет собой аминокислоту, выбранную из аргинина и лейцина;
X6e представляет собой аминокислоту, выбранную из фенилаланина, глутаминовой кислоты, гистидина, аспарагиновой кислоты и аланина;
X7e представляет собой аминокислоту, выбранную из серина и аспарагиновой кислоты;
В одном из вариантов осуществления CDR3 легкой цепи (далее обозначаемой как CDRL3) антитела против пропердина или его антигенсвязывающей части по настоящему изобретению имеет аминокислотную последовательность X1f-X2f-X3f-X4f-X5f-X6f-X7f-X8f-X9f-X10f-X11f, где
X1f представляет собой аминокислоту, выбранную из гистидина, глутамина, триптофана, аланина, глицина и метионина;
X2f представляет собой аминокислоту, выбранную из глутамина, аланина и треонина;
X3f представляет собой аминокислоту, выбранную из тирозина, глицина, лейцина, аргинина, триптофана и глутамина;
X4f представляет собой аминокислоту, выбранную из лейцина, аспарагина, треонина, тирозина, глутамина и аспарагиновой кислоты;
X5f представляет собой аминокислоту, выбранную из серина, треонина, гистидина, аланина и глицина;
X6f представляет собой аминокислоту, выбранную из серина, лейцина, фенилаланина, треонина, изолейцина и тирозина;
X7f представляет собой аминокислоту, выбранную из тирозина, пролина и лейцина;
X8f представляет собой аминокислоту, выбранную из треонина, пролина, тирозина, триптофана и аргинина;
X9f представляет собой аминокислоту, выбранную из треонина и глутаминовой кислоты, или может отсутствовать;
X10f представляет собой аминокислоту, выбранную из аланина и лейцина, или отсутствует, и
X11f представляет собой аминокислоту валин или может отсутствовать.
В одном варианте осуществления аминокислотная последовательность константной области антитела против пропердина включает константную область IgG1, IgG2, IgG3, IgG4, IgG2/G4, IgA, IgE, IgM или IgD, предпочтительно IgG1 или IgG4.
В другом варианте осуществления одно или более антител против пропердина по настоящему изобретению имеет модифицированную или сниженную активность ADCC и/или CDC или не имеет активности ADCC и/или CDC. В одном из вариантов осуществления антитело против пропердина или его антигенсвязывающая часть обладает сниженным потенциалом вызывать проблему безопасности, связанную с ADCC и CDC.
В одном из вариантов осуществления одно или более антител против пропердина по настоящему изобретению обладает сниженной активностью ADCP или не имеет ее.
В одном из вариантов осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению имеет KD 10-8 М или менее, более предпочтительно 10-10 М или менее для антигена пропердина. Значение KD представляет собой меру аффинности связывания антитела с антигеном-мишенью.
В одном из вариантов осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению перекрестно реактивно с пропердином видов, отличных от человека.
В одном из вариантов осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению обладает более высокой специфичностью связывания с пропердином человека.
В одном из вариантов осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению имеет увеличенный период полужизни у пациента по сравнению с антителом против пропердина с обычным Fc-фрагментом.
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть согласно настоящему изобретению блокирует функцию пропердина в опосредовании активации альтернативного пути комплемента.
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть согласно настоящему изобретению предотвращает связывание пропердина с клетками-мишенями.
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению может предотвращать повышенное связывание C3b с поверхностью клетки-мишени.
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению регулирует образование MAC на поверхности клеток-мишеней и тем самым предотвращает лизис клеток.
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению может минимизировать образование анафилатоксинов C3a и C5a.
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению предотвращает опосредованный комплементом лизис клеток-мишеней.
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению имеет повышенный период полужизни в кровотоке.
В другом варианте осуществления антитело против пропердина или его антигенсвязывающая часть способно связываться с пропердином обезьяны, что облегчает разработку лекарственного средства за счет создания соответствующей животной фармакологической и токсикологической модели.
В одном из вариантов осуществления настоящее изобретение относится к композиции, содержащей антитело против пропердина, которое специфически связывает пропердин человека (фактор Р), и приемлемый носитель.
В другом варианте осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению можно использовать для лечения заболеваний, при которых активность пропердина является вредной, таких как инфекции, различные виды онкологических заболеваний, аутоиммунные расстройства и другие расстройства, такие как PNH, aHUS, где активность комплемента усиливается.
Подробное описание изобретения
В одном варианте осуществления антитело против пропердина или его антигенсвязывающая часть по настоящему изобретению связывается с высокой аффинностью с пропердином человека.
Аминокислотные последовательности антитела против пропердина.
В одном из вариантов осуществления CDR1 тяжелой цепи (далее CDRH1) антитела против пропердина или его антигенсвязывающей части по настоящему изобретению имеет аминокислотную последовательность общей формулы (I): G-Y-X1a- X2a-X3a-X4a-X5a-X6a-X7a, где,
X1a представляет собой аминокислоту, выбранную из серина и треонина;
X2a представляет собой аминокислоту, выбранную из фенилаланина и изолейцина;
X3a представляет собой аминокислоту, выбранную из треонина и аланина;
X4a представляет собой аминокислоту, выбранную из аспарагиновой кислоты, серина и гистидина;
X5a представляет собой аминокислоту, выбранную из тирозина, аспарагина, глицина и треонина;
Каждый из X6a и X7a может присутствовать или отсутствовать, и, если он присутствует, он представляет собой аминокислоту тирозин.
В одном из вариантов осуществления CDR2 тяжелой цепи (далее CDRH2) антитела против пропердина или его антигенсвязывающей части по настоящему изобретению имеет аминокислотную последовательность X1b-I-X2b-X3b-X4b-X5b-X6b-X7b, где,
X1b представляет собой аминокислоту, выбранную из валина, лейцина, тирозина и глутаминовой кислоты;
X2b представляет собой аминокислоту, выбранную из серина, аспарагина и аспарагиновой кислоты;
X3b представляет собой аминокислоту, выбранную из треонина, пролина и тирозина;
X4b представляет собой аминокислоту, выбранную из тирозина, глицина, аспарагиновой кислоты и серина;
X5b представляет собой аминокислоту, выбранную из тирозина, треонина, глицина и аланина;
X6b представляет собой аминокислоту, выбранную из глицина, аспарагиновой кислоты и треонина;
X7b представляет собой аминокислоту, выбранную из аспарагиновой кислоты, тирозина и аспарагина.
В одном из вариантов осуществления CDR3 тяжелой цепи (далее CDRH3) антитела против пропердина или его антигенсвязывающей части по настоящему изобретению имеет аминокислотную последовательность X1c-X2c-X3c-X4c-X5c-X6c-X7c-X8c-X9c-X10c-X11c-X12c-X13c-X14c, где,
X1c представляет собой аминокислоту, выбранную из аспарагиновой кислоты, глутаминовой кислоты, аланина и аргинина;
X2c представляет собой аминокислоту, выбранную из лейцина, аспарагиновой кислоты, лизина и глицина;
X3c представляет собой аминокислоту, выбранную из аспарагиновой кислоты, тирозина, серина и лейцина;
X4c представляет собой аминокислоту, выбранную из глицина, аспарагиновой кислоты, аргинина, тирозина, лейцина, серина и лизина;
X5c представляет собой аминокислоту, выбранную из тирозина, аргинина, аспарагиновой кислоты и глицина;
X6c представляет собой аминокислоту, выбранную из глутаминовой кислоты, серина, фенилаланина, тирозина и аспарагина;
X7c представляет собой аминокислоту, выбранную из серина, пролина, аспарагиновой кислоты и фенилаланина, или отсутствует;
X8c представляет собой аминокислоту, выбранную из метионина, триптофана, фенилаланина и валина, или отсутствует;
X9c представляет собой аминокислоту, выбранную из аспарагиновой кислоты и фенилаланина, или отсутствует;
Каждый из X10c и X11c присутствует или отсутствует и, если присутствует, представляет собой аминокислоту, независимо выбранную из тирозина и аланина;
Каждый из X12c, X13c и X14c присутствует или отсутствует, и когда он присутствует, он представляет собой аминокислоту, независимо выбранную из метионина, аспарагиновой кислоты и тирозина.
В одном из вариантов осуществления CDR1 легкой цепи (далее CDRL1) антитела против пропердина или его антигенсвязывающей части по настоящему изобретению имеет аминокислотную последовательность X1d-X2d-X3d-X4d-X5d-X6d-X7d-X8d-X9d-X10d-X11d-X12d-X13d-X14d-X15d-X16d-X17d, где,
X1d представляет собой аминокислоту, выбранную из аргинина, лизина, лейцина, серина, тирозина и глутаминовой кислоты;
X2d представляет собой аминокислоту, выбранную из пролина, серина, аланина, лейцина, глицина и глутамима;
X3d представляет собой аминокислоту, выбранную из серина, аспарагиновой кислоты, аргинина и триптофана;
X4d представляет собой аминокислоту, выбранную из глутамина, глицина, серина и лейцина;
X5d представляет собой аминокислоту, выбранную из аспарагиновой кислоты, серина, треонина, пролина и лейцина;
X6d представляет собой аминокислоту, выбранную из изолейцина, валина, лейцина, серина, аспарагина, фенилаланина и глицина;
X7d представляет собой аминокислоту, выбранную из аспарагина, лейцина, глицина, пролина, изолейцина, лизина и гистидина;
X8d представляет собой аминокислоту, выбранную из аспарагина, аспарагиновой кислоты, треонина, глицина, аргинина, глутамина и тирозина;
X9d представляет собой аминокислоту, выбранную из тирозина, изолейцина, триптофана, аспарагина, глицина, валина, гистидина и серина;
X10d представляет собой аминокислоту, выбранную из лейцина, аспарагина, треонина, лизина, аргинина и серина;
X11d представляет собой аминокислоту, выбранную из серина, глицина, аланина, тирозина, пролина, аспарагиновой кислоты и аспарагина;
X12d представляет собой аминокислоту, выбранную из лизина, валина, треонина, аланина и глутамина, или отсутствует;
X13d представляет собой аминокислоту, выбранную из треонина, серина, аспарагина и лизина, или отсутствует;
Каждый из X14d, X15d, X16d и X17d присутствует или отсутствует, и когда он присутствует, он представляет собой аминокислоту, независимо выбранную из тирозина, лейцина, аспарагина и аланина. В одном из вариантов осуществления CDR2 легкой цепи (далее CDRL2) антитела против пропердина или его антигенсвязывающей части по настоящему изобретению имеет аминокислотную последовательность X1e-X2e-X3e-X4e-X5e-X6e-X7e, где
X1e представляет собой аминокислоту, выбранную из аспарагиновой кислоты, триптофана, тирозина, лейцина и аланина;
X2e представляет собой аминокислоту, выбранную из аспарагина, аланина, треонина и валина;
X3e представляет собой аминокислоту, выбранную из аспарагина, серина и треонина;
X4e представляет собой аминокислоту, выбранную из лизина, треонина, аргинина и серина;
X5e представляет собой аминокислоту, выбранную из аргинина и лейцина;
X6e представляет собой аминокислоту, выбранную из фенилаланина, глутаминовой кислоты, гистидина, аспарагиновой кислоты и аланина;
X7e представляет собой аминокислоту, выбранную из серина и аспарагиновой кислоты.
В одном из вариантов осуществления CDR3 легкой цепи (далее CDRL3) антитела против пропердина или его антигенсвязывающей части по настоящему изобретению имеет аминокислотную последовательность X1f-X2f-X3f-X4f-X5f-X6f-X7f-X8f-X9f-X10f-X11f
X1f представляет собой аминокислоту, выбранную из гистидина, глутамина, триптофана, аланина, глицина и метионина;
X2f представляет собой аминокислоту, выбранную из глутамина, аланина и треонина;
X3f представляет собой аминокислоту, выбранную из тирозина, глицина, лейцина, аргинина, триптофана и глутамина;
X4f представляет собой аминокислоту, выбранную из лейцина, аспарагина, треонина, тирозина, глутамина и аспарагиновой кислоты;
X5f представляет собой аминокислоту, выбранную из серина, треонина, гистидина, аланина и глицина;
X6f представляет собой аминокислоту, выбранную из серина, лейцина, фенилаланина, треонина, изолейцина и тирозина;
X7f представляет собой аминокислоту, выбранную из тирозина, пролина и лейцина;
X8f представляет собой аминокислоту, выбранную из треонина, пролина, тирозина, триптофана и аргинина;
X9f представляет собой аминокислоту, выбранную из треонина и глутаминовой кислоты, или отсутствует;
X10f представляет собой аминокислоту, выбранную из аланина и лейцина, или не содержит аминокислоты;
X11f представляет собой аминокислоту валин или не содержит аминокислоты.
В одном из вариантов осуществления CDRH1, CDRH2, CDRH3, CDRL1, CDRL2 и CDRL3 антитела против пропердина или его антигенсвязывающей части по настоящему изобретению выбирают из аминокислотных последовательностей, как указано ниже в таблице 2.
Соответственно, в другом варианте осуществления настоящее изобретение относится к антителу против пропердина или его антигенсвязывающей части, содержащему: вариабельную область тяжелой цепи, которая содержит последовательности CDRH1, CDRH2 и CDRH3; и вариабельную область легкой цепи, которая содержит последовательности CDRL1, CDRL2 и CDRL3, где:
(a) CDRH3 вариабельной области тяжелой цепи содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 12, 13, 14, 15, 16, 17 и 18, и их консервативных модификаций; (b) CDRL3 вариабельной области легкой цепи содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 35, 36, 37, 38, 39, 40, 41, 42 и 43, и их консервативных модификаций.
В другом предпочтительном варианте осуществления CDRH2 вариабельной области тяжелой цепи содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 6, 7, 8, 9, 10 и 11, и ее консервативных модификаций; и CDRL2 вариабельной области легкой цепи, содержащей аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 30, 31, 32, 33 и 34 и их консервативных модификаций.
В другом предпочтительном варианте осуществления CDRH1 вариабельной области тяжелой цепи содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 1, 2, 3, 4 и 5, и ее консервативных модификаций; и CDRL1 вариабельной области легкой цепи содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 19, 20, 21, 22, 23, 24, 25, 26, 27, 28 и 29 и их консервативных модификаций.
В другом варианте осуществления настоящее изобретение относится к антителу или его антигенсвязывающей части, содержащему:
(a) CDRH1 вариабельной области тяжелой цепи, содержащей аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 1, 2, 3, 4 и 5;
(b) CDRH2 вариабельной области тяжелой цепи, содержащей аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 6, 7, 8, 9, 10 и 11;
(c) CDRH3 вариабельной области тяжелой цепи, содержащей аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 12, 13, 14, 15, 16, 17 и 18;
(d) CDRL1 вариабельной области легкой цепи, содержащей аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 19, 20, 21, 22, 23, 24, 25, 26, 27, 28 и 29;
(e) CDRL2 вариабельной области легкой цепи, содержащей аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 30, 31, 32, 33 и 34; и
(f) CDRL3 вариабельной области легкой цепи, содержащей аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 35, 36, 37, 38, 39, 40, 41, 42 и 43; где антитело специфически связывается с пропердином, предпочтительно с пропердином человека.
В одном из вариантов осуществления HCVR и LCVR антитела против пропердина или его антигенсвязывающей части по настоящему изобретению выбирают из аминокислотных последовательностей, как указано в таблице 3 ниже.
No.
В одном из вариантов осуществления вариабельная область тяжелой цепи антитела против пропердина или его антигенсвязывающей части по настоящему изобретению содержит CDRH1, CDRH2 и CDRH3, содержащие аминокислотные последовательности, выбранные из приведенной ниже таблицы 4.
В одном из вариантов осуществления вариабельная область легкой цепи антитела против пропердина или его антигенсвязывающей части по настоящему изобретению имеет комбинацию аминокислотных последовательностей CDRL1, CDRL2 и CDRL3, выбранных из приведенной ниже таблицы 5.
LCVR
Соответственно, настоящее изобретение относится к антителу против пропердина или его антигенсвязывающей части, содержащему вариабельную область тяжелой цепи и вариабельную область легкой цепи, где:
(a) вариабельная область тяжелой цепи содержит аминокислотную последовательность, которая по меньшей мере на 80% гомологична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54 и 55;
(b) вариабельная область легкой цепи содержит аминокислотную последовательность, которая по меньшей мере на 80% гомологична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82 и 83.
Предпочтительно настоящее изобретение относится к антителу против пропердина или его антигенсвязывающей части, содержащему вариабельную область тяжелой цепи и вариабельную область легкой цепи, где:
(a) вариабельная область тяжелой цепи содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54 и 55;
(b) вариабельная область легкой цепи содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82 и 83.
Предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(a) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 1;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 7;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 13;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 20;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 31; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 37.
Другая предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(a) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 1;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 7;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 13;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 21;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 32; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 38.
Другая предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(a) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 1;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 7;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 13;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 21;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 32; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 36.
Другая предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(a) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 2;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 8;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 14;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 20;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 31; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 36.
Другая предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(a) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 3;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 9;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 15;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 22;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 33; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 39.
Другая предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 4;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 10;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 16;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 23;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 34; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 40.
Другая предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 5;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 11;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 18;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 29;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 30; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 41.
Другая предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 5;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 11;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 17;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 29;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 30; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 41.
Другая предпочтительная комбинация CDR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(a) CDRH1 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 1;
(b) CDRH2 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 6;
(c) CDRH3 вариабельной области тяжелой цепи, содержащую SEQ ID NO: 12;
(d) CDRL1 вариабельной области легкой цепи, содержащую SEQ ID NO: 19;
(e) CDRL2 вариабельной области легкой цепи, содержащую SEQ ID NO: 30; и
(f) CDRL3 вариабельной области легкой цепи, содержащую SEQ ID NO: 35.
Предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 51; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 63.
Другая предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 52; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 64.
Еще одна предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 53; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 65.
Другая предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 54; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 66.
Другая предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 45; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 57.
Еще одна предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 45; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 58.
Другая предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 45; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 59.
Еще одна предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 57.
Другая предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 47; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 60.
Другая предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 48; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 61.
Другая предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 55; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 79.
Еще одна предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 55; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 56.
Еще одна предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 50; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 62.
Другая предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 44; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 56.
Еще одна предпочтительная комбинация HCVR и LCVR антитела против пропердина или его антигенсвязывающей части согласно настоящему изобретению содержит:
(а) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 49; и
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 62.
Антитела по настоящему изобретению могут быть полноразмерными (например, антитело IgG1, lgG4 или IgG2) или могут содержать только антигенсвязывающую часть (например, фрагмент Fab, F(ab')2 или scFv), и, необязательно, могут быть модифицированными для воздействия на функциональность, например, для устранения остаточных эффекторных функций, таких как активность ADCC и CDC. Человеческие антитела могут существовать в двух формах, которые связаны с шарнирной гетерогенностью. В одной форме антитело содержит стабильную четырехцепочечную конструкцию приблизительно 150-160 кДа, в которой димеры удерживаются вместе межцепочечной дисульфидной связью тяжелой цепи. Во второй форме димеры не связаны межцепочечными дисульфидными связями, и образуется молекула примерно 75-80 кДа, состоящая из ковалентно связанных легкой и тяжелой цепей (полуантитело). Более поздние формы было чрезвычайно трудно отделить даже после аффинной очистки. Частота появления второй формы у различных интактных изотипов IgG обусловлена, помимо прочего, структурными различиями, связанными с изотипом шарнирной области антитела. Замена одной аминокислоты в шарнирной области IgG4 человека может значительно снизить появление второй формы (6) до уровня, обычно наблюдаемого при использовании шарнира IgG1 человека. Полноразмерные антитела, содержащие CDR или вариабельные области по настоящему изобретению, дополнительно содержат указанную единственную аминокислотную замену (т.е. S228P), когда они получены в форме IgG4.
В дополнительном варианте осуществления антитело или его антигенсвязывающая часть, нацеленные на антиген пропердина по настоящему изобретению, являются мышиными, химерными, человеческими или гуманизированными по своей природе, предпочтительно химерными или человеческими или гуманизированными по своей природе, более предпочтительно гуманизированными по своей природе.
Предпочтительно антитела, предпочтительно моноклональные антитела по настоящему изобретению, включают «гуманизированные» формы нечеловеческих (например, мышиных) антител. Гуманизированные или mAb с трансплантированными CDR особенно полезны в качестве терапевтических агентов для людей, поскольку они не выводятся из кровотока так же быстро, как мышиные антитела, и обычно не вызывают неблагоприятной иммунной реакции. Как правило, гуманизированное антитело имеет один или более аминокислотных остатков, введенных в него из источника, отличного от человека. Способы получения гуманизированных антител обычно хорошо известны в данной области. Например, гуманизация может быть осуществлена по существу по методу Winter и соавт. (7, 8, 9 и 10) путем замены каркасов грызунов или последовательностей CDR на соответствующие последовательности человеческого антитела. В некоторых вариантах осуществления гуманизированные формы нечеловеческих (например, мышиных) антител представляют собой человеческие антитела (реципиентное антитело), в которых аминокислотные остатки области CDR нечеловеческого антитела (например, мышиного, крысиного, кроличьего или антитела низшего примата), обладающие желаемой специфичностью, аффинностью и связывающей способностью, трансплантируют на каркас человеческого антитела.
В некоторых случаях один или более аминокислотных остатков каркасной области человеческого иммуноглобулина также заменяются соответствующими аминокислотными остатками нечеловеческого антитела (так называемые «обратные мутации»). Кроме того, библиотеки фагового дисплея можно использовать для изменения аминокислот в выбранных положениях в последовательности антитела. На свойства гуманизированного антитела также влияет выбор человеческого каркаса. Кроме того, гуманизированные и химеризованные антитела могут быть модифицированы с целью включения остатков, которых нет в реципиентном антителе или в донорном антителе, для дальнейшего улучшения свойств антитела, таких как, например, аффинность или эффекторная функция.
В другом аспекте антитело против пропердина по настоящему изобретению характеризуется повышенным связыванием FcRn и увеличенным периодом полужизни с модифицированной или сниженной активностью ADCC и/или CDC или без нее. Антитело против пропердина по настоящему изобретению может быть введено пациенту в уменьшенной дозе и с лучшим режимом дозирования из-за увеличенного времени полужизни по сравнению с известными антителами против пропердина. В одном из вариантов осуществления антитело против пропердина по настоящему изобретению имеет аминокислотные последовательности константной области IgG4 с мутацией P329G и/или M428L и N434S. Константная область антитела против пропердина с указанными всеми тремя мутациями в константной области IgG4 обозначается здесь как IgG4 (GLS). В одном из аспектов антитело против пропердина по настоящему изобретению имеет пониженную активность ADCP или не имеет ее.
В одном из вариантов осуществления антитело против пропердина по настоящему изобретению представляет собой моноклональное антитело, или биспецифическое антитело, или поликлональное антитело, предпочтительно моноклональное антитело.
Получение антител
Антитела по настоящему изобретению генерируют у мышей с использованием стандартных способов, хорошо известных в данной области. Моноклональные антитела по настоящему изобретению превращают в гуманизированную версию для терапевтического применения. Линии гибридомных клеток, обсуждаемые в настоящем документе, могут быть легко получены специалистами в данной области с учетом приведенных здесь указаний. Разработку библиотеки фагового дисплея scFv против пропердина проводили после амплификации генов вариабельной области тяжелой цепи (VH) и вариабельной области легкой цепи (VL) из РНК селезенки иммунизированных мышей. И VH, и VL соединяли пептидным линкером и клонировали в векторе фагового дисплея, как описано в настоящем документе. Выполняли пэннинг и скрининг на наличие специфических связующих пропердина.
Дальнейшие модификации в CDR и каркасной области
Настоящее изобретение охватывает антитела, имеющие одну или более мутаций в CDR и/или вариабельной области, описанных в настоящем документе, которые могут иметь аналогичные функциональные характеристики и биологическую активность, как описано для антител, представленных в настоящем документе. Эти мутации известны специалистам в данной области и входят в объем настоящего изобретения.
Дальнейшие модификации в константной области
Настоящее изобретение охватывает антитела, имеющие одну или более мутаций в шарнирной, СН2- или СН3-области, которые могут быть желательны, например, для улучшения периода полужизни антитела в кровотоке у пациента, для полной отмены иммунных эффекторных функций, для усиления эффекторных функций, и так далее. Эти мутации известны специалистам в данной области (11, 12).
Иммуноконъюгаты и биспецифические антитела
Также может быть разработан иммуноконъюгат, содержащий антитело по настоящему изобретению или его антигенсвязывающую часть, связанное с другим терапевтическим агентом, таким как цитотоксин или радиоактивный изотоп. Можно разработать биспецифическую молекулу, содержащую антитело или его антигенсвязывающую часть по настоящему изобретению, связанное со вторым функциональным фрагментом, имеющим другую специфичность связывания, чем у указанного антитела или его антигенсвязывающей части. В одном из вариантов осуществления второй функциональный фрагмент по настоящему изобретению может связываться с антигеном, выбранным из C3, C5, C5a, C5b, C3a, C3b, фактора B, фактора H и C1q. Способы получения биспецифических антител известны в данной области.
Молекулы нуклеиновой кислоты, кодирующие антитела против пропердина, векторы и клетки-хозяева
В одном варианте осуществления настоящее изобретение относится к молекулам нуклеиновых кислот, кодирующим антитела, или их антигенсвязывающие части, а также к экспрессирующим векторам, содержащим такие нуклеиновые кислоты, и к клеткам-хозяевам, содержащим такие экспрессирующие векторы. В настоящей заявке вектор pZRCIII используется для клонирования и экспрессии антител против пропердина по настоящему изобретению. Вектор pZRCIII описан в патентном документе WO 2012/046255A2. Клеткой-хозяином согласно настоящему изобретению является прокариотическая или эукариотическая клетка, предпочтительно клетка-хозяин представляет собой клетку E.coli или клетку млекопитающего, такую как клетка СНО.
Комбинация антитела против пропердина по настоящему изобретению с другими лекарственными средствами
Настоящее изобретение относится к комбинации, содержащей по меньшей мере два или более антитела или их антигенсвязывающую часть, где по меньшей мере одно антитело или его антигенсвязывающая часть представляет собой антитело против пропердина по настоящему изобретению. Комбинация по настоящему изобретению может содержать второе антитело или его антигенсвязывающую часть, выбранное из антитела против C3, антитела против C5, антитела против C5a, антитела против C5b, антитела против C3a, антитела против C3b, антитела против C3b, антитела против фактора В, экулизумаба, лампализумаба, равулизумаба, антитела против пропердина в комбинации с антителом против пропердина или его антигенсвязывающей частью по настоящему изобретению. В другом варианте осуществления настоящее изобретение относится к комбинации, содержащей антитело против пропердина или его антигенсвязывающую часть (части) и пептид, или к комбинации, содержащей антитело против пропердина или его антигенсвязывающую часть (части) и цитокин (предпочтительно интерлейкин).
Фармацевтические композиции
Можно разработать фармацевтическую композицию, содержащую одно или комбинацию моноклональных антител или их антигенсвязывающую часть(части) по настоящему изобретению, приготовленную вместе с фармацевтически приемлемым носителем. Такие композиции могут включать одно или комбинацию (например, двух или более разных) антител, или иммуноконъюгатов, или биспецифических молекул по изобретению. Например, фармацевтическая композиция по изобретению может содержать комбинацию антител (или иммуноконъюгатов, или биспецифических антител), которые связываются с разными эпитопами на антигене-мишени или с разными эпитопами на разных антигенах-мишенях, или которые обладают комплементарной активностью.
Терапевтические использования
Антитело против пропердина или его антигенсвязывающая часть или комбинация по настоящему изобретению, или биспецифические антитела, или иммуноконъюгаты по настоящему изобретению могут быть использованы в терапевтических способах лечения заболеваний, прямо или косвенно опосредованных компонентом альтернативного пути комплемента и/или фактором, генерируемым после активации альтернативного пути комплемента.
В одном варианте осуществления настоящего изобретения антитела можно использовать для ингибирования активации комплемента альтернативным путем in vivo у пациентов, включая людей, страдающих такими заболеваниями, как, но не ограничиваясь ими, гематологические заболевания, хронические заболевания почек, воспалительные заболевания глаз, различные виды онкологических заболеваний, аутоиммунные заболевания и воспаления.
В одном варианте осуществления настоящего изобретения антитела могут быть использованы для ингибирования активации комплемента через альтернативный путь in vivo у пациентов, включая людей, страдающих заболеваниями или нарушениями, такими как, но не ограничиваясь ими, атипичный гемолитико-уремический синдром, ТМА (ТА-ТМА), ассоциированная с трансплантацией гемопоэтических стволовых клеток (HSC); HELLP-синдром, связанный с беременностью (гемолиз, повышенный уровень ферментов печени, низкий уровень тромбоцитов); и ТМА, связанные с инфекцией или лекарственными средствами, атеросклероз, пароксизмальная ночная гемоглобинурия, ишемическое-реперфузионное (I/R) повреждение органов, возрастная макулярная дегенерация (AMD), географическая атрофия, ишемия-реперфузия после острого инфаркта миокарда, пурпурный нефрит Геноха-Шенлейна, геморрагический васкулит, ревматоидный артрит, артериит, аневризма, инсульт, кардиомиопатия, воспаление, связанное с сепсисом, воспаление, вызванное гемодиализом, гломерулопатии С3, геморрагический шок, размозжение, полиорганная недостаточность, гиповолемический шок и ишемия кишечника, отторжение трансплантата, кардиохирургия, чрескожная транслюминальная коронарная ангиопластика (PTCA), самопроизвольный аборт, повреждение нейронов, тяжелый острый респираторный синдром, такой как коронавирусная болезнь 2019 (COVID-19), ближневосточный респираторный синдром, вирусная пневмония, повреждение спинного мозга, тяжелая миастения, болезнь Хантингтона, боковой амиотрофический склероз, рассеянный склероз, синдром Гийена-Барре, болезнь Паркинсона, болезнь Альцгеймера, острый респираторный дистресс-синдром, астма, хроническая обструктивная болезнь легких, острое повреждение легких, связанное с переливанием крови, острое повреждение легких, болезнь Гудпасчера, инфаркт миокарда, постаортальное воспаление, трансплантация органов, пародонтоз, искусственное кровообращение, септический шок, отторжение трансплантата, ксенотрансплантация, ожоговая травма, системная красная волчанка, мембранозный нефрит, болезнь Бергера, псориаз, пемфигоид, дерматомиозит, антифосфолипидный синдром, воспалительное заболевание кишечника, гемодиализ, лейкоферез, плазмаферез, гепарин-индуцированная экстракорпоральная мембранная оксигенация с LDL преципитацией, экстракорпоральная мембранная оксигенация и макулярная дегенерация. In vivo ингибирование активации альтернативного пути комплемента достигается введением антитела пациенту.
Настоящее изобретение проиллюстрировано следующими неограничивающими примерами, которые никоим образом не следует интерпретировать как ограничивающие объем изобретения.
Примеры
Следующие примеры приведены для того, чтобы предоставить специалистам в данной области раскрытие и описание того, как осуществляются заявленные здесь способы и антитела. Они предназначены только для иллюстрации и не предназначены для ограничения объема раскрытия. Другие антитела по настоящему изобретению можно получить, используя способ, описанный в предоставленных примерах, с подходящими модификациями. Такие модификации хорошо известны специалистам в данной области.
Пример 1: Иммунизация мышей человеческим антигеном пропердин для получения связывающего агента
Для исследования иммунизации использовали четырех здоровых самок мышей BALB/c. Две из них оставляли в качестве плацебо-контроля, в которых для иммунизации использовали только PBS. Две другие мыши иммунизировали пропердином человека. Чтобы продемонстрировать, что антигенный препарат способен индуцировать антитела против пропердина у высокочувствительных видов, одного кролика использовали в качестве другого контроля для иммунизации тем же антигенным препаратом, чтобы отслеживать иммунные ответы на различных стадиях процессов иммунизации. Всех животных акклиматизировали в течение 2 дней в исследовательском центре для животных перед началом исследования иммунизации. Для иммунизации использовали коммерчески доступный белок пропердин человека (Quidel cat #A412). Белковую эмульсию готовили путем смешивания 400 мкг человеческого пропердина (в 400 мкл PBS) и 400 мкл полного адъюванта Фрейнда (CFA) в общем объеме 800 мкл в силиконизированном стеклянном флаконе с завинчивающейся крышкой на 5 мл и путем встряхивания смеси в течение 10-15 минут для приготовления эмульсии. В день 0 однократную дозу 100 мкг эмульгированного белка в объеме 200 мкл (0,5 мкг/мкл) вводили подкожно в четыре места на спине каждой мыши.
Одновременно в день 0 200 мкг белковой эмульсии в 400 мкл (0,5 мкг/мкл) вводили подкожно в 4 места на спине кролика.
Для последующих иммунизаций (бустеры) использовали неполный адъювант Фрейнда (IFA). Животным вводили четыре бустерные дозы с интервалом 15 дней после каждой иммунизации. Белковую эмульсию готовили путем смешивания 200 мкг пропердина (в 400 мкл PBS) и 400 мкл неполного адъюванта Фрейнда (IFA). Смесь встряхивали в силиконизированном стеклянном флаконе на 5 мл с завинчивающейся крышкой в течение 10-15 минут для приготовления эмульсии. Каждую бустерную дозу мышам вводили в виде разовой дозы 50 мкг белковой эмульсии в 200 мкл. Бустерную дозу кролику вводили со 100 мкг белка в 400 мкл. Состояние мышей и кроликов отслеживали ежедневно. Для проверки титров антител, генерируемых против антигена пропердина человека, кровь собирали у кроликов за 4-5 дней до каждой бустерной вакцины. Из крови готовили сыворотку и использовали в ИФА для определения антител, специфичных к пропердину человека. Мышей умерщвляли через 15 дней после 4-й бустерной дозы и собирали селезенки либо для получения гибридомы, либо для получения тотальной РНК для создания фаговой библиотеки.
Пример 2: Получение связывающих агентов против пропердина после иммунизации мышей
Генерация гибридом
После четвертой ревакцинации мышей умерщвляли, селезенки удаляли, нарезали на мелкие кусочки и пропускали через клеточное сито. Затем клетки ресуспендировали в холодной среде RPMI (Roswell Park Memorial Institute) 1640 с 10% FBS и центрифугировали при 300g в течение 5 минут. Осадок клеток ресуспендировали в RPMI 1640 с 1% FBS, фильтровали через шприцевой фильтр 50 мкМ (BD № 340603) и собирали центрифугированием.
Протокол на основе полиэтиленгликоля использовали для слияния гибридом, как описано ранее (13, 14). Суспензии отдельных клеток готовили из селезенки иммунизированных мышей, как описано выше, и использовали для слияния с клетками миеломы Sp2/0 (АТСС). Sp2/0 и клетки селезенки (соотношение 1:5) сливали с использованием полиэтиленгликоля (M.W. 1500, Sigma). После слияния клетки доводили до концентрации 0,5×106 клеток/мл в среде RPMI (Sigma), дополненной 10% фетальной бычьей сывороткой (Gibco) и 1x гипоксантин-аминоптерин-тимидиновой средой (HAT) (Sigma) для отбора гибридом. Двести микролитров этой клеточной суспензии добавляли в каждую лунку 96-луночного культурального планшета для создания минипулов. Примерно через десять дней супернатанты культур тестировали в ИФА на связывание с очищенным нативным пропердином человека. Минипулы, реагирующие с пропердином, дополнительно наращивали до 24-луночных и 6-луночных планшетов и Т-колб. Образцы антител против пропердина из культуральных супернатантов выбранных минипулов тестировали на их связывание с пропердином человека в ИФА.
На основе результатов реактивности пропердина человека в ИФА одиннадцать минипулов дополнительно включали в краткий список для проведения лимитирующего разведения отдельных клеток. Для ограничения разведения минипулы высевали при разведении 1 клетка/на лунку в 96-луночный культуральный планшет в среде RPMI с добавлением 10% эмбриональной бычьей сыворотки. Клеткам давали расти дальше и наращивали до 24-луночных и 6-луночных планшетов и Т-колб. Супернатанты клеточных культур снова тестировали на их связывание с пропердином человека в ИФА. 104 отобранных гибридомных клона выращивали в среде для продуцирования, не содержащей сыворотки (среда BD Cell mAb Medium, Quantum Yield; BD Bioscience), и культуральные супернатанты собирали для очистки антител с помощью аффинной хроматографии с Протеином А. Эти очищенные антитела-кандидаты дополнительно тестировали на их аффинность связывания в анализе на основе SPR. Четыре гибридомных клона против пропердина, обозначенные как 103B2 (VH SEQ ID 54; VL SEQ ID 66), 124F9 (VH SEQ ID 53; VL SEQ ID 65), 137D4 (VH SEQ ID 52; VL SEQ ID 64) и 149F8 (VH SEQ ID 51; VL SEQ ID 63) оказались лучшими из всей группы.
Было обнаружено, что все 4 клона демонстрируют хорошую аффинность в диапазоне нМ к пропердину человека (таблица 6). Гибридомные mAb против пропердина 103B2, 124F9, 137D4 и 149F8 вместе с другими связывающими антителами против пропердина, полученными из библиотеки фагового дисплея, были дополнительно гуманизированы и описаны в примере 6.
Создание библиотеки ScFv методом фагового дисплея
Для создания библиотеки scFv следовали протоколу иммунизации, как описано в примере 1. Селезенку от одной умерщвленной мыши собирали через 15 дней после 4-й бустерной инъекции и переносили непосредственно в 10 мл физиологического раствора в полипропиленовой пробирке объемом 50 мл. Затем селезенку взвешивали в центрифужной пробирке объемом 1,6 мл, обработанной диэтилпирокарбонатом (DEPC). Суммарную РНК выделяли тризольным методом следующим образом. Поддерживая температуру 4°С, селезенку предварительно измельчали хирургическим лезвием в чашке Петри до образования гомогенной суспензии клеток селезенки. Два мл тризола добавляли к 90-130 мг селезенки с последующим перемешиванием пипеткой, а затем встряхиванием. Процесс измельчения повторяли до образования гомогенной смеси клеток селезенки и тризола. После этого к гомогенату добавляли 200 мкл хлороформа, перемешивали и инкубировали при комнатной температуре в течение 5 минут. Затем гомогенат центрифугировали, отбирали супернатант и добавляли равное количество изопропилового спирта для осаждения РНК. Суспензию РНК снова центрифугировали и осадок промывали ледяным 70% этанолом. После трех промывок осадок, содержащий тотальную РНК, ресуспендировали в воде, обработанной DEPC, и оставляли на ночь для растворения при 4°C.
Затем мРНК выделяли из тотальной РНК в соответствии с инструкциями производителя (PolyATtract® mRNA Isolation Systems, кат.: Z5300, Promega). Выделенную мРНК использовали для получения кДНК с использованием системы синтеза первой цепи SuperScript™ III (кат. №: 18080051, Invitrogen) в соответствии с инструкциями производителя.
Амплификация VH и VL: наборы праймеров (набор праймеров для библиотеки IgG мыши, кат. №: F2010, Progen) использовали для амплификации вариабельных областей тяжелых и легких цепей из кДНК с использованием метода ПЦР в соответствии с инструкциями производителя. Амплифицированные фрагменты VH и VL анализировали на агарозном геле и очищали от геля с использованием набора для экстракции из геля QIAquick (кат. № 28706 Qiagen).
Затем очищенные VH и VL последовательно клонировали в фагмидный вектор один за другим (pSEX81, кат. №: PR3005, Progen). При первом лигировании фрагмент VH и вектор подвергали рестрикционному расщеплению NcoI и HindIII, очищали из геля и лигировали вместе. Продукт лигирования трансформировали в электрокомпетентные клетки TG1 (кат. №: 60502-1, Lucigen). Фрагмент VL и фрагмент VH, содержащие векторную ДНК, снова расщепляли рестрикционными ферментами MluI и NotI, очищали и лигировали вместе. Вектор, содержащий фрагменты VH и VL, трансформировали в электрокомпетентные клетки, как указано выше. Трансформированные клетки высевали на чашки с агаром 2xYT, содержащим ампициллин (100 мкг/мл). Полученную таким образом библиотеку трансформированных клеток соскребали с чашек с агаром и хранили в 50% глицерине при -80°С до дальнейшего использования.
Пример 3: получение фага scFv из библиотеки scFv и его очистка
В колбы, содержащие среду 2xYT с карбенициллином или ампициллином (100 мкл/мл), инокулировали клетки из глицеринового стока вышеописанной библиотеки в исходной концентрации 0,06 OD600. Культуры выращивали при 37°С при встряхивании со скоростью 250 об/мин до тех пор, пока они не достигли OD600 до ~0,4-0,6. Вспомогательные фаги, VCSM13 (Agilent, кат. № 200251) или M13KO7 (GE Healthcare, кат. № 27152401), затем добавляли к культуре при множественности заражения (MOI) 20 и инкубировали сначала без встряхивания при 37°C в течение 40 минут, а затем еще 40 минут инкубации при 37°C со встряхиванием. Затем в среду добавляли канамицин и культуру выращивали в течение ночи при 26°C при 150 об/мин. Ночную культуру фага центрифугировали при 4000 g и отбрасывали клеточный осадок. К супернатанту добавляли раствор ПЭГ (20%)/NaCl (2,5 М) в соотношении 1:5 для осаждения фагов. Ресуспендированный раствор инкубировали на льду в течение 20 минут с последующим центрифугированием при 14000g в течение 15 минут при 4°C. Супернатант удаляли, а осадок ресуспендировали в 1 мл стерильного PBS с 0,01% азида натрия. Фаги хранили при 4°С до дальнейшего использования.
Пример 4: Биопэннинг фагов, экспрессирующих scFv против пропердина
Библиотеку ScFv (1×1012 БОЕ), полученную в примере 3, подвергали скринингу на наличие связывающих агентов против пропердина. Первый раунд пэннинга антигена пропердина проводили с использованием иммунопробирок с иммобилизованным антигеном (Quidel, США), которые готовили путем инкубации их с раствором пропердина 5 мкг/мл в карбонатном буфере (0,1 М, рН 9,6) в течение ночи при 4°С. Иммунопробирки промывали 3 раза PBS, а затем инкубировали с фагами в PBS в течение 2 ч при 25°С при постоянном вращении. Пробирки промывали десять раз 4 мл PBS с tween 20 (0,1%), а затем 10 раз PBS. Связанные фаги элюировали с использованием глицин-HCl pH 2,1 (0,1 М). Элюированные фаги восстанавливали путем инфицирования клеток E. coli TG1, высевали и получали фаги, как описано в примере 3.
Второй и третий раунды пэннинга проводили на покрытых пропердиновым антигеном иммунопробирках таким образом, что выходные фаги из первого раунда пэннинга (1×1011 КОЕ) и второго (1×1010 КОЕ) раунда пэннинга использовали в качестве входных фагов для второго и третьего раундов пэннинга, соответственно. Фаги после третьего раунда пэннинга инфицировали в клетках TG1, как упоминалось ранее, и выделяли фагмидную ДНК для клонирования scFv в подходящем экспрессирующем векторе.
Пример 5: Скрининг отдельных клонов scFv в качестве растворимых белков фрагментов антител
Гены scFv из обогащенной библиотеки, полученной после 3 раундов пэннинга, клонировали в экспрессирующий вектор pOPE101 (Progen, Германия) таким образом, чтобы отдельные клоны scFv могли быть получены в виде HIS-меченых слитых продуктов. И векторную ДНК (pOPE101), и фагмидную ДНК расщепляли рестрикционными ферментами (NcoI и NotI) для выделения вектора и гена scFv, соответственно. Как рестрикционный вектор, так и ген scFv лигировали и трансформировали в электрокомпетентные клетки TG1. Трансформированные клетки высевали на чашки с агаром 2xYT, содержащим антибиотик карбенициллин. Отдельные клоны собирали с чашек с агаром 2xYT и культивировали в пробирке на 15 мл с 5 мл среды 2xYT, содержащей карбенициллин или ампициллин (100 мкл/мл), в течение ночи при 32°C при 200 об/мин. На следующий день культуры пересевали на свежую среду 2xYT, содержащую карбенициллин (100 мкг/мл) и глюкозу (0,1%) в объемном соотношении 1:200. Культуры выращивали до тех пор, пока OD600 не достигала ~0,6-0,8. После этого к культуре добавляли 1 мМ изопропил-β-D-1-тиогалактопиранозида (IPTG) и выращивали в течение ночи при 30°C при 200 об/мин. Ночную культуру центрифугировали при 10800 g в течение 15 минут и удаляли супернатант. Осадок клеток ресуспендировали в 1/20 объема исходной культуры в буфере (30 мМ Tris-Cl, pH 7, 20% сахарозы и 1 мМ EDTA) и инкубировали на льду в течение 30 минут. Ресуспендированный осадок центрифугировали при 10800 g в течение 15 минут и собирали супернатант в виде периплазматической фракции, содержащей растворимый scFv, меченный His. Эту периплазматическую фракцию использовали в иммуноанализах для изучения связывания пропердина.
Антиген пропердина наносили на полистирольные планшеты (100 нг/100 мкл/лунку) в течение ночи при 4°С. После двукратного промывания планшета добавляли периплазматическую фракцию (1/10 по объему) и инкубировали в течение 1 часа при 25°С. Планшеты промывали 4 раза PBST с последующим добавлением 100 мкл (1:5000 об./об.) мышиного антитела против HIS (GE Healthcare, кат. № 27471001) в течение 1 ч. Планшеты снова промывали, как описано ранее, и HRP-конъюгированное козье антимышиное антитело (Santa Cruz Biotech, кат. № SC-2031) добавляли (1:5000) в планшет и инкубировали еще 1 час при 25°C. В конце этой инкубации после четырех промывок добавляли субстрат 3,3',5,5'-тетраметилбензидин (ТМВ). Поглощение при 450 нм измеряли с помощью TECAN INFINITE® M1000pro после 5 минут проявления цвета. Отдельный клон считался положительным, если сигнал OD по меньшей мере в три раза превышал фон (пустые лунки) (т.е. отношение сигнал/фон ≥3).
Пример 6: Гуманизация мышиных моноклональных антител против пропердина, полученных из гибридомы и библиотеки фагового дисплея
Мышиные связывающие агенты гуманизировали методом трансплантации CDR, при котором каркасные области (FR1-4) из мышиных антител были заменены FR-областями человеческих антител. Перед трансплантацией FR из антител человека идентифицировали на основании наиболее близкого совпадения последовательности мышиного белка с антителом зародышевой линии человека. В настоящем изобретении VH и VL мышиных связывающих агентов (SEQ ID No. 49 и 62) гуманизировали, а затем рекомбинировали с константными областями IgG4 с получением полноразмерного гуманизированного антитела. После этого несколько аминокислотных остатков в FR мутировали обратно в исходные аминокислоты, присутствующие в мышиной последовательности, для восстановления их функциональной активности (SEQ ID 55 и 56). (15, 16)
Аналогично, VH (SEQ ID No 50, 51, 52, 53 и 54) и VL (SEQ ID No 63, 64, 65 и 66) других мышиных антител гуманизировали путем трансплантации CDR с использованием их наиболее подходящей матрицы человеческих FR в базе данных зародышевой линии человека для получения гуманизированных VH (SEQ ID No 44, 45, 46, 47 и 48) и VL (SEQ ID No 57, 58, 60 и 61).
Пример 7: Конструирование векторов двойной сборки pZRCIII hyg анти-пропердин IgG4
Химически синтезированные гены, включая три гена легкой каппа-цепи [P13 (SEQ ID No. 84); P14LC (SEQ ID No. 85); P15 LC (SEQ ID No. 86)]; и один ген легкой цепи лямбда [E12 LC (SEQ ID No. 87)] с выступающими концами XhoI и KpnI и пятью вариабельными областями тяжелых цепей [P12HC (SEQ ID No. 88), P13HC (SEQ ID No. 89), P14HC (SEQ ID No. 90), P15HC (SEQ ID No. 91) и E12HC (SEQ ID No. 92)] с выступающими концами SalI и ApaI, клонированными в векторах pMA/pMK, были получены от Geneart, Германия.
Все четыре гена легких цепей (SEQ ID 84, 85, 86 и 87) выделяли из этих конструкций после расщепления XhoI и KpnI. Клонирующий вектор pZRCIIIHyg-IgG4 расщепляли XhoI и KpnI, а линеаризованный вектор лигировали с расщепленными генами LC по отдельности. Вектор pZRCIII получали, как описано в патентном документе WO 2012/046255A2, с константной областью IgG4. Продукт лигирования трансформировали в E. coli Top10F', и трансформанты оценивали на основе устойчивости к антибиотикам. Клоны анализировали рестрикционным расщеплением и секвенированием ДНК по методу Сэнгера. Эти промежуточные векторы, содержащие гены легкой цепи в транскрипционной сборке № 1, были названы pZRCIII Hyg P13LC, pZRCIII Hyg P14LC, pZRCIII Hyg P15LC и pZRCIII Hyg E12LC.
Плазмидную ДНК, полученную из промежуточных LC-векторов pZRCIII Hyg P13LC, pZRCIII Hyg P14LC, pZRCIII Hyg P15LC и pZRCIII Hyg E12LC, расщепляли SalI и ApaI, чтобы сделать возможным клонирование вариабельной области тяжелой цепи в рамке с константной областью IgG4 (SEQ ID No. 93) уже присутствующей в векторах. Вариабельные области тяжелой цепи, содержащие плазмидную ДНК pMA/pMK, полученную от Geneart, расщепляли SalI и ApaI. Эти расщепленные вариабельные области тяжелой цепи лигировали с промежуточными векторами, расщепленными SalI и ApaI, в следующих комбинациях: P13 LC (SEQ ID 84) с P12 HC (SEQ ID 88); P13 LC (SEQ ID 84) с P13 HC (SEQ ID 89); P14LC (SEQ ID 85) с P14HC (SEQ ID 90); P15 LC (SEQ ID 86) с P15 HC (SEQ ID 91) и E12 LC (SEQ ID 87) с E12 HC (SEQ ID 92). Продукт лигирования трансформировали в E.coli Top10F', и трансформанты оценивали на основе устойчивости к канамицину. Клоны были подтверждены на основе рестрикционного расщепления и секвенирования по Сэнгеру и были названы pZRCIII Hyg P13LC-P12HC (соответствующая аминокислотная последовательность VH и VL: SEQ ID No. 45 и 57, соответственно; обозначено здесь как P12), pZRCIII Hyg P13LC-P13HC (соответствующая аминокислотная последовательность VH и VL: SEQ ID No. 46 и 57, соответственно; обозначено здесь как P13), pZRCIII Hyg P14LC-P14HC (соответствующая аминокислотная последовательность VH и VL: SEQ ID No. 47 и 60, соответственно; обозначено здесь как P14), pZRCIII Hyg P15LC-P15HC (соответствующая аминокислотная последовательность VH и VL: SEQ ID No. 48 и 61, соответственно; обозначено здесь как P15) и pZRCIII Hyg E12LC-E12HC (соответствующая аминокислотная последовательность VH и VL: SEQ ID No. 44 и 56, соответственно; обозначено здесь как P11).
Пример 8: Получение двойного вектора с модифицированной константной областью IgG4 (GLS)
Предполагалось, что эффекторные функции ADCC или CDC не требуются для mAb против пропердина. Чтобы иметь наименьшую из этих эффекторных функций, три мутации, а именно P329G, M428L и N434S, включали в конструкции mAb против пропердина, полученные в описанных выше примерах. mAb-кандидаты, которые содержат все три упомянутые мутации, встроенные в константную область IgG4, были названы IgG4 (GLS). Константную область векторной конструкции pZRCIII hyg P15LC-P15HC, как описано в приведенном выше примере, заменяли на IgG4 (GLS) (SEQ ID No.94). Для получения фрагмента константной области IgG4 (GLS) (SEQ ID NO.96) три мутации, а именно P329G, M428L и N434S, включали в константную область IgG4 с помощью мутагенной ПЦР. Этот очищенный ПЦР-фрагмент длиной ~976 пар оснований (SEQ ID No.94) с выступающим концом ApaI на 5'-конце и выступающим концом NotI на 3'-конце и pZRCIII Hyg P15 LC-P15HC расщепляли ферментами рестрикции ApaI и NotI. Расщепление вектора с помощью ApaI и NotI высвобождает константную область IgG4 размером ~976 п.н. и более крупный фрагмент размером ~12174 п.о., содержащий остальные элементы вектора. Фрагмент ~12174 п.о. экстрагировали из геля и очищали. Подобным образом расщепленный ПЦР-продукт очищали и лигировали с очищенным фрагментом вектора. Продукт лигирования трансформировали в E. coli Top10F'. Трансформанты оценивали на основе устойчивости к канамицину. Продукт лигирования трансформировали в E. coli Top10F'. Трансформанты оценивали на основе устойчивости к канамицину. Клоны были подтверждены рестрикционным расщеплением и секвенированием по Сэнгеру и названы pZRCIII Hyg P15LC-P15HC (IgG4 GLS) [соответствует аминокислотной последовательности VH и VL: SEQ ID No. 48 и 61 соответственно; соответствующая аминокислотная последовательность Fc-области: SEQ ID NO.96; обозначено здесь как P15 (GLS)].
Пример 9: Получение клеточных линий CHO-S, экспрессирующих гуманизированные антитела против пропердина (векторы IgG4)
В этом примере описано получение трансфецированных пулов, экспрессирующих полноразмерные гуманизированные антитела против пропердина. Для трансфекции использовали векторные конструкции, указанные ниже.
Конструкции векторов млекопитающих для IgG4
pZRCIII Hyg P13LC-P12HC, pZRCIII Hyg P13LC-P13HC, pZRCIII Hyg P14LC-P14HC, pZRCIII Hyg P15LC-P15HC, pZRCIII Hyg E12LC-E12HC и pZRCIII Hyg P15LC-P15HC (IgG4 GLS). Плазмиды перед трансфекцией линеаризовали рестрикционным ферментом AscI. Клетки яичника китайского хомячка являются одним из подходящих хозяев для экспрессии моноклональных антител. Клетки Freestyle CHO-S (Invitrogen) использовали в качестве хозяина для трансфекции. Клетки (0,5 млн/мл) высевали примерно за 24 часа до трансфекции, чтобы получить клетки в экспоненциальной фазе. Трансфекцию проводили методом электропорации с использованием системы Neon Transfection (Invitrogen) в соответствии с инструкциями производителя. После трансфекции клетки высевали в 24-луночные планшеты для культивирования клеток, содержащие 1 мл предварительно нагретой среды ProCHO5 без сыворотки (Lonza, Швейцария). Клетки инкубировали в увлажняемом инкубаторе при 37°С в присутствии 5% СО2. Эти трансфицированные пулы отбирали в присутствии 600 мкг/мл гигромицина (Invitrogen) в среде ProCHO5 в 24-луночном культуральном планшете. В процессе отбора количества клеток во всех пулах регулярно отслеживали, и осуществлялся регулярную замену среды. После завершения отбора клетки дополнительно наращивали в культуральных планшетах, Т-колбах и пробирках для культивирования (TPP).
Для всех отобранных пулов во встряхиваемых колбах (Corning) проводили периодическое культивирование с подпиткой для продуцирования рекомбинантного белка всех антител-кандидатов против пропердина. Клетки высевали с плотностью 0,3×106 клеток/мл в среду для продуцирования Power CHO-2 CD от Lonza. Колбы инкубировали во увлажняемом шейкере Kuhner при температуре 37°C, уровне СО2 5% и скорости встряхивания 110 об/мин. Фиксированный ежедневный режим подпитки соблюдали во время культивирования для всех пулов с использованием подпитки определенного химического состава от Hyclone, GE. Подпитку начинали через 72 часа культивирования и продолжали до момента сбора.
Супернатанты культуры собирали для очистки антител с помощью аффинной хроматографии с Протеином А. Эти очищенные антитела-кандидаты дополнительно тестировали in vitro.
Пример 10: Определение кинетических констант скорости связывания кандидатов против пропердина с пропердином с использованием метода поверхностного плазмонного резонанса
Кинетические константы скорости связывания антител-кандидатов против пропердина с нативным очищенным пропердином человека (Quidel; № по каталогу: A412) определяли измерением на основе поверхностного плазмонного резонанса с использованием ProteOn XPR36 (Bio-Rad). Все измерения связывания проводили в PBS (pH 7,4, 0,005% поверхностно-активного вещества P20) при 25°C. Чтобы измерить константы скорости связывания пропердина с mAb, человеческий пропердин иммобилизовали на сенсорном чипе GLC, используя химическую реакцию сочетания по аминогруппе. Готовили пять разведений аффинно очищенных mAb и вводили в проточную кювету со скоростью потока 100 мкл/мин. Данные в виде сенсограмм были анализировали с использованием программ подбора данных, интегрированных в систему ProteOn. Аффинность связывания в единицах значения KD представлена для протестированных антител в таблице 7.
Пример 11: Определение кинетических констант скорости антител против пропердина с рекомбинантным человеческим неонатальным Fc-рецептором (rhFcRn)
Кинетические константы для связывания антител с рекомбинантным неонатальным Fc-рецептором человека (hFcRn) определяли измерением на основе поверхностного плазмонного резонанса с использованием ProteOn XPR36 (Bio-Rad). Рекомбинантный рецептор rhFcRn (Sino Biologics) иммобилизовали на чипе GLC в соответствии с инструкциями производителя. Для измерения константы скорости ассоциации (ka) и константы скорости диссоциации (kd) готовили пять разведений аффинно очищенных антител и вводили их со скоростью потока 100 мкл/мин со временем ассоциации 180 с и временем диссоциации 600 с. Реакции проводили в PBS (рН 6, 0,005% ПАВ Р20) при 25°С. После каждого запуска образца поверхность чипа регенерировали с помощью PBS, pH 7,4. Данные в виде сенсограмм анализировали с помощью программ подбора данных в системе ProteOn. Кинетические константы связывания rhFcRn с антителом Р15 и антителом Р15 (GLS) показаны в таблице 8. Было обнаружено, что P15 (GLS) имеет более высокую аффинность (KD) к rhFcRn.
Пример 12: Определение связывания мышиного mAb против пропердина с нативным пропердином человека с помощью ИФА
Лунки 96-луночного микропланшетов F16 Maxisorp Immuno module (Nunc) покрывали путем добавления 100 мкл очищенного человеческого фактора P (Quidel) в концентрации 250 нг/мл в течение ночи в увлажняемой камере при 4°C. После удаления раствора покрытия путем встряхивания планшета в каждую лунку на один час добавляли 300 мкл блокирующего раствора - 5% обезжиренного молока в PBST (PBS, содержащий 0,05% Tween 20) для блокирования неспецифических участков. Через час лунки промывали промывочным буфером (0,5% обезжиренного молока в PBST). Пятьдесят микролитров культуральных супернатантов из каждой лунки для слияния собирали и разбавляли разбавителем для анализа ИФА (0,5% обезжиренного молока в PBS). Через час инкубации лунки промывали промывочным буфером. Связанные мышиные антитела затем определяли реакцией с конъюгированным с пероксидазой хрена (HRP) козьим антителом против мышиного IgG (Santa Cruz Biotechnology, Inc) в разведении 1:5000, приготовленном в разбавителе для анализа. Затем следовали 4-кратные промывки PBST. Затем в лунки добавляли раствор субстрата пероксидазы, содержащий 0,1% 3,3,5,5-тетраметилбензидина (Sigma) и 0,0003% перекиси водорода (Sigma) для проявления окраски на 30 минут. Реакцию останавливали добавлением 100 мкл 1М H2SO4. OD реакционной смеси при 450 нм считывали с помощью ИФА-ридера (Tecan Lifesciences).
Пример 13: Определение связывания гуманизированных моноклональных антител против пропердина с нативным пропердином человека с помощью ИФА
После гуманизации гены антител синтезировали, трансфецировали, экспрессировали и очищали, как описано ранее в примерах 7 и 9. Белки-кандидаты очищенного антитела, а именно Р13, Р14 и Р15, тестировали на их специфичность связывания с антигеном пропердина человека. Вкратце, для тестирования связывания антител против пропердина с нативным пропердином человека планшеты покрывали антигеном пропердина человека (Quidel Cat No. A412) в концентрации 50 нг/50 мкл/лунку в карбонатном буфере (0,1 М, рН 9,6). Планшеты инкубировали в течение ночи при 4°C, затем промывали и блокировали 5% обезжиренным молоком (1x PBS, pH-7,4). Заблокированные планшеты промывали 1x PBS, затем в лунки, покрытые антигеном, добавляли очищенные mAb (0,5-1 мкг/100 мкл на лунку) и инкубировали в течение 1 часа при 37°C при встряхивании. Планшеты трижды промывали PBST (0,1% Tween 20 в 1x PBS, pH 7,4). Конъюгированные с пероксидазой хрена (HRP) козьи антитела против человеческого IgG (SantaCruz, кат. № sc2453) добавляли в каждую лунку в разведении 1:10000 (100 мкл/лунка) с последующей инкубацией в течение 1 часа при 37°C при встряхивании. Планшеты промывали PBST (0,1% Tween 20 в 1x PBS, pH 7,4) с последующими 2 промывками 1x PBS. Затем в лунки добавляли о-фенилендиамин дигидрохлорид (OPD)/H2O2 (100 мкл/лунку) с последующей остановкой реакции через 15 минут добавлением 100 мкл 1 Н H2SO4 и измерением планшета при 450 нм. Клоны считались положительными (Фигура 1) в отношении связывания пропердина, если отношение сигнала к фону превышало 3 раза. Положительные клоны тестировали на их аффинность в SPR (поверхностный плазмонный резонанс) и сравнивали функциональную активность в гемолитическом анализе.
Пример 14: Гемолитический анализ включенных в окончательный список кандидатов против пропердина для определения функциональной активности отдельных mAb
Антитела-кандидаты против пропердина исследовали на их способность ингибировать альтернативный путь комплемента с использованием анализа лизиса эритроцитов кролика, где классический путь комплемента был заблокирован с использованием EGTA, а лизис эритроцитов кролика осуществлялся только альтернативным путем в NHS с образование MAC на поверхности эритроцитов кролика (17). Ожидается, что желаемые антитела-кандидаты против пропердина будут ингибировать этот альтернативный путь в различной степени в зависимости от их аффинности к пропердину и их сайта связывания. Таким образом, ожидается, что антитела-кандидаты против пропердина, разработанные в соответствии с настоящим изобретением, будут предотвращать лизис эритроцитов кролика в присутствии человеческой сыворотки. Процентное ингибирование гемолиза при заданном разведении/концентрации антитела для данного моноклонального антитела рассчитывали для каждого антитела и при всех испытанных разведениях и сравнивали между отдельными кандидатами.
Свежие эритроциты кролика собирали в растворе Alsever и промывали 5 раз буфером для анализа (150 мМ NaCl, 20 мМ HEPES, 25 мМ MgCl2 и 20 мМ EGTA). Промытые эритроциты ресуспендировали в буфере для анализа в необходимой концентрации. Различные концентрации каждого из образцов антител против пропердина (гуманизированные антитела против пропердина - NBE003-3, P11, P13, P14, P15 и мышиные антитела против пропердина - 124F9, 149F8, 103B20) инкубировали с сывороткой человека (25%) в течение 30 минут при 37°С. Затем их добавляли к фиксированному количеству кроличьих эритроцитов и инкубировали еще в течение 50 минут при 37°С. Обработанные клетки затем центрифугировали и в супернатантах измеряли высвободившийся гемоглобин путем измерения поглощения при длине волны 405/595. Процентное снижение высвобождаемого гемоглобина в присутствии антител против пропердина по настоящему изобретению или отрицательного контроля антител (IgG2) рассчитывали по отношению к лизису, полученному путем инкубации эритроцитов кролика с сывороткой человека без какого-либо тестируемого антитела. Этот лизис принимали за 100%. Как показано на фигурах 2a (для гуманизированных моноклональных антител против пропердина) и 2b (мышиных моноклональных антител против пропердина), все протестированные моноклональные антитела против пропердина ингибировали опосредованный сывороткой человека гемолиз эритроцитов кролика дозозависимым образом. Эти результаты показывают, что моноклональные антитела против пропердина, полученные в соответствии с настоящим изобретением, могут значительно ингибировать альтернативный путь комплемента.
Пример 15: Перекрестно-реактивный гемолитический анализ включенных в короткий список антител-кандидатов против пропердина с обезьяньей сывороткой
Антитела-кандидаты против пропердина исследовали на их способность перекрестной реактивности с пропердином обезьяны путем блокирования активности комплемента в сыворотке обезьяны в анализе лизиса эритроцитов кролика. Функциональный анализ выполняли по существу так же, как описано в примере 14, с использованием обезьяньей сыворотки вместо NHS. Ожидается, что mAb против пропердина по настоящему изобретению, обладающие способностью перекрестной реактивности с пропердином обезьяны, будут предотвращать лизис эритроцитов кролика путем блокирования образования MAC, опосредованного альтернативным путем комплемента.
Свежие эритроциты кролика собирали в растворе Alsever и промывали 5 раз буфером для анализа (150 мМ NaCl, 20 мМ HEPES, 25 мМ MgCl2 и 20 мМ EGTA). Промытые эритроциты ресуспендировали в буфере для анализа в необходимой концентрации. Различные концентрации каждого из моноклональных антител против пропердина (NBE003-3, P14 и P15) инкубировали с обезьяньей сывороткой (25%) в течение 30 минут при 37°C. Затем их добавляли к фиксированному количеству кроличьих эритроцитов и инкубировали еще в течение 50 минут при 37°С. Обработанные клетки затем центрифугировали и в супернатантах измеряли высвободившийся гемоглобин путем измерения поглощения при длине волны 405/595. Процент снижения высвобождаемого гемоглобина в присутствии антител против пропердина по настоящему изобретению рассчитывали по сравнению с лизисом эритроцитов, наблюдаемым в присутствии обезьяньей сыворотки без какого-либо антитела. Этот лизис принимали за 100%. Как показано на фигуре 3а, антитела против пропердина по настоящему изобретению предотвращают гемолиз эритроцитов кролика в присутствии обезьяньей сыворотки.
Антитело-кандидат против пропердина P15 (GLS) также исследовали на его способность блокировать активность комплемента в шести различных обезьяньих сыворотках. Функциональный анализ проводили, как описано выше, с использованием эритроцитов кролика и сыворотки шести разных обезьян. Ингибирование лизиса проводили с использованием P15 (GLS) в диапазоне концентраций от 10 до 0,17 мкг/мл. Как показано на фигуре 3b, антитело против пропердина по настоящему изобретению, P15 (GLS), предотвращает гемолиз эритроцитов кролика в присутствии обезьяньей сыворотки. Эти результаты подтвердили перекрестную реактивность моноклональных антител с пропердином обезьяны в ее сыворотке. Антитела по настоящему изобретению также можно использовать непосредственно в фармакологических и токсикологических исследованиях на животных, необходимых во время разработки лекарств, без необходимости использования суррогатных антител.
Пример 16: Анализ LPS AP для выбранных кандидатов для определения функциональности антител против пропердина
Альтернативный путь комплемента в сыворотке может быть активирован липополисахаридом (LPS) из Salmonella typhosa (18). Очищенный LPS можно использовать для получения MAC, опосредованного альтернативным путем комплемента, на поверхности планшета для ИФА (19, 20). Способность антител-кандидатов против пропердина по настоящему изобретению ингибировать такое образование МАС тестировали в этой системе анализа.
Микропланшет (Nunc) покрывали LPS из S.typhosa (Sigma, кат. №: L6386) в количестве 2 мкг/50 мкл/лунку в PBS. Затем планшет инкубировали в течение ночи при 4°C. После промывки планшет промывали 2 раза 1x PBS и блокировали на 1 час 1% BSA (300 мкл на лунку) при 37°C. Между тем, NHS (12,5% в буфере HEPES (20 мМ), содержащем 150 мМ NaCl, 20 мМ MgCl2 и 6,25 мМ EGTA) предварительно инкубировали либо с mAb против пропердина (P14, P15 или P15 (GLS)), либо с антителом изотипического контроля при 37°C при встряхивании в течение 1 часа. Затем эту предварительно инкубированную смесь сыворотки и антител переносили на описанный выше промытый планшет, покрытый LPS, и инкубировали в течение 1 часа, чтобы обеспечить активацию альтернативного пути комплемента и образование MAC. Планшет промывали 3 раза 1x PBS и добавляли мышиное антитело против MAC человека в разведении 1:5000 (объем) и инкубировали в течение 60 минут при 37°C. Планшет снова промывали 3 раза 1x PBS и 100 мкл (1:10000 по объему) HRP-конъюгированного антитела против мышиного IgG (Santa Cruz, кат.№ sc-58935) добавляли в каждую лунку и инкубировали в течение 60 минут при 37°C. Наконец, планшеты промывали 6 раз 1x PBS и окрашивание ИФА проявляли после добавления TMB/H2O2 в течение 15 минут и измеряли OD при 450 нм после остановки реакции с использованием 100 мкл/лунку 1N H2SO4.
Анализ продемонстрировал (фигура 4), что моноклональные антитела против пропердина по настоящему изобретению (P14, P15 и P15 (GLS)) ингибируют тот компонент LPS-опосредованного пути комплемента в NHS, который оставался функциональным в присутствии EGTA; и было замечено, что это зависит от дозы.
Пример 17: Анализ ингибирования лизиса эритроцитов человека с использованием антитела против CD55 и антитела против CD59
У нормального здорового человека сыворотка не вызывает лизиса собственных эритроцитов. Это происходит, несмотря на конститутивно активный характер альтернативного пути комплемента и возможно из-за присутствия естественных факторов, ингибирующих комплемент, присутствующих как в сыворотке (например, фактор Н), так и на поверхности клеток, которые вступают в контакт с плазмой (например, CD55 и CD59). Известно, что отсутствие этих ингибирующих факторов вызывает тяжелую патологию (например, PNH, aHUS и т.д.).
Гемолитические анализы с использованием эритроцитов и сыворотки, полученных от нормальных здоровых людей, были проведены по типу, как описано выше для анализа лизиса эритроцитов на кроликах, за исключением того факта, что регуляция пути комплемента ингибировалась добавлением антитела против CD55 и антитела против CD59. В присутствии этих ингибиторов сыворотка здоровых в других отношениях добровольцев была способна лизировать собственные эритроциты in vitro благодаря снятию ингибирования и активации пути комплемента.
Опубликованное (21) исследование, демонстрирующее аналогичный механизм регуляции комплемента, использовалось для описанных здесь экспериментов с небольшими изменениями. Вкратце, нормальные эритроциты человека (25 миллионов клеток на реакцию) добавляли к раствору (конечный объем 100 мкл), состоящему из желатин-вероналового буфера (GVB++) (Sigma-Aldrich) с добавлением Mg2+ (25 мМ)-EGTA (20 мМ), и антитела против CD55 человека (клон BRIC216) и антитела против CD59 человека (клон MEM-43) (конечная концентрация: 10 мкг/мл, как показано на фигуре 5; AbD Serotec) инкубировали в течение 1 часа при комнатной температуре. К эритроцитам добавляли NHS (75 мкл) с контрольным mAb или с антителом против пропердина по настоящему изобретению (оба в концентрации 5 мкг/мл), чтобы довести конечный реакционный объем до 200 мкл, и инкубировали в течение 4 часов при 37°C. Реакционные смеси центрифугировали (1500 об/мин в течение 5 минут) для осаждения нелизированных эритроцитов. Степень лизиса эритроцитов измеряли по OD при 405/595 нм аликвоты извлеченного супернатанта. Процентное ингибирование лизиса рассчитывали путем нормализации значения OD к значению OD лизированных эритроцитов (предварительно инкубированных с антителом против CD55 и антителом против CD59) в присутствии NHS. Этот лизис в отсутствие какого-либо антитела против пропердина или контрольного антитела рассматривали как 100% лизис. Гуманизированные антитела по настоящему изобретению (P14, P15 и P15 (GLS)) были способны значительно ингибировать лизис эритроцитов человека в условиях, подобных PNH, как показано на фигуре 5.
Пример 18: Анализ ингибирования лизиса эритроцитов человека, обработанных проназой
Естественная регуляция комплемента с помощью регуляторов, экспрессирующихся на клеточной поверхности нормальных клеток, также может быть устранена путем обработки клеток протеазами, такими как проназа (22). Ранее протеазы использовались для модификации нормальных эритроцитов человека с целью превращения их в PNH-подобные клетки, чувствительные к комплементу. Гемолитические анализы выполняли, как описано ранее, с использованием нормальных человеческих эритроцитов, обработанных проназой. Обработка проназой приводит к лизису нормальных эритроцитов человека их собственной сывороткой из-за активации альтернативного пути комплемента.
Нормальные эритроциты человека (25 миллионов клеток на реакцию) добавляли к раствору (конечный объем 100 мкл), состоящему из желатин-вероналового буфера (GVB++) (Sigma-Aldrich) с добавлением Mg2+ (25 мМ)-EGTA (20 мМ). Эти эритроциты обрабатывали ферментом проназой (5 мг/мл) в течение 30 минут при 37°С. NHS (50 мкл), предварительно инкубированный с антителом против пропердина или без него в различных концентрациях (5, 2,5 и 1,25) в течение 30 минут, добавляли к обработанным проназой эритроцитам, чтобы получить конечный реакционный объем 200 мкл, и инкубировали в течение 4 часов при 37°С. Реакционные смеси центрифугировали (1500 об/мин) в течение 5 минут для осаждения нелизированных эритроцитов. Степень лизиса эритроцитов измеряли по OD аликвоты извлеченного супернатанта при 405/595 нм. Процент ингибирования лизиса рассчитывали путем нормализации значения OD к значению OD лизированных эритроцитов (обработанных проназой) в присутствии NHS. Этот лизис в отсутствие какого-либо антитела против пропердина или контрольного антитела рассматривали как 100% лизис. mAb против пропердина по настоящему изобретению (P14, P15 и P15 (GLS)) были способны ингибировать опосредованный комплементом лизис эритроцитов человека, обработанных проназой, в присутствии NHS, как показано на фигуре 6.
Пример 19: Определение эффективности антител против пропердина в мышиной модели индуцированного LPS повреждения легких
Известно, что интратрахеальная инстилляция LPS вызывает повреждение легких с повышением уровня цитокинов в жидкости бронхоальвеолярного лаважа (BAL). Снижение цитокина вследствие любого терапевтического введения считается эффективным. Для эксперимента мышей Balb/c взвешивали и рандомизировали. Мыши были рандомизированы на четыре группы. Одну группу оставляли в качестве ложного контроля для определения повышения уровня цитокинов в результате процедуры интратрахеальной инстилляции. В то время как в другой группе сыворотку против пропердина (кроличье антитело против мышиного пропердина) вводили внутривенно в концентрации 400 мкг/20 г мышей. Используемое здесь кроличье антитело против мышиного пропердина называют суррогатом ZAP. В контрольном носителе (контроль заболевания) только плацебо вводили в том же объеме, что и суррогатную сыворотку против пропердина, путем внутривенного введения. Группу животных без какой-либо индукции заболевания также изучали для определения любого изменения базовых уровней цитокинов. После введения суррогатной сыворотки против пропердина или плацебо каждой мыши интратрахеально вводили LPS (Sigma). Животных гуманным образом умерщвляли через 6 часов после инстилляции LPS, и у каждой мыши собирали смывы BAL и оценивали уровни TNF-α и IL-6 с помощью ИФА. mAb против пропердина (суррогат ZAP) были способны снижать воспалительные цитокины в жидкости бронхоальвеолярного лаважа (BAL), как показано на фигурах 7 и 8. Эти результаты демонстрируют, что лечение антипропердином эффективно при лечении повреждения легких. Как для TNF-α (555268), так и для IL-6 (555240) ИФА использовали наборы, произведенные BD biosciences. Процедуры проводили в соответствии с инструкциями набора. Поглощение считывали при 450 нм с поправкой на поглощение при 570 нм.
Список нуклеотидных и аминокислотных последовательностей, используемых в настоящем изобретении:
Список нуклеотидных и аминокислотных последовательностей, используемых в настоящем изобретении:
SEQ ID No.84: P13, легкая цепь
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTGATATCGTGATGACCCAGTCTCCTGACAGCCTGGCTGTGTCTCTGGGCGAGAGAGCCACCATCAACTGCAAGTCCTCTCAGTCCCTGCTGTACTCCTCCAACCAGAAGAACTACCTGGCCTGGTATCAGCAGAAGCCCGGACAGCCTCCTAAGCTGCTGATCTACTGGGCCTCCACCAGAGAATCTGGCGTGCCAGATAGATTCTCCGGCTCTGGCTCTGGCACCGACTTTACCCTGACAATCAGCTCTCTGCAGGCCGAGGATGTGGCCGTGTACTACTGCCAGCAGTACTACAGCTACCCCTACACCTTTGGCGGAGGCACCAAGCTGGAAATCAAGAGAACCGTGGCCGCTCCTTCCGTGTTCATCTTCCCACCATCTGACGAGCAGCTGAAGTCCGGCACAGCTTCTGTCGTGTGCCTGCTGAACAACTTCTACCCTCGGGAAGCCAAGGTGCAGTGGAAGGTGGACAATGCCCTGCAGTCCGGCAACTCCCAAGAGTCTGTGACCGAGCAGGACTCCAAGGACTCTACCTACAGCCTGTCCTCCACACTGACCCTGTCTAAGGCCGACTACGAGAAGCACAAGGTGTACGCCTGTGAAGTGACCCACCAGGGACTGTCTAGCCCCGTGACCAAGTCTTTCAACAGAGGCGAGTGCTAATGA
SEQ ID No. 85: P14, легкая цепь
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTGATATCGTGATGACCCAGTCTCCTCTGAGCCTGTCTGTGACACCTGGCCAGCCTGCCTCCATCTCTTGCAAGTCATCTCAGTCCCTGCTGGACATCAACGGCAAGACCTACCTGAACTGGTATCTGCAGAAGCCCGGCCAGTCTCCACAGCTGCTGATCTACCTGGTGTCCAAGCTGGATTCTGGCGTGCCCGACAGATTTTCCGGCTCTGGCTCTGGCACCGACTTCACCCTGAAGATTTCCAGAGTGGAAGCCGAGGACGTGGGCGTGTACTACTGTTGGCAGGGCACCCACTTTCCATACACCTTCGGCCAGGGCACCAAGGTGGAAATCAAGAGAACCGTGGCCGCTCCTTCCGTGTTCATCTTCCCACCATCTGACGAGCAGCTGAAGTCCGGCACAGCTTCTGTCGTGTGCCTGCTGAACAACTTCTACCCTCGGGAAGCCAAGGTGCAGTGGAAGGTGGACAATGCCCTGCAGTCCGGCAACTCCCAAGAGTCTGTGACCGAGCAGGACTCCAAGGACTCTACCTACTCTCTGTCCTCCACACTGACCCTGTCCAAGGCCGACTACGAGAAGCACAAGGTGTACGCCTGTGAAGTGACCCACCAGGGACTGTCTAGCCCCGTGACCAAGTCTTTCAACAGAGGCGAGTGCTAATGA
SEQ ID No. 86:P15, легкая цепь
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTGATATCCAGATGACCCAGTCTCCTTCCAGCCTGTCTGCCTCTGTGGGCGACAGAGTGACCATCACCTGTCTGGCTTCTCAGACCATCGGCACCTGGCTGGCCTGGTATCAGCAGAAGCCTGGAAAGGCCCCTAAGCTGCTGATCTACGCTGCCACATCTCTGGCCGATGGCGTGCCATCTAGATTCTCTGGCTCTGGCTCCGGCACCGACTTTACCCTGACAATCAGTTCCCTGCAGCCTGAGGACTTCGCCACCTACTACTGCCAGCAGCTGTACTCTACCCCTTGGACCTTTGGCGGAGGCACCAAGCTGGAAATCAAGAGAACCGTGGCCGCTCCTTCCGTGTTCATCTTCCCACCATCTGACGAGCAGCTGAAGTCTGGCACCGCTTCTGTCGTGTGCCTGCTGAACAACTTCTACCCTCGGGAAGCCAAGGTGCAGTGGAAGGTGGACAATGCCCTGCAGTCCGGCAACTCCCAAGAGTCTGTGACCGAGCAGGACTCCAAGGACTCTACCTACAGCCTGTCCTCCACACTGACCCTGTCTAAGGCCGACTACGAGAAGCACAAGGTGTACGCCTGTGAAGTGACCCACCAGGGACTGTCTAGCCCCGTGACCAAGTCTTTCAACAGAGGCGAGTGCTAATGA
SEQ ID No. 87: E12, легкая цепь
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTCAGTCTGTTTTGACTCAGCCTCCTTCCGTGTCTGCCGCTCCTGGCCAGAAAGTGACCATCTCTTGCTCCGGCTCCTCCTCCAACATCGGCAACAACTATGTGTCTTGGTACGTGCAGCTGCCCGGCACAGCTCCTAAGCTGCTGATCTACGACAACAACAAGCGGTTCTCCGGCGTGCCCGACAGATTCTCTGGCTCTAAGTCTGGCACCAGCGCTACCCTGGGAATCACAGGATTGCAGACAGGCGACGAGGCCGATTACTACTGTGGCGCTTGGGACGGCTCTCTGAGGGAAGCTGTTTTTGGCGGAGGCACCAAAGTGACCGTGCTGAGAGCTGCTGGACAGCCTAAAGCCGCTCCTAGCGTGACCCTGTTTCCTCCATCTTCTGAGGAACTGCAGGCCAACAAGGCTACCCTCGTGTGCCTGATCTCTGACTTTTACCCTGGCGCTGTGACCGTGGCCTGGAAGGCTGATAGTTCTCCTGTGAAGGCCGGCGTGGAAACCACCACACCTTCCAAGCAGTCCAACAACAAATACGCCGCCTCCTCCTACCTGTCTCTGACCCCTGAACAGTGGAAGTCCCACCGGTCCTACTCTTGCCAAGTGACCCATGAGGGCTCCACCGTGGAAAAGACAGTGGCCCCTACCGAGTGCTCCTAATGA
SEQ ID No. 88: P12 вариабельная область тяжелой цепи
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTCAGGTTCAGTTGCAACAGTCTGGCCCTGAACTCGTCAGACCCGGCGTGTCCGTGAAGATCAGCTGTAAAGGCTCCGGCTACACCTTCACCGACTACGCCATGCACTGGGTCAAGCAGTCTCACGCCGAGTCTCTGGAATGGATCGGCCTGATCTCTACCTACTACGGCGACGCCGGCTACAACCAGAAGTTCAAGGATAAGGCCACAATGACCGTGGACATCTCCTCCTCCACCGCCTACCTGGAACTGGCTAGACTGACCTCTGAGGACTCCGCCATCTACTACTGCGCCAGAGCCGACTCCTCTGGCAACTTCTTTGATTACTGGGGCCAGGGCACCCTGGTCACCGTTTCTAGTGCTTCCACAAAGGGCCC
SEQ ID No. 89: P13, вариабельная область тяжелой цепи
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTGAAGTTCAGTTGCAGCAGTCTGGCCCCGAGCTTGTGAAACCTGGCGCCTCTGTGAAGATGTCCTGCAAGGCCTCTGGCTACACCTTCGCTCACAACTGGATTCACTGGGTCAAGCAGAAGCCAGGCCAGGGACTTGAGTGGATCGGCTACATCAACCCTGGCACCGACTACACCGAGTACTCCCAGAGATTCAAGGGCAAAGCTACCCTGACCTCCGACAAGTCCTCCTCCACCGCTTACATGGAACTGTCCAGCCTGACCTCTGAGGACTCCGCCGTGTACTACTGCGCCAGAAGAAAGCTGTACGGCAACTTCGTGGACTACGCCATGGATTATTGGGGCCAGGGCACCCTGGTTACCGTGTCTGCTGCTTCTACAAAGGGCCC
SEQ ID No. 90: P14, вариабельная область тяжелой цепи
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTCAGGTTCAGTTGCAAGAGTCTGGCCCTGGCCTGGTCAAGCCTTCTCAGACCCTGTCTCTGACCTGCACCGTGTCCGGCTATTCCATCACCTCCGGCTACTACTGGAACTGGATCAGACAGCACCCCGGCAAAGGCCTGGAATGGATCGGCTACATCTCTTACGACGGCGGCAACAAGTACAACCCCAGCCTGAAGTCCAGAGTGACCATCTCTCGGGACACCTCCAAGAACCAGTTCTCCCTGAAGCTGTCCTCTGTGACCGCTGCCGATACCGCCGTGTACTACTGTGCCCGCGATCTGGATGGCTACGAGTCTATGGATTATTGGGGCCAGGGCACCTCCGTGACCGTGTCCTCTGCTTCTACAAAGGGCCC
SEQ ID No. 91: P15, вариабельная область тяжелой цепи
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTCAGGTTCAGTTGCAAGAGTCTGGCCCTGGCCTGGTCAAGCCTTCTCAGACCCTGTCTCTGACCTGCACCGTGTCCGGCTACTCCATCACCTCCACCTACTACTGGAACTGGATCAGACAGCACCCCGGCAAAGGCCTGGAATGGATCGGCTACATCTCCTACGACGGCACCAACAAGTACAACCCCAGCCTGAAGTCCAGAGTGACCATCTCTCGGGACACCTCCAAGAACCAGTTCTCCCTGAAGCTGTCCTCTGTGACCGCTGCCGATACCGCCGTGTACTACTGCGCCAGAGATGACTACGACAGATCCCCTTGGTTTGCCTATTGGGGCCAGGGCACACTGGTCACCGTTTCCAGTGCTTCTACCAAGGGCCC
SEQ ID No. 92: E12, вариабельная область тяжелой цепи
ATGGGCTGGTCCTGCATCATTCTGTTTCTGGTGGCTACCGCCACCGGCGTGCACTCTCAGGTTCAGTTGCAACAGTCTGGCCCTGAACTCGTCAGACCCGGCGTGTCCGTGAAGATCAGCTGTAAAGGCTCCGGCTACACCTTCACCGATTACGCCCTGCACTGGGTCAAGCAGTCTCACGCTGAGTCTCTGGAATGGATCGGCGTGATCTCCACCTACTACGGCGACGCCTCCTACAACCAGAAGTTCAAGGACAAGGCCACAATGACCGTGGACATCTCCTCCTCCACCGCCTACCTGGAACTGGCTAGACTGACCTCTGAGGACTCCGCCATCTACTACTGCGCCAGAGATGGCTACCTGGATTATTGGGGCCAGGGCACACTGGTCACCGTGTCCTCTGCTTCTACAAAGGGCCC
SEQ ID No. 93: IgG4, константная область тяжелой цепи
GGGCCCCTCCGTGTTCCCTCTGGCCCCTTGCTCCCGGTCCACCTCCGAGTCTACCGCCGCTCTGGGCTGCCTGGTGAAAGACTACTTCCCCGAGCCCGTGACCGTGTCCTGGAACTCTGGCGCCCTGACCAGCGGCGTGCACACCTTCCCTGCCGTGCTGCAGTCCTCCGGCCTGTACTCCCTGTCCTCCGTGGTGACCGTGCCCTCCTCCAGCCTGGGCACCAAGACCTACACCTGTAACGTGGACCACAAGCCCTCCAACACCAAGGTGGACAAGCGGGTGGAATCTAAGTACGGCCCTCCCTGCCCCCCCTGCCCTGCCCCTGAATTTCTGGGCGGACCTTCCGTGTTCCTGTTCCCCCCAAAGCCCAAGGACACCCTGATGATCTCCCGGACCCCCGAAGTGACCTGCGTGGTGGTGGACGTGTCCCAGGAAGATCCCGAGGTGCAGTTCAATTGGTACGTGGACGGCGTGGAAGTGCACAACGCCAAGACCAAGCCCAGAGAGGAACAGTTCAACTCCACCTACCGGGTGGTGTCTGTGCTGACCGTGCTGCACCAGGACTGGCTGAACGGCAAAGAGTACAAGTGCAAGGTGTCCAACAAGGGCCTGCCCTCCAGCATCGAAAAGACCATCTCCAAGGCCAAGGGCCAGCCCCGCGAGCCCCAGGTGTACACCCTGCCCCCTAGCCAGGAAGAGATGACCAAGAACCAGGTGTCCCTGACCTGTCTGGTGAAAGGCTTCTACCCCTCCGATATCGCCGTGGAATGGGAGTCCAACGGCCAGCCCGAGAACAACTACAAGACCACCCCCCCTGTGCTGGACTCCGACGGCTCCTTCTTCCTGTACTCTCGGCTGACCGTGGACAAGTCCCGGTGGCAGGAAGGCAACGTGTTCTCCTGCTCCGTGATGCACGAGGCCCTGCACAACCACTACACCCAGAAGTCCCTGTCCCTGAGCCTGGGCAAGTGATGA
SEQ ID No. 94: IgG4 (GLS), модифицированная константная область тяжелой цепи
GGGCCCCTCCGTGTTCCCTCTGGCCCCTTGCTCCCGGTCCACCTCCGAGTCTACCGCCGCTCTGGGCTGCCTGGTGAAAGACTACTTCCCCGAGCCCGTGACCGTGTCCTGGAACTCTGGCGCCCTGACCAGCGGCGTGCACACCTTCCCTGCCGTGCTGCAGTCCTCCGGCCTGTACTCCCTGTCCTCCGTGGTGACCGTGCCCTCCTCCAGCCTGGGCACCAAGACCTACACCTGTAACGTGGACCACAAGCCCTCCAACACCAAGGTGGACAAGCGGGTGGAATCTAAGTACGGCCCTCCCTGCCCCCCCTGCCCTGCCCCTGAATTTCTGGGCGGACCTTCCGTGTTCCTGTTCCCCCCAAAGCCCAAGGACACCCTGATGATCTCCCGGACCCCCGAAGTGACCTGCGTGGTGGTGGACGTGTCCCAGGAAGATCCCGAGGTGCAGTTCAATTGGTACGTGGACGGCGTGGAAGTGCACAACGCCAAGACCAAGCCTAGAGAGGAACAGTTCAACAGCACCTACAGAGTGGTGTCCGTGCTGACCGTGCTGCACCAGGATTGGCTGAACGGCAAAGAGTACAAGTGCAAGGTGTCCAACAAGGGCCTGGGCTCCAGCATCGAAAAGACCATCTCCAAGGCTAAGGGCCAGCCTCGGGAACCTCAGGTTTACACCCTGCCTCCAAGCCAAGAGGAAATGACCAAGAATCAGGTGTCCCTGACCTGTCTCGTGAAGGGCTTCTACCCTTCTGATATCGCCGTGGAATGGGAGTCCAACGGCCAGCCTGAGAACAACTACAAGACCACACCTCCAGTGCTGGACTCCGACGGCTCTTTCTTCCTGTATTCCCGCCTGACCGTGGACAAGTCCAGATGGCAAGAGGGCAACGTGTTCTCCTGCAGCGTGCTGCATGAGGCTCTGCACTCCCACTACACCCAGAAGTCTCTGTCTCTGTCCCTGGGCAAGTGATAA
SEQ ID No. 95 белковая последовательность константной области P15 IgG4
ASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK
SEQ ID No. 96 белковая последовательность константной области P15(GLS)
ASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPSVFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVLHQDWLNGKEYKCKVSNKGLGSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAVEWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVLHEALHSHYTQKSLSLSLGK
Ссылки, включенные в настоящую патентную заявку
1. Harris CL, Pouw RB, Kavanagh D, Sun R, Ricklin D. Developments in anti-complement therapy; from disease to clinical trial. MolImmunol. 2018; 102: 89-119.
2. Mastellos DC, Ricklin D, Lambris JD. Clinical promise of next-generation complement therapeutics. Nat Rev Drug Discov. 2019; 18: 707-29.
3. Please refer FDA approved product label of Eculizumab.
4. Zelek WM, Xie L, Morgan BP, Harris CL. Compendium of current complement therapeutics. MolImmunol. 2019; 114: 341-52.
5. Ricklin D, Barratt-Due A, Mollnes TE. Complement in clinical medicine: Clinical trials, case reports and therapy monitoring. MolImmunol. 2017; 89: 10-21.
6. Angal S, King DJ, Bodmer MW, Turner A, Lawson AD, Roberts G, Pedley B, Adair JR. A single amino acid substitution abolishes the heterogeneity of chimeric mouse/human (IgG4) antibody. MolImmunol. 1993; 30(1): 105-8.
7. Jones PT, Dear PH, Foote J, Neuberger MS, Winter G. Replacing the complementarity-determining regions in a human antibody with those from a mouse. Nature. 1986; 321(6069): 522-5.
8. Riechmann L, Clark M, Waldmann H, Winter G. Reshaping human antibodies for therapy. Nature.1988; 332(6162): 323-7.
9. Verhoeyen M, Milstein C, Winter G. Reshaping human antibodies: grafting an antilysozyme activity. Science.1988; 239(4847): 1534-6.
10. Almagro JC, Fransson J. Humanization of antibodies. Front Biosci. 2008; 13: 1619-33.
11. Wang X, Mathieu M, Brezski RJ. IgG Fc engineering to modulate antibody effector functions. Protein Cell. 2018; 9(1): 63-73.
12. Schlothauer T, Herter S, Koller CF, Grau-Richards S, Steinhart V, Spick C, Kubbies M, Klein C, Umaña P, Mössner E. Novel human IgG1 and IgG4 Fc-engineered antibodies with completely abolished immune effector functions. Protein Eng Des Sel. 2016; 29(10): 457-466.
13. Wood JN. Immunization and fusion protocols for hybridoma production. Methods Mol Biol. 1984; 1: 261-70.
14. Kim HY, Stojadinovic A, Izadjoo MJ. Immunization, hybridoma generation, and selection for monoclonal antibody production. Methods Mol Biol. 2014; 1131: 33-45.
15. Honegger A, Plückthun A. Yet another numbering scheme for immunoglobulin variable domains: an automatic modeling and analysis tool. J Mol Biol. 2001; 309(3): 657-70.
16. www.vbase2.org
17. Polhill RB Jr, Pruitt KM, Johnston RB Jr. Kinetic assessment of alternative complement pathway activity in a hemolytic system. I. Experimental and mathematical analyses. J Immunol. 1978; 121(1): 363-70.
18. Douglas P. Fine. Activation of the Classic and Alternate Complement Pathways by Endotoxin. J Immunol.1974; 112(2); 763-9.
19. Clardy CW. Complement activation by whole endotoxin is blocked by a monoclonal antibody to factor B. Infect Immun.1994; 62(10): 4549-55.
20. Bansal R, Brunden K, Parent J. Process for inhibiting complement activation via the alternative pathway. US20020015701.
21. Gullipalli D, Zhang F, Sato S, Ueda Y, Kimura Y, Golla M, Miwa T, Wang J, Song WC. Antibody Inhibition of Properdin Prevents Complement-Mediated Intravascular and Extravascular Hemolysis. J Immunol. 2018; 201(3): 1021-29.
22. Yuan FF, Bryant JA, Fletcher A. Protease-modified erythrocytes: CD55 and CD59 deficient PNH-like cells. Immunol Cell Biol. 1995; 73(1): 66-72.
Включение ссылкой
Полное раскрытие каждого из патентных документов и научных статей, упомянутых в настоящем документе, включено в качестве ссылки для всех целей.
Эквиваленты
Изобретение может быть воплощено в других конкретных формах, не выходя за рамки его объема или существенных характеристик. Таким образом, вышеприведенные варианты осуществления следует рассматривать во всех отношениях как иллюстративные, а не ограничивающие изобретение, описанное в настоящем документе. Объем изобретения, таким образом, определяется с помощью формулы изобретения, а не с помощью вышеуказанного описания, и подразумевается, что все изменения, которые подпадают под значение и диапазон эквивалентности пунктов формулы изобретения, охвачены настоящим изобретением.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing nonEnglishFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="seq.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2022-10-27">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022119826</ApplicationNumberText>
<FilingDate>2022-07-20</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>574791ru</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>IN</IPOfficeCode>
<ApplicationNumberText>202021000807</ApplicationNumberText>
<FilingDate>2020-01-08</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ЗИДУС ЛАЙФСАЙЕНСИЗ
ЛИМИТЕД</ApplicantName>
<ApplicantNameLatin>ZYDUS LIFESCIENCES LIMITED</ApplicantNameLatin>
<InventionTitle languageCode="ru">АНТИТЕЛА ПРОТИВ ПРОПЕРДИНА И ИХ
ПОЛУЧЕНИЕ</InventionTitle>
<SequenceTotalQuantity>96</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GYTFTDY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GYTFAHN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q5">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GYSITSGYY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GYSITSTYY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательнсти синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GYSFTSY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанитным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VISTYYGD</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LISTYYGD</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YINPGTDY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YISYDGGN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YISYDGTN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EIDPSAGY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>6</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q23">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DGYLDY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>10</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ADSSGNFFDY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>14</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательнсти синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..14</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RKLYGNFVDYAMDY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>10</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательнсти синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DLDGYESMDY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DDYDRSPWFAY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EGYRYFDV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EGYKYFDV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SGSSSNIGNNYVS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственая
последователньость</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KSSQSLLYSSNQKNYLA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RPSQDINNYLS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>16</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..16</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KSSQSLLDINGKTYLN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q45">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LASQTIGTWLA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q47">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RLDGSSPGNNYVS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q49">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SGSSSNIRGTPTT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q51">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SGSSSNLGVKDVN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q53">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YPRLLFKGNNYVS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q55">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовтаельности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SGSSSNHQHRPAS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q57">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q58">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EQWSPGHGNNYVS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q59">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DNNKRFS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q61">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовтаельности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q62">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>WASTRES</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q63">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантым методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YTSRLHS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q65">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q66">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LVSKLDS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>7</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q67">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..7</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AATSLAD</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q69">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GAWDGSLREAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>10</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q71">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q72">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QQYYSYPYTL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q73">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантым методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>HQYLSSYT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q75">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q76">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QQGNTLPPT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q77">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q78">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовтаельность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>WQGTHFPYT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q79">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q80">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QQLYSTPWT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q81">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательнсти синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q82">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AARQHLLREAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q83">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q84">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GAWDASLREAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>11</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q85">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..11</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q86">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MTQNGILREAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>115</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..115</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q87">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..115</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QVQLQQSGPELVRPGVSVKISCKGSGYTFTDYALHWVKQSHAESLEWIG
VISTYYGDASYNQKFKDKATMTVDISSSTAYLELARLTSEDSAIYYCARDGYLDYWGQGTLVTVSS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>119</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..119</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q89">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..119</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q90">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QVQLQQSGPELVRPGVSVKISCKGSGYTFTDYAMHWVKQSHAESLEWIG
LISTYYGDAGYNQKFKDKATMTVDISSSTAYLELARLTSEDSAIYYCARADSSGNFFDYWGQGTLVTVSS
</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>123</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..123</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q91">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..123</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q92">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EVQLQQSGPELVKPGASVKMSCKASGYTFAHNWIHWVKQKPGQGLEWIG
YINPGTDYTEYSQRFKGKATLTSDKSSSTAYMELSSLTSEDSAVYYCARRKLYGNFVDYAMDYWGQGTLV
TVSA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>119</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..119</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q93">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..119</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QVQLQESGPGLVKPSQTLSLTCTVSGYSITSGYYWNWIRQHPGKGLEWI
GYISYDGGNKYNPSLKSRVTISRDTSKNQFSLKLSSVTAADTAVYYCARDLDGYESMDYWGQGTSVTVSS
</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>120</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..120</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q95">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..120</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q96">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QVQLQESGPGLVKPSQTLSLTCTVSGYSITSTYYWNWIRQHPGKGLEWI
GYISYDGTNKYNPSLKSRVTISRDTSKNQFSLKLSSVTAADTAVYYCARDDYDRSPWFAYWGQGTLVTVS
S</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>117</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..117</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q97">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..117</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QVQLQQPGAELVKPGASVILSCKASGYSFTSYWVHWVKQRPGQGLEWIG
EIDPSAGYTNFNQKFRDMATLTVDKSSSTAYMQLSSLTSEDSAVYFCTREGYRYFDVWGAGTTVTVSS</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>115</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..115</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q99">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..115</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QVQLKESGAELVRPGVSVKISCKGSGYTFTDYAMHWVKQSHAKSLEWIG
VISTYYGDASYNQKFKGKATMTVDKSSSTAYMELARLTSEDSAIYYCARDGYLDYWGQGTTLTVSS</IN
SDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>120</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..120</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q101">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..120</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q102">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GAELVRPGVSVKISCKGSGYTFTDYAMHWVKQSHAKSLEWIGLISTYYG
DARYNQKFRGKATMTVDKSSSTAYMELARLTSEDSAIYYCARADSSGNFFDYWGQGTTLTVSSAKTTPPS
V</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>128</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..128</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q103">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательнсти синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..128</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GAELAKPGASVKMSCKASGYTFAHNWMHWVKQRPGQGLEWIGYINPGTD
YTEYSQRFKDKATLTADKSSSTAYMQLSSLTSEDSAVYYCARRKLYGNFVDYAMDYWGQGTSVTVSSAKT
TPPSVYPLA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>124</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..124</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q105">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..124</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GPGLVKPSQSLSLTCSVTGYSITSGYYWNWIRQFPGNKLEWMGYISYDG
GNIYNPSLKNRISITRDTSKNQFFLKLNSVTSEDTATYYCARDLDGYESMDYWGQGTSVTVSSAKTTPPS
VYPLA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>116</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..116</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q107">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..116</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q108">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KPSQSLSLTCSVTGYSITSTYYWNWIRQFPGNKLEWMGYISYDGTNKYN
PSLKNRISITRDTSKNRFFLKLNSVTTEDTATYYCAGDDYDRSPWFAYWGQGTLVTVSAAKTTPPSV</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>117</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..117</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q109">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..117</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q110">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QVQLVQSGAEVKKPGASVKVSCKASGYSFTSYWMHWVRQAPGQGLEWMG
EIDPSAGYTNYNQKFTGRVTMTRDTSTSTVYMELSSLRSEDTAVYYCTREGYKYFDVWGAGTTVTVSS</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q111">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовтаельности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNIGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCGAWDGSLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>113</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..113</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q113">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..113</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q114">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DIVMTQSPDSLAVSLGERATINCKSSQSLLYSSNQKNYLAWYQQKPGQP
PKLLIYWASTRESGVPDRFSGSGSGTDFTLTISSLQAEDVAVYYCQQYYSYPYTFGGGTKLEIK</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>113</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..113</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q115">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..113</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q116">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DIVMTQSPDSLAVSLGERATINCKSSQSVLYSSNQKNYLAWYQQKPGQP
PKLLIYWASTRESGVPDRFSGSGSGTDFTLTISSLQAEDVAVYYCHQYLSSYTFGQGTKVEIKR</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q117">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DIQMTQSPSSLSASVGDRVTITCRPSQDINNYLSWYQQKPGKAPKLLIY
YTSRLHSGVPSRFSGSGSGTDFTFTISSLQPEDIATYYCQQGNTLPPTFGQGTKVEIKR</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>113</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..113</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q119">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..113</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q120">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DIVMTQSPLSLSVTPGQPASISCKSSQSLLDINGKTYLNWYLQKPGQSP
QLLIYLVSKLDSGVPDRFSGSGSGTDFTLKISRVEAEDVGVYYCWQGTHFPYTFGQGTKVEIKR</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q121">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовтаельности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q122">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DIQMTQSPSSLSASVGDRVTITCLASQTIGTWLAWYQQKPGKAPKLLIY
AATSLADGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQLYSTPWTFGGGTKLEIKR</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>127</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..127</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q123">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..127</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNIGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCGAWDGSLREAVFGGGTKVTVLVRPLDPKIS
ELKLLKVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>94</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..94</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q125">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..94</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q126">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SSLAVSAGEKVTMSCKSSQSVLYSSNQKNYLAWYQQKPGQSPKLLIYWA
STRESGVPDRFTGSGSGTDFTLTISSVQAEDLAVYYCHQYLSSYT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>97</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..97</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q127">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..97</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q128">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PSSLAVSVGEKVTLNCKSSQSLLYSSNQKNYLAWYQQKPGQSPKLLINW
ASTRESGVPDRCTGSGSGTDFTLTISNVKAEDLAVYYCQQYYSYPYTL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="65">
<INSDSeq>
<INSDSeq_length>94</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..94</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q129">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..94</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q130">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LTLSVTFGQPASISCKSSQSLLDINGKTYLNWLLQRPGQSPKRLIYLVS
KLDSGVPDRFTGSGSGTDFTLKISRVEAEDLGIYYCWQGTHFPYT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="66">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q131">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q132">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>DIVLTQSPASQSASLGESVTITCLASQTIGTWLAWYQQKPGKSPQLLIY
AATSLADGVPSRFSGSGSGTKFSFKISSLQAEDFVSYYCQQLYSTPWTFGGGTKLEIKR</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="67">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q133">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q134">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCRLDGSSPGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCAARQHLLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="68">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q135">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q136">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNIRGTPTTWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCGAWDGSLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="69">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q137">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q138">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNLGVKDVNWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCGAWDASLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="70">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q139">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q140">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCYPRLLFKGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCGAWDGSLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="71">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q141">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q142">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNHQHRPASWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCMTQNGILREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="72">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q143">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q144">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNIGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCMTQNGILREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="73">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q145">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q146">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCRLDGSSPGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCMTQNGILREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="74">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q147">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q148">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNIRGTPTTWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCMTQNGILREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="75">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q149">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNLGVKDVNWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCMTQNGILREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="76">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q151">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q152">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCYPRLLFKGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCMTQNGILREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="77">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q153">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q154">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNIGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCAARQHLLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="78">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q155">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q156">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNIRGTPTTWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCAARQHLLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="79">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q157">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q158">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCEQWSPGHGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCAARQHLLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="80">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q159">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q160">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCYPRLLFKGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCAARQHLLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="81">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q161">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q162">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNHQHRPASWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCAARQHLLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="82">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q163">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q164">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCRLDGSSPGNNYVSWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCGAWDGSLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="83">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q165">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q166">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSVSAAPGQKVTISCSGSSSNHQHRPASWYVQLPGTAPKLLI
YDNNKRFSGVPDRFSGSKSGTSATLGITGLQTGDEADYYCGAWDGSLREAVFGGGTKVTVLR</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="84">
<INSDSeq>
<INSDSeq_length>723</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..723</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q167">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..723</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q168">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctgatatcgtgatgacccagtctcctgacagcctggctgtgtctctgggcgagagagccaccat
caactgcaagtcctctcagtccctgctgtactcctccaaccagaagaactacctggcctggtatcagcag
aagcccggacagcctcctaagctgctgatctactgggcctccaccagagaatctggcgtgccagatagat
tctccggctctggctctggcaccgactttaccctgacaatcagctctctgcaggccgaggatgtggccgt
gtactactgccagcagtactacagctacccctacacctttggcggaggcaccaagctggaaatcaagaga
accgtggccgctccttccgtgttcatcttcccaccatctgacgagcagctgaagtccggcacagcttctg
tcgtgtgcctgctgaacaacttctaccctcgggaagccaaggtgcagtggaaggtggacaatgccctgca
gtccggcaactcccaagagtctgtgaccgagcaggactccaaggactctacctacagcctgtcctccaca
ctgaccctgtctaaggccgactacgagaagcacaaggtgtacgcctgtgaagtgacccaccagggactgt
ctagccccgtgaccaagtctttcaacagaggcgagtgctaatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="85">
<INSDSeq>
<INSDSeq_length>720</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..720</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q169">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..720</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q170">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctgatatcgtgatgacccagtctcctctgagcctgtctgtgacacctggccagcctgcctccat
ctcttgcaagtcatctcagtccctgctggacatcaacggcaagacctacctgaactggtatctgcagaag
cccggccagtctccacagctgctgatctacctggtgtccaagctggattctggcgtgcccgacagatttt
ccggctctggctctggcaccgacttcaccctgaagatttccagagtggaagccgaggacgtgggcgtgta
ctactgttggcagggcacccactttccatacaccttcggccagggcaccaaggtggaaatcaagagaacc
gtggccgctccttccgtgttcatcttcccaccatctgacgagcagctgaagtccggcacagcttctgtcg
tgtgcctgctgaacaacttctaccctcgggaagccaaggtgcagtggaaggtggacaatgccctgcagtc
cggcaactcccaagagtctgtgaccgagcaggactccaaggactctacctactctctgtcctccacactg
accctgtccaaggccgactacgagaagcacaaggtgtacgcctgtgaagtgacccaccagggactgtcta
gccccgtgaccaagtctttcaacagaggcgagtgctaatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="86">
<INSDSeq>
<INSDSeq_length>705</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..705</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q171">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..705</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q172">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственая
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctgatatccagatgacccagtctccttccagcctgtctgcctctgtgggcgacagagtgaccat
cacctgtctggcttctcagaccatcggcacctggctggcctggtatcagcagaagcctggaaaggcccct
aagctgctgatctacgctgccacatctctggccgatggcgtgccatctagattctctggctctggctccg
gcaccgactttaccctgacaatcagttccctgcagcctgaggacttcgccacctactactgccagcagct
gtactctaccccttggacctttggcggaggcaccaagctggaaatcaagagaaccgtggccgctccttcc
gtgttcatcttcccaccatctgacgagcagctgaagtctggcaccgcttctgtcgtgtgcctgctgaaca
acttctaccctcgggaagccaaggtgcagtggaaggtggacaatgccctgcagtccggcaactcccaaga
gtctgtgaccgagcaggactccaaggactctacctacagcctgtcctccacactgaccctgtctaaggcc
gactacgagaagcacaaggtgtacgcctgtgaagtgacccaccagggactgtctagccccgtgaccaagt
ctttcaacagaggcgagtgctaatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="87">
<INSDSeq>
<INSDSeq_length>720</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..720</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q173">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..720</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q174">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctcagtctgttttgactcagcctccttccgtgtctgccgctcctggccagaaagtgaccatctc
ttgctccggctcctcctccaacatcggcaacaactatgtgtcttggtacgtgcagctgcccggcacagct
cctaagctgctgatctacgacaacaacaagcggttctccggcgtgcccgacagattctctggctctaagt
ctggcaccagcgctaccctgggaatcacaggattgcagacaggcgacgaggccgattactactgtggcgc
ttgggacggctctctgagggaagctgtttttggcggaggcaccaaagtgaccgtgctgagagctgctgga
cagcctaaagccgctcctagcgtgaccctgtttcctccatcttctgaggaactgcaggccaacaaggcta
ccctcgtgtgcctgatctctgacttttaccctggcgctgtgaccgtggcctggaaggctgatagttctcc
tgtgaaggccggcgtggaaaccaccacaccttccaagcagtccaacaacaaatacgccgcctcctcctac
ctgtctctgacccctgaacagtggaagtcccaccggtcctactcttgccaagtgacccatgagggctcca
ccgtggaaaagacagtggcccctaccgagtgctcctaatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="88">
<INSDSeq>
<INSDSeq_length>431</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..431</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q175">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..431</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q176">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctcaggttcagttgcaacagtctggccctgaactcgtcagacccggcgtgtccgtgaagatcag
ctgtaaaggctccggctacaccttcaccgactacgccatgcactgggtcaagcagtctcacgccgagtct
ctggaatggatcggcctgatctctacctactacggcgacgccggctacaaccagaagttcaaggataagg
ccacaatgaccgtggacatctcctcctccaccgcctacctggaactggctagactgacctctgaggactc
cgccatctactactgcgccagagccgactcctctggcaacttctttgattactggggccagggcaccctg
gtcaccgtttctagtgcttccacaaagggccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="89">
<INSDSeq>
<INSDSeq_length>443</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..443</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q177">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..443</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q178">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctgaagttcagttgcagcagtctggccccgagcttgtgaaacctggcgcctctgtgaagatgtc
ctgcaaggcctctggctacaccttcgctcacaactggattcactgggtcaagcagaagccaggccaggga
cttgagtggatcggctacatcaaccctggcaccgactacaccgagtactcccagagattcaagggcaaag
ctaccctgacctccgacaagtcctcctccaccgcttacatggaactgtccagcctgacctctgaggactc
cgccgtgtactactgcgccagaagaaagctgtacggcaacttcgtggactacgccatggattattggggc
cagggcaccctggttaccgtgtctgctgcttctacaaagggccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="90">
<INSDSeq>
<INSDSeq_length>431</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..431</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q179">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..431</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q180">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctcaggttcagttgcaagagtctggccctggcctggtcaagccttctcagaccctgtctctgac
ctgcaccgtgtccggctattccatcacctccggctactactggaactggatcagacagcaccccggcaaa
ggcctggaatggatcggctacatctcttacgacggcggcaacaagtacaaccccagcctgaagtccagag
tgaccatctctcgggacacctccaagaaccagttctccctgaagctgtcctctgtgaccgctgccgatac
cgccgtgtactactgtgcccgcgatctggatggctacgagtctatggattattggggccagggcacctcc
gtgaccgtgtcctctgcttctacaaagggccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="91">
<INSDSeq>
<INSDSeq_length>434</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..434</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q181">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..434</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q182">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искуссвтенная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctcaggttcagttgcaagagtctggccctggcctggtcaagccttctcagaccctgtctctgac
ctgcaccgtgtccggctactccatcacctccacctactactggaactggatcagacagcaccccggcaaa
ggcctggaatggatcggctacatctcctacgacggcaccaacaagtacaaccccagcctgaagtccagag
tgaccatctctcgggacacctccaagaaccagttctccctgaagctgtcctctgtgaccgctgccgatac
cgccgtgtactactgcgccagagatgactacgacagatccccttggtttgcctattggggccagggcaca
ctggtcaccgtttccagtgcttctaccaagggccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="92">
<INSDSeq>
<INSDSeq_length>419</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..419</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q183">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..419</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q184">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggctggtcctgcatcattctgtttctggtggctaccgccaccggcg
tgcactctcaggttcagttgcaacagtctggccctgaactcgtcagacccggcgtgtccgtgaagatcag
ctgtaaaggctccggctacaccttcaccgattacgccctgcactgggtcaagcagtctcacgctgagtct
ctggaatggatcggcgtgatctccacctactacggcgacgcctcctacaaccagaagttcaaggacaagg
ccacaatgaccgtggacatctcctcctccaccgcctacctggaactggctagactgacctctgaggactc
cgccatctactactgcgccagagatggctacctggattattggggccagggcacactggtcaccgtgtcc
tctgcttctacaaagggccc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="93">
<INSDSeq>
<INSDSeq_length>976</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..976</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q185">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..976</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q186">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggcccctccgtgttccctctggccccttgctcccggtccacctccgag
tctaccgccgctctgggctgcctggtgaaagactacttccccgagcccgtgaccgtgtcctggaactctg
gcgccctgaccagcggcgtgcacaccttccctgccgtgctgcagtcctccggcctgtactccctgtcctc
cgtggtgaccgtgccctcctccagcctgggcaccaagacctacacctgtaacgtggaccacaagccctcc
aacaccaaggtggacaagcgggtggaatctaagtacggccctccctgccccccctgccctgcccctgaat
ttctgggcggaccttccgtgttcctgttccccccaaagcccaaggacaccctgatgatctcccggacccc
cgaagtgacctgcgtggtggtggacgtgtcccaggaagatcccgaggtgcagttcaattggtacgtggac
ggcgtggaagtgcacaacgccaagaccaagcccagagaggaacagttcaactccacctaccgggtggtgt
ctgtgctgaccgtgctgcaccaggactggctgaacggcaaagagtacaagtgcaaggtgtccaacaaggg
cctgccctccagcatcgaaaagaccatctccaaggccaagggccagccccgcgagccccaggtgtacacc
ctgccccctagccaggaagagatgaccaagaaccaggtgtccctgacctgtctggtgaaaggcttctacc
cctccgatatcgccgtggaatgggagtccaacggccagcccgagaacaactacaagaccaccccccctgt
gctggactccgacggctccttcttcctgtactctcggctgaccgtggacaagtcccggtggcaggaaggc
aacgtgttctcctgctccgtgatgcacgaggccctgcacaaccactacacccagaagtccctgtccctga
gcctgggcaagtgatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="94">
<INSDSeq>
<INSDSeq_length>976</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..976</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q187">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..976</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q188">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggcccctccgtgttccctctggccccttgctcccggtccacctccgag
tctaccgccgctctgggctgcctggtgaaagactacttccccgagcccgtgaccgtgtcctggaactctg
gcgccctgaccagcggcgtgcacaccttccctgccgtgctgcagtcctccggcctgtactccctgtcctc
cgtggtgaccgtgccctcctccagcctgggcaccaagacctacacctgtaacgtggaccacaagccctcc
aacaccaaggtggacaagcgggtggaatctaagtacggccctccctgccccccctgccctgcccctgaat
ttctgggcggaccttccgtgttcctgttccccccaaagcccaaggacaccctgatgatctcccggacccc
cgaagtgacctgcgtggtggtggacgtgtcccaggaagatcccgaggtgcagttcaattggtacgtggac
ggcgtggaagtgcacaacgccaagaccaagcctagagaggaacagttcaacagcacctacagagtggtgt
ccgtgctgaccgtgctgcaccaggattggctgaacggcaaagagtacaagtgcaaggtgtccaacaaggg
cctgggctccagcatcgaaaagaccatctccaaggctaagggccagcctcgggaacctcaggtttacacc
ctgcctccaagccaagaggaaatgaccaagaatcaggtgtccctgacctgtctcgtgaagggcttctacc
cttctgatatcgccgtggaatgggagtccaacggccagcctgagaacaactacaagaccacacctccagt
gctggactccgacggctctttcttcctgtattcccgcctgaccgtggacaagtccagatggcaagagggc
aacgtgttctcctgcagcgtgctgcatgaggctctgcactcccactacacccagaagtctctgtctctgt
ccctgggcaagtgataa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="95">
<INSDSeq>
<INSDSeq_length>327</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..327</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q189">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинантным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..327</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q190">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSG
VHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPS
VFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVL
HQDWLNGKEYKCKVSNKGLPSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAV
EWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVMHEALHNHYTQKSLSLSLGK</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="96">
<INSDSeq>
<INSDSeq_length>327</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..327</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q191">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sequences are synthesized by recombinant
method</INSDQualifier_value>
<NonEnglishQualifier_value>последовательности синтезированы
рекомбинанатным методом</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..327</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q192">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>Искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ASTKGPSVFPLAPCSRSTSESTAALGCLVKDYFPEPVTVSWNSGALTSG
VHTFPAVLQSSGLYSLSSVVTVPSSSLGTKTYTCNVDHKPSNTKVDKRVESKYGPPCPPCPAPEFLGGPS
VFLFPPKPKDTLMISRTPEVTCVVVDVSQEDPEVQFNWYVDGVEVHNAKTKPREEQFNSTYRVVSVLTVL
HQDWLNGKEYKCKVSNKGLGSSIEKTISKAKGQPREPQVYTLPPSQEEMTKNQVSLTCLVKGFYPSDIAV
EWESNGQPENNYKTTPPVLDSDGSFFLYSRLTVDKSRWQEGNVFSCSVLHEALHSHYTQKSLSLSLGK</
INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2018 |
|
RU2841528C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА И ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2821577C2 |
| ХИМЕРНЫЕ ЦИТОКИНОВЫЕ РЕЦЕПТОРЫ, НЕСУЩИЕ ЭКТОДОМЕН PD-1 | 2020 |
|
RU2828214C2 |
| МОНОКЛОНАЛЬНЫЕ АНТИТЕЛА ПРОТИВ РЕЦЕПТОР-СВЯЗЫВАЮЩЕГО ДОМЕНА SPIKE-БЕЛКА ВИРУСА SARS-COV-2 И ИХ АНТИГЕНСВЯЗЫВАЮЩИЕ ФРАГМЕНТЫ, КОДИРУЮЩИЕ ИХ НУКЛЕИНОВЫЕ КИСЛОТЫ, А ТАКЖЕ СПОСОБЫ ПРИМЕНЕНИЯ | 2021 |
|
RU2800649C2 |
| АНТИГЕННЫЕ ПЕПТИДЫ ДЛЯ ПРОФИЛАКТИКИ И ЛЕЧЕНИЯ В-КЛЕТОЧНЫХ ГЕМОБЛАСТОЗОВ | 2020 |
|
RU2833806C1 |
| Моноклональное антитело iC2 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2023 |
|
RU2817697C1 |
| АНТИТЕЛА ПРОТИВ IDE И ИХ ПРИМЕНЕНИЕ | 2020 |
|
RU2839026C1 |
| Моноклональное антитело iC1 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2023 |
|
RU2817696C1 |
| Штамм гибридных культивируемых клеток животных Mus musculus 5B9 - продуцент мышиного моноклонального антитела 5B9, антитело моноклональное мышиное 5В9 и антитело рекомбинантное химерное (мышь-человек) xi5В9, нейтрализующие рицин Ricinus communis | 2022 |
|
RU2802436C1 |
| ПЕПТИД, ПРИМЕНЯЕМЫЙ ДЛЯ ИММУНОТЕРАПЕВТИЧЕСКИХ СРЕДСТВ | 2021 |
|
RU2836172C1 |
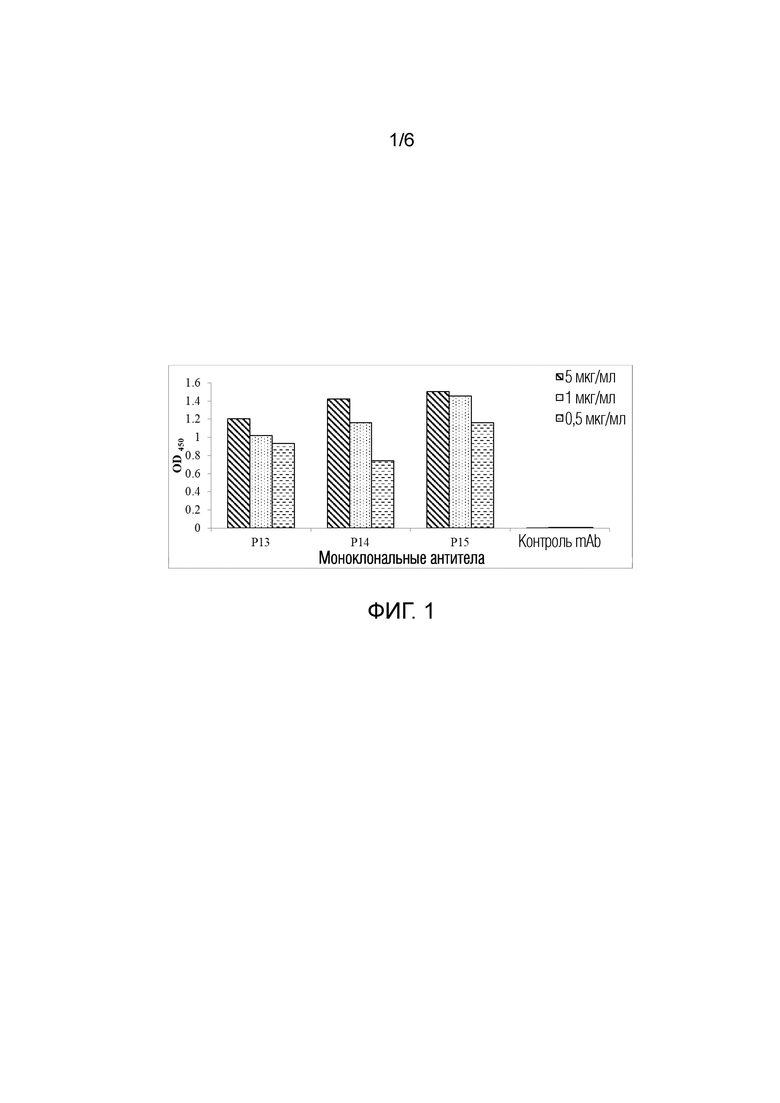
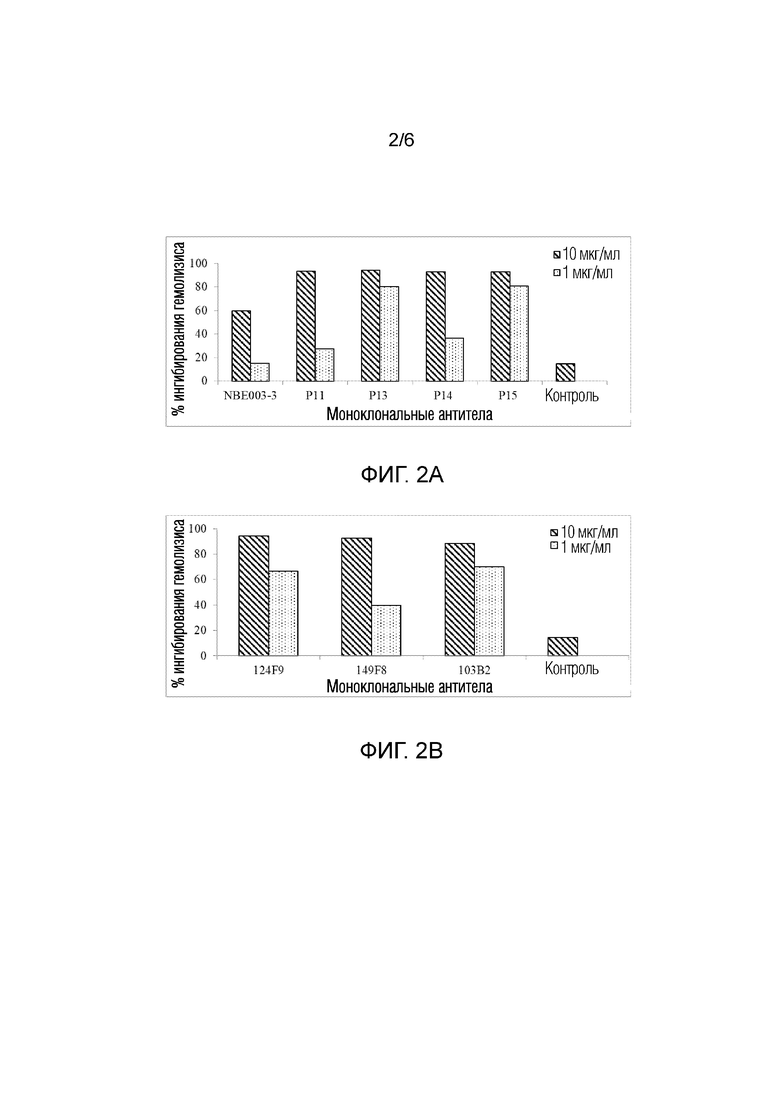
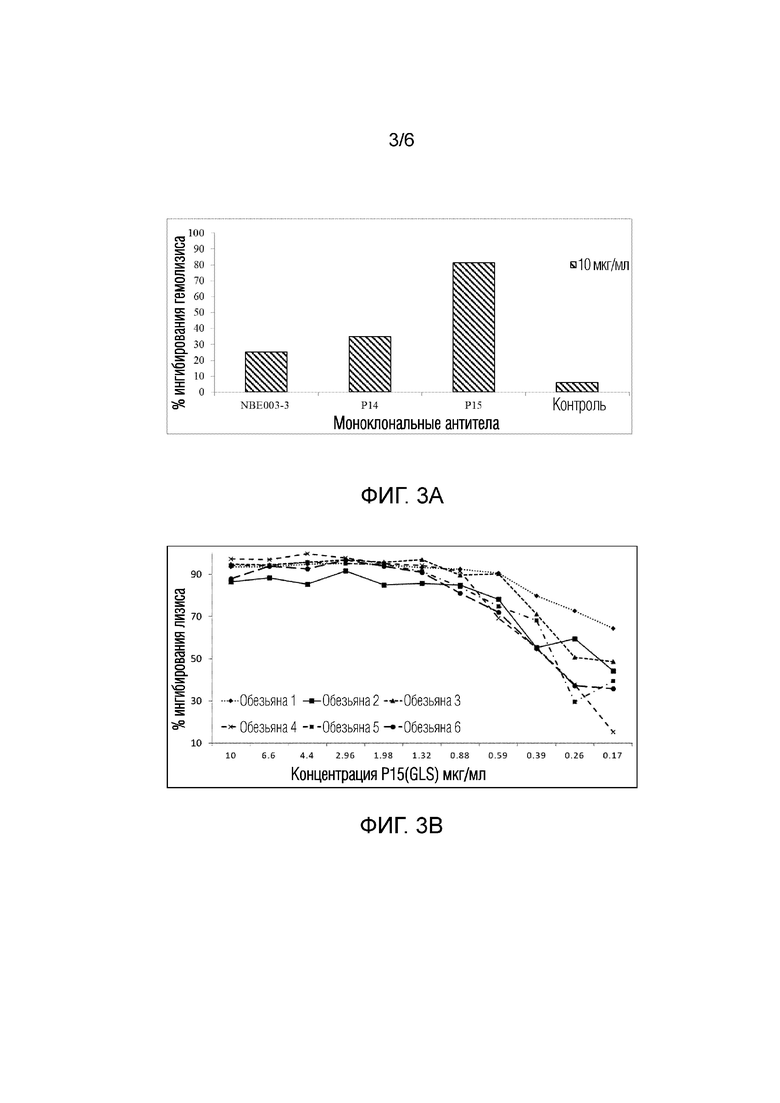
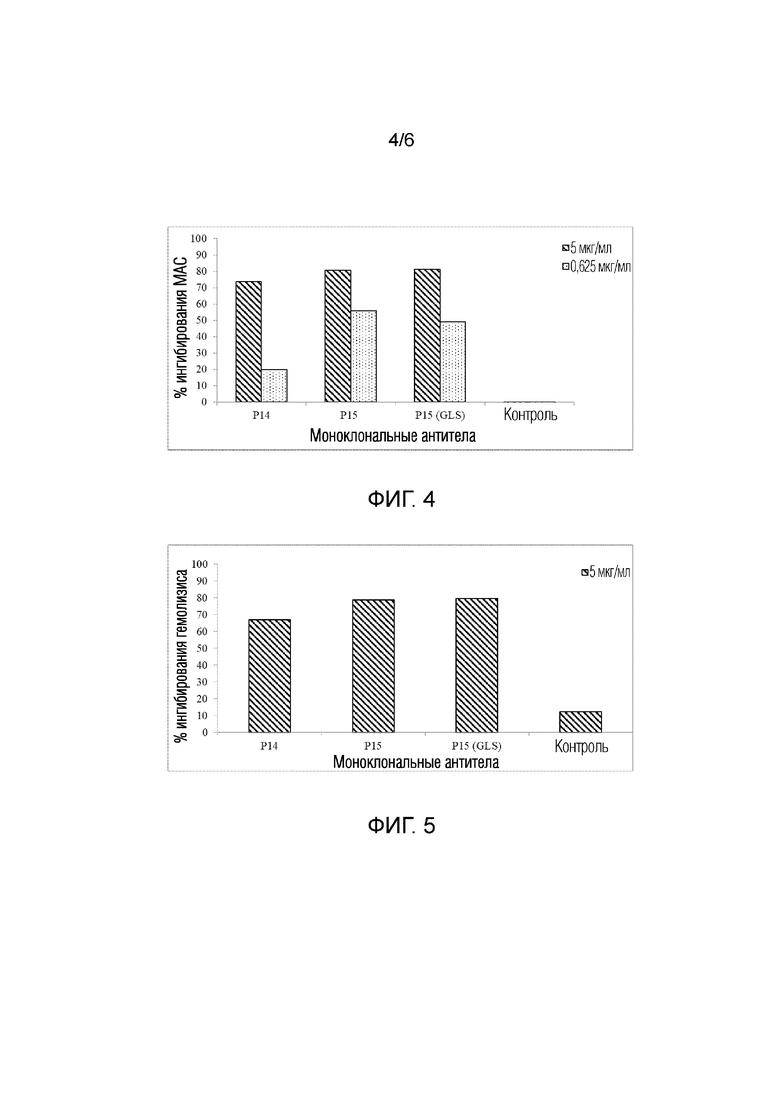
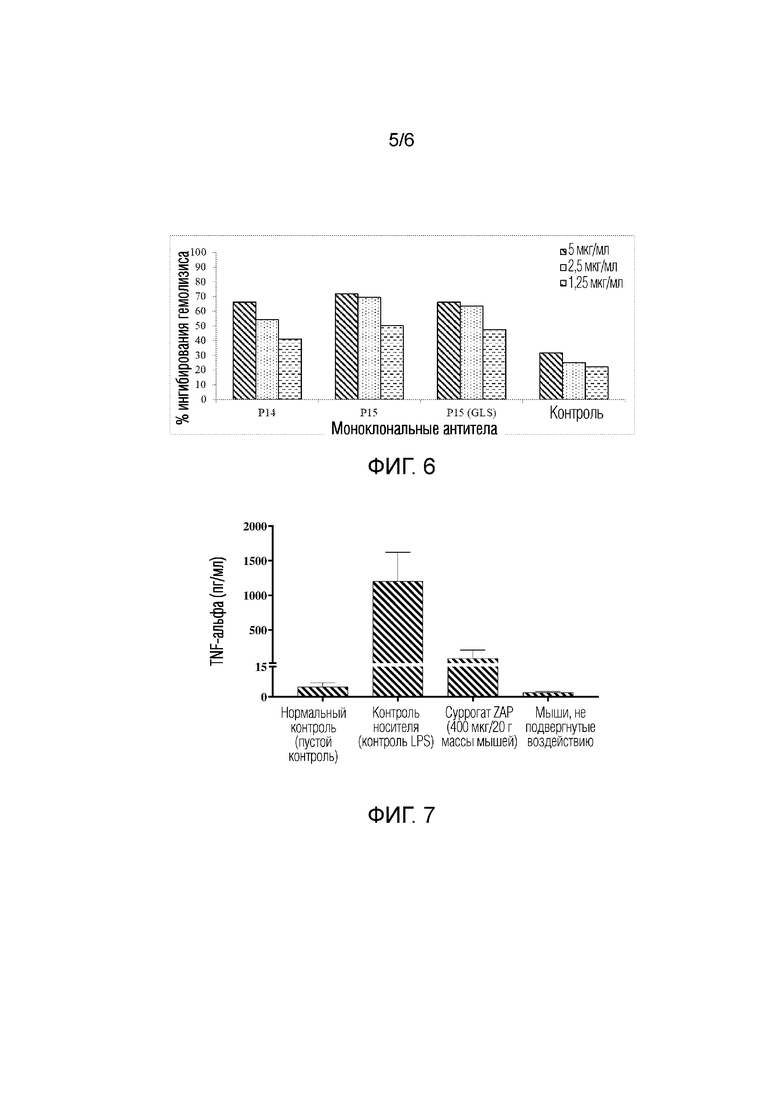
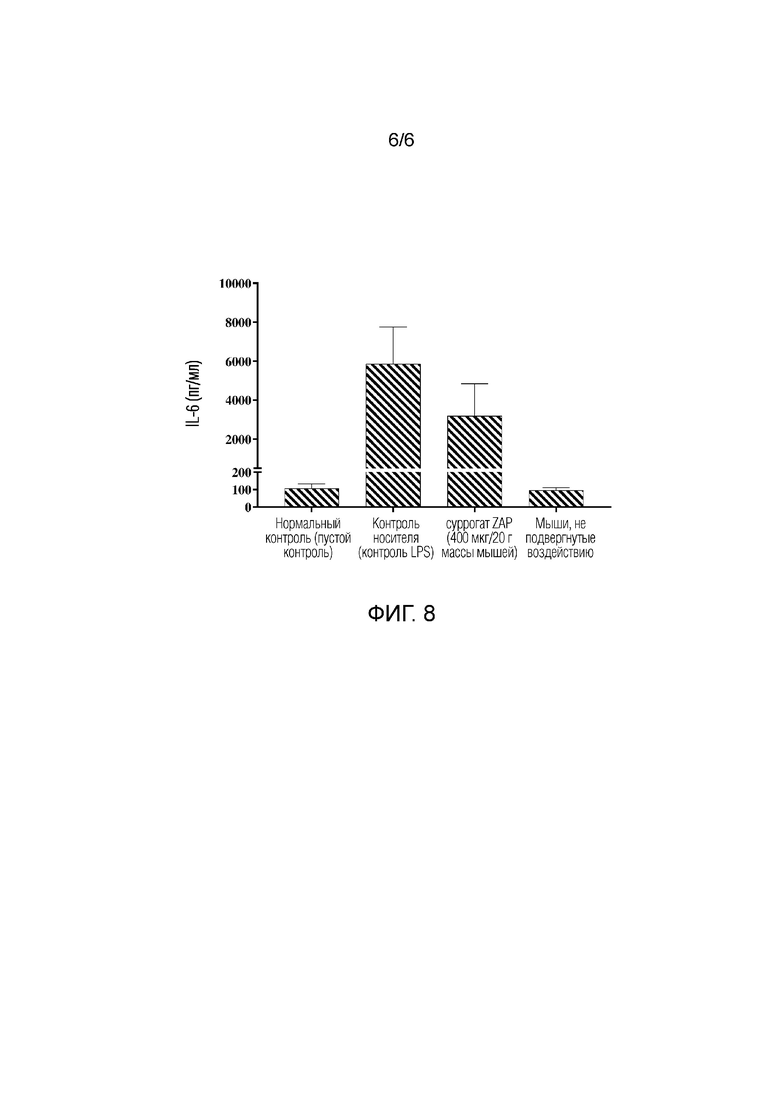
Изобретение относится к области биотехнологии. Предложены антитело против пропердина или его антигенсвязывающая часть и содержащий их иммуноконъюгат. Также предложена композиция, включающая указанные антитело или его антигенсвязывающую часть. Кроме того, изобретение относится к нуклеиновой кислоте, кодирующей указанное антитело, экспрессирующему вектору и клетке-хозяину, содержащим указанную нуклеиновую кислоту. Антитело может применяться для приготовления лекарственного средства для лечения заболеваний, опосредованных компонентом альтернативного пути комплемента и/или фактором, образующимся после активации альтернативного пути комплемента. Антитело по настоящему изобретению приводит к селективному ингибированию альтернативного пути комплемента, позволяя при этом продолжаться классическому и лектиновому путям. 7 н. и 11 з.п. ф-лы, 10 ил., 8 табл., 19 пр.
1. Антитело против пропердина или его антигенсвязывающая часть, содержащее:
(a) CDRH1, содержащую SEQ ID NO: 1, CDRH2, содержащую SEQ ID NO: 6, CDRH3, содержащую SEQ ID NO: 12, CDRL1, содержащую SEQ ID NO: 19, CDRL2, содержащую SEQ ID NO: 30 и CDRL3, содержащую SEQ ID NO: 35;
(b) CDRH1, содержащую SEQ ID NO: 3, CDRH2, содержащую SEQ ID NO: 9, CDRH3, содержащую SEQ ID NO: 15, CDRL1, содержащую SEQ ID NO: 22, CDRL2, содержащую SEQ ID NO: 33 и CDRL3, содержащую SEQ ID NO: 39;
(c) CDRH1, содержащую SEQ ID NO: 4, CDRH2, содержащую SEQ ID NO: 10, CDRH3, содержащую SEQ ID NO: 16, CDRL1, содержащую SEQ ID NO: 23, CDRL2, содержащую SEQ ID NO: 34 и CDRL3, содержащую SEQ ID NO: 40;
(d) CDRH1, содержащую SEQ ID NO: 1, CDRH2, содержащую SEQ ID NO: 7, CDRH3, содержащую SEQ ID NO: 13, CDRL1, содержащую KSSQSVLYSSNQKNYLA, CDRL2, содержащую SEQ ID NO: 31 и CDRL3, содержащую SEQ ID NO: 37; или
(e) CDRH1, содержащую SEQ ID NO: 2, CDRH2, содержащую SEQ ID NO: 8, CDRH3, содержащую SEQ ID NO: 14, CDRL1, содержащую SEQ ID NO: 20, CDRL2, содержащую SEQ ID NO: 31 и CDRL3, содержащую SEQ ID NO: 36.
2. Антитело или его антигенсвязывающая часть по п. 1, содержащее:
(a) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, которая по меньшей мере на 80% гомологична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 44, 46, 47, 48, 51, 52, 53 и 54;
(b) вариабельную область легкой цепи, содержащую аминокислотную последовательность, которая по меньшей мере на 80% гомологична аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 56, 57, 60, 61, 63, 64, 65 и 66.
3. Антитело или его антигенсвязывающую часть по п. 2 выбирают из группы, состоящей из:
(a) вариабельной области тяжелой цепи, содержащей аминокислотную последовательность SEQ ID NO: 51, и вариабельной области легкой цепи, содержащей аминокислотную последовательность SEQ ID NO: 63;
(b) вариабельной области тяжелой цепи, содержащей аминокислотную последовательность SEQ ID NO: 52, и вариабельной области легкой цепи, содержащей аминокислотную последовательность SEQ ID NO: 64;
(c) вариабельной области тяжелой цепи, содержащей аминокислотную последовательность SEQ ID NO: 53, и вариабельной области легкой цепи, содержащей аминокислотную последовательность SEQ ID NO: 65;
(d) вариабельной области тяжелой цепи, содержащей аминокислотную последовательность SEQ ID NO: 54, и вариабельной области легкой цепи, содержащей аминокислотную последовательность SEQ ID NO: 66;
(e) вариабельной области тяжелой цепи, содержащей аминокислотную последовательность SEQ ID NO: 46, и вариабельной области легкой цепи, содержащей аминокислотную последовательность SEQ ID NO: 57;
(f) вариабельной области тяжелой цепи, содержащей аминокислотную последовательность SEQ ID NO: 47, и вариабельной области легкой цепи, содержащей аминокислотную последовательность SEQ ID NO: 60;
(g) вариабельной области тяжелой цепи, содержащей аминокислотную последовательность SEQ ID NO: 48, и вариабельной области легкой цепи, содержащей аминокислотную последовательность SEQ ID NO: 61;
(h) вариабельной области тяжелой цепи, содержащей аминокислотную последовательность SEQ ID NO: 44, и вариабельной области легкой цепи, содержащей аминокислотную последовательность SEQ ID NO: 56.
4. Антитело или его антигенсвязывающая часть по любому из предыдущих пунктов, которое связывается с пропердином человека.
5. Антитело по любому из предыдущих пунктов, которое связывается с пропердином человека с KD 10-8 М или менее, предпочтительно 10-10 М или менее.
6. Антитело по любому из предыдущих пунктов, которое представляет собой изотип IgG, предпочтительно IgG1 или IgG4.
7. Антитело по любому из предыдущих пунктов, обладающее модифицированной или сниженной активностью ADCC и/или CDC или не имеет ее.
8. Антитело по любому из предыдущих пунктов, обладающее активностью ADCC и/или CDC.
9. Антитело по любому из предыдущих пунктов, содержащее одну аминокислотную замену, выбранную из S228P, P329G, M428L, N434S и их подходящей комбинации.
10. Антитело или его антигенсвязывающая часть по любому из предыдущих пунктов, где антитело или его антигенсвязывающая часть обладает по меньшей мере одной из следующих характеристик:
(a) перекрестно реактивно с пропердином других видов, кроме человека;
(b) более высокая специфичность связывания с пропердином человека;
(c) предотвращает повышенное связывание C3b с поверхностью клетки-мишени;
(d) блокирует функцию пропердина в опосредовании активации альтернативного пути комплемента;
(e) предотвращает комплемент-опосредованный лизис клеток-мишеней;
(f) регулирует образование MAC на поверхности клеток-мишеней и тем самым предотвращает лизис клеток;
(g) минимизирует образование анафилатоксинов С3а и С5а; и
(h) повышенный период полувыведения у пациента.
11. Антитело или его антигенсвязывающая часть по любому из предыдущих пунктов, которое является мышиным, химерным, рекомбинантным или гуманизированным по своей природе, предпочтительно гуманизированным по своей природе.
12. Композиция для лечения заболеваний, опосредованных, прямо или косвенно, компонентом альтернативного пути комплемента и/или фактором, образующимся после активации альтернативного пути комплемента, содержащая антитело или его антигенсвязывающую часть по любому из пп. 1-11 и фармацевтически приемлемый носитель.
13. Иммуноконъюгат против пропердина, содержащий антитело или его антигенсвязывающую часть по любому из пп. 1-11, связанное с терапевтическим средством.
14. Иммуноконъюгат по п. 13, где терапевтическое средство представляет собой цитотоксин или радиоактивный изотоп.
15. Нуклеиновая кислота, кодирующая антитело или его антигенсвязывающую часть по любому из пп. 1-11.
16. Экспрессирующий вектор, содержащий последовательность нуклеиновой кислоты по п. 15.
17. Клетка-хозяин, экспрессирующая антитело против пропердина или его антигенсвязывающую часть, содержащая экспрессирующий вектор по п. 16.
18. Применение антитела или его антигенсвязывающей части по любому из пп. 1-13 для лечения заболеваний, опосредованных, прямо или косвенно, компонентом альтернативного пути комплемента и/или фактором, образующимся после активации альтернативного пути комплемента.
| WO 2009110918 A1, 11.09.2009 | |||
| WO 2018140956 A1, 02.08.2018 | |||
| WO 2011109494 A2, 09.09.2011 | |||
| CHEN J.Y | |||
| et al | |||
| Properdin: A multifaceted molecule involved in inflammation and diseases, Molecular Immunology, 2018, v | |||
| Транспортер для перевозки товарных вагонов по трамвайным путям | 1919 |
|
SU102A1 |
| Способ окисления боковых цепей ароматических углеводородов и их производных в кислоты и альдегиды | 1921 |
|
SU58A1 |
| СЕРДОБИНЦЕВ К.В | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Механический грохот | 1922 |
|
SU41A1 |
Авторы
Даты
2024-11-18—Публикация
2021-01-08—Подача