Область изобретения
Настоящее изобретение относится к микроорганизму, содержащему указанный вариант, способу получения ретиноида с использованием указанного микроорганизма, композиции для получения ретиноида и применению указанного варианта или микроорганизма для получения ретиноида.
Предшествующий уровень техники
Ретиноиды, которые включают витамин А, жирорастворимый витамин и незаменимое питательное вещество, в настоящее время получают путем химического синтеза, и они доступны для приобретения. Однако по таким причинам, как обеспечение сырьем/потребность в сырье и т.д., проводятся различные исследования для получения ретиноидов на основе микробной ферментации (т.е. разработка микроорганизмов, способных к высокоэффективному продуцированию ретиноидов, и технологии процесса ферментации), например специфичного для целевого вещества подхода, такого как повышение экспрессии генов, кодирующих ферменты, участвующие в биосинтезе ретиноидов, или удаление генов, не требующихся для биосинтеза ретиноидов. Примером ферментов, участвующих в биосинтезе ретиноидов, является бета-каротин-15,15'оксигеназа и т.д. (US 2020-0277644 А1).
Однако бета-каротин-15,15'-оксигеназа изучена недостаточно, и сохраняется необходимость в исследовании повышения способности к эффективному продуцированию витамина А для удовлетворения возрастающей потребности в витамине А.
Краткое изложение сущности изобретения
Техническая задача
Задача настоящего изобретения состоит в предложении варианта бета-каротин-15,15'-оксигеназы и способа получения ретиноида с его использованием.
Техническое решение
Задача настоящего изобретения состоит в предложении варианта бета-каротин-15,15'-оксигеназы.
Еще одна задача настоящего изобретения состоит в предложении полинуклеотида, кодирующего указанный вариант по настоящему изобретению.
Дополнительная задача настоящего изобретения состоит в предложении микроорганизма, который содержит указанный вариант по настоящему изобретению или полинуклеотид, кодирующий указанный вариант.
Другая задача настоящего изобретения состоит в предложении способа получения ретиноида, который включает культивирование микроорганизма, содержащего указанный вариант по настоящему изобретению или полинуклеотид, кодирующий указанный вариант, в среде.
Еще одна задача настоящего изобретения состоит в предложении композиции для получения ретиноида, которая содержит указанный вариант по настоящему изобретению, микроорганизм, содержащий полинуклеотид, кодирующий указанный вариант, или его культуру.
Дополнительная задача настоящего изобретения состоит в предложении применения любого одного или более из варианта по настоящему изобретению, полинуклеотида, кодирующего указанный вариант, и микроорганизма, его содержащего.
Полезные эффекты
В случае культивирования микроорганизма, содержащего указанный вариант бета-каротин-15,15'-оксигеназы по настоящему изобретению, можно получить более высокий выход ретиноида по сравнению с микроорганизмом, содержащим существующий немодифицированный полипептид.
Подробное описание изобретения
Ниже приведено подробное описание настоящего изобретения. Тем не менее соответствующие описания и воплощения, раскрытые в настоящей заявке, также могут быть применены к другим описаниям и воплощениям. Т.е. все комбинации различных элементов, описанных в настоящей заявке, входят в объем настоящего изобретения. Дополнительно, объем настоящего изобретения не ограничен нижеприведенным специфическим описанием. Дополнительно, в тексте настоящего описания изобретения имеются ссылки на ряд статей и патентных документов, и обозначено их цитирование. Содержание цитируемых документов и патентных документов включено в настоящее описание изобретения путем ссылки в полном объеме, а уровень области техники, к которой принадлежит настоящее изобретение, и содержание настоящего изобретения описаны более ясно.
В одном аспекте настоящего изобретения предложен вариант бета-каротин-15,15'-оксигеназы, в котором любые одна или более аминокислот среди аминокислоты, соответствующей положению 43 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, аланина; аминокислоты, соответствующей положению 58 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, лейцина; и аминокислоты, соответствующей положению 74 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, изолейцина, каждая заменена на другую аминокислоту.
Вариант бета-каротин-15,15'-оксигеназы может представлять собой полипептид, обладающий активностью бета-каротин-15,15'-оксигеназы, или полипептид, в котором любые одна или более аминокислот среди аминокислот, соответствующих положениям 43, 58 и 74 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, заменены на другую аминокислоту. В частности, указанный вариант бета-каротин-15,15'-оксигеназы может представлять собой вариант, в котором аминокислоты в одном или более, двух или более или трех или более положениях из положений 43, 58 и 74 могут быть заменены, но не ограничивается этим.
В полипептиде, обладающем активностью бета-каротин-15,15'-оксигеназы, или в бета-каротин-15,15'-оксигеназе, которая является исходной для варианта, аминокислота, соответствующая положению 43 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, аминокислота, соответствующая положению 43, может представлять собой аланин; аминокислота, соответствующая положению 58, может представлять собой лейцин; и аминокислота, соответствующая положению 74, может представлять собой изолейцин, но указанный полипептид не ограничивается этим.
В одном воплощении указанный вариант может содержать одну или более замен из замены аминокислоты, соответствующей положению 43 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, на аминокислоту, отличную от аланина; замены аминокислоты, соответствующей положению 58 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, на аминокислоту, отличную от лейцина; и замены аминокислоты, соответствующей положению 74 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, на аминокислоту, отличную от изолейцина, но настоящее изобретение не ограничивается этим.
На «другую аминокислоту» не налагаются ограничения, при условии, что это аминокислота, отличная от аминокислоты до замены. При этом в настоящей заявке при использовании выражения «специфическая аминокислота заменена» очевидно, что указанная аминокислота заменена на аминокислоту, которая отличается от аминокислоты до замены, даже если это не описано как замена на другую аминокислоту.
Замена на другую аминокислоту может представлять собой замену на неполярную аминокислоту, полярную аминокислоту или положительно заряженную (основную) аминокислоту. В частности, неполярная аминокислота может быть выбрана из группы, состоящей из глицина, аланина, валина, лейцина, изолейцина, метионина и пролина, но не ограничивается этим. Полярная аминокислота может быть использована взаимозаменяемо с гидрофильной аминокислотой и может быть выбрана из группы, состоящей из серина, треонина, цистеина, тирозина, аспарагина и глутамина, и положительно заряженная (основная) аминокислота может быть выбрана из группы, состоящей из аргинина, лизина и гистидина, но не ограничивается этим.
Вариант по настоящему изобретению может представлять собой вариант, в котором любые одна или более из аминокислот, соответствующих положениям 43, 58 и 74 в аминокислотной последовательности SEQ ID NO: 1, которая является референсным белком, могут быть заменены на аминокислоту, отличную от аминокислоты до замены, из неполярных аминокислот, полярных аминокислот или положительно заряженных (основных) аминокислот, но не ограничивается этим.
В одном воплощении замена аминокислоты, соответствующей положению 43, на другую аминокислоту может представлять собой замену на неполярную аминокислоту; замена аминокислоты, соответствующей положению 58, на другую аминокислоту может представлять собой замену на полярную аминокислоту; и замена аминокислоты, соответствующей положению 74, на другую аминокислоту может представлять собой замену на неполярную аминокислоту, но не ограничивается этим.
Указанная неполярная аминокислота может представлять собой валин и указанная полярная аминокислота может представлять собой глутамин, но не ограничиваются этим.
Указанная другая аминокислота может представлять собой валин или глутамин, но не ограничивается этим.
В одном воплощении вариант по настоящему изобретению может представлять собой вариант, в котором замена выбрана из группы, состоящей из замены аминокислоты, соответствующей положению 43 аминокислотной последовательности SEQ ID NO: 1, на валин; замены аминокислоты, соответствующей положению 58 аминокислотной последовательности SEQ ID NO: 1, на глутамин; замены аминокислоты, соответствующей положению 74 аминокислотной последовательности SEQ ID NO: 1, на валин; и их комбинации, но не ограничивается этим.
В одном воплощении вариант по настоящему изобретению может содержать относительно N-конца аминокислотной последовательности SEQ ID NO: 1: 1) замену аминокислоты, соответствующей положению 43, аланина, на валин; 2) замену аминокислоты, соответствующей положению 58, лейцина, на глутамин; 3) замену аминокислоты, соответствующей положению 74, изолейцина, на валин; 4) замену аминокислоты, соответствующей положению 43, аланина, на валин; и замену аминокислоты, соответствующей положению 58, лейцина, на глутамин; 5) замену аминокислоты, соответствующей положению 58, лейцина, на глутамин; и замену аминокислоты, соответствующей положению 74, изолейцина, на валин; 6) замену аминокислоты, соответствующей положению 43, аланина, на валин; и замену аминокислоты, соответствующей положению 74, изолейцина, на валин; или 7) замену аминокислоты, соответствующей положению 43, аланина, на валин; замену аминокислоты, соответствующей положению 58, лейцина, на глутамин; и замену аминокислоты, соответствующей положению 74, изолейцина, на валин, но не ограничивается этим.
В одном воплощении вариант по настоящему изобретению может представлять собой полипептид, содержащий аминокислотную последовательность, представленную SEQ ID NO: 3, в которой аминокислота, соответствующая положению 43 аминокислотной последовательности SEQ ID NO: 1, аланин, заменена на валин; аминокислота, соответствующая положению 58 аминокислотной последовательности SEQ ID NO: 1, лейцин, заменена на глутамин; и аминокислота, соответствующая положению 74, изолейцин аминокислотной последовательности SEQ ID NO: 1, заменена на валин, но не ограничивается этим.
Вариант по настоящему изобретению может иметь, содержать, состоять из или по существу состоять из аминокислотной последовательности, представленной SEQ ID NO: 3.
Дополнительно, вариант по настоящему изобретению может содержать аминокислотную последовательность, в которой в аминокислотной последовательности, представленной SEQ ID NO: 3, аминокислота, соответствующая положению 43, установлена как валин, аминокислота, соответствующая положению 58, установлена как глутамин и аминокислота, соответствующая положению 74, установлена как валин на основе аминокислотной последовательности SEQ ID NO: 1, и которая обладает гомологией или идентичностью по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,5%, 99,7% или 99,9% или более относительно аминокислотной последовательности, представленной SEQ ID NO: 3. Дополнительно, также очевидно, что вариант, имеющий аминокислотную последовательность, содержащую делецию, модификацию, замену, консервативную замену или добавление в части последовательности, также входит в объем настоящего изобретения, если указанная аминокислотная последовательность обладает вышеназванной гомологией или идентичностью в дополнение к каждому из установленных положений последовательности и демонстрирует эффективность, соответствующую таковой у варианта по настоящему изобретению.
Примеры этого включают вариант, содержащий добавление или делецию последовательности, которые не изменяют функцию варианта по настоящему изобретению, на N-конце, С-конце аминокислотной последовательности и/или внутри аминокислотной последовательности, мутацию природного происхождения, молчащую мутацию или консервативную замену.
Термин «консервативная замена» означает замену одной аминокислоты на другую аминокислоту, обладающую схожими структурными и/или химическими свойствами. Такая аминокислотная замена может в общем случае возникать на основе сходства по полярности, заряду, растворимости, гидрофобности, гидрофильности и/или амфипатической природе остатков. Обычно консервативная замена практически не влияет или не влияет на активность белков или полипептидов.
При использовании здесь термин «вариант» означает полипептид, который имеет аминокислотную последовательность, отличную от таковой варианта до модификации посредством консервативной замены и/или модификации одной или более аминокислот, но сохраняет функции или свойства. Такой вариант может в общем случае быть идентифицирован путем модификации одной или более аминокислот аминокислотной последовательности полипептида и оценки свойств модифицированного полипептида. Т. е. способность указанного варианта может быть повышена, остаться неизменной или быть понижена по сравнению с таковой у полипептида до модификации. Дополнительно, некоторые варианты могут включать варианты, в которых одна или более частей, таких как N-концевая лидерная последовательность или трансмембранный домен, были удалены. Другие варианты могут включать варианты, в которых часть N- и/или С-конца была удалена из зрелого белка. Термин «вариант» может быть использован взаимозаменяемо с такими терминами, как модификация, модифицированный полипептид, модифицированный белок, мутант, мутеин и дивергент, и не ограничивается этими терминами, при условии, что это термин, используемый со значением вариации.
Дополнительно, вариант может содержать делеции или добавления аминокислот, которые оказывают минимальное действие на свойства и вторичную структуру полипептида. Например, сигнальная (или лидерная) последовательность, которая котрансляционно или посттрансляционно участвует в транслокации белка, может быть конъюгирована с N-концом варианта. Вариант может быть конъюгирован с другими последовательностями или линкерами таким образом, чтобы можно было подтвердить его идентичность, очистить или синтезировать.
При использовании здесь термин «гомология» или «идентичность» означает степень сходства между двумя данными аминокислотными последовательностями или нуклеотидными последовательностями и может быть выражен в процентах. Термины «гомология» и «идентичность» могут быть использованы взаимозаменяемо.
Гомология или идентичность последовательности консервативных полинуклеотидов или полипептидов может быть определена посредством стандартного алгоритма выравнивания, и вместе с ним могут быть использованы штрафы за пропуски по умолчанию, установленные в применяемой программе. По существу, гомологичные или идентичные последовательности могут в общем случае гибридизоваться друг с другом целиком или частично в умеренных или очень жестких условиях. Очевидно, что гибридизация также включает гибридизацию с полинуклеотидом, содержащим общие кодоны или кодоны, отвечающие принципам вырожденности кодонов в полинуклеотиде.
Определить, имеют ли любые две полинуклеотидные или полипептидные последовательности гомологию, сходство или идентичность, можно, например, с помощью известного компьютерного алгоритма, такого как программа «FASTA», используя параметры по умолчанию, как в публикации Pearson et al. (1988) Proc. Natl. Acad. Sci. USA 85:2444. В соответствии с другим вариантом, это можно определить с помощью алгоритма Нидлмана-Вунша (Needleman and Wunsch, 1970, J. Mot. Biol. 48:443-453), выполняемого в программе Needleman из пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al, 2000, Trends Genet. 16:276-277) (версия 5.0.0 или более поздняя) (включая пакет программ GCG (Devereux, J., et al., Nucleic Acids Research 12:387 (1984)), BLASTP, BLASTN, FASTA (Atschul, S. F., et al., J. Mot. Biol. 215:403 (1990); Guide to Huge Computers, Martin J. Bishop, ed., Academic Press, San Diego, 1994; и CARILLO et al. (1988) SI AM J Applied Math 48:1073). Например, гомология, сходство или идентичность могут быть определены с помощью BLAST от Национального центра биотехнологической информации (National Center for Biotechnology Information) или ClustalW.
Гомология, сходство или идентичность между полинуклеотидами или полипептидами могут быть определены путем сравнения информации о последовательностях с использованием компьютерной программы GAP, как предложено, например, в публикации Needleman et al. (1970), J Mol Biol. 48:443, как описано в публикации Smith and Waterman, Adv. Appl. Math (1981) 2:482. Вкратце, программа GAP определяет гомологию, сходство или идентичность как значение, полученное делением количества одинаковых выровненных символов (т.е. нуклеотидов или аминокислот) на общее количество символов в более короткой из двух последовательностей. Параметры по умолчанию для программы GAP могут включать: (1) бинарную матрицу сравнения (содержащую значение 1 для идентичности и значение 0 для неидентичности) и взвешенную матрицу сравнения по Gribskov et al. (1986) Nucl. Acids Res. 14:6745, как описано в Schwartz and Dayhoff, eds., Atlas of Protein Sequence And Structure, National Biomedical Research Foundation, pp.353-358 (1979) (или матрицу замен EDNAFULL (EMBOSS версии NCBI NUC4.4)); (2) штраф 3,0 за каждый пропуск и дополнительный штраф 0,10 за каждый символ в каждом пропуске (или штраф за открытие пропуска 10 и штраф за продление пропуска 0,5); и (3) отсутствие штрафа за концевые пропуски.
В одном воплощении настоящего изобретения вариант по настоящему изобретению может обладать ферментативной активностью бета-каротин-15,15'-оксигеназы. Дополнительно, вариант по настоящему изобретению может обладать активностью, которая повышает способность продуцировать ретиноид по сравнению с полипептидом дикого типа, обладающим активностью15,15'-оксигеназы.
При использовании здесь термин «бета-каротин-15,15'-оксигеназа» означает каталитическую функцию в отношении действия по расщеплению одной молекулы бета-каротина на две молекулы ретиналя.
Бета-каротин-15,15'-оксигеназа по настоящему изобретению может представлять собой бета-каротин-15,15'-оксигеназу или полипептид, обладающий активностью бета-каротин-15,15'-оксигеназы, к которому применена модификация для получения варианта бета-каротин-15,15'-оксигеназы, предложенного в настоящем изобретении. В частности, бета-каротин-15,15'-оксигеназа может являться полипептидом природного происхождения или полипептидом дикого типа, может являться его зрелым полипептидом и может включать его вариант или функциональный фрагмент, но любая бета-каротин-15,15'-оксигеназа может быть включена без ограничений, при условии, что она может быть исходной для варианта бета-каротин-15,15'-оксигеназы по настоящему изобретению.
В настоящем изобретении бета-каротин-15,15'-оксигеназа может представлять собой полипептид с SEQ ID NO: 1, но не ограничивается этим. Дополнительно, бета-каротин-15,15'-оксигеназа может представлять собой полипептид, имеющий идентичность последовательности около 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% или более относительно полипептида с SEQ ID NO: 1, и любой из них может быть отнесен к бета-каротин-15,15'-оксигеназе без ограничения при условии, что он обладает активностью, идентичной или соответствующей таковой полипептида, состоящего из аминокислотной последовательности SEQ ID NO: 1.
В частности, термин бета-каротин-15,15'-оксигеназа по настоящему изобретению может быть использован взаимозаменяемо с терминами ВСО или Blh. В настоящем изобретении последовательность бета-каротин-15,15'-оксигеназы может быть получена из NCBI GenBank, который является известной базой данных. В частности, бета-каротин-15,15'-оксигеназа может представлять собой полипептид, кодируемый blh и обладающий активностью бета-каротин-15,15'-оксигеназы, но не ограничивается этим.
При использовании здесь термин «соответствующий» относится к аминокислотным остаткам в положениях, перечисленных в полипептиде, или аминокислотным остаткам, которые являются сходными, идентичны или гомологичны таковым, перечисленным в полипептиде. Идентификация аминокислоты в соответствующем положении может представлять собой определение конкретной аминокислоты в последовательности, которая относится к конкретной последовательности. При использовании в этой заявке «соответствующая область» обычно означает сходное или соответствующее положение в родственном белке или референсном белке.
Например, после выравнивания случайной аминокислотной последовательности с SEQ ID NO: 1, исходя из этого, каждый аминокислотный остаток аминокислотной последовательности может быть пронумерован с учетом номера положения аминокислотного остатка, соответствующего аминокислотному остатку в SEQ ID NO: 1. Например, алгоритм выравнивания последовательностей, описанный в настоящем изобретении, может позволить определить положение аминокислоты или положение, в котором может происходить модификация, такая как замена, вставка и делеция, путем сравнения с таковым в используемой для запроса последовательности (также называемой «референсной последовательностью»).
Для таких выравниваний могут быть использованы, например, алгоритм Нидлмана-Вунша (Needleman and Wunsch, 1970, J. Mol. Biol. 48:443-453), программа Needleman из пакета EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al., 2000), Trends Genet. 16:276-277) и т.д., но не ограничиваясь этим, и соответствующим образом могут быть использованы программа выравнивания последовательностей, алгоритм попарного сравнения последовательностей и т.д., которые известны специалистам в данной области техники.
В соответствии с еще одним аспектом настоящего изобретения предложен полинуклеотид, кодирующий вариант по настоящему изобретению.
При использовании здесь термин «полинуклеотид» означает нить ДНК или РНК, обладающую определенной длиной или более, в виде полимера из нуклеотидов, в котором нуклеотидные мономеры соединены в длинную цепь ковалентными связями, и более конкретно, термин относится к полинуклеотидному фрагменту, кодирующему указанный вариант.
В одном воплощении полинуклеотид, кодирующий вариант по настоящему изобретению, может включать нуклеотидную последовательность, кодирующую аминокислотную последовательность, представленную SEQ ID NO: 3. В одном воплощении настоящего изобретения полинуклеотид по настоящему изобретению может иметь или содержать последовательность SEQ ID NO: 4. Дополнительно, полинуклеотид по настоящему изобретению может состоять или по существу состоять из последовательности SEQ ID NO: 4.
В полинуклеотид по настоящему изобретению могут быть внесены различные модификации в кодирующей области при условии, что аминокислотная последовательность варианта по настоящему изобретению не изменяется, с учетом вырожденности кодонов или кодонов, предпочтительных в организме, который, как предполагается, будет экспрессировать вариант по настоящему изобретению. В частности, полинуклеотид по настоящему изобретению может иметь или содержать нуклеотидную последовательность, обладающую 70% или более, 75% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более и менее чем 100% гомологией или идентичностью по отношению к последовательности SEQ ID NO: 4, или может состоять или по существу состоять из нуклеотидной последовательности, обладающей 70% или более, 75% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более и менее чем 100% гомологией или идентичностью по отношению к последовательности SEQ ID NO: 4, но не ограничивается этим.
В частности, в последовательности, обладающей вышеуказанной гомологией или идентичностью, кодоны, кодирующие аминокислоту, соответствующую положению 43 в SEQ ID NO: 3, могут представлять собой любой из кодонов, кодирующих валин; кодоны, кодирующие аминокислоту, соответствующую положению 58 в SEQ ID NO: 3, могут представлять собой любой из кодонов, кодирующих глутамин; и кодоны, кодирующие аминокислоту, соответствующую положению 74 в SEQ ID NO: 3, могут представлять собой любой из кодонов, кодирующих валин.
Дополнительно, полинуклеотид по настоящему изобретению может включать зонд, который может быть получен из известной последовательности гена, например, последовательности без ограничений, при условии, что она является последовательностью, которая способна гибридизоваться с последовательностью, комплементарной к целой полинуклеотид ной последовательности по настоящему изобретению или ее части в жестких условиях. Термин «жесткие условия» означает условия, которые обеспечивают специфическую гибридизацию между полинуклеотидами. Такие условия, в частности, описаны в документах (см. J. Sambrook et at, Molecular Cloning, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York, 9.50-9.51, 11.7-11.8). Их примеры включают условие, при котором полинуклеотиды, обладающие более высокой гомологией или идентичностью, т.е. полинуклеотиды, обладающие 70% или более, 75% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более или 99% или более гомологией или идентичностью, гибридизуются друг с другом, тогда как полинуклеотиды, обладающие более низкой гомологией или идентичностью, не гибридизуются друг с другом, или условие, при котором промывка проводится однократно, в частности, два или три раза при концентрации соли и температуре, эквивалентных 60°С, 1×SSC, 0,1% ДСН, в частности, 60°С, 0,1×SSC, 0,1% ДСН, и более конкретно 68°С, 0,1×SSC, 0,1% ДСН, что является условиями промывки для стандартной гибридизации по Саузерну.
Для гибридизации необходимо, чтобы две нуклеиновые кислоты имели комплементарные последовательности, хотя возможно отсутствие спаривания между основаниями, в зависимости от степени жесткости условий. Термин «комплементарность» используют для описания отношения между нуклеотидными основаниями, способными гибридизоваться друг с другом. Например, в случае ДНК, аденин комплементарен тимину, а цитозин комплементарен гуанину. Следовательно, полинуклеотид по настоящему изобретению может включать изолированные фрагменты нуклеиновой кислоты, которые комплементарны целой последовательности, а также по существу подобным последовательностям нуклеиновых кислот.
В частности, полинуклеотид, обладающий гомологией или идентичностью по отношению к полинуклеотиду по настоящему изобретению, может быть выявлен с использованием условия гибридизации, включающего этап гибридизации при значении Tm 55°С и вышеописанных условиях. Дополнительно, значение Tm может составлять 60°С, 63°С или 65°С, но не ограничивается этим, и может быть соответствующим образом скорректировано специалистами в данной области техники в соответствии с задачей.
Соответствующая степень жесткости для гибридизации полинуклеотида зависит от длины и степени комплементарности указанного полинуклеотида, и эти переменные хорошо известны специалистам в данной области техники (например, J. Sambrook et al., см. выше).
В соответствии с еще одним аспектом настоящего изобретения предложен вектор, содержащий полинуклеотид по настоящему изобретению. Указанный вектор может представлять собой экспрессионный вектор для экспрессии полинуклеотида в клетке-хозяине, но не ограничивается этим.
Вектор по настоящему изобретению может включать ДНК-конструкцию, содержащую нуклеотидную последовательность полинуклеотида, кодирующего целевой полипептид, которая функционально связана с подходящей областью регуляции экспрессии (или последовательности регуляции экспрессии) таким образом, чтобы целевой полипептид можно было экспрессировать в подходящем хозяине. Область регуляции экспрессии может включать промотор, способный инициировать транскрипцию, любую последовательность оператора для регуляции транскрипции, последовательность, кодирующую подходящий сайт связывания рибосомы на мРНК, и последовательность, регулирующую терминацию транскрипции и трансляции. Указанным вектором можно трансформировать подходящую клетку-хозяина, и затем он может реплицироваться и функционировать независимо от генома хозяина или может интегрироваться в сам геном.
Вектор, используемый в настоящем изобретении, специально не ограничен, и может быть использован любой вектор, известный специалистам в данной области техники. Примеры часто используемых векторов включают природные или рекомбинантные плазмиды, космиды, вирусы и бактериофаги. Например, pWE15, М13, MBL3, MBL4, IXII, ASHII, APII, t10, t11, Charon4A, Charon21A и т.д. могут быть использованы в качестве фагового вектора или космидного вектора, а система pDZ, система pBR, система pUC, система pBluescript II, система pGEM, система pTZ, система pCL, система рЕТ и т.д. могут быть использованы в качестве плазмидного вектора. В частности, могут быть использованы векторы pDZ, pDC, pDCM2 (публикация заявки на патент Кореи №10-2020-0136813), pACYC177, pACYC184, pCL, pECCG117, pUC19, pBR322, pMW118, pCC1BAC, pIMR53 и т.д.
В одном воплощении полинуклеотид, кодирующий целевой полипептид, может быть вставлен в хромосому посредством вектора для внутриклеточной вставки в хромосому. Вставка указанного полинуклеотида в хромосому может быть осуществлена любым способом, известным в данной области техники, например, гомологичной рекомбинацией, но не ограничивается этим. Указанный вектор может дополнительно содержать селектируемый маркер для подтверждения вставки в хромосому. Селектируемый маркер служит для отбора клеток, трансформированных вектором, т.е. для подтверждения вставки молекулы целевой нуклеиновой кислоты, и могут быть использованы маркеры, которые придают поддающиеся отбору фенотипы, такие как устойчивость к лекарственным средствам, ауксотрофия, устойчивость к цитотоксическим агентам или экспрессия поверхностных полипептидов. В среде, обработанной селективным агентом, только клетки, экспрессирующие селектируемый маркер, способны выжить или продемонстрировать другие фенотипические признаки, и таким образом можно отбирать трансформированные клетки.
При использовании здесь термин «трансформация» означает, что вектор, содержащий полинуклеотид, кодирующий целевой полипептид, введен в клетку-хозяина или микроорганизм таким образом, что указанный полипептид, кодируемый указанным полинуклеотидом, может экспрессироваться в клетке-хозяине. Трансформированный полинуклеотид может располагаться независимо от положения, либо как вставленный в хромосому клетки-хозяина и расположенный там, либо как расположенный вне хромосомы, при условии, что он может экспрессироваться в клетке-хозяине. Дополнительно, указанный полинуклеотид включает ДНК и/или РНК, кодирующие целевой полипептид. Указанный полинуклеотид может быть введен в любой форме, при условии, что она может быть введена в клетку-хозяина и экспрессирована. Например, указанный полинуклеотид может быть введен в клетку-хозяина в форме экспрессионной кассеты, которая представляет собой генную конструкцию, содержащую все элементы, необходимые для самоэкспрессии. Указанная экспрессионная кассета может обычно содержать промотор, функционально присоединенный к указанному полинуклеотид у, сигнал терминации транскрипции, сайт связывания рибосомы и сигнал терминации трансляции. Указанная экспрессионная кассета может находиться в форме экспрессионного вектора, способного к саморепликации. Дополнительно, указанный полинуклеотид может быть введен в клетку-хозяина в своей собственной форме и функционально функционально связан с последовательностью, необходимой для экспрессии в клетке-хозяине, но не ограничивается этим.
Дополнительно, термин «функционально связан» означает, что полинуклеотидная последовательность функциональным образом связана с последовательностью промотора, которая инициирует и регулирует транскрипцию полинуклеотида, кодирующего целевой вариант по настоящему изобретению.
В соответствии с еще одним аспектом настоящего изобретения предложен микроорганизм, содержащий указанный вариант по настоящему изобретению или полинуклеотид по настоящему изобретению.
Микроорганизм по настоящему изобретению может содержать любой один или более из вариантов по настоящему изобретению, полинуклеотид, кодирующий указанный вариант, и вектор, содержащий указанный полинуклеотид. Микроорганизм по настоящему изобретению может содержать модифицированный полипептид по настоящему изобретению, полинуклеотид, кодирующий указанный полипептид, или вектор, содержащий полинуклеотид по настоящему изобретению.
При использовании здесь термин «микроорганизм» или «штамм» включает все микроорганизмы дикого типа и естественным образом или искусственно генетически модифицированные микроорганизмы, и это может быть микроорганизм, в котором специфический механизм ослаблен или усилен из-за вставки чужеродного гена, усиления или инактивации активности эндогенного гена и т.д., и это может быть микроорганизм, содержащий генетическую модификацию для продуцирования целевого полипептида, белка или продукта.
Микроорганизм по настоящему изобретению может представлять собой микроорганизм, содержащий любой один или более из вариантов по настоящему изобретению, полинуклеотид по настоящему изобретению и вектор, содержащий полинуклеотид по настоящему изобретению; микроорганизм, модифицированный для экспрессии варианта по настоящему изобретению или полинуклеотида по настоящему изобретению; микроорганизм, экспрессирующий вариант по настоящему изобретению или полинуклеотид по настоящему изобретению (например, рекомбинантный микроорганизм); или микроорганизм, обладающий активностью варианта по настоящему изобретению (например, рекомбинантный микроорганизм), но не ограничивается этим. Указанный микроорганизм может представлять собой микроорганизм, который эндогенно содержит полинуклеотиды, кодирующие белки ликопинциклазу/фитоенсинтазу (crtYB) и фитоендесатуразу (crtI), или модифицирован таким образом, чтобы дополнительно содержать полинуклеотиды, кодирующие белки ликопинциклазу/фитоенсинтазу (crtYB) и фитоендесатуразу (crtI), и тем самым проявлять активности этих белков, или микроорганизм, в котором активности этих белков усилены. Дополнительно, микроорганизм по настоящему изобретению может представлять собой микроорганизм, у которого исходный штамм, не обладающий активностями белков ликопинциклазы/фитоенсинтазы (crtYB) и фитоендесатуразы (crtI), модифицирован таким образом, чтобы проявлять активности этих белков. Ликопинциклаза/фитоенсинтаза или фитоендесатураза могут представлять собой белки, происходящие из Xanthophyllomyces dendrorhous, но не ограничиваются этим. В конкретном воплощении полинуклеотид, кодирующий ликопинциклазу/фитоенсинтазу или фитоендесатуразу, в каждом случае может иметь или содержать последовательность SEQ ID NO: 13 или SEQ ID NO: 14. В указанный полинуклеотид могут быть внесены различные модификации в кодирующей области при условии, что аминокислотная последовательность не изменяется, с учетом вырожденности кодонов или кодонов, предпочтительных в микроорганизмах, которые, как предполагают, будут экспрессировать вариант по настоящему изобретению. В частности, указанный полинуклеотид может иметь или содержать нуклеотидную последовательность, обладающую 70% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более и менее чем 100% гомологией или идентичностью по отношению к последовательности SEQ ID NO: 13 или SEQ ID NO: 14, или может состоять или по существу состоять из нуклеотидной последовательности, обладающей 70% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более и менее чем 100% гомологией или идентичностью по отношению к последовательности SEQ ID NO: 13 или SEQ ID NO: 14, но не ограничивается этим.
Микроорганизм по настоящему изобретению может обладать способностью к продуцированию ретиноидов или может обладать способностью к продуцированию ретинола.
В настоящем изобретении «ретиноид» означает вещество, которое способно действовать как витамин А или родственно ему. В частности, ретиноид может быть одним или более, выбранным из группы, состоящей из ретинола, ретиналя, сложного эфира ретинола и ретиноевой кислоты. Микроорганизм по настоящему изобретению может представлять собой микроорганизм, способный продуцировать ретиналь и ретинол, а ретиноевая кислота может продуцироваться путем превращения из ретинола.
Микроорганизм по настоящему изобретению может представлять собой микроорганизм, который естественным образом обладает способностью продуцировать бета-каротин-15,15'-оксигеназу или ретиноид, или микроорганизм, не обладающий способностью продуцировать бета-каротин-15,15'-оксигеназу или ретиноид, в который введен вариант по настоящему изобретению или полинуклеотид, его кодирующий (или вектор, содержащий вектор), и/или которому придана способность продуцировать ретиноиды, но не ограничивается этим.
В одном воплощении микроорганизм по настоящему изобретению может представлять собой клетку или микроорганизм, которые трансформированы полинуклеотидом по настоящему изобретению или вектором, содержащим полинуклеотид, кодирующий вариант по настоящему изобретению, для экспрессии варианта по настоящему изобретению, и для целей настоящего изобретения микроорганизм по настоящему изобретению может включать любой микроорганизм, который содержит вариант по настоящему изобретению для продуцирования ретиноида. Например, микроорганизм по настоящему изобретению может представлять собой рекомбинантный микроорганизм, в котором полинуклеотид, кодирующий вариант по настоящему изобретению, введен в природный микроорганизм дикого типа или продуцирующий ретиноид микроорганизм для экспрессии варианта бета-каротин-15,15'-оксигеназы и достижения повышенной способности продуцировать ретиноид. Указанный рекомбинантный микроорганизм может представлять собой микроорганизм, обладающий повышенной способностью продуцировать ретиноид по сравнению с природным микроорганизмом дикого типа, или с немодифицированным микроорганизмом с бифункциональной бета-каротин-15,15'-оксигеназой (т.е. микроорганизмом дикого типа, экспрессирующим бета-каротин-15,15'-оксигеназу (SEQ ID NO: 1)), или с микроорганизмом, не экспрессирующим модифицированный белок (вариант бета-каротин-15,15'-оксигеназы SEQ ID NO: 3 или варианты бета-каротин-15,15'-оксигеназы, описанные выше), но не ограничивается этим. Например, немодифицированный микроорганизм с бета-каротин-15,15'-оксигеназой, который является целевым микроорганизмом при проведении сравнения на наличие повышенной способности продуцировать ретиноид, может представлять собой штамм СС08-1023, но не ограничивается этим.
В одном воплощении, по сравнению со способностью продуцировать ретиноид у исходного штамма до модификации или немодифицированного микроорганизма, рекомбинантный микроорганизм с повышенной способностью продуцировать ретиноид может обладать способностью, повышенной на примерно 1% или более, и, в частности, примерно 1% или более, примерно 2,5% или более, примерно 5% или более, примерно 6% или более, примерно 7% или более, примерно 8% или более, примерно 9% или более, примерно 10% или более, примерно 11% или более, примерно 11,5% или более, примерно 12% или более, примерно 12,5% или более, примерно 13% или более, примерно 13,5% или более, примерно 14% или более, примерно 14,5% или более, примерно 15% или более, примерно 15,5% или более, примерно 16% или более, примерно 16,5% или более, примерно 17% или более, примерно 17,5% или более, примерно 18% или более, примерно 20% или более, примерно 22% или более, примерно 24% или более, примерно 26% или более, примерно 28% или более, примерно 30% или более, примерно 32% или более, примерно 34% или более, примерно 36% или более, примерно 37% или более, примерно 38% или более, примерно 39% или более, примерно 40% или более, примерно 41% или более, примерно 41,5% или более, примерно 42% или более, примерно 42,5% или более, примерно 43% или более, примерно 43,5% или более, примерно 44% или более, примерно 44,5% или более, примерно 45% или более, примерно 45,5% или более либо примерно 46% или более (верхний предел точно не задан и может составлять, например, примерно 200% или менее, примерно 150% или менее, примерно 100% или менее либо примерно 50% или менее), но не ограничивается этим, при условии, что рекомбинантный микроорганизм демонстрирует количественное повышение с положительным значением по сравнению со способностью продуцировать ретиноид у исходного штамма или немодифицированного микроорганизма до модификации. В еще одном воплощении, по сравнению со способностью продуцировать ретиноид у исходного штамма до модификации или немодифицированного микроорганизма, рекомбинантный микроорганизм с повышенной способностью продуцировать ретиноид может обладать способностью, повышенной в примерно 1,01 раза или более, примерно 1,02 раза или более, примерно 1,03 раза или более, примерно 1,05 раза или более, примерно 1,06 раза или более, примерно 1,07 раза или более, примерно 1,08 раза или более, примерно 1,09 раза или более, примерно 1,10 раза или более, примерно 1,11 раза или более, примерно 1,12 раза или более, примерно 1,13 раза или более, примерно 1,14 раза или более, примерно 1,15 раза или более, примерно 1,16 раза или более, примерно 1,17 раза или более, примерно 1,18 раза или более, примерно 1,20 раза или более, примерно 1,22 раза или более, примерно 1,24 раза или более, примерно 1,26 раза или более, примерно 1,28 раза или более, примерно 1,30 раза или более, примерно 1,32 раза или более, примерно 1,34 раза или более, примерно 1,36 раза или более, примерно 1,38 раза или более, примерно 1,40 раза или более, примерно 1,41 раза или более, примерно 1,42 раза или более, примерно 1,43 раза или более, примерно 1,44 раза или более, примерно 1,45 раза или более либо примерно 1,46 раза или более (верхний предел точно не задан и может составлять, например, примерно 10 раз или менее, примерно 5 раз или менее, примерно 3 раза или менее либо примерно 2 раза или менее), но не ограничивается этим. При использовании здесь термин «примерно» относится к диапазону, который включает все значения ±0,5, ±0,4, ±0,3, ±0,2, ±0,1 и т.д. и включает все значения, которые эквивалентны или сходны со значениями, следующими за термином «примерно», но указанный диапазон не ограничивается этим.
При использовании здесь термин «немодифицированный микроорганизм» не исключает штаммы, содержащие мутацию, которая может возникнуть в микроорганизме естественным путем, и это может быть штамм дикого типа или собственно природный штамм, либо это может быть штамм до того как признак изменен генетической вариацией, вызванной природными или искусственными факторами. Например, немодифицированный микроорганизм может представлять собой штамм, в который не введен или еще не введен вариант бета-каротин-15,15'-оксигеназы по настоящему изобретению, описанный здесь. Термин «немодифицированный микроорганизм» может быть использован взаимозаменяемо с терминами «штамм до модификации», «микроорганизм до модификации», «немутированный штамм», «немодифицированный штамм», «немутированный микроорганизм» или «референсный микроорганизм».
В еще одном воплощении микроорганизм по настоящему изобретению может представлять собой микроорганизм, принадлежащий к Yarrowia sp., и в частности, Yarrowia lipolytica, но не ограничивается этим.
При использовании здесь термин «усиление» в отношении активности полипептида означает, что указанная активность полипептида повышена по сравнению с его эндогенной активностью. Термин «усиление» может быть использован взаимозаменяемо с такими терминами, как «активация», «повышающая регуляция», «сверхэкспрессия», «повышение» и т.д. В частности, активация, усиление, повышающая регуляция, сверхэкспрессия и повышение могут включать как проявление активности, которой исходно не было, так и проявление улучшенной активности по сравнению с эндогенной активностью или активностью до модификации. Термин «эндогенная активность» означает активность специфического полипептида, которой изначально обладает исходный штамм до изменения признака или немодифицированный микроорганизм, когда указанный признак изменяется в результате генетической мутации, вызванной природными или искусственными факторами. Этот термин может быть использован взаимозаменяемо с термином «активность до модификации». То, что активность полипептида «усилена», «активирована», «сверхэкспрессирована» или «повышена» по сравнению с его эндогенной активностью, означает, что указанная активность полипептида улучшена по сравнению с активностью и/или концентрацией (уровнем экспрессии) специфического полипептида, которой изначально обладает исходный штамм до изменения признака или немодифицированный микроорганизм.
Усиление может быть достигнуто посредством введения чужеродного полипептида или усиления эндогенной активности и/или концентрации (уровня экспрессии) полипептида. Усиление активности полипептида может быть подтверждено увеличением степени активности и уровня экспрессии соответствующего полипептида либо количества продукта, продуцируемого соответствующим полипептидом.
Для усиления активности полипептида могут быть использованы различные способы, хорошо известные специалистам в данной области техники, и ограничения на способ отсутствуют, при условии, что возможно усиление активности целевого полипептида по сравнению с таковой у микроорганизма до модификации. В частности, могут быть использованы способы генной инженерии и/или белковой инженерии, хорошо известные специалистам в данной области техники, которые стандартно выполняют в молекулярной биологии, но указанный способ не ограничивается этим (например, Sitnicka et al. Functional Analysis of Genes. Advances in Cell Biology. 2010, Vol.2. 1-16; Sambrook et al. Molecular Cloning 2012; etc.).
В частности, усиление полипептида по настоящему изобретению может представлять собой:
1) повышение количества внутриклеточных копий полинуклеотида, кодирующего указанный полипептид;
2) замену области, регулирующей экспрессию гена, на хромосоме, кодирующей указанный полипептид, на последовательность, обладающую сильной активностью;
3) модификацию нуклеотидной последовательности, кодирующей инициирующий кодон или область 5'-UTR в транскрипте гена, кодирующего указанный полипептид;
4) модификацию аминокислотной последовательности полипептида для усиления активности полипептида;
5) модификацию полинуклеотидной последовательности, кодирующей указанный полипептид, для усиления активности полипептида (например, модификацию полинуклеотидной последовательности гена полипептида для кодирования полипептида, который модифицирован для усиления активности указанного полипептида);
6) введение чужеродного полипептида, демонстрирующего активность указанного полипептида, или кодирующего его чужеродного полинуклеотида;
7) оптимизацию кодонов полинуклеотида, кодирующего указанный полипептид;
8) анализ третичной структуры указанного полипептида для выбора и модификации или химической модификации экспонированного сайта; или
9) комбинацию двух или более пунктов, выбранных из подпунктов 1-8 выше, но не ограничивается этим.
В частности:
1) Повышение количества внутриклеточных копий полинуклеотида, кодирующего указанный полипептид, может быть достигнуто введением в клетку-хозяина вектора, который способен реплицироваться и функционировать независимо от хозяина и с которым функционально связан полинуклеотид, кодирующий соответствующий полипептид. В соответствии с другим вариантом, повышение может быть достигнуто путем введения одной копии или двух или более копий полинуклеотида, кодирующего соответствующий полипептид, в хромосому клетки-хозяина. Введение в хромосому может быть выполнено путем введения в клетку-хозяина вектора, способного вставлять полинуклеотид в хромосому клетки-хозяина, но не ограничивается этим. Указанный вектор описан выше.
2) Замена области, регулирующей экспрессию гена (или последовательности, регулирующей экспрессию), на хромосоме, кодирующей указанный полипептид, на последовательность, демонстрирующую сильную активность, может представлять собой, например, появление мутации в последовательности вследствие делеции, вставки, неконсервативной или консервативной замены или их комбинации, или замену на последовательность, демонстрирующую сильную активность, таким образом, что активность указанной области, регулирующей экспрессию, дополнительно усиливается. Область, регулирующая экспрессию, может включать, без ограничений, промотор, последовательность оператора, последовательность, кодирующую сайт связывания рибосомы, последовательность, контролирующую терминацию транскрипции и трансляции и т.д. Например, указанная замена может состоять в замене исходного промотора на сильный промотор, но не ограничивается этим.
Примеры известных сильных промоторов включают промоторы с CJ1 по CJ7 (патент US 7662943 В2), lac-промотор, trp-промотор, trc-промотор, tac-промотор, PR-промотор фага лямбда, PL-промотор, tet-промотор, gapA-промотор, SPL7-промотор, SPL13 (sm3)-промотор (патент US 10584338 В2), O2-промотор (патент US 10273491 В2), tkt-промотор, уссА-промотор, TEFINt-промотор, ЕХР1-промотор, TEF1-промотор и т.д., но не ограничиваются этим.
3) Модификация нуклеотидной последовательности, кодирующей инициирующий кодон или область 5'-UTR в транскрипте гена, кодирующего указанный полипептид, может представлять собой, например, замену на нуклеотидную последовательность, кодирующую другой стартовый кодон, обеспечивающий более высокую скорость экспрессии полипептида по сравнению с эндогенным стартовым кодоном, но не ограничивается этим.
4) и 5) Модификация аминокислотной последовательности или полинуклеотидной последовательности может представлять собой возникновение мутации в последовательности вследствие делеции, вставки, неконсервативной или консервативной замены аминокислотной последовательности полипептида или полинуклеотидной последовательности, кодирующей указанный полипептид, или их комбинацию; либо замену на аминокислотную последовательность или полинуклеотидную последовательность, модифицированные таким образом, чтобы они демонстрировали более сильную активность, или на аминокислотную последовательность или полинуклеотидную последовательность, модифицированные таким образом, чтобы они обладали повышенной активностью так, чтобы активность указанного полипептида была усилена, но не ограничивается этим. Замена может быть, в частности, осуществлена путем вставки полинуклеотида в хромосому посредством гомологичной рекомбинации, но не ограничивается этим. Вектор, используемый в настоящем изобретении, может дополнительно содержать селектируемый маркер для подтверждения вставки в хромосому. Указанный селектируемый маркер описан выше.
6) Введение чужеродного полинуклеотида, демонстрирующего активность указанного полипептида, может представлять собой введение в клетку-хозяина чужеродного полинуклеотида, кодирующего полипептид, демонстрирующий активность, идентичную/сходную с таковой указанного полипептида. Ограничения на происхождение или последовательность указанного чужеродного полинуклеотида отсутствуют, при условии, что он демонстрирует активность, идентичную/сходную с таковой указанного полипептида. Способ, используемый при введении, может быть осуществлен путем соответствующего выбора известного способа трансформации специалистом в данной области техники. Так как введенный полинуклеотид экспрессируется в клетке-хозяине, может продуцироваться указанный полипептид, и его активность может быть повышена.
7) Оптимизация кодонов полинуклеотида, кодирующего указанный полипептид, может представлять собой оптимизацию кодонов эндогенного полинуклеотида таким образом, чтобы повысить транскрипцию или трансляцию указанного полинуклеотида в клетке-хозяине, или оптимизацию кодонов чужеродного полинуклеотида таким образом, чтобы осуществлять оптимизированную транскрипцию или трансляцию указанного полинуклеотида в клетке-хозяине.
8) Анализ третичной структуры указанного полипептида для выбора и модификации или химической модификации экспонированного сайта может представлять собой, например, определение кандидатного матричного белка в соответствии со степенью сходства последовательности путем сравнения информации о последовательности анализируемого полипептида с базой данных, в которой хранится информация о последовательностях известных белков; подтверждение структуры на основании этого; и выбор и модификацию или химическую модификацию экспонированного сайта, который необходимо модифицировать или химически модифицировать.
Такое усиление активности полипептида может представлять собой повышение активности или концентрации или уровня экспрессии соответствующего полипептида, основанное на активности или концентрации полипептида, экспрессируемого в микроорганизме дикого типа или штамме микроорганизма до модификации; или увеличение количества продукта, продуцируемого соответствующим полипептидом, но не ограничивается этим.
В микроорганизме по настоящему изобретению модификация всего или части полинуклеотида может быть индуцирована (а) гомологичной рекомбинацией, проводимой с использованием вектора для вставки в хромосому в микроорганизме или редактирования генома с использованием созданной методами тканевой инженерии нуклеазы (например, CRISPR-Cas9), и/или (б) обработкой светом (например, УФ лучами, ионизирующим облучением и т.д.) и/или химическими веществами, но не ограничивается этим. Способ модификации всего или части гена может включать способ с использованием технологии рекомбинантных ДНК. Например, весь или часть гена могут быть делетированы путем введения в микроорганизм нуклеотидной последовательности или вектора, содержащих нуклеотидную последовательность, гомологичную целевому гену, для того, чтобы вызвать гомологичную рекомбинацию. Нуклеотидная последовательность или вектор, которые будут вводить, могут содержать доминантный селектируемый маркер, но не ограничиваются этим.
В микроорганизме по настоящему изобретению указанные вариант, полинуклеотид, ретиноид и т.д. являются такими, как описано в других аспектах выше.
В еще одном аспекте настоящего изобретения предложен способ получения ретиноида, который включает культивирование микроорганизма, содержащего вариант по настоящему изобретению или полинуклеотид по настоящему изобретению, в среде.
При использовании здесь термин «культивирование» означает выращивание микроорганизма по настоящему изобретению в соответствующим образом отрегулированных условиях окружающей среды. Способ культивирования по настоящему изобретению может быть осуществлен в соответствии с подходящими условиями среды или культивирования, известными специалистам в данной области техники. Специалист в данной области техники может легко регулировать такое культивирование в соответствии с выбранным микроорганизмом. В частности, культивирование может быть периодического типа, непрерывного типа и/или подпитываемого типа, но не ограничивается этим.
При использовании здесь термин «среда» означает смесь веществ, содержащую в качестве основных ингредиентов питательные вещества, необходимые для культивирования микроорганизма по настоящему изобретению, при котором среда обеспечивает питательные вещества, включая воду, существенные для выживания и роста, факторы роста и т.д. В частности, в качестве среды и других условий культивирования, применяемых для культивирования микроорганизма по настоящему изобретению, может быть использована любая среда, в общем применяемая для культивирования микроорганизмов, без каких-либо ограничений; однако микроорганизм по настоящему изобретению может культивироваться в аэробных условиях в обычной среде, содержащей соответствующие источники углерода, источники азота, источники фосфора, неорганические соединения, аминокислоты и/или витамины и т.д., при контролировании температуры, рН и т.д.
В настоящем изобретении источник углерода может включать углеводы (например, глюкозу, сахарозу, лактозу, фруктозу, сукрозу, мальтозу и т.д.); сахарные спирты (например, маннит, сорбит и т.д.); органические кислоты (например, пировиноградную кислоту, молочную кислоту, лимонную кислоту и т.д.) и аминокислоты (например, глутаминовую кислоту, метионин, лизин и т.д.). Дополнительно, могут быть использованы природные источники органических веществ (например, гидролизаты крахмала, патока, черная патока, рисовые отруби, маниока, багасса и кукурузный экстракт), и в частности, могут быть использованы углеводы (например, глюкоза и стерилизованная предварительно обработанная патока (т.е. патока, превращенная в восстанавливающие сахара)), и могут быть использованы подходящие количества различных других источников углерода различными способами без ограничений. Эти источники углерода могут быть использованы отдельно или в комбинации из двух или более из них, но не ограничиваются этим.
В качестве источников азота могут быть использованы неорганические источники азота (например, аммиак, сульфат аммония, хлорид аммония, ацетат аммония, фосфат аммония, карбонат аммония, нитрат аммония и т.д.); аминокислоты (например, глутаминовая кислота, метионин, глутамин и т.д.); и органические источники азота (например, пептон, NZ-амин, мясные экстракты, дрожжевые экстракты, солодовые экстракты, кукурузный экстракт, гидролизаты казеина, рыба или продукты ее распада, обезжиренный соевый жмых или продукты его распада и т.д.). Эти источники азота могут быть использованы отдельно или в комбинации из двух или более из них, но не ограничиваются этим.
Источники фосфата могут включать дигидрофосфат калия, гидрофосфат калия и соответствующие натрий-содержащие соли. Могут быть использованы неорганические соединения, хлорид натрия, хлорид кальция, хлорид железа, сульфат магния, сульфат железа, сульфат марганца, карбонат кальция и т.д., и дополнительно могут быть включены аминокислоты, витамины и/или подходящие их предшественники. Эти компоненты или предшественники могут быть добавлены в среду периодически или непрерывно. Однако настоящее изобретение не ограничивается этим.
Дополнительно, значение рН среды можно регулировать путем добавления соединений, таких как гидроксид аммония, гидроксид калия, аммиак, фосфорная кислота и серная кислота, в среду соответствующим способом во время культивирования микроорганизма по настоящему изобретению. Дополнительно, может быть добавлен антивспениватель (например, полигликолевый эфир жирной кислоты) для подавления образования пены во время культивирования. Дополнительно, в среду может быть введен кислород или кислород-содержащий газ для поддержания аэробного состояния среды, или газ можно не вводить, или в среду можно вводить газообразный азот, водород или диоксид углерода для поддержания анаэробного или микроаэробного состояния среды, но настоящее изобретение не ограничивается этим.
При культивировании по настоящему изобретению температуру культивирования можно поддерживать на уровне от 20°С до 45°С, в частности, от 25°С до 40°С, от 20°С до 35°С или от 25°С до 35°С, и культивирование можно осуществлять в течение от примерно 10 часов до примерно 160 часов, но не ограничивается этим.
Ретиноид, получаемый культивированием по настоящему изобретению, может высвобождаться в среду или может оставаться в клетках.
Способ получения ретиноида по настоящему изобретению может дополнительно включать получение микроорганизма по настоящему изобретению, получение среды для культивирования указанного микроорганизма или комбинацию этого (в любом порядке), например, до или после культивирования.
Способ получения ретиноида по настоящему изобретению может дополнительно включать стадию извлечения ретиноида из среды, полученной в результате культивирования (среды, в которой осуществлялось культивирование), или из микроорганизма по настоящему изобретению. Стадия извлечения может дополнительно быть включена после стадии культивирования.
Извлечение может представлять собой сбор нужного ретиноида с использованием соответствующего способа, известного специалистам в данной области техники, в соответствии со способом культивирования микроорганизма по настоящему изобретению, например, периодический, непрерывный тип культивирования или тип культивирования с подпиткой. Например, извлечение может быть осуществлено с использованием центрифугирования, фильтрации, обработки агентом, осаждающим кристаллизованные белки (высаливания), экстракции, разрушения клеток, обработки ультразвуком, ультрафильтрации, диализа, различных типов хроматографии (например, молекулярно-ситовой хроматографии (гель-фильтрации), адсорбционной хроматографии, ионообменной хроматографии, аффинной хроматографии и т.д.), ВЭЖХ и комбинации этих способов, и нужный ретиноид может быть извлечен из среды или микроорганизма с использованием соответствующего способа, известного специалистам в данной области техники.
Также способ получения ретиноида по настоящему изобретению может дополнительно включать стадию очистки. Очистка может быть осуществлена с использованием соответствующего способа, известного специалистам в данной области техники. В одном воплощении, когда способ получения ретиноида по настоящему изобретению включает как стадию извлечения, так и стадию очистки, указанные стадия извлечения и стадия очистки могут быть осуществлены непрерывно или с интервалами, независимо от порядка, либо могут быть осуществлены одновременно или в виде одной объединенной стадии, но способ не ограничивается этим.
В настоящем изобретении в способе получения производного ретинола, которое является типом ретиноида, стадия культивирования может дополнительно включать стадию превращения ретинола в производное ретинола. В способе получения производного ретинола по настоящему изобретению стадия превращения может быть дополнительно включена после стадии культивирования или стадии извлечения. Стадия превращения может быть осуществлена с использованием соответствующего способа, известного специалистам в данной области техники (Jang et al. Microbial Cell Factories 2011, 10:59, Choi et al. Antioxidants. 2020).
В способе по настоящему изобретению указанные вариант, полинуклеотид, вектор, микроорганизм и т.д. являются такими, как описано в других воплощениях выше.
В еще одном аспекте настоящего изобретения предложена композиция для получения ретиноида, которая содержит: микроорганизм, содержащий вариант по настоящему изобретению, полинуклеотид, кодирующий указанный вариант, вектор, содержащий указанный полинуклеотид, или полинуклеотид по настоящему изобретению; среду, в которой был культивирован указанный микроорганизм; или комбинацию двух или более из этого.
Композиция по настоящему изобретению может дополнительно содержать любой подходящий эксципиент, обычно используемый в композиции для получения ретиноида, и примеры указанного эксципиента могут включать консервант, смачивающий агент, диспергирующий агент, суспендирующий агент, буфер, стабилизирующий агент, изотонический агент и т.д., но указанный эксципиент не ограничивается этим.
В композиции по настоящему изобретению указанные вариант, полинуклеотид, вектор, микроорганизм, среда, ретиноид и т.д. являются такими, как описано в других воплощениях выше.
В еще одном аспекте настоящего изобретения предложено применение любого одного или более из указанных варианта, полинуклеотида, вектора и микроорганизма по настоящему изобретению для получения ретиноида.
Указанные вариант, полинуклеотид, вектор, микроорганизм, ретиноид и т.д. являются такими, как описано в других воплощениях выше.
Примеры осуществления изобретения
Далее настоящее изобретение будет описано более подробно со ссылкой на Примеры. Однако эти примеры приведены только для иллюстрации настоящего изобретения, и не предполагается, что объем настоящего изобретения будет ограничен этими Примерами. При этом технические вопросы, не рассмотренные в этом описании изобретения, могут в достаточной степени быть понятны и легко осуществлены на практике специалистом в области техники, к которой относится настоящее изобретение, или в сходных технических областях.
Пример 1. Получение библиотеки векторов вариантов ВСО
Для амплификации гена ВСО, полученного из некультивируемой морской бактерии 66А03, получили аминокислотную последовательность SEQ ID NO: 1 и полинуклеотидную последовательность SEQ ID NO: 2, основываясь на аминокислотной последовательности (Q4PNI0), зарегистрированной в UniProtKB (UniProt Knowledgebase). Для индукции случайных мутаций посредством ПЦР пониженной точности применяли набор Diversify PCR Random Mutagenesis Kit (Takara). Условия и способы проведения ПЦР были оптимизированы в соответствии с Буферными условиями 1 и Буферными условиями 2 в руководстве пользователя, соответственно. Специфические условия проведения ПЦР пониженной точности показаны в Таблице 1 ниже.
ПЦР проводили с использованием в качестве матрицы гена ВСО дикого типа (SEQ ID NO: 2), полученного из морской бактерии 66А03, который был синтезирован компанией Macrogen Inc. по запросу, и праймеров с последовательностями SEQ ID NO: 5 и SEQ ID NO: 6. Условия проведения ПЦР были следующими: 25 циклов денатурации при 95°С в течение 30 секунд; отжига при 55°С в течение 30 секунд; и полимеризации при 68°С в течение 1 минуты. Продукты ВСО, полученные ПЦР-амплификацией в двух различных условиях, объединяли для получения библиотеки векторов вариантов ВСО на основе вектора pIMR53-TEFINtp-CYC1t.
Вектор pIMR53-TEFINtp-CYC1t получали следующим образом.
Для амплификации TEFINtp проводили ПЦР с использованием в качестве матрицы геномной ДНК из Yarrowia lipolytica PO1f и праймеров с последовательностями SEQ ID NO: 7 и SEQ ID NO: 8; а для амплификации терминатора CYC1 проводили ПЦР с использованием в качестве матрицы геномной ДНК из Saccharomyces cerevisiae и праймеров с последовательностями SEQ ID NO: 9 и SEQ ID NO: 10. Условия проведения ПЦР были следующими: 32 цикла денатурации при 95°С в течение 1 минуты; отжига при 55°С в течение 1 минуты; и полимеризации при 68°С в течение 1 минуты. Дополнительно, проводили ПЦР с использованием вектора pIMR53 в качестве матрицы и праймеров с последовательностями SEQ ID NO: 11 и SEQ ID NO: 12. Условия проведения ПЦР были следующими: 32 цикла денатурации при 95°С в течение 1 минуты; отжига при 55°С в течение 1 минуты; и полимеризации при 68°С в течение 5 минут.
В результате были получены линеаризованный pIMR53 длиной 6774 п.н., TEFINtp длиной 531 п.н. и CYC1t длиной 248 п.н. ДНК-фрагменты TEFINtp и CYC1t, полученные посредством ПЦР-амплификации, лигировали в вектор pIMR53 с использованием набора Infusion cloning kit (Invitrogen), трансформировали им Е. coli DH5α и высевали трансформантов на твердую среду LB, содержащую 30 мг/л ампициллина.
Колонии, трансформированные плазмидой, в которую были вставлены необходимые промотор TEFINt и терминатор CYC1, отбирали посредством ПЦР, и затем получили плазмиду с помощью хорошо известного способа выделения плазмид; эту плазмиду назвали pIMR53-TEFINtp-CYC1t.
Пример 2. Получение платформенного штамма Yarrowia lipolytica для получения ретиноида
Пример 2-1. Получение штамма, в который вставлены crtYB-crtI, происходящие из X. dendrorhous
Чтобы получить платформенный штамм для получения ретиноидов гены ликопинциклазы/фитоенсинтазы (crtYB) и фитоендесатуразы (crtI), происходящие из Xanthophyllomyces dendrorhous, вставляли в геном штамма Yarrowia lipolytica СС08-0125 (номер доступа KCCM12972P), который представляет собой дрожжи, накапливающие высокие уровни жиров. В случае crtYB полинуклеотид с последовательностью SEQ ID NO: 13 был получен на основе нуклеотидной последовательности (GenBank: AY 177204.1), зарегистрированной в поисковой базе данных Национального центра биотехнологической информации (NCBI), и в случае crtl полинуклеотид с последовательностью SEQ ID NO: 14 был получен на основе нуклеотидной последовательности (GenBank: на основе AY177424.1), зарегистрированной в NCBI. Полинуклеотидные последовательности crtYB и crtI были синтезированы компанией Macrogen Inc. в форме TEFINtp-crtYB-CYC1t (SEQ ID NO: 15) и TEFINtp-crtI-CYC1t (SEQ ID NO: 16), а кассета для вставки в локус гена MHY1 (YALI0B21582g) была создана с использованием гена URA3 из Y. lipolytica (SEQ ID NO: 17) в качестве селектируемого маркера. Для проведения каждой ПЦР использовали синтезированные гены crtYB и crtI и геномную ДНК KCCM12972P в качестве матриц и пары праймеров с последовательностями SEQ ID NO: 18 и SEQ ID NO: 19, SEQ ID NO: 20 и SEQ ID NO: 21, SEQ ID NO: 22 и SEQ ID NO: 23, SEQ ID NO: 24 и SEQ ID NO: 25, SEQ ID NO: 26 и SEQ ID NO: 27 и SEQ ID NO: 28 и SEQ ID NO: 29, соответственно. Условия проведения ПЦР были следующими: 35 циклов денатурации при 95°С в течение 1 минуты; отжига при 55°С в течение 1 минуты; и полимеризации при 72°С в течение 3 минут и 30 секунд. Из полученных таким образом пяти фрагментов ДНК получили одну кассету с помощью ПЦР с перекрывающимися праймерами.
Полученную таким образом кассету вводили в штамм KCCM12972P способом с тепловым шоком (D. -C. Chen et al., Appl Microbiol Biotechnol, 1997), и затем были получены колонии, сформировавшиеся на твердой среде, не содержащей урацил (YLMM1). Колонии, в которых вставка указанной кассеты в геном была подтверждена с использованием праймеров с последовательностями SEQ ID NO: 30 и SEQ ID NO: 31, наносили пятнами на твердую среду 5-FOA и культивировали при 30°С в течение 3 суток. Отбор клонов без маркера URA3 осуществляли путем получения колоний, растущих на твердой среде 5-FOA.
Пример 2-2. Получение HMGR-усиленного штамма
Была создана кассета для замены промотором TEFINt нативной промоторной (SEQ ID NO: 32) области гена 3-гидрокси-3-метилглутарил-СоА-редуктазы (HMGR) штамма, полученного в Примере 2-1, в который были вставлены гены crtYB и crtl. Для проведения каждой ПЦР использовали геномную ДНК KCCM12972P в качестве матрицы и пары праймеров с последовательностями SEQ ID NO: 33 и SEQ ID NO: 34, SEQ ID NO: 35 и SEQ ID NO: 36, SEQ ID NO: 37 и SEQ ID NO: 38, SEQ ID NO: 39 и SEQ ID NO: 40 и SEQ ID NO: 41 и SEQ ID NO: 42, соответственно. Условия проведения ПЦР были следующими: 35 циклов, состоящих из денатурации при 95°С в течение 1 минуты; отжига при 55°С в течение 1 минуты; и полимеризации при 72°С в течение 1 минуты и 30 секунд. Из полученных таким образом пяти фрагментов ДНК получили одну кассету с помощью ПЦР с перекрывающимися праймерами.
Полученную таким образом кассету вводили в штамм, полученный в Примере 2-1, способом с тепловым шоком и затем получали колонии, образованные на твердой среде (YLMM1) без урацила. Колонии, в которых вставка указанной кассеты была подтверждена с использованием праймеров с последовательностями SEQ ID NO: 43 и SEQ ID NO: 44, наносили пятнами на твердую среду 5-FOA и культивировали при 30°С в течение 3 суток. Отбор клонов без маркера URA3 осуществляли путем получения колоний, растущих на твердой среде 5-FOA. Полученный в конечном итоге платформенный штамм, продуцирующий ретиноид, назвали СС08-1023.
<Минимальная среда 1 для Yarrowia lipolytica (YLMM1)>
20 г/л глюкозы, 6,7 г/л основы азотного агара для дрожжей, без аминокислот, 2 г/л синтетических добавок в неполную среду для дрожжей без урацила и 15 г/л агара
<5-Фтороротовая кислота (5-FOA)>
20 г/л глюкозы, 6,7 г/л основы азотного агара для дрожжей, без аминокислот, 2 г/л синтетических добавок в неполную среду для дрожжей без урацила, 50 мкг/мл урацила, 1 г/л 5-фтороротовой кислоты (5-FOA) и 15 г/л агара
Пример 3. Первичный отбор штамма с усиленной активностью ВСО
Для отбора штаммов с усиленной активностью ВСО каждым из pIMR53 (отрицательный контроль, ОК), вектора pIMR53-TEFINtp-BCO (дикий тип, SEQ ID NO: 2)-CYC1t (положительный контроль, ПК) и библиотеки векторов, экспрессирующих случайные варианты ВСО, полученной в Примере 1, трансформировали штамм СС08-1023 (который представляет собой платформенный штамм для получения ретиноидов) способом с тепловым шоком. После этого были получены колонии, образованные на твердой среде без урацила.
По 350 мкл жидкой минимальной среды 2 для Yarrowia lipolytica (YLMM2) без урацила добавляли в каждую лунку 96-луночных планшетов с глубокими лунками (31 планшет), инокулировали полученные колонии и культивировали при 30°С в течение 48 часов. Затем колонии наносили пятнами на твердую среду YLMM1 без урацила и культивировали при 30°С в течение 3 суток. По завершении культивирования сначала из общего числа 3000 штаммов отобрали 17 видов штаммов, которые имели более светлое окрашивание по сравнению со штаммом, трансформированным положительным контролем (CC08-1023/pIMR53-TEFINtp-BCO-CYC1t). Способ получения вышеописанной среды YLMM2 и ее композиция следующие.
<YLMM2 (рН 7,0)>
40 г/л глюкозы, 1,7 г/л основы азотного агара для дрожжей, без аминокислот и сульфата аммония, 0,5 г/л урацила и 2,5 г/л сульфата аммония, растворенные в 0,1 М натрийфосфатном буфере (рН 7,0)
Пример 4. Вторичный отбор штамма с усиленной активностью ВСО
На 17 штаммах, полученных в результате первичного отбора в Примере 3, выполняли тестирование в колбах.
В колбу с перегородками вместимостью 250 мл, содержащую 50 мл минимальной среды 2 для Yarrowia lipolytica (YLMM2) без урацила, инокулировали каждый из 17 видов штаммов и штамм, трансформированный вектором положительного контроля (ПК), при начальной оптической плотности (OD) 1 и культивировали при 30°С в течение 48 часов со встряхиванием при 250 об/мин. По завершении культивирования 1 мл культуральной среды центрифугировали и отбрасывали супернатант. Затем добавляли к осадку 0,5 мл диметилсульфоксида (ДМСО, Sigma) и разрушали клетки встряхиванием при 2000 об/мин при 55°С в течение 10 минут. Далее добавляли 0,5 мл ацетона (Sigma) и встряхивали при 2000 об/мин при 45°С в течение 15 минут, чтобы экстрагировать ретинол и ретиналь, и определяли концентрацию экстрагированных веществ анализом на приборе для ВЭЖХ. Результаты тестирования в колбах отобранных штаммов и результаты измерения OD, концентрации и содержания ретинола и ретиналя показаны в Таблице 2 ниже.
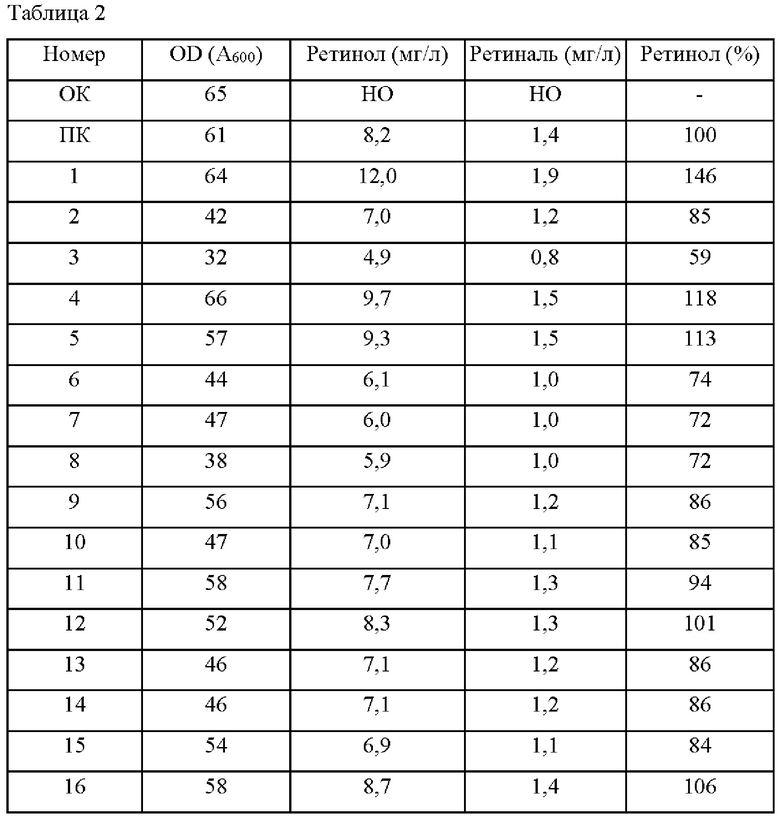

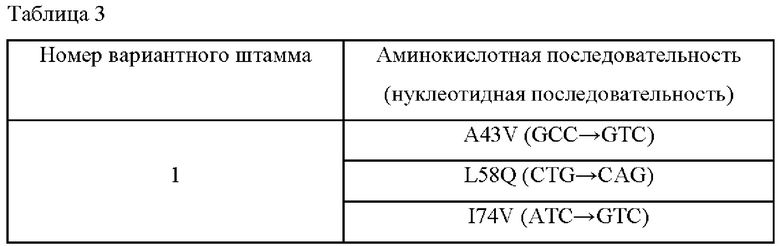
Далее концентрации ретинола и ретиналя, экстрагированных из протестированных образцов, использовали в качестве стандарта для вторичного отбора. Было подтверждено, что среди оцениваемых штаммов пять штаммов (№1, 4, 5, 12 и 16) продемонстрировали повышение концентрации ретинола (ретинол (%)) по сравнению с положительным контролем (ПК). В частности, было подтверждено, что в случае штамма №1 концентрация ретинола была повышена на примерно 46% по сравнению с концентрацией в положительном контроле (ПК). Поэтому плазмиду, которой был трансформирован штамм №1, показавшую наибольшее повышение концентрации ретинола, экстрагировали и провели анализ ее последовательности, чтобы идентифицировать случайные мутации, расположенные в открытой рамке считывания (ОРС) гена ВСО. Использованную для трансформации плазмиду ВСО экстрагировали из отобранного штамма с помощью набора Zymoprep Yeast Plasmid Miniprep Kit 2 (Zymo Research) и проводили анализ последовательности в обоих направлениях с использованием SEQ ID NO: 45 и SEQ ID NO: 46 для повышения достоверности секвенирования. В результате было подтверждено, что количество мутаций в ОРС ВСО было равно трем, и эти подтвержденные мутации показаны в Таблице 3.
Приведенные выше результаты подтвердили, что введение варианта ВСО по настоящему изобретению может количественно повышать продуцирование ретиноидов.
Из вышеприведенного описания специалисту в данной области техники будет понятно, что настоящее изобретение может быть осуществлено в другой специфической форме без изменения его технической сущности или существенных признаков. В этой связи следует понимать, что приведенное выше воплощение не является ограничивающим, а во всех аспектах является иллюстративным. Объем изобретения определяется прилагаемой формулой изобретения, а не предшествующим ей описанием, и поэтому все изменения и модификации, находящиеся в границах и пределах формулы изобретения или эквивалентах таких границ и пределов, рассматриваются как охватываемые формулой изобретения.
Перечень последовательностей
Последовательности по настоящему изобретению показаны в Таблицах 4-6 ниже.













--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="en"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="P24042RU-st26-RU.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2024-03-18">
<ApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>PCT/KR2022/010385</ApplicationNumberText>
<FilingDate>2022-07-15</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>P24042RU</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>10-2021-0093034</ApplicationNumberText>
<FilingDate>2021-07-15</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">СИДЖЕЙ ЧЕИЛДЖЕДАНГ КОРПОРЕЙШН
</ApplicantName>
<ApplicantNameLatin>CJ CHEILJEDANG CORPORATION</ApplicantNameLatin>
<InventionTitle languageCode="ru">Новый вариант
бета-каротин-15,15′-оксигеназы и способ получения ретиноида с его
использованием</InventionTitle>
<SequenceTotalQuantity>46</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>275</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..275</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MGLMLIDWCALALVVFIGLPHGALDAAISFSMISSAKRIARLAGILLIY
LLLATAFFLIWYQLPAFSLLIFLLISIIHFGMADFNASPSKLKWPHIIAHGGVVTVWLPLIQKNEVTKLF
SILTNGPTPILWDILLIFFLCWSIGVCLHTYETLRSKHYNIAFELIGLIFLAWYAPPLVTFATYFCFIHS
RRHFSFVWKQLQHMSSKKMMIGSAIILSCTSWLIGGGIYFFLNSKMIASEAALQTVFIGLAALTVPHMIL
IDFIFRPHSSRIKIKN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>828</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..828</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggcttgatgttaattgattggtgtgctttagcattggttgtgttta
ttggtttgccacatggtgccttagatgctgctatttctttttcaatgatttcttcagcaaagagaattgc
tagattagcaggaatactattaatttacctgttgttagcaaccgcattttttttaatttggtatcaatta
ccagcattttctcttcttatttttcttttgataagcataatccattttggaatggctgatttcaatgcat
ccccaagtaaacttaagtggcctcatattattgcacatggcggcgttgttactgtttggttgccgcttat
ccaaaaaaatgaagttacgaagctattttcaatattaacaaatggtccaactcccattttatgggacata
ctattgatattttttttatgttggagcataggagtatgtcttcatacctatgaaactttacgttctaaac
attataatatcgcctttgaacttattggattaatttttctagcctggtatgcacccccactcgttacttt
tgccacatacttctgctttatccacagcagacgtcactttagttttgtttggaaacagttacagcatatg
agttcaaaaaaaatgatgataggtagtgccattattttatcttgtacgagctggttgataggcggaggaa
tatattttttcctcaattcgaaaatgattgccagtgaagctgctttacaaactgtctttattggtcttgc
agctttaacagttcctcacatgatacttatcgactttatatttagaccacactcttccagaattaaaatc
aaaaattga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>275</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..275</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MGLMLIDWCALALVVFIGLPHGALDAAISFSMISSAKRIARLVGILLIY
LLLATAFFQIWYQLPAFSLLIFLLVSIIHFGMADFNASPSKLKWPHIIAHGGVVTVWLPLIQKNEVTKLF
SILTNGPTPILWDILLIFFLCWSIGVCLHTYETLRSKHYNIAFELIGLIFLAWYAPPLVTFATYFCFIHS
RRHFSFVWKQLQHMSSKKMMIGSAIILSCTSWLIGGGIYFFLNSKMIASEAALQTVFIGLAALTVPHMIL
IDFIFRPHSSRIKIKN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>828</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..828</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggcctgatgctgatcgactggtgtgccctggccctggtggtgttca
tcggcctgccccacggcgccctggacgccgccatctctttctctatgatctcttctgccaagcgaatcgc
ccgactggtcggcatcctgctgatctacctgctgctggccaccgccttcttccagatctggtaccagctg
cccgccttctctctgctgatcttcctgctggtctctatcatccacttcggcatggccgacttcaacgcct
ctccctctaagctgaagtggccccacatcatcgcccacggcggcgtggtgaccgtgtggctgcccctgat
ccagaagaacgaggtgaccaagctgttctctatcctgaccaacggccccacccccatcctgtgggacatc
ctgctgatcttcttcctgtgttggtctatcggcgtgtgtctgcacacctacgagaccctgcgatctaagc
actacaacatcgccttcgagctgatcggcctgatcttcctggcctggtacgccccccccctggtgacctt
cgccacctacttctgtttcatccactctcgacgacacttctctttcgtgtggaagcagctgcagcacatg
tcttctaagaagatgatgatcggctctgccatcatcctgtcttgtacctcttggctgatcggcggcggca
tctacttcttcctgaactctaagatgatcgcctctgaggccgccctgcagaccgtgttcatcggcctggc
cgccctgaccgtgccccacatgatcctgatcgacttcatcttccgaccccactcttctcgaatcaagatc
aagaactag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttttgcagtactaaccgcagggcctgatgctgatcgactg</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgacataactaattacatgactagttcttgatcttgattc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctttcgtcttcaaggaattagagaccgggttggcggcgc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtgacataactaattacatgactgcggttagtactgcaaaaag</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttttgcagtactaaccgcagtcatgtaattagttatgtcacg</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtcgcggccgcggcgcgccggcaaattaaagccttcgagcgt</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgccgccaacccggtctctaattccttgaagacgaaagg</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acgctcgaaggctttaatttgccggcgcgccgcggccgcgacga</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>2019</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2019</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgacggctctcgcatattaccagatccatctgatctatactctcccaa
ttcttggtcttctcggcctgctcacttccccgattttgacaaaatttgacatctacaaaatatcgatcct
cgtatttattgcgtttagtgcaaccacaccatgggactcatggatcatcagaaatggcgcatggacatat
ccatcagcggagagtggccaaggcgtgtttggaacgtttctagatgttccatatgaagagtacgctttct
ttgtcattcaaaccgtaatcaccggcttggtctacgtcttggcaactaggcaccttctcccatctctcgc
gcttcccaagactagatcgtccgccctttctctcgcgctcaaggcgctcatccctctgcccattatctac
ctatttaccgctcaccccagcccatcgcccgacccgctcgtgacagatcactacttctacatgcgggcac
tctccttactcatcaccccacctaccatgctcttggcagcattatcaggcgaatatgctttcgattggaa
aagtggccgagcaaagtcaactattgcagcaatcatgatcccgacggtgtatctgatttgggtagattat
gttgctgtcggtcaagactcttggtcgatcaacgatgagaagattgtagggtggaggcttggaggtgtac
tacccattgaggaagctatgttcttcttactgacgaatctaatgattgttctgggtctgtctgcctgcga
tcatactcaggccctatacctgctacacggtcgaactatttatggcaacaaaaagatgccatcttcattt
cccctcattacaccgcctgtgctctccctgttttttagcagccgaccatactcttctcagccaaaacgtg
acttggaactggcagtcaagttgttggaggaaaagagccggagcttttttgttgcctcggctggatttcc
tagcgaagttagggagaggctggttggactatacgcattctgccgggtgactgatgatcttatcgactct
cctgaagtatcttccaacccgcatgccacaattgacatggtctccgattttcttaccctactatttgggc
ccccgctacacccttcgcaacctgacaagatcctttcttcgcctttacttcctccttcgcacccttcccg
acccacgggaatgtatcccctcccgcctcctccttcgctctcgcctgccgagctcgttcaattccttacc
gaaagggttcccgttcaataccatttcgccttcaggttgctcgctaagttgcaagggctgatccctcgat
acccactcgacgaactccttagaggatacaccactgatcttatctttcccttatcgacagaggcagtcca
ggctcggaagacgcctatcgagaccacagctgacttgctggactatggtctatgtgtagcaggctcagtc
gccgagctattggtctatgtctcttgggcaagtgcaccaagtcaggtccctgccaccatagaagaaagag
aagctgtgttagtggcaagccgagagatgggaactgcccttcagttggtgaacattgctagggacattaa
aggggacgcaacagaagggagattttacctaccactctcattctttggtcttcgggatgaatcaaagctt
gcgatcccgactgattggacggaacctcggcctcaagatttcgacaaactcctcagtctatctccttcgt
ccacattaccatcttcaaacgcctcagaaagcttccggttcgaatggaagacgtactcgcttccattagt
cgcctacgcagaggatcttgccaaacattcttataagggaattgaccgacttcctaccgaggttcaagcg
ggaatgcgagcggcttgcgcgagctacctactgatcggccgagagatcaaagtcgtttggaaaggagacg
tcggagagagaaggacagttgccggatggaggagagtacggaaagtcttgagtgtggtcatgagcggatg
ggaagggcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>1966</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1966</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgggaaaagaacaagatcaggataaacccacagctatcatcgtgggat
gtggtatcggtggaatcgccactgccgctcgtcttgctaaagaaggtttccaggtcacggtgttcgagaa
gaacgactactccggaggtcgatgctctttaatcgagcgagatggttatcgattcgatcaggggcccagt
ttgctgctcttgccagatctcttcaagcagacattcgaagatttgggagagaagatggaagattgggtcg
atctcatcaagtgtgaacccaactatgtttgccacttccacgatgaagagactttcactctttcaaccga
catggcgttgctcaagcgggaagtcgagcgttttgaaggcaaagatggatttgatcggttcttgtcgttt
atccaagaagcccacagacattacgagcttgctgtcgttcacgtcctgcagaagaacttccctggcttcg
cagcattcttacggctacagttcattggccaaatcctggctcttcaccccttcgagtctatctggacaag
agtttgtcgatatttcaagaccgacagattacgaagagtcttctcgtttgcagtgatgtacatgggtcaa
agcccatacagtgcgcccggaacatattccttgctccaatacaccgaattgaccgagggcatctggtatc
cgagaggaggcttttggcaggttcctaatactcttcttcagatcgtcaagcgcaacaatccctcagccaa
gttcaatttcaacgctccagtttcccaggttcttctctctcctgccaaggaccgagcgactggtgttcga
cttgaatccggcgaggaacatcacgccgatgttgtgattgtcaatgctgacctcgtttacgcctccgagc
acttgattcctgacgatgccagaaacaagattggccaactgggtgaagtcaagagaagttggtgggctga
cttagttggtggaaagaagctcaagggaagttgcagtagtttgagcttctactggagcatggaccgaatc
gtggacggtctgggcggacacaatatcttcttggccgaggacttcaagggatcattcgacacaatcttcg
aggagttgggtctcccagccgatccttccttttacgtgaacgttccctcgcgaatcgatccttctgccgc
tcccgaaggcaaagatgctatcgtcattcttgtgccgtgtggccatatcgacgcttcgaaccctcaagat
tacaacaagcttgttgctcgggcaaggaagtttgtgatccacacgctttccgccaagcttggacttcccg
actttgaaaaaatgattgtggcagagaaggttcacgatgctccctcttgggagaaagaattcaacctcaa
ggacggaagcatcttgggactggctcacaactttatgcaagttcttggtttcaggccgagcaccagacat
cccaagtatgacaagttgttctttgtcggggcttcgactcatcccggaactggggttcccatcgtcttgg
ctggagccaagttaactgccaaccaagttctcgaatcctttgaccgatccccagctccagatcccaatat
gtcactctccgtaccatatggaaaacctctcaaatcaaatggaacgggtatcgattctcaggtccagctg
aagttcatggatttggagagatgggtataccttttggtgttgttgattggggccgtgatcgctcgatccg
ttggtgttcttgctttctgaagcaagacaacgatcctttcttagagttttattttagtctcttcctgtgt
tctctctatatacatactctgctcgtctgttctcttctcgagggttcctctttactttgtgtcagagtca
tacccggtctctcaacgtccgtttgagggctagacaattgttagtctcgaaatctccatcacctcaagtc
tgatgttcatcatcttttttattcgtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>2830</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2830</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagaccgggttggcggcgcatttgtgtcccaaaaaacagccccaattg
ccccaattgaccccaaattgacccagtagcgggcccaaccccggcgagagcccccttctccccacatatc
aaacctcccccggttcccacacttgccgttaagggcgtagggtactgcagtctggaatctacgcttgttc
agactttgtactagtttctttgtctggccatccgggtaacccatgccggacgcaaaatagactactgaaa
atttttttgctttgtggttgggactttagccaagggtataaaagaccaccgtccccgaattacctttcct
cttcttttctctctctccttgtcaactcacacccgaaatcgttaagcatttccttctgagtataagaatc
attcaaaatggtgagtttcagaggcagcagcaattgccacgggctttgagcacacggccgggtgtggtcc
cattcccatcgacacaagacgccacgtcatccgaccagcactttttgcagtactaaccgcagacggctct
cgcatattaccagatccatctgatctatactctcccaattcttggtcttctcggtctgctcacttccccg
attttgacaaaatttgacatctacaaaatatcgatcctcgtatttattgcgtttagtgcaaccacaccat
gggactcatggatcatcagaaatggcgcatggacatatccatcagcggagagtggccaaggcgtgtttgg
aacgtttctagatgttccatatgaagagtacgctttctttgtcattcaaaccgtaatcaccggcttggtc
tacgtcttggcaactaggcaccttctcccatctctcgcgcttcccaagactagatcgtccgccctttctc
tcgcgctcaaggcgctcatccctctgcccattatctacctatttaccgctcaccccagcccatcgcccga
cccgctcgtgacagatcactacttctacatgcgggcactctccttactcatcaccccacctaccatgctc
ttggcagcattatcaggcgaatatgctttcgattggaaaagtggccgagcaaagtcaactattgcagcaa
tcatgatcccgacggtgtatctgatttgggtagattatgttgctgtcggtcaagactcttggtcgatcaa
cgatgagaagattgtagggtggaggcttggaggtgtactacccattgaggaagctatgttcttcttactg
acgaatctaatgattgttctgggtctgtctgcctgcgatcatactcaggccctatacctgctacacggtc
gaactatttatggcaacaaaaagatgccatcttcatttcccctcattacaccgcctgtgctctccctgtt
ttttagcagccgaccatactcttctcagccaaaacgtgacttggaactggcagtcaagttgttggaggaa
aagagccggagcttttttgttgcctcggctggatttcctagcgaagttagggagaggctggttggactat
acgcattctgccgggtgactgatgatcttatcgactctcctgaagtatcttccaacccgcatgccacaat
tgacatggtctccgattttcttaccctactatttgggcccccgctacacccttcgcaacctgacaagatc
ctttcttcgcctttacttcctccttcgcacccttcccgacccacgggaatgtatcccctcccgcctcctc
cttcgctctcgcctgccgagctcgttcaattccttaccgaaagggttcccgttcaataccatttcgcctt
caggttgctcgctaagttgcaagggctgatccctcgatacccactcgacgaactccttagaggatacacc
actgatcttatctttcctttatcgacagaggcagtccaggctcggaagacgcctatcgagaccacagctg
acttgctggactatggtctatgtgtagcaggctcagtcgccgagctattggtctatgtctcttgggcaag
tgcaccaagtcaggtccctgccaccatagaagaaagagaagctgtgttagtggcaagccgagagatggga
actgcccttcagttggtgaacattgctagggacattaaaggggacgcaacagaagggagattttacctac
cactctcattctttggtcttcgggatgaatcaaagcttgcgatcccgactgattggacggaacctcggcc
tcaagatttcgacaaactcctcagtctatctccttcgtccacattaccatcttcaaacgcctcagaaagc
ttccggttcgaatggaagacgtactcgcttccattagtcgcctacgcagaggatcttgccaaacattctt
ataagggaattgaccgacttcctaccgaggttcaagcgggaatgcgagcggcttgcgcgagctacctact
gatcggccgagagatcaaagtcgtttggaaaggagacgtcggagagagaaggacagttgccggatggagg
agagtacggaaagtcttgagtgtggtcatgagcggatgggaagggcagtaactcgagtcatgtaattagt
tatgtcacgcttacattcacgccctccccccacatccgctctaaccgaaaaggaaggagttagacaacct
gaagtctaggtccctatttatttttttatagttatgttagtattaagaacgttatttatatttcaaattt
ttcttttttttctgtacagacgcgtgtacgcatgtaacattatactgaaaaccttgcttgagaaggtttt
gggacgctcgaaggctttaatttgcggcgcgcggaggccctttcgtcttca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>2555</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2555</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agagaccgggttggcggcgcatttgtgtcccaaaaaacagccccaattg
ccccaattgaccccaaattgacccagtagcgggcccaaccccggcgagagcccccttctccccacatatc
aaacctcccccggttcccacacttgccgttaagggcgtagggtactgcagtctggaatctacgcttgttc
agactttgtactagtttctttgtctggccatccgggtaacccatgccggacgcaaaatagactactgaaa
atttttttgctttgtggttgggactttagccaagggtataaaagaccaccgtccccgaattacctttcct
cttcttttctctctctccttgtcaactcacacccgaaatcgttaagcatttccttctgagtataagaatc
attcaaaatggtgagtttcagaggcagcagcaattgccacgggctttgagcacacggccgggtgtggtcc
cattcccatcgacacaagacgccacgtcatccgaccagcactttttgcagtactaaccgcagggaaaaga
acaagatcaggataaacccacagctatcatcgtgggatgtggtatcggtggaatcgccactgccgctcgt
cttgctaaagaaggtttccaggtcacggtgttcgagaagaacgactactccggaggtcgatgctctttaa
tcgagcgagatggttatcgattcgatcaggggcccagtttgctgctcttgccagatctcttcaagcagac
attcgaagatttgggagagaagatggaagattgggtcgatctcatcaagtgtgaacccaactatgtttgc
cacttccacgatgaagagactttcactctttcaaccgacatggcgttgctcaagcgggaagtcgagcgtt
ttgaaggcaaagatggatttgatcggttcttgtcgtttatccaagaagcccacagacattacgagcttgc
tgtcgttcacgtcctgcagaagaacttccctggcttcgcagcattcttacggctacagttcattggccaa
atcctggctcttcaccccttcgagtctatctggacaagagtttgtcgatatttcaagaccgacagattac
gaagagtcttctcgtttgcagtgatgtacatgggtcaaagcccatacagtgcgcccggaacatattcctt
gctccaatacaccgaattgaccgagggcatctggtatccgagaggaggcttttggcaggttcctaatact
cttcttcagatcgtcaagcgcaacaatccctcagccaagttcaatttcaacgctccagtttcccaggttc
ttctctctcctgccaaggaccgagcgactggtgttcgacttgaatccggcgaggaacatcacgccgatgt
tgtgattgtcaatgctgacctcgtttacgcctccgagcacttgattcctgacgatgccagaaacaagatt
ggccaactgggtgaagtcaagagaagttggtgggctgacttagttggtggaaagaagctcaagggaagtt
gcagtagtttgagcttctactggagcatggaccgaatcgtggacggtctgggcggacacaatatcttctt
ggccgaggacttcaagggatcattcgacacaatcttcgaggagttgggtctcccagccgatccttccttt
tacgtgaacgttccctcgcgaatcgatccttctgccgctcccgaaggcaaagatgctatcgtcattcttg
tgccgtgtggccatatcgacgcttcgaaccctcaagattacaacaagcttgttgctcgggcaaggaagtt
tgtgatccacacgctttccgccaagcttggacttcccgactttgaaaaaatgattgtggcagagaaggtt
cacgatgctccctcttgggagaaagaattcaacctcaaggacggaagcatcttgggactggctcacaact
ttatgcaagttcttggtttcaggccgagcaccagacatcccaagtatgacaagttgttctttgtcggggc
ttcgactcatcccggaactggggttcccatcgtcttggctggagccaagttaactgccaaccaagttctc
gaatcctttgaccgatccccagctccagatcccaatatgtcactctccgtaccatatggaaaacctctca
aatcaaatggaacgggtatcgattctcaggtccagctgaagttcatggatttggagagatgggtatacct
tttggtattgttgattggggccgtgatcgctcgatccgttggtgttcttgctttctgactcgagtcatgt
aattagttatgtcacgcttacattcacgccctccccccacatccgctctaaccgaaaaggaaggagttag
acaacctgaagtctaggtccctatttatttttttatagttatgttagtattaagaacgttatttatattt
caaatttttcttttttttctgtacagacgcgtgtacgcatgtaacattatactgaaaaccttgcttgaga
aggttttgggacgctcgaaggctttaatttgccggccgcgacgagtatctgtctga</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>1533</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1533</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcctcctgtctgactcgtcattgccgcctttggagtacgactccaact
atgagtgtgcttggatcactttgacgatacattcttcgttggaggctgtgggtctgacagctgcgttttc
ggcgcggttggccgacaacaatatcagctgcaacgtcattgctggctttcatcatgatcacatttttgtc
ggcaaaggcgacgcccagagagccattgacgttctttctaatttggaccgatagccgtatagtccagtct
atctataagttcaactaactcgtaactattaccataacatatacttcactgccccagataaggttccgat
aaaaagttctgcagactaaatttatttcagtctcctcttcaccaccaaaatgccctcctacgaagctcga
gctaacgtccacaagtccgcctttgccgctcgagtgctcaagctcgtggcagccaagaaaaccaacctgt
gtgcttctctggatgttaccaccaccaaggagctcattgagcttgccgataaggtcggaccttatgtgtg
catgatcaagacccatatcgacatcattgacgacttcacctacgccggcactgtgctccccctcaaggaa
cttgctcttaagcacggtttcttcctgttcgaggacagaaagttcgcagatattggcaacactgtcaagc
accagtacaagaacggtgtctaccgaatcgccgagtggtccgatatcaccaacgcccacggtgtacccgg
aaccggaatcattgctggcctgcgagctggtgccgaggaaactgtctctgaacagaagaaggaggacgtc
tctgactacgagaactcccagtacaaggagttcctggtcccctctcccaacgagaagctggccagaggtc
tgctcatgctggccgagctgtcttgcaagggctctctggccactggcgagtactccaagcagaccattga
gcttgcccgatccgaccccgagtttgtggttggcttcattgcccagaaccgacctaagggcgactctgag
gactggcttattctgacccccggggtgggtcttgacgacaagggagacgctctcggacagcagtaccgaa
ctgttgaggatgtcatgtctaccggaacggatatcataattgtcggccgaggtctgtacggccagaaccg
agatcctattgaggaggccaagcgataccagaaggctggctgggaggcttaccagaagattaactgttag
aggttagactatggatatgtcatttaactgtgtatatagagagcgtgcaagtatggagcgcttgttcagc
ttgtatgatggtcagacgacctgtctgatcgagtatgtatgatactgcacaacctgtgtatccgcatgat
ctgtccaatggggcatgttgttgtgtttctcgatacggagatgctgggtacaagtagctaatacgattga
actacttatacttatatgaggcttgaagaaagctgacttgtgtatgacttattctcaactacatccccag
tcacaataccacca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgcgcttctctcgtctcggtaaccctgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgcgccgccaacccggtctctggggtgtggtggatggggtgtg</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacaccccatccaccacaccccagagaccgggttggcggcgcat</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>39</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgccgccaacccggtctcttgaagacgaaagggcctccg</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>39</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q23">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggaggccctttcgtcttcaagagaccgggttggcggcg</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacgagtcagacaggaggcatcagacagatactcgtcgcg</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>40</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..40</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q25">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcgacgagtatctgtctgatgcctcctgtctgactcgtc</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgacgagtcagacaggaggcatggtggtattgtgactggggat</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q27">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atccccagtcacaataccaccatgcctcctgtctgactcgtcat</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cggcgtccttctcgtagtccgcttttggtggtgaagaggagact</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q29">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agtctcctcttcaccaccaaaagcggactacgagaaggacgccg</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccactcgtcaccaacagtgccgtgtgttgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q31">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcgtacgtctataccaacagatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgcatacacacacactgccggggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>550</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..550</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q33">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccacacgtcgttcttttttccttagccttttttgcagtgcgcgtgtcc
caaaccccagctctacacaccagcacaaacaaagttaagctcagggttgtcgttgaggtcgcttactgta
gtcagtgctcgtatggttcgttcaattttcgccaaaaatcgttttgcctttgtatcttgggaataacatc
aactgtggttcttcaacaggcctaaggaacgaaacaagccggaccaagatcaggttcaaggtgagtactg
agaaggaatagaaggcctaaaggcgcaaaccgacaggtggcaacagctccacaccgaccacgaaggccac
gaaatcaaggggtcctaaagttagtctttgtggcctcgacggtcagcgaaaacgcgagaccacaacgcga
tcagaaccaggacctaaacaacacaggacggggtcacaataggcttgaacagcaagtacaagctgtgatc
tctctatatttgattctcaaaccacccctgactacttcagcgcctctgtgacacagcccccctatcatcc
gactaacacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacaatgcctcgaggaggtttaaaagtaact</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q35">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgccgccaacccggtctctctgtgttagtcggatgatagg</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctatcatccgactaacacagagagaccgggttggcggcgc</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q37">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacgagtcagacaggaggcactgcggttagtactgcaaaaag</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>42</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..42</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttttgcagtactaaccgcagtgcctcctgtctgactcgtc</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q39">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgcgccgccaacccggtctcttggtggtattgtgactggggat</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>44</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..44</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atccccagtcacaataccaccaagagaccgggttggcggcgcat</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q41">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttccaatagctgcttgtagctgcggttagtactgcaaaa</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>41</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..41</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttttgcagtactaaccgcagctacaagcagctattggaaag</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q43">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcttaatgtgattgatctcaaacttgatag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgtctctgcgagagcacgtcga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q45">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggttcgcacaacttctcgggtggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttctgagtataagaatcattcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q47">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tttgaaatataaataacgttctta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к биотехнологии и представляет собой вариант бета-каротин-15,15'-оксигеназы, где аминокислота, соответствующая положению 43 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, аланин; аминокислота, соответствующая положению 58 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, лейцин; и аминокислота, соответствующая положению 74 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, изолейцин, каждая заменена на другую аминокислоту, где вариант имеет активность бета-каротин-15,15'-оксигеназы, а также микроорганизм, содержащий полинуклеотид, кодирующий вариант бета-каротин-15,15'-оксигеназы. Изобретение позволяет получать ретиноид путем культивирования такого микроорганизма с высокой степенью эффективности. 6 н. и 9 з.п. ф-лы, 4 табл., 4 пр.
1. Вариант бета-каротин-15,15'-оксигеназы, где аминокислота, соответствующая положению 43 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, аланин; аминокислота, соответствующая положению 58 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, лейцин; и аминокислота, соответствующая положению 74 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, изолейцин, каждая заменена на другую аминокислоту, где вариант имеет активность бета-каротин-15,15'-оксигеназы.
2. Вариант бета-каротин-15,15'-оксигеназы по п. 1, содержащий замену аминокислоты, соответствующей положению 43 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, на аминокислоту, отличную от аланина; замену аминокислоты, соответствующей положению 58 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, на аминокислоту, отличную от лейцина; и замену аминокислоты, соответствующей положению 74 относительно N-конца аминокислотной последовательности SEQ ID NO: 1, на аминокислоту, отличную от изолейцина.
3. Вариант бета-каротин-15,15'-оксигеназы по п. 1, где замена аминокислоты, соответствующей положению 43, на другую аминокислоту представляет собой замену на неполярную аминокислоту; замена аминокислоты, соответствующей положению 58, на другую аминокислоту представляет собой замену на полярную аминокислоту; и замена аминокислоты, соответствующей положению 74, на другую аминокислоту представляет собой замену на неполярную аминокислоту.
4. Вариант бета-каротин-15,15'-оксигеназы по п. 1, где другая аминокислота представляет собой валин или глутамин.
5. Полинуклеотид, кодирующий вариант по п. 1.
6. Микроорганизм для продуцирования ретиноида, содержащий вариант по п. 1 или полинуклеотид, кодирующий указанный вариант.
7. Микроорганизм по п. 6, где ретиноид является любым одним или более, выбранным из группы, состоящей из ретиналя, ретинола, сложного эфира ретинила и ретиноевой кислоты.
8. Микроорганизм по п. 6, принадлежащий к Yarrowia sp.
9. Микроорганизм по п. 6, представляющий собой Yarrowia lipolytica.
10. Способ получения ретиноида, включающий культивирование микроорганизма для продуцирования ретиноида, содержащего вариант по п. 1 или полинуклеотид, кодирующий указанный вариант, в среде.
11. Способ по п. 10, дополнительно включающий извлечение ретиноида из среды или микроорганизма.
12. Способ по п. 10, где микроорганизм принадлежит к Yarrowia sp.
13. Способ по п. 10, где ретиноид является любым одним или более, выбранным из группы, состоящей из ретиналя, ретинола, сложного эфира ретинила и ретиноевой кислоты.
14. Композиция для получения ретиноида, содержащая вариант по п. 1, микроорганизм по п. 7 или культуру указанного микроорганизма и эксципиент.
15. Применение микроорганизма для получения ретиноида, содержащего вариант по п. 1 или полинуклеотид, кодирующий указанный вариант.
| US 20200231993 A1, 23.07.2020 | |||
| WO 2021009194 A1, 21.01.2021 | |||
| US 20200277644 A1, 03.09.2020 | |||
| RU 2003108882 A, 10.12.2004. |
Авторы
Даты
2025-01-17—Публикация
2022-07-15—Подача