Изобретение относится к ветеринарной вирусологии, к средствам молекулярной диагностики, а именно к дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Классификация и понимание генетических вариаций между различными изолятами и штаммами микроорганизмов является важной задачей в области геномики. Быстро растет интерес к поддающимся количественной оценке методам скрининга мутаций в исследованиях в области естественных наук. Такие методы используются для обнаружения генов, ответственных за различные признаки [1, 2].
Особый интерес для генетических исследований вызывает вирус бешенства, геном которого представлен несегментированной одноцепочечной негативной молекулой РНК длиной около 12000 н.о. Нуклеиновая кислота кодирует 5 основных белков, расположенных в консервативном линейном порядке: нуклеопротеин (N-белок), фосфопротеин (Р-белок), матриксный белок (М-белок), гликопротеин (G-белок), РНК-зависимую РНК-полимеразу (L-белок) [1]. N-, М- и L-белки схожи по структуре и длине среди всех видов и штаммов лиссавирусов, в то время как длины Р- и G-белков являются переменными [3, 4].
Каждый ген состоит из внутренней кодирующей области, окруженной не кодирующими областями (NCRs), которые граничат с сигналами инициации транскрипции (TISs) и сигналами полиаденилирования завершения транскрипции (ТТР). Данные сигналы состоят примерно из 10 нуклеотидов. Последовательность участка TIS (3'-U-U-G-U-R-R-п-GA-5') строго сохраняется во всех геномах лиссавирусов. Последовательность участков ТТР (3'-A/U-C-U-U-U-U-U-U-U-U-G-5') содержит последовательность из семи остатков уридина, которые повторно копируются с помощью РНК-полимеразы для получения полиаденилированного хвоста каждой мРНК перед повторной инициацией при следующем стартовом сигнале. Гены обычно разделяются сохраненными не транскрибируемыми межгенными областями (IGRs), которые имеют длину 2-6 нуклеотидов [4]. Таким образом, единицы транскрипции разделены короткими не транслируемыми межгенными областями. Исключение составляет межгенная область G-L, которая состоит из 400-700 нуклеотидов и может представлять собой остаток гена, который потерял свою функциональность во время эволюции лиссавирусов [3].
Пять генов в РНК вируса бешенства окружены двумя короткими участками NCRs на 3'- и 5'-концах: лидерная область 3'- (leader) и трейлерная область 5' (trailer). Эти последовательности, соответственно, инициируют и завершают транскрипцию и репликацию генома, соответственно. Leader и trailer очень богаты UTP и АТР. Лидирующая область представлена 58 нуклеотидными остатками, первые девять из которых идентичны у всех представителей лиссавирусов. Длина и последовательность трейлерной области более изменчивы (около 70 нуклеотидов). Последовательности 3'- и 5'-концов генома обратно комплементарны. Эта взаимодополняемость является классической особенностью представителей порядка Mononegavirales [3].
Вирионы имеют пулевидную форму диаметром 75 нм и длиной 100-300 нм в зависимости от штамма. Один конец конический, а другой плоский. Вирусная РНК инкапсидируется N-белком (нуклеопротеином) (450 а.о.) с образованием спирального нуклеокапсида, в котором каждый N-протомер связывается с девятью нуклеотидами [9, 10, 11]. Нуклеокапсид связан со значительным количеством Р-белка (фосфопротеина) (297 а.о.). L-белок (РНК-зависимая РНК-полимераза) - белок с наибольшей длиной (2130 а.о.). Вирион состоит из следующих функциональных структур: рибонуклеопротеин (РНП), расположенный внутри вириона, и внешняя белково-липидная составляющая. РНК, N-, Р- и L-белок образуют РНП, который является компонентом, активно участвующим в транскрипции и репликации. РНП покрыт липидным бислоем, полученным из плазматической мембраны клетки-хозяина в процессе зарождения. Рибонуклеопротеин является важным иммуногенным компонентом вакцинных препаратов против бешенства. М-белок (матричный) (202 а.о.) и G-белок (гликопротеин) (505 а.о.) являются мембраносвязанными белками. М-белок расположен под вирусной мембраной и соединяет нуклеокапсид и липидный бислой. G-белок - интегральный трансмембранный белок, который участвует в проникновении вируса в клетку [4].
N-белок (нуклеопротеин) представляет собой двухдоменный белок с положительно заряженной щелью между двумя доменами с общим молекулярным весом 57 кДа и длиной 450 а.о. Данный белок упаковывает геномную РНК и формирует спиральный нуклеокапсид, который позволяет защитить РНК ВБ от клеточных РНК-аз и факторов, которые распознают чужеродные нуклеиновые кислоты, чтобы начать синтез интерферона [14, 15]. Нуклеопротеин образует комплекс с РНК вируса бешенства, формируя позитивный и негативный РНП, соответственно. При анализе нуклеотидных последовательностей N-гена вируса выявлено наличие высококонсерватиных областей. С помощью моноклональных антител на N-белке выявлены следующие линейные эпитопы антигенных сайтов: сайт I (313-337 а.о.), сайт IV (358-367 а.о.), а также конформационно зависимые сайты II и III. Обнаружено, что антигенные участки сайта I формируются на незрелой форме нуклеопротеина, а эпитопы сайта IV - после полного созревания N-белка [3]. Рибонуклеопротеин в лабораторной практике применяют для получения специфических тест-систем по качественному и количественному определению, в том числе в сырье для антирабических вакцин [4].
G-белок (гликопротеин) находится в наружном слое оболочки вириона, состоит из 524 а.о. и содержит 2 гидроформных элемента. Молекулярный вес гликопротеина составляет 70 кДа для фракции G1 и 65 кДа для фракции G2. Не гликолизированная часть белка погружена в билипидный слой. Гликопротеин составляет примерно 45% от общего количества вирусных белков. Каждая молекула G-белка содержит две олигосахаридные цепи, соединенные с гидрофильной составляющей. Данный белок имеет 4 области: сигнальный пептид, эктодомен, трансмембранный и цитоплазматический домен [3]. Гликопротеин играет ключевую роль в адгезии вириона к мембране клетки-хозяина и в связывании с вируснейтрализующими антителами, а также в нейроинвазивности, поскольку его точечная мутация в положении 333 (аргинин или лизин) полностью устраняет вирулентность, и делеция его гена устраняет транснейронное распространение [19]. Более того, у авирулентных штаммов нейроинвазивность восстанавливается путем генетической замены или трансплантации гликопротеином, полученным из вирулентных штаммов. При отсутствии положительно заряженной аминокислоты в позиции 333 вирион не поражает нервные клетки. Замена аргинина в данной позиции на глютамин или изолейцин снижает нейропатогенность. Мутанты с замененной аминокислотой не могут инфицировать нейроны в связи с отсутствием возможности распознавать рецепторы клеток. В дополнение к связыванию с нейрональными рецепторами, гликопротеин вируса бешенства способствует слиянию вируса и клеточной мембраны и придает вириону транспортные свойствам внутри клетки [3, 4].
Вторичная и третичная формы гликопротеина обуславливают наличие на его поверхности антигенных сайтов. На поверхности эктодомена находится 8 антигенных эпитопов. Экто- и эндодомены G-белка являются растворимыми фракциями, поэтому они не способны вызывать протективный иммунитет. Эктодомен (1-439 а.о.) играет важнейшую роль в патогенезе бешенства, а именно, в прикреплении вириона к цитоплазматической мембране клетки, в связывании с вируснейтрализующими антителами. Именно эктодомен G-белка включает в себя сайт, который отвечает за вирулентность вируса бешенства. Следует также отметить, что гликопротеин является мишенью для специфических Т-хелперов и цитотоксических Т-киллеров [4].
L-белок (Large) имеет молекулярную массу 190 кДа, длину около 2158 а.о. и является компонентом полимеразного комплекса. Это самый большой белок ВБ, L-ген занимает более половины вирусной РНК и включает в себя консервативные последовательности. Для поражения клетки вирус бешенства, имеющий РНК (-), должен содержать в составе своего вириона активный полимеразный комплекс для активации первичной транскрипции после внедрения РНП в цитоплазму и de novo синтезировать белки вируса. После этого происходит репликация геномных РНК и вторичная транскрипция [3].
М-белок (матриксный белок) вируса бешенства имеет небольшой молекулярный вес 20-25 кДа и длину 202 а.о. Это фосфопротеин, представленный в двух изоформах M1 и М2, которые отличаются друг от друга степенью фосфорилирования. Матриксный белок структурно представляет собой мостик между N- и G-белком. М-ген является гораздо более консервативным по сравнению с Р-белком. Считается, что этот белок образует слой между гликопротеином в оболочке вириона и спиральным ядром нуклеокапсида, состоящим из генома РНК и N-, L-, Р-белков. М-белок является основным фактором, способствующим зарождению вируса и морфогенезу вириона, в то время как G-белок играет более вспомогательную роль в этих процессах [4].
Бешенство приводит к значительным экономическим потерям, которые связаны с гибелью животных, ликвидацией последствий вспышек болезни, введением строгих ограничений, налагаемых на внутреннюю и международную торговлю продукцией животноводства, проведением профилактических и карантинных мероприятий и др. В соответствии с Кодексом наземных животных и Руководством МЭБ по диагностическим тестам и вакцинам для борьбы с бешенством должен соблюдаться комплекс мер по вакцинопрофилактике диких плотоядных животных - программа по оральной вакцинации; вакцинопрофилактика домашних животных; современная лечебно-профилактическая вакцинация людей, обратившихся за антирабической помощью, профилактическая вакцинация людей групп риска, прежде всего, профессионального риска; контроль проводимых антирабических мероприятий, включающих в себя блок задач и методик [4].
Живые аттенуированные вакцины, изготовленные на основе вируса бешенства, широко применяются во многих странах для профилактики данного заболевания. В России и странах СНГ применяют вакцину против бешенства, разработанную с применением штамма «РВ-97». Полногеномная нуклеотидная последовательность данного представителя вируса бешенства представлена в GenBank (EF 542830.1) [5].
Выбор подходов для разработки живой вакцины, способной защищать животных без наличия клинических признаков инфекции после иммунизации, является актуальным. Для реализации данной задачи создан вакцинный генно-инженерный штамм «ВНИИЗЖ-G333» вируса бешенства с использованием метода клонирования по Гибсону in vitro и плазмидных конструкции, который включает в себя редактированные локусы вирулентности.
Учитывая, что штаммы «РВ-97» и «ВНИИЗЖ-G333» применяются для производства вакцинных препаратов, но при этом отличаются друг от друга по свойствам, а именно, штамм «ВНИИЗЖ-G333» не является патогенным за счет точечных мутаций, важно использовать диагностические инструменты, которые могут быстро и специфически дифференцировать геном вируса бешенства штаммов «РВ-97» и «ВНИИЗЖ-G333», что важно при контроле качества сырья при изготовлении вакцинных препаратов и подтверждения использования для производства антирабических вакцин именно из генно-модифицированного штамма «ВНИИЗЖ-G333».
В связи с этим целесообразно провести поиск способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства. Для решения поставленной задачи требуется провести молекулярно-биологический анализ генома вируса бешенства, что даст возможность разработать метод для контроля качества и определения специфичности используемого вакцинного сырья.
В настоящее время методы молекулярно-биологического анализа находят широкое применение в диагностике различных инфекционных заболеваний. Благодаря созданию нового поколения флуоресцирующих красителей и технологическим усовершенствованиям платформ количественной ПНР анализ кривой плавления с высоким разрешением стал важным и перспективным методом генотипирования микроорганизмов, который возможно применять для дифференциации вакцинных штаммов от полевых изолятов вирусов [6, 7].
Проведено изучение истории исследований, предшествующих появлению предложенной технологии. В 1960-х годах плавление двухцепочечной ДНК контролировалось по поглощению ультрафиолетового излучения (гиперхромный эффект). Анализ требовал количества ДНК в мкг и часто занимал часы, в то время как образцы медленно нагревались со скоростью 0,1-1,0°С/мин. В современном анализе плавления ДНК используется флуоресценция, и поскольку это более чувствительный метод, для него требуется всего несколько нг ДНК (количества, которые легко получить с помощью ПЦР) [8].
Флуоресцентный анализ плавления ДНК стал популярным с появлением в 1997 году прибора для ПЦР в реальном времени LightCycler®. Данный прибор позволяет достовернее контролировать температуру и обеспечить высокую скорость плавления 0,1-1,0°С/сек, сократив время плавления до нескольких минут. Индикатором флуоресценции, использованным еще в 1997 году, был краситель SYBR®Green I, чувствительный и удобный краситель для мониторинга амплификации и плавления амплифицированных продуктов в режиме реального времени [9].
Анализ плавления с высоким разрешением - новый метод, представленный в 2003 году, который использует сбор данных с высокой плотностью и обнаруживает небольшие различия в последовательностях фрагментов ПЦР только путем прямого плавления. Кривые плавления можно использовать для сканирования мутаций, сопоставления последовательностей и анализа мультиплексного генотипирования. Благодаря своей скорости и простоте, популярность этого метода в последние годы увеличивается [8, 10].
Анализ плавления ампликонов с высоким разрешением представляет собой количественный анализ кривой плавления фрагмента ДНК после реакции амплификации. Для данного метода требуется система ПЦР-детекции в режиме реального времени с высокой термостабильностью и чувствительностью. Комбинация улучшенного инструментария количественной детекции продуктов ПЦР и насыщающих ДНК-связывающих красителей позволила идентифицировать генетические вариации в последовательностях нуклеиновых кислот путем контролируемого плавления двухцепочечного ампликона [11, 12, 13].
Анализ кривых плавления с высоким разрешением дает возможность выявлять новые варианты генов, проводить скрининг образцов ДНК на однонуклеотидные полиморфизмы, выявлять вставки/делеции и другие неизвестные мутации, и определять процент метилированной ДНК в образцах. Данный анализ проводится в амплификаторах в режиме реального времени [14, 15, 16].
Сущность представленного метода заключается в следующем: после проведения ПНР с интеркалирующим красителем смесь постепенно нагревают, и при достижении определенной температуры двухцепочечные молекулы ДНК начинают разъединяться, флуорофор постепенно высвобождается, и флуоресценция падает. Изменение свечения детектируется оптической системой амплификатора. При этом ампликоны с разной последовательностью «плавятся» при разной температуре. Чувствительность метода достигает одного нуклеотида, благодаря чему с помощью данного анализа можно проводить детекцию однонуклеотидных полиморфизмов [13].
Следует отметить, что основываясь на функциональном тестировании имеющихся в настоящее время приборов, получение 10 точек данных на градус Цельсия во время операций плавки, вероятно, является минимальным требованием для обеспечения определенного уровня сканирования мутаций. «Высокое разрешение» будет означать большее количество точек данных на °С [15].
Прототипным вариантом проведения дифференциального анализа является постановка полимеразной цепной реакции с электрофорезом с оригинальными праймерами и с последующим секвенированием по Сенгеру [16]. Однако, данный метод является очень дорогостоящим, продолжительным во времени, поскольку предполагает проведение дополнительных этапов работы с последующим расшифровыванием нуклеотидной последовательности и анализом их с помощью BioEdit и MEGA 7 или аналогов. Исходя из этого, необходимо разработать альтернативный способ исследования образцов.
Техническим решением изобретения является разработка чувствительного и специфичного, экспрессного способа дифференциации генома генетически-модифицированного вакцинного штамма «ВНИИЗЖ-G333» от производственного штамма «РВ-97» методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения с целью устранения вышеуказанных недостатков.
Данная задача решена благодаря разработке способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения. Суть метода заключается в следующем: проводится дизайн праймеров с тем расчетом, чтобы амплифицировать фрагмент ДНК содержащий мутацию, наличие которой нужно подтвердить в генетической конструкции. ПЦР проводится в присутствии интеркалирующего красителя, который встраивается в двуцепочечную ДНК, после чего ее можно обнаружить с помощью считывания флуоресцентного сигнала. Фрагмент анализируемого фрагмента ДНК отличается от генетической конструкции, содержащей мутацию, по нуклеотидному составу. Полученный участок постепенно нагревают в широком диапазоне температур. При достижении температуры плавления двуцепочечная молекула ДНК (дцДНК) распадается на отдельные цепи. При этом пропадает флуоресцентный сигнал специфичного для дцДНК красителя. Для каждого варианта нуклеотидного состава характерна своя температура плавления. Анализируя кривые плавления фрагментов ДНК можно уверенно определять нуклеотидный состав этих фрагментов, а значит - дифференцировать мутантный вариант генетической конструкции от всех прочих [1, 15].
Разработанный способ дает возможность: 1) сократить время проведения анализа до 2-3 ч; 2) применять современный интеркалирующий краситель EvaGreen, который обеспечивает более высокую флуоресценцию по сравнению с SYBR Green, что повышает точность и чувствительность проводимого анализа; 3) увеличить чувствительность и специфичность анализа за счет использования высокоспецифичных оригинальных праймеров, рассчитанных для таргетного участка ДНК вируса бешенства. Данная разработка позволит расширить арсенал средств для проведения контроля качества сырья при производстве аттенуированных вакцин против бешенства.
Сущность изобретения отражена на графических изображениях:
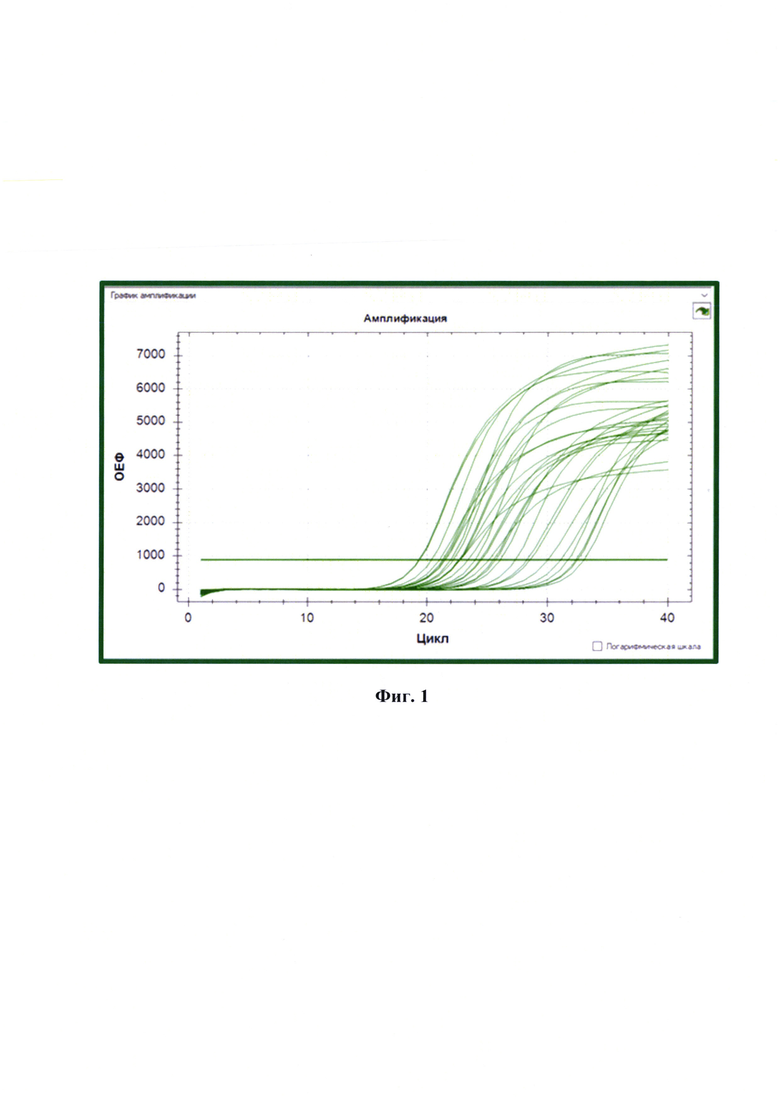
Фиг. 1 - График накопления флуоресцентного сигнала при исследовании пяти пар праймеров для вируса бешенства штаммов «ВНИИЗЖ-G333» и «РВ-97».
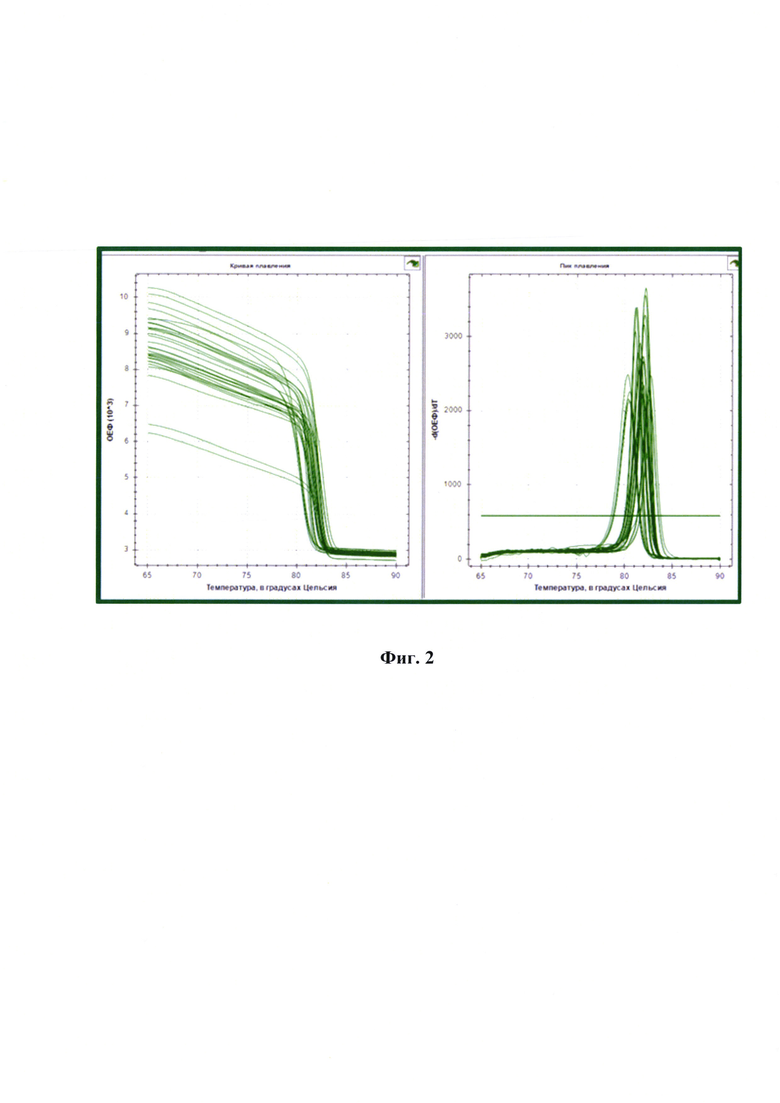
Фиг. 2 - Анализ кривых плавления при анализе пяти пар олигонуклеотидных праймеров для вируса бешенства штаммов «ВНИИЗЖ-G333» и «РВ-97».
Сущность изобретения пояснена в перечне последовательностей, в котором:
SEQ ID NO: 1 представляет последовательность нуклеотидов штамма «ВНИИЗЖ-G333» вакцинного генетически модифицированного вируса бешенства;
SEQ ID NO: 2 представляет последовательность аминокислот штамма «ВНИИЗЖ-G333» вакцинного генетически модифицированного вируса бешенства.
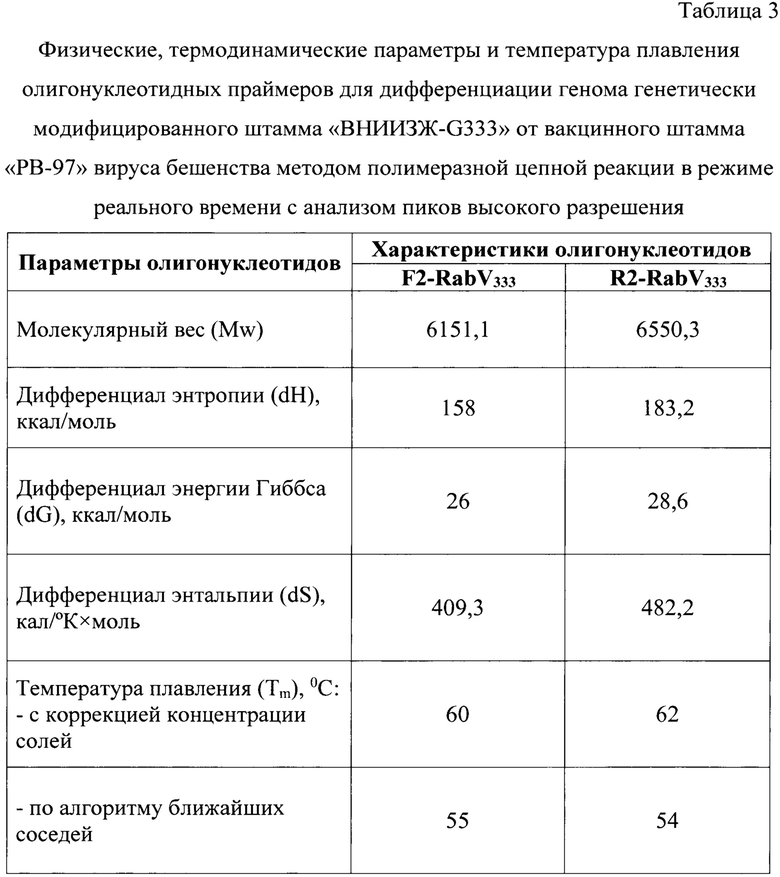
Сущность изобретения заключается в новом подходе по разработке способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения. Заявляемый способ основан на: 1) элюировании РНК вируса бешенства из исследуемого биологического материала; 2) проведении амплификации специфического фрагмента кДНК G-гена вируса бешенства размером 77 п.н. с применением разработанных оригинальных олигонуклеотидных специфических праймеров F2-RabV333 (5'-AGGCTGAGGCTCACTACAAG-3') и R2-RabV333 (5'-CCCTTCGACTCTTAAACACCCT-3') (таблицы 1, 2, 3); 3) проведении плавления ампликонов в разработанном режиме с учетов пиков высокого разрешения; 4) детекции результатов анализа с определением максимального значения температуры плавления и проведении инструментального дифференциального анализа генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
В публикациях многих авторов показана необходимость проведения дифференциации вакцинных штаммов и полевых изолятов вируса бешенства. По данным проведенного анализа литературных данных, в настоящее время доступно несколько анализов для определения вируса бешенства с помощью ПЦР и ПЦР в реальном времени [7, 8, 9, 14], но при этом ни один из них не предназначен для дифференциации генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства. При анализе публикаций способ дифференциации данных штаммов вируса бешенства с помощью молекулярно-генетических методов также не представлен. Таким образом, сведений об аналогах предлагаемого способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения авторами не обнаружено. Прототипом следует считать анализ нуклеотидных последовательностей с помощью секвенирования по Сэнгеру.
Разработанный способ дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения по сравнению с прототипом отличается экономической выгодой и экспрессностью выполнения анализа, что позволяет быстро проводить контроль сырья при изготовлении вакцинных препаратов.
В отличие от прототипа разработанный способ позволил провести анализ выравнивания множественных нуклеотидных последовательностей G-гена штаммов «ВНИИЗЖ-G333» и «РВ-97» вируса бешенства и идентифицировать область с 1054…1056-нуклеотидными заменами GAA на GAA, уникальной для вакцинного штамма «ВНИИЗЖ-G333». Данный способ предусматривает проведение реакции амплификации специфического фрагмента кДНК вируса бешенства в диапазоне 1028…1104 п.н. с применением разработанных оригинальных олигонуклеотидных специфических праймеров F2-RabV333 и R2-RabV333 для амплификации фрагмента размером 109 п.н. Разработанный способ предусматривает проведение плавления ампликонов в разработанном режиме с учетов пиков высокого разрешения, а также детекцию результатов анализа с определением максимального значения температуры плавления и проведением инструментального дифференциального анализа генома указанных штаммов вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения. Таким образом, актуально применять предложенный способ дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Ключевым элементом заявляемого способа является определение с высоким разрешением пиковых значений температур плавления амплифицированных продуктов ПЦР и дифференциация генома штаммов «ВНИИЗЖ-G333» и «РВ-97» вируса бешенства по результатам анализа максимумов графиков плавления ампликонов.
Сопоставительный анализ с прототипом позволяет сделать вывод, что новизна и изобретательский уровень заявляемого изобретения заключаются в применении способа ПЦР в режиме реального времени, оригинальных специфичных олигонуклеотидных праймеров F2-RabV333 и R2-RabV333, рассчитанных для амплификации участка кДНК G-гена вируса бешенства и технологии анализа пиков высокого разрешения для графиков плавления продуктов ПЦР.
Сущность предлагаемого изобретения пояснена на графическом материале - Анализ кривых плавления при анализе пяти пар олигонуклеотидных праймеров для вируса бешенства штаммов «ВНИИЗЖ-G333» и «РВ-97» (фиг. 2).
На основном этапе исследования проводят выделение нуклеиновой кислоты из анализируемых образцов, содержащих вирус бешенства с применением твердофазного способа с использованием набора «РИБО-сорб» в соответствии с инструкцией производителя («Интерлабсервис»).
На следующем этапе проводят ПЦР в режиме реального времени для исследования стандартных образцов и проб. Стандартными образцами являются положительные контроли, представляющие собой индивидуальные суспензии вируса бешенства штаммов «ВНИИЗЖ-G333» и «РВ-97». В качестве отрицательного контроля используют деионизированную воду без нуклеаз MilliQ. Проводят обратную транскрипцию с применением следующих реагентов: деионизированная вода - 50 мкл, буфер 5-кратный - 20 мкл, дезоксирибонуклеозидтрифосфаты - 10 мкл, праймеры - по 4 мкл, MMLV-ревертаза - 2 мкл, элюат РНК - 12 мкл.
Для постановки ПЦР в режиме реального времени готовят реакционную смесь в соответствии с наставлением к набору "SsoFast™ EvaGreen® Supermix". Кроме EvaGreen, используют дополнительные компоненты: деионизированная вода - 25 мкл, PCR buffer 10х - 5 мкл, хлорид магния 25 мМ - 5 мкл, праймеры - по 2 мкл, Taq ДНК-полимераза - 1 мкл, кДНК - 6 мкл. Итоговый объем смеси для проведения одной реакции составляет 25 мкл.
Постановку обратной транскрипции и ПЦР в режиме реального времени осуществляют в детектирующем термоциклере CFX-96 (Bio-Rad, США) при температурных и временных параметрах, сведения о которых представлены в таблице 4.
Обратную транскрипцию осуществляют при температуре 42°С в течение 20 минут. Предварительную денатурацию проводят при температуре 98°С в течение 2 минут. ПЦР в режиме реального времени включает в себя 3 подэтапа: денатурацию, отжиг олигонуклеотидов, элонгацию. Денатурацию осуществляют при температуре 95°С в течение 5 секунд, отжиг олигонуклеотидов и элонгацию - при температуре 60°С в течение 20 секунд. Далее проводят важный этап для осуществления дифференциального анализа - плавление, начиная с 65 до 90°С.Увеличение температуры составляет - 0,1°С за каждый шаг. При этом после первого шага плавления требуется ждать 90 секунд при температуре первого шага. Для каждого последующего шага время ожидания составляет 2 секунды.
Для детекции сигнала устанавливают канал HRM, для которого длина волны источника составляет 460 нм, детектора - 610 нм. В качестве красителя применяется EvaGreen. Данный флуорофор можно анализировать также в системе детекции с длиной волны 488 нм по рекомендации производителя набора "SsoFast™ EvaGreen® Supermix".
Исследование кривой плавления с высоким разрешением для анализа данных и профилей плавления образцов выполняют с использованием программного обеспечения для сканирования генов того детектирующего амплификатора, на котором осуществляют реакцию амплификации и процесс плавления (в частности, CFX 96). Проводят построение кривых плавления в виде параболических функций и детектируют пики высокого разрешения для полученных графиков. Выявляют, что для вакцинных штаммов «ВНИИЗЖ-G333» и «РВ-97» характерны определенные, очень узкие диапазоны температуры плавления, по данным которых можно судить о принадлежности исследуемого образца к одному из указанных производственных штаммов вируса бешенства.
Сущность предлагаемого изобретения пояснена примерами его исследования, которые не ограничивают объем изобретения.
Пример 1. Разработка оригинальных олигонуклеотидных праймеров для способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Для разработки оригинальных специфичных олигонуклеотидных праймеров для использования в способе дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения проводили подборку и анализ выравнивания последовательностей G-гена вируса бешенства штаммов «ВНИИЗЖ-G333» и «РВ-97», а также полевых изолятов (всего 26). Эти последовательности выравнивали с помощью сервиса BioEdit. Анализ позволил идентифицировать область с 1054…1056-нуклеотидными заменами GAA на GAA, уникальной для вакцинного штамма «ВНИИЗЖ-G333». Данная замена была обнаружена в результате сравнительного анализа, проведенного с помощью программы BioEdit и MEGA7.
В результате сравнительного анализа был выбран регион в диапазоне 950…1131 п.н. именно G-гена, поскольку именно данная область позволила разработать прямой и обратный олигонуклеотидные праймеры для однозначной дифференциации штаммов «ВНИИЗЖ-G333» и «РВ-97» вируса бешенства для оценки в анализе в ПЦР с помощью пиков высокого разрешения. Были разработаны праймеры, которые были гомологичны выбранным последовательностям и позволили проводить амплификацию фрагментов G-гена кДНК вируса бешенства. Изначально было создано 5 пар праймеров, но остановились на трех, представленных ниже:
F1-RabV333 - 5'-TGATGGAGGCTGAGGCTCAC-3' и
R1-RabV333 - 5'-ACATGAGGATGACACCTCCCT-3',
F2-RabV333 - 5'-AGGCTGAGGCTCACTACAAG-3' и
R2-RabV333 - 5'-CCCTTCGACTCTTAAACACCCT-3',
F5-RabV333 - 5'-CCCTTCGACTCTTAAACACCCT-3' и
R5-RabV333 - 5'-AACACCCTTTTGAGGGG-3'.
Для подтверждения возможности применения разработанных праймеров в анализе пиков высокого разрешения для дифференциации штаммов «ВНИИЗЖ-G333» и «РВ-97» вируса бешенства провели моделирование in-silico с предсказанными ампликонами для анализируемых последовательностей нуклеотидов, используя программное обеспечение uMelt28. Вышеупомянутые праймеры были отобраны, синтезированы и очищены. Специфичность последовательностей праймеров проверяли с помощью Базового инструмента поиска локального выравнивания (NCBI/Primer-BLAST). При анализе нуклеотидных последовательностей олигонуклеотидов установили, что для праймеров не характерно образование «шпилек», а также не выявлено 3'-комплементарности и сайтов, отжигающих сами на себя. Расчет вероятности образования «шпилек» и димеров олигонуклеотидов проводили при условии, что минимальное количество пар оснований, необходимых для димеризации, - 5, а для образования «шпилек», - 4. Таким образом, были разработаны оригинальные специфичные олигонуклеотидные праймеры, которые могут позволить проводить дифференциацию генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Пример 2. Определение пиков высокого разрешения графиков плавления ампликонов для разработки способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Для определения показателей, позволяющих проводить дифференциацию генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения, проводили следующие этапы по исследованию штаммов вируса бешенства.
На первом этапе исследования проводили выделение нуклеиновой кислоты из анализируемых образцов, содержащих РНК вируса бешенства, с применением твердофазного способа с использованием набора «РИБО-сорб» в соответствии с инструкцией производителя. Для анализа использовали суспензии вируса бешенства штаммов «ВНИИЗЖ-G333» и «РВ-97» и полевых изолятов.
На следующем этапе осуществляли обратную транскрипцию и ПЦР в режиме реального времени для исследования проб в соответствии с описанием, представленным выше. В реакции амплификации исследовали экстракты ДНК в цельном виде, а также в разведения от 10-1 до 10-4 с шагом 10. Для постановки реакции готовили реакционную смесь в соответствии с наставлением к набору "SsoFast™ EvaGreen® Supermix". Постановку реакции проводили в детектирующем амплификаторе марки CFX-96 (Bio-Rad, США).
Исследование кривой плавления с высоким разрешением для анализа данных и профилей плавления образцов выполняли с использованием программного обеспечения для сканирования генов программного обеспечения CFX-96. Проводили построение кривых плавления в виде параболических функций и детектируют пики высокого разрешения для полученных графиков. Впоследствии провели анализ ожидаемых профилей и пиков плавления прогнозируемых ампликонов для исследуемых изолятов/штаммов с использованием программного обеспечения uMelt. Полученные данные для некоторых исследуемых образцов представлены на фиг. 1 и 2.
Анализ пиков высокого разрешения проводили для образцов кДНК вируса бешенства. Изначально обратную транскрипцию и ПЦР в режиме реального времени проводили со всеми 5 парами праймеров (табл. ).
Как показал анализ, наиболее выраженная дифференциация достигается при использовании трех пар праймеров, а именно F1-RabV333 - R1-RabV333, F2-RabV333 - R2-RabV333 и F5-RabV333 - R5-RabV333. Исходя из этого, именно они были выбраны для дальнейшего исследования.
Для окончательного отбора оптимальной пары праймеров были проведены дополнительные исследования. Тестировали к ДНК вируса бешенства штаммов «ВНИИЗЖ-G333» и «РВ-97» в 30 повторностях для каждого штамма и каждой пары праймеров. Результаты эксперимента представлены в таблице 5.
Выявили, что для генно-модифицированного вакцинного штамма «ВНИИЗЖ-G333» вируса бешенства в отличии от других штаммов и изолятов характерен очень узкий диапазон температуры плавления, равный 80,28±0,07°С (n=30, p<0,005). При этом для штамма «РВ-97» вируса бешенства данные значения составили 81,40±0,09°С (n=30, р<0,005). Разность температур (ΔT) составила 1,12°±0,16°С при использовании второй пары праймеров F2-RabV333 - R2-RabV333. По полученным данным можно судить о принадлежности исследуемого образца к вакцинному штамму «ВНИИЗЖ-G333» или «РВ-97» вируса бешенства. При использовании пар праймеров F1-RabV333 - R1-RabV333 и F5-RabV333 - R5-RabV333 значения ΔТ были ниже 0,75±0,25 и 0,82±0,23°С, соответственно. При большем различии температурных пиков для двух штаммов - выше специфичность и достоверность проводимого анализа на этапе контроля вируссодержащего материала. При анализе полевых изолятов пиковые значения температур выходили за пределы рассматриваемых показаний, что подтверждало специфичность разработанного способа относительно генно-модифицированного штамма «ВНИИЗЖ-G333».
Таким образом, для штамма «ВНИИЗЖ-G333» пик график плавления отличается от штамма «РВ-97» вируса бешенства. Это указывало на возможность применения разработанного способа для дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Пример 3. Исследование аналитической специфичности разработанного способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Специфичность анализа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения со второй системой оригинальных праймеров оценивали путем тестирования 8 экстрактов ДНК от других патогенов жвачных животных (2 изолята вируса герпеса крупного рогатого скота - BOHV-1 404/2018, BOHV-2 95-5/2016; 3 биоматериала, содержащего Mycoplasma capricolum ssp., 3 суспензии кДНК от разных изолятов вируса чумы мелких жвачных животных - PPRV/Israel-2536/Hebron/1997, clone Karth99, clone Chirg98). Для проведения исследования использовали детектирующий амплификатор марки CFX-96 (Bio-Rad, США). В результате исследования амплификация с неспецифичными нуклеиновыми кислотами других инфекционных агентов не была обнаружена (табл. 6).
В результате исследования обнаружили, что для проб, не содержащих нуклеиновую кислоту вируса бешенства, и отрицательного контроля не формировали графики плавления и не были обнаружены пиковые значения при анализе высокого разрешения. При исследовании положительного контроля 1 обнаружен пик температуры, равный 80,28±0,07°С, положительного контроля 2 - 81,40±0,09°С (n=3, р<0,005). Таким образом, полученные результаты свидетельствовали о 100%-ной специфичности разработанного способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Пример 4. Определение диагностических показателей разработанного способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Для исследования разработанного способа дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения исследовали стандартные диагностические показатели. Для определения диагностической чувствительности разработанного способа анализировали 152 суспензии вируса бешенства штамма «ВНИИЗЖ-G333» с титрами инфекционной активности 5,5-8,5 lg ККИД50/см3, которые являлись заведомо положительными. Постановку обратной транскрипции и ПЦР в режиме реального времени с последующим плавлением ампликонов проводили с системой разработанных оригинальных праймеров F2-RabV333 - R2-RabV333. С помощью разработанного способа (предлагаемое изобретение) определили, что из 152 исследуемых образцов вируса бешенства штамма «ВНИИЗЖ-G333» все определены в качестве положительных и подтверждено наличие именно данного штамма в анализируемых суспензиях.
Для исследования специфичности метода тестировали 122 суспензии вируса бешенства других штаммов и изолятов, в том числе вакцинного штамма «РВ-97». С помощью метода секвенирования доказана видовая принадлежность указанных изолятов и штаммов. В результате исследования с помощью разработанного способа (предлагаемое изобретение) определили, что все 122 пробы содержали геном вируса бешенства, но при этом не включали в свой состав вакцинный штамм «ВНИИЗЖ-G333». Пользуясь представленными выше статистическими методами анализа определили, что в 95%-ном доверительном интервале диагностическая чувствительность (DSe) составила 97,60-100,00%, диагностическая специфичность (DSp) - 97,02-100,0%, k-критерий - 1,000; прогностичность положительного результата (PPV) - 100%, прогностичность отрицательного результата (NPV) - 100,00%, общая точность (DAc) - 98,66-100,00% (табл. 7).
Основными преимуществами предлагаемого изобретения является возможность проводить за короткий промежуток времени (не более 3 ч) дифференциацию генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения. Разработанный способ впервые применили для решения данной задачи. Выявили, что в анализируемом участке кДНК вируса бешенства штамма «ВНИИЗЖ-G333» в позициях 1054…1056 п.н. имеется тройная нуклеотидная замена GAA на GAA, уникальная для вакцинного штамма «ВНИИЗЖ-G333», что позволило разработать специфические олигонуклеотидные праймеры F2-RabV333 - R2-RabV333 для детекции данного участка кДНК G-гена с помощью ПЦР в режиме реального времени с последующим анализом температур плавления для анализируемых проб вируса. Впервые представлена дифференциация генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения.
Разработанный способ дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения характеризуется аналитической специфичностью, равной 100%. В 95%-ном доверительном интервале диагностическая чувствительность для данного способа составляет 97,60-100,00%, диагностическая специфичность - 97,02-100,0%.
Источники информации, принятые во внимание при составлении описания изобретения к заявке на выдачу патента РФ на изобретение «Способ дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения»:
1. Cousins MM, Swan D, Magaret СА, Hoover DR, Eshleman SH. Analysis of HIV using a high resolution melting (HRM) diversity assay: automation of HRM data analysis enhances the utility of the assay for analysis of HIV incidence. PLoS One. 2012;7(12):e51359.
2. Erali M, Voelkerding KV, Wittwer CT. High resolution melting applications for clinical laboratory medicine. Exp Mol Pathol. 2008 Aug;85(1):50-8. doi:10.1016/j.yexmp.2008.03.012. Epub 2008 Apr 13. PMID: 18502416; PMCID: PMC2606052.
3. Structural aspects of rabies virus replication / A. A. Albertini, G. Schoehn, W. Weissenhorn, R.W. Ruigrok // Cell. Mol. Life Sci. - 2008 - 65(2)- P. 282-294.
4. OIE. Manual of Diagnostic Tests and Vaccines for Terrestrial Animals. - 7th ed. - Paris, 2022. - Vol. 1, Chap. 2.1.17.
5. GenBank. URL: https://www.ncbi.nlm.nih.gov/nuccore/EF542830.l. (Дата обращения: 10.01.2023).
6. Zhou L, Wang L, Palais R, Pryor R, Wittwer CT. High-resolution DNA melting analysis for simultaneous mutation scanning and genotyping in solution. Clin Chem. 2005 Oct;51(10): 1770-7. doi: 10.1373/clinchem.2005.054924. PMID: 16189378.
7. Garritano S, Gemignani F, Voegele C, Nguyen-Dumont T, Le Calvez-Kelm F, De Silva D, Lesueur F, Landi S, Tavtigian SV. Determining the effectiveness of High Resolution Melting analysis for SNP genotyping and mutation scanning at the TP53 locus. BMC Genet. 2009 Feb 17;10:5. doi: 10.1186/1471-2156-10-5. PMID: 19222838; PMCID: PMC2648999.
8. Babiuk S, Bowden TR, Boyle DB, Wallace DB, Kitching RP. Capripoxviruses: An Emerging Worldwide Threat to Sheep, Goats and Cattle. Transbound. Emerg. Dis. 2008;55:263-272.
9. Broyles S.S.J. Vaccinia virus transcription, 84 (9) (2003), pp. 2293-2303.
10. Er TK, Chang JG. High-resolution melting: applications in genetic disorders. Clin Chim Acta. 2012 Dec 24;414:197-201.
1l .Huebner С, Weber R, Lloydd R. A HRM assay for identification of low level BRAF V600E and V600K mutations using the CADMA principle in FFPE specimens. Pathology. 2017 Dec;49(7):776-783.
12. Hussmann D, Hansen LL. Methylation-Sensitive High Resolution Melting (MS-HRM). Methods Mol Biol. 2018;1708:551-571.
13. Nikodem D, Clapa T, Narozna D. Technologia HRM-PCR w diagnostyce medycznej [HRM-PCR in medical diagnostic]. Postepy Biochem. 2021 Mar 4;67(1):59-63. Polish, doi: 10.18388/pb.2021_373. PMID: 34378898.
14. Pakbin B, Basti AA, Khanjari A, Brück WM, Azimi L, Karimi A. Development of high-resolution melting (HRM) assay to differentiate the species of Shigella isolates from stool and food samples. Sci Rep.2022 Jan 10;12(1):473.
15. Robinson CV, Uren Webster TM, Consuegra S. Data on optimisation of a multiplex HRM-qPCR assay for native and invasive crayfish as well as the crayfish plague in four river catchments. Data Brief. 2018 May 29; 19:1092-1109.
16. Dwight Z, Palais R, Wittwer CT. uMELT: prediction of high-resolution melting curves and dynamic melting profiles of PCR products in a rich web application. Bioinformatics. 2011 Apr 1;27(7):1019-1020.


Примечание: термодинамические параметры разработанных олигонуклеотидных праймеров рассчитаны при условии:
концентрация NaCl 1 М,
температура 25°С,
водородный показатель (рН) 7,0.
Примечание: «+» - наличие характерного графика плавления ампликонов,
«-» - отсутствие графика по результатам ПЦР-РВ с анализом пиков высокого разрешения.
Примечание: DSe - диагностическая чувствительность,
DSp - диагностическая специфичность,
k-критерий - индекс Каппа Коэна,
PPV - прогностичность положительного результата,
NPV - прогностичность отрицательного результата,
DAc - общая точность.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="RABV.xml"
softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2023-08-01">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-03-11</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>493</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-03-11</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБУ "Федеральный центр охраны
здоровья животных" (ФГБУ "ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin>FGBI "ARRIAH"</ApplicantNameLatin>
<InventorName languageCode="ru">Чупин Сергей
Александрович</InventorName>
<InventorNameLatin> Chupin Sergey Alexandrovich</InventorNameLatin>
<InventionTitle languageCode="ru">Способ дифференциации генома
генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного
штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в
режиме реального времени с анализом пиков высокого
разрешения</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1650</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1650</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FMDV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggaaagatggttcctcaggctctcctgtttgtaccccttctggttttt
ccattgtgttttgggaaattccctatttacacgataccagacaagcttggtccctggagcccgattgaca
tacatcacctcagctgcccaaacaatttggtagtggaggacgaaggatgcaccaacctgtcagggttctc
ctacatggaactaaaagttggatacatcttagccataaaaatgaacgggttcacttgcacaggcgttgtg
acggaggctgaaacctacactaacttcgttggttatgtcacaaccacgttcaaaagaaagcatttccgcc
caacaccagatgcatgtagagccgcgtacaactggaagatggccggtgaccccagatatgaagagtctct
acaaaatccgtaccctgactaccgctggcttcgaactgtaaaaaccaccaaggagtctctcgttatcata
tctccaagtgtggcagatttggacccatatgacagatcccttcactcgagggtcttccctagcgggaagt
gctcgggagtagcggtgtcttctacctactgctccactaaccacgattacaccatttggatgcccgagaa
tccgagactagggatgtcttgtgacatttttaccaatagtagagggaagagagcatccaaagggagtgag
acttgcggctttgtagatgaaagaggcctatataagtctttaaaaggagcatgcaaactcaagttatgtg
gagttctaggacttagacttatggatggaacatgggtcgcgatgcaaacatcaaatgaaaccaaatggtg
ccctcccgatcagttggtgaacctgcacgactttcactcagacgaaattgagcaccttgttgtagaggag
ttggtcaggaagagagaggagtgtctggatgcactagagtccatcatgacaaccaagtcagtgagtttca
gacgtctcagtcatttaagaaaacttgtccctgggtttggaaaagcatataccatattcaacaagacctt
gatggaagccgatgctcactacaagtcagtcgagacttggaatgagatcatcccttcaaaagggtgttta
agagttggggggaggtgtcatcctcatgtgaacggggtgtttttcaatggtataatattaggacctgacg
gcaatgtcttaatcccagagatgcaatcatccctcctccagcaacatatggagttgttggaatcctcggt
tatcccccttgtgcaccccctggcagacccgtctaccattttcaaggacggtgacgaggccgaggatttt
gttgaagttcaccttcccgatgtgcacaatcaggtctcaggagttgacttgggtctcccgaactggggga
agtatgtattactgagtgcaggggccctgactgccttgatgttgataattttcctgatgacatgttgtag
aagagtcaatcgatcagaacctacgcaacacaatctcagagggacagggagggaggtgtcagtcactccc
caaagcgggaagatcatatcttcatgggaatcacacaagagtgggggtgagaccagactgtgaggactgg
ccgtcctttcaacgatccaagtcctgaagatcacctccccttggggggttctttttgaaaa</INSDSeq
_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>550</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..550</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FMDV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RKDGSSGSPVCTPSGFSIVFWEIPYLHDTRQAWSLEPDHTSPQLPKQFG
SGGRRMHQPVRVLLHGTKSWIHLSHKNERVHLHRRCDGGNLHLRWLCHNHVQKKAFPPNTRCMSRVQLED
GRPQIRVSTKSVPLPLASNCKNHQGVSRYHISKCGRFGPIQIPSLEGLPREVLGSSGVFYLLLHPRLHHL
DARESETRDVLHFYQREESIQREDLRLCRKRPIVFKRSMQTQVMWSSRTTYGWNMGRDANIKNQMVPSRS
VGEPARLSSMQTQVMWSSRTTYGWNMGRDANIKNQMVLRRNAPCCRGVGQEERGVSGCTRVHHDNQVSEF
QTSQSFKKTCPWVWKSIYHIQQDGSRCSLQVSRDLEDHPFKRVFKSWGEVSSSCERGVFQWYNIRTRQCL
NPRDAIIPPPATYGVVGILGYPPCAPPGRPVYHFQGRRGRGFCSSPSRCAQSGLRSLGSPELGEVCITEC
RGPDCLDVDNFPDDMLKSQSIRTYATQSQRDREGGVSHSPKREDHIFMGITQEWGDQTVRTGRPFNDPSP
EDHLPLGGSFK</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к области молекулярной биологии. Описан способ дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения с разработанными оригинальными олигонуклеотидными праймерами. Технический результат заключается в разработке чувствительного и специфичного, экспрессного способа дифференциации генома генетически-модифицированного вакцинного штамма «ВНИИЗЖ-G333» от производственного штамма «РВ-97» методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения. 1 з.п. ф-лы, 2 ил., 7 табл., 4 пр.
1. Способ дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения с разработанными оригинальными олигонуклеотидными праймерами со следующим дизайном: F2-RabV333 - 5'-AGGCTGAGGCTCACTACAAG-3', R2-RabV333 - 5'-CCCTTCGACTCTTAAACACCCT-3' и включающий следующие этапы исследования:
- экстрагирование РНК из положительного и отрицательного контролей и исследуемых проб;
- проведение реакции амплификации в режиме реального времени с разработанными оригинальными олигонуклеотидными праймерами;
- осуществление плавления ампликонов в ПЦР-смеси, содержащей интеркалирующий краситель EvaGreen, для анализа пиков графиков высокого разрешения;
- проведение дифференциации генома генетически модифицированного штамма «ВНИИЗЖ-G333» от вакцинного штамма «РВ-97» вируса бешенства методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения, предполагающим, что максимум температуры плавления фрагмента кДНК размером 77 п.н. в пределах G-гена с мутационной заменой GAA на GAA, уникальной для вакцинного штамма «ВНИИЗЖ-G333», составляет значениям 80,28±0,07°С, что соответствует вакцинному штамму «ВНИИЗЖ-G333»; для производственного штамма «РВ-97» данный параметр имеет значения 81,40±0,09°С.
2. Способ по п. 1, отличающийся тем, что аналитическая специфичность составляет 100%, диагностическая чувствительность в 95%-ном доверительном интервале - 97,60-100,00%, диагностическая специфичность - 97,02-100,0%.
| Способ опосредованного определения количества инфекционных доз вируса бешенства штамма PB-97 в сырье для аттенуированной антирабической вакцины методом обратной транскрипции и полимеразной цепной реакции в режиме реального времени | 2020 |
|
RU2761535C1 |
| CN 101565758 B 24.08.2011 | |||
| Шишков Александр Валерьевич, Пяткина Алла Александровна, Манин Борис Леонидович ДИНАМИКА АДАПТАЦИИ ВИРУСА БЕШЕНСТВА ВАКЦИННОГО ШТАММА "RV-97" К МОНОСЛОЙНОЙ КУЛЬТУРЕ КЛЕТОК ВНК-21/13 // Вестник Ульяновской ГСХА | |||
| Способ регенерирования сульфо-кислот, употребленных при гидролизе жиров | 1924 |
|
SU2021A1 |
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
Авторы
Даты
2024-02-05—Публикация
2023-08-14—Подача