Область техники, к которой относится изобретение
Описание настоящего изобретения относится к улучшению дрожжей для увеличения содержания жира и к способу их получения, и в частности к штамму-варианту из рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (РЕМТ) или фосфолипидметилтрансферазы и, таким образом, повышено содержание жира, и к способу увеличения содержания жира в штамме путем культивирования штамма-варианта из рода Yarrowia в содержащей холин среде.
Предшествующий уровень техники
Жирообразующие дрожжи, которые продуцируют производные жирных кислот, такие как свободные жирные кислоты, жирные спирты, этиловые эфиры жирных кислот, алканы и т.д., стали предметом внимания в различных областях, таких как энергетика, производство косметики, пищевых продуктов, кормов и т.д., и проведены различные относящиеся к ним исследования. Среди них был исследован синтез больших количеств триацилглицерина (TAG), который является предшественником производных жирных кислот и представляет собой форму жира, пригодную для долгосрочного хранения.
Многие исследователи в качестве способа улучшения содержания TAG в жирообразующих дрожжах главным образом используют стратегии усиления путей биосинтеза TAG (например, dga1-2, slc1, acl1-2, acc1, sct1, lro1), ингибирования деградации TAG (например, tgl1-4, faa1, pxa1-2) и ингибирования fema-окисления (например, pox1-6, mfe1, pex1-32, pot1) (публикация заявки на патент U.S. №2013-0344548 A1).
В последние годы предприняты различные стратегии по улучшению содержания жира в дрожжах, но заметного увеличения содержания жира при этом не было. Соответственно, существует потребность в альтернативе для улучшения уровня продукции жира, позволяющей избежать ухудшения роста дрожжей.
Описание изобретения
Техническая задача
Авторы настоящего изобретения обнаружили, что продукцию жира можно увеличить с помощью штамма-варианта, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы или фосфолипидметилтрансферазы, в результате чего было выполнено настоящее изобретение.
Техническое решение
Задачей настоящего изобретения является разработка штамма-варианта из рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (РЕМТ) или фосфолипидметилтрансферазы.
Другой задачей настоящего изобретения является разработка косметической композиции, пищевой композиции, кормовой композиции или лекарственной композиции, каждая из которых включает по меньшей мере одно из указанного штамма-варианта рода Yarrowia, культуры этого штамма, экстракта этого штамма, сухого продукта этого штамма, лизата этого штамма и жира, выделенного из по меньшей мере одного из указанных штамма, культуры, экстракта, сухого продукта и лизата.
Еще одной задачей настоящего изобретения является разработка способа повышения содержания жира в штамме, включающего: культивирование указанного штамма-варианта из рода Yarrowia.
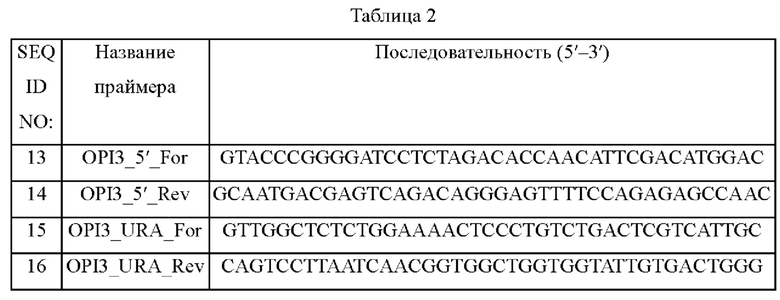
Еще одной задачей настоящего изобретения является разработка способа получения жира, где этот способ включает: культивирование указанного штамма-варианта из рода Yarrowia.
Еще одной задачей настоящего изобретения является разработка применения указанного штамма-варианта из рода Yarrowia, культуры этого штамма, экстракта этого штамма, сухого продукта этого штамма, лизата этого штамма, указанной косметической композиции, указанной пищевой композиции, указанной кормовой композиции или указанной лекарственной композиции, или применения культуры указанного штамма-варианта из рода Yarrowia в производстве жира.
Полезные эффекты изобретения
Штамм-вариант из рода Yarrowia по настоящему изобретению может увеличивать продукцию жира посредством инактивации активности фосфатидилэтаноламин-N-метилтрансферазы (РЕМТ) или фосфолипидметилтрансферазы.
Способ увеличения продукции жира в штамме и способ получения жира по настоящему изобретению может эффективно увеличивать продукцию жира, при этом, в то же время, предотвращая ухудшение роста штамма.
Косметическая композиция, пищевая композиция, кормовая композиция и лекарственная композиция по настоящему изобретению могут обеспечить улучшенную функциональность за счет увеличения содержания жира.
Лучший вариант осуществления изобретения
Ниже будет приведено подробное описание настоящего изобретения. В то же время, каждое описание и воплощение, раскрытые в описании настоящего изобретения, могут быть также применены к другим описаниям и воплощениям изобретения. То есть все комбинации различных раскрытых в настоящем описании элементов входят в объем настоящего изобретения. Кроме того, объем настоящего изобретения не ограничен приведенным ниже конкретным описанием.
Кроме того, специалисты в данной области техники поймут или смогут убедиться с использованием не более чем стандартных экспериментов, что описанные в настоящем документе конкретные воплощения могут иметь множество эквивалентов. Кроме того, эти эквиваленты должны быть интерпретированы в рамках объема описания настоящего изобретения.
Для решения описанных выше задач в одном аспекте описания настоящего изобретения предложен штамм-вариант из рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (РЕМТ) или фосфолипидметилтрансферазы.
При использовании в настоящем документе «фосфатидилэтаноламин-N-метилтрансфераза (РЕМТ)» представляет собой фермент трансферазу, которая преобразует фосфатидилэтаноламин (РЕ) в фосфатидилхолин (PC). Продуцируемый посредством РЕМТ фосфатидилхолин может утилизироваться в синтезе холина, мембранной структуре и секреции VLDL (липопротеинов очень низкой плотности) и, таким образом, играет широкий ряд физиологических ролей. В описании настоящего изобретения «фосфатидилэтаноламин-N-метилтрансфераза» может использоваться взаимозаменяемо с РЕМТ. В описании настоящего изобретения фосфатидилэтаноламин-N-метилтрансферазу может кодировать ген cho2.
При использовании в настоящем документе «фосфолипидметилтрансфераза» представляет собой фермент, который катализирует реакцию метилирования фосфатидилэтаноламина (РЕ) и может играть роль в синтезе фосфатидилхолина (PC). В описании настоящего изобретения фосфолипидметилтрансферазу может кодировать ген opi3.
Инактивация активности фосфатидилэтаноламин-N-метилтрансферазы или фосфолипидметилтрансферазы по настоящему изобретению означает инактивацию активности одного из указанных двух ферментов или инактивацию их комбинации.
Что касается задач настоящего изобретения, штамм-вариант из рода Yarrowia, в котором инактивирована активность одного из указанных двух ферментов или активности обоих ферментов, может иметь увеличенное содержание жира в штамме по сравнению с диким типом, но без ограничений.
Последовательности «фосфатидилэтаноламин-N-метилтрансферазы» или «фосфолипидметилтрансферазы» могут быть получены из опубликованной базы данных, и примеры опубликованных баз данных могут включать GenBank NCBI (Национального центра биотехнологической информации) и т.д. Например, она может представлять собой фосфатидилэтаноламин-N-метилтрансферазу и/или фосфолипидметилтрансферазу, полученную из рода Yarrowia (Yarrowia sp.), и, в частности, фосфатидилэтаноламин-N-метилтрансфераза может включать аминокислотную последовательность SEQ ID NO: 2, а фосфолипидметилтрансфераза может включать аминокислотную последовательность SEQ ID NO: 12, но без ограничений. Полипептид, включающий в себя аминокислотную последовательность SEQ ID NO: 2, может использоваться взаимозаменяемо с полипептидом, имеющим аминокислотную последовательность SEQ ID NO: 2, или полипептидом, состоящим из аминокислотной последовательности SEQ ID NO: 2, а полипептид, включающий в себя аминокислотную последовательность SEQ ID NO: 12, может использоваться взаимозаменяемо с полипептидом, имеющим аминокислотную последовательность SEQ ID NO: 12, или полипептидом, состоящим из аминокислотной последовательности SEQ ID NO: 12. Дополнительно может быть также включена любая аминокислотная последовательность, если она обладает такой же активностью, как указанная выше аминокислотная последовательность.
Дополнительно полипептид может включать в себя аминокислотную последовательность SEQ ID NO: 2 фосфатидилэтаноламин-N-метилтрансферазы и/или аминокислотную последовательность SEQ ID NO: 12 фосфолипидметилтрансферазы, либо аминокислотную последовательность, имеющую 30% или более гомологии или идентичности этой последовательности, но без ограничений. В частности, полипептид может включать аминокислотную последовательность, имеющую по меньшей мере 30%, 60%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% или более гомологии или идентичности SEQ ID NO: 2 фосфатидилэтаноламин-N-метилтрансферазы и/или SEQ ID NO: 12 фосфолипидметилтрансферазы. В дополнение к этому очевидно, что любой полипептид, имеющий аминокислотную последовательность с делецией, модификацией, заменой или добавлением в части последовательности, также может быть включен в объем настоящего изобретения, если аминокислотная последовательность имеет гомологию или идентичность, как описано выше, и проявляет эффективность, соответствующую таковой указанного полипептида.
Иными словами, хотя в описании настоящего изобретения описан «белок или полипептид, состоящий из аминокислотной последовательности конкретной SEQ ID NO», очевидно, что любой полипептид, имеющий аминокислотную последовательность с делецией, модификацией, заменой или добавлением в части последовательности, может быть также использован в настоящем изобретении, если этот полипептид может обладать активностью, идентичной или соответствующей таковой полипептида, состоящего из аминокислотной последовательности соответствующей SEQ ID NO. Например, очевидно, что любой полипептид может относиться к «полипептиду, состоящему из аминокислотной последовательностью SEQ ID NO: 2», если он обладает активностью, идентичной или соответствующей таковой «полипептида, состоящего из аминокислотной последовательности SEQ ID NO: 2».
В описании настоящего изобретения гены, кодирующие фосфатидилэтаноламин-N-метилтрансферазу и фосфолипидметилтрансферазу, могут представлять собой гены cho2 и opi3 соответственно. Полинуклеотидные последовательности, кодирующие аминокислоты по настоящему изобретению, могут представлять собой полинуклеотидную(ые) последовательность(и) SEQ ID NO: 1 и/или SEQ ID NO: 11, кодирующие фосфатидилэтаноламин-N-метилтрансферазу и фосфолипидметилтрансферазу, но без ограничений. Дополнительно полинуклеотидные последовательности могут включать любую последовательность без ограничений, если она обладает такой же активностью, как эта полинуклеотидная последовательность. Ген cho2 может представлять собой нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO: 2, и более конкретно может представлять собой последовательность, включающую в себя нуклеотидную последовательность SEQ ID NO: 1, но без ограничений. Последовательность, включающая в себя нуклеотидную последовательность SEQ ID NO: 1, и полинуклеотид, включающий в себя нуклеотидную последовательность SEQ ID NO: 1, могут использоваться взаимозаменяемо с полинуклеотидом, имеющим нуклеотидную последовательность SEQ ID NO: 1, полинуклеотидом, состоящим из нуклеотидной последовательности SEQ ID NO: 1, и геном, состоящим из полинуклеотид ной последовательности SEQ ID NO: 1.
Ген opi3 может представлять собой нуклеотидную последовательность, кодирующую аминокислотную последовательность SEQ ID NO: 12, и более конкретно может представлять собой последовательность, включающую в себя нуклеотидную последовательность SEQ ID NO: 11, но без ограничений. Последовательность, включающая в себя нуклеотидную последовательность SEQ ID NO: 11, и полинуклеотид, включающий в себя нуклеотидную последовательность SEQ ID NO: 11, может использоваться взаимозаменяемо с полинуклеотидом, имеющим нуклеотидную последовательность SEQ ID NO: 11, полинуклеотидом, состоящим из нуклеотидной последовательности SEQ ID NO: 11, и геном, состоящим из полинуклеотид ной последовательности SEQ ID NO: 11.
Используемый в настоящем документе термин «полинуклеотид», который относится к длинноцепочечному полимеру из нуклеотидов, образованному связыванием нуклеотидных мономеров посредством ковалентных связей, имеет общее значение, включающее молекулы ДНК и РНК. Нуклеотиды, которые являются основными структурными звеньями полинуклеотидов, включают не только природные нуклеотиды, но также их модифицированные аналоги, в которых группировки сахара или основания модифицированы (см. Scheit, Nucleotide Analogs, John Wiley, New York (1980); Uhlman and Peyman, Chemical Reviews, 90:543-584 (1990)).
Полинуклеотид может включать полинуклеотид, кодирующий полипептид фосфатидилэтаноламин-N-метилтрансферазу и/или фосфолипидметилтрансферазу по настоящему изобретению, или полинуклеотидную последовательность, имеющую по меньшей мере 30%, 60%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% или более гомологии или идентичности полипептиду фосфатидилэтаноламин-N-метилтрансферазе и/или фосфолипидметилтрансферазе по настоящему изобретению. В дополнение к этому очевидно, что любой полинуклеотид, имеющий полинуклеотидную последовательность с делецией, модификацией, заменой или добавлением в части последовательности, также может быть включен в объем описания настоящего изобретения, если полинуклеотидная последовательность имеет такую гомологию или идентичность, и кодирует указанный полипептид.
В дополнение к этому очевидно, что вследствие вырожденности кодонов также может быть включен полинуклеотид, который может транслироваться в полипептид, включающий в себя аминокислотную последовательность SEQ ID NO: 2 и/или 12, или полипептид, включающий в себя аминокислотную последовательность, имеющую 30% или более идентичности SEQ ID NO: 2 и/или 12, или полипептид, имеющий гомологию или идентичность этому полипептиду. Альтернативно, зонд, который может быть получен из известной последовательности гена, например любой нуклеотидной последовательности, которая гибридизуется с последовательностью, комплементарной полноразмерной нуклеотидной последовательности или ее части, в жестких условиях, для кодирования полипептида, включающего в себя аминокислотную последовательность, имеющую 30% или большую идентичности с аминокислотной последовательностью SEQ ID NO: 2, может быть включен без ограничений. «Жесткие условия» относятся к условиям, которые позволяют осуществлять специфичную гибридизацию между полинуклеотидами. Такие условия конкретно описаны в литературе (например, J. Sambrook et al., Molecular Cloning, A Laboratory Manual, 2 Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York). Например, жесткие условия, в которых гены, имеющие высокую гомологию или идентичность, гены, имеющие 30% или более, 70% или более, 80% или более, конкретно 85% или более, конкретно 90% или более, более конкретно 95% или более, значительно более конкретно 97% или более, особенно конкретно 99% или более гомологии или идентичности, гибридизуются друг с другом, при этом гены, имеющие гомологию или идентичность ниже вышеуказанной гомологии или идентичности, не гибридизуются друг с другом; либо могут включать обычные условия отмывки саузерн-гибридизации, т.е. отмывку один раз, конкретно два или три раза, при концентрации соли и температуре, соответствующих: 60°С, 1-кратный раствор цитрата и хлорида натрия (SSC), 0,1% додецилсульфат натрия (SDS), конкретно 60°С, 0,1-кратный SSC, 0,1% SDS и более конкретно 68°С, 0,1-кратный SSC, 0,1% SSD.
Для гибридизации требуется, чтобы два полинуклеотид а имели комплементарные последовательности, хотя возможны несовпадения между основаниями в зависимости от жесткости условий гибридизации. Термин «комплементарный» используют для описания взаимосвязи между азотистыми основаниями нуклеотидов, которые могут гибридизоваться друг с другом. Например, что касается ДНК, аденозин комплементарен тимину, а цитозин комплементарен гуанину. Таким образом, описание настоящего изобретения может также включать изолированный полинуклеотидный фрагмент, комплементарный полноразмерной последовательности, а также по существу подобной ей полинуклеотидной последовательности.
Конкретно полинуклеотид, имеющий гомологию или идентичность, может быть выявлен с использованием условий гибридизации, включающих стадию гибридизации при значении температуры отжига (Tm) 55°С в описанных выше условиях. Значение Tm может дополнительно составлять 60°С, 63°С или 65°С, но без ограничений, и специалист в данной области техники может соответствующим образом контролировать его в зависимости от цели.
Жесткость, подходящая для гибридизации полинуклеотидов, зависит от переменных длины и степени комплементарности полинуклеотидов, хорошо известных в данной области техники (см. Sambrook et al., выше).
Используемый в настоящем документе термин «гомология» или «идентичность» относится к степени соответствия между двумя данными аминокислотными последовательностями или нуклеотидными последовательностями и может быть выражен в процентах. Термины «гомология» или «идентичность» часто используют взаимозаменяемо друг с другом.
Гомологию или идентичность последовательностей для консервативных полинуклеотидов или полипептидов можно определить с помощью стандартных алгоритмов приведения в соответствие, и можно использовать их совместно со штрафом за гэпы по умолчанию, установленным используемой программой. Как правило, ожидается, что по существу гомологичные или идентичные последовательности гибридизуются по всей длине или по меньшей мере с приблизительно 50%, приблизительно 60%, приблизительно 70%, приблизительно 80% или приблизительно 90% или более всей длины последовательностей в условиях умеренной или высокой жесткости. Также рассматриваются полинуклеотиды, которые содержат вырожденные кодоны вместо кодонов в гибридизующихся полинуклеотидах.
Наличие или отсутствие гомологии, подобия или идентичности любых двух полинуклеотидных или полипептидных последовательностей можно определить с помощью известных компьютерных алгоритмов, таких как программа FASTA с использованием параметров по умолчанию, раскрытых в работе Pearson et al. (1988) Proc. Natl. Acad. Sci. USA 85: 2444. Альтернативно это можно определить с помощью алгоритма Нидлмана - Вунша (Needleman and Wunsch, 1970, J. Mol. Biol. 48: 443 453), который выполняют с использованием программы Нидлмана пакета программ EMBOSS (EMBOSS: The European Molecular Biology Open Software Suite, Rice et al., 2000, Trends Genet. 16: 276-277) (версия 5.0.0 или последующие) (пакет программ GCG (Devereux, J. et al., Nucleic Acids Research 12:387 (1984)), BLASTP, BLASTN, FASTA (Atschul, S. F. et al., J MOLEC BIOL 215:403 (1990); Guide to Huge Computers, Martin J. Bishop, ed., Academic Press, San Diego, 1994; и CARILLO et al. (1988) SIAM J Applied Math 48: 1073). Например, гомологию, подобие или идентичность можно определить с использованием программ BLAST или ClustalW Национального центра биотехнологической информации.
Гомологию, подобие или идентичность между полинуклеотидами или полипептидами можно определить путем сравнения информации о данной последовательности с помощью, например, компьютерной программы GAP, представленной в работе Needleman et al. (1970), J Mol Biol. 48: 443, как раскрыто в Smith and Waterman, Adv. Appl. Math (1981) 2: 482. Вкратце, программа GAP определяет их как значение, полученное в результате деления количества символов, приведенных в соответствие аналогичным образом (т.е. нуклеотидов или аминокислот), на общее количество символов в более короткой из двух последовательностей. Параметры по умолчанию для программы GAP могут включать (1) унарную матрицу сравнений (содержащую значение 1 для случаев идентичности и 0 для случаев неидентичности) и взвешенную матрицу сравнений (или EDNAFULL (версия EMBOSS NCBI NUC4.4) матрицу замен) Gribskov et al. (1986) Nucl. Acids Res. 14:6745, как раскрыто в Schwartz and Dayhoff, eds., Atlas Of Protein Sequence And Structure, National Biomedical Research Foundation, pp. 353-358 (1979); (2) штраф 3,0 за каждый гэп и дополнительный штраф 0,10 за каждый символ в каждом гэпе (или штраф на открытие гэпа 10 и штраф на удлинение гэпа 0,5); и (3) отсутствие штрафа на концевые гэпы. Соответственно, используемый в настоящем документе термин «гомология» или «идентичность» относится к родственности между последовательностями.
«Инактивация» активности полипептида или белка по настоящему изобретению относится к случаю, где полипептид или белок не экспрессируется никаким образом, или к случаю, где полипептид или белок экспрессируется, но не проявляет активность или проявляет сниженную активность по сравнению с природным штаммом дикого типа, родительским штаммом или штаммом, в котором соответствующий полипептид или белок не модифицирован. Термины «инактивация» или «аттенуация» активности могут использоваться взаимозаменяемо с терминами «аттенуация», «понижающая регуляция», «уменьшение», «снижение» активности и т.д. В этом отношении понятие инактивации активности включает случай, где активность самого полипептида снижена или устранена по сравнению с активностью полипептида, которой исходно обладает микроорганизм, вследствие модификации в гене, кодирующем этот полипептид, модификации в контрольной последовательности экспрессии, делеции части или полноразмерного гена и т.д.; случай, где снижен уровень общей активности полипептида внутри клетки по сравнению со штаммом дикого типа или штаммом до модификации вследствие ингибирования экспрессии гена, кодирующего этот полипептид, или ингибирования трансляции и т.д.; случай, где ген не экспрессируется никаким образом; или случай, где ген экспрессируется, но не проявляет активности; и их комбинацию.
В описании настоящего изобретения инактивация активности не ограничена вышеуказанным и может быть достигнута путем применения различных способов, хорошо известных в данной области техники (Nakashima N. et al., Bacterial cellular engineering by genome editing and gene silencing, hit J Mol Sci. 2014; 15(2):2773-2793, Sambrook et al. Molecular Cloning 2012, etc.). Примеры этих способов включают способ делетирования части или полноразмерного гена, кодирующего полипептид на хромосоме; способ замены гена, кодирующего полипептид на хромосоме, геном, модифицированным для снижения активности фермента; способ введения модификации в контрольную последовательность экспрессии гена, кодирующего полипептид на хромосоме; способ замены регуляторной последовательности экспрессии гена, кодирующего полипептид на хромосоме, последовательностью, не обладающей активностью или обладающей слабой активностью (например, способ замены промотора гена более слабым промотором, чем эндогенный); способ делетирования части или полноразмерного гена, кодирующего полипептид на хромосоме; способ введения антисмыслового олигонуклеотида (например, антисмысловой мРНК), который ингибирует трансляцию мРНК в белок или полипептид посредством комплементарного связывания с транскриптом гена, кодирующего полипептид на хромосоме; способ создания невозможности присоединения рибосомы посредством образования вторичной структуры в результате искусственного присоединения комплементарной последовательности к последовательности SD (Шайна - Дальгарно) на переднем конце последовательности SD гена, кодирующего полипептид; и способ обратно-транскрипционного генно-инженерного конструирования (RTE), в результате которого промотор обратной транскрипции присоединяется к 3'-концу открытой рамки считывания (ORF) полинуклеотидной последовательности гена, кодирующего этот полипептид; или их комбинацию, но без конкретных ограничений.
В частности, способ делетирования части или полноразмерного гена, кодирующего белок или полипептид, может быть выполнен путем замены полинуклеотида, кодирующего эндогенный целевой белок на хромосоме, полинуклеотидом или геном-маркером, имеющим частичную делецию в нуклеиново-кислотной последовательности, посредством вектора для встраивания в хромосому микроорганизма. Например, можно использовать способ делетирования гена посредством гомологичной рекомбинации, но способ не ограничен этим. Таким образом, используемый в настоящем документе термин «часть» может конкретно относиться к 1-300 нуклеотидам, более конкретно к 1-100 нуклеотидам и еще более конкретно к 1-50 нуклеотидам, хотя она может варьировать в зависимости от видов полинуклеотида, и соответствующее решение может принимать специалист в данной области техники. Тем не менее, часть конкретно не ограничена вышеуказанным.
Способ делетирования части или полноразмерного гена может быть выполнен путем индуцирования мутации с использованием света, такого как ультрафиолетовые лучи, или химических веществ, и отбора из полученных мутантов штамма, в котором делетирован целевой ген. Способ делетирования гена включает способ с использованием технологии рекомбинантных ДНК. Технологию рекомбинантных ДНК можно осуществлять путем, например, введения в микроорганизм нуклеотидной последовательности или вектора, включающего нуклеотидную последовательность, имеющую гомологию целевому гену, чтобы вызвать гомологичную рекомбинацию. В дополнение к этому вводимая нуклеотидная последовательность или вектор может включать в себя доминантный селективный маркер, но без ограничений.
В дополнение к этому способ модификации контрольной последовательности экспрессии можно выполнять путем индуцирования модификации в контрольной последовательности экспрессии посредством делеции, вставки, консервативной или неконсервативной замены или их комбинации таким образом, чтобы дополнительно ослабить активность контрольной последовательности экспрессии, или путем замены последовательности нуклеиново-кислотной последовательностью, обладающей ослабленной активностью. Регуляторная последовательность экспрессии может включать последовательность промотора, оператора, последовательность, кодирующую сайт связывания рибосомы, последовательности, контролирующие терминацию транскрипции и трансляции, и т.д., но контрольная последовательность экспрессии не ограничена ими.
Дополнительно способ модификации последовательности гена на хромосоме можно выполнять путем индуцирования модификации в последовательности посредством делеции, вставки, консервативной или неконсервативной замены или их комбинации таким образом, чтобы дополнительно ослабить активность полипептида; или путем замены последовательности последовательностью гена, модифицированной так, чтобы он обладал более слабой активностью, или последовательностью гена, модифицированной так, чтобы он совсем не обладал активностью, но конкретно не ограничен.
Тем не менее, приведенное выше описание предназначено только для примера, и способ не ограничен им, а микроорганизм может представлять собой микроорганизм, в котором стимулирована экспрессия генов, кодирующих ферменты различных путей биосинтеза, которые увеличивают содержание жира, инактивирована активность ферментов родственных путей биосинтеза или фермента пути, потребляющего промежуточный продукт, кофактор или источник энергии путей биосинтеза жира. Микроорганизм с увеличенным содержанием жира может быть получен путем применения различных известных способов.
В отношении задач настоящего изобретения микроорганизм по настоящему изобретению может представлять собой любой микроорганизм, если в этом микроорганизме инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (РЕМТ) или фосфолипидметилтрансферазы и увеличено содержание жира по сравнению с немодифицированным микроорганизмом или микроорганизмом дикого типа.
Используемый в настоящем документе термин «немодифицированный микроорганизм» может относиться к нативному штамму как таковому или к микроорганизму, в котором активность фосфатидилэтаноламин-N-метилтрансферазы или фосфолипидметилтрансферазы не снижена или не инактивирована, но без ограничений. «Немодифицированный микроорганизм» может использоваться взаимозаменяемо с терминами «штамм до модификации», «микроорганизм до модификации», «невариантный штамм», «немодифицированный штамм», «невариантный микроорганизм», «немодифицированный микроорганизм» или «эталонный микроорганизм».
Используемый в настоящем документе термин «дикий тип» относится к универсальному фенотипу, которым исходно обладает биологический вид, в котором наблюдается новый вариант вследствие мутации в его генотипе.
Штамм дикого типа по настоящему изобретению может представлять собой штамм рода Yarrowia. «Род Yarrowia» представляет собой род грибов семейства Dipodascaceae. Этот род представляет собой дрожжи, способные утилизировать необычные источники углерода, такие как углеводороды, и могут быть монотипическими. Штамм рода Yarrowia может конкретно представлять собой Yarrowia bubula, Yarrowia deformans, Yarrowia lipolytica, Yarrowiaporcina, Yarrowia yakushimensis и более конкретно Yarrowia lipolytica, но без ограничений.
«Штамм-вариант» по настоящему изобретению относится к штамму, имеющему новый фенотип при наблюдении нового варианта в штамме микроорганизмов вследствие мутации в генотипе.
«Штамм-вариант рода Yarrowia» по настоящему изобретению может представлять собой штамм рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы, инактивирована активность фосфолипидметилтрансферазы или инактивированы активности фосфатидилэтаноламин-N-метилтрансферазы и фосфолипидметилтрансферазы.
Дополнительно он может представлять собой штамм рода Yarrowia, в котором делетирован ген cho2, кодирующий фосфатидилэтаноламин-N-метилтрансферазу, ген opi3, кодирующий фосфолипидметилтрансферазу, или их комбинация, но без ограничений.
В частности, В отношении задач настоящего изобретения штамм-вариант рода Yarrowia не ограничен, если в этом штамме-варианте инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы или фосфолипидметилтрансферазы и увеличено содержание жира в этом штамме по сравнению с диким типом.
Используемый в настоящем документе «микроорганизм, имеющий увеличенное содержание жира по сравнению с диким типом» может использоваться взаимозаменяемо с «продуцирующим жир микроорганизмом», «микроорганизм, обладающий способностью продуцировать жир», «микроорганизм для продукции жира», «триацилглицерин (ТАО)-продуцирующий микроорганизм», «микроорганизм, обладающий способностью продуцировать триацилглицерин (TAG)», или «микроорганизм для продукции триацилглицерина (TAG)».
Штамм-вариант рода Yarrowia может представлять собой ауксотрофный по холину штамм, у которого обеспечена ауксотрофность по холину. «Холин» является бесцветным и сильнощелочным и является компонентом фосфолипидов, ацетилхолина, витаминов группы В и т.д. У живых организмов он синтезируется в пути биосинтеза холина посредством серина и этаноламина, а также синтезируется химическим путем с триэтиламином и этиленоксидом.
В отношении задач настоящего изобретения, при культивировании штамма, который представляет собой ауксотрофный по холину штамм, в содержащей холин среде возможно восстановить рост этого штамма и, в то же время, увеличить содержание жира в штамме.
Используемый в настоящем документе термин «жир», который является видом липидов, представляет собой сложный эфир, в котором три жирных кислоты связаны с одной молекулой глицерина, и является представителем органических веществ. Жир содержится в живых организмах и может использоваться в качестве источника энергии. Конкретно жир может представлять собой триацилглицерин (TAG), но без ограничений.
«Триацилглицерин (TAG)», являющийся видом жира, представляет собой триглицерид, имеющий структуру, в которой три молекулы жирной кислоты связаны с одной молекулой глицерина посредством сложноэфирной связи. Чтобы увеличить содержание TAG, можно использовать стратегию усиления пути биосинтеза TAG, ингибирования деградации TAG или ингибирования бета-окисления. Термин «жир» может использоваться взаимозаменяемо с «TAG» или «триацилглицерин». В дополнение к этому TAG может быть предшественником производных жирных кислот и формы жира, который может запасаться на длительное время.
В другом аспекте описания настоящего изобретения предложена косметическая композиция, пищевая композиция, кормовая композиция или лекарственная композиция, каждая из которых включает по меньшей мере одно из штамма-варианта рода Yarrowia по настоящему изобретению; культуры этого штамма; экстракта этого штамма; сухого продукта из этого штамма; лизата этого штамма и жира, выделенного из по меньшей мере одного из указанных штамма, культуры, экстракта, сухого продукта и лизата.
«Род Yarrowia» и «штамм-вариант рода Yarrowia» по настоящему изобретению являются такими, как описано выше.
В отношении задач настоящего изобретения композиция может включать в себя штамм-вариант рода Yarrowia как таковой, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы или фосфолипидметилтрансферазы, и, таким образом, содержание жира в этом штамме увеличено по сравнению с диким типом, и может включать в себя культуру, сухой продукт, экстракт или лизат этого штамма. В дополнение к этому композиция может включать в себя жир, выделенный из любого одного из штамма по настоящему изобретению, культуры, сухого продукта, экстракта или лизата этого штамма. Тем не менее, композиция может включать в себя любую форму без ограничений, если она способна увеличить требуемое содержание TAG в рамках объема описания настоящего изобретения.
«Косметическая композиция» по настоящему изобретению может быть изготовлена в виде препарата, выбранного из группы, состоящей из раствора, мази для наружного применения, крема, пенки, питательного лосьона, смягчающего лосьона, маски, смягчающей воды, эмульсии, основы для макияжа, эссенции, мыла, очищающей жидкости, средства для ванн, крема для загара, масла для загара, суспензии, эмульсии, пасты, геля, лосьона, порошка, содержащего поверхностно-активное вещество очищающего масла, тональной крем-пудры, тональной эмульсии, восковой основы, патча и спрея, но без ограничений.
Косметическая композиция может дополнительно включать в себя один или более косметически приемлемых носителей, которые смешивают с обычными косметическими средствами по уходу за кожей, а также при необходимости можно добавлять в смесь в качестве общепринятого компонента, например, масло, воду, поверхностно-активное вещество, увлажняющее средство, низший спирт, загуститель, хелатирующий агент, пигмент, консервант, ароматическое вещество и т.д., но носитель не ограничен ими. Косметически приемлемый носитель для включения в косметическую композицию по настоящему изобретению варьирует в зависимости от рецептуры косметической композиции.
Косметическая композиция по настоящему изобретению может включать в себя по меньшей мере один из штаммов-вариантов рода Yarrowia; культуру этого штамма; экстракт этого штамма; сухой продукт этого штамма; лизат этого штамма; и жир, выделенный из по меньшей мере одного из указанных штамма, культуры, экстракта, сухого продукта и лизата, и, таким образом, может включать в себя увеличенное содержание жира.
«Пищевая композиция» по настоящему изобретению включает все формы, такие как функциональный пищевой продукт, добавка к пище, продукт здорового питания, питательная добавка и т.д., и указанные выше виды пищевой композиции можно готовить в различных формах в соответствии с обычным способом, известным в данной области техники.
Пищевая композиция по настоящему изобретению может включать формы пилюль, порошков, гранул, растворов для инфузии, таблеток, капсул, жидкостей и т.д., и примеры пищевых продуктов, к которым добавляют композицию по настоящему изобретению, включают разнообразные пищевые продукты, такие как напитки, жевательные резинки, чаи, витаминные комплексы, добавки для здорового питания и т.д.
Пищевая композиция по настоящему изобретению может включать в себя дополнительные ингредиенты, такие как различные экстракты лекарственных растений, добавки к пище или натуральные углеводы и т.д., как в обычных пищевых продуктах. В дополнение к этому добавки к пище могут включать добавки к пище, обычно используемые в данной области техники, например ароматический агент, корригент, красящий агент, наполнитель, стабилизатор и т.д.
Примеры натурального углевода включают обычные сахара, такие как моносахариды, например глюкоза, фруктоза и т.д., дисахариды, например мальтоза, сахароза и т.д., полисахариды, например декстрин, циклодекстрин и т.д., и сахарные спирты, такие как ксилит, сорбит, эритрит и т.д. Также в качестве ароматического вещества можно предпочтительно использовать натуральное ароматическое вещество (например, ребаудиозид А, глицирризин и т.д.) и синтетическое ароматическое вещество (сахарин, аспартам и т.д.).
В дополнение к этому пищевая композиция по настоящему изобретению может включать в себя различные питательные вещества, витамины, минералы (электролиты), корригент, такой как синтетический корригент, натуральный корригент и т.д., красящий агент, наполнитель (сыр, шоколад и т.д.), пектиновую кислоту и ее соли, альгиновую кислоту и ее соли, органическую кислоту, защитный коллоидный загуститель, регулятор рН, стабилизатор, консервант, глицерин, спирт, карбонизирующий агент, используемый для газированного напитка, и т.д. В дополнение к этому функциональная пищевая композиция по настоящему изобретению может включать мякоть, которую можно использовать для приготовления натурального фруктового сока, фруктовых сокосодержащих напитков и овощных напитков. Такие компоненты можно использовать независимо или в комбинации.
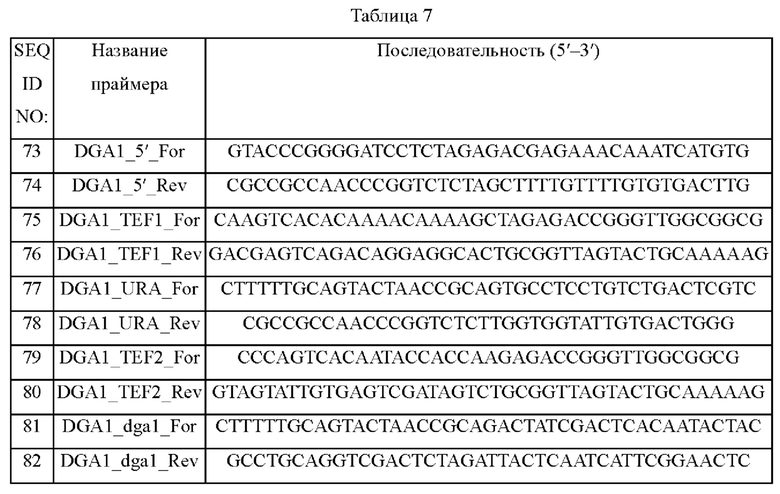
Пищевая композиция по настоящему изобретению может быть получена путем добавления исходного сырья и ингредиентов, обычно добавляемых в данной области техники во время приготовления. Дополнительно рецептуры пищевых продуктов можно готовить без ограничений, если они могут быть признаны пищевым продуктом. Пищевая композиция по настоящему изобретению может быть получена в различных типах композиций. В отличие от лекарственных средств в целом, преимущества пищевой композиции по настоящему изобретению состоят в отсутствии побочных эффектов, вызываемых долгосрочным введением, и т.д.; поскольку в пищевой композиции в качестве исходного сырья используется пищевой продукт, пищевая композиция по настоящему изобретению обладает отличной переносимостью, таким образом, пищевой продукт по настоящему изобретению можно употреблять в качестве добавки.
Пищевая композиция по настоящему изобретению может включать в себя по меньшей мере один из штаммов-вариантов рода Yarrowia; культуру этого штамма; экстракт этого штамма; сухой продукт этого штамма; лизат этого штамма; и жир, выделенный из по меньшей мере одного из указанных штамма, культуры, экстракта, сухого продукта и лизата, и, таким образом, может включать в себя увеличенное содержание жира.
«Кормовая композиция» по настоящему изобретению относится к любому натуральному или искусственному рациону, пище и т.д. или компонентам такой пищи, предназначенным или пригодным для поедания, приема внутрь или переваривания животными. Кормовую композицию можно готовить с получением различных типов кормов, известных в данной области техники, и конкретно она может включать в себя концентрированные корма, объемистые корма и/или специализированные корма.
Вид корма конкретно не ограничен, и можно использовать любой корм, известный в данной области техники. Не имеющие ограничительного характера примеры корма могут включать растительные корма, такие как зерновые культуры, орехи, пищевые побочные продукты, морские водоросли, волокна, фармацевтические побочные продукты, жиры и масла, крахмалы, бахчевые культуры, зерновые побочные продукты и т.д.; и корма животного происхождения, такие как белки, неорганические вещества, жиры и масла, минералы, белки одноклеточных организмов, зоопланктон, пищевые продукты и т.д. Их можно использовать по отдельности или в виде смеси двух или более из них.
Кормовая композиция по настоящему изобретению может включать в себя по меньшей мере один из штаммов-вариантов рода Yarrowia; культуру этого штамма; экстракт этого штамма; сухой продукт этого штамма; лизат этого штамма; и жир, выделенный из по меньшей мере одного из указанных штамма, культуры, экстракта, сухого продукта и лизата, и, таким образом, может включать в себя увеличенное содержание жира.
«Лекарственная композиция» по настоящему изобретению относится к композициям, полученным для задач предупреждения или лечения заболевания, и их можно использовать после соответствующего включения в различные лекарственные формы в соответствии с обычным способом. Например, лекарственную композицию можно включать в дозированные лекарственные формы для приема внутрь, такие как порошки, гранулы, таблетки, капсулы, суспензии, эмульсии, сиропы и т.д., и можно включать в формы препаратов для наружного применения, суппозитории и стерильные растворы для инъекций.
В дополнение к этому лекарственную композицию можно готовить путем дополнительного включения фармацевтически приемлемых носителей, таких как буферы, консерванты, анальгетики, солюбилизаторы, изотонические агенты, стабилизаторы, основы, эксципиенты, смазывающие вещества и т.д., известные в данной области техники, в соответствии с каждой лекарственной формой.
В то же время, лекарственную композицию по настоящему изобретению вводят в фармацевтически эффективном количестве. Используемый в настоящем документе термин «фармацевтически эффективное количество» означает количество, достаточное для лечения заболеваний, при разумном соотношении польза/риск, приемлемом для любого медицинского лечения, и не вызывающее каких-либо нежелательных явлений. Эффективный уровень дозировки могут определить специалисты в данной области техники в зависимости от факторов, включающих состояние здоровья пациента, тяжесть, активность лекарственного средства, чувствительность лекарственного средства, способ введения, время введения, путь введения, скорость выведения, продолжительность лечения, лекарственные средства, применяемые в комбинации или применяемые одновременно, и другие факторы, хорошо известные в области медицины.
Лекарственная композиция по настоящему изобретению может включать в себя по меньшей мере один из штаммов-вариантов рода Yarrowia; культуру этого штамма; экстракт этого штамма; сухой продукт этого штамма; лизат этого штамма; и жир, выделенный из по меньшей мере одного из указанных штамма, культуры, экстракта, сухого продукта и лизата, и, таким образом, может включать в себя увеличенное содержание жира.
В отношении задач настоящего изобретения увеличенное содержание жира в штамме может представлять собой увеличенное содержание TAG, но без ограничений.
Еще в одном аспекте описания настоящего изобретения предложен способ увеличения содержания жира в штамме, включающий: культивирование штамма-варианта из рода Yarrowia по настоящему изобретению.
«Род Yarrowia», «штамм-вариант рода Yarrowia» и «жир» являются такими, как описано выше.
Используемый в настоящем документе термин «культивирование» относится к выращиванию штамма-варианта рода Yarrowia в контролируемых надлежащим образом условиях окружающей среды. Способ культивирования по настоящему изобретению можно осуществлять в соответствии с надлежащими условиями среды и культивирования, известными в данной области техники. Такой способ культивирования могут применять специалисты в данной области техники путем простого регулирования условий в соответствии с выбранным штаммом.
Культивирование штамма можно, но без конкретных ограничений, выполнять в периодическом процессе, непрерывном процессе, процессе периодического культивирования с подпиткой и т.д., как известно в данной области техники. В этом отношении, что касается условий культивирования, рН можно регулировать до подходящего значения рН (например, рН 5-9, в частности рН 6-8 и наиболее конкретно рН 6,8) с использованием основного соединения (например, гидроксида натрия, гидроксида калия или аммиака) или кислого соединения (например, фосфорная кислота или серная кислота), но без конкретных ограничений. Дополнительно аэробные условия можно поддерживать путем введения в культуру кислорода или кислородсодержащей смеси газов. Температуру культивирования можно поддерживать при 20°С 45°С и в частности от 25°С до 40°С в течение периода от приблизительно 10 часов до 160 часов, но без ограничений.
Используемая в настоящем документе «среда» относится к культуральной среде, которая включает в себя вещество, полученное путем смешивания питательных веществ, требующихся для культивирования штамма-варианта рода Yarrowia, в качестве основного компонента и/или продукта, полученного после культивирования. Среда и другие условия культивирования, используемые при культивировании микроорганизма по настоящему изобретению, конкретно не ограничены, если среда представляет собой среду, обычно используемую при культивировании микроорганизма, но микроорганизм по настоящему изобретению можно культивировать и в обычной среде, содержащей необходимые источники углерода, источники азота, источники фосфора, неорганические соединения, аминокислоты и/или витамины и т.д., в аэробных условиях при регулировании температуры, рН и т.д.
В качестве источника углерода для культуральной среды, используемой в настоящем изобретении, можно использовать сахара и углеводы (например, глюкозу, сахарозу, лактозу, фруктозу, галактозу, маннозу, мальтозу, арабинозу, ксилозу, мелассы, крахмал и целлюлозу), масла и жиры (например, соевое масло, подсолнечное масло, арахисовое масло и кокосовое масло), жирные кислоты (например, пальмитиновую кислоту, стеариновую кислоту и линолевую кислоту), спирты (например, глицерин и этанол), органические кислоты (например, уксусную кислоту) и т.д. по отдельности или в комбинации. В частности, источник углерода может представлять собой одно или более выбранное из группы, состоящей из глюкозы, фруктозы, мальтозы, галактозы, маннозы, сахарозы, арабинозы, ксилозы и глицерина, но без ограничений. Дополнительно культуральная среда, используемая в описании настоящего изобретения, может включать в качестве источника углерода глюкозу в концентрации от 10 г/л до 50 г/л, от 10 г/л до 40 г/л, от 20 г/л до 50 г/л, от 20 г/л до 40 г/л или от 25 г/л до 35 г/л, но без ограничений.
Источник азота для культуральной среды, используемой в настоящем изобретении, можно классифицировать на органические источники азота и неорганические источники азота, но органический источник азота или неорганический источник азота можно использовать по отдельности или в смеси. В частности, источник азота может представлять собой органический источник азота, выбранный из группы, состоящей из дрожжевого экстракта, мясного экстракта, пептона и триптона, или неорганический источник азота, выбранный из группы, состоящей из ацетата аммония, нитрата аммония, хлорида аммония, сульфата аммония, нитрата натрия, мочевины и мононатрия глутамата (MSG). Кроме того, в культуральной среде, используемой в описании настоящего изобретения, примеры источника азота могут включать дрожжевой экстракт, сульфат аммония, нитрат натрия и MSG, но без ограничений.
Дрожжевой экстракт можно включать в культуральную среду в концентрации от 0,1 г/л до 10 г/л, от 0,5 г/л до 10 г/л, от 0,5 г/л до 7 г/л, от 0,5 г/л до 5 г/л, от 0,5 г/л до 3 г/л, от 0,5 г/л до 2 г/л или от 0,5 г/л до 1,5 г/л, сульфат аммония можно включать в культуральную среду в концентрации от 1 г/л до 5 г/л, от 1 г/л до 4 г/л, от 2 г/л до 5 г/л или от 2 г/л до 4 г/л, нитрат аммония можно включать в культуральную среду в концентрации от 0,1 г/л до 10 г/л, от 0,5 г/л до 9 г/л, от 1 г/л до 9 г/л, от 2 г/л до 9 г/л, от 3 г/л до 9 г/л, от 5 г/л до 9 г/л или от 7 г/л до 9 г/л, и MSG можно включать в культуральную среду в концентрации от 0,1 г/л до 2 г/л, от 0,1 г/л до 1,5 г/л, от 0,5 г/л до 2 г/л или от 0,5 г/л до 1,5 г/л, но без ограничений.
Культивирование штамма-варианта Yarrowia по настоящему изобретению можно выполнять путем культивирования штамма в среде, содержащей холин. В частности, концентрация холина в среде, содержащей холин, по настоящему изобретению может составлять от 0,05 мМ до 5 мМ, от 0,1 мМ до 5 мМ, от 0,1 мМ до 3 мМ, от 0,1 мМ до 2 мМ или от 0,1 мМ до 1 мМ, но без ограничений.
В отношении задач настоящего изобретения, при культивировании штамма, который представляет собой ауксотрофный по холину штамм, в содержащей холин среде возможно восстановить рост этого штамма и, в то же время, увеличить содержание жира в штамме.
В отношении задач настоящего изобретения среда может иметь оптимальное соотношение C/N, чтобы увеличить содержание жира в штамме.
Используемое в настоящем документе «отношение C/N (отношение углерода к азоту; отношение C:N)» относится к отношению массы углерода к массе азота в среде. По мере увеличения этого отношения источник азота в среде может уменьшаться и соответственно может уменьшаться концентрация клеток вследствие ингибирования роста.
По мере увеличения отношения C/N в среде по настоящему изобретению может уменьшаться значение оптической плотности (OD) и может увеличиваться содержание внутриклеточного жира. Таким образом, для максимальной продукции жира важно предпочтительное отношение C/N в среде.
Отношение C/N, при котором проявляется максимальная продукция жира в штамме по настоящему изобретению, можно рассчитать по формуле OD * значение содержания жира, и отношение C/N в среде может составлять от 10 до 120, в частности от 30 до 90, более конкретно от 40 до 80 и наиболее конкретно от 50 до 70, но без ограничений.
Предпочтительное отношение C/N в среде может эффективно увеличивать продукцию жира, при этом предотвращая ингибирование роста штамма по настоящему изобретению.
Еще в одном аспекте в описании настоящего изобретения предложен способ получения жира, включающий: культивирование штамма-варианта рода Yarrowia по настоящему изобретению, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (РЕМТ) или фосфолипидметилтрансферазы.
«Род Yarrowia», «штамм-вариант рода Yarrowia», «культивирование» и «жир» по настоящему изобретению являются такими, как описано выше.
Культивирование штамма может представлять собой культивирование штамма в среде, содержащей холин.
Концентрация холина в среде, содержащей холин, по настоящему изобретению может составлять от 0,05 мМ до 5 мМ, от 0,1 мМ до 5 мМ, от 0,1 мМ до 3 мМ, от 0,1 мМ до 2 мМ или от 0,1 мМ до 1 мМ.
Отношение C/N в среде может составлять от 10 до 120, в частности от 30 до 90, более конкретно от 40 до 80 и наиболее конкретно от 50 до 70, но без ограничений.
В отношении задач настоящего изобретения, при культивировании штамма, который представляет собой ауксотрофный по холину штамм, в содержащей холин среде возможно восстановить рост этого штамма и, в то же время, увеличить содержание жира в штамме и, таким образом, легко получить жир.
Способ получения жира по настоящему изобретению может дополнительно включать получение микроорганизма по настоящему изобретению, получение среды для культивирования штамма или их комбинацию (в любом порядке), например, перед культивированием.
Способ получения жира по настоящему изобретению может дополнительно включать выделение жира из указанного штамма, его культуры, его экстракта, его сухого продукта или его лизата после культивирования штамма-варианта рода Yarrowia. Иными словами, жир, продуцируемый при культивировании по настоящему изобретению, можно собрать из самого штамма или его культуры. Например, можно использовать центрифугирование, фильтрование, анионообменную хроматографию, кристаллизацию, HPLC (высокоэффективная жидкостная хроматография) и т.д., и целевой жир можно извлекать из культивируемого штамма или из культуры, его сухого продукта или его лизата, используя надлежащий способ, известный в данной области техники.
Кроме того, извлечение жира может дополнительно включать выделение и/или очистку, которые можно выполнять, используя надлежащий способ, известный в данной области техники. Таким образом, извлеченный жир может находиться в очищенной форме или в виде содержащий жир ферментационной жидкости микроорганизма.
Еще в одном аспекте описания настоящего изобретения предложено применение штамма-варианта из рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы или фосфолипидметилтрансферазы; культуры этого штамма; экстракта этого штамма; сухого продукта этого штамма; лизата этого штамма, косметической композиции, пищевой композиции, кормовой композиции или лекарственной композиции, каждая из которых включает в себя по меньшей мере одно из указанных штамма, культуры, экстракта, сухого продукта и лизата, и жира, извлеченного из одного или более указанных из штамма, культуры, экстракта, сухого продукта и лизата, в производстве жира.
«Фосфатидилэтаноламин-N-метилтрансфераза», «фосфолипидметилтрансфераза», «род Yarrowia», «штамм-вариант рода Yarrowia», «культивирование», «жир», «косметическая композиция», «пищевая композиция», «кормовая композиция» и «лекарственная композиция» по настоящему изобретению являются такими же, как описано выше.
Способ осуществления изобретения
Далее описание настоящее подробно раскрыто с помощью иллюстративных воплощений изобретения. Тем не менее, эти воплощения предназначены только для более конкретной иллюстрации настоящего изобретения, и обычному специалисту в данной области техники будет понятно, что объем настоящего изобретения не ограничен этими воплощениями.
Пример 1. Получение штамма дрожжей с дефицитом на основе штамма дикого типа
Для получения штамма, ауксотрофного по этаноламину или холину, был получен штамм, в котором был делетирован ген, кодирующий CHO2 или OPI3, на основе штамма дрожжей Yarrowia lipolytica PO1f дикого типа (АТСС MYA-2613).
1-1. Получение штамма с дефицитом cho2 (СС08-0162)
Для получения кассеты, способной к делеции гена cho2 на хромосоме Yarrowia lipolytica, была получена полинуклеотидная последовательность SEQ ID NO: 1 и аминокислотная последовательность SEQ ID NO: 2 cho2 (YALI0E06061g) на основе нуклеотидных последовательностей, зарегистрированных в KEGG (Kyoto Encyclopedia of Genes and Genomes).
Выполняли ПЦР (полимеразная цепная реакция) с использованием геномной ДНК Yarrowia lipolytica PO1f в качестве матрицы и праймеров с SEQ ID NO: 3 и 4, SEQ ID NO: 7 и 8, SEQ ID NO: 9 и 10 соответственно. Также выполняли ПЦР с использованием маркера ауксотрофности URA3 в качестве матрицы и праймеров с SEQ ID NO: 5 и 6. ПЦР выполняли в течение 35 циклов в условиях, состоящих в денатурации при 95°С в течение 1 минуты; отжига при 55°С в течение 1 минуты и полимеризации при 72°С в течение 1 минуты и 30 секунд.
В результате получили 5' UTR 1055 п. о., 5' UTR_RP 638 п. о., 3' UTR 1050 п. о. и URA3 1569 п. о. Фрагменты ДНК, амплифицированные с помощью ПЦР, были получены в одной кассете с cho2-дефицитом посредством ПЦР с перекрывающимися праймерами и удлинением, и конструирование кассеты выполняли в следующем порядке: 5' UTR-URA3-5' UTR_RP-3' UTR.
Кассетой с дефицитом cho2 трансформировали Yarrowia lipolytica PO1f методом теплового шока и отбирали на твердой среде, не содержащей урацил. Первичный штамм, отобранный таким образом, подвергали второму кроссинговеру с получением штамма с дефицитом по гену cho2. Полученный таким образом штамм с дефицитом был обозначен СС08-0162 и депонирован согласно Будапештскому договору в Корейском центре культур микроорганизмов (КССМ), международном депозитарном органе, 20 февраля 2020 г. под номером доступа КССМ12672Р.

1-2. Получение штамма с дефицитом opi3 (СС08-0123)
Для получения кассеты, способной к делеции гена opi3 на хромосоме Yarrowia lipolytica, была получена полинуклеотидная последовательность SEQ ID NO: 11 и аминокислотная последовательность SEQ ID NO: 12 opi3 (YALI0E12441g) на основе нуклеотидных последовательностей, зарегистрированных в KEGG (Kyoto Encyclopedia of Genes and Genomes).
Выполняли ПЦР с использованием геномной ДНК Yarrowia lipolytica PO1f в качестве матрицы и праймеров с SEQ ID NO: 13 и 14, SEQ ID NO: 17 и 18, SEQ ID NO: 19 и 20 соответственно. Также выполняли ПЦР с использованием маркера ауксотрофности URA3 в качестве матрицы и праймеров с SEQ ID NO: 15 и 16. ПЦР выполняли в течение 35 циклов в условиях, состоящих в денатурации при 95°С в течение 1 минуты; отжига при 55°С в течение 1 минуты и полимеризации при 72°С в течение 1 минуты и 30 секунд.
В результате получили 5' UTR 980 п. о., 5' UTR_RP 730 п. о., 3' UTR 1001 п. о. и URA3 1569 п. о. Фрагменты ДНК, амплифицированные с помощью ПЦР, были получены в одной кассете с дефицитом opi3 посредством ПЦР с перекрывающимися праймерами и удлинением, и конструирование кассеты выполняли в следующем порядке: 5' UTR-URA3-5' UTR_RP-3' UTR.
Кассетой с дефицитом opi3 трансформировали Yarrowia lipolytica PO1f методом теплового шока и отбирали на твердой среде, не содержащей урацил. Первичный штамм, отобранный таким образом, подвергали второму кроссинговеру с получением штамма с дефицитом по гену opi3. Полученный таким образом штамм с дефицитом был обозначен СС08-0123 и депонирован согласно Будапештскому договору в Корейском центре культур микроорганизмов (КССМ), международном депозитарном органе, 20 февраля 2020 г. под номером доступа КССМ 12673Р.

1-3. Получение штамма с дефицитом cho2 и opi3 (СС08-0183)
Чтобы изучить влияние одновременного дефицита генов cho2 и opi3 на Yarrowia lipolytica PO1f, был получен штамм с одновременным дефицитом cho2 и opi3 таким же образом, как в примерах 1-1 и 1-2, и штамм с комбинированной делецией обоих соответствующих генов был обозначен как СС08-0183 и депонирован согласно Будапештскому договору в Корейском центре культур микроорганизмов (КССМ), международном депозитарном органе, 20 февраля 2020 г. под номером доступа КССМ12671Р.
Пример 2. Оценка роста и накопления жира штаммов с дефицитом на основе штамма дикого типа
Была проведена оценка роста и накопления жира штаммами СС08-0162, СС08-0123 и СС08-0183 с дефицитом, полученными в примере 1.
2-1. Оценка в соответствии со средой
Два вида штаммов и контрольную группу (PO1f) высевали в колбу с угловой перегородкой вместимостью 250 мл, содержащую 50 мл YPD или YLMM1 (минимальная среда 1 для Yarrowia lipolytica) при начальной OD 0,01 и культивировали при встряхивании при 250 об/мин и 30°С в течение 72 часов. При необходимости в среду YLMM1 добавляли 0,3 мМ холина хлорида (CL). Композиции YPD и жиросодержащей среды 1 (YLMM1) представлены ниже.
<YPD>
20 г/л глюкозы, 10 г/л дрожжевого экстракта (производства BD, дрожжевой экстракт Bacto, 0,38% холина в дрожжевом экстракте Bacto), 0,5 г/л урацила, 20 г/л пептона Bacto
<YLMM1 (рН 7,2)>
40 г/л глюкозы, 1,7 г/л азотистых оснований для дрожжей без аминокислот и сульфата аммония, 0,5 г/л урацила и 2,5 г/л сульфата аммония растворяли в 0,1 М натрий-фосфатном буферном растворе (рН 7,2).
После окончания культивирования культуральную среду центрифугировали, отбрасывая супернатант, и клетки, остающиеся в нижнем слое, высушивали в сушильном шкафу при 60°С в течение 24 часов с последующей экстракцией неочищенного жира. Экстракцию неочищенного жира выполняли в соответствии со стандартным методом анализа кормов (Feed Standard Analysis Method. 2001. National Livestock Research Institute, Rural Development Administration), и результаты анализа OD и содержания внутриклеточного неочищенного жира представлены ниже в таблице 3. Результаты в таблице 3 представляют собой результаты экспериментов, выполненных в трех повторах, а увеличение содержания неочищенного жира оценивали на основании их средних значений.
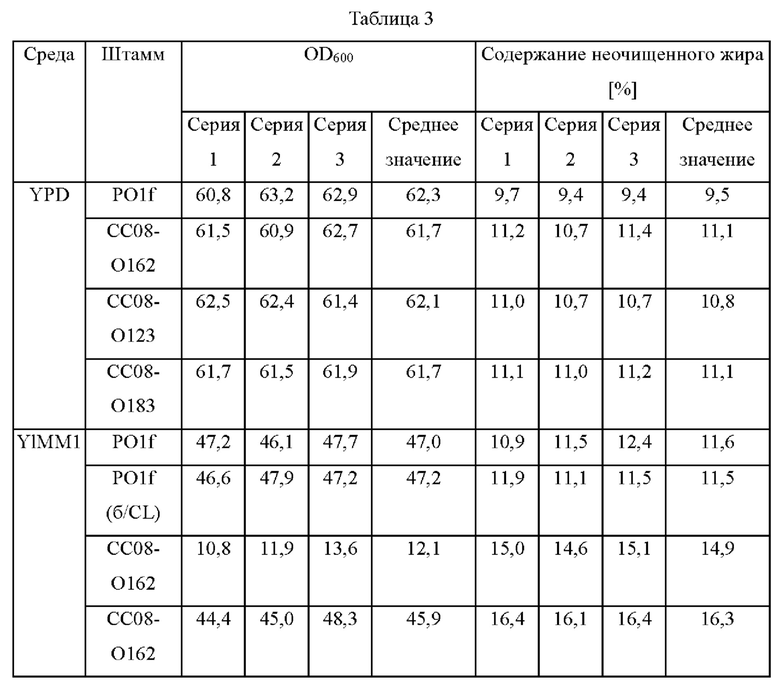
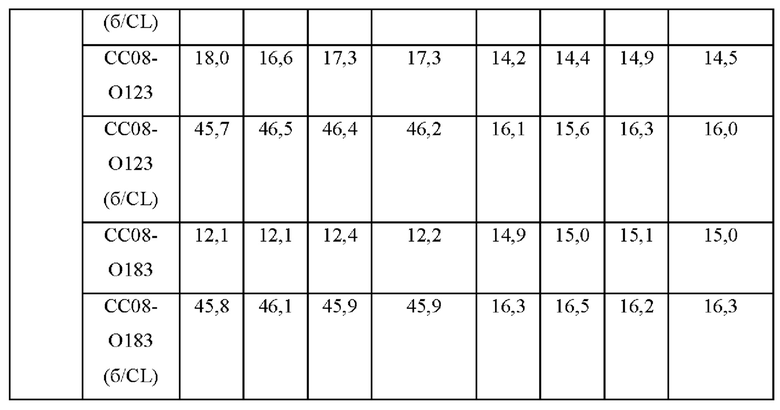
Как показано в таблице 3, подтвердили, что содержание неочищенного жира увеличивалось независимо от среды в штаммах, в которых по отдельности или одновременно были делетированы гены cho2 или оргЗ из родительского штамма PO1f. Увеличение содержания неочищенного жира также наблюдали при культивировании того же штамма в среде YLMM1 по сравнению с культивированием в среде YPD. Это увеличение составляло приблизительно 17% в штамме с дефицитом cho2 и приблизительно 14% в штамме с дефицитом opi3 по сравнению с PO1f в среде YPD. Напротив, в среде YLMM1 это увеличение составляло приблизительно 28% в штамме с дефицитом cho2 и приблизительно 25% в штамме с дефицитом opi3 по сравнению с PO1f, что указывает на большее содержание жира в среде YLMM1, чем в YPD. В дополнение к этому в штамме с одновременной делецией генов cho2 и opi3 наблюдали сходное увеличение содержания жира по сравнению со штаммом с дефицитом cho2.
Тем не менее, в отличие от YPD, среда YLMM1 не содержит холин, поэтому путь синтеза фосфатидилхолина из фосфатидилэтаноламина был блокирован, и штамм с дефицитом cho2, штамм с дефицитом opi3 и штамм с одновременным дефицитом cho2 и opi3 при условии ауксотрофности по холину показали ингибирование роста в среде YLMM1.
Таким образом, для восстановления роста в среду добавляли 0,3 мМ холина и подтвердили, что при добавлении холина в среду рост штамма, ауксотрофного по холину, восстанавливался в такой же степени, как и в контрольной группе, и содержание неочищенного жира в штамме также увеличивалось на приблизительно 10% по сравнению с ростом без добавления холина.
Рост и накопление жира в штаммах с дефицитом cho2 и opi3, полученных на основе дрожжей Yarrowia lipolytica PO1f, показали сходную тенденцию по сравнению с почкующимися дрожжами Saccharomyces cerevisiae, но эти штаммы отличались от Saccharomyces cerevisiae тем, что при добавлении холина их рост восстанавливался, а содержание жира также увеличивалось, что характерно для жирообразующих дрожжей Yarrowia lipolytica.
2-2. Оценка роста и накопления жира в соответствии с отношением C/N
При выполнении культивирования в среде YLMM1 с отношением C/N 30 по сравнению со средой YPD с отношением C/N менее 1 в примере 2-1 было подтверждено, что содержание жира в штамме увеличивается. Соответственно три вида штаммов, полученных в примере 1, и контроль PO1f культивировали в тестовой среде CN, имеющей различные отношения C/N, и оценивали рост и накопление жира в соответствии с отношением C/N. Каждый штамм высевали в колбу с угловой перегородкой вместимостью 250 мл, содержащую 50 мл тестовой среды CN, в которую был добавлен сульфат аммония в различных концентрациях, при начальной OD 0,01 и культивировали при встряхивании при 250 об/мин и 30°С в течение 72 часов. Ниже описана композиция тестовой среды CN в соответствии с отношением C/N.
<Тестовая среда CN (рН 7,2)>
40 г/л глюкозы; 1,7 г/л дрожжевых азотистых оснований без аминокислот и сульфата аммония; 0,5 г/л урацила; 0,3 мМ холина хлорида; 2,5 г/л (отношение C/N 30), 1,25 г/л (отношение C/N 60), 0,83 г/л (отношение C/N 90) или 0,63 г/л (отношение C/N 120) сульфата аммония растворяли в 0,1 М натрий-фосфатном буферном растворе (рН 7,2).
После окончания культивирования культуральную среду центрифугировали, отбрасывая супернатант, и клетки, остающиеся в нижнем слое, высушивали в сушильном шкафу при 60°С в течение 24 часов с последующей экстракцией жира. Экстракцию неочищенного жира выполняли в соответствии со стандартным методом анализа кормов (Feed Standard Analysis Method. 2001. National Livestock Research Institute, Rural Development Administration), и результаты анализа OD и содержания внутриклеточного неочищенного жира представлены ниже в таблице 4. Результаты в таблице 4 представляют собой результаты экспериментов, выполненных в трех повторах, а увеличение содержания жира оценивали на основании их средних значений.
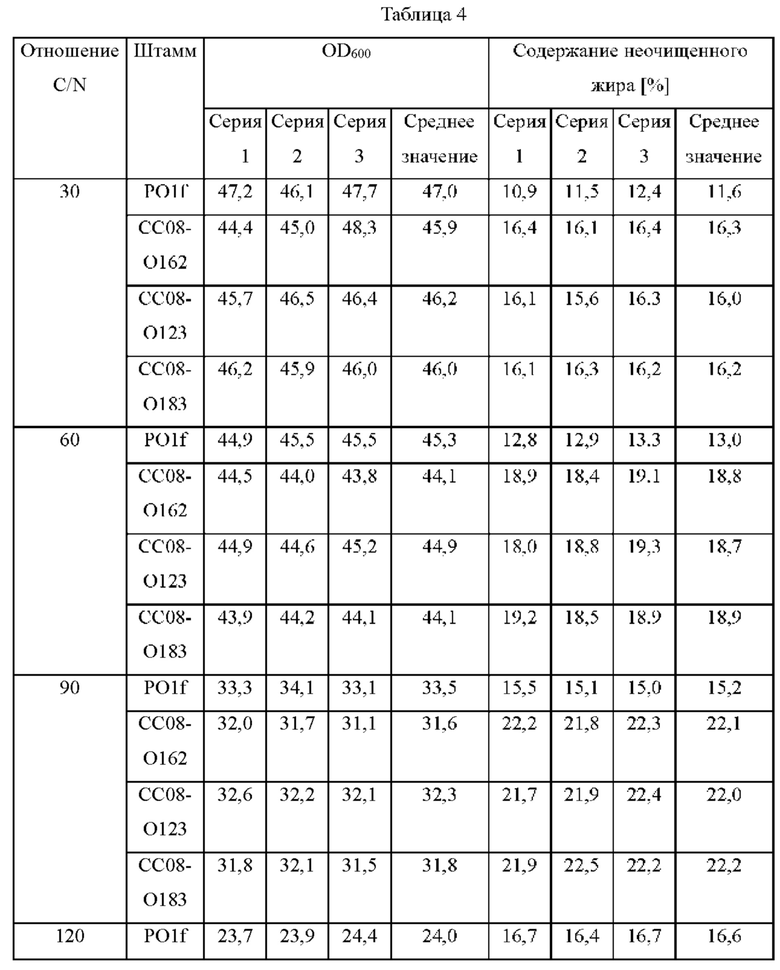
Как показано в таблице 4, подтвердили, что содержание жира увеличивалось независимо от отношения C/N среды в штаммах, в которых по отдельности или одновременно были делетированы гены cho2 или opi3 из родительского штамма PO1f, как в примере 2-1. Также наблюдали, что уровень этого увеличения был сходным и составлял приблизительно 43% независимо от отношения C/N.
Тем не менее, по мере увеличения отношения C/N в среде источник азота уменьшался, и отмечалась тенденция к снижению OD, тогда как содержание внутриклеточного жира имело тенденцию к увеличению. Чтобы получить отношение C/N, показывающее максимальную продуктивность, рассчитывали значение OD * содержание жира. Во всех штаммах при отношении C/N, равном 60, наблюдалось максимальное значение OD * содержание жира. Таким образом, определили, что оптимальным является отношение C/N, равное 60.
Пример 3. Получение и оценка накапливающего жир штамма с дефицитом на основе SCO023
3-1. Получение накапливающего жир штамма SCO023
Для изучения влияния делеции гена, кодирующего СН02 или OPI3, на накапливающий жир штамм был получен штамм дрожжей с высокой продукцией жира.
Для получения штамма дрожжей с высокой продукцией жира сначала блокировали путь деградации TAG. Путь деградации TAG приведен ниже: TAG преобразуется в жирную кислоту (FA) под действием TGL3 и 4, и FA поступает внутрь пероксисомы с помощью ацил-КоА-связывающих белков. Поступившая FA ацилируется под действием двух пероксисомальных ацил-КоА-синтаз (pxa1, 2), и ацилированная FA претерпевает десатурацию под действием ацил-КоА-оксидаз (pox1-6) в положении винила. В десатурированном сложном эфире FA-KoA участок двойной связи гидратируется под действием многофункционального фермента 2 (домен MFE2-C, mfe1), и образуется промежуточное соединение 3-гидроксиацил-КоА. В данном случае под действием домена А/В фермента MFE2 промежуточное соединение 3-гидроксиацил-КоА окисляется до промежуточного соединения 3-кетоацил-КоА, и промежуточное соединение 3-кетоацил-КоА расщепляется при альфа-атоме углерода под действием пероксисомальной 3-оксиацилтиолазы (pot1) с образованием ацетил-КоА и жирного ацил-КоА. Деградация FA происходит по мере повторения цикла от реакции рох до реакции pot1 (AIMS Bioengineering 2016. 3(4): 493-514).
Среди генов в штамме Yarrowia lipolytica PO1f были делетированы рох2 (YALI0F10857), mfe1 (YALI0E15378) и pot1 (YALI0E18568), чтобы предотвратить экспрессию каждого фермента. Кроме того, чтобы уменьшить количество пероксисом, место, где происходит бета-окисление, pex10 (YALI0C01023), который представляет собой ген, вовлеченный в образование пероксисом, был делетирован, чтобы предотвратить экспрессию этого фермента (AIMS Bioengineering 2016. 3(4): 493-514). Наконец, был делетирован ген mhyl (YALI0B21582), который представляет собой ген, связанный с морфологией. Если ген mhyl делетирован, уменьшается приток углерода в биосинтез аминокислот и увеличивается приток углерода в биосинтез жира, в результате чего увеличивается содержание внутриклеточного неочищенного жира (Biochim. Biophys. Acta. 2018. 1863(1): 81-90).
Для получения кассеты, способной к делеции каждого гена, была получена полинуклеотидная последовательность SEQ ID NO: 21 и аминокислотная последовательность SEQ ID NO: 22 рох2, полинуклеотидная последовательность SEQ ID NO: 23 и аминокислотная последовательность SEQ ID NO: 24 mfe1, полинуклеотидная последовательность SEQ ID NO: 25 и аминокислотная последовательность SEQ ID NO: 26 pexlO, полинуклеотидная последовательность SEQ ID NO: 27 и аминокислотная последовательность SEQ ID NO: 28 pot1 и полинуклеотидная последовательность SEQ ID NO: 29 и аминокислотная последовательность SEQ ID NO: 30 mhy1 на основе нуклеотидных последовательностей, зарегистрированных в KEGG.
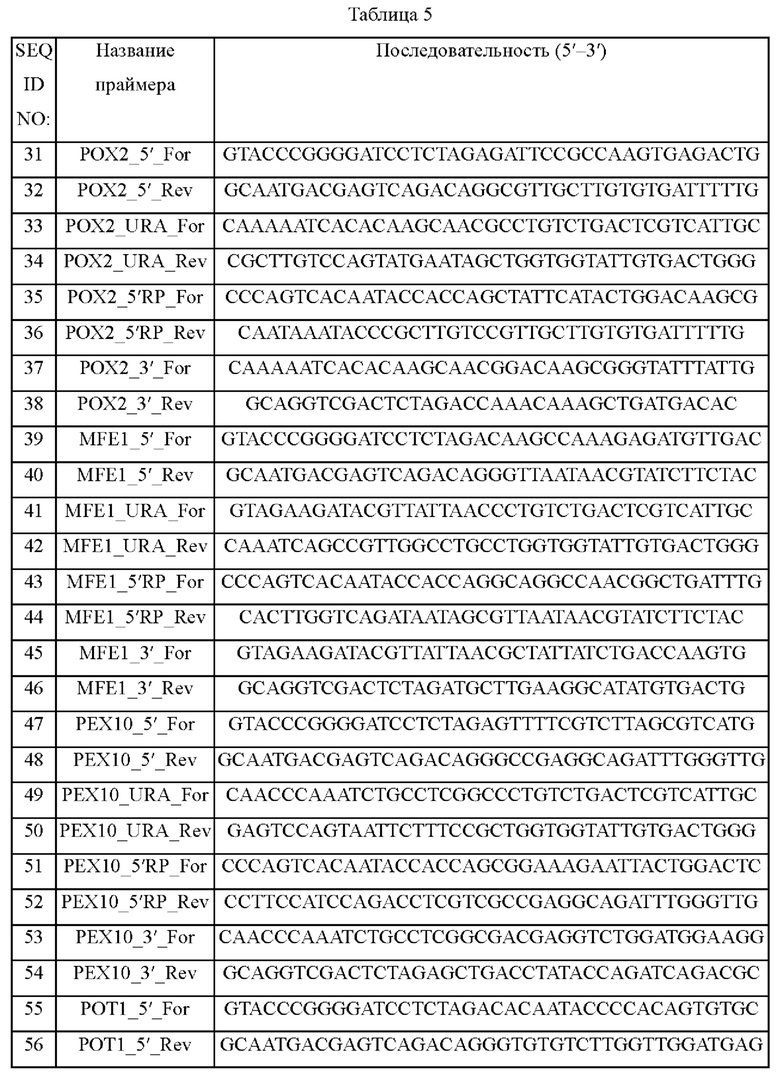
Конструирование кассет с дефицитом по каждому гену выполняли в порядке 5'UTR-URA3-5UTR_RP-3'UTR и 5'UTR, 5'UTR_RP, и 3'UTR подвергали ПЦР с использованием геномной ДНК Yarrowia lipolytica PO1f в качестве матрицы и приведенных ниже праймеров, соответственно; для получения кассеты с дефицитом по pox2 использовали праймеры SEQ ID NO: 31 и 32, SEQ ID NO: 35 и 36, SEQ ID NO: 37 и 38, для получения кассеты с дефицитом по mfe1 праймеры SEQ ID NO: 39 и 40, SEQ ID NO: 43 и 44, SEQ ID NO: 45 и 46, для получения кассеты с дефицитом по рехЮ праймеры SEQ ID NO: 47 и 48, SEQ ID NO: 51 и 52, SEQ ID NO: 53 и 54, для получения кассеты с дефицитом по pot1 праймеры SEQ ID NO: 55 и 56, SEQ ID NO: 59 и 60, SEQ ID NO: 61 и 62 и для получения кассеты с дефицитом по mhy1 праймеры SEQ ID NO: 63 и 64, SEQ ID NO: 67 и 68, SEQ ID NO: 69 и 70. Для URA3 выполняли ПЦР с использованием маркера ауксотрофности URA3 в качестве матрицы и праймеров с SEQ ID NO: 33 и 34 для получения кассеты с дефицитом по рох2, праймеры SEQ ID NO: 41 и 42 для получения кассеты с дефицитом по mfe1, праймеры SEQ ID NO: 49 и 50 для получения кассеты с дефицитом по pex10, праймеры SEQ ID NO: 57 и 58 для получения кассеты с дефицитом по pot1 и праймеры SEQ ID NO: 65 и 66 для получения кассеты с дефицитом по mhy1.
ПЦР выполняли в течение 35 циклов в условиях, состоящих в денатурации при 95°С в течение 1 минуты; отжига при 55°С в течение 1 минуты и полимеризации при 72°С в течение 1 минуты и 30 секунд. В результате получили рох2_5' UTR 1035 п.о., рох2_5' UTR_RP 720 п.о., рох2_3' UTR 1010 п.о., pox2_URA3 1569 п.о., mfe1_5' UTR 1003 п.о., mfe1_5' UTR_RP 709 п.о., mfe1_3' UTR 1036 п.о., mfe1_URA3 1569 п.о., рех10_5' UTR 1032 п.о., рех10_5' UTR_RP 735 п.о., рех10_3' UTR 1033 п.о., рех10_URA3 1569 п.о., pot1_5' UTR 1036 п.о., pot1_5' UTR_RP 735 п.о., pot1_3' UTR 1010 п.о., pot1_URA3 1569 п.о., mhy1_5' UTR 921 п.о., mhy1_5' UTR_RP 526 п.о., mhy1_3' UTR 1029 п.о. и mhy1_URA3 1569 п.о. Из фрагментов ДНК, амплифицированных с помощью ПЦР, была получена кассета с дефицитом рох2-, mfe1-, рех10-, pot1- или mhy1 посредством ПЦР с перекрывающимися праймерами и удлинением.
Сначала кассетой с дефицитом рох2 трансформировали Yarrowia lipolytica PO1f методом теплового шока и отбирали на твердой среде, не содержащей урацил. Первичный штамм, отобранный таким образом, подвергали второму кроссинговеру с получением штамма с дефицитом по гену рох2. Таким же образом были получены штаммы с дефицитом по mfe1, рех10, pot1 и mhy1 с использованием кассет с дефицитом mfe1, рех10, pot1 или mhy1. Полученные таким путем штаммы с дефицитом были обозначены как SCO023.

3-2. Получение штамма с дефицитом cho2 на основе SCO023 (SCO079)
Штамм с дефицитом гена cho2 был получен на основе штамма дрожжей SCO023 с высокой продукцией жира, полученного в примере 3-1. Кассетой с дефицитом cho2, полученной в примере 1-1, трансформировали SCO023 методом теплового шока и отбирали на твердой среде, не содержащей урацил. Первичный штамм, отобранный таким образом, подвергали второму кроссинговеру с получением штамма с дефицитом по гену cho2. Полученный таким образом штамм с дефицитом был обозначен как SCO079.
3-3. Получение штамма с дефицитом opi3 на основе SCO023 (SC0163)
Штамм с дефицитом гена opi3 был получен на основе штамма дрожжей SCO023 с высокой продукцией жира, полученного в примере 3-1. Кассетой с дефицитом opi3, полученной в примере 1-2, трансформировали SCO023 методом теплового шока и отбирали на твердой среде, не содержащей урацил. Первичный штамм, отобранный таким образом, подвергали второму кроссинговеру с получением штамма с дефицитом по гену opi3. Полученный таким образом штамм с дефицитом был обозначен как SC0163.
3-4. Тест в колбах
Тест в колбах был выполнен на штаммах SCO079 и SCO163, полученных в примерах 3-2 и 3-3.
Два вида штаммов и контрольную группу (PO1f) высевали в колбу с угловой перегородкой вместимостью 250 мл, содержащую 50 мл среды YLMM2, в которую был добавлен холин в различных концентрациях, при начальной OD 0,01 и культивировали при встряхивании при 250 об/мин и 30°С в течение 72 часов. Композиция среды YLMM2 описана ниже.
<YLMM2 (рН 7,2)>
40 г/л глюкозы, 1,7 г/л азотистых оснований для дрожжей без аминокислот и сульфата аммония, 0,5 г/л урацила и 2,5 г/л сульфата аммония растворяли в 0,1 М натрий-фосфатном буфере (рН 7,2).
После окончания культивирования культуральную среду центрифугировали, отбрасывая супернатант, и клетки, остающиеся в нижнем слое, высушивали в сушильном шкафу при 60°С в течение 24 часов с последующей экстракцией жира. Экстракцию неочищенного жира выполняли в соответствии со стандартным методом анализа кормов (Feed Standard Analysis Method. 2001. National Livestock Research Institute, Rural Development Administration), и результаты анализа OD и содержания внутриклеточного жира представлены ниже в таблице 6. Результаты в таблице 6 представляют собой результаты экспериментов, выполненных в трех повторах, а увеличение содержания жира оценивали на основании их средних значений.
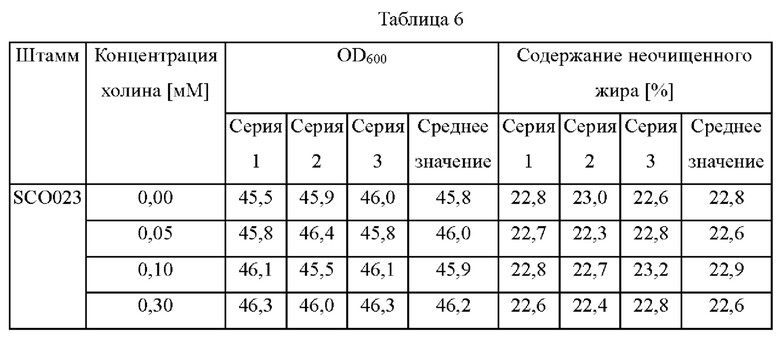
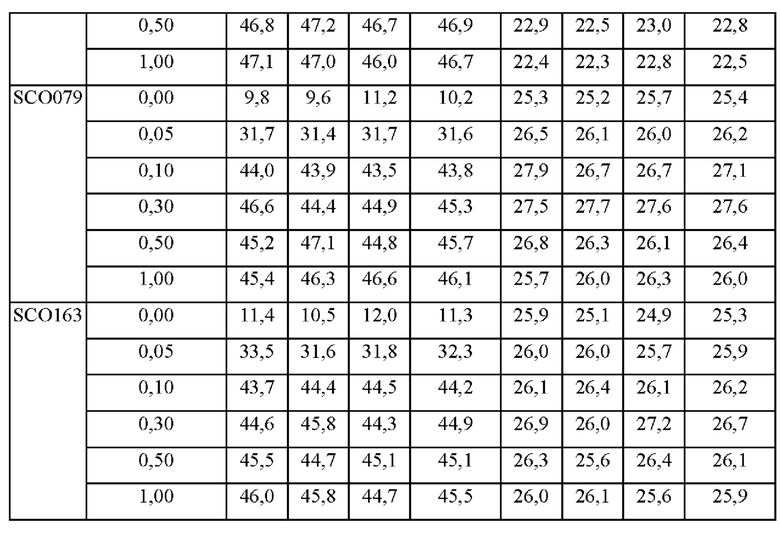
Как показано в таблице 6, штамм, в котором был делетирован ген cho2 или opi3 из родительского штамма SCO023, показал равное или сходное содержание жира по сравнению с родительским штаммом SCO023 в среде, содержащей холин в концентрации 0 мМ или 0,055 мМ, но OD была значительно снижена. При добавлении в среду холина в концентрации 0,1 мМ или более OD восстанавливалась до уровня контрольной группы, и также наблюдался эффект увеличения содержания жира в штамме. При добавлении от 0,1 мМ до 1 мМ холина в среду было показано увеличение содержания неочищенного жира в штамме SCO079 на 16-22%, а для штамма SC0163 показано увеличение содержания неочищенного жира на 14 18% по сравнению с SCO023. Тем не менее в штамме SCO023, где сохранился путь РЕМТ, изменений содержания неочищенного жира не было, независимо от концентрации холина в среде.
В результате было подтверждено, что при добавлении необходимого количества холина к штамму, в котором делетирован ген cho2 или opi3 на пути РЕМТ, рост штамма восстанавливался и содержание жира в штамме увеличивались. Тем не менее, было подтверждено, что при добавлении холина после делеции генов cho2 и opi3 на основе накапливающего жир штамма SCO023 увеличение содержания жира относительно невелико по сравнению со случаем, где гены cho2 и opi3 были делегированы на основе штамма дикого типа.
Пример 4. Получение и оценка накапливающего жир штамма с дефицитом на основе SCO028
4-1. Получение накапливающего жир штамма SCO028
Для изучения влияния делеции гена, кодирующего СН02 или OPI3, на штамм, имеющий более высокое содержание жира по сравнению со штаммом, полученным в примере 3-1, был получен другой штамм дрожжей с высокой продукцией жира.
Известно, что содержание жира увеличивается при гиперэкспрессии изофермента I ацил-КоА:диацилглицеринацилтрансферазы (DGA1), который является конечным ферментом пути биосинтеза жира, на основе штаммов с дефицитом mfe1 и рех10 (Nat. Commun. 2014. 5:3131). Таким образом, промотор dga1 (YALI0E32769) был заменен промотором TEFINt на основе штамма SCO023, полученного в примере 3-1, в котором путь деградации жира был блокирован, а приток углерода в биосинтез жира увеличен (МЕТАВ ENG 2015. 29: 56-65). Процесс получения кассеты для замены промотора выполняли, как описано ниже.
Для получения кассеты, способной к замене промотора гена dga1, была получена полинуклеотидная последовательность SEQ ID NO: 71 и аминокислотная последовательность SEQ ID NO: 72 dga1 на основе нуклеотидной последовательности, зарегистрированной в KEGG. Конструирование кассеты для замены промотора dga1 выполняли в порядке 5' UTR-TEFINt1-URA3-TEFINt2-ген (dga1), и для 5' UTR, TEFINt1 и TEFINt2 выполняли ПЦР с использованием геномной ДНК Yarrowia lipolytica PO1f в качестве матрицы и праймеров с SEQ ID NO: 73 и 74, SEQ ID NO: 75 и 76, SEQ ID NO: 79 и 80, SEQ ID NO: 81 и 82 соответственно. Далее выполняли ПЦР с использованием маркера ауксотрофности URA3 в качестве матрицы и праймеров с SEQ ID NO: 77 и 78. ПЦР выполняли в течение 35 циклов в условиях, состоящих в денатурации при 95°С в течение 1 минуты; отжига при 55°С в течение 1 минуты и полимеризации при 72°С в течение 1 минуты и 30 секунд. В результате получили dga1_5' UTR 1539 п. о., dga1_TEFINt1 573 п. о., dga1_TEFINt2 571 п. о., dga1_ген 1539 п. о. и dga1_URA3 1574 п. о. Фрагменты ДНК, амплифицированные с помощью ПЦР, подвергали ПЦР с перекрывающимися праймерами и удлинением с получением кассеты для замены промотора dga1.
Кассетой для замены промотора dga1 трансформировали SCO023 методом теплового шока и отбирали на твердой среде, не содержащей урацил. Отобранный таким путем первичный штамм подвергали второму кроссинговеру с получением штамма, в котором промотор dga1 был заменен промотором TEFINt. Полученный таким образом штамм с дефицитом был обозначен как SCO028.
4-2. Получение штамма с дефицитом cho2 на основе SCO028 (SCO048)
Штамм с дефицитом гена cho2 был получен на основе штамма дрожжей SCO028 с высокой продукцией жира, полученного в примере 4-1. Кассетой с дефицитом cho2, полученной в примере 1-1, трансформировали SCO028 методом теплового шока и отбирали на твердой среде, не содержащей урацил. Первичный штамм, отобранный таким образом, подвергали второму кроссинговеру с получением штамма с дефицитом по гену cho2. Полученный таким образом штамм с дефицитом был обозначен как SCO048.
4-3. Получение штамма с дефицитом opi3 на основе SCO028 (SCO067)
Штамм с дефицитом гена opi3 был получен на основе высокопродуктивного продуцирующего жир штамма дрожжей SCO028, полученного в примере 4-1. Кассетой с дефицитом opi3, полученной в примере 1-2, трансформировали SCO028 методом теплового шока и отбирали на твердой среде, не содержащей урацил. Первичный штамм, отобранный таким образом, подвергали второму кроссинговеру с получением штамма с дефицитом по гену opi3. Полученный таким образом штамм с дефицитом был обозначен как SCO067.
4-4. Тест в колбах
Тест в колбах был выполнен на штаммах SCO048 и SCO067, полученных в примерах 4-2 и 4-3.
Два вида штаммов и SCO028 высевали в колбу с угловой перегородкой вместимостью 250 мл, содержащую 50 мл среды YLMM2, не содержащую холин или содержащую 0,3 мМ холина, при начальной OD 0,01 и культивировали при встряхивании при 250 об/мин и 30°С в течение 72 часов. Композиция YLMM2 является такой же, как в примере 3-4.
После окончания культивирования кулыуральную среду центрифугировали, отбрасывая супернатант, и клетки, остающиеся в нижнем слое, высушивали в сушильном шкафу при 60°С в течение ночи с последующей экстракцией жира. Экстракцию неочищенного жира выполняли в соответствии со стандартным методом анализа кормов (Feed Standard Analysis Method. 2001. National Livestock Research Institute, Rural Development Administration), и результаты анализа OD и содержания внутриклеточного жира представлены ниже в таблице 8. Результаты в таблице 8 представляют собой результаты экспериментов, выполненных в трех повторах, а увеличение содержания жира оценивали на основании их средних значений.
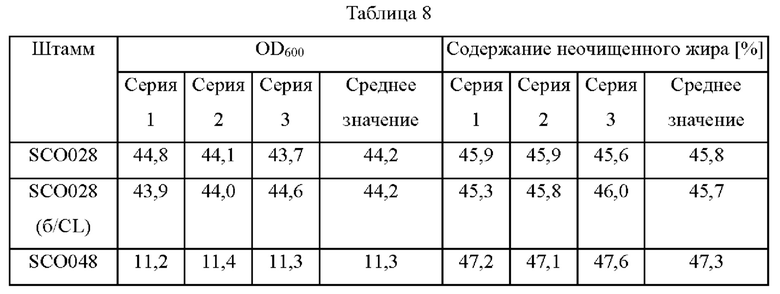
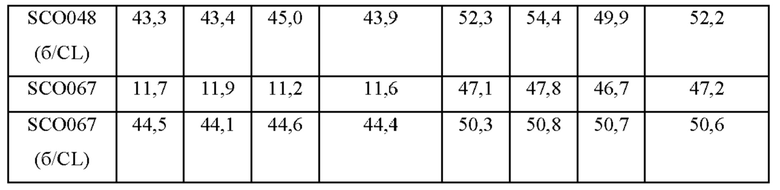
Как показано в таблице 8, штамм, в котором был делетирован ген cho2 или opi3 из родительского штамма SCO028, показал равное или сходное содержание жира по сравнению с родительским штаммом SCO028 в среде, не содержащей холин, но OD была значительно снижена.
При добавлении в среду 0,3 мМ холина OD восстанавливалась до уровня контрольной группы, и также наблюдался эффект увеличения содержания жира в штамме, как и в примере 3-4. При добавлении 0,3 мМ холина в среду было показано увеличение содержания жира в штамме SCO048 на 14%, а для штамма SCO067 показано увеличение содержания жира на 10% по сравнению с SCO028.
Соответственно, было также подтверждено, что при добавлении надлежащего количества холина к штамму, в котором делетирован ген cho2 или opi3, рост штамма восстанавливался, и в то же время содержание жира в штамме увеличивались. В дополнение к этому, как описано в примере 3-4, по мере увеличения содержания жира в штамме это увеличение содержания жира снижалось, когда гены cho2 и opi3 были делетированы.
На основании приведенного выше описания специалистам в данной области техники будет понятно, что описание настоящего изобретения можно применять на практике в другой конкретной форме без изменения его технической сущности или существенных характеристик. Таким образом, должно быть понятно, что описанное выше воплощение не имеет ограничительного характера, но является иллюстративным во всех аспектах. Объем описания изобретения определяется в большей степени прилагаемой формулой изобретения, чем предшествующим ей описанием, и, следовательно, все изменения и модификации, входящие в границы формулы изобретения или эквиваленты таких границ, предназначены для включения в объем формулы изобретения.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> CJ CheilJedang Corporation
<120> ВАРИАНТ ИЗ РОДА YARROWIA И СПОСОБ ПОЛУЧЕНИЯ ЖИРА С ЕГО
ИСПОЛЬЗОВАНИЕМ
<130> OPA20210
<150> KR 10-2020-0029137
<151> 2020-03-09
<160> 82
<170> KoPatentIn 3.0
<210> 1
<211> 2829
<212> DNA
<213> Yarrowia lipolytica
<400> 1
atggccgttc cttctgctgc catcaaggct tccggagtgg atatggggga cctcaagaag
60
agacatgaca ccaagcttga cgtggaggag agtgtgattg acgagaagat caatgagaat
120
gaaagcgacg aaaagctgac cgaaaagctg accgaaaagc tgaccgaaaa gctgactgaa
180
aagacctcca ccgagcccat tgcttacgtt ggcaagtgtc ctgatggaac gacttttatt
240
gtgccagaga cggaggatat gttgaccaac ctgtttgatc ctcgaatcac aaagtcgctg
300
acggacgtga tcatcgtatc gattctggtc acattcatgg gactcttttt tgtcgtcccc
360
aagtcctgga gagtacctct ctatctgttt ctgtttgccg catggcgtct ggcctacaac
420
ggaggaatcg gctggctact tcacaaccag tccaactacc acaaactcac caagtgggct
480
ctcaaataca aggtgtttga caaggacaac aagacgtggt ggcaccagct gattaagcgg
540
gagtttgaga cccggttcca gaaccagccc aactactcgt tctacgaggt acccgtcgag
600
ttcaacacct ggattctttt ccgacatgtg gtcgatctga ttctcatgtc agactttacc
660
tgctacttca tgctggcctg gtcctgcgct ctgtccgtga gaaccaacca gccctggtgg
720
cttgttattg gacgatggct cgccggagct ctaatgctag ccttcaacct gtgggctaag
780
actgacgccc accgagtcgt caaggattat gcctggtact ggggagactt cttcttcctc
840
aaggacatgg agttgacttt tgatggtgtc tttgagatgg cccctcatcc catgtactcc
900
attggatacg ctggtttcta cgccgcctct ctcatggcct gttcctacac cctgttcctg
960
gcctctctgg ccggccacgc tgcccagttt gtgttcctat ccatcgttga gaatcctcac
1020
attgaaaaga cttacaaccc ccatcagccc aagcagcgac gaaaggcttc tcacgtccga
1080
gaccgatctt ccatccagct ggggctcgat cagaaacaga aggatgagct gtctgctatt
1140
gttgagggcg acgagtccgt ttctattgcc tctactccca tttctactac ttttgacgag
1200
ccggagactg tccagaaaca ctcccttcct ccgttggttg tgtttagcaa cttccagatc
1260
actcgagtca cagatatcat gactgtggga gctgcagtct atacaatgct gctctacttc
1320
ctccctgcca accgtctgtg gtacactgtg acttttttca tggctctgtc tgctcgtctt
1380
ttccacactc tgggtcttgg agtcatactg cgacgacagt ccgagcgacg atccttcacc
1440
aagaccttcc tcaagtttgg aatctacccc tttgaggcat acgagcagtg gcaagtgtgg
1500
tacaacgtct ccaccgtgtt gtcctacacc acatttggtc tcttttgcct gcgacagtgg
1560
agagctcctt ccaccgatcc tctgtggccc cttaagtaca ttcttggcgc tctacttatt
1620
gctctgcatg gctggacatc gaagtccatc catgactctc ttgggcattt tggatggttc
1680
tacggtgact tcttccttga ccgaaagcag aacctcactt actccggcat ttacagatac
1740
ctcaacaacc cagagcgatt tttcggtatt gctggcatct ggggtcttgc tctgatgacc
1800
aactctccag gtgttggtat tttggccttc ttgtggactc tggagggtat ggcattcatc
1860
aagttcgtcg agcagcccca catgcagcga atctacggta ccagtatccg acacgatgct
1920
ggtgtgacca agaccattaa gaactctctc aagttgcctt ctccctttga gaaacgggtt
1980
cgacagttcc agggatccgt tgacaaggtt atcaacgata ctctgactgt ggtcgaggag
2040
ttcattggac ttgccaagcc tactctcaac gaagttgtca acgactccaa gatcctgttg
2100
cgacagtacc ctgccaaact tacccttact cgaatgctgg atgttcccga aaacattgac
2160
actgccgatt actctgtgaa gctcgtgggc ggcgagaagt gtgccgattc caaccgaatt
2220
tcgtacgcct ttggaacccc cattcatgtc aagtggcagg cttctcccaa cagatcggca
2280
aaggactgga tcggtctgta ccgagtgcac gacaacactt cccctgaggt gacttctctt
2340
ccctccaagg gccgatggtc tggaatcgac gagactggcc atgagtcgca tctcgacggt
2400
attgtgtcgt cttctaagac caatggagaa gttgttttcc gaggggacac actcttctgg
2460
gagtgtggag tctacgagtt cagataccac catgctggca agcacactgt gctcagcacc
2520
tcggaacctt tcgagattgt ggctcctaag attgatctat ctggagaggt ggatgcaaag
2580
actctggctc ttgagatcct ccccatcgtc cagcgatgct tctctctatc tatggagttc
2640
cctcctgagg aagtggacga ctactgggct cttgaggaac ccaaggtgat caacagagtg
2700
caggctgccg ttcaggcgta tttcaacgtt gagctggcgc aggaggtgct tcaatcggat
2760
gagtctgtgg agaatttggc tgagcgactg ctcagaatta gagacgccct taaaggtctt
2820
actgtataa
2829
<210> 2
<211> 942
<212> PRT
<213> Yarrowia lipolytica
<400> 2
Met Ala Val Pro Ser Ala Ala Ile Lys Ala Ser Gly Val Asp Met Gly
1 5 10 15
Asp Leu Lys Lys Arg His Asp Thr Lys Leu Asp Val Glu Glu Ser Val
20 25 30
Ile Asp Glu Lys Ile Asn Glu Asn Glu Ser Asp Glu Lys Leu Thr Glu
35 40 45
Lys Leu Thr Glu Lys Leu Thr Glu Lys Leu Thr Glu Lys Thr Ser Thr
50 55 60
Glu Pro Ile Ala Tyr Val Gly Lys Cys Pro Asp Gly Thr Thr Phe Ile
65 70 75 80
Val Pro Glu Thr Glu Asp Met Leu Thr Asn Leu Phe Asp Pro Arg Ile
85 90 95
Thr Lys Ser Leu Thr Asp Val Ile Ile Val Ser Ile Leu Val Thr Phe
100 105 110
Met Gly Leu Phe Phe Val Val Pro Lys Ser Trp Arg Val Pro Leu Tyr
115 120 125
Leu Phe Leu Phe Ala Ala Trp Arg Leu Ala Tyr Asn Gly Gly Ile Gly
130 135 140
Trp Leu Leu His Asn Gln Ser Asn Tyr His Lys Leu Thr Lys Trp Ala
145 150 155 160
Leu Lys Tyr Lys Val Phe Asp Lys Asp Asn Lys Thr Trp Trp His Gln
165 170 175
Leu Ile Lys Arg Glu Phe Glu Thr Arg Phe Gln Asn Gln Pro Asn Tyr
180 185 190
Ser Phe Tyr Glu Val Pro Val Glu Phe Asn Thr Trp Ile Leu Phe Arg
195 200 205
His Val Val Asp Leu Ile Leu Met Ser Asp Phe Thr Cys Tyr Phe Met
210 215 220
Leu Ala Trp Ser Cys Ala Leu Ser Val Arg Thr Asn Gln Pro Trp Trp
225 230 235 240
Leu Val Ile Gly Arg Trp Leu Ala Gly Ala Leu Met Leu Ala Phe Asn
245 250 255
Leu Trp Ala Lys Thr Asp Ala His Arg Val Val Lys Asp Tyr Ala Trp
260 265 270
Tyr Trp Gly Asp Phe Phe Phe Leu Lys Asp Met Glu Leu Thr Phe Asp
275 280 285
Gly Val Phe Glu Met Ala Pro His Pro Met Tyr Ser Ile Gly Tyr Ala
290 295 300
Gly Phe Tyr Ala Ala Ser Leu Met Ala Cys Ser Tyr Thr Leu Phe Leu
305 310 315 320
Ala Ser Leu Ala Gly His Ala Ala Gln Phe Val Phe Leu Ser Ile Val
325 330 335
Glu Asn Pro His Ile Glu Lys Thr Tyr Asn Pro His Gln Pro Lys Gln
340 345 350
Arg Arg Lys Ala Ser His Val Arg Asp Arg Ser Ser Ile Gln Leu Gly
355 360 365
Leu Asp Gln Lys Gln Lys Asp Glu Leu Ser Ala Ile Val Glu Gly Asp
370 375 380
Glu Ser Val Ser Ile Ala Ser Thr Pro Ile Ser Thr Thr Phe Asp Glu
385 390 395 400
Pro Glu Thr Val Gln Lys His Ser Leu Pro Pro Leu Val Val Phe Ser
405 410 415
Asn Phe Gln Ile Thr Arg Val Thr Asp Ile Met Thr Val Gly Ala Ala
420 425 430
Val Tyr Thr Met Leu Leu Tyr Phe Leu Pro Ala Asn Arg Leu Trp Tyr
435 440 445
Thr Val Thr Phe Phe Met Ala Leu Ser Ala Arg Leu Phe His Thr Leu
450 455 460
Gly Leu Gly Val Ile Leu Arg Arg Gln Ser Glu Arg Arg Ser Phe Thr
465 470 475 480
Lys Thr Phe Leu Lys Phe Gly Ile Tyr Pro Phe Glu Ala Tyr Glu Gln
485 490 495
Trp Gln Val Trp Tyr Asn Val Ser Thr Val Leu Ser Tyr Thr Thr Phe
500 505 510
Gly Leu Phe Cys Leu Arg Gln Trp Arg Ala Pro Ser Thr Asp Pro Leu
515 520 525
Trp Pro Leu Lys Tyr Ile Leu Gly Ala Leu Leu Ile Ala Leu His Gly
530 535 540
Trp Thr Ser Lys Ser Ile His Asp Ser Leu Gly His Phe Gly Trp Phe
545 550 555 560
Tyr Gly Asp Phe Phe Leu Asp Arg Lys Gln Asn Leu Thr Tyr Ser Gly
565 570 575
Ile Tyr Arg Tyr Leu Asn Asn Pro Glu Arg Phe Phe Gly Ile Ala Gly
580 585 590
Ile Trp Gly Leu Ala Leu Met Thr Asn Ser Pro Gly Val Gly Ile Leu
595 600 605
Ala Phe Leu Trp Thr Leu Glu Gly Met Ala Phe Ile Lys Phe Val Glu
610 615 620
Gln Pro His Met Gln Arg Ile Tyr Gly Thr Ser Ile Arg His Asp Ala
625 630 635 640
Gly Val Thr Lys Thr Ile Lys Asn Ser Leu Lys Leu Pro Ser Pro Phe
645 650 655
Glu Lys Arg Val Arg Gln Phe Gln Gly Ser Val Asp Lys Val Ile Asn
660 665 670
Asp Thr Leu Thr Val Val Glu Glu Phe Ile Gly Leu Ala Lys Pro Thr
675 680 685
Leu Asn Glu Val Val Asn Asp Ser Lys Ile Leu Leu Arg Gln Tyr Pro
690 695 700
Ala Lys Leu Thr Leu Thr Arg Met Leu Asp Val Pro Glu Asn Ile Asp
705 710 715 720
Thr Ala Asp Tyr Ser Val Lys Leu Val Gly Gly Glu Lys Cys Ala Asp
725 730 735
Ser Asn Arg Ile Ser Tyr Ala Phe Gly Thr Pro Ile His Val Lys Trp
740 745 750
Gln Ala Ser Pro Asn Arg Ser Ala Lys Asp Trp Ile Gly Leu Tyr Arg
755 760 765
Val His Asp Asn Thr Ser Pro Glu Val Thr Ser Leu Pro Ser Lys Gly
770 775 780
Arg Trp Ser Gly Ile Asp Glu Thr Gly His Glu Ser His Leu Asp Gly
785 790 795 800
Ile Val Ser Ser Ser Lys Thr Asn Gly Glu Val Val Phe Arg Gly Asp
805 810 815
Thr Leu Phe Trp Glu Cys Gly Val Tyr Glu Phe Arg Tyr His His Ala
820 825 830
Gly Lys His Thr Val Leu Ser Thr Ser Glu Pro Phe Glu Ile Val Ala
835 840 845
Pro Lys Ile Asp Leu Ser Gly Glu Val Asp Ala Lys Thr Leu Ala Leu
850 855 860
Glu Ile Leu Pro Ile Val Gln Arg Cys Phe Ser Leu Ser Met Glu Phe
865 870 875 880
Pro Pro Glu Glu Val Asp Asp Tyr Trp Ala Leu Glu Glu Pro Lys Val
885 890 895
Ile Asn Arg Val Gln Ala Ala Val Gln Ala Tyr Phe Asn Val Glu Leu
900 905 910
Ala Gln Glu Val Leu Gln Ser Asp Glu Ser Val Glu Asn Leu Ala Glu
915 920 925
Arg Leu Leu Arg Ile Arg Asp Ala Leu Lys Gly Leu Thr Val
930 935 940
<210> 3
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CHO2_5_For
<400> 3
gtacccgggg atcctctaga gtcatccgca aacacaacac
40
<210> 4
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CHO2_5_Rev
<400> 4
gcaatgacga gtcagacagg ggtgtttgtg gaagctggtg
40
<210> 5
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CHO2_URA_For
<400> 5
caccagcttc cacaaacacc cctgtctgac tcgtcattgc
40
<210> 6
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CHO2_URA_Rev
<400> 6
cgcattctgc gtacattttg ctggtggtat tgtgactggg
40
<210> 7
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CHO2_5RP_For
<400> 7
cccagtcaca ataccaccag caaaatgtac gcagaatgcg
40
<210> 8
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CHO2_5RP_Rev
<400> 8
cagatatgct ctctgcaaac ggtgtttgtg gaagctggtg
40
<210> 9
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CHO2_3_For
<400> 9
caccagcttc cacaaacacc gtttgcagag agcatatctg
40
<210> 10
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> CHO2_3_Rev
<400> 10
gcaggtcgac tctagagaga ggtctgttca caacatcggc
40
<210> 11
<211> 627
<212> DNA
<213> Yarrowia lipolytica
<400> 11
atgtcctttc ctgacaagat tgttgccgac ttcaacgtgg attacgacca gaagagcctc
60
tggatcgtcc tgggctccat cgccttcaac cccatctttt ggaacattgt tgctcgagcc
120
gagtacaaca cccactttct gactaagctg gctggaggag ccaagaccgg atgctatctg
180
ctggccatca ccattttctc gctcggtatc ttccgagact acctttacca ccaggctctt
240
gagcagcagc ccacctccat tctgctgtcg gtcacttacg ttgacattgt caagcctctg
300
gccattgtgc tgtttgctgt tggcaacgtg ctggtcctta cctccatgta cgccctgggc
360
gtcaccggaa cctacctcgg cgactacttt ggcattctca tgaacgagcg agtcaccggc
420
ttccccttca atgtgtccaa caaccccatg taccacggct ccactctgtg tttcctggct
480
tccgctctgt ggtacgccaa gcccgcaggc ctgctcatca ccgcctttgt gtcgctcatg
540
tactccattg ctctcaagtt cgaggaaccc ttcaccggtg agatctacgc taagcgagac
600
cgggagctcg ctgccaagca gaagtaa
627
<210> 12
<211> 208
<212> PRT
<213> Yarrowia lipolytica
<400> 12
Met Ser Phe Pro Asp Lys Ile Val Ala Asp Phe Asn Val Asp Tyr Asp
1 5 10 15
Gln Lys Ser Leu Trp Ile Val Leu Gly Ser Ile Ala Phe Asn Pro Ile
20 25 30
Phe Trp Asn Ile Val Ala Arg Ala Glu Tyr Asn Thr His Phe Leu Thr
35 40 45
Lys Leu Ala Gly Gly Ala Lys Thr Gly Cys Tyr Leu Leu Ala Ile Thr
50 55 60
Ile Phe Ser Leu Gly Ile Phe Arg Asp Tyr Leu Tyr His Gln Ala Leu
65 70 75 80
Glu Gln Gln Pro Thr Ser Ile Leu Leu Ser Val Thr Tyr Val Asp Ile
85 90 95
Val Lys Pro Leu Ala Ile Val Leu Phe Ala Val Gly Asn Val Leu Val
100 105 110
Leu Thr Ser Met Tyr Ala Leu Gly Val Thr Gly Thr Tyr Leu Gly Asp
115 120 125
Tyr Phe Gly Ile Leu Met Asn Glu Arg Val Thr Gly Phe Pro Phe Asn
130 135 140
Val Ser Asn Asn Pro Met Tyr His Gly Ser Thr Leu Cys Phe Leu Ala
145 150 155 160
Ser Ala Leu Trp Tyr Ala Lys Pro Ala Gly Leu Leu Ile Thr Ala Phe
165 170 175
Val Ser Leu Met Tyr Ser Ile Ala Leu Lys Phe Glu Glu Pro Phe Thr
180 185 190
Gly Glu Ile Tyr Ala Lys Arg Asp Arg Glu Leu Ala Ala Lys Gln Lys
195 200 205
<210> 13
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> OPI3_5_For
<400> 13
gtacccgggg atcctctaga caccaacatt cgacatggac
40
<210> 14
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> OPI3_5_Rev
<400> 14
gcaatgacga gtcagacagg gagttttcca gagagccaac
40
<210> 15
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> OPI3_URA_For
<400> 15
gttggctctc tggaaaactc cctgtctgac tcgtcattgc
40
<210> 16
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> OPI3_URA_Rev
<400> 16
cagtccttaa tcaacggtgg ctggtggtat tgtgactggg
40
<210> 17
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> OPI3_5RP_For
<400> 17
cccagtcaca ataccaccag ccaccgttga ttaaggactg
40
<210> 18
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> OPI3_5RP_Rev
<400> 18
cgtaggccgt tttctgttgc gagttttcca gagagccaac
40
<210> 19
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> OPI3_3_For
<400> 19
gttggctctc tggaaaactc gcaacagaaa acggcctacg
40
<210> 20
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> OPI3_3_Rev
<400> 20
gcaggtcgac tctagagagt gccgtctcga ttgtcacagg
40
<210> 21
<211> 2103
<212> DNA
<213> Yarrowia lipolytica
<400> 21
atgaacccca acaacactgg caccattgaa atcaacggta aggagtacaa caccttcacc
60
gagccccccg tggccatggc tcaggagcga gccaagacct ccttccccgt gcgagagatg
120
acctacttcc tcgacggtgg cgagaagaac accctcaaaa acgagcagat catggaggag
180
attgagcgag accctctttt caacaacgac aactactacg atctcaacaa ggagcagatc
240
cgagagctca ccatggagcg agtcgccaag ctgtctctgt ttgtgcgtga tcagcccgag
300
gacgacatca agaagcgatt tgctctcatt ggtatcgccg atatgggaac ctacacccga
360
cttggtgtcc actacggcct cttctttggc gccgtccgag gtaccggaac tgccgagcag
420
tttggccact ggatctccaa gggagccgga gacctgcgaa agttctacgg atgtttctcc
480
atgaccgagc tgggccatgg ctccaacctg gctggtctcg agaccaccgc catctacgat
540
gaggagaccg acgagttcat catcaacacc cctcacattg ccgccaccaa gtggtggatt
600
ggaggagccg cccacaccgc cacccacact gtcgtgttcg cccgactcat tgtcaagggc
660
aaggactacg gtgtcaagac ctttgttgtc cagctgcgaa acatcaacga ccacagcctc
720
aaggtcggta tctctattgg tgatatcgga aagaagatgg gccgagacgg tatcgataac
780
ggatggatcc agttcaccaa cgtgcgaatc ccccgacaga acctgctcat gaagtacaca
840
aaggtcgacc gagagggtaa cgtgacccag cctcctctgg ctcagcttac ctacggttct
900
cttatcactg gtcgagtctc catggcctct gattctcacc aggtcggaaa gcgattcatc
960
accattgctc tgcgatacgc ctgcattcga cgacagttct ccaccacccc cggccagccc
1020
gagaccaaga tcatcgacta cccctaccat cagcgacgac ttctgcctct tctggcctat
1080
gtctatgctc ttaagatgac tgccgatgag gttggagctc tcttctcccg aaccatgctt
1140
aagatggacg acctcaagcc cgacgacaag gccggcctca atgaggttgt ttccgacgtc
1200
aaggagctct tctccgtctc cgccggtctc aaggccttct ccacctgggc ttgtgccgac
1260
gtcattgaca agacccgaca ggcttgcggt ggccacggtt actctggata caacggtttc
1320
ggccaggcct acgccgactg ggttgtccag tgcacctggg agggtgacaa caacattctc
1380
accctttctg ccggccgagc tcttatccag tctgccgttg ctctgcgaaa gggcgagcct
1440
gttggtaacg ccgtttctta cctgaagcga tacaaggatc tggccaacgc taagctcaat
1500
ggccgatctc tcaccgaccc caaggtcctc gtcgaggcct gggaggttgc tgccggtaac
1560
atcatcaacc gagccaccga ccagtacgag aagctcattg gcgagggtct taacgccgac
1620
caggcctttg aggttctgtc tcagcagcga ttccaggccg ccaaggtcca cacacgacga
1680
cacctcattg ccgctttctt ctcccgaatt gacaccgagg ctggcgaggc catcaagcag
1740
cccctgctta acctggctct gctgtttgcc ctgtggtcca tcgaagagga ctctggtctg
1800
ttcctgcgag agggcttcct cgagcccaag gatatcgaca ccgtcaccga gctcgtcaac
1860
aagtactgca ccactgtgcg agaggaggtc attggctaca ccgatgcctt caacctgtcc
1920
gactacttca tcaacgctcc tattggatgc tacgatggtg acgcttaccg acactacttc
1980
cagaaggtca acgagcagaa ccctgcccga gacccccgac ctccttacta cgcctctact
2040
ctcaagccct tccttttccg agaggaggag gatgatgaca tttgcgagct tgatgaggaa
2100
tag
2103
<210> 22
<211> 700
<212> PRT
<213> Yarrowia lipolytica
<400> 22
Met Asn Pro Asn Asn Thr Gly Thr Ile Glu Ile Asn Gly Lys Glu Tyr
1 5 10 15
Asn Thr Phe Thr Glu Pro Pro Val Ala Met Ala Gln Glu Arg Ala Lys
20 25 30
Thr Ser Phe Pro Val Arg Glu Met Thr Tyr Phe Leu Asp Gly Gly Glu
35 40 45
Lys Asn Thr Leu Lys Asn Glu Gln Ile Met Glu Glu Ile Glu Arg Asp
50 55 60
Pro Leu Phe Asn Asn Asp Asn Tyr Tyr Asp Leu Asn Lys Glu Gln Ile
65 70 75 80
Arg Glu Leu Thr Met Glu Arg Val Ala Lys Leu Ser Leu Phe Val Arg
85 90 95
Asp Gln Pro Glu Asp Asp Ile Lys Lys Arg Phe Ala Leu Ile Gly Ile
100 105 110
Ala Asp Met Gly Thr Tyr Thr Arg Leu Gly Val His Tyr Gly Leu Phe
115 120 125
Phe Gly Ala Val Arg Gly Thr Gly Thr Ala Glu Gln Phe Gly His Trp
130 135 140
Ile Ser Lys Gly Ala Gly Asp Leu Arg Lys Phe Tyr Gly Cys Phe Ser
145 150 155 160
Met Thr Glu Leu Gly His Gly Ser Asn Leu Ala Gly Leu Glu Thr Thr
165 170 175
Ala Ile Tyr Asp Glu Glu Thr Asp Glu Phe Ile Ile Asn Thr Pro His
180 185 190
Ile Ala Ala Thr Lys Trp Trp Ile Gly Gly Ala Ala His Thr Ala Thr
195 200 205
His Thr Val Val Phe Ala Arg Leu Ile Val Lys Gly Lys Asp Tyr Gly
210 215 220
Val Lys Thr Phe Val Val Gln Leu Arg Asn Ile Asn Asp His Ser Leu
225 230 235 240
Lys Val Gly Ile Ser Ile Gly Asp Ile Gly Lys Lys Met Gly Arg Asp
245 250 255
Gly Ile Asp Asn Gly Trp Ile Gln Phe Thr Asn Val Arg Ile Pro Arg
260 265 270
Gln Asn Leu Leu Met Lys Tyr Thr Lys Val Asp Arg Glu Gly Asn Val
275 280 285
Thr Gln Pro Pro Leu Ala Gln Leu Thr Tyr Gly Ser Leu Ile Thr Gly
290 295 300
Arg Val Ser Met Ala Ser Asp Ser His Gln Val Gly Lys Arg Phe Ile
305 310 315 320
Thr Ile Ala Leu Arg Tyr Ala Cys Ile Arg Arg Gln Phe Ser Thr Thr
325 330 335
Pro Gly Gln Pro Glu Thr Lys Ile Ile Asp Tyr Pro Tyr His Gln Arg
340 345 350
Arg Leu Leu Pro Leu Leu Ala Tyr Val Tyr Ala Leu Lys Met Thr Ala
355 360 365
Asp Glu Val Gly Ala Leu Phe Ser Arg Thr Met Leu Lys Met Asp Asp
370 375 380
Leu Lys Pro Asp Asp Lys Ala Gly Leu Asn Glu Val Val Ser Asp Val
385 390 395 400
Lys Glu Leu Phe Ser Val Ser Ala Gly Leu Lys Ala Phe Ser Thr Trp
405 410 415
Ala Cys Ala Asp Val Ile Asp Lys Thr Arg Gln Ala Cys Gly Gly His
420 425 430
Gly Tyr Ser Gly Tyr Asn Gly Phe Gly Gln Ala Tyr Ala Asp Trp Val
435 440 445
Val Gln Cys Thr Trp Glu Gly Asp Asn Asn Ile Leu Thr Leu Ser Ala
450 455 460
Gly Arg Ala Leu Ile Gln Ser Ala Val Ala Leu Arg Lys Gly Glu Pro
465 470 475 480
Val Gly Asn Ala Val Ser Tyr Leu Lys Arg Tyr Lys Asp Leu Ala Asn
485 490 495
Ala Lys Leu Asn Gly Arg Ser Leu Thr Asp Pro Lys Val Leu Val Glu
500 505 510
Ala Trp Glu Val Ala Ala Gly Asn Ile Ile Asn Arg Ala Thr Asp Gln
515 520 525
Tyr Glu Lys Leu Ile Gly Glu Gly Leu Asn Ala Asp Gln Ala Phe Glu
530 535 540
Val Leu Ser Gln Gln Arg Phe Gln Ala Ala Lys Val His Thr Arg Arg
545 550 555 560
His Leu Ile Ala Ala Phe Phe Ser Arg Ile Asp Thr Glu Ala Gly Glu
565 570 575
Ala Ile Lys Gln Pro Leu Leu Asn Leu Ala Leu Leu Phe Ala Leu Trp
580 585 590
Ser Ile Glu Glu Asp Ser Gly Leu Phe Leu Arg Glu Gly Phe Leu Glu
595 600 605
Pro Lys Asp Ile Asp Thr Val Thr Glu Leu Val Asn Lys Tyr Cys Thr
610 615 620
Thr Val Arg Glu Glu Val Ile Gly Tyr Thr Asp Ala Phe Asn Leu Ser
625 630 635 640
Asp Tyr Phe Ile Asn Ala Pro Ile Gly Cys Tyr Asp Gly Asp Ala Tyr
645 650 655
Arg His Tyr Phe Gln Lys Val Asn Glu Gln Asn Pro Ala Arg Asp Pro
660 665 670
Arg Pro Pro Tyr Tyr Ala Ser Thr Leu Lys Pro Phe Leu Phe Arg Glu
675 680 685
Glu Glu Asp Asp Asp Ile Cys Glu Leu Asp Glu Glu
690 695 700
<210> 23
<211> 2706
<212> DNA
<213> Yarrowia lipolytica
<400> 23
atgtctggag aactaagata cgacggaaag gtcgtcattg ttaccggtgc cggtggcggt
60
ctcggtaagg catacgccct tttctacggc tctcgaggag cctctgttgt tgtcaacgat
120
cttggtggcg acttcaaggg cgacggtgcc caggctggca gtggcaagcg agttgccgat
180
gttgtcgtcg acgagattgt ttccaaggga ggcaaggctg ttgctaacta cgactctgtc
240
gagaacggtg acaagattgt cgagactgcc gtcaaggctt ttggctccgt ccacattgtc
300
atcaacaacg ccggtattct ccgagatatt tccttcaaga agatgaccga caaggactgg
360
gatcttgtct acaaggtcca cgttttcggt gcctacaagg ttacccgagc tgcctggcct
420
tacttccgaa agcagaagta cggtcgagtt atctctacct cttccgctgc tggtctttac
480
ggaaacttcg gccagaccaa ctactccgct gccaagctcg ccctggttgg tttcggtgag
540
actctcgcca aggagggtgc caagtacaac attacttcca acgtcatcgc tcctcttgct
600
gcttcccgaa tgaccgagac agtcatgccc gaggatatcc tcaagctcct caagcctgag
660
tacgttgttc ctctggtcgg ctacctcacc cacgactctg tcaccgagtc ttatggtatt
720
tacgaggtcg gtgctggtta catggctaaa atccgatggg agcgaggcaa cggtgctgtt
780
ttcaagggcg acgacacttt caccccgtct gctattctga agcgatggga tgaggtcacc
840
tcttttgaga gccccaccta ccctaacggc cctgctgact tcttcaaata cgctgaggag
900
tctgttaagc gacccgagaa cccccaggga cccaccgtct ccttcaagga ccaggttgtc
960
attgtcactg gagccggtgc tggcattggc cgagcttact ctcacctcct tgctaagctt
1020
ggtgccaagg tcgttgttaa cgatttcggt aaccctcaga aggttgtcga tgaaattaag
1080
gccctcggtg gtatcgccgt cgctgacaag aacaacgtca tccacggtga gaaggttgtt
1140
cagaccgcta tcgacgcctt cggtgctgtc cacgccgttg tcaacaacgc tggtattctc
1200
cgagacaagt ctttcgccaa catggatgat gagatgtggc agctgatctt tgatgtccac
1260
ctcaacggta cttactccgt taccaaggcc gcgtggcccc acttccttaa gcagaagtac
1320
ggccgtgtca tcaacaccac ctcaacttct ggtatctacg gtaacttcgg ccaggccaac
1380
tactctgccg ccaaggctgg tatcctcggt ttctcccgag ctcttgctcg agagggtgag
1440
aagtacaaca ttcttgtcaa caccattgcc cctaacgctg gtactgccat gactgcttct
1500
gtcttcactg aggagatgct cgagctcttc aagcccgatt tcatcgcacc catcaccgtc
1560
ctgcttgctt ccgatcaggc tcccgtcacc ggtgatctgt ttgagactgg ttctgcttgg
1620
atcggacaga ctcgatggca gcgagctggt ggtaaggcct tcaacaccaa gaagggtgtc
1680
acccccgaaa tggttcgaga cagctgggct aagatcgtcg acttcgatga tggtaactcc
1740
acccatccca ccactccctc cgagtctact actcagattc ttgagaacat cttcaacgtg
1800
cctgatgagg aggttgagga gactgctctc gttgctggtc ccggtggtcc cggtatcctc
1860
aacaaggagg gcgaaccttt cgactacact tacacttacc gagacctcat tctttacaac
1920
cttggtctcg gtgccaaggc taatgagctc aagtatgtct tcgagggtga tgatgacttc
1980
cagaccgtgc ccactttcgg tgttatccct tacatgggtg gcctcatcac taccaactat
2040
ggcgacttcg ttcctaactt caaccctatg atgcttctcc acggtgagca gtaccttgaa
2100
atccgacagt ggcctattcc taccaatgct acattggaga acaaggctaa ggtcatcgat
2160
gtcgttgaca agggcaaggc tgccctcctt gtcactgcta ccaccaccac gaacaaggag
2220
actggtgagg aggttttcta caacgagtct tctctcttca tccgaggctc tggtggtttc
2280
ggtggtaagt ctaccggtac tgaccgtggc gctgccactg ctgccaacaa gccccctgct
2340
cgagctcctg acttcgttaa ggagatcaag atccaggagg accaggctgc catttaccga
2400
ctttctggtg attacaaccc tcttcacatc gaccctgctt ttgctgctgt tggtaacttt
2460
gaccgaccta ttctccacgg tctctgctct tttggtgtct ccggtaaggc tctttacgat
2520
cagtttggtc ctttcaagaa cgctaaggtc cgatttgctg gtcacgtctt ccctggtgag
2580
accctgaagg ttgagggctg gaaggagggc aacaaggtca ttttccagac caaggttgtt
2640
gagcgaggta ctaccgccat cagcaatgcc gccattgagc tcttccccaa ggatgctaag
2700
ctctaa
2706
<210> 24
<211> 901
<212> PRT
<213> Yarrowia lipolytica
<400> 24
Met Ser Gly Glu Leu Arg Tyr Asp Gly Lys Val Val Ile Val Thr Gly
1 5 10 15
Ala Gly Gly Gly Leu Gly Lys Ala Tyr Ala Leu Phe Tyr Gly Ser Arg
20 25 30
Gly Ala Ser Val Val Val Asn Asp Leu Gly Gly Asp Phe Lys Gly Asp
35 40 45
Gly Ala Gln Ala Gly Ser Gly Lys Arg Val Ala Asp Val Val Val Asp
50 55 60
Glu Ile Val Ser Lys Gly Gly Lys Ala Val Ala Asn Tyr Asp Ser Val
65 70 75 80
Glu Asn Gly Asp Lys Ile Val Glu Thr Ala Val Lys Ala Phe Gly Ser
85 90 95
Val His Ile Val Ile Asn Asn Ala Gly Ile Leu Arg Asp Ile Ser Phe
100 105 110
Lys Lys Met Thr Asp Lys Asp Trp Asp Leu Val Tyr Lys Val His Val
115 120 125
Phe Gly Ala Tyr Lys Val Thr Arg Ala Ala Trp Pro Tyr Phe Arg Lys
130 135 140
Gln Lys Tyr Gly Arg Val Ile Ser Thr Ser Ser Ala Ala Gly Leu Tyr
145 150 155 160
Gly Asn Phe Gly Gln Thr Asn Tyr Ser Ala Ala Lys Leu Ala Leu Val
165 170 175
Gly Phe Gly Glu Thr Leu Ala Lys Glu Gly Ala Lys Tyr Asn Ile Thr
180 185 190
Ser Asn Val Ile Ala Pro Leu Ala Ala Ser Arg Met Thr Glu Thr Val
195 200 205
Met Pro Glu Asp Ile Leu Lys Leu Leu Lys Pro Glu Tyr Val Val Pro
210 215 220
Leu Val Gly Tyr Leu Thr His Asp Ser Val Thr Glu Ser Tyr Gly Ile
225 230 235 240
Tyr Glu Val Gly Ala Gly Tyr Met Ala Lys Ile Arg Trp Glu Arg Gly
245 250 255
Asn Gly Ala Val Phe Lys Gly Asp Asp Thr Phe Thr Pro Ser Ala Ile
260 265 270
Leu Lys Arg Trp Asp Glu Val Thr Ser Phe Glu Ser Pro Thr Tyr Pro
275 280 285
Asn Gly Pro Ala Asp Phe Phe Lys Tyr Ala Glu Glu Ser Val Lys Arg
290 295 300
Pro Glu Asn Pro Gln Gly Pro Thr Val Ser Phe Lys Asp Gln Val Val
305 310 315 320
Ile Val Thr Gly Ala Gly Ala Gly Ile Gly Arg Ala Tyr Ser His Leu
325 330 335
Leu Ala Lys Leu Gly Ala Lys Val Val Val Asn Asp Phe Gly Asn Pro
340 345 350
Gln Lys Val Val Asp Glu Ile Lys Ala Leu Gly Gly Ile Ala Val Ala
355 360 365
Asp Lys Asn Asn Val Ile His Gly Glu Lys Val Val Gln Thr Ala Ile
370 375 380
Asp Ala Phe Gly Ala Val His Ala Val Val Asn Asn Ala Gly Ile Leu
385 390 395 400
Arg Asp Lys Ser Phe Ala Asn Met Asp Asp Glu Met Trp Gln Leu Ile
405 410 415
Phe Asp Val His Leu Asn Gly Thr Tyr Ser Val Thr Lys Ala Ala Trp
420 425 430
Pro His Phe Leu Lys Gln Lys Tyr Gly Arg Val Ile Asn Thr Thr Ser
435 440 445
Thr Ser Gly Ile Tyr Gly Asn Phe Gly Gln Ala Asn Tyr Ser Ala Ala
450 455 460
Lys Ala Gly Ile Leu Gly Phe Ser Arg Ala Leu Ala Arg Glu Gly Glu
465 470 475 480
Lys Tyr Asn Ile Leu Val Asn Thr Ile Ala Pro Asn Ala Gly Thr Ala
485 490 495
Met Thr Ala Ser Val Phe Thr Glu Glu Met Leu Glu Leu Phe Lys Pro
500 505 510
Asp Phe Ile Ala Pro Ile Thr Val Leu Leu Ala Ser Asp Gln Ala Pro
515 520 525
Val Thr Gly Asp Leu Phe Glu Thr Gly Ser Ala Trp Ile Gly Gln Thr
530 535 540
Arg Trp Gln Arg Ala Gly Gly Lys Ala Phe Asn Thr Lys Lys Gly Val
545 550 555 560
Thr Pro Glu Met Val Arg Asp Ser Trp Ala Lys Ile Val Asp Phe Asp
565 570 575
Asp Gly Asn Ser Thr His Pro Thr Thr Pro Ser Glu Ser Thr Thr Gln
580 585 590
Ile Leu Glu Asn Ile Phe Asn Val Pro Asp Glu Glu Val Glu Glu Thr
595 600 605
Ala Leu Val Ala Gly Pro Gly Gly Pro Gly Ile Leu Asn Lys Glu Gly
610 615 620
Glu Pro Phe Asp Tyr Thr Tyr Thr Tyr Arg Asp Leu Ile Leu Tyr Asn
625 630 635 640
Leu Gly Leu Gly Ala Lys Ala Asn Glu Leu Lys Tyr Val Phe Glu Gly
645 650 655
Asp Asp Asp Phe Gln Thr Val Pro Thr Phe Gly Val Ile Pro Tyr Met
660 665 670
Gly Gly Leu Ile Thr Thr Asn Tyr Gly Asp Phe Val Pro Asn Phe Asn
675 680 685
Pro Met Met Leu Leu His Gly Glu Gln Tyr Leu Glu Ile Arg Gln Trp
690 695 700
Pro Ile Pro Thr Asn Ala Thr Leu Glu Asn Lys Ala Lys Val Ile Asp
705 710 715 720
Val Val Asp Lys Gly Lys Ala Ala Leu Leu Val Thr Ala Thr Thr Thr
725 730 735
Thr Asn Lys Glu Thr Gly Glu Glu Val Phe Tyr Asn Glu Ser Ser Leu
740 745 750
Phe Ile Arg Gly Ser Gly Gly Phe Gly Gly Lys Ser Thr Gly Thr Asp
755 760 765
Arg Gly Ala Ala Thr Ala Ala Asn Lys Pro Pro Ala Arg Ala Pro Asp
770 775 780
Phe Val Lys Glu Ile Lys Ile Gln Glu Asp Gln Ala Ala Ile Tyr Arg
785 790 795 800
Leu Ser Gly Asp Tyr Asn Pro Leu His Ile Asp Pro Ala Phe Ala Ala
805 810 815
Val Gly Asn Phe Asp Arg Pro Ile Leu His Gly Leu Cys Ser Phe Gly
820 825 830
Val Ser Gly Lys Ala Leu Tyr Asp Gln Phe Gly Pro Phe Lys Asn Ala
835 840 845
Lys Val Arg Phe Ala Gly His Val Phe Pro Gly Glu Thr Leu Lys Val
850 855 860
Glu Gly Trp Lys Glu Gly Asn Lys Val Ile Phe Gln Thr Lys Val Val
865 870 875 880
Glu Arg Gly Thr Thr Ala Ile Ser Asn Ala Ala Ile Glu Leu Phe Pro
885 890 895
Lys Asp Ala Lys Leu
900
<210> 25
<211> 1134
<212> DNA
<213> Yarrowia lipolytica
<400> 25
atgtggggaa gttcacatgc attcgctggt gaatctgatc tgacactaca actacacacc
60
aggtccaaca tgagcgacaa tacgacaatc aaaaagccga tccgacccaa accgatccgg
120
acggaacgcc tgccttacgc tggggccgca gaaatcatcc gagccaacca gaaagaccac
180
tactttgagt ccgtgcttga acagcatctc gtcacgtttc tgcagaaatg gaagggagta
240
cgatttatcc accagtacaa ggaggagctg gagacggcgt ccaagtttgc atatctcggt
300
ttgtgtacgc ttgtgggctc caagactctc ggagaagagt acaccaatct catgtacact
360
atcagagacc gaacagctct accgggggtg gtgagacggt ttggctacgt gctttccaac
420
actctgtttc catacctgtt tgtgcgctac atgggcaagt tgcgcgccaa actgatgcgc
480
gagtatcccc atctggtgga gtacgacgaa gatgagcctg tgcccagccc ggaaacatgg
540
aaggagcggg tcatcaagac gtttgtgaac aagtttgaca agttcacggc gctggagggg
600
tttaccgcga tccacttggc gattttctac gtctacggct cgtactacca gctcagtaag
660
cggatctggg gcatgcgtta tgtatttgga caccgactgg acaagaatga gcctcgaatc
720
ggttacgaga tgctcggtct gctgattttc gcccggtttg ccacgtcatt tgtgcagacg
780
ggaagagagt acctcggagc gctgctggaa aagagcgtgg agaaagaggc aggggagaag
840
gaagatgaaa aggaagcggt tgtgccgaaa aagaagtcgt caattccgtt cattgaggat
900
acagaagggg agacggaaga caagatcgat ctggaggacc ctcgacagct caagttcatt
960
cctgaggcgt ccagagcgtg cactctgtgt ctgtcataca ttagtgcgcc ggcatgtacg
1020
ccatgtggac actttttctg ttgggactgt atttccgaat gggtgagaga gaagcccgag
1080
tgtcccttgt gtcggcaggg tgtgagagag cagaacttgt tgcctatcag ataa
1134
<210> 26
<211> 377
<212> PRT
<213> Yarrowia lipolytica
<400> 26
Met Trp Gly Ser Ser His Ala Phe Ala Gly Glu Ser Asp Leu Thr Leu
1 5 10 15
Gln Leu His Thr Arg Ser Asn Met Ser Asp Asn Thr Thr Ile Lys Lys
20 25 30
Pro Ile Arg Pro Lys Pro Ile Arg Thr Glu Arg Leu Pro Tyr Ala Gly
35 40 45
Ala Ala Glu Ile Ile Arg Ala Asn Gln Lys Asp His Tyr Phe Glu Ser
50 55 60
Val Leu Glu Gln His Leu Val Thr Phe Leu Gln Lys Trp Lys Gly Val
65 70 75 80
Arg Phe Ile His Gln Tyr Lys Glu Glu Leu Glu Thr Ala Ser Lys Phe
85 90 95
Ala Tyr Leu Gly Leu Cys Thr Leu Val Gly Ser Lys Thr Leu Gly Glu
100 105 110
Glu Tyr Thr Asn Leu Met Tyr Thr Ile Arg Asp Arg Thr Ala Leu Pro
115 120 125
Gly Val Val Arg Arg Phe Gly Tyr Val Leu Ser Asn Thr Leu Phe Pro
130 135 140
Tyr Leu Phe Val Arg Tyr Met Gly Lys Leu Arg Ala Lys Leu Met Arg
145 150 155 160
Glu Tyr Pro His Leu Val Glu Tyr Asp Glu Asp Glu Pro Val Pro Ser
165 170 175
Pro Glu Thr Trp Lys Glu Arg Val Ile Lys Thr Phe Val Asn Lys Phe
180 185 190
Asp Lys Phe Thr Ala Leu Glu Gly Phe Thr Ala Ile His Leu Ala Ile
195 200 205
Phe Tyr Val Tyr Gly Ser Tyr Tyr Gln Leu Ser Lys Arg Ile Trp Gly
210 215 220
Met Arg Tyr Val Phe Gly His Arg Leu Asp Lys Asn Glu Pro Arg Ile
225 230 235 240
Gly Tyr Glu Met Leu Gly Leu Leu Ile Phe Ala Arg Phe Ala Thr Ser
245 250 255
Phe Val Gln Thr Gly Arg Glu Tyr Leu Gly Ala Leu Leu Glu Lys Ser
260 265 270
Val Glu Lys Glu Ala Gly Glu Lys Glu Asp Glu Lys Glu Ala Val Val
275 280 285
Pro Lys Lys Lys Ser Ser Ile Pro Phe Ile Glu Asp Thr Glu Gly Glu
290 295 300
Thr Glu Asp Lys Ile Asp Leu Glu Asp Pro Arg Gln Leu Lys Phe Ile
305 310 315 320
Pro Glu Ala Ser Arg Ala Cys Thr Leu Cys Leu Ser Tyr Ile Ser Ala
325 330 335
Pro Ala Cys Thr Pro Cys Gly His Phe Phe Cys Trp Asp Cys Ile Ser
340 345 350
Glu Trp Val Arg Glu Lys Pro Glu Cys Pro Leu Cys Arg Gln Gly Val
355 360 365
Arg Glu Gln Asn Leu Leu Pro Ile Arg
370 375
<210> 27
<211> 1245
<212> DNA
<213> Yarrowia lipolytica
<400> 27
atggaccgac ttaacaacct cgccacccag ctcgagcaga accccgccaa gggcctcgac
60
gctatcacct ccaagaaccc cgatgacgtt gtcatcaccg ccgcctaccg aactgcccac
120
accaagggag gcaagggtct gttcaaggac acctcttctt ccgagctgct cgcctctctg
180
ctggagggcc tcgtcaagga gtccaagatc gaccccaagc tcatcggtga tgtcgtctgc
240
gggaacgttc tcgctgccgg tgccggtgcc actgagcacc gagctgcctg ccttgttgcc
300
ggcatccccg agaccgttcc cttcgtcgct ctcaaccgac agtgctcctc tggtctgatg
360
gccgtcaacg acgttgccaa caagatccga gccggccaga ttgacattgg tatcggctgt
420
ggtgtcgagt ccatgtccaa ccagtacggt cccaactccg tcaccccctt ctccaacaag
480
ttccagaaca acgaggaggc taagaagtgc ctgatcccca tgggtatcac ttccgagaac
540
gttgccgcca agtacaacgt gtcccgaaag gcccaggacg cctttgctgc caagtcctac
600
gagaaggccg ccgctgccca ggccgccggc aagttcgacc aggagatcct ccccatcaag
660
accactgttc tcgatgatga tgacaacgag aaggaggtta ccgtcaacaa ggacgacggt
720
atccgacctg gtgtcaccgc cgagaagctc ggcaagctca agcctgcttt ctccgccgag
780
ggaaccaccc acgctggtaa cgcctctcag atctccgacg gtgccggagc cgttctcctc
840
atgcgacgat ctgttgccga gaagcttggc cagcccatcc ttgccaagtt tgtccactgc
900
aagaccgtcg gtgttccccc cgagctcatg ggaattggcc ccgcttacgc cattcctgct
960
gtccttgagg accttggtct gaccgtcaac gacgttgacg ttttcgagat caacgaggct
1020
ttcgcttccc aggctctgtt ctccatccag cattgtggaa tcgacgagtc caaggtcaac
1080
ccccgaggtg gtgccattgc tattggccac cctctgggag ccaccggtgc tcgacagttt
1140
gccactctgc tctccgagct taaggagtct ggcaagaagg tcggtgtcac ctccatgtgc
1200
attggtaccg gtatgggtgc cgcttctctg gttgttgccg agtaa
1245
<210> 28
<211> 414
<212> PRT
<213> Yarrowia lipolytica
<400> 28
Met Asp Arg Leu Asn Asn Leu Ala Thr Gln Leu Glu Gln Asn Pro Ala
1 5 10 15
Lys Gly Leu Asp Ala Ile Thr Ser Lys Asn Pro Asp Asp Val Val Ile
20 25 30
Thr Ala Ala Tyr Arg Thr Ala His Thr Lys Gly Gly Lys Gly Leu Phe
35 40 45
Lys Asp Thr Ser Ser Ser Glu Leu Leu Ala Ser Leu Leu Glu Gly Leu
50 55 60
Val Lys Glu Ser Lys Ile Asp Pro Lys Leu Ile Gly Asp Val Val Cys
65 70 75 80
Gly Asn Val Leu Ala Ala Gly Ala Gly Ala Thr Glu His Arg Ala Ala
85 90 95
Cys Leu Val Ala Gly Ile Pro Glu Thr Val Pro Phe Val Ala Leu Asn
100 105 110
Arg Gln Cys Ser Ser Gly Leu Met Ala Val Asn Asp Val Ala Asn Lys
115 120 125
Ile Arg Ala Gly Gln Ile Asp Ile Gly Ile Gly Cys Gly Val Glu Ser
130 135 140
Met Ser Asn Gln Tyr Gly Pro Asn Ser Val Thr Pro Phe Ser Asn Lys
145 150 155 160
Phe Gln Asn Asn Glu Glu Ala Lys Lys Cys Leu Ile Pro Met Gly Ile
165 170 175
Thr Ser Glu Asn Val Ala Ala Lys Tyr Asn Val Ser Arg Lys Ala Gln
180 185 190
Asp Ala Phe Ala Ala Lys Ser Tyr Glu Lys Ala Ala Ala Ala Gln Ala
195 200 205
Ala Gly Lys Phe Asp Gln Glu Ile Leu Pro Ile Lys Thr Thr Val Leu
210 215 220
Asp Asp Asp Asp Asn Glu Lys Glu Val Thr Val Asn Lys Asp Asp Gly
225 230 235 240
Ile Arg Pro Gly Val Thr Ala Glu Lys Leu Gly Lys Leu Lys Pro Ala
245 250 255
Phe Ser Ala Glu Gly Thr Thr His Ala Gly Asn Ala Ser Gln Ile Ser
260 265 270
Asp Gly Ala Gly Ala Val Leu Leu Met Arg Arg Ser Val Ala Glu Lys
275 280 285
Leu Gly Gln Pro Ile Leu Ala Lys Phe Val His Cys Lys Thr Val Gly
290 295 300
Val Pro Pro Glu Leu Met Gly Ile Gly Pro Ala Tyr Ala Ile Pro Ala
305 310 315 320
Val Leu Glu Asp Leu Gly Leu Thr Val Asn Asp Val Asp Val Phe Glu
325 330 335
Ile Asn Glu Ala Phe Ala Ser Gln Ala Leu Phe Ser Ile Gln His Cys
340 345 350
Gly Ile Asp Glu Ser Lys Val Asn Pro Arg Gly Gly Ala Ile Ala Ile
355 360 365
Gly His Pro Leu Gly Ala Thr Gly Ala Arg Gln Phe Ala Thr Leu Leu
370 375 380
Ser Glu Leu Lys Glu Ser Gly Lys Lys Val Gly Val Thr Ser Met Cys
385 390 395 400
Ile Gly Thr Gly Met Gly Ala Ala Ser Leu Val Val Ala Glu
405 410
<210> 29
<211> 858
<212> DNA
<213> Yarrowia lipolytica
<400> 29
atggacctcg aattggaaat tcccgtcttg cattccatgg actcgcacca ccaggtggtg
60
gactcccaca gactggcaca gcaacagttc cagtaccagc agatccacat gctgcagcag
120
acgctgtcac agcagtaccc ccacacccca tccaccacac cccccattta catgctgtcg
180
cctgcggact acgagaagga cgccgtttcc atctcaccgg taatgctgtg gcccccctcg
240
gcccactccc aggcctctta ccattacgag atgccctccg ttatctcgcc atctccttct
300
cccactagat ccttctgtaa tccgagagag ctggaggttc aggacgagct cgagcagctt
360
gaacagcagc ccgccgctct ctccgtcgaa catctgtttg acattgagaa ctcatcgatc
420
gagtatgcac acgacgagct gcatgacacc tcttcgtgct ccgactcgca gtcgagcttt
480
tcccctcagc agtcccctgc ctccccggcc tccacttact cgcctctcga ggacgagttt
540
ctcaacttgg ctggatccga gttgaagagc gagcccagcg cggacgacga gaaggatgat
600
gtggacacgg agcttcccca gcagcccgag atcatcatcc ctgtgtcgtg ccgaggccga
660
aagccgtcca tcgacgactc caaaaagact tttgtctgca cccactgcca gcgtcggttc
720
cggcgccagg agcatctcaa gcgacatttc cgatccctac acactcgaga gaagcctttc
780
aactgcgaca cgtgcggcaa gaagttttct cggtcggaca atctcgccca gcatatgcgt
840
acgcatcctc gggactag
858
<210> 30
<211> 285
<212> PRT
<213> Yarrowia lipolytica
<400> 30
Met Asp Leu Glu Leu Glu Ile Pro Val Leu His Ser Met Asp Ser His
1 5 10 15
His Gln Val Val Asp Ser His Arg Leu Ala Gln Gln Gln Phe Gln Tyr
20 25 30
Gln Gln Ile His Met Leu Gln Gln Thr Leu Ser Gln Gln Tyr Pro His
35 40 45
Thr Pro Ser Thr Thr Pro Pro Ile Tyr Met Leu Ser Pro Ala Asp Tyr
50 55 60
Glu Lys Asp Ala Val Ser Ile Ser Pro Val Met Leu Trp Pro Pro Ser
65 70 75 80
Ala His Ser Gln Ala Ser Tyr His Tyr Glu Met Pro Ser Val Ile Ser
85 90 95
Pro Ser Pro Ser Pro Thr Arg Ser Phe Cys Asn Pro Arg Glu Leu Glu
100 105 110
Val Gln Asp Glu Leu Glu Gln Leu Glu Gln Gln Pro Ala Ala Leu Ser
115 120 125
Val Glu His Leu Phe Asp Ile Glu Asn Ser Ser Ile Glu Tyr Ala His
130 135 140
Asp Glu Leu His Asp Thr Ser Ser Cys Ser Asp Ser Gln Ser Ser Phe
145 150 155 160
Ser Pro Gln Gln Ser Pro Ala Ser Pro Ala Ser Thr Tyr Ser Pro Leu
165 170 175
Glu Asp Glu Phe Leu Asn Leu Ala Gly Ser Glu Leu Lys Ser Glu Pro
180 185 190
Ser Ala Asp Asp Glu Lys Asp Asp Val Asp Thr Glu Leu Pro Gln Gln
195 200 205
Pro Glu Ile Ile Ile Pro Val Ser Cys Arg Gly Arg Lys Pro Ser Ile
210 215 220
Asp Asp Ser Lys Lys Thr Phe Val Cys Thr His Cys Gln Arg Arg Phe
225 230 235 240
Arg Arg Gln Glu His Leu Lys Arg His Phe Arg Ser Leu His Thr Arg
245 250 255
Glu Lys Pro Phe Asn Cys Asp Thr Cys Gly Lys Lys Phe Ser Arg Ser
260 265 270
Asp Asn Leu Ala Gln His Met Arg Thr His Pro Arg Asp
275 280 285
<210> 31
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POX2_5_For
<400> 31
gtacccgggg atcctctaga gattccgcca agtgagactg
40
<210> 32
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POX2_5_Rev
<400> 32
gcaatgacga gtcagacagg cgttgcttgt gtgatttttg
40
<210> 33
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POX2_URA_For
<400> 33
caaaaatcac acaagcaacg cctgtctgac tcgtcattgc
40
<210> 34
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POX2_URA_Rev
<400> 34
cgcttgtcca gtatgaatag ctggtggtat tgtgactggg
40
<210> 35
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POX2_5RP_For
<400> 35
cccagtcaca ataccaccag ctattcatac tggacaagcg
40
<210> 36
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> POX2_5RP_Rev
<400> 36
caataaatac ccgcttgtcc gttgcttgtg tgatttttg
39
<210> 37
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> POX2_3_For
<400> 37
caaaaatcac acaagcaacg gacaagcggg tatttattg
39
<210> 38
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> POX2_3_Rev
<400> 38
gcaggtcgac tctagaccaa acaaagctga tgacac
36
<210> 39
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MFE1_5_For
<400> 39
gtacccgggg atcctctaga caagccaaag agatgttgac
40
<210> 40
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MFE1_5_Rev
<400> 40
gcaatgacga gtcagacagg gttaataacg tatcttctac
40
<210> 41
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MFE1_URA_For
<400> 41
gtagaagata cgttattaac cctgtctgac tcgtcattgc
40
<210> 42
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MFE1_URA_Rev
<400> 42
caaatcagcc gttggcctgc ctggtggtat tgtgactggg
40
<210> 43
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MFE1_5RP_For
<400> 43
cccagtcaca ataccaccag gcaggccaac ggctgatttg
40
<210> 44
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> MFE1_5RP_Rev
<400> 44
cacttggtca gataatagcg ttaataacgt atcttctac
39
<210> 45
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> MFE1_3_For
<400> 45
gtagaagata cgttattaac gctattatct gaccaagtg
39
<210> 46
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> MFE1_3_Rev
<400> 46
gcaggtcgac tctagatgct tgaaggcata tgtgactg
38
<210> 47
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> PEX10_5_For
<400> 47
gtacccgggg atcctctaga gttttcgtct tagcgtcatg
40
<210> 48
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> PEX10_5_Rev
<400> 48
gcaatgacga gtcagacagg gccgaggcag atttgggttg
40
<210> 49
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> PEX10_URA_For
<400> 49
caacccaaat ctgcctcggc cctgtctgac tcgtcattgc
40
<210> 50
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> PEX10_URA_Rev
<400> 50
gagtccagta attctttccg ctggtggtat tgtgactggg
40
<210> 51
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> PEX10_5RP_For
<400> 51
cccagtcaca ataccaccag cggaaagaat tactggactc
40
<210> 52
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> PEX10_5RP_Rev
<400> 52
ccttccatcc agacctcgtc gccgaggcag atttgggttg
40
<210> 53
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> PEX10_3_For
<400> 53
caacccaaat ctgcctcggc gacgaggtct ggatggaagg
40
<210> 54
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> PEX10_3_Rev
<400> 54
gcaggtcgac tctagagctg acctatacca gatcagacgc
40
<210> 55
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POT1_5_For
<400> 55
gtacccgggg atcctctaga cacaataccc cacagtgtgc
40
<210> 56
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POT1_5_Rev
<400> 56
gcaatgacga gtcagacagg gtgtgtcttg gttggatgag
40
<210> 57
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POT1_URA_For
<400> 57
ctcatccaac caagacacac cctgtctgac tcgtcattgc
40
<210> 58
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POT1_URA_Rev
<400> 58
caggcgctcc cccattggcg ctggtggtat tgtgactggg
40
<210> 59
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POT1_5RP_For
<400> 59
cccagtcaca ataccaccag cgccaatggg ggagcgcctg
40
<210> 60
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POT1_5RP_Rev
<400> 60
gttcgatcgc gattcatttc gtgtgtcttg gttggatgag
40
<210> 61
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POT1_3_For
<400> 61
ctcatccaac caagacacac gaaatgaatc gcgatcgaac
40
<210> 62
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> POT1_3_Rev
<400> 62
gcaggtcgac tctagagagg agtgctaaaa ttagccctgc
40
<210> 63
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> MHY1_5_For
<400> 63
gtacccgggg atcctctaga tgtcagcgaa agctcaaag
39
<210> 64
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MHY1_5_Rev
<400> 64
gcaatgacga gtcagacagg caattcgagg tccattttgg
40
<210> 65
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MHY1_URA_For
<400> 65
ccaaaatgga cctcgaattg cctgtctgac tcgtcattgc
40
<210> 66
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MHY1_URA_Rev
<400> 66
gtggtgcttt tgtacttgtc ctggtggtat tgtgactggg
40
<210> 67
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MHY1_5RP_For
<400> 67
cccagtcaca ataccaccag gacaagtaca aaagcaccac
40
<210> 68
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MHY1_5RP_Rev
<400> 68
gaaggcgctc tacctctagt ccaattcgag gtccattttg
40
<210> 69
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MHY1_3_For
<400> 69
caaaatggac ctcgaattgg actagaggta gagcgccttc
40
<210> 70
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> MHY1_3_Rev
<400> 70
gcaggtcgac tctagactgt gtttgattag ctcttctcag
40
<210> 71
<211> 1545
<212> DNA
<213> Yarrowia lipolytica
<400> 71
atgactatcg actcacaata ctacaagtcg cgagacaaaa acgacacggc acccaaaatc
60
gcgggaatcc gatatgcccc gctatcgaca ccattactca accgatgtga gaccttctct
120
ctggtctggc acattttcag cattcccact ttcctcacaa ttttcatgct atgctgcgca
180
attccactgc tctggccatt tgtgattgcg tatgtagtgt acgctgttaa agacgactcc
240
ccgtccaacg gaggagtggt caagcgatac tcgcctattt caagaaactt cttcatctgg
300
aagctctttg gccgctactt ccccataact ctgcacaaga cggtggatct ggagcccacg
360
cacacatact accctctgga cgtccaggag tatcacctga ttgctgagag atactggccg
420
cagaacaagt acctccgagc aatcatctcc accatcgagt actttctgcc cgccttcatg
480
aaacggtctc tttctatcaa cgagcaggag cagcctgccg agcgagatcc tctcctgtct
540
cccgtttctc ccagctctcc gggttctcaa cctgacaagt ggattaacca cgacagcaga
600
tatagccgtg gagaatcatc tggctccaac ggccacgcct cgggctccga acttaacggc
660
aacggcaaca atggcaccac taaccgacga cctttgtcgt ccgcctctgc tggctccact
720
gcatctgatt ccacgcttct taacgggtcc ctcaactcct acgccaacca gatcattggc
780
gaaaacgacc cacagctgtc gcccacaaaa ctcaagccca ctggcagaaa atacatcttc
840
ggctaccacc cccacggcat tatcggcatg ggagcctttg gtggaattgc caccgaggga
900
gctggatggt ccaagctctt tccgggcatc cctgtttctc ttatgactct caccaacaac
960
ttccgagtgc ctctctacag agagtacctc atgagtctgg gagtcgcttc tgtctccaag
1020
aagtcctgca aggccctcct caagcgaaac cagtctatct gcattgtcgt tggtggagca
1080
caggaaagtc ttctggccag acccggtgtc atggacctgg tgctactcaa gcgaaagggt
1140
tttgttcgac ttggtatgga ggtcggaaat gtcgcccttg ttcccatcat ggcctttggt
1200
gagaacgacc tctatgacca ggttagcaac gacaagtcgt ccaagctgta ccgattccag
1260
cagtttgtca agaacttcct tggattcacc cttcctttga tgcatgcccg aggcgtcttc
1320
aactacgatg tcggtcttgt cccctacagg cgacccgtca acattgtggt tggttccccc
1380
attgacttgc cttatctccc acaccccacc gacgaagaag tgtccgaata ccacgaccga
1440
tacatcgccg agctgcagcg aatctacaac gagcacaagg atgaatattt catcgattgg
1500
accgaggagg gcaaaggagc cccagagttc cgaatgattg agtaa
1545
<210> 72
<211> 514
<212> PRT
<213> Yarrowia lipolytica
<400> 72
Met Thr Ile Asp Ser Gln Tyr Tyr Lys Ser Arg Asp Lys Asn Asp Thr
1 5 10 15
Ala Pro Lys Ile Ala Gly Ile Arg Tyr Ala Pro Leu Ser Thr Pro Leu
20 25 30
Leu Asn Arg Cys Glu Thr Phe Ser Leu Val Trp His Ile Phe Ser Ile
35 40 45
Pro Thr Phe Leu Thr Ile Phe Met Leu Cys Cys Ala Ile Pro Leu Leu
50 55 60
Trp Pro Phe Val Ile Ala Tyr Val Val Tyr Ala Val Lys Asp Asp Ser
65 70 75 80
Pro Ser Asn Gly Gly Val Val Lys Arg Tyr Ser Pro Ile Ser Arg Asn
85 90 95
Phe Phe Ile Trp Lys Leu Phe Gly Arg Tyr Phe Pro Ile Thr Leu His
100 105 110
Lys Thr Val Asp Leu Glu Pro Thr His Thr Tyr Tyr Pro Leu Asp Val
115 120 125
Gln Glu Tyr His Leu Ile Ala Glu Arg Tyr Trp Pro Gln Asn Lys Tyr
130 135 140
Leu Arg Ala Ile Ile Ser Thr Ile Glu Tyr Phe Leu Pro Ala Phe Met
145 150 155 160
Lys Arg Ser Leu Ser Ile Asn Glu Gln Glu Gln Pro Ala Glu Arg Asp
165 170 175
Pro Leu Leu Ser Pro Val Ser Pro Ser Ser Pro Gly Ser Gln Pro Asp
180 185 190
Lys Trp Ile Asn His Asp Ser Arg Tyr Ser Arg Gly Glu Ser Ser Gly
195 200 205
Ser Asn Gly His Ala Ser Gly Ser Glu Leu Asn Gly Asn Gly Asn Asn
210 215 220
Gly Thr Thr Asn Arg Arg Pro Leu Ser Ser Ala Ser Ala Gly Ser Thr
225 230 235 240
Ala Ser Asp Ser Thr Leu Leu Asn Gly Ser Leu Asn Ser Tyr Ala Asn
245 250 255
Gln Ile Ile Gly Glu Asn Asp Pro Gln Leu Ser Pro Thr Lys Leu Lys
260 265 270
Pro Thr Gly Arg Lys Tyr Ile Phe Gly Tyr His Pro His Gly Ile Ile
275 280 285
Gly Met Gly Ala Phe Gly Gly Ile Ala Thr Glu Gly Ala Gly Trp Ser
290 295 300
Lys Leu Phe Pro Gly Ile Pro Val Ser Leu Met Thr Leu Thr Asn Asn
305 310 315 320
Phe Arg Val Pro Leu Tyr Arg Glu Tyr Leu Met Ser Leu Gly Val Ala
325 330 335
Ser Val Ser Lys Lys Ser Cys Lys Ala Leu Leu Lys Arg Asn Gln Ser
340 345 350
Ile Cys Ile Val Val Gly Gly Ala Gln Glu Ser Leu Leu Ala Arg Pro
355 360 365
Gly Val Met Asp Leu Val Leu Leu Lys Arg Lys Gly Phe Val Arg Leu
370 375 380
Gly Met Glu Val Gly Asn Val Ala Leu Val Pro Ile Met Ala Phe Gly
385 390 395 400
Glu Asn Asp Leu Tyr Asp Gln Val Ser Asn Asp Lys Ser Ser Lys Leu
405 410 415
Tyr Arg Phe Gln Gln Phe Val Lys Asn Phe Leu Gly Phe Thr Leu Pro
420 425 430
Leu Met His Ala Arg Gly Val Phe Asn Tyr Asp Val Gly Leu Val Pro
435 440 445
Tyr Arg Arg Pro Val Asn Ile Val Val Gly Ser Pro Ile Asp Leu Pro
450 455 460
Tyr Leu Pro His Pro Thr Asp Glu Glu Val Ser Glu Tyr His Asp Arg
465 470 475 480
Tyr Ile Ala Glu Leu Gln Arg Ile Tyr Asn Glu His Lys Asp Glu Tyr
485 490 495
Phe Ile Asp Trp Thr Glu Glu Gly Lys Gly Ala Pro Glu Phe Arg Met
500 505 510
Ile Glu
<210> 73
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_5_For
<400> 73
gtacccgggg atcctctaga gacgagaaac aaatcatgtg
40
<210> 74
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_5_Rev
<400> 74
cgccgccaac ccggtctcta gcttttgttt tgtgtgactt g
41
<210> 75
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_TEF1_For
<400> 75
caagtcacac aaaacaaaag ctagagaccg ggttggcggc g
41
<210> 76
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_TEF1_Rev
<400> 76
gacgagtcag acaggaggca ctgcggttag tactgcaaaa ag
42
<210> 77
<211> 42
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_URA_For
<400> 77
ctttttgcag tactaaccgc agtgcctcct gtctgactcg tc
42
<210> 78
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_URA_Rev
<400> 78
cgccgccaac ccggtctctt ggtggtattg tgactggg
38
<210> 79
<211> 38
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_TEF2_For
<400> 79
cccagtcaca ataccaccaa gagaccgggt tggcggcg
38
<210> 80
<211> 43
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_TEF2_Rev
<400> 80
gtagtattgt gagtcgatag tctgcggtta gtactgcaaa aag
43
<210> 81
<211> 43
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_dga1_For
<400> 81
ctttttgcag tactaaccgc agactatcga ctcacaatac tac
43
<210> 82
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> DGA1_dga1_Rev
<400> 82
gcctgcaggt cgactctaga ttactcaatc attcggaact c
41
<---
Изобретение относится к биотехнологии. Предложен микроорганизм из рода Yarrowia, предназначенный для производства жира и в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (PEMT) или фосфолипидметилтрансферазы, где указанный микроорганизм получен на основе Yarrowia lipolytica, где указанный микроорганизм представляет собой микроорганизм, ауксотрофный по холину, и где указанный микроорганизм имеет увеличенное содержание жира по сравнению с диким типом. Также предложена композиция для производства жира, содержащая эксципиент и одно или более из указанного микроорганизма, культуры этого микроорганизма и сухого продукта этого микроорганизма. Также предложены способ увеличения содержания жира в микроорганизме, способ получения жира с использованием указанного микроорганизма и применение в производстве жира указанного микроорганизма, культуры этого микроорганизма, сухого продукта этого микроорганизма и жира, выделенного из одного или более из указанных микроорганизма, культуры и сухого продукта. Изобретение обеспечивает увеличение продукции жира в штамме Yarrowia. 5 н. и 11 з.п. ф-лы, 8 табл., 4 пр.
1. Микроорганизм для производства жира из рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (PEMT) или фосфолипидметилтрансферазы, где указанный микроорганизм получен на основе Yarrowia lipolytica, где указанный микроорганизм представляет собой микроорганизм, ауксотрофный по холину, и где указанный микроорганизм имеет увеличенное содержание жира по сравнению с диким типом.
2. Микроорганизм рода Yarrowia по п. 1, где фосфатидилэтаноламин-N-метилтрансферазу кодирует ген cho2 и указанный ген cho2 состоит из полинуклеотидной последовательности SEQ ID NO: 1.
3. Микроорганизм рода Yarrowia по п. 2, где указанный ген cho2 делетирован.
4. Микроорганизм рода Yarrowia по п. 1, где фосфолипидметилтрансферазу кодирует ген opi3 и указанный ген opi3 состоит из полинуклеотидной последовательности SEQ ID NO: 11.
5. Микроорганизм рода Yarrowia по п. 4, где указанный ген opi3 делетирован.
6. Микроорганизм рода Yarrowia по п. 1, где жир включает триацилглицерин (TAG).
7. Композиция для производства жира, содержащая эксципиент и одно или более из микроорганизма рода Yarrowia по любому из пп. 1-6, культуры этого микроорганизма и сухого продукта этого микроорганизма.
8. Способ увеличения содержания жира в микроорганизме по сравнению с диким типом, включающий: культивирование микроорганизма из рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (PEMT) или фосфолипидметилтрансферазы, где указанный микроорганизм получен на основе Yarrowia lipolytica, где указанный микроорганизм представляет собой микроорганизм, ауксотрофный по холину, и где указанный микроорганизм имеет увеличенное содержание жира по сравнению с диким типом.
9. Способ увеличения содержания жира в микроорганизме по п. 8, где культивирование микроорганизма представляет собой культивирование микроорганизма в среде, содержащей холин.
10. Способ увеличения содержания жира в микроорганизме по п. 9, где концентрация холина в среде составляет от 0,05 до 5 мМ.
11. Способ увеличения содержания жира в микроорганизме по п. 9, где отношение C/N в среде составляет от 40 до 80.
12. Способ увеличения содержания жира в микроорганизме по п. 8, где жир включает триацилглицерин (TAG).
13. Способ получения жира, включающий:
культивирование микроорганизма из рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (PEMT) или фосфолипидметилтрансферазы, где указанный микроорганизм получен на основе Yarrowia lipolytica, где указанный микроорганизм представляет собой микроорганизм, ауксотрофный по холину, и где указанный микроорганизм имеет увеличенное содержание жира по сравнению с диким типом;
сушку клеток, оставшихся в нижнем слое после центрифугирования культуральной среды микроорганизма;
и выделение жира из указанного микроорганизма, его культуры или его сухого продукта после культивирования микроорганизма.
14. Способ получения жира по п. 13, где культивирование микроорганизма представляет собой культивирование штамма в среде, содержащей холин.
15. Способ получения жира по п. 14, где отношение C/N в среде составляет от 40 до 80.
16. Применение микроорганизма из рода Yarrowia, в котором инактивирована активность фосфатидилэтаноламин-N-метилтрансферазы (PEMT) или фосфолипидметилтрансферазы, где указанный микроорганизм получен на основе Yarrowia lipolytica, где указанный микроорганизм представляет собой микроорганизм, ауксотрофный по холину, и где указанный микроорганизм имеет увеличенное содержание жира по сравнению с диким типом; культуры этого микроорганизма; сухого продукта этого микроорганизма; и жира, выделенного из одного или более из указанных микроорганизма, культуры и сухого продукта, в производстве жира.
| US 20170191073 A1, 06.07.2017 | |||
| Способ правки штампованных сварных конструкций | 1974 |
|
SU503613A1 |
| Схема обмотки ротора для пуска в ход индукционного двигателя без помощи реостата, с применением принципа противосоединения обмоток при трогании двигателя с места | 1922 |
|
SU122A1 |
| Способ получения 1,3-дизамещенных 5-амино-4цианпиразолов | 1973 |
|
SU503863A1 |
| Схема обмотки ротора для пуска в ход индукционного двигателя без помощи реостата, с применением принципа противосоединения обмоток при трогании двигателя с места | 1922 |
|
SU122A1 |
Авторы
Даты
2024-04-10—Публикация
2021-03-05—Подача