Область техники
Изобретение относится к области биотехнологии, а именно к технологии получения биологически активных веществ (БАВ) методами генной инженерии, точнее к методам получения L-аспарагиназы для использования в производстве лекарственных препаратов для терапии лимфобластных лейкозов.
Предшествующий уровень техники.
L-аспарагиназа (КФ 3.5.1.1, L-аспарагинамидогидролаза) - это фермент класса гидролаз, который катализирует гидролиз L-аспарагина с образованием L-аспарагиновой кислоты и иона аммония. Аспарагиназа применяется как противоопухолевое цитостатическое средство в терапии ряда лейкозов, а также при обработке пищевого сырья. Терапевтическое действие аспарагиназы основано на снижении уровня аспарагина в системной циркуляции и невозможности его интернализации опухолевыми клетками лимфоидного и миелоидного происхождения, которые, в отличие от остальных типов клеток человека, не способны синтезировать аспарагин.
Существует два типа бактериальных аспарагиназ. Аспарагиназы I типа - конститутивно экспрессируются в цитоплазме, обладают высоким сродством к L-аспарагину, но активны также по отношению к глутамину. Аспарагиназы II типа (asnB) локализованы в периплазматическом пространстве бактерий, экспрессируются в ответ на стресс. Их сродство к аспарагину ниже, чем у аспарагиназ I типа, но и глутаминазная активность очень низка. Только аспарагиназы II типа могут использоваться в качестве терапевтических средств, так как существенная глутаминазная активность приводила бы к постоянной интоксикации пациента ионами аммония.
В терапии в настоящее время используется аспарагиназа Escherichia coli, ее ПЭГ-производные и аспарагиназа из Dickeya dadantii (историческое название Erwinia chrysanthemi). Среди изоферментов L-аспарагиназы E. coli в качестве противоопухолевого средства используют только изоформу ЕсА2, кодируемую геном ansB (UniProtKB P00805).
Биологически активной формой аспарагиназы тип II является гомотетрамер, с молекулярной массой около 140 килодальтон, состоящий из двух более плотно взаимодействующих гомодимеров. Каждый димер содержит два активных центра, составленных из аминокислотных остатков двух разных мономеров, таким образом, димерная форма аспарагиназы способна проявлять гидролитическую активность, а мономерная форма - нет. Каждый мономер с молекулярной массой 36 кДа (около 330 аминокислотных остатков) имеет 40 β-слоев и 8 α-спиралей, уложенных в N-терминальный домен и меньший по размеру С-концевой домен. Четыре активных каталитических центра находятся между N- и С-концевыми доменами примыкающих мономеров.
Известно техническое решение по получению рекомбинантной L-аспарагиназы Erwinia cartovora в E.coli - рекомбинантная плазмидная ДНК pBADLANS и штамм E. coli TCAR-LANS ((Патент РФ № 2221868, МПК C12N, опубл. 2004). В патенте заявляется продуктивность созданного штамма как 26 единиц активности аспрагиназы на 1 мл на 6 часах индукции при плотности культуры 4,38 О.Е. Удельная активность фермента при культивации описана как 55 ЕД/мг белка через 4 часа культивации, доля целевого белка среди всех белков E.coli описана как 8-10%. Методика измерения активности аспарагиназы, определение единицы активности и результаты измерения активности для контрольного образца высокоочищенной аспарагиназы E. cartovora в патенте не приводятся. Известно, что удельная активность высокоочищенной аспарагиназы E. cartovora составляет около 90 ЕД/мг, в таком случае описанный в патенте штамм-продуцент накопил бы 288 мг/л целевого белка. Обычная культура E.coli содержит около 180 мг/л общего белка при плотности культуры 1 О.Е., в таком случае общее содержание белка в культуре с плотностью 4,38 О.Е. будет составлять около 800 мг/л, доля аспарагиназы на уровне продукции 288 мг/л составила бы 36%, что явно не соответствует указанному авторами патента значению 8-10%. Количество накопленной аспарагиназы в данной работе, исходя из заявленного содержания 8-10% от общего белка, составило бы около 65-80 мг/л, или 14-18 мг/(л*О.Е.), что представляется более реалистичной оценкой. Таким образом, недостатком данного решения является невысокое удельное и общее количество вырабатываемой штаммом аспарагиназы, а также наличие в плазмиде гена бета-лактамазы.
Известно техническое решение, включающие создание штамма-продуцента штамма E. coli на основе рекомбинантной плазмидной ДНК pACYCLANS, содержащей ген L-аспарагиназы Erwina carotovora (Патент РФ № 2224797, МПК C12N, опубл. 2004 г.) и ген устойчивости к канамицину (фосфотрансферазу). Недостатком этого метода является низкое содержание рекомбинантной L-аспарагиназы - не более 8-12% от общего клеточного белка, что составляет 14-22 мг/(л*О.Е.).
Аспарагиназа E.coli может быть получена оверэкспрессией в составе слитного белка с доменом тиоредоксина I (ген trxB) E.coli, в патенте CN103436513A заявлен уровень ферментативной активности 27980 ЕД/л. Удельная активность очищенной аспарагиназы E.coli составляет около 250 МЕ/мг, в таком случае содержание активной аспарагиназы в составе слитного белка составляло около 110 мг/л при неизвестной плотности культуры, выращенной в ферментере. Данный способ получения аспарагиназы предполагает выделение слитного белка из лизата клеток, протеолитическое отделение домена тиоредоксина и полное удаление непроцессированного слитного белка и отделенного домена тиоредоксина. В патенте не описывается такой процессинг и контроль чистоты получаемой аспарагиназы, в большинстве случаев такая обработка целевого белка многократно удорожает процесс его выделения и очистки и применяется при производстве лекарственных средств в очень редких случаях. Данный способ получения аспарагиназы также обладает недостатками - низким уровнем экспрессии целевого белка и усложненным процессом получения продукта.
Описано получение аспарагиназы E.coli в системе Pseudomonas, изложенное в патенте US20190127743A1. Были использованы синтетический ген аспарагиназы E.coli, удаление собственного гена аспарагиназы II типа бактерии-хозяина, подбор лидерного пептида, удаление гена протеазы lon и изменение системы фолдинга у бактерии-хозяина. Содержание аспарагиназы, измеренное при помощи капиллярного электрофореза в денатурирующих условиях, составило до 3000 мг/л для наиболее продуктивного штамма, однако концентрация ферментативно активной аспарагиназы не превышала 300 мг/л за 24 ч индукции, т.е. 9/10 целевого белка накапливалось в растворимом, но ферментативно неактивном виде. Основные недостатки данного способа получения аспарагиназы - наработка очень больших количеств трудноотделяемой ферментативно неактивной аспарагиназы, а также использование потенциально патогенного микроорганизма-хозяина.
Одним из основных недостатков микроорганизмов из порядка Pseudomonadales при использовании в качестве основы для создания штаммов продуцентов терапевтических белков является патогенность многих его представителей, например, упоминаемый в патенте US20190127743A1 вид Pseudomonas aeruginosa - оппортунистические болезнетворные микроорганизмы, которые часто вызывают внутрибольничные инфекции, усугубляемые множественной лекарственной устойчивостью возбудителей. В организме человека P. aeruginosa является наиболее распространенным патогеном, но заражение может произойти и от P. putida, P. fluorescens или P. аcidovorans также упомянутых в US20190127743A1. Способность P. aeruginosa и родственных видов быстро приобретать устойчивость к антибиотикам является актуальной медицинской проблемой. При промышленной культивации данных видов существует риск контаминации и инфицирования. Наконец, Pseudomonas представляет собой относительно малоизученный микроорганизм, что затрудняет оптимизацию условий экспрессии, выделения и очистки продукта.
Описано так же получение аспарагиназы E.coli в дрожжах Pichia pastoris SuperMan5 (his-), трансформированных плазмидой pJAG-s1 (Glycosylation of L-asparaginase from E. coli through yeast expression and site-directed mutagenesis, Biochemical Engineering Journal, Volume 156, 15 April 2020, 107516, Lima et al. https://doi.org/10.1016/j.bej.2020.107516). Аспарагиназа секретируется в культуральную среду в гликозилированном состоянии, что может сильно повышать ее иммуногенность и вызывать немеделенную воспалительную реакцию при инъекциях вследствие того, что дрожжи интенсивно N-гликозилируют секретируемые белки и вносят иммуногенные олигоманнозные олигосахаридные группы в такие белки.
Наиболее близкий известный способ получения рекомбинантной L-аспарагиназы (прототип изобретения) включает создание рекомбинантной плазмидной ДНК pL-ASP-08 и нового штамма бактерий Escherichia coli XL1-blue/pL-ASP-08, содержащего данную плазмиду (Патент РФ № 2397248, МПК C12N, опубл. 2010 г.). Продуктивность штамма описана как 165 мг/л суммарной внутриклеточной аспарагиназы II типа по данным электрофореза при неизвестной оптической плотности клеток, 33% от общего растворимого белка по данным денситометрии электрофореграммы. Общая ферментативная активность секретированной и внутриклеточной аспарагиназы составила 42 МЕ/мл, что соответствует общей концентрации активной аспарагиназы 168 мг/л. Среди недостатков данного решения - неоптимизированный ген, получаемый в ходе амплификации геномной ДНК E. coli, использование векторной плазмиды pQE-30, кодирующей бета-лактамазу, вызывающую аллергические реакции у пациентов при инъекции даже незначительных количеств этого белка, как примеси в лекарственном средстве, и относительно невысокий уровень продуктивности, а также использование бактерий XL-1Blue в качестве клеток-хозяев и вызываемое этим загрязнение целевого варианта аспарагиназы II типа белком, кодируемым хромосомой клеток класса K12 и отличающимся от целевого варианта белка несколькими аминокислотными остатками.
Было предположено, что продуктивность системы экспрессии может быть существенно увеличена при получении аспарагиназы с использованием оптимизированного синтетического гена и векторной плазмиды с канамициновой селекцией.
Раскрытие сущности изобретения
Целями настоящего изобретения являются:
1) предоставление экспрессионной плазмиды pET28a-AsnSYN, содержащей синтетический фрагмент ДНК, кодирующий L-аспарагиназу Escherichia coli II типа, в частности, под контролем промотора, функционирующего в бактериальной клетке, например, под контролем промотора РНК-полимеразы бактериофага Т7, либо другого индуцируемого или конститутивного промотора, например, tac-промотора, или trp-промотора, или rha-промотора, либо иного промотора.
2) предоставление бактерии, принадлежащей к роду Escherichia, трансформированной описанной выше плазмидой, - продуцента L-аспарагиназы Escherichia coli II типа,
3) предоставление описанной выше бактерии, где указанная бактерия представлена штаммом Escherichia coli BL21[DE3]/ pET28a-AsnSYN,
4) получение рекомбинантной L-аспарагиназы Escherichia coli II типа с использованием указанной выше бактерии и/или бактерий.
Технической проблемой, решаемой авторами, является создание изобретения, лишенного недостатков уровня техники, в частности, прототипа, а именно, создание технологии получения L-аспарагиназы Escherichia coli II типа с увеличением количества аспарагиназы на единицу объема ферментера и на килограмм биомассы E.coli, вследствие чего количество готового продукта тоже пропорционально возрастает, с возможностью высокого выхода и хорошей чистоты.
Техническим результатом, который обеспечивает настоящее изобретение, является улучшенная продуктивность, увеличение количества аспарагиназы на единицу объема ферментера и на килограмм биомассы E.coli, вследствие чего количество готового продукта тоже пропорционально возрастает, с возможностью высокого выхода. Также достигается хорошая чистота, и, как следствие, - потенциальное снижение частоты побочных эффектов у пациентов. Кроме того, изобретение позволяет расширить арсенал известных технических решений, направленных на продуцирование аспарагиназы.
Технический результат достигается путем создания технологии, включающей в себя новую экспрессионную плазмидную ДНК pET28a-AsnSYN, кодирующую L-аспарагиназу E. coli II типа, создание штамма продуцента E. coli на ее основе.
В основе данного решения лежит разработанная авторами плазмидная ДНК pET28a-AsnSYN длиной 6298 п.о., кодирующая L-аспарагиназу E. coli II типа. Открытая рамка считывания указанного белка L-аспарагиназы E. coli II типа содержит оптимальный для процесса трансляции набор и последовательность кодонов, позволяющие увеличить уровень экспрессии белка за счет как эффективной трансляции всех аминокислот полипептида, так и снижения количества элементов вторичной структуры РНК, что приводит к значительному увеличению выхода целевого белка.
В частности, при культивировании в колбах в культуральной среде Terrific Broth бактерий BL21[DE3]/ pET28a-AsnSYN уровень внутриклеточной аспарагиназы E.coli II типа достиг 291 мг/л за 21 ч индукции, что практически в два раза превосходит лучшие ранее достигнутые в Патенте РФ № 2397248, МПК C12N, опубл. 2010 г. результаты для E.coli -168 мг/л.
Признак «AsnSYN» указывает на код, который обозначает синтетический вариант гена аспарагиназы, где Asn - обозначение аспарагиназы, SYN - синтетический. Данный вариант гена используется с вектором pET28а (+), итоговая конструкция обозначается как «pET28a-AsnSYN».
Признак «экспрессионная плазмида» означает плазмидную ДНК, содержащую все необходимые генетические элементы для экспрессии внедренного в нее гена, например, такие как промотор и терминатор. Конкретным примером генетических элементов, необходимых для экспрессии L-аспарагиназы E. coli II типа в составе экспрессионной кассеты, согласно настоящему изобретению, является, но не ограничивается им, промотор РНК-полимеразы бактериофага Т7.
Фрагментом ДНК, кодирующим рекомбинантную L-аспарагиназу E. coli II типа, согласно настоящему изобретению, является, например, синтетический ген, кодирующий L-аспарагиназу E. coli II типа. Указанный фрагмент ДНК может быть получен методом ПЦР или с использованием технологии клонирования фирмы Sloning BioTechnology, описанной в заявке РСТ WO2005071077.
Чтобы обеспечить эффективную трансляцию клонированного гена в E. coli предпочтительно, чтобы в последовательности, кодирующей L-аспарагиназу E. coli, все редкие кодоны были заменены синонимичными часто встречающимися в генах E. coli кодонами, а также расчетная вторичная структура РНК не содержала стабильных шпилек.
Последовательность гена, кодирующего L-аспарагиназу E. coli II типа, согласно настоящему изобретению, представлена в Перечне последовательностей под номером SEQ ID NO:1.
Аминокислотная последовательность белка L-аспарагиназы E. coli II типа, согласно настоящему изобретению, представлена в Перечне последовательностей под номером SEQ ID NO: 2.
Фрагменты ДНК, которые кодируют по существу тот же белок, могут быть получены, например, путем модификации нуклеотидной последовательности фрагмента ДНК (SEQ ID NO: 1), кодирующего L-аспарагиназу E. coli II типа, например, посредством метода сайт-направленного мутагенеза, так, что один или несколько аминокислотных остатков в определенном сайте будут делетированы (удалены), заменены, вставлены или добавлены. Фрагменты ДНК, модифицированные, как описано выше, могут быть получены с помощью традиционных методов обработки с целью получения мутации. Фрагменты ДНК, которые кодируют по существу тот же белок, могут быть получены путем экспрессии фрагментов ДНК, имеющих мутацию, описанную выше, в соответствующей клетке.
Синтетический ген L-аспарагиназы E. coli II типа, приведенный в настоящем изобретении, может быть изменен, например, путем модификации нуклеотидной последовательности фрагмента ДНК (SEQ ID NO: 1), кодирующего L-аспарагиназу E. coli II типа, например, посредством метода сайт-направленного мутагенеза, так, что все аминокислотные остатки белка будут сохранены, но один или несколько кодонов будут заменены на синонимичные. Фрагменты ДНК, модифицированные, как описано выше, могут быть получены с помощью традиционных методов обработки с целью получения мутации. Фрагменты ДНК, которые кодируют тот же белок и представляют собой по существу ту же последовательность кодонов с незначительными заменами кодонов на синонимичные им, могут быть получены путем экспрессии фрагментов ДНК, имеющих мутации, описанные выше, в соответствующей клетке.
Показатели функциональной активности, при которой считается, что полученный белок обладает свойствами L-аспарагиназы E. сoli II типа, определяются по его способности гидролизовать L-аспарагин. Так, например, активность L-аспарагиназы Escherichia coli II типа можно детектировать с реактивом Несслера, как описано в Примере 3. Считается, что вариант белка обладает свойствами L-аспарангиназы E. coli II типа при условии, что указанный вариант обладает активностью не менее 5 международных единиц на 1 мг белка, предпочтительно не менее 10 международных единиц на мг белка.
Экспрессионная плазмида, согласно настоящему изобретению, содержит фрагмент ДНК, кодирующий L-аспарагиназу E. coli II типа, под контролем промотора, функционирующего в бактериальной клетке. В качестве рекомбинантной плазмиды, согласно настоящему изобретению, могут использоваться различные плазмиды, обладающие способностью к экспрессии в клетке-реципиенте, такие как плазмиды pBR322, pMW119, pUC19, pET32a, pET28b и подобные им, но список плазмид не ограничивается ими.
Конкретным вариантом реализации настоящего изобретения является плазмида pET28a-AsnSYN, размером 6298 п.о. которая состоит из:
1) фрагмента HindIII-NcoI (1-1053)-длиной 1052 п.о., содержащего участок, кодирующий синтетический ген аспарагиназы (12-1055).
2) фрагмента NcoI-HindIII (1053-6298) вектора pET28a(+) длиной 5246 п.о., содержащего область начала репликации плазмиды pBR322, ген РНК-организующего белка Rop, участок инициации репликации бактериофага f1, последовательность, кодирующую аминогликозид-3'-фосфотрансферазу, промотор РНК-полимеразы бактериофага Т7; участок терминации транскрипции; последовательность, кодирующую репрессор лактозного оперона.
Указанная плазмида содержат уникальные сайты узнавания эндонуклеазами рестрикции: HindIII (1), NcoI (1053), FspAI (2962), BstZ17I (3752), XmaI (5055).
Структуры плазмиды pET28a-AsnSYN приведена на Фиг. 7 и в SEQ ID 3.
При помощи созданной плазмиды можно трансформировать бактериальную клетку, предпочтительно бактерию рода Escherichia, восприимчивую к подобной трансформации указанной плазмидой. Выбор конкретной клетки не является критическим, поскольку методология и приемы трансформации хорошо известны специалисту в данной области техники. Хотя в зависимости от вида клетки и условий культивирования полученного трансформанта уровень экспрессии L-аспарагиназы E. coli II типа может варьироваться, факт экспрессии целевого белка будет иметь место при условии успешной трансформации клетки - реципиента.
«Трансформация клетки плазмидой» означает введение плазмиды в клетку с помощью методов, хорошо известных специалисту в данной области техники, например, кальциевым методом, химическим методом или с помощью электропорации. Трансформация этой плазмидой приводит к экспрессии гена, кодирующего белок согласно настоящему изобретению, и к синтезу белка в бактериальной клетке. Методы трансформации включают любые стандартные методы, известные специалисту в данной области техники, например метод, описанный в Jac A. Nickoloff, Electroporation Protocols for Microorganisms (Methods in Molecular Biology) // Humana Press; 1st edition (August 15, 1995).
Согласно настоящему изобретению, «бактериальная клетка - продуцент L-аспарангиназы E. coli II типа» означает бактериальную клетку, обладающую способностью к продукции и накоплению L-аспарангиназы E. coli II типа, согласно настоящему изобретению, когда бактериальная клетка, согласно настоящему изобретению, выращивается в указанной питательной среде. Используемый здесь признак «бактериальная клетка - продуцент L-аспарангиназы E. coli II типа» также означает клетку, которая способна накапливать L-аспарагиназу E. coli II типа в количестве не менее чем 2 мг/л культуры, более предпочтительно, не менее чем 20 мг/л культуры. Указанный белок L-аспарангиназа E. coli II типа накапливается в указанной клетке предпочтительно в периплазме в растворимой форме.
Предпочтительно использование бактерии, принадлежащей к роду Escherichia, для трансформации рекомбинантной плазмидой, содержащей фрагмент ДНК, кодирующий L-аспарагиназу E. coli II типа.
Признак «бактерия, принадлежащая к роду Escherichia» может означать, что бактерия относится к роду Escherichia в соответствии с классификацией, известной специалисту в области микробиологии. В качестве примера микроорганизма, принадлежащего к роду Escherichia, может быть упомянута бактерия Escherichia coli (E. coli).
Круг бактерий, принадлежащих к роду Escherichia, не ограничен каким-либо образом, однако, например, бактерии, описанные в книге Neidhardt, F.C. et al. (Escherichia coli and Salmonella typhimurium, American Society for Microbiology, Washington D.C., 1208, Таблица 1), могут быть приведены в качестве примеров.
Конкретным примером штамма - реципиента для получения продуцента L-аспарагиназы E. coli II типа, согласно настоящему изобретению, является, но не ограничиваются им, штамм Escherichia coli BL21[DE3].
Штамм Escherichia coli BL21[DE3] характеризуется следующими культурально-морфологическими, физиолого-биохимическими признаками и генетическими признаками.
Культурально-морфологические особенности штамма: грамм-отрицательные палочки, образуют нити; на агаризованной среде - беловатые крупные колонии с неровным краем. Активность штамма определяется методом денситометрии электрофореграммы. Штамм хранится в следующих условиях: среда Лурье-Бертрана, 1% глюкозы, 10% глицерина. Штамм размножается в следующих условиях - среда Лурье-Бертрана, 1% глюкозы.
Генетические особенности штамма. Генотип штамма - F- ompT gal dcm lon hsdSB(rB- mB-) λ(DE3 [lacI lacUV5-T7 gene 1 ind1 sam7 nin5]).
Трансформация штамма Escherichia coli BL21[DE3] плазмидой pET28a-AsnSYN приводит к получению штамма-продуцента BL21[DE3]/pET28a-AsnSYN, который обеспечивает синтез рекомбинантной L-аспарагиназы E. coli II типа в количестве 5-20% от суммарного содержания белка клеток.
Таким образом, сущность настоящего изобретения заключается в следующем:
- Экспрессионная плазмида pET28a-AsnSYN, предназначенная для синтеза L- аспарагиназы, содержащая фрагмент ДНК с последовательностью SEQ ID NO:3, кодирующий L-аспарагиназу Escherichia coli II типа.
- Бактерия, принадлежащая к виду Escherichia coli, трансформированная указанной плазмидой, предназначенная для синтеза L-аспарагиназы Escherichia coli II типа.
- Бактерия, которая относится к штамму Escherichia coli BL21[DE3]/pET28a-AsnSYN.
- Способ получения L-аспарагиназы, включающий культивирование штамма Escherichia coli BL21[DE3]/pET28a-AsnSYN, выделение L-аспарагиназы и ее очистку.
Особенности плазмиды и результаты их практического применения приведены на следующих Фигурах.
Краткое описание Фигур:
На Фиг. 1 показана схема получения экспрессионных плазмид pET28a-AsnWT и pET28a-AsnSYN. Используются следующие обозначения: гДНК - геномная ДНК штамма E. coli BL21 [DE3]. Пунктирная линия обозначает полимеразную цепную реакцию, сплошная - рестрикцию и лигирование фрагментов ДНК, курсивом приведены названия эндонуклеаз рестрикции, использованных для клонирования, прямым шрифтом - названия плазмид.
На Фиг. 2 показана диаграмма распределения кодонов природного и синтетического гена аспарагиназы по децилям индекса предпочтительности. Обозначения: WT - природный ген аспарагиназы; SYN - синтетический ген аспарагиназы.
На Фиг. 3 показано центроидное состояние ансамбля структур 5’-концевой области мРНК для природного гена аспарагиназы. Сайт связывания рибосомы и старт-кодон выделены жирным шрифтом.
На Фиг. 4 показано центроидное состояние ансамбля структур 5’-концевой области мРНК для синтетического гена аспарагиназы. Сайт связывания рибосомы и старт-кодон выделены жирным шрифтом.
На Фиг. 5 показано распределение индекса GC состава ДНК для природного и синтетического гена аспрагиназы. Природный ген - сплошная линия, синтетический ген - пунктирная линия, доли GC пар приведены для сегментов по 5 пар оснований.
На Фиг. 6 показана карта экспрессионной плазмиды pET28a-AsnWT. Используются следующие обозначения: «pBR322 ori» - область начала репликации плазмиды pBR322; «ROP» - ген РНК-организующего белка Rop, контролирующего копийность плазмиды; «f1 ori» - участок инициации репликации бактериофага f1; «KanR2» - ген устойчивости к канамицину; «T7 prom» - промотор РНК-полимеразы бактериофага Т7; «T7 term» - участок терминации транскрипции целевого гена; «lacI» - ген репрессора лактозного оперона; asparaginase ws - природная ОРС полипептида L-аспарагиназы Escherichia coli II типа; «2 stop» - блок стоп-кодонов. Стрелками указаны направления транскрипции генов, в скобках указаны номера первого и последнего нуклеотидов фрагментов. Курсивом выделены сайты узнавания эндонуклеаз рестрикции, в скобках указаны номера нуклеотидов в точках разрезания.
На Фиг. 7 показана карта экспрессионной плазмиды pET28a-AsnSYN. Используются следующие обозначения: «pBR322 ori» - область начала репликации плазмиды pBR322; «ROP» - ген РНК-организующего белка Rop, контролирующего копийность плазмиды; «f1 ori» - участок инициации репликации бактериофага f1; «KanR2» - ген устойчивости к канамицину; «T7 prom» - промотор РНК-полимеразы бактериофага Т7; «T7 term» - участок терминации транскрипции целевого гена; «lacI» - ген репрессора лактозного оперона; asparaginase syn - синтетическая оптимизированная ОРС полипептида L-аспарагиназы Escherichia coli II типа; «2 stop» - блок стоп-кодонов. Стрелками указаны направления транскрипции генов, в скобках указаны номера первого и последнего нуклеотидов фрагментов. Курсивом выделены сайты узнавания эндонуклеаз рестрикции, в скобках указаны номера нуклеотидов в точках разрезания.
На Фиг. 8 показана диаграмма накопления внутриклеточной аспарагиназы при индукции штаммов-продуцентов BL21[DE3]/pET28a-AsnSYN и BL21[DE3]/pET28a-AsnWT при индукции 1 мМ ИПТГ в среде Terrific broth при температуре 30°C. Обозначения: WT - штамм BL21[DE3]/pET28a-AsnWT; SYN - штамм BL21[DE3]/pET28a-AsnSYN.
На Фиг. 9 показана электрофореграмма тотального белка, растворимых белков и нерастворимых белков из клеток штамма-продуцента BL21[DE3]/pET28a-AsnSYN при индукции 1 мМ ИПТГ в среде Terrific broth при температуре 30°C.
Индукция 1 мМ ИПТГ, 37°C; 0, 3 и 21 ч. Обозначения: «0 h» - тотальный клеточный белок до внесения индуктора, «3 h SOL» - растворимые внутриклеточные белки, 3 ч индукции, «21 h SOL» - растворимые внутриклеточные белки, 21 ч индукции, «3 h RES» - нерастворимые белки, 3 ч индукции; «21 h RES» - нерастворимые белки, 21 ч индукции; «WT» - природный ген аспарагиназы, «SYN» - синтетический ген аспарагиназы; «М» - маркер молекулярных масс. Молекулярные массы полос маркера указаны в кДа. На все дорожки нанесены белки, полученные из 120 мкл культуры. Положение целевого белка указано стрелкой. Восстанавливающие условия, окраска коллоидным Кумасси синим.
На Фиг. 10 приведены электрофореграммы белковых фракций, полученных при выделении и хроматографической очистке аспарагиназы.
Обозначения: «SOL» - растворимые белки, «NaOAc» - супернатант после закисления лизата, «UF/DF» - концентрат после ультрафильтрации и диафильтрации, «CS» - элюат катионообменной хроматографии, «DEAE» - элюат анионообменной хроматографии, «PHE» - фракция проскока гидрофобной хроматографии, «HA» - элюат хроматографии на керамическом гидроксилапатите, «М» - маркер молекулярных масс. Молекулярные массы полос маркера указаны в кДа. Положение целевого белка указано стрелкой. Восстанавливающие условия, окраска коллоидным Кумасси синим.
На Фиг. 11 показана хроматограмма гель-фильтрационного анализа очищенной аспарагиназы и результаты интеграции пиков. Канал детекции 280 нм.
Осуществление изобретения
Настоящее изобретение будет более подробно описано ниже со ссылкой на следующие не ограничивающие настоящее изобретение Примеры.
Пример 1. Получение плазмидной ДНК pAL-AsnWT
Для гена аспарагиназы были подобраны адапторные праймеры AD-Asp-NcoF (SEQ ID NO:4) и AD-Asp-HindR (SEQ ID NO:5), позволяющие амплифицировать ОРС целевого бека с адапторными последовательностями, включающими сайты узнавания эндонуклеаз рестрикции NcoI и HindIII, для последующего клонирования. В последовательность ДНК также были включены дополнительные стоп-кодоны. Полученная нуклеотидная последовательность приведена в SEQ ID NO:6.
Из клеток E. coli штамма BL21 [DE3] выделяли геномную ДНК и использовали ее в качестве матрицы при проведении ПЦР-амплификации с праймерами AD-Asp-NcoF (SEQ ID NO:4) и AD-Asp-HindR (SEQ ID NO:5). Амплификацию ДНК проводили с помощью набора Tersus Plus PCR kit и Encyclo Plus PCR kit, содержащими смеси высокоточных полимераз. Режим ПЦР: предварительная денатурация - 95оС, 2 минуты, далее 35 циклов: денатурация 95оС, 15 сек, отжиг праймеров 55оС, 15 сек, элонгация 72оС, 1,5 минуты, затем финальная элонгация 72оС, 2 минуты, 10оС - охлаждение. Продукт ПЦР выделяли из 1% агарозного геля и лигировали в вектор pAL2T («Евроген», Россия). Полученными лигазными смесями трансформировали клетки E. coli штамма Top10. Колонии E. coli, отобранные в результате бело-голубого скрининга, анализировали методом ПЦР с клонов, с использованием праймеров к последовательностям реципиентной плазмиды pAL2 М13dir (SEQ ID NO:7), M13rev (SEQ ID NO:8). 5 отобранных клонов наращивали в 5 мл 2хYT-Amp и выделяли плазмидную ДНК. Для полученных плазмид pAL2-AsnWT определяли нуклеотидную последовательность области вставки методом ПЦР-секвенирования с использованием стандартных праймеров к последовательности вектора М13dir (SEQ ID NO:7), M13rev (SEQ ID NO:8).
Для создания экспрессионных плазмид использовали вектор pET28а(+) и его производные. Вектор pET28а(+) является деривативом вектора pBR322, включающим в себя фрагмент области начала репликации природной плазмиды ColE1, ген РНК-организующего белка; участок инициации репликации бактериофага f1; последовательность, кодирующую аминогликозид-3'-фосфотрансферазу, промотор и последовательность, кодирующую lac-репрессор (lacI), а также ориентированные в противоположную сторону последовательности: 1) сильного индуцибельного промотора бактериофага Т7 и 2) lac-оператора, составляющие гибридный Т7lac промотор, позволяющий строго контролировать базальный уровень экспрессии целевого гена в штаммах E. coli, лизогенизированных по фагу λ-DE3. Реципиентную плазмиду pET28a разрезали ферментами NcoI и HindIII, дефосфорилировали, переосаждали спиртом и лигировали с очищенным фрагментом NcoI - HindIII плазмиды pAL-AsnWT, соответствующим минигену аспарагиназы. Полученными лигазными смесями трансформировали клетки E. coli штамма TOP10. Колонии E. coli анализировали методом ПЦР с клонов с помощью праймеров T7prom (SEQ ID NO:9) и T7t (SEQ ID NO:10). Отобранные клоны наращивали в 5 мл среды 2хYT-Kan, проводили выделение плазмидной ДНК, секвенировали области вставки с праймеров T7prom (SEQ ID NO:9) и T7t (SEQ ID NO:10). В результате секвенирования отобрали плазмиды без мутаций в области вставки, то есть кодирующие корректную последовательность гена аспарагиназы. Карта экспрессионной конструкции pET28a-AsnWT приведена на Фиг. 6 и в SEQ ID NO:11, аминокислотная последовательность продукта экспрессии приведена в SEQ ID NO:2.
Пример 2. Получение экспрессионной плазмидной ДНК pET28a-AsnSYN
Для аминокислотной последовательности L-аспарагиназы Escherichia coli II типа была проведена обратная трансляция в последовательность нуклеотидов ДНК. При этом были использованы кодоны, оптимальные для экспрессии этого гена в E. coli, а также была проведена оптимизация структуры гена по вторичной структуре мРНК и GC составу, обеспечено отсутствие нежелательных регуляторных элементов, а также отсутствие протяженных повторов и палиндромов. В последовательность ДНК были включены стоп-кодоны и сайты узнавания эндонуклеаз рестрикции. Полученная нуклеотидная последовательность приведена в SEQ ID NO:12.
При оптимизации кодонной структуры гена была использована метрика RSCU - relative synonymous codon usage (относительная частота использования синонимичных кодонов), то есть отношение наблюдаемой частоты использования кодонов к ожидаемой частоте в предположении одинаковой частоты использования всех возможных кодонов.
Анализ кодонной структуры рассчитанной последовательности синтетического гена был проведен как ранжирование использованных кодонов против известного набора частотности (предпочтительности) кодонов в E.coli B, вычисленного, как указано в (https://bmcbioinformatics.biomedcentral.com/articles/10.1186/s12859-017-1793-7).
Использованные в природном и синтетическом генах кодоны были разделены на децильные группы по частотности, результаты приведены на Фиг. 2. Установлено, что в синтетическом гене увеличена доля кодонов с высокой предпочтительностью, от 80 до 100, за счет падения доли кодонов со средней предпочтительностью - от 40 до 60, при этом доли слабопредпочтительных кодонов, от 20 до 40, одинаковы для обоих вариантов гена, а кодоны с очень низкой предпочтительностью полностью отсутствуют в обоих вариантах гена.
Моделирование возможных вторичных структур РНК для природного и синтетического гена проводили при помощи сервиса RNAfold, расположенного на сервере http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi.
Находящуюся непосредственно на 5`-конце мРНК шпильку со значительной энергией образования, одинаковую для природного и синтетического генов, исключали из анализа, для этого вели расчет вторичных структур для 5’-концевой области мРНК, начиная с позиции +30. Для получения релевантной модели вторичных структур использовали участок мРНК 30-300. Для него в случае природного гена было предсказано практически полное сворачивание в шпилечную структуру с вероятностью образования большинства двуспиральных участков более 80% и общей энергией образования ансамбля структур -70 ккал/моль. В случае синтетического гена после варьирования кодонного состава для центроидной структуры РНК было предсказано только два двуспиральных участка с вероятностью образования около 60% и общей энергией образования ансамбля -47 ккал/моль. Сайт связывания рибосом (последовательность Шайн-Дальгарно) и старт-кодон в случае природного гена были вовлечены в образование двуспиральных структур, но с небольшой вероятностью образования, а в случае синтетического гена - находились вне участков спиралеобразования, что предпочтительно для эффективной трансляции данной мРНК. Образование шпилечной структуры мРНК вблизи старт-кодона может негативно влиять на трансляцию, такие структуры мРНК могут использоваться бактерией для метаболической регуляции работы гена аспарагиназы на уровне контроля трансляции. При оверэкспрессии гена аспарагиназы данные эффекты нежелательны, целевой ген должен обеспечивать эффективную трансляцию полипептида аспарагиназы вне зависимости от режима метаболизма бактерий.
При расчете синтетического гена также контролировали отсутствие областей с сильно увеличенным или сильно уменьшенным GC-составом для предотвращения появления участков торможения транскрипции, отклонения GC-состава от среднего значения для синтетического гена на всей длине гена не превышали отклонения GC-состава для природного гена и приведены на Фиг. 5.
Синтетический ген аспарагиназы с последовательностями для клонирования (SEQ ID NO:5) был физически получен компанией SYNBIO (КНР) и клонирован в плазмиду pUC57. Данной плазмидой трансформировали клетки E. coli штамма Top10 (ThermoFischer Scientific, США). Колонии E. coli наращивали в 5 мл 2хYT-Amp и выделяли плазмидную ДНК pUC57-AsnSYN. Для полученных плазмид pUC57-AsnSYN определяли нуклеотидную последовательность области вставки методом ПЦР-секвенирования с использованием стандартных праймеров к последовательности вектора М13dir (SEQ ID NO:7), M13rev (SEQ ID NO:8) и специфического праймера SQ-SYNASN-R (SEQ ID NO:13).
Реципиентную плазмиду pET28a гидролизовали с помощью NcoI и HindIII, дефосфорилировали, переосаждали спиртом и лигировали с очищенным фрагментом NcoI - HindIII плазмиды pUC57-AsnSYN WT, соответствующим минигену аспарагиназы. Полученными лигазными смесями трансформировали клетки E. coli штамма TOP10. Колонии E. coli анализировали методом ПЦР с клонов с праймеров T7prom (SEQ ID NO:9) и специфического праймера SQ-SYNASN-R (SEQ ID NO:13). Отобранные клоны наращивали в 5 мл среды 2хYT-Kan, проводили выделение плазмидной ДНК, секвенировали области вставки с праймеров T7prom (SEQ ID NO:9), T7t (SEQ ID NO:10) и SQ-SYNASN-R (SEQ ID NO:13). В результате секвенирования отобрали плазмиды без мутаций в области вставки, то есть кодирующие корректную последовательность гена аспарагиназы. Карта экспрессионной конструкции pET28a-AsnSYN приведена на Фиг. 7 и в SEQ ID NO:3, аминокислотная последовательность продукта экспрессии приведена в SEQ ID NO:2.
Пример 3. Метод измерения ферментативной активности L-аспарагиназы Escherichia coli II типа
Метод Несслера позволяет количественно определить концентрацию ионов аммония, выделяющихся после превращения аспарагина в аспарагиновую кислоту и вступающих в реакцию с реагентом Несслера - тетрайодомеркуратом дикалия(II). Единица активности аспарагиназы (ME) соответствует 1 мкмоль ионов аммония NH4+, образующихся в минуту при рН 7,3 и 37°C.
Водные растворы аспарагиназы и референтного препарата разводили в дистиллированной воде до концентрации 0,02-0,1 мг/мл. Для определения активности готовили реакционную смесь: к 180 мкл 0,5 М трис-буфера, рН 7,3 добавляли 20 мкл 0,094 мМ раствора L-аспарагина и 20 мкл раствора аспарагиназы, и перемешивали на вортексе. После инкубации при 37°C в течение 10 мин при контролируемой температуре, реакцию останавливали добавлением 80 мкл раствора трихлоруксусной кислоты (1,5 М). Реакционную смесь перемешивали на вортексе, выдерживали при 4°C в течение 10 мин, центрифугировали белковый осадок на 15 тыс. об/мин в течение 5 минут. Надосадочную жидкость использовали для определения аспарагиназной активности, перед нанесением на 96-луночный планшет образцы разводили в 5 раз дистиллированной водой.
Калибровочные растворы готовили путем многократного разведения исходного раствора хлорида аммония (NH4Cl, 5 мМ) в следующем диапазоне концентраций: от 0,5 до 5 мкмоль/мл NH4+.
В 96-луночный планшет вносили по 50 мкл калибровочных растворов и предварительно разведенных образцов. Затем добавляли по 50 мкл реактива Несслера, разведенного в 5 раз дистиллированной водой непосредственно перед анализом. Выдерживали 2 минуты при комнатной температуре. Концентрацию NH4+ определяли в каждом растворе методом абсорбционной спектроскопии (λmax = 450 нм) с использованием планшетного считывателя.
Калибровочную кривую строили в программе Excel, где по оси абсцисс (х) откладывали значения оптической плотности, а по оси ординат (y) - значения концентраций ионов аммония. Для определения концентрации NH4+ в мкмоль/мл реакционной смеси (Х) в образцах использовали уравнение калибровочной кривой.
Для определения концентрации NH4+ в мкмоль/мин*мл (МЕ/мл) фермента (С МЕ/мл) в образцах использовали уравнение:
С МЕ/мл = Х*А*50*15/T, где
Х - концентрация NH4+ в мкмоль/мл реакционной смеси;
А - коэффициент, учитывающий предварительное разведение фермента до концентрации 0,02-0,1 мг/мл;
50 - коэффициент пересчета на 1 мл фермента;
15 - коэффициент, учитывающий разведение фермента реакционной смесью;
Т - время инкубации при 37°C.
Для определения концентрации NH4+ в мкмоль/мин*мг (МЕ/мг) фермента (С МЕ/мг) использовали уравнение:
С МЕ/мг = С МЕ/мг /Сб, где
С МЕ/мг - концентрации NH4+ в мкмоль/мин*мл (МЕ/мл) фермента,
Сб - концентрация белка (аспарагиназы) в мг/мл.
Пример 4. Получение штаммов-продуцентов E. coli BL21[DE3]/pET28a-AsnWT и E. coli BL21[DE3]/pET28a-AsnSYN, локализация целевого белка
Для получения штаммов-продуцентов L-аспарагиназы Escherichia coli II типа, конструкции, полученные по примерам 1 и 2, использовали для трансформации компетентных клеток Escherichia coli BL21[DE3] (с генотипом F- ompT hsdSB (r-m-) gal dcm [DE3]).
Для получения штамма E. coli BL21[DE3]/pET28a-AsnWT - клетки штамма E. coli BL21[DE3] трансформировали экспрессионной плазмидой pET28a-AsnWT.
Для получения штамма E. coli BL21[DE3]/pET28a-AsnSYN - клетки штамма E. coli BL21[DE3] трансформировали экспрессионной плазмидой pET28a-AsnSYN с помощью метода химической трансформации. На 100 мкл замороженной суспензии компетентных клеток E. coli использовали 10 мкл лигазной смеси или 100 нг плазмиды, инкубировали на льду 30 минут для сорбции плазмидной ДНК, затем проводили тепловой шок, нагревая пробирки с бактериями при 42°С в течение 45 секунд, после пробирки охлаждали на льду 5 минут. После добавления 1 мл стерильной среды SOВ пробирки с трансформированными бактериями инкубировали 1 час при 37°С. Трансформированные клетки переносили на чашку Петри с твердой селективной агаризованной средой и инкубировали их в термостате при 37°С в течении ночи.
Трансформанты E. coli высевали на агаризованную среду 2хYT с добавлением глюкозы до 1% и селективного антибиотика - 50 мкг/мл канамицина. Отдельные колонии с типичным фенотипом подращивали в питательном бульоне 2xYT с 50 мкг/мл канамицина в течение 14 часов при температуре 37°C, инокулировали новую порцию питательной среды Terrific broth с 50 мкг/мл канамицина в соотношении 1:100, растили культуру до достижения оптической плотности 1-1,2 O.E., уменьшали температуру культивации до 30°C, индуцировали изопропилтио-p-D-галактозидом 1 мМ и культивировали еще 21 ч, отобрав аликвоты суспензии после 3 ч индукции. Осадок клеток отделяли от среды центрифугированием, ресуспендировали клетки в растворе 10 мМ трис-HCl, 2 мМ ЭДТА-Na, 0.1% Тритона-Х100, 10 мкг/мл лизоцима в 1/10 от исходного объема суспензии, выдерживали суспензию 30 мин на льду и проводили разрушение клеток ультразвуковым диспергатором до исчезновения видимой вязкости суспензии. Отбирали образцы для электрофоретического анализа и измерения ферментативной активности аспарагиназы, разделяли в них растворимую и нерастворимую фракцию белков центрифугированием в микроцентрифуге, дополнительно ресуспендировали осадок в том же растворе и осаждали центрифугированием. Результаты электрофоретического анализа тотального белка и белковых фракций для штамма BL21[DE3]/pET28a-AsnSYN приведены на Фиг. 9. Электрофоретическая подвижность целевого белка соответствует расчетным значениям. По данным гель-электрофореза белковых фракций целевой белок практически полностью локализован в растворимой фракции белков для обоих вариантов штамма.
Содержание ферментативно активной аспарагиназы было достоверно выше в случае штамма BL21[DE3]/pET28a-AsnSYN на 3 ч индукции и несколько выше на 21 ч индукции, данные приведены на Фиг. 8. Конечное содержание ферментативно активной аспарагиназы для штамма BL21[DE3]/pET28a-AsnSYN составляло 291±9 мг/л. Удельное содержание ферментативно активной аспарагиназы для штамма BL21[DE3]/pET28a-AsnSYN составляло 34,2 мг/(л*ОЕ) для 3 ч индукции; 44,5±2,6 мг/(л*ОЕ) для 21 ч индукции. В случае штамма BL21[DE3]/pET28a-AsnWT удельное содержание ферментативно активной аспарагиназы составляло 15,7 мг/(л*ОЕ) для 3 ч индукции и 38,4±1,6 мг/(л*ОЕ) для 3 и 21 ч индукции, что на 46% ниже, чем для синтетического гена на 3 ч индукции и на 14% меньше на 21 ч индукции.
Пример 5. Выделение и очистка L-аспарагиназы Escherichia coli II типа
Культивировали штамм-продуцент BL21[DE3]/pET28a-AsnSYN и проводили лизис клеток, как указано в Примере 4. К осветленному лизату добавляли раствор ацетата натрия, pH=4,5, до конечной концентрации 20 мМ, доводили pH уксусной кислотой до 4,5, инкубировали суспензию при перемешивании 30 мин при комнатной температуре и отделяли выпавший осадок балластного белка центрифугированием. Доводили pH супернатанта до 7,5 при помощи раствора гидроксида натрия, концентрировали раствор нейтрализованного супернатанта ультрафильтрацией до 1:40 от исходного объема культуры, используя мембрану с порогом отсечения 30 кДа из полиэфирсульфона (ПЭС). Затем проводили диафильтрацию при помощи воды очищенной до падения проводимости фильтрата ниже 0,5 мС/см.
К полученному раствору добавляли ацетат натрия, pH=4,5, до 10 мМ, доводили pH раствора до 4,5 уксусной кислотой, отделяли выпавший осадок центрифугированием. Осветленный концентрат наносили на колонку с сорбентом Capto S (Cytiva, США), уравновешенную раствором 20 мМ ацетата натрия, pH=4,5. Промывали колонку раствором 20 мМ ацетата натрия pH=4,5 до стабилизации базовой линии и элюировали аспрагиназу раствором 100 мМ NaCl, 20 мМ ацетата натрия, pH=4,5.
Обессоливали раствор полуочищенной аспарагиназы диафильтрацией, используя ПЭС мембрану с порогом отсечения 30 кДа и диафильтрующий раствор 20 мМ трис-HCl, pH=8,5. Обессоленный раствор наносили на колонку Capto DEAE (Cytiva), уравновешенную тем же раствором, после нанесения колонку промывали раствором 20 мМ NaCl, 20 мМ Трис-HCl pH=8,5. Элюировали аспарагиназу раствором 100 мМ NaCl, 20 мМ Трис-HCl pH=8,5.
К полученному элюату добавляли сухой сульфат магния до конечной концентрации 250 мМ. Пропускали полученный раствор через колонку с сорбентом Phenyl Sepharose high substitution (Cytiva), уравновешенную раствором 20 мМ трис-HCl, pH=7,5, 250 мМ MgSO4. Собирали фракцию не связавшихся с сорбентом белков (проскок), вносили фосфат калия pH=7,5 до 1 мМ и наносили на колонку с сорбентом Ceramic hydroxyapatite type I (Bio-Rad, США), уравновешенную раствором 100 мМ NaCl, 5 мМ фосфата калия, pH=7,5. После нанесения промывали колонку раствором 100 мМ NaCl, 5 мМ фосфата калия, pH=7,5, до стабилизации базовой линии и элюируют аспарагиназу раствором 100 мМ NaCl, 50 мМ фосфата калия, pH=7,5.
Выход очищенной таким способом аспарагиназы составлял не менее 25% по данным измерения ферментативной активности, удельная активность составляла более 250 МЕ/мг. Полученный препарат аспарагиназы не содержал видимых белковых примесей по данным ДСН-ПААГ (Фиг. 10). Препарат содержал менее 1% примеси мономерной аспарагиназы и менее 3% примеси мультимера аспарагиназы по данным гель-фильтрации (Фиг. 11).
Пример 6. Получение готовой лекарственной формы аспарагиназы E. coli
Полученный по условиям примера 5 раствор очищенной аспарагиназы обессоливали диафильтрацией на ПЭС мембране с порогом отсечения 30 кДа при помощи 14 диафильтрующих объемов воды для инъекций, концентрировали ультрафильтрацией до конечной концентрации 15-20 мг/мл, формулировали внесением сахарозы до 100 г/л, доводили до концентрации аспарагиназы 10 мг/мл при помощи раствора 100 г/л сахарозы в воде для инъекций, проводили стерильную фильтрацию полученного готового раствора, стерильно разливали раствор в стеклянные лиофилизационные флаконы вместимостью 25 мл по 4 мл и проводили лиофилизацию до достижения постоянной массы лиофилизата. Флаконы закрывали резиновыми пробками и укупоривали алюминиевыми колпачками.
Приведенные результаты показали, что использование синтетического гена, кодируемого оптимальными для E. coli кодонами, существенно увеличивает уровень экспрессии аспарагиназы II типа в E. coli BL21[DE3] по сравнению с природным геном, при этом накопление целевого белка в нерастворимой форме, как с непроцессированным лидерным пептидом, так и после отделения лидерного пептида, практически не происходит.
Дополнительно, преимущества предлагаемого штамма бактерии-реципиента E. coli BL21[DE3] заключаются, но не ограничиваются, использованием бактерий с фенотипом Lon OmpT, кодирующих в своей хромосоме такой же вариант аспарагиназы II типа, что и предлагаемая плазмида pET28a-AsnSYN, а также оригинальные лекарственные препараты аспарагиназы. Отсутствие протеаз lon и ompT в цитоплазме и периплазме E. coli может дополнительно уменьшить протеолитический распад целевого белка в процессе биосинтеза и выделения, а присутствие в бактериях собственной аспарагиназы II типа с такой же последовательностью, что и у кодируемой плазмидой аспарагиназы, дополнительно может позволить избежать появления в препарате очищенной аспарагиназы трудноотделяемой родственной примеси собственной аспарагиназы бактерии-реципиента с измененной относительно целевого белка аминокислотной последовательностью. Встроенный в геном штамма-реципиента ген РНК полимеразы бактериофага Т7 под контролем lacUV5 промотора при использовании T7-lac промотора и Т7 терминатора в плазмидах приводит к быстрой и эффективной продукции белка. При использовании другого индуцибельного промотора, например, tac-промотора, присутствия гена РНК полимеразы бактериофага Т7 под контролем lacUV5 промотора в геноме клеток-реципиентов не требуется, что не будет влиять на уровень биосинтеза L-аспарагиназы E.coli II типа.
Хотя указанное изобретение описано в деталях со ссылкой на Примеры, для специалиста в указанной области техники очевидно, что могут быть совершены различные изменения и произведены эквивалентные замены, и такие изменения и замены не выходят за рамки настоящего изобретения.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3"
fileName="Asparaginase_seq.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-06-27">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>-</ApplicationNumberText>
<FilingDate>2023-06-27</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>asparaginase_seq</ApplicantFileReference>
<ApplicantName languageCode="ru">ФГУП "Московский эндокринный
завод"</ApplicantName>
<ApplicantNameLatin>FGUP "Moskovskij endokrinnyj
zavod"</ApplicantNameLatin>
<InventorName languageCode="ru">Габидова Альфия Эркиновна
</InventorName>
<InventorNameLatin>Gabidova Alfiya Erkinovna</InventorNameLatin>
<InventionTitle languageCode="ru">ПРОДУЦЕНТ L-АСПАРАГИНАЗЫ E. COLI И
ЭКСПРЕССИОННАЯ ПЛАЗМИДА PET28A-ASNSYN, КОДИРУЮЩАЯ
L-АСПАРАГИНАЗУ</InventionTitle>
<SequenceTotalQuantity>13</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1047</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1047</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggaatttttcaaaaagacagcactagctgcgctggttatgggtttct
ctggtgcagcattggcccttcccaacattaccatcctggcgacgggtggcaccattgcgggtggcggtga
tagcgcgaccaagtccaattacaccgcggggaaagtcggcgtggagaacctcgtcaacgcggtgccacag
ctgaaagacatcgcgaacgtgaagggtgaacaggttgtgaacattggtagccaggacatgaatgacgacg
tgtggctcaccctggcgaagaaaatcaacacggactgtgataagaccgacggctttgtgattacccacgg
tactgacacgatggaagagacagcgtatttcctggacctgaccgttaagtgcgataagccggttgttatg
gttggtgctatgcgtccgagcaccagcatgagcgcggatggtccgttcaacctgtataacgcggttgtca
ccgcggcagataaagcaagcgccaaccgcggtgttctggtggttatgaacgataccgtgctggacggccg
tgatgttaccaagactaataccaccgacgtggccacatttaaatccgttaactacggtccgctgggttac
atccacaacggcaaaattgattatcagcgtaccccggctcgcaaacacacctctgacaccccgttcgacg
ttagcaaactgaacgagctgccgaaagtgggcatcgtgtataattacgcaaatgcgtccgacctgccggc
aaaggcgttggttgacgcgggctatgatggcattgtgtcagccggcgttggtaatggcaacttatacaaa
accgtgtttgataccttggcgacggcggctaagaacggtacggctgtggttcgttctagccgtgtcccga
ccggtgccactacgcaagatgccgaagttgatgacgctaagtacggcttcgtggcatcgggcaccttgaa
tccgcaaaaagcgcgcgtactgctgcagttggctctgacgcagaccaaggatccgcaacaaatccaacaa
atctttaatcagtactaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>348</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..348</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Escherichia coli</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MEFFKKTALAALVMGFSGAALALPNITILATGGTIAGGGDSATKSNYTA
GKVGVENLVNAVPQLKDIANVKGEQVVNIGSQDMNDDVWLTLAKKINTDCDKTDGFVITHGTDTMEETAY
FLDLTVKCDKPVVMVGAMRPSTSMSADGPFNLYNAVVTAADKASANRGVLVVMNDTVLDGRDVTKTNTTD
VATFKSVNYGPLGYIHNGKIDYQRTPARKHTSDTPFDVSKLNELPKVGIVYNYANASDLPAKALVDAGYD
GIVSAGVGNGNLYKTVFDTLATAAKNGTAVVRSSRVPTGATTQDAEVDDAKYGFVASGTLNPQKARVLLQ
LALTQTKDPQQIQQIFNQY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>6298</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6298</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcttctattagtactgattaaagatttgttggatttgttgcggatcct
tggtctgcgtcagagccaactgcagcagtacgcgcgctttttgcggattcaaggtgcccgatgccacgaa
gccgtacttagcgtcatcaacttcggcatcttgcgtagtggcaccggtcgggacacggctagaacgaacc
acagccgtaccgttcttagccgccgtcgccaaggtatcaaacacggttttgtataagttgccattaccaa
cgccggctgacacaatgccatcatagcccgcgtcaaccaacgcctttgccggcaggtcggacgcatttgc
gtaattatacacgatgcccactttcggcagctcgttcagtttgctaacgtcgaacggggtgtcagaggtg
tgtttgcgagccggggtacgctgataatcaattttgccgttgtggatgtaacccagcggaccgtagttaa
cggatttaaatgtggccacgtcggtggtattagtcttggtaacatcacggccgtccagcacggtatcgtt
cataaccaccagaacaccgcggttggcgcttgctttatctgccgcggtgacaaccgcgttatacaggttg
aacggaccatccgcgctcatgctggtgctcggacgcatagcaccaaccataacaaccggcttatcgcact
taacggtcaggtccaggaaatacgctgtctcttccatcgtgtcagtaccgtgggtaatcacaaagccgtc
ggtcttatcacagtccgtgttgattttcttcgccagggtgagccacacgtcgtcattcatgtcctggcta
ccaatgttcacaacctgttcacccttcacgttcgcgatgtctttcagctgtggcaccgcgttgacgaggt
tctccacgccgactttccccgcggtgtaattggacttggtcgcgctatcaccgccacccgcaatggtgcc
acccgtcgccaggatggtaatgttgggaagggccaatgctgcaccagagaaacccataaccagcgcagct
agtgctgtctttttgaaaaattccatggtatatctccttcttaaagttaaacaaaattatttctagaggg
gaattgttatccgctcacaattcccctatagtgagtcgtattaatttcgcgggatcgagatctcgatcct
ctacgccggacgcatcgtggccggcatcaccggcgccacaggtgcggttgctggcgcctatatcgccgac
atcaccgatggggaagatcgggctcgccacttcgggctcatgagcgcttgtttcggcgtgggtatggtgg
caggccccgtggccgggggactgttgggcgccatctccttgcatgcaccattccttgcggcggcggtgct
caacggcctcaacctactactgggctgcttcctaatgcaggagtcgcataagggagagcgtcgagatccc
ggacaccatcgaatggcgcaaaacctttcgcggtatggcatgatagcgcccggaagagagtcaattcagg
gtggtgaatgtgaaaccagtaacgttatacgatgtcgcagagtatgccggtgtctcttatcagaccgttt
cccgcgtggtgaaccaggccagccacgtttctgcgaaaacgcgggaaaaagtggaagcggcgatggcgga
gctgaattacattcccaaccgcgtggcacaacaactggcgggcaaacagtcgttgctgattggcgttgcc
acctccagtctggccctgcacgcgccgtcgcaaattgtcgcggcgattaaatctcgcgccgatcaactgg
gtgccagcgtggtggtgtcgatggtagaacgaagcggcgtcgaagcctgtaaagcggcggtgcacaatct
tctcgcgcaacgcgtcagtgggctgatcattaactatccgctggatgaccaggatgccattgctgtggaa
gctgcctgcactaatgttccggcgttatttcttgatgtctctgaccagacacccatcaacagtattattt
tctcccatgaagacggtacgcgactgggcgtggagcatctggtcgcattgggtcaccagcaaatcgcgct
gttagcgggcccattaagttctgtctcggcgcgtctgcgtctggctggctggcataaatatctcactcgc
aatcaaattcagccgatagcggaacgggaaggcgactggagtgccatgtccggttttcaacaaaccatgc
aaatgctgaatgagggcatcgttcccactgcgatgctggttgccaacgatcagatggcgctgggcgcaat
gcgcgccattaccgagtccgggctgcgcgttggtgcggatatctcggtagtgggatacgacgataccgaa
gacagctcatgttatatcccgccgttaaccaccatcaaacaggattttcgcctgctggggcaaaccagcg
tggaccgcttgctgcaactctctcagggccaggcggtgaagggcaatcagctgttgcccgtctcactggt
gaaaagaaaaaccaccctggcgcccaatacgcaaaccgcctctccccgcgcgttggccgattcattaatg
cagctggcacgacaggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatgtaagttagctc
actcattaggcaccgggatctcgaccgatgcccttgagagccttcaacccagtcagctccttccggtggg
cgcggggcatgactatcgtcgccgcacttatgactgtcttctttatcatgcaactcgtaggacaggtgcc
ggcagcgctctgggtcattttcggcgaggaccgctttcgctggagcgcgacgatgatcggcctgtcgctt
gcggtattcggaatcttgcacgccctcgctcaagccttcgtcactggtcccgccaccaaacgtttcggcg
agaagcaggccattatcgccggcatggcggccccacgggtgcgcatgatcgtgctcctgtcgttgaggac
ccggctaggctggcggggttgccttactggttagcagaatgaatcaccgatacgcgagcgaacgtgaagc
gactgctgctgcaaaacgtctgcgacctgagcaacaacatgaatggtcttcggtttccgtgtttcgtaaa
gtctggaaacgcggaagtcagcgccctgcaccattatgttccggatctgcatcgcaggatgctgctggct
accctgtggaacacctacatctgtattaacgaagcgctggcattgaccctgagtgatttttctctggtcc
cgccgcatccataccgccagttgtttaccctcacaacgttccagtaaccgggcatgttcatcatcagtaa
cccgtatcgtgagcatcctctctcgtttcatcggtatcattacccccatgaacagaaatcccccttacac
ggaggcatcagtgaccaaacaggaaaaaaccgcccttaacatggcccgctttatcagaagccagacatta
acgcttctggagaaactcaacgagctggacgcggatgaacaggcagacatctgtgaatcgcttcacgacc
acgctgatgagctttaccgcagctgcctcgcgcgtttcggtgatgacggtgaaaacctctgacacatgca
gctcccggagacggtcacagcttgtctgtaagcggatgccgggagcagacaagcccgtcagggcgcgtca
gcgggtgttggcgggtgtcggggcgcagccatgacccagtcacgtagcgatagcggagtgtatactggct
taactatgcggcatcagagcagattgtactgagagtgcaccatatatgcggtgtgaaataccgcacagat
gcgtaaggagaaaataccgcatcaggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgt
tcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataac
gcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgt
ttttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaaccc
gacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctg
ccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgta
ggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccga
ccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggca
gcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggc
ctaactacggctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaa
aagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcag
cagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagt
ggaacgaaaactcacgttaagggattttggtcatgaacaataaaactgtctgcttacataaacagtaata
caaggggtgttatgagccatattcaacgggaaacgtcttgctctaggccgcgattaaattccaacatgga
tgctgatttatatgggtataaatgggctcgcgataatgtcgggcaatcaggtgcgacaatctatcgattg
tatgggaagcccgatgcgccagagttgtttctgaaacatggcaaaggtagcgttgccaatgatgttacag
atgagatggtcagactaaactggctgacggaatttatgcctcttccgaccatcaagcattttatccgtac
tcctgatgatgcatggttactcaccactgcgatccccgggaaaacagcattccaggtattagaagaatat
cctgattcaggtgaaaatattgttgatgcgctggcagtgttcctgcgccggttgcattcgattcctgttt
gtaattgtccttttaacagcgatcgcgtatttcgtctcgctcaggcgcaatcacgaatgaataacggttt
ggttgatgcgagtgattttgatgacgagcgtaatggctggcctgttgaacaagtctggaaagaaatgcat
aaacttttgccattctcaccggattcagtcgtcactcatggtgatttctcacttgataaccttatttttg
acgaggggaaattaataggttgtattgatgttggacgagtcggaatcgcagaccgataccaggatcttgc
catcctatggaactgcctcggtgagttttctccttcattacagaaacggctttttcaaaaatatggtatt
gataatcctgatatgaataaattgcagtttcatttgatgctcgatgagtttttctaagaattaattcatg
agcggatacatatttgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaag
tgccacctgaaattgtaaacgttaatattttgttaaaattcgcgttaaatttttgttaaatcagctcatt
ttttaaccaataggccgaaatcggcaaaatcccttataaatcaaaagaatagaccgagatagggttgagt
gttgttccagtttggaacaagagtccactattaaagaacgtggactccaacgtcaaagggcgaaaaaccg
tctatcagggcgatggcccactacgtgaaccatcaccctaatcaagttttttggggtcgaggtgccgtaa
agcactaaatcggaaccctaaagggagcccccgatttagagcttgacggggaaagccggcgaacgtggcg
agaaaggaagggaagaaagcgaaaggagcgggcgctagggcgctggcaagtgtagcggtcacgctgcgcg
taaccaccacacccgccgcgcttaatgcgccgctacagggcgcgtcccattcgccaatccggatatagtt
cctcctttcagcaaaaaacccctcaagacccgtttagaggccccaaggggttatgctagttattgctcag
cggtggcagcagccaactcagcttcctttcgggctttgttagcagccggatctcagtggtggtggtggtg
gtgctcgagtgcggccgca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>30</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..30</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accatggagtttttcaaaaagacggcactt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taagcttctattagtactgattgaagatctgctgg</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>1061</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1061</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accatggagtttttcaaaaagacggcacttgccgcactggttatgggtt
ttagtggtgcagcattggcattacccaatatcaccattttagcaaccggcgggaccattgccggtggtgg
tgactccgcaaccaaatctaactacacagcgggtaaagttggcgtagaaaatctggttaatgcggtgccg
caactgaaggacattgcgaacgttaaaggcgagcaggtagtgaatattggctcccaggacatgaacgatg
atgtctggctgacactggcgaaaaaaattaacaccgactgcgataaaactgacggcttcgtcattaccca
cggtaccgacacgatggaagaaaccgcttacttcctcgacctgacggtgaaatgcgacaaaccggtggtg
atggtcggtgcaatgcgtccgtccacgtctatgagcgcagacggtccattcaacctgtataacgcggtag
tgactgcagctgataaagcctccgctaatcgtggcgtactggtagtgatgaacgacaccgtgcttgatgg
ccgtgatgtcaccaaaaccaacaccaccgatgtagcgaccttcaagtctgttaactacggtcctctgggt
tacattcacaacggtaagattgactaccaacgtaccccggcacgtaagcacaccagcgacacgccgttcg
atgtctctaagctgaatgaactgccgaaagtcggcattgtttataactacgctaacgcatccgatcttcc
ggctaaagcactggtagatgcgggctatgatggcatcgttagcgctggcgtgggtaacggcaacctgtat
aaaaccgtatttgacacccttgcaaccgctgcgaaaaacggcactgcagtagtgcgttcttcccgcgtac
cgacgggcgctaccactcaggatgccgaagtggatgatgcgaaatacggcttcgtcgcctctggcacgtt
gaacccgcaaaaagcgcgcgttctgctgcaactggctctgacgcaaactaaagatccgcagcagatccag
cagatcttcaatcagtactaatagaagcttac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gttgtaaacgacggccagtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggaaacagctatgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taatacgactcactataggg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctagttattgctcagcgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>6298</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..6298</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agcttctattagtactgattgaagatctgctggatctgctgcggatctt
tagtttgcgtcagagccagttgcagcagaacgcgcgctttttgcgggttcaacgtgccagaggcgacgaa
gccgtatttcgcatcatccacttcggcatcctgagtggtagcgcccgtcggtacgcgggaagaacgcact
actgcagtgccgtttttcgcagcggttgcaagggtgtcaaatacggttttatacaggttgccgttaccca
cgccagcgctaacgatgccatcatagcccgcatctaccagtgctttagccggaagatcggatgcgttagc
gtagttataaacaatgccgactttcggcagttcattcagcttagagacatcgaacggcgtgtcgctggtg
tgcttacgtgccggggtacgttggtagtcaatcttaccgttgtgaatgtaacccagaggaccgtagttaa
cagacttgaaggtcgctacatcggtggtgttggttttggtgacatcacggccatcaagcacggtgtcgtt
catcactaccagtacgccacgattagcggaggctttatcagctgcagtcactaccgcgttatacaggttg
aatggaccgtctgcgctcatagacgtggacggacgcattgcaccgaccatcaccaccggtttgtcgcatt
tcaccgtcaggtcgaggaagtaagcggtttcttccatcgtgtcggtaccgtgggtaatgacgaagccgtc
agttttatcgcagtcggtgttaatttttttcgccagtgtcagccagacatcatcgttcatgtcctgggag
ccaatattcactacctgctcgcctttaacgttcgcaatgtccttcagttgcggcaccgcattaaccagat
tttctacgccaactttacccgctgtgtagttagatttggttgcggagtcaccaccaccggcaatggtccc
gccggttgctaaaatggtgatattgggtaatgccaatgctgcaccactaaaacccataaccagtgcggca
agtgccgtctttttgaaaaactccatggtatatctccttcttaaagttaaacaaaattatttctagaggg
gaattgttatccgctcacaattcccctatagtgagtcgtattaatttcgcgggatcgagatctcgatcct
ctacgccggacgcatcgtggccggcatcaccggcgccacaggtgcggttgctggcgcctatatcgccgac
atcaccgatggggaagatcgggctcgccacttcgggctcatgagcgcttgtttcggcgtgggtatggtgg
caggccccgtggccgggggactgttgggcgccatctccttgcatgcaccattccttgcggcggcggtgct
caacggcctcaacctactactgggctgcttcctaatgcaggagtcgcataagggagagcgtcgagatccc
ggacaccatcgaatggcgcaaaacctttcgcggtatggcatgatagcgcccggaagagagtcaattcagg
gtggtgaatgtgaaaccagtaacgttatacgatgtcgcagagtatgccggtgtctcttatcagaccgttt
cccgcgtggtgaaccaggccagccacgtttctgcgaaaacgcgggaaaaagtggaagcggcgatggcgga
gctgaattacattcccaaccgcgtggcacaacaactggcgggcaaacagtcgttgctgattggcgttgcc
acctccagtctggccctgcacgcgccgtcgcaaattgtcgcggcgattaaatctcgcgccgatcaactgg
gtgccagcgtggtggtgtcgatggtagaacgaagcggcgtcgaagcctgtaaagcggcggtgcacaatct
tctcgcgcaacgcgtcagtgggctgatcattaactatccgctggatgaccaggatgccattgctgtggaa
gctgcctgcactaatgttccggcgttatttcttgatgtctctgaccagacacccatcaacagtattattt
tctcccatgaagacggtacgcgactgggcgtggagcatctggtcgcattgggtcaccagcaaatcgcgct
gttagcgggcccattaagttctgtctcggcgcgtctgcgtctggctggctggcataaatatctcactcgc
aatcaaattcagccgatagcggaacgggaaggcgactggagtgccatgtccggttttcaacaaaccatgc
aaatgctgaatgagggcatcgttcccactgcgatgctggttgccaacgatcagatggcgctgggcgcaat
gcgcgccattaccgagtccgggctgcgcgttggtgcggatatctcggtagtgggatacgacgataccgaa
gacagctcatgttatatcccgccgttaaccaccatcaaacaggattttcgcctgctggggcaaaccagcg
tggaccgcttgctgcaactctctcagggccaggcggtgaagggcaatcagctgttgcccgtctcactggt
gaaaagaaaaaccaccctggcgcccaatacgcaaaccgcctctccccgcgcgttggccgattcattaatg
cagctggcacgacaggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatgtaagttagctc
actcattaggcaccgggatctcgaccgatgcccttgagagccttcaacccagtcagctccttccggtggg
cgcggggcatgactatcgtcgccgcacttatgactgtcttctttatcatgcaactcgtaggacaggtgcc
ggcagcgctctgggtcattttcggcgaggaccgctttcgctggagcgcgacgatgatcggcctgtcgctt
gcggtattcggaatcttgcacgccctcgctcaagccttcgtcactggtcccgccaccaaacgtttcggcg
agaagcaggccattatcgccggcatggcggccccacgggtgcgcatgatcgtgctcctgtcgttgaggac
ccggctaggctggcggggttgccttactggttagcagaatgaatcaccgatacgcgagcgaacgtgaagc
gactgctgctgcaaaacgtctgcgacctgagcaacaacatgaatggtcttcggtttccgtgtttcgtaaa
gtctggaaacgcggaagtcagcgccctgcaccattatgttccggatctgcatcgcaggatgctgctggct
accctgtggaacacctacatctgtattaacgaagcgctggcattgaccctgagtgatttttctctggtcc
cgccgcatccataccgccagttgtttaccctcacaacgttccagtaaccgggcatgttcatcatcagtaa
cccgtatcgtgagcatcctctctcgtttcatcggtatcattacccccatgaacagaaatcccccttacac
ggaggcatcagtgaccaaacaggaaaaaaccgcccttaacatggcccgctttatcagaagccagacatta
acgcttctggagaaactcaacgagctggacgcggatgaacaggcagacatctgtgaatcgcttcacgacc
acgctgatgagctttaccgcagctgcctcgcgcgtttcggtgatgacggtgaaaacctctgacacatgca
gctcccggagacggtcacagcttgtctgtaagcggatgccgggagcagacaagcccgtcagggcgcgtca
gcgggtgttggcgggtgtcggggcgcagccatgacccagtcacgtagcgatagcggagtgtatactggct
taactatgcggcatcagagcagattgtactgagagtgcaccatatatgcggtgtgaaataccgcacagat
gcgtaaggagaaaataccgcatcaggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgt
tcggctgcggcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataac
gcaggaaagaacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgt
ttttccataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaaccc
gacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctg
ccgcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgta
ggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccga
ccgctgcgccttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggca
gcagccactggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggc
ctaactacggctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaa
aagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcag
cagattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagt
ggaacgaaaactcacgttaagggattttggtcatgaacaataaaactgtctgcttacataaacagtaata
caaggggtgttatgagccatattcaacgggaaacgtcttgctctaggccgcgattaaattccaacatgga
tgctgatttatatgggtataaatgggctcgcgataatgtcgggcaatcaggtgcgacaatctatcgattg
tatgggaagcccgatgcgccagagttgtttctgaaacatggcaaaggtagcgttgccaatgatgttacag
atgagatggtcagactaaactggctgacggaatttatgcctcttccgaccatcaagcattttatccgtac
tcctgatgatgcatggttactcaccactgcgatccccgggaaaacagcattccaggtattagaagaatat
cctgattcaggtgaaaatattgttgatgcgctggcagtgttcctgcgccggttgcattcgattcctgttt
gtaattgtccttttaacagcgatcgcgtatttcgtctcgctcaggcgcaatcacgaatgaataacggttt
ggttgatgcgagtgattttgatgacgagcgtaatggctggcctgttgaacaagtctggaaagaaatgcat
aaacttttgccattctcaccggattcagtcgtcactcatggtgatttctcacttgataaccttatttttg
acgaggggaaattaataggttgtattgatgttggacgagtcggaatcgcagaccgataccaggatcttgc
catcctatggaactgcctcggtgagttttctccttcattacagaaacggctttttcaaaaatatggtatt
gataatcctgatatgaataaattgcagtttcatttgatgctcgatgagtttttctaagaattaattcatg
agcggatacatatttgaatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaag
tgccacctgaaattgtaaacgttaatattttgttaaaattcgcgttaaatttttgttaaatcagctcatt
ttttaaccaataggccgaaatcggcaaaatcccttataaatcaaaagaatagaccgagatagggttgagt
gttgttccagtttggaacaagagtccactattaaagaacgtggactccaacgtcaaagggcgaaaaaccg
tctatcagggcgatggcccactacgtgaaccatcaccctaatcaagttttttggggtcgaggtgccgtaa
agcactaaatcggaaccctaaagggagcccccgatttagagcttgacggggaaagccggcgaacgtggcg
agaaaggaagggaagaaagcgaaaggagcgggcgctagggcgctggcaagtgtagcggtcacgctgcgcg
taaccaccacacccgccgcgcttaatgcgccgctacagggcgcgtcccattcgccaatccggatatagtt
cctcctttcagcaaaaaacccctcaagacccgtttagaggccccaaggggttatgctagttattgctcag
cggtggcagcagccaactcagcttcctttcgggctttgttagcagccggatctcagtggtggtggtggtg
gtgctcgagtgcggccgca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>1060</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1060</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accatggaatttttcaaaaagacagcactagctgcgctggttatgggtt
tctctggtgcagcattggcccttcccaacattaccatcctggcgacgggtggcaccattgcgggtggcgg
tgatagcgcgaccaagtccaattacaccgcggggaaagtcggcgtggagaacctcgtcaacgcggtgcca
cagctgaaagacatcgcgaacgtgaagggtgaacaggttgtgaacattggtagccaggacatgaatgacg
acgtgtggctcaccctggcgaagaaaatcaacacggactgtgataagaccgacggctttgtgattaccca
cggtactgacacgatggaagagacagcgtatttcctggacctgaccgttaagtgcgataagccggttgtt
atggttggtgctatgcgtccgagcaccagcatgagcgcggatggtccgttcaacctgtataacgcggttg
tcaccgcggcagataaagcaagcgccaaccgcggtgttctggtggttatgaacgataccgtgctggacgg
ccgtgatgttaccaagactaataccaccgacgtggccacatttaaatccgttaactacggtccgctgggt
tacatccacaacggcaaaattgattatcagcgtaccccggctcgcaaacacacctctgacaccccgttcg
acgttagcaaactgaacgagctgccgaaagtgggcatcgtgtataattacgcaaatgcgtccgacctgcc
ggcaaaggcgttggttgacgcgggctatgatggcattgtgtcagccggcgttggtaatggcaacttatac
aaaaccgtgtttgataccttggcgacggcggctaagaacggtacggctgtggttcgttctagccgtgtcc
cgaccggtgccactacgcaagatgccgaagttgatgacgctaagtacggcttcgtggcatcgggcacctt
gaatccgcaaaaagcgcgcgtactgctgcagttggctctgacgcagaccaaggatccgcaacaaatccaa
caaatctttaatcagtactaatagaagctta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgtccagcacggtatcgttcat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
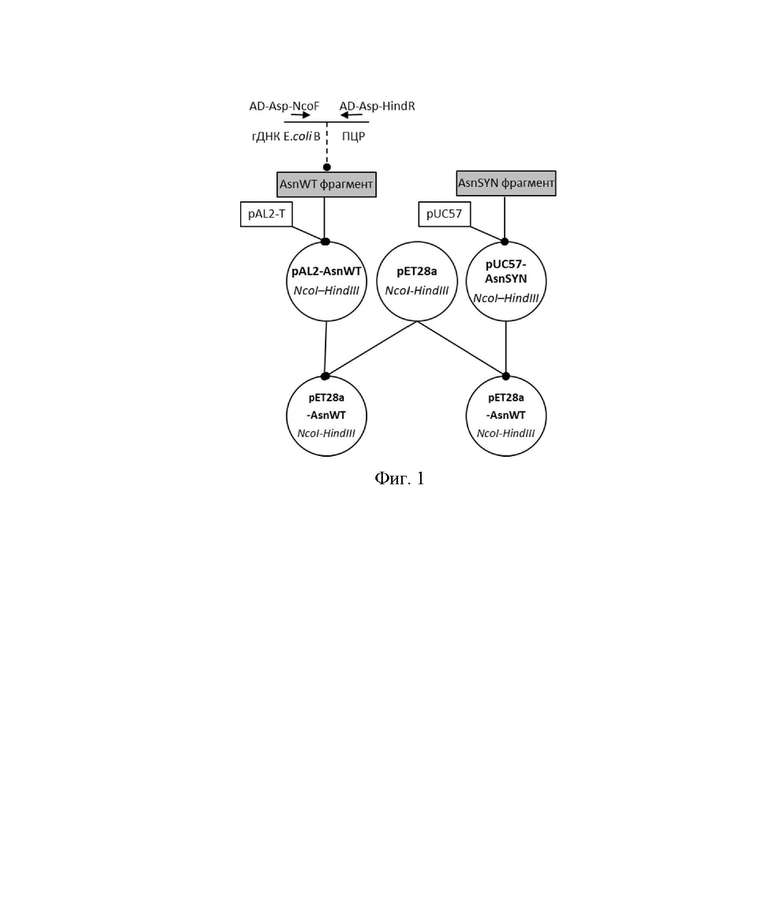
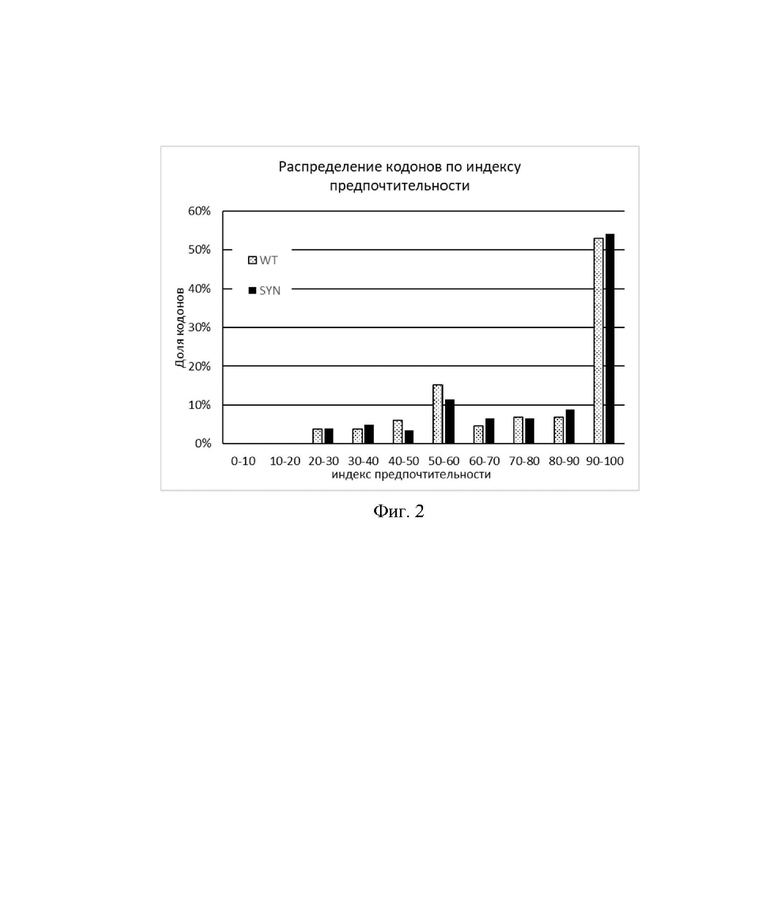
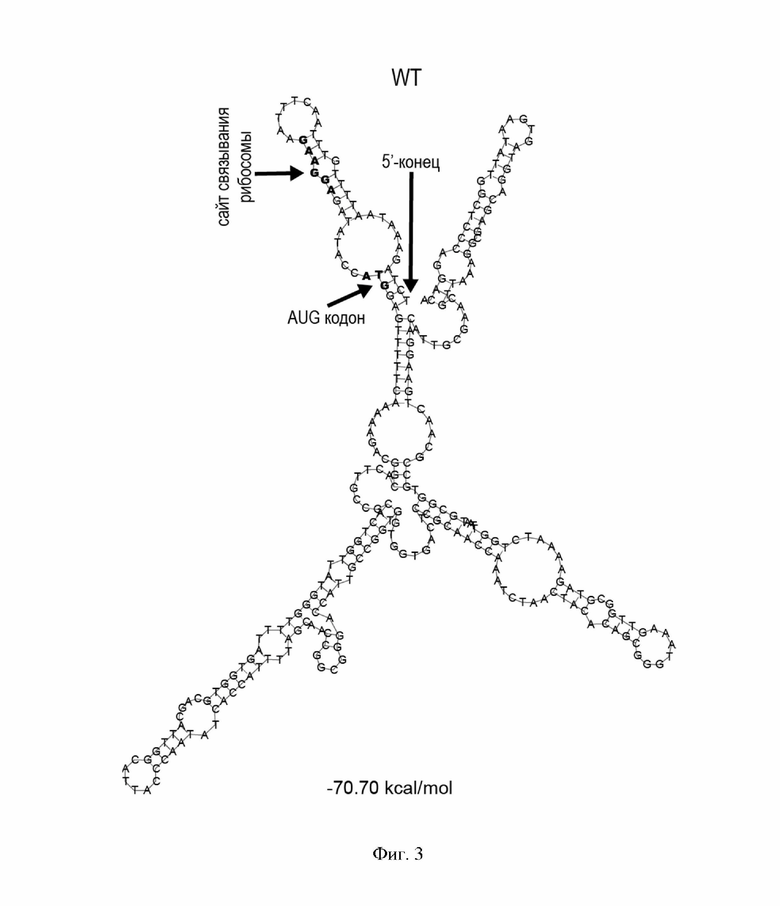
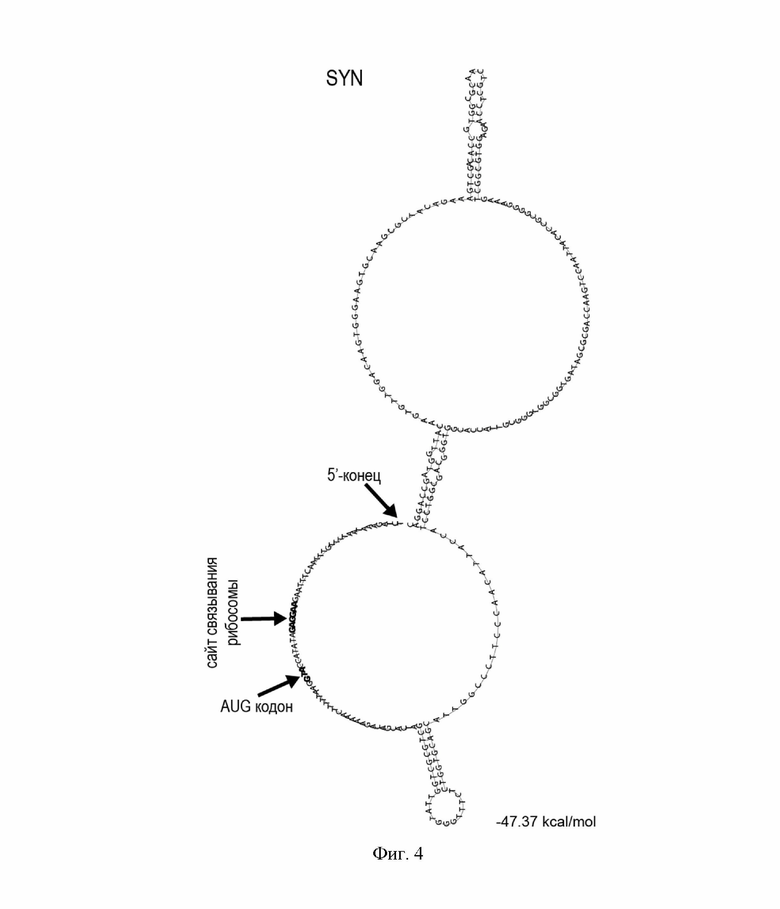
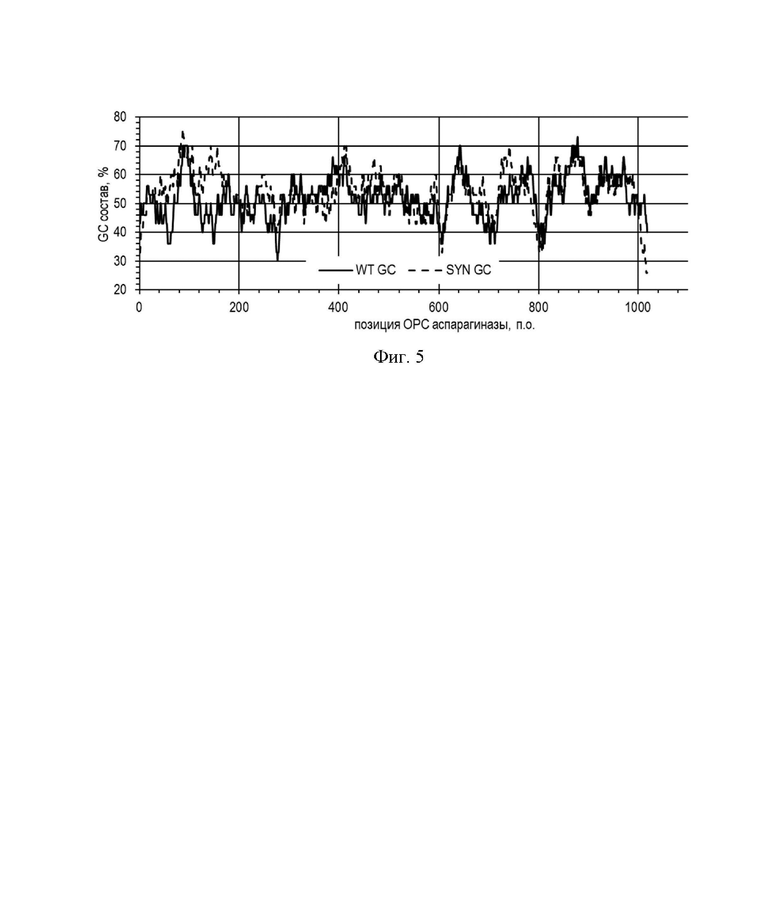
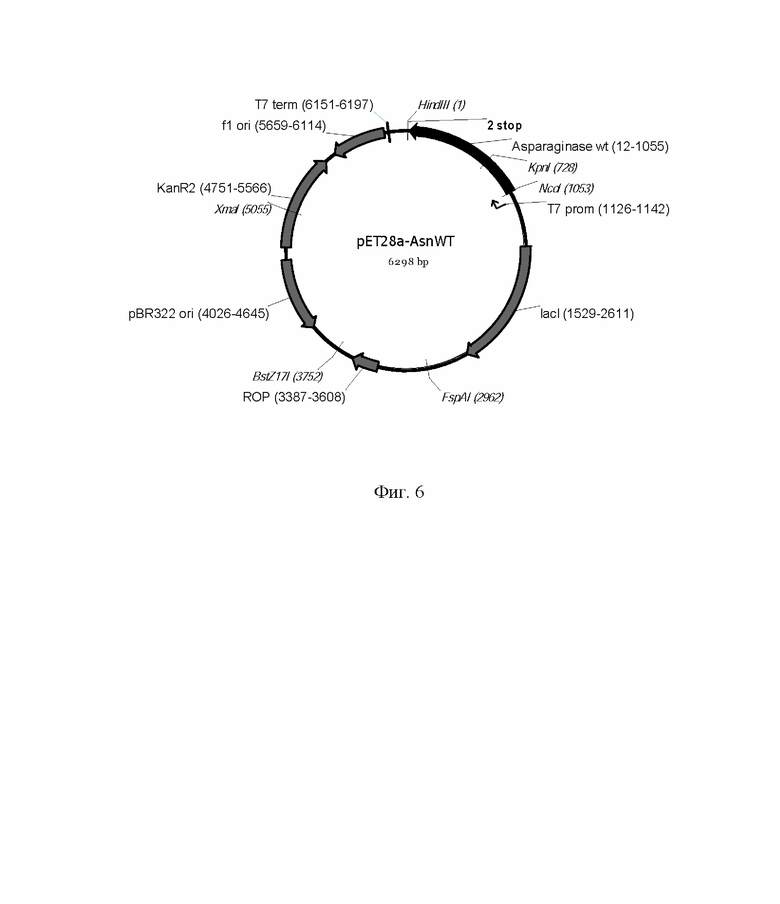
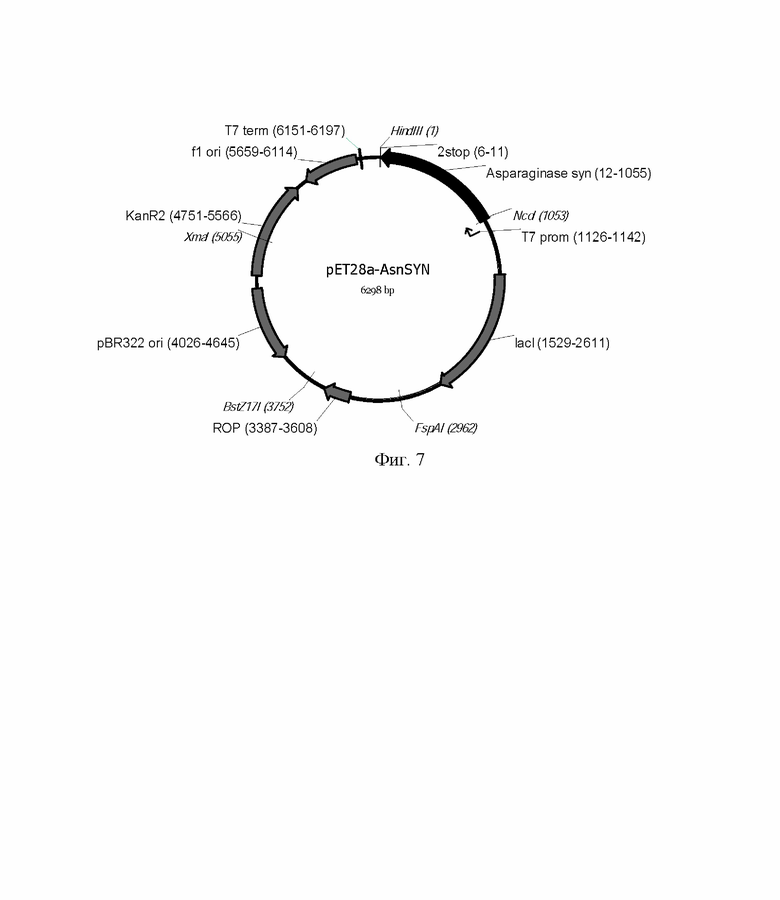
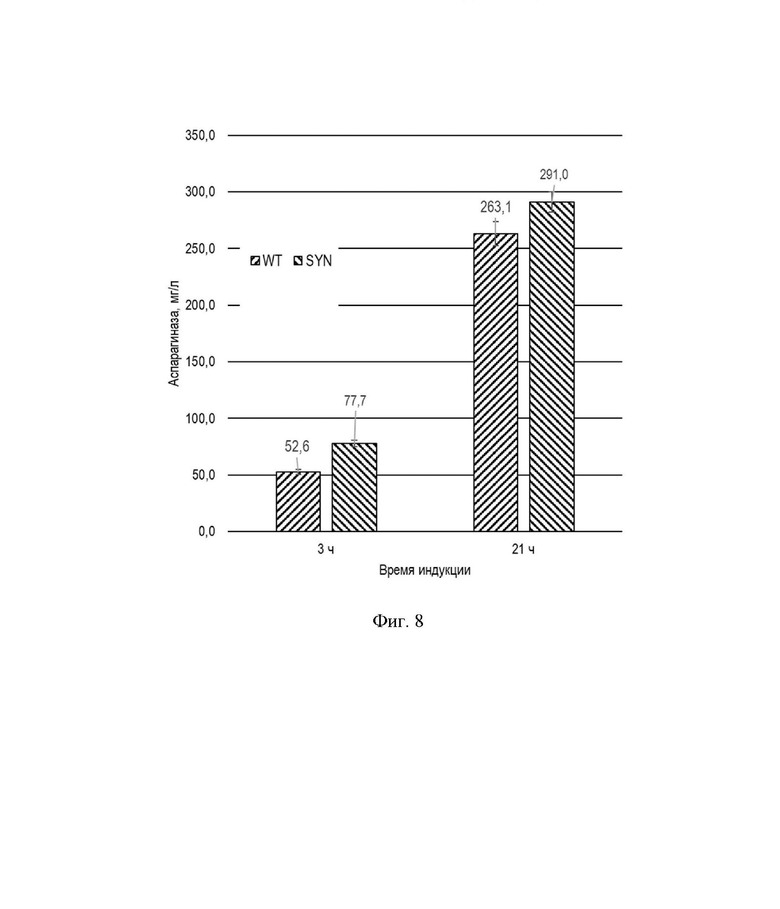
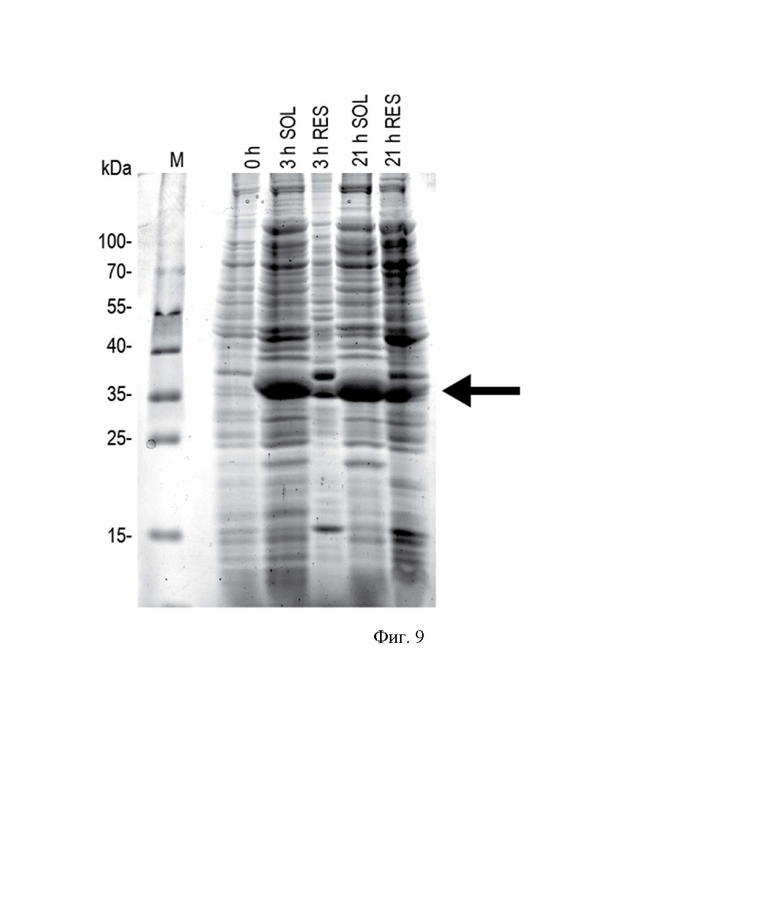
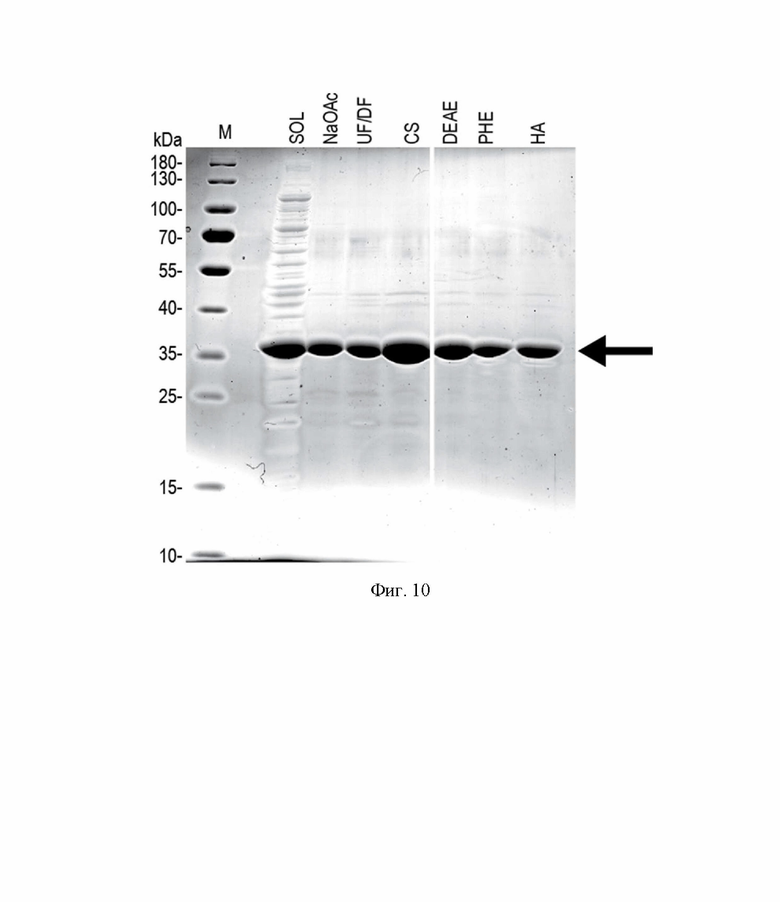
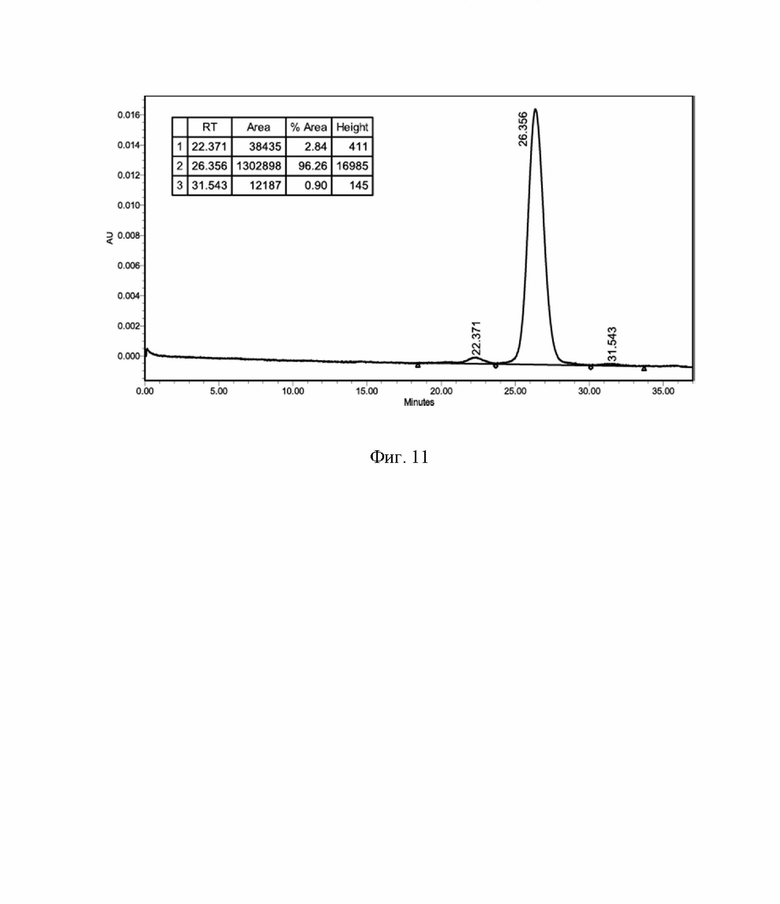
Изобретение относится к биотехнологии. Предложена экспрессионная плазмида pET28a-AsnSYN, предназначенная для синтеза L- аспарагиназы и имеющая последовательность SEQ ID NO: 3, содержащую фрагмент ДНК, кодирующий L-аспарагиназу Escherichia coli II типа. Также предложена бактерия, принадлежащая к роду Escherichia, трансформированная указанной плазмидой и предназначенная для синтеза L-аспарагиназы Escherichia coli II типа. Изобретение обеспечивает увеличение количества аспарагиназы на единицу объема ферментера и на килограмм биомассы E. coli, а также получение аспарагиназы с высоким выходом и чистотой. 2 н. и 1 з.п. ф-лы, 11 ил., 6 пр.
1. Экспрессионная плазмида pET28a-AsnSYN, предназначенная для синтеза L-аспарагиназы и имеющая последовательность SEQ ID NO: 3, содержащую фрагмент ДНК, кодирующий L-аспарагиназу Escherichia coli II типа.
2. Бактерия, принадлежащая к роду Escherichia, трансформированная плазмидой по п. 1, предназначенная для синтеза L-аспарагиназы Escherichia coli II типа.
3. Бактерия по п. 2, отличающаяся тем, что указанная бактерия относится к штамму Escherichia coli BL21[DE3]/pET28a-AsnSYN.
| РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК pL-ASP-08 И ШТАММ БАКТЕРИЙ Escherichia coli XL1-blue/pL-ASP-08 - ПРОДУЦЕНТ L-АСПАРАГИНАЗЫ | 2009 |
|
RU2397248C1 |
| РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК PACYCLANS ДЛЯ ПЕРЕНОСА И ЭКСПРЕССИИ В КЛЕТКАХ ESCHERICHIA COLI ГЕНА L-АСПАРАГИНАЗЫ ERWINIA CAROTOVORA (ECAR-LANS) И СПОСОБ ПОЛУЧЕНИЯ РЕКОМБИНАНТНОЙ ECAR-LANS ИЗ БИОМАССЫ ШТАММА E.COLI-ПРОДУЦЕНТА | 2002 |
|
RU2224797C2 |
| US 20160348085 A1, 28.10.2015 | |||
| Устройство для обеспыливания таблеток | 1980 |
|
SU939135A1 |
| Synthetic construct L-asparaginase II gene, complete cds | |||
| Устройство для обеспыливания таблеток | 1980 |
|
SU939135A1 |
Авторы
Даты
2024-04-22—Публикация
2023-06-27—Подача