Область техники
Настоящее изобретение относится к области генной инженерии, а именно к штамму-продуценту фермента ДНК-зависимой РНК-полимеразы E. coli. В частности, настоящее изобретение относится к рекомбинантному штамму E. coli BL21-T7RNA-P, предназначенному для экспрессии фермента ДНК-зависимой РНК-полимеразы фага Т7, необходимого транскрипции при in vitro синтезе РНК, обеспечивающему индуцируемую высокую экспрессию целевого белка и возможность его хроматографической очистки для дальнейшего промышленного и лабораторного применения. В другом варианте осуществления изобретение относится к рекомбинантной плазмиде pET28-T7RNAP-N6His для трансформации рекомбинантного штамма-продуцента. В еще одном варианте осуществления изобретение относится к применению рекомбинантного штамма-продуцента для in vitro синтеза РНК.
Уровень техники
ДНК-зависимая РНК-полимераза (RNAP) - основной фермент транскрипции ДНК в РНК, компонент «вилки репликации», где транскрибируются все виды РНК. Он представляет собой высоко-консервативный у прокариот и эукариот пяти-субъединичный фермент, состоящий из двух альфа-(α) субъединиц по 36 кДа каждая, одной бета-(β) субъединицы 150 кДа, бета-прайм-(β') субъединицы 155 кДа и самой маленькой омега-(ω) субъединицы. Клонирование гена ДНК-зависимой РНК-полимеразы впервые было сделано в 1978 г. из мутантов фага Т7 с его промотора [1]. Матрицей для современного клонирования РНК-полимеразы обычно служат синтетические последовательности ДНК.
Ген РНК-полимеразы активно используют для изучения антибиотикорезистентности бактерий. Продукцию РНК-полимеразы из штаммов E. coli использовали для изучения способности фермента транскрибировать мутантные гены с библиотек ДНК, в реакции ПЦР оценивали точность последовательности [2]. В ряде работ сравнивали транскрибирующую активность монопептидной РНК полимеразы из фага Т7 с наработкой в Bacillus subtilis с мульти-2αββ'ώ-субъединичной «классической» РНК полимеразой [3].
Светлов В. с коллегами раскрывают методы очистки бактериальной РНК-полимеразы [4]. В работе дано сравнение характеристик эффективности сборки 5-субъединичного фермента в разных экспрессионных векторах для продукции в E. сoli и методов его хроматографической очистки.
Основное назначение применения ДНК-зависимой РНК-полимеразы - точное транскрибирование генов целевых белков в мРНК, которая может стать матрицей для трансляции, например, при получении рекомбинантных вакцин на основе РНК-вирусов. Примером могут служить вакцины против бешенства. Ген поверхностного G-белка РНК-вируса бешенства клонируется в дефектный по инфекционной активности вектор лабораторного штамма вируса по технологии обратной транскрипции РНК-полимеразой.
В ряде перешедших в общественное достояние патентов описано применение ДНК-зависимой РНК-полимеразы для получения репортерных фрагментов РНК для метода гибридизации [5,6]. Практическое применение этих фрагментов с градиентом концентрации широко используется в наиболее распространенном современном методе клинической диагностики - ПЦР обратной транскрипции в реальном времени для количественного определения экспрессии генов [7].
Японскими авторами изложен способ изготовления тест-системы для ОТ-ПЦР с фото-деградируемыми репликами стандартов нуклеиновых кислот при помощи ряда ферментов репликации и обратной транскрипции, включая ДНК-зависимую РНК-полимеразу. Ферменты авторы изготавливают сами. При этом фрагменты РНК-стандартов синтезируются таким образом, что они защищены (кэпированы) на 3’-конце фото-чувствительным аденин-монофосфатом, и зонды вместо флуоресцентной метки содержат фото-чувствительно-меченные нуклеотиды [8].
Продолжаются исследования по созданию мутантных видов Т7-ДНК-зависимой РНК-полимеразы [9] с целью производства фермента, способного реплицировать РНК-стандарты с модифицированными нуклеотидами в своём составе. В патенте H. Zhang от 2017 года [9] описаны способ получения мутантной Т7-ДНК-зависимой РНК-полимеразы, способной синтезировать РНК-реплики с одним или несколькими химически измененными нуклеотидами.
Ближайшим аналогом настоящего изобретения является рекомбинантная плазмида для прокариотической экспрессии белка в штамме E. coli BL21 и рекомбинантный штамм-продуцент E. coli BL21-FCoV-S, разработанная учеными из университета Лонгъян и описанная ими в китайской патентной заявке CN 113072626 A. В документе раскрывается способ получения рекомбинантного S-белка кошачьего коронавируса, который включает следующие стадии: (а) проведение ПЦР амплификация путем взятия гена S-белка коронавируса кошек в качестве матрицы и клонирования фрагмента рекомбинантного S-белка; (b) проведение электрофоретического разделения фрагмента рекомбинантного S-белка, выделение и очистка, соединение с вектором и трансформация в компетентные клетки; (c) лигирование плазмиды с прокариотическим вектором экспрессии для получения рекомбинантной плазмиды pET28a-FCoV-S и (d) трансформацию рекомбинантной плазмиды в BL21 для прокариотической экспрессии с получением рекомбинантного S-белка кошачьего коронавируса.
Настоящее изобретение отличается тем, что коммерчески доступный экспрессионный вектор pET28 в составе рекомбинантной плазмиды pET28-T7RNAP-N6His трансформируется в штамм E. coli BL21 с получением рекомбинантного штамма-продуцента BL21-T7RNA-P, обеспечивающего индуцируемую высокую экспрессию целевого белка и возможность его хроматографической очистки для дальнейшего промышленного и лабораторного применения.
Создание отечественного высокоэффективного штамма-продуцента ДНК-зависимой РНК-полимеразы и её получение в диагностических и производственных лабораториях и промышленности продолжает оставаться актуальной задачей.
Краткое описание изобретения
Настоящее изобретение в общем смысле относится к области генной инженерии и в частности к штамму-продуценту фермента ДНК-зависимой РНК-полимеразы E. coli. Описанные в настоящем изобретении генетические конструкции могут быть использованы для in vitro синтеза РНК.
В частности, в настоящем изобретении описан рекомбинантный штамм-продуцент фермента ДНК-зависимой РНК-полимеразы E. coli BL21-T7RNA-P, отличающийся тем, что конструкция обеспечивает воспроизводимое с постоянными параметрами эффективное встраивание гена ДНК-зависимой РНК-полимеразы при трансформации, индуцируемую высокую экспрессию целевого белка и возможность его тонкой хроматографической очистки для дальнейшего промышленного и лабораторного применения.
Предложенный рекомбинантный штамм может применяться для осуществления работ по разработке мРНК-вакцины против бактериальных инфекций для воспроизведения одного из основных ферментов для in vitro синтеза РНК - ДНК-зависимой РНК-полимеразы.
Также описана рекомбинантная плазмида pET28-T7RNAP-N6His для трансформации рекомбинантного штамма-продуцента, необходимого для получения раскрываемого рекомбинантного штамма-продуцента BL21-T7RNA-P.
Также описано применение рекомбинантного штамма-продуцента E. coli BL21-T7RNA-P для in vitro синтеза РНК.
Краткое описание графических материалов
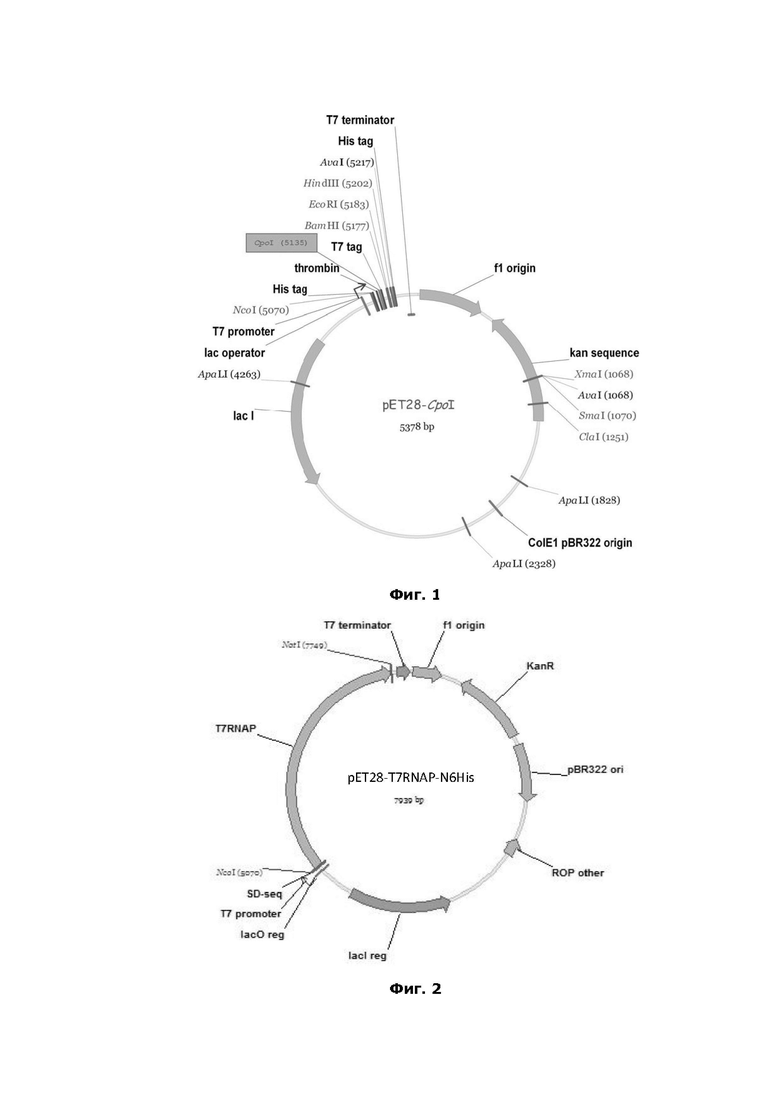
На фиг. 1 показаны карта генетической конструкции для продукции pET28 в штамме-продуценте E. coli.
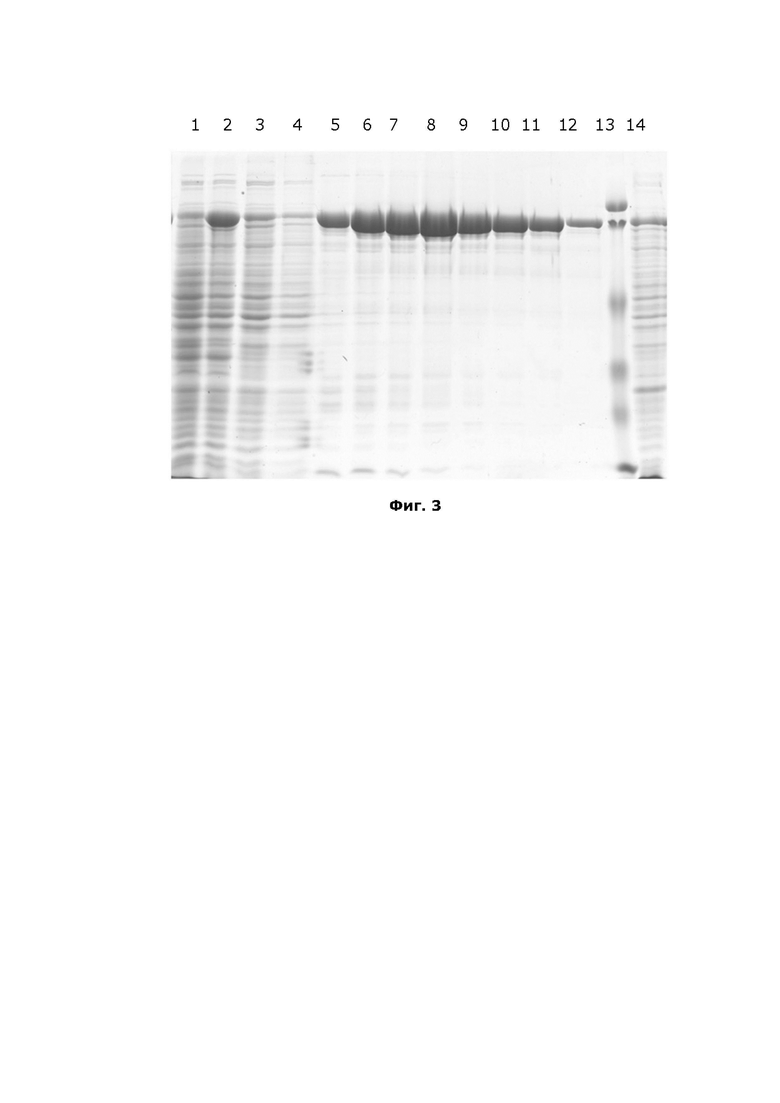
На фиг. 2 показаны карта генетической конструкции для продукции Т7РНКП в штамме-продуценте E. coli.
На фиг. 3 показаны результаты электрофоретического анализа в 10 масс.% ПААГ продукции и очистки поли(А)-полимеразы E. coli в штамме-продуценте E. coli. 1) Клетки без индукции, отрицательный контроль; 2) Супернатант, наносимый на колонку; 3) Проскок; 4) промывка; 5) 5 мМ имидазола; 6) 10 мМ имидазола; 7) 20 мМ имидазола; 8) 50 мМ имидазола; 9) 100 мМ имидазола; 10) 125 мМ имидазола; 11) 150 мМ имидазола; 12) 200 мМ имидазола; 13) 250 мМ имидазола; 14) Маркеры; 15) Тельца включения, осадок растворен в 8 М мочевине.
Описание изобретения
Определения:
Под «вектором» в настоящем изобретении понимается кольцевая молекула ДНК, построенная на основе типичном для бактерий внехромосомном репликоне - плазмиде, которая кодирует целевой фермент, используемая в генетической инженерии для передачи генетического материала внутрь клетки.
Под «рекомбинантным штаммом» в настоящем изобретении понимается штамм-продуцент, полученный в результате объединения in vitro чужеродных фрагментов и содержащий новое сочетания последовательностей нуклеотидов.
Под «РНК-вакциной» в настоящем изобретении понимается вакцина на основе матричной рибонуклеиновой кислоты - вакцина, действующая часть которой - рибонуклеиновая кислота, кодирующая белок, характерный для патогена. Помимо собственно РНК в вакцине присутствует липидная оболочка, защищающая РНК от разрушения и обеспечивающая проникновение РНК в клетку.
Под «транскрипцией» в настоящем изобретении понимается синтез молекулы РНК по матрице ДНК.
Под «штаммом-продуцентом» в настоящем изобретении понимается искусственно полученный штамм микроорганизма, который продуцирует определенное биологически активное соединение и может быть использован в биотехнологической промышленности.
Используемые сокращения:
ГИТЦ - гуанидин-изотиоцианат,
ИПТГ - изопропил-β-D1-тиогалактопиранозид,
мРНК - матричная РНК,
Т7РНКП - ДНК-зависимая РНК-полимераза фага Т7,
ПААГ - поли-акриламидный гель,
ПЦР - полимеразная цепная реакция,
E. coli - Escherichia coli,
T7RNA-P - ДНК-зависимая РНК-полимераза фага Т7.
Раскрытие сущности изобретения
Настоящее изобретение относится к штамму-продуценту фермента ДНК-зависимой РНК-полимеразы E. coli, предназначенному для экспрессии фермента ДНК-зависимой РНК-полимеразы, необходимого для транскрипции при in vitro синтезе РНК, обеспечивающему индуцируемую высокую экспрессию целевого белка и возможность его хроматографической очистки для дальнейшего промышленного и лабораторного применения.
Таким образом, целью настоящего изобретения является получение рекомбинантного штамма-продуцента фермента поли(А)-полимеразы E. coli BL21-T7RNA-P.
Еще одной целью настоящего изобретения является получение плазмидного вектора pET28-T7RNAP-N6His для трансформации рекомбинантного штамма-продуцента фермента поли(А)-полимеразы E. coli BL21-T7RNA-P.
Другой целью настоящего изобретения является обеспечение применения рекомбинантного штамма-продуцента фермента поли(А)-полимеразы E. coli BL21-T7RNA-P для in vitro синтеза РНК.
Предложенное в настоящей заявке изобретение может быть применено для синтеза одноцепочечных молекул РНК in vitro - широко используемой лабораторной процедуры, которая активно используется как для научных задач по исследованию РНК, так и для получения терапевтических препаратов на основе РНК. Этот метод универсален и позволяет исследователю или разработчику адаптировать синтез РНК и вносить модификации в кодируемые последовательности для решения разных задач. Так, этот способ применим для биохимического и молекулярного анализа РНК и взаимодействия РНК-белок, а также структурного анализа комплексов, создания РНК-аптамеров, синтеза функциональных мРНК для экспрессии и создания малых РНК для изменения экспрессии генов (например, направляющие РНК). Кроме того, использование синтезированной in vitro РНК сыграло важную роль в разработке РНК-вакцин и инструментов редактирования генома CRISPR / Cas9, создании плюрипотентных стволовых клеток, а также в разработке диагностики, основанной на амплификации РНК.
В частности, полученная при использовании раскрываемого в настоящем изобретении штамма-продуцента E. coli BL21-T7RNA-P ДНК-зависимая РНК-полимераза может применяться в ко-транскрипционное кэпирование с использованием синтетических кэп аналогов.
Способы осуществления данного изобретения
Пример 1. Получение штамма-продуцента фермента ДНК-зависимой РНК-полимеразы фага Т7
Нуклеотидную последовательность, кодирующую ДНК-зависимую РНК-полимеразу фага Т7 (Т7РНКП) (SEQ ID NO: 1), нарабатывают ПЦР со специфических праймеров, содержащих уникальные сайты рестрикции (SEQ ID NO: 2-3). В следующей реакции ПЦР к концевому участку этой последовательности амплификацией присоединяют последовательность, кодирующую 6His - поли-гистидиновую аминокислотную последовательность для аффинной хроматографической очистки. В качестве матрицы ПЦР использовали синтезированный de novo фрагмент ДНК, кодирующий Т7РНКП. Фрагмент ДНК, соответствующий требуемому гену, нарабатывают с использованием высокоточной ДНК-полимеразы Q5 (NEB).
Олигонуклеотиды 1
T7RNAP_Nco_forward:
gatataccatgggacatcatcaccaccatcacaacacgattaacatcgctaagaacgac (SEQ ID NO: 2)
T7RNAP_Not_reverse:
ctcgagtgcggccgcctattacgcgaacgcgaagtccgactctaag (SEQ ID NO: 3)
Далее этот фрагмент очищают через электрофорез в агарозном геле. Очищенный препарат ДНК-фрагмента используют в реакции рестрикции по двум уникальным эндонуклеазам рестрикции NcoI и NotI (NEB) при 37°С в течение 1 часа, после чего следует стадия переосаждения ДНК этанолом. Аналогично подготавливается плазмидная ДНК экспрессионного вектора pET28. Выделенную плазмидную ДНК гидролизуют двумя эндонуклеазами рестрикции NcoI и NotI (NEB) при 37°С в течение 1 часа. После этого гидролизованную плазмиду очищают через электрофорез в агарозном геле. Далее ставят реакцию лигирования с использованием Т4 ДНК лигазы (22°С, 1 час), реакцию лигирования останавливают прогревом при 65°С в течение 10 минут. Полученной лигазной смесью трансформируют компетентные клетки E. coli 10G, после чего инкубируют чашки с агаризованной средой LB и соответствующим антибиотиком в течение ночи при 37°С до появления отдельных колоний. На следующий день проводят ПЦР анализ отдельных колоний на содержание вектора со вставкой. Из положительных колоний выделяют плазмидную ДНК и проводят реакцию секвенирования (олигонуклеотиды-2 – SEQ ID NO: 4-9).
Олигонуклеотиды 2:
pet28_seq forward:
cacgatgcgtccggcgtagagg (SEQ ID NO: 4)
pet28_seq reverse:
gctttgttagcagccggatctc (SEQ ID NO: 5)
t7rnapseq1 forward:
cgttgaggaacaactcaacaag (SEQ ID NO: 6)
t7rnapseq2 forward:
ctagcggtcgccaacgtaatcac (SEQ ID NO: 7)
t7rnapseq3 forward:
ctgagcaagattctccgttctg (SEQ ID NO: 8)
t7rnapseq4 forward:
gaatcaggctgctggatacatg (SEQ ID NO: 9)
После анализа результатов сиквенса отбирают корректные образцы плазмидной ДНК. Далее отобранные генетические конструкции используют для трансформации штамма для продукции рекомбинантных белков в культуре E. coli. Штамм E. coli BL21 (DE3), предназначенный для продукции рекомбинантных белков, трансформируют химическим способом либо с использованием электропорации, полученной генетической конструкцией, кодирующей фермент ДНК-зависимой РНК-полимеразы E. coli. Трансформационную смесь высевают на чашки Петри с агаризованной средой LB c соответствующим антибиотиком для селекции (канамицин) и растят ночь при 37°С до появления единичных колоний. Далее единичной колонией инокулируют 5 мл жидкой среды LB с антибиотиком (канамицин) и инкубируют ночь при 37°С на шейкере (180 об/мин). На следующий день выросшей культурой заражают жидкую среду LB в колбах Эрленмейра (1/10 объема колбы) и подращивают при 37°С до оптической плотности OD = 0,4-0,6 единиц 4-5 часов. Далее в колбы добавляют индуктор экспрессии гена Т7 РНК-полимеразы (который кодируется на хромосомальной ДНК под контролем lac промотора) - синтетический аналог лактозы - 2mM ИПТГ (изопропил-β-D1-тиогалактопиранозид) и инкубируют культуру в колбах при 37°С до оптической плотности 1-1,2 3-4 часа. С полученной культуры собирают биомассу центрифугированием при 5 000 g в течение 30 мин при 4°С. Биомассу ресуспендируют в буфере для разрушения (Трис 50 мМ, рН 8, NaCl, 500 mM) на льду. Далее бактериальные клетки разрушают ультразвуком на льду до просветления культуры. Полученный лизат центрифугируют (10 000 g, 40 мин при 4°С) для осаждения клеточного дебриса. Супернатант осторожно отбирают и хранят на льду до нанесения на хроматографическую колонку для очистки белка. Аликвоту супернатанта и клеточного дебриса отбирают для дальнейшего электрофоретического анализа. Полученный сурернатант наносят на колонку с сорбентом Ni-NTA, предварительно промытую буфером (Трис 50 мМ, рН 8, NaCl 500 mM). Фермент элюируют линейным либо ступенчатым градиентом концентрации имидазола (20 мМ-500 мМ) в том же Трис-буфере. Собранные хроматографические фракции, содержащие максимальный уровень белка, объединяют, диализируют против буфера для хранения с глицерином на -20°С. Электрофорез в ПААГ проводят по стандартной методике Лэммли. Для белкового электрофореза в денатурирующих условиях используют 8-12 масс.% ПААГ. К белковому лизату добавляют двукратный объем Sample буфера (250 мМ Трис-HCl (pH 6.8), 6 масс.% SDS, 2 масс.% меркаптоэтанол, 16 масс.% глицерин, 0.05 масс.% бромфеноловый синий), раствор тщательно перемешивают и выдерживают в кипящей водяной бане в течение 5 мин. Окраска геля проводится с использованием Кумасси бриллиантового синего R-250. Электрофорез проводят в Трис-глициновом буфере (10х 1% масс. SDS 0.25М Трис- OH – 30 г/л, 1.86 М глицин – 140 г/л) на приборе фирмы «Bio-Rad» (CША).
Результаты получения очищенного хроматографически на никель-агарозе и элюированного в градиенте концентрации имидазола белка поли(А)-полимеразы (SEQ ID NO: 10) представлены на Фиг. 3.
Список литературы:
Smeekens S. P., Romano L. J. Promoter and nonspecific DNA binding by the T7 RNA polymerase // Nucleic acids research. – 1986. – Т. 14. – №. 6. – С. 2811-2827;
Biebricher C. K., Luce R. Template-free generation of RNA species that replicate with bacteriophage T7 RNA polymerase // The EMBO journal. – 1996. – Т. 15. – №. 13. – С. 3458-3465;
Forrest D. et al. Single-peptide DNA-dependent RNA polymerase homologous to multi-subunit RNA polymerase // Nature communications. – 2017. – Т. 8. – №. 1. – С. 1-8;
Svetlov V., Artsimovitch I. Purification of bacterial RNA polymerase: tools and protocols //Bacterial Transcriptional Control. – Humana Press, New York, NY, 2015. – С. 13-29;
WO 9110746 A1, (CHIRON CORPORATION), 25.07.1991;
CN 1331298 A, (BODE GENE DEVELOPMENT CO LTD, SHANGHAI), 16.01.2002;
US 6090589 A, (PROMEGA CORPORATION), 18.07.2000;
JP 2021175393 A, (TOSOH CORP; SAGAMI CHEMICAL RESEARCH INSTITUTE), 04.11.2021;
CN 107460177 A, (ZHANG HAISHENG), 12.12.2017.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="T7RNAPol.xml"
softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-03-11">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022133436</ApplicationNumberText>
<FilingDate>2022-12-20</FilingDate>
</ApplicationIdentification>
<ApplicantName languageCode="ru">Автономная некоммерческая
образовательная организация высшего образования
"Научно-Технологический Университет
"Сириус"</ApplicantName>
<ApplicantNameLatin>Sirius University of Science and
Technology</ApplicantNameLatin>
<InventionTitle languageCode="ru">Штамм-продуцент фермента
ДНК-зависимой РНК-полимеразы фага Т7</InventionTitle>
<SequenceTotalQuantity>10</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>2682</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2682</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgcatcaccatcatcatcatcatcatcatcacaacaccattaatattg
cgaaaaacgattttagcgacattgaattagccgcgattccgtttaacacgctggcggatcattacggcga
acgcctggcgcgtgaacagctggccctggaacatgaaagctacgaaatgggcgaagcgcgttttcgcaaa
atgtttgaacgccagctgaaagccggtgaagtggccgataatgcggcggccaaaccgctgattaccaccc
tgctgccgaaaatgattgcgcgtattaatgattggtttgaagaagttaaagcgaaacgtggcaaacgccc
gaccgcctttcagtttctgcaggaaattaaaccggaagccgtggcgtacattaccattaaaaccaccctg
gcctgcctgaccagcgccgataacacgaccgtgcaagcggtagcgagcgcgattggccgcgccattgaag
atgaagcacgctttggccgtattcgcgatctggaggccaaacactttaaaaaaaatgtggaagaacagct
gaataaacgcgtaggccacgtttacaaaaaagcatttatgcaggtggtggaagccgatatgttaagcaaa
ggcctgctgggcggcgaagcctggagcagctggcataaagaagatagcattcatgtgggcgtgcgttgca
ttgaaatgctgattgaatcaaccggcatggtgagcctgcaccgccagaacgccggcgttgtgggccagga
tagcgaaaccatcgaactggcgccggaatacgcggaagcgattgcaacccgcgcgggcgcactggcgggt
attagcccgatgttccagccttgcgtggttccgccgaaaccgtggaccggcattaccggcggcggctatt
gggccaacggccgccgtccgctggcgctggtgcgcacgcatagcaaaaaagcgctgatgcgttacgagga
tgtgtatatgccggaagtgtataaagcgattaacattgcccagaacaccgcctggaaaatcaacaaaaaa
gtgctggcggtggccaacgttattaccaaatggaaacattgtccggttgaagatattccggcgattgaac
gtgaagaactgccgatgaaacctgaagatattgacatgaatccggaagcactgaccgcctggaagcgcgc
ggcggcggcggtgtaccgcaaagataaagcgcgcaaaagccgtcgcatcagcctggaattcatgctggaa
caggcgaacaaatttgcgaaccacaaagcgatctggttcccgtacaacatggactggcgcggccgcgtgt
acgccgtaagcatgtttaacccgcagggtaatgatatgaccaaaggcctgctgacgctggccaaaggcaa
accgattggcaaagaaggctactactggctgaaaattcatggcgccaattgcgcgggcgtggataaagtt
ccgtttccggaacgtattaaattcatcgaagaaaaccacgaaaacattatggcgtgcgcgaaatcgccgc
tggaaaacacctggtgggcggaacaggattcgcccttctgctttctggcgttctgctttgaatatgcggg
cgtgcagcatcatggcctgagctacaactgcagcctgccgctggcgtttgatggcagctgctcaggcatt
cagcatttttcggcgatgctgcgcgatgaagttggcggtcgcgccgtgaatttactgccgagcgaaaccg
tgcaggatatttatggtattgtggcgaaaaaagtgaacgaaattctgcaggcggatgcgattaacggcac
tgataacgaagtggtgacggtgaccgatgaaaacaccggcgaaatcagtgaaaaagtgaaactgggcacc
aaagccctggcgggccagtggctggcgtatggcgtgacccgcagcgtgaccaaacgcagcgtgatgaccc
tggcctatggcagcaaagaatttggctttcgccagcaggtcctggaagacacgattcagccggccattga
tagcggcaaaggtctgatgtttacccagccgaatcaggcggcgggctacatggccaaactgatttgggaa
agcgtgagcgtcacggtggtggcggcggtggaagcgatgaactggctgaaaagcgcggcgaaactgctgg
cggcggaagtgaaagacaaaaaaaccggcgaaattctgcgtaaacgttgcgcggtgcattgggtgacccc
ggatggcttcccggtttggcaggaatataaaaaaccgattcagacccgcctgaacctgatgtttctgggc
cagtttcgtctgcagccgacgatcaacaccaataaagatagcgaaattgacgcccataaacaggaaagcg
gcattgcgccgaattttgtgcacagtcaggatggcagccatctgcgcaaaaccgtggtgtgggcccatga
aaaatatggtattgaaagctttgcgctgattcatgatagcttcggcaccattccggcggatgcggcgaac
ctgtttaaagcagtccgtgaaaccatggtggatacctacgaatcgtgcgatgtcctggccgacttttatg
atcagttcgcggaccagctgcacgaaagccaactggataaaatgccggccctgccggcgaaaggtaatct
gaatctgcgtgatattctggaaagcgattttgcgtttgcctaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>59</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..59</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatataccatgggacatcatcaccaccatcacaacacgattaacatcgc
taagaacgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>46</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..46</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcgagtgcggccgcctattacgcgaacgcgaagtccgactctaag</I
NSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacgatgcgtccggcgtagagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctttgttagcagccggatctc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q13">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgttgaggaacaactcaacaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q15">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctagcggtcgccaacgtaatcac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q17">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgagcaagattctccgttctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q19">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaatcaggctgctggatacatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>883</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..883</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q21">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Teseptimavirus T7</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MNTINIAKNDFSDIELAAIPFNTLADHYGERLAREQLALEHESYEMGEA
RFRKMFERQLKAGEVADNAAAKPLITTLLPKMIARINDWFEEVKAKRGKRPTAFQFLQEIKPEAVAYITI
KTTLACLTSADNTTVQAVASAIGRAIEDEARFGRIRDLEAKHFKKNVEEQLNKRVGHVYKKAFMQVVEAD
MLSKGLLGGEAWSSWHKEDSIHVGVRCIEMLIESTGMVSLHRQNAGVVGQDSETIELAPEYAEAIATRAG
ALAGISPMFQPCVVPPKPWTGITGGGYWANGRRPLALVRTHSKKALMRYEDVYMPEVYKAINIAQNTAWK
INKKVLAVANVITKWKHCPVEDIPAIEREELPMKPEDIDMNPEALTAWKRAAAAVYRKDKARKSRRISLE
FMLEQANKFANHKAIWFPYNMDWRGRVYAVSMFNPQGNDMTKGLLTLAKGKPIGKEGYYWLKIHGANCAG
VDKVPFPERIKFIEENHENIMACAKSPLENTWWAEQDSPFCFLAFCFEYAGVQHHGLSYNCSLPLAFDGS
CSGIQHFSAMLRDEVGGRAVNLLPSETVQDIYGIVAKKVNEILQADAINGTDNEVVTVTDENTGEISEKV
KLGTKALAGQWLAYGVTRSVTKRSVMTLAYGSKEFGFRQQVLEDTIQPAIDSGKGLMFTQPNQAAGYMAK
LIWESVSVTVVAAVEAMNWLKSAAKLLAAEVKDKKTGEILRKRCAVHWVTPDGFPVWQEYKKPIQTRLNL
MFLGQFRLQPTINTNKDSEIDAHKQESGIAPNFVHSQDGSHLRKTVVWAHEKYGIESFALIHDSFGTIPA
DAANLFKAVRETMVDTYESCDVLADFYDQFADQLHESQLDKMPALPAKGNLNLRDILESDFAFA</INSD
Seq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к биотехнологии. Предложены рекомбинантная плазмида, кодирующая ДНК-зависимую РНК-полимеразу фага Т7 (T7RNAP), рекомбинантная бактерия Escherichia coli, продуцирующая фермент ДНК-зависимую РНК-полимеразу фага Т7 (T7RNAP), кодируемый SEQ ID NO: 1, и трансформированная указанной рекомбинантной плазмидой. Также предложено применение указанной рекомбинантной бактерии в in vitro синтезе ДНК-зависимой РНК-полимеразы фага Т7 (T7RNAP). Изобретение обеспечивает эффективное получение указанных плазмиды и бактерии, а также применение бактерии в in vitro синтезе ДНК-зависимой РНК-полимеразы фага Т7 (T7RNAP). 3 н.п. ф-лы, 3 ил., 1 пр.
1. Рекомбинантная плазмида, кодирующая ДНК-зависимую РНК-полимеразу фага Т7 (T7RNAP), содержащая элементы:
- участок начала репликации вектора pBR322 origin;
- ген маркера антибиотикоустойчивости KanR;
- регулятор копийности вектора ROP;
- репрессор экспрессии гена T7RNAP lacl;
- последовательность промотора T7 (T7promoter);
- нуклеотидную последовательность SEQ ID NO: 1, кодирующую ДНК-зависимую РНК полимеразу фага Т7 (T7RNAP) с присоединенной последовательностью, кодирующей 6His;
- терминаторную последовательность транскрипции T7 terminator.
2. Рекомбинантная бактерия Escherichia coli, продуцирующая фермент ДНК-зависимую РНК-полимеразу фага Т7 (T7RNAP), кодируемый SEQ ID NO: 1, и трансформированная рекомбинантной плазмидой по п. 1.
3. Применение рекомбинантной бактерии по п. 2 в in vitro синтезе ДНК-зависимой РНК-полимеразы фага Т7 (T7RNAP).
| CN 107460177 A, 12.12.2017 | |||
| US 11066655 B2, 20.07.2021 | |||
| LI Y | |||
| et al | |||
| Способ восстановления хромовой кислоты, в частности для получения хромовых квасцов | 1921 |
|
SU7A1 |
| Protein Expr Purif | |||
| Металлический водоудерживающий щит висячей системы | 1922 |
|
SU1999A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| РЕКОМБИНАНТНАЯ ФАГОВАЯ ДНК М13 POL Т7 И РЕКОМБИНАНТНЫЙ ШТАММ ФАГА М13 - ПРОДУЦЕНТ РНК-ПОЛИМЕРАЗЫ ФАГА Т7 | 1994 |
|
RU2089613C1 |
Авторы
Даты
2024-08-26—Публикация
2022-12-20—Подача