Перекрестная ссылка на родственные заявки
[1] По настоящей заявке испрашивается приоритет согласно 35 USC §119(e) для предварительных заявок США № 62/818 577 и 62/932 725, поданных 14 марта 2019 и 8 ноября 2019 г., соответственно, содержание каждой из которых включено в настоящее изобретение посредством ссылки.
Заявление о государственной поддержке
[2] Это изобретение было сделано при государственной поддержке в рамках гранта № CA223104, предоставленного Национальным институтом здравоохранения и Национальным институтом рака (NIH/NCI). Правительство имеет определенные права на изобретение.
Список последовательностей
[3] Настоящая заявка содержит Список последовательностей, который был представлен в электронном виде в формате ASCII и включен в настоящую заявку посредством ссылки во всей полноте. Указанная копия ASCII, созданная 13 марта 2020 г., называется 106887-7660_ST25.txt и имеет размер 245014 байт.
Предшествующий уровень техники
[4] Противоопухолевая эффективность онколитических вирусов простого герпеса (oHSV), таких как Imlygic™, недавно одобренный Управлением по контролю продуктов питания и лекарственных средств для лечения меланомы, является очень многообещающей. Эти векторы имеют два основных механизма действия: (1) литическая фаза, определяемая прямым инфицированием и лизисом клеток, и (2) иммунологическая фаза, управляемая стимуляцией противоопухолевого иммунитета. Однако не все виды рака реагируют одинаково, поскольку для некоторых видов рака распространение вируса по своей природе является медленным. В культуре клетки различаются по уровню восприимчивости к вирусам. У животных вариации в составе стромы и иммунных клеток опухоли приводят к вариациям способности к распространению вируса и иммунных реакций. Следовательно, все еще необходимы стратегии повышения эффективности литической фазы для достижения оптимального терапевтического эффекта. Настоящее изобретение удовлетворяет эти потребности, а также обеспечивает связанные преимущества.
Краткое изложение сущности изобретения
[5] В настоящем описании предлагается неприродный вирус простого герпеса («HSV»), содержащий, предпочтительно состоящий по существу, или более предпочтительно состоящий из мутации в одном или нескольких из: (а) гена, кодирующего гликопротеин E («gE»), (b) гена, кодирующего белок 0 инфицированной клетки («ICP0»), (c) гена, кодирующего субъединицу 1 терминазы упаковки ДНК, (d) гена, кодирующего ICP8, или (e) гена, кодирующего ICP34.5.
[6] В другом аспекте настоящее изобретение обеспечивает композицию или фармацевтическую композицию, которая включает, предпочтительно состоит по существу, или более предпочтительно состоит из неприродного вируса простого герпеса, описанного в настоящем изобретении. В другом аспекте в настоящем изобретении представлен способ инфицирования клетки, включающий, или предпочтительно состоящий по существу, или более предпочтительно состоящий из обеспечения контакта клетки с неприродным HSV, или композицией или фармацевтической композицией, содержащей неприродный HSV.
[7] В одном из аспектов настоящее изобретение обеспечивает способ получения неприродного HSV по настоящему изобретению, включающий, или предпочтительно состоящий по существу, или более предпочтительно состоящий из мутации гена в неприродной вирусной частице HSV или введения в неприродный HSV трансгена. В другом аспекте способ получения вектора неприродного HSV включает, или предпочтительно состоит по существу, или более предпочтительно состоит из: (a) введения в клетку-хозяина вектора 17TermA HSV и вектора rRp450 HSV; (b) выращивания клетки-хозяина по меньшей мере за 3 пассажа; и (c) выделение частицы HSV, продуцируемой клеткой-хозяином.
[8] Также предложен способ ингибирования роста или метастазирования раковой клетки или метастатической раковой клетки, включающий или состоящий по существу, или более предпочтительно состоящий из обеспечения контакта клетки с эффективным количеством неприродного вектора HSV, или композиции или фармацевтической композиции, содержащей неприродный вектор HSV, как описано в настоящей заявке. Контакт осуществляют in vitro или in vivo. В одном аспекте обеспечение контакта осуществляют in vivo путем применения у субъекта неприродного HSV, или композиции или фармацевтической композиции. In vitro этот способ осуществляют путем помещения неприродного вируса простого герпеса в контакт с клеткой. Способ in vitro можно использовать для тестирования новых методов лечения или в качестве индивидуального анализа, чтобы определить, подходит ли терапия для лечения рака. Дополнительные способы лечения рака можно комбинировать с терапией, которая может быть параллельной или последовательной по отношению к раскрытым способам.
[9] Раковая клетка, подлежащая лечению, может быть солидной опухолью или гемобластозом, например, карциномой или саркомой, и неограничивающие примеры таких клеток включают рак поджелудочной железы, рак почки, мелкоклеточный рак легкого, опухоль головного мозга, нейробластому, опухоль нервной системы, рак костей, лимфому, миелому, рак толстой кишки, рак матки, рак молочной железы, лейкоз, рак печени, рак простаты, рак кожи или меланому. Клетка может относиться к любому виду, например, млекопитающему и человеку, и при проведении in vitro она может быть получена из культивируемой клеточной линии или первичной клетки, например, из биоптата ткани. Клетка может представлять собой взрослую или ювенильную клетку, или раковую стволовую клетку (т.е. раковые клетки, обладающие характеристиками, связанными с нормальными стволовыми клетками, особенно способностью давать начало всем типам клеток, обнаруживаемых в конкретном образце рака), или раковую клетку без таких характеристик, связанных с нормальными стволовыми клетками. В одном варианте осуществления клетка экспрессирует белок протоонкогена N-myc (MYCN) и/или экспрессирует MYCN на более высоком уровне, чем в нераковых клетках.
[10] В другом аспекте в настоящем изобретении также представлен способ лечения рака или метастатического рака, или ингибирования роста или метастазирования раковой клетки у субъекта, нуждающегося в этом, включающий или предпочтительно состоящий по существу, или более предпочтительно состоящий из применения у субъекта эффективного количества неприродного HSV, композиции или фармацевтической композиции по настоящему изобретению. Подлежащий лечению субъект может быть субъектом любого вида, например, млекопитающим и человеком, например, собакой, лошадью, коровой, кошкой, обезьяной, крысой или мышью. Применение может быть терапией первой линии, терапией второй линии, терапией третьей линии, терапией четвертой линии или терапией пятой линии. Дополнительные способы лечения рака можно комбинировать с терапией, которая может быть параллельной или последовательной по отношению к раскрытым способам. Подлежащий лечению рак может быть солидной опухолью или гемобластозом, например, карциномой или саркомой, и их неограничивающие примеры включают рак поджелудочной железы, рак почки, мелкоклеточный рак легкого, опухоль головного мозга, нейробластому, опухоль нервной ткани, рак костей, лимфому, миелому, рак толстой кишки, рак матки, рак молочной железы, лейкоз, рак печени, рак простаты, рак кожи или меланому.
[11] Способ из настоящего изобретения может быть объединен с соответствующей диагностикой для мониторинга ремиссии или прогрессирования заболевания. В данной области техники известно несколько методов такого мониторинга.
[12] В одном из аспектов в изобретении предложен способ индукции лизиса клеток, включающий или предпочтительно состоящий по существу, или более предпочтительно состоящий из обеспечения контакта клетки с эффективным количеством неприродного вируса простого герпеса, композиции и/или фармацевтической композиции по настоящему изобретению. Контакт осуществляют in vitro или in vivo. В одном аспекте контакт осуществляют in vivo путем введения субъекту неприродного HSV, или композиции или фармацевтической композиции. In vitro этот способ осуществляют путем помещения неприродного вируса простого герпеса в контакт с клеткой. Способ in vitro можно использовать для тестирования новых методов лечения или в качестве индивидуального анализа, чтобы определить, подходит ли терапия для пациента, подлежащего лечению. Дополнительные терапии на основе лизиса клеток можно комбинировать с терапией, которая может быть параллельной или последовательной по отношению к раскрытым способам.
[13] Клетка, подлежащая лечению, может быть клеткой солидной опухоли или гемобластоза, например, карциномы или саркомы, и неограничивающие примеры таких клеток включают рак поджелудочной железы, рак почки, мелкоклеточный рак легкого, опухоль головного мозга, нейробластому, опухоль нервной системы, рак костей, лимфому, миелому, рак толстой кишки, рак матки, рак молочной железы, лейкоз, рак печени, рак простаты, рак кожи или меланому. Клетка относится к любому виду, например, млекопитающему и человеку, и при проведении in vitro она может происходить из культивируемой клеточной линии или первичной клетки, например, из биоптата ткани. Клетка может представлять собой взрослую или ювенильную клетку, или раковую стволовую клетку, или раковую клетку, не имеющую характеристик, связанных с нормальными стволовыми клетками. Терапию можно комбинировать с соответствующим анализом для проверки эффективности терапии, например, ремиссии или прогрессирования рака.
[14] В другом аспекте в описании также предлагается набор, содержащий или предпочтительно состоящий по существу, или более предпочтительно состоящий из неприродного вируса простого герпеса, композиции и/или фармацевтической композиции по настоящему изобретению.
Краткое описание чертежей
[15] На фиг. 1A-1B показана схематическая диаграмма генерации Mut-3 и Mut-3Δ34.5 (фиг. 1A) и Mut-3 SNP (фиг. 1B). HSV Mut-3 выделяли из серийного пассажа при смешивании 17TermA и rRp450 в непермиссивной линии («Направленная эволюция»); а аттенуированный мутант Mut-3Δ34.5 конструировали посредством редактирования гена (обозначая как этап «CRISPR/Cas9») (фиг. 1A). На фиг. 1B показано сравнение последовательностей Mut-3 с его исходными вирусами. Несинонимичные мутации, которые отличаются от обоих исходных вирусов в Mut-3, заштрихованы обратной косой чертой, включая UL15, UL29, US8, RL1 и RL2. Геномные последовательности, идентичные 17TermA, показаны в виде незакрашенных прямоугольников на нижней панели; идентичные rRp450 отмечены косой чертой.
[16] На фиг. 2A-2C показаны размеры бляшек и использование рецепторов Mut-3, Mut-3Δ34.5, rRp450 и 17TermA. Автор заявки выполнил титрование методом бляшек для четырех вирусов одновременно, и сканировал и анализировал изображения бляшек через 3 дня с помощью многофункционального флуоресцентного микроскопа Keyence HS BZ-II Analyzer. (фиг. 2A) Необработанные (вверху) и маскированные (внизу, пронумерованные бляшки) изображения четырех вирусов. (фиг. 2B) Рассчитывали средний размер бляшек четырех вирусов (слева) и количество бляшек (справа). Размеры бляшек Mut-3 и Mut-3Δ34.5 значительно больше, чем у обоих исходных вирусов rRp450 и 17TermA. (фиг. 2C) Анализ цитотоксичности in vitro/MTS наборов клеток СНО. Автор заявки инфицировал CHO-K1 (как отмечено), CHO-Nectin-1 (как отмечено), CHO-Nectin-2 (как отмечено) и CHO-HVEM (как отмечено) четырьмя вирусами с разной множественностью инфицирования (MOI). Автор заявки определял выживание клеток с помощью колориметрического анализа клеточной пролиферации/MTS через три дня после вирусных инфекций (pvi) по сравнению с необработанным контролем. Только CHO-Nectin-1 и CHO-HVEM, но не CHO-K1 или CHO-Nectin-2 (в основном для проникновения HSV-2) были чувствительны к обработке четырьмя вирусами, что позволяет предположить, что Mut-3 и Mut-3Δ34.5 не обходят рецепторный барьер и все еще взаимодействуют с каноническими рецепторами проникновения HSV, чтобы инфицировать клетки.
[17] Фиг. 3A-3C - Mut-3Δ34.5 более эффективен, чем 17TermA (γ134.5-null) в отношении уничтожения клеток нейробластомы человека и мыши, что не связано с повышенным выходом инфекционных вирусных частиц in vitro. (фиг. 3A) Анализ цитотоксичности in vitro/MTS клеточных линий нейробластомы. Заявитель инфицировал как человеческие (SK-N-AS), так и мышиные (975A2) клетки нейробластомы Mut-3 (как отмечено), Mut-3Δ34.5 (как отмечено) или 17TermA (как отмечено) с разными величинами множественности инфицирования (MOI). Автор заявки измерил выживаемость клеток с помощью анализа MTS спустя 4 дня после инфицирования по сравнению с необработанным контролем. N = 6, планки погрешностей представляют стандартную ошибку среднего значения. **** p <0,0001, двусторонний дисперсионный анализ. (фиг. 3B) Анализ репликации вируса in vitro. Автор заявки инфицировал клеточные линии нейробластомы 17TermA (как отмечено) или Mut-3D34.5 (как отмечено) при MOI = 0,1 (верхняя панель, человеческая линия SK-N-AS) или MOI = 0,5 (нижняя панель, мышиная линия 975A2) и промывал клетки фосфатным буферным раствором через 2 часа после инфицирования. Автор заявки собирал лизат клеток через 2, 24, 48 и 72 часа после инфицирования и определял выход вируса с помощью анализов на бляшках. N=3. Планки погрешностей представляют стандартное отклонение. * p <0,05, ** p <0,001, **** p <0,0001, двухфакторный дисперсионный анализ. (фиг. 3C) Сравнение относительно свободно высвобожденных вирионов в культурах нейробластомы, инфицированных Mut-3Δ34.5, и инфицированных 17TermA. Автор заявки выполнил анализы, аналогичные описанным на фиг. 3B, путем сбора надосадочной жидкости и осадка для проведения анализа бляшек в двух разделенных порциях в течение времени вместо измерения в целой культуре на фиг. 3В. Автор заявки рассчитал относительный свободный вирион как долю инфекционных частиц в надосадочной жидкости во всей культуре (надосадочная жидкость + осадок). Mut-3Δ34.5 показывает значительно больше относительных высвобожденных вирионов, чем 17TermA через 48 часов. N = 3. Планки погрешностей представляют стандартное отклонение. * р <0,05.
[18] Фиг. 4A-4H - ТЭМ-анализ поглощения Mut3Δ34.5 и 17TermA клетками нейробластомы. Клетки нейробластомы SK-N-AS и 975A2 инфицировали Mut-3Δ34.5 или 17TermA при MOI 50 в течение 20 минут при 37°C. (фиг. 4A, 4B, 4E и 4F) На вставках показано, что вирионы Mut-3Δ34.5 были обнаружены в основном в эндоцитарных везикулах (стрелки, за исключением правой на фиг. 4B), и очень немногие из них были обнаружены слитыми с плазматической мембраной (правая стрелка на фиг. 4B). (фиг. 4C, 4D, 4G и 4H) Вставки показывают, что вирионы 17TermA в основном были обнаружены в эндоцитарных везикулах (стрелки, кроме нижней на фиг. 4G), очень немногие из них были обнаружены слитыми с плазматической мембраной (нижняя стрелка на фиг. 4G). ТЭМ-анализ был выполнен с помощью ТЕМ Hitachi H-7650. N: ядро. Масштабная шкала: 500 нм.
[19] Фиг. 5A-5C - аттенуированные вирионы 17Δ34.5 показали эффективность, сопоставимую с 17TermA в клетках нейробластомы. Заявитель создал 17Δ34.5 с помощью техники редактирования гена CRISPR-Cas9 для замены гена g134.5 в штамме 17+ дикого типа на кассету, экспрессирующую EGFP. (фиг. 5A) Изображения бляшек 17Δ34.5 клонов на клетках Vero. Оба клона B4 и G1 продуцируют 100% GFP-положительных (левые панели) несинцитиальных бляшек (правые панели, фазовый контраст) после 3 раундов очистки бляшек. Два дня после инфицирования. Изображения были получены с помощью системы визуализации EVOS® FL. Масштабная шкала: 400 мкм (фиг. 5B) Анализ цитотоксичности in vitro/MTS клеточных линий нейробластомы. Автор заявки инфицировал клеточные линии нейробластомы SK-N-AS и 975A2 штаммом дикого типа 17+ (как отмечено), 17TermA (как отмечено), 17Δ34.5 клоном B4 (как отмечено) или 17Δ34.5 клоном G1 (как отмечено) при разных значениях MOI. Автор заявки измерял выживаемость клеток с помощью анализа MTS через 3 дня после инфицирования относительно необработанного контроля. Эффективность обоих клонов 17Δ34.5 была значительно ниже, чем у их аналога дикого типа, штамма 17+, но сопоставима с 17TermA. Кассета EGFP и последовательность ДНК EGFP показаны на фиг. 5C (SEQ ID NO: 11).
[20] Фиг. 6A-6C - Mut-3Δ34.5 демонстрирует гораздо более быстрый перенос вирусного гена и уничтожение клеток по сравнению с 17Δ34.5. Автор заявки обработал (фиг. 6A) клетки Vero (фиг. 6B), SK-N-AS (фиг. 6C), 975A2 контролем, Mut-3Δ34.5 или 17Δ34.5 при разных значениях MOI и использовал живые клетки IncuCyte ZOOM для контроля GFP положительной области (верхние панели) и слияния клеток (нижние панели) с течением времени. N = 6 лунок на условие и n = 2 измерения на лунку для каждой временной точки. После вирусной инфекции (pvi). U-критерий Манна-Уитни использовали для сравнения времени до достижения максимальной площади GFP между вирусами (фиг. 6A). Планки погрешностей представляют стандартное отклонение.
[21] Фиг. 7 - Mut-3Δ34.5 более эффективен, чем 17TermA, для контроля роста нейробластомы человека in vivo. Бестимусным мышам nude с опухолями sub-q SK-N-AS внутриопухолевым путем вводили либо контрольный фосфатно-солевой буферный раствор (ФБР (как отмечено, n = 8), три дозы 1e7 БОЕ 17TermA (как отмечено, n = 8) или Mut-3Δ34.5 (как отмечено, n = 9). Были построены кривые выживаемости Каплана-Мейера. Логранговый тест использовали для оценки статистической значимости между 17TermA и Mut-3Δ34.5. * р <0,05.
[22] Фиг. 8A-8C - Mut-3ΔICP6, аттенуированная версия Mut-3, индуцирует более высокую цитотоксичность в клеточной линии нейробластомы человека CHP-134 по сравнению с онколитическим вирусом герпеса rRp450. (фиг. 8A) Карты Mut-3ΔICP6, построенные с помощью стратегии редактирования гена CRISPR-Cas9, где ген UL39, кодирующий ICP-6, был заменен на управляемую CMV репортерную кассету GFP. Анализы жизнеспособности MTS-клеток, сравнивающие профили цитотоксичности Mut-3, rRp450 и Mut3-ΔICP6/D7-1 в клеточных линиях нейробластомы человека. Клетки высевали в 96-луночные планшеты по 4000 клеток/лунку, инкубировали при 37°C в течение ночи и затем инфицировали каждым перечисленным вирусом при множественности инфицирования (MOI) 0,001; 0,01; 0,1 и 1 инфекционных вирусных частиц на клетку. Анализы выполняли с использованием Cell Titer96 водного нерадиоактивного анализа пролиферации клеток/ MTS (Promega, Мэдисон, Висконсин) на 3 или 4 день после инфицирования в соответствии с инструкциями производителя. Каждую группу образцов обрабатывали в четырех повторах с результатами, представленными в виде процента выживаемости клеток по сравнению с неинфицированными контролями. Планки погрешностей представляют собой стандартное отклонение. Статистическую значимость оценивали с помощью t-критерия. * p ≤ 0,05, ** p ≤ 0,01, *** p ≤ 0,001. Фиг. 8B SK-N-AS и фиг. 8С СНР-134, графическое представление результатов.
[23] Фиг. 9A-9B - Mut-3ΔICP6 индуцирует более высокую цитотоксичность в клеточных линиях нейробластомы мыши Neuro-2a и 975A2 по сравнению с rRp450. Фиг. 9A-9B показывает результаты анализов жизнеспособности клеток MTS, сравнивающих профили цитотоксичности Mut-3, rRp450 и Mut3-ΔICP6/D7-1 в клеточных линиях нейробластомы мыши. Клетки высевали в 96-луночные планшеты по 4000 клеток/лунку, инкубировали при 37°C в течение ночи и затем инфицировали каждым перечисленным вирусом при множественности инфицирования (MOI) 0,001; 0,01; 0,1 и 1 инфекционных вирусных частиц на клетку. Анализы выполняли с использованием Cell Titer96 AQuesous Non-Radioactive Cell Proliferation Assay /MTS (Promega, Мэдисон, Висконсин) на 3 или 4 день после инфицирования в соответствии с инструкциями производителя. Каждую группу образцов обрабатывали в четырех повторах с результатами, представленными в виде процента выживаемости клеток по сравнению с неинфицированными контролями. Планки погрешностей представляют собой стандартное отклонение. Статистическую значимость оценивали с помощью t-критерия. ** p ≤ 0,01, *** p ≤ 0,001. Фиг. 9A: Neuro-2a и фиг. 9B: 975A2.
Фиг. 10A-10B - Mut-3ΔICP6 обеспечивает значительно более высокий выход вируса, чем rRp450 в линии клеток нейробластомы мыши 975A2 в течение 48- и 72-часовых периодов инфицирования. Клетки нейробластомы мыши 975A2 (фиг. 10A) и человека SK-N-AS (фиг. 10B) высевали в 12-луночные чашки по 2 × 105 клеток на лунку, инкубировали при 37°C в течение ночи и инфицировали каждым вирусом в 200 мкл бессывороточной среды при MOI 0,01 для клеток SK-N-AS (фиг. 10B) или MOI 0,5 для клеток 975A2 (фиг. 10A) в течение 2 часов при осторожном встряхивании каждые 20 минут. Клетки один раз промывали ФБР и покрывали 1 мл полной среды. Затем собирали клетки и надосадочную жидкость через 2, 24, 48 и 72 часа после инфицирования, трижды замораживали-размораживали, готовили серийные разведения и титровали на клетках Vero для определения выхода инфекционного вируса с помощью стандартного анализа бляшек. Каждый образец анализировали в трех повторностях. Планки погрешностей представляют собой стандартное отклонение. Статистическую значимость оценивали с помощью t-критерия. * p ≤ 0,05, ** p ≤ 0,01.
[25] Фиг. 11A-11B - Mut-3Δ34.5 и Mut-3ΔICP6 значительно менее эффективны, чем вирус Mut-3, в отношении дифференцированных клеток кератиноцитов человека. Клетки кератиноцитов человека (HFK) выращивали в среде EpiLife (Cascade Biologics) с добавкой для роста кератиноцитов человека в соответствии с инструкциями производителя. Недифференцированные HFK (фиг. 11A) высевали в 96-луночный планшет при плотности 2000 клеток на лунку и культивировали в течение ночи. Затем культуры инфицировали Mut-3, Mut-3Δ34.5/C8G5 или Mut-3ΔICP6 D7-1 при MOI 0,0004; 0,004; 0,04; 0,4 и 4. Выживание клеток определяли с помощью анализа MTS через 3 дня после инфицирования вирусами. Дифференцированные HFK получали, добавляя 10% ЭТС и 1 ммоль/л CaCl2 к их культуральной среде через 24 часа после посева, и инкубируя их еще 48 часов до инфицирования. Выживаемость дифференцированных клеток (фиг. 11B) определяли с помощью анализа MTS спустя 4 дня после вирусной инфекции. Каждый образец анализировали в четырех повторностях. Планки погрешностей представляют собой стандартное отклонение. Статистическую значимость оценивали с помощью t-критерия. * p ≤ 0,05, ** p ≤ 0,01.
[26] Фиг. 12 - внутривенная инъекция 1e6 БОЕ штамма вируса KOS дикого типа является летальной для интактных мышей Bаlb/c. Описание исследований безопасности для Mut-3Δ34.5 и Mut-3ΔICP6. Автор заявки инициировал исследование вирусного биораспределения путем инъекции интактным мышам Balb/c, не несущим опухоли (20 самцов, 20 самок), 1e8 бляшкообразующих единиц (БОЕ) каждого вируса через хвостовую вену. После умерщвления этих животных в дни 1, 14, 28, 56 и 85 (n = 4 на момент времени) автор заявки собирал их периферическую кровь, семенники (у самцов), яичники (у самок), селезенки, легкие, почки, сердце, легкие и головной мозг для анализа. Автор заявки наблюдал за каждой мышью ежедневно в течение первых двух недель после введения вируса, а затем дважды в неделю до запланированного дня умерщвления. Регистрировали массу тела каждой мыши до инфицирования вирусом, и еженедельно после инфицирования. Параллельно, применяли вирус KOS дикого типа [диапазон доз: от 1e5 до 1e7 БОЕ на мышь (n = 3)] у групп мышей в качестве положительного (небезопасного) контроля. Автор заявки также выполнил патологический анализ собранных органов, сравнивая образцы, полученные от мышей, получавших мутантные вирусы, с образцами, полученными от мышей, получавших вирус KOS дикого типа. Автор заявки также использовал анализы бляшек для обнаружения репликативного потенциала этих вирусов. Результаты представлены кривыми выживаемости мышей, получавших однократную дозу 1e5, 1e6 или 1e7 БОЕ вируса KOS дикого типа.
[27] Фиг. 13A-13B - интактные мыши Balb/c могут переносить внутривенную инъекцию до 1-8 БОЕ Mut-3Δ34.5 (C8G5) или Mut-3ΔICP6 (D7-1). Автор заявки завершил исследование безопасности и биораспределения, и обнаружил, что мыши, получавшие до 1-8 БОЕ Mut-3Δ34.5(C8G5) или Mut-3ΔICP6(D7-1), оставались здоровыми при физикальном обследовании до назначенного дня умерщвления (до дня 85). У самок мышей анализы бляшек выявили присутствие инфекционного вируса в сердце, почках, печени, яичниках и селезенке только через 24 часа после заражения. Фиг. 13A и фиг. 13B графически представляют результаты для мышей-самок и мышей-самцов, соответственно.
[28] Фиг. 14 - реплицирующийся HSV может быть обнаружен в мозге, почках и яичниках мышей, получающих 1e6 БОЕ или более вирусов KOS дикого типа. Фиг. 14 представляет собой таблицу, суммирующую результаты анализов бляшек в тканях мышей, которым вводили 1e6 или 1e7 БОЕ вируса KOS дикого типа. Мышей умерщвляли через 5-6 дней после инъекции вируса (pvi) после того, как у них начали проявляться внешние признаки заболевания (кифоз, летаргия, паралич задних конечностей и т.д.). Знак «+» указывает на обнаруживаемые бляшки, а знак «-» указывает на их отсутствие. Серое заштрихованное поле означает, что эти данные недоступны.
[29] Фиг. 15 показывает, что репликация HSV может быть обнаружена почти во всех собранных тканях, за исключением легких мышей, получавших 1e8 БОЕ C8G5 или D7-1 24 часа pvi. Таблица, суммирующая результаты анализов бляшек в тканях мышей, которым вводили MutΔ34.5 или Mut3ΔICP6, через 24 часа после заражения вирусом. Знак «+» указывает на обнаружение бляшек, а знак «-» указывает на их отсутствие. Серое заштрихованное поле означает, что эти данные недоступны.
[30] Фиг. 16 показывает, что репликация HSV не обнаружена ни в одной из собранных тканей мышей, получавших 1e8 БОЕ C8G5 или D7-1 спустя 14 дней после инфицирования. В таблице представлены результаты анализов бляшек в тканях мышей, инфицированных вирусом MutΔ34.5 или Mut3ΔICP6, спустя 14 дней после инфицирования. Знак «-» указывает на отсутствие обнаруживаемых бляшек.
[31] Фиг. 17 показывает, что репликация HSV не обнаружена ни в одной из собранных тканей мышей, получавших 1e8 БОЕ C8G5 или D7-1, спустя 28 дней после инфицирования. В таблице обобщены результаты анализов бляшек в тканях мышей, которым вводили MutΔ34.5 или Mut3ΔICP6, спустя 28 дней после инфицирования вирусом. Знак «-» указывает на отсутствие обнаруживаемых бляшек. Серое заштрихованное поле означает, что эти данные недоступны.
[32] Фиг. 18 представляет собой таблицу, в которой суммируется тот факт, что репликация HSV не обнаруживается ни в одной из собранных тканей мышей, получавших 1e8 БОЕ C8G5 или D7-1 спустя 56 дней после инфицирования. В таблице приведены результаты анализов бляшек в тканях мышей, которым вводили MutΔ34.5 или Mut3ΔICP6, спустя 56 дней после инфицирования вирусом. Знак «-» указывает на отсутствие обнаруживаемых бляшек.
[33] Фиг. 19 показывает, что репликация HSV не выявляется ни в одной из собранных тканей мышей, получавших 1e8 БОЕ C8G5 или D7-1, спустя 85 дней после инфицирования. Таблица обобщает результаты количественной ПЦР и анализов бляшек в тканях мышей, которым вводили MutΔ34.5 или Mut3ΔICP6, спустя 85 после инфицирования вирусом. Знак «-» указывает на отсутствие обнаруживаемых бляшек.
[34] Фиг. 20A-20D представляет результаты исследования, показывающие, что размер бляшек Mut-3ΔgE намного меньше по сравнению с Mut-3 или Mut-3D34.5 (C8G5). Фиг. 20A: Mut-3ΔgE был сконструирован с помощью стратегии редактирования гена CRISPR-Cas9, в которой ген Us8, кодирующий гликопротеин gE, заменен репортерной кассетой GFP, управляемой CMV. Фиг. 20B: Несинцитиальный GFP-положительный фенотип бляшек Mut-3ΔgE клона 28D5-B4 и 28D5-H1 - клетки Vero инфицировали серийными разведениями 28D5-B4 или 28D5-H1 в течение 2 часов и покрывали слоем среды. Фотографии бляшек были сделаны через 3 дня после инфицирования. Фиг. 20C: анализ кПЦР клона Mut-3ΔgE 28D5-B4 и 28D5-H1 выявляет потерю Us8/gE. Клетки почек детенышей хомяка (BHK) инфицировали клоном Mut-3ΔgE 28D5-B4 или 28D5-H1, и собирали их лизаты, когда наблюдали цитопатический эффект ≥50%. 50-100 нг геномной ДНК, выделенной из инфицированного вирусом лизата клеток BHK, использовали в реакции кПЦР. Результаты представлены как gE/Us8 или кратность GFP относительно Us8a контрольного гена HSV. Фиг. 20D: Сравнение размеров бляшек между Mut-3ΔgE клоном 28D5-B4 и 28D5-H1 и Mut-3 или Mut-3Δ34.5/C8G5, взятым из стандартного анализа бляшек, проведенного в клетках Vero. Фотографии бляшек Mut-3, Mut-3Δ34.5, Mut-3ΔgE клона 28D5-B4 и клона 28D5-H1 были сделаны через 3 дня после инфицирования.
[35] Фиг. 21A-21D представляет результаты исследования, показывающие, что размер бляшек 17ΔgE намного меньше по сравнению с 17syn + или 17Δ34.5. Фиг. 21A: 17ΔgE был сконструирован с помощью стратегии редактирования гена CRISPR-Cas9, в которой ген Us8, кодирующий гликопротеин gE, заменен репортерной кассетой GFP, управляемой CMV. Фиг. 21B: Несинцитиальный и небольшой GFP-положительный фенотип бляшек клона 17ΔgE. Клетки 12G5- Vero инфицировали серийными разведениями 17ΔgE 12G5 в течение 2 часов и покрывали слоем среды. Фотографии бляшек были сделаны через 3 дня pvi. Фиг. 21C: анализ кПЦР 17ΔgE клона 12G5 выявляет потерю Us8/gE. Клетки почки детеныша хомяка (BHK) инфицировали клоном 17ΔgE 12G5, и лизат собирали, когда наблюдали цитопатический эффект ≥50%. 50-100 нг геномной ДНК, выделенной из инфицированного вирусом лизата клеток BHK, использовали в реакции кПЦР. Результаты представлены как gE/Us8 или кратность GFP относительно контрольного гена HSV ICP6. Фиг. 21D: Сравнение размеров бляшек между 17ΔgE клоном 12G5 17syn+ или 17Δ34.5 клоном B4, взятыми из стандартного анализа бляшек, проведенного на клетках Vero. Фотографии бляшек были сделаны через 3 дня pvi.
Подробное описание изобретения
[36] Варианты осуществления согласно настоящему изобретению будут описаны более полно ниже. Однако аспекты раскрытия могут быть воплощены в различных формах и не должны рассматриваться как ограниченные вариантами осуществления, изложенными в настоящей заявке. Скорее, эти варианты осуществления предоставлены для того, чтобы это изобретение было исчерпывающим и полным, и полностью передавало объем изобретения специалистам в данной области техники. Терминология, используемая в данном описании, предназначена только для описания конкретных вариантов осуществления и не предназначена для ограничения.
[37] Если не определено иное, все термины (включая технические и научные термины), используемые в настоящей заявке, имеют то же значение, которое обычно понимается специалистом в данной области техники, к которой принадлежит это изобретение. Далее следует понимать, что термины, такие как те, которые определены в обычно используемых словарях, должны интерпретироваться как имеющие значение, которое согласуется с их значением в контексте настоящей заявки и соответствующего уровня техники, и не должны интерпретироваться в идеализированном или чрезмерно формальном смысле, если это прямо не определено в настоящей заявке. Хотя эти термины не определены явно ниже, их следует интерпретировать в соответствии с их общим значением.
[38] Терминология, используемая в данном описании, предназначена только для описания конкретных вариантов осуществления и не предназначена для ограничения изобретения. Все публикации, заявки на патенты, патенты и другие ссылки, упомянутые в настоящей заявке, включены посредством ссылки во всей полноте.
[39] На практике настоящая технология будет использовать, если не указано иное, обычные методы культивирования тканей, иммунологии, молекулярной биологии, микробиологии, клеточной биологии и рекомбинантной ДНК, которые находятся в пределах компетенции специалистов в данной области техники.
[40] Если контекст не указывает иное, конкретно подразумевается, что различные признаки изобретения, описанные в настоящей заявке, могут использоваться в любой комбинации. Кроме того, раскрытие также предполагает, что в некоторых вариантах осуществления любая особенность или комбинация признаков, изложенных в настоящей заявке, может быть исключена или пропущена. С целью иллюстрации, если в описании указано, что комплекс включает компоненты A, B и C, конкретно подразумевается, что любой из A, B или C, или их комбинация может быть пропущена и исключена по отдельности или в любой комбинации.
[41] Если явно не указано иное, все описанные варианты осуществления, признаки и термины предназначены для включения как перечисленных вариантов осуществления, признаков или терминов, так и их биологических эквивалентов.
[42] Все числовые обозначения, например, pH, температура, время, концентрация и молекулярная масса, включая диапазоны, являются приблизительными значениями, которые изменяются (+) или (-) с шагом 1,0 или 0,1, в зависимости от ситуации, или предпочтительно, с изменением ±15%, или предпочтительно 10%, или предпочтительно 5%, или предпочтительно 2%. Следует понимать, хотя это не всегда явно указано, что всем числовым обозначениям предшествует термин «примерно». Также следует понимать, хотя не всегда явно указано, что описанные в настоящей заявке реагенты являются просто примерными и что их эквиваленты известны в данной области техники.
[43] Используемый в настоящей заявке термин «сопоставимый» относится к уровню, аналогичному контрольному, или в пределах отклонения ±50%, или предпочтительно 45%, или предпочтительно 40%, или предпочтительно 35%, или предпочтительно 30%, или предпочтительно 25%, или предпочтительно 20%, или предпочтительно 15%, или предпочтительно 10%, или предпочтительно 5%, или предпочтительно 2%, по сравнению с эталонным уровнем.
[44] В настоящем изобретении содержатся ссылки на различные публикации, патенты и опубликованные патентные спецификации с помощью идентифицирующей цитаты или арабской цифры. Полные ссылки на публикации, обозначенные арабскими цифрами, находятся непосредственно перед формулой изобретения. Раскрытия этих публикаций, патентов и опубликованных описаний патентов настоящим включены посредством ссылки в настоящее изобретение во всей их полноте, чтобы более полно описать состояние техники, к которой относится это изобретение.
Определения
[45] Практика настоящей технологии будет использовать, если не указано иное, обычные методы органической химии, фармакологии, иммунологии, молекулярной биологии, микробиологии, клеточной биологии и рекомбинантной ДНК, которые находятся в пределах компетенции специалистов в данной области техники. См., например, Sambrook, Fritsch and Maniatis, Molecular Cloning: A Laboratory Manual, 2nd edition (1989); Current Protocols In Molecular Biology (F. M. Ausubel, et al. eds., (1987)); the series Methods in Enzymology (Academic Press, Inc.): PCR 2: A Practical Approach (M.J. MacPherson, B.D. Hames and G.R. Taylor eds. (1995)), Harlow and Lane, eds. (1988) Antibodies, a Laboratory Manual, and Animal Cell Culture (R.I. Freshney, ed. (1987)).
[46] Используемые в описании изобретения и прилагаемой формуле изобретения формы единственного числа предназначены для включения и форм множественного числа, если контекст явно не указывает иное.
[47] Используемый в настоящей заявке термин «содержащий» предназначен для обозначения того, что композиции и способы включают перечисленные элементы, но не исключают другие. Как используется в данном документе, переходная фраза «состоящий по существу из» (и её грамматические варианты), должна интерпретироваться как охватывающая перечисленные материалы или этапы, а также те, которые существенно не влияют на основную и новую характеристику (характеристики) указанного варианта осуществления. Эти особенности изложены в вариантах осуществления способа. Таким образом, используемый здесь термин «состоящий по существу из» не следует интерпретировать как эквивалент «содержащий». «Состоящий из» означает исключение более чем следовых элементов других ингредиентов и существенных этапов способа введения композиций, раскрытых в настоящей заявке. Аспекты, определяемые каждым из этих переходных условий, входят в объем настоящего изобретения.
[48] Термин «примерно», используемый в настоящей заявке по отношению к измеряемой величине, такой как количество или концентрация и т.п., предназначен для охвата вариаций в 20%, 10%, 5%, 1%, 0,5% или даже 0,1% от указанного количества.
[49] Термины «приемлемый», «эффективный» или «достаточный», когда они используются для описания выбора любых компонентов, диапазонов, дозовых форм и т.д., раскрытых в настоящей заявке, подразумевают, что упомянутый компонент, диапазон, дозированная форма и т.д. подходят для раскрытой цели.
[50] Как используется в настоящей заявке, «и/или» относится и охватывает любые и все возможные комбинации одного или нескольких связанных перечисленных элементов, а также отсутствие комбинаций при интерпретации в альтернативе («или»).
[51] Используемый здесь термин «клетка» может относиться как к прокариотической, так и к эукариотической клетке, при необходимости полученной от субъекта или из коммерческого источника.
[52] «Эукариотические клетки» означают клетки всех биологических царств, кроме дробянок. Их легко отличить по мембраносвязанному ядру. Животные, растения, грибы и простейшие - это эукариоты или организмы, клетки которых организованы в сложные структуры с помощью внутренних мембран и цитоскелета. Наиболее характерной мембраносвязанной структурой является ядро. Если не указано иное, термин «хозяин» включает эукариотического хозяина, в том числе, например, дрожжи, клетки высших растений, насекомых и млекопитающих. Неограничивающие примеры эукариотических клеток или хозяев включают обезьян, коров, свиней, мышей, крыс, птиц, рептилий и человека, например, клетки HEK293 и клетки 293T.
[53] «Прокариотические клетки» - это клетки, которые обычно лишены ядра или каких-либо других мембраносвязанных органелл и делятся на два домена: бактерии и археи. Помимо хромосомной ДНК, эти клетки могут также содержать генетическую информацию в кольцевой петле, называемой эписомой. Бактериальные клетки являются очень маленькими, размером примерно с митохондрию животного (около 1-2 мкм в диаметре и 10 мкм в длину). Прокариотические клетки имеют три основные формы: палочковидную, сферическую и спиральную. Вместо прохождения сложных процессов репликации, как у эукариотических организмов, бактериальные клетки делятся путем бинарного деления. Примеры включают бактерии Bacillus, бактерии E. coli и бактерии Salmonella, но не ограничиваются ими.
[54] Термин «кодировать» в применении к последовательностям нуклеиновых кислот относится к полинуклеотиду, который, как говорят, «кодирует» полипептид, если в его нативном состоянии или при манипулировании методами, хорошо известными специалистам в данной области техники, его можно транскрибировать и/или транслировать для получения мРНК полипептида и/или его фрагмента. Антисмысловая цепь представляет собой комплемент такой нуклеиновой кислоты, и кодирующая последовательность может быть выведена из нее.
[55] Термины «эквивалент» или «биологический эквивалент» используются взаимозаменяемо, когда относятся к конкретной молекуле, биологическому или клеточному материалу, и подразумевают те эквиваленты, которые имеют минимальную гомологию, при этом сохраняя необходимую структуру или функциональность. Неограничивающие примеры эквивалентных полипептидов включают полипептид, содержащий по меньшей мере 60%, или предпочтительно по меньшей мере 65%, или предпочтительно по меньшей мере 70%, или предпочтительно по меньшей мере 75%, или предпочтительно 80%, или предпочтительно по меньшей мере 85%, или, предпочтительно по меньшей мере 90%, или предпочтительно по меньшей мере 95% идентичности для них или для полипептидных последовательностей, или для полипептида, который кодируется полинуклеотидом или его комплементом, который гибридизуется в условиях высокой жесткости с полинуклеотидом, кодирующим такие полипептидные последовательности. Условия высокой жесткости описаны здесь и включены в настоящую заявку посредством ссылки. Предпочтительно, его эквивалентом является полипептид, кодируемый полинуклеотидом или его комплементом, имеющий по меньшей мере 70%, или предпочтительно по меньшей мере 75%, или предпочтительно 80%, или предпочтительно по меньшей мере 85%, или предпочтительно по меньшей мере 90%, или предпочтительно по меньшей мере 95% идентичности, или по меньшей мере 97% идентичности последовательности с эталонным полинуклеотидом, например, полинуклеотидом дикого типа. В одном аспекте эквивалентный полипептид или полинуклеотид имеет такую же или практически аналогичную биологическую функцию, что и эталонный полипептид или полинуклеотид, соответственно, например, цитолитическую функцию, противоопухолевую, антиметастатическую или противораковую функцию, при определении соответствующим клеточным анализом или животной моделью, как описано в настоящей заявке.
[56] Неограничивающие примеры эквивалентных полипептидов включают полинуклеотид, содержащий по меньшей мере 60%, или предпочтительно по меньшей мере 65%, или предпочтительно по меньшей мере 70%, или предпочтительно по меньшей мере 75%, или предпочтительно 80%, или предпочтительно по меньшей мере 85%, или предпочтительно по меньшей мере 90%, или предпочтительно по меньшей мере 95%, или предпочтительно по меньшей мере 97% идентичности с эталонным полинуклеотидом. Эквивалент также подразумевает полинуклеотид или его комплемент, который гибридизуется в условиях высокой жесткости с эталонным полинуклеотидом.
[57] Полинуклеотид или полинуклеотидная область (или полипептид или полипептидная область), имеющая определенный процент (например, 80%, 85%, 90% или 95%) «идентичности последовательности» с другой последовательностью, означает, что при выравнивании этот процент оснований (или аминокислот) одинаков при сравнении двух последовательностей. Выравнивание и процент гомологии или идентичности последовательностей можно определить с помощью программ, известных в данной области техники, например, программ, описанных в Current Protocols in Molecular Biology (Ausubel et al., eds. 1987) Supplement 30, section 7.7.18, Table 7.7.1. В некоторых вариантах осуществления для выравнивания используют параметры по умолчанию. Неограничивающей примерной программой выравнивания является BLAST с использованием параметров по умолчанию. В частности, примерные программы включают BLASTN и BLASTP с использованием следующих параметров по умолчанию: генетический код = стандартный; фильтр = нет; цепь = обе; отсечка = 60; ожидать = 10; Матрица = BLOSUM62; Описания = 50 последовательностей; сортировать по = ВЫСОКАЯ ОЦЕНКА; Базы данных = неизбыточные, GenBank + EMBL + DDBJ + PDB + GenBank CDS-translations + SwissProtein + SPupdate + PIR. Подробную информацию об этих программах можно найти по следующему адресу в Интернете: ncbi.nlm.nih.gov/cgi-bin/BLAST. Идентичность последовательностей и процент идентичности можно определить, включив их в clustalW (доступно по веб-адресу: genome.jp/tools/clustalw/, последний доступ 13 января 2017 г.).
[58] «Гомология», или «идентичность», или «сходство» относится к сходству последовательностей между двумя пептидами или между двумя молекулами нуклеиновой кислоты. Гомология может быть определена путем сравнения положения в каждой последовательности, которая может быть выровнена для целей сравнения. Когда положение в сравниваемой последовательности занято одним и тем же основанием или аминокислотой, тогда молекулы являются гомологичными в этом положении. Степень гомологии между последовательностями является функцией количества совпадающих или гомологичных положений, общих для последовательностей. «Неродственная» или «негомологичная» последовательность имеет менее 40% идентичности или, предпочтительно, менее 25% идентичности с одной из последовательностей настоящего изобретения.
[59] «Гомология», «идентичность» или «сходство» также может относиться к двум молекулам нуклеиновой кислоты, которые гибридизуются в жестких условиях.
[60] «Гибридизация» относится к реакции, в которой один или несколько полинуклеотидов реагируют с образованием комплекса, который стабилизируется за счет водородной связи между основаниями нуклеотидных остатков. Водородная связь может происходить путем спаривания оснований Уотсона-Крика, связывания Хугстина или любым другим специфичным для последовательности способом. Комплекс может включать, или предпочтительно состоять по существу, или более предпочтительно состоять из двух цепей, образующих дуплексную структуру, трех или более цепей, образующих многоцепочечный комплекс, одного самогибридизующейся цепи, или любой их комбинации. Реакция гибридизации может составлять стадию более обширного процесса, такого как инициация реакции ПЦР или ферментативное расщепление полинуклеотида рибозимом.
[61] Примеры жестких условий гибридизации включают: температуры инкубации примерно от 25°C до 37°C; концентрации буфера для гибридизации примерно от 6×SSC до 10×SSC; концентрации формамида примерно от 0% до 25%; и промывающие растворы примерно от 4×SSC до 8×SSC. Примеры умеренных условий гибридизации включают: температуры инкубации примерно от 40°C до 50°C; концентрации буфера примерно от 9×SSC до 2×SSC; концентрации формамида примерно от 30% до 50%; и промывающие растворы примерно от 5×SSC до 2×SSC. Примеры условий высокой жесткости включают: температуры инкубации примерно от 55°C до 68°C; концентрации буфера примерно от 1×SSC до 0,1×SSC; концентрации формамида примерно от 55% до 75%; и промывающие растворы примерно 1×SSC, 0,1×SSC или деионизированная вода. Как правило, время инкубации при гибридизации составляет от 5 минут до 24 часов, с 1, 2 или более стадиями промывания, а время инкубации при промывании составляет примерно 1, 2 или 15 минут. SSC - это 0,15 М NaCl и 15 мМ цитратный буфер. Понятно, что можно использовать эквиваленты SSC с использованием других буферных систем.
[62] Используемый в настоящей заявке термин «экспрессия» относится к процессу, посредством которого полинуклеотиды транскрибируются в мРНК, и/или процессу, посредством которого транскрибируемая мРНК впоследствии транслируется в пептиды, полипептиды или белки. Если полинуклеотид происходит из геномной ДНК, экспрессия может включать сплайсинг мРНК в эукариотической клетке.
[63] «Ген» относится к полинуклеотиду, содержащему по меньшей мере одну открытую рамку считывания (ORF), которая способна кодировать конкретный полипептид или белок после транскрипции и трансляции. «Продукт гена» или, предпочтительно, «продукт экспрессии гена» относится к аминокислоте (например, пептиду или полипептиду), генерируемой при транскрипции и трансляции гена.
[64] «Под контролем транскрипции» - это термин, хорошо понятный в данной области техники, и указывает на то, что транскрипция полинуклеотидной последовательности, обычно последовательности ДНК, зависит от ее функциональной связи с элементом, который способствует инициации или способствует транскрипции. «Функционально связанные» означает, что полинуклеотиды расположены таким образом, чтобы они могли функционировать в клетке. В одном аспекте это изобретение обеспечивает промоторы, функционально связанные с нижележащими последовательностями, например, генами вирулентности HSV или их мутантами.
[65] Термин «кодировать» в применении к полинуклеотидам относится к полинуклеотиду, который, как говорят, «кодирует» полипептид, если в его нативном состоянии или при манипулировании методами, хорошо известными специалистам в данной области техники, он может транскрибироваться и/или транслироваться для получения мРНК полипептида и/или его фрагмента. Антисмысловая цепь представляет собой комплемент такой нуклеиновой кислоты, и кодирующая последовательность может быть выведена из нее.
[66] Термин «выделенный» в контексте настоящего описания относится к молекулам, биологическим или клеточным материалам, по существу не содержащим других материалов.
[67] Используемый в настоящей заявке термин «функциональный» может использоваться для модификации любой молекулы, биологического или клеточного материала с целью достижения определенного конкретного эффекта.
[68] Используемые здесь термины «нуклеиновокислотная последовательность» и «полинуклеотид» используются взаимозаменяемо для обозначения полимерной формы нуклеотидов любой длины, будь то рибонуклеотиды или дезоксирибонуклеотиды. Таким образом, этот термин включает одно-, двух- или многоцепочечную ДНК или РНК, геномную ДНК, кДНК, гибриды ДНК-РНК или полимер, содержащий или предпочтительно состоящий по существу, или более предпочтительно состоящий из пуриновых и пиримидиновых оснований или других натуральных, химически или биохимически модифицированных, неприродных или дериватизированных нуклеотидных оснований, но не ограничивается ими.
[69] Термин «дикий тип» относится к гену или продукту гена, имеющему характеристики этого гена или продукта гена, при выделении из природного источника. В некоторых вариантах осуществления гены или генные продукты дикого типа, даже для одного вирусного штамма, содержат несколько отличающиеся последовательности.
[70] Термин «мутант» относится к гену или продукту гена, который демонстрирует модификации в последовательности и/или функциональных свойствах (т.е. измененные характеристики) по сравнению с геном или продуктом гена дикого типа, или геном или продуктом гена от другого мутантного штамма (штаммов). В одном варианте осуществления другой мутантный штамм включает штамм 17TermA или rR450
[71] Термин «мутация» относится к вариации последовательности ДНК дикого типа или другого мутантного штамма (штаммов). Мутация обеспечивает или не обеспечивает функциональное свойство в организме. Существует несколько типов мутаций, включая, помимо прочего, вставку, делецию, усечение, сдвиг рамки считывания, замену или точечную мутацию.
[72] Термин «точечная мутация» относится к мутации с изменением одного нуклеотидного основания, вставкой или делецией генетического материала, ДНК или РНК.
[73] «Делеция» относится к мутации, при которой отсутствует часть хромосомы или последовательности ДНК.
[74] «Сдвиг рамки считывания» относится к мутации, вызванной изменениями (вставками или делециями) ряда нуклеотидов в последовательности ДНК, не делимой на три.
[75] «Замена» относится к мутации с заменой одного или нескольких нуклеотидов гена.
[76] «Усечение» относится к мутации с элиминацией N- или C-концевой части белка путем протеолиза или манипуляции со структурным геном, или преждевременной терминации элонгации белка из-за присутствия терминирующего кодона в его структурном гене в результате нонсенс-мутации.
[77] В некоторых вариантах осуществления мутация является несинонимичной мутацией. Термин «несинонимичная мутация» относится к мутации, изменяющей аминокислотную последовательность белка, которая контрастирует с синонимичной мутацией, не изменяющей аминокислотные последовательности.
[78] Используемый в настоящей заявке термин «промотор» относится к любой последовательности, которая регулирует экспрессию кодирующей последовательности, такой как ген. Промоторы могут быть, например, конститутивными, индуцибельными, репрессируемыми или тканеспецифичными. «Промотор» представляет собой контрольную последовательность, является областью полинуклеотидной последовательности, в которой контролируются инициация и скорость транскрипции. Он может содержать генетические элементы, с которыми могут связываться регуляторные белки и молекулы, такие как РНК-полимераза и другие факторы транскрипции. Неограничивающие иллюстративные промоторы включают промотор LTR вируса саркомы Рауса (RSV) (при необходимости с энхансером RSV), промотор цитомегаловируса (CMV), промотор SV40, промотор дигидрофолатредуктазы, промотор β-актина, промотор фосфоглицеринкиназы (PGK), промотор U6 или промотор EF1. В некоторых вариантах осуществления промотор представляет собой промотор куриного β-актина («CBA»).
[79] Дополнительные неограничивающие иллюстративные промоторы с определенной целевой специфичностью представлены ниже, включая CMV, EF1a, SV40, PGK1 (человеческий или мышиный), P5, Ubc, человеческий бета-актин, CAG, TRE, UAS, Ac5, полиэдрин, CaMKIIa, Gal1, TEF1, GDS, ADH1, CaMV35S, Ubi, H1, U6 и альфа-1-антитрипсин, но не ограничиваясь ими. Промоторы синтетического происхождения можно использовать для повсеместной или тканеспецифичной экспрессии. Кроме того, промоторы, полученные из вирусов, некоторые из которых указаны выше, могут быть полезны в способах, раскрытых в настоящей заявке, например, промоторы CMV, HIV, аденовируса и AAV. В некоторых вариантах осуществления промотор связан с энхансером для повышения эффективности транскрипции.
[80] Энхансер - это регуляторный элемент, который увеличивает экспрессию целевой последовательности. «Промотор/энхансер» представляет собой полинуклеотид, который содержит последовательности, способные обеспечивать функции как промотора, так и энхансера. Например, длинные концевые повторы ретровирусов содержат функции как промотора, так и энхансера. Энхансер/промотор может быть «эндогенным», «экзогенным» или «гетерологичным». «Эндогенный» энхансер/промотор - это энхансер/ промотор, который естественным образом связан с данным геном в геноме. «Экзогенный» или «гетерологичный» энхансер/промотор - это энхансер/промотор, который помещают рядом с геном посредством генетических манипуляций (т.е. молекулярно-биологических методов), так что транскрипция этого гена направляется связанным энхансером/ промотором.
[81] Термин «опухолеспецифический промотор или тканеспецифический промотор» в контексте настоящего описания означает промотор, обеспечивающий экспрессию гена, который находится под контролем промотора, в частности, в необходимой опухолевой клетке или ткани. Неограничивающие примеры тканеспецифических промоторов, которые можно использовать в настоящем изобретении, включают промотор простатоспецифического антигена (PSA), промотор простатоспецифического мембранного антигена (PSMA), промотор казеина, промотор IgG, промотор хорионического эмбрионального антигена, промотор эластазы, промотор порфобилиногендезаминазы, промотор инсулина, промотор фактора гормона роста, промотор рецептора ацетилхолина, промотор алкогольдегидрогеназы и промотор альфа- или бета-глобина.
[82] Неограничивающие примеры опухолеспецифических промоторов, используемых в настоящем изобретении, включают промотор обратной транскриптазы теломеразы, промотор глиального фибриллярного кислого белка, промотор E2F; промотор сурвивина, промотор СОХ-2, промотор EGD-2; промотор ELF-1; промотор, специфичный для гипоксии; промотор карциноэмбрионального антигена и промотор стромелизина 3.
[83] Термин «криоконсервант» относится к соединению или материалу, который способен защищать одну или несколько тканей, вирусов или других биологических агентов от повреждения или компрометации. Примеры криоконсервантов включают хондроитинсульфат, гликозаминогликан диметилсульфоксид, проникающие в клетки органические растворенные вещества, полисахариды, глицерин, минимальную необходимую среду Дульбекко (DMEM), глутамин, D-глюкозу, пируват натрия, эмбриональную телячью сыворотку, папаверин, глицерин, трегалозу, KH2PO4, K2HPO4, KCl, маннитол, NaHCO3, аскорбат натрия, 1,2-пропандиол, формамид, 2,3-бутандиол, пробухол, куркумин и их смеси, но не ограничиваются ими.
[84] Термины «белок», «пептид» и «полипептид» используются взаимозаменяемо и в самом широком смысле для обозначения соединения из двух или более субъединиц аминокислот, аминокислотных аналогов или пептидомиметиков. Субъединицы могут быть связаны пептидными связями. В другом аспекте субъединица может быть связана другими связями, например, сложноэфирной, простой эфирной и т.д. Белок или пептид должен содержать по меньшей мере две аминокислоты, и нет ограничений на максимальное количество аминокислот, которые могут включать, или предпочтительно по существу состоят, или более предпочтительно состоят из последовательности белка или пептида. Используемый здесь термин «аминокислота» относится либо к натуральным, либо к неприродным, либо к синтетическим аминокислотам, включая глицин и оптические изомеры D и L, аминокислотные аналоги и пептидомиметики.
[85] В контексте настоящего описания термин «вектор» относится к нехромосомной нуклеиновой кислоте, содержащей, предпочтительно состоящей по существу, или более предпочтительно состоящей из интактного репликона, так что вектор может реплицироваться при помещении в клетку, например, посредством процесса трансформации. Векторы могут быть вирусными или невирусными. Вирусные векторы включают ретровирусы, аденовирусы, вирус простого герпеса (HSV), бакуловирусы, модифицированные бакуловирусы, паповирусы или иным образом модифицированные природные вирусы. Типичные невирусные векторы для доставки нуклеиновой кислоты включают депротеинизированную ДНК; ДНК в комплексе с катионными липидами, отдельно или в комбинации с катионными полимерами; анионные и катионные липосомы; ДНК-белковые комплексы и частицы, содержащие, или предпочтительно состоящие по существу, или более предпочтительно состоящие из ДНК, конденсированной с катионными полимерами, такими как гетерогенный полилизин, олигопептиды определенной длины и полиэтиленимин, в некоторых случаях содержащиеся в липосомах; и использование тройных комплексов, содержащих или предпочтительно состоящих по существу из вируса и полилизин-ДНК, или более предпочтительно состоящих из них.
[86] «Вирусный вектор» определяется как рекомбинантно полученный вирус или вирусная частица, которые содержат, предпочтительно состоят по существу, или более предпочтительно состоят из полинуклеотида, который должен быть доставлен в клетку-хозяина in vivo, ex vivo, или in vitro. Примеры вирусных векторов включают ретровирусные векторы, векторы HSV, векторы AAV, лентивирусные векторы, аденовирусные векторы, альфавирусные векторы и т.п. Альфавирусные векторы, такие как векторы на основе вируса леса Семлики и векторы на основе вируса Синдбис, также были разработаны для использования в генной терапии и иммунотерапии. См. Schlesinger and Dubensky (1999) Curr. Opin. Biotechnol. 5:434-439 and Ying, et al. (1999) Nat. Med. 5(7):823-827.
[87] В другом варианте осуществления экспрессия вирулентного белка HSV (например, дикого типа или мутантов) регулируется промотором, который является индуцибельным промотором. В конкретном связанном варианте осуществления промотор представляет собой индуцибельный тетрациклиновый промотор. Системы экспрессии генов Tet-Off и Tet-On предоставляют исследователям легкий доступ к регулируемым системам экспрессии генов высокого уровня, описываемым как Tet-Off и Tet-On. В системе Tet-Off экспрессия генов включается, когда тетрациклин (Tc) или доксициклин (Dox; производное Tc) удаляют из культуральной среды. Напротив, экспрессия в системе Tet-On включается добавлением Dox. Обе системы позволяют жестко регулировать экспрессию генов в ответ на меняющиеся концентрации Tc или Dox. Максимальные уровни экспрессии в системах Tet очень высоки и выгодно отличаются от максимальных уровней, получаемых от сильных конститутивных промоторов млекопитающих, таких как CMV. В отличие от других индуцибельных экспрессионных систем млекопитающих, регуляция генов в Tet-системах очень специфична, поэтому интерпретация результатов не усложняется плейотропными эффектами или неспецифической индукцией. В E. coli белок-репрессор Tet (TetR) негативно регулирует гены оперона устойчивости к тетрациклину на транспозоне Tn10. TetR блокирует транскрипцию этих генов путем связывания с последовательностями оператора tet (tetO) в отсутствие Tc. TetR и tetO обеспечивают основу регуляции и индукции для использования в экспериментальных системах у млекопитающих. В системе Tet-On регуляторный белок основан на «обратном» репрессоре Tet (rTetR), который был создан в результате четырех аминокислотных изменений в TetR (Hillen & Berens, Mechanisms underlying expression of Tn10 encoded tetracycline resistance. Annu Rev Microbiol. 1994; 48:345-69; Gossen et al., Transcriptional activation by tetracyclines in mammalian cells. Science. 1995 Jun 23;268(5218):1766-9). Полученный белок, rtTA (обратный tTA, также называемый белком-активатором тетрациклина), кодируется плазмидой-регулятором pTet-On.
[88] В родственном варианте осуществления вектор дополнительно содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из нуклеиновой кислоты, кодирующей белок-активатор тетрациклина; и промотор, регулирующий экспрессию белка-активатора тетрациклина.
[89] Другие индуцибельные системы, применимые в векторах, выделенных клетках, вирусных упаковочных системах и способах, описанных в настоящей заявке, включают регуляцию экдизоном, эстрогеном, прогестероном, химическими индукторами димеризации, и изопропил-бета-D1-тиогалактопиранозидом (IPTG).
[90] Используемый в настоящей заявке термин «рекомбинантная система экспрессии» или «рекомбинантный вектор» относится к генетической конструкции или конструкциям для экспрессии определенного генетического материала, образованного путем рекомбинации.
[91] «Носитель для доставки генов» определяется как любая молекула, которая может нести встроенные полинуклеотиды в клетку-хозяина. Примерами носителей для доставки генов являются липосомы, мицеллы, биосовместимые полимеры, включая натуральные полимеры и синтетические полимеры; липопротеины; полипептиды; полисахариды; липополисахариды; искусственные вирусные оболочки; металлические частицы; и бактерии или вирусы, такие как бакуловирус, аденовирус и ретровирус, бактериофаг, космида, плазмида, грибковые векторы и другие носители рекомбинации, обычно используемые в данной области техники, которые были описаны для экспрессии в различных эукариотических и прокариотических хозяевах, и могут быть использованы для генной терапии, а также для простой экспрессии белков.
[92] Раскрытый в настоящей заявке полинуклеотид может быть доставлен в клетку или ткань с использованием носителя для доставки генов. «Доставка гена», «перенос гена», «трансдукция» и тому подобное в контексте настоящего описания - это термины, относящиеся к введению экзогенного полинуклеотида (иногда называемого «трансгеном») в клетку-хозяина, независимо от способа, используемого для введения. Такие способы включают множество хорошо известных методик, таких как вектор-опосредованный перенос генов (например, посредством вирусной инфекции/трансфекции или различных других комплексов доставки генов на основе белков или липидов), а также методики, облегчающие доставку деротеинизированных полинуклеотидов (такие как электропорация, доставка «генной пушкой» и различные другие методики, используемые для введения полинуклеотидов). Введенный полинуклеотид может стабильно или временно сохраняться в клетке-хозяине. Стабильное поддержание обычно требует, чтобы введенный полинуклеотид либо содержал точку начала репликации, совместимую с клеткой-хозяином, либо интегрировался в репликон клетки-хозяина, такой как внехромосомный репликон (например, плазмида), или ядерную или митохондриальную хромосому. Известно, что ряд векторов способен опосредовать перенос генов в клетки млекопитающих, как это известно в данной области техники и описано в настоящей заявке.
[93] «Плазмида» представляет собой молекулу внехромосомной ДНК, отдельную от хромосомной ДНК, которая способна реплицироваться независимо от хромосомной ДНК. Во многих случаях она бывает кольцевой и двухцепочечной. Плазмиды обеспечивают механизм горизонтального переноса генов в популяции микробов и обычно обеспечивают селективное преимущество при заданном состоянии окружающей среды. Плазмиды могут нести гены, которые обеспечивают устойчивость к природным антибиотикам в конкурентной экологической нише, или предпочтительно, продуцируемые белки могут действовать как токсины при аналогичных обстоятельствах.
[94] «Плазмиды», используемые в генной инженерии, называются «плазмидными векторами». Многие плазмиды коммерчески доступны для таких применений. Реплицируемый ген вставляют в копии плазмиды, содержащей гены, которые делают клетки устойчивыми к определенным антибиотикам, и сайт множественного клонирования (MCS, или полилинкер), который представляет собой короткую область, содержащую несколько часто используемых сайтов рестрикции, позволяющих легко встраивать фрагменты ДНК в этом участке. Еще одно важное применение плазмид - производство большого количества белков. В этом случае исследователи выращивают бактерии, содержащие плазмиду, несущую интересующий ген. Так же, как бактерия вырабатывает белки, придающие ей устойчивость к антибиотикам, ее также можно заставить производить большое количество белков из встроенного гена.
[95] Термин «вирус простого герпеса» или «HSV» в контексте настоящего описания означает вирус простого герпеса, который вызывает эффект из настоящего изобретения, который включает вирус простого герпеса дикого типа или мутантный вирус простого герпеса. В одном варианте осуществления мутантный неприродный HSV получают путем мутации или модификации любого из генов HSV дикого типа или путем вставки любого из экзогенных генов. Сывороточный тип HSV включает, предпочтительно состоит по существу, или более предпочтительно состоит из HSV типа 1 (или HSV-1) или HSV типа 2 (или HSV-2). HSV-1 представляет собой двухцепочечный ДНК-вирус с оболочкой. В одном варианте осуществления HSV-1 может инфицировать человеческую клетку. В другом варианте осуществления последовательность, ген или несколько генов могут быть включены в HSV-1. Размер встроенной последовательности может составлять приблизительно 1 основание, 5 оснований, 10 оснований, 100 оснований, 1 т.о., 10 т.о., 100 т.о. или 150 т.о. HSV-1 может индуцировать лизис клеток при относительно низкой множественности инфицирования (MOI), а его пролиферация может подавляться противовирусными препаратами. В одном варианте осуществления вирусная ДНК HSV остается вне хромосом, не встраиваясь в геном клеток-хозяев. HSV-1 может включать множество штаммов (например, KOS и McKrae). См. Wang et al., (2013) Virus Res. 173 (2): 436-440. В одном варианте осуществления HSV-1 представляет собой штамм KOS HSV-1. В другом варианте осуществления HSV-1 представляет собой штамм McKrae HSV-1.
[96] Существует несколько мутаций вируса простого герпеса, например, 17TermA HSV и rRp450 HSV. Термин «17TermA HSV» относится к мутантному вирусу HSV-1, который включает весь ген ICP34.5, но с кодоном терминации, вставленным перед 100 п.о. кодирующей области, что приводит к ранней терминации экспрессии белка и экспрессии белка, усеченного на 30 аминокислот. Мутант 17TermA HSV обнаруживает дефект роста из-за усеченного белка ICP34.5. См. Orvedahl et al., (2007) Cell Host & Microbe, 1:1, 23-25. Термин «rRp450» относится к аттенуированному вектору простого герпеса 1 с дефицитом кодируемой вирусом рибонуклеотидредуктазы или ICP6. См. Aghi M et al., (1999) Cancer Res., 59 (16): 3861-5.
[97] Геном HSV кодирует несколько белков вирулентности, которые включают гликопротеин E («gE»), белок 0 инфицированной клетки («ICP0»), белок 6 инфицированной клетки («ICP6»), субъединицу 1 терминазы упаковки ДНК, белок 8 инфицированной клетки («ICP8») и белок 34.5 инфицированной клетки («ICP34.5»), но не ограничиваются ими. Примерный геном HSV1 можно найти в NCBI Reference Sequence: NC_001806.2, последний доступ 13 марта 2020.
[98] Термин «ген, кодирующий gE» относится к гену или его фрагменту ДНК, кодирующему белок gE. Примерный ген, кодирующий gE, можно идентифицировать в положениях 33-2555 последовательности генома HSV-1 в эталонной последовательности NCBI: NC_001806.2. Термин «белок ICP6» относится к белку 6 инфицированной клетки, кодируемому геномом HSV. ICP6 представляет собой субъединицу рибонуклеотидредуктазы («RR») и ключевой фермент для метаболизма нуклеотидов и синтеза вирусной ДНК в неделящихся клетках.
[99] «Дисфункциональный» белок относится к белку, который имеет нарушенную функцию исходного белка или не выполняет ее. В одном варианте осуществления дисфункциональный белок обусловлен делецией или заменой в кодирующих последовательностях. Например, с дисфункциональным геном ICP6, будь то делеция или инактивация, HSV не может реплицироваться в нормальных неделящихся клетках. Однако в активно делящихся клетках с повышенной активностью RR недостаточная ферментативная активность вируса компенсируется, позволяя вирусу реплицироваться. ДНК и аминокислотные последовательности ICP 34.5 представлены в SEQ ID NO: 1, 2 и 5-10. ДНК и аминокислотные последовательности gE представлены в SEQ ID NO: 12-19. ДНК и аминокислотные последовательности ICP0 представлены в SEQ ID NO: 20-26. ДНК и аминокислотные последовательности субъединицы 1 терминазы упаковки ДНК представлены в SEQ ID NO: 35-42. ДНК и аминокислотные последовательности ICP8 представлены в SEQ ID NO: 27-34. ДНК и аминокислотные последовательности ICP6 представлены в SEQ ID NO: 43-50.
[100] Термин «ген, кодирующий ICP0» относится к гену или его фрагменту ДНК, кодирующему белок ICP0. Типичные последовательности ДНК и аминокислот ICP0 представлены в SEQ ID NO: 20-26. Термин «ген, кодирующий субъединицу 1 терминазы упаковки ДНК» относится к гену или его фрагменту ДНК, кодирующему белок или пептид субъединицы 1 терминазы упаковки ДНК. Типичные ДНК и аминокислотные последовательности субъединицы 1 терминазы упаковки ДНК представлены в SEQ ID NO: 35-42. Термин «ген, кодирующий ICP8» относится к гену или его фрагменту ДНК, кодирующему белок ICP8. Типичные последовательности ДНК и аминокислот ICP8 представлены в SEQ ID NO: 27-34. Термин «ген, кодирующий ICP34.5» относится к гену или его фрагменту ДНК, кодирующему белок ICP34.5. Типичные последовательности ДНК и аминокислот ICP34.5 представлены в SEQ ID NO: 1, 2 и 5-10. Термин «ген, кодирующий гликопротеин E («gE»)» относится к гену или его фрагменту ДНК, кодирующему белок gE. Типичные последовательности ДНК и аминокислот gE представлены в SEQ ID NO: 12-19.
[101] Термин «делеция или инактивация гена» означает делецию всего гена или его части, или подавление экспрессии гена посредством замены некоторых оснований, модификации, вставки ненужной последовательности и т.п. Делеция или инактивация гена HSV (например, gE, ICP0 и ICP8) может быть проведена специалистами в данной области техники известным способом, или основанным на нем способом. Например, можно применять метод, использующий гомологичную рекомбинацию. Например, можно разделить и инактивировать ген HSV путем клонирования фрагмента ДНК, содержащего часть гена HSV и последовательность, не связанную с геном HSV, в подходящий плазмидный вектор, а затем ввести его в HSV, чтобы вызвать гомологичную рекомбинацию в некоторой области гена HSV. Предпочтительно, мутация или делеция гена HSV может быть вызвана спонтанной мутацией в вирусном пассаже.
[102] В аспектах, где перенос гена опосредован ДНК-вирусным вектором, таким как вирус простого герпеса, векторная конструкция относится к полинуклеотиду, включающему, или предпочтительно состоящему по существу, или более предпочтительно состоящему из вирусного генома или его части, и трансгена. Таким образом, в одном аспекте неприродный HSV дополнительно содержит трансген, кодирующий терапевтический полинуклеотид или белок.
[103] Векторы, которые содержат как промотор, так и сайт клонирования, с которым может быть оперативно связан полинуклеотид, хорошо известны в данной области техники. Такие векторы способны транскрибировать РНК in vitro или in vivo и коммерчески доступны из таких источников, как Agilent Technologies (Санта-Клара, Калифорния) и Promega Biotech (Мэдисон, Висконсин). Для оптимизации экспрессии и/или транскрипции in vitro может потребоваться удаление, добавление или изменение 5'- и/или 3'-нетранслируемых частей клонов для устранения дополнительных, потенциально неподходящих альтернативных кодонов инициации трансляции или других последовательностей, которые могут препятствовать или уменьшить экспрессию на уровне транскрипции или трансляции. Предпочтительно, консенсусные сайты связывания рибосом могут быть вставлены непосредственно за 5' от стартового кодона для усиления экспрессии.
[104] Носители для доставки генов также включают комплексы ДНК/липосомы, мицеллы и целевые комплексы вирусный белок-ДНК. Липосомы, которые также содержат, или предпочтительно состоят по существу, или более предпочтительно состоят из антитела против клетки-мишени или его фрагмента, могут быть использованы в способах, раскрытых в настоящей заявке. В дополнение к доставке полинуклеотидов в клетку или популяцию клеток, прямое введение белков, описанных в настоящей заявке, в клетку или популяцию клеток, которое может быть выполнено с помощью неограничивающего метода трансфекции белков, предпочтительно в условиях культивирования, позволяющих усилить экспрессию и/или способствовать активности раскрытых в настоящей заявке белков, является другим неограничивающим способом.
[105] Используемый в настоящей заявке термин «сигнальный пептид» или «сигнальный полипептид» означает аминокислотную последовательность, обычно присутствующую на N-конце вновь синтезированных секреторных или мембранных полипептидов или белков. Он действует, направляя полипептид в конкретное клеточное местоположение, например, через клеточную мембрану, в клеточную мембрану или в ядро. В некоторых вариантах осуществления сигнальный пептид удаляют после локализации. Примеры сигнальных пептидов хорошо известны в данной области техники. Неограничивающие примеры описаны в патентах США №№ 8,853,381; 5,958,736 и 8,795,965.
[106] В одном аспекте HSV помечен детектируемой меткой. Используемый в настоящей заявке термин «метка» означает прямо или косвенно определяемое соединение или композицию, которые прямо или косвенно конъюгированы с детектируемой композицией, например, полинуклеотид или белок, такой как антитело, для создания «меченой» композиции. Термин также включает последовательности, конъюгированные с полинуклеотидом, которые будут обеспечивать сигнал при экспрессии вставленных последовательностей, таких как зеленый флуоресцентный белок (GFP) и т.п. Метка может быть обнаружена сама по себе (например, радиоизотопные метки или флуоресцентные метки) или, в случае ферментативной метки, может катализировать химическое изменение субстратного соединения или композиции, которое можно обнаружить. Метки могут быть подходящими для детекции в малом масштабе или более подходящими для высокопроизводительного скрининга. Как таковые подходящие метки включают радиоизотопы, флуорохромы, хемилюминесцентные соединения, красители и белки, в том числе ферменты, но не ограничиваются ими. Метка может быть просто обнаружена или может быть определена количественно. Отклик, который просто обнаруживается, обычно включает, предпочтительно состоит по существу, или более предпочтительно состоит из отклика, существование которого просто подтверждается, тогда как отклик, который определяют количественно, как правило, включает, предпочтительно состоит по существу, или более предпочтительно состоит из ответа, имеющего поддающееся количественной оценке (например, представляемое в численном выражении) значение, такое как интенсивность, поляризация и/или другое свойство. В анализах люминесценции или флуоресценции выявляемый ответ может быть получен непосредственно с использованием люминофора или флуорофора, связанного с компонентом анализа, фактически участвующего в связывании, или косвенно с использованием люминофора или флуорофора, связанного с другим (например, репортерным или индикаторным) компонентом.
[107] Примеры люминесцентных меток, которые производят сигналы, включают биолюминесценцию и хемилюминесценцию, но не ограничиваются ими. Обнаруживаемый люминесцентный отклик обычно включает, предпочтительно состоит по существу, или более предпочтительно состоит из изменения или появления сигнала люминесценции. Подходящие методы и люминофоры для компонентов анализа люминесцентной метки известны в данной области техники и описаны, например, в Haugland, Richard P. (1996) Handbook of Fluorescent Probes and Research Chemicals (6-е изд.). Примеры люминесцентных зондов включают экворин и люциферазы, но не ограничиваются ими.
[108] Примеры подходящих флуоресцентных меток включают флуоресцеин, родамин, тетраметилродамин, эозин, эритрозин, кумарин, метилкумарины, пирен, малахитовый зеленый, стильбен, желтый люцифер, Cascade Blue™ и техасский красный, но не ограничиваются ими. Другие подходящие оптические красители описаны в Haugland, Richard P. (1996) Handbook of Fluorescent Probes and Research Chemicals (6-е изд.).
[109] В другом аспекте флуоресцентная метка функционализирована для облегчения ковалентного прикрепления к клеточному компоненту, присутствующему на поверхности или встроенного в поверхность клетки или ткани, такого как маркер клеточной поверхности. Подходящие функциональные группы включают изотиоцианатные группы, аминогруппы, галогенацетильные группы, малеимиды, сукцинимидиловые эфиры и сульфонилгалогениды, но не ограничиваются ими, при этом все из них могут быть использованы для присоединения флуоресцентной метки ко второй молекуле. Выбор функциональной группы флуоресцентной метки будет зависеть от сайта присоединения к линкеру, агенту, маркеру или второму метящему агенту.
[110] Присоединение флуоресцентной метки может быть осуществлено либо непосредственно к клеточному компоненту или соединению, либо, предпочтительно, через линкер. Подходящие пары связывания для использования для косвенного связывания флуоресцентной метки с промежуточным продуктом включают антигены/антитела, например, родамин/анти-родамин, биотин/авидин и биотин/стрепавидин, но не ограничиваются ими.
[111] Фраза «твердая подложка» относится к неводным поверхностям, таким как «планшеты для культивирования», «генные чипы» или «микроматрицы». Такие генные чипы или микрочипы можно использовать для диагностических и терапевтических целей с использованием ряда методик, известных специалисту в данной области техники. В одной методике олигонуклеотиды присоединяют и размещают на генном чипе для определения последовательности ДНК с помощью подхода гибридизации, такого как описан в патентах США №№ 6025136 и 6018041. Полинуклеотиды по настоящему изобретению можно модифицировать в зонды, которые, в свою очередь, можно использовать для обнаружения генетической последовательности. Такие методики описаны, например, в патентах США №№ 5,968,740 и 5,858,659. Зонд также может быть прикреплен или фиксирован на поверхности электрода для электрохимической детекции последовательностей нуклеиновых кислот, как описано Kayem et al., патент США № 5,952,172 и Kelley et al. (1999) Nucleic Acids Res. 27: 4830-4837.
[112] «Композиция» предназначена для обозначения комбинации активного полипептида, полинуклеотида или антитела и другого соединения или композиции, инертных (например, детектируемая метка) или активных (например, носителя для доставки генов).
[113] «Фармацевтическая композиция» предназначена для включения комбинации активного полипептида, полинуклеотида или антитела с носителем, инертным или активным, таким как твердая подложка, что делает композицию подходящей для диагностического или терапевтического использования in vitro, in vivo или ex vivo.
[114] В контексте настоящего описания термин «фармацевтически приемлемый носитель» охватывает любой из стандартных фармацевтических носителей, например, забуференный фосфатом физиологический раствор, воду и эмульсии, такие как эмульсии масло-в-воде или вода-в-масле, а также различные типы смачивающих агентов. Композиции также могут включать стабилизаторы и консерванты. Примеры носителей, стабилизаторов и адъювантов см. в Martin (1975) Remington’s Pharm. Sci., 15th Ed. (Mack Publ. Co., Easton).
[115] «Субъектом» для диагностики или лечения является клетка или животное, такое как млекопитающее или человек. Субъект не ограничен конкретным видом и включает животных, не относящихся к человеку, подлежащих диагностике или лечению, а также животных, подверженных инфекциям, или животные модели, например, обезьян, представителей подсемейства мышиных, таких как крысы, мыши, шиншиллы, представителей семейства псовых, таких как собаки, кошек, представителей семейства зайцевых, таких как кролики, домашний скот, спортивных животных и домашних животных. В этот термин также включены пациенты-люди.
[116] Термин «ткань» используется в настоящей заявке для обозначения ткани живого или умершего организма, или любой ткани, полученной или предназначенной для имитации живого или умершего организма. Ткань может быть здоровой, больной и/или иметь генетические мутации. Биологическая ткань может включать любую отдельную ткань (например, набор клеток, которые могут быть взаимосвязаны) или группу тканей, составляющих орган, часть или область тела организма. Ткань может содержать, или предпочтительно состоять по существу, или более предпочтительно состоять из гомогенного клеточного материала, или это может быть композитная структура, такая как структура, находящаяся в областях тела, включая грудную клетку, которая, например, может включать легочную ткань, скелетную ткань и/или мышечную ткань. Примеры тканей включают ткани, полученные из печени, легких, щитовидной железы, кожи, поджелудочной железы, кровеносных сосудов, мочевого пузыря, почек, головного мозга, желчных протоков, двенадцатиперстной кишки, брюшной аорты, подвздошной вены, сердца и кишечника, включая любую их комбинацию, но не ограничиваясь этим.
[117] Используемый в настоящей заявке термин «лечение» заболевания у субъекта относится к (1) предотвращению появления симптомов или заболевания у субъекта, который предрасположен или еще не проявляет симптомы заболевания; (2) подавлению заболевания или остановке его развития; или (3) облегчению или регрессу заболевания или симптомов заболевания. Как понимается в данной области техники, «лечение» - это подход к получению полезных или необходимых результатов, включая клинические результаты. Для целей настоящей технологии полезные или необходимые результаты могут включать одно или несколько из облегчения или улучшения одного или нескольких симптомов, уменьшения степени состояния (включая заболевание), стабилизации (т.е. отсутствия ухудшения) состояния (включая заболевание), задержки или замедления прогрессирования состояния (включая заболевание), улучшения или временного облегчения состояния (включая заболевание), и ремиссии (частичной или полной), обнаруживаемой или не обнаруживаемой, но не ограничиваясь этим.
[118] Используемый в настоящей заявке термин «эффективное количество» означает количество, достаточное для достижения необходимого эффекта. В контексте терапевтических или профилактических применений эффективное количество будет зависеть от типа и тяжести рассматриваемого состояния и характеристик отдельного субъекта, таких как общее состояние здоровья, возраст, пол, масса тела и переносимость фармацевтических композиций. В контексте генной терапии в некоторых вариантах осуществления эффективное количество представляет собой количество, достаточное для восстановления частичной или полной функции гена, дефицитного у субъекта. В одном аспекте эффективное количество представляет собой количество, обеспечивающее множественность инфицирования (MOI) от 0,001 до 1 инфекционной вирусной частицы на клетку в промежутках между ними. Неограничивающие примеры включают множественность инфицирования (MOI) по меньшей мере 0,001, или по меньшей мере 0,01, или по меньшей мере 0,1, или по меньшей мере 1, или от 0,01 до 1, или от 0,1 до 1, или примерно от 0,01 до 0,1, или менее 1, или менее 0,1, или менее 0,01 инфекционных вирусных частиц на клетку. В других вариантах осуществления эффективное количество вирусной частицы HSV представляет собой количество, достаточное для того, чтобы привести к лизису клеток у субъекта. В некоторых вариантах осуществления эффективное количество представляет собой количество, необходимое для увеличения метаболизма галактозы у нуждающегося в этом субъекта. Специалист в данной области техники сможет определить подходящие количества в зависимости от этих и других факторов.
[119] В некоторых вариантах осуществления эффективное количество будет зависеть от размера и характера рассматриваемого приложения. Это также будет зависеть от характера и чувствительности целевого объекта и используемых способов. Специалист в данной области техники сможет определить эффективное количество на основе этих и других соображений. Эффективное количество может включать, или предпочтительно состоять по существу, или более предпочтительно состоять из одного или нескольких введений композиции в зависимости от варианта осуществления.
[120] Используемый в настоящей заявке термин «применять» или «применение» предназначен для обозначения доставки вещества субъекту, такому как животное или человек. Применение можно осуществлять одной дозой, непрерывно или периодически на протяжении всего курса лечения, например, внутриопухолевым или внутривенным способом. Способы определения наиболее эффективных средств и дозировки известны специалистам в данной области техники и будут варьировать в зависимости от композиции, используемой для терапии, цели терапии, а также возраста, состояния здоровья или пола субъекта, которого лечат. Однократное или многократное применение можно осуществлять с уровнем дозы и схемой, выбранными лечащим врачом, или, в случае домашних питомцев и животных, лечащим ветеринаром. Подходящие лекарственные формы и способы введения агентов известны в данной области техники. Путь введения также может быть определен, и метод определения наиболее эффективного пути введения известен специалистам в данной области техники и будет изменяться в зависимости от композиции, используемой для лечения, цели лечения, состояния здоровья или стадии заболевания, подвергаемого лечению субъекта и клетки или ткани-мишени. Неограничивающие примеры пути введения включают прямой и системный, например, внутривенный, внутриартериальный, внутримышечный, внутрисердечный, интратекальный, субвентрикулярный, эпидуральный, интрацеребральный, внутриопухолевый, интракраниальный, интрацеребровентрикулярный, субретинальный, интравитреальный, внутрисуставной, внутриглазной, интраперитонеальный, внутриматочный, внутрикожный, подкожный, трансдермальный, трансмукозальный и ингаляционный.
Онколитический вирус простого герпеса (oHSV)
[121] Эффективность oHSV против опухолей проистекает из прямого уничтожения клеток (литическая фаза) и повышения противоопухолевого иммунитета (иммунная фаза). Эти вирусы были сконструированы различными способами для избирательного воздействия на раковые клетки. Для достижения селективности рака наиболее распространенной мутацией является делеция гена нейровирулентности γ134.5/RL1. Экспрессия ICP34.5, кодируемого γ134.5/RL1, необходима для HSV-1, чтобы противодействовать пути, активируемому РНК антивирусной протеинкиназы (PKR) клетки-хозяина, которая обычно фосфорилирует e-IF2α в ответ на вирусную инфекцию и останавливает трансляцию белков. ICP34.5 перенаправляет клеточную протеинфосфатазу-1 (PP1) на дефосфорилирование e-IF2α, обеспечивая продуктивную репликацию вируса. Многие раковые клетки имеют дефект в отношении ответа PKR и, следовательно, поддерживают репликацию векторов HSV, включая γ134.5-нулевые мутанты. Некоторые векторы конструируют путем мутации критических метаболических вирусных генов, таких как рибонуклеотидредуктаза (RR, большая субъединица которой кодируется геном ICP6/UL39). Поскольку многие злокачественные клетки обладают повышенной экспрессией и активностью RR, ICP6-нулевые мутанты селективно реплицируются в высоко пролиферирующих раковых клетках из-за присутствия больших пулов рибонуклеотидов.
Проникновение HSV, распространение между клетками и образование синцития
[122] Проникновение HSV-1 включает последовательные этапы взаимодействия вирусных гликопротеинов с молекулами клеточной поверхности. Во-первых, гликопротеины B и C (gB и gC) прикрепляются к гепарансульфатным протеогликанам клеток, после чего происходит связывание gD с рецепторами проникновения вируса (т.е. нектином-1, медиатором проникновения вируса герпеса (HVEM) или 3-O-сульфатированным гепарансульфатом (3-OS-HS)). Кроме того, процесс позволяет gH/gL взаимодействовать с gB и запускать слияние вирусной оболочки с мембраной клетки-мишени, что приводит к высвобождению капсида вириона и белков тегумента в клетку. Несколько сообщений позволяют предположить, что слияние мембран также имеет решающее значение для последующего латерального распространения вируса между соседними клетками. Распространение вирусов между клетками происходит, когда вирус распространяется от инфицированных клеток к соседним неинфицированным клеткам через области контакта клеток, что может происходить даже в присутствии нейтрализующих вирус антител. Присутствие gE/gI увеличивает межклеточное распространение, тогда как гипоморфные мутанты gE уменьшают слияние клеток и размер бляшек. Синцитии являются результатом слияния нескольких соседних клеток в многоядерные гигантские клетки. Мутации, запускающие синцитии, были обнаружены по меньшей мере в четырех вирусных генах HSV (т.е. GB, gK, UL20 и UL24).
[123] В настоящем изобретении представлена стратегия объединения «направленной эволюции» и технологии CRISPR/Cas9 для разработки клинически значимых вирусных векторов. Эта эффективная комбинация представляет собой существенное отделение от подходов предшествующего уровня техники. Не углубляясь в теорию, ожидается, что неожиданные механизмы могут привести к увеличению вирусной активности в отношении уничтожения опухолей. Ожидается, что это выявит ранее неизвестные мутации, которые вызывают гиперфузогенный фенотип и повышенную эффективность, а также станут ступенькой для разработки oHSV следующего поколения. Полученные таким образом oHSV имеют усиленную литическую фазу, более длительную устойчивость и максимальный терапевтический результат, что соответствует цели клинических и трансляционных диагностических/экспериментальных исследований Национального института рака.
[124] Хотя основное внимание уделяется «литической фазе» виротерапии, более длительный устойчивый вирусный ответ будет усиливать воспалительный ответ in vivo, принося пользу последующей «иммунной фазе» виротерапии для достижения оптимальной противоопухолевой эффективности. Исследования эффективности на педиатрических моделях рака позволяют рассмотреть вопрос об использовании неприродного вируса простого герпеса для лечения рака у детей. Ожидается, что полученные в результате усовершенствованные вирусы будут активны и в отношении различных видов рака у взрослых.
Способы осуществления изобретения
[125] Несмотря на то, что HSV могут нацеливаться или инфицировать широкий спектр клеток, вызывая лизис, сама вирусная инфекция вызывала цитотоксичность и другие нарушения, например, энцефалит, эзофагит и пневмонит. Большое количество генов HSV влияет на патогенность. Например, γ34.5 (RL1) может вызывать нейропатогенность. ICP6 (UL39), рибонуклеотидредуктаза, тимидинкиназа (UL23), урацил-ДНК-гликозилаза (UL2), dUTPase (UL50) и ДНК-полимераза (UL30) участвуют в метаболизме нуклеотидов и вирулентности HSV. Следовательно, существует потребность в получении аттенуированной, но способной к репликации частицы HSV, чтобы выполнять свою функцию ингибирования опухоли, минимизируя при этом побочные эффекты.
[126] В настоящей заявке предложен новый вирус, названный Mut-3, который проявляет фенотип гигантских синцитиальных бляшек. Автор заявки выделил Mut-3 из последовательного пассажа смешивания 17TermA и rRp450 в непермиссивной линии («Направленная эволюция») и сконструировал аттенуированный мутант Mut-3Δ34.5 посредством редактирования гена (этап «CRISPR/Cas9») (фиг. 1А). Анализ последовательности всего генома показал, что Mut-3 приобрел интактные копии γ134.5/RL1 и UL39 (который кодирует ICP6), что делает его генотип похожим на генотип вируса дикого типа (WT). Не углубляясь в теорию, авторы заявки обнаружили, что литическая активность Mut-3 даже больше, чем у многих вирусов WT, что позволяет предположить, что другие геномные изменения (в дополнение к его полному геному вируса) могут быть вовлечены в его повышенную активность. Было обнаружено пять несинонимичных мутаций в Mut-3, которые отличаются от любого исходного вируса, включая мутацию аланина в треонин в положении 151 (A151T) в гене, кодирующем gE. Обеспечена аттенуированная версия вируса Mut-3, которая заменяет γ134.5/RL1 зеленым флуоресцентным белком (GFP) посредством редактирования гена CRISPR/Cas9, обозначенная как Mut-3Δ34.5 (фиг. 1A, внизу, обозначено как этап «CRISPR/Cas9»). См. фиг. 1B, показывающую сводку результатов сравнения последовательностей Mut-3 с его исходными вирусами. Несинонимические мутации, которые отличаются от любого исходного вируса в Mut-3, заштрихованы обратной косой чертой, включая UL15, UL29, US8, RL1 и RL2. Последовательности генома, идентичные 17TermA, обозначены неокрашенными прямоугольниками; идентичные rRp450 заштрихованы косой чертой.
[127] Таким образом, настоящее изобретение относится к неприродному вирусу простого герпеса («HSV»), в котором вирус содержит, предпочтительно состоит по существу, или более предпочтительно состоит из мутации в гене вирулентности, который является одним или несколькими из группы, включающей: (а) гликопротеин E («gE») - кодирующий ген; (b) ген, кодирующий белок 0 инфицированной клетки («ICP0»); (c) ген, кодирующий субъединицу 1 терминазы упаковки ДНК; (d) ген, кодирующий ICP8; или (e) ген, кодирующий ICP34.5. В одном варианте осуществления HSV дополнительно содержит, предпочтительно состоит по существу, или более предпочтительно состоит из гена, кодирующего дисфункциональный белок ICP34.5, и/или гена, кодирующего дисфункциональный белок ICP6. В другом варианте осуществления ген, кодирующий дисфункциональный белок ICP34.5, содержит, предпочтительно состоит по существу, или более предпочтительно состоит из полинуклеотида, имеющего последовательность, по меньшей мере на 95% идентичную SEQ ID № 1, 5, 7, 9 или 51, и его эквивалентов, при условии, что эквивалент сохраняет мутированную или измененную аминокислоту или нуклеотиды. В другом варианте осуществления ген, кодирующий дисфункциональный белок ICP6, включает, предпочтительно состоит по существу, или более предпочтительно состоит из полинуклеотида, имеющего последовательность, по меньшей мере на 95% идентичную SEQ ID № 43, 45, 47 или 49, и его эквивалентов при условии, что эквивалент сохраняет мутированную или измененную аминокислоту или нуклеотиды, по сравнению с последовательностью дикого типа.
[128] В конкретном варианте осуществления ген, кодирующий gE неприродного вируса простого герпеса, содержит, предпочтительно состоит по существу, или более предпочтительно состоит из полинуклеотида, имеющего последовательность, выбранную из SEQ ID NO: 12, 14, 16, 18, последовательность, по меньшей мере, на 80%, 85%, 90%, 95%, 97% или 99% идентичную любой из SEQ ID NO: 12, 14, 16 и 18, и его эквивалента, при условии, что эквивалент поддерживает мутантную или измененную аминокислоту или нуклеотиды по сравнению с последовательностью дикого типа. В другом варианте осуществления HSV, содержащий такой эквивалент, проникает в клетки и/или распространяется среди клеток и/или реплицирует ДНК на уровне, сравнимом с неприродным HSV, содержащим мутантный gE, имеющий аминокислотную последовательность SEQ ID NO: 13. Неограничивающий пример оценки проникновения HSV в клетки, распространения среди клеток и репликации ДНК можно найти в примерах. В еще одном варианте осуществления полинуклеотид гена, кодирующего gE, кодирует полипептид, имеющий аминокислотную последовательность, выбранную из SEQ ID NO: 13, 15, 17 и 19.
[129] В конкретном варианте осуществления ген, кодирующий ICP0 неприродного вируса простого герпеса, содержит, предпочтительно состоит по существу или более предпочтительно состоит из полинуклеотида, имеющего последовательность, выбранную из SEQ ID NO: 20, 22, 24, 25, и 53, последовательность любой из SEQ ID NO: 20, 22, 24, 25 и 53, свободную от одного или обоих интронов, последовательность, по меньшей мере на 80%, 85%, 90%, 95%, 97% или 99% идентичную SEQ ID NO: 20, 22, 24, 25 и 53, и его эквивалентов, при условии, что эквивалент сохраняет мутантную или измененную аминокислоту или нуклеотиды по сравнению с последовательностью дикого типа. В одном варианте осуществления интроны указаны в Списке последовательностей, а также в следующем: нуклеотиды (nt) от 58 до 861 в SEQ ID NO: 20, нуклеотиды от 1529 до 1663 в SEQ ID NO: 20, нуклеотиды от 58 до 822 в SEQ ID NO: 22, нуклеотиды от 1490 до 1625 в SEQ ID NO: 22, нуклеотиды от 58 до 862 в SEQ ID NO: 24, нуклеотиды от 1530 до 1668 в SEQ ID NO: 24, нуклеотиды от 58 до 861 в SEQ ID NO: 25, нуклеотиды от 1529 до 1663 в SEQ ID NO: 25, нуклеотиды от 58 до 822 в SEQ ID NO: 53, и нуклеотиды от 1490 до 1625 в SEQ ID NO: 53. В другом варианте осуществления эквивалент кодирует полипептид ICP0, имеющий функцию (например, стимуляцию транскрипции вирусных генов, разрушение структур в ядре, известных как ядерные точки или ядерные тельца промиелоцитарного лейкоза (PML), и изменение экспрессии генов хозяина и вирусных генов в сочетании с нейронспецифическим белком) на уровне, сравнимом с ICP0 дикого типа или мутантным ICP0, имеющим аминокислотную последовательность SEQ ID NO: 21. Примеры оценки таких функций можно найти, например, в Lee HR, Kim DJ, Lee JM, et al. (June 2004). «Ability of the human cytomegalovirus IE1 protein to modulate sumoylation of PML correlates with its functional activities in transcriptional regulation and infectivity in cultured fibroblast cells». J. Virol. 78 (12): 6527-42; Gu H, Liang Y, Mandel G, Roizman B (May 2005). «Components of the REST/CoREST/histone deacetylase repressor complex are disrupted, modified, and translocated in HSV-1-infected cells». Proc. Natl. Acad. Sci. U.S.A. 102 (21): 7571-6; and Pinnoji RC, Bedadala GR, George B, Holland TC, Hill JM, Hsia SC (2007). «Repressor element-1 silencing transcription factor/neuronal restrictive silencer factor (REST/NRSF) can regulate HSV-1 immediate-early transcription via histone modification». Virol. J. 4: 56. В дополнение или предпочтительно, HSV, содержащий такой эквивалент, проникает в клетки и/или распространяется среди клеток и/или реплицирует ДНК на уровне, сравнимом с неприродным HSV, содержащим мутантный ICP0, имеющий аминокислотную последовательность SEQ ID NO: 21. Неограничивающий пример оценки проникновения HSV в клетки, распространения среди клеток и репликации ДНК можно найти в Примерах. В еще одном варианте осуществления полинуклеотид гена, кодирующего ICP0, кодирует полипептид, имеющий аминокислотную последовательность, выбранную из SEQ ID NO: 21, 23 и 26.
[130] В конкретном варианте осуществления ген, кодирующий ICP8 неприродного вируса простого герпеса, содержит, предпочтительно состоит по существу, или более предпочтительно состоит из полинуклеотида, имеющего последовательность, выбранную из SEQ ID NO: 27, 29, 31, 33, последовательности, по меньшей мере на 80%, 85%, 90%, 95%, 97% или 99% идентичной любой из SEQ ID NO: 27, 29, 31 и 33, и его эквивалента при условии, что эквивалент сохраняет мутантную или измененную аминокислоту или нуклеотиды по сравнению с последовательностью дикого типа. В дополнительном варианте осуществления эквивалент кодирует полипептид ICP8, выполняющий функцию (например, отжига с одноцепочечной ДНК (оцДНК), плавления небольших фрагментов двухцепочечной ДНК или дестабилизации дуплексной ДНК во время инициации репликации) на уровне, сопоставимом с ICP8 дикого типа или мутантным ICP8, имеющим аминокислотную последовательность SEQ ID NO: 28. Такие функции можно оценить с помощью методов, доступных в данной области техники, например, Boehmer, PE; Lehman, IR (1993). "Herpes simplex virus type 1 ICP8: Helix-destabilizing properties". Journal of Virology. 67 (2): 711-5. В дополнение или альтернативно, HSV, содержащий такой эквивалент, проникает в клетки и/или распространяется среди клеток и/или реплицирует ДНК на уровне, сопоставимом с неприродным HSV, содержащим мутантный ICP8, имеющий аминокислотную последовательность SEQ ID NO: 28. Неограничивающий пример оценки проникновения HSV в клетки, распространения среди клеток и репликации ДНК можно найти в примерах. В еще одном варианте осуществления полинуклеотид гена, кодирующего ICP8, кодирует полипептид, имеющий аминокислотную последовательность, выбранную из SEQ ID NO: 28, 30, 32 и 34.
[131] В конкретном варианте осуществления ген, кодирующий субъединицу 1 терминазы упаковки ДНК неприродного вируса простого герпеса, включает, предпочтительно состоит по существу или более предпочтительно состоит из полинуклеотида, имеющего последовательность, выбранную из SEQ ID NO: 35, 37, 39, 41, последовательность, по меньшей мере, на 80%, 85%, 90%, 95%, 97% или 99% идентичную любой из SEQ ID NO: 35, 37, 39 и 41, и его эквивалента, при условии что эквивалент сохраняет мутантную или измененную аминокислоту или нуклеотиды по сравнению с последовательностью дикого типа. В дополнительном варианте осуществления HSV, содержащий такой эквивалент, проникает в клетки и/или распространяется среди клеток и/или реплицирует ДНК на уровне, сопоставимом с неприродным HSV, содержащим мутантную субъединицу 1 терминазы упаковки ДНК, имеющую аминокислотную последовательность SEQ ID NO: 36. Неограничивающий пример оценки проникновения HSV в клетки, распространения среди клеток и репликации ДНК можно найти в примерах. В другом варианте осуществления полинуклеотид гена, кодирующего субъединицу 1 терминазы упаковки ДНК, кодирует полипептид, имеющий аминокислотную последовательность, выбранную из SEQ ID NO: 36, 38, 40 и 42.
[132] В конкретном варианте осуществления HSV содержит, предпочтительно состоит по существу или более предпочтительно состоит из одного или нескольких полинуклеотидов, имеющих последовательность, выбранную из SEQ ID NO: 12, 20, 27 и 35. В дополнительном варианте осуществления HSV не имеет функционального ICP34.5 (т.е. кодирует дисфункциональный ICP34.5 или не кодирует ICP34.5). В дополнение или альтернативно, HSV не имеет функционального ICP6 (т.е. кодирует дисфункциональный ICP6 или не кодирует ICP6). В другом варианте осуществления мутация в гене вирулентности включает, или состоит по существу, или состоит из вставки, делеции, усечения, сдвига рамки считывания, замены или точечной мутации, например, гена ICP34.5 и/или гена ICP6. В другом варианте осуществления в HSV отсутствует ген, кодирующий функциональный белок ICP34.5 и/или функциональный белок ICP6. В другом варианте осуществления мутация представляет собой несинонимичную мутацию в гене вирулентности.
[133] В одном варианте осуществления мутация неприродного HSV согласно изобретению кодирует одно или несколько из: (a) мутации аланина в треонин в положении 151 белка gE, (b) мутации аргинина в гистидин в положении 258 белка ICP0, (c) мутации аланина в треонин в положении 376 белка субъединицы 1 терминазы упаковки ДНК, (d) мутации из треонина в метионин в положении 1155 белка ICP8, или (e) мутации пролина в гистидин в положении 119 белка ICP34.5. В другом варианте осуществления неприродный HSV содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из одной или нескольких последовательностей SEQ ID № 2, SEQ ID № 13, SEQ ID № 21, SEQ ID № 28, или SEQ ID № 36, и их эквивалентов при условии, что эквивалент поддерживает мутантную или измененную аминокислоту или нуклеотиды.
[134] В конкретном варианте осуществления обеспечен неприродный вирус простого герпеса («HSV»), который содержит, предпочтительно состоит по существу, или более предпочтительно состоит из мутации в одном или нескольких из: (a) gE, (b) ICP0, (c) субъединицы 1 терминазы упаковки ДНК, (d) ICP8 или (e) ICP34.5. В определенном варианте осуществления HSV не содержит функционального белка ICP34.5 (например, ICP34.5 штамма 17TermA или штамма rRp450). В другом варианте осуществления HSV не содержит белка ICP34.5. В дополнение или альтернативно, HSV не содержит функционального белка ICP6 (например, ICP6 штамма 17TermA или штамма rRp450). В еще одном варианте осуществления HSV не содержит белка ICP6.
[135] В одном варианте осуществления мутация (мутации) в неприродном HSV по настоящему описанию представляет собой одно или несколько из: (a) мутации из аланина в треонин в положении 151 белка gE, (b) мутации из аргинина в гистидин в положении 258 белка ICP0, (c) мутации из аланина в треонин в положении 376 белка субъединицы 1 терминазы упаковки ДНК, (d) мутации из треонина в метионин в положении 1155 белка ICP8, или (e) мутации из пролина в гистидин в положении 119 белка ICP34.5.
[136] В определенном варианте осуществления gE неприродного HSV содержит, предпочтительно состоит по существу, или более предпочтительно состоит из аминокислотной последовательности, выбранной из SEQ ID NO: 13, 15, 17 и 19. В другом варианте осуществления неприродный HSV дополнительно содержит полинуклеотид, кодирующий аминокислотную последовательность gE, например, полинуклеотид, имеющий последовательность, выбранную из SEQ ID NO: 12, 14, 16, 18, и их эквивалент. В конкретном варианте осуществления ICP0 неприродного HSV включает, предпочтительно состоит по существу, или более предпочтительно состоит из аминокислотной последовательности, выбранной из SEQ ID NO: 21, 23 и 26. В дополнительном варианте осуществления неприродный HSV дополнительно содержит полинуклеотид, кодирующий аминокислотную последовательность ICP0, например, полинуклеотид, имеющий последовательность, выбранную из SEQ ID NO: 20, 22, 24, 25, 53, и их эквивалента. В конкретном варианте осуществления ICP8 неприродного HSV содержит, предпочтительно состоит по существу, или более предпочтительно состоит из аминокислотной последовательности, выбранной из SEQ ID NO: 28, 30, 32 и 34. В дополнительном варианте осуществления неприродный HSV дополнительно включает полинуклеотид, кодирующий аминокислотную последовательность ICP8, например, полинуклеотид, имеющий последовательность, выбранную из SEQ ID NO: 27, 29, 31, 33 и их эквивалента. В конкретном варианте осуществления субъединица 1 терминазы упаковки ДНК неприродного HSV содержит, предпочтительно состоит по существу, или более предпочтительно состоит из аминокислотной последовательности, выбранной из SEQ ID NO: 36, 38, 40 и 42. В дополнительном варианте осуществления неприродный HSV дополнительно содержит полинуклеотид, кодирующий аминокислотную последовательность субъединицы 1 терминазы упаковки ДНК, например, полинуклеотид, имеющий последовательность, выбранную из SEQ ID NO: 35, 37, 39, 41, или его эквивалент.
[137] В некоторых вариантах осуществления неприродный HSV содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из одного или нескольких полипептидов, имеющих аминокислотную последовательность, выбранную из SEQ ID NO: 13, 21, 28, и 36. В дополнительном варианте осуществления неприродный HSV содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из одного или нескольких полинуклеотидов, кодирующих одну или несколько аминокислотных последовательностей, выбранных из SEQ ID NO: 13, 21, 28 и 36.
[138] В определенном варианте осуществления неприродный HSV содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из одного или нескольких из: (а) полинуклеотида, кодирующего аминокислотную последовательность, выбранную из SEQ ID № 2, 6, 8, 10 и 52, и/или полинуклеотида, имеющего последовательность, выбранную из SEQ ID № 1, 5, 7, 9 и 51; (b) полипептида, имеющего аминокислотную последовательность, выбранную из SEQ ID № 2, 6, 8, 10 и 52; (c) полинуклеотида, кодирующего аминокислотную последовательность, выбранную из SEQ ID № 13, 15, 17 и 19, и/или полинуклеотида, имеющего последовательность, выбранную из SEQ ID № 12, 14, 16 и 18; (d) полипептида, имеющего аминокислотную последовательность, выбранную из SEQ ID № 13, 15, 17 и 19; (e) полинуклеотида, кодирующего аминокислотную последовательность, выбранную из SEQ ID № 21, 23 и 26, и/или полинуклеотида, имеющего последовательность, выбранную из SEQ ID № 20, 22, 24, 25 и 53, или его последовательность, свободную от одного, двух или более интронов; (f) полипептида, имеющего аминокислотную последовательность, выбранную из SEQ ID № 21, 23 и 26; (g) полинуклеотида, кодирующего аминокислотную последовательность, выбранную из SEQ ID № 28, 30, 32 и 34, и/или полинуклеотида, имеющего последовательность, выбранную из SEQ ID № 27, 29, 31 и 33; (h) полипептида, имеющего аминокислотную последовательность, выбранную из SEQ ID № 28, 30, 32 и 34; (i) полинуклеотида, кодирующего аминокислотную последовательность, выбранную из SEQ ID № 36, 38, 40 и 42, и/или полинуклеотида, имеющего последовательность, выбранную из SEQ ID № 35, 37, 39 и 41; (j) полипептида, имеющего аминокислотную последовательность, выбранную из SEQ ID № 36, 38, 40 и 42; (k) полинуклеотида, кодирующего аминокислотную последовательность, выбранную из SEQ ID № 44, 46, 48 и 50, и/или полинуклеотида, имеющего последовательность, выбранную из SEQ ID № 43, 45, 47 и 49; (l) полипептида, имеющего аминокислотную последовательность, выбранную из SEQ ID № 44, 46, 48 и 50.
[139] В другом варианте осуществления неприродный HSV дополнительно содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из полинуклеотида, последовательность которого идентична по меньшей мере фрагменту гена вирулентности из 17TermA HSV, и его эквивалентов. В другом варианте осуществления неприродный HSV дополнительно содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из полинуклеотида, последовательность которого идентична по меньшей мере фрагменту вирулентного гена из rRp450 HSV. В некоторых вариантах осуществления неприродный HSV происходит от штамма HSV типа 1 («HSV-1») или HSV типа 2 («HSV-2»). В одном варианте осуществления неприродный HSV происходит от штамма KOS HSV-1. В другом варианте осуществления неприродный HSV дополнительно содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из трансгена.
[140] Поскольку описанные здесь вирусы простого герпеса сохраняют свою литическую функцию, в другом аспекте настоящее изобретение обеспечивает способ лечения рака или ингибирования роста или метастазирования раковых клеток у субъекта, нуждающегося в этом, включающий или состоящий по существу, или более предпочтительно состоящий из введения субъекту эффективного количества неприродного вируса простого герпеса или композиции, содержащей, или предпочтительно состоящей по существу, или более предпочтительно состоящей из неприродного вируса простого герпеса. В одном аспекте рак включает рак поджелудочной железы, рак почки, мелкоклеточный рак легкого, опухоль головного мозга, опухоль нервной системы, нейробластому, рак костей, лимфому, миелому, рак толстой кишки, рак матки, рак молочной железы, лейкоз, рак печени, рак простаты, рак кожи или меланому. Подлежащий лечению субъект может быть взрослым или педиатрическим пациентом, например, млекопитающим или пациентом-человеком. В другом варианте осуществления вектор неприродного HSV или композицию, или фармацевтическую композицию применяют местно или системно путем инъекции, инфузии, инстилляции и/или ингаляции. В другом варианте осуществления субъектом является млекопитающее. В некоторых вариантах осуществления млекопитающее представляет собой мышь, крысу, морскую свинку, примата, не являющегося человеком, собаку, кошку, лошадь, корову, свинью, козу или овцу. В другом варианте субъектом является человек.
[141] В другом аспекте в изобретении предложен способ индукции лизиса клеток, который включает, или состоит по существу, или более предпочтительно состоит из обеспечения контакта клетки с неприродным вирусом простого герпеса по настоящему изобретению или композицией, содержащей, или предпочтительно состоящей по существу, или более предпочтительно состоящей из неприродного вируса простого герпеса. В одном варианте осуществления клетка представляет собой раковую клетку. В дополнительном аспекте клетка представляет собой культивируемую клетку (для использования в качестве доклинической модели или доклинического анализа) или клетку, выделенную от субъекта. Клетка может быть культивированной или находиться в выделенной ткани. Неограничивающие примеры таких клеток включают клетки рака поджелудочной железы, рака почки, мелкоклеточного рака легкого, опухоли головного мозга, нейробластомы, опухоли нервной системы, рака костей, лимфомы, миеломы, рака толстой кишки, рака матки, рака молочной железы, лейкоза, рака печени, рака простаты, рака кожи или меланомы. Клетки могут быть выделены от млекопитающих, например, людей, могут быть взрослыми или ювенильными (педиатрическими).
[142] В другом аспекте в изобретении предложен способ инфицирования клетки, включающий, или предпочтительно состоящий по существу, или более предпочтительно состоящий из обеспечения контакта клетки с неприродным HSV. В одном варианте осуществления клетка представляет собой эукариотическую клетку. В другом варианте осуществления клетка представляет собой лимфоцит. В одном варианте осуществления клетка представляет собой раковую клетку, такую как клетка гемобластоза, или клетки солидной опухоли, например, карциномы или саркомы. В дополнительном аспекте клетка представляет собой культивируемую клетку (для использования в качестве доклинической модели или для доклинического анализа) или клетку, выделенную от субъекта. Клетка может быть культивированной или находиться в выделенной ткани. Неограничивающие примеры таких клеток включают клетки рака поджелудочной железы, рака почки, мелкоклеточного рака легкого, опухоли головного мозга, нейробластомы, опухоли нервной системы, рака костей, лимфомы, миеломы, рака толстой кишки, рака матки, рака молочной железы, лейкоза, рака печени, рака простаты, рака кожи или меланомы. Клетки могут быть выделены от млекопитающих, например, людей, которые могут быть взрослыми или детьми (педиатрическими пациентами).
[143] Авторы заявки обнаружили, что обычная группа лимфоцитов, инфицированных вирусом Эпштейна-Барр («EBV»), была устойчивой к обычным онколитическим HSV («oHSV»). Не углубляясь в теорию, устойчивость, по меньшей мере частично, обусловлена низким уровнем экспрессии рецепторов входа HSV на лимфоцитах, пораженных EBV. Неожиданно, HSV по заявленному изобретению был способен индуцировать лизис в EBV-инфицированных лимфоцитах, которые устойчивы к штамму 17 дикого типа, вирусам KOS и McKrae HSV. Следовательно, в одном варианте осуществления клетка включает, или предпочтительно состоит по существу, или более предпочтительно состоит из клетки, инфицированной EBV, или предпочтительно клетки, устойчивой к EBV. В другом варианте осуществления клетка содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из элемента вирулентности вируса Эпштейна-Барр («EBV»). В другом варианте осуществления клетка содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из лимфоцита, инфицированного EBV. В другом варианте осуществления HSV по заявленному изобретению имеет более высокий уровень инфицирования EBV-инфицированных клеток по сравнению с контролем. В одном варианте осуществления контроль включает, или предпочтительно состоит по существу, или более предпочтительно состоит из обычного oHSV. В другом варианте осуществления обычный oHSV включает штамм 17 HSV дикого типа, KOS HSV или McKrae HSV.
Получение мутантов или производных HSV
[144] Получение мутантов или производных HSV включает изменение или мутацию гена или комбинации генов, кодируемых исходным штаммом HSV. Например, производное может иметь последовательность генома HSV-1 или HSV-2, модифицированную нуклеотидными заменами, например, от 1, 2 или 3 до 10, 25, 50 или 100 замен. Геном HSV-1 или HSV-2 может предпочтительно или дополнительно быть модифицирован одной или несколькими вставками и/или делециями и/или удлинением на одном или обоих концах. Способы модификации генов известны в данной области техники, например, CRISPR, рекомбинантная конструкция или точечная мутация. Специалист в данной области техники знает, как получить мутант HSV в зависимости от необходимости.
[145] В дополнение к методам направленной генетической модификации, мутант HSV может быть получен спонтанно. Например, культивирование вирусов, таких как HSV, включает метод, известный как серийный пассаж. Для выращивания и поддержания вирусов подходящие клетки заражают вирусом, вирус реплицируется в клетке, а затем вирус собирают; затем свежие клетки повторно инфицируют, и этот процесс составляет один цикл последовательного пассажа. Каждый такой цикл может занять, например, несколько дней в случае HSV. Как обсуждалось выше, такой последовательный пассаж может привести к изменениям свойств или последовательностей генов вирусного штамма, поскольку происходит отбор свойств, которые будут благоприятствовать клиническому применению HSV. Например, улучшенные свойства могут включать быструю репликацию или способность перемещаться по аксонам для заражения клеток человека. Кроме того, спонтанная мутация может быть вызвана заражением клетки одним или несколькими HSV.
[146] Таким образом, в описании предложен способ получения HSV, или его мутантов или производных путем мутации гена HSV. В другом варианте осуществления способ включает, или предпочтительно состоит по существу, или более предпочтительно состоит из индукции трансгена неприродного HSV.
[147] В другом аспекте в настоящей заявке обеспечен способ получения вирусной частицы HSV, включающий, или предпочтительно состоящий по существу, или более предпочтительно состоящий из: (a) введения в клетку-хозяина вектора 17TermA HSV и вектора rRp450 HSV; (b) выращивания клетки-хозяина по меньшей мере за 3 пассажа; и (c) выделения частицы HSV, продуцируемой клеткой-хозяином. В одном варианте осуществления HSV вводят в клетку-хозяина путем трансфекции, инфицирования, трансформации, электропорации, инъекции, микроинъекции или их комбинации. В другом варианте осуществления клетку-хозяина выращивают по меньшей мере в течение 3 пассажей, 10 пассажей, 20 пассажей, 30 пассажей, 40 пассажей или 50 пассажей. В некоторых вариантах осуществления клетка-хозяин содержит, или предпочтительно состоит по существу или более предпочтительно состоит из продукта комплементарного гена для поддержки репликации введенных векторов HSV. В другом варианте осуществления комплементарный ген кодирует белок ICP6 и/или белок ICP34.5. В другом варианте осуществления полученная таким образом частица HSV содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из вектора HSV в настоящем изобретении.
[148] В определенном варианте осуществления в настоящей заявке обеспечен способ получения неприродной вирусной частицы HSV согласно изобретению. Способ включает, или предпочтительно состоит по существу, или более предпочтительно состоит из: (а) введения в клетку-хозяина неприродного вектора HSV; (b) выращивания клеток-хозяев; и (c) выделения частицы HSV, продуцируемой клеткой-хозяином.
[149] В определенном варианте осуществления в настоящей заявке обеспечен способ получения неприродной вирусной частицы HSV по изобретению, включающий или предпочтительно состоящий по существу, или более предпочтительно состоящий из: (а) введения в клетку-хозяина полинуклеотида, кодирующего вирусный геном неприродного вектора HSV; (b) выращивания клеток-хозяев; и (c) сбора и выделения частицы HSV, продуцируемой клеткой-хозяином. В одном варианте осуществления нуклеиновокислотную последовательность, кодирующую вирусный геном, вводят в клетку-хозяина путем трансфекции, инфицирования, трансформации, электропорации, инъекции, микроинъекции или их комбинации. В одном варианте осуществления нуклеиновокислотную последовательность, кодирующую вирусный геном, вводят в клетку-хозяина в векторе. В другом варианте осуществления вектор представляет собой вирусный вектор (такой как HSV) или невирусный вектор (такой как плазмида или наночастица). В еще одном варианте осуществления вектор представляет собой HSV. В некоторых вариантах осуществления клетка-хозяин содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из продукта комплементарного гена для поддержки репликации введенных векторов HSV. В одном варианте осуществления такой продукт комплементарного гена обеспечен в клетке-хозяине через вирус-помощник. В другом варианте осуществления комплементарный ген кодирует белок ICP6 и/или белок ICP34.5. В другом варианте осуществления полученная таким образом частица HSV содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из вектора HSV в настоящем изобретении.
[150] В одном варианте осуществления стадия выделения относится к процессу существенного отделения HSV от других материалов, таких как клетки-хозяева, клеточный детрит, культуральная среда или любой другой агент, используемый при культивировании клеток-хозяев, например, путем центрифугирования, фильтрации, хроматографии или любой их комбинации. Неограничивающий пример можно найти в Sia et al., «Optimal purification method for Herpes-based viral vectors that confers minimal cytotoxicity for systemic route of vector administration». J. Virol Methods. 2007 Feb; 139(2): 166-74.
Композиции
[151] В другом аспекте в изобретении представлена композиция, содержащая, или состоящая по существу, или более предпочтительно состоящая из неприродного вируса простого герпеса, описанного в настоящей заявке. Композиции, включая фармацевтические композиции, содержащие, или предпочтительно состоящие по существу, или более предпочтительно состоящие из агентов или вирусных частиц, описанных в настоящей заявке, могут быть изготовлены посредством обычных процессов смешивания, растворения, гранулирования, измельчения, эмульгирования, инкапсулирования, улавливания или лиофилизации. Композиции могут быть составлены обычным способом с использованием одного или нескольких физиологически приемлемых носителей, разбавителей, наполнителей или вспомогательных веществ, которые облегчают переработку вирусных частиц, представленных в настоящей заявке, в препараты, которые можно использовать в фармацевтике.
[152] Агенты и вирусные частицы из технологии можно вводить парентерально (например, внутримышечно, интраперитонеально, внутривенно, интрацеребровентрикулярно («ICV»), путем интрацистернальной инъекции или инфузии, подкожной инъекции или имплантата), перорально, с помощью ингаляционного спрея для носа, интравагинально, ректально, сублингвально, уретрально (например, с помощью уретрального суппозитория) или местным путем введения (например, с помощью геля, мази, крема, аэрозоля и т.д.), и они могут быть выполнены в виде композиций, по отдельности или вместе, в подходящих лекарственных формах, содержащих обычные нетоксичные фармацевтически приемлемые носители, адъюванты, наполнители и растворители, подходящие для каждого пути введения.
[153] В одном варианте осуществления настоящее изобретение относится к композиции, содержащей, состоящей по существу, или более предпочтительно состоящей из неприродной вирусной частицы HSV, как описано в настоящей заявке, и носителя.
[154] В другом варианте осуществления настоящее изобретение относится к фармацевтической композиции, содержащей, состоящей по существу, или более предпочтительно состоящей из неприродной вирусной частицы, как описано в настоящей заявке, и фармацевтически приемлемого носителя. В другом варианте осуществления композиция содержит, или предпочтительно состоит по существу, или более предпочтительно состоит из криоконсерванта, который способствует замораживанию и размораживанию неприродного вируса простого герпеса без потери значительной вирулентности.
[155] В другом варианте осуществления настоящее изобретение относится к фармацевтической композиции, содержащей, или предпочтительно состоящей по существу, или более предпочтительно состоящей из терапевтически эффективного количества неприродной вирусной частицы HSV, как описано в настоящей заявке, и фармацевтически приемлемого носителя.
[156] Фармацевтические композиции для применения вирусных частиц HSV могут быть удобно представлены в стандартной лекарственной форме и могут быть приготовлены любым из способов, хорошо известных в области фармацевтики. Фармацевтические композиции могут быть, например, приготовлены путем равномерного и тщательного обеспечения ассоциации представленных здесь вирусных частиц HSV с жидким носителем, тонко измельченным твердым носителем, или обоими из них, а затем, если необходимо, придания продукту формы необходимого состава. В фармацевтическую композицию представленное здесь соединение включают в количестве, достаточном для получения необходимого терапевтического эффекта. Например, фармацевтические композиции по настоящему описанию могут иметь форму, подходящую практически для любого способа введения, включая, например, местное, глазное, пероральное, трансбуккальное, системное, назальное, инъекционное, инфузионное, трансдермальное, ректальное и вагинальное, или форму, подходящую для введения путем ингаляции или инсуффляции.
[157] Для местного применения неприродные вирусные частицы HSV могут быть приготовлены в виде растворов, гелей, мазей, кремов, суспензий и т.д., как хорошо известно в данной области техники.
[158] Системные составы включают составы, предназначенные для введения путем инъекции (например, подкожной, внутривенной, инфузионной, внутримышечной, интратекальной или интраперитонеальной инъекции), а также составы, предназначенные для трансдермального, трансмукозального, перорального или легочного введения.
[159] Подходящие препараты для инъекций включают стерильные суспензии, растворы или эмульсии вирусных частиц HSV, представленные в настоящей заявке, в водных или масляных носителях. Композиции также могут содержать агенты для приготовления, такие как суспендирующие, стабилизирующие и/или диспергирующие агенты. Составы для инъекций могут быть представлены в виде стандартной лекарственной формы, например, в ампулах или в контейнерах для нескольких доз, и могут содержать добавленные консерванты.
[160] В качестве альтернативы инъекционный состав может быть предоставлен в форме порошка для восстановления подходящим носителем, включая, помимо прочего, стерильную апирогенную воду, буфер и раствор декстрозы перед использованием. С этой целью представленные здесь вирусные частицы HSV могут быть высушены любым известным в данной области техники способом, таким как лиофилизация, и восстановлены перед использованием.
[161] Для трансмукозального применения в составе используют пенетранты, подходящие для проницаемого барьера. Такие пенетранты известны в данной области техники.
[162] Для перорального введения фармацевтические композиции могут принимать форму, например, пастилок, таблеток или капсул, приготовленных обычными способами с фармацевтически приемлемыми вспомогательными веществами, такими как связывающие агенты (например, прежелатинизированный кукурузный крахмал, поливинилпирролидон или гидроксипропилметилцеллюлоза); наполнители (например, лактоза, микрокристаллическая целлюлоза или гидрофосфат кальция); любриканты (например, стеарат магния, тальк или диоксид кремния); дезинтегранты (например, картофельный крахмал или натрия крахмал-гликолят); или смачивающие агенты (например, лаурилсульфат натрия). Таблетки можно покрывать способами, хорошо известными в данной области техники, например, сахаром, пленками или энтеросолюбильными покрытиями.
[163] Композиции, предназначенные для перорального применения, могут быть приготовлены любым способом, известным в данной области техники для производства фармацевтических композиций, и такие композиции могут содержать один или несколько агентов, выбранных из группы, состоящей из подсластителей, ароматизаторов, красителей, и консервантов, для получения фармацевтически элегантных и приятных на вкус препаратов. Таблетки содержат вирусные частицы HSV, представленные в настоящей заявке, в смеси с нетоксичными фармацевтически приемлемыми вспомогательными веществами, которые подходят для производства таблеток. Эти вспомогательные вещества могут быть, например, инертными разбавителями, такими как карбонат кальция, карбонат натрия, лактоза, фосфат кальция или фосфат натрия; гранулирующими и дезинтегрирующими агенты (например, кукурузным крахмалом или альгиновой кислотой); связывающими агентами (например, крахмалом, желатином или аравийской камедью); и любрикантами (например, стеаратом магния, стеариновой кислотой или тальком). Таблетки можно оставить без покрытия или на них можно нанести покрытие с помощью известных методов, чтобы замедлить распад и абсорбцию в желудочно-кишечном тракте и, таким образом, обеспечить устойчивое действие в течение более длительного периода. Например, можно использовать материал для обеспечения пролонгированного действия, такой как глицерин моностеарат или глицерин дистеарат. На них также можно наносить покрытие с помощью методов, хорошо известных специалисту в данной области техники. Фармацевтические композиции по технологии также могут быть в форме эмульсий масло-в-воде.
[164] Жидкие препараты для перорального введения могут иметь форму, например, эликсиров, растворов, сиропов или суспензий, или они могут быть представлены в виде сухого продукта для смешивания с водой или другим подходящим носителем перед применением. Такие жидкие препараты могут быть приготовлены обычными способами с фармацевтически приемлемыми добавками, такими как суспендирующие агенты (например, сироп сорбита, производные целлюлозы или гидрогенизированные пищевые жиры); эмульгирующие агенты (например, лецитин или аравийская камедь); неводные носители (например, миндальное масло, масляные сложные эфиры, этиловый спирт, Кремофор™ или фракционированные растительные масла); и консерванты (например, метил- или пропил-п-гидроксибензоаты или сорбиновая кислота). Препараты также могут содержать буферные соли, консерванты, ароматизаторы, красители и подсластители, если это необходимо.
[165] В одном варианте осуществления обеспечен способ получения неприродного вируса простого герпеса по настоящему изобретению, включающий, или предпочтительно состоящий по существу, или более предпочтительно состоящий из мутации гена в частице неприродного HSV, или введения в неприродный HSV трансгена. В другом аспекте способ получения вектора неприродного HSV включает, или предпочтительно состоит по существу, или более предпочтительно состоит из: (a) введения в клетку-хозяина вектора 17TermA HSV и вектора rRp450 HSV; (b) выращивания клетки-хозяина по меньшей мере за 3 пассажа; и (c) выделения частиц HSV, продуцируемых клеткой-хозяином.
[166] Также обеспечен способ ингибирования роста или метастазирования раковой клетки или метастатической раковой клетки, включающий, или состоящий по существу, или более предпочтительно состоящий из обеспечения контакта клетки с эффективным количеством вектора неприродного HSV или композиции, или фармацевтической композиции, содержащей вектор неприродного HSV, как описано в настоящей заявке. Контакт осуществляют in vitro или in vivo. В одном аспекте контакт осуществляют in vivo путем введения субъекту неприродного HSV, или композиции или фармацевтической композиции. In vitro этот способ осуществляют путем приведения неприродного вируса простого герпеса в контакт с клеткой. Способ in vitro можно использовать для тестирования новых методов лечения или в качестве индивидуального анализа, чтобы определить, подходит ли терапия для лечения рака. Дополнительные методы лечения рака можно комбинировать с терапией, которая может быть параллельной или последовательной по отношению к раскрытым способам.
[167] Раковая клетка, подлежащая лечению, может быть солидной опухолью или гемобластозом, например, карциномой или саркомой, и неограничивающие примеры таких клеток включают рак поджелудочной железы, рак почки, мелкоклеточный рак легкого, опухоль головного мозга, нейробластому, опухоль нервной системы, рак костей, лимфому, миелому, рак толстой кишки, рак матки, рак молочной железы, лейкоз, рак печени, рак простаты, рак кожи или меланому. Клетка относится к любому виду, например, млекопитающему и человеку, и при проведении in vitro она может быть получена из культивируемой клеточной линии или первичной клетки, например, из биоптата ткани. Клетка может быть зрелой или ювенильной клеткой, или раковой стволовой клеткой, полученной при биопсии ткани. Клетка может быть зрелой или ювенильной клеткой, или раковой стволовой клеткой (т.е. раковой клеткой, обладающей характеристиками, связанными с нормальными стволовыми клетками, особенно способностью давать начало всем типам клеток, обнаруженным в конкретном образце рака) или раковой клеткой без таких характеристик, связанных с нормальными стволовыми клетками. В одном варианте осуществления клетка экспрессирует протеин протоонкогена N-myc (MYCN) и/или экспрессирует MYCN на более высоком уровне, чем в нераковых клетках.
[168] В настоящем изобретении дополнительно обеспечен способ лечения рака или ингибирования роста или метастазирования раковой клетки у субъекта, нуждающегося в этом, включающий или предпочтительно состоящий по существу, или более предпочтительно состоящий из введения субъекту эффективного количества неприродного HSV, композиции или фармацевтической композиции по настоящему изобретению. Подлежащий лечению субъект может быть субъектом любого вида, например, млекопитающего и человека, например, собакой, лошадью, крупным рогатым скотом, кошкой, обезьяной, крысой или мышью. Введение может быть терапией первой линии, терапией второй линии, терапией третьей линии, терапией четвертой линии или терапией пятой линии. Дополнительные методы лечения рака можно комбинировать с терапией, которая может быть параллельной или последовательной по отношению к раскрытым способам. Подлежащий лечению рак может быть солидной опухолью или гемобластозом, например, карциномой или саркомой, и неограничивающие примеры включают рак поджелудочной железы, рак почки, мелкоклеточный рак легкого, опухоль головного мозга, нейробластому, опухоль нервной ткани, рак костей, лимфому, миелому, рак толстой кишки, рак матки, рак молочной железы, лейкоз, рак печени, рак простаты, рак кожи или меланому.
[169] Способ из настоящего изобретения может быть объединен с соответствующей диагностикой для мониторинга ремиссии или прогрессирования заболевания. В данной области техники известно несколько методов такого мониторинга.
[170] Кроме того, предложен способ индукции лизиса клеток, включающий или предпочтительно состоящий по существу, или более предпочтительно состоящий из обеспечения контакта клетки с эффективным количеством неприродного вируса HSV, композиции и/или фармацевтической композиции из настоящего изобретения. Контакт осуществляют in vitro или in vivo. В одном аспекте контакт осуществляют in vivo путем введения субъекту неприродного HSV, или композиции или фармацевтической композиции. In vitro этот способ осуществляют путем помещения неприродного HSV в контакт с клеткой. Способ in vitro можно использовать для тестирования новых методов лечения или в качестве индивидуального анализа, чтобы определить, подходит ли терапия для пациента, подлежащего лечению. Дополнительные клеточнолитические терапии можно комбинировать с терапией, которая может быть параллельной или последовательной по отношению к раскрытым способам.
[171] Клетка, подлежащая лечению, может быть солидной опухолью или гемобластозом, например, карциномой или саркомой, и неограничивающие примеры таких клеток включают рак поджелудочной железы, рак почки, мелкоклеточный рак легкого, опухоль головного мозга, нейробластому, опухоль нервной ткани, рак костей, лимфому, миелому, рак толстой кишки, рак матки, рак молочной железы, лейкоз, рак печени, рак простаты, рак кожи или меланому. Клетка относится к любому виду, например, млекопитающему и человеку, и при проведении in vitro она может быть получена из культивируемой клеточной линии или первичной клетки, например, из биоптата ткани. Клетка может представлять собой зрелую или ювенильную клетку, или раковую стволовую клетку, или раковую клетку, не обладающую характеристиками, присущими нормальным стволовым клеткам. Терапия может быть в сочетании с соответствующим анализом для проверки эффективности терапии, например, ремиссии или прогрессирования рака.
Использование вирусных частиц HSV для приготовления лекарств
[172] HSV и композиции по настоящему изобретению также можно использовать при приготовлении лекарственных средств для лечения различных патологий, как описано в настоящей заявке. Способы и приемы приготовления лекарственных средств композиции известны в данной области техники. Только в целях иллюстрации здесь подробно описаны фармацевтические составы и пути доставки.
[173] Таким образом, специалист в данной области техники легко поймет, что любую одну или несколько композиций, описанных выше, включая множество конкретных вариантов осуществления, можно использовать путем применения стандартных процедур фармацевтического производства для приготовления лекарственных средств для лечения многих расстройств, описанных в настоящей заявке. Такие лекарства можно доставлять субъекту с использованием способов доставки, известных в фармацевтике.
Применение дополнительных терапевтических агентов
[174] Способы, раскрытые в настоящей заявке, могут дополнительно включать, или, предпочтительно состоять по существу, или более предпочтительно состоять из введения эффективного количества дополнительных терапевтических агентов для увеличения или усиления терапевтической эффективности раскрытых способов. В одном варианте осуществления дополнительные терапевтические агенты включают, или предпочтительно состоят по существу, или более предпочтительно состоят из хирургической резекции опухоли, применения противоопухолевого агента, такого как малая молекула, или иммунотерапии, или клеточнолитической терапии.
[175] Применение терапевтического агента или вещества по настоящему изобретению у пациента будет происходить в соответствии с общими протоколами применения этой конкретной первичной или вторичной терапии с учетом токсичности лечения, если таковая имеется. Ожидается, что циклы лечения будут повторяться по мере необходимости. Также предполагается, что различные стандартные методы лечения, а также хирургическое вмешательство могут применяться в сочетании с описанной терапией.
[176] Как очевидно специалистам в данной области техники, комбинированная терапия может принимать форму комбинированной терапии для одновременного или последовательного введения.
Наборы
[177] Агенты или неприродные HSV, описанные в настоящей заявке, в некоторых вариантах осуществления могут быть собраны в фармацевтические, диагностические или исследовательские наборы для облегчения их использования в терапевтических, диагностических или исследовательских целях. Набор может включать один или несколько контейнеров, содержащих компоненты изобретения и инструкции по применению. В частности, такие наборы могут включать один или несколько агентов, описанных в настоящей заявке, вместе с инструкциями, описывающими предполагаемое применение и правильное использование этих агентов. В некоторых вариантах осуществления агенты в наборе могут находиться в фармацевтическом составе и в дозировке, подходящих для конкретного применения и для способа введения агентов. Наборы для исследовательских целей могут содержать компоненты в соответствующих концентрациях или количествах для проведения различных экспериментов.
[178] Набор может быть разработан для облегчения использования описанных здесь способов и может принимать различные формы. Каждая из композиций набора, где это применимо, может быть предоставлена в жидкой форме (например, в растворе) или в твердой форме (например, в сухом порошке). В некоторых случаях некоторые композиции могут быть составными или обработанными иным образом (например, до активной формы), например, путем добавления подходящего растворителя или других веществ (например, воды или среды для культивирования клеток), которые могут входить или не входить в комплект. В некоторых вариантах осуществления композиции могут быть представлены в консервирующем растворе (например, в растворе для криоконсервации). Неограничивающие примеры консервирующих растворов включают ДМСО, параформальдегид и CryoStor® (Stem Cell Technologies, Ванкувер, Канада). В некоторых вариантах осуществления консервирующий раствор содержит некоторое количество ингибиторов металлопротеаз.
[179] Используемый в настоящей заявке термин «инструкции» может определять компонент инструкции и/или рекламы, и обычно включает письменные инструкции на упаковке или связанные с упаковкой по изобретению. Инструкции также могут включать любые устные или электронные инструкции, предоставленные любым способом, чтобы пользователь мог четко понять, что инструкции должны быть связаны с комплектом, например, аудиовизуальные (например, видеокассета, DVD и т.д.), Интернет и/или связи через Интернет и т.д. Письменные инструкции могут быть в форме, предписанной правительственным агентством, регулирующим производство, использование или продажу фармацевтических или биологических продуктов, эти инструкции также могут отражать одобрение агентства по производству, использованию или продаже для применения у животных.
[180] Набор может содержать один или несколько компонентов, описанных в настоящей заявке, в одном или нескольких контейнерах. В качестве примера, в одном варианте осуществления набор может включать инструкции по смешиванию одного или нескольких компонентов набора и/или по выделению и смешиванию образца и применению у субъекта. Набор может включать контейнер с описанными здесь агентами. HSV могут быть в форме жидкости, геля или твердого вещества (порошка). HSV могут быть приготовлены стерильно, упакованы в шприцы и отправлены в охлажденном состоянии. В качестве альтернативы, вирус может быть помещен во флакон или другой контейнер для хранения. Во втором контейнере могут быть стерильно приготовлены другие агенты. В качестве альтернативы, набор может включать активные агенты, предварительно смешанные и отправленные в шприце, флаконе, пробирке или другом контейнере. Набор может содержать один или несколько, или все компоненты, необходимые для введения агентов субъекту, такие как шприц, устройства для местного нанесения или трубка для внутривенной иглы и мешок.
Скрининговые анализы
[181] Настоящее изобретение также обеспечивает скрининговые анализы для идентификации потенциальных терапевтических агентов известных и новых соединений и комбинаций. Например, специалист в данной области техники может также определить, обеспечивает ли HSV терапевтический эффект in vitro, путем контакта HSV с образцом клетки или ткани, подлежащей лечению. Клетка или ткань могут быть любого вида, например, от обезьяны, собаки, коровы, овцы, крысы, мыши или человека.
[182] Контакт также может быть осуществлен in vivo на подходящей животной модели или пациенте-человеке. При проведении in vitro HSV можно напрямую добавлять в среду для культивирования клеток. При практическом применении in vitro способ можно использовать для скрининга новых комбинированных терапий, составов или схем лечения перед введением животному или пациенту-человеку.
[183] В другом аспекте анализ требует контакта первого образца, содержащего подходящие клетки или ткань («контрольный образец»), с эффективным количеством HSV, как раскрыто в настоящей заявке, и контакта второго образца подходящих клеток или ткани («тестируемый образец») с исследуемым вирусом простого герпеса, агентом или комбинацией. В одном аспекте в случае рака определяют ингибирование роста первого и второго клеточных образцов. Если ингибирование роста второго образца по существу такое же или больше, чем у первого образца, то агент является потенциальным лекарственным средством для терапии. В одном аспекте по существу такое же или большее ингибирование роста клеток представляет собой разницу менее примерно 1%, или предпочтительно менее примерно 5%, или предпочтительно менее примерно 10%, или предпочтительно более примерно 10%, или предпочтительно более примерно 20%, или предпочтительно более примерно 50%, или предпочтительно более примерно 90%. Контакт может быть осуществлен in vitro или in vivo. Способы определения ингибирования роста клеток хорошо известны в данной области техники.
[184] В дополнительном аспекте проводят контакт тестового агента с третьим образцом клеток или ткани, содержащим нормальные аналогичные клетки или ткань для контроля, и тестовым образцами, и выбирают агенты, которые воздействуют на второй образец клеток или ткани, но не оказывают отрицательного воздействия на третий образец. Для целей описанных здесь анализов в настоящей заявке указаны подходящие клетка или ткань, например, от рака или других заболеваний, как описано в настоящей заявке. Примеры таких клеток и тканей включают раковые клетки или ткани, полученные путем биопсии или из крови, но не ограничиваются этим.
185] Эффективность исследуемой композиции определяют с использованием методов, известных в данной области техники, которые включают анализы жизнеспособности клеток, но не ограничиваются ими.
[186] В еще одном аспекте для анализа требуются по меньшей мере два типа клеток, первый из которых является подходящей контрольной клеткой.
[187] Анализы также полезны для прогнозирования того, будет ли субъект лечиться соответствующим образом в соответствии с настоящим изобретением, путем доставки HSV к образцу, содержащему клетку, подлежащую лечению, и анализа лечения, который будет варьировать в зависимости от патологии, или для скрининга на новые препараты и комбинации. В одном аспекте клетку или ткань получают от субъекта или пациента путем биопсии. Настоящее изобретение также обеспечивает наборы для определения того, будет ли патологическая клетка или пациент лечиться подходящим образом с помощью этой терапии, путем обеспечения по меньшей мере одной композиции из настоящего изобретения и инструкций по применению.
[188] Тестовые клетки можно выращивать в небольших многолуночных планшетах и использовать для определения биологической активности тестируемых соединений. Для целей этого изобретения успешный HSV или другой агент будет блокировать рост или убивать раковые клетки, но оставлять контрольные клетки невредимыми.
[189] Следующие ниже примеры приведены для иллюстрации, но не для ограничения изобретения.
Примеры
Получение мутанта HSV
[190] Непермиссивную клеточную линию инфицировали 17TermA и rRp450 («направленная эволюция») и культивировали после серийного пассажа для выделения мутанта HSV Mut-3, который содержит WT-подобный генотип. Затем мутант HSV Mut-3 использовали для конструирования аттенуированного мутанта Mut-3Δ34.5 посредством редактирования гена (обозначенного как «CRISPR/Cas9») (фиг. 1A). Сравнение последовательностей Mut-3 с его исходными вирусами показано на фиг. 1В. Несинонимичные мутации, которые отличаются от обоих исходных вирусов в Mut-3, заштрихованы обратной косой чертой, включая UL15, UL29, US8, RL1 и RL2. Последовательности генома, идентичные 17TermA, обозначены незакрашенными прямоугольниками; последовательности, идентичные rRp450, заштрихованы косой чертой.
[191] Был выделен активный мутант oHSV, Mut-3, который содержит генотип, подобный WT. И Mut-3, и аттенуированная версия Mut-3Δ34.5 основаны на канонических белках входа HSV, нектине-1 или медиаторе входа вируса герпеса (HVEM), для достижения успешного заражения (не показано). Кинетика переноса вирусного гена Mut-3Δ34.5, измеренная по появлению детектируемой GFP-положительной области, является гораздо более ранней по сравнению с 17Δ34.5, γ134.5-нулевым вирусом, полученным из штамма 17 дикого типа с помощью той же стратегии редактирования гена CRISPR/Cas9. Кроме того, инфекция Mut-3Δ34.5 приводит к большему уничтожению клеток, при измерении по меньшей площади слияния клеток. Однако Mut-3Δ34.5, по-видимому, менее репликативен по сравнению с 17Δ34.5 или исходным Mut-3Δ34.5-нулевым вирусом 17TermA. Эти результаты показывают, что даже в аттенуированной версии Mut-3Δ34.5 уникальное геномное изменение (изменения) приводит к синцитиальному фенотипу (не показано), и сохраняется повышенная эффективность. В исследовании HSV, перенацеленного на рецептор эпидермального фактора роста (EGFR), было сообщено, что введение синцитиальной мутации не ухудшает специфичность проникновения и распространения. Не углубляясь в теорию, авторы заявки полагают, что основная причина синцитиальных фенотипов в Mut-3Δ34.5 может приводить к измененной кинетике при слиянии вируса с клеточной мембраной, что может повлиять на проникновение и/или опосредованное вирусом слияние клеток, что приводит к более быстрому уничтожению клеток и снижению выработки вирусных частиц.
[192] Мутации не были обнаружены в gB, gD, gH и gL, которые являются каноническими гликопротеинами, участвующими в проникновении HSV-1. Однако пять генов содержат несинонимичные (NS) мутации в Mut-3, которые отличаются от любого из исходных вирусов: RL1, RL2, UL15, UL29 и Us8/gE. Mut-3 и его аттенуированная версия Mut-3Δ34.5 проявляют фузогенный фенотип, что позволяет предположить, что это явление не связано с RL1. Полногеномный анализ также предполагает, что этот фенотип не связан с ранее описанными мутациями, поскольку аминокислотные последовательности Mut-3 либо идентичны rRp450 и геному эталонного штамма 17 (не синцитиального вируса) в gK/UL53 и UL20, либо идентичны 17TermA (не синцитиальный вирус) в gB/UL27 и UL24. Мутация A151T gE/Us8 является единственным гликопротеином (поверхностным белком вириона) в Mut-3, который не имеет значимого изменения по сравнению с его исходными вирусами. Хотя gE не был связан ни с прикреплением, ни с проникновением вируса, димеризация gE/gI опосредует распространение вируса от клетки к клетке, поскольку мутации в gE были связаны с меньшими размерами бляшек. ** Заявитель предполагает, что A151T gE в Mut-3Δ34.5 может быть основной причиной его синцитиального фенотипа и фенотипа повышенной активности. Основываясь на приоритете исследования заявителя (сверху вниз), в таблице 1 автор заявки перечисляет все пять мутаций NS, обнаруженных в пяти генах в Mut-3, а также соответствующую функцию каждого гена, возможную роль, связанную с фенотипом повышенной активности Mut-3, и предложенные заявителем исследования. Другие три мутации (кроме RL1) также могут быть определены, как это было сделано для gE A151T.
Таблица 1. Мутации белков HSV и их функции. Nt - нуклеотид; а.а. - аминокислота
Сравнение мутантов HSV
[193] Анализы методом бляшек проводили на четырех вирусах, показанных на фиг. 1A, одновременно, сканировали и анализировали изображение бляшек через 3 дня с помощью многофункционального флуоресцентного микроскопа Keyence HS BZ-II. Как показано на фиг. 1A и 1B, размеры бляшек Mut-3 и Mut-3Δ34.5 были значительно больше, чем у обоих исходных вирусов rRp450 и 17TermA. В анализе цитотоксичности/ MTS in vitro наборов клеток CHO инфицировали CHO-K1, CHO-Nectin-1, CHO-Nectin-2 и CHO-HVEM четырьмя вирусами с различной множественностью инфицирования (MOI). Анализ выживаемости клеток с помощью колориметрического теста клеточной пролиферации и теста MTS проводили через 3 дня после инфицирования вирусом (pvi) относительно необработанного контроля. Только CHO-Nectin-1 и CHO-HVEM, но не CHO-K1 или CHO-Nectin-2 (в основном для проникновения HSV-2) были чувствительны к обработке четырьмя вирусами (фиг. 2C). Не углубляясь в теорию, результаты позволяют предположить, что Mut-3 и Mut-3Δ34.5 не проходят через рецепторный барьер, по-прежнему используя канонические рецепторы входа HSV для инфицирования клеток.
[194] Повышенная эффективность Mut-3Δ34.5 в отношении уничтожения клеток нейробластомы человека и мыши по сравнению с 17TermA не была связана с повышенным выходом инфекционного вируса (фиг. 3). Анализ с помощью трансмиссионной электронной микроскопии («ТЭМ») также показал, что после инфицирования клеток нейробластомы вирионы Mut-3Δ34.5 в основном обнаруживались в эндоцитарных везикулах, в то время как вирионы 17TermA в основном обнаруживались в эндоцитарных везикулах (фиг. 4).
[195] Аттенуированный мутант 17Δ34.5 был получен методом редактирования гена CRISPR-Cas9 для замены гена g134.5 в штамме дикого типа 17+ кассетой, экспрессирующей EGFP. Мутант 17Δ34.5 имел более низкую эффективность, чем штамм дикого типа 17+, но сопоставимую с 17TermA (фиг. 5). Аттенуирование 17Δ34.5 было дополнительно подтверждено, когда Mut-3Δ34.5 проявил гораздо более быстрый перенос вирусного гена и гибель клеток по сравнению с 17Δ34.5 (фиг. 6). Кроме того, Mut-3Δ34.5 был более эффективным, чем 17TermA, для контроля роста нейробластомы человека in vivo (фиг. 7).
Ревертант Us8/gE в Mut-3Δ34.5 для исследования потери функции
[196] Внесение изменения единственного нуклеотида в геном HSV исключительно с помощью технологии CRISPR/Cas9 является сложным из-за множественных копий генома во время репликации вируса и невозможности сопровождать редактирование этого гена маркером отбора. Одна конструкция может быть результатом двухэтапного процесса в сочетании с технологией CRISPR/Cas9: замена всей кодирующей области gE репортерным геном (таким как mCherry или красный флуоресцентный белок (RFP)) для создания gE-null Mut- 3Δ34.5; затем (2) замена репортерного гена на кодирующую область gE WT, в результате чего получается ревертант gE-WT Mut-3Δ34.5. Исследование степени, в которой как gE-нулевой промежуточный продукт, так и этот gE-ревертант Mut-3Δ34.5, теряют фенотип, можно проводить, как указано выше. Настоящее изобретение также обеспечивает эту конструкцию.
Мутация Us8/gE A151T в исходном Mut-3 17TermA для приобретения функции
[197] Как указано выше, мутант gE 17TermA можно сконструировать в два этапа: (1) полностью выключить путем нокаута кодирующую область gE и заменить ее репортерным геном (например, GFP); затем (2) осуществить замену репортерного гена путем нокаута в кодирующей области gE, содержащей мутацию A151T. Фенотип Mut-3Δ34.5 будет определен. Настоящее изобретение также обеспечивает эту конструкцию.
[198] Если gE A151T отвечает только за частичный или ни один из наблюдаемых фенотипов, или даже если это так, в настоящем изобретении предусмотрены другие мутации в Mut-3. Мутанты конструируют путем систематического применения аналогичных подходов к приобретению и потере функции, как описано в настоящей заявке и как указано в таблице 1, по отдельности или в комбинации. Профиль безопасности и эффективность аттенуированного Mut-3Δ34.5 можно проверить на мышиных моделях. Таким образом, в настоящем изобретении представлены животные модели, используемые для тестирования мутантов, а также способы анализа.
[199] Штамм Mut-3, активный и WT-подобный мутант HSV, был сконструирован путем рекомбинации 17TermA и rRp450. Аттенуированная версия, Mut-3Δ34.5, была создана путем делеции γ134.5/RL1, белка вирулентности вируса, чтобы гарантировать его безопасность для клинического использования. Никаких неблагоприятных клинических признаков или значительных изменений массы тела у мышей Balb/c более чем через 85 дней после внутривенного введения до 1e8 бляшкообразующих единиц (БОЕ) в пилотном токсикологическом исследовании автора заявки не наблюдалось (не показано). Кроме того, Mut-3Δ34.5 также демонстрирует противоопухолевую эффективность на модели высокоагрессивной нейробластомы как in vitro (фиг. 3A), так и in vivo (фиг. 7), по сравнению с 17TermA.
Профиль биораспределения Mut-3Δ34.5 у интактных мышей, не несущих опухоли
[200] Собственное исследование автора заявки показывает, что интактные мыши Balb/c могут переносить до 1-8 БОЕ введенного внутривенно (в/в) вируса Mut-3Δ34.5 без каких-либо физических признаков заболевания в течение более 85 дней. Исследование биораспределения проводят, начиная с внутривенного введения ранее протестированной наивысшей дозы (1е8 БОЕ вируса Mut-3Δ34.5 на мышь) у интактных мышей Balb/c, не несущих опухоли, обоих полов (30 мышей каждого пола). Собирают периферическую кровь, а затем мышей умерщвляют. Семенники, яичники, селезенку, легкие, почки, сердце, легкие и головной мозг собирают через 24 часа, 14 дней, 28 дней, 56 дней и 85 дней pvi (n = 6 в каждой точке). Половину органов заливают формалином для сохранения с целью анализа патологии, а другую половину гомогенизируют для анализа генома HSV и анализа бляшек методом кПЦР для определения вирусной нагрузки в каждом органе. За мышами наблюдают ежедневно в течение первых двух недель после введения вируса, а затем дважды в неделю до запланированного дня умерщвления. Массу тела каждой мыши измеряют до введения вируса, а затем еженедельно. Мышей умерщвляют, чтобы показать наличие или отсутствие неблагоприятных клинических признаков или потерю массы тела >20%, и анализируют вирусную активность в органах, как описано выше. Параллельно, вирус KOS дикого типа [диапазон доз: от 1 × 105 до 1 × 107 БОЕ на мышь (n = 3)] вводят группам мышей в качестве положительного (небезопасного) контроля. Автор заявки ранее обнаружил, что доза вируса KOS 1 × 106 БОЕ была равномерно летальной в течение 2-3 дней для мышей FVBN. Тест кПЦР выполняют для определения копий генома HSV, подсчета бляшек для оценки вирусной активности и анализа патологии в органах мышей положительного контроля, у которых проявляются признаки болезни. Эти результаты могут служить в качестве положительных индикаторов/ пороговых значений для оценки данных, собранных у мышей, получавших Mut-3Δ34.5. Патологические изменения в тканях/органах в группах, получавших Mut-3Δ34.5, дополнительно оценивают, чтобы показать сопоставимые вирусные нагрузки с положительным контролем. 6 мышей на группу для каждого пола используют для оценки биораспределения, безопасности и толерантности в разные периоды времени. Биораспределение Mut-3Δ34.5 измеряют количеством копий генома HSV на нанограмм геномной ДНК в разных органах в разное время с описательной статистикой, и сравнивают с одномерными анализами (если применимо).
Цитотоксичность Mut-3Δ34.5 с другими 134.5-нулевыми вирусами (17TermA и T-VEC) в различных линиях педиатрических раковых клеток in vitro
[201] Превосходное уничтожение с помощью Mut-3Δ34.5 наблюдалось по сравнению с 17TermA в клетках нейробластомы человека и мыши (фиг. 3A). Используя анализ MTS in vitro, показанный на фиг. 3A, тот же анализ применяют к другим педиатрическим раковым клеткам, таким как саркомы, злокачественная опухоль оболочки периферических нервов (MPNST) и опухоли головного мозга (у автора заявки есть доступ к многочисленным моделям), чтобы определить, применим ли фенотип повышенной эффективности к различным типам опухолей. Наиболее эффективную линию каждого типа клеток используют для проведения исследований эффективности in vivo.
Изучение эффективности Mut-3Δ34.5 на моделях педиатрических опухолей человека по сравнению с другими терапевтическими средствами oHSV, 17TermA и T-VEC
[202] Была выбрана одна из высокочувствительных моделей каждого типа опухоли (всего три) для проведения исследований эффективности на ксенотрансплантатах с использованием самок бестимусных мышей в возрасте от 5 до 6 недель. Когда опухоль достигает 150-300 мм3, мышей объединяют и произвольно распределяют в 3 группы (n = 11 в каждой): (i) контроль с фосфатно-солевым буфером (ФБР), (ii) вирус Mut-3Δ34.5, или (iii) вирус 17TermA или T-VEC. Для сравнения эффективности между вирусами минимум 11 мышей в группе используют для обнаружения больших различий (20% против 80% на d20) в выживаемости и росте опухоли с минимальной активностью 80%. Каждой мыши вводят внутриопухолевым путем либо 1 × 107 БОЕ вируса в 100 мкл ФБР, либо только ФБР (контроль) через день с тремя инъекциями по общей схеме лечения, основанной на предыдущем исследовании автора заявки. У мышей оценивают объем опухоли (дважды в неделю) и массу тела (еженедельно) в течение 80 дней после инъекции вируса. Критерии конечной точки включают объем опухоли, превышающий 2500 мм3, диаметр опухоли, достигающий 2 см, или потерю массы тела >20%. Выживаемость животных можно отобразить с помощью кривых Каплана-Мейера, и можно сравнить между группами с помощью логранговых тестов.
Эффективность Mut-3Δ34.5 на педиатрических опухолевых моделях у мышей по сравнению с другими терапевтическими средствами oHSV (17TermA и T-VEC)
[203] Была выбрана одна из высокочувствительных моделей опухоли у мышей каждого типа опухоли (всего две) для проведения исследований эффективности автора заявки у мышей C57BL6 в возрасте 5-6 недель, совпадающих по полу. Точно так же мышей объединяют и произвольно распределяют в 3 группы по n = 11 в каждой: (i) ФБР, (ii) Mut-3Δ34, (iii) 17TermA или T-VEC. Каждой мыши вводят внутриопухолевым путем либо 1е8 БОЕ вируса в 100 мкл ФБР, либо только через день с тремя инъекциями, как это было в предыдущем исследовании автора заявки. Критерии конечной точки включают объем опухоли, превышающий 2500 мм3, диаметр опухоли, достигающий 2 см, или потерю массы тела >20%. Выживание животных будут оценивать с использованием кривых Каплана-Мейера, и сравнивать между группами по логранговым тестам.
Резюме анализа патологии
[204] Ткани от 4 мышей, которым вводили KOS дикого типа (1e6 и 1e7 БОЕ, 2 мыши на дозу), были отправлены на патологический анализ через общий ресурс OSU Comparative Pathology & Mouse Phenotyping Shared Resource. Ткани от мышей, которым вводили Mut-3D34.5/C8G5 и Mut-3DICP6/D7-1 (временные точки pvi 24 часа и 14 дней, 2 мыши на момент времени), впоследствии отправляли на анализ патологии.
Отчет о патологии (самки): мыши, которым вводили KOS
[205] Лимфоплазмоцитарный энцефалит ствола мозга согласуется с опубликованными сообщениями о патологии ЦНС у мышей Balb/c, которым вводили HSV-1. Все мыши, исследованные в этой заявке, имели это поражение, хотя у одной представленной мыши был только небольшой сегмент ствола мозга, и, таким образом, поражение выглядело более мягким, чем у трех других мышей. Представлены срезы надпочечников и почек двух мышей. У обеих этих мышей был заметный некроз как коры надпочечников, так и мозгового вещества.
Отчет о патологии (самки): 24 часа и 14 дней для мышей, которым вводили Mut-3Δ34.5/C8G5 и Mut-3ΔICP6/D7-1
[206] Все образцы от мышей, умерщвленных через 24 часа после инфицирования HSV, продемонстрировали следующие поражения от умеренной до выраженной степени тяжести: перипортальный или среднезональный некроз печени, включая отдельные гепатоциты и некроз фокально расширенных печеночных пластинок; выраженный некроз красной пульпы селезенки и умеренный некроз белой пульпы с фолликулами меньшего размера, чем ожидалось. Несколько 24-часовых образцов мышей показали повышенный апоптоз/некроз клеток в фолликулах и желтом теле в ткани яичников. Это не превышало ожидаемых количеств у контрольных мышей, оцененных несколько месяцев назад, а у мышей, указанных здесь, апоптоз/некроз превышал ожидаемые количества.
[207] У мышей на 14-й день гиперплазия овальных клеток является обычной хронической реакцией поврежденной печени, и вероятно, реакцией на инфекцию. Несколько образцов легкого показали некротические и воспалительные поражения, которые не были отмечены в 24-часовых образцах животных. В целом, у мышей на 14-й день было гораздо меньше поражений, и они были намного мягче, чем в 24-часовых образцах животных.
[208] Мутантные вирусы, разработанные в лаборатории, могут способствовать некрозу различных типов клеток, особенно через 24 часа, включая гепатоциты, спленоциты в красной и белой пульпе, и клетки яичника.
Эквиваленты
[209] Если не указано иное, все технические и научные термины, используемые в настоящей заявке, имеют то же значение, которое обычно понимается специалистом в данной области техники, к которой принадлежит это изобретение.
[210] Изобретения, иллюстративно описанные здесь, могут быть подходящим образом реализованы на практике при отсутствии какого-либо элемента или элементов, ограничений или ограничений, конкретно не раскрытых в настоящей заявке. Таким образом, например, термины «содержащий», «включающий» и т.д. следует читать широко и без ограничений. Кроме того, используемые здесь термины и выражения использовались как термины описания, а не ограничения, и при использовании таких терминов и выражений нет намерения исключать какие-либо эквиваленты показанных и описанных функций или их частей, но признается, что в объеме заявленного изобретения возможны различные модификации.
[211] Таким образом, следует понимать, что, хотя настоящее изобретение было конкретно раскрыто посредством предпочтительных вариантов осуществления и дополнительных функций, к модификациям, усовершенствованию и вариациям воплощенных в нем изобретений, раскрытых в настоящей заявке, могут прибегать специалисты в данной области техники, и что такие модификации, улучшения и вариации считаются входящими в объем настоящего изобретения. Материалы, методы и примеры, представленные в настоящей заявке, представляют предпочтительные варианты осуществления, являются иллюстративными и не предназначены для ограничения объема изобретения.
[212] Изобретение было описано здесь широко и в общих чертах. Каждый из более узких видов и подродовых групп, подпадающих под общее описание, также составляет часть изобретения. Это включает общее описание изобретения с условием или отрицательным ограничением, исключающим любой объект из рода, независимо от того, указан ли здесь исключенный материал или нет.
[213] Все публикации, заявки на патенты, патенты и другие ссылки, упомянутые в настоящей заявке, прямо включены посредством ссылки во всей их полноте, включая все формулы и чертежи, в той же степени, как если бы каждая была включена посредством ссылки по отдельности. В случае противоречия преимущественную силу имеет настоящее описание, включая определения.
[214] Другие варианты осуществления изложены в нижеследующей формуле изобретения.
SEQ ID № 1: последовательность ДНК ICP34.5 в вирусных штаммах Mut 3 и Mut-3ΔICP6. Мутация c356a выделена жирным курсивом в последовательности ниже.

SEQ ID № 2 Аминокислотная последовательность ICP34.5 в Mut3 и Mut-3ΔICP6. Мутация P119H выделена жирным курсивом в приведенной ниже последовательности.

SEQ ID № 3: ДНК, кодирующая последовательность EGFP. Кодирующая область ICP34.5 была заменена кассетой экспрессии EGFP (фиг. 5C, SEQ ID NO: 11) в Mut3ΔIC34.5.

SEQ ID № 4: аминокислотная последовательность, кодирующая EGFP.

SEQ ID № 5: последовательность ДНК ICP34.5 в штамме 17TermA.

SEQ ID № 6: аминокислотная последовательность ICP34.5 в штамме 17TermA.
(* Стоп-кодон - считается, что последовательности после * не экспрессируются).
SEQ ID № 7: последовательность ДНК ICP34.5 в штамме rRp450.

SEQ ID № 8: аминокислотная последовательность ICP34.5 в штамме rRp450.

SEQ ID № 9: последовательность ДНК ICP34.5 в штамме 17 дикого типа.

SEQ ID № 51: последовательность ICP34.5 в штамме 17 дикого типа.

SEQ ID № 10: аминокислотная последовательность ICP34.5 в штамме 17 дикого типа.

SEQ ID № 52: аминокислотная последовательность ICP34.5 в штамме 17 дикого типа.

SEQ ID NO: 11: последовательность ДНК кассеты EGFP.

SEQ ID NO: 12: последовательность ДНК gE (гликопротеина E) в вирусном штамме Mut-3, Mut-3Δ34.5 или Mut-3ΔICP6. В приведенной ниже последовательности мутация g451a выделена жирным курсивом.

SEQ ID NO: 13: аминокислотная последовательность gE (гликопротеина E) в вирусном штамме Mut-3, Mut-3Δ34.5 или Mut-3ΔICP6. Мутация A151T выделена жирным курсивом в последовательности ниже.

SEQ ID NO: 14: последовательность ДНК gE в штамме 17TermA.
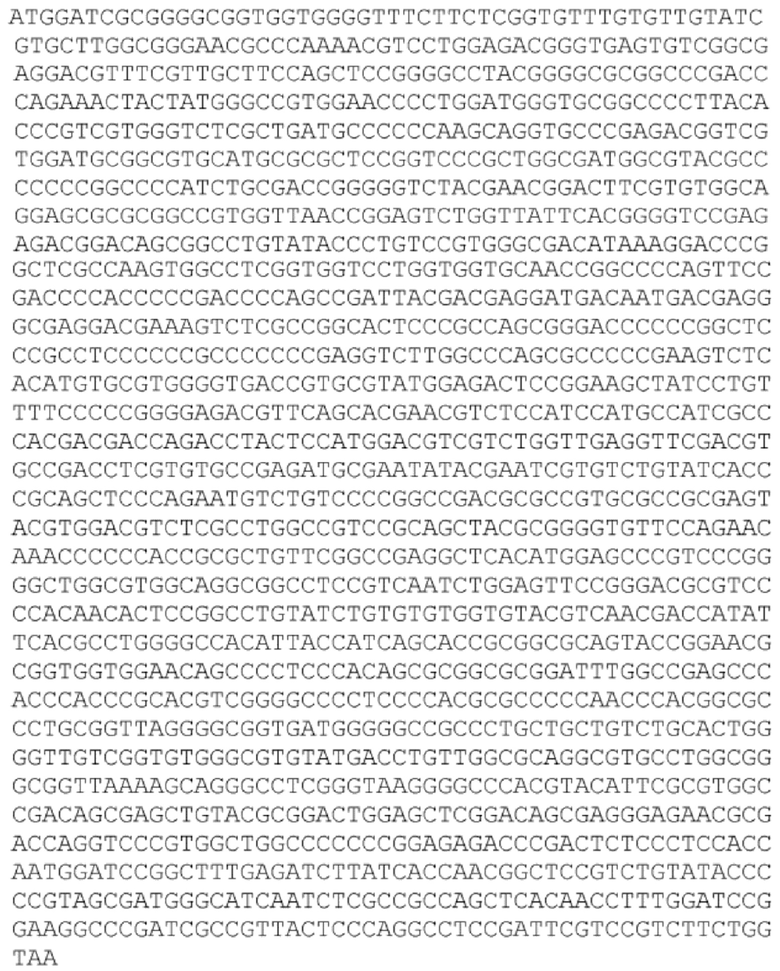
SEQ ID NO: 15: аминокислотная последовательность gE в штамме 17TermA.

SEQ ID № 16: последовательность ДНК gE в штамме rRp450.

SEQ ID № 17: аминокислотная последовательность gE в штамме rRp450.

SEQ ID № 18: последовательность ДНК gE в штамме 17 дикого типа.
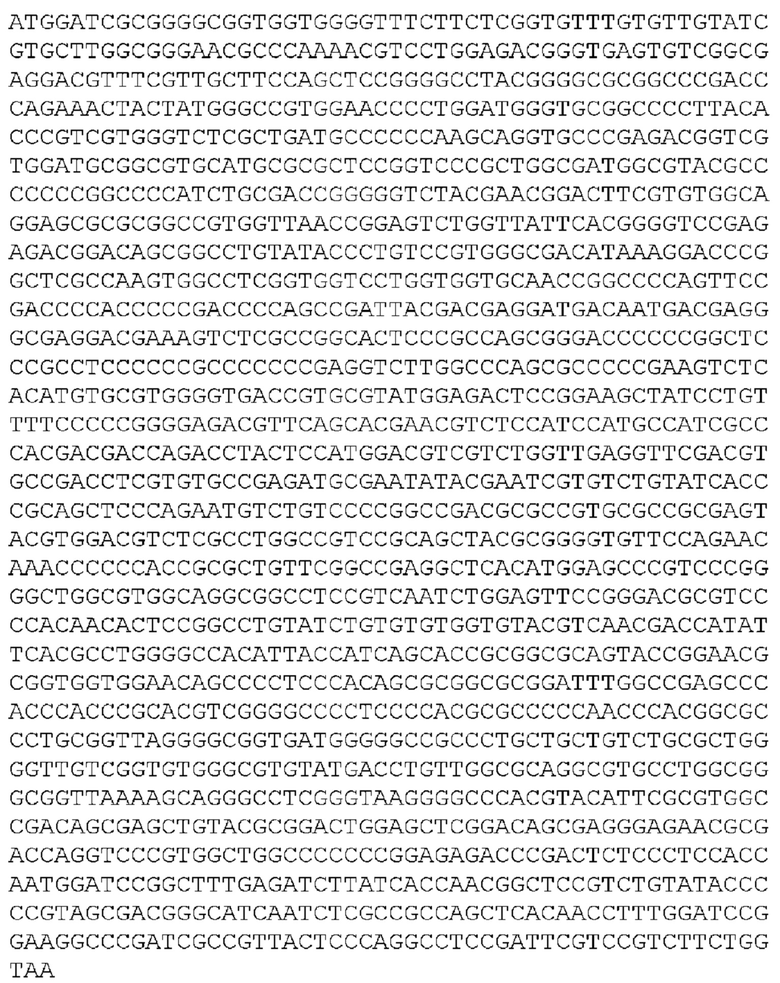
SEQ ID № 19: аминокислотная последовательность gE в штамме 17 дикого типа.

SEQ ID № 20: последовательность ДНК ICP0 в вирусных штаммах Mut 3, Mut-3Δ34.5 и Mut-3ΔICP6. Мутации a848c (внутри интрона) и g1712a (для R258H) выделены жирным курсивом в следующей последовательности. Включены два интрона (т.е. от нуклеотида (nt) 58 до nt 861 и от nt 1529 до nt 1663 из следующей последовательности, также смотрите последовательности в скобках).


SEQ ID № 21. Аминокислотная последовательность ICP0 в Mut 3, Mut-3Δ34.5 и Mut-3ΔICP6. Мутация R258H выделена курсивом и жирным шрифтом в последовательности ниже.

SEQ ID № 22: последовательность ДНК ICP0 в штамме 17TermA. Включены два интрона (т.е. от нуклеотида (nt) 58 до nt 822 и от nt 1490 до nt 1625 следующей последовательности, также смотрите последовательности в скобках)


SEQ ID № 23: аминокислотная последовательность ICP0 в штамме 17TermA.

SEQ ID № 24: последовательность ДНК ICP0 в штамме rRp450. Включены два интрона (то есть от нуклеотида (nt) 58 до nt 862, и от nt 1530 до nt 1668 следующей последовательности, также смотрите последовательности в скобках)


SEQ ID № 25: последовательность ДНК ICP0 в штамме дикого типа 17. Включены два интрона (т.е. от нуклеотида (nt) 58 до nt 861, и от nt 1529 до nt 1663 следующей последовательности, также смотрите последовательности в скобках)


SEQ ID № 53: последовательность ДНК ICP0 в штамме дикого типа 17. Включены два интрона (т.е. от нуклеотида (nt) 58 до nt 822 и от nt 1490 до nt 1625 следующей последовательности, также смотрите последовательности в скобках)
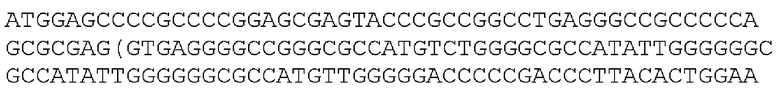


SEQ ID № 26: аминокислотная последовательность ICP0 в штамме 17 дикого типа.

SEQ ID № 27: последовательность ДНК ICP8 из штаммов вирусов Mut-3, Mut-3Δ34.5 и Mut-3ΔICP6. Мутация c3464t выделена жирным курсивом в приведенной ниже последовательности.


SEQ ID № 28: Аминокислотная последовательность ICP8 в вирусных штаммах Mut-3, Mut-3Δ34.5 и Mut-3ΔICP6. Мутация T1155M выделена жирным курсивом в приведенной ниже последовательности.


SEQ ID № 29: последовательность ДНК ICP8 в штамме 17TermA.


SEQ ID № 30: аминокислотная последовательность ICP8 в штамме 17TermA.


SEQ ID № 31: последовательность ДНК ICP8 в штамме rRp450.


SEQ ID № 32: аминокислотная последовательность ICP8 в штамме rRp450.

SEQ ID № 33: последовательность ДНК ICP8 в штамме 17 дикого типа.


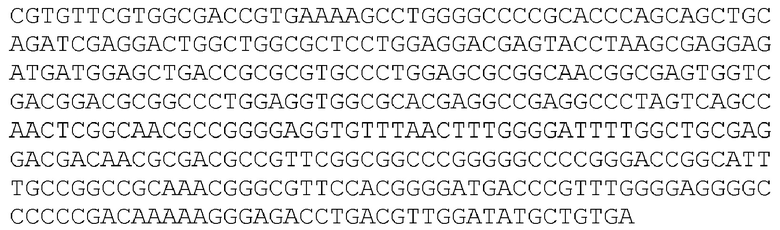
SEQ ID № 34: аминокислотная последовательность ICP8 в штамме 17 дикого типа.

SEQ ID № 35: ДНК последовательность субъединицы 1 терминазы упаковки ДНК из вирусных штаммов Mut-3, Mut-3Δ34.5 и Mut-3ΔICP6. В приведенной ниже последовательности мутация g1126a выделена жирным курсивом.


SEQ ID № 36: Аминокислотная последовательность субъединицы 1 терминазы упаковки ДНК из вирусных штаммов Mut-3, Mut-3Δ34.5 и Mut-3ΔICP6. Мутация A376T выделена жирным курсивом в приведенной ниже последовательности.

SEQ ID № 37: последовательность ДНК субъединицы 1 терминазы упаковки ДНК вирусного штамма 17TermA.

SEQ ID № 38: аминокислотная последовательность субъединицы 1 терминазы упаковки ДНК вирусного штамма 17TermA.

SEQ ID № 39: последовательность ДНК субъединицы 1 терминазы упаковки ДНК вирусного штамма rRp450.


SEQ ID № 40: аминокислотная последовательность субъединицы 1 терминазы упаковки ДНК вирусного штамма rRp450.

SEQ ID № 41: последовательность ДНК субъединицы 1 терминазы упаковки ДНК вирусного штамма 17 дикого типа.

SEQ ID № 42: аминокислотная последовательность субъединицы 1 терминазы упаковки ДНК вирусного штамма 17 дикого типа.

SEQ ID № 43: последовательность ДНК ICP6 в вирусных штаммах Mut 3 и Mut-3Δ34.5. Последовательность идентична последовательности штамма 17 дикого типа.


SEQ ID № 44: аминокислотная последовательность ICP6 в вирусных штаммах Mut 3 и Mut-3Δ34.5.

SEQ ID № 45: последовательность ДНК ICP6 в штамме 17TermA.


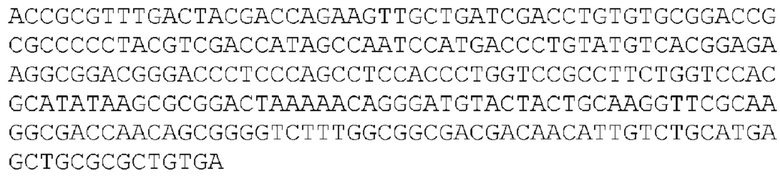
SEQ ID № 46: аминокислотная последовательность ICP6 в штамме 17TermA.

SEQ ID № 47: последовательность ДНК крысиного цитохрома P450 2B1 в штамме rRp450.



SEQ ID № 48: аминокислотная последовательность крысиного цитохрома P450 2B1 в штамме rRp450.

* Стоп-кодон - считается, что последовательности после * не экспрессируются.
SEQ ID № 49: последовательности ДНК ICP6 в штамме 17 дикого типа.


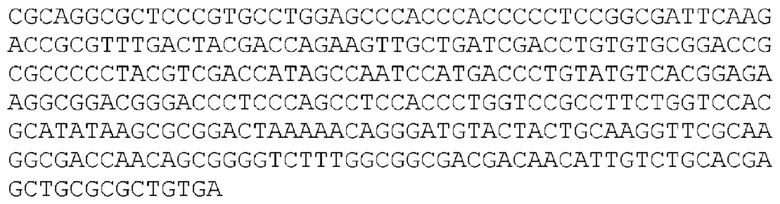
SEQ ID № 50: аминокислотная последовательность ICP6 в штамме 17 дикого типа.

--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> RESEARCH INSTITUTE AT NATIONWIDE CHILDREN'S HOSPITAL
<120> SYNCYTIAL ONCOLYTIC HERPES SIMPLEX MUTANTS AS POTENT CANCER
THERAPEUTICS
<130> 106887-7660
<150> 62/932,725
<151> 2019-11-08
<150> 62/818,577
<151> 2019-03-14
<160> 53
<170> PatentIn version 3.5
<210> 1
<211> 726
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<220>
<221> mutation
<222> (356)..(356)
<223> c356a
<400> 1
atggcccgcc gccgccgcca tcgcggcccc cgccgccccc ggccgcccgg gcccacgggc 60
gcggtcccaa ccgcacagtc ccaggtaacc tccacgccca actcggaacc cgtggtcagg 120
agcgcgcccg cggccgcccc gccgccgccc cccgccagtg ggcccccgcc ttcttgttcg 180
ctgctgctgc gccagtggct ccacgttccc gagtccgcgt ccgacgacga cgacgacgac 240
tggccggaca gccccccgcc cgagccggcg ccagaggccc ggcccaccgc cgccgccccc 300
cgcccccggt ccccaccgcc cggcgcgggc ccggggggcg gggctaaccc ctcccacccc 360
ccctcacgcc ccttccgcct tccgccgcgc ctcgccctcc gcctgcgcgt caccgcagag 420
cacctggcgc gcctgcgcct gcgacgcgcg ggcggggagg gggcgccgaa gccccccgcg 480
acccccgcga cccccgcgac ccccacgcgg gtgcgcttct cgccccacgt ccgggtgcgc 540
cacctggtgg tctgggcctc ggccgcccgc ctggcgcgcc gcggctcgtg ggcccgcgag 600
cgggccgacc gggctcggtt ccggcgccgg gtggcggagg ccgaggcggt catcgggccg 660
tgcctggggc ccgaggcccg tgcccgggcc ctggcccgcg gagccggccc ggcgaactcg 720
gtctaa 726
<210> 2
<211> 241
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<220>
<221> MISC_FEATURE
<222> (119)..(119)
<223> P119H mutation
<400> 2
Met Ala Arg Arg Arg Arg His Arg Gly Pro Arg Arg Pro Arg Pro Pro
1 5 10 15
Gly Pro Thr Gly Ala Val Pro Thr Ala Gln Ser Gln Val Thr Ser Thr
20 25 30
Pro Asn Ser Glu Pro Val Val Arg Ser Ala Pro Ala Ala Ala Pro Pro
35 40 45
Pro Pro Pro Ala Ser Gly Pro Pro Pro Ser Cys Ser Leu Leu Leu Arg
50 55 60
Gln Trp Leu His Val Pro Glu Ser Ala Ser Asp Asp Asp Asp Asp Asp
65 70 75 80
Trp Pro Asp Ser Pro Pro Pro Glu Pro Ala Pro Glu Ala Arg Pro Thr
85 90 95
Ala Ala Ala Pro Arg Pro Arg Ser Pro Pro Pro Gly Ala Gly Pro Gly
100 105 110
Gly Gly Ala Asn Pro Ser His Pro Pro Ser Arg Pro Phe Arg Leu Pro
115 120 125
Pro Arg Leu Ala Leu Arg Leu Arg Val Thr Ala Glu His Leu Ala Arg
130 135 140
Leu Arg Leu Arg Arg Ala Gly Gly Glu Gly Ala Pro Lys Pro Pro Ala
145 150 155 160
Thr Pro Ala Thr Pro Ala Thr Pro Thr Arg Val Arg Phe Ser Pro His
165 170 175
Val Arg Val Arg His Leu Val Val Trp Ala Ser Ala Ala Arg Leu Ala
180 185 190
Arg Arg Gly Ser Trp Ala Arg Glu Arg Ala Asp Arg Ala Arg Phe Arg
195 200 205
Arg Arg Val Ala Glu Ala Glu Ala Val Ile Gly Pro Cys Leu Gly Pro
210 215 220
Glu Ala Arg Ala Arg Ala Leu Ala Arg Gly Ala Gly Pro Ala Asn Ser
225 230 235 240
Val
<210> 3
<211> 846
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 3
atggtgagca agggcgagga gctgttcacc ggggtggtgc ccatcctggt cgagctggac 60
ggcgacgtaa acggccacaa gttcagcgtg tccggcgagg gcgagggcga tgccacctac 120
ggcaagctga ccctgaagtt catctgcacc accggcaagc tgcccgtgcc ctggcccacc 180
ctcgtgacca ccctgaccta cggcgtgcag tgcttcagcc gctaccccga ccacatgaag 240
cagcacgact tcttcaagtc cgccatgccc gaaggctacg tccaggagcg caccatcttc 300
ttcaaggacg acggcaacta caagacccgc gccgaggtga agttcgaggg cgacaccctg 360
gtgaaccgca tcgagctgaa gggcatcgac ttcaaggagg acggcaacat cctggggcac 420
aagctggagt acaactacaa cagccacaac gtctatatca tggccgacaa gcagaagaac 480
ggcatcaagg tgaacttcaa gatccgccac aacatcgagg acggcagcgt gcagctcgcc 540
gaccactacc agcagaacac ccccatcggc gacggccccg tgctgctgcc cgacaaccac 600
tacctgagca cccagtccgc cctgagcaaa gaccccaacg agaagcgcga tcacatggtc 660
ctgctggagt tcgtgaccgc cgccgggatc actctcggca tggacgagct gtacaagaag 720
cttagccatg gcttcccgcc ggaggtggag gagcaggatg atggcacgct gcccatgtct 780
tgtgcccagg agagcgggat ggaccgtcac cctgcagcct gtgcttctgc taggatcaat 840
gtgtag 846
<210> 4
<211> 281
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 4
Met Val Ser Lys Gly Glu Glu Leu Phe Thr Gly Val Val Pro Ile Leu
1 5 10 15
Val Glu Leu Asp Gly Asp Val Asn Gly His Lys Phe Ser Val Ser Gly
20 25 30
Glu Gly Glu Gly Asp Ala Thr Tyr Gly Lys Leu Thr Leu Lys Phe Ile
35 40 45
Cys Thr Thr Gly Lys Leu Pro Val Pro Trp Pro Thr Leu Val Thr Thr
50 55 60
Leu Thr Tyr Gly Val Gln Cys Phe Ser Arg Tyr Pro Asp His Met Lys
65 70 75 80
Gln His Asp Phe Phe Lys Ser Ala Met Pro Glu Gly Tyr Val Gln Glu
85 90 95
Arg Thr Ile Phe Phe Lys Asp Asp Gly Asn Tyr Lys Thr Arg Ala Glu
100 105 110
Val Lys Phe Glu Gly Asp Thr Leu Val Asn Arg Ile Glu Leu Lys Gly
115 120 125
Ile Asp Phe Lys Glu Asp Gly Asn Ile Leu Gly His Lys Leu Glu Tyr
130 135 140
Asn Tyr Asn Ser His Asn Val Tyr Ile Met Ala Asp Lys Gln Lys Asn
145 150 155 160
Gly Ile Lys Val Asn Phe Lys Ile Arg His Asn Ile Glu Asp Gly Ser
165 170 175
Val Gln Leu Ala Asp His Tyr Gln Gln Asn Thr Pro Ile Gly Asp Gly
180 185 190
Pro Val Leu Leu Pro Asp Asn His Tyr Leu Ser Thr Gln Ser Ala Leu
195 200 205
Ser Lys Asp Pro Asn Glu Lys Arg Asp His Met Val Leu Leu Glu Phe
210 215 220
Val Thr Ala Ala Gly Ile Thr Leu Gly Met Asp Glu Leu Tyr Lys Lys
225 230 235 240
Leu Ser His Gly Phe Pro Pro Glu Val Glu Glu Gln Asp Asp Gly Thr
245 250 255
Leu Pro Met Ser Cys Ala Gln Glu Ser Gly Met Asp Arg His Pro Ala
260 265 270
Ala Cys Ala Ser Ala Arg Ile Asn Val
275 280
<210> 5
<211> 767
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 5
atggcccgcc gccgccgcca tcgcggcccc cgccgccccc ggccgcccgg gcccacgggc 60
gccgtcccaa ccgcacagtc ccaggtaacc tagactagtc tagcgtaacc tccacgccca 120
actcggaacc cgcggtcagg agcgcgcccg cggccgcccc gccgccgccc cccgccggtg 180
ggcccccgcc ttcttgttcg ctgctgctgc gccagtggct ccacgttccc gagtccgcgt 240
ccgacaacga cgatgacgac gactggccgg acagcccccc gcccgagccg gcgccagagg 300
cccggcccac cgccgccgcc ccccggcccc ggcccccacc gcccggcgtg ggcccggggg 360
gcggggctga cccctcccac cccccctcgc gccccttccg ccttccgccg cgcctcgccc 420
tccgcctgcg cgtcaccgcg gagcacctgg cgcgcctgcg cctgcgacgc gcgggcgggg 480
agggggcgcc ggagcccccc gcgacccccg cgacccccgc gacccccgcg acccccgcga 540
cccccgcgcg ggtgcgcttc tcgccccacg tccgggtgcg ccacctggtg gtctgggcct 600
cggccgcccg cctggcgcgc cgcggctcgt gggcccgcga gcgggccgac cgggctcggt 660
tccggcgccg ggtggcggag gccgaggcgg tcatcgggcc gtgcctgggg cccgaggccc 720
gtgcccgggc cctggcccgc ggagccggcc cggcgaactc ggtctaa 767
<210> 6
<211> 30
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 6
Met Ala Arg Arg Arg Arg His Arg Gly Pro Arg Arg Pro Arg Pro Pro
1 5 10 15
Gly Pro Thr Gly Ala Val Pro Thr Ala Gln Ser Gln Val Thr
20 25 30
<210> 7
<211> 723
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 7
atggcccgcc gccgccatcg cggcccccgc cgcccccggc cgcccgggcc cacgggcgcg 60
gtcccaaccg cacagtccca ggtaacctcc acgcccaact cggaacccgt ggtcaggagc 120
gcgcccgcgg ccgccccgcc gccgcccccc gccagtgggc ccccgccttc ttgttcgctg 180
ctgctgcgcc agtggctcca cgttcccgag tccgcgtccg acgacgacga cgacgactgg 240
ccggacagcc ccccgcccga gccggcgcca gaggcccggc ccaccgccgc cgccccccgc 300
ccccggtccc caccgcccgg cgcgggcccg gggggcgggg ctaacccctc cccccccccc 360
tcacgcccct tccgccttcc gccgcgcctc gccctccgcc tgcgcgtcac cgcagagcac 420
ctggcgcgcc tgcgcctgcg acgcgcgggc ggggaggggg cgccgaagcc ccccgcgacc 480
cccgcgaccc ccgcgacccc cacgcgggtg cgcttctcgc cccacgtccg ggtgcgccac 540
ctggtggtct gggcctcggc cgcccgcctg gcgcgccgcg gctcgtgggc ccgcgagcgg 600
gccgaccggg ctcggttccg gcgccgggtg gcggaggccg aggcggtcat cgggccgtgc 660
ctggggcccg aggcccgtgc ccgggccctg gcccgcggag ccggcccggc gaactcggtc 720
taa 723
<210> 8
<211> 240
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 8
Met Ala Arg Arg Arg His Arg Gly Pro Arg Arg Pro Arg Pro Pro Gly
1 5 10 15
Pro Thr Gly Ala Val Pro Thr Ala Gln Ser Gln Val Thr Ser Thr Pro
20 25 30
Asn Ser Glu Pro Val Val Arg Ser Ala Pro Ala Ala Ala Pro Pro Pro
35 40 45
Pro Pro Ala Ser Gly Pro Pro Pro Ser Cys Ser Leu Leu Leu Arg Gln
50 55 60
Trp Leu His Val Pro Glu Ser Ala Ser Asp Asp Asp Asp Asp Asp Trp
65 70 75 80
Pro Asp Ser Pro Pro Pro Glu Pro Ala Pro Glu Ala Arg Pro Thr Ala
85 90 95
Ala Ala Pro Arg Pro Arg Ser Pro Pro Pro Gly Ala Gly Pro Gly Gly
100 105 110
Gly Ala Asn Pro Ser Pro Pro Pro Ser Arg Pro Phe Arg Leu Pro Pro
115 120 125
Arg Leu Ala Leu Arg Leu Arg Val Thr Ala Glu His Leu Ala Arg Leu
130 135 140
Arg Leu Arg Arg Ala Gly Gly Glu Gly Ala Pro Lys Pro Pro Ala Thr
145 150 155 160
Pro Ala Thr Pro Ala Thr Pro Thr Arg Val Arg Phe Ser Pro His Val
165 170 175
Arg Val Arg His Leu Val Val Trp Ala Ser Ala Ala Arg Leu Ala Arg
180 185 190
Arg Gly Ser Trp Ala Arg Glu Arg Ala Asp Arg Ala Arg Phe Arg Arg
195 200 205
Arg Val Ala Glu Ala Glu Ala Val Ile Gly Pro Cys Leu Gly Pro Glu
210 215 220
Ala Arg Ala Arg Ala Leu Ala Arg Gly Ala Gly Pro Ala Asn Ser Val
225 230 235 240
<210> 9
<211> 726
<212> DNA
<213> Herpes simplex virus type 1
<400> 9
atggcccgcc gccgccgcca tcgcggcccc cgccgccccc ggccgcccgg gcccacgggc 60
gcggtcccaa ccgcacagtc ccaggtaacc tccacgccca actcggaacc cgtggtcagg 120
agcgcgcccg cggccgcccc gccgccgccc cccgccagtg ggcccccgcc ttcttgttcg 180
ctgctgctgc gccagtggct ccacgttccc gagtccgcgt ccgacgacga cgacgacgac 240
tggccggaca gccccccgcc cgagccggcg ccagaggccc ggcccaccgc cgccgccccc 300
cgcccccggt ccccaccgcc cggcgcgggc ccggggggcg gggctaaccc ctcccacccc 360
ccctcacgcc ccttccgcct tccgccgcgc ctcgccctcc gcctgcgcgt caccgcagag 420
cacctggcgc gcctgcgcct gcgacgcgcg ggcggggagg gggcgccgaa gccccccgcg 480
acccccgcga cccccgcgac ccccacgcgg gtgcgcttct cgccccacgt ccgggtgcgc 540
cacctggtgg tctgggcctc ggccgcccgc ctggcgcgcc gcggctcgtg ggcccgcgag 600
cgggccgacc gggctcggtt ccggcgccgg gtggcggagg ccgaggcggt catcgggccg 660
tgcctggggc ccgaggcccg tgcccgggcc ctggcccgcg gagccggccc ggcgaactcg 720
gtctaa 726
<210> 10
<211> 241
<212> PRT
<213> Herpes simplex virus type 1
<400> 10
Met Ala Arg Arg Arg Arg His Arg Gly Pro Arg Arg Pro Arg Pro Pro
1 5 10 15
Gly Pro Thr Gly Ala Val Pro Thr Ala Gln Ser Gln Val Thr Ser Thr
20 25 30
Pro Asn Ser Glu Pro Val Val Arg Ser Ala Pro Ala Ala Ala Pro Pro
35 40 45
Pro Pro Pro Ala Ser Gly Pro Pro Pro Ser Cys Ser Leu Leu Leu Arg
50 55 60
Gln Trp Leu His Val Pro Glu Ser Ala Ser Asp Asp Asp Asp Asp Asp
65 70 75 80
Trp Pro Asp Ser Pro Pro Pro Glu Pro Ala Pro Glu Ala Arg Pro Thr
85 90 95
Ala Ala Ala Pro Arg Pro Arg Ser Pro Pro Pro Gly Ala Gly Pro Gly
100 105 110
Gly Gly Ala Asn Pro Ser His Pro Pro Ser Arg Pro Phe Arg Leu Pro
115 120 125
Pro Arg Leu Ala Leu Arg Leu Arg Val Thr Ala Glu His Leu Ala Arg
130 135 140
Leu Arg Leu Arg Arg Ala Gly Gly Glu Gly Ala Pro Lys Pro Pro Ala
145 150 155 160
Thr Pro Ala Thr Pro Ala Thr Pro Thr Arg Val Arg Phe Ser Pro His
165 170 175
Val Arg Val Arg His Leu Val Val Trp Ala Ser Ala Ala Arg Leu Ala
180 185 190
Arg Arg Gly Ser Trp Ala Arg Glu Arg Ala Asp Arg Ala Arg Phe Arg
195 200 205
Arg Arg Val Ala Glu Ala Glu Ala Val Ile Gly Pro Cys Leu Gly Pro
210 215 220
Glu Ala Arg Ala Arg Ala Leu Ala Arg Gly Ala Gly Pro Ala Asn Ser
225 230 235 240
Val
<210> 11
<211> 1971
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 11
aacacattaa ttaaaaaacc tcccacacct ccccctgaac ctgaaacata aaatgaatgc 60
aattgttgtt gttaacttgt ttattgcagc ttataatggt tacaaataaa gcaatagcat 120
cacaaatttc acaaataaag catttttttc actgcattct agttgtggtt tgtccaaact 180
catcaatgta tcttatcatg tctgctcgaa gcggccggcc gccccgactc tagactacac 240
attgatccta gcagaagcac aggctgcagg gtgacggtcc atcccgctct cctgggcaca 300
agacatgggc agcgtgccat catcctgctc ctccacctcc ggcgggaagc catggctaag 360
cttcttgtac agctcgtcca tgccgagagt gatcccggcg gcggtcacga actccagcag 420
gaccatgtga tcgcgcttct cgttggggtc tttgctcagg gcggactggg tgctcaggta 480
gtggttgtcg ggcagcagca cggggccgtc gccgatgggg gtgttctgct ggtagtggtc 540
ggcgagctgc acgctgccgt cctcgatgtt gtggcggatc ttgaagttca ccttgatgcc 600
gttcttctgc ttgtcggcca tgatatagac gttgtggctg ttgtagttgt actccagctt 660
gtgccccagg atgttgccgt cctccttgaa gtcgatgccc ttcagctcga tgcggttcac 720
cagggtgtcg ccctcgaact tcacctcggc gcgggtcttg tagttgccgt cgtccttgaa 780
gaagatggtg cgctcctgga cgtagccttc gggcatggcg gacttgaaga agtcgtgctg 840
cttcatgtgg tcggggtagc ggctgaagca ctgcacgccg taggtcaggg tggtcacgag 900
ggtgggccag ggcacgggca gcttgccggt ggtgcagatg aacttcaggg tcagcttgcc 960
gtaggtggca tcgccctcgc cctcgccgga cacgctgaac ttgtggccgt ttacgtcgcc 1020
gtccagctcg accaggatgg gcaccacccc ggtgaacagc tcctcgccct tgctcaccat 1080
ccgggaattg cggccgcggg tacaattccg cagcttttag agcagaagta acacttccgt 1140
acaggcctag aagtaaaggc aacatccact gaggagcagt tctttgattt gcaccaccac 1200
cggatccggg acctgaaata aaagacaaaa agactaaact taccagttaa ctttctggtt 1260
tttcagttcc tcgagtaccg gatcctctag agtccggagg ctggatcggt cccggtgtct 1320
tctatggagg tcaaaacagc gtggatggcg tctccaggcg atctgacggt tcactaaacg 1380
agctctgctt atatagacct cccaccgtac acgcctaccg cccatttgcg tcaatggggc 1440
ggagttgtta cgacattttg gaaagtcccg ttgattttgg tgccaaaaca aactcccatt 1500
gacgtcaatg gggtggagac ttggaaatcc ccgtgagtca aaccgctatc cacgcccatt 1560
gatgtactgc caaaaccgca tcaccatggt aatagcgatg actaatacgt agatgtactg 1620
ccaagtagga aagtcccata aggtcatgta ctgggcataa tgccaggcgg gccatttacc 1680
gtcattgacg tcaatagggg gcgtacttgg catatgatac acttgatgta ctgccaagtg 1740
ggcagtttac cgtaaatact ccacccattg acgtcaatgg aaagtcccta ttggcgttac 1800
tatgggaaca tacgtcatta ttgacgtcaa tgggcggggg tcgttgggcg gtcagccagg 1860
cgggccattt accgtaagtt atgtaacgac ctgcaggcat gcaagctcga attcgaacac 1920
gcagatgcag tcggggcggc agatcttaat taatggctgg ttgtttgttg t 1971
<210> 12
<211> 1653
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<220>
<221> mutation
<222> (451)..(451)
<223> g451a
<400> 12
atggatcgcg gggcggtggt ggggtttctt ctcggtgttt gtgttgtatc gtgcttggcg 60
ggaacgccca aaacgtcctg gagacgggtg agtgtcggcg aggacgtttc gttgcttcca 120
gctccggggc ctacggggcg cggcccgacc cagaaactac tatgggccgt ggaacccctg 180
gatgggtgcg gccccttaca cccgtcgtgg gtctcgctga tgccccccaa gcaggtgccc 240
gagacggtcg tggatgcggc gtgcatgcgc gctccggtcc cgctggcgat ggcgtacgcc 300
cccccggccc catctgcgac cgggggtcta cgaacggact tcgtgtggca ggagcgcgcg 360
gccgtggtta accggagtct ggttattcac ggggtccgag agacggacag cggcctgtat 420
accctgtccg tgggcgacat aaaggacccg actcgccaag tggcctcggt ggtcctggtg 480
gtgcaaccgg ccccagttcc gaccccaccc ccgaccccag ccgattacga cgaggatgac 540
aatgacgagg gcgaggacga aagtctcgcc ggcactcccg ccagcgggac cccccggctc 600
ccgcctcccc ccgccccccc gaggtcttgg cccagcgccc ccgaagtctc acatgtgcgt 660
ggggtgaccg tgcgtatgga gactccggaa gctatcctgt tttcccccgg ggagacgttc 720
agcacgaacg tctccatcca tgccatcgcc cacgacgacc agacctactc catggacgtc 780
gtctggttga ggttcgacgt gccgacctcg tgtgccgaga tgcgaatata cgaatcgtgt 840
ctgtatcacc cgcagctccc agaatgtctg tccccggccg acgcgccgtg cgccgcgagt 900
acgtggacgt ctcgcctggc cgtccgcagc tacgcggggt gttccagaac aaacccccca 960
ccgcgctgtt cggccgaggc tcacatggag cccgtcccgg ggctggcgtg gcaggcggcc 1020
tccgtcaatc tggagttccg ggacgcgtcc ccacaacact ccggcctgta tctgtgtgtg 1080
gtgtacgtca acgaccatat tcacgcctgg ggccacatta ccatcagcac cgcggcgcag 1140
taccggaacg cggtggtgga acagcccctc ccacagcgcg gcgcggattt ggccgagccc 1200
acccacccgc acgtcggggc ccctccccac gcgcccccaa cccacggcgc cctgcggtta 1260
ggggcggtga tgggggccgc cctgctgctg tctgcgctgg ggttgtcggt gtgggcgtgt 1320
atgacctgtt ggcgcaggcg tgcctggcgg gcggttaaaa gcagggcctc gggtaagggg 1380
cccacgtaca ttcgcgtggc cgacagcgag ctgtacgcgg actggagctc ggacagcgag 1440
ggagaacgcg accaggtccc gtggctggcc cccccggaga gacccgactc tccctccacc 1500
aatggatccg gctttgagat cttatcacca acggctccgt ctgtataccc ccgtagcgac 1560
gggcatcaat ctcgccgcca gctcacaacc tttggatccg gaaggcccga tcgccgttac 1620
tcccaggcct ccgattcgtc cgtcttctgg taa 1653
<210> 13
<211> 550
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<220>
<221> MISC_FEATURE
<222> (151)..(151)
<223> A151T
<400> 13
Met Asp Arg Gly Ala Val Val Gly Phe Leu Leu Gly Val Cys Val Val
1 5 10 15
Ser Cys Leu Ala Gly Thr Pro Lys Thr Ser Trp Arg Arg Val Ser Val
20 25 30
Gly Glu Asp Val Ser Leu Leu Pro Ala Pro Gly Pro Thr Gly Arg Gly
35 40 45
Pro Thr Gln Lys Leu Leu Trp Ala Val Glu Pro Leu Asp Gly Cys Gly
50 55 60
Pro Leu His Pro Ser Trp Val Ser Leu Met Pro Pro Lys Gln Val Pro
65 70 75 80
Glu Thr Val Val Asp Ala Ala Cys Met Arg Ala Pro Val Pro Leu Ala
85 90 95
Met Ala Tyr Ala Pro Pro Ala Pro Ser Ala Thr Gly Gly Leu Arg Thr
100 105 110
Asp Phe Val Trp Gln Glu Arg Ala Ala Val Val Asn Arg Ser Leu Val
115 120 125
Ile His Gly Val Arg Glu Thr Asp Ser Gly Leu Tyr Thr Leu Ser Val
130 135 140
Gly Asp Ile Lys Asp Pro Thr Arg Gln Val Ala Ser Val Val Leu Val
145 150 155 160
Val Gln Pro Ala Pro Val Pro Thr Pro Pro Pro Thr Pro Ala Asp Tyr
165 170 175
Asp Glu Asp Asp Asn Asp Glu Gly Glu Asp Glu Ser Leu Ala Gly Thr
180 185 190
Pro Ala Ser Gly Thr Pro Arg Leu Pro Pro Pro Pro Ala Pro Pro Arg
195 200 205
Ser Trp Pro Ser Ala Pro Glu Val Ser His Val Arg Gly Val Thr Val
210 215 220
Arg Met Glu Thr Pro Glu Ala Ile Leu Phe Ser Pro Gly Glu Thr Phe
225 230 235 240
Ser Thr Asn Val Ser Ile His Ala Ile Ala His Asp Asp Gln Thr Tyr
245 250 255
Ser Met Asp Val Val Trp Leu Arg Phe Asp Val Pro Thr Ser Cys Ala
260 265 270
Glu Met Arg Ile Tyr Glu Ser Cys Leu Tyr His Pro Gln Leu Pro Glu
275 280 285
Cys Leu Ser Pro Ala Asp Ala Pro Cys Ala Ala Ser Thr Trp Thr Ser
290 295 300
Arg Leu Ala Val Arg Ser Tyr Ala Gly Cys Ser Arg Thr Asn Pro Pro
305 310 315 320
Pro Arg Cys Ser Ala Glu Ala His Met Glu Pro Val Pro Gly Leu Ala
325 330 335
Trp Gln Ala Ala Ser Val Asn Leu Glu Phe Arg Asp Ala Ser Pro Gln
340 345 350
His Ser Gly Leu Tyr Leu Cys Val Val Tyr Val Asn Asp His Ile His
355 360 365
Ala Trp Gly His Ile Thr Ile Ser Thr Ala Ala Gln Tyr Arg Asn Ala
370 375 380
Val Val Glu Gln Pro Leu Pro Gln Arg Gly Ala Asp Leu Ala Glu Pro
385 390 395 400
Thr His Pro His Val Gly Ala Pro Pro His Ala Pro Pro Thr His Gly
405 410 415
Ala Leu Arg Leu Gly Ala Val Met Gly Ala Ala Leu Leu Leu Ser Ala
420 425 430
Leu Gly Leu Ser Val Trp Ala Cys Met Thr Cys Trp Arg Arg Arg Ala
435 440 445
Trp Arg Ala Val Lys Ser Arg Ala Ser Gly Lys Gly Pro Thr Tyr Ile
450 455 460
Arg Val Ala Asp Ser Glu Leu Tyr Ala Asp Trp Ser Ser Asp Ser Glu
465 470 475 480
Gly Glu Arg Asp Gln Val Pro Trp Leu Ala Pro Pro Glu Arg Pro Asp
485 490 495
Ser Pro Ser Thr Asn Gly Ser Gly Phe Glu Ile Leu Ser Pro Thr Ala
500 505 510
Pro Ser Val Tyr Pro Arg Ser Asp Gly His Gln Ser Arg Arg Gln Leu
515 520 525
Thr Thr Phe Gly Ser Gly Arg Pro Asp Arg Arg Tyr Ser Gln Ala Ser
530 535 540
Asp Ser Ser Val Phe Trp
545 550
<210> 14
<211> 1653
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 14
atggatcgcg gggcggtggt ggggtttctt ctcggtgttt gtgttgtatc gtgcttggcg 60
ggaacgccca aaacgtcctg gagacgggtg agtgtcggcg aggacgtttc gttgcttcca 120
gctccggggc ctacggggcg cggcccgacc cagaaactac tatgggccgt ggaacccctg 180
gatgggtgcg gccccttaca cccgtcgtgg gtctcgctga tgccccccaa gcaggtgccc 240
gagacggtcg tggatgcggc gtgcatgcgc gctccggtcc cgctggcgat ggcgtacgcc 300
cccccggccc catctgcgac cgggggtcta cgaacggact tcgtgtggca ggagcgcgcg 360
gccgtggtta accggagtct ggttattcac ggggtccgag agacggacag cggcctgtat 420
accctgtccg tgggcgacat aaaggacccg gctcgccaag tggcctcggt ggtcctggtg 480
gtgcaaccgg ccccagttcc gaccccaccc ccgaccccag ccgattacga cgaggatgac 540
aatgacgagg gcgaggacga aagtctcgcc ggcactcccg ccagcgggac cccccggctc 600
ccgcctcccc ccgccccccc gaggtcttgg cccagcgccc ccgaagtctc acatgtgcgt 660
ggggtgaccg tgcgtatgga gactccggaa gctatcctgt tttcccccgg ggagacgttc 720
agcacgaacg tctccatcca tgccatcgcc cacgacgacc agacctactc catggacgtc 780
gtctggttga ggttcgacgt gccgacctcg tgtgccgaga tgcgaatata cgaatcgtgt 840
ctgtatcacc cgcagctccc agaatgtctg tccccggccg acgcgccgtg cgccgcgagt 900
acgtggacgt ctcgcctggc cgtccgcagc tacgcggggt gttccagaac aaacccccca 960
ccgcgctgtt cggccgaggc tcacatggag cccgtcccgg ggctggcgtg gcaggcggcc 1020
tccgtcaatc tggagttccg ggacgcgtcc ccacaacact ccggcctgta tctgtgtgtg 1080
gtgtacgtca acgaccatat tcacgcctgg ggccacatta ccatcagcac cgcggcgcag 1140
taccggaacg cggtggtgga acagcccctc ccacagcgcg gcgcggattt ggccgagccc 1200
acccacccgc acgtcggggc ccctccccac gcgcccccaa cccacggcgc cctgcggtta 1260
ggggcggtga tgggggccgc cctgctgctg tctgcactgg ggttgtcggt gtgggcgtgt 1320
atgacctgtt ggcgcaggcg tgcctggcgg gcggttaaaa gcagggcctc gggtaagggg 1380
cccacgtaca ttcgcgtggc cgacagcgag ctgtacgcgg actggagctc ggacagcgag 1440
ggagaacgcg accaggtccc gtggctggcc cccccggaga gacccgactc tccctccacc 1500
aatggatccg gctttgagat cttatcacca acggctccgt ctgtataccc ccgtagcgat 1560
gggcatcaat ctcgccgcca gctcacaacc tttggatccg gaaggcccga tcgccgttac 1620
tcccaggcct ccgattcgtc cgtcttctgg taa 1653
<210> 15
<211> 550
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 15
Met Asp Arg Gly Ala Val Val Gly Phe Leu Leu Gly Val Cys Val Val
1 5 10 15
Ser Cys Leu Ala Gly Thr Pro Lys Thr Ser Trp Arg Arg Val Ser Val
20 25 30
Gly Glu Asp Val Ser Leu Leu Pro Ala Pro Gly Pro Thr Gly Arg Gly
35 40 45
Pro Thr Gln Lys Leu Leu Trp Ala Val Glu Pro Leu Asp Gly Cys Gly
50 55 60
Pro Leu His Pro Ser Trp Val Ser Leu Met Pro Pro Lys Gln Val Pro
65 70 75 80
Glu Thr Val Val Asp Ala Ala Cys Met Arg Ala Pro Val Pro Leu Ala
85 90 95
Met Ala Tyr Ala Pro Pro Ala Pro Ser Ala Thr Gly Gly Leu Arg Thr
100 105 110
Asp Phe Val Trp Gln Glu Arg Ala Ala Val Val Asn Arg Ser Leu Val
115 120 125
Ile His Gly Val Arg Glu Thr Asp Ser Gly Leu Tyr Thr Leu Ser Val
130 135 140
Gly Asp Ile Lys Asp Pro Ala Arg Gln Val Ala Ser Val Val Leu Val
145 150 155 160
Val Gln Pro Ala Pro Val Pro Thr Pro Pro Pro Thr Pro Ala Asp Tyr
165 170 175
Asp Glu Asp Asp Asn Asp Glu Gly Glu Asp Glu Ser Leu Ala Gly Thr
180 185 190
Pro Ala Ser Gly Thr Pro Arg Leu Pro Pro Pro Pro Ala Pro Pro Arg
195 200 205
Ser Trp Pro Ser Ala Pro Glu Val Ser His Val Arg Gly Val Thr Val
210 215 220
Arg Met Glu Thr Pro Glu Ala Ile Leu Phe Ser Pro Gly Glu Thr Phe
225 230 235 240
Ser Thr Asn Val Ser Ile His Ala Ile Ala His Asp Asp Gln Thr Tyr
245 250 255
Ser Met Asp Val Val Trp Leu Arg Phe Asp Val Pro Thr Ser Cys Ala
260 265 270
Glu Met Arg Ile Tyr Glu Ser Cys Leu Tyr His Pro Gln Leu Pro Glu
275 280 285
Cys Leu Ser Pro Ala Asp Ala Pro Cys Ala Ala Ser Thr Trp Thr Ser
290 295 300
Arg Leu Ala Val Arg Ser Tyr Ala Gly Cys Ser Arg Thr Asn Pro Pro
305 310 315 320
Pro Arg Cys Ser Ala Glu Ala His Met Glu Pro Val Pro Gly Leu Ala
325 330 335
Trp Gln Ala Ala Ser Val Asn Leu Glu Phe Arg Asp Ala Ser Pro Gln
340 345 350
His Ser Gly Leu Tyr Leu Cys Val Val Tyr Val Asn Asp His Ile His
355 360 365
Ala Trp Gly His Ile Thr Ile Ser Thr Ala Ala Gln Tyr Arg Asn Ala
370 375 380
Val Val Glu Gln Pro Leu Pro Gln Arg Gly Ala Asp Leu Ala Glu Pro
385 390 395 400
Thr His Pro His Val Gly Ala Pro Pro His Ala Pro Pro Thr His Gly
405 410 415
Ala Leu Arg Leu Gly Ala Val Met Gly Ala Ala Leu Leu Leu Ser Ala
420 425 430
Leu Gly Leu Ser Val Trp Ala Cys Met Thr Cys Trp Arg Arg Arg Ala
435 440 445
Trp Arg Ala Val Lys Ser Arg Ala Ser Gly Lys Gly Pro Thr Tyr Ile
450 455 460
Arg Val Ala Asp Ser Glu Leu Tyr Ala Asp Trp Ser Ser Asp Ser Glu
465 470 475 480
Gly Glu Arg Asp Gln Val Pro Trp Leu Ala Pro Pro Glu Arg Pro Asp
485 490 495
Ser Pro Ser Thr Asn Gly Ser Gly Phe Glu Ile Leu Ser Pro Thr Ala
500 505 510
Pro Ser Val Tyr Pro Arg Ser Asp Gly His Gln Ser Arg Arg Gln Leu
515 520 525
Thr Thr Phe Gly Ser Gly Arg Pro Asp Arg Arg Tyr Ser Gln Ala Ser
530 535 540
Asp Ser Ser Val Phe Trp
545 550
<210> 16
<211> 1653
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 16
atggatcgcg gggcggtggt ggggtttctt ctcggtgttt gtgttgtatc gtgcttggcg 60
ggaacgccca aaacgtcctg gagacgggtg agtgtcggcg aggacgtttc gttgcttcca 120
gctccggggc ctacggggcg cggcccgacc cagaaactac tatgggccgt ggaacccctg 180
gatgggtgcg gccccttaca cccgtcgtgg gtctcgctga tgccccccaa gcaggtgccc 240
gagacggtcg tggatgcggc gtgcatgcgc gctccggtcc cgctggcgat ggcgtacgcc 300
cccccggccc catctgcgac cgggggtcta cgaacggact tcgtgtggca ggagcgcgcg 360
gccgtggtta accggagtct ggttattcac ggggtccgag agacggacag cggcctgtat 420
accctgtccg tgggcgacat aaaggacccg gctcgccaag tggcctcggt ggtcctggtg 480
gtgcaaccgg ccccagttcc gaccccaccc ccgaccccag ccgattacga cgaggatgac 540
aatgacgagg gcgaggacga aagtctcgcc ggcactcccg ccagcgggac cccccggctc 600
ccgcctcccc ccgccccccc gaggtcttgg cccagcgccc ccgaagtctc acatgtgcgt 660
ggggtgaccg tgcgtatgga gactccggaa gctatcctgt tttcccccgg ggagacgttc 720
agcacgaacg tctccatcca tgccatcgcc cacgacgacc agacctactc catggacgtc 780
gtctggttga ggttcgacgt gccgacctcg tgtgccgaga tgcgaatata cgaatcgtgt 840
ctgtatcacc cgcagctccc agaatgtctg tccccggccg acgcgccgtg cgccgcgagt 900
acgtggacgt ctcgcctggc cgtccgcagc tacgcggggt gttccagaac aaacccccca 960
ccgcgctgtt cggccgaggc tcacatggag cccgtcccgg ggctggcgtg gcaggcggcc 1020
tccgtcaatc tggagttccg ggacgcgtcc ccacaacact ccggcctgta tctgtgtgtg 1080
gtgtacgtca acgaccatat tcacgcctgg ggccacatta ccatcagcac cgcggcgcag 1140
taccggaacg cggtggtgga acagcccctc ccacagcgcg gcgcggattt ggccgagccc 1200
acccacccgc acgtcggggc ccctccccac gcgcccccaa cccacggcgc cctgcggtta 1260
ggggcggtga tgggggccgc cctgctgctg tctgcgctgg ggttgtcggt gtgggcgtgt 1320
atgacctgtt ggcgcaggcg tgcctggcgg gcggttaaaa gcagggcctc gggtaagggg 1380
cccacgtaca ttcgcgtggc cgacagcgag ctgtacgcgg actggagctc ggacagcgag 1440
ggagaacgcg accaggtccc gtggctggcc cccccggaga gacccgactc tccctccacc 1500
aatggatccg gctttgagat cttatcacca acggctccgt ctgtataccc ccgtagcgac 1560
gggcatcaat ctcgccgcca gctcacaacc tttggatccg gaaggcccga tcgccgttac 1620
tcccaggcct ccgattcgtc cgtcttctgg taa 1653
<210> 17
<211> 550
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 17
Met Asp Arg Gly Ala Val Val Gly Phe Leu Leu Gly Val Cys Val Val
1 5 10 15
Ser Cys Leu Ala Gly Thr Pro Lys Thr Ser Trp Arg Arg Val Ser Val
20 25 30
Gly Glu Asp Val Ser Leu Leu Pro Ala Pro Gly Pro Thr Gly Arg Gly
35 40 45
Pro Thr Gln Lys Leu Leu Trp Ala Val Glu Pro Leu Asp Gly Cys Gly
50 55 60
Pro Leu His Pro Ser Trp Val Ser Leu Met Pro Pro Lys Gln Val Pro
65 70 75 80
Glu Thr Val Val Asp Ala Ala Cys Met Arg Ala Pro Val Pro Leu Ala
85 90 95
Met Ala Tyr Ala Pro Pro Ala Pro Ser Ala Thr Gly Gly Leu Arg Thr
100 105 110
Asp Phe Val Trp Gln Glu Arg Ala Ala Val Val Asn Arg Ser Leu Val
115 120 125
Ile His Gly Val Arg Glu Thr Asp Ser Gly Leu Tyr Thr Leu Ser Val
130 135 140
Gly Asp Ile Lys Asp Pro Ala Arg Gln Val Ala Ser Val Val Leu Val
145 150 155 160
Val Gln Pro Ala Pro Val Pro Thr Pro Pro Pro Thr Pro Ala Asp Tyr
165 170 175
Asp Glu Asp Asp Asn Asp Glu Gly Glu Asp Glu Ser Leu Ala Gly Thr
180 185 190
Pro Ala Ser Gly Thr Pro Arg Leu Pro Pro Pro Pro Ala Pro Pro Arg
195 200 205
Ser Trp Pro Ser Ala Pro Glu Val Ser His Val Arg Gly Val Thr Val
210 215 220
Arg Met Glu Thr Pro Glu Ala Ile Leu Phe Ser Pro Gly Glu Thr Phe
225 230 235 240
Ser Thr Asn Val Ser Ile His Ala Ile Ala His Asp Asp Gln Thr Tyr
245 250 255
Ser Met Asp Val Val Trp Leu Arg Phe Asp Val Pro Thr Ser Cys Ala
260 265 270
Glu Met Arg Ile Tyr Glu Ser Cys Leu Tyr His Pro Gln Leu Pro Glu
275 280 285
Cys Leu Ser Pro Ala Asp Ala Pro Cys Ala Ala Ser Thr Trp Thr Ser
290 295 300
Arg Leu Ala Val Arg Ser Tyr Ala Gly Cys Ser Arg Thr Asn Pro Pro
305 310 315 320
Pro Arg Cys Ser Ala Glu Ala His Met Glu Pro Val Pro Gly Leu Ala
325 330 335
Trp Gln Ala Ala Ser Val Asn Leu Glu Phe Arg Asp Ala Ser Pro Gln
340 345 350
His Ser Gly Leu Tyr Leu Cys Val Val Tyr Val Asn Asp His Ile His
355 360 365
Ala Trp Gly His Ile Thr Ile Ser Thr Ala Ala Gln Tyr Arg Asn Ala
370 375 380
Val Val Glu Gln Pro Leu Pro Gln Arg Gly Ala Asp Leu Ala Glu Pro
385 390 395 400
Thr His Pro His Val Gly Ala Pro Pro His Ala Pro Pro Thr His Gly
405 410 415
Ala Leu Arg Leu Gly Ala Val Met Gly Ala Ala Leu Leu Leu Ser Ala
420 425 430
Leu Gly Leu Ser Val Trp Ala Cys Met Thr Cys Trp Arg Arg Arg Ala
435 440 445
Trp Arg Ala Val Lys Ser Arg Ala Ser Gly Lys Gly Pro Thr Tyr Ile
450 455 460
Arg Val Ala Asp Ser Glu Leu Tyr Ala Asp Trp Ser Ser Asp Ser Glu
465 470 475 480
Gly Glu Arg Asp Gln Val Pro Trp Leu Ala Pro Pro Glu Arg Pro Asp
485 490 495
Ser Pro Ser Thr Asn Gly Ser Gly Phe Glu Ile Leu Ser Pro Thr Ala
500 505 510
Pro Ser Val Tyr Pro Arg Ser Asp Gly His Gln Ser Arg Arg Gln Leu
515 520 525
Thr Thr Phe Gly Ser Gly Arg Pro Asp Arg Arg Tyr Ser Gln Ala Ser
530 535 540
Asp Ser Ser Val Phe Trp
545 550
<210> 18
<211> 1653
<212> DNA
<213> Herpes simplex virus type 1
<400> 18
atggatcgcg gggcggtggt ggggtttctt ctcggtgttt gtgttgtatc gtgcttggcg 60
ggaacgccca aaacgtcctg gagacgggtg agtgtcggcg aggacgtttc gttgcttcca 120
gctccggggc ctacggggcg cggcccgacc cagaaactac tatgggccgt ggaacccctg 180
gatgggtgcg gccccttaca cccgtcgtgg gtctcgctga tgccccccaa gcaggtgccc 240
gagacggtcg tggatgcggc gtgcatgcgc gctccggtcc cgctggcgat ggcgtacgcc 300
cccccggccc catctgcgac cgggggtcta cgaacggact tcgtgtggca ggagcgcgcg 360
gccgtggtta accggagtct ggttattcac ggggtccgag agacggacag cggcctgtat 420
accctgtccg tgggcgacat aaaggacccg gctcgccaag tggcctcggt ggtcctggtg 480
gtgcaaccgg ccccagttcc gaccccaccc ccgaccccag ccgattacga cgaggatgac 540
aatgacgagg gcgaggacga aagtctcgcc ggcactcccg ccagcgggac cccccggctc 600
ccgcctcccc ccgccccccc gaggtcttgg cccagcgccc ccgaagtctc acatgtgcgt 660
ggggtgaccg tgcgtatgga gactccggaa gctatcctgt tttcccccgg ggagacgttc 720
agcacgaacg tctccatcca tgccatcgcc cacgacgacc agacctactc catggacgtc 780
gtctggttga ggttcgacgt gccgacctcg tgtgccgaga tgcgaatata cgaatcgtgt 840
ctgtatcacc cgcagctccc agaatgtctg tccccggccg acgcgccgtg cgccgcgagt 900
acgtggacgt ctcgcctggc cgtccgcagc tacgcggggt gttccagaac aaacccccca 960
ccgcgctgtt cggccgaggc tcacatggag cccgtcccgg ggctggcgtg gcaggcggcc 1020
tccgtcaatc tggagttccg ggacgcgtcc ccacaacact ccggcctgta tctgtgtgtg 1080
gtgtacgtca acgaccatat tcacgcctgg ggccacatta ccatcagcac cgcggcgcag 1140
taccggaacg cggtggtgga acagcccctc ccacagcgcg gcgcggattt ggccgagccc 1200
acccacccgc acgtcggggc ccctccccac gcgcccccaa cccacggcgc cctgcggtta 1260
ggggcggtga tgggggccgc cctgctgctg tctgcgctgg ggttgtcggt gtgggcgtgt 1320
atgacctgtt ggcgcaggcg tgcctggcgg gcggttaaaa gcagggcctc gggtaagggg 1380
cccacgtaca ttcgcgtggc cgacagcgag ctgtacgcgg actggagctc ggacagcgag 1440
ggagaacgcg accaggtccc gtggctggcc cccccggaga gacccgactc tccctccacc 1500
aatggatccg gctttgagat cttatcacca acggctccgt ctgtataccc ccgtagcgac 1560
gggcatcaat ctcgccgcca gctcacaacc tttggatccg gaaggcccga tcgccgttac 1620
tcccaggcct ccgattcgtc cgtcttctgg taa 1653
<210> 19
<211> 550
<212> PRT
<213> Herpes simplex virus type 1
<400> 19
Met Asp Arg Gly Ala Val Val Gly Phe Leu Leu Gly Val Cys Val Val
1 5 10 15
Ser Cys Leu Ala Gly Thr Pro Lys Thr Ser Trp Arg Arg Val Ser Val
20 25 30
Gly Glu Asp Val Ser Leu Leu Pro Ala Pro Gly Pro Thr Gly Arg Gly
35 40 45
Pro Thr Gln Lys Leu Leu Trp Ala Val Glu Pro Leu Asp Gly Cys Gly
50 55 60
Pro Leu His Pro Ser Trp Val Ser Leu Met Pro Pro Lys Gln Val Pro
65 70 75 80
Glu Thr Val Val Asp Ala Ala Cys Met Arg Ala Pro Val Pro Leu Ala
85 90 95
Met Ala Tyr Ala Pro Pro Ala Pro Ser Ala Thr Gly Gly Leu Arg Thr
100 105 110
Asp Phe Val Trp Gln Glu Arg Ala Ala Val Val Asn Arg Ser Leu Val
115 120 125
Ile His Gly Val Arg Glu Thr Asp Ser Gly Leu Tyr Thr Leu Ser Val
130 135 140
Gly Asp Ile Lys Asp Pro Ala Arg Gln Val Ala Ser Val Val Leu Val
145 150 155 160
Val Gln Pro Ala Pro Val Pro Thr Pro Pro Pro Thr Pro Ala Asp Tyr
165 170 175
Asp Glu Asp Asp Asn Asp Glu Gly Glu Asp Glu Ser Leu Ala Gly Thr
180 185 190
Pro Ala Ser Gly Thr Pro Arg Leu Pro Pro Pro Pro Ala Pro Pro Arg
195 200 205
Ser Trp Pro Ser Ala Pro Glu Val Ser His Val Arg Gly Val Thr Val
210 215 220
Arg Met Glu Thr Pro Glu Ala Ile Leu Phe Ser Pro Gly Glu Thr Phe
225 230 235 240
Ser Thr Asn Val Ser Ile His Ala Ile Ala His Asp Asp Gln Thr Tyr
245 250 255
Ser Met Asp Val Val Trp Leu Arg Phe Asp Val Pro Thr Ser Cys Ala
260 265 270
Glu Met Arg Ile Tyr Glu Ser Cys Leu Tyr His Pro Gln Leu Pro Glu
275 280 285
Cys Leu Ser Pro Ala Asp Ala Pro Cys Ala Ala Ser Thr Trp Thr Ser
290 295 300
Arg Leu Ala Val Arg Ser Tyr Ala Gly Cys Ser Arg Thr Asn Pro Pro
305 310 315 320
Pro Arg Cys Ser Ala Glu Ala His Met Glu Pro Val Pro Gly Leu Ala
325 330 335
Trp Gln Ala Ala Ser Val Asn Leu Glu Phe Arg Asp Ala Ser Pro Gln
340 345 350
His Ser Gly Leu Tyr Leu Cys Val Val Tyr Val Asn Asp His Ile His
355 360 365
Ala Trp Gly His Ile Thr Ile Ser Thr Ala Ala Gln Tyr Arg Asn Ala
370 375 380
Val Val Glu Gln Pro Leu Pro Gln Arg Gly Ala Asp Leu Ala Glu Pro
385 390 395 400
Thr His Pro His Val Gly Ala Pro Pro His Ala Pro Pro Thr His Gly
405 410 415
Ala Leu Arg Leu Gly Ala Val Met Gly Ala Ala Leu Leu Leu Ser Ala
420 425 430
Leu Gly Leu Ser Val Trp Ala Cys Met Thr Cys Trp Arg Arg Arg Ala
435 440 445
Trp Arg Ala Val Lys Ser Arg Ala Ser Gly Lys Gly Pro Thr Tyr Ile
450 455 460
Arg Val Ala Asp Ser Glu Leu Tyr Ala Asp Trp Ser Ser Asp Ser Glu
465 470 475 480
Gly Glu Arg Asp Gln Val Pro Trp Leu Ala Pro Pro Glu Arg Pro Asp
485 490 495
Ser Pro Ser Thr Asn Gly Ser Gly Phe Glu Ile Leu Ser Pro Thr Ala
500 505 510
Pro Ser Val Tyr Pro Arg Ser Asp Gly His Gln Ser Arg Arg Gln Leu
515 520 525
Thr Thr Phe Gly Ser Gly Arg Pro Asp Arg Arg Tyr Ser Gln Ala Ser
530 535 540
Asp Ser Ser Val Phe Trp
545 550
<210> 20
<211> 3267
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<220>
<221> Intron
<222> (58)..(861)
<223> intron
<220>
<221> mutation
<222> (848)..(848)
<223> a848c
<220>
<221> Intron
<222> (1529)..(1663)
<223> intron
<220>
<221> mutation
<222> (1712)..(1712)
<223> g1712a
<400> 20
atggagcccc gccccggagc gagtacccgc cggcctgagg gccgccccca gcgcgaggtg 60
aggggccggg cgccatgtct ggggcgccat gtctggggcg ccatgtctgg ggcgccatgt 120
ctggggcgcc atgttggggg gcgccatgtt ggggggcgcc atgttggggg acccccgacc 180
cttacactgg aaccggccgc catgttgggg gacccccact catacacggg agccgggcgc 240
catgttgggg cgccatgtta gggggcgtgg aaccccgtga cactatatat acagggaccg 300
ggggcgccat gttagggggc gcggaacccc ctgaccctat atatacaggg accggggtcg 360
ccctgttagg ggtcgccatg tgaccccctg actttatata tacagacccc caacacctac 420
acatggcccc tttgactcag acgcagggcc cggggtcgcc gtgggacccc cctgactcat 480
acacagagac acgcccccac aacaaacaca cagggaccgg ggtcgccgtg ttagggggcg 540
tggtccccac tgactcatac gcagggcccc cttactcaca cgcatctagg ggggtgggga 600
ggagccgccc gccatatttg ggggacgccg tgggaccccc gactccggtg cgtctggagg 660
gcgggagaag agggaagaag aggggtcggg atccaaagga cggacccaga ccacctttgg 720
ttgcagaccc ctttctcccc cctcttccga ggccagcagg ggggcaggac tttgtgaggc 780
ggggggggag ggggaactcg tgggcgctga ttgacgcggg aaatcccccc attcttaccc 840
gccccccctt tttcccctca gcccgccccg gatgtctggg tgtttccctg cgaccgagac 900
ctgccggaca gcagcgactc ggaggcggag accgaagtgg gggggcgggg ggacgccgac 960
caccatgacg acgactccgc ctccgaggcg gacagcacgg acacggaact gttcgagacg 1020
gggctgctgg ggccgcaggg cgtggatggg ggggcggtct cgggggggag ccccccccgc 1080
gaggaagacc ccggcagttg cgggggcgcc ccccctcgag aggacggggg gagcgacgag 1140
ggcgacgtgt gcgccgtgtg cacggatgag atcgcgcccc acctgcgctg cgacaccttc 1200
ccgtgcatgc accgcttctg catcccgtgc atgaaaacct ggatgcaatt gcgcaacacc 1260
tgcccgctgt gcaacgccaa gctggtgtac ctgatagtgg gcgtgacgcc cagcgggtcg 1320
ttcagcacca tcccgatcgt gaacgacccc cagacccgca tggaggccga ggaggccgtc 1380
agggcgggca cggccgtgga ctttatctgg acgggcaatc agcggttcgc cccgcggtac 1440
ctgaccctgg gggggcacac ggtgagggcc ctgtcgccca cccacccgga acccaccacg 1500
gacgaggatg acgacgacct ggacgacggt gaggcggggg gcggcaagga ccctggggga 1560
ggaggaggag gagggggggg agggaggaat aggcgggcgg gcgaggaaag ggcgggccgg 1620
ggagggggcg taacctgatc gcgccccccg ttgtctcttg cagcagacta cgtaccgccc 1680
gccccccgcc ggacgccccg cgccccccca cacagaggcg ccgccgcgcc ccccgtgacg 1740
ggcggggcgt ctcacgcagc cccccagccg gccgcggctc ggacagcgcc cccctcggcg 1800
cccatcgggc cacacggcag cagtaacacc aacaccacca ccaacagcag cggcggcggc 1860
ggctcccgcc agtcgcgagc cgcggcgccg cggggggcgt ctggcccctc cgggggggtt 1920
ggggttgggg ttggggttgt tgaagcggag gcggggcggc cgaggggccg gacgggcccc 1980
cttgtcaaca gacccgcccc ccttgcaaac aacagagacc ccatagtgat cagcgactcc 2040
cccccggcct ctccccacag gccccccgcg gcgcccatgc caggctccgc cccccgcccc 2100
gggccccccg cgtccgcggc cgcgtcggga cccgcgcgcc cccgcgcggc cgtggccccg 2160
tgcgtgcgag cgccgcctcc ggggcccggc ccccgcgccc cggcccccgg ggcggagccg 2220
gccgcccgcc ccgcggacgc gcgccgtgtg ccccagtcgc actcgtccct ggctcaggcc 2280
gcgaaccaag aacagagtct gtgccgggcg cgtgcgacgg tggcgcgcgg ctcggggggg 2340
ccgggcgtgg agggtgggca cgggccctcc cgcggcgccg ccccctccgg cgccgccccg 2400
ctcccctccg ccgcctctgt cgagcaggag gcggcggtgc gtccgaggaa gaggcgcggg 2460
tcgggccagg aaaacccctc cccccagtcc acgcgtcccc ccctcgcgcc ggcaggggcc 2520
aagagggcgg cgacgcaccc cccctccgac tcagggccgg gggggcgcgg ccagggtggg 2580
cccgggaccc ccctgacgtc ctcggcggcc tccgcctctt cctcctctgc ctcttcctcc 2640
tcggccccga cccccgcggg ggccgcctct tccgccgccg gggccgcgtc ctcctccgct 2700
tccgcctcct cgggcggggc cgtcggtgcc ctgggaggga gacaagagga aacctccctc 2760
ggcccccgcg ctgcttctgg gccgcggggg ccgaggaagt gtgcccggaa gacgcgccac 2820
gcggagactt ccggggccgt ccccgcgggc ggcctcacgc gctacctgcc catctcgggg 2880
gtctctagcg tggtcgccct gtcgccttac gtgaacaaga cgatcacggg ggactgcctg 2940
cccatcctgg acatggagac ggggaacatc ggggcgtacg tggtcctggt ggaccagacg 3000
ggaaacatgg cgacccggct gcgggccgcg gtccccggct ggagccgccg caccctgctc 3060
cccgagaccg cgggtaacca cgtgatgccc cccgagtacc cgacggcccc cgcgtcggag 3120
tggaacagcc tctggatgac ccccgtgggg aacatgctgt tcgaccaggg caccctagtg 3180
ggcgccctgg acttccgcag cctgcggtct cggcacccgt ggtccgggga gcagggggcg 3240
tcgacccggg acgagggaaa acaataa 3267
<210> 21
<211> 775
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<220>
<221> MISC_FEATURE
<222> (258)..(258)
<223> R258H
<400> 21
Met Glu Pro Arg Pro Gly Ala Ser Thr Arg Arg Pro Glu Gly Arg Pro
1 5 10 15
Gln Arg Glu Pro Ala Pro Asp Val Trp Val Phe Pro Cys Asp Arg Asp
20 25 30
Leu Pro Asp Ser Ser Asp Ser Glu Ala Glu Thr Glu Val Gly Gly Arg
35 40 45
Gly Asp Ala Asp His His Asp Asp Asp Ser Ala Ser Glu Ala Asp Ser
50 55 60
Thr Asp Thr Glu Leu Phe Glu Thr Gly Leu Leu Gly Pro Gln Gly Val
65 70 75 80
Asp Gly Gly Ala Val Ser Gly Gly Ser Pro Pro Arg Glu Glu Asp Pro
85 90 95
Gly Ser Cys Gly Gly Ala Pro Pro Arg Glu Asp Gly Gly Ser Asp Glu
100 105 110
Gly Asp Val Cys Ala Val Cys Thr Asp Glu Ile Ala Pro His Leu Arg
115 120 125
Cys Asp Thr Phe Pro Cys Met His Arg Phe Cys Ile Pro Cys Met Lys
130 135 140
Thr Trp Met Gln Leu Arg Asn Thr Cys Pro Leu Cys Asn Ala Lys Leu
145 150 155 160
Val Tyr Leu Ile Val Gly Val Thr Pro Ser Gly Ser Phe Ser Thr Ile
165 170 175
Pro Ile Val Asn Asp Pro Gln Thr Arg Met Glu Ala Glu Glu Ala Val
180 185 190
Arg Ala Gly Thr Ala Val Asp Phe Ile Trp Thr Gly Asn Gln Arg Phe
195 200 205
Ala Pro Arg Tyr Leu Thr Leu Gly Gly His Thr Val Arg Ala Leu Ser
210 215 220
Pro Thr His Pro Glu Pro Thr Thr Asp Glu Asp Asp Asp Asp Leu Asp
225 230 235 240
Asp Ala Asp Tyr Val Pro Pro Ala Pro Arg Arg Thr Pro Arg Ala Pro
245 250 255
Pro His Arg Gly Ala Ala Ala Pro Pro Val Thr Gly Gly Ala Ser His
260 265 270
Ala Ala Pro Gln Pro Ala Ala Ala Arg Thr Ala Pro Pro Ser Ala Pro
275 280 285
Ile Gly Pro His Gly Ser Ser Asn Thr Asn Thr Thr Thr Asn Ser Ser
290 295 300
Gly Gly Gly Gly Ser Arg Gln Ser Arg Ala Ala Ala Pro Arg Gly Ala
305 310 315 320
Ser Gly Pro Ser Gly Gly Val Gly Val Gly Val Gly Val Val Glu Ala
325 330 335
Glu Ala Gly Arg Pro Arg Gly Arg Thr Gly Pro Leu Val Asn Arg Pro
340 345 350
Ala Pro Leu Ala Asn Asn Arg Asp Pro Ile Val Ile Ser Asp Ser Pro
355 360 365
Pro Ala Ser Pro His Arg Pro Pro Ala Ala Pro Met Pro Gly Ser Ala
370 375 380
Pro Arg Pro Gly Pro Pro Ala Ser Ala Ala Ala Ser Gly Pro Ala Arg
385 390 395 400
Pro Arg Ala Ala Val Ala Pro Cys Val Arg Ala Pro Pro Pro Gly Pro
405 410 415
Gly Pro Arg Ala Pro Ala Pro Gly Ala Glu Pro Ala Ala Arg Pro Ala
420 425 430
Asp Ala Arg Arg Val Pro Gln Ser His Ser Ser Leu Ala Gln Ala Ala
435 440 445
Asn Gln Glu Gln Ser Leu Cys Arg Ala Arg Ala Thr Val Ala Arg Gly
450 455 460
Ser Gly Gly Pro Gly Val Glu Gly Gly His Gly Pro Ser Arg Gly Ala
465 470 475 480
Ala Pro Ser Gly Ala Ala Pro Leu Pro Ser Ala Ala Ser Val Glu Gln
485 490 495
Glu Ala Ala Val Arg Pro Arg Lys Arg Arg Gly Ser Gly Gln Glu Asn
500 505 510
Pro Ser Pro Gln Ser Thr Arg Pro Pro Leu Ala Pro Ala Gly Ala Lys
515 520 525
Arg Ala Ala Thr His Pro Pro Ser Asp Ser Gly Pro Gly Gly Arg Gly
530 535 540
Gln Gly Gly Pro Gly Thr Pro Leu Thr Ser Ser Ala Ala Ser Ala Ser
545 550 555 560
Ser Ser Ser Ala Ser Ser Ser Ser Ala Pro Thr Pro Ala Gly Ala Ala
565 570 575
Ser Ser Ala Ala Gly Ala Ala Ser Ser Ser Ala Ser Ala Ser Ser Gly
580 585 590
Gly Ala Val Gly Ala Leu Gly Gly Arg Gln Glu Glu Thr Ser Leu Gly
595 600 605
Pro Arg Ala Ala Ser Gly Pro Arg Gly Pro Arg Lys Cys Ala Arg Lys
610 615 620
Thr Arg His Ala Glu Thr Ser Gly Ala Val Pro Ala Gly Gly Leu Thr
625 630 635 640
Arg Tyr Leu Pro Ile Ser Gly Val Ser Ser Val Val Ala Leu Ser Pro
645 650 655
Tyr Val Asn Lys Thr Ile Thr Gly Asp Cys Leu Pro Ile Leu Asp Met
660 665 670
Glu Thr Gly Asn Ile Gly Ala Tyr Val Val Leu Val Asp Gln Thr Gly
675 680 685
Asn Met Ala Thr Arg Leu Arg Ala Ala Val Pro Gly Trp Ser Arg Arg
690 695 700
Thr Leu Leu Pro Glu Thr Ala Gly Asn His Val Met Pro Pro Glu Tyr
705 710 715 720
Pro Thr Ala Pro Ala Ser Glu Trp Asn Ser Leu Trp Met Thr Pro Val
725 730 735
Gly Asn Met Leu Phe Asp Gln Gly Thr Leu Val Gly Ala Leu Asp Phe
740 745 750
Arg Ser Leu Arg Ser Arg His Pro Trp Ser Gly Glu Gln Gly Ala Ser
755 760 765
Thr Arg Asp Glu Gly Lys Gln
770 775
<210> 22
<211> 3229
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<220>
<221> Intron
<222> (58)..(822)
<223> intron
<220>
<221> Intron
<222> (1490)..(1625)
<223> intron
<400> 22
atggagcccc gccccggagc gagtacccgc cggcctgagg gccgccccca gcgcgaggtg 60
aggggccggg cgccatgtct ggggcgccat attggggggc gccatattgg ggggcgccat 120
gttgggggac ccccgaccct tacactggaa ccggccgcca tgttggggga cccccactca 180
tacacgggag ccgggcgcca tgttggggcg ccatgttagg gggcgtggaa ccccgtgaca 240
ctatatatac agggaccggg ggcgccatgt tagggggtgc ggaaccccct gaccctatat 300
atacagggac cggggtcgcc ctgttggggg tcgccatgtg accccctgac tttatatata 360
cagaccccca acacatacac atggcccctt tgactcagac gcagggcccg gggtcgccgt 420
gggaccccct gactcataca cagagacacg cccccacaac aaacacacag ggaccggggt 480
cgccgtgttg ggggcgtggt ccccactgac tcatacgcag gcccccctta ctcacacgca 540
tctagggggg tggggaggag ccgcccgcca tatttggggg acgccgtggg acccccgact 600
ccggtgcgtc tggagggcgg gagaagaggg aagaagaggg gtcgggatcc aaaggacgga 660
cccagaccac ctttggttgc agaccccttt ctcccccctc ttccgaggcc agcagggggg 720
caggactttg tgaggcgggg gggggagagg gggaactcgt gggtgctgat tgacgcggga 780
aatccccccc cattcttacc cgcccccctt ttttcccctt agcccgcccc ggatgtctgg 840
gtgtttccct gcgaccgaga cctgccggac agcagcgact ctgaggcgga gaccgaagtg 900
ggggggcggg gggacgccga ccaccatgac gacgactccg cctccgaggc ggacagcacg 960
gacacggaac tgttcgagac ggggctgctg gggccgcagg gcgtggatgg gggggcggtc 1020
tcggggggga gccccccccg cgaggaagac cccggcagtt gcgggggcgc cccccctcga 1080
gaggacgggg ggagcgacga gggtgacgtg tgcgccgtgt gcacggatga gatcgcgccc 1140
cacctgcgct gcgacacctt cccgtgcatg caccgcttct gcatcccgtg catgaaaacc 1200
tggatgcaat tgcgcaacac ctgcccgctg tgcaacgcca agctggtgta cctgatagtg 1260
ggcgtgacgc ccagcgggtc gttcagcacc atcccgatcg tgaacgaccc ccagacccgc 1320
atggaggccg aggaggccgt cagggcgggc acggccgtgg actttatctg gacgggcaat 1380
cagcggttcg ccccgcggta cctgaccctg ggggggcaca cggtgagggc cctgtcgccc 1440
acccacccgg aacccaccac ggacgaggat gacgacgacc tggacgacgg tgaggcgggg 1500
ggcggcaagg accctggggg aggaggagga ggaggggggg ggagggagga ataggcgggc 1560
gggcgaggaa agggcgggcc ggggaggggg cgtaacctga tcgcgccccc cgttgtctct 1620
tgcagcagac tacgtaccgc ccgccccccg ccggacgccc cgcgcccccc cacgcagagg 1680
cgccgccgcg ccccccgtga cgggcggggc gtctcacgca gccccccagc cggccgcggc 1740
tcggacagcg cccccctcgg cgcccatcgg gccacacggc agcagtaaca ccaacaccac 1800
caccaacagc agcggcggcg gcggctcccg ccagtcgcga gccgcggcgc cgcggggggc 1860
gtctggcccc tccggggggg ttggggttgg ggttggggtt gttgaagcgg aggcggggcg 1920
gccgaggggc cggacgggcc cccttgtcaa cagacccgcc ccccttgcaa acaacagaga 1980
ccccatagtg atcagcgact cccccccggc ctctccccac aggccccccg cggcgcccat 2040
gccaggctcc gccccccgcc ccgggccccc cgcgtccgcg gccgcgtcgg gacccgcgcg 2100
cccccgcgcg gccgtggccc cgtgcgtgcg agcgccgcct ccggggcccg gcccccgcgc 2160
cccggccccc ggggcggagc cggccgcccg ccccgcggac gcgcgccgtg tgccccagtc 2220
gcactcgtcc ctggctcagg ccgcgaacca agaacagagt ctgtgccggg cgcgtgcgac 2280
ggtggcgcgc ggctcggggg ggccgggcgt ggagggtggg cacgggccct cccgcggcgc 2340
cgccccctcc ggcgccgccc cgctcccctc cgccgcctct gtcgagcagg aggcggcggt 2400
gcgtccgagg aagaggcgcg ggtcgggcca ggaaaacccc tccccccagt ccacgcgtcc 2460
ccccctcgcg ccggcagggg ccaagagggc ggcgacgcac cccccctccg actcagggcc 2520
gggggggcgc ggccagggtg ggcccgggac ccccctgacg tcctcggcgg cctccgcctc 2580
ttcctcctct gcctcttcct cctcggcccc gacccccgcg ggggccgcct cttccgccgc 2640
cggggccgcg tcctcctccg cttccgcctc ctcgggcggg gccgtcggtg ccctgggagg 2700
gagacaagag gaaacctccc tcggcccccg cgctgcttct gggccgcggg ggccgaggaa 2760
gtgtgcccgg aagacgcgcc acgcggagac ttccggggcc gtccccgcgg gcggcctcac 2820
gcgctacctg cccatctcgg gggtctctag cgtggtcgcc ctgtcgcctt acgtgaacaa 2880
gactatcacg ggggactgcc tgcccatcct ggacatggag acggggaaca tcggggcgta 2940
cgtggtcctg gtggaccaga cgggaaacat ggcgacccgg ctgcgggccg cggtccccgg 3000
ctggagccgc cgcaccctgc tccccgagac cgcgggtaac cacgtgatgc cccccgagta 3060
cccgacggcc cccgcgtcgg agtggaacag cctctggatg acccccgtgg ggaacatgct 3120
gttcgaccag ggcaccctag tgggcgccct ggacttccgc agcctgcggt ctcggcaccc 3180
gtggtccggg gagcaggggg cgtcgacccg ggacgaggga aaacaataa 3229
<210> 23
<211> 775
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 23
Met Glu Pro Arg Pro Gly Ala Ser Thr Arg Arg Pro Glu Gly Arg Pro
1 5 10 15
Gln Arg Glu Pro Ala Pro Asp Val Trp Val Phe Pro Cys Asp Arg Asp
20 25 30
Leu Pro Asp Ser Ser Asp Ser Glu Ala Glu Thr Glu Val Gly Gly Arg
35 40 45
Gly Asp Ala Asp His His Asp Asp Asp Ser Ala Ser Glu Ala Asp Ser
50 55 60
Thr Asp Thr Glu Leu Phe Glu Thr Gly Leu Leu Gly Pro Gln Gly Val
65 70 75 80
Asp Gly Gly Ala Val Ser Gly Gly Ser Pro Pro Arg Glu Glu Asp Pro
85 90 95
Gly Ser Cys Gly Gly Ala Pro Pro Arg Glu Asp Gly Gly Ser Asp Glu
100 105 110
Gly Asp Val Cys Ala Val Cys Thr Asp Glu Ile Ala Pro His Leu Arg
115 120 125
Cys Asp Thr Phe Pro Cys Met His Arg Phe Cys Ile Pro Cys Met Lys
130 135 140
Thr Trp Met Gln Leu Arg Asn Thr Cys Pro Leu Cys Asn Ala Lys Leu
145 150 155 160
Val Tyr Leu Ile Val Gly Val Thr Pro Ser Gly Ser Phe Ser Thr Ile
165 170 175
Pro Ile Val Asn Asp Pro Gln Thr Arg Met Glu Ala Glu Glu Ala Val
180 185 190
Arg Ala Gly Thr Ala Val Asp Phe Ile Trp Thr Gly Asn Gln Arg Phe
195 200 205
Ala Pro Arg Tyr Leu Thr Leu Gly Gly His Thr Val Arg Ala Leu Ser
210 215 220
Pro Thr His Pro Glu Pro Thr Thr Asp Glu Asp Asp Asp Asp Leu Asp
225 230 235 240
Asp Ala Asp Tyr Val Pro Pro Ala Pro Arg Arg Thr Pro Arg Ala Pro
245 250 255
Pro Arg Arg Gly Ala Ala Ala Pro Pro Val Thr Gly Gly Ala Ser His
260 265 270
Ala Ala Pro Gln Pro Ala Ala Ala Arg Thr Ala Pro Pro Ser Ala Pro
275 280 285
Ile Gly Pro His Gly Ser Ser Asn Thr Asn Thr Thr Thr Asn Ser Ser
290 295 300
Gly Gly Gly Gly Ser Arg Gln Ser Arg Ala Ala Ala Pro Arg Gly Ala
305 310 315 320
Ser Gly Pro Ser Gly Gly Val Gly Val Gly Val Gly Val Val Glu Ala
325 330 335
Glu Ala Gly Arg Pro Arg Gly Arg Thr Gly Pro Leu Val Asn Arg Pro
340 345 350
Ala Pro Leu Ala Asn Asn Arg Asp Pro Ile Val Ile Ser Asp Ser Pro
355 360 365
Pro Ala Ser Pro His Arg Pro Pro Ala Ala Pro Met Pro Gly Ser Ala
370 375 380
Pro Arg Pro Gly Pro Pro Ala Ser Ala Ala Ala Ser Gly Pro Ala Arg
385 390 395 400
Pro Arg Ala Ala Val Ala Pro Cys Val Arg Ala Pro Pro Pro Gly Pro
405 410 415
Gly Pro Arg Ala Pro Ala Pro Gly Ala Glu Pro Ala Ala Arg Pro Ala
420 425 430
Asp Ala Arg Arg Val Pro Gln Ser His Ser Ser Leu Ala Gln Ala Ala
435 440 445
Asn Gln Glu Gln Ser Leu Cys Arg Ala Arg Ala Thr Val Ala Arg Gly
450 455 460
Ser Gly Gly Pro Gly Val Glu Gly Gly His Gly Pro Ser Arg Gly Ala
465 470 475 480
Ala Pro Ser Gly Ala Ala Pro Leu Pro Ser Ala Ala Ser Val Glu Gln
485 490 495
Glu Ala Ala Val Arg Pro Arg Lys Arg Arg Gly Ser Gly Gln Glu Asn
500 505 510
Pro Ser Pro Gln Ser Thr Arg Pro Pro Leu Ala Pro Ala Gly Ala Lys
515 520 525
Arg Ala Ala Thr His Pro Pro Ser Asp Ser Gly Pro Gly Gly Arg Gly
530 535 540
Gln Gly Gly Pro Gly Thr Pro Leu Thr Ser Ser Ala Ala Ser Ala Ser
545 550 555 560
Ser Ser Ser Ala Ser Ser Ser Ser Ala Pro Thr Pro Ala Gly Ala Ala
565 570 575
Ser Ser Ala Ala Gly Ala Ala Ser Ser Ser Ala Ser Ala Ser Ser Gly
580 585 590
Gly Ala Val Gly Ala Leu Gly Gly Arg Gln Glu Glu Thr Ser Leu Gly
595 600 605
Pro Arg Ala Ala Ser Gly Pro Arg Gly Pro Arg Lys Cys Ala Arg Lys
610 615 620
Thr Arg His Ala Glu Thr Ser Gly Ala Val Pro Ala Gly Gly Leu Thr
625 630 635 640
Arg Tyr Leu Pro Ile Ser Gly Val Ser Ser Val Val Ala Leu Ser Pro
645 650 655
Tyr Val Asn Lys Thr Ile Thr Gly Asp Cys Leu Pro Ile Leu Asp Met
660 665 670
Glu Thr Gly Asn Ile Gly Ala Tyr Val Val Leu Val Asp Gln Thr Gly
675 680 685
Asn Met Ala Thr Arg Leu Arg Ala Ala Val Pro Gly Trp Ser Arg Arg
690 695 700
Thr Leu Leu Pro Glu Thr Ala Gly Asn His Val Met Pro Pro Glu Tyr
705 710 715 720
Pro Thr Ala Pro Ala Ser Glu Trp Asn Ser Leu Trp Met Thr Pro Val
725 730 735
Gly Asn Met Leu Phe Asp Gln Gly Thr Leu Val Gly Ala Leu Asp Phe
740 745 750
Arg Ser Leu Arg Ser Arg His Pro Trp Ser Gly Glu Gln Gly Ala Ser
755 760 765
Thr Arg Asp Glu Gly Lys Gln
770 775
<210> 24
<211> 3275
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<220>
<221> Intron
<222> (58)..(862)
<223> intron
<220>
<221> Intron
<222> (1530)..(1668)
<223> intron
<400> 24
atggagcccc gccccggagc gagtacccgc cggcctgagg gccgccccca gcgcgaggtg 60
aggggccggg cgccatgtct ggggcgccat gtctggggcg ccatgtctgg ggcgccatgt 120
ctggggcgcc atgttggggg gcgccatgtt ggggggcgcc atgttggggg acccccgacc 180
cttacactgg aaccggccgc catgttgggg gacccccact catacacggg agccgggcgc 240
catgttgggg cgccatgtta gggggcgtgg aaccccgtga cactatatat acagggaccg 300
ggggcgccat gttagggggc gcggaacccc ctgaccctat atatacaggg accggggtcg 360
ccctgttagg ggtcgccatg tgaccccctg actttatata tacagacccc caacacctac 420
acatggcccc tttgactcag acgcagggcc cggggtcgcc gtgggacccc cctgactcat 480
acacagagac acgcccccac aacaaacaca cagggaccgg ggtcgccgtg ttagggggcg 540
tggtccccac tgactcatac gcagggcccc cttactcaca cgcatctagg ggggtgggga 600
ggagccgccc gccatatttg ggggacgccg tgggaccccc gactccggtg cgtctggagg 660
gcgggagaag agggaagaag aggggtcggg atccaaagga cggacccaga ccacctttgg 720
ttgcagaccc ctttctcccc cctcttccga ggccagcagg ggggcaggac tttgtgaggc 780
ggggggggag ggggaactcg tgggcgctga ttgacgcggg aaatcccccc attcttaccc 840
gccccccctt ttttcccctc agcccgcccc ggatgtctgg gtgtttccct gcgaccgaga 900
cctgccggac agcagcgact cggaggcgga gaccgaagtg ggggggcggg gggacgccga 960
ccaccatgac gacgactccg cctccgaggc ggacagcacg gacacggaac tgttcgagac 1020
ggggctgctg gggccgcagg gcgtggatgg gggggcggtc tcggggggga gccccccccg 1080
cgaggaagac cccggcagtt gcgggggcgc cccccctcga gaggacgggg ggagcgacga 1140
gggcgacgtg tgcgccgtgt gcacggatga gatcgcgccc cacctgcgct gcgacacctt 1200
cccgtgcatg caccgcttct gcatcccgtg catgaaaacc tggatgcaat tgcgcaacac 1260
ctgcccgctg tgcaacgcca agctggtgta cctgatagtg ggcgtgacgc ccagcgggtc 1320
gttcagcacc atcccgatcg tgaacgaccc ccagacccgc atggaggccg aggaggccgt 1380
cagggcgggc acggccgtgg actttatctg gacgggcaat cagcggttcg ccccgcggta 1440
cctgaccctg ggggggcaca cggtgagggc cctgtcgccc acccaccctg agcccaccac 1500
ggacgaggat gacgacgacc tggacgacgg tgaggcgggg gggcggcgag gaccctgggg 1560
gaggaggagg aggggggggg gggggaggaa taggcgggcg ggcgggcgag gaaagggcgg 1620
gccggggagg gggcgtaacc tgatcgcgcc ccccgttgtc tcttgcagca gactacgtac 1680
cgcccgcccc ccgccggacg ccccgcgccc ccccacgcag aggcgccgcc gcgccccccg 1740
tgacgggcgg ggcgtctcac gcagcccccc agccggccgc ggctcggaca gcgcccccct 1800
cggcgcccat cgggccacac ggcagcagta acactaacac caccaccaac agcagcggcg 1860
gcggcggctc ccgccagtcg cgagccgcgg tgccgcgggg ggcgtctggc ccctccgggg 1920
gggttggggt tgttgaagcg gaggcggggc ggccgagggg ccggacgggc ccccttgtca 1980
acagacccgc cccccttgca aacaacagag accccatagt gatcagcgac tcccccccgg 2040
cctctcccca caggcccccc gcggcgccca tgccaggctc cgccccccgc cccggtcccc 2100
ccgcgtccgc ggccgcgtcg ggccccgcgc gcccccgcgc ggccgtggcc ccgtgtgtgc 2160
gggcgccgcc tccggggccc ggcccccgcg ccccggcccc cggggcggag ccggccgccc 2220
gccccgcgga cgcgcgccgt gtgccccagt cgcactcgtc cctggctcag gccgcgaacc 2280
aagaacagag tctgtgccgg gcgcgtgcga cggtggcgcg cggctcgggg gggccgggcg 2340
tggagggtgg acacgggccc tcccgcggcg ccgccccctc cggcgccgcc ccctccggcg 2400
cccccccgct cccctccgcc gcctctgtcg agcaggaggc ggcggtgcgt ccgaggaaga 2460
ggcgcgggtc gggccaggaa aacccctccc cccagtccac gcgtcccccc ctcgcgccgg 2520
caggggccaa gagggcggcg acgcaccccc cctccgactc agggccgggg gggcgcggcc 2580
agggagggcc cgggaccccc ctgacgtcct cggcggcctc cgcctcttcc tcctccgcct 2640
cttcctcctc ggccccgact cccgcggggg ccacctcttc cgccaccggg gccgcgtcct 2700
cctccgcttc cgcctcctcg ggcggggccg tcggtgccct gggagggaga caagaggaaa 2760
cctccctcgg cccccgcgct gcttctgggc cgcgggggcc gaggaagtgt gcccggaaga 2820
cgcgccacgc ggagacttcc ggggccgtcc ccgcgggcgg cctcacgcgc tacctgccca 2880
tctcgggggt ctctagcgtg gtcgccctgt cgccttacgt gaacaagacg atcacggggg 2940
actgcctgcc catcctggac atggagacgg ggaacatcgg ggcgtacgtg gtcctggtgg 3000
accagacggg aaacatggcg acccggctgc gggccgcggt ccccggctgg agccgccgca 3060
ccctgctccc cgagaccgcg ggtaaccacg tgacgccccc cgagtacccg acggcccccg 3120
cgtcggagtg gaacagcctc tggatgaccc ccgtggggaa catgctgttc gaccagggca 3180
ccctagtggg cgccctggac ttccgcagcc tgcggtctcg gcacccgtgg tccggggagc 3240
agggggcgtc gacccgggac gagggaaaac aataa 3275
<210> 25
<211> 3267
<212> DNA
<213> Herpes simplex virus type 1
<220>
<221> Intron
<222> (58)..(861)
<223> intron
<220>
<221> Intron
<222> (1529)..(1663)
<223> intron
<400> 25
atggagcccc gccccggagc gagtacccgc cggcctgagg gccgccccca gcgcgaggtg 60
aggggccggg cgccatgtct ggggcgccat gtctggggcg ccatgtctgg ggcgccatgt 120
ctggggcgcc atgttggggg gcgccatgtt ggggggcgcc atgttggggg acccccgacc 180
cttacactgg aaccggccgc catgttgggg gacccccact catacacggg agccgggcgc 240
catgttgggg cgccatgtta gggggcgtgg aaccccgtga cactatatat acagggaccg 300
ggggcgccat gttagggggc gcggaacccc ctgaccctat atatacaggg accggggtcg 360
ccctgttagg ggtcgccatg tgaccccctg actttatata tacagacccc caacacctac 420
acatggcccc tttgactcag acgcagggcc cggggtcgcc gtgggacccc cctgactcat 480
acacagagac acgcccccac aacaaacaca cagggaccgg ggtcgccgtg ttagggggcg 540
tggtccccac tgactcatac gcagggcccc cttactcaca cgcatctagg ggggtgggga 600
ggagccgccc gccatatttg ggggacgccg tgggaccccc gactccggtg cgtctggagg 660
gcgggagaag agggaagaag aggggtcggg atccaaagga cggacccaga ccacctttgg 720
ttgcagaccc ctttctcccc cctcttccga ggccagcagg ggggcaggac tttgtgaggc 780
ggggggggag ggggaactcg tgggcgctga ttgacgcggg aaatcccccc attcttaccc 840
gccccccttt tttcccctca gcccgccccg gatgtctggg tgtttccctg cgaccgagac 900
ctgccggaca gcagcgactc ggaggcggag accgaagtgg gggggcgggg ggacgccgac 960
caccatgacg acgactccgc ctccgaggcg gacagcacgg acacggaact gttcgagacg 1020
gggctgctgg ggccgcaggg cgtggatggg ggggcggtct cgggggggag ccccccccgc 1080
gaggaagacc ccggcagttg cgggggcgcc ccccctcgag aggacggggg gagcgacgag 1140
ggcgacgtgt gcgccgtgtg cacggatgag atcgcgcccc acctgcgctg cgacaccttc 1200
ccgtgcatgc accgcttctg catcccgtgc atgaaaacct ggatgcaatt gcgcaacacc 1260
tgcccgctgt gcaacgccaa gctggtgtac ctgatagtgg gcgtgacgcc cagcgggtcg 1320
ttcagcacca tcccgatcgt gaacgacccc cagacccgca tggaggccga ggaggccgtc 1380
agggcgggca cggccgtgga ctttatctgg acgggcaatc agcggttcgc cccgcggtac 1440
ctgaccctgg gggggcacac ggtgagggcc ctgtcgccca cccacccgga acccaccacg 1500
gacgaggatg acgacgacct ggacgacggt gaggcggggg gcggcaagga ccctggggga 1560
ggaggaggag gagggggggg agggaggaat aggcgggcgg gcgaggaaag ggcgggccgg 1620
ggagggggcg taacctgatc gcgccccccg ttgtctcttg cagcagacta cgtaccgccc 1680
gccccccgcc ggacgccccg cgccccccca cgcagaggcg ccgccgcgcc ccccgtgacg 1740
ggcggggcgt ctcacgcagc cccccagccg gccgcggctc ggacagcgcc cccctcggcg 1800
cccatcgggc cacacggcag cagtaacacc aacaccacca ccaacagcag cggcggcggc 1860
ggctcccgcc agtcgcgagc cgcggcgccg cggggggcgt ctggcccctc cgggggggtt 1920
ggggttgggg ttggggttgt tgaagcggag gcggggcggc cgaggggccg gacgggcccc 1980
cttgtcaaca gacccgcccc ccttgcaaac aacagagacc ccatagtgat cagcgactcc 2040
cccccggcct ctccccacag gccccccgcg gcgcccatgc caggctccgc cccccgcccc 2100
gggccccccg cgtccgcggc cgcgtcggga cccgcgcgcc cccgcgcggc cgtggccccg 2160
tgcgtgcgag cgccgcctcc ggggcccggc ccccgcgccc cggcccccgg ggcggagccg 2220
gccgcccgcc ccgcggacgc gcgccgtgtg ccccagtcgc actcgtccct ggctcaggcc 2280
gcgaaccaag aacagagtct gtgccgggcg cgtgcgacgg tggcgcgcgg ctcggggggg 2340
ccgggcgtgg agggtgggca cgggccctcc cgcggcgccg ccccctccgg cgccgccccg 2400
ctcccctccg ccgcctctgt cgagcaggag gcggcggtgc gtccgaggaa gaggcgcggg 2460
tcgggccagg aaaacccctc cccccagtcc acgcgtcccc ccctcgcgcc ggcaggggcc 2520
aagagggcgg cgacgcaccc cccctccgac tcagggccgg gggggcgcgg ccagggtggg 2580
cccgggaccc ccctgacgtc ctcggcggcc tccgcctctt cctcctctgc ctcttcctcc 2640
tcggccccga cccccgcggg ggccgcctct tccgccgccg gggccgcgtc ctcctccgct 2700
tccgcctcct cgggcggggc cgtcggtgcc ctgggaggga gacaagagga aacctccctc 2760
ggcccccgcg ctgcttctgg gccgcggggg ccgaggaagt gtgcccggaa gacgcgccac 2820
gcggagactt ccggggccgt ccccgcgggc ggcctcacgc gctacctgcc catctcgggg 2880
gtctctagcg tggtcgccct gtcgccttac gtgaacaaga cgatcacggg ggactgcctg 2940
cccatcctgg acatggagac ggggaacatc ggggcgtacg tggtcctggt ggaccagacg 3000
ggaaacatgg cgacccggct gcgggccgcg gtccccggct ggagccgccg caccctgctc 3060
cccgagaccg cgggtaacca cgtgatgccc cccgagtacc cgacggcccc cgcgtcggag 3120
tggaacagcc tctggatgac ccccgtgggg aacatgctgt tcgaccaggg caccctagtg 3180
ggcgccctgg acttccgcag cctgcggtct cggcacccgt ggtccgggga gcagggggcg 3240
tcgacccggg acgagggaaa acaataa 3267
<210> 26
<211> 775
<212> PRT
<213> Herpes simplex virus type 1
<400> 26
Met Glu Pro Arg Pro Gly Ala Ser Thr Arg Arg Pro Glu Gly Arg Pro
1 5 10 15
Gln Arg Glu Pro Ala Pro Asp Val Trp Val Phe Pro Cys Asp Arg Asp
20 25 30
Leu Pro Asp Ser Ser Asp Ser Glu Ala Glu Thr Glu Val Gly Gly Arg
35 40 45
Gly Asp Ala Asp His His Asp Asp Asp Ser Ala Ser Glu Ala Asp Ser
50 55 60
Thr Asp Thr Glu Leu Phe Glu Thr Gly Leu Leu Gly Pro Gln Gly Val
65 70 75 80
Asp Gly Gly Ala Val Ser Gly Gly Ser Pro Pro Arg Glu Glu Asp Pro
85 90 95
Gly Ser Cys Gly Gly Ala Pro Pro Arg Glu Asp Gly Gly Ser Asp Glu
100 105 110
Gly Asp Val Cys Ala Val Cys Thr Asp Glu Ile Ala Pro His Leu Arg
115 120 125
Cys Asp Thr Phe Pro Cys Met His Arg Phe Cys Ile Pro Cys Met Lys
130 135 140
Thr Trp Met Gln Leu Arg Asn Thr Cys Pro Leu Cys Asn Ala Lys Leu
145 150 155 160
Val Tyr Leu Ile Val Gly Val Thr Pro Ser Gly Ser Phe Ser Thr Ile
165 170 175
Pro Ile Val Asn Asp Pro Gln Thr Arg Met Glu Ala Glu Glu Ala Val
180 185 190
Arg Ala Gly Thr Ala Val Asp Phe Ile Trp Thr Gly Asn Gln Arg Phe
195 200 205
Ala Pro Arg Tyr Leu Thr Leu Gly Gly His Thr Val Arg Ala Leu Ser
210 215 220
Pro Thr His Pro Glu Pro Thr Thr Asp Glu Asp Asp Asp Asp Leu Asp
225 230 235 240
Asp Ala Asp Tyr Val Pro Pro Ala Pro Arg Arg Thr Pro Arg Ala Pro
245 250 255
Pro Arg Arg Gly Ala Ala Ala Pro Pro Val Thr Gly Gly Ala Ser His
260 265 270
Ala Ala Pro Gln Pro Ala Ala Ala Arg Thr Ala Pro Pro Ser Ala Pro
275 280 285
Ile Gly Pro His Gly Ser Ser Asn Thr Asn Thr Thr Thr Asn Ser Ser
290 295 300
Gly Gly Gly Gly Ser Arg Gln Ser Arg Ala Ala Ala Pro Arg Gly Ala
305 310 315 320
Ser Gly Pro Ser Gly Gly Val Gly Val Gly Val Gly Val Val Glu Ala
325 330 335
Glu Ala Gly Arg Pro Arg Gly Arg Thr Gly Pro Leu Val Asn Arg Pro
340 345 350
Ala Pro Leu Ala Asn Asn Arg Asp Pro Ile Val Ile Ser Asp Ser Pro
355 360 365
Pro Ala Ser Pro His Arg Pro Pro Ala Ala Pro Met Pro Gly Ser Ala
370 375 380
Pro Arg Pro Gly Pro Pro Ala Ser Ala Ala Ala Ser Gly Pro Ala Arg
385 390 395 400
Pro Arg Ala Ala Val Ala Pro Cys Val Arg Ala Pro Pro Pro Gly Pro
405 410 415
Gly Pro Arg Ala Pro Ala Pro Gly Ala Glu Pro Ala Ala Arg Pro Ala
420 425 430
Asp Ala Arg Arg Val Pro Gln Ser His Ser Ser Leu Ala Gln Ala Ala
435 440 445
Asn Gln Glu Gln Ser Leu Cys Arg Ala Arg Ala Thr Val Ala Arg Gly
450 455 460
Ser Gly Gly Pro Gly Val Glu Gly Gly His Gly Pro Ser Arg Gly Ala
465 470 475 480
Ala Pro Ser Gly Ala Ala Pro Leu Pro Ser Ala Ala Ser Val Glu Gln
485 490 495
Glu Ala Ala Val Arg Pro Arg Lys Arg Arg Gly Ser Gly Gln Glu Asn
500 505 510
Pro Ser Pro Gln Ser Thr Arg Pro Pro Leu Ala Pro Ala Gly Ala Lys
515 520 525
Arg Ala Ala Thr His Pro Pro Ser Asp Ser Gly Pro Gly Gly Arg Gly
530 535 540
Gln Gly Gly Pro Gly Thr Pro Leu Thr Ser Ser Ala Ala Ser Ala Ser
545 550 555 560
Ser Ser Ser Ala Ser Ser Ser Ser Ala Pro Thr Pro Ala Gly Ala Ala
565 570 575
Ser Ser Ala Ala Gly Ala Ala Ser Ser Ser Ala Ser Ala Ser Ser Gly
580 585 590
Gly Ala Val Gly Ala Leu Gly Gly Arg Gln Glu Glu Thr Ser Leu Gly
595 600 605
Pro Arg Ala Ala Ser Gly Pro Arg Gly Pro Arg Lys Cys Ala Arg Lys
610 615 620
Thr Arg His Ala Glu Thr Ser Gly Ala Val Pro Ala Gly Gly Leu Thr
625 630 635 640
Arg Tyr Leu Pro Ile Ser Gly Val Ser Ser Val Val Ala Leu Ser Pro
645 650 655
Tyr Val Asn Lys Thr Ile Thr Gly Asp Cys Leu Pro Ile Leu Asp Met
660 665 670
Glu Thr Gly Asn Ile Gly Ala Tyr Val Val Leu Val Asp Gln Thr Gly
675 680 685
Asn Met Ala Thr Arg Leu Arg Ala Ala Val Pro Gly Trp Ser Arg Arg
690 695 700
Thr Leu Leu Pro Glu Thr Ala Gly Asn His Val Met Pro Pro Glu Tyr
705 710 715 720
Pro Thr Ala Pro Ala Ser Glu Trp Asn Ser Leu Trp Met Thr Pro Val
725 730 735
Gly Asn Met Leu Phe Asp Gln Gly Thr Leu Val Gly Ala Leu Asp Phe
740 745 750
Arg Ser Leu Arg Ser Arg His Pro Trp Ser Gly Glu Gln Gly Ala Ser
755 760 765
Thr Arg Asp Glu Gly Lys Gln
770 775
<210> 27
<211> 3591
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<220>
<221> mutation
<222> (3464)..(3464)
<223> c3464t
<400> 27
atggagacaa agcccaagac ggcaaccacc atcaaggtcc cccccgggcc cctgggatac 60
gtgtacgctc gcgcgtgtcc gtccgaaggc atcgagcttc tggcgttact gtcggcacgc 120
agcggcgatt ccgacgtcgc cgtggcgccc ctggtcgtgg gcctgaccgt ggagagcggc 180
tttgaggcca acgtggccgt ggtcgtgggt tctcgcacga cggggctcgg gggtaccgcg 240
gtgtccctga aactgacgcc ctcgcactac agctcgtccg tgtacgtctt tcacggcggc 300
cggcacctgg accccagcac ccaggccccg aacctgacgc gactttgcga gcgggcacgc 360
cgccattttg gcttttcgga ctacaccccc cggcccggcg acctcaaaca cgagacgacg 420
ggggaggcgc tgtgtgagcg cctcggcctg gacccggacc gcgccctcct gtatctggtc 480
gttaccgagg gcttcaagga ggccgtgtgc atcaacaaca cctttctgca cctgggaggc 540
tcggacaagg taaccatagg cggggcggag gtgcaccgca tacccgtgta cccgttgcag 600
ctgttcatgc cggattttag ccgtgtcatc gcagagccgt tcaacgccaa ccaccgatcg 660
atcggggaga attttaccta cccgcttccg ttttttaacc gccccctcaa ccgcctcctg 720
ttcgaggcgg tcgtgggacc cgccgccgtg gcactgcgat gccgaaacgt ggacgccgtg 780
gcccgcgcgg ccgcccacct ggcgtttgac gaaaaccacg agggcgccgc cctccccgcc 840
gacattacgt tcacggcctt cgaagccagc cagggtaaga ccccgcgggg cgggcgcgac 900
ggcggcggca agggcccggc gggcgggttc gaacagcgcc tggcctccgt catggccgga 960
gacgccgccc tggccctcga ttctatcgtg tcgatggccg tctttgacga gccgcccacc 1020
gacatctccg cgtggccgct gttcgagggc caggacacgg ccgcggcccg cgccaacgcc 1080
gtcggggcgt acctggcgcg cgccgcggga ctcgtggggg ccatggtatt tagcaccaac 1140
tcggccctcc atctcaccga ggtggacgac gccggcccgg cggacccaaa ggaccacagc 1200
aaaccctcct tttaccgctt cttcctcgtg cccgggaccc acgtggcggc caacccacag 1260
gtggaccgcg agggacacgt ggtgcccggg ttcgagggtc ggcccaccgc gcccctcgtc 1320
ggcggaaccc aggaatttgc cggcgagcac ctggccatgc tgtgtgggtt ttccccggcg 1380
ctgctggcca agatgctgtt ttacctggag cgctgcgacg gcggcgtgat cgtcgggcgc 1440
caggagatgg acgtgtttcg atacgtcgcg gactccaacc agaccgacgt gccctgtaac 1500
ctatgcacct tcgacacgcg ccacgcctgc gtacacacga cgctcatgcg cctccgggcg 1560
cgccatccaa agttcgccag cgccgcccgc ggagccatcg gcgtcttcgg gaccatgaac 1620
agcatgtaca gcgactgcga cgtgctggga aactacgccg ccttctcggc cctgaagcgc 1680
gcggacggat ccgagaccgc ccggaccatc atgcaggaga cgtaccgcgc ggcgaccgag 1740
cgcgtcatgg ccgaactcga gaccctgcag tacgtggacc aggcggtccc cacggccatg 1800
gggcggctgg agaccatcat caccaaccgc gaggccctgc atacggtggt gaacaacgtc 1860
aggcaggtcg tggaccgcga ggtggagcag ctgatgcgca acctggtgga ggggaggaac 1920
ttcaagtttc gcgacggtct gggcgaggcc aaccacgcca tgtccctgac gctggacccg 1980
tacgcgtgcg ggccgtgccc cctgcttcag cttctcgggc ggcgatccaa cctcgccgtg 2040
taccaggacc tggccctgag tcagtgccac ggggtgttcg ccgggcagtc ggtcgagggg 2100
cgcaactttc gcaatcaatt ccaaccggtg ctgcggcggc gcgtgatgga catgtttaac 2160
aacgggtttc tgtcggccaa aacgctgacg gtcgcgctct cggagggggc ggctatctgc 2220
gcccccagcc taacggccgg ccagacggcc cccgccgaga gcagcttcga gggcgacgtt 2280
gcccgcgtga ccctggggtt tcccaaggag ctgcgcgtca agagccgcgt gttgttcgcg 2340
ggcgcgagcg ccaacgcgtc cgaggccgcc aaggcgcggg tcgccagcct ccagagcgcc 2400
taccagaagc ccgacaagcg cgtggacatc ctcctcggac cgctgggctt tctgctgaag 2460
cagttccacg cggccatctt ccccaacggc aagcccccgg ggtccaacca gccgaacccg 2520
cagtggttct ggacggccct ccaacgcaac cagcttcccg cccggctcct gtcgcgcgag 2580
gacatcgaga ccatcgcgtt cattaaaaag ttttccctgg actacggcgc gataaacttt 2640
attaacctgg cccccaacaa cgtgagcgag ctggcgatgt actacatggc aaaccagatt 2700
ctgcggtact gcgatcactc gacatacttc atcaacaccc ttacggccat catcgcgggg 2760
tcccgccgtc cccccagcgt gcaggctgcg gccgcgtggt ccgcgcaggg cggggcgggc 2820
ctggaggccg gggcccgcgc gctgatggac gccgtggacg cgcatccggg cgcgtggacg 2880
tccatgttcg ccagctgcaa cctgctgcgg cccgtcatgg cggcgcgccc catggtcgtg 2940
ttggggttga gcatcagcaa gtactacggc atggccggca acgaccgtgt gtttcaggcc 3000
gggaactggg ccagcctgat gggcggcaaa aacgcgtgcc cgctccttat ttttgaccgc 3060
acccgcaagt tcgtcctggc ctgtccccgg gccgggtttg tgtgcgcggc ctcaagcctc 3120
ggcggcggag cgcacgaaag ctcgctgtgc gagcagctcc ggggcattat ctccgagggc 3180
ggggcggccg tcgccagtag cgtgttcgtg gcgaccgtga aaagcctggg gccccgcacc 3240
cagcagctgc agatcgagga ctggctggcg ctcctggagg acgagtacct aagcgaggag 3300
atgatggagc tgaccgcgcg tgccctggag cgcggcaacg gcgagtggtc gacggacgcg 3360
gccctggagg tggcgcacga ggccgaggcc ctagtcagcc aactcggcaa cgccggggag 3420
gtgtttaact ttggggattt tggctgcgag gacgacaacg cgatgccgtt cggcggcccg 3480
ggggccccgg gaccggcatt tgccggccgc aaacgggcgt tccacgggga tgacccgttt 3540
ggggaggggc cccccgacaa aaagggagac ctgacgttgg atatgctgtg a 3591
<210> 28
<211> 1196
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<220>
<221> MISC_FEATURE
<222> (1155)..(1155)
<223> T1155M
<400> 28
Met Glu Thr Lys Pro Lys Thr Ala Thr Thr Ile Lys Val Pro Pro Gly
1 5 10 15
Pro Leu Gly Tyr Val Tyr Ala Arg Ala Cys Pro Ser Glu Gly Ile Glu
20 25 30
Leu Leu Ala Leu Leu Ser Ala Arg Ser Gly Asp Ser Asp Val Ala Val
35 40 45
Ala Pro Leu Val Val Gly Leu Thr Val Glu Ser Gly Phe Glu Ala Asn
50 55 60
Val Ala Val Val Val Gly Ser Arg Thr Thr Gly Leu Gly Gly Thr Ala
65 70 75 80
Val Ser Leu Lys Leu Thr Pro Ser His Tyr Ser Ser Ser Val Tyr Val
85 90 95
Phe His Gly Gly Arg His Leu Asp Pro Ser Thr Gln Ala Pro Asn Leu
100 105 110
Thr Arg Leu Cys Glu Arg Ala Arg Arg His Phe Gly Phe Ser Asp Tyr
115 120 125
Thr Pro Arg Pro Gly Asp Leu Lys His Glu Thr Thr Gly Glu Ala Leu
130 135 140
Cys Glu Arg Leu Gly Leu Asp Pro Asp Arg Ala Leu Leu Tyr Leu Val
145 150 155 160
Val Thr Glu Gly Phe Lys Glu Ala Val Cys Ile Asn Asn Thr Phe Leu
165 170 175
His Leu Gly Gly Ser Asp Lys Val Thr Ile Gly Gly Ala Glu Val His
180 185 190
Arg Ile Pro Val Tyr Pro Leu Gln Leu Phe Met Pro Asp Phe Ser Arg
195 200 205
Val Ile Ala Glu Pro Phe Asn Ala Asn His Arg Ser Ile Gly Glu Asn
210 215 220
Phe Thr Tyr Pro Leu Pro Phe Phe Asn Arg Pro Leu Asn Arg Leu Leu
225 230 235 240
Phe Glu Ala Val Val Gly Pro Ala Ala Val Ala Leu Arg Cys Arg Asn
245 250 255
Val Asp Ala Val Ala Arg Ala Ala Ala His Leu Ala Phe Asp Glu Asn
260 265 270
His Glu Gly Ala Ala Leu Pro Ala Asp Ile Thr Phe Thr Ala Phe Glu
275 280 285
Ala Ser Gln Gly Lys Thr Pro Arg Gly Gly Arg Asp Gly Gly Gly Lys
290 295 300
Gly Pro Ala Gly Gly Phe Glu Gln Arg Leu Ala Ser Val Met Ala Gly
305 310 315 320
Asp Ala Ala Leu Ala Leu Asp Ser Ile Val Ser Met Ala Val Phe Asp
325 330 335
Glu Pro Pro Thr Asp Ile Ser Ala Trp Pro Leu Phe Glu Gly Gln Asp
340 345 350
Thr Ala Ala Ala Arg Ala Asn Ala Val Gly Ala Tyr Leu Ala Arg Ala
355 360 365
Ala Gly Leu Val Gly Ala Met Val Phe Ser Thr Asn Ser Ala Leu His
370 375 380
Leu Thr Glu Val Asp Asp Ala Gly Pro Ala Asp Pro Lys Asp His Ser
385 390 395 400
Lys Pro Ser Phe Tyr Arg Phe Phe Leu Val Pro Gly Thr His Val Ala
405 410 415
Ala Asn Pro Gln Val Asp Arg Glu Gly His Val Val Pro Gly Phe Glu
420 425 430
Gly Arg Pro Thr Ala Pro Leu Val Gly Gly Thr Gln Glu Phe Ala Gly
435 440 445
Glu His Leu Ala Met Leu Cys Gly Phe Ser Pro Ala Leu Leu Ala Lys
450 455 460
Met Leu Phe Tyr Leu Glu Arg Cys Asp Gly Gly Val Ile Val Gly Arg
465 470 475 480
Gln Glu Met Asp Val Phe Arg Tyr Val Ala Asp Ser Asn Gln Thr Asp
485 490 495
Val Pro Cys Asn Leu Cys Thr Phe Asp Thr Arg His Ala Cys Val His
500 505 510
Thr Thr Leu Met Arg Leu Arg Ala Arg His Pro Lys Phe Ala Ser Ala
515 520 525
Ala Arg Gly Ala Ile Gly Val Phe Gly Thr Met Asn Ser Met Tyr Ser
530 535 540
Asp Cys Asp Val Leu Gly Asn Tyr Ala Ala Phe Ser Ala Leu Lys Arg
545 550 555 560
Ala Asp Gly Ser Glu Thr Ala Arg Thr Ile Met Gln Glu Thr Tyr Arg
565 570 575
Ala Ala Thr Glu Arg Val Met Ala Glu Leu Glu Thr Leu Gln Tyr Val
580 585 590
Asp Gln Ala Val Pro Thr Ala Met Gly Arg Leu Glu Thr Ile Ile Thr
595 600 605
Asn Arg Glu Ala Leu His Thr Val Val Asn Asn Val Arg Gln Val Val
610 615 620
Asp Arg Glu Val Glu Gln Leu Met Arg Asn Leu Val Glu Gly Arg Asn
625 630 635 640
Phe Lys Phe Arg Asp Gly Leu Gly Glu Ala Asn His Ala Met Ser Leu
645 650 655
Thr Leu Asp Pro Tyr Ala Cys Gly Pro Cys Pro Leu Leu Gln Leu Leu
660 665 670
Gly Arg Arg Ser Asn Leu Ala Val Tyr Gln Asp Leu Ala Leu Ser Gln
675 680 685
Cys His Gly Val Phe Ala Gly Gln Ser Val Glu Gly Arg Asn Phe Arg
690 695 700
Asn Gln Phe Gln Pro Val Leu Arg Arg Arg Val Met Asp Met Phe Asn
705 710 715 720
Asn Gly Phe Leu Ser Ala Lys Thr Leu Thr Val Ala Leu Ser Glu Gly
725 730 735
Ala Ala Ile Cys Ala Pro Ser Leu Thr Ala Gly Gln Thr Ala Pro Ala
740 745 750
Glu Ser Ser Phe Glu Gly Asp Val Ala Arg Val Thr Leu Gly Phe Pro
755 760 765
Lys Glu Leu Arg Val Lys Ser Arg Val Leu Phe Ala Gly Ala Ser Ala
770 775 780
Asn Ala Ser Glu Ala Ala Lys Ala Arg Val Ala Ser Leu Gln Ser Ala
785 790 795 800
Tyr Gln Lys Pro Asp Lys Arg Val Asp Ile Leu Leu Gly Pro Leu Gly
805 810 815
Phe Leu Leu Lys Gln Phe His Ala Ala Ile Phe Pro Asn Gly Lys Pro
820 825 830
Pro Gly Ser Asn Gln Pro Asn Pro Gln Trp Phe Trp Thr Ala Leu Gln
835 840 845
Arg Asn Gln Leu Pro Ala Arg Leu Leu Ser Arg Glu Asp Ile Glu Thr
850 855 860
Ile Ala Phe Ile Lys Lys Phe Ser Leu Asp Tyr Gly Ala Ile Asn Phe
865 870 875 880
Ile Asn Leu Ala Pro Asn Asn Val Ser Glu Leu Ala Met Tyr Tyr Met
885 890 895
Ala Asn Gln Ile Leu Arg Tyr Cys Asp His Ser Thr Tyr Phe Ile Asn
900 905 910
Thr Leu Thr Ala Ile Ile Ala Gly Ser Arg Arg Pro Pro Ser Val Gln
915 920 925
Ala Ala Ala Ala Trp Ser Ala Gln Gly Gly Ala Gly Leu Glu Ala Gly
930 935 940
Ala Arg Ala Leu Met Asp Ala Val Asp Ala His Pro Gly Ala Trp Thr
945 950 955 960
Ser Met Phe Ala Ser Cys Asn Leu Leu Arg Pro Val Met Ala Ala Arg
965 970 975
Pro Met Val Val Leu Gly Leu Ser Ile Ser Lys Tyr Tyr Gly Met Ala
980 985 990
Gly Asn Asp Arg Val Phe Gln Ala Gly Asn Trp Ala Ser Leu Met Gly
995 1000 1005
Gly Lys Asn Ala Cys Pro Leu Leu Ile Phe Asp Arg Thr Arg Lys
1010 1015 1020
Phe Val Leu Ala Cys Pro Arg Ala Gly Phe Val Cys Ala Ala Ser
1025 1030 1035
Ser Leu Gly Gly Gly Ala His Glu Ser Ser Leu Cys Glu Gln Leu
1040 1045 1050
Arg Gly Ile Ile Ser Glu Gly Gly Ala Ala Val Ala Ser Ser Val
1055 1060 1065
Phe Val Ala Thr Val Lys Ser Leu Gly Pro Arg Thr Gln Gln Leu
1070 1075 1080
Gln Ile Glu Asp Trp Leu Ala Leu Leu Glu Asp Glu Tyr Leu Ser
1085 1090 1095
Glu Glu Met Met Glu Leu Thr Ala Arg Ala Leu Glu Arg Gly Asn
1100 1105 1110
Gly Glu Trp Ser Thr Asp Ala Ala Leu Glu Val Ala His Glu Ala
1115 1120 1125
Glu Ala Leu Val Ser Gln Leu Gly Asn Ala Gly Glu Val Phe Asn
1130 1135 1140
Phe Gly Asp Phe Gly Cys Glu Asp Asp Asn Ala Met Pro Phe Gly
1145 1150 1155
Gly Pro Gly Ala Pro Gly Pro Ala Phe Ala Gly Arg Lys Arg Ala
1160 1165 1170
Phe His Gly Asp Asp Pro Phe Gly Glu Gly Pro Pro Asp Lys Lys
1175 1180 1185
Gly Asp Leu Thr Leu Asp Met Leu
1190 1195
<210> 29
<211> 3591
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 29
atggagacaa agcccaagac ggcaaccacc atcaaggtcc cccccgggcc cctgggatac 60
gtgtacgctc gcgcgtgtcc gtccgaaggc atcgagcttc tggcgttact gtcggcacgc 120
agcggcgatt ccgacgtcgc cgtggcgccc ctggtcgtgg gcctgaccgt ggagagcggc 180
tttgaggcca acgtggccgt ggtcgtgggt tctcgcacga cggggctcgg gggtaccgcg 240
gtgtccctga aactgacgcc ctcgcactac agctcgtccg tgtacgtctt tcacggcggc 300
cggcacctgg accccagcac ccaggccccg aacctgacgc gactttgcga gcgggcacgc 360
cgccattttg gcttttcgga ctacaccccc cggcccggcg acctcaaaca cgagacgacg 420
ggggaggcgc tgtgtgagcg cctcggcctg gacccggacc gcgccctcct gtatctggtc 480
gttaccgagg gcttcaagga ggccgtgtgc atcaacaaca cctttctgca cctgggaggc 540
tcggacaagg taaccatagg cggggcggag gtgcaccgca tacccgtgta cccgttgcag 600
ctgttcatgc cggattttag ccgtgtcatc gcagagccgt tcaacgccaa ccaccgatcg 660
atcggggaga attttaccta cccgcttccg ttttttaacc gccccctcaa ccgcctcctg 720
ttcgaggcgg tcgtgggacc cgccgccgtg gcactgcgat gccgaaacgt ggacgccgtg 780
gcccgcgcgg ccgcccacct ggcgtttgac gaaaaccacg agggcgccgc cctccccgcc 840
gacattacgt tcacggcctt cgaagccagc cagggtaaga ccccgcgggg cgggcgcgac 900
ggcggcggca agggcccggc gggcgggttc gaacagcgcc tggcctccgt catggccgga 960
gacgccgccc tggccctcga ttctatcgtg tcgatggccg tctttgacga gccgcccacc 1020
gacatctccg cgtggccgct gttcgagggc caggacacgg ccgcggcccg cgccaacgcc 1080
gtcggggcgt acctggcgcg cgccgcggga ctcgtggggg ccatggtatt tagcaccaac 1140
tcggccctcc atctcaccga ggtggacgac gccggcccgg cggacccaaa ggaccacagc 1200
aaaccctcct tttaccgctt cttcctcgtg cccgggaccc acgtggcggc caacccacag 1260
gtggaccgcg agggacacgt ggtgcccggg ttcgagggtc ggcccaccgc gcccctcgtc 1320
ggcggaaccc aggaatttgc cggcgagcac ctggccatgc tgtgtgggtt ttccccggcg 1380
ctgctggcca agatgctgtt ttacctggag cgctgcgacg gcggcgtgat cgtcgggcgc 1440
caggagatgg acgtgtttcg atacgtcgcg gactccaacc agaccgacgt gccctgtaac 1500
ctatgcacct tcgacacgcg ccacgcctgc gtacacacga cgctcatgcg cctccgggcg 1560
cgccatccaa agttcgccag cgccgcccgc ggagccatcg gcgtcttcgg gaccatgaac 1620
agcatgtaca gcgactgcga cgtgctggga aactacgccg ccttctcggc cctgaagcgc 1680
gcggacggat ccgagaccgc ccggaccatc atgcaggaga cgtaccgcgc ggcgaccgag 1740
cgcgtcatgg ccgaactcga gaccctgcag tacgtggacc aggcggtccc cacggccatg 1800
gggcggctgg agaccatcat caccaaccgc gaggccctgc atacggtggt gaacaacgtc 1860
aggcaggtcg tggaccgcga ggtggagcag ctgatgcgca acctggtgga ggggaggaac 1920
ttcaagtttc gcgacggtct gggcgaggcc aaccacgcca tgtccctgac gctggacccg 1980
tacgcgtgcg ggccgtgccc cctgcttcag cttctcgggc ggcgatccaa cctcgccgtg 2040
taccaggacc tggccctgag tcagtgccac ggggtgttcg ccgggcagtc ggtcgagggg 2100
cgcaactttc gcaatcaatt ccaaccggtg ctgcggcggc gcgtgatgga catgtttaac 2160
aacgggtttc tgtcggccaa aacgctgacg gtcgcgctct cggagggggc ggctatctgc 2220
gcccccagcc taacggccgg ccagacggcc cccgccgaga gcagcttcga gggcgacgtt 2280
gcccgcgtga ccctggggtt tcccaaggag ctgcgcgtca agagccgcgt gttgttcgcg 2340
ggcgcgagcg ccaacgcgtc cgaggccgcc aaggcgcggg tcgccagcct ccagagcgcc 2400
taccagaagc ccgacaagcg cgtggacatc ctcctcggac cgctgggctt tctgctgaag 2460
cagttccacg cggccatctt ccccaacggc aagcccccgg ggtccaacca gccgaacccg 2520
cagtggttct ggacggccct ccaacgcaac cagcttcccg cccggctcct gtcgcgcgag 2580
gacatcgaga ccatcgcgtt cattaaaaag ttttccctgg actacggcgc gataaacttt 2640
attaacctgg cccccaacaa cgtgagcgag ctggcgatgt actacatggc aaaccagatt 2700
ctgcggtact gcgatcactc gacatacttc atcaacaccc ttacggccat catcgcgggg 2760
tcccgccgtc cccccagcgt gcaggctgcg gccgcgtggt ccgcgcaggg cggggcgggc 2820
ctggaggccg gggcccgcgc gctgatggac gccgtggacg cgcatccggg cgcgtggacg 2880
tccatgttcg ccagctgcaa cctgctgcgg cccgtcatgg cggcgcgccc catggtcgtg 2940
ttggggttga gcatcagcaa gtactacggc atggccggca acgaccgtgt gtttcaggcc 3000
gggaactggg ccagcctgat gggcggcaaa aacgcgtgcc cgctccttat ttttgaccgc 3060
acccgcaagt tcgtcctggc ctgtccccgg gccgggtttg tgtgcgcggc ctcaagcctc 3120
ggcggcggag cgcacgaaag ctcgctgtgc gagcagctcc ggggcattat ctccgagggc 3180
ggggcggccg tcgccagtag cgtgttcgtg gcgaccgtga aaagcctggg gccccgcacc 3240
cagcagctgc agatcgagga ctggctggcg ctcctggagg acgagtacct aagcgaggag 3300
atgatggagc tgaccgcgcg tgccctggag cgcggcaacg gcgagtggtc gacggacgcg 3360
gccctggagg tggcgcacga ggccgaggcc ctagtcagcc aactcggcaa cgccggggag 3420
gtgtttaact ttggggattt tggctgcgag gacgacaacg cgacgccgtt cggcggcccg 3480
ggggccccgg gaccggcatt tgccggccgc aaacgggcgt tccacgggga tgacccgttt 3540
ggggaggggc cccccgacaa aaagggagac ctgacgttgg atatgctgtg a 3591
<210> 30
<211> 1196
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 30
Met Glu Thr Lys Pro Lys Thr Ala Thr Thr Ile Lys Val Pro Pro Gly
1 5 10 15
Pro Leu Gly Tyr Val Tyr Ala Arg Ala Cys Pro Ser Glu Gly Ile Glu
20 25 30
Leu Leu Ala Leu Leu Ser Ala Arg Ser Gly Asp Ser Asp Val Ala Val
35 40 45
Ala Pro Leu Val Val Gly Leu Thr Val Glu Ser Gly Phe Glu Ala Asn
50 55 60
Val Ala Val Val Val Gly Ser Arg Thr Thr Gly Leu Gly Gly Thr Ala
65 70 75 80
Val Ser Leu Lys Leu Thr Pro Ser His Tyr Ser Ser Ser Val Tyr Val
85 90 95
Phe His Gly Gly Arg His Leu Asp Pro Ser Thr Gln Ala Pro Asn Leu
100 105 110
Thr Arg Leu Cys Glu Arg Ala Arg Arg His Phe Gly Phe Ser Asp Tyr
115 120 125
Thr Pro Arg Pro Gly Asp Leu Lys His Glu Thr Thr Gly Glu Ala Leu
130 135 140
Cys Glu Arg Leu Gly Leu Asp Pro Asp Arg Ala Leu Leu Tyr Leu Val
145 150 155 160
Val Thr Glu Gly Phe Lys Glu Ala Val Cys Ile Asn Asn Thr Phe Leu
165 170 175
His Leu Gly Gly Ser Asp Lys Val Thr Ile Gly Gly Ala Glu Val His
180 185 190
Arg Ile Pro Val Tyr Pro Leu Gln Leu Phe Met Pro Asp Phe Ser Arg
195 200 205
Val Ile Ala Glu Pro Phe Asn Ala Asn His Arg Ser Ile Gly Glu Asn
210 215 220
Phe Thr Tyr Pro Leu Pro Phe Phe Asn Arg Pro Leu Asn Arg Leu Leu
225 230 235 240
Phe Glu Ala Val Val Gly Pro Ala Ala Val Ala Leu Arg Cys Arg Asn
245 250 255
Val Asp Ala Val Ala Arg Ala Ala Ala His Leu Ala Phe Asp Glu Asn
260 265 270
His Glu Gly Ala Ala Leu Pro Ala Asp Ile Thr Phe Thr Ala Phe Glu
275 280 285
Ala Ser Gln Gly Lys Thr Pro Arg Gly Gly Arg Asp Gly Gly Gly Lys
290 295 300
Gly Pro Ala Gly Gly Phe Glu Gln Arg Leu Ala Ser Val Met Ala Gly
305 310 315 320
Asp Ala Ala Leu Ala Leu Asp Ser Ile Val Ser Met Ala Val Phe Asp
325 330 335
Glu Pro Pro Thr Asp Ile Ser Ala Trp Pro Leu Phe Glu Gly Gln Asp
340 345 350
Thr Ala Ala Ala Arg Ala Asn Ala Val Gly Ala Tyr Leu Ala Arg Ala
355 360 365
Ala Gly Leu Val Gly Ala Met Val Phe Ser Thr Asn Ser Ala Leu His
370 375 380
Leu Thr Glu Val Asp Asp Ala Gly Pro Ala Asp Pro Lys Asp His Ser
385 390 395 400
Lys Pro Ser Phe Tyr Arg Phe Phe Leu Val Pro Gly Thr His Val Ala
405 410 415
Ala Asn Pro Gln Val Asp Arg Glu Gly His Val Val Pro Gly Phe Glu
420 425 430
Gly Arg Pro Thr Ala Pro Leu Val Gly Gly Thr Gln Glu Phe Ala Gly
435 440 445
Glu His Leu Ala Met Leu Cys Gly Phe Ser Pro Ala Leu Leu Ala Lys
450 455 460
Met Leu Phe Tyr Leu Glu Arg Cys Asp Gly Gly Val Ile Val Gly Arg
465 470 475 480
Gln Glu Met Asp Val Phe Arg Tyr Val Ala Asp Ser Asn Gln Thr Asp
485 490 495
Val Pro Cys Asn Leu Cys Thr Phe Asp Thr Arg His Ala Cys Val His
500 505 510
Thr Thr Leu Met Arg Leu Arg Ala Arg His Pro Lys Phe Ala Ser Ala
515 520 525
Ala Arg Gly Ala Ile Gly Val Phe Gly Thr Met Asn Ser Met Tyr Ser
530 535 540
Asp Cys Asp Val Leu Gly Asn Tyr Ala Ala Phe Ser Ala Leu Lys Arg
545 550 555 560
Ala Asp Gly Ser Glu Thr Ala Arg Thr Ile Met Gln Glu Thr Tyr Arg
565 570 575
Ala Ala Thr Glu Arg Val Met Ala Glu Leu Glu Thr Leu Gln Tyr Val
580 585 590
Asp Gln Ala Val Pro Thr Ala Met Gly Arg Leu Glu Thr Ile Ile Thr
595 600 605
Asn Arg Glu Ala Leu His Thr Val Val Asn Asn Val Arg Gln Val Val
610 615 620
Asp Arg Glu Val Glu Gln Leu Met Arg Asn Leu Val Glu Gly Arg Asn
625 630 635 640
Phe Lys Phe Arg Asp Gly Leu Gly Glu Ala Asn His Ala Met Ser Leu
645 650 655
Thr Leu Asp Pro Tyr Ala Cys Gly Pro Cys Pro Leu Leu Gln Leu Leu
660 665 670
Gly Arg Arg Ser Asn Leu Ala Val Tyr Gln Asp Leu Ala Leu Ser Gln
675 680 685
Cys His Gly Val Phe Ala Gly Gln Ser Val Glu Gly Arg Asn Phe Arg
690 695 700
Asn Gln Phe Gln Pro Val Leu Arg Arg Arg Val Met Asp Met Phe Asn
705 710 715 720
Asn Gly Phe Leu Ser Ala Lys Thr Leu Thr Val Ala Leu Ser Glu Gly
725 730 735
Ala Ala Ile Cys Ala Pro Ser Leu Thr Ala Gly Gln Thr Ala Pro Ala
740 745 750
Glu Ser Ser Phe Glu Gly Asp Val Ala Arg Val Thr Leu Gly Phe Pro
755 760 765
Lys Glu Leu Arg Val Lys Ser Arg Val Leu Phe Ala Gly Ala Ser Ala
770 775 780
Asn Ala Ser Glu Ala Ala Lys Ala Arg Val Ala Ser Leu Gln Ser Ala
785 790 795 800
Tyr Gln Lys Pro Asp Lys Arg Val Asp Ile Leu Leu Gly Pro Leu Gly
805 810 815
Phe Leu Leu Lys Gln Phe His Ala Ala Ile Phe Pro Asn Gly Lys Pro
820 825 830
Pro Gly Ser Asn Gln Pro Asn Pro Gln Trp Phe Trp Thr Ala Leu Gln
835 840 845
Arg Asn Gln Leu Pro Ala Arg Leu Leu Ser Arg Glu Asp Ile Glu Thr
850 855 860
Ile Ala Phe Ile Lys Lys Phe Ser Leu Asp Tyr Gly Ala Ile Asn Phe
865 870 875 880
Ile Asn Leu Ala Pro Asn Asn Val Ser Glu Leu Ala Met Tyr Tyr Met
885 890 895
Ala Asn Gln Ile Leu Arg Tyr Cys Asp His Ser Thr Tyr Phe Ile Asn
900 905 910
Thr Leu Thr Ala Ile Ile Ala Gly Ser Arg Arg Pro Pro Ser Val Gln
915 920 925
Ala Ala Ala Ala Trp Ser Ala Gln Gly Gly Ala Gly Leu Glu Ala Gly
930 935 940
Ala Arg Ala Leu Met Asp Ala Val Asp Ala His Pro Gly Ala Trp Thr
945 950 955 960
Ser Met Phe Ala Ser Cys Asn Leu Leu Arg Pro Val Met Ala Ala Arg
965 970 975
Pro Met Val Val Leu Gly Leu Ser Ile Ser Lys Tyr Tyr Gly Met Ala
980 985 990
Gly Asn Asp Arg Val Phe Gln Ala Gly Asn Trp Ala Ser Leu Met Gly
995 1000 1005
Gly Lys Asn Ala Cys Pro Leu Leu Ile Phe Asp Arg Thr Arg Lys
1010 1015 1020
Phe Val Leu Ala Cys Pro Arg Ala Gly Phe Val Cys Ala Ala Ser
1025 1030 1035
Ser Leu Gly Gly Gly Ala His Glu Ser Ser Leu Cys Glu Gln Leu
1040 1045 1050
Arg Gly Ile Ile Ser Glu Gly Gly Ala Ala Val Ala Ser Ser Val
1055 1060 1065
Phe Val Ala Thr Val Lys Ser Leu Gly Pro Arg Thr Gln Gln Leu
1070 1075 1080
Gln Ile Glu Asp Trp Leu Ala Leu Leu Glu Asp Glu Tyr Leu Ser
1085 1090 1095
Glu Glu Met Met Glu Leu Thr Ala Arg Ala Leu Glu Arg Gly Asn
1100 1105 1110
Gly Glu Trp Ser Thr Asp Ala Ala Leu Glu Val Ala His Glu Ala
1115 1120 1125
Glu Ala Leu Val Ser Gln Leu Gly Asn Ala Gly Glu Val Phe Asn
1130 1135 1140
Phe Gly Asp Phe Gly Cys Glu Asp Asp Asn Ala Thr Pro Phe Gly
1145 1150 1155
Gly Pro Gly Ala Pro Gly Pro Ala Phe Ala Gly Arg Lys Arg Ala
1160 1165 1170
Phe His Gly Asp Asp Pro Phe Gly Glu Gly Pro Pro Asp Lys Lys
1175 1180 1185
Gly Asp Leu Thr Leu Asp Met Leu
1190 1195
<210> 31
<211> 3591
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 31
atggagacaa agcccaagac ggcaaccacc atcaaggtcc cccccgggcc cctgggatac 60
gtgtacgctc gcgcgtgtcc gtccgaaggc atcgagcttc tggcgttact gtcggcgcgc 120
agcggcgatg ccgacgtcgc cgtggcgccc ctggtcgtgg gcctgaccgt ggagagcggc 180
tttgaggcca acgtagccgt ggtcgtgggt tctcgcacga cggggctcgg gggtaccgcg 240
gtgtccctga aactgacgcc atcgcactac agctcgtccg tgtacgtctt tcacggcggc 300
cggcacctgg accccagcac ccaggcccca aacctgacgc gactctgcga gcgggcacgc 360
cgccattttg gcttttcgga ctacaccccc cggcccggcg acctcaaaca cgagacgacg 420
ggggaggcgc tgtgtgagcg cctcggcctg gacccggacc gcgccctcct gtatctggtc 480
gttaccgagg gcttcaagga ggccgtgtgc atcaacaaca cctttctgca cctgggaggc 540
tcggacaagg taaccatagg cggggcggag gtgcaccgca tacccgtgta tccgttgcag 600
ctgttcatgc cggattttag ccgggtcatc gccgagccgt tcaacgccaa ccaccgatcg 660
atcggggaga attttaccta cccgcttccg ttttttaacc gccccctcaa ccgcctcctg 720
ttcgaggcgg tcgtgggacc cgccgccgtg gcactgcgat gccgaaacgt ggacgccgtg 780
gcccgcgcgg ccgcccacct ggcgtttgac gaaaaccacg agggcgccgc cctccccgcc 840
gacattacgt tcacggcctt cgaagccagc cagggtaaga ccccgcgggg tgggcgcgac 900
ggcggcggca agggcccggc gggcgggttc gaacagcgcc tggcctccgt catggccgga 960
gacgccgccc tggccctcga gtctatcgtg tcgatggccg tcttcgacga gccgcccacc 1020
gacatctccg cgtggccgct gtgcgagggc caggacacgg ccgcggcccg cgacaacgcc 1080
gtcggggcgt acctggcgcg cgccgcggga ctcgtggggg ccatggtatt tagcaccaac 1140
tcggccctcc atctcaccga ggtggacgac gccggtccgg cggacccaaa ggaccacagc 1200
aaaccctcct tttaccgctt cttcctcgtg cccgggaccc acgtggcggc caacccacag 1260
gtggaccgcg agggacacgt ggtgcccggg ttcgagggtc ggcccaccgc gcccctcgtc 1320
ggcggaaccc aggaatttgc cggcgagcac ctggccatgc tgtgtgggtt ttccccggcg 1380
ctgctggcca agatgctgtt ttacctggag cgctgcgacg gcggcgtgat cgtcgggcgc 1440
caggagatgg acgtgtttcg atacgtcgcg gactccaacc agaccgacgt gccctgcaac 1500
ctgtgcacct tcgacacgcg ccacgcctgc gtacacacga cgctcatgcg cctccgggcg 1560
cgccatccca agttcgccag cgccgcccgc ggagccatcg gcgtcttcgg gaccatgaac 1620
agcatgtaca gcgactgcga cgtgctggga aactacgccg ccttctcggc cctgaagcgc 1680
gcggacggat ccgagaccgc ccggaccatc atgcaggaga cgtaccgcgc ggcgaccgag 1740
cgcgtcatgg ccgaactcga gaccctgcag tacgtggacc aggcggtccc cacggccatg 1800
gggcggctgg agaccatcat caccaaccgc gaggccctgc atacggtggt gaacaacgtc 1860
aggcaggtcg tggaccgcga ggtggagcag ctgatgcgca acctggtgga ggggaggaac 1920
ttcaagtttc gcgacggtct gggcgaggcc aaccacgcca tgtccctgac gctggacccg 1980
tacgcgtgcg ggccatgccc cctgcttcag cttctcgggc ggcgatccaa cctcgccgtg 2040
tatcaggacc tggccctgag ccagtgccac ggggtgttcg ccgggcagtc ggtcgagggg 2100
cgcaactttc gcaatcaatt ccaaccggtg ctgcggcggc gcgtgatgga catgtttaac 2160
aacgggtttc tgtcggccaa aacgctgacg gtcgcgctct cggagggggc ggctatctgc 2220
gcccccagcc taacggccgg ccagacggcc cccgccgaga gcagcttcga gggcgacgtt 2280
gcccgcgtga ccctggggtt tcccaaggag ctgcgcgtca agagccgcgt gttgttcgcg 2340
ggcgcgagcg ccaacgcgtc cgaggccgcc aaggcgcggg tcgccagcct ccagagcgcc 2400
taccagaagc ccgacaagcg cgtggacatc ctcctcggac cgctgggctt tctgctgaag 2460
cagttccacg cggccatctt ccccaacggc aagcccccgg ggtccaacca gccgaacccg 2520
cagtggttct ggacggccct ccaacgcaac cagcttcccg cccggctcct gtcgcgcgag 2580
gacatcgaga ccatcgcgtt cattaaaaag ttttccctgg actacggcgc gataaacttt 2640
attaacctgg cccccaacaa cgtgagcgag ctggcgatgt actacatggc aaaccagatt 2700
ctgcggtact gcgatcactc gacatacttc atcaacaccc tcacggccat catcgcgggg 2760
tcccgccgtc cccccagcgt gcaggcggcg gccgcgtggt ccgcgcaggg cggggcgggc 2820
ctggaggccg gggcccgcgc gctgatggac gccgtggacg cgcatccggg cgcgtggacg 2880
tccatgttcg ccagctgcaa cctgctgcgg cccgtcatgg cggcgcgccc catggtcgtg 2940
ttggggttga gcatcagcaa atactacggc atggccggca acgaccgtgt gtttcaggcc 3000
gggaactggg ccagcctgat gggcggcaaa aacgcgtgcc cgctccttat ttttgaccgc 3060
acccgcaagt tcgtcctggc ctgtccccgg gccgggtttg tgtgcgcggc ctcgaacctc 3120
ggcggcggag cgcacgaaag ctcgctgtgc gagcagctcc ggggcattat ctccgagggc 3180
ggggcggccg tcgccagtag cgtgttcgtg gcgaccgtga aaagcctggg gccccgcacc 3240
cagcagctgc agatcgagga ctggctggcg ctcctggagg acgagtacct aagcgaggag 3300
atgatggagc tgaccgcgcg tgccctggag cgcggcaacg gcgagtggtc gacggacgcg 3360
gccctggagg tggcgcacga ggccgaggcc ctagtcagcc aactcggcaa cgccggggag 3420
gtgtttaact ttggggattt tggctgcgag gacgacaacg cgacgccgtt cggcggcccg 3480
ggggccccgg gaccggcatt tgccggccgc aaacgggcgt tccacgggga tgacccgttt 3540
ggggaggggc cccccgacaa aaagggagac ctgacgttgg atatgctgtg a 3591
<210> 32
<211> 1196
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 32
Met Glu Thr Lys Pro Lys Thr Ala Thr Thr Ile Lys Val Pro Pro Gly
1 5 10 15
Pro Leu Gly Tyr Val Tyr Ala Arg Ala Cys Pro Ser Glu Gly Ile Glu
20 25 30
Leu Leu Ala Leu Leu Ser Ala Arg Ser Gly Asp Ala Asp Val Ala Val
35 40 45
Ala Pro Leu Val Val Gly Leu Thr Val Glu Ser Gly Phe Glu Ala Asn
50 55 60
Val Ala Val Val Val Gly Ser Arg Thr Thr Gly Leu Gly Gly Thr Ala
65 70 75 80
Val Ser Leu Lys Leu Thr Pro Ser His Tyr Ser Ser Ser Val Tyr Val
85 90 95
Phe His Gly Gly Arg His Leu Asp Pro Ser Thr Gln Ala Pro Asn Leu
100 105 110
Thr Arg Leu Cys Glu Arg Ala Arg Arg His Phe Gly Phe Ser Asp Tyr
115 120 125
Thr Pro Arg Pro Gly Asp Leu Lys His Glu Thr Thr Gly Glu Ala Leu
130 135 140
Cys Glu Arg Leu Gly Leu Asp Pro Asp Arg Ala Leu Leu Tyr Leu Val
145 150 155 160
Val Thr Glu Gly Phe Lys Glu Ala Val Cys Ile Asn Asn Thr Phe Leu
165 170 175
His Leu Gly Gly Ser Asp Lys Val Thr Ile Gly Gly Ala Glu Val His
180 185 190
Arg Ile Pro Val Tyr Pro Leu Gln Leu Phe Met Pro Asp Phe Ser Arg
195 200 205
Val Ile Ala Glu Pro Phe Asn Ala Asn His Arg Ser Ile Gly Glu Asn
210 215 220
Phe Thr Tyr Pro Leu Pro Phe Phe Asn Arg Pro Leu Asn Arg Leu Leu
225 230 235 240
Phe Glu Ala Val Val Gly Pro Ala Ala Val Ala Leu Arg Cys Arg Asn
245 250 255
Val Asp Ala Val Ala Arg Ala Ala Ala His Leu Ala Phe Asp Glu Asn
260 265 270
His Glu Gly Ala Ala Leu Pro Ala Asp Ile Thr Phe Thr Ala Phe Glu
275 280 285
Ala Ser Gln Gly Lys Thr Pro Arg Gly Gly Arg Asp Gly Gly Gly Lys
290 295 300
Gly Pro Ala Gly Gly Phe Glu Gln Arg Leu Ala Ser Val Met Ala Gly
305 310 315 320
Asp Ala Ala Leu Ala Leu Glu Ser Ile Val Ser Met Ala Val Phe Asp
325 330 335
Glu Pro Pro Thr Asp Ile Ser Ala Trp Pro Leu Cys Glu Gly Gln Asp
340 345 350
Thr Ala Ala Ala Arg Asp Asn Ala Val Gly Ala Tyr Leu Ala Arg Ala
355 360 365
Ala Gly Leu Val Gly Ala Met Val Phe Ser Thr Asn Ser Ala Leu His
370 375 380
Leu Thr Glu Val Asp Asp Ala Gly Pro Ala Asp Pro Lys Asp His Ser
385 390 395 400
Lys Pro Ser Phe Tyr Arg Phe Phe Leu Val Pro Gly Thr His Val Ala
405 410 415
Ala Asn Pro Gln Val Asp Arg Glu Gly His Val Val Pro Gly Phe Glu
420 425 430
Gly Arg Pro Thr Ala Pro Leu Val Gly Gly Thr Gln Glu Phe Ala Gly
435 440 445
Glu His Leu Ala Met Leu Cys Gly Phe Ser Pro Ala Leu Leu Ala Lys
450 455 460
Met Leu Phe Tyr Leu Glu Arg Cys Asp Gly Gly Val Ile Val Gly Arg
465 470 475 480
Gln Glu Met Asp Val Phe Arg Tyr Val Ala Asp Ser Asn Gln Thr Asp
485 490 495
Val Pro Cys Asn Leu Cys Thr Phe Asp Thr Arg His Ala Cys Val His
500 505 510
Thr Thr Leu Met Arg Leu Arg Ala Arg His Pro Lys Phe Ala Ser Ala
515 520 525
Ala Arg Gly Ala Ile Gly Val Phe Gly Thr Met Asn Ser Met Tyr Ser
530 535 540
Asp Cys Asp Val Leu Gly Asn Tyr Ala Ala Phe Ser Ala Leu Lys Arg
545 550 555 560
Ala Asp Gly Ser Glu Thr Ala Arg Thr Ile Met Gln Glu Thr Tyr Arg
565 570 575
Ala Ala Thr Glu Arg Val Met Ala Glu Leu Glu Thr Leu Gln Tyr Val
580 585 590
Asp Gln Ala Val Pro Thr Ala Met Gly Arg Leu Glu Thr Ile Ile Thr
595 600 605
Asn Arg Glu Ala Leu His Thr Val Val Asn Asn Val Arg Gln Val Val
610 615 620
Asp Arg Glu Val Glu Gln Leu Met Arg Asn Leu Val Glu Gly Arg Asn
625 630 635 640
Phe Lys Phe Arg Asp Gly Leu Gly Glu Ala Asn His Ala Met Ser Leu
645 650 655
Thr Leu Asp Pro Tyr Ala Cys Gly Pro Cys Pro Leu Leu Gln Leu Leu
660 665 670
Gly Arg Arg Ser Asn Leu Ala Val Tyr Gln Asp Leu Ala Leu Ser Gln
675 680 685
Cys His Gly Val Phe Ala Gly Gln Ser Val Glu Gly Arg Asn Phe Arg
690 695 700
Asn Gln Phe Gln Pro Val Leu Arg Arg Arg Val Met Asp Met Phe Asn
705 710 715 720
Asn Gly Phe Leu Ser Ala Lys Thr Leu Thr Val Ala Leu Ser Glu Gly
725 730 735
Ala Ala Ile Cys Ala Pro Ser Leu Thr Ala Gly Gln Thr Ala Pro Ala
740 745 750
Glu Ser Ser Phe Glu Gly Asp Val Ala Arg Val Thr Leu Gly Phe Pro
755 760 765
Lys Glu Leu Arg Val Lys Ser Arg Val Leu Phe Ala Gly Ala Ser Ala
770 775 780
Asn Ala Ser Glu Ala Ala Lys Ala Arg Val Ala Ser Leu Gln Ser Ala
785 790 795 800
Tyr Gln Lys Pro Asp Lys Arg Val Asp Ile Leu Leu Gly Pro Leu Gly
805 810 815
Phe Leu Leu Lys Gln Phe His Ala Ala Ile Phe Pro Asn Gly Lys Pro
820 825 830
Pro Gly Ser Asn Gln Pro Asn Pro Gln Trp Phe Trp Thr Ala Leu Gln
835 840 845
Arg Asn Gln Leu Pro Ala Arg Leu Leu Ser Arg Glu Asp Ile Glu Thr
850 855 860
Ile Ala Phe Ile Lys Lys Phe Ser Leu Asp Tyr Gly Ala Ile Asn Phe
865 870 875 880
Ile Asn Leu Ala Pro Asn Asn Val Ser Glu Leu Ala Met Tyr Tyr Met
885 890 895
Ala Asn Gln Ile Leu Arg Tyr Cys Asp His Ser Thr Tyr Phe Ile Asn
900 905 910
Thr Leu Thr Ala Ile Ile Ala Gly Ser Arg Arg Pro Pro Ser Val Gln
915 920 925
Ala Ala Ala Ala Trp Ser Ala Gln Gly Gly Ala Gly Leu Glu Ala Gly
930 935 940
Ala Arg Ala Leu Met Asp Ala Val Asp Ala His Pro Gly Ala Trp Thr
945 950 955 960
Ser Met Phe Ala Ser Cys Asn Leu Leu Arg Pro Val Met Ala Ala Arg
965 970 975
Pro Met Val Val Leu Gly Leu Ser Ile Ser Lys Tyr Tyr Gly Met Ala
980 985 990
Gly Asn Asp Arg Val Phe Gln Ala Gly Asn Trp Ala Ser Leu Met Gly
995 1000 1005
Gly Lys Asn Ala Cys Pro Leu Leu Ile Phe Asp Arg Thr Arg Lys
1010 1015 1020
Phe Val Leu Ala Cys Pro Arg Ala Gly Phe Val Cys Ala Ala Ser
1025 1030 1035
Asn Leu Gly Gly Gly Ala His Glu Ser Ser Leu Cys Glu Gln Leu
1040 1045 1050
Arg Gly Ile Ile Ser Glu Gly Gly Ala Ala Val Ala Ser Ser Val
1055 1060 1065
Phe Val Ala Thr Val Lys Ser Leu Gly Pro Arg Thr Gln Gln Leu
1070 1075 1080
Gln Ile Glu Asp Trp Leu Ala Leu Leu Glu Asp Glu Tyr Leu Ser
1085 1090 1095
Glu Glu Met Met Glu Leu Thr Ala Arg Ala Leu Glu Arg Gly Asn
1100 1105 1110
Gly Glu Trp Ser Thr Asp Ala Ala Leu Glu Val Ala His Glu Ala
1115 1120 1125
Glu Ala Leu Val Ser Gln Leu Gly Asn Ala Gly Glu Val Phe Asn
1130 1135 1140
Phe Gly Asp Phe Gly Cys Glu Asp Asp Asn Ala Thr Pro Phe Gly
1145 1150 1155
Gly Pro Gly Ala Pro Gly Pro Ala Phe Ala Gly Arg Lys Arg Ala
1160 1165 1170
Phe His Gly Asp Asp Pro Phe Gly Glu Gly Pro Pro Asp Lys Lys
1175 1180 1185
Gly Asp Leu Thr Leu Asp Met Leu
1190 1195
<210> 33
<211> 3591
<212> DNA
<213> Herpes simplex virus type 1
<400> 33
atggagacaa agcccaagac ggcaaccacc atcaaggtcc cccccgggcc cctgggatac 60
gtgtacgctc gcgcgtgtcc gtccgaaggc atcgagcttc tggcgttact gtcggcacgc 120
agcggcgatt ccgacgtcgc cgtggcgccc ctggtcgtgg gcctgaccgt ggagagcggc 180
tttgaggcca acgtggccgt ggtcgtgggt tctcgcacga cggggctcgg gggtaccgcg 240
gtgtccctga aactgacgcc ctcgcactac agctcgtccg tgtacgtctt tcacggcggc 300
cggcacctgg accccagcac ccaggccccg aacctgacgc gactttgcga gcgggcacgc 360
cgccattttg gcttttcgga ctacaccccc cggcccggcg acctcaaaca cgagacgacg 420
ggggaggcgc tgtgtgagcg cctcggcctg gacccggacc gcgccctcct gtatctggtc 480
gttaccgagg gcttcaagga ggccgtgtgc atcaacaaca cctttctgca cctgggaggc 540
tcggacaagg taaccatagg cggggcggag gtgcaccgca tacccgtgta cccgttgcag 600
ctgttcatgc cggattttag ccgtgtcatc gcagagccgt tcaacgccaa ccaccgatcg 660
atcggggaga attttaccta cccgcttccg ttttttaacc gccccctcaa ccgcctcctg 720
ttcgaggcgg tcgtgggacc cgccgccgtg gcactgcgat gccgaaacgt ggacgccgtg 780
gcccgcgcgg ccgcccacct ggcgtttgac gaaaaccacg agggcgccgc cctccccgcc 840
gacattacgt tcacggcctt cgaagccagc cagggtaaga ccccgcgggg cgggcgcgac 900
ggcggcggca agggcccggc gggcgggttc gaacagcgcc tggcctccgt catggccgga 960
gacgccgccc tggccctcga ttctatcgtg tcgatggccg tctttgacga gccgcccacc 1020
gacatctccg cgtggccgct gttcgagggc caggacacgg ccgcggcccg cgccaacgcc 1080
gtcggggcgt acctggcgcg cgccgcggga ctcgtggggg ccatggtatt tagcaccaac 1140
tcggccctcc atctcaccga ggtggacgac gccggcccgg cggacccaaa ggaccacagc 1200
aaaccctcct tttaccgctt cttcctcgtg cccgggaccc acgtggcggc caacccacag 1260
gtggaccgcg agggacacgt ggtgcccggg ttcgagggtc ggcccaccgc gcccctcgtc 1320
ggcggaaccc aggaatttgc cggcgagcac ctggccatgc tgtgtgggtt ttccccggcg 1380
ctgctggcca agatgctgtt ttacctggag cgctgcgacg gcggcgtgat cgtcgggcgc 1440
caggagatgg acgtgtttcg atacgtcgcg gactccaacc agaccgacgt gccctgtaac 1500
ctatgcacct tcgacacgcg ccacgcctgc gtacacacga cgctcatgcg cctccgggcg 1560
cgccatccaa agttcgccag cgccgcccgc ggagccatcg gcgtcttcgg gaccatgaac 1620
agcatgtaca gcgactgcga cgtgctggga aactacgccg ccttctcggc cctgaagcgc 1680
gcggacggat ccgagaccgc ccggaccatc atgcaggaga cgtaccgcgc ggcgaccgag 1740
cgcgtcatgg ccgaactcga gaccctgcag tacgtggacc aggcggtccc cacggccatg 1800
gggcggctgg agaccatcat caccaaccgc gaggccctgc atacggtggt gaacaacgtc 1860
aggcaggtcg tggaccgcga ggtggagcag ctgatgcgca acctggtgga ggggaggaac 1920
ttcaagtttc gcgacggtct gggcgaggcc aaccacgcca tgtccctgac gctggacccg 1980
tacgcgtgcg ggccgtgccc cctgcttcag cttctcgggc ggcgatccaa cctcgccgtg 2040
taccaggacc tggccctgag tcagtgccac ggggtgttcg ccgggcagtc ggtcgagggg 2100
cgcaactttc gcaatcaatt ccaaccggtg ctgcggcggc gcgtgatgga catgtttaac 2160
aacgggtttc tgtcggccaa aacgctgacg gtcgcgctct cggagggggc ggctatctgc 2220
gcccccagcc taacggccgg ccagacggcc cccgccgaga gcagcttcga gggcgacgtt 2280
gcccgcgtga ccctggggtt tcccaaggag ctgcgcgtca agagccgcgt gttgttcgcg 2340
ggcgcgagcg ccaacgcgtc cgaggccgcc aaggcgcggg tcgccagcct ccagagcgcc 2400
taccagaagc ccgacaagcg cgtggacatc ctcctcggac cgctgggctt tctgctgaag 2460
cagttccacg cggccatctt ccccaacggc aagcccccgg ggtccaacca gccgaacccg 2520
cagtggttct ggacggccct ccaacgcaac cagcttcccg cccggctcct gtcgcgcgag 2580
gacatcgaga ccatcgcgtt cattaaaaag ttttccctgg actacggcgc gataaacttt 2640
attaacctgg cccccaacaa cgtgagcgag ctggcgatgt actacatggc aaaccagatt 2700
ctgcggtact gcgatcactc gacatacttc atcaacaccc ttacggccat catcgcgggg 2760
tcccgccgtc cccccagcgt gcaggctgcg gccgcgtggt ccgcgcaggg cggggcgggc 2820
ctggaggccg gggcccgcgc gctgatggac gccgtggacg cgcatccggg cgcgtggacg 2880
tccatgttcg ccagctgcaa cctgctgcgg cccgtcatgg cggcgcgccc catggtcgtg 2940
ttggggttga gcatcagcaa gtactacggc atggccggca acgaccgtgt gtttcaggcc 3000
gggaactggg ccagcctgat gggcggcaaa aacgcgtgcc cgctccttat ttttgaccgc 3060
acccgcaagt tcgtcctggc ctgtccccgg gccgggtttg tgtgcgcggc ctcaagcctc 3120
ggcggcggag cgcacgaaag ctcgctgtgc gagcagctcc ggggcattat ctccgagggc 3180
ggggcggccg tcgccagtag cgtgttcgtg gcgaccgtga aaagcctggg gccccgcacc 3240
cagcagctgc agatcgagga ctggctggcg ctcctggagg acgagtacct aagcgaggag 3300
atgatggagc tgaccgcgcg tgccctggag cgcggcaacg gcgagtggtc gacggacgcg 3360
gccctggagg tggcgcacga ggccgaggcc ctagtcagcc aactcggcaa cgccggggag 3420
gtgtttaact ttggggattt tggctgcgag gacgacaacg cgacgccgtt cggcggcccg 3480
ggggccccgg gaccggcatt tgccggccgc aaacgggcgt tccacgggga tgacccgttt 3540
ggggaggggc cccccgacaa aaagggagac ctgacgttgg atatgctgtg a 3591
<210> 34
<211> 1196
<212> PRT
<213> Herpes simplex virus type 1
<400> 34
Met Glu Thr Lys Pro Lys Thr Ala Thr Thr Ile Lys Val Pro Pro Gly
1 5 10 15
Pro Leu Gly Tyr Val Tyr Ala Arg Ala Cys Pro Ser Glu Gly Ile Glu
20 25 30
Leu Leu Ala Leu Leu Ser Ala Arg Ser Gly Asp Ser Asp Val Ala Val
35 40 45
Ala Pro Leu Val Val Gly Leu Thr Val Glu Ser Gly Phe Glu Ala Asn
50 55 60
Val Ala Val Val Val Gly Ser Arg Thr Thr Gly Leu Gly Gly Thr Ala
65 70 75 80
Val Ser Leu Lys Leu Thr Pro Ser His Tyr Ser Ser Ser Val Tyr Val
85 90 95
Phe His Gly Gly Arg His Leu Asp Pro Ser Thr Gln Ala Pro Asn Leu
100 105 110
Thr Arg Leu Cys Glu Arg Ala Arg Arg His Phe Gly Phe Ser Asp Tyr
115 120 125
Thr Pro Arg Pro Gly Asp Leu Lys His Glu Thr Thr Gly Glu Ala Leu
130 135 140
Cys Glu Arg Leu Gly Leu Asp Pro Asp Arg Ala Leu Leu Tyr Leu Val
145 150 155 160
Val Thr Glu Gly Phe Lys Glu Ala Val Cys Ile Asn Asn Thr Phe Leu
165 170 175
His Leu Gly Gly Ser Asp Lys Val Thr Ile Gly Gly Ala Glu Val His
180 185 190
Arg Ile Pro Val Tyr Pro Leu Gln Leu Phe Met Pro Asp Phe Ser Arg
195 200 205
Val Ile Ala Glu Pro Phe Asn Ala Asn His Arg Ser Ile Gly Glu Asn
210 215 220
Phe Thr Tyr Pro Leu Pro Phe Phe Asn Arg Pro Leu Asn Arg Leu Leu
225 230 235 240
Phe Glu Ala Val Val Gly Pro Ala Ala Val Ala Leu Arg Cys Arg Asn
245 250 255
Val Asp Ala Val Ala Arg Ala Ala Ala His Leu Ala Phe Asp Glu Asn
260 265 270
His Glu Gly Ala Ala Leu Pro Ala Asp Ile Thr Phe Thr Ala Phe Glu
275 280 285
Ala Ser Gln Gly Lys Thr Pro Arg Gly Gly Arg Asp Gly Gly Gly Lys
290 295 300
Gly Pro Ala Gly Gly Phe Glu Gln Arg Leu Ala Ser Val Met Ala Gly
305 310 315 320
Asp Ala Ala Leu Ala Leu Asp Ser Ile Val Ser Met Ala Val Phe Asp
325 330 335
Glu Pro Pro Thr Asp Ile Ser Ala Trp Pro Leu Phe Glu Gly Gln Asp
340 345 350
Thr Ala Ala Ala Arg Ala Asn Ala Val Gly Ala Tyr Leu Ala Arg Ala
355 360 365
Ala Gly Leu Val Gly Ala Met Val Phe Ser Thr Asn Ser Ala Leu His
370 375 380
Leu Thr Glu Val Asp Asp Ala Gly Pro Ala Asp Pro Lys Asp His Ser
385 390 395 400
Lys Pro Ser Phe Tyr Arg Phe Phe Leu Val Pro Gly Thr His Val Ala
405 410 415
Ala Asn Pro Gln Val Asp Arg Glu Gly His Val Val Pro Gly Phe Glu
420 425 430
Gly Arg Pro Thr Ala Pro Leu Val Gly Gly Thr Gln Glu Phe Ala Gly
435 440 445
Glu His Leu Ala Met Leu Cys Gly Phe Ser Pro Ala Leu Leu Ala Lys
450 455 460
Met Leu Phe Tyr Leu Glu Arg Cys Asp Gly Gly Val Ile Val Gly Arg
465 470 475 480
Gln Glu Met Asp Val Phe Arg Tyr Val Ala Asp Ser Asn Gln Thr Asp
485 490 495
Val Pro Cys Asn Leu Cys Thr Phe Asp Thr Arg His Ala Cys Val His
500 505 510
Thr Thr Leu Met Arg Leu Arg Ala Arg His Pro Lys Phe Ala Ser Ala
515 520 525
Ala Arg Gly Ala Ile Gly Val Phe Gly Thr Met Asn Ser Met Tyr Ser
530 535 540
Asp Cys Asp Val Leu Gly Asn Tyr Ala Ala Phe Ser Ala Leu Lys Arg
545 550 555 560
Ala Asp Gly Ser Glu Thr Ala Arg Thr Ile Met Gln Glu Thr Tyr Arg
565 570 575
Ala Ala Thr Glu Arg Val Met Ala Glu Leu Glu Thr Leu Gln Tyr Val
580 585 590
Asp Gln Ala Val Pro Thr Ala Met Gly Arg Leu Glu Thr Ile Ile Thr
595 600 605
Asn Arg Glu Ala Leu His Thr Val Val Asn Asn Val Arg Gln Val Val
610 615 620
Asp Arg Glu Val Glu Gln Leu Met Arg Asn Leu Val Glu Gly Arg Asn
625 630 635 640
Phe Lys Phe Arg Asp Gly Leu Gly Glu Ala Asn His Ala Met Ser Leu
645 650 655
Thr Leu Asp Pro Tyr Ala Cys Gly Pro Cys Pro Leu Leu Gln Leu Leu
660 665 670
Gly Arg Arg Ser Asn Leu Ala Val Tyr Gln Asp Leu Ala Leu Ser Gln
675 680 685
Cys His Gly Val Phe Ala Gly Gln Ser Val Glu Gly Arg Asn Phe Arg
690 695 700
Asn Gln Phe Gln Pro Val Leu Arg Arg Arg Val Met Asp Met Phe Asn
705 710 715 720
Asn Gly Phe Leu Ser Ala Lys Thr Leu Thr Val Ala Leu Ser Glu Gly
725 730 735
Ala Ala Ile Cys Ala Pro Ser Leu Thr Ala Gly Gln Thr Ala Pro Ala
740 745 750
Glu Ser Ser Phe Glu Gly Asp Val Ala Arg Val Thr Leu Gly Phe Pro
755 760 765
Lys Glu Leu Arg Val Lys Ser Arg Val Leu Phe Ala Gly Ala Ser Ala
770 775 780
Asn Ala Ser Glu Ala Ala Lys Ala Arg Val Ala Ser Leu Gln Ser Ala
785 790 795 800
Tyr Gln Lys Pro Asp Lys Arg Val Asp Ile Leu Leu Gly Pro Leu Gly
805 810 815
Phe Leu Leu Lys Gln Phe His Ala Ala Ile Phe Pro Asn Gly Lys Pro
820 825 830
Pro Gly Ser Asn Gln Pro Asn Pro Gln Trp Phe Trp Thr Ala Leu Gln
835 840 845
Arg Asn Gln Leu Pro Ala Arg Leu Leu Ser Arg Glu Asp Ile Glu Thr
850 855 860
Ile Ala Phe Ile Lys Lys Phe Ser Leu Asp Tyr Gly Ala Ile Asn Phe
865 870 875 880
Ile Asn Leu Ala Pro Asn Asn Val Ser Glu Leu Ala Met Tyr Tyr Met
885 890 895
Ala Asn Gln Ile Leu Arg Tyr Cys Asp His Ser Thr Tyr Phe Ile Asn
900 905 910
Thr Leu Thr Ala Ile Ile Ala Gly Ser Arg Arg Pro Pro Ser Val Gln
915 920 925
Ala Ala Ala Ala Trp Ser Ala Gln Gly Gly Ala Gly Leu Glu Ala Gly
930 935 940
Ala Arg Ala Leu Met Asp Ala Val Asp Ala His Pro Gly Ala Trp Thr
945 950 955 960
Ser Met Phe Ala Ser Cys Asn Leu Leu Arg Pro Val Met Ala Ala Arg
965 970 975
Pro Met Val Val Leu Gly Leu Ser Ile Ser Lys Tyr Tyr Gly Met Ala
980 985 990
Gly Asn Asp Arg Val Phe Gln Ala Gly Asn Trp Ala Ser Leu Met Gly
995 1000 1005
Gly Lys Asn Ala Cys Pro Leu Leu Ile Phe Asp Arg Thr Arg Lys
1010 1015 1020
Phe Val Leu Ala Cys Pro Arg Ala Gly Phe Val Cys Ala Ala Ser
1025 1030 1035
Ser Leu Gly Gly Gly Ala His Glu Ser Ser Leu Cys Glu Gln Leu
1040 1045 1050
Arg Gly Ile Ile Ser Glu Gly Gly Ala Ala Val Ala Ser Ser Val
1055 1060 1065
Phe Val Ala Thr Val Lys Ser Leu Gly Pro Arg Thr Gln Gln Leu
1070 1075 1080
Gln Ile Glu Asp Trp Leu Ala Leu Leu Glu Asp Glu Tyr Leu Ser
1085 1090 1095
Glu Glu Met Met Glu Leu Thr Ala Arg Ala Leu Glu Arg Gly Asn
1100 1105 1110
Gly Glu Trp Ser Thr Asp Ala Ala Leu Glu Val Ala His Glu Ala
1115 1120 1125
Glu Ala Leu Val Ser Gln Leu Gly Asn Ala Gly Glu Val Phe Asn
1130 1135 1140
Phe Gly Asp Phe Gly Cys Glu Asp Asp Asn Ala Thr Pro Phe Gly
1145 1150 1155
Gly Pro Gly Ala Pro Gly Pro Ala Phe Ala Gly Arg Lys Arg Ala
1160 1165 1170
Phe His Gly Asp Asp Pro Phe Gly Glu Gly Pro Pro Asp Lys Lys
1175 1180 1185
Gly Asp Leu Thr Leu Asp Met Leu
1190 1195
<210> 35
<211> 2208
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<220>
<221> mutation
<222> (1126)..(1126)
<223> g1126a
<400> 35
atgtttggtc agcagctggc gtccgacgtc cagcagtacc tggagcgcct cgagaaacag 60
aggcaactta aggtgggcgc ggacgaggcg tcggcgggcc tcaccatggg cggcgatgcc 120
ctacgagtgc cctttttaga tttcgcgacc gcgaccccca agcgccacca gaccgtggtc 180
cctggcgtcg ggacgctcca cgactgctgc gagcactcgc cgctcttctc ggccgtggcg 240
cggcggctgc tgtttaatag cctggtgccg gcgcaactaa aggggcgtga tttcgggggc 300
gaccacacgg ccaagctgga attcctggcc cccgagttgg tacgggcggt ggcgcgactg 360
cggtttaagg agtgcgcgcc ggcggacgtg gtgcctcagc gtaacgccta ctatagcgtt 420
ctgaatacgt ttcaggccct ccaccgctcc gaagcctttc gccagctggt gcactttgtg 480
cgggactttg cccagctgct caaaacctcc ttccgggcct ccagcctcac ggagaccacg 540
ggccccccca aaaaacgggc caaggtggac gtggccaccc acggccggac gtacggcacg 600
ctggagctgt tccaaaaaat gatccttatg cacgccacct actttctggc cgccgtgctc 660
ctcggggacc acgcggagca ggtcaacacg ttcctgcgtc tcgtgtttga gatccccctg 720
tttagcgacg cggccgtgcg ccacttccgc cagcgcgcca ccgtgtttct cgtcccccgg 780
cgccacggca agacctggtt tctggtgccc ctcatcgcgc tgtcgctggc ctcctttcgg 840
gggatcaaga tcggctacac ggcgcacatc cgcaaggcga ccgagccggt gtttgaggag 900
atcgacgcct gcctgcgggg ctggttcggt tcggcccgag tggaccacgt taaaggggaa 960
accatctcct tctcgtttcc ggacgggtcg cgcagtacca tcgtgtttgc ctccagccac 1020
aacacaaacg gaatccgagg ccaggacttt aacctgctct ttgtcgacga ggccaacttt 1080
attcgcccgg atgcggtcca gacgattatg ggctttctca accagaccaa ctgcaagatt 1140
atcttcgtgt cgtccaccaa caccgggaag gccagtacga gctttttgta caacctccgc 1200
ggggccgcag acgagcttct caacgtggtg acctatatat gcgatgatca catgccgagg 1260
gtggtgacgc acacaaacgc cacggcctgt tcttgttata tcctcaacaa gcccgttttc 1320
atcacgatgg acggggcggt tcgccggacc gccgatttgt ttctggccga ttccttcatg 1380
caggagatca tcgggggcca ggccagggag accggcgacg accggcccgt tctgaccaag 1440
tctgcggggg agcggtttct gttgtaccgc ccctcgacca ccaccaacag cggcctcatg 1500
gcccccgatt tgtacgtgta cgtggatccc gcgttcacgg ccaacacccg agcctccggg 1560
accggcgtcg ctgtcgtcgg gcggtaccgc gacgattata tcatcttcgc cctggagcac 1620
ttttttctcc gcgcgctcac gggctcggcc cccgccgaca tcgcccgctg cgtcgtccac 1680
agtctgacgc aggtcctggc cctgcatccc ggggcgtttc gcggcgtccg ggtggcggtc 1740
gagggaaata gcagccagga ctcggccgtc gccatcgcca cgcacgtgca cacagagatg 1800
caccgcctac tggcctcgga gggggccgac gcgggctcgg gccccgagct tctcttctac 1860
cactgcgagc ctcccgggag cgcggtgctg tacccctttt tcctgctcaa caaacagaag 1920
acgcccgcct ttgaacactt tattaaaaag tttaactccg ggggcgtcat ggcctcccag 1980
gagatcgttt ccgcgacggt gcgcctgcag accgacccgg tcgagtatct gctcgagcag 2040
ctaaataacc tcaccgaaac cgtctccccc aacactgacg tccgtacgta ttccggaaaa 2100
cggaacggcg cctcggatga ccttatggtc gccgtcatta tggccatcta cctcgcggcc 2160
caggccggac ctccgcacac attcgctcct atcatacgcg tctcgtga 2208
<210> 36
<211> 735
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<220>
<221> MISC_FEATURE
<222> (376)..(376)
<223> A376T
<400> 36
Met Phe Gly Gln Gln Leu Ala Ser Asp Val Gln Gln Tyr Leu Glu Arg
1 5 10 15
Leu Glu Lys Gln Arg Gln Leu Lys Val Gly Ala Asp Glu Ala Ser Ala
20 25 30
Gly Leu Thr Met Gly Gly Asp Ala Leu Arg Val Pro Phe Leu Asp Phe
35 40 45
Ala Thr Ala Thr Pro Lys Arg His Gln Thr Val Val Pro Gly Val Gly
50 55 60
Thr Leu His Asp Cys Cys Glu His Ser Pro Leu Phe Ser Ala Val Ala
65 70 75 80
Arg Arg Leu Leu Phe Asn Ser Leu Val Pro Ala Gln Leu Lys Gly Arg
85 90 95
Asp Phe Gly Gly Asp His Thr Ala Lys Leu Glu Phe Leu Ala Pro Glu
100 105 110
Leu Val Arg Ala Val Ala Arg Leu Arg Phe Lys Glu Cys Ala Pro Ala
115 120 125
Asp Val Val Pro Gln Arg Asn Ala Tyr Tyr Ser Val Leu Asn Thr Phe
130 135 140
Gln Ala Leu His Arg Ser Glu Ala Phe Arg Gln Leu Val His Phe Val
145 150 155 160
Arg Asp Phe Ala Gln Leu Leu Lys Thr Ser Phe Arg Ala Ser Ser Leu
165 170 175
Thr Glu Thr Thr Gly Pro Pro Lys Lys Arg Ala Lys Val Asp Val Ala
180 185 190
Thr His Gly Arg Thr Tyr Gly Thr Leu Glu Leu Phe Gln Lys Met Ile
195 200 205
Leu Met His Ala Thr Tyr Phe Leu Ala Ala Val Leu Leu Gly Asp His
210 215 220
Ala Glu Gln Val Asn Thr Phe Leu Arg Leu Val Phe Glu Ile Pro Leu
225 230 235 240
Phe Ser Asp Ala Ala Val Arg His Phe Arg Gln Arg Ala Thr Val Phe
245 250 255
Leu Val Pro Arg Arg His Gly Lys Thr Trp Phe Leu Val Pro Leu Ile
260 265 270
Ala Leu Ser Leu Ala Ser Phe Arg Gly Ile Lys Ile Gly Tyr Thr Ala
275 280 285
His Ile Arg Lys Ala Thr Glu Pro Val Phe Glu Glu Ile Asp Ala Cys
290 295 300
Leu Arg Gly Trp Phe Gly Ser Ala Arg Val Asp His Val Lys Gly Glu
305 310 315 320
Thr Ile Ser Phe Ser Phe Pro Asp Gly Ser Arg Ser Thr Ile Val Phe
325 330 335
Ala Ser Ser His Asn Thr Asn Gly Ile Arg Gly Gln Asp Phe Asn Leu
340 345 350
Leu Phe Val Asp Glu Ala Asn Phe Ile Arg Pro Asp Ala Val Gln Thr
355 360 365
Ile Met Gly Phe Leu Asn Gln Thr Asn Cys Lys Ile Ile Phe Val Ser
370 375 380
Ser Thr Asn Thr Gly Lys Ala Ser Thr Ser Phe Leu Tyr Asn Leu Arg
385 390 395 400
Gly Ala Ala Asp Glu Leu Leu Asn Val Val Thr Tyr Ile Cys Asp Asp
405 410 415
His Met Pro Arg Val Val Thr His Thr Asn Ala Thr Ala Cys Ser Cys
420 425 430
Tyr Ile Leu Asn Lys Pro Val Phe Ile Thr Met Asp Gly Ala Val Arg
435 440 445
Arg Thr Ala Asp Leu Phe Leu Ala Asp Ser Phe Met Gln Glu Ile Ile
450 455 460
Gly Gly Gln Ala Arg Glu Thr Gly Asp Asp Arg Pro Val Leu Thr Lys
465 470 475 480
Ser Ala Gly Glu Arg Phe Leu Leu Tyr Arg Pro Ser Thr Thr Thr Asn
485 490 495
Ser Gly Leu Met Ala Pro Asp Leu Tyr Val Tyr Val Asp Pro Ala Phe
500 505 510
Thr Ala Asn Thr Arg Ala Ser Gly Thr Gly Val Ala Val Val Gly Arg
515 520 525
Tyr Arg Asp Asp Tyr Ile Ile Phe Ala Leu Glu His Phe Phe Leu Arg
530 535 540
Ala Leu Thr Gly Ser Ala Pro Ala Asp Ile Ala Arg Cys Val Val His
545 550 555 560
Ser Leu Thr Gln Val Leu Ala Leu His Pro Gly Ala Phe Arg Gly Val
565 570 575
Arg Val Ala Val Glu Gly Asn Ser Ser Gln Asp Ser Ala Val Ala Ile
580 585 590
Ala Thr His Val His Thr Glu Met His Arg Leu Leu Ala Ser Glu Gly
595 600 605
Ala Asp Ala Gly Ser Gly Pro Glu Leu Leu Phe Tyr His Cys Glu Pro
610 615 620
Pro Gly Ser Ala Val Leu Tyr Pro Phe Phe Leu Leu Asn Lys Gln Lys
625 630 635 640
Thr Pro Ala Phe Glu His Phe Ile Lys Lys Phe Asn Ser Gly Gly Val
645 650 655
Met Ala Ser Gln Glu Ile Val Ser Ala Thr Val Arg Leu Gln Thr Asp
660 665 670
Pro Val Glu Tyr Leu Leu Glu Gln Leu Asn Asn Leu Thr Glu Thr Val
675 680 685
Ser Pro Asn Thr Asp Val Arg Thr Tyr Ser Gly Lys Arg Asn Gly Ala
690 695 700
Ser Asp Asp Leu Met Val Ala Val Ile Met Ala Ile Tyr Leu Ala Ala
705 710 715 720
Gln Ala Gly Pro Pro His Thr Phe Ala Pro Ile Ile Arg Val Ser
725 730 735
<210> 37
<211> 2208
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 37
atgtttggtc agcagctggc gtccgacgtc cagcagtacc tggagcgcct cgagaaacag 60
aggcaactta aggtgggcgc ggacgaggcg tcggcgggcc tcaccatggg cggcgatgcc 120
ctacgagtgc cctttttaga tttcgcgacc gcgaccccca agcgccacca gaccgtggtc 180
cctggcgtcg ggacgctcca cgactgctgc gagcactcgc cgctcttctc ggccgtggcg 240
cggcggctgc tgtttaatag cctggtgccg gcgcaactaa aggggcgtga tttcgggggc 300
gaccacacgg ccaagctgga attcctggcc cccgagttgg tacgggcggt ggcgcgactg 360
cggtttaagg agtgcgcgcc ggcggacgtg gtgcctcagc gtaacgccta ctatagcgtt 420
ctgaatacgt ttcaggccct ccaccgctcc gaagcctttc gccagctggt gcactttgtg 480
cgggactttg cccagctgct caaaacctcc ttccgggcct ccagcctcac ggagaccacg 540
ggccccccca aaaaacgggc caaggtggac gtggccaccc acggccggac gtacggcacg 600
ctggagctgt tccaaaaaat gatccttatg cacgccacct actttctggc cgccgtgctc 660
ctcggggacc acgcggagca ggtcaacacg ttcctgcgtc tcgtgtttga gatccccctg 720
tttagcgacg cggccgtgcg ccacttccgc cagcgcgcca ccgtgtttct cgtcccccgg 780
cgccacggca agacctggtt tctggtgccc ctcatcgcgc tgtcgctggc ctcctttcgg 840
gggatcaaga tcggctacac ggcgcacatc cgcaaggcga ccgagccggt gtttgaggag 900
atcgacgcct gcctgcgggg ctggttcggt tcggcccgag tggaccacgt taaaggggaa 960
accatctcct tctcgtttcc ggacgggtcg cgcagtacca tcgtgtttgc ctccagccac 1020
aacacaaacg gaatccgagg ccaggacttt aacctgctct ttgtcgacga ggccaacttt 1080
attcgcccgg atgcggtcca gacgattatg ggctttctca accaggccaa ctgcaagatt 1140
atcttcgtgt cgtccaccaa caccgggaag gccagtacga gctttttgta caacctccgc 1200
ggggccgcag acgagcttct caacgtggtg acctatatat gcgatgatca catgccgagg 1260
gtggtgacgc acacaaacgc cacggcctgt tcttgttata tcctcaacaa gcccgttttc 1320
atcacgatgg acggggcggt tcgccggacc gccgatttgt ttctggccga ttccttcatg 1380
caggagatca tcgggggcca ggccagggag accggcgacg accggcccgt tctgaccaag 1440
tctgcggggg agcggtttct gttgtaccgc ccctcgacca ccaccaacag cggcctcatg 1500
gcccccgatt tgtacgtgta cgtggatccc gcgttcacgg ccaacacccg agcctccggg 1560
accggcgtcg ctgtcgtcgg gcggtaccgc gacgattata tcatcttcgc cctggagcac 1620
ttttttctcc gcgcgctcac gggctcggcc cccgccgaca tcgcccgctg cgtcgtccac 1680
agtctgacgc aggtcctggc cctgcatccc ggggcgtttc gcggcgtccg ggtggcggtc 1740
gagggaaata gcagccagga ctcggccgtc gccatcgcca cgcacgtgca cacagagatg 1800
caccgcctac tggcctcgga gggggccgac gcgggctcgg gccccgagct tctcttctac 1860
cactgcgagc ctcccgggag cgcggtgctg tacccctttt tcctgctcaa caaacagaag 1920
acgcccgcct ttgaacactt tattaaaaag tttaactccg ggggcgtcat ggcctcccag 1980
gagatcgttt ccgcgacggt gcgcctgcag accgacccgg tcgagtatct gctcgagcag 2040
ctaaataacc tcaccgaaac cgtctccccc aacactgacg tccgtacgta ttccggaaaa 2100
cggaacggcg cctcggatga ccttatggtc gccgtcatta tggccatcta cctcgcggcc 2160
caggccggac ctccgcacac attcgctcct atcatacgcg tctcgtga 2208
<210> 38
<211> 735
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 38
Met Phe Gly Gln Gln Leu Ala Ser Asp Val Gln Gln Tyr Leu Glu Arg
1 5 10 15
Leu Glu Lys Gln Arg Gln Leu Lys Val Gly Ala Asp Glu Ala Ser Ala
20 25 30
Gly Leu Thr Met Gly Gly Asp Ala Leu Arg Val Pro Phe Leu Asp Phe
35 40 45
Ala Thr Ala Thr Pro Lys Arg His Gln Thr Val Val Pro Gly Val Gly
50 55 60
Thr Leu His Asp Cys Cys Glu His Ser Pro Leu Phe Ser Ala Val Ala
65 70 75 80
Arg Arg Leu Leu Phe Asn Ser Leu Val Pro Ala Gln Leu Lys Gly Arg
85 90 95
Asp Phe Gly Gly Asp His Thr Ala Lys Leu Glu Phe Leu Ala Pro Glu
100 105 110
Leu Val Arg Ala Val Ala Arg Leu Arg Phe Lys Glu Cys Ala Pro Ala
115 120 125
Asp Val Val Pro Gln Arg Asn Ala Tyr Tyr Ser Val Leu Asn Thr Phe
130 135 140
Gln Ala Leu His Arg Ser Glu Ala Phe Arg Gln Leu Val His Phe Val
145 150 155 160
Arg Asp Phe Ala Gln Leu Leu Lys Thr Ser Phe Arg Ala Ser Ser Leu
165 170 175
Thr Glu Thr Thr Gly Pro Pro Lys Lys Arg Ala Lys Val Asp Val Ala
180 185 190
Thr His Gly Arg Thr Tyr Gly Thr Leu Glu Leu Phe Gln Lys Met Ile
195 200 205
Leu Met His Ala Thr Tyr Phe Leu Ala Ala Val Leu Leu Gly Asp His
210 215 220
Ala Glu Gln Val Asn Thr Phe Leu Arg Leu Val Phe Glu Ile Pro Leu
225 230 235 240
Phe Ser Asp Ala Ala Val Arg His Phe Arg Gln Arg Ala Thr Val Phe
245 250 255
Leu Val Pro Arg Arg His Gly Lys Thr Trp Phe Leu Val Pro Leu Ile
260 265 270
Ala Leu Ser Leu Ala Ser Phe Arg Gly Ile Lys Ile Gly Tyr Thr Ala
275 280 285
His Ile Arg Lys Ala Thr Glu Pro Val Phe Glu Glu Ile Asp Ala Cys
290 295 300
Leu Arg Gly Trp Phe Gly Ser Ala Arg Val Asp His Val Lys Gly Glu
305 310 315 320
Thr Ile Ser Phe Ser Phe Pro Asp Gly Ser Arg Ser Thr Ile Val Phe
325 330 335
Ala Ser Ser His Asn Thr Asn Gly Ile Arg Gly Gln Asp Phe Asn Leu
340 345 350
Leu Phe Val Asp Glu Ala Asn Phe Ile Arg Pro Asp Ala Val Gln Thr
355 360 365
Ile Met Gly Phe Leu Asn Gln Ala Asn Cys Lys Ile Ile Phe Val Ser
370 375 380
Ser Thr Asn Thr Gly Lys Ala Ser Thr Ser Phe Leu Tyr Asn Leu Arg
385 390 395 400
Gly Ala Ala Asp Glu Leu Leu Asn Val Val Thr Tyr Ile Cys Asp Asp
405 410 415
His Met Pro Arg Val Val Thr His Thr Asn Ala Thr Ala Cys Ser Cys
420 425 430
Tyr Ile Leu Asn Lys Pro Val Phe Ile Thr Met Asp Gly Ala Val Arg
435 440 445
Arg Thr Ala Asp Leu Phe Leu Ala Asp Ser Phe Met Gln Glu Ile Ile
450 455 460
Gly Gly Gln Ala Arg Glu Thr Gly Asp Asp Arg Pro Val Leu Thr Lys
465 470 475 480
Ser Ala Gly Glu Arg Phe Leu Leu Tyr Arg Pro Ser Thr Thr Thr Asn
485 490 495
Ser Gly Leu Met Ala Pro Asp Leu Tyr Val Tyr Val Asp Pro Ala Phe
500 505 510
Thr Ala Asn Thr Arg Ala Ser Gly Thr Gly Val Ala Val Val Gly Arg
515 520 525
Tyr Arg Asp Asp Tyr Ile Ile Phe Ala Leu Glu His Phe Phe Leu Arg
530 535 540
Ala Leu Thr Gly Ser Ala Pro Ala Asp Ile Ala Arg Cys Val Val His
545 550 555 560
Ser Leu Thr Gln Val Leu Ala Leu His Pro Gly Ala Phe Arg Gly Val
565 570 575
Arg Val Ala Val Glu Gly Asn Ser Ser Gln Asp Ser Ala Val Ala Ile
580 585 590
Ala Thr His Val His Thr Glu Met His Arg Leu Leu Ala Ser Glu Gly
595 600 605
Ala Asp Ala Gly Ser Gly Pro Glu Leu Leu Phe Tyr His Cys Glu Pro
610 615 620
Pro Gly Ser Ala Val Leu Tyr Pro Phe Phe Leu Leu Asn Lys Gln Lys
625 630 635 640
Thr Pro Ala Phe Glu His Phe Ile Lys Lys Phe Asn Ser Gly Gly Val
645 650 655
Met Ala Ser Gln Glu Ile Val Ser Ala Thr Val Arg Leu Gln Thr Asp
660 665 670
Pro Val Glu Tyr Leu Leu Glu Gln Leu Asn Asn Leu Thr Glu Thr Val
675 680 685
Ser Pro Asn Thr Asp Val Arg Thr Tyr Ser Gly Lys Arg Asn Gly Ala
690 695 700
Ser Asp Asp Leu Met Val Ala Val Ile Met Ala Ile Tyr Leu Ala Ala
705 710 715 720
Gln Ala Gly Pro Pro His Thr Phe Ala Pro Ile Ile Arg Val Ser
725 730 735
<210> 39
<211> 2208
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 39
atgtttggtc agcagctggc gtccgacgtc cagcagtacc tggagcgcct cgagaaacag 60
aggcaactta aggtgggcgc ggacgaggcg tcggcgggcc tcacaatggg cggcgatgcc 120
ctacgagtgc cctttttaga tttcgcgacc gcgaccccca agcgccacca gaccgtggtc 180
ccgggcgtcg ggacgctcca cgactgctgc gagcactcgc cgctcttctc ggccgtggcg 240
cggcggctgc tgtttaatag cctggtgccg gcgcaactaa aggggcgtga tttcgggggc 300
gaccacacgg ccaagctgga attcctggcc cccgagttgg tacgggcggt ggcgcgactg 360
cggtttaagg agtgcgcgcc ggcggacgtg gtgcctcagc gtaacgccta ctatagcgtt 420
ctgaacacgt ttcaggccct ccaccgctcc gaagcctttc gccagctggt gcactttgtg 480
cgggactttg cccagctgct taaaacctcc ttccgggcct ccagcctcac ggagaccacg 540
ggccccccaa aaaaacgggc caaggtggac gtggccaccc acggccggac gtacggcacg 600
ctggagctgt tccaaaaaat gatccttatg cacgccacct actttctggc cgccgtgctc 660
ctcggggacc acgcggagca ggtcaacacg ttcctgcgtc tcgtgtttga gatccccctg 720
tttagcgacg cggccgtgcg ccacttccgc cagcgcgcca ccgtgtttct cgtcccccgg 780
cgccacggca agacctggtt tctagtgccc ctcatcgcgc tgtcgctggc ctcctttcgg 840
gggatcaaga tcggctacac ggcgcacatc cgcaaggcga ccgagccggt gtttgaggag 900
atcgacgcct gcctgcgggg ctggttcggt tcggcccgag tggaccacgt taaaggggaa 960
accatctcct tctcgtttcc ggacgggtcg cgcagtacca tcgtgtttgc ctccagccac 1020
aacacaaacg gaatccgagg ccaggacttt aacctgctct ttgtcgacga ggccaacttt 1080
attcgcccgg atgcggtcca gacgattatg ggctttctca accaggccaa ctgcaagatt 1140
atcttcgtgt cgtccaccaa caccgggaag gccagtacga gctttttgta caacctccgc 1200
ggggccgccg acgagcttct caacgtggtg acctatatat gcgatgatca catgccgcgg 1260
gtggtgacgc acacaaacgc cacggcctgt tcttgttata tcctcaacaa gcccgttttc 1320
atcacgatgg acggggcggt tcgccggacc gccgatttgt ttctggccga ttccttcatg 1380
caggagatca tcgggggcca ggccagggag accggcgacg accggcccgt tctgaccaag 1440
tctgcggggg agcggtttct gttgtaccgc ccctcgacca ccaccaacag cggcctcatg 1500
gcccccgatt tgtacgtgta cgtggatccc gcgttcacgg ccaacacccg agcctccggg 1560
accggcgtcg ctgtcgtcgg gcggtaccgc gacgattata tcatcttcgc cctggagcac 1620
ttttttctcc gcgcgctcac gggctcggcc cccgccgaca tcgcccgctg cgtcgtccac 1680
agtctgacgc aggtcctggc cctgcatccc ggggcgtttc gcggcgtccg ggtggcggtc 1740
gagggaaata gcagccagga ctcggccgtc gccatcgcca cgcacgtgca cacagagatg 1800
caccgcctac tggcctcgga gggggccgac gcgggctcgg gccccgagct tctcttctac 1860
cactgcgagc ctcccgggag cgcggtgctg tacccctttt tcctgctcaa caaacagaag 1920
acgcccgcct ttgaacactt tattaaaaag tttaactccg ggggcgtcat ggcctcccag 1980
gagatcgttt ccgcgacggt gcgcctgcag accgacccgg tcgagtatct gctcgagcag 2040
ctgaataacc tcaccgaaac cgtctccccc aacacggacg tccgtacgta ttccggaaaa 2100
cggaacggcg cctcggatga ccttatggtc gccgtcatta tggccatcta ccttgcggcc 2160
caggccggac ctccgcacac attcgctccc atcacacgcg tttcgtga 2208
<210> 40
<211> 735
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 40
Met Phe Gly Gln Gln Leu Ala Ser Asp Val Gln Gln Tyr Leu Glu Arg
1 5 10 15
Leu Glu Lys Gln Arg Gln Leu Lys Val Gly Ala Asp Glu Ala Ser Ala
20 25 30
Gly Leu Thr Met Gly Gly Asp Ala Leu Arg Val Pro Phe Leu Asp Phe
35 40 45
Ala Thr Ala Thr Pro Lys Arg His Gln Thr Val Val Pro Gly Val Gly
50 55 60
Thr Leu His Asp Cys Cys Glu His Ser Pro Leu Phe Ser Ala Val Ala
65 70 75 80
Arg Arg Leu Leu Phe Asn Ser Leu Val Pro Ala Gln Leu Lys Gly Arg
85 90 95
Asp Phe Gly Gly Asp His Thr Ala Lys Leu Glu Phe Leu Ala Pro Glu
100 105 110
Leu Val Arg Ala Val Ala Arg Leu Arg Phe Lys Glu Cys Ala Pro Ala
115 120 125
Asp Val Val Pro Gln Arg Asn Ala Tyr Tyr Ser Val Leu Asn Thr Phe
130 135 140
Gln Ala Leu His Arg Ser Glu Ala Phe Arg Gln Leu Val His Phe Val
145 150 155 160
Arg Asp Phe Ala Gln Leu Leu Lys Thr Ser Phe Arg Ala Ser Ser Leu
165 170 175
Thr Glu Thr Thr Gly Pro Pro Lys Lys Arg Ala Lys Val Asp Val Ala
180 185 190
Thr His Gly Arg Thr Tyr Gly Thr Leu Glu Leu Phe Gln Lys Met Ile
195 200 205
Leu Met His Ala Thr Tyr Phe Leu Ala Ala Val Leu Leu Gly Asp His
210 215 220
Ala Glu Gln Val Asn Thr Phe Leu Arg Leu Val Phe Glu Ile Pro Leu
225 230 235 240
Phe Ser Asp Ala Ala Val Arg His Phe Arg Gln Arg Ala Thr Val Phe
245 250 255
Leu Val Pro Arg Arg His Gly Lys Thr Trp Phe Leu Val Pro Leu Ile
260 265 270
Ala Leu Ser Leu Ala Ser Phe Arg Gly Ile Lys Ile Gly Tyr Thr Ala
275 280 285
His Ile Arg Lys Ala Thr Glu Pro Val Phe Glu Glu Ile Asp Ala Cys
290 295 300
Leu Arg Gly Trp Phe Gly Ser Ala Arg Val Asp His Val Lys Gly Glu
305 310 315 320
Thr Ile Ser Phe Ser Phe Pro Asp Gly Ser Arg Ser Thr Ile Val Phe
325 330 335
Ala Ser Ser His Asn Thr Asn Gly Ile Arg Gly Gln Asp Phe Asn Leu
340 345 350
Leu Phe Val Asp Glu Ala Asn Phe Ile Arg Pro Asp Ala Val Gln Thr
355 360 365
Ile Met Gly Phe Leu Asn Gln Ala Asn Cys Lys Ile Ile Phe Val Ser
370 375 380
Ser Thr Asn Thr Gly Lys Ala Ser Thr Ser Phe Leu Tyr Asn Leu Arg
385 390 395 400
Gly Ala Ala Asp Glu Leu Leu Asn Val Val Thr Tyr Ile Cys Asp Asp
405 410 415
His Met Pro Arg Val Val Thr His Thr Asn Ala Thr Ala Cys Ser Cys
420 425 430
Tyr Ile Leu Asn Lys Pro Val Phe Ile Thr Met Asp Gly Ala Val Arg
435 440 445
Arg Thr Ala Asp Leu Phe Leu Ala Asp Ser Phe Met Gln Glu Ile Ile
450 455 460
Gly Gly Gln Ala Arg Glu Thr Gly Asp Asp Arg Pro Val Leu Thr Lys
465 470 475 480
Ser Ala Gly Glu Arg Phe Leu Leu Tyr Arg Pro Ser Thr Thr Thr Asn
485 490 495
Ser Gly Leu Met Ala Pro Asp Leu Tyr Val Tyr Val Asp Pro Ala Phe
500 505 510
Thr Ala Asn Thr Arg Ala Ser Gly Thr Gly Val Ala Val Val Gly Arg
515 520 525
Tyr Arg Asp Asp Tyr Ile Ile Phe Ala Leu Glu His Phe Phe Leu Arg
530 535 540
Ala Leu Thr Gly Ser Ala Pro Ala Asp Ile Ala Arg Cys Val Val His
545 550 555 560
Ser Leu Thr Gln Val Leu Ala Leu His Pro Gly Ala Phe Arg Gly Val
565 570 575
Arg Val Ala Val Glu Gly Asn Ser Ser Gln Asp Ser Ala Val Ala Ile
580 585 590
Ala Thr His Val His Thr Glu Met His Arg Leu Leu Ala Ser Glu Gly
595 600 605
Ala Asp Ala Gly Ser Gly Pro Glu Leu Leu Phe Tyr His Cys Glu Pro
610 615 620
Pro Gly Ser Ala Val Leu Tyr Pro Phe Phe Leu Leu Asn Lys Gln Lys
625 630 635 640
Thr Pro Ala Phe Glu His Phe Ile Lys Lys Phe Asn Ser Gly Gly Val
645 650 655
Met Ala Ser Gln Glu Ile Val Ser Ala Thr Val Arg Leu Gln Thr Asp
660 665 670
Pro Val Glu Tyr Leu Leu Glu Gln Leu Asn Asn Leu Thr Glu Thr Val
675 680 685
Ser Pro Asn Thr Asp Val Arg Thr Tyr Ser Gly Lys Arg Asn Gly Ala
690 695 700
Ser Asp Asp Leu Met Val Ala Val Ile Met Ala Ile Tyr Leu Ala Ala
705 710 715 720
Gln Ala Gly Pro Pro His Thr Phe Ala Pro Ile Thr Arg Val Ser
725 730 735
<210> 41
<211> 2208
<212> DNA
<213> Herpes simplex virus type 1
<400> 41
atgtttggtc agcagctggc gtccgacgtc cagcagtacc tggagcgcct cgagaaacag 60
aggcaactta aggtgggcgc ggacgaggcg tcggcgggcc tcaccatggg cggcgatgcc 120
ctacgagtgc cctttttaga tttcgcgacc gcgaccccca agcgccacca gaccgtggtc 180
cctggcgtcg ggacgctcca cgactgctgc gagcactcgc cgctcttctc ggccgtggcg 240
cggcggctgc tgtttaatag cctggtgccg gcgcaactaa aggggcgtga tttcgggggc 300
gaccacacgg ccaagctgga attcctggcc cccgagttgg tacgggcggt ggcgcgactg 360
cggtttaagg agtgcgcgcc ggcggacgtg gtgcctcagc gtaacgccta ctatagcgtt 420
ctgaatacgt ttcaggccct ccaccgctcc gaagcctttc gccagctggt gcactttgtg 480
cgggactttg cccagctgct caaaacctcc ttccgggcct ccagcctcac ggagaccacg 540
ggccccccca aaaaacgggc caaggtggac gtggccaccc acggccggac gtacggcacg 600
ctggagctgt tccaaaaaat gatccttatg cacgccacct actttctggc cgccgtgctc 660
ctcggggacc acgcggagca ggtcaacacg ttcctgcgtc tcgtgtttga gatccccctg 720
tttagcgacg cggccgtgcg ccacttccgc cagcgcgcca ccgtgtttct cgtcccccgg 780
cgccacggca agacctggtt tctggtgccc ctcatcgcgc tgtcgctggc ctcctttcgg 840
gggatcaaga tcggctacac ggcgcacatc cgcaaggcga ccgagccggt gtttgaggag 900
atcgacgcct gcctgcgggg ctggttcggt tcggcccgag tggaccacgt taaaggggaa 960
accatctcct tctcgtttcc ggacgggtcg cgcagtacca tcgtgtttgc ctccagccac 1020
aacacaaacg gaatccgagg ccaggacttt aacctgctct ttgtcgacga ggccaacttt 1080
attcgcccgg atgcggtcca gacgattatg ggctttctca accaggccaa ctgcaagatt 1140
atcttcgtgt cgtccaccaa caccgggaag gccagtacga gctttttgta caacctccgc 1200
ggggccgcag acgagcttct caacgtggtg acctatatat gcgatgatca catgccgagg 1260
gtggtgacgc acacaaacgc cacggcctgt tcttgttata tcctcaacaa gcccgttttc 1320
atcacgatgg acggggcggt tcgccggacc gccgatttgt ttctggccga ttccttcatg 1380
caggagatca tcgggggcca ggccagggag accggcgacg accggcccgt tctgaccaag 1440
tctgcggggg agcggtttct gttgtaccgc ccctcgacca ccaccaacag cggcctcatg 1500
gcccccgatt tgtacgtgta cgtggatccc gcgttcacgg ccaacacccg agcctccggg 1560
accggcgtcg ctgtcgtcgg gcggtaccgc gacgattata tcatcttcgc cctggagcac 1620
ttttttctcc gcgcgctcac gggctcggcc cccgccgaca tcgcccgctg cgtcgtccac 1680
agtctgacgc aggtcctggc cctgcatccc ggggcgtttc gcggcgtccg ggtggcggtc 1740
gagggaaata gcagccagga ctcggccgtc gccatcgcca cgcacgtgca cacagagatg 1800
caccgcctac tggcctcgga gggggccgac gcgggctcgg gccccgagct tctcttctac 1860
cactgcgagc ctcccgggag cgcggtgctg tacccctttt tcctgctcaa caaacagaag 1920
acgcccgcct ttgaacactt tattaaaaag tttaactccg ggggcgtcat ggcctcccag 1980
gagatcgttt ccgcgacggt gcgcctgcag accgacccgg tcgagtatct gctcgagcag 2040
ctaaataacc tcaccgaaac cgtctccccc aacactgacg tccgtacgta ttccggaaaa 2100
cggaacggcg cctcggatga ccttatggtc gccgtcatta tggccatcta cctcgcggcc 2160
caggccggac ctccgcacac attcgctcct atcatacgcg tctcgtga 2208
<210> 42
<211> 735
<212> PRT
<213> Herpes simplex virus type 1
<400> 42
Met Phe Gly Gln Gln Leu Ala Ser Asp Val Gln Gln Tyr Leu Glu Arg
1 5 10 15
Leu Glu Lys Gln Arg Gln Leu Lys Val Gly Ala Asp Glu Ala Ser Ala
20 25 30
Gly Leu Thr Met Gly Gly Asp Ala Leu Arg Val Pro Phe Leu Asp Phe
35 40 45
Ala Thr Ala Thr Pro Lys Arg His Gln Thr Val Val Pro Gly Val Gly
50 55 60
Thr Leu His Asp Cys Cys Glu His Ser Pro Leu Phe Ser Ala Val Ala
65 70 75 80
Arg Arg Leu Leu Phe Asn Ser Leu Val Pro Ala Gln Leu Lys Gly Arg
85 90 95
Asp Phe Gly Gly Asp His Thr Ala Lys Leu Glu Phe Leu Ala Pro Glu
100 105 110
Leu Val Arg Ala Val Ala Arg Leu Arg Phe Lys Glu Cys Ala Pro Ala
115 120 125
Asp Val Val Pro Gln Arg Asn Ala Tyr Tyr Ser Val Leu Asn Thr Phe
130 135 140
Gln Ala Leu His Arg Ser Glu Ala Phe Arg Gln Leu Val His Phe Val
145 150 155 160
Arg Asp Phe Ala Gln Leu Leu Lys Thr Ser Phe Arg Ala Ser Ser Leu
165 170 175
Thr Glu Thr Thr Gly Pro Pro Lys Lys Arg Ala Lys Val Asp Val Ala
180 185 190
Thr His Gly Arg Thr Tyr Gly Thr Leu Glu Leu Phe Gln Lys Met Ile
195 200 205
Leu Met His Ala Thr Tyr Phe Leu Ala Ala Val Leu Leu Gly Asp His
210 215 220
Ala Glu Gln Val Asn Thr Phe Leu Arg Leu Val Phe Glu Ile Pro Leu
225 230 235 240
Phe Ser Asp Ala Ala Val Arg His Phe Arg Gln Arg Ala Thr Val Phe
245 250 255
Leu Val Pro Arg Arg His Gly Lys Thr Trp Phe Leu Val Pro Leu Ile
260 265 270
Ala Leu Ser Leu Ala Ser Phe Arg Gly Ile Lys Ile Gly Tyr Thr Ala
275 280 285
His Ile Arg Lys Ala Thr Glu Pro Val Phe Glu Glu Ile Asp Ala Cys
290 295 300
Leu Arg Gly Trp Phe Gly Ser Ala Arg Val Asp His Val Lys Gly Glu
305 310 315 320
Thr Ile Ser Phe Ser Phe Pro Asp Gly Ser Arg Ser Thr Ile Val Phe
325 330 335
Ala Ser Ser His Asn Thr Asn Gly Ile Arg Gly Gln Asp Phe Asn Leu
340 345 350
Leu Phe Val Asp Glu Ala Asn Phe Ile Arg Pro Asp Ala Val Gln Thr
355 360 365
Ile Met Gly Phe Leu Asn Gln Ala Asn Cys Lys Ile Ile Phe Val Ser
370 375 380
Ser Thr Asn Thr Gly Lys Ala Ser Thr Ser Phe Leu Tyr Asn Leu Arg
385 390 395 400
Gly Ala Ala Asp Glu Leu Leu Asn Val Val Thr Tyr Ile Cys Asp Asp
405 410 415
His Met Pro Arg Val Val Thr His Thr Asn Ala Thr Ala Cys Ser Cys
420 425 430
Tyr Ile Leu Asn Lys Pro Val Phe Ile Thr Met Asp Gly Ala Val Arg
435 440 445
Arg Thr Ala Asp Leu Phe Leu Ala Asp Ser Phe Met Gln Glu Ile Ile
450 455 460
Gly Gly Gln Ala Arg Glu Thr Gly Asp Asp Arg Pro Val Leu Thr Lys
465 470 475 480
Ser Ala Gly Glu Arg Phe Leu Leu Tyr Arg Pro Ser Thr Thr Thr Asn
485 490 495
Ser Gly Leu Met Ala Pro Asp Leu Tyr Val Tyr Val Asp Pro Ala Phe
500 505 510
Thr Ala Asn Thr Arg Ala Ser Gly Thr Gly Val Ala Val Val Gly Arg
515 520 525
Tyr Arg Asp Asp Tyr Ile Ile Phe Ala Leu Glu His Phe Phe Leu Arg
530 535 540
Ala Leu Thr Gly Ser Ala Pro Ala Asp Ile Ala Arg Cys Val Val His
545 550 555 560
Ser Leu Thr Gln Val Leu Ala Leu His Pro Gly Ala Phe Arg Gly Val
565 570 575
Arg Val Ala Val Glu Gly Asn Ser Ser Gln Asp Ser Ala Val Ala Ile
580 585 590
Ala Thr His Val His Thr Glu Met His Arg Leu Leu Ala Ser Glu Gly
595 600 605
Ala Asp Ala Gly Ser Gly Pro Glu Leu Leu Phe Tyr His Cys Glu Pro
610 615 620
Pro Gly Ser Ala Val Leu Tyr Pro Phe Phe Leu Leu Asn Lys Gln Lys
625 630 635 640
Thr Pro Ala Phe Glu His Phe Ile Lys Lys Phe Asn Ser Gly Gly Val
645 650 655
Met Ala Ser Gln Glu Ile Val Ser Ala Thr Val Arg Leu Gln Thr Asp
660 665 670
Pro Val Glu Tyr Leu Leu Glu Gln Leu Asn Asn Leu Thr Glu Thr Val
675 680 685
Ser Pro Asn Thr Asp Val Arg Thr Tyr Ser Gly Lys Arg Asn Gly Ala
690 695 700
Ser Asp Asp Leu Met Val Ala Val Ile Met Ala Ile Tyr Leu Ala Ala
705 710 715 720
Gln Ala Gly Pro Pro His Thr Phe Ala Pro Ile Ile Arg Val Ser
725 730 735
<210> 43
<211> 3414
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 43
atggccagcc gcccagccgc atcctctccc gtcgaagcgc gggccccggt tgggggacag 60
gaggccggcg gccccagcgc agccacccag ggggaggccg ccggggcccc tctcgcccac 120
ggccaccacg tgtactgcca gcgagtcaat ggcgtgatgg tgctttccga caagacgccc 180
gggtccgcgt cctaccgcat cagcgatagc aactttgtcc aatgtggttc caactgcacc 240
atgatcatcg acggagacgt ggtgcgcggg cgcccccagg acccgggggc cgcggcatcc 300
cccgctccct tcgttgcggt gacaaacatc ggagccggca gcgacggcgg gaccgccgtc 360
gtggcattcg ggggaacccc acgtcgctcg gcggggacgt ctaccggtac ccagacggcc 420
gacgtcccca ccgaggccct tgggggcccc cctcctcctc cccgcttcac cctgggtggc 480
ggctgttgtt cctgtcgcga cacacggcgc cgctctgcgg tattcggggg ggagggggat 540
ccagtcggcc ccgcggagtt cgtctcggac gaccggtcgt ccgattccga ctcggatgac 600
tcggaggaca cggactcgga gacgctgtca cacgcctcct cggacgtgtc cggcggggcc 660
acgtacgacg acgcccttga ctccgattcg tcatcggatg actccctgca gatagatggc 720
cccgtgtgtc gcccgtggag caatgacacc gcgcccctgg atgtttgccc cgggaccccc 780
ggcccgggcg ccgacgccgg tggtccctca gcggtagacc cacacgcgcc gacgccagag 840
gccggcgctg gtcttgcggc cgatcccgcc gtggcccggg acgacgcgga ggggctttcg 900
gacccccggc cacgtctggg aacgggcacg gcctaccccg tccccctgga actcacgccc 960
gagaacgcgg aggccgtggc gcgctttctg ggagatgccg tgaaccgcga acccgcgctc 1020
atgctggagt acttttgccg gtgcgcccgc gaggaaacca agcgtgtccc ccccaggaca 1080
ttcggcagcc cccctcgcct cacggaggac gactttgggc ttctcaacta cgcgctcgtg 1140
gagatgcagc gcctgtgtct ggacgttcct ccggtcccgc cgaacgcata catgccctat 1200
tatctcaggg agtatgtgac gcggctggtc aacgggttca agccgctggt gagccggtcc 1260
gctcgccttt accgcatcct gggggttctg gtgcacctgc ggatccggac ccgggaggcc 1320
tcctttgagg agtggctgcg atccaaggaa gtggccctgg attttggcct gacggaaagg 1380
cttcgcgagc acgaagccca gctggtgatc ctggcccagg ctctggacca ttacgactgt 1440
ctgatccaca gcacaccgca cacgctggtc gagcgggggc tgcaatcggc cctgaagtat 1500
gaggagtttt acctaaagcg ttttggcggg cactacatgg agtccgtctt ccagatgtac 1560
acccgcatcg ccggcttttt ggcctgccgg gccacgcgcg gcatgcgcca catcgccctg 1620
gggcgagagg ggtcgtggtg ggaaatgttc aagttctttt tccaccgcct ctacgaccac 1680
cagatcgtac cgtcgacccc cgccatgctg aacctgggga cccgcaacta ctacacctcc 1740
agctgctacc tggtaaaccc ccaggccacc acaaacaagg cgaccctgcg ggccatcacc 1800
agcaacgtca gtgccatcct cgcccgcaac gggggcatcg ggctatgcgt gcaggcgttt 1860
aacgactccg gccccgggac cgccagcgtc atgcccgccc tcaaggtcct tgactcgctg 1920
gtggcggcgc acaacaaaga gagcgcgcgt ccgaccggcg cgtgcgtgta cctggagccg 1980
tggcacaccg acgtgcgggc cgtgctccgg atgaaggggg tcctcgccgg cgaagaggcc 2040
cagcgctgcg acaatatctt cagcgccctc tggatgccag acctgttttt caagcgcctg 2100
attcgccacc tggacggcga gaagaacgtc acatggaccc tgttcgaccg ggacaccagc 2160
atgtcgctcg ccgactttca cggggaggag ttcgagaagc tctaccagca cctcgaggtc 2220
atggggttcg gcgagcagat acccatccag gagctggcct atggcattgt gcgcagtgcg 2280
gccacgaccg ggagcccctt cgtcatgttc aaagacgcgg tgaaccgcca ctacatctac 2340
gacacccagg gggcggccat cgccggctcc aacctctgca ccgagatcgt ccatccggcc 2400
tccaagcgat ccagtggggt ctgcaacctg ggaagcgtga atctggcccg atgcgtctcc 2460
aggcagacgt ttgactttgg gcggctccgc gacgccgtgc aggcgtgcgt gctgatggtg 2520
aacatcatga tcgacagcac gctacaaccc acgccccagt gcacccgcgg caacgacaac 2580
ctgcggtcca tgggaatcgg catgcagggc ctgcacacgg cctgcctgaa gctggggctg 2640
gatctggagt ctgtcgaatt tcaggacctg aacaaacaca tcgccgaggt gatgctgctg 2700
tcggcgatga agaccagcaa cgcgctgtgc gttcgcgggg cccgtccctt caaccacttt 2760
aagcgcagca tgtatcgcgc cggccgcttt cactgggagc gctttccgga cgcccggccg 2820
cggtacgagg gcgagtggga gatgctacgc cagagcatga tgaaacacgg cctgcgcaac 2880
agccagtttg tcgcgctgat gcccaccgcc gcctcggcgc agatctcgga cgtcagcgag 2940
ggctttgccc ccctgttcac caacctgttc agcaaggtga cccgggacgg cgagacgctg 3000
cgccccaaca cgctcctgct aaaggaactg gaacgcacgt ttagcgggaa gcgcctcctg 3060
gaggtgatgg acagtctcga cgccaagcag tggtccgtgg cgcaggcgct cccgtgcctg 3120
gagcccaccc accccctccg gcgattcaag accgcgtttg actacgacca gaagttgctg 3180
atcgacctgt gtgcggaccg cgccccctac gtcgaccata gccaatccat gaccctgtat 3240
gtcacggaga aggcggacgg gaccctccca gcctccaccc tggtccgcct tctggtccac 3300
gcatataagc gcggactaaa aacagggatg tactactgca aggttcgcaa ggcgaccaac 3360
agcggggtct ttggcggcga cgacaacatt gtctgcacga gctgcgcgct gtga 3414
<210> 44
<211> 1137
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 44
Met Ala Ser Arg Pro Ala Ala Ser Ser Pro Val Glu Ala Arg Ala Pro
1 5 10 15
Val Gly Gly Gln Glu Ala Gly Gly Pro Ser Ala Ala Thr Gln Gly Glu
20 25 30
Ala Ala Gly Ala Pro Leu Ala His Gly His His Val Tyr Cys Gln Arg
35 40 45
Val Asn Gly Val Met Val Leu Ser Asp Lys Thr Pro Gly Ser Ala Ser
50 55 60
Tyr Arg Ile Ser Asp Ser Asn Phe Val Gln Cys Gly Ser Asn Cys Thr
65 70 75 80
Met Ile Ile Asp Gly Asp Val Val Arg Gly Arg Pro Gln Asp Pro Gly
85 90 95
Ala Ala Ala Ser Pro Ala Pro Phe Val Ala Val Thr Asn Ile Gly Ala
100 105 110
Gly Ser Asp Gly Gly Thr Ala Val Val Ala Phe Gly Gly Thr Pro Arg
115 120 125
Arg Ser Ala Gly Thr Ser Thr Gly Thr Gln Thr Ala Asp Val Pro Thr
130 135 140
Glu Ala Leu Gly Gly Pro Pro Pro Pro Pro Arg Phe Thr Leu Gly Gly
145 150 155 160
Gly Cys Cys Ser Cys Arg Asp Thr Arg Arg Arg Ser Ala Val Phe Gly
165 170 175
Gly Glu Gly Asp Pro Val Gly Pro Ala Glu Phe Val Ser Asp Asp Arg
180 185 190
Ser Ser Asp Ser Asp Ser Asp Asp Ser Glu Asp Thr Asp Ser Glu Thr
195 200 205
Leu Ser His Ala Ser Ser Asp Val Ser Gly Gly Ala Thr Tyr Asp Asp
210 215 220
Ala Leu Asp Ser Asp Ser Ser Ser Asp Asp Ser Leu Gln Ile Asp Gly
225 230 235 240
Pro Val Cys Arg Pro Trp Ser Asn Asp Thr Ala Pro Leu Asp Val Cys
245 250 255
Pro Gly Thr Pro Gly Pro Gly Ala Asp Ala Gly Gly Pro Ser Ala Val
260 265 270
Asp Pro His Ala Pro Thr Pro Glu Ala Gly Ala Gly Leu Ala Ala Asp
275 280 285
Pro Ala Val Ala Arg Asp Asp Ala Glu Gly Leu Ser Asp Pro Arg Pro
290 295 300
Arg Leu Gly Thr Gly Thr Ala Tyr Pro Val Pro Leu Glu Leu Thr Pro
305 310 315 320
Glu Asn Ala Glu Ala Val Ala Arg Phe Leu Gly Asp Ala Val Asn Arg
325 330 335
Glu Pro Ala Leu Met Leu Glu Tyr Phe Cys Arg Cys Ala Arg Glu Glu
340 345 350
Thr Lys Arg Val Pro Pro Arg Thr Phe Gly Ser Pro Pro Arg Leu Thr
355 360 365
Glu Asp Asp Phe Gly Leu Leu Asn Tyr Ala Leu Val Glu Met Gln Arg
370 375 380
Leu Cys Leu Asp Val Pro Pro Val Pro Pro Asn Ala Tyr Met Pro Tyr
385 390 395 400
Tyr Leu Arg Glu Tyr Val Thr Arg Leu Val Asn Gly Phe Lys Pro Leu
405 410 415
Val Ser Arg Ser Ala Arg Leu Tyr Arg Ile Leu Gly Val Leu Val His
420 425 430
Leu Arg Ile Arg Thr Arg Glu Ala Ser Phe Glu Glu Trp Leu Arg Ser
435 440 445
Lys Glu Val Ala Leu Asp Phe Gly Leu Thr Glu Arg Leu Arg Glu His
450 455 460
Glu Ala Gln Leu Val Ile Leu Ala Gln Ala Leu Asp His Tyr Asp Cys
465 470 475 480
Leu Ile His Ser Thr Pro His Thr Leu Val Glu Arg Gly Leu Gln Ser
485 490 495
Ala Leu Lys Tyr Glu Glu Phe Tyr Leu Lys Arg Phe Gly Gly His Tyr
500 505 510
Met Glu Ser Val Phe Gln Met Tyr Thr Arg Ile Ala Gly Phe Leu Ala
515 520 525
Cys Arg Ala Thr Arg Gly Met Arg His Ile Ala Leu Gly Arg Glu Gly
530 535 540
Ser Trp Trp Glu Met Phe Lys Phe Phe Phe His Arg Leu Tyr Asp His
545 550 555 560
Gln Ile Val Pro Ser Thr Pro Ala Met Leu Asn Leu Gly Thr Arg Asn
565 570 575
Tyr Tyr Thr Ser Ser Cys Tyr Leu Val Asn Pro Gln Ala Thr Thr Asn
580 585 590
Lys Ala Thr Leu Arg Ala Ile Thr Ser Asn Val Ser Ala Ile Leu Ala
595 600 605
Arg Asn Gly Gly Ile Gly Leu Cys Val Gln Ala Phe Asn Asp Ser Gly
610 615 620
Pro Gly Thr Ala Ser Val Met Pro Ala Leu Lys Val Leu Asp Ser Leu
625 630 635 640
Val Ala Ala His Asn Lys Glu Ser Ala Arg Pro Thr Gly Ala Cys Val
645 650 655
Tyr Leu Glu Pro Trp His Thr Asp Val Arg Ala Val Leu Arg Met Lys
660 665 670
Gly Val Leu Ala Gly Glu Glu Ala Gln Arg Cys Asp Asn Ile Phe Ser
675 680 685
Ala Leu Trp Met Pro Asp Leu Phe Phe Lys Arg Leu Ile Arg His Leu
690 695 700
Asp Gly Glu Lys Asn Val Thr Trp Thr Leu Phe Asp Arg Asp Thr Ser
705 710 715 720
Met Ser Leu Ala Asp Phe His Gly Glu Glu Phe Glu Lys Leu Tyr Gln
725 730 735
His Leu Glu Val Met Gly Phe Gly Glu Gln Ile Pro Ile Gln Glu Leu
740 745 750
Ala Tyr Gly Ile Val Arg Ser Ala Ala Thr Thr Gly Ser Pro Phe Val
755 760 765
Met Phe Lys Asp Ala Val Asn Arg His Tyr Ile Tyr Asp Thr Gln Gly
770 775 780
Ala Ala Ile Ala Gly Ser Asn Leu Cys Thr Glu Ile Val His Pro Ala
785 790 795 800
Ser Lys Arg Ser Ser Gly Val Cys Asn Leu Gly Ser Val Asn Leu Ala
805 810 815
Arg Cys Val Ser Arg Gln Thr Phe Asp Phe Gly Arg Leu Arg Asp Ala
820 825 830
Val Gln Ala Cys Val Leu Met Val Asn Ile Met Ile Asp Ser Thr Leu
835 840 845
Gln Pro Thr Pro Gln Cys Thr Arg Gly Asn Asp Asn Leu Arg Ser Met
850 855 860
Gly Ile Gly Met Gln Gly Leu His Thr Ala Cys Leu Lys Leu Gly Leu
865 870 875 880
Asp Leu Glu Ser Val Glu Phe Gln Asp Leu Asn Lys His Ile Ala Glu
885 890 895
Val Met Leu Leu Ser Ala Met Lys Thr Ser Asn Ala Leu Cys Val Arg
900 905 910
Gly Ala Arg Pro Phe Asn His Phe Lys Arg Ser Met Tyr Arg Ala Gly
915 920 925
Arg Phe His Trp Glu Arg Phe Pro Asp Ala Arg Pro Arg Tyr Glu Gly
930 935 940
Glu Trp Glu Met Leu Arg Gln Ser Met Met Lys His Gly Leu Arg Asn
945 950 955 960
Ser Gln Phe Val Ala Leu Met Pro Thr Ala Ala Ser Ala Gln Ile Ser
965 970 975
Asp Val Ser Glu Gly Phe Ala Pro Leu Phe Thr Asn Leu Phe Ser Lys
980 985 990
Val Thr Arg Asp Gly Glu Thr Leu Arg Pro Asn Thr Leu Leu Leu Lys
995 1000 1005
Glu Leu Glu Arg Thr Phe Ser Gly Lys Arg Leu Leu Glu Val Met
1010 1015 1020
Asp Ser Leu Asp Ala Lys Gln Trp Ser Val Ala Gln Ala Leu Pro
1025 1030 1035
Cys Leu Glu Pro Thr His Pro Leu Arg Arg Phe Lys Thr Ala Phe
1040 1045 1050
Asp Tyr Asp Gln Lys Leu Leu Ile Asp Leu Cys Ala Asp Arg Ala
1055 1060 1065
Pro Tyr Val Asp His Ser Gln Ser Met Thr Leu Tyr Val Thr Glu
1070 1075 1080
Lys Ala Asp Gly Thr Leu Pro Ala Ser Thr Leu Val Arg Leu Leu
1085 1090 1095
Val His Ala Tyr Lys Arg Gly Leu Lys Thr Gly Met Tyr Tyr Cys
1100 1105 1110
Lys Val Arg Lys Ala Thr Asn Ser Gly Val Phe Gly Gly Asp Asp
1115 1120 1125
Asn Ile Val Cys Thr Ser Cys Ala Leu
1130 1135
<210> 45
<211> 3414
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 45
atggccagcc gcccagccgc atcctctccc gtcgaagcgc gggccccggt tgggggacag 60
gaggccggcg gccccagcgc agccacccag ggggaggccg ccggggcccc tctcgcccac 120
ggccaccacg tgtactgcca gcgagtcaat ggcgtgatgg tgctttccga caagacgccc 180
gggtccgcgt cctaccgcat cagcgatagc aactttgtcc aatgtggttc caactgcacc 240
atgatcatcg acggagacgt ggtgcgcggg cgcccccagg acccgggggc cgcggcatcc 300
cccgctccct tcgttgcggt gacaaacatc ggagccggca gcgacggcgg gaccgccgtc 360
gtggcattcg ggggaacccc acgtcgctcg gcggggacgt ctaccggtac ccagacggcc 420
gacgtcccca ccgaggccct tgggggcccc cctcctcctc cccgcttcac cctgggtggc 480
ggctgttgtt cctgtcgcga cacacggcgc cgctctgcgg tattcggggg ggagggggat 540
ccagtcggcc ccgcggagtt cgtctcggac gaccggtcgt ccgattccga ctcggatgac 600
tcggaggaca cggactcgga gacgctgtca cacgcctcct cggacgtgtc cggcggggcc 660
acgtacgacg acgcccttga ctccgattcg tcatcggatg actccctgca gatagatggc 720
cccgtgtgtc gcccgtggag caatgacacc gcgcccctgg atgtttgccc cgggaccccc 780
ggcccgggcg ccgacgccgg tggtccctca gcggtagacc cacacgcgcc gacgccagag 840
gccggcgctg gtcttgcggc cgatcccgcc gtggcccggg acgacgcgga ggggctttcg 900
gacccccggc cacgtctggg aacgggcacg gcctaccccg tccccctgga actcacgccc 960
gagaacgcgg aggccgtggc gcgctttctg ggagatgccg tgaaccgcga acccgcgctc 1020
atgctggagt acttttgccg gtgcgcccgc gaggaaacca agcgtgtccc ccccaggaca 1080
ttcggcagcc cccctcgcct cacggaggac gactttgggc ttctcaacta cgcgctcgtg 1140
gagatgcagc gcctgtgtct ggacgttcct ccggtcccgc cgaacgcata catgccctat 1200
tatctcaggg agtatgtgac gcggctggtc aacgggttca agccgctggt gagccggtcc 1260
gctcgccttt accgcatcct gggggttctg gtgcacctgc ggatccggac ccgggaggcc 1320
tcctttgagg agtggctgcg atccaaggaa gtggccctgg attttggcct gacggaaagg 1380
cttcgcgagc acgaagccca gctggtgatc ctggcccagg ctctggacca ttacgactgt 1440
ctgatccaca gcacaccgca cacgctggtc gagcgggggc tgcaatcggc cctgaagtat 1500
gaggagtttt acctaaagcg ttttggcggg cactacatgg agtccgtctt ccagatgtac 1560
acccgcatcg ccggcttttt ggcctgccgg gccacgcgcg gcatgcgcca catcgccctg 1620
gggcgagagg ggtcgtggtg ggaaatgttc aagttctttt tccaccgcct ctacgaccac 1680
cagatcgtac cgtcgacccc cgccatgctg aacctgggga cccgcaacta ctacacctcc 1740
agctgctacc tggtaaaccc ccaggccacc acaaacaagg cgaccctgcg ggccatcacc 1800
agcaacgtca gtgccatcct cgcccgcaac gggggcatcg ggctatgcgt gcaggcgttt 1860
aacgactccg gccccgggac cgccagcgtc atgcccgccc tcaaggtcct tgactcgctg 1920
gtggcggcgc acaacaaaga gagcgcgcgt ccgaccggcg cgtgcgtgta cctggagccg 1980
tggcacaccg acgtgcgggc cgtgctccgg atgaaggggg tcctcgccgg cgaagaggcc 2040
cagcgctgcg acaatatctt cagcgccctc tggatgccag acctgttttt caagcgcctg 2100
attcgccacc tggacggcga gaagaacgtc acatggaccc tgttcgaccg ggacaccagc 2160
atgtcgctcg ccgactttca cggggaggag ttcgagaagc tctaccagca cctcgaggtc 2220
atggggttcg gcgagcagat acccatccag gagctggcct atggcattgt gcgcagtgcg 2280
gccacgaccg ggagcccctt cgtcatgttc aaagacgcgg tgaaccgcca ctacatctac 2340
gacacccagg gggcggccat cgccggctcc aacctctgca ccgagatcgt ccatccggcc 2400
tccaagcgat ccagtggggt ctgcaacctg ggaagcgtga atctggcccg atgcgtctcc 2460
aggcagacgt ttgactttgg gcggctccgc gacgccgtgc aggcgtgcgt gctgatggtg 2520
aacatcatga tcgacagcac gctacaaccc acgccccagt gcacccgcgg caacgacaac 2580
ctgcggtcca tgggaatcgg catgcagggc ctgcacacgg cctgcctgaa gctggggctg 2640
gatctggagt ctgccgaatt tcaggacctg aacaaacaca tcgccgaggt gatgctgctg 2700
tcggcgatga agaccagcaa cgcgctgtgc gttcgcgggg cccgtccctt caaccacttt 2760
aagcgcagca tgtatcgcgc cggccgcttt cactgggagc gctttccgga cgcccggccg 2820
cggtacgagg gcgagtggga gatgctacgc cagagcatga tgaaacacgg cctgcgcaac 2880
agccagtttg tcgcgctgat gcccaccgcc gcctcggcgc agatctcgga cgtcagcgag 2940
ggctttgccc ccctgttcac caacctgttc agcaaggtga cccgggacgg cgagacgctg 3000
cgccccaaca cgctcctgct aaaggaactg gaacgcacgt ttagcgggaa gcgcctcctg 3060
gaggtgatgg acagtctcga cgccaagcag tggtccgtgg cgcaggcgct cccgtgcctg 3120
gagcccaccc accccctccg gcgattcaag accgcgtttg actacgacca gaagttgctg 3180
atcgacctgt gtgcggaccg cgccccctac gtcgaccata gccaatccat gaccctgtat 3240
gtcacggaga aggcggacgg gaccctccca gcctccaccc tggtccgcct tctggtccac 3300
gcatataagc gcggactaaa aacagggatg tactactgca aggttcgcaa ggcgaccaac 3360
agcggggtct ttggcggcga cgacaacatt gtctgcatga gctgcgcgct gtga 3414
<210> 46
<211> 1137
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 46
Met Ala Ser Arg Pro Ala Ala Ser Ser Pro Val Glu Ala Arg Ala Pro
1 5 10 15
Val Gly Gly Gln Glu Ala Gly Gly Pro Ser Ala Ala Thr Gln Gly Glu
20 25 30
Ala Ala Gly Ala Pro Leu Ala His Gly His His Val Tyr Cys Gln Arg
35 40 45
Val Asn Gly Val Met Val Leu Ser Asp Lys Thr Pro Gly Ser Ala Ser
50 55 60
Tyr Arg Ile Ser Asp Ser Asn Phe Val Gln Cys Gly Ser Asn Cys Thr
65 70 75 80
Met Ile Ile Asp Gly Asp Val Val Arg Gly Arg Pro Gln Asp Pro Gly
85 90 95
Ala Ala Ala Ser Pro Ala Pro Phe Val Ala Val Thr Asn Ile Gly Ala
100 105 110
Gly Ser Asp Gly Gly Thr Ala Val Val Ala Phe Gly Gly Thr Pro Arg
115 120 125
Arg Ser Ala Gly Thr Ser Thr Gly Thr Gln Thr Ala Asp Val Pro Thr
130 135 140
Glu Ala Leu Gly Gly Pro Pro Pro Pro Pro Arg Phe Thr Leu Gly Gly
145 150 155 160
Gly Cys Cys Ser Cys Arg Asp Thr Arg Arg Arg Ser Ala Val Phe Gly
165 170 175
Gly Glu Gly Asp Pro Val Gly Pro Ala Glu Phe Val Ser Asp Asp Arg
180 185 190
Ser Ser Asp Ser Asp Ser Asp Asp Ser Glu Asp Thr Asp Ser Glu Thr
195 200 205
Leu Ser His Ala Ser Ser Asp Val Ser Gly Gly Ala Thr Tyr Asp Asp
210 215 220
Ala Leu Asp Ser Asp Ser Ser Ser Asp Asp Ser Leu Gln Ile Asp Gly
225 230 235 240
Pro Val Cys Arg Pro Trp Ser Asn Asp Thr Ala Pro Leu Asp Val Cys
245 250 255
Pro Gly Thr Pro Gly Pro Gly Ala Asp Ala Gly Gly Pro Ser Ala Val
260 265 270
Asp Pro His Ala Pro Thr Pro Glu Ala Gly Ala Gly Leu Ala Ala Asp
275 280 285
Pro Ala Val Ala Arg Asp Asp Ala Glu Gly Leu Ser Asp Pro Arg Pro
290 295 300
Arg Leu Gly Thr Gly Thr Ala Tyr Pro Val Pro Leu Glu Leu Thr Pro
305 310 315 320
Glu Asn Ala Glu Ala Val Ala Arg Phe Leu Gly Asp Ala Val Asn Arg
325 330 335
Glu Pro Ala Leu Met Leu Glu Tyr Phe Cys Arg Cys Ala Arg Glu Glu
340 345 350
Thr Lys Arg Val Pro Pro Arg Thr Phe Gly Ser Pro Pro Arg Leu Thr
355 360 365
Glu Asp Asp Phe Gly Leu Leu Asn Tyr Ala Leu Val Glu Met Gln Arg
370 375 380
Leu Cys Leu Asp Val Pro Pro Val Pro Pro Asn Ala Tyr Met Pro Tyr
385 390 395 400
Tyr Leu Arg Glu Tyr Val Thr Arg Leu Val Asn Gly Phe Lys Pro Leu
405 410 415
Val Ser Arg Ser Ala Arg Leu Tyr Arg Ile Leu Gly Val Leu Val His
420 425 430
Leu Arg Ile Arg Thr Arg Glu Ala Ser Phe Glu Glu Trp Leu Arg Ser
435 440 445
Lys Glu Val Ala Leu Asp Phe Gly Leu Thr Glu Arg Leu Arg Glu His
450 455 460
Glu Ala Gln Leu Val Ile Leu Ala Gln Ala Leu Asp His Tyr Asp Cys
465 470 475 480
Leu Ile His Ser Thr Pro His Thr Leu Val Glu Arg Gly Leu Gln Ser
485 490 495
Ala Leu Lys Tyr Glu Glu Phe Tyr Leu Lys Arg Phe Gly Gly His Tyr
500 505 510
Met Glu Ser Val Phe Gln Met Tyr Thr Arg Ile Ala Gly Phe Leu Ala
515 520 525
Cys Arg Ala Thr Arg Gly Met Arg His Ile Ala Leu Gly Arg Glu Gly
530 535 540
Ser Trp Trp Glu Met Phe Lys Phe Phe Phe His Arg Leu Tyr Asp His
545 550 555 560
Gln Ile Val Pro Ser Thr Pro Ala Met Leu Asn Leu Gly Thr Arg Asn
565 570 575
Tyr Tyr Thr Ser Ser Cys Tyr Leu Val Asn Pro Gln Ala Thr Thr Asn
580 585 590
Lys Ala Thr Leu Arg Ala Ile Thr Ser Asn Val Ser Ala Ile Leu Ala
595 600 605
Arg Asn Gly Gly Ile Gly Leu Cys Val Gln Ala Phe Asn Asp Ser Gly
610 615 620
Pro Gly Thr Ala Ser Val Met Pro Ala Leu Lys Val Leu Asp Ser Leu
625 630 635 640
Val Ala Ala His Asn Lys Glu Ser Ala Arg Pro Thr Gly Ala Cys Val
645 650 655
Tyr Leu Glu Pro Trp His Thr Asp Val Arg Ala Val Leu Arg Met Lys
660 665 670
Gly Val Leu Ala Gly Glu Glu Ala Gln Arg Cys Asp Asn Ile Phe Ser
675 680 685
Ala Leu Trp Met Pro Asp Leu Phe Phe Lys Arg Leu Ile Arg His Leu
690 695 700
Asp Gly Glu Lys Asn Val Thr Trp Thr Leu Phe Asp Arg Asp Thr Ser
705 710 715 720
Met Ser Leu Ala Asp Phe His Gly Glu Glu Phe Glu Lys Leu Tyr Gln
725 730 735
His Leu Glu Val Met Gly Phe Gly Glu Gln Ile Pro Ile Gln Glu Leu
740 745 750
Ala Tyr Gly Ile Val Arg Ser Ala Ala Thr Thr Gly Ser Pro Phe Val
755 760 765
Met Phe Lys Asp Ala Val Asn Arg His Tyr Ile Tyr Asp Thr Gln Gly
770 775 780
Ala Ala Ile Ala Gly Ser Asn Leu Cys Thr Glu Ile Val His Pro Ala
785 790 795 800
Ser Lys Arg Ser Ser Gly Val Cys Asn Leu Gly Ser Val Asn Leu Ala
805 810 815
Arg Cys Val Ser Arg Gln Thr Phe Asp Phe Gly Arg Leu Arg Asp Ala
820 825 830
Val Gln Ala Cys Val Leu Met Val Asn Ile Met Ile Asp Ser Thr Leu
835 840 845
Gln Pro Thr Pro Gln Cys Thr Arg Gly Asn Asp Asn Leu Arg Ser Met
850 855 860
Gly Ile Gly Met Gln Gly Leu His Thr Ala Cys Leu Lys Leu Gly Leu
865 870 875 880
Asp Leu Glu Ser Ala Glu Phe Gln Asp Leu Asn Lys His Ile Ala Glu
885 890 895
Val Met Leu Leu Ser Ala Met Lys Thr Ser Asn Ala Leu Cys Val Arg
900 905 910
Gly Ala Arg Pro Phe Asn His Phe Lys Arg Ser Met Tyr Arg Ala Gly
915 920 925
Arg Phe His Trp Glu Arg Phe Pro Asp Ala Arg Pro Arg Tyr Glu Gly
930 935 940
Glu Trp Glu Met Leu Arg Gln Ser Met Met Lys His Gly Leu Arg Asn
945 950 955 960
Ser Gln Phe Val Ala Leu Met Pro Thr Ala Ala Ser Ala Gln Ile Ser
965 970 975
Asp Val Ser Glu Gly Phe Ala Pro Leu Phe Thr Asn Leu Phe Ser Lys
980 985 990
Val Thr Arg Asp Gly Glu Thr Leu Arg Pro Asn Thr Leu Leu Leu Lys
995 1000 1005
Glu Leu Glu Arg Thr Phe Ser Gly Lys Arg Leu Leu Glu Val Met
1010 1015 1020
Asp Ser Leu Asp Ala Lys Gln Trp Ser Val Ala Gln Ala Leu Pro
1025 1030 1035
Cys Leu Glu Pro Thr His Pro Leu Arg Arg Phe Lys Thr Ala Phe
1040 1045 1050
Asp Tyr Asp Gln Lys Leu Leu Ile Asp Leu Cys Ala Asp Arg Ala
1055 1060 1065
Pro Tyr Val Asp His Ser Gln Ser Met Thr Leu Tyr Val Thr Glu
1070 1075 1080
Lys Ala Asp Gly Thr Leu Pro Ala Ser Thr Leu Val Arg Leu Leu
1085 1090 1095
Val His Ala Tyr Lys Arg Gly Leu Lys Thr Gly Met Tyr Tyr Cys
1100 1105 1110
Lys Val Arg Lys Ala Thr Asn Ser Gly Val Phe Gly Gly Asp Asp
1115 1120 1125
Asn Ile Val Cys Met Ser Cys Ala Leu
1130 1135
<210> 47
<211> 3780
<212> DNA
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polynucleotide
<400> 47
gaaccccttc gccatggagc ccagtatctt gctcctcctt gctctccttg tgggcttctt 60
gttactctta gtcaggggac acccaaagtc ccgtggcaac ttcccaccag gacctcgtcc 120
ccttcccctc ttggggaacc tcctgcagtt ggacagaggg ggcctcctca attccttcat 180
gcagcttcga gaaaaatatg gagatgtgtt cacagtacac ctgggaccaa ggcctgtggt 240
catgctatgt gggacagaca ccataaagga ggctctggtg ggccaagctg aggatttctc 300
tggtcgggga acaatcgctg tgattgagcc aatcttcaag gaatatggtg tgatctttgc 360
caatggggaa cgctggaagg cccttcggcg attctctctg gctaccatga gagactttgg 420
gatgggaaag aggagtgtgg aagaacggat tcaggaggaa gcccaatgtt tggtggagga 480
actgcggaaa tcccagggag ccccactgga tcccaccttc ctcttccagt gcatcacagc 540
caacatcatc tgctccattg tgtttggaga gcgctttgac tacacagacc gccagttcct 600
gcgcctgttg gagctgttct accggacctt ttccctccta agttcattct ccagccaggt 660
gtttgagttc ttctctgggt tcctgaaata ctttcctggt gcccacagac aaatctccaa 720
aaacctccag gaaatcctcg attacattgg ccatattgtg gagaagcaca gggccacctt 780
agacccaagc gctccacgag acttcatcga cacttacctt ctgcgcatgg agaaggagaa 840
gtcgaaccac cacacagagt tccatcatga gaacctcatg atctccctgc tctctctctt 900
ctttgctggc actgagacca gcagcaccac actccgctat ggtttcctgc tgatgctcaa 960
gtacccccat gtcgcagaga aagtccaaaa ggagattgat caggtgatcg gctcacaccg 1020
gctaccaacc cttgatgacc gcagtaaaat gccatacact gatgcagtta tccatgagat 1080
tcagaggttt tcagatcttg tccctattgg agtaccacac agagtcacca aagacaccat 1140
gttccgaggg tacctgcttc ccaagaacac tgaagtgtac cccatcctga gttcagctct 1200
ccatgaccca cagtactttg accacccaga cagcttcaat cctgaacact tcctggatgc 1260
caatggggca ctgaaaaaga gtgaagcttt catgcccttc tccacaggaa agcgcatttg 1320
tcttggcgaa ggcattgccc gaaatgaatt gttcctcttc ttcaccacca tcctccagaa 1380
cttctctgtg tcaagccatt tggctcccaa ggacattgac ctcacgccca aggagagtgg 1440
cattggaaaa atacctccaa cgtaccagat ctgcttctca gctcggtgat ccggctgagg 1500
cagccatgtg ccccagttct gttgggaatg gaacttgttt attgcagctt ataatggtta 1560
caaataaagc aatagcatca caaatttcac aaataaagca tttttttcac tgcattctag 1620
ttgtggtttg tccaaactca tcaatgtatc ttatcatgtc tggatccccg ggcgagctcg 1680
aattcctcct ttgaggagtg gctgcgatcc aaggaagtgg ccctggactt tggcctgacg 1740
gaaaggcttc gcgagcacga agcccagctg gtgatcctgg cccaggctct ggaccattac 1800
gactgtctga tccacagcac accgcacacg ctggtcgagc gggggctgca atcggccctg 1860
aagtatgagg agttttacct aaagcgcttt ggcgggcact acatggagtc cgtcttccag 1920
atgtacaccc gcatcgccgg ctttttggcc tgccgggcca cgcgcggcat gcgccacatc 1980
gccctggggc gagaggggtc gtggtgggaa atgttcaagt tctttttcca ccgcctctac 2040
gaccaccaga tcgtaccgtc gacccccgcc atgctgaacc tggggacccg caactactac 2100
acctccagct gctacctggt aaacccccag gccaccacaa acaaggcgac cctgcgggcc 2160
atcaccagca acgtcagcgc catcctcgcc cgcaacgggg gcatcgggct atgcgtgcag 2220
gcgtttaacg actccggccc cgggaccgct agcgtcatac ccgccctcaa ggtcctcgac 2280
tcgctggtgg cggcgcacaa caaagagagc gcgcgtccaa ccggcgcgtg cgtgtacctg 2340
gagccgtggc acaccgacgt gcgggccgtg ctccggatga agggggtcct cgccggcgaa 2400
gaggcccagc gctgcgacaa tatcttcagc gccctctgga tgccagacct gtttttcaag 2460
cgcctgattc gccacctgga cggcgagaag aacgtcacat ggaccctgtt cgaccgggac 2520
accagcatgt cgctcgccga ctttcacggg gaggagttcg agaagctcta ccagcacctc 2580
gaggtcatgg ggttcggcga gcagataccc atccaggagc tggcctatgg cattgtgcgc 2640
agtgcggcca cgaccgggag ccccttcgtc atgttcaaag acgcggtgaa ccgccactac 2700
atctacgaca cccagggggc ggccatcgcc ggctccaacc tctgcaccga gatcgtccat 2760
ccggcctcca agcgatccag tggggtctgc aatctgggaa gcgtgaatct ggcccgatgc 2820
gtctccaggc agacgtttga ctttgggcgg ctccgcgacg ccgtgcaggc gtgcgtgctg 2880
atggtgaaca tcatgatcga cagcacgcta caacccacgc cccagtgcac ccgcggcaac 2940
gacaacctgc ggtccatggg aatcggcatg cagggcctgc acacggcctg cctgaagctg 3000
gggctggatc tggagtctgt cgaatttcag gacctgaaca aacacatcgc cgaggtgatg 3060
ctgctgtcgg cgatgaagac cagcaacgcg ctgtgcgttc gcggggcccg tcccttcaac 3120
cactttaagc gcagcatgta tcgcgccggc cgctttcact gggagcgctt tccggacgcc 3180
cggccgcggt acgagggcga gtgggagatg ctacgccaga gcatgatgaa acacggcctg 3240
cgcaacagcc agtttgtcgc gctgatgccc accgccgcct cggcgcagat ctcggacgtc 3300
agcgagggct ttgcccccct gttcaccaac ctgttcagca aggtgacccg ggacggcgag 3360
acgctgcgcc ccaacacgct cctgctaaag gaactggaac gcacgtttag cgggaagcgc 3420
ctcctggagg tgatggacag tctcgacgcc aagcagtggt ccgtggcgca ggcgctcccg 3480
tgcctggagc ccacccaccc cctccggcga ttcaagaccg cgtttgacta cgaccagaag 3540
ttgctgatcg acctgtgtgc ggaccgcgcc ccctacgtcg accatagcca atccatgacc 3600
ctgtatgtca cggagaaggc ggacgggacc ctcccagcct ccaccctggt ccgccttctg 3660
gtccacgcat ataagcgcgg actaaaaaca gggatgtact actgcaaggt tcgcaaggcg 3720
accaacagcg gggtctttgg cggcgacgac aacattgtct gcacgagctg cgcgctgtga 3780
<210> 48
<211> 491
<212> PRT
<213> Artificial Sequence
<220>
<223> Description of Artificial Sequence: Synthetic polypeptide
<400> 48
Met Glu Pro Ser Ile Leu Leu Leu Leu Ala Leu Leu Val Gly Phe Leu
1 5 10 15
Leu Leu Leu Val Arg Gly His Pro Lys Ser Arg Gly Asn Phe Pro Pro
20 25 30
Gly Pro Arg Pro Leu Pro Leu Leu Gly Asn Leu Leu Gln Leu Asp Arg
35 40 45
Gly Gly Leu Leu Asn Ser Phe Met Gln Leu Arg Glu Lys Tyr Gly Asp
50 55 60
Val Phe Thr Val His Leu Gly Pro Arg Pro Val Val Met Leu Cys Gly
65 70 75 80
Thr Asp Thr Ile Lys Glu Ala Leu Val Gly Gln Ala Glu Asp Phe Ser
85 90 95
Gly Arg Gly Thr Ile Ala Val Ile Glu Pro Ile Phe Lys Glu Tyr Gly
100 105 110
Val Ile Phe Ala Asn Gly Glu Arg Trp Lys Ala Leu Arg Arg Phe Ser
115 120 125
Leu Ala Thr Met Arg Asp Phe Gly Met Gly Lys Arg Ser Val Glu Glu
130 135 140
Arg Ile Gln Glu Glu Ala Gln Cys Leu Val Glu Glu Leu Arg Lys Ser
145 150 155 160
Gln Gly Ala Pro Leu Asp Pro Thr Phe Leu Phe Gln Cys Ile Thr Ala
165 170 175
Asn Ile Ile Cys Ser Ile Val Phe Gly Glu Arg Phe Asp Tyr Thr Asp
180 185 190
Arg Gln Phe Leu Arg Leu Leu Glu Leu Phe Tyr Arg Thr Phe Ser Leu
195 200 205
Leu Ser Ser Phe Ser Ser Gln Val Phe Glu Phe Phe Ser Gly Phe Leu
210 215 220
Lys Tyr Phe Pro Gly Ala His Arg Gln Ile Ser Lys Asn Leu Gln Glu
225 230 235 240
Ile Leu Asp Tyr Ile Gly His Ile Val Glu Lys His Arg Ala Thr Leu
245 250 255
Asp Pro Ser Ala Pro Arg Asp Phe Ile Asp Thr Tyr Leu Leu Arg Met
260 265 270
Glu Lys Glu Lys Ser Asn His His Thr Glu Phe His His Glu Asn Leu
275 280 285
Met Ile Ser Leu Leu Ser Leu Phe Phe Ala Gly Thr Glu Thr Ser Ser
290 295 300
Thr Thr Leu Arg Tyr Gly Phe Leu Leu Met Leu Lys Tyr Pro His Val
305 310 315 320
Ala Glu Lys Val Gln Lys Glu Ile Asp Gln Val Ile Gly Ser His Arg
325 330 335
Leu Pro Thr Leu Asp Asp Arg Ser Lys Met Pro Tyr Thr Asp Ala Val
340 345 350
Ile His Glu Ile Gln Arg Phe Ser Asp Leu Val Pro Ile Gly Val Pro
355 360 365
His Arg Val Thr Lys Asp Thr Met Phe Arg Gly Tyr Leu Leu Pro Lys
370 375 380
Asn Thr Glu Val Tyr Pro Ile Leu Ser Ser Ala Leu His Asp Pro Gln
385 390 395 400
Tyr Phe Asp His Pro Asp Ser Phe Asn Pro Glu His Phe Leu Asp Ala
405 410 415
Asn Gly Ala Leu Lys Lys Ser Glu Ala Phe Met Pro Phe Ser Thr Gly
420 425 430
Lys Arg Ile Cys Leu Gly Glu Gly Ile Ala Arg Asn Glu Leu Phe Leu
435 440 445
Phe Phe Thr Thr Ile Leu Gln Asn Phe Ser Val Ser Ser His Leu Ala
450 455 460
Pro Lys Asp Ile Asp Leu Thr Pro Lys Glu Ser Gly Ile Gly Lys Ile
465 470 475 480
Pro Pro Thr Tyr Gln Ile Cys Phe Ser Ala Arg
485 490
<210> 49
<211> 3414
<212> DNA
<213> Herpes simplex virus type 1
<400> 49
atggccagcc gcccagccgc atcctctccc gtcgaagcgc gggccccggt tgggggacag 60
gaggccggcg gccccagcgc agccacccag ggggaggccg ccggggcccc tctcgcccac 120
ggccaccacg tgtactgcca gcgagtcaat ggcgtgatgg tgctttccga caagacgccc 180
gggtccgcgt cctaccgcat cagcgatagc aactttgtcc aatgtggttc caactgcacc 240
atgatcatcg acggagacgt ggtgcgcggg cgcccccagg acccgggggc cgcggcatcc 300
cccgctccct tcgttgcggt gacaaacatc ggagccggca gcgacggcgg gaccgccgtc 360
gtggcattcg ggggaacccc acgtcgctcg gcggggacgt ctaccggtac ccagacggcc 420
gacgtcccca ccgaggccct tgggggcccc cctcctcctc cccgcttcac cctgggtggc 480
ggctgttgtt cctgtcgcga cacacggcgc cgctctgcgg tattcggggg ggagggggat 540
ccagtcggcc ccgcggagtt cgtctcggac gaccggtcgt ccgattccga ctcggatgac 600
tcggaggaca cggactcgga gacgctgtca cacgcctcct cggacgtgtc cggcggggcc 660
acgtacgacg acgcccttga ctccgattcg tcatcggatg actccctgca gatagatggc 720
cccgtgtgtc gcccgtggag caatgacacc gcgcccctgg atgtttgccc cgggaccccc 780
ggcccgggcg ccgacgccgg tggtccctca gcggtagacc cacacgcgcc gacgccagag 840
gccggcgctg gtcttgcggc cgatcccgcc gtggcccggg acgacgcgga ggggctttcg 900
gacccccggc cacgtctggg aacgggcacg gcctaccccg tccccctgga actcacgccc 960
gagaacgcgg aggccgtggc gcgctttctg ggagatgccg tgaaccgcga acccgcgctc 1020
atgctggagt acttttgccg gtgcgcccgc gaggaaacca agcgtgtccc ccccaggaca 1080
ttcggcagcc cccctcgcct cacggaggac gactttgggc ttctcaacta cgcgctcgtg 1140
gagatgcagc gcctgtgtct ggacgttcct ccggtcccgc cgaacgcata catgccctat 1200
tatctcaggg agtatgtgac gcggctggtc aacgggttca agccgctggt gagccggtcc 1260
gctcgccttt accgcatcct gggggttctg gtgcacctgc ggatccggac ccgggaggcc 1320
tcctttgagg agtggctgcg atccaaggaa gtggccctgg attttggcct gacggaaagg 1380
cttcgcgagc acgaagccca gctggtgatc ctggcccagg ctctggacca ttacgactgt 1440
ctgatccaca gcacaccgca cacgctggtc gagcgggggc tgcaatcggc cctgaagtat 1500
gaggagtttt acctaaagcg ttttggcggg cactacatgg agtccgtctt ccagatgtac 1560
acccgcatcg ccggcttttt ggcctgccgg gccacgcgcg gcatgcgcca catcgccctg 1620
gggcgagagg ggtcgtggtg ggaaatgttc aagttctttt tccaccgcct ctacgaccac 1680
cagatcgtac cgtcgacccc cgccatgctg aacctgggga cccgcaacta ctacacctcc 1740
agctgctacc tggtaaaccc ccaggccacc acaaacaagg cgaccctgcg ggccatcacc 1800
agcaacgtca gtgccatcct cgcccgcaac gggggcatcg ggctatgcgt gcaggcgttt 1860
aacgactccg gccccgggac cgccagcgtc atgcccgccc tcaaggtcct tgactcgctg 1920
gtggcggcgc acaacaaaga gagcgcgcgt ccgaccggcg cgtgcgtgta cctggagccg 1980
tggcacaccg acgtgcgggc cgtgctccgg atgaaggggg tcctcgccgg cgaagaggcc 2040
cagcgctgcg acaatatctt cagcgccctc tggatgccag acctgttttt caagcgcctg 2100
attcgccacc tggacggcga gaagaacgtc acatggaccc tgttcgaccg ggacaccagc 2160
atgtcgctcg ccgactttca cggggaggag ttcgagaagc tctaccagca cctcgaggtc 2220
atggggttcg gcgagcagat acccatccag gagctggcct atggcattgt gcgcagtgcg 2280
gccacgaccg ggagcccctt cgtcatgttc aaagacgcgg tgaaccgcca ctacatctac 2340
gacacccagg gggcggccat cgccggctcc aacctctgca ccgagatcgt ccatccggcc 2400
tccaagcgat ccagtggggt ctgcaacctg ggaagcgtga atctggcccg atgcgtctcc 2460
aggcagacgt ttgactttgg gcggctccgc gacgccgtgc aggcgtgcgt gctgatggtg 2520
aacatcatga tcgacagcac gctacaaccc acgccccagt gcacccgcgg caacgacaac 2580
ctgcggtcca tgggaatcgg catgcagggc ctgcacacgg cctgcctgaa gctggggctg 2640
gatctggagt ctgtcgaatt tcaggacctg aacaaacaca tcgccgaggt gatgctgctg 2700
tcggcgatga agaccagcaa cgcgctgtgc gttcgcgggg cccgtccctt caaccacttt 2760
aagcgcagca tgtatcgcgc cggccgcttt cactgggagc gctttccgga cgcccggccg 2820
cggtacgagg gcgagtggga gatgctacgc cagagcatga tgaaacacgg cctgcgcaac 2880
agccagtttg tcgcgctgat gcccaccgcc gcctcggcgc agatctcgga cgtcagcgag 2940
ggctttgccc ccctgttcac caacctgttc agcaaggtga cccgggacgg cgagacgctg 3000
cgccccaaca cgctcctgct aaaggaactg gaacgcacgt ttagcgggaa gcgcctcctg 3060
gaggtgatgg acagtctcga cgccaagcag tggtccgtgg cgcaggcgct cccgtgcctg 3120
gagcccaccc accccctccg gcgattcaag accgcgtttg actacgacca gaagttgctg 3180
atcgacctgt gtgcggaccg cgccccctac gtcgaccata gccaatccat gaccctgtat 3240
gtcacggaga aggcggacgg gaccctccca gcctccaccc tggtccgcct tctggtccac 3300
gcatataagc gcggactaaa aacagggatg tactactgca aggttcgcaa ggcgaccaac 3360
agcggggtct ttggcggcga cgacaacatt gtctgcacga gctgcgcgct gtga 3414
<210> 50
<211> 1137
<212> PRT
<213> Herpes simplex virus type 1
<400> 50
Met Ala Ser Arg Pro Ala Ala Ser Ser Pro Val Glu Ala Arg Ala Pro
1 5 10 15
Val Gly Gly Gln Glu Ala Gly Gly Pro Ser Ala Ala Thr Gln Gly Glu
20 25 30
Ala Ala Gly Ala Pro Leu Ala His Gly His His Val Tyr Cys Gln Arg
35 40 45
Val Asn Gly Val Met Val Leu Ser Asp Lys Thr Pro Gly Ser Ala Ser
50 55 60
Tyr Arg Ile Ser Asp Ser Asn Phe Val Gln Cys Gly Ser Asn Cys Thr
65 70 75 80
Met Ile Ile Asp Gly Asp Val Val Arg Gly Arg Pro Gln Asp Pro Gly
85 90 95
Ala Ala Ala Ser Pro Ala Pro Phe Val Ala Val Thr Asn Ile Gly Ala
100 105 110
Gly Ser Asp Gly Gly Thr Ala Val Val Ala Phe Gly Gly Thr Pro Arg
115 120 125
Arg Ser Ala Gly Thr Ser Thr Gly Thr Gln Thr Ala Asp Val Pro Thr
130 135 140
Glu Ala Leu Gly Gly Pro Pro Pro Pro Pro Arg Phe Thr Leu Gly Gly
145 150 155 160
Gly Cys Cys Ser Cys Arg Asp Thr Arg Arg Arg Ser Ala Val Phe Gly
165 170 175
Gly Glu Gly Asp Pro Val Gly Pro Ala Glu Phe Val Ser Asp Asp Arg
180 185 190
Ser Ser Asp Ser Asp Ser Asp Asp Ser Glu Asp Thr Asp Ser Glu Thr
195 200 205
Leu Ser His Ala Ser Ser Asp Val Ser Gly Gly Ala Thr Tyr Asp Asp
210 215 220
Ala Leu Asp Ser Asp Ser Ser Ser Asp Asp Ser Leu Gln Ile Asp Gly
225 230 235 240
Pro Val Cys Arg Pro Trp Ser Asn Asp Thr Ala Pro Leu Asp Val Cys
245 250 255
Pro Gly Thr Pro Gly Pro Gly Ala Asp Ala Gly Gly Pro Ser Ala Val
260 265 270
Asp Pro His Ala Pro Thr Pro Glu Ala Gly Ala Gly Leu Ala Ala Asp
275 280 285
Pro Ala Val Ala Arg Asp Asp Ala Glu Gly Leu Ser Asp Pro Arg Pro
290 295 300
Arg Leu Gly Thr Gly Thr Ala Tyr Pro Val Pro Leu Glu Leu Thr Pro
305 310 315 320
Glu Asn Ala Glu Ala Val Ala Arg Phe Leu Gly Asp Ala Val Asn Arg
325 330 335
Glu Pro Ala Leu Met Leu Glu Tyr Phe Cys Arg Cys Ala Arg Glu Glu
340 345 350
Thr Lys Arg Val Pro Pro Arg Thr Phe Gly Ser Pro Pro Arg Leu Thr
355 360 365
Glu Asp Asp Phe Gly Leu Leu Asn Tyr Ala Leu Val Glu Met Gln Arg
370 375 380
Leu Cys Leu Asp Val Pro Pro Val Pro Pro Asn Ala Tyr Met Pro Tyr
385 390 395 400
Tyr Leu Arg Glu Tyr Val Thr Arg Leu Val Asn Gly Phe Lys Pro Leu
405 410 415
Val Ser Arg Ser Ala Arg Leu Tyr Arg Ile Leu Gly Val Leu Val His
420 425 430
Leu Arg Ile Arg Thr Arg Glu Ala Ser Phe Glu Glu Trp Leu Arg Ser
435 440 445
Lys Glu Val Ala Leu Asp Phe Gly Leu Thr Glu Arg Leu Arg Glu His
450 455 460
Glu Ala Gln Leu Val Ile Leu Ala Gln Ala Leu Asp His Tyr Asp Cys
465 470 475 480
Leu Ile His Ser Thr Pro His Thr Leu Val Glu Arg Gly Leu Gln Ser
485 490 495
Ala Leu Lys Tyr Glu Glu Phe Tyr Leu Lys Arg Phe Gly Gly His Tyr
500 505 510
Met Glu Ser Val Phe Gln Met Tyr Thr Arg Ile Ala Gly Phe Leu Ala
515 520 525
Cys Arg Ala Thr Arg Gly Met Arg His Ile Ala Leu Gly Arg Glu Gly
530 535 540
Ser Trp Trp Glu Met Phe Lys Phe Phe Phe His Arg Leu Tyr Asp His
545 550 555 560
Gln Ile Val Pro Ser Thr Pro Ala Met Leu Asn Leu Gly Thr Arg Asn
565 570 575
Tyr Tyr Thr Ser Ser Cys Tyr Leu Val Asn Pro Gln Ala Thr Thr Asn
580 585 590
Lys Ala Thr Leu Arg Ala Ile Thr Ser Asn Val Ser Ala Ile Leu Ala
595 600 605
Arg Asn Gly Gly Ile Gly Leu Cys Val Gln Ala Phe Asn Asp Ser Gly
610 615 620
Pro Gly Thr Ala Ser Val Met Pro Ala Leu Lys Val Leu Asp Ser Leu
625 630 635 640
Val Ala Ala His Asn Lys Glu Ser Ala Arg Pro Thr Gly Ala Cys Val
645 650 655
Tyr Leu Glu Pro Trp His Thr Asp Val Arg Ala Val Leu Arg Met Lys
660 665 670
Gly Val Leu Ala Gly Glu Glu Ala Gln Arg Cys Asp Asn Ile Phe Ser
675 680 685
Ala Leu Trp Met Pro Asp Leu Phe Phe Lys Arg Leu Ile Arg His Leu
690 695 700
Asp Gly Glu Lys Asn Val Thr Trp Thr Leu Phe Asp Arg Asp Thr Ser
705 710 715 720
Met Ser Leu Ala Asp Phe His Gly Glu Glu Phe Glu Lys Leu Tyr Gln
725 730 735
His Leu Glu Val Met Gly Phe Gly Glu Gln Ile Pro Ile Gln Glu Leu
740 745 750
Ala Tyr Gly Ile Val Arg Ser Ala Ala Thr Thr Gly Ser Pro Phe Val
755 760 765
Met Phe Lys Asp Ala Val Asn Arg His Tyr Ile Tyr Asp Thr Gln Gly
770 775 780
Ala Ala Ile Ala Gly Ser Asn Leu Cys Thr Glu Ile Val His Pro Ala
785 790 795 800
Ser Lys Arg Ser Ser Gly Val Cys Asn Leu Gly Ser Val Asn Leu Ala
805 810 815
Arg Cys Val Ser Arg Gln Thr Phe Asp Phe Gly Arg Leu Arg Asp Ala
820 825 830
Val Gln Ala Cys Val Leu Met Val Asn Ile Met Ile Asp Ser Thr Leu
835 840 845
Gln Pro Thr Pro Gln Cys Thr Arg Gly Asn Asp Asn Leu Arg Ser Met
850 855 860
Gly Ile Gly Met Gln Gly Leu His Thr Ala Cys Leu Lys Leu Gly Leu
865 870 875 880
Asp Leu Glu Ser Val Glu Phe Gln Asp Leu Asn Lys His Ile Ala Glu
885 890 895
Val Met Leu Leu Ser Ala Met Lys Thr Ser Asn Ala Leu Cys Val Arg
900 905 910
Gly Ala Arg Pro Phe Asn His Phe Lys Arg Ser Met Tyr Arg Ala Gly
915 920 925
Arg Phe His Trp Glu Arg Phe Pro Asp Ala Arg Pro Arg Tyr Glu Gly
930 935 940
Glu Trp Glu Met Leu Arg Gln Ser Met Met Lys His Gly Leu Arg Asn
945 950 955 960
Ser Gln Phe Val Ala Leu Met Pro Thr Ala Ala Ser Ala Gln Ile Ser
965 970 975
Asp Val Ser Glu Gly Phe Ala Pro Leu Phe Thr Asn Leu Phe Ser Lys
980 985 990
Val Thr Arg Asp Gly Glu Thr Leu Arg Pro Asn Thr Leu Leu Leu Lys
995 1000 1005
Glu Leu Glu Arg Thr Phe Ser Gly Lys Arg Leu Leu Glu Val Met
1010 1015 1020
Asp Ser Leu Asp Ala Lys Gln Trp Ser Val Ala Gln Ala Leu Pro
1025 1030 1035
Cys Leu Glu Pro Thr His Pro Leu Arg Arg Phe Lys Thr Ala Phe
1040 1045 1050
Asp Tyr Asp Gln Lys Leu Leu Ile Asp Leu Cys Ala Asp Arg Ala
1055 1060 1065
Pro Tyr Val Asp His Ser Gln Ser Met Thr Leu Tyr Val Thr Glu
1070 1075 1080
Lys Ala Asp Gly Thr Leu Pro Ala Ser Thr Leu Val Arg Leu Leu
1085 1090 1095
Val His Ala Tyr Lys Arg Gly Leu Lys Thr Gly Met Tyr Tyr Cys
1100 1105 1110
Lys Val Arg Lys Ala Thr Asn Ser Gly Val Phe Gly Gly Asp Asp
1115 1120 1125
Asn Ile Val Cys Thr Ser Cys Ala Leu
1130 1135
<210> 51
<211> 747
<212> DNA
<213> Herpes simplex virus type 1
<400> 51
atggcccgcc gccgccgcca tcgcggcccc cgccgccccc ggccgcccgg gcccacgggc 60
gccgtcccaa ccgcacagtc ccaggtaacc tccacgccca actcggaacc cgcggtcagg 120
agcgcgcccg cggccgcccc gccgccgccc cccgccggtg ggcccccgcc ttcttgttcg 180
ctgctgctgc gccagtggct ccacgttccc gagtccgcgt ccgacgacga cgatgacgac 240
gactggccgg acagcccccc gcccgagccg gcgccagagg cccggcccac cgccgccgcc 300
ccccggcccc ggcccccacc gcccggcgtg ggcccggggg gcggggctga cccctcccac 360
cccccctcgc gccccttccg ccttccgccg cgcctcgccc tccgcctgcg cgtcaccgcg 420
gagcacctgg cgcgcctgcg cctgcgacgc gcgggcgggg agggggcgcc ggagcccccc 480
gcgacccccg cgacccccgc gacccccgcg acccccgcga cccccgcgcg ggtgcgcttc 540
tcgccccacg tccgggtgcg ccacctggtg gtctgggcct cggccgcccg cctggcgcgc 600
cgcggctcgt gggcccgcga gcgggccgac cgggctcggt tccggcgccg ggtggcggag 660
gccgaggcgg tcatcgggcc gtgcctgggg cccgaggccc gtgcccgggc cctggcccgc 720
ggagccggcc cggcgaactc ggtctaa 747
<210> 52
<211> 248
<212> PRT
<213> Herpes simplex virus type 1
<400> 52
Met Ala Arg Arg Arg Arg His Arg Gly Pro Arg Arg Pro Arg Pro Pro
1 5 10 15
Gly Pro Thr Gly Ala Val Pro Thr Ala Gln Ser Gln Val Thr Ser Thr
20 25 30
Pro Asn Ser Glu Pro Ala Val Arg Ser Ala Pro Ala Ala Ala Pro Pro
35 40 45
Pro Pro Pro Ala Gly Gly Pro Pro Pro Ser Cys Ser Leu Leu Leu Arg
50 55 60
Gln Trp Leu His Val Pro Glu Ser Ala Ser Asp Asp Asp Asp Asp Asp
65 70 75 80
Asp Trp Pro Asp Ser Pro Pro Pro Glu Pro Ala Pro Glu Ala Arg Pro
85 90 95
Thr Ala Ala Ala Pro Arg Pro Arg Pro Pro Pro Pro Gly Val Gly Pro
100 105 110
Gly Gly Gly Ala Asp Pro Ser His Pro Pro Ser Arg Pro Phe Arg Leu
115 120 125
Pro Pro Arg Leu Ala Leu Arg Leu Arg Val Thr Ala Glu His Leu Ala
130 135 140
Arg Leu Arg Leu Arg Arg Ala Gly Gly Glu Gly Ala Pro Glu Pro Pro
145 150 155 160
Ala Thr Pro Ala Thr Pro Ala Thr Pro Ala Thr Pro Ala Thr Pro Ala
165 170 175
Arg Val Arg Phe Ser Pro His Val Arg Val Arg His Leu Val Val Trp
180 185 190
Ala Ser Ala Ala Arg Leu Ala Arg Arg Gly Ser Trp Ala Arg Glu Arg
195 200 205
Ala Asp Arg Ala Arg Phe Arg Arg Arg Val Ala Glu Ala Glu Ala Val
210 215 220
Ile Gly Pro Cys Leu Gly Pro Glu Ala Arg Ala Arg Ala Leu Ala Arg
225 230 235 240
Gly Ala Gly Pro Ala Asn Ser Val
245
<210> 53
<211> 3229
<212> DNA
<213> Herpes simplex virus type 1
<220>
<221> Intron
<222> (58)..(822)
<223> intron
<220>
<221> Intron
<222> (1490)..(1625)
<223> intron
<400> 53
atggagcccc gccccggagc gagtacccgc cggcctgagg gccgccccca gcgcgaggtg 60
aggggccggg cgccatgtct ggggcgccat attggggggc gccatattgg ggggcgccat 120
gttgggggac ccccgaccct tacactggaa ccggccgcca tgttggggga cccccactca 180
tacacgggag ccgggcgcca tgttggggcg ccatgttagg gggcgtggaa ccccgtgaca 240
ctatatatac agggaccggg ggcgccatgt tagggggtgc ggaaccccct gaccctatat 300
atacagggac cggggtcgcc ctgttggggg tcgccatgtg accccctgac tttatatata 360
cagaccccca acacatacac atggcccctt tgactcagac gcagggcccg gggtcgccgt 420
gggaccccct gactcataca cagagacacg cccccacaac aaacacacag ggaccggggt 480
cgccgtgttg ggggcgtggt ccccactgac tcatacgcag gcccccctta ctcacacgca 540
tctagggggg tggggaggag ccgcccgcca tatttggggg acgccgtggg acccccgact 600
ccggtgcgtc tggagggcgg gagaagaggg aagaagaggg gtcgggatcc aaaggacgga 660
cccagaccac ctttggttgc agaccccttt ctcccccctc ttccgaggcc agcagggggg 720
caggactttg tgaggcgggg gggggagagg gggaactcgt gggtgctgat tgacgcggga 780
aatccccccc cattcttacc cgcccccctt ttttcccctt agcccgcccc ggatgtctgg 840
gtgtttccct gcgaccgaga cctgccggac agcagcgact ctgaggcgga gaccgaagtg 900
ggggggcggg gggacgccga ccaccatgac gacgactccg cctccgaggc ggacagcacg 960
gacacggaac tgttcgagac ggggctgctg gggccgcagg gcgtggatgg gggggcggtc 1020
tcggggggga gccccccccg cgaggaagac cccggcagtt gcgggggcgc cccccctcga 1080
gaggacgggg ggagcgacga gggcgacgtg tgcgccgtgt gcacggatga gatcgcgccc 1140
cacctgcgct gcgacacctt cccgtgcatg caccgcttct gcatcccgtg catgaaaacc 1200
tggatgcaat tgcgcaacac ctgcccgctg tgcaacgcca agctggtgta cctgatagtg 1260
ggcgtgacgc ccagcgggtc gttcagcacc atcccgatcg tgaacgaccc ccagacccgc 1320
atggaggccg aggaggccgt cagggcgggc acggccgtgg actttatctg gacgggcaat 1380
cagcggttcg ccccgcggta cctgaccctg ggggggcaca cggtgagggc cctgtcgccc 1440
acccacccgg agcccaccac ggacgaggat gacgacgacc tggacgacgg tgaggcgggg 1500
ggcggcaagg accctggggg aggaggagga ggaggggggg ggagggagga ataggcgggc 1560
gggcgaggaa agggcgggcc ggggaggggg cgtaacctga tcgcgccccc cgttgtctct 1620
tgcagcagac tacgtaccgc ccgccccccg ccggacgccc cgcgcccccc cacgcagagg 1680
cgccgccgcg ccccccgtga cgggcggggc gtctcacgca gccccccagc cggccgcggc 1740
tcggacagcg cccccctcgg cgcccatcgg gccacacggc agcagtaaca ccaacaccac 1800
caccaacagc agcggcggcg gcggctcccg ccagtcgcga gccgcggcgc cgcggggggc 1860
gtctggcccc tccggggggg ttggggttgg ggttggggtt gttgaagcgg aggcggggcg 1920
gccgaggggc cggacgggcc cccttgtcaa cagacccgcc ccccttgcaa acaacagaga 1980
ccccatagtg atcagcgact cccccccggc ctctccccac aggccccccg cggcgcccat 2040
gccaggctcc gccccccgcc ccgggccccc cgcgtccgcg gccgcgtcgg gacccgcgcg 2100
cccccgcgcg gccgtggccc cgtgcgtgcg agcgccgcct ccggggcccg gcccccgcgc 2160
cccggccccc ggggcggagc cggccgcccg ccccgcggac gcgcgccgtg tgccccagtc 2220
gcactcgtcc ctggctcagg ccgcgaacca agaacagagt ctgtgccggg cgcgtgcgac 2280
ggtggcgcgc ggctcggggg ggccgggcgt ggagggtggg cacgggccct cccgcggcgc 2340
cgccccctcc ggcgccgccc cgctcccctc cgccgcctct gtcgagcagg aggcggcggt 2400
gcgtccgagg aagaggcgcg ggtcgggcca ggaaaacccc tccccccagt ccacgcgtcc 2460
ccccctcgcg ccggcagggg ccaagagggc ggcgacgcac cccccctccg actcagggcc 2520
gggggggcgc ggccagggtg ggcccgggac ccccctgacg tcctcggcgg cctccgcctc 2580
ttcctcctct gcctcttcct cctcggcccc gacccccgcg ggggccgcct cttccgccgc 2640
cggggccgcg tcctcctccg cttccgcctc ctcgggcggg gccgtcggtg ccctgggagg 2700
gagacaagag gaaacctccc tcggcccccg cgctgcttct gggccgcggg ggccgaggaa 2760
gtgtgcccgg aagacgcgcc acgcggagac ttccggggcc gtccccgcgg gcggcctcac 2820
gcgctacctg cccatctcgg gggtctctag cgtggtcgcc ctgtcgcctt acgtgaacaa 2880
gactatcacg ggggactgcc tgcccatcct ggacatggag acggggaaca tcggggcgta 2940
cgtggtcctg gtggaccaga cgggaaacat ggcgacccgg ctgcgggccg cggtccccgg 3000
ctggagccgc cgcaccctgc tccccgagac cgcgggtaac cacgtgatgc cccccgagta 3060
cccgacggcc cccgcgtcgg agtggaacag cctctggatg acccccgtgg ggaacatgct 3120
gttcgaccag ggcaccctag tgggcgccct ggacttccgc agcctgcggt ctcggcaccc 3180
gtggtccggg gagcaggggg cgtcgacccg ggacgaggga aaacaataa 3229
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| УЛУЧШЕННЫЙ СИНТЕТИЧЕСКИЙ Т-КЛЕТОЧНЫЙ РЕЦЕПТОР И АНТИГЕННЫЙ РЕЦЕПТОР | 2021 |
|
RU2820497C1 |
| T-КЛЕТОЧНЫЕ РЕЦЕПТОРЫ, РАСПОЗНАЮЩИЕ МУТАЦИЮ R175H ИЛИ Y220C В P53 | 2020 |
|
RU2830061C2 |
| НЕДЕСТРУКТИВНАЯ ГЕННАЯ ТЕРАПИЯ ДЛЯ ЛЕЧЕНИЯ MMA | 2018 |
|
RU2820602C2 |
| ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР | 2018 |
|
RU2770002C2 |
| СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ ПРЕДУПРЕЖДЕНИЯ ИЛИ ЛЕЧЕНИЯ ЗЛОКАЧЕСТВЕННОЙ ОПУХОЛИ | 2016 |
|
RU2718499C2 |
| Выделенная нуклеиновая кислота, которая кодирует слитый белок на основе FVIII-BDD и гетерологичного сигнального пептида, и ее применение | 2022 |
|
RU2818229C2 |
| АНТИ-CLL1-СПЕЦИФИЧЕСКИЕ ОДНОЦЕПОЧЕЧНЫЕ ХИМЕРНЫЕ АНТИГЕННЫЕ РЕЦЕПТОРЫ (scCAR) ДЛЯ ИММУНОТЕРАПИИ РАКА | 2016 |
|
RU2731543C2 |
| МНОГОФУНКЦИОНАЛЬНЫЕ СЛИТЫЕ БЕЛКИ И ИХ ПРИМЕНЕНИЯ | 2020 |
|
RU2815388C2 |
| РЕКОМБИНАНТНЫЕ ВЕКТОРЫ, ЭКСПРЕССИРУЮЩИЕ АНТИГЕНЫ ВИРУСА ПТИЧЬЕГО ГРИППА, И ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2761869C2 |
| Конструкции для направленного редактирования генов и способы с их применением | 2020 |
|
RU2832109C2 |
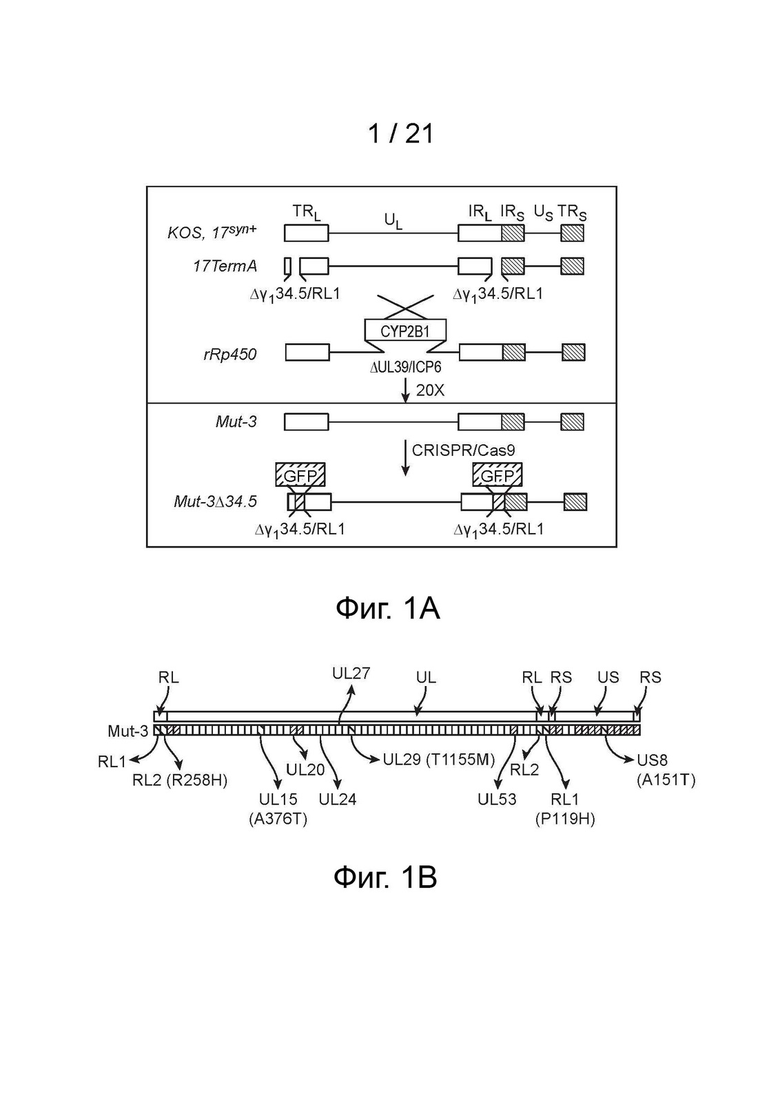
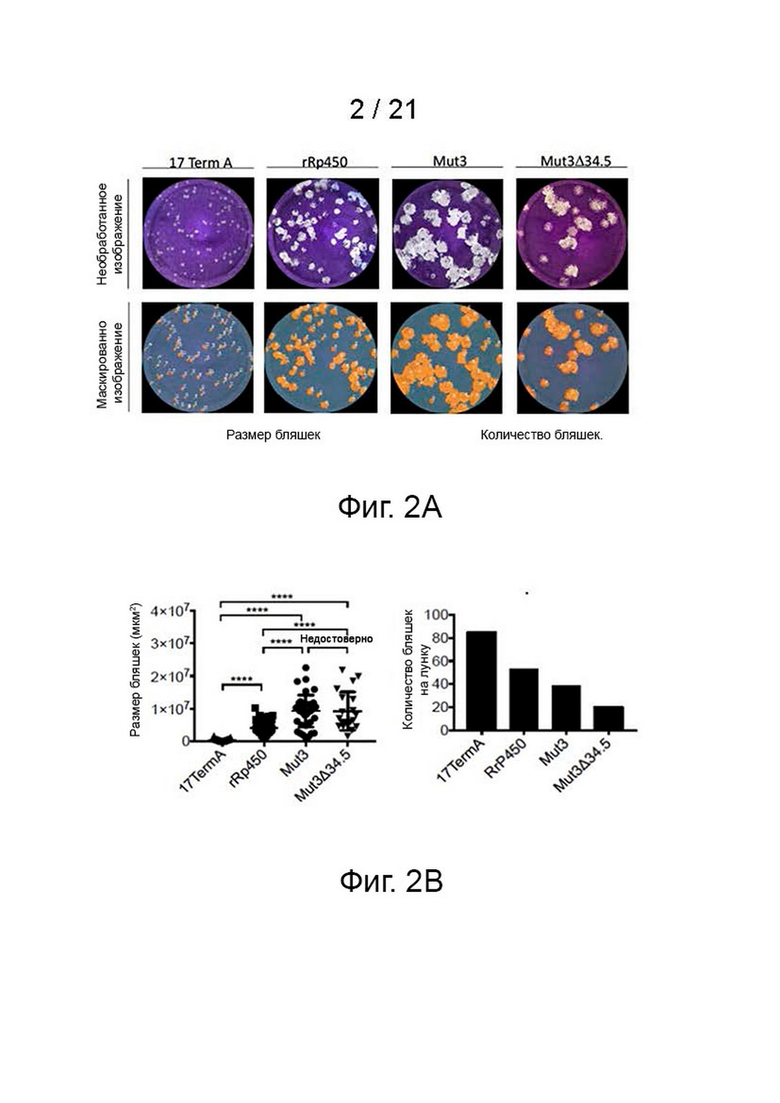
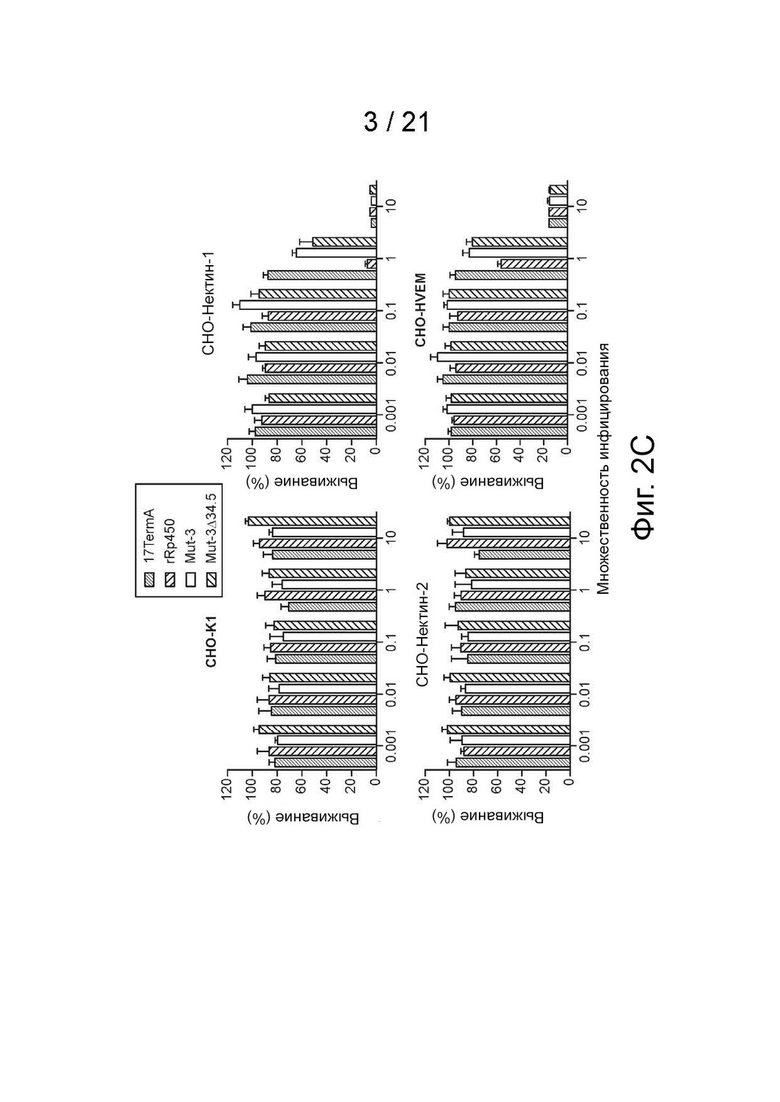
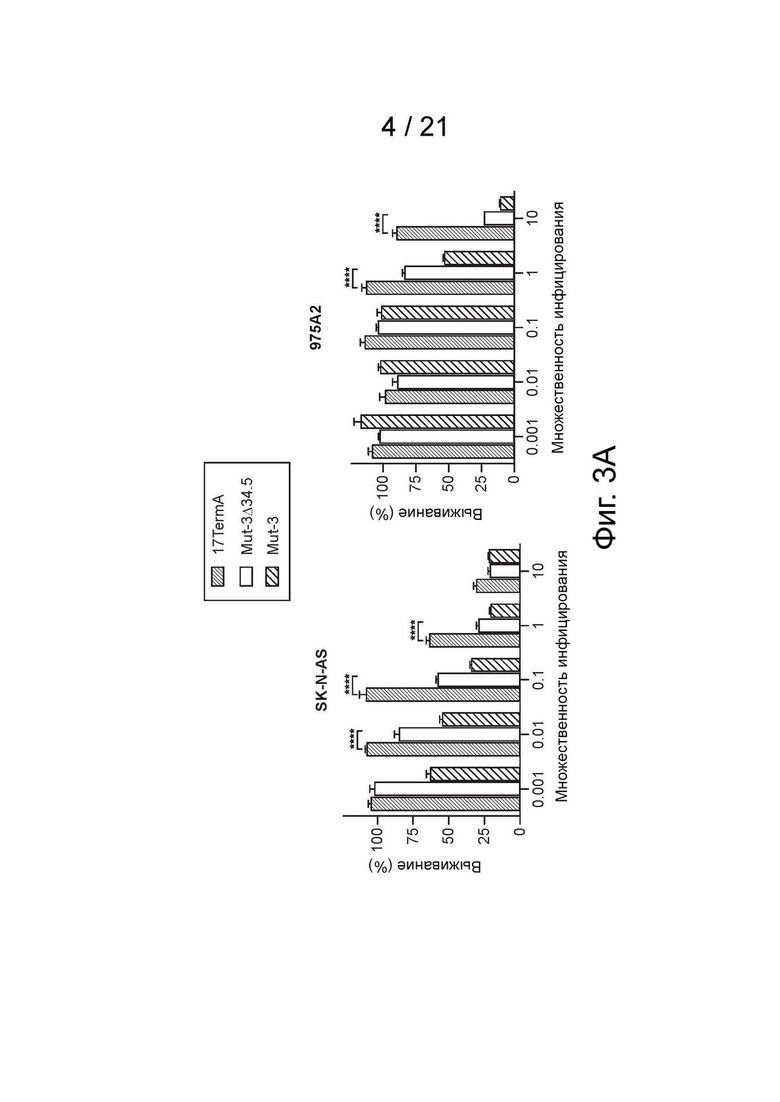
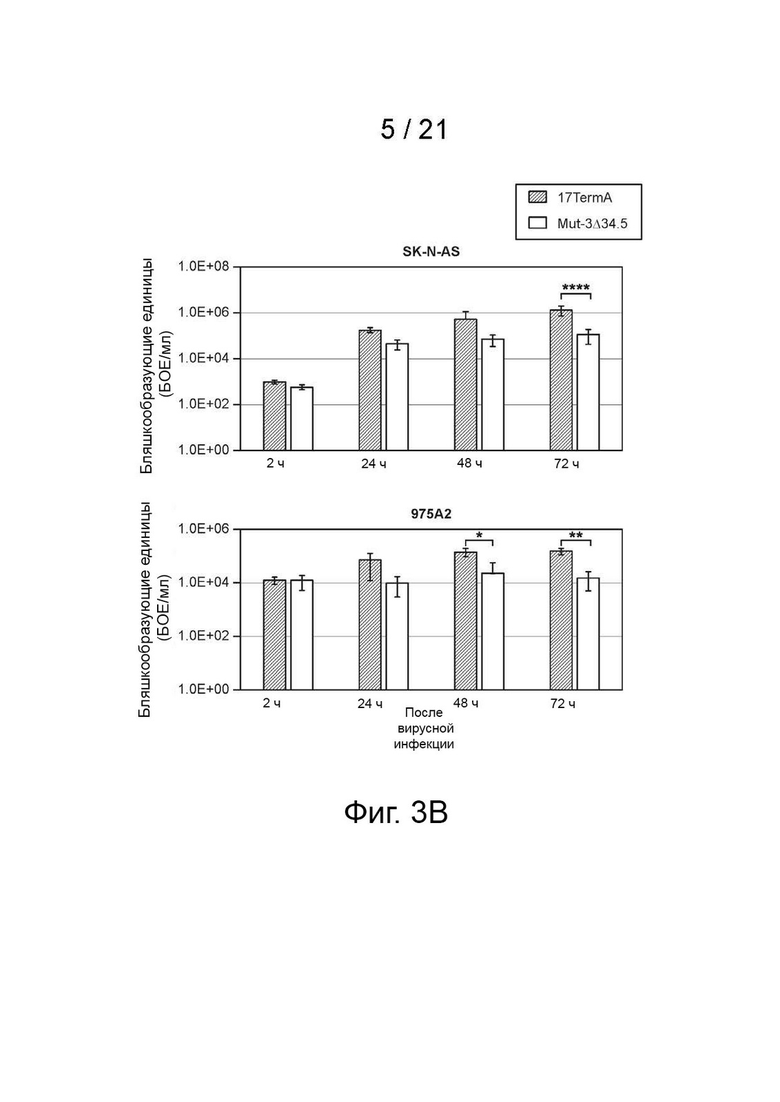
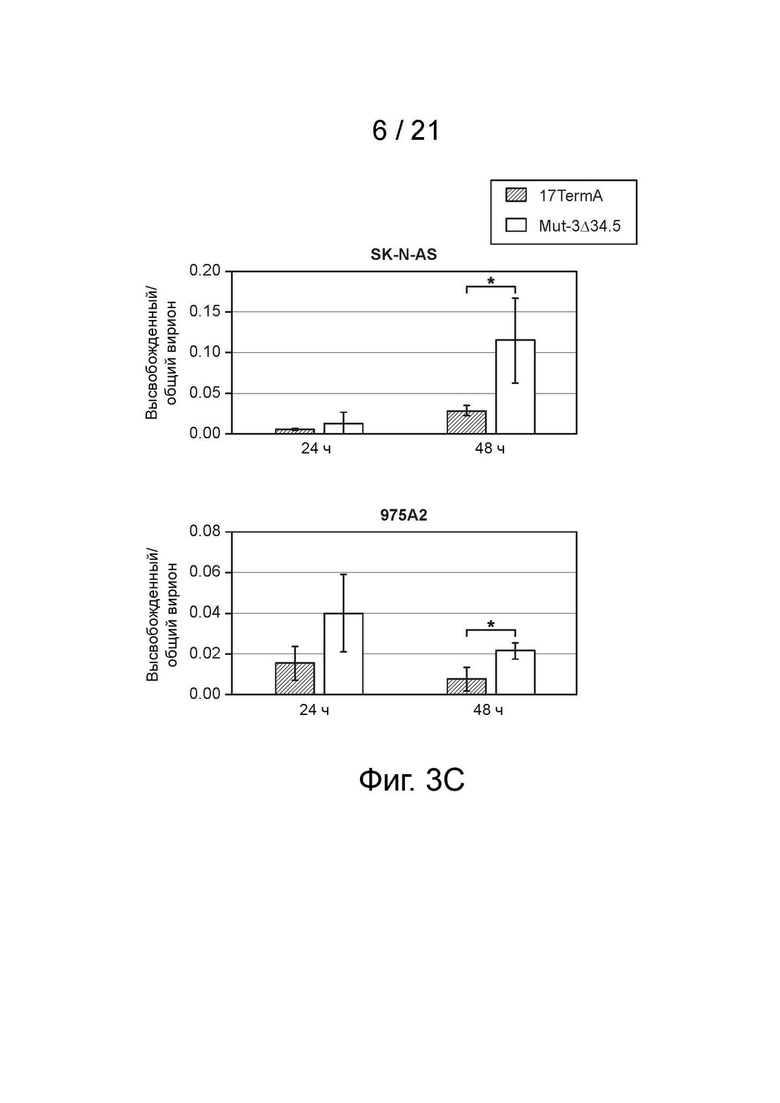
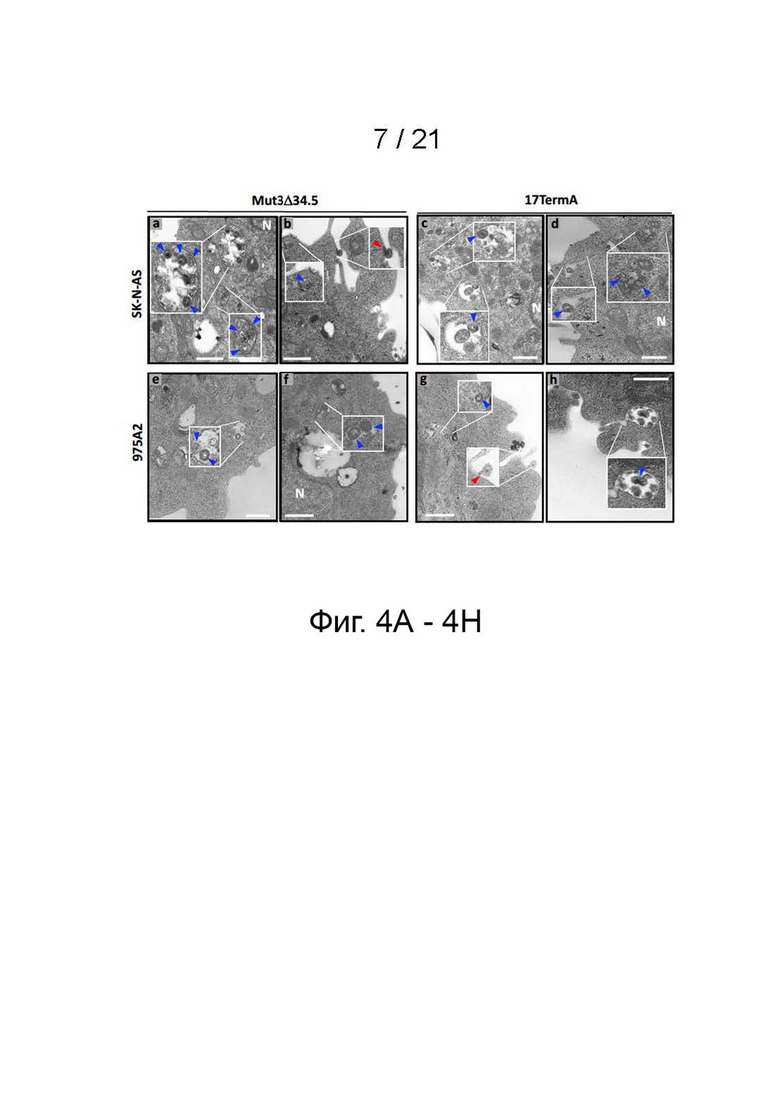
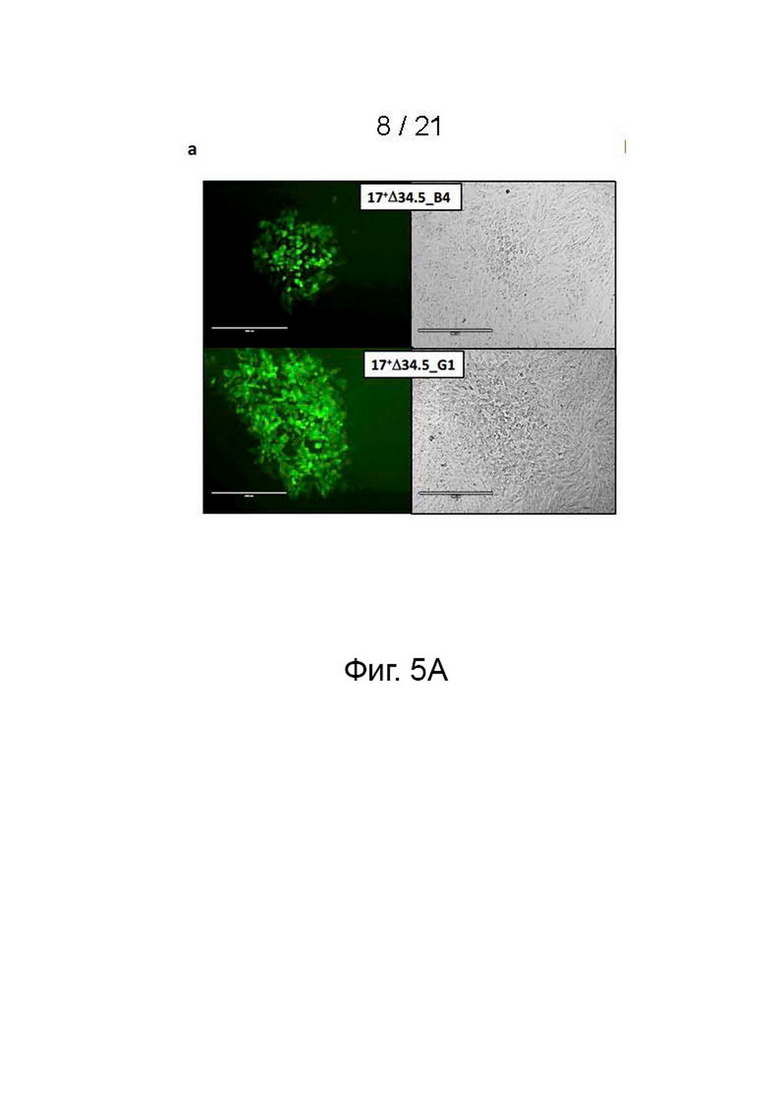
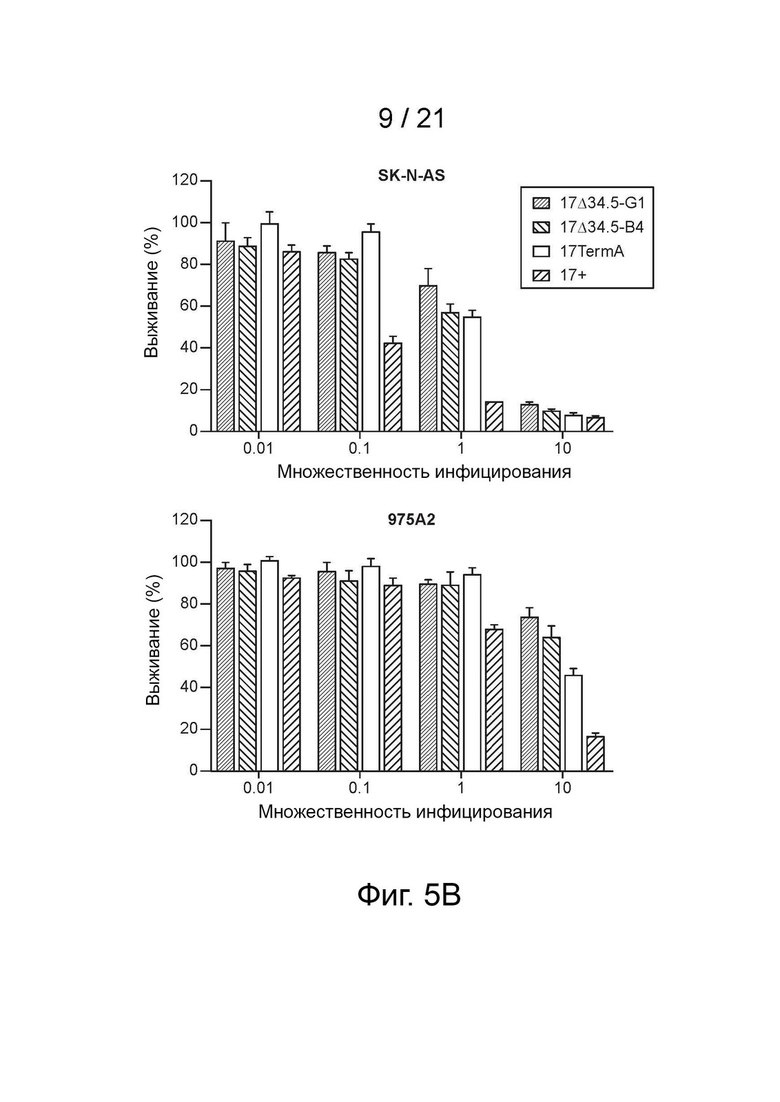
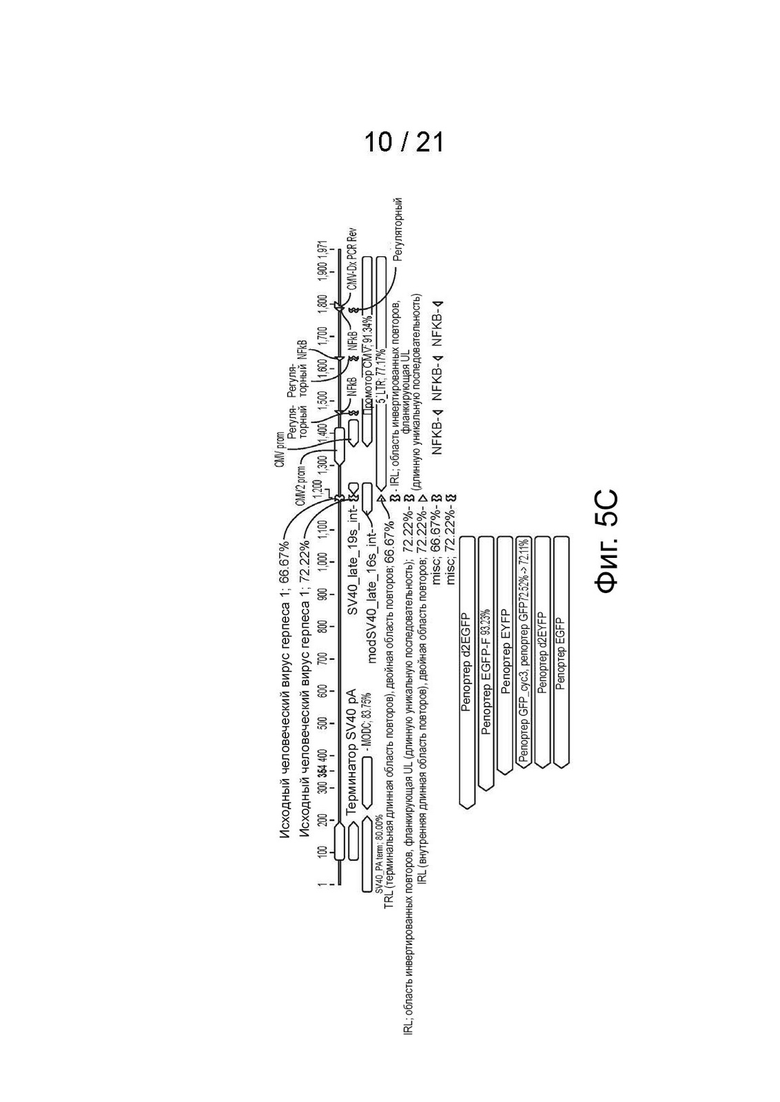


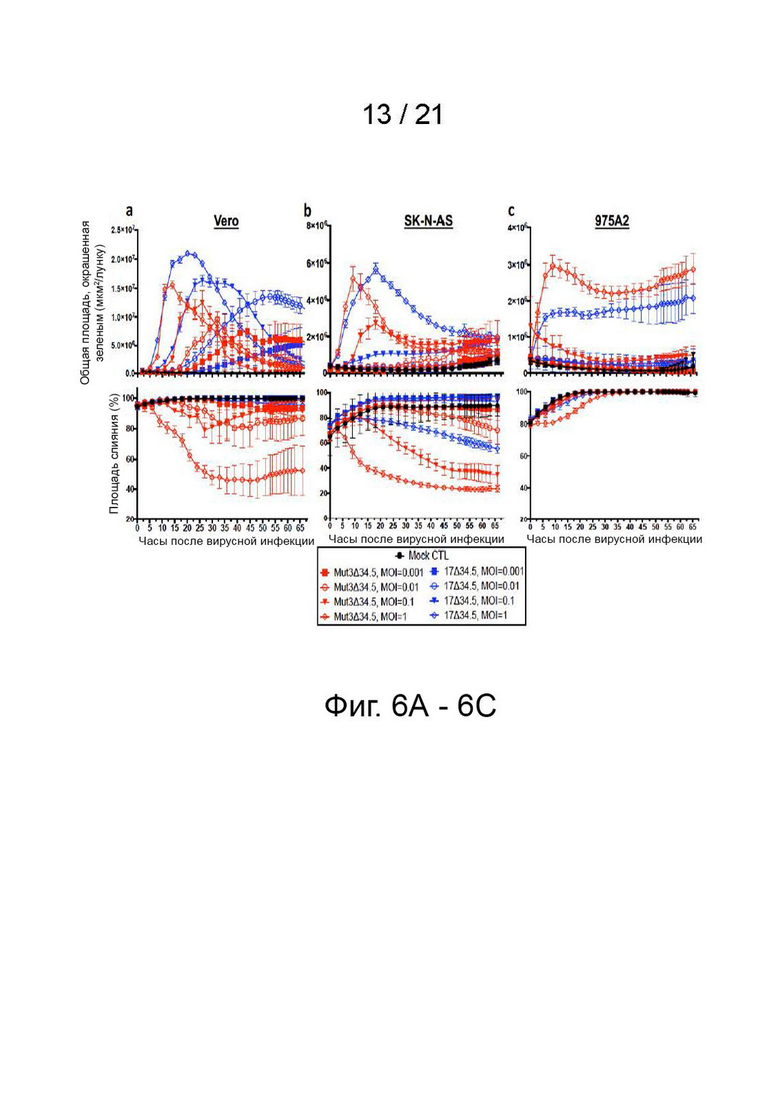
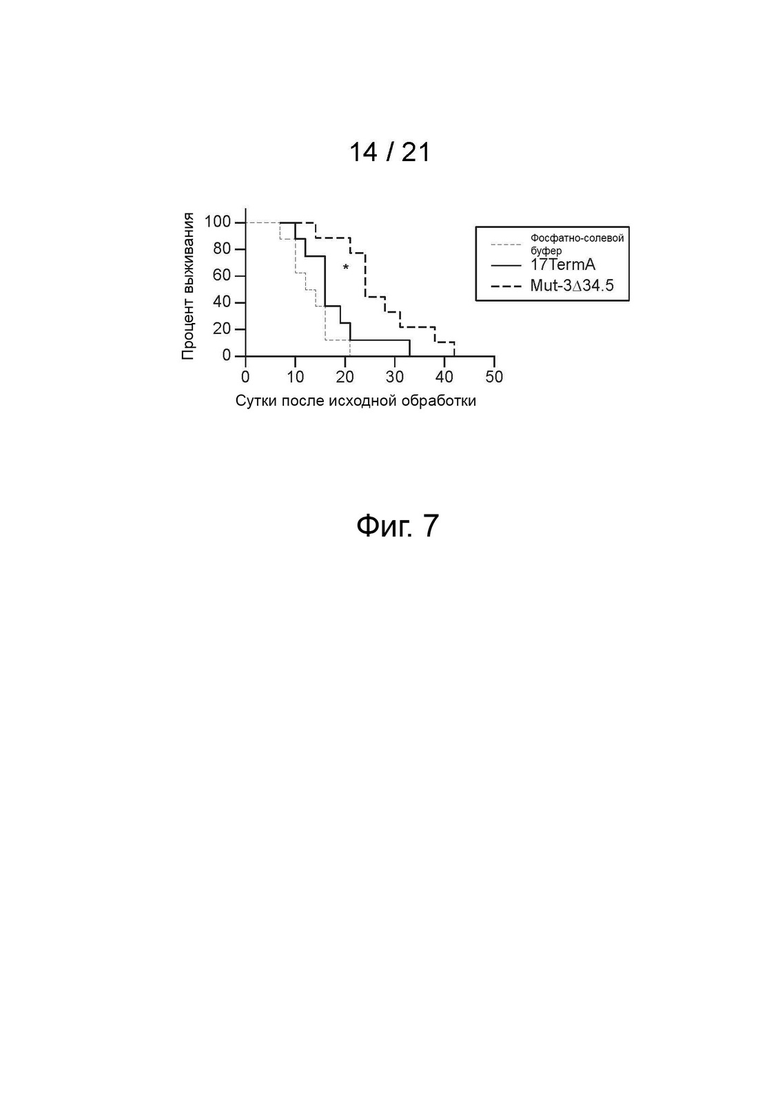
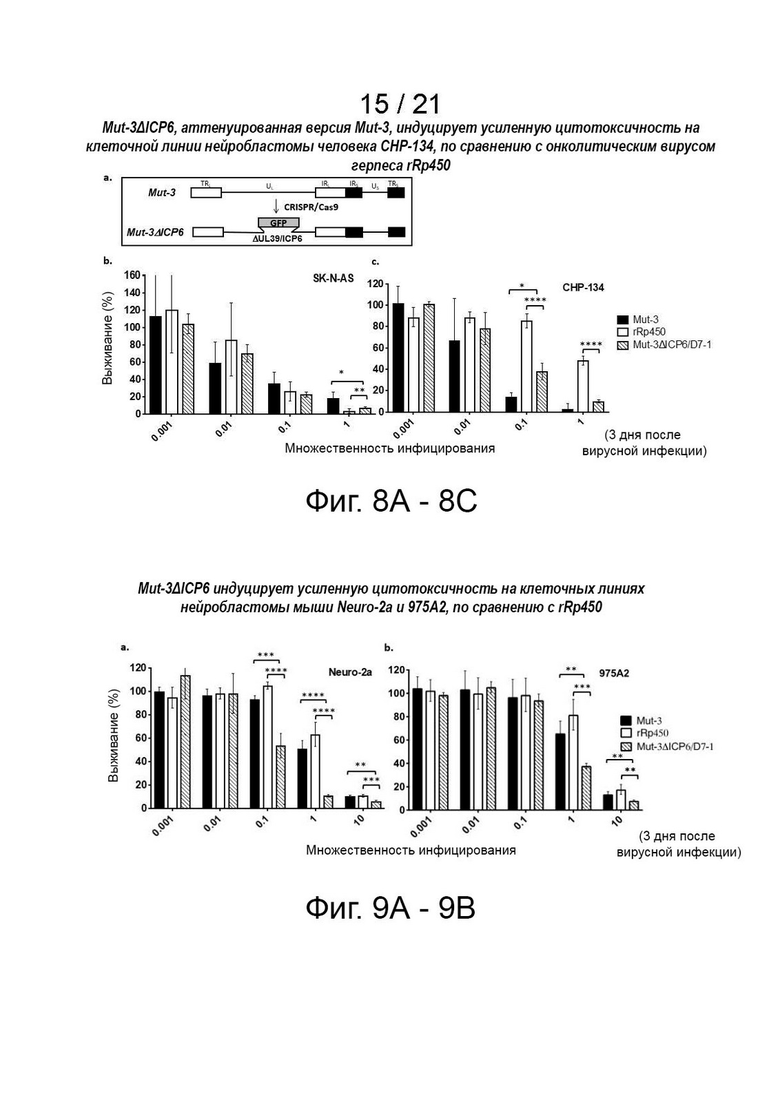
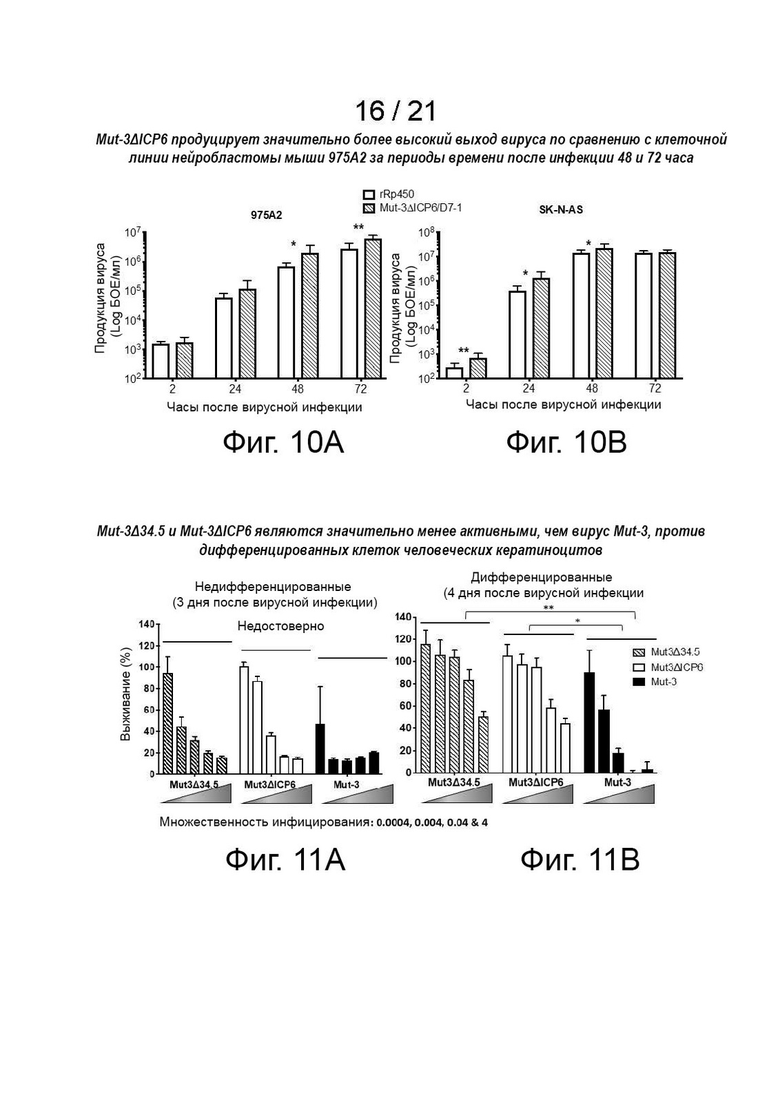
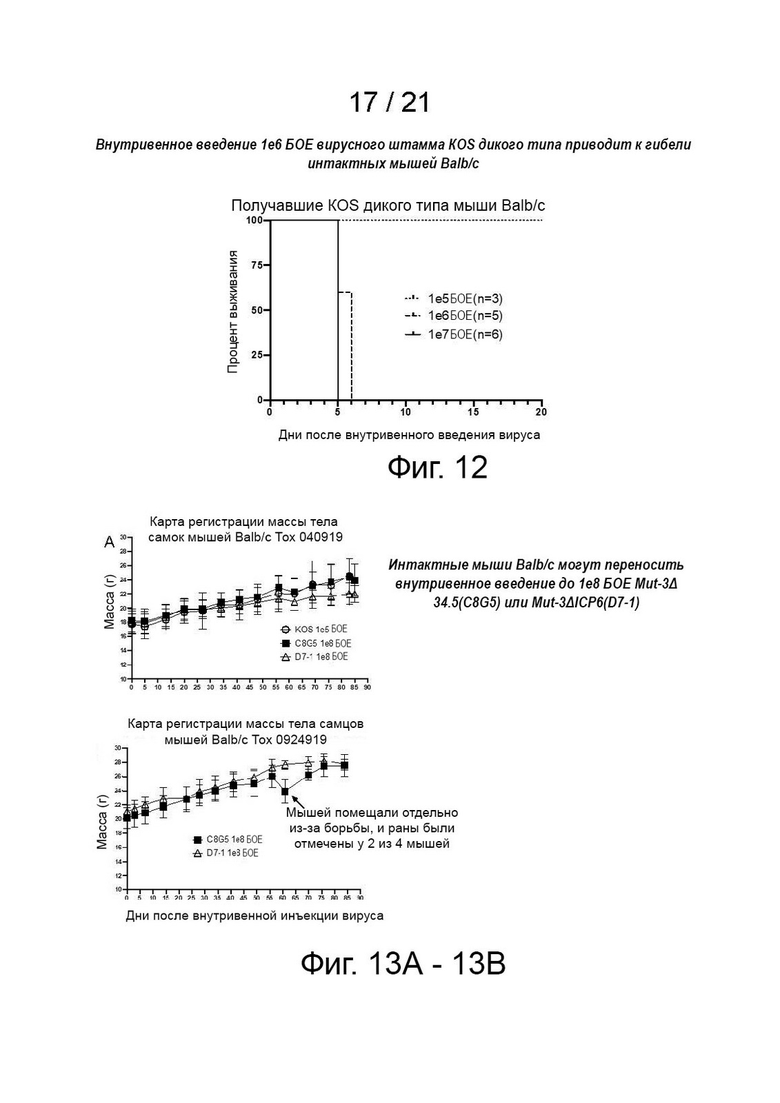
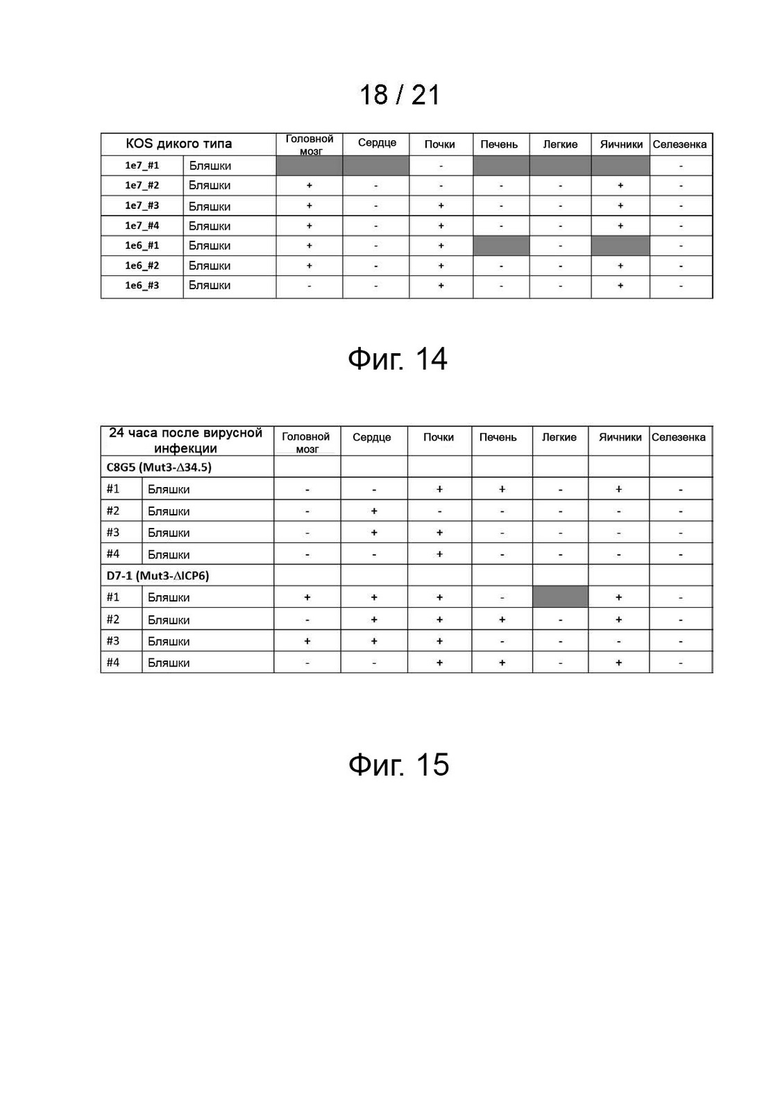
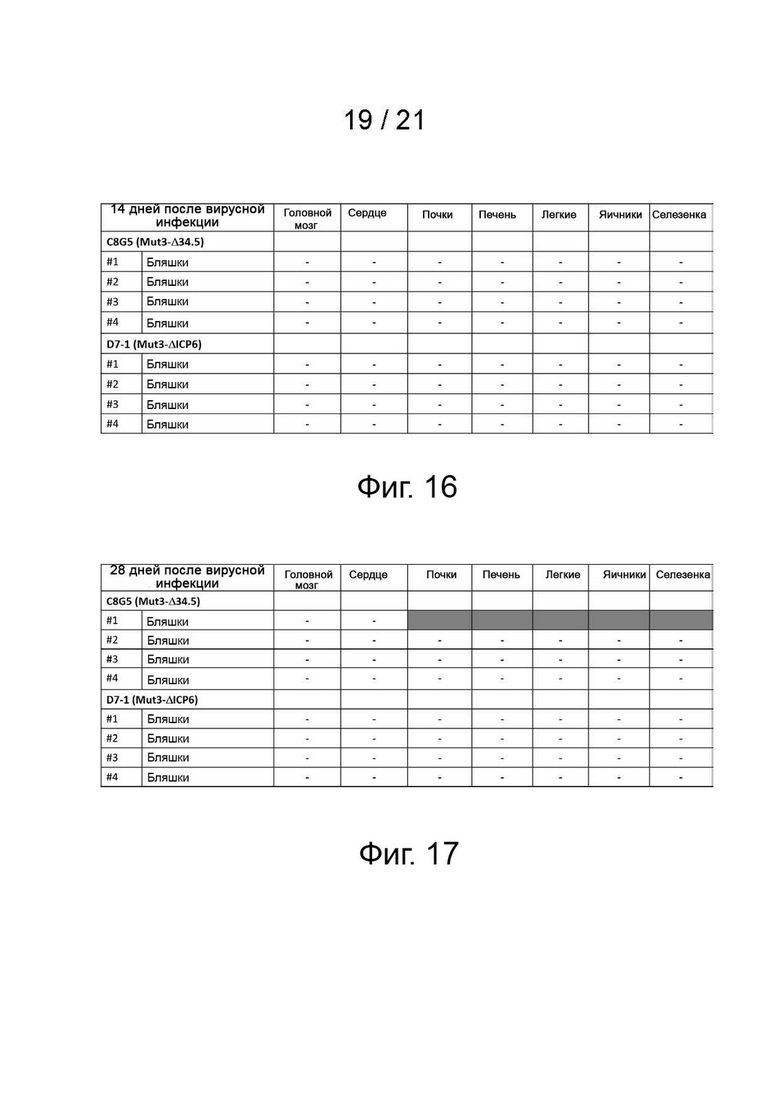
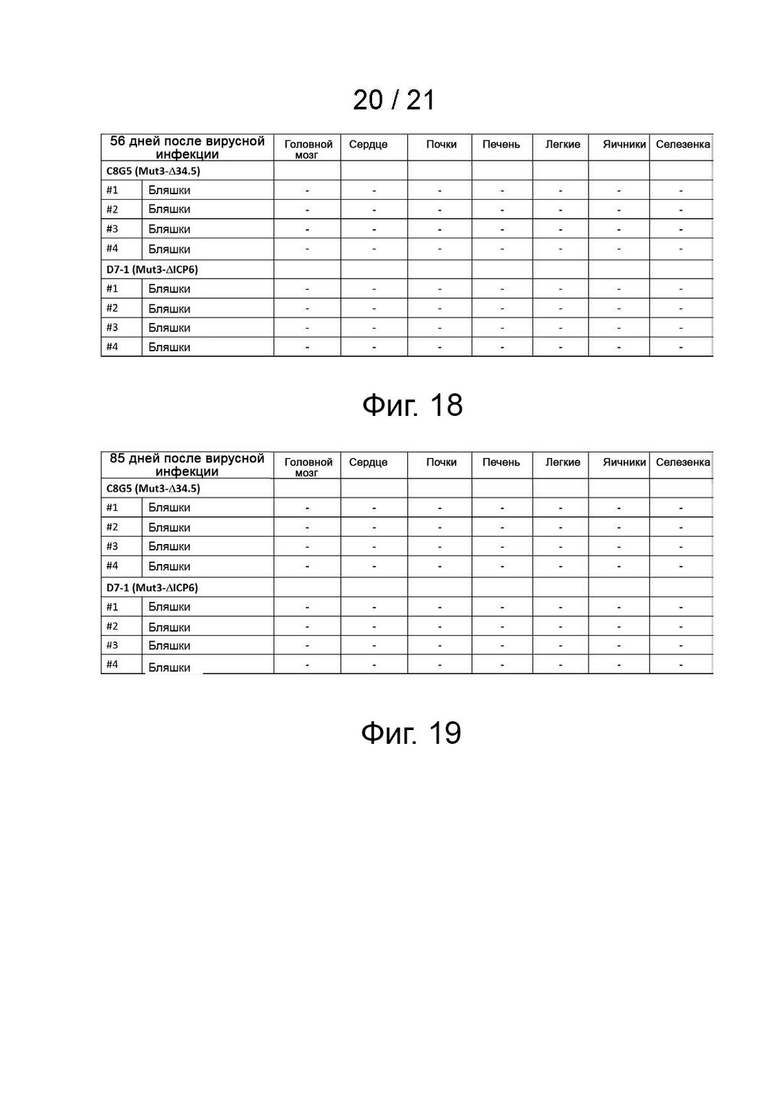
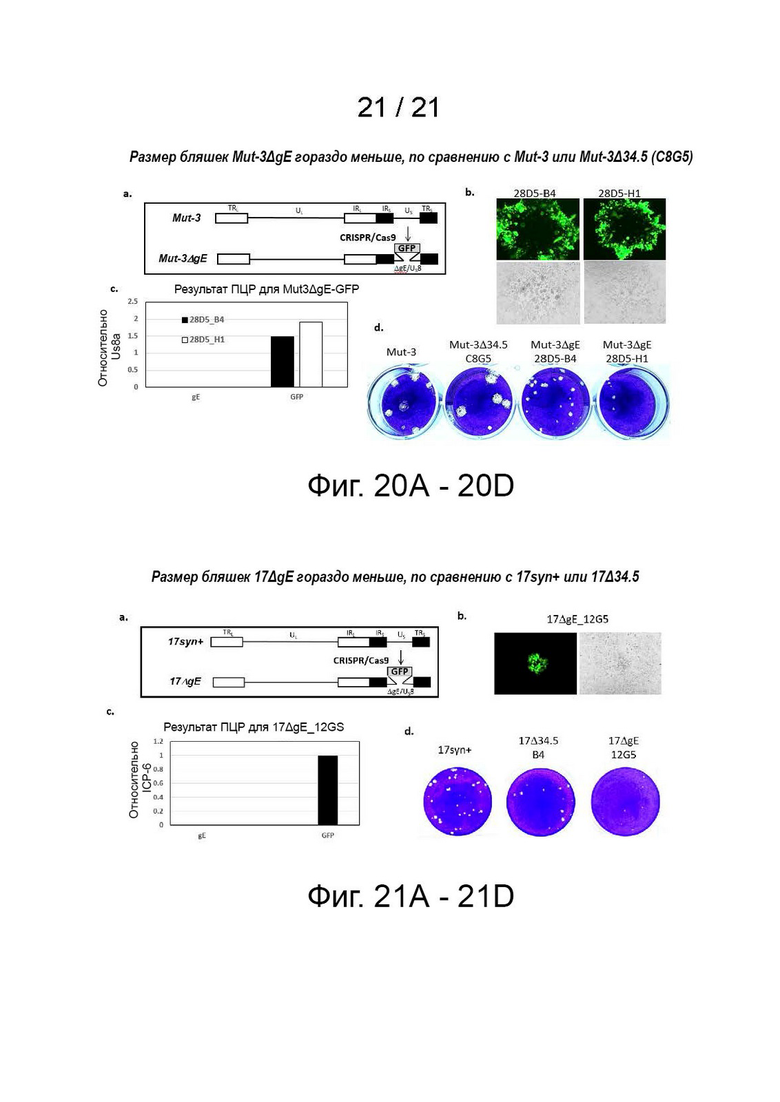
Изобретение относится к биотехнологии. Описан неприродный вирус простого герпеса («HSV») для лечения рака, содержащий одну или несколько мутаций в гене, кодирующем гликопротеин E («gE»), причем мутация кодирует мутацию аланина в треонин в положении 151 белка gE. Также описана композиция для лечения рака, содержащая неприродный HSV и носитель, такой как фармацевтически приемлемый носитель. Раскрыто не относящееся к человеку млекопитающее для лечения рака, инфицированное неприродным HSV. Описана клетка для лечения рака, инфицированная неприродным HSV. Раскрыт способ инфицирования клетки или индукции клеточного лизиса, включающий обеспечение контакта клетки с неприродным HSV или композицией. Раскрыт способ получения вирусного вектора, включающий введение трансгена в неприродный HSV. Описан способ ингибирования роста или метастазирования раковой клетки, включающий обеспечение контакта клетки с эффективным количеством неприродного HSV или композиции. Раскрыт способ лечения рака у субъекта, причем способ включает введение эффективного количества неприродного HSV или композиции субъекту. Описан способ получения HSV, включающий (а) введение в клетку-хозяина 17TermA HSV и rRp450 HSV; (b) выращивание клетки-хозяина по меньшей мере за 3 пассажа; и (c) выделение частицы HSV, продуцируемой клеткой-хозяином. Представлена частица HSV для лечения рака, полученная способом. Представлен способ получения HSV по любому из пп. 1-5, включающий: (а) введение в клетку-хозяина полинуклеотида, кодирующего вирусный геном HSV; (b) выращивание клетки-хозяина; и (c) выделение частицы HSV, продуцируемой клеткой-хозяином. Изобретение расширяет арсенал средств для лечения рака. 11 н. и 10 з.п. ф-лы, 5 ил., 1 табл.
1. Неприродный вирус простого герпеса («HSV») для лечения рака, содержащий одну или несколько мутаций в гене, кодирующем гликопротеин E («gE»), причем мутация кодирует мутацию аланина в треонин в положении 151 белка gE.
2. Неприродный HSV по п. 1, дополнительно содержащий ген, кодирующий дисфункциональный белок ICP34.5, и/или ген, кодирующий дисфункциональный белок ICP6, например, где ген, кодирующий дисфункциональный белок ICP34.5, включает полинуклеотид, имеющий последовательность, выбранную из SEQ ID NO: 5, SEQ ID NO: 7 и последовательности, по меньшей мере на 95% идентичной SEQ ID NO: 5 или 7 и сохраняющей мутацию в нуклеотиде в любой из SEQ ID NO: 5 или 7; или
где ген, кодирующий дисфункциональный белок ICP6, включает полинуклеотид, имеющий последовательность, выбранную из SEQ ID NO: 45, SEQ ID NO: 47 и последовательности, по меньшей мере на 95% идентичной SEQ ID NO: 45 или 47 и сохраняющей мутацию в нуклеотиде в любой из SEQ ID NO: 45 или 47.
3. Неприродный HSV по п. 1 или 2, где неприродный HSV утратил ген, кодирующий функциональный белок ICP34.5 и/или функциональный белок ICP6.
4. Неприродный HSV по любому из пп. 1-3, содержащий полинуклеотид, кодирующий аминокислотную последовательность, показанную в SEQ ID NO: 13, и/или полинуклеотид, показанный в SEQ ID NO: 12.
5. Неприродный HSV по любому из пп. 1-4, дополнительно содержащий полинуклеотид, имеющий последовательность, идентичную по меньшей мере фрагменту гена вирулентности из 17TermA HSV; и/или
дополнительно содержащий полинуклеотид, имеющий последовательность, идентичную по меньшей мере фрагменту гена вирулентности из rRp450 HSV; и/или
где HSV происходит от штамма HSV типа 1 («HSV-1») или штамма HSV типа 2 («HSV-2»); и/или
где HSV происходит от штамма KOS HSV-1; и/или
дополнительно содержащий трансген.
6. Композиция для лечения рака, содержащая неприродный HSV по любому из пп. 1-5 и носитель, такой как фармацевтически приемлемый носитель.
7. Композиция по п. 6, дополнительно содержащая криоконсервант, облегчающий замораживание и размораживание неприродного HSV без потери существенной вирулентности.
8. Не относящееся к человеку млекопитающее для лечения рака, инфицированное неприродным HSV по любому из пп. 1-5.
9. Клетка для лечения рака, инфицированная неприродным HSV по любому из пп. 1-5.
10. Клетка по п. 9, которая является лимфоцитом.
11. Способ инфицирования клетки или индукции клеточного лизиса, включающий обеспечение контакта клетки с неприродным HSV по любому из пп. 1-5 или композицией по п. 6 или 7.
12. Способ по п. 11, где клетка представляет собой лимфоцит и/или где клетка инфицирована вирусом Эпштейна-Барр («EBV»).
13. Способ получения вирусного вектора, включающий введение трансгена в неприродный HSV по любому из пп. 1-5.
14. Способ ингибирования роста или метастазирования раковой клетки, включающий обеспечение контакта клетки с эффективным количеством неприродного HSV по любому из пп. 1-5 или композиции по п. 6 или 7.
15. Способ по п. 14, где раковая клетка относится к типу, выбранному из: рака поджелудочной железы, рака почки, мелкоклеточного рака легкого, опухоли головного мозга, опухоли нервной системы, рака костей, лимфомы, миеломы, рака толстой кишки, рака матки, рака молочной железы, лейкоза, рака печени, рака простаты, рака кожи или меланомы.
16. Способ лечения рака у субъекта, причем способ включает введение эффективного количества неприродного HSV любого из пп. 1-5 или композиции по п. 6 или 7 субъекту.
17. Способ по п. 16, где рак представляет собой рак поджелудочной железы, рак почки, мелкоклеточный рак легкого, рак мозга, нервный рак, рак кости, лимфому, миелому, рак толстой кишки, рак матки, рак молочной железы, лейкоз, рак печени, рак простаты, рак кожи, или меланому; и/или
где HSV или фармацевтическую композицию вводят путем инъекции, инфузии, инстилляции и/или ингаляции; и/или
где способ дополнительно включает применение у субъекта эффективного количества противоопухолевой терапии; и/или
где субъектом является млекопитающее, например человек, мышь, крыса, морская свинка, не являющийся человеком примат, собака, кошка, лошадь, корова, свинья, коза или овца, причем предпочтительно субъектом является человек.
18. Способ получения HSV по любому из пп. 1-5, включающий:
(а) введение в клетку-хозяина 17TermA HSV и rRp450 HSV;
(b) выращивание клетки-хозяина по меньшей мере за 3 пассажа; и
(c) выделение частицы HSV, продуцируемой клеткой-хозяином;
необязательно, где HSV вводят в клетку-хозяина путем трансфекции, инфицирования, трансформации, электропорации, инъекции, микроинъекции или их комбинации, также необязательно, где клетку-хозяина выращивают по меньшей мере за 3 пассажа, 10 пассажей, 20 пассажей, 30 пассажей, 40 пассажей или 50 пассажей; кроме того, необязательно, где клетка-хозяин содержит продукт комплементарного гена для поддержки репликации введенных HSV, например, где комплементарный ген кодирует белок ICP6 и/или белок ICP34.5;
где, необязательно, способ дополнительно включает введение трансгена в HSV.
19. Частица HSV для лечения рака, полученная способом по п. 18.
20. Способ получения HSV по любому из пп. 1-5, включающий:
(а) введение в клетку-хозяина полинуклеотида, кодирующего вирусный геном HSV;
(b) выращивание клетки-хозяина; и
(c) выделение частицы HSV, продуцируемой клеткой-хозяином.
21. Способ по п. 20, где полинуклеотид вводят в клетку-хозяина путем трансфекции, инфицирования, трансформации, электропорации, инъекции, микроинъекции или их комбинации; и/или
где последовательность нуклеиновой кислоты, кодирующую вирусный геном, вводят в клетку-хозяина в векторе, например, HSV или плазмиде; и/или
где клетка-хозяин содержит продукт комплементарного гена для поддержки репликации введенного HSV; и/или
где выделенный HSV практически не содержит клеток-хозяев, клеточного детрита, питательной среды или любого другого агента, используемого при культивировании клеток-хозяев; и/или
где выделение проводят центрифугированием, фильтрацией, хроматографией или любой их комбинацией.
| WO 2005005637 A2, 10.02.2016 | |||
| WO 2006072620 A1, 13.07.2006 | |||
| ВАКЦИНЫ ПРОТИВ ВИРУСА ПРОСТОГО ГЕРПЕСА | 2010 |
|
RU2575067C2 |
Авторы
Даты
2024-06-28—Публикация
2020-03-13—Подача