Изобретение относится к области биотехнологии, а именно к способу опосредованного определения количества колониеобразующих единиц Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации.
Mycoplasma spp. представляют серьезную проблему для молочных ферм, поскольку связаны с несколькими заболеваниями крупного рогатого скота, включая мастит, пневмонию, отит, артрит и нарушения репродуктивной функции [1]. Политика содержания КРС на фермах в РФ и многих странах мира заключается в выбраковывании больных микоплазменным маститом коров, поскольку он может быть очень заразным и не поддаваться лечению [2]. Комнатные мухи представляют угрозу для молочных ферм, приводя к значительным потерям, связанным с передачей заболеваний, включая мастит [2].
Возбудитель заболевания - Mycoplasma arginini, которая относится к плевропневмонийной группе микроорганизмов. Данный микроорганизм принадлежит к мелкоклеточным прокариотам семейства Mycoplasmataceae, рода Mycoplasma, вида Mycoplasma arginini [2].
У М. arginini имеется трехслойная липопротеиновую цитоплазматическую мембрану. В состав клеточной мембраны данного вида микоплазм входит до 1/3 протеинов от массы мембраны, которые обеспечивают прикрепление и внедрение в клетку-хозяина. Внутри клетки находятся ДНК, РНК, рибосомы и цитоплазма [1, 2].
Геномная ДНК М. arginini имеет длину около 631000 п.н. Общее содержание GC в каркасах составило 26,3%. Размер генома и содержание GC были сопоставимы с таковыми в эталонном геноме эталонного штамма «HAZ145» [3, 4]. В геноме бактерии находится 879 генов: 816 из них кодируют структурные и функциональные протеины, а 58 - разнообразные РНК. Среди указанного количества имеются 5 псевдогенов. 432 белка кодируются генами прямой цепи (+), 384 - комплементарной цепи (-). Информацию о 39 разновидностях РНК данного штамма микоплазм несет цепь (-), а о 19 - цепь (+). При оценке протеома данного микроорганизма определено, что максимальная длина транскрибируемого белка данным штаммом составляет 2019 а.о., а минимальная - 30 а.о. Прямая цепь несет в небе информацию большего количества генов белков [4].
Микоплазмы характеризуются выраженным антигенным полиморфизмом. Среди микоплазм выделяют различные серовары. Антигенами микоплазм являются белки-адгезины, фосфолипиды, гликолипиды, полисахаридные компоненты. Адгезины микоплазм имеют высокую гомологию к структурным белкам млекопитающих, что позволяет им уклоняться от иммунной системы хозяина (антигенная мимикрия) [5].
Для определения количества колониеобразующих единиц (КОЕ) Mycoplasma arginini используют стандарт ОФС.1.7.2.0008.15 «Определение концентрации микробных клеток» кондуктометрическим методом {прототип) [6]. Кондуктометрические методы основаны на зависимости сопротивления или проводимости среды от концентрации клеток. При росте и размножении в соответствующей жидкой питательной среде микроорганизмы продуцируют высоко заряженные ионные метаболиты, что приводит к изменению электрохимических свойств питательной среды. Эти изменения полного сопротивления, измеряемые проводимостью или емкостным сопротивлением, регистрируются электродами, находящимися в контакте с культуральной средой. Время определения обратно пропорционально концентрации микробных клеток в суспензии [6].
Одним из таких методов является подсчет числа клеток в электронном счетчике Култера. Суспендированные в электролите клетки проходят через апертуру малого диаметра, расположенную между двумя электродами. Когда клетки проходят через апертуру («электрочувствительную зону»), каждая из них вытесняет определенное количество электролита, вызывая возрастание сопротивления. В результате происходит небольшое изменение величины электрического тока в усилителе, который преобразует колебания силы тока в импульсы напряжения, которые находятся в прямой зависимости от размера прошедшей через апертуру клетки [7, 8].
При этом данный метод имеет ряд ограничений: 1) относительно невысокая чувствительность и специфичность; 2) погрешности измерений в результате «слипания» клеток; 3) получение недостоверных результатов анализа.
В связи с этим целесообразно провести поиск альтернативного способа опосредованного определения количества колониеобразующих единиц Mycoplasma arginini на основе молекулярно-генетических методов.
Задачей настоящего изобретения является разработка высокочувствительного, высокоспецифичного, объективного, достоверного способа опосредованного определения количества колониеобразующих единиц Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации с целью устранения вышеуказанных недостатков.
Данная задача решена благодаря созданию способа опосредованного определения количества колониеобразующих единиц Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации.
Предложенный метод является высокочувствительным и специфичным, объективным, достоверным и позволяет опосредованно определять количество колониеобразующих единиц Mycoplasma arginini в суспензиях в течение короткого периода времени (2,5 ч).
Технический результат изобретения заключается в том, что разработанный способ имеет следующие преимущества по сравнению с прототипом: 1) повысить чувствительность и специфичность за счет применения высокоспецифичных оригинальных праймеров и ДНК-зонда, рассчитанных для argE-гена М. arginine, который кодирует белок Xaa-His dipeptidase (амплифицируемый участок в диапазоне 238250…238529 п.н., размер ампликона составляет 149 п.н.); 2) повысить достоверность проводимого анализа благодаря установлению зависимости между количеством КОЕ М. arginini (КОЕМ.а.) и циклом количественной оценки (Cq), представленной в виде логарифмической функции:
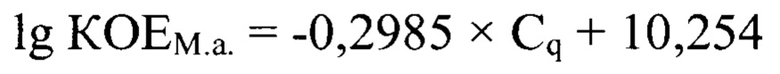
с высокой достоверностью аппроксимации (R2=0,9996) и эффективностью амплификации 98,84%. Предложенная модель позволяет опосредованно определять количество КОЕ М. arginini в исследуемом материале.
Сущность изобретения отражена на графических изображениях:
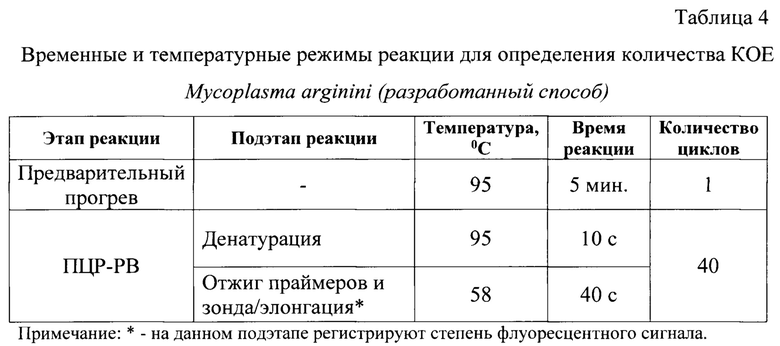
Фиг. 1 - Дизайн олигонуклеотидных праймеров и зонда, рассчитанных для argE-гена М. arginine. Примечание: А - прямой праймер, Б - ДНК-зонд, В - обратный праймер, RV - перевернутая комплементарная цепь.
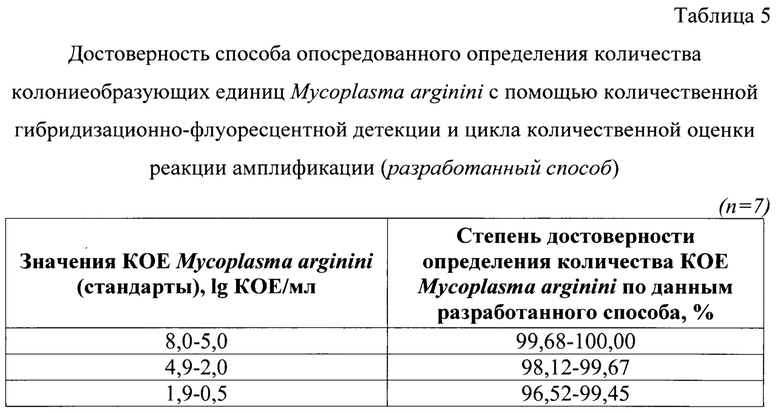
Фиг. 2 - Зависимость количества колониеобразующих частиц М. arginini и цикла количественной оценки с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации (n=7, р<0,005).
SEQ ID NO: 1 представляет последовательность argE-гена М. arginine штамма «HAZ145»;
SEQ ID NO: 2 представляет последовательность аминокислот, кодируемых argE-гена М. arginine штамма «HAZ145»;
SEQ ID NO: 3 представляет последовательность праймера F-M.argE238381;
SEQ ID NO: 4 представляет последовательность праймера F-M.argE238511;
SEQ ID NO: 5 представляет последовательность праймера F-M.argE238457-FAM/RTQ1.
Сущность изобретения заключается в подходе по опосредованному определению количества КОЕ М. arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации.
Заявляемый способ основан на: 1) элюировании ДНК М. arginini с применением набора «ДНК-сорб» (ООО «Интерлабсервис»); 2) амплификации специфического фрагмента argE-гена М. arginini с применение оригинальных специфических прямого и обратного праймеров, а также молекулярного зонда (фиг. 1), меченого флуоресцентным красителем FAM (λmax флуоресценции = 520 нм) и тушителем свечения RTQ-1 (λmax поглощения = 520 нм); 3) детекции ампликонов с помощью флуоресцентного свечения и отображения накопления сигнала в виде сигмоиды, стремящейся к экспоненте; 4) определении активности М. arginini с применением логарифмической функции, выраженной в виде логарифмического уравнения:
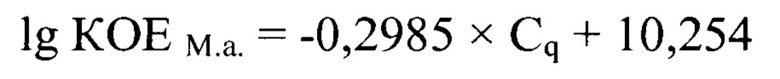
с высокой достоверностью аппроксимации (R2=0,9996) и эффективностью амплификации 98,84%.
В настоящее время метод ПЦР в режиме реального времени применяют для индикации нуклеиновых кислот различных инфекционных агентов, в том числе микоплазм [8-13]. Для определения количества КОЕ М. arginini ранее данный метод с применением разработанной системы оригинальных праймеров и молекулярного зонда не использовался. Таким образом, сведений об аналогах предлагаемого способа опосредованного определения количества КОЕ М. arginini авторами не обнаружено.
Разработанный способ опосредованного определения количества КОЕ М. arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации с прототипом отличается более высокой чувствительностью и специфичностью, достоверностью получаемых результатов.
В отличие от прототипа разработанный способ включает этап элюирования ДНК микоплазмы; амплификацию специфического фрагмента argE-гена М. arginini с применение оригинальных специфических прямого и обратного праймеров, а также молекулярного зонда, меченого флуоресцентным красителем FAM и тушителем свечения RTQ-1; детекции флуоресцентного сигнала в виде сигмоиды, стремящейся к экспоненте; новый подход к методике расчета количества КОЕ М. arginini с применением модели зависимости цикла количественной оценки для кривой флуоресценции. Применение предложенного способа позволит повысить специфичность и чувствительность анализа, увеличить достоверность проводимого анализа. Таким образом, актуально применять предложенный способ для опосредованного определения количества КОЕ М. arginini в исследуемом материале.
Ключевым элементом заявляемого способа является определение значений цикла количественной оценки в реакции амплификации ДНК М. arginini в ПЦР режиме реального времени и расчет количества КОЕ с использованием разработанной математической модели.
Сопоставительный анализ с прототипом позволяет сделать вывод, что новизна и изобретательский уровень заявляемого изобретения заключается в применении количественной гибридизационно-флуоресцентной детекции, определение цикла количественной оценки реакции амплификации, оригинальных специфичных праймеров и молекулярного зонда, рассчитанных на участок argE-гена М. arginini, и разработанной математической функции зависимости величины цикла количественной оценки и количества КОЕ М. arginini.
Сущность предлагаемого изобретения пояснена на графическом материале - Зависимость количества колониеобразующих частиц М. arginini и цикла количественной оценки с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации (n=7, р<0,005) (фиг. 2).
С целью определения количества КОЕ М. arginini подготавливают контрольную панель стандартов М. arginini, в качестве которых используют лиофильно высушенные суспензии со следующими значениями активности: 8,0; 7,0; 6,0; 5,0; 4,0; 3,0; 2,0; 1,0; 0,0 lg КОЕ/мл. В качестве отрицательного контроля применяют деионизированную воду.
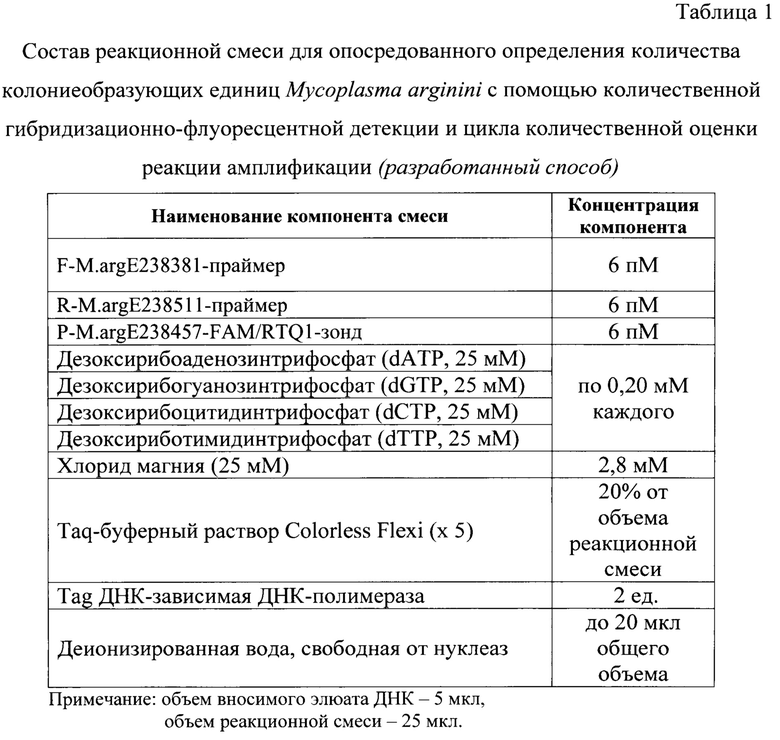
Из всех контролей и тестируемых проб выделяют ДНК. Полученный экстракт нуклеиновой кислоты хранят при температуре минус 20±2°С или сразу используют в дальнейшей работе. Проводят ПЦР в режиме реального времени для исследования контрольных образцов и проб. Для постановки реакции готовят реакционную смесь, рецептура которой представлена в таблице 1. Дизайн праймеров и молекулярного зонда отражены в таблице 2. Расчет праймеров и зонда проводили на основании нуклеотидных последовательностей argE-гена М. arginini различных штаммов и изолятов, опубликованных в базах данных GenBank, в частности, штамма «HAZ145» [3, 4].
В качестве гомологичных участку argE-гена М. arginini олигонуклеотидов используют:
F-M.argE238381-праймер (5'-AAATGTTGATGATGCTTTAGAAT-3'),
R-M.argE238511-праймер (5'-GCACACCTTGATGTTGTTC-3'),
P-M.argE238457-FAM/RTQ1-зонд (5'-FAM-AATAGATATGGATATGCCCAAA-RTQ1-3')
в концентрации 6 пМ на реакцию. Для элонгации применяют дезоксирибонуклеозидтрифосфаты с их концентрацией в реакционной смеси по 0,20 мМ каждого. В качестве основы используют буферный раствор (5х), содержание которого составляет 20% от общего объема реакционной смеси. В смесь добавляют хлорид магния до концентрации 2,8 мМ. В качестве катализатора реакции применяют Taq ДНК-зависимую ДНК-полимеразу (2 ед.). Элюаты суммарной ДНК каждого образца добавляют к реакционной смеси по 5 мкл. Итоговый объем смеси для проведения одной реакции составляет 25 мкл.
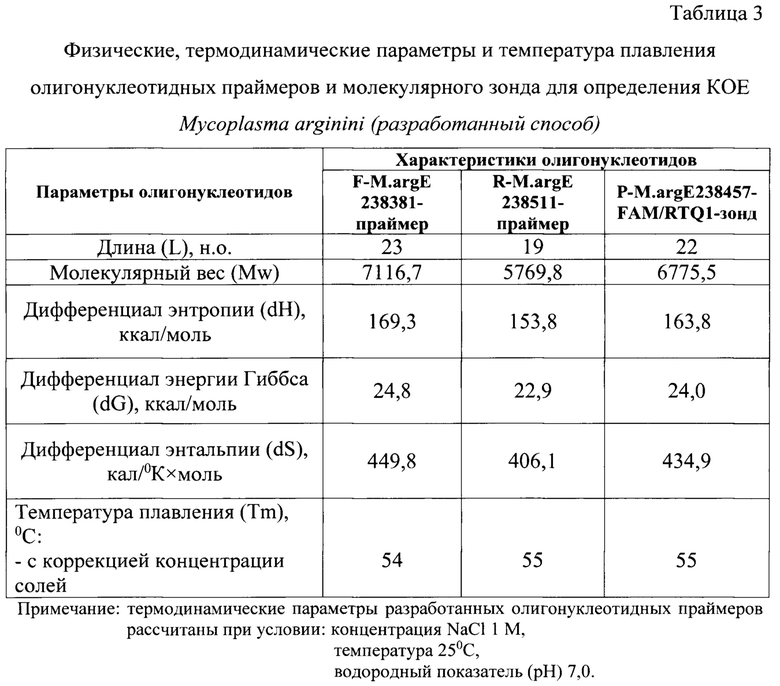
Олигонуклеотиды для ДНК-матрицы подбирали в соответствии с рядом общих правил, которые отражены в работах В. Deiman и R. Sooknanan. Длины прямого, обратного праймеров и зонда составляют 23, 19 и 22 н.о. каждый, что соответствует требованиям. Молекулярный вес олигонуклеотидов прямого, обратного праймеров и зонда равен 7116,7; 5769,8; 6775,5, соответственно. Праймеры и зонд очищены в полиакриламидном геле и с помощью высокоэффективной жидкостной хроматографии, соответственно. Нуклеотидная последовательность зонда не комплементарна олигонуклеотидным праймерам. Отсутствуют 4 и более подряд одинаковых нуклеотидов в цепи праймеров и зонда. Флуорофор FAM присоединен к 5'-концу, а гаситель флуоресценции RTQ1 - к 3'-концу. Данные условия соответствуют требованиям, предъявляемым к олигонуклеотидным праймерам и молекулярному зонду, которые участвуют в ПЦР в режиме реального времени. В качестве флуоресцентного красителя был выбран FAM с длиной волны максимальной флуоресценцией 520 нм. Для тушения свечения использовали гаситель флуоресценции RTQ1 с длиной волны максимального поглощения при 520 нм и возможном диапазоне гашения 470-570 нм. Иными словами, была выбрана подходящая пара «флуорофор-гаситель».
При анализе нуклеотидных последовательностей олигонуклеотидов установили, что для праймеров и зонда не характерно образование «шпилек», а также не выявлено 3'-комплементарности и сайтов, отжигающих сами на себя. Расчет вероятности образования «шпилек» и димеров олигонуклеотидов проводили при условии, что минимальное количество пар оснований, необходимых для димеризации, - 5, а для образования «шпилек», - 4.
Проведено определение температур плавления (Tm) для олигонуклеотидных праймеров и зонда. Для оценки температуры плавления олигонуклеотидов применяли метод, учитывающий концентрации ионов K+ и диметилсульфооксида (DMSO).
Физические, термодинамические константы и расчет температур плавления разработанных олигонуклеотидных праймеров и молекулярного зонда представлены в таблице 3. Из нее следует, что энтропия, энергия Гиббса и энтальпия для прямого праймера составили 169,3 ккал/моль, 24,8 ккал/моль, 449,8 ккал/(°K×моль), соответственно. Энтропия, энергия Гиббса и энтальпия для обратного праймера составили 153,8 ккал/моль, 22,9 ккал/моль, 406,1 ккал/(°K×моль), соответственно. Энтропия, энергия Гиббса и энтальпия для молекулярного зонда - 163,8 ккал/моль, 24,0 ккал/моль, 434,9 кал/(°K×моль), соответственно. Данные значения необходимы для расчета температур плавления представленных олигонуклеотидов. Tm для прямого, обратного праймеров и зонда теоретически составили 54, 55 и 55°С, соответственно.
Экспериментально было выявлено, что оптимальная температура отжига рассматриваемых олигонуклеотидов составляет 58°С. Для проведения ПЦР в режиме реального времени было решено проводить гибридизацию праймеров и зонда с участком argE-гена М. arginini при температуре 58°С.
Последовательности разработанных праймеров и молекулярного зонда проверили на наличие нежелательных совпадений с другими последовательностями нуклеиновых кислот с использованием Банка данных последовательности нуклеиновых кислот М. arginini. Последовательности праймеров и зонда также проанализировали на наличие внутренних вторичных структур с помощью программы сворачивания нуклеиновых кислот с помощью программы Mfold. Выявлено, что для разработанных олигонуклеотидов нежелательных совпадений с другими последовательностями нуклеиновых кислот, а также наличия внутренних вторичных структур не обнаружено.
Постановку реакции осуществляли в детектирующем термоциклере любой марки при температурных и временных параметрах, сведения о которых представлены в таблице 4.
Перед проведением реакции амплификации в режиме реального времени осуществляют предварительный прогрев смеси при температуре 95°С в течение 5 мин. для активации фермента Taq ДНК-полимеразы за 1 цикл.
Реакция амплификации в режиме реального времени включает в себя 3 подэтапа: денатурацию, отжиг олигонуклеотидов, элонгацию. Денатурацию осуществляют при температуре 95°С в течение 10 с, отжиг олигонуклеотидов и элонгацию проводят в совмещенном режиме при температуре 58°С в течение 40 с, что оптимизирует реакцию по времени анализа (табл. 4). Количество циклов - 40.
Результаты реакции анализируют, оценивая и сравнивая графики накопления флуоресцентного сигнала по значениям циклов количественной оценки Cq, определенных с помощью пересечения пороговой линии и логарифмическим отображением функции Fl=f(Cq). Учет результатов в реакции происходит на каждом цикле. Флуориметр определяет уровень флуоресценции и строит кинетическую кривую в координатах: уровень флуоресценции - цикл реакции амплификации. В случае присутствия в исследуемой пробе специфической ДНК-матрицы М. arginini кинетическая кривая имеет экспоненциальную зависимость (график представлен в виде логистической кривой). Положительными считаются пробы, которые соответствуют логистической кривой, полученные при анализе флуоресценции красителя FAM, входящего в состав молекулярного зонда. Пробы считаются отрицательными, если при их анализе отсутствует логистическая кривая в виде графика, приближенного к экспоненте.
Устанавливают зависимость между циклом количественной оценки и количеством КОЕ М. arginini в сырье для вакцин в процессе построения логарифмической функции. Оценивают величину эффективности реакции амплификации (Е) по формуле: 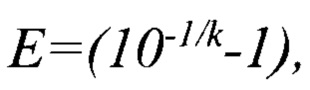
где k - угловой коэффициент в зависимости: 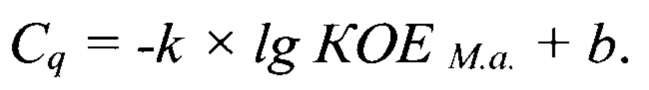
На основе разработанной модели рассчитывают количество КОЕ М. arginini в исследуемом материале.
Сущность предлагаемого изобретения пояснена примерами его использования, которые не ограничивают объем изобретения.
Пример 1. Выявление зависимости количества КОЕ М. arginini и цикла количественной оценки ПЦР в виде логарифмической функции
Для выявления зависимости количества КОЕ М. arginini и цикла количественной оценки реакции амплификации подготавливали контрольную панель стандартов М. arginini, в качестве которых использовали лиофилизированные суспензии бактерий с количеством КОЕ активности: 0,0; 1,0; 2,0; 3,0; 4,0; 5,0; 6,0; 7,0; 8,0 lg КОЕ/мл, которые растворяли в 1/15 М фосфатном буферном растворе до требуемого объема. В качестве отрицательного контроля применяли деионизированную, не контаминированную микроорганизмами.
Из всех стандартных положительных образцов и отрицательных контролей, а также тестируемых проб выделяли ДНК в соответствии с методикой, представленной выше.
Проводили реакцию амплификации в режиме реального времени для исследования стандартов, как описано выше. Результаты реакции анализировали, оценивая и сравнивая графики накопления флуоресцентного сигнала по значениям циклов количественной оценки Cq, определенных с помощью пересечения пороговой линии и логарифмическим отображением функции 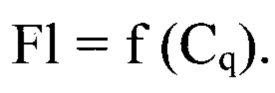 Устанавливали зависимость между циклом количественной оценки и количеством КОЕ М. arginini в процессе построения логарифмической функции. Полученные данные отражены на фиг. 2 и выражены в виде логарифмической функции:
Устанавливали зависимость между циклом количественной оценки и количеством КОЕ М. arginini в процессе построения логарифмической функции. Полученные данные отражены на фиг. 2 и выражены в виде логарифмической функции:
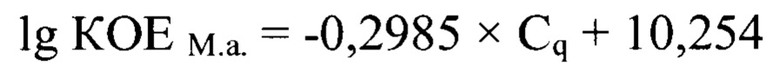
с высокой достоверностью аппроксимации (R2=0,9996) и эффективностью амплификации 98,84%.
Таким образом, предложенная модель позволяет опосредованного определять количество КОЕ М. arginini с помощью разработанного способа.
Пример 2. Применение способа опосредованного определения количества колониеобразующих единиц Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации
В исследовании использовали шесть бактериальных суспензий М. arginini со следующими показателями активности: 6,50; 6,75; 7,00; 7,25; 7,50; 7,75 lg КОЕ/мл, соответственно (пробы №1-6). В качестве положительного контроля применяли культуральную суспензию М. arginini с активностью 7,00 lg КОЕ/мл. В качестве отрицательного контроля применяли деионизированную воду, не зараженную микроорганизмами. Испытуемые пробы и контрольные образцы исследовали в 7 повторностях. Этап элюирования ДНК и постановку реакции проводили, как описано выше.
Средние значения циклов количественной оценки для проб №1-6 составляли 12,43±0,01, 11,80±0,01, 10,86±0,01, 10,06±0,01, 9,15±0,01, 8,35±0,01, соответственно. Пользуясь разработанной логарифмической функцией, представленной выше, рассчитали средние значения КОЕ М. arginini для проб №1-6, которые составили 6,53; 6,72; 7,00; 7,24; 7,51; 7,75 lg КОЕ/мл, соответственно. Для положительного контроля значение цикла количественной оценки составило 10,86±0,01, что соответствовало активности М. arginini, равной 7,00 lg КОЕ/мл. Для отрицательных контролей экспоненциальные графики не были сформированы, что означало отсутствие М. arginini в данных образцах. Полученные результаты свидетельствовали о высокой степени точности разработанного способа. Таким образом, предложенный способ позволяет опосредованно определять количество колониеобразующих единиц Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации.
Пример 3. Выявление степени достоверности определения количества КОЕ М. arginini с применением разработанного способа
Для анализа использовали 315 бактериальных суспензий М. arginini с активностью от 1,00 до 8,00 lg КОЕ/мл. Этапы экстрагирования нуклеиновых кислот и постановку ПЦР в режиме реального времени проводили, как отражено выше. При анализе применяли контроли, представленные выше. Результаты анализа представлены в таблице 5.
Интерпретацию результатов проводили, пользуясь разработанной логарифмической функцией: lg КОЕМ.а.=-0,2985×Cq+10,254, с получением значений КОЕМ.а. для каждой из 315 проб. Для положительного контроля значение цикла количественной оценки составило 10,86±0,01, что соответствовало активности М. arginini, равной 7,00 lg КОЕ/мл. Для отрицательного контроля экспоненциальный график не был сформирован, что означало отсутствие М. arginini в данных образцах.
Анализируемые пробы и контроли также тестировали в реакции и определили, что данные, полученные с помощью разработанного способа, коррелировали с со значениями стандартов на 99,68-100,00% для 8,0-5,0 lg КОЕ/мл (n=105), на 98,12-99,67% для 4,9-2,0 lg КОЕ/мл (n=105), на 96,52-99,45% для 1,9-0,5 lg КОЕ/мл (n=105). Полученные результаты свидетельствовали о высокой степени точности разработанного способа опосредованного определения количества КОЕ М. arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации. Таким образом, разработанный способ позволяет с высокой степенью достоверности определять активность М. arginini предложенным молекулярно-биологическим методом.
Пример 4. Оценка аналитической чувствительности способа опосредованного определения количества КОЕ Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации
При определении аналитической чувствительности разработанного способа подготавливали серию стандартов М. arginini с активностью в диапазоне 0,10-8,00 lg КОЕ/мл с шагом 0,1 lg КОЕ/мл. Контрольные образцы тестировали в 7 повторностях. Этапы элюирования нуклеиновой кислоты и постановку реакции проводили, как описано выше. Выявлено, что с достоверностью 96,01-100,00% разработанным способом определены значения КОЕ М. arginini со значениями от 0,5 до 8,0 lg КОЕ/мл.
Аналитическая чувствительность способа опосредованного определения количества КОЕ Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации составляет не менее 0,5 lg КОЕ/мл с достоверностью результатов исследования не менее 96,01%.
Пример 5. Исследование аналитической специфичности способа опосредованного определения количества колониеобразующих единиц Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации
При анализе аналитической специфичности разработанного способа исследовали бактериальные суспензии М. arginini, а также штамма «ВНИИЗЖ» вируса бешенства, штамма «Рич» возбудителя коронавирусного энтерита собак, штамма «Rockborn» возбудителя чумы плотоядных. Количество инфекционных доз вирусов в суспензиях составлял не менее 4,0 lg ТЦД50/см3. Исследования проводили в 5 повторностях.
Этапы исследования проводили, как описано выше. Для проб, содержащих другие патогены, не наблюдалось формирования графиков экспоненты, и они не выходили за пороговый уровень флуоресцентного сигнала (0,01 у.е.). Таким образом, разработанный способ является специфичным по отношению к М. arginini и может быть использован для его количественного определения.
Основными преимуществами предлагаемого изобретения является возможность повысить чувствительность и специфичность за счет применения высокоспецифичных оригинальных праймеров и ДНК-зонда, рассчитанных для argE-гена М. arginine, который кодирует белок Xaa-His dipeptidase; повысить достоверность проводимого анализа благодаря установлению зависимости между КОЕ М. arginini (КОЕМ.а.) и циклом количественной оценки (Cq), представленной в виде логарифмической функции: lg КОЕМ.а.=-0,2985×Cq+10,254 с высокой достоверностью аппроксимации (R2=0,9996) и эффективностью амплификации 98,84%. Предложенная модель позволяет опосредованно определять количество КОЕ М. arginini в исследуемом материале.
Предлагаемое изобретение позволяет быстро и с высокой степенью достоверности рассчитывать количество КОЕ М. arginini в исследуемом материале на основе разработанного способа с применением оригинальных специфических олигонуклеотидных праймеров и молекулярного зонда для участка argE-гена М. arginini и разработанной логарифмической модели.
Источники информации
1. Maunsell FP, Woolums AR, Francoz D, Rosenbusch RF, Step DL, Wilson DJ, Janzen ED. 2011. Mycoplasma bovis infections in cattle. J Vet Intern Med-25:772-783.
2. Punyapornwithaya V, Fox LK, Hancock DD, Gay JM, Alldredge JR. 2012. Time to clearance of mycoplasma mastitis: the effect of management factors including milking time hygiene and preferential culling. Can Vet J - 53:1119-1122.
3. GenBank. Mycoplasma arginini DNA, complete genome, strain: HAZ145_1. URL: https://www.ncbi.nlm.nih.gov/nuccore/AP014657.1. (Дата обращения: 07.06.2023).
4. Hata E. Complete genome sequence of Mycoplasma arginini strain HAZ 145_1 from bovine mastitic milk in Japan. Genome Announc 3:e00265-15.
5. Yiwen CW, Yueyue WY, Qin LM, Zhu CM, You XX. 2021. Infection strategies of mycoplasmas: unraveling the panoply of virulence factors. Virulence 12:788-817.
6. ОФС.1.7.2.0008.15 «Определение концентрации микробных клеток». URL: https://pharmacopoeia.ru/ofs-1-7-2-0008-15-opredelenie-kontsentratsii-mikrobnyh-kletok/ (Дата обращения: 08.07.2023).
7. Gioia G, Freeman J, Sipka A, Santisteban C, Wieland M, Gallardo VA, Monistero V, Scott JG, Moroni P. 2022. Pathogens associated with houseflies from different areas within a New York State dairy. J Dairy Sci Communications 3:285-290.
8. Gioia G, Werner B, Nydam DV, Moroni P. 2016. Validation of a mycoplasma molecular diagnostic test and distribution of mycoplasma species in bovine milk among New York State dairy farms. J Dairy Sci 99:4668-4677.
9. Thomas A, Ball H, Dizier I, Trolin A, Bell C, Mainil J, Linden A. 2002. Isolation of Mycoplasma species from the lower respiratory tract of healthy cattle and cattle with respiratory disease in Belgium. Vet Rec 151:472-476.
10. Stipkovits L, Somogyi M, Asvanyi B, Toth A, Szathmary S. 2013. Short communication: role of Mycoplasma arginini in mastitis caused by Streptococcus dysgalactiae. J Dairy Sci 96:1661-1667.
11. Bao SJ, Chen DQ, Yu SQ, Chen HJ, Tan L, Hu MR, Qiu XS, Song CP, Ding C. 2015. Characterization of triosephosphate isomerase from Mycoplasma gallisepticum. FEMS Microbiol Lett 362:fhv140.
12. Chen S, Hao H, Zhao P, Ji W, Li M, Liu Y, Chu Y. 2018. Differential immunoreactivity to bovine convalescent serum between Mycoplasma bovis biofilms and planktonic cells revealed by comparative immunoproteomic analysis. Front Microbiol 9:379.
13. Rice P, Longden I, Bleasby A. 2000. EMBOSS: the European Molecular Biology Open Software Suite. Trends Genet 16:276-277.

--->
Перечень последовательностей
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="1710914670697.xml"
softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2024-03-20">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-08-04</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>530</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-08-04</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБУ "Федеральный центр охраны
здоровья животных" (ФГБУ "ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin>Federal State-Financed Institution Federal
Centre for Animal Health (FGBI ARRIAH)</ApplicantNameLatin>
<InventorName languageCode="ru">Доронин Максим
Игоревич</InventorName>
<InventorNameLatin>Doronin Maksim Igorevich </InventorNameLatin>
<InventionTitle languageCode="ru">Способ опосредованного определения
количества колониеобразующих единиц Mycoplasma arginini с помощью
количественной гибридизационно-флуоресцентной детекции и цикла
количественной оценки реакции амплификации</InventionTitle>
<SequenceTotalQuantity>5</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1311</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1311</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>M.arginini</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaaaaaatatattaaatatcaacaaaataaaagagaatttgatgaaa
tgattcaacacatagctaatttatgtagaattccatcaattagcatcgaacaagaaaatgatgaaaaata
tccttttagtaaaaatgttgatgatgctttagaatatgcattaaaactagcaaaaaattttggatttgaa
acatataaagattcaaaaaatagatatggatatgcccaaattggaaatggaactaaaattctaggcattt
tagcacaccttgatgttgttcctgccggtgatgagaatcaatgaaaaactagtgcctttgttccagtaat
tactgaaaatgaaataatcggtagaggttcattagacgataagggccctgcaattatcaacctttatgct
atgaagtatattttagataataacttaattcataaagattgaacaattcgtttagttttcggtatttctg
aagaaacaacaatgaaatcaatgaaatattacttaaatgattttggtcatccttttgtttcttatacacc
tgatggtgaatgacctttaatttttgcagaaaaattgatttaccaagcaaatcttatttttaataaaata
gaaggtttggaaattaatgcaggtgatgtagctaaccaaatacctgataaggttaattatcaattcaata
ataatgattttcttgttttaggaaaaggtggccatggttcaacacctgaaaaaggtgataatgcaataat
aaaagctttattagaattaagtaaaaattgtccagacttaaaaaatcaagatttaattaaatttattgaa
aataatttatcaaataaagactatacactaaaaaacattttcaaagatttcgatgatttttcaggtaaac
tatcagccaatttaggaattataagaactttcgataataagcacgttgtaacatttgattttagagtacc
agcgacaagaagcgttgaagaggttacaaaagcaataaattcatatttagatgctaatttacctaacgta
aaattagaaatagtaggaactaaaaattcaatgtttatggatccagatagcaaattaataaaactattga
tggaaacatataatgaaggcatgcaaattgaagaaaaacctttagcaattggtgggggtacatacgcgcg
tattgtaaaaaactgtgtcgcttttggttcaacaaaatatatgcacttaatgcatggtcctaatgaatac
tttacttttaaagaaattagagaatctttagaaatatatattaatgccttaaacagaatgcaagatgaac
ta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>437</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..437</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>M.arginini</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MKKYIKYQQNKREFDEMIQHIANLCRIPSISIEQENDEKYPFSKNVDDA
LEYALKLAKNFGFETYKDSKNRYGYAQIGNGTKILGILAHLDVVPAGDENQWKTSAFVPVITENEIIGRG
SLDDKGPAIINLYAMKYILDNNLIHKDWTIRLVFGISEETTMKSMKYYLNDFGHPFVSYTPDGEWPLIFA
EKLIYQANLIFNKIEGLEINAGDVANQIPDKVNYQFNNNDFLVLGKGGHGSTPEKGDNAIIKALLELSKN
CPDLKNQDLIKFIENNLSNKDYTLKNIFKDFDDFSGKLSANLGIIRTFDNKHVVTFDFRVPATRSVEEVT
KAINSYLDANLPNVKLEIVGTKNSMFMDPDSKLIKLLMETYNEGMQIEEKPLAIGGGTYARIVKNCVAFG
STKYMHLMHGPNEYFTFKEIRESLEIYINALNRMQDEL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>M.arginini</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaatgttgatgatgctttagaat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>M.arginini</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcacaccttgatgttgttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>M.arginini</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aatagatatggatatgcccaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в режиме реального времени | 2023 |
|
RU2823778C1 |
| Способ опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК | 2023 |
|
RU2821027C1 |
| Способ опосредованного определения титра инфекционной активности вируса инфекционного гепатита собак генотипа CAV-1 в сырье для культуральных вакцин с помощью полимеразной цепной реакции в режиме реального времени по циклу количественной оценки Cq | 2023 |
|
RU2812441C1 |
| Способ опосредованного определения титра инфекционной активности возбудителя инфекционного ринотрахеита кошек FHV в неинактивированном сырье для вакцин с применением полимеразной цепной реакции и количественной гибридизационно-флуоресцентной детекции в режиме реального времени | 2023 |
|
RU2808641C1 |
| Способ опосредованного определения титра инфекционной активности мастаденовируса собак в сырье для вакцин методом ПЦР в режиме реального времени | 2023 |
|
RU2808585C1 |
| Способ опосредованного определения концентрации иммуногенного RNP-комплекса вируса бешенства в сырье для антирабических вакцин методом амплификации и гибридизационно-флуоресцентной детекции ДНК-ампликонов | 2020 |
|
RU2760436C1 |
| Способ опосредованного определения титра инфекционной активности возбудителя чумы плотоядных в сырье для вакцин методом обратной транскрипции и реакции амплификации в режиме реального времени | 2023 |
|
RU2815533C1 |
| Способ опосредованного определения инфекционного титра вируса бешенства в неинактивированном сырье для антирабических вакцин при транскрипционной амплификации и детекции продуктов реакции с применением beacon-технологии | 2020 |
|
RU2756472C1 |
| Способ определения титра инфекционной активности возбудителя чумы плотоядных животных в сырье для изготовления культуральных инактивированных вакцин | 2024 |
|
RU2831062C1 |
| Способ опосредованного определения титра инфекционной активности вобудителя парвовирусного энтерита собак в сырье для вакцин методом ПЦР в режиме реального времени | 2023 |
|
RU2812858C1 |
Изобретение относится к области биотехнологии. Описан способ опосредованного определения количества колониеобразующих единиц Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации. Значение КОЕ Mycoplasma arginini определяют по величине цикла количественной оценки. На основании установленной величины данного показателя рассчитывают значение КОЕ данного возбудителя. Предлагаемое изобретение позволяет быстро и с высокой степенью достоверности рассчитывать титр инфекционной активности М. arginini в исследуемом материале за счет применения специфических олигонуклеотидных праймеров и молекулярного зонда для участка regE-гена М. arginini и разработанной логарифмической модели. 1 з.п. ф-лы, 2 ил., 5 табл., 5 пр.
1. Способ опосредованного определения количества колониеобразующих единиц Mycoplasma arginini с помощью количественной гибридизационно-флуоресцентной детекции и цикла количественной оценки реакции амплификации, использующий следующие олигонуклеотиды: F-M.argE238381-праймера с дизайном 5’-AAATGTTGATGATGCTTTAGAAT-3’, R-M.argE238511-праймера с дизайном 5’-GCACACCTTGATGTTGTTC-3', P-M.argE238457-FAM/RTQ1-зонда с дизайном 5’-FAM-AATAGATATGGATATGCCCAAA-RTQ1-3’, рассчитанные для argE-гена М. arginine, в диапазоне 238250…238529 п.н., который кодирует белок Xaa-His дипептидазы; и функцию зависимости количества КОЕ М. arginini от цикла количественной оценки, представленной в виде логарифмической функции:
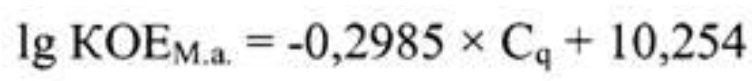
с достоверностью аппроксимации, равной 0,9996, и эффективностью амплификации 98,84%.
2. Способ по п. 1, где время проведения анализа сокращается до 2,5 ч, а аналитическая чувствительность составляет не менее 0,5 lg КОЕ/мл.
| Gioia G., Freeman J., Sipka A., Santisteban C., Wieland M., Gallardo V.A., Monistero V., Scott J.G., Moroni P | |||
| Способ получения продуктов конденсации фенолов с формальдегидом | 1924 |
|
SU2022A1 |
| Pathogens associated with houseflies from different areas within a New York State dairy | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| WO 2020223259 A1, 05.11.2020 | |||
| Способ опосредованного определения титра вируса ящура в неинактивированном сырье для вакцины при амплификации вирусной нуклеиновой кислоты и детекции РНК-ампликонов с применением технологии молекулярных биконов | 2020 |
|
RU2756557C1 |
Авторы
Даты
2024-07-29—Публикация
2023-08-29—Подача