Изобретение относится к области биотехнологии, а именно к способу опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПНР в режиме реального времени с применением разработанной системы специфичных олигонуклеотидов и математической функции зависимости величины цикла количественной оценки и титра инфекционной активности Mycoplasma gallisepticum.
Респираторный микоплазмоз птиц - это преимущественно хронически протекающее инфекционное заболевание кур, индеек, фазанов, цесарок, сопровождающаяся поражением органов дыхания, истощением и снижением продуктивности [1].
Возбудитель заболевания - Mycoplasma gallisepticum, который относится к плевропневмонийной группе микроорганизмов. М. gallisepticum - полиморфный кокк, величиной от 0,5 до 1 мкм. Данный микроорганизм принадлежит к мелкоклеточным прокариотам царства Bacteria, типа Tenericutes, класса Mollicutes, порядка Mycoplasmatales, семейства Mycoplasmataceae, рода Mycoplasma, вида Mycoplasma gallisepticum [2].
У М. gallisepticum отсутствует ригидная клеточная стенка по причине отсутствия собственной системы ферментов, которые обеспечивают процесс синтеза стенки. Вместо нее микоплазмы имеют трехслойную липопротеиновую цитоплазматическую мембрану. В состав клеточной мембраны данного вида микоплазм входит до 1/3 протеинов от массы мембраны, которые обеспечивает прикрепление и внедрение в клетку-хозяина. Внутри клетки находятся ДНК, РНК, рибосомы и цитоплазма [1, 2].
Геном М. gallisepticum представлен молекулой ДНК длиной около 985430-1100000 п.н. В частности, для производственного штамма «S6» размер нуклеиновой кислоты составляет 985433 п.н. [3].
В геноме бактерии находится 879 генов: 816 из них кодируют структурные и функциональные протеины, а 58 - разнообразные РНК. Среди указанного количества имеются 5 псевдогенов. 432 белка кодируются генами прямой цепи (+), 384 - комплементарной цепи (-). Информацию о 39 разновидностях РНК данного штамма микоплазм несет цепь (-), а о 19 - цепь (+). При оценке протеома данного микроорганизма определено, что максимальная длина транскрибируемого белка данным штаммом составляет 2019 а.о., а минимальная - 30 а.о. Прямая цепь несет в небе информацию большего количества генов белков и молекул РНК [4].
Проведен анализ молекул РНК, которые кодируются геномом М. gallisepticum. Информацию об antisense_RNA несут 3 гена прямой цепи и 13 генов комплементарной цепи ДНК, rRNA - 6 генов прямой цепи и 1 ген комплементарной цепи, tRNA - 28 генов прямой цепи и 4 гена комплементарной цепи, tmRNA - 1 ген отрицательной цепи, ncRNA - 1 ген положительной цепи, RNase_P_RNA - 1 ген прямой цепи. Antisense_RNA - антисмысловой транскриптом, представляющий собой одноцепочечную РНК, комплементарную белку, кодирующему матричную РНК (мРНК) микоплазмы, с которой она гибридизуется, и, тем самым, блокирует ее трансляцию. Иными словами, данная РНК регулирует экспрессию генов данного микроорганизма. rRNA - рибосомальная РНК микоплазмы, гены которой включают в себя высоко консервативные и вариабельные последовательности, в частности, 16S rRNA позволяющие идентифицировать М. gallisepticum от других микоплазм. tRNA - транспортная РНК, которая доставляет активированные остатки аминокислот в рибосому и обеспечивает их включение в белковую цепь, tmRNA - небольшая молекула РНК длиной от 260 до 430 и.о., участвующая в репарации проблемных участков белковых соединений. ncRNA - не кодирующая РНК (non-coding), которые выполняют функции факторов транскрипции с помощью различных механизмов. RNase_P_RNA - ферментативная РНК, которая играет роль рибозима, который катализирует расщепление молекул РНК [5].
Геном М. gallisepticum кодирует белки длиной от 30 до 2019 а.о. При этом самыми мелкими являются рибосомальные белки, а наиболее крупные - ДНК-полимеразы, а также гиразы, эндонуклеазы и т.д. [1, 4].
В состав М. gallisepticum входит белок Р1-адгезии с молекулярной массой 168 кДа, который участвует в прикреплении микоплазмы к поверхности эпителия. Анти-Р 1-антитела блокируют прикрепление микоплазм к клеткам. Белки группы HMW1 - HMW3 контролируют распределение белка Р1 на поверхности микоплазмы [6].
Микоплазмы характеризуются выраженным антигенным полиморфизмом. Среди микоплазм выделяют различные серовары. Антигенами микоплазм являются белки-адгезины, фосфолипиды, гликолипиды, полисахаридные компоненты. Адгезины микоплазм имеют высокую гомологию к структурным белкам млекопитающих, что позволяет им уклоняться от иммунной системы хозяина (антигенная мимикрия) [7].
Филогения микоплазмы интересна с точки зрения дегенерации генома. В течение их эволюционной истории произошло многократное уменьшение размера генома, обычный генетический код был изменен, и общая скорость эволюции была не характерно высокой. Одна из предполагаемых причин уменьшения размера генома - эволюция в сторону строгого паразитизма, в результате чего большая часть их метаболических механизмов устарела. Микоплазма также уникально использует аминокислотный кодон UGA, в качестве дополнительного кодона для триптофана, в то время как другие организмы применяют его в качестве стоп-кодона [6, 8].
Гибель клетки чаще всего происходит из-за длительной персистенции М. gallisepticum в ней. В клетке микоплазма может довольно долго персистировать, что может привести к смерти клетки. Данный микроорганизм вызывает хронические респираторные заболевания у большинства видов птиц. Лечение осложнено тем, что эти бактерии длительное время живут в клетках хозяина [9].
M. gallisepticum приводит к заболеванию цыплят, к снижению продуктивности взрослых птиц, а также к уменьшению общего количества яиц, их размеров и к ухудшению показателей бройлеров [2].
Для определения титра инфекционной активности М. gallisepticum используют реакцию гемагглютинации (РГА) (прототип) [5]. Данный анализ применяется для идентификации микроорганизмов по гемагглютинирующей активности, т.е. способности гемагглютинировать эритроциты животных. Гемагглютинация - склеивание эритроцитов под влиянием гемагглютинина микоплазмы. Данный метод хорошо зарекомендовал себя, однако он имеет некоторые недостатки: 1) невысокая чувствительность и специфичность; 2) определенная степень субъективности при оценке результатов исследования; 3) необходимость применения свежих эритроцитов крови барана.
В связи с этим целесообразно провести поиск альтернативного способа опосредованного определения инфекционной активности М. gallisepticum на основе метода ПЦР в режиме реального времени.
Предложенный метод является высокочувствительным и специфичным, объективным и позволяет определять титр инфекционной активности М. gallisepticum в суспензиях в течение 2,5 часов, не требует использования эритроцитов барана для анализа.
Задачей настоящего изобретения является разработка высокочувствительного, высокоспецифичного, объективного, не предполагающего применения эритроцитов барана способа опосредованного определения титра инфекционной активности М. gallisepticum методом ПЦР в режиме реального времени с целью устранения вышеуказанных недостатков.
Данная задача решена благодаря созданию способа опосредованного определения титра инфекционной активности М. gallisepticum при использовании метода ПЦР в режиме реального времени.
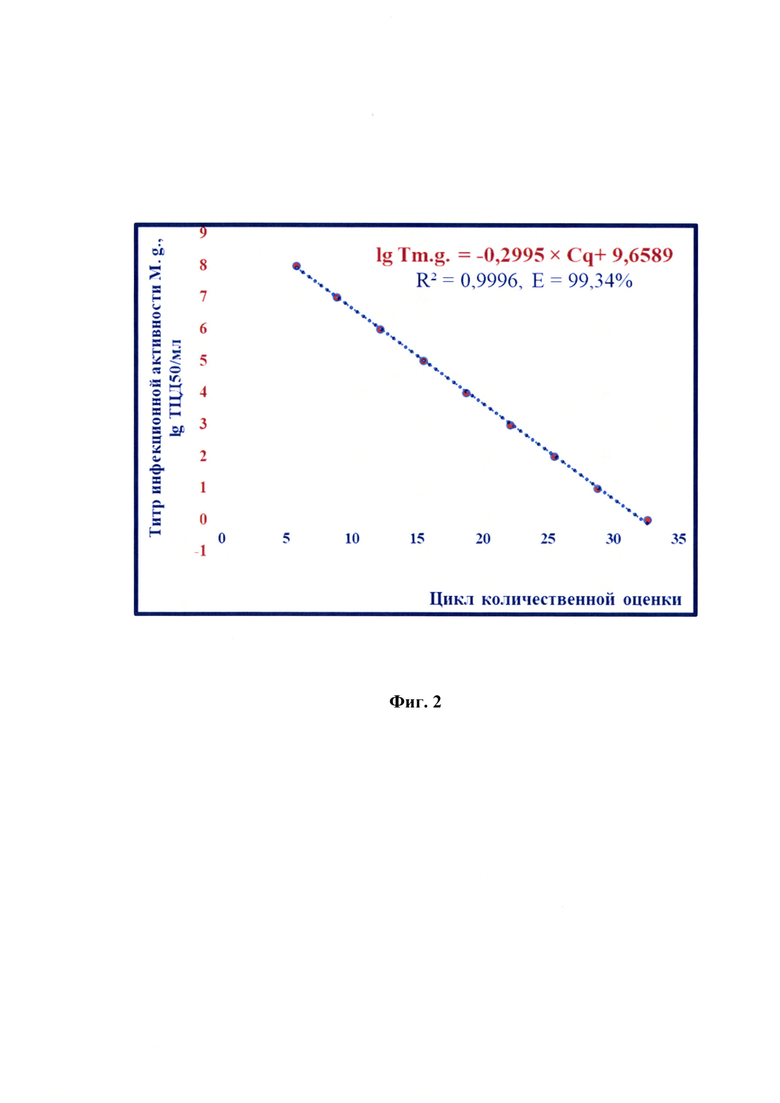
Технический результат изобретения заключается в том, что разработанный способ дает возможность: 1) повысить чувствительность и специфичность за счет применения высокоспецифичных оригинальных праймеров и ДНК-зонда, рассчитанных для 16S rRNA-гена М. gallisepticum; 2) повысить достоверность проводимого анализа благодаря установлению зависимости между титром инфекционной активности М. gallisepticum (TM.g.) и циклом количественной оценки (Cq), представленной в виде логарифмической функции:
lg TM.g.=-0,2995×Cq+9,6589
с высокой достоверностью аппроксимации (R2=0,9996) и эффективностью амплификации 99,34%. Предложенная модель позволяет опосредованно определять титр инфекционной активности М. gallisepticum в исследуемом материале.
Сущность изобретения отражена на графических изображениях:
Фиг. 1 - Дизайн олигонуклеотидных праймеров и зонда, рассчитанных для 16S rRNA-гена М. gallisepticum. Примечание: А - прямой праймер и зонд, Б - обратный праймер.
Фиг. 2 - Зависимость титра инфекционной активности М. gallisepticum (TM.g.) с помощью метода ПЦР в режиме реального времени и величины цикла количественной оценки (Cq) (n=7).
SEQ ID NO: 1 представляет последовательность 16S rRNA-гена ДНК М. gallisepticum;
SEQ ID No: 2 представляет последовательность аминокислот, кодируемых 16S rRNA-геном М. gallisepticum;
SEQ ID No: 3 представляет последовательность праймера MG-F-314650;
SEQ ID No: 4 представляет последовательность праймера MG-R-314817;
SEQ ID No: 5 представляет последовательность праймера MG-Р-314687.
Сущность изобретения заключается в новом подходе к опосредованному определению титра инфекционной активности М. gallisepticum с применением метода ПЦР в режиме реального времени. Заявляемый способ основан на: 1) элюировании ДНК М. gallisepticum с применением гуанидинизотиоцианата, пропанола-2 с выделением на стекловолокнистых фильтрах; 2) амплификации специфического фрагмента 16S rRNA-гена М. gallisepticum с применение оригинальных специфических прямого и обратного праймеров, а также молекулярного зонда, меченого флуоресцентным красителем FAM (λmax флуоресценции=520 нм) и тушителем свечения RTQ-1 (λmaxпоглощения=520 нм); 3) детекции ампликонов с помощью флуоресцентного свечения и отображения накопления сигнала в виде сигмоиды, стремящейся к экспоненте; 4) определении активности М. gallisepticum с применением логарифмической функции, выраженной в виде логарифмического уравнения:
lg TM.g.=-0,2995×Cq+9,6589
с высокой достоверностью аппроксимации (R2=0,9996) и эффективностью амплификации 99,34%.
В настоящее время метод ПЦР в режиме реального времени применяют для индикации нуклеиновых кислот различных инфекционных агентов, в том числе микоплазм [4-10]. Для определения активности М. gallisepticum ранее данный метод с применением разработанной системы оригинальных праймеров и молекулярного зонда не использовался. Таким образом, сведений об аналогах предлагаемого способа опосредованного определения активности М. gallisepticum авторами не обнаружено.
Разработанный способ опосредованного определения титра инфекционной активности М. gallisepticum с помощью метода ПЦР в режиме реального времени по сравнению с прототипом отличается более высокой чувствительностью и специфичностью, объективностью получаемых результатов.
В отличие от прототипа разработанный способ включает этап элюирования ДНК микоплазмы с помощью ГТЦ и пропанола-2 на стекловолокнистых фильтрах; амплификацию специфического фрагмента 16S rRNA-гена М. gallisepticum с применение оригинальных специфических прямого и обратного праймеров, а также молекулярного зонда, меченого флуоресцентным красителем FAM и тушителем свечения RTQ-1; детекции флуоресцентного сигнала в виде сигмоиды, стремящейся к экспоненте; новый подход к методике расчета активности М. gallisepticum с применением модели зависимости цикла количественной оценки для кривой флуоресценции и активности микоплазмы. Применение предложенного способа позволит повысить специфичность и чувствительность анализа; удешевить исследование; увеличить достоверность проводимого анализа. Таким образом, актуально применять предложенный способ для опосредованного определения инфекционной активности М. gallisepticum в исследуемом материале.
Ключевым элементом заявляемого способа является определение значений цикла количественной оценки в реакции амплификации ДНК М. gallisepticum в ПЦР режиме реального времени и расчет титра инфекционной активности данного микроорганизма с использованием разработанной логарифмической модели.
Сопоставительный анализ с прототипом позволяет сделать вывод, что новизна и изобретательский уровень заявляемого изобретения заключается в применении способа количественной ПЦР в режиме реального времени, оригинальных специфичных праймеров и молекулярного зонда, рассчитанных на участок 16S rRNA-гена М. gallisepticum, и разработанной математической функции зависимости величины цикла количественной оценки и титра инфекционной активности М. gallisepticum.
Сущность предлагаемого изобретения пояснена на графическом материале - графике зависимости величины цикла количественной оценки (Cq) и титра инфекционной активности М. gallisepticum (TM.g.) (n=7) (фиг. 2).
С целью определения титра инфекционной активности М. gallisepticum подготавливают контрольную панель стандартов М. gallisepticum, в качестве которых используют лиофильно высушенные суспензии со следующими значениями активности в РГА: 8,0; 7,0; 6,0; 5,0; 4,0; 3,0; 2,0; 1,0; 0,0 log2. Титр выражается в виде разведений возбудителя и обозначается как log2. В качестве отрицательного контроля применяют деионизированную воду.
Из всех контролей и тестируемых проб выделяют ДНК. Полученный экстракт нуклеиновой кислоты хранят при температуре минус 20±2°С или сразу используют в дальнейшей работе. Проводят ПЦР в режиме реального времени для исследования контрольных образцов и проб. Для постановки реакции готовят реакционную смесь, рецептура которой представлена в таблице 1. Дизайн праймеров и молекулярного зонда отражены в таблице 2 и фиг. 1. Расчет праймеров и зонда проводили на основании нуклеотидных последовательностей 16S rRNA-гена М. gallisepticum различных штаммов и изолятов, опубликованных в базах данных GenBank [3].
В качестве гомологичных участку 16S rRNA-гена М. gallisepticum олигонуклеотидов используют:
MG-F-314650-праймер (5'-GAGCTAATCTGTAAAGTTGGTCT-3'), MG-R-314817-праймер (5'-GGCTGCAATTCGCCCTCA-3') и MG-P-314687-FAM/RTQ1-зонд (5'-FAM-GCTTCCTTGCGGTTAGCAA-RTQ1-3') в концентрации 7 пМ на реакцию. Для элонгации применяют дезоксирибонуклеозидтрифосфаты с их концентрацией в реакционной смеси по 0,25 мМ каждого. В качестве основы используют буферный раствор (5х), содержание которого составляет 20% от общего объема реакционной смеси. Буферный раствор включает в свой состав ионы калия (К+) (5×10-2 М) и диметилсульфооксид (DMSO) (1%). В смесь также добавляют хлорида магния до концентрации 3,5 мМ. В качестве катализатора реакции применяют Taq ДНК-зависимую ДНК-полимеразу (10 ед.). Элюаты суммарной ДНК каждого образца добавляют к реакционной смеси по 5 мкл. Итоговый объем смеси для проведения одной реакции составляет 25 мкл.
Олигонуклеотиды для ДНК-матрицы подбирали в соответствии с рядом общих правил, которые отражены в работах В. Deiman и R. Sooknanan. Длины MG-F-314650-праймер, MG-R-314817-праймер и MG-P-314687-FAM/RTQ1-зонд составляют 23, 18, 19 н.о. каждый, что соответствует требованиям. Молекулярный вес олигонуклеотидов прямого, обратного праймеров и зонда равен 7093,7; 5435,6; 5794,8, соответственно. Праймеры и зонд очищены в полиакриламидном геле и с помощью высокоэффективной жидкостной хроматографии, соответственно. Нуклеотидная последовательность зонда не комплементарна олигонуклеотидным праймерам. Отсутствуют 4 и более подряд одинаковых нуклеотидов в цепи праймеров и зонда. Флуорофор FAM присоединен к 5'-концу, а гаситель флуоресценции RTQ1 - к 3'-концу. Данные условия соответствуют требованиям, предъявляемым к олигонуклеотидным праймерам и молекулярному зонду, которые участвуют в ПЦР в режиме реального времени. В качестве флуоресцентного красителя был выбран FAM с длиной волны максимальной флуоресценцией 520 нм. Для тушения свечения использовали гаситель флуоресценции RTQ1 с длиной волны максимального поглощения при 520 нм и возможном диапазоне гашения 470-570 нм. Иными словами, была выбрана подходящая пара «флуорофор-гаситель».
При анализе нуклеотидных последовательностей олигонуклеотидов установили, что для праймеров и зонда не характерно образование «шпилек», а также не выявлено 3'-комплементарности и сайтов, отжигающих сами на себя. Расчет вероятности образования «шпилек» и димеров олигонуклеотидов проводили при условии, что минимальное количество пар оснований, необходимых для димеризации, - 5, а для образования «шпилек», - 4.
Проведено определение температур плавления (Tm) для олигонуклеотидных праймеров и зонда. Для оценки температуры плавления олигонуклеотидов применяли метод, учитывающий концентрации ионов К+ и диметилсульфооксида (DMSO).
Физические, термодинамические константы и расчет температур плавления разработанных олигонуклеотидных праймеров и молекулярного зонда представлены в таблице 3. Из нее следует, что энтропия, энергия Гиббса и энтальпия для прямого праймера составили 176,2 ккал/моль, 26,8 ккал/моль, 465,4 кал/(K×моль), соответственно. Энтропия, энергия Гиббса и энтальпия для обратного праймера составили 153,0 ккал/моль, 25,4 ккал/моль, 395,2 кал/(K×моль), соответственно. Энтропия, энергия Гиббса и энтальпия для молекулярного зонда - 158,1 ккал/моль, 25,5 ккал/моль, 411,2 кал/(K×моль), соответственно. Данные значения необходимы для расчета температур плавления представленных олигонуклеотидов. Tm для прямого, обратного праймеров и зонда теоретически составили 59, 58, 60°С, соответственно.
Экспериментально было выявлено, что оптимальная температура отжига рассматриваемых олигонуклеотидов составляет 62°С. Для проведения ПЦР в режиме реального времени было решено проводить гибридизацию праймеров и зонда с участком 16S rRNA-гена М. gallisepticum при температуре 62°С.
Последовательности разработанных праймеров и молекулярного зонда проверили на наличие нежелательных совпадений с другими последовательностями нуклеиновых кислот с использованием Банка данных последовательности нуклеиновых кислот М. gallisepticum. Последовательности праймеров и зонда также проанализировали на наличие внутренних вторичных структур с помощью программы сворачивания нуклеиновых кислот с помощью программы Mfold. Выявлено, что для разработанных олигонуклеотидов нежелательных совпадений с другими последовательностями нуклеиновых кислот, а также наличия внутренних вторичных структур не обнаружено.
Постановку реакции осуществляли в детектирующем термоциклере любой марки при температурных и временных параметрах, сведения о которых представлены в таблице 4.
Перед проведением ПЦР в режиме реального времени осуществляют предварительный прогрев смеси при температуре 95°С в течение 8 мин. для активации фермента Taq ДНК-полимеразы.
ПЦР в режиме реального времени включает в себя 3 подэтапа: денатурацию, отжиг олигонуклеотидов, элонгацию. Денатурацию осуществляют при температуре 95°С в течение 10 с, отжиг олигонуклеотидов и элонгацию проводят в совмещенном режиме при температуре 62°С в течение 45 с, что оптимизирует реакцию по времени анализа (табл. 4).
Результаты ПЦР в режиме реального времени анализируют, оценивая и сравнивая графики накопления флуоресцентного сигнала по значениям циклов количественной оценки Cq, определенных с помощью пересечения пороговой линии и логарифмическим отображением функции F1=f(Cq). Учет результатов в реакции происходит на каждом цикле. Флуориметр определяет уровень флуоресценции и строит кинетическую кривую в координатах: уровень флуоресценции - цикл реакции амплификации. В случае присутствия в исследуемой пробе специфической ДНК-матрицы М. gallisepticum кинетическая кривая имеет экспоненциальную зависимость (график представлен в виде сигмоиды). Положительными считаются пробы, которые соответствуют логистической кривой, полученные при анализе флуоресценции красителя FAM, входящего в состав молекулярного зонда. Пробы считаются отрицательными, если при их анализе отсутствует логистическая кривая в виде графика, приближенного к экспоненте.
Устанавливают зависимость между циклом количественной оценки и титром инфекционной активности М. gallisepticum в сырье для вакцин в процессе построения логарифмической функции. Оценивают величину эффективности реакции амплификации (Е) по формуле:
Е=(10-1/k-1),
где k - угловой коэффициент в зависимости:
Cq=-k×lg TM.g.+Ь.
На основе разработанной модели рассчитывают значение титра инфекционной активности М. gallisepticum в исследуемом материале.
Сущность предлагаемого изобретения пояснена примерами его использования, которые не ограничивают объем изобретения.
Пример 1. Выявление зависимости титра инфекционной активности М. gallisepticum и цикла количественной оценки ПЦР в виде логарифмической функции.
Для выявления зависимости титра инфекционной активности М. gallisepticum и цикла количественной оценки ПЦР подготавливали контрольную панель стандартов М. gallisepticum, в качестве которых использовали лиофилизированные бактериальные суспензии с титрами активности: 0,0; 1,0; 2,0; 3,0; 4,0; 5,0; 6,0; 7,0; 8,0 log2, которые растворяли в 1/15 М фосфатном буферном растворе до требуемого объема. В качестве отрицательного контроля применяли суспензию клеток ВНК-21/SUSP/ARRIAH, не контаминированную микроорганизмами.
Из всех стандартных положительных образцов и отрицательных контролей, а также тестируемых проб выделяли ДНК в соответствии с методикой, представленной выше.
Проводили ПЦР в режиме реального времени для исследования стандартов, как описано выше. Результаты реакции анализировали, оценивая и сравнивая графики накопления флуоресцентного сигнала по значениям циклов количественной оценки Cq, определенных с помощью пересечения пороговой линии и логарифмическим отображением функции F1=f(Cq). Устанавливали зависимость между циклом количественной оценки и титром инфекционной активности М. gallisepticum в процессе построения логарифмической функции. Полученные данные отражены на фиг.2 и выражены в виде логарифмической функции: lg TM.g.=-0,2995×Cq+9,6589.
Предложенная модель позволяет опосредованного определять титр инфекционной активности М. gallisepticum с помощью разработанного способа.
Пример 2. Применение способа опосредованного определения титра инфекционной активности М. gallisepticum с использованием ПЦР в режиме реального времени.
В исследовании использовали 6 бактериальных суспензий М. gallisepticum с титрами инфекционной активности 6,50; 6,75; 7,00; 7,25; 7,50; 7,75 log2, соответственно (пробы №1-6). В качестве положительного контроля применяли культуральную суспензию М. gallisepticum с титром инфекционной активности 7,00 log2. В качестве отрицательного контроля применяли суспензию клеток BHK-21/SUSP/ARRIAH, не зараженную микроорганизмами. Испытуемые пробы и контрольные образцы исследовали в 7 повторностях. Этап элюирования ДНК и постановку ПЦР в режиме реального времени проводили, как описано выше.
Средние значения циклов количественной оценки для проб №1-6 составляли 10,59±0,01, 9,69±0,01, 8,82±0,01, 7,98±0,01, 7,26±0,01, 6,39±0,01, соответственно. Пользуясь разработанной логарифмической функцией:
lg TM.g.=-0,2995×Cq+9,6589,
рассчитали средние значения титра инфекционной активности М. gallisepticum для проб №1-6, которые составили 6,49; 6,76; 7,01; 7,27; 7,48; 7,75 log2, соответственно. Для положительного контроля значение цикла количественной оценки составило 8,88±0,01, что соответствовало титру инфекционной активности М. gallisepticum, равному 7,00 log2. Для отрицательных контролей экспоненциальные графики не были сформированы, что означало отсутствие М. gallisepticum в данных образцах. Полученные результаты свидетельствовали о высокой степени точности разработанного способа опосредованного определения титра инфекционной активности М. gallisepticum с помощью ПЦР в режиме реального времени. Таким образом, разработанный способ позволяет рассчитывать титр инфекционной активности М. gallisepticum методом ПЦР в режиме реального времени.
Пример 3. Выявление степени достоверности определения титра инфекционной активности М. gallisepticum с применением разработанного способа.
Для анализа использовали 294 культуральной суспензии М. gallisepticum с титрами инфекционной активности от 1,00 до 8,00 log2. В качестве положительного контроля применяли культуральную суспензию М. gallisepticum с титром инфекционной активности вируса 7,00 log2. Испытуемые пробы и контрольные образцы исследовали в трех повторностях. Этапы экстрагирования нуклеиновых кислот и постановку ПЦР в режиме реального времени проводили, как отражено выше. Результаты анализа представлены в таблице 5.
Интерпретацию результатов проводили, пользуясь разработанной логарифмической функцией: lg TM.g.=-0,2995×Cq+9,6589, с получением значений TM.g. для каждой из 294 проб. Для положительного контроля значение цикла количественной оценки составило 8,88±0,01, что соответствовало титру инфекционной активности М. gallisepticum, равному 7,00 log2. Для отрицательного контроля экспоненциальный график не был сформирован, что означало отсутствие М. gallisepticum в данных образцах.
Анализируемые пробы и контроли также тестировали в ПЦР в режиме реального времени. Выявили, что данные, полученные с помощью разработанного способа, коррелировали со значениями стандартов на 99,02-100,00% для 8,0-5,0 log2 (n=98), на 97,98-99,68% для 4,9-2,0 log2 (n=98), на 96,36-99,01% для 1,9-0,5 log2 (n=98). Полученные результаты свидетельствовали о высокой степени точности разработанного способа опосредованного определения титра инфекционной активности М. gallisepticum с помощью ПЦР в режиме реального времени. Таким образом, разработанный способ позволяет с высокой степенью достоверности определять титр инфекционной активности М. gallisepticum методом ПЦР в режиме реального времени.
Пример 4. Определение аналитической чувствительности способа опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в режиме реального времени.
При определении аналитической чувствительности способа опосредованного определения титра инфекционной активности М. gallisepticum методом ПЦР в режиме реального времени подготавливали серию стандартов М. gallisepticum с титрами инфекционной активности, равными 1,00-8,00 log2 с шагом 0,1 log2. Контрольные образцы тестировали в 7 повторностях. Этапы элюирования нуклеиновой кислоты и постановку ПЦР в режиме реального времени, как описано выше. Выявлено, что с достоверностью 96,36-100,00% разработанным способом определены титры инфекционной активности М. gallisepticum со значениями от 0,5 до 8,0 log2.
Аналитическая чувствительность способа опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в режиме реального времени составляет не менее 0,5 log2 с достоверностью результатов исследования не менее 96,36%.
Пример 5. Исследование специфичности способа опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в режиме реального времени.
При анализе специфичности способа опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в режиме реального времени, исследовали бактериальные суспензии М. gallisepticum и Mycoplasma sinoviae, а также вирусные суспензии: штамма «ВНИИЗЖ» вируса бешенства, штамма «Рич» возбудителя коронавирусного энтерита собак, штамма «Rockborn» возбудителя чумы плотоядных. Количество инфекционных доз вирусов в суспензиях составляло не менее 4,0 lg ТЦД5о/см3. Исследования проводили в 5 повторностях.
Этапы исследования проводили, как описано выше. Для проб, содержащих другие патогены, не наблюдалось формирования графиков экспоненты, и они не выходили за пороговый уровень флуоресцентного сигнала (0,005 у.е.) (табл. 6). Таким образом, разработанный способ является специфичным по отношению к М. gallisepticum и может быть использован для его количественного определения.
Основными преимуществами предлагаемого изобретения является возможность исключить вероятность контаминации в ходе реакции и субъективность при учете результатов анализа; снизить время проведения анализа бактериального сырья для вакцин до 2,5 ч; увеличить специфичность и чувствительность анализа по определению титра инфекционной активности М. gallisepticum. В предлагаемом изобретении между титром инфекционной активности М. gallisepticum (TM.g.) и циклом количественной оценки (Cq) установлена зависимость, отраженная в виде логарифмической функции: lg TM.g.=-0,2995×Cq+9,6589, (R2=0,9996, E=99,34%).
Разработанная логарифмическая модель дает возможность определять титр инфекционной активности М. gallisepticum.
Предлагаемое изобретение позволяет быстро и с высокой степенью достоверности рассчитывать титр инфекционной активности М. gallisepticum в исследуемом материале (сырье для вакцин) на основе разработанного способа с применением оригинальных специфических олигонуклеотидных праймеров и молекулярного зонда для участка 16S rRNA-гена М. gallisepticum и разработанной логарифмической модели.
Источники информации, принятые во внимание при составлении описания изобретения к заявке на выдачу патента РФ на изобретение «Способ опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в режиме реального времени»:
1. Ali В.Н, Ali A.J, Yosif Е.Н. Isolation and molecular characterization of Mycoplasma synoviae from infected chickens with respiratory signs. Iraqi J. Agri. Sci. 2020;51(5):1466-1473.
2. Abed A.A, Al-Iedani A.A, Neamah A.J. Sequencing based phylogenetic analysis of local Mycoplasma gallisepticum of broiler chickens in Al-Dewaniyah Province / Iraq. Ann. Rom. Soc. Cell Bio. 2021;25(5):2719-2738.
3. GenBank. Mycoplasma gallisepticum. URL: https://www.ncbi.nlm.nih.gov/nuccore/?term=Mycoplasma+gallisepticum. (Дата обращения: 05.06.2023).
4. Mahmmoud EN, Hamad MA, Khudhur ZN. Detection of Mycoplasma gallisepticum in broiler chickens by PCR. Open Vet J. 2022 May-Jun;12(3):329-334. doi: 10.5455/OVJ.2022.v12.i3.4. Epub 2022 May 21. PMID: 35821769; PMCID: PMC9270947.
5. Ishfaq M, Hu W, Khan MZ, Ahmad I, Guo W, Li J. Current status of vaccine research, development, and challenges of vaccines for Mycoplasma gallisepticum. Poult Sci. 2020 Sep;99(9):4195-4202. doi: 10.1016/j.psj.2020.06.014. Epub 2020 Jun 27. PMID: 32867963; PMCID: PMC7598112.
6. Abbas N, Suleman M, Khan N.A, АН I, Rauf M, Rahman S. Prevalence of Mycoplasma gallisepticum in poultry and wild life birds suspected of chronic respiratory disease in Northern Pakistan. Pakistan J. Zool. 2018;50(3): 1071-1077.
7. Aghabalaei E, Hedaiati M.H. Detection of urogenital mycoplasmas using culture and PCR: a descriptive pilot study. J. Anim. Vet. Adv. 2012; 11 (16):2905-2909.
8. Al-dabbagh S.Y.A, Rasheed B.Y, AL-Jumaa Z.M. Molecular diagnosis of Mycoplasma gallisepticum in Turkey in Mosul city. Vet. Pract. 2021;22(1):1-3.
9. Ipoutcha T, Gourgues G, Lartigue C, Blanchard A, Sirand-Pugnet P. Genome Engineering in Mycoplasma gallisepticum Using Exogenous Recombination Systems. ACS Synth Biol. 2022 Mar 18;11(3): 1060-1067. doi: 10.1021/acssynbio.lc00541. Epub 2022 Feb 15. PMID: 35167277.
10. Semashko ТА, Arzamasov AA, Evsyutina DV, Garanina IA, Matyushkina DS, Ladygina VG, Pobeguts OV, Fisunov GY, Govorun VM. Role of DNA modifications in Mycoplasma gallisepticum. PLoS One. 2022 Nov 22;17(11):e0277819. doi: 10.1371/journal.pone.0277819. PMID: 36413541; PMCID: PMC9681074.
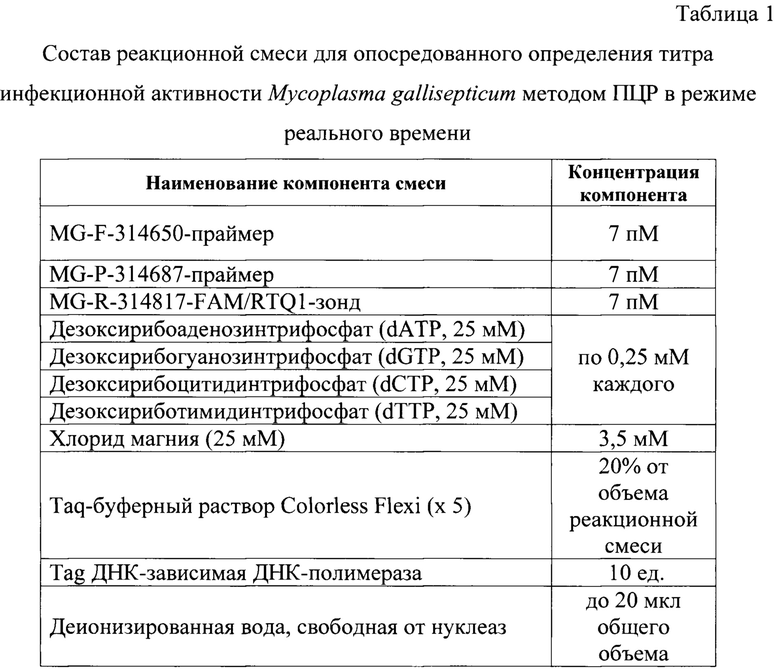
Примечание: объем вносимого элюата ДНК - 5 мкл,
объем реакционной смеси - 25 мкл. 
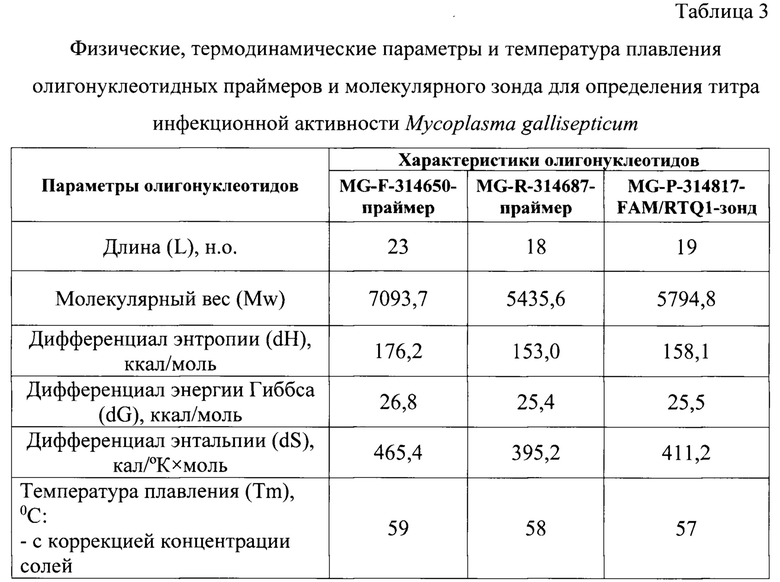
Примечание: термодинамические параметры разработанных олигонуклеотидных праймеров рассчитаны при условии: концентрация NaCl 1 М,
температура 25°С,
водородный показатель (рН) 7,0.

Примечание: * - на данном подэтапе регистрируют степень флуоресцентного сигнала.

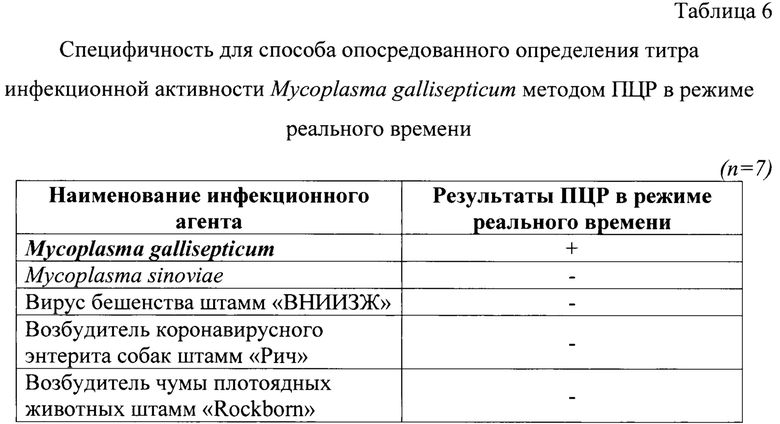
Примечание: «+» - наличие экспоненциального графика (положительный образец),
«-» - отсутствие экспоненциального графика (отрицательный образец).
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Mycoplasma
gallisepticum titre xml.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2024-03-26">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-06-21</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>516</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-06-21</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБУ "Федеральный центр охраны
здоровья животных" (ФГБУ "ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin>Federal State-Financed Institution Federal
Centre for Animal Health (FGBI ARRIAH)</ApplicantNameLatin>
<InventorName languageCode="ru">Доронин Максим
Игоревич</InventorName>
<InventorNameLatin>Doronin Maksim Igorevich </InventorNameLatin>
<InventionTitle languageCode="ru">Способ опосредованного определения
титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в
режиме реального времени</InventionTitle>
<SequenceTotalQuantity>5</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>2814</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..2814</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>M. gallisepticum </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaaaaaaactaaagctcaaaaagatcaacaaaaaaaatcgcaattta
aaaagagattttgatttacctttacatttttaatcggaagcataacaattataagttcttttttaattcc
gctttcattaaaatgaatttgagctcctcttaatatagaagaggatcttgaaattaatattaaagatgat
ttaaatggttcaagatttatctcagagataaatagtgataaccttccagagtttgttgatgcatttttta
acaacaaacaatattcaattgatgatttaaatttaaaactagcaggagttaattacattactggttgagt
ttatgttcaagcttcagtaaaacctaatgttaaagtagctaaaaaatatcaagatgcagttaaactattt
aaactagaatttaaaaactcacaagttaaattattcgatcctaacgtgacaacatttttaattaaacctg
aaaataacactaaagaattcactagcgagtttcaaactagagaagatcttgaaagctttttaaataatgc
agataatatttcactatcagagcttcaagataagcttaatttaactttttttgataaaaataacgttccg
ctaaatttatataccggagctgtagttacgctaagcaatcctgttaaattcactatcgcatcatatgtaa
tggattataaaattacatgacaaataccggcagctaatgacgtattttacggaaacttatattcatttag
cttaaatggaaaattaaatctatgactaaaaaaaataaattcaatttcaagtttatacagtgatgttgat
gacattgactttaaaaaacttcaatcaaaagaaatttttaaaaaccttgaagaggttcaaaacttttcaa
ccagtattgaaactttagatcaattctataacgttcctgatgatttagtagttaaagggcaagactttaa
actaggatactacgttaataaaacagcacaaacctttgacaacgttgaatcatttttagataacagcaac
ttaatatcaagcggttacttaactagaatgacctacgatgataaatataaagttgttatatctaaaccta
atttagatacctttactaaagcatcatctgaaacccaaagaaaatacctacaaagctttaaacaaaataa
agctccagttaaagtaactatcgcaaataccgtcgatagaactgtagaagtattttataccgacttttca
tttaataaaagtccgcttgttaacggaaaatatttattctttaacatgtctcctccaggtgataaattcg
tctactatacaaaagatgtaaatacttgaagaatacctggagttataaaaactcatgataataaactaat
ttttaacgctgataaaagggttaacaataaagatgaccgtgggcatctagaacaagacataagagtatct
gaagatggtggaaaaatctgatcaaatcctcaaactattgttagaatttcagctaaaaataccaaagatg
gtgcaaataaaggtcaagtaattgacggaaccatgttagaggtatttgataaaagcattaacaagcataa
acttttatatgtttttggagtttctagttacttacatacagttcacgatgaatgaaccattagaggtgat
ggaaatacttcagggctagtaaataacaataagtgaatgctatttattgataaagaaaatctaaagcaaa
aatttgtagcaaaacctattacagtcgatggtgatagtaactgatttaaagtatttaaagttaaaaatag
ttctcgtgttcaatgaaaagatattgatgatagaacccagcttgacgaagctaatataataattgataac
tcattgaaaaatggaactataactggaaatgtctacgataaagttccaagtaatatctttagcactttcg
gtgagcctggtagaaattcaagatcatcagaaatttcagaatatatatcaaaatactcaatttgagatca
aagatgaccaaattactttgtttttaatagaatgcaaaaaaatgcttactttgcaaattctagatactac
tttgataacattttatgaactttccaagttgaaagtatcgatggcggaaaaacctgaactaatttaagaa
acgtatctccatttttaaatagaaaagatcaaagatgatacataaatggtgttggaaacggaattcaact
taatcatcaacgtaattcaaatggagatgttatagcagttttcccattctattttgctaacaaaagaaag
ctagaacgttctaaattaatggcaactaaagatggcggaaaaacctgatatgaacttgcagattatccat
ttaaacttcttcctcctggagcttctgaatcatctataagtgaagatagtaaaggtaatttatgatgact
tgccagaagatctgatggaagagttttcacacttattaaatcagaagacggtggaaaaaactgaaaagta
atctttaacgctaaaagaggaatttttacaggacaatttattggatctactatctttaacattaaatcac
taggagagaaattaattttctcagctagtcataatggtggtggaaaaatttatctagttgatataaataa
tcctagaacccttgaaccaatttacaccataaatagaggttcatatggttattcaactacagtaactttt
aacgaaaatgataactactttgacgtattaactttttacgaggtaaatagccctcaaaaaggaagagaca
tcaacctaaaaagacttagagtctttgtggttagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>938</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..938</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>M. gallisepticum </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MKKTKAQKDQQKKSQFKKRFWFTFTFLIGSITIISSFLIPLSLKWIWAP
LNIEEDLEINIKDDLNGSRFISEINSDNLPEFVDAFFNNKQYSIDDLNLKLAGVNYITGWVYVQASVKPN
VKVAKKYQDAVKLFKLEFKNSQVKLFDPNVTTFLIKPENNTKEFTSEFQTREDLESFLNNADNISLSELQ
DKLNLTFFDKNNVPLNLYTGAVVTLSNPVKFTIASYVMDYKITWQIPAANDVFYGNLYSFSLNGKLNLWL
KKINSISSLYSDVDDIDFKKLQSKEIFKNLEEVQNFSTSIETLDQFYNVPDDLVVKGQDFKLGYYVNKTA
QTFDNVESFLDNSNLISSGYLTRMTYDDKYKVVISKPNLDTFTKASSETQRKYLQSFKQNKAPVKVTIAN
TVDRTVEVFYTDFSFNKSPLVNGKYLFFNMSPPGDKFVYYTKDVNTWRIPGVIKTHDNKLIFNADKRVNN
KDDRGHLEQDIRVSEDGGKIWSNPQTIVRISAKNTKDGANKGQVIDGTMLEVFDKSINKHKLLYVFGVSS
YLHTVHDEWTIRGDGNTSGLVNNNKWMLFIDKENLKQKFVAKPITVDGDSNWFKVFKVKNSSRVQWKDID
DRTQLDEANIIIDNSLKNGTITGNVYDKVPSNIFSTFGEPGRNSRSSEISEYISKYSIWDQRWPNYFVFN
RMQKNAYFANSRYYFDNILWTFQVESIDGGKTWTNLRNVSPFLNRKDQRWYINGVGNGIQLNHQRNSNGD
VIAVFPFYFANKRKLERSKLMATKDGGKTWYELADYPFKLLPPGASESSISEDSKGNLWWLARRSDGRVF
TLIKSEDGGKNWKVIFNAKRGIFTGQFIGSTIFNIKSLGEKLIFSASHNGGGKIYLVDINNPRTLEPIYT
INRGSYGYSTTVTFNENDNYFDVLTFYEVNSPQKGRDINLKRLRVFVVS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mycoplasma
gallisepticum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagctaatctgtaaagttggtct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mycoplasma
gallisepticum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggctgcaattcgccctca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Mycoplasma
gallinaceum</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcttccttgcggttagcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

Изобретение относится к области биотехнологии. Описан способ опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в режиме реального времени с применением разработанной системы специфичных олигонуклеотидов и математической функции зависимости величины цикла количественной оценки и титра инфекционной активности Mycoplasma gallisepticum. Значение титра инфекционной активности Mycoplasma gallisepticum определяют по величине цикла количественной оценки. На основании установленной величины данного показателя рассчитывают значение титра инфекционной активности возбудителя микоплазмоза. Изобретение позволяет быстро и с высокой степенью достоверности рассчитывать титр инфекционной активности М. gallisepticum в исследуемом материале на основе разработанного способа с применением оригинальных специфических олигонуклеотидных праймеров и молекулярного зонда для участка 16S rRNA-гена М. gallisepticum и разработанной логарифмической модели. 1 з.п. ф-лы, 2 ил., 6 табл., 5 пр.
1. Способ опосредованного определения титра инфекционной активности Mycoplasma gallisepticum методом ПЦР в режиме реального времени, с использованием специфических олигонуклеотидов: MG-F-314650-праймер с дизайном 5’-GAGCTAATCTGTAAAGTTGGTCT-3’, MG-R-314817-праймер с дизайном 5’-GGCTGCAATTCGCCCTCA-3’ и MG-P-314687-FAM/RTQ1-зонд с дизайном 5’-FAM-GCTTCCTTGCGGTTAGCAA-RTQ1-3’, включающий следующие этапы:
- выделение ДНК Mycoplasma gallisepticum с применением гуанидинизотиоцианата, пропанола-2 с выделением на стекловолокнистых фильтрах;
- проведение ПЦР в режиме реального времени с детекцией флуоресцентного сигнала логистических кривых для исследуемых образцов с использованием олигонуклеотидов, которые рассчитаны на участок 16S rRNA-гена Mycoplasma gallisepticum;
- определение цикла количественной оценки по данным ПЦР в режиме реального времени;
- расчет титра инфекционной активности Mycoplasma gallisepticum на основе логарифмической регрессионной функции: lg TM.g.=-0,2995×Cq+9,6589, с достоверностью аппроксимации 0,9996% и эффективностью амплификации 99,34%.
2. Способ по п. 1, где время проведения анализа сокращается до 2,5 ч, аналитическая чувствительность составляет не менее 0,5 log2.
| Ishfaq M, Hu W, Khan MZ, Ahmad I, Guo W, Li J | |||
| Current status of vaccine research, development, and challenges of vaccines for Mycoplasma gallisepticum | |||
| Poult Sci | |||
| Способ восстановления спиралей из вольфрамовой проволоки для электрических ламп накаливания, наполненных газом | 1924 |
|
SU2020A1 |
| WO 2016027135 A1, 25.02.2016 | |||
| СПОСОБ СПЕЦИФИЧНОЙ ДЕТЕКЦИИ МАЛОПРЕДСТАВЛЕННЫХ ФРАКЦИЙ РНК В В БИОЛОГИЧЕСКОМ ОБРАЗЦЕ | 2009 |
|
RU2524115C2 |
Авторы
Даты
2024-07-30—Публикация
2023-08-18—Подача