Изобретение относится к области биотехнологии и производству культуральных вакцин, а именно к способу опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК.
Калицивирусная инфекция кошек является высококонтагиозным вирусным заболеванием, характеризующимся поражением слизистой оболочки ротовой полости и верхних дыхательных путей [1, 2]. Вирус распространен повсеместно и поражает домашних кошек всех пород и возрастов, а также зоопарковых и диких представителей семейства кошачьих.
Вирионы калицивируса кошек имеют пулевидную форму длиной 100-300 нм, диаметром 45-100 нм. На наружной поверхности вирусной частицы имеются выступы в виде шипов длиной 10-12 нм, которые прикреплены к двуслойной липидной оболочке [1].
Возбудитель заболевания Feline calicivirus (FCV), относится к порядку Picornavirales, семейству Caliciviridae, роду Vesivirus. Нуклеиновая кислота вируса представлена одноцепочечной РНК (+) размером около 7690 н.о. 5’-NTR (не транслируемый регион) соответствует 1…19 н.о., ORF1 (2C, протеаза, РНК-полимераза) - 20…5311 н.о., ORF2 (структурный белок VP1) - 5314…7329 н.о., ORF3 (белок VP2 с неизвестной функцией) - 7617…7643 н.о. [3-5].
В различных источниках описана системная инфекция, вызывающая гибель до 60% заболевших животных [6-8]. Калицивирус характеризуется высокой степенью изменчивости и большим антигенным разнообразием штаммов, что значительно снижает эффективность существующих вакцин.
Система мер для борьбы с калицивирозом кошек и его профилактика предусматривает иммунизацию домашних животных [8]. Для этой цели применяют вакцинные препараты. При их изготовлении вируссодержащее сырье исследуют на определение титра инфекционной активности калицивируса кошек для оценки его активности в клетках. В 1,0 см3 суспензии вируса определяют количество клеточных культуральных инфекционных доз, вызывающих 50%-ное поражение клеток, что фактически отражает концентрацию полных вирусных частиц, содержащих РНК калицивируса кошек в активном состоянии.
Как правило, для определения титра инфекционной активности калицивируса кошек применяют метод титрования в перевиваемой монослойной культуре клеток почки кошки (CrFK), с помощью которой вычисляют минимальную дозу вируса, способную вызвать лизис 50% клеток (прототип) [6]. Данный метод имеет некоторые недостатки: 1) длительная процедура титрования, связанная с поражением клеток вирусом (не менее 72 ч); 2) определенная степень субъективности при оценке результатов исследования; 3) высокая стоимость клеточной линии как тест-системы и затраты на ее поддержание; 4) высокая вероятность риска контаминации культуры клеток.
В связи с этим целесообразно провести поиск способа определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для вакцины.
Существует метод, моделирующий в условиях in vitro репликацию ретровирусов, где в качестве мишени для амплификации служит одноцепочечная молекула РНК. Методика, названная «Self-Sustained Sequence Replication» или 3SR, базируется на конкурентном действии трех ферментов, участвующих в ретровирусной репликации - ревертазы, РНКазы Н и ДНК-зависимой РНК-полимеразы. Реакция протекает с участием специфичных праймеров при постоянной температуре. В результате реакции амплификации за 60-90 минут образуется до 109-1012 копий РНК [9, 10].
Данный метод является объективным, высокочувствительным и высокоспецифичным, более дешевым по сравнению с прототипом, характеризуется применением стандартизированных компонентов реакции, не создает ситуаций риска контаминации, отличается высокими значениями правильности и позволяет определять титр инфекционной активности, в частности, вирусов ящура и бешенства в течение 2-3 часов. Исходя из этого, целесообразно разработать способ опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации вирусной РНК.
Задачей настоящего изобретения является разработка высокочувствительного и высокоспецифичного способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК с целью устранения вышеуказанных недостатков.
Данная задача решена благодаря разработке способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК. Предложенный способ позволяет: 1) сократить время проведения анализа вируссодержащих суспензий для определения титра инфекционной активности калицивируса кошек до 2,5 ч; 2) исключить вероятность контаминации; 3) повысить объективность анализа; 4) увеличить специфичность анализа за счет применения высокоспецифичных оригинальных РНК-праймеров и молекулярного зонда, меченого флуорофором FAM (карбоксифлуоресцеином) (максимальная длина волны поглощения - 520 нм) и гасителем свечения RTQ1 (максимальная длина волны поглощения - 520 нм); 5) увеличить чувствительность анализа за счет амплификации только молекул вирусной РНК; 6) удешевить способ анализа за счет отсутствия использования клеточных культур в качестве тест-систем; 7) применение фермента Т7 ДНК-зависимой РНК-полимеразы позволяет повторно использовать молекулы комплементарной ДНК (кДНК), что приводит к увеличению концентрации РНК-ампликонов не менее, чем на 1 порядок по сравнению с концентрацией РНК-праймеров; 8) повысить достоверность проводимого анализа благодаря установлению зависимости между значениями титра инфекционной активности калицивируса кошек (ТFCV) и циклами количественной оценки реакции амплификации РНК (CQ-РНК-FCV), представленной в виде логарифмической функции:
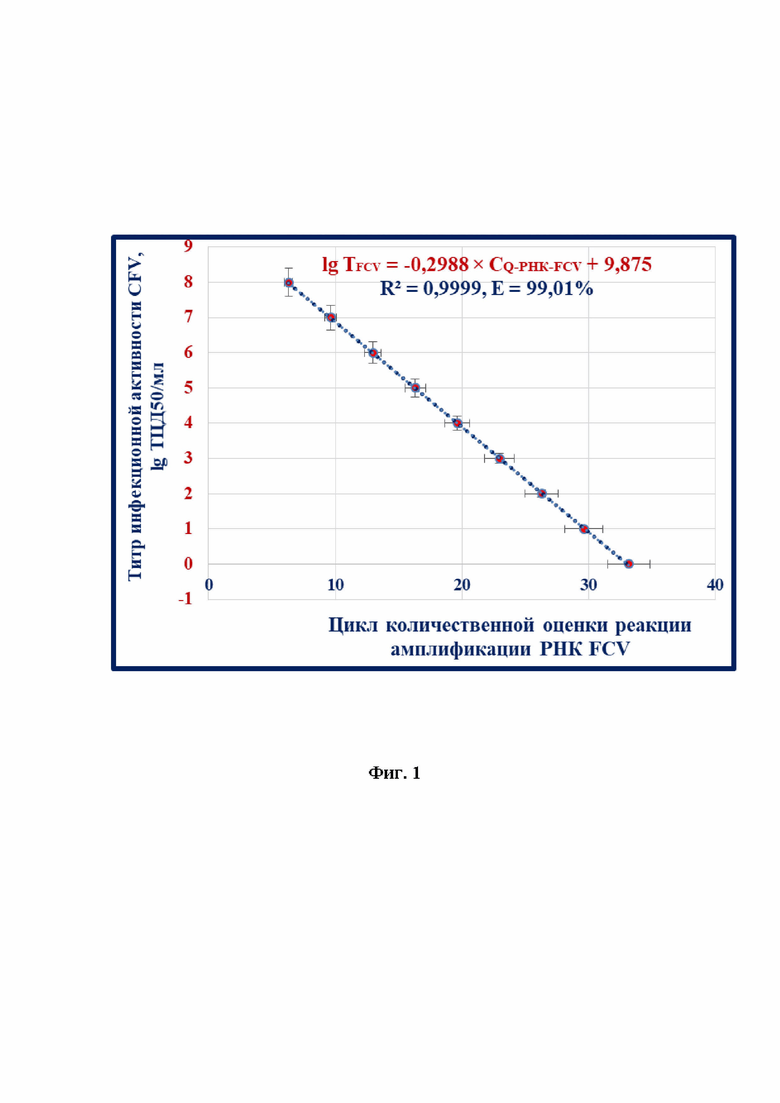
lg ТFCV=-0,2988 × CQ-РНК-FCV+9,875
с высокой достоверностью аппроксимации (R2=0,9999) и эффективностью амплификации 99,01%.
Предложенная модель позволяет количественно определить инфекционный титр калицивируса кошек в неинактивированном сырье для культуральных вакцин.
Сущность изобретения отражена на графических изображениях:
Фиг.1 - Зависимость цикла количественной оценки реакции амплификации вирусной РНК и титра инфекционной активности калицивируса кошек (n=3, отмечены точки, отображающие средние значения цикла количественной оценки реакции амплификации вирусной РНК).
Сущность изобретения пояснена в перечне последовательностей, в котором:
SEQ ID NO:1 представляет последовательность нуклеотидов ORF1-гена кДНК калицивируса кошек штамма «F9»;
SEQ ID NO:2 представляет последовательность аминокислот, соответствующих участку ORF1-гена кДНК коронавируса кошек штамма «F9».
Сущность изобретения заключается в подходе по определению титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации вирусной РНК.
Заявляемый способ основан на: 1) элюировании РНК калицивируса кошек; 2) амплификации специфического фрагмента ORF1-гена РНК калицивируса кошек с применение специфического олигонуклеотида P1 и обратного праймера P2, а также молекулярного зонда-beacon, меченого флуорофором FAM и тушителем свечения RTQ1; 3) обнаружении РНК-ампликонов с помощью флуоресцентного свечения и отображения накопления сигнала в виде сигмоиды; 4) расчете титра инфекционной активности калицивируса кошек в сырье неинактивированном сырье для культуральных вакцин с применением логарифмической функции, выраженной в виде уравнения:
lg ТFCV=-0,2988 × CQ-РНК-FCV+9,875
с высокой достоверностью аппроксимации (R2=0,9999) и эффективностью амплификации 99,01%.
В настоящее время реакцию транскрипционной амплификации применяют для выявления генома возбудителей различных инфекционных агентов, в частности, возбудителя парагриппа-1, 2, 3, 4, гриппа птиц А, энтеровирусной инфекции, цитомегаловирусной инфекции, аспергиллеза, кандидоза, хламидиоза, микобактериоза КРС, сальмонеллеза животных и др. патогенов [9, 10]. Данный метод применяется для определения титра инфекционной активности вируса ящура и бешенства в сырье для вакцин [11, 12]. При этом сведений об аналогах предлагаемого способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК авторами не обнаружено.
Разработанный способ опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК по сравнению с прототипом отличается быстротой выполнения анализа, его объективностью, более высокой чувствительностью и специфичностью.
Применение разработанного способа позволит сократить время проведения анализа вируссодержащего сырья для культуральных вакцин при определения титра инфекционной активности калицивируса кошек до 2,5 ч; повысить степень объективности получаемого результата; исключить вероятность контаминации; увеличить специфичность и чувствительность анализа; повторно использовать молекулы кДНК, что приводит к увеличению концентрации РНК-ампликонов не менее чем на 1 порядок по сравнению с концентрацией РНК-праймеров; использовать молекулярный зонд-beacon, меченый флуорофором FAM и тушителем свечения RTQ1; повысить достоверность и правильность проводимого анализа. Исходя их этого, актуально применять разработанный способ для опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье при изготовлении культуральных вакцин посредством количественного учета реакции амплификации участка ORF1-гена вирусной РНК.
Ключевым элементом заявляемого способа является проведение этапов денатурации, обратной транскрипции, разрушения гетеродуплекса РНК/кДНК и амплификации вирусной РНК с последующим отжигом молекулярного зонда-beacon, детектирования пороговых циклов сигмоид для исследуемых проб и опосредованного определения титра инфекционной активности калицивируса кошек с применением разработанной логарифмической модели зависимости цикла количественной оценки реакции амплификации РНК-мишени для сигмоиды накопления сигнала флуоресценции и титра инфекционной активности калицивируса кошек.
Сопоставительный анализ с прототипом позволяет сделать вывод, что новизна и изобретательский уровень заявляемого изобретения заключается в разработке и апробации способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК.
Технический результат изобретения заключается в том, что разработанный способ дает возможность: 1) повысить чувствительность и специфичность за счет применения высокоспецифичных оригинальных праймеров и молекулярного зонда, рассчитанных для целевого участка ORF1-гена РНК FCV; 2) увеличить достоверность проводимого анализа благодаря подбору оптимальных температурного и временного режимов термоциклирования; 3) в 28,8 раз быстрее по сравнению с прототипом опосредованно определять титр инфекционной активности FCV в сырье для культуральных вакцин, что в рамках производственного процесса важно с экономической точки зрения.
На первом этапе работы подготавливают панель положительных стандартов калицивируса кошек, в качестве которых используют не инактивированные вируссодержащие суспензии с инфекционными титрами: 0,0; 1,0; 2,0; 3,0; 4,0; 5,0; 6,0; 7,0; 8,0 lg ТЦД50/см3. Для получения данных разведений применют охарактеризованную очищенную с помощью фильтра на 0,8 мкм суспензию калицивируса кошек с титром инфекционной активности 8,0 lg ТЦД50/см3. Репродукция вируса проводится в монослойной клеточной линии CrFK. В данном анализе взят широкий диапазон возможных значений титра инфекционной активности калицивируса кошек, применяемого при исследовании. Для производства культуральных вакцин используют сырье с титрами (6,0 lg ТЦД50/см3. Отрицательным контролем служила суспензия клеток CrFK, не зараженная FCV.
Из всех стандартных положительных образцов и отрицательного контроля выделяют РНК калицивируса кошек с помощью набора «РИБО-сорб» («Интерлабсервис», РФ).
На следующем этапе исследования проводят реакцию амплификации РНК для исследования контрольных образцов и исследуемых проб. Для постановки реакции готовят реакционную смесь, рецептура приготовления которой представлена в таблице 1. Дизайн олигонуклеотидов отражены в таблице 2. Расчет олигонуклеотидных праймеров и зонда-beacon осуществляли на основании нуклеотидных последовательностей ORF1-гена калицивируса кошек, опубликованных в базах данных GenBank и полученных в рамках исследований в «Федеральном центре охраны здоровья животных» (ФГБУ «ВНИИЗЖ»).
В качестве гомологичных ORF1-гену калицивируса кошек олигонуклеотидов используют:
Oligo-FCV-ORF1-P1 (5'-AAT-TCT-AAT-ACG-ACT-CAC-TAT-AGG-G- CGACAATGTCGATTCCATCATG-3') (курсивом обозначена 5'-промоторная часть фермента T7 ДНК-зависимой РНК-полимеразы),
Downstream-FCV-ORF1-P2 (5'- TCAGGGCATAACTCGTCGG-3') и
FCV-ORF1-FAM/RHQ1-beacon (5'-FAM-CAA-AGC-GTC-TTC-AAC-CTA-GCU-UUG -RTQ1-3') в концентрации 0,25 пМ на реакцию. Для синтеза нуклеотидных цепей РНК-ампликонов применяют дезоксирибонуклеозидтрифосфаты (dNTP) и рибонуклеозидтрифосфаты (NTP) с их концентрацией в реакционной смеси по 2,3 мМ. В качестве основы используют буферный раствор (5х), содержание которого составляет 20% от общего объема реакционной смеси. Буферный раствор включает в свой состав ионы калия (К+) (5⋅10−2 M) и диметилсульфооксид (DMSO) (1,5%). В качестве ко-фактора добавляют 12 мМ хлорид магния. В качестве катализаторов реакции амплификации РНК применяют следующие ферменты: AMV-обратную транскриптазу (2 ед.), РНКаза Н E. сoli (2 ед.) и Т7 ДНК-зависимую РНК-полимеразу (2 ед.). Данные ферменты добавляют в реакционную смесь после прогревания до температуры 65°С и снижения температуры до 41°С, поскольку эти компоненты реакции термолабильны.
Элюаты РНК калицивируса кошек каждого образца добавляют к реакционной смеси по 5 мкл. Итоговый объем реакционной смеси для проведения одной реакции составляет 25 мкл.
При анализе нуклеотидных последовательностей гибридизационной части установили, что для олигонуклеотидов не характерно образование «шпилек» (за исключением «стеблевой» части зонда), а также не выявлено 3'-комплементарности и сайтов, отжигающих сами на себя при условии, когда минимальное количество пар оснований, необходимое для димеризации праймера и минимальное количество пар оснований, необходимое для образования шпильки - 4 [13-18].
Проведено определение температур плавления (Tm) для гибридизационной части олигонуклеотидов. Точное определение температуры плавления играет очень важную роль в молекулярно-биологических исследованиях, в том числе при подборе РНК-праймеров и бикона для реакции амплификации РНК калицивируса кошек. В соответствии с требованиями к реакции транскрипционной амплификации с последующей детекцией РНК-ампликонов температура плавления олигонуклеотидов (гибридной их части) должна быть выше температуры реакции (41°С) не менее чем на 7-10°С [15-18].
Tm при использовании алгоритма ближайших соседей для прямого, обратного праймеров и зонда-beacon составили 60-61°С, что более чем на 7-10°С ниже, чем температура амплификации РНК (4°С) и соответствует общепринятым требованиям, предъявляемым к олигонуклеотидам, используемым для данной реакции [14-17].
Последовательности разработанных оригинальных олигонуклеотидов исследованы на наличие нежелательных совпадений с другими последовательностями нуклеиновых кислот с использованием Банка данных последовательности РНК. Последовательности праймеров также проанализировали на наличие внутренних вторичных структур с помощью программы сворачивания нуклеиновых кислот с помощью программы Mfold [18]. Было выявлено, что для разработанных олигонуклеотидов нежелательных совпадений с другими последовательностями нуклеиновых кислот, а также наличия внутренних вторичных структур не обнаружено.
Постановку реакции осуществляют в амплификаторе с наличием флуориметра любой марки при температурных и временных параметрах, сведения о которых отражены в таблице 3. Стадию денатурации РНК проводят при температуре 65°С в течение 5 мин за 1 цикл. Реакцию амплификации РНК осуществляют в течение 35 циклов в изотермических условиях при температуре 41°С в течение 105 минут.Каждый условный цикл длится 3 минуты и складывается из 7 подэтапов: отжиг Oligo-FCV-ORF1-P1 на вирусной РНК, элонгация комплементарной ДНК (кДНК), разрушение гибрида РНК/кДНК, отжиг Downstream-FCV-ORF1-P2, элонгация второй цепи кДНК, активация промотора Т7 ДНК-зависимой РНК-полимеразы, синтез вирусной РНК, отжиг FCV-ORF1-FAM/RHQ1-beacon.
Принцип применяемого метода основан на проведении реакции транскрипционной амплификации нуклеиновой кислоты при фиксированной температуре 41°С (изотермические условия) с участием трех ферментов: AMV-ревертазы (ревертаза вируса миелобластоза птиц), РНКазы (RNase) Н E.coli и Т7 ДНК-зависимой РНК-полимеразы, полученной рекомбинантным способом из бактериофага Т7, специфических прямого и обратного олигонуклеотидных праймеров и молекулярного зонда-beacon для амплификации РНК калицивируса кошек. В результате этого процесса в ходе реакции происходит накопление миллиардов специфических фрагментов вирусной РНК-мишени. После инкубации РНК калицивируса кошек при температуре 65°С в течение 5 мин, при котором осуществляется денатурация нуклеиновой кислоты, начинается линейная стадия реакции амплификации, при которой специфический Oligo-FCV-ORF1-P1 гибридизируется с участком вирусной РНК. Данный олигонуклеотидный праймер включает в свой состав специфическую гибридизационную часть, а также промоторную последовательность T7 ДНК-зависимой РНК-полимеразы. При температуре 41°С AMV-ревертаза осуществляет элонгацию, создавая комплементарную ДНК с вирусной РНК-мишени. В результате формируется гетеродуплекс РНК/кДНК. Для РНКазы Н E.coli данный гибрид выступает в качестве субстрата. Фермент гидролизует РНК, тем самым разрушает гибрид РНК/кДНК, оставляя одноцепочечную кДНК. С кДНК гибридизуется Downstream-FCV-ORF1-P2. AMV-ревертаза вновь удлиняет кДНК до 5´-конца с образованием двуцепочечной кДНК (дц кДНК), что приводит промотор T7 ДНК-зависимой РНК-полимеразы в функциональное состояние. Данный фермент воспринимает дц кДНК с активным промотором в качестве субстрата. В результате производится множество копий фрагмента вирусной РНК калицивируса кошек. Молекулярный зонд-beacon, комплементарный участку ORF1-гена РНК-мишени калицивируса кошек, гибридизутся с ним. В отсутствии мишени флуорофор и гаситель флуоресценции в составе молекулярного зонда сближены за счет максимального использования водородных связей между атомами H, O и N олигонуклеотида. Благодаря механизму флуоресцентно-резонансного переноса энергии свечение подавлено. После линейной стадии реакция амплификации РНК вступает в циклический процесс.Downstream-FCV-ORF1-P2 гибридизуется с вновь синтезированной молекулой РНК калицивируса кошек. Фермент AMV-ревертаза проводит элонгацию кДНК. В результате образуется гетеродуплекс РНК/кДНК. За счет активности РНКазы Н E.coli после отжига beacon происходит разрушение гибрида РНК/кДНК за счет гидролиза РНК, наблюдается пространственное разделение флуорофора и гасителя свечения, что приводит к росту детектируемого сигнала при длине волны 520 нм. Oligo-FCV-ORF1-P1 гибридизируется с кДНК, а AMV-ревертаза удлиняет его с образованием дц кДНК. Фермент T7 ДНК-зависимая РНК-полимераза синтезирует РНК калицивируса кошек. После этого запускается следующий цикл амплификации РНК.
Результаты реакции амплификации РНК в режиме реального времени с каждого цикла анализируют, оценивая и сравнивая графики накопления флуоресцентного сигнала по значениям циклов количественной оценки реакции амплификации вирусной РНК CQ-РНК-FCV, определенных с помощью пересечения пороговой линии и логарифмическим отображением функции Fl=f (CQ-РНК-FCV). Флуориметр определяет уровень свечения и строит кинетическую кривую в координатах: уровень флуоресценции - цикл амплификации РНК. В случае присутствия в исследуемой пробе специфической РНК-матрицы кинетическая кривая имеет экспоненциальную зависимость в виде графика сигмоиды. Положительными считаются пробы, которым соответствуют экспонециальные кривые, полученные при анализе флуоресценции красителя, входящего в состав молекулярного зонда, и при значении цикла количественной оценки реакции амплификации менее 33,20. Пробы считаются отрицательными, если при их анализе отсутствует экспоненциальная кривая, или график начинает формироваться после 33,20 цикла амплификации. Данное значение цикла количественной оценки реакции амплификации определено экспериментально. При его достижении исследуемый образец имеет значение титра инфекционной активности калицивируса кошек 0,0 lg ТЦД50/см3 или 1,0 ТЦД50/см3.
Выявляют зависимость между CQ-РНК-FCV и значением десятичного логарифма титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин. Оценивают величину эффективности реакции амплификации (Е), а также достоверность аппроксимации (R2). На основе разработанной модели рассчитывают значение титра инфекционной активности калицивируса кошек в неинактивированном сырье для вакцин.
Сущность предлагаемого изобретения пояснена примерами его использования, которые не ограничивают объем изобретения.
Пример 1. Выражение функции зависимости титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин и цикла количественной оценки реакции амплификации вирусной РНК.
Для определения значения титра инфекционной активности калицивируса кошек подготавливали серию разведений положительных стандартных образцов калицивируса кошек, в качестве которых применяли не инактивированные суспензии калицивируса кошек с титрами: 0,0; 1,0; 2,0; 3,0; 4,0; 5,0; 6,0; 7,0; 8,0 lg ТЦД50/см3. Для получения данных разведений применяли охарактеризованную очищенную с помощью фильтра на 0,8 мкм суспензию калицивируса кошек с титром инфекционной активности 8,0 lg ТЦД50/см3.
Выделение нуклеиновой кислоты осуществляли, как представлено выше. Проводили постановку реакции амплификации РНК для исследования контролей, как описано выше. Получены данные реакции амплификации РНК, которые анализировали, оценивая и сравнивая графики накопления флуоресцентного сигнала по значениям циклов количественной оценки реакции амплификации вирусной РНК. Установили зависимость между CQ-РНК-FCV и значением десятичного логарифма титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин. Полученные результаты отражены на фиг.1 и выражены в виде логарифмической функции:
lg ТFCV=-0,2988 (CQ-РНК-FCV+9,875)
с высокой достоверностью аппроксимации (R2=0,9999) и эффективностью амплификации 99,01%, что соответствовало общепринятым требованиям, предъявляемым к реакции транскрипционной амплификации [11].
Пример 2. Применение способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК.
В исследовании использовали 6 суспензий культурального FCV с титрами инфекционной активности 6,50; 6,75; 7,00; 7,25; 7,50; 7,75 lg ТЦД50/см3, соответственно (пробы №1-6). В качестве положительного контроля применяли суспензию культурального FCV с титром инфекционной активности 7,00 lg ТЦД50/см3. В качестве отрицательных контролей применяли суспензию клеток CrFK, не зараженную микроорганизмами. Испытуемые пробы и контрольные образцы исследовали в 7 повторностях. Этап элюирования РНК, и постановку реакции амплификации вирусной РНК проводили, как описано выше.
Средние значения циклов количественной оценки реакции амплификации вирусной РНК для проб №1-6 составляли 11,23±0,01, 10,39±0,01, 9,62±0,01, 8,79±0,01, 7,81±0,01, 7,01±0,02, соответственно. Пользуясь разработанной логарифмической функцией, рассчитали средние значения титра инфекционной активности FCV для проб №1-6, которые составили 6,52; 6,77; 7,00; 7,25; 7,54; 7,78 lg ТЦД50/см3, соответственно. Для положительного контроля значение порогового цикла амплификации составило 9,62±0,00, что соответствовало титру инфекционной активности FCV, равному 7,00 lg ТЦД50/см3. Для отрицательных контролей экспоненциальные графики не были сформированы, что означало отсутствие FCV в данных образцах. Полученные результаты свидетельствовали о высокой степени точности разработанного способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК. Таким образом, разработанный способ позволяет рассчитывать титр инфекционной активности FCV в сырье для культуральных вакцин.
Пример 3. Выявление степени достоверности определения титра инфекционной активности FCV в сырье для вакцин с применением разработанного способа.
Для анализа использовали 300 суспензий культурального FCV с титрами инфекционной активности от 1,00 до 8,00 lg ТЦД50/см3. В качестве положительного контроля применяли суспензию культурального FCV с титром инфекционной активности вируса 7,00 lg ТЦД50/см3. В качестве отрицательных контролей применяли суспензию клеток CrFK, не зараженную микроорганизмами. Испытуемые пробы и контрольные образцы исследовали в трех повторностях. Этапы анализа проводили, как отражено выше. По результатам исследования, для положительного контроля значение порогового цикла амплификации составило 9,62±0,00, что соответствовало титру инфекционной активности FCV, равному 7,00 lg ТЦД50/см3. Для отрицательных контролей экспоненциальные графики не были сформированы, что означало отсутствие FCV в данных образцах.
Выявили, что данные, полученные с помощью разработанного способа, коррелировали со значениями стандартов на 99,12-100,00% для 8,0-6,0 lg ТЦД50/см3 (n=75), на 98,33-99,11% для 5,9-4,0 lg ТЦД50/см3 (n=75), на 96,33-98,32% для 3,9-1,5 lg ТЦД50/см3 (n=75), на 96,05-96,32% для 1,4-1,0 lg ТЦД50/см3 (n=75) (табл.4). Полученные результаты свидетельствовали о высокой степени точности разработанного способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК. Таким образом, разработанный способ позволяет с высокой степенью достоверности рассчитывать титр инфекционной активности FCV в сырье для вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК.
Пример 4. Определение аналитической чувствительности способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК.
При определении аналитической чувствительности способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК подготавливали серию стандартов FCV разных штаммов с титрами инфекционной активности, равными 0,00-8,00 lg ТЦД50/см3 с шагом 0,1 lg ТЦД50/см3. Контрольные образцы тестировали в 5 повторностях. Этапы анализа проводили, как описано выше.
Выявлено, что с достоверностью 96,05-100,00% разработанным способом определены титры инфекционной активности калицивируса кошек со значениями от 1,0 до 8,0 lg ТЦД50/см3. При исследовании вируссодержащего сырья с титрами от 0,0 до 1,0 lg ТЦД50/см3 обнаружили, что аналитическая чувствительность разработанного способа составляет 0,2 lg ТЦД50/см3 с достоверностью определения аналита, равной 95,56% (n=75, p<0,05).
Пример 5. Исследование специфичности способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК.
При анализе специфичности предлагаемого изобретнния, исследовали суспензии FCV, а также вирусы бешенства, ящура, инфекционного ринотрахеита кошек, чумы плотоядных, возбудителя парвовирусного энтерита, коронавирусного энтерита собак, аденовируса собак первого серотипа. Количество инфекционных доз вирусов в суспензиях составлял не менее 6,0 lg ТЦД50/см3. Исследования проводили в 5 повторностях.
Этапы анализа проводили, как описано выше. Для проб, содержащих другие вирусы, не наблюдалось формирования графиков экспоненты, и они не выходили за пороговый уровень флуоресцентного сигнала (0,005 у.е.). Таким образом, разработанный способ является специфичным по отношению к калицивирусу кошек и может быть использован для его количественного определения.
Пример 6. Определение диагностических показателей разработанного способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для кулътуральных вакцин с помощью количественного учета реакции амплификации участка 0RF1-гена вирусной РНК.
Для определения диагностической чувствительности разработанного способа анализировали 350 кулътуральных суспензий FCV с разными значениями инфекционной активности вируса (1,0-8,0 lg ТЦДзо/см3). Данные пробы являлись заведомо положительными. Проведение анализа осуществляли, как отражено выше. С помощью разработанного способа (предлагаемое изобретение) определили, что из 350 исследуемых образцов в 348 пробах концентрация определена верно, в 3 - отличия были существенными (значения титра инфекционной активности составили ниже аналитической чувствительности способа).
Для исследования специфичности метода тестировали 210 отрицательных суспензий клеток линии CrFK, не содержащих FCV. В результате исследования с помощью разработанного способа (предлагаемое изобретение) определили, что все 210 проб были отрицательными. Пользуясь представленными выше статистическими методами анализа определили, что в 95%-ном доверительном интервале диагностическая чувствительность (DSe) составила 97,96-99,93%, диагностическая специфичность (DSp) - 98,26-100,00%, k-критерий - 0,992; общая точность (DAc) - 98,72-99,96%.
Основными преимуществами предлагаемого изобретения является возможность сократить время проведения анализа вируссодержащих суспензий для определения титра инфекционной активности калицивируса кошек до 2,5 ч; исключить вероятность контаминации; повысить объективность анализа; увеличить специфичность анализа за счет применения высокоспецифичных оригинальных РНК-праймеров и молекулярного зонда-beacon; увеличить чувствительность анализа за счет амплификации только молекул вирусной РНК; удешевить способ анализа за счет отсутствия использования клеточных культур в качестве тест-систем; применение фермента Т7 ДНК-зависимой РНК-полимеразы позволяет повторно использовать молекулы комплементарной ДНК (кДНК), что приводит к увеличению концентрации РНК-ампликонов не менее, чем на 1 порядок по сравнению с концентрацией РНК-праймеров; повысить достоверность проводимого анализа благодаря установлению зависимости между значениями титра инфекционной активности калицивируса кошек (ТFCV) и циклами количественной оценки реакции амплификации РНК (CQ-РНК-FCV), представленной в виде логарифмической функции:
lg ТFCV=-0,2988 (CQ-РНК-FCV+9,875
с высокой достоверностью аппроксимации (R2=0,9999) и эффективностью амплификации 99,01%.
Источники информации, принятые во внимание при составлении описания изобретения к заявке на выдачу патента РФ на изобретение «Способ опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК»:
1. Abd-Eldaim MM, Wilkes RP, Thomas KV, Kennedy MA. Development and validation of a TaqMan real-time reverse transcription-PCR for rapid detection of feline calicivirus. Arch Virol. 2009;154(4):555-60.
2. Алипер Т. И., Непоклонов Е. А., Мухин А. Н. и др. Диагностика и профилактика инфекционных болезней кошек и кошек: руководство для практикующих ветеринарных врачей. Под ред. Т. И. Алипера. М.: ЗооВетКнига; 2017. 300 с.
3. Komina A, Krasnikov N, Kucheruk O, Zhukova E, Yuzhakov A, Gulyukin A. Distribution and genetic diversity of Feline calicivirus in Moscow metropolitan area. J Vet Sci. 2022 Nov;23(6):e92.
4. Глотова Т.И., Семенова О.В., Никонова А.А., Глотов А.Г., Вяткин Ю.В., Бондарь А.А. Выделение и филогенетический анализ калицивируса кошек в Сибири. Вопросы вирусологии. 2018; 63(6):268-274.
5. Hou J,  McGahie D, Lesbros C, Almeras T, Howarth D, O'Hara V, Dawson S, Radford AD. European molecular epidemiology and strain diversity of feline calicivirus. Vet Rec. 2016 Jan 30;178(5):114-5.
McGahie D, Lesbros C, Almeras T, Howarth D, O'Hara V, Dawson S, Radford AD. European molecular epidemiology and strain diversity of feline calicivirus. Vet Rec. 2016 Jan 30;178(5):114-5.
6. Рахманина М.М. Калицивирусная инфекция кошек: биологические свойства возбудителя, эпизоотология, специфические средства и методы профилактики: Автореф. диcс. д-ра ветеринар. наук. М.; 2005.
7. Hofmann-Lehmann R, Hosie MJ, Hartmann K, et al. Infection in Cats. Viruses. 2022 Apr 29;14(5):937.
8. Вакцина против калицивироза, вирусного ринотрахеита и панлейкопении кошек Нобивак Tricat Trio. - URL: https://www.vetlek.ru/directions/?id=112 (Дата обращения 11.08.2023).
9. Morre S., Sillekens P., Jacobs M.V., et al. RNA amplification by nucleic acid sequence-based amplification with an internal standard enables reliable detection of Chlamydia trachomatis in cervical scrapings and urine samples // J. Clin. Microbiol. - 1996. - Vol.34. - P. 3108-3114.
10. Шипицина Е.В. Применение метода NUCLEIC ACID SEQUENCE -BASED AMPLIFICATION в реальном времени (NASBA-REAL-TIME) для диагностики урогенитальной хламидийной инфекции // Журнал Акушерства и женских болезней. - Т. LIV выпуск 4/2005. - С.17-21.
11. Патент РФ №2 756 557, 10.01.2021 Способ опосредованного определения титра вируса ящура в неинактивированном сырье для вакцины при амплификации вирусной нуклеиновой кислоты и детекции РНК-ампликонов с применением технологии молекулярных биконов» // Заявка 2020136076, Бюл. 28 от 02.11.2020 / Доронин М.И., Михалишин Д.В., Стариков В.А., Борисов А.В., Гусева М.Н.
12. Патент РФ №2 756 472, 10.09.2020 Способ опосредованного определения инфекционного титра вируса бешенства в неинактивированном сырье для антирабических вакцин при транскрипционной амплификации и детекции продуктов реакции с применением beacon-технологии // Заявка 2020129954, Бюл. №28 от 30.09.2021 / Доронин М.И., Михалишин Д.В., Борисов А.В., Мудрак Н.С.
13. Deiman B., van Aarle P., Sillekens P. Characteristics and applications of nucleic acid sequence based amplification // Mol. Biotech. - 2002. - Vol.20. - P. 163-179.
14. Sooknanan R., van Gemen B., Malek L. Nucleis acid sequence-based amplification // Molecular methods for virus detection-London: Academic press, 1995. - P. 261-285.
15. Nicolas von Ahsen, Carl T. Wittwer, Ekkehard  Oligonucleotide melting temperatures under PCR conditions: nearest-neighbor corrections for Mg2+, deoxynucleotide triphosphate, and dimethyl sulfoxide concentrations with comparison to alternative empirical formulas (англ.) // Clinical Chemistry: journal. - 2001. - Vol.47, no. 11. - P. 1956-1961.
Oligonucleotide melting temperatures under PCR conditions: nearest-neighbor corrections for Mg2+, deoxynucleotide triphosphate, and dimethyl sulfoxide concentrations with comparison to alternative empirical formulas (англ.) // Clinical Chemistry: journal. - 2001. - Vol.47, no. 11. - P. 1956-1961.
16. SantaLucia J. J. A unified view of polymer, dumbbell, and oligonucleotide DNA nearest-neighbor thermodynamics (англ.) // Proceedings of the National Academy of Sciences of the United States of America: journal. - 1998. - Vol.95, no. 4. - P. 1460-1465.
17. SantaLucia J. J., Hicks D. The thermodynamics of DNA structural motifs // Annual Review of Biophysics and Biomolecular Structure: journal. - 2004. - Vol.33. - P. 11-14.
18. The RNA Institute college of arts and science university at Albany. The mfold Web Server. RNA Folding Form. [Электронный ресурс] / URL: http://bioinfo.math.rpi.edu/~mfold/dna/form1.cgi (Дата обращения: 14.08.2023).
Таблица 1
Состав реакционной смеси для опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК (разработанный способ)
Примечание: объем вносимого элюата РНК - 5 мкл,
объем реакционной смеси - 25 мкл.
Таблица 2
Дизайн оригинальных олигонуклеотидов для разработанного способа
Примечание: * - курсивом обозначена 5'-промоторная часть T7 ДНК-зависимой РНК-полимеразы
** - выделены флуорофор и гаситель свечения, подчеркнута гибридизационная часть в центре.
Таблица 3
Временные и температурные режимы для опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК (разработанный способ)
Таблица 4
Достоверность способа опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК (разработанный способ)
(nкаждого измерения=3, p<0,001)
--->
<ST26SequenceListing originalFreeTextLanguageCode="ru"
dtdVersion="V1_3" fileName="1.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2024-03-14">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2024-03-12</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>544</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2024-03-12</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБУ "Федеральный центр охраны
здоровья животных" (ФГБУ "ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin>Federal State-Financed Institution Federal Centre
for Animal Health (FGBI ARRIAH)</ApplicantNameLatin>
<InventorName languageCode="ru">Доронин Максим
Игоревич</InventorName>
<InventorNameLatin>Doronin Maksim Igorevich </InventorNameLatin>
<InventionTitle languageCode="ru">Способ опосредованного определения
титра инфекционной активности калицивируса кошек в неинактивированном
сырье для культуральных вакцин с помощью количественного учета
реакции амплификации участка ORF1-гена вирусной РНК </InventionTitle>
<SequenceTotalQuantity>5</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>5289</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5289</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtctcaaactctgagcttcgtgcttaaaactcacagtgtccgaaaggact
ttgtgcactctgtcaagttaacacttgcacggaggcgcgatcttcagtatatttataacaagctctcacg
cactatacgtgctgaggcttgcccttcttgtgctagttacgacgtatgtcctaactgcacctctggtgac
gtcccggatgacgggtcttcgacaatgtcgattccatcatgggaggatgtcacaaagtcttcaacctact
ctctcctgctctctgaggacacctccgacgagttatgccctgaggacttggttaatgtggctgctcatat
ccgtaaggcgctatccactcagtcccacccggctaatgctgaaatgtgcaaagaacagctcactttctta
ttagtcatggctgaggcgatgctgccccaacgatcccgagcgtcaatcccactgcaccaacaacacacgg
ctgcacggttggaatggagggagaaattcttctctaaacctcttgactttctccttgaaagagttggtgt
gtcaaaagatattctccaaactactgcgatttggaaaattatcttggaaaaagcatgctattgtaaatcc
tatggagagcagtggttcactgctgccaaacaaaagttaagggaaatgaaaaactttgagagtgatacgc
taaaacctcttattggtggatttatagatggtctacggttcttgaccgtggacaacccaaacccgatggg
tttcctcccaaaactcatagggcttgtaaaacccctgaatttggcgatgattattgataatcatgaaaac
acaatatctggctggatcatcacattaaccgcaataatggagctatacaacatcaccgaatgcaccatag
atattataacatcagtcattacggctttctatgataaaattggcaaggcaaccaaattttacagttgtgt
taaggcgctgttcactggatttagatctgaggacgtagcgaattccttttggtacatggcagcagcgatt
ctatgttacctgatcactgggttaatcccaaacaacggcagattttcaaagataaaagcttgcctggccg
gagcgacaactcttgtatcaggtatagttgccacacaaaagctagctgcaatgttcgcaacatggaactc
tgagtccattgttaatgagttgtcagcaagaactgttgccctatcagagctaaacaatccaacaacaacg
tctgacacggattcggtagaacgactgctagaattggctaagatcttgcatgaggagatcaagattcata
ctctaaaccccatcatgcaatcatacaatccaatcttgagaaatttaatgtcaaccttggacggtgttat
aacatcatgcaacaagaggaaagctattgctcggaagagacaggtgccagtttgttacatattaactggc
ccacctggatgtgggaaaacaaccgcagctcaagcattagctaagaaattgtctgatcaagagccgtcgg
taattaaccttgatgttgatcatcatgacacatatactggaaatgaggtatgtatcattgatgagtttga
ttcgtctgacaaggtagactatgcaaattttgtgattgggatggtaaactctgctcctatggtgttaaat
tgtgacatgcttgagaacaagggaaagctcttcacctcaaagtatataattatgacttccaactctgaaa
ctccagtgaaaccctcttccaagcgcgcaggtgcattctaccgaagggttactatcatcgatgtaactaa
cccttttgtggagtcgcacaagcgtgcaaggcctgggacgtctgttcctcgtagctgctataagaaaaac
ttctcccatctctcacttgctaaacgaggggccgagtgctggtgcaaggaatacgttcttgaccctaagg
ggcttcaacaccaaagcatgaaggctccccctcctacctttcttaacattgactctttagctcagacgat
gaagcaagacttcttgctcaagaacatggcttttgaggctgaagatgggtgcgcagaacatcgatatggg
tttgtgtgtcaacaggaagaggttgaaactgttcgcaggctcctcaacgcggttagggcaaggatgaatg
ctaccttcactgtgtgtgttggacccgagacctcgcactcgattggttgcactgcgcacgtgttaactcc
caatgagacctttaatggaaagaagtttgttgtgtcacgatgtaacgaagcatcactttctgcccttgaa
ggtaactgtgtaaagtcagctttgggcgtgtgtatgtcagataaggatctcactcatttatgccacttca
ttaaagggaaaattgtcaatgacagtgttaggttggatgaactacccgccaatcagcatgtggtaaccgt
taattcggtgtttgatttggcctgggctgttcgtcgtcatctcacactggcagggcagtttcaagctatc
agagccgcatatgatgtgcttactgtccccgacaagatccctgcaatgttgcgtcactggatggatgaga
cctccttttctgatgaccacgttgtaacacaatttgtaacacctggtggcatagtcatcctggagtcttg
cggtggtgcacgcatctgggctttaggtcgcaacgtgatccgagcaggaggtgtcaccgcaaccccaacc
ggaggatgtgttaggttaatgggattatctgcgccaaccatgccatggtccgagatctttcgtgagctct
tctctctcttaggtagaatttggtcatctgtcaaagtatcagccctagtactaactgctcttggaatgta
cgcatctagatttagaccaaaatcggaagcaaaaggaaaaaccaaattgaagattgggacatacaggggt
cgcggtgtagcgctgactgatgacgagtacgatgagtggcgcgaacacaacgcctccagaaaattggatt
tgtcagtggaggatttcttaatgttgcgccatcgtgccgctctaggagccgacgacaatgatgcagtaaa
attccggtcgtggtggaactccagaaccaaaatggccaatgattatgaggatgtcaccgtaattggcaaa
ggtggcgtcaaacatgaaaagatcagaaccaataccctaaaagctgtggatcgtggttatgacgtcagct
ttgctgaggaatcaggaccaggcaccaagttccacaagaatgcaattggatctgtcacagatgtttgtgg
ggagcacaaaggctattgcatccacatgggccacggtgtttacgcatccgtagctcacgtggtgaaaggg
gattcatttttcttgggtgaaaggatttttgatcttaagactaatggtgaattttgctgctttcgcagca
cgaaaattctacctagtgctgcacctttcttttctgggaagcccactcgtgatccgtggggatcccccgt
ggcaactgagtggaagcctaaaatgtacacaacaacctctggaaagattctggggtgctttgcaacaact
tcaactgaaactcacccgggagactgtggcctcccatatattgatgacaacgggagggtgaccggcctcc
acactggctctgggggacccaaaaccccaagtgccaagttggtggtgccatatgtgcatattgacatgaa
gactaaatccgtcactgctcaaaagtatgacgtaacaaagcctgatataagttacaaaggcttaatttgt
aagcaattggatgagattaggattataccaaaaggcacacgtctccatgtctccccagcccacactgagg
attatcaagaatgctcacaccaacccgcatcacttggaagcggggatccccgctgtccaaaatctctcac
tgctatagttgttgattctctaaaaccatactgtgagaacgttgagggtcctccacatgatgttttgcac
agagttcaaaagatgcttatcgaccacctttcaggctttgtccctatgaacatttcctcggaaacctcta
tgctctcagctttccacaaactcaatcatgatacttcctgtggaccatacttgggtggcagaaagaaaga
tcacatggctaacggtgagccggacaagcagttattggatctcctgtctgcaaaatggaaattggcaacc
caaggcatagcactaccacatgagtacacaattgggctaaaggacgagttaaggcccgtggagaaggtta
gtgaagggaagagaaggatgatttggggttgtgatgttggcgtcgctactgtctgtgcagctgcgttcaa
gggtgttagcgatgccatcacagcaaaccaccagtacgggcctatacaggttggtatcaacatggatagc
cccagcgtcgaagcgctgttccaaaggatcaaaagcgcggccaaggtatttgcggtcgattattccaaat
gggattcgacccaatcgcctcgtgtcagtgcagcttcaattgacatccttcgttacttttccgatcgctc
tccaattgttgactcagcctctaacacactgaagagccctcctgttgcaatctttaatggtgttgctgta
aaagtgtcctctggcttaccatctggaatgcctcttacctcagtaatcaattcccttaatcattgtctgt
atgttgggtgtgccattcttcaatccctagaagctaaggccattcccgtcacttggaaccttttctcaac
ttttgatatcatgacttacggggatgatggtgtctacatgtttcctattatgtatgcaagtattagtgac
caaatttttggaaatctttcttcctatggcctgaaaccaactcgggttgacaagtccgttggagcaattg
agcctattgatcctgactctgttgttttcttgaagagaacaatcacaaggacacctcaggggataagggg
tttacttgatcgcagctctataataagacaattctattatattaaaggtgagaactccgatgactggaag
agccccccaaaacatattgacccaacatctcgagggcaacagctttggaatgcctgtctgtacgctagcc
aacatggcttggagtttttcaacaaggtttacaggctggccgagagggctgttgaatatgaagagctgca
ctttgaacccccaacatatgcttcggctttggatcattacaacagccagttcaatggcgtggaggcgcgg
tctgaccagatcgactcgagtggcatgaccgccctacactgtgatgtgttcgaagtt</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1763</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1763</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSQTLSFVLKTHSVRKDFVHSVKLTLARRRDLQYIYNKLSRTIRAEACPSCA
SYDVCPNCTSGDVPDDGSSTMSIPSWEDVTKSSTYSLLLSEDTSDELCPEDLVNVAAHIRKALSTQSHPA
NAEMCKEQLTFLLVMAEAMLPQRSRASIPLHQQHTAARLEWREKFFSKPLDFLLERVGVSKDILQTTAIW
KIILEKACYCKSYGEQWFTAAKQKLREMKNFESDTLKPLIGGFIDGLRFLTVDNPNPMGFLPKLIGLVKP
LNLAMIIDNHENTISGWIITLTAIMELYNITECTIDIITSVITAFYDKIGKATKFYSCVKALFTGFRSED
VANSFWYMAAAILCYLITGLIPNNGRFSKIKACLAGATTLVSGIVATQKLAAMFATWNSESIVNELSART
VALSELNNPTTTSDTDSVERLLELAKILHEEIKIHTLNPIMQSYNPILRNLMSTLDGVITSCNKRKAIAR
KRQVPVCYILTGPPGCGKTTAAQALAKKLSDQEPSVINLDVDHHDTYTGNEVCIIDEFDSSDKVDYANFV
IGMVNSAPMVLNCDMLENKGKLFTSKYIIMTSNSETPVKPSSKRAGAFYRRVTIIDVTNPFVESHKRARP
GTSVPRSCYKKNFSHLSLAKRGAECWCKEYVLDPKGLQHQSMKAPPPTFLNIDSLAQTMKQDFLLKNMAF
EAEDGCAEHRYGFVCQQEEVETVRRLLNAVRARMNATFTVCVGPETSHSIGCTAHVLTPNETFNGKKFVV
SRCNEASLSALEGNCVKSALGVCMSDKDLTHLCHFIKGKIVNDSVRLDELPANQHVVTVNSVFDLAWAVR
RHLTLAGQFQAIRAAYDVLTVPDKIPAMLRHWMDETSFSDDHVVTQFVTPGGIVILESCGGARIWALGRN
VIRAGGVTATPTGGCVRLMGLSAPTMPWSEIFRELFSLLGRIWSSVKVSALVLTALGMYASRFRPKSEAK
GKTKLKIGTYRGRGVALTDDEYDEWREHNASRKLDLSVEDFLMLRHRAALGADDNDAVKFRSWWNSRTKM
ANDYEDVTVIGKGGVKHEKIRTNTLKAVDRGYDVSFAEESGPGTKFHKNAIGSVTDVCGEHKGYCIHMGH
GVYASVAHVVKGDSFFLGERIFDLKTNGEFCCFRSTKILPSAAPFFSGKPTRDPWGSPVATEWKPKMYTT
TSGKILGCFATTSTETHPGDCGLPYIDDNGRVTGLHTGSGGPKTPSAKLVVPYVHIDMKTKSVTAQKYDV
TKPDISYKGLICKQLDEIRIIPKGTRLHVSPAHTEDYQECSHQPASLGSGDPRCPKSLTAIVVDSLKPYC
ENVEGPPHDVLHRVQKMLIDHLSGFVPMNISSETSMLSAFHKLNHDTSCGPYLGGRKKDHMANGEPDKQL
LDLLSAKWKLATQGIALPHEYTIGLKDELRPVEKVSEGKRRMIWGCDVGVATVCAAAFKGVSDAITANHQ
YGPIQVGINMDSPSVEALFQRIKSAAKVFAVDYSKWDSTQSPRVSAASIDILRYFSDRSPIVDSASNTLK
SPPVAIFNGVAVKVSSGLPSGMPLTSVINSLNHCLYVGCAILQSLEAKAIPVTWNLFSTFDIMTYGDDGV
YMFPIMYASISDQIFGNLSSYGLKPTRVDKSVGAIEPIDPDSVVFLKRTITRTPQGIRGLLDRSSIIRQF
YYIKGENSDDWKSPPKHIDPTSRGQQLWNACLYASQHGLEFFNKVYRLAERAVEYEELHFEPPTYASALD
HYNSQFNGVEARSDQIDSSGMTALHCDVFEV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>47</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..47</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aattctaatacgactcactatagggcgacaatgtcgattccatcatg</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcagggcataactcgtcgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>RNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>viral cRNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaagcgtcttcaacctagcnnng</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ опосредованного определения титра инфекционной активности калицивируса кошек в сырье для вакцины методом ПЦР в режиме реального времени | 2023 |
|
RU2809221C1 |
| Способ определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин | 2024 |
|
RU2838181C1 |
| Способ определения титра инфекционной активности возбудителя чумы плотоядных животных в сырье для изготовления культуральных инактивированных вакцин | 2024 |
|
RU2831062C1 |
| Способ опосредованного определения титра инфекционной активности возбудителя инфекционного ринотрахеита кошек в неинактивированном сырье для культуральных вакцин с помощью реакции амплификации целевого участка UL26.5-гена вирусной ДНК | 2024 |
|
RU2834248C1 |
| Способ опосредованного определения титра инфекционной активности возбудителя инфекционного ринотрахеита кошек в сырье для вакцины с помощью дифференциала второго порядка точки пересечения логистической кривой реакции амплификации участка UL35-гена | 2023 |
|
RU2823781C1 |
| Способ дифференциации штамма "Мира" калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1 | 2024 |
|
RU2839481C1 |
| Способ опосредованного определения титра инфекционной активности возбудителя инфекционного ринотрахеита кошек FHV в неинактивированном сырье для вакцин с применением полимеразной цепной реакции и количественной гибридизационно-флуоресцентной детекции в режиме реального времени | 2023 |
|
RU2808641C1 |
| Способ опосредованного определения инфекционного титра вируса бешенства в неинактивированном сырье для антирабических вакцин при транскрипционной амплификации и детекции продуктов реакции с применением beacon-технологии | 2020 |
|
RU2756472C1 |
| Способ опосредованного определения титра инфекционной активности вакцинного штамма "Лавр" возбудителя инфекционного ринотрахеита кошек в сырье для вакцины на основе анализа кривых плавления ампликонов высокого разрешения гена UL47 с применением флуоресцентного красителя dsSafe | 2024 |
|
RU2834233C1 |
| Способ опосредованного определения титра инфекционной активности вируса ящура в неинактивированном сырье для вакцин с помощью математического двойного дифференциала данных точки crossing point при амплификации вирусной кДНК | 2022 |
|
RU2793900C1 |
Изобретение относится к области биотехнологии и производству культуральных вакцин. Описан способ опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК. Основными преимуществами предлагаемого изобретения является возможность сократить время проведения анализа вируссодержащих суспензий для определения титра инфекционной активности калицивируса кошек до 2,5 ч; исключить вероятность контаминации; повысить объективность анализа; увеличить специфичность анализа за счет применения высокоспецифичных оригинальных РНК-праймеров и молекулярного зонда-beacon; увеличить чувствительность анализа за счет амплификации только молекул вирусной РНК; удешевить способ анализа за счет отсутствия использования клеточных культур в качестве тест-систем; повысить достоверность проводимого анализа благодаря установлению зависимости между значениями титра инфекционной активности калицивируса кошек и циклами количественной оценки реакции амплификации РНК. Аналитическая чувствительность разработанного способа составляет 0,2 lg ТЦД50/см3 с достоверностью определения аналита, равной 95,56%. В 95%-ном доверительном интервале диагностическая чувствительность составила 97,96-99,93%, диагностическая специфичность - 98,26-100,00%, общая точность - 98,72-99,96%. 1 з.п. ф-лы, 1 ил., 4 табл., 6 пр.
1. Способ опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК, включающий применение олигонуклеотидов:
- Oligo-FCV-ORF1-P1 с дизайном 5'-AAT-TCT-AAT-ACG-ACT-CAC-TAT-AGG-G-CGACAATGTCGATTCCATCATG-3',
- Downstream-FCV-ORF1-P2 с дизайном 5'- TCAGGGCATAACTCGTCGG-3',
- FCV-ORF1-FAM/RHQ1-beacon с дизайном 5'-FAM-CAA-AGC-GTC-TTC-AAC-CTA-GCU-UUG-RTQ1-3',
которые рассчитаны для целевого участка ORF1-гена калицивируса кошек,
и использование логарифмической функции, выраженной в виде уравнения:
lg ТFCV=-0,2988×CQ-РНК-FCV+9,875,
с достоверностью аппроксимации 0,9999 и эффективностью амплификации 99,01%.
2. Способ по п. 1, где его аналитическая чувствительность составляет 0,2 lg ТЦД50/см3 с достоверностью определения титра инфекционной активности FCV, равной 95,56% при n=75 и p<0,05, в 95%-ном доверительном интервале диагностическая чувствительность составляет 97,96-99,93%, диагностическая специфичность – 98,26-100,00%, k-критерий – 0,992; общая точность – 98,72-99,96%.
| Способ опосредованного определения титра инфекционной активности вируса бешенства штамма ВНИИЗЖ в сырье для инактивированной антирабической вакцины методом ПЦР в режиме реального времени | 2020 |
|
RU2755925C1 |
| Способ опосредованного определения титра инфекционной активности вируса ящура в неинактивированном сырье для вакцин с помощью математического двойного дифференциала данных точки crossing point при амплификации вирусной кДНК | 2022 |
|
RU2793900C1 |
| EP 1156333 B1, 01.08.2007. | |||
Авторы
Даты
2024-06-17—Публикация
2023-11-03—Подача