Изобретение относится к универсальному интеграционному вектору SB7G_HIGH, рекомбинантной плазмиде SB7G_HIGH_DEV_57, обеспечивающей синтез и секрецию рекомбинантного человеческого антитела к рецептор-связывающему домену (RBD) θкороновируса SARS-CoV-2 в клетках млекопитающих, полученному с использованием вектора SB7G_HIGH и моноклональное антитело DEV_K57, обладающее вируснейтрализующей активностью в отношении SARS-CoV-2 и может быть использовано в биотехнологии и генетической инженерии.
Терапия антителами обусловлена высокой селективностью и аффинностью связывания с вирусными антигенами. Существует несколько технологий отбора антигенсвязывающих доменов, таких как гибридомные технологии, фаговый дисплей или изоляция единичных клеток методом клеточного сортинга. Выделение антиген-специфичных В-клеток из периферической крови человека методом клеточного сортинга позволяет отбирать варианты иммуноглобулинов после естественной соматической рекомбинации с нативно реаранжированными вариабельными областями при большем генетическом разнообразии.
Вирус таксономического семейства Coronaviridae тяжелого острого респираторного синдрома 2 (SARS-CoV-2) относится к порядку Nidovirales и причислен к роду Betacoronavirus подсемейства Orthocoronavirinae. Вирус SARS-CoV-2 относится к оболочечным вирусам полиморфной сферической формы и содержит 4 основных структурных белка, а именно мембранные белки (M), белок оболочки (E), белок нуклеокапсида (N) и тримерный поверхностный гликопротеин (белок S), содержащий на поверхности рецептор-связывающий домен (RBD) необходимый для связывания с рецептором – ангиотензинпревращающий фермент 2 (АСЕ2) клетки-хозяина [1, 2].
RBD считается ключевой мишенью для разработки вакцинных препаратов и поиска вируснейтрализующих антител [3]. Показано, что иммуноглобулины на RBD проявляют вируснейтрализующие свойства, предотвращая взаимодействие вирионов SARS-CoV-2 с клеточным рецептором ACE2. Накопления мутаций в RBD, вызванное адаптационной изменчивостью вируса SARS-COV-2, приводило как к появлению новых вариантов, так и к снижению нейтрализующей активности ранее отобранных антител или к ее полному исчезновению относительно исходных вариантов вируса [4]. Поэтому получение моноклональных антител с перекрестной вируснейтрализующей активностью к нескольким актуальным вариантам позволит снизить вероятность потери нейтрализующей активности разрабатываемых препаратов на основе моноклональных антител. Нейтрализующую активность возможно восполнить использованием комбинации антител (коктейли), взаимодействующих с различными высококонсервативными последовательностями рецептор-связывающего домена.
Во время пандемии COVID-19 FDA одобрило несколько коктейлей нейтрализующих антител, обнаруженных в процессе исследования В-лимфоцитов из периферической мононуклеарной крови реконвалесцентов. Бамланивимаб (LY-CoV555, разработчик: Eli Lilly) первое антитело, получившие разрешение FDA на экстренное использование для лечения инфекции COVID-19, которое отозвали в связи с неоднозначными результатами исследований [5].
Известно решение по патенту (RU2765731C1, МПК C07K 16/10, опубл 2019.04.29) [6], где предложено гуманизированное моноклональное антитело, полученное в клетках-продуцентах CHO, имеющее высокую аффиность (константа диссоциации 4*10-9М) к рекомбинантному RBD гликопротеина SARS-CoV-2 варианта Delta и обладающее вируснейтрализующей активностью в концентрации 3,3 нг/мл. Экспрессирующая конструкция не отражена в патенте, однако заявлено, что в качестве основы антитела последовательность иммуноглобулина G1 человека, которое содержит гипервариабельные антигенсвязывающие участки, полученные слиянием спленоцитов иммунизированной мыши BalB/c с линией мышиной миеломы Sp2/0-Ag-14.
В статье Ju et al. [7] кристаллизованы и охарактеризованы RBD-специфические нейтрализующие антитела, полученные из крови инфицированных доноров. Для вложенной полимеразной цепной реакцией (ПЦР) использовали праймеры, описанные в статье Tiller et al. [8], и использовали коммерческий вектор pcDNA3.4 для проведения транзиентной трансфекции клеточной линии HEK293. Известно решение по патенту (RU2800649C2) [11] с описанием получения 27 рекомбинантных антител (iB0-iB26) с высокой аффинностью к RBD коронавируса, часть из которых обладала вируснейтрализующей активностью, и создание комбинаций антител с широкой вируснейтрализующей активностью по отношению SARS-CoV-2 вариантов Beta и Delta. Существенным недостатком данного патента является отсутствие данных о исследованиях нейтрализующей активности в отношении циркулирующих в настоящее время вариантов SARS-CoV-2 (Omicron и д.р.).
Известно решение по международной заявке (WO2022036788, РСТ/CN2020/115480, МПК C07K 16/10, опубл. 24.02.2022), где описывалось антитело CQTS126, полученное транзиентной котрансфекцией линеаризованными кассетами экспрессии в эукариотической линии. Каждая кассета состоит из промотора CMV, лидерной последовательности, ДНК вариабельной и константной областей цепи иммуноглобулина, выделенного с помощью амплификации продукта ОТ-ПЦР из мРНК RBD-специфической В-клетки памяти, посттранскрипционного регуляторного элемент вируса гепатита сурка (WPRE) и сигнала полиаденилирования.
В статье Han X. еt al. [10] получали и изучали антитела аналогичной технологией – транзиентной трансфекцией клеточной линии линеаризованными кассетами. 198 антигенсвязывающих антител, 96 из которых показали нейтрализующую активность в отношении псевдовируса SARS-CoV-2 и блокировали псевдовирусную инфекцию со степенью ингибирования более 75 %. Для наработки антител использовали клеточную культуру Expi293F, трансфицированную двумя экспрессирующими конструкциями на основе вектора pcDNA3.4 и ДНК фрагментов VH и VL.
Наиболее близким аналогом (прототипом) является решение по заявке на патент Китая (CN116284362A, МПК С07К16/10, С12N15/85, опубл. 23.06.2023 г.), в котором описывалось моноклональное антитело VacBB-548, обладающее перекрестной нейтрализующей активностью в отношении множественных сублиний Омикрона. Последовательности, кодирующие легкие и тяжелые домены иммуноглобулина, выделяли из единичных иммортализованных В-лимфоцитов добровольцев, привитых инактивированной вакциной BBIBP-CorV против SARS-CoV-2. Антитела нарабатывались в эукариотических клетках 293F за счет экспрессирующей кассеты из промотора CMV, лидерной последовательности Ig, константной область тяжелой или легкой цепи IgG1, поли(А)-хвост и заменяемых генов VH или VL.
Недостатком аналога по патенту RU2765731C1 является получение гуманизированного мышиного антитела XR19, так как данный метод требует больших затрат по времени и ресурсам. Гуманизированные антитела могут вызвать образование HAHA-ответ (HAHA – human anti-human antibody) с большей вероятностью, чем моноклональные человеческие антитела.
Недостатком в межд. заявке WO2022036788 и наиболее близкого аналога (прототипа) по межд. заявке CN116284362A является проведение транзиентной трансфекции с кратковременной экспрессией интересующих генов в клеточных линиях 293T или 293F. В нашем изобретении интегрировали экспрессирующую кассету в геном клеточной линии CHO-S для получения клеток, стабильно продуцирующих иммуноглобулины. Интеграционный вектор SB7G_HIGH применим к разным эукариотическим клеточным линиям, содержит селективные гены и два участка для интеграции целевых генов, кодирующих легкий и тяжелый домены иммуноглобулинов. Вектор SB7G_HIGH более стабилен к воздействию нуклеаз, чем линейные экспрессионные кассеты, описанные в межд. заявке WO2022036788.
Существенным отличиями от данного прототипа, обеспечивающими достижение технического результата, отражены в пунктах:
1. Сконструирована генно-инженерная кассета, фланкированная сайтами распознавания интегразы (ITR), которая содержит Fab и Fс-фрагмент, регуляторные элементы и селективные маркеры. Транспозаза Sleeping Beauty расщепляет по сайтам распознавания и интегрирует интересующий ген в геном клетки, позволяя получить клетки-трансформанты, стабильно продуцирующие иммуноглобулины;
2. Последовательность Р2А-пептида, располагающаяся между геном флуоресцентного белка EGFP и геном пуромицин-N-ацетил-трансферазы (PuroR) после сайта альтернативной посадки рибосомы IRES в конструкции позволяет производить визуализацию и отбор высокопродуктивных стабильных трансформантов через давление селективным анибиотиком, не влияя на экспрессию иммуноглобулина;
3. Единичные сайты узнавания рестриктазы MluI, NheI, EcoRI и XhoI для клонирования ДНК вариабельного и константного домена цепи моноклонального антитела гарантировали правильную ориентацию вставки;
4. Особенностью полной транскрипционной кассеты являются разные промоторы: сильный промотор с CAG обеспечивает экспрессию гена легкой цепи, а CMV промотор – ген тяжелой цепи иммуноглобулина. Конструкционные варианты промоторов CAG и CMV имеют различные регуляторные области и потенциальные сайты метилирования, что обуславливает разный уровень экспрессии генов [5]. Строение иммуноглобулинов IgG1 обусловлено равным соотношением легких и тяжелых цепей, при этом аффинная хроматография с использованием протеина A позволяет выделить иммуноглобулины, имеющие высококонсервативный Fc фрагмент, таким образом для получения полноразмерных антител требуется обеспечить более высокий уровень экспрессии гена легкой цепи (избыток) относительно гена тяжелой цепи;
5. Нуклеотидные последовательности, кодирующие константные части иммуноглобулинов тяжелой цепи СН и легких цепей CLk/CLl оптимизированы по кодонному составу для повышения эффективности трансляции в клеточной системе CHO;
6. В отличие от наиболее близкого аналога (прототипа) в качестве клеток-продуцентов используется клеточная линии CHO-S, которая характеризуется эффективной посттрансляционной модификацией, устойчивым ростом в суспензионной культуре (улучшенная масштабированность) и более высоким выходом рекомбинантных антител.
Таким образом, существует потребность в создании новых рекомбинантных иммуноглобулинов, специфичных к нескольким актуальным вариантам короновируса SARS-CoV-2 для профилактики и терапии тяжелых форм COVID-19.
Раскрытие изобретения
Техническим результатом заявленного изобретения является получение таких генетических конструкций, которые обеспечивают более эффективный синтез и секрецию рекомбинантных моноклональных антител в эукариотической клеточной линии-продуцента CHO-S, которая характеризуется более эффективной посттрансляционной модификацией и более высоким выходом указанных рекомбинантных антител, обладающих вируснейтрализующей активностью в отношении вируса SARS-CoV-2 вариантов Delta и Omicron.
Указанный технический результат достигается получением рекомбинантного вектора SB7G_HIGH для клонирования кДНК, кодирующих вариабельные домены антител, содержащего экспрессионную кассету, фланкированную инвертированными повторами для интеграции транспозазой Sleeping Beauty (SB) в геном эукариотической клетки, имеющего размер 9906 п.н, нуклеотидную последовательность SEQ NO:1 (фиг. 8) и состоящего в соответствии с физической и генетической картой, представленной на фиг 1, из следующих элементов:
• 5'-ITR и 3'-ITR инвертированные повторы, имеющие координаты с 14 по 238 п.н. и с 7623 по 7847 соответственно, обеспечивают интеграцию участка плазмиды, заключенного между ними, в геном клеток млекопитающих транспозазой Sleeping Beauty (SB);
• CAG промотор – сильный синтетический промотор, состоящий из раннего энхансерного элемента цитомегаловируса (CMV), промотора, первого экзона и первого интрона ген бета-актина курицы, сплайс-акцептор гена бета-глобина кролика, имеющий координаты c 846 по 1503 п.н.;
• LC – область для встраивания гена, кодирующего вариабельный и константный домены легкой цепи каппа/лямбда, содержащая специфические сайты узнавания рестрикции EcoRI и XhoI с координатами 2519 и 2542 п.н. соответственно;
• SV40 poly(A) signal – сигнал полиаденелирования, имеющий координаты c 2543 по 2781 п.н.;
• промотор и энхансер цитомегаловируса человека (CMV) – обеспечивает экспрессию гена в клетках млекопитающих, имеющий координаты c 2784 по 3510 п.н.;
• Inr с Transcription start (+1) – последовательность инициатора транскрипции РНК-полимеразы II, усиливающие активность промотора и имеющие координаты c 3525 по 3531 п.н.;
• Т7 промотер – нуклеотидная последовательность промотора бактериофага Т7, необходимая для in vitro транскрипции, имеющий координаты c 3823 по 3841 п.н.;
• HC – область для встраивания гена, кодирующего вариабельную и константную тяжелую цепь иммуноглобулина, содержащая специфические сайты узнавания рестрикции NheI и MluI с координатами 3842 и 3864 п.н. соответственно;
• IRES2 – внутренний сайт посадки рибосом вируса энцефаломиокардита человека (EMCV), обеспечивающий экспрессию гена устойчивости к пуромицину, имеющий координаты c 3868 по 4454 п.н.;
• EGFP – ген, кодирующий белок с усиленной зеленой флуоресценцией, имеющий координаты c 4455 по 5174 п.н.;
• P2A – 2А пептид из полипротеина тешовируса-1, обеспечивающий расщепление полипротеина на этапе трансляции, имеющий координаты c 5184 по 5240 п.н.;
• PuroR – ген устойчивости к пуромицину, кодирующий N-ацетил трансферазу, имеющий координаты c 5247 по 5849 п.н.;
• WPRE – посттранскрипционный регуляторный элемент вируса гепатита сурка, необходимый для увеличения уровня экспрессии белка, имеющий координаты c 5879 по 6467 п.н.;
• bGH poly(A) signal - сигнал полиаденелирования, имеющий координаты c 6507 по 6731 п.н.;
• ORI (origin of replication) – точка начала репликации плазмидного вектора SB7G, имеющая координаты с 8092 по 8680 п.н.;
• AmpR – ген β-лактамазы в качестве генетического маркера, имеющего координаты c 8851 по 9711 п.н. и определяющего устойчивость к ампициллину клеток бактерии E. Coli.
Указанный технический результат достигается также тем, что получена рекомбинантная плазмида SB7G_HIGH_DEV_K57, предназначенная для создания рекомбинантной клеточной линии, стабильно экспрессирующей полноразмерное рекомбинантное человеческое антитело DEV_K57, размером 12062 п.н, имеющая нуклеотидную последовательность, описанную SEQ NO:6 (фиг. 13), и содержащая в соответствии с физической и генетической картой, представленной на фиг 2, кодон-оптимизированные последовательности, кодирующие тяжелую цепь (IgG1) и легкую цепь каппа (IgK), и фрагменты кДНК, характеризующиеся нуклеотидными последовательностями SEQ ID NO:2 (фиг. 9) и SEQ ID NO:3 (фиг. 10), которые кодируют вариабельные домены легкой и тяжелой цепи антитела соответственно, состоящая из следующих элементов:
• 5'-ITR и 3'-ITR инвертированные повторы, имеющие координаты с 14 по 238 п.н. и с 9779 по 10003 п.н. соответственно, обеспечивают интеграцию участка плазмиды, заключенного между ними, в геном клеток млекопитающих транспозазой Sleeping Beauty (SB);
• CAG промотор – сильный синтетический промотор, состоящий из раннего энхансерного элемента цитомегаловируса (CMV), промотора, первого экзона и первого интрона ген бета-актина курицы, сплайс-акцептор гена бета-глобина кролика, имеющий координаты c 846 по 1503 п.н.
• VK_DEV_57 – последовательность кДНК, имеющая координаты c 2528 по 2932 п.н. и кодирующая вариабельный домен легкой цепи антитела DEV_K57;
• IgK – нуклеотидная кодон-оптимизированная последовательность, имеющая координаты c 2933 по 3247 п.н. и кодирующая константный домен легкой цепи каппа CLk;
• SV40 poly(A) signal – сигнал полиаденелирования, имеющий координаты c 3257 по 3495 п.н.;
• энхансер и промотор цитомегаловируса человека (CMV) – обеспечивают экспрессию гена в клетках млекопитающих и имеют координаты c 3498 и 4224 п.н.;
• Inr с Transcription start (+1) – последовательность инициатора транскрипции РНК-полимеразы II, имеющие координаты c 4239 по 4245 п.н. и усиливающие активность промотора;
• Т7 промотер – нуклеотидная последовательность промотора бактериофага Т7, необходимая для in vitro транскрипции и имеющая координаты c 4537 по 4555 п.н.;
• VH_DEV_57 – последовательность кДНК, кодирующая вариабельный домен легкой цепи антитела DEV_K57 и имеющая координаты c 4564 по 5019 п.н.;
• hIgG1 – нуклеотидная кодон оптимизированная последовательность, кодирующая константный домен тяжелой цепи и имеющая координаты c 5020 по 6012 п.н.;
• IRES2 – внутренний сайт посадки рибосом вируса энцефаломиокардита человека (EMCV), обеспечивающий экспрессию гена устойчивости и имеющий координаты с 6024 по 6610 п.н.;
• EGFP – ген, кодирующий белок с усиленной зеленой флуоресценцией и имеющий координаты c 6611 по 7330 п.н.;
• P2A – 2А пептид из полипротеина тешовируса-1, обеспечивающий расщепление полипротеина на этапе трансляции и имеющий координаты c 7340 по 7396 п.н.;
• PuroR – ген устойчивости к пуромицину, кодирующий N-ацетил трансферазу и имеющий координаты c 7403 по 8005 п.н.;
• WPRE – посттранскрипционный регуляторный элемент вируса гепатита сурка, необходимый для увеличения уровня экспрессии белка и имеющий координаты c 8035 по 8623 п.н.;
• bGH poly(A) signal - сигнал полиаденелирования, имеющий координаты c 8663 по 8887 п.н.;
• ORI (origin of replication) – точка начала репликации плазмидного вектора SB7G, имеющая координаты c 10248 по 10836 п.н.;
• AmpR – ген β-лактамазы в качестве генетического маркера, имеющий координаты c 11007 по 11867 п.н. и определяющего устойчивость к ампициллину клеток бактерии E. Coli.
Указанный результат также достигается получением рекомбинантного моноклонального антитела DEV_K57, обладающего вируснейтрализующей активностью в отношении SARS-CoV-2 вариантов Delta и Omicron и содержащего константную область человеческого IgG1 и антигенсвязывающий центр с аминокислотной последовательностью вариабельной области легкой κ-цепи SEQ ID NO: 4 (фиг. 11) и тяжелой цепи SEQ ID NO: 5 (фиг. 12).
Моноклональное антитело DEV_K57 продуцируется с использованием эукариотической клеточной линией-продуцента CHO-S, полученной котрансфекцией экспрессирующей конструкции (табл. 1) и плазмиды, несущей ген транспозазы Sleeping Beauty.
Таблица 1. Перечень нуклеотидных и аминокислотных вариабельных последовательностей тяжелой и легкой цепей антитела DEV_K57, специфичных к RBD SARS-CoV-2
домен
После введения ДНК в клетку транспозаза Sleeping Beauty распознает инвертированные повторы, фланкирующие экспрессирующую кассету, и специфически вырезает целевой ген, встраивая его в геном клетки. Тяжелые (HC) и легкие (LC) цепи иммуноглобулинов синтезируются внутри клетки и котрансляционно перемещаются в просвет эндоплазматического ретикулума. HC cобираются в димеры, которые совмещаются с LC ковалентными дисульфидными связями, затем подвергаются посттрансляционным модификациям.
Геном-интегрированные клетки-трансформанты CHO-S, продуцирующие полноразмерный рекомбинантный иммуноглобулин в культуральную жидкость, отбирали методом селекции пуромицином вовремя пассирования клеток. Культуральную жидкость, содержащую иммуноглобулины, собирали со стабильных клеток-трансформантов и очищали антитело аффинной хроматографией. После аффинной экстракции изучали специфическую и вируснейтрализующую активность в отношении вируса SARS-CoV-2 вариантов Delta и Omicron методами микронейтрализации, а также ТФ ИФА и вестерн-блоттинг. Полученный иммуноглобулин DEV_K57 может быть использован в качестве компонента при разработке диагностических и терапевтических препаратов.
Сущность заявленного изобретения поясняется чертежами.
Фиг. 1 – физическая и генетическая карта универсального интеграционного вектора SB7G_HIGH. Фиг.2 - физическая и генетическая карта рекомбинантной плазмиды SB7G_HIGH_DEV_K57. Фиг. 3 – схема амплификации последовательностей константных доменов СH(А), CLk(Б), CLl(С) и последовательностей вариабельных доменов VL (Д) и VH (Е) антитела DEV_K57. Фиг. 4 – схемы получения продуктов VK_CLk_DEV_57 (А) и VH_CH_DEV_57 (Б) с использованием праймеров, обеспечивающих перекрытие последовательностей константного и вариабельного доменов, для последующей ПЦР с перекрывающимися концами (overlap extension PCR на схеме). Фиг. 5 – схема последовательного клонирования нуклеотидных последовательностей, кодирующих тяжелую и легкую цепь антитела DEV_K57 в интеграционный вектор SB7G_HIGH для получения экспрессирующей конструкции. Фиг. 6 – хроматограмма аффинной экстракции, отражающая нанесение культуральной среды и элюцию антитела DEV_K57. Фиг.7 – электрофореграмма (A) разделения в SDS PAGE хроматографически очищенных антител в восстанавливающих (+) и невосстанавливающих условиях (–) и нитроцеллюлозная мембрана (Б), полученная в результате постановки вестерн-блот анализа с использованием рекомбинантного рецептор-связывающего домена RBD. Фиг. 8 - нуклеотидная последовательность интеграционного вектора SB7G_HIGH. Фиг. 9 - нуклеотидная последовательность кДНК, кодирующая вариабельный домен легкой цепи VK_DEV_57 антитела DEV_K57. Фиг. 10 - нуклеотидная последовательность кДНК, кодирующая вариабельный домен тяжелой цепи VH_DEV_57 антитела DEV_K57. Фиг. 11 - аминокислотная последовательность вариабельной области легкой κ-цепи рекомбинантного моноклонального антитела DEV_K57. Фиг. 12 - аминокислотная последовательность вариабельной области тяжелой цепи рекомбинантного моноклонального антитела DEV_K57. Фиг. 13 - нуклеотидная последовательность рекомбинантной плазмиды SB7G_HIGH_DEV_57, обеспечивающая синтез и секрецию рекомбинантного человеческого антитела к рецептор-связывающему домену (RBD) короновируса SARS-CoV-2.
Осуществление изобретения
Для осуществления настоящего изобретения на первом этапе получили экспрессирующие конструкции. Провели амплификацию фрагментов ДНК константных доменов CH и CL. Константные и вариабельные последовательности объединяли методом ПЦР с перекрывающимися концами, внеся концевые уникальные сайты рестрикции. Вариабельную последовательности лёгкой цепи встраивали в интеграционный вектор по сайтам EcoRI и XhoI, а тяжелую по сайтам NheI и MluI.
Полученную экспрессирующую конструкцию использовали для трансфекции перевиваемой линии клеток СHO-S с использованием полиэтиленимина (PEI), далее отбирали стабильный клон давлением селективного антибиотика пуромицина. Культуральную жидкость после культивирования клеток-продуцентов, содержащую рекомбинантный иммуноглобулин, собирали и осветляли для последующей хроматографической очистки.
Рекомбинантный иммуноглобулин экстрагировали аффинной хроматографией на иммобилизованном протеине А, далее фракцию анализировали методами SDS PAGE, вестерн-блоттинг и непрямого твердофазного ИФА. Вируснейтрализующую активность изучали в реакции нейтрализации и оценивали рабочую концентрацию антитела при 50 % и 100% ингибировании (IC50 и IC100).
Осуществление изобретения подтверждается следующими примерами.
Пример 1. Получение последовательностей, кодирующей легкие и тяжелые вариабельные домены антитела
1.1. Амплификация последовательностей константных доменов CH, CLk, CLl иммуноглобулина человека
Олигонуклеотиды (Таблица 2 и 3) для амплификации методом ПЦР подобраны с помощью ПО SnapGene v.5.3.2 и синтезированы в компании «ЗАО Евроген» (г. Москва). Постановку ПЦР проводили с использованием ДНК-полимеразы Q5 High Fidelity DNA Polymerase (New England Biolabs, США) согласно рекомендациям производителя. Компоненты реакции смешивали на холодовом штативе и помещали в амплификатор в Thermal Cycler С1000 (BioRad, США). Для реакциии использовались праймеры, указанные в таб. 2.
Таблица 2. Праймеры используемые для амплификации последовательностей константных доменов CH, CLk, CLl иммуноглобулина человека
Условия реакции: предварительная денатурация при 98 °С в течение 45 сек; денатурация при 98 °С в течение 15 с, отжиг при 57 °С в течение 30 с, удлинение при 72 °С в течение 1 мин, 28 циклов; элонгация при 72 °C в течение 5 минут. Подробная схема амплификации указана на фигуре 3.
1.2. Амплификация последовательностей вариабельных доменов VK_DEV_57 и VH_DEV_57 иммуноглобулина человека
Для получения последовательностей полноразмерных доменов иммуноглобулинов и последующего клонирования в экспрессирующий вектор использовали плазмидные конструкции, кодирующие вариабельные домены иммуноглобулинов. Вариабельные последовательности ранее получены методом ОТ-ПЦР из изолированных RBD-специфичных В-лимфоцитов донора-реконвалесцента COVID-19 и субклонированы в вектор pJet1.2.
На одну реакцию использовали 10 нг плазмиды и добавляли в реакционную смесь, состоящую из буфера для из Q5 High Fidelity DNA Polymerase, 25 мкМ франкирующих олигонуклеотидов, и 1 е.а. Q5 High Fidelity DNA Polymerase. Реакцию проводили в амплификаторе в Thermal Cycler С1000 (BioRad, США) с использованием олигонуклеотидных праймеров из таб. 3. Условия реакции: предварительная денатурация при 98°С в течение 45 сек; денатурация при 98°С в течение 15 с, отжиг при 57°С в течение 30 с, удлинение при 72°С в течение 1 мин, 28 циклов; элонгация при 72°C в течение 5 минут.
Таблица 3. Праймеры используемые для амплификации последовательностей вариабельных доменов VH и VK иммуноглобулина
1.3. Выделение ДНК из агарозного геля
Очистку продуктов амплификации проводили методом гель-электрофореза в 1,5 % агарозном геле в трис-ацетатном буфере (ТАЕ). Для оценки длины последовательности ДНК использовали маркер длин ДНК 1 т.п.н. (1 kb DNA Ladder, SibEnzyme). Гель окрашивали раствором бромистого этидия в течение 10 мин и проявляли с использованием УФ-трансиллюминатора «UV Transilluminator 2000» (Bio-Rad, США). Фрагменты необходимой длины вырезали из геля и экстрагировали ДНК набором QIAquick Gel Extraction kit (Qiagen, Германия) в соответствии с рекомендациями производителя. Концентрацию выделенных ДНК фрагментов измеряли спектрофотометром NanoDrop One (Thermo Scientific, США).
1.4. Объединение последовательностей вариабельного и константного доменов легкой цепи методом ПЦР с перекрывающимися концами
Для получения гена VK_CLk_DEV_57, кодирующего вариабельный домен VK_DEV_57 и константный домен легкой цепи каппа CLk, использовали метод ПЦР с перекрывающимися концами (фиг. 4А). Выделенные амплифицированные последовательности смешивали в эквимолярных соотношениях и добавляли в реакционную смесь со специфическими олигонуклеотидными праймерами R-VL_EcoRI и R-hIGK_XhoI. В зависимости от типа легкой цепи: каппа (kappa, κ) или лямбда (lambda, λ), выбирали кодон-оптимизированные гены CLk или CLl. В качестве специфический праймеров использовали
Реакцию осуществляли с использованием ДНК-полимеразы Q5 High Fidelity DNA Polymerase (New England Biolabs, США) согласно рекомендациям производителя. Условия реакции, следующие: предварительная денатурация при 98 °С в течение 45 сек; денатурация при 98 °С в течение 15 с, отжиг при 60°С в течение 30 с, удлинение при 72°С в течение 1 мин, 28 циклов; элонгация при 72 °C в течение 5 минут. Продукты ПЦР реакции разделяли в 1,5 % агарозном геле, как описано в пункте 1.3. После визуализации под УФ-излучением фрагмент VK_CLk_DEV_57 (727 п.н.) вырезали и выделяли из геля ДНК набором QIAquick Gel Extraction kit (Qiagen, Германия).
1.5. Объединение последовательностей вариабельного и константного доменов тяжелой цепи
Для формирования гена VH_CH_DEV_57, кодирующего полноразмерную тяжелую цепь иммуноглобулина, проводили постановку реакции ПЦР с перекрывающимися концами. Объединяли фрагменты VH_DEV_57 и CH, кодирующие только вариабельную часть тяжелой цепи и константную тяжелую цепь иммуноглобулина, соответственно с использованием олигонуклеотидных специфических праймеров R-hIGH_MluI и R-VH_NheI. В реакционную смесь добавляли фрагменты в эквимолярных соотношениях согласно фиг. 4Б и вносили в стандартную реакционную смесь.
Реакцию осуществляли с использованием ДНК-полимеразы Q5 High Fidelity DNA Polymerase (New England Biolabs, США) согласно рекомендациям производителя. Условия реакции: предварительная денатурация при 98 °С в течение 45 сек; денатурация при 98 °С в течение 15 с, отжиг при 60 °С в течение 30 с, удлинение при 72 °С в течение 90 сек, 28 циклов; элонгация при 72 °C в течение 5 минут. Продукты ПЦР реакции разделяли в 1,5 % агарозном геле, как описано в пункте 1.3. После визуализации под УФ-излучением фрагмент VH_CH_DEV_57 (1482 п.н.) вырезали и выделяли из геля ДНК набором QIAquick Gel Extraction kit (Qiagen, Германия).
Пример 2. Получение экспрессирующей конструкции SB7G_HIGH_DEV_K57
Получение экспрессирующей конструкции, кодирующей полноразмерное антитело DEV_K57, проводили в два этапа. Сначала в интеграционный вектор SB7G_HIGH клонировали ген VK_CLk_DEV_57, кодирующий легкий полноразмерный домен иммуноглобулина, полученный в пункте 1.4. В полученную промежуточную плазмидную конструкцию SB7G_HIGH_VK_CLk_DEV_57 клонировали последовательность VH_CH_DEV_57, кодирующую тяжелый полноразмерный домен этого же иммуноглобулина, полученную в пункте 1.5.
2.1 Гидролиз ДНК эндонуклеазами рестрикции
Для клонирования гена VK_CLk_DEV_57 в плазмиду SB7G_HIGH осуществляли ферментативный гидролиз интеграционного вектора и ПЦР продукта эндонуклеазами рестрикции EcoRI и XhoI (NEB, США). Для этого 2 мкг плазмидной ДНК SB7G_HIGH гидролизовали эндонуклеазами рестрикции 10 е.а. EcoRI и 10 е.а. XhoI (NEB) в буфере CutSmart в течение 90 минут при температуре 37 °C. Аналогично проводили гидролиз ПЦР продукта VK_CLk_DEV_57.
Гидролизаты разделяли методом электрофореза с последующим выделением гидролизованных продуктов, как описано в пункте 1.3.
2.2 Клонирование гена VK_CLk_DEV_57 в интеграционный вектор SB7G_HIGH
На первом этапе клонирования (фиг. 5) подготовили смесь для лигирования, состоящую из однократного буфера для T4 ДНК-лигазы (Thermo Scientific, США), 1 е.а. T4 ДНК-лигазы (Thermo Scientific, США) с добавлением гидролизованного вектора SB7G_HIGH и ПЦР продукта VK_CLk_DEV_57 в эквимолярном соотношении. Смесь инкубировали при 22 °C в течение 30 минут. На втором этапе трансформировали химически компетентные клетки E.coli штамм NEB Stable (NEB, США) 2 мкл лигазной смеси. Клетки высевали на твердую агаризованную среду SOB (среда SOB с 1,5 % агара) с добавлением селективного антибиотика ампициллина (50 мкг/мл) и инкубировали 18 часов при 30 °С. Далее единичные ампициллин-резистентные колонии ссаживали в 5 мл питательного бульона SOB с добавлением ампициллина в концентрации 50 мкг/мл и культивировали до оптической плотности OD=0,6. Бактериальную массу центрифугировали при 6000 g в течение 5 минут и из осадка выделяли плазмиду SB7G_HIGH_VK_CLk_DEV_57 набором QIAprep Spin Miniprep Kit (Qiagen, Германия) согласно рекомендациям производителя.
2.3 Гидролиз гена VH_CH_DEV_57 и промежуточной плазмиды SB7G_HIGH_VK_CLk_DEV_57
Следующим шагом было клонирование гена VH_CH_DEV_57 в промежуточную плазмиду SB7G_HIGH_VK_CLk_DEV_57 (фиг. 5). ПЦР продукт VH_CH_DEV_57 и промежуточную плазмиду SB7G_HIGH_VK_CLk_DEV_57 обрабатывали эндонуклеазами рестрикции NheI и MluI (NEB, США) из расчета 10 а.е. фермента на 1 мкг ДНК. Реакционную смесь с плазмидной ДНК инкубировали в течение 90 минут, а ПЦР продукт – 45 минут при температуре 37°C.
Фрагменты реакционной смеси разделяли методом электрофореза с последующим выделением, как описано в пункте 1.3.
2.4 Клонирование ПЦР продукта VH_CH_DEV_57 в промежуточную плазмидную конструкцию SB7G_HIGH_VK_CLk_DEV_57
Реакцию лигирования проводили 60 минут при 20°С с использованием ДНК-лигазы фага Т4 (ThermoFisher, США) в соответствии рекомендациям производителя. Лигазную смесь использовали для трансформации культуры компетентных клеток E.coli штамм NEB Stable (NEB, США). Клетки высевали на твердую агаризованную среду SOB с добавлением ампициллина. Колонии анализировали на наличие целевых фрагментов стандартным методом ПЦР-скрининга. Положительные колонии ссаживали в жидкую среду SOB с добавлением 50 мг/мл ампициллина и инкубировали при 30 °С до оптической плотности OD=0,6, затем выделяли плазмидную ДНК набором QIAprep Spin Miniprep Kit (Qiagen, Германия) в соответствии с рекомендациями производителя.
2.4 Подтверждение структуры экспрессирующей конструкции SB7G_HIGH_DEV_K57 секвенированием по методу Сэнгера
Скрининг рекомбинантных плазмид проводили методом рестрикционного анализа с последующим подтверждением корректности нуклеотидных последовательностей секвенированием по методу Сэнгера.
С использованием программного обеспечения SnapGene 5.3.2 рассчитывали длины фрагментов, возникающих при гидролизе плазмиды SB7G_HIGH_DEV_K57 (4 фрагмента с длинами 8279 п.н., 1997 п.н., 1000 п.н. и 786 п.н.). Плазмидную ДНК гидролизовали эндонуклеазами рестрикции HindIII, XhoI, XbaI. Гидролизаты анализировали разделением в 1% агарозном геле. Плазмидные конструкции отбирали после сравнения полученных гидролизатов плазмид с маркером молекулярных масс и с ожидаемой рестрикционной картиной.
Корректность нуклеотидной последовательности отобранных плазмид подтверждали секвенированием по методу Сэнгера.
Для постановки реакции использовали набор BrilliantDye Terminator (v3.1) Cycle Sequencing kit (Nimagen, Нидерланды), очищали образцы через Sephadex G-50 Superfine (Cytiva, Швеция) колоночным методом согласно рекомендациям производителя, затем проводили капиллярный гель-электрофорез на автоматическом секвенаторе 3500XL Applied Biosystems (США). Полученные хроматограммы анализировали с использованием программного обеспечения SnapGene 5.3.2.
Полученная рекомбинантная плазмида SB7G_HIGH_DEV_K57 содержала экспрессирующие кассеты для генов VH_CH_DEV_57 и VK_CLk_DEV_57. Так как интеграционный вектор содержит специфческие сайты гидролиза для последовательного встраивания последовательностей полноразмерных генов легкой и тяжелой цепей соответственно, то на его основе могут быть получены рекомбинантные конструкции, кодирующие иммуноглобулины с легкими цепями как типа каппа (kappa, κ), так и типа лямбда (lambda, λ ).
Пример 3. Трансфекция клеток линии CHO-S плазмидами SB7G_HIGH_DEV_K57 и SB100X
Для получения клеточной линии, стабильно экспрессирующей полноразмерное рекомбинантное антитело DEV_K57, котрансфекцировали клеточную линию CHO-S рекомбинантной плазмидой SB7G_HIGH_DEV_K57 совместно с конструкцией SB100X, экспрессирующей транспозазу Sleeping Beauty, с использованием полиэтиленимина (PEI).
Для проведения трансфекции смешали рекомбинантную плазмиду SB7G_HIGH_DEV_K57, плазмиду SB100X в среде OptiMEM (Gibco, США), далее добавили катионный полимерный носитель PEI (Sigma, США). Смесь инкубировали 10 минут при комнатной температуре. Суммарное количество плазмидной ДНК для трансфекции составило 27 мкг на 25 мл клеточной суспензии в соотношении 1 часть плазмиды SB100X к 9 частям экспрессирующей конструкции SB7G_HIGH_DEV_K57.
Клеточную линию CHO-S (8×106 – 1,5×107 кл) центрифугировали при 300×g, аккуратно ресуспендировали в 25 мл среды OptiMEM и переносили в 125 мл колбу Эрленмейера. К суспензии клеток вносили по каплям 700 мкл комплексов ДНК/PEI и инкубировали сутки при непрерывном перемешивании 130 об/мин на орбитальном шейкере при 37 °C в атмосфере 8,0 % СО2 и относительной влажности ≥ 90 %. Затем проводили замену среды для трансфекции на среду Hy Clone ADCF-MAb (Cytiva, США) с добавлением однократного раствора аминокслот МЕМ (Gibco), 200 мМ до конечной концентрации 8 мМ раствора L-Глутамина и 0,18% Pluronic F-68 (Invitrogen).
Эффективность трансфекции оценивали через 72 часа по количеству клеток с зеленой флуоресценцией ЕGFP с использованием системы визуализации Evos M5000. В качестве положительного контроля использовали клетки CHO-S, трансфицированные плазмидой phMGFP (Promega, Великобритания).
Через 3-4 суток вносили в клетки селективный антибиотик пуромицин в концентрации 10 мкг/мл. Концентрацию пуромицина увеличивали на 10 мкг/мл при пассировании клеток-трансформантов.
Суспензию из геном-интегрированных клеток-трансформантов CHO-S, устойчивых к воздействию 50 мкг/мл пуромицина, культивировали в среде в течение 7-14 суток при постоянном помешивании на орбитальном шейкере с частотой 130 об/мин при 37 °C, относительной влажности ≥ 80 % и 8 % содержании CO2,, периодически добавляя свежую ростовую среду для поддержания жизнеспособности 95-98 %. Жизнеспособность клеток определяли окрашиванием мертвых клеток витальным красителем трипановым синим. Для этого отбирали 50 мкл клеточной суспензии и смешивали с равным объемом 0,1 % раствора красителя и проводили автоатический почет клеток на приборе Luna (Logos Biosystems, Корея).
Пример 4. Очистка и характеризация рекомбинантных иммуноглобулинов с клеточных линий-продуцентов CHO-S_DEV_K57
После селекции стабильных клеток-продуцентов производили масскультивирование со сбором культуральной жидкости каждые 7-14 суток. Условия культивирования: температура 37°С, перемешивание на орбитальном шейкере 140 об/мин, 98 % влажности, в атмосфере 8 % CO2, среда HyClonе ADCF-MAb (Cytiva, США) с добавлением 0,18% (v/v) Pluronic F-68 (Invitrogen). После накопления 500 мл ростовой среды проводили аффинную хроматографию.
4.1. Аффинная хроматография
Культуральную жидкость, содержащую рекомбинантное антитело изотипа IgG1, центрифугировали для удаления клеточного дебриса при 10000×g в течение 15 мин. Супернатант наносили на колонку с 10 мл сорбента MabSure Select (Cytiva, США) с использованием хроматографа AKTA Pure 150 (GE, США). Сорбент отмывали от не связавшихся компонентов среды 7-10 объемами колонки буфером для нанесения (0,3 М NaCl, 1,76 мM KH2PO4, 8,1 мM Na2HPO4, 2,7 мM KCl pH 7,8) до выхода оптической плотности на базовую линию и заполняли колонку объемом буфера для элюции (0,1М глицин, pH 3,0). Элюцию проводили при скорости непрерывного потока 3 мл/мин и собирали фракцию элюата, соответствующую пику иммуноглобулинов на длине волне 280 нм. Для нейтрализации буфера для элюции и предотвращения деградации антител доводили pH элюата до 7,0-7,4 добавлением буфера для нейтрализации (1М Трис-HCl, рН 8,0) в соотношении буфера к элюату 1:9. Полученный элюат диализовали против ФСБ (фосфатно-солевой буфер, 137 мМ NaCl, 2,7 мМ KCl, 10 мМ Na2HPO4, 1,76 мМ KH2PO4, рН 7,4) в течение 12 часов и хранили при температуре + 4 °С.
Оценку гомогенности и степени очистки препарата проводили с методом SDS PAGE в денатурирующих условиях (фиг. 6).
4.2. Белковый электрофорез белковых фракций в полиакриламидном геле в присутствии додецилсульфата натрия (SDS-PAGE)
Молекулярную массу иммуноглобулинов определяли электрофоретическим анализом по методу Лэммли в 10 %-ном полиакриламидном геле. Образцы для SDS PAGE в невосстанавливающих условиях готовили, смешивая 10 мкл фракции элюата с равным объемом буфера для внесения (25 мM Tris-HCl, pH 6,8, 1 % SDS, 0,05 % бромфеноловый синий, 5 % глицерин). Для изучения вклада дисульфидных связей готовили образцы, смешивая 10 мкл фракцию элюата буфер с 10 мкл буфера для внесения с добавлением 2 % 2-меркаптоэтанола для восстановления дисульфидных связей. Все образцы инкубировали при 96 °C в течение 5 мин. В качестве белкового маркера с известными молекулярными массами использовали PageRuler Prestained Protein Ladder (Thermo Scientific, США).
Электрофорез проводили в трис-глициновом буфере (25 мM Трис, 250 мM глицин, 0,1 % SDS, pH 8,3), затем гель инкубировали в буфере для окрашивания (0,25% Coomassie Brilliant Blue R-250, 45% этанол, 10 % уксусная кислота) в течение 40 минут при комнатной температуре. Буфер для окрашивания удаляли и отмывали гель смесью 10 % бутанола и 5 % уксусной кислоты на водяной бане при температуре. Анализ геля проводили на системе гель-документирования ImageQuant 800 Fluor (GE, США).
Электрофорезом в невосстанавливающих условиях показано, что исследованная фракция антитела содержала белки с высокой молекулярной массой (~148кДа). Подтверждена чистота фракций – не менее 95 % (фиг. 6). В восстанавливающих условиях антитело разделялось на лёгкие и тяжёлые цепи с молекулярной массой 25 кДа и 50 кДа, что соответствовало легкой и тяжелой цепи иммуноглобулина после восстановления дисульфидных связей между ними (фиг. 7A).
4.3. Вестерн-блоттинг анализ
Вестерн-блоттинг анализ использовали для определения специфичности антитела к рекомбинантному RBD определенной молекулярной массы.
Антиген RBD разделяли методом SDS-PAGE в денатурирующих условиях в 10% полиакриламидном геле. Электроперенос разделенных в геле белков на нитроцеллюлозную мембрану с величиной пор 0,22 нм проводили полусухим методом на аппарате Western Blot Semi-Dry Transfer Device (Invirogen, США) в течение 10 минут. Неспецифические сайты связывания блокировали в блокирующем растворе (ФСБ, 3 % БСА, бычьего сывороточного альбумина) в течение 12 часов при + 4 °C. Затем мембрану помещали в ФСБ-Т (ФСБ с 0,01% Tween 20) для удаления излишек БСА, инкубировали в течение 30 минут при непрерывном перемешивании. Мембрану инкубировали с антителами в блокирующем буфере (разведение 1:100) в течение 60 минут при 37 °C. Не связавшиеся антитела удаляли, трижды инкубируя мембрану в ФСБ-Т по 10 минут. Для выявления связавшихся иммунных комплексов мембраны инкубировали с конъюгатом Protein G, меченный пероксидазой хрена в разведении 1:10000 (Abcam) в течение 60 минут при 37 °C. Не связавшийся конъюгат удаляли пятикратной промывкой мембраны в ФСБ-Т. Визуализацию иммунных комплексов проводили в проявочном буфере (1M Tris-HCl, 100мM имидазол, 1М 3,3′-Диаминобензидина) с добавлением 0,001 % пероксида водорода, выступавшего окислителем 3,3′-Диаминобензидина в реакции, катализируемой пероксидазой хрена, придавая иммунокомплексам характерную коричневую окраску.
На фиг. 7Б видно, что рекомбинантное антитело из очищенной фракции взаимодействовало с рекомбинантным RBD SARS-CoV-2. Яркие коричневые полосы на мембране свидетельствовали о том, что антитело распознает линейный эпитоп.
4.4. Иммуноферментный анализ
Специфичность рекомбинантного антитела к поверхностному гликопротеину вируса SARS-CoV-2 вариантов Delta или Omicron оценивали методом ТФ ИФА. Перед постановкой реакции вирусный материал SARS-CoV-2, содержащийся в культуральной жидкости, инактивировали b-пропиолактоном в лаборатории уровня биобезопасности BSL-3
Инактивированную вируссодержащую жидкость разводили ФСБ и сорбировали вирус в 96-луночный полистирольный планшет с плоским дном в течение 14 часов при 25 °C. Культуральную вируссодержащую жидкость удаляли и трехкратно промывали лунки ФСБ-T. Неспецифические сайты связывания блокировали однократным раствором рыбьего желатина (10×Fish Gelatin Blocking Agent, Biotium, США) в ФБР-Т 2 ч при температуре 37 °C, затем трижды отмывали ФСБ-T для удаления излишек блокирующего буфера.
Очищенные рекомбинантные антитела разводили ФСБ-Т до 1:1000. Разведения антител инкубировали с сорбированным вирусным материалом в течение 12 часов при + 4 °C. Не связавшиеся антитела удаляли и трижды промывали лунки планшета ФСБ-Т. Антитела, селективно связавшиеся с гликопротеином на поверхности вириона, выявляли иммуноконъюгатом, состоящим из антитела кролика против Fc-фрагмента антител IgG1 человека, меченного пероксидазой хрена (Rabbit Anti-Human IgG H&L (HRP), Abcam, Великобритания) в разведении 1:10000. Разведение инкубировали 60 минут при 37 °C и удаляли не связавшийся иммунокоъюгат пятикратной промывкой ФСБ-Т. Затем добавляли в лунки хромогенный субстрат TMB (Abcam, Великобритания) и инкубировали 15 минут, наблюдая окрашивание лунок в синий цвет. Реакцию окисления TMB H2O2 (содержится в хромогенном субстрате), катализируемую пероксидазой хрена, останавливали 0,1М раствором соляной кислоты и измеряли оптическую плотность на длине волны 450 нм (ОП450) с использованием многофункционального анализатора Varioskan LUX (TermoFisher, США)
Отрицательный и положительный контроль ТФ ИФА учитывали для каждого планшета индивидуально. Лунки отрицательного контроля инкубировали с ФСБ вместо анализируемых разведений антител, а лунки положительного контроля – инактивированной сывороткой донора-реконвалесцента в разведении 1:300 соответственно. Положительным сигналом считали значение ОП450 с превышением ОП исследуемого антитела над значением отрицательного контроля более чем 3 раз.
Таблица 4. Оптические плотности при длине волны 450 нм в ТФ ИФА к короновирусу SARS-CoV-2
(Omicron)
(Omicron)
Показано, что иммуноглобулин взаимодействовал с эпитопами вирусного антигена. Положительные сигналы выделены в таб. 4 жирным шрифтом. ОП450 антитела DEV_K57 превышало значение отрицательного контроля в 3 раза и соответствовало положительному сигналу в ТФ ИФА как для варианта Omicron, так и для варианта Delta. Предполагалось, что антитело DEV_K57 специфично к поверхностным гликопротенам вируса SARS-CoV-2 вариантов Delta и Omicron может проявлять и вируснейтрализующие свойства в отношении вируса.
Пример 5. Определение вируснейтрализующей активности рекомбинантных антител в отношении различных вариантов вируса SARS-CoV-2
Выявление вируснейтрализующей активности рекомбинантного антитела по отношению к актуальным вариантам SARS-CoV-2 проводили с помощью постановки реакции нейтрализации.
При постановке реакции нейтрализации использовали клеточную линию Vero Е6, выращенную в 96-луночных культуральных планшетах. Для получения монослоя клеток с конфлюэнтностью 90-95 %, суспензию клеток Vero Е6 высевали из расчета 2×104 клеток на лунку и инкубировали 24 часа в CO2-инкубаторе (температура - 37 °С, влажность ≥ 95 %, CO2 - 5 %).
Готовили серию последовательных двукратных разведений рекомбинантных антител в культуральной среде, где первое разведение соответствовало 1:10. Далее каждое разведение иммуноглобулинов и положительный контроль (питательная среда) смешивали с равным объёмом вируссодержащего материала (не менее 3,0 lg ТЦД50/мл вируса SARS-CoV-2 варианта Delta или Omicron) и инкубировали 1 ч при 37 °C в СО2- инкубаторе.
Из 96-луночного планшета с клетками Vero E6 удаляли ростовую среду и двукратно обмывали монослой клеток ФСБ. Прединкубированный вируссодержащий материал с разведениями анализируемого антитела, а также положительный контроль вносили в лунки с клетками по 100 мкл из расчета 4 повтора на каждое разведение и инкубировали в CO2-инкубаторе в течение 48 ч при 37 °C. В качестве отрицательного контроля использовали интактную культуру клеток Vero E6.
По окончанию инкубации удалили содержимое лунок и фиксировали клетки 30 % ацетоном в ФСБ (pH 7.4) в течение 10 минут с последующей двукратной отмывкой фиксированных клеток ФСБ.
Активность эндогенной пероксидазы блокировали раствором 0,3 % H2O2 в ФСБ в течение 30 минут для уменьшения количества ложноположительных сигналов. После инкубации лунки двукратно промывали ФСБ и блокировали остаточные сайты сорбции в течении 60 минут однократным раствором желатина (10x Fish Gelatin Blocking Agent), разведенным в ФСБ с последующей двукратной отмывкой ФСБ с добавлением 0,2 % Tween 20 (ФСБ-Т).
Затем в лунки вносили по 50 мкл раствора моноклонального антитела к антигенам N или S (AtaGenix Laboratories, КНР), разведенного в ФСБ-Т (разведение 1:500), и инкубировали в течение 60 мин при 37 °C. Не связавшиеся моноклональные антитела удаляли и четырехкратно отмывали клеточный монослой раствором ФСБ-Т. В отмытые лунки вносили конъюгат Recombinant Protein G (HRP) (Abcam), меченный пероксидазой хрена в разведении 1:1000 и инкубировали 60 минут при комнатной температуре с последующей пятикратной отмывкой ФСБ-Т. Для визуализации в лунки с клетками вносили фильтрованный раствор АЭК (0,05% 3-амино-9-этилкарбазола, 0,015 % H2O2, 50 мM CH3COONa, pH 5.5) и инкубировали до 20 минут при комнатной температуре. Тщательно промывали лунки в ФСБ.
Учет реакции проводили с использованием системы визуализации Evos M5000. Значения контрольных показателей учитывали следующим образом: в лунке с клеточным контролем наблюдали полное сохранение клеточного монослоя, в лунках с вирусным контролем наблюдали деструкцию монослоя под влиянием ЦПД вируса, а также окрашивание участков зараженного монослоя клеток хромогенным субстратом визуализирующее местоположение антигена N или S вируса SARS-Cov-2. Анализ результатов проводили индивидуально для каждого планшета. При визуальном учёте титр нейтрализующих антител считали наибольшее разведение сыворотки, в котором признаков ЦПД не обнаруживалось в 2 или более лунках. Результаты выражали в концентрации иммуноглобулина, подавляющей репродукцию вируса на 50% и 99% относительно контролей (IC50 и IC99), представленные в таблице 5.
Таблица 5. Ингибирующая (IC99) и полуингибирующая (IC50) доза иммуноглобулинов
Постановка реакции выявила вируснейтрализующие свойства и позволила оценить полуингибирующую и ингибирующую концентрацию (IC50 и IC99) рекомбинантного антитела, снижающего развитие вирусиндуцированного ЦПД. Антитело DEV_K57 перспективно для разработки диагностических и фармацевтических препаратов, так как проявляет вируснейтрализующую активность как в отношении варианта Delta, так и в отношении варианта Omicron вируса SARS-CoV-2.
Источники патентной и научно-технической информации:
1. Wu C. et al. Structure genomics of SARS-CoV-2 and its Omicron variant: drug design templates for COVID-19 //Acta Pharmacologica Sinica. – 2022. – Т. 43. – №. 12. – С. 3021-3033.
2. Liang J. G. et al. S-Trimer, a COVID-19 subunit vaccine candidate, induces protective immunity in nonhuman primates //Nature communications. – 2021. – Т. 12. – №. 1. – С. 1346.
3. Thakur V., Ratho R. K. OMICRON (B. 1.1. 529): a new SARS-CoV-2 variant of concern mounting worldwide fear //Journal of medical virology. – 2022. – Т. 94. – №. 5. – С. 1821-1824.
4. Hajizadeh F. et al. SARS-COV-2 RBD (Receptor binding domain) mutations and variants (A sectional-analytical study) //Microbial Pathogenesis. – 2022. – Т. 168. – С. 105595.
5. Lyu X. et al. The global landscape of approved antibody therapies //Antibody therapeutics. – 2022. – Т. 5. – №. 4. – С. 233-257.
6. RU2765731C1, МПК C07K 16/10, опубл 2019.04.29 (аналог).
7. Ju B. et al. Human neutralizing antibodies elicited by SARS-CoV-2 infection //Nature. – 2020. – Т. 584. – №. 7819. – С. 115-119.
8. Tiller T. et al. Efficient generation of monoclonal antibodies from single human B cells by single cell RT-PCR and expression vector cloning //Journal of immunological methods. – 2008. – Т. 329. – №. 1-2. – С. 112-124.
9. WO2022036788, РСТ/CN2020/115480, МПК C07K 16/10, опубл. 24.02.2022 (аналог).
10. Han X. et al. A rapid and efficient screening system for neutralizing antibodies and its application for SARS-CoV-2 //Frontiers in Immunology. – 2021. – Т. 12. – С. 653189.
11. RU2800649C2, МПК C07K 16/10, опубл. 25.07.2023 (аналог).
12. CN116284362A, C07K 16/10, опуб 23.06.2023(прототип).
Приложение
Последовательности, представленные в соответствии со стандартом ST26 ВОИС
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Приложение_Рекомб
моноклональное антитело DEV_K57.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-10-25">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>1234567</ApplicationNumberText>
<FilingDate>2023-10-19</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>123456</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>12345678</ApplicationNumberText>
<FilingDate>2023-10-25</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Государственный научный центр вирусологии и биотехнологии
«Вектор» Федеральной службы по надзору в сфере защиты прав
потребителей и благополучия человека (ФБУН ГНЦ ВБ «Вектор»
Роспотребнадзора)</ApplicantName>
<ApplicantNameLatin>Federalnoe byudzhetnoe uchrezhdenie nauki
"Gosudarstvennyj nauchnyj tsentr virusologii i biotekhnologii
"Vektor" Federalnoj sluzhby po nadzoru v sfere zashchity
prav potrebitelej i blagopoluchiya cheloveka (FBUN GNTS VB
"Vektor" Rospotrebnadzora) (RU)</ApplicantNameLatin>
<InventionTitle languageCode="ru">Универсальный интеграционный
вектор SB7G_HIGH и рекомбинантная плазмида SB7G_HIGH_DEV_57,
обеспечивающая синтез и секрецию рекомбинантного человеческого
антитела к рецептор-связывающему домену (RBD) короновируса SARS-CoV-2
в клетках млекопитающих и рекомбинантное моноклональное антитело
DEV_K57, обладающее вируснейтрализующей активностью в отношении
SARS-CoV-2 </InventionTitle>
<SequenceTotalQuantity>6</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>9906</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9906</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgcaggctatacagttgaagtcggaagtttacatacacttaagttgg
agtcattaaaactcgtttttcaactactccacaaatttcttgttaacaaacaatagttttggcaagtcag
ttaggacatctactttgtgcatgacacaagtcatttttccaacaattgtttacagacagattatttcact
tataattcactgtatcacaattccagtgggtcagaagtttacatacactaagttcgactcctctgcagaa
tgcggcgatgtttcggtaaggggtccgcggccgcaaaacatacgctctccatcaaaacaaaacgaaacaa
aacaaactagcaaaataggctgtccccagtgcaagtgcaggtgccagaacatttctctattcgataataa
aatatctttattttcattacatctgtgtgttggttttttgtgtgctaataatcgattatcacgggcccgg
tgctttgctctgagccagccctccagtttggaatgactcctttttatgacttgaattttcaagtataaag
tctagtgctaaatttaatttgaacaactgtatagtttttgctggttgggggaaggaaaaaaaatggtggc
agtgtttttttcagaattagaagtgaaatgaaaattgttgtgtgtgaggatttctaatgacatgtggtgg
ttgcatactgagtgaagccggtgagcattctgccatgtcaccccctcgtgctcagtaatgtactttacag
aaatcctaaactcaaaagattgatataaaccatgcttcttgtgtatatccggtctcttctctgggtagtc
tcactcagcctgcatttctgccactcgacattgattattgactagttattaatagtaatcaattacgggg
tcattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgac
cgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggacttt
ccattgacgtcaatgggtggactatttacggtaaactgcccacttggcagtacatcaagtgtatcatatg
ccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgacct
tatgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatgggtcgaggtgagcc
ccacgttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttatttttta
attattttgtgcagcgatgggggcggggggggggggggcgcgcgccaggcggggcggggcggggcgaggg
gcggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccgaaagtttcctttt
atggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcggcgggcgggagtcgctgcgttgc
cttcgccccgtgccccgctccgcgccgcctcgcgccgcccgccccggctctgactgaccgcgttactccc
acaggtgagcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctcgtt
tcttttctgtggctgcgtgaaagccttaaagggctccgggagggccctttgtgcgggggggagcggctcg
gggggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggcccgcgctgcccggcggctgtgagcg
ctgcgggcgcggcgcggggctttgtgcgctccgcgtgtgcgcgaggggagcgcggccgggggcggtgccc
cgcggtgcgggggggctgcgaggggaacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcaggg
ggtgtgggcgcggcggtcgggctgtaacccccccctgcacccccctccccgagttgctgagcacggcccg
gcttcgggtgcggggctccgtgcggggcgtggcgcggggctcgccgtgccgggcggggggtggcggcagg
tgggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggaggggcgcggcggccccgg
agcgccggcggctgtcgaggcgcggcgagccgcagccattgccttttatggtaatcgtgcgagagggcgc
agggacttcctttgtcccaaatctggcggagccgaaatctgggaggcgccgccgcaccccctctagcggg
cgcgggcgaagcggtgcggcgccggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccg
ccgtccccttctccatctccagcctcggggctgccgcagggggacggctgccttcgggggggacggggca
gggcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcatgccttctt
ctttttcctacagctcctggaattcgttcaattacagctcgagtgatcataatcagccataccacatttg
tagaggttttacttgctttaaaaaacctcccacacctccccctgaacctgaaacataaaatgaatgcaat
tgttgttgttaacttgtttattgcagcttataatggttacaaataaagcaatagcatcacaaatttcaca
aataaagcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgtatcttatcatgtct
ggacagatcttcaatattggccattagccatattattcattggttatatagcataaatcaatattggcta
ttggccattgcatacgttgtatctatatcataatatgtacatttatattggctcatgtccaatatgaccg
ccatgttggcattgattattgactagttattaatagtaatcaattacggggtcattagttcatagcccat
atatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaacgacccccgccc
attgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccattgacgtcaatgggtg
gagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgccaagtccgccccctattg
acgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgaccttacgggactttcctacttg
gcagtacatctacgtattagtcatcgctattaccatggtgatgcggttttggcagtacaccaatgggcgt
ggatagcggtttgactcacggggatttccaagtctccaccccattgacgtcaatgggagtttgttttggc
accaaaatcaacgggactttccaaaatgtcgtaataaccccgccccgttgacgcaaatgggcggtaggcg
tgtacggtgggaggtctatataagcagagctcgtttagtgaaccgtcagatcactagaagctttattgcg
gtagtttatcacagttaaattgctaacgcagtcagtgcttctgacacaacagtctcgaacttaagctgca
gaagttggtcgtgaggcactgggcaggtaagtatcaaggttacaagacaggtttaaggagaccaatagaa
actgggcttgtcgagacagagaagactcttgcgtttctgataggcacctattggtcttactgacatccac
tttgcctttctctccacaggtgtccactcccagttcaattacagctcttaaggctagagtacttaatacg
actcactataggctagcgttcaattacaaacgcgttagcccctctccctcccccccccctaacgttactg
gccgaagccgcttggaataaggccggtgtgcgtttgtctatatgttattttccaccatattgccgtcttt
tggcaatgtgagggcccggaaacctggccctgtcttcttgacgagcattcctaggggtctttcccctctc
gccaaaggaatgcaaggtctgttgaatgtcgtgaaggaagcagttcctctggaagcttcttgaagacaaa
caacgtctgtagcgaccctttgcaggcagcggaaccccccacctggcgacaggtgcctctgcggccaaaa
gccacgtgtataagatacacctgcaaaggcggcacaaccccagtgccacgttgtgagttggatagttgtg
gaaagagtcaaatggctctcctcaagcgtattcaacaaggggctgaaggatgcccagaaggtaccccatt
gtatgggatctgatctggggcctcggtacacatgctttacatgtgtttagtcgaggttaaaaaaacgtct
aggccccccgaaccacggggacgtggttttcctttgaaaaacacgatgataatatggccacaaccatggg
cgtgagcaagggcgaggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaac
ggccacaagttcagcgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagttca
tctgcaccaccggcaagctgcccgtgccctggcccaccctcgtgaccaccctgacctacggcgtgcagtg
cttcagccgctaccccgaccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtc
caggagcgcaccatcttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcg
acaccctggtgaaccgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaa
gctggagtacaactacaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtg
aacttcaagatccgccacaacatcgaggacggcagcgtgcagctcgccgaccactaccagcagaacaccc
ccatcggcgacggccccgtgctgctgcccgacaaccactacctgagcacccagtccgccctgagcaaaga
ccccaacgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcactctcggcatg
gacgagctgtacaagtctggaggcgccacgaacttctctctgttaaagcaagcaggagacgtggaagaaa
accctggtcccacaaccatgggcaccgagtacaagcctaccgtgcgcctggccactagagatgatgtgcc
ccgcgccgtcaggactctggccgccgctttcgccgactaccccgctacccggcacaccgtggaccccgac
cggcacatcgagcgggtgacagagttgcaggagctcttcctgacccgcgtcgggctggacatcggcaagg
tgtgggtagccgacgacggcgctgccgtggccgtgtggactacccccgagagcgttgaggccggcgccgt
gttcgccgagatcggccccagaatggccgagctgagcggcagccgcctggccgcccagcagcaaatggag
ggcctgcttgccccccatcggcccaaggagcctgcctggtttctggccactgttggagtgagccccgacc
accagggcaagggcttgggcagcgccgtcgtgttgcccggcgtagaggccgccgaacgcgccggtgtgcc
cgcctttctcgaaacaagcgcaccaagaaaccttccattctacgagcgcctgggcttcaccgtgaccgcc
gatgtcgaggtgcccgagggacctaggacctggtgtatgacacggaaacctggcgcctgagatccgcgta
ccggttagtaatgatcgacaatcaacctctggattacaaaatttgtgaaagattgactggtattcttaac
tatgttgctccttttacgctatgtggatacgctgctttaatgcctttgtatcatgctattgcttcccgta
tggctttcattttctcctccttgtataaatcctggttgctgtctctttatgaggagttgtggcccgttgt
caggcaacgtggcgtggtgtgcactgtgtttgctgacgcaacccccactggttggggcattgccaccacc
tgtcagctcctttccgggactttcgctttccccctccctattgccacggcggaactcatcgccgcctgcc
ttgcccgctgctggacaggggctcggctgttgggcactgacaattccgtggtgttgtcggggaagctgac
gtcctttccatggctgctcgcctgtgttgccacctggattctgcgcgggacgtccttctgctacgtccct
tcggccctcaatccagcggaccttccttcccgcggcctgctgccggctctgcggcctcttccgcgtcttc
gccttcgccctcagacgagtcggatctccctttgggccgcctccccgcctggcgatggatctaagtgagt
aggtgagtaagtaggccctgtgccttctagttgccagccatctgttgtttgcccctcccccgtgccttcc
ttgaccctggaaggtgccactcccactgtcctttcctaataaaatgaggaaattgcatcgcattgtctga
gtaggtgtcattctattctggggggtggggtggggcaggacagcaagggggaggattgggaagacaatag
caggcatgctggggatgcggtgggctctatggttcctcaggtaccaagcatggggtaaagtactgttctc
atcacatcatatcaaggttatataccatcaatattgccacagatgttacttagccttttaatatttctct
aatttagtgtatatgcaatgatagttctctgatttctgagattgagtttctcatgtgtaatgattattta
gagtttctctttcatctgttcaaatttttgtctagttttattttttactgatttgtaagacttcttttta
taatctgcatattacaattctctttactggggtgttgcaaatattttctgtcattctatggcctgacttt
tcttaatggttttttaattttaaaaataagtcttaatattcatgcaatctaattaacaatcttttctttg
tggttaggactttgagtcataagaaatttttctctacactgaagtcatgatggcatgcttctatattatt
ttctaaaagatttaaagttttgccttctccatttagacttataattcactggaatttttttgtgtgtatg
gtatgacatatgggttcccttttattttttacatataaatatatttccctgtttttctaaaaaagaaaaa
gatcatcattttcccattgtaaaatgccatatttttttcataggtcacttacatatatcaatgggtctgt
ttctgagctctactctattttatcagcctcactgtctatccccacacatctcatgctttgctctaaatct
tgatatttagtggaacattctttcccattttgttctacaagaatatttttgttattgtcttttgggcttc
tatatacattttagaatgaggttggcaagttcgaatactcgggaccccttaccgaaacatcgccgcattc
tgcagaggagtcgagtgtatgtaaacttctgacccactgggaatgtgatgaaagaaataaaagctgaaat
gaatcattctctctactattattctgatatttcacattcttaaaataaagtggtgatcctaactgaccta
agacagggaatttttactaggattaaatgtcaggaattgtgaaaaagtgagtttaaatgtatttggctaa
ggtgtatgtaaacttccgacttcaactgtatagcagctgcattaatgaatcggccaacgcgcggggagag
gcggtttgcgtattgggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcg
gcgagcggtatcagctcactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaag
aacatgtgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccata
ggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggact
ataaagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttacc
ggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctca
gttcggtgtaggtcgttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgc
cttatccggtaactatcgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccact
ggtaacaggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacg
gctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttgg
tagctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacg
cgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaa
actcacgttaagggattttggtcatgagattatcaaaaaggatcttcacctagatccttttaaattaaaa
atgaagttttaaatcaatctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagt
gaggcacctatctcagcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataa
ctacgatacgggagggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctcaccggc
tccagatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggtcctgcaactttatcc
gcctccatccagtctattaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgca
acgttgttgccattgctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattcagctccgg
ttcccaacgatcaaggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcct
ccgatcgttgtcagaagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctc
ttactgtcatgccatccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaata
gtgtatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcgccacatagcagaact
ttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagat
ccagttcgatgtaacccactcgtgcacccaactgatcttcagcatcttttactttcaccagcgtttctgg
gtgagcaaaaacaggaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactc
atactcttcctttttcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatttg
aatgtatttagaaaaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctgacgtcta
agaaaccattattatcatgacattaacctataaaaataggcgtatcacgaggccgcc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>404</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..404</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgaggctccctgctcagctcctggggctgctaatgctctgggtctctg
gatccagtggggatattgtgatgactcagtctccactctccctgcccgtcacccctggagagccggcctc
catctcctgcacgtctagtcagagcctcctgcatagtaatggaaacaattattggattggtacctgcaga
agccagggcagtctccacagctcctgatctatttggggtctaatcgggcctccggggtccctgagaggtt
cagtggcagtggatcaggcacagattttacactgaaaatcagcagagtggaggctgaggatgttggggtt
tattactgcatgcaagctctacaaactcctcctagttttggccaggggaccaagctggagatcaaacgaa
ctgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>456</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..456</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggactggacctggaggttcctctttgtggtggcagcagctacaggtg
ttcagtcccaggtgcagctggtgcagtctggggctgaggtgaagaagcctgggtcctcggtgaaggtctc
ctgcaaggcttctggaggcagcttcaacagttatgctattagttgggtgcgacaggcccctggacaaggg
cttgagtgggtgggagggctcatccatgtccttggtatagcaaactacgcacagaagttccagggcagag
tcacgattaccgcggacgagtaccccagtgcagcagcctacctggagctgagcagcctgaggtctgagga
cacggccgtatattattgtgcgagagtgggggggcccctagaaaacaactatggttcccaacaccgcttc
tactactacggtatggacgtctggggccaagggaccaccgtcaccgtctcctcagcc</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>135</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..135</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MRLPAQLLGLLMLWVSGSSGDIVMTQSPLSLPVTPGEPASISCTSSQSL
LHSNGNNYLDWYLQKPGQSPQLLIYLGSNRASGVPERFSGSGSGTDFTLKISRVEAEDVGVYYCMQALQT
PPSFGQGTKLEIKRTV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>152</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..152</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MDWTWRFLFVVAAATGVQSQVQLVQSGAEVKKPGSSVKVSCKASGGSFN
SYAISWVRQAPGQGLEWVGGLIHVLGIANYAQKFQGRVTITADEYPSAAAYLELSSLRSEDTAVYYCARV
GGPLENNYGSQHRFYYYGMDVWGQGTTVTVSSA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>12062</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12062</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgcaggctatacagttgaagtcggaagtttacatacacttaagttgg
agtcattaaaactcgtttttcaactactccacaaatttcttgttaacaaacaatagttttggcaagtcag
ttaggacatctactttgtgcatgacacaagtcatttttccaacaattgtttacagacagattatttcact
tataattcactgtatcacaattccagtgggtcagaagtttacatacactaagttcgactcctctgcagaa
tgcggcgatgtttcggtaaggggtccgcggccgcaaaacatacgctctccatcaaaacaaaacgaaacaa
aacaaactagcaaaataggctgtccccagtgcaagtgcaggtgccagaacatttctctattcgataataa
aatatctttattttcattacatctgtgtgttggttttttgtgtgctaataatcgattatcacgggcccgg
tgctttgctctgagccagccctccagtttggaatgactcctttttatgacttgaattttcaagtataaag
tctagtgctaaatttaatttgaacaactgtatagtttttgctggttgggggaaggaaaaaaaatggtggc
agtgtttttttcagaattagaagtgaaatgaaaattgttgtgtgtgaggatttctaatgacatgtggtgg
ttgcatactgagtgaagccggtgagcattctgccatgtcaccccctcgtgctcagtaatgtactttacag
aaatcctaaactcaaaagattgatataaaccatgcttcttgtgtatatccggtctcttctctgggtagtc
tcactcagcctgcatttctgccactcgacattgattattgactagttattaatagtaatcaattacgggg
tcattagttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgac
cgcccaacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggacttt
ccattgacgtcaatgggtggactatttacggtaaactgcccacttggcagtacatcaagtgtatcatatg
ccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgacct
tatgggactttcctacttggcagtacatctacgtattagtcatcgctattaccatgggtcgaggtgagcc
ccacgttctgcttcactctccccatctcccccccctccccacccccaattttgtatttatttatttttta
attattttgtgcagcgatgggggcggggggggggggggcgcgcgccaggcggggcggggcggggcgaggg
gcggggcggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccgaaagtttcctttt
atggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcggcgggcgggagtcgctgcgttgc
cttcgccccgtgccccgctccgcgccgcctcgcgccgcccgccccggctctgactgaccgcgttactccc
acaggtgagcgggcgggacggcccttctcctccgggctgtaattagcgcttggtttaatgacggctcgtt
tcttttctgtggctgcgtgaaagccttaaagggctccgggagggccctttgtgcgggggggagcggctcg
gggggtgcgtgcgtgtgtgtgtgcgtggggagcgccgcgtgcggcccgcgctgcccggcggctgtgagcg
ctgcgggcgcggcgcggggctttgtgcgctccgcgtgtgcgcgaggggagcgcggccgggggcggtgccc
cgcggtgcgggggggctgcgaggggaacaaaggctgcgtgcggggtgtgtgcgtgggggggtgagcaggg
ggtgtgggcgcggcggtcgggctgtaacccccccctgcacccccctccccgagttgctgagcacggcccg
gcttcgggtgcggggctccgtgcggggcgtggcgcggggctcgccgtgccgggcggggggtggcggcagg
tgggggtgccgggcggggcggggccgcctcgggccggggagggctcgggggaggggcgcggcggccccgg
agcgccggcggctgtcgaggcgcggcgagccgcagccattgccttttatggtaatcgtgcgagagggcgc
agggacttcctttgtcccaaatctggcggagccgaaatctgggaggcgccgccgcaccccctctagcggg
cgcgggcgaagcggtgcggcgccggcaggaaggaaatgggcggggagggccttcgtgcgtcgccgcgccg
ccgtccccttctccatctccagcctcggggctgccgcagggggacggctgccttcgggggggacggggca
gggcggggttcggcttctggcgtgtgaccggcggctctagagcctctgctaaccatgttcatgccttctt
ctttttcctacagctcctggaattcaccatgaggctccctgctcagctcctggggctgctaatgctctgg
gtctctggatccagtggggatattgtgatgactcagtctccactctccctgcccgtcacccctggagagc
cggcctccatctcctgcacgtctagtcagagcctcctgcatagtaatggaaacaattatttggattggta
cctgcagaagccagggcagtctccacagctcctgatctatttggggtctaatcgggcctccggggtccct
gagaggttcagtggcagtggatcaggcacagattttacactgaaaatcagcagagtggaggctgaggatg
ttggggtttattactgcatgcaagctctacaaactcctcctagttttggccaggggaccaagctggagat
caaacgaactgtggctgcaccatctgtcttcatcttcccaccatctgacgagcagttgaaatctggaact
gcctctgttgtgtgcctgctgaataacttctaccctcgggaagccaaggtgcagtggaaggtggacaatg
ccctgcagtccggcaactcccaagagtctgtgaccgagcaggactccaaggacagcacctacagcctgtc
ctccacactgaccctgtccaaggccgactacgagaagcacaaggtgtacgcctgcgaagtgacccatcag
ggcctgtctagccctgtgaccaagtctttcaaccggggcgagtgctgatagctcgagtgatcataatcag
ccataccacatttgtagaggttttacttgctttaaaaaacctcccacacctccccctgaacctgaaacat
aaaatgaatgcaattgttgttgttaacttgtttattgcagcttataatggttacaaataaagcaatagca
tcacaaatttcacaaataaagcatttttttcactgcattctagttgtggtttgtccaaactcatcaatgt
atcttatcatgtctggacagatcttcaatattggccattagccatattattcattggttatatagcataa
atcaatattggctattggccattgcatacgttgtatctatatcataatatgtacatttatattggctcat
gtccaatatgaccgccatgttggcattgattattgactagttattaatagtaatcaattacggggtcatt
agttcatagcccatatatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgccc
aacgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggactttccatt
gacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatcaagtgtatcatatgccaag
tccgccccctattgacgtcaatgacggtaaatggcccgcctggcattatgcccagtacatgaccttacgg
gactttcctacttggcagtacatctacgtattagtcatcgctattaccatggtgatgcggttttggcagt
acaccaatgggcgtggatagcggtttgactcacggggatttccaagtctccaccccattgacgtcaatgg
gagtttgttttggcaccaaaatcaacgggactttccaaaatgtcgtaataaccccgccccgttgacgcaa
atgggcggtaggcgtgtacggtgggaggtctatataagcagagctcgtttagtgaaccgtcagatcacta
gaagctttattgcggtagtttatcacagttaaattgctaacgcagtcagtgcttctgacacaacagtctc
gaacttaagctgcagaagttggtcgtgaggcactgggcaggtaagtatcaaggttacaagacaggtttaa
ggagaccaatagaaactgggcttgtcgagacagagaagactcttgcgtttctgataggcacctattggtc
ttactgacatccactttgcctttctctccacaggtgtccactcccagttcaattacagctcttaaggcta
gagtacttaatacgactcactataggctagcaccatggactggacctggaggttcctctttgtggtggca
gcagctacaggtgttcagtcccaggtgcagctggtgcagtctggggctgaggtgaagaagcctgggtcct
cggtgaaggtctcctgcaaggcttctggaggcagcttcaacagttatgctattagttgggtgcgacaggc
ccctggacaagggcttgagtgggtgggagggctcatccatgtccttggtatagcaaactacgcacagaag
ttccagggcagagtcacgattaccgcggacgagtaccccagtgcagcagcctacctggagctgagcagcc
tgaggtctgaggacacggccgtatattattgtgcgagagtgggggggcccctagaaaacaactatggttc
ccaacaccgcttctactactacggtatggacgtctggggccaagggaccaccgtcaccgtctcctcagcc
tccaccaagggcccatccgtcttccccctggctccatcctctaagtctacctctggcggcacagctgctc
tgggctgtctggtcaaggactacttccctgagcctgtgaccgtgtcctggaactctggcgctctgacatc
tggcgtgcacacctttccagctgtgctgcagtcctccggcctgtactctctgtcctctgtcgtgaccgtg
ccttccagctctctgggaacccagacctacatctgcaatgtgaaccacaagccttccaacaccaaggtgg
acaagaaggtggaacccaagtcctgcgacaagacccacacctgtcctccatgtcctgctccagaactgct
cggcggaccttccgtgttcctgtttcctccaaagcctaaggacaccctgatgatctctcggacccctgaa
gtgacctgcgtggtggtggatgtgtctcacgaggatcccgaagtgaagttcaattggtacgtggacggcg
tggaagtgcacaacgccaagaccaagcctagagaggaacagtacaactccacctacagagtggtgtccgt
gctgaccgtgctgcaccaggattggctgaacggcaaagagtacaagtgcaaggtgtccaacaaggccctg
cctgctcctatcgaaaagaccatctccaaggccaagggccagcctagggaaccccaggtttacaccttgc
ctccatctcgggacgagctgaccaagaaccaggtgtccctgacctgtctcgtgaagggcttctacccctc
cgatatcgccgtggaatgggagtctaatggccagcctgagaacaactacaagacaacccctcctgtgctg
gactccgacggctcattcttcctgtactccaagctgacagtggacaagtccagatggcagcagggcaacg
tgttctcctgctccgtgatgcacgaggccctgcacaatcactacacccagaagtccctgtctctgtcccc
tggcaagtgatgataacgcgttagcccctctccctcccccccccctaacgttactggccgaagccgcttg
gaataaggccggtgtgcgtttgtctatatgttattttccaccatattgccgtcttttggcaatgtgaggg
cccggaaacctggccctgtcttcttgacgagcattcctaggggtctttcccctctcgccaaaggaatgca
aggtctgttgaatgtcgtgaaggaagcagttcctctggaagcttcttgaagacaaacaacgtctgtagcg
accctttgcaggcagcggaaccccccacctggcgacaggtgcctctgcggccaaaagccacgtgtataag
atacacctgcaaaggcggcacaaccccagtgccacgttgtgagttggatagttgtggaaagagtcaaatg
gctctcctcaagcgtattcaacaaggggctgaaggatgcccagaaggtaccccattgtatgggatctgat
ctggggcctcggtacacatgctttacatgtgtttagtcgaggttaaaaaaacgtctaggccccccgaacc
acggggacgtggttttcctttgaaaaacacgatgataatatggccacaaccatgggcgtgagcaagggcg
aggagctgttcaccggggtggtgcccatcctggtcgagctggacggcgacgtaaacggccacaagttcag
cgtgtccggcgagggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggc
aagctgcccgtgccctggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttcagccgctacc
ccgaccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggctacgtccaggagcgcaccat
cttcttcaaggacgacggcaactacaagacccgcgccgaggtgaagttcgagggcgacaccctggtgaac
cgcatcgagctgaagggcatcgacttcaaggaggacggcaacatcctggggcacaagctggagtacaact
acaacagccacaacgtctatatcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatccg
ccacaacatcgaggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgacggc
cccgtgctgctgcccgacaaccactacctgagcacccagtccgccctgagcaaagaccccaacgagaagc
gcgatcacatggtcctgctggagttcgtgaccgccgccgggatcactctcggcatggacgagctgtacaa
gtctggaggcgccacgaacttctctctgttaaagcaagcaggagacgtggaagaaaaccctggtcccaca
accatgggcaccgagtacaagcctaccgtgcgcctggccactagagatgatgtgccccgcgccgtcagga
ctctggccgccgctttcgccgactaccccgctacccggcacaccgtggaccccgaccggcacatcgagcg
ggtgacagagttgcaggagctcttcctgacccgcgtcgggctggacatcggcaaggtgtgggtagccgac
gacggcgctgccgtggccgtgtggactacccccgagagcgttgaggccggcgccgtgttcgccgagatcg
gccccagaatggccgagctgagcggcagccgcctggccgcccagcagcaaatggagggcctgcttgcccc
ccatcggcccaaggagcctgcctggtttctggccactgttggagtgagccccgaccaccagggcaagggc
ttgggcagcgccgtcgtgttgcccggcgtagaggccgccgaacgcgccggtgtgcccgcctttctcgaaa
caagcgcaccaagaaaccttccattctacgagcgcctgggcttcaccgtgaccgccgatgtcgaggtgcc
cgagggacctaggacctggtgtatgacacggaaacctggcgcctgagatccgcgtaccggttagtaatga
tcgacaatcaacctctggattacaaaatttgtgaaagattgactggtattcttaactatgttgctccttt
tacgctatgtggatacgctgctttaatgcctttgtatcatgctattgcttcccgtatggctttcattttc
tcctccttgtataaatcctggttgctgtctctttatgaggagttgtggcccgttgtcaggcaacgtggcg
tggtgtgcactgtgtttgctgacgcaacccccactggttggggcattgccaccacctgtcagctcctttc
cgggactttcgctttccccctccctattgccacggcggaactcatcgccgcctgccttgcccgctgctgg
acaggggctcggctgttgggcactgacaattccgtggtgttgtcggggaagctgacgtcctttccatggc
tgctcgcctgtgttgccacctggattctgcgcgggacgtccttctgctacgtcccttcggccctcaatcc
agcggaccttccttcccgcggcctgctgccggctctgcggcctcttccgcgtcttcgccttcgccctcag
acgagtcggatctccctttgggccgcctccccgcctggcgatggatctaagtgagtaggtgagtaagtag
gccctgtgccttctagttgccagccatctgttgtttgcccctcccccgtgccttccttgaccctggaagg
tgccactcccactgtcctttcctaataaaatgaggaaattgcatcgcattgtctgagtaggtgtcattct
attctggggggtggggtggggcaggacagcaagggggaggattgggaagacaatagcaggcatgctgggg
atgcggtgggctctatggttcctcaggtaccaagcatggggtaaagtactgttctcatcacatcatatca
aggttatataccatcaatattgccacagatgttacttagccttttaatatttctctaatttagtgtatat
gcaatgatagttctctgatttctgagattgagtttctcatgtgtaatgattatttagagtttctctttca
tctgttcaaatttttgtctagttttattttttactgatttgtaagacttctttttataatctgcatatta
caattctctttactggggtgttgcaaatattttctgtcattctatggcctgacttttcttaatggttttt
taattttaaaaataagtcttaatattcatgcaatctaattaacaatcttttctttgtggttaggactttg
agtcataagaaatttttctctacactgaagtcatgatggcatgcttctatattattttctaaaagattta
aagttttgccttctccatttagacttataattcactggaatttttttgtgtgtatggtatgacatatggg
ttcccttttattttttacatataaatatatttccctgtttttctaaaaaagaaaaagatcatcattttcc
cattgtaaaatgccatatttttttcataggtcacttacatatatcaatgggtctgtttctgagctctact
ctattttatcagcctcactgtctatccccacacatctcatgctttgctctaaatcttgatatttagtgga
acattctttcccattttgttctacaagaatatttttgttattgtcttttgggcttctatatacattttag
aatgaggttggcaagttcgaatactcgggaccccttaccgaaacatcgccgcattctgcagaggagtcga
gtgtatgtaaacttctgacccactgggaatgtgatgaaagaaataaaagctgaaatgaatcattctctct
actattattctgatatttcacattcttaaaataaagtggtgatcctaactgacctaagacagggaatttt
tactaggattaaatgtcaggaattgtgaaaaagtgagtttaaatgtatttggctaaggtgtatgtaaact
tccgacttcaactgtatagcagctgcattaatgaatcggccaacgcgcggggagaggcggtttgcgtatt
gggcgctcttccgcttcctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcag
ctcactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaa
aggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccct
gacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactataaagataccagg
cgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgccgcttaccggatacctgtccgc
ctttctcccttcgggaagcgtggcgctttctcatagctcacgctgtaggtatctcagttcggtgtaggtc
gttcgctccaagctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaact
atcgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaacaggattag
cagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggctacactagaagg
acagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttggtagctcttgatccg
gcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagcagattacgcgcagaaaaaaagg
atctcaagaagatcctttgatcttttctacggggtctgacgctcagtggaacgaaaactcacgttaaggg
attttggtcatgagattatcaaaaaggatcttcacctagatccttttaaattaaaaatgaagttttaaat
caatctaaagtatatatgagtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctc
agcgatctgtctatttcgttcatccatagttgcctgactccccgtcgtgtagataactacgatacgggag
ggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctcaccggctccagatttatcag
caataaaccagccagccggaagggccgagcgcagaagtggtcctgcaactttatccgcctccatccagtc
tattaattgttgccgggaagctagagtaagtagttcgccagttaatagtttgcgcaacgttgttgccatt
gctacaggcatcgtggtgtcacgctcgtcgtttggtatggcttcattcagctccggttcccaacgatcaa
ggcgagttacatgatcccccatgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcag
aagtaagttggccgcagtgttatcactcatggttatggcagcactgcataattctcttactgtcatgcca
tccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaatagtgtatgcggcgac
cgagttgctcttgcccggcgtcaatacgggataataccgcgccacatagcagaactttaaaagtgctcat
cattggaaaacgttcttcggggcgaaaactctcaaggatcttaccgctgttgagatccagttcgatgtaa
cccactcgtgcacccaactgatcttcagcatcttttactttcaccagcgtttctgggtgagcaaaaacag
gaaggcaaaatgccgcaaaaaagggaataagggcgacacggaaatgttgaatactcatactcttcctttt
tcaatattattgaagcatttatcagggttattgtctcatgagcggatacatatttgaatgtatttagaaa
aataaacaaataggggttccgcgcacatttccccgaaaagtgccacctgacgtctaagaaaccattatta
tcatgacattaacctataaaaataggcgtatcacgaggccgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Широко нейтрализующее антитело против SARS-CoV-2 | 2022 |
|
RU2810476C1 |
| Искусственный ген, кодирующий бицистронную структуру, образованную последовательностями рецептор-связующего домена гликопротеина S коронавируса SARS-CoV-2, P2A-пептида и гликопротеина G VSV, рекомбинантная плазмида pStem-rVSV-Stbl_RBD_SC2, обеспечивающая экспрессию искусственного гена и рекомбинантный штамм вируса везикулярного стоматита rVSV-Stbl_RBD_SC2, используемого для создания вакцины против коронавируса SARS-CoV-2 | 2020 |
|
RU2733831C1 |
| Искусственный ген Stbl_RBD_TrM_SC2, кодирующий бицистронную структуру, образованную последовательностями рецепторсвязывающего домена гликопротеина S коронавируса SARS-CoV-2, трансмембранного региона, P2A-пептида и гликопротеина G VSV, рекомбинантная плазмида pStem-rVSV-Stbl_RBD_TrM_SC2, обеспечивающая экспрессию искусственного гена, и рекомбинантный штамм вируса везикулярного стоматита rVSV-Stbl_RBD_TrM_SC2, используемый для создания вакцины против коронавируса SARS-CoV-2 | 2020 |
|
RU2733832C1 |
| Искусственный ген EctoS_SC2, кодирующий эктодомен гликопротеина S коронавируса SARS-CoV-2 с C-концевым тримеризующим доменом, рекомбинантная плазмида pStem-rVSV-EctoS_SC2, обеспечивающая экспрессию искусственного гена, и рекомбинантный штамм вируса везикулярного стоматита rVSV-EctoS_SC2, используемый для создания вакцины против коронавируса SARS-CoV-2 | 2020 |
|
RU2733834C1 |
| Плазмидная генетическая конструкция pET21a_SKP, обеспечивающая экспрессию в прокариотической системе E.coli рекомбинантного белка SKP, и рекомбинантный белок SKP, обладающий широконейтрализующими свойствами однодоменного наноантитела против SARS-CoV-2 | 2024 |
|
RU2839376C1 |
| Интегративный плазмидный вектор pVEAL3-RBDdel, обеспечивающий синтез и секрецию рекомбинантного белка рецепторсвязывающего домена RBDdelta коронавируса SARS-CoV-2 в клетках млекопитающих, рекомбинантный штамм клеточной линии CHO-K1- RBDdelta и рекомбинантный белок RBDdelta SARS-CoV-2, продуцируемый штаммом клеточной линии | 2023 |
|
RU2816175C1 |
| Моноклональное антитело к RBD фрагменту в составе S белка вируса SARS-CoV-2 | 2020 |
|
RU2744274C1 |
| Моноклональное антитело iC2 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2023 |
|
RU2817697C1 |
| Моноклональное антитело iC1 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2023 |
|
RU2817696C1 |
| Средство и способ терапии и экстренной профилактики заболеваний, вызываемых вирусом SARS-CoV-2 на основе рекомбинантного антитела и гуманизированного моноклонального антитела | 2021 |
|
RU2769223C1 |
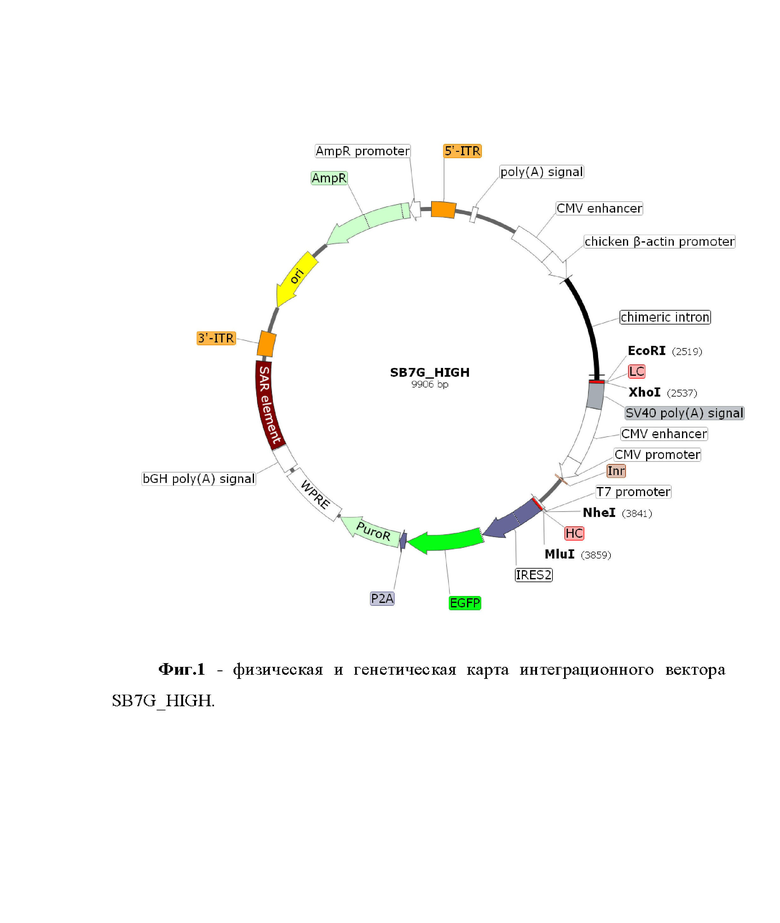
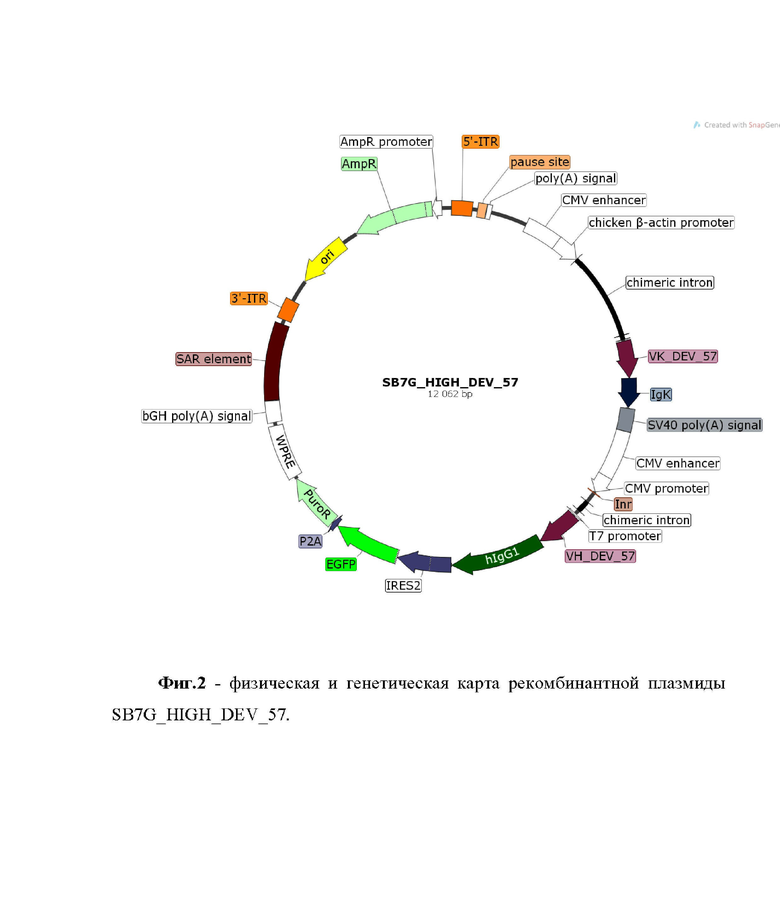
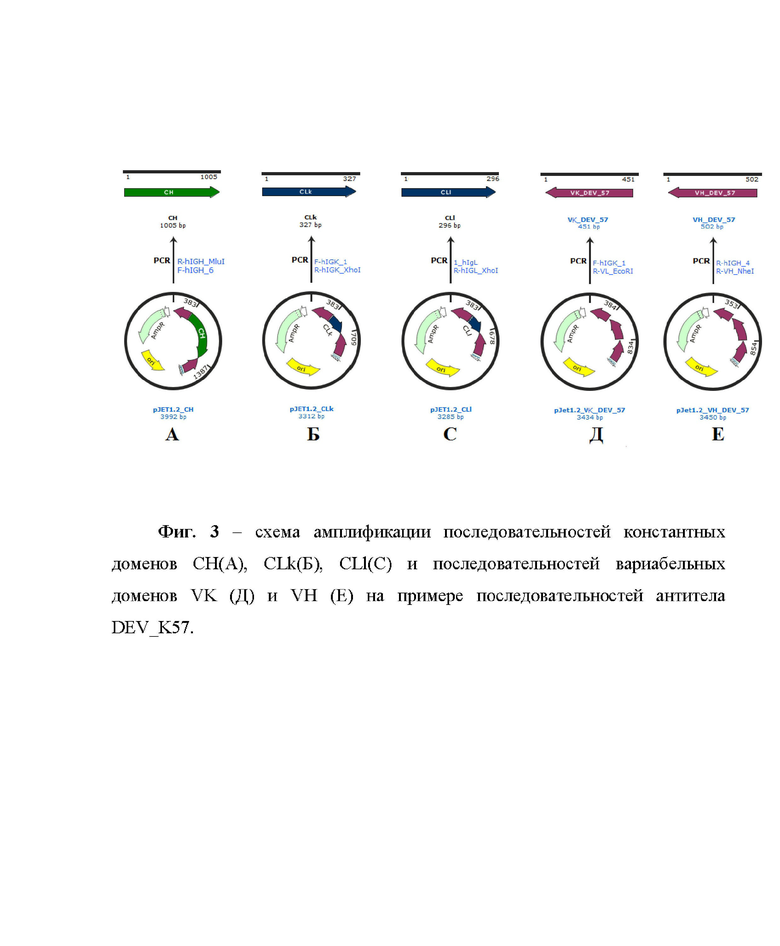
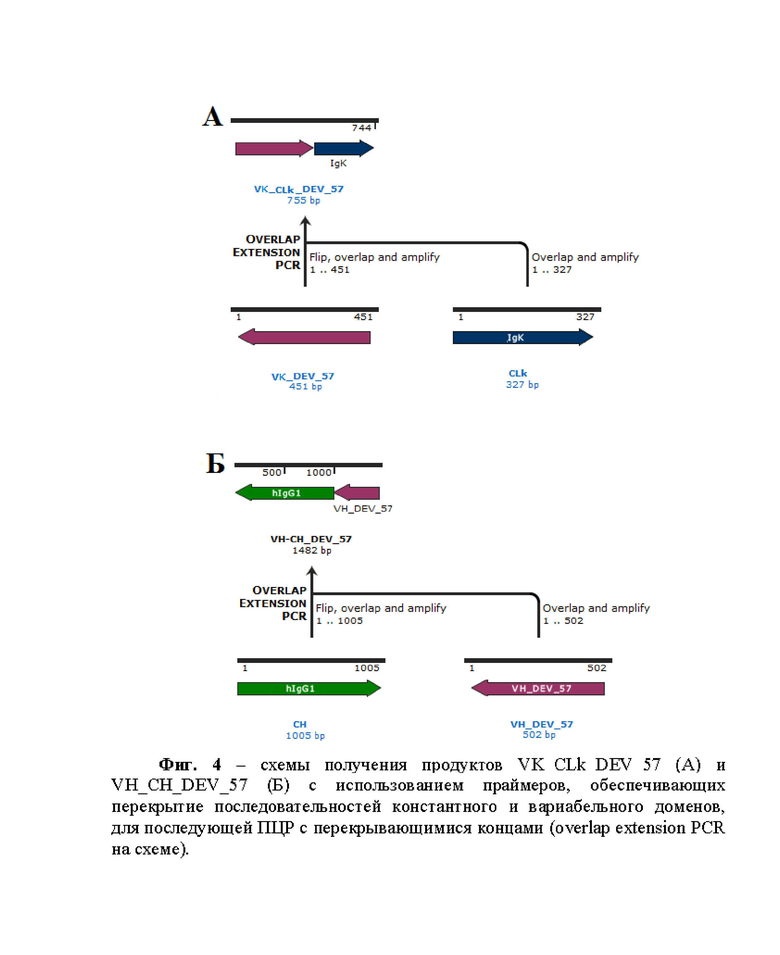
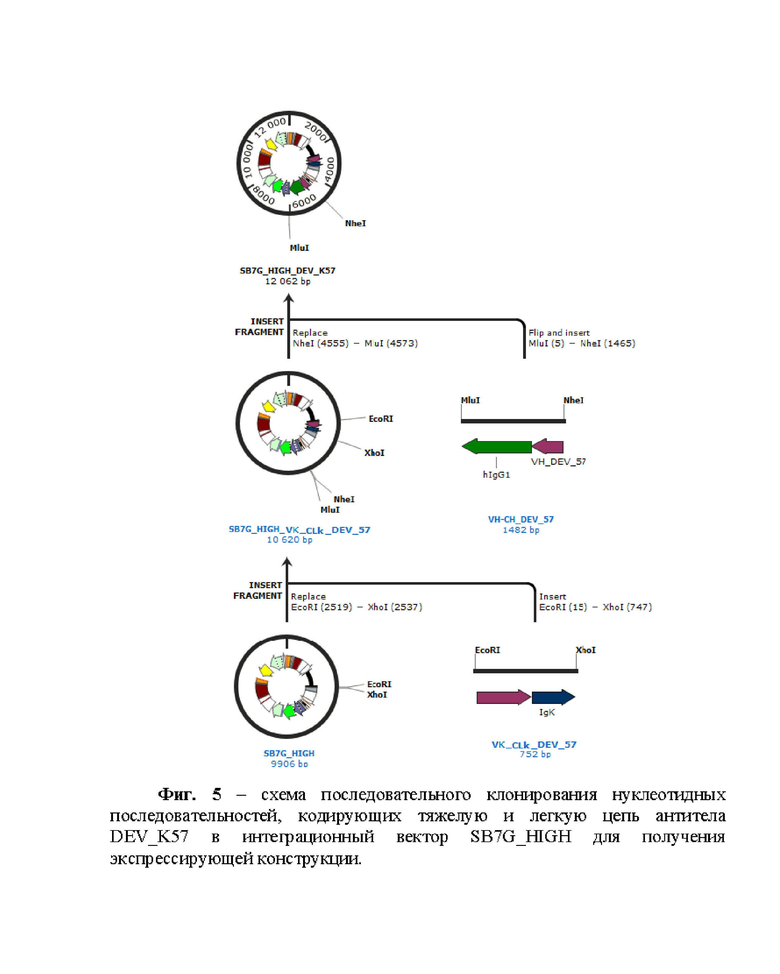
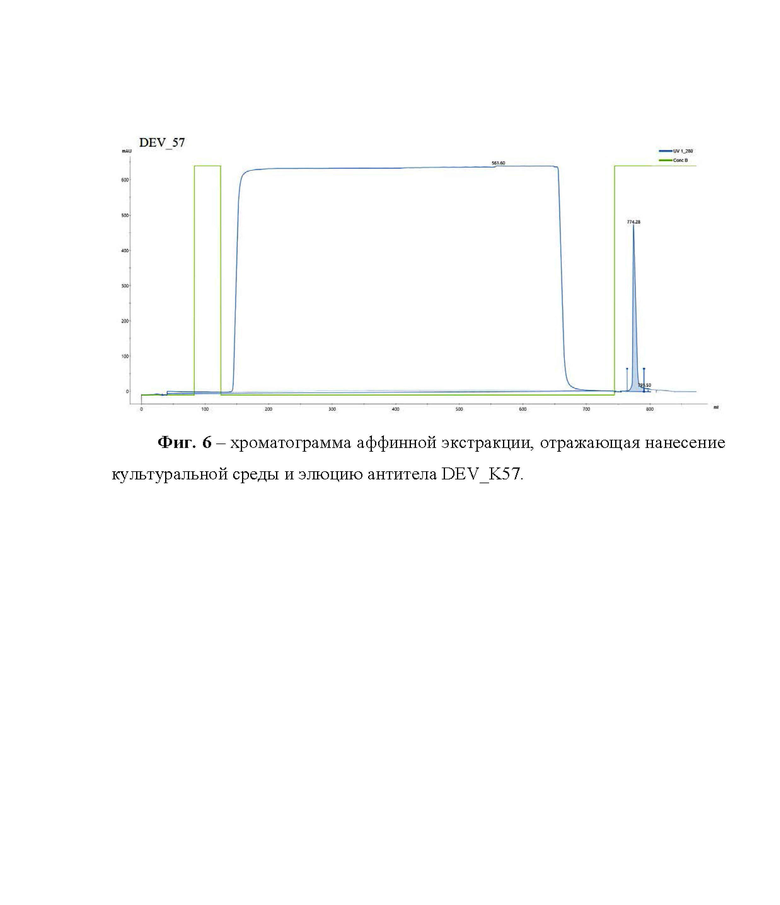
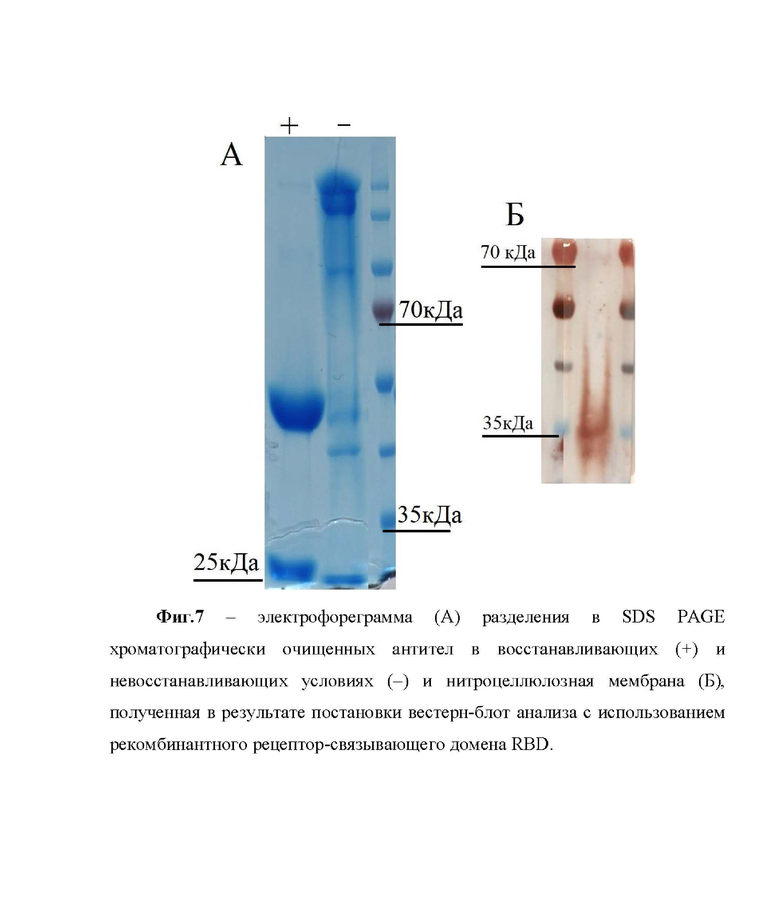










Изобретение относится к области биотехнологии, в частности к универсальному интеграционному вектору SB7G_HIGH для клонирования кДНК, кодирующих вариабельные домены антител, содержащему экспрессионную кассету, фланкированную инвертированными повторами для интеграции транспозазой Sleeping Beauty (SB) в геном эукариотической клетки, а также к рекомбинантной плазмиде SB7G_HIGH_DEV_K57, полученной на его основе. Также раскрыто рекомбинантное моноклональное антитело DEV_K57, обладающее вируснейтрализующей активностью в отношении SARS-CoV-2 вариантов Delta и Omicron. Изобретение эффективно для получения генетических конструкций, которые обеспечивают более эффективный синтез и секрецию рекомбинантных моноклональных антител в эукариотической клеточной линии-продуцента CHO-S, которая характеризуется более эффективной посттрансляционной модификацией и более высоким выходом указанных рекомбинантных антител, обладающих вируснейтрализующей активностью в отношении вируса SARS CoV 2 вариантов Delta и Omicron. 3 н.п. ф-лы, 13 ил., 5 табл., 5 пр.
1. Универсальный интеграционный вектор SB7G_HIGH для клонирования кДНК, кодирующих вариабельные домены антител, содержащий экспрессионную кассету, фланкированную инвертированными повторами для интеграции транспозазой Sleeping Beauty (SB) в геном эукариотической клетки, имеющий размер 9906 п.н, нуклеотидную последовательность SEQ NO: 1 и состоящий в соответствии с физической и генетической картой из следующих элементов:
• 5-ITR и 3'-ITR инвертированные повторы, имеющие координаты с 14 по 238 п.н. и с 7623 по 7847 соответственно, обеспечивают интеграцию участка плазмиды, заключенного между ними, в геном клеток млекопитающих транспозазой Sleeping Beauty (SB);
• CAG промотор - сильный синтетический промотор, состоящий из раннего энхансерного элемента цитомегаловируса (CMV), промотора, первого экзона и первого интрона ген бета-актина курицы, сплайс-акцептор гена бета-глобина кролика и имеющий координаты с 846 по 1503 п.н.;
• LC - область для встраивания гена, кодирующего вариабельный и константный домены легкой цепи каппа/лямбда, содержащая специфические сайты узнавания рестрикции EcoRI и XhoI с координатами 2519 и 2542 п.н. соответственно;
• SV40 poly(A) signal - сигнал полиаденелирования, имеющий координаты с 2543 по 2781 п.н.;
• промотор и энхансер цитомегаловируса человека (CMV), обеспечивающий экспрессию гена в клетках млекопитающих и имеющий координаты с 2784 по 3510 п.н.;
• Inr с Transcription start (+1) - последовательность инициатора транскрипции РНК-полимеразы II, усиливающая активность промотора и имеющая координаты с 3525 по 3531 п.н.;
• Т7 промотер - нуклеотидная последовательность промотора бактериофага Т7, необходимая для in vitro транскрипции и имеющая координаты с 3823 по 3841 п.н.;
• НС - область для встраивания гена, кодирующего вариабельный и константный домены тяжелой цепи, содержащая специфические сайты узнавания рестрикции NheI и MluI с координатами 3842 и 3864 п.н. соответственно;
• IRES2 - внутренний сайт посадки рибосом вируса энцефаломиокардита человека (EMCV), обеспечивающий экспрессию гена устойчивости к пуромицину и имеющий координаты с 3868 по 4454 п.н.;
• EGFP - ген, кодирующий белок с усиленной зеленой флуоресценцией, имеющий координаты с 4455 по 5174 п.н.;
• Р2А - 2А пептид из полипротеина тешовируса-1, обеспечивающий расщепление полипротеина на этапе трансляции и имеющий координаты с 5184 по 5240 п.н.;
• PuroR - ген устойчивости к пуромицину, кодирующий N-ацетил трансферазу и имеющий координаты с 5247 по 5849 п.н.;
• WPRE - посттранскрипционный регуляторный элемент вируса гепатита сурка, необходимый для увеличения уровня экспрессии белка и имеющий координаты с 5879 по 6467 п.н.;
• bGH poly(A) signal - сигнал полиаденелирования, имеющий координаты с 6507 по 6731;
• ORI (origin of replication) - точка начала репликации плазмидного вектора SB7G, имеющая координаты с 8092 по 8680 п.н.;
• AmpR - ген β-лактамазы, используемый в качестве генетического маркера, определяющего устойчивость к ампициллину клеток бактерии Е. Coli и имеющий координаты с 8851 по 9711 п.н.
2. Рекомбинантная плазмида SB7G_HIGH_DEV_K57, полученная на основе универсального интеграционного вектора по п. 1., и имеющая размер 12062 п.н с нуклеотидной последовательностью, описанной SEQ NO: 6, состоящая в соответствии с физической и генетической картой из следующих элементов:
• 5-ITR и 3'-ITR инвертированные повторы, имеющие координаты с 14 по 238 п.н. и с 9779 по 10003 п.н. соответственно, обеспечивают интеграцию участка плазмиды, заключенного между ними, в геном клеток млекопитающих транспозазой Sleeping Beauty (SB);
• CAG промотор - сильный синтетический промотор, состоящий из раннего энхансерного элемента цитомегаловируса (CMV), промотора, первого экзона и первого интрона ген бета-актина курицы, сплайс-акцептор гена бета-глобина кролика, имеющий координаты с 846 по 1503 п.н.;
• VK_DEV_57 - последовательность SEQ ID NO: 2 кДНК, кодирующая вариабельный домен легкой цепи антитела DEV_K57, имеющая координаты с по 2932 п.н.;
• IgK - нуклеотидная кодон-оптимизированная последовательность, кодирующая константный домен легкой цепи каппа CLk, имеющая координаты с 2933 по 3247 п.н.;
• SV40 poly(A) signal - сигнал полиаденелирования, имеющий координаты с 3257 по 3495 п.н.;
• энхансер и промотор цитомегаловируса человека (CMV), обеспечивающие экспрессию гена в клетках млекопитающих и имеющие координаты с 3498 и 4224 п.н.;
• Inr с Transcription start (+1) - последовательность инициатора транскрипции РНК-полимеразы II, усиливающая активность промотора и имеющая координаты с 4239 по 4245 п.н.;
• Т7 промотер - нуклеотидная последовательность промотора бактериофага Т7, необходимая для in vitro транскрипции и имеющая координаты с 4537 по 4555 п.н.;
• VH_DEV_57 - последовательность кДНК, кодирующая вариабельный домен тяжелой цепи антитела DEV_K57 и имеющая координаты с 4564 по 5019 п.н., описанная нуклеотидной последовательностью в SEQ ID NO: 3;
• hlgG1 - нуклеотидная кодон-оптимизированная последовательность, кодирующая константный домен тяжелой цепи и имеющая координаты с 5020 по 6012 п.н.;
• IRES2 - внутренний сайт посадки рибосом вируса энцефаломиокардита человека (EMCV), обеспечивающий экспрессию гена устойчивости к пуромицину и имеющий координаты с 6024 по 6610 п.н.;
• EGFP - ген, кодирующий белок с усиленной зеленой флуоресценцией, имеющий координаты с 6611 по 7330 п.н.;
• Р2А - 2А пептид из полипротеина тешовируса-1, обеспечивающий расщепление полипротеина на этапе трансляции, имеющий координаты с 7340 по 7396 п.н.;
• PuroR - ген устойчивости к пуромицину, кодирующий N-ацетил трансферазу, имеющий координаты с 7403 по 8005 п.н.;
• WPRE - посттранскрипционный регуляторный элемент вируса гепатита сурка, необходимый для увеличения уровня экспрессии белка, имеющий координаты с 8035 по 8623 п.н.;
• bGH poly(A) signal - сигнал полиаденелирования, имеющий координаты с 8663 по 8887 п.н.;
• ORI (origin of replication) - точка начала репликации плазмидного вектора SB7G, имеющая координаты с 10248 по 10836 п.н.;
• AmpR - ген β-лактамазы в качестве генетического маркера, определяющего устойчивость к ампициллину клеток бактерии Е. Coli, имеющий координаты с 11007 по 11867 п.н.
3. Рекомбинантное моноклональное антитело DEV_K57, обладающее вируснейтрализующей активностью в отношении SARS-CoV-2 вариантов Delta и Omicron и содержащее константную область человеческого IgG1 и антигенсвязывающий центр с аминокислотной последовательностью вариабельной области легкой κ-цепи SEQ ID NO: 4 и тяжелой цепи SEQ ID NO: 5.
| CN 116284362 A, 23.06.2023 | |||
| WO 2022036788 A1, 24.02.2022 | |||
| Универсальный интеграционный вектор pVEAL и рекомбинантная плазмида pVEAL-15742, обеспечивающая синтез и секрецию scFv-Fc антител ADI-15742 против вируса Эбола в клетках млекопитающих и полученная с использованием вектора pVEAL | 2020 |
|
RU2749459C1 |
| Гуманизированное моноклональное антитело, специфически связывающиеся с RBD S белка вируса SARS-CoV-2, средство и способ для терапии и экстренной профилактики заболеваний, вызываемых вирусом SARS-CoV-2 | 2021 |
|
RU2765731C1 |
Авторы
Даты
2024-10-30—Публикация
2023-11-20—Подача