Группа изобретений относится к области биотехнологии, иммунологии и вирусологии, а именно к моноклональным антителам и их антигенсвязывающим фрагментам, которые связываются с рецептор-связывающим доменом (RBD) Spike-белка вируса SARS-CoV-2, нейтрализующие вирус SARS-CoV-2, к фрагментам ДНК, кодирующим указанные антитела.
За три года с начала пандемии, вызванной бета-коронавирусом SARS-CoV-2, была зарегистрирована гибель почти 7 млн. человек. Использование ряда средств профилактики и терапии коронавирусной инфекции, вызванной SARS-CoV-2 (также называемой в данном документе ковид-19), позволяет снизить вероятность заражения, госпитализации и летального исхода. К таким средствам относятся вакцинопрофилактика, использование вирус-специфических малых молекул, препаратов плазмы реконвалесцентов, рекомбинантных Spike-специфичных белков (в том числе антител) и их производных. Несмотря на повсеместное проведение кампаний по иммунизации населения и идентификации нейтрализующих моноклональных антител против Spike-белка вируса SARS-CoV-2, существует значительная потребность в получении новых вариантов таких антител для расширения арсенала диагностических, профилактических и терапевтических средств особенно с учетом появления новых вариантов вируса. Кроме того, существуют группы населения, которым вакцинация либо не показана по медицинским соображениям, либо у которых не может быть выработан протективный иммунный ответ. Таким образом, проведение пассивной иммунизации - введение препаратов моноклональных антител в качестве пред- или постэкспозиционной профилактики или терапии позволяет защитить таких людей, обеспечить менее длительное и тяжелое течение болезни, в том числе предотвратить развитие постковидных осложнений, и снизить вероятность дальнейшей передачи вируса.
Используемый в настоящей заявке термин «антитело» предназначен для обозначения молекулы иммуноглобулина, составленной из четырех полипептидных цепей, при этом две тяжелые (Н) цепи и две легкие (L) цепи связываются друг с другом дисульфидными связями. Каждая тяжелая цепь содержит вариабельную область тяжелой цепи (сокращенно называемую в настоящем документе VH) и константную область тяжелой цепи. Константная область тяжелой цепи состоит из трех доменов: CH1, СН2 и СН3. Каждая легкая цепь содержит вариабельную область легкой цепи (сокращенно называемую в настоящем документе VL) и константную область легкой цепи. Константная область легкой цепи состоит из одного домена: CL.
Области VH и VL могут далее подразделяться на гипервариабельные районы, которые называются определяющими комплементарность областями (CDR), перемежаемые областями с более высоким уровнем консервативности, называемыми каркасными областями (FR). Каждая область VH и VL образована тремя CDR и четырьмя FR, расположенными от амино-терминального конца к карбокси-терминальному концу в следующем порядке: FR1, CDR1, FR2, CDR2, FR3, CDR3, FR4.
Четыре каркасных участка формируют конформацию типа бета-складчатого слоя. Участки CDR расположены в близком соседстве друг с другом благодаря каркасным участкам и вносят вклад в образование антигенсвязывающего участка. Участки CDR и каркасные участки антител могут быть определены путем ссылки на нумерационную систему Кабата (Kabat numberings ystem, Kabat et al., 1987 "Sequences of Proteins of Immunological Interest", US Dept. Of Health and Human Services, US Government Printing Office) в сочетании с данными рентгеноструктурного анализа, как указано в заявке WO 91/09967. Участки CDR и каркасные участки антител могут также быть определены по номенклатуре Международной информационной системы по иммуногенетике (IMGT, International Immunogenetics Information System, www.imgt.org) (Lefranc, M.P. and Lefranc, G. Methods Mol Biol. 2552:3-59 (2023)).
Если не указано иное, любая представленная в описании полипептидная цепь имеет аминокислотную последовательность, которая начинается на N-конце и заканчивается на С-конце. Если антигенсвязывающий центр содержит и VH-, и VL-области, то они могут быть расположены на одной и той же полипептидной молекуле или предпочтительно каждая область может располагаться на разных цепях, VH-область может быть частью тяжелой цепи иммуноглобулина или ее фрагмента, a VL-область - частью легкой цепи иммуноглобулина или ее фрагмента.
В настоящем изобретении термин «антитело, обладающее вируснейтрализующей активностью» означает молекулу, которая связывается с SARS-CoV-2, образует стабильный комплекс и препятствует прикреплению и/или проникновению вируса SARS-CoV-2 в клетку. Способность антитела к связыванию с антигеном может быть определена специалистом в данной области с использованием методов, включающих, но не ограничивающихся, методом иммуноферментного анализа (ИФА), равновесным диализом, с использованием плазмон-поверхностного резонанса и биослойной интерферометрии (ВЫ). Методы определения аффинности хорошо известны специалисту в данной области техники, подробно описаны Janeway и соавт.(Immunobiology: The Immune System in Health and Disease (Garland Publishing Company, 1996)).
Используемый в настоящем изобретении термин «антигенсвязывающий фрагмент» антитела обозначает фрагмент антитела, который сохраняет способность к специфическому связыванию с антигеном. К примерам таких фрагментов, включенных в термин «антигенсвязывающий фрагмент» антитела, относятся: (i) фрагмент Fab, моновалентный фрагмент, состоящий из доменов VL, VH, CL и СН1; (ii) фрагмент F(ab')2, бивалентный фрагмент, состоящий из двух фрагментов Fab, связанных дисульфидным мостиком в шарнирной области; (iii) фрагмент Fd, состоящий из доменов VH и CH1; (iv) фрагмент Fv, состоящий из доменов VL и VH отдельной области антитела; (v) фрагмент dAb (Ward, E.S. etal. Nature. 241, 544-546, (1989)), состоящий из домена VH; и (vi) отдельная определяющая комплементарность область (CDR). Более того, несмотря на то, что два домена во фрагменте Fv, VL и VH кодируются различными генами, их можно объединить, используя рекомбинантные технологии, посредством синтетического линкера, который позволяет построить из них единую белковую цепочку, в которой области VL и VH связываются с образованием моновалентных молекул (известных как одноцепочечные антитела или scFv; см., например, Bird, R.E. et al. Science. 24, 423-426 (1988); Huston, J.S. et al. Proc. Natl. Acad. Sci. USA 85:5879-5883 (1988)). Такие одноцепочечные антитела также включаются в сферу охвата термина «антигенсвязывающий фрагмент» антитела.
Специалисту в данной области известно, что за взаимодействие антитела с антигеном наибольший вклад вносит тяжелая цепь (Н). Однако известны случаи, когда в распознавании антигена участвует только одна цепь (или Н, или L), а вторую цепь можно заменить без потери антигенсвязывающих свойств антителом (Shiraiwa, Н. et al. Engineering a bispecific antibody with a common light chain: Identification and optimization of an anti-CD3 epsilon and anti-GPC3 bispecific antibody, ERY974. Methods. 154:10-20 (2009); Senn, B.M. et al. Combinatorial immunoglobulin light chain variability creates sufficient В cell diversity to mount protective antibody responses against pathogen infections. Eur J Immunol. 33(4):950-61 (2003)). Таким образом, в широком понимании в сферу охвата термина «антигенсвязывающий фрагмент» антитела можно включить любые белковые молекулы, содержащие хотя бы один из VH и VL доменов заданного антитела.
Известно, что из трех поверхностных белков SARS-CoV-2 лишь один, Spike, вызывает образование вируснейтрализующих антител. Также было показано, что подавляющее число полученных к настоящему моменту Spike-специфических моноклональных антител, обладающих способностью нейтрализовать вирус, направлены против RBD-домена Spike, ответственного за связывание с основным входным рецептором вируса, белком АСЕ2.
Идентификация SARS-CoV-2-специфических нейтрализующих моноклональных антител, в основном, осуществляется методом секвенирования генов иммуноглобулинов из единичных В-клеток людей, перенесших ковид-19 (Pinto, D. et al. Cross-neutralization of SARS-CoV-2 by a human monoclonal SARS-CoV antibody. Nature 583, 290-295 (2020)).
В настоящий момент уже созданы сотни различных SARS-CoV-2-специфических, в том числе и вируснейтрализующих антител, обладающих различными профилями потентности и широты нейтрализации. Однако разработанные моноклональные антитела могут быть неактивными в отношении появившихся новых вариантов SARS-CoV-2. Так, например, моноклональное антитело бамланивимаб не способно нейтрализовать дельта-вариант SARS-CoV-2 (Planas, D. etal. Reduced sensitivity of SARS-CoV-2 variant Delta to antibody neutralization. Nature 596, 276-280 (2021)). В отношении бета-варианта бамланивимаб и этесивимаб полностью неактивны, а активность касиривимаба значительно снижена (Wang, P. Et al. Antibody resistance of SARS-CoV-2 variants B. 1.351 and B.l.1.7. Nature 593, 130-135 (2021)).
Антитела REGN10987 и REGN10933 (имдевимаб и касиривимаб) на 2-3 порядка хуже нейтрализуют вирус штаммов Омикрон ВАЛ и ВА.2, а сотровимаб и антитело S2X259 - чувствительны к этим же штаммам, но в меньшей степени (Iketani, S. et al, Antibody evasion properties of SARS-CoV-2 Omicron sublineages. Nature 604, 553-556. (2022)).
Таким образом, идентификация и всесторонняя характеризация новых SARS-CoV-2-специфических антител является актуальной задачей и позволяет создавать композиции антител, против новых вирусных вариантов SARS-CoV-2 и потенциально против других, в том числе, неописанных коронавирусов.
Существует множество публикаций, описывающих получение и характеризацию RBD-специфических моноклональных антител человека, в том числе и обладающих вируснейтрализующей активностью.
Например, известны человеческие RBD-специфические нейтрализующие антитела бамланивимаб (LY-CoV555) (Jones, В. et al. The neutralizing antibody, LY-CoV555, protects against SARS-CoV-2 infection in nonhuman primates. Sci. Transl. Med. 13(593), 1-17 (2021) и этесивимаб (LY-C0VOI6/JSOI6/CB6) (Shi, R. et al. A human neutralizing antibody targets the receptor-binding site of SARS-CoV-2. Nature 584, 120-124 (2020)), последовательности которых известны. Однако существенным недостатком данных антител является их неспособность нейтрализовать ряд циркулирующих вирусных вариантов SARS-CoV-2 (https://covdb.stanford.edu/page/susceptibility-data/), что ограничивает их профилактическое и терапевтическое использование.
Известна панель моноклональных антител человека iBl-iB26, которая нейтрализует Wu-1 и Delta варианты SARS-CoV-2 (патент RU 2800649 С2, опубл. 25.07.2023). Недостатком данного решения является отсутствие информации о нейтрализации вариантов вируса более поздних вариантов.
Известна общедоступная база данных CoV-AbDab (http://opig.stats.ox.ac.uk/webapps/covabdab/), содержащая информацию о последовательностях и свойствах сотен RBD-специфических моноклональных антител различного происхождения (в том числе и человеческих), многие из которых обладают вируснейтрализующей активностью в отношении SARS-CoV-2.
Недостатками большинства таких антител является их неполная характеризация в плане устойчивости к циркулирующим вариантам SARS-CoV-2, поскольку такие варианты появились позже, чем были получены антитела, а также в плане эпитопной специфичности, широты нейтрализации, наличия или отсутствия ауто- и полиреактивности и т.д. Таким образом, создание новых детально охарактеризованных RBD-специфических моноклональных антител, как нейтрализующих так и ненейтрализующих, является крайне актуальной задачей, поскольку может обеспечить рациональный дизайн композиций антител, предотвращающих мутационное ускользание вируса, а также более точную диагностику, эффективную терапию и профилактику коронавирусной инфекции.
Наиболее близкими к заявляемой группе изобретений - прототипом, являются моноклональное антитело мыши IgM-изотипа и его антигенсвязывающий фрагмент, селективно взаимодействующие с RBD фрагментом в составе S белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью к SARS-CoV-2. Моноклональное антитело получено путем иммунизации мышей Balb/C человеческим рекомбинантным RBD фрагментом S-белка вируса SARS-CoV-2, экспрессированным в E.coli и полноразмерным S-белком вируса SARS-CoV-2 в составе клеток линии НЕК293, получения и селекции линий гибридом, продуцирующих моноклональные антитела к рекомбинантному RBD фрагменту в составе S белка вируса SARS-CoV-2, анализа аффинности и специфичности отобранного МАт, определения нуклеотидной последовательности его вариабельных доменов. Специфичность моноклонального антитела к RBD фрагменту в составе S белка вируса SARS-CoV-2 подтверждена методами иммуноферментного анализа, иммуноблоттинга, иммуноцитохимического и иммуногистохимического окрашивания. Установлена нуклеотидная последовательность вариабельных доменов моноклонального антитела (заявка WO 2021211775 А1, опубл. 21.10.2021).
Недостатками данного мышиного антитела и его антигенсвязывающего фрагмента для целей терапии и профилактики коронавирусной инфекции, вызванной SARS-CoV-2, являются его чужеродность для организма человека, вследствие чего введение такого антитела пациентам может быть иммуногенным и сопровождаться нежелательными побочными реакциями. Также не известен уровень (ауто)реактивности данного антитела по отношению к клеткам человека, поскольку оно было получено в иммунизированных мышах и в процессе созревания В-клетки проходило негативную селекцию против антигенов мыши, а не человека.
Также недостатком данного мышиного антитела является его принадлежность к IgM классу: (i) это снижает время его полужизни, а, значит, будет требовать дополнительных инъекций антитела пациенту; (ii) производство антител IgM изотипа является крайне трудоемким и сопровождается низким выходом, а сохранение всех заявленных свойств данного антитела при его возможной конверсии в IgG1-формат для преодоления данного ограничения является сомнительным, поскольку будет связано, как минимум, со снижением его авидности.
Также недостатком данного изобретения является то, что нейтрализующая активность мышиного антитела была показана только in vitro на клетках Vero Е6 и с использованием раннего штамма SARS-CoV-2 Wuhan-Hu-1. Таким образом, уровень и механизм нейтрализующей активности данного антитела в отношении SARS-CoV-2, а также детали эпитопной специфичности (следовательно, и узнавание им вирусных вариантов, среди новых циркулирующих изолятов) - неизвестны.
Задачей заявляемой группы изобретений является получение новых моноклональных антител человека IgG1 подкласса, способных к связыванию с RBD Spike-белка вируса SARS-CoV-2, а также антигенсвязывающих фрагментов таких антител, обладающих вируснейтрализующей активностью широкого спектра, а также получение ДНК фрагментов, кодирующих антитело и его антигенсвязывающий фрагмент.
Техническим результатом группы изобретений является создание нового моноклонального антитела человека iC1, отличающегося по аминокислотной последовательности вариабельных доменов легкой и тяжелой цепей от известных моноклональных антител к RBD фрагменту в составе Spike-белка вируса SARS-CoV-2 и его антигенсвязывающего фрагмента, способного к связыванию с RBD Spike-белка вируса SARS-CoV-2, обладающего вируснейтрализующей активностью широкого спектра.
Также техническим результатом является повышение аффинности созданного антитела, что позволяет снизить его дозировку при применении, что в значительной степени снижает его стоимость при производстве, а значит - повышает привлекательность для коммерциализации.
Также техническим результатом является способность созданного антитела высокоэффективно нейтрализовать вирус SARS-CoV-2 как в in vitro схемах, так и in vivo в профилактических и терапевтических схемах, что позволяет использовать его для профилактики и терапии заболевания, вызываемого вирусом SARS-CoV-2.
Также техническим результатом является способность созданного антитела нейтрализовать вирус SARS-CoV-2 разных вариантов, что позволяет использовать его для профилактики и терапии заболеваний, вызываемых вирусом SARS-CoV-2 разных штаммов.
Поставленная задача решается созданием моноклонального антитела человека iC1, селективно связывающего RBD фрагмент Spike-белка вируса SARS-CoV-2, состоящего из тяжелой (Н) и легкой (L) цепей, имеющих аминокислотные последовательности SEQ ID NO: 1 и 2, а также созданием кодирующих их фрагментов ДНК, имеющих нуклеотидные последовательности SEQ ID NO: 5 и 6, соответственно.
В аминокислотных последовательностях цепей антитела iC1 можно выделить вариабельный домен тяжелой цепи (VH) и вариабельный домен легкой цепи (VL), имеющих аминокислотные последовательности SEQ ID NO: 3 и 4, кодируемые нуклеотидными последовательностями SEQ ID NO: 7 и 8 соответственно.
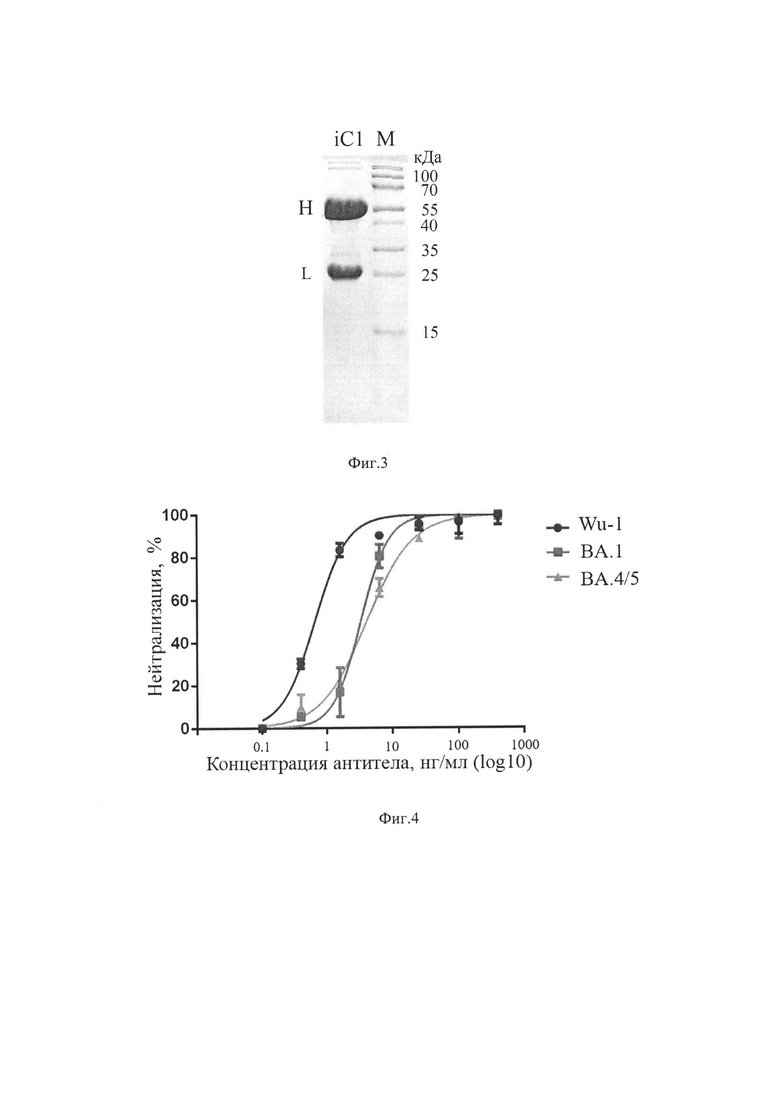
Вариабельный домен тяжелой цепи (VH) содержит последовательности гипервариабельных районов: CDRH1, имеющего аминокислотную последовательность SEQ ID NO: 9 (GFTFDHYA), CDRH2, имеющего аминокислотную последовательность SEQ ID NO: 10 (ASWNSGTI), и CDRH3, имеющего аминокислотную последовательность SEQ ID NO: 11 (VLLAGFTPFDFTAARPDIDY). Вариабельный домен легких цепей (VL) содержит последовательности гипервариабельных районов: CDRL1, имеющего аминокислотную последовательность SEQ ID NO: 12 (SSNIGSNT), CDRL2, имеющего аминокислотную последовательность SEQ ID NO: 13 (NNDQ) и CDRL3, имеющего аминокислотную последовательность SEQ ID NO: 14 (ATWDDSLDGPWV). Участки CDR в VH и VL были определены по номенклатуре IMGT (Lefranc, М.Р. and Lefranc, G. Methods Mol Biol. 2552:3-59 (2023)).
Как хорошо известно, минорные изменения аминокислотной последовательности, такие как делеция, добавление или замена одной, небольшого количества или даже нескольких аминокислот, могут приводить к получению аллельной формы исходного белка, которая обладает практически идентичными свойствами. Также известен способ «перевивки» CDR-петель одного антитела в контекст каркасных последовательностей другого антитела, также с сохранением исходных свойств, что в частности позволяет производить гуманизацию антител, полученных в других млекопитающих. Таким образом, антитело изобретения включает те варианты, аминокислотная последовательность которых вне CDR петель отличается от приведенных в настоящей заявке последовательностей, но которые сохранили ту же антигенсвязывающую активность.
Антитело iC1 было получено способом, включающим следующие стадии: (i) выделение В-лимфоцитов периферической крови доноров, перенесших вакцинацию и коронавирусную инфекцию ковид-19 и сформировавших высокие нейтрализующие титры антител в сыворотке крови; (ii) окрашивание В-лимфоцитов флуоресцентно меченными препаратами RBD SARS-CoV-1; (iii) сортировка единичных окрашенных RBD+В-лимфоцитов; (iv) проведение обратной транскрипции и ПЦР с наборами вырожденных праймеров, комплементарных лидерным и константным последовательностям генов иммуноглобулинов; (v) секвенирование полученных ПЦР продуктов с целью выяснения нуклеотидной и аминокислотной последовательностей, экспрессируемых такими В-клетками антител; (vi) клонирование нуклеотидных последовательностей таких антител в экспрессионные векторы для наработки в культуре клеток млекопитающих; (vii) очистка, анализ специфичности и активности рекомбинантных моноклональных антител человека. В последующем также предусмотрено определение аминокислотной последовательности вариабельных доменов таких антител, измерение аффинности и специфичности, нейтрализующих свойств in vitro и in vivo.
Использованный метод является общепринятым, однако был модифицирован таким образом, чтобы отбираемые моноклональные антитела человека были обогащены антителами, способными распознавать консервативные участки RBD Spike-белка SARS-CoV-2. Достигается это тем, что окрашивание В-лимфоцитов (пункт ii) проводят флуоресцентно меченными препаратами RBD SARS-CoV-1, а не SARS-CoV-2, как это делается обычно. Данная модификация метода серьезно влияет на специфичность получаемых антител. Суть замены меченного препарата заключается в том, что тем самым производится отбор антител, распознающих SARS-CoV-1, но из крови реконвалесцентов, которые были вакцинированы прививкой на основе RBDSARS-CoV-2 и переболели ковид-19, а значит обладают антителами, распознающими SARS-CoV-2. Таким образом, происходит отбор только тех антител, которые распознают эпитопы, присутствующие в Spike-белках обоих вирусов (SARS-CoV-1 и SARS-CoV-2). Учитывая, что данные белки идентичны только на 76% по аминокислотной последовательности, отбираемые антитела с высокой долей вероятности будут распознавать эпитопы, которые являются консервативными, то есть не будут меняться в ходе дальнейшего мутагенеза SARS-CoV-2.
Также настоящим изобретением создан антигенсвязывающий фрагмент моноклонального антитела iC1, селективно связывающий RBD Spike-белка вируса SARS-CoV-2, обладающий вируснейтрализующей активностью, имеющий аминокислотные последовательности вариабельной области тяжелой цепи (VH) - SEQ ID NO: 3, и вариабельной области легкой цепи (VL) - SEQ ID NO: 4, кодируемые нуклеотидными последовательностями SEQ ID NO: 7 и 8, соответственно.
У белков, созданных в рамках заявляемого технического решения, CDR участки являются идентичными, то есть CDRH1 участок антитела iC1 совпадает с CDRH1 участком антигенсвязывающего фрагмента моноклонального антитела iC1, и т.д.
Антитело iC1 человека селективно связывается с RBD Spike-белка SARS-CoV-2, содержит приведенные выше последовательности вариабельных областей и может содержать консервативные участки обеих цепей, необходимые для функционирования указанного антитела. Консервативные участки тяжелых цепей иммуноглобулинов классов IgG, IgM, IgA, IgD или IgE человека и других млекопитающих могут быть использованы в качестве консервативных участков для тяжелых цепей согласно настоящему изобретению. Например, антитело или его антигенсвязывающий фрагмент, согласно изобретению, могут содержать консервативные участки тяжелых цепей, принадлежащие к изотипу IgG1, IgG2, IgG3 или IgG4. Консервативные участки легкой цепи иммуноглобулинов изотипов каппа или лямбда человека и других млекопитающих могут быть использованы в качестве консервативных участков для легких цепей антител согласно настоящему изобретению.
Антитело настоящего изобретения является человеческим антителом. В частном случае антитело может быть гуманизованным антителом, мышиным антителом, химерным антителом или другим генно-инженерным антителом.
В ходе решения поставленной задачи были установлены последовательности нуклеиновых кислот, кодирующие тяжелую (Н) цепь антитела iC1 (нуклеотидная последовательность SEQ ID NO: 5), легкую (L) цепь антитела iC1 (нуклеотидная последовательность SEQ ID NO: 6), вариабельный VH домен антитела iC1 (нуклеотидная последовательность SEQ ID NO: 7) и вариабельный VL домен антитела iC1 (нуклеотидная последовательность SEQ ID NO: 8).
Специалистам в данной области техники известно, что ввиду вырожденности генетического кода могут быть различия в последовательности ДНК кодирующей одну и ту же аминокислотную последовательность (Льюин Б. Гены. Москва: Мир, 1987). Поэтому последовательности нуклеиновых кислот, согласно настоящему изобретению, могут отличаться, при условии, что они кодируют участки цепей антитела iC1, содержащего последовательность аминокислот, не менее чем на 90% гомологичную SEQ ID NO: 3, и последовательность аминокислот, не менее чем на 90% гомологичную SEQ ID NO: 4.
Вышеназванные аминокислотные последовательности антитела iC1 (SEQ ID NO: 1-4), кодируемые нуклеотидными последовательностями (SEQ ID NO: 5-8), а также аминокислотные последовательности антигенсвязывающего фрагмента (SEQ ID NO: 3-4), кодируемые нуклеотидными последовательностями (SEQ ID NO: 7-8) представлены в перечне последовательностей.
Коктейли и комбинация антител. Для SARS-CoV-2 описано несколько основных механизмов прямой вируснейтрализующей активности антител. Первый основан на экранировании сайтов связывания S-белка с рецептором Асе2 за счет конкурентного узнавания антителом так называемого рецептор-связывающего мотива (RBM) в пределах RBD. Второй механизм также предотвращает связывание S-белка с Асе2, но за счет узнавания антителом эпитопов, отличных от RBM, при котором молекула антитела стерически мешает взаимодействию Асе2 с RBM. Третьим является дестабилизация антителом предфьюжн структуры Spike-белка и индукция его необратимого перехода в постфьюжн конформацию. Четвертый, наоборот, основан на стабилизации пред-фьюжн конформации Spike-белка.
Очевидно, что площадь контакта антитела с интерфейсом RBD-Ace2 прямо коррелирует с потенциальной устойчивостью антитела к аминокислотным заменам в Spike-белке. Так как максимальная площадь контакта антитела и антигена ограничена базовыми особенностями структуры вариабельных доменов тяжелой и легкой цепи, она не может быть существенно увеличена для каждого конкретного антитела. Однако, тот же эффект может достигаться за счет одновременного применения различных антител, связывающихся с независимыми или частично перекрывающимися эпитопами на поверхности Spike-белка, поскольку возникновение двух и более мутаций, инактивирующих сразу несколько вируснейтрализующих антител, без негативных последствий для приспособленности вируса крайне маловероятно. Таким образом, оправдано применение коктейлей антител, сформулированных на основе информации об эпитопах связывания со Spike-белком.
Полученная в данной работе информация о конкуренции антитела iC1 с референсными антителами за связывание с RBD, а также данные о чувствительности антитела к аминокислотным заменам в RBD, позволяют осуществлять рациональный дизайн коктейлей антител друг с другом и с известными коммерческими нейтрализующими антителами. Например, антитело id можно комбинировать с антителами, распознающими непересекающиеся эпитопы RBD: iC1+S2H97+S2X259+S2K146/iB14 или iC1+S2K146/iB14+iB20 (см. пример 5). Таким образом, представляется возможным формулировать коктейль, целиком состоящий из неконкурирующих нейтрализующих антител против района взаимодействия АСЕ2 и RBD, и обеспечивающий большую устойчивость к возникающим заменам в структуре Spike-белка SARS-CoV-2. Наличие нескольких непересекающихся эпитопов позволяет получить как минимум 10 вариантов трех компонентного коктейля (8 вариантов с S2H97 и/или S2X259 и 2 варианта с iB20), являющегося наиболее оптимальным. Отсутствие критической чувствительности антитела iC1 к аминокислотным заменам в RBD позволяют не учитывать этот параметр при подборе состава коктейля антител.
Для целей терапии RBD-специфические моноклональные антитела и их комбинации могут быть использованы при введении человеку или другому млекопитающему в короткий срок после появления первых симптомов заболевания, вызванного SARS-CoV-2. Пути введения антител могут включать внутривенную и внутримышечную инъекцию, доставку аэрозолем в дыхательные пути или подкожное введение. Антитело или его антигенсвязывающий фрагмент изобретения могут быть введены системно, назально или путем прямой доставки кодирующих его нуклеиновых кислот в организм млекопитающих. Помимо терапевтического применения антитела могут быть введены заранее в качестве меры профилактики. В таком случае целесообразно использование антител для лиц, имеющих высокий риск заражения вирусом SARS-CoV-2, тяжелого течения заболевания и не подлежащих вакцинации по медицинским или иным причинам. При профилактическом использовании необходимо учитывать период полужизни антител в кровотоке, который в среднем для антител изотипа IgG1 составляет 21 день, но может быть значительно увеличен за счет модификации последовательности аминокислотных остатков в Fc-части.
Краткое описание чертежей и таблиц, иллюстрирующих изобретение
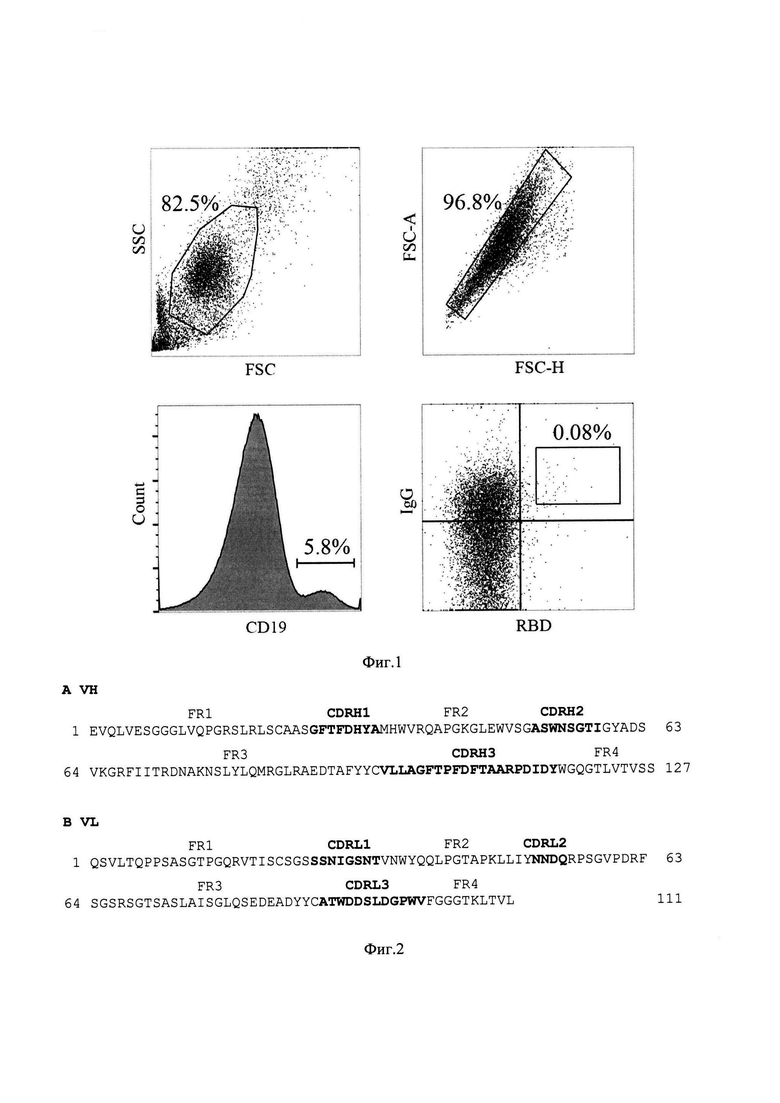
На фиг. 1 показана стратегия гейтирования и результаты FACS-окрашивания В-лимфоцитов донора-реконвалесцента С.
На фиг. 2 представлены аминокислотные последовательности вариабельных доменов тяжелой VH (А) и легкой VL (В) цепей моноклонального антитела человека iC1. CDR районы отмечены жирным шрифтом.
На фиг. 3 показаны результаты разделения очищенного рекомбинантного антитела iC1 в SDS-полиакриламидном геле, окрашенном Кумасси, где М - маркер молекулярного веса, L - легкая цепь антитела, Н - тяжелая цепь антитела.
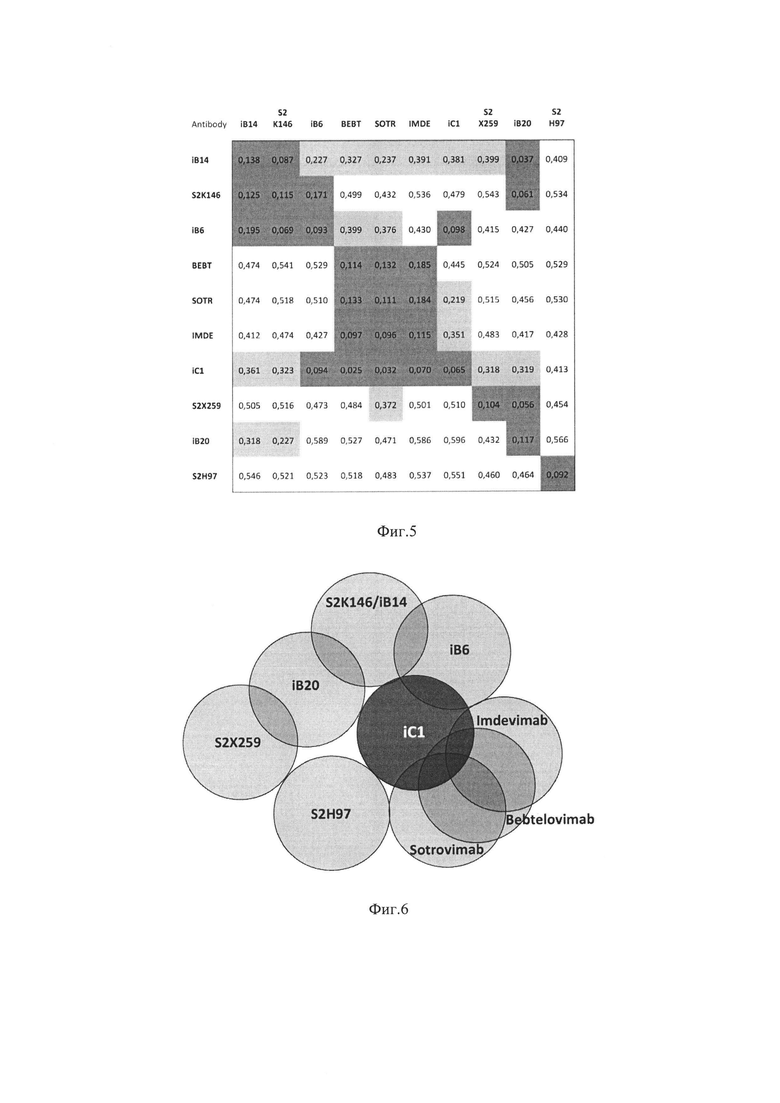
На фиг. 4 показаны результаты нейтрализационных тестов моноклонального антитела iC1 в псевдовирусной вирусной системе с использованием цитометрического теста детекции зараженных клеток. Лентивирусные частицы были псевдотипированы S-белками с RBD от Wu-1, ВАЛ и ВА.4/5 штаммов SARS-CoV-2.
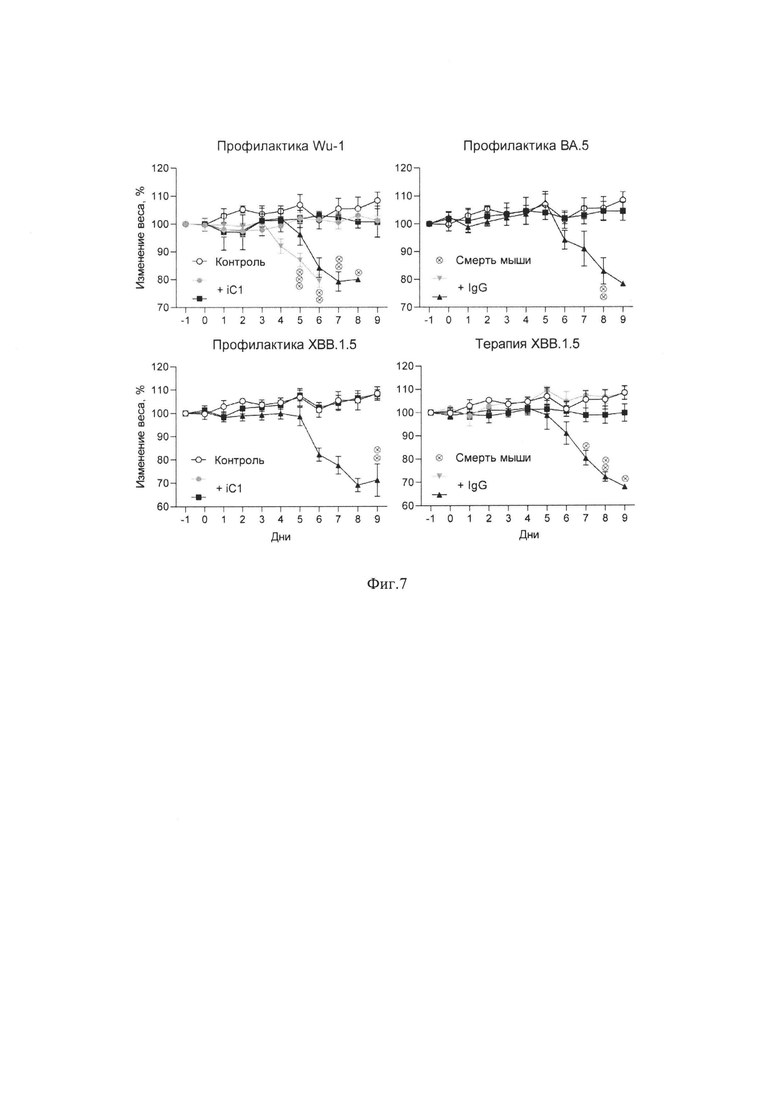
На фиг. 5 показаны данные анализа попарной конкуренции моноклональных антител за связывание с RBD SARS-CoV-2, полученные при помощи биослойной интерферометрии (ВЫ). Черный цвет означает высокую конкуренцию антител за связывание с эпитопом, серый - умеренную конкуренцию, белый - отсутствие конкуренции. ВЕВТ - Бебтеловимаб; SOTR - Сотровимаб; IMDE - Имдевимаб.
На фиг. 6 представлена Венн-диаграмма перекрывания эпитопов, распознаваемых антителом iC1 и референсными моноклональными антителами.
На фиг. 7 приведены данные динамики изменения веса АСЕ2-трансгенных мышей, зараженных Wu-1, ВА.5 или ХВВ.1.5 штаммами SARS-CoV-2. Моноклональное антитело iC1 или нерелевантные IgG человека вводилось в терапевтической (6 часов после заражения) и профилактической (24 часа до) схемах.
Последующие примеры приведены исключительно для целей объяснения и не ограничивают каким-либо образом объема настоящего изобретения.
Пример 1. Выделение В-лимфоцитов человека, специфических к RBD Spike-белка вируса SARS-CoV-1
У доноров был произведены забор периферической венозной крови и выделение живых СD19+IgG+В-лимфоцитов (окрашивание DAPI, конъюгатами anti-human CD19-PE (ООО «Сорбент»), anti-human IgG-FITC (ООО «Сорбент»), 5 мкл/106 мононуклеарных клеток крови). При отборе доноров выбирали только тех, кто был вакцинирован и ревакцинирован (вакциной на основе Wu-1 штамма), а также переболел ковид-19 хотя бы один раз после начала циркуляции штаммов Дельта или Омикрон. В-лимфоциты были дополнительно окрашены биотинилированным рекомбинантным RBD SARS-CoV-1 (2 мкг/мл) с последующей детекцией при помощи конъюгата АРС-стрептавидин (Thermo Fisher Scientific). Поверхностный фенотип целевой популяции В-лимфоцитов для сортировки единичных клеток в индивидуальные ПЦР-пробирки, таким образом, был RBD+CD19+IgG+DAPI- (фиг. 1). Окрашенные аналогичным образом В-лимфоциты здоровых доноров были использованы в качестве негативного контроля.
Пример 2. Получение моноклонального антитела человека против RBD Spike-белка вируса SARS-CoV-1
Процедуры синтеза кДНК с единичных В-лимфоцитов и ПЦР с обратной транскрипцией проводили, как описано в работе Guselnikov и соавт.(Guselnikov, S. V. et al. A simple way to increase recovery of the expressed VH and VL genes in single-sorted human В cells. BioTechniques 67, 184-187 (2019)), за исключением того, что для амплификации последовательностей, кодирующих γ- и λ-цепи антител, был использован протокол для μ-цепей. В частности, для амплификации последовательностей генов VHγ, Vκ и Vλ был использован набор вырожденных праймеров из работы Tiller и соавт.(Tiller, Т. et al. Efficient generation of monoclonal antibodies from single human В cells by single cell RT-PCR and expression vector cloning. J. Immunol. Methods 329, 112-124 (2008)). Парные образцы ПЦР-ампликонов (VHγ+Vκ или Vλ) секвенировали по Сэнгеру и клонировали в векторы для эукариотической экспрессии, кодирующие константные районы γ1, к или λ-цепей иммуноглобулинов человека.
В результате была расшифрована пара нуклеотидных последовательностей VH (SEQ ID NO: 7) и VL (SEQ ID NO: 8) генов из одиночного В-лимфоцита. С учетом генетического кода были определены аминокислотные последовательности VH (SEQ ID NO: 3) и VL (SEQ ID NO: 4) участков моноклонального антитела iC1, специфичного к RBD Spike-белка SARS-CoV-2. Анализ последовательности VL, а также ассоциированного с ним CL, показал принадлежность легкой цепи iC1 антитела к лямбда типу (λ). Участки CDRs VH (CDRH1 - SEQ ID NO: 9; CDRH2 - SEQ ID NO: 10; CDRH3 - SEQ ID NO: 11) и VL (CDRL1 - SEQ ID NO: 12; CDRL2 - SEQ ID NO: 13; CDRL3 - SEQ ID NO: 14) были определены по номенклатуре IMGT (LefrancM.P., LefrancG. MethodsMolBiol. 2552:3-59 (2023)) (фиг. 2).
Проведенный биоинформатический анализ последовательности iC1 антитела против известных последовательностей антител к SARS-CoV-2 не выявил антител, на 100% совпадающих по последовательностям легкой и тяжелой цепи с антителом изобретения.
В дальнейшем нуклеотидная последовательность VH была клонирована в вектор для эукариотической экспрессии в одной рамке с последовательностью, кодирующей константную область тяжелой цепи иммуноглобулина человека (Fc(IgG1)), в результате чего была получена нуклеотидная последовательность (SEQ ID NO: 5), кодирующая аминокислотную последовательность (SEQ ID NO: 1). Аналогичным образом, VL последовательность была клонирована в одной рамке с константной областью лямбда легкой цепи иммуноглобулина человека (Сλ), в результате чего была получена нуклеотидная последовательность (SEQ ID NO: 6), кодирующая аминокислотную последовательность (SEQ ID NO: 2).
Смесь плазмидных ДНК экспрессионных конструкций, кодирующих легкую или тяжелую цепь антитела iC1, была доставлена в клетки линии НЕК293Т, используя метод кальций-фосфатной трансфекции (Chenuet, S. et al. Calcium phosphate transfection generates mammalian recombinant cell lines with higher specific productivity than polyfection. Biotechnol. Bioeng. 101, 937-945 (2008)). Как правило, за 12 часов до проведения трансфекции 107 клеток рассаживали в культуральные флаконы площадью 150 см2 в среде IMDM (Gibco, Thermo Fisher Scientific) с добавлением 10% FBS (Gibco, Thermo Fisher Scientific) и смеси пенициллина/стрептомицина (Gibco, Thermo Fisher Scientific). Плазмидные ДНК смешивали в молярном соотношении 1:1, к полученной смеси добавляли 2 М СаСl2 до финальной концентрации 25 мМ и доводили суммарный объем смеси стерильной водой до 3 мл. Затем смесь медленно добавляли к 3 мл раствора 2×HBS (50 мМ HEPES, 1,5 мМ Na2HPO4, 280 мМ NaCl, 10 мМ КСl, 12 мМ сахароза, рН=7.11) при постоянном интенсивном перемешивании. Полученный раствор добавляли по каплям к клеткам. Спустя 8 часов, культуральную среду заменяли на бессывороточную среду EX-CELL® 293 Serum-Free Medium (Sigma). Трансфицированные клетки росли в течение 6 дней в СОг-инкубаторе при 37°С, 5% СО2. Супернатантны, очищенные от клеточного дебриса при помощи центрифугирования (4000×g, 10 мин), фильтровали через 0.22 мкм PES-фильтр (ТРР). Антитела очищали на колонке с Protein А агарозой (McLab, USA, #РРА-503) в соответствии с инструкциями производителя. Вкратце, после загрузки колонку промывали дважды PBS. Антитело элюировали раствором 0,1 М глицина, рН 2,7, 150 мМ NaCl в нейтрализующий буфер состава 1 М Tris, рН 8,0, в соотношении 1:10. Затем проводили диализ в течение ночи в PBS и концентрировали с использованием колонок Ultra-15 UItracel-100К (Amicon) до финальной концентрации 2 мг/мл. Чистоту и выход препарата антитела контролировали посредством разделения в 15% SDS-полиакриламидном геле (фиг. 3).
Пример 3. Определение аффинности связывания моноклонального антитела iC1 человека и разных вариантов рекомбинантого RBD SARS-CoV-2
Для измерения кинетики взаимодействия антитела iC1 с RBD вариантами (Wu-1, ВА.4/5, ХВВ) была использована биослойная интерферометрия на приборе Octet (ForteBio). С этой целью была вначале проведена иммобилизация антитела iC1 на поверхности биосенсора АНС (45 мкг/мл). На референсном сенсоре в качестве нерелевантного антитела было использовано рекомбинантное моноклональное антитело человека glVRCOl (Georgiev, I. S. et al. Antibodies VRC01 and 10E8 neutralize HIV-1 with high breadth and potency even with Ig-framework regions substantially reverted to germline. J Immunol. 192(3), 1100-1106 (2014)). Характеризация связывания была проведена в диапазоне трех концентраций RBD (10 нМ, 20 нМ, 40 нМ). Для обработки данных были использованы параметры полного глобального фиттинга, бивалентная модель, R2>0,9, Х2<3 и последовательный переход между режимами: Baseline 60 с, Loading 120 с, Association 300 с, Dissociation 1800 с, Regeneration Зраза по5 с. В Табл. 1 представлены результаты измерения аффинности связывания разных вариантов рекомбинантного RBD с антителом iC1 методом биослойной интерферометрии, где KD - равновесная константа диссоциации, ка - константа скорости ассоциации и kdis - константа скорости диссоциации.
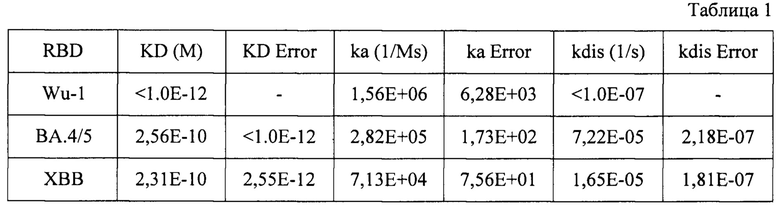
Стоит отметить, что KD имеет высокое значение не только для RBD "дикого типа", но и для разных вариантов RBD SARS-CoV-2, что делает возможным использование iC1 в диагностических приложениях для детекции вируса, например, в образцах слюны человека.
Пример 4. Определение вируснейтрализующей активности моноклонального антитела iC1 человека in vitro
Для оценки уровня вируснейтрализующей активности моноклонального антитела iC1 использовали лентивирусные частицы, псевдотипированные разными вариантами S-белка SARS-CoV-2. Детекцию уровня заражения клеток НЕК293Т проводили двумя способами: цитометрическим тестом (фиг. 4) или люминометрическим тестом (Табл. 2).
Для получения лентивирусных частиц, псевдотипированных Spike-белком SARS-CoV-2, проводили кальций-фосфатную трансфекцию клеток линии НЕК293Т смесью плазмид psPAX2, pLV-EGFP (или pLV-Luc) и pCAGGS-SpikeA19 в молярном соотношении 4:6:3. Плазмида pCAGGS-SpikeA19 кодировала укороченный с С-конца на 19 аминокислотных остатков Spike-белок, соответствующий исходному штамму Wuhan-Hu-1 SARS-CoV-2 или мутантным вариантам Spike SARS-CoV-2 (SA19). Через восемь часов после трансфекции среду заменяли на Opti-Mem (Gibco, Thermo Fisher Scientific) с добавлением 2,5% FBS (Gibco, Thermo Fisher Scientific). Супернатанты собирали два дня спустя, центрифугировали при 4000 об/мин в течение 5 минут, после чего лентивирусные частицы концентрировали путем центрифугирования при 20000×g, 4°С в течение 90 минут. Для проведения нейтрализационного теста в качестве мишеней использовали клетки линии АСЕ2-НЕК293Т со стабильной поверхностной экспрессией белка АСЕ2, рассаженные с плотностью 30000 клеток/лунку в 96-луночном штативе в день проведения теста. Антитело iC1 серийно двукратно разводили в среде Opti-MEMc добавлением 2,5% FBS в диапазоне от 0,5 нг/мл до 1 мкг/мл и ко-инкубировали с 20000 лентивирусных частиц, псевдотипированных Spike-белком SARS-CoV-2 в течение 30 мин при 37°С в объеме 100 мкл. Полученную смесь антител и лентивирусных частиц добавляли к клеткам АСЕ2-НЕК293Т и возвращали в СО2-инкубатор. Спустя 48-72 часа после трансдукции измеряли процент трансдуцированных (GFP+) клеток, используя проточный цитометр (фиг. 4) или уровень люминесценции клеточного лизата, используя люминометр. Значение концентрации полумаксимального ингибирования IC50 определяли, используя нелинейную регрессию, как концентрацию антитела, обеспечивавшую нейтрализацию 50% псевдотипированных лентивирусных частиц.
В Табл. 2 продемонстрирована способность антитела iC1 нейтрализовать лентивирусы, псевдотипированные разными вариантами Spike-белка SARS-CoV-2 с помощью люминометрического теста.
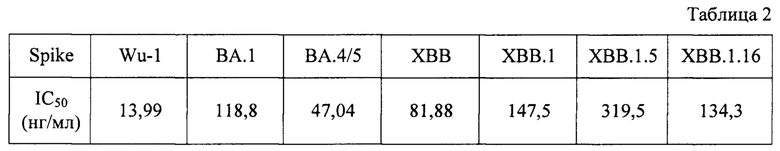
Таким образом, было установлено, что моноклональное антитело iC1 является ультрапотентным нейтрализатором SARS-CoV-2 дикого типа (IС50<16 нг/мл), при этом оставаясь высокопотентным в отношении прочих исследованных вариантов RBD. Хуже всего антитело iC1 нейтрализовало лентивирус псевдотипированный RBD ХВВ.1.5, для которого IС50~300 нг/мл.
Пример 5. Определение конкурентных взаимоотношений между моноклональными антителами за связывание с эпитопами RBD
Для выяснения конкуренции антитела iC1 с референсными моноклональными антителами человека за связывание с RBD был проведен их попарный анализ с помощью биослойной интерферометрии. С этой целью рекомбинантный RBD-His6 (30 мкг/мл) был иммобилизован на NTA-биосенсор, после чего к нему было добавлено насыщающее количество первого антитела (Аb1, 200 нМ). Сформированный таким образом комплекс RBD-Ab1 затем инкубировали со вторым антителом, Аb2 (200 нМ). В целом была использована следующая 6-шаговая схема: Baseline 60 с, Loading 120 с, Baseline 230 с, Saturation АS 500 с, Competing Аb2 500 с, Regeneration 3 раза по 5 с.
Стоит отметить, что степень конкуренции, а значит перекрываемость эпитопов, между антителами была разной. Так, iB14/S2K146 конкурировали на высоком уровне с iB20 и на низком со всеми остальными антителами, за исключением iB6, с которым конкуренция была на среднем уровне (фиг. 5). Также, некоторые антитела вели себя не одинаково в прямой и обратной постановке эксперимента по конкуренции - например, S2X259 конкурирует с iB20, но не наоборот.
Тем не менее, на основе полученных данных удалось получить интегральную картину и картировать эпитопы связывания для всех исследованных антител. Так, все антитела, одобренные для использования в медицине (Бебтеловимаб, Сотровимаб, Имдевимаб), сформировали с антителом iC1 единый кластер. Остальные антитела конкурировали друг с другом попарно, за исключением S2H97, образовавшего полностью неперекрывающийся эпитоп (фиг. 6).
Таким образом, проведенный конкурентный анализ свидетельствует о том, что 10 нейтрализующих моноклональных антител образуют 6 эпитопных групп: S2H97, S2X259/iB20, iB20/S2K146/iB14, S2K146/iB14/iB6, iB6/iC1, iC1/Сотровимаб/Бебтеловимаб/Имдевимаб (фиг. 6), что открывает значительный простор для создания различных композиций (комбинаций) неконкурирующих друг с другом вируснейтрализующих антител. Например, трехкомпонентные композиции, включающие в себя антитело iC1, могут быть образованы сочетаниями: (1) iC1+S2H97+iB14; (2) iC1+S2H97+iB20; (3) iC1+S2H97+S2X259; (4) iC1+S2H97+S2K146; (5) iC1+S2K146+S2X259; (6) iC1+iB14+S2X259.
Пример 6. Определение вируснейтрализующей активности антитела in vivo
Для оценки вируснейтрализующей активности антитела iC1 in vivo была использована модель инфекции SARS-CoV-2 на АСЕ2-трансгенных мышах. Данная модель является более чувствительной в отношении "слабых" штаммов SARS-CoV-2, например, В А.5 или ХВВ.1.5, заражение которыми практически не влияет на состояние сирийских хомячков. Для трансгенной мыши, в которую введен ген АСЕ2 человека, заражение любым штаммом SARS-CoV-2 приводит к системным последствиям - инфицированию практически всех органов и, чаще всего, к летальному исходу. Таким образом, на мышиной модели очень удобно детектировать профилактический и терапевтический эффект противовирусных препаратов: в случае их эффективного действия подопытные мыши сохранят вес и выживают, а контрольные - теряют вес и погибают.
Было изучена нейтрализующая активность антитела iC1 в отношении Wu-1 (дикий тип), ВА.5 и ХВВ.1.5 штаммов SARS-CoV-2 в профилактической схеме введения. Дополнительно для ХВВ.1.5 была использована также и терапевтическая схема.
Всего было сформировано несколько групп животных (n=3-7), которым внутрибрюшинно вводили моноклональное антитело iC1 (эксперимент) или суммарный иммуноглобулин IgG человека (негативный контроль) в количестве 10 мг/кг за 24 часа до (День -1, профилактика) или через 6 часов после (терапия) инфицирования. Животные, не получившие ни антител, ни вируса, были использованы в качестве дополнительного контроля (интактные). На следующий день (День 0) животных интраназально зараражали SARS-CoV-2 в суммарной дозе 2,0×103 БОЕ. Животных взвешивали ежедневно. В некоторых случаях эксперимент проводили в 2 повторах. В Таблице 3 продемонстрировано количество повторов экспериментов по оценке вируснейтрализующей активности антитела iC1 in vivo.
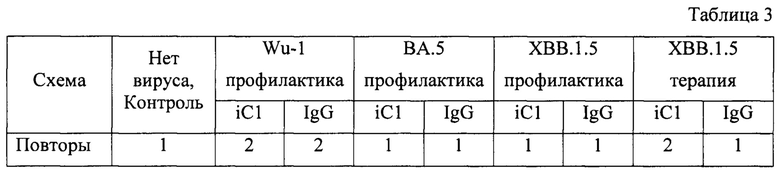
Как профилактическое, так и терапевтическое введение антител обладало выраженным эффектом. В то время контрольные животные ("+IgG") прогрессивно теряли в весе (снижение на 20-25% к 9 дню после заражения) и погибали (16 смертей во всех группах), все животные из контрольной (интактные, "Контроль") и экспериментальных групп ("+iC1") выживали и прибавляли в весе (фиг. 7). Эксперименты в двух повторах показали близкие результаты.
Как и ожидалось, эффект заражения Wu-1 штаммом был более выраженный -контрольные мыши быстрее теряли вес и погибали, начиная с 5 дня, не доживая до дня окончания эксперимента. При заражении ВА.5 и ХВВ.1.5 штаммами эффект потери веса был также явно выражен, но запаздывал на 1-2 дня относительно дикого типа. Гибель животных также наблюдалась позже - начиная с 7 дня (фиг. 7). Часть мышей дожила до конца эксперимента (день 9), однако продолжающаяся потеря веса и их критическое состояние не оставляли сомнения, что в итоге они погибнут в течение нескольких дней.
Также экспериментальные животные, зараженные Wu-1, набирали вес медленнее, чем зараженные ВА.5. Экспериментальные мыши, зараженные ХВВ.1.5, в свою очередь, набирали вес быстрее, на уровне интактных мышей (фиг. 7).
Таким образом, полученные результаты однозначно свидетельствуют о наличии у антитела iC1 явно выраженной нейтрализующей активности в отношении разных штаммов SARS-CoV-2 в профилактической и терапевтической схемах применения in vivo.
Пример 7. Изучение чувствительности антитела iC1 к единичным аминокислотным заменам в Spike-белка SARS-CoV-2
С целью выяснения возможной чувствительности нейтрализующих моноклональных антител к заменам в RBD Spike-белка SARS-CoV-2 с использованием направленного мутагенеза нами была получена панель экспрессионных конструкций, кодирующих 14 вариантов Spike-белка с одноаминокислотными заменами, присутствующими, в том числе, в циркулирующих в настоящий момент штаммах SARS-CoV-2 (N439K, R346G, A352D, V367F, E406W, K444Q, V445A, N450K, Y453F, N460T, A475V, F486L, F490L, S494P, P499L). Такие конструкции были использованы для получения псевдотипированных лентивирусных частиц и проведения люминометрического теста на вируснейтрализацию с антителом iC1.
В Таблице 4 представлены данные по влиянию одиночных аминокислотных замен в Spike-белке SARS-CoV-2 на вируснейтрализующую активность антитела iC1 с помощью люминометрического теста.
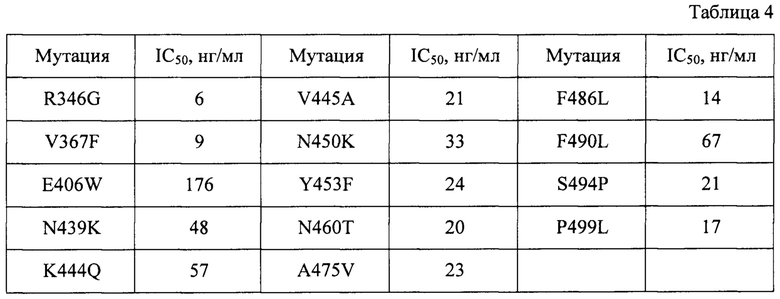
Из полученных данных (Табл. 4) видно, что хотя некоторые замены и приводят к падению IC50, это падение не является критическим - не более одного порядка - что говорит, о том, что вируснейтрализующие свойства у антитела iC1 сохраняются. Самой чувствительной является замена глутаминовой кислоты на триптофан (E406W). Однако, эта же замена приводит и к снижению аффинности взаимодействия Spike-белка SARS-CoV-2 с АСЕ2 рецептором также на 1 порядок (Addetia, A. et al. Structural changes in the SARS-CoV-2 spike E406W mutant escaping a clinical monoclonal antibody cocktail. CellRep. 42, 112621 (2023)). Таким образом, при наличии E406W замены в RBD циркулирующего SARS-CoV-2 будет происходить хуже связывание как антитела и вирусом, так и вируса с клеткой-хозяина и, в итоге, падение IС50 будет компенсировано.
Полученные результаты свидетельствуют об устойчивости антитела iC1 к мутационному ускользанию вируса, что выгодно отличает заявляемое антитело от иных антител, полученных ранее с использованием обычной схемы селекции, то есть с окраской на RBD SARS-CoV-2 Wu-1. Например, каждое из 7 топовых iB антител (Gorchakov, А. А. et al. Isolation of a panel of ultra-potent human antibodies neutralizing SARS-CoV-2 and viral variants of concern. Cell Discov 7, 96 (2021)) имеет уязвимость: 5 чувствительны к Е484К замене, из них 3 чувствительны еще к F486L и F490L заменам; еще по одному антителу чувствительны только к F486L или N460T заменам. На активность REGN10987, в свою очередь, негативно влияют варианты K444Q и V445A, а на активность REGN 10933 -F486K (Starr, Т. N. et al. Prospective mapping of viral mutations that escape antibodies used to treat COVID-19. Science 371, 850-854 (2021); Baum, A. et al. Antibody cocktail to SARS-CoV-2 spike protein prevents rapid mutational escape seen with individual antibodies. Science 369, 1014-1018 (2020)).
Высокая аффинность заявляемого антитела позволит снизить его дозировку при диагностическом, профилактическом и терапевтическом применении, а способность нейтрализовать различные варианты вируса SARS-CoV-2 и устойчивость к появлению аминокислотных замен в Spike-белке вируса говорят о его широком спектре действия и наличии потенциала для нейтрализации новых вариантов SARS-CoV-2.
--->
Перечень последовательностей
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Перечень
последовательностей iC1.xml" softwareName="WIPO Sequence"
softwareVersion="2.0.0" productionDate="2022-06-08">
<ApplicantFileReference>1</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
бюджетное учреждение науки Институт молекулярной и клеточной биологии
Сибирского отделения Российской академии наук (ИМКБ СО
РАН)</ApplicantName>
<ApplicantNameLatin>Institute of Molecular and Cellular Biology,
Siberian Branch of Russian Academy of Sciences</ApplicantNameLatin>
<InventionTitle languageCode="ru">Моноклональное антитело iC1 и его
антигенсвязывающий фрагмент, селективно связывающие
рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие
вируснейтрализующей активностью </InventionTitle>
<SequenceTotalQuantity>14</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>457</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..457</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EVQLVESGGGLVQPGRSLRLSCAASGFTFDHYAMHWVRQAPGKGLEWVS
GASWNSGTIGYADSVKGRFIITRDNAKNSLYLQMRGLRAEDTAFYYCVLLAGFTPFDFTAARPDIDYWGQ
GTLVTVSSASTKGPSVFPLAPSSKSTSGGTAALGCLVKDYFPEPVTVSWNSGALTSGVHTFPAVLQSSGL
YSLSSVVTVPSSSLGTQTYICNVNHKPSNTKVDKKVEPKSCDKTHTCPPCPAPELLGGPSVFLFPPKPKD
TLMISRTPEVTCVVVDVSHEDPEVKFNWYVDGVEVHNAKTKPREEQYNSTYRVVSVLTVLHQDWLNGKEY
KCKVSNKALPAPIEKTISKAKGQPREPQVYTLPPSRDELTKNQVSLTCLVKGFYPSDIAVEWESNGQPEN
NYKTTPPVLDSDGSFFLYSKLTVDKSRWQQGNVFSCSVMHEALHNHYTQKSLSLSPGK</INSDSeq_se
quence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>217</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..217</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSASGTPGQRVTISCSGSSSNIGSNTVNWYQQLPGTAPKLLI
YNNDQRPSGVPDRFSGSRSGTSASLAISGLQSEDEADYYCATWDDSLDGPWVFGGGTKLTVLGQPKAAPS
VTLFPPSSEELQANKATLVCLISDFYPGAVTVAWKADSSPVKAGVETTTPSKQSNNKYAASSYLSLTPEQ
WKSHRSYSCQVTHEGSTVEKTVAPTECS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>127</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..127</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>EVQLVESGGGLVQPGRSLRLSCAASGFTFDHYAMHWVRQAPGKGLEWVSGAS
WNSGTIGYADSVKGRFIITRDNAKNSLYLQMRGLRAEDTAFYYCVLLAGFTPFDFTAARPDIDYWGQGTL
VTVSS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>111</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..111</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QSVLTQPPSASGTPGQRVTISCSGSSSNIGSNTVNWYQQLPGTAPKLLIYNN
DQRPSGVPDRFSGSRSGTSASLAISGLQSEDEADYYCATWDDSLDGPWVFGGGTKLTVL</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>1374</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1374</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GAAGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGCAGGTCCC
TGAGACTCTCCTGTGCAGCCTCTGGATTCACCTTTGATCATTATGCCATGCACTGGGTCCGGCAGGCTCC
AGGGAAGGGCCTGGAGTGGGTCTCAGGTGCTAGTTGGAATAGTGGAACCATAGGCTATGCGGACTCTGTG
AAGGGCCGATTCATCATTACCAGAGACAACGCCAAGAACTCCCTGTATTTGCAAATGAGAGGTCTGAGAG
CTGAGGACACGGCCTTCTATTATTGTGTGCTTCTAGCTGGATTTACTCCTTTCGACTTCACGGCAGCCCG
TCCCGACATTGACTACTGGGGCCAGGGGACCCTGGTCACCGTCTCCTCAGCGTCGACCAAGGGCCCATCG
GTCTTCCCCCTGGCACCCTCCTCCAAGAGCACCTCTGGGGGCACAGCGGCCCTGGGCTGCCTGGTCAAGG
ACTACTTCCCCGAACCTGTGACGGTCTCGTGGAACTCAGGCGCCCTGACCAGCGGCGTGCACACCTTCCC
GGCTGTCCTACAGTCCTCAGGACTCTACTCCCTCAGCAGCGTGGTGACCGTGCCCTCCAGCAGCTTGGGC
ACCCAGACCTACATCTGCAACGTGAATCACAAGCCCAGCAACACCAAGGTGGACAAGAAAGTTGAGCCCA
AATCTTGTGACAAAACTCACACATGCCCACCGTGCCCAGCACCTGAACTCCTGGGGGGACCGTCAGTCTT
CCTCTTCCCCCCAAAACCCAAGGACACCCTCATGATCTCCCGGACCCCTGAGGTCACATGCGTGGTGGTG
GACGTGAGCCACGAAGACCCTGAGGTCAAGTTCAACTGGTACGTGGACGGCGTGGAGGTGCATAATGCCA
AGACAAAGCCGCGGGAGGAGCAGTACAACAGCACGTACCGTGTGGTCAGCGTCCTCACCGTCCTGCACCA
GGACTGGCTGAATGGCAAGGAGTACAAGTGCAAGGTCTCCAACAAAGCCCTCCCAGCCCCCATCGAGAAA
ACCATCTCCAAAGCCAAAGGGCAGCCCCGAGAACCACAGGTGTACACCCTGCCCCCATCCCGGGATGAGC
TGACCAAGAACCAGGTCAGCCTGACCTGCCTGGTCAAAGGCTTCTATCCCAGCGACATCGCCGTGGAGTG
GGAGAGCAATGGGCAGCCGGAGAACAACTACAAGACCACGCCTCCCGTGCTGGACTCCGACGGCTCCTTC
TTCCTCTACAGCAAGCTCACCGTGGACAAGAGCAGGTGGCAGCAGGGGAACGTCTTCTCATGCTCCGTGA
TGCATGAGGCTCTGCACAACCACTACACGCAGAAGAGCCTCTCCCTGTCTCCGGGTAAATGA</INSDSe
q_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>654</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..654</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CAGTCTGTGCTGACTCAGCCACCCTCAGCGTCTGGGACCCCCGGGCAGAGGG
TCACCATCTCTTGTTCTGGAAGCAGCTCCAACATCGGAAGTAATACTGTAAACTGGTACCAGCAACTCCC
AGGAACGGCCCCCAAACTCCTCATCTATAATAATGATCAGCGGCCCTCAGGGGTCCCTGACCGATTCTCT
GGCTCCAGGTCTGGCACCTCAGCCTCCCTGGCCATCAGTGGGCTCCAGTCTGAGGATGAGGCTGATTATT
ACTGTGCAACATGGGATGACAGCCTGGATGGTCCCTGGGTGTTCGGCGGAGGGACCAAGCTGACCGTCCT
AGGTCAGCCCAAGGCTGCCCCCTCGGTCACTCTGTTCCCACCCTCGAGTGAGGAGCTTCAAGCCAACAAG
GCCACACTGGTGTGTCTCATAAGTGACTTCTACCCGGGAGCCGTGACAGTGGCCTGGAAGGCAGATAGCA
GCCCCGTCAAGGCGGGAGTGGAGACCACCACACCCTCCAAACAAAGCAACAACAAGTACGCGGCCAGCAG
CTACCTGAGCCTGACGCCTGAGCAGTGGAAGTCCCACAGAAGCTACAGCTGCCAGGTCACGCATGAAGGG
AGCACCGTGGAGAAGACAGTCGCCCCAACTGAGTGTTCTTGA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>381</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..381</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GAAGTGCAGCTGGTGGAGTCTGGGGGAGGCTTGGTACAGCCTGGCAGGTCCC
TGAGACTCTCCTGTGCAGCCTCTGGATTCACCTTTGATCATTATGCCATGCACTGGGTCCGGCAGGCTCC
AGGGAAGGGCCTGGAGTGGGTCTCAGGTGCTAGTTGGAATAGTGGAACCATAGGCTATGCGGACTCTGTG
AAGGGCCGATTCATCATTACCAGAGACAACGCCAAGAACTCCCTGTATTTGCAAATGAGAGGTCTGAGAG
CTGAGGACACGGCCTTCTATTATTGTGTGCTTCTAGCTGGATTTACTCCTTTCGACTTCACGGCAGCCCG
TCCCGACATTGACTACTGGGGCCAGGGGACCCTGGTCACCGTCTCCTCA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>333</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..333</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CAGTCTGTGCTGACTCAGCCACCCTCAGCGTCTGGGACCCCCGGGCAGAGGG
TCACCATCTCTTGTTCTGGAAGCAGCTCCAACATCGGAAGTAATACTGTAAACTGGTACCAGCAACTCCC
AGGAACGGCCCCCAAACTCCTCATCTATAATAATGATCAGCGGCCCTCAGGGGTCCCTGACCGATTCTCT
GGCTCCAGGTCTGGCACCTCAGCCTCCCTGGCCATCAGTGGGCTCCAGTCTGAGGATGAGGCTGATTATT
ACTGTGCAACATGGGATGACAGCCTGGATGGTCCCTGGGTGTTCGGCGGAGGGACCAAGCTGACCGTCCT
A</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GFTFDHYA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ASWNSGTI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
/INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLLAGFTPFDFTAARPDIDY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>8</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SSNIGSNT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>4</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>NNDQ</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>12</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..12</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier>
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ATWDDSLDGPWV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Моноклональное антитело iC2 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2023 |
|
RU2817697C1 |
| МОНОКЛОНАЛЬНЫЕ АНТИТЕЛА ПРОТИВ РЕЦЕПТОР-СВЯЗЫВАЮЩЕГО ДОМЕНА SPIKE-БЕЛКА ВИРУСА SARS-COV-2 И ИХ АНТИГЕНСВЯЗЫВАЮЩИЕ ФРАГМЕНТЫ, КОДИРУЮЩИЕ ИХ НУКЛЕИНОВЫЕ КИСЛОТЫ, А ТАКЖЕ СПОСОБЫ ПРИМЕНЕНИЯ | 2021 |
|
RU2800649C2 |
| Однодоменное антитело ламы Н5 и его производное H5-Fc, специфически связывающие RBD-домен S-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью | 2022 |
|
RU2793967C1 |
| БИСПЕЦИФИЧЕСКОЕ МОНОКЛОНАЛЬНОЕ АНТИТЕЛО ПРОТИВ SARS-COV-2 | 2022 |
|
RU2791749C1 |
| Моноклональное антитело к RBD фрагменту в составе S белка вируса SARS-CoV-2 | 2020 |
|
RU2744274C1 |
| Универсальный интеграционный вектор SB7G_HIGH и рекомбинантная плазмида SB7G_HIGH_DEV_57, обеспечивающая синтез и секрецию рекомбинантного человеческого антитела к рецептор-связывающему домену (RBD) коронавируса SARS-CoV-2 в клетках млекопитающих и полученная с использованием универсального вектора SB7G_HIGH, и рекомбинантное моноклональное антитело DEV_K57, обладающее вируснейтрализующей активностью в отношении SARS-CoV-2 | 2023 |
|
RU2829359C1 |
| ПОЛИПЕПТИДНЫЙ МОДУЛЬ ДЛЯ СВЯЗЫВАНИЯ КОНСЕРВАТИВНОГО ЭПИТОПА РЕЦЕПТОР-СВЯЗЫВАЮЩЕГО ДОМЕНА БЕЛКА SPIKE КОРОНАВИРУСА SARS-COV-2 | 2022 |
|
RU2809183C1 |
| Широко нейтрализующее антитело против SARS-CoV-2 | 2022 |
|
RU2810476C1 |
| Тяжелоцепочечные моноклональные антитела, специфически связывающиеся с S белком вируса SARS-CoV-2, и способ их применения для терапии заболеваний, вызываемых различными вариантами вируса SARS-CoV-2 | 2024 |
|
RU2836313C1 |
| Однодоменные наноантитела против шиповидного белка вируса SARS-CoV-2 | 2021 |
|
RU2794141C2 |
Изобретение относится к области биотехнологии. Описана группа изобретений, включающая моноклональное антитело iC1 и его антигенсвязывающий фрагмент, селективно связывающие рецептор-связывающий домен (RBD) Spike-белка вируса SARS-CoV-2, обладающие вируснейтрализующей активностью. В одном из вариантов реализации изобретения моноклональное антитело или его антигенсвязывающий фрагмент включают вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот SEQ ID NO: 3, и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот SEQ ID NO: 4. Изобретение направлено на разработку нового моноклонального антитела, связывающегося с RBD Spike-белка вируса SARS-CoV-2, имеющего повышенную аффинность и способного нейтрализовать вирус SARS-CoV-2. 2 н. и 5 з.п. ф-лы, 7 ил., 4 табл., 7 пр.
1. Моноклональное антитело iC1, селективно связывающее рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающее вируснейтрализующей активностью, состоящее из тяжелой (Н) цепи, имеющей последовательность аминокислот SEQ ID NO: 1, и легкой (L) цепи, имеющей последовательность аминокислот SEQ ID NO: 2, включающее вариабельную область тяжелой цепи (VH) указанного антитела, содержащую последовательность аминокислот SEQ ID NO: 3, и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот SEQ ID NO: 4, кодируемые нуклеотидными последовательностями SEQ ID NO: 5-8 соответственно.
2. Антигенсвязывающий фрагмент моноклонального антитела iC1, селективно связывающий рецептор-связывающий домен Spike-белка вируса SARS-CoV-2, обладающий вируснейтрализующей активностью, включающий вариабельную область тяжелой цепи (VH) указанного антитела, содержащую последовательность аминокислот SEQ ID NO: 3, и включающий вариабельную область легкой цепи (VL), содержащую последовательность аминокислот SEQ ID NO: 4, кодируемые нуклеотидными последовательностями SEQ ID NO: 7 и 8 соответственно.
3. Моноклональное антитело iC1 или его антигенсвязывающий фрагмент по пп. 1, 2, имеющие определяющие комплементарность области (CDR) со следующими аминокислотными последовательностями:
(i) CDRH1 - SEQIDNO: 9;
(ii) CDRH2 - SEQIDNO: 10;
(iii) CDRH3 - SEQIDNO: 11;
(iv) CDRL1 - SEQIDNO: 12;
(v) CDRL2 - SEQIDNO: 13;
(vi) CDRL3 -SEQIDNO: 14.
4. Моноклональное антитело iC1 или его антигенсвязывающий фрагмент по пп. 1, 2, содержащее вариабельную область тяжелой цепи (VH), содержащую последовательность аминокислот, не менее чем на 90% сходной с SEQ ID NO: 3, и вариабельную область легкой цепи (VL), содержащую последовательность аминокислот, не менее чем на 90% сходной с SEQ ID NO: 4.
5. Моноклональное антитело iC1 или его антигенсвязывающий фрагмент по пп. 1, 2, где антитело или его антигенсвязывающий фрагмент содержат консервативные участки тяжелых цепей, относящиеся к классу IgG, IgM, IgA, IgD или IgE.
6. Моноклональное антитело iC1 или его антигенсвязывающий фрагмент по пп. 1, 2, где антитело или его антигенсвязывающий фрагмент содержат консервативные участки тяжелых цепей, принадлежащие к изотипу IgG1, IgG2, IgG3 или IgG4.
7. Антигенсвязывающий фрагмент по п. 2, где антигенсвязывающий фрагмент представляет собой Fv, scFv, Fab, F(ab')2, Fab' или любой иной белок, содержащий VH и/или VL последовательности антитела iC1.
| Моноклональное антитело к RBD фрагменту в составе S белка вируса SARS-CoV-2 | 2020 |
|
RU2744274C1 |
| Гуманизированное моноклональное антитело, специфически связывающиеся с RBD S белка вируса SARS-CoV-2, средство и способ для терапии и экстренной профилактики заболеваний, вызываемых вирусом SARS-CoV-2 | 2021 |
|
RU2765731C1 |
| Рекомбинантный белок, связывающийся с RBD S-белка SARS-CoV-2 | 2022 |
|
RU2778942C1 |
| МОНОКЛОНАЛЬНЫЕ АНТИТЕЛА ПРОТИВ РЕЦЕПТОР-СВЯЗЫВАЮЩЕГО ДОМЕНА SPIKE-БЕЛКА ВИРУСА SARS-COV-2 И ИХ АНТИГЕНСВЯЗЫВАЮЩИЕ ФРАГМЕНТЫ, КОДИРУЮЩИЕ ИХ НУКЛЕИНОВЫЕ КИСЛОТЫ, А ТАКЖЕ СПОСОБЫ ПРИМЕНЕНИЯ | 2021 |
|
RU2800649C2 |
| WO 2021045836 A1, 11.03.2021 | |||
| EP 3889177 B1, 11.01.2023 | |||
| Bordoloi D | |||
| et al | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Прибор для определения долготы места | 1923 |
|
SU1349A1 |
Авторы
Даты
2024-04-18—Публикация
2023-11-22—Подача