Изобретение относится к биотехнологии и медицине, а именно к мониторингу носительства вируса обезьяньей оспы вида monkeypox у здоровых лиц, диагностике лиц с подозрением на обезьянью оспу и больных обезьяньей оспой.
Обезьянья оспа относится к зоонозным инфекционным заболеваниям вирусной этиологии. Вирус обезьяньей оспы филогенетически близок вирусу натуральной оспы (оба входят в род Orthopoxvirus, семейство Poxviridae) и вызывает схожие симптомы, но обезьянья оспа у людей протекает значительно легче. Симптомы включают высокую температуру, головную боль, миалгию, боль в спине, озноб, истощение, увеличение лимфоузлов и характерные высыпания. В большинстве случаев обезьянья оспа - самокупируемое заболевание, и ее симптомы исчезают через 2-4 недели после начала заболевания, но в некоторых случаев возникают осложнения, которые включают вторичные инфекции, бронхопневмонию, заражение крови, энцефалит, а также инфекцию роговицы, которая может привести к потере зрения. Летальность варьируется от 0 до 11%. Наибольший уровень смертности наблюдается среди детей младшего возраста.
Зоонозная передача вируса может происходить при прямом контакте с кровью, биологическими жидкостями или пораженными участками кожи или слизистой зараженного животного. В Африке признаки инфицирования оспой обезьян были обнаружены у целого ряда животных, в том числе полосатых белок, древесных белок, гамбийских крыс, соней, а также различных видов обезьян. К настоящему моменту естественный резервуар вируса не выявлен, однако с наибольшей долей вероятности им выступают грызуны.
Передача инфекции от человека к человеку происходит воздушно-капельным путем и при тесном контакте с кожными поражениями инфицированного или предметами, недавно загрязненными вирусными частицами. Так как для передачи инфекции воздушно-капельным путем обычно требуется длительный контакт «лицом к лицу», в зоне повышенного риска находятся медицинские работники, домочадцы и другие люди, имевшие тесный физический контакт с инфицированными людьми. Вирус также может передаваться от матери к плоду через плаценту (что может привести к врожденной оспе обезьян) или при тесном контакте во время или после родов. Распространение обезьяньей оспы в человеческой популяции объясняться общим снижением иммунитета к вирусу из-за прекращения вакцинации от натуральной оспы.
В течение 1-3 дней (иногда позже) после появления лихорадки у больного появляется сыпь, часто начинающаяся на лице, затем распространяющаяся на другие части тела. Поражает лицо (в 95% случаев), ладони и подошвы стоп (в 75% случаев). Поражаются также слизистые оболочки полости рта (в 70% случаев), половых органов (30%) и конъюнктивы (20%), а также роговица. Сыпь развивается последовательно от пятен (поражения с плоским основанием) к папулам, везикулам, пустулам и, наконец, коркам, которые подсыхают и отпадают. Количество поражений колеблется от нескольких до нескольких тысяч. В тяжелых случаях поражения могут слоиться до тех пор, пока не отслоятся большие участки кожи.
Диагностируют оспу обезьян по наличию вирусной ДНК в биологических образцах методом ПНР. В качестве анализируемых образцов могут выступать смывы с участков кожных поражений, соскоб со дна пузырьков, корочки, мазки с задней стенки глотки или миндалин, образцы крови (с ЭДТА), аутопаты легких, печени, почек и селезенки (https://www.who.int/ru). Метод диагностики предусматривает экстракцию ДНК из биологических образцов с последующим проведением полимеразной цепной реакции для целевых фрагментов ДНК генома вируса обезьяньей оспы с использованием набора праймеров и зонда, комплементарных участку гена анкирин-подобного белка вируса обезьяньей оспы.
Однако не существует готовых наборов для выявления ДНК вируса обезьяньей оспы вида monkeypox методом ПНР в реальном времени. В настоящее время пандемия обезьяньей оспой объявлена чрезвычайной ситуацией мирового масштаба. Для того чтобы сдержать распространения болезни, требуются инструменты для своевременной диагностики заболевших, а также для выявления носителей вируса оспы обезьян еще до появления первых симптомов. Создание такого инструмента и является целью нашего изобретения.
Технической задачей изобретения является разработка высокочувствительного способа идентификации ДНК вируса обезьяньей оспы в биологических образцах и других вируссодержащих пробах (культуральная вируссодержащая жидкость, и т.д.).
Поставленная задача решалась путем: конструирования диагностических праймеров и флуоресцентно-меченного зонда на специфические для вируса оспы обезьян фрагменты ДНК; конструирования рекомбинантной плазмидной ДНК, несущей специфический участок ДНК-матрицы; оптимизации концентраций компонентов реакционной смеси и условий проведения ПЦР.
Авторами предложен способ выявления вируса обезьяньей оспы, согласно которому выделенную из биологических образцов ДНК анализируют в одной пробирке при помощи полимеразной цепной реакции, при помощи олигонуклеотидных праймеров и соответствующего флуоресцентно меченого зонда.
Анализ результатов проводят с помощью программного обеспечения используемого прибора для проведения ПЦР с гибридизационно-флуоресцентной детекцией в режиме «реального времени». Результаты интерпретируют на основании наличия или отсутствия пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию или отсутствию значения порогового цикла Ct, причем результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию.
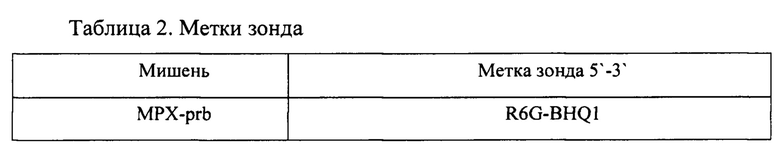
На начальном этапе были подобраны и синтезированы пара специфических олигонуклеотидных праймеров и зонд для гибридизационно-флуоресцентной детекции продуктов ПЦР. Для этого в базе данных NCBI Mega BLAST (http://www.ncbi.nlm.nih. gov/) был выбран наиболее консервативный участок генома вируса обезьяньей оспы. Были проанализированы все имеющиеся в базе данных последовательности. Также были подобраны праймеры и флуоресцентный зонд для ВКО в качестве внутреннего контроля ПНР. Последовательности олигонуклеотидных праймеров и флуоресцентного зонда представлены в Таблице 1 и Таблице 2.

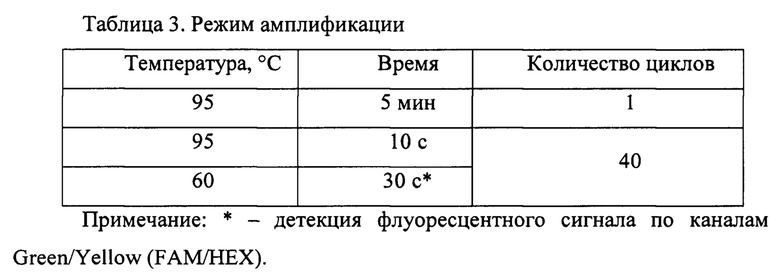
Сущность изобретения поясняется чертежом, где на Фиг. 1 представлены кривые флуоресценции, отражающие динамику образования продукта реакции при анализе контрольных образцов. Каждая кривая выше пороговой линии отображает положительный результат на выявление в образце ДНК вируса обезьяньей оспы, каждая кривая ниже пороговой линии отображает отрицательные контроли, не содержащие ДНК вируса.
Подбор и анализ свойств олигонуклеотидных праймеров и флуоресцентного зонда проводился с использованием программного обеспечения Oligonucleotide Properties Calculator и MFold.
Для контроля качества прохождения ПНР в состав методики были введены рекомбинантные положительные контрольные образцы K+ и ПКО и внутренний контрольный образец (ВКО). Матрицу для создания рекомбинантных положительных контрольных образцов получали синтетическим методом на основе ампликона, включающего в себя диагностическую область-мишень и фланкирующие последовательности нуклеотидов. Ампликон получали методом ПЦР в два шага. Конечный ПНР-продукт лигировали в плазмидный вектор pGEM-T («Promega», USA) и трансформировали им Escherichia coli (штамм XLl-Blue). Рекомбинантные плазмиды из индивидуальных клонов проверяли на правильность ориентации целевой последовательности и отсутствие мутаций в области посадки праймеров и зонда. Проверку осуществляли методом секвенирования по Сэнгеру с помощью прибора для автоматического капиллярного секвенирования ABI PRISM 3500x1 («Applied Biosystems», США).
Соответствующие заданным критериям рекомбинантные плазмиды использовали для приготовления положительных контрольных образцов этапа ПЦР (К+) и выделения (ПКО). Для этого определяли концентрацию ДНК в растворе рекомбинантной плазмиды и разводили стерильной водой до рабочей концентрации 1*107 копий/мл для K+, и 1*105 копий/мл для ПКО.
Оценку аналитической чувствительности метода производили на разведениях раствора рекомбинантной плазмиды, которую проводили через все стадии выделения с помощью набора для выделения нуклеиновых кислот «Рибо-Преп» (ФБУН ЦНИИ Эпидемиологии, Россия), а затем с помощью специфических праймеров и флуоресцентных зондов определяли минимальное разведение, детектируемое как положительное в 100% случаев.
Определенная таким образом аналитическая чувствительность метода составила 1*102 копий/мл.
Свойство изобретения специфически определять ДНК вируса обезьяньей оспы достигнуто путем подбора праймеров и зондов к высококонсервативным фрагментам ДНК вируса, что минимизирует возможность перекрестных реакций с геномом человека, близкородственных микроорганизмов, других инфекционных возбудителей.
Аналитическую специфичность оценивали при помощи тестирования образцов генома человека и следующих микроорганизмов: вирус коровьей оспы, вирус ветряной оспы, вакцинный штамм. Неспецифические реакции отсутствуют.
Таким образом, в результате проведенных исследований был разработан и апробирован способ выявления ДНК вируса обезьяньей оспы в различных видах биологического материала. Технический результат достигается путем определения ДНК вируса, включающим выделение ДНК исследуемой пробы и проведение ПЦР с учетом результатов в режиме реального времени, согласно изобретению.
Диагностика проводится следующим образом: Подготовку проб проводят согласно МУ 1.3.2569-09 «Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I-IV групп патогенности». Материалом для исследования могут служить: клинические и биологические образцы.
Экстракция производится из 100 мкл полученной суспензии образца с лизирующим буфером на основе 6 моль гуанидинизотиоцианата в объеме, указанном в инструкции к набору для выделения нуклеиновых кислот, и последующим инкубированием 5 минут при температуре (65±1)°С. Выделение ДНК осуществляют с помощью наборов «Рибо-преп» и «Рибо-Сорб» (ФБУН ЦНИИ Эпидемиологии, Россия) в соответствии с инструкциями к наборам. Во все пробирки, включая контроль выделения, до этапа прогревания в лизирующем буфере добавляют 10 мкл внутреннего контрольного образца (ВКО). После выделения ДНК приступают к постановке ПЦР.
Для упрощения подготовки и стандартизации выполнения анализа в тест-систему входят 7 реактивов:
1) Amp 1 МРХ, представляющий собой буферный раствор 100 mМ Трис-HCl, рН 8,5, 100 mМ KСl, 0,4 мМ каждого нуклеозидтрифосфата, 10 мМ MgCl2, 0.1 ед. акт./мкл HS-Taq ДНК-полимеразы, 0,025% Tween 20, стабилизаторы Taq ДНК-полимеразы;
2) Amp 2 МРХ - содержит олигонуклеотидные праймеры и флуоресцентно-меченые олигонуклеотидные зонды к ДНК вируса обезьяньей оспы и к ВКО;
3) K+- представляет собой смесь двух плазмидных ДНК:
a) Плазмида, содержащая фрагмент вируса обезьяньей оспы;
b) Плазмида, содержащая фрагмент ВКО;
с которых, при помощи реактива Amp 2 МРХ, амплифицируются специфические фрагменты;
4) K- - представляет собой дистиллированную стерильную воду;
5) ПКО - плазмида, содержащая фрагмент вируса обезьяньей оспы, который амплифицируются при помощи реактива Amp 2 МРХ;
6) ОКО - дистиллированная стерильная вода;
7) ВКО - представляет собой плазмиду, содержащую фрагмент ВКО, которая амплифицируются при помощи реактива Amp 2 МРХ.
Все реактивы хранятся при температуре -20°С.
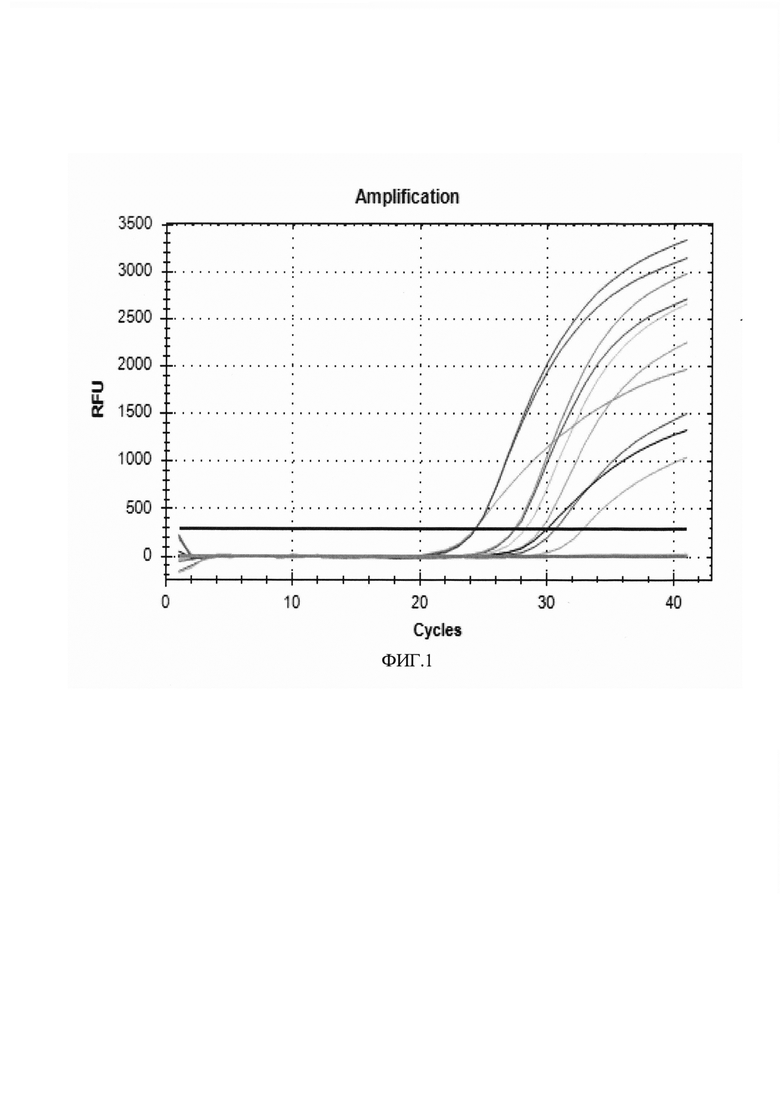
Для постановки реакции ПЦР в реальном времени нужно взять необходимое количество микропробирок объемом 0,2 мл, соответствующее числу исследуемых проб; плюс четыре пробирки для положительных и отрицательных контролей. Подготовить ПЦР-смесь из расчета на одну реакцию: 12,5 мкл реактив Ampl МРХ, 2,5 мкл реактив Аmр2 МРХ. По 15 мкл полученной смеси вносят в пробирки, затем добавляют в каждую по 10 мкл ДНК-пробы, экстрагированной из исследуемого материала. Готовят 4 контрольные реакции. Для этого в пробирку для положительного контроля ПЦР вносят 10 мкл K+, в пробирку для положительного контроля экстракции вносят 10 мкл образца, экстрагированного из ПКО. В пробирку для отрицательного контроля ПЦР вносят 10 мкл K-, в пробирку для отрицательного контроля экстракции вносят 10 мкл пробы, экстрагированной из ОКО. Конечный объем реакционной смеси составляет 25 мкл. Микропробирки переносят в программируемый амплификатор с функцией детекции флуоресценции в режиме «реального времени» по каналам (FAM/SybrGreen/Green и JOE/HEX/Yellow). Режим амплификации представлен в таблице 3.
Детекцию флуоресцентного сигнала производят при 60°С по каналам Green (FAM) и Yellow (JOE/HEX). Учет и анализ результатов проводят с помощью программного обеспечения прибора на основании отсутствия или наличия сигнала флуоресценции в исследуемых пробах по детектируемым каналам. Результаты интерпретируют на основании наличия (или отсутствия) пересечения кривой флуоресценции с установленной на соответствующем уровне пороговой линией, что соответствует наличию (или отсутствию) значения порогового цикла Ct в соответствующей графе в таблице результатов. Результат считают положительным в случае, если кривая накопления флуоресценции для соответствующего образца имеет характерную «сигмовидную» форму и пересекает пороговую линию. Накопление флуоресцентного сигнала по каналу HEX свидетельствует о наличии в исследуемом материале ДНК вируса обезьяньей оспы. При этом результат считается валидным, если параллельно в пробе по каналу FAM регистрируется накопление флуоресцентного сигнала ВКО, а в образцах K+ и ПКО - по каналу HEX. Сигнал по каналу HEX в образцах K- и ОКО должен отсутствовать.
Таким образом, заявляемый способ выявления позволяет достоверно выявлять ДНК вируса обезьяньей оспы в биологических образцах.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="ru"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="21032023MPX.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2023-03-21">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>00000</ApplicationNumberText>
<FilingDate>2023-03-13</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>00000</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022128990</ApplicationNumberText>
<FilingDate>2022-11-08</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки "Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия
человека"(ФБУН НИИ эпидемиологии и микробиологии имени
Пастера),ул.Мира,14,Санкт-Петербург,197101,Российская Федерация
</ApplicantName>
<ApplicantNameLatin>Saint-Petersburg Pasteur Institute
</ApplicantNameLatin>
<InventorName languageCode="ru">Долгова Анна Сергеевна
</InventorName>
<InventorNameLatin>Dolgova Anna Sergeevna </InventorNameLatin>
<InventionTitle languageCode="ru">Способ выявления вируса обезьяньей
оспы вида Monkeypox методом ПЦР в реальном времени (MPX AMP
PS)</InventionTitle>
<InventionTitle languageCode="en">A method for detecting monkeypox
virus by Real-Time PCR (MPX AMP PS).</InventionTitle>
<SequenceTotalQuantity>3</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atagatagatagaatatgcattagttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная последовательность
</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacttatcgtgaacgtaccatac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>искусственная
последовательность</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location><1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q8">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Rhodamine 6G
(9-[2-(ethoxycarbonyl)phenyl)-N-ethyl-6-(ethylamino)-2,7-dimethyl-3H-x
anthen-3-iminium chloride) fluorophore at
5'end.</INSDQualifier_value>
<NonEnglishQualifier_value>Флуорофор Родамин 6Ж
(9-[2-(этоксикарбонил)фенил]-N-этил-6-(этиламино)-2,7-диметил-3H-ксант
ен-3-иминия хлорид) на 5'-конце.</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>>31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Non fluorescent Black Hole Quencher 1
(2-[N-(2-hydroxyethyl)-4-[[2-methoxy-5-methyl-4-[(4-methyl-2-nitrophen
yl)diazenyl]phenyl]diazenyl]anilino)ethanol at 3' end
.</INSDQualifier_value>
<NonEnglishQualifier_value>Не флуоресцентный тушитель Black
Hole Quencher 1
(2-[N-(2-гидроксиэтил)-4-[[2-метокси-5-метил-4-[(4-метил-2-нитрофенил)
диазенил]фенил]диазенил]анилино]этанол] на 3'-конце
.</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>taccgctatagttactatcgaataacacggt</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ выявления вируса кори методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822430C1 |
| Способ выявления РНК вируса Bandavirus dabieense (SFTSV) методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2831410C1 |
| Способ выявления РНК вируса Хендра вида Hendra henipavirus методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822161C1 |
| Способ выявления РНК вируса геморрагической лихорадки Крым-Конго методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2834908C1 |
| Способ выявления вируса Nipah методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2816270C2 |
| Способ выявления РНК вируса Хунин методом ОТ-ПЦР в реальном времени | 2023 |
|
RU2822164C1 |
| Способ выявления РНК вируса Mammarenavirus guanaritoense методом ОТ-ПЦР в реальном времени | 2024 |
|
RU2839982C1 |
| Способ выявления РНК модифицированного вакцинного полиовируса типа 2 (nOPV2) методом ОТ-ПЦР в реальном времени | 2022 |
|
RU2795703C1 |
| Набор для выявления вируса SARS-CoV методом ОТ-ПЦР в реальном времени | 2020 |
|
RU2744198C1 |
| Способ выявления вируса клещевого энцефалита методом ОТ-ПЦР в реальном времени | 2019 |
|
RU2744187C1 |
Изобретение относится к биотехнологии и медицине. Описан способ выявления вируса обезьяньей оспы у здоровых лиц, диагностики лиц с подозрением на оспу, и у людей, больных оспой обезьян. Диагностику проводят с использованием ДНК, праймеров и зонда: ATAGATAGATAGAATATGCATTAGTTC, TACTTATCGTGAACGTACCATAC, R6G-TACCGCTATAGTTACTATCGAATAACACGGT-BHQ1. Изобретение позволяет осуществлять эпидемиологический мониторинг, а также для решения научно-исследовательских задач по изучению свойств вируса обезьяньей оспы. 1 ил., 3 табл.
Способ выявления вируса обезьяньей оспы вида monkeypox методом ПЦР в реальном времени, предусматривающий экстракцию ДНК из биологических образцов, с последующим проведением полимеразной цепной реакции для целевых фрагментов ДНК генома вируса обезьяньей оспы с использованием набора праймеров и зонда, комплементарных участку гена анкирин-подобного белка вируса обезьяньей оспы, отличающийся тем, что в качестве праймеров используются последовательности
MPX-f (SEQ ID NO. 1) ata gat aga tag aat atg cat tag ttc
MPX-r (SEQ ID NO. 2) tac tta teg tga acg tac cat ac
а в качестве зонда
MPX-pr (SEQ ID NO. 3) R6G - tac cgc tat agt tac tat cga ata аса egg t - BHQ1.
| НАБОР ОЛИГОНУКЛЕОТИДНЫХ ПРАЙМЕРОВ И ФЛУОРЕСЦЕНТНОМЕЧЕНЫХ ЗОНДОВ ДЛЯ ВИДОСПЕЦИФИЧНОЙ ЭКСПРЕСС-ИДЕНТИФИКАЦИИ ОРТОПОКСВИРУСОВ НА ОСНОВЕ МУЛЬТИПЛЕКСНОЙ ПЦР В РЕАЛЬНОМ ВРЕМЕНИ | 2010 |
|
RU2427648C1 |
| CN 101407850 A, 15.04.2009 | |||
| СПОСОБ ИЗГОТОВЛЕНИЯ РАБОЧИХ ОРГАНОВ ПОЧВООБРАБАТЫВАЮЩИХ МАШИН | 2013 |
|
RU2528687C1 |
Авторы
Даты
2023-09-21—Публикация
2023-03-23—Подача