ОБЛАСТЬ ТЕХНИКИ
[1] Настоящее изобретение относится к новым антисмысловым олигонуклеотидам и их применению, и более конкретно к антисмысловым олигонуклеотидам, нацеленным на ген Cav 3.1, и их применениям для лечения таких состояний, как болезнь Паркинсона, эссенциальный тремор, эпилепсия, депрессия и потеря сознания.
Уровень техники
[2] Болезнь Паркинсона представляет собой расстройство движения, вызванное разрушением дофаминергических нервов в черной субстанции, расположенной в центре ствола головного мозга. Дофамин представляет собой важный нейромедиатор, который действует на базальные ганглии мозга и позволяет нам совершать точные движения по своему желанию. Симптомы болезни Паркинсона становятся очевидными после того, как 60-80 процентов дофаминергических нервов в черной субстанции утрачены, а патологоанатомическое исследование выявляет тельца Леви, вызванные отложением патогенного белка альфа-синуклеина в различных частях мозга и периферических нервах. Болезнь Паркинсона является вторым по распространенности дегенеративным заболеванием головного мозга после болезни Альцгеймера, ее распространенность составляет 1% у людей старше 60 лет, а заболеваемость увеличивается с возрастом.
[3] Основные или классические двигательные характеристики болезни Паркинсона включают брадикинезию, тремор и ригидность, но существует множество других симптомов, связанных с двигательной деятельностью, включая изменения походки и равновесия, контроля движений глаз, речи и глотания, а также контроля мочевого пузыря. У людей с болезнью Паркинсона брадикинезия проявляется снижением амплитуды и скорости движений, затруднением начала движений. Это может влиять на произвольный контроль над многими группами мышц, включая мышцы глаз (приводя к более медленному началу саккадических движений), речевые мышцы (приводя к более мягкой, а иногда и невнятной речи) и мышцы конечностей (приводя к снижению ловкости). Пациенты жалуются на трудности с выполнением задач мелкой моторики, таких как нажатие кнопок, письмо и использование инструментов, а также тремор носит вариабельный характер. У большинства, но не у всех пациентов наблюдаются симптомы тремора (McGregor and Alexandra B. Nelson, Neuron 101: 1042-1056, 2019).
[4] По этиологии болезнь Паркинсона наследуется лишь в 5-10% случаев, при этом большинство случаев носит идиопатический характер. Исследования факторов окружающей среды при болезни Паркинсона указали на такие факторы, как воздействие токсинов, таких как 1-метил-4-фенил-1,2,3,6-тетрагидропиридин (MPTP), пестицидов (ротенон, паракват), тяжелых металлов (марганец, свинец, медь), монооксид углерода, органические растворители, следы металлов и травмы головы.
[5] Точная причина разрушения дофаминергических нейронов черной субстанции до сих пор неизвестна.
[6] С конца 1960-х годов и по сей день для лечения болезни Паркинсона применяется средство леводопа, которое стало не только прорывом в лечении, но и до сих пор признается препаратом с наибольшим противопаркинсоническим действием. Сегодня при правильном лечении люди с болезнью Паркинсона могут поддерживать хорошее качество жизни в течение многих лет, а уровень смертности снизился. Однако длительное лечение леводопой связано со многими проблемами по мере прогрессирования заболевания: примерно у 75% пациентов наблюдаются двигательные флуктуации, дискинезии и другие осложнения после более чем шести лет применения леводопы (Imet al., J. Korean Neurol. Assoc. 19(4): 315-336, 2001).
[7] Недавно авторы настоящего изобретения показали, что оптогенетическая фотостимуляция ингибирующих входов базальных ганглиев от бледного шара головного мозга индуцирует потенциалы действия в вентролатеральных нейронах таламуса и мышечные сокращения в период после ингибирования, и эти потенциалы действия и мышечные сокращения уменьшаются за счет уменьшение популяции нейронов, демонстрирующих пост-ингибирующий рикошетный импульс за счет локализованного нокаута кальциевых каналов Т-типа в вентральном таламусе, тем самым связывая кальциевые каналы Т-типа с патологией болезни Паркинсона (Kim et al., Neuron 95: 1181-1196, 2017).
[8] Потенциалзависимые кальциевые каналы ответственны за увеличение внутриклеточной концентрации кальция в ответ на активность нейронов (Tsien, R.W., Annu. Rev. Physiol. 45: 341-358, 1983) и подразделяются на высокопороговые и низкопороговые каналы на основании их зависимости от потенциала (Tsien, R.W. et al., Trends Neurosci. 18: 52-54, 1995). Кальциевые каналы Т-типа являются типичными низкопороговыми кальциевыми каналами, и у млекопитающих существует три типа: Cav3.1 (α1G), Cav3.2 (α1H) и Cav3.3 (α1I), в зависимости от генотипа субъединицы α1 (Perez-Reyes, E., Physiol. Rev. 83: 117-161, 2003). Среди них кальциевые каналы α1G участвуют в генерации вспышки активности нейронов в таламическом ядре и, как недавно было показано, выполняют важные патологические функции (Kim, D. et al., Science 302: 117-119, 2003; Kim, D. et al., Neuron 31: 35-45, 2001). Патологии, связанные с кальциевыми каналами α1G, включают боль в животе (Корейский патент № 868735), эпилепсию (Корейский патент № 534556), тревожные расстройства (Корейский патент № 958291), синдром Драве (публикация Корейского патента № 10-2019-0139302), дисфункцию лобных долей после необратимого повреждения головного мозга (публикация Корейского патента № 10-2013-0030011), нейропатическую боль (Choi et al., Proc. Natl. Acad. Sci. U.S.A., 113(8): 2270-2275, 2016; WO2006064981A1), синдром Ангельмана и/или синдром Прадера-Вилли (WO2017070680A1), депрессию (CN108853510A), ревматоидный артрит (WO2020203610A1), расстройства концентрации (Корейский Патент № 1625575), диабет, (CN10264172A), и эссенциальный тремор (Корейский Патент № 958286). Интересно, что трансгенные мыши, у которых нокаутирован кальциевый канал Т-типа Cav3.1 (Cav3.1-/-), не летальны и внешне развиваются нормально.
[9] Для лечения этих патологий необходимы препараты, блокирующие функцию кальциевых каналов Т-типа, но есть несколько проблем. Во-первых, различным блокаторам кальциевых каналов Т-типа, включая этосуксимид, трудно избирательно ингибировать только Cav3.1 из-за высокой гомологии между тремя типами кальциевых каналов Т-типа, описанными выше. Во-вторых, этосуксимид, который используется в клинике под торговой маркой Заронтин®, также ингибирует потенциал-зависимые каналы Na+ и K+, которые важны для генерации нервных сигналов (Lerescheet al., J. Neurosci. 18(13): 4842-4853, 1998), с известными побочными эффектами, включая головную боль, головокружение и рвоту. В-третьих, структурное сходство кальциевых каналов приводит к перекрестным помехам между ингибиторами кальциевых каналов и другими типами кальциевых каналов, такими как каналы N-типа и P/Q-типа, которые важны для высвобождения нейромедиаторов во всех нервах и, при блокировке, вызывают значительные нарушения мозговых и жизненных функций. В-четвертых, даже блокаторы, селективные в отношении каналов Cav3.1, могут влиять на функцию Cav3.1, экспрессируемых в сердечных нервах, что приводит к нарушениям сердечной функции (Mangoniet al., Circ. Res. 98: 1422-1430, 2006).
[10] По этой причине были разработаны лекарственные средства на основе молекул нуклеиновой кислоты, такие как киРНК и кшРНК, которые нацелены только на ген Cav3.1, но у них есть серьезные проблемы при реальном клиническом применении, такие как необходимость введения в отдельный вектор, такой как лентивирус и необходимость локальной и точной инъекции в нужную область мозга с помощью высокоинвазивного устройства, такого как стереотаксический инжектор.
[11] Поэтому существует острая необходимость в разработке нового класса препаратов, которые избирательно ингибируют только кальциевые каналы α1G Т-типа (Cav3.1) без других побочных эффектов.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
ТЕХНИЧЕСКАЯ ПРОБЛЕМА
[12] Соответственно, настоящее изобретение направлено на решение описанных выше проблем, и целью настоящего изобретения является создание лекарственного средства, которое более эффективно ингибирует экспрессию кальциевых каналов α1G Т-типа, обеспечивая тем самым терапевтический агент для ингибирования кальциевых каналов α1G Т-типа, например, при болезни Паркинсона. Однако эти цели являются иллюстративными и не ограничивают объем настоящего изобретения.
ТЕХНИЧЕСКОЕ РЕШЕНИЕ
[13] В одном из аспектов настоящего изобретения предложен антисмысловой олигонуклеотид, нацеленный на последовательность нуклеиновой кислоты, представленную SEQ ID NO: 1, для ингибирования экспрессии гена Cav3.1.
[14] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения болезни Паркинсона, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[15] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения депрессии, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[16] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения эссенциального тремора, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[17] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения эпилепсии, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[18] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения тревожных расстройств, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[19] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для восстановления сознания у пациента, находящегося в бессознательном состоянии, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[20] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего болезнью Паркинсона, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[21] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего депрессией, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[22] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего эссенциальным тремором, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[23] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего эпилепсией, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[24] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего тревожным расстройством, включающий введение пациенту, страдающему тревожным расстройством, терапевтически эффективного количества антисмыслового олигонуклеотида.
[25] В другом аспекте настоящего изобретения предложен способ восстановления сознания у пациента, находящегося в бессознательном состоянии, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
ПРЕИМУЩЕСТВЕННЫЕ ЭФФЕКТЫ
[26] В отличие от обычных блокаторов каналов Т-типа, антисмысловые олигонуклеотиды согласно варианту осуществления настоящего изобретения не только специфически ингибируют экспрессию гена Cav3.1, но также имеют мало побочных эффектов при введении in vivo, что делает их высокоэффективными при лечении различных состояний, связанных с активностью Cav3.1, таких как болезнь Паркинсона, нейропатическая боль, проблемы с вниманием и обучением, а также тревожные расстройства. Однако эффекты настоящего изобретения не ограничиваются этим.
Описание чертежей
[27] ФИГ. 1 представляет собой геномную карту, показывающую структуру геномной ДНК изоформы 13 гена CACNA1G, мишени антисмысловых олигонуклеотидов по настоящему изобретению и целевых местоположений кандидатных антисмысловых олигонуклеотидов, предназначенных для нацеливания на ген CACNA1G.
[28] ФИГ. 2 представляет собой схематическую иллюстрацию структуры гэпмера, антисмыслового олигонуклеотида, полученного в соответствии с одним из вариантов осуществления настоящего изобретения, и типов используемых модификаций.
[29] ФИГ. 3 представляет собой график, показывающий эффективность нокдауна гена CACNA1G, когда клетки трансфецированы кандидатными антисмысловыми олигонуклеотидами согласно одному из вариантов осуществления настоящего изобретения.
[30] ФИГ. 4 представляет собой график, представляющий результаты сравнения степени ингибирования экспрессии гена CACNA1G в зависимости от концентрации обработки, когда клетки обрабатывают кандидатными антисмысловыми олигонуклеотидами согласно одному из вариантов осуществления настоящего изобретения.
[31] ФИГ. 5а представляет собой схематическое изображение экспериментальной схемы анализа токсичности антисмысловых олигонуклеотидов согласно одному из вариантов осуществления настоящего изобретения и их способности регулировать экспрессию гена CACNA1G in vivo; ФИГ. 5b представляет собой график, показывающий выживаемость до 7 дней после введения восьми кандидатных антисмысловых олигонуклеотидов согласно одному из вариантов осуществления настоящего изобретения экспериментальным животным; и ФИГ. 5c представляет собой график, показывающий экспрессию гена CACNA1G в мозге, удаленном у экспериментальных животных, измеренную с помощью кОТ-ПЦР.
[32] ФИГ. 6а представляет собой график, показывающий результаты анализа кОТ-ПЦР, выполненного для измерения перекрестной реактивности антисмысловых олигонуклеотидов согласно одному из вариантов осуществления настоящего изобретения против Cav3.2; и ФИГ.6b представляет собой график, показывающий результаты анализа кОТ-ПЦР, выполненного для измерения перекрестной реактивности антисмысловых олигонуклеотидов согласно одному из вариантов осуществления настоящего изобретения против Cav3.3.
[33] ФИГ. 7а представляет собой схематическое изображение, представляющее схему экспериментов на животных для проверки терапевтического эффекта антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения в отношении болезни Паркинсона; ФИГ. ФИГ.7b представляет собой график, представляющий результаты измерения расстояния перемещения при боковом вращении в тесте в открытом поле, выполненном через неделю после введения антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения; и ФИГ. 7c представляет собой график, представляющий результаты измерения расстояния перемещения в тесте в открытом поле; ФИГ. 7d представляет собой график, показывающий результаты измерения скорости передвижения в тесте в открытом поле; ФИГ. 7e представляет собой график, показывающий результаты измерения времени падения, измеренного путем проведения ускоренного теста с вращающимся стержнем в течение 5 дней с 1-дневными интервалами, после 1 недели введения антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения у животных, как описано выше; и ФИГ. 7f представляет собой серию фигур, показывающих результаты гистохимических анализов, выполненных для определения механизма действия антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения.
[34] ФИГ. 8а представляет собой схематическое изображение, иллюстрирующее график экспериментов на животных для проверки эффективности антисмыслового олигонуклеотида при лечении эссенциального тремора согласно одному из вариантов осуществления настоящего изобретения; и ФИГ. 8b представляет собой схематическое изображение, иллюстрирующее устройство для измерения симптомов тремора, вызванного эссенциальным тремором; ФИГ. 8c представляет собой схематическое изображение, представляющее результаты измерения тяжести тремора в форме спектрограммы после введения антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения; и ФИГ. 8d представляет собой серию графиков, количественно отражающих результаты ФИГ. 8c, где левая панель представляет собой измерение продолжительности тремора, а правая панель представляет собой количественную оценку интенсивности тремора с течением времени.
[35] ФИГ.9a представляет собой схематическую диаграмму, иллюстрирующую схему эксперимента на животных с депрессией, вызванной социальным стрессом, для проверки терапевтического эффекта антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения в отношении хронической устойчивой депрессии, вызванной стрессом; ФИГ.9b представляет собой общую диаграмму, схематически изображающую способ тестирования депрессии, вызванной хроническим социальным поражением, вызванным стрессом, выполняемый в течение 10-дневного курса социального стресса с 5-го по 17-й день после введения антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения; ФИГ. 9c представляет собой серию графиков, представляющих количественную оценку степени хронической депрессии при введении антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения, где левая панель изображает экспрессию мРНК Cav3.1, определенную количественно с помощью кОТ-ПЦР, центральная панель отображает показатель времени социального взаимодействия, а правая панель представляет собой показатель индекса общительности.
[36] ФИГ. 10а представляет собой схематическую диаграмму, иллюстрирующую схему теста принудительного плавания на животных для проверки терапевтического эффекта антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения в отношении кратковременной острой депрессии, вызванной стрессом; и ФИГ. 10b представляет собой график, показывающий количественную оценку шкалы острой депрессии, вызванной вынужденным плаванием, при введении антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения, где на левой панели показана экспрессия мРНК Cav3.1, определенная количественно с помощью кОТ-ПЦР, а правая панель показывает результаты измерения времени неподвижности, которое является мерой стресса.
[37] ФИГ.11a представляет собой схематическую диаграмму, иллюстрирующую схему экспериментов на животных для проверки терапевтического эффекта антисмысловых олигонуклеотидов в отношении эпилепсии согласно одному из вариантов осуществления настоящего изобретения, в котором введение зонда электроэнцефалографии (ЭЭГ) выполняют через 12 дней после введения антисмыслового олигонуклеотида и измеряют ЭЭГ за 30 минут до и после обработки GBL (70 мг/кг), который индуцирует абсанс на 14-й день; ФИГ. 11b представляет собой серию графиков, показывающих измерения ЭЭГ, количественно определяющие степень снижения спайк-волновых разрядов (SWD) в волновых диапазонах ЭЭГ, специфичных для приступов, после введения антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения вышеуказанным животным, где левая панель представляет продолжительность SWD, общее время SWD, а правая панель представляет количество SWD.
[38] ФИГ. 12а представляет собой схематическую диаграмму, иллюстрирующую схему экспериментов на животных для проверки терапевтического эффекта антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения в отношении анестезирующей потери сознания, вызванной арбутином; и ФИГ. 12b представляет собой схематическую диаграмму, показывающую время восстановления после вызванной арбутином потери сознания после введения антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения; ФИГ. 12c представляет собой график, представляющий результаты проверки эффективности лечения потери сознания путем измерения времени восстановления после потери сознания (потеря установочного рефлекса, LORR); и ФИГ. 12d представляет собой схематическую диаграмму, иллюстрирующую другую схему испытаний на животных для подтверждения эффекта пробуждения антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения после обработки этанолом; ФИГ. 12d представляет собой серию графиков, представляющих результаты проверки терапевтического эффекта в отношении потери сознания путем измерения степени пробуждения, вызванного низкими концентрациями этанола, при введении антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения, где левая панель показывает результат измерения скорости экспериментального животного, на центральной панели показан результат измерения пройденного экспериментальным животным расстояния, а на правой панели - результат измерения частоты вращения экспериментального животного; и ФИГ. 12e представляет собой график, показывающий время до потери сознания (задержка LORR) после воздействия высокой концентрации (3. 5 г/кг) этанола.
[39] ФИГ. 13а представляет собой схематическую диаграмму, представляющую схему экспериментов на животных с использованием антисмыслового олигонуклеотида, нацеленного на область вблизи целевой последовательности антисмыслового олигонуклеотида А91, согласно одному из вариантов осуществления настоящего изобретения; ФИГ. 13b представляет собой график, показывающий выживаемость экспериментальных животных до 7 дней после введения различных антисмысловых олигонуклеотидов согласно одному из вариантов осуществления настоящего изобретения; и ФИГ. 13c представляет собой график, показывающий результаты измерения экспрессии Cav3.1 в головном мозге, удаленном у экспериментальных животных, умерщвленных после 7 дней лечения различными антисмысловыми олигонуклеотидами согласно одному из вариантов осуществления изобретения.
ЛУЧШИЙ ВАРИАНТ ОСУЩЕСТВЛЕНИЯ
[40] Определение Терминов:
[41] Используемый в настоящем описании термин «олигонуклеотид» относится к полинуклеотиду, образованному из природного азотистого основания и пентафуранозильной группы (сахара), образующих нуклеозиды, которые связаны между собой природными фосфодиэфирными связями. Таким образом, термин «олигонуклеотид» относится к природным видам или синтетическим видам, образованным из их природных субъединиц или близких гомологов.
[42] Используемый в настоящем описании термин «олигонуклеотид» представляет собой концепцию, которая включает любой нуклеотид, предпочтительно дезоксирибонуклеотид (ДНК), рибонуклеотид (РНК) или гибридную молекулу нуклеиновой кислоты ДНК/РНК, которая представляет собой смесь ДНК и РНК. Этот термин относится только к первичной структуре молекулы; следовательно, этот термин включает двухцепочечную или одноцепочечную ДНК и двухцепочечную или одноцепочечную РНК.
[43] Кроме того, термин «олигонуклеотид» включает сегмент, который является искусственным, а не природным, и выполняет функцию, аналогичную функции природного олигонуклеотида. Олигонуклеотид может иметь сахарную часть, нуклеиновую часть или модифицированную межнуклеотидную связь. Среди возможных модификаций предпочтительными модификациями являются 2'-O-алкильные производные сахарной части, в частности 2'-O-этилоксиметил или 2'-O-метил, и/или фосфоротиоат, фосфородитиоат или метилфосфонат для межнуклеотидного остова.
[44] Используемый в настоящем описании термин «межнуклеозидный линкер» относится к группе, способной ковалентно связывать два нуклеозида, например, между единицами ДНК, между единицами ДНК и нуклеотидными аналогами, между двумя единицами, не являющимися LNA, между единицей, не являющейся LNA, и единицей LNA или между двумя единицами LNA. Во встречающихся в природе молекулах нуклеиновой кислоты в качестве такого межнуклеозидного линкера используется фосфодиэфир, но такой фосфодиэфир можно заменить аналогичными линкерами, такими как фосфоротиоат, как описано выше. Такой линкер может представлять собой любой один или более, выбранных из группы, состоящей из -O-P(O)2-O-, -O-P(O, S)-O-, -O-P(S)2-O-, -S-P(O)2-O-, -S-P(O, S)-O-, -S-P(S)2-O-, -O-P(O)2-S-, -O-P(O, S)-S-, -S-P(O)2-S-, -O-PO(R)-O-, O-PO(OCH3)-O-, -O-PO(NR)-O-, -O-PO(OCH2CH2S-R)-O-, -O-PO(BH3)-O-, -O-PO(NR)-O-, -O-P(O)2-NR-, -NR-P(O)2-O-, -NR-CO-O-, -NR-CO-NR-, -O-CO-O-, -O-CO-NR-, -NR-CO-CH2-, -O-CH2-CO-NR-, -O-CH2-CH2-NR-, -CO-NR-CH2-, -CH2-NR-CO-, -OCH2-CH2-S-, -S-CH2-CH2-O-, -S-CH2-CH2-S-, -CH2-SO2-CH2-, -CH2-CO-NR-, -O-CH2-CH2-NR-CO-, -CH2-NCH3-O-CH2-, где R представляет собой водород или алкильную группу, имеющую от 1 до 4 атомов углерода.
[45] Используемый в настоящем описании термин «нацеленный» означает способность к комплементарной гибридизации со специфической последовательностью нуклеиновой кислоты.
[46] Используемые в настоящем описании термины «способный к гибридизации» или «гибридизация» обычно используются для обозначения образования водородных связей, также известных как связи Уотсона-Крика, между комплементарными основаниями на двух цепях нуклеиновой кислоты с образованием дуплекса или триплекса двойной спирали (если олигонуклеотид двухцепочечный).
[47] Используемый в настоящем описании термин «ген, кодирующий Cav3.1» относится к геномной последовательности cacna1g, которая включает интроны и экзоны гена, кодирующего подтип α1G кальциевого канала Т-типа. Известно, что ген cacna1g расположен на хромосоме 17 человека и имеет размер 66 760 п.о., согласно эталонной последовательности NCBI: NC_000017.11.
[48] Используемый в настоящем описании термин «продукт гена, кодирующего Cav3.1» относится к последовательности мРНК, полученной путем транскрипции из геномной последовательности cacna1g. Указанная мРНК может содержать транскрипты как до, так и после сплайсинга.
[49] Антисмысловой олигонуклеотид согласно одному из вариантов осуществления изобретения предпочтительно гибридизуется специфически с геном, кодирующим Cav3.1 или его продуктом. Антисмысловые олигонуклеотиды по настоящему изобретению могут гибридизоваться с ДНК генов, кодирующих Cav3.1, и/или мРНК, полученной из этого гена. В частности, антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения может нацеливаться на нуклеотидную последовательность, описанную SEQ ID NO: 1, которая включает области интрона 37 и экзона 38 мРНК, кодирующей Cav3.1. Другими словами, антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения может ингибировать экспрессию Cav3.1 путем гибридизации с мРНК как до, так и после сплайсинга, кодирующей Cav3.1, при условии, что местоположение является подходящим. Антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения содержат нуклеотиды, которые достаточно идентичны для специфичной гибридизации.
[50] Используемый в настоящем описании термин «специфическая гибридизация» означает, что существует достаточная степень комплементарности, чтобы избежать неспецифической гибридизации олигонуклеотида с нецелевыми последовательностями, особенно в условиях, требующих специфической гибридизации. Для специфичной гибридизации олигонуклеотид не обязательно должен быть на 100% комплементарен целевой последовательности нуклеиновой кислоты. В частности, олигонуклеотид, имеющий степень комплементарности по меньшей мере примерно 80%, может специфически гибридизоваться с нуклеиновой кислотой, выбранной в качестве мишени.
[51] Используемые в настоящем описании термины «единица LNA» или «мономер LNA», «остаток LNA», «единица замкнутой нуклеиновой кислоты», «мономер замкнутой нуклеиновой кислоты» или «остаток замкнутой нуклеиновой кислоты» относятся к аналогу бициклического нуклеозида. Единицы LNA подробно описаны в WO1999014226A, WO2000056746A, WO2000056748A, WO200125248A, WO2002028875A, WO2003006475A и WO2003095467A. Единицу LNA также можно определить по ее химической формуле. Следовательно, используемый в настоящем описании термин «единица LNA» означает соединение, формула которого представлена следующей структурной формулой:
[52] 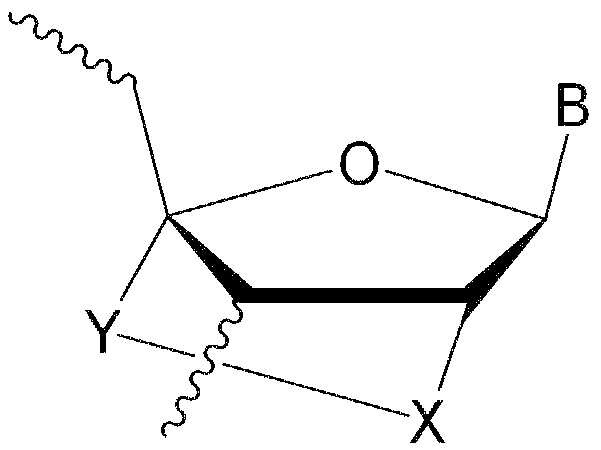 (Структурная формула 1a) или
(Структурная формула 1a) или
[53] 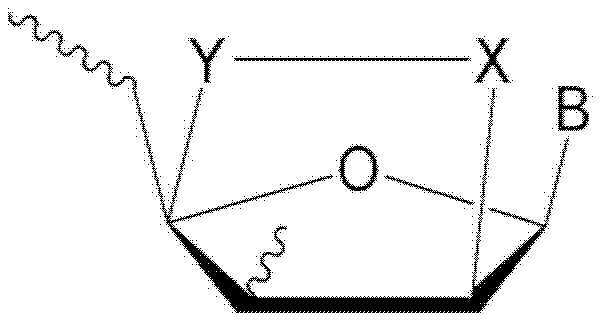 (Структурная формула 1b)
(Структурная формула 1b)
[54] (В приведенной выше структурной формуле X выбран из группы, состоящей из O, S и NR, R представляет собой H или алкильную группу, имеющую от 1 до 4 атомов углерода, и Y представляет собой CH2).
[55] В приведенных выше структурных формулах 1a и 1b, когда X представляет собой S, его называют «единицей тио-LNA», когда X представляет собой NR, его называют «единицей амино-LNA», а когда X представляет собой O , его называют «единицей окси-LNA».
[56] Используемый в настоящем описании термин «гэпмерный антисмысловой олигонуклеотид» относится к короткому одноцепочечному антисмысловому олигонуклеотиду, содержащему центральную последовательность ДНК между последовательностями LNA на обоих концах, который поглощается клеткой без агента трансфекции и препятствует экспрессии целевой мРНК посредством индукция активации РНКазы Н посредством процесса, называемого гимнозом (gymnosis) (Hvamet al., Mol. Ther. 25: 1710-1717, 2017). Таким образом, это своего рода гибридная молекула нуклеиновой кислоты, в которой LNA в виде РНК и ДНК связаны друг с другом. LNA на обоих концах делает ее устойчивой к нуклеазам и обладает высокой аффинностью к целевой последовательности, в то время как последовательность ДНК внутри действует как сайт активации РНКазы H, вызывая специфическую деградацию РНК в гибриде ДНК/РНК. Обычно LNA на конце «носка» имеет длину от 3 до 5 нт, а ДНК в центре имеет длину примерно 10 нт. Однако в одном из вариантов осуществления настоящего изобретения РНК на обоих концах антисмыслового олигонуклеотида может использовать в качестве основания тимин (Т) вместо урацила (U).
[57] Подробное описание изобретения:
[58] В одном из аспектов настоящего изобретения предложен антисмысловой олигонуклеотид, нацеленный на последовательность нуклеиновой кислоты, представленную SEQ ID NO: 1, для ингибирования экспрессии гена, кодирующего Cav3.1.
[59] Антисмысловой олигонуклеотид представляет собой молекулу нуклеиновой кислоты, способную гибридизоваться с последовательностью нуклеиновой кислоты, представленной SEQ ID NO: 1, и может иметь длину, например, от 11 до 30 нуклеотидов или от 12 до 29 нуклеотидов. В предпочтительном варианте осуществления антисмысловой олигонуклеотид по настоящему изобретению может иметь длину от 13 до 28 нт, более предпочтительно длину от 14 до 27 нт, от 15 до 26 нт, от 16 до 25 нт, от 17 до 24 нт, от 18 до 23 нт или от 19 до 22 нт. Более конкретно, антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения может иметь длину 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29 или 30 нт.
[60] Антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения может представлять собой молекулу нуклеиновой кислоты длиной от 7 до 30 нт. Последовательность нуклеиновой кислоты SEQ ID NO: 1 содержит экзон 38 гена, кодирующего Cav3.1, и прилегающую к нему 3'-концевую часть интрона 37. Предпочтительно антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения может содержать последовательность нуклеиновой кислоты, обозначенную SEQ ID NO: 20 или SEQ ID NO: 21.
[61] Антисмысловой олигонуклеотид может представлять собой молекулу гибридной нуклеиновой кислоты ДНК, РНК или ДНК/РНК, в которой некоторая часть или весь фосфодиэфирный (РО) остов заменен другой формой, например фосфоротиоатом (PS), фосфородитиоатом или метилфосфонатом, некоторая часть или вся 2'-ОН часть сахара может быть модифицирована, примеры таких модификаций 2'-ОН включают нуклеотидные аналоги, замещенные -O-CH3 (-OMe), -O-CH2-CH3 (-OEt), -O-CH2-CH2-O-CH3 (-MOE), -O-CH2-CH2-CH2-NH2, -O-CH2-CH2-CH2-OH, -OF или -F.
[62] Необязательно, антисмысловой олигонуклеотид согласно одному из вариантов осуществления изобретения может содержать нуклеотидный аналог, в котором сахарная часть модифицирована, например, так называемая единица замкнутой нуклеиновой кислоты (LNA), в которой на сахарной части образуется дополнительное кольцо. Такие LNA описаны выше. По меньшей мере примерно 30% от общего количества нуклеотидов могут быть нуклеотидными аналогами, например, по меньшей мере, примерно 33%, например, по меньшей мере, примерно 40%, например, по меньшей мере, примерно 50%, например, по меньшей мере, примерно 60%, например, по меньшей мере примерно 66%, например, по меньшей мере, примерно 70%, например, по меньшей мере, примерно 80% или, например, по меньшей мере, примерно 90%. Антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения может содержать нуклеотидную последовательность, содержащую только последовательности нуклеотидных аналогов.
[63] Модификация остова и сахарной части таких олигонуклеотидов предназначена для улучшения стабильности и фармакокинетических свойств лекарственного средства за счет повышения стабильности гибридизации с целевой последовательностью или за счет ингибирования значительного уменьшения периода полувыведения в организме вследствие деградации нуклеазами и т. п.
[64] Предпочтительные модификации изобретения включают химерные олигонуклеотиды. Олигонуклеотиды содержат по меньшей мере две химически различных области, каждая из которых содержит один или более нуклеотидов. В частности, они содержат одну или более областей, содержащих модифицированные нуклеотиды, придающие одно или более благоприятных свойств, таких как лучшая биологическая стабильность, повышенная биодоступность, повышенная клеточная интернализация или повышенная аффинность к целевой РНК.
[65] Степень комплементарности между двумя последовательностями нуклеиновых кислот одинаковой длины определяют путем выравнивания двух последовательностей и сравнения последовательностей, которые комплементарны первой и второй последовательностям. Степень комплементарности рассчитывают путем определения количества идентичных положений между двумя последовательностями, нуклеотиды которых сравниваются, деления числа идентичных положений на общее количество положений и умножения полученного результата на 100 для получения степени комплементарности между этими двумя последовательностями.
[66] Антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения может представлять собой гэпмер, причем указанный гэпмер представляет собой молекулу нуклеиновой кислоты, содержащую единицу РНК длиной 3-5 нуклеотидов на каждом конце и ДНК длиной 8-12 нуклеотидов, расположенную между единицами РНК и LNA. В этом случае указанная единица РНК может содержать по меньшей мере одну LNA.
[67] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения болезни Паркинсона, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[68] Связь болезни Паркинсона с Cav3.1 хорошо описана в уровне техники (Park et al., Front. Neural Circuits, 7: 172, 2013; Rubitet al., Eur. J. Neurosci: 36(2): 2213-2228, 2012; Xiang et al., ASC Chem. Neurosci. 2(12): 730-742, 2012). Кроме того, авторы настоящего изобретения экспериментально продемонстрировали, что антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения, которые специфически связываются с Cav3.1, особенно с областью, включающей интрон 37-экзон 38, ингибируют экспрессию Cav3.1, но без других побочных эффектов, могут значительно облегчить аномальные мышечные симптомы у животных на модели болезни Паркинсона. Таким образом, антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения, которые могут специфически ингибировать экспрессию Cav3.1, могут быть высокоэффективным терапевтическим средством при лечении болезни Паркинсона.
[69] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения депрессии, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[70] Недавние исследования показали, что кальциевые каналы Т-типа коррелируют с поведением, связанным с тревожностью. В частности, сообщалось, что открытие кальциевых каналов Т-типа вызывает поведение, связанное с тревожностью, и, наоборот, введение блокаторов кальциевых каналов Т-типа уменьшает симптомы тревожности (Kaur et al., J. Basic Clin. Physiol. Pharmacol., 31(3):20190067, 2020). Более того, авторы настоящего изобретения экспериментально подтвердили, что симптомы, связанные с депрессией, облегчаются у трансгенных мышей, у которых нокаутирован ген Cav3.1. Таким образом, антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения, нацеленные на ген Cav3.1, можно использовать для лечения депрессии. Кроме того, авторы изобретения продемонстрировали в исследованиях на животных, что антисмысловые олигонуклеотиды в одном варианте осуществления настоящего изобретения эффективны при лечении депрессии.
[71] Согласно одному аспекту настоящего изобретения предложена фармацевтическая композиция для лечения эссенциального тремора, содержащая указанный антисмысловой олигонуклеотид в качестве активного ингредиента.
[72] Авторы настоящего изобретения показали, что эссенциальный тремор можно лечить путем специфического ингибирования Cav3.1 (субъединицы α1G кальциевого канала Т-типа) (Корейский патент № 958286). Следовательно, антисмысловой олигонуклеотид, способный специфически ингибировать экспрессию Cav3.1, согласно одному из вариантов осуществления настоящего изобретения, может быть использован для лечения эссенциального тремора. Кроме того, авторы изобретения продемонстрировали в исследованиях на животных, что антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения можно использовать для лечения эссенциального тремора.
[73] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения эпилепсии, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[74] Известно, что блокаторы кальциевых каналов Т-типа, которые специфически ингибируют кальциевые каналы Т-типа, уменьшают активацию таламуса и тем самым подавляют симптомы судорог (Powell et al., Br. J. Clin. Pharmacol., 77: 729-739, 2014; Casillas-Espinosa et al., PLoS One, 10: 30130012, 2015). Таким образом, антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения можно использовать для лечения эпилепсии. Кроме того, авторы изобретения продемонстрировали в исследованиях на животных, что антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения можно использовать для лечения эпилепсии.
[75] В фармацевтической композиции эпилепсия может представлять собой абсанс.
[76] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для лечения тревожных расстройств, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента.
[77] Авторы настоящего изобретения показали, что введение мибефрадила, селективного блокатора кальциевых каналов Т-типа, мышам с нокаутным геном фосфолипазы β4 (PLCβ4), модельному животному тревожных расстройств, нормализует способность к угашению памяти, которая была нарушена у указанных мышей, тогда как введение мибефрадила и эфонидипина MD нормальным мышам быстро гасит воспоминания о страхе у указанных мышей и снижает способность возврата указанных погашенных воспоминаний (Корейский патент № 958291). Таким образом, антисмысловые олигонуклеотиды, способные специфически ингибировать экспрессию Cav3.1, согласно одному из вариантов осуществления настоящего изобретения, могут быть использованы для лечения тревожных расстройств.
[78] В другом аспекте настоящего изобретения предложена фармацевтическая композиция для восстановления сознания у пациента, находящегося в бессознательном состоянии, содержащая антисмысловой олигонуклеотид в качестве активного ингредиента. Авторы настоящего изобретения экспериментально продемонстрировали, что введение антисмысловых олигонуклеотидов в одном варианте осуществления настоящего изобретения экспериментальным животным, у которых была вызвана потеря сознания, способствует восстановлению сознания. Таким образом, антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения можно использовать в качестве лекарственного средства для восстановления сознания у пациентов без сознания, включая пациентов, находящихся в коме.
[79] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего болезнью Паркинсона, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[80] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего депрессией, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[81] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего эссенциальным тремором, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[82] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего эпилепсией, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[83] В указанном способе указанная эпилепсия может представлять собой абсанс.
[84] В другом аспекте настоящего изобретения предложен способ лечения пациента, страдающего тревожным расстройством, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[85] В другом аспекте настоящего изобретения предложен способ восстановления сознания у пациента, находящегося в бессознательном состоянии, включающий введение пациенту терапевтически эффективного количества антисмыслового олигонуклеотида.
[86] Композиция может содержать фармацевтически приемлемый носитель и может дополнительно содержать фармацевтически приемлемые адъюванты, вспомогательные вещества или разбавители в дополнение к носителю.
[87] Используемый в настоящем описании термин «фармацевтически приемлемый» относится к композиции, которая является физиологически приемлемой и обычно не вызывает желудочно-кишечных расстройств, аллергических реакций, таких как головокружение, или подобных реакций при введении людям. Примеры указанных носителей, вспомогательных веществ или разбавителей включают лактозу, декстрозу, сахарозу, сорбит, маннит, ксилит, эритрит, мальтит, крахмал, акациевую камедь, алгинат, желатин, фосфат кальция, силикат кальция, целлюлоза, метилцеллюлозу, поливинилпирролидон, воду, метилгидроксибензоат, пропилгидроксибензоат, тальк, стерат магния и минеральные масла. Они могут дополнительно включать наполнители, антифлокулянты, смазывающие вещества, увлажняющие агенты, ароматизаторы, эмульгаторы и консерванты.
[88] Кроме того, фармацевтическая композиция согласно одному из вариантов осуществления настоящего изобретения может быть приготовлена с использованием способов, известных в данной области техники, обеспечивающих быстрое высвобождение или замедленное или отсроченное высвобождение активного ингредиента при введении млекопитающему. Составы включают порошки, гранулы, таблетки, эмульсии, сиропы, аэрозоли, мягкие или твердые желатиновые капсулы, стерильные растворы для инъекций и стерильные порошковые формы.
[89] Композицию согласно одному из вариантов осуществления настоящего изобретения можно вводить различными путями, но предпочтительно путем интратекальной инъекции, внутричерепной инъекции или интрацеребровентрикулярной инъекции (ICV) через резервуар Оммайя. Учитывая несколько инвазивный характер способа введения, автоматическое введение через заданные интервалы также возможно с помощью дозирующего устройства, содержащего резервуар и насос, подходящим образом соединенных с катетером, вставленным в точку введения. Необязательно, вместо автоматического дозирующего устройства, дозу можно вводить вручную с подходящими интервалами, например, один раз в день или несколько раз в день, один раз в неделю, два раза в неделю, ежемесячно и т. д.
[90] Композиция согласно одному из вариантов осуществления настоящего изобретения может быть приготовлена в любой подходящей форме с обычно используемым фармацевтически приемлемым носителем. Фармацевтически приемлемые носители включают, например, воду, подходящие масла, физиологический раствор, носители для парентерального введения, такие как водный раствор глюкозы и гликоли и т. п., и могут дополнительно включать стабилизаторы и консерванты. Подходящие стабилизаторы включают антиоксиданты, такие как гидросульфит натрия, сульфит натрия или аскорбиновая кислота. Подходящие консерванты включают хлорид бензалкония, метил- или пропилпарабен и хлорбутанол. Композиция по настоящему изобретению может также содержать суспендирующие агенты, солюбилизирующие агенты, стабилизаторы, изотонические агенты, консерванты, антиадсорбенты, поверхностно-активные вещества, разбавители, вспомогательные вещества, регуляторы pH, неактивные агенты, буферные агенты, антиоксиданты и т. п. при необходимости, в зависимости от способа введения или состава. Фармацевтически приемлемые носители и составы, подходящие для настоящего изобретения, включая те, которые проиллюстрированы выше, подробно описаны в литературе [Remington's Pharmaceutical Sciences, последнее издание].
[91] Дозировка композиции для пациента зависит от многих факторов, включая рост пациента, площадь поверхности тела, возраст, конкретное вводимое соединение, пол, время и способ введения, общее состояние здоровья и другие лекарственные средства, вводимые одновременно. Фармацевтически активный антисмысловой олигонуклеотид можно вводить в количестве от 100 нг/кг массы тела (кг) до 10 мг/кг массы тела (кг), более предпочтительно в количестве от 1 до 500 мкг/кг массы тела (кг), и наиболее предпочтительно в количестве от 5 до 50 мкг/кг массы тела (кг), но дозировку корректируют с учетом вышеуказанных факторов.
[92] Используемый в настоящем описании термин «терапевтически эффективное количество» означает количество, достаточное для лечения состояния с разумным соотношением польза/риск, применимым к медицинскому лечению, а уровень эффективной дозы может быть определен на основе факторов, включая тип и тяжесть индивидуального заболевания, возраст, пол, активность лекарственного средства, чувствительность к лекарственному средству, время введения, путь введения и скорость выведения, продолжительность лечения, сопутствующие лекарственные средства и другие факторы, хорошо известные в области медицины. Терапевтически эффективные количества композиции по настоящему изобретению могут составлять от 0,5 мкг/кг до 1 г/кг, более предпочтительно от 10 мкг/кг до 500 мг/кг, но эффективная дозировка может быть соответствующим образом скорректирована в зависимости от возраста, пола и состояние пациента.
[93] Используемый в настоящем описании термин «лечение» относится к систематическому процессу и действиям, предназначенным для лечения, излечения или облегчения любого заболевания, расстройства или проблемы. Хотя конечной целью является устранение этиологии заболевания или возвращение состояния к исходному состоянию, в объем лечения также входит симптоматическое облегчение, устраняющее или облегчающее симптомы заболевания, даже если оно не устраняет этиологии заболевания или не восстанавливает состояние до его исходной точки. Кроме того, также в категорию лечения включено профилактическое действие по введению лекарственного средства согласно одному из вариантов осуществления настоящего изобретения до появления симптомов, чтобы симптомы не появлялись или, если они появляются, были смягчены, так чтобы повседневная жизнь пациента существенно не ухудшалась. Таким образом, фармацевтическую композицию согласно одному из вариантов осуществления изобретения можно использовать для предотвращения заболевания, а способы лечения согласно одному из вариантов осуществления изобретения включают предотвращение симптомов заболевания.
СПОСОБ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
[94] Настоящее изобретение будет описано более подробно ниже с помощью примеров и экспериментального примера. Однако настоящее изобретение не ограничивается примерами и экспериментальными примерами, раскрытыми ниже, но может быть реализовано во многих различных вариантах осуществления, и следующие примеры и экспериментальные примеры представлены для того, чтобы сделать раскрытие изобретения полным и полностью донести информацию об объеме изобретения специалисту в данной области.
[95] Пример: Дизайн антисмысловых олигонуклеотидов, нацеленных на Cacna1g.
[96] Авторы настоящего изобретения разработали 20 потенциальных антисмысловых олигонуклеотидов (ASO), нацеленных на cacna1g, ген, кодирующий субъединицу α1G (Cav3.1) среди трех изоформ кальциевого канала Т-типа (ФИГ. 1 и Таблица 1), с учетом целевой специфичности и стабильности. Кроме того, авторы настоящего изобретения использовали гэпмеры, содержащие внутреннюю последовательность ДНК длиной 10 нт и последовательность РНК длиной 5 нт, каждая с модифицированным 2'-MOE скелетом на 5'-конце и 3'-конце, соответственно, которые, как известно в данной области техники, стабильны и эффективны в качестве антисмысловых олигонуклеотидных структур. В ДНК-части гэпмера вместо обычных фосфодиэфирных связей (PS-связи) в качестве связей сахар-сахар использовались фосфортиоатные связи (PO-связи), а в РНК-части - PS-связи только на обоих концах последней последовательности, тогда как связи PO использовались в оставшейся части РНК (ФИГ. 2). Кроме того, весь урацил (U) в части РНК был заменен тимином (Т), а весь цитозин (С) заменен метилированным цитозином для дальнейшего повышения стабильности антисмыслового олигонуклеотида.
[97] Таблица 1: Антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения
Интрон 10
Экзон 38
20мер
[98] Экспериментальный пример 1: Первичный скрининг
[99] Авторы настоящего изобретения разработали кандидатные антисмысловые олигонуклеотиды, нацеленные на мРНК cacna1g, как описано в Таблице 1 выше, и поручили компании Mycrosynth (Германия) подготовить их. Вышеуказанные антисмысловые олигонуклеотиды трансфецировали в клетки А549 в концентрации 200 нМ с использованием реагента липофектамина, а через 48 часов после трансфекции клетки лизировали с помощью набора для лизиса и RT (TOYOBO, Япония) и проводили реакцию обратной транскрипции. Затем проводили кОТ-ПЦР с использованием зонда Taqman и премикса кОТ-ПЦР (TOYOBO, Япония) для измерения уровней экспрессии гена cacna1g. Информация о зонде и праймере для специфической кОТ-ПЦР указана в таблице 2 ниже. В результате были отобраны восемь антисмысловых олигонуклеотидов, включая А80, А82, А91 и А92, которые неоднократно снижали экспрессию генов более чем на 50%, как показано на ФИГ. 3.
[100] Экспериментальный пример 2: Вторичный скрининг
[101] Затем авторы настоящего изобретения исследовали, могут ли восемь антисмысловых олигонуклеотидов, выбранных в экспериментальном примере 1, поглощаться клетками и приводить к нокдауну гена cacna1g в двух концентрациях (100 нМ и 400 нМ) с использованием реагента липофектамина.
[102] Как показано на ФИГ. 4, все восемь антисмысловых олигонуклеотидов продемонстрировали очень хорошую эффективность нокдауна при концентрации 400 нМ. Однако для а80, а82 и а84 эффективность нокдауна была ниже, чем у других антисмысловых олигонуклеотидов в концентрации 100 нМ.
[103] Экспериментальный пример 3: Анализ токсичности и анализ регуляции экспрессии генов in vivo
[104] На основании результатов клеточных экспериментов в экспериментальных примерах 1 и 2 авторы настоящего изобретения провели эксперименты на животных для проверки стабильности и фармакологического действия восьми кандидатных антисмысловых олигонуклеотидов согласно одному из вариантов осуществления настоящего изобретения.
[105] В частности, авторы настоящего изобретения разделили бестимусных мышей C57BL/6J дикого типа P0-недельного возраста на 4-6 групп по 4-6 мышей в каждой группе и вводили в общей сложности 4 мкл (25 мкг) каждого антисмыслового олигонуклеотида (1 мМ) путем интрацеребровентрикулярной (ICV) инъекции по 2 мкл в каждое полушарие головного мозга. Через 7 дней измеряли выживаемость, собирали ткани мозга выживших мышей и измеряли уровень экспрессии гена cacna1g в тканях головного мозга с использованием кОТ-ПЦР, как описано в экспериментальном примере (ФИГ. 5a).
[106] В результате, как показано на ФИГ. 5b, все мыши, обработанные А88, умерли в течение 6 часов, А86 убил 4 из 6 мышей, А82 убил 2 из 5 мышей, а А84 убил 2 из 6 мышей, что указывает на значительную токсичность. С другой стороны, было подтверждено, что A89, A91 и 92 являются относительно безопасными кандидатами в антисмысловые олигонуклеотиды, при этом только 1 из 6 животных умирает или не умирает вообще. С другой стороны, AS-Cav3.1_20мер, положительный контроль, используемый для подавления Cav3.1 в предшествующем уровне техники (Bourinetet al., EMBO J. 24: 315-324, 2005), использовали в качестве положительного контроля только в первоначальном эксперименте по скринингу и исключали из дальнейшего скрининга in vivo, поскольку его последовательность комплементарна только у мышей и крыс, а не у генов приматов, включая обезьяну и человека, и имеет высокую вероятность перекрестной реакции с другими генами.
[107] Кроме того, в результате подтверждения эффекта ингибирования экспрессии генов в экспериментах in vivo было обнаружено, что A82 и A84 демонстрируют более низкий эффект ингибирования экспрессии генов CACNA1G в условиях in vivo, чем в клеточных экспериментах, тогда как A86, A89, A91, и А92 продемонстрировали более высокий эффект ингибирования экспрессии генов, как показано на ФИГ. 5c.
[108] Поэтому, основываясь на результатах ФИГ. 5b и 5c, авторы настоящего изобретения выбрали А91 и А92 в качестве окончательных кандидатов.
[109] Экспериментальный пример 4: Анализ перекрестной реакции
[110] Существует три изоформы альфа-субъединицы кальциевых каналов Т-типа, и гомология между ними очень высока. В частности, альфа-субъединицы этих трех изотипов экспрессируются в разных органах и тканях, и известно, что недискриминационные блокаторы этих изотипов вызывают различные побочные эффекты при системном применении, блокируя кальциевые каналы Т-типа в органах, не являющихся мишенями. Из-за этой перекрестной реактивности разработанные на сегодняшний день блокаторы кальциевых каналов Т-типа связаны с серьезными побочными эффектами при клиническом использовании.
[111] Поэтому разработка антисмысловых олигонуклеотидов, способных избирательно ингибировать экспрессию только альфа-субъединицы (Cav3.1), не ингибируя при этом экспрессию других изоформ, является предпосылкой разработки более безопасных препаратов.
[112] Соответственно, авторы изобретения исследовали, избирательно ли ингибируют a91 и a92, которые ранее были проверены в качестве окончательных кандидатов, в отдельности Cav3.1 на животных моделях.
[113] В частности, в качестве контроля авторы изобретения вводили в общей сложности 4 мкл (25 мкг) a91 и a92 (1 мМ) бестимусным мышам C57BL/6J дикого типа P0-недельного возраста путем интрацеребровентрикулярной (ICV) инъекции, 2 мкл в каждого полушария мозга, а затем через 7 дней собирали ткань мозга мышей, чтобы определить экспрессию генов cacna1g (Cav3. 1), cacna1h (Cav3.2) и cacna1i (Cav3.3), которую измеряли с помощью кОТ-ПЦР с использованием праймеров и зондов, перечисленных в таблице 2 и описанных в экспериментальном примере 1 выше (ФИГ. 5a).
[114] Таблица 2: Информация о праймерах и зондах, используемых в qPCR
[115] В результате было установлено, что антисмысловые олигонуклеотиды А91 и А92 согласно одному из вариантов осуществления настоящего изобретения специфически ингибируют экспрессию CACNA1G, не ингибируя при этом экспрессию генов, кодирующих другие изоформы, как показано на ФИГ. 6a и 6b. Таким образом, можно видеть, что антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения можно эффективно использовать для лечения различных заболеваний, в которых участвует субъединица альфа 1G, путем селективного ингибирования экспрессии только субъединицы альфа 1G, не влияя на экспрессию других изоформ.
[116] Пример 5: Анализ терапевтического эффекта при болезни Паркинсона
[117] На основе результатов экспериментальных примеров 1-4 авторы настоящего изобретения исследовали, будет ли антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения проявлять терапевтическое действие в отношении болезни Паркинсона, которая, как известно, является репрезентативным заболеванием среди заболеваний, связанных с CACANA1G.
[118] 5-1: Подтверждение терапевтического эффекта
[119] В частности, авторы настоящего изобретения использовали мышей, получавших 6-гидроксидофамин (6-OHDA), в качестве модельного животного болезни Паркинсона, а затем вводили 200 мкг антисмыслового олигонуклеотида (а91) согласно одному из вариантов осуществления настоящего изобретения 7-8-недельным бестимусным мышам дикого типа C57BL/6J посредством интрацеребровентрикулярной инъекции и через неделю проводили анализ «открытое поле» (ФИГ. 7а).
[120] В результате, как показано на ФИГ. 7b, анализ «открытое поле» через 1 неделю после инъекции антисмысловых олигонуклеотидов согласно одному из вариантов осуществления настоящего изобретения выявил снижение ипсилатерального вращения при введении антисмыслового олигонуклеотида (А91) согласно одному из вариантов осуществления настоящего изобретения.
[121] Модель болезни Паркинсона у мышей, вызванная односторонней инъекцией 6-OHDA в полосатое тело, характеризующаяся фенотипом повторяющегося вращения в направлении инъекции (Thiele et al., J. Vis. Exp., 60: e3234, 2012). В случае мышей, использованных на ФИГ. 7b и 7c, 6-OHDA вводили в полосатое тело, расположенное в левом полушарии, что приводило к фенотипу вращения влево. Этот фенотип вращения в том же направлении, что и инъекция, известен как ипсилатеральное вращение. На Фигуре 7b соотношение коаксиального вращения к пройденному расстоянию увеличивается в группе мышей, которым инъецировали только 6-OHDA, тогда как соотношение ипсилатерального вращения снижается у мышей, которым интрацеребровентрикулярно инъецировали 200 мкг Cav3.1-ASO (a91) в дополнение к модели 6-OHDA. Это позволяет предположить, что инъекция 200 мкг Cav3.1-ASO снижает индуцированный 6-OHDA патологический фенотип болезни Паркинсона.
[122] На ФИГ. 7c расстояние, пройденное мышами, измерялось за 30-минутный период. Хотя у мышей, получавших дополнительные 200 мкг Cav3.1-ASO, может показаться, что движение снизилось до уровня отрицательного контроля, не было статистически значимой разницы в пройденном расстоянии ни в одной из групп. Кроме того, на ФИГ. 7d показана скорость движения этих мышей. Подобно пройденному выше расстоянию, мыши, обработанные дополнительными 200 мкг Cav3.1-ASO, показали снижение локомоции до уровня отрицательного контроля, но не было статистически значимой разницы в локомоции ни в одной из групп.
[123] Затем авторы настоящего изобретения проводили тест с ускоренным вращающимся стержнем в качестве конечной точки двигательной активности. Тест с вращающимся стержнем с ускорением проводили на вышеуказанных животных через 1 неделю после инъекции антисмыслового олигонуклеотида и с интервалом в 1 день.
[124] В результате, как показано на ФИГ. 7e, антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения улучшал физическую работоспособность дозозависимым образом и с увеличением дней испытаний. Напротив, в группе отрицательного контроля, получавшей только 6-OHDA, атаксия со временем не улучшилась.
[125] 5-2: Идентификация механизма
[126] Авторы настоящего изобретения определяли, действительно ли механизм действия антисмысловых олигонуклеотидов согласно одному из вариантов осуществления настоящего изобретения действует после гибели дофаминовых нейронов.
[127] Для этого авторы изобретения специально вводили препарат 6-OHDA, известный как препарат, вызывающий болезнь Паркинсона, в полосатое тело головного мозга экспериментальных животных из примера 5-1, и в тот же день одновременно вводили антисмысловой олигонуклеотид. Поскольку 6-OHDA индуцирует гибель клеток в течение недели, а антисмысловому олигонуклеотиду требуется примерно неделя для работы, трудно ожидать ингибирующего эффекта в отношении гибели дофаминовых нейронов при использовании этого метода. Для измерения гибели дофаминергических нейронов животных умерщвляли через 5 недель, мозг вырезали, срезы тканей готовили с помощью криотома и проводили гистохимический анализ полосатого тела и черной субстанции с использованием антитела, специфически связывающегося с тирозингидроксилазой (ТН), маркера, специфического для дофаминергических нейронов. Как показано на ФИГ. 7f, дофаминергические нейроны в контрольной группе без какого-либо лекарственного средства выглядели нормальными, но гибель дофаминергических нейронов наблюдалась во всех группах, получавших 6-OHDA. Это показывает, что антисмысловой олигонуклеотид не влияет на гибель дофаминергических нейронов в экспериментальном методе, используемом в этом варианте осуществления, но действует путем блокирования аномальной передачи сигналов двигательных нейронов, которая возникает после гибели дофаминергических нейронов, что следует за потерей дофаминергических нейронов.
[128] Пример 6: Анализ эффективности лечения эссенциального тремора
[129] Затем авторы изобретения исследовали, эффективен ли антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения при лечении эссенциального тремора, проводя эксперименты на животных моделях эссенциального тремора, индуцированного гармалином.
[130] С этой целью, в частности, как показано на ФИГ. 8а, 200 мкг антисмыслового олигонуклеотида (а91) согласно одному из вариантов осуществления настоящего изобретения вводили интрацеребровентрикулярно бестимусным мышам C57BL/6J дикого типа в возрасте 7-8 недель, с последующим через 2 недели внутрибрюшинным введением гармалина, индуктора тремора, в дозе 9 мг/кг и через 8-10 минут проводили тест на тремор с использованием того же устройства, что показано на ФИГ. 8b.
[131] В результате, как показано на ФИГ. 8c и 8d, антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения значительно ингибировал тремор по сравнению с контролем (дикий тип) или носителем (контрольная обработка ASO).
[132] Приведенные выше результаты демонстрируют, что антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения эффективен при лечении эссенциального тремора.
[133] Пример 7: Анализ эффективности лечения депрессии
[134] Затем авторы изобретения исследовали, эффективен ли антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения при лечении ET, путем проведения экспериментов на мышах как на животных моделях депрессии, вызванной стрессом.
[135] 7-1: Эффективность на модели хронического социального стресса
[136] Сначала авторы настоящего изобретения исследовали шкалу депрессии, используя экспериментальную схему, показанную на ФИГ. 9a и конкретный способ, показанный на ФИГ. 9b. В частности, 200 мкг антисмыслового олигонуклеотида (а91) согласно одному из вариантов осуществления настоящего изобретения инъецировали интрацеребровентрикулярно 7-8-недельным бестимусным мышам C57BL/6J дикого типа с последующим стрессом социального поражения с 5-го по 10-й день путем введения другого агрессивного индивидуума в клетку, в которой содержались животные, а затем степень вызванной стрессом депрессии измеряли путем измерения социальных взаимодействий на 17-й день.
[137] В результате, как показано на ФИГ. 9c, у экспериментальных животных, обработанных антисмысловым олигонуклеотидом согласно одному из вариантов осуществления настоящего изобретения, наблюдалось снижение экспрессии Cav3.1, в то время как социальное взаимодействие восстанавливалось почти до контрольного уровня. Это указывает на то, что антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения является высокоэффективным агентом для снижения социального стресса.
[138] 7-2: Эффективность на модели краткосрочного острого стресса
[139] Затем авторы настоящего изобретения провели тест принудительного плавания, чтобы определить, оказывает ли антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения терапевтический эффект при острой депрессии.
[140] В частности, 200 мкг антисмыслового олигонуклеотида (а91) согласно одному из вариантов осуществления настоящего изобретения вводили интрацеребровентрикулярно бестимусным мышам C57BL/6J дикого типа в возрасте 7-8 недель и через 3 недели мышей помещали в водяную ванну, как показано на ФИГ. 10а, а индекс напряжения определяли путем измерения времени неподвижности, вызванного стрессом.
[141] Результаты подтвердили, что антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения не только значительно снижает экспрессию Cav3.1 в головном мозге, как показано на ФИГ. 10b, но и значительно сокращает время неподвижности, являющееся признаком стресса. Эти результаты демонстрируют, что антисмысловой олигонуклеотид согласно одному из вариантов осуществления изобретения представляет собой вещество, которое придает толерантность к кратковременному стрессу, а также к хроническому стрессу.
[142] Пример 8: Эффективность при лечении эпилепсии
[143] Авторы настоящего изобретения исследовали, можно ли использовать антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения для лечения эпилепсии.
[144] В частности, как показано на ФИГ. 11а, авторы изобретения вводили 200 мкг антисмыслового олигонуклеотида (а91) согласно одному из вариантов осуществления настоящего изобретения интрацеребровентрикулярно бестимусным мышам C57BL/6J дикого типа в возрасте 7-8 недель с последующей процедурой введения зонда EEG через 12 дней и вызывающего абсанс гамма-бутиролактона (GBL) в дозе 70 мг/кг через 14 дней. Электроэнцефалограмму (EEG) измеряли в течение 30 минут до и после введения GBL, а через 42 дня после введения антисмыслового олигонуклеотида животных умерщвляли и собирали мозг для измерения уровней экспрессии мРНК Cav3.1.
[145] В результате, как показано на ФИГ. 11b, антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения значительно снижает как частоту, так и количество спайк-волновых разрядов (SWD) за 10 минут.
[146] Эти результаты демонстрируют, что антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения эффективны при лечении эпилепсии, особенно абсансов.
[147] Пример 9: Восстановление сознания
[148] Кома - медицинский термин, обозначающий состояние глубокого бессознательного состояния. Пациент в коме, также известный как «вегетативный пациент», не может быть разбужен и не реагирует на внешние раздражители, такие как боль, свет или звук. Существует много известных причин комы, в том числе наркотическая интоксикация, нарушение обмена веществ, заболевания центральной нервной системы, гипоксия и инсульт, но кома также может быть вызвана травмой головного мозга, например, автомобильной аварией или падением. К сожалению, не сообщалось ни об одном лекарственном средстве, способном вернуть сознание таким пациентам, находящимся в коме.
[149] Потеря сознания является основным симптомом эпилепсии, как обсуждалось в Примере 8. Кроме того, учитывая хорошо известную корреляцию эпилепсии с кальциевыми каналами Т-типа и судорогами, а также результаты Примера 8, демонстрирующие, что ингибирование экспрессии субъединицы альфа1G кальциевых каналов Т-типа может облегчить симптомы судорог, авторы настоящего изобретения стремились определить, эффективны ли антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения для восстановления сознания.
[150] С этой целью авторы настоящего изобретения специально вводили 200 мкг антисмыслового олигонуклеотида (а91) согласно одному из вариантов осуществления настоящего изобретения интрацеребровентрикулярно бестимусным мышам C57BL/6J дикого типа в возрасте 7-8 недель, как показано на ФИГ. 12а, и индуцировали потерю выпрямительного рефлекса (LORR) через 11 дней внутрибрюшинным введением анестетика авертина в дозе 400 мг/кг. Затем измеряли продолжительность потери выпрямительного рефлекса (LORR). В результате, как показано на ФИГ. 12b, продолжительность LORR была значительно снижена в группе обработки антисмысловым олигонуклеотидом (А91) согласно одному из вариантов осуществления настоящего изобретения.
[151] Авторы настоящего изобретения дополнительно исследовали, может ли антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения быстро восстанавливать потерю сознания, вызванную алкоголем, с использованием модельного животного с потерей сознания, вызванной алкоголем.
[152] В частности, 200 мкг антисмыслового олигонуклеотида (а91) согласно одному из вариантов осуществления настоящего изобретения вводили интрацеребровентрикулярно бестимусным мышам C57BL/6J дикого типа в возрасте 7-8 недель в соответствии со схемой экспериментов, показанной на ФИГ. 12C, а через 12-13 дней мышам вводили 20% водный раствор этанола в дозе 0,6 г/кг внутрибрюшинно в дозе 0,6 г/кг, поведение животных снимали на видео в открытом поле в течение 30 мин, измеряли скорость движения, пройденное расстояние и частоту поворотов с помощью компьютерного программного обеспечения (EthoVision, Noldus, Нидерланды). Через два дня потерю сознания вызывали внутрибрюшинным введением высокой концентрации (3,5 г/кг) этанола и измеряли период задержки до потери сознания (период задержки LORR). Кроме того, через 21 день после введения олигонуклеотида (А91) согласно одному из вариантов осуществления настоящего изобретения животных умерщвляли и собирали их мозг для измерения экспрессии гена Cav3.1.
[153] В результате, как показано на ФИГ. 12d, более энергичные движения наблюдались в группе обработки антисмысловым олигонуклеотидом согласно одному из вариантов осуществления настоящего изобретения, а в случае периода задержки потери сознания группа обработки антисмысловым олигонуклеотидом согласно одному из вариантов осуществления настоящего изобретения показала более длительное время по сравнению с контрольной группой.
[154] Это указывает на то, что антисмысловой олигонуклеотид согласно одному из вариантов осуществления настоящего изобретения может способствовать восстановлению сознания у пациентов в состоянии потери сознания путем специфического ингибирования экспрессии гена Cav3.1, и показывает, что антисмысловой олигонуклеотид по настоящему изобретению представляет собой многообещающее вещество, которое может быть разработано в качестве стимулятора восстановления сознания у пациентов, находящихся в коме, для которых не существует подходящего лечения.
[155] Примеры 21-23: Идентификация целевого сайта ASO
[155] На основании результатов приведенных выше примеров авторы настоящего изобретения стремились более точно идентифицировать целевой сайт антисмыслового олигонуклеотида согласно одному из вариантов осуществления настоящего изобретения.
[157] С этой целью определили множество целевых последовательностей (А4, А7 и А11), выбранных в пределах SEQ ID NO: 1 и расположенных вблизи антисмыслового олигонуклеотида (А91) согласно одному из вариантов осуществления настоящего изобретения, принимая во внимание геномное местоположение гена CACNA1G, как показано в таблице 3, и исследовали их безопасность и ингибирование экспрессии Cav3.1.
[158] Таблица 3: Последовательности, окружающие антисмысловой олигонуклеотид, нацеленный на SEQ ID NO: 1.
(Примеры)
(SEQ ID No)
(SEQ ID NO)
[159] Геномные местоположения в Таблице 3 основаны на геномной последовательности нуклеиновой кислоты гена cacna1g, как описано в Эталонной последовательности GenBank NC_000017.11 REGION: 50560715..50627474.
[160] В частности, авторы настоящего изобретения разделили мышей C57BL/6J дикого типа в возрасте P0 недель на четыре-шесть на группу и в общей сложности вводили 4 мкл (25 мкг) антисмыслового олигонуклеотида (1 мМ), указанного в Таблице 3, внутрицеребровентрикулярной (ICV) инъекцией, 2 мкл в каждое полушарие головного мозга. Уровень выживаемости измеряли через 7 дней, ткани мозга выживших мышей собирали, а уровень экспрессии гена cacna1g в тканях головного мозга измеряли с помощью кОТ-ПЦР, как описано в примере 1 (ФИГ. 13а).
[161] В результате, как показано на ФИГ. 13b, вновь созданные антисмысловые олигонуклеотиды по настоящему изобретению продемонстрировали аналогичные или более высокие показатели выживаемости по сравнению с контрольной группой и антисмысловым олигонуклеотидом (А91) согласно одному из вариантов осуществления настоящего изобретения, за исключением А4, что указывает на высокую безопасность.
[162] Кроме того, исследовали влияние на экспрессию Cav3.1, и, как показано на ФИГ. 13c, среди вновь созданных антисмысловых олигонуклеотидов a11 продемонстрировал значительное, хотя и несколько более слабое, ингибирование экспрессии мРНК Cav3.1, чем a91, в то время как a4 и a7 также показали ингибирование экспрессии мРНК Cav3.1, хотя и не было статистически значимым. Причина статистической незначительности заключается в том, что одна особь в каждой экспериментальной группе показала более высокую степень экспрессии генов, чем в контрольной группе, что, как считается, связано с экспериментальной ошибкой, и считается, что увеличение количества экспериментальных голов будет показывать статистически значительное снижение экспрессии генов.
[163] Как можно видеть из приведенных выше результатов, геномное местоположение гена cacna1g, выбранного в настоящем изобретении, вопреки ожиданиям, находится близко к 3'-концу предварительно сплайсированной мРНК, и можно видеть, что любой сайт внутри SEQ ID NO 1, включая интрон 37 и экзон 38, может эффективно снижать экспрессию мРНК Cav3.1 и быть полезным для лечения неврологических заболеваний, связанных с кальциевыми каналами альфа 1G Т-типа.
[164] Как описано выше, антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения не только избирательно ингибируют экспрессию субъединицы альфа 1G, не влияя на экспрессию других изоформ кальциевых каналов Т-типа, но также демонстрируют замечательную эффективность при лечении заболеваний, связанных с субъединицей альфа 1G, в частности, при болезни Паркинсона, без каких-либо других побочных эффектов. Таким образом, антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения можно использовать в качестве терапевтических средств для лечения различных заболеваний, связанных с субъединицей альфа 1G кальциевых каналов Т-типа, включая болезнь Паркинсона, а также различных нейродегенеративных заболеваний, таких как депрессия, эссенциальный тремор, эпилепсия, тревожные расстройства и потеря сознания.
[165] Изобретение было описано со ссылкой на вышеописанные варианты осуществления и экспериментальные примеры, но они являются лишь иллюстративными, и специалисту в данной области техники будет понятно, что на его основе возможны различные модификации и другие равноценные варианты осуществления. Следовательно, истинный объем изобретения должен определяться техническими особенностями прилагаемой формулы изобретения.
ПРОМЫШЛЕННАЯ ПРИМЕНИМОСТЬ
[166] Антисмысловые олигонуклеотиды согласно одному из вариантов осуществления настоящего изобретения избирательно ингибируют экспрессию кальциевых каналов альфа 1G Т-типа и могут использоваться в качестве лекарственных средств для лечения нервно-психических расстройств, таких как болезнь Паркинсона, депрессия, эссенциальный тремор, эпилепсия и потеря сознания, а также для восстановления сознания у пациентов без сознания.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="PO24-5282_sequence
listing.xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-02-08">
<ApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>PCT/KR2022/012269</ApplicationNumberText>
<FilingDate>2022-08-17</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>PT22-5269</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>10-2021-0108274</ApplicationNumberText>
<FilingDate>2021-08-17</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="en">Korea Advanced Institute of Science
and Technology</ApplicantName>
<InventionTitle languageCode="en">ANTISENSE OLIGONUCLEOTIDE
TARGETING CAV3.1 GENE AND USES THEREOF</InventionTitle>
<SequenceTotalQuantity>64</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1660</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1660</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggagagaggaggcatggtggacaacagaggtcaggctgcaatgacccc
tatctactagtaagagcggctaccaggacctctgcttaggatagtcctgctgtgcctggtggtggtgggc
agggggcacagccagcttgacaggagggctagtccatgctggagaggagactgctgggctgagccctccc
ccaccccatataggcagcaataaggactgactccttggacgttcagggtctgggcagccgggaagacctg
ctggcagaggtgagtgggccctccccgcccctggcccgggcctactctttctggggccagtcaagtaccc
aggcacagcagcactcccgcagccacagcaagatctccaagcacatgaccccgccagccccttgcccagg
cccagaacccaactggggcaagggccctccagagaccagaagcagcttagagttggacacggagctgagc
tggatttcaggagacctcctgccccctggcggccaggaggagcccccatccccacgggacctgaagaagt
gctacagcgtggaggcccagagctgccagcgccggcctacgtcctggctggatgagcagaggagacactc
tatcgccgtcagctgcctggacagcggctcccaaccccacctgggcacagacccctctaaccttgggggc
cagcctcttggggggcctgggagccggcccaagaaaaaactcagcccgcctagtatcaccatagaccccc
ccgagagccaaggtcctcggaccccgcccagccctggtatctgcctccggaggagggctccgtccagcga
ctccaaggatcccttggcctctggcccccctgacagcatggctgcctcgccctccccaaagaaagatgtg
ctgagtctctccggtttatcctctgacccagcagacctggacccctgagtcctgccccactttcccactc
acctttctccactgggtgccaagtcctagctcctcctcctgggctatattcctgacaaaagttccatata
gacaccaaggaggcggaggcgctcctccctgcctcagtggctctgggtacctgcaagcagaacttccaaa
gagagttaaaagcagcagccccggcaactctggctccaggcagaaggagaggcccggtgcagctgaggtt
cccgacaccagaagctgttgggagaaagcaatacgtttgtgcagaatctctatgtatattctattttatt
aaattaattgaatctagtatatgcgggatgtacgacattttgtgactgaagagacttgtttccttctact
tttatgtgtctcagaatatttttgaggcgaaggcgtctgtctcttggctattttaacctaaaataacagt
ctagttatattccctcttcttgcaaagcacaagctgggaccgcgagcacattgcagccccaacggtggcc
catcttcagcggagagcgagaaccattttggaaactgtaatgtaacttattttttcctttaacctcgtca
tcattttctgtagggaaaaaaaaaagaaaaagaaaaaatgagattttacaagtgaaatggaaccttttta
tatatacatacatacatatctatctatctatctatatatatataaaataaagtaattttcctaaataaaa
a</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q2">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_73_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q138">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribunucleotides but alll uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q139">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaagtagaagaaaaccacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q140">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_74_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q141">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q142">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q143">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcagtaaaaacctcagcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q144">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_75_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q145">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q146">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q147">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atctccttgttgtgcctcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q148">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_76_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q149">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q151">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccaaatgagaaaaagctgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q152">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_77_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q153">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q154">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q155">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgactatgagagacctgtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q156">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_78_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q157">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q158">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q159">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgtgtgatgttcttctgtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q160">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_79_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q161">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q162">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q163">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catagccaatgttgtcaaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q164">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_80_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q165">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q166">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q167">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctagtgtgattccagagagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q168">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_81_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q169">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q170">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q171">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacctgatgaccacaatgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q172">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_82_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q173">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q174">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q175">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacttgattgctacttttga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q176">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_83_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q177">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q178">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q179">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggttccagttgacacaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q180">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_84_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q181">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q182">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q183">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccattggcatccctgtcctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q184">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_85_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q185">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q186">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q187">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacagcttaactgtccactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q188">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_86_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>mat_peptide</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q189">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q190">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q191">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcttagcaggtcagtaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q192">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_87_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q193">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>nibonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q194">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q195">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accttcactgtcatttcagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q196">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_88_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q197">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q198">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q199">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggagattggaaattggacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q200">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_89_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q201">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q202">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q203">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgttgtagcaggtggactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q204">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_90_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q205">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q206">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q207">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcatcaggcaaagagtcatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q208">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_91_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q209">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q210">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q211">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcagtccttattgctgcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q212">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_92_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q213">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q214">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q215">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tattgctttctcccaacagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q216">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sh3.1_20mer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q217">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q218">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q219">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acttgctgtccacaatcttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q220">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>AS-CAv3.1_20mer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q221">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q222">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q223">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgggacccattggcatccct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q224">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>scrambled</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q225">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q226">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q227">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctataggactatccaggaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q228">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>HTTRX_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q229">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q230">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q231">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcagtaacattgacaccac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q232">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_73_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q233">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtggttttcttctacttga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q234">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_74_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q235">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggctgaggtttttactgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q236">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_75_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q237">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaggcacaacaaggagat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q238">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_76_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q239">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacagctttttctcatttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q240">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_77_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q241">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaggcacaacaaggagat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q242">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_78_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q243">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacagaagaacatcacacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q244">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_79_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q245">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttgacaacattggctatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q246">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_80_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q247">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctctctggaatcacactag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q315">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcattgtggtcatcaggta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q249">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_82_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q250">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaaaagtagcaatcaagta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q251">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_83_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q252">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acctgtgtcaactggaacca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q253">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_84_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q254">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggacagggatgccaatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q255">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_85_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q256">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagtggacagttaagctgta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q257">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_86_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q258">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acctactgacctgctaagaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q259">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_87_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q260">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgaaatgacagtgaaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q261">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_88_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q262">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtccaatttccaatctcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q263">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_89_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q264">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagtccacctgctacaacac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q265">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_90_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q266">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatgactctttgcctgatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q267">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_91_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q268">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggcagcaataaggactgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q269">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_92_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q270">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgttgggagaaagcaata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q271">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sh3.1_20mer_target</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q272">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaagattgtggacagcaagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q273">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>AS-CAv3.1_20mer_target</INSDQualifier_valu
e>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q274">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agggatgccaatgggtcccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q275">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>scrambled_target</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q276">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcctggatagtcctatagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q277">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>HTTRX_target</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q278">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtggtgtcaatgttactgag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q279">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.1 probe</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q280">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtcattgggcagagagtgtgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q281">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.1 qPCR-F</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q282">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaggaagtctggtgtcagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q283">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.1 qPCR-R</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q284">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtcctagggatctctcggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q285">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.2 probe</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q286">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accttggtcttcttttcatgctcctgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q287">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.2 qPCR-F</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q288">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacactgtggttcaagctct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q289">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.2 qPCR-R</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q290">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttatcctcgctgcattctagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q291">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.3 probe</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q292">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acactgtgtcctcagagcaagcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q293">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.3 qPCR-F</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q294">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggagggatgttcaggcatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q295">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.3 qPCR-R</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q296">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcttccttcatccccacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q297">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q298">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Target sequence of
CACNA1G_gapmer_4_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggacacggagctgagctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q299">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q300">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Target sequence of
CACNA_gapmer_7_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaagcaatacgtttgtgca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q301">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q302">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Target sequence of
CACNA1G_gapmer_11_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacattttgtgactgaagag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q303">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>All uracils are substituted with
thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q304">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q305">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>All uracils are substituted with
thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q306">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_4_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagctcagctccgtgtcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q307">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q308">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q309">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q310">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_7_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcacaaacgtattgctttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q311">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q312">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>all uracils are substituted with
thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q313">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>all uracils are substituted with
thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q314">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_11_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcttcagtcacaaaatgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="PO24-5282_sequence
listing.xml" softwareName="WIPO Sequence" softwareVersion="2.3.0"
productionDate="2024-02-08">
<ApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>PCT/KR2022/012269</ApplicationNumberText>
<FilingDate>2022-08-17</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>PT22-5269</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>10-2021-0108274</ApplicationNumberText>
<FilingDate>2021-08-17</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="en">Korea Advanced Institute of Science
and Technology</ApplicantName>
<InventionTitle languageCode="en">ANTISENSE OLIGONUCLEOTIDE
TARGETING CAV3.1 GENE AND USES THEREOF</InventionTitle>
<SequenceTotalQuantity>64</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>1660</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1660</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggagagaggaggcatggtggacaacagaggtcaggctgcaatgacccc
tatctactagtaagagcggctaccaggacctctgcttaggatagtcctgctgtgcctggtggtggtgggc
agggggcacagccagcttgacaggagggctagtccatgctggagaggagactgctgggctgagccctccc
ccaccccatataggcagcaataaggactgactccttggacgttcagggtctgggcagccgggaagacctg
ctggcagaggtgagtgggccctccccgcccctggcccgggcctactctttctggggccagtcaagtaccc
aggcacagcagcactcccgcagccacagcaagatctccaagcacatgaccccgccagccccttgcccagg
cccagaacccaactggggcaagggccctccagagaccagaagcagcttagagttggacacggagctgagc
tggatttcaggagacctcctgccccctggcggccaggaggagcccccatccccacgggacctgaagaagt
gctacagcgtggaggcccagagctgccagcgccggcctacgtcctggctggatgagcagaggagacactc
tatcgccgtcagctgcctggacagcggctcccaaccccacctgggcacagacccctctaaccttgggggc
cagcctcttggggggcctgggagccggcccaagaaaaaactcagcccgcctagtatcaccatagaccccc
ccgagagccaaggtcctcggaccccgcccagccctggtatctgcctccggaggagggctccgtccagcga
ctccaaggatcccttggcctctggcccccctgacagcatggctgcctcgccctccccaaagaaagatgtg
ctgagtctctccggtttatcctctgacccagcagacctggacccctgagtcctgccccactttcccactc
acctttctccactgggtgccaagtcctagctcctcctcctgggctatattcctgacaaaagttccatata
gacaccaaggaggcggaggcgctcctccctgcctcagtggctctgggtacctgcaagcagaacttccaaa
gagagttaaaagcagcagccccggcaactctggctccaggcagaaggagaggcccggtgcagctgaggtt
cccgacaccagaagctgttgggagaaagcaatacgtttgtgcagaatctctatgtatattctattttatt
aaattaattgaatctagtatatgcgggatgtacgacattttgtgactgaagagacttgtttccttctact
tttatgtgtctcagaatatttttgaggcgaaggcgtctgtctcttggctattttaacctaaaataacagt
ctagttatattccctcttcttgcaaagcacaagctgggaccgcgagcacattgcagccccaacggtggcc
catcttcagcggagagcgagaaccattttggaaactgtaatgtaacttattttttcctttaacctcgtca
tcattttctgtagggaaaaaaaaaagaaaaagaaaaaatgagattttacaagtgaaatggaaccttttta
tatatacatacatacatatctatctatctatctatatatatataaaataaagtaattttcctaaataaaa
a</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q2">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_73_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q138">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribunucleotides but alll uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q139">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaagtagaagaaaaccacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q140">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_74_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q141">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q142">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q143">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcagtaaaaacctcagcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q144">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_75_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q145">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q146">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q147">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atctccttgttgtgcctcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q148">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_76_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q149">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q151">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccaaatgagaaaaagctgtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q152">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_77_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q153">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q154">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q155">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgactatgagagacctgtgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q156">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_78_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q157">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q158">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q159">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgtgtgatgttcttctgtt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q160">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_79_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q161">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q162">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q163">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>catagccaatgttgtcaaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q164">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_80_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q165">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q166">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q167">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctagtgtgattccagagagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q168">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_81_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q169">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q170">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q171">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacctgatgaccacaatgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q172">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_82_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q173">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q174">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q175">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacttgattgctacttttga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q176">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_83_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q177">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q178">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q179">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggttccagttgacacaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q180">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_84_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q181">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q182">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q183">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccattggcatccctgtcctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q184">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_85_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q185">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q186">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q187">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tacagcttaactgtccactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q188">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_86_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>mat_peptide</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q189">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q190">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q191">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcttagcaggtcagtaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q192">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_87_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q193">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>nibonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q194">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q195">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accttcactgtcatttcagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q196">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_88_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q197">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q198">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q199">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggagattggaaattggacat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q200">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_89_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q201">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q202">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q203">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgttgtagcaggtggactc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q204">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_90_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q205">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q206">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q207">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcatcaggcaaagagtcatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q208">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_91_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q209">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q210">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q211">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcagtccttattgctgcct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q212">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_92_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q213">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q214">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q215">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tattgctttctcccaacagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q216">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sh3.1_20mer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q217">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q218">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q219">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acttgctgtccacaatcttt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q220">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>AS-CAv3.1_20mer</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q221">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q222">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q223">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgggacccattggcatccct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q224">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>scrambled</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q225">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q226">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q227">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctataggactatccaggaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q228">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>HTTRX_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q229">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q230">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q231">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcagtaacattgacaccac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q232">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_73_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q233">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggtggttttcttctacttga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q234">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_74_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q235">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggctgaggtttttactgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q236">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_75_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q237">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaggcacaacaaggagat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q238">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_76_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q239">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cacagctttttctcatttgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q240">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_77_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q241">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggaggcacaacaaggagat</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q242">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_78_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q243">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aacagaagaacatcacacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q244">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_79_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q245">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctttgacaacattggctatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q246">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_80_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q247">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctctctggaatcacactag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q315">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcattgtggtcatcaggta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q249">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_82_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q250">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcaaaagtagcaatcaagta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q251">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_83_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q252">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acctgtgtcaactggaacca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q253">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_84_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q254">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggacagggatgccaatgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q255">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_85_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q256">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagtggacagttaagctgta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q257">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_86_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q258">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acctactgacctgctaagaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q259">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_87_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q260">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgaaatgacagtgaaggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q261">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_88_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q262">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtccaatttccaatctcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q263">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_89_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q264">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagtccacctgctacaacac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q265">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_90_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q266">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatgactctttgcctgatga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q267">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_91_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q268">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aggcagcaataaggactgac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q269">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_92_target</INSDQualifier_va
lue>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q270">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgttgggagaaagcaata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q271">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>sh3.1_20mer_target</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q272">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aaagattgtggacagcaagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q273">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>AS-CAv3.1_20mer_target</INSDQualifier_valu
e>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q274">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agggatgccaatgggtcccg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q275">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>scrambled_target</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q276">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttcctggatagtcctatagg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q277">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>HTTRX_target</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q278">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtggtgtcaatgttactgag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q279">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.1 probe</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q280">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtcattgggcagagagtgtgtcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q281">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.1 qPCR-F</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q282">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaggaagtctggtgtcagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q283">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.1 qPCR-R</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q284">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtcctagggatctctcggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q285">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.2 probe</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q286">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>accttggtcttcttttcatgctcctgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q287">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.2 qPCR-F</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q288">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacactgtggttcaagctct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q289">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.2 qPCR-R</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q290">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ttatcctcgctgcattctagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q291">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.3 probe</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q292">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>acactgtgtcctcagagcaagcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q293">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.3 qPCR-F</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q294">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gggagggatgttcaggcatc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q295">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Cav3.3 qPCR-R</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q296">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gagcttccttcatccccacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q297">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q298">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Target sequence of
CACNA1G_gapmer_4_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggacacggagctgagctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q299">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q300">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Target sequence of
CACNA_gapmer_7_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaaagcaatacgtttgtgca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q301">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q302">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Target sequence of
CACNA1G_gapmer_11_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gacattttgtgactgaagag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q303">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>All uracils are substituted with
thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q304">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q305">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>All uracils are substituted with
thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q306">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_4_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagctcagctccgtgtcca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q307">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q308">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q309">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>ribonucleotides but all uracils are
substituted with thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q310">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_7_ASO</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcacaaacgtattgctttc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q311">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q312">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>all uracils are substituted with
thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_RNA</INSDFeature_key>
<INSDFeature_location>16..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q313">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>all uracils are substituted with
thymines.</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q314">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>CACNA1G_gapmer_11_ASO</INSDQualifier_value
>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcttcagtcacaaaatgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
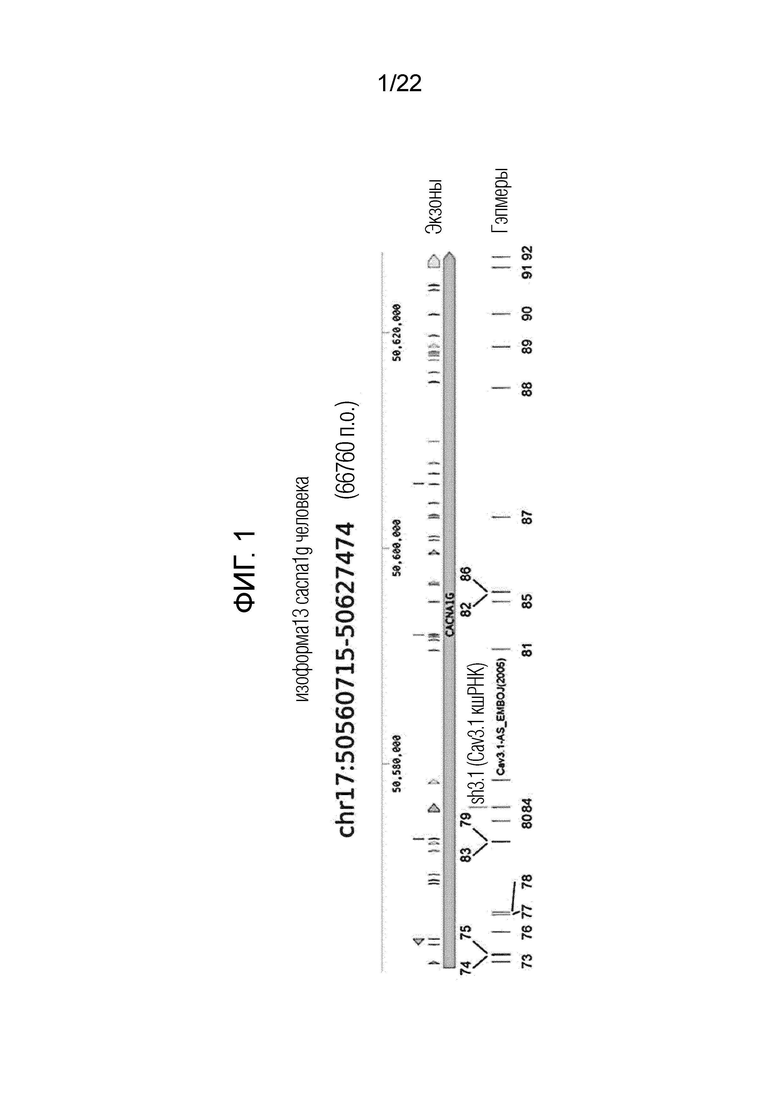
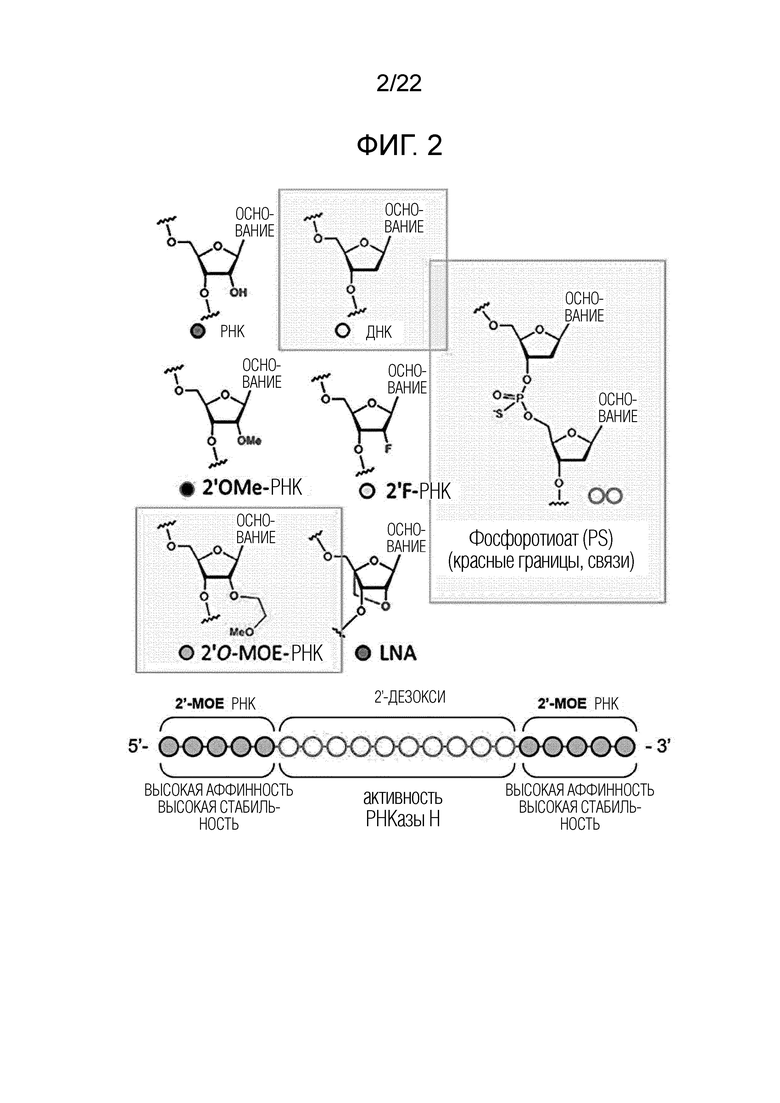
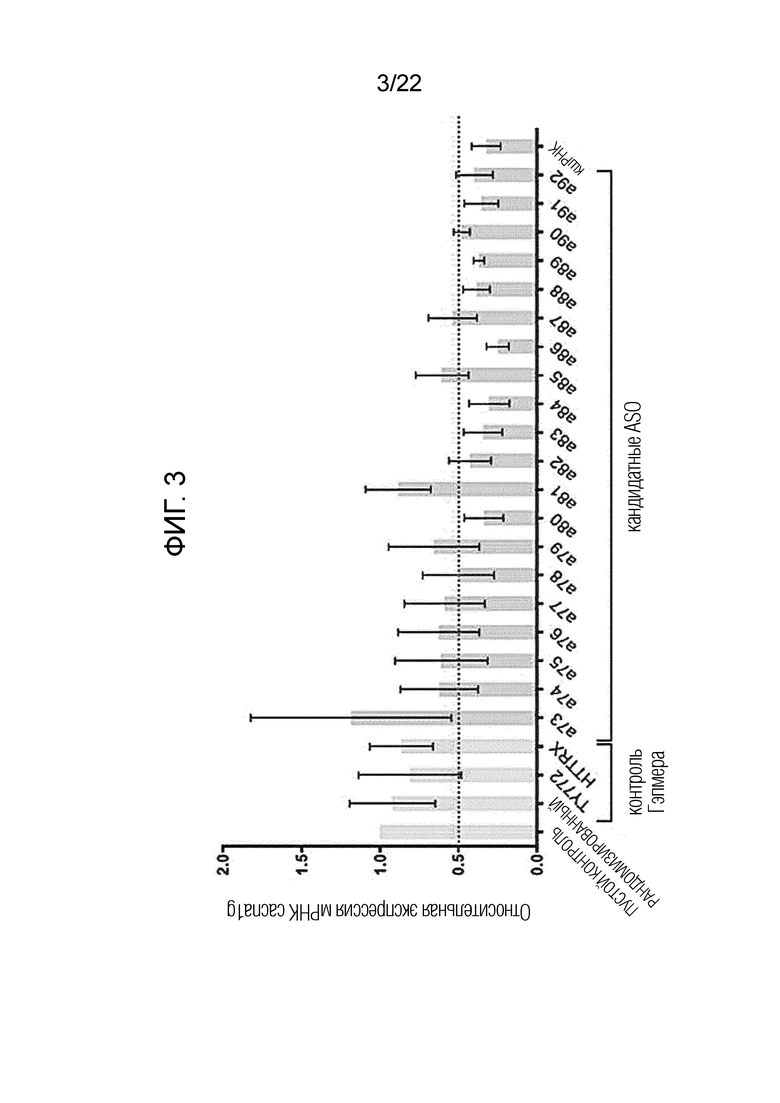
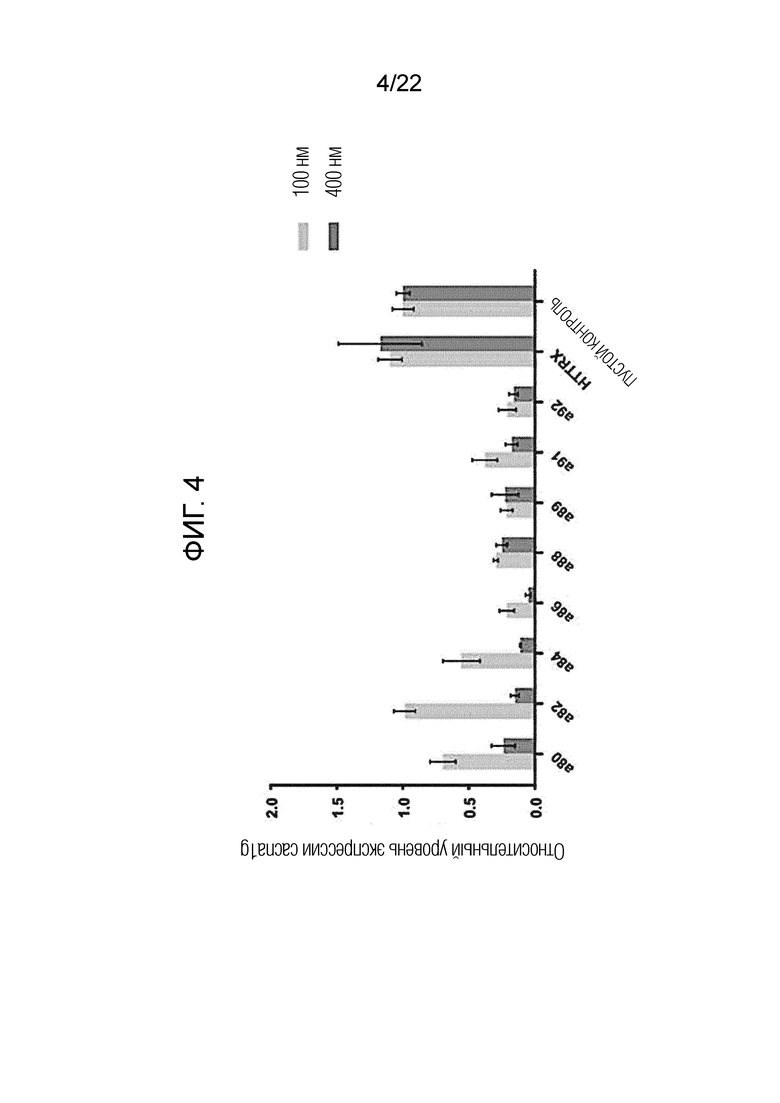
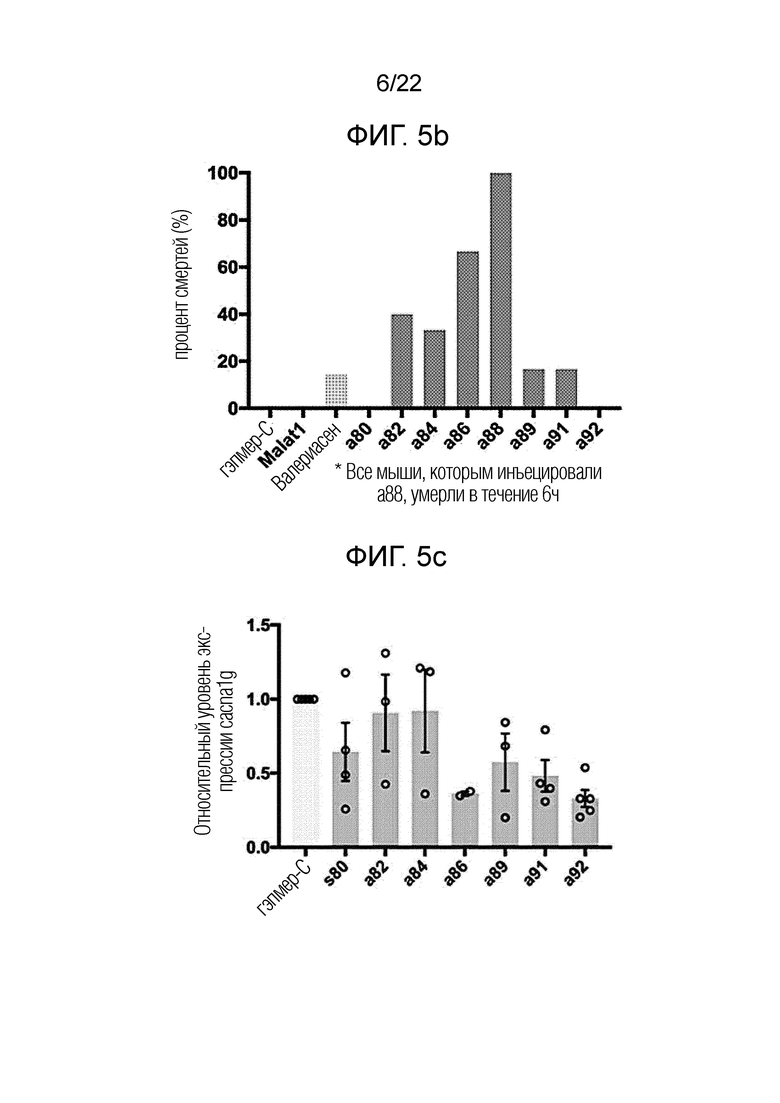

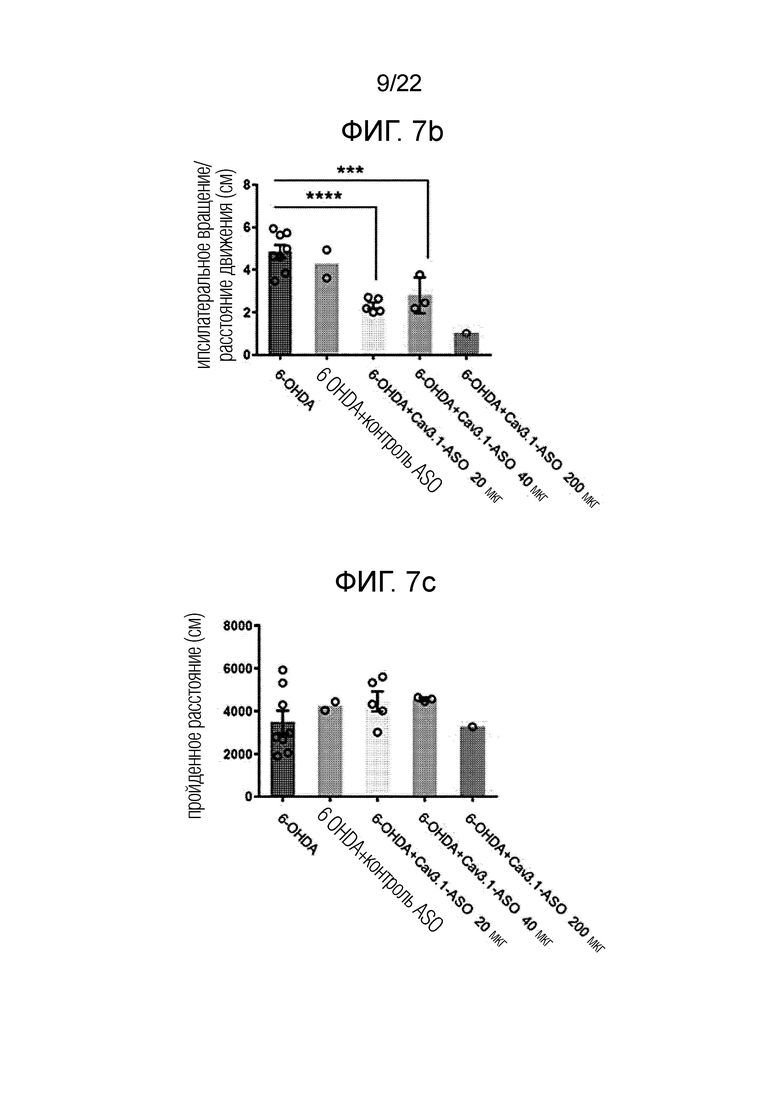
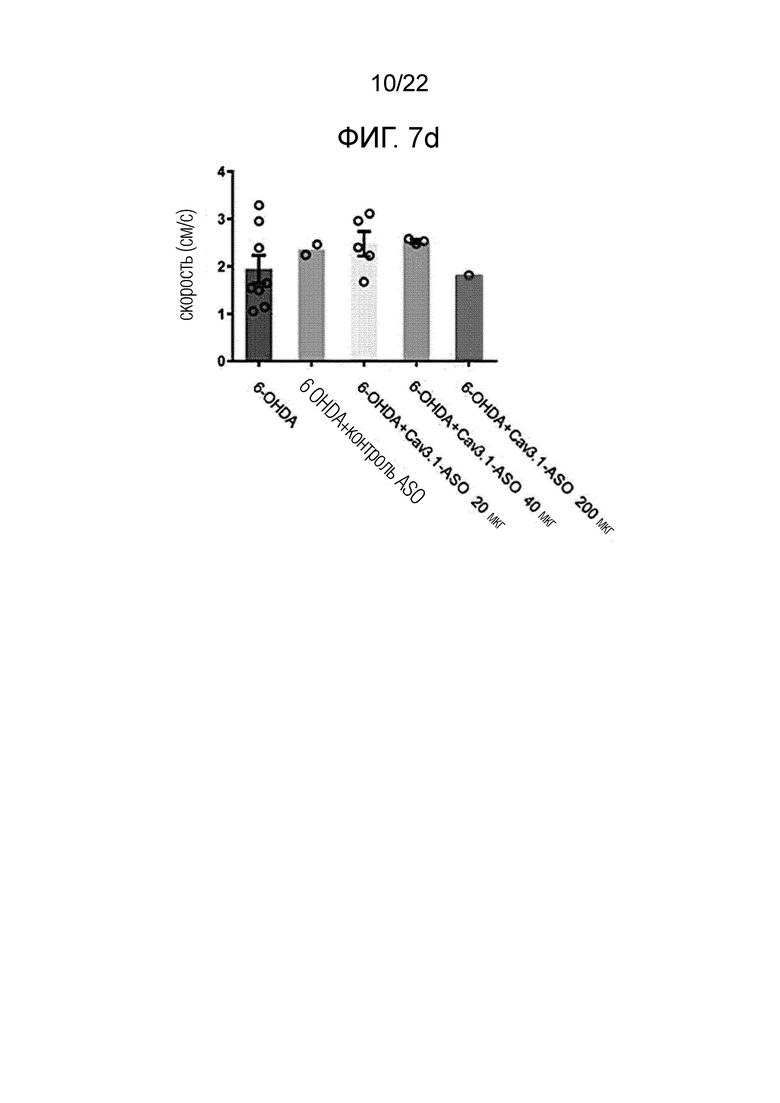
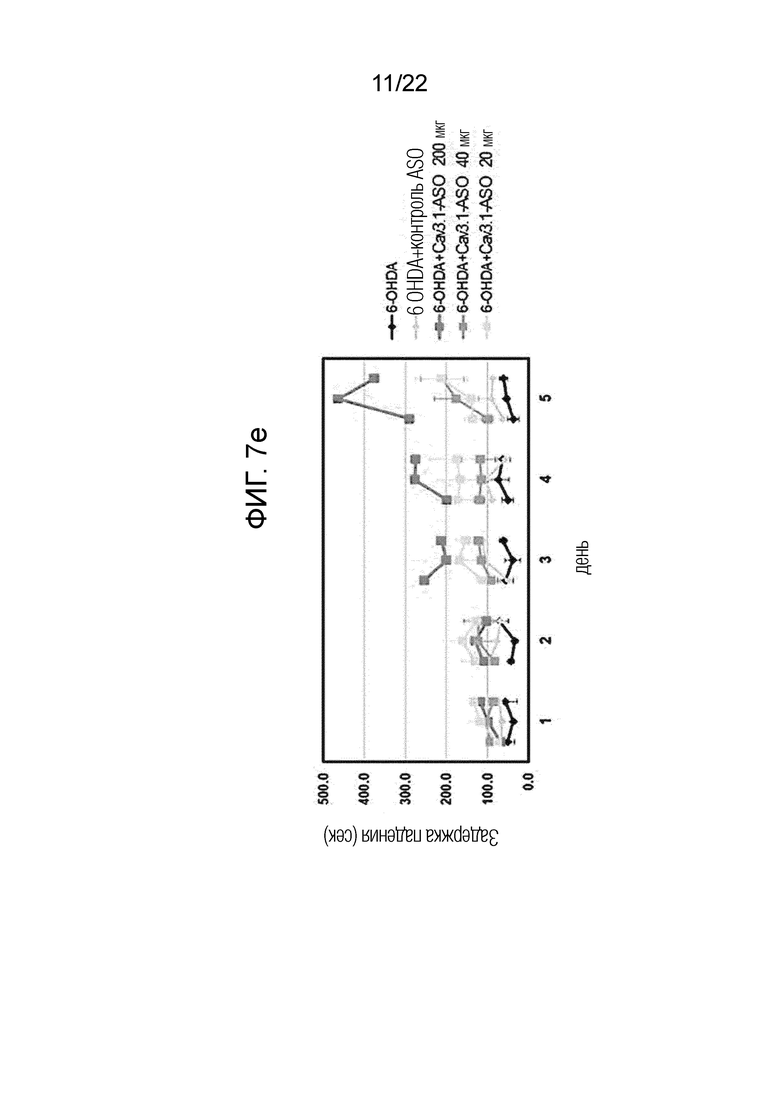
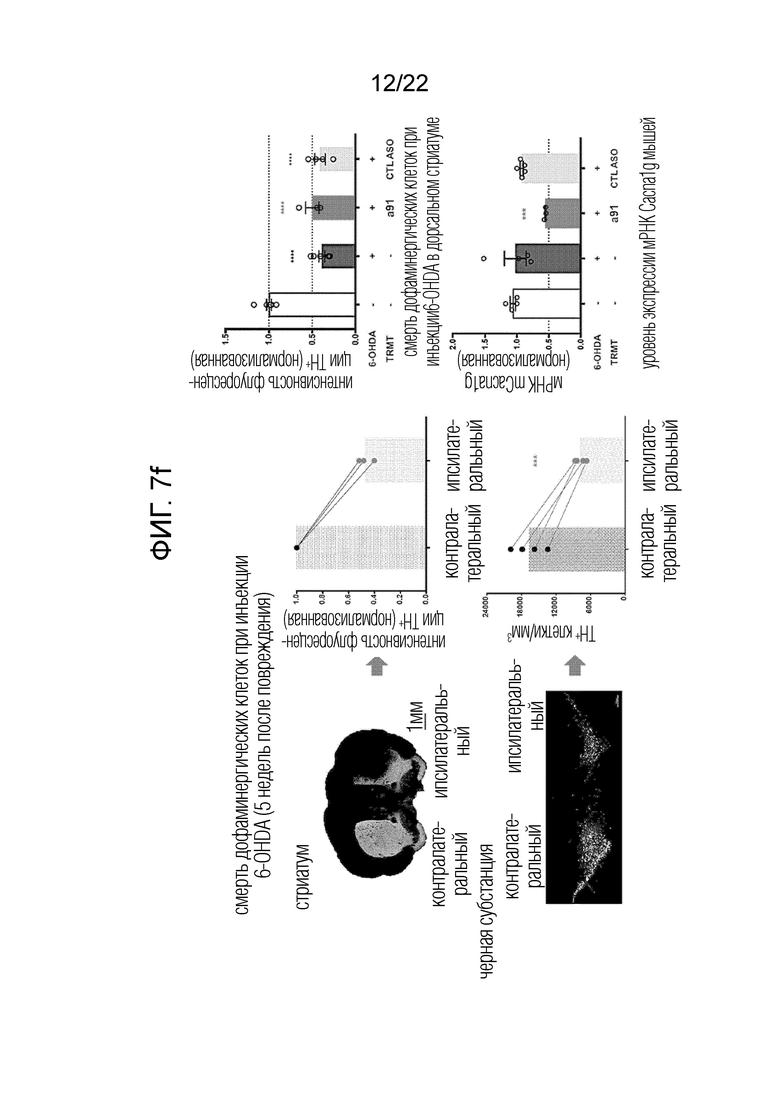
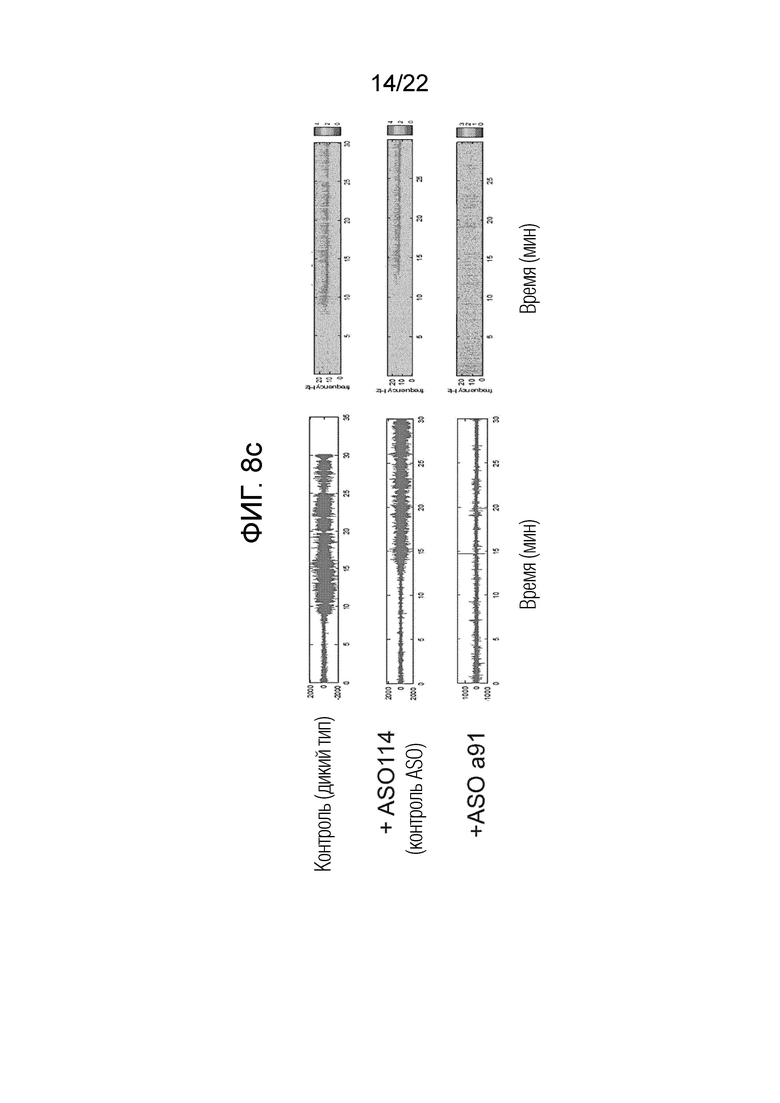
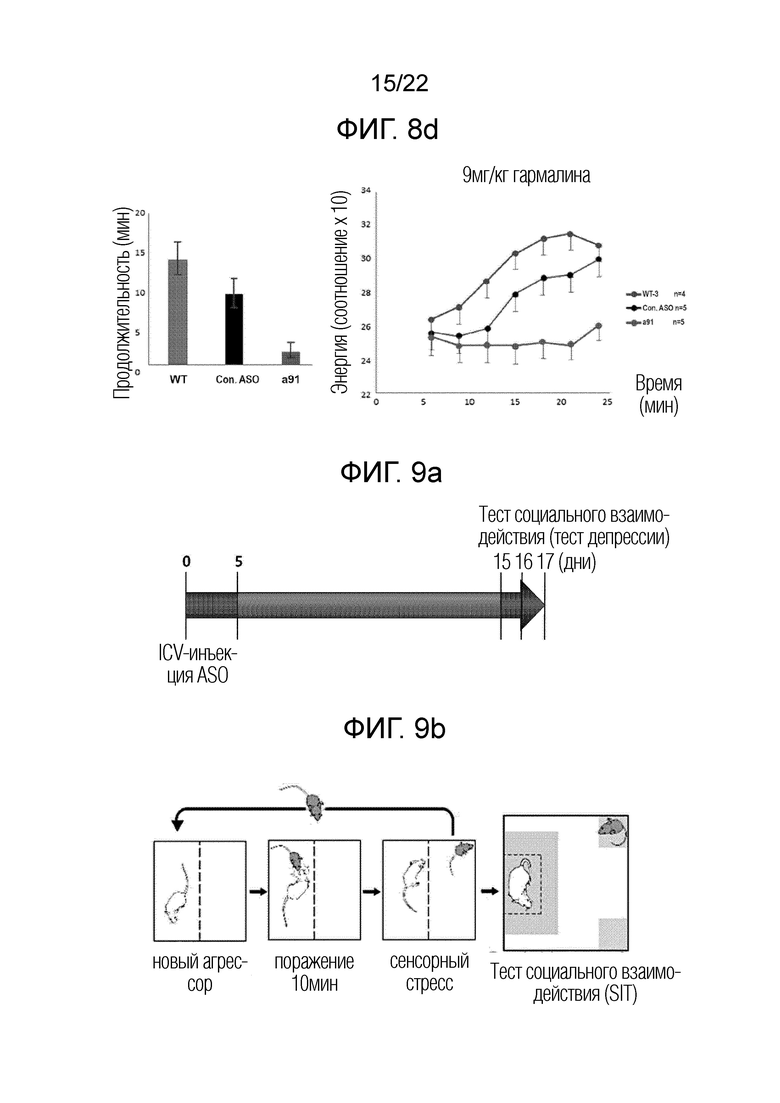
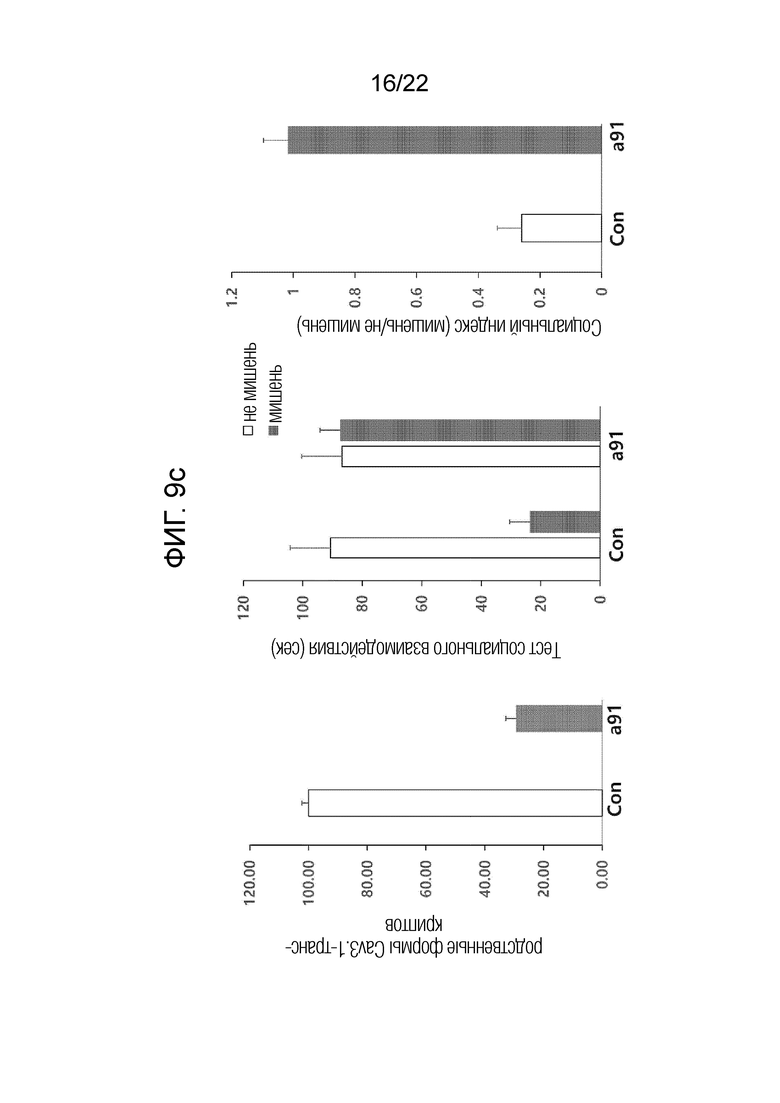
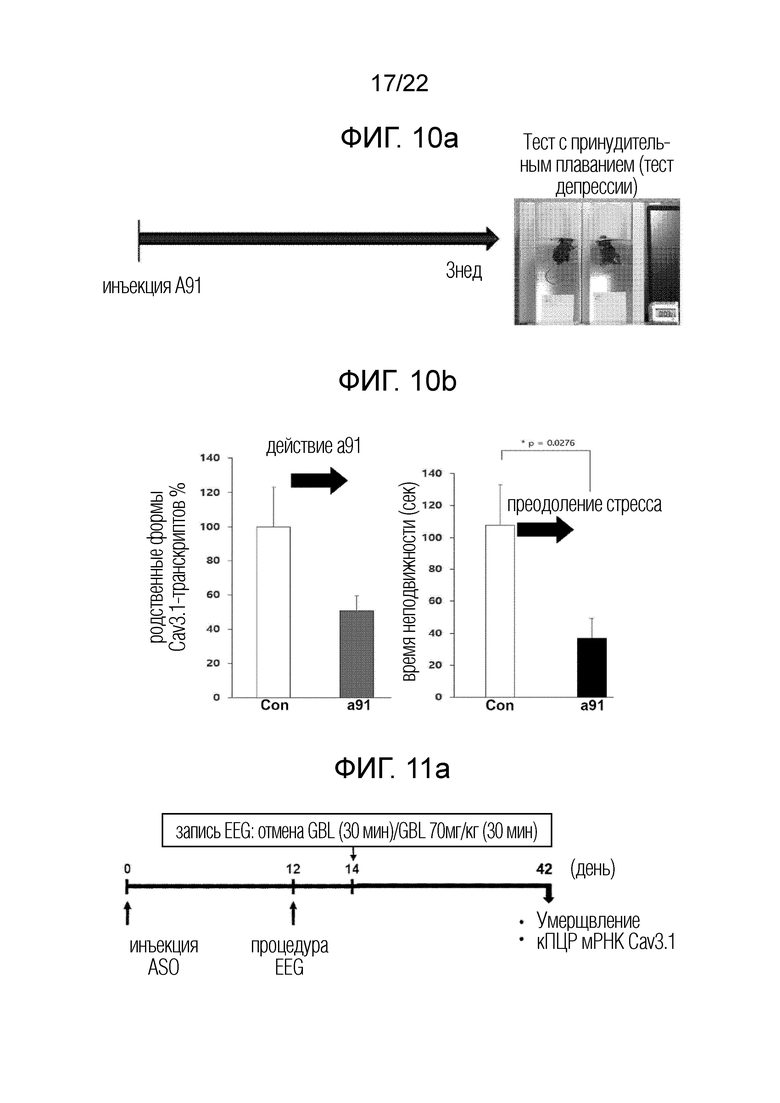
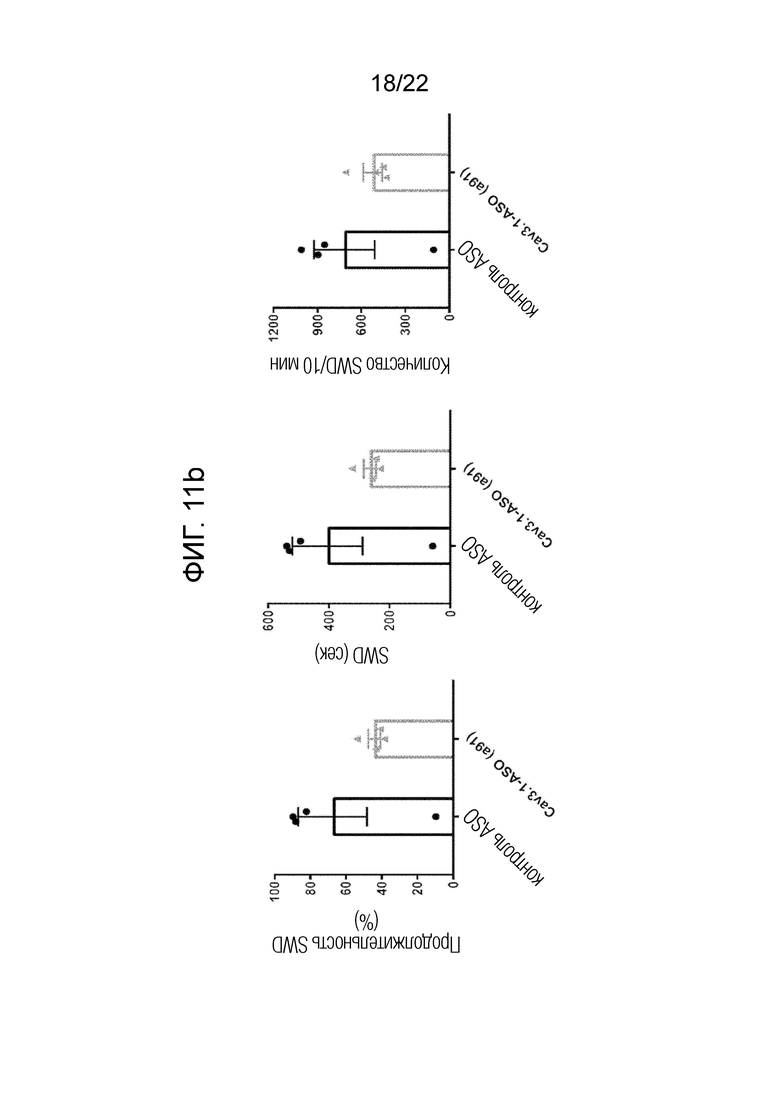
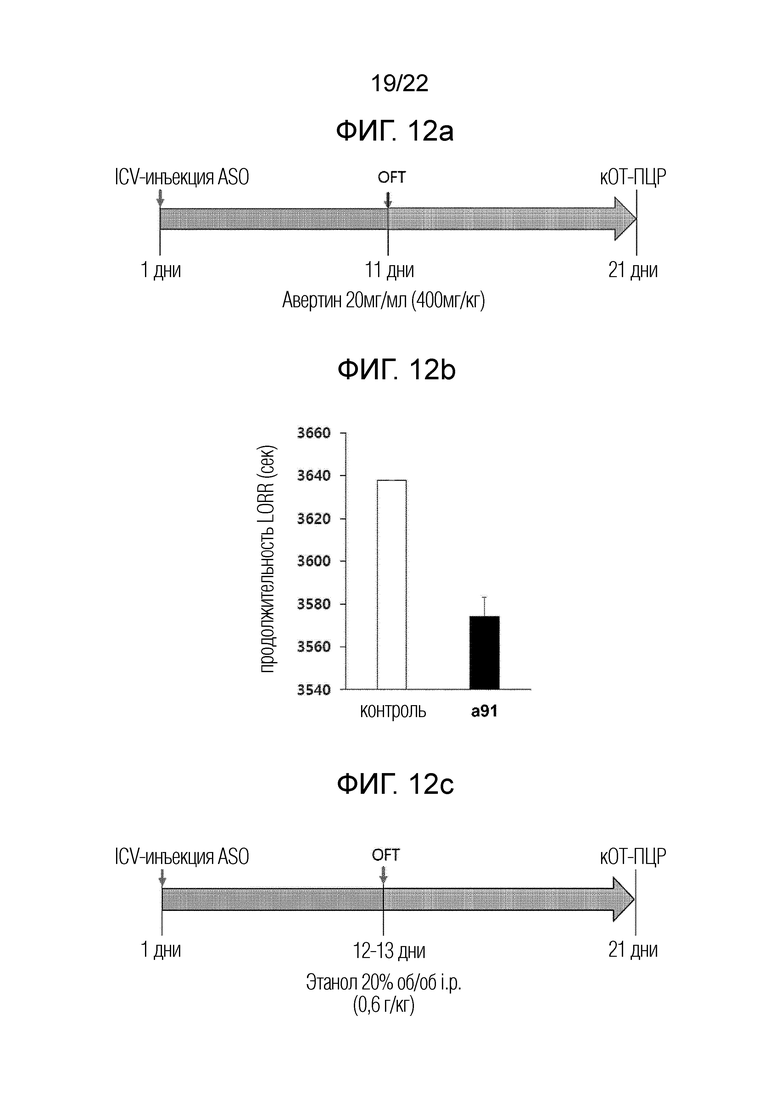
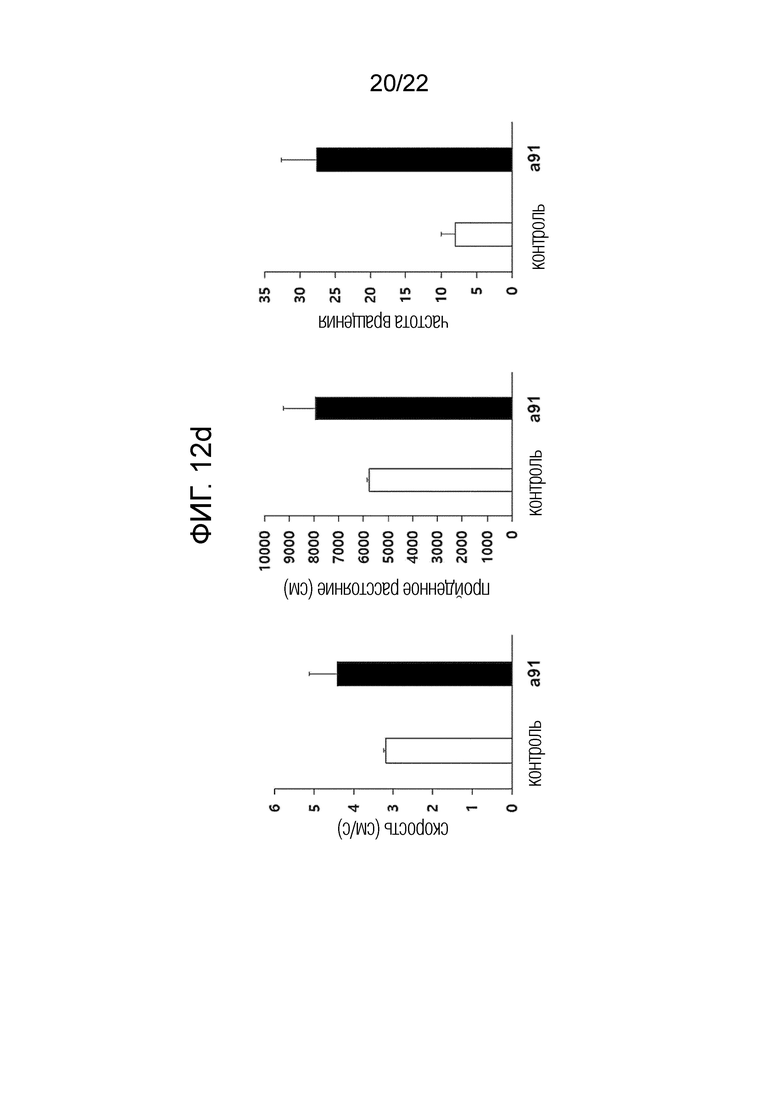
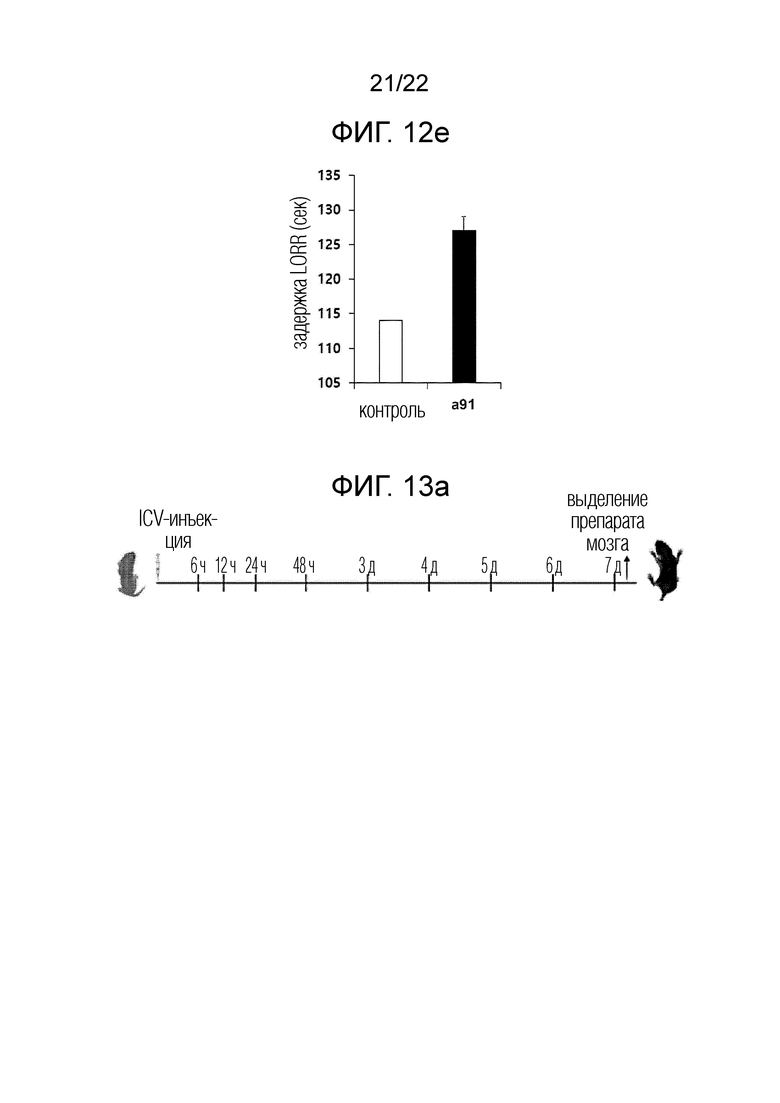

Группа изобретений относится к области химии и фармацевтики. 1 объект представляет собой антисмысловой олигонуклеотид, нацеленный на последовательность нуклеиновой кислоты SEQ ID NO: 1, для ингибирования экспрессии гена, кодирующего Cav3.1, где антисмысловой олигонуклеотид способен к гибридизации с последовательностью нуклеиновой кислоты SEQ ID NO: 1 и имеет длину от 16 до 25 нт. 2-7 объекты – фармацевтические композиции, содержащие указанный антисмысловой олигонуклеотид, для лечения болезни Паркинсона, депрессии, эссенциального тремора, эпилепсии, тревожного расстройства, восстановления сознания у пациента, находящегося в бессознательном состоянии. Технический результат заключается в специфическом ингибировании антисмысловым олигонуклеотидом экспрессии гена Cav3.1 при малых побочных эффектах при введении in vivo. 7 н. и 11 з.п. ф-лы, 13 ил., 3 табл., 9 пр.
1. Антисмысловой олигонуклеотид, нацеленный на последовательность нуклеиновой кислоты SEQ ID NO: 1, для ингибирования экспрессии гена, кодирующего Cav3.1, где антисмысловой олигонуклеотид способен к гибридизации с последовательностью нуклеиновой кислоты SEQ ID NO: 1 и имеет длину от 16 до 25 нт.
2. Антисмысловой олигонуклеотид по п. 1, где антисмысловой олигонуклеотид представляет собой ДНК, РНК или химерную молекулу нуклеиновой кислоты ДНК/РНК.
3. Антисмысловой олигонуклеотид по п. 1, где антисмысловой олигонуклеотид содержит последовательность нуклеиновой кислоты SEQ ID NO: 20 или SEQ ID NO: 21.
4. Антисмысловой олигонуклеотид по п. 1, где часть или весь фосфодиэфирный (РО) остов замещены фосфоротиоатом (PS), фосфородитиоатом или метилфосфонатом.
5. Антисмысловой олигонуклеотид по п. 1, где часть или все группы 2'-ОН сахарной части модифицированы.
6. Антисмысловой олигонуклеотид по п. 5, где группа 2'-ОН сахарной части замещена на -O-CH3 (-OMe), -O-CH2-CH3 (-OEt), -O-CH2-CH2-O-CH3 (-MOE), -O-CH2-CH2-CH2-NH2, -O-CH2-CH2-CH2-OH, -OF или -F.
7. Антисмысловой олигонуклеотид по п. 1, где сахарная часть содержит по меньшей мере один нуклеотидный аналог, имеющий образованное на нем дополнительное кольцо.
8. Антисмысловой олигонуклеотид по п. 7, где нуклеотидный аналог представляет собой замкнутую нуклеиновую кислоту (LNA).
9. Антисмысловой олигонуклеотид по п. 7, где нуклеотидные аналоги составляют по меньшей мере 33% от общего количества нуклеотидов.
10. Антисмысловой олигонуклеотид по п. 1, где антисмысловой олигонуклеотид представляет собой гэпмер.
11. Антисмысловой олигонуклеотид по п. 10, где указанный гэпмер представляет собой молекулу нуклеиновой кислоты, содержащую единицу РНК длиной 3-7 нт на каждом конце и ДНК длиной 8-12 нт, расположенную между указанными единицами РНК.
12. Антисмысловой олигонуклеотид по п. 11, где часть или вся указанная РНК представляет собой LNA.
13. Фармацевтическая композиция для лечения болезни Паркинсона, содержащая антисмысловой олигонуклеотид по любому из пп. 1-12 и по меньшей мере один фармацевтически приемлемый носитель.
14. Фармацевтическая композиция для лечения депрессии, содержащая антисмысловой олигонуклеотид по любому из пп. 1-12 в качестве активного ингредиента и по меньшей мере один фармацевтически приемлемый носитель.
15. Фармацевтическая композиция для лечения эссенциального тремора, содержащая антисмысловой олигонуклеотид по любому из пп. 1-12 в качестве активного ингредиента и по меньшей мере один фармацевтически приемлемый носитель.
16. Фармацевтическая композиция для лечения эпилепсии, содержащая антисмысловой олигонуклеотид по любому из пп. 1-12 в качестве активного ингредиента и по меньшей мере один фармацевтически приемлемый носитель.
17. Фармацевтическая композиция для лечения тревожного расстройства, содержащая антисмысловой олигонуклеотид по любому из пп. 1-12 в качестве активного ингредиента и по меньшей мере один фармацевтически приемлемый носитель.
18. Фармацевтическая композиция для восстановления сознания у пациента, находящегося в бессознательном состоянии, содержащая антисмысловой олигонуклеотид по любому из пп. 1-12 в качестве активного ингредиента и по меньшей мере один фармацевтически приемлемый носитель.
| US 2020197377 A1, 25.06.2020 | |||
| WO 2013039271 A1, 21.03.2013 | |||
| WO 2021007487 A1, 14.01.2021 | |||
| US 2009131495 A1, 21.05.2009 | |||
| CHOI S | |||
| et al | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Устройство для закрепления лыж на раме мотоциклов и велосипедов взамен переднего колеса | 1924 |
|
SU2015A1 |
| - Vol | |||
| Прялка для изготовления крученой нити | 1920 |
|
SU112A1 |
| - No | |||
| Видоизменение пишущей машины для тюркско-арабского шрифта | 1923 |
|
SU25A1 |
| - P | |||
| Четырехтактный двигатель внутреннего горения | 1925 |
|
SU7839A1 |
Авторы
Даты
2025-05-12—Публикация
2022-08-17—Подача