ОБЛАСТЬ ТЕХНИКИ
Перекрестная ссылка на родственную(ые) заявку(и)
Настоящая заявка испрашивает преимущество и приоритет на основании заявки на патент Кореи №10-2021-0098072, поданной 26 июля 2021 г., и все содержание, раскрытое в документе соответствующей заявки на патент Кореи, включено как часть описания настоящего изобретения.
Настоящая заявка относится к микроорганизму, имеющему ДНК-связывающий регулятор транскрипции семейства LacI с ослабленной активностью, и способу получения L-глутаминовой кислоты с его использованием.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
С целью получения L-аминокислот и других полезных веществ проводятся различные исследования по разработке высокоэффективных микроорганизмов-продуцентов и технологии процесса ферментации. Например, в основном применяют способ, подходящий для специфичного получения целевого вещества, такой как усиление экспрессии гена, кодирующего фермент, вовлеченный в биосинтез L-глутаминовой кислоты, или удаление гена, не являющегося необходимым для биосинтеза.
Тем не менее, в связи с возросшим спросом на L-глутаминовую кислоту по-прежнему сохраняется потребность в исследованиях по повышению способности продуцировать полезную L-глутаминовую кислоту.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
ПАТЕНТНЫЙ ДОКУМЕНТ
Публикация патента США №2011-0027840.
ТЕХНИЧЕСКАЯ ПРОБЛЕМА
Одной из целей настоящей заявки является получение микроорганизма Corynebacterium sp., имеющего ДНК-связывающий регулятор транскрипции семейства LacI с ослабленной активностью и обладающего повышенной способностью продуцировать L-глутаминовую кислоту.
Другой целью настоящей заявки является разработка способа получения L-глутаминовой кислоты, включающего культивирование микроорганизма Corynebacterium sp. в среде.
ТЕХНИЧЕСКОЕ РЕШЕНИЕ
Далее будет приведено подробное описание. При этом, каждое описание и воплощение, раскрытые в настоящей заявке, могут быть применены в случае каждого другого описания и воплощения. Другими словами, все комбинации различных элементов, описанные в настоящей заявке, попадают в объем настоящей заявки. Помимо этого, нельзя считать, что объем настоящей заявки ограничен приведенным ниже подробным описанием. Кроме того, по всему описанию настоящего изобретения даются ссылки на ряд статей и патентных документов и приводятся цитаты из них. Описания цитируемых статей и патентных документов включены в данное описание посредством ссылки во всей своей полноте для более четкого представления уровня той области техники, к которой относится настоящее изобретение, и содержания настоящего изобретения.
Согласно одному из аспектов настоящей заявки предложен микроорганизм Corynebacterium sp. (или штамм, рекомбинантная клетка), с ослабленной активностью ДНК-связывающего регулятора транскрипции семейства LacI.
В настоящей заявке ДНК-связывающий белок-регулятор транскрипции семейства LacI может представлять собой белок, обладающий активностью регулятора транскрипции семейства LacI (например, регулятор транскрипции семейства LacI), и, например, может происходить из штамма Corynebacterium glutamicum с номером от Американской коллекции типовых культур (АТСС) 13869, из штамма Corynebacterium glutamicum с АТСС13032 или из штамма Corynebacterium glutamicum с АТСС14067, но этим не ограничиваться. В одном из воплощений ДНК-связывающий белок-регулятор транскрипции семейства LacI может происходить из штамма Corynebacterium glutamicum с АТСС 13869 (последовательность может быть получена из GenBank, известной базы данных Национального центра биотехнологической информации (NCBI) и, например, может иметь в GenBank № доступа WP_060564415.1). В одном из воплощений ДНК-связывающий белок-регулятор транскрипции семейства LacI, происходящий из Corynebacterium glutamicum, может иметь или содержать аминокислотную последовательность с SEQ ID NO: 3 либо состоять из данной аминокислотной последовательности или по существу состоять из данной аминокислотной последовательности. В частности, этот белок может состоять из полипептида, описываемого аминокислотной последовательностью с SEQ ID NO: 3.
В одном из воплощений ДНК-связывающий белок-регулятор транскрипции семейства LacI может представлять собой полипептид, обладающий активностью ДНК-связывающего регулятора транскрипции семейства LacI, который кодируется геном ДНК-связывающего регулятора транскрипции семейства LacI, но этим не ограничивается. В одном из воплощений ген ДНК-связывающего регулятора транскрипции семейства LacI может происходить из штамма Corynebacterium glutamicum АТСС13869 и, в частности, он может содержать последовательность нуклеиновой кислоты SEQ ID NO: 4 (последовательность от 1421016-ого до 1422125-ого основания в последовательности нуклеиновой кислоты с идентификационным (ID) № доступа в GenBank для последовательности: СР016335.1, например, последовательность гена BBD29_06680).
В одном из воплощений ДНК-связывающий белок-регулятор транскрипции семейства LacI может содержать аминокислотную последовательность, имеющую гомологию или идентичность, составляющую по меньшей мере 70%, 75%, 80%, 85%, 90%, 95%, 96%, 97%, 98%, 99%, 99,5%, 99,7% либо 99,9% или более, с аминокислотной последовательностью, описанной как SEQ ID NO: 3, либо может состоять из данной последовательности. Помимо этого, если существует аминокислотная последовательность, которая имеет данную гомологию или идентичность и демонстрирует эффективность, соответствующую ДНК-связывающему белку-регулятору транскрипции семейства LacI, то ее вариант, имеющий аминокислотную последовательность, в которой некоторые участки последовательности делетированы, модифицированы, заменены, консервативно заменены или добавлены, может рассматриваться как ДНК-связывающий белок-регулятор транскрипции семейства LacI. Примером этого является случай, когда в последовательности на N-конце, С-конце и/или внутри аминокислотной последовательности имеются добавление или делеция, встречающаяся в природе мутация, молчащая мутация или консервативная замена, не приводящие к изменению функции данного варианта по настоящей заявке.
Консервативная замена означает замену одной аминокислоты на другую аминокислоту, имеющую сходные структурные и/или химические свойства. Основой для такой аминокислотной замены, как правило, может быть сходство полярности, заряда, растворимости, гидрофобности, гидрофильности и/или амфипатической природы остатка. Обычно консервативная замена может оказывать незначительное влияние или не оказывает влияния на активность белка или полипептида.
В настоящей заявке тот факт, что полинуклеотид или полипептид «имеет, содержит конкретную последовательность нуклеиновой кислоты (нуклеотидную последовательность) или аминокислотную последовательность либо состоит из данной последовательности или по существу состоит из данной последовательности» может означать, что полинуклеотид или полипептид по существу содержит конкретную последовательность нуклеиновой кислоты (нуклеотидную последовательность) или аминокислотную последовательность, и может истолковываться как содержащий «по существу эквивалентную последовательность», при этом в данную конкретную последовательность нуклеиновой кислоты (нуклеотидную последовательность) или аминокислотную последовательность внесена мутация (делеция, замена, модификация и/или добавление) с сохранением исходной функции и/или желаемой функции данного полинуклеотида или полипептида. В одном из воплощений тот факт, что полинуклеотид или полипептид «имеет или содержит конкретную последовательность нуклеиновой кислоты (нуклеотидную последовательность) или аминокислотную последовательность, состоит из данной последовательности или по существу состоит из данной последовательности», может означать, что полинуклеотид или полипептид: (1) по существу содержит данную конкретную последовательность нуклеиновой кислоты (нуклеотидную последовательность) или аминокислотную последовательность или (2) состоит из последовательности нуклеиновой кислоты или аминокислотной последовательности, имеющей гомологию или идентичность, составляющую 70% или более, 80% или более, 85% или более, 90% или более, 95% или более, 96% или более, 97% или более, 98% или более, 99% или более, 99,5% или более, 99,7% или более либо 99,9% или более с данной конкретной последовательностью или аминокислотной последовательностью, либо по существу содержит ее, и сохраняет исходную функцию и/или желаемую функцию. В одном из воплощений термин «желаемая функция» может означать функцию, связанную с повышением (усилением) у микроорганизма или приданием микроорганизму способности продуцировать L-глутаминовую кислоту.
Использованный в настоящей заявке термин «гомология» или «идентичность» означает степень сходства между двумя указанными аминокислотными последовательностями или нуклеотидными последовательностями, которая может быть выражена в процентном отношении. Термины «гомология» и «идентичность» часто могут быть использованы взаимозаменяемо.
Томологию или идентичность последовательностей для консервативных полинуклеотидов или полипептидов можно определить при сочетанном применении стандартного алгоритма выравнивания и штрафа за разрыв по умолчанию, установленных используемой программой. По существу, гомологичная или идентичная последовательность обычно может гибридизоваться со всей последовательностью или ее частью в условиях умеренной или высокой жесткости. Очевидно, что гибридизация также включает гибридизацию с полинуклеотидом, содержащим общий кодон или кодон, рассматриваемый как вырожденный кодон в этом полинуклеотиде.
Имеют ли любые две полинуклеотидные или полипептидные последовательности гомологию, сходство или идентичность, можно определить, применяя известные компьютерные алгоритмы, такие как, например, программа в формате «FASTA», использующая параметры по умолчанию как в работе Pearson et al. (1988) Proc. Natl. Acad. Sci. USA, 85: 2444. Это можно определить иным образом, используя алгоритм Нидлмана-Вунша (Needleman and Wunsch, 1970, J. Mol. Biol., 48: 443-453), который реализован в программе Нидлмана из Европейского пакета открытого программного обеспечения по молекулярной биологии (EMBOSS) (Rice et al., 2000, Trends Genet., 16: 276-277) (версия 5.0.0 или более поздняя версия) (в том числе пакет программ GCG (Devereux J., et al., Nucleic Acids Research, 12: 387 (1984)), BLASTP (средство поиска основного локального выравнивания (BLAST) для белков), BLASTN (BLAST для нуклеотидов), FASTA (Atschul, S.F., et al., J. Molec. Biol., 215: 403 (1990); Guide to Huge Computers, Martin J. Bishop, Ed., Academic Press, San Diego, 1994 и Carillo et al., (1988) SIAM J. Applied Math., 48: 1073). Например, для определения гомологии, сходства или идентичности можно использовать BLAST или ClustalW (кластерный анализ W множественных выравниваний) из Национального центра биотехнологической информации.
Томологию, сходство или идентичность полинуклеотидов или полипептидов можно определить путем сравнения информации о последовательностях, используя, например, компьютерную программу GAP, например, Needleman et al. (1970), J. Mol. Biol., 48: 443, которая описана, например, в Smith and Waterman, Adv. Appl. Math. (1981), 2: 482. Кратко, с использованием программы GAP гомологию, сходство или идентичность можно определить в виде значения, полученного в результате деления числа аналогично выравненных символов (а именно, нуклеотидов или аминокислот) на общее число символов в более короткой из этих двух последовательностей. Параметры по умолчанию для программы GAP могут включать (1) бинарную матрицу сравнения (содержащую значения 1 в случае идентичности и 0 в случае отсутствия идентичности) и не отягощенную вычислительными погрешностями матрицу сравнения согласно Gribskov et al. (1986) Nucl. Acids Res., 14: 6745, описанную в Schwartz and Dayhoff, eds., Atlas Of Protein Sequence And Structure, National Biomedical Research Foundation, pp. 353-358 (1979) (или матрицу замен EDNAFULL (EMBOSS версия NCBI NUC4.4)); (2) штраф 3,0 за каждый разрыв и дополнительный штраф 0,10 за каждый символ в каждом разрыве (или штраф за внесение разрыва 10 и штраф за удлинение разрыва 0,5); и (3) отсутствие штрафа за внесение концевых разрывов.
Использованный в настоящей заявке термин «микроорганизм (или штамм)» включает в себя все микроорганизмы дикого типа или микроорганизмы, генетически модифицированные естественным либо искусственным образом, и может означать микроорганизм, содержащий генетическую модификацию с целью продуцирования желаемого полипептида, белка или продукта, как, например, микроорганизм со специфическим механизмом, ослабленным или усиленным таким путем, как вставка чужеродного гена или ослабление активности собственного гена.
В настоящей заявке термин «ослабление» полипептида представляет собой понятие, включающее оба случая, а именно, снижение активности или отсутствие активности. Термин «ослабление» можно использовать наряду с такими терминами, как инактивация, недостаточность, отрицательная регуляция, снижение, уменьшение, аттенуация и тому подобное.
Ослабление также может означать случай, когда активность самого полипептида снижена или устранена по сравнению с активностью полипептида, который имеется в исходном микроорганизме, вследствие мутации в полинуклеотиде, кодирующем данный полипептид (или белок, например, ДНК-связывающий белок-регулятор транскрипции семейства LacI), и тому подобного, случай, когда общий уровень активности и/или концентрация (уровень экспрессии) полипептида в клетке ниже по сравнению с таковыми у природного штамма в клетке вследствие подавления экспрессии гена, кодирующего полипептид, или подавления трансляции с образованием полипептида, случай, когда экспрессии полинуклеотид а не происходит вообще, и/или случай, когда активность полипептида не проявляется даже при экспрессии полинуклеотид а. Термин «эндогенная активность» означает активность конкретного полипептида, который имелся в родительском штамме до осуществления трансформации либо изначально имелся в микроорганизме дикого типа или немодифицированном микроорганизме, когда признак изменен вследствие генетической мутации, вызванной природным или искусственным фактором. Этот термин можно использовать взаимозаменяемо с термином «активность до модификации». Тот факт, что полипептид в плане его активности является «инактивированным, обладает недостаточной, более низкой, подвергнутой отрицательной регуляции, сниженной или аттенуированной» по сравнению с эндогенной активностью означает то, что она понижена по сравнению с активностью конкретного полипептида, который имеется в родительском штамме до осуществления трансформации или изначально имеется в немодифицированном микроорганизме.
Ослабление активности этого полипептида (или белка, например, ДНК-связывающего белка-регулятора транскрипции семейства LacI) может быть осуществлено любым способом, известным в данной области техники, но этим не ограничивается, и может быть достигнуто путем применения различных способов, хорошо известных в данной области техники (например, описанных в Nakashima N. et al., Bacterial cellular engineering by genome editing and gene silencing. Int. J. Mol. Sci., 2014, 15(2): 2773-2793, Sambrook et al., Molecular Cloning, 2012, и т.д.).
В частности, ослабление полипептида (или белка, например, ДНК-связывающего белка-регулятора транскрипции семейства LacI; далее называемого полипептидом) может быть результатом:
1) делеции всего или части гена, кодирующего полипептид;
2) модификации регулирующей экспрессию области (или регулирующей экспрессию последовательности) с тем, чтобы уровень экспрессии гена, кодирующего полипептид, снижался;
3) модификации аминокислотной последовательности, составляющей полипептид, с тем, чтобы активность полипептида устранялась или ослаблялась (например, в результате делеции/замены/добавления одной или более чем одной из аминокислот в аминокислотной последовательности);
4) модификации последовательности гена, кодирующего полипептид, с тем, чтобы активность данного полипептида устранялась или ослаблялась (например, в результате делеции/замены/добавления одного или нескольких нуклеотидов в нуклеотидной последовательности нуклеиновой кислоты кодирующего полипептид гена с тем, чтобы данный полипептид модифицировался с устранением или ослаблением его активности);
5) модификации нуклеотидной последовательности инициирующего трансляцию кодона или нетранслируемого участка на 5'-конце (5'-UTR) транскрипта гена, кодирующего полипептид;
6) введения антисмыслового олигонуклеотида (например, антисмысловой РНК), который комплементарно связывается с транскриптом гена, кодирующего полипептид;
7) добавления последовательности, комплементарной последовательности Шайна-Дальгарно, перед последовательностью Шайна-Дальгарно гена, кодирующего данный полипептид, с целью образования вторичной структуры, к которой прикрепление рибосомы невозможно;
8) добавления промотора для обратной транскрипции на 3'-конец открытой рамки считывания (ORF) последовательности гена, кодирующего данный полипептид (метод инженерии с обратной транскрипцией, RTE); или
9) комбинации из двух или более пунктов, выбранных из приведенных выше пунктов (1)-(8), но конкретно ими не ограничиваясь.
Например, 1) делеция всего или части гена, кодирующего полипептид, может представлять собой делетирование всего полинуклеотида, кодирующего собственный целевой полипептид в хромосоме, замену на полинуклеотид, в котором часть нуклеотидов делетирована, или замену на маркерный ген.
Помимо этого, 2) модификация регулирующей экспрессию области (или регулирующей экспрессию последовательности) может представлять собой результат возникновения мутации в регулирующей экспрессию области (или регулирующей экспрессию последовательности) вследствие делеции, вставки, неконсервативной или консервативной замены или их комбинации, либо замены на последовательность, характеризующуюся гораздо более слабой активностью. Регулирующая экспрессию область включает промотор, последовательность оператора, последовательность, кодирующую сайт связывания рибосомы, и последовательность, регулирующую терминацию транскрипции и трансляции, но этим не ограничивается.
Кроме того, 3) модификация нуклеотидной последовательности инициирующего трансляцию кодона или 5'-UTR участка транскрипта гена, кодирующего полипептид, может представлять собой, например, замену на нуклеотидную последовательность другого инициирующего кодона с более низким уровнем экспрессии полипептида по сравнению с собственным инициирующим кодоном, но этим не ограничивается.
Помимо этого, модификация аминокислотной последовательности или полинуклеотидной последовательности согласно пунктам (4) и (5) может представлять собой результат возникновения мутации в последовательности вследствие делеции, вставки, неконсервативной или консервативной замены или их комбинации в аминокислотной последовательности полипептида или полинуклеотидной последовательности, кодирующей данный полипептид, с последующим ослаблением активности полипептида, либо представлять собой замену на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную так, чтобы она имела гораздо более слабую активность, либо на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную так, чтобы она не имела никакой активности, но этим не ограничивается. Например, экспрессию гена можно подавлять или ослаблять посредством введения мутации в полинуклеотидную последовательность с образованием стоп-кодона, но этим не ограничиваясь. Термин «стоп-кодон» относится к кодону, который действует как сигнал, указывающий на то, что он не кодирует какую-либо аминокислоту, и что процесс синтеза белка в соответствии с кодонами на мРНК завершен, и обычно в качестве стоп-кодона могут быть использованы 3 вида кодонов - UAA, UAG и UGA.
Микроорганизм, соответствующий одному из примеров, может быть заменен на (замещен) микроорганизм, у которого в последовательность ORF (открытой рамки считывания) собственного гена с SEQ ID NO: 4 введена мутация с образованием стоп-кодона, и, например, кодон, соответствующий аминокислоте в положении 310 (например, соответствующий глутамину Gin, Q) ДНК-связывающего белка-регулятора транскрипции семейства LacI, может быть заменен на (замещен) стоп-кодон. Например, полинуклеотид, кодирующий аминокислоту глутамин в положении 310 в последовательности нуклеиновой кислоты с SEQ ID NO: 4, представляет собой «CAG», и его можно использовать в качестве кодона, в который вводят мутацию с заменой (замещением) «CAG» на «ТАА», «TAG» или «TGA», соответственно, для замены его на стоп-кодон. В одном из воплощений последовательность нуклеиновой кислоты, в которую введена мутация с образованием стоп-кодона, может представлять собой последовательность нуклеиновой кислоты с SEQ ID NO: 2 (в полинуклеотиде дикого типа с SEQ ID NO: 4 в положении 928 С заменен на Т; С928Т).
6) Ссылку на введение антисмыслового олигонуклеотида (например, антисмысловой РНК), который комплементарно связывается с транскриптом гена, кодирующего полипептид, может найти в литературе, например, в работе Weintraub, Н. et al., Antisense-RNA as a molecular tool for genetic analysis, Reviews - Trends in Genetics, Vol.1(1), 1986.
7) Добавление последовательности, комплементарной последовательности Шайна-Дальгарно, перед последовательностью Шайна-Дальгарно гена, кодирующего данный полипептид, с целью образования вторичной структуры, к которой прикрепление рибосомы невозможно, может сделать трансляцию мРНК невозможной или снизить ее скорость.
8) Добавление промотора для обратной транскрипции на 3'-конец открытой рамки считывания (ORF) последовательности гена, кодирующего данный полипептид (метод инженерии с обратной транскрипцией, RTE), может сделать антисмысловой нуклеотид комплементарным транскрипту гена, кодирующему полипептид, с ослаблением его активности.
В настоящей заявке термин «усиление» активности полипептида означает то, что активность полипептида возрастает по сравнению с эндогенной активностью. Термин «усиление» можно использовать взаимозаменяемо с такими терминами, как активация, положительная регуляция, сверхэкспрессия и возрастание и тому подобное. При этом активация, усиление, положительная регуляция, сверхэкспрессия и возрастание могут включать в себя все виды проявления активности, которой изначально полипептид не обладал, либо проявление улучшенной активности по сравнению с эндогенной активностью или активностью до модификации. Термин «эндогенная активность» означает активность конкретного полипептида, которую исходно имеет родительский штамм до преобразования или имеет немодифицированный микроорганизм, когда признак изменяется в результате генетической мутации под действием природного или искусственного фактора. Этот термин можно использовать взаимозаменяемо с термином «активность до модификации». Тот факт, что активность полипептида «усиливается», «претерпевает положительную регуляцию», «сверхэкспрессируется» или «возрастает» по сравнению с эндогенной активностью, означает то, что она улучшается по сравнению с активностью и/или концентрацией (уровнем экспрессии) конкретного полипептида, которую исходно имеет родительский штамм до преобразования или имеет немодифицированный микроорганизм.
Данное усиление может быть достигнуто посредством введения чужеродного полипептида или повышения активности и/или концентрации (уровня экспрессии) собственного полипептида. Усиление активности полипептида может быть подтверждено данными по возрастанию уровня активности или уровня экспрессии соответствующего полипептида либо количества продукта, выделенного в случае наличия соответствующего полипептида.
Для усиления активности полипептида можно применять различные способы, хорошо известные в данной области техники, без ограничения, при условии, что это может усиливать активность целевого полипептида по сравнению с активностью в микроорганизме до модификации. В частности, можно использовать метод генной инженерии и/или белковой инженерии, хорошо известный специалистам в данной области техники, который представляет собой обычный метод молекулярной биологии, но этим не ограничиваясь (например, Sitnicka et al., Functional Analysis of Genes. Advances in Cell Biology. 2010, Vol.2, 1-16; Sambrook et al., Molecular Cloning, 2012; и т.д.).
Конкретно, усиление активности полипептида по настоящей заявке может быть результатом:
1) увеличения в клетке числа копий полинуклеотида, кодирующего полипептид;
2) замены области, регулирующей экспрессию гена в хромосоме, кодирующего полипептид, на последовательность с высокой активностью;
3) модификации нуклеотидной последовательности инициирующего трансляцию кодона или S'-UTR участка транскрипта гена, кодирующего полипептид;
4) модификации аминокислотной последовательности полипептида для усиления активности данного полипептида;
5) модификации полинуклеотидной последовательности, кодирующей полипептид, для усиления активности данного полипептида (например, модификации полинуклеотидной последовательности кодирующего полипептид гена для кодирования полипептида, модифицированного с целью усиления активности данного полипептида);
6) введения чужеродного полипептида, проявляющего активность данного полипептида, или кодирующего его чужеродного полинуклеотида;
7) оптимизации кодонов полинуклеотида, кодирующего полипептид;
8) деформации или химической модификации подлежащего воздействию сайта, выбираемого на основании анализа третичной структуры полипептида; или
9) комбинации из двух или более пунктов, выбранных из пунктов (1)-(8), но конкретно этим не ограничиваясь.
Более конкретно:
1) Увеличение в клетке числа копий полинуклеотида, кодирующего полипептид, может быть достигнуто посредством введения в клетку-хозяина вектора, который может реплицироваться и функционировать независимо от хозяина, и в котором полинуклеотид, кодирующий соответствующий полипептид, связан функциональным образом. Иным образом, полинуклеотид, кодирующий соответствующий полипептид, может быть получен путем введения 1 копии или 2 копий либо большего числа копий в хромосому клетки-хозяина. Введение в хромосому может быть осуществлено путем введения в клетку-хозяина вектора, способного встраивать полинуклеотид в хромосому, но этим не ограничивается.
2) Замена области, регулирующей экспрессию (или последовательности, регулирующей экспрессию) гена в хромосоме, кодирующей полипептид, на последовательность с высокой активностью может происходить, например, в результате возникновения мутации в данной последовательности посредством делеции, вставки, неконсервативной или консервативной замены либо их комбинации, или в результате замены на последовательность, характеризующуюся гораздо более высокой активностью, для дополнительного усиления активности регулирующей экспрессию области. Регулирующая экспрессию область, хотя конкретно не ограничивается этим, может содержать промотор, последовательность оператора, последовательность, кодирующую сайт связывания рибосомы, последовательность, регулирующую терминацию транскрипции и трансляции, и тому подобное. В качестве одного из воплощений возможна замена исходного промотора на сильный промотор, но этим не ограничиваясь.
Примеры известных сильных промоторов включают промоторы CJ1-CJ7 (патент США US 7662943 В2), lac промотор, trp промотор, trc промотор, tac промотор, PR промотор фага лямбда, PL промотор, tet промотор, gapA промотор, SPL7 промотор, SPL13 (sm3) (патент США US 10584338 В2), O2 промотор (патент США US 10273491 В2), tkt промотор, усс А промотор и тому подобные, но этим не ограничиваются.
3) Модификация нуклеотидной последовательности инициирующего трансляцию кодона или 5'-UTR участка транскрипта гена, кодирующего полипептид, может представлять собой, например, замену на нуклеотидную последовательность другого инициирующего кодона, имеющего более высокий уровень экспрессии полипептида по сравнению с собственным инициирующим кодоном, но этим не ограничивается.
Модификация аминокислотной последовательности или полинуклеотидной последовательности согласно пунктам 4) и 5) может представлять собой результат возникновения мутации в последовательности вследствие делеции, вставки, неконсервативной или консервативной замены или их комбинации в аминокислотной последовательности полипептида или полинуклеотидной последовательности, кодирующей данный полипептид, с последующим усилением активности полипептида, либо представлять собой замену на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную так, чтобы она имела гораздо более сильную активность, либо на аминокислотную последовательность или полинуклеотидную последовательность, модифицированную с возрастанием активности, но этим не ограничивается. Данную замену можно осуществлять, в частности, посредством вставки полинуклеотида в хромосому в результате гомологической рекомбинации, но этим не ограничиваясь. В этом случае, используемый вектор может дополнительно содержать селективный маркер для подтверждения вставки в хромосому.
6) Введение чужеродного полипептида, проявляющего активность данного полипептида, может представлять собой введение в клетку-хозяина чужеродного полинуклеотида, кодирующего полипептид, проявляющий такую же или аналогичную данному полипептиду активность. Выбор чужеродного полинуклеотида не ограничивается его происхождением или последовательностью, при условии, что он проявляет такую же или аналогичную данному полипептиду активность. Используемый для введения способ может быть осуществлен после надлежащего выбора способа трансформации, известного специалистам в данной области техники, и, поскольку полипептид получают путем экспрессии введенного полинуклеотида, то его активность может быть увеличена.
7) Оптимизация кодонов полинуклеотида, кодирующего полипептид, может представлять собой оптимизацию кодонов эндогенного полинуклеотида с целью усиления транскрипции или трансляции в клетке хозяина, или оптимизацию кодонов для чужеродного полинуклеотида с целью достижения оптимизированной транскрипции и трансляции в клетке хозяина.
8) Деформация или химическая модификация подлежащего воздействию сайта, выбираемого на основании анализа третичной структуры полипептида, может представлять собой, например, деформацию или модификацию для определения белка-кандидата на роль матрицы в зависимости от степени сходства последовательности посредством сравнения информации по последовательности полипептида, подлежащего анализу, с базой данных, в которой хранится информация по последовательностям основных белков, и подтверждения на основе этого структуры и, таким образом, выбора подлежащего воздействию сайта для осуществления деформации или химической модификации.
Такое усиление активности полипептида может представлять собой повышение уровня экспрессии либо активности или концентрации соответствующего полипептида на основании данных по активности или концентрации полипептида, экспрессируемого в штамме микроорганизма дикого типа или в штамме микроорганизма до модификации либо возрастание количества продукта, продуцируемого с участием соответствующего полипептида, но этим не ограничивается.
Модификация (например, модификация для кодирования вышеупомянутого варианта белка) части или всего полинуклеотида в микроорганизме по настоящей заявке может быть обусловлена (а) гомологической рекомбинацией с использованием вектора для вставки в хромосому в микроорганизме или редактированием генома с использованием генетических ножниц (генетически сконструированной нуклеазы, например, CRISPR-Cas9), и/или (б) обработкой светом, таким как ультрафиолетовые лучи и радиоактивное излучение, и/или химическим веществом, но этим не ограничивается. Способ модификации части или всего гена может включать способ с применением технологии рекомбинантной ДНК. Например, посредством введения в микроорганизм нуклеотидной последовательности или вектора, содержащего нуклеотидную последовательность, имеющую гомологию с целевым геном, с целью вызывания гомологической рекомбинации можно осуществить делецию части или всего гена. Нуклеотидная последовательность или вектор, подлежащие введению, могут содержать доминантный селективный маркер, но этим не ограничиваются.
В одном из воплощений причиной ослабления полипептида (или белка, например, ДНК-связывающего белка-регулятора транскрипции семейства LacI; далее описываемого как полипептид) может быть применение метода рекомбинации. Метод рекомбинации может включать гомологическую рекомбинацию. Когда вектор, содержащий часть последовательности гена, кодирующего полипептид, используют для трансформации микроорганизма и осуществляют культивирование в присутствии продукта - селективного маркера, применение метода гомологической рекомбинации может привести к гомологической рекомбинации части последовательности данного гена с собственным геном в микроорганизме.
Микроорганизм по настоящей заявке может представлять собой микроорганизм, в котором ДНК-связывающий белок-регулятор транскрипции семейства LacI или кодирующий его полинуклеотид инактивирован или ослаблен; или микроорганизм (например, рекомбинантный микроорганизм), генетически модифицированный с использованием вектора, в результате чего ДНК-связывающий белок-регулятор транскрипции семейства LacI или полинуклеотид инактивируется или ослабляется, но без ограничения этим.
Микроогранизм (или штамм, рекомбинантная клетка) по настоящей заявке может представлять собой микроорганизм со способностью продуцировать L-глутаминовую кислоту или с усиленной способностью продуцировать L-глутаминовую кислоту (или с повышенным выходом L-глутаминовой кислоты).
Микроорганизм по настоящей заявке может представлять собой микроорганизм, естественным образом обладающий способностью продуцировать L-глутаминовую кислоту, или микроорганизм, в котором активность ДНК-связывающего белка-регулятора транскрипции семейства LacI ослаблена, и/или когда родительскому штамму, не обладающему способностью продуцировать L-глутаминовую кислоту придана или улучшена способность продуцировать L-глутаминовую кислоту, но без ограничения этим.
Тот факт, что микроорганизм (или штамм, рекомбинантная клетка) обладает улучшенной способностью продуцировать L-глутаминовую кислоту (или улучшенным выходом L-глутаминовой кислоты) или обладает способностью продуцировать L-глутаминовую кислоту, может означать, что в данном микроорганизме (или штамме, рекомбинантной клетке) способность продуцировать L-глутаминовую кислоту улучшена по сравнению с немодифицированным микроорганизмом, клеткой до рекомбинации, родительским штаммом и/или штаммом дикого типа, либо указано, что способность продуцировать L-глутаминовую кислоту отличается от таковой у немодифицированного микроорганизма, клетки до рекомбинации, родительского штамма и/или штамма дикого типа, которые не обладают способностью продуцировать L-глутаминовую кислоту.
Микроорганизм, имеющий ДНК-связывающий регулятор транскрипции семейства LacI с ослабленной активностью, по одному из воплощений может обладать улучшенной (повышенной) способностью продуцировать L-глутаминовую кислоту по сравнению с гомологичным немодифицированным микроорганизмом. В настоящей заявке «немодифицированный» микроорганизм не исключает штамма, содержащего мутацию, которая может возникать в микроорганизме естественным образом, но это может быть штамм дикого типа или сам природный штамм либо может означать штамм до изменения признака вследствие генетической мутации, вызываемой природным или искусственным фактором. Например, термин «немодифицированный микроорганизм» может означать штамм, в котором активность ДНК-связывающего белка-регулятора транскрипции семейства LacI не ослаблена, или штамм до момента ее ослабления согласно одному из воплощений (или штамм, в котором не введена мутация, вызывающая ослабление активности ДНК-связывающего белка-регулятора транскрипции семейства LacI, или штамм до момента ее введения). Термин «немодифицированный микроорганизм» может быть использован взаимозаменяемо с терминами «штамм до модификации», «микроорганизм до модификации», «штамм без введенной мутации», «немодифицированный штамм», «микроорганизм без введенной мутации» или «стандартный микроорганизм». То, что активность ДНК-связывающего белка-регулятора транскрипции семейства LacI ослаблена, понимается так, как упомянуто выше. В одном из воплощений целевым штаммом для сравнения, является ли способность продуцировать L-глутаминовую кислоту повышенной, то есть немодифицированным микроорганизмом, может быть штамм Corynebacterium glutamicum АТСС13032, штамм Corynebacterium glutamicum АТСС13869, штамм Corynebacterium glutamicum АТСС14067, штамм дикого типа Corynebacterium glutamicum, в котором ген odhA делетирован (например, штамм ATCC13869ΔodhA) или штамм Corynebacterium glutamicum BL2, известный как продуцирующий L-глутаминовую кислоту штамм с индуцированной N-метил-N'-нитро-N-нитрозогуанидином (NTG) мутацией (KFCC11074, патент Кореи №10-0292299), но этим не ограничиваясь.
Микроорганизм (или штамм, рекомбинантная клетка) может дополнительно содержать мутацию, приводящую к усилению продуцирования L-глутаминовой кислоты, и положение мутации и/или тип гена и/или подлежащий введению мутации белок могут быть включены без ограничения, при условии, что в результате этого будет усиливаться продуцирование L-глутаминовой кислоты. Рекомбинантная клетка может быть использована без ограничения, при условии, что она представляет собой клетку, которую можно трансформировать.
Согласно одному из воплощений в микроорганизме (или штамме, рекомбинантной клетке) с улучшенной (повышенной) способностью продуцирования (или повышенным выходом) по сравнению с родительским штаммом до введения мутации или немодифицированным микроорганизмом способность продуцировать L-глутаминовую кислоту может быть повышена примерно на 1% или более, примерно на 2,5% или более, примерно на 5% или более, примерно на 6% или более, примерно на 7% или более, примерно на 8% или более, примерно на 9% или более, примерно на 10% или более, примерно на 10,5% или более, примерно на 11% или более, примерно на 11,5% или более, примерно на 12% или более, примерно на 12,5% или более, примерно на 13% или более, примерно на 13,5% или более, примерно на 14% или более, примерно на 14,5% или более, примерно на 15% или более, примерно на 15,5% или более, примерно на 16% или более, примерно на 16,5% или более, примерно на 17% или более, примерно на 17,4% или более, примерно на 17,5% или более, примерно на 18% или более, примерно на 18,5% или более, примерно на 19% или более, примерно на 19,5% или более, примерно на 20% или более, примерно на 20,5% или более, примерно на 21% или более, примерно на 21,1% или более, примерно на 21,5% или более, примерно на 21,5% или более, примерно на 22% или более, примерно на 22,5% или более, примерно на 23% или более, примерно на 23,5% или более, примерно на 24% или более, примерно на 24,5% или более, примерно на 25% или более, примерно на 25,5% или более, примерно на 26% или более, примерно на 26,5% или более, примерно на 27% или более, примерно на 27,5% или более, примерно на 28% или более, примерно на 28,5% или более, примерно на 29% или более, примерно на 29,5% или более, примерно на 30% или более, примерно на 31% или более, примерно на 32% или более, примерно на 33% или более, примерно на 34% или более или примерно на 35% или более (при этом верхний предел особо не ограничен и, например, может составлять приблизительно 200% или менее, приблизительно 150% или менее, приблизительно 100% или менее, приблизительно 50% или менее, приблизительно 45% или менее, приблизительно 40% или менее либо приблизительно 35% или менее), и в одном из воплощений способность продуцирования может быть повышена примерно на 17,2% или более, примерно на 21,4% или более либо примерно на 21,6% или более. В другом воплощении в микроорганизме (или штамме, рекомбинантной клетке) с повышенной способностью продуцирования (или повышенным выходом) по сравнению с родительским штаммом до введения мутации или немодифицированным микроорганизмом способность продуцировать L-глутаминовую кислоту (или выход L-глутаминовой кислоты) могут быть повышены примерно в 1,1 раза или более, примерно в 1,12 раза или более, примерно в 1,13 раза или более, в 1,15 раза или более, в 1,16 раза или более, в 1,17 раза или более, в 1,18 раза или более, в 1,19 раза или более, примерно в 1,2 раза или более, примерно в 1,21 раза или более, примерно в 1,22 раза или более, в 1,25 раза или более либо примерно в 1,3 раза или более (при этом верхний предел особо не ограничен и, например, может соответствовать повышению приблизительно в 10 раз или менее, приблизительно в 5 раз или менее, приблизительно в 3 раза или менее либо приблизительно в 2 раза или менее), и в одном из воплощений способность продуцирования может быть повышена примерно в 1,172 раза или более, примерно в 1,214 раза или более, примерно в 1,216 раза или более. Более конкретно, в рекомбинантном штамме с повышенной способностью продуцирования (или повышенным выходом) по сравнению с родительским штаммом до введения мутации или немодифицированным микроорганизмом способность продуцировать L-глутаминовую кислоту может быть повышена примерно на 17,2%, примерно на 21,4% или примерно на 21,6% (или примерно в 1,17 раза, примерно в 1,21 раза или примерно в 1,22 раза), но этим не ограничиваясь. Термин «примерно» означает диапазон, включающий в себя все значения ±0,5, ±0,4,±0,3, ±0,2, ±0,1 и тому подобные, и включает в себя все значения в пределах диапазона, равные или подобные значению, следующему за термином «примерно», но этим не ограничивается.
В одном из воплощений микроорганизм Corynebacterium sp. может представлять собой Corynebacterium glutamicum, Corynebacterium crudilactis, Corynebacterium deserti, Corynebacterium efficiens, Corynebacterium callunae, Corynebacterium stationis, Corynebacterium singulare, Corynebacterium halotolerans, Corynebacterium striatum, Corynebacterium ammoniagenes, Corynebacterium pollutisoli, Corynebacterium imitans, Corynebacterium testudinoris и/или Corynebacterium flavescens.
Согласно другому воплощению рекомбинантный микроорганизм по настоящей заявке может представлять собой микроорганизм, в котором активность части белка в пути биосинтеза L-глутаминовой кислоты в свою очередь усилена или активность части белка в пути деградации L-глутаминовой кислоты дополнительно подавлена для повышения способности продуцировать L-глутаминовую кислоту.
В частности, микроорганизм по настоящей заявке может представлять собой микроорганизм, в котором белок 2-оксоглутаратдегидрогеназа A (OdhA) в свою очередь инактивирован или ген odhA в свою очередь делетирован. Более конкретно, микроорганизм по настоящей заявке может представлять собой Corynebacterium glutamicum АТСС 13869, в котором инактивирован белок OdhA, или микроорганизм Corynebacterium glutamicum АТСС 13869, в котором делетирован ген odhA. Белок OdhA может содержать аминокислотную последовательность с ID последовательности в NCBI WP_060564343.1 (например, аминокислотную последовательность с SEQ ID NO: 23). Белок OdhA может представлять собой белок, обладающий активностью многофункциональной оксоглутаратдекарбоксилазы/тиаминпирофосфат-связывающей субъединицы оксоглутаратдегидрогеназы/субъединицы сукцинилтрансферазы с дигидролипоиллизиновым остатком, происходящий из штамма Corynebacterium glutamicum. Ген odhA может происходить из штамма Corynebacterium glutamicum АТСС13869 и, в частности, может содержать последовательность нуклеиновой кислоты с SEQ ID NO: 24 (последовательность от нуклеотида 1276170 до нуклеотида 1279787 в нуклеиновокислотной последовательности с №доступа в GenBank для последовательности ID: СР016335.1, например, ген BBD29_06050).
Однако инактивация белка OdhA или делеция гена odhA представляет собой один из видов примеров и этим не ограничивается, и микроорганизм по настоящей заявке может представлять собой микроорганизм, в котором усилена активность белка в различных известных путях биосинтеза L-глутаминовой кислоты либо подавлена или ослаблена активность белка в путях ее деградации.
Согласно другому аспекту настоящей заявки предложен способ получения L-аминокислоты, включающий культивирование микроорганизма по настоящей заявке в среде.
Способ получения L-аминокислоты по настоящей заявке может включать культивирование микроорганизма по настоящей заявке в среде. Микроорганизм по настоящей заявке упомянут выше.
Помимо этого, в настоящей заявке L-аминокислота может представлять собой L-глутаминовую кислоту.
Использованный в настоящей заявке термин «культивирование» означает выращивание штамма Corynebacterium glutamicum по настоящей заявке в условиях среды, корректируемых соответствующим образом. Способ культивирования по настоящей заявке может быть осуществлен в соответствующей среде и в соответствующих условиях культивирования, известных в данной области техники. Специалисты в данной области техники могут использовать такой способ культивирования, легко корректируя его в зависимости от выбранного штамма. В частности, культивирование может быть периодического типа, непрерывного типа и/или периодического типа с подпиткой, но этим не ограничивается.
Использованный в настоящей заявке термин «среда» означает смесь веществ, содержащую питательные вещества, необходимые для культивирования штамма Corynebacterium glutamicum по настоящей заявке в качестве основного компонента, и данная среда обеспечивает наличие питательных веществ, факторов роста и тому подобного, включая воду, которые являются незаменимыми для выживания и роста. В частности, данную среду и другие условия культивирования, применяемые для культивирования штамма Corynebacterium glutamicum по настоящей заявке, можно использовать без конкретного ограничения, при условии, что она представляет собой среду, применяемую для культивирования обычного микроорганизма, однако штамм Corynebacterium glutamicum по настоящей заявке можно культивировать, корректируя температуру, рН и тому подобное, в аэробных условиях в обычной среде, содержащей подходящие источники углерода, источники азота, источники фосфора, неорганические соединения, аминокислоты и/или витамины и тому подобное.
В частности, состав среды для культивирования штамма Corynebacterium sp.можно найти в литературном источнике ("Manual of Methods for General Bacteriology" Американского общества бактериологов (Washington, D.C., USA, 1981)).
В настоящей заявке источник углерода может включать углевод, такой как глюкоза, сахароза, лактоза, фруктоза, сахароза, мальтоза и тому подобное; сахарный спирт, такой как маннит, сорбит и тому подобное; органическую кислоту, такую как пировиноградная кислота, молочная кислота, лимонная кислота и тому подобное; и аминокислоту, такую как глутаминовая кислота, метионин, лизин и тому подобное. Помимо этого можно использовать природные органические питательные вещества, такие как гидролизат крахмала, меласса, сырая меласса, рисовые отруби, маниок, стебли сахарного тростника и жидкий кукурузный экстракт и, в частности, можно использовать такой углевод, как глюкоза, и стерилизованную предварительно обработанную мелассу (а именно, мелассу, превращенную в редуцирующий сахар) и тому подобное, а также по-разному и без ограничения можно использовать другие источники углерода в соответствующих количествах. Эти источники углерода можно использовать по отдельности или в комбинации из двух или более видов, но этим не ограничиваясь.
В качестве источника азота можно использовать источники неорганического азота, такие как аммиак, сульфат аммония, хлорид аммония, ацетат аммония, фосфат аммония, карбонат аммония, нитрат аммония и тому подобное; и источники органического азота, такие как аминокислота, в том числе глутаминовая кислота, метионин, глутамин и тому подобное, пептон, NZ-амин, мясной экстракт, дрожжевой экстракт, солодовый экстракт, жидкий кукурузный экстракт, гидролизат казеина, рыба или продукты ее разложения, обезжиренный соевый жмых или продукты его разложения и тому подобное. Эти источники азота можно использовать по отдельности или в комбинации из двух или более видов, но этим не ограничиваясь.
Источники фосфора могут включать моногидрофосфат калия, дигидрофосфат калия или соответствующую им натрийсодержащую соль или тому подобное. В качестве неорганических соединений можно использовать хлорид натрия, хлорид кальция, хлорид железа, сульфат магния, сульфат железа, сульфат марганца, карбонат кальция и тому подобное, а также могут быть включены аминокислоты, витамины и/или соответствующие предшественники и тому подобное. Эти компоненты или предшественники можно добавлять в среду порционно или непрерывным образом. Однако этим не ограничиваясь.
Помимо этого, в процессе культивирования штамма Corynebacterium sp.по настоящей заявке рН среды можно корректировать соответствующим добавлением в данную среду таких соединений, как гидроксид аммония, гидроксид калия, аммиак, фосфорная кислота, серная кислота и тому подобное. Кроме того, в процессе культивирования для подавления пенообразования можно применять противовспенивающий агент, такой как сложный полигликоле вый эфир жирной кислоты. Помимо этого, для поддержания в среде аэробных условий в нее можно подавать кислород или кислородсодержащий газ, либо для поддержания анаэробных и неаэробных условий газ можно не подавать или можно подавать азот, водород или углекислый газ, но этим не ограничиваясь.
При культивировании по настоящей заявке температуру культивирования можно поддерживать при 20-45°С, в частности, 25-40°С, и культивирование можно проводить в течение примерно 10-160 часов, но этим не ограничиваясь.
L-аминокислота (например, L-глутаминовая кислота), получаемая посредством культивирования по настоящей заявке, может секретироваться в среду или оставаться в клетке.
Способ получения L-аминокислоты по настоящей заявке может дополнительно включать стадию получения микроорганизма (штамма) по настоящей заявке, стадию приготовления среды для культивирования данного микроорганизма или их комбинацию (в любом порядке), например, перед проведением культивирования.
Способ получения L-аминокислоты по настоящей заявке может дополнительно включать стадию извлечения L-аминокислоты из среды (среды, в которой проводится культивирование) или из микроорганизма (штамма Corynebacterium sp.) в соответствии со способом культивирования. После стадии культивирования может быть дополнительно включена стадия извлечения.
Стадия извлечения может служить для сбора целевой L-аминокислоты с использованием способа, известного в данной области техники, соответствующего способу культивирования микроорганизма по настоящей заявке, например, способу периодического, непрерывного культивирования или периодического культивирования с подпиткой и тому подобным. Например, можно использовать центрифугирование, фильтрование, обработку агентом, вызывающим осаждение кристаллов белка (метод высаливания), экстракцию, разрушение ультразвуком, ультрафильтрацию, диализ, различные виды хроматографии, такие как хроматография на молекулярный ситах (гель-фильтрация), адсорбционная хроматография, ионообменная хроматография, аффинная хроматография и тому подобное, высокоэффективную жидкостную хроматографию (HPLC) или использовать комбинацию этих способов, и целевую L-аминокислоту можно извлекать из среды или микроорганизма с применением соответствующего способа, известного в данной области техники.
Помимо этого, способ получения L-аминокислоты по настоящей заявке может дополнительно включать стадию очистки. Очистку можно осуществлять с использованием подходящего способа, известного в данной области техники. В одном из воплощений, когда способ получения L-аминокислоты по настоящей заявке включает как извлечение, так и очистку, данные стадии извлечения и очистки можно осуществлять в непрерывном режиме или с перерывами в любом порядке либо можно осуществлять одновременно или посредством объединения в одну стадию, но этим не ограничиваясь.
Согласно другому аспекту настоящей заявки предложена композиция для получения L-аминокислоты (например, L-глутаминовой кислоты), содержащая микроорганизм по настоящей заявке; среду для его культивирования; или комбинацию из 2-х или более чем двух из них.
Композиция по настоящей заявке может дополнительно содержать любой соответствующий эксципиент, обычно используемый в композиции для получения аминокислоты, и этот эксципиент может представлять собой, например, консервант, увлажняющий агент, диспергирующий агент, суспендирующий агент, буфер, стабилизатор или придающий изотоничность агент либо тому подобное, но этим не ограничивается.
В композиции по настоящей заявке микроорганизм (штамм), среда и L-аминокислота и тому подобное являются такими, как они описаны выше для других аспектов.
Согласно другому аспекту настоящей заявки предложено применение микроорганизма по настоящей заявке; среды для его культивирования; или комбинации из 2-х или более чем двух из них для получения L-аминокислоты (например, L-глутаминовой кислоты).
Согласно другому аспекту настоящей заявки предложено применение микроорганизма по настоящей заявке; среды для его культивирования; или комбинации из 2-х или более чем двух из них для приготовления композиции для получения L-аминокислоты (например, L-глутаминовой кислоты).
ПОЛЕЗНЫЕ ЭФФЕКТЫ
Микроорганизм, имеющий ДНК-связывающий регулятор транскрипции семейства LacI с ослабленной активностью, по настоящей заявке характеризуется значительно более высокой способностью продуцировать L-глутаминовую кислоту, и поэтому с его использованием можно получать L-глутаминовую кислоту с более высоким выходом по сравнению с традиционными микроорганизмами.
СПОСОБ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Далее настоящая заявка будет описана более подробно посредством примеров. Однако приведенные ниже примеры являются лишь предпочтительными воплощениями для иллюстрации настоящей заявки и поэтому не предназначены для ограничения объема настоящей заявки. С другой стороны, технические вопросы, не описанные в настоящем изобретении, могут быть в достаточной степени поняты и легко воплощены специалистами в данной области техники или в областях техники, аналогичных описанным в настоящей заявке.
Пример 1. Получение вектора для экспрессии варианта ДНК-связывающего белка-регулятора транскрипции семейства LacI в микроорганизме
В настоящем примере для подтверждения влияния на продуцирование L-глутаминовой кислоты, оказываемого вариантом (Q310*; SEQ ID NO: 1, 309 последовательностей), в котором кодон, соответствующий глутамину (Gin, Q) в положении 310 аминокислотной последовательности ДНК-связывающего белка-регулятора транскрипции семейства LacI (аминокислотной последовательности SEQ ID NO: 3), заменен на стоп-кодон (*), конструировали вектор для экспрессии его в штамме так, как приведено ниже.
С использованием гДНК (геномной ДНК) Corynebacterium glutamicum дикого типа (АТСС 13869) в качестве матрицы проводили полимеразную цепную реакцию (ПЦР), применяя по отдельности пару праймеров с последовательностями, описанными как SEQ ID NO: 5 и 6, и пару праймеров с последовательностями, описанными как SEQ ID NO: 7 и 8, соответственно. С использованием смеси этих двух полученных выше фрагментов в качестве матрицы проводили ПЦР с перекрывающимися праймерами, вновь применяя пару праймеров с последовательностями SEQ ID NO: 5 и SEQ ID NO: 8, для получения фрагмента. В качестве полимеразы использовали ДНК-полимеразу Solg™ Pfu-X, и ПЦР проводили, применяя денатурацию при 95°С в течение 5 минут, затем повторяли 30 раз денатурацию при 95°С в течение 30 секунд, отжиг при 55°С в течение 30 секунд и полимеризацию при 72°С в течение 1 минуты и 30 секунд, а затем полимеризацию при 72°С в течение 5 минут.
Амплифицированный фрагмент гена и вектор pDCM2 для трансформации в хромосому, разрезанный ферментом рестрикции SmaI (публикация на патент Кореи №10-2020-0136813), клонировали, используя метод сборки Гибсона (DG Gibson et al., NATURE METHODS, VOL. 6, №5, MAY 2009; мастер-микс для сборки ДНК-конструкций NEBuilder HiFi), и затем осуществляли клонирование путем смешивания реагента Гибсона для сборки и каждого фрагмента гена с расчетным количеством молей и далее выдерживания их при 50°С в течение 1 часа. Штамм наносили мазками на твердую среду Луриа-Бертани (LB), содержащую канамицин (25 мг/л). После отбора колонии, в которую встраивался целевой ген, получали вектор путем использования общеизвестного метода экстракции плазмид (векторов). Вектору присваивали название pDCM2-BBD29_06680 (Q310*). Последовательности праймеров, использованных в настоящем примере, описаны в приведенной ниже Таблице 1.
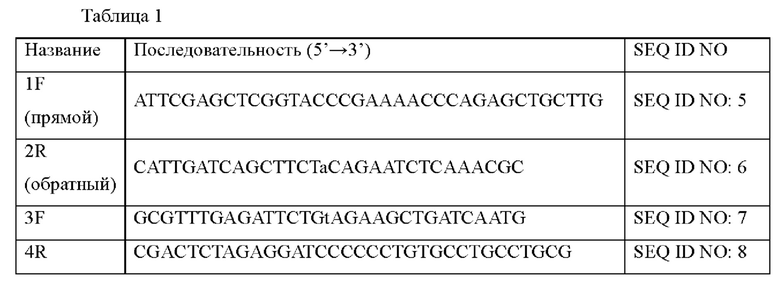
Пример 2. Получение штамма, продуцирующего L-глутаминовую кислоту, происходящего из Corynebacterium glutamicum дикого типа, и введение варианта ДНК-связывающего белка-регулятора транскрипции семейства LacI в микроорганизм
Пример 2-1. Получение штамма Corynebacterium glutamicum, обладающего способностью продуцировать L-глутаминовую кислоту, происходящего из Corynebacterium glutamicum дикого типа
Для получения штамма, обладающего способностью продуцировать L-глутаминовую кислоту, происходящего из Corynebacterium glutamicum АТСС 13869, на основании предшествующего уровня техники (Appl. Environ. Microbiol., 2007, Feb, 73(4): 1308-19. Epub, 2006, Dec 8), получали штамм Corynebacterium glutamicum ATCC13869 ΔodhA, в котором был делетирован ген odhA (№доступа в GenBank: WP 060564343.1, SEQ ID NO: 24).
Конкретно, чтобы делетировать ген odhA с использованием хромосомной ДНК Corynebacterium glutamicum АТСС 13869 в качестве матрицы, получали участок «вверх по течению» и «вниз по течению» относительно гена odhA, проводя ПЦР с применением пары праймеров с SEQ ID NO: 17 и 18 и пары праймеров с SEQ ID NO: 19 и 20, соответственно. В качестве полимеразы использовали ДНК-полимеразу Solg™ Pfu-X, а ПЦР-амплификацию проводили в следующих условиях: денатурация при 95°С в течение 5 минут, затем повторяли 30 раз денатурацию при 95°С в течение 30 секунд, отжиг при 58°С в течение 30 секунд и полимеризацию при 72°С в течение 60 секунд, и затем полимеризацию при 72°С в течение 5 минут.
Рекомбинантный вектор получали путем клонирования амплифицированных участков «вверх по течению» и «вниз по течению» относительно гена odhA и вектора pDCM2 для трансформации в хромосому, разрезанного ферментом рестрикции SmaI, с использованием метода сборки Гибсона, и называли pDCM2-ΔodhA. Клонирование осуществляли путем смешивания реагента Гибсона для сборки и каждого фрагмента гена с расчетным количеством молей и далее выдерживания их при 50°С в течение 1 часа.
Полученным вектором pDCM2-ΔodhA трансформировали штамм Corynebacterium glutamicum АТСС13869 посредством эле ктр опор ации, с последующим вторичным кроссинговером для получения таким образом штамма с делецией гена odhA в хромосоме. Наличие делеции гена odhA подтверждали результатами ПЦР с использованием пары праймеров с SEQ ID NO: 21 и 22 и геномного секвенирования, и полученному штамму присваивали название АТСС13869 ΔodhA. Последовательности праймеров, использованных в настоящем примере, описаны в приведенной ниже Таблице 2.

Пример 2-2. Получение штамма, в который введен вариант ДНК-связывающего белка-регулятора транскрипции семейства LacI
Вектором pDCM2-BBD29_06680 (Q310*), полученным так, как указано выше в примере 1, трансформировали штамм АТСС13869 ΔodhA, полученного так, как указано выше в примере 2-1, посредством электропорации, с последующим вторичным кроссинговером для получения таким образом штамма, в хромосому которого была введена мутация BBD29_06680 (Q310*). Наличие в штамме введенной мутации BBD29_06680 (Q310*) подтверждали результатами ПЦР с использованием пары праймеров SEQ ID NO: 9 и 10 и геномного секвенирования, и полученному штамму присваивали название СА02-1626. Штамму СА02-1626 присваивали название Corynebacterium glutamicum СА02-1626 и депонировали в Корейский центр культур микроорганизмов (KCCM) 18 января 2021 г. с номером доступа KCCM12930P.
Последовательности праймеров, использованных в настоящем примере, описаны в приведенной ниже Таблице 3.
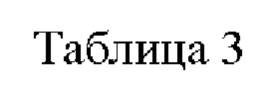
Пример 2-3. Сравнение способности штамма, экспрессирующего вариант ДНК-связывающего белка-регулятора транскрипции семейства LacI, продуцировать L-глутаминовую кислоту
Используя полученный в примере 2-2 штамм АТСС13869ΔodhA в качестве контрольного штамма, необходимо было подтвердить способность продуцировать L-глутаминовую кислоту. Контрольный штамм и штамм СА02-1626 культивировали описанным ниже способом.
Каждый штамм высевали в колбу с угловыми перегородками емкостью 250 мл, содержащую 25 мл среды для посева, и культивировали со встряхиванием при 200 об./мин и 30°С в течение 20 часов. После этого 1 мл раствора затравочной культуры высевали в колбу с угловыми перегородками емкостью 250 мл, содержащую 25 мл среды для продуцирования, и культивировали со встряхиванием при 200 об./мин и 30°С в течение 40 часов. По завершении культивирования измеряли выход L-глутаминовой кислоты, используя высокоэффективную жидкостную хроматографию (HPLC), и результат измерения показан в приведенной ниже Таблице 4.
Среда для посева
Глюкоза 1%; мясной экстракт 0,5%; полипептон 1%; хлорид натрия 0,25%; дрожжевой экстракт 0,5%; мочевина 0,2%; рН 7,2.
Среда для продуцирования
Нерафинированный сахар 6%; карбонат кальция 5%; сульфат аммония 2,25%; монофосфат калия 0,1%; сульфат магния 0,04%; сульфат железа 10 мг/л; тиамин гидрохлорид 0,2 мг/л; биотин 50 мкг/л.
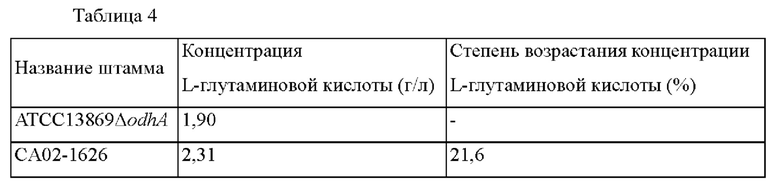
Как показано выше в Таблице 4, было подтверждено, что концентрация L-глутаминовой кислоты возрастала примерно на 21,6% в случае штамма СА02-1626, в котором была введена мутация BBD29_06680 (Q310*), по сравнению с контрольным штаммом АТСС13869 ΔodhA.
Пример 3. Получение штамма с делетированным геном ДНК-связывающего белка-регулятора транскрипции семейства LacI и измерение способности продуцировать L-глутаминовую кислоту
Пример 3-1. Получение вектора с делетированным геном ДНК-связывающего белка-регулятора транскрипции семейства LacI
В этом примере было подтверждено, что способность продуцировать L-глутаминовую кислоту улучшалась, когда кодон, соответствующий глутамину (Gin, Q), который является аминокислотным остатком в положении 310 ДНК-связывающего белка-регулятора транскрипции семейства LacI, был заменен на стоп-кодон. Соответственно, в настоящем примере необходимо было подтвердить влияние делетирования гена ДНК-связывающего белка-регулятора транскрипции семейства LacI (BBD29_06680) на продуцирование L-глутаминовой кислоты.
Конкретно, чтобы делетировать ген BBD29_06680 с использованием гДНК (геномной ДНК) Corynebacterium glutamicum АТСС13869 в качестве матрицы, получали участок «вверх по течению» и участок «вниз по течению» относительно гена BBD29_06680, проводя ПЦР с применением пары праймеров с SEQ ID NO: 11 и 12 и пары праймеров с SEQ ID NO: 13 и 14. В качестве полимеразы использовали ДНК-полимеразу Solg™ Pfu-X, а ПЦР-амплификацию проводили в следующих условиях: денатурация при 95°С в течение 5 минут, затем повторяли 30 раз денатурацию при 95°С в течение 30 секунд, отжиг при 58°С в течение 30 секунд и полимеризацию при 72°С в течение 60 секунд, и затем полимеризация при 72°С в течение 5 минут. Рекомбинантный вектор получали путем клонирования амплифицированного фрагмента ДНК с использованием вектора pDCM2 для трансформации в хромосому, разрезанного ферментом рестрикции SmaI, и с использованием метода сборки Гибсона, и этому вектору присваивали название pDCM2-ABBD29_06680. Клонирование осуществляли путем смешивания реагента Гибсона для сборки и каждого фрагмента гена с расчетным количеством молей и далее выдерживания их при 50°С в течение 1 часа.
Последовательности праймеров, использованных в настоящем примере, описаны в приведенной ниже Таблице 5.
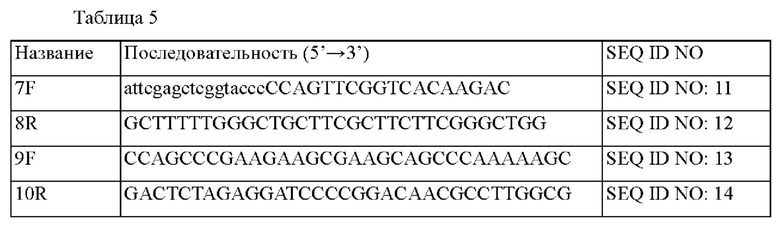
Пример 3-2. Получение штамма с делетированным геном ДНК-связывающего белка-регулятора транскрипции семейства LacI
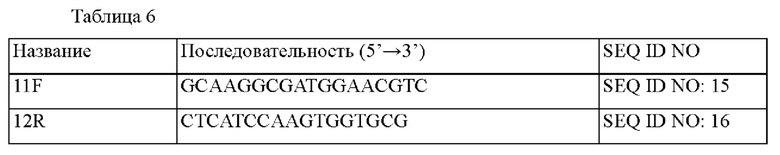
Вектором pDCM2-ABBD29_06680, полученным так, как указано выше в примере 3-1, трансформировали штамм АТСС13869 ΔodhA, полученного так, как указано выше в примере 2-1, посредством электропорации, с последующим вторичным кроссинговером для получения таким образом штамма, в котором ген BBD29_06680 был делетирован из хромосомы, и это подтверждали результатами ПЦР с использованием пары праймеров с SEQ ID NO: 15 и 16 и геномного секвенирования. Отобранному штамму присваивали название СА02-1627. Последовательности праймеров, использованных в настоящих примерах, описаны в приведенной ниже Таблице 6.
Пример 3-3. Измерение способности штамма с делетированным геном ДНК-связывающего белка-регулятора транскрипции семейства LacI продуцировать L-глутаминовую кислоту
Чтобы подтвердить способность продуцировать L-глутаминовую кислоту с использованием штамма АТСС13869ΔodhA, полученного так, как указано выше в примере 2-1 в качестве контрольного штамма, проводили оценку согласно методу оценки титра после ферментации из примера 2-3 и по завершении культивирования выход L-глутаминовой кислоты измеряли с использованием высокоэффективной жидкостной хроматографии (HPLC), и результат измерения показан в приведенной ниже Таблице 7.

Как показано выше в Таблице 7, было подтверждено, что концентрация L-глутаминовой кислоты возрастала примерно на 21,4% в случае штамма СА02-1627, в котором был делетирован ген BBD29_06680, по сравнению с контрольным штаммом АТСС13869 ΔodhA.
Пример 4. Получение штамма с введенным геном ДНК-связывающего белка-регулятора транскрипции семейства LacI с NTG-индуцированной мутацией и измерение способности продуцировать L-глутаминовую кислоту
Чтобы подтвердить, будет ли при внесении варианта гена BBD29_06680 (Q310*) проявляться тот же самый эффект даже в штамме, полученном из Corynebacterium sp.с индуцированной NTG (N-метил-N'-нитро-N-нитрозогуанидином) мутацией, обладающем повышенной способностью продуцировать L-глутаминовую кислоту, этот вариант гена вводили в штамм Corynebacterium glutamicum BL2, который, как известно, является продуцирующим L-глутаминовую кислоту штаммом с NTG-индуцированной мутацией (KFCC11074, патент Кореи №10-0292299).
Вектором pDCM2-BBD29_06680 (Q310*), полученным так, как в примере 1, трансформировали штамма KFCC11074 посредством электропорации с последующим вторичным кроссинговером с целью отбора таким образом штамма, в хромосому которого был встроен вариант гена BBD29_06680 (Q310*). Это подтверждали результатами ПЦР с использованием пары праймеров с SEQ ID NO: 9 и 10 и геномного секвенирования, и полученному штамму присваивали название СА02-1630.
Для полученного штамма СА02-1630 и штамма Corynebacterium glutamicum KFCC11074 проводили эксперимент по оценке титра после ферментации методом, приведенным ниже.
Каждый штамм высевали в колбу с угловыми перегородками емкостью 250 мл, содержащую 25 мл среды для посева, и культивировали со встряхиванием при 200 об./мин и 30°С в течение 20 часов. После этого 1 мл раствора затравочной культуры высевали в колбу с угловыми перегородками емкостью 250 мл, содержащую 25 мл среды для продуцирования, и культивировали со встряхиванием при 200 об./мин и 30°С в течение 40 часов. По завершении культивирования выход L-глутаминовой кислоты измеряли методом с использованием высокоэффективной жидкостной хроматографии (HPLC), и результат измерения показан в приведенной ниже Таблице 8.
Среда для посева
Глюкоза 1%; мясной экстракт 0,5%; полипептон 1%; хлорид натрия 0,25%; дрожжевой экстракт 0,5%; мочевина 0,2%; рН 7,2.
Среда для продуцирования
Нерафинированный сахар 6%; карбонат кальция 5%; сульфат аммония 2,25%; монофосфат калия 0,1%; сульфат магния 0,04%; сульфат железа 10 мг/л; тиамин гидрохлорид 0,2 мг/л; биотин 500 мкг/л.
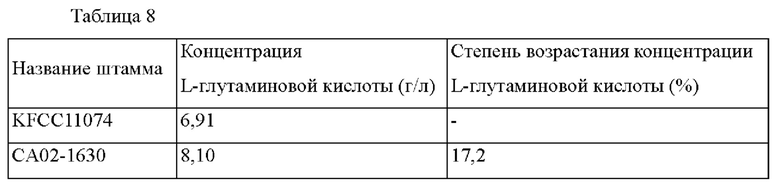
Как показано выше в Таблице 8, было подтверждено, что концентрация L-глутаминовой кислоты возрастала примерно на 17,2% в случае штамма СА02-1630 по сравнению с контрольным штаммом KFCC11074.
Из приведенного выше описания специалисты в области техники, к которой относится настоящая заявка, смогут понять, что настоящая заявка может быть реализована в других конкретных формах без изменения ее технической сущности или существенных признаков. В связи с этим следует понимать, что описанные выше примеры являются иллюстративными, а не ограничительными во всех отношениях. Объем настоящей заявки следует рассматривать как включающий все измененные или модифицированные формы, вытекающие из значения и объема формулы изобретения, которая будет приведена ниже, и эквивалентных понятий, а не из приведенного выше подробного описания.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing originalFreeTextLanguageCode="en"
nonEnglishFreeTextLanguageCode="ru" dtdVersion="V1_3"
fileName="P23966RU-st26-RU.xml" softwareName="WIPO Sequence"
softwareVersion="2.2.0" productionDate="2024-02-21">
<ApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>PCT/KR2022/010905</ApplicationNumberText>
<FilingDate>2022-07-25</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>P23966RU</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>KR</IPOfficeCode>
<ApplicationNumberText>10-2021-0098072</ApplicationNumberText>
<FilingDate>2021-07-26</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">СИДЖЕЙ ЧЕИЛДЖЕДАНГ КОРПОРЕЙШН
</ApplicantName>
<ApplicantNameLatin>CJ CHEILJEDANG CORPORATION</ApplicantNameLatin>
<InventionTitle languageCode="ru">Микроорганизм, обладающий
ослабленной активностью ДНК-связывающего транскрипционного регулятора
семейства LACI и способ получения l-глутаминовой кислоты с его
использованием</InventionTitle>
<SequenceTotalQuantity>24</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>309</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..309</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_LacI family DNA-binding
transcriptional regulator (Q310var.)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..309</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MASETSSPKKRATTLKDIAQATQLSVSTVSRALANNASIPESTRIRVVE
AAQKLNYRPNAQARALRKSRTDTIGVIIPNIENPYFSSLAASIQKAAREAGVSTILSNSEENPELLGQTL
AIMDDQRLDGIIVVPHIQSEEQVTDLVDRGVPVVLADRSFVNSSIPSVTSDPVPGMTEAVDLLLAADVQL
GYLAGPQDTSTGQLRLNTFEKLCVDRGIVGASVYYGGYRQESGYDGIKVLIKQGANAIIAGDSMMTIGAL
LALHEMNLKIGEDVQLIGFDNNPIFRLQNPPLSIIDQHVQEIGKRAFEIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1110</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..1110</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_coding gene LacI family
DNA-binding transcriptionalregulator (Q310
var.)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1110</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggcttccgaaacctccagcccgaagaagcgggccaccacgctcaaag
acatcgcgcaagcaacacagctttcagtcagcacggtgtcccgggcattggccaacaacgcgagcattcc
ggaatccacacgcatccgagtggttgaagccgctcaaaagctgaactaccgtcccaatgcccaagctcgt
gcattgcggaagtcgaggacagacaccatcggtgtcatcattccaaacattgagaacccatatttctcct
cactagcagcatcgattcaaaaagctgctcgtgaagctggggtgtccaccattttgtccaactctgaaga
aaacccagagctgcttggtcagactttggcgatcatggatgaccaacgcctcgatggaatcatcgtggtg
ccacacattcagtcagaggaacaagtcactgacttggttgacaggggagtgccagtagtgctggcagacc
gtagttttgttaactcgtctattccttcggttacctcagatccagttccgggcatgactgaagctgtgga
cttactcctggcagctgacgtgcaattgggctaccttgccggcccgcaggatacttccactggtcagctg
cgtcttaacacttttgaaaaactatgcgtggaccgcggcatcgtcggagcatctgtctattacggtggct
accgccaagaatctggatatgacggcatcaaggtgctgatcaagcagggagccaatgcgattatcgctgg
tgactccatgatgaccatcggtgcgttgttggctcttcatgagatgaatttgaagatcggtgaggatgtg
cagctcattgggtttgataacaacccaattttccggctgcagaatccaccgctgagcatcattgaccagc
acgtacaagagatcggtaagcgtgcgtttgagattctgtagaagctgatcaatggggacaccgcgcaaaa
atctgtggtgattccaacgcagctcagcatcaatggatcaacggcggtttcccaaaaggccgccgcaaaa
gcagcaaaagcagcccaaaaagcagccgcgaaagccgcacagaacacgcaacacgaggtgagcctagatg
gtgaactctga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>369</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..369</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q5">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_LacI family DNA-binding
transcriptional regulator(wild)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..369</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MASETSSPKKRATTLKDIAQATQLSVSTVSRALANNASIPESTRIRVVE
AAQKLNYRPNAQARALRKSRTDTIGVIIPNIENPYFSSLAASIQKAAREAGVSTILSNSEENPELLGQTL
AIMDDQRLDGIIVVPHIQSEEQVTDLVDRGVPVVLADRSFVNSSIPSVTSDPVPGMTEAVDLLLAADVQL
GYLAGPQDTSTGQLRLNTFEKLCVDRGIVGASVYYGGYRQESGYDGIKVLIKQGANAIIAGDSMMTIGAL
LALHEMNLKIGEDVQLIGFDNNPIFRLQNPPLSIIDQHVQEIGKRAFEILQKLINGDTAQKSVVIPTQLS
INGSTAVSQKAAAKAAKAAQKAAAKAAQNTQHEVSLDGEL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>1110</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..1110</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_gene coding LacI family
DNA-binding transcriptionalregulator (wild)</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1110</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggcttccgaaacctccagcccgaagaagcgggccaccacgctcaaag
acatcgcgcaagcaacacagctttcagtcagcacggtgtcccgggcattggccaacaacgcgagcattcc
ggaatccacacgcatccgagtggttgaagccgctcaaaagctgaactaccgtcccaatgcccaagctcgt
gcattgcggaagtcgaggacagacaccatcggtgtcatcattccaaacattgagaacccatatttctcct
cactagcagcatcgattcaaaaagctgctcgtgaagctggggtgtccaccattttgtccaactctgaaga
aaacccagagctgcttggtcagactttggcgatcatggatgaccaacgcctcgatggaatcatcgtggtg
ccacacattcagtcagaggaacaagtcactgacttggttgacaggggagtgccagtagtgctggcagacc
gtagttttgttaactcgtctattccttcggttacctcagatccagttccgggcatgactgaagctgtgga
cttactcctggcagctgacgtgcaattgggctaccttgccggcccgcaggatacttccactggtcagctg
cgtcttaacacttttgaaaaactatgcgtggaccgcggcatcgtcggagcatctgtctattacggtggct
accgccaagaatctggatatgacggcatcaaggtgctgatcaagcagggagccaatgcgattatcgctgg
tgactccatgatgaccatcggtgcgttgttggctcttcatgagatgaatttgaagatcggtgaggatgtg
cagctcattgggtttgataacaacccaattttccggctgcagaatccaccgctgagcatcattgaccagc
acgtacaagagatcggtaagcgtgcgtttgagattctgcagaagctgatcaatggggacaccgcgcaaaa
atctgtggtgattccaacgcagctcagcatcaatggatcaacggcggtttcccaaaaggccgccgcaaaa
gcagcaaaagcagcccaaaaagcagccgcgaaagccgcacagaacacgcaacacgaggtgagcctagatg
gtgaactctga</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>36</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_1F primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_1F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..36</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attcgagctcggtacccgaaaacccagagctgcttg</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_2R primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_2R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cattgatcagcttctacagaatctcaaacgc</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>31</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_3F primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_3F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..31</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgtttgagattctgtagaagctgatcaatg</INSDSeq_sequence
>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>34</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_4R primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_4R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..34</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgactctagaggatcccccctgtgcctgcctgcg</INSDSeq_seque
nce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_5F primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_5F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgctcgtgaagctgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_6R primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_6R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaacccacctgagcattc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>35</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_7F primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_7F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..35</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>attcgagctcggtacccccagttcggtcacaagac</INSDSeq_sequ
ence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q23">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_8R primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_8R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctttttgggctgcttcgcttcttcgggctgg</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>32</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_9F primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_9F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..32</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccagcccgaagaagcgaagcagcccaaaaagc</INSDSeq_sequenc
e>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>33</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_10R
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_10R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..33</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gactctagaggatccccggacaacgccttggcg</INSDSeq_sequen
ce>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_11F
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_11F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcaaggcgatggaacgtc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>17</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_12R
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_12R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..17</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcatccaagtggtgcg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>38</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_13F
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_13F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgaattcgagctcggtacccttgaacggaattgggtgg</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>38</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_14R
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_14R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccaggtggcatcggtaccttcacccagcgccacgcag</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>39</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_15F
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_15F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..39</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cgctgggtgaaggtaccgatgccacctgggttggtcaag</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>38</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_16R
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_16R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..38</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtcgactctagaggatccccggacaaggaatggagaga</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_17F
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_17F
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttaccgttgttgccctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_18R
primer</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_18R
праймер</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctccttcacccacatcatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>1205</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..1205</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q45">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_odhA protein
(wild)</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_белок odhA (дикого
типа)</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1205</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MFQQFQKDPKSVDKEWRELFEAQGGPNATPATTEAQPSAPKESAKPAPK
AAPAAKAAPRVETKPAAKTAPKAKESSVPQQPKLPEPGQTPIRGIFKSIAKNMDISLEIPTATSVRDMPA
RLMFENRAMVNDQLKRTRGGKISFTHIIGYAMVKAVMAHPDMNNSYDVIDGKPTLIVPEHINLGLAIDLP
QKDGSRALVVAAIKETEKMNFSEFLAAYEDIVTRSRKGKLTMDDYQGVTVSLTNPGGIGTRHSVPRLTKG
QGTIIGVGSMDYPAEFQGASEDRLAELGVGKLVTITSTYDHRVIQGAVSGEFLRTMSRLLTDDSFWDEIF
DAMNVPYTPMRWAQDVPNTGVDKNTRVMQLIEAYRSRGHLIADTNPLSWVQPGMPVPDHRDLDIETHSLT
IWDLDRTFSVGGFGGKETMTLREVLSRLRAAYTLKVGSEYTHILDRDERTWLQDRLEAGMPKPTQAEQKY
ILQKLNAAEAFENFLQTKYVGQKRFSLEGAEALIPLMDSAIDTAAGQGLDEVVIGMPHRGRLNVLFNIVG
KPLASIFNEFEGQMEQGQIGGSGDVKYHLGSEGQHLQMFGDGEIKVSLTANPSHLEAVNPVMEGIVRAKQ
DYLDKGVDGKTVVPLLLHGDAAFAGLGIVPETINLAKLRGYDVGGTIHIVVNNQIGFTTTPDSSRSMHYA
TDYAKAFGCPVFHVNGDDPEAVVWVGQLATEYRRRFGKDVFIDLVCYRLRGHNEADDPSMTQPKMYELIT
GRETVRAQYTEDLLGRGDLSNEDAEAVVRDFHDQMESVFNEVKEGGKKQAEAQTGITGSQKLPHGLETNI
SREELLELGQAFANTPEGFNYHPRVAPVAKKRVSSVTEGGIDWAWGELLAFGSLANSGRLVRLAGEDSRR
GTFTQRHAVAIDPATAEEFNPLHELAQSKGNNGKFLVYNSALTEYAGMGFEYGYSVGNEDSVVAWEAQFG
DFANGAQTIIDEYVSSGEAKWGQTSKLILLLPHGYEGQGPDHSSARIERFLQLCAEGSMTVAQPSTPANH
FHLLRRHALSDLKRPLVIFTPKSMLRNKAAASAPEDFTEVTKFQSVIDDPNVADAAKVKKVMLVSGKLYY
ELAKRKEKDGRDDIAIVRIEMLHPIPFNRISEALAGYPNAEEVLFVQDEPANQGPWPFYQEHLPELIPNM
PKMRRVSRRAQSSTATGVAKVHQLEEKQLIDEAFEA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>3618</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>misc_feature</INSDFeature_key>
<INSDFeature_location>1..3618</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q47">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>snythetic_gene coding odhA protein
(wild)</INSDQualifier_value>
<NonEnglishQualifier_value>синтетический_ген, кодирующий белок
odhA (дикого типа)</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..3618</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
<NonEnglishQualifier_value>синтетическая
конструкция</NonEnglishQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgttccagcagttccagaaggaccccaagtccgtggacaaggaatgga
gagaactctttgaggcgcaggggggaccaaatgctacccccgctacaacagaagcacagccttcagcgcc
caaggagtctgcgaaaccagcaccaaaggctgcccctgcagccaaggcagcaccgcgcgtagaaaccaag
ccggccgccaagaccgcccctaaggccaaggagtcctcagtgccacagcaacctaagcttccggagccag
gacaaaccccaatcaggggtattttcaagtccatcgcgaagaacatggatatctccctggaaatcccaac
cgcaacctcggttcgcgatatgccagctcgcctcatgttcgaaaaccgcgcgatggtcaacgatcagctc
aagcgcacccgcggtggcaagatctccttcacccacatcattggctacgccatggtgaaggcagtcatgg
ctcacccggacatgaacaactcctacgacgtcatcgacggcaagccaaccctgatcgtgcctgagcacat
caacctgggccttgccatcgaccttcctcagaaggacggctcccgcgcacttgtcgtagcagccatcaag
gaaaccgagaagatgaacttctccgagttcctcgcagcatacgaagacatcgtgacacgctcccgcaagg
gcaagctcaccatggatgactaccagggcgttaccgtttccttgaccaacccaggtggcatcggtacccg
ccactctgtcccacgtctgaccaagggccagggcaccatcatcggtgtcggttccatggattacccagca
gagttccagggcgcttccgaagaccgccttgcagagctcggcgttggcaagcttgtcaccatcacctcca
cctacgatcaccgcgtgatccagggtgctgtgtccggtgaattcctgcgtaccatgtctcgcctgctcac
cgatgattccttctgggatgagatcttcgacgcaatgaacgttccttacaccccaatgcgttgggcacag
gacgttccaaacaccggtgttgataagaacacccgcgtcatgcagctcattgaggcataccgctcccgtg
gacacctcatcgctgacaccaacccactttcatgggttcagcctggcatgccagttccagaccaccgcga
cctcgacatcgagacccacagcctgaccatctgggatctggaccgtaccttcagcgtcggtggcttcggc
ggcaaggagaccatgaccctgcgcgaggtactgtcccgcctgcgcgctgcctacaccttgaaggtcggct
ccgaatacacccacatcctggaccgcgacgagcgcacctggctgcaggaccgcctcgaagccggaatgcc
aaagccaacccaggcagagcagaagtacatcctgcagaagctgaacgccgcagaggctttcgagaacttc
ctgcagaccaagtacgtcggccagaagcgcttctccctcgaaggtgcagaagctctcatcccactgatgg
actccgccatcgacaccgccgcaggccagggcctcgacgaagttgtcatcggtatgccacaccgtggtcg
cctcaacgtgctgttcaacatcgtgggcaagccactggcatccatcttcaacgagtttgaaggccaaatg
gagcagggccagatcggtggctccggtgacgtgaagtaccacctcggttccgaaggccagcacctgcaga
tgttcggcgacggcgagatcaaggtctccctgactgctaacccgtcccacctggaagctgttaacccagt
gatggaaggtatcgtccgcgcaaagcaggactacctggacaagggcgtagacggcaagactgttgtgcca
ctgctgctccacggtgacgctgcattcgcaggcctgggcatcgtgccagaaaccatcaacctggctaagc
tgcgtggctacgacgtcggaggcaccatccacatcgtggtgaacaaccagatcggcttcaccaccacccc
agactccagccgctccatgcactacgcaaccgactacgccaaggcattcggctgcccagtcttccacgtc
aatggtgatgacccagaggcagttgtctgggttggccagctggcaaccgagtaccgtcgtcgcttcggca
aggacgtcttcatcgacctcgtttgctaccgcctccgcggccacaacgaagctgatgatccttccatgac
ccagccaaagatgtatgagctcatcaccggccgcgagaccgttcgtgctcagtacaccgaagacctgctc
ggacgtggagacctctccaacgaagatgcagaagcagtcgtccgcgacttccacgaccagatggaatctg
tgttcaacgaagtcaaggaaggcggcaagaagcaggctgaggcacagaccggcatcaccggctcccagaa
gcttccacacggccttgagaccaacatctcccgtgaagagctcctggaactgggacaggctttcgccaac
accccagaaggcttcaactaccacccacgtgtggctccagttgctaagaagcgcgtctcctctgtcaccg
aaggtggcatcgactgggcatggggcgagctcctcgccttcggttccctggctaactccggccgcttggt
tcgccttgcaggtgaagattcccgccgcggtaccttcacccagcgccacgcagttgccatcgacccagcg
accgctgaagagttcaacccactccacgagcttgcacagtccaagggcaacaacggtaagttcctggtct
acaactccgcactgaccgagtacgcaggcatgggcttcgagtacggctactccgtaggaaacgaagactc
cgtcgttgcatgggaagcacagttcggcgacttcgccaacggcgctcagaccatcatcgatgagtacgtc
tcctcaggcgaagctaagtggggccagacctccaagctgatccttctgctgcctcacggctacgaaggcc
agggcccagaccactcttccgcacgtatcgagcgcttcctgcagctgtgcgctgagggttccatgactgt
tgctcagccatccaccccagcaaaccacttccacctgctgcgtcgtcacgctctgtccgacctgaagcgt
ccactggttatcttcaccccgaagtccatgctgcgtaacaaggctgctgcctccgcaccagaagacttca
ctgaggtcaccaagttccaatccgtgatcgacgatccaaacgttgcagatgcagccaaggtgaagaaggt
catgctggtctccggcaagctgtactacgaattggcaaagcgcaaggagaaggacggacgcgacgacatc
gcgatcgttcgtatcgaaatgctccacccaattccgttcaaccgcatctccgaggctcttgccggctacc
ctaacgctgaggaagtcctcttcgttcaggatgagccagcaaaccagggcccatggccgttctaccagga
gcacctcccagagctgatcccgaacatgccaaagatgcgccgcgtttcccgccgcgctcagtcctccacc
gcaactggtgttgctaaggtgcaccagctggaggagaagcagcttatcgacgaggctttcgaggcttaa<
/INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к биотехнологии и представляет собой микроорганизм, имеющий ДНК-связывающий белок-регулятор транскрипции семейства LacI с ослабленной активностью. В микроорганизме Corynebacterium sp., имеющем ДНК-связывающий белок-регулятор транскрипции семейства LacI с ослабленной активностью, значительно повышена способность продуцировать L-глутаминовую кислоту, и в результате этого данный микроорганизм при своем использовании может продуцировать L-глутаминовую кислоту с более высоким выходом, чем традиционный микроорганизм. 2 н. и 11 з.п. ф-лы, 8 табл., 3 пр.
1. Микроорганизм Corynebacterium sp. для получения L-глутаминовой кислоты, имеющий ДНК-связывающий белок-регулятор транскрипции семейства LacI с ослабленной активностью, где ДНК-связывающий белок-регулятор транскрипции семейства LacI содержит аминокислотную последовательность с SEQ ID NO: 3.
2. Микроорганизм по п. 1, где ДНК-связывающий белок-регулятор транскрипции семейства LacI происходит из рода Corynebacterium.
3. Микроорганизм по п. 1, где ослабленная активность ДНК-связывающего белка-регулятора транскрипции семейства LacI достигается посредством: (1) делеции гена, кодирующего ДНК-связывающий белок-регулятор транскрипции семейства LacI, или (2) мутации кодона, соответствующего аминокислоте в положении 310 в аминокислотной последовательности SEQ ID NO: 3 с образованием стоп-кодона.
4. Микроорганизм по п. 1, где ДНК-связывающий белок-регулятор транскрипции семейства LacI кодируется полипептидом, описанным нуклеотидной последовательностью с SEQ ID NO: 4.
5. Микроорганизм по п. 1, представляющий собой Corynebacterium glutamicum.
6. Микроорганизм по п. 1, обладающий повышенной способностью продуцировать L-глутаминовую кислоту по сравнению с родительским штаммом или штаммом дикого типа, в котором активность ДНК-связывающего белка-регулятора транскрипции семейства LacI не ослаблена.
7. Способ получения L-глутаминовой кислоты, включающий культивирование микроорганизма Corynebacterium sp., имеющего ДНК-связывающий белок-регулятор транскрипции семейства LacI с ослабленной активностью, в среде, где ДНК-связывающий белок-регулятор транскрипции семейства LacI содержит аминокислотную последовательность с SEQ ID NO: 3.
8. Способ получения L-глутаминовой кислоты по п. 7, дополнительно включающий стадию извлечения L-глутаминовой кислоты из среды или из микроорганизма в соответствии со способом культивирования.
9. Способ получения L-глутаминовой кислоты по п. 7, где ДНК-связывающий белок-регулятор транскрипции семейства LacI происходит из рода Corynebacterium.
10. Способ получения L-глутаминовой кислоты по п. 7, где ослабленная активность ДНК-связывающего белка-регулятора транскрипции семейства LacI достигается посредством: (1) делеции гена, кодирующего ДНК-связывающий белок-регулятор транскрипции семейства LacI, или (2) мутации кодона, соответствующего аминокислоте в положении 310 в аминокислотной последовательности SEQ ID NO: 3 с образованием стоп-кодона.
11. Способ получения L-глутаминовой кислоты по п. 7, где ДНК-связывающий белок-регулятор транскрипции семейства LacI кодируется полипептидом, описанным нуклеотидной последовательностью с SEQ ID NO: 4.
12. Способ получения L-глутаминовой кислоты по п. 7, где микроорганизм Corynebacterium sp. представляет собой Corynebacterium glutamicum.
13. Способ получения L-глутаминовой кислоты по п. 7, где микроорганизм Corynebacterium sp. обладает повышенной способностью продуцировать L-глутаминовую кислоту по сравнению с родительским штаммом или штаммом дикого типа, в котором активность ДНК-связывающего белка-регулятора транскрипции семейства LacI не ослаблена.
| US 20020151001 A1, 17.10.2002 | |||
| СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТ С ИСПОЛЬЗОВАНИЕМ БАКТЕРИЙ С ОПТИМИЗИРОВАННЫМ УРОВНЕМ ГЕННОЙ ЭКСПРЕССИИ | 2003 |
|
RU2268305C2 |
| СПОСОБ ПОЛУЧЕНИЯ L-ГЛУТАМИНОВОЙ КИСЛОТЫ | 1999 |
|
RU2194076C2 |
| СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТЫ МЕТОДОМ ФЕРМЕНТАЦИИ, ШТАММ БАКТЕРИИ ESCHERICHIA COLI - ПРОДУЦЕНТ L-АМИНОКИСЛОТЫ (ВАРИАНТЫ) | 1999 |
|
RU2207376C2 |
Авторы
Даты
2025-01-17—Публикация
2022-07-25—Подача