Изобретение относится к биотехнологии, а именно к штамму Saccharomyces cerevisiae KEP1/pYES2-Amy, обеспечивающему продукцию рекомбинантного белка «альфа-амилаза» и обладающему регулируемой мутабельностью, а также к генетической конструкции (плазмиде), обеспечивающей синтез рекомбинантной альфа-амилазы из Bacillus sp.Н3.
Альфа-амилаза - это фермент, катализирующий гидролиз 1,4-α-D-глюкозидные связи в полисахаридной цепи крахмала и гликогена до олигосахаридов различной длины. Синонимы - 1,4-α-D-глюкан-глюканогидролаза, гликогеназа; шифр КФ: 3.2.1.1.
Альфа-амилазы имеют применение в пивоварении, хлебопечении, производстве сахара, спиртных напитков и моющих средств, а также практически полностью заменили собой химический гидролиз в крахмалоперерабатывающей промышленности. В хлебопечении и кондитерской промышленности амилазы широко используются для улучшения хлебопекарных (диастатических) свойств пшеничной и ржаной муки, а именно пористости и аромата изделий, изменения режима тестоведения за счет увеличения содержания сахара в них. В спиртовой промышленности применение альфа-амилаз позволяет повысить выход спирта. При производстве различных изделий из крупяных продуктов амилазы применяют для предварительной обработки (частичного расщепления крахмала) во всех тех случаях, когда из сырья вырабатывают пищевой продукт в виде хлопьев или зерен. Таким образом, очевидна необходимость в насыщении рынка ферментными препаратами альфа-амилаз, способных функционировать в различных условиях (широкий диапазон температур, кислотности и присутствие сильно окисляющихся соединений) для удовлетворения растущего спроса на них в различных областях производств. Для этого необходимы безопасные микробные штаммы, способные производить альфа-амилазу с нужными характеристиками и высоким выходом, что можно обеспечить путем создания эффективных штаммов-продуцентов генетическими методами.
Для удовлетворения растущего спроса на рекомбинантные белки, в широком смысле, необходимо использовать эффективные системы экспрессии, обеспечивающие высокую продуктивность фермента. Для этих целей дрожжи Saccharomyces cerevisiae активно применяются для производства рекомбинантных белков в промышленных масштабах: Methods Mol. Biol. 2012. Т. 824. С.329-358 [1]; Bioengineered. 2013. Т. 4. С.207-211 [2]; Braz. J. Microbiol. 2016. Т. 47. №1. С.51-63 [3], благодаря простоте генетических манипуляций, возможности посттрансляционных модификаций, хорошо изученному пути секреции белка и общепринятому статусу безопасности по системе GRAS: Appl. Microbiol. Biotechnol. 2013. Т. 97. С.8955-8962 [4]; Microorganisms. 2018. Т. 6. С.38 [5]. Так, S. cerevisiae обладают способностью выполнять посттрансляционные модификации и секрецию, что резко снижает затраты на постферментационную очистку и модификацию in vitro: Appl. Microbiol. Biotechnol. 2004. Т. 65. №4. С. 363-372 [6].
Известны штаммы дрожжей Saccharomyces cerevisiae, продуцирующие альфа-амилазы, сконструированные для конкретных производственных процессов, происходящих при заранее установленных условиях: патент ЕР 0252774А1 [7], патент US 20130244295 А1 [8]). Известно, что недостатком используемых в промышленности штаммов дрожжей S. cerevisiae в качестве продуцентов рекомбинантных белков является низкое содержание внеклеточных целевых ферментов: Biotechnol Bioeng. 2012. Т. 109. №5. С.1259-1268 [9].
Для различных областей промышленного применения альфа-амилаз (пивоварении, хлебопечении, производстве сахара, спиртных напитков и моющих средств) и, соответственно, при разных условиях (кислотность и температурный режим среды, наличие потенциально дезактивирующих веществ) наряду с неустойчивыми секретирующими способностями S. cerevisiae, в целом, обуславливает потребность в создании универсального штамма дрожжей S. cerevisiae, который бы обеспечил возможность модификаций генетической конструкции альфа-амилазы для изменения ее свойств в зависимости от нужд области применения, т.е. расширения области применения, а также для получения вариантов с высоким устойчивым уровнем продукции и секреции фермента.
Наиболее близким аналогом (прототипом) такого штамма является штамм S. cerevisiae AAC icrobiol с генотипом МАТа URA3HIS3 LEU2 TRP1 SUC2 MAL2-8c tpil(41-707)::loxP-KanMX4-loxP, экспрессирующий альфа-амилазу из мицелиального гриба в плазмиде CPOTud и секретирующий фермент в питательную среду. Штамм описан в работе: Appl. Environ. Microbiol. Т. 80 №17 С.5542-5550 [10].
Данный штамм не обладает регулируемой мутабельностью, что означает, что для изменения его свойств необходимо проведение многократного отбора после УФ-облучения и анализа получаемых вариантов. Таким образом, данный штамм не может обеспечить быстрый и эффективный отбор целевых вариантов, продуцентов альфа-амилазы.
Технический результат настоящего изобретения заключается в конструировании штамма-продуцента альфа-амилазы на основе дрожжей S. cerevisiae с регулируемой мутабельностью.
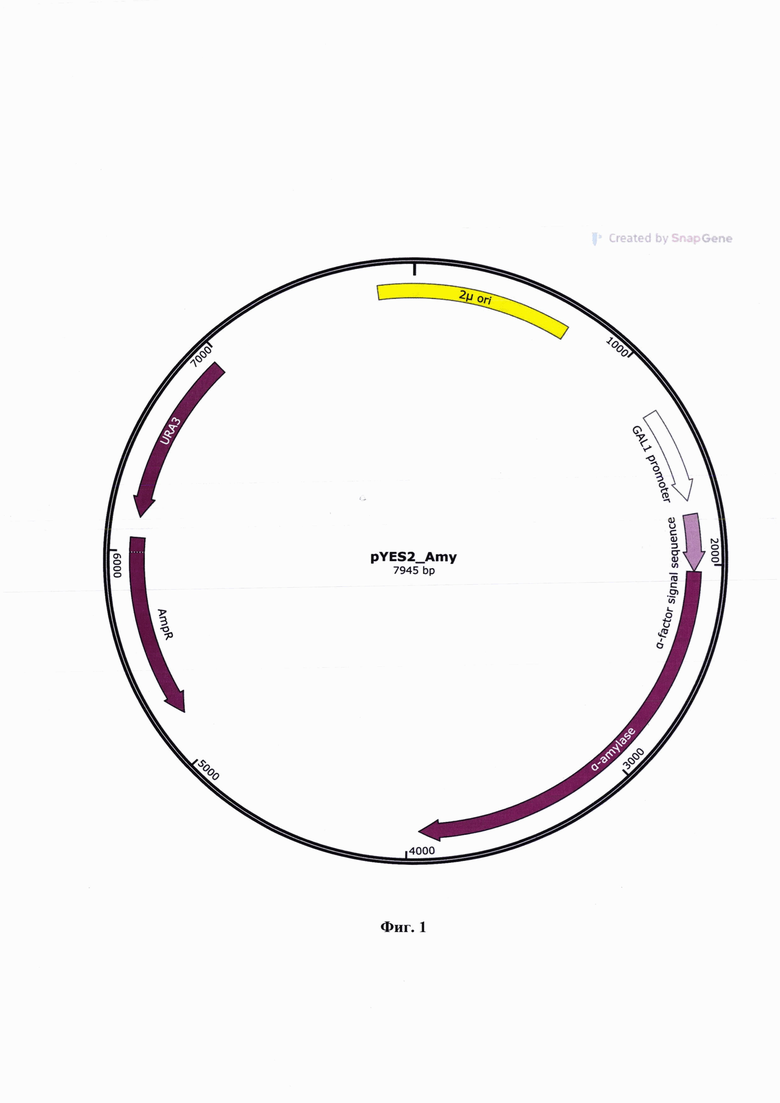
Технический результат достигается тем, что создан штамм Saccharomyces cerevisiae KEP1/pYES2-Amy-продуцент белка альфа-амилазы из Bacillus sp.Н3 с регулируемой мутабельностью, а также рекомбинантная плазмида pYES2-Amy для конструирования штамма Saccharomyces cerevisiae KEP1/pYES2-Amy, несущая ген, кодирующий белок альфа-амилазы из Bacillus sp.Н3, размером 7945 пар оснований (и.о.), характеризующаяся физической и генетической картой, представленной на фиг. 1.
Заявляемая совокупность признаков позволяет при каждой генерации клеток многократно повысить вероятность появления варианта продуцента альфа амилазы с нужными свойствами, т.е. обеспечивает эффективный отбор целевых вариантов.
Изобретение иллюстрируется следующими графическими фигурами.
На фиг. 1 приведена физическая и генетическая карта рекомбинантной плазмиды pYES2-Amy, содержащая последовательность альфа-амилазы Bacillus sp.НЗ.
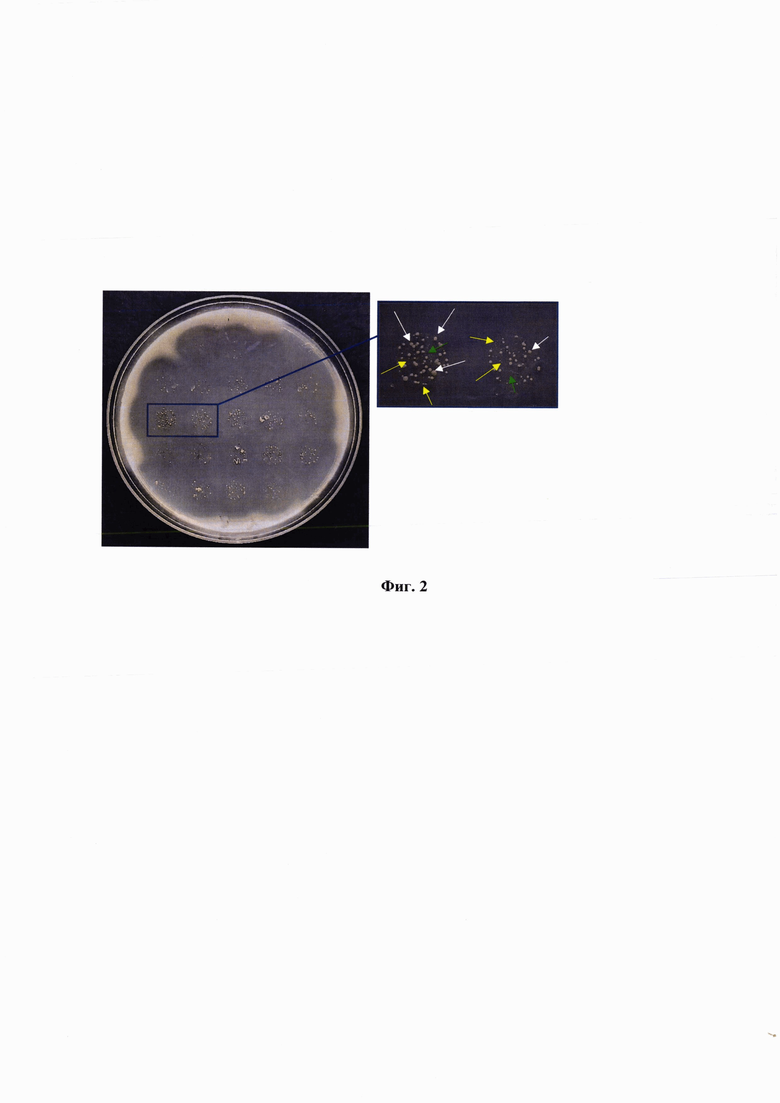
Фиг. 2 демонстрирует появление спонтанно возникших, активно растущих, колоний клеток среди медленно растущей исходной культуры KEP1/pYES2-Amy на селективной крахмалсодержащей среде.
Получение штамма KEP1/pYES2 включат следующие стадии.
1. Получение штамма ТАЕ366
Для создания штамма ТАЕ-366 использовали штамм дикого типа 11D-3031 (МАТα ade2Δ-248 leu2-3,112 ига3-160.188 trp1Δ) из коллекции дрожжевых штаммов Лаборатории генетики эукариот ПИЯФ. На первом этапе в этом штамме был делетирован ген RAD1. Плазмида pJH552 (любезно предоставленная Dr. R. Keil) была разрезана рестрикиазой SalI. Полученные фрагменты трансформировали в клетки штамма 11D-3031 и клетки высевали на селективную среду без урацила. Через четыре дня отбирали выросшие колонии, в которых ген RAD1 был заменен на ген LEU2. Полученные такимобразом штаммы проверяли на чувствительность к УФ-лучам. Штаммы с высокой УФ-чувствительностью несли мутацию rad1. На следующем этапе использовали один из полученных на первом этапе штаммов с высокой УФ-чувствительностью для делетирования гена HMO1. Для этой цели использовали плазмиду pJL15, любезно предоставленную др. S.J. Brill. Плазмида была разрезана рестрикиазой Sal1. Полученные фрагменты трансформировали в клетки отобранного штамма и клетки высевали на селективную среду без триптофана. Через четыре дня отбирали выросшие колонии, в которых ген HMO1 был заменен на ген TRP1. Полученные таким образом штаммы проверяли с помощью ПЦР на правильность встраивания маркерного гена TRP1. Один из полученных трансформантов, удовлетворяющих заданным свойствам, назван ТАЕ-366 (генотип МАТα ade2-248 leu2-3,112 ura3-160.188 rad1Δ hmo1Δ).
Свойства штамма TAE366: Штамм ТАЕ366 имеет генотип: МАТα ade2-248 1еи2-3,112 ига3-160.188 rad1Δ hsm2Δ. Мутация ade2-248 является делецией гена, контролирующего одну из стадий биосинтеза аденина; leu2-3,112 мутация блокирует синтез аминокислоты лейцина; ura3-160.188 блокирует синтез урацила; rad1Δ нарушает репарацию наиболее опасных для выживания клетки повреждений ДНК; hsm2Δ увеличивает вероятность репарационного мутагенеза. В течение роста клеток на питательной среде в их ДНК возникают спонтанные повреждения химической структуры. По оценкам, основанным на данных литературы, в течение одной генерации на богатой среде в ДНК дрожжей возникает около 100 спонтанных повреждений, которые в клетках дикого типа с большой вероятностью удаляются репарационными системами клетки. В мутанте rad1Δ многие из этих повреждений остаются и служат источником повышенного спонтанного мутагенеза; в штамме ТАЕ366 частота спонтанного мутагенеза в нормальных условиях повышается примерно в 10 раз. Если снизить скорость деления клеток, т.е. увеличить время генерации, количество спонтанных повреждений в ДНК клетки увеличится пропорционально увеличению этого времени. Мутация hsm2Δ в ходе незатронутых мутацией rad1Δ путей репарации, способных безошибочно обойти повреждение ДНК, приводит в условиях медленного роста к смене точных полимераз на часто ошибающиеся репаративные полимеразы, что еще раз многократно повышает выход мутаций. Таким образом, использование в штамме ТАЕ366 двух целевых мутаций rad1Δ и hsm2Δ позволяет управлять темпом спонтанного мутагенеза, регулируя условия роста.
2. Конструирования плазмиды pYES2-Amy
а. Амплификация гена альфа-амилазы из Bacillus sp.Н3
Штамм Bacillus sp.Н3 выращивали в 5 мл среды LB (Amresco) при температуре 37°С в аэробных условия при перемешивании 120 об/мин в течение 16 часов. Геномную ДНК выделяли с использованием набора реактивов Monarch® Genomic DNA Purification Kit (NEB) согласно инструкции производителя. Фрагмент, содержащий ген альфа-амилазы, получали при помощи полимеразной цепной реакции с использованием 100 нг геномной ДНК Bacillus sp.Н3 в качестве матрицы и праймеров
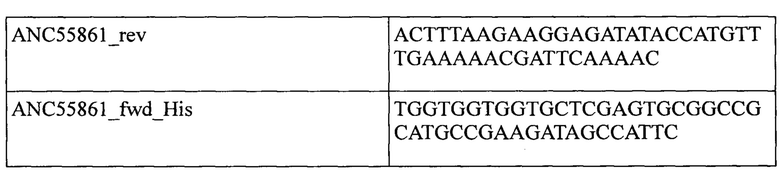
Для амплификации использовали высокоточную полимеразу Q5 (NEB) согласно инструкции производителя. Процесс амплификации состоял из следующих стадий: прогрев 3 мин при 96°С, 25 циклов ПЦР (30 сек при 96°С, 30 сек при 62°С, 1 мин при 72°С), инкубация 3 мин при 72°С. После амплификации фрагмент ДНК очищали электрофоретически в 1% агарозном геле с использованием набора реактивов Cleanup Standard (Евроген) согласно инструкции производителя.
b. Конструирование плазмиды pET24d(+)-Amy
Плазмидную ДНК pET24d(+) обрабатывали эндонуклеазами рестрикции EcoRI и NcoI (ThermoFisher scientific). Реакция ферментативного гидролиза проводилась в условиях, рекомендованных производителем. 50 мкл реакционной смеси содержали 2 мкг плазмидной ДНК и по 10 е.а. каждой эндонуклеазы рестрикции в буфере Tango 2х (ThermoFisher scientific). Векторную часть плазмидной ДНК очищали электрофоретически в 1% агарозном геле с использованием набора реактивов Cleanup Standard (Евроген) согласно инструкции производителя.
Векторную часть pET24d(+) и амплифицированный фрагмент ДНК сшивали с использованием набора реактивов NEBuilder® HiFi DNA Assembly (NEB) согласно инструкции производителя. Реакционная смесь содержала 50 нг векторной части pET24d(+) и 40 нг амплифицированного фрагмента ДНК. Полученной смесью трансформировали Е. coli штамм DH5α. Трансформантов высевали на твердую среду LB, содержащую 50 мкг/мл канамицина.
Отбор клонов на наличие вставки проводили при помощи ПЦР с колонии. Для амплификации использовали полимеразу Taq (ThermoFisher scientific) согласно инструкции производителя. Процесс амплификации состоял из следующих стадий: прогрев 3 мин при 95°С, 35 циклов ПЦР (30 сек при 95°С, 30 сек при 55°С, 2 мин при 72°С). Разделение продуктов амплификации проводили в 1% агарозном геле с бромистым этидием (0.5 мкг/мл). Положительные колонии выращивали в 5 мл среды LB, содержащей 50 мкг/мл канамицина, в течение 16 часов при 37°С при 120 об/мин, после чего выделяли из бактериальных клеток плазмидную ДНК с использованием набора реактивов Plasmid Miniprep (Евроген) согласно инструкции производителя. Структуру полученных плазмид подтверждали секвенированием. Секвенирование проводили по методу Сэнгера с использованием набора реактивов BigDye™ Terminator v3.1 согласно инструкции производителя. В 5 мкл реакционной смеси содержалось 80 нг плазмидной ДНК, 3.2 пмоль праймера и 0,25 мкл BigDye™ Terminator 3.1 Ready Reaction Mix. Для секвенирования использовались следующие праймеры:

Очистка продуктов реакции секвенирования проводилась с использованием D-Pure™ DyeTerminator Removal kit (Nimagen) согласно инструкции производителя. Электрофоретический анализ продуктов реакции проводился на приборе Нанофор-05 (Синтол) с последующей обработкой данных с использованием программы Mutation Surveyor (SoftGenetics). Выравнивание нуклеотидных последовательностей проводилось с использованием программы Ugene (Унипро). Полученная консенсусная нуклеотидная последовательность гена альфа-амилазы из Bacillus sp.Н3 была депонирована в базу данных GenBank под номером OR713115.
с. Конструирование рекомбинантной плазмиды pYES2-Amy
Плазмидную ДНК pYES2 (Invitrogen) последовательно обрабатывали эндонуклеазами рестрикции Hindlll и XbaI (ThermoFisher scientific). Реакция ферментативного гидролиза проводилась в условиях, рекомендованных производителем. 50 мкл реакционной смеси содержали 2 мкг плазмидной ДНК и 10 е.а. эндонуклеазы Hindlll. После окончания реакции ДНК очищалась с использованием набора реактивов CleanMag DNA PCR (Евроген) согласно инструкции производителя. ДНК элюировали 50 мкл воды и обрабатывали 10 е.а. эндонукдеазы рестрикции XbaI. Векторную часть плазмидной ДНК очищали электрофоретически в 1% агарозном геле с использованием набора реактивов Cleanup Standard (Евроген) согласно инструкции производителя.
Сигнальный пептид в соответствующей аминокислотной последовательности альфа-амилазы был определен с использованием программы SignalP - 5.0. Ген альфа-амилазы без сигнального пептида был получен при помощи ПЦР с использованием 1 нг плазмидной ДНК pET24d(+)-Amy в качестве матрицы и праймеров.
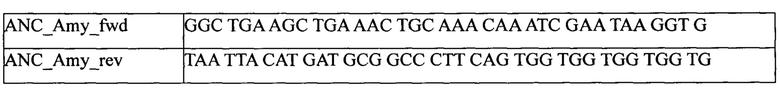
Для амплификации использовали высокоточную полимеразу Q5 (NEB) согласно инструкции производителя. Процесс амплификации состоял из следующих стадий: прогрев 3 мин при 96°С, 25 циклов ПЦР (10 сек при 96°С, 10 сек при 62°С, 1 мин при 72°С), инкубация 3 мин при 72°С. После амплификации фрагмент ДНК очищали с использованием набора реактивов CleanMag DNA PCR.
Фрагмент ДНК, содержащий сигнальную последовательность альфа-фактора дрожжей, был получен при помощи ПЦР с использованием 1 нг плазмидной ДНК pPICZa (ThermoFisher scientific) в качестве матрицы и праймеров

Для амплификации использовали высокоточную полимеразу Q5 (NEB) согласно инструкции производителя. Процесс амплификации состоял из следующих стадий: прогрев 3 мин при 96°С, 25 циклов ПЦР (10 сек при 96°С, 10 сек при 62°С, 20 сек при 72°С), инкубация 1 мин при 72°С. После амплификации фрагмент ДНК очищали с использованием набора реактивов CleanMag DNA PCR.
Векторную часть pYES2 и амплифицированные фрагменты ДНК сшивали с использованием набора реактивов NEBuilder® HiFi DNA Assembly (NEB) согласно инструкции производителя. Реакционная смесь содержала 50 нг векторной части pYES2, 35 нг фрагмента ДНК, содержащего ген альфа-амилазы, и 5 нг фрагмента ДНК, содержащего сигнальный пептид альфа-фактора дрожжей. Полученной смесью трансформировали Е. coli штамм DH5α. Трансформантов высевали на твердую среду LB, содержащую 50 мкг/мл ампициллина.
Отбор клонов на наличие вставки проводили при помощи ПЦР с колонии. Для амплификации использовали полимеразу Taq (ThermoFisher scientific) согласно инструкции производителя. Процесс амплификации состоял из следующих стадий: прогрев 3 мин при 95°С, 35 циклов ПЦР (30 сек при 95°С, 30 сек при 55°С, 2 мин при 72°С). Разделение продуктов амплификации проводили в 1% агарозном геле с бромистым этидием (0.5 мкг/мл). Положительные колонии выращивали в 5 мл среды LB, содержащей 50 мкг/мл ампициллина, течение 16 часов при 37°С при 120 об/мин, после чего выделяли из бактериальных клеток плазмидную ДНК с использованием набора реактивов Plasmid Miniprep (Евроген) согласно инструкции производителя. Структуру полученных плазмид подтверждали секвенированием. Секвенирование проводили по методу Сэнгера с использованием набора реактивов BigDye™ Terminator v3.1 согласно инструкции производителя. В 5 мкл реакционной смеси содержалось 80 нг плазмидной ДНК, 3.2 пмоль праймера и 0.25 мкл BigDye™ Terminator 3.1 Ready Reaction Mix. Для секвенирования использовались следующие праймеры:

Очистка продуктов реакции секвенирования проводилась с использованием D-Pure™ DyeTerminator Removal kit (Nimagen) согласно инструкции производителя. Электрофоретический анализ продуктов реакции проводился на приборе Нанофор-05 (Синтол) с последующей обработкой данных с использованием программы Mutation Surveyor (SofxGenetics). Выравнивание нуклеотидных последовательностей проводилось с использованием программы Ugene (Унипро).
Полученная плазмидная ДНК pYES2-Amy использовалась для трансформации дрожжей S. cerevisiae штамм ТАЕ366. Выросшие на селективной среде трансформанты проверяли с помощью ПЦР-реакции на правильность вставки. Среди полученных трансформантов отбирался наиболее активно растущий, который был назван KEP1/pYES2-Amy, для дальнейшей проверки на амилазную активность.
Характеристика плазмиды (фиг. 1): Размер плазмиды pYES2-Amy 7945 пар оснований. Плазмида pYES2-Amy содержит ряд структурных элементов. Участок 2μ ori обеспечивает инициацию репликации в штаммах Saccharomyces cerevisiae. Дрожжевой промотор GAL1 обеспечивает эффективную экспрессию рекомбинантного белка в дрожжах, индуцируемую галактозой. Ген α-амилазы Bacillus sp.Н3 кодирует целевой рекомбинантный белок. Сигнальная последовательность а-фактора дрожжей обеспечивает внеклеточную экспрессию рекомбинантного белка. Ген URA3 кодирует оротидин-5'-фосфатдекарбоксилазу, необходимую для биосинтеза урацила, и является селективным маркером для трансформации штаммов дрожжей с генотипом ura3. Ген AmpR кодирует белок бета-лактамазу, способствующий устойчивости к ампициллину, карбенициллину и родственным антибиотикам, и является селективным маркером для трансформации штаммов Е. coli.
3. Проверка альфа-амилазной активности полученного штамма KEP1/pYES2-Amy
Для этого клетки штамма KEP1/pYES2-Amy были выращены в жидкой среде состава: среда Yeast Nitrogen Base w/o amino acids, 6,7 г/л; крахмал, 20 г/л; галактоза, 20 г/л; аминокислоты и аденин (без урацила), по 20 мг/л. Для изготовления плотных сред добавлялся агар-агар, 25 г/л. Любая питательная среда приготавливалась с использованием дистиллированной воды. Клетки в жидкой среде выращивались в термостате на шейкере (120 об/мин) при температуре 28,5°С в течение 3-х суток, после чего культуральную жидкость отделяли центрифугированием.
Для оценки амилазной активности 20 мкл культуральной жидкости, предварительного отдиализованной против 20 ммоль раствора калий-фосфатного буфера, рН 6,5, смешали с 40 мкл 1%-ного раствора крахмала, выдержали реакционную смесь 15 минут при 40°С. Затем амилазную активность оценивали по количеству редуцирующих Сахаров, согласно описанной методике: J. Biol. Eng. 2017. Т. 11, №1 [11]. Содержание белка определяли с помощью анализа связывания красителя по методу Брэдфорда: Anal. Biochem. 1976. Т. 72. С.248-254 [12] с бычьим сывороточным альбумином в качестве эталонного белка, измеряемого по поглощению света при 595 нм.
В результате альфа-амилазная активность составила 1,3 ед./мг белка.
4. Проверка регулируемой мутабельности у штамма KEP1/pYES2-Amy
Экспериментальным путем установлен медленный рост полученного штамма KEP1/pYES2-Amy на твердой среде с крахмалом, что включает его высокую мутабельность.
Суспензия клеток штамма KEP1/pYES2-Amy с концентрацией 107 кл/мл наносилась с помощью репликатора на чашки Петри с плотной средой с крахмалом (объем перенесенных капель -0,02 мл). Через 10 дней видно (фиг. 2), что исходные клетки практически не растут на этой среде (фоновый рост на отпечатке капли), но появляются быстро растущие клетки, которые образуют видимые колонии разного размера. Крупные колонии образовались за счет мутантов, возникших при первых делениях клеток и последующих делениях уже в составе растущей колонии, более мелкие образовались при последующих делениях исходных клеток и претерпели значительно меньше генераций при росте колоний, что ограничило появление эффективной комбинации мутаций, необходимых для успешного роста на селективной среде. Белыми стрелками обозначены клетки 1-й генерации, желтыми - 2-й, зелеными - 3-й.
Выводы: В результате настоящего изобретения полученный штамм KEP1/pYES2-Amy при выращивании на селективной крахмалсодержащей питательной среде растет исключительно медленно и при этом с высокой скоростью мутирует. Это обеспечивает при каждой генерации клеток многократное повышение вероятности появления варианта с нужными свойствами и приводит к быстрому отбору мутаций, приводящих к более активному расщеплению крахмала до глюкозы и, как следствие, образованию колоний мутантных клеток на этой среде.
Таким образом, данный штамм обеспечивает быстрый и эффективный отбор целевых вариантов без многократных отборов вариантов и анализов требуемых свойств.
Список использованной литературы
1. Mattanovich D., Branduardi P., Dato L. et al. Recombinant protein production in yeasts. Methods Mol. Biol. 2012. T. 824. C. 329-358.
2. Nielsen J. Production of biopharmaceutical proteins by yeast: advances through metabolic engineering. Bioengineered. 2013. T. 4. C. 207-211.
3. Jozala A.F., Geraldes D.C., Tundisi L.L. et al. Biopharmaceuticals from microorganisms: from production to purification. Braz. J. Microbiol. 2016. T. 47. №1. C. 51-63.
4. Liu Z., Hou J., Martinez J.L. et al. Correlation of cell growth and heterologous protein production by Saccharomyces cerevisiae. Appl. Microbiol. Biotechnol. 2013. T. 97. C. 8955-8962.
5. Vieira Gomes A.M., Souza Carmo Т., Silva Carvalho L. et al. Comparison of yeasts as hosts for recombinant protein production. Microorganisms. 2018. T. 6. C. 38.
6. Schmidt F. Recombinant expression systems in the pharmaceutical industry. Appl. Microbiol. Biotechnol. 2004. T. 65. №4. C. 363-372.
7. Патент ЕР 0252774A1. Gloeckler R., Loison G., LemoineY. Saccharomyces strains producing alpha-amylase. Опубл.1988-01-13.
8. Патент US 20130244295 Al. Van Zyl et al. Biofuel production. Опубл. 2013-09-13.
9. Liu Z., Tyo K.E.J., Martinez J.L., Petranovic D., Nielsen J. Different expression systems for production of recombinant proteins in Saccharomyces cerevisiae. Biotechnol Bioeng. 2012. T. 109. №5. C. 1259-1268.
10. Liu Z., Liu L., Osterlund Т., Hou J. et al. Improved production of a heterologous amylase in Saccharomyces cerevisiae by inverse metabolic engineering. Appl. Environ. Microbiol. T. 80. №17. C. 5542-5550 – прототип.
11. Muhlmann M., Kunze M., Ribeiro J., Geinitz В., Lehmann C, Schwaneberg U., et al. Cellulolytic RoboLector - towards an automated high-throughput screening platform for recombinant cellulase expression. J. Biol. Eng. 2017. T. 11, №1.
12. Bradford M.M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Anal. Biochem. 1976. T. 72. C. 248-254.
| название | год | авторы | номер документа |
|---|---|---|---|
| Рекомбинантная плазмида pUSB2-AmQ, обеспечивающая синтез белка альфа-амилазы Bacillus amyloliquefaciens, и штамм Bacillus subtilis/pUSB2-AmQ - продуцент белка альфа-амилазы Bacillus amyloliquefaciens | 2020 |
|
RU2747627C1 |
| СПОСОБ МИКРОБИОЛОГИЧЕСКОГО СИНТЕЗА ЗРЕЛОГО ИНТЕРФЕРОНА АЛЬФА-2 ЧЕЛОВЕКА, ШТАММ Saccharomyces cerevisiae - ПРОДУЦЕНТ ЗРЕЛОГО ИНТЕРФЕРОНА АЛЬФА-2 ЧЕЛОВЕКА (ВАРИАНТЫ) | 2010 |
|
RU2427645C1 |
| ШТАММ ДРОЖЖЕЙ SACCHAROMYCES CEREVISIAE 1-60-Д578 (MSIL), - ПРОДУЦЕНТ РЕКОМБИНАНТНОГО ИНТЕРЛЕЙКИНА-2 ЧЕЛОВЕКА И СПОСОБ ЕГО ПОЛУЧЕНИЯ | 2002 |
|
RU2230781C1 |
| РЕКОМБИНАНТНАЯ ПЛАЗМИДНАЯ ДНК pPBS-St9, КОДИРУЮЩАЯ ПОЛИПЕПТИД СОМАТОТРОПИНА, И ШТАММ ДРОЖЖЕЙ SACCHAROMYCES CEREVISIAE ДЛЯ ПРОДУКЦИИ РЕКОМБИНАНТНОГО СОМАТОТРОПИНА | 2011 |
|
RU2465315C1 |
| Способ получения альфа-амилазы | 1982 |
|
SU1838410A3 |
| Штамм бактерий BacILLUS амYLоLIQUеFасIеNS-продуцент @ -амилазы BacILLUS LIснеNIFоRмIS | 1991 |
|
SU1788966A3 |
| Способ получения штамма-продуцента фосфолипазы А2 Komagataella phaffii (Pichia pastoris) YIB Δleu2_PLA2S | 2020 |
|
RU2746817C1 |
| ВАРИАНТЫ АЛЬФА-АМИЛАЗЫ | 2000 |
|
RU2231547C2 |
| ПРОТИВОМИКРОБНЫЕ ПОЛИПЕПТИДЫ ИЗ PSEUDOPLECTANIA NIGRELLA | 2002 |
|
RU2336278C2 |
| Фрагмент ДНК ALV 7, кодирующий синтез альфа-амилазы, способ его конструирования и штамм бактерий BacILLUS SUвтILIS - продуцент альфа-амилазы | 1990 |
|
SU1717633A1 |
Изобретение относится к биотехнологии. Предложена рекомбинантная плазмида для экспрессии альфа-амилазы из Bacillus sp.Н3, несущая ген, кодирующий альфа-амилазу из Bacillus sp.Н3, имеющая размером 7945 пар оснований, полученная на основе pYES2 и характеризующаяся физической и генетической картой, представленной на фиг. 1. Также предложен штамм дрожжей Saccharomyces cerevisiae - продуцент альфа-амилазы из Bacillus sp.Н3, обладающий регулируемой мутабельностью, полученный путем трансформации штамма Saccharomyces cerevisiae, имеющего генотип MATα ade2-248 leu2-3,112 ura3-160.188 radlΔ hmolΔ, вышеуказанной рекомбинантной плазмидой. Изобретение позволяет получить штамм-продуцент альфа-амилазы из Bacillus sp.Н3 на основе дрожжей S. cerevisiae с регулируемой мутабельностью. 2 н.п. ф-лы, 2 ил., 5 табл.
1. Рекомбинантная плазмида для экспрессии альфа-амилазы из Bacillus sp.Н3, несущая ген, кодирующий альфа-амилазу из Bacillus sp.Н3, имеющая размером 7945 пар оснований, полученная на основе pYES2 и характеризующаяся физической и генетической картой, представленной на фиг. 1.
2. Штамм дрожжей Saccharomyces cerevisiae - продуцент альфа-амилазы из Bacillus sp.Н3, обладающий регулируемой мутабельностью, полученный путем трансформации штамма Saccharomyces cerevisiae, имеющего генотип MATα ade2-248 leu2-3,112 ura3-160.188 radlΔ hmolΔ, рекомбинантной плазмидой по п. 1.
| SUE-ANN O’CALLAGHAN, B.Sc | |||
| Saccharomyces cerevisiae: a model host for antisense gene control? A thesis presented for the Degree of Ph.D | |||
| ЩИТОВОЙ ДЛЯ ВОДОЕМОВ ЗАТВОР | 1922 |
|
SU2000A1 |
| Клозетный промывной бак | 1929 |
|
SU19149A1 |
| Taxonomy Bacillus sp | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Найдено по адресу: | |||
Авторы
Даты
2025-01-29—Публикация
2023-11-29—Подача