Изобретение относится к ветеринарной вирусологии, к средствам молекулярной диагностики, а именно к дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ собак с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
Возбудитель парвовирусного энтерита Canine parvovirus (CPV) вызывает острый, иногда с летальным исходом, гастроэнтерит у собак.
Собачий парвовирус (ПВЭ) является патогеном для домашних собак и некоторых видов диких хищников. Он принадлежит к роду Protoparvovirus семейства Parvoviridae. Вирус автономно реплицируется в клетках-хозяевах и генетически связан с парвовирусом кошек (FPV) и вирусом энтерита норок (MEV), которые инфицируют разных животных-хозяев. Оригинальный CPV был впервые идентифицирован в 1978 году и быстро распространился по всему миру [1, 2].
Геном возбудителя парвовирусного энтерита представлен двуцепочечной молекулой ДНК длиной около 4200-4300 п.н. В результате проведения его картирования определили, что участок ДНК в диапазоне 1…2007 п.н. кодирует два неструктурных белка NS1 и NS2. Структурный белок VP1 кодируется участками в диапазонах 2014…2044 и 2117…2514 п.н., а VP2 - 2515…4269 п.н. [3].
В настоящее время известны следующие генотипы CPV: 1, 2а, 2b, 2c, 3, которые по-разному распространены в популяции собак во всем мире [4-6].
Система мер для борьбы с парвовирусным энтеритом собак и его профилактики предусматривает иммунизацию домашних животных. Для этой цели применяют вакцинные препараты [7, 8, 9].
Для иммунизации щенков применяют различные вакцины против парвовирусного энтерита, преимущественно состоящие из антигена CPV производственного штамма «Арчи».
В случае вспышек данного заболевания важно правильно дифференцировать вакцинный штамм «Арчи» и полевые изоляты вируса друг от друга при проведении лабораторной диагностики для подтверждения безопасности производимого вакцинного лекарственного препарата и принятия дальнейших мер по лечению и борьбе с заболеванием, поскольку штаммы существенно отличаются друг от друга генетически и по строению и функциям вирусных белков. Следует отметить, что данный анализ также необходим на этапе контроля качества вируссодержащего сырья при изготовлении вакцинных препаратов и подтверждения использования требуемого штамма для производства вакцин против парвовирусного энтерита собак.
Штамм «Арчи» возбудителя парвовирусного энтерита собак депонирован во Всероссийской государственной коллекции экзотических типов вируса ящура и других патогенов животных (ГКШМ) ФГБУ «ВНИИЗЖ», под регистрационным номером: № 454-деп / 23-4 - ГКШМ ФГБУ «ВНИИЗЖ».
Учитывая, что в настоящее время производственный штамм «Арчи» применяется для изготовления вакцин, целесообразно провести разработку способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ.
В настоящее время методы молекулярно-биологического анализа находят широкое применение в диагностике различных инфекционных заболеваний. Благодаря созданию последнего поколения флуоресцирующих красителей и технологическим усовершенствованиям платформ количественной полимеразной цепной реакции (ПЦР) исследование пиков температур плавления продуктов реакции в HRM-анализе (High-resolution melting, HRM) стало перспективным методом генотипирования микроорганизмов, который возможно применять для дифференциации вакцинных штаммов вирусов [10-16].
Анализ максимальных значений температур плавления для различных изолятов/штаммов вирусов и других микроорганизмов представляет собой исследование кривой плавления ПЦР-продуктов в широком диапазоне температур с узким шагом. Сочетание улучшенного инструментария количественной детекции ампликонов и флуоресцирующих красителей последнего поколения позволила выявлять генетические вариации в последовательностях нуклеиновых кислот путем контролируемого плавления двухцепочечных ДНК [15-17].
Сущность представленного метода заключается в том, что после проведения ПЦР с применением флуоресцирующего красителя смесь постепенно нагревают, и при достижении определённой температуры ампликоны начинают разъединяться, флуорофор постепенно высвобождается, и флуоресценция, которая детектируется оптической системой термоциклера, снижается. Чувствительность анализа достигает одного нуклеотида, благодаря чему с помощью данного метода можно достичь высокой общей точности разработанного способа [14].
Прототипным вариантом проведения дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ является ПЦР с применением специфических праймеров, гель-электрофорез и секвенирование методом Фредерика Сенгера [17]. При этом указанный метод является крайне дорогостоящим, трудоемким и продолжительным во времени проводимого исследования. Следовательно, необходимо разработать альтернативный способ дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
Задачей настоящего изобретения является разработка чувствительного и специфичного, быстрого способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428 с целью устранения вышеуказанных недостатков.
Данная задача решена благодаря разработке способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
Сущность разработанного способа заключается в следующем:
1) создание дизайна оригинальных высокоспецифичных олигонуклеотидных прямого и обратного праймеров для синтеза фрагмента ДНК с мутациями, уникальными для вакцинного штамма «Арчи» CPV; 2) проведение ПЦР с применением тандемного флуоресцирующего красителя SN v428; 3) полученные ПЦР-продукты постепенно нагревают в широком диапазоне температур, и при достижении пика температуры плавления двуцепочечная молекула ДНК распадается на отдельные цепи, что вызывает яркий флуоресцентный сигнал, специфичный для красителя. Для каждого варианта нуклеотидного состава характерна своя температура плавления. Анализируя кривые плавления ПЦР-продуктов, возможно проводить дифференциацию вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ.
Разработанный способ дает возможность: 1) сократить время проведения анализа до 2 ч; 2) применять тандемный флуоресцирующий краситель последнего поколения SN v428, который не ингибирует реакцию, и характеризующийся яркой флуоресценцией после встраивания в ампликоны, что позволяет достичь высокую точность и чувствительность проводимого анализа; 3) увеличить чувствительность и специфичность анализа за счет использования высокоспецифичных оригинальных олигонуклеотидных праймеров, рассчитанных для целевого участка ДНК гена VP1 возбудителя парвовирусного энтерита собак (таргетный участок указанного гена в диапазоне 2524…2699 п.н., размер ПЦР-продукта составляет 176 п.н.). Данная разработка позволит расширить арсенал средств для проведения контроля качества сырья при производстве вакцин против парвовирусного энтерита собак из производственного штамма «Арчи».
Технический результат изобретения заключается в том, что разработанный способ дает возможность: 1) повысить чувствительность и специфичность за счет применения высокоспецифичных оригинальных праймеров CPV-F2524 с дизайном 3'-GGAGCAGTTCAACCAGACGGT-5' и CPV-R2676 с дизайном 3'-GTGATTTCCACCCATCCGTTTTCC-5', рассчитанных для целевого участка VP1-гена CPV c размером ПЦР-продукта 176 п.н.; 2) повысить достоверность проводимого анализа благодаря применению тандемного флуоресцирующего красителя последнего поколения SN v428; 3) с помощью высокочувствительного HRM-анализа по найденным константам проводить дифференциацию вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ.
Сущность изобретения отражена на графических изображениях:
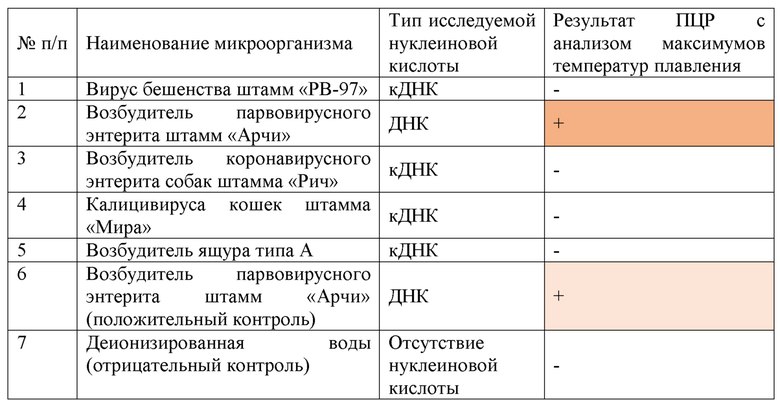
Фиг. 1 - Дизайн оригинальных высокоспецифичных олигонуклеотидных праймеров для дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428. Примечание: rev-compl - перевернутая комплементарная форма обратного праймера.
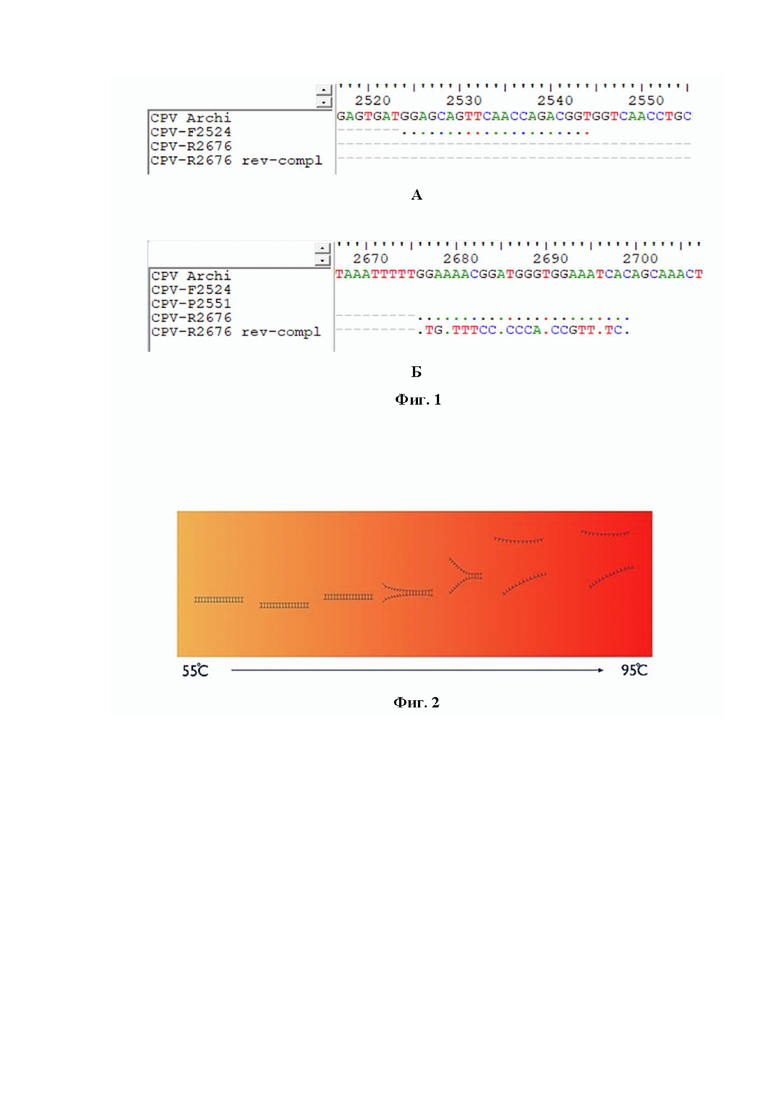
Фиг. 2 - Сущность HRM-анализа.
Фиг. 3 - Карта нуклеотидных замен для участка VP1-гена вакцинного штамма «Арчи» по сравнению с другими штаммами/изолятами. Примечание: анализировали диапазон 2524…2699 п.н.; для исследования использовали следующие вакцинные штаммы/полевые изоляты возбудителя парвовирусного энтерита собак: «HD-97», «Fine-5», «DDH-5», «Klin-22», «Ballu-2», «JIO-144», «Kine26».
Фиг. 4 - Диаграмма максимумов температур плавления ПЦР-продуктов при анализе вакцинных штаммов/полевых изолятов возбудителя парвовирусного собак. Примечание: для исследования использовали следующие вакцинные штаммы возбудителя парвовирусного энтерита собак: «HD-97», «Fine-5», «DDH-5», «Klin-22», «Ballu-2», «JIO-144», «Kine26».
Сущность изобретения пояснена в перечне последовательностей, в котором:
SEQ ID NO:1 представляет последовательность нуклеотидов ДНК VP1 гена вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак;
SEQ ID NO:2 представляет последовательность аминокислот, кодируемых VP1 геном ДНК вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак;
SEQ ID NO:3 представляет последовательность нуклеотидов прямого праймера CPV-F2524;
SEQ ID NO:4 представляет последовательность нуклеотидов обратного праймера CPV-R2676.
Заявляемый способ основан на проведении следующих этапов анализа: 1) экстрагирование ДНК CPV из образца; 2) проведение ПЦР специфического фрагмента ДНК гена VP1 возбудителя парвовирусного энтерита собак размером 176 п.н. с применением оригинальных олигонуклеотидных высокоспецифичных праймеров CPV-F2524 с дизайном 3'-GGAGCAGTTCAACCAGACGGT-5' и CPV-R2676 с дизайном 3'-GTGATTTCCACCCATCCGTTTTCC-5' (фиг. 1, таблицы 1 и 2) и тандемного флуоресцирующего красителя SN v428; 3) детекция результатов анализа с определением максимумов температуры плавления и проведение HRM-анализа геномов вакцинного штамма «Арчи» от других производственных штаммов/полевых изолятов возбудителя парвовирусного энтерита собак (фиг. 2).
По результатам анализа опубликованных в научной литературе данных, в настоящее время используется только прототипный способ дифференциации штаммов и изолятов CPV с помощью секвенирования по Ф. Сенгеру [17], однако, как указано выше, он очень дорогостоящий, трудоемкий и продолжительный по времени проводимого исследования. При анализе публикаций способ дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428 не представлен. Таким образом, сведений об аналогах предлагаемого способа авторами не обнаружено.
Разработанный способ дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428 по сравнению с прототипом отличается экономической выгодой, легкостью и экспрессностью выполнения анализа, что позволяет быстро и качественно проводить анализ. Тандемный флуоресцирующий краситель SN v428 более чувствителен своих аналогов, в частности, чем бромистый этидий, SYBR Green I и SYBR Green II, для обнаружения различных типов нуклеиновых кислот. Максимум возбуждения для красителя SN v428 отмечается при длине волны 414 нм, а максимум излучения - при 428 нм [18].
В отличие от прототипа разработанный способ позволяет провести анализ выравнивания множественных нуклеотидных последовательностей целевого участка ДНК гена VP1 вакцинных штаммов/полевых изолятов возбудителя парвовирусного энтерита собак. Разработанный способ предусматривает проведение ПЦР специфического фрагмента гена VP1 ДНК в диапазоне 2524…2699 п.н. с применением олигонуклеотидных высокоспецифичных праймеров CPV-F2524 с дизайном 3'-GGAGCAGTTCAACCAGACGGT-5' и CPV-R2676 с дизайном 3'-GTGATTTCCACCCATCCGTTTTCC-5' для амплификации фрагмента размером 176 п.н. Разработанный способ предусматривает проведение плавления ампликонов в разработанном режиме с учетов пиков температур плавления двуцепочечных ДНК и применение тандемного флуоресцирующего красителя SN v428, а также детекцию результатов анализа с определением максимальных значений температур плавления для решения задачи по дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ. Таким образом, разработан способ дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
Обоснованием выбранного участка гена VP1 возбудителя парвовирусного энтерита является наличие двух уникальных нуклеотидных мутаций, характерных для вакцинного штамма «Арчи» CPV в сравнении со штаммами и изолятами CPV, а именно в позиции 2606 п.н. замена T на G, 2620 п.н. - A на G (фиг. 3).
Ключевым элементом заявляемого способа является применение олигонуклеотидных праймеров CPV-F2524 с дизайном 3'-GGAGCAGTTCAACCAGACGGT-5' и CPV-R2676 с дизайном 3'-GTGATTTCCACCCATCCGTTTTCC-5' и HRM-анализа с применением тандемного флуоресцирующего красителя SN v428 для дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ.
Сопоставительный анализ с прототипом позволяет сделать вывод, что новизна и изобретательский уровень заявляемого изобретения заключаются в применении олигонуклеотидных праймеров CPV-F2524 и CPV-R2676, рассчитанных для амплификации целевого участка ДНК гена VP1 возбудителя парвовирусного энтерита размером 176 п.н. и HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
На основном этапе исследования проводят выделение ДНК из образцов с использованием набора «ДНК-сорб» в соответствии с инструкцией производителя («Интерлабсервис», РФ).
На следующем этапе проводят ПЦР с применением олигонуклеотидных праймеров и тандемного флуоресцирующего красителя SN v428 для исследования стандартного образца и проб. Стандартным образцом является культуральная суспензия CPV вакцинного штамма «Арчи», которая изначально исследована с помощью секвенирования по Сенгеру для подтверждения наличия именно требуемого штамма в стандарте (n = 5). В качестве отрицательного контроля используют деионизированную воду без нуклеаз MilliQ.
Для постановки ПЦР готовят реакционную смесь, которая включает в свой состав следующие компоненты: PCR buffer 10х - 3 мкл, хлорид магния 25 мМ - 3 мкл, олигонуклеотидыне праймеры - по 5 мкл, краситель SN v428 - 1 мкл, Taq ДНК-полимераза - 1 е.а., элюат ДНК - 6 мкл, деионизированная вода - 6,9 мкл (0,1 мкл). Итоговый объем смеси для проведения одной реакции составляет 30 мкл.
Постановку ПЦР осуществляют в детектирующем термоциклере CFX-96 (Bio-Rad, США) или аналоге при температурных и временных параметрах, сведения о которых представлены в таблице 3.
Предварительную денатурацию проводят при температуре 95°С в течение 5 минут. ПЦР в режиме реального времени включает в себя 3 подэтапа: денатурацию, отжиг олигонуклеотидов, элонгацию. Денатурацию осуществляют при температуре 98°С в течение 7 секунд, отжиг олигонуклеотидов и элонгацию - при температуре 58°С в течение 45 секунд. Далее проводят плавление при температуре, начиная с 65 до 90°С. Увеличение температуры составляет - 0,1°С за каждый шаг. При этом после первого шага плавления требуется ждать 90 секунд при температуре первого шага. Для каждого последующего шага время ожидания составляет 2 секунды.
Исследование кривой плавления двуцепочечных фрагментов ДНК представленных выше вакцинных штаммов/полевых изолятов возбудителя парвовирусного энтерита для анализа данных и профилей плавления образцов выполняют с использованием программного обеспечения для сканирования с помощью детектирующего амплификатора. Проводят построение кривых плавления в виде параболы и детектируют пики температур плавления для полученных графиков. Выявляют, какие максимумы температур плавления характерны для указанных выше штаммов/изолятов возбудителя парвовирусного энтерита собак, что позволяет проводить дифференциацию вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
Сущность предлагаемого изобретения пояснена примерами его использования, которые не ограничивают объем изобретения.
Пример 1. Разработка способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428
Для определения показателей, позволяющих проводить дифференциацию вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428, осуществляли этапы работы, представленные ниже.
На первом этапе исследования проводили выделение нуклеиновой кислоты из вируссодержащего образца, как указано выше.
На следующем этапе проводили ПЦР для исследования проб в соответствии с описанием, представленным выше. Для постановки реакции готовили реакционную смесь, как отражено выше. Проведение анализа осуществляли, как отражено выше.
Исследование кривой плавления для HRM-анализа данных пиков температуры плавления ампликонов участка гена VP1 ДНК вакцинных штаммов/полевых изолятов возбудителя парвовирусного энтерита и профилей плавления образцов выполняли с использованием программного обеспечения CFX-96, или аналога. Проводили построение кривых плавления в виде параболы и детектировали максимумы температур плавления для полученных графиков. Тестировали ДНК CPV указанных вакцинных штаммов и полевых изолятов в 32 повторностях для каждого образца для получения результатов с высокой степенью достоверности (таблица 4).
Выявили, что для вакцинного штамма «Арчи» данная температура составила 73,86±0,04°С (n = 32, p < 0,001), «HD-97», «Fine-5», «DDH-5», «Klin-22», «Ballu-2», «JIO-144», «Kine26» - 76,21±0,04°С (n = 32, p < 0,001) (фиг. 4, таблица 4).
Следовательно, для каждого из исследуемых штаммов/изолятов возбудителя парвовирусного энтерита характерен индивидуальный пик температуры плавления. При этом значительно отличается именно вакцинный штамм «Арчи» CPV. Таким образом, разработан способ дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
Пример 2. Исследование аналитической специфичности разработанного способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428
Специфичность анализа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428 оценивали при исследовании суспензий штамма «РВ-97» вируса бешенства, штамма «Рич» возбудителя коронавирусного энтерита собак, штамма «Мира» калицивируса кошек, серотипа А вируса ящура. Количество инфекционных доз вирусов в суспензиях составлял не менее 6,0 lg ТЦД50/см3 или 6,0 lg ККИД50/см3. Исследования проводили в 5 повторностях.
В качестве положительного контроля использовали суспензию вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак. Отрицательным контролем служила деионизированная вода. Для проведения исследования использовали детектирующий амплификатор марки CFX-96 (Bio-Rad, США). В результате исследования амплификация с неспецифичными нуклеиновыми кислотами других инфекционных агентов графики не были сформированы, что подтверждало высокую специфичность компонентов реакционной смеси данного способа для возбудителя парвовирусного энтерита (таблица 5).
В результате исследования обнаружили, что для проб, не содержащих нуклеиновую кислоту возбудителя парвовирусного энтерита, и отрицательного контроля не формировали графики плавления и не были обнаружены пиковые значения при анализе высокого разрешения. При исследовании положительного контроля получены следующие значения температур плавления: 73,86±0,03°С (n = 3, p < 0,005). Результаты анализа контролей подтверждают стандартные данные, отраженные в примере 1.
Таким образом, полученные данные свидетельствовали о 100%-ной специфичности разработанного способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
Пример 3. Определение диагностических показателей разработанного способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428
Для определения диагностической чувствительности разработанного способа анализировали 385 заведомо положительных проб суспензий возбудителя парвовирусного энтерита штамма «Арчи» с титрами инфекционной активности не ниже 6,0 lg ТЦД50/см3.
Проведение ПЦР с последующим плавлением ампликонов осуществляли, как отражено выше. С помощью разработанного способа (предлагаемое изобретение) определили, что из 385 исследуемых проб CPV все определены в качестве положительных и подтверждено наличие именно того штамма, который содержался в анализируемых суспензиях.
Для исследования специфичности метода тестировали 265 суспензии возбудителя парвовирусного энтерита различных штаммов и изолятов, за исключением вакцинного штамма «Арчи». В результате исследования с помощью разработанного способа (предлагаемое изобретение) определили, что все 265 пробы содержали геном возбудителя парвовирусного энтерита CPV, но не штамма «Арчи». Пользуясь представленными выше статистическими методами анализа, определили, что в 95%-ном доверительном интервале диагностическая чувствительность (DSe) составила 99,05-100,00%, диагностическая специфичность (DSp) - 98,62-100,0%, k-критерий - 1,000; общая точность (DAc) - 99,43-100,00% (таблица 6).
Основными преимуществами предлагаемого изобретения является возможность проводить за короткий промежуток времени (не более 2 ч) дифференциацию вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428. Разработанный способ впервые применили для решения данной задачи. Выявили, что в целевом участке ДНК гена VP1 возбудителя парвовирусного энтерита имеется две нуклеотидных мутации, уникальные для вакцинного штамма «Арчи», что позволило применить олигонуклеотидные праймеры CPV-F2524 и CPV-R2676, рассчитанных для амплификации целевого участка ДНК гена VP1 возбудителя парвовирусного энтерита размером 176 п.н. и HRM-анализа с применением тандемного флуоресцирующего красителя SN v428.
Разработанный способ характеризуется 100%-ной аналитической специфичностью. В 95%-ном доверительном интервале диагностическая чувствительность для данного способа составляет 99,05-100,00%, диагностическая специфичность - 98,62-100,0%, точность - 99,43-100,00%.
Источники информации, принятые во внимание при составлении описания изобретения к заявке на выдачу патента РФ на изобретение «Способ дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428»:
1. Decaro N., Buonavoglia C., Barrs V.R. Canine parvovirus vaccination and immunisation failures: Are we far from disease eradication? Vet Microbiol. 2020 Aug; 247: 108760. doi: 10.1016/j.vetmic.2020.108760. Epub 2020 Jun 15. PMID: 32768213; PMCID: PMC7295477.
2. Kang B.K., Song D.S., Lee C.S., Jung K.I., Park S.J., Kim E.M., Park B.K. Prevalence and genetic characterization of canine parvoviruses in Korea. Virus Genes. 2008; 36: 127-33. doi: 10.1007/s11262-007-0189-6.
3. GenBank. Canine parvovirus. URL: https://www.ncbi.nlm.nih.gov/nuccore/? term=Canine+parvovirus (Дата обращения: 11.10.2023).
4. Wang H.C., Chen W.D., Lin S.L., Chan J.P., Wong M.L. Phylogenetic analysis of canine parvovirus VP2 gene in Taiwan. Virus Genes. 2005; 31: 171-4. doi: 10.1007/s11262-005-1791-0.
5. Mohan Raj J., Mukhopadhyay H.K., Thanislass J., Antony P.X., Pillai R.M. Isolation, molecular characterization and phylogenetic analysis of canine parvovirus. Infect Genet Evol. 2010; 10: 1237-41. doi: 10.1016/j.meegid.2010.08.005.
6. Phromnoi S., Sirinarumitr K., Sirinarumitr T. Sequence analysis of VP2 gene of canine parvovirus isolates in Thailand. Virus Genes. 2010; 41: 23-9. doi: 10.1007/s11262-010-0475-6.
7. Hao X., Li Y., Xiao X., Chen B., Zhou P., Li S. The Changes in Canine Parvovirus Variants over the Years. Int J Mol Sci. 2022 Sep 29; 23 (19): 11540. doi: 10.3390/ijms231911540. PMID: 36232841; PMCID: PMC9569878.
8. Decaro N., Elia G., Desario C., Roperto S., Martella V., Campolo M., Lorusso A., Cavalli A., Buonavoglia C. A minor groove binder probe real-time PCR assay for discrimination between type 2-based vaccines and field strains of canine parvovirus. J Virol Methods. 2006 Sep; 136 (1-2): 65-70.
9. Miranda C., Thompson G. Canine parvovirus: the worldwide occurrence of antigenic variants. J Gen Virol. 2016 Sep; 97 (9): 2043-2057. doi: 10.1099/jgv.0.000540. Epub 2016 Jul 7. PMID: 27389721.
10. Decaro N.., Buonavoglia C. Canine parvovirus - a review of epidemiological and diagnostic aspects, with emphasis on type 2c. Vet Microbiol. 2012 Feb 24; 155 (1): 1-12. doi: 10.1016/j.vetmic.2011.09.007. Epub 2011 Sep 12. PMID: 21962408; PMCID: PMC7173204.
11. Erali M., Voelkerding K.V., Wittwer C.T. High resolution melting applications for clinical laboratory medicine. Exp Mol Pathol. 2008 Aug; 85 (1): 50-8. doi: 10.1016/j.yexmp.2008.03.012. Epub 2008 Apr 13. PMID: 18502416; PMCID: PMC2606052.
12. Lung O., Fisher M., Beeston A., Hughes K.B., Clavijo A., Goolia M., Pasick J., Mauro W., Deregt D. Multiplex RT-PCR detection and microarray typing of vesicular disease viruses. J Virol Methods. 2011 Aug; 175 (2): 236-45. doi: 10.1016/j.jviromet.2011.05.023. Epub 2011 May 19. PMID: 21620898.
13. Zhou L., Wang L., Palais R., Pryor R., Wittwer C.T. High-resolution DNA melting analysis for simultaneous mutation scanning and genotyping in solution. Clin Chem. 2005 Oct; 51 (10): 1770-7. doi: 10.1373/clinchem.2005.054924. PMID: 16189378.
14. Shi X., Liu X., Wang Q., Das A., Ma G., Xu L., Sun Q., Peddireddi L., Jia W., Liu Y., Anderson G., Bai J., Shi J. A multiplex real-time PCR panel assay for simultaneous detection and differentiation of 12 common swine viruses. J Virol Methods. 2016 Oct; 236: 258-265. doi: 10.1016/j.jviromet.2016.08.005. Epub 2016 Aug 6. PMID: 27506582; PMCID: PMC7119729.
15. Ambagala A., Fisher M., Goolia M., Nfon C., Furukawa-Stoffer T., Ortega Polo R., Lung O. Field-Deployable Reverse Transcription-Insulated Isothermal PCR (RT-iiPCR) Assay for Rapid and Sensitive Detection of Foot-and-Mouth Disease Virus. Transbound Emerg Dis. 2017 Oct; 64 (5): 1610-1623. doi: 10.1111/tbed.12554. Epub 2016 Sep 3. PMID: 27589902; PMCID: PMC7169878.
16. Reid S.M., Pierce K.E., Mistry R., Bharya S., Dukes J.P., Volpe C., Wangh L.J., King D.P. Pan-serotypic detection of foot-and-mouth disease virus by RT linear-after-the-exponential PCR. Mol Cell Probes. 2010 Oct; 24 (5): 250-5. doi: 10.1016/j.mcp.2010.04.004. Epub 2010 Apr 28. PMID: 20433917.
17. Liu Y.L., Ding Y.Z., Dai J.F., Ma B., He J.J., Ma W.M., Lv J.L., Ma X.Y., Ou Y.W., Wang J., Liu Y.S., Chang H.Y., Wang Y.L., Zhang Q., Liu X.T., Zhang Y.G., Zhang J. Development of a New RT-PCR with Multiple Primers for Detecting Southern African Territories Foot-and-mouth Disease Viruses. J Vet Res. 2018 Dec 31; 62 (4): 431-437. doi: 10.2478/jvetres-2018-0064. PMID: 30729199; PMCID: PMC6364153.
18. Estrada-Rivadeneyra D. Sanger sequencing. FEBS J. 2017 Dec; 284 (24): 4174. doi: 10.1111/febs.14319. Epub 2017 Nov 24. PMID: 29171728.
19. Флуоресцентные полимерные красители SuperNova. URL: https://www.beckman.co.il/ru/reagents/coulter-flow-cytometry/supernova-fluorescent-polymer-dyes. (Дата обращения: 14.11.2023).
Таблица 1
Дизайн оригинальных специфичных олигонуклеотидных праймеров, используемых при разработке предлагаемого изобретения
Таблица 2
Физические, термодинамические параметры и температура плавления олигонуклеотидных праймеров для дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа ПВЭ с применением тандемного флуоресцирующего красителя SN v428 (предлагаемое изобретение)
Примечание: термодинамические параметры разработанных олигонуклеотидных праймеров рассчитаны при условии: концентрация NaCl 1 М,
температура 25°С,
водородный показатель (рН) 7,0.
Таблица 3
Временные и температурные режимы обратной транскрипции и ПЦР в режиме реального времени дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов с ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428 (предлагаемое изобретение)
Примечание: градиент температур составлен на этапе плавления с шагом 0,1°С,
* - детекция сигнала.
Таблица 4
Средние показания температур плавления, при которых достигались пиковые значения для исследуемых ампликонов штаммов и изолятов возбудителя парвовирусного энтерита (предлагаемое изобретение)
(n = 32, p < 0,001)
Примечание: анализ проводили на приборе CFX-96 (Bio-Rad, США), отражены средние значения пиков температур и среднеквадратичное отклонение.
Таблица 5
Исследование специфичности способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428 (предлагаемое изобретение)
(n = 3)
Примечание: «+» - наличие характерного графика плавления ампликонов,
«-» - отсутствие графика по результатам реакции амплификации с анализом пиков температур плавления.
Таблица 6
Пределы диагностических возможностей способа дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов с помощью высокочувствительного HRM-анализа ПВЭ с применением тандемного флуоресцирующего красителя SN v428 (предлагаемое изобретение)
Примечание: DSe - диагностическая чувствительность,
DSp - диагностическая специфичность,
k-критерий - индекс Каппа Коэна,
DAc - общая точность.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="CPV Archi
diff.xml" softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2024-07-30">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-12-05</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>559</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-12-05</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБУ "Федеральный центр охраны
здоровья животных" (ФГБУ "ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin> Federal State-Financed Institution Federal
Centre for Animal Health (FGBI ARRIAH)</ApplicantNameLatin>
<InventorName languageCode="ru">Доронин Максим
Игоревич</InventorName>
<InventorNameLatin>Doronin Maksim Igorevich </InventorNameLatin>
<InventionTitle languageCode="ru">Способ дифференциации вакцинного
штамма «Арчи» возбудителя парвовирусного энтерита собак от других
штаммов и полевых изолятов с высокочувствительного HRM-анализа с
применением тандемного флуоресцирующего красителя SN
v428</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>4260</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..4260</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>CPV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtctggcaaccagtatactgaggaagttatggagggagtaaattggt
taaagaaacatgcagaaaatgaagcattttcgtttgtttttaaatgtgacaacgtccaactaaatggaaa
ggatgttcgctggaacaactataccaaaccaattcaaaatgaagagctaacatctttaattagaggagca
caaacagcaatggatcaaaccgaagaagaagaaatggactgggaatcggaagttgatagtctcgccaaaa
agcaagtacaaacttttgatgcattaattaaaaaatgtctttttgaagtctttgtttctaaaaatataga
accaaatgaatgtgtttggtttattcaacatgaatggggaaaagatcaaggctggcattgtcatgtttta
cttcatagtaagaacttacaacaagcaactggtaaatggctacgcagacaaatgaatatgtattggagta
gatggttagtgactctttgttcggtaaacttaacaccaactgaaaagattaagctcagagaaattgcaga
agatagtgaatgggtgactatattaacatacagacataagcaaacaaaaaaagactatgttaaaatggtt
cattttggaaatatgatagcatattactttttaacaaagaaaaaaattgtccacatgacaaaagaaagtg
gctattttttaagtactgattctggttggaaatttaactttatgaagtatcaagacagacaaattgtcag
tacactttacactgaacaaatgaaaccagaaaccgttgaaaccacagtgacgacagcacaggaaacaaag
cgcgggagaattcaaactaaaaaggaagtgtcaatcaaatgtactttgcgggacttggttagtaaaagag
taacatcacctgaagactggatgatgttacaaccagatagttatattgaaatgatggcacaaccaggagg
tgaaaatcttttaaaaaatacacttgaaatttgtactttgactttagcaagaacaaaaacagcatttgaa
ttaatacttgaaaaagcagataagactaaactaactaactttgatcttgcaaattctagaacatgtcaaa
tttttaggatgcacggatggaattggattaaagtttgtcatgctatagcatgtgttttaaatagacaagg
tggtaaaagaaatacagttctttttcatggaccagcaagtacaggaaaatctatcattgctcaagccata
gcacaagctgtgggtaatgttggttgttataatgcagcaaatgtaaattttccatttaatgactgtacca
ataaaaatttaatttggattgaagaagctggtaactttggtcaacaagttaatcaatttaaagcaatctg
ttctggacaaacaattagaattgatcaaaaaggtaaaggaagtaagcaaattgaaccaactccagtaatt
atgacaactaatgaaaatataacaattgtgagaattggatgtgaagaaagacctgaacatacacaaccaa
taagagacagaatgttgaacattaagttagtatgtaagcttccaggagactttggtttggttgataaaga
agaatggcctttaatatgtgcatggttagttaaacatggttttgaatcaaccatggctaactatacacat
cattggggaaaagtaccagaatgggatgaaaactgggcggagcctaaaatacaagaaggtataaattcac
caggttgcaaagacttagagacacaagcggcaagcaatcctcagagtcaagaccaagttccaactcctct
gactccggacgtagtggaccttgcactggaaccgtggagtactccagatacgcctattgcagaaactgca
aatcaacaatcaaaccaacttggcgttactcgcaaagacgtgcaagcgagtccgacgtggtccgaaatag
aggcagacctgagagccatctttacttctgaacaattggaagaagattttcgagacgacttggattaagg
tacgatggcacctccggcaaagagagccaggagaggtaagggtgtgttagtaaagtggggggaggggaaa
gatttaataacttaactaagtatgtgtttttttataggacttgtgcctccaggttataaatatcttgggc
ctgggaacagtcttgaccaaggagaaccaactaacccttctgacgccgctgcaaaagaacacgacgaagc
ttacgctgcttatcttcgctctggtaaaaacccatacttatatttctcgccagcagatcaacgctttata
gatcaaactaaggacgctaaagattggggggggaaaataggacattatttttttagagctaaaaaggcaa
ttgctccagtattaactgatacaccagatcatccatcaacatcaagaccaacaaaaccaactaaaagaag
taaaccaccacctcatattttcatcaatcttgcaaaaaaaaaaaaagccggtgcaggacaagtaaaaaga
gacaatcttgcaccaatgagtgatggagcagttcaaccagacggtggtcaacctgctgtcagaaatgaaa
gagctacaggatctgggaacgggtctggaggcggggttggtggtggttctaggggtgtggggatttctac
gggtactttcaataatcagacggaatttaaatttttggaaaacggatgggtggaaatcacagcaaactca
agcagacttgtacatttaaatatgccagaaagtgaaaattatagaagagtggttgtaaataatttggata
aaactgcagttaacggaaacatggctttagatgatactcatgcacaaattgtaacaccttggtcattggt
tgatgcaaatgcttggggagtttggtttaatccaggagattggcaactaattgttaatactatgagtgag
ttgcatttagtcagttttgaacaagaaatttttaatgttgttttaaagactgtttcagaatctgctactc
agccaccaactaaagtttataataatgatttaactgcatcattgatggttgcattagatagcaataatac
tatgccatttactccagcagctatgagatctgagacattgggtttttatccatggaaaccaaccatacca
actccatggagatattattttcaatgggatagaacattaataccatctcatactggaactagtggcacac
caacaaatatataccatggtacagatccagatgatgttcaattttatactattgaaaattctgtgccagt
acacttactaagaacaggtgatgaatttgctacaggaacatttttttttgattgtaaaccatgtagacta
acacatacatggcaaacaaatagagcattgggcttaccaccatttctaaattctttgcctcaagctgaag
gaggtactaactttggttatataggagttcaacaagataaaagacgtggtgtaactcaaatgggaaatac
aaactatattactgaagctactattatgagaccagctgaggttggttatagtgcaccatattattctttt
gaggcgtctacacaagggccatttaaaacacctattgcagcaggacgggggggagcgcaaacagatgaaa
atcaagcagcagatggtgatccaagatatgcatttggtagacaacatggtcaaaaaactaccacaacagg
agaaacacctgagagatttacatatatagcacatcaagatacaggaagatatccagaaggagattggatt
caaaatattaactttaaccttcctgtaacaaatgataatgtattgctaccaacagatccaattggaggta
aaacaggaattaactatactaatatatttaatacttatggtcctttaactgcattaaataatgtaccacc
agtttatccaaatggtcaaatttgggataaagaatttgatactgacttaaaaccaagacttcatgtaaat
gcaccatttgtttgtcaaaataattgtcctggtcaattatttgtaaaagttgcgcctaatttaacaaatg
aatatgatcctgatgcatctgctaatatgtcaagaattgtaacttactcagatttttggtggaaaggtaa
attagtatttaaagctaaactaagagcctctcatacttggaatccaattcaacaaatgagtattaatgta
gataaccaatttaactatgtaccaagtaatattggaggtatgaagattgtatatgaaaaatctcaactag
cacctagaaaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1420</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1420</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>CPV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSGNQYTEEVMEGVNWLKKHAENEAFSFVFKCDNVQLNGKDVRWNNYTK
PIQNEELTSLIRGAQTAMDQTEEEEMDWESEVDSLAKKQVQTFDALIKKCLFEVFVSKNIEPNECVWFIQ
HEWGKDQGWHCHVLLHSKNLQQATGKWLRRQMNMYWSRWLVTLCSVNLTPTEKIKLREIAEDSEWVTILT
YRHKQTKKDYVKMVHFGNMIAYYFLTKKKIVHMTKESGYFLSTDSGWKFNFMKYQDRQIVSTLYTEQMKP
ETVETTVTTAQETKRGRIQTKKEVSIKCTLRDLVSKRVTSPEDWMMLQPDSYIEMMAQPGGENLLKNTLE
ICTLTLARTKTAFELILEKADKTKLTNFDLANSRTCQIFRMHGWNWIKVCHAIACVLNRQGGKRNTVLFH
GPASTGKSIIAQAIAQAVGNVGCYNAANVNFPFNDCTNKNLIWIEEAGNFGQQVNQFKAICSGQTIRIDQ
KGKGSKQIEPTPVIMTTNENITIVRIGCEERPEHTQPIRDRMLNIKLVCKLPGDFGLVDKEEWPLICAWL
VKHGFESTMANYTHHWGKVPEWDENWAEPKIQEGINSPGCKDLETQAASNPQSQDQVPTPLTPDVVDLAL
EPWSTPDTPIAETANQQSNQLGVTRKDVQASPTWSEIEADLRAIFTSEQLEEDFRDDLDGTMAPPAKRAR
RGKGVLVKWGEGKDLITLSMCFFIGLVPPGYKYLGPGNSLDQGEPTNPSDAAAKEHDEAYAAYLRSGKNP
YLYFSPADQRFIDQTKDAKDWGGKIGHYFFRAKKAIAPVLTDTPDHPSTSRPTKPTKRSKPPPHIFINLA
KKKKAGAGQVKRDNLAPMSDGAVQPDGGQPAVRNERATGSGNGSGGGVGGGSRGVGISTGTFNNQTEFKF
LENGWVEITANSSRLVHLNMPESENYRRVVVNNLDKTAVNGNMALDDTHAQIVTPWSLVDANAWGVWFNP
GDWQLIVNTMSELHLVSFEQEIFNVVLKTVSESATQPPTKVYNNDLTASLMVALDSNNTMPFTPAAMRSE
TLGFYPWKPTIPTPWRYYFQWDRTLIPSHTGTSGTPTNIYHGTDPDDVQFYTIENSVPVHLLRTGDEFAT
GTFFFDCKPCRLTHTWQTNRALGLPPFLNSLPQAEGGTNFGYIGVQQDKRRGVTQMGNTNYITEATIMRP
AEVGYSAPYYSFEASTQGPFKTPIAAGRGGAQTDENQAADGDPRYAFGRQHGQKTTTTGETPERFTYIAH
QDTGRYPEGDWIQNINFNLPVTNDNVLLPTDPIGGKTGINYTNIFNTYGPLTALNNVPPVYPNGQIWDKE
FDTDLKPRLHVNAPFVCQNNCPGQLFVKVAPNLTNEYDPDASANMSRIVTYSDFWWKGKLVFKAKLRASH
TWNPIQQMSINVDNQFNYVPSNIGGMKIVYEKSQLAPRKLY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>CPV </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggagcagttcaaccagacggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>CPV </INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgatttccacccatccgttttcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

Изобретение относится к ветеринарной вирусологии, к средствам молекулярной диагностики, а именно к дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ с помощью высокочувствительного HRM-анализа с применением тандемного флуоресцирующего красителя SN v428. Основным преимуществом предлагаемого изобретения является возможность проводить дифференциальный анализ за короткий промежуток времени - не более 2 ч. Разработанный способ впервые применили для решения данной задачи. В целевом участке ДНК гена VP1 возбудителя парвовирусного энтерита имеются две нуклеотидные мутации, уникальные для вакцинного штамма «Арчи», что позволило разработать высокоспецифичные оригинальные олигонуклеотидные праймеры CPV-F2524 и CPV-R2676, рассчитанные для амплификации целевого участка ДНК гена VP1 возбудителя парвовирусного энтерита размером 176 п.н. и анализа максимальных значений температур плавления ПЦР-продуктов с применением тандемного флуоресцирующего красителя SN v428. Разработанный способ характеризуется 100%-ной аналитической специфичностью. В 95%-ном доверительном интервале диагностическая чувствительность для данного способа составляет 99,05-100,00%, диагностическая специфичность - 98,62-100,0%. 1 з.п. ф-лы, 4 ил., 6 табл., 3 пр.
1. Способ дифференциации вакцинного штамма «Арчи» возбудителя парвовирусного энтерита собак от других штаммов и полевых изолятов ПВЭ, включающий высокочувствительный HRM анализ с применением тандемного флуоресцирующего красителя SN v428 и олигонуклеотидных праймеров CPV-F2524 с дизайном 3'-GGAGCAGTTCAACCAGACGGT-5' и CPV-R2676 с дизайном 3'-GTGATTTCCACCCATCCGTTTTCC-5' для амплификации целевого участка ДНК гена VP1 возбудителя парвовирусного энтерита в диапазоне 2524…2699 п.н., при проведении которого определено, что для вакцинного штамма «Арчи» CPV температура плавления ампликонов имеет значение 73,86±0,04°С.
2. Способ по п. 1, где время проведения анализа сокращается до 2 ч.
| СПОСОБ ИНДИКАЦИИ И ДИФФЕРЕНЦИАЦИИ ВАКЦИННЫХ ШТАММОВ И ПОЛЕВЫХ ИЗОЛЯТОВ ВИРУСА КЛАССИЧЕСКОЙ ЧУМЫ СВИНЕЙ | 2001 |
|
RU2196992C2 |
| Способ дифференциации генома вакцинного штамма НИСХИ от полевых изолятов вируса оспы овец методом полимеразной цепной реакции в режиме реального времени с анализом пиков высокого разрешения | 2022 |
|
RU2786213C1 |
| Штамм "Арчи" вируса Carnivore protoparvovirus 1 парвовирусного энтерита собак для изготовления биопрепаратов для диагностики и специфической профилактики парвовирусного энтерита собак | 2023 |
|
RU2817267C1 |
Авторы
Даты
2025-03-05—Публикация
2024-07-31—Подача