ОБЛАСТЬ ТЕХНИКИ, К КОТОРОЙ ОТНОСИТСЯ ИЗОБРЕТЕНИЕ
Настоящее изобретение относится к области сельского хозяйства. В частности, настоящее изобретение относится к белкам CenH3 и полинуклеотидам, их кодирующим, способам получения гаплоидных, а также последующих удвоенных гаплоидных растений и получаемым из них растениям и семенам, в частности, у видов пшеницы.
ИСПРАШИВАНИЕ ПРИОРИТЕТА
Настоящая заявка испрашивает приоритет согласно Парижской конвенции по PCT/CN 2018/110063, поданной 12 октября 2018 г., которая включена в данный документ во всей своей полноте.
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
К настоящей заявке прилагается перечень последовательностей под названием 81696WOPCT_ST25.txt, созданный 30 сентября 2019 г., который имеет размер примерно 112 килобайтов. Этот перечень последовательностей включен в данный документ посредством ссылки во всей своей полноте. Данный перечень последовательностей подается вместе с настоящей заявкой посредством EFS-Web и соответствует §1.824(а)(2)-(6) и (b) раздела 37 C.F.R.
ПРЕДПОСЫЛКИ ИЗОБРЕТЕНИЯ
Высокая степень гетерозиготности селекционного материала может сделать селекцию растений и отбор полезных признаков очень времязатратным процессом. Широкомасштабный скрининг популяции, даже с использованием самых современных инструментов молекулярной селекции, является одновременно и трудоемким, и дорогостоящим. Создание гаплоидных растений с последующим химическим или спонтанным удвоением генома доказало свою эффективность в качестве пути решения проблемы высокой гетерозиготности и ускорения процесса селекции. Такая технология также называется системой получения удвоенных гаплоидов. Применение системы получения удвоенных гаплоидов позволило селекционерам достичь гомозиготности во всех локусах за одно поколение с помощью полногеномного удвоения. Это позволяет фактически исключить необходимость самоопыления или возвратного скрещивания, при которых обычно потребовалось бы по меньшей мере 7 поколений самоопыления или возвратного скрещивания для снижения гетерозиготности до приемлемого уровня.
Гаплоидные растения могут быть получены в соответствии с разными методиками. Например, гаплоидные растения в некоторых сельскохозяйственных культурах могут быть получены путем применения способа, называемого культивированием микроспор. Однако данный способ является дорогостоящим, времязатратным и работает не для всех сельскохозяйственных культур. У некоторых видов сельскохозяйственных культур (удвоенные) гаплоидные растения могут быть получены путем партеногенеза яйцеклетки или путем элиминации одного из родительских геномов. Однако такие способы не являются оптимальными, так как они работают только у некоторых отдельных видов сельскохозяйственных культур и характеризуются довольно низкими показателями выхода (удвоенных) гаплоидных растений.
В WO 2011/044132 раскрыт способ получения гаплоидных растений, заключающийся в инактивации, или изменении, или осуществлении нокаута центромероспецифического белка Н3 (CenH3) у растения. На первой стадии способ заключается в элиминации или осуществлении нокдауна эндогенного гена CenH3 у растения. На второй стадии в растение вводится кассета экспрессии, кодирующая мутантный или измененный белок CenH3. Мутантный или измененный белок CenH3 образуется путем слияния N-концевого домена Н3.3, необязательно меченного GFP, с эндогенным доменом гистоновой складки CenH3. Такая методика также известна как "обмен хвостами с GFP" или "обмен хвостами" (обзор которой также приведен в Britt and Kuppu, Front Plant Sci. 2016; 7: 357). Скрещивание растения, несущего такой "обмененный хвост", с растением дикого типа (т.е. имеющим функциональный эндогенный белок CenH3 без "обмененного хвоста"), вызывает однородительскую элиминацию генома, что, в свою очередь, приводит к получению гаплоидного растения. Некоторая индукция гаплоидии, хоть и реже встречающаяся, также была обнаружена при добавлении GFP к N-концу эндогенного CenH3 (без "обмененного хвоста"). Однако данная методика не идеальна, так как она является трудоемкой, времязатратной и требует получения трансгенного растения.
В WO 2014/110274 описан способ получения гаплоидных растений, заключающийся в скрещивании первого растения, экспрессирующего эндогенный ген CenH3, со вторым растением, называемым растением, являющимся индуктором гаплоидии, имеющим геном из по меньшей мере двух видов, где большая часть генома получена из первого вида, и геном содержит гетерологичную геномную область из второго вида, где гетерологичная геномная область кодирует полипептид CenH3, отличный от CenH3 первого вида (также описано в Maheshwari et al, PLoS Genet. 2015 Jan 26; 11(1):е1004970). Однако данная методика не является оптимальной, так как она страдает от тех же недостатков, что и вышеупомянутые - она является трудоемкой, времязатратной и требует получения трансгенного растения. Кроме того, данный способ сопряжен с низким выходом гаплоидных растений.
Другие способы заключаются во введении одной или нескольких точечных мутаций, приводящих к замене одной аминокислоты в С-концевом домене гистоновой складки белка CenH3 или в гене CenH3, кодирующем белок CenH3. О примерах таких мутаций в С-концевом домене гистоновой складки белка CenH3 сообщалось в Karimi-Ashtiyani et al (2015) Proc Natl Acad Sci USA. 2015 Sep 8; 112(36):11211-16; Kuppu, et al. (2015) PLoS Genet. 2015 Sep 9; 11(9):е1005494. Однако успех таких способов затмевается тем, что было обнаружено, что некоторые из таких мутаций, но не все из них, являются достаточными для того, чтобы индуцировать однородительскую элиминацию генома после скрещивания с растением дикого типа для получения гаплоидного растения.
Пшеница (Triticum aestivum) является особенно сложным организмом для редактирования или осуществления мутаций ее генов, так как она представляет собой гексаплоидный организм. Эволюционировавшая на протяжении нескольких тысяч лет и подвергшаяся нескольким межвидовым скрещиваниям с предковыми видами пшеницы, Triticum aestivum содержит три генома: А (вероятно, от Т. топососсит или пшеницы-однозернянки), В (вероятно, от Т. searsii) и D (вероятно, от Т. tauschii). Каждый геном имеет по 7 хромосом. Triticum aestivum имеет две копии каждого из геномов, т.е., АА ВВ DD; таким образом, она имеет всего 42 хромосомы (6 полных геномов по 7 хромосом в каждом). См. в целом The Evolution of Wheat по адресу www.cerealsdb.uk.net/cerealgenomics/WheatBP/Documents/DOC_Evolution.php, последний доступ 10 июля 2019 г. Кроме того, редактирование или мутация в одной из копий одного гена у Triticum aestivum может не проявиться в виде наблюдаемых эффектов, так как дополнительные 5 его копий, вероятно, будут компенсировать изменения в мутантной копии. Для того чтобы на самом деле наблюдать эффект нокаут-мутации, необходимо было бы подвергнуть мутации все 6 копий.
Следовательно, остается весьма труднодостижимым определение того, какая(какие) мутация(мутации) или модификация(модификации) в белке CenH3 или гене CenH3, кодирующем белок CenH3, способны индуцировать или являются достаточными для того, чтобы индуцировать однородительскую элиминацию генома для получения гаплоидных растений. Таким образом, в данной области техники остается необходимость в альтернативных или улучшенных способах, обеспечивающих возможность эффективного получения гаплоидных растений (например, менее трудоемких, менее времязатратных, менее дорогостоящих и/или не обязательно требующих получения трансгенного растения), которые впоследствии можно подвергнуть удвоению с получением удвоенных гаплоидных растений. При использовании систем получения удвоенных гаплоидов гомозиготность может быть достигнута за одно поколение.
КРАТКОЕ ОПИСАНИЕ
Для удовлетворения этой потребности один вариант осуществления настоящего изобретения представляет собой растение пшеницы, содержащее по меньшей мере геном А, геном В и геном D, где геном В содержит нокаут-мутацию в гене CENH3, и где необязательно геном D содержит нокаут-мутацию в гене CENH3, и где дополнительно геном А содержит мутантный ген CENH3, содержащий по меньшей мере одну нокдаун-мутацию в 5'-сайте сплайсинга интрона. В одном аспекте нокдаун-мутация представляет собой мутацию по типу восстановления сдвинутой рамки считывания или мутацию по типу большой делеции. В другом варианте осуществления растение пшеницы является гомозиготным по нокаут-мутации в гене CENH3 в геноме В. В альтернативном варианте осуществления растение пшеницы является биаллельным по нокаут-мутации в гене CENH3 в геноме В. В другом варианте осуществления растение пшеницы является гомозиготным по нокаут-мутации в гене CENH3 в геноме D. В альтернативном варианте осуществления растение пшеницы является биаллельным по нокаут-мутации в гене CENH3 в геноме D. В еще одном варианте осуществления растение пшеницы является гомозиготным, биаллельным или характеризуется комбинацией этих свойств по нокаут-мутации в гене CENH3 в геноме В и геноме D. В другом варианте осуществления растение пшеницы является гомозиготным по мутации по типу восстановления сдвинутой рамки считывания в CENH3; или оно является гетерозиготным по мутации по типу восстановления сдвинутой рамки считывания в CENH3; или оно является биаллельным по мутации по типу восстановления сдвинутой рамки считывания в CENH3.
Другим аспектом настоящего изобретения является способ получения растения пшеницы, индуцирующего гаплоидию, при этом способ включает: (а) получение по меньшей мере клетки растения пшеницы, содержащей по меньшей мере три генома; (b) осуществление мутаций в двух из трех геномов с получением гомозиготных нокаут-мутаций в гене CENH3; (с) осуществление мутаций в третьем геноме с получением гомозиготной нокдаун-мутации в гене CENH3 и (d) получение в результате этого растения пшеницы, содержащего гомозиготные нокаут-мутации в гене CENH3 в двух из трех геномов и дополнительно содержащего гомозиготную нокдаун-мутацию в гене CENH3 в третьем геноме; вследствие чего растение пшеницы, полученное на стадии (d), производит гаплоидное потомство при скрещивании с растением пшеницы дикого типа. В одном варианте осуществления три генома включают в себя геном А, геном В и геном D. В другом варианте осуществления нокаут-мутации в гене CENH3 имеют место в геномах В и D. В еще одном варианте осуществления нокдаун-мутация в гене CENH3 имеет место в геноме А. В одном аспекте нокдаун-мутации в гене CENH3 в геноме А представляют собой мутации по типу восстановления сдвинутой рамки считывания. В другом аспекте мутации по типу восстановления сдвинутой рамки считывания выбраны из группы, состоящей из SEQ ID NO: 56, последовательности нуклеиновой кислоты, на 70% идентичной SEQ ID NO: 56, SEQ ID NO: 63, последовательности нуклеиновой кислоты, на 70% идентичной SEQ ID NO: 63, SEQ ID NO: 69 и последовательности нуклеиновой кислоты, на 70% идентичной SEQ ID NO: 69.
Другим аспектом настоящего изобретения является растение пшеницы, содержащее мутантный ген CENH3, содержащий по меньшей мере одну делеционную мутацию в N-концевом домене, которая приводит к сдвигу рамки считывания, восстановлению сдвинутой рамки считывания или большой делеции. Еще одним аспектом является растение пшеницы, содержащее мутантный ген CENH3, содержащий по меньшей мере одну инсерционную мутацию в N-концевом домене, которая приводит к сдвигу рамки считывания, восстановлению сдвинутой рамки считывания или большой делеции.
Другим аспектом настоящего изобретения является способ получения сконструированного восстановления сдвинутой рамки считывания в гене в клетке, включающий: (а) приведение генома в контакт с сайт-направленной нуклеазой ("SDN") и по меньшей мере двумя направляющими нуклеиновыми кислотами, где по меньшей мере две направляющие нуклеиновые кислоты нацеливаются на по меньшей мере две последовательности-мишени в гене; (b) предоставление для SDN возможности разрезания гена в по меньшей мере двух последовательностях-мишенях с утратой таким образом вставочной последовательности между по меньшей мере двумя последовательностями-мишенями; и обеспечение возможности прохождения эндогенной репарации ДНК; вследствие чего эндогенная репарация ДНК приводит к получению гена со сконструированным восстановлением сдвинутой рамки считывания. В одном варианте осуществления утрачиваемая вставочная последовательность из стадии (b) содержит (N) пар оснований, где (N) является числом, кратным 3.
Еще одним аспектом настоящего изобретения является способ получения гаплоидного растения пшеницы, включающий: (а) получение растения пшеницы; (b) скрещивание растения пшеницы с растением пшеницы, содержащим мутантный ген CENH3; и (с) отбор потомка, полученного в результате стадии скрещивания; где потомок представляет собой гаплоидное растение пшеницы. В одном варианте осуществления растение пшеницы из стадии (а) представляет собой отцовское родительское растение. В другом варианте осуществления растение пшеницы из стадии (а) представляет собой материнское родительское растение. В другом варианте осуществления способ включает дополнительную стадию превращения растения пшеницы, являющегося потомком, в удвоенное гаплоидное растение пшеницы.
Другим аспектом настоящего изобретения является получение растения пшеницы, содержащего мутантный аллель CENH3, содержащий последовательность нуклеиновой кислоты, на по меньшей мере 70% идентичную последовательности, выбранной из группы, состоящей из SEQ ID NO: 53-73, где мутация представляет собой мутацию по типу восстановления сдвинутой рамки считывания, и где растение пшеницы образует гаплоидное потомство при скрещивании с диплоидным растением пшеницы дикого типа. В одном варианте осуществления растение пшеницы содержит по меньшей мере одну копию мутантного аллеля CENH3; в другом варианте осуществления растение пшеницы содержит по меньшей мере две копии мутантного аллеля CENH3; в еще одном варианте осуществления растение пшеницы содержит по меньшей мере три копии мутантного аллеля CENH3. В одном варианте осуществления мутантный аллель CENH3 содержит последовательность нуклеиновой кислоты, на 80, 90, 95 или 100% идентичную SEQ ID NO: 53-73.

КРАТКОЕ ОПИСАНИЕ ФИГУР
На фигуре 1 показана структура гена TaCenH3α и относительные местоположения gRNA. Экзоны пронумерованы и представлены закрашенными прямоугольниками. Интроны представлены тонкими линиями. Длина и тех, и других представлена их шириной.
ОПРЕДЕЛЕНИЯ
Настоящее изобретение не ограничено конкретной методикой, протоколами, линиями клеток, видами или родами растений, конструкциями и реагентами, описанными в данном документе. Терминология, используемая в данном документе, представлена исключительно с целью описания конкретных вариантов осуществления, а не предназначена для ограничения объема настоящего изобретения, который будет ограничиваться лишь прилагаемой формулой изобретения. Необходимо отметить, что, как используется в данном документе и в прилагаемой формуле изобретения, формы единственного числа включают ссылки на множественное число, если в контексте явно не указано иное. Таким образом, например, ссылка на "растение" является ссылкой на одно или несколько растений и включает их эквиваленты, известные специалистам в данной области, и т.д. Используемое в данном документе слово "или" означает любой элемент из конкретного перечня, а также включает любую комбинацию элементов из такого перечня (то есть включает также "и").
Используемый в данном документе термин "приблизительно" означает примерно, ориентировочно, около или в районе. Если термин "приблизительно" используется в сочетании с числовым диапазоном, он модифицирует этот диапазон, расширяя границы выше и ниже указанных числовых значений. В целом, используемый в данном документе термин "приблизительно" модифицирует числовое значение путем его варьирования выше и ниже указанного значения на 20 процентов, предпочтительно на 10 процентов выше или ниже (больше или меньше). Что касается температуры, термин "приблизительно" означает ±1°С, предпочтительно ±0,5°С. Если термин "приблизительно" используется в контексте настоящего изобретения (например, в комбинациях со значениями температуры или молекулярной массы), то предпочтительным является точное значение (т.е. без "приблизительно").
Используемый в данном документе термин "амплифицированный" означает конструирование нескольких копий молекулы нуклеиновой кислоты или нескольких копий, комплементарных молекуле нуклеиновой кислоты, с использованием по меньшей мере одной из молекул нуклеиновых кислот в качестве матрицы. Системы амплификации включают систему полимеразной цепной реакции (ПЦР), систему лигазной цепной реакции (LCR), систему амплификации, основанной на последовательности нуклеиновых кислот (NASBA, Cangene, Миссиссога, Онтарио), систему на основе Q-бета-репликазы, систему амплификации, основанной на транскрипции (TAS), и амплификацию с замещением нитей (SDA). См., например, Diagnostic Molecular Microbiology: Principles and Applications, PERSING et al., Ed., American Society for Microbiology, Washington, D.C. (1993). Продукт амплификации называется "ампликоном".
Термин "биаллельный" относится к паре генов, которая не является ни гомозиготной (АА или аа), ни гетерозиготной (Аа). Вернее говоря, оба гена в паре были отредактированы, но не идентичным образом. Например, пара генов CenH3 в хромосоме А в настоящем изобретении может содержать одну мутацию по типу RFS в одном аллеле, приводящую к нокдауну гена при экспрессии, тогда как другой аллель содержит нокаут-мутацию. Это может быть указано символами как "А*а" и свидетельствует о биаллельной мутации.
Термин "специфичная последовательность ДНК" указывает на полинуклеотидную последовательность, обладающую гомологией нуклеотидной последовательности, составляющей более 80%, предпочтительно более 85%, более предпочтительно более 90%, еще более предпочтительно более 95%, и еще более предпочтительно более 97%, наиболее предпочтительно более 99%, с другой названной последовательностью.
"кДНК" относится к однонитевой или двухнитевой ДНК, которая комплементарна мРНК и получена из нее. Термин "центромероспецифический вариант гистонового белка Н3" ("белок CenH3" или просто "CENH3"), используемый в данном документе, относится к белку, который является членом комплекса кинетохора. Белок CenH3 также известен как белок CENP-A. Комплекс кинетохора располагается на хроматидах там, где нити веретена деления прикрепляются в процессе клеточного деления для отделения сестринских хроматид друг от друга. Белки CenH3 принадлежат к хорошо изученному классу белков, которые представляют собой варианты гистоновых белков Н3. Такие белки являются существенно важными для правильного образования и функционирования кинетохора и содействуют связыванию кинетохора с ДНК. В клетках, которые являются дефицитными по CenH3, наблюдается неспособность к локализации белков кинетохора на хроматидах и демонстрируются серьезные дефекты хромосомного расщепления (т.е. все хромосомы растения, экспрессирующего дефицитный белок CenH3, элиминируются или утрачиваются, что приводит к изменению плоидности соматических клеток (например, к редукции числа хромосомных наборов, как, например, от диплоидного к гаплоидному состоянию)). По этой причине белки CenH3 были предметом интенсивного изучения в отношении их потенциального применения в системе получения удвоенных гаплоидов. Белки CenH3 характеризуются наличием вариабельного хвостового домена (также называемого "N-концевым доменом" или "N-концевым хвостовым доменом") и консервативного домена гистоновой складки (также называемого "С-концевым доменом"), состоящего из трех альфа-спиральных областей, соединенных петлевыми участками. Домен гистоновой складки CenH3 является относительно высококонсервативным среди белков CenH3 у разных видов. Домен гистоновой складки располагается на карбоксильном конце эндогенного белка CenH3. В отличие от домена гистоновой складки, N-концевой хвостовой домен CenH3 является высоковариабельным даже у близкородственных видов.
Термин "полинуклеотид, кодирующий CenH3, имеющий одну или несколько активных мутаций" относится к неэндогенному или эндогенному мутантному полинуклеотиду, кодирующему CenH3, который кодирует белок CenH3, имеющему одну или несколько активных мутаций, которые в случае наличия в растении при отсутствии его эндогенного полинуклеотида, кодирующего CenH3, и/или эндогенного белка CenH3 позволяют растению быть жизнеспособным и позволяют образовывать гаплоидное потомство или потомство с аберрантной плоидностью при скрещивании растения с растением дикого типа. Растение, содержащее полинуклеотид, кодирующий CenH3, имеющий одну или несколько активных мутаций, может называться "модифицированным растением". Процентная доля гаплоидного потомства или потомства с аберрантной плоидностью, которое образуется при скрещивании с растением дикого типа, может, например, составлять по меньшей мере 0,1, 0,5, 1, 5, 10, 20 процентов или больше. Мутация, которая вызывает переход из эндогенного полинуклеотида, кодирующего CenH3, в полинуклеотид, кодирующий CenH3, имеющий одну или несколько активных мутаций, в данном документе называется активной мутацией. Активная мутация применительно к белку CenH3 может приводить, помимо прочего, к сниженной загрузке в центромеры, менее функциональному белку CenH3 и/или сниженной функциональности при разделении хромосом в процессе клеточного деления. Одна или несколько активных мутаций могут быть введены в полинуклеотид, кодирующий CenH3, с помощью любого из нескольких способов, хорошо известных специалисту в данной области, например, с помощью случайного мутагенеза, как, например, индуцированного обработкой семян или растительных клеток химическими соединениями или радиоактивным излучением, целенаправленного мутагенеза, применения эндонуклеаз, путем получения частичных или полных делеций доменов белков или путем слияния с гетерологичными последовательностями.
Посредством нокаута или инактивации эндогенного полинуклеотида, кодирующего CenH3, можно добиться отсутствия эндогенного полинуклеотида, кодирующего CenH3, у растения. В качестве альтернативы, эндогенный полинуклеотид, кодирующий CenH3, может быть модифицирован таким образом, чтобы он кодировал неактивный или нефункциональный белок CenH3.
Модифицированное растение, содержащее полинуклеотид, кодирующий CenH3, имеющий одну или несколько активных мутаций, указанное в данном документе, может быть скрещено с растением дикого типа в качестве родительского растения, образующего пыльцу, либо в качестве родительского растения, образующего семязачатки. В одном варианте осуществления белок CenH3, имеющий одну или несколько активных мутаций, может содержать 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 12, 15, 20 или больше аминокислотных замен относительно эндогенного белка CenH3. В одном варианте осуществления полинуклеотид, кодирующий CenH3, имеющий одну или несколько активных мутаций, обладает 70-, 75-, 80-, 85-, 90-, 95-, 96-, 97-, 98-, 99-, 99,5-процентной идентичностью последовательности с эндогенным полинуклеотидом, кодирующим CenH3, предпочтительно по всей длине. Специалист в данной области будет в состоянии легко удостовериться, содержит или не содержит модифицированное растение, указанное в данном документе, одну или несколько активных мутаций. Например, специалист в данной области может воспользоваться прогностическими инструментами, такими как SIFT (Kumar Р, Henikoff S, Ng PC. (2009) Predicting the effects of coding non-synonymous variants on protein function using the SIFT algorithm. Nat Protoc; 4(7): 1073-81. doi: 10.1038/nprot.2009.86), чтобы сделать предположение о такой активной мутации. Затем в растении могут быть произведены одна или несколько активных мутаций, и должен произойти нокаут экспрессии эндогенного белка CenH3 в растении. Растение может рассматриваться как содержащее одну или несколько активных мутаций, если процентная доля гаплоидного потомства или потомства с аберрантной плоидностью, которое образуется при скрещивании с растением дикого типа, составляет по меньшей мере 0,1, 0,5, 1, 5, 10, 20 процентов или больше.
Скрещивание растения, у которого отсутствует эндогенный полинуклеотид, кодирующий CenH3, или у которого отсутствует экспрессия эндогенного белка CenH3 и которое экспрессирует белок CenH3, имеющий одну или несколько активных мутаций, в качестве родительского растения, образующего пыльцу, либо родительского растения, образующего семязачатки, с растением дикого типа (т.е. экспрессирующим эндогенный белок CenH3) приводит к получению потомства, которое является гаплоидным или демонстрирует аберрантную плоидность. Такое растение содержит только хромосомы родительского растения, которое экспрессирует эндогенный белок CenH3, и не содержит хромосомы растения, экспрессирующего белок CenH3, имеющий одну или несколько активных мутаций.
Термин "аберрантная плоидность", используемый в данном документе, относится к ситуации, в которой клетка содержит аберрантное или аномальное число наборов хромосом. Например, клетка, имеющая один или три набора хромосом на клетку, тогда как обычное число равно двум, является клеткой с аберрантной плоидностью. В настоящем изобретении активные мутантные белки CenH3 и способы их применения могут применяться для получения мутантных растений с аберрантной плоидностью, например, для получения гаплоидных растений, тогда как немутантное растение является диплоидным. Гаплоидные растения могут использоваться для ускорения селекционных программ по созданию гомозиготных линий и для исключения необходимости в инбридинге.
Термины "химерная конструкция", "химерный ген", "химерный полинуклеотид" или "химерная нуклеиновая кислота" (и аналогичные термины), используемые в данном документе, относятся к конструкции или молекуле, содержащей два или более полинуклеотида разного происхождения, собранных в виде одной молекулы нуклеиновой кислоты. Термины "химерная конструкция", "химерный ген", "химерный полинуклеотид" или "химерная нуклеиновая кислота" относятся к любой конструкции или молекуле, которая содержит (1) полинуклеотиды (например, ДНК), в том числе регуляторные и кодирующие полинуклеотиды, которые вместе не встречаются в природе (т.е. по меньшей мере один из полинуклеотидов является гетерологичным по отношению к по меньшей мере одному из других ее полинуклеотидов), или (2) полинуклеотиды, кодирующие части белков, которые в естественных условиях не соединены, или (3) части промоторов, которые в естественных условиях не соединены. Кроме того, химерная конструкция, химерный ген, химерный полинуклеотид или химерная нуклеиновая кислота могут содержать регуляторные полинуклеотиды и кодирующие полинуклеотиды, полученные из разных источников, или могут содержать регуляторные полинуклеотиды и кодирующие полинуклеотиды, полученные из одного и того же источника, но расположенные иным способом, чем встречающийся в природе. В предпочтительном аспекте настоящего изобретения химерная конструкция, химерный ген, химерный полинуклеотид или химерная нуклеиновая кислота содержат кассету экспрессии, содержащую полинуклеотиды согласно настоящему изобретению, под контролем регуляторных полинуклеотидов, в частности, под контролем регуляторных полинуклеотидов, функционирующих в растениях.
Термин "хромосома" используется в данном документе как общепризнанный в данной области техники, означающий самовоспроизводящуюся генетическую структуру в клеточном ядре, содержащую клеточную ДНК и несущую линейный набор генов.
"Кодирующий полинуклеотид" представляет собой полинуклеотид, который транскрибируется в РНК, такую как мРНК, рРНК, тРНК, snRNA, смысловая РНК или антисмысловая РНК. РНК затем предпочтительно транслируется в организме с продуцированием белка. Она может представлять собой "непрерывающийся кодирующий полинуклеотид", т.е. не иметь интронов, как, например, в кДНК, или она может содержать один или несколько интронов, ограниченных соответствующими границами сплайсинга. "Интрон" представляет собой поли(рибо)нуклеотид, который содержится в первичном транскрипте, но который удаляется посредством расщепления и повторного лигирования РНК в клетке с получением зрелой мРНК, которая может транслироваться в белок.
Термин "удвоенное гаплоидное растение", используемый в данном документе, относится к генотипу, образующемуся в случае, когда гаплоидные клетки подвергаются удвоению числа хромосом. Искусственное получение удвоенных гаплоидов является важным в селекции растений. Удвоенные гаплоиды могут быть получены in vivo или in vitro. Гаплоидные зародыши образуются in vivo в результате партеногенеза, псевдогамии или элиминации хромосом. Известен целый ряд способов получения in vitro удвоенных гаплоидных организмов из гаплоидных организмов. Неограничивающий пример способа получения удвоенных гаплоидов in vitro заключается в обработке соматических гаплоидных клеток, гаплоидных зародышей, гаплоидных семян или гаплоидных растений, полученных из гаплоидных семян, средством для удвоения числа хромосом, таким как колхицин. В настоящем изобретении гомозиготные двойные гаплоидные растения могут быть регенерированы из гаплоидных клеток путем приведения гаплоидных клеток в контакт со средствами для удвоения числа хромосом, такими как колхицин, гербициды, являющиеся ингибиторами образования микротрубочек, или оксид одновалентного азота, для создания гомозиготных удвоенных гаплоидных клеток. Способы удвоения числа хромосом раскрыты, например, в патентах США №№5770788, 7135615 и публикациях заявок на патент США №№2004/0210959 и 2005/0289673; Antoine-Michard, S. et al., Plant Cell, Tissue Organ Cult., Dordrecht, the Netherlands, Kluwer Academic Publishers 48(3):203-207 (1997); Kato, A., Maize Genetics Cooperation Newsletter 1997, 36-37; a также Wan, Y. et al., Trends Genetics 77: 889-892 (1989), Wan, Y. et al., Trends Genetics 81: 205-21 1 (1991), раскрытия которых включены в данный документ посредством ссылки. Двойные гаплоидные растения могут быть дополнительно скрещены с другими растениями с получением F1, F2 или последующих поколений растений с желаемыми признаками. Традиционные процедуры инбридинга занимают семь поколений до достижения практически полной гомозиготности, тогда как при использовании удвоенных гаплоидов это достигается за одно поколение.
Термин "Е0" относится к первому экземпляру растения, подвергнутого редактированию. Иными словами, растительная клетка, которая подвергается редактированию с помощью, например, CRISPR и которой затем предоставляется возможность развиться до растения, становится растением Е0. Растение Е1 является потомком Е0 (обычно, но не обязательно, полученным путем самооплодотворения), содержащим редактирование. Аналогичным образом, растение Е2 является потомком Е1 (обычно, но не обязательно, полученным путем самооплодотворения), содержащим редактирование. Растения Е3, Е4, Е5 и т.д. аналогичным образом отдалены в поколениях от растения Е0.
Термины "редактирование гена", "редактирование", "редактирование генома", "GE" и т.п.относятся к сайт-специфическим мутациям, произведенным в последовательности-мишени. Это также может называться "целенаправленным мутагенезом". Используемый в данном документе термин "целенаправленный мутагенез" или "стратегия мутагенеза" относится к любому способу мутагенеза, который приводит в результате к преднамеренному мутагенезу выбранного гена. Целенаправленный мутагенез включает способы с использованием CRISPR, TILLING, TALEN и другие способы, которые еще не разработаны, но которые можно применять для достижения такого же результата. Мутагенез можно осуществлять в соответствии с любыми методиками, известными из уровня техники, такими как без ограничения синтез олигонуклеотида, имеющего одну или несколько мутаций в пределах последовательности в конкретной регуляторной последовательности. В частности, сайт-специфический мутагенез представляет собой методику, применимую при получении мутантных промоторов посредством специфического мутагенеза ДНК, лежащей в их основе. Также можно использовать РНК-направляемые эндонуклеазы ("RGEN", например, CRISPR/Cas9). Данная методика дополнительно обеспечивает легкодоступную возможность получения и тестирования вариантов последовательностей, например, с учетом одного или нескольких из вышеуказанных соображений, посредством введения одного или нескольких изменений нуклеотидной последовательности в ДНК. Сайт-специфический мутагенез обеспечивает получение мутантных форм посредством использования специфических олигонуклеотидных последовательностей, которые кодируют последовательность ДНК с требуемой мутацией, а также достаточного количества смежных нуклеотидов для обеспечения получения последовательности праймера, имеющей достаточные размер и сложность последовательности для образования стабильного дуплекса по обе стороны от пересекаемой границы делеции. Как правило, предпочтительным является праймер, длина которого составляет от приблизительно 17 до приблизительно 75 нуклеотидов или больше, содержащий от приблизительно 10 до приблизительно 25 или больше остатков по обе стороны от границы в изменяемой последовательности. См. в целом патент США №10285348, включенный в данный документ посредством ссылки во всей своей полноте.
Термины "отредактированный N-концевой хвост" или "отредактированный N-концевой домен" используются взаимозаменяемо во всем в данном документе.
Термин "эндогенный", используемый в контексте настоящего изобретения в комбинации с белком или геном, означает, что указанный белок или ген происходит из растения, в котором он по-прежнему содержится. Зачастую эндогенный ген будет присутствовать в растении в его нормальном генетическом окружении. В другом контексте термин "эндогенный" может относится к нормальным функциям клетки. Для примера и не в качестве ограничения, "эндогенная репарация ДНК" относится к нормальным клеточным механизмам, ферментам или процессам репарации ДНК.
Термин "экспрессия" при использовании в отношении полинуклеотида, такого как ген, ORF или их часть, или трансгена в растениях относится к процессу преобразования генетической информации, закодированной в гене, в РНК (например, мРНК, рРНК, тРНК или snRNA) посредством "транскрипции" гена (т.е. благодаря ферментативному действию РНК-полимеразы) и в белок в соответствующих случаях (например, если ген кодирует белок) посредством "трансляции" мРНК. Экспрессия гена может регулироваться на многих стадиях в ходе этого процесса. Например, в случае антисмысловых конструкций или dsRNA-конструкций соответственно экспрессия может относиться к транскрипции только антисмысловой РНК или только dsRNA. В вариантах осуществления "экспрессия" относится к транскрипции и стабильному накоплению смысловой (мРНК) или функциональной РНК. "Экспрессия" может также относиться к продуцированию белка.
Термины "гомология", "сходство последовательностей" или "идентичность последовательностей" для нуклеотидных или аминокислотных последовательностей означают степень идентичности или сходства двух или более последовательностей и могут быть определены традиционными способами с использованием известного программного обеспечения или компьютерных программ, таких как программы для попарного сравнения BestFit или Gap (GCG Wisconsin Package, Genetics Computer Group, 575 Science Drive, Мэдисон, Висконсин 53711). В BestFit используется алгоритм поиска локальной гомологии из Smith and Waterman, Advances in Applied Mathematics 2:482-489 (1981), для обнаружения сегмента с наилучшими идентичностью или сходством между двумя последовательностями. Сравнение последовательностей двух или более полинуклеотидов или полипептидов обычно осуществляют путем сравнения частей двух последовательностей в окне сравнения для выявления и сравнения локальных областей сходства последовательностей. Размер окна сравнения обычно составляет от приблизительно 20 до 200 смежных нуклеотидов. Gap осуществляет глобальные выравнивания: выравнивание одной полной последовательности с другой полной сходной последовательностью с использованием способа из Needleman and Wunsch, J. Mol. Biol. 48:443-453 (1970). При использовании программы для выравнивания последовательностей, такой как BestFit, для определения степени гомологии, сходства или идентичности последовательностей ДНК можно использовать настройки по умолчанию, или для оптимизации баллов идентичности, сходства или гомологии можно выбрать соответствующую матрицу замен. Аналогично, при использовании программы, такой как BestFit, для определения степени идентичности, сходства или гомологии последовательностей между двумя отдельными аминокислотными последовательностями можно использовать настройки по умолчанию, или для оптимизации баллов идентичности, сходства или гомологии можно выбрать соответствующую матрицу замен, такую как blosum45 или blosum80.
Термин "локус" относится к положению (например, гена, генетического маркера или т.п.) в хромосоме данного вида.
Термин "праймер", используемый в данном документе, относится к олигонуклеотиду, который способен отжигаться с мишенью для амплификации, что позволяет прикрепиться ДНК-полимеразе, и тем самым служить в качестве точки начала синтеза ДНК при помещении в условия, в которых индуцируется синтез продукта удлинения праймера, например, в присутствии нуклеотидов и средства для полимеризации, такого как ДНК-полимераза, и при подходящих температуре и рН. Праймер (для амплификации) предпочтительно является однонитевым для максимальной эффективности амплификации. Праймер предпочтительно представляет собой олигодезоксирибонуклеотид. Праймер обычно является достаточно длинным, чтобы служить затравкой для синтеза продуктов удлинения в присутствии средства для полимеризации. Точные значения длины праймера будут зависеть от многих факторов, в том числе от температуры и состава (содержания А/Т и G/C) праймера. Пара двунаправленных праймеров состоит из одного прямого и одного обратного праймера, обычно используемых в области амплификации ДНК, как, например, при ПЦР-амплификации. Будет понятно, что используемый в данном документе термин "праймер" может относиться более чем к одному праймеру, в частности, в случае, когда существует некоторая неясность в информации о концевой(концевых) по следовательности(последовательностях) области-мишени, по дл ежащей амплификации. Следовательно, "праймер" включает совокупность праймерных олигонуклеотидов, содержащих последовательности, представляющие возможные вариации в последовательности, или содержит нуклеотиды, обеспечивающие возможность обычного спаривания оснований. Олигонуклеотидные праймеры можно получить любым подходящим способом. Способы получения олигонуклеотидов с конкретной последовательностью известны из уровня техники и включают, например, клонирование и рестрикцию соответствующих последовательностей, а также прямой химический синтез. Способы химического синтеза могут включать, например, фосфодиэфирный или фосфотриэфирный способ, диэтилфосфорамидатный способ и способ с использованием твердых подложек, например, раскрытые в US 4458066. При необходимости праймеры можно пометить путем включения в их состав средств, выявляемых, например, с помощью спектроскопических, флуоресцентных, фотохимических, биохимических, иммунохимических или химических способов. Матрично-зависимое удлинение олигонуклеотидного(олигонуклеотидных) праймера(праймеров) катализируется средством для полимеризации в присутствии достаточных количеств четырех дезоксирибонуклеотидтрифосфатов (dATP, dGTP, dCTP и dTTP; т.е. dNTP) или их аналогов в реакционной среде, которая состоит из подходящих солей, катионов металлов и буферной системы для поддержания рН. Подходящие средства для полимеризации представляют собой ферменты, которые, как известно, катализируют праймер- и матрично-зависимый синтез ДНК. Известные ДНК-полимеразы включают, например, ДНК-полимеразу I из Е. coli или ее фрагмент Кленова, ДНК-полимеразу Т4 и ДНК-полимеразу Taq. Условия реакции для катализа синтеза ДНК с помощью этих ДНК-полимераз известны из уровня техники. Продуктами синтеза являются дуплексные молекулы, состоящие из нитей-матриц и нитей удлинения праймеров, которые содержат последовательность-мишень. Эти продукты, в свою очередь, служат в качестве матрицы для следующего раунда репликации. Во втором раунде репликации нить удлинения праймера из первого цикла отжигается с комплементарным ей праймером; при этом синтез дает "короткий" продукт, который ограничен на обоих из 5'- и 3'-конца праймерными последовательностями или комплементарными им последовательностями. В результате повторяющихся циклов денатурации, отжига и удлинения праймеров происходит экспоненциальное накопление области-мишени, определяемой праймерами. Для достижения требуемого количества полинуклеотида, содержащего область-мишень нуклеиновой кислоты, проводят достаточное число циклов. Требуемое количество может варьировать, и оно определяется функцией, для которой служит полинуклеотидный продукт. Способ ПЦР хорошо описан в руководствах и известен специалисту в данной области. После амплификации путем ПЦР полинуклеотиды-мишени можно выявлять посредством гибридизации с полинуклеотидным зондом, который образует стабильный гибрид с полинуклеотидом с последовательностью-мишенью в условиях гибридизации и отмывки низкой, умеренной или даже высокой жесткости. Если ожидается, что зонды будут фактически полностью комплементарными (т.е. на приблизительно 99% или больше) последовательности-мишени, то можно применять условия высокой жесткости. Если ожидается некоторое несовпадение, например, если ожидается наличие вариантов штаммов, вследствие чего зонд не будет полностью комплементарен, то жесткость гибридизации может быть снижена. Однако, как правило, подбираются такие условия, которые исключают неспецифическое/случайное связывание. Условия, которые влияют на гибридизацию и которые выбирают для предупреждения неспецифического связывания, известны из уровня техники и описаны, например, в Sambrook and Russell, 2001. В целом, более низкая концентрация солей и более высокая температура повышают жесткость условий гибридизации. "Праймер для ПЦР", как предпочтительно понимают в рамках настоящего изобретения, относится к относительно коротким фрагментам однонитевой ДНК, используемым в ПЦР-амплификации специфических областей ДНК.
Термины "белок", "пептид" и "полипептид" используются в данном документе взаимозаменяемо.
Термин "промотор" относится к полинуклеотиду, как правило, расположенному выше (5') от его кодирующего полинуклеотида, который осуществляет контроль экспрессии кодирующего полинуклеотида, обеспечивая узнавание РНК-полимеразой и другими факторами, необходимыми для правильной транскрипции.
Термин "сайт-направленная нуклеаза" относится к любому ферменту, направляемому нуклеотидной последовательностью к последовательности-мишени в цепи ДНК. Сайт-направленная нуклеаза предпочтительно является используемой в CRISPR, но также может представлять собой мегануклеазу, эффекторную нуклеазу, подобную активатору транскрипции (TALEN), или нуклеазу с "цинковыми пальцами". Сайт-направленная(сайт-направленные) нуклеаза(нуклеазы) могут быть обозначены аббревиатурой "SDN". SDN включают без ограничения мегануклеазы (MN), нуклеазы с "цинковыми пальцами" (ZFN), эффекторные нуклеазы, подобные активаторам транскрипции (TALEN), нуклеазу Cas9, нуклеазу Cpf1 (Cas12a), dCas9-FokI, dCpf1-FokI, химерную Cas9-цитозиндезаминазу, химерную Cas9-адениндезаминазу, химерную FEN1-FokI и Mega-TAL, никазу Cas9 (nCas9), химерную нуклеазу dCas9, не содержащую FokI, и нуклеазу dCpf1, не содержащую FokI; и где дополнительно направляющая нуклеиновая кислота представляет собой направляющую РНК.
Термины "жесткие условия" или "жесткие условия гибридизации" включают ссылку на условия, при которых полинуклеотид будет гибридизироваться со своей последовательностью-мишенью в заметно большей степени, чем с другими последовательностями (например, на уровне, в по меньшей мере 2 раза превышающем фоновый). Жесткие условия зависят от последовательности и будут отличаться при разных обстоятельствах. Путем контроля жесткости условий гибридизации и/или отмывки можно идентифицировать полинуклеотиды-мишени, которые на 100% комплементарны зонду (гомологичные зонды). В качестве альтернативы, жесткость условий можно корректировать для обеспечения возможности некоторого несовпадения в последовательностях, чтобы выявлять более низкие степени их подобия (гетерологичные зонды). Как правило, жесткие условия будут представлять собой условия, при которых концентрация солей составляет менее чем примерно 1,5 М ионов Na, как правило, приблизительно 0,01-1,0 М ионов Na (или других солей) при рН 7,0-8,3, а температура составляет по меньшей мере приблизительно 30°С для коротких зондов (например, 10-50 нуклеотидов) и по меньшей мере приблизительно 60°С для длинных зондов (например, более 50 нуклеотидов). Жесткие условия также могут достигаться при добавлении дестабилизирующих средств, таких как формамид. Типичные условия низкой жесткости включают гибридизацию с буферным раствором, содержащим 30-35% формамид, 1 М NaCl, 1% SDS (вес/об.; додецилсульфат натрия) при 37°С и отмывку в 1-2×SSC (20×SSC=3,0 М NaCl/0,3M цитрата натрия) при 50-55°С. Типичные условия умеренной жесткости включают гибридизацию в 40-45% формамиде, 1 М NaCl, 1% SDS при 37°С и отмывку в 0,5-1×SSC при 55-60°С. Типичные условия высокой жесткости включают гибридизацию в 50% формамиде, 1 М NaCl, 1% SDS при 37°С и отмывку в 0,1×SSC при 60-65°С. Специфичность, как правило, зависит от отмывок после гибридизации, при которых ключевыми факторами являются ионная сила и температура конечного раствора для отмывки. Для гибридных молекул ДНК-ДНК Tm можно примерно вычислить из уравнения согласно Meinkoth and Wahl (Anal. Biochem., 138:267-284, 1984): Tm=81,5°C+16,6 (log M)+0,41 (% GC) - 0,61 (% форм.) - 500/L; где М представляет собой молярность одновалентных катионов, % GC представляет собой процентную долю гуанозиновых и цитозиновых нуклеотидов в ДНК, % форм, представляет собой процентную долю формамида в растворе для гибридизации, a L представляет собой длину гибрида в парах оснований. Tm представляет собой температуру (при заданных ионной силе и рН), при которой 50% комплементарной последовательности-мишени гибридизируется с абсолютно совпадающим зондом. Tm снижается на приблизительно 1°С на каждый 1% несовпадения, таким образом, Tm, условия гибридизации и/или отмывки можно регулировать, чтобы обеспечить гибридизацию с последовательностями с требуемой идентичностью. Например, если осуществляют поиск последовательностей с примерно 90% идентичностью, Tm можно снизить на 10°С. В целом жесткие условия выбирают таким образом, чтобы температура была на приблизительно 5°С ниже температуры точки плавления (Tm) для специфической последовательности и комплементарной ей последовательности при заданных ионной силе и рН. Однако при условиях сильной жесткости можно использовать гибридизацию и/или отмывку при температуре на 1, 2, 3 или 4°С ниже, чем температура точки плавления (Tm), при условиях умеренной жесткости можно использовать гибридизацию и/или отмывку при температуре на 6, 7, 8, 9 или 10°С ниже, чем температура точки плавления (Tm); при условиях низкой жесткости можно использовать гибридизацию и/или отмывку при температуре на 11, 12, 13, 14, 15 или 20°С ниже, чем температура точки плавления (Tm). Рядовые специалисты в данной области будут понимать, что вариации жесткости растворов для гибридизации и/или отмывки по существу описаны с помощью уравнения, составов для гибридизации и отмывки и требуемой Tm. Если требуемая степень несовпадения приводит к Tm, составляющей менее 45°С (водный раствор) или 32°С (раствор формамида), предпочтительно повысить концентрацию SSC для того, чтобы можно было использовать более высокую температуру. Подробное руководство по гибридизации нуклеиновых кислот можно найти в Tijssen, Laboratory Techniques in Biochemistry and Molecular Biology - Hybridization with Nucleic Acid Probes, Part I, Chapter 2 "Overview of principles of hybridization and the strategy of nucleic acid probe assays", Elsevier, N.Y. (1993); и в Current Protocols in Molecular Biology, Chapter 2, Ausubel, et al., eds., Greene Publishing and Wiley-Interscience, New York (1995). Из уровня техники известны способы жесткой гибридизации, условия которых могут быть рассчитаны с помощью средств, известных из уровня техники. Они раскрыты в Sambrook et al., Molecular Cloning: A Laboratory Manual, 2nd Ed., Cold Spring Harbor Laboratory Press, 1989, Cold Spring Harbor, N.Y., и в Current Protocols in Molecular Biology, Ausubel et al, eds., John Wiley and Sons, Inc., 2000. Из уровня техники известны способы определения процентной идентичности последовательностей, примером которых является программное обеспечение для компьютерного анализа последовательностей GCG (GCG, Inc, Мэдисон, Висконсин).
Используемый в данном документе термин "восстановление сдвинутой рамки считывания" ("RFS") относится к мутации или серии мутаций в гене, которые, по отдельности или в комбинации, прерывают кодирующую последовательность гена, но не изменяют рамку считывания кодирующей последовательности. Это также может называться "синхронизацией, восстанавливающей рамку считывания". Например, кодирующая последовательность ДНК содержит серию кодонов. Каждый кодон содержит три нуклеотида, и каждый кодон при его транскрипции в РНК кодирует одну аминокислоту после трансляции. Мутация по типу вставки/делеции ("вставка/делеция") одного или двух нуклеотидов в кодирующей последовательности вызывает сдвиг кодирующей рамки считывания ("сдвиг рамки считывания"). Однако вставки или делеции, будь то по отдельности или в комбинации, которые в совокупности имеют место в количестве, кратном трем, восстанавливают кодоны до их первоначальной рамки считывания, даже если кодирующая последовательность сама по себе является измененной. См., например, B.N. Ames and H.J. Whitfield, Jr., Frameshift Mutagenesis in Salmonella, Cold Spring Harb. Symp.Quant. Biol. 31:221-225 (1966). Например, и в пределах объема данного определения, последовательность, которая содержит по меньшей мере две мутации по типу вставки/делеции, представляющие собой делеции, будь то последовательные или нет, и в которой мутации по типу вставки/делеции вызывают восстановление рамки считывания до первоначальной рамки считывания, представляет собой последовательность, содержащую мутацию по типу восстановления сдвинутой рамки считывания. Термин "сконструированное восстановление сдвинутой рамки считывания" также может использоваться для описания мутации по типу RFS, которая была создана с помощью редактирования генома или модификации генома.
Используемый в данном документе термин "большая делеция" ("LD") относится к мутации, которая вызывает утрату нескольких последовательных нуклеотидов. В частности, большая делеция относится к утрате 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 40, 50, 60, 70, 80, 90, или 100 или больше нуклеотидов. В некоторых вариантах осуществления последовательность, утрачиваемая в результате LD, имеет размер, кратный 3 (т.е. 3, 6, 9, 12, 15, 18, 21, 24, 27, 30 и т.д.) В других вариантах осуществления мутация по типу LD может также иметь место в сочетании с мутацией по типу вставки/делеции где-либо еще в той же последовательности, что тем самым вызывает мутацию по типу восстановления сдвинутой рамки считывания.
В контексте настоящего изобретения использование терминов "дикий тип" или "растение дикого типа" относится к растению, которое не несет мутантный белок или ген CenH3 (т.е. не содержит одну или несколько активных мутаций, указанных в данном документе) и которое эндогенно экспрессирует или продуцирует функциональные гены и белки CenH3.
ПОДРОБНОЕ ОПИСАНИЕ
В данной ситуации авторы настоящего изобретения индуцировали альтернативный сплайсинг у пшеницы (Triticum aestivum) путем применения CRISPR-Cas9 для редактирования последовательностей цис-сплайсинга, содержащих 5'- и 3'-сайты сплайсинга. Авторы настоящего изобретения выбрали пшеницу в качестве модельного организма-мишени, поскольку пшеница является гексаплоидной, что обеспечивает функциональную избыточность генома пшеницы. В качестве гена-мишени авторы настоящего изобретения выбрали ген центромерного гистона 3 ("CenH3"), кодирующий центромерный белок, поскольку модификации данного гена должны приводить к получению растений, обладающих ценностью для селекции сельскохозяйственных культур. CenH3 отвечает за точное расщепление хромосом в процессе клеточного деления. В отличие от Н3 и других традиционных гистонов, CENH3 имеет длинный, гипервариабельный N-концевой "хвост". См. J. Monen, et al. Separase Cleaves the N-Tail of the CENP-A Related Protein CPAR-1 at the Meiosis I Metaphase-Anaphase Transition in C. elegans, PLoS One 10:e0125382 (2015). Направленная или естественная модификация "хвоста" запускает компенсаторные изменения в кинетохоре, которые могут обеспечить для CENH3 возможность управления видообразованием посредством нарушений мейоза или ингибирования зиготического расщепления хромосом. См. I. Lermontova, et al., Knockdown of CENH3 in Arabidopsis reduces mitotic divisions and causes sterility by disturbed meiotic chromosome segregation, Plant J 68:40-50 (2011), и M. Ravi and R. Bondada, Genome Elimination by Tailswap CenH3: In Vivo Haploid Production in Arabidopsis thaliana, Methods Mol Biol 1469:77-99 (2016). Обмен N-концевого "хвоста" и "хвоста" Н3 приводил к индукции гаплоидии у Arabidopsis (М. Ravi and S. Chan, Haploid plants produced by centromere-mediated genome elimination, Nature 464:615-618 (2010)) и кукурузы (Т. Kelliher, et al., Maternal Haploids are Preferentially Induced by CENH3-tailswap Transgenic Complementation in Maize, Front. Plant Scl, doi.org/10.3389/fpls.2016.00414, 31(7):414 (2016). Индукция гаплоидии представляет собой аберрантный репродуктивный процесс, который приводит к уменьшению плоидности от одного поколения к следующему. Гаплоиды можно подвергнуть удвоению для получения инбредных линий, исключая необходимость в шести поколениях самоопыления, обычно требуемых для получения новых чистосортных линий. Реализация подхода с "обменом хвостами" у сельскохозяйственных культур требует многих поколений для осуществления сборки нокаутного нативного аллеля и стабильной вставки трансгенов. Авторы настоящего изобретения смогли индуцировать AS путем непосредственного редактирования N-концевых последовательностей CenH3 пшеницы. Такие новые последовательности CenH3 были изучены для того, чтобы определить, могут ли мутантные белки CENH3 вызывать индукцию гаплоидии у пшеницы и в какой комбинации они это делают. В описанных авторами настоящего изобретения обстоятельствах это действительно происходит.
Следовательно, один вариант осуществления настоящего изобретения представляет собой растение пшеницы, содержащее по меньшей мере геном А, геном В и геном D, где геном В содержит нокаут-мутацию в гене CENH3, и где необязательно геном D содержит нокаут-мутацию в гене CENH3, и где дополнительно геном А содержит мутантный ген CENH3, содержащий по меньшей мере одну нокдаун-мутацию в 5'-сайте сплайсинга интрона. В одном аспекте нокдаун-мутация представляет собой мутацию по типу восстановления сдвинутой рамки считывания или мутацию по типу большой делеции. В другом варианте осуществления растение пшеницы является гомозиготным по нокаут-мутации в гене CENH3 в геноме В. В альтернативном варианте осуществления растение пшеницы является биаллельным по нокаут-мутации в гене CENH3 в геноме В. В другом варианте осуществления растение пшеницы является гомозиготным по нокаут-мутации в гене CENH3 в геноме D. В альтернативном варианте осуществления растение пшеницы является биаллельным по нокаут-мутации в гене CENH3 в геноме D. В еще одном варианте осуществления растение пшеницы является гомозиготным, биаллельным или характеризуется комбинацией этих свойств по нокаут-мутации в гене CENH3 в геноме В и геноме D. В другом варианте осуществления растение пшеницы является гомозиготным по мутации по типу восстановления сдвинутой рамки считывания в CENH3; или оно является гетерозиготным по мутации по типу восстановления сдвинутой рамки считывания в CENH3; или оно является биаллельным по мутации по типу восстановления сдвинутой рамки считывания в CENH3.
Другим аспектом настоящего изобретения является способ получения растения пшеницы, индуцирующего гаплоидию, при этом способ включает: (а) получение по меньшей мере клетки растения пшеницы, содержащей по меньшей мере три генома; (b) осуществление мутаций в двух из трех геномов с получением гомозиготных нокаут-мутаций в гене CENH3; (с) осуществление мутаций в третьем геноме с получением гомозиготной нокдаун-мутации в гене CENH3 и (d) получение в результате этого растения пшеницы, содержащего гомозиготные нокаут-мутации в гене CENH3 в двух из трех геномов и дополнительно содержащего гомозиготную нокдаун-мутацию в гене CENH3 в третьем геноме; вследствие чего растение пшеницы, полученное на стадии (d), производит гаплоидное потомство при скрещивании с растением пшеницы дикого типа. В одном варианте осуществления три генома включают в себя геном А, геном В и геном D. В другом варианте осуществления нокаут-мутации в гене CENH3 имеют место в геномах В и D. В еще одном варианте осуществления нокдаун-мутация в гене CENH3 имеет место в геноме А. В одном аспекте нокдаун-мутации в гене CENH3 в геноме А представляют собой мутации по типу восстановления сдвинутой рамки считывания. В другом аспекте мутации по типу восстановления сдвинутой рамки считывания выбраны из группы, состоящей из SEQ ID NO: 56, последовательности нуклеиновой кислоты, на 70% идентичной SEQ ID NO: 56, SEQ ID NO: 63, последовательности нуклеиновой кислоты, на 70% идентичной SEQ ID NO: 63, SEQ ID NO: 69 и последовательности нуклеиновой кислоты, на 70% идентичной SEQ ID NO: 69.
Другим аспектом настоящего изобретения является растение пшеницы, содержащее мутантный ген CENH3, содержащий по меньшей мере одну делеционную мутацию в N-концевом домене, которая приводит к сдвигу рамки считывания, восстановлению сдвинутой рамки считывания или большой делеции. Еще одним аспектом является растение пшеницы, содержащее мутантный ген CENH3, содержащий по меньшей мере одну инсерционную мутацию в N-концевом домене, которая приводит к сдвигу рамки считывания, восстановлению сдвинутой рамки считывания или большой делеции.
Другим аспектом настоящего изобретения является способ получения сконструированного восстановления сдвинутой рамки считывания в гене в клетке, включающий: (а) приведение генома в контакт с сайт-направленной нуклеазой ("SDN") и по меньшей мере двумя направляющими нуклеиновыми кислотами, где по меньшей мере две направляющие нуклеиновые кислоты нацеливаются на по меньшей мере две последовательности-мишени в гене; (b) предоставление для SDN возможности разрезания гена в по меньшей мере двух последовательностях-мишенях с утратой таким образом вставочной последовательности между по меньшей мере двумя последовательностями-мишенями; и обеспечение возможности прохождения эндогенной репарации ДНК; вследствие чего эндогенная репарация ДНК приводит к получению гена со сконструированным восстановлением сдвинутой рамки считывания. В одном варианте осуществления утрачиваемая вставочная последовательность из стадии (b) содержит (N) пар оснований, где (N) является числом, кратным 3.
Еще одним аспектом настоящего изобретения является способ получения гаплоидного растения пшеницы, включающий: (а) получение растения пшеницы; (b) скрещивание растения пшеницы с растением пшеницы, содержащим мутантный ген CENH3; и (с) отбор потомка, полученного в результате стадии скрещивания; где потомок представляет собой гаплоидное растение пшеницы. В одном варианте осуществления растение пшеницы из стадии (а) представляет собой отцовское родительское растение. В другом варианте осуществления растение пшеницы из стадии (а) представляет собой материнское родительское растение. В другом варианте осуществления способ включает дополнительную стадию превращения растения пшеницы, являющегося потомком, в удвоенное гаплоидное растение пшеницы.
Другим аспектом настоящего изобретения является получение растения пшеницы, содержащего мутантный аллель CENH3, содержащий последовательность нуклеиновой кислоты, на по меньшей мере 70% идентичную последовательности, выбранной из группы, состоящей из SEQ ID NO: 56-73, где мутация представляет собой мутацию по типу восстановления сдвинутой рамки считывания, и где растение пшеницы образует гаплоидное потомство при скрещивании с диплоидным растением пшеницы дикого типа. В одном варианте осуществления растение пшеницы содержит по меньшей мере одну копию мутантного аллеля CENH3; в другом варианте осуществления растение пшеницы содержит по меньшей мере две копии мутантного аллеля CENH3; в еще одном варианте осуществления растение пшеницы содержит по меньшей мере три копии мутантного аллеля CENH3. В одном варианте осуществления мутантный аллель CENH3 содержит последовательность нуклеиновой кислоты, на 80, 90, 95 или 100% идентичную SEQ ID NO: 56-73.
ПРИМЕРЫ
Пример 1. Теория, лежащая в основе применения двух направляющих РНК для N-концов
Трансгены CENH3 с "обмененными хвостами" при гетерологичной экспрессии в линии, в которой нативные гены CENH3 являются нокаутными, приводят к индукции гаплоидии. См., например, публикацию заявки на патент США №2019/0136250, включенную в данный документ посредством ссылки. Это называется подходом с "обменом хвостами". Важно, что у линий, являющихся индукторами гаплоидии, с "обмененными хвостами" отсутствуют аллели дикого типа. Предполагается, что трансгены обладают частичной функциональностью и способны образовывать центромеры, которые являются достаточно стабильными для получения нормально развивающихся растений в гомозиготном состоянии. Однако, если трансгены с "обмененными хвостами" являются гетерологичными по отношению к CENH3 дикого типа в клетке, трансгены с "обмененными хвостами" являются нестабильными и приводят к успешной индукции гаплоидов в процессе ауткроссинга. При такой разработке растений с "обмененными хвостами" чрезвычайно важно, чтобы нативные гены CENH3 были нокаутными и чтобы трансгены с "обмененными хвостами" имели значительные изменения в N-концевом домене в сочетании с лишь незначительными либо предпочтительно отсутствующими изменениями в С-концевом домене. Индукции гаплоидии не произойдет, даже если мутантные гены CENH3 будут кодировать белки CENH3, которые сохраняют нормальную или близкую к нормальной функциональность.
Для того чтобы достичь индукции гаплоидии у пшеницы, авторы настоящего изобретения непосредственно отредактировали шесть генов CENH3a для нокаута нескольких копий и создания модификаций в N-концевом домене (оставляя интактным С-концевой домен) в еще нескольких других копиях. Исходя из экспериментов авторов настоящего изобретения по измерению экспрессии генов для генов CENH3a из геномов А, В и D, авторы настоящего изобретения сосредоточились на создании N-концевых модификаций в геноме А и нокаут-мутаций в геномах В и D. В случае, если редактирования, произведенные авторами настоящего изобретения, были успешными, то не останется ни одной нормальной (интактной) копии CENH3α: все гены будут отредактированы, но результаты редактирования будут различаться. Важно, что план редактирования, разработанный авторами настоящего изобретения, не включал какие-либо трансгены CENH3 - авторы настоящего изобретения всего лишь хотели путем непосредственного редактирования создать частично функциональный вариант CENH3α с N-концевой модификацией в геноме А.
Как правило, значительные изменения в областях, кодирующих белки, могут быть достигнуты путем редактирования генома с помощью SDN II CRISPR (также называемого заменой аллеля ("AR") или гомологичной рекомбинацией ("HR")), но эффективность этой технологии у растений является крайне низкой и у пшеницы достигается очень редко. Следовательно, авторы настоящего изобретения разработали стратегию редактирования с использованием двух направляющих РНК ("gRNA"), которые обладали способностью модифицировать CENH3α для создания аллеля с частичной потерей функции, который имел значительное изменение (изменение более чем 5 аминокислот) в N-концевом домене и нативную (неизмененную) последовательность С-концевого домена. Это будет требовать специфичного разрезания в обоих сайтах для направляющей РНК. Авторы настоящего изобретения знали, что выбранные направляющие РНК также будут редактировать копии CENH3α в геномах В и D, и это было преднамеренным. На самом деле из-за непредсказуемости каждого конкретного результата редактирования для двух направляющих РНК авторы настоящего изобретения ожидали, что большая часть отредактированных аллелей будет аллелями с полной потерей функции (в копиях в геномах А, В и D) вследствие сдвигов рамок считывания в кодирующих последовательностях, которые приводили бы к появлению преждевременных стоп-кодонов, обуславливающих усечение белкового продукта CENH3α, и таким образом, были бы полностью нокаутными формами нативного гена. Однако авторы настоящего изобретения также знали, что если бы все копии гена CENH3α были нокаутными, растения погибли бы, поскольку частичное функционирование CENH3α необходимо для развития растения. Если бы те же направляющие последовательности, которые приводили к созданию модифицированного аллеля, являющегося индуктором гаплоидии, в геноме А, могли одновременно (в одних и тех же растениях) осуществлять нокаут аллелей в геномах В и D, это помогло бы авторам настоящего изобретения: результатом была бы идеально сконструированная линия, являющаяся индуктором гаплоидии. Таким образом, целью авторов настоящего изобретения было использование двух gRNA для имитации трансгенной системы с "обмененными хвостами" путем непосредственного редактирования, но ключевой стадией настоящего изобретения было то, что вместо выполнения какой-либо замены аллеля или вставки трансгена CENH3α авторы настоящего изобретения получили новые модифицированные варианты посредством небольших мутаций по типу вставки/делеции, создаваемых с помощью разрезания нуклеазой сайтов для двух направляющих РНК в N-концевом домене.
Однако не каждая пара возможных направляющих последовательностей обладала способностью совмещать в себе создание измененного N-концевого домена вкупе с функциональным С-концевым доменом: не каждая пара направляющих последовательностей могла производить эти измененные копии "отредактированного N-концевого хвоста" с частичной функциональностью и способностью к индукции гаплоидии. После проверки того, к чему могут приводить редактирования в отношении аминокислотной последовательности, выяснилось, что многие из направляющих последовательностей будут приводить к появлению преждевременных стоп-кодонов. Другими словами, авторы настоящего изобретения поняли, что они должны специально отбирать направляющие РНК, для которых авторы настоящего изобретения предсказали, что они могут производить такую комбинацию редактирований в сайтах-мишенях, которая приводила бы к образованию аминокислотных последовательностей в зрелом белковом продукте CENH3α, содержащих существенные изменения в N-концевом домене, но остающихся нетронутыми в С-концевом домене. В частности, авторы настоящего изобретения планировали провести скрининг растений и идентифицировать те из них, которые содержали такие продуктивные аллели с N-концевыми модификациями для копий CENH3α в геноме А и которые также имели нокаутные аллели для копий CENH3α в геномах В и D. Зная, что редактирование, опосредованное сайт-направленной нуклеазой, не всегда происходит непосредственно в процессе трансформации, авторы настоящего изобретения заключили, что в поколении Е0 они могут создавать нокаутные аллели (с полной потерей функции) для некоторых копий, а также некоторое количество аллелей с частичной потерей функции (индукторов гаплоидии) для еще нескольких других копий гена CENH3α, и что такие материалы могут быть способны производить гаплоиды путем самоопыления-при условии, что мужские и женские гаметы (спермин и яйцеклетки) имеют разные редактирования или разные комбинации аллелей и, следовательно, потенциально разные центромеры. Другими словами, авторы настоящего изобретения полагали, что они могут обнаружить некоторое количество гаплоидов среди растений поколения Е1. Авторы настоящего изобретения надеялись, что эти наблюдения позволят направить их на отбор определенных растений Е1 для того, чтобы генотипировать (т.е. генотипировать диплоидных сибсов из популяций, в которых появилось некоторое количество гаплоидов в результате самоопыления) и идентифицировать растения Е1, которые имели аллели с частичной потерей функции в гомозиготном состоянии. Если бы авторы настоящего изобретения могли так сделать, то затем они бы использовали эти конкретные растения для тестирования частоты индукции гаплоидии посредством ауткроссинга. Подводя итог, авторы настоящего изобретения полагали, что после создания ими линий со стабильными мутациями они могли бы протестировать то, являются ли они действительно индукторами гаплоидии, но авторы настоящего изобретения знали, что они не могли сделать это в первом поколении, поскольку редактирование может не быть "полным" на тот момент времени, и что им было необходимо тестировать и повторно тестировать индукцию гаплоидии для различных генотипов в поколениях Е1 и Е2.
Имелось множество направляющих РНК, которые авторы настоящего изобретения могли выбрать из тех, для которых имелись подходящие РАМ-сайты, но только несколько отборных пар могли создавать желаемые аллели, являющиеся индукторами гаплоидии. Что касается желаемых аллелей, то одним из способов, посредством которого пара направляющих РНК могла бы создавать такие аллели, является редактирование первого сайта-мишени для gRNA с образованием сдвига рамки считывания, восстановление которого затем происходит посредством компенсаторной мутации во втором сайте-мишени, которая приводит к возвращению транскрипта в правильную (нативную) рамку считывания. Это будет приводить к изменению аминокислотной последовательности N-концевого домена по вставочной нити из аминокислот между двумя сайтами-мишенями для направляющей РНК и будет обеспечивать восстановление "нативной" последовательности С-концевого домена. Такие аллели авторы настоящего изобретения решили называть аллелями с восстановлением сдвинутой рамки считывания ("RFS"). В качестве альтернативы, одновременное или практически одновременное разрезание в обоих сайтах-мишенях для направляющей РНК может приводить к делеции вставочной последовательности нуклеиновой кислоты. Во многих случаях эта делеция будет приводить к сдвигу рамки считывания в нижерасположенной последовательности, но в некоторых случаях может произойти так, что при такой делеции 3'-последовательность транскрипта остается в нормальной рамке считывания, так что значительная часть аминокислотной последовательности N-концевого домена отсутствует в образующемся в результате белковом продукте, но С-концевой домен опять-таки остается интактным. Авторы настоящего изобретения решили называть такие аллели аллелями с большой делецией ("LD"). Наконец, в некоторых случаях авторы настоящего изобретения разработали gRNA для нацеливания на границы сайтов сплайсинга, и редактирование в этих сайтах-мишенях может приводить к формированию паттернов альтернативного сплайсинга (например, оно может приводить к удержанию интрона или пропуску экзона). Такие аллели, подвергнутые альтернативному сплайсингу ("AS"), в большинстве случаев будут приводить к появлению преждевременных стоп-кодонов и генетическим нокаутам, но авторы настоящего изобретения также обнаружили одну направляющую РНК, которая в случае, если происходит правильное редактирование и правильный сплайсинг, не будет приводить к появлению каких-либо преждевременных стоп-кодонов в рамке считывания. Вместо этого она может приводить к большой вставке в зрелый транскрипт, что приводит к значительным изменениям в N-концевом домене путем вставки длинного аминокислотного фрагмента, но затем специфичное редактирование во втором сайте-мишени может приводить к возвращению последовательности в рамку считывания для С-концевого домена. Другими словами, благодаря продуманной разработке gRNA может быть предсказана мутация в сайте сплайсинга, приводящая к образованию вариантов зрелых мРНК-транскриптов, в которых изменен аминокислотный код N-концевого домена, но восстановлены нормальная рамка считывания и последовательность С-концевого домена.
В следующих примерах авторы настоящего изобретения подробно описывают обнаруженные ими конкретные направляющие РНК и редактирования, а также комбинации редактирований в конкретных растениях. Авторы настоящего изобретения подробно демонстрируют то, как они создали линии пшеницы, являющиеся индукторами гаплоидии, путем непосредственного редактирования нативных генов CENH3 с использованием двух направляющих РНК, нацеливающихся на гипервариабельный N-концевой домен. В соответствии с запланированным растения, имеющие мутации в обоих сайтах-мишенях, в некоторых случаях продуцировали белковые продукты, которые содержали значительные изменения в аминокислотной последовательности N-концевого домена, не затрагивающие аминокислотную последовательность С-концевого домена. Авторы настоящего изобретения отбирали и поддерживали линии, подвергнутые редактированию, которые имели такие типы измененных аллелей CENH3 с отредактированным N-концевым "хвостом", и удостоверились в том, что другие копии генов CenH3 (из геномов А, В и D) были подвергнуты нокауту посредством мутаций, произведенных теми же самыми направляющими РНК. Авторы настоящего изобретения обнаружили и протестировали индукцию гаплоидии в линиях, которые имели желаемые мутации, включающие комбинацию RFS-аллелей из генома А с нокаутными аллелями из геномов В и D. В тех же самых линиях правильная комбинация редактирований, которая согласно предсказанию авторов настоящего изобретения приводила к образованию гаплоидов, действительно приводила к индукции гаплоидии.
Пример 2. Определение последовательностей gRNA для редактирования генов CENH3α в геноме Fielder
У гексаплоидной пшеницы имеется два гена CenH3 - TaCen3α и TaCenH3β. Копии обоих генов из геномов А, В и D были клонированы в сорте пшеницы "Fielder" с праймерами, разработанными для геномной последовательности сорта "Chinese Spring_v2". Последовательности приведены в (SEQ ID NO: 1-12). Предыдущие исследования показали, что вирус-индуцированный сайленсинг гена ("VIGS") TaCenH3α приводил к карликовости и недоразвитости корневой системы, тогда как сайленсинг TaCenH3β приводил к снижению степени завязывания семян (Yuan et al., New Phytol. 206(2):839-51. 2015). Как и в случае с большинством генов пшеницы, специфические паттерны экспрессии и функции каждого из гомологов из геномов А, В и D являются недостаточно хорошо изученными. Для редактирования генома авторы настоящего изобретения решили модифицировать TaCenH3α у яровой пшеницы сорта Fielder, полагая, что мутации в данном гене не должны оказывать такое значительное влияние на завязывание семян, как мутации в TaCenH3β. Специфичные для гомологов qPCR-анализы TaqMan применяли для сбора информации об уровне экспрессии TaCENH3α-A, -В и -D (SEQ ID NO: 13-21) в репродуктивных тканях (пыльце, завязи и пыльнике), а также ткани молодых листьев. TaCenH3α-А и -В экспрессировались на высоких уровнях в пыльниках, пыльце и завязях, тогда как экспрессионный транскрипт TaCenH3α-D практически отсутствовал (таблица 2). В листе TaCenH3α-А был преобладающим транскриптом, что может указывать на то, что потеря функции данного гена способствует фенотипу карликовости после сайленсинга TaCenH3α.
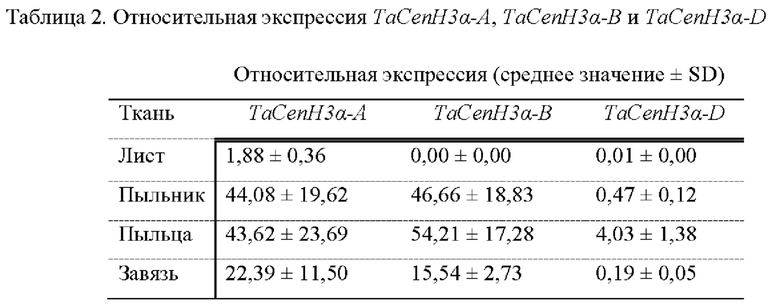
Направляющие РНК подбирались с использованием РАМ-сайтов NGG и согласно сделанным авторами настоящего изобретения предсказаниям типов аминокислотных последовательностей, которые будут приводить к получению белкового продукта CENH3, если определенные редактирования будут произведены в этих сайтах-мишенях. Важно, что большая часть направляющих РНК, которые рассматривались авторами настоящего изобретения, не была способна производить комбинации редактирований, которые приводили бы к получению RFS- или AS-аллелей. Авторы настоящего изобретения сосредоточились на отборе немногих пар gRNA, которые предположительно могли бы это сделать. Направляющая РНК 1 (ACGTCGGCGACACCGGTGCG; SEQ ID NO: 25) (подчеркнут примерный сайт разрезания с образованием двухнитевого разрыва, индуцированного комплексом CRISPR-Cas9) локализуется в области границы экзона 2 и интрона 2. Данная gRNA1 находилась под управлением промотора TaU6. Направляющая РНК 2 (CTTGTGGGAGCAGGGGCAAC; SEQ ID NO: 26) нацеливается на область, расположенную сразу после границы интрона 3 и экзона 4, под управлением TaU6. Направляющая РНК 2 в большинстве случаев не будет редактировать 3'-акцепторный сайт сплайсинга интрона 3. Был сделан выбор в пользу использования двух направляющих РНК, так что авторы настоящего изобретения могли производить значительные изменения, например, RFS, LD или AS-аллели, в N-концевом домене, оставляя в то же время С-концевой домен в рамке считывания. Например, в некоторых растениях и отредактированных аллелях обе gRNA будут производить разрезание одновременно, приводя к делеции вставочной последовательности. В некоторых случаях в ходе происходящей в результате репарации образуется сдвиг рамки считывания, который приведет к нокауту белка. В других случаях будет образовываться укороченный LD-транскрипт, у которого отсутствуют интрон 2, весь экзон 3 и часть экзона 4, с удалением примерно аминокислотной последовательности RAGRAAAPGGAQGA (SEQ ID NO: 76) из белкового продукта, что составляет значительное изменение N-концевого домена.
В качестве альтернативы, неодновременное разрезание в обоих сайтах может приводить к образованию сдвига рамки считывания в сайте для gRNA1 (например, любой мутации по типу вставки/делеции, которая встречается в кодирующей последовательности и имеет размер, не кратный 3), восстановление которого происходит в сайте для gRNA2 посредством дополняющей мутации по типу вставки/делеции, что таким образом приводит к возвращению кодирующей последовательности в ее нормальную рамку считывания. Например, делеция размером 1 нуклеотид ("нт") в сайте для gRNA1 и вставка размером 1 нт в сайте для gRNA2 восстанавливают рамку считывания, что приводит к образованию RFS-аллеля. Данный аллель, по всей вероятности, не будет аллелем с потерей функции при предположении о том, что во вставочной последовательности со сдвигом рамки считывания не образуются стоп-кодоны. На основании оценки авторами настоящего изобретения потенциальных изменений можно видеть, что по меньшей мере одна комбинация редактирований будет приводить к образованию RFS между сайтами для gRNA1 и gRNA2, который не имеет какого-либо стоп-кодона во вставочной последовательности. Таким образом, авторы настоящего изобретения могут предсказать, какие пары небольших мутаций по типу вставки/делеции в сайтах для gRNA1 и gRNA2 будут в комбинации давать функциональный RFS-аллель.
Две направляющие РНК могут приводить к образованию аллелей, подвергнутых альтернативному сплайсингу ("AS"), которые также могут выступать в качестве RFS-аллелей или аллелей с большой вставкой. Направляющая РНК 1 производит разрезание между G и Т (SEQ ID NO: 25; подчеркнуто выше). Это 5'-донорный сайт сплайсинга на конце экзона 2. AS-аллели могут образовываться в случае модификации GT таким образом, что сплайсинг интрона 2 происходит ненадлежащим образом, что приводит к удержанию интрона 2 в кодирующей последовательности. При трансляции рибосома будет осуществлять сквозное прочитывание этого интрона и создавать новую вставку из 44-47 аминокислот в зависимости от природы мутаций по типу вставки/делеции в сайтах для gRNA1 и gRNA2. Эта новая вставка может быть предсказана путем прочитывания новой кодирующей рамки считывания после учета фактора мутаций по типу вставки/делеции и трансляции новой зрелой мРНК. Например, если gRNA1 и gRNA2 создают вставки одиночного нуклеотида А в обоих сайтах TaCENH3α-A, транскрипт может быть подвергнут альтернативному сплайсингу, что приводит к вставке аминокислотной последовательности
"VARDLPGSLPFRFVLFSVFWSDLLVTCSTECRGEPGGRRPQGGLKGQ" (SEQ ID NO: 77) с удалением WT-последовательности ''RRAGRAAAPGGAQGA" (SEQ ID NO: 76) из экзона 3 перед восстановлением нормальной последовательности с помощью мутации, опосредованной gRNA. Аналогично, можно подобным образом предсказать, что другая мутация в сайте для gRNA1 (например, делеция GTG) в комбинации с мутацией в сайте для gRNA2 (делецией С) вызывают альтернативный сплайсинг с образованием новой вставки последовательности "GTFPGRFLFVSSCFLFFGLTCSSPVRRNAEASRAGGGPRGGSRG" (SEQ ID NO: 78) и удалением нативной последовательности "RAGRAAAPGGAQGA" (SEQ ID NO: 76). Авторы настоящего изобретения также могут предсказать, что другие комбинации мутаций в сайтах для gRNA1 и gRNA2 будут приводить к образованию сдвигов рамки считывания, которые не подвергаются возвращению в рамку считывания, что приводит к появлению аллелей с потерей функции. Подобным образом, аллели, подвергнутые альтернативному сплайсингу, индуцированному в других сайтах N-концевой области CenH3, будут неспособны производить модифицированные последовательности мРНК с восстановленной до нормального состояния аминокислотной последовательностью С-концевого домена, поскольку там будут присутствовать стоп-кодоны, образовавшиеся в удержанных интронах или после пропуска одного или нескольких экзонов. Следовательно, gRNA могут быть специально отобраны по их способности производить большие изменения в N-концевом домене, оставляя при этом С-концевой домен нетронутым и нормально транслируемым.
Пример 3. Разработка конструкций и трансформация растений
После клонирования специфических последовательностей TaCENH3α-A, -В и -D в сорте Fielder с праймерами, разработанными для геномной последовательности сорта Chinese Spring, авторы настоящего изобретения отобрали gRNA в соответствии с РАМ-сайтами. gRNA1 (ACGTCGGCGACACCGGTGCG; SEQ ID NO: 25) локализуется в области границы экзона 2 и интрона 2 (фигура 1). gRNA2 (CTTGTGGGAGCAGGGGCAAC; SEQ ID NO: 26) нацеливается на область, расположенную сразу после границы интрона 3 и экзона 4. Ген SpCas9 был кодон-оптимизированным для пшеницы с помощью двух NLS на обоих концах и находился под управлением промотора и двух энхансеров Ubi сахарного тростника. Кассеты gRNA, содержащие промотор U6 пшеницы и каркасные последовательности gRNA, были синтезированы в GenScript (www.genscript.com) и клонированы в бинарный вектор - конструкцию 24194 (SEQ ID NO: 74).
Для трансформации использовали Fielder - инбредный сорт яровой пшеницы. Незрелые зародыши диаметром приблизительно 2,0-2,5 мм собирали, стерилизовали 70% этанолом в течение 1 мин и 1% гипохлоритом натрия в течение 10 мин. После стерилизации незрелые зародыши выделяли с помощью скальпеля и лабораторного шпателя в небольшую пробирку и центрифугировали при 20000 × g при 4°С в течение 10 мин в среде для инокуляции. Выделенные зародыши инфицировали Agrobacterium в течение 5 мин, затем переносили в среду для совместного культивирования при 23°С в темноте в течение 2 дней. Зародышевую ось вырезали из незрелых зародышей перед переносом в среду для покоя, культивировали при 25°С в темноте в течение 5 дней, затем переносили в селективную среду, содержащую маннозу в концентрации 15 г/л. См. Y. Ishida, et al., Wheat (Triticum aestivum L.) Transformation Using Immature Embryos, Methods in Molecular Biology 1223: 189-198 (2015). Через 4 недели интенсивно растущие каллюсы переносили в среду для регенерации для образования зеленых растений. Выжившие растения проходили контроль по методу TaqMan, с помощью которого анализировали присутствия или отсутствия сегментов ДНК из трансгенной вставки ДНК; среди них в теплицу были отправлены только растения, положительные согласно анализам по методу TaqMan в отношении 35S и PMI.
Пример 4. Секвенирование у растений Е0, подвергнутых редактированию
Геномную ДНК выделяли из молодых листьев растений Е0, положительных согласно TaqMan. Секвенирование осуществляли с помощью высокоточной ДНК-полимеразы, а именно KOD-Plus-Neo (поставщик: TOYOBO Life Science). Использовали аллель-специфические праймеры для TaCenH3α-А (FA, SEQ ID NO: 50; R3, SEQ ID NO: 51). ПЦР осуществляли следующим образом: 5 мин при 95°С; 35 циклов по 30 с при 95°С, 30 с при 65°С, 1 мин при 68°С; 10 мин при 68°С. Реакционная смесь для ПЦР содержит 11,5 мкл дистиллированной воды, 2,5 мкл 10Х ПЦР-буфера для KOD-Plus-Neo, 1 мкл 2 мМ dNTP, 1 мкл 25 мМ MgSO4, 1 мкл ДНК-полимер азы KOD-Plus-Neo, 1 мкл прямого праймера FA (10 мкМ), 1 мкл обратного праймера R3 (10 мкМ) и 1 мкл геномной ДНК. Продукты ПЦР секвенировали напрямую с помощью праймера SQ-1 (SEQ ID NO: 79) или клонировали в клонирующий вектор pEASY-Blunt Zero (Transgen Biotech). M13R (SEQ ID NO: 52) и M13F (SEQ ID NO: 53) использовали для секвенирования по методу молекулярных колоний.
Пример 5. Трансформант пшеницы А004А
Трансформант пшеницы А004А демонстрировал индукцию гаплоидии. Трансформант пшеницы №А004А представляет собой одно из сотен трансгенных растений, которые получали посредством трансформации конструкцией 24194. Анализ по методу TaqMan с последующим прямым секвенированием указывал на то, что генотип по генам TaCENH3α представлял собой AA*BBdd на стадии прорастания Е0. В данном случае заглавная буква указывает на аллель TaCENH3α дикого типа без редактирования, буква нижнего регистра указывает на аллель с потерей функции, и заглавная буква со звездочкой (*) указывает на аллель с восстановлением сдвинутой рамки считывания (RFS), большой делецией (LD) или аллель, подвергнутый альтернативному сплайсингу (AS), что означает, что аллель является предполагаемым индуктором гаплоидии. Аллель А* растения А004А содержит вставку аденина в сайте-мишени для gRNA1 и другую вставку аденина в сайте-мишени для gRNA2 (SEQ ID NO: 56). Вставка аденина в сайте для gRNA1 фактически находится в интроне на 3 п. о. ниже конца экзона 2 и сразу после 5'-границы сплайсинга. Сама по себе она не нарушает кодирующую последовательность, но она может в некоторых случаях изменять паттерн сплайсинга. Вставка аденина в сайте для gRNA2 находится в экзоне 3 и приводит к сдвигу рамки считывания кодирующей последовательности. Предсказание паттерна сплайсинга, индуцированного вставкой аденина в сайт-мишень для gRNA1, указывает на то, что это может быть AS-аллель, который демонстрирует удержание интрона (IR) для интрона №2, поскольку наличие дополнительного аденина после 5'-донорного сайта сплайсинга может изменять инициацию удаления интрона, запуская альтернативный сплайсинг. Альтернативный сплайсинг в данном случае будет приводить к вставке многих аминокислот в экзон 3. При наличии альтернативного сплайсинга анализ результатов указывает на то, что последовательности в экзоне 3 будут находиться вне рамки считывания до тех пор, пока редактирование с помощью gRNA2, являющееся еще одной вставкой аденина, не восстановит нормальную рамку считывания и аминокислотный код для С-концевого домена.
Чтобы удостовериться в том, что аллель А* у А004А подвергнут альтернативному сплайсингу и представляет собой аллель, являющийся предполагаемым индуктором гаплоидии, авторы настоящего изобретения изучили последовательности зрелой мРНК гена CENH3α-A в листе растения А004А Е0 на ювенильной стадии. Общую РНК из молодых листьев экстрагировали с использованием TRIzol от INVITROGEN, следуя инструкциям производителя. кДНК синтезировали из 1 мг общей РНК с помощью системы для синтеза первой нити Superscript III (Invitrogen) с праймером олиго-dT. KOD-Plus-Neo (TOYOBO) использовали для амплификации транскриптов TaCenH3α-А с праймерами F1 (SEQ ID NO: 54) и R1 (SEQ ID NO: 55). ПЦР осуществляли в соответствии с инструкциями производителя и следующим образом: 5 мин при 95°С; 35 циклов по 30 с при 95°С, 20 с при 62°С, 20 с при 68°С, 35 циклов; 10 мин при 68°С. Реакционная смесь для ПЦР содержит 11,5 мкл дистиллированной воды, 2,5 мкл 10Х ПЦР-буфера для KOD-Plus-Neo, 1 мкл 2 мМ dNTP, 1 мкл 25 мМ MgSO4, 1 мкл KOD-Plus-Neo, 1 мкл праймера F1 (10 мкМ), 1 мкл праймера R1 (10 мкМ) и 1 мкл кДНК. Продукт ПЦР очищали с помощью набора для очистки продуктов ПЦР GeneJET (Thermo Scientific) и клонировали в клонирующий вектор pEASY-Blunt Zero (Transgen Biotech). Праймеры M13R и M13F использовали для секвенирования по методу молекулярных колоний. Несколько клонов на один продукт ПЦР секвенировали и анализировали с использованием программного обеспечения Vector NTI (Invitrogen). Относительную экспрессию сплайс-вариантов рассчитывали по числу клонов. Анализ ПЦР-секвенирования по методу молекулярных колоний указывал на то, что мРНК TaCENH3α-A в А004А имеет два транскрипта, что свидетельствует об альтернативном сплайсинге. Один из транскриптов (SEQ ID NO: 58), обнаруживаемый в 8 из 18 (44%) колоний, был подвергнут сплайсингу с использованием канонического 5'-сайта сплайсинга. Для этих транскриптов нормальный сплайсинг интрона 2 означает, что редактирование с помощью gRNA1 не влияло на трансляцию аминокислот со зрелой мРНК; однако редактирование с помощью gRNA2 вызывало сдвиг рамки считывания. Таким образом, в этом случае зрелые мРНК, подвергнутые конститутивному сплайсингу, фактически представляют собой нокаутные транскрипты. С другой стороны, 10 из 18 (56%) колоний имели зрелые мРНК-транскрипты с удержанным интроном 2 (SEQ ID NO: 57), что приводило к появлению аллеля с N-концевым RFS, который содержал 47 новых аминокислот - так, 32 аминокислоты были вставлены из интрона 2, а также 15 аминокислот были изменены вследствие сдвига рамки считывания в экзоне 3. Данная вставка 32 а.к. и изменение в целом 47 а.к. существенно изменяет N-концевой домен белков, полученных в результате трансляции мРНК с RFS. Важно, что последовательность зрелой мРНК, которую авторы настоящего изобретения получили в AS-аллеле TaCENH3α-A* у А004А, является точным результатом прогнозирования авторами настоящего изобретения последовательности зрелой мРНК, который должен был произойти. Растение А004А Е0 поддерживали в течение цветения. Авторы настоящего изобретения не наблюдали каких-либо аномальных фенотипов, и семена Е1 (после самоопыления) были собраны с колосьев, образованных данным растением.
Проверяли уровень плоидности семян-потомков Е1, полученных при самоопылении растения А004А. Семена высаживали, и из проросших всходов отбирали образцы и анализировали в отношении содержания ДНК с помощью проточной цитометрии. Гаплоиды получали в первой партии высеянных растений-потомков: частота индукции гаплоидии ("HIR") составляла 3,8%. Во второй партии всходов также наблюдались гаплоиды, и HIR составляла 4,2%. Гаплоиды пшеницы, подобно гаплоидам риса и кукурузы, имеют меньший размер, чем соответствующие им диплоиды. Важно, что эти растения либо не имели нокаута обеих копий аллеля "В", либо не имели восстановления сдвинутой рамки считывания, индуцированного мутацией в сайте-мишени для gRNA2. Это позволяет предположить, что только правильная комбинация редактирований в обоих сайтах-мишенях для gRNA1 и gRNA2 в гене CENH3α-A вкупе с нокаутом обеих копий аллеля "В" является достаточной для запуска индукции гаплоидии.
В первой партии авторы настоящего изобретения наблюдали два растения, являющихся проростками-близнецами; оба проростка были гаплоидами согласно проверке с помощью проточной цитометрии. Это указывает на то, что в одном семени имеется два гаплоидных зародыша. Образование зародышей-близнецов может быть вызвано нарушением развития семязачатков, которое отчасти может запускаться редактированиями в CENH3α, хотя для подтверждения этого необходимо больше экспериментальной работы.
Хотя авторы настоящего изобретения были немного удивлены, что они наблюдали гаплоидов после самоопыления (поскольку обычно гаплоиды индуцируются только при модификациях CENH3 в процессе ауткроссинга), непрерывная возможность редактирования в растениях Е0 означает, что мужские и женские половые клетки могут унаследовать разные последовательности (редактирования) и, таким образом, обладать разными по сравнению друг с другом свойствами связывания с центромерами и строением кинетохора, что приводит к гаплоидии после самоопыления.
Пример 6. Трансформант пшеницы С003А
Растение С003А подвергнуто редактированию таким образом, что генотип TaCenH3α представляет собой A*abbdd на стадии растения Е0. А* вводится путем делеции гуанина в сайте для gRNA1 и вставки аденина в сайте для gRNA2. На уровне белка наблюдается отличие в одиннадцать аминокислот в N-концевом домене по сравнению с последовательностью дикого типа. Под SEQ ID NO: 63-65 показаны соответственно геномная последовательность CenH3 А*, CDS-последовательность А* и последовательность белка А* для С003А.
Семена Е1 получали путем самоопыления у растения С003А Е0. Растения Е1 с генотипом A*A*bbdd выращивали в теплице для определения их способности к индукции гаплоидии при ауткроссинге. Растение дикого типа (тестерная линия 03S0352-22) было выбрано в качестве донора пыльцы. У С003А Е1 вручную удаляли несозревшие пестики, и его опыляли пыльцой от растений дикого типа. Гаплоиды выявляли с помощью маркеров SNP (SEQ ID NO: 29-43), по которым можно определить различия между Fielder и 03S0352-22, впоследствии подтверждаемые проверкой с помощью проточной цитометрии. Среди 208 растений F1 авторы настоящего изобретения получили один гаплоид. Это демонстрировало наличие только отцовских генотипов по четырем маркерам и вместе с тем материнского генотипа по одному маркеру (KW11091).
Пример 7. Трансформант пшеницы А073А
Растение А073А имело генотип AA*B*bdd по TaCENH3α на стадии прорастания Е0. Аллель А* в А073А имел вставку аденина, вызванную gRNA3, и делецию гуанина, вызванную gRNA4, в геномной ДНК. Это приводит к запуску восстановления сдвинутой рамки считывания на уровне белка с отличием в тридцать одну аминокислоту между вариантом дикого типа и отредактированным вариантом N-концевого домена. Данное растение было отмечено в качестве потенциального предшественника линий, являющихся индукторами гаплоидии, благодаря его способности к образованию потомства, которое имело генотип A*A*bbdd - идеальную генетическую комбинацию для запуска индукции гаплоидии. Под SEQ ID NO: 69-71 показаны соответственно геномная последовательность CenH3 A*, CDS-последовательность А* и последовательность белка А* для А073А.
Семена Е1 получали путем обеспечения возможности самоопыления у растения А073А Е0. Растения Е1 подвергали секвенированию, и тех, которые имели генотипическую комбинацию AA*bbdd, отбирали для дальнейшего выращивания в теплице для определения способности к индукции гаплоидии при ауткроссинге. Используя тестерную линию 03S0352-22, выбранную в качестве донора пыльцы, у растений Е1, подвергнутых редактированию, вручную удаляли несозревшие пестики, и их опыляли ручным способом. Гаплоиды выявляли с помощью маркеров SNP, по которым можно провести генотипические различия между Fielder и 03S0352-22. Предполагаемые гаплоиды, идентифицированные по гомозиготности по этим маркерам, затем были подтверждены путем проверки общего содержания ДНК с помощью проточной цитометрии. Среди 57 растений F1 53 были преимущественно гетерозиготными по маркерам SNP и были диплоидными согласно проверке плоидности, что указывает на то, что они были гибридами. В противоположность этому, четыре растения имели только отцовские геномные маркеры SNP и были гаплоидами согласно данным проточной цитометрии, что было эквивалентно частоте индукции гаплоидии при ауткроссинге, составляющей 7%.
Растения Е1 с генотипом AA*bbdd также давали гаплоиды Е2 в процессе самоопыления (таблица 3). Авторы настоящего изобретения наблюдали один гаплоид среди 13 растений Е2 (частота индукции гаплоидии 7,7%). В то же время авторы настоящего изобретения наблюдали несколько растений с частичной элиминацией хромосом.


--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Syngenta Participations AG
Syngenta Biotechnology China Co., Ltd.
Lv, Jian
Yu, Kun
Wei, Juan
Liu, Chunxia
Zhou, Hongju
Kelliher, Timothy
<120> NOVEL WHEAT CENH3 ALLELES
<130> 81696-WO-REG-ORG-P-1
<150> PCT/CN2018/110063
<151> 2018-10-12
<160> 79
<170> PatentIn version 3.5
<210> 1
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 1
agcgaatgaa acaacacacg 20
<210> 2
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 2
gaagtcggtg acctccttga 20
<210> 3
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 3
caaggaggtc accgacttct 20
<210> 4
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 4
gccttgcaag ctgtatgtcc 20
<210> 5
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 5
actagacgag tcgggacgaa 20
<210> 6
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 6
gaagtcggtg acctccttga 20
<210> 7
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 7
caaggaggtc accgacttct 20
<210> 8
<211> 18
<212> DNA
<213> Triticum aestivum
<400> 8
acgcctcgcg agctgtat 18
<210> 9
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 9
gggtccaaga aagacacacg 20
<210> 10
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 10
gaagtcggtg acctccttga 20
<210> 11
<211> 20
<212> DNA
<213> Triticum aestivum
<400> 11
caaggaggtc accgacttct 20
<210> 12
<211> 18
<212> DNA
<213> Triticum aestivum
<400> 12
acgcctcgcg agctgtat 18
<210> 13
<211> 17
<212> DNA
<213> Artificial sequence
<220>
<223> primer/probe
<400> 13
ggttcaggcc aggcacg 17
<210> 14
<211> 19
<212> DNA
<213> Artificial sequence
<220>
<223> primer/probe
<400> 14
caaatggtgc aaacgggat 19
<210> 15
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 15
aggtatcaga agtcg 15
<210> 16
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 16
gtcgttagaa agtattgtag gtgtatcatt 30
<210> 17
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 17
ctgaatgcaa aagtcgaatg atc 23
<210> 18
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 18
tagatgtgtc ttcaaagtt 19
<210> 19
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 19
cgttcgactc gctggagtag t 21
<210> 20
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 20
cctgccgatt gtttgtttta ctg 23
<210> 21
<211> 24
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 21
tagtagtaac cgcgcctccc gcac 24
<210> 22
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 22
tcgatgccgc tggtaagac 19
<210> 23
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 23
tccgatggtt gggatggt 18
<210> 24
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> primer/probe
<400> 24
ctacaagctc aagctcggag agatcgtca 29
<210> 25
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> sgRNA targetting exon2-intron2 junction
<400> 25
acgtcggcga caccggtgcg 20
<210> 26
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> sgRNA targetting intron3-exon4 junction
<400> 26
cttgtgggag caggggcaac 20
<210> 27
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> sgRNA targetting exon1
<400> 27
gccttggtct tcctgacggc 20
<210> 28
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> sgRNA targetting intron2-exon3 junction
<400> 28
gacggaatgc agaggcgagc 20
<210> 29
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 29
ggatcttgct ctgcctgcct gtt 23
<210> 30
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 30
gcagctcatt tcagttacaa acttag 26
<210> 31
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 31
gcagctcatt tcagttacaa acttac 26
<210> 32
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 32
tcggagttca ctattgcctc aggaa 25
<210> 33
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 33
gcccatcact tggctgatct ctt 23
<210> 34
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 34
cccatcactt ggctgatctc tc 22
<210> 35
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 35
cggaccagtt aatgtgattc gacagat 27
<210> 36
<211> 27
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 36
aaatcagcaa acccaaaatg gcatgat 27
<210> 37
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 37
atcagcaaac ccaaaatggc atgac 25
<210> 38
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 38
gtggttgttc tgctcggcca ct 22
<210> 39
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 39
cagactgaat caagtgcatc cct 23
<210> 40
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 40
cagactgaat caagtgcatc ccc 23
<210> 41
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 41
tgttttcctt ggcgctggcg atttt 25
<210> 42
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 42
cagagacaag aatcaggagc a 21
<210> 43
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 43
ctcagagaca agaatcagga gcg 23
<210> 44
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 44
gacagtggtc ccaaagatgg a 21
<210> 45
<211> 22
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 45
cgtggttgga acgtcttctt tt 22
<210> 46
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> probe
<400> 46
ccccacccac gaggagcatc g 21
<210> 47
<211> 19
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 47
ccctatcccc gttgacgac 19
<210> 48
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 48
ctgatagtgg tctccttgtc gct 23
<210> 49
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> probe
<400> 49
ttcgccttca gcctgcacga cct 23
<210> 50
<211> 31
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 50
caaaacagat aaaacaaaat gtagggaatg t 31
<210> 51
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 51
ggtgcaaacg ggatgagaaa gtc 23
<210> 52
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 52
gtaaaacgac ggccagt 17
<210> 53
<211> 17
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 53
caggaaacag ctatgac 17
<210> 54
<211> 15
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 54
tggcccgcac caagc 15
<210> 55
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 55
gctgtatgtc cttttgcatg acg 23
<210> 56
<211> 4401
<212> DNA
<213> Triticum aestivum
<400> 56
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgccgcccaa gaagcagctc 60
gggccccgcc ccgcgcagcg gcggcaggag acaggtcagc ccttccctcc ttcctccccc 120
cgtctcccgc ctctcgagcg aacgccgtcg ccatttcgtc gcgagagatg gacggacgga 180
cgccccacgc atgacgctaa tgtgctcttc ctctcccttt gcagatggcg cgggcacgtc 240
ggcgacaccg gtagcgcggg accttcccgg gtcgcttcct tttcgtttcg tcttgttttc 300
tgttttttgg tctgacttgc tcgtcacctg ttcgacggaa tgcagaggcg agccgggcgg 360
gcggcggccc cagggggggc tcaaggtgcg gccttctttg cgcttttcgg ttttccgccg 420
cgtgtttagg gccatttccg tcttgtttgg gggtgcgcgg ggcgggggct tgtttttttt 480
cctcccccct tcgttgttgc gcacattgct cgggaatgct gccaggagcg gttgcggttc 540
ttctttgacc cttcgggagg gctggatcgg cagtttcttc gcttccttgc tccagatttt 600
agttcatctt gtaccagtac agtagcaaga tgatggatgg gggcttgttt tttctttctc 660
cttccttgtt ccggacattt ctctggagcg acttttatgc atttcccagt attgtccttt 720
gtccttagag ggtagtggat cggcagtttt cttcgcctca ttggtccaga ttttagttta 780
tcttgtacgg taccaagatg atggatgtgg cacaaagttc tcagtttggg ggttgcgctc 840
ttccgggcag ttgttatttt ggtctgtgat gactaactcg tatctattct tgtgggagca 900
ggggcaaact gggcaaccca agcagaggaa accacaccgg ttcaggccag gcacggtggc 960
actgcgggag atcaggaggt atcagaagtc ggtcgacttt ctcatcccgt ttgcaccatt 1020
tgtccgtctg gtgggtacct ctgtctgtca tatcctctcg ctctctctac aaacgatctg 1080
cagtgcagag tgtaattgga atattttgtt cctgacaaat ttgcagatca aggaggtcac 1140
cgacttcttc tgtcctgaaa tcagccgctg gactccccaa gcgctcgtcg cgattcaaga 1200
ggtcagtgct aaaacctggc atgtactatt agatctgatg gtttgattag agtactacaa 1260
tgcagatgaa ttcaatatcc gaaaaccatg aactgtgggg tagatacatg tatcgcctta 1320
attcatggtt tctgaatgct ctgctattaa ttcagtttga tatatttatt tagcagcatg 1380
gtattgtttt ggtggctggt aaatcaaaac tgaaatgtga ttacgagcaa aacggtatcg 1440
attgtcgatc ctgtgtgttt ttgtgcacat ctggttgttt ggtcaagatg tgtttgtgca 1500
catcttgcaa catgatcctg cccacacact caaaactgac tattggttag gttccatttg 1560
tcttatggaa tttagggtgt aactgagagg tgagcaagtg gtagtaacgt tcaattttga 1620
ttcaggatga ggatattgtg atccagaaaa ttgcatgtta tggttatgtg tccaaacgcc 1680
aaatgattat gtctatatcc agtactttag aaccagtaca acaacaaaaa gtactttaga 1740
accagtcaag tttattgtgc atttatacaa gagtgttgtt tgcacaatag acttgcttta 1800
gtcgtctctt gccagaaatg ccttcttctg cacaacgagc aaaaataaca taaagttgac 1860
tatactcagt gtggcctaag aaatgagttg tactttttag tcactggcct gtgtatttgc 1920
tttgaattga cacataattc ccttttcctt tccctctgca ggctgcagag tatcacctcg 1980
tcgacgtatt tgaaagggca aatcactgtg ccatccatgc aaagcgtgtt accgtcagta 2040
agttctcact gaatgaaaac tccctttctt ttacaatatt gcgcagaagg aaacatgcca 2100
gttatgaaag agtttcaatt acaggatcac ctttgctttc atttgatgtg atatctagtt 2160
ttgatgttgt ttcaaggttc aagaattcta atgataaatg ataggatcca caattgttat 2220
atctctgcag ctcctcgtat ctgttgtcca cgaacaaaca tatcaaacaa ttcattaaaa 2280
agatgaagaa gtcaaacaaa cagtatatgt gcactgcata ttagatatca aacctggtta 2340
ctatacgact gatctggcct gacccccgcg tcgcctctcc ctggcggcac ggggggaacc 2400
actccggcgc cgccaccttc cctcctccct ccacccccca cctcgccgcc gcctgaggag 2460
ttcgccggcg aagcccggtc tggctccagg gagggtggcg gcggggcatc tctctgcgag 2520
gcgtgagggc gcatctcgtg cgcgggcgcg gcgagcttgg gcgggatcgc gggcgcggct 2580
ctggctcggg ctggtgaggc tccggcggtc cgaggggtgt cgcggcgcgg cgggggtctc 2640
gctccggcgc gggcggtccg agggcgcgcg ggatctggcg ctccagcagg ggcgccgtca 2700
aggggagccg ggcagggaag ctcgtcaggc gggctcgtca ggcaggcgtg acgcggggcg 2760
gcggccgcgg gctggaagcc atggcgtttt ggccatggtg tcgtgtgcgc tcgcttctca 2820
ttcgcagctt gaggtgctgc tgctgagggt ggtgcgcggt gaagcttggt ggtcggcgtt 2880
ggagagtgca aggcgcaaca ggaatggctc caatgatctc cacgcttctg ggtggatcag 2940
atctgcggcc ctccataggg gtgtgttccg ggcgaaagcc ttgacccgac tttgtcggtg 3000
ccgtcgacgg cggcgctttc gggcgtcgtt tccctccttg gaggcgtcgt tgtggaactc 3060
atcttcttct atgtggggct cgggctctcc gggtgaaaac ctaagctcca gattttccgg 3120
agcgggcgat ggcggcgtct tcgtcgtttc cctcttgggg gcgttgcttg ggagagttag 3180
cttgtgcttg gtgcgtttgg ttttctccta cgtcgggttt ggtggatgcc ggggcagcgg 3240
ccccggacgg ctgatgaacg ccgaggcggc ggcctcggaa agtgatgcgc ggtgcggctc 3300
catggggcgg ggcggtggct cggccttcac ttgggtggca atcttgggcc acttgggtgg 3360
caggcttgtc ggtccggtcg acgcgttcca gagggggcgg tctgactttg cgtcggggcg 3420
gcggccccgg atgtggtgcg acgttcgtgg tctgcgagcg gttgccttga gcagcgtggg 3480
ctgcgggcag ctgggtcgcg cggcgttgct gctcgagcgg agcggtggta cgtcggggcg 3540
gcggccccgg aaggtgatgc cggttgattg cgctgggcgt gacggagcgg tggatgtcgg 3600
ggcggcggcc ccgagaaaat cactgtggcg tccagatttc tgtggcaacg atgatggtgg 3660
gagcgatgtc ggcgacgcgg caatggttgc gatagtcggc tcttctccgg cgtgtccacg 3720
atattgcctc ggtttgtttg ttgctgtgga gtcgaagctg cggcggcgag gccctgtggt 3780
atacgatgac tggttccagg tgtcctttcg tcgatcttcc gtggcgccag ccgtgcctgg 3840
tttcgttctt ccgagttctc cgtcagaatc ggagctgcgt tgtctgtccg caggtcgaca 3900
tgttgtcgag aagggtgggc tttgccctgt gtgtttcagt ctatgcgagt gggctcggcc 3960
cttgttgttc tggtttttgc ccggttttcc gtaattaact gggcaattct cttctgctta 4020
attaatagat gaggcaatct ttgcctccct ttcaaaaaaa aacctggtta ctatagcagg 4080
aaattcaggg ttgattactt tatttcttat ctgaaggata aacattgtat caaatcagaa 4140
ttttatttgt aagttacatt tttttttact tataaaactt ggaaactgtt ttactgtgac 4200
aaatagatgc cactagaatc atgatcacat cgtggctgtt gctattctaa caaataaatg 4260
ctcctgaaca aatgggaact atatatgaag atgtatggac cagcatgttc ctgttaacct 4320
gacctttttc ctttttttgc tgctgcagtg caaaaggaca tacagcttgc aaggcgtatc 4380
ggcgggagga ggctttggtg a 4401
<210> 57
<211> 588
<212> DNA
<213> Triticum aestivum
<400> 57
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgccgcccaa gaagcagctc 60
gggccccgcc ccgcgcagcg gcggcaggag acagatggcg cgggcacgtc ggcgacaccg 120
gtagcgcggg accttcccgg gtcgcttcct tttcgtttcg tcttgttttc tgttttttgg 180
tctgacttgc tcgtcacctg ttcgacggaa tgcagaggcg agccgggcgg gcggcggccc 240
cagggggggc tcaaggggca aactgggcaa cccaagcaga ggaaaccaca ccggttcagg 300
ccaggcacgg tggcactgcg ggagatcagg aggtatcaga agtcggtcga ctttctcatc 360
ccgtttgcac catttgtccg tctgatcaag gaggtcaccg acttcttctg tcctgaaatc 420
agccgctgga ctccccaagc gctcgtcgcg attcaagagg ctgcagagta tcacctcgtc 480
gacgtatttg aaagggcaaa tcactgtgcc atccatgcaa agcgtgttac cgtcatgcaa 540
aaggacatac agcttgcaag gcgtatcggc gggaggaggc tttggtga 588
<210> 58
<211> 493
<212> DNA
<213> Triticum aestivum
<400> 58
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgccgcccaa gaagcagctc 60
gggccccgcc ccgcgcagcg gcggcaggag acagatggcg cgggcacgtc ggcgacaccg 120
aggcgagccg ggcgggcggc ggccccaggg ggggctcaag gggcaaactg ggcaacccaa 180
gcagaggaaa ccacaccggt tcaggccagg cacggtggca ctgcgggaga tcaggaggta 240
tcagaagtcg gtcgactttc tcatcccgtt tgcaccattt gtccgtctga tcaaggaggt 300
caccgacttc ttctgtcctg aaatcagccg ctggactccc caagcgctcg tcgcgattca 360
agaggctgca gagtatcacc tcgtcgacgt atttgaaagg gcaaatcact gtgccatcca 420
tgcaaagcgt gttaccgtca tgcaaaagga catacagctt gcaaggcgta tcggcgggag 480
gaggctttgg tga 493
<210> 59
<211> 195
<212> PRT
<213> Triticum aestivum
<400> 59
Met Ala Arg Thr Lys His Pro Ala Val Arg Lys Thr Lys Ala Pro Pro
1 5 10 15
Lys Lys Gln Leu Gly Pro Arg Pro Ala Gln Arg Arg Gln Glu Thr Asp
20 25 30
Gly Ala Gly Thr Ser Ala Thr Pro Val Ala Arg Asp Leu Pro Gly Ser
35 40 45
Leu Pro Phe Arg Phe Val Leu Phe Ser Val Phe Trp Ser Asp Leu Leu
50 55 60
Val Thr Cys Ser Thr Glu Cys Arg Gly Glu Pro Gly Gly Arg Arg Pro
65 70 75 80
Gln Gly Gly Leu Lys Gly Gln Thr Gly Gln Pro Lys Gln Arg Lys Pro
85 90 95
His Arg Phe Arg Pro Gly Thr Val Ala Leu Arg Glu Ile Arg Arg Tyr
100 105 110
Gln Lys Ser Val Asp Phe Leu Ile Pro Phe Ala Pro Phe Val Arg Leu
115 120 125
Ile Lys Glu Val Thr Asp Phe Phe Cys Pro Glu Ile Ser Arg Trp Thr
130 135 140
Pro Gln Ala Leu Val Ala Ile Gln Glu Ala Ala Glu Tyr His Leu Val
145 150 155 160
Asp Val Phe Glu Arg Ala Asn His Cys Ala Ile His Ala Lys Arg Val
165 170 175
Thr Val Met Gln Lys Asp Ile Gln Leu Ala Arg Arg Ile Gly Gly Arg
180 185 190
Arg Leu Trp
195
<210> 60
<211> 106
<212> PRT
<213> Triticum aestivum
<400> 60
Met Ala Arg Thr Lys His Pro Ala Val Arg Lys Thr Lys Ala Pro Pro
1 5 10 15
Lys Lys Gln Leu Gly Pro Arg Pro Ala Gln Arg Arg Gln Glu Thr Asp
20 25 30
Gly Ala Gly Thr Ser Ala Thr Pro Arg Arg Ala Gly Arg Ala Ala Ala
35 40 45
Pro Gly Gly Ala Gln Gly Ala Asn Trp Ala Thr Gln Ala Glu Glu Thr
50 55 60
Thr Pro Val Gln Ala Arg His Gly Gly Thr Ala Gly Asp Gln Glu Val
65 70 75 80
Ser Glu Val Gly Arg Leu Ser His Pro Val Cys Thr Ile Cys Pro Ser
85 90 95
Asp Gln Gly Gly His Arg Leu Leu Leu Ser
100 105
<210> 61
<211> 2618
<212> DNA
<213> Triticum aestivum
<400> 61
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgctgcccaa gaagcagctc 60
gggacgcgcc cctcggccgg gacgccgcgg cggcaggaga caggtgagcc cgcccctcct 120
tccttcctcc cccccatctc gagcgaacgc cgtcgcctcg tcgcgagaga tggacgcccc 180
acgcgtgatg ctaatgtgtt cttcctctcc ctttgcagat ggcgcgggca cgtcggcgac 240
tccggtgcgc gggccttcct gggtcacttc cttttgtttc gtcttgtttt ctgtttttgg 300
tctgacttgc tcgtctcctg ttcgacggaa tgcagaggcg agccgggcgg gcggcggccc 360
caggggcggc tgaaggtgcg cccttctttg tgcttttcgg ttttccgccg cgtgtttagg 420
gctatttccg tcttgtttgg ggcggggact tgtatcttct ccccccttcg ttgttgcgca 480
catttctcgg gaatgctacc aggagcagtt gcggttcttt ggcccttagg gagggctgca 540
ccggcaattt cttcgcttcc ttgctccaga ttttagttta tcttctacag tagcaagatg 600
atggatgggg acttgcgttt ttctttcccc ttccttgttc cgggcatttc tctggagcaa 660
tgtttatgca tttcccagta ttgtcctttg ccctcagagg ggaggggatc ggcagttttc 720
ttcgcttcat cggtccagat tttagtttat cttgtacagt agcaagatga tggatgtgac 780
acaaagttct cagtttgggg gttgcgctct tccgggtagt tgttaatttc gtctgtgact 840
gactcgtatc tattcttgtg ggagcagggg caactgggca acccaagcag aggaagccac 900
accggttcag gccaggcacg gtggcactgc gggagatcag gaagtaccag aaatcggtcg 960
actttctcat cccgtttgca ccatttgttc gtctggtggg tacctctgtc atatcctctc 1020
gctctctcta ccaacgatct ggagtgcgga gtgtaaattg gaatcttttg ttcctgacaa 1080
atttgtagat caaggaggtc accgacttct tctgtcctga aatcagccgc tggactcccc 1140
aagcgctcgt tgcaattcaa gaggtcagtg ctcaaacctg gcatgtacta ttagatctga 1200
tggtttgatt agagtactac aatgcagatg aattcaagat ctgaaaacca tgaactgtgg 1260
ggtagatgca tgtatcatag tgtctgaatg ctctggtatt aattcagttt gatatattta 1320
tttagcagca tggtattgtt ttggtggctg gtaacccaaa actgaaacgt gattacgagc 1380
aaaatggtat caattgtctt ttctgtgtgt ttgtgcaaat ctggttgttt ggtcaagata 1440
tacagtcttg caacatgaat acatgatcct gcccacacac tcaaaactga actattggtt 1500
agatgccata tgtcttatgg aatttagggt gtaactgaga ggtgagcaag tggtagtgac 1560
gttcaatttt gattcaggat gaggatattg tgatccagaa aattgcatgt tatggttatg 1620
tgtccgaacg ccaaatgatt atgtctatat ccactacatg cttatgtgtc ccaaccccca 1680
aatgattatc tctgtttcca gtacaacaac aaaaagtact ttagaaccag tcaagtttat 1740
tgtgcattta tacgagattg ttgtttgcac aatagactta ctttagtcgc ctcttgccag 1800
aaatgccttc ttctgcacaa cgagcaaaaa taacataaag ttgactatac tcagtgtggc 1860
ctaagaaatg agttagtact tttagtcact ggcctgtgta tttgctttga attgacgcat 1920
aattcccttt tcctttccct ctgcaggctg cagagtatca cctcgtcgac gtatttgaaa 1980
gggcaaatca ctgtgccatc catgcaaagc gtgttaccgt cagtaagttc tcactgaatg 2040
aaaacttcct tccatttaca atattatgca gaaggaaaca tgccagttat gaaagagttt 2100
caattacagg atcacctttg ctttcatttg atgtgatatc tagttttgat gttgtttcaa 2160
ggttcaagaa ttctaacgat taatgatagg atccacaatt gttatatctc tgcagctcct 2220
cgtatctgtt gtcaacgaac aaacatgtca aacaattcat taaaaaaaac gaagatgtca 2280
aaccgggtta ctatagcagg aaattcaggg ttgattacct atttattcag ggttgattac 2340
ctatttattt cttatctgaa ggataaacat tatataaaat cagaatttta tttgtaggtt 2400
acattttttg tttccttata aaacttggaa actgttgaga atcatcatca tcatggctgt 2460
tgctattcta acaaataaaa cggatgctcc tgaataaatg gaactatata tgaagatgta 2520
tttactagca tgttcctgtt aacctgactt tttttgctgc tacagtgcaa aaggacatac 2580
agctcgcgag gcgtatcggc gggaggaggc tttggtga 2618
<210> 62
<211> 2676
<212> DNA
<213> Triticum aestivum
<400> 62
atggcccgta ccaagcaccc ggccgtcagg aagaccaagg cgccgcccaa gaagcagctc 60
gggccccgtc ccgcgcagcg gcggcaggag acaggtcagc ccgcccgccc gtcgctcctc 120
ccttccttcc cccccccgtc tcccgcctct cgaccgaacg ccgccgcctt gtttgttgcg 180
gtacagaggt gcccgtccgt gatgctaaca ggttcttcct ctcctttgca gatggcgcgg 240
gcacgtcggc gacaccggtg cgcgggtctt cccgggtcgc ttccttttgt ttcgtcttgt 300
tttctgtttt ttggtctgac ttgctcgtca cctgttcgac ggaatgcaga ggcgagccgg 360
gcgggcggcg gccccagggg gcgctgaagg tgcggccttc tttgcgcttt tcggttttcc 420
gccgcgtgtt tagggccatt tccgtcttgt ttgggggtgg gtggggcggg ggcttgattt 480
ttttcctcct cccttcgttg ttgcgcacat ttctcgggaa tgcttccagg agcggttgca 540
gttcttcttt ggccttaggg agggctggat cggcagtttc ttcgcttcct tgctccagat 600
tttagtttat cttgtagtag tacagtagca agatgatgga tggggacttg ttttttcttt 660
ccccttcctt gttccggaca tttctttgga gcaactttta tgcatttccc ggtattgtcc 720
tttgccctta gagtggagtg gatcggcagt tttcttcgct tccctggtcc agattttagt 780
ttatctttac agtagcaaga tgatgtatgt gacacaaagt tctcagtttg ggggttgcgc 840
tcttccaggt gtagttgtta tttttcgtct gtgactgact cgtatctatt cttgtgggag 900
caggggcaaa ctgggcaacc caagcagagg aagccacacc ggttcaggcc aggcacggtg 960
gcactgcggg agatcaggag gtaccagaag tcggtcgact ttctcatccc gtttgcacca 1020
tttgtccgtc tggtgggtac ctctgtctgt catatcctct cgctctctct accaacaatc 1080
tggagtgcgg ggtgtaaact ggaatctttt gttcctgaca aatttgcaga tcaaggaggt 1140
caccgacttc ttctgtcctg aaatcagccg ctggactccc caagcgctcg tcgcgattca 1200
agaggtcagt gctcaaacct ggcatgtact attagatctg atggtttgat tagagtacta 1260
caatgcagat gaattcaaga tctgaaaacc atgaactgtg gggtagatgc atgtatcgcc 1320
ttaattcata gtttctgaat gctctggtat taattcagtt tgatatattt atttagctgc 1380
atggtattgt tttggtggct ggtaattcaa aactgaaatg tgattacgag caaaatggta 1440
tcaattgtct tttctgtgca catctggttg tttggtcaag atatacagtc ttgcaacatg 1500
atcctgccca cacactcaaa actgaactat tggttagatt ccattttgct tatggaattt 1560
agggtgtaac tgagaggtga gcaggtggca gtgaccagag ctacgttcaa ttttgattca 1620
ggaagaggat cttgcgatcc agaaaattgc atgttatggt tatgtgtccg aaggccaaat 1680
gattatctct atatccagta catgcttatg tgtccgaacc cccaaatgat tatctctata 1740
tccagtacat gcttatgtgt ccgaaccatt caagttattg tgcatttatt caagattgtt 1800
gtttgcacaa tagacttact ttagtcgcct cttgccagaa atgcctcctt ctgcacaatg 1860
agcaaaaata acataaagtt gattatgctc agtgtggcct aattgtactt tcagttactg 1920
gcctgtgtat ttgctttgaa ttgacacata attccctttt cctttccctc tgcaggctgc 1980
agagtatcac ctcgtcgacg tatttgaaag ggcaaatcac tgtgccatcc atgcaaagcg 2040
tgttaccgtc agtaagttct cactgaatga aaacttcctt tcttttacaa tattatgcag 2100
aaggaaacat gccagttatg aaagagtttc aattacaaga tcacctttgc tttcatttga 2160
tgtggtatct agttttgatg ttgtttcaag gttcaagaat tctaatgatt aatgatagga 2220
tccacaattg ttatatctct gcagctcctc gtatctgacg aacaaacatg tcaaacaatt 2280
cattaaaaaa tgaagatgtc aaacaaacag tatatgcact gcatattgga tatcaaacct 2340
ggttactaca gcaggaagtt cagggttgat tactttattt cttatctgaa ggataaacat 2400
tgtatcaaat cagaatttta tttgtaagtt gcattttttg tttactcata aaacttggaa 2460
actgttttac tgtgagaaat agatgcccac tagaatcatg atcatcatca tggctgttgc 2520
tattctaaca aataaaacgg atgctcctga acaaatggaa ctatgtatga agatgtatgg 2580
actagcatgt tcctgttaac ctgacttttt tttgctgcta cagtgcaaaa ggacatacag 2640
ctcgcgaggc gtatcggtgg gaggaggctt tggtga 2676
<210> 63
<211> 4399
<212> DNA
<213> Triticum aestivum
<400> 63
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgccgcccaa gaagcagctc 60
gggccccgcc ccgcgcagcg gcggcaggag acaggtcagc ccttccctcc ttcctccccc 120
cgtctcccgc ctctcgagcg aacgccgtcg ccatttcgtc gcgagagatg gacggacgga 180
cgccccacgc atgacgctaa tgtgctcttc ctctcccttt gcagatggcg cgggcacgtc 240
ggcgacaccg tgcgcgggac cttcccgggt cgcttccttt tcgtttcgtc ttgttttctg 300
ttttttggtc tgacttgctc gtcacctgtt cgacggaatg cagaggcgag ccgggcgggc 360
ggcggcccca gggggggctc aaggtgcggc cttctttgcg cttttcggtt ttccgccgcg 420
tgtttagggc catttccgtc ttgtttgggg gtgcgcgggg cgggggcttg ttttttttcc 480
tccccccttc gttgttgcgc acattgctcg ggaatgctgc caggagcggt tgcggttctt 540
ctttgaccct tcgggagggc tggatcggca gtttcttcgc ttccttgctc cagattttag 600
ttcatcttgt accagtacag tagcaagatg atggatgggg gcttgttttt tctttctcct 660
tccttgttcc ggacatttct ctggagcgac ttttatgcat ttcccagtat tgtcctttgt 720
ccttagaggg tagtggatcg gcagttttct tcgcctcatt ggtccagatt ttagtttatc 780
ttgtacggta ccaagatgat ggatgtggca caaagttctc agtttggggg ttgcgctctt 840
ccgggcagtt gttattttgg tctgtgatga ctaactcgta tctattcttg tgggagcagg 900
ggcaaactgg gcaacccaag cagaggaaac cacaccggtt caggccaggc acggtggcac 960
tgcgggagat caggaggtat cagaagtcgg tcgactttct catcccgttt gcaccatttg 1020
tccgtctggt gggtacctct gtctgtcata tcctctcgct ctctctacaa acgatctgca 1080
gtgcagagtg taattggaat attttgttcc tgacaaattt gcagatcaag gaggtcaccg 1140
acttcttctg tcctgaaatc agccgctgga ctccccaagc gctcgtcgcg attcaagagg 1200
tcagtgctaa aacctggcat gtactattag atctgatggt ttgattagag tactacaatg 1260
cagatgaatt caatatccga aaaccatgaa ctgtggggta gatacatgta tcgccttaat 1320
tcatggtttc tgaatgctct gctattaatt cagtttgata tatttattta gcagcatggt 1380
attgttttgg tggctggtaa atcaaaactg aaatgtgatt acgagcaaaa cggtatcgat 1440
tgtcgatcct gtgtgttttt gtgcacatct ggttgtttgg tcaagatgtg tttgtgcaca 1500
tcttgcaaca tgatcctgcc cacacactca aaactgacta ttggttaggt tccatttgtc 1560
ttatggaatt tagggtgtaa ctgagaggtg agcaagtggt agtaacgttc aattttgatt 1620
caggatgagg atattgtgat ccagaaaatt gcatgttatg gttatgtgtc caaacgccaa 1680
atgattatgt ctatatccag tactttagaa ccagtacaac aacaaaaagt actttagaac 1740
cagtcaagtt tattgtgcat ttatacaaga gtgttgtttg cacaatagac ttgctttagt 1800
cgtctcttgc cagaaatgcc ttcttctgca caacgagcaa aaataacata aagttgacta 1860
tactcagtgt ggcctaagaa atgagttgta ctttttagtc actggcctgt gtatttgctt 1920
tgaattgaca cataattccc ttttcctttc cctctgcagg ctgcagagta tcacctcgtc 1980
gacgtatttg aaagggcaaa tcactgtgcc atccatgcaa agcgtgttac cgtcagtaag 2040
ttctcactga atgaaaactc cctttctttt acaatattgc gcagaaggaa acatgccagt 2100
tatgaaagag tttcaattac aggatcacct ttgctttcat ttgatgtgat atctagtttt 2160
gatgttgttt caaggttcaa gaattctaat gataaatgat aggatccaca attgttatat 2220
ctctgcagct cctcgtatct gttgtccacg aacaaacata tcaaacaatt cattaaaaag 2280
atgaagaagt caaacaaaca gtatatgtgc actgcatatt agatatcaaa cctggttact 2340
atacgactga tctggcctga cccccgcgtc gcctctccct ggcggcacgg ggggaaccac 2400
tccggcgccg ccaccttccc tcctccctcc accccccacc tcgccgccgc ctgaggagtt 2460
cgccggcgaa gcccggtctg gctccaggga gggtggcggc ggggcatctc tctgcgaggc 2520
gtgagggcgc atctcgtgcg cgggcgcggc gagcttgggc gggatcgcgg gcgcggctct 2580
ggctcgggct ggtgaggctc cggcggtccg aggggtgtcg cggcgcggcg ggggtctcgc 2640
tccggcgcgg gcggtccgag ggcgcgcggg atctggcgct ccagcagggg cgccgtcaag 2700
gggagccggg cagggaagct cgtcaggcgg gctcgtcagg caggcgtgac gcggggcggc 2760
ggccgcgggc tggaagccat ggcgttttgg ccatggtgtc gtgtgcgctc gcttctcatt 2820
cgcagcttga ggtgctgctg ctgagggtgg tgcgcggtga agcttggtgg tcggcgttgg 2880
agagtgcaag gcgcaacagg aatggctcca atgatctcca cgcttctggg tggatcagat 2940
ctgcggccct ccataggggt gtgttccggg cgaaagcctt gacccgactt tgtcggtgcc 3000
gtcgacggcg gcgctttcgg gcgtcgtttc cctccttgga ggcgtcgttg tggaactcat 3060
cttcttctat gtggggctcg ggctctccgg gtgaaaacct aagctccaga ttttccggag 3120
cgggcgatgg cggcgtcttc gtcgtttccc tcttgggggc gttgcttggg agagttagct 3180
tgtgcttggt gcgtttggtt ttctcctacg tcgggtttgg tggatgccgg ggcagcggcc 3240
ccggacggct gatgaacgcc gaggcggcgg cctcggaaag tgatgcgcgg tgcggctcca 3300
tggggcgggg cggtggctcg gccttcactt gggtggcaat cttgggccac ttgggtggca 3360
ggcttgtcgg tccggtcgac gcgttccaga gggggcggtc tgactttgcg tcggggcggc 3420
ggccccggat gtggtgcgac gttcgtggtc tgcgagcggt tgccttgagc agcgtgggct 3480
gcgggcagct gggtcgcgcg gcgttgctgc tcgagcggag cggtggtacg tcggggcggc 3540
ggccccggaa ggtgatgccg gttgattgcg ctgggcgtga cggagcggtg gatgtcgggg 3600
cggcggcccc gagaaaatca ctgtggcgtc cagatttctg tggcaacgat gatggtggga 3660
gcgatgtcgg cgacgcggca atggttgcga tagtcggctc ttctccggcg tgtccacgat 3720
attgcctcgg tttgtttgtt gctgtggagt cgaagctgcg gcggcgaggc cctgtggtat 3780
acgatgactg gttccaggtg tcctttcgtc gatcttccgt ggcgccagcc gtgcctggtt 3840
tcgttcttcc gagttctccg tcagaatcgg agctgcgttg tctgtccgca ggtcgacatg 3900
ttgtcgagaa gggtgggctt tgccctgtgt gtttcagtct atgcgagtgg gctcggccct 3960
tgttgttctg gtttttgccc ggttttccgt aattaactgg gcaattctct tctgcttaat 4020
taatagatga ggcaatcttt gcctcccttt caaaaaaaaa cctggttact atagcaggaa 4080
attcagggtt gattacttta tttcttatct gaaggataaa cattgtatca aatcagaatt 4140
ttatttgtaa gttacatttt tttttactta taaaacttgg aaactgtttt actgtgacaa 4200
atagatgcca ctagaatcat gatcacatcg tggctgttgc tattctaaca aataaatgct 4260
cctgaacaaa tgggaactat atatgaagat gtatggacca gcatgttcct gttaacctga 4320
cctttttcct ttttttgctg ctgcagtgca aaaggacata cagcttgcaa ggcgtatcgg 4380
cgggaggagg ctttggtga 4399
<210> 64
<211> 492
<212> DNA
<213> Triticum aestivum
<400> 64
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgccgcccaa gaagcagctc 60
gggccccgcc ccgcgcagcg gcggcaggag acagatggcg cgggcacgtc ggcgacacca 120
ggcgagccgg gcgggcggcg gccccagggg gggctcaagg ggcaaactgg gcaacccaag 180
cagaggaaac cacaccggtt caggccaggc acggtggcac tgcgggagat caggaggtat 240
cagaagtcgg tcgactttct catcccgttt gcaccatttg tccgtctgat caaggaggtc 300
accgacttct tctgtcctga aatcagccgc tggactcccc aagcgctcgt cgcgattcaa 360
gaggctgcag agtatcacct cgtcgacgta tttgaaaggg caaatcactg tgccatccat 420
gcaaagcgtg ttaccgtcat gcaaaaggac atacagcttg caaggcgtat cggcgggagg 480
aggctttggt ga 492
<210> 65
<211> 163
<212> PRT
<213> Triticum aestivum
<400> 65
Met Ala Arg Thr Lys His Pro Ala Val Arg Lys Thr Lys Ala Pro Pro
1 5 10 15
Lys Lys Gln Leu Gly Pro Arg Pro Ala Gln Arg Arg Gln Glu Thr Asp
20 25 30
Gly Ala Gly Thr Ser Ala Thr Pro Gly Glu Pro Gly Gly Arg Arg Pro
35 40 45
Gln Gly Gly Leu Lys Gly Gln Thr Gly Gln Pro Lys Gln Arg Lys Pro
50 55 60
His Arg Phe Arg Pro Gly Thr Val Ala Leu Arg Glu Ile Arg Arg Tyr
65 70 75 80
Gln Lys Ser Val Asp Phe Leu Ile Pro Phe Ala Pro Phe Val Arg Leu
85 90 95
Ile Lys Glu Val Thr Asp Phe Phe Cys Pro Glu Ile Ser Arg Trp Thr
100 105 110
Pro Gln Ala Leu Val Ala Ile Gln Glu Ala Ala Glu Tyr His Leu Val
115 120 125
Asp Val Phe Glu Arg Ala Asn His Cys Ala Ile His Ala Lys Arg Val
130 135 140
Thr Val Met Gln Lys Asp Ile Gln Leu Ala Arg Arg Ile Gly Gly Arg
145 150 155 160
Arg Leu Trp
<210> 66
<211> 2619
<212> DNA
<213> Triticum aestivum
<400> 66
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgctgcccaa gaagcagctc 60
gggacgcgcc cctcggccgg gacgccgcgg cggcaggaga caggtgagcc cgcccctcct 120
tccttcctcc cccccatctc gagcgaacgc cgtcgcctcg tcgcgagaga tggacgcccc 180
acgcgtgatg ctaatgtgtt cttcctctcc ctttgcagat ggcgcgggca cgtcggcgac 240
tccggtgcgc gggccttcct gggtcacttc cttttgtttc gtcttgtttt ctgtttttgg 300
tctgacttgc tcgtctcctg ttcgacggaa tgcagaggcg agccgggcgg gcggcggccc 360
caggggcggc tgaaggtgcg cccttctttg tgcttttcgg ttttccgccg cgtgtttagg 420
gctatttccg tcttgtttgg ggcggggact tgtatcttct ccccccttcg ttgttgcgca 480
catttctcgg gaatgctacc aggagcagtt gcggttcttt ggcccttagg gagggctgca 540
ccggcaattt cttcgcttcc ttgctccaga ttttagttta tcttctacag tagcaagatg 600
atggatgggg acttgcgttt ttctttcccc ttccttgttc cgggcatttc tctggagcaa 660
tgtttatgca tttcccagta ttgtcctttg ccctcagagg ggaggggatc ggcagttttc 720
ttcgcttcat cggtccagat tttagtttat cttgtacagt agcaagatga tggatgtgac 780
acaaagttct cagtttgggg gttgcgctct tccgggtagt tgttaatttc gtctgtgact 840
gactcgtatc tattcttgtg ggagcagggg caaactgggc aacccaagca gaggaagcca 900
caccggttca ggccaggcac ggtggcactg cgggagatca ggaagtacca gaaatcggtc 960
gactttctca tcccgtttgc accatttgtt cgtctggtgg gtacctctgt catatcctct 1020
cgctctctct accaacgatc tggagtgcgg agtgtaaatt ggaatctttt gttcctgaca 1080
aatttgtaga tcaaggaggt caccgacttc ttctgtcctg aaatcagccg ctggactccc 1140
caagcgctcg ttgcaattca agaggtcagt gctcaaacct ggcatgtact attagatctg 1200
atggtttgat tagagtacta caatgcagat gaattcaaga tctgaaaacc atgaactgtg 1260
gggtagatgc atgtatcata gtgtctgaat gctctggtat taattcagtt tgatatattt 1320
atttagcagc atggtattgt tttggtggct ggtaacccaa aactgaaacg tgattacgag 1380
caaaatggta tcaattgtct tttctgtgtg tttgtgcaaa tctggttgtt tggtcaagat 1440
atacagtctt gcaacatgaa tacatgatcc tgcccacaca ctcaaaactg aactattggt 1500
tagatgccat atgtcttatg gaatttaggg tgtaactgag aggtgagcaa gtggtagtga 1560
cgttcaattt tgattcagga tgaggatatt gtgatccaga aaattgcatg ttatggttat 1620
gtgtccgaac gccaaatgat tatgtctata tccactacat gcttatgtgt cccaaccccc 1680
aaatgattat ctctgtttcc agtacaacaa caaaaagtac tttagaacca gtcaagttta 1740
ttgtgcattt atacgagatt gttgtttgca caatagactt actttagtcg cctcttgcca 1800
gaaatgcctt cttctgcaca acgagcaaaa ataacataaa gttgactata ctcagtgtgg 1860
cctaagaaat gagttagtac ttttagtcac tggcctgtgt atttgctttg aattgacgca 1920
taattccctt ttcctttccc tctgcaggct gcagagtatc acctcgtcga cgtatttgaa 1980
agggcaaatc actgtgccat ccatgcaaag cgtgttaccg tcagtaagtt ctcactgaat 2040
gaaaacttcc ttccatttac aatattatgc agaaggaaac atgccagtta tgaaagagtt 2100
tcaattacag gatcaccttt gctttcattt gatgtgatat ctagttttga tgttgtttca 2160
aggttcaaga attctaacga ttaatgatag gatccacaat tgttatatct ctgcagctcc 2220
tcgtatctgt tgtcaacgaa caaacatgtc aaacaattca ttaaaaaaaa cgaagatgtc 2280
aaaccgggtt actatagcag gaaattcagg gttgattacc tatttattca gggttgatta 2340
cctatttatt tcttatctga aggataaaca ttatataaaa tcagaatttt atttgtaggt 2400
tacatttttt gtttccttat aaaacttgga aactgttgag aatcatcatc atcatggctg 2460
ttgctattct aacaaataaa acggatgctc ctgaataaat ggaactatat atgaagatgt 2520
atttactagc atgttcctgt taacctgact ttttttgctg ctacagtgca aaaggacata 2580
cagctcgcga ggcgtatcgg cgggaggagg ctttggtga 2619
<210> 67
<211> 502
<212> DNA
<213> Triticum aestivum
<400> 67
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgctgcccaa gaagcagctc 60
gggacgcgcc cctcggccgg gacgccgcgg cggcaggaga cagatggcgc gggcacgtcg 120
gcgactccga ggcgagccgg gcgggcggcg gccccagggg cggctgaagg ggcaaactgg 180
gcaacccaag cagaggaagc cacaccggtt caggccaggc acggtggcac tgcgggagat 240
caggaagtac cagaaatcgg tcgactttct catcccgttt gcaccatttg ttcgtctgat 300
caaggaggtc accgacttct tctgtcctga aatcagccgc tggactcccc aagcgctcgt 360
tgcaattcaa gaggctgcag agtatcacct cgtcgacgta tttgaaaggg caaatcactg 420
tgccatccat gcaaagcgtg ttaccgtcat gcaaaaggac atacagctcg cgaggcgtat 480
cggcgggagg aggctttggt ga 502
<210> 68
<211> 2676
<212> DNA
<213> Triticum aestivum
<400> 68
atggcccgta ccaagcaccc ggccgtcagg aagaccaagg cgccgcccaa gaagcagctc 60
gggccccgtc ccgcgcagcg gcggcaggag acaggtcagc ccgcccgccc gtcgctcctc 120
ccttccttcc cccccccgtc tcccgcctct cgaccgaacg ccgccgcctt gtttgttgcg 180
gtacagaggt gcccgtccgt gatgctaaca ggttcttcct ctcctttgca gatggcgcgg 240
gcacgtcggc gacaccggtg cgcgggtctt cccgggtcgc ttccttttgt ttcgtcttgt 300
tttctgtttt ttggtctgac ttgctcgtca cctgttcgac ggaatgcaga ggcgagccgg 360
gcgggcggcg gccccagggg gcgctgaagg tgcggccttc tttgcgcttt tcggttttcc 420
gccgcgtgtt tagggccatt tccgtcttgt ttgggggtgg gtggggcggg ggcttgattt 480
ttttcctcct cccttcgttg ttgcgcacat ttctcgggaa tgcttccagg agcggttgca 540
gttcttcttt ggccttaggg agggctggat cggcagtttc ttcgcttcct tgctccagat 600
tttagtttat cttgtagtag tacagtagca agatgatgga tggggacttg ttttttcttt 660
ccccttcctt gttccggaca tttctttgga gcaactttta tgcatttccc ggtattgtcc 720
tttgccctta gagtggagtg gatcggcagt tttcttcgct tccctggtcc agattttagt 780
ttatctttac agtagcaaga tgatgtatgt gacacaaagt tctcagtttg ggggttgcgc 840
tcttccaggt gtagttgtta tttttcgtct gtgactgact cgtatctatt cttgtgggag 900
caggggcaaa ctgggcaacc caagcagagg aagccacacc ggttcaggcc aggcacggtg 960
gcactgcggg agatcaggag gtaccagaag tcggtcgact ttctcatccc gtttgcacca 1020
tttgtccgtc tggtgggtac ctctgtctgt catatcctct cgctctctct accaacaatc 1080
tggagtgcgg ggtgtaaact ggaatctttt gttcctgaca aatttgcaga tcaaggaggt 1140
caccgacttc ttctgtcctg aaatcagccg ctggactccc caagcgctcg tcgcgattca 1200
agaggtcagt gctcaaacct ggcatgtact attagatctg atggtttgat tagagtacta 1260
caatgcagat gaattcaaga tctgaaaacc atgaactgtg gggtagatgc atgtatcgcc 1320
ttaattcata gtttctgaat gctctggtat taattcagtt tgatatattt atttagctgc 1380
atggtattgt tttggtggct ggtaattcaa aactgaaatg tgattacgag caaaatggta 1440
tcaattgtct tttctgtgca catctggttg tttggtcaag atatacagtc ttgcaacatg 1500
atcctgccca cacactcaaa actgaactat tggttagatt ccattttgct tatggaattt 1560
agggtgtaac tgagaggtga gcaggtggca gtgaccagag ctacgttcaa ttttgattca 1620
ggaagaggat cttgcgatcc agaaaattgc atgttatggt tatgtgtccg aaggccaaat 1680
gattatctct atatccagta catgcttatg tgtccgaacc cccaaatgat tatctctata 1740
tccagtacat gcttatgtgt ccgaaccatt caagttattg tgcatttatt caagattgtt 1800
gtttgcacaa tagacttact ttagtcgcct cttgccagaa atgcctcctt ctgcacaatg 1860
agcaaaaata acataaagtt gattatgctc agtgtggcct aattgtactt tcagttactg 1920
gcctgtgtat ttgctttgaa ttgacacata attccctttt cctttccctc tgcaggctgc 1980
agagtatcac ctcgtcgacg tatttgaaag ggcaaatcac tgtgccatcc atgcaaagcg 2040
tgttaccgtc agtaagttct cactgaatga aaacttcctt tcttttacaa tattatgcag 2100
aaggaaacat gccagttatg aaagagtttc aattacaaga tcacctttgc tttcatttga 2160
tgtggtatct agttttgatg ttgtttcaag gttcaagaat tctaatgatt aatgatagga 2220
tccacaattg ttatatctct gcagctcctc gtatctgacg aacaaacatg tcaaacaatt 2280
cattaaaaaa tgaagatgtc aaacaaacag tatatgcact gcatattgga tatcaaacct 2340
ggttactaca gcaggaagtt cagggttgat tactttattt cttatctgaa ggataaacat 2400
tgtatcaaat cagaatttta tttgtaagtt gcattttttg tttactcata aaacttggaa 2460
actgttttac tgtgagaaat agatgcccac tagaatcatg atcatcatca tggctgttgc 2520
tattctaaca aataaaacgg atgctcctga acaaatggaa ctatgtatga agatgtatgg 2580
actagcatgt tcctgttaac ctgacttttt tttgctgcta cagtgcaaaa ggacatacag 2640
ctcgcgaggc gtatcggtgg gaggaggctt tggtga 2676
<210> 69
<211> 4399
<212> DNA
<213> Triticum aestivum
<400> 69
atggcccgca ccaagcaccc ggccagtcag gaagaccaag gcgccgccca agaagcagct 60
cgggccccgc cccgcgcagc ggcggcagga gacaggtcag cccttccctc cttcctcccc 120
ccgtctcccg cctctcgagc gaacgccgtc gccatttcgt cgcgagagat ggacggacgg 180
acgccccacg catgacgcta atgtgctctt cctctccctt tgcagatggc gcgggcacgt 240
cggcgacacc ggtgcgcggg accttcccgg gtcgcttcct tttcgtttcg tcttgttttc 300
tgttttttgg tctgacttgc tcgtcacctg ttcgacggaa tgcagaggca gccgggcggg 360
cggcggcccc agggggggct caaggtgcgg ccttctttgc gcttttcggt tttccgccgc 420
gtgtttaggg ccatttccgt cttgtttggg ggtgcgcggg gcgggggctt gttttttttc 480
ctcccccctt cgttgttgcg cacattgctc gggaatgctg ccaggagcgg ttgcggttct 540
tctttgaccc ttcgggaggg ctggatcggc agtttcttcg cttccttgct ccagatttta 600
gttcatcttg taccagtaca gtagcaagat gatggatggg ggcttgtttt ttctttctcc 660
ttccttgttc cggacatttc tctggagcga cttttatgca tttcccagta ttgtcctttg 720
tccttagagg gtagtggatc ggcagttttc ttcgcctcat tggtccagat tttagtttat 780
cttgtacggt accaagatga tggatgtggc acaaagttct cagtttgggg gttgcgctct 840
tccgggcagt tgttattttg gtctgtgatg actaactcgt atctattctt gtgggagcag 900
gggcaactgg gcaacccaag cagaggaaac cacaccggtt caggccaggc acggtggcac 960
tgcgggagat caggaggtat cagaagtcgg tcgactttct catcccgttt gcaccatttg 1020
tccgtctggt gggtacctct gtctgtcata tcctctcgct ctctctacaa acgatctgca 1080
gtgcagagtg taattggaat attttgttcc tgacaaattt gcagatcaag gaggtcaccg 1140
acttcttctg tcctgaaatc agccgctgga ctccccaagc gctcgtcgcg attcaagagg 1200
tcagtgctaa aacctggcat gtactattag atctgatggt ttgattagag tactacaatg 1260
cagatgaatt caatatccga aaaccatgaa ctgtggggta gatacatgta tcgccttaat 1320
tcatggtttc tgaatgctct gctattaatt cagtttgata tatttattta gcagcatggt 1380
attgttttgg tggctggtaa atcaaaactg aaatgtgatt acgagcaaaa cggtatcgat 1440
tgtcgatcct gtgtgttttt gtgcacatct ggttgtttgg tcaagatgtg tttgtgcaca 1500
tcttgcaaca tgatcctgcc cacacactca aaactgacta ttggttaggt tccatttgtc 1560
ttatggaatt tagggtgtaa ctgagaggtg agcaagtggt agtaacgttc aattttgatt 1620
caggatgagg atattgtgat ccagaaaatt gcatgttatg gttatgtgtc caaacgccaa 1680
atgattatgt ctatatccag tactttagaa ccagtacaac aacaaaaagt actttagaac 1740
cagtcaagtt tattgtgcat ttatacaaga gtgttgtttg cacaatagac ttgctttagt 1800
cgtctcttgc cagaaatgcc ttcttctgca caacgagcaa aaataacata aagttgacta 1860
tactcagtgt ggcctaagaa atgagttgta ctttttagtc actggcctgt gtatttgctt 1920
tgaattgaca cataattccc ttttcctttc cctctgcagg ctgcagagta tcacctcgtc 1980
gacgtatttg aaagggcaaa tcactgtgcc atccatgcaa agcgtgttac cgtcagtaag 2040
ttctcactga atgaaaactc cctttctttt acaatattgc gcagaaggaa acatgccagt 2100
tatgaaagag tttcaattac aggatcacct ttgctttcat ttgatgtgat atctagtttt 2160
gatgttgttt caaggttcaa gaattctaat gataaatgat aggatccaca attgttatat 2220
ctctgcagct cctcgtatct gttgtccacg aacaaacata tcaaacaatt cattaaaaag 2280
atgaagaagt caaacaaaca gtatatgtgc actgcatatt agatatcaaa cctggttact 2340
atacgactga tctggcctga cccccgcgtc gcctctccct ggcggcacgg ggggaaccac 2400
tccggcgccg ccaccttccc tcctccctcc accccccacc tcgccgccgc ctgaggagtt 2460
cgccggcgaa gcccggtctg gctccaggga gggtggcggc ggggcatctc tctgcgaggc 2520
gtgagggcgc atctcgtgcg cgggcgcggc gagcttgggc gggatcgcgg gcgcggctct 2580
ggctcgggct ggtgaggctc cggcggtccg aggggtgtcg cggcgcggcg ggggtctcgc 2640
tccggcgcgg gcggtccgag ggcgcgcggg atctggcgct ccagcagggg cgccgtcaag 2700
gggagccggg cagggaagct cgtcaggcgg gctcgtcagg caggcgtgac gcggggcggc 2760
ggccgcgggc tggaagccat ggcgttttgg ccatggtgtc gtgtgcgctc gcttctcatt 2820
cgcagcttga ggtgctgctg ctgagggtgg tgcgcggtga agcttggtgg tcggcgttgg 2880
agagtgcaag gcgcaacagg aatggctcca atgatctcca cgcttctggg tggatcagat 2940
ctgcggccct ccataggggt gtgttccggg cgaaagcctt gacccgactt tgtcggtgcc 3000
gtcgacggcg gcgctttcgg gcgtcgtttc cctccttgga ggcgtcgttg tggaactcat 3060
cttcttctat gtggggctcg ggctctccgg gtgaaaacct aagctccaga ttttccggag 3120
cgggcgatgg cggcgtcttc gtcgtttccc tcttgggggc gttgcttggg agagttagct 3180
tgtgcttggt gcgtttggtt ttctcctacg tcgggtttgg tggatgccgg ggcagcggcc 3240
ccggacggct gatgaacgcc gaggcggcgg cctcggaaag tgatgcgcgg tgcggctcca 3300
tggggcgggg cggtggctcg gccttcactt gggtggcaat cttgggccac ttgggtggca 3360
ggcttgtcgg tccggtcgac gcgttccaga gggggcggtc tgactttgcg tcggggcggc 3420
ggccccggat gtggtgcgac gttcgtggtc tgcgagcggt tgccttgagc agcgtgggct 3480
gcgggcagct gggtcgcgcg gcgttgctgc tcgagcggag cggtggtacg tcggggcggc 3540
ggccccggaa ggtgatgccg gttgattgcg ctgggcgtga cggagcggtg gatgtcgggg 3600
cggcggcccc gagaaaatca ctgtggcgtc cagatttctg tggcaacgat gatggtggga 3660
gcgatgtcgg cgacgcggca atggttgcga tagtcggctc ttctccggcg tgtccacgat 3720
attgcctcgg tttgtttgtt gctgtggagt cgaagctgcg gcggcgaggc cctgtggtat 3780
acgatgactg gttccaggtg tcctttcgtc gatcttccgt ggcgccagcc gtgcctggtt 3840
tcgttcttcc gagttctccg tcagaatcgg agctgcgttg tctgtccgca ggtcgacatg 3900
ttgtcgagaa gggtgggctt tgccctgtgt gtttcagtct atgcgagtgg gctcggccct 3960
tgttgttctg gtttttgccc ggttttccgt aattaactgg gcaattctct tctgcttaat 4020
taatagatga ggcaatcttt gcctcccttt caaaaaaaaa cctggttact atagcaggaa 4080
attcagggtt gattacttta tttcttatct gaaggataaa cattgtatca aatcagaatt 4140
ttatttgtaa gttacatttt tttttactta taaaacttgg aaactgtttt actgtgacaa 4200
atagatgcca ctagaatcat gatcacatcg tggctgttgc tattctaaca aataaatgct 4260
cctgaacaaa tgggaactat atatgaagat gtatggacca gcatgttcct gttaacctga 4320
cctttttcct ttttttgctg ctgcagtgca aaaggacata cagcttgcaa ggcgtatcgg 4380
cgggaggagg ctttggtga 4399
<210> 70
<211> 492
<212> DNA
<213> Triticum aestivum
<400> 70
atggcccgca ccaagcaccc ggccagtcag gaagaccaag gcgccgccca agaagcagct 60
cgggccccgc cccgcgcagc ggcggcagga gacagatggc gcgggcacgt cggcgacacc 120
gaggcagccg ggcgggcggc ggccccaggg ggggctcaag gggcaactgg gcaacccaag 180
cagaggaaac cacaccggtt caggccaggc acggtggcac tgcgggagat caggaggtat 240
cagaagtcgg tcgactttct catcccgttt gcaccatttg tccgtctgat caaggaggtc 300
accgacttct tctgtcctga aatcagccgc tggactcccc aagcgctcgt cgcgattcaa 360
gaggctgcag agtatcacct cgtcgacgta tttgaaaggg caaatcactg tgccatccat 420
gcaaagcgtg ttaccgtcat gcaaaaggac atacagcttg caaggcgtat cggcgggagg 480
aggctttggt ga 492
<210> 71
<211> 163
<212> PRT
<213> Triticum aestivum
<400> 71
Met Ala Arg Thr Lys His Pro Ala Ser Gln Glu Asp Gln Gly Ala Ala
1 5 10 15
Gln Glu Ala Ala Arg Ala Pro Pro Arg Ala Ala Ala Ala Gly Asp Arg
20 25 30
Trp Arg Gly His Val Gly Asp Thr Glu Ala Ala Gly Arg Ala Ala Ala
35 40 45
Pro Gly Gly Ala Gln Gly Ala Thr Gly Gln Pro Lys Gln Arg Lys Pro
50 55 60
His Arg Phe Arg Pro Gly Thr Val Ala Leu Arg Glu Ile Arg Arg Tyr
65 70 75 80
Gln Lys Ser Val Asp Phe Leu Ile Pro Phe Ala Pro Phe Val Arg Leu
85 90 95
Ile Lys Glu Val Thr Asp Phe Phe Cys Pro Glu Ile Ser Arg Trp Thr
100 105 110
Pro Gln Ala Leu Val Ala Ile Gln Glu Ala Ala Glu Tyr His Leu Val
115 120 125
Asp Val Phe Glu Arg Ala Asn His Cys Ala Ile His Ala Lys Arg Val
130 135 140
Thr Val Met Gln Lys Asp Ile Gln Leu Ala Arg Arg Ile Gly Gly Arg
145 150 155 160
Arg Leu Trp
<210> 72
<211> 2619
<212> DNA
<213> Triticum aestivum
<400> 72
atggcccgca ccaagcaccc ggccgtcagg aagaccaagg cgctgcccaa gaagcagctc 60
gggacgcgcc cctcggccgg gacgccgcgg cggcaggaga caggtgagcc cgcccctcct 120
tccttcctcc cccccatctc gagcgaacgc cgtcgcctcg tcgcgagaga tggacgcccc 180
acgcgtgatg ctaatgtgtt cttcctctcc ctttgcagat ggcgcgggca cgtcggcgac 240
tccggtgcgc gggccttcct gggtcacttc cttttgtttc gtcttgtttt ctgtttttgg 300
tctgacttgc tcgtctcctg ttcgacggaa tgcagaggcg aagccgggcg ggcggcggcc 360
ccaggggcgg ctgaaggtgc gcccttcttt gtgcttttcg gttttccgcc gcgtgtttag 420
ggctatttcc gtcttgtttg gggcggggac ttgtatcttc tccccccttc gttgttgcgc 480
acatttctcg ggaatgctac caggagcagt tgcggttctt tggcccttag ggagggctgc 540
accggcaatt tcttcgcttc cttgctccag attttagttt atcttctaca gtagcaagat 600
gatggatggg gacttgcgtt tttctttccc cttccttgtt ccgggcattt ctctggagca 660
atgtttatgc atttcccagt attgtccttt gccctcagag gggaggggat cggcagtttt 720
cttcgcttca tcggtccaga ttttagttta tcttgtacag tagcaagatg atggatgtga 780
cacaaagttc tcagtttggg ggttgcgctc ttccgggtag ttgttaattt cgtctgtgac 840
tgactcgtat ctattcttgt gggagcaggg gcaactgggc aacccaagca gaggaagcca 900
caccggttca ggccaggcac ggtggcactg cgggagatca ggaagtacca gaaatcggtc 960
gactttctca tcccgtttgc accatttgtt cgtctggtgg gtacctctgt catatcctct 1020
cgctctctct accaacgatc tggagtgcgg agtgtaaatt ggaatctttt gttcctgaca 1080
aatttgtaga tcaaggaggt caccgacttc ttctgtcctg aaatcagccg ctggactccc 1140
caagcgctcg ttgcaattca agaggtcagt gctcaaacct ggcatgtact attagatctg 1200
atggtttgat tagagtacta caatgcagat gaattcaaga tctgaaaacc atgaactgtg 1260
gggtagatgc atgtatcata gtgtctgaat gctctggtat taattcagtt tgatatattt 1320
atttagcagc atggtattgt tttggtggct ggtaacccaa aactgaaacg tgattacgag 1380
caaaatggta tcaattgtct tttctgtgtg tttgtgcaaa tctggttgtt tggtcaagat 1440
atacagtctt gcaacatgaa tacatgatcc tgcccacaca ctcaaaactg aactattggt 1500
tagatgccat atgtcttatg gaatttaggg tgtaactgag aggtgagcaa gtggtagtga 1560
cgttcaattt tgattcagga tgaggatatt gtgatccaga aaattgcatg ttatggttat 1620
gtgtccgaac gccaaatgat tatgtctata tccactacat gcttatgtgt cccaaccccc 1680
aaatgattat ctctgtttcc agtacaacaa caaaaagtac tttagaacca gtcaagttta 1740
ttgtgcattt atacgagatt gttgtttgca caatagactt actttagtcg cctcttgcca 1800
gaaatgcctt cttctgcaca acgagcaaaa ataacataaa gttgactata ctcagtgtgg 1860
cctaagaaat gagttagtac ttttagtcac tggcctgtgt atttgctttg aattgacgca 1920
taattccctt ttcctttccc tctgcaggct gcagagtatc acctcgtcga cgtatttgaa 1980
agggcaaatc actgtgccat ccatgcaaag cgtgttaccg tcagtaagtt ctcactgaat 2040
gaaaacttcc ttccatttac aatattatgc agaaggaaac atgccagtta tgaaagagtt 2100
tcaattacag gatcaccttt gctttcattt gatgtgatat ctagttttga tgttgtttca 2160
aggttcaaga attctaacga ttaatgatag gatccacaat tgttatatct ctgcagctcc 2220
tcgtatctgt tgtcaacgaa caaacatgtc aaacaattca ttaaaaaaaa cgaagatgtc 2280
aaaccgggtt actatagcag gaaattcagg gttgattacc tatttattca gggttgatta 2340
cctatttatt tcttatctga aggataaaca ttatataaaa tcagaatttt atttgtaggt 2400
tacatttttt gtttccttat aaaacttgga aactgttgag aatcatcatc atcatggctg 2460
ttgctattct aacaaataaa acggatgctc ctgaataaat ggaactatat atgaagatgt 2520
atttactagc atgttcctgt taacctgact ttttttgctg ctacagtgca aaaggacata 2580
cagctcgcga ggcgtatcgg cgggaggagg ctttggtga 2619
<210> 73
<211> 2673
<212> DNA
<213> Triticum aestivum
<400> 73
atggcccgta ccaagcaccc ggccaggaag accaaggcgc cgcccaagaa gcagctcggg 60
ccccgtcccg cgcagcggcg gcaggagaca ggtcagcccg cccgcccgtc gctcctccct 120
tccttccccc ccccgtctcc cgcctctcga ccgaacgccg ccgccttgtt tgttgcggta 180
cagaggtgcc cgtccgtgat gctaacaggt tcttcctctc ctttgcagat ggcgcgggca 240
cgtcggcgac accggtgcgc gggtcttccc gggtcgcttc cttttgtttc gtcttgtttt 300
ctgttttttg gtctgacttg ctcgtcacct gttcgacgga atgcagaggc gaagccgggc 360
gggcggcggc cccagggggc gctgaaggtg cggccttctt tgcgcttttc ggttttccgc 420
cgcgtgttta gggccatttc cgtcttgttt gggggtgggt ggggcggggg cttgattttt 480
ttcctcctcc cttcgttgtt gcgcacattt ctcgggaatg cttccaggag cggttgcagt 540
tcttctttgg ccttagggag ggctggatcg gcagtttctt cgcttccttg ctccagattt 600
tagtttatct tgtagtagta cagtagcaag atgatggatg gggacttgtt ttttctttcc 660
ccttccttgt tccggacatt tctttggagc aacttttatg catttcccgg tattgtcctt 720
tgcccttaga gtggagtgga tcggcagttt tcttcgcttc cctggtccag attttagttt 780
atctttacag tagcaagatg atgtatgtga cacaaagttc tcagtttggg ggttgcgctc 840
ttccaggtgt agttgttatt tttcgtctgt gactgactcg tatctattct tgtgggagca 900
ggggcaactg ggcaacccaa gcagaggaag ccacaccggt tcaggccagg cacggtggca 960
ctgcgggaga tcaggaggta ccagaagtcg gtcgactttc tcatcccgtt tgcaccattt 1020
gtccgtctgg tgggtacctc tgtctgtcat atcctctcgc tctctctacc aacaatctgg 1080
agtgcggggt gtaaactgga atcttttgtt cctgacaaat ttgcagatca aggaggtcac 1140
cgacttcttc tgtcctgaaa tcagccgctg gactccccaa gcgctcgtcg cgattcaaga 1200
ggtcagtgct caaacctggc atgtactatt agatctgatg gtttgattag agtactacaa 1260
tgcagatgaa ttcaagatct gaaaaccatg aactgtgggg tagatgcatg tatcgcctta 1320
attcatagtt tctgaatgct ctggtattaa ttcagtttga tatatttatt tagctgcatg 1380
gtattgtttt ggtggctggt aattcaaaac tgaaatgtga ttacgagcaa aatggtatca 1440
attgtctttt ctgtgcacat ctggttgttt ggtcaagata tacagtcttg caacatgatc 1500
ctgcccacac actcaaaact gaactattgg ttagattcca ttttgcttat ggaatttagg 1560
gtgtaactga gaggtgagca ggtggcagtg accagagcta cgttcaattt tgattcagga 1620
agaggatctt gcgatccaga aaattgcatg ttatggttat gtgtccgaag gccaaatgat 1680
tatctctata tccagtacat gcttatgtgt ccgaaccccc aaatgattat ctctatatcc 1740
agtacatgct tatgtgtccg aaccattcaa gttattgtgc atttattcaa gattgttgtt 1800
tgcacaatag acttacttta gtcgcctctt gccagaaatg cctccttctg cacaatgagc 1860
aaaaataaca taaagttgat tatgctcagt gtggcctaat tgtactttca gttactggcc 1920
tgtgtatttg ctttgaattg acacataatt cccttttcct ttccctctgc aggctgcaga 1980
gtatcacctc gtcgacgtat ttgaaagggc aaatcactgt gccatccatg caaagcgtgt 2040
taccgtcagt aagttctcac tgaatgaaaa cttcctttct tttacaatat tatgcagaag 2100
gaaacatgcc agttatgaaa gagtttcaat tacaagatca cctttgcttt catttgatgt 2160
ggtatctagt tttgatgttg tttcaaggtt caagaattct aatgattaat gataggatcc 2220
acaattgtta tatctctgca gctcctcgta tctgacgaac aaacatgtca aacaattcat 2280
taaaaaatga agatgtcaaa caaacagtat atgcactgca tattggatat caaacctggt 2340
tactacagca ggaagttcag ggttgattac tttatttctt atctgaagga taaacattgt 2400
atcaaatcag aattttattt gtaagttgca ttttttgttt actcataaaa cttggaaact 2460
gttttactgt gagaaataga tgcccactag aatcatgatc atcatcatgg ctgttgctat 2520
tctaacaaat aaaacggatg ctcctgaaca aatggaacta tgtatgaaga tgtatggact 2580
agcatgttcc tgttaacctg actttttttt gctgctacag tgcaaaagga catacagctc 2640
gcgaggcgta tcggtgggag gaggctttgg tga 2673
<210> 74
<211> 18400
<212> DNA
<213> Artificial Sequence
<220>
<223> Construct 24195
<220>
<221> misc_feature
<222> (4)..(259)
<223> bNRB-05
<220>
<221> misc_feature
<222> (101)..(125)
<223> bNRB-01-01
<220>
<221> enhancer
<222> (168)..(259)
<223> eNOS-01
<220>
<221> enhancer
<222> (292)..(485)
<223> eFMV-06
<220>
<221> enhancer
<222> (492)..(784)
<223> e35S-11
<220>
<221> promoter
<222> (791)..(2592)
<223> prSoUbi4-02
<220>
<221> misc_feature
<222> (1170)..(1234)
<223> u5SoUbi4-02
<220>
<221> Intron
<222> (1235)..(2592)
<223> iSoUbi4-02
<220>
<221> misc_feature
<222> (2607)..(2657)
<223> NLS
<220>
<221> gene
<222> (2607)..(6827)
<223> CRISPR associated 9
<220>
<221> misc_feature
<222> (2610)..(2610)
<223> +4G
<220>
<221> misc_feature
<222> (2611)..(2611)
<223> +5C
<220>
<221> mutation
<222> (6147)..(6149)
<223> L to V mutation
<220>
<221> mutation
<222> (6192)..(6194)
<223> I to V mutation
<220>
<221> misc_feature
<222> (6762)..(6824)
<223> NLS
<220>
<221> terminator
<222> (6834)..(7835)
<223> tZmMTL-01
<220>
<221> misc_feature
<222> (7836)..(7847)
<223> xSTOPS-01
<220>
<221> promoter
<222> (7855)..(8216)
<223> prTau6-01
<220>
<221> misc_feature
<222> (8217)..(8236)
<223> xTaCenh3 target-03
<220>
<221> misc_feature
<222> (8217)..(8321)
<223> gRNA TaCenH3-03
<220>
<221> misc_feature
<222> (8237)..(8248)
<223> crRNA-01
<220>
<221> misc_feature
<222> (8253)..(8321)
<223> tracrRNA-01
<220>
<221> promoter
<222> (8322)..(8683)
<223> prTaU6-01
<220>
<221> misc_feature
<222> (8684)..(8703)
<223> xTaCenH3 target-04
<220>
<221> misc_feature
<222> (8684)..(8788)
<223> guide RNA TaCenH3-04
<220>
<221> misc_feature
<222> (8704)..(8715)
<223> crRNA-01
<220>
<221> misc_feature
<222> (8720)..(8788)
<223> tracrRNA-01
<220>
<221> misc_feature
<222> (8795)..(8806)
<223> xSTOPS-01
<220>
<221> promoter
<222> (8807)..(10799)
<223> prUbi1-10
<220>
<221> TATA_signal
<222> (9677)..(9685)
<223> TATA box
<220>
<221> Intron
<222> (9790)..(10799)
<223> iUbi1-02-01
<220>
<221> gene
<222> (10811)..(11989)
<223> cPMI-12
<220>
<221> terminator
<222> (11994)..(13028)
<223> tUbi1-01
<220>
<221> misc_feature
<222> (13072)..(13083)
<223> xSTOPS-01
<220>
<221> misc_feature
<222> (13084)..(13123)
<223> xTAG-02
<220>
<221> misc_feature
<222> (13132)..(13261)
<223> bNLB-05
<220>
<221> misc_feature
<222> (13167)..(13191)
<223> bNLB-01-01
<220>
<221> gene
<222> (13541)..(14329)
<223> cSpec-03
<220>
<221> promoter
<222> (14424)..(14554)
<223> prVirG-01
<220>
<221> gene
<222> (14629)..(15354)
<223> cVirG-01
<220>
<221> gene
<222> (15384)..(16457)
<223> cRepA-03
<220>
<221> misc_feature
<222> (16500)..(16904)
<223> oVS1-02
<220>
<221> misc_feature
<222> (17582)..(18388)
<223> oCOLE-06
<400> 74
attcctgtgg ttggcatgca catacaaatg gacgaacgga taaacctttt cacgcccttt 60
taaatatccg attattctaa taaacgctct tttctcttag gtttacccgc caatatatcc 120
tgtcaaacac tgatagttta aactgaaggc gggaaacgac aatctgatca tgagcggaga 180
attaagggag tcacgttatg acccccgccg atgacgcggg acaagccgtt ttacgtttgg 240
aactgacaga accgcaacgc tgcaggaatt ggccgcagcg gccatttaaa tagctgcttg 300
tggggaccag acaaaaaagg aatggtgcag aattgttagg cgcacctacc aaaagcaact 360
ttgcctttat tgcaaagata aagcagattc ctctagtaca agtggggaac aaaataacgt 420
ggaaaagagc tgtcctgaca gcccactcac tattgcgttt gacgaacgca gtgacgacca 480
caaaactcga gacttttcaa caaagggtat tatccggaaa cctcctcgga ttccattgcc 540
cagctatctg tcactttatt gtgaagatag tggaaaagga aggtggctcc tacaaatgcc 600
atcattgcga taaaggaaag gctatcgttg aagatgcctc tgccgacagt ggtcccaaag 660
atggaccccc acccacgagg agcatcgtgg aaaaagaaga cgttccaacc acgtcttcaa 720
agcaagtgga ttgatgtgat atctccactg acgtaagggt tgacgaacaa tcccactatc 780
cttcggaccc gaattcatta tgtggtctag gtaggttcta tatataagaa aacttgaaat 840
gttctaaaaa aaaattcaag cccatgcatg attgaagcaa acggtatagc aacggtgtta 900
acctgatcta gtgatctctt gcaatcctta acggccacct accgcaggta gcaaacggcg 960
tccccctcct cgatatctcc gcggcgacct ctggcttttt ccgcggaatt gcgcggtggg 1020
gacggattcc acgagaccgc gacgcaaccg cctctcgccg ctgggcccca caccgctcgg 1080
tgccgtagcc tcacgggact ctttctccct cctcccccgt tataaattgg cttcatcccc 1140
tccttgcctc atccatccaa atcccagtcc ccaatcccat cccttcgtag gagaaattca 1200
tcgaagctaa gcgaatcctc gcgatcctct caaggtactg cgagttttcg atccccctct 1260
cgacccctcg tatgtttgtg tttgtcgtag cgtttgatta ggtatgcttt ccctgtttgt 1320
gttcgtcgta gcgtttgatt aggtatgctt tccctgttcg tgttcatcgt agtgtttgat 1380
taggtcgtgt gaggcgatgg cctgctcgcg tccttcgatc tgtagtcgat ttgcgggtcg 1440
tggtgtagat ctgcgggctg tgatgaagtt atttggtgtg atctgctcgc ctgattctgc 1500
gggttggctc gagtagatat gatggttgga ccggttggtt cgtttaccgc gctagggttg 1560
ggctgggatg atgttgcatg cgccgttgcg cgtgatcccg cagcaggact tgcgtttgat 1620
tgccagatct cgttacgatt atgtgatttg gtttggactt tttagatctg tagcttctgc 1680
ttatgtgcca gatgcgccta ctgctcatat gcctgatgat aatcataaat ggctgtggaa 1740
ctaactagtt gattgcggag tcatgtatca gctacaggtg tagggactag ctacaggtgt 1800
agggacttgc gtctaattgt ttggtccttt actcatgttg caattatgca atttagttta 1860
gattgtttgt tccactcatc taggctgtaa aagggacact gcttagattg ctgtttaatc 1920
tttttagtag attatattat attggtaact tattacccct attacatgcc atacgtgact 1980
tctgctcatg cctgatgata atcatagatc actgtggaat taattagttg attgttgaat 2040
catgtttcat gtacatacca cggcacaatt gcttagttcc ttaacaaatg caaattttac 2100
tgatccatgt atgatttgcg tggttctcta atgtgaaata ctatagctac ttgttagtaa 2160
gaatcaggtt cgtatgctta atgctgtatg tgccttctgc tcatgcctga tgataatcat 2220
atatcactgg aattaattag ttgatcgttt aatcatatat caagtacata ccatgccaca 2280
atttttagtc acttaaccca tgcagattga actggtccct gcatgttttg ctaaattgtt 2340
ctattctgat tagaccatat atcatgtatt tttttttggt aatggttctc ttattttaaa 2400
tgctatatag ttctggtact tgttagaaag atctgcttca tagtttagtt gcctatccct 2460
cgaattagga tgctgagcag ctgatcctat agctttgttt catgtatcaa ttcttttgtg 2520
ttcaacagtc agtttttgtt agattcattg taacttatgg tcgcttactc ttctggtcct 2580
caatgcttgc agcctagtaa gccgccatgg ccccaaagaa gaagaggaag gtcggcatcc 2640
acggcgtccc agctgcgatg gacaagaagt actccatcgg cctcgacatc ggcaccaaca 2700
gcgtgggctg ggccgtcatc accgacgagt acaaggtgcc atccaagaag ttcaaggtcc 2760
tgggcaacac cgaccgccac agcatcaaga agaacctcat cggcgctctc ctgttcgact 2820
ccggcgagac ggctgaggct accaggctca agcgcaccgc caggaggagg tacaccagga 2880
ggaagaacag gatctgctac ctccaagaga tcttctccaa cgagatggcc aaggtggacg 2940
actccttctt ccaccgcctg gaggagagct tcctcgtcga ggaggacaag aagcacgaga 3000
ggcacccaat cttcggcaac atcgtggacg aggtcgccta ccacgagaag tacccaacca 3060
tctaccacct gaggaagaag ctcgtggact ccaccgacaa ggccgacctc cgcctgatct 3120
acctcgccct ggcccacatg atcaagttca ggggccactt cctgatcgag ggcgacctca 3180
acccagacaa cagcgacgtg gacaagctgt tcatccaact cgtccagacc tacaaccagc 3240
tcttcgagga gaacccgatc aacgcttccg gcgtggacgc taaggctatc ctgagcgcta 3300
ggctctccaa gagcaggagg ctcgagaacc tgatcgccca gctcccaggc gagaagaaga 3360
acggcctgtt cggcaacctc atcgctctct ccctgggcct caccccaaac ttcaagagca 3420
acttcgacct cgccgaggac gccaagctgc aactctccaa ggacacctac gacgacgacc 3480
tggacaacct cctggcccag atcggcgacc aatacgccga cctgttcctc gccgccaaga 3540
acctgtccga cgccatcctc ctgagcgaca tcctccgcgt gaacaccgag atcaccaagg 3600
ccccactctc cgccagcatg atcaagcgct acgacgagca ccaccaggac ctgaccctcc 3660
tgaaggccct ggtcaggcaa cagctcccag agaagtacaa ggagatcttc ttcgaccaga 3720
gcaagaacgg ctacgctggc tacatcgacg gcggcgcctc ccaagaggag ttctacaagt 3780
tcatcaagcc aatcctggag aagatggacg gcaccgagga gctcctggtg aagctcaaca 3840
gggaggacct cctgaggaag cagcgcacct tcgacaacgg cagcatccca caccaaatcc 3900
acctcggcga gctgcacgct atcctgagga ggcaagagga cttctaccca ttcctcaagg 3960
acaacaggga gaagatcgag aagatcctga ccttccgcat cccctactac gtcggcccac 4020
tcgctagggg caactccagg ttcgcttgga tgacccgcaa gagcgaggag acgatcaccc 4080
cgtggaactt cgaggaggtc gtcgacaagg gcgcttccgc tcagagcttc atcgagagga 4140
tgaccaactt cgacaagaac ctgccaaacg agaaggtgct cccaaagcac tccctcctgt 4200
acgagtactt caccgtctac aacgagctca ccaaggtgaa gtatgtgacc gagggcatgc 4260
gcaagccagc cttcctgagc ggcgagcaga agaaggccat cgtggacctc ctgttcaaga 4320
ccaacaggaa ggtgaccgtc aagcaactca aggaggacta cttcaagaag atcgagtgct 4380
tcgactccgt ggagatcagc ggcgtcgagg accgcttcaa cgcctccctc ggcacctacc 4440
acgacctcct gaagatcatc aaggacaagg acttcctgga caacgaggag aacgaggaca 4500
tcctcgagga catcgtgctg accctcaccc tgttcgagga cagggagatg atcgaggagc 4560
gcctgaagac ctacgcccac ctcttcgacg acaaggtcat gaagcaactc aagaggagga 4620
ggtacaccgg ctggggcagg ctgagccgca agctcatcaa cggcatccgc gacaagcagt 4680
ccggcaagac catcctcgac ttcctgaaga gcgacggctt cgccaacagg aacttcatgc 4740
aactgatcca cgacgactcc ctcaccttca aggaggacat ccaaaaggct caggtgtccg 4800
gccagggcga cagcctgcac gagcacatcg ctaacctcgc tggcagccca gccatcaaga 4860
agggcatcct gcagaccgtg aaggtcgtcg acgagctcgt gaaggtcatg ggcaggcaca 4920
agccagagaa catcgtcatc gagatggccc gcgagaacca gaccacccag aagggccaaa 4980
agaactccag ggagcgcatg aagcgcatcg aggagggcat caaggagctg ggcagccaaa 5040
tcctcaagga gcacccagtg gagaacaccc aactgcagaa cgagaagctc tacctgtact 5100
acctccagaa cggcagggac atgtatgtgg accaagagct ggacatcaac cgcctctccg 5160
actacgacgt ggaccacatc gtcccacagt ccttcctgaa ggacgacagc atcgacaaca 5220
aggtgctcac caggagcgac aagaaccgcg gcaagtccga caacgtccca agcgaggagg 5280
tggtcaagaa gatgaaaaac tactggaggc agctcctgaa cgccaagctg atcacccaaa 5340
ggaagttcga caacctcacc aaggctgaga ggggcggcct ctccgagctg gacaaggccg 5400
gcttcattaa aaggcagctg gtggagacgc gccaaatcac caagcacgtc gcccaaatcc 5460
tcgacagccg catgaacacc aagtacgacg agaacgacaa gctgatcagg gaggtgaagg 5520
tcatcaccct gaagtccaag ctcgtgagcg acttcaggaa ggacttccag ttctacaagg 5580
tccgcgagat caataattac caccacgccc acgacgctta cctcaacgct gtggtcggca 5640
ccgccctgat taaaaagtac ccaaagctcg agtccgagtt cgtgtacggc gactacaagg 5700
tgtacgacgt ccgcaagatg atcgccaagt ccgagcaaga gatcggcaag gccaccgcca 5760
agtacttctt ctacagcaac atcatgaact tcttcaagac cgagatcacc ctggccaacg 5820
gcgagatcag gaagcgccca ctcatcgaga cgaacggcga gacgggcgag atcgtgtggg 5880
acaagggcag ggacttcgcc accgtgcgca aggtcctctc catgccacag gtgaacatcg 5940
tcaagaagac cgaggtccaa accggcggct tctccaagga gagcatcctg ccaaagagga 6000
acagcgacaa gctcatcgcc cgcaagaagg actgggatcc aaagaagtac ggcggcttcg 6060
actccccaac cgtggcctac agcgtcctcg tggtcgccaa ggtggagaag ggcaagtcca 6120
agaagctgaa gagcgtgaag gagctcgtcg gcatcaccat catggagagg tccagcttcg 6180
agaagaaccc agtggacttc ctcgaggcca agggctacaa ggaggtcaag aaggacctga 6240
tcattaaact cccaaagtac agcctcttcg agctggagaa cggcaggaag cgcatgctgg 6300
cttccgctgg cgagctccaa aagggcaacg agctcgccct gccatccaag tatgtgaact 6360
tcctctacct ggcctcccac tacgagaagc tcaagggcag cccagaggac aacgagcaaa 6420
agcagctgtt cgtcgagcag cacaagcact acctcgacga gatcatcgag caaatctccg 6480
agttcagcaa gcgcgtgatc ctcgccgacg ccaacctgga caaggtcctc tccgcctaca 6540
acaagcacag ggacaagcca atccgcgagc aggccgagaa catcatccac ctcttcaccc 6600
tgaccaacct cggcgctcca gctgccttca agtacttcga caccaccatc gacaggaagc 6660
gctacacctc caccaaggag gtgctggacg ccaccctcat ccaccagtcc atcaccggcc 6720
tctacgagac gaggatcgac ctgagccaac tcggcggcga ctccagccca ccaaagaaga 6780
agaggaaggt cagctggaag gacgcttccg gctggagccg catgtgaggt acctcacatc 6840
gatcgacgac caaggatatg attattatct atctagcttg tggtggtggt tgaacaataa 6900
taagcgaggc cgagctggct gccatacata ggtattgtgt ggtgtgtgtg agagagagag 6960
aaacagagtt cttcagtttg ctatctctct ctgcatgttt ggcgtcagtc tttgtgctca 7020
tgtacgtacg tgtgtctacc tgcatgttgg ttgatccgat tgcatctgct gtaaccatat 7080
attaattggt ccacgatgat atgatttgat actatatata tactaaaacc ggacttctta 7140
ttataatact tgtagtatat aagtttctta cgcccgcaat tgatcgattc agaaggagtt 7200
ctagctagct aaaacatgca gattcagaat atcagatttt taggactact ggagatccag 7260
aaccttcgtg tccttgtacc cgtgattttg gatccccttt ctccccacta caatcgttgg 7320
cgcaatcttg ttctgctcgc ctagaatggt acgccctcac cgaccatgtc ctcgccgatg 7380
cctaccccgc cacgccatct ggatggtgca tgaatgtcgt cgttttgtcc tggatgctag 7440
gcacactctc ccccgagttg atggagcctc ctcgcacctc tggtggcact gcgccgcgcc 7500
tggcttgcca tcgaggagca attcctcggc aatcgcgaag ctcgcacact tcgtctcgac 7560
gccgagttcc atgtcttcat gtaggcgatc tcttcgtcag cgactattgt ctattgacgc 7620
aagatgaagg ggatggacga ggcccttggt gatcttggtg aggtcatcca tgaccgtacc 7680
cttgtcctaa acgtgttgtg tggtctgaat gagaggtttg cccacataaa ggtccacttc 7740
aagcactcga atccgttccc ctccttcacc gacgtttgta atgatctcat ccttgaggag 7800
atcgactcca gcgcgcctcc tccgcctccg accacctaat tagctaaggg acccgaccaa 7860
gcccgttatt ctgacagttc tggtgctcaa cacatttata tttatcaagg agcacattgt 7920
tactcactgc taggagggaa tcgaactagg aatattgatc agaggaacta cgagagagct 7980
gaagataact gccctctagc tctcactgat ctgggtcgca tagtgagatg cagcccacgt 8040
gagttcagca acggtctagc gctgggcttt taggcccgca tgatcgggct tttgtcgggt 8100
ggtcgacgtg ttcacgattg gggagagcaa cgcagcagtt cctcttagtt tagtcccacc 8160
tcgcctgtcc agcagagttc tgaccggttt ataaactcgc ttgctgcatc agacttgcct 8220
tggtcttcct gacggcgttt tagagctaga aatagcaagt taaaataagg ctagtccgtt 8280
atcaacttga aaaagtggca ccgagtcggt gctttttttt tgaccaagcc cgttattctg 8340
acagttctgg tgctcaacac atttatattt atcaaggagc acattgttac tcactgctag 8400
gagggaatcg aactaggaat attgatcaga ggaactacga gagagctgaa gataactgcc 8460
ctctagctct cactgatctg ggtcgcatag tgagatgcag cccacgtgag ttcagcaacg 8520
gtctagcgct gggcttttag gcccgcatga tcgggctttt gtcgggtggt cgacgtgttc 8580
acgattgggg agagcaacgc agcagttcct cttagtttag tcccacctcg cctgtccagc 8640
agagttctga ccggtttata aactcgcttg ctgcatcaga cttgacggaa tgcagaggcg 8700
agcgttttag agctagaaat agcaagttaa aataaggcta gtccgttatc aacttgaaaa 8760
agtggcaccg agtcggtgct ttttttttcc taggctaatt agctaactgc agtgcagcgt 8820
gacccggtcg tgcccctctc tagagataat gagcattgca tgtctaagtt ataaaaaatt 8880
accacatatt ttttttgtca cacttgtttg aagtgcagtt tatctatctt tatacatata 8940
tttaaacttt actctacgaa taatataatc tatagtacta caataatatc agtgttttag 9000
agaatcatat aaatgaacag ttagacatgg tctaaaggac aattgagtat tttgacaaca 9060
ggactctaca gttttatctt tttagtgtgc atgtgttctc cttttttttt gcaaatagct 9120
tcacctatat aatacttcat ccattttatt agtacatcca tttagggttt agggttaatg 9180
gtttttatag actaattttt ttagtacatc tattttattc tattttagcc tctaaattaa 9240
gaaaactaaa actctatttt agttttttta tttaataatt tagatataaa atagaataaa 9300
ataaagtgac taaaaattaa acaaataccc tttaagaaat taaaaaaact aaggaaacat 9360
ttttcttgtt tcgagtagat aatgccagcc tgttaaacgc cgtcgacgag tctaacggac 9420
accaaccagc gaaccagcag cgtcgcgtcg ggccaagcga agcagacggc acggcatctc 9480
tgtcgctgcc tctggacccc tctcgagagt tccgctccac cgttggactt gctccgctgt 9540
cggcatccag aaattgcgtg gcggagcggc agacgtgagc cggcacggca ggcggcctcc 9600
tcctcctctc acggcaccgg cagctacggg ggattccttt cccaccgctc cttcgctttc 9660
ccttcctcgc ccgccgtaat aaatagacac cccctccaca ccctctttcc ccaacctcgt 9720
gttgttcgga gcgcacacac acacaaccag atctccccca aatccacccg tcggcacctc 9780
cgcttcaagg tacgccgctc gtcctccccc cccccccctc tctaccttct ctagatcggc 9840
gttccggtcc atggttaggg cccggtagtt ctacttctgt tcatgtttgt gttagatccg 9900
tgtttgtgtt agatccgtgc tgctagcgtt cgtacacgga tgcgacctgt acgtcagaca 9960
cgttctgatt gctaacttgc cagtgtttct ctttggggaa tcctgggatg gctctagccg 10020
ttccgcagac gggatcgatt tcatgatttt ttttgtttcg ttgcataggg tttggtttgc 10080
ccttttcctt tatttcaata tatgccgtgc acttgtttgt cgggtcatct tttcatgctt 10140
ttttttgtct tggttgtgat gatgtggtct ggttgggcgg tcgttctaga tcggagtaga 10200
attctgtttc aaactacctg gtggatttat taattttgga tctgtatgtg tgtgccatac 10260
atattcatag ttacgaattg aagatgatgg atggaaatat cgatctagga taggtataca 10320
tgttgatgcg ggttttactg atgcatatac agagatgctt tttgttcgct tggttgtgat 10380
gatgtggtgt ggttgggcgg tcgttcattc gttctagatc ggagtagaat actgtttcaa 10440
actacctggt gtatttatta attttggaac tgtatgtgtg tgtcatacat cttcatagtt 10500
acgagtttaa gatggatgga aatatcgatc taggataggt atacatgttg atgtgggttt 10560
tactgatgca tatacatgat ggcatatgca gcatctattc atatgctcta accttgagta 10620
cctatctatt ataataaaca agtatgtttt ataattattt tgatcttgat atacttggat 10680
gatggcatat gcagcagcta tatgtggatt tttttagccc tgccttcata cgctatttat 10740
ttgcttggta ctgtttcttt tgtcgatgct caccctgttg tttggtgtta cttctgcagt 10800
gactaaatag atgcagaagc tgatcaacag cgtgcagaac tacgcctggg gcagcaagac 10860
cgccctgacc gagctgtacg gcatggagaa ccccagcagc cagcccatgg ccgagctgtg 10920
gatgggcgcc caccccaaga gctcaagccg cgtgcagaac gccgccggcg atatcgttag 10980
cctgcgcgac gtgatcgaga gcgacaagag caccctgctg ggcgaggccg tggccaagcg 11040
cttcggcgag ctgcccttcc tgttcaaggt gctgtgcgcc gctcagcccc tgagcatcca 11100
ggtgcaccct aacaagcaca acagcgagat cggcttcgcc aaggagaacg ccgccggcat 11160
ccccatggac gccgccgagc gcaactacaa ggaccccaac cacaagcccg agctggtgtt 11220
cgccctgacc cccttcctgg ccatgaacgc cttccgcgag ttcagcgaga tcgttagcct 11280
gctgcagccc gtggccggcg cccaccccgc tatcgcccac ttccttcagc agcccgacgc 11340
cgagcgcctg agcgagctgt tcgccagcct gctgaacatg cagggtgagg agaagtcacg 11400
cgccctggcc atcctgaaga gcgccctgga cagccagcag ggcgagccct ggcagacaat 11460
ccgcctgatc agcgagttct accccgagga tagcggcctg ttcagccccc tgctgctgaa 11520
cgtggtgaag ctgaaccccg gcgaggccat gttcctgttc gccgagaccc cccacgccta 11580
cctgcagggc gtggccctgg aggtgatggc caacagcgac aacgtgctgc gcgccggcct 11640
gacccccaag tacatcgaca tccccgagct ggtggccaac gtgaagttcg aggctaagcc 11700
cgccaaccag ctgctgaccc agcccgtgaa gcagggcgcc gagctggact tccctatccc 11760
cgttgacgac ttcgccttca gcctgcacga cctgagcgac aaggagacca ctatcagcca 11820
gcagagcgcc gcgatcctgt tctgcgtgga gggcgacgcc accctgtgga agggcagcca 11880
gcagctgcag ctgaagcccg gcgagagcgc ctttatcgcc gccaacgaga gccccgtgac 11940
cgtgaagggc cacggccgcc tggcccgcgt gtacaacaag ctgtgatagc tacgtcatgg 12000
gtcgtttaag ctgccgatgt gcctgcgtcg tctggtgccc tctctccata tggaggttgt 12060
caaagtatct gctgttcgtg tcatgagtcg tgtcagtgtt ggtttaataa tggaccggtt 12120
gtgttgtgtg tgcgtactac ccagaactat gacaaatcat gaataagttt gatgtttgaa 12180
attaaagcct gtgctcatta tgttctgtct ttcagttgtc tcctaatatt tgcctgcagg 12240
tactggctat ctaccgtttc ttacttagga ggtgtttgaa tgcactaaaa ctaatagtta 12300
gtggctaaaa ttagttaaaa catccaaaca ccatagctaa tagttgaact attagctatt 12360
tttggaaaat tagttaatag tgaggtagtt atttgttagc tagctaattc aactaacaat 12420
ttttagccaa ctaacaatta gtttcagtgc attcaaacac ccccttaatg ttaacgtggt 12480
tctatctacc gtctcctaat atatggttga ttgttcggtt tgttgctatg ctattgggtt 12540
ctgattgctg ctagttcttg ctgaatccag aagttctcgt agtatagctc agattcatat 12600
tatttatttg agtgataagt gatccaggtt attactatgt tagctaggtt ttttttacaa 12660
ggataaatta tctgtgatca taattcttat gaaagcttta tgtttcctgg aggcagtggc 12720
atgcaatgca tgacagcaac ttgatcacac cagctgaggt agatacggta acaaggttct 12780
taaatctgtt caccaaatca ttggagaaca cacatacaca ttcttgccag tcttggttag 12840
agaaatttca tgacaaaatg ccaaagctgt cttgactctt cacttttggc catgagtcgt 12900
gacttagttt ggtttaatgg accggttctc ctagcttgtt ctactcaaaa ctgttgttga 12960
tgcgaataag ttgtgatggt tgatctctgg attttgtttt gctctcaata gtggacgaga 13020
ttagatagct taagcctgca ggcggaccgc ctgcaggccc gggggcgcgc cctaattagc 13080
taacggccag gatcgccgcg tgagccttta gcaactagct agattaatta acgcaatctg 13140
ttattaagtt gtctaagcgt caatttgttt acaccacaat atatcctgcc accagccagc 13200
caacagctcc ccgaccggca gctcggcaca aaatcaccac tcgatacagg cagcccatca 13260
gaattaattc tcatgtttga cagcttatca tcgactgcac ggtgcaccaa tgcttctggc 13320
gtcaggcagc catcggaagc tgtggtatgg ctgtgcaggt cgtaaatcac tgcataattc 13380
gtgtcgctca aggcgcactc ccgttctgga taatgttttt tgcgccgaca tcataacggt 13440
tctggcaaat attctgaaat gagctgttga caattaatca tccggctcgt ataatgtgtg 13500
gaattgtgag cggataacaa tttcacacag gaaacagacc atgagggaag cgttgatcgc 13560
cgaagtatcg actcaactat cagaggtagt tggcgtcatc gagcgccatc tcgaaccgac 13620
gttgctggcc gtacatttgt acggctccgc agtggatggc ggcctgaagc cacacagtga 13680
tattgatttg ctggttacgg tgaccgtaag gcttgatgaa acaacgcggc gagctttgat 13740
caacgacctt ttggaaactt cggcttcccc tggagagagc gagattctcc gcgctgtaga 13800
agtcaccatt gttgtgcacg acgacatcat tccgtggcgt tatccagcta agcgcgaact 13860
gcaatttgga gaatggcagc gcaatgacat tcttgcaggt atcttcgagc cagccacgat 13920
cgacattgat ctggctatct tgctgacaaa agcaagagaa catagcgttg ccttggtagg 13980
tccagcggcg gaggaactct ttgatccggt tcctgaacag gatctatttg aggcgctaaa 14040
tgaaacctta acgctatgga actcgccgcc cgactgggct ggcgatgagc gaaatgtagt 14100
gcttacgttg tcccgcattt ggtacagcgc agtaaccggc aaaatcgcgc cgaaggatgt 14160
cgctgccgac tgggcaatgg agcgcctgcc ggcccagtat cagcccgtca tacttgaagc 14220
taggcaggct tatcttggac aagaagatcg cttggcctcg cgcgcagatc agttggaaga 14280
atttgttcac tacgtgaaag gcgagatcac caaagtagtc ggcaaataaa gctctagtgg 14340
atctccgtac ccagggatct ggctcgcggc ggacgcacga cgccggggcg agaccatagg 14400
cgatctccta aatcaatagt agctgtaacc tcgaagcgtt tcacttgtaa caacgattga 14460
gaatttttgt cataaaattg aaatacttgg ttcgcatttt tgtcatccgc ggtcagccgc 14520
aattctgacg aactgcccat ttagctggag atgattgtac atccttcacg tgaaaatttc 14580
tcaagcgctg tgaacaaggg ttcagatttt agattgaaag gtgagccgtt gaaacacgtt 14640
cttcttgtcg atgacgacgt cgctatgcgg catcttatta ttgaatacct tacgatccac 14700
gccttcaaag tgaccgcggt agccgacagc acccagttca caagagtact ctcttccgcg 14760
acggtcgatg tcgtggttgt tgatctagat ttaggtcgtg aagatgggct cgagatcgtt 14820
cgtaatctgg cggcaaagtc tgatattcca atcataatta tcagtggcga ccgccttgag 14880
gagacggata aagttgttgc actcgagcta ggagcaagtg attttatcgc taagccgttc 14940
agtatcagag agtttctagc acgcattcgg gttgccttgc gcgtgcgccc caacgttgtc 15000
cgctccaaag accgacggtc tttttgtttt actgactgga cacttaatct caggcaacgt 15060
cgcttgatgt ccgaagctgg cggtgaggtg aaacttacgg caggtgagtt caatcttctc 15120
ctcgcgtttt tagagaaacc ccgcgacgtt ctatcgcgcg agcaacttct cattgccagt 15180
cgagtacgcg acgaggaggt ttatgacagg agtatagatg ttctcatttt gaggctgcgc 15240
cgcaaacttg aggcagatcc gtcaagccct caactgataa aaacagcaag aggtgccggt 15300
tatttctttg acgcggacgt gcaggtttcg cacgggggga cgatggcagc ctgagccaat 15360
tcccagatcc ccgaggaatc ggcgtgagcg gtcgcaaacc atccggcccg gtacaaatcg 15420
gcgcggcgct gggtgatgac ctggtggaga agttgaaggc cgcgcaggcc gcccagcggc 15480
aacgcatcga ggcagaagca cgccccggtg aatcgtggca agcggccgct gatcgaatcc 15540
gcaaagaatc ccggcaaccg ccggcagccg gtgcgccgtc gattaggaag ccgcccaagg 15600
gcgacgagca accagatttt ttcgttccga tgctctatga cgtgggcacc cgcgatagtc 15660
gcagcatcat ggacgtggcc gttttccgtc tgtcgaagcg tgaccgacga gctggcgagg 15720
tgatccgcta cgagcttcca gacgggcacg tagaggtttc cgcagggccg gccggcatgg 15780
ccagtgtgtg ggattacgac ctggtactga tggcggtttc ccatctaacc gaatccatga 15840
accgataccg ggaagggaag ggagacaagc ccggccgcgt gttccgtcca cacgttgcgg 15900
acgtactcaa gttctgccgg cgagccgatg gcggaaagca gaaagacgac ctggtagaaa 15960
cctgcattcg gttaaacacc acgcacgttg ccatgcagcg tacgaagaag gccaagaacg 16020
gccgcctggt gacggtatcc gagggtgaag ccttgattag ccgctacaag atcgtaaaga 16080
gcgaaaccgg gcggccggag tacatcgaga tcgagctggc tgattggatg taccgcgaga 16140
tcacagaagg caagaacccg gacgtgctga cggttcaccc cgattacttt ttgatcgatc 16200
ccggcatcgg ccgttttctc taccgcctgg cacgccgcgc cgcaggcaag gcagaagcca 16260
gatggttgtt caagacgatc tacgaacgca gtggcagcgc cggagagttc aagaagttct 16320
gtttcaccgt gcgcaagctg atcgggtcaa atgacctgcc ggagtacgat ttgaaggagg 16380
aggcggggca ggctggcccg atcctagtca tgcgctaccg caacctgatc gagggcgaag 16440
catccgccgg ttcctaatgt acggagcaga tgctagggca aattgcccta gcaggggaaa 16500
aaggtcgaaa aggtctcttt cctgtggata gcacgtacat tgggaaccca aagccgtaca 16560
ttgggaaccg gaacccgtac attgggaacc caaagccgta cattgggaac cggtcacaca 16620
tgtaagtgac tgatataaaa gagaaaaaag gcgatttttc cgcctaaaac tctttaaaac 16680
ttattaaaac tcttaaaacc cgcctggcct gtgcataact gtctggccag cgcacagccg 16740
aagagctgca aaaagcgcct acccttcggt cgctgcgctc cctacgcccc gccgcttcgc 16800
gtcggcctat cgcggccgct ggccgctcaa aaatggctgg cctacggcca ggcaatctac 16860
cagggcgcgg acaagccgcg ccgtcgccac tcgaccgccg gcgctgaggt ctgcctcgtg 16920
aagaaggtgt tgctgactca taccaggcct gaatcgcccc atcatccagc cagaaagtga 16980
gggagccacg gttgatgaga gctttgttgt aggtggacca gttggtgatt ttgaactttt 17040
gctttgccac ggaacggtct gcgttgtcgg gaagatgcgt gatctgatcc ttcaactcag 17100
caaaagttcg atttattcaa caaagccgcc gtcccgtcaa gtcagcgtaa tgctctgcca 17160
gtgttacaac caattaacca attctgatta gaaaaactca tcgagcatca aatgaaactg 17220
caatttattc atatcaggat tatcaatacc atatttttga aaaagccgtt tctgtaatga 17280
aggagaaaac tcaccgaggc agttccatag gatggcaaga tcctggtatc ggtctgcgat 17340
tccgactcgt ccaacatcaa tacaacctat taatttcccc tcgtcaaaaa taaggttatc 17400
aagtgagaaa tcaccatgag tgacgactga atccggtgag aatggcaaaa gctctgcatt 17460
aatgaatcgg ccaacgcgcg gggagaggcg gtttgcgtat tgggcgctct tccgcttcct 17520
cgctcactga ctcgctgcgc tcggtcgttc ggctgcggcg agcggtatca gctcactcaa 17580
aggcggtaat acggttatcc acagaatcag gggataacgc aggaaagaac atgtgagcaa 17640
aaggccagca aaaggccagg aaccgtaaaa aggccgcgtt gctggcgttt ttccataggc 17700
tccgcccccc tgacgagcat cacaaaaatc gacgctcaag tcagaggtgg cgaaacccga 17760
caggactata aagataccag gcgtttcccc ctggaagctc cctcgtgcgc tctcctgttc 17820
cgaccctgcc gcttaccgga tacctgtccg cctttctccc ttcgggaagc gtggcgcttt 17880
ctcatagctc acgctgtagg tatctcagtt cggtgtaggt cgttcgctcc aagctgggct 17940
gtgtgcacga accccccgtt cagcccgacc gctgcgcctt atccggtaac tatcgtcttg 18000
agtccaaccc ggtaagacac gacttatcgc cactggcagc agccactggt aacaggatta 18060
gcagagcgag gtatgtaggc ggtgctacag agttcttgaa gtggtggcct aactacggct 18120
acactagaag aacagtattt ggtatctgcg ctctgctgaa gccagttacc ttcggaaaaa 18180
gagttggtag ctcttgatcc ggcaaacaaa ccaccgctgg tagcggtggt ttttttgttt 18240
gcaagcagca gattacgcgc agaaaaaaag gatctcaaga agatcctttg atcttttcta 18300
cggggtctga cgctcagtgg aacgaaaact cacgttaagg gattttggtc atgagattat 18360
caaaaaggat cttcacctag atccttttga tccggaatta 18400
<210> 75
<211> 18402
<212> DNA
<213> Artificial Sequence
<220>
<223> Construct 24194
<220>
<221> misc_feature
<222> (4)..(259)
<223> bNRB-05
<220>
<221> misc_feature
<222> (101)..(125)
<223> bNRB-01-01
<220>
<221> enhancer
<222> (168)..(259)
<223> eNOS-01
<220>
<221> enhancer
<222> (292)..(485)
<223> eFMV-06
<220>
<221> enhancer
<222> (492)..(784)
<223> e35S-11
<220>
<221> promoter
<222> (791)..(2592)
<223> prSoUbi4-02
<220>
<221> misc_feature
<222> (1170)..(1234)
<223> u5SoUbi4-02
<220>
<221> Intron
<222> (1235)..(2592)
<223> iSoUbi4-02
<220>
<221> misc_feature
<222> (2607)..(2657)
<223> NLS
<220>
<221> gene
<222> (2607)..(6827)
<223> CRISPR associated 9
<220>
<221> misc_feature
<222> (2610)..(2610)
<223> +4G
<220>
<221> misc_feature
<222> (2611)..(2611)
<223> +5C
<220>
<221> mutation
<222> (6147)..(6149)
<223> L to V mutation
<220>
<221> mutation
<222> (6192)..(6194)
<223> I to V mutation
<220>
<221> misc_feature
<222> (6762)..(6824)
<223> NLS
<220>
<221> terminator
<222> (6834)..(7835)
<223> tZmMTL-01
<220>
<221> misc_feature
<222> (7836)..(7847)
<223> xSTOPS-01
<220>
<221> promoter
<222> (7855)..(8216)
<223> prTaU6-01
<220>
<221> misc_feature
<222> (8217)..(8322)
<223> guide RNA TaCenH3-01
<220>
<221> misc_feature
<222> (8218)..(8237)
<223> xTaCenH3 target-01
<220>
<221> misc_feature
<222> (8238)..(8249)
<223> crRNA-01
<220>
<221> misc_feature
<222> (8254)..(8322)
<223> tracrRNA-01
<220>
<221> promoter
<222> (8323)..(8684)
<223> prTaU6-01
<220>
<221> misc_feature
<222> (8685)..(8790)
<223> guide RNA TaCenH3-02
<220>
<221> misc_feature
<222> (8686)..(8705)
<223> xTaCenH3 target-02
<220>
<221> misc_feature
<222> (8706)..(8717)
<223> crRNA-01
<220>
<221> misc_feature
<222> (8722)..(8790)
<223> tracrRNA-01
<220>
<221> misc_feature
<222> (8797)..(8808)
<223> xSTOPS-01
<220>
<221> promoter
<222> (8809)..(10801)
<223> prUbi1-10
<220>
<221> TATA_signal
<222> (9679)..(9687)
<223> TATA box
<220>
<221> Intron
<222> (9792)..(10801)
<223> iUbi1-02-01
<220>
<221> gene
<222> (10813)..(11991)
<223> cPMI-12
<220>
<221> terminator
<222> (11996)..(13030)
<223> tUbi1-01
<220>
<221> misc_feature
<222> (13074)..(13085)
<223> xSTOPS-01
<220>
<221> misc_feature
<222> (13086)..(13125)
<223> xTAG-02
<220>
<221> misc_feature
<222> (13134)..(13263)
<223> bNLB-05
<220>
<221> misc_feature
<222> (13169)..(13193)
<223> bNLB-01-01
<220>
<221> gene
<222> (13543)..(14331)
<223> cSpec-03
<220>
<221> promoter
<222> (14426)..(14556)
<223> prVirG-01
<220>
<221> gene
<222> (14631)..(15356)
<223> cVirG-01
<220>
<221> gene
<222> (15386)..(16459)
<223> cRepA-03
<220>
<221> misc_feature
<222> (16502)..(16906)
<223> oVS1-02
<220>
<221> misc_feature
<222> (17584)..(18390)
<223> oCOLE-06
<400> 75
attcctgtgg ttggcatgca catacaaatg gacgaacgga taaacctttt cacgcccttt 60
taaatatccg attattctaa taaacgctct tttctcttag gtttacccgc caatatatcc 120
tgtcaaacac tgatagttta aactgaaggc gggaaacgac aatctgatca tgagcggaga 180
attaagggag tcacgttatg acccccgccg atgacgcggg acaagccgtt ttacgtttgg 240
aactgacaga accgcaacgc tgcaggaatt ggccgcagcg gccatttaaa tagctgcttg 300
tggggaccag acaaaaaagg aatggtgcag aattgttagg cgcacctacc aaaagcaact 360
ttgcctttat tgcaaagata aagcagattc ctctagtaca agtggggaac aaaataacgt 420
ggaaaagagc tgtcctgaca gcccactcac tattgcgttt gacgaacgca gtgacgacca 480
caaaactcga gacttttcaa caaagggtat tatccggaaa cctcctcgga ttccattgcc 540
cagctatctg tcactttatt gtgaagatag tggaaaagga aggtggctcc tacaaatgcc 600
atcattgcga taaaggaaag gctatcgttg aagatgcctc tgccgacagt ggtcccaaag 660
atggaccccc acccacgagg agcatcgtgg aaaaagaaga cgttccaacc acgtcttcaa 720
agcaagtgga ttgatgtgat atctccactg acgtaagggt tgacgaacaa tcccactatc 780
cttcggaccc gaattcatta tgtggtctag gtaggttcta tatataagaa aacttgaaat 840
gttctaaaaa aaaattcaag cccatgcatg attgaagcaa acggtatagc aacggtgtta 900
acctgatcta gtgatctctt gcaatcctta acggccacct accgcaggta gcaaacggcg 960
tccccctcct cgatatctcc gcggcgacct ctggcttttt ccgcggaatt gcgcggtggg 1020
gacggattcc acgagaccgc gacgcaaccg cctctcgccg ctgggcccca caccgctcgg 1080
tgccgtagcc tcacgggact ctttctccct cctcccccgt tataaattgg cttcatcccc 1140
tccttgcctc atccatccaa atcccagtcc ccaatcccat cccttcgtag gagaaattca 1200
tcgaagctaa gcgaatcctc gcgatcctct caaggtactg cgagttttcg atccccctct 1260
cgacccctcg tatgtttgtg tttgtcgtag cgtttgatta ggtatgcttt ccctgtttgt 1320
gttcgtcgta gcgtttgatt aggtatgctt tccctgttcg tgttcatcgt agtgtttgat 1380
taggtcgtgt gaggcgatgg cctgctcgcg tccttcgatc tgtagtcgat ttgcgggtcg 1440
tggtgtagat ctgcgggctg tgatgaagtt atttggtgtg atctgctcgc ctgattctgc 1500
gggttggctc gagtagatat gatggttgga ccggttggtt cgtttaccgc gctagggttg 1560
ggctgggatg atgttgcatg cgccgttgcg cgtgatcccg cagcaggact tgcgtttgat 1620
tgccagatct cgttacgatt atgtgatttg gtttggactt tttagatctg tagcttctgc 1680
ttatgtgcca gatgcgccta ctgctcatat gcctgatgat aatcataaat ggctgtggaa 1740
ctaactagtt gattgcggag tcatgtatca gctacaggtg tagggactag ctacaggtgt 1800
agggacttgc gtctaattgt ttggtccttt actcatgttg caattatgca atttagttta 1860
gattgtttgt tccactcatc taggctgtaa aagggacact gcttagattg ctgtttaatc 1920
tttttagtag attatattat attggtaact tattacccct attacatgcc atacgtgact 1980
tctgctcatg cctgatgata atcatagatc actgtggaat taattagttg attgttgaat 2040
catgtttcat gtacatacca cggcacaatt gcttagttcc ttaacaaatg caaattttac 2100
tgatccatgt atgatttgcg tggttctcta atgtgaaata ctatagctac ttgttagtaa 2160
gaatcaggtt cgtatgctta atgctgtatg tgccttctgc tcatgcctga tgataatcat 2220
atatcactgg aattaattag ttgatcgttt aatcatatat caagtacata ccatgccaca 2280
atttttagtc acttaaccca tgcagattga actggtccct gcatgttttg ctaaattgtt 2340
ctattctgat tagaccatat atcatgtatt tttttttggt aatggttctc ttattttaaa 2400
tgctatatag ttctggtact tgttagaaag atctgcttca tagtttagtt gcctatccct 2460
cgaattagga tgctgagcag ctgatcctat agctttgttt catgtatcaa ttcttttgtg 2520
ttcaacagtc agtttttgtt agattcattg taacttatgg tcgcttactc ttctggtcct 2580
caatgcttgc agcctagtaa gccgccatgg ccccaaagaa gaagaggaag gtcggcatcc 2640
acggcgtccc agctgcgatg gacaagaagt actccatcgg cctcgacatc ggcaccaaca 2700
gcgtgggctg ggccgtcatc accgacgagt acaaggtgcc atccaagaag ttcaaggtcc 2760
tgggcaacac cgaccgccac agcatcaaga agaacctcat cggcgctctc ctgttcgact 2820
ccggcgagac ggctgaggct accaggctca agcgcaccgc caggaggagg tacaccagga 2880
ggaagaacag gatctgctac ctccaagaga tcttctccaa cgagatggcc aaggtggacg 2940
actccttctt ccaccgcctg gaggagagct tcctcgtcga ggaggacaag aagcacgaga 3000
ggcacccaat cttcggcaac atcgtggacg aggtcgccta ccacgagaag tacccaacca 3060
tctaccacct gaggaagaag ctcgtggact ccaccgacaa ggccgacctc cgcctgatct 3120
acctcgccct ggcccacatg atcaagttca ggggccactt cctgatcgag ggcgacctca 3180
acccagacaa cagcgacgtg gacaagctgt tcatccaact cgtccagacc tacaaccagc 3240
tcttcgagga gaacccgatc aacgcttccg gcgtggacgc taaggctatc ctgagcgcta 3300
ggctctccaa gagcaggagg ctcgagaacc tgatcgccca gctcccaggc gagaagaaga 3360
acggcctgtt cggcaacctc atcgctctct ccctgggcct caccccaaac ttcaagagca 3420
acttcgacct cgccgaggac gccaagctgc aactctccaa ggacacctac gacgacgacc 3480
tggacaacct cctggcccag atcggcgacc aatacgccga cctgttcctc gccgccaaga 3540
acctgtccga cgccatcctc ctgagcgaca tcctccgcgt gaacaccgag atcaccaagg 3600
ccccactctc cgccagcatg atcaagcgct acgacgagca ccaccaggac ctgaccctcc 3660
tgaaggccct ggtcaggcaa cagctcccag agaagtacaa ggagatcttc ttcgaccaga 3720
gcaagaacgg ctacgctggc tacatcgacg gcggcgcctc ccaagaggag ttctacaagt 3780
tcatcaagcc aatcctggag aagatggacg gcaccgagga gctcctggtg aagctcaaca 3840
gggaggacct cctgaggaag cagcgcacct tcgacaacgg cagcatccca caccaaatcc 3900
acctcggcga gctgcacgct atcctgagga ggcaagagga cttctaccca ttcctcaagg 3960
acaacaggga gaagatcgag aagatcctga ccttccgcat cccctactac gtcggcccac 4020
tcgctagggg caactccagg ttcgcttgga tgacccgcaa gagcgaggag acgatcaccc 4080
cgtggaactt cgaggaggtc gtcgacaagg gcgcttccgc tcagagcttc atcgagagga 4140
tgaccaactt cgacaagaac ctgccaaacg agaaggtgct cccaaagcac tccctcctgt 4200
acgagtactt caccgtctac aacgagctca ccaaggtgaa gtatgtgacc gagggcatgc 4260
gcaagccagc cttcctgagc ggcgagcaga agaaggccat cgtggacctc ctgttcaaga 4320
ccaacaggaa ggtgaccgtc aagcaactca aggaggacta cttcaagaag atcgagtgct 4380
tcgactccgt ggagatcagc ggcgtcgagg accgcttcaa cgcctccctc ggcacctacc 4440
acgacctcct gaagatcatc aaggacaagg acttcctgga caacgaggag aacgaggaca 4500
tcctcgagga catcgtgctg accctcaccc tgttcgagga cagggagatg atcgaggagc 4560
gcctgaagac ctacgcccac ctcttcgacg acaaggtcat gaagcaactc aagaggagga 4620
ggtacaccgg ctggggcagg ctgagccgca agctcatcaa cggcatccgc gacaagcagt 4680
ccggcaagac catcctcgac ttcctgaaga gcgacggctt cgccaacagg aacttcatgc 4740
aactgatcca cgacgactcc ctcaccttca aggaggacat ccaaaaggct caggtgtccg 4800
gccagggcga cagcctgcac gagcacatcg ctaacctcgc tggcagccca gccatcaaga 4860
agggcatcct gcagaccgtg aaggtcgtcg acgagctcgt gaaggtcatg ggcaggcaca 4920
agccagagaa catcgtcatc gagatggccc gcgagaacca gaccacccag aagggccaaa 4980
agaactccag ggagcgcatg aagcgcatcg aggagggcat caaggagctg ggcagccaaa 5040
tcctcaagga gcacccagtg gagaacaccc aactgcagaa cgagaagctc tacctgtact 5100
acctccagaa cggcagggac atgtatgtgg accaagagct ggacatcaac cgcctctccg 5160
actacgacgt ggaccacatc gtcccacagt ccttcctgaa ggacgacagc atcgacaaca 5220
aggtgctcac caggagcgac aagaaccgcg gcaagtccga caacgtccca agcgaggagg 5280
tggtcaagaa gatgaaaaac tactggaggc agctcctgaa cgccaagctg atcacccaaa 5340
ggaagttcga caacctcacc aaggctgaga ggggcggcct ctccgagctg gacaaggccg 5400
gcttcattaa aaggcagctg gtggagacgc gccaaatcac caagcacgtc gcccaaatcc 5460
tcgacagccg catgaacacc aagtacgacg agaacgacaa gctgatcagg gaggtgaagg 5520
tcatcaccct gaagtccaag ctcgtgagcg acttcaggaa ggacttccag ttctacaagg 5580
tccgcgagat caataattac caccacgccc acgacgctta cctcaacgct gtggtcggca 5640
ccgccctgat taaaaagtac ccaaagctcg agtccgagtt cgtgtacggc gactacaagg 5700
tgtacgacgt ccgcaagatg atcgccaagt ccgagcaaga gatcggcaag gccaccgcca 5760
agtacttctt ctacagcaac atcatgaact tcttcaagac cgagatcacc ctggccaacg 5820
gcgagatcag gaagcgccca ctcatcgaga cgaacggcga gacgggcgag atcgtgtggg 5880
acaagggcag ggacttcgcc accgtgcgca aggtcctctc catgccacag gtgaacatcg 5940
tcaagaagac cgaggtccaa accggcggct tctccaagga gagcatcctg ccaaagagga 6000
acagcgacaa gctcatcgcc cgcaagaagg actgggatcc aaagaagtac ggcggcttcg 6060
actccccaac cgtggcctac agcgtcctcg tggtcgccaa ggtggagaag ggcaagtcca 6120
agaagctgaa gagcgtgaag gagctcgtcg gcatcaccat catggagagg tccagcttcg 6180
agaagaaccc agtggacttc ctcgaggcca agggctacaa ggaggtcaag aaggacctga 6240
tcattaaact cccaaagtac agcctcttcg agctggagaa cggcaggaag cgcatgctgg 6300
cttccgctgg cgagctccaa aagggcaacg agctcgccct gccatccaag tatgtgaact 6360
tcctctacct ggcctcccac tacgagaagc tcaagggcag cccagaggac aacgagcaaa 6420
agcagctgtt cgtcgagcag cacaagcact acctcgacga gatcatcgag caaatctccg 6480
agttcagcaa gcgcgtgatc ctcgccgacg ccaacctgga caaggtcctc tccgcctaca 6540
acaagcacag ggacaagcca atccgcgagc aggccgagaa catcatccac ctcttcaccc 6600
tgaccaacct cggcgctcca gctgccttca agtacttcga caccaccatc gacaggaagc 6660
gctacacctc caccaaggag gtgctggacg ccaccctcat ccaccagtcc atcaccggcc 6720
tctacgagac gaggatcgac ctgagccaac tcggcggcga ctccagccca ccaaagaaga 6780
agaggaaggt cagctggaag gacgcttccg gctggagccg catgtgaggt acctcacatc 6840
gatcgacgac caaggatatg attattatct atctagcttg tggtggtggt tgaacaataa 6900
taagcgaggc cgagctggct gccatacata ggtattgtgt ggtgtgtgtg agagagagag 6960
aaacagagtt cttcagtttg ctatctctct ctgcatgttt ggcgtcagtc tttgtgctca 7020
tgtacgtacg tgtgtctacc tgcatgttgg ttgatccgat tgcatctgct gtaaccatat 7080
attaattggt ccacgatgat atgatttgat actatatata tactaaaacc ggacttctta 7140
ttataatact tgtagtatat aagtttctta cgcccgcaat tgatcgattc agaaggagtt 7200
ctagctagct aaaacatgca gattcagaat atcagatttt taggactact ggagatccag 7260
aaccttcgtg tccttgtacc cgtgattttg gatccccttt ctccccacta caatcgttgg 7320
cgcaatcttg ttctgctcgc ctagaatggt acgccctcac cgaccatgtc ctcgccgatg 7380
cctaccccgc cacgccatct ggatggtgca tgaatgtcgt cgttttgtcc tggatgctag 7440
gcacactctc ccccgagttg atggagcctc ctcgcacctc tggtggcact gcgccgcgcc 7500
tggcttgcca tcgaggagca attcctcggc aatcgcgaag ctcgcacact tcgtctcgac 7560
gccgagttcc atgtcttcat gtaggcgatc tcttcgtcag cgactattgt ctattgacgc 7620
aagatgaagg ggatggacga ggcccttggt gatcttggtg aggtcatcca tgaccgtacc 7680
cttgtcctaa acgtgttgtg tggtctgaat gagaggtttg cccacataaa ggtccacttc 7740
aagcactcga atccgttccc ctccttcacc gacgtttgta atgatctcat ccttgaggag 7800
atcgactcca gcgcgcctcc tccgcctccg accacctaat tagctaaggg acccgaccaa 7860
gcccgttatt ctgacagttc tggtgctcaa cacatttata tttatcaagg agcacattgt 7920
tactcactgc taggagggaa tcgaactagg aatattgatc agaggaacta cgagagagct 7980
gaagataact gccctctagc tctcactgat ctgggtcgca tagtgagatg cagcccacgt 8040
gagttcagca acggtctagc gctgggcttt taggcccgca tgatcgggct tttgtcgggt 8100
ggtcgacgtg ttcacgattg gggagagcaa cgcagcagtt cctcttagtt tagtcccacc 8160
tcgcctgtcc agcagagttc tgaccggttt ataaactcgc ttgctgcatc agacttgacg 8220
tcggcgacac cggtgcggtt ttagagctag aaatagcaag ttaaaataag gctagtccgt 8280
tatcaacttg aaaaagtggc accgagtcgg tgcttttttt ttgaccaagc ccgttattct 8340
gacagttctg gtgctcaaca catttatatt tatcaaggag cacattgtta ctcactgcta 8400
ggagggaatc gaactaggaa tattgatcag aggaactacg agagagctga agataactgc 8460
cctctagctc tcactgatct gggtcgcata gtgagatgca gcccacgtga gttcagcaac 8520
ggtctagcgc tgggctttta ggcccgcatg atcgggcttt tgtcgggtgg tcgacgtgtt 8580
cacgattggg gagagcaacg cagcagttcc tcttagttta gtcccacctc gcctgtccag 8640
cagagttctg accggtttat aaactcgctt gctgcatcag acttgcttgt gggagcaggg 8700
gcaacgtttt agagctagaa atagcaagtt aaaataaggc tagtccgtta tcaacttgaa 8760
aaagtggcac cgagtcggtg cttttttttt cctaggctaa ttagctaact gcagtgcagc 8820
gtgacccggt cgtgcccctc tctagagata atgagcattg catgtctaag ttataaaaaa 8880
ttaccacata ttttttttgt cacacttgtt tgaagtgcag tttatctatc tttatacata 8940
tatttaaact ttactctacg aataatataa tctatagtac tacaataata tcagtgtttt 9000
agagaatcat ataaatgaac agttagacat ggtctaaagg acaattgagt attttgacaa 9060
caggactcta cagttttatc tttttagtgt gcatgtgttc tccttttttt ttgcaaatag 9120
cttcacctat ataatacttc atccatttta ttagtacatc catttagggt ttagggttaa 9180
tggtttttat agactaattt ttttagtaca tctattttat tctattttag cctctaaatt 9240
aagaaaacta aaactctatt ttagtttttt tatttaataa tttagatata aaatagaata 9300
aaataaagtg actaaaaatt aaacaaatac cctttaagaa attaaaaaaa ctaaggaaac 9360
atttttcttg tttcgagtag ataatgccag cctgttaaac gccgtcgacg agtctaacgg 9420
acaccaacca gcgaaccagc agcgtcgcgt cgggccaagc gaagcagacg gcacggcatc 9480
tctgtcgctg cctctggacc cctctcgaga gttccgctcc accgttggac ttgctccgct 9540
gtcggcatcc agaaattgcg tggcggagcg gcagacgtga gccggcacgg caggcggcct 9600
cctcctcctc tcacggcacc ggcagctacg ggggattcct ttcccaccgc tccttcgctt 9660
tcccttcctc gcccgccgta ataaatagac accccctcca caccctcttt ccccaacctc 9720
gtgttgttcg gagcgcacac acacacaacc agatctcccc caaatccacc cgtcggcacc 9780
tccgcttcaa ggtacgccgc tcgtcctccc cccccccccc tctctacctt ctctagatcg 9840
gcgttccggt ccatggttag ggcccggtag ttctacttct gttcatgttt gtgttagatc 9900
cgtgtttgtg ttagatccgt gctgctagcg ttcgtacacg gatgcgacct gtacgtcaga 9960
cacgttctga ttgctaactt gccagtgttt ctctttgggg aatcctggga tggctctagc 10020
cgttccgcag acgggatcga tttcatgatt ttttttgttt cgttgcatag ggtttggttt 10080
gcccttttcc tttatttcaa tatatgccgt gcacttgttt gtcgggtcat cttttcatgc 10140
ttttttttgt cttggttgtg atgatgtggt ctggttgggc ggtcgttcta gatcggagta 10200
gaattctgtt tcaaactacc tggtggattt attaattttg gatctgtatg tgtgtgccat 10260
acatattcat agttacgaat tgaagatgat ggatggaaat atcgatctag gataggtata 10320
catgttgatg cgggttttac tgatgcatat acagagatgc tttttgttcg cttggttgtg 10380
atgatgtggt gtggttgggc ggtcgttcat tcgttctaga tcggagtaga atactgtttc 10440
aaactacctg gtgtatttat taattttgga actgtatgtg tgtgtcatac atcttcatag 10500
ttacgagttt aagatggatg gaaatatcga tctaggatag gtatacatgt tgatgtgggt 10560
tttactgatg catatacatg atggcatatg cagcatctat tcatatgctc taaccttgag 10620
tacctatcta ttataataaa caagtatgtt ttataattat tttgatcttg atatacttgg 10680
atgatggcat atgcagcagc tatatgtgga tttttttagc cctgccttca tacgctattt 10740
atttgcttgg tactgtttct tttgtcgatg ctcaccctgt tgtttggtgt tacttctgca 10800
gtgactaaat agatgcagaa gctgatcaac agcgtgcaga actacgcctg gggcagcaag 10860
accgccctga ccgagctgta cggcatggag aaccccagca gccagcccat ggccgagctg 10920
tggatgggcg cccaccccaa gagctcaagc cgcgtgcaga acgccgccgg cgatatcgtt 10980
agcctgcgcg acgtgatcga gagcgacaag agcaccctgc tgggcgaggc cgtggccaag 11040
cgcttcggcg agctgccctt cctgttcaag gtgctgtgcg ccgctcagcc cctgagcatc 11100
caggtgcacc ctaacaagca caacagcgag atcggcttcg ccaaggagaa cgccgccggc 11160
atccccatgg acgccgccga gcgcaactac aaggacccca accacaagcc cgagctggtg 11220
ttcgccctga cccccttcct ggccatgaac gccttccgcg agttcagcga gatcgttagc 11280
ctgctgcagc ccgtggccgg cgcccacccc gctatcgccc acttccttca gcagcccgac 11340
gccgagcgcc tgagcgagct gttcgccagc ctgctgaaca tgcagggtga ggagaagtca 11400
cgcgccctgg ccatcctgaa gagcgccctg gacagccagc agggcgagcc ctggcagaca 11460
atccgcctga tcagcgagtt ctaccccgag gatagcggcc tgttcagccc cctgctgctg 11520
aacgtggtga agctgaaccc cggcgaggcc atgttcctgt tcgccgagac cccccacgcc 11580
tacctgcagg gcgtggccct ggaggtgatg gccaacagcg acaacgtgct gcgcgccggc 11640
ctgaccccca agtacatcga catccccgag ctggtggcca acgtgaagtt cgaggctaag 11700
cccgccaacc agctgctgac ccagcccgtg aagcagggcg ccgagctgga cttccctatc 11760
cccgttgacg acttcgcctt cagcctgcac gacctgagcg acaaggagac cactatcagc 11820
cagcagagcg ccgcgatcct gttctgcgtg gagggcgacg ccaccctgtg gaagggcagc 11880
cagcagctgc agctgaagcc cggcgagagc gcctttatcg ccgccaacga gagccccgtg 11940
accgtgaagg gccacggccg cctggcccgc gtgtacaaca agctgtgata gctacgtcat 12000
gggtcgttta agctgccgat gtgcctgcgt cgtctggtgc cctctctcca tatggaggtt 12060
gtcaaagtat ctgctgttcg tgtcatgagt cgtgtcagtg ttggtttaat aatggaccgg 12120
ttgtgttgtg tgtgcgtact acccagaact atgacaaatc atgaataagt ttgatgtttg 12180
aaattaaagc ctgtgctcat tatgttctgt ctttcagttg tctcctaata tttgcctgca 12240
ggtactggct atctaccgtt tcttacttag gaggtgtttg aatgcactaa aactaatagt 12300
tagtggctaa aattagttaa aacatccaaa caccatagct aatagttgaa ctattagcta 12360
tttttggaaa attagttaat agtgaggtag ttatttgtta gctagctaat tcaactaaca 12420
atttttagcc aactaacaat tagtttcagt gcattcaaac acccccttaa tgttaacgtg 12480
gttctatcta ccgtctccta atatatggtt gattgttcgg tttgttgcta tgctattggg 12540
ttctgattgc tgctagttct tgctgaatcc agaagttctc gtagtatagc tcagattcat 12600
attatttatt tgagtgataa gtgatccagg ttattactat gttagctagg ttttttttac 12660
aaggataaat tatctgtgat cataattctt atgaaagctt tatgtttcct ggaggcagtg 12720
gcatgcaatg catgacagca acttgatcac accagctgag gtagatacgg taacaaggtt 12780
cttaaatctg ttcaccaaat cattggagaa cacacataca cattcttgcc agtcttggtt 12840
agagaaattt catgacaaaa tgccaaagct gtcttgactc ttcacttttg gccatgagtc 12900
gtgacttagt ttggtttaat ggaccggttc tcctagcttg ttctactcaa aactgttgtt 12960
gatgcgaata agttgtgatg gttgatctct ggattttgtt ttgctctcaa tagtggacga 13020
gattagatag cttaagcctg caggcggacc gcctgcaggc ccgggggcgc gccctaatta 13080
gctaacggcc aggatcgccg cgtgagcctt tagcaactag ctagattaat taacgcaatc 13140
tgttattaag ttgtctaagc gtcaatttgt ttacaccaca atatatcctg ccaccagcca 13200
gccaacagct ccccgaccgg cagctcggca caaaatcacc actcgataca ggcagcccat 13260
cagaattaat tctcatgttt gacagcttat catcgactgc acggtgcacc aatgcttctg 13320
gcgtcaggca gccatcggaa gctgtggtat ggctgtgcag gtcgtaaatc actgcataat 13380
tcgtgtcgct caaggcgcac tcccgttctg gataatgttt tttgcgccga catcataacg 13440
gttctggcaa atattctgaa atgagctgtt gacaattaat catccggctc gtataatgtg 13500
tggaattgtg agcggataac aatttcacac aggaaacaga ccatgaggga agcgttgatc 13560
gccgaagtat cgactcaact atcagaggta gttggcgtca tcgagcgcca tctcgaaccg 13620
acgttgctgg ccgtacattt gtacggctcc gcagtggatg gcggcctgaa gccacacagt 13680
gatattgatt tgctggttac ggtgaccgta aggcttgatg aaacaacgcg gcgagctttg 13740
atcaacgacc ttttggaaac ttcggcttcc cctggagaga gcgagattct ccgcgctgta 13800
gaagtcacca ttgttgtgca cgacgacatc attccgtggc gttatccagc taagcgcgaa 13860
ctgcaatttg gagaatggca gcgcaatgac attcttgcag gtatcttcga gccagccacg 13920
atcgacattg atctggctat cttgctgaca aaagcaagag aacatagcgt tgccttggta 13980
ggtccagcgg cggaggaact ctttgatccg gttcctgaac aggatctatt tgaggcgcta 14040
aatgaaacct taacgctatg gaactcgccg cccgactggg ctggcgatga gcgaaatgta 14100
gtgcttacgt tgtcccgcat ttggtacagc gcagtaaccg gcaaaatcgc gccgaaggat 14160
gtcgctgccg actgggcaat ggagcgcctg ccggcccagt atcagcccgt catacttgaa 14220
gctaggcagg cttatcttgg acaagaagat cgcttggcct cgcgcgcaga tcagttggaa 14280
gaatttgttc actacgtgaa aggcgagatc accaaagtag tcggcaaata aagctctagt 14340
ggatctccgt acccagggat ctggctcgcg gcggacgcac gacgccgggg cgagaccata 14400
ggcgatctcc taaatcaata gtagctgtaa cctcgaagcg tttcacttgt aacaacgatt 14460
gagaattttt gtcataaaat tgaaatactt ggttcgcatt tttgtcatcc gcggtcagcc 14520
gcaattctga cgaactgccc atttagctgg agatgattgt acatccttca cgtgaaaatt 14580
tctcaagcgc tgtgaacaag ggttcagatt ttagattgaa aggtgagccg ttgaaacacg 14640
ttcttcttgt cgatgacgac gtcgctatgc ggcatcttat tattgaatac cttacgatcc 14700
acgccttcaa agtgaccgcg gtagccgaca gcacccagtt cacaagagta ctctcttccg 14760
cgacggtcga tgtcgtggtt gttgatctag atttaggtcg tgaagatggg ctcgagatcg 14820
ttcgtaatct ggcggcaaag tctgatattc caatcataat tatcagtggc gaccgccttg 14880
aggagacgga taaagttgtt gcactcgagc taggagcaag tgattttatc gctaagccgt 14940
tcagtatcag agagtttcta gcacgcattc gggttgcctt gcgcgtgcgc cccaacgttg 15000
tccgctccaa agaccgacgg tctttttgtt ttactgactg gacacttaat ctcaggcaac 15060
gtcgcttgat gtccgaagct ggcggtgagg tgaaacttac ggcaggtgag ttcaatcttc 15120
tcctcgcgtt tttagagaaa ccccgcgacg ttctatcgcg cgagcaactt ctcattgcca 15180
gtcgagtacg cgacgaggag gtttatgaca ggagtataga tgttctcatt ttgaggctgc 15240
gccgcaaact tgaggcagat ccgtcaagcc ctcaactgat aaaaacagca agaggtgccg 15300
gttatttctt tgacgcggac gtgcaggttt cgcacggggg gacgatggca gcctgagcca 15360
attcccagat ccccgaggaa tcggcgtgag cggtcgcaaa ccatccggcc cggtacaaat 15420
cggcgcggcg ctgggtgatg acctggtgga gaagttgaag gccgcgcagg ccgcccagcg 15480
gcaacgcatc gaggcagaag cacgccccgg tgaatcgtgg caagcggccg ctgatcgaat 15540
ccgcaaagaa tcccggcaac cgccggcagc cggtgcgccg tcgattagga agccgcccaa 15600
gggcgacgag caaccagatt ttttcgttcc gatgctctat gacgtgggca cccgcgatag 15660
tcgcagcatc atggacgtgg ccgttttccg tctgtcgaag cgtgaccgac gagctggcga 15720
ggtgatccgc tacgagcttc cagacgggca cgtagaggtt tccgcagggc cggccggcat 15780
ggccagtgtg tgggattacg acctggtact gatggcggtt tcccatctaa ccgaatccat 15840
gaaccgatac cgggaaggga agggagacaa gcccggccgc gtgttccgtc cacacgttgc 15900
ggacgtactc aagttctgcc ggcgagccga tggcggaaag cagaaagacg acctggtaga 15960
aacctgcatt cggttaaaca ccacgcacgt tgccatgcag cgtacgaaga aggccaagaa 16020
cggccgcctg gtgacggtat ccgagggtga agccttgatt agccgctaca agatcgtaaa 16080
gagcgaaacc gggcggccgg agtacatcga gatcgagctg gctgattgga tgtaccgcga 16140
gatcacagaa ggcaagaacc cggacgtgct gacggttcac cccgattact ttttgatcga 16200
tcccggcatc ggccgttttc tctaccgcct ggcacgccgc gccgcaggca aggcagaagc 16260
cagatggttg ttcaagacga tctacgaacg cagtggcagc gccggagagt tcaagaagtt 16320
ctgtttcacc gtgcgcaagc tgatcgggtc aaatgacctg ccggagtacg atttgaagga 16380
ggaggcgggg caggctggcc cgatcctagt catgcgctac cgcaacctga tcgagggcga 16440
agcatccgcc ggttcctaat gtacggagca gatgctaggg caaattgccc tagcagggga 16500
aaaaggtcga aaaggtctct ttcctgtgga tagcacgtac attgggaacc caaagccgta 16560
cattgggaac cggaacccgt acattgggaa cccaaagccg tacattggga accggtcaca 16620
catgtaagtg actgatataa aagagaaaaa aggcgatttt tccgcctaaa actctttaaa 16680
acttattaaa actcttaaaa cccgcctggc ctgtgcataa ctgtctggcc agcgcacagc 16740
cgaagagctg caaaaagcgc ctacccttcg gtcgctgcgc tccctacgcc ccgccgcttc 16800
gcgtcggcct atcgcggccg ctggccgctc aaaaatggct ggcctacggc caggcaatct 16860
accagggcgc ggacaagccg cgccgtcgcc actcgaccgc cggcgctgag gtctgcctcg 16920
tgaagaaggt gttgctgact cataccaggc ctgaatcgcc ccatcatcca gccagaaagt 16980
gagggagcca cggttgatga gagctttgtt gtaggtggac cagttggtga ttttgaactt 17040
ttgctttgcc acggaacggt ctgcgttgtc gggaagatgc gtgatctgat ccttcaactc 17100
agcaaaagtt cgatttattc aacaaagccg ccgtcccgtc aagtcagcgt aatgctctgc 17160
cagtgttaca accaattaac caattctgat tagaaaaact catcgagcat caaatgaaac 17220
tgcaatttat tcatatcagg attatcaata ccatattttt gaaaaagccg tttctgtaat 17280
gaaggagaaa actcaccgag gcagttccat aggatggcaa gatcctggta tcggtctgcg 17340
attccgactc gtccaacatc aatacaacct attaatttcc cctcgtcaaa aataaggtta 17400
tcaagtgaga aatcaccatg agtgacgact gaatccggtg agaatggcaa aagctctgca 17460
ttaatgaatc ggccaacgcg cggggagagg cggtttgcgt attgggcgct cttccgcttc 17520
ctcgctcact gactcgctgc gctcggtcgt tcggctgcgg cgagcggtat cagctcactc 17580
aaaggcggta atacggttat ccacagaatc aggggataac gcaggaaaga acatgtgagc 17640
aaaaggccag caaaaggcca ggaaccgtaa aaaggccgcg ttgctggcgt ttttccatag 17700
gctccgcccc cctgacgagc atcacaaaaa tcgacgctca agtcagaggt ggcgaaaccc 17760
gacaggacta taaagatacc aggcgtttcc ccctggaagc tccctcgtgc gctctcctgt 17820
tccgaccctg ccgcttaccg gatacctgtc cgcctttctc ccttcgggaa gcgtggcgct 17880
ttctcatagc tcacgctgta ggtatctcag ttcggtgtag gtcgttcgct ccaagctggg 17940
ctgtgtgcac gaaccccccg ttcagcccga ccgctgcgcc ttatccggta actatcgtct 18000
tgagtccaac ccggtaagac acgacttatc gccactggca gcagccactg gtaacaggat 18060
tagcagagcg aggtatgtag gcggtgctac agagttcttg aagtggtggc ctaactacgg 18120
ctacactaga agaacagtat ttggtatctg cgctctgctg aagccagtta ccttcggaaa 18180
aagagttggt agctcttgat ccggcaaaca aaccaccgct ggtagcggtg gtttttttgt 18240
ttgcaagcag cagattacgc gcagaaaaaa aggatctcaa gaagatcctt tgatcttttc 18300
tacggggtct gacgctcagt ggaacgaaaa ctcacgttaa gggattttgg tcatgagatt 18360
atcaaaaagg atcttcacct agatcctttt gatccggaat ta 18402
<210> 76
<211> 14
<212> PRT
<213> Triticum aestivum
<400> 76
Arg Ala Gly Arg Ala Ala Ala Pro Gly Gly Ala Gln Gly Ala
1 5 10
<210> 77
<211> 47
<212> PRT
<213> Triticum aestivum
<400> 77
Val Ala Arg Asp Leu Pro Gly Ser Leu Pro Phe Arg Phe Val Leu Phe
1 5 10 15
Ser Val Phe Trp Ser Asp Leu Leu Val Thr Cys Ser Thr Glu Cys Arg
20 25 30
Gly Glu Pro Gly Gly Arg Arg Pro Gln Gly Gly Leu Lys Gly Gln
35 40 45
<210> 78
<211> 44
<212> PRT
<213> Triticum aestivum
<400> 78
Gly Thr Phe Pro Gly Arg Phe Leu Phe Val Ser Ser Cys Phe Leu Phe
1 5 10 15
Phe Gly Leu Thr Cys Ser Ser Pro Val Arg Arg Asn Ala Glu Ala Ser
20 25 30
Arg Ala Gly Gly Gly Pro Arg Gly Gly Ser Arg Gly
35 40
<210> 79
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> primer
<400> 79
cgactcgcct cgcgctagta 20
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ГЕТЕРОЗИГОТНЫЕ ПО CENH3 ОДНОДОЛЬНЫЕ РАСТЕНИЯ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ ДЛЯ ИНДУКЦИИ ГАПЛОИДОВ И ОДНОВРЕМЕННОГО РЕДАКТИРОВАНИЯ ГЕНОМА | 2021 |
|
RU2832578C1 |
| КОМПОЗИЦИИ ДЛЯ ИНДУКЦИИ ГАПЛОИДИИ И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2016 |
|
RU2816649C1 |
| ГЕНОМНАЯ ИНЖЕНЕРИЯ | 2014 |
|
RU2764757C2 |
| ГЕНОМНАЯ ИНЖЕНЕРИЯ | 2021 |
|
RU2812848C2 |
| ПЕПТИДЫ И ИХ ПРИМЕНЕНИЕ В ЛЕЧЕНИИ ЗАБОЛЕВАНИЙ, НАРУШЕНИЙ ИЛИ СОСТОЯНИЙ, АССОЦИИРОВАННЫХ С МУТАНТНЫМ р53 | 2017 |
|
RU2762089C2 |
| КОМПОНЕНТЫ СИСТЕМЫ CRISPR-CAS, СПОСОБЫ И КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796549C2 |
| ДНК-СОДЕРЖАЩИЕ ПОЛИНУКЛЕОТИДЫ И РУКОВОДЯЩИЕ ПОСЛЕДОВАТЕЛЬНОСТИ ДЛЯ СИСТЕМ CRISPR ТИПА V И СПОСОБЫ ИХ ПОЛУЧЕНИЯ И ПРИМЕНЕНИЯ | 2021 |
|
RU2832314C1 |
| ДОСТАВКА, КОНСТРУИРОВАНИЕ И ОПТИМИЗАЦИЯ СИСТЕМ, СПОСОБОВ И КОМПОЗИЦИЙ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ И ПРИМЕНЕНИЯ В ТЕРАПИИ | 2013 |
|
RU2721275C2 |
| НОВЫЕ ФЕРМЕНТЫ CRISPR И СИСТЕМЫ | 2016 |
|
RU2771826C2 |
| КОНСТРУИРОВАНИЕ СИСТЕМ, СПОСОБЫ И ОПТИМИЗИРОВАННЫЕ НАПРАВЛЯЮЩИЕ КОМПОЗИЦИИ ДЛЯ МАНИПУЛЯЦИИ С ПОСЛЕДОВАТЕЛЬНОСТЯМИ | 2013 |
|
RU2796017C2 |
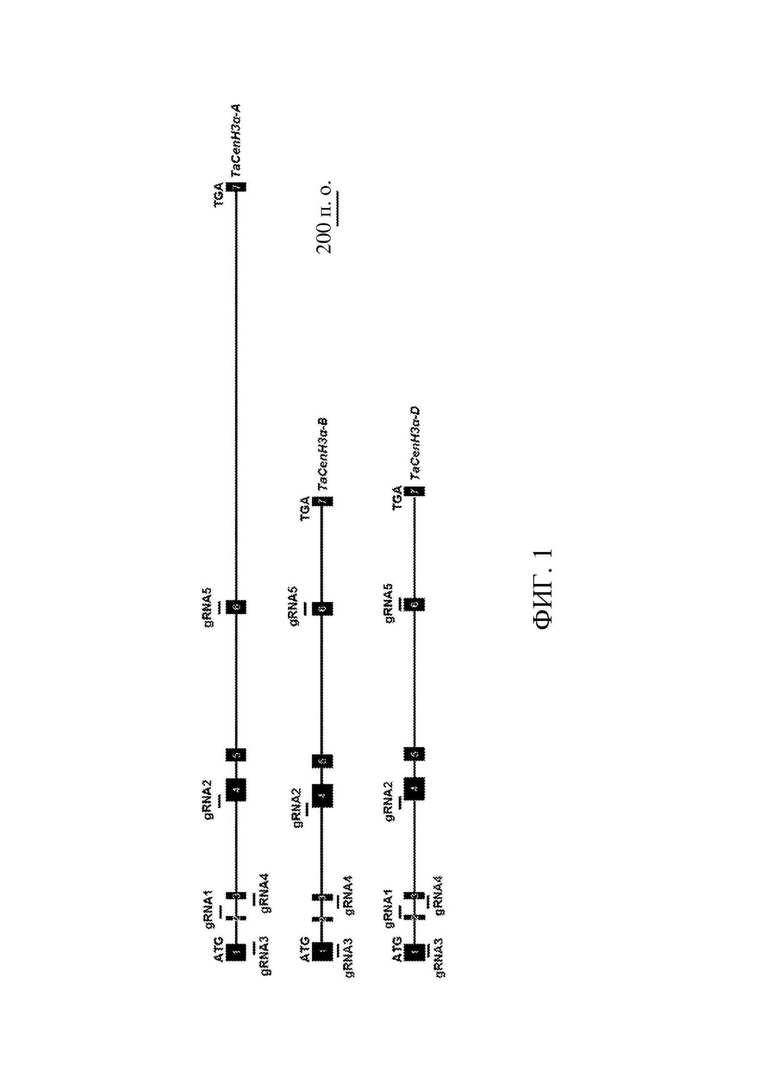
Предложенная группа изобретений относится к области биотехнологии и может быть применима в сельском хозяйстве. Предусмотрены растения пшеницы, содержащие мутацию, вызывающую изменение аминокислотной последовательности центромерного гистона Н3 (CENH3), которые обладают биологической активностью индукторов гаплоидии. Кроме того, предусмотрены способы получения растений пшеницы и гаплоидных и удвоенных гаплоидных растений пшеницы, получаемых путем скрещивания растений пшеницы с растениями пшеницы дикого типа. Изобретение может быть применимо в сельском хозяйстве в качестве пути решения проблемы высокой гетерозиготности растений пшеницы дикого типа и для ускорения процесса селекции. 4 н. и 18 з.п. ф-лы, 1 ил., 3 табл., 7 пр.
1. Гаплоид-индуцирующее растение пшеницы, содержащее геном A, геном B и геном D,
где геном B содержит нокаут-мутацию в каждом гене CENH3, и
где геном D содержит нокаут-мутацию в каждом гене CENH3, и
где дополнительно геном A содержит мутантный ген CENH3, содержащий одну нокдаун-мутацию в 5'-сплайсинг-сайте интрона.
2. Гаплоид-индуцирующее растение пшеницы по п. 1, где нокдаун-мутация представляет собой мутацию по типу восстановления сдвинутой рамки считывания.
3. Гаплоид-индуцирующее растение пшеницы по п. 1, где растение пшеницы является гомозиготным по нокаут-мутации в гене CENH3 в геноме B.
4. Гаплоид-индуцирующее растение пшеницы по п. 1, где растение пшеницы является биаллельным по нокаут-мутации в гене CENH3 в геноме B.
5. Гаплоид-индуцирующее растение пшеницы по п. 1, где растение пшеницы является гомозиготным по нокаут-мутации в гене CENH3 в геноме D.
6. Гаплоид-индуцирующее растение пшеницы по п. 1, где растение пшеницы является биаллельным по нокаут-мутации в гене CENH3 в геноме D.
7. Гаплоид-индуцирующее растение пшеницы по п. 1, где растение пшеницы является гомозиготным, биаллельным или характеризуется комбинацией этих свойств по нокаут-мутации в гене CENH3 в геноме B и геноме D.
8. Гаплоид-индуцирующее растение пшеницы по п. 2, где растение пшеницы является гомозиготным по мутации по типу восстановления сдвинутой рамки считывания в CENH3.
9. Гаплоид-индуцирующее растение пшеницы по п. 2, где растение пшеницы является гетерозиготным по мутации по типу восстановления сдвинутой рамки считывания в CENH3.
10. Гаплоид-индуцирующее растение пшеницы по пп. 1-9, где растение пшеницы является гомозиготным по нокдаун-мутации в гене CENH3 в геноме A и гомозиготным по нокаут-мутации в гене CENH3 в геноме B и геноме D.
11. Способ получения растения пшеницы, индуцирующего гаплоидию, при этом способ включает:
(a) получение клетки растения пшеницы, содержащей три генома, где три генома включают геном А, геном В и геном D;
(b) осуществление мутаций в двух из трех геномов с получением гомозиготных нокаут-мутаций в гене CENH3, где нокаут-мутации в гене CENH3 произведены в геномах В и D;
(c) осуществление мутаций в третьем геноме с получением гомозиготной нокдаун-мутации в гене CENH3, где нокдаун-мутации в гене CENH3 произведены в геноме А; и
(d) получение в результате этого растения пшеницы, содержащего гомозиготные нокаут-мутации в гене CENH3 в двух из трех геномов и дополнительно содержащего гомозиготную нокдаун-мутацию в гене CENH3 в третьем геноме;
вследствие чего растение пшеницы, полученное на стадии (d), производит гаплоидное потомство при скрещивании с растением пшеницы дикого типа.
12. Способ по п. 11, где нокдаун-мутация в гене CENH3 имеет место в геноме A.
13. Способ по п. 12, где нокдаун-мутации в гене CENH3 представляют собой мутации по типу восстановления сдвинутой рамки считывания.
14. Способ по п. 13, где мутации по типу восстановления сдвинутой рамки считывания выбраны из группы, состоящей из SEQ ID NO: 56, SEQ ID NO: 63, SEQ ID NO: 69.
15. Способ получения гаплоидного растения пшеницы, включающий:
(a) получение растения пшеницы;
(b) скрещивание растения пшеницы с гаплоид-индуцирующим растением пшеницы по пп. 1-10; и
(c) отбор потомка, полученного на стадии скрещивания,
где потомок представляет собой гаплоидное растение пшеницы.
16. Способ по п. 15, где гаплоид-индуцирующее растение пшеницы из стадии (a) представляет собой отцовское родительское растение.
17. Способ по п. 15, где растение гаплоид-индуцирующее пшеницы из стадии (a) представляет собой материнское родительское растение.
18. Способ по п. 15, дополнительно включающий превращение растения пшеницы, являющегося потомком, в удвоенное гаплоидное растение пшеницы.
19. Гаплоид-индуцирующее растение пшеницы, содержащее мутантный аллель CENH3, содержащий последовательность нуклеиновой кислоты, выбранную из группы, состоящей из SEQ ID NO: 56-73,
где мутация представляет собой мутацию по типу восстановления сдвинутой рамки считывания и где растение пшеницы образует гаплоидное потомство при скрещивании с диплоидным растением пшеницы дикого типа.
20. Гаплоид-индуцирующее растение пшеницы по п. 19, где растение пшеницы содержит по меньшей мере одну копию мутантного аллеля CENH3.
21. Гаплоид-индуцирующее растение пшеницы по п. 19, где растение пшеницы содержит по меньшей мере две копии мутантного аллеля CENH3.
22. Гаплоид-индуцирующее растение пшеницы по п. 19, где растение пшеницы содержит по меньшей мере три копии мутантного аллеля CENH3.
| WO 2016138021 A1, 01.09.2016 | |||
| WO 2009068313 A2, 04.06.2009 | |||
| УСТОЙЧИВЫЕ К ГЕРБИЦИДАМ РАСТЕНИЯ ПОДСОЛНЕЧНИКА | 2009 |
|
RU2551781C2 |
Авторы
Даты
2025-03-11—Публикация
2019-10-10—Подача