ОБЛАСТЬ ТЕХНИКИ
[001] Настоящая заявка содержит перечень последовательностей в машиночитаемой форме, который включен в настоящий документ посредством ссылки.
[002] Настоящее изобретение относится к композициям, содержащим один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и носитель, а также к применениям и способам, включающим эти полипептиды.
ОПИСАНИЕ
[003] Микотоксины представляют собой вторичные метаболиты, продуцируемые мицелиальными грибами. Одним из представителей микотоксинов является дезоксиниваленол (ДОН), представляющий собой трихотецен типа В, или вомитоксин, который продуцируется различными грибами рода Fusarium и встречается во всем мире. Эти грибы заражают культурные растения, такие как, среди прочих, различные виды зерновых, при этом заражение грибами обычно возникает до сбора урожая, когда рост грибов и/или выработка микотоксинов могут происходить до помещения на хранение, или они могут происходить и после сбора урожая, либо перед помещением на хранение, либо при ненадлежащих условиях хранения. Согласно оценкам Продовольственной и сельскохозяйственной организации Объединенных Наций (FAO), 25% сельскохозяйственных продуктов во всем мире загрязнены микотоксинами, что приводит к существенным экономическим потерям. В ходе международного исследования продолжительностью 8 лет с января 2004 г. по декабрь 2011 г. были проанализированы 19757 образцов; из них 72% продемонстрировали положительный результат на наличие по меньшей мере одного микотоксина, 39% оказались загрязненными более чем одним микотоксином и 56% продемонстрировали положительный результат на наличие ДОН (Schatzmayr and Streit (2013)). Трихотецены и, таким образом, в том числе ДОН были обнаружены во всех регионах мира и во всех исследованных видах зерновых и кормовых культур, таких как кукуруза, соевая мука, пшеница, пшеничные отруби, DDGS (сухая дробина с растворимыми веществами), а также в готовых кормовых смесях для животных с частотой встречаемости до 100%.
[004] Основная стратегия снижения загрязнения пищевых продуктов и кормовых продуктов для животных трихотеценами заключается в ограничении роста грибов, например, путем обеспечения «надлежащей сельскохозяйственной практики». Эта практика включает, среди прочего, обеспечение отсутствия заражения семян вредителями и грибами или незамедлительное удаление сельскохозяйственных отходов с поля. Кроме того, рост грибов в поле можно уменьшить с помощью фунгицидов. После сбора урожая собранный материал следует хранить при остаточной влажности менее 15% и при низкой температуре, чтобы предотвратить рост грибов. Аналогичным образом, материал, зараженный грибами, должен быть удален перед дальнейшей обработкой. Несмотря на этот длинный перечень профилактических мер, даже в регионах с самыми высокими сельскохозяйственными стандартами, таких как Северная Америка и Центральная Европа, в период с 2004 по 2011 гг.до 68% исследованных образцов оказались загрязненными представителем трихотеценов ДОН (Schatzmayr and Streit (2013)).
[005] Известно, что токсичность трихотеценов (по меньшей мере) частично обусловлена присутствием гидроксильной (-ОН) группы при атоме С-3. Одним из наиболее распространенных трихотеценов, содержащих такую 3-гидроксигруппу, является дезоксиниваленол (ДОН).
[006] Кроме того, гидроксильная группа ДОН и других трихотеценов может присутствовать в двух изомерных состояниях, а именно в S-конформации (ДОН) или R-конформации (3-эпи-ДОН). Некоторые публикации включают Не et al. (2015), Payros et al. (2016) и Pierron et al. (2016).
[007] В публикации Hassan et al. (2017) утверждается, что эпимеризация ДОН в 3-эпи-ДОН протекает в виде двухстадийного процесса через образование 3-кето-ДОН. 3-Кето-ДОН содержит 3-оксогруппу вместо 3-гидроксигруппы, присутствующей в упомянутых изомерах. Наконец, авторы публикации Carere et al. (2018) идентифицировали фермент, а именно DmDepB из D, mutatis 17-2-Е-8, который катализирует восстановление 3-кето-ДОН до 3-эпи-ДОН и ДОН. В WO2019/046954 описан тот же самый фермент, что и в публикации Hassan et al. (2017), и другой фермент DepB, полученный из Rhizobium leguminosarum (RIDepB).
[008] Однако все еще остается потребность в обеспечении ферментов, которые характеризуются лучшей эффективностью превращения 3-кето-ДОН в нетоксичный изомер 3-эпи-ДОН, или в превращении трихотеценов, содержащих 3-оксогруппу, в трихотецены, содержащие 3-гидроксигруппу в S-конфигурации.
[009] Решение, предложенное в настоящем изобретении, описано ниже, проиллюстрировано в примерах, проиллюстрировано с помощью фигур и отражено в формуле изобретения.
[0010] Настоящее изобретение относится к композиции, содержащей а) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1; и b) носитель.
[0011] Настоящее изобретение также относится к композиции, содержащей
a) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, и
b) носитель.
[0012] Настоящее изобретение также относится к полипептиду, содержащему последовательность SEQ ID NO. 1 или последовательность, которая на по меньшей мере 75% идентична SEQ ID NO. 1, при этом указанный полипептид способен превращать 3-кето-ДОН в 3-эпи-ДОН. Настоящее изобретение также относится к полипептиду, содержащему любую из последовательностей SEQ ID NO. 63-101.
[0013] Кроме того, настоящее изобретение относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, для превращения 3-кето-ДОН в 3-эпи-ДОН.
[0014] Настоящее изобретение также относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, при получении кормовой добавки или кормовой композиции.
[0015] Кроме того, настоящее изобретение относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, при получении кормовой добавки или кормовой композиции.
[0016] Настоящее изобретение относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением по меньшей мере 25:1 (3-эпи-ДОН:ДОН), при получении биогаза, биоэтанола или сахара, предпочтительно из сахарного тростника или сахарной свеклы.
[0017] Кроме того, настоящее изобретение относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, при получении биогаза, биоэтанола или сахара, предпочтительно из сахарного тростника или сахарной свеклы.
[0018] Настоящее изобретение также охватывает кормовые добавки или пищевые добавки, а также кормовые или пищевые продукты, содержащие один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
[0019] Кроме того, настоящее изобретение относится к кормовым добавкам или пищевым добавкам или кормовым или пищевым продуктам, содержащим один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН.
[0020] Настоящее изобретение также охватывает добавку для применения в композициях, предпочтительно композициях сельскохозяйственных продуктов, содержащих 3-кето-ДОН или 3-ДОН, при этом указанная добавка содержит один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
[0021] Настоящее изобретение также относится к добавке для применения в композициях, предпочтительно композициях сельскохозяйственных продуктов, содержащих 3-кето-ДОН или ДОН, при этом указанная добавка содержит один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН.
[0022] Кроме того, настоящее изобретение относится к способу превращения трихотецена, содержащего 3-оксогруппу, в трихотецен, содержащий 3-гидроксигруппу, включающему приведение в контакт одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, с трихотеценом, содержащим 3-оксогруппу.
[0023] Кроме того, настоящее изобретение относится к способу превращения 3-кето-ДОН в 3-эпи-ДОН, включающему приведение в контакт одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, с 3-кето-ДОН.
[0024] Кроме того, настоящее изобретение относится к способу снижения содержания ДОН в композиции, содержащей ДОН, или снижения токсичности композиции, содержащей ДОН, путем превращения ДОН в 3-эпи-ДОН, включающему
a) приведение в контакт указанной композиции с ферментом, способным превращать ДОН в 3-кето-ДОН; и
b) последующее или одновременное приведение в контакт указанной композиции с одним или более полипептидами, содержащими последовательность или состоящими из последовательности SEQ ID NO. 1-14 или содержащими последовательность или состоящими из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащими последовательность или состоящими из последовательности SEQ ID NO. 63-101.
[0025] Кроме того, настоящее изобретение относится к клетке-хозяину, содержащей один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101.
[0026] Настоящее изобретение также охватывает растение, генетически модифицированное для экспрессии одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101.
[0027] Настоящее изобретение относится к семени растения согласно настоящему изобретению.
[0028] Настоящее изобретение также относится к полипептиду, содержащему последовательность или состоящему из последовательности SEQ ID NO. 1-14 или содержащему последовательность или состоящему из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащему последовательность или состоящему из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением по меньшей мере 25:1 (3-эпи-ДОН/ДОН), для применения при предотвращении и/или лечении микотоксикоза.
[0029] Кроме того, настоящее изобретение относится к полипептиду, содержащему последовательность или состоящему из последовательности SEQ ID NO. 1-14 или содержащему последовательность или состоящему из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащему последовательность или состоящему из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, для применения при предотвращении и/или лечении микотоксикоза.
[0030] На фигурах представлено:
[0031] На фиг. 1А показана идентичность последовательностей в пределах всей длины белка, представленная в виде процента идентичности (100% покрытие последовательности белка), для SEQ ID NO. 1-16. SEQ ID NO. 1-14 представляют собой последовательности 3-кето-ДОН-редуктаз, описанных в настоящем документе, тогда как SEQ ID NO. 15 и SEQ ID NO. 16 относятся к последовательностям известных из уровня техники ферментов DepB. На фиг. 1В показаны идентичности последовательностей в пределах всей длины белка, представленные в виде процента идентичности (100% покрытие последовательности белка), для SEQ ID NO. 1, 3, 6, 63-101.
[0032] На фиг. 2 показаны данные для различных ферментов относительно выхода 3-эпи-ДОН. Следует отметить, что данные для SEQ ID NO. 16 не показаны, поскольку фермент, имеющий последовательность SEQ ID NO. 16, не мог быть получен в количествах, достаточных для проведения необходимых аналитических исследований. Однако, без привязки к какой-либо теории, следует ожидать, что SEQ ID NO. 16 будет демонстрировать сходную с SEQ ID NO. 15 эффективность. Причина этого заключается в том, что SEQ ID NO. 15 и 16 являются в большой степени сходными последовательностями, в частности в областях петли А и петли В.
[0033] На фиг. 3 показаны данные для различных ферментов относительно выхода ДОН. Следует отметить, что данные для SEQ ID NO. 16 не показаны, поскольку фермент, имеющий последовательность SEQ ID NO. 16, не мог быть получен в количествах, достаточных для проведения необходимых аналитических исследований. Однако, без привязки к какой-либо теории, следует ожидать, что SEQ ID NO. 16 будет демонстрировать сходную с SEQ ID NO. 15 эффективность. Причина этого заключается в том, что SEQ ID NO. 15 и 16 являются в большой степени сходными последовательностями, в частности в областях петли А и петли В.
[0034] На фиг. 4 показаны петли А и В, определяющие субстратную специфичность, представленные в полной последовательности различных ферментов, описанных в настоящем документе. Черные прямоугольники обозначают альфа-спирали, и белые прямоугольники обозначают бета-листы. Рамки со штриховыми линиями обозначают расположение остатков, входящих в предполагаемый активный центр, а остатки, образующие каталитическую тетраду, заключены в рамки с точечными линиями. Эти данные также обобщенно представлены в приведенной в настоящем документе таблице последовательностей.
[0035] На фиг. 5 показано выравнивание последовательностей, выполненное с помощью программы Clustal W. А) представляет собой выравнивание последовательностей SEQ ID NO. 1-14 с SEQ ID NO. 15 и 16 для петли А. В) представляет собой выравнивание последовательностей SEQ ID NO. 1-14 с SEQ ID NO. 15 и 16 для петли В. С) представляет собой выравнивание последовательностей SEQ ID NO. 1-14 с SEQ ID NO. 15 и 16 для части петли В, которая, без привязки к какой-либо теории, считается опосредующей контакт с субстратом.
[0036] Неожиданно было обнаружено, что редуктазы, имеющие любую из последовательностей SEQ ID NO. 1-14 и 63-101, более эффективно превращают 3-кето-ДОН в 3-эпи-ДОН, чем известные из уровня техники ферменты. В ходе этого превращения 3-оксогруппа трихотеценов (например, ДОН) превращается в 3-гидроксигруппу более эффективно по сравнению с известными из уровня техники ферментами, имеющими последовательности SEQ ID NO. 15 и 16. Эта более высокая эффективность проиллюстрирована в примерах. Поскольку большинство трихотеценов имеют общее свойство, заключающееся в том, что они содержат 3-гидроксигруппу в положении 3 (что также будет обсуждаться далее в настоящем документе), то эта группа может быть превращена в 3-оксогруппу аналогично реакции, наблюдаемой для ДОН. Таким образом, ферменты, имеющие любую из последовательностей SEQ ID NO. 1-14 и 63-101, способны превращать любые трихотецены, содержащие 3-оксогруппу, в трихотецены, содержащие гидроксильную группу.
[0037] Следует отметить, что ферменты согласно настоящему изобретению способны к высокостереоселективному превращению. Это означает, что 3-оксогруппа превращается в 3-гидроксигруппу конкретного изомера с более высокой эффективностью, чем другого 3-гидроксиизомера. В частности, как проиллюстрировано в примерах, полипептиды, применяемые в настоящем изобретении, могут превращать 3-оксогруппу в гидроксильную группу в S-конформации (3-эпи-ДОН) с более высокой эффективностью, чем в гидроксильную группу в R-конформации (ДОН).
[0038] Кроме того, неожиданно было обнаружено, что ферменты, имеющие любую из последовательностей SEQ ID NO. 1-14, 63-101, характеризуются идентичностью последовательностей в областях двух петель, далее называемых петля А и петля В, что отличает их от известных из уровня техники ферментов, как показано в приведенной в настоящем документе таблице последовательностей. Кроме того, полипептиды, применяемые в настоящем изобретении, содержат общий мотив, который взаимодействует с субстратом, что также показано в приведенной в настоящем документе таблице последовательностей.
[0039] Настоящее изобретение относится к композиции, содержащей
а) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1; и b) носитель.
[0040] В качестве альтернативы или дополнения, настоящее изобретение относится к композиции, содержащей
a) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, и
b) носитель.
[0041] Композиция может представлять собой любую композицию. Например, композиция может представлять собой композицию для снижения количества 3-кето-ДОН. Композиция также может представлять собой композицию для снижения количества трихотеценов, содержащих 3-оксогруппу. Композиция может представлять собой добавку, например, для кормового или пищевого продукта. Композиция также может представлять собой кормовой или пищевой продукт.Композиция также может представлять собой добавку для применения в композициях сельскохозяйственных продуктов или композиции сельскохозяйственного продукта. Композиция сельскохозяйственного продукта также может представлять собой композицию, содержащую трихотецены, такие как ДОН.
[0042] Термин «полипептид» в настоящем документе означает пептид, белок или полипептид, и эти термины используются взаимозаменяемо и охватывают аминокислотные цепи заданной длины, в которых аминокислотные остатки связаны посредством ковалентных пептидных связей. Настоящее изобретение также охватывает аминокислоты, отличные от 20 протеиногенных аминокислот, кодируемых с помощью стандартного генетического кода, известные специалисту в данной области техники, такие какселеноцистеин. Такие полипептиды включают любую H3 SEQ ID NO. 1-14 и/или SEQ ID NO. 63-101.
[0043] Термин «полипептид» также относится к модификациям полипептида и не исключает их. Модификации включают гликозилирование, ацетилирование, ацилирование, фосфорилирование, АДФ-рибозилирование, амидирование, ковалентное присоединение флавина, ковалентное присоединение фрагмента гема, ковалентное присоединение нуклеотида или производного нуклеотида, ковалентное присоединение липида или производного липида, ковалентное присоединение фосфатидилинозитола, поперечное сшивание, циклизацию, образование дисульфидных связей, деметилирование, образование ковалентных поперечных связей, образование цистеина, образование пироглутамата, формирование, гамма-карбоксилирование, гликозилирование, образование ГФИ-якоря, гидроксилирование, иодирование, метилирование, миристоилирование, окисление, пегилирование, протеолитический процессинг, фосфорилирование, пренилирование, рацемизацию, селеноилирование, сульфатирование, опосредованное тРНК добавление аминокислот к белкам, такое какаргинилирование, и убиквитинирование; см., например, PROTEINS -STRUCTURE AND MOLECULAR PROPERTIES, 2nd Ed., Т. E. Creighton, W. H. Freeman and Company, New York (1993); POST-TRANSLATIONAL COVALENT MODIFICATION OF PROTEINS, В. C. Johnson, Ed., Academic Press, New York (1983), pgs. 1-12; Seifter, Meth. Enzymol. 182 (1990); 626-646, Rattan, Ann. NY Acad. Sci. 663 (1992); 48-62.
[0044] Предполагается, что один или более полипептидов, описанных в настоящем документе, содержат последовательность или состоят из последовательности, которая на по меньшей мере 80%, по меньшей мере 83%, по меньшей мере 85%, по меньшей мере 87%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или более идентична любой из SEQ ID NO. 1-14. Также предполагается, что один или более полипептидов, описанных в настоящем документе, содержат последовательность или состоят из последовательности, которая на по меньшей мере 80%, по меньшей мере 83%, по меньшей мере 85%, по меньшей мере 87%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или более идентична любой из SEQ ID NO. 1. Например, один или более полипептидов, описанных в настоящем документе, содержат последовательность или состоят из последовательности, имеющей последовательность SEQ ID NO. 63-75.
[0045] Также предполагается, что один или более полипептидов, описанных в настоящем документе, содержат последовательность или состоят из последовательности, которая на по меньшей мере 80%, по меньшей мере 83%, по меньшей мере 85%, по меньшей мере 87%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или более идентична любой из SEQ ID NO. 3. Например, один или более полипептидов, описанных в настоящем документе, содержат последовательность или состоят из последовательности, имеющей последовательность SEQ ID NO. 76-88.
[0046] Также предполагается, что один или более полипептидов, описанных в настоящем документе, содержат последовательность или состоят из последовательности, которая на по меньшей мере 80%, по меньшей мере 83%, по меньшей мере 85%, по меньшей мере 87%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или более идентична любой из SEQ ID NO. 6. Например, один или более полипептидов, описанных в настоящем документе, содержат последовательность или состоят из последовательности, имеющей последовательность SEQ ID NO. 89-101.
[0047] Настоящее изобретение также охватывает, что один или более полипептидов, описанных в настоящем документе, содержат последовательность или состоят из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и содержат одну или более аминокислотных замен по сравнению с любой из последовательностей SEQ ID NO. 1-14. Для определения идентичности двух последовательностей, например, представляющую интерес последовательность сравнивают с любой из SEQ ID NO 1-14. Например, один или более полипептидов, описанных в настоящем документе, могут содержать последовательность или состоять из последовательности, имеющей последовательность SEQ ID NO. 63-101, или содержать последовательность или состоять из последовательности, которая на по меньшей мере 75%, по меньшей мере 80%, по меньшей мере 83%, по меньшей мере 85%, по меньшей мере 87%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или более идентична любой из SEQ ID NO. 63-101.
[0048] В соответствии с настоящим изобретением, термин «идентичный» или «процент идентичности» в контексте двух или более полипептидных последовательностей, таких как SEQ ID NO. 1-14 и/или 63-101, относится к двум или более последовательностям или частям последовательностей, которые являются одинаковыми или которые имеют определенный процент одинаковых нуклеотидов (например, являются идентичными на по меньшей мере 85%, 90%, 95%, 96%, 97%, 98% или 99%) при сравнении и выравнивании для максимального соответствия в пределах окна сравнения или в пределах заданной области, определенный с использованием алгоритма сравнения последовательностей, известного в данной области техники, либо путем выравнивания вручную и визуальной оценки. Последовательности, которые, например, идентичны на 80%-95% или более, считают по существу идентичными. Такое определение также применимо к последовательности, комплементарной исследуемой последовательности. Специалистам в данной области техники будут известны способы определения процента идентичности между последовательностями с использованием, например, таких алгоритмов как алгоритмы на основе компьютерной программы CLUSTALW (Thompson Nucl. Acids Res. 2 (1994), 4673-4680) или FASTDВ (Brutlag Сотр. App.Biosci. 6 (1990), 237-245), которые известны в данной области техники.
[0049] Также для специалистов в данной области техники доступны алгоритмы BLAST и BLAST 2.6 (Altschul Nucl. Acids Res. 25 (1977), 3389-3402). В программе BLASTP для аминокислотных последовательностей по умолчанию используются длина сегмента (W), равная 6, ожидаемое пороговое значение 10 и сравнение обеих цепей. Кроме того, можно использовать матрицу замен BLOSUM62 (Henikoff Proc. Natl. Acad. Sci., USA, 89, (1989), 10915; Henikoff and Henikoff (1992) 'Amino acid substitution matrices from protein blocks.' Proc Natl Acad Sci USA. 1992 Nov 15;89(22): 10915-9).
[0050] Например, для поиска локального выравнивания последовательностей можно использовать программу BLAST2.6, которая расшифровывается как «средство поиска основного локального выравнивания» (Basic Local Alignment Search Tool) (Altschul, Nucl. Acids Res. 25 (1997), 3389-3402; Altschul, J. Mol. Evol. 36 (1993), 290-300; Altschul, J. Mol. Biol. 215 (1990), 403-410).
[0051] Как описано в настоящем документе, один или более полипептидов могут иметь некоторую идентичность последовательности с любой из SEQ ID NO. 1-14. Это подразумевает, что указанные полипептиды также могут представлять собой фрагменты и, таким образом, содержать аминокислотные делеции относительно любой из SEQ ID NO. 1-14.
[0052] Как описано в настоящем документе, один или более полипептидов могут содержать аминокислотные замены относительно любой из SEQ ID NO. 1 -14. Например, полипептид может содержать одну или более консервативных аминокислотных замен или содержать только одну или более консервативных аминокислотных замен по сравнению с любой из SEQ ID NO. 1-14. Также предполагается, что полипептид может содержать одну или более высококонсервативных аминокислотных замен или содержать только одну или более высококонсервативных аминокислотных замен по сравнению слюбой из SEQ ID NO. 1-14.
[0053] В настоящем документе «консервативные» замены означают замены, перечисленные как «типичные замены» в таблице 1 ниже. В настоящем документе «высококонсервативные» замены означают замены, приведенные под заголовком «Предпочтительные замены» в таблице 1 ниже.
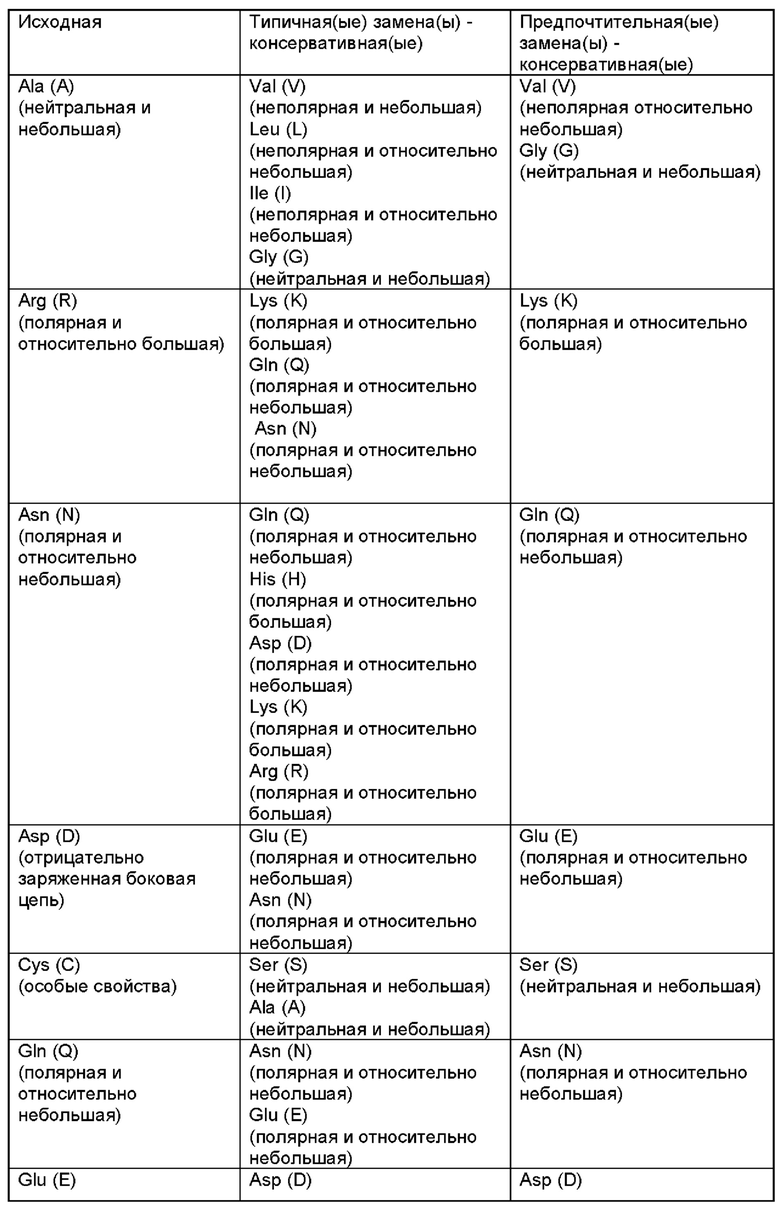
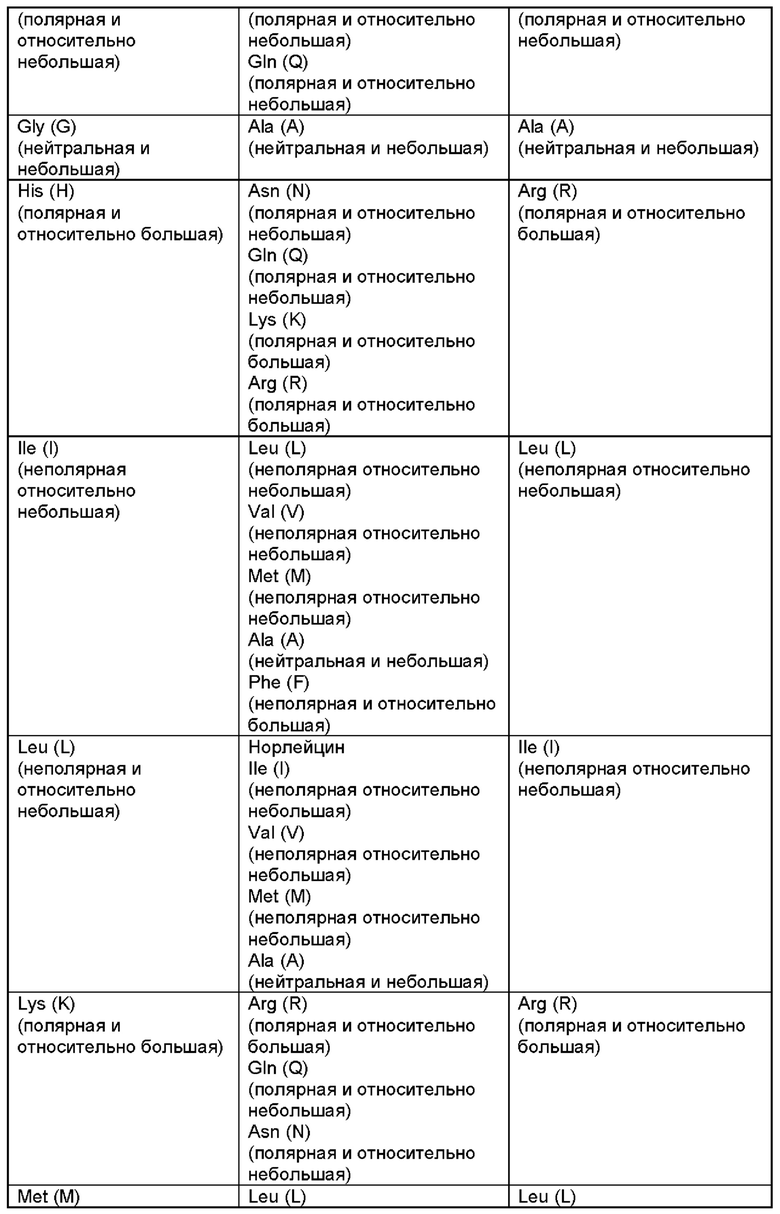
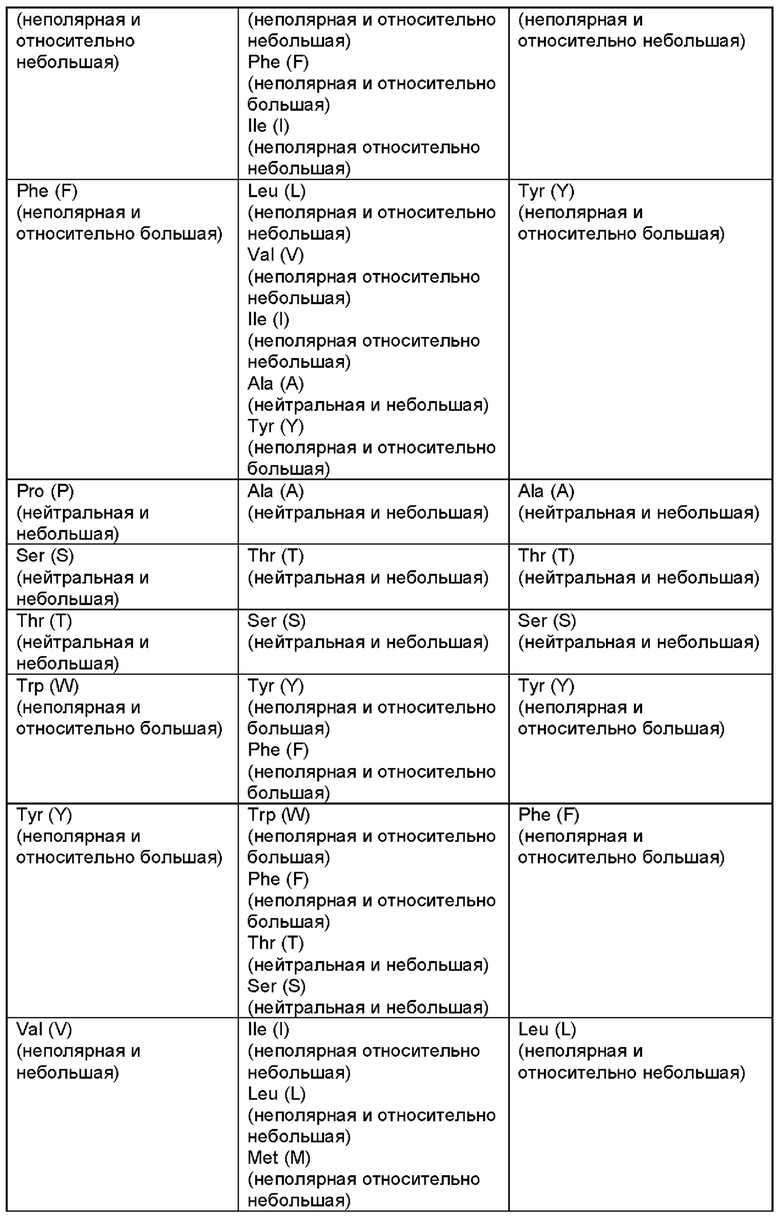

[0054] Полипептид, содержащий консервативные аминокислотные замены, может представлять собой любой полипептид, имеющий последовательность SEQ ID NO. 63-101.
[0055] Полипептиды согласно настоящему изобретению способны превращать или превращают субстрат 3-кето-ДОН в продукты 3-эпи-ДОН (в настоящем документе сокращенно обозначен как 3-эпи-ДОН) и ДОН (в настоящем документе сокращенно обозначен как ДОН) с соотношением по меньшей мере 25:1 (3-эпи-ДОН/ДОН). Реакция, описанная в настоящем документе, представлена ниже на схеме реакции, взятой из публикации Hassan et al. (2017) "The enzymatic epimerization of DON by Devosia mutans proceeds through the formation of 3-keto-DOM intermediate." Scientific Reports 7, article number 6929.
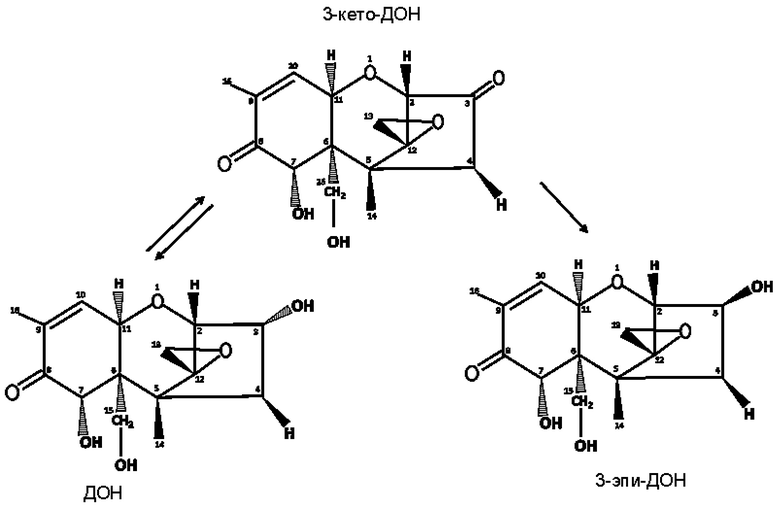
Схема реакции: Превращение 3-кето-ДОН в два изомера 3-эпи-ДОН и ДОН, взято из Hassan et al. (2017).
[0056] Как видно из приведенной выше схемы реакции, 3-кето-ДОН может быть превращен в два изомера, а именно ДОН (R-конфигурация) и 3-эпи-ДОН (S-конфигурация). Соотношение, составляющее по меньшей мере 25:1, указывает на то, что с помощью полипептидов согласно настоящему изобретению изомер 3-эпи-ДОН получают из 3-кето-ДОН в большем количестве, чем изомер ДОН. Следовательно, соотношение указывает на то, что изомера 3-эпи-ДОН получают в 50 раз больше, чем изомера ДОН. Соотношение рассчитывают путем деления полученного количества изомера 3-эпи-ДОН на полученное количество изомера ДОН.
[0057] Настоящее изобретение охватывает, что полипептид(ы), описанный(ые) в настоящем документе, способен/способны превращать или превращает (ют) субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, по меньшей мере 30:1, по меньшей мере 40:1, по меньшей мере 50:1, по меньшей мере 60:1, по меньшей мере 70:1, по меньшей мере 80:1, по меньшей мере 90:1, по меньшей мере 100:1, по меньшей мере 150:1, по меньшей мере 200:1, 250:1, 300:1, 350:1, 400:1, 500:1, 600:1, 700:1, 800:1, 900:1, 1000:1 или более.
[0058] Очевидно, что соотношение не поддается вычислению в случае, если получен только 3-эпи-ДОН и не получен ДОН. Это связано с тем, что математически деление на число 0 невозможно. Кроме того, настоящее изобретение также относится к полипептидам, которые являются на 100% стереоселективными для 3-эпи-ДОН. Таким образом, в случае, если деление представляет собой деление на число 0, то такой полипептид все еще охватывается настоящим изобретением. Однако если результат деления равен 0, поскольку получен только ДОН, то такие полипептиды не охватываются настоящим изобретением.
[0059] Один из способов определения того, способен ли конкретный полипептид превращать или превращает ли он субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, подробно описан в примерах.
[0060] Согласно настоящему изобретению также предполагается, что полипептид(ы), применяемый(ые) в настоящем изобретении, способен/способны превращать или превращает(ют) субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН. Как видно из приведенной выше схемы реакции, 3-кето-ДОН может быть превращен в два изомера, а именно 3-эпи-ДОН и ДОН. Таким образом, эти два изомера представляют собой общее количество продукта. Общее количество продукта по определению равно 100%. Поскольку полипептиды согласно настоящему изобретению являются высокостереоселективными, один изомер (в данном случае 3-эпи-ДОН) получают с более высокой эффективностью, чем другой (в данном случае ДОН). В частности, по меньшей мере 96% от общего количества полученного продукта (равного 100%) представляют собой 3-эпи-ДОН.
[0061] Настоящее изобретение также охватывает, что по меньшей мере 96,5%, 97%, 97,5%, 98%, 98,5%, 99%, 99,5%, 99,9% или 100% от общего количества продукта (равного 100%) представляют собой 3-эпи-ДОН.
[0062] Таким образом, полипептиды, имеющие любую из последовательностей SEQ ID NO. 1-14 и/или SEQ ID NO. 63-101, являются высокоэффективными в отношении превращения 3-кето-ДОН в 3-эпи-ДОН. Об этом также можно судить по тому факту, что для превращения 1 мкмоль 3-эпи-ДОН в минуту необходимо лишь небольшое количество полипептида. Результаты этого измерения указывают на то, что полипептиды, описанные в настоящем документе, обладают высокой активностью.
[0063] Таким образом, настоящее изобретение охватывает, что, когда 3-эпи-ДОН получают из 3-кето-ДОН со скоростью 1 мкмоль 3-эпи-ДОН в минуту, то количество полипептида составляет не более 7 мг, не более 6 мг, не более 5 мг, не более 3 мг, не более 2 мг, не более 1 мг или менее. Аналогичным образом, полипептиды согласно настоящему изобретению способны превращать 3-кето-ДОН в 3-эпи-ДОН с удельной активностью, составляющей по меньшей мере 0,1 мкмоль/мин/мг, по меньшей мере 0,2 мкмоль/мин/мг, по меньшей мере 0,3 мкмоль/мин/мг, по меньшей мере 0,4 мкмоль/мин/мг, по меньшей мере 0,5 мкмоль/мин/мг, по меньшей мере 0,6 мкмоль/мин/мг, по меньшей мере 0,8 мкмоль/мин/мг, по меньшей мере 1,0 мкмоль/мин/мг, по меньшей мере 1,2 мкмоль/мин/мг, по меньшей мере 1,4 мкмоль/мин/мг или по меньшей мере 1,6 мкмоль/мин/мг.
[0064] С другой стороны, активность полипептидов, описанных в настоящем документе, в отношении превращения 3-эпи-ДОН в ДОН является низкой. Таким образом, для получения ДОН необходимо большое количество полипептида. В частности, когда ДОН получают из 3-кето-ДОН со скоростью 1 мкмоль ДОН в минуту, то количество полипептида составляет по меньшей мере 600 мг, по меньшей мере 700 мг, по меньшей мере 800 мг, по меньшей мере 900 мг, по меньшей мере 1000 мг, по меньшей мере 1100 мг, по меньшей мере 1300 мг, по меньшей мере 1500 мг, по меньшей мере 1600 мг, по меньшей мере 1700 мг, по меньшей мере 1800 мг, по меньшей мере 1900 мг, по меньшей мере 2000 мг или более.
[0065] Настоящее изобретение также охватывает, что полипептид(ы), описанный(ые) в настоящем документе, способен/способны превращать или превращает (ют) 3-кето-ДОН в ДОН с удельной активностью, составляющей не более 0,0015 мкмоль/мин/мг, не более 0,0010 мкмоль/мин/мг, не более 0,0009 мкмоль/мин/мг, не более 0,0008 мкмоль/мин/мг, не более 0,0007 мкмоль/мин/мг, не более 0,0006 мкмоль/мин/мг или менее.
[0066] Полипептид, имеющий любую из последовательностей SEQ ID NO. 1-14 и/или SEQ ID NO. 63-101, который способен превращать или превращает 3-кето-ДОН в 3-эпи-ДОН и, необязательно, помимо этого в ДОН, может представлять собой редуктазу. Например, полипептид(ы) может(могут) представлять собой альдокеторедуктазу.
[0067] Полипептиды, описанные в настоящем документе, не только имеют одинаковые функциональные признаки, но также и общие структурные признаки. Кроме того, эти структурные признаки отличаются от структурных признаков известных из уровня техники ферментов, как показано в приведенной в настоящем документе таблице последовательностей. Без привязки к какой-либо теории, авторы настоящего изобретения ожидают, что эти структурные признаки (петля А и/или петля В) важны для субстратной специфичности и активности ферментов вследствие того, что петли А и В содержат части активного центра. Поскольку, помимо прочего, аминокислота гистидин (Н) из каталитической тетрады расположена в петле А, то эта петля, вероятно, также вносит больший вклад в активность ферментов согласно настоящему изобретению. Кроме того, без привязки к какой-либо теории, полагают, что петля В имеет большую роль, чем петля А, в определении субстратной специфичности. В частности, мотив Х8-G-X9-X10-Х11-Х12-Х13-Х14-Х15-Х16 в петле В, как описано в настоящем документе, взаимодействует с субстратом. Субстрат может, помимо прочего, представлять собой 3-оксотрихотецен, такой как 3-кето-ДОН или окислительно-восстановительный фактор.
[0068] Таким образом, вполне вероятно, что эти общие структурные признаки, наблюдаемые во всех полипептидах, применяемых в настоящем изобретении, ответственны за высокую стереоселективность, а также за высокую удельную активность превращения, например, 3-кето-ДОН в 3-эпи-ДОН.
[0069] Например, настоящее изобретение охватывает, что полипептид(ы), применяемый(ые) в настоящем изобретении, содержит(ат) более чем или состоит(ят) из более чем 353, 350, 348 или 351 аминокислот.В противоположность этому, известные из уровня техники ферменты, имеющие последовательности SEQ ID NO. 15 и 16, содержат 343 аминокислоты. Также предполагается, что указанный(ые) полипептид(ы) содержит(ат) более чем или состоит(ят) из более чем 345, 346, 347, 348, 349, 350, 351, 352, 353 или более аминокислот.Полипептид(ы) может(могут) содержать 348, 350, 351, 353 аминокислоты или состоять из них. Полипептид(ы) может(могут) содержать 344-353 аминокислоты, предпочтительно 348-353 аминокислоты, или состоять из них.
[0070] Полипептид(ы), применяемый(ые) в настоящем изобретении, может(могут) содержать одну или более петель в пределах аминокислотной последовательности. В настоящем документе «петля» представляет собой нерегулярную структуру в белках/полипептидах, ответственную за изменения направления и соединение основных вторичных структур альфа-спиралей и бета-тяжей. Петли могут быть неструктурированными и образовывать статистические клубки, витки и нити. Отличие от основных вторичных структур заключается в отсутствии регулярного геометрического повторения углов пси и фи в остатках в пределах структуры. В ферментах петли образуют сайты связывания субстрата и активные центры или являются их частями. Часто последовательность этих структур не является консервативной. Структурные особенности петель также описаны в публикации Kundert K, Kortemme Т. (2019).
[0071] Настоящее изобретение также охватывает, что полипептид(ы), описанный(ые) в настоящем документе, содержит(ат) петлю в пределах положений аминокислот, соответствующих положениям аминокислот
125-133 в SEQ ID NO. 1-10 или 63-101;
127-135 в SEQ ID NO. 13 или 14;
124-132 в SEQ ID NO. 11 или 12.
Эта петля также описана в настоящем документе как петля А. Петля А, раскрытая в настоящем документе, охватывает последовательность, расположенную после мотива I-D/E-LYQ, содержащегося в бета-листе 4, и до мотива I/M/L-E/D-E/D-T-L/М, содержащегося в альфа-спирали 4, например, в последовательностях SEQ ID NO. 1-14, 63-101.
[0072] Также предполагается, что полипептид в пределах положений аминокислот, соответствующих положениям аминокислот
125-133 в SEQ ID NO. 1-10 или 63-101;
127-135 в SEQ ID NO. 13 или 14;
124-132 в SEQ ID NO. 11 или 12, содержит последовательность или состоит из последовательности, которая не содержит аланина (Ala/A). Таким образом, полипептиды, описанные в настоящем документе, не содержат аланина в области петли А.
[0073] Настоящее изобретение также охватывает, что полипептид(ы), применяемый(ые) в настоящем изобретении, в пределах положений аминокислот, соответствующих положениям аминокислот
125-133 в SEQ ID NO. 1-10 или 63-101;
127-135 в SEQ ID NO. 13 или 14;
124-132 в SEQ ID NO. 11 или 12, может(могут) содержать последовательность,
содержащую последовательность
X1-H-X2-W-D-X3-X4-T-P, где
Х1 не является М;
Х2 не является А;
Х3 не является А;
Х4 не является V.
[0074] Таким образом, полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать последовательность, содержащую последовательность Х1-Н-X2-W-D-X3-X4-T-P, где
Х1 не является М;
Х2 не является А;
Х3 не является А;
Х4 не является V.
[0075] Например, полипептид(ы), описанный(ые) в настоящем документе, в пределах положений аминокислот, соответствующих положениям аминокислот
125-133 в SEQ ID NO. 1-10 или 63-101;
127-135 в SEQ ID NO. 13 или 14;
124-132 в SEQ ID NO. 11 или 12, может(могут) содержать последовательность или состоять из последовательности, содержащей последовательность X1-H-X2-W-D-X3-Х4-Т-Р, где
Х1 представляет собой V, I, L, М, F или А, предпочтительно V;
Х2 представляет собой Е, D, Q или N, предпочтительно Е или Q;
Х3 представляет собой G или А, предпочтительно G;
Х4 представляет собой М, L, F, I, R, K, Q, N, Е, V, А или норлейцин, предпочтительно М, R, Q или L.
[0076] Таким образом, полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать последовательность, содержащую последовательность
Х1-Н-X2-W-D-X3-X4-T-P, где
Х1 представляет собой V, I, L, М, F или А, предпочтительно V;
Х2 представляет собой Е, D, Q или N, предпочтительно Е или Q;
Х3 представляет собой G или А, предпочтительно G;
Х4 представляет собой М, L, F, I, R, K, Q, N, Е, V, А или норлейцин, предпочтительно М, R, Q или L.
[0077] Также предполагается, что полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать любую из последовательностей или состоять из любой из последовательностей SEQ ID NO. 20-24 или содержать последовательность или состоять из последовательности, которая на по меньшей мере 80%, 85%, 90%, 95%, 98%, 99% или более идентична любой из SEQ ID NO. 20-24.
[0078] В качестве дополнения или альтернативы, полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать петлю в пределах положений аминокислот, соответствующих положениям аминокислот 211-244 в SEQ ID NO. 1-8, 10 или 63-101; 211-245 в SEQ ID NO. 9; 210-243 в SEQ ID NO. 11 или 12;
213-247 в SEQ ID NO. 13 или 14. Эта петля также описана в настоящем документе как петля В. Петля В, раскрытая в настоящем документе, начинается с остатка серина, расположенного в общем мотиве WSPL-X-G, и заканчивается перед альфа-спиралью с последовательностью X-X-R/K/H-T/L-W/Y-X-I/V/L-I/V/L и состоит из 35 или меньшего количества остатков, например, в последовательностях SEQ ID NO. 1-14, 63-101.
[0079] Также предполагается, что полипептид(ы), описанный(ые) в настоящем документе, в пределах положений аминокислот, соответствующих положениям аминокислот
211-244 в SEQ ID NO. 1-8, 10 или 63-101;
211-245 в SEQ ID NO. 9;
210-243 в SEQ ID NO. 11 или 12;
213-247 SEQ ID NO. 13 или 14, может(могут) содержать последовательность, которая не содержит метионина (Met/M).
[0080] Например, полипептид(ы), описанный(ые) в настоящем документе, в пределах положений аминокислот, соответствующих положениям аминокислот
211-244 в SEQ ID NO. 1-8, 10 или 63-101;
211-245 в SEQ ID NO. 9;
210-243 в SEQ ID NO. 11 или 12;
213-247 в SEQ ID NO. 13 или 14, может(могут) содержать последовательность, содержащую последовательность S-P-L-X5-G-G-X6-L-X7-G-K, где
Х5 не является G;
Х6 представляет собой L, I, V, М, A, Y, F, норлейцин или W;
Х7 не является А или Т.
[0081] Таким образом, полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать последовательность, содержащую последовательность S-P-L-X5-G-G-X6-L-X7-G-K, где
Х5 не является G;
Х6 представляет собой L, I, V, М, A, Y, F, норлейцин или W;
Х7 не является А или Т.
[0082] Полипептид(ы), описанный(ые) в настоящем документе, в пределах положений аминокислот, соответствующих положениям аминокислот
211- 244 в SEQ ID NO. 1-8, 10 или 63-101;
211-245 в SEQ ID NO. 9;
210-243 в SEQ ID NO. 11 или 12;
213-247 в SEQ ID NO. 13 или 14, может(могут) содержать последовательность, содержащую последовательность S-P-L-X5-G-G-X6-L-X7-G-K, где
Х5 представляет собой А, V, L, G или I, предпочтительно А;
Х6 представляет собой L, I, V, М, A, Y, F, норлейцин или W, предпочтительно L или W;
Х7 представляет собой S или Т, предпочтительно S.
[0083] Таким образом, полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать последовательность, содержащую последовательность S-P-L-X5-G-G-X6-L-X7-G-K, где
Х5 представляет собой А, V, L или I, предпочтительно А;
Х6 представляет собой L, I, V, М, A, Y, F, норлейцин или W, предпочтительно L или W;
Х7 представляет собой S или Т, предпочтительно S.
[0084] Также предполагается, что полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать любую из последовательностей или состоять из любой из последовательностей SEQ ID NO. 33-46 или содержать последовательность или состоять из последовательности, которая на по меньшей мере 80%, 85%, 90%, 95%, 98%, 99% или более идентична любой из SEQ ID NO. 33-46.
[0085] Например, полипептид(ы), описанный(ые) в настоящем документе, в пределах положений аминокислот, соответствующих положениям аминокислот 211-244 в SEQ ID NO. 1-8, 10 или 63-101;
211-245 в SEQ ID NO. 9;
210-243 в SEQ ID NO. 11 или 12;
213-247 в SEQ ID NO. 13 или 14, может(могут) содержать последовательность,
содержащую последовательность
X8-G-X9-X10-Х11-Х12-Х13-Х14-Х15-Х16, где
Х8 представляет собой G, R, K, Q, N, А, V, L, I, S, Т, Е, D;
Х9 не является М или G;
Х10 не является Е;
Х11 не является S;
Х12 не является Y;
Х13 не является G или А;
Х14 не является Р;
Х15 представляет собой R, K, Q, N, Р, A, Y, W, F, Т, S, Н, Х16 не является N.
[0086] Таким образом, полипептид(ы), описанный(ые) в настоящем документе,
может(могут) содержать последовательность, содержащую последовательность X8-G-Х9-Х10-Х11-Х12-Х13-Х14-Х15-Х16, где
Х8 представляет собой G, R, K, Q, N, А, V, L, I, S, Т, Е, D;
Х9 не является Е;
Х10 не является Е;
Х11 не является S;
Х12 не является Y;
Х13 не является G или А;
Х14 не является Р;
Х15 представляет собой R, K, Q, N, Р, A, Y, W, F, Т, S, Н; и
Х16 не является N.
[0087] Также предполагается, что полипептид(ы), описанный(ые) в настоящем документе, в пределах положений аминокислот, соответствующих положениям аминокислот
211-244 в SEQ ID NO. 1-8, 10 или 63-101;
211-245 в SEQ ID NO. 9;
210-243 в SEQ ID NO. 11 или 12;
213-247 в SEQ ID NO. 13 или 14, может(могут) содержать последовательность, содержащую последовательность X8-G-X9-X10-Х11-Х12-Х13-Х14-Х15-Х16, где
Х8 представляет собой G, R, K, Q, N, А, V, L, I, S, Т, Е, D предпочтительно G, R, А, Е, более предпочтительно G;
Х9 представляет собой W, S, Т, F, L, V, I, А или Y, предпочтительно W, S, F, более предпочтительно F;
Х10 представляет собой Т, S, R, Q, N или K, предпочтительно Т, R или K, более предпочтительно R;
Х11 представляет собой Е, D или Q, предпочтительно Е;
Х12 представляет собой Р или А, предпочтительно Р;
Х13 представляет собой Р или А, предпочтительно Р;
Х14 представляет собой I, L, М, A, F или V, предпочтительно I или V, более предпочтительно V;
Х15 представляет собой R, K, Q, N, Р, А, Н, W, F, Т, S или Y, предпочтительно R, Р, Н или Y, более предпочтительно Р;
Х16 представляет собой D, Е или N, предпочтительно D.
Таким образом, полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать последовательность, содержащую последовательность X8-G-X9-X10-Х11-Х12-Х13-Х14-Х15-Х16, где
Х8 представляет собой G, R, K, Q, N, А, V, L, I, S, Т, Е, D предпочтительно G, R, А, Е, более предпочтительно G;
Х9 представляет собой W, S, Т, F, L, V, I, А или Y, предпочтительно W, S, F, более предпочтительно F;
Х10 представляет собой Т, S, R, Q, норлейцин, K, предпочтительно Т, R или K, более предпочтительно R;
Х11 представляет собой Е, D или Q, предпочтительно Е;
Х12 представляет собой Р или А, предпочтительно Р;
Х13 представляет собой Р или А, предпочтительно Р;
Х14 представляет собой I, L, М, A, F или V, предпочтительно I или V, более предпочтительно V;
Х15 представляет собой R, K, Q, N, Р, А, Н, W, F, Т, S или Y, предпочтительно R, Р, Н или Y, более предпочтительно Р;
Х16 представляет собой D, Е или N, предпочтительно D.
[0088] Также предполагается, что полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать любую из последовательностей или состоять из любой из последовательностей SEQ ID NO. 25-32 или содержать последовательность или состоять из последовательности, которая на по меньшей мере 80%, 85%, 90%, 95%, 98%, 99% или более идентична любой из SEQ ID NO. 25-32.Эти последовательности представляют собой последовательности, которые могут контактировать с субстратом.
[0089] Также предполагается, что полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать последовательность, содержащую любую из последовательностей SEQ ID NO. 25-32, и/или последовательность, представленную в любой из SEQ ID NO. 33-46.
[0090] Настоящее изобретение также охватывает, что полипептид(ы), описанный(ые) в настоящем документе, может(могут) содержать последовательность, содержащую любую из последовательностей SEQ ID NO. 25-32 и/или 33-46, и, необязательно, помимо этого содержать последовательность, представленную в любой из SEQ ID NO. 20-24.
[0091] Также предполагается, что полипептид(ы), описанный(ые) в настоящем документе, содержит(ат) последовательность или состоит(ят) из последовательности, представленной в любой из SEQ ID NO. 1-14, 63-101.
[0092] Полипептид(ы), описанный(ые) в настоящем документе, может(могут) присутствовать в композиции вместе с носителем. Носитель может представлять собой любой подходящий носитель. Кроме того, композиция может содержать 1, 2, 3, 4, 5, 6 или более носителей. Носитель может представлять собой съедобный компонент, предпочтительно нетоксичный компонент и/или компонент, обеспечивающий консистенцию.
[0093] Например, носитель может представлять собой твердое вещество или жидкость. Примеры твердых веществ включают пищевую(ые)/кормовую(ые) добавку(и), биологически активные добавки, нутрицевтики и/или фармацевтическое(ие) средство(а). Примеры кормовых/пищевых добавок включают, помимо прочего, кормовые/пищевые добавки, витамины, минеральные вещества, аминокислоты, незаменимые жирные кислоты, клетчатку, микроэлементы, минеральные вещества, антиоксиданты, растительные экстракты и травяные экстракты.
[0094] Носитель также может представлять собой носитель для фермента. Носители для ферментов могут иметь как неорганическое, так и органическое происхождение. Неорганические материалы, используемые для иммобилизации ферментов, могут представлять собой диоксид кремния (диоксид кремния, полученный с помощью технологии золь-гель, высокодисперсный диоксид кремния, наночастицы коллоидного диоксида кремния и силикагели) и различные оксиды, такие как оксид титана, оксид алюминия и оксид циркония. Кроме того, применяют глинистые материалы, такие как бентонит, галлуазит, каолинит, монтмориллонит, сепиолит и апатит кальция. Кроме того, материалы на основе углерода, такие как активированный уголь и древесный уголь, известны в качестве эффективных иммобилизаторов для ферментов. Органические носители для ферментов могут представлять собой не только биополимеры, но также и синтетические полимеры. Биополимеры включают углеводы и белки. Типичными примерами являются мальтодекстрин, трегалоза, инулин, коллаген, целлюлоза, кератины, каррагинан, хитин, хитозан и альгинат.В качестве примеров синтетических полимеров можно привести полианилин, полиамиды, полистирол, полиуретан, полипропилен, поливиниловый спирт и ионообменные смолы. Носитель для фермента также может представлять собой носитель, описанный в Zdarta et al. (2018).
[0095] Носитель также может представлять собой жидкость. Примеры жидкостей включают Н2О, водные растворы, солевые растворы (например, буферы), гели, вязкие препараты, жиры или масла. Предпочтительными являются водные растворы, содержащие Н2О и другие вещества, такие как буферные вещества и/или полиспирты, такие как полиалкиленоксиды (ПАО), поливиниловые спирты (ПВС), сополимеры этилена и малеинового ангидрида, сополимеры стирола и малеинового ангидрида, декстраны, целлюлозы, гидролизаты хитозана, крахмалы, гликоген, сорбит, агароза и их производные, гуаровая камедь, пуллулан, инулин, ксантановая камедь, каррагинан, пектин, гидролизаты альгиновой кислоты, биополимеры, сорбит, глицерин, целлобиоза и монопропиленгликоль (МПГ).
[0096] Композиция может представлять собой композицию для снижения количества ДОН. В таких случаях композиция дополнительно содержит фермент, способный превращать ДОН в 3-кето-ДОН. Композиция также может представлять собой композицию для снижения количества трихотеценов. В таких случаях композиция дополнительно содержит фермент, способный превращать трихотецены в трихотецены, содержащие 3-оксогруппу. Композиции, описанные в настоящем документе, дополнительно могут содержать один или более ферментов, способных превращать или превращающих ДОН в 3-кето-ДОН и/или трихотецены, содержащие 3-оксогруппу, в трихотецены, содержащие 3-гидроксильную группу. Типичные ферменты, которые могут быть применены в композиции, описаны, помимо прочего, в WO2016/154640 или WO2019/046954. Таким образом, ферменты, превращающие ДОН в 3-кето-ДОН, могут содержать последовательность или состоять из последовательности, раскрытой в SEQ ID NO. 1 в WO2016/154640 (SEQ ID NO: 61 в настоящем документе) или в SEQ ID NO. 7 в WO2019/046954 (SEQ ID NO. 62 в настоящем документе). Таким образом, композиция(и), описанная(ые) в настоящем документе, дополнительно может(могут) содержать фермент, содержащий SEQ ID NO. 61 и/или 62 или состоящий из них.
[0097] Настоящее изобретение также относится к полипептиду, содержащему последовательность SEQ ID NO. 1 или последовательность, которая на по меньшей мере 75% идентична SEQ ID NO. 1. Кроме того, настоящее изобретение относится к полипептиду, содержащему последовательность SEQ ID NO. 3 или последовательность, которая на по меньшей мере 75% идентична SEQ ID NO. 3. Кроме того, настоящее изобретение относится к полипептиду, содержащему последовательность SEQ ID NO. 6 или последовательность, которая на по меньшей мере 75% идентична SEQ ID NO. 6. Кроме того, настоящее изобретение относится к полипептиду, содержащему любую из последовательностей SEQ ID NO. 63-101 или последовательность, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 63-101.
[0098] Настоящее изобретение также относится к применению одного или более полипептидов, описанных в настоящем документе, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, для превращения 3-кето-ДОН в 3-эпи-ДОН.
[0099] Кроме того, настоящее изобретение относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением по меньшей мере 25:1 (3-эпи-ДОНДОН), при получении кормовой/пищевой добавки или кормовой/пищевой композиции.
[00100] В качестве дополнения или альтернативы, настоящее изобретение относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, при получении кормовой/пищевой добавки или кормовой/пищевой композиции.
[00101] Кормовая/пищевая добавка или кормовые/пищевые композиции согласно настоящему изобретению дополнительно могут содержать фермент, способный превращать или превращающий ДОН в 3-кето-ДОН и/или трихотецены, содержащие 3-оксогруппу, в трихотецены, содержащие 3-гидроксильную группу, как описано в настоящем документе.
[00102] Способы получения таких кормовых/пищевых добавок или кормовых/пищевых композиций известны специалисту в данной области техники и описаны, помимо прочего, в WO 99/35240.
[00103] Настоящее изобретение также относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, при получении биогаза, биоэтанола или сахара, предпочтительно из сахарного тростника или сахарной свеклы. В случае таких применений композиции или полипептиды согласно настоящему изобретению могут быть добавлены к композициям, содержащим трихотецены, такие как ДОН.
[00104] В качестве дополнения или альтернативы, настоящее изобретение относится к применению одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, при получении биогаза, биоэтанола или сахара, предпочтительно из сахарного тростника или сахарной свеклы. В случае таких применений композиции или полипептиды согласно настоящему изобретению добавляют к композициям, содержащим трихотецены, такие как ДОН.
[00105] Настоящее изобретение также относится к кормовым добавкам или пищевым добавкам или кормовым или пищевым продуктам, содержащим один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
[00106] В качестве альтернативы или дополнения, к кормовым добавкам или пищевым добавкам или кормовым или пищевым продуктам, содержащим один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН.
[00107] Кормовые/пищевые добавки согласно настоящему изобретению дополнительно могут содержать один или более ферментов, способных превращать или превращающих ДОН в 3-кето-ДОН и/или трихотецены, содержащие 3-оксогруппу, в трихотецены, содержащие 3-гидроксильную группу, как описано в настоящем документе.
[00108] Настоящее изобретение также относится к добавке для применения в композициях, предпочтительно композициях сельскохозяйственных продуктов, содержащих 3-кето-ДОН или ДОН, при этом указанная добавка содержит один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать или превращает субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
[00109] В качестве альтернативы или дополнения, настоящее изобретение также относится к добавке для применения в композициях, предпочтительно композициях сельскохозяйственных продуктов, содержащих 3-кето-ДОН и/или ДОН, при этом указанная добавка содержит один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН.
[00110] Композиция сельскохозяйственного продукта может представлять собой любую композицию, содержащую растение или части растения, такие как семя или дерево. Такие композиции сельскохозяйственных продуктов могут содержать 3-кето-ДОН и/или ДОН. Добавка в соответствии с настоящим изобретением дополнительно может содержать один или более ферментов, способных превращать или превращающих ДОН в 3-кето-ДОН и/или трихотецены, содержащие 3-оксогруппу, в трихотецены, содержащие 3-гидроксильную группу, как описано в настоящем документе.
[00111] Настоящее изобретение также относится к способу превращения трихотецена, содержащего 3-оксогруппу, в трихотецен, содержащий 3-гидроксигруппу, включающему приведение в контакт одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, с трихотеценом, содержащим 3-оксогруппу.
[00112] Трихотецены имеют следующую общую структуру:
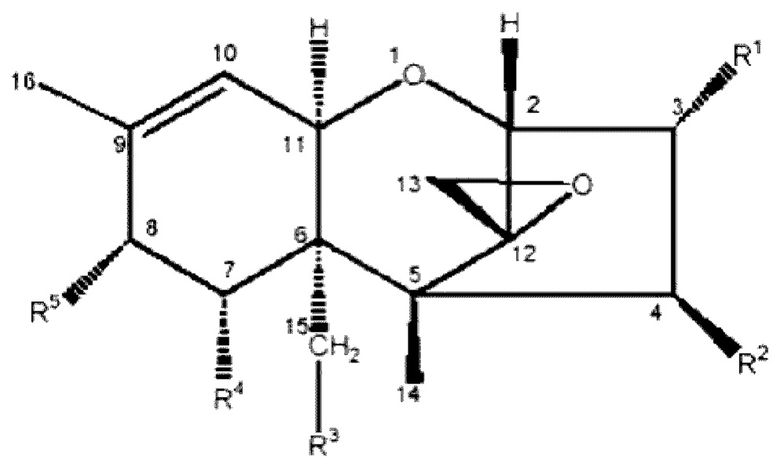
[00113] Специалисту в данной области техники известно, какие замены могут присутствовать в положениях R1, R2, R3, R4 и R5. Известно, что токсичность трихотеценов (по меньшей мере) частично обусловлена присутствием гидроксильной (-ОН) группы при атоме С-3. Таким образом, в некоторых трихотеценах С-3 соединен с гидроксильной группой (R1=-ОН/-гидроксигруппа).
[00114] Специалисту в данной области техники известны трихотецены, содержащие 3-гидроксигруппу (3-ОН) (в положении R1 в приведенной выше формуле). Неограничивающие примеры таких трихотеценов включают ДОН (ДОН, номер в реестре CAS 51481-10-8), токсин Т-2 (номер в реестре CAS 21259-20-1), токсин НТ-2 (номер в реестре CAS 26934-87-2), ниваленол (номер в реестре CAS 23282-20-4), фузеранон X (fuseranon X) (номер в реестре CAS 23255-69-8), сцирпенетриол (scirpenetriol) (номер в реестре CAS 2270-41-9), 15-ацетоксисцирпенол (номер в реестре CAS 2623-22-5), 4,15-диацетоксисцирпенол (номер в реестре CAS 2270-40-8), деацетилнеосоланиол (номер в реестре CAS 74833-39-9), неосоланиол (номер в реестре CAS 36519-25-2), споротрихиол (sporotrichiol) (номер в реестре CAS 101401-89-2) и самбуцинол (номер в реестре CAS 90044-33-0).
[00115] Эти трихотецены, содержащие 3-гидроксигруппу (3-ОН), могут быть окислены в трихотецены, содержащие 3-оксогруппу (=O; в положении R1). Ферменты, способные к такому превращению, описаны в настоящем документе. Далее полипептиды могут превращать эти трихотецены, содержащие 3-оксогруппу, в предпочтительно один из двух изомеров трихотеценов, содержащих 3-гидроксигруппу (3-ОН), а именно изомеров с R- и S-конфигурацией.
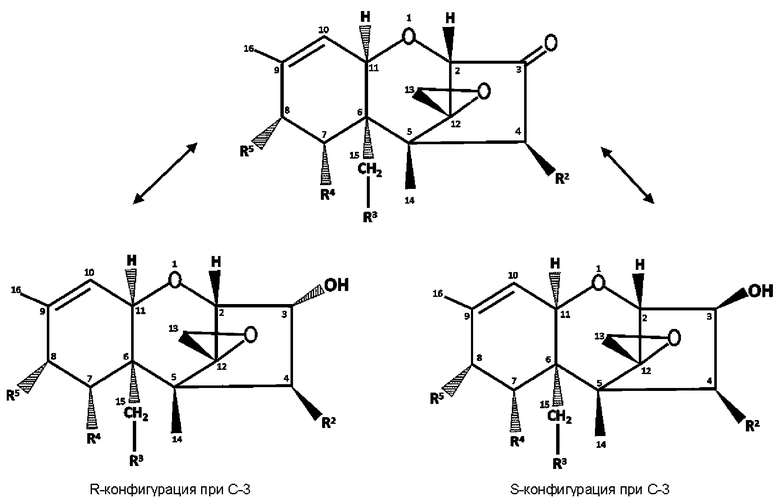
[00116] Настоящее изобретение охватывает, что трихотецен, содержащий 3-гидроксигруппу, присутствует в S-конфигурации.
[00117] В качестве дополнения или альтернативы, настоящее изобретение также относится к способу превращения 3-кето-ДОН в 3-эпи-ДОН, включающему приведение в контакт одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, с 3-кето-ДОН.
[00118] Настоящее изобретение также относится к способу снижения содержания ДОН в композиции, содержащей ДОН, или кспособу снижения токсичности композиции, содержащей ДОН, путем превращения ДОН в 3-эпи-ДОН, включающему
a) приведение в контакт указанной композиции с ферментом, способным превращать ДОН в 3-кето-ДОН, как описано, помимо прочего, в настоящем документе; и
b) последующее или одновременное приведение в контакт указанной композиции с одним или более полипептидами, содержащими последовательность или состоящими из последовательности SEQ ID NO. 1-14 или содержащими последовательность или состоящими из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14. Этот способ дополнительно может включать стадию приведения в контакт композиции с по меньшей мере одним хиноновым кофактором и/или по меньшей мере одним ионом металла и/или по меньшей мере одним окислительно-восстановительным кофактором. По меньшей мере один хиноновый кофактор может быть выбран из группы, состоящей из PQQ (пирролохинолинхинона (номер в реестре CAS 72909-34-3), триптофантриптофилхинона (TTQ, номер в реестре CAS 134645-25-3), топахинона (TPQ, номер в реестре CAS 64192-68-3), лизинтирозилхинона (LTQ, номер в реестре CAS 178989-72-5) и цистеинтриптофилхинона (CTQ, номер в реестре CAS 400616-72-0). Ион металла, обеспечивающий быстрое и полное связывание хинонового кофактора с ферментом, способным превращать ДОН в 3-кето-ДОН, который предпочтительно может представлять собой ал ко гол ьде гид роге на зу, имеющую последовательность SEQ ID NO. 1 -3 и описанную в WO2016/154640, может быть выбран из группы, состоящей из Li+, Na+, K+, Mg2+, Са2+, Zn2+, Zn3+, Mn2+, Mn3+, Fe2+, Fe3+, Cu2+, Cu3+, Co2+ и Co3+, предпочтительно Ca2+и Mg2+. По меньшей мере один окислительно-восстановительный кофактор может быть выбран из группы, состоящей из НАД+, НАДФ+, феназинметосульфатной группы (PMS, номер в реестре CAS 299-11-6), производных PMS, гексацианоферрата калия (III), гексацианоферрата натрия (III), цитохрома С, кофермента Q1, кофермента Q10, метиленового синего и N,N,N',N'-тетраметил-п-фенилендиамина (TMPD). Примерами производных PMS являются: 1-гидроксифеназин, 2-(пентафенилокси)дигидрофеназин, 5,10-дигидро-9-диметилаллилфеназин-1-карбоновая кислота, 5,10-дигидрофеназин-1-карбоновая кислота, метилсульфат 5-метилфеназиния, 6-ацетофеназин-1-карбоновая кислота, бентофоенин (benthophoenin), кпофазимин, дигидрометанофеназин, эсмеральдиновая кислота, эсмеральдин В, изумифеназин А-С, катион красителя Зеленый янус В (Janus Green В), метанофеназин, пелагиомицин А, феназин, феназин-1,6-дикарбоновая кислота, феназин-1-карбоксамид, феназин-1-карбоновая кислота, феносафранин, пиоцианин, сафенамицин или метиловый эфир сафеновой кислоты.
[00119] Настоящее изобретение также относится к молекулам нуклеиновой кислоты, кодирующим полипептид, описанный в настоящем документе. Нуклеиновая кислота может быть введена или встроена в вектор экспрессии. Термин «вектор экспрессии» относится к конструкции молекулы нуклеиновой кислоты, которая способна экспрессировать ген in vivo или in vitro. В частности, он может охватывать конструкции ДНК, подходящие для переноса кодирующей полипептид нуклеотидной последовательности в клетку-хозяина таким образом, чтобы эта последовательность была интегрирована в геном или свободно располагалась во внехромосомном пространстве, и для внутриклеточной экспрессии кодирующей полипептид нуклеотидной последовательности и, необязательно, транспорта полипептида из клетки.
[00120] Кроме того, настоящее изобретение также относится к клетке-хозяину (например, рекомбинантной выделенной клетке-хозяину), содержащей один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14. Настоящее изобретение также относится к клетке-хозяину, содержащей последовательность нуклеиновой кислоты, кодирующую полипептид, описанный в настоящем документе.
[00121] Термин «клетка-хозяин» относится ко всем клеткам (например, рекомбинантной клетке-хозяину, выделенной клетке-хозяину или выделенной рекомбинантной клетке-хозяину), содержащим либо подлежащую экспрессии нуклеотидную последовательность, либо вектор экспрессии, которые способны обеспечивать выработку фермента или полипептида в соответствии с настоящим изобретением. Указанный термин также охватывает любое потомство родительской клетки, которое не является идентичным родительской клетке вследствие мутаций, появляющихся в процессе репликации, а также рекомбинантной клетки-хозяина, выделенной клетки-хозяина (например, выделенной рекомбинантной клетки-хозяина). В частности, это относится к прокариотическим и/или эукариотическим клеткам, предпочтительно Pichia pastoris, Escherichia coii, Bacillus subtilis, Streptomyces, Hansenula, Trichoderma, Lactobacillus, Aspergillus, растительным клеткам и/или спорам Bacillus, Trichoderma или Aspergillus. Название P. pastoris, используемое в настоящем документе, является синонимом названия Komagataella pastoris, при этом P. pastoris является более старым, а К pastoris - систематически более новым названием (Yamada et al. (1995)). Следует отметить, что виды Komagataella pastoris недавно были переопределены как Komagataella phaffii (Kurtzman (2009)). В настоящем документе Komagataella phaffii может, например, относиться к штаммам Komagataella phaffii CBS 7435, Komagataella phaffii GS115 или Komagataella phaffii JC308.
[00122] В настоящее изобретение также включено, что клетка-хозяин дополнительно может экспрессировать кофактор для полипептида(ов), описанного(ых) в настоящем документе. Кофактор может представлять собой любой подходящий кофактор. Например, кофактор представляет собой НАД/Н или НАДФ/Н.
[00123] Настоящее изобретение также относится к растению, генетически модифицированному для экспрессии одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14. Типичные растения включают, помимо прочего, зерновую культуру (кукурузу), пшеницу, ячмень, рожь и овес.
[00124] Настоящее изобретение также относится к семени растения, описанного в настоящем документе.
[00125] Настоящее изобретение также относится к препарату, содержащему
a) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1; и
b) носитель.
[00126] В качестве дополнения или альтернативы, настоящее изобретение также относится к препарату, содержащему
a) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН;
b) носитель.
[00127] «Препарат» в соответствии с настоящим изобретением может быть получен с применением полипептида(ов), описанного(ых) в настоящем документе, в композиции, описанной в настоящем документе. Таким образом, в одном варианте реализации препарат может содержать полипептиды или части полипептида(ов), описанные в настоящем документе, а также другие компоненты, такие как носитель, экстракты из сельскохозяйственных продуктов и т.д. Препарат также может содержать другие молекулы и/или белки и/или вещества, например, остаточное количество буфера, применяемого в способе согласно настоящему изобретению, вследствие, например, менее эффективной очистки полипептида(ов), описанного(ых) в настоящем документе.
[00128] Настоящее изобретение также относится к полипептиду, содержащему последовательность или состоящему из последовательности SEQ ID NO. 1-14 или содержащему последовательность или состоящему из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением по меньшей мере 25:1 (3-эпи-ДОН/ДОН), для применения при предотвращении и/или лечении микотоксикоза.
[00129] Настоящее изобретение также относится к полипептиду, содержащему последовательность или состоящему из последовательности SEQ ID NO. 1-14 или содержащему последовательность или состоящему из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, для применения при предотвращении и/или лечении микотоксикоза.
[00130] Настоящее изобретение также относится к фармацевтической композиции, содержащей полипептид, содержащий последовательность или состоящий из последовательности SEQ ID NO. 1-14 или содержащий последовательность или состоящий из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, для применения при предотвращении и/или лечении микотоксикоза.
[00131] Фармацевтическая композиция дополнительно может содержать фармацевтически приемлемый носитель.
[00132] Настоящее изобретение также относится к способу лечения или предотвращения микотоксикоза у субъекта, включающему введение одного или более полипептидов, описанных в настоящем документе. Предпочтительно вводят терапевтически эффективное количество одного или более полипептидов, описанных в настоящем документе. Субъект может быть поражен микотоксикозом.
[00133] В некоторых предпочтительных вариантах реализации способ или применение согласно настоящему изобретению (например, включающие SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно 4,5 до примерно 10, например, при рН примерно 6,5) и/или осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С), предпочтительно как описано в разделе «Примеры» в настоящем документе (например, в примерах 4-5).
[00134] Следует отметить, что в настоящем документе неопределенная и определенная формы единственного числа включают определяемые объекты во множественном числе, если контекст явно не предписывает иное. Таким образом, например, ссылка на «полипептид» включает один или более таких различных полипептидов, и ссылка на «способ» включает ссылку на эквивалентные стадии и способы, известные специалистам в данной области техники, которые могут быть модифицированы или заменены на способы, описанные в настоящем документе.
[00135] Если не указано иное, термин «по меньшей мере», предшествующий ряду элементов, следует понимать как относящийся к каждому элементу в данном ряду. Специалисты в данной области техники будут способны понять или установить, с использованием лишь обычных экспериментов, многие эквиваленты конкретных вариантов реализации настоящего изобретения, описанных в настоящем документе. Подразумевается, что такие эквиваленты охватываются настоящим изобретением.
[00136] Термин «и/или», где бы он ни использовался в настоящем документе, включает значения «и», «или» и «все или любая другая комбинация элементов, связанных указанным термином».
[00137] Термин «менее чем» или, в свою очередь, «более чем» не включает конкретное число.
[00138] Например, менее 20 означает менее чем указанное число. Аналогичным образом, более чем или больше чем означает более чем или больше чем указанное число, например, более 80% означает более чем или больше чем указанное число 80%.
[00139] Во всем тексте настоящего описания и прилагаемой формуле изобретения, если контекст не требует иного, слово «содержать» и его варианты, такие как «содержит» и «содержащий», подразумевают включение указанного целого числа или стадии или группы целых чисел или стадий, но не исключают любые другие целое число или стадию или группу целых чисел или стадий. В настоящем документе термин «содержащий» может быть заменен терминами «содержащий» или «включающий» или иногда термином «имеющий». В настоящем документе термин «состоящий из» исключает любые неуказанные элемент, стадию или ингредиент.
[00140] Термин «включающий» означает «включающий, но не ограничивающийся». Термины «включающий» и «включающий, но не ограничивающийся» используются взаимозаменяемо.
[00141] Следует понимать, что настоящее изобретение не ограничивается конкретной методологией, протоколами, материалами, реагентами и веществами и т.д., описанными в настоящем документе, и таким образом может варьировать. Терминология, используемая в настоящем документе, предназначена только для описания конкретных вариантов реализации и не ограничивает объем настоящего изобретения, который определяется исключительно формулой изобретения.
[00142] Все публикации, цитируемые как выше, так и ниже во всем тексте настоящего описания (включая все патенты, заявки на патенты, научные публикации, инструкции и т.д.), полностью включены в настоящий документ посредством ссылки. В настоящем документе ничего не следует толковать как признание того, что настоящее изобретение не датировано более ранним числом на основании предшествующего изобретения. В той степени, в которой материал, включенный посредством ссылки, противоречит настоящему описанию или не согласуется с ним, настоящее описание заменяет собой любой такой материал.
[00143] Содержание всех документов и патентных документов, цитируемых в настоящем документе, полностью включено посредством ссылки.
[00144] Настоящее изобретение дополнительно характеризуется следующими аспектами.
[00145] 1. Композиция, содержащая
a) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением по меньшей мере 25:1 (3-эпи-ДОН ДОН); и
b) носитель.
[00146] 2. Композиция, содержащая
а) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, и b) носитель.
[00147] 3. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид представляет собой редуктазу.
[00148] 4. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит более 344 аминокислот.
[00149] 5. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит петлю в пределах положений аминокислот, соответствующих положениям аминокислот
125-133 в SEQ ID NO. 1-10 или 63-101;
127-135 в SEQ ID NO. 13 или 14;
125-132 в SEQ ID NO. 11 или 12.
[00150] 6. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит в пределах положений аминокислот, соответствующих положениям аминокислот
125-133 в SEQ ID NO. 1-10 или 63-101;
127-135 в SEQ ID NO. 13 или 14;
124-132 последовательности SEQ ID NO. 11 или 12, последовательность, которая не содержит метионина (Met/M).
[00151] 7. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит последовательность, содержащую последовательность X1-H-X2-W-D-X3-X4-T-P, где
Х1 не является М; Х2 не является А;
Х3 не является А;
Х4 не является V.
[00152] 8. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит последовательность, содержащую последовательность X1-H-X2-W-D-X3-X4-T-P, где
Х1 представляет собой V, I, L, М, F или А, предпочтительно V;
Х2 представляет собой Е, D, Q или N, предпочтительно Е или Q;
Х3 представляет собой G или А, предпочтительно G;
Х4 представляет собой М, L, F, I, R, K, Q, N, Е, V, А или норлейцин, предпочтительно М, R, Q или L.
[00153] 9. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит петлю в пределах положений аминокислот, соответствующих положениям аминокислот
211-244 в SEQ ID NO. 1-8, 10 или 63-101;
211-245 в SEQ ID NO. 9;
210-243 в SEQ ID NO. 11 или 12;
213-247 в SEQ ID NO. 13 или 14.
[00154] 10. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит в пределах положений аминокислот, соответствующих положениям аминокислот
211-244 в SEQ ID NO. 1-8, 10 или 63-101;
211-245 в SEQ ID NO. 9;
210-243 в SEQ ID NO. 11 или 12;
213-247 в SEQ ID NO. 13 или 14, последовательность, которая не содержит метионина (Met/M).
11. Композиция в соответствии с любым из предшествующих пунктов,
отличающаяся тем, что указанный полипептид содержит последовательность, содержащую последовательность S-P-L-X5-G-G-X6-L-X7-G-K, где
Х5 не является G;
Х6 представляет собой L, I, V, М, A, Y, F, норлейцин или W;
Х7 не является А или Т.
[00155] 12. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит последовательность, содержащую последовательность S-P-L-X5-G-G-X6-L-X7-G-K, где Х5 представляет собой А, V, G, L или I, предпочтительно А;
Х6 представляет собой L, I, V, М, A, Y, F, норлейцин или W, предпочтительно L или W;
Х7 представляет собой S или Т, предпочтительно S.
[00156] 13. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит последовательность, содержащую последовательность
X8-G-X9-X10-Х11-Х12-Х13-Х14-Х15-Х16, где
Х8 представляет собой G, R, K, Q, N, А, V, L, I, S, Т, Е, D;
Х9 не является М или G;
Х10 не является Е;
Х11 не является S;
Х12 не является Y;
Х13 не является G или А;
Х14 не является Р;
Х15 представляет собой R, K, Q, N, Р, A, Y, W, F, Т, S, Н;
Х16 не является N.
[00157] 14. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит последовательность, содержащую последовательность X8-G-X9-X10-Х11-Х12-Х13-Х14-Х15-Х16, где Х8 представляет собой G, R, K, Q, N, А, V, L, I, S, Т, Е, D предпочтительно G, R, А, Е, более предпочтительно G;
Х9 представляет собой W, S, Т, F, L, V, I, А или Y, предпочтительно W, S, F, более предпочтительно F;
Х10 представляет собой Т, S, R, Q, N или K, предпочтительно Т, R или K, более предпочтительно R;
Х11 представляет собой Е, D или Q, предпочтительно Е;
Х12 представляет собой Р или А, предпочтительно Р;
Х13 представляет собой Р или А, предпочтительно Р;
Х14 представляет собой I, L, М, A, F или V, предпочтительно I или V, более предпочтительно V;
Х15 представляет собой R, K, Q, N, Р, А, Н, W, F, Т, S или Y, предпочтительно R, Р, Н или Y, более предпочтительно Р;
Х16 представляет собой D, Е или N, предпочтительно D.
[00158] 15. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанная последовательность на по меньшей мере 80%, по меньшей мере 83%, по меньшей мере 85%, по меньшей мере 87%, по меньшей мере 90%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или более идентична любой из SEQ ID NO. 1-14, предпочтительно указанная композиция состоит из полипептида, имеющего любую из последовательностей SEQ ID NO. 1-14 или их комбинаций.
[00159] 16. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что один или более полипептидов, содержащих последовательность, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, содержат одну или более аминокислотных замен по сравнению с любой из последовательностей SEQ ID NO. 1-14.
[00160] 17. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанные одна или более аминокислотных замен включают консервативные аминокислотные замены или состоят из них.
[00161] 18. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением по меньшей мере 50:1, по меньшей мере 100:1, по меньшей мере 150:1, по меньшей мере 200:1, 250:1, 300:1, 350:1, 400:1, 500:1, 600:1, 700:1, 800:1, 900:1, 1000:1 или более.
[00162] 19. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что, когда 3-эпи-ДОН получают со скоростью 1 мкмоль 3-эпи-ДОН в минуту, то количество указанного полипептида составляет не более 7 мг, не более 6 мг, не более 5 мг, не более 3 мг, не более 2 мг, не более 1 мг или менее.
[00163] 20. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид способен превращать 3-кето-ДОН в 3-эпи-ДОН с удельной активностью, составляющей по меньшей мере 0,1 мкмоль/мин/мг, по меньшей мере 0,2 мкмоль/мин/мг, по меньшей мере 0,3 мкмоль/мин/мг, по меньшей мере 0,4 мкмоль/мин/мг, по меньшей мере 0,5 мкмоль/мин/мг, по меньшей мере 0,6 мкмоль/мин/мг, по меньшей мере 0,8 мкмоль/мин/мг, по меньшей мере 1,0 мкмоль/мин/мг, по меньшей мере 1,2 мкмоль/мин/мг, по меньшей мере 1,4 мкмоль/мин/мг или по меньшей мере 1,6 мкмоль/мин/мг.
[00164] 21. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что, когда ДОН получают со скоростью 1 мкмоль ДОН в минуту, то количество указанного полипептида составляет по меньшей мере 600 мг, по меньшей мере 700 мг, по меньшей мере 800 мг, по меньшей мере 900 мг, по меньшей мере 1000 мг, по меньшей мере 1100 мг, по меньшей мере 1300 мг, по меньшей мере 1500 мг, по меньшей мере 1600 мг, по меньшей мере 1700 мг, по меньшей мере 1800 мг, по меньшей мере 1900 мг, по меньшей мере 2000 мг или более.
[00165] 22. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид, присутствующий в композиции, способен превращать 3-кето-ДОН в ДОН с удельной активностью, составляющей не более 0,0015 мкмоль/мин/мг, не более 0,0010 мкмоль/мин/мг, не более 0,0009 мкмоль/мин/мг, не более 0,0008 мкмоль/мин/мг, не более 0,0007 мкмоль/мин/мг, не более 0,0006 мкмоль/мин/мг или менее.
[00166] 23. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный носитель представляет собой жидкость, предпочтительно Н2О, или твердое вещество, предпочтительно нутрицевтик и/или фармацевтическое средство.
[00167] 24. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный носитель представляет собой съедобный компонент, предпочтительно нетоксичный компонент и/или компонент, обеспечивающий консистенцию.
[00168] 25. Полипептид, содержащий последовательность SEQ ID NO. 1 или последовательность, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1, при этом указанный полипептид способен превращать 3-кето-ДОН в 3-эпи-ДОН.
[00169] 26. Применение одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, для превращения 3-кето-ДОН в 3-эпи-ДОН; предпочтительно при этом: (i) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно 4,5 до примерно 10, например, при рН примерно 6,5) и/или (ii) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно от 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С).
[00170] 27. Применение одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, при получении кормовой добавки или кормовой композиции или фармацевтической композиции; предпочтительно при этом: (i) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно 4,5 до примерно 10, например, при рН примерно 6,5) и/или (и) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С).
[00171] 27. Применение одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, при получении кормовой добавки или кормовой композиции или фармацевтической композиции; предпочтительно при этом: (i) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно 4,5 до примерно 10, например, при рН примерно 6,5) и/или (ii) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С).
[00172] 28. Применение одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, при получении биогаза, биоэтанола или сахара, предпочтительно из сахарного тростника или сахарной свеклы; также предпочтительно при этом: (i) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно от 4,5 до примерно 10, например, при рН примерно 6,5) и/или (и) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С).
[00173] 29. Применение одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, при получении биогаза, биоэтанола или сахара, предпочтительно из сахарного тростника или сахарной свеклы; предпочтительно при этом: (i) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно 4,5 до примерно 10, например, при рН примерно 6,5) и/или (ii) указанное применение (например, включающее SEQ ID NO: 1) осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С).
[00174] 30. Кормовые или пищевые добавки или кормовые или пищевые продукты, содержащие один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
[00175] 31. Кормовые или пищевые добавки или кормовые или пищевые продукты, содержащие один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН.
[00176] 32. Добавка для применения в композициях, предпочтительно композициях сельскохозяйственных продуктов, содержащих 3-кето-ДОН или ДОН, при этом указанная добавка содержит один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
[00177] 33. Добавка для применения в композициях, предпочтительно композициях сельскохозяйственных продуктов, содержащих 3-кето-ДОН или ДОН, при этом указанная добавка содержит один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН.
[00178] 34. Способ превращения трихотецена, содержащего 3-оксогруппу, в трихотецен, содержащий 3-гидроксигруппу, включающий приведение в контакт одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, с трихотеценом, содержащим 3-оксогруппу; предпочтительно при этом: (i) указанный способ (например, включающий SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно 4,5 до примерно 10, например, при рН примерно 6,5) и/или (ii) указанный способ (например, включающий SEQ ID NO: 1) осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С).
[00179] 35. Способ в соответствии с аспектом 29, отличающийся тем, что указанный трихотецен, содержащий 3-гидроксигруппу, присутствует в S-конфигурации.
[00180] 36. Способ превращения 3-кето-ДОН в 3-эпи-ДОН, включающий приведение в контакт одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, с 3-кето-ДОН; предпочтительно при этом: (i) указанный способ (например, включающий SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно 4,5 до примерно 10, например, при рН примерно 6,5) и/или (ii) указанный способ (например, включающий SEQ ID NO: 1) осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С).
[00181] 37. Способ снижения содержания ДОН в композиции, содержащей ДОН, или снижения токсичности композиции, содержащей ДОН, путем превращения ДОН в 3-эпи-ДОН, включающий
a) приведение в контакт указанной композиции с ферментом, способным превращать ДОН в 3-кето-ДОН; и
b) последующее или одновременное приведение в контакт указанной композиции с одним или более полипептидами, содержащими последовательность или состоящими из последовательности SEQ ID NO. 1-14 или содержащими последовательность или состоящими из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и/или содержащими последовательность или состоящими из последовательности SEQ ID NO. 63-101; предпочтительно при этом: (i) указанный способ (например, включающий SEQ ID NO: 1) осуществляют в диапазоне рН от примерно 4 до примерно 10 (например, рН от примерно 4,5 до примерно 10, например, при рН примерно 6,5) и/или (ii) указанный способ (например, включающий SEQ ID NO: 1) осуществляют в диапазоне температур (Т) от примерно 14°С до примерно 60°С (например, Т от примерно 14,9°С до примерно 36°С, например, Т от примерно 25°С до примерно 31°С).
[00182] 38. Клетка-хозяин, содержащая один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101.
[00183] 39. Клетка-хозяин в соответствии с аспектом 14, дополнительно экспрессирующая кофактор для полипептида, предпочтительно сверхэкспрессирующая НАДН или НАДФН.
[00184] 40. Растение, генетически модифицированное для экспрессии одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101.
[00185] 41. Семя растения, определенного в аспекте 40.
[00186] 42. Препарат, содержащий
а) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1; и
b) носитель.
[00187] 43. Препарат, содержащий
a) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащих или состоящих из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН;
b) носитель.
[00188] 43. Полипептид, содержащий последовательность или состоящий из последовательности SEQ ID NO. 1-14 или содержащий последовательность или состоящий из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащий последовательность или состоящий из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1;
для применения при предотвращении и/или лечении микотоксикоза.
[00189] 44. Полипептид, содержащий последовательность или состоящий из последовательности SEQ ID NO. 1-14 или содержащий последовательность или состоящий из последовательности, которая на по меньшей мере 75% (например, по меньшей мере 90,7%, по меньшей мере 90,8%, по меньшей мере 90,9%, по меньшей мере 91%, по меньшей мере 92%, по меньшей мере 93%, по меньшей мере 93,5%, по меньшей мере 93,6%, по меньшей мере 93,7%, по меньшей мере 93,8%, по меньшей мере 93,9%, по меньшей мере 94%, по меньшей мере 95%, по меньшей мере 96%, по меньшей мере 97%, по меньшей мере 98%, по меньшей мере 99% или 100%) идентична любой из SEQ ID NO. 1-14, и/или содержащий последовательность или состоящий из последовательности SEQ ID NO. 63-101, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукт 3-эпи-ДОН и, необязательно, помимо этого в продукт ДОН с получением 100% общего количества продукта, причем по меньшей мере 96% от общего количества продукта представляют собой 3-эпи-ДОН, для применения при предотвращении и/или лечении микотоксикоза.
[00190] 45. Композиция в соответствии с любым из предшествующих пунктов, отличающаяся тем, что указанный полипептид содержит петлю В, имеющую длину не более 39 аминокислот, не более 38 аминокислот, не более 37 аминокислот, не более 35 аминокислот, не более 34 аминокислот или содержащую меньшее количество аминокислот.
[00191] 46. Полипептид, содержащий последовательность SEQ ID NO. 63-101 или последовательность, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 63-101, при этом указанный полипептид способен превращать 3-кето-ДОН в 3-эпи-ДОН.
[00192] В настоящей заявке применяются следующие последовательности.

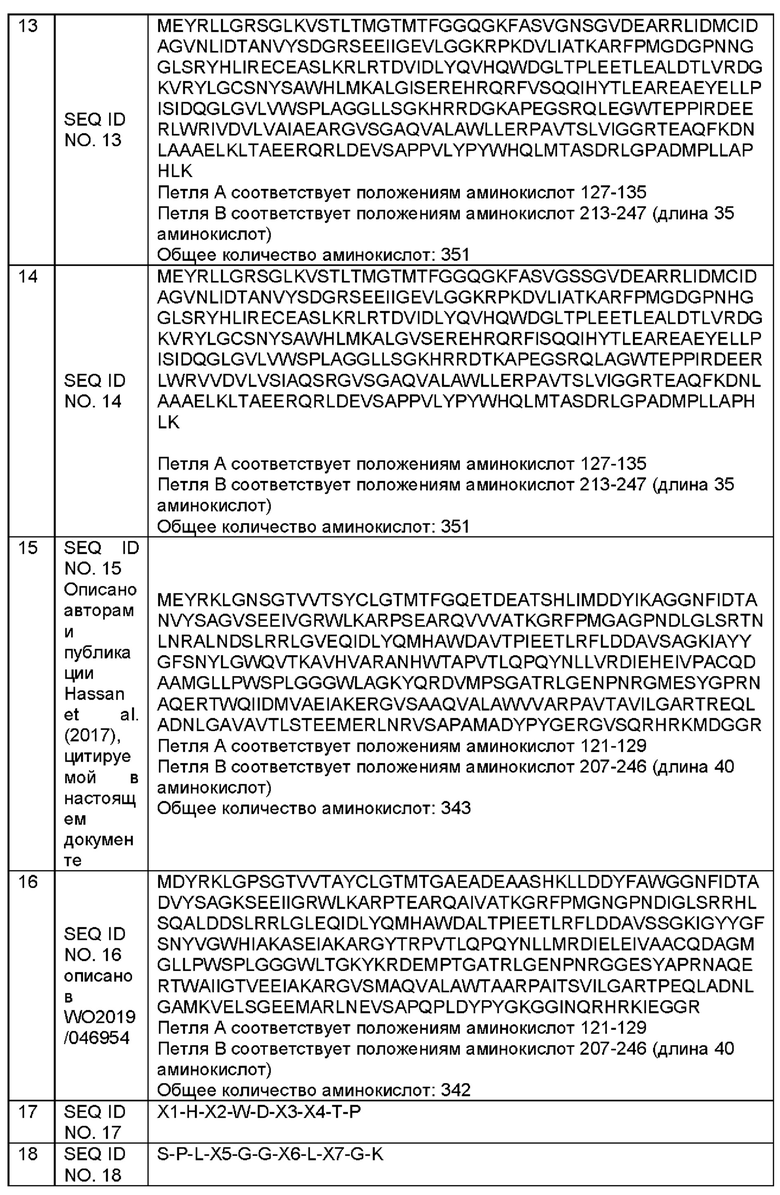
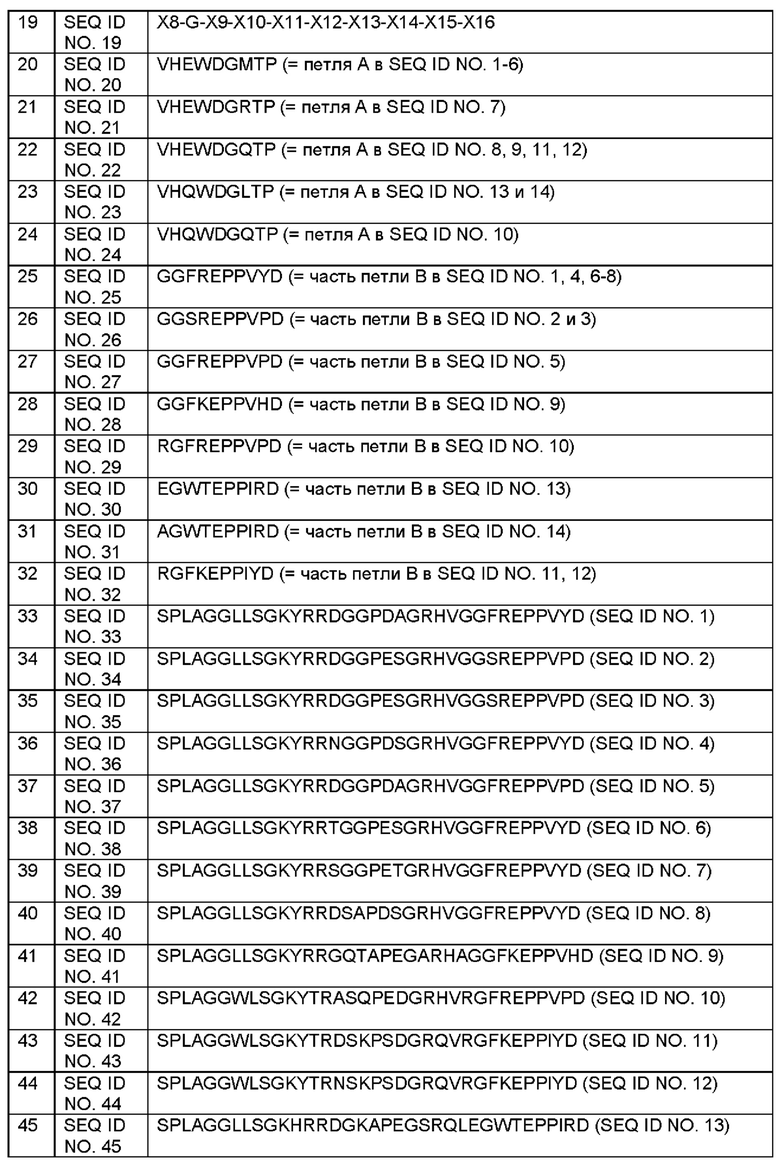
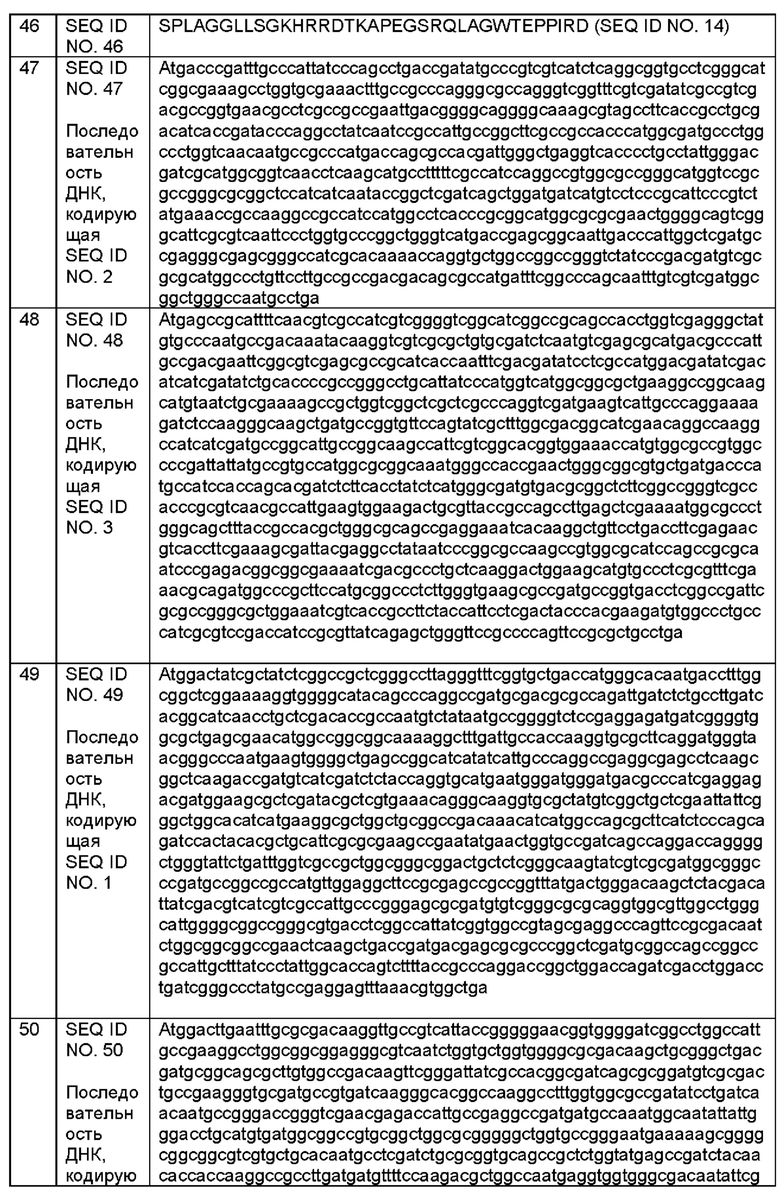
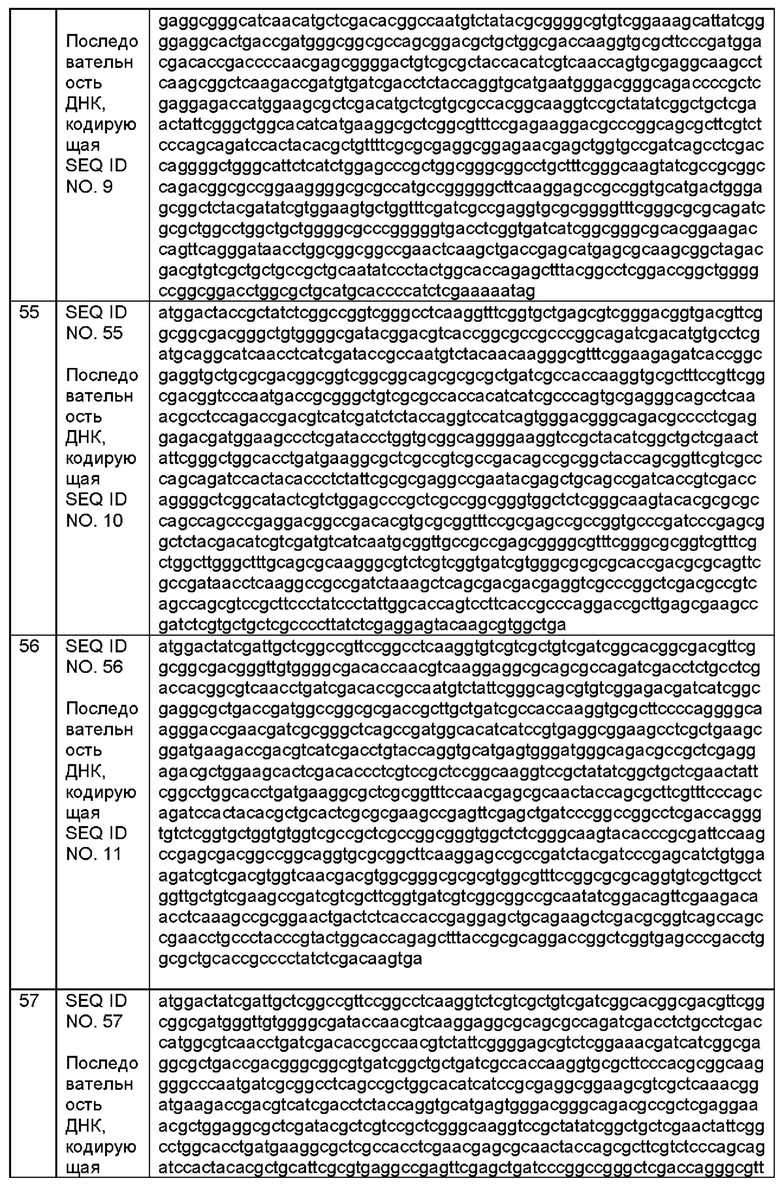
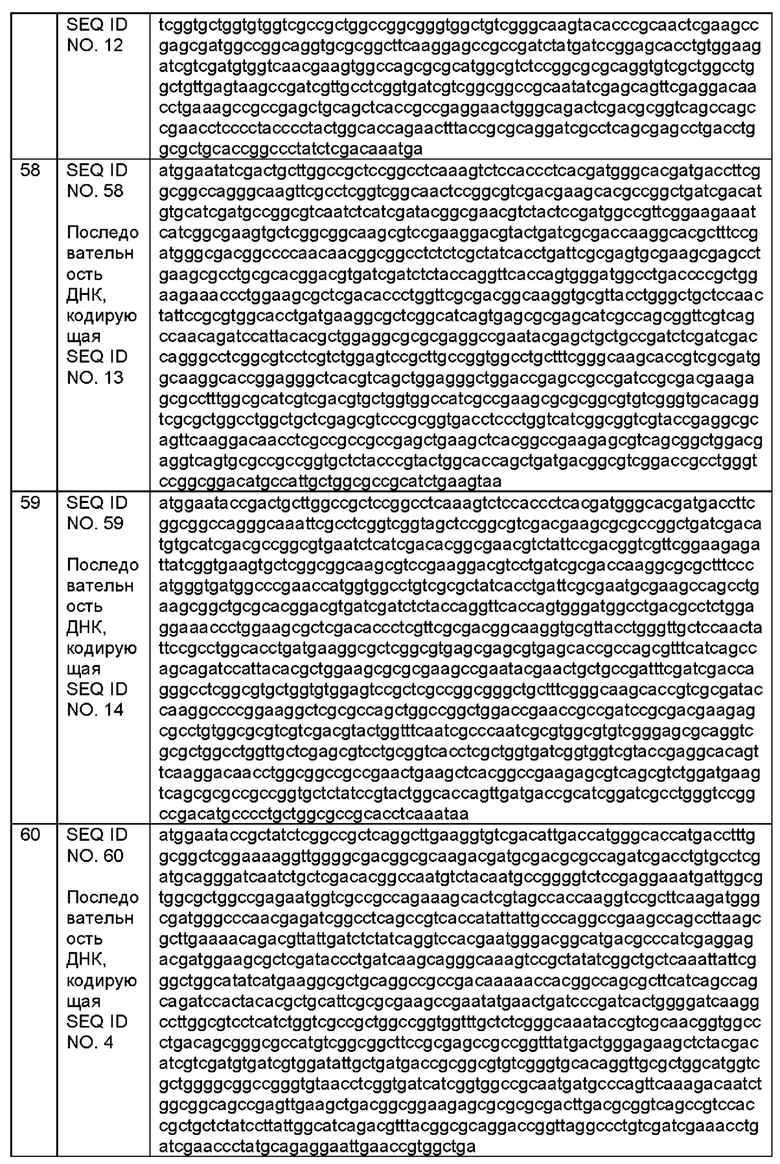
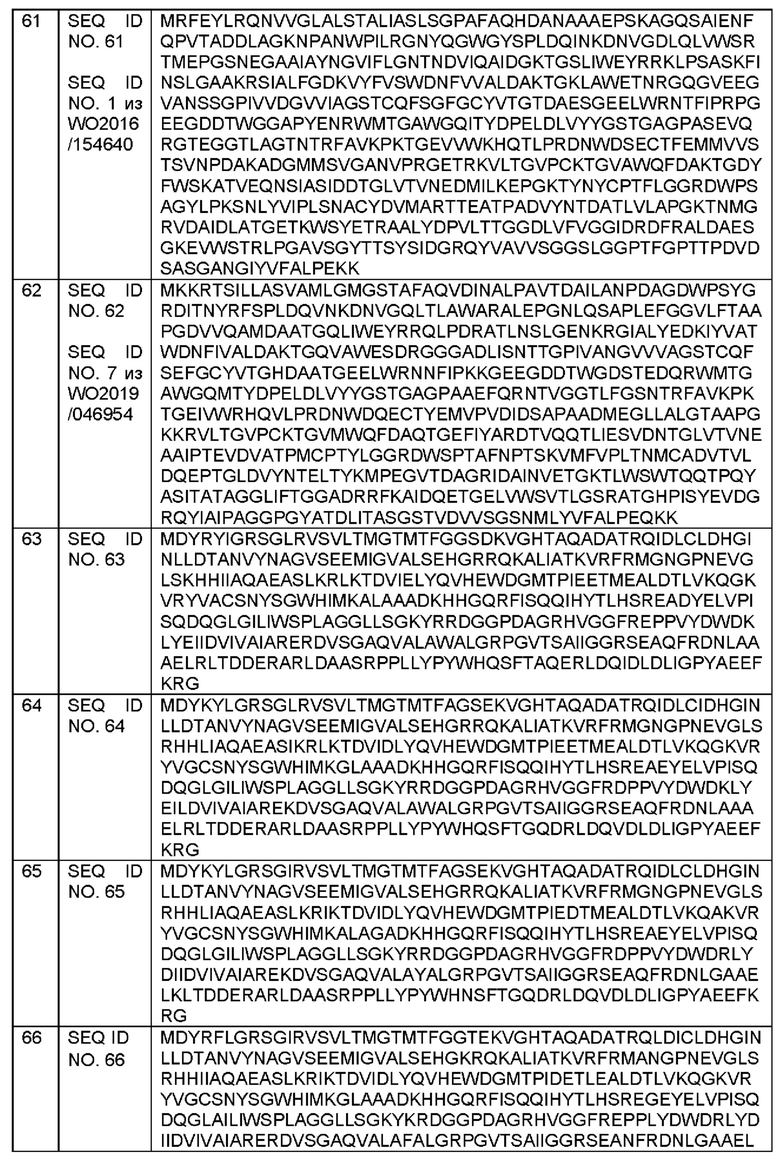


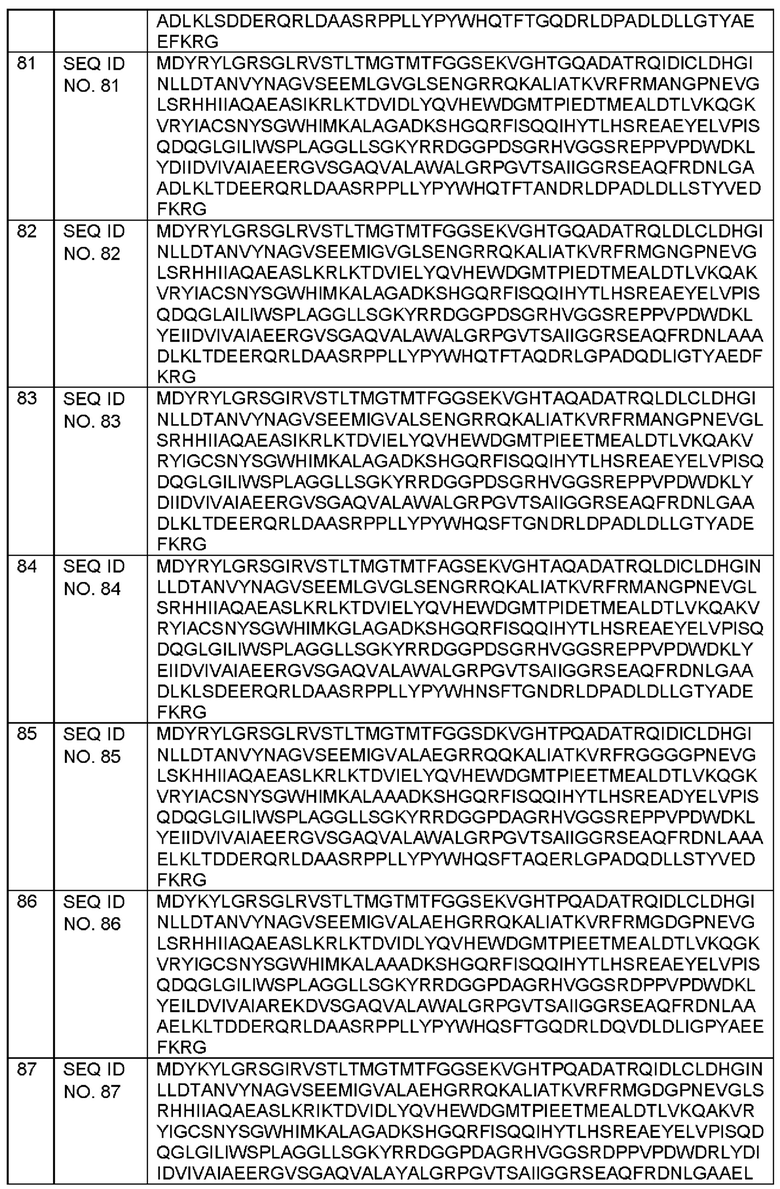


ПРИМЕРЫ СОГЛАСНО НАСТОЯЩЕМУ ИЗОБРЕТЕНИЮ
[00193] Пример 1: Клонирование, экспрессия и очистка ферментов
[00194] Авторы настоящего изобретения обнаружили различные гены-кандидаты, которые могли бы кодировать ферменты, обладающие способностью восстанавливать 3-оксотрихотецены, такие как кето-ДОН, до трихотеценов, содержащих 3-гидроксигруппу, таких какэпи-ДОН. Для экспрессии и очистки белков, кодируемых всеми генами-кандидатами, нуклеотидные последовательности, кодирующие полипептиды ферментов, имеющих последовательности SEQ ID №1-14 (а именно SEQ ID №47-60), а также последовательности, кодирующие известные из уровня техники ферменты, имеющие последовательности SEQ ID №15 и SEQ ID №16, а также последовательности, кодирующие варианты ферментов, имеющих последовательность либо SEQ ID №1 (SEQ ID №63-75), либо SEQ ID №3 (SEQ ID №76-88), либо SEQ ID №6 (SEQ ID №89-101), которые содержат аминокислотные замены в соответствии с таблицей 1, раскрытой в настоящем документе, были встроены в плазмиды для экспрессии в подходящем организме-хозяине, таком как Е, coll. Рекомбинантные векторы, содержащие соответствующие кодирующие последовательности, были собраны с помощью инструментов TWIST bioscience.com (США) в вектор рЕТ28а для экспрессии в Е, coli, при этом нуклеотидные последовательности содержали сайт рестрикции Ncol на 5'-конце и сайт рестрикции Xhol на 3'-конце, а также метку 6xHis либо на С-конце, либо на N-конце. Идентичность последовательностей для последовательностей SEQ ID NO. 1-16, 63-101 показана на фигуре 1А и фигуре 1 В, а также в приведенной в настоящем документе таблице последовательностей.
[00195] Для экспрессии рекомбинантные векторы на основе рЕТ28а переносили в Е, coli или любой другой подходящий организм-хозяин. Экспрессию белка индуцировали путем добавления изопропил-бета-D-1-тиогалактопиранозида (IPTG). Клетки собирали путем центрифугирования и затем разрушали их с помощью ультразвука, френч-пресса или любого другого подходящего способа разрушения бактериальных клеток и получения неочищенных клеточных лизатов. Эти лизаты очищали, и растворимый рекомбинантный белок использовали для определения каталитических свойств полученных редуктаз. Для определения специфических свойств редуктаз ферменты селективно очищали посредством аффинной хроматографии на ионах никеля с использованием колонок с никель-сефарозой. К сожалению, полученные количества фермента, имеющего последовательность SEQ ID NO. 16, были слишком малы для проведения анализа чистоты и анализов ферментативных свойств. С целью определения чистоты рекомбинантных ферментов их разделяли на геле для белков посредством электрофореза в полиакриламидном геле в присутствии додецилсульфата натрия для визуализации белков с помощью стандартного окрашивания Кумасси синим. Концентрации различных белков определяли фотометрически на спектрофотометре для прочтения планшетов (Biotek, Synergy НТ) при длине волны 595 нм с использованием реагента Брэдфорд (Sigma, № по каталогу В6916) и/или посредством флуориметрии с использованием набора для определения количества белка Qubit (Thermofischer Life technologies, №по каталогу Q33211). При проведении анализа методом Брэдфорд в качестве основы для определения концентрации белка строили стандартную кривую, полученную путем проведения измерений для бычьего сывороточного альбумина (БСА, Sigma, № по каталогу А4919) в концентрациях от 0 мкг/мл до максимум 1500 мкг/мл. При проведении анализа с использованием набора Qubit стандартная кривая была получена путем проведения измерений для предложенных производителем стандартных образцов с концентрациями от 0 мкг/мл до 400 мкг/мл и далее использована для определения концентрации белка в образцах.
[00196] Пример 2: Превращение 3-кето-ДОН in vitro
[00197] Определяли способность редуктаз, полученных в соответствии с примером 1, использовать 3-кето-ДОН в качестве субстрата, и ферменты-редуктазы с SEQ ID NO. 15 и 16 использовали в качестве примеров, известных из уровня техники. Все ферменты получали в соответствии с примером 1 и использовали в анализе активности, описанном ниже.
[00198] Следующий анализ ферментативной активности должен быть определен как эталонный способ определения активности, в частности специфической активности ферментов, заявленных в настоящем изобретении. Активность ферментов, восстанавливающих 3-кето-ДОН, исследовали с помощью стандартной системы для анализа разложения 3-кето-ДОН, состоящей из: 100 мМ буфера на основе триса с рН 8,0, содержащего 0,5 мМ НАДФН, 0,5 мМ НАДН, 25 м.д. 3-кето-ДОН и 100 нМ соответствующего фермента. Все компоненты реакции, за исключением НАДН и НАДФН, смешивали на льду и отбирали образец в момент времени 0 минут (смешивали образец 1:1 (об./об.) со 100% метанолом для остановки всех реакций). После этого реакционные смеси незамедлительно переносили в термостат и запускали реакцию путем добавления смеси кофакторов (то есть НАДН и НАДФН), и оставляли для продолжения реакции на 2 часа при 30°С. С целью контроля протекания реакции образцы отбирали с регулярными интервалами и незамедлительно смешивали 1:1 (об./об.) со 100% метанолом для остановки реакции. Затем образцы подвергали анализу с использованием метода ВЭЖХ-МС/МС, описанного ниже.
[00199] Для анализа содержания ДОН, 3-кето-ДОН и 3-эпи-ДОН посредством ВЭЖХ использовали разделительную колонку Phenomenex Gemini-NX С18 150 мм × 4,6 мм с размером частиц 5 мкм. Смесь ацетонитрила и ультрачистой воды использовали в качестве подвижной фазы, и для определения концентраций анализируемых веществ регистрировали и оценивали сигнал УФ детектора при длине волны 220 нм. Для анализа посредством ЖХ-МС/МС, ДОН, 3-кето-ДОН и 3-эпи-ДОН разделяли на колонке Phenomenex Kinetex Biphenyl 150 мм × 2,1 мм с размером частиц 2,6 мкм. Подвижная фаза состояла из смеси метанола и ультрачистой воды с 0,1% (об./об.) уксусной кислоты. Ионы генерировали с использованием ионизации электрораспылением (ИЭР) в режиме регистрации отрицательно заряженных ионов. Количественное определение проводили с использованием системы QTrap и/или тройного квадрупольного масс-спектрометрического детектора. Образцы требовали разведения для попадания в линейный диапазон этого метода, который варьирует от концентрации 1 млрд-1 анализируемых веществ до концентрации 500 млрд-1 во введенном образце. Предел количественного определения (ПКО) представляет собой предел надежного обнаружения, и концентрации ниже 1 млрд-1 соединения, обнаруженные в образце, не считаются надежными, таким образом ПКО составляет 1 млрд-1. В случае, когда концентрация анализируемого вещества ниже ПКО, все дальнейшие расчеты (например, для определения удельной активности или соотношения анализируемых веществ) выполняют для значения 1 млрд1.
[00200] Перед анализом посредством ВЭЖХ-МС/МС все образцы разбавляли 40% раствором метанола в воде до теоретической конечной концентрации 3-кето-ДОН в образце, составляющей 200 млрд-1.
[00201] Результаты использовали для расчета удельной активности (мкмольпродукта/мин/мгфермента) ферментов на основе выработки 3-эпи-ДОН (или ДОН) в линейном диапазоне реакции в мкмольпродукта/мин/мгфермента. Расчет проводили путем перевода абсолютного количества 3-эпи-ДОН (или ДОН), полученного в ходе реакции до выбранного момента времени (то есть 10 минут для 3-эпи-ДОН и 120 минут для ДОН), из м.д. в мкмоль (то есть концентрации 3-эпи-ДОН в м.д. (=мкг/мл) умножали на объем реакционной смеси в мл и делили на молекулярную массу 3-эпи-ДОН в мкг/мкмоль с получением количества мкмоль 3-эпи-ДОН в реакционной смеси в выбранный момент времени). Затем результат делили на выбранный промежуток времени в мин. Полученное значение далее делили на общее количество фермента в реакции в мг для расчета удельной активности (мкмольпродукта/мин/мгфермента), которая соответствует числу единиц активности на мг фермента (Ед/мг). Результаты анализов активности показаны на фигуре 2 для превращения 3-кето-ДОН в 3-эпи-ДОН и на фигуре 3 для превращения 3-кето-ДОН в ДОН.
[00202] Пример 3: Анализ последовательностей для консервативных доменов и уникальных структурных признаков
[00203] Ферменты, имеющие последовательности SEQ ID NO. 1-14 и 63-101, и известные из уровня техники ферменты (SEQ ID NO. 15 и 16) относятся к оксидоредуктазам и часто являются предполагаемыми представителями альдокеторедуктаз, суперсемейства НАД(Ф)(Н)-зависимых оксидоредуктаз, участвующих в выработке первичных и вторичных спиртов посредством катализа восстановления альдегидов и кетонов, соответственно. Альдокеторедуктазы характеризуются общей структурой (α/β)8, консервативным каталитическим механизмом с участием каталитической тетрады, состоящей из остатков тирозина (Туг, Y), аспартата (Asp, D), гистидина (His, Н) и лизина (Lys, K). В ферментах, имеющих SEQ ID NO. 15 и 16, оба из которых известны из уровня техники, каталитическая тетрада состоит из Asp48, Туг 53, Lys81 и His122. В редуктазе, имеющей последовательность SEQ ID NO. 1, каталитическая тетрада соответствует положениям Asp53, Tyr58, Lys85 и His126. Однако указанные ферменты значительно отличаются друг от друга по меньшей мере в областях петель А и В, которые экспонированы на задней части структуры бочонка. Эти петли определяют субстратную специфичность вместе с вариациями в субстратсвязывающем кармане (Jez and Penning (1998)).
[00204] Суперсемейство характеризуется признаком cd06660 в справочной базе данных «Национального центра биотехнологической информации» (NCBI; http://www.ncbi.nlm.nih.gov/). Для дальнейшего анализа доменов последовательностей был выполнен пакетный поиск в базе данных консервативных доменов «CDD» (NCBI; https://www.ncbi.nlm.nih.gov/cdd/) с конкретной заявкой на множественные последовательности https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi. Поиск выполняли со стандартными параметрами: автоматический режим поиска по базе данных CDD-52910 PSSMs, ожидаемое пороговое значение 0,01 и использование функции «Composition-corrected scoring». PSSM= позиционная матрица замен (position-specific scoring matrix), которая рассчитывается для степени консенсусной последовательности. Профили PSSM описывают, насколько часто определенные остатки обнаруживаются в заданном положении при множественном выравнивании последовательностей для модели домена. Все анализируемые последовательности относятся к суперсемейству cl30127 (Marchler-Bauer et al. (2011), Marchler-Bauer and Bryant (2004), Marchler-Bauer et al. (2017).
[00205] Альдокеторедуктазы различаются по петлевым структурам, которые определяют субстратную специфичность. Таким образом, петли А и В определены как петли А и В, определяющие субстратную специфичность. Петли А и В, определяющие субстратную специфичность в ферментах, имеющих последовательности SEQ ID NO. 1-14 и 63-101, значительно отличаются от петель А и В в известных из уровня техники ферментах, в частности имеющих последовательности SEQ ID NO. 15 и 16. Фигура 4. Во всех случаях петля А состоит из 9 остатков и содержит гистидин каталитической тетрады, но вместо неполярного аланина (положение 123 в известных из уровня техники ферментах), расположенного после гистидина 122 в известных из уровня техники ферментах, соответствующий остаток в SEQ ID NO. 1-14 и 63-101, непосредственно следующий за каталитическим гистидином, не является аланином. Петля А, определяющая субстратную специфичность, охватывает последовательность, расположенную после общего мотива I-D/E-LYQ и до мотива l/M/L-E/D-E/D-T-L/М. Петля В, определяющая субстратную специфичность, начинается с остатка серина в общем мотиве WSPL-X-G и заканчивается до общего мотива X-X-R/K/H-T/L-W/Y-X-I/V/L-I/V/L. Петля В значительно различается по длине и содержит 35 или меньшее количество остатков.
[00206] Вариабельность в петлях А и В с точки зрения остатков и длины способствует почти 100% стереоселективности в отношении 3-эпи-ДОН и повышенной удельной активности ферментов, имеющих SEQ ID NO. 1-14 и 63-101, по сравнению с ферментами, имеющими SEQ ID NO. 15-16.
[00207] Фигура 5. Петли А и В, определяющие субстратную специфичность. Начальные остатки, концевые остатки и длина аминокислотной последовательности. Эти данные также обобщенно представлены в приведенной в настоящем документе таблице последовательностей.
[00208] Пример 4: Определение оптимального рН
[00209] Для определения рН, оптимального для катализа с участием полипептида, как указано в настоящем документе, готовили реакционные смеси для превращения 3-кето-ДОН при различных значениях рН. С этой целью готовили 16 реакционных смесей с различными значениями рН, при этом конечный объем каждой смеси составлял 200 мкп.Полипептид, имеющий аминокислотную последовательность SEQ ID NO: 1, использовали в конечной концентрации 200 нМ. В качестве субстрата использовали 3-кето-ДОН в конечной концентрации 30 м.д., и НАДФН использовали в конечной концентрации 1 мМ. В качестве буфера использовали буфер Teorell Stenhagen (TS) (Stenhagen & Teorell. 1938. Nature 141, 415) и доводили рН до значений 2,5, 3,0, 3,5, 4,0, 4,5, 5,0, 5,5, 6,0, 6,5, 7,0, 7,5, 8,0, 8,5, 9,0, 9,5, 10,0. Реакции превращения инкубировали при 30°С в течение 120 мин и запускали путем добавления НАДФН. На протяжении всей инкубации отбирали 20 мкл образцов в моменты времени 0 мин, 10 мин, 20 мин, 30 мин, 60 мин и 120 мин. Образец, соответствующий моменту времени 0 мин, отбирали перед добавлением НАДФН. 20 мкл образцов незамедлительно смешивали с 20 мкл чистого метанола для остановки реакции превращения и затем держали на льду до конца инкубации.
[00210] Перед анализом посредством ЖХ-МС/МС все образцы разбавляли 40% раствором метанола в воде до теоретической конечной концентрации 3-кето-ДОН 0,3 м.д. Для анализа посредством ЖХ-МС/МС, ДОН, 3-кето-ДОН и 3-эпи-ДОН разделяли на колонке Phenomenex Kinetex Biphenyl 150 мм × 2,1 мм с размером частиц 2,6 мкм. Подвижная фаза состояла из смеси метанола и ультрачистой воды с 0,1% (об./об.) уксусной кислоты. Ионы генерировали с использованием ионизации электрораспылением (ИЭР) в режиме регистрации отрицательно заряженных ионов. Количественное определение проводили с использованием системы QTrap и/или тройного квадрупольного масс-спектрометрического детектора. При необходимости образцы разбавляли для обеспечения попадания в линейный диапазон этого метода, который варьирует от концентрации 1 млрд-1 анализируемых веществ до концентрации 500 млрд-1 во введенном образце. Предел количественного определения (ПКО) представлял собой предел надежного обнаружения, и концентрации ниже 1 млрд-1 соединения, обнаруженного в образце, не считались надежными, таким образом ПКО составлял 1 млрд-1. В случае, когда концентрация анализируемого вещества была ниже ПКО, все дальнейшие расчеты (например, для определения удельной активности или соотношения анализируемых веществ) выполняли для значения 1 млрд1. Кинетические параметры рассчитывали на основе количеств 3-эпи-ДОН и/или ДОН, определенных в образцах в отдельные моменты времени. Значения удельной активности рассчитывали по максимальной линейной скорости образования продукта в каждой реакции. Значения удельной активности для образования 3-эпи-ДОН при различных значениях рН показаны в таблице 3 ниже.
[00211] 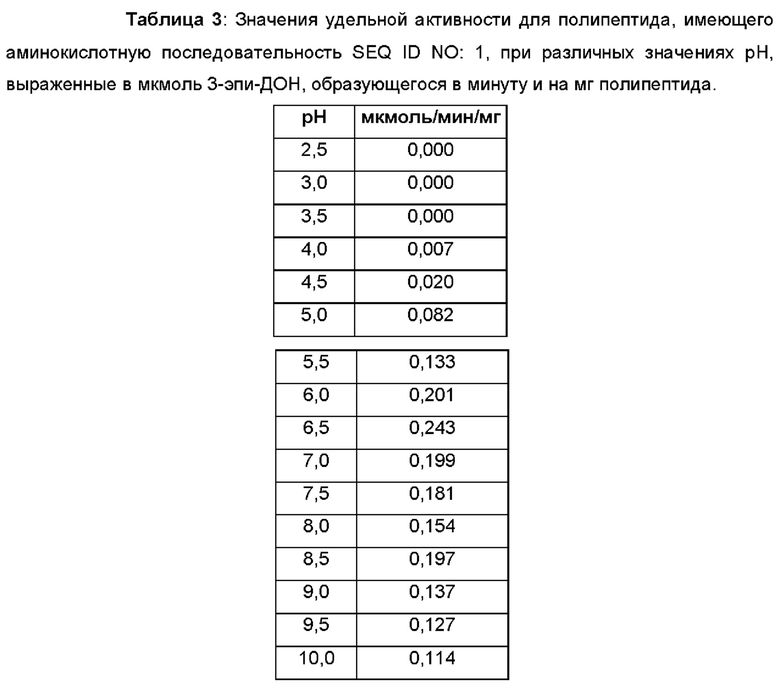
[00212] Было обнаружено, что полипептид обладал активностью в диапазоне рН от рН 4,5 до рН 10, при этом оптимальный для образования 3-эпи-ДОН рН составлял 6,5.
[00213] Пример 5: Определение оптимальной температуры
[00214] Для определения температуры, оптимальной для катализа с участием полипептида, как указано в настоящем документе, готовили реакционные смеси для превращения 3-кето-ДОН при различных температурах. С этой целью готовили 24 реакционные смеси, при этом конечный объем каждой смеси составлял 180 мкл. Полипептид, имеющий аминокислотную последовательность SEQ ID NO: 1, использовали в конечной концентрации 200 нМ. В качестве субстрата использовали 3-кето-ДОН в конечной концентрации 30 м.д., и НАДФН использовали в конечной концентрации 1 мМ. В качестве буфера использовали буфер TS при рН 7,0. Реакции превращения инкубировали в лабораторном ПЦР-термоциклере при различных температурах в течение 120 мин и запускали путем добавления НАДФН. На протяжении всей инкубации отбирали 20 мкл образцов в моменты времени 0 мин, 5 мин, 10 мин, 20 мин, 30 мин, 60 мин и 120 мин. Образец, соответствующий моменту времени 0 мин, отбирали из реакционных смесей перед добавлением НАДФН. 20 мкл образцов незамедлительно смешивали со 180 мкл чистого метанола для остановки реакции превращения и затем держали на льду до конца инкубации. Анализы проводили посредством ЖХ-МС/МС, как описано в примере 4. Значения удельной активности для образования 3-эпи-ДОН при различных температурах показаны в таблице 4 ниже.
[00215] 
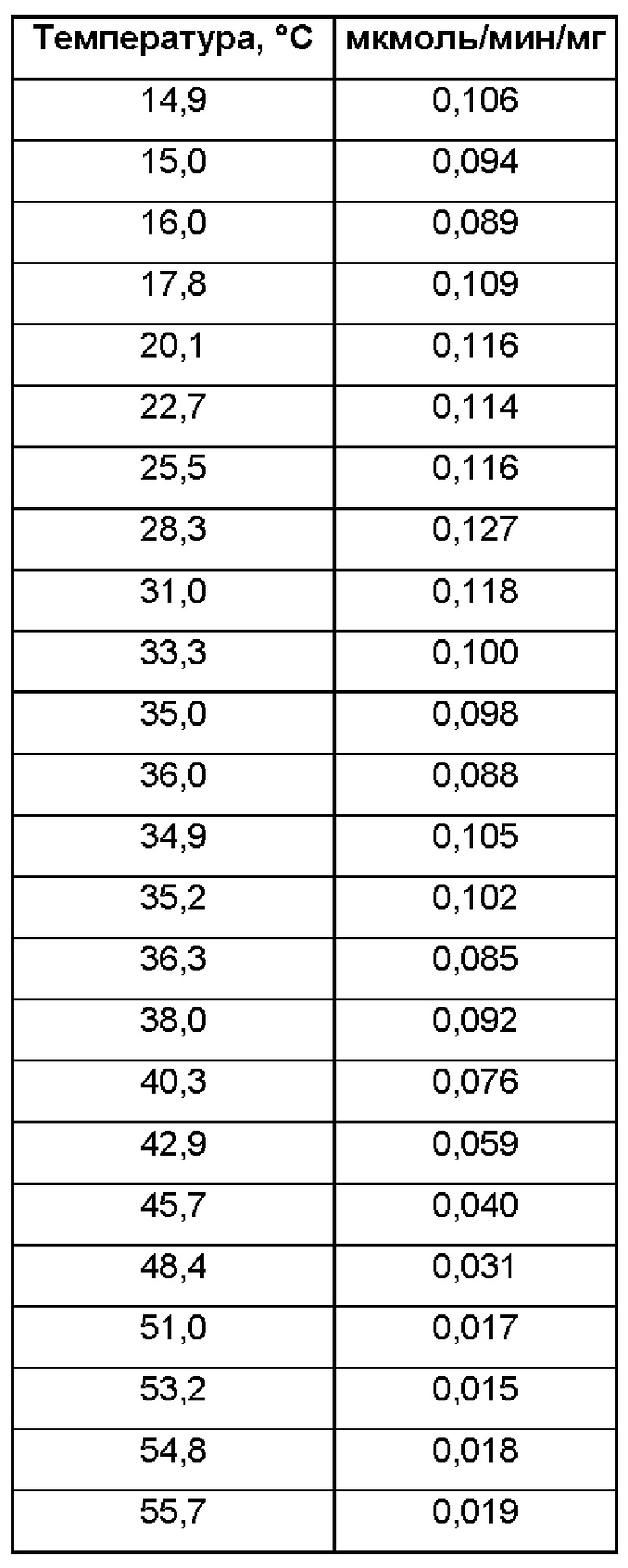
[00216] Наибольшее количество 3-эпи-ДОН образовалось под действием полипептида при температурах от 14,9°С до 36°С, при этом оптимальная температура составляла от 25°С до 31°С.
БИБЛИОГРАФИЧЕСКИЙ СПИСОК
Altschul, J. Mol. Evol. 36 (1993), 290-300.
Altschul, J. Mol. Biol. 215 (1990), 403-410.
Altschul et al., 1997 (Nucleic Acids Res. (1997) 25:3389-3402.
Carere et al. 2018 (Carere (2018) "The identification of DepB: An enzyme responsible for the final detroxification Step in the deoxynivalenol epimerization pathway in Devosia mutans 17-2-E-8." Frontiers in Microbiology. 9:1573.
Cregg, Pichia Protocols, second Edition, ISBN-10: 1588294293, 2007.
Ellis (2002) "Microbial aldo-keto reductases" FEMS Microbiol Lett. 5;216(2):123-31.
Hassan et al. (2017) "The enzymatic epimerization of deoxynivalenol by Devosia mutans proceeds through the formation of 3-/cefo-DON intermediate." Scientific Reports 7, article number 6929.
He et al. (2015) "Toxicology of 3-epi-deoxynivalenol, a deoxynivalenol-transformation product by Devosia mutans 17-2-E-8." Food and Chemical Toxicology 84: 250-259.
Henikoff Proc. Natl. Acad. Sci., USA, 89, (1989), 10915.
Henikoff and Henikoff (1992) 'Amino acid substitution matrices from protein blocks.' Proc Natl Acad Sci USA. 1992 Nov 15;89(22):10915-9.
Jez and Penning (1998) "Engineering steroid 5 beta-reductase activity into rat liver 3 alpha-hydroxysteroid dehydrogenase" Biochemistry.37(27):9695-703.
Kundert K, Kortemme T. (2019) "Computational design of structured loops for new protein functions." Biol Chem. 2019 Feb 25;400(3):275-288.
Kurtzman (2009) "Biotechnological strains of Komagataella (Pichia) pastoris are Komagataella phaffii as determined from multigene sequence analysis." J Ind Microbiol Biotechnol. 36(11): 1435-8.
Marchler-Bauer et al. (2011) "CDD: a Conserved Domain Database for the functional annotation of proteins." Nucleic Acids Res.39(D)225-9.
Marchler-Bauer and Bryant (2004), "CD-Search: protein domain annotations on the fly.", Nucleic Acids Res.32(W)327-331.
Marchler-Bauer et al. (2017), "CDD/SPARCLE: functional classification of proteins via subfamily domain architectures.", Nucleic Acids Res.45(D)200-3.
Needleman and Wunsch, 1970 (Journal of Molecular Biology. 1970 Mar;48:443-453.
Payros et al., (2016) "Toxicology of deoxynivalenol and its acetylated and modified forms." Archives of Toxicology 90(12): 2931-2957).
Penning (2015) "The aldo-keto reductases (AKRs): Overview" Chem Biol Interact. 234:236-46.
Pierron et al., (2016) "Microbial biotransformation of DON: molecular basis for reduced toxicity." Scientific Reports 6(1)).
POST-TRANSLATIONAL COVALENT MODIFICATION OF PROTEINS, В.C. Johnson, Ed., Academic Press, New York (1983), p. 1-12.
PROTEINS - STRUCTURE AND MOLECULAR PROPERTIES, 2nd Ed., Т.E. Creighton, W. H. Freeman and Company, New York (1993).
Rattan, Ann. NY Acad. Sci. 663 (1992); 48-62.
Sambrooket al. 2012, Molecular Cloning, A Laboratory Manual 4th Edition, Cold Spring Harbor.
Schatzmayr and Streit (2013) 'Global occurrence of mycotoxins in the food and feed chain: Facts and figures.'World Mycotoxin Journal 6(3):213-222.
Seifter, Meth. Enzymol. 182 (1990); 626-646.
Thompson Nucl. Acids Res. 2 (1994), 4673-4680) or FASTDВ (Brutlag Сотр. App.Biosci. 6 (1990), 237-245.
WO2019/046954.
Yamada et al. (1995) 'The Phylogenetic Relationships of Methanol-assimilating Yeasts Based on the Partial Sequences of 18S and 26S Ribosomal RNAs: The Proposal of Komagataella Gen. Nov. (Saccharomycetaceae)' Bioscience, Biotechnology and Biochemistry, Vol.59, issue 3, pp.439-444.
Zdarta et al. (2018) "A general overview of support materials for enzyme immobilization: characteristics, properties, practical utility" Catalysts 8, 92, p.1-27.
Zhu Y., Hassan Y. I., Lepp D., Shao S., Zhou T. (2017) "Strategies and Methodologies for Developing Microbial Detoxification Systems to Mitigate Mycotoxins." Toxins. 9 (4): 130.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Erber Aktiengesellschaft
<120> Улучшенные полипептиды, способные превращать субстрат
3-кетодезоксиниваленол в 3-эпидезоксиниваленол
<130> ERB16790PCT
<150> EP 19194632
<151> 2019-08-30
<160> 101
<170> PatentIn version 3.5
<210> 1
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 1
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Glu Leu Lys Leu
290 295 300
Thr Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 2
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 2
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ser Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Pro Gln Ala Glu Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Arg Met Gly Asp Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Val Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys Asn His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Gly Lys Leu Tyr Asp Ile Val Glu Val Ile Val
245 250 255
Ala Ile Ala Glu Ala Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Val Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Lys Asp Asn Leu Ala Ala Ala Asp Leu Lys Leu
290 295 300
Thr Pro Glu Glu Arg Gln Lys Leu Asp Ala Val Ser Gln Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Arg Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 3
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 3
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Pro Gln Ala Glu Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asp Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Asp Ile Val Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Val Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Lys Asp Asn Leu Ala Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Gln Lys Leu Asp Ala Val Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Thr Phe Thr Ala Asn Asp Arg Leu Gly
325 330 335
Pro Ala Asp Gln Asp Leu Leu Ser Thr Tyr Val Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 4
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 4
Met Glu Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly Ala Thr
20 25 30
Ala Gln Asp Asp Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Lys Met Gly Asp Gly Pro Asn Glu
85 90 95
Ile Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Ile Lys Gln Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Gln Ala Ala Asp Lys Asn His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Ile Pro Ile Thr Gly Asp Gln Gly Leu Gly Val Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asn Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Glu Lys Leu Tyr Asp Ile Val Asp Val Ile Val
245 250 255
Asp Ile Ala Asp Asp Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ser Leu Gly Arg Pro Gly Val Thr Ser Val Ile Ile Gly Gly Arg
275 280 285
Asn Asp Ala Gln Phe Lys Asp Asn Leu Ala Ala Ala Glu Leu Lys Leu
290 295 300
Thr Ala Glu Glu Arg Ala Arg Leu Asp Ala Val Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Thr Phe Thr Ala Gln Asp Arg Leu Gly
325 330 335
Pro Val Asp Arg Asn Leu Ile Glu Pro Tyr Ala Glu Glu Leu Asn Arg
340 345 350
Gly
<210> 5
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 5
Met Gln Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Gly Glu Lys Ile Gly Gly Thr
20 25 30
Gly Gln Asp Glu Ala Asn Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Lys Met Gly Glu Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Ser Arg His Gln Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Thr Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asn Arg Leu Tyr Asp Ile Val Asp Ala Leu Val
245 250 255
Ala Ile Ala Glu Ala Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Leu Leu Gly Arg Pro Gly Val Thr Ser Val Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Asp Leu Gln Leu
290 295 300
Thr Ala Asp Glu Arg Gln Arg Leu Asp Ala Val Ser Gln Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Thr Phe Thr Ala Asn Asp Arg Leu Gly
325 330 335
Pro Ala Asp Arg Asp Leu Leu Thr Pro Tyr Val Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 6
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 6
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Asp Leu Ala Gly Ala Ala Arg Gln Val Asp Leu Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Tyr Asp Ser Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Leu Gly Gln Val Leu Ala Glu Asn Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Ala Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Ala Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Thr Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Leu Ile Asp Val Ile Val
245 250 255
Asp Ile Ala Ala Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ser
260 265 270
Trp Leu Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Asn Glu Ala Gln Phe Ala Asp Asn Ile Ala Ala Ile His Leu Lys Leu
290 295 300
Ser Ser Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Arg Ile Asp Leu Asp Leu Ile Glu Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 7
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 7
Met Glu Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Ile Gly Asn Thr
20 25 30
Ser Pro Ala Asp Ala Ala Arg Gln Val Asp Met Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Tyr Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Ile Leu Gly Ala Ile Leu Ala Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Lys Met Gly Asp Gly Pro Asn Glu
85 90 95
Ala Gly Leu Ser Arg His His Ile Ile Glu Gln Cys Lys Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Arg Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg His Gly His Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ser Ala Asp Gln Arg His Asn Ala
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Ser Gly Gly Pro Glu Thr Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Gln Glu Lys Leu Tyr Asp Val Ile Glu Glu Ile Val
245 250 255
Ala Ile Ala Glu Asp Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Leu Leu Gly Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Asn Glu Lys Gln Phe Ala Asp Asn Ile Ala Ala Ala Asp Phe Lys Leu
290 295 300
Ser Ala Glu Glu Arg Ala Arg Leu Asp Lys Val Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Ser Ser Gln Asp Arg Leu Ser
325 330 335
Pro Val Asp Met Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 8
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 8
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ser Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Ile Gly Asn Thr
20 25 30
Ser Arg Gln Asp Ala Ala Arg Gln Val Asp Met Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Tyr Asp Ser Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Ile Leu Gly Asp Ile Leu Ala Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Lys Met Gly Asp Gly Pro Asn Glu
85 90 95
Ala Gly Leu Ser Arg His His Ile Ile Glu Gln Cys Lys Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Gln Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Ile Leu
130 135 140
Val Arg His Gly His Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Ser Lys Ala Leu Ala Ala Ala Asp Gln Arg His Asn Ala
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Ser Ala Pro Asp Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Ser Val Ile Asp Val Leu Val
245 250 255
Ala Val Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ser
260 265 270
Trp Leu Met Gly Arg Pro Gly Val Thr Ser Val Ile Ile Gly Gly Arg
275 280 285
Asn Glu Lys Gln Phe Ala Asp Asn Ile Ala Ala Ala Gln Leu Val Leu
290 295 300
Ser Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Gly
325 330 335
Lys Ile Asp Met Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 9
<211> 350
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 9
Met Glu Tyr Arg Leu Leu Gly Arg Ser Gly Leu Lys Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Asn Pro Lys Val Gly Asn Thr
20 25 30
Ser Leu Lys Asp Ala Gln Arg Gln Ile Asp Met Cys Leu Glu Ala Gly
35 40 45
Ile Asn Met Leu Asp Thr Ala Asn Val Tyr Thr Arg Gly Val Ser Glu
50 55 60
Ser Ile Ile Gly Glu Ala Leu Thr Asp Gly Arg Arg Gln Arg Thr Leu
65 70 75 80
Leu Ala Thr Lys Val Arg Phe Pro Met Asp Asp Thr Asp Pro Asn Glu
85 90 95
Arg Gly Leu Ser Arg Tyr His Ile Val Asn Gln Cys Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Gln Thr Pro Leu Glu Glu Thr Met Glu Ala Leu Asp Met Leu
130 135 140
Val Arg His Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Gly Val Ser Glu Lys Asp Ala Arg Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Thr Leu Phe Ser Arg Glu Ala
180 185 190
Glu Asn Glu Leu Val Pro Ile Ser Leu Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Gly Gln Thr Ala Pro Glu Gly Ala Arg His Ala Gly Gly Phe Lys Glu
225 230 235 240
Pro Pro Val His Asp Trp Glu Arg Leu Tyr Asp Ile Val Glu Val Leu
245 250 255
Val Ser Ile Ala Glu Val Arg Gly Val Ser Gly Ala Gln Ile Ala Leu
260 265 270
Ala Trp Leu Leu Gly Arg Pro Gly Val Thr Ser Val Ile Ile Gly Gly
275 280 285
Arg Thr Glu Asp Gln Phe Arg Asp Asn Leu Ala Ala Ala Glu Leu Lys
290 295 300
Leu Thr Glu His Glu Arg Lys Arg Leu Asp Asp Val Ser Leu Leu Pro
305 310 315 320
Leu Gln Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Ser Asp Arg Leu
325 330 335
Gly Pro Ala Asp Leu Ala Leu His Ala Pro His Leu Glu Lys
340 345 350
<210> 10
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 10
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Val Leu
1 5 10 15
Ser Val Gly Thr Val Thr Phe Gly Gly Asp Gly Leu Trp Gly Asp Thr
20 25 30
Asp Val Thr Gly Ala Ala Arg Gln Ile Asp Met Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Ile Asp Thr Ala Asn Val Tyr Asn Lys Gly Val Ser Glu
50 55 60
Glu Ile Thr Gly Glu Val Leu Arg Asp Gly Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Pro Phe Gly Asp Gly Pro Asn Asp
85 90 95
Arg Gly Leu Ser Arg His His Ile Ile Ala Gln Cys Glu Gly Ser Leu
100 105 110
Lys Arg Leu Gln Thr Asp Val Ile Asp Leu Tyr Gln Val His Gln Trp
115 120 125
Asp Gly Gln Thr Pro Leu Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Leu Met Lys Ala Leu Ala Val Ala Asp Ser Arg Gly Tyr Gln
165 170 175
Arg Phe Val Ala Gln Gln Ile His Tyr Thr Leu Tyr Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Gln Pro Ile Thr Val Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Trp Leu Ser Gly Lys Tyr Thr Arg
210 215 220
Ala Ser Gln Pro Glu Asp Gly Arg His Val Arg Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Pro Glu Arg Leu Tyr Asp Ile Val Asp Val Ile Asn
245 250 255
Ala Val Ala Ala Glu Arg Gly Val Ser Gly Ala Val Val Ser Leu Ala
260 265 270
Trp Ala Leu Gln Arg Lys Gly Val Ser Ser Val Ile Val Gly Ala Arg
275 280 285
Thr Asp Ala Gln Phe Ala Asp Asn Leu Lys Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Asp Glu Val Ala Arg Leu Asp Ala Val Ser Gln Arg Pro Leu
305 310 315 320
Pro Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Glu Ala Asp Leu Val Leu Leu Ala Pro Tyr Leu Glu Glu Tyr Lys Arg
340 345 350
Gly
<210> 11
<211> 348
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 11
Met Asp Tyr Arg Leu Leu Gly Arg Ser Gly Leu Lys Val Ser Ser Leu
1 5 10 15
Ser Ile Gly Thr Ala Thr Phe Gly Gly Asp Gly Leu Trp Gly Asp Thr
20 25 30
Asn Val Lys Glu Ala Gln Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Val Asn Leu Ile Asp Thr Ala Asn Val Tyr Ser Gly Ser Val Ser Glu
50 55 60
Thr Ile Ile Gly Glu Ala Leu Thr Asp Gly Arg Arg Asp Arg Leu Leu
65 70 75 80
Ile Ala Thr Lys Val Arg Phe Pro Arg Gly Lys Gly Pro Asn Asp Arg
85 90 95
Gly Leu Ser Arg Trp His Ile Ile Arg Glu Ala Glu Ala Ser Leu Lys
100 105 110
Arg Met Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp Asp
115 120 125
Gly Gln Thr Pro Leu Glu Glu Thr Leu Glu Ala Leu Asp Thr Leu Val
130 135 140
Arg Ser Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Ala Trp
145 150 155 160
His Leu Met Lys Ala Leu Ala Val Ser Asn Glu Arg Asn Tyr Gln Arg
165 170 175
Phe Val Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala Glu
180 185 190
Phe Glu Leu Ile Pro Ala Gly Leu Asp Gln Gly Val Ser Val Leu Val
195 200 205
Trp Ser Pro Leu Ala Gly Gly Trp Leu Ser Gly Lys Tyr Thr Arg Asp
210 215 220
Ser Lys Pro Ser Asp Gly Arg Gln Val Arg Gly Phe Lys Glu Pro Pro
225 230 235 240
Ile Tyr Asp Pro Glu His Leu Trp Lys Ile Val Asp Val Val Asn Asp
245 250 255
Val Ala Gly Ala Arg Gly Val Ser Gly Ala Gln Val Ser Leu Ala Trp
260 265 270
Leu Leu Ser Lys Pro Ile Val Ala Ser Val Ile Val Gly Gly Arg Asn
275 280 285
Ile Gly Gln Phe Glu Asp Asn Leu Lys Ala Ala Glu Leu Thr Leu Thr
290 295 300
Thr Glu Glu Leu Gln Lys Leu Asp Ala Val Ser Gln Pro Asn Leu Pro
305 310 315 320
Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Gly Glu
325 330 335
Pro Asp Leu Ala Leu His Arg Pro Tyr Leu Asp Lys
340 345
<210> 12
<211> 348
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 12
Met Asp Tyr Arg Leu Leu Gly Arg Ser Gly Leu Lys Val Ser Ser Leu
1 5 10 15
Ser Ile Gly Thr Ala Thr Phe Gly Gly Asp Gly Leu Trp Gly Asp Thr
20 25 30
Asn Val Lys Glu Ala Gln Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Val Asn Leu Ile Asp Thr Ala Asn Val Tyr Ser Gly Ser Val Ser Glu
50 55 60
Thr Ile Ile Gly Glu Ala Leu Thr Asp Gly Arg Arg Asp Arg Leu Leu
65 70 75 80
Ile Ala Thr Lys Val Arg Phe Pro Arg Gly Lys Gly Pro Asn Asp Arg
85 90 95
Gly Leu Ser Arg Trp His Ile Ile Arg Glu Ala Glu Ala Ser Leu Lys
100 105 110
Arg Met Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp Asp
115 120 125
Gly Gln Thr Pro Leu Glu Glu Thr Leu Glu Ala Leu Asp Thr Leu Val
130 135 140
Arg Ser Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Ala Trp
145 150 155 160
His Leu Met Lys Ala Leu Ala Thr Ser Asn Glu Arg Asn Tyr Gln Arg
165 170 175
Phe Val Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala Glu
180 185 190
Phe Glu Leu Ile Pro Ala Gly Leu Asp Gln Gly Val Ser Val Leu Val
195 200 205
Trp Ser Pro Leu Ala Gly Gly Trp Leu Ser Gly Lys Tyr Thr Arg Asn
210 215 220
Ser Lys Pro Ser Asp Gly Arg Gln Val Arg Gly Phe Lys Glu Pro Pro
225 230 235 240
Ile Tyr Asp Pro Glu His Leu Trp Lys Ile Val Asp Val Val Asn Glu
245 250 255
Val Ala Ser Ala His Gly Val Ser Gly Ala Gln Val Ser Leu Ala Trp
260 265 270
Leu Leu Ser Lys Pro Ile Val Ala Ser Val Ile Val Gly Gly Arg Asn
275 280 285
Ile Glu Gln Phe Glu Asp Asn Leu Lys Ala Ala Glu Leu Gln Leu Thr
290 295 300
Ala Glu Glu Leu Gly Arg Leu Asp Ala Val Ser Gln Pro Asn Leu Pro
305 310 315 320
Tyr Pro Tyr Trp His Gln Asn Phe Thr Ala Gln Asp Arg Leu Ser Glu
325 330 335
Pro Asp Leu Ala Leu His Arg Pro Tyr Leu Asp Lys
340 345
<210> 13
<211> 351
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 13
Met Glu Tyr Arg Leu Leu Gly Arg Ser Gly Leu Lys Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Gln Gly Lys Phe Ala Ser Val
20 25 30
Gly Asn Ser Gly Val Asp Glu Ala Arg Arg Leu Ile Asp Met Cys Ile
35 40 45
Asp Ala Gly Val Asn Leu Ile Asp Thr Ala Asn Val Tyr Ser Asp Gly
50 55 60
Arg Ser Glu Glu Ile Ile Gly Glu Val Leu Gly Gly Lys Arg Pro Lys
65 70 75 80
Asp Val Leu Ile Ala Thr Lys Ala Arg Phe Pro Met Gly Asp Gly Pro
85 90 95
Asn Asn Gly Gly Leu Ser Arg Tyr His Leu Ile Arg Glu Cys Glu Ala
100 105 110
Ser Leu Lys Arg Leu Arg Thr Asp Val Ile Asp Leu Tyr Gln Val His
115 120 125
Gln Trp Asp Gly Leu Thr Pro Leu Glu Glu Thr Leu Glu Ala Leu Asp
130 135 140
Thr Leu Val Arg Asp Gly Lys Val Arg Tyr Leu Gly Cys Ser Asn Tyr
145 150 155 160
Ser Ala Trp His Leu Met Lys Ala Leu Gly Ile Ser Glu Arg Glu His
165 170 175
Arg Gln Arg Phe Val Ser Gln Gln Ile His Tyr Thr Leu Glu Ala Arg
180 185 190
Glu Ala Glu Tyr Glu Leu Leu Pro Ile Ser Ile Asp Gln Gly Leu Gly
195 200 205
Val Leu Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys His
210 215 220
Arg Arg Asp Gly Lys Ala Pro Glu Gly Ser Arg Gln Leu Glu Gly Trp
225 230 235 240
Thr Glu Pro Pro Ile Arg Asp Glu Glu Arg Leu Trp Arg Ile Val Asp
245 250 255
Val Leu Val Ala Ile Ala Glu Ala Arg Gly Val Ser Gly Ala Gln Val
260 265 270
Ala Leu Ala Trp Leu Leu Glu Arg Pro Ala Val Thr Ser Leu Val Ile
275 280 285
Gly Gly Arg Thr Glu Ala Gln Phe Lys Asp Asn Leu Ala Ala Ala Glu
290 295 300
Leu Lys Leu Thr Ala Glu Glu Arg Gln Arg Leu Asp Glu Val Ser Ala
305 310 315 320
Pro Pro Val Leu Tyr Pro Tyr Trp His Gln Leu Met Thr Ala Ser Asp
325 330 335
Arg Leu Gly Pro Ala Asp Met Pro Leu Leu Ala Pro His Leu Lys
340 345 350
<210> 14
<211> 351
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 14
Met Glu Tyr Arg Leu Leu Gly Arg Ser Gly Leu Lys Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Gln Gly Lys Phe Ala Ser Val
20 25 30
Gly Ser Ser Gly Val Asp Glu Ala Arg Arg Leu Ile Asp Met Cys Ile
35 40 45
Asp Ala Gly Val Asn Leu Ile Asp Thr Ala Asn Val Tyr Ser Asp Gly
50 55 60
Arg Ser Glu Glu Ile Ile Gly Glu Val Leu Gly Gly Lys Arg Pro Lys
65 70 75 80
Asp Val Leu Ile Ala Thr Lys Ala Arg Phe Pro Met Gly Asp Gly Pro
85 90 95
Asn His Gly Gly Leu Ser Arg Tyr His Leu Ile Arg Glu Cys Glu Ala
100 105 110
Ser Leu Lys Arg Leu Arg Thr Asp Val Ile Asp Leu Tyr Gln Val His
115 120 125
Gln Trp Asp Gly Leu Thr Pro Leu Glu Glu Thr Leu Glu Ala Leu Asp
130 135 140
Thr Leu Val Arg Asp Gly Lys Val Arg Tyr Leu Gly Cys Ser Asn Tyr
145 150 155 160
Ser Ala Trp His Leu Met Lys Ala Leu Gly Val Ser Glu Arg Glu His
165 170 175
Arg Gln Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu Glu Ala Arg
180 185 190
Glu Ala Glu Tyr Glu Leu Leu Pro Ile Ser Ile Asp Gln Gly Leu Gly
195 200 205
Val Leu Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys His
210 215 220
Arg Arg Asp Thr Lys Ala Pro Glu Gly Ser Arg Gln Leu Ala Gly Trp
225 230 235 240
Thr Glu Pro Pro Ile Arg Asp Glu Glu Arg Leu Trp Arg Val Val Asp
245 250 255
Val Leu Val Ser Ile Ala Gln Ser Arg Gly Val Ser Gly Ala Gln Val
260 265 270
Ala Leu Ala Trp Leu Leu Glu Arg Pro Ala Val Thr Ser Leu Val Ile
275 280 285
Gly Gly Arg Thr Glu Ala Gln Phe Lys Asp Asn Leu Ala Ala Ala Glu
290 295 300
Leu Lys Leu Thr Ala Glu Glu Arg Gln Arg Leu Asp Glu Val Ser Ala
305 310 315 320
Pro Pro Val Leu Tyr Pro Tyr Trp His Gln Leu Met Thr Ala Ser Asp
325 330 335
Arg Leu Gly Pro Ala Asp Met Pro Leu Leu Ala Pro His Leu Lys
340 345 350
<210> 15
<211> 343
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 15
Met Glu Tyr Arg Lys Leu Gly Asn Ser Gly Thr Val Val Thr Ser Tyr
1 5 10 15
Cys Leu Gly Thr Met Thr Phe Gly Gln Glu Thr Asp Glu Ala Thr Ser
20 25 30
His Leu Ile Met Asp Asp Tyr Ile Lys Ala Gly Gly Asn Phe Ile Asp
35 40 45
Thr Ala Asn Val Tyr Ser Ala Gly Val Ser Glu Glu Ile Val Gly Arg
50 55 60
Trp Leu Lys Ala Arg Pro Ser Glu Ala Arg Gln Val Val Val Ala Thr
65 70 75 80
Lys Gly Arg Phe Pro Met Gly Ala Gly Pro Asn Asp Leu Gly Leu Ser
85 90 95
Arg Thr Asn Leu Asn Arg Ala Leu Asn Asp Ser Leu Arg Arg Leu Gly
100 105 110
Val Glu Gln Ile Asp Leu Tyr Gln Met His Ala Trp Asp Ala Val Thr
115 120 125
Pro Ile Glu Glu Thr Leu Arg Phe Leu Asp Asp Ala Val Ser Ala Gly
130 135 140
Lys Ile Ala Tyr Tyr Gly Phe Ser Asn Tyr Leu Gly Trp Gln Val Thr
145 150 155 160
Lys Ala Val His Val Ala Arg Ala Asn His Trp Thr Ala Pro Val Thr
165 170 175
Leu Gln Pro Gln Tyr Asn Leu Leu Val Arg Asp Ile Glu His Glu Ile
180 185 190
Val Pro Ala Cys Gln Asp Ala Ala Met Gly Leu Leu Pro Trp Ser Pro
195 200 205
Leu Gly Gly Gly Trp Leu Ala Gly Lys Tyr Gln Arg Asp Val Met Pro
210 215 220
Ser Gly Ala Thr Arg Leu Gly Glu Asn Pro Asn Arg Gly Met Glu Ser
225 230 235 240
Tyr Gly Pro Arg Asn Ala Gln Glu Arg Thr Trp Gln Ile Ile Asp Met
245 250 255
Val Ala Glu Ile Ala Lys Glu Arg Gly Val Ser Ala Ala Gln Val Ala
260 265 270
Leu Ala Trp Val Val Ala Arg Pro Ala Val Thr Ala Val Ile Leu Gly
275 280 285
Ala Arg Thr Arg Glu Gln Leu Ala Asp Asn Leu Gly Ala Val Ala Val
290 295 300
Thr Leu Ser Thr Glu Glu Met Glu Arg Leu Asn Arg Val Ser Ala Pro
305 310 315 320
Ala Met Ala Asp Tyr Pro Tyr Gly Glu Arg Gly Val Ser Gln Arg His
325 330 335
Arg Lys Met Asp Gly Gly Arg
340
<210> 16
<211> 342
<212> PRT
<213> Artificial Sequence
<220>
<223> Enzyme
<400> 16
Met Asp Tyr Arg Lys Leu Gly Pro Ser Gly Thr Val Val Thr Ala Tyr
1 5 10 15
Cys Leu Gly Thr Met Thr Gly Ala Glu Ala Asp Glu Ala Ala Ser His
20 25 30
Lys Leu Leu Asp Asp Tyr Phe Ala Trp Gly Gly Asn Phe Ile Asp Thr
35 40 45
Ala Asp Val Tyr Ser Ala Gly Lys Ser Glu Glu Ile Ile Gly Arg Trp
50 55 60
Leu Lys Ala Arg Pro Thr Glu Ala Arg Gln Ala Ile Val Ala Thr Lys
65 70 75 80
Gly Arg Phe Pro Met Gly Asn Gly Pro Asn Asp Ile Gly Leu Ser Arg
85 90 95
Arg His Leu Ser Gln Ala Leu Asp Asp Ser Leu Arg Arg Leu Gly Leu
100 105 110
Glu Gln Ile Asp Leu Tyr Gln Met His Ala Trp Asp Ala Leu Thr Pro
115 120 125
Ile Glu Glu Thr Leu Arg Phe Leu Asp Asp Ala Val Ser Ser Gly Lys
130 135 140
Ile Gly Tyr Tyr Gly Phe Ser Asn Tyr Val Gly Trp His Ile Ala Lys
145 150 155 160
Ala Ser Glu Ile Ala Lys Ala Arg Gly Tyr Thr Arg Pro Val Thr Leu
165 170 175
Gln Pro Gln Tyr Asn Leu Leu Met Arg Asp Ile Glu Leu Glu Ile Val
180 185 190
Ala Ala Cys Gln Asp Ala Gly Met Gly Leu Leu Pro Trp Ser Pro Leu
195 200 205
Gly Gly Gly Trp Leu Thr Gly Lys Tyr Lys Arg Asp Glu Met Pro Thr
210 215 220
Gly Ala Thr Arg Leu Gly Glu Asn Pro Asn Arg Gly Gly Glu Ser Tyr
225 230 235 240
Ala Pro Arg Asn Ala Gln Glu Arg Thr Trp Ala Ile Ile Gly Thr Val
245 250 255
Glu Glu Ile Ala Lys Ala Arg Gly Val Ser Met Ala Gln Val Ala Leu
260 265 270
Ala Trp Thr Ala Ala Arg Pro Ala Ile Thr Ser Val Ile Leu Gly Ala
275 280 285
Arg Thr Pro Glu Gln Leu Ala Asp Asn Leu Gly Ala Met Lys Val Glu
290 295 300
Leu Ser Gly Glu Glu Met Ala Arg Leu Asn Glu Val Ser Ala Pro Gln
305 310 315 320
Pro Leu Asp Tyr Pro Tyr Gly Lys Gly Gly Ile Asn Gln Arg His Arg
325 330 335
Lys Ile Glu Gly Gly Arg
340
<210> 17
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> X is not M or X is V, I, L, M, F or A.
<220>
<221> MISC_FEATURE
<222> (3)..(3)
<223> X is not A or X is E, D, Q or N.
<220>
<221> MISC_FEATURE
<222> (6)..(6)
<223> X is not A or X is G or A.
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> X is not V or X is M, L, F, I, R, K, Q, N, E, V, A, or Norleucin.
<220>
<221> MISC_FEATURE
<222> (7)..(7)
<223> X is not V and/or X is M, L, F, I, R, K, Q, N, E, V, A, or
Norleucin.
<400> 17
Xaa His Xaa Trp Asp Xaa Xaa Thr Pro
1 5
<210> 18
<400> 18
000
<210> 19
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<220>
<221> MISC_FEATURE
<222> (1)..(1)
<223> X is G, R, K, Q, N, A, V, L, I, S, T, E, D
<220>
<221> MISC_FEATURE
<222> (3)..(3)
<223> X is not M or G or X is W, S, T, F, L, V, I, A or Y
<220>
<221> MISC_FEATURE
<222> (4)..(4)
<223> X is not E or X is T, S, R, Q, N or K.
<220>
<221> MISC_FEATURE
<222> (4)..(4)
<223> X is not E and/or X is T, S, R, Q, N or K.
<220>
<221> MISC_FEATURE
<222> (5)..(5)
<223> X is not S and/or X is E, D or Q.
<220>
<221> MISC_FEATURE
<222> (5)..(5)
<223> X is not S and/or X is E, D or Q.
<220>
<221> MISC_FEATURE
<222> (5)..(5)
<223> X is not S and/or X is P or A.
<220>
<221> misc_feature
<222> (6)..(7)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> MISC_FEATURE
<222> (8)..(8)
<223> X is not P and/or X is I, L, M, A, F or V.
<220>
<221> MISC_FEATURE
<222> (9)..(9)
<223> X is R, K, Q, N, P, A, Y, W, F, T, S, H.
<220>
<221> MISC_FEATURE
<222> (10)..(10)
<223> X is not N and/or X is D, E or N.
<400> 19
Xaa Gly Xaa Xaa Xaa Xaa Xaa Xaa Xaa Xaa
1 5 10
<210> 20
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 20
Val His Glu Trp Asp Gly Met Thr Pro
1 5
<210> 21
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 21
Val His Glu Trp Asp Gly Arg Thr Pro
1 5
<210> 22
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 22
Val His Glu Trp Asp Gly Gln Thr Pro
1 5
<210> 23
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 23
Val His Gln Trp Asp Gly Leu Thr Pro
1 5
<210> 24
<211> 9
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 24
Val His Gln Trp Asp Gly Gln Thr Pro
1 5
<210> 25
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 25
Gly Gly Phe Arg Glu Pro Pro Val Tyr Asp
1 5 10
<210> 26
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 26
Gly Gly Ser Arg Glu Pro Pro Val Pro Asp
1 5 10
<210> 27
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 27
Gly Gly Phe Arg Glu Pro Pro Val Pro Asp
1 5 10
<210> 28
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 28
Gly Gly Phe Lys Glu Pro Pro Val His Asp
1 5 10
<210> 29
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 29
Arg Gly Phe Arg Glu Pro Pro Val Pro Asp
1 5 10
<210> 30
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 30
Glu Gly Trp Thr Glu Pro Pro Ile Arg Asp
1 5 10
<210> 31
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 31
Ala Gly Trp Thr Glu Pro Pro Ile Arg Asp
1 5 10
<210> 32
<211> 10
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 32
Arg Gly Phe Lys Glu Pro Pro Ile Tyr Asp
1 5 10
<210> 33
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 33
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Asp Gly
1 5 10 15
Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro Pro Val
20 25 30
Tyr Asp
<210> 34
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 34
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Asp Gly
1 5 10 15
Gly Pro Glu Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro Pro Val
20 25 30
Pro Asp
<210> 35
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 35
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Asp Gly
1 5 10 15
Gly Pro Glu Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro Pro Val
20 25 30
Pro Asp
<210> 36
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 36
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Asn Gly
1 5 10 15
Gly Pro Asp Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro Pro Val
20 25 30
Tyr Asp
<210> 37
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 37
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Asp Gly
1 5 10 15
Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro Pro Val
20 25 30
Pro Asp
<210> 38
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 38
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Thr Gly
1 5 10 15
Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro Pro Val
20 25 30
Tyr Asp
<210> 39
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 39
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Ser Gly
1 5 10 15
Gly Pro Glu Thr Gly Arg His Val Gly Gly Phe Arg Glu Pro Pro Val
20 25 30
Tyr Asp
<210> 40
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 40
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Asp Ser
1 5 10 15
Ala Pro Asp Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro Pro Val
20 25 30
Tyr Asp
<210> 41
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 41
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg Gly Gln
1 5 10 15
Thr Ala Pro Glu Gly Ala Arg His Ala Gly Gly Phe Lys Glu Pro Pro
20 25 30
Val His Asp
35
<210> 42
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 42
Ser Pro Leu Ala Gly Gly Trp Leu Ser Gly Lys Tyr Thr Arg Ala Ser
1 5 10 15
Gln Pro Glu Asp Gly Arg His Val Arg Gly Phe Arg Glu Pro Pro Val
20 25 30
Pro Asp
<210> 43
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 43
Ser Pro Leu Ala Gly Gly Trp Leu Ser Gly Lys Tyr Thr Arg Asp Ser
1 5 10 15
Lys Pro Ser Asp Gly Arg Gln Val Arg Gly Phe Lys Glu Pro Pro Ile
20 25 30
Tyr Asp
<210> 44
<211> 34
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 44
Ser Pro Leu Ala Gly Gly Trp Leu Ser Gly Lys Tyr Thr Arg Asn Ser
1 5 10 15
Lys Pro Ser Asp Gly Arg Gln Val Arg Gly Phe Lys Glu Pro Pro Ile
20 25 30
Tyr Asp
<210> 45
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 45
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys His Arg Arg Asp Gly
1 5 10 15
Lys Ala Pro Glu Gly Ser Arg Gln Leu Glu Gly Trp Thr Glu Pro Pro
20 25 30
Ile Arg Asp
35
<210> 46
<211> 35
<212> PRT
<213> Artificial Sequence
<220>
<223> motif
<400> 46
Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys His Arg Arg Asp Thr
1 5 10 15
Lys Ala Pro Glu Gly Ser Arg Gln Leu Ala Gly Trp Thr Glu Pro Pro
20 25 30
Ile Arg Asp
35
<210> 47
<211> 771
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 47
atgacccgat ttgcccatta tcccagcctg accgatatgc ccgtcgtcat ctcaggcggt 60
gcctcgggca tcggcgaaag cctggtgcga aactttgccg cccagggcgc cagggtcggt 120
ttcgtcgata tcgccgtcga cgccggtgaa cgcctcgccg ccgaattgac ggggcagggg 180
caaagcgtag ccttcaccgc ctgcgacatc accgataccc aggcctatca atccgccatt 240
gccggcttcg ccgccaccca tggcgatgcc ctggccctgg tcaacaatgc cgcccatgac 300
cagcgccacg attgggctga ggtcacccct gcctattggg acgatcgcat ggcggtcaac 360
ctcaagcatg cctttttcgc catccaggcc gtggcgccgg gcatggtccg cgccgggcgc 420
ggctccatca tcaataccgg ctcgatcagc tggatgatca tgtcctcccg cattcccgtc 480
tatgaaaccg ccaaggccgc catccatggc ctcacccgcg gcatggcgcg cgaactgggg 540
cagtcgggca ttcgcgtcaa ttccctggtg cccggctggg tcatgaccga gcggcaattg 600
acccattggc tcgatgccga gggcgagcgg gccatcgcac aaaaccaggt gctggccggc 660
cgggtctatc ccgacgatgt cgcgcgcatg gccctgttcc ttgccgccga cgacagcgcc 720
atgatttcgg cccagcaatt tgtcgtcgat ggcggctggg ccaatgcctg a 771
<210> 48
<211> 1086
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 48
atgagccgca ttttcaacgt cgccatcgtc ggggtcggca tcggccgcag ccacctggtc 60
gagggctatg tgcccaatgc cgacaaatac aaggtcgtcg cgctgtgcga tctcaatgtc 120
gagcgcatga cgcccattgc cgacgaattc ggcgtcgagc gccgcatcac caatttcgac 180
gatatcctcg ccatggacga tatcgacatc atcgatatct gcaccccgcc gggcctgcat 240
tatcccatgg tcatggcggc gctgaaggcc ggcaagcatg taatctgcga aaagccgctg 300
gtcggctcgc tcgcccaggt cgatgaagtc attgcccagg aaaagatctc caagggcaag 360
ctgatgccgg tgttccagta tcgctttggc gacggcatcg aacaggccaa ggccatcatc 420
gatgccggca ttgccggcaa gccattcgtc ggcacggtgg aaaccatgtg gcgccgtggc 480
cccgattatt atgccgtgcc atggcgcggc aaatgggcca ccgaactggg cggcgtgctg 540
atgacccatg ccatccacca gcacgatctc ttcacctatc tcatgggcga tgtgacgcgg 600
ctcttcggcc gggtcgccac ccgcgtcaac gccattgaag tggaagactg cgttaccgcc 660
agccttgagc tcgaaaatgg cgccctgggc agctttaccg ccacgctggg cgcagccgag 720
gaaatcacaa ggctgttcct gaccttcgag aacgtcacct tcgaaagcga ttacgaggcc 780
tataatcccg gcgccaagcc gtggcgcatc cagccgcgca atcccgagac ggcggcgaaa 840
atcgacgccc tgctcaagga ctggaagcat gtgccctcgc gtttcgaaac gcagatggcc 900
cgcttccatg cggccctctt gggtgaagcg ccgatgccgg tgacctcggc cgattcgcgc 960
cgggcgctgg aaatcgtcac cgccttctac cattcctcga ctacccacga agatgtggcc 1020
ctgcccatcg cgtccgacca tccgcgttat cagagctggg ttccgcccca gttccgcgct 1080
gcctga 1086
<210> 49
<211> 1062
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 49
atggactatc gctatctcgg ccgctcgggc cttagggttt cggtgctgac catgggcaca 60
atgacctttg gcggctcgga aaaggtgggg catacagccc aggccgatgc gacgcgccag 120
attgatctct gccttgatca cggcatcaac ctgctcgaca ccgccaatgt ctataatgcc 180
ggggtctccg aggagatgat cggggtggcg ctgagcgaac atggccggcg gcaaaaggct 240
ttgattgcca ccaaggtgcg cttcaggatg ggtaacgggc ccaatgaagt ggggctgagc 300
cggcatcata tcattgccca ggccgaggcg agcctcaagc ggctcaagac cgatgtcatc 360
gatctctacc aggtgcatga atgggatggg atgacgccca tcgaggagac gatggaagcg 420
ctcgatacgc tcgtgaaaca gggcaaggtg cgctatgtcg gctgctcgaa ttattcgggc 480
tggcacatca tgaaggcgct ggctgcggcc gacaaacatc atggccagcg cttcatctcc 540
cagcagatcc actacacgct gcattcgcgc gaagccgaat atgaactggt gccgatcagc 600
caggaccagg ggctgggtat tctgatttgg tcgccgctgg cgggcggact gctctcgggc 660
aagtatcgtc gcgatggcgg gcccgatgcc ggccgccatg ttggaggctt ccgcgagccg 720
ccggtttatg actgggacaa gctctacgac attatcgacg tcatcgtcgc cattgcccgg 780
gagcgcgatg tgtcgggcgc gcaggtggcg ttggcctggg cattggggcg gccgggcgtg 840
acctcggcca ttatcggtgg ccgtagcgag gcccagttcc gcgacaatct ggcggcggcc 900
gaactcaagc tgaccgatga cgagcgcgcc cggctcgatg cggccagccg gccgccattg 960
ctttatccct attggcacca gtcttttacc gcccaggacc ggctggacca gatcgacctg 1020
gacctgatcg ggccctatgc cgaggagttt aaacgtggct ga 1062
<210> 50
<211> 786
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 50
atggacttga atttgcgcga caaggttgcc gtcattaccg ggggaacggt ggggatcggc 60
ctggccattg ccgaaggcct ggcggcggag ggcgtcaatc tggtgctggt ggggcgcgac 120
aagctgcggg ctgacgatgc ggcagcgctt gtggccgaca agttcgggat tatcgccacg 180
gcgatcagcg cggatgtcgc gactgccgaa gggtgcgatg ccgtgatcaa gggcacggcc 240
aaggcctttg gtggcgccga tatcctgatc aacaatgccg ggaccgggtc gaacgagacc 300
attgccgagg ccgatgatgc caaatggcaa tattattggg acctgcatgt gatggcggcc 360
gtgcggctgg cgcgggggct ggtgccggga atgaaaaagc ggggcggcgg cgtcgtgctg 420
cacaatgcct cgatctgcgc ggtgcagccg ctctggtatg agccgatcta caacaccacc 480
aaggccgcct tgatgatgtt ttccaagacg ctggccaatg aggtggtggg cgacaatatt 540
cgcgtcaata ccatcaatcc cgggctggtg ctgacgcccg attgggtcaa gaccgccaag 600
cagattgccg gcgaagatgg ctggcaggcg catctgcaag gggtggccga cgaggcgggc 660
ggcatgaagc gcttcgcgac gcccgaggaa ttggccaatt tcttcgtctt catgtgttcg 720
gacagagcca gctattccac cggctcgacc tattttgtcg atggcggctg gctcaagacg 780
gtttga 786
<210> 51
<211> 1038
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 51
atggaactcg gactctattc cttcgccgaa aacacgcccg atcccctgaa cggccgccgc 60
ctgcaaagcc cggccgaacg tctcaaggac ctgctggaag aaatcgaact ggccgaccag 120
ctcggcctat ccttttacgg cctgggcgaa caccaccgcc ccgactacac cgcttccgcg 180
cccgtcacca tcctggccgc cgccgccgcg cgcaccaagt cgatccgcct ctctaccgcg 240
gtcaccgtgc tcagctcgga agaccccatc cgcgtctatc agcaattcgc cacgctcgat 300
aatctcagca atggccgcgc cgagatcatg gccggccgcg gctccttcat cgagagcttc 360
cccctcttcg ggctcgacct caacgattac gacgcgctgt tcgaggaaaa gctcgaaatg 420
ctgctcaaaa tccgcgaagg cggcaaggtc acctggcccg gcagcaccca taccaagccc 480
atccccggca ttgccatcta tcccatcccg gtccagcagc ccctgccgct ctggatcgcc 540
gtcggcggca cgccaaattc cgttgcccgc gcggcctatt acggcctgcc cctgatgatc 600
gccatcatcg gcggtcagcc gcgccagttt gcgccgctgg tcgatttcta ccgcgaaacc 660
gctgaaaagg tcggccgcga ccccaaatcg ctgcccgtcg gcatcagctc gcacggcttc 720
atcgccgccg acagccagga cgcccgcgac atcgccttcc ccgcccatag cgaggccatg 780
agccgcatcg gcaaggaacg cggttggccg cccaccaccc gcgcccaatt cgacgccggc 840
gccacgccca acggcgccta tttcatgggg tctccgcagg aagtcatcga caagattctg 900
atgcagcacg aatggttcaa acacgaccgc ttcggcctgc aattcagcgt cggcaccctg 960
ccccacgaca aggtcatgaa agccatcgag ctttacggca ccgtggtcaa gccggcggtg 1020
gacaaggcgc taggctaa 1038
<210> 52
<211> 1026
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 52
atgagcactg aaatcgtcac cgatgtcgtt gtgattggcg cgggcccctg cgggctgttt 60
gcggccttcg aactcgggct gctcgacctc aaatgccatt tcatcgacat tctggaccgc 120
cccggcgggc agtgcgccga gctttatccc gaaaagccga tctacgatat tcccggcttc 180
ccggtggtga ccggccagga tctgaccgac aatctgatgg cccagatcgc gcccttcgcc 240
cccgaattcc acttcaaccg catggtcaat tcgatcgaga agcaggaaga cggctcgttt 300
aagctcgaaa ccgacgccga cgagattttc cacgcgaagg tcgtggtgat cgcggccggc 360
ggcggctcgt tccagcccaa gcgcccgccg gtcgacaata tcgaagccta tgaaaacaag 420
tcggttttct attcggtccg caagatggac gatttccgtg accaggacgt ggtgatcgtc 480
ggcggcggcg attccgccct cgactggacg ctcaacctgc agcccatcgt gcgctcgctg 540
accctggtac accgccgcga tgccttcaag gccgcgcccg cttcggtcaa caagatgaag 600
gaaatggtcg ccgagggtaa gatcaatttc ctgctcggcc aggtggccaa gctcgatggc 660
gaaaacggcc agatcaacca cgtgcatctg accaccgatg ccggcgatct ctcggtgccc 720
gccacccgcc tgctgccctt cttcggcctc accatgaagc tgggtccggt ggccgattgg 780
ggccttgagc tcaatgacaa tacgatcaag gtcgataccg aaaagttcga gacctcggtg 840
cccggcattt tcgccattgg cgacatcaac tggtatccgg gcaagctcaa gctgatcctc 900
tccggcttcc acgaggctgc cctcatggcc caggccgcca agaagattgt gtcgcccgac 960
gagcgcgtgg tgttccaata caccaccagc tccaccaagc tgcagaagaa gctgggcgtc 1020
gcctag 1026
<210> 53
<211> 1062
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 53
atggactatc gctatctcgg gcgctcgggc ctgaaagtct cgagcatgac catgggcacc 60
atgacctttg gcggcagcga aaagatcggc aataccagcc gacaggacgc ggcgcggcag 120
gtggatatgt gtctcgatgc gggcatcaac ctctatgaca gcgccaatgt ctataatgcg 180
ggggtgtccg aggaaatcct gggcgacatt ctcgccgaaa acgggcggcg gcagaaagcg 240
ctgatcgcca ccaaggtacg cttcaagatg ggcgatggcc ccaacgaggc cgggctcagc 300
cgacaccata tcatcgagca gtgcaaggcg agcctcaaac gcctcaagac cgatgtgatc 360
gacctctatc aggtgcatga atgggatggg cagaccccca tcgaggaaac gatggaagcg 420
ctcgacattc tcgtgcgcca cggccatgtg cgctatatcg ggtgctccaa ttattcgggc 480
tggcatattt ccaaggccct tgccgccgcc gaccagcggc acaatgcgcg ctttgtgtcc 540
cagcagatcc attattcgct gcatagccgc gacgccgaat atgagctggt gccgatttcc 600
caggaccagg ggctgggcat tctcgtctgg tcgccgctgg cgggcgggtt gctttcgggc 660
aagtatcgcc gggacagcgc gccggacagc gggcggcatg tcggcgggtt ccgcgagccg 720
ccggtctatg actgggacaa gctctattcg gtcatcgacg tgctggtggc ggtggccgag 780
gagcgtggcg tttcgggggc gcaggtggcg ctgagctggc tgatgggacg cccgggcgtc 840
acttccgtca tcatcggcgg acgtaacgaa aagcagtttg ccgacaatat cgcagcggcc 900
cagctcgtgc tcagtgacga ggaacgcgcc cggctcgatg cggccagccg accgccgttg 960
ctctatccct actggcacca gtccttcacc gcgcaggatc ggctgggcaa gatcgacatg 1020
gatttgatcg gtccttatgc cgaggagttc aagcgtggct ga 1062
<210> 54
<211> 1053
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 54
atggaatacc gcttgctcgg gcgctcggga ctgaaggtct cgacccttac catgggcacg 60
atgacgttcg gggggaaccc gaaggtcggc aataccagcc tcaaggacgc gcagcgccag 120
atcgacatgt gtctggaggc gggcatcaac atgctcgaca cggccaatgt ctatacgcgg 180
ggcgtgtcgg aaagcattat cggggaggca ctgaccgatg ggcggcgcca gcggacgctg 240
ctggcgacca aggtgcgctt cccgatggac gacaccgacc ccaacgagcg gggactgtcg 300
cgctaccaca tcgtcaacca gtgcgaggca agcctcaagc ggctcaagac cgatgtgatc 360
gacctctacc aggtgcatga atgggacggg cagaccccgc tcgaggagac catggaagcg 420
ctcgacatgc tcgtgcgcca cggcaaggtc cgctatatcg gctgctcgaa ctattcgggc 480
tggcacatca tgaaggcgct cggcgtttcc gagaaggacg cccggcagcg cttcgtctcc 540
cagcagatcc actacacgct gttttcgcgc gaggcggaga acgagctggt gccgatcagc 600
ctcgaccagg ggctgggcat tctcatctgg agcccgctgg cgggcggcct gctttcgggc 660
aagtatcgcc gcggccagac ggcgccggaa ggggcgcgcc atgccggggg cttcaaggag 720
ccgccggtgc atgactggga gcggctctac gatatcgtgg aagtgctggt ttcgatcgcc 780
gaggtgcgcg gggtttcggg cgcgcagatc gcgctggcct ggctgctggg gcgcccgggg 840
gtgacctcgg tgatcatcgg cgggcgcacg gaagaccagt tcagggataa cctggcggcg 900
gccgaactca agctgaccga gcatgagcgc aagcggctag acgacgtgtc gctgctgccg 960
ctgcaatatc cctactggca ccagagcttt acggcctcgg accggctggg gccggcggac 1020
ctggcgctgc atgcacccca tctcgaaaaa tag 1053
<210> 55
<211> 1062
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 55
atggactacc gctatctcgg ccggtcgggc ctcaaggttt cggtgctgag cgtcgggacg 60
gtgacgttcg gcggcgacgg gctgtggggc gatacggacg tcaccggcgc cgcccggcag 120
atcgacatgt gcctcgatgc aggcatcaac ctcatcgata ccgccaatgt ctacaacaag 180
ggcgtttcgg aagagatcac cggcgaggtg ctgcgcgacg gcggtcggcg gcagcgcgcg 240
ctgatcgcca ccaaggtgcg ctttccgttc ggcgacggtc ccaatgaccg cgggctgtcg 300
cgccaccaca tcatcgccca gtgcgagggc agcctcaaac gcctccagac cgacgtcatc 360
gatctctacc aggtccatca gtgggacggg cagacgcccc tcgaggagac gatggaagcc 420
ctcgataccc tggtgcggca ggggaaggtc cgctacatcg gctgctcgaa ctattcgggc 480
tggcacctga tgaaggcgct cgccgtcgcc gacagccgcg gctaccagcg gttcgtcgcc 540
cagcagatcc actacaccct ctattcgcgc gaggccgaat acgagctgca gccgatcacc 600
gtcgaccagg ggctcggcat actcgtctgg agcccgctcg ccggcgggtg gctctcgggc 660
aagtacacgc gcgccagcca gcccgaggac ggccgacacg tgcgcggttt ccgcgagccg 720
ccggtgcccg atcccgagcg gctctacgac atcgtcgatg tcatcaatgc ggttgccgcc 780
gagcggggcg tttcgggcgc ggtcgtttcg ctggcttggg ctttgcagcg caagggcgtc 840
tcgtcggtga tcgtgggcgc gcgcaccgac gcgcagttcg ccgataacct caaggccgcc 900
gatctaaagc tcagcgacga cgaggtcgcc cggctcgacg ccgtcagcca gcgtccgctt 960
ccctatccct attggcacca gtccttcacc gcccaggacc gcttgagcga agccgatctc 1020
gtgctgctcg ccccttatct cgaggagtac aagcgtggct ga 1062
<210> 56
<211> 1047
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 56
atggactatc gattgctcgg ccgttccggc ctcaaggtgt cgtcgctgtc gatcggcacg 60
gcgacgttcg gcggcgacgg gttgtggggc gacaccaacg tcaaggaggc gcagcgccag 120
atcgacctct gcctcgacca cggcgtcaac ctgatcgaca ccgccaatgt ctattcgggc 180
agcgtgtcgg agacgatcat cggcgaggcg ctgaccgatg gccggcgcga ccgcttgctg 240
atcgccacca aggtgcgctt ccccaggggc aagggaccga acgatcgcgg gctcagccga 300
tggcacatca tccgtgaggc ggaagcctcg ctgaagcgga tgaagaccga cgtcatcgac 360
ctgtaccagg tgcatgagtg ggatgggcag acgccgctcg aggagacgct ggaagcactc 420
gacaccctcg tccgctccgg caaggtccgc tatatcggct gctcgaacta ttcggcctgg 480
cacctgatga aggcgctcgc ggtttccaac gagcgcaact accagcgctt cgtttcccag 540
cagatccact acacgctgca ctcgcgcgaa gccgagttcg agctgatccc ggccggcctc 600
gaccagggtg tctcggtgct ggtgtggtcg ccgctcgccg gcgggtggct ctcgggcaag 660
tacacccgcg attccaagcc gagcgacggc cggcaggtgc gcggcttcaa ggagccgccg 720
atctacgatc ccgagcatct gtggaagatc gtcgacgtgg tcaacgacgt ggcgggcgcg 780
cgtggcgttt ccggcgcgca ggtgtcgctt gcctggttgc tgtcgaagcc gatcgtcgct 840
tcggtgatcg tcggcggccg caatatcgga cagttcgaag acaacctcaa agccgcggaa 900
ctgactctca ccaccgagga gctgcagaag ctcgacgcgg tcagccagcc gaacctgccc 960
tacccgtact ggcaccagag ctttaccgcg caggaccggc tcggtgagcc cgacctggcg 1020
ctgcaccgcc cctatctcga caagtga 1047
<210> 57
<211> 1047
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 57
atggactatc gattgctcgg ccgttccggc ctcaaggtct cgtcgctgtc gatcggcacg 60
gcgacgttcg gcggcgatgg gttgtggggc gataccaacg tcaaggaggc gcagcgccag 120
atcgacctct gcctcgacca tggcgtcaac ctgatcgaca ccgccaacgt ctattcgggg 180
agcgtctcgg aaacgatcat cggcgaggcg ctgaccgacg ggcggcgtga tcggctgctg 240
atcgccacca aggtgcgctt cccacgcggc aaggggccca atgatcgcgg cctcagccgc 300
tggcacatca tccgcgaggc ggaagcgtcg ctcaaacgga tgaagaccga cgtcatcgac 360
ctctaccagg tgcatgagtg ggacgggcag acgccgctcg aggaaacgct ggaggcgctc 420
gatacgctcg tccgctcggg caaggtccgc tatatcggct gctcgaacta ttcggcctgg 480
cacctgatga aggcgctcgc cacctcgaac gagcgcaact accagcgctt cgtctcccag 540
cagatccact acacgctgca ttcgcgtgag gccgagttcg agctgatccc ggccgggctc 600
gaccagggcg tttcggtgct ggtgtggtcg ccgctggccg gcgggtggct gtcgggcaag 660
tacacccgca actcgaagcc gagcgatggc cggcaggtgc gcggcttcaa ggagccgccg 720
atctatgatc cggagcacct gtggaagatc gtcgatgtgg tcaacgaagt ggccagcgcg 780
catggcgtct ccggcgcgca ggtgtcgctg gcctggctgt tgagtaagcc gatcgttgcc 840
tcggtgatcg tcggcggccg caatatcgag cagttcgagg acaacctgaa agccgccgag 900
ctgcagctca ccgccgagga actgggcaga ctcgacgcgg tcagccagcc gaacctcccc 960
tacccctact ggcaccagaa ctttaccgcg caggatcgcc tcagcgagcc tgacctggcg 1020
ctgcaccggc cctatctcga caaatga 1047
<210> 58
<211> 1056
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 58
atggaatatc gactgcttgg ccgctccggc ctcaaagtct ccaccctcac gatgggcacg 60
atgaccttcg gcggccaggg caagttcgcc tcggtcggca actccggcgt cgacgaagca 120
cgccggctga tcgacatgtg catcgatgcc ggcgtcaatc tcatcgatac ggcgaacgtc 180
tactccgatg gccgttcgga agaaatcatc ggcgaagtgc tcggcggcaa gcgtccgaag 240
gacgtactga tcgcgaccaa ggcacgcttt ccgatgggcg acggccccaa caacggcggc 300
ctctctcgct atcacctgat tcgcgagtgc gaagcgagcc tgaagcgcct gcgcacggac 360
gtgatcgatc tctaccaggt tcaccagtgg gatggcctga ccccgctgga agaaaccctg 420
gaagcgctcg acaccctggt tcgcgacggc aaggtgcgtt acctgggctg ctccaactat 480
tccgcgtggc acctgatgaa ggcgctcggc atcagtgagc gcgagcatcg ccagcggttc 540
gtcagccaac agatccatta cacgctggag gcgcgcgagg ccgaatacga gctgctgccg 600
atctcgatcg accagggcct cggcgtcctc gtctggagtc cgcttgccgg tggcctgctt 660
tcgggcaagc accgtcgcga tggcaaggca ccggagggct cacgtcagct ggagggctgg 720
accgagccgc cgatccgcga cgaagagcgc ctttggcgca tcgtcgacgt gctggtggcc 780
atcgccgaag cgcgcggcgt gtcgggtgca caggtcgcgc tggcctggct gctcgagcgt 840
cccgcggtga cctccctggt catcggcggt cgtaccgagg cgcagttcaa ggacaacctc 900
gccgccgccg agctgaagct cacggccgaa gagcgtcagc ggctggacga ggtcagtgcg 960
ccgccggtgc tctacccgta ctggcaccag ctgatgacgg cgtcggaccg cctgggtccg 1020
gcggacatgc cattgctggc gccgcatctg aagtaa 1056
<210> 59
<211> 1056
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 59
atggaatacc gactgcttgg ccgctccggc ctcaaagtct ccaccctcac gatgggcacg 60
atgaccttcg gcggccaggg caaattcgcc tcggtcggta gctccggcgt cgacgaagcg 120
cgccggctga tcgacatgtg catcgacgcc ggcgtgaatc tcatcgacac ggcgaacgtc 180
tattccgacg gtcgttcgga agagattatc ggtgaagtgc tcggcggcaa gcgtccgaag 240
gacgtcctga tcgcgaccaa ggcgcgcttt cccatgggtg atggcccgaa ccatggtggc 300
ctgtcgcgct atcacctgat tcgcgaatgc gaagccagcc tgaagcggct gcgcacggac 360
gtgatcgatc tctaccaggt tcaccagtgg gatggcctga cgcctctgga ggaaaccctg 420
gaagcgctcg acaccctcgt tcgcgacggc aaggtgcgtt acctgggttg ctccaactat 480
tccgcctggc acctgatgaa ggcgctcggc gtgagcgagc gtgagcaccg ccagcgtttc 540
atcagccagc agatccatta cacgctggaa gcgcgcgaag ccgaatacga actgctgccg 600
atttcgatcg accagggcct cggcgtgctg gtgtggagtc cgctcgccgg cgggctgctt 660
tcgggcaagc accgtcgcga taccaaggcc ccggaaggct cgcgccagct ggccggctgg 720
accgaaccgc cgatccgcga cgaagagcgc ctgtggcgcg tcgtcgacgt actggtttca 780
atcgcccaat cgcgtggcgt gtcgggagcg caggtcgcgc tggcctggtt gctcgagcgt 840
cctgcggtca cctcgctggt gatcggtggt cgtaccgagg cacagttcaa ggacaacctg 900
gcggccgccg aactgaagct cacggccgaa gagcgtcagc gtctggatga agtcagcgcg 960
ccgccggtgc tctatccgta ctggcaccag ttgatgaccg catcggatcg cctgggtccg 1020
gccgacatgc ccctgctggc gccgcacctc aaataa 1056
<210> 60
<211> 1062
<212> DNA
<213> Artificial Sequence
<220>
<223> enzyme
<400> 60
atggaatacc gctatctcgg ccgctcaggc ttgaaggtgt cgacattgac catgggcacc 60
atgacctttg gcggctcgga aaaggttggg gcgacggcgc aagacgatgc gacgcgccag 120
atcgacctgt gcctcgatgc agggatcaat ctgctcgaca cggccaatgt ctacaatgcc 180
ggggtctccg aggaaatgat tggcgtggcg ctggccgaga atggtcgccg ccagaaagca 240
ctcgtagcca ccaaggtccg cttcaagatg ggcgatgggc ccaacgagat cggcctcagc 300
cgtcaccata ttattgccca ggccgaagcc agccttaagc gcttgaaaac agacgttatt 360
gatctctatc aggtccacga atgggacggc atgacgccca tcgaggagac gatggaagcg 420
ctcgataccc tgatcaagca gggcaaagtc cgctatatcg gctgctcaaa ttattcgggc 480
tggcatatca tgaaggcgct gcaggccgcc gacaaaaacc acggccagcg cttcatcagc 540
cagcagatcc actacacgct gcattcgcgc gaagccgaat atgaactgat cccgatcact 600
ggggatcaag gccttggcgt cctcatctgg tcgccgctgg ccggtggttt gctctcgggc 660
aaataccgtc gcaacggtgg ccctgacagc gggcgccatg tcggcggctt ccgcgagccg 720
ccggtttatg actgggagaa gctctacgac atcgtcgatg tgatcgtgga tattgctgat 780
gaccgcggcg tgtcgggtgc acaggttgcg ctggcatggt cgctggggcg gccgggtgta 840
acctcggtga tcatcggtgg ccgcaatgat gcccagttca aagacaatct ggcggcagcc 900
gagttgaagc tgacggcgga agagcgcgcg cgacttgacg cggtcagccg tccaccgctg 960
ctctatcctt attggcatca gacgtttacg gcgcaggacc ggttaggccc tgtcgatcga 1020
aacctgatcg aaccctatgc agaggaattg aaccgtggct ga 1062
<210> 61
<211> 608
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 61
Met Arg Phe Glu Tyr Leu Arg Gln Asn Val Val Gly Leu Ala Leu Ser
1 5 10 15
Thr Ala Leu Ile Ala Ser Leu Ser Gly Pro Ala Phe Ala Gln His Asp
20 25 30
Ala Asn Ala Ala Ala Glu Pro Ser Lys Ala Gly Gln Ser Ala Ile Glu
35 40 45
Asn Phe Gln Pro Val Thr Ala Asp Asp Leu Ala Gly Lys Asn Pro Ala
50 55 60
Asn Trp Pro Ile Leu Arg Gly Asn Tyr Gln Gly Trp Gly Tyr Ser Pro
65 70 75 80
Leu Asp Gln Ile Asn Lys Asp Asn Val Gly Asp Leu Gln Leu Val Trp
85 90 95
Ser Arg Thr Met Glu Pro Gly Ser Asn Glu Gly Ala Ala Ile Ala Tyr
100 105 110
Asn Gly Val Ile Phe Leu Gly Asn Thr Asn Asp Val Ile Gln Ala Ile
115 120 125
Asp Gly Lys Thr Gly Ser Leu Ile Trp Glu Tyr Arg Arg Lys Leu Pro
130 135 140
Ser Ala Ser Lys Phe Ile Asn Ser Leu Gly Ala Ala Lys Arg Ser Ile
145 150 155 160
Ala Leu Phe Gly Asp Lys Val Tyr Phe Val Ser Trp Asp Asn Phe Val
165 170 175
Val Ala Leu Asp Ala Lys Thr Gly Lys Leu Ala Trp Glu Thr Asn Arg
180 185 190
Gly Gln Gly Val Glu Glu Gly Val Ala Asn Ser Ser Gly Pro Ile Val
195 200 205
Val Asp Gly Val Val Ile Ala Gly Ser Thr Cys Gln Phe Ser Gly Phe
210 215 220
Gly Cys Tyr Val Thr Gly Thr Asp Ala Glu Ser Gly Glu Glu Leu Trp
225 230 235 240
Arg Asn Thr Phe Ile Pro Arg Pro Gly Glu Glu Gly Asp Asp Thr Trp
245 250 255
Gly Gly Ala Pro Tyr Glu Asn Arg Trp Met Thr Gly Ala Trp Gly Gln
260 265 270
Ile Thr Tyr Asp Pro Glu Leu Asp Leu Val Tyr Tyr Gly Ser Thr Gly
275 280 285
Ala Gly Pro Ala Ser Glu Val Gln Arg Gly Thr Glu Gly Gly Thr Leu
290 295 300
Ala Gly Thr Asn Thr Arg Phe Ala Val Lys Pro Lys Thr Gly Glu Val
305 310 315 320
Val Trp Lys His Gln Thr Leu Pro Arg Asp Asn Trp Asp Ser Glu Cys
325 330 335
Thr Phe Glu Met Met Val Val Ser Thr Ser Val Asn Pro Asp Ala Lys
340 345 350
Ala Asp Gly Met Met Ser Val Gly Ala Asn Val Pro Arg Gly Glu Thr
355 360 365
Arg Lys Val Leu Thr Gly Val Pro Cys Lys Thr Gly Val Ala Trp Gln
370 375 380
Phe Asp Ala Lys Thr Gly Asp Tyr Phe Trp Ser Lys Ala Thr Val Glu
385 390 395 400
Gln Asn Ser Ile Ala Ser Ile Asp Asp Thr Gly Leu Val Thr Val Asn
405 410 415
Glu Asp Met Ile Leu Lys Glu Pro Gly Lys Thr Tyr Asn Tyr Cys Pro
420 425 430
Thr Phe Leu Gly Gly Arg Asp Trp Pro Ser Ala Gly Tyr Leu Pro Lys
435 440 445
Ser Asn Leu Tyr Val Ile Pro Leu Ser Asn Ala Cys Tyr Asp Val Met
450 455 460
Ala Arg Thr Thr Glu Ala Thr Pro Ala Asp Val Tyr Asn Thr Asp Ala
465 470 475 480
Thr Leu Val Leu Ala Pro Gly Lys Thr Asn Met Gly Arg Val Asp Ala
485 490 495
Ile Asp Leu Ala Thr Gly Glu Thr Lys Trp Ser Tyr Glu Thr Arg Ala
500 505 510
Ala Leu Tyr Asp Pro Val Leu Thr Thr Gly Gly Asp Leu Val Phe Val
515 520 525
Gly Gly Ile Asp Arg Asp Phe Arg Ala Leu Asp Ala Glu Ser Gly Lys
530 535 540
Glu Val Trp Ser Thr Arg Leu Pro Gly Ala Val Ser Gly Tyr Thr Thr
545 550 555 560
Ser Tyr Ser Ile Asp Gly Arg Gln Tyr Val Ala Val Val Ser Gly Gly
565 570 575
Ser Leu Gly Gly Pro Thr Phe Gly Pro Thr Thr Pro Asp Val Asp Ser
580 585 590
Ala Ser Gly Ala Asn Gly Ile Tyr Val Phe Ala Leu Pro Glu Lys Lys
595 600 605
<210> 62
<211> 586
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 62
Met Lys Lys Arg Thr Ser Ile Leu Leu Ala Ser Val Ala Met Leu Gly
1 5 10 15
Met Gly Ser Thr Ala Phe Ala Gln Val Asp Ile Asn Ala Leu Pro Ala
20 25 30
Val Thr Asp Ala Ile Leu Ala Asn Pro Asp Ala Gly Asp Trp Pro Ser
35 40 45
Tyr Gly Arg Asp Ile Thr Asn Tyr Arg Phe Ser Pro Leu Asp Gln Val
50 55 60
Asn Lys Asp Asn Val Gly Gln Leu Thr Leu Ala Trp Ala Arg Ala Leu
65 70 75 80
Glu Pro Gly Asn Leu Gln Ser Ala Pro Leu Glu Phe Gly Gly Val Leu
85 90 95
Phe Thr Ala Ala Pro Gly Asp Val Val Gln Ala Met Asp Ala Ala Thr
100 105 110
Gly Gln Leu Ile Trp Glu Tyr Arg Arg Gln Leu Pro Asp Arg Ala Thr
115 120 125
Leu Asn Ser Leu Gly Glu Asn Lys Arg Gly Ile Ala Leu Tyr Glu Asp
130 135 140
Lys Ile Tyr Val Ala Thr Trp Asp Asn Phe Ile Val Ala Leu Asp Ala
145 150 155 160
Lys Thr Gly Gln Val Ala Trp Glu Ser Asp Arg Gly Gly Gly Ala Asp
165 170 175
Leu Ile Ser Asn Thr Thr Gly Pro Ile Val Ala Asn Gly Val Val Val
180 185 190
Ala Gly Ser Thr Cys Gln Phe Ser Glu Phe Gly Cys Tyr Val Thr Gly
195 200 205
His Asp Ala Ala Thr Gly Glu Glu Leu Trp Arg Asn Asn Phe Ile Pro
210 215 220
Lys Lys Gly Glu Glu Gly Asp Asp Thr Trp Gly Asp Ser Thr Glu Asp
225 230 235 240
Gln Arg Trp Met Thr Gly Ala Trp Gly Gln Met Thr Tyr Asp Pro Glu
245 250 255
Leu Asp Leu Val Tyr Tyr Gly Ser Thr Gly Ala Gly Pro Ala Ala Glu
260 265 270
Phe Gln Arg Asn Thr Val Gly Gly Thr Leu Phe Gly Ser Asn Thr Arg
275 280 285
Phe Ala Val Lys Pro Lys Thr Gly Glu Ile Val Trp Arg His Gln Val
290 295 300
Leu Pro Arg Asp Asn Trp Asp Gln Glu Cys Thr Tyr Glu Met Val Pro
305 310 315 320
Val Asp Ile Asp Ser Ala Pro Ala Ala Asp Met Glu Gly Leu Leu Ala
325 330 335
Leu Gly Thr Ala Ala Pro Gly Lys Lys Arg Val Leu Thr Gly Val Pro
340 345 350
Cys Lys Thr Gly Val Met Trp Gln Phe Asp Ala Gln Thr Gly Glu Phe
355 360 365
Ile Tyr Ala Arg Asp Thr Val Gln Gln Thr Leu Ile Glu Ser Val Asp
370 375 380
Asn Thr Gly Leu Val Thr Val Asn Glu Ala Ala Ile Pro Thr Glu Val
385 390 395 400
Asp Val Ala Thr Pro Met Cys Pro Thr Tyr Leu Gly Gly Arg Asp Trp
405 410 415
Ser Pro Thr Ala Phe Asn Pro Thr Ser Lys Val Met Phe Val Pro Leu
420 425 430
Thr Asn Met Cys Ala Asp Val Thr Val Leu Asp Gln Glu Pro Thr Gly
435 440 445
Leu Asp Val Tyr Asn Thr Glu Leu Thr Tyr Lys Met Pro Glu Gly Val
450 455 460
Thr Asp Ala Gly Arg Ile Asp Ala Ile Asn Val Glu Thr Gly Lys Thr
465 470 475 480
Leu Trp Ser Trp Thr Gln Gln Thr Pro Gln Tyr Ala Ser Ile Thr Ala
485 490 495
Thr Ala Gly Gly Leu Ile Phe Thr Gly Gly Ala Asp Arg Arg Phe Lys
500 505 510
Ala Ile Asp Gln Glu Thr Gly Glu Leu Val Trp Ser Val Thr Leu Gly
515 520 525
Ser Arg Ala Thr Gly His Pro Ile Ser Tyr Glu Val Asp Gly Arg Gln
530 535 540
Tyr Ile Ala Ile Pro Ala Gly Gly Pro Gly Tyr Ala Thr Asp Leu Ile
545 550 555 560
Thr Ala Ser Gly Ser Thr Val Asp Val Val Ser Gly Ser Asn Met Leu
565 570 575
Tyr Val Phe Ala Leu Pro Glu Gln Lys Lys
580 585
<210> 63
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 63
Met Asp Tyr Arg Tyr Ile Gly Arg Ser Gly Leu Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Asp Lys Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Lys His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Asp Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Glu Leu Arg Leu
290 295 300
Thr Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Glu Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 64
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 64
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Lys Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Ile Asp Leu Cys Ile Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Leu Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Ala Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Asp Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Lys Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Glu Leu Arg Leu
290 295 300
Thr Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Gln Asp Arg Leu Asp
325 330 335
Gln Val Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 65
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 65
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Lys Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Leu Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Ile Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Asp Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Arg Leu Tyr Asp Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Lys Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Tyr Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Glu Leu Lys Leu
290 295 300
Thr Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Gln Asp Arg Leu Asp
325 330 335
Gln Val Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 66
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 66
Met Asp Tyr Arg Phe Leu Gly Arg Ser Gly Ile Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Thr Glu Lys Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Leu Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ser Glu His Gly Lys Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Ile Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Leu Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Ala Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Lys Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Leu Tyr Asp Trp Asp Arg Leu Tyr Asp Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Phe Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Asn Phe Arg Asp Asn Leu Gly Ala Ala Glu Leu Arg Leu
290 295 300
Ser Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Asn Asp Arg Leu Asp
325 330 335
Gln Leu Asp Leu Asp Leu Ile Gly Pro Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 67
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 67
Met Asp Tyr Lys Tyr Ile Gly Arg Ser Gly Ile Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Arg Val Gly His Thr
20 25 30
Gly Gln Ala Glu Ala Thr Arg Gln Leu Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Gly Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Asp Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Gly Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Asn Asp Arg Leu Asp
325 330 335
Gln Leu Asp Leu Asp Leu Leu Gly Pro Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 68
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 68
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Arg Val Gly His Thr
20 25 30
Gly Gln Ala Glu Ala Thr Arg Gln Leu Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Gly Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Leu Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Ala Gly Ala Glu Leu Lys Leu
290 295 300
Ser Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Asn Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Leu Gly Pro Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 69
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 69
Met Asp Tyr Lys Tyr Ile Gly Arg Ser Gly Ile Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Asp Ala Thr Arg Gln Ile Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Ala Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Ala Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Gly Ala Ala Glu Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Gln Asp Arg Leu Asp
325 330 335
Gln Leu Asp Leu Asp Leu Leu Gly Pro Tyr Ala Asp Asp Phe Lys Arg
340 345 350
Gly
<210> 70
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 70
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Ala Gln Ala Glu Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Ala Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Ala Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Gly Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Asn Asp Arg Leu Asp
325 330 335
Gln Leu Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 71
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 71
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Asp Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Gly Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Ala Ala Ala Glu Leu Lys Leu
290 295 300
Ser Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Gln Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Leu Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 72
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 72
Met Asp Tyr Arg Tyr Ile Gly Arg Ser Gly Leu Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Asp Ala Thr Arg Gln Ile Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Gly Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Leu Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Gly Ala Ala Asp Leu Lys Leu
290 295 300
Thr Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Ala Asn Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 73
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 73
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Asp Ala Thr Arg Gln Leu Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Gly Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Glu Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Asp
325 330 335
Gln Leu Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 74
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 74
Met Asp Tyr Arg Tyr Ile Gly Arg Ser Gly Ile Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Leu Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Leu Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Glu Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Asn Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Leu Gly Pro Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 75
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 75
Met Asp Tyr Arg Tyr Ile Gly Arg Ser Gly Ile Arg Val Ser Val Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Arg Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Leu Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Gly Leu Ser Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Asp Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Leu Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Gly Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Asn Asp Arg Leu Asp
325 330 335
Gln Leu Asp Leu Asp Leu Leu Gly Pro Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 76
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 76
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Glu Ala Thr Arg Gln Leu Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Gly Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Ile Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Gly Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Glu Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Asn Asp Arg Leu Asp
325 330 335
Pro Ala Asp Leu Asp Leu Leu Gly Thr Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 77
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 77
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Lys Val Gly His Thr
20 25 30
Pro Gln Ala Glu Ala Thr Arg Gln Leu Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Asp Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Val Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Thr Phe Thr Gly Asn Asp Arg Leu Gly
325 330 335
Pro Ala Asp Gln Asp Leu Leu Ser Thr Tyr Val Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 78
<211> 354
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 78
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Asp Ala Thr Arg Gln Ile Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Ala Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Ile Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Ala Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Asp Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Asp Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Gln Asp Arg Leu Gly
325 330 335
Pro Ala Asp Gln Asp Leu Leu Leu Ser Thr Tyr Val Glu Asp Phe Lys
340 345 350
Arg Gly
<210> 79
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 79
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Ala Gln Ala Glu Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Ile Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Ala Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Glu Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Asn Asp Arg Leu Gly
325 330 335
Pro Ala Asp Gln Asp Leu Ile Gly Thr Tyr Ala Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 80
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 80
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Asp Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Gly Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Ile Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Gly Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Asp Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Asp Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Thr Phe Thr Gly Gln Asp Arg Leu Asp
325 330 335
Pro Ala Asp Leu Asp Leu Leu Gly Thr Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 81
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 81
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Asp Ala Thr Arg Gln Ile Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Gly Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Ile Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Asp Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Asp Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Thr Phe Thr Ala Asn Asp Arg Leu Asp
325 330 335
Pro Ala Asp Leu Asp Leu Leu Ser Thr Tyr Val Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 82
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 82
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Asp Ala Thr Arg Gln Leu Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Gly Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Ile Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Glu Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Asp Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Thr Phe Thr Ala Gln Asp Arg Leu Gly
325 330 335
Pro Ala Asp Gln Asp Leu Ile Gly Thr Tyr Ala Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 83
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 83
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Leu Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Ile
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Asp Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Asp Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Asn Asp Arg Leu Asp
325 330 335
Pro Ala Asp Leu Asp Leu Leu Gly Thr Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 84
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 84
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Ala Gly Ser Glu Lys Val Gly His Thr
20 25 30
Ala Gln Ala Asp Ala Thr Arg Gln Leu Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Gly Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Ala Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Ile Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Gly Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ser Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Glu Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Asp Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Asn Asp Arg Leu Asp
325 330 335
Pro Ala Asp Leu Asp Leu Leu Gly Thr Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 85
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 85
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Asp Lys Val Gly His Thr
20 25 30
Pro Gln Ala Asp Ala Thr Arg Gln Ile Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu Gly Arg Arg Gln Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Gly Gly Gly Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Lys His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Ile Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Asp Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Glu Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Glu Leu Lys Leu
290 295 300
Thr Asp Asp Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Glu Arg Leu Gly
325 330 335
Pro Ala Asp Gln Asp Leu Leu Ser Thr Tyr Val Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 86
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 86
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Leu Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Pro Gln Ala Asp Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asp Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Ser Arg Asp Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Lys Leu Tyr Glu Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Lys Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala Glu Leu Lys Leu
290 295 300
Thr Asp Asp Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Gln Asp Arg Leu Asp
325 330 335
Gln Val Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 87
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 87
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Pro Gln Ala Asp Ala Thr Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asp Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Ile Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Ser Arg Asp Pro
225 230 235 240
Pro Val Pro Asp Trp Asp Arg Leu Tyr Asp Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Glu Glu Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Tyr Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Gly Ala Ala Glu Leu Lys Leu
290 295 300
Thr Asp Asp Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Gln Asp Arg Leu Gly
325 330 335
Pro Ala Asp Gln Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 88
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 88
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Ile Arg Val Ser Thr Leu
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Pro Gln Ala Asp Ala Thr Arg Gln Ile Asp Ile Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Ala Leu Ala Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asp Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Ile Lys Thr Asp Val Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Ile Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys Ser His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Ala Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Lys Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Ser Arg Glu Pro
225 230 235 240
Pro Leu Pro Asp Trp Asp Arg Leu Tyr Asp Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Glu Arg Asp Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Phe Ala Leu Gly Arg Pro Gly Val Thr Ser Ala Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Asn Phe Arg Asp Asn Leu Gly Ala Ala Glu Leu Lys Leu
290 295 300
Ser Asp Asp Glu Arg Gln Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Asn Asp Arg Leu Asp
325 330 335
Gln Leu Asp Leu Asp Leu Ile Gly Pro Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 89
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 89
Met Asp Tyr Arg Tyr Ile Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Asp Leu Ala Gly Ala Ala Arg Gln Val Asp Leu Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Tyr Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Leu Gly Gln Val Leu Ala Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Ala Gly Leu Ser Lys His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Ala Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Thr Gly Gly Pro Glu Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Leu Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Ala Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Leu Leu Gly Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala His Leu Arg Leu
290 295 300
Thr Ser Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Arg Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 90
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 90
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Asp Leu Ala Gly Ala Ala Arg Gln Ile Asp Leu Cys Ile Asp Ala Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Ile Gly Val Ala Leu Ala Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Ala Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Ile Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Gln Lys Gly Val Ser Gly Ala Gln Val Ala Leu Ser
260 265 270
Trp Leu Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Ala Asp Asn Leu Ala Ala Ile His Leu Arg Leu
290 295 300
Thr Ser Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Gln Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 91
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 91
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Asp Leu Ala Gly Ala Ala Arg Gln Ile Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Tyr Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Ile Gly Gln Ala Leu Ala Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Ala Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Arg Leu Tyr Asp Leu Ile Asp Val Ile Val
245 250 255
Asp Ile Ala Ala Gln Lys Gly Val Ser Gly Ala Gln Val Ala Leu Ser
260 265 270
Tyr Ala Leu Gly Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Asn Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ile His Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Arg Ile Val Asp Leu Asp Leu Glu Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 92
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 92
Met Asp Tyr Arg Phe Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Ala Gln Ala Gly Ala Ala Arg Gln Val Asp Leu Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Leu Gly Val Val Leu Ala Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Ala Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Ala Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Lys Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Leu Tyr Asp Trp Asp Lys Leu Tyr Asp Leu Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ser
260 265 270
Phe Leu Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Asn Phe Ala Asp Asn Leu Ala Ala Ala His Leu Lys Leu
290 295 300
Ser Ser Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Gln Ile Asp Leu Asp Leu Ile Glu Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 93
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 93
Met Asp Tyr Lys Tyr Ile Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Asp Leu Ala Gly Ala Ala Arg Gln Val Asp Ile Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Leu Gly Val Val Leu Ala Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Gly Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Ile Ile Asp Val Ile Val
245 250 255
Asp Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Ala Asp Asn Ile Ala Ala Ile His Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Gln Asp Arg Leu Ser
325 330 335
Gln Ile Asp Leu Asp Leu Ile Glu Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 94
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 94
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Gly Gln Ala Gly Ala Thr Arg Gln Val Asp Ile Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Val Leu Ala Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Ala Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Gly Leu Ala Gly Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Leu Ile Asp Val Ile Val
245 250 255
Asp Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ser
260 265 270
Trp Ala Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Ala Asp Asn Ile Ala Gly Ala His Leu Lys Leu
290 295 300
Ser Ser Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Gln Asp Arg Leu Ser
325 330 335
Gln Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 95
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 95
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Asp Leu Ala Gly Ala Ala Arg Gln Val Asp Leu Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Tyr Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Met Leu Gly Val Val Leu Ala Glu Asn Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Ala Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Ala Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Thr Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Leu Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Leu Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Ala Ala Ile His Leu Lys Leu
290 295 300
Ser Ser Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Gly Gln Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Ile Glu Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 96
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 96
Met Asp Tyr Lys Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Ala Gln Ala Gly Ala Ala Arg Gln Val Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Leu Gly Val Val Leu Ala Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Gly Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Ala Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Ala Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Ile Leu Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Leu Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Ala Asp Asn Ile Ala Ala Ile His Leu Lys Leu
290 295 300
Ser Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Asn Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Ile Glu Pro Tyr Ala Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 97
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 97
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Asp Leu Ala Gly Ala Thr Arg Gln Val Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Val Leu Ser Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Gly Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Asp Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Gly Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Asp Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Leu Leu Asp Val Ile Val
245 250 255
Asp Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ser
260 265 270
Trp Leu Leu Gly Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Ala Ala Ala His Leu Lys Leu
290 295 300
Ser Ser Asp Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Leu Glu Pro Tyr Ala Glu Glu Phe Lys Arg
340 345 350
Gly
<210> 98
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 98
Met Asp Tyr Arg Tyr Ile Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Lys Val Gly His Thr
20 25 30
Gly Gln Ala Gly Ala Ala Arg Gln Val Asp Leu Cys Leu Asp His Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Leu Gly Val Val Leu Ala Glu His Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Ile Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Ala Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Ile Ser Gln Gln Ile His Tyr Thr Leu His Ser Arg Glu Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Leu Ile Asp Val Ile Val
245 250 255
Asp Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ser
260 265 270
Trp Leu Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Ile Ala Ala Ile His Leu Lys Leu
290 295 300
Thr Ser Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Asn Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Gln Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 99
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 99
Met Asp Tyr Arg Tyr Leu Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Gly Gln Ala Gly Ala Ala Arg Gln Leu Asp Leu Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Leu Gly Val Val Leu Ser Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Gly Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Val Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Asp Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Lys Gln Ala Lys Val Arg Tyr Val Ala Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Gly Ala Asp Lys His His Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Ile Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Leu Ile Asp Val Ile Val
245 250 255
Ala Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Leu Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Arg Asp Asn Leu Ala Ala Ala His Leu Lys Leu
290 295 300
Thr Asp Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Ala Gln Asp Arg Leu Ser
325 330 335
Gln Ile Asp Leu Asp Leu Ile Glu Pro Tyr Ala Glu Asp Phe Lys Arg
340 345 350
Gly
<210> 100
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 100
Met Asp Tyr Arg Tyr Ile Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Asp Leu Ala Gly Ala Thr Arg Gln Val Asp Leu Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Leu Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Glu Met Ile Gly Val Val Leu Ala Glu His Gly Arg Arg Gln Arg Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Arg Met Gly Asn Gly Pro Asn Glu
85 90 95
Val Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Arg Thr Asp Ala Ile Glu Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Ala Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Gly Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Gly
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Asp Gly Gly Pro Asp Ala Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Asp Leu Ile Asp Val Ile Val
245 250 255
Asp Ile Ala Arg Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Ala Leu Glu Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Ser Glu Ala Gln Phe Ala Asp Asn Leu Ala Ala Ile His Leu Lys Leu
290 295 300
Thr Ser Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Asn Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<210> 101
<211> 353
<212> PRT
<213> Artificial Sequence
<220>
<223> enzyme
<400> 101
Met Asp Tyr Arg Tyr Ile Gly Arg Ser Gly Leu Lys Val Ser Ala Met
1 5 10 15
Thr Met Gly Thr Met Thr Phe Gly Gly Ser Glu Arg Val Gly His Thr
20 25 30
Ala Gln Ala Gly Ala Ala Arg Gln Val Asp Leu Cys Leu Asp Ala Gly
35 40 45
Ile Asn Leu Tyr Asp Thr Ala Asn Val Tyr Asn Ala Gly Val Ser Glu
50 55 60
Asp Ile Leu Gly Val Val Leu Ser Glu Asn Gly Arg Arg Gln Lys Ala
65 70 75 80
Leu Val Ala Thr Lys Val Arg Phe Lys Met Gly Asn Gly Pro Asn Glu
85 90 95
Ala Gly Leu Ser Arg His His Ile Ile Ala Gln Ala Glu Ala Ser Leu
100 105 110
Lys Arg Leu Lys Thr Asp Ala Ile Asp Leu Tyr Gln Val His Glu Trp
115 120 125
Asp Gly Met Thr Pro Ile Glu Glu Thr Met Glu Ala Leu Asp Thr Leu
130 135 140
Val Arg Gln Gly Lys Val Arg Tyr Val Gly Cys Ser Asn Tyr Ser Gly
145 150 155 160
Trp His Ile Met Lys Ala Leu Met Ala Ala Asp Lys Arg Gln Gly Gln
165 170 175
Arg Phe Val Ser Gln Gln Ile His Tyr Ser Leu His Ser Arg Asp Ala
180 185 190
Glu Tyr Glu Leu Val Pro Ile Ser Gln Asp Gln Gly Leu Gly Ile Leu
195 200 205
Val Trp Ser Pro Leu Ala Gly Gly Leu Leu Ser Gly Lys Tyr Arg Arg
210 215 220
Thr Gly Gly Pro Glu Ser Gly Arg His Val Gly Gly Phe Arg Glu Pro
225 230 235 240
Pro Val Tyr Asp Trp Asp Lys Leu Tyr Glu Leu Ile Asp Val Ile Val
245 250 255
Asp Ile Ala Ala Gln Arg Gly Val Ser Gly Ala Gln Val Ala Leu Ala
260 265 270
Trp Leu Leu Gly Arg Pro Gly Val Thr Ser Leu Ile Ile Gly Gly Arg
275 280 285
Asn Glu Ala Gln Phe Arg Asp Asn Ile Ala Ala Ile His Leu Lys Leu
290 295 300
Ser Ser Glu Glu Arg Ala Arg Leu Asp Ala Ala Ser Arg Pro Pro Leu
305 310 315 320
Leu Tyr Pro Tyr Trp His Gln Ser Phe Thr Gly Gln Asp Arg Leu Asp
325 330 335
Gln Ile Asp Leu Asp Leu Ile Gly Pro Tyr Ala Asp Glu Phe Lys Arg
340 345 350
Gly
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| АНТИТЕЛА, СОДЕРЖАЩИЕ ПОЛИПЕПТИД, ВСТРОЕННЫЙ В УЧАСТОК КАРКАСНОЙ ОБЛАСТИ 3 | 2019 |
|
RU2796254C2 |
| ПОЛИПЕПТИДЫ, ЯВЛЯЮЩИЕСЯ РАСТВОРИМЫМИ РЕЦЕПТОРАМИ 3 ФАКТОРА РОСТА ФИБРОБЛАСТОВ (SFGFR3), И ПУТИ ИХ ПРИМЕНЕНИЯ | 2017 |
|
RU2751483C2 |
| АНТАГОНИСТЫ NGF ДЛЯ МЕДИЦИНСКОГО ИСПОЛЬЗОВАНИЯ | 2020 |
|
RU2829812C2 |
| ВАРИАНТЫ FC-ОБЛАСТИ С ИЗМЕНЕННЫМ СВЯЗЫВАНИЕМ С НЕОНАТАЛЬНЫМ FC-РЕЦЕПТОРОМ (FCRN) ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2019 |
|
RU2830231C2 |
| ЛЕЧЕНИЕ АНОМАЛЬНОГО ОТЛОЖЕНИЯ ВИСЦЕРАЛЬНОГО ЖИРА С ИСПОЛЬЗОВАНИЕМ РАСТВОРИМЫХ ПОЛИПЕПТИДОВ РЕЦЕПТОРА ФАКТОРА РОСТА ФИБРОБЛАСТОВ 3 (sFGFR3) | 2018 |
|
RU2794170C2 |
| ВАРИАНТЫ IgG-FC ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2018 |
|
RU2814952C2 |
| Композиции для получения глюкозных сиропов | 2015 |
|
RU2706297C2 |
| МОЛЕКУЛА РЕЦЕПТОРА IL4/IL13 ДЛЯ ВЕТЕРИНАРНОГО ПРИМЕНЕНИЯ | 2018 |
|
RU2795591C2 |
| АНТИТЕЛА К РЕЦЕПТОРУ IL4 ДЛЯ ПРИМЕНЕНИЯ В ВЕТЕРИНАРИИ | 2019 |
|
RU2837141C2 |
| КОНСТРУКЦИИ СЛИТОГО БЕЛКА ДЛЯ ЗАБОЛЕВАНИЯ, СВЯЗАННОГО С КОМПЛЕМЕНТОМ | 2019 |
|
RU2824402C2 |
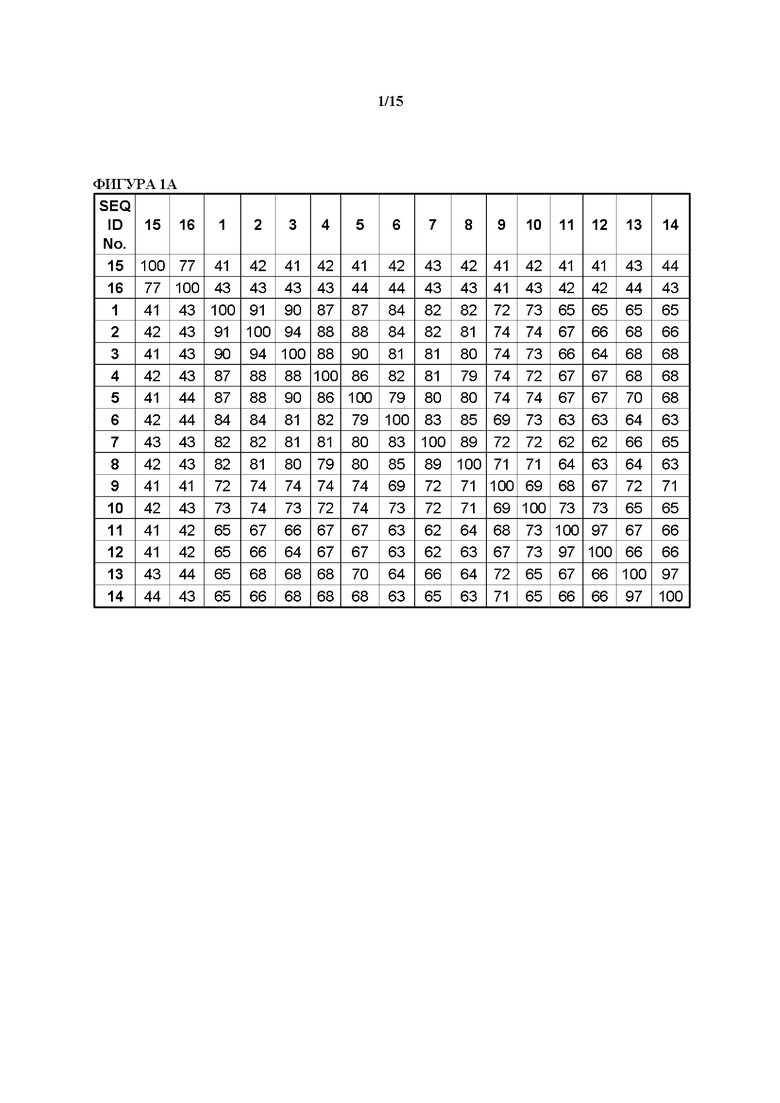
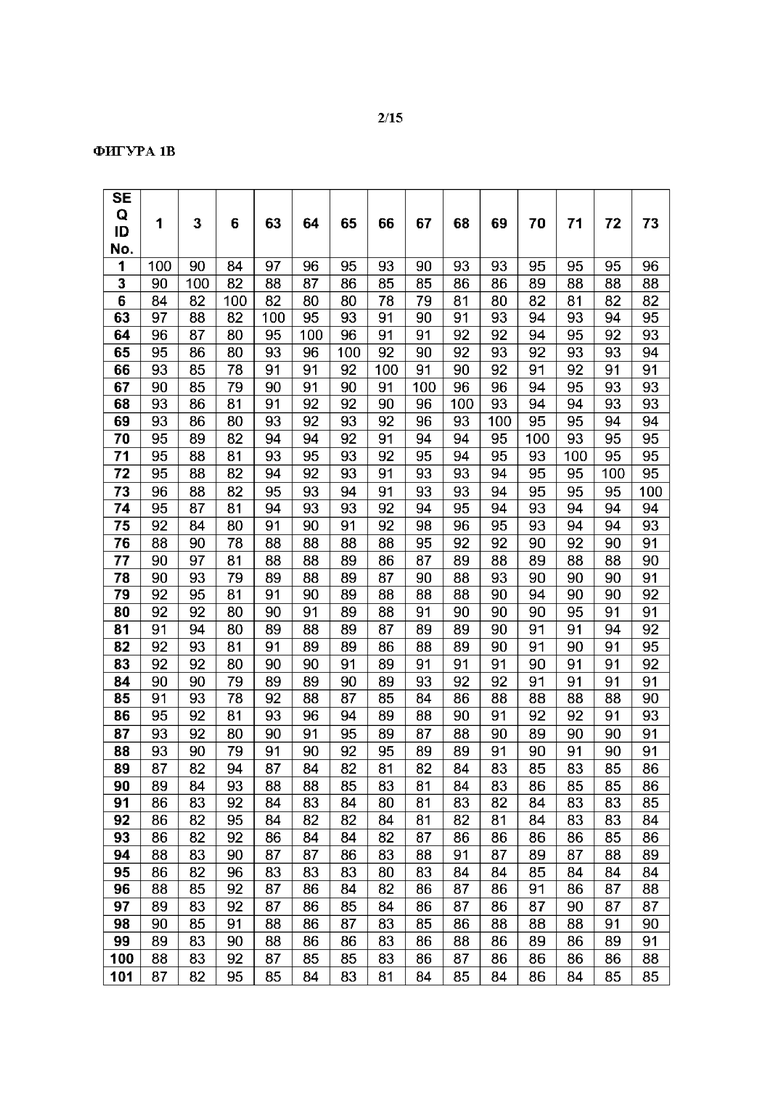
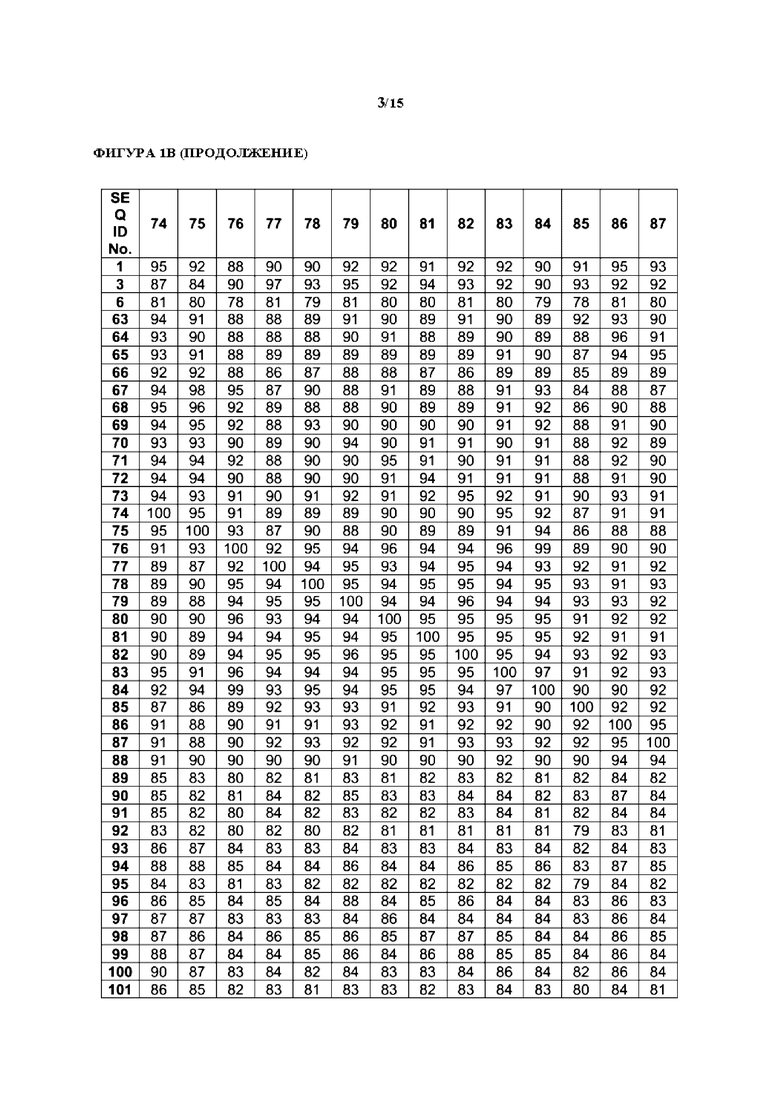
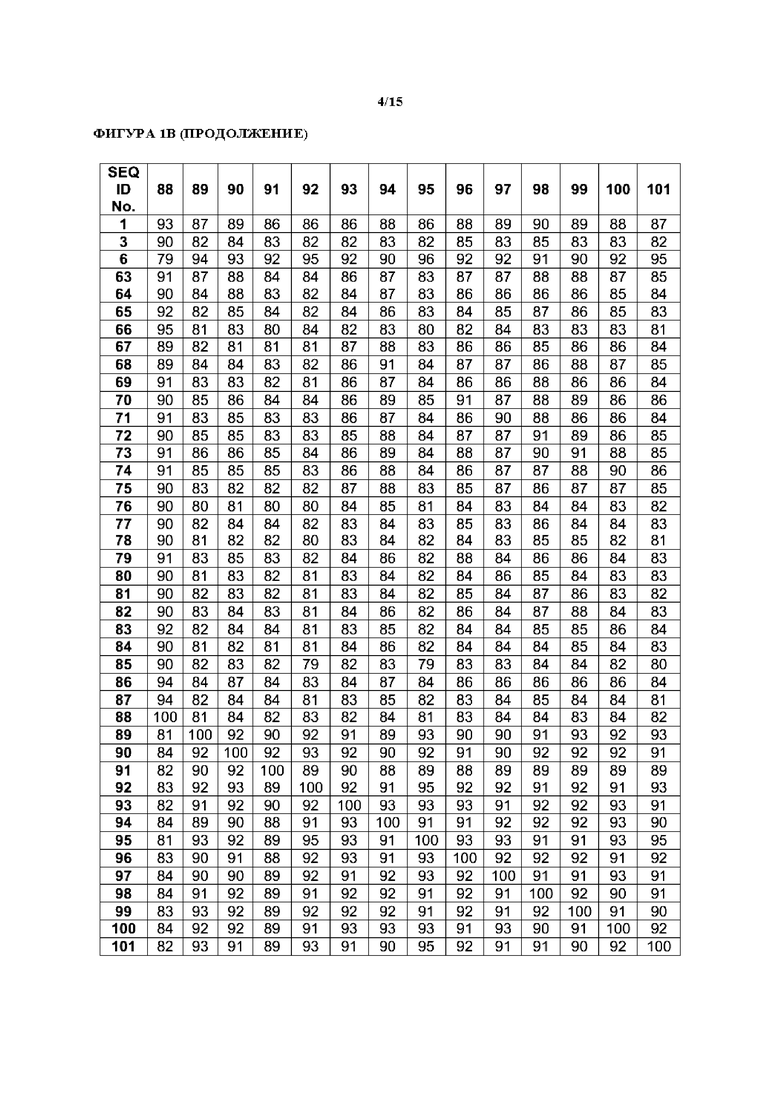
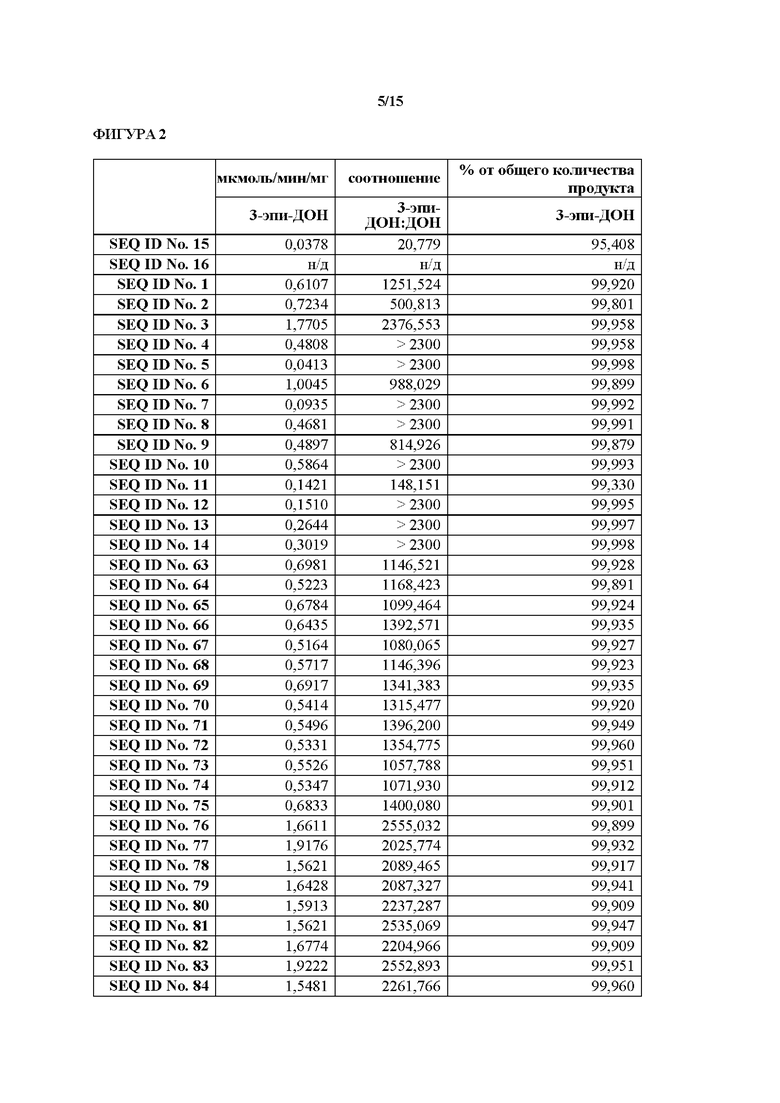
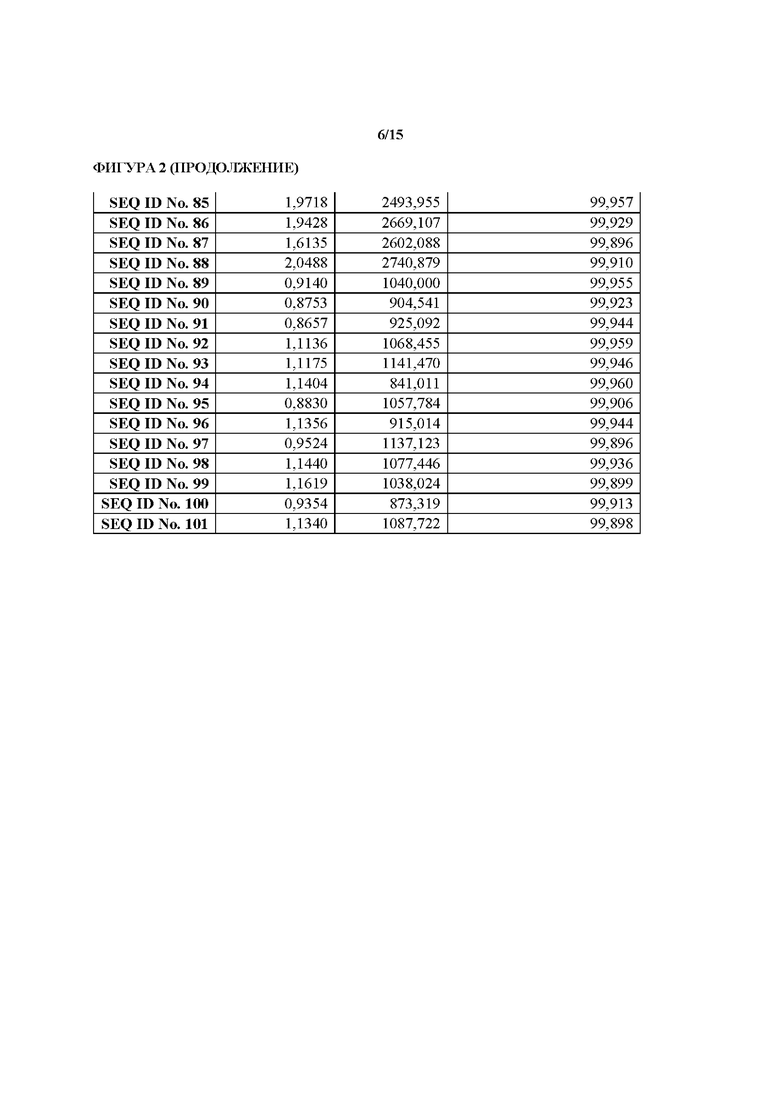
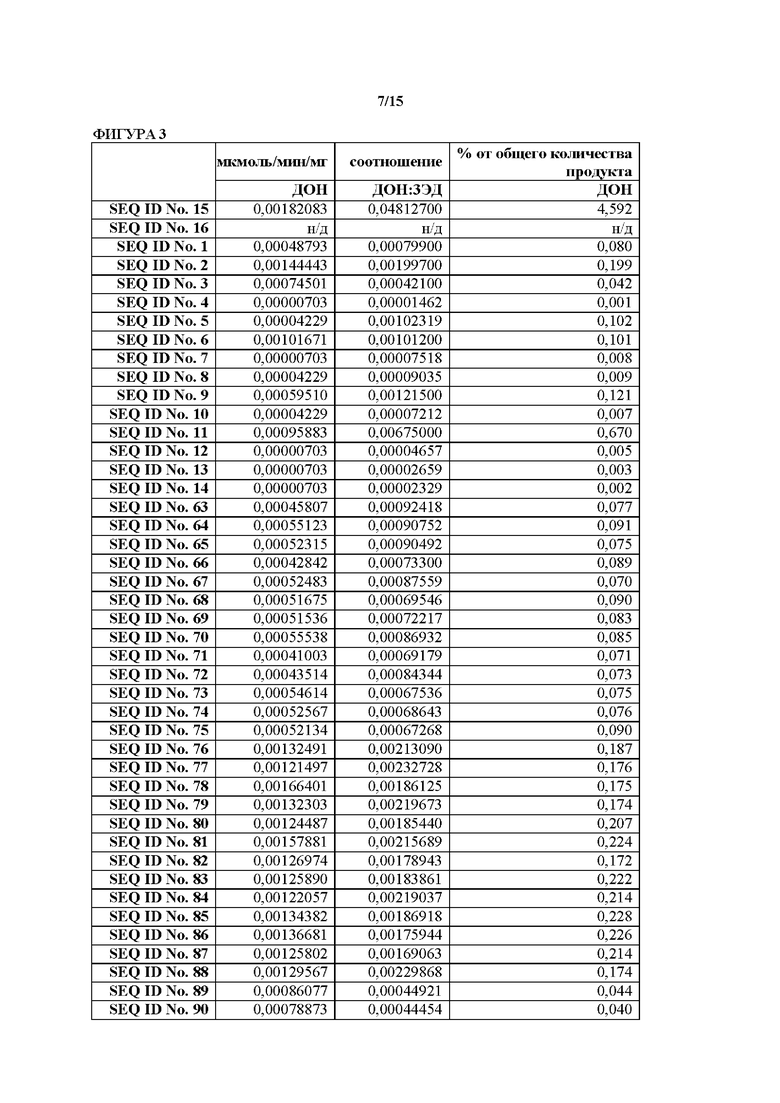
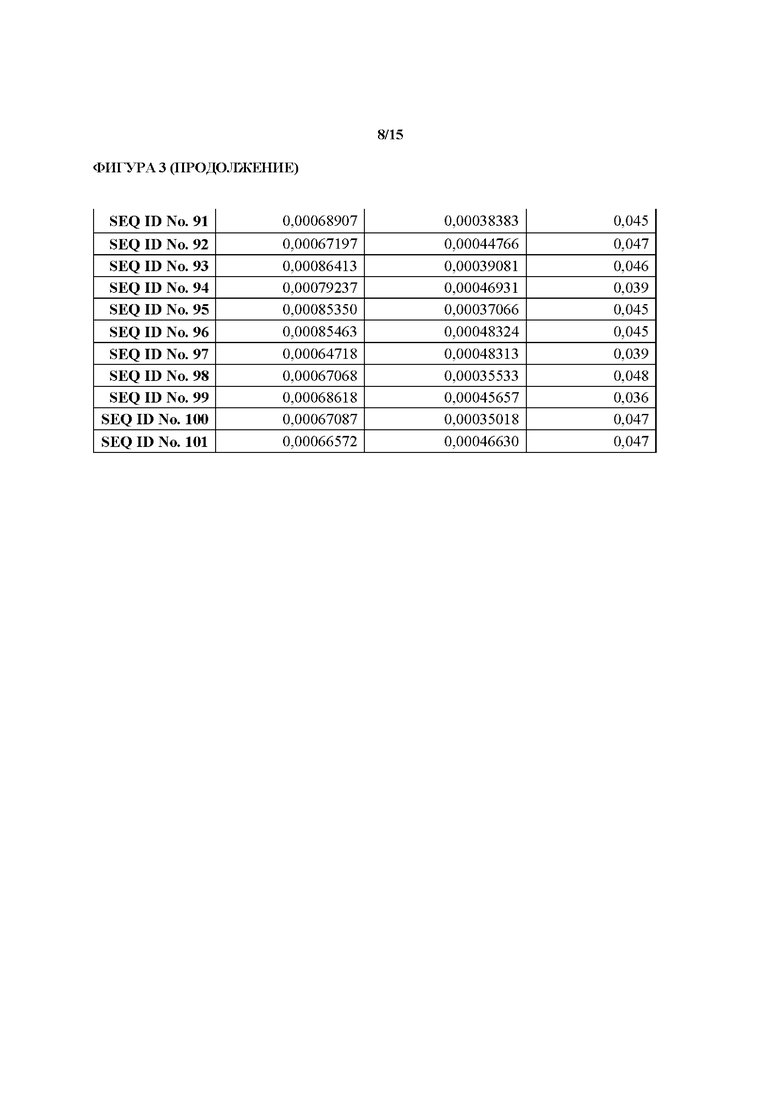


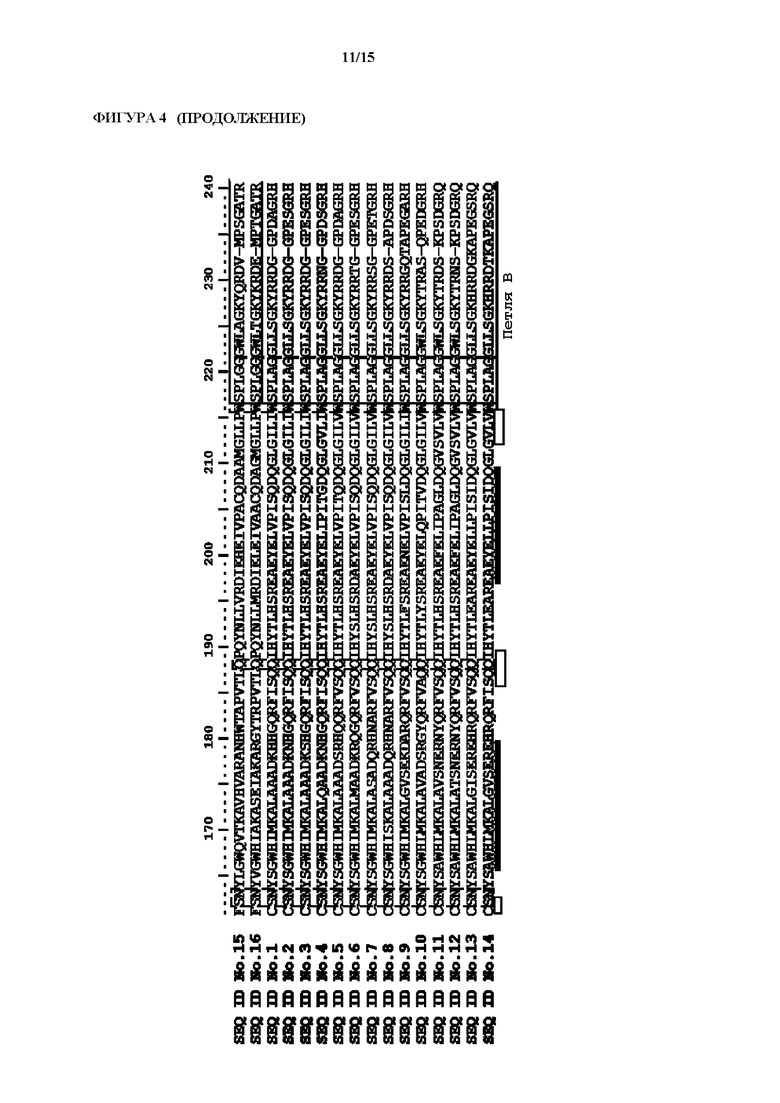
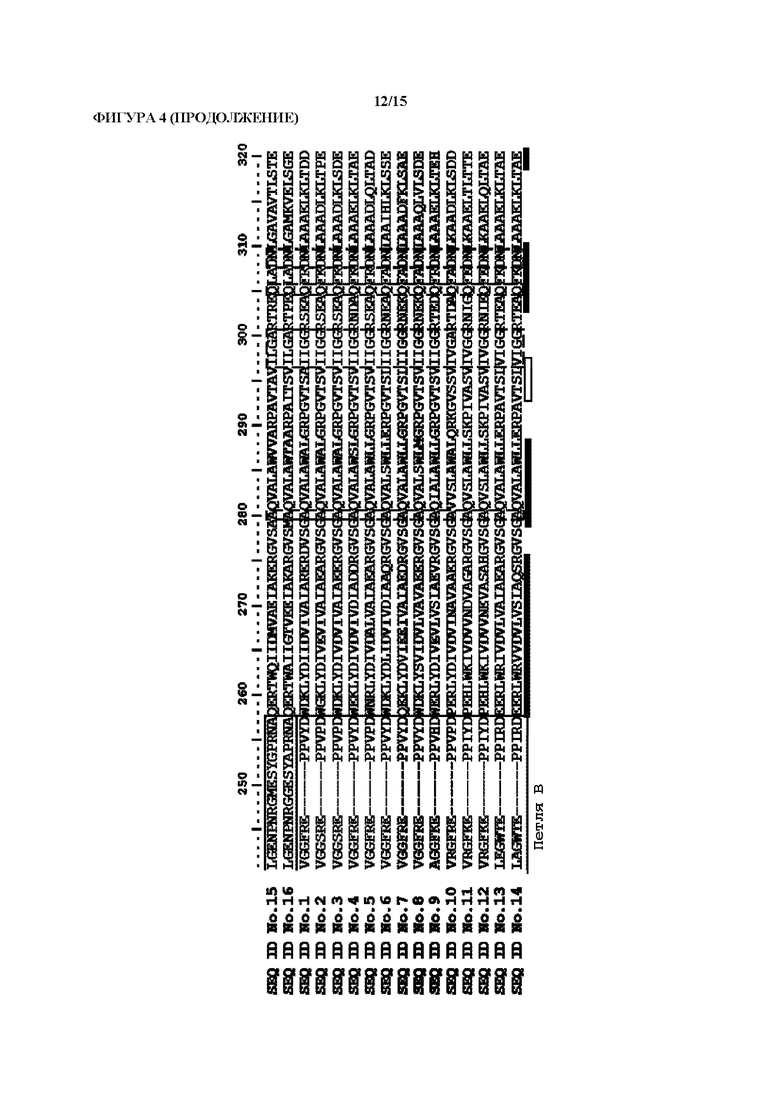



Изобретение относится к области биотехнологии, в частности к композиции сельскохозяйственного продукта для превращения субстрата 3-кето-ДОН в 3-эпи-ДОН. Указанная композиция сельскохозяйственного продукта содержит один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и носитель. Кроме того, изобретение также относится к полипептиду, содержащему последовательность SEQ ID NO. 1 или идентичную ей на по меньшей мере 92%, кормовым и пищевым добавкам и продуктам, способу превращения трихотецена, содержащего 3-оксогруппу, в трихотецен, способу снижения содержания ДОН, клетке-хозяину и применениям, в том числе для предотвращения или лечения микотоксикоза. Настоящее изобретение обеспечивает превращение субстрата 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1. 13 н. и 5 з.п. ф-лы, 5 ил., 4 табл., 5 пр.
1. Композиция сельскохозяйственного продукта, содержащая растение или части растения, для превращения субстрата 3-кето-ДОН в 3-эпи-ДОН, при этом указанная композиция сельскохозяйственного продукта содержит
a) один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14,
при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1; и
b) носитель.
2. Композиция по любому из предшествующих пунктов, отличающаяся тем, что указанный полипептид представляет собой редуктазу.
3. Композиция по любому из предшествующих пунктов, отличающаяся тем, что указанный полипептид дополнительно содержит последовательность, содержащую последовательность X1-H-X2-W-D-X3-X4-T-P, где
X1 не является M;
X2 не является A;
X3 не является A;
X4 не является V.
4. Композиция по любому из предшествующих пунктов, отличающаяся тем, что указанный полипептид дополнительно содержит последовательность, содержащую последовательность:
(а) S-P-L-X5-G-G-X6-L-X7-G, где
X5 представляет собой A, V, L, G или I, предпочтительно A;
X6 представляет собой L, I, V, M, A, Y, F, норлейцин или W, предпочтительно L или W;
X7 представляет собой S или T, предпочтительно S;
и/или
(b) X8-G-X9-X10-X11-X12-X13-X14-X15-X16, где
X8 представляет собой G, R, K, Q, N, A, V, L, I, S, T, E, D;
X9 не является M или G;
X10 не является E;
X11 не является S;
X12 не является Y;
X13 не является G или A;
X14 не является P;
X15 представляет собой R, K, Q, N, P, A, Y, W, F, T, S, H,
X16 не является N.
5. Композиция по любому из предшествующих пунктов, отличающаяся тем, что один или более полипептидов содержат последовательность, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, и содержат одну или более аминокислотных замен по сравнению с любой из последовательностей SEQ ID NO. 1-14, предпочтительно указанные одна или более аминокислотных замен представляют собой консервативные аминокислотные замены.
6. Композиция по любому из предшествующих пунктов, отличающаяся тем, что указанный носитель представляет собой жидкость, предпочтительно H2O, или твердое вещество, предпочтительно нутрицевтик и/или фармацевтическое средство.
7. Полипептид, содержащий последовательность SEQ ID NO. 1 или последовательность, которая на по меньшей мере 92% идентична SEQ ID NO. 1, для превращения субстрата 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
8. Применение одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, для превращения 3-кето-ДОН в 3-эпи-ДОН.
9. Применение одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, при получении: (a) кормовой добавки или кормовой композиции; или
(b) биогаза, биоэтанола или сахара.
10. Кормовая добавка, содержащая один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
11. Пищевая добавка, содержащая один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
12. Кормовой продукт, содержащий один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
13. Пищевой продукт, содержащий один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
14. Добавка для превращения субстрата 3-кето-ДОН в 3-эпи-ДОН, для применения в композициях, предпочтительно композициях сельскохозяйственных продуктов, содержащих 3-кето-ДОН или ДОН, при этом указанная добавка содержит один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1.
15. Способ превращения трихотецена, содержащего 3-оксогруппу, в трихотецен, содержащий 3-гидроксигруппу, включающий
приведение в контакт одного или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, с одним или более трихотеценами, содержащими 3-оксогруппу.
16. Способ снижения содержания ДОН в композиции, содержащей ДОН, или снижения токсичности композиции, содержащей ДОН, путем превращения ДОН в 3-эпи-ДОН, включающий
a) приведение в контакт указанной композиции с ферментом, способным превращать ДОН в 3-кето-ДОН; и
b) последующее или одновременное приведение в контакт указанной композиции с одним или более полипептидами, содержащими последовательность или состоящими из последовательности SEQ ID NO. 1-14 или содержащими последовательность или состоящими из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14.
17. Рекомбинантная клетка-хозяин, способная превращать субстрат 3-кето-ДОН в 3-эпи-ДОН, содержащая один или более полипептидов, содержащих или состоящих из последовательности SEQ ID NO. 1-14 или содержащих или состоящих из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, при этом указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, при этом в указанную рекомбинантную клетку-хозяин перенесен вектор экспрессии, способный обеспечивать выработку одного или более полипептидов.
18. Применение полипептида, содержащего последовательность или состоящего из последовательности SEQ ID NO. 1-14 или содержащего последовательность или состоящего из последовательности, которая на по меньшей мере 75% идентична любой из SEQ ID NO. 1-14, где указанный полипептид способен превращать субстрат 3-кето-ДОН в продукты 3-эпи-ДОН и ДОН с соотношением 3-эпи-ДОН:ДОН по меньшей мере 25:1, в предотвращении или лечении микотоксикоза.
| База данных UNIPROT: A0A085FJY9, 29.10.2014 | |||
| База данных UNIPROT: A0A0F5Q4N8, 24.06.2015 | |||
| База данных UNIPROT: A0A0F5LPU3, 24.06.2015 | |||
| База данных UNIPROT: A0A2L1S5V0, 25.04.2018 | |||
| База данных UNIPROT: A0A3S4GI93, 10.04.2019 | |||
| База данных UNIPROT: A0A0F5FI82, 24.06.2015 | |||
| База данных UNIPROT: A0A0F5FQD0, 24.06.2015 | |||
| База данных UNIPROT: A0A0Q7EN47, |
Авторы
Даты
2025-03-18—Публикация
2020-08-28—Подача