Изобретение относится к компьютерным системам, использующим генетические модели при разработке новых соединений для фармацевтики, биотехнологии и других областей промышленности, для которых существенно использование новых белковых соединений на основе аминокислот.
Метионин является одной из незаменимых аминокислот и, кроме того, первой аминокислотой в большинстве белковых молекул, поэтому большое внимание уделяется возможности его синтеза в промышленных масштабах, что, в свою очередь, требует детального изучения пути биосинтеза метионина.
Известно, что некоторые мутанты Escherichia coli, устойчивые к аналогам метионина, могут продуцировать метионин.
Было установлено, что мутанты Escherichia coli, устойчивые к этионину и утратившие способность к ингибированию конечным продуктом, способны производить метионин (Abelberg E.A., J.Bacteriol., 76, 326-328 (1958)).
Также была описана продукция метионина мутантами Escherichia coli, устойчивыми к норлейцину (Rowbury R.J., Nature, 206, 962 (1965)).
Но были получены только следовые количества метионина.
Описан микроорганизм, полученный введением в реципиентный штамм Escherichia гибридной плазмиды, содержащей фрагмент ДНК, выделенный из донорного штамма Escherichia, устойчивого к этионину (патент Великобритании GB2075055). Также описан вариационный тип гена metJ со специфической последовательностью, кодирующей связанный с биосинтезом метионина белок-репрессор из Escherichia coli, у которого активность к репрессии понижена и который полезен в продукции метионина (JP 157267 A2). Но выходы в процессе продукции метионина указанными мутантами (около 0,3-0,5 г/л) не могут удовлетворить растущие потребности в данной аминокислоте.
Известен способ получения метионина, в котором его получают путем выращивания бактерии Escherichia coli, обладающей способностью к продукции по крайней мере 0,5 г/л метионина в процессе выращивания на минимальной среде и устойчивой к аналогу метионина. Такими свойствами обладает штамм Escherichia coli 218 (ВКПМ В-8125). Накопленную аминокислоту выделяют из культуральной жидкости. Данное изобретение позволяет получать метионин с относительно высокой степенью эффективности (RU 2209248, 2001). Однако и этот способ не может удовлетворить потребности промышленности в метионине.
Все указанные выше способы получения метионина, помимо невысокой производительности, трудоемки и являются дорогостоящими.
В связи с этим насущной необходимостью является изыскание генетических моделей и прогнозирование новых путей получения метионина с использованием накопленного опыта, информационных технологий и сравнительной геномики.
Вид Corynebacterium glutamicum представлен свободно-живущими почвенными бактериями, которые используются в промышленном производстве большого количества аминокислот, в основном лизина, аргинина, треонина, изолейцина, валина, серина, триптофана, фенилаланина и гистидина [1], но не метионина, несмотря на общность путей биосинтеза лизина и треонина с биосинтезом метионина. В последние годы биосинтез метионина в С.glutamicum детально изучался с целью использования этой бактерии как промышленного продуцента метионина.
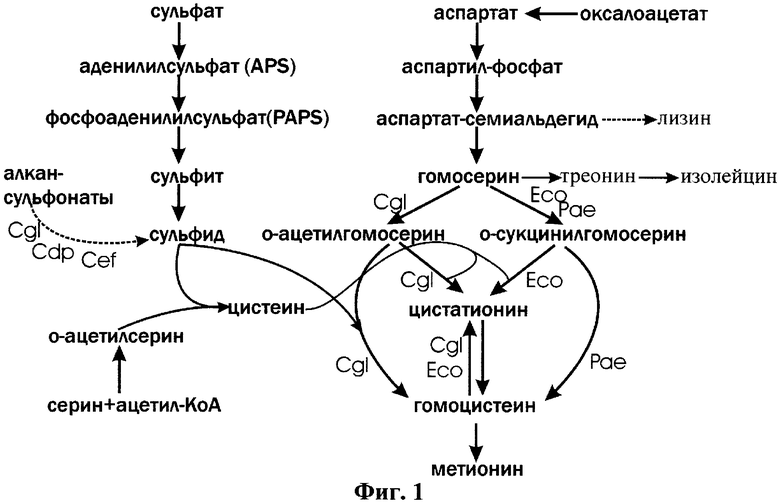
Структурные гены генетической модели пути синтеза метионина можно разделить на две части - гены пути включения серы в углеродный скелет и пути биосинтеза непосредственно метионина (см. фиг.1 - схема биосинтеза). Основной путь ассимиляции серы представляет собой путь восстановления сульфата до сульфида. Сера, входящая в состав сульфида, затем входит в путь биосинтеза метионина либо в составе цистеина, либо в составе цистатионина в зависимости от микроорганизма [2]. Гены ферментов восстановления сульфата формируют в геноме C.glutamicum единый оперон (фиг.4, позиция A, C.glutamicum). Помимо сульфата, в качестве источника серы для почвенных бактерий могут выступать и другие соединения, такие как сульфатные эфиры и сульфонаты, утилизация которых требует наличия соответствующих ферментов. Они также организованы в единые опероны.
Для генов модели синтеза непосредственно метионина объединение в крупные опероны не характерно. Биосинтез метионина идет от аспартата с последующим образованием гомосерина, который, в свою очередь, преобразуется либо в ацетилгомосерин в Neurospora crassa, Saccharomyces cerevisiae и C.glutamicum, либо в сукцинилгомосерин в Escherichia coli и Pseudomonas aeruginosa. Затем идет образование гомоцистеина, которое может происходить двумя путями: по прямому пути сульфгидрилирования (C.glutamicum, Pseudomonas aeruginosa и Saccharomyces cerevisiae) или по пути транс-сульфурилирования с образованием промежуточного продукта - цистатионина, этот путь характерен для Escherichia coli, Neurospora crassa и C.glutamicum [2; 3]. Таким образом, в отличие от большинства микроорганизмов, которые используют один из путей синтеза гомоцистеина, для C.glutamicum характерно наличие обоих полностью функциональных путей модели биосинтеза [3] (см. фиг.1).
Гены биосинтеза метионина в геноме C.glutamicum находятся под регуляцией как минимум двух регуляторов. Одним из них является непрямой регулятор, кодируемый геном cg3031. Соответствующий белок cg3031p подавляет биосинтез метионина, но при этом не способен к связыванию с ДНК [4].
Известен для Corynebacterium glutamicum [5; 6] транскрипционный регулятор генов пути биосинтеза метионина, mcbR (methionine and cysteine biosynthesis represser gene) и предполагаемая оперонная структура регулона mcbR.
Это димерный транскрипционный репрессор, принадлежащий к TetR-семейству, связывающийся с палиндромной последовательностью TAGAC-N6-GTCTA [6] в регуляторных областях генов метаболического пути. Большая часть сайтов связывания данного регулятора в C.glutamicum была предсказана на основании выравнивания регуляторных областей генов-мишеней и предсказание было экспериментально проверено и подтверждено для пяти регулируемых генов, таких как hom, cysI, cysK, metK и mcbR [6]. В целом в работе [6] была предсказана оперонная организация генов пути биосинтеза метионина (см. фиг.4, C.glutamicim) и были предъявлены потенциальные сайты связывания транскрипционного репрессора перед большей частью оперонов в геноме C.glutamicum (см. фиг.4, C.glutamicum - оперонная структура из [6] и соответствующие сайты).
Несмотря на то, что биосинтез метионина детально исследован в большинстве таксономических групп бактерий, очень немногое известно об этом метаболическом пути для организмов класса актиномицет.
Задачей предлагаемого изобретения является расширение функциональных возможностей способа прогнозирования, увеличение его оперативности, а также, в перспективе, уменьшение затрат на промышленное производство метионина.
Указанная задача решается в способе исследования и прогнозирования пути биосинтеза метионина в родственных геномах коринебактерий, включающем этапы, на которых:
а) составляют исходную обучающую выборку на основе предсказанных ранее и подтвержденных экспериментально регуляторных сигналов перед ферментами, участвующими в биосинтезе метионина, и записывают ее на информационный носитель;
б) вводят в компьютер записанную на информационный носитель исходную обучающую выборку и обрабатывают ее заранее записанной в память компьютера программой SignalX для построения матриц позиционных весов нуклеотидов;
в) обрабатывают исходную обучающую выборку заранее записанными в память компьютера алгоритмами из пакета программ Genome Explorer соответственно для поиска потенциальных сайтов связывания регулона McbR и анализа ортологов белков, при этом область поиска потенциальных сайтов связывания ограничивают диапазоном - 400...+100 пар нуклеотидов относительно старта трансляции гена, а сам ген считают принадлежащим оперону в случае одинакового направления считывания и расстояния не более 100 пар нуклеотидов от других генов этого оперона и записывают результаты обработки в память компьютера;
г) производят поиск ортологов белков пути биосинтеза метионина во всех родственных геномах, а результаты поиска и обработки по пункту в) разделяют на два выходных текстовых файла, первый из которых выводят на информационный носитель в виде таблицы соответствия генов пути биосинтеза метионина потенциальным сайтам связывания регулона McbR и родственным геномам коринебактерий, а второй - в виде диаграммы структуры оперонов регулона McbR;
д) на основе позиционного сравнения содержания обоих выходных текстовых файлов с учетом результатов поиска регуляторных сайтов связывания в более филогенетически отдаленных геномах для первого выходного текстового файла и с учетом характерных эволюционных событий формирования структуры оперонов для второго выходного текстового файла прогнозируют направление пути биосинтеза метионина.
Кроме того, в качестве эволюционных событий фиксируют неортологичные замещения, оперонные перестройки, паралоги и дупликации, утрату ортологов или полную консервативность в структуре оперона.
Кроме того, при фиксировании наличия паралогов и дупликаций используют эволюционные деревья, построение которых производят по методу максимального правдоподобия (maximum likelyhood) с помощью программы из программного пакета PHYLIP 3.63.
Кроме того, при составлении исходной обучающей выборки формируют полные последовательности геномов актинобактерий на основе базы данных Gen Bank, поиск гомологов в базе данных производят с помощью программы BLAST, а построение множественных выравниваний аминокислот и нуклеотидных последовательностей осуществляют посредством алгоритмов программы ClustalX.
Предлагаемый способ был применен для регулона mcbR во всех геномах, где присутствует данный регулятор, - это семь геномов порядка Actinomycetales.
На фиг.1. представлена генетическая модель схемы биосинтеза метионина, отмечены используемые различными микроорганизмами (альтернативные) пути прямого сульфгидрилирования и транс-сульфурилирования (сокращения названий геномов: Cgl - C.glutamicum, Cdp - C.diphtheriae, Cef - C.effiiciens, Eco - Escherichia coli, Рае - Pseudomonas aeruginosa);
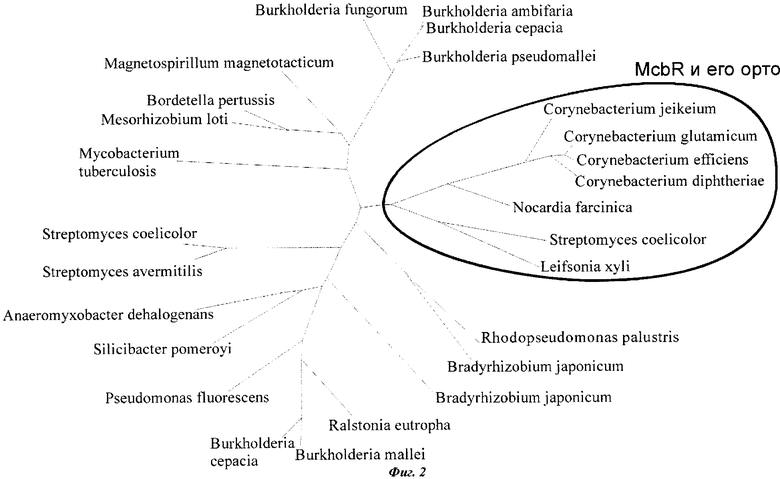
на фиг.2 показано эволюционное дерево McbR, выделена ветка, на которой находится McbR C.glutamicum и его ортологи;
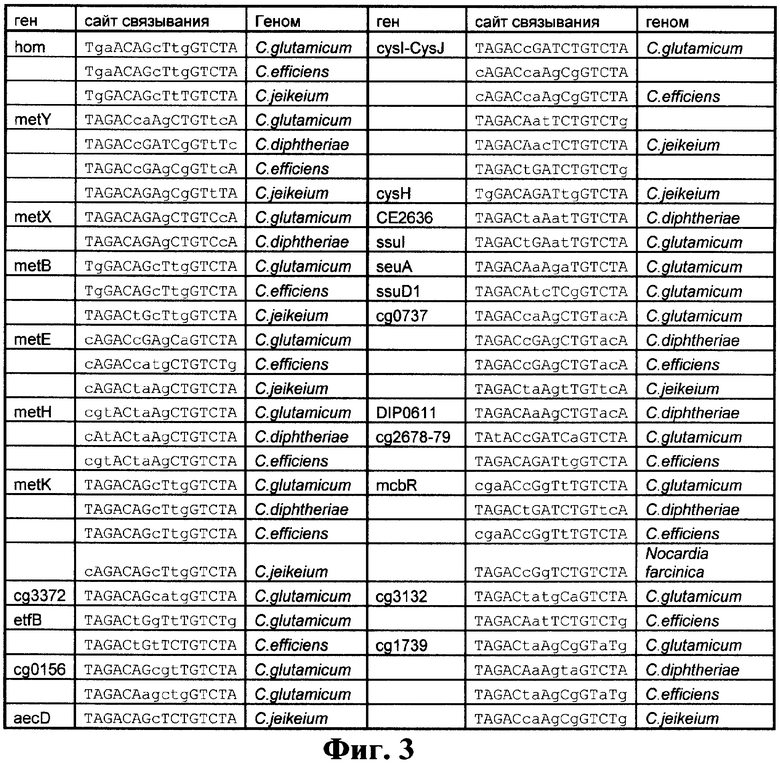
на фиг.3 представлен выход первого текстового файла в виде таблицы потенциальных сайтов связывания McbR перед генами пути биосинтеза метионина C.glutamicum и их ортологами в родственных геномах, имена генов и ортологов соответствуют названиям генов в геноме C.glutamicum за исключением генов DIP0611 C.diptheriae, у которого отсутствует ортолог в геноме C.glutamicum, и ортолога гена СЕ2636 C.efficiens в C.diphtheriae;
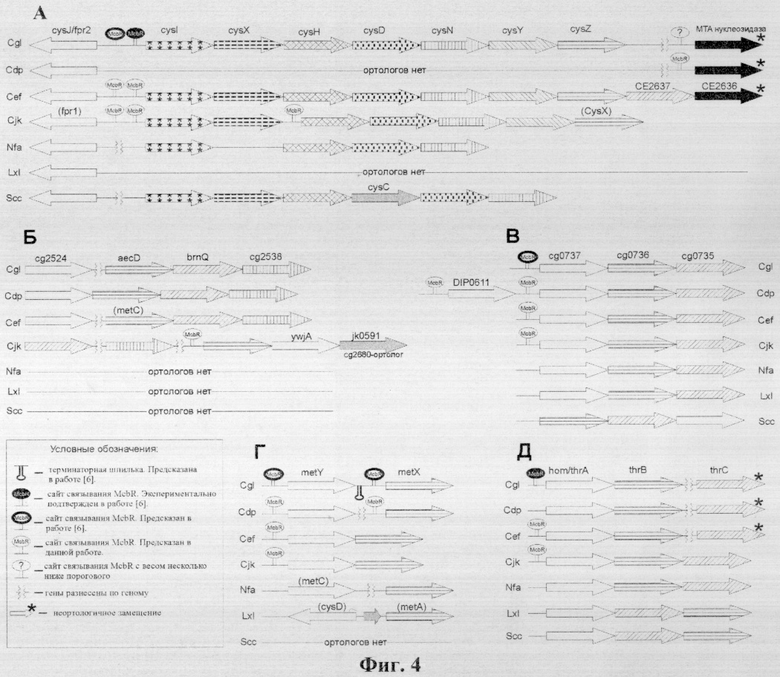
на фиг.4. представлен выход второго текстового файла в виде структуры пяти оперонов регулона McbR, претерпевших наибольшие эволюционные перестройки, тривиальные имена генов приведены для C.glutamicum, для ортологов, имеющих в GenBank другие названия, имена генов приведены в скобках над соответствующим геном, ортологичные гены имеют одинаковую контрастность (сокращения названий геномов: Cgl - C.glutamicum, Cdp - C.diphtheriae, Cef - C.effiiciens, Cjk - C.jeikeium, Nfa - Nocardia faricnica, Lxl - Leifsonia xyli и Scc - Streptomyces coelicolor);
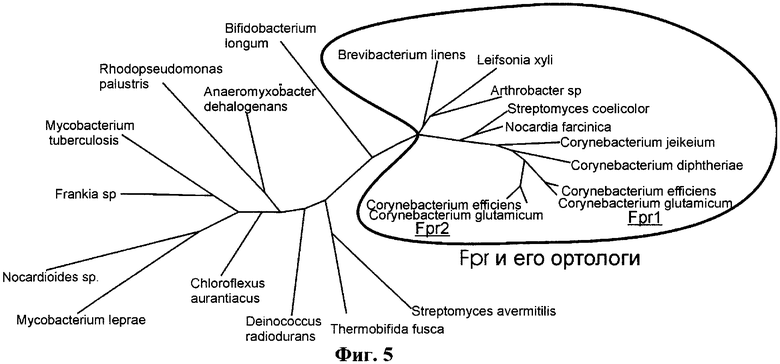
на фиг.5 показано эволюционное дерево Fpr, выделена ветка, на которой находятся Fpr C.jeikeium и его ортологи;
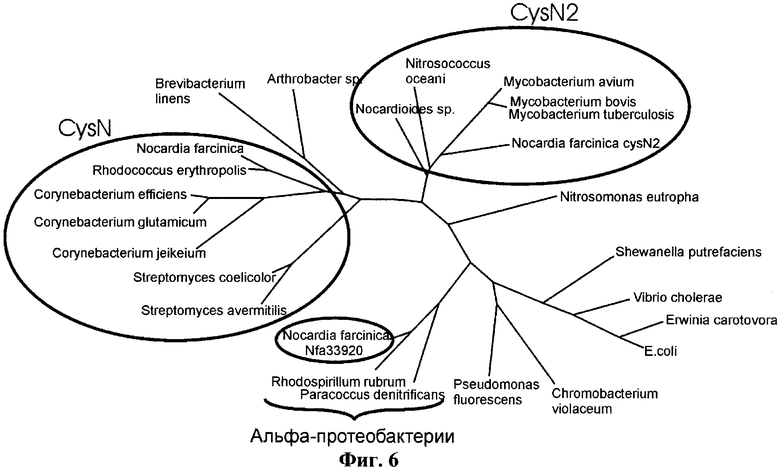
на фиг.6 показано эволюционное дерево CysN, выделены ветки ортологов белков CysN и CysN2, белок Nfa33920 и его ближайшие гомологи из группы альфа-протеобактерий;
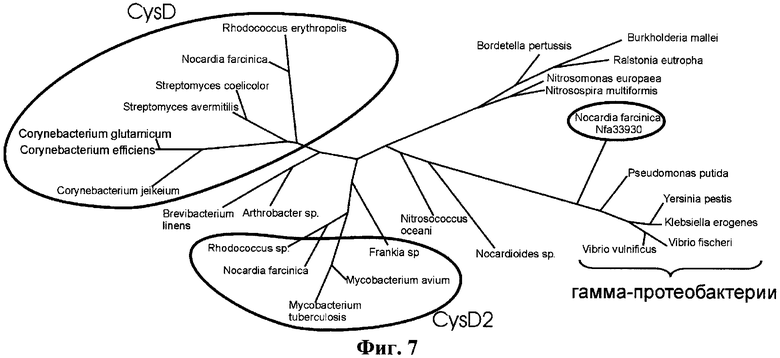
на фиг.7 показано эволюционное дерево CysD. Выделены ветки ортологов белков CysD и CysD2, белок Nfa33930 и его ближайшие гомологи из группы гамма-протеобактерий.
Способ осуществляется следующим образом.
Определение членов регулона
Для поиска потенциальных сайтов связывания mcbR использовался метод матриц позиционных весов нуклеотидов [Mironov A.A., Koonin E.V., Roytberg M.A., Gelfand M.S. 1999. Computer analysis of transcription regulatory patterns in completely sequenced bacterial genomes. Nucleic Acid Res. 27,2981-2989]. Процедура построения такой матрицы для поиска mcbR-сайтов подробно описана ниже.
Для определения принадлежности гена к регулону был применен метод проверки соответствия. Согласно данному методу ген считается членом регулона, если в его регуляторной области обнаружен потенциальный сайт, который сохраняется перед ортологичными генами в родственных геномах (там же).
Поиск потенциальных сайтов производился в области -400...+100 пар нуклеотидов относительно старта трансляции гена. В случае, когда оперонная структура данного фрагмента последовательности неизвестна, гены считались принадлежащими к одному оперону, если они имели одинаковое направление считывания, а расстояние между ними не превышало 100 пар нуклеотидов
Программное обеспечение
Для поиска потенциальных сайтов связывания mcbR, а также анализа ортологов использовался пакет программ GenomeExplorer [Миронов А.А., Винокурова Н.П., Гельфанд М.С. 2000. Программное обеспечение анализа бактериальных геномов. Мол. Биол. 34, 253-262]. Для построения матриц позиционных весов была использована программа SignalX [там же]. Поиск гомологов в базах данных проводился с помощью программы BLAST [Altschul S.F., Madden T.L., Schaffer А.А., Zhang J., Zhang Z M.W., Lipman D.J. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs. Nucleic Acid Res. 25, 3389-3402]. Для построения множественных выравниваний аминокислотных и нуклеотидных последовательностей была использована программа ClustalX [Thompson J.D., Gibson T.J., Plewniak F., Jeanmougin F., Higgins D.G. 1997. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acid Res. 25, 4876-4882]. Построение эволюционных деревьев производилось по методу максимального правдоподобия (maximum likelyhood) [Felsenstein J. 1996. Inferring phylogenies from protein sequences by parsimony, distance and likelihood methods. Methods Enzymol. 266, 418-427] с помощью программы proml из программного пакета PHYLIP 3.63 (http://evolution.genetics.washington.edu/).
В общей сложности было рассмотрено 7 геномов актинобактерий. Полные последовательности геномов:
Corynebacterium glutamicum ATCC 13032 [Kalinowski J., Bathe В., Bartels D., Bischoff N., Bott M., Burkovski A., Dusch N., Eggeling L., Eikmanns B.J., Gaigalat L., Goesmann A., Hartmann M., Huthmacher K., Kramer R., Linke В., McHardy A.C., Meyer F., Mockel В., Pfefferle W., Puhler A., Rey D.A., Ruckert C., Rupp О., Sahm H., Wendisch V.F., Wiegrabe I., Tauch A. 2003. The complete Corynebacterium glutamicum ATCC 13032 genome sequence and its impact on the production of L-aspartate-derived amino acids and vitamins. J. Biotechnol. 104, 5-25],
Corynebacterium diphtheriae NCTC 13129 [Cerdeno-Tarraga A.M., Efstratiou A., Dover L.G., Holden M.T., Pallen M., Bentley S.D., Besra G.S., Churcher C., James K.D., De Zoysa A., Chillingworth Т., Cronin A., Dowd L., Feltwell Т., Hamlin N., Holroyd S., Jagels K., Moule S., Quail M.A., Rabbinowitsch E., Rutherford K.M., Thomson N.R., Unwin L., Whitehead S., Barrell B.G., Parkhill J. 2003. The complete genome sequence and analysis of Corynebacterium diphtheriae NCTC13129. Nucleic Acids Res. 31, 6516-6523],
Corynebacterium efficiens YS-314 [Nishio Y., Nakamura Y., Kawarabayasi Y., Usuda Y., Kimura E., Sugimoto S., Matsui K., Yamagishi A., Kikuchi H., Ikeo K., Gojobori T. 2003. Comparative complete genome sequence analysis of the amino acid replacements responsible for the thermostability of Corynebacterium efficiens. Genome Res. 13, 1572-1579],
Corynebacterium jeikeium K411 [Tauch A., Kaiser O., Hain Т., Goesmann A., Weisshaar В., Albersmeier A., Bekel Т., Bischoff N., Brune I., Chakraborty Т., Kalinowski J., Meyer F., Rupp O., Schneiker S., Viehoever P., Puhler A. 2005. Complete genome sequence and analysis of the multiresistant nosocomial pathogen Corynebacterium jeikeium K411, a lipid-requiring bacterium of the human skin flora. J. Bacteriol. 187, 4671-4682],
Nocardia farcinica IFM 10152 [Ishikawa J., Yamashita A., Mikami Y., Hoshino Y., Kurita H., Hotta K., Shiba Т., Hattori M. 2004. The complete genomic sequence of Nocardia farcinica IFM 10152. Proc. Natl. Acad. Sci. USA. 101, 14925-14930],
Leifsonia xyli subsp. xyli str. CTCB07 [Monteiro-Vitorello C.B., Camargo L.E., Van Sluys M.A., Kitajima J.P., Truffi D., do Amaral A.M., Harakava R., de Oliveira J.C., Wood D., de Oliveira M.C., Miyaki C., Takita M.A., da Silva A.C., Furlan L.R., Carraro D.M., Camarotte G., Almeida N.F. Jr., Carrer H., Coutinho L.L., El-Dorry H.A., Ferro M.I., Gagliardi P.R., Giglioti E., Goldman M.H., Goldman G.H., Kimura E.T., Ferro E.S., Kuramae E.E., Lemos E.G., Lemos M.V., Mauro S.M., Machado M.A., Marino C.L., Menck C.F., Nunes L.R., Oliveira R.C., Pereira G.G., Siqueira W., de Souza A.A., Tsai S.M., Zanca A.S., Simpson A.J., Brumbley S.M., Setubal J.C. 2004. The genome sequence of the gram-positive sugarcane pathogen Leifsonia xyli subsp. xyli. Mol. Plant. Microbe. Interact. 17, 827-836] и
Streptomyces coelicolor A3(2) [Redenbach M., Kieser H.M., Denapaite D., Eichner A., Cullum J., Kinashi H., Hopwood D.A. 1996. A set of ordered cosmids and a detailed genetic and physical map for the 8 Mb Streptomyces coelicolor A3(2) chromosome. Mol. Microbiol. 21, 77-96, а также Bentley S.D., Chater K.F., Cerdeno-Tarraga A.M., Challis G.L., Thomson N.R., James K.D., Harris D.E., Quail M.A., Kieser H., Harper D., Bateman A., Brown S., Chandra G., Chen C.W., Collins M., Cronin A., Fraser A., Goble A., Hidalgo J., Homsby Т., Howarth S., Huang C.H., Kieser Т., Larke L., Murphy L., Oliver K., O'Neil S., Rabbinowitsch E., Rajandream M.A., Rutherford K., Rutter S., Seeger K., Saunders D., Sharp S., Squares R., Squares S., Taylor K., Warren Т., Wietzorrek A., Woodward J., Barrell B.G., Parkhill J., Hopwood D.A. 2002. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3(2). Nature. 417, 141-147]
были взяты из базы данных GenBank [Benson D.A., Boguski M.S., Lipman D.J., Ostell J., Ouellette B.F., Rapp B.A., Wheeler D.L. 1999. GenBank. Nucleic Acids Res. 27, 12-17].
Эволюционное дерево регулятора mcbR (фиг.2) свидетельствует, что данный регулятор сохраняется в геномах рода Corynebacterium (C.glutamicum, C.diphtheriae, C.efficiens и C.jeikeium), а также в других геномах порядка Actinomycetales, таких как Nocardia farcinica, Leifsonia xyli и Streptomyces coelicolor. Поэтому этапы способа применялись только для этих геномов.
Позиционное сравнение
Для C.glutamicum уже были показаны гены метаболического пути и их организация в опероны [6]. При реализации предлагаемого способа был произведен поиск ортологов белков пути биосинтеза метионина в родственных геномах и предсказание их организации в опероны (см. фиг.4). Анализ предсказанных оперонных структур позволил выделить несколько типов эволюционных событий:
1. Неортологичные замещения. C.jeikeium, Nocardia farcinica, Leifsonia xyli и Streptomyces coelicolor используют треонин-синтазу (thrC), характерную для большинства актиномицет. Треонин-синтаза при этом входит в состав оперона с генами гомосерин-дегидрогеназы {hom или thrA) и гомосерин-киназы thrB. Последовательность генов в опероне может меняться, так в C.jeikeium и Nocardia farcinica оперон выглядит как hom-thrB-thrC, а в Leifsonia xyli и Streptomyces coelicolor- как hom-thrC-thrB (фиг.4, позиция Д).
В то же время в геномах C.glutamicum, C.diphtheriae и C.efficiens функции треонин-синтазы выполняет белок, не ортологичный thrC актиномицет. Его картирование и функциональность были проверены экспериментально, хотя происхождение этого белка не очевидно. Характерно, что эта треонин-синтаза уже не входит в состав оперона и таким образом образуется два независимых оперона: hom-thrB и нерегулируемый моноцистронный оперон thrC, расположенные в разных частях генома (фиг.4, позиция Д).
Для почвенных бактерий одними из основных источников серы являются сульфатные эфиры и сульфонаты. Сульфатные эфиры утилизуются посредством сульфатаз, которые присутствуют практически в каждом микроорганизме. Тем не менее, в геноме C.glutamicum для усвоения сульфатных эфиров есть отдельный оперон seuABC, кодирующий три потенциальных фермента десульфуризации [6], в состав которого также входит ssuD2 (см. ниже). Утилизация же сульфонатов требует обязательного наличия специфических ферментов. Во всех описанных случаях гены утилизации сульфонатов формируют один оперон, включающий в себя АВС-транспортер и NAD(Р)Н-зависимую монооксигеназу. Для C.glutamicum это оперон ssuABC-D1 (ssu - sulfonate-sulfur utilization) описан в [Koch D.J., Ruckert С., Rey D.A., Mix A., Puhler A., Kalinowski J. 2005. Role of the ssu and seu genes of Corynebacterium glulamicum ATCC 13032 in utilization of sulfonates and sulfonate esters as sulfur sources. Appl. Environ. Microbiol. 71, 6104-6114]. Интересно, что во всех исследованных геномах оба оперона практически полностью утрачены (см. ниже), однако в геноме C.efficiens в опероне утилизации сульфата cysIXHDNYZ (фиг.4, позиция А) появляется новый потенциальный член - СЕ2637, ближайшими ортологами которого являются члены белкового семейства люцифераз из геномов протеобактерий. Поиск консервативных белковых доменов в GenBank позволяет более точно предсказать функцию белка СЕ2637 как алкансульфонат-монооксигеназы, которая, по всей видимости, появилась в геноме C.efficiens вследствие неортологичного замещения. В опероне утилизации сульфата нет АВС-транспортера, однако субстрат для алкансульфонат-монооксигеназы, возможно, поставляется одним из двух АВС-транспортеров (cg0735-cg0737 и cg2675-cg2678 в геноме C.glutamicum) с неизвестной специфичностью. Оба транспортера и регуляторные сигналы перед кодирующими их оперонами (см. ниже) сохраняются в геноме C.efficiens (фиг.4, позиция А).
2. Оперонные перестройки. Выше уже упоминалась ситуация с перестановкой генов в опероне thrABC. Аналогичные случаи характерны и для других оперонов. Так, гены гомосерин-ацетилтрансферазы (metX) и ацетилгомосерин-лиазы (metY) в разных геномах по-разному расположены относительно друг друга (фиг.4, позиция Г). В геноме C.glutamicum они находятся рядом, транскрибируются в одном направлении, расстояние между генами относительно невелико - 144 пары нуклеотидов и, тем не менее, гены разделены потенциальным терминатором [6]. В геномах C.diphtheriae и Nocardia farcinica эти гены также транскрибируются в одном направлении, однако уже сильно разнесены по геному. Для геномов C.efficiens и C.jeikeium характерно формирование единого оперона metXY. Кроме того, возможно и дивергентное расположение этих генов, когда metY (здесь cysD) расположен конвергентно с геном потенциальной дегидрогеназы, а дивергентно к ним расположен ген metX (здесь metA). Такая ситуация наблюдается в геноме Leifsoma xyli (фиг.4, позиция Г).
Внутриоперонные перестановки генов характерны и для оперона cg0735-cg0737, кодирующего АВС-переносчик неизвестного субстрата (фиг.4, позиция В). В геномах рода Corynebacterium (кроме C.diphtheriae) так же, как и в геномах Nocardia farcinica и Leifsonia xyli, структура оперона сохраняется, тогда как в геноме Streptomyces coelicolor он изменен на cg0737-cg0735-cg0736. В геноме C.diphtheriae оперон удлинен за счет появления паралога белка cg0737 перед опероном. Возможно, удлинение оперона наблюдается и на примере регулятора mcbR (см. ниже).
В уже упоминавшемся выше опероне утилизации сульфата с геноме Streptomyces coelicolor в центре оперона cysIXHCDN возникает новый член, cysC, потенциально кодирующий аденилилсульфат-киназу (фиг.4, позиция А). Происхождение этого белка не очевидно, поскольку его ортологи присутствуют только в геномах рода Streptomyces. В гомологичном опероне cysIXHDNYZB C.efficiens, помимо алкансульфонат-монооксигеназы (см. выше), появляется еще один новый потенциальный член регулона (фиг.4, позиция А), кодирующий метилтиоаденозин-нуклеозидазу (см. ниже). Ортологи этого белка среди рассматриваемых геномов находятся также в C.glutamicum и C.diphtheriae, где образуют моноцистронные опероны.
Оперон cg2679 C.glutamicum, дивергентно расположенный к оперону cg2674-cg2678 [6], может включать в себя и ген cg2680 (кодирующий аминотрансферазу), поскольку межгенное расстояние составляет всего 52 пары нуклеотидов и потенциального Rho-независимого терминатора между этими генами не было предсказано [6]. Дивергон cg2574-cg2680 консервативен только в геноме C.efficiens и полностью утрачен во всех остальных геномах. Только для гена cg2680 присутствует ортолог в геноме C.jeikeium, где он входит в состав другого регулируемого оперона (фиг.4, позиция Б), также включающего в себя aecD (цистатионин-бегалиаза) и ywjA (АТФаза АВС-транспортера). В геномах C.glutamicum и C.efficiens ортологи гена ywjA формируют моноцистронные нерегулируемые опероны, а ортологи гена aecD входят в состав нерегулируемого оперона, также включающего в себя ген brnQ (переносчик аминокислот с разветвленной цепью) и алканал-монооксигеназу, кодируемую геном сg2538 (фиг.4, позиция Б). В геноме С.diphtheriae ортолог гена ywjA входит в состав нерегулируемого оперона aecD-brnQ-DIP1738 [DIP 1738 кодирует алканал-монооксигеназу). В геноме C.jeikeium ортологичные гены алканал-монооксигеназы и транспортера аминокислот с разветвленной цепью формируют нерегулируемые моноцистронные опероны (фиг.4, позиция Б).
3. Паралоги и дупликации. Выше уже описана ситуация с появлением в С.diphtheriae паралога белка cg0737 в опероне, ортологичном cg0735-cg0737 C.glutamicum (фиг.4, позиция В). Кроме того, в геноме C.glufamicum произошла дупликация белка ssuD Nocardia farcinica, кодирующего алкансульфонат-монооксигеназу, с образованием паралогов ssuD1 и ssuD2, вероятно, выполняющих ту же функцию. Ген ssuDl входит в состав оперона ssuABC-D1, отвечающего за утилизацию сульфонатов. Ген ssuD2 включен в оперон утилизации сульфатных эфиров seuABC-ssuD1. Оба оперона полностью утрачены во всех исследованных геномах, кроме Nocardia farcinica, где, помимо белка ssuD, сохраняется и ортолог гена seuA, кодирующего фермент десульфуризации. Оба гена формируют оперон, в состав которого также, вероятно, входит fadE14, кодирующий потенциальную ацилКоА-дегидрогеназу.
Дивергентно к оперону утилизации сульфата (cysIXHDNYZ) расположен ген cysJ/fpr2 (сульфат-редуктаза), причем он консервативен во всех исследованных геномах, несмотря на то, что основной оперон может быть полностью утрачен (фиг.4, позиция А). Нужно отметить, что на эволюционном дереве (фиг.5) все ортологи расположены на одной ветке за исключением fpr белка Leifsonia xyli, который расположен ближе к Arthobacter и Brevibacterium. В каждом из исследованных геномов белок fpr представлен в виде одной копии, тогда как в геномах С.efficiens и C.glutamicum - в виде двух паралогов, fpr1 и frp2 (фиг.5). В каждом из двух геномов один из паралогов входит в состав дивергона утилизации сульфата, второй формирует моноцистронный нерегулируемый оперон.
Для того же оперона утилизации сульфата (cysIHDNY) в разных частях генома Nocardia farcinica наблюдается наличие двух дополнительных пар генов, ортологичных генам cysDN, кодирующим АТФ-сульфурилазу, - cysD2-N2 и nfa33930-nfa33920. На эволюционных деревьях белков CysN (фиг.6) и CysD (фиг.7) видно, что ближайшими гомологами белков CysD2 и CysN2 у Nocardia farcinica являются белки бактерий рода Mycobacterium и, по всей видимости, пара генов cysD2-N2 в геноме Nocardia farcinica появилась вследствие горизонтального переноса от других бактерий порядка Actinomycetales (вероятнее всего, от бактерий рода Mycobacterium). Аналогично, для белков, кодируемых генами nfa33930-nfa33920, ближайшими гомологами являются белки протеобактерий (фиг.6 и 7) и, скорее всего, пара генов nfa33930-nfa33920 в геноме Nocardia farcinica также появилась в результате горизонтального переноса, но в данном случае от геномов протеобактерий. В обоих случаях (cysD2-N2 и nfa33930-nfa33920) гены предположительно формируют опероны (о регуляции см. ниже).
4. Утрата ортологов. Полное отсутствие ортологов наблюдается в случае следующих генов C.glutamicum: ssuR (регулятор генов утилизации сульфонатов и сульфатных эфиров), cg3372 (белок с неизвестной функцией), ssul (флавин-редуктаза), seuBC и ssuABC (см. выше). Многие ортологи утрачены только в филогенетически более далеких геномах. Так, в геномах Nocardia farcinica, Leifsonia xyli и Streptomyces coelicolor отсутствуют ортологи генов cysK (ацетилсерин-лиаза), cysE (серин-ацетилтрансфераза), aecD (см. выше) и brnQ (см. выше) C.glutamicum и ортологи гена ywjA C.jeikeium. В далеких геномах также отсутствует ортолог гена cg1739 (см. ниже). Некоторые ортологи были утрачены не только в более далеких геномах, но также в геномах C.diphtheriae и C.jeikeium, - это почти все гены оперона, ортологичного оперону cg2674-2680 C.glutamicum (см. выше), и ортологи гена cg3132 с неизвестной функцией. В геномах всех далеких организмов и в C.jeikeium отсутствуют ортологи потенциального транскрипционного регулятора cg0156.
Среди более частных случаев можно выделить утрату ортологов генов seuA и ssuD (см. выше) во всех изученных геномах, кроме Nocardia farcinica. Также не найдены ортологи генов metX и metY в геноме Strepromyces coelicolor, гена metB (цистатионин-гамма-синтаза) в геноме C.diphtheriae, гена metH (гомоцистеин-метилтрансфераза) в геномах C.jeikeium и Leifsonia xyli, генов etfAB (флавопротеин) в геноме Leifsonia xyli.
Ортологи гена metE (гомоцистеин-метилтрансфераза) и оперон утилизации сульфата cysIXHDNYZ (фиг.4, позиция А) полностью утрачены в геномах C.diphtheriae и Leifsonia xyli. В состав оперона cysIXHDNYZ C.glutamicum входит короткий белок cysX (потенциально участвует в переносе электронов), ортологи которого сохраняются в геномах C.efficiens, C.jeikeium и Streptomyces coelicolor (фиг.4, позиция А). Среди геномов, сохранивших оперон утилизации сульфата, в геноме Streptomyces coelicolor отсутствуют ортологи генов cysYZ (cys Y принимает участие в биосинтезе кофактора, cysZ - потенциальная сульфат-пермеаза), а в геноме Nocardia farcinica отсутствуют ортологи белков cysX и cysZ.
5. Полная консервативность в структуре оперона во всех изученных геномах наблюдается только в случае гена metK (аденозилметионин-синтаза) и, возможно, гена mcbR (см. ниже).
В работе [6] были предсказаны регуляторные мотивы перед практически всеми членами регулона McbR в C.glutamicum и некоторые их них были подтверждены экспериментально. Был также представлен консенсус палиндромного сайта связывания регулятора вида TAGAC-N6-GTCTA. При осуществлении предлагаемого способа поиск по консенсусу подтвердил предсказание регуляторных сигналов перед ферментами, участвующими в биосинтезе метионина, и на основании предсказанных и подтвержденных экспериментально сигналов была составлена исходная обучающая выборка и построена матрица для поиска сайтов связывания McbR.
В геномах рода Corynebacterium проводился поиск сайтов с весом выше порогового значения 4,1. Все найденные потенциальные сайты связывания McbR представлены на фиг.3.
В геноме C.glutamicum, помимо уже предсказанных [6] сигналов, был обнаружен новый потенциальный сайт связывания McbR - перед геном cg1739, кодирующим белок с консервативным доменом глутамин-амидотрансферазы. Ортологи данного белка представлены во всех геномах рода Corynebacterium и отсутствуют в более филогенетически далеких организмах. Перед всеми ортологами этого белка был предсказан сайт связывания с весом выше заданного порогового значения (фиг.3), таким образом, согласно методу процедуры проверки соответствия ген cg1739 является новым потенциальным членом регулона McbR. Косвенным экспериментальным подтверждением этого предсказания является активация экспрессии этого гена в делеционном mcbR-мутанте C.glutamicum [6].
Перед основной частью регулируемых ортологов генов C.glutamicum в других геномах рода Corynebacterium также предсказаны потенциальные сайты связывания регулятора McbR (фиг.3). Так, сигнал сохраняется перед всеми найденными в этих геномах ортологами генов metK, metH, metE, metB, cysK и cg3132 и перед генами дивергона cg2674-cg2680.
Однако иногда даже в близких геномах рода Corynebacterium регуляторные сигналы отсутствуют перед ортологичными генами. Так, оперон hom-thrB, регулируемый в C.glutamicum, содержит потенциальные сигналы перед гомологичными оперонами только в геномах С.efficiens и C.jeikeium, но не в C.diphtheriae (фиг.4, позиция Д). Для гена cg0156 ситуация обратная: ортолог данного гена в C.diphtheriae сохраняет сайт связывания регулятора, тогда как ортолог в C.efficiens - нет (фиг.3). В случае оперона etfAB регуляция сохраняется только перед ортологичным опероном в геноме C.efficiens.
К более частным случаям относится регуляция генов гомосерин-ацетилтрансферазы (metX) и ацетилгомосерин-лиазы (metY). Гены metX и metY в геноме C.glutamicum расположены тандемно, но разделены потенциальным Rho-независимым терминатором [6] и перед каждым из генов находится сайт связывания McbR. В геноме C.diphtheriae эти гены формируют моноцистронные опероны, разнесенные по геному, перед каждым из генов сохраняется регуляторный сигнал (фиг.3). В геномах C.efficiens и C.jeikeium присутствует уже единый регулируемый бицистронный оперон metYX, сайт связывания McbR находится перед первым геном оперона metY (фиг.4, позиция Г).
В геноме C.diphtheriae перед опероном DIP0608-DIP0610, гомологичным оперону cg0735-cg0737 C.glitamicum, появляется паралог гена DIP0610-DIP0611, который образует независимый моноцистронный оперон. Сайты связывания регулятора расположены перед всеми ортологами гена cg0737 в геномах рода Corynebacterium, a также перед геном DJP0611 (фиг.4, позиция В, фиг.3).
Для транскрипционного регулятора MbR была предсказана и экспериментально подтверждена авторегуляция. Среди ортологов регулятора в геномах рода Corynebacterium авторегуляция сохраняется в геноме C.diphtheriae и утрачивается в геноме C.jeikeium. Для генома C.efficiens перед геном mcbR предсказана тандемная, перекрывающаяся с ним на 28 пар нуклеотидов, короткая открытая рамка считывания, кодирующая гипотетический белок из 57 аминокислотных остатков. Потенциальный сайт связывания mcbR при этом расположен в регуляторной области этого гипотетического гена. Однако для этого гипотетического белка не существует ни одного ортолога среди всех белков базы данных GenBank и, возможно, он является результатом перепредсказания при картировании генома С.efficiens. Если это верно, оперон mcbR полностью консервативен во всех исследованных геномах и сайт связывания регулятора в геноме C.efficiens расположен на расстоянии - 143 пар нуклеотидов от точки старта трансляции гена mcbR.
Выше были описаны оперонные перестройки ортологов, в которых участвуют ортологи генов aecD, brnQ и монооксигеназы C.glutamicum и ортологи гена ywjA C.jeikeium (фиг.4, позиция Б). В геномах C.glutamicum, C.diphtheriae и C.efficiens все эти гены (и их ортологи) не регулируются McbR. В геноме C.jeikeium гены, кодирующие монооксигеназу и белок brnQ, формируют два нерегулируемых моноцистронных оперона, тогда как гены aecD, ywjA и ортолог гена cg2680 C.glutamicum находятся в едином, потенциально регулирующемся опероне - перед геном aecD находится предсказанный сайт связывания McbR (фиг.4, позиция Б, фиг.3).
Дивергоны, гомологичные дивергону утилизации сульфата cysJIXHDNZY C.glutamicum, сохраняют регуляцию со стороны McbR в геномах C.efficiens и C.jeikeium, в которых этот дивергон сохранился (фиг.4, позиция А). Структура оперона консервативна во всех трех геномах, однако межгенные расстояния отличаются. В геномах C.glutamicum и C.efficiens расстояние между генами cysXvi cysH составляет - 3 пары нуклеотидов, тогда как в геноме C.jeikeium - 247 пар нуклеотидов. То, что гены cysHDNYZ образуют самостоятельный оперон, подтверждается наличием перед геном cysH потенциального сайта связывания McbR (фиг.4, позиция А, фиг.3). Тем самым гены утилизации сульфата в C.jeikeium образуют дивергон cysJIX и оперон cysHDNYZ.
Как уже упоминалось, в геноме C.efficiens оперон cysIXHDNZY включает два дополнительных члена, кодирующих алкансульфонат-монооксигеназу (СЕ2637) и метилтиоаденозин-нуклеозидазу (СЕ2636). Ген СЕ2637 не имеет ортологов ни в одном из рассматриваемых геномов (см. выше). Ортологи гена СЕ2636 присутствуют в геномах C.glutamicum и C.diphtheriae в виде моноцистронных оперонов, причем перед ортологом в геноме C.diphtheriae находится сайт связывания McbR (фиг.4, позиция А, фиг.3). Регуляторная область перед ортологичным геном в геноме C.glutamicum также содержит потенциальный сайт связывания, но вес данного сигнала ниже заданного порогового значения и составляет 3,79. Таким образом, принадлежность гена СЕ2636 регулону McbR является сильно гипотетической, хотя и заслуживающей экспериментальной проверки.
Матрица позиционных весов, созданная на основе сайтов связывания McbR в геноме C.glutamicum, позволяла проводить поиск регуляторных сайтов в геномах рода Corynebacterium. Однако при поиске сигналов в более филогенетически далеких геномах эта матрица находила только один сильный сайт связывания регулятора (вес сайта 5,02) перед генами исследуемого метаболического пути, - сайт авторегуляции mcbR в геноме Nocardia farcinica (фиг.3). Все остальные гены пути биосинтеза метионина в этих геномах имеют сигналы со слишком слабым весом (меньше 3,0). Возможное объяснение этому наблюдению может состоять в том, что у регулятора мог измениться ДНК-связывающий домен и, соответственно, распознаваемый им сигнал. Для поиска сигнала, специфичного для более далеких геномов, в каждом геноме проверялись следующие предположения:
- возможность небольшого изменения сигнала. Была сделана попытка построить новую матрицу на основании выборки тех слабых сайтов, которые распознавала исходная матрица;
- возможность значительного изменения сигнала. Учитывая принадлежность McbR к TetR-семейству транскрипционных регуляторов, можно было предположить, что структура сигнала осталась палиндромной. Был проведен поиск более или менее строгого палиндромного сигнала разной четности и длины в 5'-областях основных и, отдельно, "вторичных" генов метаболического пути.
Ни один из подходов к созданию новой матрицы не дал результатов. Возможным объяснением этому является крайне высокий GC состав всех трех далеких геномов: средний уровень GC-пар по геному составляет в среднем 70% (для C.glutamicum - 54%), что значительно затрудняет поиск специфичного сигнала.
В итоге проанализирована структура и потенциальная регуляция регулона McbR во всех семи геномах актинобактерий, в которых сохраняются близкие ортологи данного регулятора.
Несмотря на невозможность изучения регуляции в филогенетически более далеких геномах вследствие значительно повышенного GC-состава этих геномов, исследование потенциальных сигналов в четырех геномах рода Corynebacterium позволяет заключить, что большинство генов регулона McbR сохраняет регуляцию со стороны этого репрессора. В большей части случаев потенциальный сигнал сохраняется перед исследуемым ортологичным геном, однако в некоторых ситуациях вследствие различного относительного расположения генов потенциальный сайт связывания может находиться в регуляторной области другого гена. Например, гены metX и metY могут формировать независимо регулирующиеся моноцистронные локусы или же единый оперон с сигналом перед геном metY (фиг.4, позиция Г), а дивергон утилизации сульфата cysJIHDNZY C.glutamicum в геноме C.jeikeium разрывается на независимо регулирующиеся дивергон cysJIX и оперон cysHDNYZ (фиг.4, позиция А).
Позиционное сравнение и исследование регуляции ортологичных регулонов позволили прогнозировать наличие нового потенциального члена регулона McbR в геномах рода Corynebacterium, - ген cg1739 C.glutamicum. Поиск консервативных доменов в GenBank выявляет сходство кодируемого белка с доменом глутамин-амидотрансферазы guaA Bacillus subtilis [1312531] и отдаленную гомологию белка с гомосерин-сукцинилтрансферазным доменом. Гомосерин-сукцинилтрансфераза выполняет функцию, аналогичную функции гомосерин-ацетилтрансферазы в геномах некоторых бактерий, таких как Escherichia coli и Pseudomonas aeruginosa [Lee H.S., Hwang B.J. 2003. Methionine biosynthesis and its regulation in Corynebacterium glutamicum: parallel pathways oftranssulfuration and direct sulfhydrylation. Appl. Microbiol. Biotechnol. 62, 459-467]. Одновременное наличие этих двух ферментов еще не было показано и, более того, было экспериментально показано, что единственным ферментом утилизации гомосерина в геноме C.glutamicum является гомосерин-ацетилтрансфераза [Park S.D., Lee J.Y., Kim Y., Kim J.H., Lee H.S. 1998. Isolation and analysis of metA, a methionine biosynthetic gene encoding homoserine acetyltransferase in Corynebacterium glutamicum. Mol. Cells. 8, 286-294.]. Возможная роль белка, гомологичного белку guaA B.subtilis, в биосинтезе метионина также не очевидна. Функция этого фермента состоит в гидролизе иона аммония (NH4+) от молекулы глутамина с образованием молекулы глутамата и последующем присоединении иона аммония к акцепторной молекуле. Объяснением роли данной ферментативной активности в биосинтезе метионина/цистеина могла бы явиться возможная регуляция биосинтеза на стадии образования аспартата из глутамата и оксалоцетата. Но такая интерпретация представляется маловероятной в силу того, что глутамат вовлечен в большую часть процессов биосинтеза и регуляция его уровня в клетке со стороны специфичного регулятора биосинтеза метионина не целесообразна.
Кроме того, полученные результаты свидетельствуют о том, что белок СЕ2636р C.efficiens и его ортологи в C.glutamicum и C.diphtheriae также могут входить в регулон McbR. Этот белок имеет значительную степень сходства с представителями семейства нуклеозид-фосфорилаз, которое включает в себя 5'-метилтиоаденозин-нуклеозидазы (МТА-нуклеозидазы). В процессе биосинтеза полиаминов одним из побочных продуктов является МТА. Процесс восстановления метионина из МТА состоит из нескольких реакций, первой и лимитирующей из которых является расщепление МТА на аденин и 5'-метилтиорибозу, катализируемое МТА-нуклеозидазами. Таким образом, увеличение пула метионина за счет МТА потенциально также находится под регуляцией со стороны McbR, по крайней мере, в геномах C.diphtheriae и C.efficiens.
Несмотря на общую высокую стабильность геномов коринебактерий [Nakamura Y., Nishio Y,, Ikeo K,, Gojobori Т. 2003. The genome stability in Corynebacterium species due to lack of the recombinational repair system. Gene. 317, 149-155], во многих случаях фиксировалась утрата ортологичных генов как в далеких, так и в близкородственных геномах. Большая часть таких ситуаций связана с особенностями паразитического образа жизни, характерного для четырех микроорганизмов из описываемых семи, - C.diphtheriae, C.jeikeium, Nocardia farcinica и Leifsonia xyli. По-видимому, эти микроорганизмы способны получать серосодержащие органические вещества из организмов хозяев, в силу чего отсутствует острая необходимость в ферментах пути биосинтеза метионина, и, как следствие, путь биосинтеза метионина в этих микроорганизмах редуцирован. Так, к примеру, в геноме C.diphtheriae полностью утрачен оперон утилизации сульфата и оба оперона утилизации алкансульфонатов, что только частично возмещается появлением новой алкансульфонат-монооксигеназы в результате неортологичного замещения. Кроме того, утрачен ген metB (цистатионин гамма-синтаза), кодирующий первый фермент пути транс-сульфурилирования, и единственно возможным способом синтеза гомоцистеина остается путь прямого сульфгидрилирования с участием белка MetY (ацетилгомосерин-лиаза). Утрачен ген metE, кодирующий высокоэффективную метионин-синтазу. Тем не менее, путь биосинтеза метионина остается функциональным благодаря наличию метионин-синтазы MetH, эффективность которой ниже.
Позиционное сравнение генов биосинтеза аминокислот в геномах C.glutamicum, C.efficiens и C.diphtheriae выявило отсутствие в геноме C.diphtheriae большого количества ортологов, что привело к заметно меньшему размеру генома этой бактерии. Среди изученных микроорганизмов для ведущих паразитический образ жизни также характерен заметно уменьшенный размер генома (за исключением генома Nocardia farcinica), что может свидетельствовать об аналогичной массовой утрате ортологов и, возможно, косвенно свидетельствовать об использовании необходимых соединений организма хозяина.
В некоторых случаях отсутствие ортологов возмещается наличием гомологичных, но не ортологичных белков, как это было описано для функций треонин-синтазы и алкансульфонат-монооксигеназы. Так, геномы C.glutamicum, C.efficiens и C.diphtheriae несут треонин-синтазу, не характерную для большинства актиномицет, полученную, по всей видимости, общим предком этих трех бактерий за счет горизонтального переноса. Более частным аналогичным случаем горизонтального переноса генов является появление в геноме C.efficiens алкансульфонат-монооксигеназы, характерной для протеобактерий.
Применение предлагаемого способа к регулону mcbR продемонстрировало многочисленные и разнообразные оперонные перестройки, несмотря на показанную ранее геномную стабильность, и, кроме того, позволило прогнозировать и предъявить несколько новых потенциальных членов данного регулона на основании позиционного сравнения геномов и исследования сайтов связывания регулятора mcbR. Большинству новых потенциальных членов регулона ранее были приписаны ферментативные активности, которые позволили предсказать роли кодируемых белков в пути биосинтеза метионина в геномах порядка Actinomycetales.
Таким образом, предлагаемое изобретение позволяет решить задачу расширения функциональных возможностей способа прогнозирования, увеличения его оперативности, а также, в перспективе, уменьшения затрат на промышленное производство метионина.
Источники информации
1. Nishio Y., Nakamura Y., Usuda Y., Sugimoto S., Matsui K., Kawarabayasi Y., Kikuchi H., Gojobori Т., Ikeo K. 2004. Evolutionary process of amino acid biosynthesis in Corynebacterium at the whole genome level. Mol. Biol. Evol. 21, 1683-1691.
2. Lee H.S., Hwang B.J. 2003. Methionine biosynthesis and its regulation in Corynebacterium glutamicum: parallel pathways oftranssulfuration and direct sulfhydrylation. Appl. Microbiol. Biotechnol. 62, 459-467.
3. Hwang B.J., Yeom H.J., Kim Y., Lee H.S. 2002. Corynebacterium glutamicum utilizes both transsulfuration and direct sulfhydrylation pathways for methionine biosynthesis. J. Bacteriol. 184, 1277-1286.
4. Mampel J., Schroder H., Haefner S., Sauer U. 2005. Single-gene knockout of a novel regulatory element confers ethionine resistance and elevates methionine production in Corynebacterium glutamicum. Appl. Microbiol. Biotechnol. 68, 228-236.
5. Rey D.A., Puhler A., Kalinowski J. 2003. The putative transcriptional represser McbR, member of the TetR-family, is involved in the regulation of the metabolic network directing the synthesis of sulfur containing amino acids in Corynebacterium glutamicum. J. Biotechnol. 103, 51-65.
6. Rey D.A., Nentwich S.S., Koch D.J., Ruckert C., Puhler A., Tauch A., Kalinowski J. 2005. The McbR repressor modulated by the effector substance S-adenosylhomocysteine controls directly the transcription of a regulon involved in sulphur metabolism of Corynebacterium glutamicum ATCC 13032. Mol. Microbiol. 56, 871-887.
Изобретение относится к области биоинформатики и может быть использовано в фармацевтической и биотехнологической отраслях промышленности. Пути биосинтеза метионина в родственных геномах коринебактерий прогнозируют на основе результатов поиска потенциальных сайтов связывания регулона McbR среди предсказанных ранее и подтвержденных экспериментально регуляторных областей перед генами ферментов, участвующих в биосинтезе метионина, и ортологов белков пути биосинтеза метионина в геномах коринебактерий, а также определения состава оперонов. Указанные результаты представляют в виде таблицы соответствия генов потенциальным сайтам связывания регулона McbR и родственным геномам коринебактерий и диаграммы структуры оперонов регулона McbR. А участников и направления пути биосинтеза метионина в родственных геномах коринебактерий прогнозируют путем позиционного сравнения содержания указанных таблицы и диаграммы. Применение изобретения позволяет увеличить оперативность прогнозирования пути биосинтеза метионина в коринебактериях и уменьшить затраты на промышленное производство метионина. 3 з.п. ф-лы, 7 ил.
а) составление исходной обучающей выборки, для чего формируют полные последовательности геномов коринебактерий на основе базы данных Gen Bank и предсказанных ранее и подтвержденных экспериментально регуляторных областей перед генами ферментов, участвующих в биосинтезе метионина, и записывают ее на информационный носитель;
б) введение в компьютер записанной на информационный носитель исходной обучающей выборки и обработка ее заранее записанной в память компьютера программой SignalX для построения матриц позиционных весов нуклеотидов;
в) поиск потенциальных сайтов связывания регулятора McbR и предсказание состава оперонов, если оперонная структура неизвестна, путем обработки исходной обучающей выборки заранее записанными в память компьютера алгоритмами из пакета программ Genome Explorer, причем область поиска потенциальных сайтов связывания ограничивают диапазоном -400...+100 пар нуклеотидов относительно старта трансляции гена, а гены считают принадлежащими оперону в случае одинакового направления считывания и расстояния между ними не более 100 пар нуклеотидов;
г) записывание результатов, полученных на стадии по п/п (в) в память компьютера;
д) поиск ортологов белков пути биосинтеза метионина во всех родственных геномах с использованием пакета программ GenomeExplorer, представление результатов этого поиска и стадии по п/п (в) в двух выходных текстовых файлах, первый из которых выводят на информационный носитель в виде таблицы соответствия генов пути биосинтеза метионина потенциальным сайтам связывания регулона McbR и родственным геномам коринебактерий, а второй - в виде диаграммы структуры оперонов регулона McbR;
д) прогнозирование участников и направления пути биосинтеза метионина в родственных геномах коринебактерий на основе позиционного сравнения содержания обоих выходных текстовых файлов с учетом результатов поиска регуляторных сайтов связывания в более филогенетически отдаленных геномах, для первого выходного текстового файла, и с учетом характерных эволюционных событий формирования структуры оперонов, для второго выходного текстового файла.
| J | |||
| Biotechnol | |||
| Способ и приспособление для нагревания хлебопекарных камер | 1923 |
|
SU2003A1 |
| Mol Microbiol | |||
| Способ обработки целлюлозных материалов, с целью тонкого измельчения или переведения в коллоидальный раствор | 1923 |
|
SU2005A1 |
| Appl Microbiol Biotechnol | |||
| Способ и приспособление для нагревания хлебопекарных камер | 1923 |
|
SU2003A1 |
| Mol Biol Evol | |||
| Способ приготовления мыла | 1923 |
|
SU2004A1 |
| RU 2002102452 A, 20.08.2003 | |||
| СПОСОБ ПОЛУЧЕНИЯ АНСОЛЬВО-КИСЛОТЫ | 0 |
|
SU185915A1 |
| US 2005026189, 03.02.2005 | |||
| WO 03027262, 04.03.2003. | |||
Авторы
Даты
2007-09-27—Публикация
2006-07-27—Подача