ОБЛАСТЬ ТЕХНИКИ
Описание настоящего изобретения относится к микроорганизму, продуцирующему L-аминокислоту или ее предшественник, и к способу получения L-аминокислоты или ее предшественника с использованием этого микроорганизма.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
L-аминокислоты, основные составляющие единицы белка, используют в качестве главных сырьевых материалов в лекарственных средствах, пищевых добавках, кормах для животных, питательных добавках, пестицидах, стерилизаторах и тому подобном. Были проведены обширные исследования по разработке микроорганизмов и способов ферментации для получения L-аминокислот и других полезных веществ с высокими выходами. Например, главным образом использовали специфические в отношении мишени подходы, такие как способ повышения экспрессии гена, кодирующего фермент, вовлеченный в биосинтез L-лизина, и способ удаления гена, не являющегося необходимым для биосинтеза (патент Кореи №10-0838038).
В то же время, штаммы рода Corynebacterium, в частности Corynebacterium glutamicum, представляют собой грамположительные микроорганизмы, широко используемые для получения L-аминокислот и других полезных веществ. Были выполнены интенсивные исследования по разработке микроорганизмов и способов ферментации для получения аминокислот с высокими выходами. Например, широко использовали специфические в отношении мишени подходы, такие как способ повышения экспрессии гена, кодирующего фермент, вовлеченный в биосинтез аминокислот, или способ удаления гена, не являющегося необходимым для биосинтеза, в штаммах рода Corynebacterium (публикации патентов Кореи №№10-0924065 и 10-1208480). В дополнение к этим способам также был использован способ удаления гена, не вовлеченного в продуцирование аминокислот, и способ удаления гена, конкретные функции которого в отношении продуцирования аминокислот неизвестны. Тем не менее еще существует потребность в исследованиях способов эффективного получения L-аминокислот с высокими выходами.
ОПИСАНИЕ ВОПЛОЩЕНИЙ ИЗОБРЕТЕНИЯ ТЕХНИЧЕСКАЯ ЗАДАЧА
Авторы настоящего изобретения приложили интенсивные усилия к разработке микроорганизма, способного продуцировать L-аминокислоты с высокими выходами, и обнаружили, что продуктивность в отношении L-аминокислот можно повысить путем введения в него белка, имеющего происхождение из другого микроорганизма, и, таким образом, было выполнено настоящее изобретение.
РЕШЕНИЕ ЗАДАЧИ
Задачей настоящего изобретения является обеспечение микроорганизма, продуцирующего L-аминокислоту или ее предшественник, модифицированного для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента.
Другой задачей настоящего изобретения является обеспечение композиции для получения L-аминокислоты или ее предшественника, содержащей микроорганизм, модифицированный для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента, или указанный белок.
Еще одной задачей настоящего изобретения является обеспечение способа получения L-аминокислоты или ее предшественника, включающего: культивирование указанного микроорганизма в культуральной среде; и выделение L-аминокислоты или ее предшественника из культивированного микроорганизма или из культуральной среды.
Еще одной задачей настоящего изобретения является обеспечение применения белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента, для повышения продуцирования L-аминокислоты или ее предшественника.
ПРЕДПОЧТИТЕЛЬНЫЕ ЭФФЕКТЫ ОПИСАНИЯ ИЗОБРЕТЕНИЯ
Микроорганизм в соответствии с настоящим изобретением, продуцирующий L-аминокислоту или ее предшественник, где микроорганизм модифицирован для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента, может продуцировать L-серин, L-триптофан, L-гистидин, L-метионин, L-цистеин и/или О-фосфосерин.
ЛУЧШИЙ ВАРИАНТ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Далее настоящее изобретение будет описано более подробно.
При этом каждое описание и воплощение изобретения, представленное в описании настоящего изобретения, может быть применено в настоящем документе к другим описаниям и воплощениям. Иными словами, все комбинации различных компонентов, раскрытых в описании настоящего изобретения, включены в объем описания настоящего изобретения. Кроме того, объем описания настоящего изобретения не должен быть ограничен представленными ниже подробными описаниями.
Дополнительно специалисты в данной области техники, используя обычные эксперименты, могут распознать или подтвердить многие эквиваленты конкретных аспектов настоящего изобретения. Такие эквиваленты предназначены для включения в объем настоящего изобретения.
Для достижения описанных выше объектов аспект настоящего изобретения обеспечивает микроорганизм, продуцирующий L-аминокислоту или ее предшественник, где указанный микроорганизм модифицирован для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента.
Белок, содержащий аминокислотную последовательность SEQ ID NO: 1, или его функциональный фрагмент может представлять собой белок, обладающий активностью D-3-фосфоглицератдегидрогеназы.
В описании настоящего изобретения «D-3-фосфоглицератдегидрогеназа» представляет собой фермент, преимущественно катализирующий описанные ниже химические реакции.
3-фосфо-D-глицерат+NAD+ (никотинамидадениндинуклеотид)↔3-фосфонооксипируват+NADH+Н+
2-гидроксиглутарат+NAD+↔2-оксоглутарат+NADH+Н+
Для задачи описания настоящего изобретения D-3-фосфоглицератдегидрогеназа может представлять собой SerA, и ее последовательность может быть идентифицирована в известной базе данных Genbank NCBI (Национального института биотехнологической информации). Дополнительно можно также, без ограничений, использовать любой другой белок, обладающий эквивалентной ей активностью и выделенный из микроорганизмов, которые отличаются от описанного выше микроорганизма, продуцирующих L-аминокислоту или ее предшественник и включающих этот белок.
Конкретно белок может представлять собой белок, содержащий аминокислотную последовательность SEQ ID NO: 1, и может, без ограничений, использоваться взаимозаменяемо с белком, в состав которого входит аминокислотная последовательность SEQ ID NO: 1, белком, состоящим из аминокислотной последовательности SEQ ID NO: 1, или белком, имеющим аминокислотную последовательность SEQ ID NO: 1.
Белок может иметь аминокислотную последовательность SEQ ID NO: 1 и/или по меньшей мере 70%, 80%, 85%, 90%, 95%, 96%, 97%, 98% или 99% гомологии или идентичности с аминокислотной последовательностью SEQ ID NO: 1. Дополнительно будет очевидно, что любой акцессорный белок, имеющий аминокислотную последовательность, включающую делецию, модификацию, замену или добавление одной или нескольких аминокислот, находятся в пределах объема настоящего изобретения, если эта аминокислотная последовательность сохраняет описанную выше гомологию или идентичность и эффект, эквивалентный эффекту указанного белка.
В дополнение к этому можно также, без ограничений, использовать любой полипептид, обладающий активностью D-3-фосфоглицератдегидрогеназы и кодируемый полинуклеотидом, который гибридизуется в жестких условиях с зондом, сконструированным с использованием известных генетических последовательностей, например нуклеотидной последовательности, полностью или частично комплементарной нуклеотидной последовательности, кодирующей полипептид.
Дополнительно для задач описания настоящего изобретения белок может иметь происхождение из других микроорганизмов, отличающихся от описанного выше микроорганизма, продуцирующих L-аминокислоту или ее предшественник и включающих белок, и конкретно этот белок может, без ограничений, представлять собой белок, имеющий происхождение из рода Azotobacter, белок, идентичный белку, имеющему происхождение из рода Azotobacter, или любой белок, способный к повышению продуцирования L-аминокислоты или ее предшественника. Более конкретно микроорганизм рода Azotobacter может представлять собой Azotobacter agilis, Azotobacter armeniacus, Azotobacter beijerinckii, Azotobacter chroococcum, Azotobacter sp.DCU26, Azotobacter sp.FA8, Azotobacter nigricans, Azotobacter paspali, Azotobacter salinestris, Azotobacter tropicalis или Azotobacter vinelandii, и в воплощении настоящего изобретения может представлять собой белок, имеющий происхождение из Azotobacter vinelandii, но микроорганизм не ограничен этим.
Используемый в настоящем документе термин «функциональный фрагмент» относится к аминокислотной последовательности, обладающей эффектом, эквивалентным эффекту указанного белка, и очевидно, что любой белок, имеющий аминокислотную последовательность, включающую делецию, модификацию, замену или добавление одной или нескольких аминокислот, и сохраняющий эффект, эквивалентный эффекту указанного белка, находится в пределах объема настоящего изобретения и может быть рассмотрен как функциональный фрагмент для задач описания настоящего изобретения.
Хотя в настоящем документе используется выражение «белок или полипептид, содержащий аминокислотную последовательность конкретной SEQ ID NO:», «белок или полипептид, состоящий из аминокислотной последовательности конкретной SEQ ID NO:» или «белок или полипептид, имеющий аминокислотную последовательность конкретной SEQ ID NO:», очевидно, что в описании настоящего изобретения можно также использовать любой белок, имеющий аминокислотную последовательность, включающую делецию, модификацию, замену, консервативную замену или добавление одной или нескольких аминокислот, если этот белок обладает активностью, идентичной или эквивалентной полипептиду, состоящему из аминокислотной последовательности конкретной SEQ ID NO:. Например, белок может иметь добавление последовательности на N-конце и/или С-конце аминокислотной последовательности, не вызывающее изменения функций белка, природную мутацию, молчащую мутацию или консервативную замену.
Термин «консервативная замена» относится к замене одной аминокислоты на другую аминокислоту, обладающую сходными структурными и/или химическими свойствами. Такая замена аминокислоты может, как правило, произойти на основе сходства полярности, заряда, растворимости, гидрофобности, гидрофильности и/или амфипатической природы остатка. Например, положительно заряженные (основные) аминокислоты включают аргинин, лизин и гистидин; отрицательно заряженные (кислые) аминокислоты включают глутаминовую кислоту и аспарагиновую кислоту; ароматические аминокислоты включают фенилаланин, триптофан и тирозин; и гидрофобные аминокислоты включают аланин, валин, изолейцин, лейцин, метионин, фенилаланин, тирозин и триптофан.
Другой аспект настоящего изобретения обеспечивает полинуклеотид, кодирующий белок, содержащий аминокислотную последовательность SEQ ID NO: 1.
Используемый в настоящем документе термин «полинуклеотид» имеет широкое значение, включая молекулы ДНК и РНК, и нуклеотид, который является основным структурным звеном в полинуклеотиде, может включать не только природный нуклеотид, но также аналог, в котором сахар или азотистое основание модифицированы (Scheit, Nucleotide Analogs, John Wiley, New York (1980); Uhlman and Peyman, Chemical Reviews, 90:543-584 (1990)).
Полинуклеотид, кодирующий белок, содержащий аминокислотную последовательность SEQ ID NO: 1, может иметь, без ограничений, любую последовательность, способную кодировать белок, обладающий активностью D-3-фосфоглицератдегидрогеназы, имеющий происхождение из Azotobacter vinelandii. Альтернативно, полинуклеотид может иметь, без ограничений, любую последовательность, кодирующую белок, обладающий активностью повышения продуцирования L-аминокислоты или ее предшественника, содержащий аминокислотную последовательность SEQ ID NO: 1.
Полинуклеотид может, например, представлять собой полинуклеотид, кодирующий полипептид, имеющий по меньшей мере 70%, 75%, 80%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% гомологии с аминокислотной последовательностью SEQ ID NO: 1. Конкретно, например, полинуклеотид, кодирующий белок, содержащий аминокислотную последовательность SEQ ID NO: 1 или аминокислотную последовательность, имеющую по меньшей мере 70% гомологии или идентичности с аминокислотной последовательностью SEQ ID NO: 1, может представлять собой полинуклеотидную последовательность SEQ ID NO: 95 или полинуклеотид, имеющий по меньшей мере 70%, 75%, 80%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98% или 99% гомологии или идентичности последовательности с нуклеотидной последовательностью SEQ ID NO: 95.
В дополнение к этому очевидно, что полинуклеотид может также представлять собой полинуклеотид, который может транслироваться в белок, содержащий аминокислотную последовательность SEQ ID NO: 1 или аминокислотную последовательность, имеющую по меньшей мере 70% гомологии или идентичности с аминокислотной последовательностью SEQ ID NO: 1, или в белок, имеющий гомологию или идентичность с ней за счет вырожденности кодонов. Альтернативно, полинуклеотид может иметь, без ограничений, нуклеотидную последовательность, которая может гибридизоваться с зондом, сконструированным с использованием известных генетических последовательностей, например нуклеотидной последовательности, полностью или частично комплементарной этой нуклеотидной последовательности, в жестких условиях, чтобы кодировать белок, содержащий аминокислотную последовательность, имеющую 70% идентичности с SEQ ID NO: 1. Термин «жесткие условия» относится к условиям, дающим возможность для специфичной гибридизации между полинуклеотидами. Такие условия подробно описаны в известных документах (например, J. Sambrook et al., Molecular Cloning, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York). Например, жесткие условия могут включать условия, в которых гены, имеющие высокую гомологию или идентичность, например по меньшей мере 70%, 80%, конкретно 85%, конкретно 90%, более конкретно 95%, более конкретно 97% или еще более конкретно 99% гомологии или идентичности, гибридизуются друг с другом, при этом гены, имеющие более низкую гомологию или идентичность, чем описано выше, не гибридизуются друг с другом; или условия, в которых отмывку осуществляют один раз, а конкретно два или три раза, в обычных условиях отмывки для гибридизации по Саузерну при концентрации соли и температуре, соответствующих 60°С, 1×SSC (буфер физиологический раствор-цитрат натрия), 0,1% SDS (додецилсульфат натрия), конкретно 60 °С, 0.1×SSC, 0,1% SDS, и более конкретно 68°С, 0.1×SSC, 0,1% SDS. Для гибридизации требуется, чтобы два полинуклеотида имели комплементарные последовательности, хотя из-за жестких условий гибридизации азотистые основания могут не совпадать. Термин «комплементарный» используют для описания соотношения между азотистыми основаниями нуклеотидов, способных гибридизоваться друг с другом. Например, в отношении ДНК, аденозин комплементарен тимину, а цитозин комплементарен гуанину. Таким образом, настоящее изобретение может включать не только по существу схожие полинуклеотидные последовательности, но также их выделенный полинуклеотидный фрагмент, комплементарный полноразмерной последовательности.
Конкретно полинуклеотид, имеющий гомологию или идентичность, можно выявлять, используя описанные выше условия гибридизации, включая процесс гибридизации со значением Tm (температуры плавления) 55°С. Дополнительно значение Tm может составлять 60°С, 63°С или 65°С, но не ограничено ими, и может быть соответствующим образом скорректировано специалистами в данной области техники в соответствии с задачей.
«Гомология» и «идентичность» относятся к степени соответствия между двумя аминокислотными последовательностями или нуклеотидными последовательностями и могут быть выражены в процентах.
Термины «гомология» и «идентичность» могут часто быть использованы взаимозаменяемо.
Гомологию или идентичность последовательности консервативных полинуклеотидов или полипептидов можно определить с использованием стандартного алгоритма выравнивания, и вместе с этим могут быть использованы установленные программой штрафы за пробелы по умолчанию. По существу гомологичные или идентичные последовательности могут обычно гибридизоваться друг с другом по всей длине последовательности или по меньшей мере 50%, 60%, 70%, 80% или 90% полноразмерной последовательности в условиях средней или высокой степени жесткости. В гибридизуемых полинуклеотидах могут также рассматриваться полинуклеотиды, включающие вырожденный кодон вместо кодона.
Гомологию или идентичность между полипептидами или полинуклеотидными последовательностями можно определить с использованием любого известного в данной области техники алгоритма, например BLAST (см.: Karlin and Altschul, Proc. Natl. Acad. Sci. USA, 90, 5873, (1993)) или FASTA, введенного Пирсоном (см.: Methods Enzymol., 183, 63, 1990). На основании алгоритма BLAST разработаны программы, известные как BLASTN или BLASTX (см.: http://www.ncbi.nlm.nih.gov). В дополнение к этому наличие гомологии, сходства или идентичности между аминокислотными или полинуклеотидными последовательностями можно подтверждать путем сравнения этих последовательностей посредством экспериментов гибридизации по Саузерну в определенных жестких условиях, и эти определенные жесткие условия гибридизации находятся в пределах объема технологии объекта изобретения и могут быть определены способом, известным среднему специалисту в данной области техники (например, J. Sambrook et al., Molecular Cloning, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York).
Используемый в настоящем документе термин
«экспрессироваться/экспрессируемый» в отношении белка означает состояние, в котором целевой белок вводят в микроорганизм, либо в случае присутствия этого белка в микроорганизме активность этого белка усиливается по сравнению с его эндогенной активностью или активностью до модификации.
Конкретно, термин «введение белка» относится к обеспечению активности конкретного белка в микроорганизме, который исходно не имеет этого белка, или к усилению активности белка по сравнению с его эндогенной активностью или активностью до модификации. Например, введение белка может относиться к введению кодирующего конкретный белок полинуклеотида в хромосому или введение в микроорганизм фрагмента или вектора, включающего в себя кодирующий конкретный белок полинуклеотид, тем самым способного к экспрессии активности этого белка. «Эндогенная активность» относится к активности белка, которой исходно обладает родительский штамм микроорганизма до трансформации, если микроорганизм трансформируют посредством генетической модификации, вызванной естественным или искусственным фактором.
При использовании в настоящем документе термин «аминокислота или ее предшественник» относится к аминокислоте или ее предшественнику, которые могут быть продуцированы с использованием указанного белка и могут включать серин, триптофан, гистидин, метионин, цистеин, L-цистатионин, L-гомоцистеин, О-ацетилгомосерин, О-сукцинилгомосерин, L-гомосерин и/или О-фосфосерин, без ограничения ими. В описании настоящего изобретения аминокислота может представлять собой L-аминокислоту, конкретно L-серин, L-триптофан, L-гистидин, L-метионин или L-цистеин, но может включать все L-аминокислоты, продуцируемые микроорганизмами из различных источников углерода посредством метаболических процессов. Предшественник может представлять собой О-ацетилгомосерин или О-сукцинилгомосерин, который представляет собой предшественник, конвертируемый в метионин посредством О-ацетилгомосеринсульфгидрилазы (KR10-1048593); L-гомосерин, L-гомоцистеин или L-цистатионин, который представляет собой предшественник метионина; и ацетилсерин, который представляет собой предшественник L-цистеина; и/или О-фосфосерин, который представляет собой предшественник, конвертируемый в цистеин посредством О-фосфосеринсульфгидрилазы, без ограничения ими. Более конкретно, аминокислота или ее предшественник могут представлять собой L-серин, L-триптофан, L-гистидин, L-метионин, О-фосфосерин или L-цистеин, но не ограничены ими.
Для усиления биосинтеза L-аминокислот или их предшественников можно использовать белок, содержащий аминокислотную последовательность SEQ ID NO: 1 или ее функциональный фрагмент в соответствии с описанием настоящего изобретения. Например, для усиления биосинтеза L-серина, L-триптофана, L-гистидина, L-метионина, L-цистеина, L-гомоцистеина, L-цистатионина, ацетилсерина, О-ацетилгомоцистеина, О-сукцинилгомосерина, L-гомосерина и/или О-фосферина микроорганизм можно модифицировать для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функциональный фрагмент в соответствии с описанием настоящего изобретения. В качестве конкретного примера, можно вводить белок, содержащий аминокислотную последовательность SEQ ID NO: 1, либо можно усиливать активность указанного белка. Дополнительно способность к продуцированию L-аминокислоты или ее предшественника можно дополнительно усилить путем дополнительного введения или усиления активности конкретного белка, либо инактивации активности конкретного белка.
Конкретно, микроорганизм может продуцировать L-аминокислоты или их предшественники в результате дополнительного включения (1) фосфосеринфосфатазы, имеющей ослабленную активность, (2) 3-фосфосеринаминотрансферазы, имеющей усиленную активность или (3) как фосфосеринфосфатазы, имеющей ослабленную активность, так и 3-фосфосеринаминотрансферазы, имеющей усиленную активность, но без ограничения ими.
Микроорганизм может быть, без ограничений, дополнительно модифицирован путем усиления trp (триптофанового) оперона, инактивации триптофаназы (TnaA), инактивации мембранного белка Mtr (Mtr) или любой их комбинации.
Конкретно, микроорганизм может быть, без ограничений, дополнительно модифицирован путем усиления trp оперона посредством инактивации TrpR, который ингибирует экспрессию генов (trpEDCBA), ассоциированных с биосинтезом L-триптофана, вовлеченных в продуцирование L-триптофана, путем инактивации триптофаназы (TnaA), которая играет роль при введении внеклеточного L-триптофана в клетку, и путем инактивации мембранного белка Mtr, который играет роль в распаде внутриклеточного L-триптофана и молекул воды до индола, пирувата и аммиака (NH3).
Дополнительно для задач описания настоящего изобретения микроорганизм может быть, без ограничений, дополнительно модифицирован путем усиления his (гистидинового) оперона.
Конкретно, гены биосинтеза, разделенные в общей сложности на 4 оперона, можно вводить в микроорганизм в форме кластера, в котором был заменен промотор, чтобы усилить путь биосинтеза L-гистидина, и кластер биосинтеза L-гистидина разделяется в общей сложности на 4 оперона (hisE-hisG, hisA-impA-hisF-hisI, hisD-hisC-hisB и cg0911-hisN). His оперон можно усилить, без ограничений, с использованием вектора, который может одновременно вводить в микроорганизм гены биосинтеза.
Дополнительно для задач описания настоящего изобретения микроорганизм может быть, без ограничений, дополнительно модифицирован путем инактивации регулятора транскрипции (McbR), усиления метионинсинтазы (meth), усиления сульфитредуктазного [NADPH] гемопротеинового бета-компонента (cysI) или любой их комбинации.
Конкретно, микроорганизм может быть, без ограничений, дополнительно модифицирован путем инактивации McbR, который представляет собой метиониновый/цистеиновый регулятор транскрипции, усиления метионинсинтазы (Meth), усиления сульфитредуктазного [NADPH] бета-компонента гемопротеина или любой их комбинации.
Введение, усиление или инактивацию активности конкретного белка и/или гена можно выполнять, используя любой подходящий способ, известный в данной области техники.
Используемый в настоящем документе термин «усиление» активности белка означает, что активность белка вводят или повышают по сравнению с его эндогенной активностью. «Введение» активности означает, что микроорганизм приобретает активность конкретного полипептида, которой микроорганизм не обладал ни естественным, ни искусственным путем.
Используемый в настоящем документе термин «повышение» активности белка относительно его эндогенной активности означает, что активность белка, включенного в микроорганизм, усилена по сравнению с эндогенной активностью белка или его активностью до модификации. Термин «эндогенная активность» относится к активности белка, которой исходно обладал родительский штамм микроорганизма или немодифицированный микроорганизм до трансформации, когда микроорганизм трансформируют посредством генетической модификации, вызванной естественным или искусственным фактором. «Эндогенная активность» может также использоваться взаимозаменяемо с «активностью до модификации». Повышение активности может включать как введение чужеродного белка, так и усиление эндогенной активности белка.
Повышение/усиление активности белка может быть достигнуто путем повышения/усиления экспрессии гена.
Конкретно, повышение активности белка в соответствии с настоящим изобретением может быть достигнуто одним из следующих способов, без ограничения ими:
(1) способ увеличения числа копий кодирующего белок полинуклеотида,
(2) способ модификации последовательности контроля экспрессии для повышения экспрессии полинуклеотида,
(3) способ модификации полинуклеотидной последовательности в хромосоме для усиления активности белка,
(4) способ введения чужеродного полинуклеотида, кодирующего обладающий активностью белок, или полинуклеотида с модификацией оптимизации кодонов, кодирующего обладающий активностью белок, или
(5) способ усиления активности посредством любой их комбинации.
Способ увеличения числа копий полинуклеотида, описанный в п. (1) выше, конкретно не ограничен, но может быть выполнен в форме, функционально связанной с вектором, или в интегрированной форме в хромосоме клетки-хозяина. Конкретно, этот способ может быть выполнен путем введения в клетку-хозяина вектора, который реплицируется и функционирует независимо от клетки-хозяина и функционально связан с полинуклеотидом, кодирующим белок по описанию настоящего изобретения; или путем введения в клетку-хозяина вектора, который встраивает полинуклеотид в хромосому клетки-хозяина и функционально связан с полинуклеотидом, в результате чего увеличивается число копий полинуклеотида в хромосоме клетки-хозяина.
Следующий способ модификации последовательности контроля экспрессии для повышения экспрессии полинуклеотида, описанный в п. (2) выше, может быть, без ограничений, выполнен путем индуцирования модификации нуклеиновокислотной последовательности посредством делеции, инсерции, неконсервативной замены, консервативной замены или любой их комбинации, чтобы дополнительно усилить активность последовательности контроля экспрессии, либо путем замены нуклеотидной последовательности нуклеотидной последовательностью, обладающей более сильной активностью. Последовательность контроля экспрессии может включать, без ограничений, промотор, последовательность оператора, последовательность, кодирующую сайт связывания рибосомы, и последовательность регуляции терминации транскрипции и трансляции.
Выше экспрессионной единицы полинуклеотида вместо собственного промотора может быть связан сильный гетерологичный промотор, и примеры этого сильного промотора могут включать, без ограничений, промоторы CJ1-CJ7 (патент Кореи №0620092 и Международная публикация №WO 2006/065095), промотор lysCP1 (Международная публикация №WO 2009/096689), промотор EF-Tu, промотор groEL, промотор асеА, промотор асеВ, промотор lac, промотор trp, промотор trc, промотор tac, промотор PR фага лямбда, промотор PL, промотор tet, промотор gapA, промотор SPL1, SPL7 или SPL13 (патент Кореи №10-1783170) или промотор O2 (патент Кореи №10-1632642). В дополнение к этому способ модификации полинуклеотидной последовательности в хромосоме, описанный в п. (3) выше, может быть выполнен, без ограничений, путем индуцирования вариации в последовательности контроля экспрессии посредством делеции, инсерции, неконсервативной замены, консервативной замены или любой их комбинации, чтобы дополнительно усилить активность полинуклеотидной последовательности, либо путем замены нуклеотидной последовательности нуклеотидной последовательностью, модифицированной для обладания более сильной активностью.
В дополнение к этому способ введения чужеродной полинуклеотидной последовательности, описанный в п. (4) выше, может быть выполнен путем введения в клетку-хозяина чужеродного полинуклеотида, кодирующего белок, обладающий активностью, идентичной/подобной активности указанного белка, либо его оптимизированного по кодонам полинуклеотидного варианта. Этот чужеродный полинуклеотид может представлять собой любой полинуклеотид, без ограничений, кодирующий белок, обладающий идентичной/подобной активности указанного белка. В дополнение к этому его оптимизированный по кодонам вариант можно вводить в клетку-хозяина для выполнения оптимизированной транскрипции и трансляции введенного в клетку-хозяина чужеродного полинуклеотида. Это введение может быть выполнено любым известным способом трансформации, подходящим образом выбранным специалистами в данной области техники. Когда введенный полинуклеотид экспрессируется в клетке-хозяине, продуцируется белок, и его активность может быть повышена.
Наконец, способ усиления активности посредством любой комбинации способов (1)-(4), описанный в п. (5) выше, может быть выполнен путем комбинирования по меньшей мере одного из способов увеличения числа копий кодирующего белок полинуклеотида, модификации последовательности контроля экспрессии для повышения его экспрессии, модификации полинуклеотидной последовательности в хромосоме, введения чужеродного полинуклеотида, кодирующего обладающий активностью белок, или его модифицированного по кодонам полинуклеотидного варианта.
Используемый в настоящем документе термин «ослабление» активности белка представляет собой понятие, включающее как снижение, так и удаление активности по сравнению с эндогенной активностью.
Ослабление активности белка может быть достигнуто посредством ряда способов, хорошо известных в данной области техники. Примеры этого способа могут включать: способ делетирования части или полноразмерного гена, кодирующего белок в хромосоме, включая случай удаления этой активности; способ замены гена, кодирующего белок в хромосоме, геном, мутированным для снижения активности белка; способ введения мутации в последовательность контроля экспрессии гена, кодирующего белок; замена последовательности контроля экспрессии гена последовательностью, обладающей более слабой активностью или не обладающей активностью (например, замещение эндогенного промотора гена более слабым промотором); способ делетирования части или полноразмерного гена, кодирующего белок в хромосоме; способ введения антисмыслового олигонуклеотида (например, антисмысловой РНК), который комплементарно связывается с транскриптом гена на хромосоме с ингибированием трансляции мРНК в белок; способ искусственного добавления последовательности, комплементарной последовательности SD (Шайна-Дальгарно), выше SD последовательности гена, кодирующего белок, для образования вторичной структуры, тем самым ингибирующей связывание с ней рибосомы, и способ встраивания промотора в 3'-конец открытой рамки считывания (ORF) с индуцированием обратной транскрипции (инженерия обратной транскрипции (RTE)), либо любую их комбинацию, без ограничений.
Конкретно, способ делетирования части или полноразмерного гена, кодирующего белок, может быть выполнен путем замены полинуклеотида, кодирующего эндогенный целевой белок, полнинуклеотидом, кодирующим эндогенный целевой белок внутри хромосомы, где полинуклеотид или маркерный ген имеет частичную делецию последовательности нуклеиновой кислоты, с использованием вектора для вставки в хромосому микроорганизма. Например, можно, без ограничений, использовать способ делетирования части или полноразмерного гена посредством гомологичной рекомбинации. В дополнение к этому термин «часть», хотя он может варьировать в соответствии с типом полинуклеотида и может быть подходящим образом определен средним специалистом в данной области техники, относится к от 1 нуклеотида до 300 нуклеотидов, конкретно от 1 нуклеотида до 100 нуклеотидов и более конкретно от 1 нуклеотида до 50 нуклеотидов соответственно, без ограничения этим.
Дополнительно, способ модификации последовательности контроля экспрессии может быть осуществлен путем индуцирования модификации последовательности контроля экспрессии посредством делеции, вставки, консервативной замены или неконсервативной замены, либо любой их комбинации, для дополнительного ослабления активности последовательности контроля экспрессии, либо осуществлен путем замены последовательности контроля экспрессии нуклеотидной последовательностью, обладающей более слабой активностью. Последовательность контроля экспрессии может включать, без ограничений, промотор, последовательность оператора, последовательность, кодирующую сайт связывания рибосомы, и последовательность для регуляции терминации транскрипции и трансляции.
В дополнение к этому способ модификации последовательности гена в хромосоме может быть осуществлен, без ограничений, путем индуцирования модификации посредством делеции, инсерции, консервативной замены или неконсервативной замены в последовательности, либо любой их комбинации, для дополнительного ослабления активности белка, либо осуществлен путем замены последовательности гена последовательностью гена, модифицированной таким образом, чтобы она обладала более слабой активностью или не обладала активностью.
Используемое в настоящем документе выражение «микроорганизм, продуцирующий L-аминокислоту или ее предшественник» относится к микроорганизму, способному продуцировать L-аминокислоту или ее предшественник в больших количествах из источников углерода, содержащихся в культуральной среде, по сравнению с микроорганизмами дикого типа или немодифицированными микроорганизмами. Дополнительно, «микроорганизм» может относиться к микроорганизму, естественным образом обладающему способностью продуцировать L-аминокислоту или ее предшественник, или к микроорганизму, полученному путем обеспечения способности продуцировать L-аминокислоту или ее предшественник в родительском штамме микроорганизма, который неспособен продуцировать L-аминокислоту или ее предшественник. Конкретно, этот микроорганизм может, без ограничений, представлять собой микроорганизм, модифицированный для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента, для продуцирования L-аминокислоты или ее предшественника.
Дополнительно «микроорганизм, продуцирующий L-аминокислоту или ее предшественник» включает как микроорганизмы дикого типа, так и микроорганизмы, в которых произошла естественная или искусственная генетическая модификация, такие как микроорганизмы, в которых ослаблен или усилен конкретный механизм посредством введения экзогенного гена, усиления или инактивации эндогенного гена и так далее, и в которых произошла генетическая модификация или усилена активность для продуцирования целевой L-аминокислоты или ее предшественника. В частности, типы микроорганизмов конкретно не ограничены, если микроорганизм способен продуцировать L-аминокислоту или ее предшественник, но этот микроорганизм может принадлежать к роду Enterobacier, роду Escherichia, роду Erwinia, роду Serratia, роду Providencia, роду Corynebacterium или роду Brevibacterium. Более конкретно, этот микроорганизм может представлять собой любой микроорганизм, принадлежащий к роду Corynebacterium или роду Escherichia. Микроорганизм рода Corynebacterium может представлять собой Corynebacterium glutamicum, Corynebacterium ammoniagenes, Brevibacterium lactofermentum, Brevibacterium flavum, Corynebacterium thermoaminogenes, Corynebacterium efficiens, Corynebacterium stationis и тому подобные, но не ограничен ими. Более конкретно, микроорганизм рода Escherichia может представлять собой Escherichia coli, а микроорганизм рода Corynebacterium может представлять собой Corynebacterium glutamicum, без ограничения ими.
Для задач описания настоящего изобретения микроорганизм может представлять собой любой микроорганизм, включающий белок и, таким образом, способный продуцировать L-аминокислоту или ее предшественник.
Используемое в настоящем документе выражение «микроорганизм, способный продуцировать L-аминокислоту или ее предшественник», может использоваться взаимозаменяемо с выражениями «микроорганизм, продуцирующий L-аминокислоту или ее предшественник» и «микроорганизм, обладающий способностью продуцировать L-аминокислоту или ее предшественник».
Другой аспект настоящего изобретения обеспечивает композицию для получения L-аминокислоты или ее предшественника, содержащая микроорганизм, модифицированный для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента, или указанный белок.
Композиция для получения L-аминокислоты или ее предшественника относится к композиции, способной продуцировать L-аминокислоту или ее предшественник посредством белка в соответствии с настоящим изобретением. Композиция может, без ограничений, содержать указанный белок, его функциональный фрагмент или любые компоненты, используемые для работы с белком.
Другой аспект настоящего изобретения обеспечивает способ получения L-аминокислоты или ее предшественника, включающий культивирование указанного микроорганизма в культуральной среде.
Способ может дополнительно включать выделение L-аминокислоты или ее предшественника из их культуральной среды или культуры.
В описанном выше способе культивирование микроорганизма можно осуществлять, без ограничений, методом периодической культуры, непрерывной культуры, культуры с подпиткой и тому подобного, как известно в данной области техники. В связи с этим условия культивирования конкретно не ограничены, но оптимальный рН (например, рН от 5 до 9, конкретно рН от 6 до 8 и наиболее конкретно рН 6,8) можно поддерживать путем использования основного соединения (например, гидроксида натрия, гидроксида калия или аммиака) или кислотного соединения (например, фосфорной кислоты или серной кислоты). Дополнительно можно поддерживать аэробные условия путем добавления в культуру кислорода или кислородсодержащей газовой смеси. Температуру культивирования можно поддерживать при 20°С-45°С, конкретно 25°С-40°С, и культивирование можно осуществлять, без ограничений, в течение от приблизительно 10 часов до приблизительно 160 часов. Продуцируемые при культивировании аминокислоты могут высвобождаться в культуральную среду или оставаться в клетках.
Примеры источника углерода, который должен содержаться в культуральной среде, могут включать, без ограничений, сахариды и углеводы (например, глюкозу, сахарозу, лактозу, фруктозу, мальтозу, мелассу, крахмал и целлюлозу), масла и жиры (например, соевое масло, подсолнечное масло, арахисовое масло, кокосовое масло), жирные кислоты (например, пальмитиновую кислоту, стеариновую кислоту и линолевую кислоту), спирты (например, глицерин и этанол) и органические кислоты (уксусную кислоту), которые можно использовать по отдельности или в комбинации, и тому подобное. Примерами источника азота, который должен содержаться в культуральной среде, могут быть, без ограничений, азотсодержащее органические соединение (например, пептон, дрожжевой экстракт, мясной сок, солодовый экстракт, кукурузный сироп, соевая мука и мочевина), неорганическое соединение (например, сульфат аммония, хлорид аммония, фосфат аммония, карбонат аммония и нитрат аммония), которые можно использовать по отдельности или в комбинации, и тому подобное. В качестве источника фосфора можно использовать, без ограничений, дигидрофосфат калия, гидрофосфат дикалия и соответствующие им натрийсодержащие соли по отдельности или в комбинации. В дополнение к этому культуральная среда может включать, без ограничений, необходимые стимулирующие рост вещества, такие как соль металла (например, сульфат натрия и сульфат железа), аминокислоты и витамины.
Аминокислоты, продуцируемые на описанной выше стадии культивирования по настоящему изобретению, могут быть извлечены путем сбора целевых аминокислот из культурального раствора с использованием любого известного способа, выбранного в соответствии со способом культивирования. Например, можно использовать центрифугирование, фильтрование, анионообменную хроматографию, кристаллизацию и высокоэффективную жидкостную хроматографию (HPLC), и целевые аминокислоты можно выделять из культуральной среды или микроорганизма с использованием любого подходящего способа в данной области техники, без ограничений.
Дополнительно стадия извлечения может включать процесс очистки, который можно осуществлять, используя любой подходящий способ, хорошо известный в данной области техники. Так, выделенная аминокислота может представлять собой очищенную аминокислоту или ферментационный бульон микроорганизма, включающий аминокислоту (Introduction to Biotechnology and Genetic Engineering, A. J. Nair., 2008).
В дополнение к этому для задач описания настоящего изобретения в случае микроорганизма, модифицированного для экспрессии D-3-фосфоглицератдегидрогеназы, имеющей происхождение из рода Azotobacter, увеличиваются выходы L-аминокислот и их предшественников, включая серин, триптофан, гистидин, метионин и О-фосфосерин. Важно, что модифицировванный микроорганизм увеличивает выходы L-аминокислот и их предшественников, в то время как штаммы дикого типа рода Corynebacterium неспособны продуцировать L-аминокислоты или их предшественники или способны продуцировать их в очень малом количестве.
Еще один аспект настоящего изобретения обеспечивает способ получения L-аминокислоты или ее предшественника с использованием композиции, которая содержит микроорганизм, модифицированный для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента, или указанный белок.
Микроорганизм, модифицированный для экспрессии белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента, и включающий ее микроорганизм являются такими, как описано выше.
Еще один аспект настоящего изобретения обеспечивает применение белка, содержащего аминокислотную последовательность SEQ ID NO: 1, или его функционального фрагмента, для повышения продукции L-аминокислоты или ее предшественника.
SEQ ID NO: 1 или ее функциональный фрагмент, L-аминокислот и ее предшественник являются такими, как описано выше.
ПРИМЕРЫ ОСУЩЕСТВЛЕНИЯ ИЗОБРЕТЕНИЯ
Далее настоящее изобретения будет описано более подробно со ссылкой на следующие примеры. Однако эти примеры предназначены только для иллюстративных целей и не предназначены для ограничения объема настоящего изобретения. В то же время, техническая сущность, не раскрытая в данном описании, может быть в достаточной степени понятна и легко выполнена специалистами в области техники, к которой относится настоящая заявка, или в подобной области техники.
Пример 1: Получение вектора сверхэкспрессии гена D-3-фосфоглицератдегидрогеназы (serA(Avn)), имеющего происхождение из Azotobacter
Чтобы идентифицировать, улучшается ли способность продуцировать серин и О-фосфосерин (OPS) в результате усиления D-3-фосфоглицератдегидрогеназы (далее обозначена в настоящем документе как SerA(Avn)), имеющей происхождение из Azotobacter vinelandii, был получен экспрессионный вектор.
Вектор pCL1920 (GenBank № АВ236930) использовали для экспрессии гена serA(Avn) (SEQ ID NO: 1), кодирующего SerA(Avn), а промотор trc (Ptrc) использовали в качестве экспрессионного промотора, в результате чего был сконструирован вектор в форме pCL-Ptrc-serA(Avn).
В качестве контроля был получен вектор, включающий в себя D-3-фосфоглицератдегидрогеназу, имеющую происхождение из Е. coli, в котором устранено ингибирование серином по принципу обратной связи, и назван pCL-Ptrc-serA*(G336V). Последовательности праймеров, использованных для получения векторов, показаны в Таблице 1 ниже.
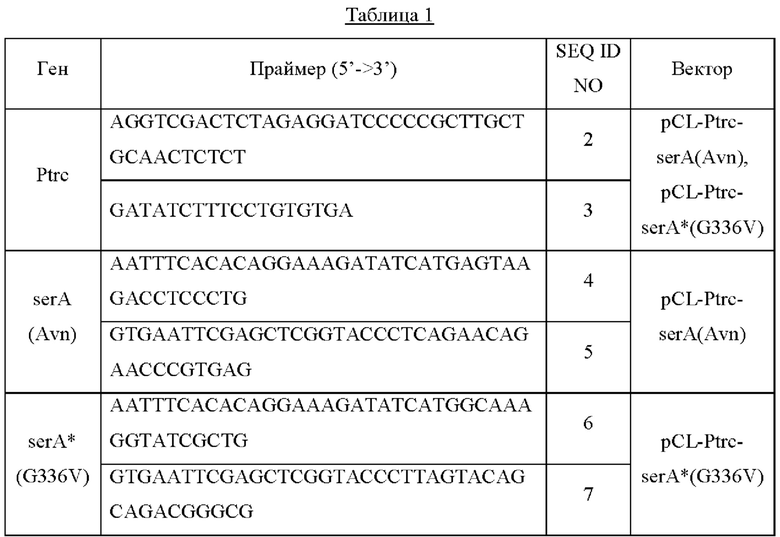
PCR (полимеразная цепная реакция) для Ptrc, которую использовали в получении обоих векторов, выполняли с использованием праймеров с SEQ ID NO: 2 и 3. Конкретно, PCR для чужеродного гена serA(Avn) выполняли с использованием праймеров с SEQ ID NO: 4 и 5, a PCR для serA*(G336V) выполняли с использованием праймеров с SEQ ID NO: 6 и 7. Амплифицированные фрагменты Ptrc, и serA(Avn), и serA*(G336V) соответствующих генов клонировали в вектор pCL1920, обработанный рестрикционным ферментом Sma1, методом сборки Гибсона, соответственно, в результате чего сконструировали pCL-Ptrc-serA(Avn) и pCL-Ptrc-serA*(G336V).
Пример 2: Получение штамма путем введения serA(Avn), имеющего происхождение из Azotobacter, в Е. coli дикого типа и оценка способности продуцировать серин
С использованием штамма E.coli W3110 дикого типа в качестве несущего штамма получали штаммы путем введения в штамм W3110 каждого из двух типов плазмид, полученных в Примере 1, а затем оценивали их способность продуцировать серин.
Каждый из штаммов высевали на твердую среду LB (Лурия - Бертани) и культивировали в течение ночи в инкубаторе при 33°С. Штаммом, который культивировали в течение ночи на твердой среде LB, инокулировали 25 мл среды для титрования, как показано в Таблице 2 ниже, и культивировали в инкубаторе при 34,5°С при 200 об/мин в течение 40 часов. Результаты показаны в Таблице 3 ниже.
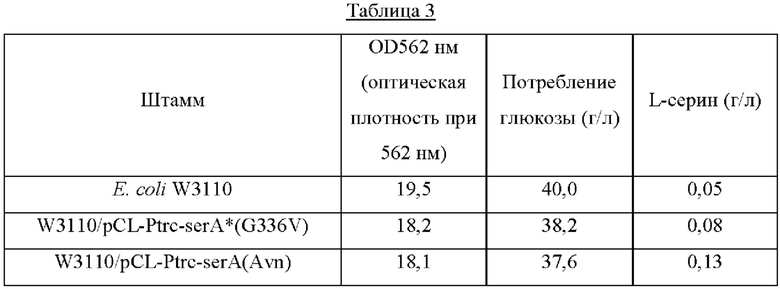
Как показано в Таблице 3, штамм W3110/pCL-Ptrc-serA*(G336V), в котором устранено ингибирование серином по принципу обратной связи и усилена активность serA, продемонстрировал повышение продукции серина на 60% по сравнению со штаммом дикого типа. Для сравнения было подтверждено, что штамм W3110/pCL-Ptrc-serA(Avn), включающий в себя serA(Avn), имеющий происхождение из Azotobacter, продемонстрировал повышение продукции серина на 160% по сравнению со штаммом W3110 дикого типа, а также продемонстрировал повышение на 62,5% по сравнению со штаммом W3110/pCL-Ptrc-serA*(G336V).
Пример 3: Получение штамма, в котором ослаблена активность serB и введен чужеродный ген serA(Avn), имеющий происхождение из Azotobacter, и оценка способности этого штамма продуцировать OPS
Продуцирующий О-фосфосерин (OPS) микроорганизм получали путем ослабления эндогенной фосфосеринфосфатазы (SerB) в штамме Е. coli W3110 дикого типа (также назван «СА07-0012», номер доступа: KCCM11212P, описан в патенте Кореи №10-1381048 и публикации заявки на патент США №2012-0190081).
Каждый из двух типов плазмид, полученных в Примере 1, вводили в СА07-0012 и оценивали способность полученных штаммов продуцировать OPS.
Каждый из штаммов высевали на твердую среду LB и культивировали в течение ночи в инкубаторе при 33°С. Штаммом, который культивировали в течение ночи на твердой среде LB, инокулировали 25 мл среды для титрования, как показано в Таблице 4 ниже, и культивировали в инкубаторе при 34,5°С при 200 об/мин в течение 40 часов. Результаты показаны в Таблице 5 ниже.


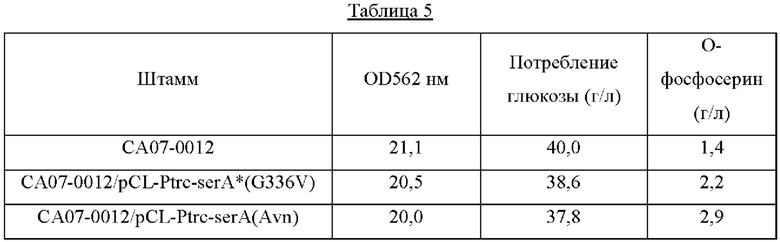
Как показано в Таблице 5 выше, CA07-0012/pCL-Ptrc-serA*(G336V), в котором устранено ингибирование серином по принципу обратной связи и усилена активность serA, продемонстрировал увеличение продукции OPS на 57% по сравнению со штаммом дикого типа. Было подтверждено, что штамм CA07-0012/pCL-Ptrc-serA(Avn), включающий serA(Avn), имеющий происхождение из Azotobacter, продемонстрировал увеличение продукции OPS на 107% по сравнению со штаммом дикого типа, а также продемонстрировал повышение на 32% по сравнению со штаммом CA07-0012/pCL-Ptrc-serA*(G336V).
Пример 4: Получение вектора для совместной сверхэкспрессии serA(Avn), имеющего происхождение из Azotobacter, и serC, имеющего происхождение из Е.coli
Чтобы идентифицировать, улучшается ли дополнительно способность к продуцированию серина и OPS за счет введения гена serA(Avn) в штамм, в котором была сверхэкспрессирована 3-фосфосеринаминотрансфераза, имеющая происхождение из Е. coli (serC), получали вектор в форме pCL-Ptrc-serA(Avn)-(RBS)serC для экспрессии serA(Avn) и serC в виде оперонов.
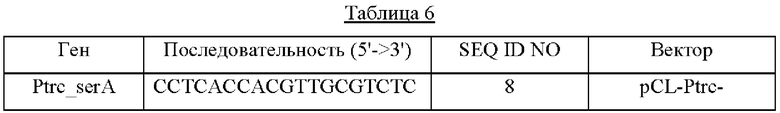
В качестве его положительного контроля конструировали вектор pCL-Ptrc-serA*(G336V)-(RBS)serC для получения микроорганизма, совместно экспрессирующего гены serA*(G336V) и serC, имеющие происхождение из Е. coli. Последовательности праймеров, используемых для получения векторов, представлены в Таблице 6 ниже.

PCR для Ptrc_serA(Avn) выполняли, используя в качестве матрицы pCL-Ptrc-serA(Avn), полученный в Примере 1, и праймеры с SEQ ID NO: 2 и 8, a PCR для Ptrc_serA*(G336V) выполняли, используя в качестве матрицы pCL-Ptrc-serA*(G336V) и праймеры с SEQ ID NO: 2 и 11. Имеющий происхождение из Е. coli (RBS)serC, используемый в обоих векторах, получали посредством PCR, выполненной с использованием в качестве матрицы ДНК w3110 и праймеров с SEQ ID NO: 9 и 10.
Амплифицированные фрагменты Ptrc_serA(Avn) и (RBS)serC и фрагменты Ptrc_serA*(G336V) и (RBS)serC клонировали в вектор pCL1920, обработанный рестрикционным ферментом SmaI, методом сборки Гибсона (DG Gibson et al., NATURE METHODS, VOL. 6, NO. 5, MAY 2009, NEBuilder HiFi DNA Assembly Master Mix), соответственно, в результате чего сконструировали векторы pCL-Ptrc-serA(Avn)-(RBS)serC и pCL-Ptrc-serA*(G336V)-(RBS)serC.
Пример 5: Получение штамма, в котором усилена активность serC и введен ген serA(Avn), имеющий происхождение из Azotobacter, и оценка способности этого штамма продуцировать серин
Чтобы оценить способность к продуцированию серина при введении гена serA(Avn), имеющего происхождение из Azotobacter, в штамм, в котором был сверхэкспрессирован serC, в W3110 вводили два типа плазмид, полученных в Примере 4, соответственно.
Каждый из штаммов высевали на твердую среду LB и культивировали в течение ночи в инкубаторе при 33°С. Штаммом, который культивировали в течение ночи на твердой среде LB, инокулировали 25 мл среды для титрования, как показано в Таблице 7 ниже, и культивировали в инкубаторе при 34,5°С при 200 об/мин в течение 40 часов. Результаты показаны в Таблице 8 ниже.
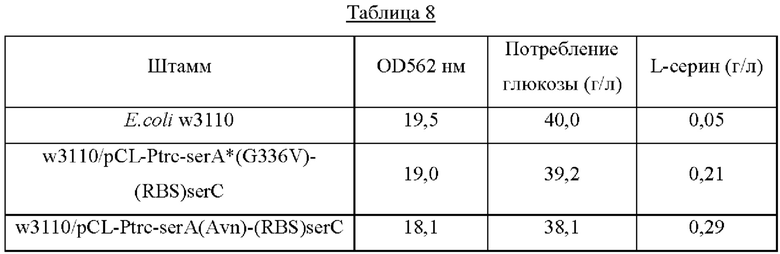
Как показано в Таблице 8 выше, было подтверждено, что штамм w3110/pCL-Ptrc-serA(Avn)-(RBS)serC, включающий в себя ген serA(Avn), имеющий происхождение из Azotobacter, продемонстрировал повышение продукции L-серина по сравнению со штаммом w3110/pCL-Ptrc-serA*(G336V)-(RBS)serC, включающим в себя serA*(G336V). Таким образом, было подтверждено, что способность к продуцированию L-серина дополнительно повысилась в результате включения гена serA(Avn), имеющего происхождение из Azotobacter, в штамм, в котором была повышена способность к продуцированию L-серина.
Штамм w3110/pCL-Ptrc-serA(Avn)-(RBS)serC был назван СА07-4383 и депонирован в Корейском центре культур микроорганизмов (KCCM) согласно Будапештскому договору, и ему был присвоен номер доступа KCCM12381P 9 ноября 2018 г.
Пример 6: Получение штамма, в котором ослаблена активность гена serB, усилена активность гена serC и введен ген serA(Avn), имеющий происхождение из Azotobacter, и оценка способности этого штамма продуцировать QPS
Чтобы оценить способность к продуцированию серина в случае введения гена serA(Avn), имеющего происхождение из Azotobacter, в штамм с ослабленной активностью serB и сверхэкспрессией serC, в штамм СА07-0012 вводили два типа плазмид, полученных в Примере 4, соответственно, и оценивали способность этих штаммов продуцировать OPS.
Каждый из штаммов высевали на твердую среду LB и культивировали в течение ночи в инкубаторе при 33°С. Штаммом, который культивировали в течение ночи на твердой среде LB, инокулировали 25 мл среды для титрования, как показано в Таблице 9 ниже, и культивировали в инкубаторе при 34,5°С при 200 об/мин в течение 40 часов. Результаты показаны в Таблице 10 ниже.

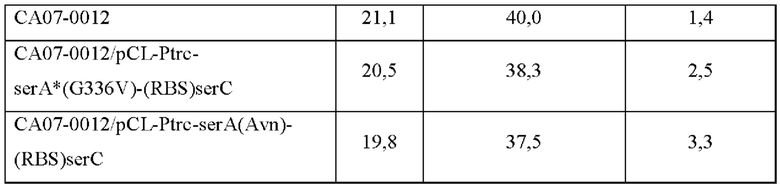
Как показано в Таблице 10 выше, было подтверждено, что штамм СА07-0012/pCL-Ptrc-serA(Avn)-(RBS)serC, включающий в себя ген serA(Avn), имеющий происхождение из Azotobacter, имел более высокую продукцию OPS, чем штамм СА07-0012/pCL-Ptrc-serA*(G336V)-(RBS)serC, включающий в себя serA*(G336V). Таким образом, было подтверждено, что способность к продуцированию OPS дополнительно повысилась в результате включения гена serA(Avn), имеющего происхождение из Azotobacter, в штамм, в котором была повышена способность к продуцированию OPS.
Пример 7: Получение штамма рода Escherichia, в который введен ген serA(Avn), имеющий происхождение из Azotobacter, и оценка способности этого штамма продуцировать триптофан
Пример 7-1: Получение микроорганизма рода Escherichia, продуцирующего L-триптофан
Штамм рода Escherichia, продуцирующий L-триптофан, был разработан на основе Е. coli W3110 дикого типа. Чтобы идентифицировать, значимо ли повышается способность к продуцированию L-триптофана в результате модификации для экспрессии белка, обладающего способностью экспортировать L-триптофан, в качестве родительского штамма использовали штамм, полученный для продуцирования L-триптофана. Конкретно, TrpR ингибирует экспрессию генов биосинтеза L-триптофана (trpEDCBA), которые вовлечены в продуцирование L-триптофана из хоризмата. Таким образом, ген trpR, кодирующий TrpR, был удален. В дополнение к этому для устранения ингибирования полипептидом TrpE по принципу обратной связи в соответствии с повышенной продукцией L-триптофана, пролин, который является 21-ой аминокислотой с N-конца TrpE, был заменен серином (J. Biochem. Mol. Biol. 32, 20-24 (1999)).
Мембранный белок Mtr играет роль в транспортировке внеклеточного L-триптофана в клетку, а белок TnaA играет роль в деградации внутриклеточного L-триптофана и молекул воды до индола, пирувата и аммиака (NH3). Таким образом, были удалены гены mtr и tnaA, которые ингибируют продуцирование L-триптофана и осуществляют деградацию L-триптофана.
Для удаления этих генов использовали метод рекомбинации λ-red (лямбда-ред) (One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products, Datsenko KA, Wanner BL., Proc Natl Acad Sci USA. 2000 Jun 6;97(12):6640-5). Для удаления гена mtr выполняли PCR, используя вектор pKD4 в качестве матрицы и праймеры с SEQ ID NO: 12 и 13 с получением генного фрагмента (1580 п. о. (пар оснований)), в котором связаны кассета FRT-канамицин-FRT и гомологичные пары оснований 50 п. о., фланкирующие ген mtr, где между ними происходит хромосомная гомологичная рекомбинация. Для подтверждения удаления гена-мишени и инсерции гена устойчивости к антибиотику использовали маркер антибиотика канамицина вектора pKD4, а область FRT играет роль в удалении маркера антибиотика после удаления гена-мишени. В качестве полимеразы использовали ДНК-полимеразу Solg™TM Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 2 минут; 27 циклов денатурации при 95 °С в течение 20 секунд, отжига при 62°С в течение 40 секунд и полимеризации при 72 °С в течение 1 минуты; и полимеризация при 72°С в течение 5 минут.

Штамм Е. coli W3110 трансформировали вектором pKD46, который экспрессирует рекомбиназу λ-red (gam, bet и ехо), посредством электропорации и высевали на твердую среду LB, содержащую 50 мг/л канамицина. В штамме Е. coli W3110 с подтвержденной трансформацией вектором pKD46 индуцировали экспрессию рекомбинантного фермента путем добавления к нему 10 мМ L-арабинозы при достижении OD600 (оптической плотности при 600 нм) приблизительно 0,1 при 30°С.
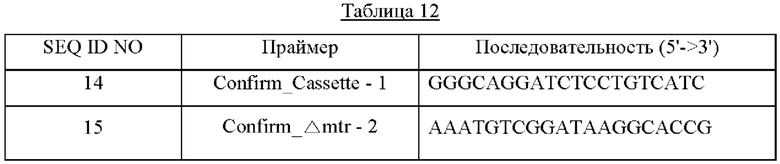
При достижении OD600 приблизительно 0,6 готовили этот штамм в виде компетентных клеток и трансформировали посредством электропорации линейным генным фрагментом, полученным в описанном выше процессе, в котором были связаны кассета FRT-канамицин-FRT и гомологичные пары оснований 50 п. о., фланкирующие ген mtr. Для колоний, выращенных на твердой среде LB, содержащей 25 мг/л канамицина, выполняли PCR колоний, используя праймеры с SEQ ID NO: 14 и 15, и отбирали колонии, где был получен генный фрагмент 782 п. о.
Штамм, из которого был удален ген mtr путем гомологичной рекомбинации, готовили в виде компетентных клеток для удаления маркера антибиотика канамицина, а затем трансформировали их вектором рСР20 посредством электропорации. Вектор рСР20 предназначен для распознавания сайтов FRT, фланкирующих ген устойчивости к антибиотику канамицину, и связывания с ними на хромосоме посредством экспрессии белка FLP, в результате чего он удаляет маркер антибиотика между сайтами FRT. Штамм, трансформированный вектором рСР20 и выращенный на твердой среде LB, содержащей 100 мг/л ампициллина и 25 мг/л хлорамфеникола, культивировали в жидкой среде LB при 30°С в течение 1 часа, дополнительно культивировали при 42°С в течение 15 часов и высевали на твердую среду LB. Выросшие колонии культивировали на твердой среде LB, содержащей 100 мг/л ампициллина и 25 мг/л хлорамфеникола, твердой среде LB, содержащей 12,5 мг/л канамицина, и твердой среде LB, не содержащей антибиотик. Отбирали только колонии, выросшие на твердой среде LB, не содержащей антибиотик. Наконец, удаление гена mtr подтверждали путем секвенирования генома, и штамм был назван СА04-9300.
Для удаления гена tnaA выполняли генетические манипуляции таким же способом, как описано выше. PCR выполняли, используя вектор pKD4 в качестве матрицы и праймеры с SEQ ID NO: 16 и 17 с получением генного фрагмента (1580 п. о.), в котором связаны кассета FRT-канамицин-FRT и гомологичные пары оснований 50 п. о., фланкирующие ген tnaA, где между ними происходит хромосомная гомологичная рекомбинация. В качестве полимеразы использовали ДНК-полимер азу Solg™ Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 2 минут; 27 циклов денатурации при 95°С в течение 20 секунд, отжига при 62°С в течение 40 секунд и полимеризации при 72°С в течение 1 минуты; и полимеризация при 72°С в течение 5 минут.

Трансформацию вектором pKD46 подтверждали, и штамм СА04-9300, в котором экспрессировали рекомбиназы путем добавления 10 мМ L-арабинозы, трансформировали посредством электропорации линейным генным фрагментом, в котором связаны кассета FRT-канамицин-FRT и гомологичные пары оснований 50 п. о., фланкирующие ген tnaA. Для колоний, выращенных на твердой среде LB, содержащей 25 мг/л канамицина, выполняли PCR колоний, используя праймеры с SEQ ID NO: 14 и 18, и отбирали колонии, где был получен генный фрагмент 787 п. о.
Штамм, из которого был удален ген tnaA путем гомологичной рекомбинации, готовили в виде компетентных клеток и трансформировали их вектором рСР20 для удаления маркера антибиотика канамицина, и получили штамм, из которого был удален маркер устойчивости к антибиотику канамицину, путем экспрессии белка FLP. Наконец, удаление гена tnaA подтверждали путем секвенирования генома, и штамм был назван СА04-9301.
Для удаления гена trpR выполняли PCR, используя вектор pKD4 в качестве матрицы и праймеры с SEQ ID NO: 19 и 20 с получением генного фрагмента (1580 п. о.), в котором были связаны кассета FRT-канамицин-FRT и гомологичные пары оснований 50 п. о., фланкирующие ген trpR, где между ними происходит хромосомная гомологичная рекомбинация. В качестве полимеразы использовали ДНК-полимеразу Solg™ Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 2 минут; 27 циклов денатурации при 95°С в течение 20 секунд, отжига при 62°С в течение 40 секунд и полимеризации при 72°С в течение 1 минуты; и полимеризация при 72°С в течение 5 минут.
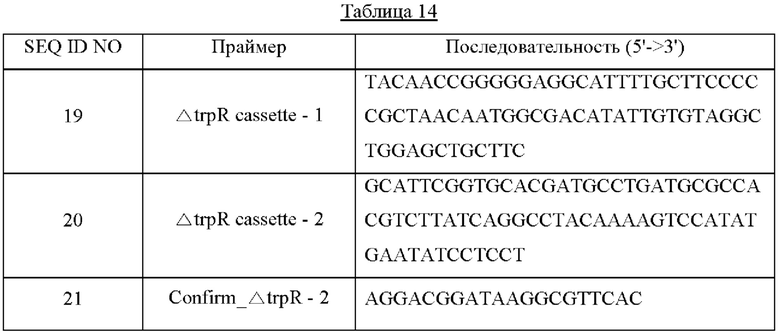
Трансформацию вектором pKD46 подтверждали, и штамм СА04-9301, в котором экспрессировали рекомбиназы путем добавления 10 мМ L-арабинозы, трансформировали посредством электропорации линейным генным фрагментом, полученным в описанном выше процессе, в котором связаны кассета FRT-канамицин-FRT и гомологичные пары оснований 50 п. о., фланкирующие ген trpR. Для колоний, выращенных на твердой среде LB, содержащей 25 мг/л канамицина, выполняли PCR колоний, используя праймеры с SEQ ID NO: 14 и 21, и отбирали колонии, где был получен генный фрагмент 838 п. о.
Штамм, из которого был удален ген trpR путем гомологичной рекомбинации, готовили в виде компетентных клеток и трансформировали их вектором рСР20 для удаления маркера антибиотика канамицина, и получили штамм, из которого был удален маркер устойчивости к антибиотику канамицину, путем экспрессии белка FLP. Наконец, удаление гена trpR подтверждали путем секвенирования генома, и штамм был назван СА04-9307.
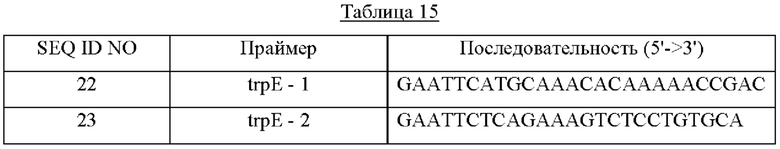
Для обеспечения штамма СА04-9307 признаком устойчивости к ингибированию trpE по принципу обратной связи выполняли PCR, используя в качестве матрицы гДНК (геномную ДНК) Е. coli W3110 и праймеры с SEQ ID NO: 22 и 23, содержащие сайт фермента рестрикции EcoRI, в результате чего получили генный фрагмент trpE, содержащий последовательность EcoRI (1575 п. о.). В качестве полимеразы использовали ДНК-полимеразу Solg™ Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 2 минут; 27 циклов денатурации при 95°С в течение 20 секунд, отжига при 62°С в течение 1 минуты и полимеризации при 72°С в течение 1 минуты; и полимеризация при 72°С в течение 5 минут.
Ген trpE, полученный описанным выше способом, и плазмиду pSG76-C (JOURNAL OF BACTERIOLOGY, July 1997, p.4426-4428) обрабатывали ферментом рестрикции EcoRI и клонировали. Е. coli DH5α трансформировали клонированной плазмидой посредством электропорации, и трансформированный штамм Е. coli DH5α отбирали с чашки со средой LB, содержащей 25 мкг/мл хлорамфеникола, с получением плазмиды pSG76-C-trpE.
Сайт-направленный мутагенез (Stratagene, США) выполняли, используя полученную плазмиду pSG76-C-trpE и праймеры с SEQ ID NO: 24 и 25, с получением pSG76-C-trpE(P21S).

Штамм СА04-9307 трансформировали плазмидой pSG76-C-trpE(P21S) и культивировали в среде LB-Cm (10 г/л дрожжевого экстракта, 5 г/л NaCl, 10 г/л триптона и 25 мкг/л хлорамфеникола) и отбирали колонии, устойчивые к хлорамфениколу. Отобранные трансформанты представляют собой штаммы, в которых плазмида pSG76-C-trpE(P21S) включена в область trpE генома в результате первой инсерции. Штамм, в который встроен полученный ген trpE(P21S), трансформировали плазмидой pAScep (Journal of Bacteriology, July 1997, p.4426 4428), экспрессирующей фермент рестрикции I-SceI, который расщепляет область I-SceI, присутствующую в плазмиде pSG76-C, и отбирали штамм, растущий в среде LB-Ap (10 г/л дрожжевого экстракта, 5 г/л NaCl, 10 г/л триптона и 100 мкг/л ампициллина). Ген trpE амплифицировали в отобранном штамме, используя праймеры с SEQ ID NO: 22 и 23, и подтверждали замену гена trpE(P21S) путем секвенирования. Полученный штамм был назван СА04-4303.
Пример 7-2: Получение микроорганизма рода Escherichia, в который введен ген serA(Avn), имеющий происхождение из Azotobacter, и оценка способности этого микроорганизма продуцировать триптофан
Вектор pCL-Ptrc-serA(Avn), полученный в Примере 1, и вектор pCL1920 в качестве контрольного вводили в СА04-4303, полученный в Примере 1, соответственно, с получением штаммов СА04-4303/pCL1920 и CA04-4303/pCL-Ptrc-serA(Avn). Для изучения продуцирования L-триптофана штаммами CA04-4303/pCL1920 и СА04-4303/pCL-Ptrc-serA(Avn) эти два штамма культивировали в жидкой среде LB, содержащей 50 мг/л спектиномицина, в течение 12 часов. Впоследствии каждый из штаммов инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл среды для продуцирования так, чтобы исходное значение OD600 достигло 0,01, и культивировали при 37°С в течение 48 часов при встряхивании со скоростью 200 об/мин. После завершения культивирования измеряли количество продукции L-триптофана методом HPLC.
Результаты продуцирования L-триптофана штаммами CA04-4303/pCL1920 и CA04-4303/pCL-Ptrc-serA(Avn) в культуральной среде показаны в Таблице 17 ниже. Штамм CA04-4303/pCL1920 продемонстрировал продукцию L-триптофана 1,2 г/л и накопление индола, который является промежуточным продуктом, в количестве 37 мг/л. Тем не менее штамм, в который был введен serA(Avn), продемонстрировал продукцию L-триптофана 1,7 г/л без накопления индола.
<Среда для продуцирования (рН 7,0)>
70 г глюкозы, 20 г (NH4)2SO4, 1 г MgSO4⋅7H2O, 2 г KH2PO4, 2,5 г дрожжевого экстракта, 5 г Na-цитрата, 1 г NaCl и 40 г СаСО3 (в расчете на 1 л дистиллированной воды).

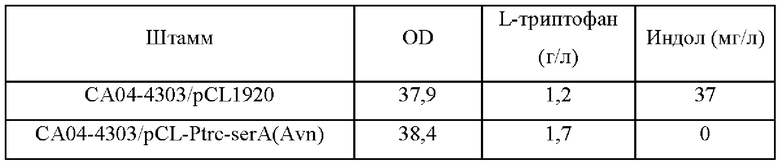
Как видно из представленных выше результатов, было установлено, что запас L-серина был достаточным за счет введения serA(Avn), и было подтверждено, что выход L-триптофана повышался без накопления индола, который является промежуточным продуктом, на конечной стадии биосинтеза L-триптофана.
Пример 7-3: Получение штамма Corynebacterium glutamicum, продуцирующего триптофан, в который введен чужеродный ген serA(Avn), имеющий происхождение из Azotobacter
Чтобы идентифицировать влияние гена serA(Avn), имеющего происхождение из Azotobacter, на штамм рода Corynebacterium, продуцирующий триптофан, использовали KCCM12218P (публикация патента Кореи №2018-0089329) в качестве штамма рода Corynebacterium, продуцирующего L-триптофан.
Этот штамм был получен путем замены гена Corynebacterium glutamicum serA (далее обозначенного как serA(Cgl)) геном serA(Avn), имеющим происхождение из Azotobacter, который должен экспрессироваться под действием промотора gapA.
Для этих генетических манипуляций сначала была получена расположенная выше по ходу транскрипции промоторная область и расположенная ниже по ходу транскрипции область ORF гена serA (Cgl), где происходит хромосомная гомологичная рекомбинация. Конкретно, генный фрагмент промоторной области, расположенной выше по ходу транскрипции, был получен путем выполнения PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum и праймеров с SEQ ID NO: 26 и 27, а генный фрагмент расположенной ниже по ходу транскрипции области был получен путем выполнения PCR с использованием праймеров с SEQ ID NO: 28 и 29. Дополнительно была получена промоторная область gapA путем выполнения PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum и праймеров с SEQ ID NO: 30 и 31.
В качестве полимеразы использовали ДНК-полимеразу Solg™ Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 5 минут; 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 58°С в течение 30 секунд и полимеризации при 72°С в течение 60 секунд; и полимеризация при 72°С в течение 5 минут.
Область гена serA(Avn), имеющего происхождение из Azotobacter, была получена путем выполнения PCR с использованием в качестве матрицы вектора pCL-Ptrc-serA(Avn), полученного в Примере 1, и праймеров с SEQ ID NO: 32 и 33.
В качестве полимеразы использовали ДНК-полимеразу Solg™ Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 5 минут; 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 58°С в течение 30 секунд и полимеризации при 72°С в течение 30 секунд; и полимеризация при 72°С в течение 5 минут.
Рекомбинантную плазмиду получили посредством клонирования, используя амплифицированные расположенную выше по ходу транскрипции и расположенную ниже по ходу транскрипции области для хромосомной гомологичной рекомбинации, промотор gapA, ген serA(Avn), имеющий происхождение из Azotobacter, и вектор pDZ для хромосомной трансформации, расщепленный ферментом рестрикции SmaI методом сборки Гибсона, и назвали ее pDZ-PgapA-serA(Avn). Клонирование выполняли путем смешивания реактива для сборки Гибсона и рассчитанного числа моль каждого из генных фрагментов с последующим инкубированием при 50°С в течение 1 часа.
Штамм Corynebacterium glutamicum KCCM12218P, продуцирующий L-триптофан, трансформировали полученным вектором pDZ-PgapA-serA(Avn) посредством электропорации и подвергали второму процессу кроссинговера с получением штамма, в котором ген serA(cgl) был заменен геном serA Azotobacter, экспрессируемым под действием промотора gapA. Эти генетические манипуляции были подтверждены путем проведения PCR и геномного секвенирования с использованием праймеров с SEQ ID NO: 34 и 35, соответственно, амплифицирующих внешние области расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей гомологичной рекомбинации, в которые был встроен ген, и полученный в результате штамм был назван KCCM12218P-PgapA-serA(Avn).
Последовательности праймеров, используемых в этом примере, показаны в Таблице 18 ниже.
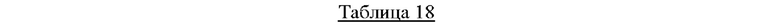
Пример 7-4: Оценка способности продуцировать триптофан штамма Corynebacterium glutamicum, в который введен ген serA(Avn), имеющий происхождение из Azotobacter
Штамм KCCM12218P-PgapA-serA(Avn), полученный в Примере 7-3, и родительский штамм KCCM12218P культивировали описанным ниже способом для идентификации продуциования ими триптофана. Каждый из штаммов инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл посевной среды, и культивировали при 30°С в течение 20 часов при встряхивании со скоростью 200 об/мин. Затем 1 мл посевной среды инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл среды для продуцирования, и культивировали при 30°С в течение 24 часов при встряхивании со скоростью 200 об/мин. После завершения культивирования для каждого штамма измеряли продукцию L-триптофана методом HPLC.
<Посевная среда (рН 7,0)>
20 г глюкозы, 10 г пептона, 5 г дрожжевого экстракта, 1,5 г мочевины, 4 г KH2PO4, 8 г K2HPO4, 0,5 г MgSO4⋅7H2O, 100 мкг биотина, 1000 мкг тиамина HCl, 2000 мкг пантотената кальция и 2000 мкг никотинамида (в расчете на 1 л дистиллированной воды).
<Среда для продуцирования (рН 7,0)>
30 г глюкозы, 15 г (NH4)2SO4, 1,2 г MgSO4⋅7H2O, 1 г KH2PO4, 5 г дрожжевого экстракта, 900 мкг биотина, 4500 мкг тиамина HCl, 4500 мкг пантотената кальция и 30 г СаСО3 (в расчете на 1 л дистиллированной воды)

Результаты оценки продукции L-триптофана штаммами KCCM12218P и KCCM12218P-PgapA-serA(Avn) показаны в Таблице 19 выше.
Если родительский штамм KCCM12218P продемонстрировал продукцию L-триптофана 2,5 г/л и накопление промежуточного продукта индола в количестве 59 мг/л, штамм, в который был введен serA(Avn), продемонстрировал продукцию L-триптофана 3,1 г/л без накопления индола.
На основании этих результатов было установлено, что запас L-серина был также достаточным за счет введения serA(Avn), имеющего происхождение из Azotobacter, в Corynebacterium glutamicum, продуцирующий L-триптофан, и было подтверждено, что выход L-триптофана также повышался без накопления индола, который является промежуточным продуктом на конечной стадии биосинтеза L-триптофана. Таким образом, видно, что синергетические эффекты в отношении продукции триптофана улучшаются при одновременном улучшении продукции предшественника.
Штамм KCCM12218P-PgapA-serA(Avn) был назван СМ05-8935 и депонирован в Корейском центре культур микроорганизмов (KCCM) согласно Будапештскому договору, и ему был присвоен номер доступа KCCM12414P 27 ноября 2018 г.
Пример 8: Получение штамма Corynebacterium glutamicum, в который введен ген serA(Avn), имеющий происхождение из Azotobacter, и оценка способности этого штамма продуцировать гистидин
Пример 8-1: Получение продуцирующего гистидин штамма Corynebacterium glutamicum
Продуцирующий L-гистидин штамм Corynebacterium glutamicum был разработан на основе штамма дикого типа АТСС13032. Для устранения ингибирования по принципу обратной связи полипептидом HisG, который является первым ферментом пути биосинтеза L-гистидина, глицин в 233-м положении с N-конца HisG был заменен гистидином, а треонин в 235-м положении с N-конца был одновременно заменен глутамином (SEQ ID NO: 88) (ACS Synth. Biol., 2014, 3 (1), pp 21-29). Дополнительно, чтобы усилить путь биосинтеза L-гистидина, гены биосинтеза (hisD-hisC-hisB-hisN), в общей сложности разделенные на 4 оперона, были получены в форме кластера, где промотор был заменен, и введены в штамм (SEQ ID NO: 89).
Для этих генетических манипуляций сначала были получены расположенная выше по ходу транскрипции и расположенная ниже по ходу транскрипции области модификаций 233-й и 235-й аминокислот гена hisG, где происходит хромосомная гомологичная рекомбинация. Конкретно, генный фрагмент расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей модификаций 233-й и 235-й аминокислот hisG был получен путем выполнения PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum АТСС13032 и праймеров с SEQ ID NO: 36 и 37, и генный фрагмент расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей модификаций 233-й и 235-й аминокислот hisG был получен путем выполнения PCR с использованием праймеров с SEQ ID NO: 38 и 39.
В качестве полимеразы использовали ДНК-полимер азу Solg™ Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 5 минут; 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 60°С в течение 30 секунд и полимеризации при 72°С в течение 60 секунд; и полимеризация при 72°С в течение 5 минут.
Рекомбинантная плазмида была получена посредством клонирования с использованием амплифицированных расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей модификаций 233-й и 235-й аминокислот hisG и вектора pDZ (патент Кореи №10-0924065) для хромосомной трансформации, расщепленного ферментом рестрикции SmaI, методом сборки Гибсона (DG Gibson et al., NATURE METHODS, VOL. 6, NO. 5, MAY 2009, NEBuilder HiFi DNA Assembly Master Mix) и названа pDZ-hisG(G233H, T235Q). Клонирование выполняли путем смешивания реактива для сборки Гибсона и рассчитанного числа моль каждого из генных фрагментов с последующим инкубированием при 50°С в течение 1 часа.
Штамм Corynebacterium glutamicum дикого типа АТСС13032 трансформировали полученным вектором pDZ-hisG(G233H, T235Q) посредством электропорации и подвергали второму процессу кроссинговера с получением штамма, имеющего замены аминокислот HisG с глицина на гистидин в 233-м положении и с треонина на глутамин в 235-м положении на хромосоме (SEQ ID NO: 88). Эти генетические манипуляции были подтверждены путем проведения PCR и геномного секвенирования с использованием праймеров с SEQ ID NO: 40 и 41, соответственно, амплифицирующих внешние области расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей гомологичной рекомбинации, в которые был встроен ген, и полученный в результате штамм был назван СА14-0011.
Последовательности праймеров, используемых в этом примере, показаны в Таблице 20 ниже.


Дополнительно для усиления пути биосинтеза L-гистидина гены биосинтеза, в общей сложности разделенные на 4 оперона, вводили в форме кластера, где промотор был заменен. Конкретно, кластер биосинтеза L-гистидина был в общей сложности разделен на четыре оперона (hisE-hisG, hisA-impA-hisF-hisI, hisD-hisC-hisB и cg0911-hisN), и получен вектор, который одновременно вводил эти гены биосинтеза в микроорганизм.
В дополнение к этому ген Ncgl1108, кодирующий гамма-аминобутиратпермеазу (Microb Biotechnol. 2014 Jan; 7 (1): 5-25)), был использован в качестве сайта инсерции кластера биосинтеза.
Для этих генетических манипуляций сначала были получены расположенная выше по ходу транскрипции и расположенная ниже по ходу транскрипции области гена Ncgl1108, где происходит хромосомная гомологичная рекомбинация. Конкретно, генный фрагмент расположенной выше по ходу транскрипции области гена Ncgl1108 был получен путем выполнения PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum АТСС13032 и праймеров с SEQ ID NO: 42 и 43, а генный фрагмент расположенной ниже по ходу транскрипции области гена Ncgl1108 был получен путем выполнения PCR с использованием праймеров с SEQ ID NO: 44 и 45.
В качестве полимеразы использовали ДНК-полимер азу Solg™ Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 5 минут; 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 60°С в течение 30 секунд и полимеризации при 72°С в течение 60 секунд; и полимеризация при 72°С в течение 5 минут.
Рекомбинантная плазмида была получена посредством клонирования с использованием амплифицированных расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей гена NCgl1108 и вектора pDZ (патент Кореи №10-0924065) для хромосомной трансформации, расщепленного ферментом рестрикции SmaI, методом сборки Гибсона (DG Gibson et al., NATURE METHODS, VOL. 6, NO. 5, MAY 2009, NEBuilder HiFi DNA Assembly Master Mix) и названа pDZ-ΔNcgl1108. Клонирование выполняли путем смешивания реактива для сборки Гибсона и рассчитанного числа моль генных фрагментов с последующим инкубированием при 50°С в течение 1 часа.
Штамм СА14-0011 трансформировали полученным вектором pDZ-ΔNcgl1108 посредством электропорации и подвергали второму процессу кроссинговера с получением штамма, в котором нарушен ген Ncg1108. Эти генетические манипуляции были подтверждены путем проведения PCR и геномного секвенирования с использованием праймеров с SEQ ID NO: 46 и 47, соответственно, амплифицирующих внешние области расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей гомологичной рекомбинации, где ген был нарушен, и полученный в результате штамм был назван СА14-0736.
Последовательности праймеров, используемых в этом примере, показаны в Таблице 21 ниже.

Дополнительно для усиления кластера биосинтеза была получена промоторная область для замены с группой генов 4 оперонов. Были получены: усиленная промоторная область lysC (далее обозначена как lysCP1, патент Кореи №10-0930203) и область hisE-hisG, промоторная область gapA и область hisA-impA-hisF-hisI, синтезированная промоторная область SPL13 (патент Кореи №10-1783170) и область hisD-hisC-hisB и синтезированная промоторная область CJ7 (патент Кореи №10-0620092 и WO 2006/065095) и область cg0911-hisN. Конкретно, PCR выполняли, используя хромосому штамма KCCM10919P (патент Кореи №10-0930203) в качестве матрицы и праймеры с SEQ ID NO: 48 и 49. В качестве полимеразы для PCR использовали высокоточную ДНК-полимеразу PfuUltraTM (Stratagene), и амплифицированные таким путем продукты PCR очищали, используя набор реактивов для очистки продуктов PCR производства QIAGEN, с получением промоторной области lysCP1. Генный фрагмент hisE-hisG был получен путем выполнения PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum СА14-0011 и праймеров с SEQ ID NO: 50 и 51. Генный фрагмент промоторной области gapA был получен путем выполнения PCR с использованием праймеров с SEQ ID NO: 52 и 53, а генный фрагмент области hisA-impA-hisF-hisI был получен путем выполнения PCR с использованием праймеров с SEQ ID NO: 54 и 55. Дополнительно PCR выполняли, используя синтезированный промотор SPL13 в качестве матрицы и праймеры с SEQ ID NO: 56 и 57, а генный фрагмент области hisD-hisC-hisB был получен путем выполнения PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum СА14-0011 и праймеров с SEQ ID NO: 58 и 59. Затем PCR выполняли, используя синтезированный промотор CJ7 в качестве матрицы и праймеры с SEQ ID NO: 60 и 61, а генный фрагмент области cg0911-hisN был получен путем выполнения PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum СА14-0011 и праймеров с SEQ ID NO: 62 и 63.
Последовательности праймеров, используемых в этом примере, показаны в Таблице 22 ниже.

В качестве полимеразы использовали ДНК-полимер азу Solg™ Pfu-X и выполняли PCR в следующих условиях амплификации: денатурация при 95°С в течение 5 минут; 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 60°С в течение 30 секунд и полимеризации при 72°С в течение 180 секунд; и полимеризация при 72°С в течение 5 минут.
Рекомбинантная плазмида была получена посредством клонирования с использованием амплифицированных: области lysCP1 и области hisE-hisG, промоторной области gapA и области hisA-impA-hisF-hisI, синтезированной промоторной области SPL13 и области hisD-hisC-hisB, синтезированной промоторной области CJ7 и области cg0911-hisN и вектора pDZ-вектора ΔNCgl1108 для хромосомной трансформации, расщепленных ферментом рестрикции Seal, методом сборки Гибсона (DG Gibson et al., NATURE METHODS, VOL. 6, NO. 5, MAY 2009, NEBuilder HiFi DNA Assembly Master Mix) и была названа pDZ-ΔNCgl1108::lysCP1_hisEG-PgapA_hisA-impA-hisFI-SPL13_HisDCB-CJ7_cg0911-hisN. Клонирование выполняли путем смешивания реактива для сборки Гибсона и рассчитанного числа моль каждого из генных фрагментов с последующим инкубированием при 50°С в течение 1 часа.
Штамм СА14-0011 трансформировали полученным вектором pDZ-ΔNcgl1108::PlysCm1_hisEG-PgapA_hisA-impA-hisFI-SPL13_HisDCB-CJ7_cg0911-hisN посредством электропорации и подвергали второму процессу кроссинговера с получением штамма, в котором были встроены гены биосинтеза. Эти генетические манипуляции были подтверждены путем выполнения PCR и геномного секвенирования с использованием праймеров с SEQ ID NO: 46 и 47, соответственно, амплифицирующих внешние области расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей гомологичной рекомбинации, в которые был встроен ген, и трансформированный штамм был назван СА14-0737.
Штамм СА14-0737 был депонирован в Корейском центре культур микроорганизмов (KCCM) согласно Будапештскому договору, и ему был присвоен номер доступа KCCM 12411Р 27 ноября 2018 г.
Пример 8-2: Получение продуцирующего His штамма Corynebacterium glutamicum, в который введен чужеродный ген serA(Avn), имеющий происхождение из Azotobacter
Чтобы идентифицировать эффект гена serA(Avn), имеющего происхождение из Azotobacter, в отношении повышения продукции L-гистидина, использовали штамм СА14-0737.
Штамм был получен путем замены гена serA(Cgl) геном serA(Avn), имеющим происхождение из Azotobacter, для экспрессии под действием промотора gapA, с использованием pDZ-PgapA-serA(Avn), полученного в Примере 7-3.
Штамм Corynebacterium glutamicum СА14-0737, продуцирующий L-гистидин, трансформировали вектором pDZ-PgapA-serA(Avn) посредством электропорации и подвергали второму процессу кроссинговера с получением штамма, в котором ген serA(Cgl) был заменен геном serA Azotobacter, экспрессируемым под действием сильного промотора gapA. Эти генетические манипуляции были подтверждены путем проведения PCRh геномного секвенирования с использованием праймеров с SEQ ID NO: 34 и 35, соответственно, амплифицирующих внешние области расположенной выше по ходу транскрипции и расположенной ниже по ходу транскрипции областей гомологичной рекомбинации, в которые был встроен ген, и полученный в результате штамм был назван СА14-0738.
Пример 8-3: Оценка продуцирующего L-гистидин штамма Corynebacterium glutamicum, в который введен чужеродный ген serA(Avn), имеющий происхождение из Azotobacter
Штаммы СА14-0011, СА14-0736, СА14-0737 и СА14-0738, полученные в Примерах 8-1 и 8-2 выше, культивировали в соответствии с описанным ниже способом для идентификации способности продуцировать L-гистидин. Каждый из штаммов инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл посевной среды, и культивировали при 30°С в течение 20 часов при встряхивании со скоростью 200 об/мин. Затем 1 мл посевной среды инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл среды для продуцирования, и культивировали при 30°С в течение 24 часов при встряхивании со скоростью 200 об/мин. После завершения культивирования измеряли продукцию L-гистидина методом HPLC.
<Посевная среда (рН 7,0)>
20 г глюкозы, 10 г пептона, 5 г дрожжевого экстракта, 1,5 г мочевины, 4 г KH2PO4, 8 г K2HPO4, 0,5 г MgSO4⋅7H2O, 100 мкг биотина, 1000 мкг тиамина HCl, 2000 мкг пантотената кальция и 2000 мкг никотинамида (в расчете на 1 л дистиллированной воды).
<Среда для продуцирования (рН 7,0)>
100 г глюкозы, 40 г (NH4)2SO4, 3 г дрожжевого экстракта, 1 г KH2PO4, 1 г MgSO4⋅7H2O, 0,01 г FeSO4⋅7H2O, 50 мкг биотина, 100 мкг тиамина и 30 г СаСО3 (в расчете на 1 л дистиллированной воды).
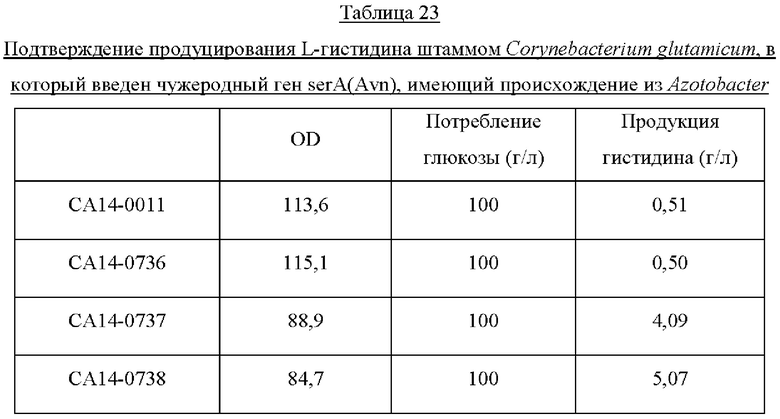
Результаты оценки продукции L-гистидина продуцирующими L-гистидин штаммами Corynebacterium glutamicum показаны в Таблице 24 выше.
В то время как родительский штамм СА14-0737, обладающий усиленной способностью продуцировать гистидин, продемонстрировал продукцию L-гистидина 4,09 г/л, штамм СА14-0738, в который был введен serA(Avn), продемонстрировал продукцию L-гистидина 5,07 г/л, что указывает на повышение продукции L-гистидина на 20% по сравнению с родительским штаммом СА14-0737.
На основании этих результатов было подтверждено, что способность продуцировать L-гистидин была усилена в результате введения serA(Avn), имеющего происхождение из Azotobacter. Штамм СА14-0738 был депонирован в Корейском центре культур микроорганизмов (KCCM) согласно Будапештскому договору, и ему был присвоен номер доступа KCCM 12412Р 27 ноября 2018 г.
Пример 9: Получение и оценка продуцирующего метионин (Met) штамма, в который введен serA Azotobacter
Пример 9-1: Получение рекомбинантного вектора для делеции гена mcbR
Чтобы получить продуцирующий метионин штамм, использовали штамм АТСС13032 для получения вектора для инактивации гена mcbR, кодирующий регулятор транскрипции метионина/цистеина (J. Biotechnol. 103:51 65, 2003).
В частности, для делетирования гена mcbR из хромосомы штамма АТСС13032 Corynebacterium glutamicum был получен рекомбинантный плазмидный вектор в соответствии с описанным ниже способом. На основе нуклеотидных последовательностей, депонированных в GenBank Национальных институтов здоровья США (NIH), был получен ген mcbR и фланкирующие последовательности Corynebacterium glutamicum (SEQ ID NO: 91).
Чтобы получить делетированный ген mcbR, выполняли PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum АТСС13032 и праймеры с SEQ ID NO: 64, 65, 66 и 67. PCR выполняли в следующих условиях амплификации: денатурация при 95°С в течение 5 минут; 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 53°С в течение 30 секунд и полимеризации при 72°С в течение 30 секунд; и полимеризация при 72°С в течение 7 минут. В результате были получены фрагменты ДНК 700 п. о. соответственно.
Вектор pDZ (патент Кореи №10-0924065), не способный реплицироваться в Corynebacterium glutamicum, и амплифицированные фрагменты гена mcbR обрабатывали ферментом рестрикции SmaI для введения в хромосому и лигировали, используя ДНК-лигазу. Е. coli DH5α трансформировали этим вектором и высевали на твердую среду LB, содержащую 25 мг/л канамицина. Отбирали колонии, трансформированные вектором, в который был встроен фрагмент, имеющий делецию гена-мишени. Затем получили плазмиду методом выделения плазмид и назвали ее pDZ-ΔmcbR.
Последовательности праймеров, используемых в этом примере, показаны в Таблице 24 ниже.

Пример 9-2: Получение рекомбинантного вектора, в котором одновременно усилены metH и cysI
Чтобы получить продуцирующий метионин штамм, использовали штамм АТСС13032 для получения вектора, в котором были усилены как ген metH (Ncgl1450), кодирующий метионинсинтазу, так и ген cysI (Ncgl2718), кодирующий сульфитредуктазу, хорошо известные в данной области техники.
Конкретно, для дополнительной инсерции генов metH и cysI в хромосому штамма АТСС13032 Corynebacterium glutamicum был получен рекомбинантный плазмидный вектор в соответствии с описанным ниже способом. На основе нуклеотидных последовательностей, депонированных в GenBank Национальных институтов здоровья США (NIH), был получен ген metH и фланкирующие последовательности (SEQ ID NO: 92) и ген cysI и фланкирующие последовательности (SEQ ID NO: 93) Corynebacterium glutamicum.
Сначала для инсерции этих генов был получен вектор для удаления Ncgl1021 (транспозазы). На основе нуклеотидных последовательностей, депонированных в GenBank Национальных институтов здоровья США (NIH), был получен ген Ncgl1021 и фланкирующие последовательности (SEQ ID NO: 94) Corynebacterium glutamicum. Чтобы получить делетированный ген Ncgl1021, выполняли PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum АТСС13032 и праймеры с SEQ ID NO: 68, 69, 70 и 71. PCR выполняли в следующих условиях амплификации: денатурация при 95°С в течение 5 минут; 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 53°С в течение 30 секунд и полимеризации при 72°С в течение 30 секунд; и полимеризация при 72°С в течение 7 минут. В результате были получены фрагменты ДНК. Вектор pDZ, не способный реплицироваться в Corynebacterium glutamicum (патент Кореи №10-0924065), и амплифицированные генные фрагменты Ncgl1021 обрабатывали ферментом рестрикции XbaI для введения в хромосому и клонировали методом сборки Гибсона. Е. coli DH5α трансформировали этим вектором и высевали на твердую среду LB, содержащую 25 мг/л канамицина. Отбирали колонии, трансформированные вектором, в который был встроен фрагмент, имеющий делецию гена-мишени. Затем получили плазмиду методом выделения плазмид и назвали ее pDZ-ΔNcgl1021.
Впоследствии, чтобы получить гены metH и cysI, выполняли PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium glutamicum АТСС13032 и праймеров с SEQ ID NO: 72, 73, 74 и 75. Дополнительно для усиления экспрессии гена metH использовали промотор Pcj7, а для усиления экспрессии гена cysI использовали промотор Pspl1. Для получения этих генов сначала был получен промотор Pcj7 путем выполнения PCR с использованием в качестве матрицы хромосомной ДНК Corynebacterium ammoniagenes АТСС 6872 и праймеров с SEQ ID NO: 76 и 77, а промотор Pspl1 был получен путем выполнения PCR с использованием в качестве матрицы ДНК вектора spl1-GFP, известного в данной области техники (патент Кореи №10-1783170) и праймеров с SEQ ID NO: 78 и 79. PCR выполняли в следующих условиях амплификации: денатурация при 95°С в течение 5 минут; 30 циклов денатурации при 95°С в течение 30 секунд, отжига при 53°С в течение 30 секунд и полимеризации при 72°С в течение 30 секунд; и полимеризация при 72°С в течение 7 минут. В результате были получены фрагменты ДНК гена metH, гена cysI, промотора Pcj7 и промотора Pspl1.
После этого вектор pDZ-ΔNcgl1021, не способный реплицироваться в Corynebacterium glutamicum, обрабатывали ферментом рестрикции SeaI и обрабатывали 4 амплифицированных фрагмента ДНК ферментом рестрикции SeaI и клонировали методом сборки Гибсона. Е. coli DH5α трансформировали этим вектором и высевали на твердую среду LB, содержащую 25 мг/л канамицина. Отбирали колонии, трансформированные вектором, в который был встроен фрагмент, имеющий делецию гена-мишени. Затем получили плазмиду методом выделения плазмид и назвали ее pDZ-ΔNcgl1021 -Pcj7metH-Pspl1cysI.
Последовательности праймеров, используемых в этом примере, показаны в Таблице 25 ниже.

Пример 9-3: Разработка продуцирующего L-метионин штамма и продуцирование L-метионина с использованием этого штамма
Штамм АТСС13032 трансформировали каждым из векторов pDC-ΔmcBR, pDZ-ΔNcgl1021 и pDZ-ΔNcgl1021-Pcj7metH-Pspl1cysI, полученных, как описано выше, путем электропорации посредством хромосомной гомологичной рекомбинации (van der Rest et al., Appl Microbiol Biotechnol 52:541 545, 1999). Затем выполняли вторую рекомбинацию на твердой среде, содержащей сахарозу. После завершения второй рекомбинации трансформированный штамм Corynebacterium glutamicum, имеющий делецию гена mcBR, идентифицировали путем выполнения PCR с использованием праймеров с SEQ ID NO: 80 и 81, а трансформированный штамм, имеющий делецию гена Ncgl1021 и инсерцию гена Pcj7-metH-Pspl1cysI в сайте Ncgl1021, идентифицировали путем выполнения PCR с использованием праймеров с SEQ ID NO: 82 и 83. Каждый из этих рекомбинантных штаммов получил название - Corynebacterium glutamicum 13032/ΔmcbR, 13032/ΔNcgl1021 и 13032/ΔNcgl102l-Pcj7metH-Pspl1cysI.
Последовательности праймеров, используемых в этом примере, показаны в Таблице 26 ниже.

Для оценки способности продуцировать L-метионин полученных штаммов 13032/ΔmcbR, 13032/ΔNcgl1021 и CJP13032/ΔNcgl1021-Pcj7metH-Pspl1cysI эти штаммы и родительский штамм Corynebacterium glutamicum АТСС13032 культивировали в соответствии со следующим способом.
Каждый из штаммов Corynebacterium glutamicum АТСС13032 и Corynebacterium glutamicum 13032/ΔmcbR, 13032/ΔNcgl1021 и 13032/ΔNcgl1021-Pcj7metH-Pspl1cysI no настоящему изобретению инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл описанной ниже посевной среды и культивировали при 30°С в течение 20 часов при встряхивании со скоростью 200 об/мин. Затем 1 мл посевной культуры инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 24 мл среды для продуцирования, и культивировали при 30°С в течение 48 часов при встряхивании со скоростью 200 об/мин. Композиции посевной среды и среды для продуцирования описаны ниже.
<Посевная среда (рН 7,0)>
20 г глюкозы, 10 г пептона, 5 г дрожжевого экстракта, 1,5 г мочевины, 4 г KH2PO4, 8 г K2HPO4, 0,5 г MgSO4⋅7H2O, 100 мкг биотина, 1000 мкг тиамина HCl, 2000 мкг пантотената кальция и 2000 мкг никотинамида (в расчете на 1 л дистиллированной воды).
<Среда для продуцирования (рН 8,0)>
50 г глюкозы, 12 г (NH4)2SO2, 5 г дрожжевого экстракта, 1 г KH2PO4, 1,2 г MgSO4⋅7H2O, 100 мкг биотина, 1000 мкг тиамина HCl, 2000 мкг пантотената кальция, 3000 мкг никотинамида и 30 г СаСО3 (в расчете на 1 л дистиллированной воды).
Концентрации L-метионина, содержащиеся в культурах, полученных путем культивирования штаммов в соответствии с описанным выше способом, были проанализированы и показаны в Таблице 27 ниже.

В результате было подтверждено, что штамм, в котором был только делетирован ген mcbR, продемонстрировал продукцию L-метионина 0,12 г/л, что указывает на повышение по сравнению с контрольным штаммом. В дополнение к этому штамм с сверхэкспрессией генов metH и cysI без делеции mcBR продемонстрировал продукцию L-метионина 0,18 г/л, что указывает на повышение по сравнению с контрольным штаммом.
Пример 9-4: Получение вектора с сверхэкспрессией имеющего происхождение из Azotobacter гена D-3-фосфоглицератдегидрогеназы (serA(Avn))
Экспрессионный вектор был получен, чтобы идентифицировать, улучшается ли способность продуцировать метионин в результате усиления имеющего происхождение из Azotobacter гена D-3-фосфоглицератдегидрогеназы (далее обозначенного serA(Avn)).
Для экспрессии гена serA(Avn) (SEQ ID NO: 1), кодирующего SerA(Avn), использовали челночный вектор pECCG117 (Biotechnology letters vol 13, No. 10, p.721-726 1991 или публикация патента Кореи №92-7401), доступный для трансформации Corynebacterium glutamicum. В качестве промотора экспрессии использовали промотор spl1 (далее Pspl1) с получением вектора pECCG117-Pspl1-serA(Avn). PCR для Pspl1 выполняли, используя праймеры с SEQ ID NO: 84 и 85, a PCR для чужеродного serA(Avn) выполняли, используя праймеры с SEQ ID NO: 86 и 87. Амплифицированные генные фрагменты Pspl1 и serA(Avn) клонировали методом сборки Гибсона, используя вектор pECCG117, обработанный ферментом рестрикции EcoRV, в результате чего получили pECCG117-Pspl1-serA(Avn).
Последовательности праймеров, используемых в этом примере, показаны в Таблице 28 ниже.
Пример 9-5: Получение продуцирующего L-метионин штамма, в который введен serA(Avn), имеющий происхождение из Azotobacter, с использованием штамма Е. coli дикого типа и оценка способности продуцировать L-метионин
Штаммы 13032/ΔmcbR и 13032/ΔNcgl1021-Pcj7metH-Pspl1cysI трансформировали описанным выше вектором pECCG117-Pspl1-serA(Avn) посредством электропорации, соответственно (van der Rest et al., Appl Microbiol Biotechnol 52:541-545, 1999). Рекомбинантные штаммы были названы Corynebacterium glutamicum 13032/ΔmcbR (pECCG117-Pspl1-serA(Avn)) и 13032/ΔNcgl1021-Pcj7metH-Pspl1cysI (pECCG117-Pspl1-serA(Avn)), соответственно.
Чтобы оценить способность полученных рекомбинантных штаммов 13032/ΔmcbR (pECCG117-Pspl1-serA(Avn)) и 13032/ΔNcgl1021-Pcj7metH-Pspl1cysI (pECCG117-Pspl1-serA(Avn)) продуцировать L-метионин, эти штаммы и их родительские штаммы (13032/ΔmcbR и 13032/ΔNcgl1021-Pcj7metH-Pspl1cysI) культивировали в соответствии со следующим способом.
Каждый из штаммов Corynebacterium glutamicum АТСС13032 и полученных штаммов Corynebacterium glutamicum 13032/ΔmcbR, 13032/ΔNcgl1021 и 13032/ΔNcgl1021-Pcj7metH-Pspl1cysI в соответствии с описанием настоящего изобретения инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 25 мл описанной ниже посевной среды и культивировали при 30°С в течение 20 часов при встряхивании со скоростью 200 об/мин. Затем 1 мл посевной культуры инокулировали в колбу с угловыми перегородками объемом 250 мл, содержащую 24 мл среды для продуцирования, и культивировали при 30°С в течение 48 часов при встряхивании со скоростью 200 об/мин. В частности, штаммы, в которые был включен вектор, культивировали после дополнительного добавления к ним канамицина (25 мг/л). Композиции посевной среды и среды для продуцирования описаны ниже.
<Посевная среда (рН 7,0)>
20 г глюкозы, 10 г пептона, 5 г дрожжевого экстракта, 1,5 г мочевины, 4 г KH2PO4, 8 г K2HPO4, 0,5 г MgSO4⋅7H2O, 100 мкг биотина, 1000 мкг тиамина HCl, 2000 мкг пантотената кальция и 2000 мкг никотинамида (в расчете на 1 л дистиллированной воды).
<Среда для продуцирования (рН 8,0)>
50 г глюкозы, 12 г (NH4)2SO2, 5 г дрожжевого экстракта, 1 г KH2PO4, 1,2 г MgSO4⋅7H2O, 100 мкг биотина, 1000 мкг тиамина HCl, 2000 мкг пантотената кальция, 3000 мкг никотинамида и 30 г СаСО3 (в расчете на 1 л дистиллированной воды).
Концентрации L-метионина, содержащиеся в культуре, полученной путем культивирования штаммов в соответствии с описанным выше способом, были проанализированы и показаны в Таблице 29 ниже.

В результате было подтверждено, что оба штамма, трансформированные pECCG117-Pspl1-serA(Avn), продемонстрировали повышение продукции L-метионина по сравнению с контрольным штаммом. В дополнение к этому штамм 13032/ΔmcbR (pECCG117-Pspl1-serA(Avn)) продемонстрировал повышение продукции L-метионина на 83% по сравнению с контрольным штаммом, а штамм 13032/ΔNcgl1021-Pcj7metH-Pspl1cysI pECCG117-Pspl1-serA(Avn)) продемонстрировал повышение продукции L-метионина на 78% по сравнению с контрольным штаммом. Таким образом, в соответствии с данным примером было подтверждено, что способность микроорганизмов продуцировать L-метионин была улучшена в результате введения в них serA(Avn), имеющего происхождение из Azotobacter.
Штамм 13032/ΔmcbR был назван СМ02-0618 и депонирован в Корейском центре культур микроорганизмов (KCCM) согласно Будапештскому договору, и ему был присвоен номер доступа KCCM12425P 4 января 2019 г. В дополнение к этому штамм 13032/ΔmcbR (pECCG117-Pspl1-serA(Avn)) был назван СМ02-0693 и депонирован в Корейском центре культур микроорганизмов (KCCM) согласно Будапештскому договору, и ему был присвоен номер доступа KCCM12413P 27 ноября 2018 г.
Хотя описание настоящего изобретения приведено со ссылкой на конкретные иллюстративные воплощения, специалистам в области техники, к которой относится описание настоящего изобретения, будет понятно, что настоящее изобретение может быть воплощено в других конкретных формах без отклонения от технической сущности или существенных характеристик настоящего изобретения. Таким образом, описанные выше воплощения рассматривают как иллюстративные и во всех отношениях не имеющие ограничительного характера. Кроме того, объем настоящего изобретения должен быть определен прилагаемой формулой изобретения, а не его подробным описанием, и следует понимать, что все модификации или варианты, выведенные из значений и объема настоящего изобретения, и их эквиваленты включены в объем настоящего изобретения.
--->
<110> CJ CheilJedang Corporation
<120> MICROORGANISM PRODUCING L-AMINO ACID AND METHOD OF PRODUCING
L-AMINO ACID USING THE SAME
<130> OPA19298
<150> KR 10-2019-0054430
<151> 2019-05-09
<160> 95
<170> KoPatentIn 3.0
<210> 1
<211> 409
<212> PRT
<213> Unknown
<220>
<223> полипептид
<400> 1
Met Ser Lys Thr Ser Leu Asp Lys Ser Lys Ile Arg Phe Leu Leu Leu
1 5 10 15
Glu Gly Val His Gln Thr Ala Leu Asp Thr Leu Lys Ala Ala Gly Tyr
20 25 30
Thr Asn Ile Glu Tyr Leu Thr Gly Ser Leu Pro Glu Glu Gln Leu Lys
35 40 45
Glu Lys Ile Ala Asp Ala His Phe Ile Gly Ile Arg Ser Arg Thr Gln
50 55 60
Leu Thr Glu Glu Val Phe Asp Arg Ala Lys Lys Leu Val Ala Val Gly
65 70 75 80
Cys Phe Cys Ile Gly Thr Asn Gln Val Asp Leu Glu Ala Ala Arg Glu
85 90 95
Arg Gly Ile Ala Val Phe Asn Ala Pro Tyr Ser Asn Thr Arg Ser Val
100 105 110
Ala Glu Leu Val Leu Ala Glu Ala Ile Leu Leu Leu Arg Gly Ile Pro
115 120 125
Glu Lys Asn Ala Ala Ser His Arg Gly Gly Trp Leu Lys Ser Ala Ser
130 135 140
Asn Ser Tyr Glu Ile Arg Gly Lys Lys Leu Gly Ile Ile Gly Tyr Gly
145 150 155 160
Ser Ile Gly Thr Gln Leu Ser Val Leu Ala Glu Ser Leu Gly Met Gln
165 170 175
Val Leu Phe Tyr Asp Val Val Thr Lys Leu Pro Leu Gly Asn Ala Ala
180 185 190
Gln Val Gly Asn Leu Tyr Asp Leu Leu Gly Gln Ala Asp Ile Val Thr
195 200 205
Leu His Val Pro Glu Thr Ala Ala Thr Lys Trp Met Ile Gly Glu Lys
210 215 220
Glu Ile Arg Ala Met Lys Lys Gly Ala Ile Leu Leu Asn Ala Ala Arg
225 230 235 240
Gly Thr Val Val Asp Ile Asp Ala Leu Ala Ala Ala Leu Arg Asp Lys
245 250 255
His Leu Asn Gly Ala Ala Ile Asp Val Phe Pro Val Glu Pro Arg Ser
260 265 270
Asn Asn Asp Glu Phe Val Ser Pro Leu Arg Glu Phe Asp Asn Val Ile
275 280 285
Leu Thr Pro His Val Gly Gly Ser Thr Met Glu Ala Gln Ala Asn Ile
290 295 300
Gly Ser Glu Val Ala Glu Lys Leu Val Lys Tyr Ser Asp Asn Gly Thr
305 310 315 320
Ser Val Ser Ser Val Asn Phe Pro Glu Val Ala Leu Pro Ser His Pro
325 330 335
Gly Lys His Arg Leu Leu His Ile His Lys Asn Ile Pro Gly Val Met
340 345 350
Ser Glu Ile Asn Lys Val Phe Ala Glu Asn Gly Ile Asn Ile Ser Gly
355 360 365
Gln Phe Leu Gln Thr Asn Glu Thr Val Gly Tyr Val Val Ile Asp Val
370 375 380
Asp Ala Glu Tyr Ser Glu Met Ala Leu Glu Lys Leu Gln Gln Val Asn
385 390 395 400
Gly Thr Ile Arg Ser Arg Val Leu Phe
405
<210> 2
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 2
aggtcgactc tagaggatcc cccgcttgct gcaactctct 40
<210> 3
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 3
gatatctttc ctgtgtga 18
<210> 4
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 4
aatttcacac aggaaagata tcatgagtaa gacctccctg 40
<210> 5
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 5
gtgaattcga gctcggtacc ctcagaacag aacccgtgag 40
<210> 6
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 6
aatttcacac aggaaagata tcatggcaaa ggtatcgctg 40
<210> 7
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 7
gtgaattcga gctcggtacc cttagtacag cagacgggcg 40
<210> 8
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 8
cctcaccacg ttgcgtctcg agtcagaaca gaacccgtga 40
<210> 9
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 9
ctcgagacgc aacgtggtga 20
<210> 10
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 10
agtgaattcg agctcggtac ccttaaccgt gacggcgttc 40
<210> 11
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 11
ctcaccacgt tgcgtctcga gttagtacag cagacgggcg 40
<210> 12
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 12
tgcaatgcat aacaacgcag tcgcactatt tttcactgga gagaagccct gtgtaggctg 60
gagctgcttc 70
<210> 13
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 13
tgcaatgcat aacaacgcag tcgcactatt tttcactgga gagaagccct gtccatatga 60
atatcctcct 70
<210> 14
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 14
gggcaggatc tcctgtcatc 20
<210> 15
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 15
aaatgtcgga taaggcaccg 20
<210> 16
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 16
tgtaatattc acagggatca ctgtaattaa aataaatgaa ggattatgta gtgtaggctg 60
gagctgcttc 70
<210> 17
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 17
tgtagggtaa gagagtggct aacatcctta tagccactct gtagtattaa gtccatatga 60
atatcctcct 70
<210> 18
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 18
acatccttat agccactctg 20
<210> 19
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 19
tacaaccggg ggaggcattt tgcttccccc gctaacaatg gcgacatatt gtgtaggctg 60
gagctgcttc 70
<210> 20
<211> 70
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 20
gcattcggtg cacgatgcct gatgcgccac gtcttatcag gcctacaaaa gtccatatga 60
atatcctcct 70
<210> 21
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 21
aggacggata aggcgttcac 20
<210> 22
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 22
gaattcatgc aaacacaaaa accgac 26
<210> 23
<211> 25
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 23
gaattctcag aaagtctcct gtgca 25
<210> 24
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 24
cgcttatcgc gacaattcca ccgcgctttt tcaccag 37
<210> 25
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 25
ctggtgaaaa agcgcggtgg aattgtcgcg ataagcg 37
<210> 26
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 26
tcgagctcgg tacccggaag atctagtcgg atacg 35
<210> 27
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 27
tcgtttttag gcctccgact actttgggca atcct 35
<210> 28
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 28
tctgttctga ttagagatcc atttgcttga ac 32
<210> 29
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 29
ctctagagga tcccctcacc cagctcaaag ctgat 35
<210> 30
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 30
tgcccaaagt agtcggaggc ctaaaaacga ccgag 35
<210> 31
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 31
tcttactcat gttgtgtctc ctctaaag 28
<210> 32
<211> 28
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 32
gagacacaac atgagtaaga cctccctg 28
<210> 33
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 33
ggatctctaa tcagaacaga acccgtgag 29
<210> 34
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 34
accaagagtt cgaagaccag 20
<210> 35
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 35
ttcagtggct tccacatcgc 20
<210> 36
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 36
tcgagctcgg tacccatcgc catctacgtt gctgg 35
<210> 37
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 37
gtgccagtgg ggatacctgt gggtgggata agcctggggt tactg 45
<210> 38
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 38
aaccccaggc ttatcccacc cacaggtatc cccactggca cgcga 45
<210> 39
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 39
ctctagagga tccccgggac gtggttgatg gtggt 35
<210> 40
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 40
atggaaatcc tcgccgaagc 20
<210> 41
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 41
atcgatgggg aactgatcca 20
<210> 42
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 42
tcgagctcgg tacccatcgc catctacgtt gctgg 35
<210> 43
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 43
gagtctagaa gtactcgaga tgctgacctc gtttc 35
<210> 44
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 44
agcatctcga gtacttctag actcgcacga aaaag 35
<210> 45
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 45
ctctagagga tcccctttgg gcagagctca aattc 35
<210> 46
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 46
agtttcgtaa cccaccttgc 20
<210> 47
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 47
cgcttctcaa tctgatgaga 20
<210> 48
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 48
gtcagcatct cgagtgctcc ttagggagcc atctt 35
<210> 49
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 49
gtcaaatgtc ttcacatgtg tgcacctttc gatct 35
<210> 50
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 50
gaaaggtgca cacatgtgaa gacatttgac tcgct 35
<210> 51
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 51
tcgtttttag gcctcctaga tgcgggcgat gcgga 35
<210> 52
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 52
atcgcccgca tctaggaggc ctaaaaacga ccgag 35
<210> 53
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 53
gacagttttg gtcatgttgt gtctcctcta aagat 35
<210> 54
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 54
tagaggagac acaacatgac caaaactgtc gccct 35
<210> 55
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 55
tgaagcgccg gtaccgctta cagcaaaacg tcatt 35
<210> 56
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 56
cgttttgctg taagcggtac cggcgcttca tgtca 35
<210> 57
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 57
agtgacattc aacattgttt tgatctcctc caata 35
<210> 58
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 58
gaggagatca aaacaatgtt gaatgtcact gacct 35
<210> 59
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 59
cgctgggatg tttctctaga gcgctccctt agtgg 35
<210> 60
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 60
aagggagcgc tctagagaaa catcccagcg ctact 35
<210> 61
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 61
agtcatgcct tccatgagtg tttcctttcg ttggg 35
<210> 62
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 62
cgaaaggaaa cactcatgga aggcatgact aatcc 35
<210> 63
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 63
cgagtctaga agtgcctatt ttaaacgatc cagcg 35
<210> 64
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 64
tcgagctcgg tacccctgcc tggtttgtct tgta 34
<210> 65
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 65
cggaaaatga agaaagttcg gccacgtcct ttcgg 35
<210> 66
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 66
aggacgtggc cgaactttct tcattttccg aaggg 35
<210> 67
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 67
ctctagagga tccccgtttc gatgcccact gagca 35
<210> 68
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 68
acccggggat cctctagaat gtttgtgatg cgcag 35
<210> 69
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 69
gtcagagagt acttacgctg atcgggaggg aaagc 35
<210> 70
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 70
atcagcgtaa gtactctctg actagcgtca ccctc 35
<210> 71
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 71
ctgcaggtcg actctagaaa agggattgga gtgtt 35
<210> 72
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 72
caacgaaagg aaacaatgtc tacttcagtt acttc 35
<210> 73
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 73
tcgagctcgg tacccctgcg acagcatgga actc 34
<210> 74
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 74
atcaaaacag atatcatgac aacaaccacc ggaag 35
<210> 75
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 75
cgctagtcag agagttcaca ccaaatcttc ctcag 35
<210> 76
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 76
ccgatcagcg taagtagaaa catcccagcg ctact 35
<210> 77
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 77
aactgaagta gacattgttt cctttcgttg ggtac 35
<210> 78
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 78
tactttaacg tctaaggtac cggcgcttca tgtca 35
<210> 79
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 79
ggtggttgtt gtcatgatat ctgttttgat ctcct 35
<210> 80
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 80
aatctggatt tccgccaggt 20
<210> 81
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 81
cttcctaact cctgaggaag 20
<210> 82
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 82
atccccatcg gcatctttat 20
<210> 83
<211> 20
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 83
cgatcacact gggctgatct 20
<210> 84
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 84
atcgataagc ttgatggtac cggcgcttca tgtca 35
<210> 85
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 85
ggaggtctta ctcatgatat ctgttttgat ctcct 35
<210> 86
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 86
atcaaaacag atatcatgag taagacctcc ctgga 35
<210> 87
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> праймер
<400> 87
ctgcaggaat tcgattcaga acagaacccg tgagc 35
<210> 88
<211> 281
<212> PRT
<213> Unknown
<220>
<223> полипептид
<400> 88
Met Leu Lys Ile Ala Val Pro Asn Lys Gly Ser Leu Ser Glu Arg Ala
1 5 10 15
Met Glu Ile Leu Ala Glu Ala Gly Tyr Ala Gly Arg Gly Asp Ser Lys
20 25 30
Ser Leu Asn Val Phe Asp Glu Ala Asn Asn Val Glu Phe Phe Phe Leu
35 40 45
Arg Pro Lys Asp Ile Ala Ile Tyr Val Ala Gly Gly Gln Leu Asp Leu
50 55 60
Gly Ile Thr Gly Arg Asp Leu Ala Arg Asp Ser Gln Ala Asp Val His
65 70 75 80
Glu Val Leu Ser Leu Gly Phe Gly Ser Ser Thr Phe Arg Tyr Ala Ala
85 90 95
Pro Ala Asp Glu Glu Trp Ser Ile Glu Lys Leu Asp Gly Lys Arg Ile
100 105 110
Ala Thr Ser Tyr Pro Asn Leu Val Arg Asp Asp Leu Ala Ala Arg Gly
115 120 125
Leu Ser Ala Glu Val Leu Arg Leu Asp Gly Ala Val Glu Val Ser Ile
130 135 140
Lys Leu Gly Val Ala Asp Ala Ile Ala Asp Val Val Ser Thr Gly Arg
145 150 155 160
Thr Leu Arg Gln Gln Gly Leu Ala Pro Phe Gly Glu Val Leu Cys Thr
165 170 175
Ser Glu Ala Val Ile Val Gly Arg Lys Asp Glu Lys Val Thr Pro Glu
180 185 190
Gln Gln Ile Leu Leu Arg Arg Ile Gln Gly Ile Leu His Ala Gln Asn
195 200 205
Phe Leu Met Leu Asp Tyr Asn Val Asp Arg Asp Asn Leu Asp Ala Ala
210 215 220
Thr Ala Val Thr Pro Gly Leu Ser His Pro Gln Val Ser Pro Leu Ala
225 230 235 240
Arg Asp Asn Trp Val Ala Val Arg Ala Met Val Pro Arg Arg Ser Ala
245 250 255
Asn Ala Ile Met Asp Lys Leu Ala Gly Leu Gly Ala Glu Ala Ile Leu
260 265 270
Ala Ser Glu Ile Arg Ile Ala Arg Ile
275 280
<210> 89
<211> 10676
<212> DNA
<213> Unknown
<220>
<223> полинуклеотид
<400> 89
gctccttagg gagccatctt ttggggtgcg gagcgcgatc cggtgtctga ccacggtgcc 60
ccatgcgatt gttaatgccg atgctagggc gaaaagcacg gcgagcagat tgctttgcac 120
ttgattcagg gtagttgact aaagagttgc tcgcgaagta gcacctgtca cttttgtctc 180
aaatattaaa tcgaatatca atatatggtc tgtttattgg aacgcgtccc agtggctgag 240
acgcatccgc taaagcccca ggaaccctgt gcagaaagaa aacactcctc tggctaggta 300
gacacagttt attgtggtag agttgagcgg gtaactgtca gcacgtagat cgaaaggtgc 360
acacatgtga agacatttga ctcgctgtac gaagaacttc ttaaccgtgc tcagacccgc 420
cctgaagggt ctggaaccgt ggccgccttg gataaaggca tccatcatct aggtaagaag 480
gtcatcgaag aagccggaga ggtctggatt gcagccgagt atgagaccga tgaagagcta 540
gccggagaaa tctcccagct catttattgg acccaggtca tcatggttgc tcgcggcctg 600
aagccagaag atatctacaa gaacctgtag gagttttaaa gcaatcatgt tgaaaatcgc 660
tgtcccaaac aaaggctcgc tgtccgagcg cgccatggaa atcctcgccg aagcaggcta 720
cgcaggccgt ggagattcca aatccctcaa cgtttttgat gaagcaaaca acgttgaatt 780
cttcttcctt cgccctaaag atatcgccat ctacgttgct ggtggccagc tcgatttggg 840
tatcaccggc cgcgaccttg ctcgcgattc ccaggctgat gtccacgaag ttctttccct 900
cggcttcggt tcctccactt tccgttacgc agcaccagct gatgaagagt ggagcatcga 960
aaagctcgac ggcaagcgca tcgctacctc ttaccccaac cttgttcgcg atgacctcgc 1020
agcacgtggg ctttccgctg aggtgctccg cctcgacggt gcagtagagg tatccatcaa 1080
gcttggtgtc gcagatgcca tcgccgatgt tgtatccacc ggccgcacgc tgcgtcagca 1140
aggtcttgca cctttcggcg aggttctgtg cacctctgag gctgtcattg ttggccgcaa 1200
ggatgaaaag gtcaccccag agcagcagat cctgcttcgc cgcatccagg gaattttgca 1260
cgcgcagaac ttcctcatgc tggattacaa cgtcgaccgc gacaacctgg acgctgccac 1320
tgcagtaacc ccaggcttat cccacccaca ggtatcccca ctggcacgcg acaactgggt 1380
tgctgtacgc gccatggtgc cacgcaggtc agctaacgcc atcatggata agcttgctgg 1440
actcggcgct gaagccatcc tggcttctga aatccgcatc gcccgcatct aggaggccta 1500
aaaacgaccg agcctattgg gattaccatt gaagccagtg tgagttgcat cacattggct 1560
tcaaatctga gactttaatt tgtggattca cgggggtgta atgtagttca taattaaccc 1620
cattcggggg agcagatcgt agtgcgaacg atttcaggtt cgttccctgc aaaaactatt 1680
tagcgcaagt gttggaaatg cccccgtttg gggtcaatgt ccatttttga atgtgtctgt 1740
atgattttgc atctgctgcg aaatctttgt ttccccgcta aagttgagga caggttgaca 1800
cggagttgac tcgacgaatt atccaatgtg agtaggtttg gtgcgtgagt tggaaaaatt 1860
cgccatactc gcccttgggt tctgtcagct caagaattct tgagtgaccg atgctctgat 1920
tgacctaact gcttgacaca ttgcatttcc tacaatcttt agaggagaca caacatgacc 1980
aaaactgtcg cccttctcga ctacggatct ggaaaccttc gttctgctca acgcgcacta 2040
gagcgtgccg gtgcagaagt tatcgtgagc tccgatccag aagtttgcac caacgctgat 2100
ggcctcctag ttcctggagt gggcgcattt gatgcctgca tgaagggttt gaaaaacgtc 2160
ttcggacatc gcattatcgg acagcgtctt gctggtggac gtccagtgat gggtatttgt 2220
gtgggcatgc agatcctgtt cgatgaaggc gatgagcacg gcattaagtc agctggttgc 2280
ggcgagtggc ctggcaaagt ggaacgcctc caagcggaga tcctgcctca catggggtgg 2340
aacacacttg aaatgcctac caactcacca atgtttgagg gaatttcacc tgatgagcgt 2400
ttctacttcg tgcactccta tggtgtgcgc aagtggacgt tggaaaccga cgatctgacc 2460
acgcctccag aggttgtgtg ggcgaagcac gaaaatgatc gttttgtggc agctgtggaa 2520
aacggcacgc tgtgggctac tcaattccac ccagaaaaat caggtgacgc aggcgcacag 2580
ctactgcgaa actggatcaa ctacatctaa cagataggat caatattcat gaccttcact 2640
attcttcctg cagtcgatgt agttaacgga caagcagttc gcctagatca gggcgaggcc 2700
ggcactgaaa agtcttatgg cacccctttg gaatccgcac tgaagtggca ggagcagggt 2760
gcaaagtggt tgcactttgt ggacctggac gcagcgttca accgtggttc caaccatgag 2820
atgatggcgg aaattgtcgg caagctcgat gttgatgtgg agctcactgg cggtatccgt 2880
gatgatgagt ctctggagcg cgcgctggca accggtgcac gtcgtgtaaa cattggtacc 2940
gctgctctgg agaagccaga gtggattgct tctgcgattc aacgctatgg cgagaagatt 3000
gctgtcgata tcgctgtgcg tttggaagat ggtgaatggc gcacccgtgg aaacggttgg 3060
gtctccgatg gtggcgatct gtgggaagtt ctcgagcgtt tggattccca aggttgtgca 3120
cgtttcgtgg ttaccgatgt gtccaaggac ggcaccttga gtggtccaaa tgttgagctg 3180
ctgcgtgagg ttgctgcagc tacagacgca cctatcgtgg catctggtgg aatttctgtt 3240
ttggaagatg ttttggaact agccaagtac caggatgagg gcattgattc cgtcatcatt 3300
ggcaaggcac tttatgagca caagttcacc ctcgaagagg ctttggctgc agtagaaaag 3360
ctcggttaat acatggatgc tcgtgggatg ttggccattg cggaggccgt tgtagatgat 3420
gccgaagccc tcttcatgca gggcttcgga gctgcacctg cccatatgaa atccccgggg 3480
gattttgcca cggaagtgga tatggccatc gaatcccata tgcgttcgat gctgaacatg 3540
atgacaggca ttgctgtcat cggtgaagaa ggtggcggtg cgacctccgg cacgcgctgg 3600
gtgattgatc ccatcgacgg caccgccaac ttcgcggcgt ccaacccgat gagcgcgatc 3660
ctggtgtctt tgcttgtcga cgaccagccc gtcctgggta ttacctccat gcccatgctg 3720
ggtaaacgcc tcaccgcttt tgaaggttca ccgctgatga tcaacggtga acctcaggaa 3780
ccattgcaag aacaatccag tttggtatcc cacattggtt ttagttccat ggcctccccg 3840
cgcaatacag cgtttcctgt ggagttgcgt cgggatcttc tgaccgagct cacggaatcg 3900
tatcttcgtc cccgcattac aggttcggtg ggtgttgatc tcgcgttcac tgcgcagggc 3960
atttttggag catgcgtatc gtttagtcct catgtttggg acaattccgc aggcgtgatg 4020
ttgatgcgcg ctgctggtgc acaagttact gacaccgaag gccatccgtg ggcaccaggt 4080
aggggagtcg tggccggaac aaaaagggct cacgatgtgc tgttaagtaa gattgaaaaa 4140
gttcggttga tgcatgcaga tgcaggtaat gaccagtcgt taaatgagga gtacaagtaa 4200
aatgggcgtg gcaattcgag ttattccttg cctggacgtg gacaacggcc gggttgttaa 4260
aggcgtgaac tttgaaaacc tccgcgatgc tggcgatcct gtggagttgg caaagcgcta 4320
tgacgaggaa ggggcagatg agctgacctt cctggatgtc accgcctcga agcatggtcg 4380
cggcaccatg ctggatgttg ttcgacgcac cgctgatcag gtgttcatcc ctctgactgt 4440
cggtggcggc gtgcgcagcg aagaagatgt tgatcaattg ctgcgcgctg gcgccgacaa 4500
ggtttcggtg aacacgtctg cgattgcccg tccagaactg ctgtcagagc tgtccaagcg 4560
ttttggtgct cagtgcatcg tgttgtctgt ggatgccagg cgcgttcctg aaggtggaac 4620
tcctcagcca tctggttttg aagtcaccac ccacggcggt tccaagtccg cagaacttga 4680
tgcaatcgag tgggcaaagc gcggcgaaga gctgggcgtt ggcgaaattc tgctcaactc 4740
catggacggc gacggcacca aaaacggctt tgacctagag ctgctggaaa aagttcgcgc 4800
agccgtatcc attcctgtaa tcgcctccgg cggcgctggc aaggcggagc atttcccacc 4860
agctgttgca gctggcgcca acgcagtgct tgccgcgacc attttccact tccgcgaagt 4920
aaccatcgcc gaagtaaagg gagccattaa agatgcagga tttgaggtgc ggaaatgagt 4980
gacaatccac aagagtatga gctggattgg gacgtcgaaa agcgattaaa gcttaacgac 5040
gccggcctgg tgccggcaat cgtccaggcc gacgggacca acgaggtcct catgatggcc 5100
tggatggata cccacgcgct agcctatact ttggcgaccc gccgtggaac ctatttttct 5160
aggtcccgca acgagtactg gatcaagggc ctgacctctg gaaacgtcca agaagtcacc 5220
ggacttgccc tcgactgcga cggcgacacc gtccttctga ccgtgaaaca aaccggcggt 5280
gcgtgccaca ctggtgccca cacatgtttc gacaatgacg ttttgctgta agcggtaccg 5340
gcgcttcatg tcaacaatct ttaacgtttt caagttcaca agtcgtgttc aaatggtgac 5400
aagattggac actgtgctga attggcacca agccctcata aatgatagat ctaaatcgaa 5460
tatcaatata tggtctgttt attggaacgc gtcccagtgg ctgagacgca tccgctaaag 5520
ccccaggaac cctgtgcaga aagaacaaat aatcgtgaat tttggcagca acagcggggc 5580
ctggtataat tgaaaacgtg caaaagcata gattattgga ggagatcaaa acaatgttga 5640
atgtcactga cctgcgaggt caaacaccat ccaagagcga catccgacgt gctttgccac 5700
gtggtggcac tgacgtgtgg tctgtgcttc ccatagtgca gcctgttgta gaagatgtcc 5760
aaaaccgcgg cgctgaagct gctttggatt acggcgagaa gttcgaccat attcgccccg 5820
cctcggtgcg ggtgccagct gaggttattg ctgcagcaga aaacacctta gatccgttgg 5880
tgcgtgaatc gattgaagag tcgattcgtc gcgtccgcaa ggttcacgct gagcaaaagc 5940
catccgagca caccactgaa ctttcaccag gtggcaccgt cactgagcgt ttcatgccga 6000
ttgatcgcgt gggactgtac gttccaggcg gcaatgcggt gtacccatca agcgtgatta 6060
tgaatactgt cccagctcaa gaggctggtg tgaactccct tgtggttgcg tcgcctcctc 6120
aggctgagca cggtggctgg cctcacccca ccattttggc ggcgtgttcc atcttgggtg 6180
ttgatgaggt gtgggctgtc ggcggcggtc aggccgtggc gttgctggct tatggtgatg 6240
acgctgcagg tctcgagcct gtggatatga tcactggacc tggcaatatc tttgtcaccg 6300
ctgcgaagcg cctggtcagg ggagtggtag gtactgattc tgaggctggc cctacagaaa 6360
tcgctgtgct tgctgatgcc tctgccaacg ccgtcaacgt tgcctacgat ctgatcagcc 6420
aagcagaaca cgatgtcatg gctgcgtccg tgctcatcac tgactccgag cagcttgcca 6480
aggacgtaaa cagggaaatc gaggcgcgtt actcaatcac gcgcaacgcc gagcgcgtcg 6540
cagaagcttt gcgcggggcc cagagtggca tcgtgcttgt cgacgacatt tccgtgggta 6600
tccaagtagc cgatcaatac gcagcggaac acctggaaat ccacactgag aacgcgcgcg 6660
ccgtagcaga gcagatcacc aacgcgggtg cgatcttcgt gggcgatttc tcaccagtac 6720
cactgggtga ttactccgca ggatccaacc acgtgctgcc aacctctgga tccgctcgtt 6780
tctccgcagg tctatccacg cacacgttcc ttcgcccagt caacctcatt gaatacgatg 6840
aggctgctct gaaggacgtc tcgcaggttg tcatcaactt tgccaacgcc gaagatcttc 6900
cagcgcacgg cgaagcaatc cgtgcacgct ttgaaaacct ccccaccacc gacgaggcct 6960
aagaaaaatg accaaaatta ctttgagcga tttgccattg cgtgaagaac tgcgcggtga 7020
gcacgcttac ggcgcacccc agctcaacgt tgatattcgc ctcaacacca acgaaaaccc 7080
ttacccaccg tcagaggcat tggtcgctga cttggttgcc accgtggata agatcgccac 7140
cgagctgaac cgctacccag agcgcgatgc tgtggaactg cgtgatgagt tggctgcgta 7200
catcaccaag caaaccggcg tggctgtcac cagggataac ctgtgggctg ccaatggttc 7260
caatgaaatt ctgcagcagc tgctgcaggc ttttggtgga cctggacgca ccgcgttggg 7320
attccaaccc agctattcca tgcacccaat tttggctaaa ggcacccaca ctgaattcat 7380
tgcggtgtcc cgaggtgctg atttccgcat cgatatggat gtggcgctgg aagaaattcg 7440
tgcaaagcag cctgacattg tttttgtcac caccccgaac aacccgaccg gtgatgtgac 7500
ctcgctggac gatgttgagc gcatcatcaa cgttgcccca ggcatcgtga tcgtggatga 7560
agcttatgcg gaattctccc catcaccttc agcaaccact cttctggaga agtacccaac 7620
caagctggtg gtgtcccgca ccatgagtaa ggcttttgat ttcgcaggtg gacgcctcgg 7680
ctacttcgtg gccaacccag cgtttatcga cgccgtgatg ctagtccgcc ttccgtatca 7740
tctttcagcg ctgagccaag cagccgcaat cgtagcgctg cgtcactccg ctgacacgct 7800
gggaaccgtc gaaaagctct ctgtagagcg tgttcgcgtg gcagcacgct tggaggaact 7860
gggctacgct gtggttccaa gtgagtccaa ctttgtgttc tttggagatt tctccgatca 7920
gcacgcggca tggcaggcat ttttggatag gggagtgctc atccgcgatg tgggaatcgc 7980
tgggcacttg cgcactacca ttggtgtgcc tgaggaaaat gatgcgtttt tggacgcagc 8040
tgcagagatc atcaagctga acctgtaaga gagaagaatt tttcatgact gtcgcaccaa 8100
gaattggtac cgcaacccgc accaccagcg aatccgacat caccgtcgag atcaacctgg 8160
acggcaccgg caaagtagat atcgataccg gcctgccatt tttcgaccac atgctcactg 8220
cattcggcgt gcacggcagt tttgatctga aagtccatgc caagggcgac atcgagatcg 8280
acgcacacca caccgtggaa gataccgcca tcgtgctcgg ccaagcactc cttgacgcta 8340
ttggcgacaa gaaaggcatc cgccgtttcg catcctgcca gctgcccatg gatgaggcat 8400
tagtggagtc cgtggtggat atctccggtc gcccatactt cgtgatctcc ggcgaaccag 8460
accacatgat cacctccgtg atcggtggac actacgcaac cgtgatcaac gagcacttct 8520
ttgaaaccct cgcgctcaac tcccgaatca ccctccacgt gatctgccac tacggccgcg 8580
accctcacca catcaccgaa gcagagtaca aggctgttgc ccgtgcgctg cgcggtgccg 8640
tagagatgga tcctcgtcaa acaggaatcc catccactaa gggagcgctc tagagaaaca 8700
tcccagcgct actaataggg agcgttgacc ttccttccac ggaccggtaa tcggagtgcc 8760
taaaaccgca tgcggcttag gctccaagat aggttctgcg cggccgggta atgcatcttc 8820
tttagcaaca agttgagggg taggtgcaaa taagaacgac atagaaatcg tctcctttct 8880
gtttttaatc aacatacacc accacctaaa aattccccga ccagcaagtt cacagtattc 8940
gggcacaata tcgttgccaa aatattgttt cggaatatca tgggatacgt acccaacgaa 9000
aggaaacact catggaaggc atgactaatc cagagcagac acatcccgct gcaagcctcg 9060
aagacatgat caaaaccatc acaaagacct tcgtgattgc tcacgatcag gattctgatg 9120
agcatcttgc gcaggcactg gtgtacaacg ctggacgttt ggcatggcgc atgcgcgaaa 9180
acggtgtgga tacggattac aagacttctg tgtctgatgt ggtcacggat gccgatcgtg 9240
cggccgaggc cttcgtcgca ggcgttcttg aagcgttgcg gcctgaggac ggcgtgcttg 9300
gcgaggaagg cgcggaccgg gcgtcgaaaa gcggaaaaac ctgggtcatc gacccggttg 9360
atggcaccta caacttcacc cagggctcag attattggtg ctcggcgctc gcgctggtcg 9420
agggcgatcc atccgcgcca tcgcgcgtgc ttttcggcgc cgtacaccgc ccagccatgg 9480
gttatacgtg gttcggtggc ccgggaatcc gcaccacgct cgacggcaag gagctagatt 9540
tgcttgtcga cgcccccctc aatcaaatct ccctggccac ctacatccac ccgtcacgca 9600
tcgcggaacc tgatattcaa aaggcgtgga tgagcgttgc cacccaccct gcaacgctgc 9660
gcatgttcgg cgccggctcc atcgatttgg ccaacatcgc cgacggcagc atcggcgcat 9720
gggtgcagca cagcgtcgca gattgggact ggctacccgg ccgcgcactc atcgaaggcg 9780
tcggcggagc atgcatcaaa gtgaccgccg gcggcgtcga atggtccgtt gcaggaaacg 9840
cggaagcagt tagtgagatc tccgaaactt taagcgcact agactaacaa cacatgagca 9900
aatatgcaga cgatttagcc ttagccctcg aacttgccga acttgccgat tccatcaccc 9960
tcgaccgctt cgaagcctct gacctggaag tatcctccaa gccagacatg actcccgtca 10020
gcgatgccga cctggcgacc gaagaagcac tccgtgagaa aatcgccacc gcccgccccg 10080
ccgactccat cctcggtgaa gaattcggtg gcgacgtaga attcagcggc cgccagtgga 10140
tcatcgaccc catcgacggc accaaaaact acgtccgcgg cgtccccgta tgggcaaccc 10200
tgatcgcgct gctcgacaac ggcaaacccg tcgcaggtgt catctccgca cccgcactgg 10260
ctaggcgttg gtgggcatcc gaaggggccg gcgcatggcg caccttcaac ggcagctccc 10320
cacgcaaact gtccgtgtcc caggtgtcca agcttgacga cgcctccctc tccttctcct 10380
ccctctccgg ctgggccgaa cgagatttgc gcgatcagtt cgtctcccta actgatacca 10440
cctggcgact ccgcggctac ggcgacttct tctcctactg cctcgtcgcc gaaggtgccg 10500
tcgatatcgc cgctgaacca gaagtcagcc tctgggatct tgctcccctg tccatcctgg 10560
tcaccgaagc cggaggaaag ttcacctcac tggctggcgt cgatggacca cacggtggcg 10620
atgcagtagc caccaacggc atcctgcacg atgagacgct ggatcgttta aaatag 10676
<210> 90
<211> 281
<212> PRT
<213> Unknown
<220>
<223> полипептид
<400> 90
Met Leu Lys Ile Ala Val Pro Asn Lys Gly Ser Leu Ser Glu Arg Ala
1 5 10 15
Met Glu Ile Leu Ala Glu Ala Gly Tyr Ala Gly Arg Gly Asp Ser Lys
20 25 30
Ser Leu Asn Val Phe Asp Glu Ala Asn Asn Val Glu Phe Phe Phe Leu
35 40 45
Arg Pro Lys Asp Ile Ala Ile Tyr Val Ala Gly Gly Gln Leu Asp Leu
50 55 60
Gly Ile Thr Gly Arg Asp Leu Ala Arg Asp Ser Gln Ala Asp Val His
65 70 75 80
Glu Val Leu Ser Leu Gly Phe Gly Ser Ser Thr Phe Arg Tyr Ala Ala
85 90 95
Pro Ala Asp Glu Glu Trp Ser Ile Glu Lys Leu Asp Gly Lys Arg Ile
100 105 110
Ala Thr Ser Tyr Pro Asn Leu Val Arg Asp Asp Leu Ala Ala Arg Gly
115 120 125
Leu Ser Ala Glu Val Leu Arg Leu Asp Gly Ala Val Glu Val Ser Ile
130 135 140
Lys Leu Gly Val Ala Asp Ala Ile Ala Asp Val Val Ser Thr Gly Arg
145 150 155 160
Thr Leu Arg Gln Gln Gly Leu Ala Pro Phe Gly Glu Val Leu Cys Thr
165 170 175
Ser Glu Ala Val Ile Val Gly Arg Lys Asp Glu Lys Val Thr Pro Glu
180 185 190
Gln Gln Ile Leu Leu Arg Arg Ile Gln Gly Ile Leu His Ala Gln Asn
195 200 205
Phe Leu Met Leu Asp Tyr Arg Val Asp Arg Asp Asn Leu Asp Ala Ala
210 215 220
Thr Ala Val Thr Pro Gly Leu Ser His Pro Gln Val Ser Pro Leu Ala
225 230 235 240
Arg Asp Asn Trp Val Ala Val Arg Ala Met Val Pro Arg Arg Ser Ala
245 250 255
Asn Ala Ile Met Asp Lys Leu Ala Gly Leu Gly Ala Glu Ala Ile Leu
260 265 270
Ala Ser Glu Ile Arg Ile Ala Arg Ile
275 280
<210> 91
<211> 2642
<212> DNA
<213> Unknown
<220>
<223> полинуклеотид
<400> 91
ctcccgcgca ctgctgcaat ccgcaccgtg cccaatgatg gtggttcgcc cacctgagaa 60
gattaagaag tagtttcttt taagtttcga tgccccggtt tcctgatttt gtgcagggag 120
gccggggcat tggtgtttgc gggttagttc gggccattcg aaagggagaa accaagggca 180
gccagacaga cgtgccaaga atctggattt ccgccaggtt ttggcacgcc cgtctggttt 240
aggcaatgag ataccgaaca cacgtgccaa aagttcggct ttttcgccga tcttgtcacg 300
cctgcctggt ttgtcttgta aagagtgatt tcatggccga gactcctaaa agtttgacct 360
cacaggattg cttctaaggg cctctccaat ctccactgag gtacttaatc cttccgggga 420
attcgggcgc ttaaatcgag aaattaggcc atcacctttt aataacaata caatgaataa 480
ttggaatagg tcgacacctt tggagcggag ccggttaaaa ttggcagcat tcaccgaaag 540
aaaaggagaa ccacatgctt gccctaggtt ggattacatg gatcattatt ggtggtctag 600
ctggttggat tgcctccaag attaaaggca ctgatgctca gcaaggaatt ttgctgaaca 660
tagtcgtcgg tattatcggt ggtttgttag gcggctggct gcttggaatc ttcggagtgg 720
atgttgccgg tggcggcttg atcttcagct tcatcacatg tctgattggt gctgtcattt 780
tgctgacgat cgtgcagttc ttcactcgga agaagtaatc tgctttaaat ccgtagggcc 840
tgttgatatt tcgatatcaa caggcctttt ggtcattttg gggtggaaaa agcgctagac 900
ttgcctgtgg attaaaacta tacgaaccgg tttgtctata ttggtgttag acagttcgtc 960
gtatcttgaa acagaccaac ccgaaaggac gtggccgaac gtggctgcta gcgcttcagg 1020
caagagtaaa acaagtgccg gggcaaaccg tcgtcgcaat cgaccaagcc cccgacagcg 1080
tctcctcgat agcgcaacca accttttcac cacagaaggt attcgcgtca tcggtattga 1140
tcgtatcctc cgtgaagctg acgtggcgaa ggcgagcctc tattcccttt tcggatcgaa 1200
ggacgccttg gttattgcat acctggagaa cctcgatcag ctgtggcgtg aagcgtggcg 1260
tgagcgcacc gtcggtatga aggatccgga agataaaatc atcgcgttct ttgatcagtg 1320
cattgaggaa gaaccagaaa aagatttccg cggctcgcac tttcagaatg cggctagtga 1380
gtaccctcgc cccgaaactg atagcgaaaa gggcattgtt gcagcagtgt tagagcaccg 1440
cgagtggtgt cataagactc tgactgattt gctcactgag aagaacggct acccaggcac 1500
cacccaggcg aatcagctgt tggtgttcct tgatggtgga cttgctggat ctcgattggt 1560
ccacaacatc agtcctcttg agacggctcg cgatttggct cggcagttgt tgtcggctcc 1620
acctgcggac tactcaattt agtttcttca ttttccgaag gggtatcttc gttgggggag 1680
gcgtcgataa gccccttctt tttagcttta acctcagcgc gacgctgctt taagcgctgc 1740
atggcggcgc ggttcatttc acgttgcgtt tcgcgcctct tgttcgcgat ttctttgcgg 1800
gcctgttttg cttcgttgat ttcggcagta cgggttttgg tgagttccac gtttgttgcg 1860
tgaagcgttg aggcgttcca tggggtgaga atcatcaggg cgcggttttt gcgtcgtgtc 1920
cacaggaaga tgcgcttttc tttttgtttt gcgcggtaga tgtcgcgctg ctctaggtgg 1980
tgcactttga aatcgtcggt aagtgggtat ttgcgttcca aaatgaccat catgatgatt 2040
gtttggagga gcgtccacag gttgttgctg acccaataga gtgcgattgc tgtggggaat 2100
ggtcctgtga ggccaaggga cagtgggaag atcggcgcga ggatcgacat cacgatcatg 2160
aacttcagca tgccgttaga gaatccggat gcgtaatcgt tggtttggaa gctgcggtac 2220
atggacatcg ccatgttgat tgcggtgagg attgcggctg tgatgaacag tggcaaaacg 2280
aaactaagaa cttccgcctg cgtggtgctc aaatatttta gctgctcagt gggcatcgaa 2340
acataagcgg gcagaggcac attgctcacg cgaccagcga ggaaagattc cacttcctca 2400
ggagttagga agccgatcga ctggaagacg ggattttcca aaccaccttc agggcgagcc 2460
atgcggagaa gtgcccagta aagaccaagg acaatcggta tctggatcag cccaggcaca 2520
caacctgcca gcgggttaat gccgtattcc ttattcaaat cattctggcg cttctgcaac 2580
tcccgaatgg acgcttcatc gtactttccc ttgtattctt cccggagcgc agcgcggtga 2640
gg 2642
<210> 92
<211> 5666
<212> DNA
<213> Unknown
<220>
<223> полинуклеотид
<400> 92
tgtcatgctt ccggaggtgc gcagggctcg agactccgga aagctatttg ccactccgat 60
gtttgggtca ctcgacgaga tacgtgctga tcacctaatt tggtgcacag ggtttcggcc 120
ggcgattagg ccagttcgtc aacttctcaa acacggacaa ccaaaggttc ctggtcttta 180
tttagtaggc tacggagatt ggacgggacc tgggtctgcg actatcacag gggtcgggct 240
ttatgccaag cgagcagcca aagagattgc cgcgtcagtc ggcaaagtcg ttaaatagtt 300
tgaaggctaa gaacttaatg ttaaagcgaa aattgttttg acacctcaac taatgcagcg 360
atgcgttctt tccagaatgc tttcatgaca gggatgctgt cttgatcagg caggcgtctg 420
tgctggatgc cgaagctgga tttattgtcg cctttggagg tgaagttgac gctcactcga 480
gaatcatcgg ccaaccattt ggcattgaat gttctaggtt cggaggcgga ggttttctca 540
attagtgcgg gatcgagcca ctgcgcccgc aggtcatcgt ctccgaagag cttccacact 600
ttttcgaccg gcaggttaag ggttttggag gcattggccg cgaacccatc gctggtcatc 660
ccgggtttgc gcatgccacg ttcgtattca taaccaatcg cgatgccttg agcccaccag 720
ccactgacat caaagttgtc cacgatgtgc tttgcgatgt gggtgtgagt ccaagaggtg 780
gcttttacgt cgtcaagcaa ttttagccac tcttcccacg gctttccggt gccgttgagg 840
atagcttcag gggacatgcc tggtgttgag ccttgcggag tggagtcagt catgcgaccg 900
agactagtgg cgctttgcct gtgttgctta ggcggcgttg aaaatgaact acgaatgaaa 960
agttcgggaa ttgtctaatc cgtactaagc tgtctacaca atgtctactt cagttacttc 1020
accagcccac aacaacgcac attcctccga atttttggat gcgttggcaa accatgtgtt 1080
gatcggcgac ggcgccatgg gcacccagct ccaaggcttt gacctggacg tggaaaagga 1140
tttccttgat ctggaggggt gtaatgagat tctcaacgac acccgccctg atgtgttgag 1200
gcagattcac cgcgcctact ttgaggcggg agctgacttg gttgagacca atacttttgg 1260
ttgcaacctg ccgaacttgg cggattatga catcgctgat cgttgccgtg agcttgccta 1320
caagggcact gcagtggcta gggaagtggc tgatgagatg gggccgggcc gaaacggcat 1380
gcggcgtttc gtggttggtt ccctgggacc tggaacgaag cttccatcgc tgggccatgc 1440
accgtatgca gatttgcgtg ggcactacaa ggaagcagcg cttggcatca tcgacggtgg 1500
tggcgatgcc tttttgattg agactgctca ggacttgctt caggtcaagg ctgcggttca 1560
cggcgttcaa gatgccatgg ctgaacttga tacattcttg cccattattt gccacgtcac 1620
cgtagagacc accggcacca tgctcatggg ttctgagatc ggtgccgcgt tgacagcgct 1680
gcagccactg ggtatcgaca tgattggtct gaactgcgcc accggcccag atgagatgag 1740
cgagcacctg cgttacctgt ccaagcacgc cgatattcct gtgtcggtga tgcctaacgc 1800
aggtcttcct gtcctgggta aaaacggtgc agaataccca cttgaggctg aggatttggc 1860
gcaggcgctg gctggattcg tctccgaata tggcctgtcc atggtgggtg gttgttgtgg 1920
caccacacct gagcacatcc gtgcggtccg cgatgcggtg gttggtgttc cagagcagga 1980
aacctccaca ctgaccaaga tccctgcagg ccctgttgag caggcctccc gcgaggtgga 2040
gaaagaggac tccgtcgcgt cgctgtacac ctcggtgcca ttgtcccagg aaaccggcat 2100
ttccatgatc ggtgagcgca ccaactccaa cggttccaag gcattccgtg aggcaatgct 2160
gtctggcgat tgggaaaagt gtgtggatat tgccaagcag caaacccgcg atggtgcaca 2220
catgctggat ctttgtgtgg attacgtggg acgagacggc accgccgata tggcgacctt 2280
ggcagcactt cttgctacca gctccacttt gccaatcatg attgactcca ccgagccaga 2340
ggttattcgc acaggccttg agcacttggg tggacgaagc atcgttaact ccgtcaactt 2400
tgaagacggc gatggccctg agtcccgcta ccagcgcatc atgaaactgg taaagcagca 2460
cggtgcggcc gtggttgcgc tgaccattga tgaggaaggc caggcacgta ccgctgagca 2520
caaggtgcgc attgctaaac gactgattga cgatatcacc ggcagctacg gcctggatat 2580
caaagacatc gttgtggact gcctgacctt cccgatctct actggccagg aagaaaccag 2640
gcgagatggc attgaaacca tcgaagccat ccgcgagctg aagaagctct acccagaaat 2700
ccacaccacc ctgggtctgt ccaatatttc cttcggcctg aaccctgctg cacgccaggt 2760
tcttaactct gtgttcctca atgagtgcat tgaggctggt ctggactctg cgattgcgca 2820
cagctccaag attttgccga tgaaccgcat tgatgatcgc cagcgcgaag tggcgttgga 2880
tatggtctat gatcgccgca ccgaggatta cgatccgctg caggaattca tgcagctgtt 2940
tgagggcgtt tctgctgccg atgccaagga tgctcgcgct gaacagctgg ccgctatgcc 3000
tttgtttgag cgtttggcac agcgcatcat cgacggcgat aagaatggcc ttgaggatga 3060
tctggaagca ggcatgaagg agaagtctcc tattgcgatc atcaacgagg accttctcaa 3120
cggcatgaag accgtgggtg agctgtttgg ttccggacag atgcagctgc cattcgtgct 3180
gcaatcggca gaaaccatga aaactgcggt ggcctatttg gaaccgttca tggaagagga 3240
agcagaagct accggatctg cgcaggcaga gggcaagggc aaaatcgtcg tggccaccgt 3300
caagggtgac gtgcacgata tcggcaagaa cttggtggac atcattttgt ccaacaacgg 3360
ttacgacgtg gtgaacttgg gcatcaagca gccactgtcc gccatgttgg aagcagcgga 3420
agaacacaaa gcagacgtca tcggcatgtc gggacttctt gtgaagtcca ccgtggtgat 3480
gaaggaaaac cttgaggaga tgaacaacgc cggcgcatcc aattacccag tcattttggg 3540
tggcgctgcg ctgacgcgta cctacgtgga aaacgatctc aacgaggtgt acaccggtga 3600
ggtgtactac gcccgtgatg ctttcgaggg cctgcgcctg atggatgagg tgatggcaga 3660
aaagcgtggt gaaggacttg atcccaactc accagaagct attgagcagg cgaagaagaa 3720
ggcggaacgt aaggctcgta atgagcgttc ccgcaagatt gccgcggagc gtaaagctaa 3780
tgcggctccc gtgattgttc cggagcgttc tgatgtctcc accgatactc caaccgcggc 3840
accaccgttc tggggaaccc gcattgtcaa gggtctgccc ttggcggagt tcttgggcaa 3900
ccttgatgag cgcgccttgt tcatggggca gtggggtctg aaatccaccc gcggcaacga 3960
gggtccaagc tatgaggatt tggtggaaac tgaaggccga ccacgcctgc gctactggct 4020
ggatcgcctg aagtctgagg gcattttgga ccacgtggcc ttggtgtatg gctacttccc 4080
agcggtcgcg gaaggcgatg acgtggtgat cttggaatcc ccggatccac acgcagccga 4140
acgcatgcgc tttagcttcc cacgccagca gcgcggcagg ttcttgtgca tcgcggattt 4200
cattcgccca cgcgagcaag ctgtcaagga cggccaagtg gacgtcatgc cattccagct 4260
ggtcaccatg ggtaatccta ttgctgattt cgccaacgag ttgttcgcag ccaatgaata 4320
ccgcgagtac ttggaagttc acggcatcgg cgtgcagctc accgaagcat tggccgagta 4380
ctggcactcc cgagtgcgca gcgaactcaa gctgaacgac ggtggatctg tcgctgattt 4440
tgatccagaa gacaagacca agttcttcga cctggattac cgcggcgccc gcttctcctt 4500
tggttacggt tcttgccctg atctggaaga ccgcgcaaag ctggtggaat tgctcgagcc 4560
aggccgtatc ggcgtggagt tgtccgagga actccagctg cacccagagc agtccacaga 4620
cgcgtttgtg ctctaccacc cagaggcaaa gtactttaac gtctaacacc tttgagaggg 4680
aaaactttcc cgcacattgc agatcgtgcc actttaacta aggttgacgg catgattaag 4740
gcgattttct gggacatgga cggcacgatg gtggactctg agccacagtg gggcattgct 4800
acctacgagc tcagcgaagc catgggccgc cgcctcaccc cggagctccg ggaactcacc 4860
gtcggctcga gcctgccgcg caccatgcgc ttatgcgcag agcacgcagg cattacattg 4920
agcgacgcgg actacgagcg ctaccgggct ggcatgttcg cccgggtcca tgagcttttc 4980
gacgaatccc tcgtcccaaa tccaggcgtc accgaactcc tgacagagtt gaaggccctc 5040
gagatcccca tgttggtcac caccaacaca gagcgcgatc tcgcgacccg ttcagtcgca 5100
gccgtgggaa atgagttctt catcggttct atcgctggtg atgaagtccc aacagcaaag 5160
ccagcccccg acatgtacct cgaagcagca cgacgtgtgg gctttgaccc atcagagtgc 5220
ctcgtgttcg aagattccta caacggcatg ctgggcgctg ttactgcagg ttgccgcgtc 5280
attggtctgc acccagaaga agtccaagcg ccagaaggtg tagtgccttt gcgttccctc 5340
cacggtaaaa actctttcga aggtgtcacc gctgagatgg tcactgcctg gtaccaccag 5400
atcgagccgg caggtgtcgc aaaataaaac caggtggggg agtgaaatta ttcgactaat 5460
atcctccccc aaacacacat tgataactgt tgtgtggaag aatgtaccga gtgaagacat 5520
ttgactcgct gtacgaagaa cttcttaacc gtgctcagac ccgccctgaa gggtctggaa 5580
ccgtggccgc cttggataaa ggcatccatc atctaggtaa gaaggtcatc gaagaagccg 5640
gagaggtctg gattgcagcc gagtat 5666
<210> 93
<211> 11332
<212> DNA
<213> Unknown
<220>
<223> полинуклеотид
<400> 93
tgtcatgctt ccggaggtgc gcagggctcg agactccgga aagctatttg ccactccgat 60
gtttgggtca ctcgacgaga tacgtgctga tcacctaatt tggtgcacag ggtttcggcc 120
ggcgattagg ccagttcgtc aacttctcaa acacggacaa ccaaaggttc ctggtcttta 180
tttagtaggc tacggagatt ggacgggacc tgggtctgcg actatcacag gggtcgggct 240
ttatgccaag cgagcagcca aagagattgc cgcgtcagtc ggcaaagtcg ttaaatagtt 300
tgaaggctaa gaacttaatg ttaaagcgaa aattgttttg acacctcaac taatgcagcg 360
atgcgttctt tccagaatgc tttcatgaca gggatgctgt cttgatcagg caggcgtctg 420
tgctggatgc cgaagctgga tttattgtcg cctttggagg tgaagttgac gctcactcga 480
gaatcatcgg ccaaccattt ggcattgaat gttctaggtt cggaggcgga ggttttctca 540
attagtgcgg gatcgagcca ctgcgcccgc aggtcatcgt ctccgaagag cttccacact 600
ttttcgaccg gcaggttaag ggttttggag gcattggccg cgaacccatc gctggtcatc 660
ccgggtttgc gcatgccacg ttcgtattca taaccaatcg cgatgccttg agcccaccag 720
ccactgacat caaagttgtc cacgatgtgc tttgcgatgt gggtgtgagt ccaagaggtg 780
gcttttacgt cgtcaagcaa ttttagccac tcttcccacg gctttccggt gccgttgagg 840
atagcttcag gggacatgcc tggtgttgag ccttgcggag tggagtcagt catgcgaccg 900
agactagtgg cgctttgcct gtgttgctta ggcggcgttg aaaatgaact acgaatgaaa 960
agttcgggaa ttgtctaatc cgtactaagc tgtctacaca atgtctactt cagttacttc 1020
accagcccac aacaacgcac attcctccga atttttggat gcgttggcaa accatgtgtt 1080
gatcggcgac ggcgccatgg gcacccagct ccaaggcttt gacctggacg tggaaaagga 1140
tttccttgat ctggaggggt gtaatgagat tctcaacgac acccgccctg atgtgttgag 1200
gcagattcac cgcgcctact ttgaggcggg agctgacttg gttgagacca atacttttgg 1260
ttgcaacctg ccgaacttgg cggattatga catcgctgat cgttgccgtg agcttgccta 1320
caagggcact gcagtggcta gggaagtggc tgatgagatg gggccgggcc gaaacggcat 1380
gcggcgtttc gtggttggtt ccctgggacc tggaacgaag cttccatcgc tgggccatgc 1440
accgtatgca gatttgcgtg ggcactacaa ggaagcagcg cttggcatca tcgacggtgg 1500
tggcgatgcc tttttgattg agactgctca ggacttgctt caggtcaagg ctgcggttca 1560
cggcgttcaa gatgccatgg ctgaacttga tacattcttg cccattattt gccacgtcac 1620
cgtagagacc accggcacca tgctcatggg ttctgagatc ggtgccgcgt tgacagcgct 1680
gcagccactg ggtatcgaca tgattggtct gaactgcgcc accggcccag atgagatgag 1740
cgagcacctg cgttacctgt ccaagcacgc cgatattcct gtgtcggtga tgcctaacgc 1800
aggtcttcct gtcctgggta aaaacggtgc agaataccca cttgaggctg aggatttggc 1860
gcaggcgctg gctggattcg tctccgaata tggcctgtcc atggtgggtg gttgttgtgg 1920
caccacacct gagcacatcc gtgcggtccg cgatgcggtg gttggtgttc cagagcagga 1980
aacctccaca ctgaccaaga tccctgcagg ccctgttgag caggcctccc gcgaggtgga 2040
gaaagaggac tccgtcgcgt cgctgtacac ctcggtgcca ttgtcccagg aaaccggcat 2100
ttccatgatc ggtgagcgca ccaactccaa cggttccaag gcattccgtg aggcaatgct 2160
gtctggcgat tgggaaaagt gtgtggatat tgccaagcag caaacccgcg atggtgcaca 2220
catgctggat ctttgtgtgg attacgtggg acgagacggc accgccgata tggcgacctt 2280
ggcagcactt cttgctacca gctccacttt gccaatcatg attgactcca ccgagccaga 2340
ggttattcgc acaggccttg agcacttggg tggacgaagc atcgttaact ccgtcaactt 2400
tgaagacggc gatggccctg agtcccgcta ccagcgcatc atgaaactgg taaagcagca 2460
cggtgcggcc gtggttgcgc tgaccattga tgaggaaggc caggcacgta ccgctgagca 2520
caaggtgcgc attgctaaac gactgattga cgatatcacc ggcagctacg gcctggatat 2580
caaagacatc gttgtggact gcctgacctt cccgatctct actggccagg aagaaaccag 2640
gcgagatggc attgaaacca tcgaagccat ccgcgagctg aagaagctct acccagaaat 2700
ccacaccacc ctgggtctgt ccaatatttc cttcggcctg aaccctgctg cacgccaggt 2760
tcttaactct gtgttcctca atgagtgcat tgaggctggt ctggactctg cgattgcgca 2820
cagctccaag attttgccga tgaaccgcat tgatgatcgc cagcgcgaag tggcgttgga 2880
tatggtctat gatcgccgca ccgaggatta cgatccgctg caggaattca tgcagctgtt 2940
tgagggcgtt tctgctgccg atgccaagga tgctcgcgct gaacagctgg ccgctatgcc 3000
tttgtttgag cgtttggcac agcgcatcat cgacggcgat aagaatggcc ttgaggatga 3060
tctggaagca ggcatgaagg agaagtctcc tattgcgatc atcaacgagg accttctcaa 3120
cggcatgaag accgtgggtg agctgtttgg ttccggacag atgcagctgc cattcgtgct 3180
gcaatcggca gaaaccatga aaactgcggt ggcctatttg gaaccgttca tggaagagga 3240
agcagaagct accggatctg cgcaggcaga gggcaagggc aaaatcgtcg tggccaccgt 3300
caagggtgac gtgcacgata tcggcaagaa cttggtggac atcattttgt ccaacaacgg 3360
ttacgacgtg gtgaacttgg gcatcaagca gccactgtcc gccatgttgg aagcagcgga 3420
agaacacaaa gcagacgtca tcggcatgtc gggacttctt gtgaagtcca ccgtggtgat 3480
gaaggaaaac cttgaggaga tgaacaacgc cggcgcatcc aattacccag tcattttggg 3540
tggcgctgcg ctgacgcgta cctacgtgga aaacgatctc aacgaggtgt acaccggtga 3600
ggtgtactac gcccgtgatg ctttcgaggg cctgcgcctg atggatgagg tgatggcaga 3660
aaagcgtggt gaaggacttg atcccaactc accagaagct attgagcagg cgaagaagaa 3720
ggcggaacgt aaggctcgta atgagcgttc ccgcaagatt gccgcggagc gtaaagctaa 3780
tgcggctccc gtgattgttc cggagcgttc tgatgtctcc accgatactc caaccgcggc 3840
accaccgttc tggggaaccc gcattgtcaa gggtctgccc ttggcggagt tcttgggcaa 3900
ccttgatgag cgcgccttgt tcatggggca gtggggtctg aaatccaccc gcggcaacga 3960
gggtccaagc tatgaggatt tggtggaaac tgaaggccga ccacgcctgc gctactggct 4020
ggatcgcctg aagtctgagg gcattttgga ccacgtggcc ttggtgtatg gctacttccc 4080
agcggtcgcg gaaggcgatg acgtggtgat cttggaatcc ccggatccac acgcagccga 4140
acgcatgcgc tttagcttcc cacgccagca gcgcggcagg ttcttgtgca tcgcggattt 4200
cattcgccca cgcgagcaag ctgtcaagga cggccaagtg gacgtcatgc cattccagct 4260
ggtcaccatg ggtaatccta ttgctgattt cgccaacgag ttgttcgcag ccaatgaata 4320
ccgcgagtac ttggaagttc acggcatcgg cgtgcagctc accgaagcat tggccgagta 4380
ctggcactcc cgagtgcgca gcgaactcaa gctgaacgac ggtggatctg tcgctgattt 4440
tgatccagaa gacaagacca agttcttcga cctggattac cgcggcgccc gcttctcctt 4500
tggttacggt tcttgccctg atctggaaga ccgcgcaaag ctggtggaat tgctcgagcc 4560
aggccgtatc ggcgtggagt tgtccgagga actccagctg cacccagagc agtccacaga 4620
cgcgtttgtg ctctaccacc cagaggcaaa gtactttaac gtctaacacc tttgagaggg 4680
aaaactttcc cgcacattgc agatcgtgcc actttaacta aggttgacgg catgattaag 4740
gcgattttct gggacatgga cggcacgatg gtggactctg agccacagtg gggcattgct 4800
acctacgagc tcagcgaagc catgggccgc cgcctcaccc cggagctccg ggaactcacc 4860
gtcggctcga gcctgccgcg caccatgcgc ttatgcgcag agcacgcagg cattacattg 4920
agcgacgcgg actacgagcg ctaccgggct ggcatgttcg cccgggtcca tgagcttttc 4980
gacgaatccc tcgtcccaaa tccaggcgtc accgaactcc tgacagagtt gaaggccctc 5040
gagatcccca tgttggtcac caccaacaca gagcgcgatc tcgcgacccg ttcagtcgca 5100
gccgtgggaa atgagttctt catcggttct atcgctggtg atgaagtccc aacagcaaag 5160
ccagcccccg acatgtacct cgaagcagca cgacgtgtgg gctttgaccc atcagagtgc 5220
ctcgtgttcg aagattccta caacggcatg ctgggcgctg ttactgcagg ttgccgcgtc 5280
attggtctgc acccagaaga agtccaagcg ccagaaggtg tagtgccttt gcgttccctc 5340
cacggtaaaa actctttcga aggtgtcacc gctgagatgg tcactgcctg gtaccaccag 5400
atcgagccgg caggtgtcgc aaaataaaac caggtggggg agtgaaatta ttcgactaat 5460
atcctccccc aaacacacat tgataactgt tgtgtggaag aatgtaccga gtgaagacat 5520
ttgactcgct gtacgaagaa cttcttaacc gtgctcagac ccgccctgaa gggtctggaa 5580
ccgtggccgc cttggataaa ggcatccatc atctaggtaa gaaggtcatc gaagaagccg 5640
gagaggtctg gattgcagcc gagtattgtc atgcttccgg aggtgcgcag ggctcgagac 5700
tccggaaagc tatttgccac tccgatgttt gggtcactcg acgagatacg tgctgatcac 5760
ctaatttggt gcacagggtt tcggccggcg attaggccag ttcgtcaact tctcaaacac 5820
ggacaaccaa aggttcctgg tctttattta gtaggctacg gagattggac gggacctggg 5880
tctgcgacta tcacaggggt cgggctttat gccaagcgag cagccaaaga gattgccgcg 5940
tcagtcggca aagtcgttaa atagtttgaa ggctaagaac ttaatgttaa agcgaaaatt 6000
gttttgacac ctcaactaat gcagcgatgc gttctttcca gaatgctttc atgacaggga 6060
tgctgtcttg atcaggcagg cgtctgtgct ggatgccgaa gctggattta ttgtcgcctt 6120
tggaggtgaa gttgacgctc actcgagaat catcggccaa ccatttggca ttgaatgttc 6180
taggttcgga ggcggaggtt ttctcaatta gtgcgggatc gagccactgc gcccgcaggt 6240
catcgtctcc gaagagcttc cacacttttt cgaccggcag gttaagggtt ttggaggcat 6300
tggccgcgaa cccatcgctg gtcatcccgg gtttgcgcat gccacgttcg tattcataac 6360
caatcgcgat gccttgagcc caccagccac tgacatcaaa gttgtccacg atgtgctttg 6420
cgatgtgggt gtgagtccaa gaggtggctt ttacgtcgtc aagcaatttt agccactctt 6480
cccacggctt tccggtgccg ttgaggatag cttcagggga catgcctggt gttgagcctt 6540
gcggagtgga gtcagtcatg cgaccgagac tagtggcgct ttgcctgtgt tgcttaggcg 6600
gcgttgaaaa tgaactacga atgaaaagtt cgggaattgt ctaatccgta ctaagctgtc 6660
tacacaatgt ctacttcagt tacttcacca gcccacaaca acgcacattc ctccgaattt 6720
ttggatgcgt tggcaaacca tgtgttgatc ggcgacggcg ccatgggcac ccagctccaa 6780
ggctttgacc tggacgtgga aaaggatttc cttgatctgg aggggtgtaa tgagattctc 6840
aacgacaccc gccctgatgt gttgaggcag attcaccgcg cctactttga ggcgggagct 6900
gacttggttg agaccaatac ttttggttgc aacctgccga acttggcgga ttatgacatc 6960
gctgatcgtt gccgtgagct tgcctacaag ggcactgcag tggctaggga agtggctgat 7020
gagatggggc cgggccgaaa cggcatgcgg cgtttcgtgg ttggttccct gggacctgga 7080
acgaagcttc catcgctggg ccatgcaccg tatgcagatt tgcgtgggca ctacaaggaa 7140
gcagcgcttg gcatcatcga cggtggtggc gatgcctttt tgattgagac tgctcaggac 7200
ttgcttcagg tcaaggctgc ggttcacggc gttcaagatg ccatggctga acttgataca 7260
ttcttgccca ttatttgcca cgtcaccgta gagaccaccg gcaccatgct catgggttct 7320
gagatcggtg ccgcgttgac agcgctgcag ccactgggta tcgacatgat tggtctgaac 7380
tgcgccaccg gcccagatga gatgagcgag cacctgcgtt acctgtccaa gcacgccgat 7440
attcctgtgt cggtgatgcc taacgcaggt cttcctgtcc tgggtaaaaa cggtgcagaa 7500
tacccacttg aggctgagga tttggcgcag gcgctggctg gattcgtctc cgaatatggc 7560
ctgtccatgg tgggtggttg ttgtggcacc acacctgagc acatccgtgc ggtccgcgat 7620
gcggtggttg gtgttccaga gcaggaaacc tccacactga ccaagatccc tgcaggccct 7680
gttgagcagg cctcccgcga ggtggagaaa gaggactccg tcgcgtcgct gtacacctcg 7740
gtgccattgt cccaggaaac cggcatttcc atgatcggtg agcgcaccaa ctccaacggt 7800
tccaaggcat tccgtgaggc aatgctgtct ggcgattggg aaaagtgtgt ggatattgcc 7860
aagcagcaaa cccgcgatgg tgcacacatg ctggatcttt gtgtggatta cgtgggacga 7920
gacggcaccg ccgatatggc gaccttggca gcacttcttg ctaccagctc cactttgcca 7980
atcatgattg actccaccga gccagaggtt attcgcacag gccttgagca cttgggtgga 8040
cgaagcatcg ttaactccgt caactttgaa gacggcgatg gccctgagtc ccgctaccag 8100
cgcatcatga aactggtaaa gcagcacggt gcggccgtgg ttgcgctgac cattgatgag 8160
gaaggccagg cacgtaccgc tgagcacaag gtgcgcattg ctaaacgact gattgacgat 8220
atcaccggca gctacggcct ggatatcaaa gacatcgttg tggactgcct gaccttcccg 8280
atctctactg gccaggaaga aaccaggcga gatggcattg aaaccatcga agccatccgc 8340
gagctgaaga agctctaccc agaaatccac accaccctgg gtctgtccaa tatttccttc 8400
ggcctgaacc ctgctgcacg ccaggttctt aactctgtgt tcctcaatga gtgcattgag 8460
gctggtctgg actctgcgat tgcgcacagc tccaagattt tgccgatgaa ccgcattgat 8520
gatcgccagc gcgaagtggc gttggatatg gtctatgatc gccgcaccga ggattacgat 8580
ccgctgcagg aattcatgca gctgtttgag ggcgtttctg ctgccgatgc caaggatgct 8640
cgcgctgaac agctggccgc tatgcctttg tttgagcgtt tggcacagcg catcatcgac 8700
ggcgataaga atggccttga ggatgatctg gaagcaggca tgaaggagaa gtctcctatt 8760
gcgatcatca acgaggacct tctcaacggc atgaagaccg tgggtgagct gtttggttcc 8820
ggacagatgc agctgccatt cgtgctgcaa tcggcagaaa ccatgaaaac tgcggtggcc 8880
tatttggaac cgttcatgga agaggaagca gaagctaccg gatctgcgca ggcagagggc 8940
aagggcaaaa tcgtcgtggc caccgtcaag ggtgacgtgc acgatatcgg caagaacttg 9000
gtggacatca ttttgtccaa caacggttac gacgtggtga acttgggcat caagcagcca 9060
ctgtccgcca tgttggaagc agcggaagaa cacaaagcag acgtcatcgg catgtcggga 9120
cttcttgtga agtccaccgt ggtgatgaag gaaaaccttg aggagatgaa caacgccggc 9180
gcatccaatt acccagtcat tttgggtggc gctgcgctga cgcgtaccta cgtggaaaac 9240
gatctcaacg aggtgtacac cggtgaggtg tactacgccc gtgatgcttt cgagggcctg 9300
cgcctgatgg atgaggtgat ggcagaaaag cgtggtgaag gacttgatcc caactcacca 9360
gaagctattg agcaggcgaa gaagaaggcg gaacgtaagg ctcgtaatga gcgttcccgc 9420
aagattgccg cggagcgtaa agctaatgcg gctcccgtga ttgttccgga gcgttctgat 9480
gtctccaccg atactccaac cgcggcacca ccgttctggg gaacccgcat tgtcaagggt 9540
ctgcccttgg cggagttctt gggcaacctt gatgagcgcg ccttgttcat ggggcagtgg 9600
ggtctgaaat ccacccgcgg caacgagggt ccaagctatg aggatttggt ggaaactgaa 9660
ggccgaccac gcctgcgcta ctggctggat cgcctgaagt ctgagggcat tttggaccac 9720
gtggccttgg tgtatggcta cttcccagcg gtcgcggaag gcgatgacgt ggtgatcttg 9780
gaatccccgg atccacacgc agccgaacgc atgcgcttta gcttcccacg ccagcagcgc 9840
ggcaggttct tgtgcatcgc ggatttcatt cgcccacgcg agcaagctgt caaggacggc 9900
caagtggacg tcatgccatt ccagctggtc accatgggta atcctattgc tgatttcgcc 9960
aacgagttgt tcgcagccaa tgaataccgc gagtacttgg aagttcacgg catcggcgtg 10020
cagctcaccg aagcattggc cgagtactgg cactcccgag tgcgcagcga actcaagctg 10080
aacgacggtg gatctgtcgc tgattttgat ccagaagaca agaccaagtt cttcgacctg 10140
gattaccgcg gcgcccgctt ctcctttggt tacggttctt gccctgatct ggaagaccgc 10200
gcaaagctgg tggaattgct cgagccaggc cgtatcggcg tggagttgtc cgaggaactc 10260
cagctgcacc cagagcagtc cacagacgcg tttgtgctct accacccaga ggcaaagtac 10320
tttaacgtct aacacctttg agagggaaaa ctttcccgca cattgcagat cgtgccactt 10380
taactaaggt tgacggcatg attaaggcga ttttctggga catggacggc acgatggtgg 10440
actctgagcc acagtggggc attgctacct acgagctcag cgaagccatg ggccgccgcc 10500
tcaccccgga gctccgggaa ctcaccgtcg gctcgagcct gccgcgcacc atgcgcttat 10560
gcgcagagca cgcaggcatt acattgagcg acgcggacta cgagcgctac cgggctggca 10620
tgttcgcccg ggtccatgag cttttcgacg aatccctcgt cccaaatcca ggcgtcaccg 10680
aactcctgac agagttgaag gccctcgaga tccccatgtt ggtcaccacc aacacagagc 10740
gcgatctcgc gacccgttca gtcgcagccg tgggaaatga gttcttcatc ggttctatcg 10800
ctggtgatga agtcccaaca gcaaagccag cccccgacat gtacctcgaa gcagcacgac 10860
gtgtgggctt tgacccatca gagtgcctcg tgttcgaaga ttcctacaac ggcatgctgg 10920
gcgctgttac tgcaggttgc cgcgtcattg gtctgcaccc agaagaagtc caagcgccag 10980
aaggtgtagt gcctttgcgt tccctccacg gtaaaaactc tttcgaaggt gtcaccgctg 11040
agatggtcac tgcctggtac caccagatcg agccggcagg tgtcgcaaaa taaaaccagg 11100
tgggggagtg aaattattcg actaatatcc tcccccaaac acacattgat aactgttgtg 11160
tggaagaatg taccgagtga agacatttga ctcgctgtac gaagaacttc ttaaccgtgc 11220
tcagacccgc cctgaagggt ctggaaccgt ggccgccttg gataaaggca tccatcatct 11280
aggtaagaag gtcatcgaag aagccggaga ggtctggatt gcagccgagt at 11332
<210> 94
<211> 3311
<212> DNA
<213> Unknown
<220>
<223> полинуклеотид
<400> 94
ctcattccag cgtcacgacg ttccgaaggt actggttacc tggcattggg cactaccgtt 60
tctgcagcac ttggaccagc cctagcactt tttgtcctag gaacatttga ttacgacatg 120
ctgtttatcg tggtcttggc aacctcggtc atctctttga tcgccgtcgt gttcatgtac 180
tttaagacca gcgaccctga gccttctggg gaaccagcca agttcagctt caaatctatt 240
atgaacccaa agatcatccc catcggcatc tttatcttgc ttatttgctt tgcttactct 300
ggcgtcattg cctacatcaa cgcatttgct gaagaacgcg atctgattac gggtgctgga 360
ttgttcttca ttgcctacgc agtatcaatg tttgtgatgc gcagcttcct tggcaaactg 420
caggaccgtc gcggagacaa cgtcgttatt tactttggat tgttcttctt cgttatttcc 480
ttgacgattt tgtcctttgc cacttccaac tggcacgttg tgttgtccgg agtcattgca 540
ggtctgggat acggcacttt gatgccagca gtgcagtcca tcgctgttgg tgtagtagac 600
aaaaccgaat tcggtacggc cttctccact ttgttcctgt ttgtggactt aggttttggc 660
tttggaccta ttatcctggg agcagtttct gcggcaattg gtttcggacc tatgtatgca 720
gcactggcag gtgtgggtgt gattgccgga atcttctacc tgttcacaca cgctcgcacc 780
gatcgagcta agaatggctt tgttaaacac ccagagcctg tcgctttagt tagctagttc 840
tttcagcttt ccctcccgat cagcgtaaac cggcccttcc ggttttgggg tacatcacag 900
aacctgggct agcggtgtag acccgaaaat aaacgagcct tttgtcaggg ttaaggttta 960
ggtatctaag ctaaccaaac accaacaaaa ggctctaccc atgaagtcta ccggcaacat 1020
catcgctgac accatctgcc gcactgcgga actaggactc accatcaccg gcgcttccga 1080
tgcaggtgat tacaccctga tcgaagcaga cgcactcgac tacacctcca cctgcccaga 1140
atgctcccaa cctggggtgt ttcgtcatca cacccaccgg atgctcattg atttacccat 1200
cgtcgggttt cccaccaaac tgtttatccg tctacctcgc taccgctgca ccaaccccac 1260
atgtaagcaa aagtatttcc aagcagaact aagctgcgct gaccacggta aaaaggtcac 1320
ccaccgggtc acccgctgga ttttacaacg ccttgctatt gaccggatga gtgttcacgc 1380
aaccgcgaaa gcacttgggc tagggtggga tttaacctgc caactagccc tcgatatgtg 1440
ccgtgagctg gtctataacg atcctcacca tcttgatgga gtgtatgtca ttggggtgga 1500
tgagcataag tggtcacata atagggctaa gcatggtgat gggtttgtca ccgtgattgt 1560
cgatatgacc gggcatcggt atgactcacg gtgtcctgcc cggttattag atgtcgtccc 1620
aggtcgtagt gctgatgctt tacggtcctg gcttggctcc cgcggtgaac agttccgcaa 1680
tcagatacgg atcgtgtcca tggatggatt ccaaggctac gccacagcaa gtaaagaact 1740
cattccttct gctcgtcgcg tgatggatcc attccatgtt gtgcggcttg ctggtgacaa 1800
gctcaccgcc tgccggcaac gcctccagcg ggagaaatac cagcgtcgtg gtttaagcca 1860
ggatccgttg tataaaaacc ggaagacctt gttgaccacg cacaagtggt tgagtcctcg 1920
tcagcaagaa agcttggagc agttgtgggc gtatgacaaa gactacgggg cgttaaagct 1980
tgcgtggctt gcgtatcagg cgattattga ttgttatcag atgggtaata agcgtgaagc 2040
gaagaagaaa atgcggacca ttattgatca gcttcgggtg ttgaaggggc cgaataagga 2100
actcgcgcag ttgggtcgta gtttgtttaa acgacttggt gatgtgttgg cgtatttcga 2160
tgttggtgtc tccaacggtc cggtcgaagc gatcaacgga cggttggagc atttgcgtgg 2220
gattgctcta ggtttccgta atttgaacca ctacattctg cggtgcctta tccattcagg 2280
gcagttggtc cataagatca atgcactcta aaacaggaag agcccgtaaa cctctgacta 2340
gcgtcaccct ctgattaagg cgaccgcgga tttaagagca gaggctgcca cgagcgcatc 2400
ttcacggctg tgtgttgtac taaaagtaca gcgcacagcc gttcgtgctt gatcctcctc 2460
aagccccaac gccagcaaca catgggatac ctctccggaa ccacaggcag aaccagggga 2520
gcacacaatg ccttggcgtt ccaattccag aagaacagtt tcagatccta tgctgtcgaa 2580
gagaaaagat gcgtgtccat caatgcgcat cctaggatgt ccagtcaggt gtgctcccgg 2640
gatagtgaga acttcctcga tgaattcgcc aagatctgga taggattccg ccctggccaa 2700
ttccaaggca gtggcaaagg cgatagcccc cgcaacgttt tccgtgccac tacgccgccc 2760
tttttcctgg ccgccgccat ggattaccgg ctccagggga agctttgacc ataacactcc 2820
aatcccttta ggcgcaccga atttatgacc cgacaaactt aacgcgtcaa ctcccaagtc 2880
aaaggttaaa tgtgcagctt gcactgcatc ggtgtgaaaa ggcgtactgc ttaccgccgc 2940
caactcagct atcggctgaa tggttcccac ctcattgttg gcataaccaa tgctgatcaa 3000
tgtggtgtcc ggcctgactg ctttgcggag accctccggg gagatcagcc cagtgtgatc 3060
gggggatagg taggtgatct cgaaatcatg aaacctttca agataagcag cagtttctag 3120
gacactgtca tgctcgatcg gggtggtgat gaggtgccgg ccacgaggat tagctaagca 3180
cgctcctttg atagcgaggt tgttggcttc tgatccaccc gacgtaaacg tcacctgtgt 3240
ggggcgtcct ccgataatgc gggccacccg agttcgagca tcctccagcc ccgcagaggc 3300
gagtcttccc a 3311
<210> 95
<211> 1230
<212> DNA
<213> Unknown
<220>
<223> полинуклеотид
<400> 95
atgagcaaga cctctctcga caagagcaag atccgcttcc ttctgctgga aggcgtccac 60
cagaccgcac tggatacgct caaggctgcc ggctacacca acatcgagta cctgaccggc 120
tcgctgcccg aggaacagtt gaaagagaag atcgccgacg cccacttcat cggcatccgc 180
tcgcgcaccc aactgaccga ggaggtcttc gaccgcgcga agaagctggt cgcggtcggc 240
tgtttctgca tcggcaccaa ccaggtcgac ctggaggccg ctcgcgagcg cggcatcgcg 300
gtgttcaacg ccccctactc gaacacccgc tcggtggccg agctggtgct cgccgaggcg 360
atcctgctgc tgcgcggcat tcccgagaag aacgcggcca gccaccgcgg cggctggctg 420
aagagcgcca gcaactccta cgagatccgc ggcaagaagc tcggcatcat cggctatggc 480
tcgatcggta cccagctctc ggtgctggcc gaaagcctgg gcatgcaggt gctgttctac 540
gacgtggtga ccaagctgcc gctgggcaac gccgcccagg tgggcaacct ctacgacttg 600
ctcggccagg ccgacatcgt caccctgcac gtgccggaaa ccgccgcgac caagtggatg 660
atcggcgaga aggaaatccg cgccatgaag aaaggcgcca tcctgctcaa cgccgcccgc 720
ggcaccgtgg tggacatcga cgcgctggcc gccgccctcc gcgacaagca cctcaacggc 780
gcggccatcg acgtgttccc ggtggaaccg cgctccaaca acgacgagtt cgtcagcccg 840
ctgcgcgagt tcgacaacgt catcctcacc ccgcacgtcg gcggctcgac catggaggcc 900
caggccaaca tcggttcgga agtggccgag aagctggtca agtacagcga caacggcacc 960
tcggtgtcct cggtcaactt cccggaagtg gccctgccct cccacccggg caagcaccgc 1020
ctgctgcaca tccacaagaa catccccgga gtgatgagcg agatcaacaa ggtcttcgcc 1080
gagaacggca tcaacatttc cggccagttc ctgcagacca acgagacggt cggctacgtg 1140
gtgatcgatg tcgatgccga gtactcggaa atggccctgg agaaactgca gcaggtgaac 1200
ggcaccatcc gcagccgcgt gctgttctga 1230
<---
Изобретение относится к биотехнологии. Предложен микроорганизм, продуцирующий L-аминокислоту и ее предшественник, где микроорганизм модифицирован посредством включения белка, содержащего аминокислотную последовательность SEQ ID NO: 1. Также предложены способ получения L-аминокислоты или предшественника L-аминокислоты с использованием указанного микроорганизма, композиция для получения L-аминокислоты или предшественника L-аминокислоты, применение указанного микроорганизма для продукции L- аминокислоты или предшественника L-аминокислоты. Изобретение обеспечивает продукцию L-аминокислоты или предшественника L-аминокислоты с высокими выходами. 7 н. и 10 з.п. ф-лы, 29 табл., 9 пр.
1. Микроорганизм, продуцирующий L-аминокислоту и ее предшественник, где микроорганизм модифицирован посредством включения белка, содержащего аминокислотную последовательность SEQ ID NO: 1.
2. Микроорганизм по п. 1, где указанный белок имеет происхождение из Azotobacter vinelandii.
3. Микроорганизм по п. 1, который дополнительно имеет (1) ослабленную активность фосфосеринфосфатазы, по сравнению с эндогенной активностью фосфосеринфосфатазы (2) усиленную активность 3-фосфосеринаминотрансферазы, по сравнению с эндогенной активностью фосфосеринфосфатазы, или (3) как ослабленную активность фосфосеринфосфатазы, по сравнению с эндогенной активностью фосфосеринфосфатазы, так и усиленную активность 3-фосфосеринаминотрансферазы, по сравнению с эндогенной активностью фосфосеринфосфатазы.
4. Микроорганизм по п. 1, который дополнительно модифицирован путем усиления trp (триптофанового) оперона, по сравнению с эндогенным trp опероном, инактивации триптофаназы (TnaA), инактивации мембранного белка Mtr (Mtr) или любой их комбинации.
5. Микроорганизм по п. 1, который дополнительно имеет усиленный his (гистидиновый) оперон, по сравнению с эндогенным his опероном.
6. Микроорганизм по п. 1, который дополнительно модифицирован путем инактивации McbR (регулятор транскрипции; mcbR), усиления метионинсинтазы (meth), по сравнению с эндогенной активностью метионинсинтазы, усиления сульфитредуктазного [NADPH] гемопротеинового бета-компонента (cysI), по сравнению с эндогенной активностью сульфитредуктазного [NADPH] гемопротеинового бета-компонента, или любой их комбинации.
7. Микроорганизм по п. 1, который принадлежит к роду Corynebacterium или роду Escherichia.
8. Микроорганизм по п. 7, который представляет собой Corynebacterium glutamicum или Escherichia coli.
9. Микроорганизм по п. 1, где L-аминокислота и ее предшественник выбраны из группы, состоящей из L-серина, L-триптофана, L-гистидина, L-метионина, L-цистеина, О-сукцинилгомосерина, О-ацетилгомосерина, L-гомосерина, ацетилсерина, L-цистотионина, L-гомоцистеина и О-фосфосерина.
10. Способ получения L-аминокислоты, включающий культивирование микроорганизма по любому из пп. 1-9 в среде.
11. Способ по п. 10, дополнительно включающий выделение L-аминокислоты из культивированного микроорганизма или из культуральной среды.
12. Способ по п. 10, где L-аминокислота выбрана из группы, состоящей из серина, триптофана, гистидина, метионина или L-цистеина.
13. Применение микроорганизма по любому из пп. 1-9 для продукции L-аминокислоты.
14. Композиция для получения L-аминокислоты, которая содержит микроорганизм по любому из пп. 1-9, или белок, содержащий аминокислотную последовательность SEQ ID NO: 1, и буфер.
15. Способ получения предшественника L-аминокислоты, включающий культивирование микроорганизма по любому из пп. 1-9 в среде.
16. Применение микроорганизма по любому из пп. 1-9 для получения предшественника L-аминокислоты.
17. Композиция для получения предшественника L-аминокислоты, которая содержит микроорганизм по любому из пп. 1-9, или белок, содержащий аминокислотную последовательность SEQ ID NO: 1, и буфер.
| JP 11253187 A, 21.09.1999 | |||
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| СПОСОБ ПОЛУЧЕНИЯ L-АМИНОКИСЛОТ МЕТОДОМ ФЕРМЕНТАЦИИ СМЕСИ ГЛЮКОЗЫ И ПЕНТОЗ | 2003 |
|
RU2273666C2 |
| US 2014134680 A1, 15.05.2014 | |||
| KR 101381048 B1, 14.04.2014 | |||
| KR 20180089329 A, 08.08.2018. | |||
Авторы
Даты
2023-04-28—Публикация
2020-04-29—Подача