Настоящее изобретение относится к области медицины, в частности к кардиологии и молекулярной биологии, и может быть использовано для выявления мутаций кодирующей части гена десмина (DES), ассоциированных с кардиомиопатиями различной природы, для определения тактики ведения больных десминовой кардиомиопатией и оценки рисков развития заболевания при выявлении семейных десминовых кардиомиопатий.
Мутации кодирующей части гена DES являются причиной приблизительно 2% всех кардиомиопатий, до 70% десминовых кардиомиопатий имеют семейный характер. Ранняя диагностика таких состояний позволит подбирать своевременное и адекватное лечение.
Кардиомиопатии - хронические заболевания миокарда, сопровождающиеся нарушением функции сердца. Одним из генов, описанных в связи с развитием дилатационной кардиомиоптии, является ген десмина (DES). Десмин, белок цитоскелета, относится к группе промежуточных филаментов. Он экспрессируется во всех трех типах мышечной ткани - гладкой, сердечной и поперечно-полосатой скелетной, являясь основным представителем группы промежуточных филаментов в зрелых мышечных клетках. Фенотипическим проявлением мутаций гена DES может являться кардиомиопатия или дистальная миопатия, однако в ряде случаев встречается сочетанный фенотип [Goldfarb LG, Vicart Ρ, Goebel EH and Dalakas MC (2004) Desmin myopathy. Brain, Vol.127, Pages 723-734].
Мутации гена DES могут являться причиной развития дилатационной кардиомиопатии и обусловливают развитие заболевания приблизительно в 2% случаев [Taylor MR, Slavov D, Ku L, Di Lenarda A, Sinagra G, Carniel E, Haubold K, Boucek MM, Ferguson D, Graw SL, Zhu X, Cavanaugh J, Sucharov CC, Long CS, Bristow MR, Lavori P, Mestroni L (2007) Prevalence of desmin mutations in dilated cardiomyopathy. Circulation, Vol.115, No 10, Pages 1244- 1251; Kostareva A, Gudkova A, Sjoberg G, Kiselev I, Moiseeva O, Karelkina E, Goldfarb L, Schlyakhto E, Sejersen T. (2006) Desmin mutations in a St. Petersburg cohort of cardiomyopathies. Acta Myologica, Vol.25, No 3, Pages 109-115].
Выявление генетической детерминированности, своевременная диагностика и лечение асимптоматических пациентов с кардиомиопатиями - тяжелыми хроническими заболеваниями сердца, имеющими плохой прогноз и высокую летальность, - являются важными для улучшения прогноза заболевания и выживаемости больных.
Поскольку одним из проявлений кардиомиопатий, вызванных мутациями кодирующей части гена DES, является нарушение работы проводящей системы сердца и возникновение атриовентрикулярных блокад, своевременное выявление мутаций гена DES с помощью генетических методов исследования позволит своевременно определить тактику ведения больного и определить показания к постановке электрокардиостимулятора.
Десмин - цитоскелетный белок с молекулярной массой 53 кДа, относящийся к классу промежуточных филаментов и являющийся специфичным для мышечной ткани. Ген десмина (DES) располагается на длинном плече 2-й хромосомы (2q35). В 1996 году последовательность гена была расшифрована, а позже была доказана связь мутаций десмина с дистальной миопатией и кардиомиопатией. Ген состоит из 9 экзонов, которые расположены на участке 8363 пар оснований, и является высококонсервативным среди позвоночных. Десмин экспрессируется в поперечнополосатых и гладких мышцах, а также в миокарде.
К настоящему времени описано более 40 мутаций кодирующей части гена DES, большинство из которых локализованы в С-терминальной части белка, что соответствует 5-му и 6-му экзонам.
Выявление мутаций кодирующей части гена DES рекомендовано для использования в практическом здравоохранении у больных различными видами кардиомиопатий для определения генетической причины заболевания. Однако в настоящее время в рутинной практике медицинских лабораторий не проводится скрининг мутаций кодирующей части гена DES, поскольку все известные способы определения последовательности кодирующей части гена DES требуют больших затрат времени, включают большое количество манипуляций и приводят к большому количеству ошибок из-за сложности постановки реакций.
Из уровня техники известен способ определения последовательности кодирующей части гена DES, описанный в: Li D, Tapscoft Τ, Gonzalez О, Burch PE, Quiñones MA, Zoghbi WA, Hill R, Bachinski LL, Mann DL, Roberts R (1999) Desmin Mutation Responsible for Idiopathic Dilated Cardiomyopathy. Circulation; Vol.100, No 5, Pages 461-464. При осуществлении этого способа геномную ДНК выделяют из образцов крови пациентов, применяя стандартную методику выделения ДНК. На выделенной ДНК проводят амплификацию методом полимеразной цепной реакции (ПЦР) с использованием 7 пар синтетических нуклеотидов, называемых также праймерами, сконструированных для 9 экзонов гена десмина. Последовательности праймеров приведены ниже:
exon 1F, CTGATGTCAGGAGGGATACA;
exon 1R, AGAAGGCAGGTGGTGACAG;
exon 2-3F, CTTTATCACCCGCAACTGTC;
exon 2-3R, TATTCCCAGCCAGAGCCTC;
exon 4-5F, AGGCTCTGGCTGGGAATAG;
exon 4-5R, ATGGCCAAGGTCACAAAGTG;
exon 6F, TTTGGGCTGCTAGTGTCCTC;
exon 6R, ATCAGTAATCTCGAGCCTCC;
exon 7F, ATGGTCTCGATCTCCTGACC;
exon 7R, CCCTTTTCTTCCCTAGCTC;
exon 8F, GAAGTAACAAGCCTGTCTTG;
exon 8R, GATCTCTCTTGCCCACTAGC;
exon 9F, CGTCATCCTGCTAGCACATG;
exon 9R, CTGGCAGTCAAGACAACAGG.
ПНР проводят с использованием 200 нг геномной ДНК, в присутствии 20 ммоль/л Tris-HCl (рН 8,4), 50 ммоль/л KCl, 1,5 ммоль/л MgCl2, 50 мкмоль/л каждого деоксинуклеотидтрифосфата, 0,2 мкмоль/л прямого праймера, 0,2 мкмоль/л обратного праймера, 2,5 ед. Taq-полимеразы в конечном объеме 50 мкл. Для каждой пары праймеров подбирают свои условия ПЦР - время и температуру отжига. В результате ПЦР получают фрагменты ДНК размером 709, 385, 621, 361, 299, 329, и 859 пар оснований соответственно. Продукты ПЦР очищают с помощью стандартной методики. Очищенные продукты ПЦР секвенируют на генетическом анализаторе ABI 310 (Applied Biosystems) с помощью набора реагентов «Big Dye Terminator sequencing kit» компании Perkin-Elmer и указанных выше праймеров.
Однако этот способ очень трудоемкий и занимает много времени: из-за разной температуры и времени отжига, ПЦР для каждой пары праймеров нужно проводить отдельно. На стадии внесения реагентов в микропланшет для проведения секвенирования много времени уходит на проверку соответствия внесенных праймеров и продуктов амплификации. На этом этапе допускается основная масса ошибок, приводящих к необходимости проведения повторного определения последовательности кодирующей части гена DES. Эти особенности делают невозможным широкое применение упомянутого способа в рутинной лабораторной практике для скрининга мутаций кодирующей части гена DES.
Из уровня техники не выявлен прототип данного изобретения.
Техническим результатом, на достижение которого направлено изобретение, является сокращение времени, затрачиваемого в процессе амплификации, уменьшение количества манипуляций на стадии секвенирования, а также повышение точности и достоверности диагностики путем снижения вероятности ошибок при определении последовательности экзонов гена DES.
Уменьшение затраченного времени достигается в результате проведения амплификации всех экзонов гена DES одновременно при одной и той же температуре и времени отжига праймеров за счет использования предлагаемого набора праймеров с последовательностями Seq ID NO:1-14.
Дополнительно, уменьшение количества манипуляций и вследствие этого дополнительное уменьшение затраченного времени и снижение вероятности ошибок достигается в результате последующего секвенирования полученных продуктов амплификации с использования одной пары универсальных праймеров за счет присоединения к последовательностям Seq ID NO:1-14 с 5'-конца последовательности одного из упомянутой пары универсальных праймеров.
В частности, в качестве таких универсальных праймеров могут быть использованы прямой праймер pUC/M13(-21) Seq ID NO:15 и обратный праймер М13(-29) Seq ID NO:16.
В настоящем изобретении предлагается набор праймеров для определения последовательности экзонов гена DES, с использованием которого амплификацию всех экзонов можно проводить одновременно при одной температуре и времени отжига праймеров, а последующее секвенирование продуктов амплификации можно проводить с использованием одной пары универсальных праймеров.
Создание специфических праймеров для амплификации
Выбор специфических праймеров для амплификации интересующего фрагмента ДНК осуществляют способом, хорошо известным специалистам в данной области. Работа над созданием праймеров строится обычно следующим образом:
1) С помощью открытых и коммерческих баз данных нуклеотидных последовательностей в целевом геноме выбирают уникальный участок, содержащий область интереса исследователя.
2) На основании выбранного участка генома с помощью специального программного обеспечения подбирают последовательность олигонуклеотидов, используемых для проведения ПНР, как правило, 2 праймера. Многие из таких программ находятся в свободном доступе, например, Primer3 (http://frodo.wi.mit.edu/), Primer designing tool (NCBI), PerlPrimer (http://perlprimer.sourceforge.net/).
3) Пары праймеров дополнительно проверяют на полиморфизмы целевой последовательности в области, комплементарной 3'-концевому участку праймера.
4) К последовательностям прямых праймеров с 5'-конца присоединяют последовательность одного из универсальных праймеров, а к последовательностям обратных праймеров с 5'-конца присоединяют последовательность другого универсального праймера, отличную от той, которая присоединена к прямым праймерам.
4) Праймеры синтезируют на заказ в специализированном сервисном центре, например НПК Синтол.
5) С помощью практических экспериментов доказывают пригодность подобранных последовательностей праймеров для конкретных целей.
Выбор праймеров, являющихся предметом настоящего изобретения, характеризуется тем, что праймеры подбирали таким образом, чтобы оптимальные температура и время отжига всех праймеров для экзонов гена DES была одинаковой.
Предлагаемый набор праймеров для амплификации экзонов гена DES характеризуется тем, что содержит
1) праймеры следующего нуклеотидного состава:
2) дополнительно, к последовательностям праймеров Seq ID NO:1, Seq ID NO:3, Seq ID NO:5, Seq ID NO:7, Seq ID NO:9, Seq ID NO:11, Seq ID NO:13, называемых «прямыми праймерами», с 5'-конца присоединена последовательность одного универсального праймера, например прямого праймера pUC/M13(-21) Seq ID NO: 15, а к последовательностям праймеров Seq ID NO:2, Seq ID NO:4, Seq ID NO:6, Seq ID NO:8, Seq ID NO:10, Seq ID NO:12, Seq ID NO:14, называемых «обратными праймерами», с 5'-конца присоединена последовательность другого универсального праймера, например обратного праймера М13(-29) Seq ID NO: 16.
Помимо предлагаемых праймеров, в реакционную смесь для амплификации входят следующие компоненты:
- смесь дезоксирибонуклеотидтрифосфатов четырех типов (dATP, dTTP, dGTP, dCTP);
- фермент Taq-полимераза;
- реакционный буфер: 100 ммоль Tris-HCl, рН 8,8 при 25°С, 500 ммоль KCl, 20 ммоль MgCl2.
Амплификация экзонов гена DES
Кодирующая часть гена DES длиной 2268 пар нуклеотидов состоит из 9 фрагментов, или экзонов. В случае небольших экзонов предлагаемый набор праймеров позволяет амплифицировать 2 целых экзона с находящимся между фрагментами некодирующим участком гена, или интроном. В случае больших экзонов, например экзона 1, амплификацию проводят в виде двух перекрывающихся фрагментов. Амплификацию проводят из расчета 7 реакций на один образец геномной ДНК по следующей схеме:
реакционной
смеси
Реакцию амплификации проводят одновременно для всех реакционных смесей с использованием специализированного оборудования - амплификатора Veriti® Thermal Cycler, Applied Biosystems, США согласно следующему температурному режиму:
Секренирование экзонов гена DES
После окончания реакции амплификации полученные на первом этапе анализа продукты амплификации используют для постановки реакции секвенирования. В реакции секвенирования продуктов амплификации, полученных в результате первого этапа анализа, используют универсальные праймеры, например прямой праймер pUC/M13(-21) Seq ID NO: 15 и обратный праймер М13(-29) Seq ID NO:16. При этом праймеры смешивают с реакционным буфером перед работой, и таким образом исключают стадию внесения разных праймеров в лунки микропланшета, в которых будет проходить реакция секвенирования, это существенно снижает риск допустить ошибку во время внесения компонентов и уменьшает время внесения реактивов более чем в два раза.
Реакцию секвенирования проводят с применением набора меченых дидезоксирибонуклеотидтрифосфатов (BigDye Terminator Cycle Sequencing Kit, версия 3.0, Applied Biosystems, США) и специализированного оборудования - амплификатора Veriti® Thermal Cycler, Applied Biosystems, США, согласно следующему температурному режиму:
Электрофоретический анализ продуктов секвенирования производят на секвенаторе ABI PRISM 377, Applied Biosystems, США. Обработку полученных данных производят с помощью программы BioEdit или другого аналогичного программного обеспечения.
Изобретение иллюстрируется следующим графическим материалом.
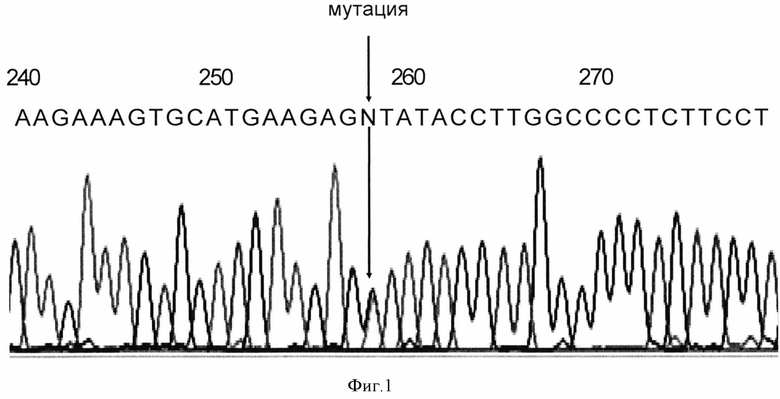
На фиг.1 представлено графическое изображение результатов секвенирования, полученных при использовании заявляемых праймеров для амплификации и определения нуклеотидной последовательности 2 экзона гена DES. Обнаружена IVS3+1G→А мутация гена DES.
Заявляемый набор праймеров применяют следующим образом:
1) Получение биологического материала от пациента. В качестве образцов для проведения анализа могут быть использованы биоптаты тканей, кровь, соскобы буккального эпителия.
2) Выделение и очистка препаратов ДНК из исходных образцов. Способы выделения ДНК из биологического материала хорошо известны специалистам и, как правило, включают стадии лизиса клеток, выделения ДНК и ее очистки. Быстрое и качественное выделение ДНК можно проводить с использованием коммерчески доступных наборов реагентов, например «Flexi Gene DNA kit» (не являются предметом данного патента).
3) Амплификация полученных препаратов геномной ДНК с применением предлагаемого набора праймеров, специфичных к экзонам гена DES, 7 реакций при одинаковых условиях, 1 загрузка амплификатора. Электрофоретическое разделение и последующая очистка продуктов амплификации.
4) Секвенирование продуктов амплификации, полученных в п.3 с применением пары коммерчески доступных универсальных праймеров, 7 реакций. Очистка продуктов реакции секвенирования и последующий капиллярный электрофорез.
5) Анализ результатов реакции секвенирования с использованием специализированного программного обеспечения, например BioEdit.






| название | год | авторы | номер документа |
|---|---|---|---|
| НАБОР СИНТЕТИЧЕСКИХ ОЛИГОНУКЛЕОТИДОВ ДЛЯ ОПРЕДЕЛЕНИЯ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ КОДИРУЮЩЕЙ ЧАСТИ ГЕНОВ NKX2.5, CFC1, GATA4 И ВЫЯВЛЕНИЯ МУТАЦИЙ, АССОЦИИРОВАННЫХ С ОРФАННОЙ МОНОГЕННОЙ ПАТОЛОГИЕЙ, ЛЕЖАЩЕЙ В ОСНОВЕ СЕМЕЙНЫХ ФОРМ ВРОЖДЕННЫХ ПОРОКОВ СЕРДЦА | 2013 |
|
RU2554056C1 |
| Способ генотипирования полиморфного локуса rs139794067 (G>T) гена MYL3 | 2025 |
|
RU2837876C1 |
| Способ поиска мутаций гена ATP7B для постановки диагноза болезни Вильсона (Вильсона-Коновалова) | 2023 |
|
RU2821573C1 |
| Способ анализа крови ВИЧ-инфицированных пациентов | 2024 |
|
RU2830447C1 |
| СПОСОБ АНАЛИЗА СОМАТИЧЕСКИХ МУТАЦИЙ В ГЕНЕ PI3K С ИСПОЛЬЗОВАНИЕМ LNA-БЛОКИРУЮЩЕЙ МУЛЬТИПЛЕКСНОЙ ПЦР И ПОСЛЕДУЮЩЕЙ ГИБРИДИЗАЦИЕЙ С ОЛИГОНУКЛЕОТИДНЫМ БИОЛОГИЧЕСКИМ МИКРОЧИПОМ (БИОЧИПОМ) | 2013 |
|
RU2549682C1 |
| СПОСОБ ВЫЯВЛЕНИЯ МУТАЦИЙ В СЛОЖНЫХ СМЕСЯХ ДНК | 2014 |
|
RU2613489C2 |
| Способ анализа терминальных мутаций в генах BRCA1, BRCA2, ATM и PALB2 с использованием мультиплексной ПЦР и последующей гибридизацией с олигонуклеотидным биологическим микрочипом (биочипом) | 2020 |
|
RU2729360C1 |
| Олигонуклеотиды для определения мутации S:L452R SARS-CoV-2 | 2022 |
|
RU2795018C1 |
| Способ генотипирования полиморфного локуса rs36210422 (G>A) гена KCNH2 | 2025 |
|
RU2837877C1 |
| НОВЫЕ ПРАЙМЕРЫ ДЛЯ СКРИНИРОВАНИЯ ШИЗОФРЕНИИ И СПОСОБ ЕЕ СКРИНИРОВАНИЯ | 2002 |
|
RU2338788C2 |
Изобретение относится к области молекулярной биологии и может быть использовано в диагностике кардиомиопатий различной природы. Предложен набор синтетических олигонуклеотидов для выявления мутаций кодирующей части гена десмина (DES), ассоциированных с кардиомиопатиями. Мутации выявляют путем определения полной нуклеотидной последовательности кодирующей части гена DES. Осуществляют амплификацию кодирующей части гена DES с помощью 7 пар синтетических олигонуклеотидов при одной температуре и времени отжига, с последующим секвенированием полученных продуктов амплификации с помощью одной пары универсальных праймеров. Предложенное изобретение позволяет чувствительно и специфично определять мутации гена DES, при этом сокращаются время реакции амплификации, количество манипуляций, время внесения реактивов для реакции секвенирования и снижается вероятность ошибки при постановке реакции. 2 з.п. ф-лы, 1 ил.
1. Набор синтетических олигонуклеотидов для определения нуклеотидной последовательности кодирующей части гена DES и выявления мутаций, ассоциированных с кардиомиопатиями, включающий праймеры следующей нуклеотидной последовательности:
необязательно соединенные с универсальными праймерами.
2. Набор синтетических нуклеотидов по п.1, в котором к последовательностям Seq ID NO:1, Seq ID NO:3, Seq ID NO:5, Seq ID NO:7, Seq ID NO:9, Seq ID N0:11, Seq ID NO:13 с 5'-конца присоединена последовательность первого универсального праймера, а к последовательностям Seq ID NO:2, Seq ID NO:4, Seq ID NO:6, Seq ID NO:8, Seq ID NO:10, Seq ID NO:12, Seq ID NO:14 с 5'-конца присоединена последовательность второго универсального праймера, при этом в качестве первого и второго универсального праймера используют разные универсальные праймеры.
3. Набор синтетических нуклеотидов по п.2, в котором в качестве одного из первого или второго универсальных праймеров используют прямой праймер pUC/M13(-21) Seq ID NO:15, а в качестве другого универсального праймера используют обратный праймер М13(-29) Seq ID NO:16.
| НАБОР СИНТЕТИЧЕСКИХ ОЛИГОНУКЛЕОТИДОВ ДЛЯ ОПРЕДЕЛЕНИЯ НУКЛЕОТИДНОЙ ПОСЛЕДОВАТЕЛЬНОСТИ КОДИРУЮЩЕЙ ЧАСТИ ГЕНА BRCA1 И ВЫЯВЛЕНИЯ МУТАЦИЙ, АССОЦИИРОВАННЫХ С НАСЛЕДСТВЕННЫМИ ФОРМАМИ РАКА МОЛОЧНОЙ ЖЕЛЕЗЫ | 2010 |
|
RU2440415C1 |
| LI D | |||
| et al., Desmin Mutation Responsible for Idiopathic Dilated Cardiomyopathy, Circulation, 1999, v.100, p | |||
| Рельсовое стыковое скрепление | 1922 |
|
SU461A1 |
| GOUDEAU B | |||
| et al., Structural and Functional Analysis of a New Desmin Variant Causing Desmin-Related Myopathy, Hyman Mutation, 2001, v | |||
| Способ использования делительного аппарата ровничных (чесальных) машин, предназначенных для мериносовой шерсти, с целью переработки на них грубых шерстей | 1921 |
|
SU18A1 |
| Уровень с пузырьком | 1922 |
|
SU388A1 |
| TESSON F | |||
| et al., Epidemiology of Desmin and Cardiac | |||
Авторы
Даты
2015-07-20—Публикация
2013-06-06—Подача