Перекрестная ссылка на родственные заявки
[0001] Данная заявка испрашивает приоритет на основании предварительной заявки на патент США № 62/535,107, поданной 20 июля 2017 г.; 62/575,820, поданной 23 октября 2017 г.; и 62/648,072, поданной 26 марта 2018 г., содержание каждой из которых включено в настоящий документ посредством ссылки в полном объеме.
Описание текстового файла, поданного в электронном виде
[0002] Содержание текстового файла, поданного в электронном виде вместе с настоящей заявкой, включено в настоящий документ посредством ссылки в полном объеме. Копия перечня последовательностей в машиночитаемом формате (имя файла: APVO_057_03WO_SeqList_ST25.txt, дата записи: 20 июля 2018 г., размер файла: 260 килобайт).
Область техники, к которой относится изобретение
[0003] Настоящее изобретение относится к молекулам, специфично связывающимся с трофобластическим гликопротеином (5T4). Данные молекулы могут иметь по меньшей мере один гуманизированный 5T4-связывающий домен. Эти молекулы пригодны для характеристики или лечения расстройств, характеризующихся экспрессией 5T4, таких как рак. Белковое терапевтическое средство, связывающееся с 5Т4, может представлять собой моноспецифичное белковое терапевтическое средство или полиспецифичное белковое терапевтическое средство. Данное изобретение также относится к белковым терапевтическим средствам, специфично связывающимся с членом 9 суперсемейства рецепторов фактора некроза опухоли (4-1BB или CD137). Полиспецифичное белковое терапевтическое средство может связывать как 5T4-экспрессирующие клетки, так и 4-1BB, экспрессируемый на эффекторных клетках, для усиления активации, пролиферации эффекторных клеток, и/или опосредованной эффекторными клетками цитотоксичности.
Уровень техники
[0004] 5T4 (также называемый трофобластическим гликопротеином, TPBG, M6P1 и Waif1) представляет собой хорошо охарактеризованный опухолеассоциированный антиген (TAA), впервые идентифицированный профессором Peter Stern из Манчестерского университета (Hole and Stern, 1988). Он представляет собой онкофетальный антиген, экспрессируемый у значительной части пациентов при различных злокачественных новообразованиях, включая немелкоклеточный рак легкого, рак почки, поджелудочной железы, предстательной железы, молочной железы, колоректальный рак, рак желудка, яичника и шейки матки, а также при остром лимфоцитарном лейкозе, и также было показано, что он экспрессируется в опухоль-инициирующих клетках (Castro et al., 2012; Damelin et al., 2011; Elkord et al., 2009; Southall et al., 1990).
[0005] Хотя низкие уровни экспрессии 5T4 были выявлены в некоторых здоровых тканях, таких как плацента и специализированный эпителий, уровни экспрессии в опухолях значительно выше.
[0006] Данные дают основания полагать, что 5T4 регулирует функциональную активность CXCR4 (Castro et al., 2012; Southgate et al., 2010). Связывающие 5T4 антитела или нокдаун 5T4 приводили к ингибированию опосредованной CXCR4 клеточной миграции - пути, задействованного в росте и метастазировании опухолей. Следовательно, нацеливание на 5T4 может обеспечить терапевтические преимущества при лечении различных видов рака. В настоящее время не существует одобренных FDA (Управление по санитарному надзору за качеством пищевых продуктов и медикаментов США) терапевтических средств, специфично нацеленных на 5T4 или связывающих его. Существует потребность в новых терапевтических средствах для лечения злокачественных новообразований, характеризующихся экспрессией 5T4.
[0007] 4-1BB (также известный как CD137 или TNFRSF9) является членом суперсемейства рецепторов фактора некроза опухолей (TNF) (TNFR). 4-1BB экспрессируется на клетках различных клеточных популяций, включая активированные CD4+ и CD8+ T-клетки, регуляторные T-клетки (Treg), дендритные клетки (ДК), моноциты, тучные клетки, эозинофилы и опухолевые эндотелиальные клетки. Активация 4-1BB зависит от олигомеризации рецептора (Rabu et al., 2005; Wyzgol et al., 2009), вызванной связыванием с 4-1BBL (также известным как CD137L), который экспрессируется в виде тримера на клеточной поверхности антигенпрезентирующих клеток (АПК) и других типов клеток. Активация 4-1BB на CD8+ T-клетках способствует эффекторным функциям CD8+ T-клеток и усиливает их, и избирательно способствует выработке цитокинов Th1 (Shuford et al., 1997; Lee et al., 2002; Pulle et al., 2006). Активация 4-1BB на CD4+ T-клетках, стимуляция 4-1BB вначале приводит к активации, а затем - к индуцированной активацией гибели клеток, что, как полагают, объясняет, почему антитела-агонисты 4-1BB показали терапевтический эффект в отношении опухолевого иммунитета, а также аутоиммунитета (Zhang, JCI, 2007; Sun, Trends Mol Med, 2003). Также сообщалось, что активация 4-1BB подавляет функцию Treg или превращает Treg в цитотоксические CD4+ T-клетки (Akhmetzyanova et al., 2016; So et al., 2008).
[0008] Помимо экспрессии на Т-клетках и модуляции их эффекторной функции, экспрессия 4-1BB повышена на CD16- и цитокин-активированных естественных киллерах (NK). Было показано, что активация 4-1BB повышает активность антителозависимой клеточной цитотоксичности (ADCC) NK-клеток как в мышиных, так и в клетках человека (Kohrt 2012 and 2014 J Clin Invest, reviewed by Hout 2012, Oncoimm). Кроме того, активация 4-1BB, экспрессируемого на АПК, таких как ДК и макрофаги, также может индуцировать и/или модулировать иммунную активацию.
[0009] Роль 4-1BB в модуляции активации иммунных клеток дает основания полагать, что он может быть подходящей мишенью для иммунотерапии при лечении множества типов рака. И действительно, два антитела к 4-1BB проходят клинические испытания: урелумаб (BMS-66513), разработанное Bristol-Myers Squibb, и PF-05082566, разработанное Pfizer. Продолжаются исследования фазы I и II по различным показаниям для каждого из антител. Однако при активации 4-1BB у пациентов и на мышиных моделях наблюдали токсичность для печени и кожи (Ascierto et al., 2010; Dubrot et al., 2010; Niu et al., 2007). Кроме того, исследование фазы II урелумаба в качестве терапии второй линии при метастатической меланоме было прекращено в 2009 году из-за токсичности для печени (Garber et al., 2011; Li and Liu, 2013).
[0010] Следовательно, в данной области техники остается потребность в терапевтических средствах, безопасно и эффективно активирующих 4-1BB для применения при лечении онкологических заболеваний.
Краткое описание изобретения
[0011] В некоторых аспектах настоящее изобретение относится к полиспецифичным полипептидам, специфично связывающимся с 5T4 и/или 4-1BB. В некоторых вариантах реализации 5T4-связывающий домен согласно изобретению связывается с внеклеточным доменом человеческого 5T4. В отдельных вариантах реализации 4-1BB-связывающий домен согласно изобретению связывается с внеклеточным доменом человеческого 4-1BB. В некоторых вариантах реализации изобретение относится к полиспецифичному полипептиду, содержащему 5T4-связывающий домен, специфично связывающийся с 5T4 человека, и 4-1BB-связывающий домен, где указанный 5T4-связывающий домен содержит: (i) вариабельную область тяжелой цепи иммуноглобулина, содержащую HCDR1, HCDR2 и HCDR3, и (ii) вариабельную область легкой цепи иммуноглобулина, содержащую LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность SEQ ID NO: 30; (b) HCDR2 содержит аминокислотную последовательность SEQ ID NO: 32; (c) HCDR3 содержит аминокислотную последовательность SEQ ID NO: 34; (d) LCDR1 содержит аминокислотную последовательность SEQ ID NO: 42; (e) LCDR2 содержит аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3 содержит аминокислотную последовательность SEQ ID NO: 36. В некоторых вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID NO: 38 и 46. В отдельных вариантах реализации 5T4-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 38 и 46. В других вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID NO: 44, 48 и 50. В некоторых вариантах реализации 5T4-связывающий домен содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 44, 48 и 50. В других вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 44; или ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 48; или iii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 5. В некоторых вариантах реализации 5T4-связывающий домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 44; или ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 48; или iii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 50.
[0012] В отдельных аспектах изобретение включает полиспецифичный полипептид, содержащий 5T4-связывающий домен, специфично связывающийся с 5T4 человека, и 4-1BB-связывающий домен, специфично связывающийся с 4-1BB человека, где каждый из указанных 4-1BB-связывающего домена и 5T4-связывающего домена содержит (i) вариабельную область тяжелой цепи (VH) иммуноглобулина, содержащую HCDR1, HCDR2 и HCDR3; и (ii) вариабельную область легкой цепи (VL) иммуноглобулина, содержащую LCDR1, LCDR2 и LCDR3, где область VH и/или VL 5T4-связывающего домена содержит одну или более мутаций в каркасной области; и где 5T4-связывающий домен содержит (а) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 30; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 32; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 34; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 8; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 36. В некоторых вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 38. В одном из вариантов реализации 5T4-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38. В некоторых вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 40. В отдельных вариантах реализации 5T4-связывающий домен содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 40. В некоторых вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 40. В некоторых вариантах реализации 5T4-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 40.
[0013] Изобретение также относится к полиспецифичному полипептиду, содержащему 5T4-связывающий домен, специфично связывающийся с 5T4 человека, и 4-1BB-связывающий домен, специфично связывающийся с 4-1BB человека, где каждый из указанных 4-1BB-связывающего домена и 5T4-связывающего домена содержит (i) вариабельную область тяжелой цепи (VH) иммуноглобулина, содержащую HCDR1, HCDR2 и HCDR3; и (ii) вариабельную область легкой цепи (VL) иммуноглобулина, содержащую LCDR1, LCDR2 и LCDR3, где область VH и/или VL 4-1BB-связывающего домена содержит одну или более мутаций в каркасной области; и где 5T4-связывающий домен содержит (а) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 30; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 32; c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 34; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 42; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 36. В некоторых вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID NO: 38 и 46. В отдельных вариантах реализации 5T4-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 38 и 46. В некоторых вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID NO: 44, 48 и 50. В некоторых аспектах 5T4-связывающий домен содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 44, 48 и 50. В отдельных вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 44; или ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 48; или iii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 50. В некоторых вариантах реализации 5T4-связывающий домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 44; или ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 48; или iii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 50.
[0014] В некоторых аспектах изобретение относится к полиспецифичному полипептиду, содержащему 5T4-связывающий домен, специфично связывающийся с 5T4 человека, и 4-1BB-связывающий домен, специфично связывающийся с 4-1BB человека, где каждый из указанных 4-1BB-связывающего домена и 5T4-связывающего домена содержит (i) вариабельную область тяжелой цепи (VH) иммуноглобулина, содержащую HCDR1, HCDR2 и HCDR3; и (ii) вариабельную область легкой цепи (VL) иммуноглобулина, содержащую LCDR1, LCDR2 и LCDR3, где область VH и/или VL 4-1BB-связывающего домена содержит одну или более мутаций в каркасной области; и где 5T4-связывающий домен содержит (а) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 52; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 32; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 34; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 54; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 36. В отдельных вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 56. В некоторых аспектах 5T4-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 56. В отдельных вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 58. В некоторых вариантах реализации 5T4-связывающий домен содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 58. В отдельных вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 56, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 58. В некоторых вариантах реализации 5T4-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 56, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 58.
[0015] Изобретение также относится к полиспецифичному полипептиду, содержащему 5T4-связывающий домен, специфично связывающийся с 5T4 человека, и 4-1BB-связывающий домен, специфично связывающийся с 4-1BB человека, где каждый из указанных 4-1BB-связывающего домена и 5T4-связывающего домена содержит (i) вариабельную область тяжелой цепи (VH) иммуноглобулина, содержащую HCDR1, HCDR2 и HCDR3; и (ii) вариабельную область легкой цепи (VL) иммуноглобулина, содержащую LCDR1, LCDR2 и LCDR3, где область VH и/или VL 4-1BB-связывающего домена содержит одну или более мутаций в каркасной области; и где 5T4-связывающий домен содержит (а) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 60; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 62; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 34; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 54; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 36. В отдельных вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 64. В некоторых вариантах реализации 5T4-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 64. В отдельных аспектах 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 58. В некоторых вариантах реализации 5T4-связывающий домен содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 58. В отдельных вариантах реализации 5T4-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 64, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 58. В некоторых вариантах реализации 5T4-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 58.
[0016] В отдельных аспектах настоящее изобретение относится к полиспецифичному полипептиду, содержащему 4-1BB-связывающий домен, специфично связывающийся с 4-1BB человека. Изобретение также относится к полиспецифичному полипептиду, содержащему 4-1BB-связывающий домен и 5T4-связывающий домен, где указанный 4-1BB-связывающий домен связан с указанным 5T4-связывающим доменом посредством линкера связывающих доменов. В некоторых вариантах реализации полиспецифичный полипептид содержит, в направлении от аминоконца к карбоксиконцу, (i) 5T4-связывающий домен, (ii) линкер связывающих доменов и (iii) 4-1BB-связывающий домен. В отдельных аспектах 5T4-связывающий домен представляет собой одноцепочечный вариабельный фрагмент (scFv). В некоторых вариантах реализации вариабельная область легкой цепи указанного scFv является карбоксиконцевой относительно вариабельной области тяжелой цепи указанного scFv. В других вариантах реализации вариабельная область легкой цепи указанного scFv является аминоконцевой относительно вариабельной области тяжелой цепи указанного scFv. В отдельных аспектах scFv содержит линкерный полипептид. В некоторых примерах линкерный полипептид находится между вариабельной областью легкой цепи и вариабельной областью тяжелой цепи указанного scFv. В отдельных вариантах реализации линкерный полипептид содержит линкер Gly4Ser. В некоторых вариантах реализации линкерный полипептид содержит структуру формулы (Gly4Ser)n, где n = 1-5. В некоторых аспектах линкерный полипептид содержит аминокислотную последовательность, выбранную из SEQ ID NO: 85-108. В отдельных вариантах реализации линкерный полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 98 (GGGGSGGGGSGGGGSGGGGS).
[0017] В отдельных вариантах реализации scFv, содержащий последовательности CDR, указанные выше, содержит последовательность, обладающую по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90% или по меньшей мере 95% идентичностью аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 118, 120, 122, 124, 126, 128, 130, 132, 134 и 170. В некоторых вариантах реализации scFv содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 118, 120, 122, 124, 126, 128, 130, 132, 134 и 170.
[0018] В некоторых аспектах изобретение относится к полиспецифичному полипептиду, содержащему 4-1BB-связывающий домен, специфично связывающийся с 4-1BB человека, и 5T4-связывающий домен, где указанный 4-1BB-связывающий домен содержит: (i) вариабельную область тяжелой цепи (VH) иммуноглобулина, содержащую HCDR1, HCDR2 и HCDR3, и (ii) вариабельную область легкой цепи (VL) иммуноглобулина, содержащую LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность SEQ ID NO: 2; (b) HCDR2 содержит аминокислотную последовательность SEQ ID NO: 4; (c) HCDR3 содержит аминокислотную последовательность SEQ ID NO: 6; (d) LCDR1 содержит аминокислотную последовательность SEQ ID NO: 8; (e) LCDR2 содержит аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3 содержит аминокислотную последовательность SEQ ID NO: 12. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 14. В некоторых вариантах реализации 4-1BB-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 14. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 16. В некоторых вариантах реализации 4-1BB-связывающий домен содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 16. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 14, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 16. В некоторых аспектах 4-1BB-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 16.
[0019] Изобретение также относится к полиспецифичному полипептиду, содержащему 5T4-связывающий домен, специфично связывающийся с 5T4 человека, и 4-1BB-связывающий домен, где указанный 4-1BB-связывающий домен содержит (i) вариабельную область тяжелой цепи (VH) иммуноглобулина, содержащую HCDR1, HCDR2 и HCDR3; и (ii) вариабельную область легкой цепи (VL) иммуноглобулина, содержащую LCDR1, LCDR2 и LCDR3, и где указанная область VH и/или VL 4-1BB-связывающего домена содержит одну или более мутаций в каркасной области, и где указанный 4-1BB-связывающий домен содержит (а) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 18; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 4; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 6; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 8; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 12. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 20. В некоторых вариантах реализации 4-1BB-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 20. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 22. В некоторых вариантах реализации 4-1BB-связывающий домен содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 22. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 20, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 22. В некоторых вариантах реализации 4-1BB-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 20, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 22.
[0020] Изобретение также относится к полиспецифичному полипептиду, содержащему 5T4-связывающий домен, специфично связывающийся с 5T4 человека, и 4-1BB-связывающий домен, где указанный 4-1BB-связывающий домен содержит (i) вариабельную область тяжелой цепи (VH) иммуноглобулина, содержащую HCDR1, HCDR2 и HCDR3; и (ii) вариабельную область легкой цепи (VL) иммуноглобулина, содержащую LCDR1, LCDR2 и LCDR3, и где указанная область VH и/или VL 4-1BB-связывающего домена содержит одну или более мутаций в каркасной области, и где указанный 4-1BB-связывающий домен содержит (а) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 24; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 4; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 6; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 8; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 12. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 26. В некоторых вариантах реализации 4-1BB-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 26. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 16. В некоторых вариантах реализации 4-1BB-связывающий домен содержит вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 16. В отдельных вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 26, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, обладающую по меньшей мере 90%, по меньшей мере 95% или по меньшей мере 97% идентичностью SEQ ID NO: 16. В некоторых вариантах реализации 4-1BB-связывающий домен содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 26, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 16.
[0021] В отдельных вариантах реализации изобретение относится к полиспецифичному полипептиду, содержащему 4-1BB-связывающий домен, где указанный 4-1BB-связывающий домен представляет собой одноцепочечный вариабельный фрагмент (scFv). В некоторых аспектах вариабельная область легкой цепи указанного scFv является карбоксиконцевой относительно вариабельной области тяжелой цепи указанного scFv. В других аспектах вариабельная область легкой цепи указанного scFv является аминоконцевой относительно вариабельной области тяжелой цепи указанного scFv. В отдельных аспектах scFv содержит линкерный полипептид. В некоторых примерах линкерный полипептид находится между вариабельной областью легкой цепи и вариабельной областью тяжелой цепи указанного scFv. В отдельных вариантах реализации линкерный полипептид содержит линкер Gly4Ser. В некоторых вариантах реализации линкерный полипептид содержит структуру формулы (Gly4Ser)n, где n = 1-5. В некоторых аспектах линкерный полипептид содержит аминокислотную последовательность, выбранную из SEQ ID NO: 85-108. В отдельных вариантах реализации линкерный полипептид содержит аминокислотную последовательность, представленную в SEQ ID NO: 98 (GGGGSGGGGSGGGGSGGGGS). В отдельных аспектах полиспецифичный полипептид согласно изобретению содержит 4-1BB-связывающий домен, связанный с 5T4-связывающим доменом посредством линкера связывающих доменов. В некоторых вариантах реализации полиспецифичный полипептид содержит, в направлении от аминоконца к карбоксиконцу, (i) 5T4-связывающий домен, (ii) линкер связывающих доменов и (iii) 4-1BB-связывающий домен.
[0022] В некоторых вариантах реализации 4-1BB-связывающий домен, содержащий последовательности CDR, указанные выше, содержит последовательность, обладающую по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90% или по меньшей мере 95% идентичностью аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 110, 112, 114 и 116. В отдельных вариантах реализации 4-1BB-связывающий домен содержит последовательность, выбранную из группы, состоящей из SEQ ID NO: 110, 112, 114 и 116.
[0023] В отдельных аспектах полиспецифичный полипептид согласно изобретению содержит 4-1BB-связывающий домен, конъюгированный с лекарственным средством или токсином
[0024] Настоящее изобретение также относится к полиспецифичному полипептиду, содержащему 5T4-связывающий домен, слитый или конъюгированный с константной областью иммуноглобулина. Константная область иммуноглобулина может представлять собой Fc-домен иммуноглобулин человека. В некоторых вариантах реализации Fc-домен иммуноглобулина человека содержит последовательность, представленную в SEQ ID NO: 158 или SEQ ID NO: 160. В некоторых вариантах реализации настоящее изобретение относится к полиспецифичному полипептиду, содержащему, в направлении от аминоконца к карбоксиконцу: (i) 5T4-связывающий домен, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) 4-1BB-связывающий домен. В некоторых вариантах реализации линкер связывающих доменов содержит последовательность Gly4Ser. В некоторых примерах линкер связывающих доменов содержит структуру формулы (Gly4Ser)n, где n = 1-5. В некоторых аспектах линкер связывающих доменов содержит аминокислотную последовательность, выбранную из SEQ ID NO: 85-108. В отдельных вариантах реализации линкер связывающих доменов содержит аминокислотную последовательность, представленную в SEQ ID NO: 107 (SGGGGSGGGGSGGGGSPS).
[0025] В некоторых аспектах настоящее изобретение также относится к полиспецифичному полипептиду, содержащему первый scFv-домен и второй scFv-домен, где указанные первый и второй scFv-домены связаны друг с другом посредством линкера связывающих доменов или линкера связывающих доменов и Fc-домена иммуноглобулина, где указанный Fc-домен иммуноглобулина содержит шарнирную область и константную область иммуноглобулина; и где указанный второй scFv-домен специфично связывается с 4-1BB человека и содержит: (i) вариабельную область тяжелой цепи иммуноглобулина, содержащую аминокислотную последовательность HCDR1, выбранную из группы, состоящей из SEQ ID NO: 2, 18, 24, аминокислотную последовательность HCDR2, представляющую собой SEQ ID NO: 4, и аминокислотную последовательность HCDR3, представляющую собой SEQ ID NO: 6; и (ii) вариабельную область легкой цепи иммуноглобулина, содержащую аминокислотную последовательность LCDR1, представляющую собой SEQ ID NO: 8, аминокислотную последовательность LCDR2, представляющую собой SEQ ID NO: 10, и аминокислотную последовательность LCDR3, представляющую собой SEQ ID NO: 12, где указанные первый и второй scFv-домены связаны друг с другом посредством линкера связывающих доменов или линкера связывающих доменов и Fc-домена иммуноглобулина, где указанный Fc-домен иммуноглобулина содержит шарнирную область и константную область иммуноглобулина. В отдельных вариантах реализации данный полипептид содержит аминокислотную последовательность, обладающую по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90% или по меньшей мере 95% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154 и 156. В некоторых аспектах полипептид содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154 и 156. В некоторых вариантах реализации полипептид содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 136, 138 и 146.
[0026] В некоторых вариантах реализации полиспецифичный полипептид содержит, в направлении от аминоконца к карбоксиконцу, (i) первый scFv-домен, (ii) линкер связывающих доменов и (iii) второй связывающий домен. В других вариантах реализации полиспецифичный полипептид содержит, в направлении от аминоконца к карбоксиконцу, (i) второй scFv-домен, (ii) линкер связывающих доменов и (iii) первый scFv-домен. В отдельных аспектах полиспецифичный полипептид содержит, в направлении от аминоконца к карбоксиконцу: (i) первый scFv-домен, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) второй scFv-домен. В некоторых вариантах реализации линкер связывающих доменов содержит последовательность Gly4Ser. В некоторых вариантах реализации линкер связывающих доменов содержит структуру формулы (Gly4Ser)n, где n = 1-5. В отдельных аспектах линкер связывающих доменов содержит аминокислотную последовательность, выбранную из SEQ ID NO: 85-108. В отдельных вариантах реализации линкер связывающих доменов содержит аминокислотную последовательность, представленную в SEQ ID NO: 107 (SGGGGSGGGGSGGGGSPS). В некоторых вариантах реализации второй scFv-домен полиспецифичного полипептида содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 2; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 4; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 6; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 8; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 12. В других вариантах реализации второй scFv-домен полиспецифичного полипептида содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 24; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 4; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 6; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 8; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 12. В некоторых вариантах реализации второй scFv-домен полиспецифичного полипептида содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16. В других вариантах реализации второй scFv-домен полиспецифичного полипептида содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 20, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 22. В некоторых вариантах реализации второй scFv-домен полиспецифичного полипептида содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 26 и вариабельную область легкой цепи SEQ ID NO: 16. В некоторых вариантах реализации второй scFv-домен полиспецифичного полипептида содержит вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16.
[0027] Изобретение также относится к полиспецифичному полипептиду, содержащему первый scFv-домен и второй scFv-домен, где указанные первый и второй scFv-домены связаны друг с другом посредством линкера связывающих доменов или линкера связывающих доменов и Fc-домена иммуноглобулина, где указанный Fc-домен иммуноглобулина содержит шарнирную область и константную область иммуноглобулина; где указанный второй scFv-домен специфично связывается с 4-1BB человека; и где указанный первый scFv-домен содержит (i) вариабельную область тяжелой цепи иммуноглобулина, содержащую аминокислотную последовательность HCDR1, выбранную из группы, состоящей из SEQ ID NO: 30, 52 и 60, аминокислотную последовательность HCDR2, выбранную из группы, состоящей из SEQ ID NO: 32 и 62, и аминокислотную последовательность HCDR3, представляющую собой SEQ ID NO: 34; и (ii) вариабельную область легкой цепи иммуноглобулина, содержащую аминокислотную последовательность LCDR1, выбранную из группы, состоящей из SEQ ID NO: 8, 42 и 54, аминокислотную последовательность LCDR2, представляющую собой SEQ ID NO: 10, и аминокислотную последовательность LCDR3, представляющую собой SEQ ID NO: 36; и где указанный второй scFv-домен содержит (i) вариабельную область тяжелой цепи иммуноглобулина, содержащую аминокислотную последовательность HCDR1, выбранную из группы, состоящей из SEQ ID NO: 2, 18, 24, аминокислотную последовательность HCDR2, выбранную из SEQ ID NO: 4, и аминокислотную последовательность HCDR3, представляющую собой SEQ ID NO: 6; и (ii) вариабельную область легкой цепи иммуноглобулина, содержащую аминокислотную последовательность LCDR1, представляющую собой SEQ ID NO: 8, аминокислотную последовательность LCDR2, представляющую собой SEQ ID NO: 10, и аминокислотную последовательность LCDR3, представляющую собой SEQ ID NO: 12, где указанные первый и второй scFv-домены связаны вдруг с другом посредством линкера связывающих доменов или линкера связывающих доменов и Fc-домена иммуноглобулина, где указанный Fc-домен иммуноглобулина содержит шарнирную область и константную область иммуноглобулина. В некоторых вариантах реализации второй scFv-домен содержит (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 2; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 4; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 6; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 8; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 12. В отдельных вариантах реализации первый scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 30; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 32; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 34; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 8; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 36. В некоторых вариантах реализации первый scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 30; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 32; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 34; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 42; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 36. В отдельных аспектах первый scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 30; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 32; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 34; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 42; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 36. В некоторых вариантах реализации первый scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 52; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 32; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 34; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 54; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 36. В некоторых вариантах реализации первый scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 60; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 62; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 34; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 54; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 36. В отдельных вариантах реализации второй scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 18; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 4; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 6; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 8; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 12. В некоторых вариантах реализации первый scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 30; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 32; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 34; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 42; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 36. В некоторых вариантах реализации второй scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 24; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 4; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 6; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 8; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 12. В некоторых вариантах реализации первый scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 52; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 32; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 34; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 54; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 36. В некоторых вариантах реализации второй scFv-домен содержит: (а) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 24; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 4; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 6; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 8; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 12, и, необязательно, первый scFv-домен содержит: (a) аминокислотную последовательность HCDR1, представленную в SEQ ID NO: 30; (b) аминокислотную последовательность HCDR2, представленную в SEQ ID NO: 32; (c) аминокислотную последовательность HCDR3, представленную в SEQ ID NO: 34; (d) аминокислотную последовательность LCDR1, представленную в SEQ ID NO: 8; (e) аминокислотную последовательность LCDR2, представленную в SEQ ID NO: 10; и (f) аминокислотную последовательность LCDR3, представленную в SEQ ID NO: 36.
[0028] Настоящее изобретение также относится к полиспецифичному полипептиду, в котором первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 40, и где второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16. В некоторых вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 44, и второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16. В отдельных вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 48, и второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16. В других вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 56, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 58, и второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16. В отдельных вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 64, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 58, и второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 14, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16. В других вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 50, и второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 20, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 22. В других вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 56, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 58, и второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 26, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16. В некоторых вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 68, и второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16. В некоторых вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 70, и второй scFv-домен содержит ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 28, и вариабельную область легкой цепи, представляющую собой SEQ ID NO: 16.
[0029] В некоторых вариантах реализации настоящее изобретение относится к полиспецифичному полипептиду, содержащему первый scFv-домен и второй scFv-домен, где указанные первый и второй scFv-домены связаны друг с другом посредством линкера связывающих доменов или линкера связывающих доменов и Fc-домена иммуноглобулина, где указанный Fc-домен иммуноглобулина содержит шарнирную область и константную область иммуноглобулина; и где указанный первый scFv-домен специфично связывается с 5Т4 человека и содержит: (i) вариабельную область тяжелой цепи иммуноглобулина, содержащую аминокислотную последовательность HCDR1, представляющую собой SEQ ID NO: 30, аминокислотную последовательность HCDR2, представляющую собой SEQ ID NO: 32, и аминокислотную последовательность HCDR3, представляющую собой SEQ ID NO: 34; и (ii) вариабельную область легкой цепи иммуноглобулина, содержащую аминокислотную последовательность LCDR1, представляющую собой SEQ ID NO: 42, аминокислотную последовательность LCDR2, представляющую собой SEQ ID NO: 10, и аминокислотную последовательность LCDR3, представляющую собой SEQ ID NO: 36.
[0030] В некоторых вариантах реализации первый scFv-домен содержит: i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 44; или ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 48; или iii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 50.
[0031] В некоторых вариантах реализации настоящее изобретение относится к полиспецифичному полипептиду, содержащему первый scFv-домен и второй scFv-домен, где указанные первый и второй scFv-домены связаны друг с другом посредством линкера связывающих доменов или линкера связывающих доменов и Fc-домена иммуноглобулина, где указанный Fc-домен иммуноглобулина содержит шарнирную область и константную область иммуноглобулина; и где указанный первый scFv-домен специфично связывается с 5Т4 человека, где область VH и/или VL указанного второго scFv-домена содержит одну или более мутаций в каркасной области; и где указанный первый scFv-домен содержит: (а) HCDR1, содержащую аминокислотную последовательность SEQ ID NO: 30; (b) HCDR2, содержащую аминокислотную последовательность SEQ ID NO: 32; (c) HCDR3, содержащую аминокислотную последовательность SEQ ID NO: 34; (d) LCDR1, содержащую аминокислотную последовательность SEQ ID NO: 42; (e) LCDR2, содержащую аминокислотную последовательность SEQ ID NO: 10; и (f) LCDR3, содержащую аминокислотную последовательность SEQ ID NO: 36. В некоторых вариантах реализации первый scFv-домен содержит i) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 38, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 44; или ii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 48; или iii) вариабельную область тяжелой цепи, содержащую аминокислотную последовательность SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую аминокислотную последовательность SEQ ID NO: 50.
[0032] В некоторых вариантах реализации полиспецифичные полипептиды, описанные в настоящем документе, содержат, в направлении от аминоконца к карбоксиконцу, (i) первый scFv-домен, (ii) линкер связывающих доменов и (iii) второй связывающий домен. В некоторых вариантах реализации полиспецифичные полипептиды содержат, в направлении от аминоконца к карбоксиконцу, (i) второй scFv-домен, (ii) линкер связывающих доменов и (iii) первый scFv-домен. В некоторых вариантах реализации полиспецифичные полипептиды содержат, в направлении от аминоконца к карбоксиконцу: (i) первый scFv-домен, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) второй scFv-домен. В некоторых вариантах реализации линкер связывающих доменов содержит последовательность Gly4Ser. В некоторых вариантах реализации линкер связывающих доменов содержит структуру формулы (Gly4Ser)n, где n = 1-5. В некоторых вариантах реализации линкер связывающих доменов содержит аминокислотную последовательность, выбранную из SEQ ID NO: 85-108. В некоторых вариантах реализации линкер связывающих доменов содержит аминокислотную последовательность, представленную в SEQ ID NO: 107 (SGGGGSGGGGSGGGGSPS).
[0033] В отдельных аспектах первый scFv-домен полиспецифичного полипептида согласно изобретению специфично связывается с 5Т4 человека и содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 118, 120, 122, 124, 126, 128, 130, 132, 134 и 170; и второй scFv-домен специфично связывается с 4-1BB человека и содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 110, 112, 114 и 116. В некоторых вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 118, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 110. В других вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 120, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 110. В отдельных вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 122, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 110. В некоторых вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 124, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 112. В некоторых вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 126, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 114. В других вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 126, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 110. В отдельных вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 128, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 110. В некоторых вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 170, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 116. В некоторых вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 130, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 116. В некоторых вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 132, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 116. В других вариантах реализации первый scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 134, и второй scFv-домен содержит аминокислотную последовательность, представленную в SEQ ID NO: 116. В некоторых вариантах реализации изобретение относится к полиспецифичному полипептиду, содержащему аминокислотную последовательность, обладающую по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90% или по меньшей мере 95% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176. В некоторых вариантах реализации полипептид содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176.
[0034] В некоторых вариантах реализации изобретение относится к полиспецифичному полипептиду, специфично связывающемуся с 5T4 и 4-1BB, где указанный полипептид содержит аминокислотную последовательность, обладающую по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90% или по меньшей мере 95% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176. В отдельных вариантах реализации полипептид, специфично связывающийся с 5T4 и 4-1BB, содержит аминокислотную последовательность, обладающую по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90% или по меньшей мере 95% идентичностью с последовательностью, выбранной из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176, где указанный полипептид содержит те же аминокислотные последовательности CDR, что и соответствующая SEQ ID NO:, или указанный полипептид содержит аминокислотные последовательности CDR, которые отличаются от соответствующей SEQ ID NO: не более чем одной аминокислотой. В некоторых вариантах реализации полипептид содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176.
[0035] Настоящее изобретение также относится к полиспецифичному полипептиду, где первый scFv-домен связывается с внеклеточным доменом 5T4, и где второй scFv-домен связывается с внеклеточным доменом 4-1BB. В некоторых аспектах полипептид приводит к усилению активации эффекторных клеток. В некоторых аспектах полиспецифичный полипептид согласно изобретению повышает активацию эффекторных клеток и/или пролиферацию эффекторных клеток. В других аспектах полиспецифичный полипептид усиливает зависимый от эффекторных клеток лизис 5Т4-экспрессирующих клеток. В некоторых вариантах реализации 5T4-связывающий домен согласно изобретению способен связывать 5T4 со значением kD менее 50 нМ. В некоторых аспектах вариабельная область легкой цепи и/или вариабельная область тяжелой цепи 5T4-связывающего домена и/или 4-1BB-связывающего домена является гуманизированной.
[0036] В некоторых вариантах реализации настоящее изобретение относится к полиспецифичному полипептиду, содержащему первый домен, представляющий собой одноцепочечный вариабельный фрагмент (scFv), и второй scFv-домен, связанные друг с другом посредством линкера связывающих доменов и Fc-домена иммуноглобулина, где указанный Fc-домен иммуноглобулина содержит шарнирную область и константную область иммуноглобулина; где указанный полиспецифичный полипептид способен образовывать гомодимер путем ассоциации со вторым идентичным полиспецифичным полипептидом; где указанный первый scFv-домен специфично связывается с 5Т4 человека и содержит: (i) вариабельную область тяжелой цепи иммуноглобулина, содержащую аминокислотную последовательность определяющей комплементарность области тяжелой цепи (HCDR)-1, представляющую собой SEQ ID NO: 30, аминокислотную последовательность HCDR2, представляющую собой SEQ ID NO: 32, и аминокислотную последовательность HCDR3, представляющую собой SEQ ID NO: 34; и (ii) вариабельную область легкой цепи иммуноглобулина, содержащую аминокислотную последовательность определяющей комплементарность области легкой цепи (LCDR)-1, представляющую собой SEQ ID NO: 42, аминокислотную последовательность LCDR2, представляющую собой SEQ ID NO: 10, и аминокислотную последовательность LCDR3, представляющую собой SEQ ID NO: 36; и где указанный второй scFv-домен специфично связывается с 4-1BB человека и содержит: (i) вариабельную область тяжелой цепи иммуноглобулина, содержащую аминокислотную последовательность HCDR1, представляющую собой SEQ ID NO: 2, аминокислотную последовательность HCDR2, представляющую собой SEQ ID NO: 4, и аминокислотную последовательность HCDR3, представляющую собой SEQ ID NO: 6; и (ii) вариабельную область легкой цепи иммуноглобулина, содержащую аминокислотную последовательность LCDR1, представляющую собой SEQ ID NO: 8, аминокислотную последовательность LCDR2, представляющую собой SEQ ID NO: 10, и аминокислотную последовательность LCDR3, представляющую собой SEQ ID NO: 12. 2. Полиспецифичный полипептид по п. 1, содержащий, в направлении от аминоконца к карбоксиконцу: (i) первый scFv-домен, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) второй scFv-домен. В некоторых вариантах реализации первый scFv-домен содержит мутацию в каркасной области по сравнению с каркасной областью SEQ ID NO: 130 или 170. В некоторых вариантах реализации мутация вводит стабилизирующую дисульфидную связь.
[0037] В некоторых вариантах реализации первый scFv-домен полиспецифичного полипептида содержит вариабельную область тяжелой цепи иммуноглобулина, представляющую собой SEQ ID NO: 38, и вариабельную область легкой цепи иммуноглобулина, представляющую собой SEQ ID NO: 44. В некоторых вариантах реализации первый scFv-домен полиспецифичного полипептида содержит вариабельную область тяжелой цепи иммуноглобулина, представляющую собой SEQ ID NO: 46, и вариабельную область легкой цепи иммуноглобулина, представляющую собой SEQ ID NO: 48. В некоторых вариантах реализации второй scFv-домен полиспецифичного полипептида содержит вариабельную область тяжелой цепи иммуноглобулина, представляющую собой SEQ ID NO: 14, и вариабельную область легкой цепи иммуноглобулина, представляющую собой SEQ ID NO: 16. В некоторых вариантах реализации первый scFv-домен полиспецифичного полипептида содержит вариабельную область тяжелой цепи иммуноглобулина, представляющую собой SEQ ID NO: 46, и вариабельную область легкой цепи иммуноглобулина, представляющую собой SEQ ID NO: 48, и второй scFv-домен содержит вариабельную область цепь тяжелой цепи иммуноглобулина, представляющую собой SEQ ID NO: 14, и вариабельную область легкой цепи иммуноглобулина, представляющую собой SEQ ID NO: 16. В некоторых вариантах реализации аминокислотная последовательность первого scFv-домена по меньшей мере на 97% идентична SEQ ID NO: 120, и аминокислотная последовательность второго scFv-домена по меньшей мере на 97% идентична SEQ ID NO: 110. В некоторых вариантах реализации полипептид содержит аминокислотную последовательность, по меньшей мере на 95% идентичную последовательности SEQ ID NO: 172, или содержит аминокислотную последовательность, идентичную последовательности SEQ ID NO: 172. В некоторых вариантах реализации аминокислотная последовательность первого домена scFv по меньшей мере на 97% идентична SEQ ID NO: 122, а аминокислотная последовательность второго домена scFv по меньшей мере на 97% идентична SEQ ID NO: 110. В некоторых вариантах реализации полипептид содержит аминокислотную последовательность, по меньшей мере на 95% идентичную последовательности SEQ ID NO: 174, или содержит аминокислотную последовательность SEQ ID NO: 174.
[0038] В некоторых вариантах реализации связывание полиспецифичного полипептида с эффекторной клеткой приводит к повышенной активации эффекторной клетки, повышенной пролиферации эффекторной клетки, или связывание полиспецифичного полипептида с эффекторной клеткой и 5T4-экспрессирующей клеткой приводит к усиленному зависимому от эффекторных клеток лизису указанной 5T4-экспрессирующей клетки.
[0039] Изобретение также включает димер, содержащий два идентичных полипептида, где каждый из двух полипептидов представляет собой полиспецифичный полипептид, описанный в настоящем документе.
[0040] В отдельных аспектах раскрытие изобретение относится к фармацевтической композиции, содержащей полипептид или белок, описанные в настоящем документе, и фармацевтически приемлемый носитель, разбавитель или вспомогательное вещество. В некоторых вариантах реализации фармацевтическая композиция может быть выполнена в дозированной форме, выбранной из группы, состоящей из: пероральной формы однократной дозы, внутривенной формы однократной дозы, интраназальной формы однократной дозы, суппозиторной формы однократной дозы, внутрикожной формы однократной дозы, внутримышечной формы однократной дозы, внутрибрюшинной формы однократной дозы, подкожной формы однократной дозы, эпидуральной формы однократной дозы, сублингвальной формы однократной дозы и интрацеребральной формы однократной дозы. Неограничивающие примеры пероральной формы однократной дозы включают таблетки, пилюли, пеллеты, капсулы, порошки, таблетки для рассасывания, гранулы, растворы, суспензии, эмульсии, сиропы, эликсиры, составы с замедленным высвобождением, аэрозоли и спреи.
[0041] Изобретение также включает способ усиления активации эффекторных клеток, направленной против клетки, экспрессирующей 5T4, включающий: приведение указанной 5T4-экспрессирующей клетки в контакт с полипептидом или белком согласно изобретению, где указанное приведение в контакт проводят в условиях, при которых индуцируется усиленная активация эффекторных клеток, направленная против указанной 5T4-экспрессирующей клетки. В некоторых аспектах изобретение относится к способу лечения расстройства у субъекта, где указанное расстройство характеризуется экспрессией 5T4, включающему введение указанному субъекту терапевтически эффективного количества полипептида, или белка, или фармацевтической композиции согласно изобретению. Изобретение также включает применение полипептида или белка согласно изобретению для изготовления лекарственного средства для лечения расстройства у субъекта, где указанное расстройство характеризуется экспрессией 5T4. В некоторых вариантах реализации изобретение относится к полипептиду или белку согласно изобретению для применения для лечения расстройства у субъекта, где указанное расстройство характеризуется экспрессией 5T4. В некоторых вариантах реализации расстройство представляет собой рак. В отдельных аспектах рак представляет собой рак молочной железы, рак поджелудочной железы, рак яичника, немелкоклеточный рак легкого, мезотелиому, хронический лимфоцитарный лейкоз (ХЛЛ), мантийноклеточный лейкоз (МКЛ), острый лимфобластный лейкоз (ОЛЛ), плоскоклеточную карциному, меланому, рак надпочечника, рак мочевого пузыря, рак шейки матки, рак почки, рак желудка, рак предстательной железы, рак щитовидной железы, рак печени, рак матки, нейрофиброму, саркому или рак головы и шеи.
[0042] В некоторых вариантах реализации полиспецифичный полипептид для применения в таких способах содержит, в направлении от аминоконца к карбоксиконцу: (i) первый scFv-домен, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) второй scFv-домен. В некоторых вариантах реализации полиспецифичный полипептид для применения в таких способах содержит второй scFv-домен, содержащий вариабельную область тяжелой цепи иммуноглобулина, представляющую собой SEQ ID NO: 14, и вариабельную область легкой цепи иммуноглобулина, представляющую собой SEQ ID NO: 16, и первый scFv-домен, содержащий (i) вариабельную область тяжелой цепи иммуноглобулина, представляющую собой SEQ ID NO: 38, и вариабельную область легкой цепи иммуноглобулина, представляющую собой SEQ ID NO: 44; или (ii) вариабельную область тяжелой цепи иммуноглобулина, представляющую собой SEQ ID NO: 46, и вариабельную область легкой цепи иммуноглобулина, представляющую собой SEQ ID NO: 48. В некоторых вариантах реализации аминокислотная последовательность второго scFv-домена полиспецифичного полипептида по меньшей мере на 97% идентична SEQ ID NO: 110, и аминокислотная последовательность первого scFv-домена полиспецифичного полипептида по меньшей мере на 97% идентична SEQ ID NO: 120 (scFv к 5T4 для 209) или SEQ ID NO: 122. В некоторых вариантах реализации указанный полипептид демонстрирует статистически значимо усиленную активацию эффекторных клеток по сравнению со вторым полиспецифичным полипептидом, где указанный второй полиспецифичный полипептид представляет собой структуру IgG-scFv, содержащую антитело к 4-1BB, содержащее вариабельную тяжелую цепь, содержащую SEQ ID NO: 28, и вариабельную легкую цепь, содержащую SEQ ID NO: 16, и scFv к 5T4, содержащую вариабельную тяжелую цепь, содержащую SEQ ID NO: 46, и вариабельную легкую цепь, содержащую SEQ ID NO: 66. В некоторых вариантах реализации указанный полипептид индуцирует статистически значимо повышенную пролиферацию эффекторных клеток по сравнению со вторым полиспецифичным полипептидом, где указанный второй полиспецифичный полипептид представляет собой структуру IgG-scFv, содержащую антитело против 4-1BB, содержащее вариабельную тяжелую цепь, содержащую SEQ ID NO: 28, и вариабельную легкую цепь, содержащую SEQ ID NO: 16, и scFv против 5T4, содержащую вариабельную тяжелую цепь, содержащую SEQ ID NO: 46, и вариабельную легкую цепь, содержащую SEQ ID NO: 66.
[0043] Некоторые аспекты изобретения включают выделенную молекулу нуклеиновой кислоты, кодирующую полипептид согласно изобретению. В некоторых вариантах реализации молекула нуклеиновой кислоты содержит нуклеотидную последовательность, выбранную из группы, состоящей из SEQ ID NO: 109, 111, 113, 115, 117, 119, 121, 123, 125, 127, 129, 131, 133, 135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 171, 173 и 175. Изобретение включает вектор экспрессии, содержащий выделенную молекулу нуклеиновой кислоты, описанную в настоящем документе. В некоторых вариантах реализации молекула нуклеиновой кислоты в векторе экспрессии функционально связана с регуляторными последовательностями, подходящими для экспрессии сегмента нуклеиновой кислоты в клетке-хозяине. Изобретение относится к рекомбинантной клетке-хозяину, содержащей вектор экспрессии, описанный в настоящем документе. В некоторых аспектах изобретение относится к способу получения полипептида, содержащего 5T4-связывающий домен, включающему культивирование рекомбинантной клетки-хозяина, содержащей вектор экспрессии, описанный в настоящем документе, в условиях, при которых экспрессируется сегмент нуклеиновой кислоты, с получением тем самым полипептида, содержащего 5T4-связывающий домен.
Краткое описание графических материалов
[0044] На ФИГ. 1 проиллюстрирована агонистическая функция семи конструкций (ALG.APV-004, ALG.APV-006, ALG.APV-099, ALG.APV-127, ALG.APV-148 и ALG.APV-150) в присутствии 5T4(+) клеток.
[0045] На ФИГ. 2 проиллюстрированы кривые связывания семи молекул scFv-Fc-scFv (ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198) с линией клеток CHO-K1, экспрессирующей 4-1BB человека.
[0046] На ФИГ. 3 проиллюстрированы кривые связывания семи молекул scFv-Fc-scFv (ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198) с линией клеток CHO-K1, экспрессирующей 4-1BB яванского макака.
[0047] На ФИГ. 4 проиллюстрированы кривые связывания семи молекул scFv-Fc-scFv (ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198) с линией клеток CHO-K1, экспрессирующей 5T4 человека.
[0048] На ФИГ. 5 проиллюстрированы уровни экспрессии человеческого 5T4 в линиях клеток CHO-K1 и SKOV-3.
[0049] На ФИГ. 6 проиллюстрированы кривые связывания конструкций ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-196 и ALG.APV-198 на клетках SKOV-3.
[0050] На ФИГ. 7 проиллюстрированы кривые связывания ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198 на клетках линии CHO-K1, экспрессирующих 5T4 яванского макака.
[0051] На ФИГ. 8 проиллюстрирована активность конструкций к 5Т4 x 4-1ВВ в репортерном анализе 4-1ВВ.
[0052] На ФИГ. 9 проиллюстрировано действие конструкций к 5Т4 x 4-1ВВ на высвобождение ИФН-γ из совместных культур МНПК и CHO-K1.
[0053] На ФИГ. 10 проиллюстрирован иллюстративный вариант реализации белка в формате scFv-Fc-scFv (формат ADAPTIR™).
[0054] На ФИГ. 11A - ФИГ. 11B показан ответ интерферона-гамма (ИФН-γ) в CD8+ T-клетках человека, культивированных с биспецифичными конструкциями scFv-Fc-scFv. На ФИГ. 11А показаны Т-клеточные ответы при культивировании на планшетах, покрытых антигеном 5T4-Fc. На ФИГ. 11B показаны Т-клеточные ответы при культивировании без антигена 5T4-Fc. На фигуре показаны средние значения для двух доноров.
[0055] На ФИГ. 12 показаны кривые связывания ALG.APV-004, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210 с линией клеток CHO-K1/ CD137 человека.
[0056] На ФИГ. 13 показаны кривые связывания ALG.APV-004, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210 с мышиными клетками CT26 (ATCC), экспрессирующими 5T4 человека.
[0057] На ФИГ. 14 показаны кривые связывания ALG.APV-004, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210 на трансфектантах CHO-K1/5T4 яванского макака.
[0058] На ФИГ. 15 показаны кривые связывания ALG.APV-004, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210 на линии клеток человека, не экспрессирующих ни CD137, ни 5T4 (клетки MOLM13, ATCC).
[0059] На ФИГ. 16 показана активность биспецифичных конструкций в репортерных клетках Jurkat/NF-κB после 5 часов инкубации в присутствии 5T4(+) клеток (HCC1143) или 5T4(-) клеток (MOLM13). Каждая точка на кривой представляет собой среднее значение для параллельных лунок. На оси y показаны значения в относительных единицах флуоресценции (RLU).
[0060] На ФИГ. 17 показаны уровни ИФН-γ, индуцированные в первичных культурах МНПК через 72 часа посредством ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209, ALG.APV-210, и контрольной конструкцией в формате Моррисона (Morrison), ALG.APV-004. Каждая точка на кривой представляет собой среднее значение для параллельных лунок.
[0061] На ФИГ. 18A - ФИГ. 18B проиллюстрирован анализ методом FACS 5T4-зависимой локализации биспецифичных конструкций ALG.APV-209, ALG.APV-210 и ALG.APV-004 в антигенпрезентирующие опухоли. Локализацию определяли путем окрашивания антителом к человеческому Fc (ФИГ. 18A) или биотинилированным 4-1BB (ФИГ. 18B).
[0062] На ФИГ. 19A - ФИГ. 19B проиллюстрирован иммуногистохимический анализ 5T4-зависимой локализации биспецифичных конструкций ALG.APV-209, ALG.APV-210 и ALG.APV-004 в 5T4+ (ФИГ. 19A) и 5T4- (ФИГ. 19B) опухоли.
[0063] На ФИГ. 20 показан ИФН-g-ответ CD8 T-клеток человека, культивированных с биспецифичным антителом ALG.APV-210 в присутствии клеток CT26, экспрессирующих различные уровни 5T4.
[0064] На ФИГ. 21А - ФИГ. 21B показано максимальное высвобождение ИФН-γ CD8 Т-клетками человека, культивированными с биспецифичным антителом ALG.APV-210 в присутствии клеток СТ26, экспрессирующих различные уровни 5Т4.
[0065] На ФИГ. 22А - ФИГ. 22C показано связывание ALG.APV-210 или ANC107 (изотипический контроль) с первичными CD8 Т-клетками, отобранными из стимулированных CD3 или нестимулированных МНПК человека и яванского макака. MFI (и SEM) из 2 независимых экспериментов показаны на ФИГ. 22A и ФИГ. 22B. На ФИГ. 22C показаны нормированные объединенные данные для ALG.APV-210 из 2 независимых экспериментов. n = 3 донора/группу/эксперимент.
[0066] На ФИГ. 23А - ФИГ. 23C показано связывание ALG.APV-210 или ANC107 (изотипический контроль) с первичными CD8 Т-клетками, гейтированными из стимулированных CD3 или нестимулированных МНПК человека и яванского макака. Процент и SEM связывания ALG.APV-210 с CD8 Т-клетками из 2 независимых экспериментов показаны на ФИГ. 23А и 23В. На ФИГ. 23C показаны нормированные объединенные данные из экспериментов 1 и 2. n = 3 донора/группу/эксперимент.
[0067] На ФИГ. 24 показана агонистическая функция ALG.APV-210 в отношении CD8 Т-клеток человека.
[0068] На ФИГ. 25A - ФИГ. 25B показана агонистическая функция ALG.APV-210 в отношении CD8 T-клеток человека от отдельных репрезентативных доноров.
[0069] На ФИГ. 26 показана агонистическая функция ALG.APV-210 в отношении CD8 T-клеток яванского макака.
[0070] На ФИГ. 27A - ФИГ. 27C показана агонистическая функция ALG.APV-210 в отношении CD8 T-клеток яванского макака от отдельных репрезентативных доноров. На фигурах показана дозозависимая выработка ИФН-g CD8 T-клетками яванского макака, активированными ALG.APV-210, в присутствии 5T4-Fc.
[0071] На ФИГ. 28 показана противоопухолевая эффективность биспецифичного антитела ALG.APV-210 в HCT-116 - 5T4-позитивной ксенотрансплантатной модели карциномы толстой кишки человека у мышей SCID beige.
[0072] На ФИГ. 29 показан анализ ФК свойств ALG.APV-210 или ALG.APV-209.
[0073] На ФИГ. 30А - ФИГ. 30B показано связывание Fc-части ALG.APV-210 с FcγR. ALG.APV-210 с определенным титром инкубировали с FcγR-экспрессирующими клетками и зондировали флуоресцентно-меченым вторичным антителом к человеческому IgG (ФИГ. 30А). Молекулу человеческого IgG1 использовали в качестве положительного контроля (ФИГ. 30B).
[0074] На ФИГ. 31А - ФИГ. 31B показана пролиферация CD8+ и CD4+ T-клеток, индуцированная биспецифичными молекулами ALG.APV-209 и ALG.APV-210 в присутствии клеток CHO-K1, экспрессирующих 5T4 человека, и антител к CD3.
[0075] На ФИГ. 32А - ФИГ. 32B показана индукция секреции ИФН-γ биспецифичной молекулой ALG.APV-210 в культурах МНПК из цельной крови от двух разных людей-доноров МНПК (A и B).
[0076] На ФИГ. 33А - FIG. 33B показана пролиферация CD8+ T-клеток, индуцированная биспецифичной молекулой ALG.APV-210 в культурах МНПК из цельной крови от двух разных людей-доноров МНПК.
[0077] На ФИГ. 34 показано связывание ALG.APV-210 с 5T4-экспрессирующими линиями клеток.
[0078] На ФИГ. 35 показана агонистическая функция биспецифичной конструкции ALG.APV-210 в репортерных клетках Jurkat/NF-κB после инкубации в присутствии клеток, экспрессирующих белок 5T4 в диапазоне плотностей на своей поверхности.
[0079] На ФИГ. 36 показан ИФН-γ-ответ CD8+ T-клеток человека, культивированных с биспецифичным антителом ALG.APV-210 в присутствии клеток HCT116 человека, экспрессирующих 5T4.
[0080] На ФИГ. 37 показан ИФН-γ-ответ CD8+ T-клеток человека, культивированных с биспецифичным антителом ALG.APV-210 в присутствии клеток HCT116 человека, экспрессирующих 5T4.
[0081] На ФИГ. 38 показано максимальное высвобождение ИФН-γ CD8+ T-клетками человека, культивированными с биспецифичным антителом ALG.APV-210 в присутствии клеток HCT116, экспрессирующих 5T4 человека.
[0082] На ФИГ. 39А - ФИГ. 39D показано связывание ALG.APV-210 с опухолевыми клетками человека, экспрессирующими 5T4, или линиями трансфицированных клеток.
[0083] На ФИГ. 40А - FIG. 40D показана нормированная MFI для экспериментов, демонстрирующих связывание ALG.APV-210 с опухолевыми клетками человека, экспрессирующими 5T4, или линиями трансфицированных клеток.
Подробное описание изобретения
[0084] Настоящее изобретение относится к полипептидам, содержащим связывающие домены, специфично связывающиеся с трофобластическим гликопротеином (5T4), и полипептидам, специфично связывающимся с 5T4. В некоторых вариантах реализации полипептиды представляют собой полиспецифичные полипептиды, способные специфично связываться с 5Т4 и с другой мишенью. В некоторых вариантах реализации полипептиды, описанные в настоящем документе, представляют собой полиспецифичные полипептиды, специфично связывающиеся с 5Т4, а также специфично связывающиеся с мишенью на эффекторной клетке. Изобретение также относится к полипептидам, содержащим связывающие домены, специфично связывающиеся с членом 9 суперсемейства рецепторов фактора некроза опухоли (41BB или CD137), и полипептидам, специфично связывающимся с 4-1BB. В некоторых вариантах реализации полипептиды представляют собой полиспецифичные полипептиды, способные специфично связываться с 4-1BB и с другой мишенью (например, опухолеассоциированным антигеном). В некоторых вариантах реализации полипептиды, описанные в настоящем документе, представляют собой полиспецифичные полипептиды, специфично связывающиеся с 4-1BB, а также специфично связывающиеся с мишенью на клетке-мишени. В некоторых вариантах реализации полиспецифичные полипептиды представляют собой биспецифичные полипептиды, специфично связывающиеся с 5T4 и специфично связывающиеся с 4-1BB. В некоторых вариантах реализации биспецифичные полипептиды связываются с 5T4, экспрессируемым на клетке-мишени, и 4-1BB, экспрессируемым на эффекторной клетке, тем самым приводя к усилению активации эффекторных клеток и усиливая опосредованную эффекторными клетками цитотоксичность в отношении клетки-мишени.
[0085] В некоторых вариантах реализации настоящее изобретение относится к 5T4-связывающим доменам и/или 4-1BB-связывающим доменам (и полипептидам или белкам, содержащим такие связывающие домены), демонстрирующим меньшее нецелевое связывание по сравнению с ранее известными 5T4-связывающими доменами и/или 4-1BB-связывающими доменами. В отдельных аспектах описанные в настоящем документе связывающие домены и/или полипептиды, содержащие связывающие домены, связываются с 5T4 и/или 4-1BB более эффективно в определенных форматах и/или определенных ориентациях (например, VH-VL по сравнению с VL-VH), что приводит к более высокой активности и/или улучшенной эффективности при лечении расстройств, связанных с экспрессией 5Т4.
[0086] В некоторых вариантах реализации введение терапевтически эффективного количества полипептида или белка, описанного в настоящем документе, нуждающемуся в этом пациенту, эффективно для лечения определенных расстройств, связанных с экспрессией 5T4, включая определенные виды рака. В одном из вариантов реализации полипептид или белок связывает как клетку-мишень, экспрессирующую 5T4, так и эффекторную клетку, тем самым «сшивая» клетку-мишень, экспрессирующую 5T4, и эффекторную клетку. Связывание обоих доменов с их мишенями усиливает активацию эффекторных клеток, приводя к длительному и/или более устойчивому ответу эффекторных клеток (например, цитотоксичности, опосредованной эффекторными клетками). Полипептиды и белки согласно настоящему изобретению обеспечивают различные преимущества при лечении пациентов, например, эффективное связывание с 5Т4, эффективное усиление активности эффекторных клеток, сниженные уровни высвобождения цитокинов и/или более низкий риск нежелательных явлений (например, токсичности). В некоторых вариантах реализации клетка-мишень экспрессирует 5T4 на более высоком уровне, чем клетка, не являющаяся мишенью (например, нормальная клетка или незлокачественная клетка того же субъекта, органа или ткани). В других вариантах реализации клетка-мишень экспрессирует 5T4, тогда как клетка, не являющаяся мишенью (например, нормальная клетка или незлокачественная клетка того же субъекта, органа или ткани), не экспрессирует 5T4.
[0087] Заголовки разделов, используемые в настоящем документе, предназначены только для целей упорядочения и не должны рассматриваться как ограничивающие описанное изобретение. Все документы или части документов, цитируемые в настоящем документе, включая, не ограничиваясь перечисленным, патенты, патентные заявки, статьи, книги и трактаты, настоящим явным образом включены посредством ссылки в полном объеме для любых целей. В случае, если один или более из включенных документов или частей документов определяют термин, который противоречит определению этого термина в заявке, определение, приведенное в данной заявке, имеет преимущественную силу. Однако упоминание любого источника, статьи, публикации, патента, патентной публикации и патентной заявки, цитируемых в настоящем документе, не является и не должно быть истолковано как признание или любая форма предположения о том, что они представляют собой релевантный уровень техники или составляют часть общеизвестных сведений в какой-либо стране мира.
[0088] В настоящем описании любой диапазон концентраций, процентный диапазон, диапазон отношений или целочисленный диапазон следует понимать как включающие значение любого целого числа в пределах указанного диапазона и, когда применимо, его долей (таких как одна десятая и одна сотая целого числа), если не указано иное. Следует понимать, что термины, используемые в настоящем документе в единственном числе, относятся к «одному или более» из перечисленных компонентов, если не указано иное. Применение альтернативы (например, «или») следует понимать как одну, обе или любую комбинацию этих альтернатив. В контексте настоящего документа термины «включать» и «содержать» являются синонимами. Кроме того, следует понимать, что полипептиды, содержащие различные комбинации компонентов (например, доменов или областей) и заместителей, описанных в настоящем документе, раскрыты в настоящей заявке в той же степени, как если бы каждый полипептид был описан по отдельности. Таким образом, выбор конкретных компонентов отдельных полипептидов находится в пределах объема настоящего изобретения.
Определения
[0089] В контексте настоящего документа термин «связывающий домен» или «связывающая область» относится к домену, области, части или сайту белка, полипептида, олигопептида, пептида, антитела или связывающего домена, полученного из антитела, обладающим способностью специфично распознавать и связываться с молекулой-мишенью, такой как антиген, лиганд, рецептор, субстрат или ингибитор (например, 5T4 или 4-1BB). Иллюстративные связывающие домены включают антитела и антителоподобные белки или домены, вариабельные области тяжелой и легкой цепи антитела и вариабельные области одноцепочечного антитела (например, доменные антитела, sFv, scFv, scFab). В отдельных вариантах реализации связывающий домен содержит или состоит из антигенсвязывающего сайта (например, содержащего вариабельную последовательность тяжелой цепи и вариабельную последовательность легкой цепи, или три определяющие комплементарность области (CDR) легкой цепи и три CDR тяжелой цепи антитела, размещенные на альтернативных каркасных областях (FR) (например, FR человека, необязательно содержащих одну или более аминокислотных замен)). Известны различные анализы для идентификации связывающих доменов согласно настоящему изобретению, которые специфично связывают определенную мишень, включая вестерн-блоттинг, ELISA, скрининг библиотеки фагового дисплея и анализ взаимодействия BIACORE®. В некоторых вариантах реализации полипептиды согласно настоящему изобретению содержат связывающий домен, специфично связывающийся с антигеном-мишенью, экспрессируемым клеткой-мишенью (например, опухолеассоциированным антигеном, таким как 5T4). В некоторых вариантах реализации полипептиды согласно настоящему изобретению содержат связывающий домен, специфично связывающийся с антигеном-мишенью, экспрессируемым эффекторной клеткой (например, 4-1BB). В некоторых вариантах реализации полипептиды согласно настоящему изобретению представляют собой полиспецифичные полипептиды и содержат два или более связывающих доменов.
[0090] Связывающий домен или белок, содержащий связывающий домен, «специфично связывает» мишень, если он связывает указанную мишень с аффинностью или Ka (то есть, равновесной константой ассоциации конкретного связывающего взаимодействия, выражаемой в 1/М), равной или большей 105 M-1, и при этом в незначительной степени связывает другие компоненты, присутствующие в тестируемом образце. Связывающие домены могут быть классифицированы как «высокоаффинные» и «низкоаффинные» связывающие домены. «Высокоаффинные» связывающие домены относятся к связывающим доменам, Ka которых составляет по меньшей мере 107 M-1, по меньшей мере 108 M-1, по меньшей мере 109 M-1, по меньшей мере 1010 M-1, по меньшей мере 1011 M-1, по меньшей мере 1012 M-1 или по меньшей мере 1013 M-1. «Низкоаффинные» связывающие домены относятся связывающим доменам, Ka которых составляет вплоть до 107 M-1, вплоть до 106 M-1, вплоть до 105 M-1. В качестве альтернативы, аффинность может быть определена как равновесная константа диссоциации (Kd) конкретного связывающего взаимодействия, выражаемая в М (например, от 10-5 M до 10-13, или приблизительно 500 нМ, приблизительно 300 нМ, приблизительно 250 нМ, приблизительно 200 нМ, приблизительно 150 нМ, приблизительно 100 нМ, приблизительно 50 нМ, приблизительно 25 нМ, приблизительно 10 нМ или приблизительно 5 нМ). Аффиности связывающего домена полипептидов и одноцепочечных полипептидов согласно настоящему изобретению можно без труда определить с использованием общепринятых методик (см., например, Scatchard et al. (1949) Ann. N.Y. Acad. Sci. 51:660; и патенты США №№ 5,283,173, 5,468,614, или их эквивалент).
[0091] В контексте настоящей заявки термин «консервативная замена» в данной области техники известен как замена одной аминокислоты другой аминокислотой, обладающей схожими свойствами. Типичные консервативные замены хорошо известны в данной области техники (см., например, публикацию заявки PCT № WO 97/09433, стр. 10, опубликовано 13 марта 1997 г.; Lehninger, Biochemistry, Second Edition; Worth Publishers, Inc. NY:NY (1975), стр.71-77; Lewin, Genes IV, Oxford University Press, NY and Cell Press, Cambridge, MA (1990), стр. 8).
[0092] В контексте настоящей заявки термин «производное» относится к модификации одного или более аминокислотных остатков пептида химическими или биологическими средствами, с использованием фермента либо без него, например, путем гликозилирования, алкилирования, ацилирования, образования сложного эфира или образования амида.
[0093] В контексте настоящего документа полипептид или аминокислотная последовательность, «полученные из» указанного полипептида или белка, относится к происхождению полипептида. В отдельных вариантах реализации полипептид или аминокислотная последовательность, полученная из конкретной последовательности (иногда называемой «исходной» или «родительской» последовательностью) и имеющая аминокислотную последовательность, по существу идентичную исходной последовательности или ее части, где часть состоит из по меньшей мере 10-20 аминокислот, по меньшей мере 20-30 аминокислот, или по меньшей мере 30-50 аминокислот, или по меньшей мере 50-150 аминокислот, или иным образом может быть идентифицирована специалистом в данной области техники как происходящая от исходной последовательности. Например, связывающий домен (например, Fab, F(ab’)2, Fab’, scFv, однодоменное антитело (sdAb) и т. д.) может быть получен из антитела. В некоторых вариантах реализации последовательность связывающего домена (например, 5T4- или 4-1BB-связывающего домена) получена из антитела или белка посредством компьютерного алгоритма или in silico.
[0094] Полипептиды, полученные из другого полипептида, могут иметь одну или более мутаций или изменений относительно исходного полипептида, например, один или более аминокислотных остатков, которые были заменены другим аминокислотным остатком, или имеющие одну или более инсерций или делеций аминокислот. В таких вариантах реализации полипептиды, полученные из исходного полипептида и содержащие одну или более мутаций или изменений, называются «вариантами». В контексте настоящего документа термин «вариант» или «варианты» относится к полинуклеотиду или полипептиду с последовательностью, отличающейся от последовательности референсного полинуклеотида или полипептида, но сохранившему его существенные свойства. Как правило, вариантные полинуклеотидные или полипептидные последовательности в целом очень похожи и во многих областях идентичны референсному полинуклеотиду или полипептиду. Например, вариантный полинуклеотид или полипептид может обладать по меньшей мере приблизительно 70%, по меньшей мере приблизительно 80%, по меньшей мере приблизительно 90%, по меньшей мере приблизительно 91%, по меньшей мере приблизительно 92%, по меньшей мере приблизительно 93%, по меньшей мере приблизительно 94%, по меньшей мере приблизительно 95%, по меньшей мере приблизительно 96%, по меньшей мере приблизительно 97%, по меньшей мере приблизительно 98% или по меньшей мере приблизительно 99% идентичностью с последовательностью активной части или полноразмерному референсному полинуклеотиду или полипептиду. Полипептид может содержать аминокислотную последовательность, которая не встречается в природе. Такие вариации обязательно обладают менее чем 100% идентичностью или сходством последовательности с исходным полипептидом. В одном из вариантов реализации вариант будет иметь аминокислотную последовательность, обладающую от приблизительно 60% до менее чем 100% идентичностью или сходством аминокислотной последовательности исходного полипептида. В еще одном варианте реализации вариант будет иметь аминокислотную последовательность, обладающую от приблизительно 75% до менее чем 100%, от приблизительно 80% до менее чем 100%, от приблизительно 85% до менее чем 100%, от приблизительно 90% до менее чем 100%, от приблизительно 95% до менее чем 100% идентичностью или сходством аминокислотной последовательности исходного полипептида.
[0095] В контексте настоящего документа термин «идентичность последовательности» относится к взаимосвязи между двумя или более полинуклеотидными последовательностями или между двумя или более полипептидными последовательностями. Когда положение в одной последовательности занято тем же основанием нуклеиновой кислоты или аминокислотным остатком, что и в соответствующем положении последовательности сравнительной последовательности, говорят, что последовательности «идентичны» в этом положении. Процент идентичности последовательностей рассчитывают путем определения количества положений, в которых идентичное основание нуклеиновой кислоты или аминокислотный остаток встречается в обеих последовательностях, с получением количества идентичных положений. Количество идентичных положений затем делят на общее количество положений в окне сравнения и умножают на 100 с получением процента идентичности последовательностей. Процент идентичности последовательностей определяют путем сравнения двух оптимально выровненных последовательностей в окне сравнения. Окно сравнения для полинуклеотидных последовательностей может иметь длину, например, по меньшей мере 20, 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 300, 400, 500, 600, 700, 800, 900 или 1000 или более нуклеиновых кислот. Окно сравнения для полипептидных последовательностей может иметь длину, например, по меньшей мере 20, 30, 40, 50, 60, 70, 80, 90, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 300 или более аминокислот. Чтобы оптимально выровнять последовательности для сравнения, часть полинуклеотидной или полипептидной последовательности в окне сравнения может содержать добавления или делеции, называемые гэпами, в то время как референсная последовательность не изменяется. Оптимальное выравнивание представляет собой такое выравнивание, которое, даже с гэпами, обеспечивает максимально возможное количество «идентичных» положений между референсной и сравнительной последовательностями. Процент «идентичности последовательностей» между двумя последовательностями может быть определен с использованием версии программы «BLAST 2 Sequences», доступной от Национального центра биотехнологической информации на 1 сентября 2004 г., и которая включает программы BLASTN (для сравнения нуклеотидных последовательностей) и BLASTP (для сравнения полипептидных последовательностей), основанные на алгоритме Karlin и Altschul (Proc. Natl. Acad. Sci. USA 90(12):5873-5877, 1993). При использовании «BLAST 2 Sequences» параметры, которые были параметрами по умолчанию на 1 сентября 2004 г., могут быть использованы для длины сегмента (3), штрафа за внесение гэпа (11), штрафа за продление гэпа (1), выброса гэпа (50), ожидаемого значения (10) и любого другого необходимого параметра, включая, не ограничиваясь перечисленным, опцию матрицы. Считается, что две нуклеотидные или аминокислотные последовательности имеют «по существу схожую идентичность последовательности» или являются «по существу идентичными», если эти две последовательности обладают по меньшей мере 80%, по меньшей мере 85%, по меньшей мере 90%, по меньшей мере 95%, по меньшей мере 96 %, по меньшей мере 97%, по меньшей мере 98% или по меньшей мере 99% идентичностью относительно друг друга.
[0096] В контексте настоящего документа, если не указано иное, положение аминокислотного остатка в вариабельной области молекулы иммуноглобулина пронумеровано в соответствии с критериями IMGT (Brochet et al., Nucl. Acids Res. (2008) 36, W503-508), или в соответствии с номенклатурой EU (Ward et al., 1995 Therap. Immunol. 2:77-94), а положение аминокислотного остатка в константной области молекулы иммуноглобулина пронумеровано в соответствии с номенклатурой EU (Ward et al., 1995 Therap. Immunol. 2:77-94). Система нумерации Kabat (Kabat, Sequences of Proteins of Immunological Interest, 5th ed. Bethesda, MD: Public Health Service, National Institutes of Health (1991)) представляет собой альтернативную систему, используемую для обозначения положения аминокислотного остатка в вариабельной области молекулы иммуноглобулина, и в некоторых случаях она используется для обозначения положения аминокислотного остатка в вариабельной области молекулы иммуноглобулина в настоящем документе.
[0097] В контексте настоящего документа термин «димер» относится к биологическому объекту, который состоит из двух субъединиц, связанных друг с другом посредством одного или более видов внутримолекулярных сил, включая ковалентные связи (например, дисульфидные связи) и другие взаимодействия (например, электростатические взаимодействия, солевые мостики, водородные связи и гидрофобные взаимодействия), и является стабильным в соответствующих условиях (например, в физиологических условиях, в водном растворе, подходящем для экспрессии, очистки и/или хранения рекомбинантных белков, или в условиях для неденатурирующего и/или невосстанавливающего электрофореза). В контексте настоящего документа термины «гетеродимер» или «гетеродимерный белок» относятся к димеру, образованному из двух разных полипептидов. Гетеродимер может содержать молекулу к 5Т4 x 4-1ВВ, как описано в настоящем документе. Гетеродимер не включает антитело, образованное из четырех полипептидов (то есть, двух легких цепей и двух тяжелых цепей). Термины «гомодимер» или «гомодимерный белок» в контексте настоящего документа относятся к димеру, образованному из двух идентичных полипептидов.
[0098] «Fc-область» или «Fc-домен» относится к полипептидной последовательности, соответствующей или полученной из части исходного антитела, способной связываться с Fc-рецепторами на клетках и/или компонентом комплемента C1q, тем самым опосредуя эффекторную функцию антитела. Fc означает «кристаллизующийся фрагмент» - фрагмент антитела, который легко образует кристалл белка. Отдельные фрагменты белка, которые были первоначально описаны посредством протеолитического расщепления, могут определять общую структуру белка иммуноглобулина. Как изначально было определено в литературе, Fc-область представляет собой гомодимерный белок, содержащий два полипептида, связанные дисульфидными связями, каждый из которых содержит шарнирную область, домен CH2 и домен CH3. Однако в последнее время этот термин применяют к одноцепочечному мономерному компоненту, состоящему из СН3, СН2 и по меньшей мере части шарнира, достаточной для образования дисульфидно-связанного димера со второй такой цепью. По этой причине в зависимости от контекста использование терминов «Fc-область» или «Fc-домен» в настоящем документе относится либо к димерной форме, либо к отдельным мономерам, связывающимся с образованием димерного белка. Обзор структуры и функции иммуноглобулинов см. в Putnam, The Plasma Proteins, Vol. V (Academic Press, Inc., 1987), стр. 49-140; и Padlan, Mol. Immunol. 31:169-217, 1994. В контексте настоящего документа термин Fc включает варианты встречающихся в природе последовательностей.
[0099] «Константная область иммуноглобулина» или «константная область» представляет собой термин, определенный в настоящем документе для обозначения пептидной или полипептидной последовательности, полученной из части или всей последовательности одного или более константных доменов иммуноглобулина, или соответствующей им. В отдельных вариантах реализации константная область содержит домены CH2 и CH3 IgG, например, домены CH2 и CH3 IgG1. В отдельных вариантах реализации константная область не содержит домен CH1. В отдельных вариантах реализации константные домены, составляющие константную область, являются областью иммуноглобулина человека. В некоторых вариантах реализации (например, в отдельных вариациях 41BB-связывающего полипептида, 5T4-связывающего полипептида или содержащих их полиспецифичных полипептидов) константная область слитого белка согласно данному изобретению лишена эффекторных функций или имеет минимальные эффекторные функции, сохраняя при этом способность связывать некоторые Fc-рецепторы, такие как неонатальный Fc-рецептор (FcRn), и сохраняя относительно длительный период полувыведения in vivo. Например, константная область слитого белка согласно данному изобретению не приводит к индукции антителозависимой клеточно-опосредованной цитотоксичности (ADCC), антителозависимого клеточно-опосредованного фагоцитоза (ADCP), активации комплемента и/или комплемент-зависимой цитотоксичности (CDC), или существенно снижает ее. В других вариациях слитый белок согласно данному изобретению содержит константные домены, сохранившие одну или более эффекторных функций, например, одну или обе из ADCC и CDC. В отдельных вариантах реализации связывающий домен согласно данному изобретению слит с константной областью человеческого IgG1, где константная область IgG1 имеет одну или более мутаций из следующих аминокислот: лейцин в положении 234 (L234), лейцин в положении 235 (L235), глицин в положении 237 (G237), глутамат в положении 318 (E318), лизин в положении 320 (K320), лизин в положении 322 (K322) или любая их комбинация (нумерация согласно EU). Например, любая одна или более из этих аминокислот могут быть заменены на аланин. В дополнительном варианте реализации в Fc-домене IgG1 каждый из L234, L235, G237, E318, K320 и K322 (согласно нумерации EU) мутирован в аланин (то есть, L234A, L235A, G237A, E318A, K320A и K322A, соответственно) и, необязательно, он также содержит мутацию N297A (т. е., по существу, устраняющую гликозилирование домена CH2).
[00100] Термины «вариабельная область легкой цепи» (также называемая «вариабельным доменом легкой цепи» или «VL») и «вариабельная область тяжелой цепи» (также называемая «вариабельным доменом тяжелой цепи» или «VH») относятся к вариабельной связывающей области из легкой и тяжелой цепи антитела, соответственно. Вариабельные связывающие области состоят из обособленных, четко определенных субобластей, известных как «определяющие комплементарность области» (CDR) и «каркасные области» (FR). В одном из вариантов реализации FR являются гуманизированными. Термин «CL» относится к «константной области легкой цепи иммуноглобулина» или «константной области легкой цепи», то есть, константной области из легкой цепи антитела. Термин «СН» относится к «константной области тяжелой цепи иммуноглобулина» или «константной области тяжелой цепи», которая дополнительно подразделяется, в зависимости от изотипа антитела, на домены СН1, СН2 и СН3 (IgA, IgD, IgG), или CH1, CH2, CH3 и CH4 (IgE, IgM). «Fab» (антигенсвязывающий фрагмент) представляет собой часть антитела, связывающуюся с антигенами, и включает вариабельную область и домен CH1 тяжелой цепи, связанный с легкой цепью посредством межцепочечной дисульфидной связи.
[00101] В контексте настоящего документа термин «линкер» в общем случае относится к короткой полипептидной последовательности, соединяющей два субдомена полипептида. Неограничивающие примеры линкеров включают гибкие линкеры, содержащие глицин-сериновые повторы, и линкеры, полученные из (а) междоменной области трансмембранного белка (например, трансмембранного белка I типа); (b) стеблевой области лектина C-типа II группы; или (с) шарнира иммуноглобулина. В некоторых вариантах реализации линкер обеспечивает функцию спейсера, совместимую с взаимодействием двух субсвязывающих доменов, так что получаемый полипептид сохраняет специфичную аффинность связывания с той же молекулой-мишенью, что и антитело, которое содержит те же вариабельные области легкой и тяжелой цепи. В отдельных вариантах реализации линкер содержит от пяти до приблизительно 35 аминокислот, например, от приблизительно 15 до приблизительно 25 аминокислот. В контексте настоящего документа выражение «линкер между CH3 и CH1 или CL» относится к одному или более аминокислотным остаткам (например, приблизительно 2-12, приблизительно 2-10, приблизительно 4-10, приблизительно 5-10, приблизительно 6-10, приблизительно 7-10, приблизительно 8-10, приблизительно 9-10, приблизительно 8-12, приблизительно 9-12 или приблизительно 10-12) между С-концом домена СН3 (например, СН3 дикого типа или мутированного CH3) и N-концом домена CH1 или домена CL (например, Cκ).
[00102] В некоторых вариантах реализации, в зависимости от контекста, линкер может относиться к (1) полипептидной области между областями VH и VL в одноцепочечном Fv (scFv) или (2) полипептидной области между первым связывающим доменом и вторым связывающим доменом в полиспецифичном полипептиде, содержащем два связывающих домена. В последнем примере когда линкер соединяет два или более связывающих доменов, такой линкер называется в настоящем документе «линкером связывающих доменов». В некоторых вариантах реализации линкер связывающих доменов может непосредственно связывать или соединять два или более связывающих доменов, в результате чего образуется конструкция, содержащая следующую структуру: связывающий домен - линкер связывающих доменов - связывающий домен. В некоторых вариантах реализации полиспецифичные полипептиды, описанные в настоящем документе, содержат, в порядке от аминоконца к карбоксиконцу, (i) первый связывающий домен, (ii) линкер связывающих доменов и (iii) второй связывающий домен. В некоторых вариантах реализации полиспецифичный полипептид содержит, в порядке от аминоконца к карбоксиконцу, (i) второй связывающий домен, (ii) линкер связывающих доменов и (iii) первый связывающий домен. В некоторых вариантах реализации линкер связывающих доменов может связывать или соединять два или более связывающих доменов путем связывания по меньшей мере одного связывающего домена с полипептидом несвязывающего домена, таким как Fc-домен иммуноглобулина (то есть, полипептидом, содержащим структуру: шарнир Ig - константная область Ig). В таких вариантах реализации полученные конструкции могут содержать следующую структуру: связывающий домен - Fc-домен - линкер связывающих доменов - связывающий домен. В некоторых вариантах реализации полиспецифичные полипептиды, описанные в настоящем документе, содержат, в порядке от аминоконца к карбоксиконцу: (i) первый связывающий домен, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) второй связывающий домен. В некоторых вариантах реализации полиспецифичный полипептид содержит, в порядке от аминоконца к карбоксиконцу, (i) второй связывающий домен, (ii) линкер связывающих доменов, (iii) константную область иммуноглобулина, (iv) шарнирную область и (v) первый связывающий домен. Полипептидная область между константной областью иммуноглобулина и вторым связывающим доменом в полиспецифичном полипептиде, содержащем два связывающих домена (например, линкер связывающих доменов), также может называться «карбоксиконцевым линкером» или «аминоконцевым линкером», в зависимости от ориентации доменов в молекуле полиспецифичного полипептида. Неограничивающие примеры линкеров приведены в таблице 1.
[00103] В некоторых вариантах реализации «шарнир» или «шарнирная область» относится к полипептиду, полученному из шарнирной области иммуноглобулина и расположенному в описанном в настоящем документе полипептиде между связывающим доменом (например, 5T4-связывающим доменом или 4-1BB-связывающим доменом) и константной областью иммуноглобулина. «Шарнирная область иммуноглобулина дикого типа» относится к встречающимся в природе аминокислотным последовательностям верхнего и среднего шарниров, расположенным между доменами CH1 и CH2 (для IgG, IgA и IgD) и соединяющим их, или расположенным между доменами CH1 и CH3 (для IgE и IgM) и соединяющим их, находящимся в тяжелой цепи антитела. В отдельных вариантах реализации последовательность шарнирной области иммуноглобулина дикого типа является человеческой и может содержать шарнирную область человеческого IgG (например, шарнирную область IgG1, IgG2, IgG3 или IgG4).
[00104] «Измененная шарнирная область иммуноглобулина» или «вариантная шарнирная область иммуноглобулина» относится к полипептиду шарнирной области с одной или более мутациями, заменами, инсерциями или делециями по сравнению с соответствующей исходной шарнирной областью иммуноглобулина дикого типа. В отдельных вариантах реализации измененная шарнирная область иммуноглобулина по меньшей мере на 70% гомологична шарнирной области иммуноглобулина дикого типа (например, гомологична по меньшей мере на 70%, 75%, 80%, 85%, 90%, 95%, 97%, 98% или 99%). В отдельных вариантах реализации измененная шарнирная область иммуноглобулина представляет собой фрагмент шарнирной области иммуноглобулина дикого типа, имеющий длину от приблизительно 5 аминокислот (например, приблизительно 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20 или более аминокислот) до приблизительно 120 аминокислот (например, имеющий длину от приблизительно 10 до приблизительно 40 аминокислот, или от приблизительно 15 до приблизительно 30 аминокислот, или от приблизительно 15 до приблизительно 20 аминокислот, или от приблизительно 20 до приблизительно 25 аминокислот). Как правило, измененная шарнирная область иммуноглобулина, которая является фрагментом шарнирной области иммуноглобулина дикого типа, содержит коровую шарнирную область IgG (например, полипептид, содержащий последовательность C-X-X-C, где X представляет собой любую аминокислоту), как раскрыто в публикациях патентных заявок США №№ 2013/0129723 и 2013/0095097. Неограничивающие примеры шарниров приведены в таблице 1.
[00105] В контексте настоящего документа термин «гуманизированный» относится к процессу придания антителам или иммуноглобулинсвязывающим белкам и полипептидам, полученным от вида, отличного от человека (например, мыши или крысы), меньшей иммуногенности для человека, при сохранении ими при этом антигенсвязывающих свойств исходного антитела, с помощью методов генной инженерии. В некоторых вариантах реализации связывающий домен(ы) антитела или иммуноглобулинсвязывающих белков и полипептидов (например, вариабельные области легкой и тяжелой цепи, Fab, scFv) являются гуманизированными. Не являющиеся человеческими связывающие домены могут быть гуманизированы с использованием методов, известных как привитие CDR (Jones et al., Nature 321:522 (1986)), и их вариантов, включая «реконструирование» (Verhoeyen, et al., 1988 Science 239:1534-1536; Riechmann, et al., 1988 Nature 332:323-337; Tempest, et al., Bio/Technol 1991 9:266-271), «гиперхимеризацию» (Queen, et al., 1989 Proc Natl Acad Sci USA 86:10029-10033; Co, et al., 1991 Proc Natl Acad Sci USA 88:2869-2873; Co, et al., 1992 J Immunol 148:1149-1154) и «венирование» (Mark, et al., "Derivation of therapeutically active humanized and veneered anti-CD18 antibodies.” B: Metcalf BW, Dalton BJ, eds. Cellular adhesion: molecular definition to therapeutic potential. New York: Plenum Press, 1994: 291-312). Другие области антитела или иммуноглобулинсвязывающих белков и полипептидов, такие как шарнирная область и домены константной области, если они получены из отличного от человеческого источника, также могут быть гуманизированы. Сведения о гуманизированных антителах в данной области техники применимы к полипептидам согласно изобретению, даже если эти полипептиды не являются антителами.
[00106] В контексте настоящего документа термин «нуждающийся пациент» относится к пациенту, подверженному риску развития заболевания, расстройства или состояния, которое поддается лечению или уменьшению интенсивности с помощью 5T4-связывающего белка или полиспецифичного полипептида, или содержащей его композиции, предложенных в настоящем документе, или страдающему от такого заболевания, расстройства или состояния.
[00107] В контексте настоящего документа термин «фармацевтически приемлемый» относится к химическим соединениям и композициям, которые обычно не вызывают аллергические или другие серьезные нежелательные реакции при введении способами, хорошо известными в данной области техники. Химические соединения и композиции, одобренные надзорным органом Федерального правительства или правительства штата, или приведенные в Фармакопее США или другой общепризнанной фармакопее для применения у животных и, более конкретно, у человека, считаются «фармацевтически приемлемыми».
[00108] В контексте настоящего документа термин «промотор» относится к области ДНК, участвующей в связывании РНК-полимеразы для инициации транскрипции.
[00109] В контексте настоящего документа термины «нуклеиновая кислота», «молекула нуклеиновой кислоты» или «полинуклеотид» относятся к дезоксирибонуклеотидам или рибонуклеотидам и их полимерам в одно- или двухцепочечной форме. При отсутствии специальных ограничений данные термины охватывают нуклеиновые кислоты, содержащие аналоги природных нуклеотидов, обладающие свойствами связывания, аналогичными свойствам референсной нуклеиновой кислоты, и метаболизирующиеся аналогично встречающимся в природе нуклеотидам. Если не указано иное, конкретная нуклеотидная последовательность помимо явным образом указанной последовательности также подразумевает ее консервативно модифицированные варианты (например, замены из-за вырожденности кодонов) и комплементарные последовательности. В частности, замены из-за вырожденности кодонов можно получить, генерируя последовательности, в которых третье положение одного или более из выбранных (или всех) кодонов заменено остатками смешанных оснований и/или дезоксиинозина (Batzer et al. (1991) Nucleic Acid Res. 19:5081; Ohtsuka et al. (1985) J. Biol. Chem. 260:2605-2608; Cassol et al. (1992); Rossolini et al. (1994) Mol. Cell. Probes 8:91-98). Термин «нуклеиновая кислота» используется взаимозаменяемо с геном, кДНК (комплементарной ДНК) и мРНК (матричной РНК), кодируемой геном. В контексте настоящего документа подразумевается, что термины «нуклеиновая кислота», «молекула нуклеиновой кислоты» или «полинуклеотид» включают молекулы ДНК (например, кДНК или геномной ДНК), молекулы РНК (например, мРНК), аналоги ДНК или РНК, полученные с использованием нуклеотидных аналогов, и их производные, фрагменты и гомологи.
[00110] Термин «экспрессия» относится к биосинтезу продукта, кодируемого нуклеиновой кислотой. Например, в случае сегмента нуклеиновой кислоты, кодирующего представляющий интерес полипептид, экспрессия включает транскрипцию сегмента нуклеиновой кислоты в мРНК и трансляцию мРНК в один или более полипептидов.
[00111] Термины «единица экспрессии» и «кассета экспрессии» используются в настоящем документе взаимозаменяемо и обозначают сегмент нуклеиновой кислоты, кодирующий представляющий интерес полипептид и способный обеспечивать экспрессию сегмента нуклеиновой кислоты в клетке-хозяине. Единица экспрессии обычно содержит промотор транскрипции, открытую рамку считывания, кодирующую представляющий интерес полипептид, и терминатор транскрипции, все из которых находятся в работоспособной конфигурации. Помимо промотора и терминатора транскрипции единица экспрессии может дополнительно включать другие сегменты нуклеиновой кислоты, такие как, например, энхансер или сигнал полиаденилирования.
[00112] В контексте настоящего документа термин «вектор экспрессии» относится к молекуле нуклеиновой кислоты, линейной или циклической, содержащей одну или более единиц экспрессии. Помимо одной или более единиц экспрессии вектор экспрессии может также включать дополнительные сегменты нуклеиновой кислоты, такие как, например, одна или более точек начала репликации или один или более селектируемых маркеров. Векторы экспрессии обычно получают из плазмидной или вирусной ДНК, или они могут содержать элементы обеих.
[00113] В контексте настоящего документа термин «полипептид», «полипептидная цепь» или «белок» относится к одному линейному и непрерывному расположению ковалентно связанных аминокислот. Полипептиды могут образовывать одну или более внутрицепочечных дисульфидных связей. Что касается полипептидов, как описано в настоящем документе, ссылка на модификации или изменения аминокислотных остатков, соответствующих тем, которые указаны в SEQ ID NO:, включает посттрансляционные модификации таких остатков. Термины «полипептид» и «белок» также охватывают варианты реализации, где две полипептидные цепи связаны друг с другом нелинейным образом, например, посредством межцепочечной дисульфидной связи. Например, нативная молекула иммуноглобулина состоит из двух полипептидов тяжелой цепи и двух полипептидов легкой цепи. Каждый из полипептидов тяжелой цепи связывается с полипептидом легкой цепи благодаря межцепочечным дисульфидным связям между полипептидами тяжелой и легкой цепи с образованием двух гетеродимерных белков или полипептидов (то есть, белка, состоящего из двух гетерологичных полипептидных цепей). Затем два гетеродимерных белка связываются посредством дополнительных межцепочечных дисульфидных связей между полипептидами тяжелой цепи с образованием белка или полипептида иммуноглобулина. В настоящем документе белок или полипептид может представлять собой антитело или антигенсвязывающий фрагмент антитела. В некоторых вариантах реализации белок может представлять собой белок scFv-Fc-scFv, димер scFv-scFv или диатело.
[00114] Как будет ясно специалисту в данной области техники, белки и полипептиды определены в настоящем документе посредством аминокислотных последовательностей отдельных полипептидных цепей, которые обозначены ссылкой на SEQ ID NO: во всем настоящем описании. Например, в некоторых вариантах реализации белок или полипептид scFv-Fc-scFv, описанный в настоящем документе, состоит из двух полипептидных цепей scFv-Fc-scFv, связанных межцепочечными связями (например, межцепочечными дисульфидными связями) с образованием димерного белка scFv-Fc-scFv (например, гомодимерного или гетеродимерного белка scFv-Fc-scFv) (см., например, ФИГ. 10). В таких вариантах реализации белок scFv-Fc-scFv определен аминокислотными последовательностями отдельных полипептидных цепей scFv-Fc-scFv. Полипептиды и белки также могут содержать непептидные компоненты, такие как углеводные группы. Углеводы и другие непептидные заместители могут быть добавлены к белку или полипептиду клеткой, в которой продуцируется белок, и их состав будет варьироваться в зависимости от типа клетки. Белки и полипептиды определены в настоящем документе посредством структуры их аминокислотного остова; заместители, такие как углеводные группы, обычно не указаны, но, тем не менее, могут присутствовать.
[00115] Термины «аминоконцевой» и «карбоксиконцевой» используются в настоящем документе для обозначения положений в полипептидах. Там, где позволяет контекст, эти термины используются со ссылкой на конкретную последовательность или часть полипептида для обозначения близости или относительного положения. Например, отдельно взятая последовательность, являющаяся карбоксиконцевой относительно референсной последовательности в полипептиде, расположена вблизи карбоксиконца указанной референсной последовательности, но необязательно находится на карбоксиконце всей последовательности полипептида.
[00116] В контексте настоящего документа термин «трансформация», «трансфекция» и «трансдукция» относится к переносу нуклеиновой кислоты (то есть, нуклеотидного полимера) в клетку. В контексте настоящего документа термин «генетическая трансформация» относится к переносу и включению в клетку ДНК, в частности, рекомбинантной ДНК. Перенесенная нуклеиновая кислота может быть введена в клетку посредством вектора экспрессии.
[00117] «Антителозависимая клеточно-опосредованная цитотоксичность» и «ADCC» в контексте настоящего документа относятся к клеточно-опосредованному процессу, в котором неспецифичные цитотоксические клетки, экспрессирующие FcγR (например, моноцитарные клетки, такие как естественные киллеры (NK) и макрофаги), распознают связанное антитело (или другой белок, способный связывать FcγR) на клетке-мишени и затем вызывают лизис указанной клетки-мишени. В принципе, любая эффекторная клетка с активирующим FcγR может быть стимулирована для опосредования ADCC. Первичными клетками для опосредования ADCC являются NK-клетки, экспрессирующие только FcγRIII, тогда как моноциты, в зависимости от их состояния активации, локализации или дифференцировки, могут экспрессировать FcγRI, FcγRII и FcγRIII. Обзор экспрессии FcγR на гемопоэтических клетках см., например, в Ravetch et al., 1991, Annu. Rev. Immunol., 9:457-92.
[00118] Термин «обладающий активностью ADCC», используемый в настоящем документе применительно к полипептиду или белку, означает, что указанный полипептид или белок, например, содержащий Fc-домен (например, шарнирную область иммуноглобулина и константную область иммуноглобулина, имеющую домены СН2 и СН3), такой как полученный из IgG (например, IgG1), способен опосредовать антителозависимую клеточно-опосредованную цитотоксичность (ADCC) посредством связывания цитолитического Fc-рецептора (например, FcγRIII) на цитолитической иммунной эффекторной клетке, экспрессирующей Fc-рецептор (например, NK-клетке). В некоторых вариантах реализации полиспецифичный полипептид или белок (например, молекула к 5T4 x 4-1BB, как описано в настоящем документе), содержащий Fc-домен, может не иметь эффекторной функции (например, иметь нулевую активность ADCC) в результате мутаций в домене СН2 и/или CH3.
[00119] «Комплементзависимая цитотоксичность» и «CDC» в контексте настоящего документа относятся к процессу, в котором компоненты в нормальной сыворотке («комплемент») вместе с антителом или другим связывающим комплемент C1q белком, связанным с антигеном-мишенью, осуществляют лизис клетки-мишени, экспрессирующей антиген-мишень. Комплемент состоит из группы сывороточных белков, которые действуют согласованно и в упорядоченной последовательности, чтобы оказывать свое действие.
[00120] Термины «классический путь комплемента» и «классическая система комплемента» в контексте настоящего документа являются синонимами и относятся к конкретному пути активации комплемента. Для инициации классического пути необходимы комплексы антиген-антитело, и он включает упорядоченную активацию девяти основных белков компонента, обозначаемых как С1-С9. На нескольких стадиях процесса активации продукт представляет собой фермент, катализирующий последующую стадию. Этот каскад обеспечивает усиление и активацию большого количества комплемента с помощью относительно небольшого исходного сигнала.
[00121] Термин «обладающий активностью CDC», используемый в настоящем документе применительно к полипептиду или белку, означает, что указанный полипептид или белок, например, содержащий Fc-домен (например, шарнирную область иммуноглобулина и константную область иммуноглобулина, имеющую домены СН2 и СН3), такой как полученный из IgG (например, IgG1), способен опосредовать комплементзависимую цитотоксичность (CDC) посредством связывания белка комплемента C1q и активации классической системы комплемента. В некоторых вариантах реализации полиспецифичный полипептид или белок (например, молекула к 5T4 x 4-1BB, как описано в настоящем документе) может не иметь эффекторной функции (например, иметь нулевую активность CDC) в результате одной или более мутаций в доменах СН2 и/или CH3.
[00122] «Усиленная активация эффекторных клеток» в контексте настоящего документа относится к повышению, пролонгированию и/или усилению ответа эффекторных клеток полипептидами или белками, описанными в настоящем документе. В некоторых вариантах реализации усиленная активация эффекторных клеток относится к повышению цитотоксической активности эффекторной клетки. В некоторых вариантах реализации усиленная активация эффекторных клеток относится к повышению выработки цитокинов, пролиферации клеток или изменению экспрессии молекулы на клеточной поверхности, так что усиливается способность эффекторной клетки осуществлять лизис клетки-мишени. В отдельных вариантах реализации полипептиды и белки, описанные в настоящем документе, усиливают активацию эффекторных клеток путем модуляции передачи сигналов по пути Wnt/β-катенина.
[00123] В контексте настоящего документа термин «эффекторная клетка» относится к клетке иммунной системы, способной осуществлять лизис или уничтожать клетку-мишень, такую как опухолевая клетка. В настоящем документе эффекторная клетка может относиться к лимфоциту, такому как Т-клетка, естественный киллер (NK) или NKT-клетка, моноциту, макрофагу, дендритной клетке или гранулоциту. В частных вариантах реализации термин «эффекторная клетка» относится к T-клетке, NK-клетке или NKT-клетке.
[00124] В контексте настоящего документа термины «лечение» или «уменьшение интенсивности» относятся либо к терапевтическому лечению, либо к профилактическому/превентивному лечению. Лечение является терапевтическим, если по меньшей мере один симптом заболевания у индивидуума, получающего лечение, улучшается, или лечение может отсрочить ухудшение прогрессирующего заболевания у индивидуума или предотвратить возникновение дополнительных сопутствующих заболеваний.
[00125] В контексте настоящего документа термин «терапевтически эффективное количество (или доза)» или «эффективное количество (или доза)» описанного в настоящем документе полипептида или белка, или содержащей его композиции, относится к такому количеству соединения, которое достаточно для уменьшения интенсивности одного или более симптомов заболевания, подлежащего лечению, статистически значимым образом, или статистически значимого улучшения функции органа. Применительно к отдельному активному ингредиенту, вводимому по отдельности, терапевтически эффективная доза относится только к одному этому ингредиенту. Применительно к комбинации терапевтически эффективная доза относится к объединенным количествам активных ингредиентов, которые приводят к терапевтическому эффекту, независимо от того, вводятся ли они последовательно или одновременно (в составе одного и того же препарата или одновременно в отдельных препаратах).
[00126] В настоящем документе термин «статистическая значимость» относится к вероятности получения результата теста, который происходит случайно. Например, результат наблюдения или теста считается статистически значимым, если вероятность того, что он произойдет по чистой случайности (p), будет меньше, чем заданный статистический порог (α), или p < α. Статистический порог для конкретного теста может быть установлен в соответствии с характеристиками данных и правилами, известными в данной области техники. Например, α обычно устанавливается равным 5% (0,05), так что для того, чтобы отдельно взятый результат считался статистически значимым, должно выполняться р < 0,05.
[00127] В контексте настоящего документа «5T4-связывающий домен» относится к домену полипептида, описанного в настоящем документе, который специфично связывается с онкофетальным трофобластическим гликопротеином (5T4) (например, 5T4 человека), также известным как TPBG и Wnt-активированный ингибирующий фактор 1 (WAIF1). 5T4 представляет собой онкофетальный гликопротеин размером 72 кДа, который представляет собой в высокой степени N-гликозилированные белки с несколькими богатыми лейцином повторами, связанными белок-белковыми взаимодействиями. Термин «5T4» может относиться к любой изоформе 5T4. Иллюстративные нуклеотидные последовательности человеческого 5Т4 представлены в приведенной ниже таблице 1, а также в SEQ ID NO: 163 и 167, а иллюстративные аминокислотные последовательности человеческого 5Т4 представлены в SEQ ID NO: 164 и 168.
[00128] В контексте настоящего документа «4-1BB-связывающий домен» относится к связывающему домену белка или полипептида, описанного в настоящем документе, который способен специфично связываться с 4-1BB человека (также известным как CD137). Термин «4-1BB» может относиться к любой изоформе 4-1BB. Иллюстративные нуклеотидные последовательности человеческого 4-1BB представлены в приведенной ниже таблице 1, а также в SEQ ID NO: 161 и 165, а иллюстративные аминокислотные последовательности человеческого 4-1BB представлены в SEQ ID NO: 162 и 166.
Таблица 1. Иллюстративные последовательности ДНК и аминокислотные последовательности 4-1BB и 5T4
*ECD = внеклеточный домен
[00129] В контексте настоящего документа термин «полиспецифичный полипептид» относится к полипептиду, содержащему два или более связывающих домена, каждый из которых способен специфично связываться с антигеном-мишенью. Например, полипептиды, описанные в настоящем документе, могут содержать 2, 3, 4 или более связывающих доменов и могут быть способны связывать 2, 3, 4 или более антигенов-мишеней. В некоторых вариантах реализации полиспецифичный полипептид представляет собой биспецифичный полипептид. В настоящем документе «биспецифичный полипептид» содержит два связывающих домена и способен связываться с двумя различными антигенами-мишенями. В некоторых вариантах реализации биспецифичные полипептиды, описанные в настоящем документе, содержат первый связывающий домен, специфично связывающийся с антигеном клеточной поверхности, экспрессируемым на клетке-мишени, таким как 5T4. В некоторых вариантах реализации биспецифичные полипептиды, описанные в настоящем документе, содержат связывающий домен, специфично связывающийся с антигеном клеточной поверхности, экспрессируемым на эффекторной клетке, таким как 4-1BB. В частных вариантах реализации полиспецифичный полипептид представляет собой гомодимер биспецифичного полипептида ADAPTIR™ в формате scFv-Fc-scFv.
[00130] Полиспецифичные полипептиды, в которых используются скаффолды, раскрыты, например, в публикациях заявок PCT №№ WO 2007/146968; WO 2010/040105; WO 2010/003108; WO 2016/094873; WO 2017/053469; публикации патентной заявки США № 2006/0051844; и патентах США №№ 7,166,707; и 8,409,577, каждый из которых включен в настоящий документ посредством ссылки в полном объеме. В отдельных вариантах реализации полиспецифичные полипептиды, описанные в настоящем документе, представляют собой биспецифичные полипептиды и могут содержать структуру scFv-Fc-scFv, также называемую в настоящем документе полипептидом ADAPTIR™, или формат 1, иллюстративный вариант реализации которого показан на ФИГ. 10. Структура полипептида, содержащего структуру формата 1, содержит, в направлении от N-конца к C-концу: первый связывающий scFv-домен - шарнирную область иммуноглобулина (Ig) - константную область Ig - второй связывающий scFv-домен (ФИГ. 10). В отдельных вариантах реализации полиспецифичные полипептиды, описанные в настоящем документе, могут содержать структуру IgG-scFv (также называемую в настоящем документе форматом Моррисона или форматом 2). Структура полипептида, содержащего структуру формата 2, содержит, в направлении от N-конца к C-концу: связывающий scFv-домен - константную область Ig - шарнирную область Ig - вариабельную область Ig. Формат 2 по существу представляет собой интактную молекулу Ig, содержащую С-концевой scFv-домен.
Связывающие полипептиды и белки
[00131] В некоторых вариантах реализации настоящее изобретение относится к полипептидам, способным специфично связываться с 5Т4 (например, 5Т4-связывающим полипептидам), а также полиспецифичным полипептидам и белкам, содержащим эти связывающие домены. Такие варианты реализации могут называться 5T4-связывающими полипептидами. В частных вариантах реализации настоящее изобретение относится к полиспецифичным связывающим белкам, содержащим 5T4-связывающий домен и второй связывающий домен, способный связываться с молекулой клеточной поверхности на эффекторной клетке. В частных вариантах реализации настоящее изобретение относится к биспецифичным связывающим белкам, содержащим 5T4-связывающий домен и второй связывающий домен, способный связываться с 4-1BB (например, 4-1BB-связывающий домен). В некоторых вариантах реализации 5T4-связывающие полипептиды содержат следующую структуру, в направлении от N-конца к C-концу: 5T4-связывающий домен - Fc-домен.
[00132] В некоторых вариантах реализации настоящее изобретение относится к полипептидам, способным специфично связываться с 4-1BB, а также полиспецифичным полипептидам и белкам, содержащим эти связывающие домены. Такие варианты реализации могут называться 4-1BB-связывающими полипептидами. В частных вариантах реализации настоящее изобретение относится к полиспецифичным полипептидам, содержащим 4-1BB-связывающий домен и второй связывающий домен, способный связываться с молекулой клеточной поверхности на клетке-мишени. В частных вариантах реализации настоящее изобретение относится к биспецифичным полипептидам, содержащим 4-1BB-связывающий домен и второй связывающий домен, способный связываться с 5T4 (например, 5T4-связывающий домен). В некоторых вариантах реализации 4-1BB-связывающие полипептиды содержат следующую структуру, в направлении от N-конца к C-концу: 4-1BB-связывающий домен - Fc-домен.
[00133] В некоторых вариантах реализации полипептиды, описанные в настоящем документе, могут дополнительно содержать константные области иммуноглобулина, линкерные пептиды, шарнирные области, домены димеризации/гетеродимеризации иммуноглобулина, соединительные аминокислоты, метки и т. д. Эти компоненты раскрытых полипептидов и белков более подробно описаны ниже.
[00134] Полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), содержат один или более связывающих доменов, способных специфично связывать антиген-мишень. Связывающие домены, описанные в настоящем документе, могут иметь форму антитела или слитого белка любого из множества различных форматов (например, слитый белок может иметь форму биспецифичной или полиспецифичной молекулы). Неограничивающие примеры биспецифичных молекул включают молекулу scFv-Fc-scFv (например, биспецифичный белок, содержащий структуру формата 1), молекулу scFv-Ig (например, биспецифичный белок, содержащий структуру формата 2) и молекулу scFv-scFv. В некоторых вариантах реализации биспецифичные молекулы, описанные в настоящем документе, содержат или состоят из первого связывающего scFv-домена, связанного со вторым связывающим scFv-доменом, и не включают другие последовательности, такие как константная область иммуноглобулина. В других вариантах реализации биспецифичный белок, описанный в настоящем документе, представляет собой диатела.
[00135] В некоторых вариантах реализации полипептиды и белки, описанные в настоящем документе, конъюгированы с лекарственным средством или токсическим фрагментом.
[00136] В некоторых вариантах реализации полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) могут содержать последовательности аминокислот и/или нуклеиновых кислот, представленные в таблицах 2-10. В отдельных вариантах реализации связывающие домены полипептидов, описанных в настоящем документе, содержат (i) вариабельную область легкой цепи (VL) иммуноглобулина, содержащую CDR LCDR1, LCDR2 и LCDR3, и (ii) вариабельную область тяжелой цепи (VH) иммуноглобулина, содержащую CDR HCDR1, HCDR2 и HCDR3. В некоторых вариантах реализации аминокислотные последовательности, приведенные для полипептидных конструкций, не включают лидерные последовательности человеческого иммуноглобулина. Приведенные последовательности CDR и положения аминокислотных замен определены в соответствии с критериями IMGT (Brochet et al, Nucl. Acids Res. (2008) 36, W503-508). В некоторых случаях последовательности, приведенные в описании, в том числе в таблицах 2-10, содержат аминокислотные замены относительно исходной последовательности (например, антитела к 4-1BB или его связывающего фрагмента, такого как клон 1618/1619, раскрытый в публикации заявки PCT № WO 2016/185016, или антитела к 5T4 или его связывающего фрагмента, такого как раскрытый в публикации заявки PCT № WO 2016/185016). В некоторых вариантах реализации исходная последовательность связывающего домена к 5T4 содержит аминокислотную последовательность, представленную в SEQ ID NO: 38 и 68. В некоторых вариантах реализации исходная последовательность связывающего домена к 4-1BB содержит аминокислотную последовательность, представленную в SEQ ID NO: 28 и 16. Иллюстративные исходные последовательности связывающих доменов к 4-1BB и к 5T4, описанных в настоящем документе, представлены ниже в таблице 2. Подчеркнутый текст обозначает последовательности CDR.
Таблица 2. Иллюстративные исходные последовательности связывающих доменов
[00137] В отдельных вариантах реализации область VL и/или VH связывающего домена согласно настоящему изобретению получена из VL и/ или VH исходной области VL и/ или VH (например, 1618/1619, как описано в публикации заявки PCT № WO 2016/185016) и необязательно содержит приблизительно одну или более (например, приблизительно 2, 3, 4, 5, 6, 7, 8, 9, 10) инсерций, приблизительно одну или более (например, приблизительно 2, 3, 4, 5, 6, 7, 8, 9, 10) делеций, приблизительно одну или более (например, приблизительно 2, 3, 4, 5, 6, 7, 8, 9, 10) аминокислотных замен (например, консервативных аминокислотных замен или неконсервативных аминокислотных замен), или комбинацию вышеупомянутых изменений по сравнению с последовательностью VL и/ или VH известного моноклонального антитела. Инсерция(ии), делеция(ии) или замена(ы) могут иметь место в любом месте области VL и/ или VH, в том числе на амино- или карбоксиконце, или на обоих концах этой области, при условии, что каждая CDR содержит ноль изменений или не более одного, двух или трех изменений. В некоторых вариантах реализации связывающий домен, содержащий модифицированную область VL и/или VH, сохраняет способность специфично связывать свою мишень с аффинностью, аналогичной или большей, чем у исходного связывающего домена.
[00138] В некоторых вариантах реализации связывающий домен, описанный в настоящем документе, получен из антитела и содержит вариабельную тяжелую цепь (VH) и вариабельную легкую цепь (VL). Например, scFv, содержащий цепи VH и VL. Эти связывающие домены и вариабельные цепи могут быть расположены в любом порядке, при котором сохраняется некоторая степень связывания с мишенью(ями). Например, вариабельные домены могут быть расположены следующем порядке: VH 5T4 - VL 5T4 - VH 4-1BB - VL 4-1BB; VL 5T4 - VH 5T4 - VH 4-1BB - VL 4-1BB; VH 5T4 - VL 5T4 - VL 4-1BB - VH 4-1BB; VL 5T4 - VH 5T4 - VL 4-1BB - VH 4-1BB; VH 4-1BB - VL 4-1BB - VH 5T4 - VL 5T4; VL 4-1BB - VH 4-1BB - VL 5T4 - VH 5T4; VH 4-1BB - VL 4-1BB - VL 5T4 - VH 5T4; или VL 4-1BB - VH 4-1BB - VH 5T4 - VL 5T4. Пары областей VH и областей VL в связывающем домене, связывающемся с 4-1BB, и/или связывающем домене, связывающемся с 5T4, могут иметь формат одноцепочечного антитела (scFv).
[00139] В некоторых вариантах реализации полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), содержат связывающие домены, представляющие собой scFv. В таких вариантах реализации связывающие домены могут называться scFv-доменами. В некоторых вариантах реализации связывающий домен представляет собой одноцепочечный Fv-фрагмент (scFv), содержащий области VH и VL, специфичные к представляющей интерес мишени. В отдельных вариантах реализации области VH и VL являются человеческими или гуманизированными. В некоторых вариациях связывающий домен представляет собой одноцепочечный Fv (scFv), содержащий области VL и VH, соединенные пептидным линкером.
[00140] Применение пептидных линкеров для соединения областей VL и VH хорошо известно в данной области техники, и в этой конкретной области существует большое количество публикаций. В некоторых вариантах реализации пептидный линкер является 15-мерным и состоит из трех повторов аминокислотной последовательности Gly-Gly-Gly-Gly-Ser ((Gly4Ser)3) (SEQ ID NO: 96). Были использованы и другие линкеры, и для диверсификации и выбора подходящих линкерных последовательностей была использована технология фагового дисплея, а также технология селективно инфицирующего фага (Tang et al., J. Biol. Chem. 271, 15682-15686, 1996; Hennecke et al., Protein Eng. 11, 405-410, 1998). В отдельных вариантах реализации области VL и VH соединены пептидным линкером, имеющим аминокислотную последовательность, содержащую структуру формулы (Gly4Ser)n, где n = 1-5. Например, в одном из вариантов реализации изобретения линкер содержит (Gly4Ser)4. Другие подходящие линкеры могут быть получены путем оптимизации простого линкера (например, (Gly4Ser)n) посредством случайного мутагенеза. В некоторых вариантах реализации область VH scFv, описанного в настоящем документе, может быть расположена с N-конца относительно линкерной последовательности. В некоторых вариантах реализации область VL scFv, описанного в настоящем документе, может быть расположена с С-конца относительно линкерной последовательности. В некоторых вариантах реализации scFv может связываться с 5T4 и/или 4-1BB более эффективно, чем антитело, содержащее такие же последовательности областей VH и VL в такой же ориентации. В отдельных вариантах реализации scFv может более эффективно связываться с 5T4 и/или 4-1BB в ориентации VL-VH по сравнению с ориентацией VH-VL, или наоборот.
[00141] В некоторых вариантах реализации полипептид содержит линкер связывающих доменов, соединяющий связывающие домены (например, соединяющий scFv-домены). В некоторых вариантах реализации линкер связывающих доменов представляет собой линкер Gly4Ser. В некоторых вариантах реализации линкер связывающих доменов является 20-мерным и состоит из четырех повторов аминокислотной последовательности Gly-Gly-Gly-Gly-Ser ((Gly4Ser)3) (SEQ ID NO: 98). В некоторых вариантах реализации линкер связывающих доменов содержит аминокислотную последовательность, выбранную из SEQ ID NO: 86-108. Были использованы и другие линкеры, и для диверсификации и выбора подходящих линкерных последовательностей была использована технология фагового дисплея, а также технология селективно инфицирующего фага (Tang et al., J. Biol. Chem. 271, 15682-15686, 1996; Hennecke et al., Protein Eng. 11, 405-410, 1998). В отдельных вариантах реализации области VL и VH соединены пептидным линкером, имеющим аминокислотную последовательность, содержащую структуру формулы (Gly4Ser)n, где n = 1-5. Другие подходящие линкеры могут быть получены путем оптимизации простого линкера (например, (Gly4Ser)n) посредством случайного мутагенеза. В некоторых случаях биспецифичная молекула может содержать scFv, связывающийся с 5T4, связанный с scFv, связывающимся с 4-1BB. В некоторых вариантах реализации биспецифичные молекулы не содержат шарнирную область или константную область.
[00142] В некоторых вариантах реализации полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), дополнительно содержат шарнир. В некоторых вариантах реализации шарнир представляет собой измененный шарнир иммуноглобулина, в котором один или более остатков цистеина в шарнирной области иммуноглобулина дикого типа заменен одним или более другими аминокислотными остатками (например, серином или аланином). Иллюстративные измененные шарниры иммуноглобулина, карбоксиконцевые линкеры и аминоконцевые линкеры включают шарнирную область человеческого иммуноглобулина IgG1, в которой один, два или три остатка цистеина, содержащихся в шарнире человеческого IgG1 дикого типа, заменены одним, двумя или тремя различными аминокислотными остатками (например, серином или аланином). Измененный шарнир иммуноглобулина может дополнительно содержать замену пролина другой аминокислотой (например, серином или аланином). Например, описанный выше измененный шарнир человеческого IgG1 может дополнительно содержать замену пролина, расположенного с карбоксиконца относительно трех цистеинов шарнирной области человеческого IgG1 дикого типа, другим аминокислотным остатком (например, серином, аланином). В одном из вариантов реализации пролины коровой шарнирной области не заменены. В отдельных вариантах реализации полипептид шарнира, карбоксиконцевого линкера или аминоконцевого линкера содержит или представляет собой последовательность, по меньшей мере на 80%, по меньшей мере на 81%, по меньшей мере на 82%, по меньшей мере на 83%, по меньшей мере на 84%, по меньшей мере на 85%, по меньшей мере на 86%, по меньшей мере на 87%, по меньшей мере на 88%, по меньшей мере на 89%, по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98% или по меньшей мере на 99% идентичную шарнирной области иммуноглобулина дикого типа, такой как шарнир человеческого IgG1 дикого типа, шарнир человеческого IgG2 дикого типа или шарнир человеческого IgG4 дикого типа.
[00143] В отдельных вариантах реализации шарнир, присутствующий в полипептиде, который образует гетеродимер с другой полипептидной цепью, может представлять собой шарнир иммуноглобулина, такой как шарнирная область иммуноглобулина дикого типа или его измененная шарнирная область. В отдельных вариантах реализации шарнир одной полипептидной цепи гетеродимерного белка идентичен соответствующему шарниру другой полипептидной цепи указанного гетеродимера. В некоторых других вариантах реализации шарнир одной цепи отличается от шарнира другой цепи (по длине или последовательности). Разные шарниры в разных цепях позволяют по-разному влиять на аффинности связывания связывающих доменов, с которыми связаны шарниры, так что гетеродимер способен избирательно связываться с мишенью одного связывающего домена по сравнению с мишенью другого связывающего домена. Например, в отдельных вариантах реализации гетеродимерный белок имеет 4-1BB-связывающий домен в одной цепи и 5T4-связывающий домен в другой цепи. Наличие двух разных шарниров в двух цепях может позволить гетеродимеру связываться сначала с 5T4, а затем с 4-1BB. Таким образом, гетеродимер может рекрутировать 4-1BB-экспрессирующие эффекторные клетки в 5T4-экспрессирующие клетки-мишени (например, 5T4-экспрессирующие опухолевые или раковые клетки), которые, в свою очередь, могут повреждать или уничтожать 5T4-экспрессирующие клетки.
[00144] В отдельных вариантах реализации карбоксиконцевой линкер или аминоконцевой линкер представляет собой гибкую линкерную последовательность, содержащую повторы глицин-серин (например, Gly4Ser). В отдельных вариантах реализации линкер содержит три повтора Gly4Ser (SEQ ID NO: 96), за которыми следует остаток пролина. В отдельных вариантах реализации за остатком пролина следует аминокислота, выбранная из группы, состоящей из глицина, аргинина и серина. В некоторых вариантах реализации карбоксиконцевой линкер или аминоконцевой линкер содержит или состоит из последовательности, выбранной из SEQ ID NO: 86 - 108.
[00145] Некоторые иллюстративные последовательности шарнира, карбоксиконцевого линкера и аминоконцевого линкера, подходящие для применения в соответствии с настоящим изобретением, представлены ниже в таблице 3. Дополнительные иллюстративные шарнирные и линкерные области представлены в SEQ ID NO: 241-244, 601, 78, 763-791, 228, 379-434, 618-749, содержащихся в публикации патента США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки).
Таблица 3. Иллюстративные петли и линкеры
[00146] В других вариантах реализации полипептиды и белки, описанные в настоящем документе, включают домен гетеродимеризации, способный гетеродимеризоваться с другим доменом гетеродимеризации во второй, неидентичной полипептидной цепи. В отдельных вариациях вторая полипептидная цепь для гетеродимеризации включает второй связывающий домен. Соответственно, в отдельных вариантах реализации настоящего изобретения две неидентичные полипептидные цепи, одна из которых содержит полипептид, содержащий первый связывающий домен, а вторая необязательно содержит второй связывающий домен, димеризуются с образованием гетеродимерного связывающего белка. Домены димеризации/гетеродимеризации могут быть использованы в случаях, когда желательно образование гетеродимеров из двух неидентичных полипептидных цепей, где одна или обе полипептидные цепи содержат связывающий домен. В отдельных вариантах реализации одна полипептидная цепь-член отдельных гетеродимеров, описанных в настоящем документе, не содержит связывающий домен. Примеры типов гетеродимеров включают описанные в публикациях патентных заявок США №№ и 2013/0095097 и 2013/0129723, и публикации международной заявки PCT № WO 2016/094873.
[00147] В отдельных вариантах реализации первая и вторая полипептидные цепи димеризуются посредством включения «домена димеризации иммуноглобулина» или «домена гетеродимеризации иммуноглобулина». Термин «домен димеризации иммуноглобулина» или «домен гетеродимеризации иммуноглобулина» в настоящем документе относится к домену иммуноглобулина первой полипептидной цепи, который избирательно взаимодействует или связывается с другим доменом иммуноглобулина второй полипептидной цепи, где взаимодействие разных доменов иммуноглобулинов в значительной степени способствует гетеродимеризации первой и второй полипептидных цепей (т. е., образование димера между двумя разными полипептидными цепями, который также называют «гетеродимером»), или эффективно стимулирует ее. Домены гетеродимеризации иммуноглобулина в полипептидных цепях гетеродимера отличаются друг от друга и, таким образом, могут быть выборочно модифицированы, чтобы способствовать гетеродимеризации обеих цепей и минимизировать гомодимеризацию любой из цепей. Домены гетеродимеризации иммуноглобулина, предложенные в настоящем документе, обеспечивают эффективную гетеродимеризацию между различными полипептидами и облегчают очистку полученного гетеродимерного белка.
[00148] Как указано в настоящем документе, домены гетеродимеризации иммуноглобулина, пригодные для стимулирования гетеродимеризации двух разных полипептидных цепей согласно настоящему изобретению, включают домены СН1 и CL иммуноглобулина, как дикого типа, так и измененные, например, домены СН1 и CL иммуноглобулина человека. В отдельных вариантах реализации домен гетеродимеризации иммуноглобулина представляет собой домен CH1 дикого типа, такой как домен CH1 IgG1, IgG2, IgG3, IgG4, IgA1, IgA2, IgD, IgE или IgM дикого типа, например, как представлено в SEQ ID NO: 114, 186-192 и 194, соответственно, из публикации патентной заявки США № 2013/0129723, или SEQ ID NO: 114 из публикации патентной заявки США № 2013/0129723 (указанная последовательность включена в настоящий документ посредством ссылки). В дополнительных вариантах реализации остаток цистеина домена CH1 дикого типа (например, человеческого CH1), участвующий в образовании дисульфидной связи с доменом CL иммуноглобулина дикого типа (например, CL человека), удален или заменен в измененном домене CH1 иммуноглобулина, так что дисульфидная связь между измененным доменом CH1 и доменом CL дикого типа не образуется.
[00149] В отдельных вариантах реализации домен гетеродимеризации иммуноглобулина представляет собой домен CL дикого типа, такой как домен Cκ дикого типа или домен Cλ дикого типа, например, как представлено в SEQ ID NO: 112 и 113, соответственно, из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки). В дополнительных вариантах реализации домен гетеродимеризации иммуноглобулина представляет собой измененный домен CL иммуноглобулина, такой как измененный домен Cκ или Cλ, например, измененный й домен Cκ или Cλ иммуноглобудина человека. В отдельных вариантах реализации остаток цистеина в домене CL дикого типа, участвующий в образовании дисульфидной связи с доменом CH1 иммуноглобулина дикого типа, удален или заменен в измененном домене CL иммуноглобулина, например, в домене Cκ, как представлено в SEQ ID NO: 141 из публикации патентной заявки США № 2013/0129723, или домене Cλ, как представлено в SEQ ID NO: 140 из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки). В отдельных вариантах реализации в измененном домене Cκ удален только последний цистеин человеческого домена Cκ дикого типа, поскольку первый аргинин, удаленный из человеческого домена Cκ дикого типа, может быть обеспечен с помощью линкера, содержащего аргинин на его карбоксиконце и связывающего аминоконец измененного домена Cκ с другим доменом (например, субобластью иммуноглобулина, такой как субобласть, содержащая домены CH2 и CH3 иммуноглобулина).
[00150] В дополнительных вариантах реализации домен гетеродимеризации иммуноглобулина представляет собой измененный домен Cκ, содержащий одну или более аминокислотных замен по сравнению с доменом Cκ дикого типа, в положениях, которые могут участвовать в образовании сети межцепочечных водородных связей на поверхности контакта Cκ-Cκ. Например, в отдельных вариантах реализации домен гетеродимеризации иммуноглобулина представляет собой измененный домен Cκ человека, в котором одна или более аминокислот в положениях N29, N30, Q52, V55, T56, S68 или T70 заменены другой аминокислотой. Нумерация аминокислот основана на их положениях в измененной последовательности человеческого Cκ, представленной в SEQ ID NO: 141 из публикации патентной заявки США № 2013/0129723 (указанная последовательность включена в настоящий документ посредством ссылки). В отдельных вариантах реализации домен гетеродимеризации иммуноглобулина представляет собой измененный домен Cκ человека, имеющий одну, две, три или четыре аминокислотные замены в положениях N29, N30, V55 или T70. Аминокислота, используемая в качестве заменителя в вышеуказанных положениях, может представлять собой аланин или аминокислотный остаток с объемным фрагментом боковой цепи, такой как аргинин, триптофан, тирозин, глутамат, глутамин, лизин, аспартат, метионин, серин или фенилаланин. Измененные домены Cκ человека представляют собой домены, способствующие гетеродимеризации с доменом CH1, но минимизирующие гомодимеризацию с другим доменом Cκ. Иллюстративные измененные Cκ домены иммуноглобулина человека представлены в SEQ ID NO: 142-178 публикации патентной заявки США № 2013/0129723; SEQ ID NO: 160 (N29W V55A T70A), 161 (N29Y V55A T70A), 202 (T70E N29A N30A V55A), 167 (N30R V55A T70A), 168 (N30K V55A T70A), 170 (N30E V55A T70A), 172 (V55R N29A N30A), 175 (N29W N30Y V55A T70E), 176 (N29Y N30Y V55A T70E), 177 (N30E V55A T70E), 178 (N30Y V55A T70E), 838 (N30D V55A T70E), 839 (N30M V55A T70E), 840 (N30S V55A T70E) и 841 (N30F V55A T70E) публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки).
[00151] В отдельных вариантах реализации в дополнение к мутациям в доменах Cκ, описанным в настоящем документе, или в качестве альтернативы этим мутациям, оба домена гетеродимеризации иммуноглобулина (т. е. домены CH1 и CL иммуноглобулина) полипептидного гетеродимера имеют мутации, так что получаемые в результате домены гетеродимеризации иммуноглобулина образуют солевые мостики (т. е., ионные взаимодействия) между аминокислотными остатками в мутированных сайтах. Например, домены гетеродимеризации иммуноглобулина полипептидного гетеродимера могут представлять собой мутированный домен CH1 в комбинации с мутированным доменом Cκ. В мутированном домене CH1 валин в положении 68 (V68) человеческого домена CH1 дикого типа заменен аминокислотным остатком, имеющим отрицательный заряд (например, аспартатом или глутаматом), тогда как лейцин в положении 29 (L29) мутированного человеческого домена Cκ, в котором были удалены первый аргинин и последний цистеин, заменен аминокислотным остатком, имеющим положительный заряд (например, лизином, аргинином или гистидином). Взаимодействие зарядов между аминокислотным остатком полученного мутированного домена CH1, имеющим отрицательный заряд, и аминокислотным остатком полученного мутированного домена Cκ, имеющим положительный заряд, образует солевой мостик, стабилизирующий гетеродимерную поверхность контакта между мутированными доменами CH1 и Cκ. В качестве альтернативы, V68 CH1 дикого типа может быть заменен аминокислотным остатком, имеющим положительный заряд, тогда как L29 мутированного человеческого домена Cκ, в котором были удалены первый аргинин и последний цистеин, может быть заменен аминокислотным остатком, имеющим отрицательный заряд. Иллюстративные мутированные последовательности CH1, в которых V68 заменен аминокислотой с отрицательным или положительным зарядом, представлены в SEQ ID NO: 844 и 845 из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки). Иллюстративные мутированные последовательности Cκ, в которых L29 замещен аминокислотой с отрицательным или положительным зарядом, представлены в SEQ ID NO: 842 и 843 из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки).
[00152] Положения человеческого домена CH1 и L29 человеческого домена Cκ, отличные от V68, могут быть заменены аминокислотами, имеющими противоположные заряды, для создания ионных взаимодействий между аминокислотами в дополнение или в качестве альтернативы мутациям в V68 домена CH1 и L29 домена Cκ. Такие положения могут быть идентифицированы любым подходящим способом, включая случайный мутагенез, анализ кристаллической структуры пары CH1-Cκ для идентификации аминокислотных остатков на поверхности контакта CH1-Cκ, а также идентификацию подходящих положений среди аминокислотных остатков на поверхности контакта CH1-Cκ с использованием набора критериев (например, склонности вступать в ионные взаимодействия, близости к потенциальному партнерскому остатку и т. д.).
[00153] В отдельных вариантах реализации полипептидные гетеродимеры согласно настоящему изобретению содержат только одну пару доменов гетеродимеризации иммуноглобулина. Например, первая цепь полипептидного гетеродимера может содержать домен CH1 в качестве домена гетеродимеризации иммуноглобулина, тогда как вторая цепь может содержать домен CL (например, Cκ или Cλ) в качестве домена гетеродимеризации иммуноглобулина. В качестве альтернативы, первая цепь может содержать домен CL (например, Cκ или Cλ) в качестве домена гетеродимеризации иммуноглобулина, тогда как вторая цепь может содержать домен CH1 в качестве домена гетеродимеризации иммуноглобулина. Как указано в настоящем документе, домены гетеродимеризации иммуноглобулина первой и второй цепей способны связываться с образованием гетеродимерного белка согласно настоящему изобретению.
[00154] В некоторых других вариантах реализации гетеродимерные белки согласно настоящему изобретению могут иметь две пары доменов гетеродимеризации иммуноглобулина. Например, первая цепь гетеродимера может содержать два домена CH1, тогда как вторая цепь может иметь два домена CL, связывающихся с двумя доменами CH1 в первой цепи. В качестве альтернативы, первая цепь может содержать два домена CL, тогда как вторая цепь может иметь два домена CH1, связывающихся с двумя доменами CL в первой цепи. В отдельных вариантах реализации первая полипептидная цепь содержит домен CH1 и домен CL, тогда как вторая полипептидная цепь содержит домен CL и домен CH1, связывающиеся с доменом CH1 и доменом CL, соответственно, первой полипептидной цепи.
[00155] В вариантах реализации, в которых гетеродимерный белок содержит только одну гетеродимеризационную пару (то есть, по одному домену гетеродимеризации иммуноглобулина в каждой цепи), домен гетеродимеризации иммуноглобулина каждой из цепей может являться аминоконцевым относительно константной области иммуноглобулина этой цепи. В качестве альтернативы, домен гетеродимеризации иммуноглобулина в каждой из цепей может являться карбоксиконцевым относительно константной области иммуноглобулина этой цепи.
[00156] В вариантах реализации, в которых гетеродимерный белок содержит две гетеродимеризационные пары (то есть, по два домена гетеродимеризации иммуноглобулина в каждой цепи), оба домена гетеродимеризации иммуноглобулина в каждой из цепей могут являться аминоконцевыми относительно константной области иммуноглобулина этой цепи. В качестве альтернативы, оба домена гетеродимеризации иммуноглобулина в каждой из цепей могут являться карбоксиконцевыми относительно константной области иммуноглобулина этой цепи. В дополнительных вариантах реализации один домен гетеродимеризации иммуноглобулина в каждой из цепей может являться аминоконцевым относительно константной области иммуноглобулина этой цепи, тогда как другой домен гетеродимеризации иммуноглобулина каждой из цепей может являться карбоксиконцевым относительно константной области иммуноглобулина этой цепи. Другими словами, в этих вариантах реализации константная область иммуноглобулина расположена между двумя доменами гетеродимеризации иммуноглобулина каждой из цепей.
[00157] Как указано в настоящем документе, в некоторых вариантах реализации полипептиды и белки (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) согласно настоящему изобретению дополнительно содержат константную область иммуноглобулина (также называемую в настоящем документе константной областью, Fc-доменом, Fc-областью и т.п.) в полипептидной цепи. В некоторых вариантах реализации константная область иммуноглобулина содержит аминокислотную последовательность в соответствии с SEQ ID NO: 158, 160, или ее вариант. Включение константной области иммуноглобулина замедляет клиренс полипептидов и белков согласно настоящему изобретению из кровотока после введения субъекту. Посредством мутаций или других изменений константная область иммуноглобулина дополнительно позволяет относительно легко модулировать эффекторные функции полипептида (например, ADCC, ADCP, CDC, фиксацию комплемента и связывание с Fc-рецепторами), которые могут быть повышены или снижены в зависимости от подлежащего лечению заболевания, как известно в данной области техники и описано в настоящем документе. В отдельных вариантах реализации полипептиды и белки, описанные в настоящем документе, содержат константную область иммуноглобулина, способную опосредовать одну или более из этих эффекторных функций. В других вариантах реализации одна или более из этих эффекторных функций снижены или отсутствуют в константной области иммуноглобулина полипептида или белка, описанного в настоящем документе, по сравнению с соответствующей константной областью иммуноглобулина дикого типа. Например, для димерных 5T4-связывающих или 4-1BB-связывающих полипептидов, предназначенных для усиления активации эффекторных клеток, например, путем включения 4-1BB-связывающего домена, константная область иммуноглобулина предпочтительно имеет сниженную эффекторную функцию относительно соответствующей константной области иммуноглобулина дикого типа, или не имеет такой эффекторной функции.
[00158] Константная область иммуноглобулина, присутствующая в полипептидах и белках согласно настоящему изобретению (например, 5T4-связывающих полипептидах, 4-1BB-связывающих полипептидах и/или содержащих их полиспецифичных связывающих белках) может содержать или может быть получена из части или всех из: домена CH2, домена CH3, домена CH4 или любой их комбинации. Например, константная область иммуноглобулина может содержать домен CH2, домен CH3, оба домена CH2 и CH3, оба домена CH3 и CH4, два домена CH3, домен CH4, два домена CH4, и домен CH2 и часть домена CH3. В отдельных вариантах реализации полипептиды или белки, описанные в настоящем документе, не содержат домен CH1.
[00159] Полипептид или белок, описанный в настоящем документе, может содержать домен СН2 иммуноглобулина дикого типа или измененный домен СН2 иммуноглобулина из определенных классов или подклассов иммуноглобулинов (например, IgG1, IgG2, IgG3, IgG4, IgA1, IgA2 или IgD) и от различных видов (включая человека, мышь, крысу и других млекопитающих). В отдельных вариантах реализации домен CH2 полипептида или белка, описанного в настоящем документе, представляет собой домен CH2 человеческого иммуноглобулина дикого типа, такой как домены CH2 дикого типа человеческого IgG1, IgG2, IgG3, IgG4, IgA1, IgA2 или IgD, как представлено в SEQ ID NO: 115, 199-201 и 195-197, соответственно, из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки). В отдельных вариантах реализации домен CH2 представляет собой домен CH2 человеческого IgG1 дикого типа, как представлено в SEQ ID NO: 115 из публикации патентной заявки США № US 2013/0129723 (указанная последовательность включена в настоящий документ посредством ссылки).
[00160] В отдельных вариантах реализации измененная область СН2 в полипептиде или белке согласно настоящему изобретению содержит или представляет собой последовательность, по меньшей мере на 90%, по меньшей мере на 91%, по меньшей мере на 92%, по меньшей мере на 93%, по меньшей мере на 94%, по меньшей мере на 95%, по меньшей мере на 96%, по меньшей мере на 97%, по меньшей мере на 98%, по меньшей мере на 99% идентичную области CH2 иммуноглобулина дикого типа, такой как область CH2 человеческого IgG1, IgG2 или IgG4 дикого типа, или мышиного IgG2a (например, IGHG2c).
[00161] Измененная область CH2 иммуноглобулина в полипептиде или белке согласно настоящему изобретению (например, 5T4-связывающих полипептидах, 4-1BB-связывающих полипептидах и/или содержащих их полиспецифичных связывающих белках) может быть получена из области CH2 различных изотипов иммуноглобулина, таких как IgG1, IgG2, IgG3, IgG4, IgA1, IgA2 и IgD, от различных видов (включая человека, мышь, крысу и других млекопитающих). В отдельных вариантах реализации измененная область CH2 иммуноглобулина в слитом белке согласно настоящему изобретению может быть получена из области CH2 человеческого IgG1, IgG2 или IgG4 или мышиного IgG2a (например, IGHG2c), последовательности которых представлены в SEQ ID NO: 115, 199, 201 и 320 из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки). В отдельных вариантах реализации измененный домен CH2 полипептида или белка, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), представляет собой измененный домен CH2 человеческого IgG1, содержащий мутации, которые, как известно в данной области техники, которое усиливают или снижают иммунологические активности (т. е., эффекторные функции), такие как ADCC, ADCP, CDC, фиксация комплемента, связывание Fc-рецептора или любая их комбинация.
[00162] В отдельных вариантах реализации домен CH2 полипептида или белка, описанного в настоящем документе, представляет собой измененную область CH2 иммуноглобулина (например, измененный домен CH2 человеческого IgG1), содержащую одну или более делеций или замен аминокислот. В некоторых вариантах реализации домен CH2 содержит аминокислотную замену аспарагина в положении 297 (например, замену аспарагина на аланин). Такая аминокислотная замещена снижает или устраняет гликозилирование в этом сайте и подавляет эффективное связывание Fc с FcγR и C1q. Последовательность измененного домена CH2 человеческого IgG1 с заменой Asn на Ala в положении 297 представлена в SEQ ID NO: 324 публикации патентной заявки США № 2013/0129723 (указанная последовательность включена в настоящий документ посредством ссылки). В некоторых вариантах реализации измененный домен CH2 содержит по меньшей мере одну замену или делецию в положениях с 234 по 238. Например, область CH2 иммуноглобулина может содержать замену в положении 234, 235, 236, 237 или 238; положениях 234 и 235; положениях 234 и 236; положениях 234 и 237; положениях 234 и 238; положениях 234-236; положениях 234, 235 и 237; положениях 234, 236 и 238; положениях 234, 235, 237 и 238; положениях 236-238; или любой другой комбинации из двух, трех, четырех или пяти аминокислот в положениях 234-238. В некоторых вариантах реализации измененная область СН2 содержит одну или более (например, две, три, четыре или пять) делеций аминокислот в положениях 234-238, например, в одном из положения 236 или положения 237, в то время как в другом положении осуществлена замена. В отдельных вариантах реализации аминокислотные остатки в одном или более положениях 234-238 были заменены одним или более остатками аланина. В дополнительных вариантах реализации только один из аминокислотных остатков в положениях 234-238 был удален, тогда как одна или более оставшихся аминокислот в положениях 234-238 могут быть заменены другой аминокислотой (например, аланином или серином).
[00163] В некоторых вариантах реализации указанная выше мутация(ии) снижают или устраняют активность ADCC или способность связывания Fc-рецептора полипептида, содержащего измененный домен CH2.
[00164] В некоторых других вариантах реализации домен CH2 полипептида или белка, описанного в настоящем документе, представляет собой измененную область CH2 иммуноглобулина (например, измененный домен CH2 человеческого IgG1), содержащую одну или более аминокислотных замен в положениях 253, 310, 318, 320, 322 и 331. Например, область CH2 иммуноглобулина может содержать замену в положении 253, 310, 318, 320, 322 или 331, положениях 318 и 320, положениях 318 и 322, положениях 318, 320 и 322, или любой другой комбинации из двух, трех, четырех, пяти или шести аминокислот в положениях 253, 310, 318, 320, 322 и 331. В таких вариантах реализации указанная выше мутация(ии) снижают или устраняют активность CDC полипептида, содержащего измененный домен CH2.
[00165] В некоторых других вариантах реализации в дополнение к аминокислотной замене в положении 297 измененная область СН2 полипептида или белка, описанного в настоящем документе (например, измененный домен СН2 человеческого IgG1), может дополнительно содержать одну или более (например, две, три, четыре или пять) дополнительных замен в положениях 234-238. Например, область CH2 иммуноглобулина может содержать замену в положениях 234 и 297, положениях 234, 235 и 297, положениях 234, 236 и 297, положениях 234-236 и 297, положениях 234, 235, 237 и 297, положениях 234, 236, 238 и 297, положениях 234, 235, 237, 238 и 297, положениях 236-238 и 297, или любой комбинации из двух, трех, четырех или пяти аминокислот в положениях 234-238 в дополнение к положению 297. В дополнение или в качестве альтернативы, измененная область СН2 может содержать одну или более (например, две, три, четыре или пять) делеций аминокислот в положениях 234-238, например, в положении 236 или положении 237. Дополнительная мутация(ии) снижает или устраняет активность ADCC или способность связывания Fc-рецептора полипептида, содержащего измененный домен CH2. В отдельных вариантах реализации аминокислотные остатки в одном или более положениях 234-238 были заменены одним или более остатками аланина. В дополнительных вариантах реализации только один из аминокислотных остатков в положениях 234-238 был удален, тогда как одна или более оставшихся аминокислот в положениях 234-238 могут быть заменены другой аминокислотой (например, аланином или серином).
[00166] В отдельных вариантах реализации, в дополнение к одной или более (например, 2, 3, 4 или 5) аминокислотным заменам в положениях 234-238, мутированная область CH2 полипептида или белка, описанного в настоящем документе (например, измененный домен СН2 человеческого IgG1) в слитом белке согласно настоящему изобретению может содержать одну или более (например, 2, 3, 4, 5 или 6) дополнительных аминокислотных замен (например, замены аланином) в одном или более положениях, участвующих в фиксации комплемента (например, в положениях I253, H310, E318, K320, K322 или P331). Примеры мутированных областей CH2 иммуноглобулина включают области CH2 человеческого IgG1, IgG2, IgG4 и мышиного IgG2a с заменами на аланин в положениях 234, 235, 237 (если присутствует), 318, 320 и 322. Примером мутированной области CH2 иммуноглобулина является область CH2 мышиного IGHG2c с заменами на аланин в L234, L235, G237, E318, K320 и K322.
[00167] В некоторых других вариантах реализации в дополнение к аминокислотной замене в положении 297 и жополнительной делеции(ях) или замене(ах) в положениях 234-238 измененная область СН2 полипептида или белка, описанного в настоящем документе (например, измененный домен СН2 человеческого IgG1), может дополнительно содержать одну или более (например, две, три, четыре, пять или шесть) дополнительных замен в положениях 253, 310, 318, 320, 322 и 331. Например, область CH2 иммуноглобулина может содержать (1) замену в положении 297, (2) одну или более замен или делеций, или их комбинацию в положениях 234-238, и одну или более (например, 2, 3, 4, 5 или 6) аминокислотных замен в положениях I253, H310, E318, K320, K322 и P331, например, одну, две, три замены в положениях E318, K320 и K322. Аминокислоты в вышеуказанных положениях могут быть заменены аланином или серином.
[00168] В отдельных вариантах реализации область CH2 иммуноглобулина полипептида или белка, описанного в настоящем документе, содержит: (i) аминокислотную замену аспарагина в положении 297 и одну аминокислотную замену в положении 234, 235, 236 или 237; (ii) аминокислотную замену аспарагина в положении 297 и аминокислотные замены в двух положениях из 234-237; (iii) аминокислотную замену аспарагина в положении 297 и аминокислотные замены в трех из положений 234-237; (iv) аминокислотную замену аспарагина в положении 297, аминокислотные замены в положениях 234, 235 и 237, и делецию аминокислоты в положении 236; (v) аминокислотные замены в трех положениях из 234-237 и аминокислотные замены в положениях 318, 320 и 322; или (vi) аминокислотные замены в трех положениях из 234-237, делецию аминокислоты в положении 236 и аминокислотные замены в положениях 318, 320 и 322.
[00196] Иллюстративные измененные области CH2 иммуноглобулина с аминокислотными заменами аспарагина в положении 297 включают: область CH2 человеческого IgG1 с заменами на аланин в L234, L235, G237 и N297 и делецией в G236 (SEQ ID NO: 325 из публикации патентной заявки № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки), область CH2 человеческого IgG2 с заменами на аланин в V234, G236 и N297 (SEQ ID NO: 326 из публикации патентной заявки США № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки), область CH2 человеческого IgG4 с заменами на аланин в F234, L235, G237 и N297 и делецией в G236 (SEQ ID NO: 322 из публикации патентной заявки США № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки), область CH2 человеческого IgG4 с заменами на аланин в F234 и N297 (SEQ ID NO: 343 из публикации патентной заявки США № US 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки), область CH2 человеческого IgG4 с заменами на аланин в L235 и N297 (SEQ ID NO: 344 из публикации патентной заявки США № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки), область CH2 человеческого IgG4 с заменами на аланин в G236 и N297 (SEQ ID NO: 345 из публикации патентной заявки США № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки) и область CH2 человеческого IgG4 с заменами на аланин в G237 и N297 (SEQ ID № 346 публикации патентной заявки США № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки). Эти области CH2 могут быть использованы в полипептиде согласно настоящему изобретению (например, 5T4-связывающем полипептиде и/или биспецифичном 4-1BB полипептиде).
[00170] В отдельных вариантах реализации в дополнение к аминокислотным заменам, описанным выше, измененная область СН2 полипептида или белка, описанного в настоящем документе (например, измененный домен СН2 человеческого IgG1), может содержать одну или более дополнительных аминокислотных замен в одном или более положениях, отличных от вышеуказанных положений. Такие аминокислотные замены могут представлять собой консервативные или неконсервативные аминокислотные замены. Например, в отдельных вариантах реализации P233 может быть заменен на E233 в измененной области CH2 IgG2 (см., например, SEQ ID NO: 326 из публикации патентной заявки США № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки). В дополнение или в качестве альтернативы, в отдельных вариантах реализации измененная область CH2 может содержать одну или более инсерций, делеций аминокислот, или обе. Инсерция(ии), делеция(ии) или замена(ы) могут иметь место в любом положении области CH2 иммуноглобулина, например, на N- или C-конце области CH2 иммуноглобулина дикого типа, являющейся результатом связывания области CH2 с другой областью (например, связывающим доменом или доменом гетеродимеризации иммуноглобулина) посредством шарнира.
[00171] В отдельных вариантах реализации измененный домен CH2 полипептида или белка, описанного в настоящем документе, представляет собой домен CH2 человеческого IgG1 с заменами на аланин в положениях 235, 318, 320 и 322 (то есть, домен CH2 человеческого IgG1 с заменами L235A, E318A, K320A и K322A) (SEQ ID NO: 595 из публикации патентной заявки США № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки) и, необязательно, мутацией N297 (например, в аланин). В некоторых других вариантах реализации измененный домен CH2 представляет собой домен CH2 человеческого IgG1 с заменами на аланин в положениях 234, 235, 237, 318, 320 и 322 (то есть, домен CH2 человеческого IgG1 с заменами L234A, L235A, G237A, E318A, K320A и K322A) (SEQ ID NO: 596 из публикации патентной заявки США № 2013/0129723, указанная последовательность включена в настоящий документ посредством ссылки) и, необязательно, мутацией N297 (например, в аланин).
[00172] В некоторых вариантах реализации константная область иммуноглобулина полипептида или белка, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), содержит домен CH2 человеческого IgG1, содержащий замены L234A, L235A, G237A и K322A, в соответствии с системой нумерации EU.
[00173] Домен CH3, который может образовывать константную область иммуноглобулина полипептида или белка, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), может представлять собой домен CH3 иммуноглобулина дикого типа или измененный домен СН3 иммуноглобулина из определенных классов или подклассов иммуноглобулинов (например, IgG1, IgG2, IgG3, IgG4, IgA1, IgA2, IgD, IgE, IgM) от различных видов (включая человека, мышь, крысу и других млекопитающих). В некоторых вариантах реализации домен CH3 полипептида, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), представляет собой домен CH3 человеческого иммуноглобулина дикого типа, такой как домены CH3 дикого типа человеческого IgG1, IgG2, IgG3, IgG4, IgA1, IgA2, IgD, IgE или IgM, как представлено в SEQ ID NO: 116, 208-210, 204-207 и 212, соответственно, из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки). В отдельных вариантах реализации домен CH3 представляет собой домен CH3 человеческого IgG1 дикого типа, как представлено в SEQ ID NO: 116 публикации патентной заявки США № 2013/0129723 (указанная последовательность включена в настоящий документ посредством ссылки).
[00174] В отдельных вариантах реализации домен CH3 полипептида, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), представляет собой измененный домен CH3 человеческого иммуноглобулина, такой как измененный домен CH3, основанный на или полученный из домена СН3 дикого типа человеческих антител IgG1, IgG2, IgG3, IgG4, IgA1, IgA2, IgD, IgE или IgM. Например, измененный домен CH3 может представлять собой домен CH3 человеческого IgG1 с одной или двумя мутациями в положениях H433 и N434 (положения пронумерованы в соответствии с системой EU). Мутации в таких положениях могут участвовать в фиксации комплемента. В некоторых других вариантах реализации измененный домен СН3 полипептида, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), может представлять собой домен CH3 человеческого IgG1, но с одной или двумя аминокислотными заменами в положении F405 или Y407. Аминокислоты в таких положениях участвуют во взаимодействии с другим доменом СН3. В отдельных вариантах реализации измененный домен СН3 полипептида, описанного в настоящем документе, может представлять собой измененный домен СН3 человеческого IgG1, последний лизин которого был удален. Последовательность этого измененного домена СН3 представлена в SEQ ID NO: 761 из публикации патентной заявки США № 2013/0129723 (указанная последовательность включена в настоящий документ посредством ссылки).
[00175] В отдельных вариантах реализации полипептид или белок, описанный в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), содержит домен CH3, содержащий так называемые мутации по типу «выступы во впадины» (см. Marvin and Zhu, Acta Pharmacologica Sinica 26:649-58, 2005; Ridgway et al., Protein Engineering 9:617-21, 1966). Более конкретно, мутации могут быть введены в каждый из доменов СН3 каждой полипептидной цепи, так что стерическая комплементарность, необходимая для связывания СН3/СН3, обязывает эти два домена СН3 спариваться друг с другом. Например, домен CH3 в одном одноцепочечном полипептиде полипептидного гетеродимера может содержать мутацию T366W (мутацию «выступ», которая заменяет небольшую аминокислоту более крупной), и домен CH3 в другом одноцепочечном полипептиде полипептидного гетеродимера может содержать мутацию Y407A (мутация «впадина», которая заменяет большую аминокислоту на менее крупную). Другие иллюстративные мутации по типу «выступы во впадины» включают (1) мутацию T366Y в одном домене CH3 и Y407T в другом домене CH3, и (2) мутацию T366W в одном домене CH3 и мутации T366S, L368A и Y407V в другом домене CH3.
[00176] Домен CH4, который может образовывать константную область иммуноглобулина полипептида или белка, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), может представлять собой домен CH4 иммуноглобулина дикого типа или измененный домен СН4 иммуноглобулина из молекул IgE или IgM. В отдельных вариантах реализации домен CH4 полипептида, описанного в настоящем документе, представляет собой домен CH4 человеческого иммуноглобулина дикого типа, такой как домены CH4 дикого типа молекул человеческих IgE и IgM, как представлено в SEQ ID NO: 213 и 214, соответственно, из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки). В отдельных вариантах реализации домен CH4 полипептида, описанного в настоящем документе, представляет собой измененный домен CH4 человеческого иммуноглобулина, такой как измененный домен CH4, основанный на или полученный из домена CH4 молекул человеческого IgE или IgM, имеющих мутации, повышающие или снижающие иммунологическую активность, связанную, как известно, с Fc-областью IgE или IgM.
[00177] В отдельных вариантах реализации константная область иммуноглобулина полипептида или белка, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), содержит комбинацию доменов CH2, CH3 или CH4 (т. е. более чем один домен константной области выбран из СН2, СН3 и СН4). Например, константная область иммуноглобулина может содержать домены СН2 и СН3 или домены СН3 и СН4. В некоторых других вариантах реализации константная область иммуноглобулина может содержать два домена СН3 и не содержать доменов СН2 или СН4 (т. е., только два или более СН3). Несколько доменов константной области, образующих константную область иммуноглобулина полипептидов, описанных в настоящем документе, могут быть основаны на или получены из одной и той же молекулы иммуноглобулина, или из молекул иммуноглобулина того же класса или подкласса. В отдельных вариантах реализации константная область иммуноглобулина представляет собой CH2-CH3 IgG (например, CH2-CH3 IgG1, CH2-CH3 IgG2 и CH2-CH3 IgG4) и может представлять собой человеческую CH2CH3 (например, человеческого IgG1, IgG2 и IgG4). Например, в отдельных вариантах реализации константная область иммуноглобулина полипептида, описанного в настоящем документе, содержит (1) домены CH2 и CH3 человеческого IgG1 дикого типа, (2) CH2 человеческого IgG1 с заменой N297A (т. е., CH2(N297A)) и CH3 человеческого IgG1 дикого типа, или (3) CH2(N297A) человеческого IgG1 и измененный CH3 человеческого IgG1, последний лизин которого был удален. В качестве альтернативы, несколько доменов константной области полипептида или белка, описанного в данном документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков) могут быть основаны на или получены из разных молекул иммуноглобулинов, или из молекул иммуноглобулинов разных классов или подклассов. Например, в отдельных вариантах реализации константная область иммуноглобулина содержит как домен CH3 человеческого IgM, так и домен CH3 человеческого IgG1. Несколько доменов константной области, образующих константную область иммуноглобулина полипептида, описанного в настоящем документе, могут быть непосредственно связаны друг с другом или могут быть связаны друг с другом посредством одной или нескольких (например, от приблизительно 2 до приблизительно 10) аминокислот.
[00178] Иллюстративные константные области иммуноглобулина, которые могут применяться в полипептиде или белке, описанном в настоящем документе (например, 5T4-связывающих полипептидах, 4-1BB-связывающих полипептидах и/или содержащих их полиспецифичных связывающих белках), представлены в SEQ ID NO: 305-309, 321, 323, 341, 342 и 762 из публикации патентной заявки США № 2013/0129723 (указанные последовательности включены в настоящий документ посредством ссылки). Дополнительные иллюстративные константные области иммуноглобулина, которые могут применяться в полипептиде или белке, описанном в настоящем документе, представлены ниже в таблице 4.
Таблица 4. Иллюстративные константные области иммуноглобулина
[00179] В отдельных вариантах реализации константные области иммуноглобулина каждой из полипептиднах цепей гомодимерного или гетеродимерного белка, описанного в настоящем документе (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков) идентичны друг другу. В некоторых других вариантах константная область иммуноглобулина одной полипептидной цепи гетеродимерного белка отличается от константной области иммуноглобулина другой полипептидной цепи гетеродимера. Например, одна константная область иммуноглобулина гетеродимерного белка может содержать домен СН3 с мутацией «выступ», тогда как другая константная область иммуноглобулина гетеродимерного белка может содержать домен СН3 с мутацией «впадина».
[00180] В некоторых вариантах реализации полипептиды согласно настоящему изобретению могут содержать константную область иммуноглобулина, содержащую любую из описанных выше мутаций, и связывающий домен, содержащий одну или более аминокислотных мутаций по сравнению с аминокислотной последовательностью исходного связывающего домена. Например, в некоторых вариантах реализации полипептиды согласно настоящему изобретению могут содержать константную область иммуноглобулина, содержащую одну или более мутаций L234A, L235A, G237A и K322A в домене CH2 человеческого IgG1, и 5T4-связывающий домен, содержащий домен VH, выбранный из группы, состоящей из SEQ ID NO: 38 и 46, и домен VL, выбранный из группы, состоящей из SEQ ID NO: 44, 48 и 50. В некоторых вариантах реализации полипептиды согласно настоящему изобретению могут содержать константную область иммуноглобулина, содержащую одну или более мутаций L234A, L235A, G237A и K322A в домене CH2 человеческого IgG1, и 41BB-связывающий домен, содержащий аминокислотную последовательность домена VH, представляющую собой SEQ ID NO: 14, и аминокислотную последовательность домена VL, представляющую собой SEQ ID NO: 16. В некоторых вариантах реализации полипептиды согласно настоящему изобретению могут содержать константную область иммуноглобулина, содержащую одну или более мутаций L234A, L235A, G237A и K322A в домене CH2 человеческого IgG1, и 5T4-связывающий домен, содержащий домен VH, выбранный из группы, состоящей из SEQ ID NO: 38 и 46, и домена VL, выбранного из группы, состоящей из SEQ ID NO: 44, 48 и 50; и 41BB-связывающий домен, содержащий аминокислотную последовательность домена VH, представляющую собой SEQ ID NO: 14, и аминокислотную последовательность домена VL, представляющую собой SEQ ID NO: 16.
[00181] Полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), могут быть получены с использованием скаффолдинга, как в общем раскрыто в публикациях патентных заявок США №№ 2013/0129723 и 2013/0095097, каждая из которых включена в настоящий документ посредством ссылки в полном объеме. Полипептиды, описанные в настоящем документе, могут содержать две неидентичные полипептидные цепи, каждая из которых содержит домен гетеродимеризации иммуноглобулина. Домены гетеродимеризации иммуноглобулина на поверхности контакта различаются. В одном из вариантов реализации домен гетеродимеризации иммуноглобулина содержит домен CH1 или его производное. В еще одном варианте реализации домен гетеродимеризации иммуноглобулина содержит домен CL или его производное. В одном из вариантов реализации домен CL представляет собой изотип Cκ или Cλ, или его производное.
[00182] В некоторых вариантах реализации полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) могут обладать улучшенными характеристиками по сравнению с другими 5T4-связывающими полипептидами или 4-1BB-связывающими полипептидами. Например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды или содержащие их полиспецифичные белки согласно настоящему изобретению могут обладать сниженной изоэлектрической точкой по сравнению с изоэлектрической точкой другого 5T4-связывающего полипептида и/или 4-1BB-связывающего полипептид. «Изоэлектрическая точка» или «pI» представляет собой pH, при котором суммарный заряд равен нулю. Изоэлектрическая точка белка может быть измерена любым подходящим способом, например, с помощью аналитической капиллярной хроматографии методом изоэлектрического фокусирования.
[00183] В некоторых вариантах реализации полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) могут связываться с 5T4 (например, 5T4 человека) и/или 4-1BB с более высокая аффинностью по сравнению с известными 5T4- и/или 4-1BB-связывающими доменами и/или исходным 5T4- и/или 4-1BB-связывающим доменом или белком. В некоторых вариантах реализации константа диссоциации 5T4- и/или 4-1BB-связывающего домена или полипептида может составлять приблизительно 2-5 нМ. В отдельных вариантах реализации скорость диссоциации 5T4- и/или 4-1BB-связывающего домена или полипептида может быть снижена в 4-10 раз по сравнению со скоростью диссоциации известного антитела или конструкции scFv, или исходного 5T4- и/или 4-1BB-связывающего домена или белка.
[00184] В некоторых вариантах реализации полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) могут характеризоваться низким уровнем высокомолекулярных агрегатов, образуемых в ходе рекомбинантной экспрессии полипептида или белка. В некоторых вариантах реализации полипептиды и белки, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) могут характеризоваться более длительной стабильностью в человеческой сыворотке, в зависимости от комбинации доменов, присутствующих в полипептиде или белке.
5T4-связывающие домены и содержащие их белки
[00185] В некоторых вариантах реализации настоящее изобретение относится к 5T4-связывающим доменам, специфично связывающимся с 5T4 (например, 5T4 человека). В некоторых вариантах реализации настоящее изобретение дополнительно относится к полипептидам, содержащим 5T4-связывающий домен (например, 5T4-связывающим полипептидам). В отдельных вариациях 5T4-связывающий полипептид содержит шарнирную область, являющуюся карбоксиконцевой относительно 5T4-связывающего домена, и константную область иммуноглобулина. В дополнительных вариациях 5T4-связывающий полипептид содержит карбоксиконцевой связывающий домен, линкер, являющийся карбоксиконцевым относительно константной области иммуноглобулина, и второй связывающий домен, являющийся карбоксиконцевым относительно карбоксиконцевого линкера. В других вариациях 5T4-связывающий полипептид содержит шарнирную область, аминоконцевую относительно полипептида, содержащего 5T4-связывающий домен, и константную область иммуноглобулина, аминоконцевую относительно шарнирной области.
[00186] В некоторых вариантах реализации 5T4-связывающие домены, описанные в настоящем документе, связывают эпитоп, расположенный на внеклеточном домене 5T4 (например, эпитоп, содержащийся в SEQ ID NO: 168). В отдельных аспектах этот эпитоп является прерывистым и/или конформационным.
[00187] Полипептид 5T4-связывающего домена может специфично связываться с 5T4 человека и содержать CDR1 тяжелой цепи (HCDR1), HCDR2, HCDR3, CDR1 легкой цепи (LCDR1), LCDR2 и LCDR3, где HCDR1 содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 30, 52 и 60; HCDR2 содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 32 и 62; HCDR3 содержит аминокислотную последовательность SEQ ID NO: 34; LCDR1 содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 8, 42 и 54; LCDR2 содержит аминокислотную последовательность SEQ ID NO: 10; и LCDR3 содержит аминокислотную последовательность SEQ ID NO: 36.
[00188] В частных вариантах реализации полипептид 5T4-связывающего домена может специфично связываться с 5T4 человека и содержать HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 30; (b) HCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 32; (c) HCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 34; (d) LCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 8; LCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 10; и (f) LCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 36. В других частных вариантах реализации полипептид 5T4-связывающего домена специфично связывается с 5T4 человека и содержит HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 30; (b) HCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 32; (c) HCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 34; (d) LCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 42; (e) LCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 10; и (f) LCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 36. В других частных вариантах реализации полипептид 5T4-связывающего домена специфично связывается с 5T4 человека и содержит HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 52; (b) HCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 32; (c) HCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 34; (d) LCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 54; (e) LCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 10; и (f) LCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 36. В других частных вариантах реализации полипептид 5T4-связывающего домена специфично связывается с 5T4 человека и содержит HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 60; (b) HCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 62; (c) HCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 34; (d) LCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 54; (e) LCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 10; и (f) LCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 36.
[00189] В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит или представляет собой последовательность, по меньшей мере приблизительно на 85%, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98%, по меньшей мере приблизительно на 99%, по меньшей мере приблизительно на 99,5% или на 100% идентичную аминокислотной последовательности вариабельной области легкой цепи (VL), выбранной из группы, состоящей из SEQ ID NO: 40, 44, 48, 50, 58, 68 и 70, где указанный полипептид представляет собой полиспецифичный полипептид в формате scFv-Fc-scFv и содержит замену Y на F в LCDR1 в положении 99 VL и/или замену F на S в FR3 в положении 148 VL. В отдельных вариантах реализации полипептид, содержащий 5T4-связывающий домен, содержит аминокислотную последовательность вариабельной области легкой цепи (VL), выбранную из группы, состоящей из SEQ ID NO: 40, 44, 48, 50, 58, 68 и 70. В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит аминокислотную последовательность вариабельной области тяжелой цепи (VH), выбранную из группы, состоящей из SEQ ID NO: 38, 46, 56 и 64.
[00190] В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 38, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 40. В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 38, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 44. В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 46, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 48. В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 46, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 50. В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 56, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 58. В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 64, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 58. В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 38, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 68. В отдельных вариантах реализации полипептид 5T4-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 46, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 70.
[00191] Иллюстративные последовательности связывающих доменов к 5Т4 представлены в таблице 5.
Таблица 5. Аминокислотные последовательности и последовательности ДНК CDR и вариабельной области полипептида 5T4-связывающего домена
ALG.APV-208
ALG.APV-209
ALG.APV-222
ALG.APV-210
ALG.APV-223
ALG.APV-198
ALG.APV-010
ALG.APV-018
[00192] В некоторых вариантах реализации настоящее изобретение относится к полиспецифичным полипептидам (например, биспецифичным полипептидам), содержащим 5T4-связывающий домен. В таких вариантах реализации 5T4-связывающий белок или полипептид может содержать один или более дополнительных связывающих доменов (например, второй связывающий домен), связывающих мишень, отличную от 5T4. Эти другие связывающие домены могут содержать, например, определенный цитокин или молекулу, которая нацеливает полипептид связывающего домена, например, на конкретный тип клеток, токсин, дополнительный клеточный рецептор или антитело. Полиспецифичный 5T4-связывающий полипептид или белок может содержать два связывающих домена (домены могут быть сконструированы для специфичного связывания одной и той же или разными мишенями), шарнирную область, линкер (например, карбоксиконцевой или аминоконцевой линкер) и константную область иммуноглобулина. Полиспецифичный 5T4-связывающий белок может представлять собой гомодимерный белок, содержащий два идентичных полипептида, связанных дисульфидной связью. В некоторых вариантах реализации 5T4-связывающие домены могут быть получены из моноклонального антитела, связывающегося с 5T4.
[00193] В некоторых вариантах реализации изобретения 5T4-связывающий полипептид способен образовывать гетеродимер со второй полипептидной цепью и содержит шарнирную область, (а) непосредственно примыкающую с аминоконца к константной области иммуноглобулина (например, аминоконцевую относительно домена СН2, если константная область иммуноглобулина включает домены CH2 и CH3, или аминоконцевую относительно домена CH3, если субобласти иммуноглобулина включают домены CH3 и CH4), (b) расположенную между связывающим доменом (например, scFv) и доменом гетеродимеризации иммуноглобулина, и соединяющую их, (c) расположенную между доменом гетеродимеризации иммуноглобулина и константной областью иммуноглобулина (например, когда константная область иммуноглобулина включает домены CH2 и CH3 или домены CH3 и CH4), и соединяющую их, (d) расположенную между константной областью иммуноглобулина и связывающим доменом, и соединяющую их, (е) на аминоконце полипептидной цепи или (f) на карбоксиконце полипептидной цепи. Полипептидная цепь, содержащая шарнирную область, как описано в настоящем документе, будет способна связываться с другой полипептидной цепью с образованием гетеродимерного белка, предложенного в настоящем документе, и образованный гетеродимер будет содержать связывающий домен, сохраняющий свою специфичность к мишени или свою специфичную аффинность связывания с мишенью.
[00194] В некоторых вариантах реализации 5T4-связывающий полипептид, предложенный в настоящем документе, представляет собой полипептид, содержащий два scFv. В некоторых вариантах реализации два scFv содержат 5T4-связывающие домены. В отдельных вариантах реализации два scFv содержат идентичные 5T4-связывающие домены. В других вариантах реализации два scFv содержат разные 5T4-связывающие домены. В других вариантах реализации полипептид содержит 5T4-связывающий домен в качестве первого scFv и связывающий для домен эффекторных клеток в качестве второго scFv. Например, связывающий домен для эффекторных клеток может представлять собой scFv, специфичный к 4-1BB.
[00195] В некоторых вариантах реализации 5T4-связывающий полипептид, предложенный в настоящем документе, содержит scFv к 5T4, по меньшей мере приблизительно на 82%, по меньшей мере приблизительно на 85%, по меньшей мере приблизительно на 87%, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98%, по меньшей мере приблизительно на 99% или на 100% идентичный аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 118, 120, 122, 124, 126, 128, 130, 132, 134 и 170, где указанный полипептид представляет собой полиспецифичный полипептид в формате scFv-Fc -scFv и содержит замену Y на F в LCDR1 в положении 99 VL к 5T4 и/или замену F на S в FR3 в положении 148 VL к 5T4. В некоторых вариантах реализации 5T4-связывающий полипептид, предложенный в настоящем документе, содержит scFv к 5T4, содержащий аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 118, 120, 122, 124, 126, 128, 130, 132, 134 и 170. Аминокислотные и нуклеотидные последовательности иллюстративных scFv к 5T4 представлены ниже в таблице 6.
Таблица 6. Аминокислотные последовательности и последовательности ДНК иллюстративных scFv к 5T4
ALG.APV-208
к 5T4
ALG.APV.209
ALG.APV222
к 5T4
ALG.APV.210
ALG.APV223
к 5T4
к 5T4
к 5T4
ALG.APV-198
к 5T4
к 5T4
к 5T4
к 5T4
к 5T4
к 5T4
[00196] В некоторых вариантах реализации 5T4-связывающий полипептид содержит, в порядке от аминоконца к карбоксиконцу (или в порядке от карбоксиконца к аминоконцу), (i) 5T4-связывающий домен, (ii) шарнирную область (iii) константную область иммуноглобулина, (iv) карбоксиконцевой линкер (или аминоконцевой линкер) и (v) второй связывающий домен. В дополнительных вариантах реализации второй связывающий домен представляет собой scFv, специфичный к 4-1BB. В некоторых вариантах реализации 5T4-связывающий полипептид содержит, в порядке от аминоконца к карбоксиконцу (или в порядке от карбоксиконца к аминоконцу), (i) второй связывающий домен, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) карбоксиконцевой линкер (или аминоконцевой линкер) и (v) 5T4-связывающий домен. В дополнительных вариантах реализации второй связывающий домен содержит или представляет собой 4-1BB-связывающий домен. В отдельных вариантах реализации 5T4-связывающий белок может содержать связывающий домен для эффекторных клеток для рекрутирования эффектора к клеткам-мишеням, экспрессирующим 5T4. В отдельных вариантах реализации связывающий домен для эффекторных клеток специфично связывается с 4-1BB.
[00197] В некоторых вариантах реализации второй связывающий домен 5T4-связывающего полипептида, описанного в настоящем документе, представляет собой 4-1BB-связывающий домен и содержит одну или более 4-1BB-связывающих последовательностей (например, CDR или вариабельных областей), раскрытых в публикации заявки PCT № WO 2016/185016; заявке PCT № PCT/EP2017/059656; Dubrot et al., 2010; Gauttier et al., 2014; Kim et al., 2001; McMillin et al., 2006; Melero et al., 1997; Miller et al., 2002; Sallin et al., 2014; Taraban et al., 2002; Uno et al., 2006; Vinay and Kwon, 2012; Wilcox et al., 2002, каждый из которых включен в настоящий документ посредством ссылки в полном объеме.
[00198] В некоторых вариантах реализации второй связывающий домен специфично связывает 4-1BB и содержит вариабельную область легкой цепи (VL) иммуноглобулина и вариабельную область тяжелой цепи иммуноглобулина (VH); где VL содержит аминокислотную последовательность, по меньшей мере приблизительно на 93% идентичную, по меньшей мере приблизительно на 95% идентичную, по меньшей мере приблизительно на 97% идентичную, по меньшей мере приблизительно на 98% идентичную, по меньшей мере приблизительно на 99% идентичную или на 100% идентичную аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 16 и 22; и где VH содержит аминокислотную последовательность, по меньшей мере приблизительно на 93% идентичную, по меньшей мере приблизительно на 95% идентичную, по меньшей мере приблизительно на 97% идентичную, по меньшей мере приблизительно на 98% идентичную, по меньшей мере приблизительно на 99% идентичную или на 100% идентичную аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 14, 20, 26 и 28, где полипептид представляет собой полиспецифичный полипептид в формате scFv-Fc-scFv и содержит замену Y на H в HCDR1 в положении 150 VH и/или замену R на H в FR3 в положении 127 VH. В некоторых вариантах реализации второй связывающий домен специфично связывает 4-1BB и содержит VL и VH; где VL содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 16 и 22; и где VH содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 14, 20, 26 и 28.
[00199] В некоторых вариантах реализации второй связывающий домен представляет собой полипептид 4-1BB-связывающего домена, где указанный полипептид представляет собой scFv, содержащий последовательность, по меньшей мере приблизительно на 82%, по меньшей мере приблизительно на 85%, по меньшей мере приблизительно на 87%, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98%, по меньшей мере приблизительно на 99% или на 100% идентичную аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 110, 112, 114 и 116. Иллюстративные последовательности связывающего домена к 4-1BB представлены в таблицах 6 и 7 и обсуждаются ниже.
4T1-связывающие домены и содержащие их белки
[00200] В некоторых вариантах реализации настоящее изобретение относится к 4-1BB-связывающим доменам, специфично связывающимся с 4-1BB (например, 4-1BB человека). 4-1BB также известен как CD137 или TNFRSF9 и является членом семейства рецепторов фактора некроза опухолей (TNF). 4-1BB экспрессируется на множестве типов клеток, включая активированные субпопуляции T-клеток (например, CD4+ и CD8+ T-клетки и регуляторные T-клетки (Treg)), естественные киллеры (NK), дендритные клетки, моноциты, тучные клетки и эозинофилы. Активация 4-1BB на CD8+ T-клетках поддерживает и/или усиливает активацию клеток, тогда как активация 4-1BB на CD4+ T-клетках может инициировать активацию клеток и, в некоторых случаях, приводить к гибели клеток, вызванной активацией. Активация 4-1BB на Treg обычно подавляет функцию Treg. В нескольких исследованиях была продемонстрирована индукция опухолевого иммунитета посредством применения антител-антагонистов 4-1ВВ. Иллюстративные нуклеотидные и аминокислотные последовательности человеческого 4-1BB представлены в SEQ ID NO: 163, 167 и SEQ ID NO: 164 и 166, соответственно.
[00201] В некоторых вариантах реализации настоящее изобретение дополнительно относится к полипептидам, содержащим 4-1BB-связывающий домен (например, 4-1BB-связывающим полипептидам). В отдельных вариациях 4-1BB-связывающий полипептид содержит шарнирную область, являющуюся карбоксиконцевой относительно 4-1BB-связывающего домена, и константную область иммуноглобулина. В дополнительных вариациях 4-1BB-связывающий полипептид содержит карбоксиконцевой связывающий домен, линкер, являющийся карбоксиконцевым относительно константной области иммуноглобулина, и второй связывающий домен, являющийся карбоксиконцевым относительно карбоксиконцевого линкера.
[00202] В других вариациях 4-1BB-связывающий полипептид содержит шарнирную область, аминоконцевую относительно полипептида, содержащего 4-1BB-связывающий домен, и константную область иммуноглобулина, аминоконцевую относительно шарнирной области.
[00203] В некоторых вариантах реализации 4-1BB-связывающие домены, описанные в настоящем документе, связывают эпитоп, расположенный на внеклеточном домене 4-1BB (например, эпитоп, содержащийся в SEQ ID NO: 166). В отдельных аспектах этот эпитоп является прерывистым и/или конформационным.
[00204] Полипептид 4-1BB-связывающего домена может специфично связываться с 4-1BB человека и содержать CDR1 тяжелой цепи (HCDR1), HCDR2, HCDR3, CDR1 легкой цепи (LCDR1), LCDR2 и LCDR3, где HCDR1 содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 2, 18, 24; HCDR2 содержит аминокислотную последовательность SEQ ID NO: 4; HCDR3 содержит аминокислотную последовательность SEQ ID NO: 6; LCDR1 содержит аминокислотную последовательность SEQ ID NO: 8; LCDR2 содержит аминокислотную последовательность SEQ ID NO: 10; и LCDR3 содержит аминокислотную последовательность SEQ ID NO: 12, где указанный полипептид представляет собой полиспецифичный полипептид в формате scFv-Fc-scFv и содержит замену Y на H в HCDR1 в положении 150 VH к 4-1BB и/или замену R на H в FR3 в положении 127 VH к 4-1BB.
[00205] В частных вариантах реализации полипептид 4-1BB-связывающего домена может специфично связываться с 4-1BB человека и содержать HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 2; (b) HCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 4; (c) HCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 6; (d) LCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 8; LCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 10; и (f) LCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 12. В других частных вариантах реализации полипептид 5T4-связывающего домена специфично связывается с 5T4 человека и содержит HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 18; (b) HCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 4; (c) HCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 6; (d) LCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 8; (e) LCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 10, и (f) LCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 12. В других частных вариантах реализации полипептид 5T4-связывающего домена специфично связывается с 5T4 человека и содержит HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3, где (a) HCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 24; (b) HCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 4; (c) HCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 6; (d) LCDR1 содержит аминокислотную последовательность, представленную в SEQ ID NO: 8; (e) LCDR2 содержит аминокислотную последовательность, представленную в SEQ ID NO: 10; и (f) LCDR3 содержит аминокислотную последовательность, представленную в SEQ ID NO: 12.
[00206] В отдельных вариантах реализации полипептид 4-1BB-связывающего домена содержит аминокислотную последовательность вариабельной области легкой цепи (VL), выбранную из группы, состоящей из SEQ ID NO: 16 и 22. В отдельных вариантах реализации полипептид 4-1BB-связывающего домена содержит аминокислотную последовательность, по меньшей мере приблизительно на 85%, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98%, по меньшей мере приблизительно на 99%, по меньшей мере приблизительно на 99,5% или на 100% идентичную аминокислотной последовательности вариабельной области тяжелой цепи (VH), выбранной из группы, состоящей из SEQ ID NO: 14, 20, 26 и 28, где указанный полипептид представляет собой полиспецифичный полипептид в формате scFv-Fc-scFv и содержит замену Y на H в HCDR1 в положении 150 VH к 4-1BB и/или замену R на H в FR3 в положении 127 VH к 4-1BB. В отдельных вариантах реализации полипептид 4-1BB-связывающего домена содержит аминокислотную последовательность вариабельной области тяжелой цепи (VH), выбранную из группы, состоящей из SEQ ID NO: 14, 20, 26 и 28.
[00207] В отдельных вариантах реализации полипептид 4-1BB-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 14, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 16. В отдельных вариантах реализации полипептид 4-1BB-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 20, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 22. В отдельных вариантах реализации полипептид 4-1BB-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 26, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 16. В отдельных вариантах реализации полипептид 4-1BB-связывающего домена содержит область VH, содержащую аминокислотную последовательность SEQ ID NO: 28, и область VL, содержащую аминокислотную последовательность SEQ ID NO: 16.
[00208] Иллюстративные последовательности связывающих доменов к 4-1BB представлены в таблице 7.
Таблица 7. Иллюстративные полипептидные и нуклеотидные последовательности 4-1BB-связывающего домена
ALG.APV-179
ALG.APV-187
ALG.APV-198
ALG.APV-199
ALG.APV-208
ALG.APV-209
ALG.APV-210
ALG.APV-222
ALG.APV-223
ALG.APV-006
ALG.APV-010
ALG.APV-014
ALG.APV-018
[00209] В некоторых вариантах реализации настоящее изобретение относится к полиспецифичным полипептидам (например, биспецифичным полипептидам), содержащим 4-1BB-связывающий домен. Полиспецифичный 4-1BB-связывающий полипептид или белок может содержать два связывающих домена (домены могут быть сконструированы для специфичного связывания одной и той же или разными мишенями), шарнирную область, линкер (например, карбоксиконцевой или аминоконцевой линкер) и константную область иммуноглобулина. Полиспецифичный 4-1BB-связывающий белок может представлять собой гомодимерный белок, содержащий два идентичных полипептида, связанных дисульфидной связью. В некоторых вариантах реализации 4-1BB-связывающие домены могут быть получены из моноклонального антитела, связывающегося с 4-1BB (например, урелумаба (BMS-66513) или PF-05082566 (Pfizer)), получены из 4-1BB-связывающего домена, описанного в публикации заявки PCT № WO 2016/185016, или получены из 4-1BB-связывающего домена согласно заявке PCT № PCT/EP2017/059656.
[00210] В некоторых вариантах реализации полиспецифичный полипептид, содержащий 4-1BB-связывающий домен, может содержать, в порядке от аминоконца к карбоксиконцу: (i) первый связывающий домен, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) 4-1BB-связывающий домен. В некоторых вариантах реализации полиспецифичный полипептид, содержащий 4-1BB-связывающий домен, может содержать, в порядке от аминоконца к карбоксиконцу: (i) 4-1BB-связывающий домен, (ii) линкер связывающих доменов, (iii) константную область иммуноглобулина, (iv) шарнирную область и (v) первый связывающий домен. Аминокислотные и нуклеотидные последовательности для иллюстративных последовательностей связывающего домена к 4-1BB представлены в таблице 7.
[00211] В отдельных вариантах реализации 4-1BB-связывающий белок или полипептид может содержать один или более дополнительных связывающих доменов (например, второй связывающий домен), связывающих мишень, отличную от 4-1BB. Эти другие связывающие домены могут содержать, например, определенный цитокин или молекулу, которая нацеливает полипептид связывающего домена, например, на конкретный тип клеток, токсин, дополнительный клеточный рецептор или антитело.
[00212] В некоторых вариантах реализации изобретения 4-1BB-связывающий полипептид способен образовывать гетеродимер со второй полипептидной цепью и содержит шарнирную область, (а) непосредственно примыкающую с аминоконца к константной области иммуноглобулина (например, аминоконцевую относительно домена СН2, если константная область иммуноглобулина включает домены CH2 и CH3, или аминоконцевую относительно домена CH3, если субобласти иммуноглобулина включают домены CH3 и CH4), (b) расположенную между связывающим доменом (например, scFv) и доменом гетеродимеризации иммуноглобулина, и соединяющую их, (c) расположенную между доменом гетеродимеризации иммуноглобулина и константной областью иммуноглобулина (например, когда константная область иммуноглобулина включает домены CH2 и CH3 или домены CH3 и CH4), и соединяющую их, (d) расположенную между константной областью иммуноглобулина и связывающим доменом, и соединяющую их, (е) на аминоконце полипептидной цепи или (f) на карбоксиконце полипептидной цепи. Полипептидная цепь, содержащая шарнирную область, как описано в настоящем документе, будет способна связываться с другой полипептидной цепью с образованием гетеродимерного белка, предложенного в настоящем документе, и образованный гетеродимер будет содержать связывающий домен, сохраняющий свою специфичность к мишени или свою специфичную аффинность связывания с мишенью.
[00213] В отдельных вариантах реализации 4-1BB-связывающий полипептид может содержать связывающий домен клеток-мишеней для рекрутирования клеток-мишеней к эффекторным клеткам, экспрессирующим 4-1BB. В отдельных вариантах реализации связывающий домен клеток-мишеней специфично связывается с 5T4. В отдельных вариантах реализации 4-1BB-связывающий белок, как описано в настоящем документе, может содержать (i) связывающий домен, специфично связывающий 4-1BB, и (ii) другой связывающий домен, специфично связывающийся с 5T4. Неограничивающие примеры антител к 5T4, из которых может быть получен 5T4-связывающий домен, включают описанные в публикации заявки PCT №. WO 2016/185016 и заявке PCT № PCT/EP2017/059656.
[00214] В некоторых вариантах реализации 4-1BB-связывающий полипептид, предложенный в настоящем документе, представляет собой полипептид, содержащий два scFv. В некоторых вариантах реализации два scFv содержат 4-1BB-связывающие домены. В отдельных вариантах реализации два scFv содержат идентичные 4-1BB-связывающие домены. В других вариантах реализации два scFv содержат разные 4-1BB-связывающие домены. В других вариантах реализации полипептид содержит 4-1BB-связывающий домен в качестве первого scFv и связывающий домен клеток-мишеней в качестве второго scFv. Например, связывающий домен клеток-мишеней может представлять собой scFv, специфичный к 5T4.
[00215] В некоторых вариантах реализации биспецифичные 4-1BB-связывающие полипептиды и белки, предложенные в настоящем документе, содержат scFv к 4-1BB, по меньшей мере приблизительно на 82%, по меньшей мере приблизительно на 85%, по меньшей мере приблизительно на 87%, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98%, по меньшей мере приблизительно на 99% или на 100% идентичный аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 110, 112, 114 и 116, где указанный полипептид представляет собой полиспецифичный полипептид в формате scFv-Fc-scFv и содержит замену Y на H в HCDR1 в положении 150 VH к 4-1BB и/или замену R на H в FR3 в положении 127 VH к 4-1BB. В отдельных вариантах реализации биспецифичные 4-1BB-связывающие полипептиды и белки, предложенные в настоящем документе, содержат scFv к 4-1BB, содержащий аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 110, 112, 114 и 116. Аминокислотные и нуклеотидные последовательности иллюстративных scFv к 4-1BB представлены ниже в таблице 8.
Таблица 8. Аминокислотные последовательности и последовательности ДНК иллюстративных scFv к 4-1BB
ALG.APV-179-4-1BB
ALG.APV-187-4-1BB
ALG.APV-198-4-1BB
ALG.APV-199-4-1BB
ALG.APV-208-4-1BB
ALG.APV-209-4-1BB
ALG.APV-210-4-1BB
ALG.APV-222-4-1BB
ALG.APV-223-4-1BB
ADSVKGRFTISHDNSKNTLYLQMNSLRAEDTAVYYCARSSYYGSYYSIDYWGQGTLVTVS
SGGGGSGGGGSGGGGSGGGGSDIQMTQSPSSLSASVGDRVTITCRASQSISSYLNWYQQK
PGKAPKLLIYAASSLQSGVPSRFSGSGSGTDFTLTISSLQPEDFATYYCQQYYDNLPTFG
QGTKLEIK
ALG.APV-010-4-1BB
ALG.APV-014-4-1BB
ALG.APV-018-4-1BB
Биспецифичные молекулы
[00216] В некоторых вариантах реализации настоящее изобретение относится к полиспецифичным 5T4-связывающим полипептидам, содержащим два связывающих домена, где один из связывающих доменов специфичен к 5T4, а второй связывающий домен специфичен к другому антигену-мишени. В некоторых вариантах реализации настоящее изобретение относится к биспецифичным 4-1BB-связывающим полипептидам, содержащим два связывающих домена, где связывающий домен специфичен к 4-1BB, а второй связывающий домен специфичен к другому антигену-мишени. В частных вариантах реализации настоящее изобретение относится к биспецифичным полипептидам, содержащим два связывающих домена, где один из связывающих доменов специфичен к 4-1BB, а второй связывающий домен специфичен к 5T4. Такие варианты реализации называются в настоящем документе молекулами к 5T4 x 4-1BB или полипептидами или белками к 5T4 x 4-1BB. В частных вариантах реализации 5T4- и 4-1BB-связывающие домены молекулы к 5T4 x 4-1BB представляют собой scFv-домены, специфично связывающиеся с 5T4 и 4-1BB, соответственно.
[00217] В некоторых вариантах реализации молекулы к 5T4 x 4-1BB, описанные в настоящем документе, содержат, в порядке от аминоконца к карбоксиконцу, (i) 5T4-связывающий домен; (ii) линкер связывающих доменов; и (iii) 4-1BB-связывающий домен. В некоторых вариантах реализации молекулы к 5T4 x 4-1BB, описанные в настоящем документе, содержат, в порядке от аминоконца к карбоксиконцу, (i) 4-1BB-связывающий домен; (ii) линкер связывающих доменов; и (iii) 5T4-связывающий домен. В некоторых вариантах реализации молекулы к 5T4 x 4-1BB, описанные в настоящем документе, содержат, в порядке от аминоконца к карбоксиконцу (или в порядке от карбоксиконца к аминоконцу), (i) 5T4-связывающий домен, (ii) шарнирную область (iii) константную область иммуноглобулина, (iv) карбоксиконцевой линкер (или аминоконцевой линкер) и (v) второй связывающий домен.
[00218] Молекулы к 5Т4 х 4-1BB, описанные в настоящем документе, могут содержать любую комбинацию последовательностей связывающих доменов к 5T4 и к 4-1BB, представленных в таблицах 4-7. В частных вариантах реализации молекулы к 5T4 x 4-1BB согласно настоящему изобретению содержат 5T4-связывающий домен и 4-1BB-связывающий домен, каждый из которых содержит HCDR1, HCDR2, HCDR3, LCDR1, LCDR2 и LCDR3. В некоторых вариантах реализации молекулы к 5Т4 х 4-1ВВ содержат (а) первый scFv-домен, содержащий: (i) VH, содержащую аминокислотную последовательность HCDR1, выбранную из группы, состоящей из SEQ ID NO: 30, 52 и 60, аминокислотную последовательность HCDR2, выбранную из группы, состоящей из SEQ ID NO: 32 и 62, и аминокислотную последовательность HCDR3, представляющую собой SEQ ID NO: 34; и (ii) VL, содержащую аминокислотную последовательность LCDR1, выбранную из группы, состоящей из SEQ ID NO: 8, 42 и 54, аминокислотную последовательность LCDR2, представляющую собой SEQ ID NO: 10, и аминокислотную последовательность LCDR3, представляющую собой SEQ ID NO: 36; и (b) второй scFv-домен, содержащий (i) VH, содержащую аминокислотную последовательность HCDR1, выбранную из группы, состоящей из SEQ ID NO: 2, 18 и 24, аминокислотную последовательность HCDR2, представляющую собой SEQ ID NO: 4, и аминокислотную последовательность HCDR3, представляющую собой SEQ ID NO: 6; и (ii) VL, содержащую аминокислотную последовательность LCDR1 SEQ ID NO: 8, аминокислотную последовательность LCDR2, представляющую собой SEQ ID NO: 10, и аминокислотную последовательность LCDR3, представляющую собой SEQ ID NO: 12.
[00219] В некоторых вариантах реализации молекулы к 5T4 x 4-1BB согласно настоящему изобретению содержат 5T4-связывающий домен и 4-1BB-связывающий домен, каждый из которых содержит домен VH и VL. В некоторых вариантах реализации молекулы к 5T4 x 4-1BB содержат первый scFv-домен, содержащий i) VH, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 38, 46, 56 и 64, и VL, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 40, 44, 48, 50 58, 66, 68 и 70; и второй scFv-домен, содержащий ii) VH, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 14, 20, 26 и 28, и VL, содержащую аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 16 и 22.
[00220] В отдельных вариантах реализации 5T4-связывающий домен молекулы к 5T4 x 4-1BB содержит или представляет собой scFv, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98%, по меньшей мере приблизительно на 99%, по меньшей мере приблизительно на 99,5% или на 100% идентичный аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 118, 120, 122, 124, 126, 128, 130, 132, 134 и 170, где указанная молекула представляет собой полиспецифичный полипептид в формате scFv-Fc-scFv и содержит замену Y на F в LCDR1 в положении 99 VL к 5T4 и/или замену F на S в FR3 в положении 148 VL к 5T4. В некоторых вариантах реализации 5T4-связывающий домен молекулы к 5T4 x 4-1BB содержит scFv, содержащий аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 118, 120, 122, 124, 126, 128, 130, 132, 134 и 170.
[00221] В отдельных вариантах реализации 4-1BB-связывающий домен молекулы 5T4 x 4-1BB содержит или представляет собой scFv, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98%, по меньшей мере приблизительно на 99%, по меньшей мере приблизительно на 99,5% или на 100% идентичный аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 110, 112, 114, и 116, где указанная молекула представляет собой полиспецифичный полипептид в формате scFv-Fc-scFv и содержит замену Y на H в HCDR1 в положении 150 VH к 4-1BB и/или замену R на H в FR3 в положении 127 VH к 4-1BB. В отдельных вариантах реализации 4-1BB-связывающий домен молекулы к 5T4 x 4-1BB содержит scFv, содержащий аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 110, 112, 114 и 116.
[00222] В частных вариантах реализации молекула к 5T4 x 4-1BB содержит комбинацию последовательностей CDR, последовательностей VH/VL и/или последовательностей scFv, как представлено ниже в таблице 9.
Таблица 9. Иллюстративные комбинации связывающих последовательностей к 5Т4 и к 4-1BB
[00223] В некоторых вариантах реализации молекулы к 5Т4 х 4-1BB, описанные в настоящем документе, могут содержать аминокислотную последовательность, по меньшей мере приблизительно на 90%, по меньшей мере приблизительно на 91%, по меньшей мере приблизительно на 92%, по меньшей мере приблизительно на 93%, по меньшей мере приблизительно на 94%, по меньшей мере приблизительно на 95%, по меньшей мере приблизительно на 96%, по меньшей мере приблизительно на 97%, по меньшей мере приблизительно на 98%, по меньшей мере приблизительно на 99%, по меньшей мере приблизительно на 99,5% или на 100% идентичную аминокислотной последовательности, выбранной из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176. В некоторых вариантах реализации молекулы к 5Т4 х 4-1ВВ, описанные в настоящем документе, могут содержать аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176. Иллюстративные аминокислотные и нуклеотидные последовательности молекул к 5Т4 х 4-1BB, описанных в настоящем документе, представлены в таблицах 10 и 11. В каждой из таблиц 10 и 11 Fc-домены иммуноглобулина (шарнир - CH1 - CH2) обозначены подчеркнутым текстом, а последовательности линкеров связывающих доменов обозначены жирным шрифтом.
Таблица 10. Иллюстративные последовательности ДНК молекулы к 5Т4 х 4-1BB
ALG.APV-222
ALG.APV-223
Таблица 11. Иллюстративные аминокислотные последовательности молекулы к 5Т4 х 4-1BB
ALG.APV-222
ALG.APV-223
Полинуклеотиды и способы экспрессии белка
[00224] Изобретение также относится к нуклеиновым кислотам (например, ДНК или РНК), кодирующим полипептиды согласно настоящему изобретению (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) или одну или более полипептидных цепей полипептида, как описано в настоящем документе. Нуклеиновые кислоты согласно изобретению включают нуклеиновые кислоты, имеющие область, по существу идентичную полинуклеотиду из перечисленных в таблицах 1-10 ниже. В отдельных вариантах реализации нуклеиновая кислота согласно настоящему изобретению обладает по меньшей мере 80%, обычно по меньшей мере приблизительно 90%, и чаще по меньшей мере приблизительно 95% или по меньшей мере приблизительно 98% идентичностью полинуклеотиду, кодирующему полипептид, из перечисленных в таблицах 1-10, где нуклеиновая кислота кодирует полипептид, представляющий собой полиспецифичный полипептид в формате scFv-Fc-scFv, и где кодируемый полипептид содержит одну или более из (1) замены Y на F в LCDR1 в положении 99 VL к 5T4; (2) замены F на S в FR3 в положении 148 VL к 5T4; (3) замены Y на H в HCDR1 в положении 150 VH к 4-1BB; и (4) замены R на H в FR3 в положении 127 VH к 4-1BB. Нуклеиновые кислоты согласно изобретению также включают комплементарные нуклеиновые кислоты. В некоторых случаях последовательности будут полностью комплементарными (без несовпадений) при выравнивании. В других случаях последовательности могут не совпадать вплоть до приблизительно 20%. В некоторых вариантах реализации изобретения предложены нуклеиновые кислоты, кодирующие как первую, так и вторую полипептидные цепи биспецифичного белка согласно изобретению. Последовательности нуклеиновых кислот, предложенные в настоящем документе, могут быть использованы с использованием оптимизации кодонов, вырожденных последовательностей, молчащих мутаций и других методик в отношении ДНК для оптимизации экспрессии в конкретном хозяине, и настоящее изобретение включает такие модификации последовательности.
[00225] Изобретение относится к выделенной молекуле нуклеиновой кислоты, кодирующей полипептиды согласно настоящему изобретению (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), где указанная молекула нуклеиновой кислоты содержит нуклеотидную последовательность, выбранную из SEQ ID NO: 109, 111, 113, 115, 117, 119, 121, 123, 125, 127, 129, 131, 133, 135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 171, 173 и 175.
[00226] Молекулы полинуклеотидов, содержащие желаемую полинуклеотидную последовательность, репродуцируют, помещая молекулу в вектор. Используют вирусные и невирусные векторы, включая плазмиды. Выбор плазмиды будет зависеть от типа клетки, в которой желательно репродуцирование, и цели репродуцирования. Некоторые векторы используются для усиления и получения больших количеств желаемой последовательности ДНК. Другие векторы подходят для экспрессии в клетках в культуре. Другие векторы подходят для переноса и экспрессии в клетках в животном или человеке в целом. Выбор подходящего вектора находится в пределах компетенции специалиста в данной области техники. Многие такие векторы являются коммерчески доступными. Частичный или полноразмерный полинуклеотид обычно вставляют в вектор посредством присоединения ДНК-лигазы к расщепленному сайту рестрикции в векторе. В качестве альтернативы, желаемая нуклеотидная последовательность может быть вставлена путем гомологичной рекомбинации in vivo. Обычно это осуществляют путем присоединения областей гомологии к вектору по краям желаемой нуклеотидной последовательности. Области гомологии добавляют путем лигирования олигонуклеотидов или, например, с помощью полимеразной цепной реакции с использованием праймеров, содержащих как область гомологии, так и часть желаемой нуклеотидной последовательности.
[00227] Для экспрессии может быть использована кассета или система экспрессии. Для экспрессии нуклеиновой кислоты, кодирующей полипептид, раскрытый в настоящем документе, молекулу нуклеиновой кислоты, кодирующую указанный полипептид, функционально связанную с регуляторными последовательностями, контролирующими транскрипционную экспрессию в векторе экспрессии, вводят в клетку-хозяина. Помимо регуляторных последовательностей транскрипции, таких как промоторы и энхансеры, векторы экспрессии могут включать регуляторные последовательности трансляции и маркерный ген, подходящий для отбора клеток, несущих вектор экспрессии. Генный продукт, кодируемый полинуклеотидом согласно изобретению, экспрессируется в любой подходящей системе экспрессии, включая, например, систему бактерии, дрожжей, насекомого, амфибии и млекопитающего. В векторе экспрессии полинуклеотид, кодирующий полипептид, связан с регуляторной последовательностью соответствующим образом для получения желаемых свойств экспрессии. Такие последовательности могут включать промоторы, энхансеры, терминаторы, операторы, репрессоры и индукторы. Промоторы могут быть регулируемыми (например, промотор из индуцируемого стероидами вектора pIND (Invitrogen)) или конститутивными (например, промоторы из последовательностей CMV, SV40, фактора элонгации или LTR). Они связаны с желаемой нуклеотидной последовательностью с помощью методов, описанных выше для связывания с векторами. Могут использоваться любые методы, известные в данной области техники. Соответственно, вектор экспрессии обычно будет обеспечивать область инициации транскрипции и трансляции, которая может быть индуцибельной или конститутивной, где кодирующая область функционально связана под контролем транскрипции области инициации транскрипции и области терминации транскрипции и трансляции.
[00228] Кассета экспрессии («единица экспрессии») может быть введена в различные векторы, например, плазмиду, ВАС, YAC, бактериофаг, такой как лямбда, P1, M13 и т. д., векторы на основе вирусов растений или животных (например, векторы на основе ретровирусов, аденовирусные векторы) и тому подобное, где векторы обычно характеризуются способностью обеспечивать отбор клеток, содержащих векторы экспрессии. Векторы могут обеспечивать поддержание во внехромосомном состоянии, особенно в виде плазмид или вирусов, или встраивание в хромосому хозяина. Если требуется внехромосомное поддержание, для репликации плазмиды обеспечивается последовательность точки начала репликации, которая может иметь низкое или высокое число копий. Для выбора доступно большое количество различных маркеров, в частности обеспечивающих защиту от токсинов, в частности, от антибиотиков. Выбор конкретного маркера осуществляют в зависимости от природы хозяина, где в некоторых случаях с ауксотрофными хозяевами может использоваться комплементация. Для введения конструкции ДНК может быть использован любой удобный способ, включая, например, конъюгацию, бактериальную трансформацию, осаждение ДНК с помощью кальция, электропорацию, слияние, трансфекцию, инфицирование вирусными векторами, биолистическую трансфекцию и тому подобное. Изобретение относится к вектору экспрессии, содержащему сегмент нуклеиновой кислоты, где указанный сегмент нуклеиновой кислоты может содержать нуклеотидную последовательность, выбранную из SEQ ID NO: 109, 111, 113, 115, 117, 119, 121, 123, 125, 127, 129, 131, 133, 135, 137, 139, 141, 143, 145, 147, 149, 151, 153, 155, 171, 173 и 175.
[00229] Соответственно, белки для применения в настоящем изобретении могут быть получены в генетически сконструированных клетках-хозяевах в соответствии с общепринятыми методами. Подходящие клетки-хозяева относятся к типам клеток, которые могут быть трансформированы или трансфицированы экзогенной ДНК и выращены в культуре, и включают бактерии, клетки грибов и культивируемые клетки высших эукариот (включая культивируемые клетки многоклеточных организмов), в частности, культивируемые клетки млекопитающих. Методы манипулирования клонированными молекулами ДНК и введения экзогенной ДНК в различные клетки-хозяева описаны Sambrook and Russell, Molecular Cloning: A Laboratory Manual (3rd ed., Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 2001), и Ausubel et al., Short Protocols in Molecular Biology (4th ed., John Wiley & Sons, 1999).
[00230] Например, для рекомбинантной экспрессии гомодимерного связывающего белка, содержащего два идентичных связывающих полипептида, как описано в настоящем документе, вектор экспрессии обычно будет включать сегмент нуклеиновой кислоты, кодирующий связывающий полипептид, функционально связанный с промотором. Для рекомбинантной экспрессии гетеродимерного связывающего белка, содержащего разные первую и вторую полипептидные цепи, первая и вторая полипептидные цепи могут быть совместно экспрессированы из отдельных векторов в клетке-хозяине для экспрессии целого гетеродимерного белка. В качестве альтернативы, для экспрессии гетеродимерных связывающих белков первая и вторая полипептидные цепи совместно экспрессируют из отдельных единиц экспрессии в одном и том же векторе в клетке-хозяине для экспрессии целого гетеродимерного белка. Вектор(ы) экспрессии переносят в клетку-хозяина общепринятыми методами, и затем трансфицированные клетки культивируют общепринятыми методами с получением кодируемого полипептида(ов) для продуцирования соответствующих связывающих белков (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков).
[00231] Для направления рекомбинантного белка в секреторный путь клетки-хозяина в векторе экспрессии обеспечивают секреторную сигнальную последовательность (также известную как лидерная последовательность). Секреторная сигнальная последовательность может представлять собой последовательность нативной формы рекомбинантного белка или может быть получена из другого секретируемого белка, или синтезирована de novo. Указанная секреторная сигнальная последовательность функционально связана с последовательностью ДНК, кодирующей полипептид, то есть, две последовательности соединены в правильной рамке считывания и расположены так, чтобы направлять вновь синтезированный полипептид в секреторный путь клетки-хозяина. Секреторные сигнальные последовательности обычно являются 5'-концевыми относительно последовательности ДНК, кодирующей представляющий интерес полипептид, хотя некоторые сигнальные последовательности могут быть расположены в других местах представляющей интерес последовательности ДНК (см., например, патенты США №№ 5,037,743 и 5,143,830).
[00232] Культивируемые клетки млекопитающих являются подходящими хозяевами для продуцирования рекомбинантных полипептидов и белков согласно настоящему изобретению (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков) для применения в настоящем изобретении. Способы введения экзогенной ДНК в клетки-хозяева млекопитающих включают трансфекцию, опосредованную фосфатом кальция (Wigler et al., Cell 14:725, 1978; Corsaro and Pearson, Somatic Cell Genetics 7:603, 1981: Graham and Van der Eb, Virology 52:456, 1973), электропорацию (Neumann et al., EMBO J. 1:841-845, 1982), трансфекцию, опосредованную ДЭАЭ-декстраном (Ausubel et al., supra) и трансфекцию, опосредованную липосомами (Hawley-Nelson et al., Focus 15:73, 1993; Ciccarone et al., Focus 15:80, 1993). Получение рекомбинантных полипептидов в культивируемых клетках млекопитающих раскрыто, например, в патентах США №№ 4,713,339; 4,784,950; 4,579,821 и 4,656,134. Примеры подходящих клеток-хозяев млекопитающих включают клетки почек африканской зеленой мартышки (Vero; ATCC CRL 1587), клетки почки человеческого эмбриона (293-HEK; ATCC CRL 1573), клетки почки новорожденного хомяка (BHK-21, BHK-570; ATCC CRL 8544, ATCC CRL 10314), клетки почек собак (MDCK; ATCC CCL 34), клетки яичника китайского хомячка (CHO-K1; ATCC CCL61; CHO DG44; CHO DXB11 (Hyclone, Логан, Юта); см. также, например, Chasin et al., Som. Cell. Molec. Genet. 12:555, 1986)), клетки гипофиза крысы (GH1; ATCC CCL82), клетки HeLa S3 (ATCC CCL2.2), клетки гепатомы крысы (H-4-II-E; ATCC CRL 1548), трансформированные SV40 клетки почки обезьяны (COS-1; ATCC CRL 1650) и мышиные эмбриональные клетки (NIH-3T3; ATCC CRL 1658). Другие подходящие линии клеток известны в данной области техники и доступны из публичных депозитариев, таких как Американская коллекция типовых культур, Манассас, Виргиния. Могут быть использованы сильные промоторы транскрипции, такие как промоторы из SV-40 или цитомегаловируса. См., например, патент США № 4,956,288. Другие подходящие промоторы включают промоторы из генов металлотионеина (патенты США №№ 4,579,821 и 4,601,978) и главный поздний промотор аденовируса.
[00233] Для отбора культивируемых клеток млекопитающих, в которые была вставлена чужеродная ДНК, обычно применяют отбор по чувствительности к лекарственному средству. Такие клетки обычно называют «трансфектантами». Клетки, культивированные в присутствии селективного агента и способные передавать представляющий интерес ген своему потомству, называются «стабильными трансфектантами». Иллюстративные селектируемые маркеры включают ген, кодирующий устойчивость к антибиотику неомицину, который позволяет проводить отбор в присутствии лекарственного средства типа неомицина, такого как G-418 или тому подобного; ген ксантин-гуанин-фосфорибозилтрансферазы gpt, который обеспечивает рост клеток-хозяев в присутствии микофеноловой кислоты/ксантина; и маркеры, обеспечивающие устойчивость к зеоцину, блеомицину, бластицидину и гигромицину (см., например, Gatignol et al., Mol. Gen. Genet. 207:342, 1987; Drocourt et al., Nucl. Acids Res. 18:4009, 1990). Системы отбора также могут применяться для повышения уровня экспрессии представляющего интерес гена - процесс, называемый «амплификацией». Амплификацию проводят путем культивирования трансфектантов в присутствии низкого уровня селективного агента и затем увеличения количества селективного агента для отбора клеток, продуцирующих высокие уровни продуктов введенных генов. Иллюстративный амплифицируемый селектируемый маркер представляет собой дигидрофолатредуктазу, придающую устойчивость к метотрексату. Также могут применяться другие гены, придающие устойчивость к лекарственному средству (например, устойчивость к гигромицину, устойчивость к нескольким лекарственным средствам, пуромицин-ацетилтрансфераза).
[00234] Другие клетки высших эукариот также могут быть использованы в качестве хозяев, включая клетки насекомых, клетки растений и клетки птиц. Применение Agrobacterium rhizogenes в качестве вектора для экспрессии генов в клетках растений было рассмотрено Sinkar et al., J. Biosci. (Bangalore) 11:47-58, 1987. Трансформация клеток насекомых и продуцирование в них чужеродных полипептидов раскрыты в патенте США № 5,162,222 и публикации PCT № WO 94/06463.
[00235] Клетки насекомых могут быть инфицированы рекомбинантным бакуловирусом, обычно получаемым из вируса ядерного полиэдроза Autographa californica (AcNPV). См. King and Possee, The Baculovirus Expression System: A Laboratory Guide (Chapman & Hall, London); O’Reilly et al., Baculovirus Expression Vectors: A Laboratory Manual (Oxford University Press., New York 1994); и Baculovirus Expression Protocols. Methods in Molecular Biology (Richardson ed., Humana Press, Totowa, NJ, 1995). Рекомбинантный бакуловирус также может быть получен путем использования системы на основе транспозонов, описанной Luckow et al. (J. Virol. 67:4566-4579, 1993). Эта система, в которой используются векторы переноса, коммерчески доступна в форме набора (набор BAC-TO-BAC; Life Technologies, Гейтерсберг, MD). Вектор переноса (например, PFASTBAC1; Life Technologies) содержит транспозон Tn7 для перемещения ДНК, кодирующей представляющий интерес белок, в геном бакуловируса, поддерживаемый в E. coli в виде большой плазмиды, называемой «бакмидой». см. Hill-Perkins and Possee, J. Gen. Virol. 71:971-976, 1990; Bonning et al., J. Gen. Virol. 75:1551-1556, 1994; и Chazenbalk and Rapoport, J. Biol. Chem. 270:1543-1549, 1995. Кроме того, векторы переноса могут включать внутрирамочное слияние с ДНК, кодирующей удлинение полипептида или аффинную метку, как описано выше. С помощью методов, известных в данной области техники, вектор переноса, содержащий последовательность ДНК, кодирующую белок, трансформируют в клетки-хозяева E. coli, и клетки подвергают скринингу на наличие бакмид, содержащих прерванный ген lacZ, свидтельствующий о рекомбинантном бакуловирусе. Бакмидную ДНК, содержащую рекомбинантный бакуловирусный геном, выделяют, используя общепринятые методы, и используют для трансфекции клеток Spodoptera frugiperda, таких как клетки Sf9. Затем получают рекомбинантный вирус, экспрессирующий представляющий интерес белок. Исходные рекомбинантные вирусы готовят способами, обычно используемыми в данной области техники.
[00236] Для получения белка рекомбинантный вирус используют для инфицирования клеток-хозяев, как правило, линии клеток, полученной из совки травяной, Spodoptera frugiperda (например, клеток Sf9 или Sf21) или Trichoplusia ni (например, клеток HIGH FIVE™; Invitrogen, Карлсбад, Калифорния). Общий обзор см. в Glick and Pasternak, Molecular Biotechnology, Principles & Applications of Recombinant DNA (ASM Press, Washington, D.C., 1994). См. также патент США № 5,300,435. Для выращивания и поддержания клеток используют бессывороточные среды. Подходящие составы сред известны в данной области техники и могут быть получены от коммерческих поставщиков. Клетки выращивают от плотности инокуляции, равной приблизительно 2-5 × 105 клеток, до плотности 1-2 × 106 клеток, и в это время добавляют исходный рекомбинантный вирус при множественности заражения (MOI) от 0,1 до 10, как правило, приблизительно 3. Используемые процедуры обычно описаны в доступных лабораторных руководствах (см., например, источники King and Possee, цитируемый выше; O’Reilly et al., цитируемый выше; Richardson, цитируемый выше).
[00237] Клетки грибов, включая дрожжевые клетки, также могут быть использованы в настоящем изобретении для получения полипептидов согласно настоящему изобретению (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков). Подходящие виды дрожжей включают, например, Saccharomyces cerevisiae, Pichia pastoris и Pichia methanolica. Способы трансформации клеток S. cerevisiae экзогенной ДНК и получения из них рекомбинантных полипептидов описаны, например, в патентах США №№ 4,599,311; 4,931,373; 4,870,008; 5,037,743; и 4,845,075. Трансформированные клетки отбирают по фенотипу, определяемому селектируемым маркером, обычно представляющим собой устойчивость к лекарственному средству или способность роста в отсутствие определенного питательного вещества (например, лейцина). Иллюстративная векторная система для применения в Saccharomyces cerevisiae представляет собой векторную систему POT1, раскрытую Kawasaki et al. (патент США № 4,931,373), позволяющую отбирать трансформированные клетки путем выращивания в средах, содержащих глюкозу. Подходящие промоторы и терминаторы для применения в дрожжах включают промоторы из генов гликолитического фермента (см., например, патенты США №№ 4,599,311; 4,615,974 и 4,977,092) и генов алкогольдегидрогеназы. См. также патенты США №№ 4,990,446; 5,063,154; 5,139,936 и 4,661,454. В данной области техники известны системы трансформации для других дрожжей, включая Hansenula polymorpha, Schizosaccharomyces pombe, Kluyveromyces lactis, Kluyveromyces fragilis, Ustilago maydis, Pichia pastoris, Pichia methanolica, Pichia guillermondii и Candida maltosa. См., например, Gleeson et al., J. Gen. Microbiol. 132:3459-3465, 1986; патент США № 4,882,279; и Raymond et al., Yeast 14:11-23, 1998. Клетки Aspergillus могут быть использованы способами согласно McKnight et al., патент США № 4,935,349. Способы трансформации Acremonium chrysogenum раскрыты Sumino et al., патент США № 5,162,228. Способы трансформации Neurospora раскрыты Lambowitz, патент США № 4,486,533. Получение рекомбинантных белков в Pichia methanolica описано в патентах США №№ 5,716,808; 5,736,383; 5,854,039 и 5,888,768.
[00238] Прокариотические клетки-хозяева, включая штаммы бактерий Escherichia coli, Bacillus и других родов, также походят для применения в качестве клеток-хозяев в рамках настоящего изобретения для получения, например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков, включая молекулы к 5Т4 х 4-1ВВ. Методы трансформации этих хозяев и экспрессии клонированных в них чужеродных последовательностей ДНК хорошо известны в данной области техники (см., например, источник Sambrook and Russell, цитируемый выше). При экспрессии рекомбинантного белка в бактериях, таких как Е. coli, белок может удерживаться в цитоплазме, обычно в виде нерастворимых гранул, или может быть направлен в периплазматическое пространство посредством бактериальной последовательности секреции. В первом случае клетки лизируют, а гранулы выделяют и денатурируют, используя, например, изотиоцианат гуанидина или мочевину. Затем денатурированный белок может быть повторно свернут и димеризован путем разбавления денатурирующего агента, например, путем диализа против раствора мочевины и комбинации восстановленного и окисленного глутатиона, с последующим диализом против забуференного физиологического раствора. В качестве альтернативы, белок может быть извлечен из цитоплазмы в растворимой форме и выделен без использования денатурирующих агентов. Белок извлекают из клетки в виде водного экстракта, например, в натрий-фосфатном буфере. Для захвата представляющего интерес белка экстракт наносят непосредственно на хроматографическую среду, такую как иммобилизованное антитело или гепарин-сефарозная колонка. Секретируемые белки могут быть извлечены из периплазматического пространства в растворимой и функциональной форме путем разрушения клеток (например, ультразвуком или осмотическим шоком) для высвобождения содержимого периплазматического пространства и выделения белка, тем самым устраняя необходимость в денатурации и повторном сворачивании. Антитела, включая одноцепочечные антитела, могут быть получены в бактериальных клетках-хозяевах в соответствии с известными способами. См., например, Bird et al., Science 242:423-426, 1988; Huston et al., Proc. Natl. Acad. Sci. USA 85:5879-5883, 1988; и Pantoliano et al., Biochem. 30:10117-10125, 1991.
[00239] Трансформированные или трансфицированные клетки-хозяева для продуцирования полипептидов и белков согласно настоящему изобретению (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков) культивируют в соответствии с общепринятыми методиками в культуральной среде, содержащей питательные вещества и другие компоненты, необходимые для роста выбранных клеток-хозяев. В данной области техники известны различные подходящие среды, включая среды с определенным составом и сложные среды, и обычно они включают источник углерода, источник азота, незаменимые аминокислоты, витамины и минералы. Среды также могут по необходимости содержать такие компоненты, как факторы роста или сыворотка. Питательная среда обычно позволяет осуществлять отбор клеток, содержащих экзогенно добавленную ДНК, например, путем отбора по чувствительности к лекарственному средству или недостатка необходимого питательного вещества, который дополняется селектируемым маркером, переносимым на векторе экспрессии или совместно трансфицированным в клетку-хозяина.
[00240] Белки и полипептиды согласно настоящему изобретению (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) могут быть очищены общепринятыми способами очистки белков, как правило, с помощью комбинации хроматографических методов. Общий обзор см. в Affinity Chromatography: Principles & Methods (Pharmacia LKB Biotechnology, Uppsala, Sweden, 1988); Scopes, Protein Purification: Principles and Practice (Springer-Verlag, New York 1994). Белки, содержащие Fc-область иммуноглобулина, могут быть очищены методом аффинной хроматографии на иммобилизованном белке А или белке G. Дополнительные стадии очистки, такие как гель-фильтрация, могут использоваться для получения желаемого уровня чистоты или для обессоливания, замены буфера и тому подобного.
Композиции и способы применения
[00241] Настоящее изобретение относится к способам лечения субъекта, страдающего расстройством, характеризующимся экспрессией 5Т4. Обычно такие способы включают введение субъекту, нуждающемуся в таком лечении, белка согласно настоящему изобретению (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков). В некоторых вариантах реализации 5T4-связывающий полипептид, 4-1BB-связывающий полипептид и/или содержащие их полиспецифичные связывающие белки (например, молекула к 5T4 x 4-1BB) не индуцируют или индуцирует минимальную антителозависимую клеточно-опосредованную цитотоксическую активность (ADCC) и/или комплементзависимую цитотоксическую активность (CDC).
[00242] В других вариантах реализации, где связывающие белки (например, полиспецифичные связывающие белки) содержат второй связывающий домен, специфично связывающий эффекторную клетку (например, 4-1BB), связывающие белки и/или полиспецифичные связывающие белки приводят к усиленной активации эффекторных клеток против 5T4-экспрессирующих клеток у субъекта. Например, в некоторых вариантах реализации связывающие белки и/или полиспецифичные связывающие белки приводят к повышенной пролиферации эффекторных клеток, повышению выработки эффекторными клетками одного или более цитокинов (например, IFNγ, TNFα, ИЛ-6, ИЛ-8, ИЛ-12, ИЛ-1α и т. д.) или повышенной экспрессии одного или более маркеров активации клеточной поверхности (например, CD137, ГКГС-II или CD69). Активация эффекторных клеток может быть измерена различными способами, известными в данной области техники, включая проточную цитометрию, иммунофлуоресценцию и иммуногистохимию для оценки изменений в экспрессии маркеров на клеточной поверхности, ELISA для оценки выработки цитокинов и других факторов, методы подсчета клеток для оценки пролиферации, кПЦР для оценки изменений в экспрессии генов и тому подобное.
[00243] В некоторых вариантах реализации полиспецифичные связывающие белки, описанные в настоящем документе, характеризуются повышенной активацией эффекторных клеток по сравнению со вторым полиспецифичным полипептидом другой структуры. Например, в некоторых вариантах реализации первый полиспецифичный полипептид содержит первый scFv-домен, специфично связывающийся с 5T4, и второй scFv, специфично связывающийся с 4-1BB, связанные вместе посредством линкера связывающих доменов, и Fc-домен иммуноглобулина в следующей конфигурации, в направлении от аминоконца к карбоксиконцу: (i) первый scFv-домен, (ii) шарнирная область, (iii) константная область иммуноглобулина, (iv) линкер связывающих доменов и (v) второй scFv-домен. В этом примере второй полиспецифичный связывающий белок содержит структуру IgG-scFv, содержащую антитело к 4-1BB и scFv к 5T4. В некоторых вариантах реализации первый полиспецифичный связывающий белок демонстрирует статистически значимое усиление активации эффекторных клеток по сравнению со вторым полиспецифичным полипептидом. Например, в некоторых вариантах реализации первый полиспецифичный связывающий белок демонстрирует более низкое значение EC50 в анализе на репортерных клетках Jurkat/NF-κB, чем наблюдается для второго полиспецифичного связывающего белка (см., например, пример 10). В некоторых вариантах реализации ЕС50 первого полиспецифичного связывающего белка снижено приблизительно в 2 раза, приблизительно в 3 раза, приблизительно в 4 раза, приблизительно в 5 раз, приблизительно в 6 раз, приблизительно в 7 раз, приблизительно в 8 раз, приблизительно в 9 раз, приблизительно в 10 раз или более по сравнению с ЕС50 второго полиспецифичного связывающего белка. Значения EC50 могут быть определены с помощью нелинейного регрессионного анализа и других статистических методов, известных в данной области техники.
[00244] В некоторых вариантах реализации первый полиспецифичный связывающий белок индуцирует статистически значимое повышение пролиферации эффекторных клеток по сравнению со вторым полиспецифичным полипептидом. Например, в некоторых вариантах реализации первый полиспецифичный связывающий белок индуцирует статистически значимое повышение пролиферации примированных CD8+ Т-клеток человека по сравнению с пролиферацией, индуцируемой вторым полиспецифичным связывающим белком (см., например, пример 22). В некоторых вариантах реализации первый полиспецифичный связывающий белок индуцирует повышение пролиферации эффекторных клеток, приблизительно в 2 раза, приблизительно в 3 раза, приблизительно в 4 раза, приблизительно в 5 раз, приблизительно в 6 раз, приблизительно в 7 раз, приблизительно в 8 раз, приблизительно в 9 раз, приблизительно в 10 раз или более превышающее пролиферацию эффекторных клеток, индуцированную вторым полиспецифичным связывающим белком.
[00245] В отдельных вариациях способа лечения субъекта белком согласно настоящему изобретению (например, 5T4-связывающими полипептидами, 4-1BB-связывающими полипептидами и/или содержащими их полиспецифичными связывающими белками) расстройство представляет собой рак. Иллюстративные виды рака, поддающиеся лечению белком согласно настоящему изобретению, включают, например, рак молочной железы (например, тройной негативный рак молочной железы (ТНРМЖ)), рак поджелудочной железы, рак яичника, рак легкого (например, немелкоклеточный рак легкого), гемобластозы (например, хронический лимфоцитарный лейкоз (ХЛЛ), мантийноклеточный лейкоз (МКЛ) или острый лимфобластный лейкоз (ОЛЛ)), рак кожи (например, плоскоклеточную карциному или меланому), рак надпочечника, рак мочевого пузыря, рак шейки матки, рак почки, рак желудка, рак предстательной железы, рак щитовидной железы, рак печени, рак матки, опухоль, образованную на нейронах или оболочке нервных волокон (например, нейрофиброму), саркому или рак головы и шеи. ТНРМЖ определен как рак молочной железы с отсутствием окрашивания на рецептор эстрогена, рецептор прогестерона и HER2/neu. В некоторых вариантах реализации белок согласно настоящему изобретению может быть введен субъекту для лечения у указанного субъекта мезотелиомы. В одном из вариантов реализации белок согласно настоящему изобретению может быть введен субъекту для лечения у указанного субъекта светлоклеточной карциномы. В одном из вариантов реализации белок согласно настоящему изобретению может быть введен субъекту для лечения у указанного субъекта опухоли из поперечно-полосатой мышцы.
[00246] В дополнительном варианте реализации изобретение включает способ усиления активации эффекторных клеток, направленной против клетки, экспрессирующей 5T4, включающий приведение указанной 5T4-экспрессирующей клетки в контакт с белком согласно настоящему изобретению (например, 5T4-связывающими полипептидами, 4-1BB-связывающими полипептидами и/или содержащими их полиспецифичными связывающими белками), где указанное приведение в контакт проводят в условиях, при которых индуцируется усиленная активация эффекторных клеток, направленная против указанной 5T4-экспрессирующей клетки. В некоторых вариантах реализации изобретение относится к способу усиления активации эффекторных клеток, направленной против клетки, экспрессирующей 5T4, включающему приведение указанной 5T4-экспрессирующей клетки в контакт с белком согласно настоящему изобретению (например, 5T4-связывающими полипептидами, 4-1BB-связывающими полипептидами и/или содержащими их полиспецифичными связывающими белками), содержащим первый связывающий домен, специфично связывающий эпитоп человеческого 5T4, и второй связывающий домен, специфично связывающий 4-1BB (например, молекулой к 5T4 x 4-1BB); где указанное приведение в контакт проводят в условиях, при которых индуцируется усиленная активация эффекторных клеток, направленная против указанной 5T4-экспрессирующей клетки. Изобретение включает способ индуцирования зависимого от эффекторных клеток лизиса клетки, экспрессирующей 5T4, включающий приведение указанной 5T4-экспрессирующей клетки в контакт с белком согласно настоящему изобретению (например, 5T4-связывающими полипептидами, 4-1BB-связывающими полипептидами и/или содержащими их полиспецифичными связывающими белками), где второй связывающий домен специфично связывает 4-1BB (например, молекулой к 5T4 x 4-1BB); и где указанное приведение в контакт проводят в условиях, при которых индуцируется усиленная активация эффекторных клеток, направленная против указанной 5T4-экспрессирующей клетки, что приводит к лизису указанной 5T4-экспрессирующей клетки. В некоторых вариантах реализации изобретение относится к способу индукции зависимого от эффекторных клеток лизиса клетки, экспрессирующей 5T4, включающему приведение указанной 5T4-экспрессирующей клетки в контакт с белком согласно настоящему изобретению, содержащим первый связывающий домен, специфично связывающий эпитоп человеческого 5T4, и второй связывающий домен, специфично связывающий 4-1BB (например, молекулой к 5T4 x 4-1BB); где указанное приведение в контакт проводят в условиях, при которых индуцируется усиленная активация эффекторных клеток, направленная против указанной 5T4-экспрессирующей клетки, что приводит к лизису указанной 5T4-экспрессирующей клетки.
[00247] Изобретение также включает белки и полипептиды (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) для производства лекарственного средства для лечения расстройства (например, рака), характеризующегося экспрессией 5Т4. В одном из вариантов реализации белок или полипептид содержит связывающий домен к 5T4 и к 4-1BB (например, молекулу к 5T4 x 4-1BB), и обладает повышенной активностью активации эффекторных клеток. В одном из вариантов реализации изобретение относится к белкам и полипептидам (например, молекуле к 5T4 x 4-1BB) для применения для лечения расстройства (например, рака), характеризующегося экспрессией 5T4. В отдельных вариантах реализации изобретение относится к способу лечения расстройства у субъекта, где указанное расстройство характеризуется экспрессией 5T4, включающему введение указанному субъекту терапевтически эффективного количества белка или полипептида согласно настоящему изобретению, содержащего 5T4-связывающий домен, специфично связывающий эпитоп человеческого 5T4 (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков).
[00248] В некоторых вариантах реализации изобретение относится к способу лечения пациента, страдающего раком, включающему введение указанному пациенту полипептида, содержащего аминокислотную последовательность, представленную в настоящем документе (например, аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176). В дополнительных вариантах реализации полипептид содержит Fc. Например, в некоторых вариантах реализации настоящее изобретение относится к способу лечения пациента, страдающего раком, включающему введение указанному пациенту полипептида, содержащего 5T4-связывающий домен и Fc, где указанный полипептид содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176. В дополнительных вариантах реализации настоящее изобретение относится к способу лечения пациента, страдающего раком, включающему введение указанному пациенту полипептида, содержащего 5T4-связывающий домен и 4-1BB-связывающий домен, где указанный полипептид содержит аминокислотную последовательность, выбранную из группы, состоящей из SEQ ID NO: 136, 138, 140, 142, 144, 146, 148, 150, 152, 154, 156, 172, 174 и 176. В одном из вариантов реализации настоящее изобретение относится к способу лечения пациента, страдающего раком, включающему введение указанному пациенту полипептида, содержащего 5T4-связывающий домен и 4-1BB-связывающий домен, где указанный полипептид содержит SEQ ID NO: 172. В одном из вариантов реализации настоящее изобретение относится к способу лечения пациента, страдающего раком, включающему введение указанному пациенту полипептида, содержащего 5T4-связывающий домен и 4-1BB-связывающий домен, где указанный полипептид содержит SEQ ID NO: 174.
[00249] В некоторых вариантах реализации для способов лечения и применений, описанных в настоящем документе, белок или полипептид, описанный в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), доставляют способом, согласующимся с общепринятыми принципами, связанными с контролем заболевания или расстройства, подлежащего лечению. В соответствии с приведенным в настоящем документе описанием терапевтически эффективное количество белка полипептида вводят субъекту, нуждающемуся в таком лечении, в течение времени и в условиях, достаточных для предупреждения или лечения заболевания или расстройства.
[00250] Субъекты, подлежащие введению белка согласно настоящему изобретению (например, 5T4-связывающих полипептидов, 4-1BB-связывающих полипептидов и/или содержащих их полиспецифичных связывающих белков), включают пациентов с высоким риском развития определенного расстройства, характеризующегося экспрессией 5T4, а также пациентов, уже страдающих от такого расстройства. Как правило, у субъекта было диагностировано расстройство, подлежащее лечению. Кроме того, субъекты в ходе лечения могут наблюдаться на предмет любого изменения расстройства (например, увеличения или уменьшения клинических симптомов расстройства). Кроме того, в некоторых вариациях субъект не страдает от другого расстройства, требующего лечения, связанного с нацеливанием на 5T4-экспрессирующие клетки.
[00251] В профилактических целях фармацевтические композиции или лекарственные средства, содержащие белок согласно настоящему изобретению (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), вводят пациенту, уязвимого или иным образом подверженного риску развития определенного расстройства, в количестве, достаточном для устранения или уменьшения риска или задержки возникновения указанного расстройства. В терапевтических целях композиции или лекарственные средства, содержащие белок согласно настоящему изобретению, вводят пациенту, предположительно страдающему или уже страдающему таким расстройством, в количестве, достаточном для излечения или по меньшей мере частичного прекращения симптомов расстройства и его осложнений. Количество, достаточное для достижения этой цели, называется терапевтически эффективной дозой или количеством. Как в схемах профилактики, так и в схемах лечения агенты обычно вводят в нескольких дозах до тех пор, пока не будет достигнут достаточный ответ (например, ингибирование аномальной активности ангиогенеза). Как правило, ответ контролируют и вводят повторные дозы, если желаемый ответ начинает ослабевать.
[00252] Для идентификации пациентов, подлежащих лечению способами согласно изобретению (например, 5T4-связывающими полипептидами, 4-1BB-связывающими полипептидами и/или содержащими их полиспецифичными связывающими белками), могут быть использованы принятые способы скрининга для определения факторов риска, связанных с конкретными расстройствами, или для определения статуса существующего расстройства, выявленного у субъекта. Такие способы могут включать, например, определение того, есть ли у индивидуума родственники, у которых было диагностировано конкретное заболевание. Способы скрининга могут также включать, например, обычные обследования для определения семейного статуса конкретного заболевания, о котором известно, что оно имеет наследственный компонент. Например, известно, что различные виды рака имеют определенные наследственные компоненты. Наследственные компоненты раковых заболеваний включают, например, трансформирующие мутации в нескольких генах (например, Ras, Raf, EGFR, cMet и др.), наличие или отсутствие отдельных молекул человеческого лейкоцитарного антигена (HLA) и рецептора подавления цитотоксичности (KIR), или механизмы, посредством которых раковые клетки способны модулировать подавление иммунного ответа таких клеток, как NK-клетки и Т-клетки, напрямую или опосредованно (см., например, Ljunggren and Malmberg, Nature Rev. Immunol. 7:329-339, 2007; Boyton and Altmann, Clin. Exp. Immunol. 149:1-8, 2007). С этой целью в соответствии со стандартной практикой могут быть использованы нуклеотидные зонды для идентификации индивидуумов, несущих генетические маркеры, связанные с конкретным представляющим интерес расстройством. Кроме того, в данной области техники известно множество разнообразных иммунологических методов, подходящих для идентификации маркеров конкретного расстройства. Например, в данной области техники доступны и хорошо известны различные методы иммуноферментного анализа ELISA, в которых используются зонды на основе моноклональных антител для детекции антигенов, связанных с определенными опухолями. Скрининг может быть осуществлен в соответствии с известной симптоматикой пациента, возрастными факторами, связанными факторами риска и тому подобным. Эти способы позволяют лечащему врачу отбирать пациентов, нуждающихся в описанных в настоящем документе способах лечения, по стандартной методике. В соответствии с этими способами нацеливание на патологические клетки, экспрессирующие 5T4, может быть реализовано в качестве независимой программы лечения или в качестве последующей, вспомогательной или координирующей схемы лечения относительно других видов лечения.
[00253] Для введения белок согласно настоящему изобретению (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) может быть введен в состав фармацевтической композиции. Фармацевтическая композиция может содержать: (i) 5T4-связывающий полипептид, 4-1BB-связывающий полипептид и/или содержащий их полиспецифичный связывающий белок (например, молекулу к 5T4 x 4-1BB); и (ii) фармацевтически приемлемый носитель, разбавитель или вспомогательное вещество. Фармацевтическая композиция, содержащая 5T4-связывающий полипептид, 4-1BB-связывающий полипептид и/или содержащий их полиспецифичный связывающий белок (например, молекулу к 5T4 x 4-1BB), может быть приготовлена в соответствии с известными способами приготовления фармацевтически пригодных композиций, в которых терапевтическую молекулу объединяют в смеси с фармацевтически приемлемым носителем, разбавителем или вспомогательным веществом. Считается, что носитель является «фармацевтически приемлемым носителем», если его введение переносится пациентом-реципиентом. Стерильный натрий-фосфатный буфер является одним из примеров фармацевтически приемлемого носителя. Другие подходящие носители, разбавители или вспомогательные вещества хорошо известны специалистам в данной области техники. (См., например, Gennaro (ed.), Remington's Pharmaceutical Sciences (Mack Publishing Company, 19th ed. 1995).) Композиции могут дополнительно включать одно или более вспомогательных веществ, консервантов, солюбилизаторов, буферных агентов, альбумина для предотвращения потери белка на поверхностях ампул и т. д. В отдельных вариантах реализации фармацевтическая композиция содержит 5T4-связывающий полипептид, 4-1BB-связывающий полипептид и/или содержащий их полиспецифичный связывающий белок (например, молекулу к 5T4 x 4-1BB), представляющий собой гомодимер или гетеродимер. «Гомодимер» может представлять собой димер, образованный из двух идентичных полипептидов (например, молекулы к 5Т4 х 4-1BB, как описано в настоящем документе).
[00254] Фармацевтическая композиция, содержащая полипептид или белок, описанные в настоящем документе, может быть выполнена в дозированной форме, выбранной из группы, состоящей из: пероральной формы однократной дозы, внутривенной формы однократной дозы, интраназальной формы однократной дозы, суппозиторной формы однократной дозы, внутрикожной формы однократной дозы, внутримышечной формы однократной дозы, внутрибрюшинной формы однократной дозы, подкожной формы однократной дозы, эпидуральной формы однократной дозы, сублингвальной формы однократной дозы и интрацеребральной формы однократной дозы. Пероральная форма однократной дозы может быть выбрана из группы, состоящей из таблеток, пилюль, пеллетов, капсул, порошков, таблеток для рассасывания, гранул, растворов, суспензий, эмульсий, сиропов, эликсиров, составов с замедленным высвобождением, аэрозолей и спреев.
[00255] Фармацевтическая композиция, содержащая полипептид или белок, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), может быть введена субъекту в терапевтически эффективном количестве. В соответствии со способами согласно настоящему изобретению полипептид или белок, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), могут быть введены субъектам различными способами введения, включая, например, внутримышечный, подкожный, внутривенный, внутрипредсердный, внутрисуставной, парентеральный, интраназальный, внутрилегочный, чрескожный, внутриплевральный, интратекальный и пероральный способы введения. В целях предупреждения и лечения агонист (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) может быть введен субъекту посредством однократной болюсной доставки, посредством непрерывной доставки (например, непрерывной чрескожной доставки) в течение длительного периода времени, или по схеме повторного введения (например, ежечасного, ежесуточного, еженедельного или ежемесячного).
[00256] Определение эффективных дозировок в этом контексте обычно основывается на модельных исследованиях на животных, после которых проводят клинические исследования с участием людей, и определяется в соответствии с эффективными дозировками и схемами введения, значительно снижающими частоту возникновения или тяжесть подлежащего лечению расстройства у модельных субъектов. Эффективные дозы композиций согласно настоящему изобретению варьируются в зависимости от многих различных факторов, включая способы введения, целевой сайт, физиологическое состояние пациента, является ли пациент человеком или животным, другие вводимые лекарственные средства, является ли лечение профилактическим или терапевтическим, а также конкретную активность самой композиции и ее способность вызывать желаемый ответ у индивидуума. Обычно пациент является человеком, но при некоторых заболеваниях пациент может представлять собой отличное от человека млекопитающее. Как правило, схемы дозирования корректируют для обеспечения оптимального терапевтического ответа, то есть, для оптимизации безопасности и эффективности. Соответственно, терапевтически эффективное количество также представляет собой такое, при котором любые нежелательные побочные действия перевешиваются положительными эффектами от введения 5Т4-связывающего полипептида, 4-1ВВ-связывающего полипептида и/или содержащего их полиспецифичного связывающего белка (например, молекулы к 5T4 x 4-1BB), как описано в настоящем документе. Для введения белка или полипептида, описанного в настоящем документе, дозировка может составлять от приблизительно 0,1 мкг до 100 мг/кг или от 1 мкг/кг до приблизительно 50 мг/кг и чаще от 10 мкг до 5 мг/кг массы тела субъекта. В частных вариантах реализации эффективное количество агента составляет от приблизительно 1 мкг/кг до приблизительно 20 мг/кг, от приблизительно 10 мкг/кг до приблизительно 10 мг/кг или от приблизительно 0,1 мг/кг до приблизительно 5 мг/кг. Дозировки в этом диапазоне могут быть достигнуты путем однократного или многократного введения, включая, например, многократного введения несколько раз в день или ежесуточно, еженедельно, раз в две недели или ежемесячно. Например, в отдельных вариациях схема состоит из первоначального введения с последующими многократными последовательными введениями с недельными или двухнедельными интервалами. Еще одна схема состоит из первоначального введения с последующими многократными последовательными введениями с месячными или двухмесячными интервалами. В качестве альтернативы введение может осуществляться нерегулярно, в зависимости от наблюдаемых клинических симптомов расстройства.
[00257] Дозировка фармацевтической композиции, содержащей полипептид или белок, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки), может варьироваться лечащим врачом для поддержания желаемой концентрации в целевом сайте. Например, если выбран внутривенный способ доставки, локальная концентрация агента в кровотоке в целевой ткани может составлять приблизительно от 0,01-50 наномолей композиции на литр, иногда от приблизительно 1,0 наномоля на литр до 10, 15, или 25 наномолей на литр, в зависимости от состояния субъекта и прогнозируемого измеренного ответа. Могут быть выбраны более высокие или более низкие концентрации в зависимости от способа доставки, например, трансэпидермальной доставки в сравнении с доставкой на поверхность слизистой оболочки. Дозировка также должна корректироваться в зависимости от скорости высвобождения введенного препарата, например, назального спрея в сравнении с порошком, пероральных или инъецируемых частиц с замедленным высвобождением, трансдермальных препаратов и т. д. Например, для достижения того же уровня концентрации в сыворотке требуется в два раза более высокая докировка частиц с медленным высвобождением со скоростью высвобождения 5 наномолярных единиц (в стандартных условиях) по сравнению с дозировкой частиц со скоростью высвобождения 10 наномолярных единиц.
[00258] Белки и полипептиды, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки) (например, молекула к 5T4 x 4-1BB), также могут быть введены в суточной дозе от приблизительно 0,001 до приблизительно 10 миллиграммов (мг) на килограмм (мг на кг) массы тела, предпочтительно в виде разовой суточной дозы или в виде дробных доз приблизительно от двух до шести раз в сутки. Для введения взрослому пациенту, являющемуся человеком, терапевтически эффективное количество может быть введено в дозах от 0,2 до 800 мг на дозу, включая, не ограничиваясь перечисленным, 0,2 мг на дозу, 0,5 мг на дозу, 1 мг на дозу, 5 мг на дозу, 10 мг на дозу, 25 мг на дозу, 100 мг на дозу, 200 мг на дозу и 400 мг на дозу, и в ходе лечения могут быть введены многократные, обычно последовательные ежесуточные дозы. Белки и полипептиды, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки, такие как молекула к 5T4 x 4-1BB), могут быть введены в разное время суток. В одном из вариантов реализации оптимальная терапевтическая доза может быть введена вечером. В еще одном варианте реализации оптимальная терапевтическая доза может быть введена утром. Таким образом, общая суточная доза белков и полипептидов, описанных в настоящем документе, в одном из вариантов реализации может составлять от приблизительно 1 мг до приблизительно 2 г, и часто составляет от приблизительно 100 мг до приблизительно 1,5 г, и наиболее часто составляет от приблизительно 200 мг до приблизительно 1200 мг. В случае типичного взрослого человека весом 70 кг общая суточная доза терапевтического средства к 5Т4 может составлять от приблизительно 2 мг до приблизительно 1200 мг и часто будет составлять, как отмечено выше, от приблизительно 0,2 мг до приблизительно 800 мг.
[00259] В отношении лечения солидных опухолей протоколы для оценки конечных точек и противоопухолевой активности хорошо известны в данной области техники. Хотя каждый протокол может определять оценки ответа опухоли по-разному, критерии RECIST (Критерии оценки ответа солидных опухолей) в настоящее время считаются рекомендованными руководящими принципами для оценки ответа опухоли Национальным институтом онкологии (см. Therasse et al., J. Natl. Cancer Inst. 92:205-216, 2000). В соответствии с критериями RECIST ответ опухоли означает уменьшение или устранение всех измеримых очагов или метастазов. Заболевание обычно считают измеримым, если оно включает очаги, которые могут быть точно измерены по меньшей мере в одном направлении как равные или превышающие 20 мм с помощью общепринятых методов, или равные или превышающие 10 мм с помощью спиральной компьютерной томографии с четко определенными границами, с помощью медицинской фотографии или рентгенографии, компьютерной осевой томографии (КТ), магнитно-резонансной томографии (МРТ) или клинического осмотра (если очаги расположены на поверхности). Неизмеримое заболевание означает, что заболевание включает очаги, имеющие размер менее 20 мм при измерении с помощью общепринятых методов или менее 10 мм при изменении с помощью спиральной компьютерной томографии, и принципиально неизмеримые очаги (слишком маленькие, чтобы их можно было точно измерить). К неизмеримым заболеваниям относятся плевральные выпоты, асцит и заболевание, подтвержденное косвенными данными.
[00260] Критерии объективного статуса необходимы для протоколов для оценки ответа солидной опухоли. Иллюстративные критерии включают следующее: (1) полный ответ (CR), определенный как полное исчезновение всех измеримых заболеваний; отсутствие новых очагов; отсутствие связанных с заболеванием симптомов; отсутствие свидетельств неизмеримого заболевания; (2) частичный ответ (PR), определяемый как уменьшение на 30% суммы самого длинного диаметра целевых очагов; (3) прогрессирующее заболевание (PD), определяемое как увеличение на 20% суммы самого длинного диаметра целевых очагов или появление любого нового очага; (4) стабильное или отсутствие ответа, определяемое как не отвечающее критериям CR, PR или прогрессирующего заболевания (см. Therasse et al., цитируемый выше).
[00261] Дополнительные конечные точки, которые принимаются в области онкологии, включают общую выживаемость (OS), безрецидивную выживаемость (DFS), частоту объективного ответа (ORR), время до прогрессирования (TTP) и выживаемость без прогрессирования (PFS) (см. Guidance for Industry: Clinical Trial Endpoints for the Approval of Cancer Drugs and Biologics, April 2005, Center for Drug Evaluation and Research, FDA, Rockville, MD.)
[00262] Фармацевтические композиции, содержащие белки и полипептиды, описанные в настоящем документе (например, 5T4-связывающие полипептиды, 4-1BB-связывающие полипептиды и/или содержащие их полиспецифичные связывающие белки, такие как молекула к 5T4 x 4-1BB), могут поставляться в виде набора, включающего контейнер, содержащий фармацевтическую композицию, как описано в настоящем документе. Фармацевтическая композиция может быть предоставлена, например, в форме раствора для инъекций для однократной или многократных доз или в виде стерильного порошка, который необходимо разводить перед инъекцией. В качестве альтернативы, такой набор может включать прибор для распыления сухого порошка, генератор жидкого аэрозоля или небулайзер для введения фармацевтической композиции. Такой набор может дополнительно включать письменную информацию о показаниях и применении фармацевтической композиции.
[00263] Изобретение дополнительно поясняется следующими примерами, которые предназначены исключительно для иллюстрации изобретения и не имеют никакого ограничительного характера.
Примеры
Пример 1. Преобразование моноклонального антитела к CD137 «1618» в формат scFv и получение биспецифичных молекул к CD137 x 5T4 в формате ADAPTIR™ (scFv-Fc-scFv)
[00264] Для создания нуклеотидной последовательности, кодирующей scFv 1618, нуклеотидные последовательности, кодирующие вариабельные домены тяжелой цепи (VH) и легкой цепи (VL) (SEQ ID NO: 28 и SEQ ID NO: 16, соответственно) из моноклонального антитела к 4-1BB 1618 синтезировали и/или амплифицировали из существующей плазмидной ДНК и связывали вместе с помощью линкера на основе (Gly)4Ser, используя стандартные методы молекулярной биологии, описанные, не ограничиваясь перечисленным, например, в публикациях заявок PCT №№ WO 2007/146968, WO 2010/040105 и WO 2010/003108; публикации патентной заявки США № 2006/0051844; и патенте США № 7,166,707. Затем нуклеотидную последовательность scFv 1618 подвергали слиянию с модифицированной последовательностью Fc-области человеческого IgG1, содержащей точечные мутации для устранения эффекторной функциональной активности Fc-области. Аналогичным образом нуклеотидные последовательности, кодирующие домены VH и VL моноклонального антитела к 5T4 1210 (SEQ ID NO: 38 и 68, соответственно), связывали друг с другом дополнительным линкером на основе (Gly)4Ser. Полученные биспецифичные молекулы экспрессировали путем временной трансфекции клеток HEK-293 или клеток яичника китайского хомячка (CHO) и очищали от кондиционированных сред с использованием аффинной очистки с белком A (ProA) и эксклюзионной хроматографии (SEC). Чистоту белка определяли с помощью аналитической SEC после каждой из стадий очистки с белком A и SEC. Уровни эндотоксина определяли с использованием прибора Endosafe PTS в соответствии с инструкциями производителя, чтобы гарантировать, что результаты анализа активности in vitro не будут искажены из-за присутствия эндотоксина. Буфер каждой партии белка заменяли на PBS как часть процесса очистки методом SEC, концентрировали до 1 мг/мл, подвергали стерилизующей фильтрации и хранили при 4°C до следующего использования. Концентрацию белка определяли по величине поглощения при 280 нм, используя теоретический коэффициент экстинкции.
[00265] В некоторых случаях биспецифичные молекулы состояли из 2 scFv и 1 Fc-домена (scFv-Fc-scFv) со следующей структурой, в направлении от N-конца к C-концу: scFv 1618 - Fc - scFv 1210. Были получены дополнительные биспецифичные молекулы, в которых scFv 1618 был помещен на С-конец конструкции, а scFv 1210 - на N-конец (то есть, scFv 1210 - Fc - scFv 1618). Однако молекулы с этой альтернативной ориентацией обладали менее желательными свойствами по сравнению с молекулами scFv 1618 - Fc - scFv 1210-Fc-scFv.
[00266] Большинство, но не все, из молекул 1618-Fc-scFv 1210-Fc-scFv ADAPTIRTM (например, ALG.APV-006, ALG.APV-010, ALG.APV-014 и ALG.APV-018) имели значительно улучшенные уровни экспрессии при временной трансфекции в клетки HEK-293 по сравнению с форматом Моррисона (т. е., ALG.APV-004). В таблице 12 содержатся иллюстративные данные, демонстрирующие улучшенные уровни экспрессии биспецифичных молекул ADAPTIR™ (например, ALG.APV-006, ALG.APV-010, ALG.APV-014 и ALG.APV-018) по сравнению с биспецфическими конструкциями Моррисона (ALG.APV-004). Этот результат был неожиданным, поскольку аминокислотная последовательность вариабельных доменов 1618 и 1210 не была модифицирована для достижения этого улучшения. Эти результаты показывают, что биспецифичные молекулы ADAPTIRTM могут обладать преимуществами для производства терапевтических белковых лекарственных препаратов; более высокие уровни экспрессии, как правило, считаются преимуществом при производстве терапевтических белковых лекарственных средств, поскольку это может обеспечить более низкую стоимость товаров и другие виды эффективности производственного процесса, например, меньшее количество производственных циклов, необходимых для удовлетворения рыночного спроса.
[00267] Помимо явного улучшения экспрессии в HEK-293 уровни агрегатов, количественно определенные после очистки с белком A биспецифичной молекулы из кондиционированной среды, были значительно улучшены в биспецифичных конструкциях ADAPTIRTM (ALG.APV-006, ALG.APV-010, ALG.APV-014 и ALG.APV-018) по сравнению с биспецифичным форматом Моррисона (ALG.APV-004), как показано в таблице 12.
Таблица 12. Уровни временной экспрессии в HEK-293 и чистота после очистки с белком A биспецифичных конструкций ADAPTIR™ по сравнению с биспецифичными конструкциями Моррисона
[00268] Порядок доменов VH и VL также оценивали с scFv как 1210, так и 1618 (VH-VL или VL-VH), и было определено, что предпочтительной ориентацией scFv 1618 является VH-VL, поскольку уровни экспрессии молекулы scFv-Fc-scFv были значительно снижены при использовании ориентации VL-VH. Предпочтительной ориентацией scFv 1210 была VH-VL, обеспечивавшая улучшенное связывание с 5T4-экспрессирующими клетками и активность in vitro в репортерном анализе CD137 по сравнению с биспецифичными молекулами, содержавшими scFv 1210 в формате VL-VH, как показано в таблице 13.
Таблица 13. Сравнение связывания с клетками (EC50) и активности in vitro в репортерном анализе CD137 в случаях, когда scFv 1210 находится в ориентации VH-VL или VL-VH
[00269] Также была оценена длина линкера между доменами VH и VL в случае scFv 1618 и 1210. Изменение длины линкера, соединяющего VH и VL с 4-кратным или 3-кратным повтором линкера Gly4Ser, по-видимому, не приводило к значительной разнице в активности или стабильности (данные не показаны), поэтому обычно использовали 4-кратный повтор, если не указано иное.
Пример 2. Оптимизация связывающих scFv-доменов к CD137 1618 и к 5T4
[00270] После первоначальной характеристики биспецифичной конструкции ADAPTIR™ (ALG.APV-006) было желательно увеличить 1) аффинности связывания связывающих scFv-доменов с их соответствующими мишенями; 2) биофизическую стабильность; и 3) активность in vitro. Ожидается, что улучшение этих параметров приведет к улучшению стоимости товаров, простоте изготовления и снижению величины клинических доз. Для оптимизации связывающих доменов были созданы фаговые библиотеки случайного мутагенеза для scFV 1618 и 1210 с использованием подверженной ошибкам ПЦР. Вкратце, каждый scFv использовали в качестве матрицы в подверженной ошибкам реакции ПЦР с использованием коммерчески доступного набора для мутагенеза (GeneMorph II Random Mutagenesis Kit, Agilent Technologies) в соответствии с протоколом производителя. Продукты подверженной ошибкам ПЦР расщепляли, лигировали в вектор фагемида-scFV и трансформировали в компетентные клетки E. coli SS320/M13K07 для создания фаговых библиотек. Для каждой библиотеки проводили пять циклов пэннинга, используя биотинилированный ECD 4-1BB или 5T4 в качестве антигена (для связывающих доменов 1618 или 1210, соответственно). Для каждого последующего цикла использовали увеличенную строгость пэннинга, уменьшая концентрацию антигена и увеличивая время промывки. После последнего цикла пэннинга полученных фагов высевали и готовили для массового клонирования пула scFv в подготовленный вектор экспрессии ADAPTIR™. Было отобрано приблизительно 400 отдельных колоний, фагемиды были выделены и секвенированы, и очищенную ДНК использовали для высокопроизводительной временной трансфекции в клетки 293 малом масштабе (в объеме культуры ~0,6 мл). 5-дневные очищенные супернатанты анализировали на связывание с помощью проточной цитометрии и SPR. В отношении наиболее эффективных вариантов затем проводили ряд тестов, включая связывание с клетками, активность in vitro и различные анализы стабильности. Эти биспецифичные варианты ADAPTIRTM были получены с изменениями в scFv либо 1210, либо 1618, при одновременном сохранении другого scFv в виде неизмененной исходной последовательности. Это упростило интерпретацию данных и позволило авторам оценить влияние этих отдельных изменений на связывание, активность и стабильность. На основе улучшения стабильности, аффинности или биологической активности были выявлены полезные точечные мутации. Затем их объединяли для получения дополнительного набора конструкций ДНК ADAPTIR. Эти конструкции включали несколько изменений в scFv 1210 и/или 1618. Каждую конструкцию экспрессировали в клетках HEK-293 путем временной трансфекции в объеме культуры 250 мл и очищали с использованием стадий очистки с белком A и SEC. Конечную чистоту белка подтверждали с помощью аналитической SEC, а уровни эндотоксина определяли с использованием прибора Endosafe PTS в соответствии с инструкциями производителя, чтобы гарантировать, что результаты анализа активности in vitro не будут искажены из-за присутствия эндотоксина. Буфер полученного белка заменяли на PBS как часть процесса очистки методом SEC, концентрировали до 1 мг/мл, подвергали стерилизующей фильтрации и хранили при 4°C до следующего использования. Концентрацию белка определяли по величине поглощения при 280 нм, используя теоретический коэффициент экстинкции.
Пример 3. Оценка полученных из фагов оптимизированных вариантов
[00271] В процессе пэннинга фагов было идентифицировано несколько полезных изменений аминокислот в scFv 1210 и 1618 по сравнению с исходной конструкцией ALG.APV-006. Одним из показателей коллоидной стабильности белка является исследование количества белка, который выпадает в осадок из раствора при добавлении сульфата аммония до конечной концентрации приблизительно 1 М. В этих условиях исходная конструкция ALG.APV-006 теряла почти 89% белка в растворе (таблица 14). Для сравнения, все изменения, представленные ALG.APV-099, ALG.APV-127, ALG.APV-148 и ALG.APV-150, демонстрируют снижение потери белка в этих условиях, что дает основания полагать, что отдельные изменения аминокислот, сделанные в этих конструкциях, улучшили коллоидную стабильность биспецифичных конструкций ADAPTIR™. Еще одной мерой стабильности белка является изучение влияния приложения силы сдвига к образцу белка и исследование этого образца на предмет потери в результате осаждения или образования растворимых агрегированных форм биспецифичного белка. При оценке в сдвиговом анализе, в котором раствор белка помещали в 96-луночный планшет и встряхивали при 2000 об/мин в течение двух часов, мутация ALG.APV-127 показала значительно сниженную величину потери белка, тогда как другие варианты показали минимальный эффект или несколько усугубленную потерю (таблица 14). Еще одна мера термической стабильности может быть определена при использовании дифференциальной сканирующей калориметрии (DSC) или дифференциальной сканирующей флуориметрии (DSF) для определения температуры в средней точке кривой плавления, иначе известной как Tm. После определения Tm вариантов 1618 и 1210 авторы отметили, что варианты ALG.APV-127 и ALG.APV-150 показали повышенную на три-четыре градуса Tm scFv 1618 по сравнению с исходной последовательностью ALG.APV-006 (таблица 14). Повышение значения Tm, как правило, может быть интерпретировано как улучшение стабильности свернутого белка, поскольку это означает, что белок более устойчив к тепловому разворачиванию/денатурации. ALG.APV-148 повысила термическую стабильность scFv 1210, так как Tm была повышена почти на три градуса по сравнению с исходной последовательностью 1210, используемой в ALG.APV-006.
[00272] Были оценены различные комбинации мутаций ALG.APV-099, ALG.APV-127, ALG.APV-148 и ALG.APV-150, чтобы понять, обеспечивают ли комбинированные мутации аддитивный положительный эффект в отношении биофизической стабильности биспецифичных конструкций ADAPTIR™. Они были экспрессированы и очищены с использованием того же метода и оценены в тех же анализах, что и отдельные точечные мутации. Результаты в таблице 14 показывают, что комбинация мутаций, включенных в ALG.APV-178 и ALG.APV-179, в качестве иллюстративных примеров, привела к повышению стабильности, которое в целом было эквивалентным или лучшим, чем достигнутое в результате отдельных изменений.
Таблица 14. Данные биофизической оценки для неоптимизированных и оптимизированных биспецифичных конструкций ADAPTIR™
% потери белка
scFv 1618
scFv 1210
[00273] В дополнение к проведенным оценкам стабильности определяли аффинность связывания ALG.APV-006 и полученных из фагов вариантов с помощью поверхностного плазмонного резонанса (SPR) с использованием Biacore T-200, используя рекомбинантные внеклеточные домены человеческого 5T4 и человеческого CD137. Модификации ALG.APV-127 и ALG.APV-150 привели к значительно более высокой аффинности связывания с CD137 (таблица 15). Аналогичным образом было отмечено, что ALG.APV-099 связывается в значительно более высокой степени, чем ALG.APV-006 (13 нМ по сравнению со 149 нМ, соответственно).
Таблица 15. Сводные данные в отношении оценки связывания и активности для неоптимизированных и оптимизированных биспецифичных конструкций ADAPTIR™
CD137 методом SPR
KD (нМ)
5T4 методом SPR
KD (нМ)
ЕС50 (нМ)
[00274] Связывание вариантов ALG.APV-099, ALG.APV-127, ALG.APV-148 и ALG.APV-150 с CD137 и 5T4, экспрессируемыми на поверхности клеток, сравнивали со связыванием ALG.APV-006. Несмотря на улучшенную аффинность к растворимому CD137 или 5T4, проявляемую некоторыми из этих мутаций, в экспериментах по связыванию на поверхности клетки значительных различий не наблюдалось (данные не показаны). Для сравнения эффективности вариантов, полученных из фагового дисплея, в индукции зависимой от мишени активации CD137, варианты сравнивали в репортерном анализе CD137.
[00275] Репортерный анализ CD137: трансфектанты Jurkat/CD137, несущие репортерный ген люциферазы под контролем промотора NF-κB (Promega), культивировали в соответствии с протоколами производителя. Репортерные клетки Jurkat/NF-κB культивировали с клетками CHO-K1, трансфицированными 5T4 человека, в количестве приблизительно 15 000 репортерных клеток на 30 000 клеток-мишеней в 96-луночных планшетах. Добавляли концентрации биспецифичных молекул с конечной концентрацией от 10 нМ до 0,002 нМ. Клетки культивировали в общем объеме 100 мкл среды RPMI 1640 с добавками 1% эмбриональной бычьей сыворотки, пирувата натрия, антибиотиков и заменимых аминокислот. Планшеты инкубировали при 37°C, 5% CO2 в инкубаторах с увлажненной средой в течение 5-6 часов. Сто микролитров буфера Bio-Glow (Promega) добавляли в каждую лунку и инкубировали в течение 5-10 минут перед измерением флуоресценции. Люминесценцию измеряли на счетчике для микропланшетов MicroBeta2 2450 (Perkin Elmer). Нелинейный регрессионный анализ для определения значений EC50 проводили с помощью программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
[00276] На ФИГ. 1 показано, что все конструкции показали агонистическую функцию в присутствии 5T4(+) клеток; в отсутствие 5Т4(+) клеток репортерной активности не наблюдалось (не показано). Конструкции ALG.APV-099, ALG.APV-127, ALG.APV-148 и ALG.APV-150 показали лучшую агонистическую функцию (более низкие значения EC50) по сравнению с исходной конструкцией ALG.APV-006. Каждая точка на кривой представляет собой среднее значение для параллельных лунок. Молекула в формате Моррисона ALG.APV-004 была включена для сравнения. На оси y показаны значения в относительных единицах флуоресценции (RLU), представленные в процентах от максимальной флуоресценции.
[00277] Так как посредством отдельных точечных мутаций, представленных ALG.APV-099, ALG.APV-127, ALG.APV-148 и ALG.APV-150, были достигнуты улучшения, указанные мутации были объединены различным образом, чтобы определить, будут ли характеристики отдельных мутаций обеспечивать аддитивный положительный эффект при включении в один и тот же белок. Эти уникальные мутации были оценены в комбинации с получением вариантов scFv 1618 и 1210 со значительно улучшенными свойствами, таких как ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196, ALG.APV-198 и ALG.APV-199. Для создания конструкций ALG.APV-191, ALG.APV-196, ALG.APV-198 и ALG.APV-199 отдельные точечные мутации дополнительно комбинировали с мутациями LO1, LO2 и LO11, описанными в примерах 22 и 23 заявки PCT № PCT/EP2017/059656. Кроме того, были созданы целевые фаговые библиотеки после анализа областей scFv 1618 и 1210 для выявления областей, которые могут способствовать нестабильности. Однако пэннинг этих библиотек не дал scFv с улучшенными свойствами (данные не показаны).
Пример 4. Оценка стабилизирующих дисульфидных связей
[00278] Были сделаны точечные мутации для включения дополнительного остатка цистеина в домены VH и VL scFv как 1618, так и 1210. При экспрессии этих биспецифичных молекул эти цистеины реагируют с образованием дисульфидной связи и могут способствовать повышению стабильности scFv и придавать полезные свойства для улучшения производства и хранения этих продуктов. Были проведены эксперименты, демонстрирующие, что при хранении оптимизированных биспецифичных молекул со стабилизирующей дисульфидной связью в scFv 1210 в течение одной недели при 4 или 25°C или при подвергании их трем циклам замораживания-оттаивания от -80°C до комнатной температуры они сохраняют свою чистоту лучше, чем биспецифичные молекулы без дисульфидной связи. Иллюстративные данные представлены в таблице 16. Также было оценено добавление стабилизирующей дисульфидной связи в scFv 1618. Хотя дисульфидная связь, по-видимому, обеспечивает преимущество в части стабильности, данные связывания Biacore дают основания полагать, что образец отличался неоднородностью, приводящей к нетипичной кинетике связывания (данные не показаны).
Таблица 16. Улучшение стабильности при хранении и при замораживании-оттаивании биспецифичных молекул, содержащих стабилизирующую дисульфидную связь в scFv 1210
7 день при 4˚C
7 день при 25˚C
-80oC до комнатной температуры
[00279] Улучшение стабильности биспецифичной молекулы с включением стабилизирующей дисульфидной связи в scFv 1210 также было понятно при последующей оценке, проведенной при 40°C. Две конструкции, ALG.APV-209 и ALG.APV-210, отличаются добавлением стабилизирующий дисульфидной связи в scFv 1210 в ALG.APV-210. Через одну неделю при этой повышенной температуре ALG.APV-210 образовала значительно меньше агрегированного вещества (таблица 17).
Таблица 17. Данные по улучшенной стабильности при 40°C для биспецифичной молекулы со стабилизирующей дисульфидной связью в scFv 1210
7 день при 40˚C
[00280] В то время как включение дисульфидной связи в 1210 обеспечило значительное преимущество в отношении стабильности при хранении, другие параметры стабильности, которые были оценены, не обнаружили значительного различия между двумя конструкциями (таблица 18). Обе конструкции показали значительно лучшие результаты в этих анализах по сравнению с исходной биспецифичной молекулой ALG-006 (таблица 14).
Таблица 18. Сравнение данных по стабильности ALG.APV-209 с ALG.APV-210
% потери белка
Пример 5. Оценка аффинности связывания биспецифичных белков scFv2Fc с 5T4 человека и CD137 человека
Методы
[00281] Экспрессия и очистка рекомбинантных внеклеточных доменов (ECD) человеческого 5T4 и человеческого CD137: с использованием стандартных методов молекулярной биологии и начиная с вектора, кодирующего полноразмерную последовательность белков 5T4 и CD137, были разработаны нуклеотидные праймеры для амплификации внеклеточных областей человеческого 5T4 и человеческого CD137. Дополнительная пептидная последовательность была добавлена к c-концу ECD в положении, в котором, как ожидается, природный белок входит в мембрану. Этот пептид содержал последовательности распознавания, которые могут быть использованы для целей аффинной очистки. Векторы экспрессии временно трансфицировали в клетки HEK-293. Рекомбинантный ECD очищали из кондиционированной среды с использованием металл-аффинной хроматографии с иммобилизованными ионами металлов (IMAC) и эксклюзионной хроматографии. Чистоту белка подтверждали с помощью аналитической SEC.
[00282] Анализы аффинности биспецифичных белков к рекомбинантному эктодомену CD137 и 5T4 методом поверхностного плазмонного резонанса (SPR): исследования аффинности связывания методом SPR биспецифичных белков к 5T4 x CD137 с рекомбинантным мономерным эктодоменом (ECD) 5T4 и CD137 проводили при 25°C в буфере HBS-EP+ в системе Biacore T200. Козье антитело к фрагменту F(ab')2 человеческого IgG (Jackson ImmunoResearch) в концентрации 20 мкг/мл в 10 мМ ацетате натрия (pH 4,5) иммобилизовали с плотностью 2000 единиц ответа (RU) в проточной ячейке сенсорного чипа для исследований CM5 (GE) посредством стандартной иммобилизации по аминогруппе. Каждый биспецифичный вариант в концентрации 200 нМ в буфере HBS-EP+ был захвачен в проточной ячейке иммобилизованным антителом к человеческому IgG при скорости потока 50 мкл/мин в течение 60 секунд для достижения ответа ~500 RU, при этом поверхность одной проточной ячейки оставляли без изменений в качестве контроля. После 30-секундного периода стабилизации четыре различные концентрации каждого ECD (0, 20, 60 и 180 нМ) вводили со скоростью 50 мкл/мин в течение либо 120 секунд с последующим периодом диссоциации 240 секунд (для 5T4), либо 90 секунд с последующим периодом диссоциации 180 секунд (для 4-1BB). Регенерацию осуществляли путем двукратных инъекций 10 мМ глицина (рН 1,5) при скорости потока 50 мкл/мин в течение 30 с последующей стабилизацией буфером HBS-EP+ в течение 1 мин.
[00283] Сенсограммы, полученные из измерений кинетики в SPR, были проанализированы методом двойного вычитания. Сигнал от контрольной проточной ячейки вычитали из ответа связывания аналита, полученного из проточных ячеек с иммобилизованными или захваченными лигандами. Контрольные ответы буфера затем усредняли по нескольким введениям. Усредненные контрольные ответы буфера затем вычитали из ответов связывания аналита, и окончательные данные с двойным контролем анализировали с помощью программного обеспечения Biacore T200 Evaluation (2.0, GE), проводя глобальную аппроксимацию данных с получением кинетических параметров. Все сенсограммы были аппрокисимрованы с использованием простой модели связывания один к одному.
Результаты
[00284] В таблице 19 показаны параметры связывания, определенные с помощью SPR для биспецифичных конструкций ADAPTIR™, связывающихся с CD137 и 5T4. Следует отметить, что проведение измерений аффинности в этом формате с захватом биспецифичной молекулы на чипе и введением рекомбинантного мономерного ECD CD137 или 5T4 должно привести к истинным значениям аффинности, которые не искажаются эффектами авидности. Как показано в таблице 19, путем тщательного скрининга рандомизированных фаговых библиотек удалось выделить варианты связывающего домена, которые привели к значительному улучшению аффинности связывания scFv к CD137 и/или к 5Т4. По сравнению с неоптимизированной исходной конструкцией ALG.APV-006 оптимизированными биспецифичными конструкциями ADAPTIR™ ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-209 и ALG.APV-210 было достигнуто большое снижение значения KD к человеческому CD137. Аналогичным образом ALG.APV-179, ALG.APV-187, ALG.APV-209 и ALG.APV-210 демонстрировали улучшенные значения аффинности к человеческому 5T4 по сравнению с неоптимизированной ALG.APV-006.
Таблица 19. Константы аффинности связывания (KD), диссоциации и ассоциации для биспецифичных молекул, связывающихся с рекомбинантным CD137 человека или 5T4 человека
методом SPR
методом SPR
Пример 6. Оценка термической стабильности биспецифичных белков scFv-Fc-scFv к человеческому 5T4 и человеческому CD137
[00285] Дифференциальная сканирующая калориметрия (DSC) и дифференциальная сканирующая флуориметрия (DSF) являются инструментами, обычно используемыми для измерения стабильности структуры рекомбинантных белков. Измеряя энергию, необходимую для повышения температуры образца белка, можно определить среднюю температуру плавления (Tm) отдельных белковых доменов. Общепринято, что белковые домены с более высокими значениями Tm считаются более стабильными. Анализ DSF проводили с использованием набора оптимизированных биспецифичных белков ADAPTIRTM (ALG.APV-178, ALG.APV-179 и ALG.APV-187) и сравнивали с неоптимизированной биспецифичной версией ADAPTIRTM (ALG.APV-006) (таблица 20). Значительное повышение Tm 1618 к CD137 и 1210 к 5T4 наблюдалось после включения полезных мутаций, которые были идентифицированы с помощью описанного выше подхода фагового дисплея случайного мутагенеза.
Таблица 20. Средняя температура плавления, Tm, оптимизированных биспецифичных конструкций ADAPTIR™ по сравнению с неоптимизированными конструкциями
Пример 7. Связывание молекул биспецифичных белковых молекул scFv-Fc-scFv с линиями CD137(+) клеток человека и отличного от человека примата.
[00286] Чтобы подтвердить, что связывание с CD137 на поверхности клеток не было потеряно после преобразования вариабельных доменов в scFv или введения мутаций, была использована проточная цитометрия для количественного определения связывания сконструированных биспецифичных молекул к CD137 x 5T4 ADAPTIR™ с линиями клеток, экспрессирующих CD137 человека или яванского макака.
[00287] Исследования связывания на клетках линии CHO-K1, экспрессирующих CD137, проводили с помощью стандартных процедур окрашивания на основе проточной цитометрии. Клетки CHO-K1 (полученные от Alligator) трансфицировали CD137 человека или яванского макака. В типичном эксперименте метили приблизительно 100 000 клеток на лунку в 96-луночных планшетах с помощью от 100 нМ до 0,05 нМ связывающей молекулы в 50 мкл солевого буфера с 2% BSA и 2 мМ ЭДТА в течение 30 мин на льду с последующими промывками и инкубацией с меченым РЕ вторичным антителом, козьим антителом к Fcγ человеческого IgG (Jackson Laboratory), в течение 20 минут на льду. Сигнал от связанных молекул детектировали с использованием LSR-IITM или проточного цитометра Symphony A3 (BD Biosciences) и анализировали с помощью программного обеспечения для анализа данных проточной цитометрии FlowJo. Медианную интенсивность флуоресценции (медианная MFI) связанных молекул на живых клетках определяли после исключения дублетов клеток. Нелинейный регрессионный анализ для определения значений EC50 проводили с помощью программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
[00288] На ФИГ. 2 показаны кривые связывания семи молекул ADAPTIR™ (ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198) с линией клеток CHO-K1/ CD137 человека. Все молекулы показали схожие уровни связывания (значения EC50) и насыщения. На ФИГ. 3 показаны кривые связывания тех же семи конструкций с линией клеток CHO-K1/CD137 яванского макака. Схожие уровни связывания и насыщения наблюдались для всех семи конструкций для мишени яванского макака. На ФИГ. 12 показаны кривые связывания шести молекул ADAPTIR™ (ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210) и контроля в формате Моррисона ALG.APV-004 с линией клеток CHO-K1/ CD137 человека. Все молекулы связывались со схожими значениями EC50 от 0,3 до 0,6 нМ и схожими уровнями насыщения.
Пример 8. Связывание молекул биспецифичных белков scFv-Fc-scFv с линиями 5T4(+) клеток человека и отличного от человека примата.
[00289] Для сравнения связывания 5Т4 на поверхности раковых клеток после внесения изменений в scFv-домены использовали проточную цитометрию для количественного определения связывания сконструированных биспецифичных связывающих молекул к CD137 × 5Т4 ADAPTIR™ с линиями клеток, экспрессирующими 5Т4.
[00290] Связывание биспецифичных молекул CD137 x 5T4 с линиями 5T4(+) клеток: сравнивали характеристики связывания биспецифичных молекул ADAPTIR™ (ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-196, ALG.APV-208, ALG.APV-209 и ALG.APV-210). Исследования связывания проводили с помощью стандартных процедур окрашивания на основе проточной цитометрии двух линий клеток, экспрессирующих 5T4: клеток CHO-K1 (полученных от Alligator), трансдуцированных 5T4 человека или яванского макака, и клеток аденокарциномы яичника человека SKOV-3, экспрессирующих (человеческий) 5T4. Все мечения и промывки проводили в 96-луночных планшетах с U-образным дном в солевом буфере с 0,1% BSA и 2 мМ ЭДТА. Клетки высевали с плотностью приблизительно 100 000 клеток на лунку и инкубировали с исследуемыми молекулами в концентрации от 100 нМ до 0,05 нМ в объеме 50 мкл/лунка в течение 30 минут на льду. Клетки трижды промывали, затем инкубировали в течение еще 30 мин на льду с флуоресцентно-меченым вторичным поликлональным козьим антителом к F(ab’)2 человеческого IgG (Jackson ImmunoResearch Laboratories). Затем клетки дважды промывали и вводили образцы в проточный цитометр BD LSRII или Symphony A3. Файлы образцов анализировали с использованием программного обеспечения FlowJo; среднюю интенсивность флуоресценции (средняя MFI) или медианную интенсивность флуоресценции (медианная MFI) для живой популяции клеток в каждой лунке рассчитывали после гейтинга живых клеток (по сигналам прямого и бокового светорассеяния). Нелинейный регрессионный анализ для определения значений EC50 проводили с помощью программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
[00291] На ФИГ. 4 показаны кривые связывания семи молекул ADAPTIR™ (ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198) с линией клеток CHO-K1/ 5T4 человека. Конструкция ALG.APV-006 связывалась со значением EC50 > 10 нМ, тогда как остальные конструкции ADAPTIR™ (ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198) связывались со значениями EC50 от 3 до 10 нМ. Различия в значениях EC50 между конструкциями были более явными, когда анализы связывания проводили на клетках SKOV-3. Клетки SKOV-3 экспрессируют 5T4 человека на уровне приблизительно в 10 раз более низком, чем трансфектанты CHO-K1/ 5T4 человека (ФИГ. 5). На ФИГ. 6 показаны кривые связывания конструкций ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-196 и ALG.APV-198, полученные на клетках SKOV-3. Конструкции ALG.APV-006, ALG.APV-078, ALG.APV-179 и ALG.APV-187 связывались с немного более низкой аффинностью (более высокими значениями EC50) и более высокими максимальными уровнями связывания, чем конструкции ALG.APV-196 и ALG.APV-198. На ФИГ. 7 показаны кривые связывания ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198 на трансфектантах CHO-K1/5T4 яванского макака. Аналогично профилю связывания на человеческих 5T4(+) клетках конструкции ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198 показали более низкую аффинность и более высокие максимальные уровни связывания, чем конструкции ALG.APV-196 и ALG.APV-198.
[00292] На ФИГ. 13 показаны кривые связывания шести молекул ADAPTIR™ (ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210) и контроля в формате Моррисона ALG.APV-004 с мышиными клетками CT26 (ATCC), экспрессирующими 5T4 человека. Эти клетки экспрессируют 5Т4 человека на уровнях, сопоставимых с наблюдаемыми для клеток SKOV-3 (ФИГ. 5). Все конструкции связывались со значениями EC50 от 1 до 3 нМ. Конструкции ALG.APV-179, ALG.APV-187, ALG.APV-209 и ALG.APV-210 показали более высокие уровни максимального связывания, чем ALG.APV-178 и ALG.APV-208. Контрольная молекула в формате Моррисона ALG.APV-004 показала самые низкие уровни максимального связывания. На ФИГ. 14 показаны кривые связывания ALG.APV-004, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210 на трансфектантах CHO-K1/5T4 яванского макака. Все молекулы связывались со схожими значениями EC50 от 6 до 10 нМ и схожими уровнями насыщения. На ФИГ. 15 показаны кривые связывания ALG.APV-004, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210 с линией клеток человека, не экспрессирующих ни CD137, ни 5T4 (клетки MOLM13, ATCC). Ни одна из молекул не показала связывания при какой-либо концентрации.
Пример 9. Связывание биспецифичного антитела к 5T4-CD137 с линиями 5T4-экспрессирующих клеток.
[00293] Проводили эксперименты для определения связывания ALG.APV-210 с 5T4 человека, экспрессируемым на клетках при различной плотности рецепторов. Как линию опухолевых клеток человека с эндогенными уровнями 5T4, так и клетки, трансфицированные для продуцирования 5T4, оценивали на предмет целевого связывания ALG.APV-210.
Материалы и методы
[00294] Биотинилирование: для детекции связывания ALG.APV-210 с линиями 5T4-экспрессирующих клеток ALG.APV-210 был сначала биотинилирован. Качество биотинилированного белка и влияние на связывание с его мишенями оценивали с помощью ELISA.
[00295] Окрашивание для FACS и анализ линий 5T4-экспрессирующих клеток: перед окрашиванием для FACS все линии клеток характеризовали по их уровням экспрессии 5T4 с использованием набора для определения плотности рецепторов (Quantum Simply Cellular, античеловеческое IgG, Bangs Laboratories, Inc). Информация об использованных линиях клеток и их соответствующих плотностях экспрессии 5T4 представлена в таблице 21. Прикрепленные клеточные линии культивировали в соответствии с инструкциями их поставщика и собирали из колб, в которых их культивировали, с использованием трипсина/ЭДТА, подсчитывали в среде R10 в счетной камере Бюркера и разводили буфером FACS (PBS + 0,5% BSA, 0,05% NaN3) до концентрации 4х 106 клеток/мл. 50 мкл (0,20 x 106) раствора клеток добавляли в 96-луночный планшет для окрашивания FACS (Falcon № 351190). Клетки инкубировали в течение 20 мин при 4°С с 50 мкл биотинилированного ALG.APV-210 (двукратная конечная концентрация). Добавляли последовательные разведения антител, начиная с титра 20 мкг/мл, с 12-ступенчатым разведением в 2 раза до 0,010 мкг/мл. После двухкратной промывки буфером FACS клетки инкубировали в течение 30 мин при 4°С со 100 мкл вторичного антитела (стрептавидин-РЕ, BD № 554061, разведенное 1/500). После двух стадий промывки буфером FACS клетки повторно суспендировали в 200 мкл реагента для фиксации клеток (BD CellFIX 10x BD, № 340181, разведенный в MQ H2O). Клетки анализировали на связывание ALG.APV-210 с использованием проточной цитометрии (FACSVerse) путем гейтинга отдельных клеток. Значения MFI (вычитали только вторичное антитело) в 1 из 2 независимых экспериментов, а также нормированные данные и значения EC50 объединенных данных из 2 экспериментов определяли и наносили на график с использованием GraphPad Prism 7, нелинейная регрессионная зависимость log (агонист) от нормированного ответа - переменный угловой коэффициент.
Таблица 21. Линии клеток, экспрессирующих 5T4, и плотности рецептора/клетка
Результаты и выводы
[0029] С помощью ELISA было установлено, что связывание биотинилированного ALG.APV-210 с 5T4 аналогично связыванию небиотинилированного ALG.APV-210 (данные не показаны).
[00297] Было оценено связывание (ЕС50) биотинилированного ALG.APV-210 с 5Т4 при экспрессии на клетках, либо на эндогенных уровнях на опухолевых клетках человека, либо на клетках, трансфицированных для экспрессии 5Т4. Как показано на фиг. 39 с помощью MFI, ALG.APV-210 дозозависимым образом связывается как с человечекими опухолевыми клетками, экспрессирующими эндогенные уровни 5T4, так и с трансфицированными линиями клеток.
[00298] В приведенной выше таблице 21 показана EC50 связывания ALG.APV-210 с 5T4-положительной линией опухолевых клеток человека и 5T4-трансфицированными клетками, определенная по нормированным значениям MFI (за вычетом фоновых значений) из 2 объединенных экспериментов, построенных на графике доза-ответ, показанном на фиг. 40. Средние значения EC50 составляли 8-23 нМ для разных линий 5T4-положительных клеток. Таким образом, ALG.APV-210 связывается с 5T4-экспрессирующими клетками при EC50, составляющей 8-23 нМ.
Пример 10. Агонистическая функция биспецифичных белков scFv-Fc-scFv в репортерных анализах
[00299] Чтобы сравнить эффективность различных биспецифичных CD137-связывающих молекул в отношении индукции мишень-зависимой активации CD137, семь разных биспецифичных молекул к CD137 x 5T4 сравнивали в репортерном анализе CD137, как описано в примере 3.
Репортерный анализ CD137
[00300] Трансфектанты Jurkat/CD137, несущие репортерный ген люциферазы под контролем промотора NF-κB (Promega), культивировали в соответствии с протоколами производителя. Репортерные клетки Jurkat/NF-κB культивировали с клетками первичной протоковой карциномы молочной железы человека HCC1143 (ATCC), экспрессирующими 5T4 человека, в количестве приблизительно 15 000 репортерных клеток на 30 000 клеток-мишеней в 96-луночных планшетах. Добавляли концентрации биспецифичных молекул с конечной концентрацией от 10 нМ до 0,002 нМ. Клетки культивировали в общем объеме 100 мкл среды RPMI 1640 с добавками 10% эмбриональной бычьей сыворотки, пирувата натрия, антибиотиков и заменимых аминокислот. Планшеты инкубировали при 37°C, 5% CO2 в инкубаторах с увлажненной средой в течение 5-6 часов. Сто микролитров буфера Bio-Glow (Promega) добавляли в каждую лунку и инкубировали в течение 5-10 минут перед измерением флуоресценции. Люминесценцию измеряли на счетчике для микропланшетов MicroBeta2 2450 (Perkin Elmer). Нелинейный регрессионный анализ для определения значений EC50 проводили с помощью программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
Результаты
[00301] На ФИГ. 8 все конструкции показывают агонистическую функциональную активность в присутствии 5T4(+) клеток; в отсутствие 5Т4(+) клеток репортерной активности не наблюдалось (данные не показаны). Конструкции ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-191, ALG.APV-196 и ALG.APV-198 показали лучшую агонистическую функциональную активность (вплоть до 6-кратно более низкие значения EC50) по сравнению с неоптимизированной конструкцией ADAPTIRTM, ALG.APV-006 и контрольной конструкцией в формате Моррисона ALG.APV-004. Каждая точка на кривой представляет собой среднее значение для параллельных лунок. На оси y показаны значения в относительных единицах флуоресценции (RLU). На ФИГ. 16 показана активность биспецифичных конструкций в репортерных клетках Jurkat/NF-κB после 5 часов инкубации в присутствии 5T4(+) клеток (HCC1143) или 5T4(-) клеток (MOLM13). Конструкции ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210 показали лучшую агонистическую функциональную активность (вплоть до 10-кратно более низкие значения EC50) по сравнению с контрольной конструкцией в формате Моррисона ALG.APV-004. Репортерной активности не наблюдалось в присутствии 5T4(-) клеток (MOLM13). Каждая точка на кривой представляет собой среднее значение для параллельных лунок. На оси y показаны значения в относительных единицах флуоресценции (RLU).
Пример 11. Агонистическая функция биспецифичных белков scFv-Fc-scFv в анализах с первичными T-клетками
[00302] Агонистическую функцию различных биспецифичных молекул к CD137 x 5T4 в отношении индукции мишень-зависимой активации CD137 тестировали в культурах мононуклеарных клеток периферической крови (МНПК). Для тестирования функции биспецифичных конструкций к CD137 первичные МНПК стимулировали антителами к CD3 для повышения экспрессии CD137, который не экспрессируется на покоящихся Т-клетках. Совместная стимуляция первичных Т-клеток посредством TCR и CD137 усиливает секрецию Т-клетками цитокина интерферона-гамма (ИФН-γ).
Стимуляция МНПК посредством CD137:
[00303] Мононуклеарные клетки периферической крови (МНПК) выделяли из человеческой крови с использованием стандартных градиентов фиколла. Выделенные клетки промывали в солевом буфере. МНПК культивировали с клетками CHO-K1/ 5T4 человека в количестве приблизительно 120 000 МНПК на 30 000 5T4(+) клеток в 96-луночных планшетах. Во все лунки добавляли антитело к CD3 OKT3 (eBioscience) в концентрации 0,1 мкг/мл. Клетки культивировали в общем объеме 200 мкл среды RPMI 1640 с добавками 1% эмбриональной бычьей сыворотки, пирувата натрия, антибиотиков и заменимых аминокислот. Планшеты инкубировали при 37°C, 5% CO2 в инкубаторах с увлажненной средой в течение 72 часов. Через 72 часа собирали сто микролитров среды. В супернатанте измеряли уровни ИФН-γ с использованием наборов Milliplex® (Millipore) в соответствии с инструкциями производителя. Данные собирали в системе Bio-Plex Reader 200 (Bio-Rad). Нелинейный регрессионный анализ для определения значений EC50 проводили с помощью программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
[00304] На ФИГ. 9 показаны уровни ИФН-γ, индуцированные ALG.APV-006, ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-196, ALG.APV-198, и контрольной конструкцией в формате Моррисона ALG.APV-004. Все конструкции индуцировали более высокие уровни ИФН-γ, чем антитело к CD3 по отдельности, дозозависимым образом. Каждая точка на кривой представляет собой среднее значение для параллельных лунок. На ФИГ. 17 показаны уровни ИФН-γ, индуцированные в первичных культурах МНПК через 72 часа посредством ALG.APV-178, ALG.APV-179, ALG.APV-187, ALG.APV-208, ALG.APV-209 и ALG.APV-210, и контрольной конструкцией в формате Моррисона, ALG.APV-004, в присутствии клеток CHO-K1/ 5T4 человека. Все конструкции индуцировали более высокие уровни ИФН-γ, чем антитело к CD3 по отдельности, дозозависимым образом. Каждая точка на кривой представляет собой среднее значение для параллельных лунок.
[00305] Функциональную активность биспецифичных конструкций к 5Т4 х CD137 также оценивали в анализе с CD8+ Т-клетками, в котором клетки культивировали в планшетах для микротитрования, покрытых конструкцией 5Т4-Fc и антителом к CD3.
Стимуляция очищенных Т-клеток посредством 5T4:
[00306] МНПК выделяли центрифугированием в градиенте плотности с использованием Ficoll-Paque (ρ 1,077 г/мл) (GE Healthcare № 17-1440-02) из концентратов лейкоцитов, полученных от здоровых доноров (Clinical Immunology and Transfusion Medicine,  Лунд, Швеция). CD8+ Т-клетки обогащали посредством отрицательного отбора с использованием набора для выделения CD8+ Т-клеток (Miltenyi 130-096-495). Планшеты покрывали в течение ночи при 4°С 3 мкг/мл α-CD3, клоном OKT3 (Affymetrix eBioscience № 16-0037-85), промывали и покрывали 5 мкг/мл 5T4-Fc в течение 2 ч при 37°С. После покрытия 5T4-Fc планшеты промывали и блокировали в течение минимум 30 минут с помощью RPMI (Gibco № 61870010), содержащей 10% FCS (инактивированной нагреванием, Gibco № 10270-106, лот 41Q9248K) и 10 мМ Hepes (Gibco № 15630056). Биспецифичные конструкции разводили в RPMI, содержащей 10% FCS и 10 мМ Hepes, и добавляли в планшеты за 30 минут до добавления CD8+ T-клеток (0,07 × 106 клеток/лунку). Планшеты для анализа инкубировали в течение 72 ч при 37°С, после чего собирали культуральные супернатанты. Уровни ИФН-γ в супернатантах измеряли методом ELISA (BD OptiEIA № 555142). Эксперимент повторяли дважды в общей сложности с 3 донорами. Результаты первого эксперимента показаны на ФИГ. 11А. Результаты для контрольных образцов, в которых клетки и биспецифичные конструкции культивировали в планшетах, которые не были покрыты 5T4-Fc, показаны на ФИГ. 11В.
Лунд, Швеция). CD8+ Т-клетки обогащали посредством отрицательного отбора с использованием набора для выделения CD8+ Т-клеток (Miltenyi 130-096-495). Планшеты покрывали в течение ночи при 4°С 3 мкг/мл α-CD3, клоном OKT3 (Affymetrix eBioscience № 16-0037-85), промывали и покрывали 5 мкг/мл 5T4-Fc в течение 2 ч при 37°С. После покрытия 5T4-Fc планшеты промывали и блокировали в течение минимум 30 минут с помощью RPMI (Gibco № 61870010), содержащей 10% FCS (инактивированной нагреванием, Gibco № 10270-106, лот 41Q9248K) и 10 мМ Hepes (Gibco № 15630056). Биспецифичные конструкции разводили в RPMI, содержащей 10% FCS и 10 мМ Hepes, и добавляли в планшеты за 30 минут до добавления CD8+ T-клеток (0,07 × 106 клеток/лунку). Планшеты для анализа инкубировали в течение 72 ч при 37°С, после чего собирали культуральные супернатанты. Уровни ИФН-γ в супернатантах измеряли методом ELISA (BD OptiEIA № 555142). Эксперимент повторяли дважды в общей сложности с 3 донорами. Результаты первого эксперимента показаны на ФИГ. 11А. Результаты для контрольных образцов, в которых клетки и биспецифичные конструкции культивировали в планшетах, которые не были покрыты 5T4-Fc, показаны на ФИГ. 11В.
[00307] На ФИГ. 11А показано, что биспецифичные конструкции к 5Т4 х CD137 в формате ADAPTIR™ (ALG.APV-178, ALG.APV-179, ALG.APV-196 и ALG.APV-198) обладают повышенной функциональной активностью по сравнению с биспецифичными конструкциями в формате Моррисона (ALG.APV-004), о чем свидетельствует повышение уровней ИФН-γ, присутствующих в культуральных супернатантах в присутствии 5T4. Фактически, конструкции в формате ADAPTIR™ индуцировали уровни ИФН-γ, которые были в 2-4,5 раза выше, чем индуцируемые конструкциями в формате Моррисона (ФИГ. 11A).
Пример 12. Антигензависимая локализация биспецифичных антител в 5T4-позитивные опухоли in vivo
[00308] Антигензависимую локализацию биспецифичных конструкций, нацеленных на 5T4 и 4-1BB, в 5T4-позитивные опухоли B16-5T4, оценивали на мышах C57BL/6 дикого типа.
Материалы и методы
[00309] Мыши: для экспериментов использовали 8-недельных самок мышей C57BL/6 из Janvier, Франция. Все эксперименты проводили с одобрения этического комитета Мальмё/Лунда.
[00310] Биспецифичные конструкции: использовали две оптимизированные нацеленные на 5T4 и CD137 биспецифичные конструкции в формате ADAPTIRTM: ALG.APV-209 и ALG.APV-210. ALG.APV-004, биспецифичная конструкция в формате Моррисона, служила положительным контролем. Введение носителя служило отрицательным контролем.
[00311] Клетки: клетки B16.F10 дикого типа (B16) были получены от ATCC и культивированы в соответствии с их рекомендациями. Клетки B16, экспрессирующие 5Т4 человека (далее В16-5Т4), были получены от профессора Peter Stern и выращены в селективной среде G418 в концентрации 1,2 мг/мл.
[00312] Методы: для антигензависимой локализации биспецифичных конструкций использовали модель опухолей-близнецов из клеток меланомы B16, в которой каждая мышь получала одну 5T4-негативную и одну 5T4-позитивную опухоль на каждую сторону заднего бока/спины. Линии опухолевых клеток в логарифмической фазе роста вводили подкожно (1x105 клеток в 100 мкл) в 0 день. Конструкцию внутрибрюшинно (100 мкг) вводили на 6 и 13 день, и мышей умерщвляли на 14 день (через 24 часа после последнего введения).
[00313] Анализ методом FACS: опухоли для анализа методом проточной цитометрии механически и ферментативно расщепляли с использованием либеразы TL и ДНКазы I (Roche), и пропускали через 70 мкм клеточное сито. Полученную суспензию отдельных клеток окрашивали для анализа FACS. Вкратце, неспецифичное связывание конструкций блокировали с использованием блокирующего мышиного IgG или Fc. Мертвые клетки исключали с использованием Fixable Viability Stain 450 (Molecular probes) в соответствии с инструкциями производителя. Связывание двух биспецифичных конструкций ADAPTIR™ или контрольных конструкций с опухолевыми клетками анализировали с помощью вторичного антитела: козьего к человеческому Fc-PE (Jackson Immunoresearch). В качестве альтернативы, детекцию биспецифичных конструкций проводили с использованием биотинилированного антигена 4-1BB, а затем стрептавидина-PE/PerCP-Cy5.5. Образцы анализировали на BD FACSVerse, а данные анализировали с использованием программного обеспечения FlowJo. Статистический анализ проводили с использованием непараметрического 2-стороннего T-критерия Манна-Уитни и программы GraphPad prism.
[00314] Иммуногистохимия: опухоли для иммуногистохимии мгновенно замораживали в изопропаноле на сухом льду. Срезы замороженной ткани (8 мм) окрашивали на экспрессию 5T4 с использованием кроличьего антитела к человеческому 5T4 (Abcam) или кроличьего антитела к человеческому Fc (Jackson Immunoresearch) с последующим окрашиванием антикроличьим Brightvision-HRP (Immunologic) и DAPI. Иммуногистохимическое окрашивание оценивали следующим образом: отрицательное (0), слабое окрашивание (1+), умеренное окрашивание (2+) или сильное окрашивание (3+).
Результаты
[00315] Обе биспецифичные конструкции в формате ADAPTIRTM, ALG.APV-209 и ALG.APV-210, и положительный контроль, ALG.APV-004, локализовались в 5T4-экспрессирующие опухоли B16, но не в 5T4-негативные опухоли (B16.F10). Это было продемонстрировано как с помощью проточной цитометрии (ФИГ. 18A и 18B), так и с помощью иммуногистохимии (ФИГ. 19A и 19B).
[00316] Как биспецифичные конструкции (ALG.APV-209, ALG.APV-210), так и положительный контроль (ALG.APV-004) продемонстрировали статистически значимую 5T4-зависимую локализацию в антиген-позитивные опухоли по сравнению с отрицательным контролем носителем, согласно данным проточной цитометрии (ФИГ. 18А). Связывание с 5T4-экспрессирующими опухолями также могло быть обнаружено с использованием биотинилированного 4-1BB для детекции (ФИГ. 18B), что подтверждает, что биспецифичные молекулы были интактными.
[00317] Локализация была дополнительно продемонстрирована иммуногистохимией (ФИГ. 19A и 19B и таблица 22). Биспецифичные антитела в высокой степени связывались с 5T4-позитивными опухолями (B16-5T4, ФИГ. 19A), но не с 5T4-негативными опухолями (B16.F10, ФИГ. 19B).
Таблица 22. In vivo локализация биспецифичных конструкций и контролей носителем
Пример 13. Активность биспецифичного антитела к 5T4-CD137 in vitro в анализе высвобождения ИФН-γ с использованием CD8 Т-клеток человека и опухолевых клеток 5T4-CT26
[00318] Функциональную активность биспецифичного антитела к 5T4-CD137 ALG.APV-210 оценивали в анализе с CD8 Т-клетками, в котором CD8 Т-клетки, стимулированные антителами к CD3, нанесенными на гранулы, совместно культивировали в планшетах с опухолевыми клетками СТ26, экспрессирующими различные уровни 5Т4.
[00319] Опухолевые клетки CT26, линию клеток карциномы толстой кишки мыши (ATCC® CRL-2638), предварительно трансфицировали для экспрессии человеческого 5T4, а затем сортировали на отдельные клетки с использованием проточной цитометрии для создания клонов отдельных клеток, экспрессирующих либо высокие, либо низкие уровни человеческого 5T4 (плотность рецептора см. в таблице 23). Опухолевые клетки 5T4-CT26 или нетрансфицированные клетки дикого типа (CT26 дикого типа) облучали УФ-излучением с использованием сшивающего агента УФ-B (AnalytikJena, источник излучения: 254 нм). После облучения планшета клетки однократно промывали в среде и затем повторно суспендировали в концентрации 4×106 клеток/мл в новой питательной среде R10 (RPMI, Gibco № 61870010, содержащей 10% инактивированной нагреванием FBS, Hyclone № SV30160, партия RB35944, и 10 мМ Hepes, Gibco № 15630056). 50 мкл (2×105 клеток/лунку) облученных УФ-излучением клеток CT26-h5T4высокий, CT26-h5T4низкий или CT26 дикого типа добавляли в планшеты для анализа, обработанные тканевыми культурами (Eppendorf № 0030 730.119), и инкубировали при 37°C в течение ночи.
Таблица 23. Плотность рецептора 5T4 на трансфицированных 5Т4 человека и отсортированных на отдельные клетки клетках CT26
[00320] На следующий день после облучения УФ-излучением клеток CT26 МНПК человека выделяли центрифугированием в градиенте плотности с использованием Ficoll-Paque (GE Healthcare № 17-1440-02) из концентратов лейкоцитов, полученных от здоровых доноров (Clinical Immunology and Transfusion Medicine,  Лунд, Швеция). CD8+ клетки обогащали посредством отрицательного отбора с использованием набора для выделения CD8+ Т-клеток (Miltenyi 130-096-495).
Лунд, Швеция). CD8+ клетки обогащали посредством отрицательного отбора с использованием набора для выделения CD8+ Т-клеток (Miltenyi 130-096-495).
[00321] Готовили последовательные разведения биспецифичного антитела ALG.APV-210 в среде R10, и в каждую лунку планшетов для анализа добавляли 50 мкл и инкубировали при 37°С в течение 30 минут перед добавлением 50 мкл гранул с αCD3 (4 x 108/мл клона антител к CD3: OKT-3, Affymetrix eBioscience № 16-0037-85, нанесенный на гранулы, разведенный до 2×106/мл) при соотношении CD8+ T-клеток к гранулам 1:1, с последующим добавлением 50 мкл эффекторных клеток (обогащенные клетки CD8+, 2 × 106/мл или 1 × 105/лунку). Планшеты для анализа инкубировали в течение 72 ч при 37°С и собирали культуральный супернатант. Уровни ИФН-γ в супернатантах измеряли методом ELISA (BD OptiEIA № 555142). EC50 определяли с помощью нелинейной регрессионной зависимости log (агонист) от нормированного ответа (переменный угловой коэффициент) с использованием GraphPad Prism 7. Величину TOP (максимальное функциональное действие) определяли путем расчета среднего двух самых высоких значений выработки ИФН-γ в культуральном супернатанте. Эксперимент проводили 5 раз, включая в общей сложности 12 доноров.
[00322] В исследовании установили функциональную активность CD137-5T4 биспецифичного антитела ALG.APV-210 с использованием CD8 T-клеток человека (эффекторных клеток), совместно культивированных в планшетах с клетками CT26, экспрессирующими различные уровни 5T4 (для сшивания CD137 посредством связывания с 5T4). Как показано на ФИГ. 20, биспецифичное антитело к CD137-5T4 ALG.APV-210 индуцирует сильную активацию Т-клеток, измеренную по высвобождению ИФН-γ, дозозависимым образом в присутствии клеток CT26, экспрессирующих либо высокий (CT26-h5T4высокий), либо низкий (CT26-h5T4низкий) уровни 5T4. Высвобождение ИФН-g представлено на ФИГ. 20 в виде нормированных данных ALG.APV-210; показаны средние значения для 12 доноров из 5 объединенных экспериментов.
[00323] В таблице 24 представлены EC50 для ALG.APV-210 в анализе с CD8 Т-клетками в присутствии клеток CT26, экспрессирующих 5Т4 человека. Средние значения log (агонист) в зависимости от средних значений нормированного ответа (переменный угловой коэффициент) и 95% CI для 12 доноров из 5 объединенных экспериментов представлены в данной таблице. Как показано на ФИГ. 21A и 21B, максимальная функциональная активность ALG.APV-210 зависела от уровня экспрессии 5T4 на клетках CT26, о чем свидетельствует немного более низкая секреция максимального высвобождения ИФН-γ в совместной культуре с клетками CT26-h5T4низкий по сравнению с совместными культурами с клетками CT26-h5T4высокий (максимальная активность CT26-h5T4низкий составила 82% от CT26-h5T4высокий). В отсутствие 5T4-индуцированного сшивания с использованием клеток CT26 дикого типа активность T-клеток отсутствует или является очень низкой (7% от максимальной активности CT26-h5T4высокий). Максимальное высвобождение ИФН-γ представлено на ФИГ. 21 в виде абсолютных значений (нг/мл) (ФИГ. 21A) или нормированных значений относительно CT26-h5T4высокий (ФИГ. 21B). Показаны средние значения и значения SEM для 12 доноров из 5 объединенных экспериментов.
Таблица 24. Связывание (EC50) ALG.APV-210 в присутствии человеческих 5T4+ клеток CT26
Пример 14. Связывание биспецифичного антитела к 5T4-CD137 с CD137 на первичных стимулированных CD3 CD8 T-клетках человека и яванского макака.
[00324] Было проведено исследование для сравнения относительного связывания ALG.APV-210 с CD137 человека и яванского макака, экспрессируемым на активированных первичных CD8 Т-клетках (гейтированных из стимулированных CD3 МНПК).
Материалы и методы
[00325] Для детекции связывания ALG.APV-210 с первичными клетками человека и яванского макака в FACS ALG.APV-210 сначала биотинилировали. Качество биотинилированного белка оценивали с помощью ВЭЖХ, и влияние на связывание с его мишенями оценивали с помощью ELISA. ANC017, антитело в формате ADAPTIR с тем же каркасом, что и ALG.APV-210 (CDR зародышевой линии), использовали в качестве отрицательного контроля и также биотинилировали.
[00326] Выделение и стимуляция МНПК человека: МНПК человека выделяли центрифугированием в градиенте плотности с использованием Ficoll-Paque (GE Healthcare № 17-1440-02) из концентратов лейкоцитов, полученных от 3 здоровых людей-доноров (Clinical Immunology and Transfusion Medicine,  Лунд, Швеция). 96-луночные планшеты, не обработанные тканевыми культурами (Nunc № 268200), предварительно покрывали в течение ночи при 4 ºC 10 мкг/мл αCD3, клоном OKT3 (Affymetrix eBioscience № 16-0037-85). На следующий день планшеты промывали PBS и добавляли МНПК человека, разведенные в R10 (RPMI, Gibco № 61870010, содержащая 10% инактивированной нагреванием FBS, Hyclone № SV30160, серия RB35944, и 10 мМ Hepes, Gibco № 15630056), в концентрации 0,2 х 106 клеток/лунку, и инкубировали при 37°С в лунках со стимуляцией CD3 или без нее в течение 48 часов.
Лунд, Швеция). 96-луночные планшеты, не обработанные тканевыми культурами (Nunc № 268200), предварительно покрывали в течение ночи при 4 ºC 10 мкг/мл αCD3, клоном OKT3 (Affymetrix eBioscience № 16-0037-85). На следующий день планшеты промывали PBS и добавляли МНПК человека, разведенные в R10 (RPMI, Gibco № 61870010, содержащая 10% инактивированной нагреванием FBS, Hyclone № SV30160, серия RB35944, и 10 мМ Hepes, Gibco № 15630056), в концентрации 0,2 х 106 клеток/лунку, и инкубировали при 37°С в лунках со стимуляцией CD3 или без нее в течение 48 часов.
[00327] Выделение и стимуляция МНПК яванского макака: МНПК яванского макака выделяли из 20 мл крови яванского макака от 3 разных доноров, полученной от Silabe, Франция, путем центрифугирования в градиенте плотности с использованием Lympholyte-mammal (Cedarlane labs) в соответствии с инструкциями производителя. 96-луночные планшеты, не обработанные тканевыми культурами, предварительно покрывали в течение ночи при 4°С 3 мкг/мл антитела к обезьяньему CD3, клоном FN-18 (Invitrogen № APS0301). Затем МНПК яванского макака стимулировали таким же образом, как описано выше для МНПК человека.
[00328] Окрашивание для FACS и анализ CD8 Т-клеток: после инкубации в течение 48 ч с CD3 или без него МНПК человека и яванского макака собирали, объединяли, пересчитывали и разводили буфером FACS (PBS + 0,5% BSA, 0,05% NaN3) до концентрации 5×106 клеток/мл. 50 мкл (0,25 x 106 клеток) раствора клеток добавляли в 96-луночный планшет для окрашивания FACS (Falcon № 351190). После 10 мин Fc-блокирования при комнатной температуре с использованием Beriglobin (hIgG, 200 мкг/мл) клетки промывали в буфере FACS и затем инкубировали в течение 1 часа при 4°C со 100 мкл биотинилированного ALG.APV-210 или отрицательного контроля. Добавляли последовательные разведения антител, начиная либо с титра 5 мкг/мл с 12-ступенчатым разведением в 3 раза до 0,0003 мкг/мл (эксп. 1), либо с титра 1 мкг/мл с 12-ступенчатым разведением в 2 раза до 0,0005 мкг/мл (эксп. 2). После двукратной промывки буфером FACS клетки инкубировали в течение 30 мин при 4°С с 50 мкл вторичного антитела (стрептавидин-APC, BD № 554067) и 50 мкл флуоресцентно-конъюгированных антител против различных маркеров на поверхности Т-клеток (CD4-FITC № 550628, CD3-PECy7 № 557749 и CD8-APC-H7 № 5601797, BD). После промывки в PBS клетки окрашивали в течение 15 мин при 4°C с помощью 50 мкл фиксируемого красителя на жизнеспособность клеток BV510 (для того, чтобы отделить нежизнеспособные клетки), снова промывали и затем повторно суспендировали в 130 мкл реагента для фиксации клеток (BD CellFIX 10x BD, № 340181, разведенный в MQ H2O). Клетки анализировали на связывание ALG.APV-210 с использованием проточной цитометрии путем гейтинга отдельных жизнеспособных клеток, экспрессирующих CD3 и CD8. Значения MFI (за вычетом FMO) из 2 независимых экспериментов, а также нормированные объединенные данные из 2 экспериментов и значения EC50 определяли и наносили на график с использованием GraphPad Prism 7, нелинейная регрессионная зависимость log (агонист) от нормированного ответа - переменный угловой коэффициент, n = 6 доноров/группа.
Результаты
[00329] В тестах ELISA связывание биотинилированного ALG.APV-210 с CD137 было схожим со связыванием небиотинилированного ALG.APV-210 (0,6 нМ против 0,7 нМ). Связывание (EC50) биотинилированного ALG.APV-210 с его иммуномодулирующей мишенью CD137 при экспрессии на активированных первичных CD8 T-клетках человека и яванского макака из стимулированных CD3 МНПК оценивали и сравнивали между двумя видами, а также с нестимулированными клетками. Как показано с использованием MFI всех CD3+ CD8+ клеток на ФИГ. 22A, 22B и 22C и в таблице 25, ALG.APV-210 связывается с CD8 T-клетками как человека, так и яванского макака дозозависимым образом, но не с нестимулированными клетками. Связывание с отрицательным контролем, ANC107, было низким/необнаружимым для стимулированных CD3 или нестимулированных МНПК человека или яванского макака. Максимальное количество (MFI) ALG.APV-210, связанного со стимулированными CD3 МНПК яванского макака, было выше по сравнению с МНПК человека, вероятнее всего из-за более высокой доли стимулированных CD3 МНПК яванского макака, экспрессирующей CD137, или из-за в целом более высокой экспрессии CD137 на CD8 Т-клетках яванского макака. Неясно, были ли клетки яванского макака более чувствительными к стимуляции посредством CD3 по сравнению с клетками человека, и, следовательно, экспрессировали ли они CD137 в более высокой степени, так как клетки были стимулированы разными клонами антитела к CD3 из-за отсутствия перекрестной реактивности реагентов к CD3 между видами.
Таблица 25. Связывание ALG.APV-210 (EC50) с CD8 T-клетками человека и яванского макака
[00330] Связывание ALG.APV-210 также оценивали по проценту CD8 Т-клеток, связанных ALG.APV-210. Наблюдали более высокий процент CD8 T-клеток яванского макака, связанных ALG.APV-210, по сравнению с CD8 T-клетками человека (71-96% для клеток яванского макака, 65-76% для клеток человека, см. ФИГ. 23A, 23B и 23C). Значения связывания EC50 также рассчитывали по MFI CD137-положительных клеток, и в результате было получено значение EC50, равное 0,17 нМ для связывания с CD137 человека, и 0,20 нМ для CD137 яванского макака.
[00331] В совокупности результаты исследования показали, что ALG.APV-210 связывается со схожими значениями ЕС50: 0,2 нМ со стимулированными CD3 первичными CD8 Т-клетками человека и яванского макака, но не с нестимулированными клетками.
Пример 15. Активность биспецифичного антитела к 5T4-CD137 in vitro в анализе высвобождения ИФН-γ с использованием CD8 Т-клеток человека или яванского макака в присутствии 5T4
[00332] Было проведено исследование для сравнения функциональной активности и определения EC50 ALG.APV-210 в анализе агонистической активности с использованием CD8 Т-клеток человека или яванского макака в присутствии иммобилизованного на планшете 5Т4.
Материалы и методы
[00333] CD8 Т-клетки субоптимально стимулировали с помощью иммобилизованных на планшете антител к CD3 и активировали путем сшивания CD137 посредством связывания ALG.APV-210 с иммобилизованным на планшете 5T4-Fc. Активацию CD8 Т-клеток измеряли путем определения высвобождения ИФН-γ в супернатант клеточной культуры.
[00334] Выделение и схема анализа с CD8 T-клетками человека: МНПК человека выделяли центрифугированием в градиенте плотности с использованием Ficoll-Paque (GE Healthcare № 17-1440-02) из концентратов лейкоцитов, полученных от здоровых доноров (Clinical Immunology and Transfusion Medicine,  Лунд, Швеция). CD8+ клетки обогащали посредством отрицательного отбора с использованием набора для выделения CD8+ Т-клеток (Miltenyi № 130-096-495). 96-луночные планшеты для анализа, не обработанные тканевыми культурами, покрывали в течение ночи при 4 ºC 3 мкг/мл αCD3, антитела к CD3 (клон: OKT-3, Affymetrix eBioscience № 16-0037-85). На следующий день планшеты промывали в PBS и покрывали 5 мкг/мл 5T4-Fc в течение 2 часов при 37 °C. После иммобилизации 5T4-Fc планшеты для анализа промывали в PBS и блокировали в течение как минимум 30 минут с помощью R10 (RPMI, Gibco № 61870010, содержащая 10% инактивированной нагреванием FBS, Hyclone № SV30160, партия RB35944, и 10 мМ Hepes, Gibco № 15630056). Готовили последовательные разведения ALG.APV-210 в R10 и добавляли 50 мкл в трех экземплярах на планшет для анализа за 30 минут до добавления 50 мкл эффекторных клеток (обогащенные CD8 Т-клетки, 1,4 × 106 клеток/мл или 0,07 × 105 клеток/лунка). Планшеты для анализа инкубировали в течение 72 ч при 37°С и собирали культуральный супернатант. Уровни ИФН-γ в супернатантах измеряли методом ELISA (BD OptiEIA № 555142). Эксперимент проводили 4 раза, включая в общей сложности 8 доноров.
Лунд, Швеция). CD8+ клетки обогащали посредством отрицательного отбора с использованием набора для выделения CD8+ Т-клеток (Miltenyi № 130-096-495). 96-луночные планшеты для анализа, не обработанные тканевыми культурами, покрывали в течение ночи при 4 ºC 3 мкг/мл αCD3, антитела к CD3 (клон: OKT-3, Affymetrix eBioscience № 16-0037-85). На следующий день планшеты промывали в PBS и покрывали 5 мкг/мл 5T4-Fc в течение 2 часов при 37 °C. После иммобилизации 5T4-Fc планшеты для анализа промывали в PBS и блокировали в течение как минимум 30 минут с помощью R10 (RPMI, Gibco № 61870010, содержащая 10% инактивированной нагреванием FBS, Hyclone № SV30160, партия RB35944, и 10 мМ Hepes, Gibco № 15630056). Готовили последовательные разведения ALG.APV-210 в R10 и добавляли 50 мкл в трех экземплярах на планшет для анализа за 30 минут до добавления 50 мкл эффекторных клеток (обогащенные CD8 Т-клетки, 1,4 × 106 клеток/мл или 0,07 × 105 клеток/лунка). Планшеты для анализа инкубировали в течение 72 ч при 37°С и собирали культуральный супернатант. Уровни ИФН-γ в супернатантах измеряли методом ELISA (BD OptiEIA № 555142). Эксперимент проводили 4 раза, включая в общей сложности 8 доноров.
[00335] Выделение и схема анализа с CD8 T-клетками яванского макака: использовали цельную кровь яванского макака (Macaca flavicularis, взрослые самцы, 3-15 лет), полученную из Silabe, Франция. Эритроциты удаляли лизисом эритроцитов (BD Pharma-lyse, BD Biosciences, № 555899) в соответствии с протоколом производителя. CD8+ клетки обогащали посредством положительного отбора с использованием набора CD8 MicroBead (Miltenyi Biotec, № 130-091-112). Планшеты покрывали в течение ночи при 4°C 1 мкг/мл антитела к CD3 яванского макака, клоном FN-18 (Invitrogen, № APS0301), промывали и покрывали 5 мкг/мл 5T4-Fc в течение 2 часов при 37°C. После покрытия 5T4-Fc планшеты промывали и блокировали в течение как минимум 30 минут с помощью R10. ALG.APV-210 разводили в R10 и добавляли 50 мкл в трех экземплярах в планшеты за 30 минут до добавления 50 мкл CD8 Т-клеток (0,07 × 106 клеток/лунку). Планшеты для анализа инкубировали в течение 72 ч при 37°С и собирали культуральный супернатант. Уровни ИФН-γ в супернатантах измеряли с помощью ELISA (набор для визуализации ИФН-γ обезьян в ELISA, MABTECH, № 3421M-1H-20). Эксперимент проводили 3 раза, включая в общей сложности 8 доноров. Полученные уровни ИФН-γ от каждого донора, представляющего собой человека и яванского макака, были нормированы, и были рассчитаны средние значения. Среднее значение нормированных уровней ИФН-γ от всех 8 доноров, представляющих собой людей или яванских макаков, были объединены, и EC50 была определена с помощью нелинейной регрессии (зависимость log агониста от нормированного ответа, переменный угловой коэффициент) с использованием GraphPad 7.
Результаты
[00336] Как показано на ФИГ. 24 и в таблице 26, в анализе с CD8 Т-клетками человека биспецифичное антитело к CD137-5T4 ALG.APV-210 имело высокую функциональную активность с ЕС50, равной 0,2 нМ. Нормированные уровни ИФН-γ из 4 экспериментов и в общей сложности 8 доноров были объединены, и EC50 определена путем нелинейной регрессии с использованием GraphPad 7. Среднее значение и SEM представлены на ФИГ. 24. Абсолютные средние значения ИФН-γ от двух доноров из одного иллюстративного эксперимента представлены на ФИГ. 25А и 25В. Максимальные значения реагентов к CD3, а также фоновые уровни ИФН-γ варьируются от донора к донору в рамках одного эксперимента. Из-за наблюдаемых различий между донорами данные нормировали до анализа объединенного набора данных. Активацию CD8 Т-клеток посредством ALG.APV-210 оценивали как в присутствии, так и в отсутствие 5T4-Fc, чтобы проверить зависимость от сшивания 5T4. Результаты показали, что биспецифичное антитело к CD137-5T4 ALG.APV-210 индуцирует высокую активацию Т-клеток, измеренную по высвобождению ИФН-γ, при сшивании 5T4; в отсутствие 5Т4 активность Т-клеток отсутствует (ФИГ. 25А и 25В).
[00337] Также была оценена функциональная активность биспецифичного антитела к CD137-5T4 ALG.APV-210 при использовании CD8 T-клеток яванского макака. На основании объединенных данных из 3 экспериментов и в общей сложности 8 доноров было определено, что EC50 ALG.APV-210 в анализе с CD8 Т-клетками яванского макака составило 0,4 нМ (ФИГ. 26 и таблица 26). Абсолютные средние значения от 3 доноров из одного иллюстративного эксперимента также представлены на ФИГ. 27A, 27B и 27C. Аналогично эксперименту с клетками человека абсолютные значения (включая максимальные значения и фоновые уровни ИФН-γ) также варьируются между отдельными яванскими макаками-донорами в рамках одного эксперимента. Поэтому данные нормировали до проведения анализа EC50 объединенного набора данных. Активацию CD8 Т-клеток посредством ALG.APV-210 оценивали как в присутствии, так и в отсутствие 5T4-Fc, чтобы проверить зависимость от вызванного 5T4 сшивания CD137. Как показано на ФИГ. 27A-27C, агонистическое действие ALG.APV-210 является 5T4-зависимым в анализе с CD8 T-клетками яванского макака, аналогично результату, наблюдаемому в эксперименте с клетками человека. На каждой из ФИГ. 27А, 27В и 27С показаны абсолютные средние значения и значения SEM для ИФН-g для 3 отдельных доноров из одного иллюстративного эксперимента. В качестве отрицательного контроля были включены лунки без 5T4.
Таблица 26. Связывание ALG.APV-210 в анализе с CD8 T-клетками человека и яванского макака
[00338] Исследование показало, что ЕС50 биспецифичного антитела к CD137-5T4 ALG.APV-210 в функциональном анализе активации CD8 Т-клеток было сопоставимым для человека (0,2 нМ) и яванского макака (0,4 нМ). Активация Т-клеток была опосредована сшиванием CD137, зависимым от 5Т4, в анализах с CD8 Т-клетками как человека, так и яванского макака.
Пример 16. Активность биспецифичного антитела к 5T4-CD137 ALG.APV-210 in vitro в анализах секреции Т-клетками ИФН-γ и пролиферации Т-клеток
[00339] Агонистическую функциональную активность биспецифичной молекулы к 5T4-CD137 ALG.APV-210 в отношении индукции мишень-зависимой активации CD137 оценивали в анализе секреции Т-клетками ИФН-γ и анализе пролиферации Т-клеток с использованием неотделенных мононуклеарных клеток периферической крови (МНПК). Для тестирования функциональной активности биспецифичных конструкций к CD137 первичные МНПК стимулировали антителами к CD3 для повышения экспрессии CD137, который не экспрессируется, или экспрессируется на низких уровнях, на покоящихся Т-клетках. Совместная стимуляция первичных Т-клеток посредством TCR и CD137 усиливает секрецию Т-клетками цитокина интерферона-гамма (ИФН-γ).
Методы
Анализ секреции ИФН-γ:
[00340] Мононуклеарные клетки периферической крови (МНПК) выделяли из крови здоровых людей с использованием стандартных градиентов фиколла. Выделенные клетки промывали в солевом буфере для удаления тромбоцитов. МНПК культивировали с клетками CHO-K1, трансфицированными 5T4 человека или пустым вектором, в количестве приблизительно 120 000 МНПК на 30 000 клеток CHO-K1 в 96-луночных планшетах. Во все лунки добавляли антитело к CD3 OKT3 (eBioscience) в концентрации 1 нг/мл. Клетки культивировали в общем объеме 200 мкл среды RPMI 1640 с добавками 10% эмбриональной бычьей сыворотки, пирувата натрия, антибиотиков и заменимых аминокислот. Планшеты инкубировали при 37°C, 5% CO2 в инкубаторах с увлажненной средой в течение 72 часов. 100 мкл среды собирали после 72 часов стимуляции культур МНПК, примированных субоптимальной концентрацией антител к CD3 для индукции активации CD137. В супернатанте измеряли уровни ИФН-γ с использованием наборов Milliplex® (Millipore) в соответствии с инструкциями производителя. Данные собирали в системе Bio-Plex Reader 200 (Bio-Rad). Нелинейный регрессионный анализ для определения значений EC50 проводили с помощью программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
Анализ пролиферации Т-клеток:
[00341] МНПК выделяли из крови здоровых людей с использованием стандартных градиентов фиколла. После выделения клетки промывали в солевом буфере для удаления тромбоцитов. PBMC метили фиолетовым CellTraceTM (Thermofisher), промывали и культивировали с облученными клетками CHO-K1, трансфицированными 5T4 человека, в количестве приблизительно 120 000 МНПК до 30 000 клеток CHO-K1 в 96-луночных планшетах. Клетки CHO-K1 облучали (рентгеновский облучатель Cell Rad, Faxitron Bioptics, LLC) и промывали в среде перед высеванием с МНПК. Во все лунки добавляли антитело к CD3 OKT3 (eBioscience) в концентрации 5 нг/мл. Клетки культивировали в общем объеме 200 мкл среды RPMI 1640 (GIBCO) с добавками 10% человеческой сыворотки (SIGMA), пирувата натрия, антибиотиков и заменимых аминокислот. Планшеты инкубировали при 37°C, 5% CO2 в инкубаторах с увлажненной средой в течение 96 часов.
[00342] Супернатанты удаляли и клетки метили в тех же планшетах для анализа антителами к CD4, CD8 и CD5 в буфере PBS с 2% BSA и 2 мМ ЭДТА в течение 30 минут на льду. Добавляли 7AAD (SIGMA), чтобы исключить из анализа мертвые клетки. После промывки клетки повторно суспендировали в концентрации 120 мкл/лунку, и из каждой лунки отбирали по 70 мкл и анализировали в проточном цитометре (LSR-IITM, BD Biosciences). Анализ данных проводили с использованием программного обеспечения Flowjo двумя способами: вычисляя % событий CD8+ живых Т-клеток (CD4- CD8+ CD5+, 7AAD-), претерпевших хотя бы одно деление клеток, или путем подсчета количества событий CD8+ живых Т-клеток (CD4- CD8+ CD5+, 7AAD-). В обоих случаях анализ проводили после отбора лимфоцитов по сигналам прямого (FSC) и бокового (SSC) светорассеяния. Проводили два параллельных испытания каждого условия и строили графики средних значений из каждого двойного набора были построены с использованием программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
Результаты и выводы
[00343] Результаты экспериментов по секреции ИФН-γ показаны на ФИГ. 32. Каждая точка на графиках представляет собой среднее значение для параллельных лунок. Добавление одного лишь антитела к CD3 без биспецифичных антител индуцировало очень низкие уровни ИФН-γ. Добавление биспецифичной молекулы ALG.APV-210 повышало секрецию ИФН-γ дозозависимым образом. Исходные уровни ИФН-γ, индуцированного антителом к CD3, а также общее количество секретированного ИФН-γ, индуцированное ALG.APV-210, варьировалось в зависимости от донора МНПК (ФИГ. 32A по сравнению с ФИГ. 32B). Усиленная секреция ИФН-γ наблюдалась в присутствии клеток CHO-K1, экспрессирующих 5T4, но не в присутствии клеток CHO-K1, трансфицированных пустым векторным контролем. Следовательно, биспецифичная молекула ALG.APV-210 требует включения 5T4 для стимуляции функции CD137, измеряемой по секреции ИФН-γ.
[00344] Результаты экспериментов по пролиферации Т-клеток показаны на ФИГ. 33. Каждая точка на графиках представляет собой среднее значение для параллельных лунок. Поликлональную пролиферацию CD8+ T-клеток оценивали после 96 часов стимуляции примированием при субоптимальной концентрации антитела к CD3. Добавление одного лишь антитела к CD3 без биспецифичного антитела индуцировало низкие уровни пролиферации CD8+ Т-клеток. Добавление ALG.APV-210 повышало пролиферацию CD8+ T-клеток дозозависимым образом (ФИГ. 33A и ФИГ. 33B). Как процент клеток, претерпевающих деление (верхние секции), так и общее количество клеток (нижние секции), собранных на лунку, соответствовали устойчивому делению клеток и накоплению поделившихся CD8+ Т-клеток в лунках. Результаты согласовались среди нескольких доноров.
Пример 17. Функциональная репортерная активность ALG.APV-210 в присутствии различных линий опухолевых клеток человека, экспрессирующих белок 5Т4 в диапазоне плотностей
[00345] Проводили эксперименты для определения того, обладает ли конструкция ALG.APV-210 связывающей и функциональной репортерной активностью, на нескольких линиях опухолевых клеток человека, экспрессирующих 5T4 в диапазоне плотностей на своей поверхности. Максимальная связывающая способность ALG.APV-210 будет варьироваться в зависимости от уровня экспрессии клетками опухолевого антигена 5T4. Поэтому связывание ALG.APV-210 тестировали на 3 линиях опухолевых клеток (MDA-MB-231, H1975 и TF-1), естественным образом экспрессирующих 5T4 в диапазоне плотностей, а также клеток CHO-K1, стабильно трансфицированных 5T4 (CHO/5T4).
Материалы и методы
[00346] Анализ связывания 5T4: 3-кратный титр конструкции ALG.APV-210 (в диапазоне от 100 нМ до 0,14 нМ) инкубировали с линиями клеток, экспрессирующими 5T4 человека, в течение 40 минут в буфере FACS при 4°C, промывали 3 раза и детектировали с помощью PE-меченого вторичного козьего антитела к человеческому Fc (Jackson ImmunoResearch). Связывание ALG.APV-210 детектировали методом проточной цитометрии. Все образцы тестировали в двух экземплярах. Нелинейный регрессионный анализ для определения значений EC50 проводили с помощью программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
[00347] Плотность белка 5T4: поверхностную плотность белка 5T4, присутствующего на каждой линии клеток, исследовали с использованием гранул Quantibrite™ (BD Pharmingen). Гранулы Quantibrite™ были приготовлены с использованием точных настроек цитометра, использованных для получения клеток для кривых связывания с ALG.APV-210. Гейтировали 4 популяции гранул и для каждого пика определяли среднюю геометрическую величину флуоресценции. Строили зависимость линейной регрессии Log10 молекул PE на гранулу от Log10 флуоресценции с использованием уравнения y = mx + c, где y равно Log10 флуоресценции, а x равно Log10 молекул PE на гранулу. Исходя из этого можно рассчитать количество молекул PE (5T4) на клетку. Для этого конкретного анализа уравнение представляло собой y = 1,036x - 1,478. Все средние значения флуоресценции рассчитывали, используя среднее геометрическое для образцов, как рекомендовано протоколом производителя.
[00348] Репортерный анализ CD137: трансфектанты Jurkat/CD137, несущие репортерный ген люциферазы под контролем промотора NF-κB (Promega), культивировали с опухолевыми клетками H1975, TF-1, MDA-MB-231 или CHO/5T4 в соотношении 30 000 репортерных клеток на 60 000 клеток-мишеней в 96-луночных планшетах. Добавляли пятикратные концентрации молекулы ALG.APV-210 с конечной концентрацией в диапазоне от 10 нМ до 0,0006 нМ. Клетки культивировали в общем объеме 100 мкл среды RPMI 1640 с добавками 1% эмбриональной бычьей сыворотки, пирувата натрия, антибиотиков и заменимых аминокислот. Планшеты инкубировали при 37°C, 5% CO2 в инкубаторах с увлажненной средой в течение 5 часов. 100 мкл буфера Bio-Glow (Promega) добавляли в каждую лунку, перемешивали и инкубировали в течение 10 минут перед измерением люминесценции. Люминесценцию измеряли на счетчике для микропланшетов MicroBeta2 2450 (Perkin Elmer). Все точки представляют собой образцы в двух экземплярах. Нелинейный регрессионный анализ для определения значений EC50 проводили с помощью программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®. Каждая точка на кривой представляет собой среднее значение для параллельных лунок. На оси y показаны значения в относительных единицах флуоресценции (RLU).
Результаты и выводы
[00349] Каждая из протестированных линий опухолевых клеток отличалась по количеству белка 5Т4, экспрессируемого на клеточной поверхности, как было определено с использованием гранул Quantibrite™. Таким образом может быть исследована способность ALG.APV-210 связываться и быть функционально активной в диапазоне экспрессии 5T4.
[00350] ALG.APV-210 связывалась с рядом линий опухолевых клеток, экспрессирующих 5T4 на своей поверхности. Как показано на ФИГ. 34, CHO/5T4 имеют в 10 раз более высокую экспрессию 5T4, чем линии опухолевых клеток, эндогенно экспрессирующие 5T4. Из трех нетрансфицированных линий опухолевых клеток H1975 обладали самой высокой экспрессией 5T4, тогда как клетки TF-1 имели самую низкую плотность белка 5T4. Клетки MDA-MB-231 продемонстрировали промежуточную экспрессию 5T4. Плотности белка 5T4 на поверхности клеток рассчитывали по кривым связывания с получением диапазона молекул/клетка от 17 000 до более 250 000. В таблице 27 представлены плотности белка 5T4 на клеточной поверхности и значения связывания EC50, полученные из кривых связывания. Плотность экспрессии 5T4 на линиях опухолевых клеток рассчитывали с использованием стандарта флуоресценции.
Таблица 27. Сводные данные в отношении плотностей 5T4 и EC50 ALG.APV-210
[00351] На ФИГ. 35 показаны результаты функционального репортерного анализа CD137. Как показано, ALG.APV-210 была в достаточной степени способна индуцировать передачу сигналов NF-κB при сшивании CD137 посредством связывания с линиями опухолевых клеток CHO/hu5T4, H1975, MDA.MB.231 TF-1, несмотря на диапазон экспрессии 5T4 на клеточной поверхности от 17 000 до более 250 000. Плотность белка 5T4 на клеточной поверхности оказывала влияние на максимальную функциональную активность (максимальные RLU), причем клетки CHO/hu5T4 демонстрировали самые высокие значения RLU. Однако три линии опухолевых клеток индуцировали устойчивую репортерную функциональную активность. Результаты свидетельствуют о том, что ALG.APV-210 играет роль агониста в присутствии 5T4-положительных клеток, экспрессирующих 5T4 в диапазоне плотностей. В таблице 28 представлены значения EC50 и максимальные RLU, индуцированные ALG.APV-210 в репортерном анализе CD137 в присутствии различных линий опухолевых клеток, экспрессирующих 5T4 человека.
Таблица 28. Сводные данные в отношении EC50 и максимальных RLU для ALG.APV-210
Пример 18. Активность биспецифичного антитела к 5T4-CD137 in vitro в анализе высвобождения ИФН-γ с использованием CD8 Т-клеток человека и опухолевых клеток HCT116 человека
[00352] Функциональную активность биспецифичного антитела к 5T4-CD137 ALG.APV-210 оценивали в анализе с CD8 Т-клетками, в котором CD8+ Т-клетки, стимулированные антителами к CD3, нанесенными на гранулы, совместно культивировали в планшетах с опухолевыми клетками HCT116 человека, экспрессирующими эндогенные уровни 5Т4.
Материалы и методы
[00353] Обработка митомицином С клеток НСТ116: опухолевые клетки НСТ116, линию клеток карциномы толстой кишки человека (ATCC® CCL-247™), экспрессирующие эндогенные уровни человеческого 5Т4 (плотность рецептора = 6,2 × 104/клетка, таблица 21), предварительно обрабатывали митомицином С (0,5 мг/мл, Sigma-Aldrich) в концентрации 50 мкг/мл, чтобы препятствовать избыточному росту опухолевых клеток. После 45 мин инкубации с митомицином С при 37°С клетки трижды промывали в среде R10 (RPMI, Gibco № 61870010, содержащая 10% инактивированной нагреванием FBS, Hyclone № SV30160, партия RB35944, и 10 мМ Hepes, Gibco № 15630056), а затем повторно суспендировали в концентрации 10 × 106 клеток/мл в новой среде R10. 50 мкл (5 × 105 клеток/лунку) клеток HCT116, обработанных митомицином С, добавляли в планшеты для анализа, обработанные тканевой культурой (Eppendorf № 0030 730.119), и инкубировали при 37°С в течение ночи.
[00354] Схема анализа с CD8 T-клетками: на следующий день МНПК человека выделяли центрифугированием в градиенте плотности с использованием Ficoll-Paque (GE Healthcare № 17-1440-02) из концентратов лейкоцитов, полученных от здоровых доноров (Clinical Immunology and Transfusion Medicine,  Лунд, Швеция). CD8+ клетки обогащали посредством отрицательного отбора с использованием набора для выделения CD8+ Т-клеток (Miltenyi 130-096-495). Готовили последовательные разведения биспецифичного антитела ALG.APV-210 или изотипического контроля в формате Adaptir, ANC017 (последовательности вариабельных областей тяжелой и легкой цепи для ANC017 представлены ниже в таблицах 29A и 29B), в среде R10, и в каждую лунку планшетов для анализа добавляли 50 мкл и инкубировали при 37°С в течение 30 минут перед добавлением 50 мкл гранул с αCD3 (4 × 108/мл клона антител к CD3: OKT-3, Affymetrix eBioscience № 16-0037-85, нанесенный на гранулы, разведенный до 2 × 106/мл) при соотношении CD8+ T-клеток к гранулам 1:1, с последующим добавлением 50 мкл эффекторных клеток (обогащенные клетки CD8+, 2 × 106/мл или 1 × 105/лунку). Планшеты для анализа инкубировали в течение 72 ч при 37°С и собирали культуральный супернатант. Уровни ИФН-γ в супернатантах измеряли с помощью ELISA (BD OptiEIA № 555142), и они представлены на ФИГ. 36 в виде нормированных данных (средние значения и СКО). Значения EC50 определяли с помощью нелинейной регрессионной зависимости log (агонист) от нормированного ответа (переменный угловой коэффициент) с использованием GraphPad Prism 7. Величину TOP (максимальная функциональная активность) определяли путем расчета среднего двух самых высоких значений выработки ИФН-γ в культуральном супернатанте. Эксперимент проводили 4 раза, включая в общей сложности 12 доноров.
Лунд, Швеция). CD8+ клетки обогащали посредством отрицательного отбора с использованием набора для выделения CD8+ Т-клеток (Miltenyi 130-096-495). Готовили последовательные разведения биспецифичного антитела ALG.APV-210 или изотипического контроля в формате Adaptir, ANC017 (последовательности вариабельных областей тяжелой и легкой цепи для ANC017 представлены ниже в таблицах 29A и 29B), в среде R10, и в каждую лунку планшетов для анализа добавляли 50 мкл и инкубировали при 37°С в течение 30 минут перед добавлением 50 мкл гранул с αCD3 (4 × 108/мл клона антител к CD3: OKT-3, Affymetrix eBioscience № 16-0037-85, нанесенный на гранулы, разведенный до 2 × 106/мл) при соотношении CD8+ T-клеток к гранулам 1:1, с последующим добавлением 50 мкл эффекторных клеток (обогащенные клетки CD8+, 2 × 106/мл или 1 × 105/лунку). Планшеты для анализа инкубировали в течение 72 ч при 37°С и собирали культуральный супернатант. Уровни ИФН-γ в супернатантах измеряли с помощью ELISA (BD OptiEIA № 555142), и они представлены на ФИГ. 36 в виде нормированных данных (средние значения и СКО). Значения EC50 определяли с помощью нелинейной регрессионной зависимости log (агонист) от нормированного ответа (переменный угловой коэффициент) с использованием GraphPad Prism 7. Величину TOP (максимальная функциональная активность) определяли путем расчета среднего двух самых высоких значений выработки ИФН-γ в культуральном супернатанте. Эксперимент проводили 4 раза, включая в общей сложности 12 доноров.
Таблица 29А. Последовательности вариабельной области ACN017
Таблица 29B. Полноразмерная последовательность ACN017
Результаты и выводы
[00355] Была определена функциональная активность биспецифичного антитела к CD137-5T4 ALG.APV-210 с использованием CD8+ T-клеток человека (эффекторных клеток), культивируемых в планшетах с клетками HCT116. Как показано на ФИГ. 36, биспецифичное антитело к CD137-5T4 ALG.APV-210 индуцировало высокую активацию Т-клеток, измеренную по высвобождению ИФН-γ, дозозависимым образом в присутствии клеток HCT116, экспрессирующих эндогенные уровни 5T4. Определенные значения EC50 см. в таблице 30, где приведены средние значения и 95% CI для 12 доноров из 4 объединенных экспериментов. Эндогенные уровни 5T4, экспрессируемые клетками HCT116, достаточны для индукции высокой агонистической функциональной активности ALG.APV-210, как показано на ФИГ. 37, где уровни ИФН-γ для 6 отдельных доноров после обработки ALG.APV-210 сравниваются с изотипическим контролем (ANC017). Эффективность (максимальный ИФН-γ-ответ) также представлена на ФИГ. 38 (кратное изменение по сравнению с изотипическим контролем, среднее значение и СКО для 12 доноров).
Таблица 30. EC50 ALG.APV-210 в анализе с CD8+ T-клетками в присутствии клеток HCT116, экспрессирующих 5T4 человека
Пример 19. Противоопухолевая эффективность in vivo биспецифичного антитела в ксенотрансплантатной модели 5Т4-позитивной опухоли HCT-116 у мышей SCID beige
[00356] Было проведено исследование для оценки противоопухолевой эффективности ALG.APV-210 in vivo.
Материалы и методы
[00357] В эксперименте использовали самок мышей SCID beige (6-8 недель) из Taconic, Дания. Все процедуры на животных проводили в соответствии с законодательством Швеции о правах и защите животных и с одобрения Этического комитета по экспериментам на животных в Лунде/Мальмё (номер заявки на этическое одобрение: M151-13). МНПК выделяли из концентратов лейкоцитов, полученных от 4 здоровых доноров (Clinical Immunology and Transfusion Medicine,  Лунд, Швеция) центрифугированием в градиенте плотности с использованием Ficoll-Paque (GE Healthcare № 17-1440-02) в соответствии с инструкциями производителя. Линия клеток HCT-116, линия клеток карциномы толстой кишки человека, положительных на опухолеассоциированный антиген 5T4, была приобретена у ATCC (ATCC® CCL-247™, № партии 62765668) и культивировалась в соответствии с их рекомендациями.
Лунд, Швеция) центрифугированием в градиенте плотности с использованием Ficoll-Paque (GE Healthcare № 17-1440-02) в соответствии с инструкциями производителя. Линия клеток HCT-116, линия клеток карциномы толстой кишки человека, положительных на опухолеассоциированный антиген 5T4, была приобретена у ATCC (ATCC® CCL-247™, № партии 62765668) и культивировалась в соответствии с их рекомендациями.
[00358] В 0 день опухолевые клетки HCT-116 в логарифмической фазе роста подкожно инокулировали в правый задний бок мышей (3 × 106 в 110 мкл PBS). На следующий день (1 день) внутрибрюшинно инъецировали 7 × 106 МНПК человека от 4 доноров в 100 мкл PBS. Внутрибрюшинное введение антител (100 мкл в PBS), начиная с 6-го дня, либо с носителем (PBS), либо с 10 мкг/мышь ALG.APV-210, либо со 100 мкг/мышь ALG.APV-210, осуществляли дважды в неделю, проведя в общей сложности 5 инъекций (n = 5 мышей/введение/донора). Осуществляли наблюдение за ростом опухоли и дважды в неделю проводили измерения с помощью штангенциркуля по ширине, длине и высоте, из которых рассчитывали объем опухоли (ш/2x д/2xв/2x pi x (4/3)). Конечной точкой эксперимента был объем опухоли ≥2 см3, ранение или ухудшенное состояние здоровья мышей. Рост опухоли статистически анализировали с использованием непараметрического 2-стороннего Т-критерия Манна-Уитни с использованием GraphPad Prism 7 в разные моменты времени (9, 13, 16, 20 и 23 день).
Результаты
[00359] Как показано на ФИГ. 28, ALG.APV-210 в дозе 10 мкг/мышь или 100 мкг/мышь продемонстрировала статистически значимую противоопухолевую эффективность (например, ингибирование объема опухоли) по сравнению с группой, получавшей носитель, с 13 по 23 день (объединенные данные для 4 доноров, данные статистического анализа и р-значения см. в таблице 31).
Таблица 31. Статистический анализ противоопухолевой эффективности после лечения ALG.APV-210 на ксенотрансплантатной модели HCT-116
[00360] Таким образом, исследование показало, что ALG.APV-210 обладает противоопухолевыми свойствами в 5T4-положительной ксенотрансплантатной модели карциномы толстой кишки человека.
Пример 20. Фармакокинетические свойства ALG.APV-209 у мышей BALB/c
[00361] Было проведено исследование для оценки фармакокинетических (ФК) свойств ALG.APV-209 и ALG.APV-210 у мышей. Нормальным самкам мышей BALB/c внутривенно (в/в) в момент времени 0 вводили однократную дозу 200 мкг ALG.APV-209 или ALG.APV-210 и брали кровь у 1-3 мышей в каждый момент времени. Мышей под анестезией обескровливали с помощью пункции сердца и собирали сыворотку через t = 15 минут и через 2, 6, 24, 48, 72, 96, 168, 336 и 504 часа после инъекции. Концентрации ALG.APV-209 и ALG.APV-210 в сыворотках определяли с помощью метода ELISA, разработанного для детекции только интактного белка, с использованием huCD137 ECD-AFH (ALG027) для захвата BD к CD137 и биотинилированного 5T4 ECD-mFc (ALG029) для детекции BD к 5T4. Расчетные параметры ФК распределения на основе некомпартментного анализа (NCA), полученные с использованием программного обеспечения Phoenix 64 (лицензия v6.4 WinNonlin™) перечислены в таблице 32, а расчетные периоды полувыведения вместе со статистикой соответствия показаны на ФИГ. 29. Для NCA использовали предварительно скомпилированную модель для внутривенного введения с разреженной выборкой и равномерным взвешиванием показателей.
[00362] У нормальных самок мышей BALB/c, которым в/в вводили болюсную дозу 200 мкг ALG.APV-209 или ALG.APV-210, кажущийся конечный период полувыведения, определенный с помощью NCA, составлял 142 и 215 часов, соответственно (за исключением образцов, на которые могли оказать влияние ADA). Расчетные параметры для периода полувыведения, определенные с использованием компартментного анализа (2-компартментный с в/в введением дозы), были схожими для ALG.APV-209 и ALG.APV-210. Согласно оценкам NCA клиренс и объем для ALG.APV-209 и ALG.APV-210 составляли: 0,263 и 0,204 мл/ч/кг и 54 и 63 мл/кг, соответственно. Согласно обоим методам анализа ALG.APV-210 имел самый длинный конечный период полувыведения и немного лучшие значения клиренса. Внезапные падения сывороточных концентраций, свидетельствующие об антителах против лекарственного средства (ADA), были обнаружены в поздние моменты времени у мышей, которым вводили ALG.APV-210, однако присутствие ADA не могло быть подтверждено методами ELISA, поскольку лекарственное средство все еще присутствовало в образцах, что также, вероятно, привело к быстрому клиренсу ADA.
Таблица 32. Рассчитанные с помощью NCA параметры ФК распределения ALG.APV-209 и ALG.APV-210
- Rsq: статистика соответствия для фазы конечного выведения
- HL лямбда z: кажущийся конечный период полувыведения
- Tmax: время максимальной наблюдаемой концентрации
- Cmax: максимальная наблюдаемая концентрация, возникающая при Tmax
- Cmax SE: среднеквадратическая ошибка данных при Tmax (время максимальной средней концентрации)
- Cmax/D: максимальная наблюдаемая концентрация, деленная на дозу
- C0: экстраполированная к начальному значению начальная концентрация в момент времени 0
- _obs: на основе наблюдаемых концентраций
- AUClast: площадь под кривой от момента введения дозы до последней измеримой концентрации
- AUCINF: площадь под кривой от момента введения дозы, экстраполированная на бесконечность
- AUCINF/D: AUCINF, деленная на дозу
- V: объем распределения на основе конечной фазы
- CL: сывороточный клиренс
- Vss: расчетный объем распределения в равновесном состоянии
- MRTlast: среднее время пребывания до последней измеримой концентрации
- MRTINF: среднее время пребывания, экстраполированное на бесконечность
Пример 21. Fc-часть ALG.APV-210 не взаимодействует с Fcγ-рецепторами
[00363] ALG.APV-210 содержит мутации, введенные для предотвращения взаимодействия с Fcγ-рецепторами (FcγR), которые могут привести к FcγR-опосредованной активации T-клеток или ADCC, ADCP, CDC и т. д. Поэтому связывание ALG.APV-210 было протестировано на трансфектантах клеток CHO, стабильно экспрессирующих следующие FcγR: FcγRI (CD64), FcγRIIa (CD32a His131), FcγRIIb (CD32b), FcγRIIIa (CD16a Val158 и CD16a Phe158) и FcγRIIIb (CD16b). Нетрансфицированные клетки СНО использовали в качестве отрицательного контроля. Контрольную молекулу человеческого IgG1 использовали в качестве положительного контроля. Различные концентрации ALG.APV-210 или контрольного IgG1 инкубировали с линиями клеток, экспрессирующих FcγR, промывали и метили вторичным реагентом к человеческому IgG. Связывание ALG.APV-210 и контрольного IgG1 определяли методом проточной цитометрии.
[00364] Результаты представлены на ФИГ. 30А и 30В. На ФИГ. 30B показано, что контрольное IgG1 связывалось со всеми протестированными линиями клеток, экспрессирующих FcγR: FcγRI (CD64), FcγRIIa (CD32a His131), FcγRIIb (CD32b), FcγRIIIa (CD16a Val158 и CD16a Phe158) и FcγRIIIb (CD16b), но не связывалось с нетрансфицированными клетками CHO (отрицательный контроль). Значительное связывание контрольного IgG1 с некоторыми линиями клеток, экспрессирующих FcγR, наблюдалось при концентрациях всего 4 нМ. На ФИГ. 30А показано, что не наблюдалось детектируемого связывания конструкции ALG.APV-210 с тестируемым FcγR: FcγRI (CD64), FcγRIIa (CD32a His131), FcγRIIb (CD32b), FcγRIIIa (CD16a Val158 и CD16a Phe158) и FcγRIIIb (CD16b).
Пример 22. Активность биспецифичного антитела к 5T4-CD137 in vitro в анализе пролиферации Т-клеток с использованием МНПК человека и клеток СНО-5Т4
[00365] Функциональную активность биспецифичных молекул к 5T4-CD137 ALG.APV-209 и ALG.APV-210 оценивали в анализе пролиферации Т-клеток с использованием МНПК. МНПК выделяли из крови здоровых людей с использованием стандартных градиентов фиколла. После выделения клетки промывали в солевом буфере для удаления тромбоцитов. МНПК культивировали с облученными клетками CHO-K1/ 5T4 человека в количестве приблизительно 120 000 МНПК на 30 000 5T4(+) клеток в 96-луночных планшетах. Клетки CHO-K1/5T4 облучали (рентгеновский облучатель Cell Rad, Faxitron Bioptics, LLC) и промывали в среде перед высеванием с МНПК. Во все лунки добавляли антитело к CD3 OKT3 (eBioscience) в концентрации 1 нг/мл. Клетки культивировали в общем объеме 200 мкл среды RPMI 1640 (GIBCO) с добавками 10% фетальной бычьей сыворотки (SIGMA), пирувата натрия, антибиотиков и заменимых аминокислот. Планшеты инкубировали при 37 ºC, 5% CO2 в инкубаторах с увлажненной средой в течение 72 часов. Супернатанты удаляли и клетки метили в тех же планшетах для анализа антителами к CD4, CD8 и CD5 в буфере PBS с 2% BSA и 2 мМ ЭДТА в течение 30 минут на льду, добавляли 7AAD для исключения мертвых клеток. После промывки клетки повторно суспендировали в концентрации 120 мкл/лунку, и из каждой лунки отбирали по 70 мкл в проточном цитометре (LSR-IITM, BD Biosciences). Анализ клеток проводили с использованием программного обеспечения Flowjo и анализировали путем подсчета количества событий CD4+ или CD8+ живых Т-клеток (CD4+ CD8- CD5+, 7AAD- или CD4- CD8+ CD5+ 7AAD-) после гейтинга лимфоцитов по сигналам прямого (FSC) и бокового (SSC) светорассеяния. Проводили два параллельных испытания каждого условия и строили графики средних значений из каждого двойного набора с использованием программного обеспечения для построения графиков и статистических вычислений GraphPad Prism 6®.
[00366] Результаты исследования представлены на ФИГ. 31А и 31В. Антигенспецифичную пролиферацию CD8+ T-клеток оценивали после 72 часов стимуляции T-клеток, примированных низкой концентрацией антител к CD3. Стимуляция антителами к CD3 индуцирует активацию CD137. Добавление только антител к CD3 без биспецифичных антител индуцировало некоторую пролиферацию CD8+ и CD4+ Т-клеток. Добавление ALG.APV-209 или ALG.APV-210 повышало пролиферацию CD8+ T-клеток (ФИГ. 31A). Напротив, и, как и ожидалось, исходя из избирательной экспрессии CD137 на CD8+ T-клетках, биспецифичные антитела оказывали ограниченное влияние на индуцированную антителами к CD3 пролиферацию CD4+ T-клеток (ФИГ. 31B). Результаты согласовались среди нескольких экспериментов. Контрольная молекула в формате Моррисона (ALG.APV-004) была включена для сравнения.
[00367] В совокупности результаты показали, что и ALG.APV-209, и ALG.APV-210 усиливали пролиферацию CD8+ T-клеток. ALG.APV-209 и ALG.APV-210 индуцировали более высокую степень пролиферации клеток, чем конструкция в формате Моррисона ALG.APV-004.
Пример 23. Экспрессия 5T4 в нормальных и опухолевых тканях, оцененная с помощью иммуногистохимического анализа.
[00368] Экспрессию опухолевого антигена 5T4 в нормальной человеческой ткани и в ряде различных типов опухолей оценивали с помощью иммуногистохимического анализа (IHC).
[00369] Разработка метода окрашивания: пять коммерчески доступных антител к 5Т4 и два разработанных в собственной лаборатории антитела к 5Т4 оценивали на связывание 5Т4 в срезах замороженной ткани по сравнению с фиксированными формалином, залитыми парафином (FFPE) срезами контрольных клеток и тестируемых человеческих тканей. Также было включено окрашивание изотипическим контролем. Мышиное моноклональное антитело (клон: MAB4975, R&D Systems) было выбрано в качестве антитела с наилучшими характеристиками, поскольку оно было высокоспецифичным и функциональным как в срезах замороженной ткани, так и в срезах FFPE. Способ демаскирования антигена и титр антител были оптимизированы для этого антитела.
[00370] Ткани и тканевые матрицы: в качестве положительных контролей использовали ткань человеческой плаценты и линию клеток карциномы толстой кишки мыши CT26 (от ATCC® CRL-2638), трансфицированных для экспрессии человеческого 5T4. Человеческий головной мозг, печень и нетрансфицированные клетки CT26 использовали в качестве отрицательных контролей. Человеческие ткани, использованные в качестве положительных и отрицательных контролей, были взяты из архивов Charles River Laboratories. Микроматрицы тканей FFPE (TMA) были приобретены у US Biomax Inc. Все окрашивания оценивались лаборантом-исследователем в PAI, Charles River Laboratories.
[00371] Окрашивание для IHC микроматриц нормальных человеческих тканей: на выборке нормальных человеческих тканей с 24 типами тканей от 3 доноров (FDA808j-1) экспрессия 5T4 не была обнаружена ни в одном из основных органов.
[00372] Окрашивание для IHC типов человеческой опухолевой ткани: осуществляли скрининг на экспрессию 5T4 в матрице опухолевых тканей нескольких органов, полученных от 72 больных (FDA808j-2), представленных одним ядром на опухоль. Экспрессия 5T4 была обнаружена в опухолях поджелудочной железы, щитовидной железы, молочной железы, печени, матки, шейки матки, поперечно-полосатой мышцы, кожи (плоскоклеточная карцинома), нервной ткани (нейрофиброма) и мочевого пузыря.
[00373] Экспрессию 5T4 оценивали в TMA, содержащих опухоли от 10-50 больных для каждого из выбранных видов опухолей, приведенных в таблице 33, представленных двойными или тройными ядрами. При раке мочевого пузыря, раке головы и шеи (ГиШ), немелкоклеточном раке легкого (НМРЛ) и мезотелиоме 50-56% исследованных опухолей были 5T4-позитивными. Значительное количество опухолей поджелудочной железы также были 5T4-позитивными (36%). 50% исследованных светлоклеточных карцином были 5T4-позитивными.
Таблица 33. Экспрессия 5T4 в семи выбранных видах опухолей
*****
Включение посредством ссылки
[00374] Все источники, статьи, публикации, патенты, патентные публикации и патентные заявки, цитируемые в настоящем документе, включены посредством ссылки в полном объеме для любых целей. Однако упоминание любого источника, статьи, публикации, патента, патентной публикации и патентной заявки, цитируемых в настоящем документе, не является и не должно быть истолковано как признание или любая форма предположения о том, что они представляют собой релевантный уровень техники или составляют часть общеизвестных сведений в какой-либо стране мира.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> Aptevo Research and Development LLC
Alligator Bioscience AB
Bienvenue, David
Misher, Lynda
Hoyos, Gabriela H.
Mitchell, Danielle
Ellmark, Peter
Sall, Anna
Furebring, Christina
Von Schantz, Laura
Fritzell, Sara
Varas, Laura
<120> БЕЛКИ, СВЯЗЫВАЮЩИЕ ОНКОФЕТАЛЬНЫЙ АНТИГЕН, И ОТНОСЯЩИЕСЯ
К НИМ КОМПОЗИЦИИ И СПОСОБЫ
<130> APVO-057/03WO 327714-2734
<150> US 62/535,017
<151> 2017-07-20
<150> US 62/575,820
<151> 2017-10-23
<150> US 62/648,072
<151> 2018-03-26
<160> 181
<170> PatentIn версия 3.5
<210> 1
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 1
ggattcacct tttctcacgg ttct 24
<210> 2
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 2
Gly Phe Thr Phe Ser His Gly Ser
1 5
<210> 3
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 3
atttcttctg gttctggttc taca 24
<210> 4
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 4
Ile Ser Ser Gly Ser Gly Ser Thr
1 5
<210> 5
<211> 42
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 5
gcgcgctctt cttactacgg ttcttactac tctattgact at 42
<210> 6
<211> 14
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 6
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr
1 5 10
<210> 7
<211> 18
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 7
cagagcatta gcagctat 18
<210> 8
<211> 6
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 8
Gln Ser Ile Ser Ser Tyr
1 5
<210> 9
<211> 9
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 9
gctgcatcc 9
<210> 10
<211> 3
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 10
Ala Ala Ser
1
<210> 11
<211> 27
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 11
caacagtact acgacaacct gcccact 27
<210> 12
<211> 9
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 12
Gln Gln Tyr Tyr Asp Asn Leu Pro Thr
1 5
<210> 13
<211> 363
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 13
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 300
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 14
<211> 121
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 14
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser His Gly
20 25 30
Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 15
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 15
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctacaacct 240
gaagattttg caacttatta ctgtcaacag tactacgaca acctgcccac ttttggccag 300
gggaccaagc tggagatcaa a 321
<210> 16
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 16
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 17
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 17
ggattcacct ttgattacgg ttct 24
<210> 18
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 18
Gly Phe Thr Phe Asp Tyr Gly Ser
1 5
<210> 19
<211> 363
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 19
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt cacctttgat tacggttcta tgtactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccacgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 300
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 20
<211> 121
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 20
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Tyr Gly
20 25 30
Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 21
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 21
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcacagtgg ggtcccatca 180
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttatta ctgtcaacag tactacgaca acctgcccac ttttggccag 300
gggaccaagc tggagatcaa a 321
<210> 22
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 22
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu Pro
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 23
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 23
ggattcacct tttcttacgg ttct 24
<210> 24
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 24
Gly Phe Thr Phe Ser Tyr Gly Ser
1 5
<210> 25
<211> 363
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 25
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 300
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 26
<211> 121
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 26
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Tyr Gly
20 25 30
Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 27
<211> 363
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 27
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 300
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 360
tca 363
<210> 28
<211> 121
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагменты антител
<400> 28
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Tyr Gly
20 25 30
Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 29
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 29
ggcttcacat tcagcagcta tgct 24
<210> 30
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 30
Gly Phe Thr Phe Ser Ser Tyr Ala
1 5
<210> 31
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 31
atctccggca gcggcggaag cacc 24
<210> 32
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 32
Ile Ser Gly Ser Gly Gly Ser Thr
1 5
<210> 33
<211> 42
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 33
gccaggtact atggcggcta ctactccgcc tggatggact ac 42
<210> 34
<211> 14
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 34
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr
1 5 10
<210> 35
<211> 27
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 35
cagcagacct atggctacct gcacacc 27
<210> 36
<211> 9
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 36
Gln Gln Thr Tyr Gly Tyr Leu His Thr
1 5
<210> 37
<211> 363
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 37
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cacattcagc agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagg gcctggagtg ggtgtccgct atctccggca gcggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agc 363
<210> 38
<211> 121
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 38
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 39
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 39
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 60
atcacctgca gggccagcca gtccatctcc agctatttaa actggtacca gcagaagcct 120
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 180
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 240
gaggactccg ctacctacta ctgccagcag acctatggct acctgcacac cttcggccag 300
ggcacaaagc tggagatcaa g 321
<210> 40
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 40
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 41
<211> 18
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 41
cagtccatct ccagcttc 18
<210> 42
<211> 6
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 42
Gln Ser Ile Ser Ser Phe
1 5
<210> 43
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 43
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 60
atcacctgca gggccagcca gtccatctcc agcttcttaa actggtacca gcagaagcct 120
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 180
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 240
gaggactccg ctacctacta ctgccagcag acctatggct acctgcacac cttcggccag 300
ggcacaaagc tggagatcaa g 321
<210> 44
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 44
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 45
<211> 363
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 45
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cacattcagc agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagt gcctggagtg ggtgtccgct atctccggca gcggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agc 363
<210> 46
<211> 121
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 46
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 47
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 47
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 60
atcacctgca gggccagcca gtccatctcc agcttcttaa actggtacca gcagaagcct 120
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 180
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 240
gaggactccg ctacctacta ctgccagcag acctatggct acctgcacac cttcggctgc 300
ggcacaaagc tggagatcaa g 321
<210> 48
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 48
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 49
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 49
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 60
atcacctgca gggccagcca gtccatctcc agcttcttaa actggtacca gcagaagcct 120
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 180
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 240
gaggacttcg ctacctacta ctgccagcag acctatggct acctgcacac cttcggctgc 300
ggcacaaagc tggagatcaa g 321
<210> 50
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 50
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Phe
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 51
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 51
ggcttcgact tcgagagcta tgct 24
<210> 52
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 52
Gly Phe Asp Phe Glu Ser Tyr Ala
1 5
<210> 53
<211> 18
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 53
cagtccatca ggagcgcc 18
<210> 54
<211> 6
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 54
Gln Ser Ile Arg Ser Ala
1 5
<210> 55
<211> 363
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 55
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cgacttcgag agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagt gcctggagtg ggtgtccgct atctccggca gcggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agc 363
<210> 56
<211> 121
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 56
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Glu Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 57
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 57
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 60
atcacctgca gggccagcca gtccatcagg agcgccctga actggtacca gcagaagcct 120
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 180
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 240
gaggacttcg ctacctacta ctgccagcag acctatggct acctgcacac cttcggctgc 300
ggcacaaagc tggagatcaa g 321
<210> 58
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 58
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Arg Ser Ala
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 59
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 59
ggcttcgact tcgacagcta tgct 24
<210> 60
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 60
Gly Phe Asp Phe Asp Ser Tyr Ala
1 5
<210> 61
<211> 24
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 61
atctccggca ggggcggaag cacc 24
<210> 62
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 62
Ile Ser Gly Arg Gly Gly Ser Thr
1 5
<210> 63
<211> 363
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 63
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cgacttcgac agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagt gcctggagtg ggtgtccgct atctccggca ggggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agc 363
<210> 64
<211> 121
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 64
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Asp Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Arg Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser
115 120
<210> 65
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 65
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttatta ctgtcaacag acttacggtt acctgcacac ttttggctgt 300
gggaccaggc tggagatcaa a 321
<210> 66
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 66
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Cys Gly Thr Arg Leu Glu Ile Lys
100 105
<210> 67
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 67
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttatta ctgtcaacag acttacggtt acctgcacac ttttggccag 300
gggaccaagc tggagatcaa a 321
<210> 68
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 68
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 69
<211> 321
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 69
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttatta ctgtcaacag acttacggtt acctgcacac ttttggctgc 300
gggaccaagc tggagatcaa a 321
<210> 70
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 70
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 71
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 71
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Ser Pro Pro Ser Ser
1 5 10 15
<210> 72
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 72
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Ser Pro Pro Cys Ser
1 5 10 15
<210> 73
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 73
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Ser Pro Pro Cys Ser
1 5 10 15
<210> 74
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 74
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Ser
1 5 10 15
<210> 75
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 75
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Ser Pro Pro Ser Ser
1 5 10 15
<210> 76
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 76
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Ser Ser
1 5 10 15
<210> 77
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 77
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Ser Pro Pro Cys Ser
1 5 10 15
<210> 78
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 78
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Ser Pro Pro Cys Pro
1 5 10 15
<210> 79
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 79
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Ser Pro Pro Ser Pro
1 5 10 15
<210> 80
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 80
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Ser Pro Pro Cys Pro
1 5 10 15
<210> 81
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 81
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Ser Pro Pro Cys Pro
1 5 10 15
<210> 82
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 82
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro
1 5 10 15
<210> 83
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 83
Glu Pro Lys Ser Cys Asp Lys Thr His Thr Ser Pro Pro Ser Pro
1 5 10 15
<210> 84
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 84
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Ser Pro
1 5 10 15
<210> 85
<211> 6
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 85
Ser Cys Pro Pro Cys Pro
1 5
<210> 86
<211> 20
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 86
Asn Tyr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10 15
Ser Gly Asn Ser
20
<210> 87
<211> 38
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 87
Asn Tyr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10 15
Ser Gly Asn Tyr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
20 25 30
Gly Gly Ser Gly Asn Ser
35
<210> 88
<211> 2
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 88
Asn Ser
1
<210> 89
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 89
Gly Gly Gly Gly Ser Gly Asn Ser
1 5
<210> 90
<211> 10
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 90
Asn Tyr Gly Gly Gly Gly Ser Gly Asn Ser
1 5 10
<210> 91
<211> 13
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 91
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Asn Ser
1 5 10
<210> 92
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 92
Asn Tyr Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Asn Ser
1 5 10 15
<210> 93
<211> 18
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 93
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Asn Ser
<210> 94
<211> 8
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 94
Gly Cys Pro Pro Cys Pro Asn Ser
1 5
<210> 95
<211> 5
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 95
Gly Gly Gly Gly Ser
1 5
<210> 96
<211> 15
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 96
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 97
<211> 16
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 97
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
<210> 98
<211> 20
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 98
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 99
<211> 21
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 99
Gln Arg His Asn Asn Ser Ser Leu Asn Thr Gly Thr Gln Met Ala Gly
1 5 10 15
His Ser Pro Asn Ser
20
<210> 100
<211> 16
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 100
Ser Ser Leu Asn Thr Gly Thr Gln Met Ala Gly His Ser Pro Asn Ser
1 5 10 15
<210> 101
<211> 18
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 101
Gln Arg His Asn Asn Ser Ser Leu Asn Thr Gly Thr Gln Met Ala Gly
1 5 10 15
His Ser
<210> 102
<211> 14
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 102
Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr Ser Pro Asn Ser
1 5 10
<210> 103
<211> 20
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 103
Asn Ser Leu Ala Asn Gln Glu Val Gln Ile Pro Leu Thr Glu Ser Tyr
1 5 10 15
Ser Pro Asn Ser
20
<210> 104
<211> 19
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 104
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
Pro Asn Ser
<210> 105
<211> 19
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 105
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
Pro Gly Ser
<210> 106
<211> 19
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 106
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
Pro Ala Ser
<210> 107
<211> 18
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 107
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
Pro Ser
<210> 108
<211> 19
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - шарнирная/линкерная последовательность
<400> 108
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10 15
Pro Ser Ser
<210> 109
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 109
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 300
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 360
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 420
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 480
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 540
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 600
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 660
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 720
caggggacca agctggagat caaa 744
<210> 110
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 110
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser His Gly
20 25 30
Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu Pro Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Leu Glu Ile Lys
245
<210> 111
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 111
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt cacctttgat tacggttcta tgtactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccacgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 300
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 360
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 420
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 480
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 540
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcacag tggggtccca 600
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 660
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 720
caggggacca agctggagat caaa 744
<210> 112
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 112
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asp Tyr Gly
20 25 30
Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu His Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu Pro Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Leu Glu Ile Lys
245
<210> 113
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 113
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 300
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 360
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 420
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 480
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 540
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 600
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 660
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 720
caggggacca agctggagat caaa 744
<210> 114
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 114
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Tyr Gly
20 25 30
Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu Pro Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Leu Glu Ile Lys
245
<210> 115
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 115
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 300
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 360
tcaggcggcg gcggcagcgg cggcggcggc agcggcggcg gaggctccgg cggcggcggc 420
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 480
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 540
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 600
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 660
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 720
caggggacca agctggagat caaa 744
<210> 116
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 116
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Tyr Gly
20 25 30
Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu Pro Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Leu Glu Ile Lys
245
<210> 117
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 117
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cacattcagc agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagg gcctggagtg ggtgtccgct atctccggca gcggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agcggcggag gcggctccgg aggcggtggc tccggaggag gcggaagcgg aggaggaggc 420
tccgatattc agatgacaca gtcccctagc tccctgtccg ccagcgtggg agatcgggtg 480
accatcacct gcagggccag ccagtccatc tccagctatt taaactggta ccagcagaag 540
cctggaaagg ctcccaagct gctgatctac gccgcttcca gcctccagag cggcgtgcct 600
agcaggttct ccggctccgg aagcggaaca gacttcaccc tgaccatcag ctccctgcag 660
cccgaggact ccgctaccta ctactgccag cagacctatg gctacctgca caccttcggc 720
cagggcacaa agctggagat caag 744
<210> 118
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 118
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Ser
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Leu Glu Ile Lys
245
<210> 119
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 119
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cacattcagc agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagg gcctggagtg ggtgtccgct atctccggca gcggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agcggcggag gcggctccgg aggcggtggc tccggaggag gcggaagcgg aggaggaggc 420
tccgatattc agatgacaca gtcccctagc tccctgtccg ccagcgtggg agatcgggtg 480
accatcacct gcagggccag ccagtccatc tccagcttct taaactggta ccagcagaag 540
cctggaaagg ctcccaagct gctgatctac gccgcttcca gcctccagag cggcgtgcct 600
agcaggttct ccggctccgg aagcggaaca gacttcaccc tgaccatcag ctccctgcag 660
cccgaggact ccgctaccta ctactgccag cagacctatg gctacctgca caccttcggc 720
cagggcacaa agctggagat caag 744
<210> 120
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 120
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Phe Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Ser
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Leu Glu Ile Lys
245
<210> 121
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 121
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cacattcagc agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagt gcctggagtg ggtgtccgct atctccggca gcggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agcggcggag gcggctccgg aggcggtggc tccggaggag gcggaagcgg aggaggaggc 420
tccgatattc agatgacaca gtcccctagc tccctgtccg ccagcgtggg agatcgggtg 480
accatcacct gcagggccag ccagtccatc tccagcttct taaactggta ccagcagaag 540
cctggaaagg ctcccaagct gctgatctac gccgcttcca gcctccagag cggcgtgcct 600
agcaggttct ccggctccgg aagcggaaca gacttcaccc tgaccatcag ctccctgcag 660
cccgaggact ccgctaccta ctactgccag cagacctatg gctacctgca caccttcggc 720
tgcggcacaa agctggagat caag 744
<210> 122
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 122
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Phe Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Ser
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly
225 230 235 240
Cys Gly Thr Lys Leu Glu Ile Lys
245
<210> 123
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 123
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cacattcagc agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagt gcctggagtg ggtgtccgct atctccggca gcggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agcggcggag gcggctccgg aggcggtggc tccggaggag gcggaagcgg aggaggaggc 420
tccgatattc agatgacaca gtcccctagc tccctgtccg ccagcgtggg agatcgggtg 480
accatcacct gcagggccag ccagtccatc tccagcttct taaactggta ccagcagaag 540
cctggaaagg ctcccaagct gctgatctac gccgcttcca gcctccagag cggcgtgcct 600
agcaggttct ccggctccgg aagcggaaca gacttcaccc tgaccatcag ctccctgcag 660
cccgaggact tcgctaccta ctactgccag cagacctatg gctacctgca caccttcggc 720
tgcggcacaa agctggagat caag 744
<210> 124
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 124
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Phe Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly
225 230 235 240
Cys Gly Thr Lys Leu Glu Ile Lys
245
<210> 125
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 125
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cgacttcgag agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagt gcctggagtg ggtgtccgct atctccggca gcggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agcggcggag gcggctccgg aggcggtggc tccggaggag gcggaagcgg aggaggaggc 420
tccgatattc agatgacaca gtcccctagc tccctgtccg ccagcgtggg agatcgggtg 480
accatcacct gcagggccag ccagtccatc aggagcgccc tgaactggta ccagcagaag 540
cctggaaagg ctcccaagct gctgatctac gccgcttcca gcctccagag cggcgtgcct 600
agcaggttct ccggctccgg aagcggaaca gacttcaccc tgaccatcag ctccctgcag 660
cccgaggact tcgctaccta ctactgccag cagacctatg gctacctgca caccttcggc 720
tgcggcacaa agctggagat caag 744
<210> 126
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 126
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Glu Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Arg Ser Ala Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly
225 230 235 240
Cys Gly Thr Lys Leu Glu Ile Lys
245
<210> 127
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 127
gaagtgcagc tgctggagtc cggaggagga ctggtgcagc ctggcggaag cctgaggctg 60
agctgcgctg cctccggctt cgacttcgac agctatgcta tgagctgggt gaggcaagcc 120
cctggaaagt gcctggagtg ggtgtccgct atctccggca ggggcggaag cacctactac 180
gctgactccg tcaagggcag gttcaccatc agccgggaca acagcaagaa caccctgtac 240
ctgcagatga atagcctcag ggctgaagac accgctgtgt actactgcgc caggtactat 300
ggcggctact actccgcctg gatggactac tggggacagg gcacactggt gaccgtgtcc 360
agcggcggag gcggctccgg aggcggtggc tccggaggag gcggaagcgg aggaggaggc 420
tccgatattc agatgacaca gtcccctagc tccctgtccg ccagcgtggg agatcgggtg 480
accatcacct gcagggccag ccagtccatc aggagcgccc tgaactggta ccagcagaag 540
cctggaaagg ctcccaagct gctgatctac gccgcttcca gcctccagag cggcgtgcct 600
agcaggttct ccggctccgg aagcggaaca gacttcaccc tgaccatcag ctccctgcag 660
cccgaggact tcgctaccta ctactgccag cagacctatg gctacctgca caccttcggc 720
tgcggcacaa agctggagat caag 744
<210> 128
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 128
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Asp Phe Asp Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Arg Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Arg Ser Ala Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly
225 230 235 240
Cys Gly Thr Lys Leu Glu Ile Lys
245
<210> 129
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 129
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt cacctttagc agctatgcca tgagctgggt ccgccaggct 120
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctactac 300
ggtggttact actctgcttg gatggactat tggggccagg gaaccctggt caccgtctcc 360
tcaggcggtg gaggcagcgg tgggggtggg tctggaggcg gtggcagtgg cggcggaggc 420
tctgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 480
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 540
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 600
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 660
cctgaagatt ttgcaactta ttactgtcaa cagacttacg gttacctgca cacttttggc 720
caggggacca agctggagat caaa 744
<210> 130
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 130
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly
225 230 235 240
Gln Gly Thr Lys Leu Glu Ile Lys
245
<210> 131
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 131
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 60
tcctgtgcag ccagcggatt cacctttagc agctatgcca tgagctgggt ccgccaggct 120
ccagggaagt gcctggagtg ggtctcagct attagtggta gtggtggtag cacatactat 180
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 240
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctactac 300
ggtggttact actctgcttg gatggactat tggggccagg gaaccctggt caccgtctcc 360
tcaggcggtg gaggcagcgg tgggggtggg tctggaggcg gtggcagtgg cggcggaggc 420
tctgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 480
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 540
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 600
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 660
cctgaagatt ttgcaactta ttactgtcaa cagacttacg gttacctgca cacttttggc 720
tgcgggacca agctggagat caaa 744
<210> 132
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 132
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly
100 105 110
Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln
130 135 140
Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val
145 150 155 160
Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp
165 170 175
Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala
180 185 190
Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser
195 200 205
Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe
210 215 220
Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly
225 230 235 240
Cys Gly Thr Lys Leu Glu Ile Lys
245
<210> 133
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 133
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttatta ctgtcaacag acttacggtt acctgcacac ttttggctgc 300
gggaccaagc tggagatcaa aggcggtgga ggcagcggtg ggggtgggtc tggaggcggt 360
ggcagtggcg gcggaggctc tgaggtgcag ctgttggaga gcgggggagg cttggtacag 420
cctggggggt ccctgcgcct ctcctgtgca gccagcggat tcacctttag cagctatgcc 480
atgagctggg tccgccaggc tccagggaag tgcctggagt gggtctcagc tattagtggt 540
agtggtggta gcacatacta tgcagactcc gtgaagggcc ggttcaccat ctcccgtgac 600
aattccaaga acacgctgta tctgcaaatg aacagcctgc gtgccgagga cacggctgta 660
tattattgtg cgcgctacta cggtggttac tactctgctt ggatggacta ttggggccag 720
ggaaccctgg tcaccgtctc ctca 744
<210> 134
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 134
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
115 120 125
Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
130 135 140
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala
145 150 155 160
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Cys Leu Glu Trp Val Ser
165 170 175
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
180 185 190
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
195 200 205
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
210 215 220
Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 135
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 135
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcgagt gagcccaaat cttctgacaa aactcacaca 840
tgcccaccgt gcccagcacc tgaagccgcg ggtgcaccgt cagtcttcct cttcccccca 900
aaacccaagg acaccctcat gatctcccgg acccctgagg tcacatgcgt ggtggtggac 960
gtgagccacg aagaccctga ggtcaagttc aactggtacg tggacggcgt ggaggtgcat 1020
aatgccaaga caaagccgcg ggaggagcag tacaacagca cgtaccgtgt ggtcagcgtc 1080
ctcaccgtcc tgcaccagga ctggctgaat ggcaaggaat acaagtgcgc ggtctccaac 1140
aaagccctcc cagcccccat cgagaaaacc atctccaaag ccaaagggca gccccgagaa 1200
ccacaggtgt acaccctgcc cccatcccgg gatgagctga ccaagaacca ggtcagcctg 1260
acctgcctgg tcaaaggctt ctatccaagc gacatcgccg tggagtggga gagcaatggg 1320
cagccggaga acaactacaa gaccacgcct cccgtgctgg actccgacgg ctccttcttc 1380
ctctacagca agctcaccgt ggacaagagc aggtggcagc aggggaacgt cttctcatgc 1440
tccgtgatgc atgaggctct gcacaaccac tacacgcaga agagcctctc cctgtctccg 1500
ggttccggag gtggcggttc gggaggtggc gggtcaggag gtgggggatc cccttcagaa 1560
gtgcagctgc tggagtccgg aggaggactg gtgcagcctg gcggaagcct gaggctgagc 1620
tgcgctgcct ccggcttcac attcagcagc tatgctatga gctgggtgag gcaagcccct 1680
ggaaagggcc tggagtgggt gtccgctatc tccggcagcg gcggaagcac ctactacgct 1740
gactccgtca agggcaggtt caccatcagc cgggacaaca gcaagaacac cctgtacctg 1800
cagatgaata gcctcagggc tgaagacacc gctgtgtact actgcgccag gtactatggc 1860
ggctactact ccgcctggat ggactactgg ggacagggca cactggtgac cgtgtccagc 1920
ggcggaggcg gctccggagg cggtggctcc ggaggaggcg gaagcggagg aggaggctcc 1980
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 2040
atcacctgca gggccagcca gtccatctcc agctatttaa actggtacca gcagaagcct 2100
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 2160
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 2220
gaggactccg ctacctacta ctgccagcag acctatggct acctgcacac cttcggccag 2280
ggcacaaagc tggagatcaa gcgc 2304
<210> 136
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 136
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser His Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Glu Pro
260 265 270
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
275 280 285
Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
290 295 300
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
305 310 315 320
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
325 330 335
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
340 345 350
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
355 360 365
Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro
370 375 380
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
385 390 395 400
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
405 410 415
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
420 425 430
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
435 440 445
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
450 455 460
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
465 470 475 480
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
485 490 495
Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
500 505 510
Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
515 520 525
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
530 535 540
Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
545 550 555 560
Gly Lys Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser
565 570 575
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
580 585 590
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
595 600 605
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
610 615 620
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
625 630 635 640
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
645 650 655
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
660 665 670
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
675 680 685
Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
690 695 700
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
705 710 715 720
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
725 730 735
Ser Leu Gln Pro Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr
740 745 750
Gly Tyr Leu His Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 137
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 137
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcgagt gagcccaaat cttctgacaa aactcacaca 840
tgcccaccgt gcccagcacc tgaagccgcg ggtgcaccgt cagtcttcct cttcccccca 900
aaacccaagg acaccctcat gatctcccgg acccctgagg tcacatgcgt ggtggtggac 960
gtgagccacg aagaccctga ggtcaagttc aactggtacg tggacggcgt ggaggtgcat 1020
aatgccaaga caaagccgcg ggaggagcag tacaacagca cgtaccgtgt ggtcagcgtc 1080
ctcaccgtcc tgcaccagga ctggctgaat ggcaaggaat acaagtgcgc ggtctccaac 1140
aaagccctcc cagcccccat cgagaaaacc atctccaaag ccaaagggca gccccgagaa 1200
ccacaggtgt acaccctgcc cccatcccgg gatgagctga ccaagaacca ggtcagcctg 1260
acctgcctgg tcaaaggctt ctatccaagc gacatcgccg tggagtggga gagcaatggg 1320
cagccggaga acaactacaa gaccacgcct cccgtgctgg actccgacgg ctccttcttc 1380
ctctacagca agctcaccgt ggacaagagc aggtggcagc aggggaacgt cttctcatgc 1440
tccgtgatgc atgaggctct gcacaaccac tacacgcaga agagcctctc cctgtctccg 1500
ggttccggag gtggcggttc gggaggtggc gggtcaggag gtgggggatc cccttcagaa 1560
gtgcagctgc tggagtccgg aggaggactg gtgcagcctg gcggaagcct gaggctgagc 1620
tgcgctgcct ccggcttcac attcagcagc tatgctatga gctgggtgag gcaagcccct 1680
ggaaagggcc tggagtgggt gtccgctatc tccggcagcg gcggaagcac ctactacgct 1740
gactccgtca agggcaggtt caccatcagc cgggacaaca gcaagaacac cctgtacctg 1800
cagatgaata gcctcagggc tgaagacacc gctgtgtact actgcgccag gtactatggc 1860
ggctactact ccgcctggat ggactactgg ggacagggca cactggtgac cgtgtccagc 1920
ggcggaggcg gctccggagg cggtggctcc ggaggaggcg gaagcggagg aggaggctcc 1980
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 2040
atcacctgca gggccagcca gtccatctcc agcttcttaa actggtacca gcagaagcct 2100
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 2160
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 2220
gaggactccg ctacctacta ctgccagcag acctatggct acctgcacac cttcggccag 2280
ggcacaaagc tggagatcaa gcgc 2304
<210> 138
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 138
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser His Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Glu Pro
260 265 270
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
275 280 285
Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
290 295 300
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
305 310 315 320
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
325 330 335
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
340 345 350
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
355 360 365
Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro
370 375 380
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
385 390 395 400
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
405 410 415
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
420 425 430
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
435 440 445
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
450 455 460
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
465 470 475 480
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
485 490 495
Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
500 505 510
Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
515 520 525
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
530 535 540
Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
545 550 555 560
Gly Lys Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser
565 570 575
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
580 585 590
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
595 600 605
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
610 615 620
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
625 630 635 640
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
645 650 655
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
660 665 670
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
675 680 685
Ile Ser Ser Phe Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
690 695 700
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
705 710 715 720
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
725 730 735
Ser Leu Gln Pro Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr
740 745 750
Gly Tyr Leu His Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 139
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 139
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcgagt gagcccaaat cttctgacaa aactcacaca 840
tgcccaccgt gcccagcacc tgaagccgcg ggtgcaccgt cagtcttcct cttcccccca 900
aaacccaagg acaccctcat gatctcccgg acccctgagg tcacatgcgt ggtggtggac 960
gtgagccacg aagaccctga ggtcaagttc aactggtacg tggacggcgt ggaggtgcat 1020
aatgccaaga caaagccgcg ggaggagcag tacaacagca cgtaccgtgt ggtcagcgtc 1080
ctcaccgtcc tgcaccagga ctggctgaat ggcaaggaat acaagtgcgc ggtctccaac 1140
aaagccctcc cagcccccat cgagaaaacc atctccaaag ccaaagggca gccccgagaa 1200
ccacaggtgt acaccctgcc cccatcccgg gatgagctga ccaagaacca ggtcagcctg 1260
acctgcctgg tcaaaggctt ctatccaagc gacatcgccg tggagtggga gagcaatggg 1320
cagccggaga acaactacaa gaccacgcct cccgtgctgg actccgacgg ctccttcttc 1380
ctctacagca agctcaccgt ggacaagagc aggtggcagc aggggaacgt cttctcatgc 1440
tccgtgatgc atgaggctct gcacaaccac tacacgcaga agagcctctc cctgtctccg 1500
ggttccggag gtggcggttc gggaggtggc gggtcaggag gtgggggatc cccttcagaa 1560
gtgcagctgc tggagtccgg aggaggactg gtgcagcctg gcggaagcct gaggctgagc 1620
tgcgctgcct ccggcttcac attcagcagc tatgctatga gctgggtgag gcaagcccct 1680
ggaaagtgcc tggagtgggt gtccgctatc tccggcagcg gcggaagcac ctactacgct 1740
gactccgtca agggcaggtt caccatcagc cgggacaaca gcaagaacac cctgtacctg 1800
cagatgaata gcctcagggc tgaagacacc gctgtgtact actgcgccag gtactatggc 1860
ggctactact ccgcctggat ggactactgg ggacagggca cactggtgac cgtgtccagc 1920
ggcggaggcg gctccggagg cggtggctcc ggaggaggcg gaagcggagg aggaggctcc 1980
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 2040
atcacctgca gggccagcca gtccatctcc agcttcttaa actggtacca gcagaagcct 2100
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 2160
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 2220
gaggactccg ctacctacta ctgccagcag acctatggct acctgcacac cttcggctgc 2280
ggcacaaagc tggagatcaa gcgc 2304
<210> 140
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 140
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser His Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Glu Pro
260 265 270
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
275 280 285
Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
290 295 300
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
305 310 315 320
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
325 330 335
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
340 345 350
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
355 360 365
Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro
370 375 380
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
385 390 395 400
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
405 410 415
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
420 425 430
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
435 440 445
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
450 455 460
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
465 470 475 480
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
485 490 495
Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
500 505 510
Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
515 520 525
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
530 535 540
Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
545 550 555 560
Gly Lys Cys Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser
565 570 575
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
580 585 590
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
595 600 605
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
610 615 620
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
625 630 635 640
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
645 650 655
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
660 665 670
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
675 680 685
Ile Ser Ser Phe Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
690 695 700
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
705 710 715 720
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
725 730 735
Ser Leu Gln Pro Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr
740 745 750
Gly Tyr Leu His Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 141
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 141
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt cacctttgat tacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccacgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcacag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcctcg gagcccaaat cttctgacaa aactcacaca 840
tgcccaccgt gcccagcacc tgaagccgcg ggtgcaccgt cagtcttcct cttcccccca 900
aaacccaagg acaccctcat gatctcccgg acccctgagg tcacatgcgt ggtggtggac 960
gtgagccacg aagaccctga ggtcaagttc aactggtacg tggacggcgt ggaggtgcat 1020
aatgccaaga caaagccgcg ggaggagcag tacaacagca cgtaccgtgt ggtcagcgtc 1080
ctcaccgtcc tgcaccagga ctggctgaat ggcaaggaat acaagtgcgc ggtctccaac 1140
aaagccctcc cagcccccat cgagaaaacc atctccaaag ccaaagggca gccccgagaa 1200
ccacaggtgt acaccctgcc cccatcccgg gatgagctga ccaagaacca ggtcagcctg 1260
acctgcctgg tcaaaggctt ctatccaagc gacatcgccg tggagtggga gagcaatggg 1320
cagccggaga acaactacaa gaccacgcct cccgtgctgg actccgacgg ctccttcttc 1380
ctctacagca agctcaccgt ggacaagagc aggtggcagc aggggaacgt cttctcatgc 1440
tccgtgatgc atgaggctct gcacaaccac tacacgcaga agagcctctc cctgtctccg 1500
ggttccggag gtggcggttc gggaggtggc gggtcaggag gtgggggatc cccttcagaa 1560
gtgcagctgc tggagtccgg aggaggactg gtgcagcctg gcggaagcct gaggctgagc 1620
tgcgctgcct ccggcttcac attcagcagc tatgctatga gctgggtgag gcaagcccct 1680
ggaaagtgcc tggagtgggt gtccgctatc tccggcagcg gcggaagcac ctactacgct 1740
gactccgtca agggcaggtt caccatcagc cgggacaaca gcaagaacac cctgtacctg 1800
cagatgaata gcctcagggc tgaagacacc gctgtgtact actgcgccag gtactatggc 1860
ggctactact ccgcctggat ggactactgg ggacagggca cactggtgac cgtgtccagc 1920
ggcggaggcg gctccggagg cggtggctcc ggaggaggcg gaagcggagg aggaggctcc 1980
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 2040
atcacctgca gggccagcca gtccatctcc agcttcttaa actggtacca gcagaagcct 2100
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 2160
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 2220
gaggacttcg ctacctacta ctgccagcag acctatggct acctgcacac cttcggctgc 2280
ggcacaaagc tggagatcaa gagc 2304
<210> 142
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 142
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Asp Tyr Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu His Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Glu Pro
260 265 270
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
275 280 285
Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
290 295 300
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
305 310 315 320
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
325 330 335
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
340 345 350
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
355 360 365
Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro
370 375 380
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
385 390 395 400
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
405 410 415
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
420 425 430
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
435 440 445
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
450 455 460
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
465 470 475 480
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
485 490 495
Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
500 505 510
Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
515 520 525
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
530 535 540
Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
545 550 555 560
Gly Lys Cys Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser
565 570 575
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
580 585 590
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
595 600 605
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
610 615 620
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
625 630 635 640
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
645 650 655
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
660 665 670
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
675 680 685
Ile Ser Ser Phe Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
690 695 700
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
705 710 715 720
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
725 730 735
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr
740 745 750
Gly Tyr Leu His Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Ser
755 760 765
<210> 143
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 143
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcctcg gagcccaaat cttctgacaa aactcacaca 840
tgcccaccgt gcccagcacc tgaagccgcg ggtgcaccgt cagtcttcct cttcccccca 900
aaacccaagg acaccctcat gatctcccgg acccctgagg tcacatgcgt ggtggtggac 960
gtgagccacg aagaccctga ggtcaagttc aactggtacg tggacggcgt ggaggtgcat 1020
aatgccaaga caaagccgcg ggaggagcag tacaacagca cgtaccgtgt ggtcagcgtc 1080
ctcaccgtcc tgcaccagga ctggctgaat ggcaaggaat acaagtgcgc ggtctccaac 1140
aaagccctcc cagcccccat cgagaaaacc atctccaaag ccaaagggca gccccgagaa 1200
ccacaggtgt acaccctgcc cccatcccgg gatgagctga ccaagaacca ggtcagcctg 1260
acctgcctgg tcaaaggctt ctatccaagc gacatcgccg tggagtggga gagcaatggg 1320
cagccggaga acaactacaa gaccacgcct cccgtgctgg actccgacgg ctccttcttc 1380
ctctacagca agctcaccgt ggacaagagc aggtggcagc aggggaacgt cttctcatgc 1440
tccgtgatgc atgaggctct gcacaaccac tacacgcaga agagcctctc cctgtctccg 1500
ggttccggag gtggcggttc gggaggtggc gggtcaggag gtgggggatc cccttcagaa 1560
gtgcagctgc tggagtccgg aggaggactg gtgcagcctg gcggaagcct gaggctgagc 1620
tgcgctgcct ccggcttcga cttcgagagc tatgctatga gctgggtgag gcaagcccct 1680
ggaaagtgcc tggagtgggt gtccgctatc tccggcagcg gcggaagcac ctactacgct 1740
gactccgtca agggcaggtt caccatcagc cgggacaaca gcaagaacac cctgtacctg 1800
cagatgaata gcctcagggc tgaagacacc gctgtgtact actgcgccag gtactatggc 1860
ggctactact ccgcctggat ggactactgg ggacagggca cactggtgac cgtgtccagc 1920
ggcggaggcg gctccggagg cggtggctcc ggaggaggcg gaagcggagg aggaggctcc 1980
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 2040
atcacctgca gggccagcca gtccatcagg agcgccctga actggtacca gcagaagcct 2100
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 2160
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 2220
gaggacttcg ctacctacta ctgccagcag acctatggct acctgcacac cttcggctgc 2280
ggcacaaagc tggagatcaa gcgc 2304
<210> 144
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 144
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser Tyr Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Glu Pro
260 265 270
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
275 280 285
Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
290 295 300
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
305 310 315 320
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
325 330 335
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
340 345 350
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
355 360 365
Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro
370 375 380
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
385 390 395 400
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
405 410 415
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
420 425 430
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
435 440 445
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
450 455 460
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
465 470 475 480
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
485 490 495
Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
500 505 510
Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
515 520 525
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
530 535 540
Gly Phe Asp Phe Glu Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
545 550 555 560
Gly Lys Cys Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser
565 570 575
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
580 585 590
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
595 600 605
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
610 615 620
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
625 630 635 640
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
645 650 655
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
660 665 670
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
675 680 685
Ile Arg Ser Ala Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
690 695 700
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
705 710 715 720
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
725 730 735
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr
740 745 750
Gly Tyr Leu His Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 145
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 145
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcgagt gagcccaaat cttctgacaa aactcacaca 840
tgcccaccgt gcccagcacc tgaagccgcg ggtgcaccgt cagtcttcct cttcccccca 900
aaacccaagg acaccctcat gatctcccgg acccctgagg tcacatgcgt ggtggtggac 960
gtgagccacg aagaccctga ggtcaagttc aactggtacg tggacggcgt ggaggtgcat 1020
aatgccaaga caaagccgcg ggaggagcag tacaacagca cgtaccgtgt ggtcagcgtc 1080
ctcaccgtcc tgcaccagga ctggctgaat ggcaaggaat acaagtgcgc ggtctccaac 1140
aaagccctcc cagcccccat cgagaaaacc atctccaaag ccaaagggca gccccgagaa 1200
ccacaggtgt acaccctgcc cccatcccgg gatgagctga ccaagaacca ggtcagcctg 1260
acctgcctgg tcaaaggctt ctatccaagc gacatcgccg tggagtggga gagcaatggg 1320
cagccggaga acaactacaa gaccacgcct cccgtgctgg actccgacgg ctccttcttc 1380
ctctacagca agctcaccgt ggacaagagc aggtggcagc aggggaacgt cttctcatgc 1440
tccgtgatgc atgaggctct gcacaaccac tacacgcaga agagcctctc cctgtctccg 1500
ggttccggag gtggcggttc gggaggtggc gggtcaggag gtgggggatc cccttcagaa 1560
gtgcagctgc tggagtccgg aggaggactg gtgcagcctg gcggaagcct gaggctgagc 1620
tgcgctgcct ccggcttcga cttcgagagc tatgctatga gctgggtgag gcaagcccct 1680
ggaaagtgcc tggagtgggt gtccgctatc tccggcagcg gcggaagcac ctactacgct 1740
gactccgtca agggcaggtt caccatcagc cgggacaaca gcaagaacac cctgtacctg 1800
cagatgaata gcctcagggc tgaagacacc gctgtgtact actgcgccag gtactatggc 1860
ggctactact ccgcctggat ggactactgg ggacagggca cactggtgac cgtgtccagc 1920
ggcggaggcg gctccggagg cggtggctcc ggaggaggcg gaagcggagg aggaggctcc 1980
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 2040
atcacctgca gggccagcca gtccatcagg agcgccctga actggtacca gcagaagcct 2100
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 2160
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 2220
gaggacttcg ctacctacta ctgccagcag acctatggct acctgcacac cttcggctgc 2280
ggcacaaagc tggagatcaa gcgc 2304
<210> 146
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 146
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser His Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Glu Pro
260 265 270
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
275 280 285
Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
290 295 300
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
305 310 315 320
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
325 330 335
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
340 345 350
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
355 360 365
Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro
370 375 380
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
385 390 395 400
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
405 410 415
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
420 425 430
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
435 440 445
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
450 455 460
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
465 470 475 480
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
485 490 495
Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
500 505 510
Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
515 520 525
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
530 535 540
Gly Phe Asp Phe Glu Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
545 550 555 560
Gly Lys Cys Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser
565 570 575
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
580 585 590
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
595 600 605
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
610 615 620
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
625 630 635 640
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
645 650 655
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
660 665 670
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
675 680 685
Ile Arg Ser Ala Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
690 695 700
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
705 710 715 720
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
725 730 735
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr
740 745 750
Gly Tyr Leu His Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 147
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 147
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcgagt gagcccaaat cttctgacaa aactcacaca 840
tgcccaccgt gcccagcacc tgaagccgcg ggtgcaccgt cagtcttcct cttcccccca 900
aaacccaagg acaccctcat gatctcccgg acccctgagg tcacatgcgt ggtggtggac 960
gtgagccacg aagaccctga ggtcaagttc aactggtacg tggacggcgt ggaggtgcat 1020
aatgccaaga caaagccgcg ggaggagcag tacaacagca cgtaccgtgt ggtcagcgtc 1080
ctcaccgtcc tgcaccagga ctggctgaat ggcaaggaat acaagtgcgc ggtctccaac 1140
aaagccctcc cagcccccat cgagaaaacc atctccaaag ccaaagggca gccccgagaa 1200
ccacaggtgt acaccctgcc cccatcccgg gatgagctga ccaagaacca ggtcagcctg 1260
acctgcctgg tcaaaggctt ctatccaagc gacatcgccg tggagtggga gagcaatggg 1320
cagccggaga acaactacaa gaccacgcct cccgtgctgg actccgacgg ctccttcttc 1380
ctctacagca agctcaccgt ggacaagagc aggtggcagc aggggaacgt cttctcatgc 1440
tccgtgatgc atgaggctct gcacaaccac tacacgcaga agagcctctc cctgtctccg 1500
ggttccggag gtggcggttc gggaggtggc gggtcaggag gtgggggatc cccttcagaa 1560
gtgcagctgc tggagtccgg aggaggactg gtgcagcctg gcggaagcct gaggctgagc 1620
tgcgctgcct ccggcttcga cttcgacagc tatgctatga gctgggtgag gcaagcccct 1680
ggaaagtgcc tggagtgggt gtccgctatc tccggcaggg gcggaagcac ctactacgct 1740
gactccgtca agggcaggtt caccatcagc cgggacaaca gcaagaacac cctgtacctg 1800
cagatgaata gcctcagggc tgaagacacc gctgtgtact actgcgccag gtactatggc 1860
ggctactact ccgcctggat ggactactgg ggacagggca cactggtgac cgtgtccagc 1920
ggcggaggcg gctccggagg cggtggctcc ggaggaggcg gaagcggagg aggaggctcc 1980
gatattcaga tgacacagtc ccctagctcc ctgtccgcca gcgtgggaga tcgggtgacc 2040
atcacctgca gggccagcca gtccatcagg agcgccctga actggtacca gcagaagcct 2100
ggaaaggctc ccaagctgct gatctacgcc gcttccagcc tccagagcgg cgtgcctagc 2160
aggttctccg gctccggaag cggaacagac ttcaccctga ccatcagctc cctgcagccc 2220
gaggacttcg ctacctacta ctgccagcag acctatggct acctgcacac cttcggctgc 2280
ggcacaaagc tggagatcaa gcgc 2304
<210> 148
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 148
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser His Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Glu Pro
260 265 270
Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu
275 280 285
Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
290 295 300
Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
305 310 315 320
Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly
325 330 335
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn
340 345 350
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
355 360 365
Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro
370 375 380
Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
385 390 395 400
Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn
405 410 415
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
420 425 430
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
435 440 445
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys
450 455 460
Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys
465 470 475 480
Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
485 490 495
Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
500 505 510
Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
515 520 525
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
530 535 540
Gly Phe Asp Phe Asp Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
545 550 555 560
Gly Lys Cys Leu Glu Trp Val Ser Ala Ile Ser Gly Arg Gly Gly Ser
565 570 575
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
580 585 590
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
595 600 605
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
610 615 620
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
625 630 635 640
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
645 650 655
Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser
660 665 670
Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser
675 680 685
Ile Arg Ser Ala Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro
690 695 700
Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser
705 710 715 720
Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser
725 730 735
Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr
740 745 750
Gly Tyr Leu His Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 149
<211> 2307
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 149
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggcggcg gcggcagcgg cggcggcggc agcggcggcg gaggctccgg cggcggcggc 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcctcg agtgagccca aatcttctga caaaactcac 840
acatgcccac cgtgcccagc acctgaagcc gcgggtgcac cgtcagtctt cctcttcccc 900
ccaaaaccca aggacaccct catgatctcc cggacccctg aggtcacatg cgtggtggtg 960
gacgtgagcc acgaagaccc tgaggtcaag ttcaactggt acgtggacgg cgtggaggtg 1020
cataatgcca agacaaagcc gcgggaggag cagtacaaca gcacgtaccg tgtggtcagc 1080
gtcctcaccg tcctgcacca ggactggctg aatggcaagg aatacaagtg cgcggtctcc 1140
aacaaagccc tcccagcccc catcgagaaa accatctcca aagccaaagg gcagccccga 1200
gaaccacagg tgtacaccct gcccccatcc cgggatgagc tgaccaagaa ccaggtcagc 1260
ctgacctgcc tggtcaaagg cttctatcca agcgacatcg ccgtggagtg ggagagcaat 1320
gggcagccgg agaacaacta caagaccacg cctcccgtgc tggactccga cggctccttc 1380
ttcctctaca gcaagctcac cgtggacaag agcaggtggc agcaggggaa cgtcttctca 1440
tgctccgtga tgcatgaggc tctgcacaac cactacacgc agaagagcct ctccctgtct 1500
ccgggttccg gaggtggcgg ttcgggaggt ggcgggtcag gaggtggggg atccccttca 1560
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 1620
tcctgtgcag ccagcggatt cacctttagc agctatgcca tgagctgggt ccgccaggct 1680
ccagggaagg ggctggagtg ggtctcagct attagtggta gtggtggtag cacatactat 1740
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 1800
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctactac 1860
ggtggttact actctgcttg gatggactat tggggccagg gaaccctggt caccgtctcc 1920
tcaggcggtg gaggcagcgg tgggggtggg tctggaggcg gtggcagtgg cggcggaggc 1980
tctgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 2040
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 2100
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 2160
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 2220
cctgaagatt ttgcaactta ttactgtcaa cagacttacg gttacctgca cacttttggc 2280
caggggacca agctggagat caaatca 2307
<210> 150
<211> 769
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 150
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser Tyr Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ser Glu
260 265 270
Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
275 280 285
Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
290 295 300
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
305 310 315 320
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
325 330 335
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
340 345 350
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
355 360 365
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu
370 375 380
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
385 390 395 400
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
405 410 415
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
420 425 430
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
435 440 445
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
450 455 460
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
465 470 475 480
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
485 490 495
Leu Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
500 505 510
Ser Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly
515 520 525
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala
530 535 540
Ser Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala
545 550 555 560
Pro Gly Lys Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly
565 570 575
Ser Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
580 585 590
Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala
595 600 605
Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr
610 615 620
Ser Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
625 630 635 640
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
645 650 655
Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu
660 665 670
Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln
675 680 685
Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala
690 695 700
Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro
705 710 715 720
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
725 730 735
Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr
740 745 750
Tyr Gly Tyr Leu His Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
755 760 765
Ser
<210> 151
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 151
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggcggcg gcggcagcgg cggcggcggc agcggcggcg gaggctccgg cggcggcggc 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcctcg agtgagccca aatcttctga caaaactcac 840
acatgcccac cgtgcccagc acctgaagcc gcgggtgcac cgtcagtctt cctcttcccc 900
ccaaaaccca aggacaccct catgatctcc cggacccctg aggtcacatg cgtggtggtg 960
gacgtgagcc acgaagaccc tgaggtcaag ttcaactggt acgtggacgg cgtggaggtg 1020
cataatgcca agacaaagcc gcgggaggag cagtacaaca gcacgtaccg tgtggtcagc 1080
gtcctcaccg tcctgcacca ggactggctg aatggcaagg aatacaagtg cgcggtctcc 1140
aacaaagccc tcccagcccc catcgagaaa accatctcca aagccaaagg gcagccccga 1200
gaaccacagg tgtacaccct gcccccatcc cgggatgagc tgaccaagaa ccaggtcagc 1260
ctgacctgcc tggtcaaagg cttctatcca agcgacatcg ccgtggagtg ggagagcaat 1320
gggcagccgg agaacaacta caagaccacg cctcccgtgc tggactccga cggctccttc 1380
ttcctctaca gcaagctcac cgtggacaag agcaggtggc agcaggggaa cgtcttctca 1440
tgctccgtga tgcatgaggc tctgcacaac cactacacgc agaagagcct ctccctgtct 1500
ccgggttccg gaggtggcgg ttcgggaggt ggcgggtcag gaggtggggg atccccttca 1560
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 1620
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 1680
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 1740
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctgcaacct 1800
gaagattttg caacttatta ctgtcaacag acttacggtt acctgcacac ttttggccag 1860
gggaccaagc tggagatcaa aggcggtgga ggcagcggtg ggggtgggtc tggaggcggt 1920
ggcagtggcg gcggaggctc tgaggtgcag ctgttggaga gcgggggagg cttggtacag 1980
cctggggggt ccctgcgcct ctcctgtgca gccagcggat tcacctttag cagctatgcc 2040
atgagctggg tccgccaggc tccagggaag gggctggagt gggtctcagc tattagtggt 2100
agtggtggta gcacatacta tgcagactcc gtgaagggcc ggttcaccat ctcccgtgac 2160
aattccaaga acacgctgta tctgcaaatg aacagcctgc gtgccgagga cacggctgta 2220
tattattgtg cgcgctacta cggtggttac tactctgctt ggatggacta ttggggccag 2280
ggaaccctgg tcaccgtctc ctca 2304
<210> 152
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 152
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser Tyr Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ser Glu
260 265 270
Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
275 280 285
Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
290 295 300
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
305 310 315 320
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
325 330 335
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
340 345 350
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
355 360 365
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu
370 375 380
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
385 390 395 400
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
405 410 415
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
420 425 430
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
435 440 445
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
450 455 460
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
465 470 475 480
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
485 490 495
Leu Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
500 505 510
Ser Gly Gly Gly Gly Ser Pro Ser Asp Ile Gln Met Thr Gln Ser Pro
515 520 525
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
530 535 540
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
545 550 555 560
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
565 570 575
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
580 585 590
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
595 600 605
Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly Gln Gly Thr Lys Leu
610 615 620
Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
625 630 635 640
Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
645 650 655
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
660 665 670
Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
675 680 685
Gly Lys Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser
690 695 700
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
705 710 715 720
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
725 730 735
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
740 745 750
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
755 760 765
<210> 153
<211> 2307
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 153
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggcggcg gcggcagcgg cggcggcggc agcggcggcg gaggctccgg cggcggcggc 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcctcg agtgagccca aatcttctga caaaactcac 840
acatgcccac cgtgcccagc acctgaagcc gcgggtgcac cgtcagtctt cctcttcccc 900
ccaaaaccca aggacaccct catgatctcc cggacccctg aggtcacatg cgtggtggtg 960
gacgtgagcc acgaagaccc tgaggtcaag ttcaactggt acgtggacgg cgtggaggtg 1020
cataatgcca agacaaagcc gcgggaggag cagtacaaca gcacgtaccg tgtggtcagc 1080
gtcctcaccg tcctgcacca ggactggctg aatggcaagg aatacaagtg cgcggtctcc 1140
aacaaagccc tcccagcccc catcgagaaa accatctcca aagccaaagg gcagccccga 1200
gaaccacagg tgtacaccct gcccccatcc cgggatgagc tgaccaagaa ccaggtcagc 1260
ctgacctgcc tggtcaaagg cttctatcca agcgacatcg ccgtggagtg ggagagcaat 1320
gggcagccgg agaacaacta caagaccacg cctcccgtgc tggactccga cggctccttc 1380
ttcctctaca gcaagctcac cgtggacaag agcaggtggc agcaggggaa cgtcttctca 1440
tgctccgtga tgcatgaggc tctgcacaac cactacacgc agaagagcct ctccctgtct 1500
ccgggttccg gaggtggcgg ttcgggaggt ggcgggtcag gaggtggggg atccccttca 1560
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 1620
tcctgtgcag ccagcggatt cacctttagc agctatgcca tgagctgggt ccgccaggct 1680
ccagggaagt gcctggagtg ggtctcagct attagtggta gtggtggtag cacatactat 1740
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 1800
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctactac 1860
ggtggttact actctgcttg gatggactat tggggccagg gaaccctggt caccgtctcc 1920
tcaggcggtg gaggcagcgg tgggggtggg tctggaggcg gtggcagtgg cggcggaggc 1980
tctgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 2040
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 2100
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 2160
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 2220
cctgaagatt ttgcaactta ttactgtcaa cagacttacg gttacctgca cacttttggc 2280
tgcgggacca agctggagat caaatca 2307
<210> 154
<211> 769
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 154
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser Tyr Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ser Glu
260 265 270
Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
275 280 285
Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
290 295 300
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
305 310 315 320
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
325 330 335
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
340 345 350
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
355 360 365
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu
370 375 380
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
385 390 395 400
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
405 410 415
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
420 425 430
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
435 440 445
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
450 455 460
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
465 470 475 480
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
485 490 495
Leu Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
500 505 510
Ser Gly Gly Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly
515 520 525
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala
530 535 540
Ser Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala
545 550 555 560
Pro Gly Lys Cys Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly
565 570 575
Ser Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
580 585 590
Asp Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala
595 600 605
Glu Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr
610 615 620
Ser Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser
625 630 635 640
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
645 650 655
Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu
660 665 670
Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln
675 680 685
Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala
690 695 700
Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro
705 710 715 720
Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile
725 730 735
Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr
740 745 750
Tyr Gly Tyr Leu His Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys
755 760 765
Ser
<210> 155
<211> 2304
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 155
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct tacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccgtgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggcggcg gcggcagcgg cggcggcggc agcggcggcg gaggctccgg cggcggcggc 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctgcaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaatcctcg agtgagccca aatcttctga caaaactcac 840
acatgcccac cgtgcccagc acctgaagcc gcgggtgcac cgtcagtctt cctcttcccc 900
ccaaaaccca aggacaccct catgatctcc cggacccctg aggtcacatg cgtggtggtg 960
gacgtgagcc acgaagaccc tgaggtcaag ttcaactggt acgtggacgg cgtggaggtg 1020
cataatgcca agacaaagcc gcgggaggag cagtacaaca gcacgtaccg tgtggtcagc 1080
gtcctcaccg tcctgcacca ggactggctg aatggcaagg aatacaagtg cgcggtctcc 1140
aacaaagccc tcccagcccc catcgagaaa accatctcca aagccaaagg gcagccccga 1200
gaaccacagg tgtacaccct gcccccatcc cgggatgagc tgaccaagaa ccaggtcagc 1260
ctgacctgcc tggtcaaagg cttctatcca agcgacatcg ccgtggagtg ggagagcaat 1320
gggcagccgg agaacaacta caagaccacg cctcccgtgc tggactccga cggctccttc 1380
ttcctctaca gcaagctcac cgtggacaag agcaggtggc agcaggggaa cgtcttctca 1440
tgctccgtga tgcatgaggc tctgcacaac cactacacgc agaagagcct ctccctgtct 1500
ccgggttccg gaggtggcgg ttcgggaggt ggcgggtcag gaggtggggg atccccttca 1560
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 1620
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 1680
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 1740
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctgcaacct 1800
gaagattttg caacttatta ctgtcaacag acttacggtt acctgcacac ttttggctgc 1860
gggaccaagc tggagatcaa aggcggtgga ggcagcggtg ggggtgggtc tggaggcggt 1920
ggcagtggcg gcggaggctc tgaggtgcag ctgttggaga gcgggggagg cttggtacag 1980
cctggggggt ccctgcgcct ctcctgtgca gccagcggat tcacctttag cagctatgcc 2040
atgagctggg tccgccaggc tccagggaag tgcctggagt gggtctcagc tattagtggt 2100
agtggtggta gcacatacta tgcagactcc gtgaagggcc ggttcaccat ctcccgtgac 2160
aattccaaga acacgctgta tctgcaaatg aacagcctgc gtgccgagga cacggctgta 2220
tattattgtg cgcgctacta cggtggttac tactctgctt ggatggacta ttggggccag 2280
ggaaccctgg tcaccgtctc ctca 2304
<210> 156
<211> 768
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 156
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser Tyr Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Ser Ser Ser Glu
260 265 270
Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro
275 280 285
Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
290 295 300
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
305 310 315 320
Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp
325 330 335
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr
340 345 350
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
355 360 365
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu
370 375 380
Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
385 390 395 400
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys
405 410 415
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
420 425 430
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
435 440 445
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
450 455 460
Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser
465 470 475 480
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
485 490 495
Leu Ser Leu Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
500 505 510
Ser Gly Gly Gly Gly Ser Pro Ser Asp Ile Gln Met Thr Gln Ser Pro
515 520 525
Ser Ser Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
530 535 540
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
545 550 555 560
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
565 570 575
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
580 585 590
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
595 600 605
Gln Gln Thr Tyr Gly Tyr Leu His Thr Phe Gly Cys Gly Thr Lys Leu
610 615 620
Glu Ile Lys Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
625 630 635 640
Gly Ser Gly Gly Gly Gly Ser Glu Val Gln Leu Leu Glu Ser Gly Gly
645 650 655
Gly Leu Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser
660 665 670
Gly Phe Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro
675 680 685
Gly Lys Cys Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser
690 695 700
Thr Tyr Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
705 710 715 720
Asn Ser Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu
725 730 735
Asp Thr Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser
740 745 750
Ala Trp Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
755 760 765
<210> 157
<211> 699
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 157
tcgagtgagc ccaaatcttc tgacaaaact cacacatgcc caccgtgccc agcacctgaa 60
gccgcgggtg caccgtcagt cttcctcttc cccccaaaac ccaaggacac cctcatgatc 120
tcccggaccc ctgaggtcac atgcgtggtg gtggacgtga gccacgaaga ccctgaggtc 180
aagttcaact ggtacgtgga cggcgtggag gtgcataatg ccaagacaaa gccgcgggag 240
gagcagtaca acagcacgta ccgtgtggtc agcgtcctca ccgtcctgca ccaggactgg 300
ctgaatggca aggaatacaa gtgcgcggtc tccaacaaag ccctcccagc ccccatcgag 360
aaaaccatct ccaaagccaa agggcagccc cgagaaccac aggtgtacac cctgccccca 420
tcccgggatg agctgaccaa gaaccaggtc agcctgacct gcctggtcaa aggcttctat 480
ccaagcgaca tcgccgtgga gtgggagagc aatgggcagc cggagaacaa ctacaagacc 540
acgcctcccg tgctggactc cgacggctcc ttcttcctct acagcaagct caccgtggac 600
aagagcaggt ggcagcaggg gaacgtcttc tcatgctccg tgatgcatga ggctctgcac 660
aaccactaca cgcagaagag cctctccctg tctccgggt 699
<210> 158
<211> 233
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 158
Ser Ser Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys
1 5 10 15
Pro Ala Pro Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro
20 25 30
Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys
35 40 45
Val Val Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp
50 55 60
Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu
65 70 75 80
Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu
85 90 95
His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn
100 105 110
Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly
115 120 125
Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu
130 135 140
Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr
145 150 155 160
Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn
165 170 175
Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe
180 185 190
Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn
195 200 205
Val Phe Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr
210 215 220
Gln Lys Ser Leu Ser Leu Ser Pro Gly
225 230
<210> 159
<211> 693
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 159
gagcccaaat cttctgacaa aactcacaca tgcccaccgt gcccagcacc tgaagccgcg 60
ggtgcaccgt cagtcttcct cttcccccca aaacccaagg acaccctcat gatctcccgg 120
acccctgagg tcacatgcgt ggtggtggac gtgagccacg aagaccctga ggtcaagttc 180
aactggtacg tggacggcgt ggaggtgcat aatgccaaga caaagccgcg ggaggagcag 240
tacaacagca cgtaccgtgt ggtcagcgtc ctcaccgtcc tgcaccagga ctggctgaat 300
ggcaaggaat acaagtgcgc ggtctccaac aaagccctcc cagcccccat cgagaaaacc 360
atctccaaag ccaaagggca gccccgagaa ccacaggtgt acaccctgcc cccatcccgg 420
gatgagctga ccaagaacca ggtcagcctg acctgcctgg tcaaaggctt ctatccaagc 480
gacatcgccg tggagtggga gagcaatggg cagccggaga acaactacaa gaccacgcct 540
cccgtgctgg actccgacgg ctccttcttc ctctacagca agctcaccgt ggacaagagc 600
aggtggcagc aggggaacgt cttctcatgc tccgtgatgc atgaggctct gcacaaccac 660
tacacgcaga agagcctctc cctgtctccg ggt 693
<210> 160
<211> 231
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - фрагмент антитела
<400> 160
Glu Pro Lys Ser Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala
1 5 10 15
Pro Glu Ala Ala Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro
20 25 30
Lys Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val
35 40 45
Val Asp Val Ser His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val
50 55 60
Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln
65 70 75 80
Tyr Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln
85 90 95
Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala
100 105 110
Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro
115 120 125
Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr
130 135 140
Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser
145 150 155 160
Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr
165 170 175
Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr
180 185 190
Ser Lys Leu Thr Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe
195 200 205
Ser Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys
210 215 220
Ser Leu Ser Leu Ser Pro Gly
225 230
<210> 161
<211> 768
<212> ДНК
<213> Homo sapiens
<400> 161
atgggaaaca gctgttacaa catagtagcc actctgttgc tggtcctcaa ctttgagagg 60
acaagatcat tgcaggatcc ttgtagtaac tgcccagctg gtacattctg tgataataac 120
aggaatcaga tttgcagtcc ctgtcctcca aatagtttct ccagcgcagg tggacaaagg 180
acctgtgaca tatgcaggca gtgtaaaggt gttttcagga ccaggaagga gtgttcctcc 240
accagcaatg cagagtgtga ctgcactcca gggtttcact gcctgggggc aggatgcagc 300
atgtgtgaac aggattgtaa acaaggtcaa gaactgacaa aaaaaggttg taaagactgt 360
tgctttggga catttaacga tcagaaacgt ggcatctgtc gaccctggac aaactgttct 420
ttggatggaa agtctgtgct tgtgaatggg acgaaggaga gggacgtggt ctgtggacca 480
tctccagccg acctctctcc gggagcatcc tctgtgaccc cgcctgcccc tgcgagagag 540
ccaggacact ctccgcagat catctccttc tttcttgcgc tgacgtcgac tgcgttgctc 600
ttcctgctgt tcttcctcac gctccgtttc tctgttgtta aacggggcag aaagaaactc 660
ctgtatatat tcaaacaacc atttatgaga ccagtacaaa ctactcaaga ggaagatggc 720
tgtagctgcc gatttccaga agaagaagaa ggaggatgtg aactgtga 768
<210> 162
<211> 255
<212> ПРТ
<213> Homo sapiens
<400> 162
Met Gly Asn Ser Cys Tyr Asn Ile Val Ala Thr Leu Leu Leu Val Leu
1 5 10 15
Asn Phe Glu Arg Thr Arg Ser Leu Gln Asp Pro Cys Ser Asn Cys Pro
20 25 30
Ala Gly Thr Phe Cys Asp Asn Asn Arg Asn Gln Ile Cys Ser Pro Cys
35 40 45
Pro Pro Asn Ser Phe Ser Ser Ala Gly Gly Gln Arg Thr Cys Asp Ile
50 55 60
Cys Arg Gln Cys Lys Gly Val Phe Arg Thr Arg Lys Glu Cys Ser Ser
65 70 75 80
Thr Ser Asn Ala Glu Cys Asp Cys Thr Pro Gly Phe His Cys Leu Gly
85 90 95
Ala Gly Cys Ser Met Cys Glu Gln Asp Cys Lys Gln Gly Gln Glu Leu
100 105 110
Thr Lys Lys Gly Cys Lys Asp Cys Cys Phe Gly Thr Phe Asn Asp Gln
115 120 125
Lys Arg Gly Ile Cys Arg Pro Trp Thr Asn Cys Ser Leu Asp Gly Lys
130 135 140
Ser Val Leu Val Asn Gly Thr Lys Glu Arg Asp Val Val Cys Gly Pro
145 150 155 160
Ser Pro Ala Asp Leu Ser Pro Gly Ala Ser Ser Val Thr Pro Pro Ala
165 170 175
Pro Ala Arg Glu Pro Gly His Ser Pro Gln Ile Ile Ser Phe Phe Leu
180 185 190
Ala Leu Thr Ser Thr Ala Leu Leu Phe Leu Leu Phe Phe Leu Thr Leu
195 200 205
Arg Phe Ser Val Val Lys Arg Gly Arg Lys Lys Leu Leu Tyr Ile Phe
210 215 220
Lys Gln Pro Phe Met Arg Pro Val Gln Thr Thr Gln Glu Glu Asp Gly
225 230 235 240
Cys Ser Cys Arg Phe Pro Glu Glu Glu Glu Gly Gly Cys Glu Leu
245 250 255
<210> 163
<211> 1263
<212> ДНК
<213> Homo sapiens
<400> 163
atgcctgggg ggtgctcccg gggccccgcc gccggggacg ggcgtctgcg gctggcgcga 60
ctagcgctgg tactcctggg ctgggtctcc tcgtcttctc ccacctcctc ggcatcctcc 120
ttctcctcct cggcgccgtt cctggcttcc gccgtgtccg cccagccccc gctgccggac 180
cagtgccccg cgctgtgcga gtgctccgag gcagcgcgca cagtcaagtg cgttaaccgc 240
aatctgaccg aggtgcccac ggacctgccc gcctacgtgc gcaacctctt ccttaccggc 300
aaccagctgg ccgtgctccc tgccggcgcc ttcgcccgcc ggccgccgct ggcggagctg 360
gccgcgctca acctcagcgg cagccgcctg gacgaggtgc gcgcgggcgc cttcgagcat 420
ctgcccagcc tgcgccagct cgacctcagc cacaacccac tggccgacct cagtcccttc 480
gctttctcgg gcagcaatgc cagcgtctcg gcccccagtc cccttgtgga actgatcctg 540
aaccacatcg tgccccctga agatgagcgg cagaaccgga gcttcgaggg catggtggtg 600
gcggccctgc tggcgggccg tgcactgcag gggctccgcc gcttggagct ggccagcaac 660
cacttccttt acctgccgcg ggatgtgctg gcccaactgc ccagcctcag gcacctggac 720
ttaagtaata attcgctggt gagcctgacc tacgtgtcct tccgcaacct gacacatcta 780
gaaagcctcc acctggagga caatgccctc aaggtccttc acaatggcac cctggctgag 840
ttgcaaggtc taccccacat tagggttttc ctggacaaca atccctgggt ctgcgactgc 900
cacatggcag acatggtgac ctggctcaag gaaacagagg tagtgcaggg caaagaccgg 960
ctcacctgtg catatccgga aaaaatgagg aatcgggtcc tcttggaact caacagtgct 1020
gacctggact gtgacccgat tcttccccca tccctgcaaa cctcttatgt cttcctgggt 1080
attgttttag ccctgatagg cgctattttc ctcctggttt tgtatttgaa ccgcaagggg 1140
ataaaaaagt ggatgcataa catcagagat gcctgcaggg atcacatgga agggtatcat 1200
tacagatatg aaatcaatgc ggaccccaga ttaacgaacc tcagttctaa ctcggatgtc 1260
tga 1263
<210> 164
<211> 420
<212> ПРТ
<213> Homo sapiens
<400> 164
Met Pro Gly Gly Cys Ser Arg Gly Pro Ala Ala Gly Asp Gly Arg Leu
1 5 10 15
Arg Leu Ala Arg Leu Ala Leu Val Leu Leu Gly Trp Val Ser Ser Ser
20 25 30
Ser Pro Thr Ser Ser Ala Ser Ser Phe Ser Ser Ser Ala Pro Phe Leu
35 40 45
Ala Ser Ala Val Ser Ala Gln Pro Pro Leu Pro Asp Gln Cys Pro Ala
50 55 60
Leu Cys Glu Cys Ser Glu Ala Ala Arg Thr Val Lys Cys Val Asn Arg
65 70 75 80
Asn Leu Thr Glu Val Pro Thr Asp Leu Pro Ala Tyr Val Arg Asn Leu
85 90 95
Phe Leu Thr Gly Asn Gln Leu Ala Val Leu Pro Ala Gly Ala Phe Ala
100 105 110
Arg Arg Pro Pro Leu Ala Glu Leu Ala Ala Leu Asn Leu Ser Gly Ser
115 120 125
Arg Leu Asp Glu Val Arg Ala Gly Ala Phe Glu His Leu Pro Ser Leu
130 135 140
Arg Gln Leu Asp Leu Ser His Asn Pro Leu Ala Asp Leu Ser Pro Phe
145 150 155 160
Ala Phe Ser Gly Ser Asn Ala Ser Val Ser Ala Pro Ser Pro Leu Val
165 170 175
Glu Leu Ile Leu Asn His Ile Val Pro Pro Glu Asp Glu Arg Gln Asn
180 185 190
Arg Ser Phe Glu Gly Met Val Val Ala Ala Leu Leu Ala Gly Arg Ala
195 200 205
Leu Gln Gly Leu Arg Arg Leu Glu Leu Ala Ser Asn His Phe Leu Tyr
210 215 220
Leu Pro Arg Asp Val Leu Ala Gln Leu Pro Ser Leu Arg His Leu Asp
225 230 235 240
Leu Ser Asn Asn Ser Leu Val Ser Leu Thr Tyr Val Ser Phe Arg Asn
245 250 255
Leu Thr His Leu Glu Ser Leu His Leu Glu Asp Asn Ala Leu Lys Val
260 265 270
Leu His Asn Gly Thr Leu Ala Glu Leu Gln Gly Leu Pro His Ile Arg
275 280 285
Val Phe Leu Asp Asn Asn Pro Trp Val Cys Asp Cys His Met Ala Asp
290 295 300
Met Val Thr Trp Leu Lys Glu Thr Glu Val Val Gln Gly Lys Asp Arg
305 310 315 320
Leu Thr Cys Ala Tyr Pro Glu Lys Met Arg Asn Arg Val Leu Leu Glu
325 330 335
Leu Asn Ser Ala Asp Leu Asp Cys Asp Pro Ile Leu Pro Pro Ser Leu
340 345 350
Gln Thr Ser Tyr Val Phe Leu Gly Ile Val Leu Ala Leu Ile Gly Ala
355 360 365
Ile Phe Leu Leu Val Leu Tyr Leu Asn Arg Lys Gly Ile Lys Lys Trp
370 375 380
Met His Asn Ile Arg Asp Ala Cys Arg Asp His Met Glu Gly Tyr His
385 390 395 400
Tyr Arg Tyr Glu Ile Asn Ala Asp Pro Arg Leu Thr Asn Leu Ser Ser
405 410 415
Asn Ser Asp Val
420
<210> 165
<211> 558
<212> ДНК
<213> Homo sapiens
<400> 165
atgggaaaca gctgttacaa catagtagcc actctgttgc tggtcctcaa ctttgagagg 60
acaagatcat tgcaggatcc ttgtagtaac tgcccagctg gtacattctg tgataataac 120
aggaatcaga tttgcagtcc ctgtcctcca aatagtttct ccagcgcagg tggacaaagg 180
acctgtgaca tatgcaggca gtgtaaaggt gttttcagga ccaggaagga gtgttcctcc 240
accagcaatg cagagtgtga ctgcactcca gggtttcact gcctgggggc aggatgcagc 300
atgtgtgaac aggattgtaa acaaggtcaa gaactgacaa aaaaaggttg taaagactgt 360
tgctttggga catttaacga tcagaaacgt ggcatctgtc gaccctggac aaactgttct 420
ttggatggaa agtctgtgct tgtgaatggg acgaaggaga gggacgtggt ctgtggacca 480
tctccagccg acctctctcc gggagcatcc tctgtgaccc cgcctgcccc tgcgagagag 540
ccaggacact ctccgcag 558
<210> 166
<211> 186
<212> ПРТ
<213> Homo sapiens
<400> 166
Met Gly Asn Ser Cys Tyr Asn Ile Val Ala Thr Leu Leu Leu Val Leu
1 5 10 15
Asn Phe Glu Arg Thr Arg Ser Leu Gln Asp Pro Cys Ser Asn Cys Pro
20 25 30
Ala Gly Thr Phe Cys Asp Asn Asn Arg Asn Gln Ile Cys Ser Pro Cys
35 40 45
Pro Pro Asn Ser Phe Ser Ser Ala Gly Gly Gln Arg Thr Cys Asp Ile
50 55 60
Cys Arg Gln Cys Lys Gly Val Phe Arg Thr Arg Lys Glu Cys Ser Ser
65 70 75 80
Thr Ser Asn Ala Glu Cys Asp Cys Thr Pro Gly Phe His Cys Leu Gly
85 90 95
Ala Gly Cys Ser Met Cys Glu Gln Asp Cys Lys Gln Gly Gln Glu Leu
100 105 110
Thr Lys Lys Gly Cys Lys Asp Cys Cys Phe Gly Thr Phe Asn Asp Gln
115 120 125
Lys Arg Gly Ile Cys Arg Pro Trp Thr Asn Cys Ser Leu Asp Gly Lys
130 135 140
Ser Val Leu Val Asn Gly Thr Lys Glu Arg Asp Val Val Cys Gly Pro
145 150 155 160
Ser Pro Ala Asp Leu Ser Pro Gly Ala Ser Ser Val Thr Pro Pro Ala
165 170 175
Pro Ala Arg Glu Pro Gly His Ser Pro Gln
180 185
<210> 167
<211> 1065
<212> ДНК
<213> Homo sapiens
<400> 167
atgcctgggg ggtgctcccg gggccccgcc gccggggacg ggcgtctgcg gctggcgcga 60
ctagcgctgg tactcctggg ctgggtctcc tcgtcttctc ccacctcctc ggcatcctcc 120
ttctcctcct cggcgccgtt cctggcttcc gccgtgtccg cccagccccc gctgccggac 180
cagtgccccg cgctgtgcga gtgctccgag gcagcgcgca cagtcaagtg cgttaaccgc 240
aatctgaccg aggtgcccac ggacctgccc gcctacgtgc gcaacctctt ccttaccggc 300
aaccagctgg ccgtgctccc tgccggcgcc ttcgcccgcc ggccgccgct ggcggagctg 360
gccgcgctca acctcagcgg cagccgcctg gacgaggtgc gcgcgggcgc cttcgagcat 420
ctgcccagcc tgcgccagct cgacctcagc cacaacccac tggccgacct cagtcccttc 480
gctttctcgg gcagcaatgc cagcgtctcg gcccccagtc cccttgtgga actgatcctg 540
aaccacatcg tgccccctga agatgagcgg cagaaccgga gcttcgaggg catggtggtg 600
gcggccctgc tggcgggccg tgcactgcag gggctccgcc gcttggagct ggccagcaac 660
cacttccttt acctgccgcg ggatgtgctg gcccaactgc ccagcctcag gcacctggac 720
ttaagtaata attcgctggt gagcctgacc tacgtgtcct tccgcaacct gacacatcta 780
gaaagcctcc acctggagga caatgccctc aaggtccttc acaatggcac cctggctgag 840
ttgcaaggtc taccccacat tagggttttc ctggacaaca atccctgggt ctgcgactgc 900
cacatggcag acatggtgac ctggctcaag gaaacagagg tagtgcaggg caaagaccgg 960
ctcacctgtg catatccgga aaaaatgagg aatcgggtcc tcttggaact caacagtgct 1020
gacctggact gtgacccgat tcttccccca tccctgcaaa cctct 1065
<210> 168
<211> 355
<212> ПРТ
<213> Homo sapiens
<400> 168
Met Pro Gly Gly Cys Ser Arg Gly Pro Ala Ala Gly Asp Gly Arg Leu
1 5 10 15
Arg Leu Ala Arg Leu Ala Leu Val Leu Leu Gly Trp Val Ser Ser Ser
20 25 30
Ser Pro Thr Ser Ser Ala Ser Ser Phe Ser Ser Ser Ala Pro Phe Leu
35 40 45
Ala Ser Ala Val Ser Ala Gln Pro Pro Leu Pro Asp Gln Cys Pro Ala
50 55 60
Leu Cys Glu Cys Ser Glu Ala Ala Arg Thr Val Lys Cys Val Asn Arg
65 70 75 80
Asn Leu Thr Glu Val Pro Thr Asp Leu Pro Ala Tyr Val Arg Asn Leu
85 90 95
Phe Leu Thr Gly Asn Gln Leu Ala Val Leu Pro Ala Gly Ala Phe Ala
100 105 110
Arg Arg Pro Pro Leu Ala Glu Leu Ala Ala Leu Asn Leu Ser Gly Ser
115 120 125
Arg Leu Asp Glu Val Arg Ala Gly Ala Phe Glu His Leu Pro Ser Leu
130 135 140
Arg Gln Leu Asp Leu Ser His Asn Pro Leu Ala Asp Leu Ser Pro Phe
145 150 155 160
Ala Phe Ser Gly Ser Asn Ala Ser Val Ser Ala Pro Ser Pro Leu Val
165 170 175
Glu Leu Ile Leu Asn His Ile Val Pro Pro Glu Asp Glu Arg Gln Asn
180 185 190
Arg Ser Phe Glu Gly Met Val Val Ala Ala Leu Leu Ala Gly Arg Ala
195 200 205
Leu Gln Gly Leu Arg Arg Leu Glu Leu Ala Ser Asn His Phe Leu Tyr
210 215 220
Leu Pro Arg Asp Val Leu Ala Gln Leu Pro Ser Leu Arg His Leu Asp
225 230 235 240
Leu Ser Asn Asn Ser Leu Val Ser Leu Thr Tyr Val Ser Phe Arg Asn
245 250 255
Leu Thr His Leu Glu Ser Leu His Leu Glu Asp Asn Ala Leu Lys Val
260 265 270
Leu His Asn Gly Thr Leu Ala Glu Leu Gln Gly Leu Pro His Ile Arg
275 280 285
Val Phe Leu Asp Asn Asn Pro Trp Val Cys Asp Cys His Met Ala Asp
290 295 300
Met Val Thr Trp Leu Lys Glu Thr Glu Val Val Gln Gly Lys Asp Arg
305 310 315 320
Leu Thr Cys Ala Tyr Pro Glu Lys Met Arg Asn Arg Val Leu Leu Glu
325 330 335
Leu Asn Ser Ala Asp Leu Asp Cys Asp Pro Ile Leu Pro Pro Ser Leu
340 345 350
Gln Thr Ser
355
<210> 169
<211> 744
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 169
gacatccaga tgacccagtc tccatcctcc ctgagcgcat ctgtaggaga ccgcgtcacc 60
atcacttgcc gggcaagtca gagcattagc agctatttaa attggtatca gcagaaacca 120
gggaaagccc ctaagctcct gatctatgct gcatccagtt tgcaaagtgg ggtcccatca 180
cgtttcagtg gcagtggaag cgggacagat ttcactctca ccatcagcag tctgcaacct 240
gaagattttg caacttatta ctgtcaacag acttacggtt acctgcacac ttttggccag 300
gggaccaagc tggagatcaa aggcggtgga ggcagcggtg ggggtgggtc tggaggcggt 360
ggcagtggcg gcggaggctc tgaggtgcag ctgttggaga gcgggggagg cttggtacag 420
cctggggggt ccctgcgcct ctcctgtgca gccagcggat tcacctttag cagctatgcc 480
atgagctggg tccgccaggc tccagggaag gggctggagt gggtctcagc tattagtggt 540
agtggtggta gcacatacta tgcagactcc gtgaagggcc ggttcaccat ctcccgtgac 600
aattccaaga acacgctgta tctgcaaatg aacagcctgc gtgccgagga cacggctgta 660
tattattgtg cgcgctacta cggtggttac tactctgctt ggatggacta ttggggccag 720
ggaaccctgg tcaccgtctc ctca 744
<210> 170
<211> 248
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - синтезированная последовательность scFv
<400> 170
Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr Leu His
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Gly Gly Gly Gly Ser
100 105 110
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Glu
115 120 125
Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
130 135 140
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala
145 150 155 160
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
165 170 175
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
180 185 190
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
195 200 205
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
210 215 220
Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp Met Asp Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser
245
<210> 171
<211> 2298
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 171
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaagagccc aaatcttctg acaaaactca cacatgccca 840
ccgtgcccag cacctgaagc cgcgggtgca ccgtcagtct tcctcttccc cccaaaaccc 900
aaggacaccc tcatgatctc ccggacccct gaggtcacat gcgtggtggt ggacgtgagc 960
cacgaagacc ctgaggtcaa gttcaactgg tacgtggacg gcgtggaggt gcataatgcc 1020
aagacaaagc cgcgggagga gcagtacaac agcacgtacc gtgtggtcag cgtcctcacc 1080
gtcctgcacc aggactggct gaatggcaag gaatacaagt gcgcggtctc caacaaagcc 1140
ctcccagccc ccatcgagaa aaccatctcc aaagccaaag ggcagccccg agaaccacag 1200
gtgtacaccc tgcccccatc ccgggatgag ctgaccaaga accaggtcag cctgacctgc 1260
ctggtcaaag gcttctatcc aagcgacatc gccgtggagt gggagagcaa tgggcagccg 1320
gagaacaact acaagaccac gcctcccgtg ctggactccg acggctcctt cttcctctac 1380
agcaagctca ccgtggacaa gagcaggtgg cagcagggga acgtcttctc atgctccgtg 1440
atgcatgagg ctctgcacaa ccactacacg cagaagagcc tctccctgtc tccgggttcc 1500
ggaggtggcg gttcgggagg tggcgggtca ggaggtgggg gatccccttc agaagtgcag 1560
ctgctggagt ccggaggagg actggtgcag cctggcggaa gcctgaggct gagctgcgct 1620
gcctccggct tcacattcag cagctatgct atgagctggg tgaggcaagc ccctggaaag 1680
ggcctggagt gggtgtccgc tatctccggc agcggcggaa gcacctacta cgctgactcc 1740
gtcaagggca ggttcaccat cagccgggac aacagcaaga acaccctgta cctgcagatg 1800
aatagcctca gggctgaaga caccgctgtg tactactgcg ccaggtacta tggcggctac 1860
tactccgcct ggatggacta ctggggacag ggcacactgg tgaccgtgtc cagcggcgga 1920
ggcggctccg gaggcggtgg ctccggagga ggcggaagcg gaggaggagg ctccgatatt 1980
cagatgacac agtcccctag ctccctgtcc gccagcgtgg gagatcgggt gaccatcacc 2040
tgcagggcca gccagtccat ctccagcttc ttaaactggt accagcagaa gcctggaaag 2100
gctcccaagc tgctgatcta cgccgcttcc agcctccaga gcggcgtgcc tagcaggttc 2160
tccggctccg gaagcggaac agacttcacc ctgaccatca gctccctgca gcccgaggac 2220
tccgctacct actactgcca gcagacctat ggctacctgc acaccttcgg ccagggcaca 2280
aagctggaga tcaagcgc 2298
<210> 172
<211> 766
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 172
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser His Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser
260 265 270
Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
275 280 285
Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
290 295 300
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
305 310 315 320
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
325 330 335
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
340 345 350
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
355 360 365
Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro Ala Pro
370 375 380
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
385 390 395 400
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
405 410 415
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
420 425 430
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
435 440 445
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
450 455 460
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
465 470 475 480
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
485 490 495
Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
500 505 510
Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
515 520 525
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
530 535 540
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
545 550 555 560
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
565 570 575
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
580 585 590
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
595 600 605
Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp
610 615 620
Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
625 630 635 640
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
645 650 655
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
660 665 670
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
675 680 685
Ser Phe Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
690 695 700
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
705 710 715 720
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
725 730 735
Gln Pro Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr
740 745 750
Leu His Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 173
<211> 2298
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 173
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaagagccc aaatcttctg acaaaactca cacatgccca 840
ccgtgcccag cacctgaagc cgcgggtgca ccgtcagtct tcctcttccc cccaaaaccc 900
aaggacaccc tcatgatctc ccggacccct gaggtcacat gcgtggtggt ggacgtgagc 960
cacgaagacc ctgaggtcaa gttcaactgg tacgtggacg gcgtggaggt gcataatgcc 1020
aagacaaagc cgcgggagga gcagtacaac agcacgtacc gtgtggtcag cgtcctcacc 1080
gtcctgcacc aggactggct gaatggcaag gaatacaagt gcgcggtctc caacaaagcc 1140
ctcccagccc ccatcgagaa aaccatctcc aaagccaaag ggcagccccg agaaccacag 1200
gtgtacaccc tgcccccatc ccgggatgag ctgaccaaga accaggtcag cctgacctgc 1260
ctggtcaaag gcttctatcc aagcgacatc gccgtggagt gggagagcaa tgggcagccg 1320
gagaacaact acaagaccac gcctcccgtg ctggactccg acggctcctt cttcctctac 1380
agcaagctca ccgtggacaa gagcaggtgg cagcagggga acgtcttctc atgctccgtg 1440
atgcatgagg ctctgcacaa ccactacacg cagaagagcc tctccctgtc tccgggttcc 1500
ggaggtggcg gttcgggagg tggcgggtca ggaggtgggg gatccccttc agaagtgcag 1560
ctgctggagt ccggaggagg actggtgcag cctggcggaa gcctgaggct gagctgcgct 1620
gcctccggct tcacattcag cagctatgct atgagctggg tgaggcaagc ccctggaaag 1680
tgcctggagt gggtgtccgc tatctccggc agcggcggaa gcacctacta cgctgactcc 1740
gtcaagggca ggttcaccat cagccgggac aacagcaaga acaccctgta cctgcagatg 1800
aatagcctca gggctgaaga caccgctgtg tactactgcg ccaggtacta tggcggctac 1860
tactccgcct ggatggacta ctggggacag ggcacactgg tgaccgtgtc cagcggcgga 1920
ggcggctccg gaggcggtgg ctccggagga ggcggaagcg gaggaggagg ctccgatatt 1980
cagatgacac agtcccctag ctccctgtcc gccagcgtgg gagatcgggt gaccatcacc 2040
tgcagggcca gccagtccat ctccagcttc ttaaactggt accagcagaa gcctggaaag 2100
gctcccaagc tgctgatcta cgccgcttcc agcctccaga gcggcgtgcc tagcaggttc 2160
tccggctccg gaagcggaac agacttcacc ctgaccatca gctccctgca gcccgaggac 2220
tccgctacct actactgcca gcagacctat ggctacctgc acaccttcgg ctgcggcaca 2280
aagctggaga tcaagcgc 2298
<210> 174
<211> 766
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 174
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser His Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser
260 265 270
Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
275 280 285
Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
290 295 300
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
305 310 315 320
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
325 330 335
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
340 345 350
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
355 360 365
Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro Ala Pro
370 375 380
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
385 390 395 400
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
405 410 415
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
420 425 430
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
435 440 445
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
450 455 460
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
465 470 475 480
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
485 490 495
Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
500 505 510
Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
515 520 525
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
530 535 540
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
545 550 555 560
Cys Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
565 570 575
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
580 585 590
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
595 600 605
Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp
610 615 620
Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
625 630 635 640
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
645 650 655
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
660 665 670
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
675 680 685
Ser Phe Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
690 695 700
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
705 710 715 720
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
725 730 735
Gln Pro Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr
740 745 750
Leu His Thr Phe Gly Cys Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 175
<211> 2298
<212> ДНК
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полинуклеотидная конструкция к 5T4 x 4-1BB
<400> 175
atggaagcac cagcgcagct tctcttcctc ctgctactct ggctcccaga taccaccggt 60
gaggtgcagc tgttggagag cgggggaggc ttggtacagc ctggggggtc cctgcgcctc 120
tcctgtgcag ccagcggatt caccttttct cacggttcta tgtactgggt ccgccaggct 180
ccagggaagg ggctggagtg ggtctcatct atttcttctg gttctggttc tacatactat 240
gcagactccg tgaagggccg gttcaccatc tcccatgaca attccaagaa cacgctgtat 300
ctgcaaatga acagcctgcg tgccgaggac acggctgtat attattgtgc gcgctcttct 360
tactacggtt cttactactc tattgactat tggggccagg gaaccctggt caccgtctcc 420
tcaggtggag gtggctccgg gggtggaggt tccggaggag gcggatcagg tggaggcgga 480
agcgacatcc agatgaccca gtctccatcc tccctgagcg catctgtagg agaccgcgtc 540
accatcactt gccgggcaag tcagagcatt agcagctatt taaattggta tcagcagaaa 600
ccagggaaag cccctaagct cctgatctat gctgcatcca gtttgcaaag tggggtccca 660
tcacgtttca gtggcagtgg aagcgggaca gatttcactc tcaccatcag cagtctacaa 720
cctgaagatt ttgcaactta ttactgtcaa cagtactacg acaacctgcc cacttttggc 780
caggggacca agctggagat caaagagccc aaatcttctg acaaaactca cacatgccca 840
ccgtgcccag cacctgaagc cgcgggtgca ccgtcagtct tcctcttccc cccaaaaccc 900
aaggacaccc tcatgatctc ccggacccct gaggtcacat gcgtggtggt ggacgtgagc 960
cacgaagacc ctgaggtcaa gttcaactgg tacgtggacg gcgtggaggt gcataatgcc 1020
aagacaaagc cgcgggagga gcagtacaac agcacgtacc gtgtggtcag cgtcctcacc 1080
gtcctgcacc aggactggct gaatggcaag gaatacaagt gcgcggtctc caacaaagcc 1140
ctcccagccc ccatcgagaa aaccatctcc aaagccaaag ggcagccccg agaaccacag 1200
gtgtacaccc tgcccccatc ccgggatgag ctgaccaaga accaggtcag cctgacctgc 1260
ctggtcaaag gcttctatcc aagcgacatc gccgtggagt gggagagcaa tgggcagccg 1320
gagaacaact acaagaccac gcctcccgtg ctggactccg acggctcctt cttcctctac 1380
agcaagctca ccgtggacaa gagcaggtgg cagcagggga acgtcttctc atgctccgtg 1440
atgcatgagg ctctgcacaa ccactacacg cagaagagcc tctccctgtc tccgggttcc 1500
ggaggtggcg gttcgggagg tggcgggtca ggaggtgggg gatccccttc agaagtgcag 1560
ctgctggagt ccggaggagg actggtgcag cctggcggaa gcctgaggct gagctgcgct 1620
gcctccggct tcacattcag cagctatgct atgagctggg tgaggcaagc ccctggaaag 1680
ggcctggagt gggtgtccgc tatctccggc agcggcggaa gcacctacta cgctgactcc 1740
gtcaagggca ggttcaccat cagccgggac aacagcaaga acaccctgta cctgcagatg 1800
aatagcctca gggctgaaga caccgctgtg tactactgcg ccaggtacta tggcggctac 1860
tactccgcct ggatggacta ctggggacag ggcacactgg tgaccgtgtc cagcggcgga 1920
ggcggctccg gaggcggtgg ctccggagga ggcggaagcg gaggaggagg ctccgatatt 1980
cagatgacac agtcccctag ctccctgtcc gccagcgtgg gagatcgggt gaccatcacc 2040
tgcagggcca gccagtccat ctccagctat ttaaactggt accagcagaa gcctggaaag 2100
gctcccaagc tgctgatcta cgccgcttcc agcctccaga gcggcgtgcc tagcaggttc 2160
tccggctccg gaagcggaac agacttcacc ctgaccatca gctccctgca gcccgaggac 2220
tccgctacct actactgcca gcagacctat ggctacctgc acaccttcgg ccagggcaca 2280
aagctggaga tcaagcgc 2298
<210> 176
<211> 766
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Получено в лаборатории - полипептидная конструкция к 5T4 x 4-1BB
<400> 176
Met Glu Ala Pro Ala Gln Leu Leu Phe Leu Leu Leu Leu Trp Leu Pro
1 5 10 15
Asp Thr Thr Gly Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val
20 25 30
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr
35 40 45
Phe Ser His Gly Ser Met Tyr Trp Val Arg Gln Ala Pro Gly Lys Gly
50 55 60
Leu Glu Trp Val Ser Ser Ile Ser Ser Gly Ser Gly Ser Thr Tyr Tyr
65 70 75 80
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser His Asp Asn Ser Lys
85 90 95
Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala
100 105 110
Val Tyr Tyr Cys Ala Arg Ser Ser Tyr Tyr Gly Ser Tyr Tyr Ser Ile
115 120 125
Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly
130 135 140
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
145 150 155 160
Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val
165 170 175
Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser
180 185 190
Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu
195 200 205
Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser
210 215 220
Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln
225 230 235 240
Pro Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Tyr Asp Asn Leu
245 250 255
Pro Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Glu Pro Lys Ser
260 265 270
Ser Asp Lys Thr His Thr Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala
275 280 285
Gly Ala Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu
290 295 300
Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser
305 310 315 320
His Glu Asp Pro Glu Val Lys Phe Asn Trp Tyr Val Asp Gly Val Glu
325 330 335
Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr
340 345 350
Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn
355 360 365
Gly Lys Glu Tyr Lys Cys Ala Val Ser Asn Lys Ala Leu Pro Ala Pro
370 375 380
Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln
385 390 395 400
Val Tyr Thr Leu Pro Pro Ser Arg Asp Glu Leu Thr Lys Asn Gln Val
405 410 415
Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val
420 425 430
Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro
435 440 445
Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr
450 455 460
Val Asp Lys Ser Arg Trp Gln Gln Gly Asn Val Phe Ser Cys Ser Val
465 470 475 480
Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu
485 490 495
Ser Pro Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
500 505 510
Gly Gly Ser Pro Ser Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu
515 520 525
Val Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe
530 535 540
Thr Phe Ser Ser Tyr Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys
545 550 555 560
Gly Leu Glu Trp Val Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr
565 570 575
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser
580 585 590
Lys Asn Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr
595 600 605
Ala Val Tyr Tyr Cys Ala Arg Tyr Tyr Gly Gly Tyr Tyr Ser Ala Trp
610 615 620
Met Asp Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
625 630 635 640
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
645 650 655
Gly Ser Asp Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser
660 665 670
Val Gly Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser
675 680 685
Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu
690 695 700
Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe
705 710 715 720
Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu
725 730 735
Gln Pro Glu Asp Ser Ala Thr Tyr Tyr Cys Gln Gln Thr Tyr Gly Tyr
740 745 750
Leu His Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys Arg
755 760 765
<210> 177
<211> 10
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Последовательность CDRH1 ACN017
<400> 177
Ala Lys Gly Ser Gly Ser Tyr Phe Asp Leu
1 5 10
<210> 178
<211> 9
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> CDRL3 ACN017
<400> 178
Gln Gln Tyr Ser Gly Tyr Pro Tyr Thr
1 5
<210> 179
<211> 117
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> VH ACN017
<400> 179
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Ser Gly Ser Tyr Phe Asp Leu Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser
115
<210> 180
<211> 107
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> VL ACN017
<400> 180
Asp Ile Gln Met Thr Gln Ser Pro Ser Phe Leu Ser Ala Ser Val Gly
1 5 10 15
Asp Arg Val Thr Ile Thr Cys Arg Ala Ser Gln Ser Ile Ser Ser Tyr
20 25 30
Leu Asn Trp Tyr Gln Gln Lys Pro Gly Lys Ala Pro Lys Leu Leu Ile
35 40 45
Tyr Ala Ala Ser Ser Leu Gln Ser Gly Val Pro Ser Arg Phe Ser Gly
50 55 60
Ser Gly Ser Gly Thr Asp Phe Thr Leu Thr Ile Ser Ser Leu Gln Pro
65 70 75 80
Glu Asp Phe Ala Thr Tyr Tyr Cys Gln Gln Tyr Ser Gly Tyr Pro Tyr
85 90 95
Thr Phe Gly Gln Gly Thr Lys Leu Glu Ile Lys
100 105
<210> 181
<211> 741
<212> ПРТ
<213> ИСКУССТВЕННАЯ ПОСЛЕДОВАТЕЛЬНОСТЬ
<220>
<223> Полноразмерное ACN017
<400> 181
Glu Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr
20 25 30
Ala Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Lys Gly Ser Gly Ser Tyr Phe Asp Leu Trp Gly Gln Gly Thr Leu
100 105 110
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser
130 135 140
Pro Ser Phe Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys
145 150 155 160
Arg Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys
165 170 175
Pro Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln
180 185 190
Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe
195 200 205
Thr Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr
210 215 220
Cys Gln Gln Tyr Ser Gly Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys
225 230 235 240
Leu Glu Ile Lys Ser Ser Glu Pro Lys Ser Ser Asp Lys Thr His Thr
245 250 255
Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Ala Pro Ser Val Phe
260 265 270
Leu Phe Pro Pro Lys Pro Lys Asp Thr Leu Met Ile Ser Arg Thr Pro
275 280 285
Glu Val Thr Cys Val Val Val Asp Val Ser His Glu Asp Pro Glu Val
290 295 300
Lys Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys Thr
305 310 315 320
Lys Pro Arg Glu Glu Gln Tyr Asn Ser Thr Tyr Arg Val Val Ser Val
325 330 335
Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys
340 345 350
Ala Val Ser Asn Lys Ala Leu Pro Ala Pro Ile Glu Lys Thr Ile Ser
355 360 365
Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro
370 375 380
Ser Arg Asp Glu Leu Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val
385 390 395 400
Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly
405 410 415
Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp
420 425 430
Gly Ser Phe Phe Leu Tyr Ser Lys Leu Thr Val Asp Lys Ser Arg Trp
435 440 445
Gln Gln Gly Asn Val Phe Ser Cys Ser Val Met His Glu Ala Leu His
450 455 460
Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Pro Gly Ser Gly Gly
465 470 475 480
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Pro Ser Glu
485 490 495
Val Gln Leu Leu Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser
500 505 510
Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Ser Tyr Ala
515 520 525
Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser
530 535 540
Ala Ile Ser Gly Ser Gly Gly Ser Thr Tyr Tyr Ala Asp Ser Val Lys
545 550 555 560
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ser Lys Asn Thr Leu Tyr Leu
565 570 575
Gln Met Asn Ser Leu Arg Ala Glu Asp Thr Ala Val Tyr Tyr Cys Ala
580 585 590
Lys Gly Ser Gly Ser Tyr Phe Asp Leu Trp Gly Gln Gly Thr Leu Val
595 600 605
Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
610 615 620
Gly Gly Ser Gly Gly Gly Gly Ser Asp Ile Gln Met Thr Gln Ser Pro
625 630 635 640
Ser Phe Leu Ser Ala Ser Val Gly Asp Arg Val Thr Ile Thr Cys Arg
645 650 655
Ala Ser Gln Ser Ile Ser Ser Tyr Leu Asn Trp Tyr Gln Gln Lys Pro
660 665 670
Gly Lys Ala Pro Lys Leu Leu Ile Tyr Ala Ala Ser Ser Leu Gln Ser
675 680 685
Gly Val Pro Ser Arg Phe Ser Gly Ser Gly Ser Gly Thr Asp Phe Thr
690 695 700
Leu Thr Ile Ser Ser Leu Gln Pro Glu Asp Phe Ala Thr Tyr Tyr Cys
705 710 715 720
Gln Gln Tyr Ser Gly Tyr Pro Tyr Thr Phe Gly Gln Gly Thr Lys Leu
725 730 735
Glu Ile Lys Arg Ser
740
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| БЕЛОК, СВЯЗЫВАЮЩИЙСЯ С NKG2D, CD16 И С ОПУХОЛЕСПЕЦИФИЧЕСКИМ АНТИГЕНОМ | 2018 |
|
RU2788531C2 |
| КОНСТРУКЦИИ, ИМЕЮЩИЕ SIRP-АЛЬФА ДОМЕН ИЛИ ЕГО ВАРИАНТ | 2016 |
|
RU2740672C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА, СПЕЦИФИЧЕСКИ СВЯЗЫВАЮЩИЕСЯ С PD1 И LAG3 | 2018 |
|
RU2778805C2 |
| ЛЕЧЕНИЕ РАКА С ИСПОЛЬЗОВАНИЕМ ХИМЕРНОГО АНТИГЕНСПЕЦИФИЧЕСКОГО РЕЦЕПТОРА НА ОСНОВЕ ГУМАНИЗИРОВАННОГО АНТИТЕЛА ПРОТИВ EGFRvIII | 2014 |
|
RU2831418C2 |
| ВАРИАНТНЫЕ СВЯЗЫВАЮЩИЕ CD3 ДОМЕНЫ И ИХ ПРИМЕНЕНИЕ В КОМБИНИРОВАННОЙ ТЕРАПИИ ПРИ ЛЕЧЕНИИ ЗАБОЛЕВАНИЙ | 2019 |
|
RU2810222C2 |
| HER-2-НАПРАВЛЕННЫЕ АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ, СОДЕРЖАЩИЕ 4-1BBL | 2019 |
|
RU2815451C2 |
| АНТИТЕЛА ПРОТИВ TNFRSF9 И ИХ ПРИМЕНЕНИЕ | 2019 |
|
RU2812200C2 |
| ПОЛИСПЕЦИФИЧЕСКИЕ ПОЛИПЕПТИДНЫЕ КОНСТРУКЦИИ, ОБЛАДАЮЩИЕ ОГРАНИЧЕННЫМ СВЯЗЫВАНИЕМ С СD3, И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2018 |
|
RU2813816C2 |
| НОВЫЕ КОНСТРУКЦИИ ХИМЕРНОГО АНТИГЕННОГО РЕЦЕПТОРА, СОДЕРЖАЩИЕ ДОМЕНЫ TNFR2 | 2019 |
|
RU2808254C2 |
| БИСПЕЦИФИЧЕСКИЕ 2+1 КОНТОРСТЕЛА | 2018 |
|
RU2797305C2 |
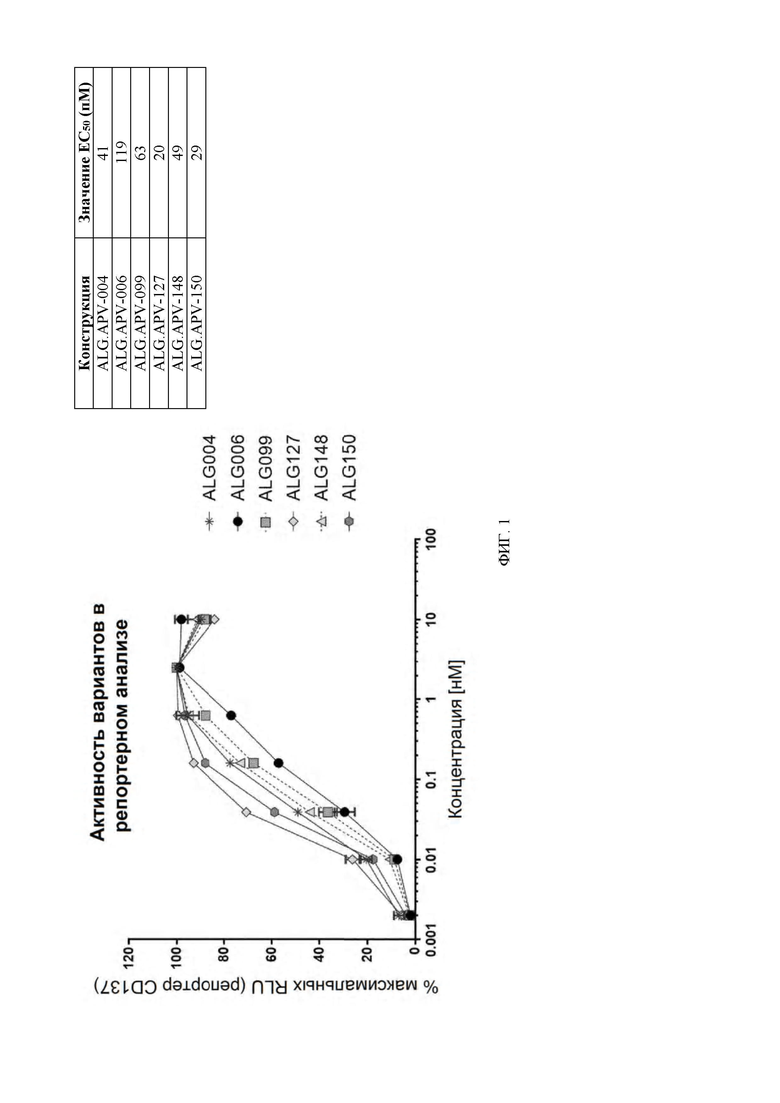
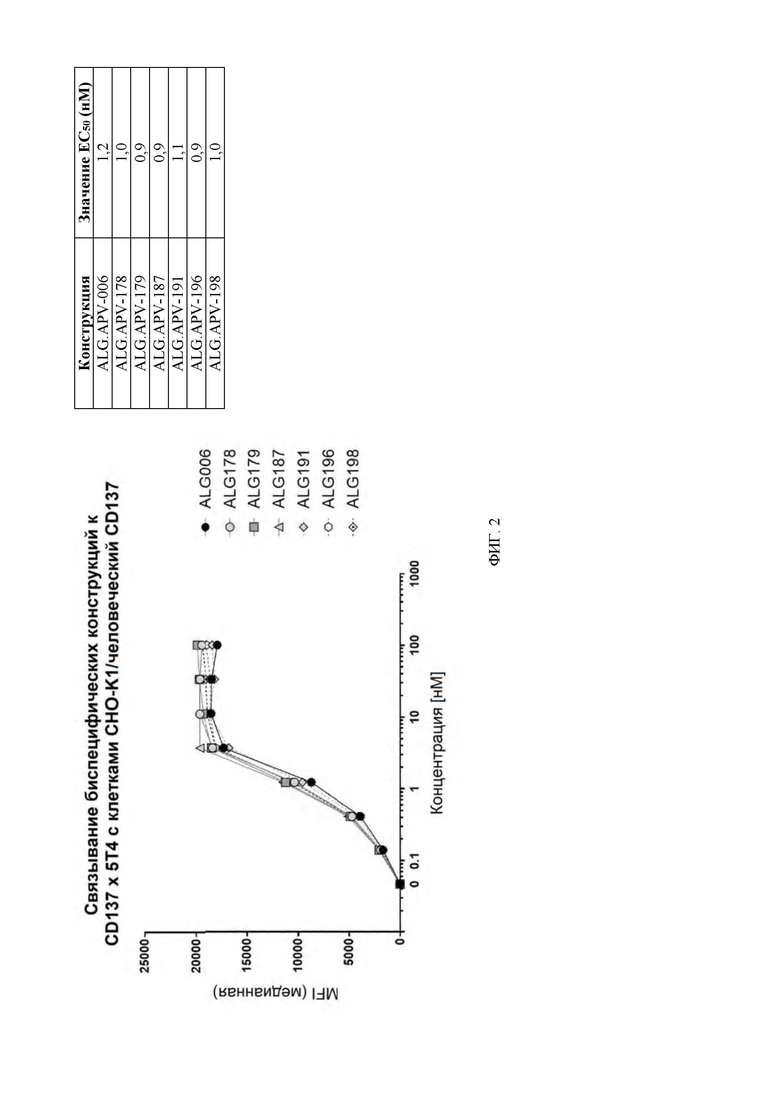
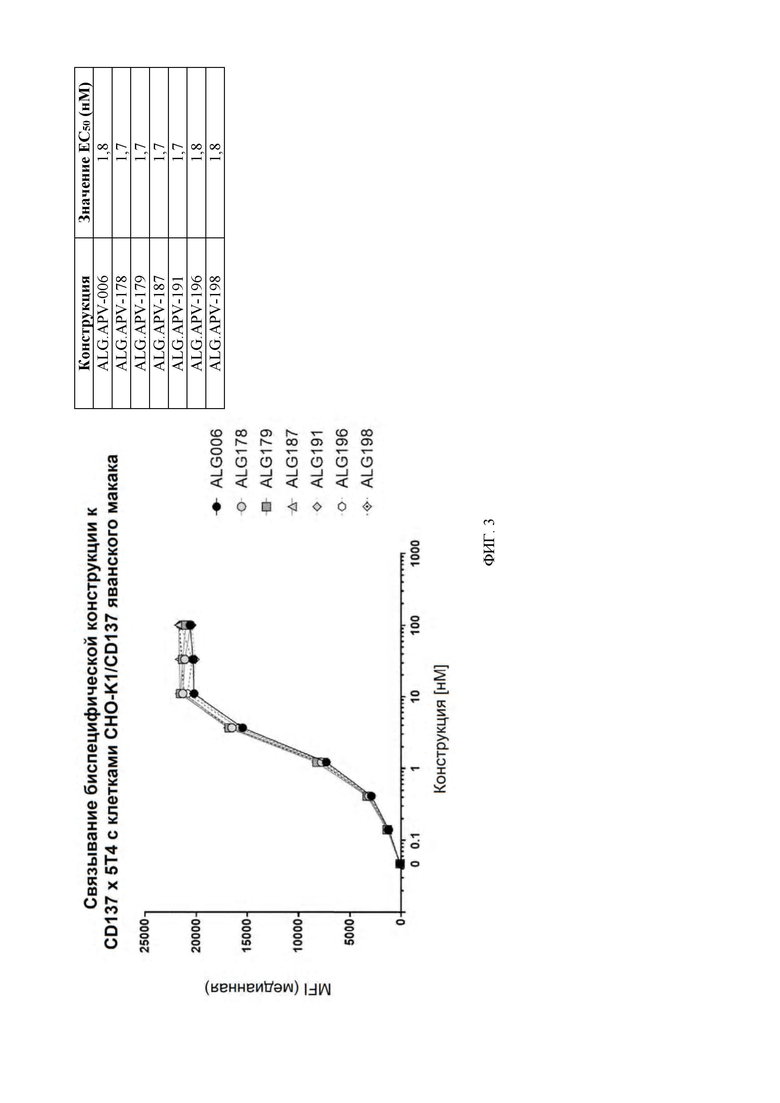
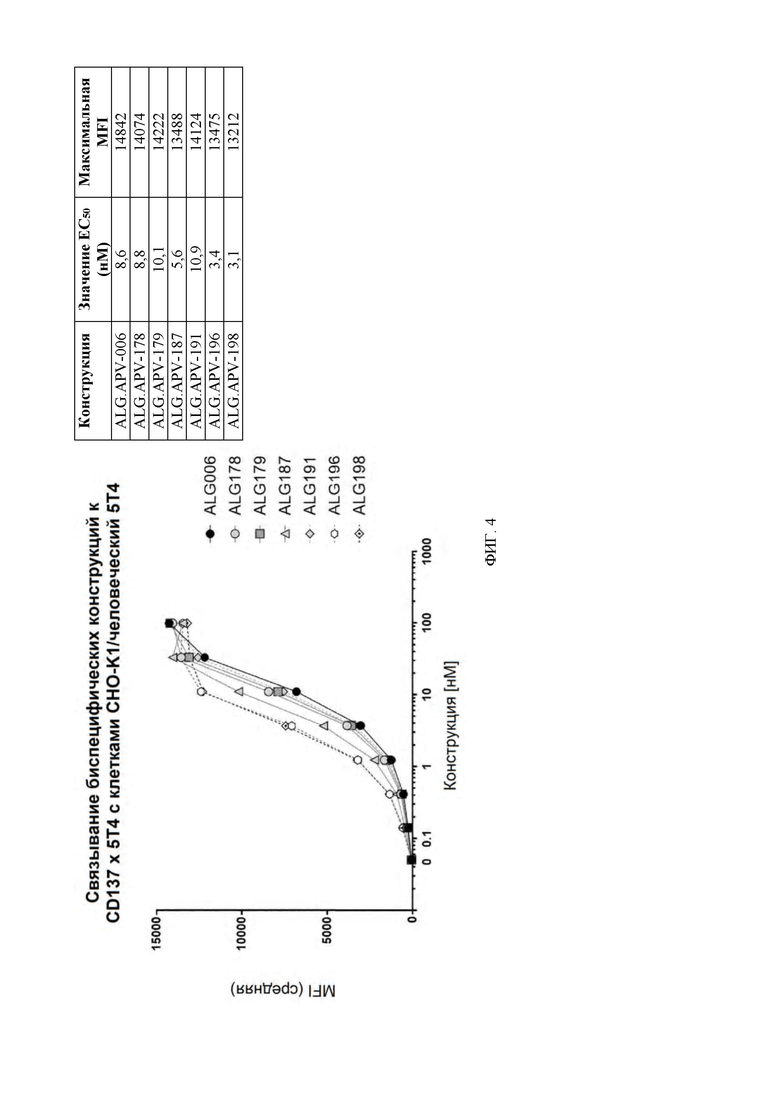
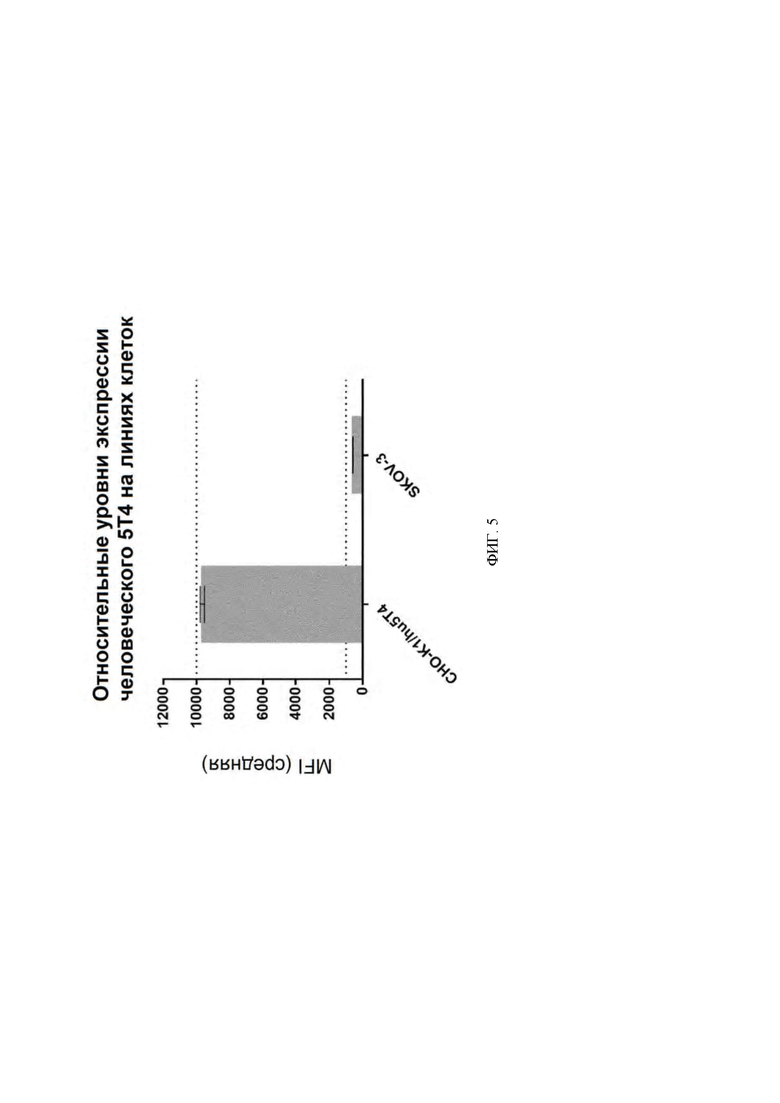
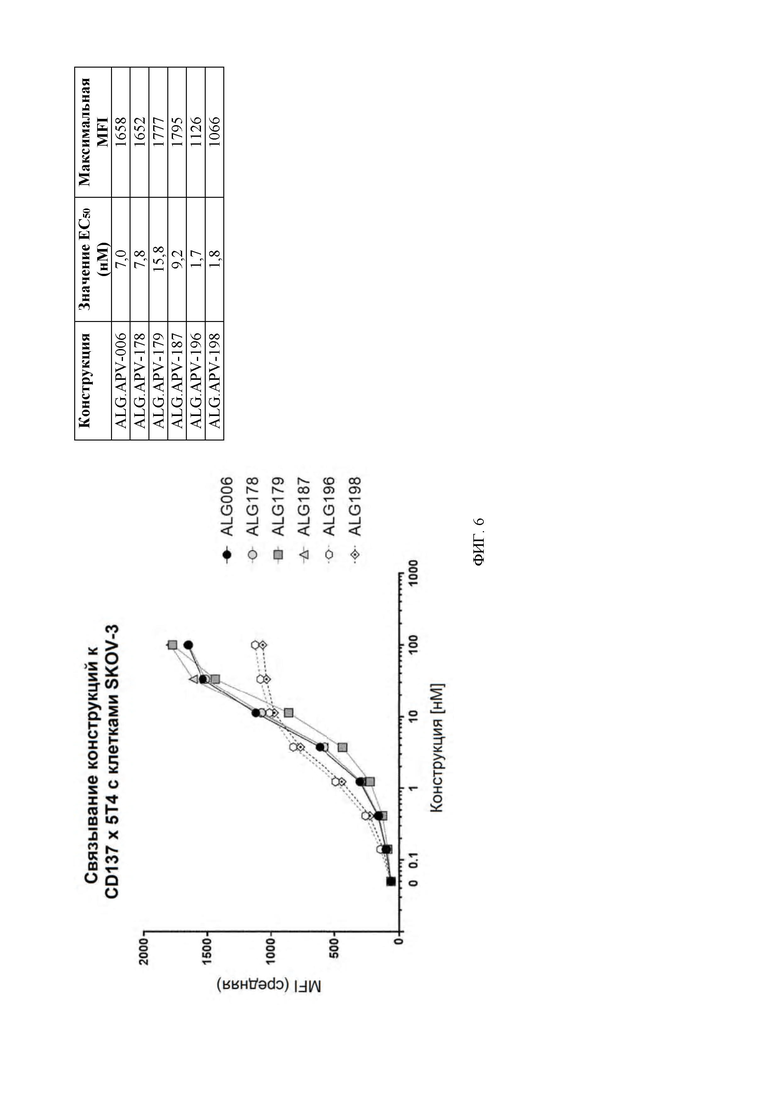
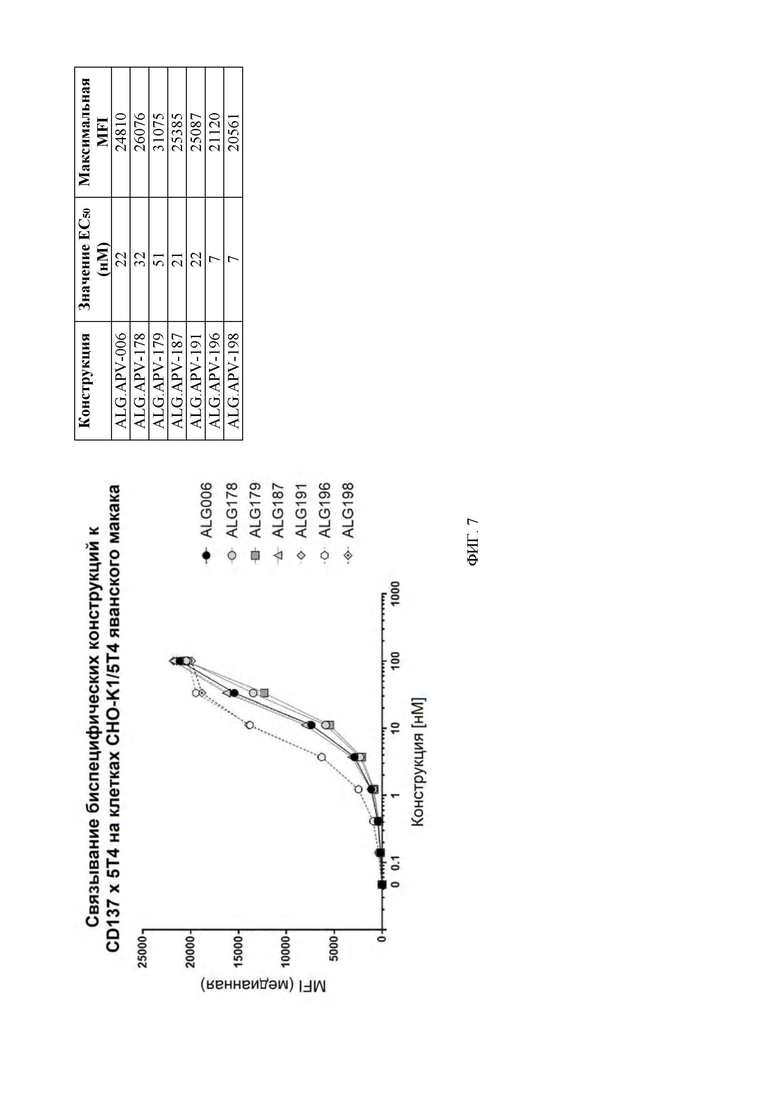
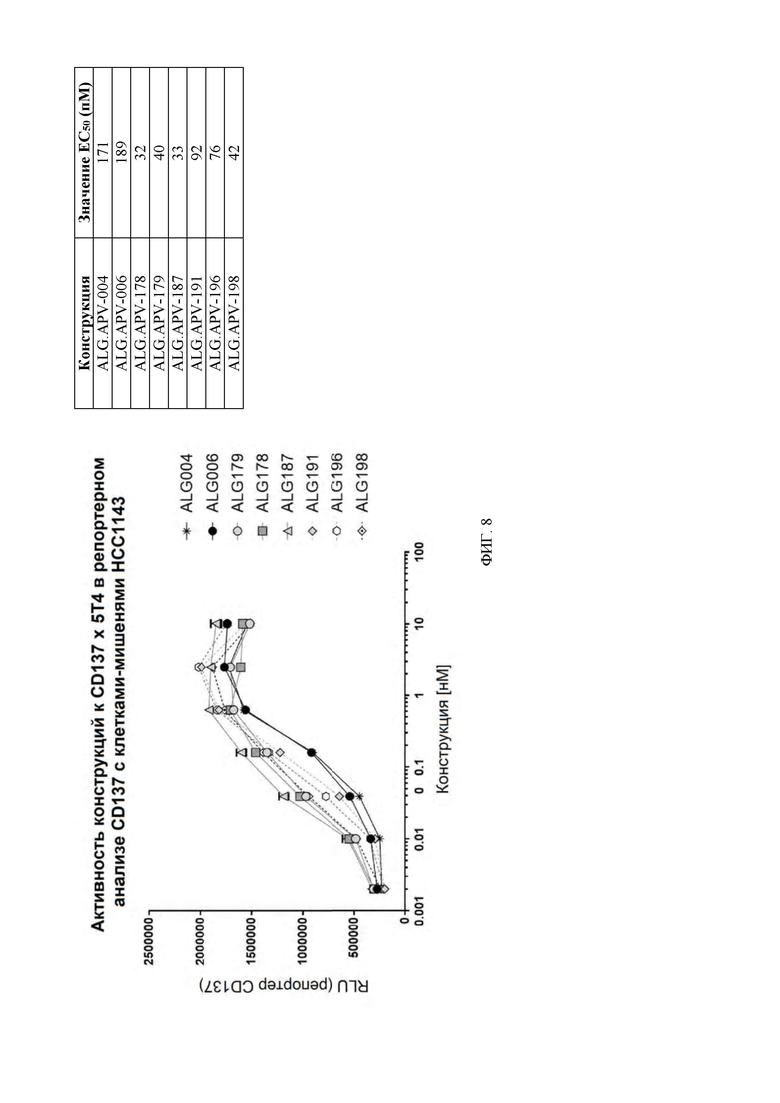
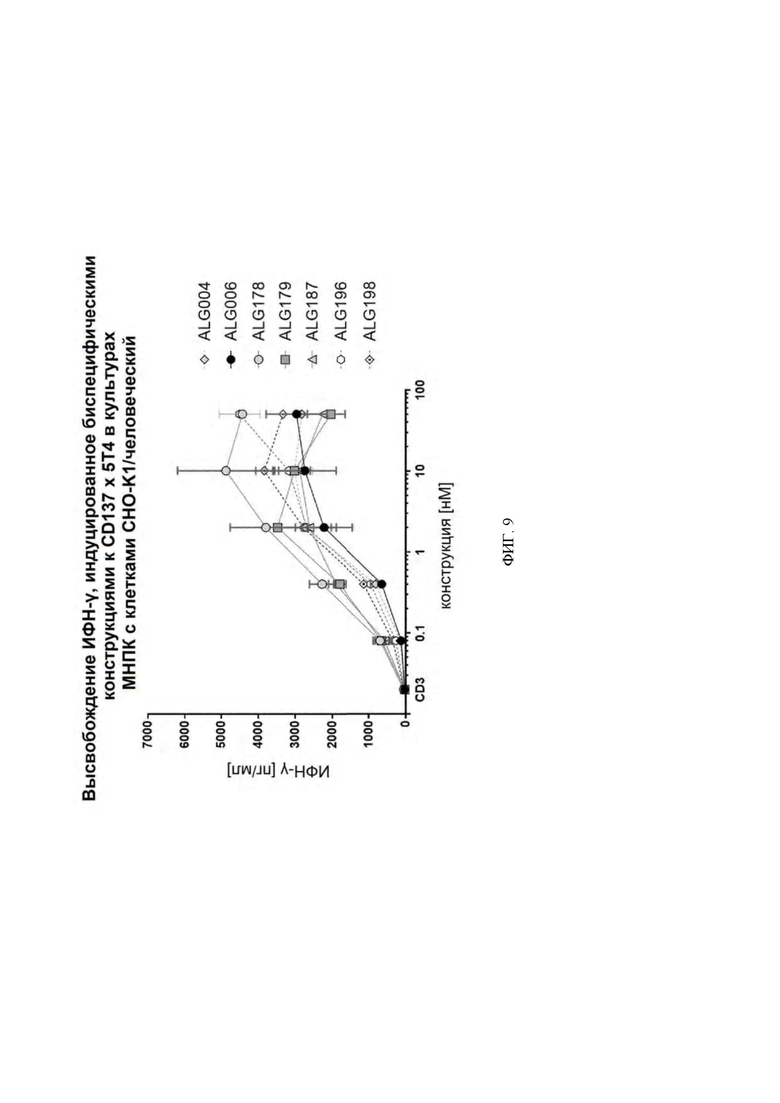
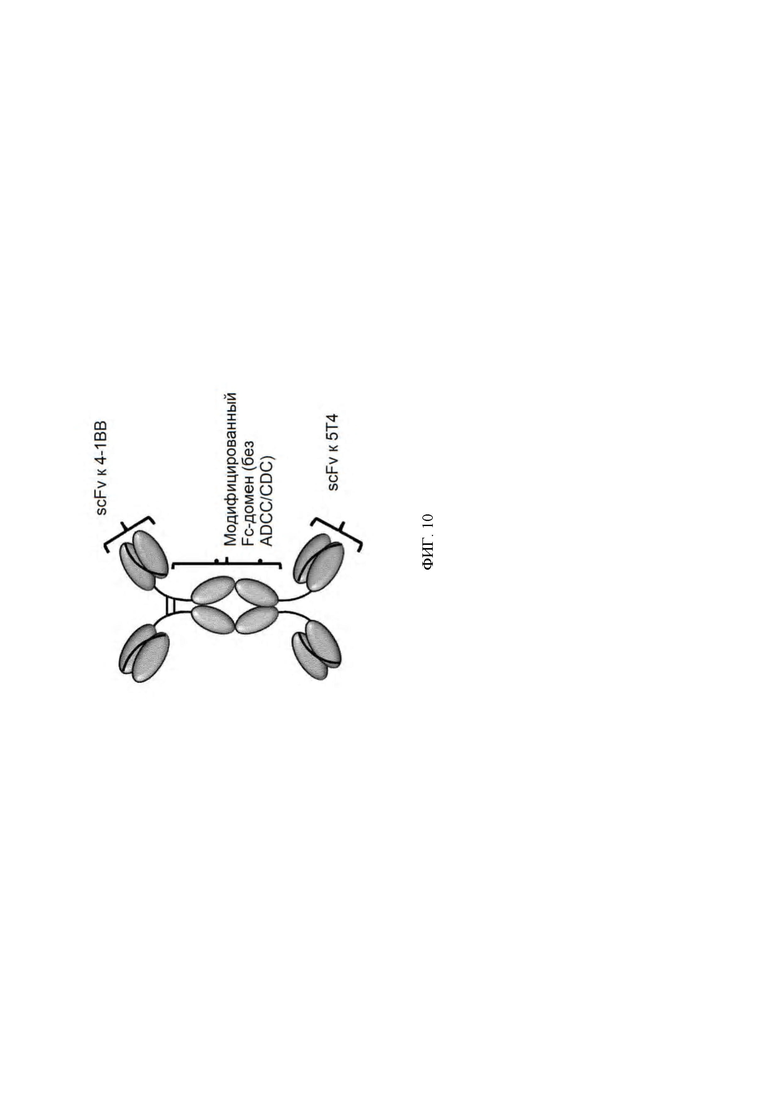

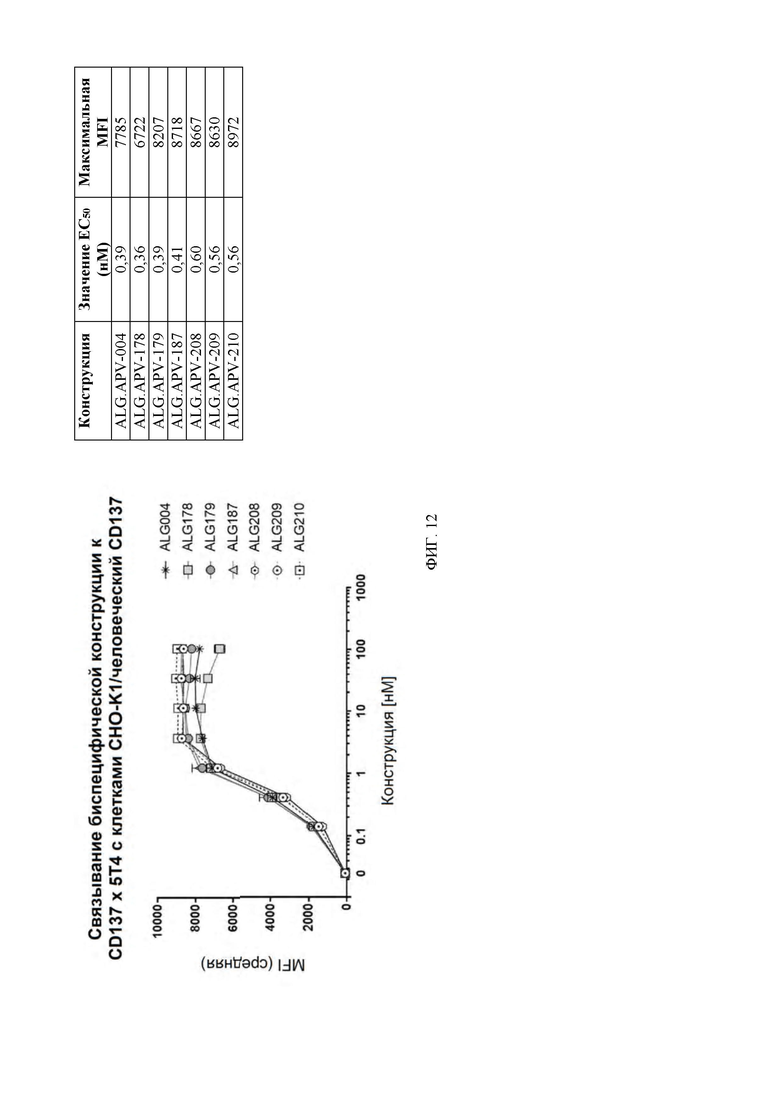
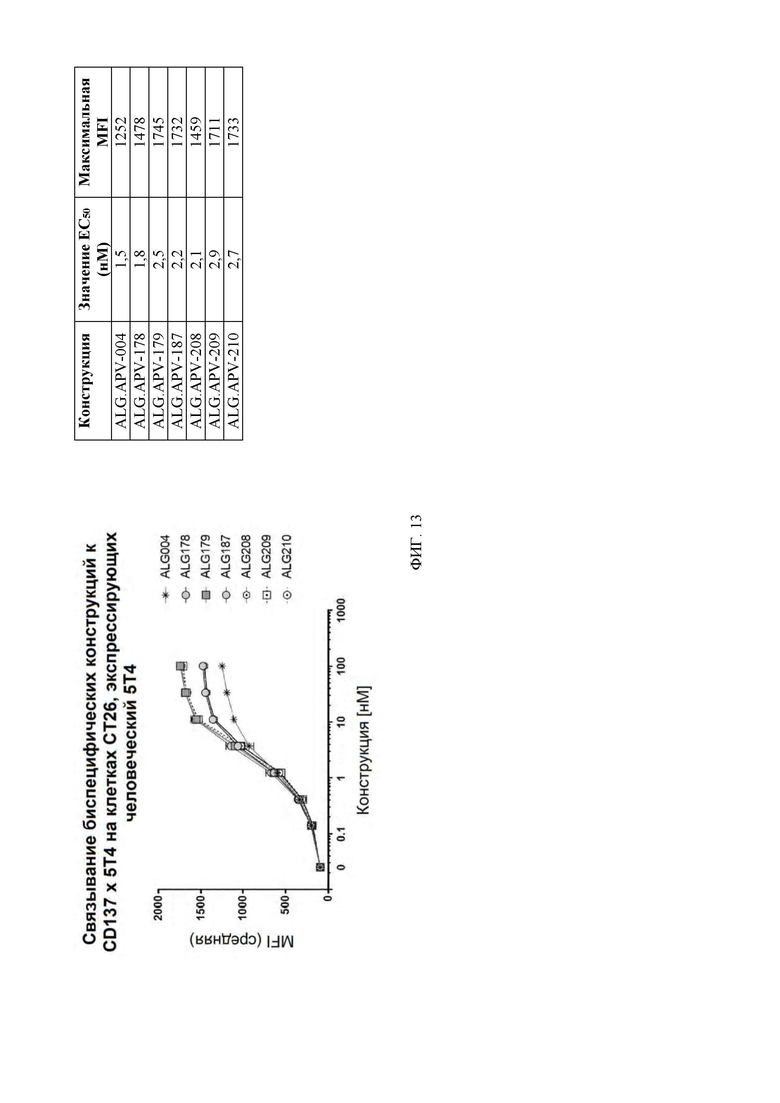
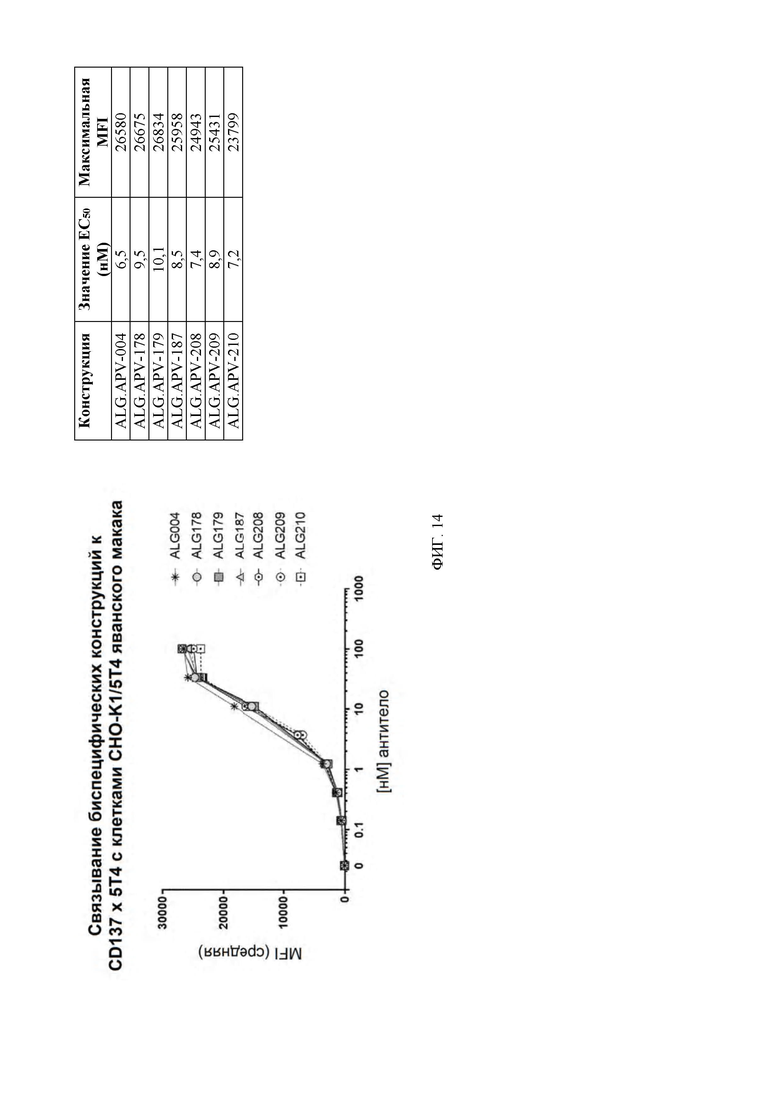

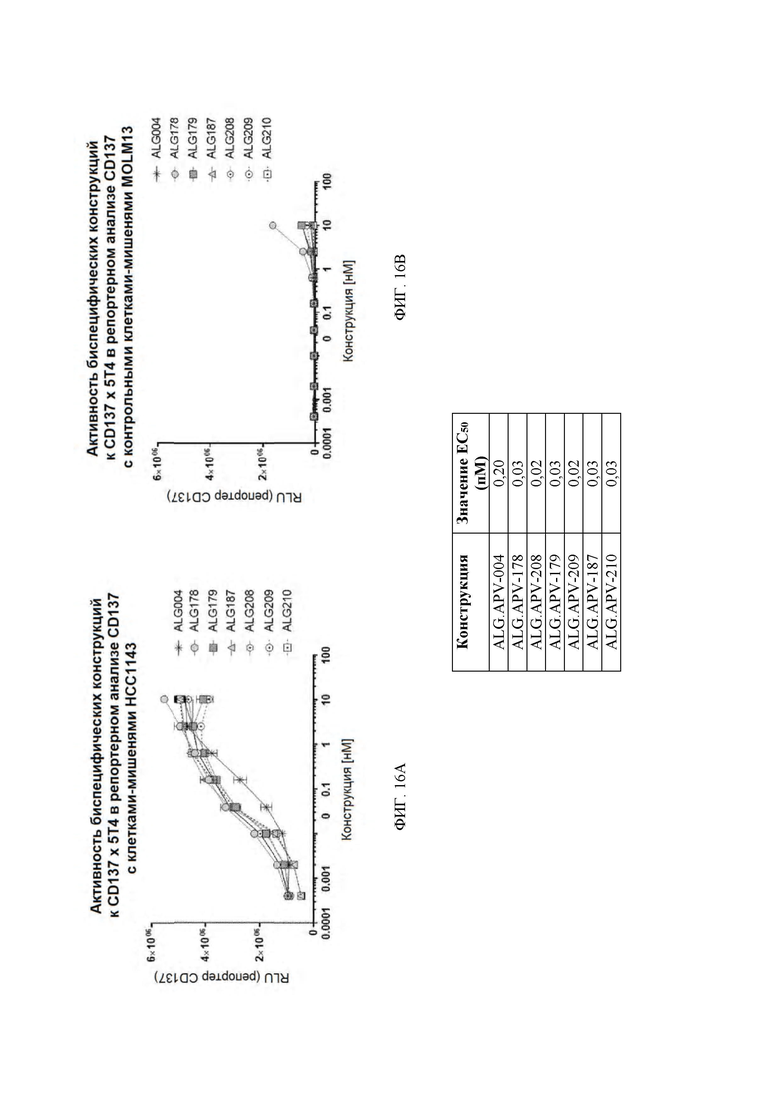
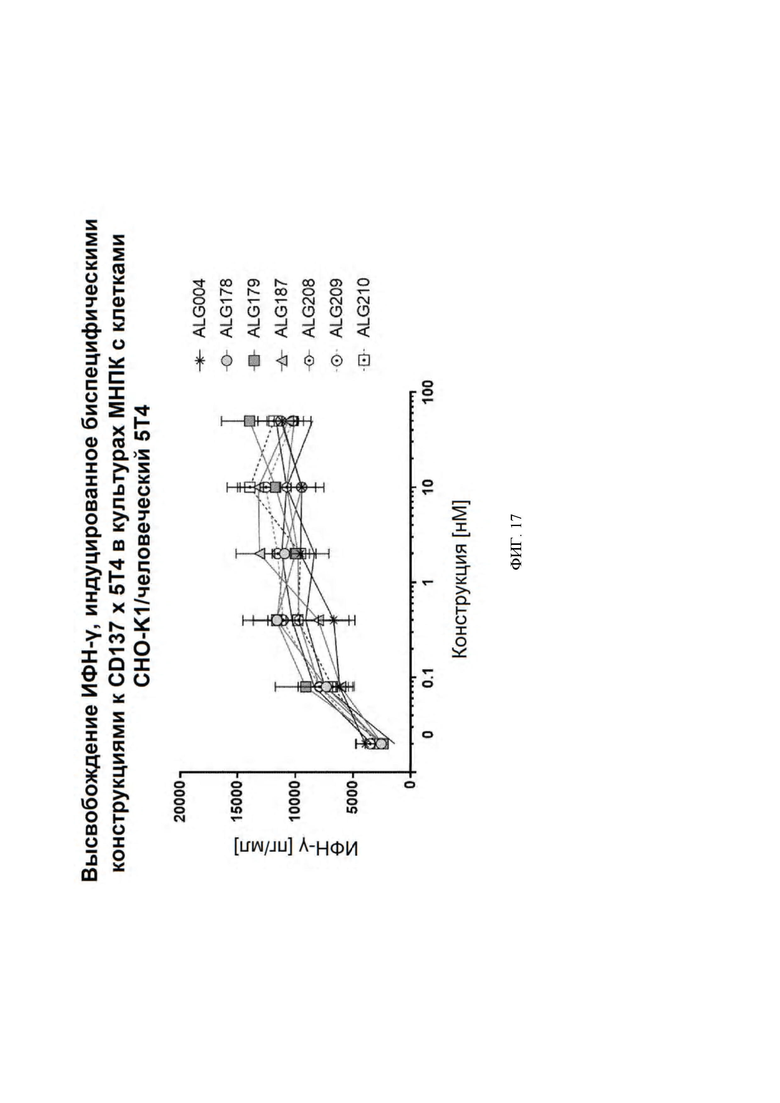
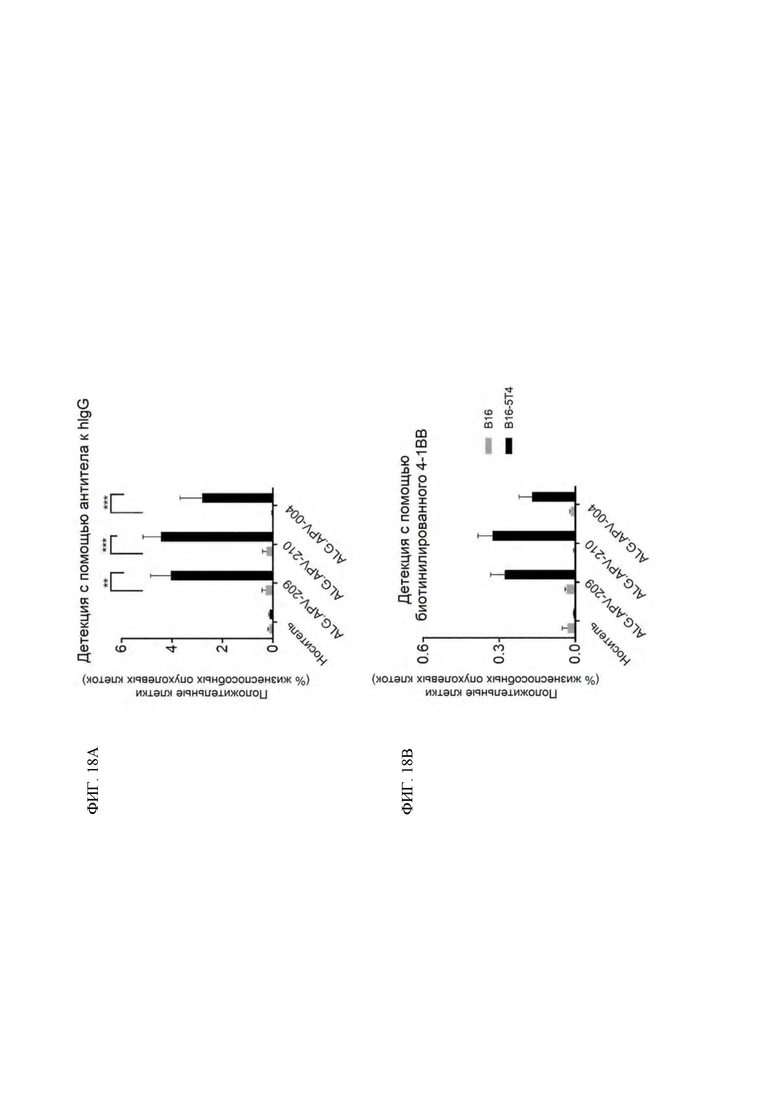

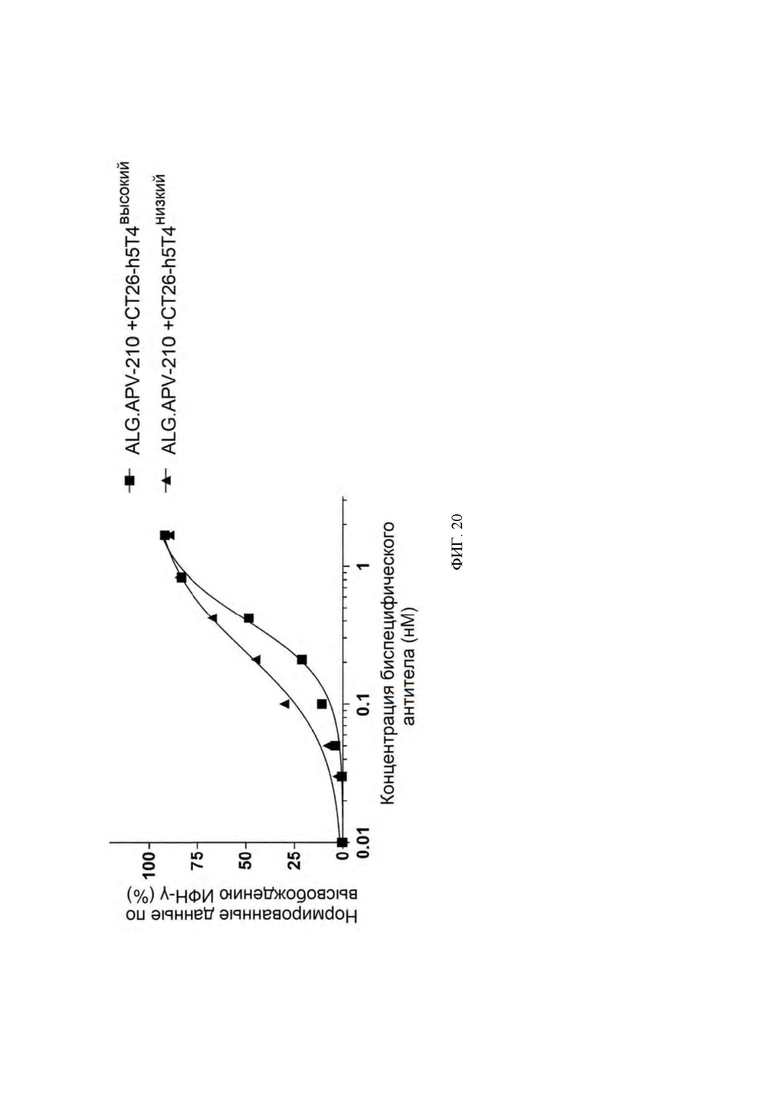
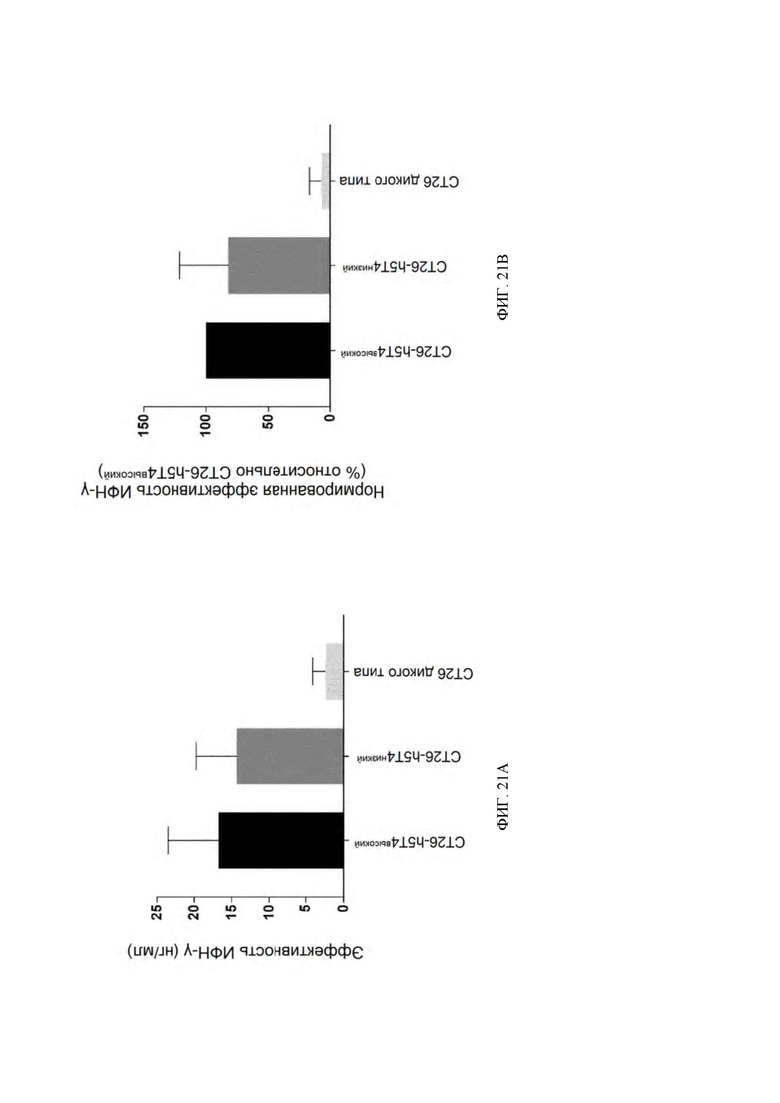


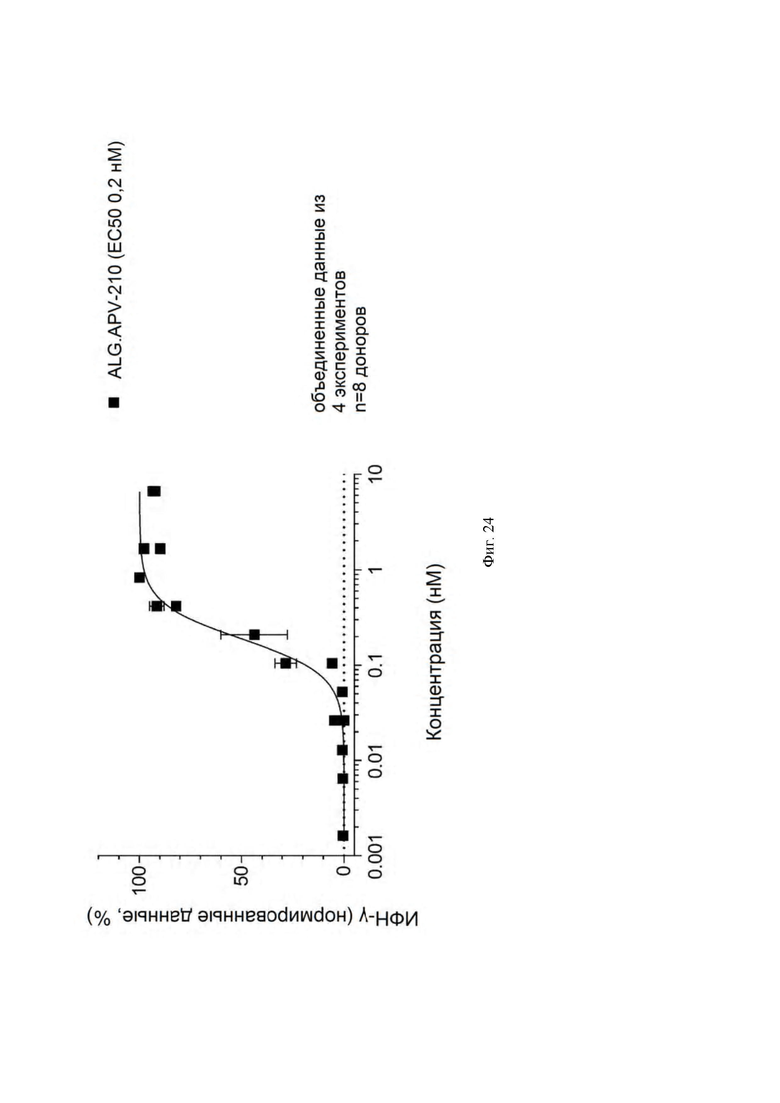
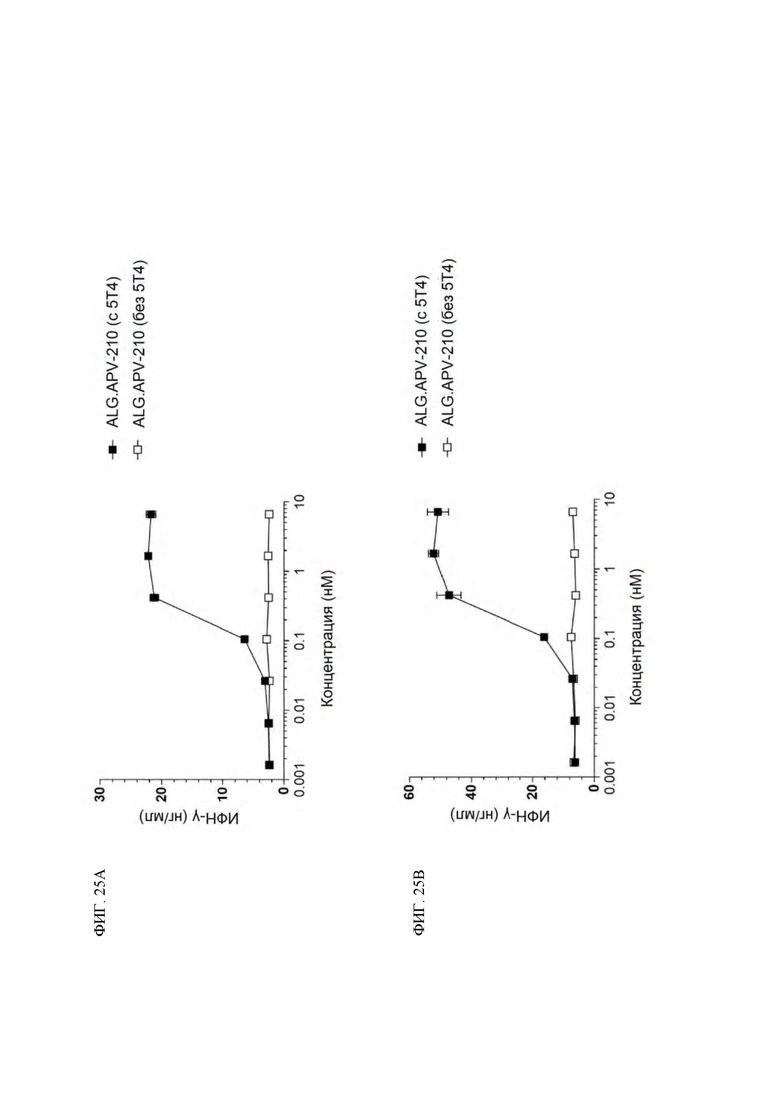
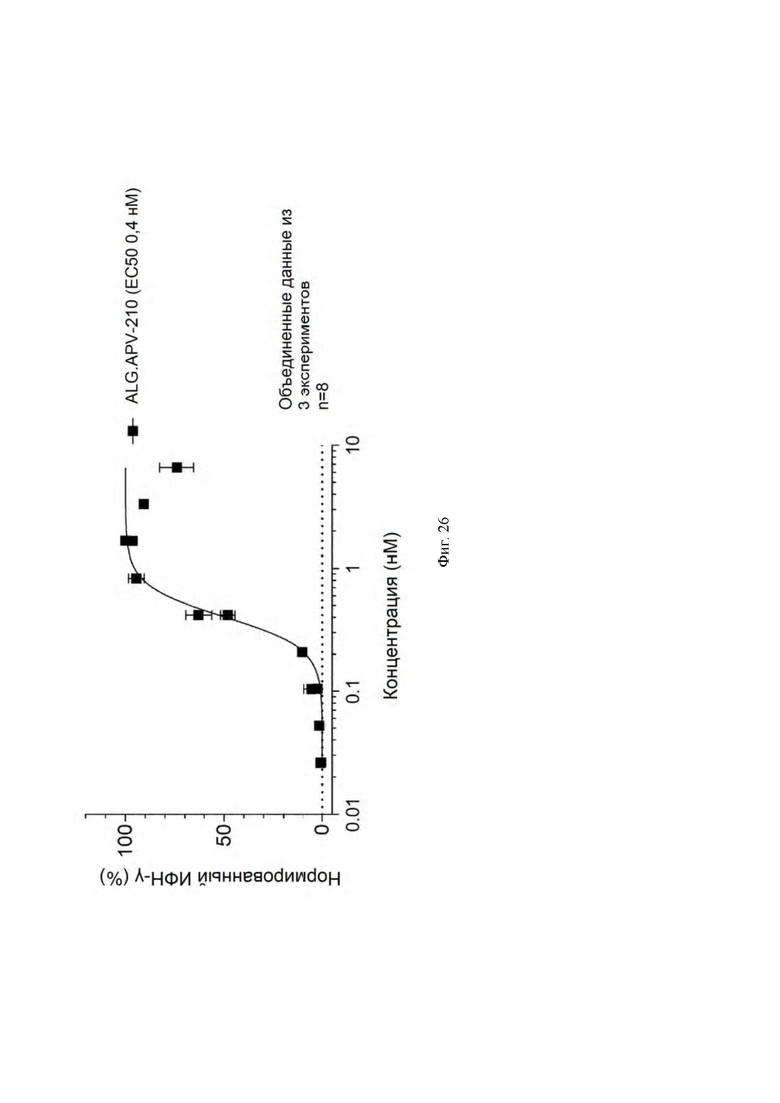

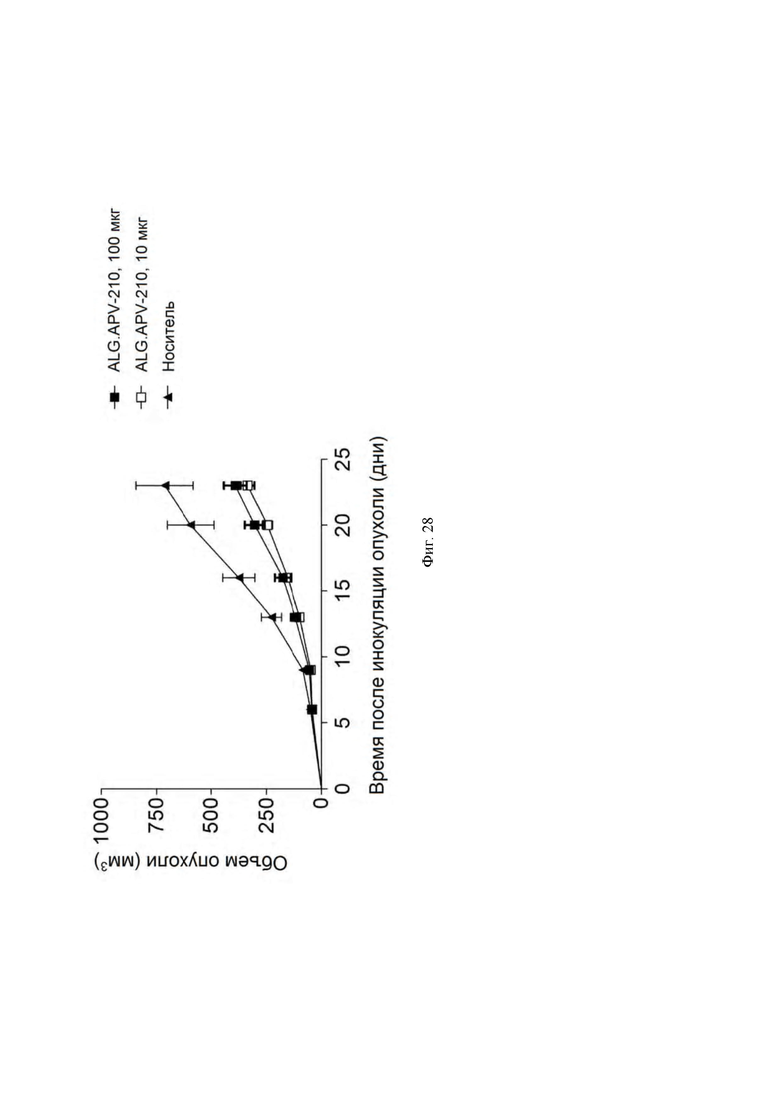
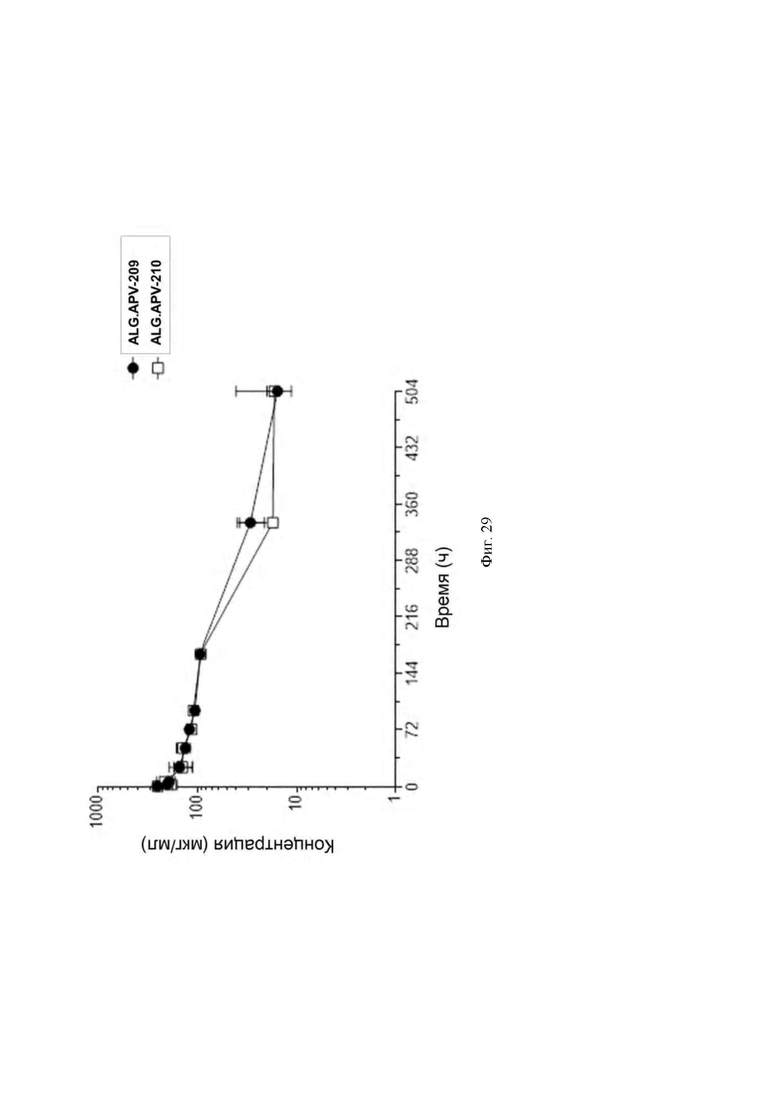
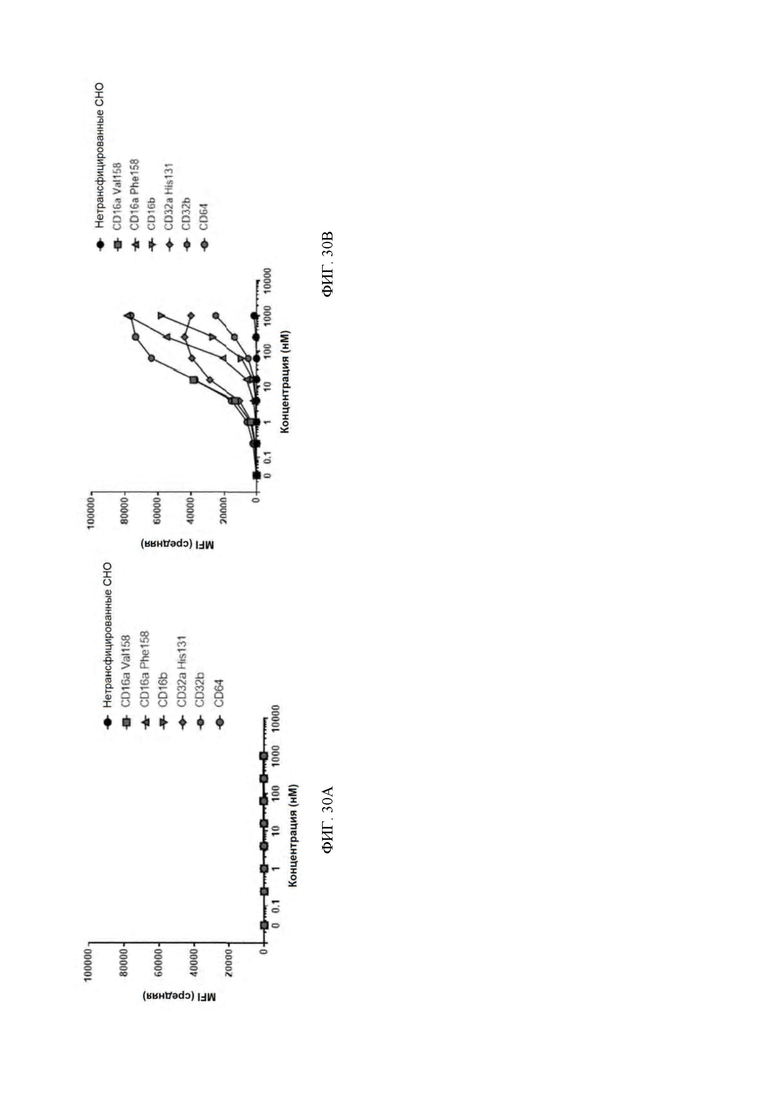
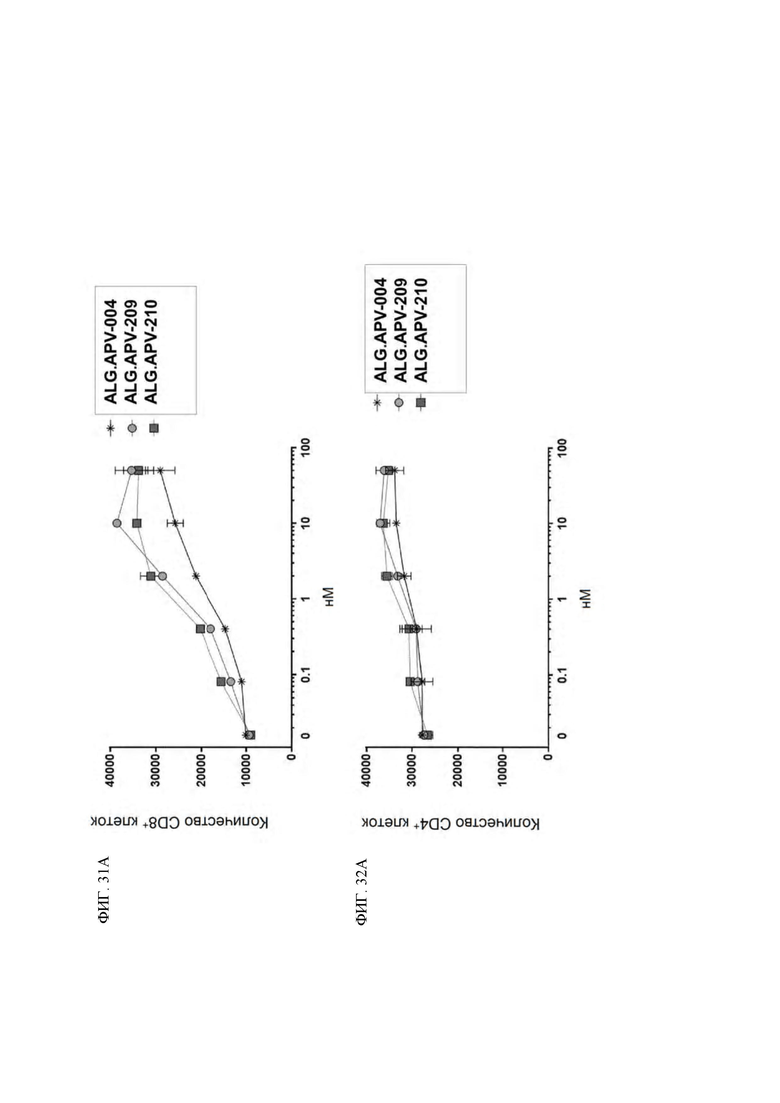
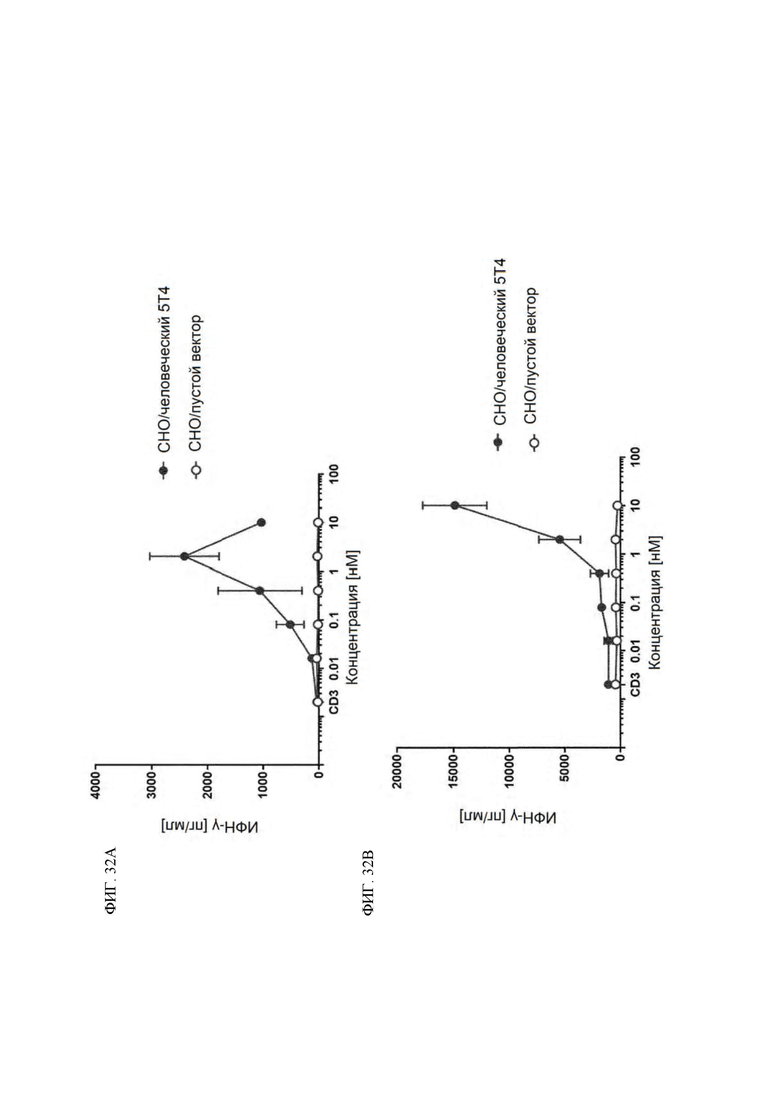
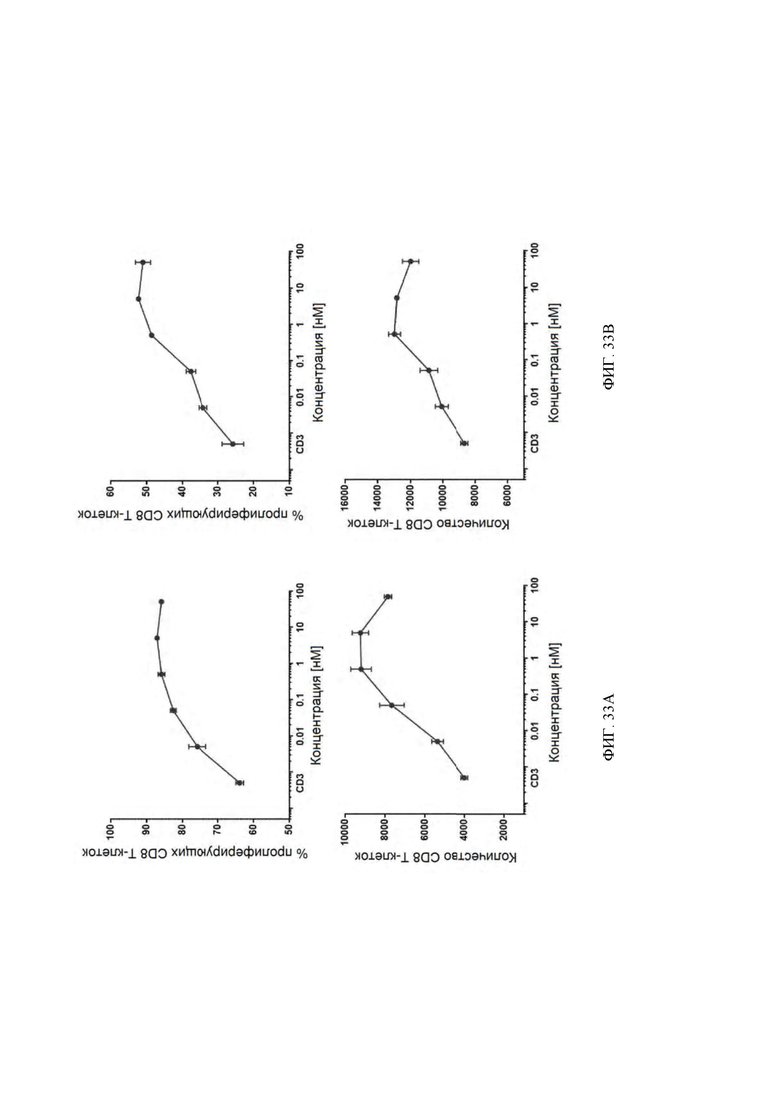
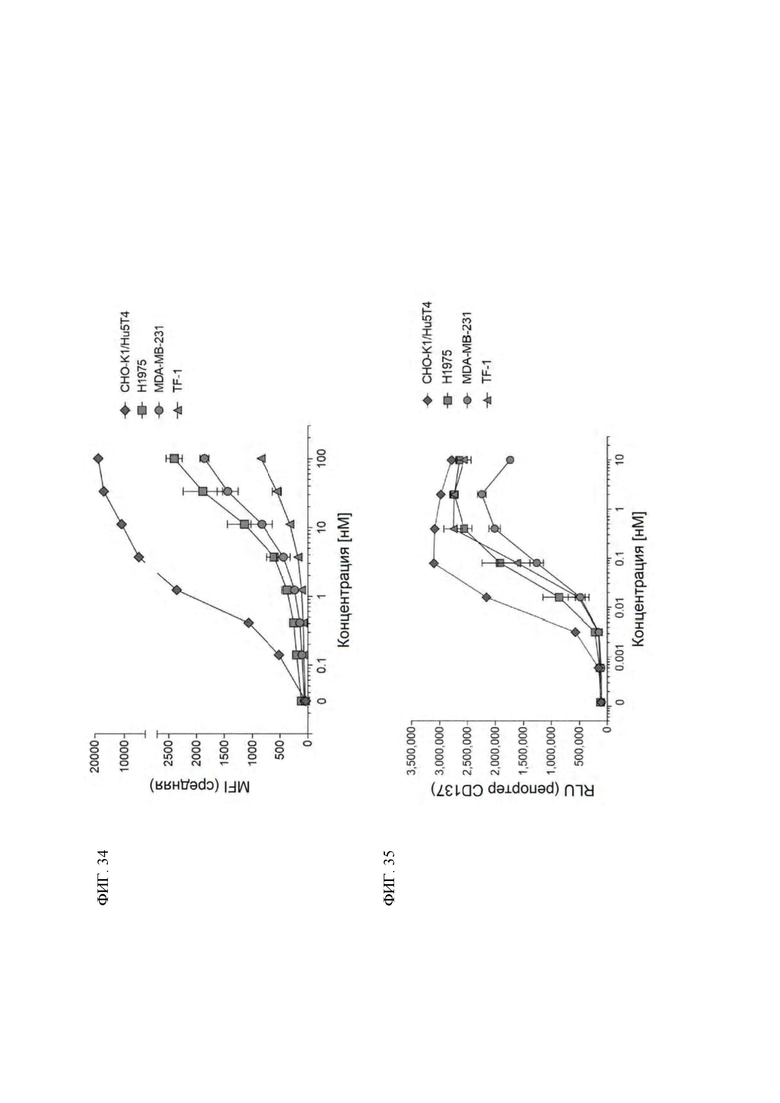
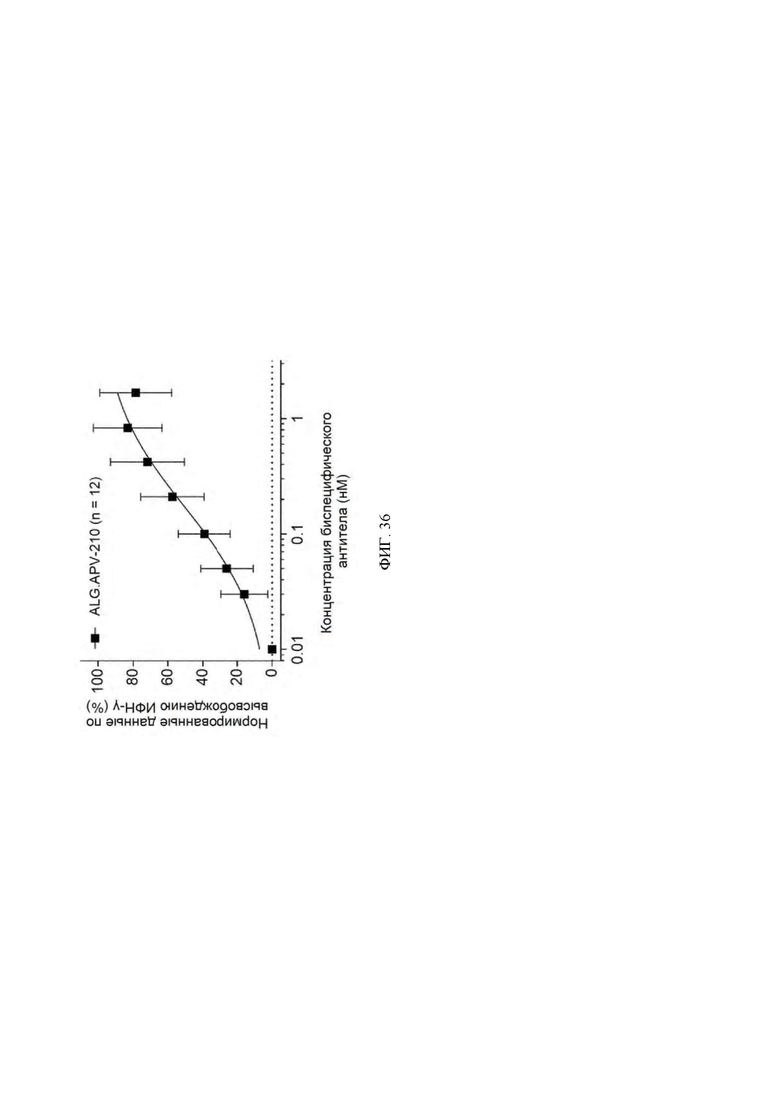
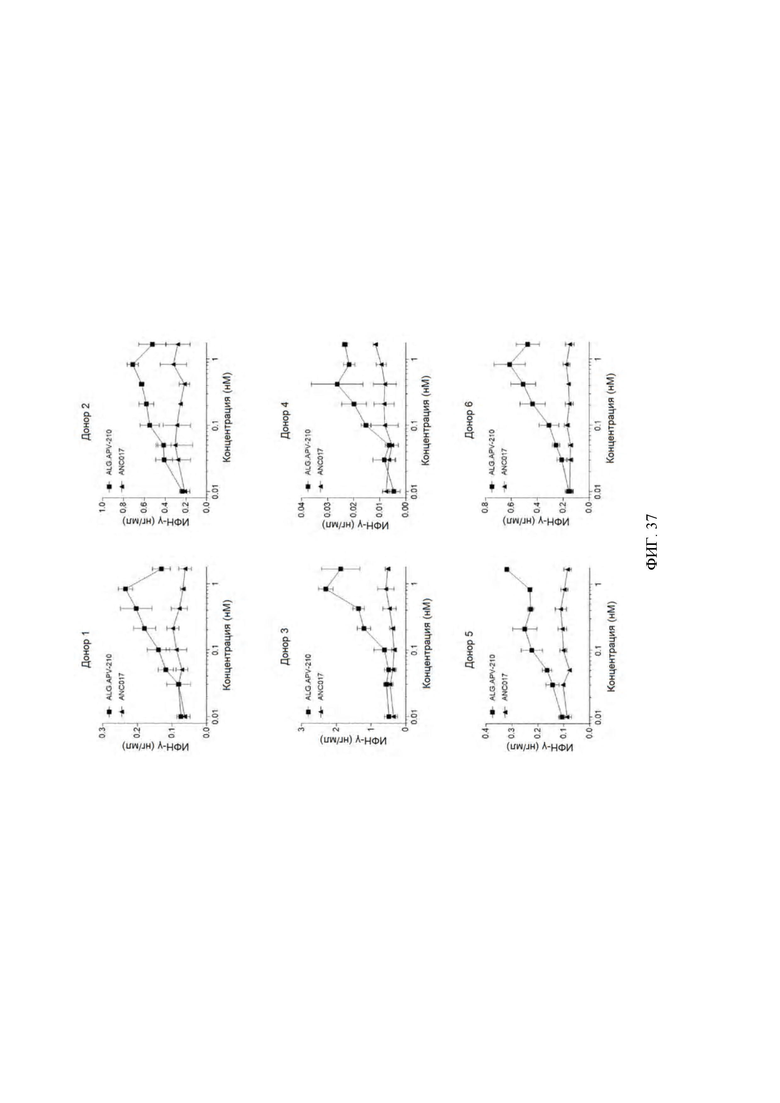
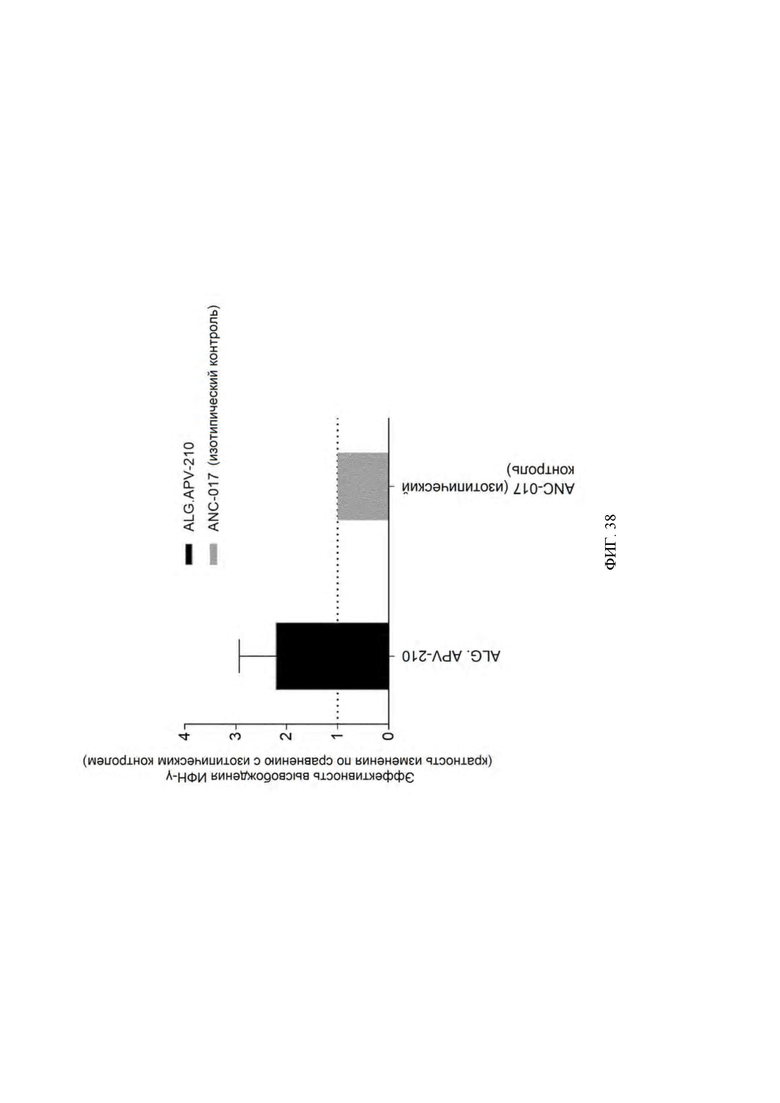
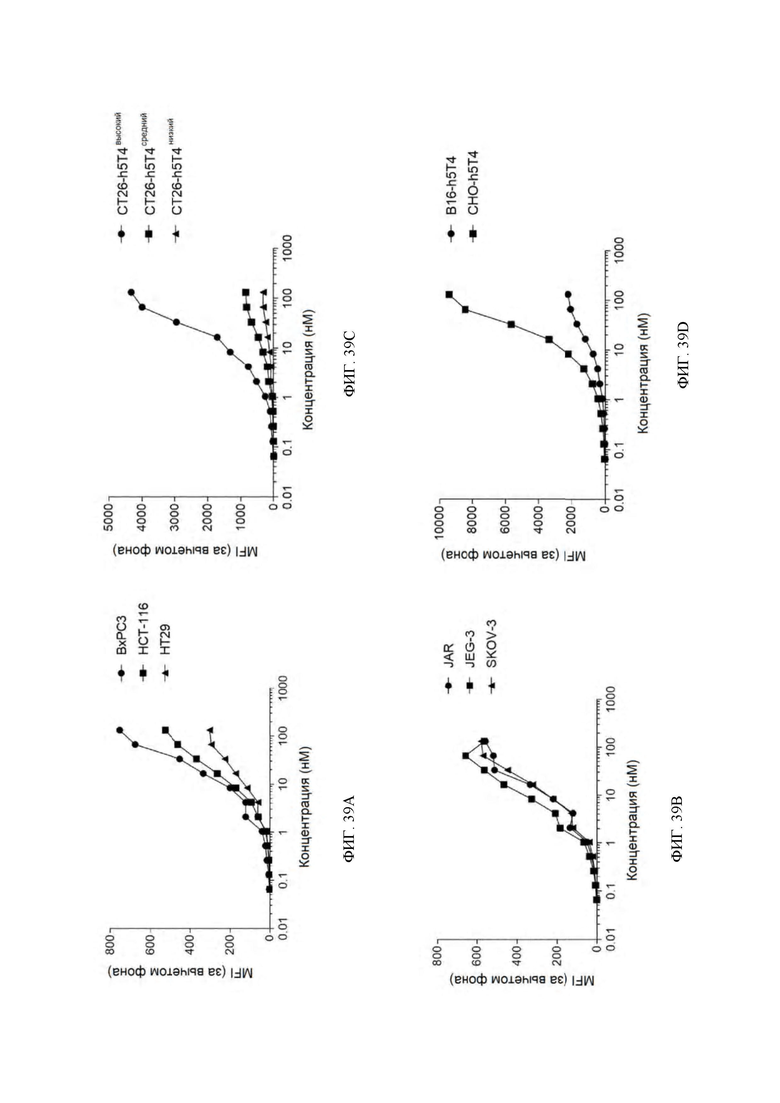
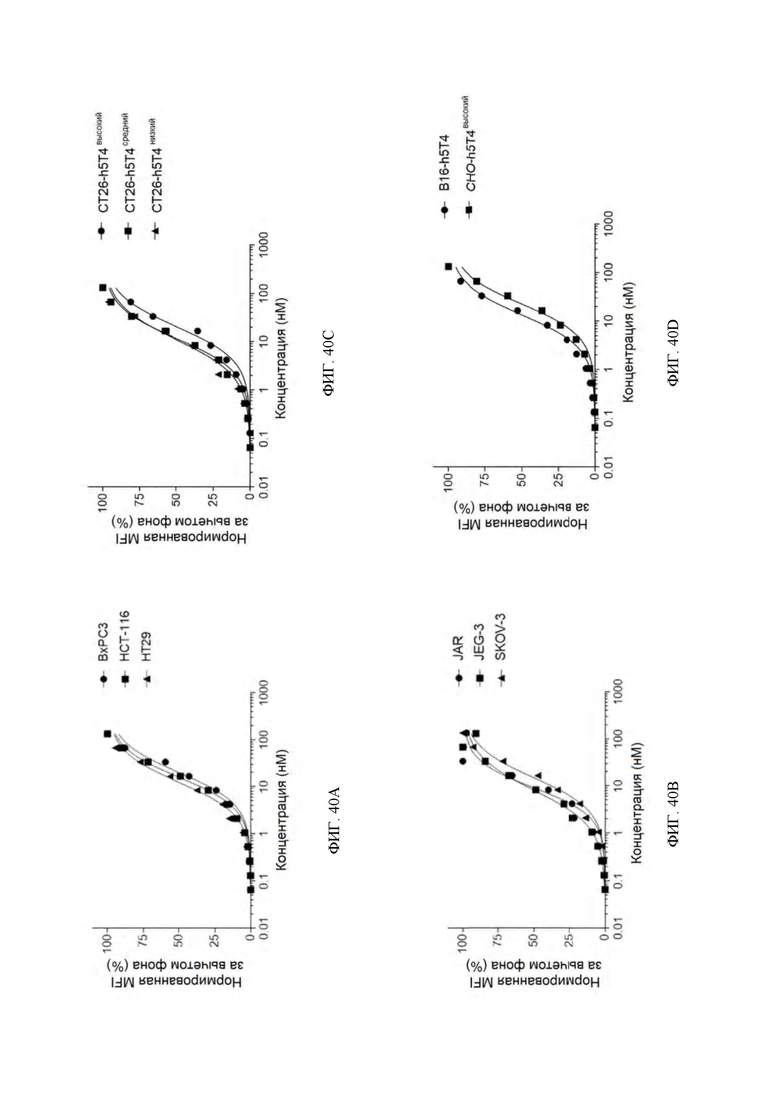
Изобретение относится к области биохимии, в частности к биспецифичному полипептиду для применения при лечении расстройства, ассоциированного с экспрессией 5T4. Также раскрыт способ лечения рака у субъекта с помощью указанного антитела. Изобретение позволяет эффективно лечить рак, характеризующийся экспрессией 5T4. 2 н. и 18 з.п. ф-лы, 40 ил., 23 пр., 33 табл.
1. Биспецифичный полипептид для применения при лечении расстройства, ассоциированного с экспрессией 5T4, содержащий первый домен одноцепочечного вариабельного фрагмента (scFv) и второй домен одноцепочечного вариабельного фрагмента (scFv), соединённые вместе с помощью линкера связывающего домена, и Fc домен иммуноглобулина, где указанный Fc домен иммуноглобулина содержит шарнирную область и константную область иммуноглобулина, причём указанный биспецифичный полипептид способен образовывать гомодимер посредством ассоциации со вторым идентичным биспецифичным полипептидом,
при этом указанный первый домен scFv специфичным образом связывается с 5T4 человека и содержит:
(i) вариабельную область тяжелой цепи иммуноглобулина, содержащую определяющий комплементарность участок тяжелой цепи (HCDR) 1 с аминокислотной последовательностью SEQ ID NO: 30, HCDR2 с аминокислотной последовательностью SEQ ID NO: 32 и HCDR3 с аминокислотной последовательностью SEQ ID NO: 34; и
(ii) вариабельную область легкой цепи иммуноглобулина, содержащую определяющий комплементарность участок легкой цепи (LCDR) 1 с аминокислотной последовательностью SEQ ID NO: 42, LCDR2 с аминокислотной последовательностью SEQ ID NO: 10 и LCDR3 с аминокислотной последовательностью SEQ ID NO: 36; и
при этом указанный второй домен scFv специфичным образом связывается с 4-1BB человека и содержит:
(i) вариабельную область тяжелой цепи иммуноглобулина, содержащую HCDR1 с аминокислотной последовательностью SEQ ID NO: 2, HCDR2 с аминокислотной последовательностью SEQ ID NO: 4 и HCDR3 с аминокислотной последовательностью SEQ ID NO: 6; и
(ii) вариабельную область легкой цепи иммуноглобулина, содержащую LCDR1 с аминокислотной последовательностью SEQ ID NO: 8, LCDR2 с аминокислотной последовательностью SEQ ID NO: 10 и LCDR3 с аминокислотной последовательностью SEQ ID NO: 12.
2. Биспецифичный полипептид по п. 1, отличающийся тем, что указанный биспецифичный полипептид содержит, в направлении от аминоконца к карбоксиконцу, (i) первый домен scFv, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) второй домен scFv.
3. Биспецифичный полипептид по п. 1, где первый домен scFv содержит мутацию в каркасной области по сравнению с каркасной областью с последовательностью SEQ ID NO: 130 или SEQ ID NO: 170.
4. Биспецифичный полипептид по п. 3, где указанная мутация приводит к появлению стабилизирующей дисульфидной связи.
5. Биспецифичный полипептид по п. 1, где указанный первый домен scFv содержит вариабельную область тяжелой цепи иммуноглобулина с последовательностью SEQ ID NO: 38 и вариабельную область легкой цепи иммуноглобулина с последовательностью SEQ ID NO: 44.
6. Биспецифичный полипептид по п. 1, где указанный первый домен scFv содержит вариабельную область тяжелой цепи иммуноглобулина с последовательностью SEQ ID NO: 46 и вариабельную область легкой цепи иммуноглобулина с последовательностью SEQ ID NO: 48.
7. Биспецифичный полипептид по п. 1, где указанный второй домен scFv содержит вариабельную область тяжелой цепи иммуноглобулина с последовательностью SEQ ID NO: 14 и вариабельную область легкой цепи иммуноглобулина с последовательностью SEQ ID NO: 16.
8. Биспецифичный полипептид по п. 1, где указанный первый домен scFv содержит вариабельную область тяжелой цепи иммуноглобулина с последовательностью SEQ ID NO: 46 и вариабельную область легкой цепи иммуноглобулина с последовательностью SEQ ID NO: 48; и при этом указанный второй домен scFv содержит вариабельную область тяжелой цепи иммуноглобулина с последовательностью SEQ ID NO: 14 и вариабельную область легкой цепи иммуноглобулина с последовательностью SEQ ID NO: 16.
9. Биспецифичный полипептид по п. 1, где аминокислотная последовательность указанного первого домена scFv содержит SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 42, SEQ ID NO: 10 и SEQ ID NO: 36 и имеет по меньшей мере 97% идентичности с SEQ ID NO: 120; и аминокислотная последовательность указанного второго домена scFv содержит SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10 и SEQ ID NO: 12 и имеет по меньшей мере 97% идентичности с SEQ ID NO: 110.
10. Биспецифичный полипептид по п. 9, где указанный полипептид содержит первый домен scFv, содержащий SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 42, SEQ ID NO: 10 и SEQ ID NO: 36, и второй домен scFv, содержащий SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10 и SEQ ID NO: 12, и содержит аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности SEQ ID NO: 172; или содержит аминокислотную последовательность, которая идентична последовательности SEQ ID NO: 172.
11. Биспецифичный полипептид по п. 1, где аминокислотная последовательность указанного первого домена scFv содержит SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 42, SEQ ID NO: 10 и SEQ ID NO: 36 и имеет по меньшей мере 97% идентичности с SEQ ID NO: 122; и аминокислотная последовательность указанного второго домена scFv содержит SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10 и SEQ ID NO: 12 и имеет по меньшей мере 97% идентичности с SEQ ID NO: 110.
12. Биспецифичный полипептид по п. 11, где указанный полипептид содержит первый домен scFv, содержащий SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 42, SEQ ID NO: 10, и SEQ ID NO: 36 и второй домен scFv, содержащий SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10 и SEQ ID NO: 12, и содержит аминокислотную последовательность, которая по меньшей мере на 95% идентична последовательности SEQ ID NO: 174; или содержит аминокислотную последовательность SEQ ID NO: 174.
13. Биспецифичный полипептид по п. 1, где связывание биспецифичного полипептида с эффекторной клеткой приводит к увеличению активации эффекторной клетки, увеличению пролиферации эффекторной клетки, или где связывание биспецифичного полипептида с эффекторной клеткой и 5T4-экспрессирующей клеткой усиливает зависимый от эффекторной клетки лизис указанной 5T4-экспрессирующей клетки.
14. Биспецифичный полипептид по п. 1, где указанный полипептид демонстрирует статистически значимо усиленную активацию эффекторных клеток по сравнению со вторым биспецифичным полипептидом, причём указанный второй биспецифичный полипептид представляет собой структуру IgG-scFv, содержащую антитело к 4-1BB, содержащее вариабельную область тяжелой цепи, содержащую SEQ ID NO: 28, и вариабельную область легкой цепи, содержащую SEQ ID NO: 16, и scFv к 5T4, содержащую вариабельную область тяжелой цепи, содержащую SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую SEQ ID NO: 66.
15. Биспецифичный полипептид по п. 1, где указанный полипептид индуцирует статистически значимо увеличенную пролиферацию эффекторных клеток по сравнению со вторым биспецифичным полипептидом, причём указанный второй биспецифичный полипептид представляет собой структуру IgG-scFv, содержащую антитело к 4-1BB, содержащее вариабельную область тяжелой цепи, содержащую SEQ ID NO: 28, и вариабельную область легкой цепи, содержащую SEQ ID NO: 16, и scFv к 5T4, содержащую вариабельную область тяжелой цепи, содержащую SEQ ID NO: 46, и вариабельную область легкой цепи, содержащую SEQ ID NO: 66.
16. Способ лечения рака у нуждающегося в этом субъекта, включающий введение указанному субъекту терапевтически эффективного количества биспецифичного полипептида по п. 1, где указанный рак характеризуется экспрессией 5T4.
17. Способ по п. 16, где указанный рак представляет собой рак молочной железы, рак поджелудочной железы, рак яичника, немелкоклеточный рак легкого, мезотелиому, хронический лимфоцитарный лейкоз (ХЛЛ), мантийноклеточный лейкоз (МКЛ), острый лимфобластный лейкоз (ОЛЛ), плоскоклеточную карциному, меланому, рак надпочечника, рак мочевого пузыря, рак шейки матки, рак почки, рак желудка, рак предстательной железы, рак щитовидной железы, рак печени, рак матки, нейрофиброму, саркому, карциному или рак головы и шеи.
18. Способ по п. 16, где указанный биспецифичный полипептид содержит в направлении от аминоконца к карбоксиконцу: (i) первый домен scFv, (ii) шарнирную область, (iii) константную область иммуноглобулина, (iv) линкер связывающих доменов и (v) второй домен scFv.
19. Способ по п. 16, где указанный второй домен scFv содержит вариабельную область тяжелой цепи иммуноглобулина с последовательностью SEQ ID NO: 14 и вариабельную область легкой цепи иммуноглобулина с последовательностью SEQ ID NO: 16, и при этом указанный первый домен scFv содержит:
(i) вариабельную область тяжелой цепи иммуноглобулина с последовательностью SEQ ID NO: 38, и вариабельную область легкой цепи иммуноглобулина с последовательностью SEQ ID NO: 44; или
(ii) вариабельную область тяжелой цепи иммуноглобулина с последовательностью SEQ ID NO: 46, и вариабельную область легкой цепи иммуноглобулина с последовательностью SEQ ID NO: 48.
20. Способ по п. 16, где аминокислотная последовательность указанного второго домена scFv указанного биспецифичного полипептида содержит SEQ ID NO: 2, SEQ ID NO: 4, SEQ ID NO: 6, SEQ ID NO: 8, SEQ ID NO: 10 и SEQ ID NO: 12 и имеет по меньшей мере 97% идентичности с SEQ ID NO: 110; и при этом аминокислотная последовательность указанного первого домена scFv указанного биспецифичного полипептида содержит SEQ ID NO: 30, SEQ ID NO: 32, SEQ ID NO: 34, SEQ ID NO: 42, SEQ ID NO: 10 и SEQ ID NO: 36 и имеет по меньшей мере 97% идентичности с SEQ ID NO: 120 или SEQ ID NO: 122.
| WO 2013123061 A1, 22.08.2013 | |||
| US 20150322155 A1, 12.11.2015 | |||
| WO 2016033331 A1, 03.03.2016 | |||
| АНТИТЕЛА ПРОТИВ 5Т4 И ИХ ПРИМЕНЕНИЕ | 2007 |
|
RU2487889C2 |
Авторы
Даты
2023-10-02—Публикация
2018-07-20—Подача