На сегодняшний день, согласно данным The Joint United Nations Programme on HIV/AIDS (UNAIDS), в мире зарегистрированы 31,6 - 44,5 миллиона человек, живущих с вирусом иммунодефицита человека (ВИЧ), при этом число новых заражений за 2022 год составило 1,2-2,2 млн. случаев 1,3 млн. случаев [UNAIDS Data 2023. Geneva: Joint United Nations Programme on HIV/AIDS (UNAIDS)]. Российская Федерация (РФ) на сегодняшний день является одним из регионов с максимальными темпами развития эпидемии. По данным Министерства Здравоохранения РФ количество случаев ВИЧ-инфекции в стране превышает 408 тыс.чел. Однако Минздрав учитывает только тех пациентов, которые состоят на диспансерном наблюдении. Реальное число лиц с ВИЧ в стране оценивается UNAIDS в пределах 0,8-1,2 млн. чел. Эпидемия носит концентрированный характер, что означает распространенность ВИЧ преимущественно в группах риска. ВИЧ-инфекция в России относится к так называемым социально значимым заболеваниям, опасным для окружающих, а также признана угрозой национальной безопасности. На 31 декабря 2022 г. в стране проживало 1168076 россиян с лабораторно подтвержденным диагнозом ВИЧ-инфекции. В 2022 г. было зарегистрировано 63150 новых случая болезни, вызванной вирусом иммунодефицита человека (ВИЧ), в том числе 615 случаев среди детей и 12913 - у жителей сельских поселений [Федеральный научно-методический центр по профилактике и борьбе со СПИДом. Справка ВИЧ в России на 31.12.2022.].
В рамках государственного эпидемиологического надзора за ВИЧ-инфекцией, должно проводиться обязательное эпидрасследование каждого нового случая ВИЧ-инфекции. Оно направлено на выявление источника инфекции и проведение мероприятий, с целью предотвращения дальнейшего распространения ВИЧ-инфекции [Методические указания МУ 3.1.3342-16 "Эпидемиологический надзор за ВИЧ-инфекцией" (утв. Главным государственным санитарным врачом РФ 26 февраля 2016 г.)]. В рамках проводимого расследования часто должна быть решена задача по установлению эпидемиологической связи между пациентом и предполагаемым источником инфекции. В настоящее время данная задача решается при помощи сбора информации, содержащейся в официальных документах и определения факторов риска заражения ВИЧ-инфекцией. Полученные сведения позволяют установить круг контактных лиц и предположить вероятный источник инфекции.
В тоже время, в связи с активным развитием таких молекулярных методов, как определение нуклеотидных последовательностей участков генома ВИЧ, появился новый инструмент, позволяющий предоставить дополнительную информацию о взаимосвязи между пациентом и предполагаемым источником инфекции. Как известно, ВИЧ обладает высоким уровнем генетической изменчивости. Поэтому применение эволюционного филогенетического анализа последовательностей позволяет определить степень генетического родства, выявляя минимальные различия в генах ВИЧ и определяя генетическую дистанцию между штаммами. Зачастую вследствие невозможности обнаружения штаммов вируса с идентичным геномом, анализ в первую очередь применятся для исключения возможных источников инфекции. Однако в ряде случаев использование молекулярных методов вместе с анализом эпидемиологической информации, позволяет с большей долей вероятности установить предполагаемый источник инфекции.
Филогенетический анализ является одним из важных инструментов получения объективных данных о наличии/отсутствии эпидемиологической связи, в том числе и при расследовании криминальных случаев заражения ВИЧ-инфекцией.
Использование филогенетического анализа при эпидемиологических расследованиях в России регламентируется методическими указаниями МУ 3.1.3342-16 "Эпидемиологический надзор за ВИЧ-инфекцией" от 26.02.2016 г.
Для определения связи между биологическими образцами от пациентов предположительно из одной эпидемиологической цепи (исследуемая группа), необходимо провести генотипирование образцов крови всех инфицированных ВИЧ из предполагаемого очага передачи ВИЧ-инфекции, а также образцов крови от инфицированных лиц из группы сравнения. В исследуемую группу необходимо включить пострадавшего/их и лицо/лиц, потенциально являющихся для него источником инфекции. В группу сравнения (контрольная группа) необходимо включить не менее 20-30 образцов крови, полученных от ВИЧ-инфицированных из того же географического региона, социальной группы, группы риска заражения и инфицированных ВИЧ в близкие (к исследуемой группе) сроки. Дополнительно в качестве контрольной группы возможно использовать охарактеризованные нуклеотидные последовательности из баз данных, подобранных по аналогичным критериям [Методические указания МУ 3.1.3342-16 "Эпидемиологический надзор за ВИЧ-инфекцией" (утв. Главным государственным санитарным врачом РФ 26 февраля 2016 г.)]. Данный алгоритм выбран за прототип.
По причине большей стандартизации рекомендуется использование наборов реагентов, предназначенных для определения лекарственной устойчивости ВИЧ, в том случае если они позволяют получить нуклеотидные последовательности в необходимом для анализа объеме [Методические указания МУ 3.1.3342-16 "Эпидемиологический надзор за ВИЧ-инфекцией" (утв. Главным государственным санитарным врачом РФ 26 февраля 2016 г.)].
Основными недостатками описанного в нормативных документах алгоритма является труднодоступность достаточного количества референсных последовательностей из того же географического региона, а также ограниченность нуклеотидной последовательности, получаемой при помощи коммерческих наборов, используемых для определения лекарственной устойчивости. Следствием этого может быть недостаточная разрешающая способность используемых методов, что сделает затруднительным определение филогенетической связи между штаммами ВИЧ, полученными от разных пацеинтов.
Авторами предложен способ определения возможного источника ВИЧ-инфекции при молекулярно-эпидемиологических расследованиях с использованием набора олигодезоксирибонуклеотидных праймеров и разработанной региональной базы данных, зарегистрированной авторами ранее (RU 2022622009).
Технический результат - повышение разрешающей способности филогенетического анализа при определении возможного источника ВИЧ-инфекции.
Этот результат достигается за счет секвенирования расширенного фрагмента генома ВИЧ и использования в качестве дополнительных референсных последовательностей фрагментов генома ВИЧ из региональной базы данных, полученных при анализе собранных ранее образцов из того же географического региона и с известными анамнестическими характеристиками
Сущность метода заключается в том, что на первом этапе проводят амплификацию кДНК вируса с использованием олигонуклеотидных праймеров (Таблица 1), комплементарных областям наибольшего сходства геномов различных изолятов ВИЧ, продуктом амлификации является фрагмент генома ВИЧ, включающий гены gag и pol протяженностью 4,8×103 пнт.

Для повышения чувствительности проводят вторую полимеразную цепную реакцию с использованием продукта амплификации первой реакции, внутренних (вложенных, гнездовых) праймеров для ВИЧ, комплементарных участкам амплифицируемых на втором этапе фрагментов (Таблица 2).

Состав амплификационной смеси на первом этапе:
0,1 μМ прямого и обратного праймеров, 0.2 mM dNTP's, 2,5 ммоль/л MgCl2, 1 ед. рекомбинантной Taq ДНК-полимеразы (Fermentas), буфер для Taq ДНК-полимеразы (750 ммоль/л Трис-HCl, (рН 8,8), 200 ммоль/л (NH4)2So4, 0,1% (v/v) твин 20), глицерин 5% от конечного объема, 20 мкл раствора ДНК, вода без нуклеаз до конечного объема 40 мкл. Условия проведения ПНР представлены в таблице 3.

Состав амплификационной смеси на втором этапе:
0,1 μМ прямого и обратного праймеров, 0.2 mM dNTP's, 2,5 ммоль/л MgCl2, 1 ед. рекомбинантной Taq ДНК-полимеразы (Fermentas), буфер для Taq ДНК-полимеразы (750 ммоль/л Трис-HCl, (рН 8,8), 200 ммоль/л (NH4)2SO4, 0,1% (v/v) твин 20), глицерин 5% от конечного объема, 5 мкл продукта амплификации из первой ПЦР, вода без нуклеаз до конечного объема 25 мкл.
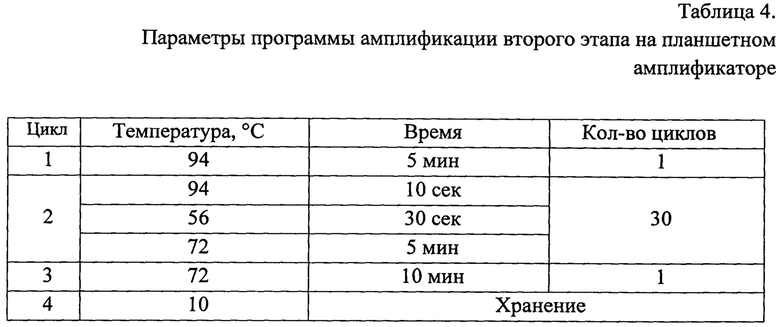
Далее следует этап очищения продуктов амплификации методом спиртового осаждения в присутствии ацетата натрия, для чего смесь из 2 мкл 3 М ацетата натрия, 2 мкл 0,125 М EDTA и 1 мкл гликогена вносили в 20 мкл продукта амплификации и инкубировали при комнатной температуре в присутствии охлажденного 96% этилового спирта 15 минут. Центрифугировали при 14000 об/мин, 4°С 15 мин супернатан удаляли и дважды промывали осадок охлажденным 70% этиловым спиртом, повторяя процедуру центрифугирования на холоде. Промытый осадок сушили.
Для анализа качества очищения продуктов амплификации осадок растворяли в 30 мкл ТЕ-буфера и визуализировали в агарозном геле. Концентрацию ПК измеряли на флюориметре Qubit 2.0 по стандартной методике, рекомендованной производителем.
Очищенный фрагмент с концентрацией 50-100 нг в зависимости от нуклеотидного состава анализируемого участка использовали для постановки секвенирующих реакций с использованием праймеров для секвенирования (таблица 5).


Для проведения секвенирующей реакции использовали набор реагентов ABI PRISM BigDye Terminator v3.1. (Applied Biosystems, США), согласно инструкции производителя. Для этого готовили смесь для секвенирующей реакции следующего состава: матрица 0,2-8,0 мкл, праймер для секвенирования 3,0 мкл (концентрация 1,6 пмол/мкл), ABI PRISM BigDye Terminator v 3.1 Kit 8 мкл, деионизованная вода до конечного объема смеси 20 мкл. Секвенирующую реакцию проводили при условиях: устанавливали 25 циклов амплификации в режиме: 96°С - 10 сек, 50°С - 5 сек, 60°С - 4 мин. Продукты секвенирующей реакции очищали с использованием вышеприведенных методов, очищенный осадок растворяли в растворе формамида помещали в генетический анализатор ABI PRISM 3500 (Applied Biosystems, США)
Выравнивание нуклеотидных последовательностей проводили в программе MEGA версия X, используя алгоритм ClustalW. Поскольку для выбранных для секвенирования регионов показана высокая скорость эволюции, для построения филогенетических деревьев и последующего филогенетического анализа использовали метод присоединения соседей (Neighbor-joining), позволяющий оптимизацию дерева в соответствии с критерием «сбалансированной минимальной эволюции». Визуализацию и оформление филогенетических деревьев осуществляли в программе MEGA версия X. Для оценки достоверности реконструированной топологии построенных филогенетических деревьев проводили бутстреп (bootstrap) анализ (1000 повторностей) [Kumar S, Stecher G, Li M, Knyaz С, Tamura K. MEGA X: Molecular Evolutionary Genetics Analysis across Computing Platforms. Mol Biol Evol. 2018. 35(6): 1547-1549. doi: 10.1093/molbev/msy096].
Для расширения возможностей филогенетического анализа, кроме последовательностей, полученных из образцов, направленных для генотипирования, в анализ включаются последовательности из региональной базы данных, зарегистрированной ранее.
Сущность изобретения поясняется чертежом, где на фиг.1 представлены результаты филогенетического анализа генетических последовательностей ВИЧ, полученных с использованием предложенного способа совместно с образцами из региональной базы последовательностей. На филогенетическом дереве тестируемые образцы обведены прямоугольником. Образцы, полученные из одного региона с тестируемыми образцами, обозначены маркировкой VG, последовательности ВИЧ, взятые из региональной базы данных, отмечены маркировкой LO.
Ниже приведен пример конкретного использования предложенного способа.
Пример 1.
В 2023 году были получены образцы плазмы крови от ВИЧ-инфицированных матери и дочери для установления возможности инфицирования из одного источника. Для исследования были также привлечены дополнительные образцы плазмы крови ВИЧ-инфицированных из того же региона.
Применение набора олигонуклеотидных праймеров позволило успешно провести ПЦР с накоплением необходимого фрагмента кДНК ВИЧ, а также выполнить секвенирование и получить последовательности исследуемых фрагментов для тестируемых и контрольных образцов.
Для увеличения разрешающей способности исследования из Базы данных пациентов с вирусологической неэффективностью АРВТ в СЗФО были взяты дополнительные контрольные последовательности ВИЧ от пациентов из Ленинградской области.
Проведение филогенетического анализа позволило установить, что с высокой степенью вероятности инфицирование произошло из одного источника. Полученные данные филогенетического анализа соответствовали другим эпидемиологическим данным, что подтверждает работоспособность используемого метода.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Перечень
последовательностей.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2023-11-26">
<ApplicationIdentification>
<IPOfficeCode></IPOfficeCode>
<ApplicationNumberText>-</ApplicationNumberText>
<FilingDate></FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>-</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное бюджетное учреждение
науки «Санкт-Петербургский научно-исследовательский институт
эпидемиологии и микробиологии им. Пастера» Федеральной службы по
надзору в сфере защиты прав потребителей и благополучия
человека</ApplicantName>
<ApplicantNameLatin>Saint-Petersburg Pasteur
Institute</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ определения источника
ВИЧ-инфекции при молекулярно-эпидемиологических расследованиях с
использованием набора олигодезоксирибонуклеотидных праймеров и
разработанной региональной базы данных</InventionTitle>
<SequenceTotalQuantity>8</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccctcaaatcactctttggc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccacacaatcatcacctgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcgactggtgagtacgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcttctggggcttgttcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgaatgggagtttgtcaatacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctccctggcagaatgcctatt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaagccatgcatggacaag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atcctcatcctgtctacttgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

Изобретение относится к медицине и, в частности, к вирусологии. Предложен способ определения источника ВИЧ-инфекции при молекулярно-эпидемиологических расследованиях. Способ включает проведение вложенной двухэтапной ПЦР с набором праймеров для 1 этапа hivoutf 5'-gcgactggtgagtacgcc-3', 5'-tcttctggggcttgttcc-3' и для 2 этапа hivinf 5'-ccctcaaatcactctttggc-3' и hivinr 5'-ccacacaatcatcacctgcc-3' с получением длинного фрагмента генома ВИЧ, включающего гены gag и pol, секвенирование и получение последовательности исследуемых фрагментов, проведение филогенетического анализа с включением последовательностей из Базы данных пациентов с вирусологической неэффективностью АРВТ в СЗФО с последующим установлением возможного источника инфицирования. Изобретение позволяет повысить разрешающую способность филогенетического анализа при определении возможного источника ВИЧ-инфекции. 1 ил., 5 табл., 1 пр.
Способ определения источника ВИЧ-инфекции при молекулярно-эпидемиологических расследованиях, включающий проведение вложенной двухэтапной ПЦР с набором праймеров для 1 этапа hivoutf 5'-gcgactggtgagtacgcc-3', 5'-tcttctggggcttgttcc-3' и для 2 этапа hivinf 5'-ccctcaaatcactctttggc-3' и hivinr 5'-ccacacaatcatcacctgcc-3' с получением длинного фрагмента генома ВИЧ, включающего гены gag и pol, секвенирование и получение последовательности исследуемых фрагментов, проведение филогенетического анализа с включением последовательностей из Базы данных пациентов с вирусологической неэффективностью АРВТ в СЗФО с последующим установлением возможного источника инфицирования.
| Переносная печь для варки пищи и отопления в окопах, походных помещениях и т.п. | 1921 |
|
SU3A1 |
| Прибор для получения стереоскопических впечатлений от двух изображений различного масштаба | 1917 |
|
SU26A1 |
| Sergei L Kosakovsky Pond et al., HIV-TRACE (TRAnsmission Cluster Engine): a Tool for Large Scale Molecular Epidemiology of HIV-1 and Other Rapidly Evolving Pathogens, Molecular | |||
Авторы
Даты
2024-11-22—Публикация
2023-12-04—Подача