[Область изобретения]
Описание настоящего изобретения относится к микроорганизму, продуцирующему микоспорин-подобную аминокислоту, и к способу получения микоспорин-подобной аминокислоты с использованием микроорганизма.
[Предшествующий уровень техники]
Ультрафиолетовые (УФ) лучи, излучаемые солнцем, состоят из УФ-А (с длиной волны от приблизительно 320 нм до приблизительно 400 нм), УФ-В (с длиной волны от приблизительно 290 нм до приблизительно 320 нм) и УФ-С (с длиной волны от приблизительно 100 нм до приблизительно 280 нм). Известно, что лучи УФ-А проникают в слой кожи, в основном вызывая пигментацию и старение кожи, и вовлечены в возникновение светочувствительного заболевания кожи, тогда как лучи УФ-В представляют собой лучи, несущие высокий уровень энергии, которые проникают в эпидермис и базальный слой дермы и вовлечены в возникновение солнечных ожогов, пигментацию и возникновение рака кожи.
Для того, чтобы предотвратить эти побочные действия УФ лучей, предпринимаются попытки блокировать УФ лучи. Типы солнцезащитных агентов включают химические солнцезащитные агенты и физические солнцезащитные агенты. Химические солнцезащитные агенты предотвращают проникновение УФ лучей в основном путем поглощения УФ лучей, тогда как физические солнцезащитные агенты предотвращают проникновение УФ лучей путем отражения и рассеяния.
Компоненты, которые, как известно, содержатся в химических солнцезащитных агентах, могут включать агенты, которые в основном поглощают лучи УФ-В (например РАВА (пара-аминобензойная кислота), сложные эфиры РАВА (амилдиметил-РАВА, октилдиметил-РАВА), циннаматы (циноксат), салицилаты (гомоментил-салицилат), камфора и т.п.); и агенты, которые в основном поглощают лучи УФ-А (например бензофеноны (оксибензон, диоксибензон и сульфобензол), дибензоилметан, антранилат и т.п.). Хотя эти химические солнцезащитные агенты могут обеспечить действия, поглощающие и блокирующие УФ лучи, известно, что некоторые из этих химических солнцезащитных агентов могут раздражать кожу или глаза, и, в частности, РАВА, сложные эфиры РАВА, бензофеноны и циннаматы и т.п.могут вызывать контактный дерматит.Дополнительно, сообщалось о том, что другие из этих химических солнцезащитных агентов вызывают светочувствительную реакцию на коже и т.п.Соответственно, в некоторых странах применение или объем использования этих химических солнцезащитных агентов ограничен.
Компоненты, которые, как известно, содержатся в физических солнцезащитных агентах, могут включать диоксид титана, тальк (силикат магния), оксид магния, оксид цинка, каолин и т.п. Физические солнцезащитные агенты обладают преимуществами, заключающимися в том, что они не вызывают побочные действия, такие как контактный дерматит, и что они не удаляются легко водой. Тем не менее, они обладают недостатками, заключающимися в том, что сложно с их помощью поддерживать эффективное содержание при использовании желаемых композиций и что возникает белый налет и т.п. при их нанесении на кожу.
Микоспорин-подобные аминокислоты (МАА) представляют собой вещества, представленные в организмах в природе и они, как известно, эффективно поглощают УФ-А и УФ-В. Известно, что в природе существует более 35 видов МАА {Mar. Biol., 1991, 108: 157-166; Planta Med., 2015, 81: 813-820). Недавно сообщали о том, что МАА, к которым прикреплены различные типы Сахаров, существуют в микроводорослях, и они обладают превосходной антиоксидантной функцией (Journal of Photochemistry and Photobiology, 2015, 142: 154-168). Дополнительно, МАА, как известно, обеспечивают не только способность блокировать УФ лучи, но также устойчивость к окислению, осмосу, температурному стрессу и т.п. (Comp. Biochem. Physiol.C Toxicol. Pharmacol., 2007, 146: 60-78; J. Photochem. Photobiol. В., 2007, 89: 29-35).
Тем не менее, количество МАА, продуцируемое в микроводорослях, является очень низким, находясь на уровне нескольких микрограмм, и условия для разделения, экстракции и очистки МАА после культивирования микроводорослей затруднены. Таким образом, сложно массово продуцировать материал МАА.
[Документы предшествующего уровня техники]
[Непатентные документы]
(Непатентный документ 1) Comp. Biochem. Physiol. В 1995, 112: 105-114.
(Непатентный документ 2) FEMSMicrobiol Lett. 2007,269: 1-10.
(Непатентный документ 3) Ann.Rev. Physiol. 2002, 64: 223-262.
Непатентный документ 4) Mar. Biol. 1991, 108: 157-166.
(Непатентный документ 5) Journal of Photochemistry and Photobiology В: Biology. 2015, 142: 154-168
(Непатентный документ 6) Biol. Rev. 1999, 74: 311-345.
(Непатентный документ 7) Mol. Biol. Evol. 2006, 23: 1437-1443.
(Непатентный документ 8) Science, 2010, 329: 1653-1656.
(Непатентный документ 9) Genomics 2010, 95: 120-128.
(Непатентный документ 10) Geomicrobiol. J. 1997. 14: 231-241.
(Непатентный документ 11) Comp. Biochem. Physiol. С Toxicol. Pharmacol. 2007. 146: 60-78.
(Непатентный документ 12) Can. J. Bot. 2003. 81: 131-138.
(Непатентный документ 13) J. Photochem. Photobiol. B. 2007, 89: 29-35.
(Непатентный документ 14) J. Bacteriol. 2011. 193(21): 5923-5928.
(Непатентный документ 15) Planta Med. 2015. 81: 813-820
(Непатентный документ 16) ACS Appl. Mater. Interfaces. 2015. 7: 16558-16564
(Непатентный документ 11) Appl Environ Microbiol. 2016, 82(20): 6167-6173
(Непатентный документ 18) ChemBioChem. 2015, 16: 320-327
(Непатентный документ 19) MethodsMolBiol. 2013, 1073: 43-7
(Непатентный документ 20) Nature Review, 2011, 9: 791-802
[Описание изобретения]
[Техническая проблема]
Авторы настоящего изобретения предприняли множество попыток для увеличения продукции МАА в микроорганизмах. В результате они подтвердили то, что продукция МАА может быть увеличена в микроорганизмах, продуцирующих МАА, при помощи различных исследований, ассоциированных с усилением активности белков 2-дегидро-3-дезоксифосфогептонатальдолазы, фосфоенолпируватсинтетазы и транскетолазы в микроорганизмах, таким образом, завершая описание настоящего изобретения.
[Техническое решение]
Задача в соответствии с описанием настоящего изобретения заключается в том, чтобы предложить микроорганизм, продуцирующий микоспорин-подобную аминокислоту (ММА), в котором усилена активность по меньшей мере одного белка, выбранного из группы, состоящей из 2-дегидро-3-дезоксифосфогептонатальдолазы,
фосфоенолпируватсинтетазы и транскетолазы.
Еще одна задача в соответствии с описанием настоящего изобретения заключается в том, чтобы предложить способ получения микоспорин-подобной аминокислоты, который включает культивирование микроорганизма в среде; и выделение микоспорин-подобной аминокислоты из культивированного микроорганизма или среды.
Еще одна задача в соответствии с описанием настоящего изобретения заключается в том, чтобы предложить применение микроорганизма для продуцирования микоспорин-подобной аминокислоты.
[Благоприятные эффекты]
Поскольку микроорганизм в соответствии с описанием настоящего изобретения обладает увеличенной способностью продуцировать микоспорин-подобную аминокислоту (ММА), он может быть эффективно использован в получении микоспорин-подобных аминокислот.
[Наилучший способ осуществления изобретения]
Описание настоящего изобретения подробно изложено ниже. В то же самое время, соответствующие описания и воплощения, раскрытые в описании настоящего изобретения, также могут быть применены в отношении других описаний и воплощений. То есть, все комбинации различных элементов, раскрытые в описании настоящего изобретения, оказываются в объеме изобретения в соответствии с описанием настоящего изобретения. Кроме того, объем изобретения в соответствии с описанием настоящего изобретения не ограничивается приведенным ниже конкретным описанием изобретения. Кроме того, специалист в данной области техники может обнаружить или идентифицировать множество эквивалентов некоторых аспектов в соответствии с описанием настоящего изобретения исключительно при помощи стандартных экспериментов. Кроме того, предполагается, что такие эквиваленты включены в описание настоящего изобретения.
Для достижения вышеприведенных задач в аспекте в соответствии с описанием настоящего изобретения предложен микроорганизм, продуцирующий микоспорин-подобную аминокислоту (ММА), в котором усилена активность по меньшей мере одного белка, выбранного из группы, состоящей из 2-дегидро-3-дезоксифосфогептонатальдолазы, фосфоенолпируватсинтетазы и транскетолазы.
Используемый здесь термин "2-дегидро-3-дезоксифосфогептонатальдолаза" относится к ферменту, который катализирует обратимую реакцию на следующей реакционной схеме, и, в частности, может относиться к ферменту, который синтезирует 3-дезокси-D-арабино-гептулозонат-7-фосфат (DAHP), но не ограничивается ими.
[Реакционная схема]
Фосфоенолпируват + D-эритроза-4-фосфат + Н2О
↔ 3-дезокси-D-арабиногептулозонат-7-фосфат + фосфат
В описании настоящего изобретения 2-дегидро-3-дезоксифосфогептонатальдолаза может быть использована взаимозаменяемо с 3-дезокси-D-арабино-гептулозонат-7-фосфат (DAHP) синтазой.
Используемый здесь термин "фосфоенолпируватсинтетаза" относится к ферменту, который катализирует обратимую реакцию на следующей реакционной схеме, и, в частности, может относиться к ферменту, который синтезирует фосфоенолпируват, но не ограничивается им.
[Реакционная схема]
АТФ + пируват +Н2О
↔ AMP + фосфоенолпируват + фосфат
Используемый здесь термин "транскетолаза" относится к ферменту, который катализирует обратимую реакцию на следующей реакционной схеме.
[Реакционная схема]
Седогептулозо-7-фосфат+ D-глицеральдегид-3-фосфат
↔ D-рибозо-5-фосфат + D-ксилулозо-5-фосфат
или
Фруктозо-6-фосфат + D-глицеральдегид-3-фосфат
↔ эритрозо-4-фосфат + D-ксилулозо-5-фосфат
Генетическая информация о 2-дегидро-3-дезоксифосфогептонатальдолазе, фосфоенолпируватсинтетазе и транскетолазе могут быть получены в известной базе данных (например GenBank database в National Center for Biotechnology Information (Национальном центре биотехнологической информации) (NCBI) и т.п.), но не ограничивается ей.
2-Дегидро-3-дезоксифосфогептонатальдолаза, фосфоенолпируватсинтетаза и транскетолаза не ограничиваются своим происхождением или последовательностями, поскольку существуют случаи, когда белки, демонстрирующие активности, отличаются по своим аминокислотным последовательностям в зависимости от видов микроорганизма или самого микроорганизма.
В частности, 2-дегидро-3-дезоксифосфогептонатальдолаза может представлять собой белок, включающий аминокислотную последовательность в соответствии с SEQ ID NO: 2, 37 или 124; фосфоенолпируватсинтетаза может представлять собой белок, включающий аминокислотную последовательность в соответствии с SEQ ID NO: 19 или 98; и транскетолаза может представлять собой белок, включающий аминокислотную последовательность в соответствии с SEQ ID NO: 24, 96 или 123, но аминокислотные последовательности этих белков не ограничиваются ими. Используемый здесь термин "белок, включающий аминокислотную последовательность" может быть использован взаимозаменяемо с выражением "белок, имеющий аминокислотную последовательность", или "белок, состоящий из аминокислотной последовательности".
Дополнительно, в описании настоящего изобретения эти ферменты могут включать белки, которые имеют аминокислотные последовательности в соответствии с SEQ ID NO, описанными выше, а также 80% или более высокую, 85% или более высокую, 90% или более высокую, 91% или более высокую, 92% или более высокую, 93% или более высокую, 94% или более высокую, 95% или более высокую, 96% или более высокую, 97% или более высокую, 98% или более высокую, 99% или более высокую гомологию или идентичность с вышеприведенными аминокислотными последовательностями, до тех пор, пока белки обладают биологической активностью, идентичной или соответствующей каждому из этих ферментов.
Дополнительно, понятно, что любой белок, который имеет аминокислотную последовательность с делецией, модификацией, заменой или добавлением части последовательности, также может быть включен в объем изобретения в соответствии с описанием настоящего изобретения, до тех пор, пока аминокислотная последовательность обладает гомологией или идентичностью с SEQ ID NO, описанными выше, и обладает биологической активностью, по существу идентичной или соответствующей белкам ферментов в соответствии с SEQ ID NO, описанными выше.
Используемый здесь термин "гомология или идентичность" относится к степени соответствия между заданными аминокислотными последовательностями или нуклеотидными последовательностями, и могут быть выражены в процентных долях. В описании настоящего изобретения гомологичная последовательность, обладающая активностью, идентичной или похожей на активность для заданной аминокислотной последовательности или нуклеотидной последовательности, представлена как "% гомологии" или "% идентичности". Например, гомология может быть подтверждена с использованием стандартного программного обеспечения, в частности BLAST 2.0, для расчета параметров, таких как оценка, идентичность и сходство, или путем сравнения последовательностей путем гибридизации по Саузерну при определенных жестких условиях. Определенные подходящие условия гибридизации могут находиться в объеме уровня техники и могут быть определены при помощи способа, хорошо известного специалистам в данной области техники (например J. Sambrook et al., Molecular Cloning, A Laboratory Manual, 2nd Edition, Cold Spring Harbor Laboratory press, Cold Spring Harbor, New York, 1989; F.M. Ausubel et al., Current Protocols in Molecular Biology, John Wiley & Sons, Inc., New York). Термин "жесткие условия" относится к условиям, которые обеспечивают специфическую гибридизацию между полинуклеотидами. Например, такие условия, в частности, раскрыты в литературе (например J. Sambrook et al., выше).
2-Дегидро-3-дезоксифосфогептонатальдолаза, фосфоенолпируватсинтетаза и транскетолаза в соответствии с описанием настоящего изобретения могут включать полинуклеотиды, кодирующие белки, которые имеют аминокислотные последовательности в соответствии с описанными выше SEQ ID NO, или 80% или более высокую, 85% или более высокую, 90% или более высокую, 91% или более высокую, 92% или более высокую, 93% или более высокую, 94% или более высокую, 95% или более высокую, 96% или более высокую, 97% или более высокую, 98% или более высокую, 99% или более высокую гомологию или идентичность с аминокислотными последовательностями в соответствии с SEQ ID NO, описанными выше, до тех пор, пока эти полинуклеотиды обладают биологической активностью, идентичной или соответствующей каждому из этих ферментов.
Дополнительно, рассматривая кодоны, предпочтительные в организме, когда должен экспрессироваться белок, вследствие вырожденности кодонов различные модификации могут быть осуществлены в кодирующей области нуклеотидной последовательности в объеме изобретения, не изменяющие аминокислотную последовательность белка, экспрессирующегося с кодирующей области. Таким образом, любой полинуклеотид, имеющий последовательность, кодирующую каждый из белков-ферментов, может быть включен без ограничения.
Дополнительно, любая последовательность, которая кодирует белок, обладающий активностью белков-ферментов 2-дегидро-3-дезоксифосфогептонатальдолазы,
фосфоенолпируватсинтетазы или транскетолазы, путем гибридизации с любым зондом, который могут быть получен из известных последовательностей генов (например последовательности, комплементарные всей или части вышеприведенной полинуклеотидной последовательности), в жестких условиях, может быть включена без ограничения.
Термин "жесткие условия" относится к условиям, которые обеспечивают специфическую гибридизацию между полинуклеотидами. Такие условия, в частности, описаны в литературе (например J Sambrook et al., выше). Например условия гибридизации между генами, имеющими высокую гомологию или идентичность, гомология или идентичность 40% или выше, в частности, 90% или выше, в частности, 95% или выше, в частности, 97% или выше, и, в частности, 99% или выше, без гибридизации между генами, имеющими гомологию или идентичность ниже, чем гомологии или идентичности, описанные выше; или могут быть перечислены обычные условия отмывания для гибридизации по Саузерну, т.е. однократное отмывание, в частности, два или три раза при концентрации соли и температуре, соответствующей 60°С, 1×SSC, 0,1% SDS (додецилсульфат натрия), в частности 60°С, 0,1×SSC, 0,1% SDS, и, в частности, 68°С, 0,1×SSC, 0,1% SDS.
Гибридизация требует того, что два полинуклеотида обладают комплементарными последовательностями, хотя некомплементарности между основаниями возможны в зависимости от жесткости гибридизации. Термин "комплементарный" используют для описания взаимосвязи между нуклеотидными основаниями, которые могут гибрид изо в аться друг с другом. Например, в отношении ДНК аденозин комплементарен тимину и цитозин комплементарен гуанину. Соответственно, описание настоящего изобретения также может включать выделенные полинуклеотидные фрагменты, комплементарные полноразмерной последовательности, а также, по существу, похожие полинуклеотидные последовательности.
В частности, полинуклеотиды, обладающие гомологией или идентичностью, могут быть обнаружены с использованием условий гибридизации, которые включают стадию гибридизации при величине Tm 55°С и с использованием условий, описанных выше. Дополнительно, величина Tm может составлять 60°С, 63°С или 65°С, но температура не ограничивается ими, и может быть подходящим образом скорректирована специалистом в данной области техники в зависимости от задачи.
Жесткость, подходящая для гибридизации полинуклеотидов, зависит от длины и степени комплементарности полинуклеотидов, и варианты хорошо известны в области техники (смотри Sambrook et al., выше, 9.50-9.51 и 11.7-11.8). Используемый здесь термин "усиление активности" означает, что введена активность белка-фермента, или активность усилена по сравнению с собственной эндогенной активностью или активностью до его модификации, которую демонстрирует микроорганизм. "Введение" активности означает, что микроорганизм естественным или искусственным образом демонстрирует активность конкретного белка, которая исходно не демонстрируется микроорганизмом. В частности, микроорганизм с усиленной активностью белка-фермента относится к микроорганизму, в котором активность белка-фермента усилена по сравнению с активностью природного микроорганизма дикого типа или немодифицированного микроорганизма. Усиление активности может включать, например как усиление активности путем введения экзогенной 2-дегидро-3-дезоксифосфогептонатальдолазы, фосфоенолпируватсинтетазы и/или транскетолазы в микроорганизм; так и усиление активности эндогенных 2-дегидро-3-дезоксифосфогептонатальдолазы, фосфоенолпируватсинтетазы и/или транскетолазы.
В частности, в описании настоящего изобретения способ усиления активности может включать:
(1) способ увеличения количества копий полинуклеотидов, кодирующих ферменты;
(2) способ модификации последовательности, контролирующей экспрессию, для увеличения экспрессии полинуклеотидов;
(3) способ модификации полинуклеотидных последовательностей на хромосоме для усиления активностей ферментов; или
(4) способ модификации для усиления путем комбинирования вышеприведенных способов (1)-(3) и т.п, но способы не ограничиваются ими.
Способ (1) увеличения количества копий полинуклеотидов может быть осуществлен в форме, при которой полинуклеотид функционально связан с вектором, или путем встраивания полинуклеотида в хромосому клетки-хозяина, но не ограничен конкретно ими. Дополнительно, в качестве альтернативы, количество копий может быть увеличено путем введения экзогенного полинуклеотида, демонстрирующего активность фермента, или оптимизированного по кодонам модифицированного полинуклеотида относительно вышеприведенного полинуклеотида в клетку-хозяина. Экзогенный полинуклеотид может быть использован без ограничения своего происхождения или последовательности до тех пор, пока полинуклеотид демонстрирует активность, идентичную или похожую на активность фермента. Введение может быть осуществлено специалистом в данной области техники путем надлежащего выбора известного способа трансформации, и активность фермента может быть усилена таким образом, что введенный полинуклеотид экспрессируется в клетке-хозяине, таким образом, продуцируя фермент.
Затем, способ (2) модификации последовательности, контролирующей экспрессию, для увеличения экспрессии полинуклеотидов, может быть осуществлен путем индукции модификации в последовательности путем делеции, вставки, неконсервативной или консервативной замены или их комбинации для дополнительного усиления активности последовательности, контролирующей экспрессию; или путем замены последовательности нуклеиновой кислоты на последовательность нуклеиновой кислоты, обладающую более сильной активностью, но способ по существу не ограничивается ими. Последовательность, контролирующая экспрессию, может включать промотор, операторную последовательность, последовательность, кодирующую сайт связывания с рибосомой, последовательности, контролирующие прекращение транскрипции и трансляции и т.п., но последовательность, контролирующая экспрессию, не ограничена конкретно ими.
В частности, сильный гетерологический промотор вместо исходного промотора может быть связан выше относительно полинуклеотидного экспрессирующегося фрагмента, и примеры сильного промотора могут включать промотор CJ7, промотор lysCP1, промотор EF-Tu, промотор groEL, промотор асе А или асеВ и т.п. В частности, полинуклеотидный экспрессирующийся фрагмент может быть функционально связан с промотором, происходящим из Corynebacterium, таким как промотор lysCP1 (WO 2009/096689), промотор CJ7 (WO 2006/065095), промотор SPL (KR 10-1783170 В) или промотор о2 (KR 10-1632642 В), для улучшения уровня экспрессии полинуклеотида, кодирующего фермент, но способ не ограничивается ими.
Дополнительно, способ (3) модификации полинуклеотидной последовательности на хромосоме может быть осуществлен путем индукции модификации в последовательности, контролирующей экспрессию, путем делеции, вставки, неконсервативной или консервативной замены или их комбинации для дополнительного усиления активности полинуклеотидной последовательности; или путем замены последовательности нуклеиновой кислоты на улучшенную полинуклеотидную последовательность, обладающую более сильной активностью, но способ не ограничивается конкретно ими.
Наконец, способ (4) модификации для усиления путем комбинирования способов (1)-(3) может быть осуществлен путем применения вместе одного или более чем одного способа из числа способов: способа увеличения числа копий полинуклеотидов, кодирующих ферменты; способа модификации последовательности, контролирующей экспрессию, для увеличения экспрессии; и способа модификации полинуклеотидных последовательностей на хромосоме или способа модификации экзогенных полинуклеотидов, демонстрирующих активность фермента или его оптимизированного по кодонам модифицированного полинуклеотида.
Полинуклеотиды могут быть описаны как гены, когда они представляют собой ансамбль из полинуклеотидов, способных функционировать. В описании настоящего изобретения полинуклеотиды и гены могут быть использованы взаимозаменяемо, и полинуклеотидные последовательности и нуклеотидные последовательности могут быть использованы взаимозаменяемо.
Используемый здесь термин "вектор" относится к конструкции ДНК, включающей нуклеотидную последовательность полинуклеотида, кодирующего желаемый белок, в которой желаемый белок функционально связан с подходящей контролирующей последовательностью таким образом, что он может экспрессироваться в подходящем хозяине. Контролирующая последовательность может включать промотор, способный инициировать транскрипцию, любую операторную последовательность для контроля транскрипции, последовательность, кодирующую подходящий сайт связывания мРНК с рибосомой, и последовательности, контролирующие прекращение транскрипции и трансляции. Вектор после трансформации в подходящую клетку-хозяина может реплицироваться или функционировать независимо от генома хозяина, или может интегрироваться в геном самого хозяина.
Вектор, используемый в описании настоящего изобретения, не ограничен конкретным образом, пока вектор может реплицироваться в клетке-хозяине, и может быть использован любой вектор, известный в области техники. Примеры обычных векторов могут включать природную или рекомбинантную плазмиду, космиду, вирус и бактериофаг.Например pWE15, М13, MBL3, MBL4, IXII, ASHII, APII, t10, t11, Charon4A и Charon21A и т.п. могут быть использованы в качестве фагового вектора или космидного вектора; и pBR, pUC, pBluescriptII, pGEM, pTZ, pCL и pET и т.п.могут быть использованы в качестве плазмидного вектора. В частности, могут быть использованы векторы, такие как pDZ, pACYC177, pACYC184, pCL, pECCG117, pUC19, pBR322, pMW118, pCC1BAC, pSKH, pRS-413, pRS-414 и pRS-415 и т.п., но векторы не ограничиваются ими.
Векторы, используемые в описании настоящего изобретения, не ограничены конкретным образом, и может быть использован любой экспрессирующий вектор. Дополнительно, полинуклеотид, кодирующий желаемый белок на хромосоме, может быть встроен в хромосому при помощи вектора для хромосомного встраивания в клетку. Встраивание полинуклеотида в хромосому может быть осуществлено с использованием любого способа, известного в области техники (например, путем гомологичной рекомбинации), но способ встраивания не ограничивается ими. Также может быть включен селективный маркер для подтверждения встраивания в хромосому. Селективный маркер используется для отбора клеток, трансформированных вектором (т.е. для подтверждения того, встроилась ли желаемая молекула нуклеиновой кислоты), и могут быть использованы маркеры, способные обозначать отбираемые фенотипы (например лекарственная устойчивость, ауксотрофность, устойчивость к цитотоксическим агентам и экспрессия поверхностных белков). В условиях, когда обрабатывают селективными агентами, тогда только клетки, способные экспрессировать селективные маркеры, могут выживать или экспрессировать другие фенотипические признаки, и, таким образом, легко могут быть отобраны трансформированные клетки.
Используемый здесь термин "трансформация" относится к введению вектора, включающего полинуклеотид, кодирующий желаемый белок, в клетку-хозяина, таким образом, что белок, кодируемый полинуклеотидом, может экспрессироваться в клетке-хозяине. Трансформированный полинуклеотид может не ограничиваться конкретным образом, пока он может экспрессироваться в клетке-хозяине независимо от того, встроен ли трансформированный полинуклеотид в хромосому клетки-хозяина, располагаясь в ней, или располагается за пределами хромосомы. Дополнительно, полинуклеотид включает ДНК и РНК, кодирующие желаемый белок. До тех пор, пока полинуклеотид может быть введен в клетку-хозяина и экспрессироваться в ней, не имеет значения, в какой форме встроен полинуклеотид. Например полинуклеотид может быть введен в клетку-хозяина в форме экспрессионной кассеты, которая представляет собой генную конструкцию, включающую все элементы, необходимые для ее экспрессии. Экспрессионная кассета может включать промотор, который функционально связан с полинуклеотидом, сигнал прекращения транскрипции, сайт связывания с рибосомой и сигнал прекращения трансляции. Экспрессионная кассета может представлять собой самореплицирующийся экспрессирующий вектор. Дополнительно, полинуклеотид может представлять собой полинуклеотид, который вводят в клетку-хозяина в виде самого полинуклеотида и который связан с последовательностью, необходимой для экспрессии в клетке-хозяине, но не ограничивается ими. Способы трансформации включают любой способ введения в клетку нуклеиновой кислоты, и могут быть осуществлены путем выбора подходящего стандартного способа, известного в области техники, в зависимости от клетки-хозяина. Например, способы трансформации могут включать электропорацию, осаждение при помощи фосфата кальция (CaPO4), осаждение при помощи хлорида кальция (CaCl2), микроинъекцию, полиэтиленгликолевый способ (PEG), способ с использованием ЕАЕ-декстрана, катионный липосомальный способ, способ с использованием ацетата лития-DMSO (диметилсульфоксид) и т.п., но способы не ограничиваются ими.
Дополнительно, используемый здесь темин "функционально связанный" обозначает функциональную связь между промоторной последовательностью, которая инициирует и опосредует транскрипцию полинуклеотида, кодирующего желаемый белок в соответствии с описанием настоящего изобретения, и полинуклеотидной последовательностью. Функциональная связь может быть получена при помощи способа генетической рекомбинации, известного в области техники, и сайт-специфическое расщепление ДНК и лигирование могут быть осуществлены с использованием рестрикционного фермента, лигазы и т.п., известных в области техники, не ограничиваясь ими.
В микроорганизме в соответствии с описанием настоящего изобретения активность 3-дегидрохинатдегидратазы может быть дополнительно инактивирована.
Используемый здесь термин "3-дегидрохинатдегидратаза" относится к ферменту, который катализирует обратимую реакцию в приведенной ниже реакционной схеме, и, в частности, она может превращать 3-дегидрохинат в 3-дегидрошикимат, но не ограничивается ими.
[Реакционная схема]
3-дегидрохинат ↔ 3-дегидрошикимат + Н2О
3-Дегидрохинатдегидратаза не ограничена в своем происхождении или последовательности, поскольку существуют случаи, когда белки, демонстрирующие активность 3-дегидрохинатдегидратазы, отличаются по своей аминокислотной последовательности в зависимости от вида микроорганизма или микроорганизмов. В частности, 3-дегидрохинатдегидратаза может представлять собой белок, включающий аминокислотную последовательность в соответствии с SEQ ID NO: 90, но не ограничивается ей. Дополнительно, 3-дегидрохинатдегидратаза может включать аминокислотную последовательность в соответствии с SEQ ID NO: 90 или аминокислотную последовательность, обладающую гомологией или идентичностью по меньшей мере на 80%, 90%, 95%, 96%, 97%, 98% или 99% с SEQ ID NO: 90. Дополнительно, понятно, что любая аминокислотная последовательность с делецией, модификацией, заменой или добавлением части последовательности также может быть включена в объем изобретения в соответствии с описанием настоящего изобретения до тех пор, пока аминокислотная последовательность обладает гомологией или идентичностью, описанными выше, и обладает биологической активностью, идентичной или соответствующей вышеприведенному белку.
Используемый здесь термин "инактивация" относится к случаю, когда активность белка фермента ослаблена по сравнению с эндогенной активностью белка фермента или его активностью до модификации, исходно демонстрируемой микроорганизмом; случаю, когда белок не экспрессируется вовсе; или случаю, когда белок экспрессируется, но не обладает активностью. Инактивация представляет собой ситуацию, которая включает: случай, когда активность самого фермента ослаблена по сравнению с его эндогенной активностью, демонстрируемой микроорганизмом, вследствие модификации полинуклеотида, кодирующего фермент, и т.п, или активность удалена; случай, когда степень общей внутриклеточной активности фермента ниже по сравнению с активностью в микроорганизме дикого типа или активность удалена вследствие ингибирования экспрессии или трансляции гена, кодирующего фермент, и т.п; случай, когда весь ген или часть гена, кодирующего фермент, удален; и их комбинация, но инактивация не ограничивается ими. То есть, микроорганизм, в котором активность фермента инактивирована, относится к микроорганизму, в котором активность белка-фермента ниже по сравнению с активностью у его природного микроорганизма дикого типа или немодифицированного микроорганизма или в котором активность удалена.
Инактивация активности фермента может быть достигнута путем применения различных способов, известных в области техники. Примеры вышеприведенных способов могут включать: 1) способ делеции на хромосоме всего гена или части гена, кодирующего фермент; 2) способ модификации на хромосоме последовательности, контролирующей экспрессию, для уменьшения экспрессии гена, кодирующего белок; 3) способ модификации на хромосоме последовательности гена, кодирующего белок, таким образом, что активность белка удаляется или ослабляется; 4) способ введения антисмыслового олигонуклеотида (например антисмысловой РНК), который комплементарно связывается с транскриптом гена на хромосоме, кодирующего белок; 5) способ, приводящий к невозможности прикрепления рибосомы за счет вторичной структуры, образующейся при добавлении последовательности, которая комплементарна последовательности Шайна-Дальгарно (SD), на переднем конце последовательности SD гена на хромосоме, кодирующего белок; 6) способ инженерии с обратной транскрипцией (RTE), при котором промотор, который транскрибируется в обратном направлении добавляется по 3'-концу открытой рамки считывания (ORF) полинуклеотидной последовательности, кодирующей белок, и т.п; и инактивация может быть осуществлена путем их комбинирования, но способы не ограничены конкретно ими.
Способ делеции всего гена или части гена на хромосоме, кодирующего фермент, может быть осуществлен путем замены полинуклеотида на хромосоме, кодирующего эндогенный желаемый белок, на полинуклеотид или маркерный ген, имеющий частично подвергнутую делеции нуклеотидную последовательность, с использованием вектора для встраивания в хромосому. В качестве примера способа делеции всего или части полинуклеотида может быть использован способ делеции полинуклеотида путем гомологичной рекомбинации, но способ не ограничивается им.
Способ модификации последовательности, контролирующей экспрессию, может быть осуществлен путем индукции модификации последовательности нуклеиновой кислоты в последовательности, контролирующей экспрессию, путем делеции, вставки, консервативной или неконсервативной замены или их комбинации таким образом, чтобы дополнительно ослабить активность последовательности, контролирующей экспрессию; или путем замены последовательности нуклеиновой кислоты на последовательность нуклеиновой кислоты, обладающую более слабой активностью. Последовательность, контролирующая экспрессию, может включать промотор, операторную последовательность, последовательность, кодирующую сайт связывания с рибосомой, и последовательность для регулирования транскрипции и трансляции, но не ограничивается ими.
Способ модификации последовательности гена на хромосоме может быть осуществлен путем индукции модификации в последовательности гена путем делеции, вставки, консервативной или неконсервативной замены или их комбинации таким образом, чтобы дополнительно ослабить активность фермента; или путем замены последовательности гена на последовательность гена, модифицированного таким образом, что он обладает более слабой активностью, или на последовательность гена, модифицированного таким образом, что он вовсе не обладает активностью, но способ не ограничивается ими.
В вышеприведенном описании термин "часть", независимо от того, что может варьировать в зависимости от типа полинуклеотида, может составлять, в частности, от 1 нуклеотида до 300 нуклеотидов, в частности, от 1 нуклеотида до 100 нуклеотидов, и, в частности, от 1 нуклеотида до 50 нуклеотидов, но не ограничивается конкретно ими.
В микроорганизме в соответствии с описанием настоящего изобретения активность белка 3-дегидрохинатсинтазы может быть дополнительно усилена по сравнению с активностью у немодифицированного микроорганизма.
3-Дегидрохинатсинтаза относится к ферменту, который катализирует обратимую реакцию следующей реакционной схемы, и, в частности, может синтезировать 3-дегидрохинат (3-DHQ), но не ограничивается ими.
[Реакционная схема]
3-дезокси-арабино-гептулозонат-7-фосфат
↔ 3-дегидрохинат + фосфат
Используемый здесь термин "микоспорин-подобная аминокислота (МАА)" относится к циклическому соединению, которое поглощает ультрафиолетовые (УФ) лучи. В описании настоящего изобретения микоспорин-подобная аминокислота не ограничена до тех пор, пока она может поглощать УФ лучи, и в частности, она может представлять собой соединение, которое имеет центральное кольцо циклогексанона или циклогексенимина; или может представлять собой соединение, в котором различные вещества (например аминокислоты и т.п.) связаны с центральным кольцом, и, в частности, оно может представлять собой микоспорин-2-глицин, палитинол, палитеновую кислоту, дезоксигадузол, микоспорин-метиламин-треонин, микоспорин-глицин-валин, палитин, астерину-330 (asterina-330), шинорин, порфиру-334 porphyra-334), эухалотец-362 (euhalothece-362), микоспорин-глицин, микоспорин-орнитин, микоспорин-лизин, микоспорин-глутаминовая кислота-глицин, микоспорин-метиламин-серин, микоспорин-таурин, палитен, палитин-серин, палитин-серин-сульфат, палитинол, усуджирен или их комбинацию.
В описании настоящего изобретения термин микоспорин-подобная аминокислота может быть использован взаимозаменяемо с МАА и МАА.
Используемый здесь термин "микроорганизм, продуцирующий микоспорин-подобную аминокислоту (МАА)" может относиться к микроорганизму, который включает ген фермента, вовлеченного в биосинтез МАА, или кластер этих генов, или микроорганизму, в котором кластер введен или усилен. Дополнительно, используемый здесь термин "кластер генов биосинтеза микоспорин-подобной аминокислоты (МАА)" может относиться к группе генов, кодирующих ферменты, вовлеченные в биосинтез МАА, и, в частности, может включать ген биосинтеза МАА, ген фермента, обладающего активностью, присоединяющей дополнительный аминокислотный остаток к МАА, или кластер вышеприведенных генов. Ген биосинтеза МАА включает как экзогенные гены, так и/или эндогенные гены микроорганизма, до тех пор, пока микроорганизм, включающий такой ген, может продуцировать МАА. Экзогенные гены могут быть гомогенными или гетерогенными.
Виды микроорганизмов, из которых происходит ген биосинтеза МАА, не ограничены, пока микроорганизмы, включающие этот ген, могут продуцировать ферменты, вовлеченные в биосинтез МАА, и следовательно могут продуцировать МАА. В частности, виды микроорганизмов, из которых происходит ген биосинтеза МАА, могут представлять собой цианобактерии (например Anabaena variabilis, Nostoc punctiforme, Nodularia spumigena, Cyanothece sp. PCC 7424, Lyngbya sp. PCC 8106, Microcystis aeruginosa, Microcoleus chthonoplastes, Cyanothece sp. ATCC 51142, Crocosphaera watsonii, Cyanothece sp. CCY 0110, Cylindrospermum stagnate sp. PCC 7417, Aphanothece halophytica или Trichodesmium erythraeum); грибы (например Magnaporthe orzyae, Pyrenophora tritici-repentis, Aspergillus clavatus, Nectria haematococca, Aspergillus nidulans, Gibberella zeae, Verticillium albo-atrum, Botryotinia fuckeliana или Phaeosphaeria nodorum); или Nematostella vectensis; Heterocapsa triquetra, Oxyrrhis marina, Karlodinium micrum, Actinosynnema mirum и т.п, но не ограничиваться ими.
В соответствии с воплощением микроорганизм в соответствии с описанием настоящего изобретения, который продуцирует МАА, включает ген биосинтеза МАА или его кластер. В частности, в микроорганизм может быть введен генный кластер биосинтеза МАА или активности белков, кодируемых генами, могут быть усилены по сравнению с эндогенными активностями или активностями до модификации, но микроорганизм не ограничивается ими.
Дополнительно, хотя ген биосинтеза МАА не ограничен в своем названии ферментами или микроорганизмами, из которых происходят эти гены, пока микроорганизмы могут продуцировать МАА, этот ген биосинтеза МАА может включать ген, кодирующий белок-фермент, обладающий активностью, идентичной и/или похожей на один или более чем один, в частности, один или более чем один, два или более чем два, три или более чем три, или все из белков-ферментов, выбранных из группы, состоящей из 2-деметил-4-дезоксигадузолсинтазы, О-метилтрансферазы и C-N-лигазы.
Например, 2-деметил-4-дезоксигадузолсинтаза представляет собой фермент, который превращает седогептулозу-7-фосфат в 2-деметил-4-дезоксигадузол. О-Метилтрансфераза представляет собой фермент, который превращает 2-деметил-4-дезоксигадузол в 4-дезоксигадузол, глицилирование 4-дезоксигадузола катализируется С-N-лигазой.
Дополнительно, микроорганизм, продуцирующий МАА, может включать ген фермента, который обладает активностью, присоединяющей дополнительный аминокислотный остаток к МАА, или кластер генов. Хотя вышеприведенный ген или кластер генов не ограничен в своих названиях ферментами или микроорганизмами, из которых происходят эти гены, пока микроорганизмы, продуцирующие МАА, могут продуцировать МАА, к которым присоединены два или более чем два аминокислотных остатка. Микроорганизмы, продуцирующие МАА, могут включать ген, кодирующий белок-фермент, обладающий активностью, идентичной и/или похожей на один или более чем один, в частности, один или более чем один, два или более чем два, три или более чем три или все из белков-ферментов, выбранных из группы, состоящей из нерибосомальной пептидсинтетазы (NRPS), фермента, подобного нерибосомальной пептидсинтетазе (NRPS-подобный фермент), и D-аланин D-аланин-лигазы (D-Ala-D-Ala лигаза; DDL).
Некоторые из МАА включают второй аминокислотный остаток в микоспорин-глицине. Один или более чем один фермент, которые выбраны из группы, состоящей из нерибосомальной пептидсинтетазы, фермента, подобного нерибосомальной пептидсинтетазе, и D-Ala-D-Ala-лигазы, могут присоединять второй аминокислотный остаток к микоспорин-глицину.
В соответствии с воплощением микроорганизм, продуцирующий МАА, может включать без ограничения названия ферментов или виды микроорганизмов, из которых происходят гены биосинтеза МАА, ферменты, пока они обладают активностью, способной присоединять вторую аминокислоту к микоспорин-глицину, как в нерибосомальной пептидсинтетазе, ферменте, подобном нерибосомальной пептидсинтетазе и D-Ala-D-Ala-лигазе.
Например, фермент, подобный нерибосомальной пептидсинтетазе (Ava_3855), в АпаЬаепа variabilis или D-Ala-D-Ala-лигаза (NpF5597) в Nostoc punctiforme могут присоединять сериновый остаток к микоспорин-глицину с образованием шинорина. В еще одном примере микоспорин-2-глицин может быть получен путем присоединения второго глицинового остатка при помощи гомолога D-Ala-D-Ala-лигазы (Ар_3855) в Aphanothece halophytica. Аналогично, в Actinosynnema mirum серии или аланин могут быть присоединены при помощи D-Ala-D-Ala-лигазы и, таким образом, может образовываться шинорин или микоспорин-глицин-аланин. Микроорганизм в соответствии с воплощением в соответствии с описанием настоящего изобретения может быть выбран и включать ферменты, которые подходят для получения желаемых МАА, среди вышеописанных ферментов или ферментов, обладающими активностями, идентичными и/или похожими на активность вышеописанных ферментов.
2-Деметил-4-дезоксигадузолсинтаза, О-метилтрансфераза, C-N-лигаза, не рибосомальная пептидсинтетаза, фермент, подобный нерибосомальной пептидсинтетазе, и/или D-Ala-D-Ala-лигаза, которые могут быть использованы в описании настоящего изобретения, не ограничены видами микроорганизмов, из которых происходят эти ферменты, пока ферменты, которые, как известно, способны выполнять функции и роли, идентичные и/или похожие на функции и роли для вышеописанных ферментов, и диапазон гомологии или идентичности среди них также не ограничен. Например MylA, MylB, MylD, MylE и MylC из С. stagnale PCC 7417 гомологичны с 2-деметил-4-дезоксигадузолсинтазой, О-метилтрансферазой, C-N-лигазой и D-Ala-D-Ala-лигазой, происходящими из Anabaena variabilis и Nostoc punctiforme, и степень сходства между ними находится в диапазоне от приблизительно 61% до приблизительно 88% (Appl Environ Microbiol, 2016, 82(20), 6167-6173; J Bacteriol, 2011, 193(21), 5923-5928). To есть, ферменты, которые могут быть использованы в описании настоящего изобретения, не ограничены существенно видами, из которых происходят ферменты, гомологией последовательностей или идентичностью последовательностей, до тех пор, пока они, как известно, демонстрируют идентичные и/или похожие функции и действия. Дополнительно, непатентные документы, описанные в предшествующем уровне техники, включены в полном объеме, как ссылка на описание настоящего изобретения в целом.
Дополнительно, ген биосинтеза МАА может представлять собой полинуклеотид, кодирующий белок, который включает аминокислотную последовательность в соответствии с SEQ ID NO: 115, 116, 117, 118, 119, 120, 121 или 122, но ген биосинтеза МАА не ограничивается ими.
Дополнительно, ген биосинтеза МАА может включать нуклеотидную последовательность, кодирующую белок, который включает аминокислтную последовательность, имеющую 50%, 60% или 70% или более высокую, в частности, 80% или более высокую, в частности, 90% или более высокую, в частности, 95% или более высокую, и, в частности, 99% или более высокую гомологию или идентичность с аминокислотной последовательностью в соответствии с SEQ ID NO: 115, 116, 117, 118, 119, 120, 121 или 122, и может включать без ограничения нуклеотидную последовательность, кодирующую белок, который выходит за пределы диапазона вышеприведенной гомологии или идентичности, до тех пор, пока микроорганизм может продуцировать ММА. В частности, ген биосинтеза МАА может включать нуклеотидную последовательность в соответствии с SEQ ID NO: 102, 103, 104, 105, 106, 107, 108 или 109, но не ограничивается ими.
Дополнительно, понятно, что любая аминокислотная последовательность с делецией, модификацией, заменой или добавлением части последовательности, также может быть включена в описание настоящего изобретения, до тех пор, пока аминокислотная последовательность обладает гомологией или идентичностью с вышеприведенными последовательностями и по существу обладает биологической активностью, идентичной или соответствующей белкам в соответствии с SEQ ID NO, описанными выше.
Дополнительно, рассматривая кодоны, предпочтительные в организме, в котором должен экспрессироваться белок, вследствие вырожденности кодонов различные модификации могут быть осуществлены в кодирующей области нуклеотидной последовательности в объеме изобретения, не изменяющие аминокислотную последовательность белка, экспрессируемого с кодирующей области. Таким образом, в отношении гена биосинтеза МАА, любая нуклеотидная последовательность может быть включена в описание настоящего изобретения без ограничения, пока нуклеотидная последовательность представляет собой нуклеотидную последовательность, которая кодирует белок, вовлеченный в биосинтез МАА.
Альтернативно, любая последовательность, которая кодирует белок, вовлеченный в биосинтез МАА, путем гибридизации с любым зондом, который может быть получен из известных последовательностей генов (например последовательности, комплементарные всей или части полинуклеотидной последовательности) в жестких условиях, может быть включена в описание настоящего изобретения без ограничения.
В соответствии с воплощением микроорганизм, продуцирующий МАА, может включать гены биосинтеза МАА, имеющие различное происхождение.
В описании настоящего изобретения усиление активности белка и/или введение генов может быть осуществлено одновременно, последовательно и в обратном порядке независимо от самого порядка.
Микроорганизм, продуцирующий МАА, может продуцировать МАА за счет включения генного кластера биосинтеза МАА, и дополнительно, может представлять собой микроорганизм, в котором способность продуцировать МАА увеличена путем усиления активности одного или более чем одного белка, выбранного из группы, состоящей из 2-дегидро-3-дезоксифосфогептонатальдолазы, фосфоенолпируватсинтетазы и транскетолазы. Дополнительно, микроорганизм в соответствии с описанием настоящего изобретения не ограничен до тех пор, пока он представляет собой микроорганизм, в котором способность продуцировать МАА увеличена путем усиления активности одного или более чем одного белка, выбранного из группы, состоящей из 2-дегидро-3-дезоксифосфогептонатальдолазы, фосфоенолпируватсинтетазы. В частности, микроорганизм в соответствии с описанием настоящего изобретения может представлять собой микроорганизм рода Corynebacterium, микроорганизм рода Escherichia или дрожжи.
Микроорганизм рода Corynebacterium может представлять собой, в частности, Corynebacterium glutamicum, Corynebacterium ammoniagenes, Brevibacterium lactofermentum, Brevibacterium flavum, Corynebacterium thermoaminogenes, Corynebacterium efficiens и т.п., и, в частности, может представлять собой Corynebacterium glutamicum, но микроорганизм не ограничивается ими.
Микроорганизм рода Escherichia может представлять собой, в частности, Escherichia albertii, Escherichia coli, Escherichia fergusonii, Escherichia hermannii, Escherichia vulneris и т.п., и, в частности, может представлять собой Escherichia coli, но микроорганизм не ограничивается ими.
Дрожжи могут представлять собой, в частности, Saccharomycotina и Taphrinomycotina типа Ascomycota, Agar i corny с otina типа Basidiomycota, микроорганизм, относящийся к типу Pucciniomycotina и т.п., в частности, микроорганизм рода Saccharomyces, микроорганизм рода Schizosaccharomyces, микроорганизм рода Phaffia, микроорганизм рода Kluyveromyces, микроорганизм рода Pichia и микроорганизм рода Candida, и, в частности, Saccharomyces cerevisiae, но микроорганизм не ограничивается ими.
В еще одном аспекте в соответствии с описанием настоящего изобретения предложен способ получения микоспорин-подобной аминокислоты, который включает культивирование микроорганизма в соответствии с описанием настоящего изобретения в среде; и выделение микоспорин-подобной аминокислоты (МАА) из культивированного микроорганизма или среды.
"Микроорганизм" и "микоспорин-подобная аминокислота (МАА)" являются такими, как описано выше.
Используемый здесь термин "культивирование" означает, что микроорганизм выращивается при надлежащим образом контролируемых условиях окружающей среды. Процесс культивирования в соответствии с описанием настоящего изобретения может быть осуществлен в подходящей культуральной среде и условиях культивирования, известных в области техники. Такой процесс культивирования могут быть легко скорректирован для применения специалистом в данной области техники в зависимости от выбранного микроорганизма. Стадия культивирования микроорганизма может быть осуществлена в периодической культуре, непрерывной культуре, культуре с подпиткой и т.п., известными в области техники, но стадия культивирования микроорганизма не ограничивается конкретно ими. Среда и другие условия культивирования, используемые для культивирования микроорганизма в соответствии с описанием настоящего изобретения, не ограничены конкретно ими, и может быть использована любая среда, используемая для обычного выращивания микроорганизма. В частности, микроорганизм в соответствии с описанием настоящего изобретения, может быть выращен в аэробных условиях в обычной среде, содержащей подходящий источник углерода, источник азота, источник фосфора, неорганическое соединение, аминокислоту и/или витамин и т.п, путем корректирования температуры, рН и т.п. В частности, рН могут быть скорректирован с использованием основного соединения (например гидроксида натрия, гидроксида калия или аммиака) или кислотного соединения (например фосфорная кислота или серная кислота) с получением оптимального рН (например рН от 5 до 9, в частности, рН от 6 до 8, и, в частности, рН 6,8), но способ корректирования рН не ограничивается ими. Дополнительно, кислород или содержащий кислород газ может быть инжектирован в культуру для поддержания аэробного состояния в культуре; или газ азот, водород или диоксид углерода может быть инжектирован в культуру без инжекции газа для поддержания анаэробного или микроаэробного состояния в культуре, но газ не ограничивается ими. Дополнительно, температура в культуре может поддерживаться на уровне от 20°С до 45°С, и, в частности, от 25°С до 40°С, и культивирование может быть осуществлено в течение от приблизительно 10 часов до приблизительно 160 часов, не ограничиваясь ими. Дополнительно, во время культивирования антивспенивающий агент (например полигликолевый эфир жирной кислоты) может быть добавлен для предупреждения образования пены, но не ограничивается им.
Дополнительно, в качестве источника углерода, используемого в среде для культивирования, сахариды и углеводы (например глюкоза, сахароза, лактоза, фруктоза, мальтоза, меласса, крахмал и целлюлоза), масла и жиры (например соевое масло, подсолнечное масло, арахисовое масло и кокосовое масло), жирные кислоты (например пальмитиновая кислота, стеариновая кислота и линолевая кислота), спирты (например глицерин и этанол), органические кислоты (например уксусная кислота) и т.п. могут быть использованы сами по себе или в комбинации, но источник углерода не ограничивается ими. В качестве источника азота азотсодержащее органическое соединение (например пептон, дрожжевой экстракт, мясная подливка, солодовый экстракт, кукурузный сироп, бобовая мука и мочевина) и неорганическое соединение (например сульфат аммония, хлорида аммония, фосфат аммония, карбонат аммония и нитрат аммония) и т.п. могут быть использованы сами по себе или в комбинации, но источник азота не ограничивается ими. В качестве источника фосфора дигидрофосфат калия, гидрофосфат калия и натрийсодержащие соли, соответствующие им, могут быть использованы сами по себе или в комбинации, но источник фосфора не ограничивается ими. Дополнительно, среда может включать не заменимые способствующие росту вещества, такие как соль металла (например сульфат магния или сульфат железа), аминокислоты и витамины.
МАА, продуцируемые путем культивирования, могут быть секретированы в среду или остаются в клетках.
Используемый здесь термин "среда" относится к культуральной среде для выращивания микроорганизма в соответствии с описанием настоящего изобретения и/или продукту, получаемому после выращивания. Среда представляет собой то, что включает форму, в которую включен микроорганизм, и форму, в которой микроорганизм удаляется из содержащего микроорганизм культурального раствора путем центрифугирования, фильтрования и т.п.
На стадии выделения МАА, продуцируемых на вышеизложенной стадии культивирования в соответствии с описанием настоящего изобретения, желаемые МАА могут быть собраны из культурального раствора с использованием подходящего способа, известного в области техники, в соответствии со способом культивирования. Например может быть использовано центрифугирование, фильтрование, анионообменная хроматография, кристаллизация, высокоэффективная жидкостная хроматография (HPLC) и т.п., и желаемые МАА могут быть выделены из культивируемого микроорганизма или среды с использованием подходящего способа, известного в области техники. Стадия выделения МАА может дополнительно включать стадию разделения и/или стадию очистки.
В еще одном аспекте в соответствии с описанием настоящего изобретения предложено применение микроорганизма в соответствии с описанием настоящего изобретения для получения микоспорин-подобной аминокислоты (ММА).
"Микроорганизм" и "микоспорин-подобная аминокислота" являются такими, как описано выше.
[Способ осуществления изобретения]
Далее описание настоящего изобретения будет раскрыто более подробно со ссылкой на следующие примеры. Тем не менее, эти примеры приведены исключительно для иллюстративных задач, и объем описания не ограничен этими примерами.
Получение рекомбинантного микроорганизма на основе Е. сой, продуцирующего МАА, и продукция МАА с его использованием
Пример 1: Получение штамма, в котором усилена активность 2-дегидро-3-дезоксифосфогептонатальдолазы
Для увеличения способности микроорганизма продуцировать МАА получали штаммы Е. coli, в которых усилена активность 2-дегидро-3-дезоксифосфогептонатальдолазы. В частности, дополнительно вводили ген aroG (2-дегидро-3-дезоксифосфогептонатальдолаза; SEQ ID NO: 1 и 2) на основе штамма Е. coli W3110. Матрицы и праймеры, используемые для получения плазмид, представлены в таблице 1 ниже.
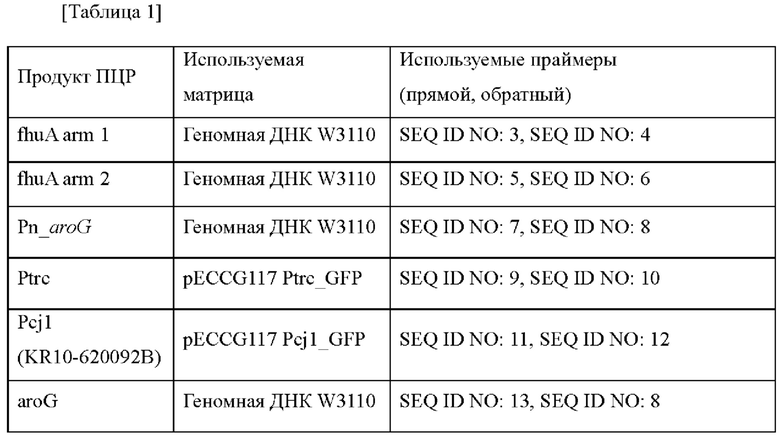
После амплификации фрагментов гена путем ПЦР с использованием вышеприведенных матриц и праймеров амплифицированные фрагменты лигировали с вектором pSKH, в котором фрагменты генов fhuA arm 1 и fhuA arm 2 обрабатывали ферментами рестрикции BamH1-SpeI с использованием набора для клонирования In-FusionR HD (Clontech), и полученный вектор был назван pSKH-ΔfhuA. Вследствие делеции renajhuA фаговое инфицирование Е. coli подавлялось.
Фрагмент гена Pn_aroG лигировали с вектором pSKH-ΔfhuA, который разрезали ферментами рестрикции Spel-EcoRV с использованием набора для клонирования In-FusionR HD (Clontech). После разрезания фрагментов гена Ptrc и Pcj1, которые известны как усиленные промоторы, при помощи ферментов рестрикции Spe1-Nde1 и разрушения фрагмента гена aroG ферментами рестрикции Nde1-EcoRV, фрагменты гена Ptrc и aroG или фрагменты гена Pcj1 (Корейский патент №10-620092) и aroG соответственно лигировали с вектором pSKH-ΔfhuA, который разрезали ферментами рестрикции Spel-EcoRV, с использованием набора для клонирования In-Fusion HDR (Clontech). Полученные векторы были названы соответственно pSKH-ΔfhuA-Pn-aroG, pSKH-ΔfhuA-Ptrc-aroG и pSKH-ΔfhuA-Pcjl-aroG.
Вышеприведенные векторы подтверждали путем секвенирования в отношении успеха клонирования и последовательности гена в каждом векторе, и затем трансформировали в каждом штамме дикого типа Е. coli W3110 путем электропорации. Каждый трансформированный ген вводили в хромосому путем первичной рекомбинации (кроссовер), и область плазмиды вырезали из хромосомы путем вторичной рекомбинации (кроссовер). Для каждого из трансформированного штамма Е. coli, в котором вторичная рекомбинация завершена, введение гена aroG подтверждали путем ПЦР с использованием праймеров SEQ ID NO: 14 (прямой) и 8 (обратный).
Пример 2: Получение вектора, сверхэкспрессирующего полученный из микроводорослей ген биосинтеза шинорина
Основанный на A. variabilis генный кластер для биосинтеза шинорина состоит из четырех генов (Ava_ABCD), который кодирует 2-деметил-4-дезоксигадузолсинтазу, О-метилтрансферазу, C-N-лигазу и нерибосомальную пептид с интетазу. Генный кластер для биосинтеза шинорина идентифицировали с использованием геномной ДНК A. variabilis АТСС29413. Вектор, включающий ген биосинтеза шинорина, полученный из A. variabilis АТСС29413, получали с использованием вектора pECCG117_Pcj1_GFP терминатор. Название вектора, экспрессирующего ген биосинтеза шинорина, и соответствующая матрица и праймеры для получения вектора представлены в таблице 2 ниже.

Фрагменты гена получали с использованием вышеприведенных матрицы и праймеров, и каждый фрагмент гена лигировали с вектором pECCG 117_Pcj1_GFP терминатор, который обрабатывали ферментами рестрикции EcoRY-Xbal, с использованием набора для клонирования In-FusionR HD (Clontech). Полученный вектор был назван pECCG117_Pcj1_Ava_ABCD, и успех клонирования и последовательности гена вектора подтверждали путем секвенирования. Нуклеотидная последовательность гена Ava_ABCD представлена в SEQ ID NO: 17.
Пример 3: Оценка способности продуцировать шинорин штаммом, в котором активность 2-дегидро-3-дезоксифосфогептонатальдолазы усилена
Плазмиду pECCG117_Pcj1_Ava_ABCD, полученную в примере 2, путем электропорации вводили в каждый из штаммов, полученных в примере 1, в котором усилен ген aroG, и штамм дикого типа W3110, и каждый трансформированный штамм высевали на твердую среду LB. Штаммы выращивали в инкубаторе в течение ночи при 37°С и затем одну платиновую петлю со штаммом инокулировали в 25 мл среды для титрования [состав среды: глюкоза 40 г/л, KH2PO4 0,3 г/л, K2HPO4 0,6 г/л, (NH4)2SO4 15 г/л, MgSO4⋅7H2O 1 г/л, NaCl 2,5 г/л, цитрат натрия 1,2 г/л, дрожжевой экстракт 2,5 г/л, карбонат кальция 40 г/л: рН 7,0], которую инкубировали в инкубаторе при 37°С при 200 об./мин в течение 48 часов. Получающиеся в результате штаммы анализировали при помощи HPLC (Waters Corp.) и результаты представлены в таблице 3 ниже.
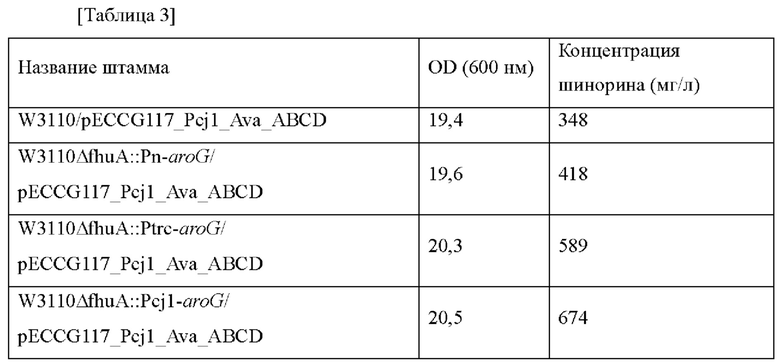
Как представлено в таблице 3 выше, концентрация шинорина продуцируемого в штамме (W3110ΔfhuA::Pn-arcG/pECCG117_Pcj1_Ava_ABCD), в котором усилен ген aroG, увеличивалась приблизительно на 20% по сравнению с контрольной группой. В частности, в случае штаммов, в которых усилен ген aroG путем усиления промотора (т.е. W3110ΔfhuA::Ptrc-araG/pECCG117_Pcj1_Ava_ABCD и W3110ΔfhuA::Pcj1-aroG/pECCG117_Pcj1_Ava_ABCD), концентрация шинорина увеличивалась на величину, составляющую 69% и 94% соответственно.
Пример 4: Получение штамма, в котором усилена активность фосфоенолпируватсинтетазы
Для увеличения способности микроорганизма продуцировать МАА получали штаммы Е. coli, в которых усилена активность фосфоенолпируватсинтетазы. В частности, ген pps (фосфоенолпируватсинтетаза; SEQ ID NO: 18 и 19) дополнительно вводили на основе штамма Е. coli W3110. Матрица и праймеры, используемые для получения плазмиды, представлены в таблице 4 ниже.
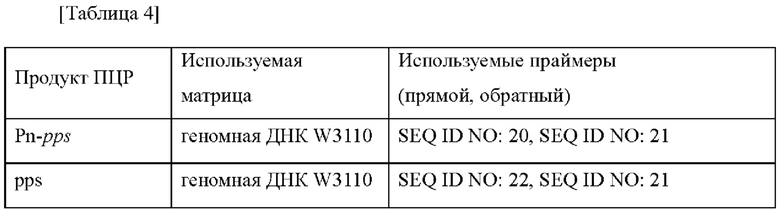
После амплификации фрагментов гена с использованием вышеприведенных матрицы и праймеров фрагмент гена Pn_pps лигировали с вектором pSKH-ΔfhuA, который разрезали при помощи ферментов рестрикции Spel-EcoRY, с использованием набора для клонирования m-FusionR HD (Clontech). После разрушения фрагментов гена Ptrc и Pcj1, полученных в примере 1, при помощи ферментов рестрикции Spe1-Nde1, и разрушения фрагмента гена pps при помощи ферментов рестрикции NdeI-EcoRV, фрагменты гена. Ptrc и pps или фрагменты тепа. Pcj1 и pps соответственно лигировали с вектором pSKH-ΔfhuA, который разрезали при помощи ферментов рестрикции Spel-EcoRV, с использованием набора для клонирования In-Fusion HDR (Clontech). Полученные векторы были названы pSKH-ΔfhuA-Pn-pps, pSKH-ΔfhuA-Ptrc-pps и pSKH-ΔfhuA-Pcj1-pps, соответственно.
Вышеприведенные векторы подтверждали путем секвенирования в отношении успеха клонирования и последовательности гена в каждом векторе, и затем трансформировали в каждый штамм дикого типа Е. coli W3110 путем электропорации. Каждый трансформированный ген вводили в хромосому путем первичной рекомбинации (кроссовер), и область плазмиды вырезали из хромосомы путем вторичной рекомбинации (кроссовер). Для каждого из трансформированных штаммов Е. coli, в котором вторичная рекомбинация завершена, введение гена pps подтверждали путем ПЦР с использованием праймеров SEQ ID NO: 14 (прямой) и 21 (обратный).
Пример 5: Оценка способности продуцировать шинорин штаммом, в котором усилена активность фосфоенолпируватсинтетазы
Плазмиду pECCG117_Pcj1_Ava_ABCD, полученную в примере 2, путем электропорации вводили в каждый из штаммов, полученных в примере 4, в которые введен ген pps, и штамм дикого типа W3110, и каждый трансформированный штамм высевали на твердую среду LB. Штаммы выращивали в инкубаторе в течение ночи при 37°С, и одну платиновую петлю со штаммом инокулировали в 25 мл среды для титрования в соответствии с примером 3, которую затем инкубировали в инкубаторе при 37°С при 200 об./мин в течение 48 часов. Результаты представлены в таблице 5 ниже.
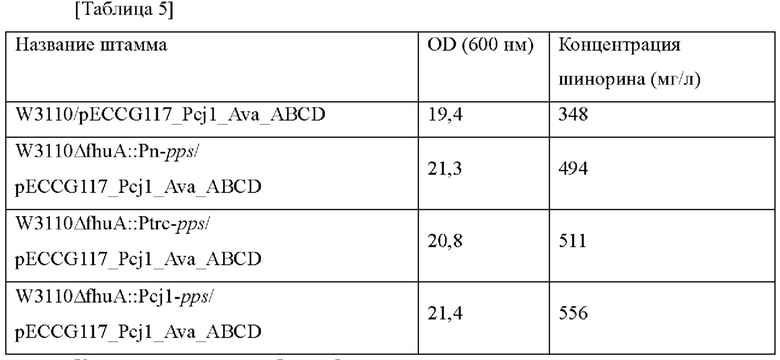
Как представлено в таблице 5 выше, концентрация шинорина, продуцируемого в штамме, в котором усилен ген pps, увеличивалась на 41%, и в случае, когда его активность усилена путем замены на сильный промотор, концентрация шинорина увеличивалась на величину, составляющую до 60%, по сравнению с контрольной группой.
Пример 6: Получение штамма, в котором усилена активность транскетолазы
Для увеличения способности микроорганизма продуцировать МАА получали штаммы Е. coli, в которых усилена активность транскетолазы. В частности, в штамм Е. coli W3110 вводили ген tktA (транскетолаза; SEQ ID NO: 23 и 24). Матрица и праймеры, используемые для получения плазмиды, представлены в таблице 6 ниже.
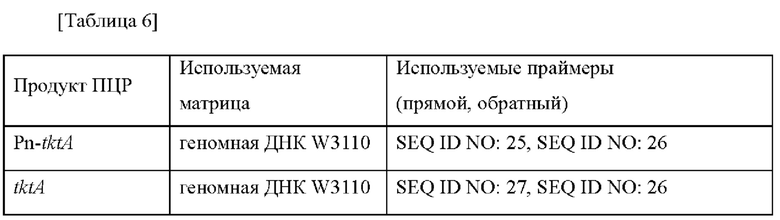
После амплификации фрагментовгена путем ПЦР с использованием вышеприведенных матрицы и праймеров фрагмент гена Vn_tktA лигировали с вектором pSKH-ΔfhuA, который разрезали при помощи ферментов рестрикции Spel-EcoRY, с использованием набора для клонирования In-FusionR HD (Clontech). После разрезания фрагментов гена Ptrc и Pcj1, полученных в примере 1, при помощи ферментов рестрикции Spe1, Nde1 и разрушения фрагмента гена tktA при помощи ферментов рестрикции Nde1-EcoRY, фрагменты гена Ptrc и tktA или фрагменты гена Pcj1 и tktA соответственно лигировали с вектором pSKH-ΔfhuA, который разрезали при помощи ферментов рестрикции Spel-EcoRV, с использованием набора для клонирования ш-Fusion HDR (Clontech). Полученные векторы названы соответственно pSKH-ΔfhuA-Pn-tktA, pSKH-ΔfhuA-Ptrc-tAtA и pSKH-ΔfhuA-Pcj1-tktA.
Вышеприведенные векторы подтверждали путем секвенирования в отношении успеха клонирования и последовательности гена в каждом векторе, и затем трансформировали в каждый штамм дикого типа Е. coli W3110 путем электропорации. Каждый трансформированный ген вводили в хромосому путем первичной рекомбинации (кроссовер), и область плазмиды вырезали из хромосомы путем вторичной рекомбинации (кроссовер). Для каждого из трансформированных штаммов Е. coli, в котором вторичная рекомбинация завершена, введение гена tktA подтверждали путем ПЦР с использованием праймеров SEQ ID NO: 14 (прямой) и 26 (обратный).
Пример 7: Оценка способности продуцировать шинорин в штамме, в котором усилена активность транскетолазы
Плазмиду pECCG117_Pcj1_Ava_ABCD, полученную в примере 2, путем электропорации вводили в каждый из штаммов, полученных в примере 6, в который введен ген tktA, и штамм дикого типа W3110, и каждый трансформированный штамм высевали на твердую среду LB. Штаммы выращивали в инкубаторе в течение ночи при 37°С, и одну платиновую петлю с ночной культурой каждого штамма инокулировали в 25 мл среды для титрования в соответствии с примером 3, которую затем инкубировали в инкубаторе при 37°С при 200 об./мин в течение 48 часов. Результаты представлены в таблице 7 ниже.
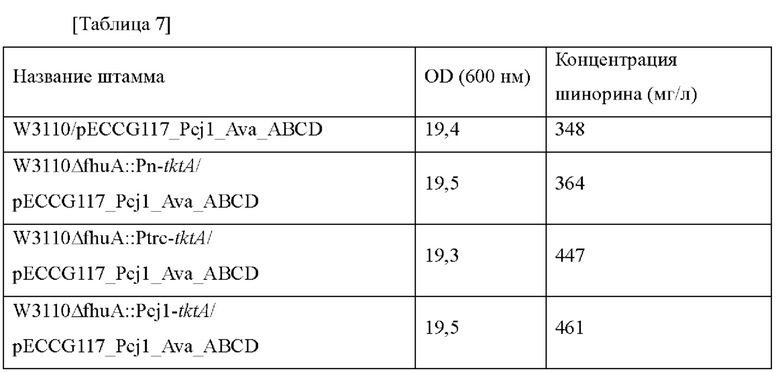
Как представлено в таблице 7 выше, концентрация шинорина продуцируемого в штамме, в котором усилен ген tktA, увеличивалась на 4,5%, и в случае, когда его активность усилена путем замены на сильный промотор, тогда концентрация шинорина увеличивалась на величину, составляющую до 32%, по сравнению с контрольной группой.
Пример 8: Получение штамма, в котором усилены активности 2-дегидро-3-дезоксифосфогептонатальдолазы/фосфоенолпируватсинтетазы/транскетолазы
Для увеличения способности микроорганизма продуцировать МАА получали штаммы Е. coli, в которых усилена активность каждой из 2-дегидро-3-дезоксифосфогептонатальдолазы/фосфоенолпируватсинтетазы/транскетолазы. В частности, в штамм Е. coli W3110 дополнительно вводили каждый из гена aroG, гена pps и гена tktA. Матрицы и праймеры, используемые для получения плазмид, представлены в таблице 8 ниже.
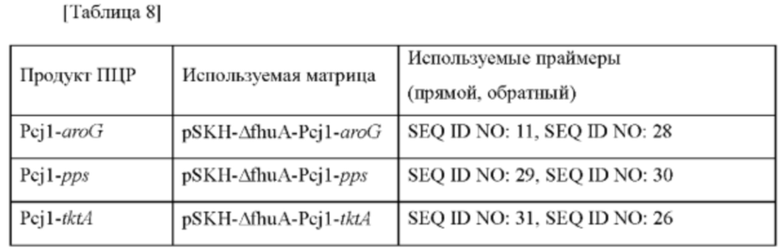
После амплификации фрагментов гена с использованием вышеприведенных матриц и праймеров каждый фрагмент гена лигировали с вектором pSKH-ΔfhuA, который разрезали при помощи ферментов рестрикции Spel-EcoRY, с использованием набора для клонирования In-FusionR HD (Clontech). Полученный вектор назван pSKH-ΔfhuA-Pcj1-aroG-Pcj1-ppsA-Pcj1-tktA.
Вышеприведенный вектор подтверждали путем секвенирования в отношении успеха клонирования и последовательностей генов в векторе и затем путем электропорации трансформировали в штамм дикого типа Е. coli W3110. Трансформированные гены вводили в хромосому путем первичной рекомбинации (кроссовер), и область плазмиды вырезали из хромосомы путем вторичной рекомбинации (кроссовер). Для трансформированных штаммов Е. coli, в которых вторичная рекомбинация завершена, введение генов aroG, pps и tktA подтверждали путем ПЦР с использованием праймеров SEQ ID NO: 14 (прямой) и 26 (обратный).
Пример 9: Оценка способности продуцировать шинорин в штамме, в котором усилены активности 2-дегидро-3-дезоксифосфогептонатальдолазы, фосфоенолпируватсинтетазы и транскетолазы
Плазмиду pECCG117_Pcj1_Ava_ABCD, полученную в примере 2, путем электропорации вводили в каждый из штаммов, полученных в примере 8, в который введены гены aroG, pps и tktA, и штамм дикого типа W3110, и каждый трансформированный штамм высевали на твердую среду LB. Штаммы выращивали в инкубаторе в течение ночи при 37°С, и одну платиновую петлю с ночной культурой каждого штамма инокулировали в 25 мл среды для титрования в соответствии с примером 3, которую затем инкубировали в инкубаторе при 37°С при 200 об./мин в течение 48 часов. Результаты представлены в таблице 9 ниже.
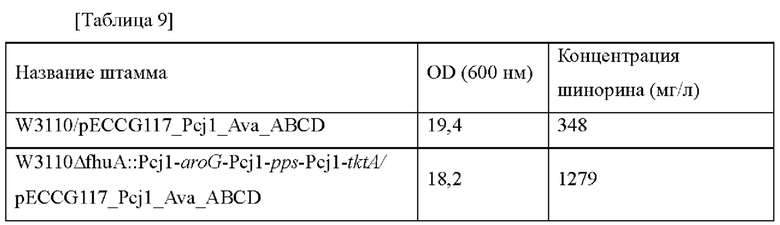
Как представлено в таблице 9 выше, концентрация шинорина, продуцируемого в штамме, в котором три типа генов (т.е. aroG, pps и tktA) комбинированы и усилены, увеличивалась на 267% по сравнению с контрольной группой. Этот результат является неожиданным, демонстрируя улучшение, превосходящее ожидание по сравнение с суммой максимальных увеличений, обнаруженных при замене промотора каждого гена на сильный промотор. То есть, последнее подтверждает то, что когда комбинируют три гена (т.е. aroG, pps и tktA), тогда возможно продуцировать шинорин в более высокой концентрации.
Пример 10: Получение штамма, в котором инактивирована активность 3-дегидрохинатдегидратазы
Для увеличения способности микроорганизма продуцировать МАА получали штаммы Е. coli, в которых инактивирована активность 3-дегидрохинатдегидратазы (aroD).
В частности, ген резистентности к хлорамфениколу плазмиды pKD3 использовали в качестве маркера встраивания гена, и кассету с делецией по aroD, которая включает часть гена aroD и ген резистентности к хлорамфениколу пазмиды pKD3, получали путем ПЦР с использованием праймеров SEQ ID NO: 32 (прямой) и 33 (обратный). Компетентные клетки получали путем трансформации штамма дикого типа Е. coli W3110 и штамма, полученного в примере 8, в который введены гены aroG, pps и tktA, плазмидой pKD46, включающей ген рекомбиназы фага лямбда-ред, а затем путем индукции экспрессии соответствующего гена с использованием арабинозы. После введения кассеты с делецией по aroD в компетентные клетки путем электропорации получающиеся в результате компетентные клетки высевали на твердую среду LB, содержащую 30 мг/л хлорамфеникола. Полученный таким образом штамм подвергали ПЦР с использованием праймеров SEQ ID NO: 34 (прямой) и 35 (обратный), и делецию по гену aroD подтверждали путем обнаружения амплифицированного фрагмента 1300 п.о.
Пример 11: Оценка способности продуцировать шинорин штаммом, в котором инактивир ована 3 -дегидрохинатдегидр атаза
Плазмиду pECCG117 Pcj1_Ava_ABCD, полученную в примере 2, путем электропорации вводили в штамм, полученный в примере 10, в котором осуществлена делеция по гену aroD, и получающийся в результате штамм высевали на твердую среду LB. Штамм выращивали в инкубаторе в течение ночи при 37°С, и одну платиновую петлю со штаммом инокулировали в 25 мл среды для титрования в соответствии с примером 3, которую инкубировали в инкубаторе при 37°С при 200 об./мин в течение 48 часов. Результаты представлены в таблице 10 ниже.
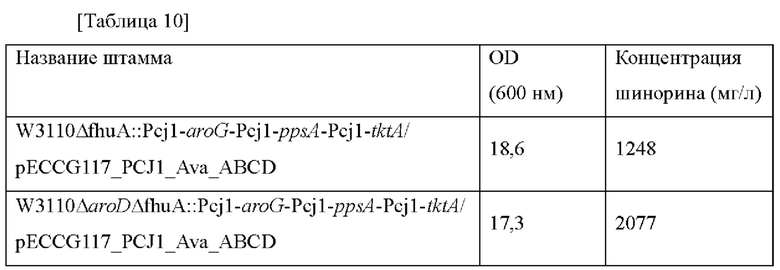
Как представлено в таблице 10 выше, концентрация шинорина, продуцируемого в штамме, в котором ген aroD дополнительно подвергнут делеции, увеличивалась на 66% по сравнению со штаммом, продуцирующим шинорин, в котором усилены гены aroG, pps и tktA. Штамм W3110ΔfhuA::Pcj1-aroG-Pcj1-ppsA-Pcj1-tktA/pECCG117_PCJ1_Ava_ABCD, который представляет собой штамм, в котором усилены гены aroG, pps и tktA, назван СВ06-0020 и депонирован в соответствии с Будапештским договором о международном признании депонирования микроорганизмов для целей патентной процедуры 14 февраля 2018 года в Корейском центре культур микроорганизмов (KCCM) и под идентификационным номером КССМ12224Р.
Получение рекомбинантного микроорганизма на основе Corynebacterium gtutamicum, продуцирующего МАА, и продукция МАА с его использованием
Пример 12: Получение вектора, в котором усилена активность 2-дегидро-3-дезоксифосфогептонатальдолазы, и оценка его способности продуцировать шинорин
Для увеличения способности микроорганизма продуцировать МАА получали штаммы Е. coli, в которых усилена активность 2-дегидро-3-дезоксифосфогептонатальдолазы. В частности, ген aroG (2-дегидро-3-дезоксифосфогептонатальдолаза; SEQ ID NO: 36 и 37) дополнительно вводили на основе штамма Corynebacterium glutamicum АТСС13032. Матрица и праймеры, используемые для получения плазмиды, представлены в таблице 11 ниже.
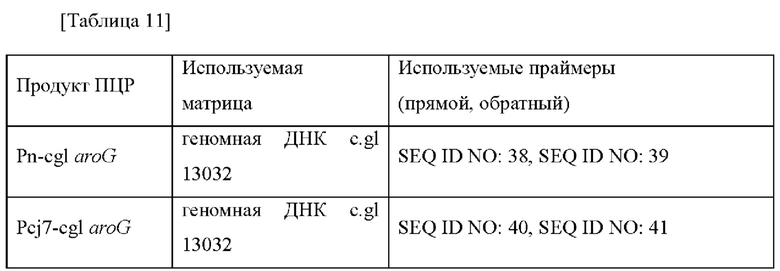
После получения фрагментов гена с использованием вышеприведенных матрицы и праймеров каждый фрагмент гена лигировали с векторами ECCG 117 и pECCG 117_Pcj7_GFP_терминатор (Корейский патент №10-620092, p117-cj7-gfp), которые обрабатывали ферментами рестрикции EcoRV/XbaI, с использованием набора для клонирования m-FusionR HD (Clontech). Полученные векторы названы pECCG117 Pn_cg1_aroG и pECCG117 Pcj7_cg1_aroG, соответственно. Вышеприведенные векторы подтверждали путем секвенирования в отношении успеха клонирования и последовательности гена в каждом векторе.
Во-первых, поскольку микроорганизм рода Corynebacterium не может продуцировать шинорин, получали штамм, в который введен путь биосинтеза шинорина. В частности, ген Ava_ABCD подвергали ПЦР с использованием pECCG117_Ptrc_Ava_ABCD в качестве матрицы вместе с парой праймеров SEQ ID NO: 42 (прямой) и 43 (обратный). pDZTn_Ava_ABCD получали путем лигирования приблизительно 7 т.п.о. фрагмента ПЦР с вектором pDZTn (WO 2009-125992А), который обрабатывали ферментом рестрикции NdeI, с использованием набора для клонирования In-FusionR HD (Clontech). Затем фрагмент промотора O2 (Корейский патент №10-1632642) подвергали ПЦР с использованием пары праймеров SEQ ID NO: 44 (прямой) и 45 (обратный), и лигировали в pDZTn_Ava_ABCD, который обрабатывали ферментом рестрикции NdeI, с использованием набора для клонирования ш-Fusion HD (Clontech), таким образом получая pDZTn_PO2_Ava_ABCD.
Рекомбинантную плазмиду трансформировали в штамм дикого типа АТСС13032 путем электропорации (van der Rest et al. 1999) и плазмиду вводили в хромосому путем первичной рекомбинации (кроссовер), и область плазмиды вырезали из хромосомы путем вторичной рекомбинации (кроссовер).
Для каждого из трансформированных штаммов Corynebacterium glutamicum, в котором вторичная рекомбинация завершена, введение гена Ava_ABCD подтверждали путем ПЦР с использованием специфической для гена пары праймеров SEQ ID NO: 42 (прямой) и 43 (обратный). Полученный штамм назван Corynebacterium glutamicum 13032 ΔN1021PO2_Ava_ABCD.
Каждый из векторов pECCG117_Pn_cgl aroG и pECCG117_Pcj7_cgl aroG путем электропорации трансформировали в штамм Corynebacterium glutamicum 13032 ΔN1021_PO2_Ava_ABCD.
Полученные выше штаммы и контрольную группу Corynebacterium glutamicum АТСС13032 (cgl 13032) выращивали в течение ночи в твердой среде BHIS, содержащей канамицин, и одну платиновую петлю со штаммом инокулировали в 25 мл среды для титрования [состав среды: глюкоза 40 г/л, KH2PO4 1 г/л, (NH4)2SO4 10 г/л, MgSO47H2O 5 г/л, NaCl 5 г/л, дрожжевой экстракт 5 г/л, карбонат кальция 30 г/л: рН 7,0], которую инкубировали в инкубаторе при 37°С при 200 об./мин в течение 48 часов. Результаты представлены в таблице 12 ниже.
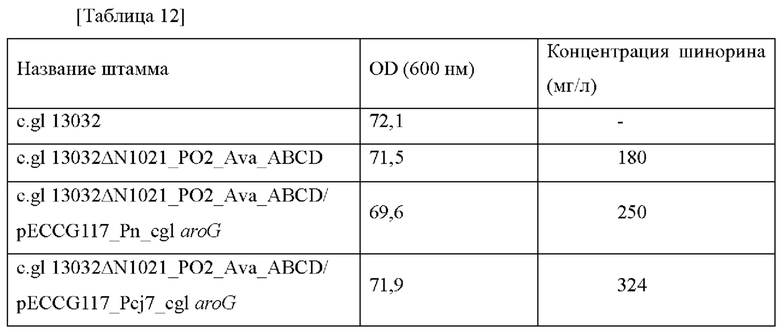
Как представлено в таблице 12 выше, когда уровень экспрессии aroG увеличивался в штамме, содержащем ген биосинтеза шинорина, тогда концентрация шинорина увеличивалась на 39%. В частности, когда промотор был усилен, тогда это подтверждало то, что концентрация шинорина может увеличиваться на величину, составляющую до 80%.
Пример 13: Получение вектора, в котором усилены активности фосфоенолпируватсинтетазы/транскетолазы, и оценка его способности продуцировать шинорин
Для увеличения способности микроорганизма продуцировать МАА получали штаммы Corynebacterium glutamicum, в которых усилена активность tkt или pps. В частности, дополнительно вводили tkt (транскетолаза; SEQ ID NO: 95 и 96) или pps (фосфоенолпируватсинтетаза; SEQ ID NO: 97 и 98) на основе штамма Corynebacterium glutamicum АТСС13032. Матрица и праймеры, используемые для получения плазмиды, представлены в таблице 13 ниже.
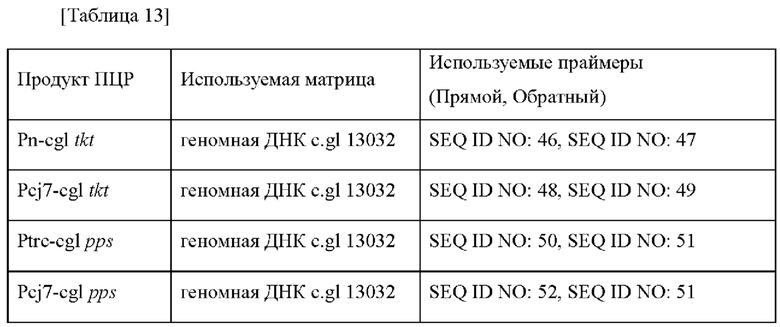
Векторы получали путем лигирования фрагментов гена, которые получали при помощи технологии ПЦР, в которой матрица соответствовала комбинации праймеров, с векторами pECCG117, pECCG117_Ptrc_GFP_терминатор и pECCG117_Pcj7_GFP терминатор, которые обрабатывали ферментами рестрикции EcoRY/XbaI, с использованием набора для клонирования In-Fusion HDR (Clontech). Полученные векторы названы pECCG117-Pn-tkt/pECCG117-Pcj7-tkt и pECCG117-Ptrc-pps/pECCG117-Pcj7-pps соответственно. Вышеприведенные векторы подтверждали путем секвенирования в отношении успеха клонирования и последовательности гена в каждом векторе и затем путем электропорации трансформировали в штамм Corynebacterium glutamicum 13032 ΔN1021_PO2_Ava_ABCD. Каждый штамм выращивали на содержащей канамицин твердой среде BHIS в течение ночи, и платиновую петлю со средой инокулировали в 25 мл среды для титрования в соответствии с примером 12, которую инкубировали в инкубаторе при 37°С при 200 об./мин в течение 48 часов. Результаты представлены в таблице 14 ниже.
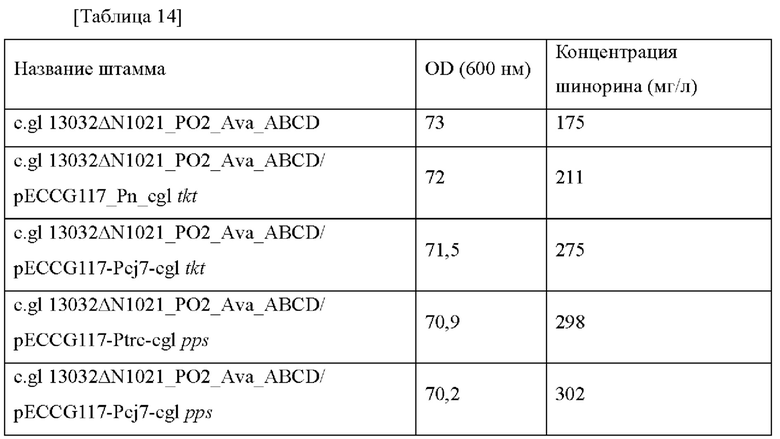
Как представлено в таблице 14 выше, подтверждено, что когда ген tkt или ген pps усилен, тогда продукция шинорина увеличивалась на величину, составляющую до 57% или 72% соответственно.
Пример 14: Получение штамма, в котором усилены активности 2-дегидро-3-дезоксифосфогептонатальдолазы/фосфоенолпируватсинтетазы/тр анскетолазы, и его оценка
Для увеличения способности микроорганизма продуцировать МАА получали штаммы Е. coli, в которых усилены активности генов aroG, pps и tkt, и для подтверждения большей продукции МАА дополнительно инактивировали 3-дегидрохинатдегидратазу (aroD). В частности, для усиления генов aroG, pps и tkt получали плазмиду pDZ-ΔaroD-Pcj7-aroG-Pcj7-pps-Pcj7-tktA. Матрицы и праймеры, используемые для получения плазмиды pDZ-ΔaroD-Pcj7-aroG-Pcj7-pps-Pcj7-tktA, представлены в таблице 15 ниже.
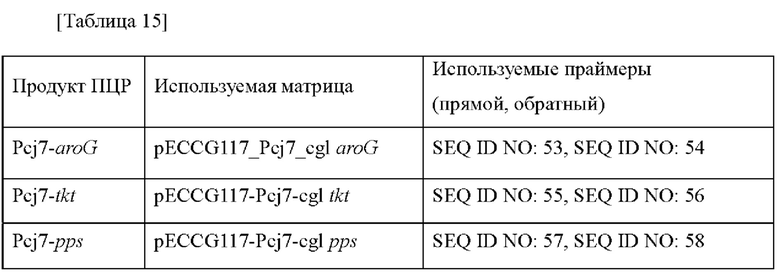
Во-первых, для получения штамма Corynebacterium glutamicum, в котором осуществлена делеция по гену aroD (SEQ ID NO: 89 и 90), получали плазмиду pDZ-AaroD, в которой открытая рамка считывания гена aroD подвергнута внутренней делеции. Внутреннюю делецию гена в плазмиде pDZ-AaroD осуществляли путем проведения перекрестной ПЦР с использованием геномной ДНК штамма Corynebacterium glutamicum АТСС 13032 в качестве матрицы вместе с SEQ ID NO: 91 и 92 и SEQ ID NO: 93 и 94 в качестве пары прямого и обратного праймеров в последующим введением получающихся в результате фрагментов гена в вектор pDZ.
Затем каждый фрагмент генов aroG, pps и tkt амплифицировали при помощи ПЦР с использованием матриц и праймеров, представленных в таблице 15 выше, и затем вводили в вектор pDZ-AaroD, соответственно расщепленный при помощи фермента рестрикции SpeI. Вышеприведенные два типа векторов подтверждали путем секвенирования в отношении успеха клонирования и последовательности гена в каждом векторе и затем трансформировали путем электропорации в штамм Corynebacterium glutamicum 13032ΔN1021_PO2_Ava_ABCD. Каждый штамм выращивали на содержащей канамицин твердой среде BHIS в течение ночи, и платиновую петлю с ночной культурой каждого штамма инокулировали в 25 мл среды для титрования в соответствии с примером 12, которую инкубировали в инкубаторе при 37°С при 200 об./мин в течение 48 часов. Результаты представлены в таблице 16 ниже.
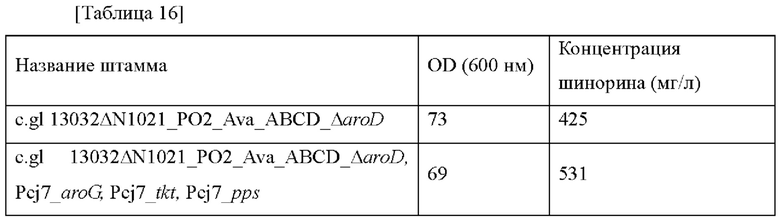
Как представлено в таблице 16 выше, концентрация шинорина, продуцируемого в штамме, в котором усилены три типа генов {aroG, pps и tktA), увеличивалась на приблизительно 25%. Подтверждено, что даже в штамме, в котором способность продуцировать шинорин увеличена путем делеции по гену aroD, шинорин может продуцироваться в высокой концентрации путем комбинации трех типов генов. Дополнительно, можно интерпретировать, что когда ген aroD дополнительно инактивирован в штамме, в котором комбинированы три типа генов, тогда шинорин может продуцироваться при еще более высокой концентрации.
Получение рекомбинантного микроорганизма на основе дрожжей, продуцирующего МАА, и продукция МАА с его использованием
Пример 15: Получение штамма Saccharomyces cerevisiae (S. cerevisiae), продуцирующего шинорин
Для использования штамма Saccharomyces cerevisiae (S. cerevisiae) в качестве штамма, продуцирующего шинорин, ген биосинтеза шинорина, полученный из А. variabilis АТСС29413, вводили в вектор для экспрессии в дрожжах. Гены Ava_A и Ava_B встраивали в вектор pRS-413 с использованием промотора GPD. В частности, области pGPD-Ava_A и pGPD-Ava_B лигировали при помощи ПЦР с перекрывающимися праймерами. Векторы и продукты ПЦР обрабатывали ферментами рестрикции BamIIl и SalI и затем лигировали с использованием лигазы Т4 для получения вектора pRS-413-pGPD-Ava_A-pGPD-Ava_В.
Затем гены Ava_A и Ava_B встраивали в вектор pRS-414 с использованием промотора GPD. В частности, области pGPD-Ava_С и pGPD-Ava_D лигировали при помощи ПЦР с перекрывающимися праймерами, и затем векторы и продукты ПЦР обрабатывали BamHI и SalI и лигировали с использованием лигазы Т4 для получения вектора pRS-414-pGPD-Ava_C-pGPD-Ava_D. Праймеры и матрицы ДНК, используемые для получения вектора, представлены в таблице 17 ниже.
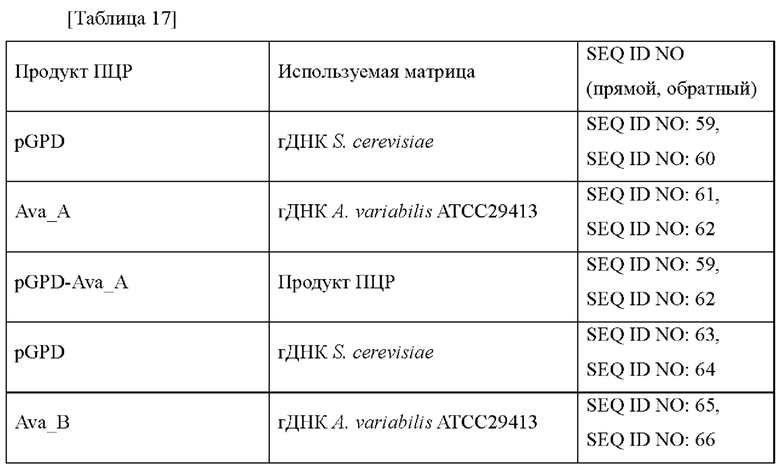

Вектор pRS-413-pGPD-Ava_A-pGPD-Ava_B и вектор pRS-414-pGPD-Ava_C-pGPD-Ava_D вводили в штамм Saccharomyces cerevisiae CEN.PK-1D (S. cerevisiae CEN.PK-1D) путем трансформации при помощи ацетата лития, и затем подтверждали продукцию шинорина. Этот штамм высевали на синтетической полной (SC) твердой среде, из которой исключены Trp и His (т.е. ауксотрофные маркеры), и выращивали в течение ночи в инкубаторе при 30°С Одну платиновую петлю со штаммом, выращенным в течение ночи в синтетической полной (SC) твердой среде, из которой исключены Trp и His, инокулировали в 25 мл среды для титрования в соответствии с таблицей 18, и затем выращивали в инкубаторе при 30°С при 150 об./мин в течение 24 часов. Результаты представлены в таблице 19 ниже.
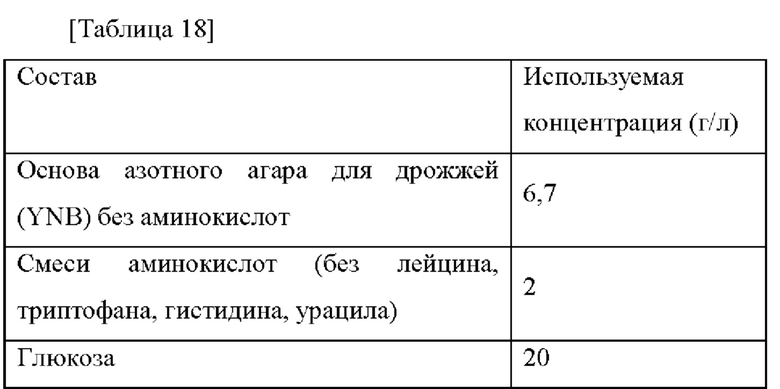
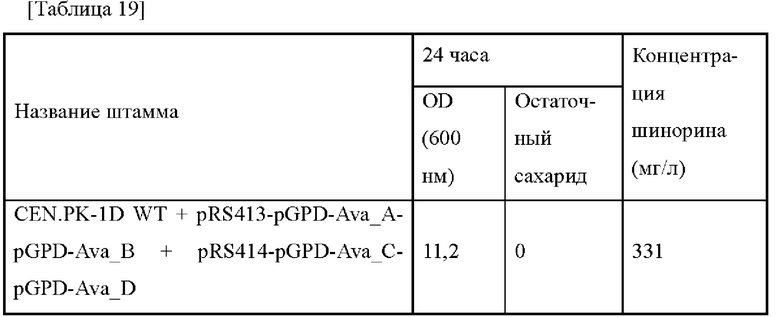
В качестве результата эксперимента подтвердили, что штамм дикого типа Saccharomyces cerevisiae, не продуцирующий шинорин, продуцировал 331 мг/л шинорина вследствие введения гена биосинтеза шинорина.
Пример 16: Увеличение продукции шинорина путем усиления TKL1 (транскетолазы) Saccharomyces cerevisiae
Для увеличения способности продуцировать МАА получали штамм Saccharomyces cerevisiae, в котором усилена активность TKL1. Для этой задачи экспрессию гена TKL1 усиливали путем клонирования гена TKL1 (SEQ ID NO: 110 и 123) в векторы pRS-415-pGPD, pRS-415-pADH и pRS-415-pTEF.
Промотор GPD, включенный в вектор pRS-415-pGPD, представляет собой промотор генов глицеральдегид-3-фосфатдегидрогеназы (GAPDH) и изозима 3 (TDH3), и он включает последовательность от -674 п.о. до -1 п.о. относительно кодона инициации ORF гена THD3.
Промотор ADH, включенный в вектор pRS-415-pADH, представляет собой промотор гена алкогольдегидрогеназы (ADH1), и он включает последовательность от -1500 п.о. до -1 п.о. относительно кодона инициации ORF гена.ADH1.
Промотор TEF, включенный в вектор pRS-415-pTEF, представляет собой промотор фактора элонгации трансляции гена EF-1 альфа (TEFL), и он включает последовательность от -500 п. о. до -1 п. о. относительно кодона инициации ORF гена TEFL
В частности, ген TEF1 подвергали ПЦР с использованием праймеров в соответствии с таблицей 20 ниже, и продукты ПЦР и векторы pRS-415-pGPD, pRS-415-pADH и pRS-415-pTEF обрабатывали ферментами рестрикции BamHI и SalI, и затем лигировали с использованием лигазы Т4 с получением векторов pRS-415-pGPD-TKL1, pRS-415-pADH-TKL1 и pRS-415-pTEF-TKL1.

Затем, плазмиду для биосинтеза шинорина, полученную в примере 15, вводили в штамм Saccharomyces cerevisiae CEN.PK-1D вместе с pRS-415-pGPD-TKL1, pRS-415-pADH-TKL1 и pRS-415-pTEF-TKL1, и каждый получающийся в результате штамм высевали на синтетической полной твердой среде (SC), из которой исключены Trp, Ura и His, и выращивали в течение ночи в инкубаторе при 30°С Одну платиновую петлю со штаммом, выращиваемым в течение ночи в синтетической полной твердой среде (SC), из которой исключены Trp, Ura и His, инокулировали в 25 мл среды для титрования, и затем выращивали в инкубаторе при 30°С при 150 об./мин в течение 24 часов. Результаты представлены в таблице 21 ниже.
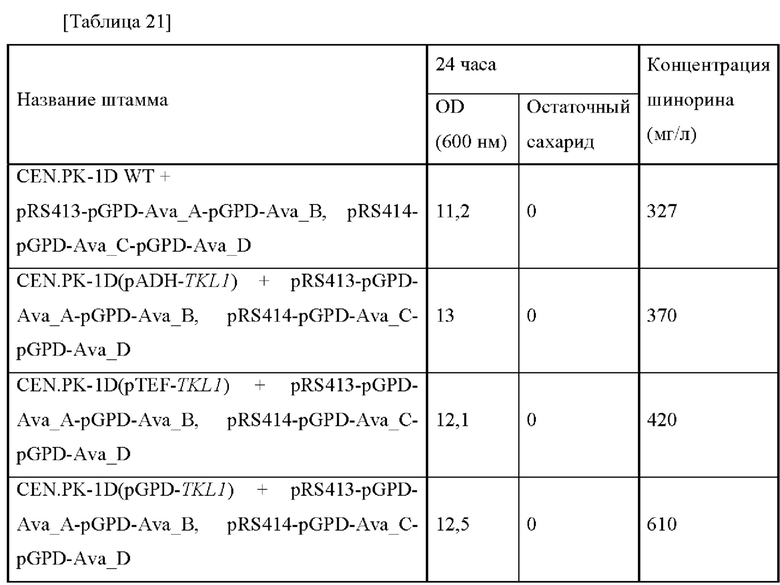
Как представлено в таблице 21 выше, подтверждено, что продукция шинорина увеличивалась в штамме, в котором экспрессия гена TKL1 усилена с использованием промотора GPD, по сравнению со штаммом WT. Дополнительно, также подтверждено, что по мере усиления силы промотора (т.е. pGPD>pTEF>pADH) увеличивается продукция шинорина.
Пример 17: Увеличение продукции шинорина путем усиления ARQ4 (3-дезокси-D-арабино-гептулозонат-7-фосфат (DAHP) синтазы) в Saccharomyces cerevisiae
Для увеличения способности продуцировать МАА получали штаммы Saccharomyces cerevisiae, в которых усилена активность ARO4. Для этой задачи использовали стратегию, при которой экспрессию гена ARO4 увеличивали путем клонирования гена AR04 (SEQ ID NO: 111 и 124) в векторы pRS-415-pGPD, pRS-415-pADH и pRS-415-pTEF. В частности, ген ARO4 подвергали ПЦР с использованием праймеров в соответствии с таблицей 22 ниже, и продукты ПЦР векторов ARO4 и pRS-415-pGPD, pRS-415-pADH и pRS-415-pTEF обрабатывали ферментами рестрикции BamHI и SalI, и затем лигировали с использованием лигазы Т4 с получением векторов pRS-415-pGPD-ARO4, pRS-415-pADH-ARO4 и pRS-415-pTEF-ARO4.

Затем, плазмиду для биосинтеза шинорина, полученную в примере 15, вводили в штамм Saccharomyces cerevisiae CEN.PK-1D вместе с pRS-415-pGPD-ARO4, pRS-415-pADH-ARO4 и pRS-415-pTEF-ARO4, и каждый получающийся в результате штамм высевали на синтетическую полную (SC) твердую среду, из которой исключены Trp, Ura и His, и выращивали в течение ночи в инкубаторе при 30°С. Одну платиновую петлю со штаммом, выращиваемым в течение ночи в синтетической полной (SC) твердой среде, из которой исключены Trp, Ura и His, инокулировали в 25 мл среды для титрования, и затем выращивали в инкубаторе при 30°С при 150 об./мин в течение 24 часов. Результаты представлены в таблице 23 ниже.
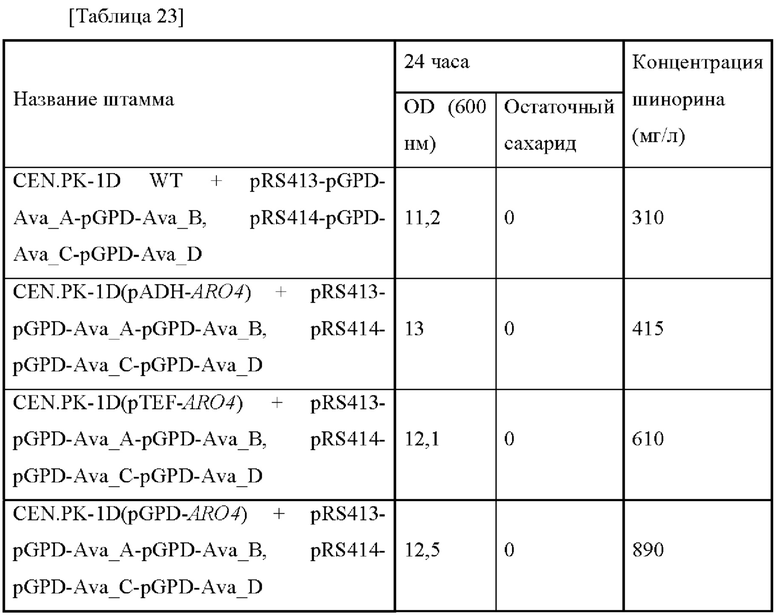
Как представлено в таблице 23 выше, подтверждено, что продукция шинорина увеличилась на 187% в штамме, в котором усилена экспрессия гена ARO4 с использованием промотора GPD по сравнению со штаммом WT.
Пример 18: Увеличение продукции шинорина путем усиления фосфоенолпируватсинтетазы (pps) в Saccharomyces cerevisiae
Для увеличения способности продуцировать МАА получали штаммы Saccharomyces cerevisiae, в которых усилена активность pps. Для этой задачи использовали стратегию, при которой экспрессия гена pps усилена путем клонирования гена pps в векторы pRS-415-pGPD, pRS-415-pADH и pRS-415-pTEF, и усилена экспрессия гена pps.
В частности, ген pps подвергали ПЦР с использованием праймеров в соответствии с таблицей 24 ниже и продукты ПЦР векторов pps и pRS-415-pGPD, pRS-415-pADH и pRS-415-pTEF обрабатывали ферментами рестрикции BamHI и SalI и затем лигировали с использованием лигазы Т4 с получением векторов pRS-415-pGPD-pps, pRS-415-pADH-pps npRS-415-pTEF-pps.
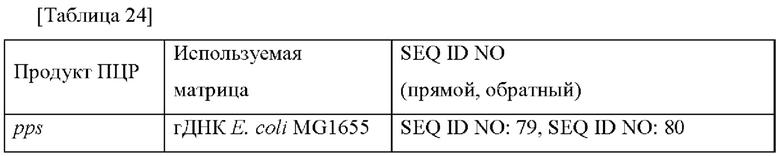
Затем плазмиду для биосинтеза шинорина, полученную в примере 15, вводили в штамм Saccharomyces cerevisiae CEN.PK-1D вместе с pRS-415-pGPD-pps, pRS-415-pADH-pps и pRS-415-pTEF-pps, и каждый получающийся в результате штамм высевали на синтетической полной (SC) твердой среде, из которой исключены Trp, Ura и His, и выращивали в течение ночи при 30°С в инкубаторе. Одну платиновую петлю со штаммом, выращиваемым в течение ночи на синтетической полной (SC) твердой среде, из которой исключены Trp, Ura и His, инокулировали в 25 мл среды для титрования, и затем выращивали в инкубаторе при 30°С при 150 об./мин в течение 24 часов. Результаты представлены в таблице 25 ниже.
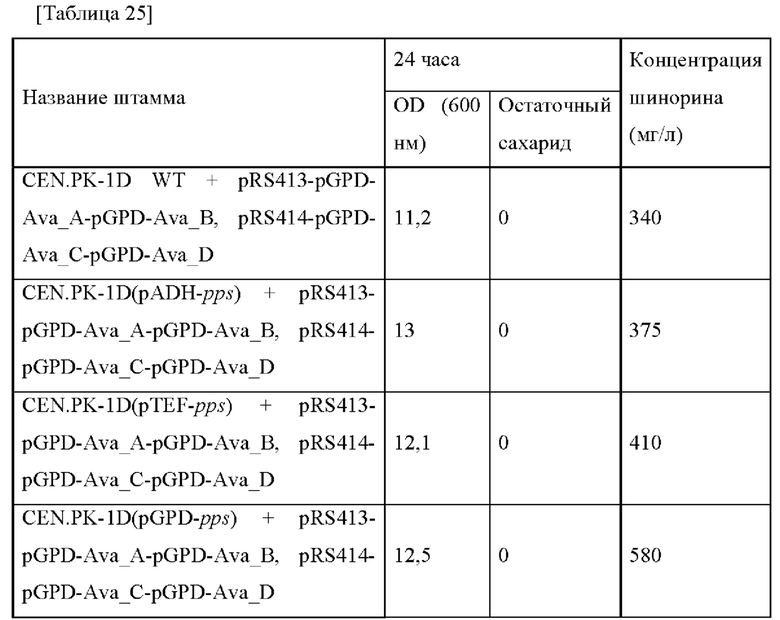
Как представлено в таблице 25 выше, подтверждено, что продукция шинорина увеличивалась на 70% в штамме, в котором происходит сверэкспрессия гена pps, по сравнению со штаммом WT. Дополнительно, также подтвердили, что по мере увеличения силы промотора (т.е. pGPD>pTEF>pADH) увеличивается продукция шинорина.
Пример 19: Увеличение продукции шинорина путем усиления TKL1, усиления ARO4 и введения гена pps в штамм Saccharomyces cerevisiae
На основе результатов примеров 16, 17 и 18 гены TKL1, ARO4 и pps (Е. coli) отобраны в качестве эффективных факторов, которые оказывают влияние на биосинтез шинорина в Saccharomyces cerevisiae, и сделаны попытки увеличить биосинтез шинорина путем одновременного усиления этих трех типов генов. Для введения этих трех типов генов получали векторы pRS-415-pGVD-TKLl-pGVD-ARO4 и pRS-416-pGPD-pps. В частности, после связывания областей pGFD-TKL1 и pGFD-ARO4 при помощи ПЦР с перекрывающимися праймерами векторы и продукты ПЦР обрабатывали ферментами рестрикции BamHI и SalI, и затем лигировали с использованием лигазы Т4 для получения вектора pRS-415-pGRD-TKL1-pGPD-ARO4.
Затем, ген pps, полученный из Е. coli, подвергали ПЦР. Продукты ПЦР гена pps и вектор pRS-416-pGPD обрабатывали ферментами рестрикции BamHI и SailI и затем лигировали с использованием лигазы Т4 для получения вектора pRS-416-pGPD-pps. Праймеры и матрицы ДНК, используемые для получения векторов, представлены в таблице 26 ниже.
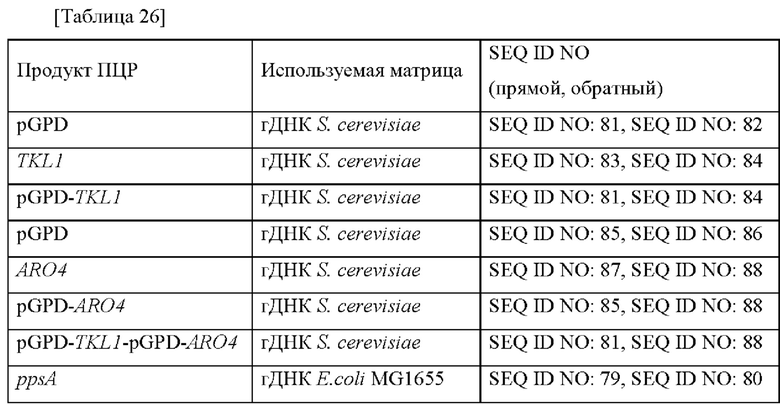
Затем, плазмиду для биосинтеза шинорина, полученную в примере 15, вводили в штамм Saccharomyces cerevisiae CEN.PK-1D вместе с векторами pRS-415-pGPD-TKL1-pGPD-ARO4 и pRS-416-pGPD-pps, и каждый получающийся в результате штамм высевали на синтетическую полную твердую среду (SC), из которой исключены Leu, Trp, Ura и His, и выращивали в течение ночи в инкубаторе при 30°С Одну платиновую петлю со штаммом, выращенным в течение ночи в синтетической полной твердой среде (SC), из которой исключены Leu, Trp, Ura и His, инокулировали в 25 мл среды для титрования, и затем выращивали в инкубаторе при 30°С при 150 об./мин в течение 24 часов. Результаты представлены в таблице 27 ниже.
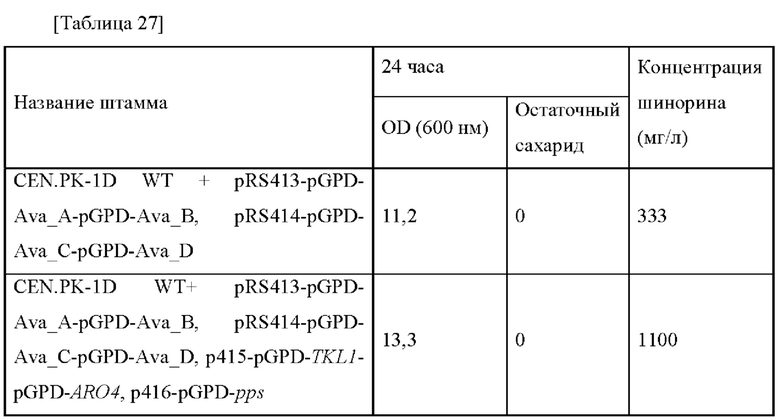
Как представлено в таблице 27 выше, подтверждено, что продукция шинорина значительно увеличивалась на 230% в штамме, в котором осуществляется сверхэкспрессия трех типов эффективных генов (т.е. pps, TKL1 и ARO4), по сравнению со штаммом WT.
В этом описании изобретения подробные изложения содержания, которое может полностью быть понято и принято специалистом в данной области техники, опущены. Дополнительно к конкретным воплощениям, изложенным в этом описании изобретения, возможны различные модификации без изменения технической сущности или важных составляющих описания настоящего изобретения. Таким образом, описание настоящего изобретения может быть реализовано образом, отличающимся от конкретно описанного и подкрепленного примерами в данном описании изобретения, который может быть понятен специалистам в данной области техники.
--->
Последовательности
<110> CJ CheilJedang Corporation
<120> A microorganism for producing a Mycosporine-like amino acid and a
method for preparing a Mycosporine-like amino acid using the same
<130> OPA18547-PCT
<150> KR10-2018-0022185
<151> 2018-02-23
<160> 124
<170> KoPatentIn 3.0
<210> 1
<211> 1053
<212> DNA
<213> Escherichia coli W3110
<400> 1
atgaattatc agaacgacga tttacgcatc aaagaaatca aagagttact tcctcctgtc 60
gcattgctgg aaaaattccc cgctactgaa aatgccgcga atacggttgc ccatgcccga 120
aaagcgatcc ataagatcct gaaaggtaat gatgatcgcc tgttggttgt gattggccca 180
tgctcaattc atgatcctgt cgcggcaaaa gagtatgcca ctcgcttgct ggcgctgcgt 240
gaagagctga aagatgagct ggaaatcgta atgcgcgtct attttgaaaa gccgcgtacc 300
acggtgggct ggaaagggct gattaacgat ccgcatatgg ataatagctt ccagatcaac 360
gacggtctgc gtatagcccg taaattgctg cttgatatta acgacagcgg tctgccagcg 420
gcaggtgagt ttctcgatat gatcacccca caatatctcg ctgacctgat gagctggggc 480
gcaattggcg cacgtaccac cgaatcgcag gtgcaccgcg aactggcatc agggctttct 540
tgtccggtcg gcttcaaaaa tggcaccgac ggtacgatta aagtggctat cgatgccatt 600
aatgccgccg gtgcgccgca ctgcttcctg tccgtaacga aatgggggca ttcggcgatt 660
gtgaatacca gcggtaacgg cgattgccat atcattctgc gcggcggtaa agagcctaac 720
tacagcgcga agcacgttgc tgaagtgaaa gaagggctga acaaagcagg cctgccagca 780
caggtgatga tcgatttcag ccatgctaac tcgtccaaac aattcaaaaa gcagatggat 840
gtttgtgctg acgtttgcca gcagattgcc ggtggcgaaa aggccattat tggcgtgatg 900
gtggaaagcc atctggtgga aggcaatcag agcctcgaga gcggggagcc gctggcctac 960
ggtaagagca tcaccgatgc ctgcatcggc tgggaagata ccgatgctct gttacgtcaa 1020
ctggcgaatg cagtaaaagc gcgtcgcggg taa 1053
<210> 2
<211> 350
<212> PRT
<213> Escherichia coli W3110
<400> 2
Met Asn Tyr Gln Asn Asp Asp Leu Arg Ile Lys Glu Ile Lys Glu Leu
1 5 10 15
Leu Pro Pro Val Ala Leu Leu Glu Lys Phe Pro Ala Thr Glu Asn Ala
20 25 30
Ala Asn Thr Val Ala His Ala Arg Lys Ala Ile His Lys Ile Leu Lys
35 40 45
Gly Asn Asp Asp Arg Leu Leu Val Val Ile Gly Pro Cys Ser Ile His
50 55 60
Asp Pro Val Ala Ala Lys Glu Tyr Ala Thr Arg Leu Leu Ala Leu Arg
65 70 75 80
Glu Glu Leu Lys Asp Glu Leu Glu Ile Val Met Arg Val Tyr Phe Glu
85 90 95
Lys Pro Arg Thr Thr Val Gly Trp Lys Gly Leu Ile Asn Asp Pro His
100 105 110
Met Asp Asn Ser Phe Gln Ile Asn Asp Gly Leu Arg Ile Ala Arg Lys
115 120 125
Leu Leu Leu Asp Ile Asn Asp Ser Gly Leu Pro Ala Ala Gly Glu Phe
130 135 140
Leu Asp Met Ile Thr Pro Gln Tyr Leu Ala Asp Leu Met Ser Trp Gly
145 150 155 160
Ala Ile Gly Ala Arg Thr Thr Glu Ser Gln Val His Arg Glu Leu Ala
165 170 175
Ser Gly Leu Ser Cys Pro Val Gly Phe Lys Asn Gly Thr Asp Gly Thr
180 185 190
Ile Lys Val Ala Ile Asp Ala Ile Asn Ala Ala Gly Ala Pro His Cys
195 200 205
Phe Leu Ser Val Thr Lys Trp Gly His Ser Ala Ile Val Asn Thr Ser
210 215 220
Gly Asn Gly Asp Cys His Ile Ile Leu Arg Gly Gly Lys Glu Pro Asn
225 230 235 240
Tyr Ser Ala Lys His Val Ala Glu Val Lys Glu Gly Leu Asn Lys Ala
245 250 255
Gly Leu Pro Ala Gln Val Met Ile Asp Phe Ser His Ala Asn Ser Ser
260 265 270
Lys Gln Phe Lys Lys Gln Met Asp Val Cys Ala Asp Val Cys Gln Gln
275 280 285
Ile Ala Gly Gly Glu Lys Ala Ile Ile Gly Val Met Val Glu Ser His
290 295 300
Leu Val Glu Gly Asn Gln Ser Leu Glu Ser Gly Glu Pro Leu Ala Tyr
305 310 315 320
Gly Lys Ser Ile Thr Asp Ala Cys Ile Gly Trp Glu Asp Thr Asp Ala
325 330 335
Leu Leu Arg Gln Leu Ala Asn Ala Val Lys Ala Arg Arg Gly
340 345 350
<210> 3
<211> 49
<212> DNA
<213> Artificial Sequence
<220>
<223> fhuA arm 1_F
<400> 3
ccaagcgacg cccaacctgc catcatggcg cgttccaaaa ctgctcagc 49
<210> 4
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> fhuA arm 1_R
<400> 4
tatcctcgag actagtgagc tcctttcagc ggttcggtgg 40
<210> 5
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> fhuA arm 2_F
<400> 5
ctagtctcga ggatatcgac cacaccctgc tgaccggtgt 40
<210> 6
<211> 48
<212> DNA
<213> Artificial Sequence
<220>
<223> fhuA arm 2_R
<400> 6
atagaatacc aattggcatg ctttttagaa acggaaggtt gcggttgc 48
<210> 7
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn_aroG_F
<400> 7
aaaggagctc actagtagga tgctcctgtt atgg 34
<210> 8
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn_aroG_R
<400> 8
cagggtgtgg tcgatatctt acccgcgacg cgcttt 36
<210> 9
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Ptrc_F
<400> 9
aaaggagctc actagtccgc ttgctgcaac tctc 34
<210> 10
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Ptrc_R
<400> 10
tggacgatac tcatatgttt cctgtgtgaa attgttatcc 40
<210> 11
<211> 35
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj1_F
<400> 11
aaaggagctc actagtaccg cgggcttatt ccatt 35
<210> 12
<211> 41
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj1_R
<400> 12
atctatagtc tgcatatgtt aatctcctag attgggtttc a 41
<210> 13
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> aroG_F
<400> 13
taggagatta acatatgaat tatcagaacg ac 32
<210> 14
<211> 18
<212> DNA
<213> Artificial Sequence
<220>
<223> certification primer
<400> 14
ggaacgctca gattgcgt 18
<210> 15
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_ABCD_F
<400> 15
acaatttcac acaggaaaga tatcatgagt atcgtccaag caaag 45
<210> 16
<211> 45
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_ABCD_R
<400> 16
ctcatccgcc aaaacagctc tagattatga attattttcc agaca 45
<210> 17
<211> 6461
<212> DNA
<213> Anabaena variabilis ATCC29413
<400> 17
atgagtatcg tccaagcaaa gtttgaagct aaggaaacat cttttcatgt agaaggttac 60
gaaaagattg agtatgattt ggtgtatgta gatggtattt ttgaaatcca gaattctgca 120
ctagcagatg tatatcaagg ttttggacga tgcttggcga ttgtagatgc taacgtcagt 180
cggttgtatg gtaatcaaat tcaggcatat ttccagtatt atggtataga actgaggcta 240
tttcctatta ccattactga accagataag actattcaaa ctttcgagag agttatagat 300
gtctttgcag atttcaaatt agtccgcaaa gaaccagtat tagtcgtggg tggcggttta 360
attacagatg ttgtcggctt tgcttgttct acatatcgtc gcagcagcaa ttacatccgc 420
attcctacta cattgattgg attaattgat gccagtgtag caattaaggt agcagttaat 480
catcgcaaac tgaaaaaccg tttgggtgct tatcatgctt ctcgcaaagt atttttagat 540
ttctccttgt tgcgtactct ccctacagac caagtacgta acgggatggc ggaattggta 600
aaaatcgctg tagtagcgca tcaagaagtt tttgaattgt tggagaagta cggcgaagaa 660
ttactacgta ctcattttgg caatatagat gcaactccag agattaaaga aatagcccat 720
cgtttgactt acaaagctat ccataagatg ttggaattgg aagttcccaa cctgcatgag 780
ttagacctag atagggtgat tgcttacggt cacacttgga gtcccacctt ggaacttgcg 840
cctcgtctac ccatgttcca cggacacgcc gttaatgtag atatggcttt ctcggcaacg 900
atcgccgccc gtagaggata tattacaatt gcagaacgcg atcgtatttt aggattaatg 960
agtcgcgttg gtctatccct cgaccatccc atgttggata tagatatttt gtggcgtggt 1020
actgaatcta tcacattaac tcgtgatggt ttgttaagag ctgctatgcc aaaacccatt 1080
ggtgattgtg tcttcgtcaa tgacctgaca agagaagaat tagcagccgc attagctgac 1140
cacaaagaac tttgtaccag ttatccccgt ggtggtgaag gtgtggatgt gtatcccgtt 1200
tatcaaaaag aattaatcgg gagtgttaaa taatgacttt tttgaattca aaatgcaaaa 1260
tactccacgg atacactgcg cgagcgcggt agcatttctg ttcgcggagc gtcccgtagg 1320
gaaagagaag gctacgcaaa taatcggaca ctaattgtct ttaattttga attttgaatt 1380
ttgaattttg aattggagcg aagcgacttg acaaatgtga ttgtccaacc aacagctaga 1440
cctgttacac cattgggaat tttaaccaag cagttagaag ccatagtcca agaggttaag 1500
caacatccag atttacctgg ggaattgata gcaaacatcc atcaggcttg gcgtttagcc 1560
gcaggtatag acccttattt ggaagaatgc accactccag aatctcctga actcgctgca 1620
ttggcaaaaa ccacagccac cgaagcctgg ggagaacact tccacggagg tacaaccgtc 1680
cgtcctctag aacaagagat gctttctggt catatcgaag gacaaacctt aaagatgttt 1740
gttcacatga ccaaagctaa aaaagtctta gaaattggga tgtttaccgg ttattcggcg 1800
ctggcgatgg cggaagcatt accagaggat ggactgcttg tggcttgtga agttgaccct 1860
tacgcggcgg aaattggaca gaaagccttt caacaatctc cccacggtgg aaagattcgt 1920
gtggaattgg atgcagcctt agcaactctt gataagttag cagaagctgg ggagtctttt 1980
gacttggtat ttatcgacgc agataaaaaa gagtatgtag cctattttca caagttgcta 2040
ggtagcagtt tgttagcacc agatggcttt atttgtgtag ataacacctt attacaaggg 2100
gaagtttatc taccagcaga ggaacgtagc gtcaatggtg aagcgatcgc gcaatttaat 2160
catacagtag ctatagaccc ccgtgtagaa caggttttgt tgccgttgcg agatggttta 2220
acaattatcc gcagaataca accttaattg tccaatcgac tatggcacaa tcccttcccc 2280
tttcttccgc acctgctaca ccgtctcttc cttcccagac gaaaatagcc gcaattatcc 2340
aaaatatctg cactttggct ttgttattac tagcattgcc cattaatgcc accattgttt 2400
ttatatcctt gttagtcttc cgaccgcaaa aggtcaaagc agcaaacccc caaaccattc 2460
ttatcagtgg cggtaagatg accaaagctt tacaactagc aaggtcattc cacgcggctg 2520
gacatagagt tgtcttggtg gaaacccata aatactggtt gactggtcat cgtttttccc 2580
aagcagtgga taagttttac acagtccccg caccccagga caatccccaa gcttacattc 2640
aggctttggt agatatcgtc aaacaagaaa acatcgatgt ttatattccc gtcaccagtc 2700
cagtgggtag ctactacgac tcattagcca aaccagagtt atcccattat tgcgaagtgt 2760
ttcactttga cgcagatatt acccaaatgt tggatgataa atttgcgttg acacaaaaag 2820
cgcgatcgct tggtttatca gtacccaaat cctttaaaat tacctcacca gaacaagtca 2880
tcaacttcga tttttctgga gagacacgta aatacatcct caaaagcatt ccctacgact 2940
cagtgcggcg gttggactta accaaactcc cctgtgctac tccagaggaa acagcagcat 3000
tcgtcagaag tttgccaatt actcccgaaa aaccgtggat tatgcaggaa tttatccccg 3060
gtaaggaatt ctgcacccat agcaccgttc ggaatgggga actcagactg cattgctgtt 3120
gcgaatcttc agccttccaa gttaattatg agaatgtaaa taacccgcaa attaccgaat 3180
gggtacagca ttttgtcaag gaactgaaac tgacaggaca gatttccttt gactttatcc 3240
aagccgaaga cggaacagtt tacgccatcg agtgtaaccc ccgcacacat tcagcaatta 3300
ccacatttta cgaccacccc caggtagcag aagcgtactt gagtcaagca ccgacgactg 3360
aaaccataca accactaacg acaagcaagc ctacctattg gacttatcac gaagtttggc 3420
gtttaactgg tatccgttct ttcacccagt tgcaaagatg gctggggaat atttggcgcg 3480
ggactgatgc gatttatcag ccagatgacc ccttaccgtt tttgatggta catcattggc 3540
aaattcccct actgttattg aataatttgc gtcgtcttaa aggttggacg cggatagatt 3600
tcaatattgg gaagttggtg gaattggggg gagattagtt tttaaacgca gagggacgct 3660
gaggttagcg cagcgaaaag ttctggagga gggtttccct ccgtaggaaa cttttcaaga 3720
gagagggacg cggagtgtgt tttctctgcg tctctgcgtg agaaattttt tattattgag 3780
caaagttaga agatatgcag actatagatt ttaatattcg taagttactt gtagagtgga 3840
acgcgaccca cagagattat gatctttccc agagtttaca tgaactaatt gtagctcaag 3900
tagaacgaac acctgaggcg atcgctgtca cctttgacaa gcaacaacta acttatcaag 3960
aactaaatca taaagcaaac cagctaggac attatttaca aacattagga gtccagccag 4020
aaaccctggt aggcgtttgt ttagaacgtt ccttagaaat ggttatctgt cttttaggaa 4080
tcctcaaagc tgggggtgct tatgttccta ttgaccctga atatcctcaa gaacgcatag 4140
cttatatgct agaagattct caggtgaagg tactactaac tcaagaaaaa ttactcaatc 4200
aaattcccca ccatcaagca caaactatct gtgtagatag ggaatgggag aaaatttcca 4260
cacaagctaa taccaatccc aaaagtaata taaaaacgga taatcttgct tatgtaattt 4320
acacctctgg ttccactggt aaaccaaaag gtgcaatgaa cacccacaaa ggtatctgta 4380
atcgcttatt gtggatgcag gaagcttatc aaatcgattc cacagatagc attttacaaa 4440
aaaccccctt tagttttgat gtttccgttt gggagttctt ttggacttta ttaactggcg 4500
cacgtttggt aatagccaaa ccaggcggac ataaagatag tgcttacctc atcgatttaa 4560
ttactcaaga acaaatcact acgttgcatt ttgtcccctc aatgctgcaa gtgtttttac 4620
aaaatcgcca tgtaagcaaa tgcagctctc taaaaagagt tatttgtagc ggtgaagctt 4680
tatctataga tttacaaaat agatttttcc agcatttgca atgtgaatta cataacctct 4740
atggcccgac agaagcagca attgatgtca cattttggca atgtagaaaa gatagtaatt 4800
taaagagtgt acctattggt cgtcccattg ctaatactca aatttatatt cttgatgccg 4860
atttacaacc agtaaatatt ggtgtcactg gtgaaattta tattggtggt gtaggggttg 4920
ctcgtggtta tttgaataaa gaagaattga ccaaagaaaa atttattatt aatccctttc 4980
ccaattctga gtttaagcga ctttataaaa caggtgattt agctcgttat ttacccgatg 5040
gaaatattga atatcttggt agaacagatt atcaagtaaa aattcggggt tatagaattg 5100
aaattggcga gattgaaaat gttttatctt cacacccaca agtcagagaa gctgtagtca 5160
tagcgcggga tgataacgct caagaaaaac aaatcatcgc ttatattacc tataactcca 5220
tcaaacctca gcttgataat ctgcgtgatt tcctaaaagc aaggctacct gattttatga 5280
ttccagccgc ttttgtgatg ctggagcatc ttcctttaac tcccagtggt aaagtagacc 5340
gtaaggcatt acctaagcct gatttattta attatagtga acataattcc tatgtagcgc 5400
ctcggaatga agttgaagaa aaattagtac aaatctggtc gaatattctg catttaccta 5460
aagtaggtgt gacagaaaac tttttcgcta ttggtggtaa ttccctcaaa gctctacatt 5520
taatttctca aattgaagag ttatttgcta aagagatatc cttagcaaca cttttaacaa 5580
atccagtaat tgcagattta gccaaggtta ttcaagcaaa caaccaaatc cataattcac 5640
ccctagttcc aattcaacca caaggtaagc agcagccttt cttttgtata catcctgctg 5700
gtggtcatgt tttatgctat tttaaactcg cacaatatat aggaactgac caaccatttt 5760
atggcttaca agctcaagga ttttatggag atgaagcacc cttgacgcga gttgaagata 5820
tggctagtct ctacgtcaaa actattagag aatttcaacc ccaagggcct tatcgtgtcg 5880
gggggtggtc atttggtgga gtcgtagctt atgaagtagc acagcagtta catagacaag 5940
gacaagaagt atctttacta gcaatattag attcttacgt accgattctg ctggataaac 6000
aaaaacccat tgatgacgtt tatttagttg gtgttctctc cagagttttt ggcggtatgt 6060
ttggtcaaga taatctagtc acacctgaag aaatagaaaa tttaactgta gaagaaaaaa 6120
ttaattacat cattgataaa gcacggagcg ctagaatatt cccgcctggt gtagaacgtc 6180
aaaataatcg ccgtattctt gatgttttgg tgggaacttt aaaagcaact tattcctata 6240
taagacaacc atatccagga aaagtcactg tatttcgagc cagggaaaaa catattatgg 6300
ctcctgaccc gaccttagtt tgggtagaat tattttctgt aatggcggct caagaaatta 6360
agattattga tgtccctgga aaccattatt cgtttgttct agaaccccat gtacaggttt 6420
tagcacagcg tttacaagat tgtctggaaa ataattcata a 6461
<210> 18
<211> 2379
<212> DNA
<213> Escherichia coli W3110
<400> 18
atgtccaaca atggctcgtc accgctggtg ctttggtata accaactcgg catgaatgat 60
gtagacaggg ttgggggcaa aaatgcctcc ctgggtgaaa tgattactaa tctttccgga 120
atgggtgttt ccgttccgaa tggtttcgcc acaaccgccg acgcgtttaa ccagtttctg 180
gaccaaagcg gcgtaaacca gcgcatttat gaactgctgg ataaaacgga tattgacgat 240
gttactcagc ttgcgaaagc gggcgcgcaa atccgccagt ggattatcga cactcccttc 300
cagcctgagc tggaaaacgc catccgcgaa gcctatgcac agctttccgc cgatgacgaa 360
aacgcctctt ttgcggtgcg ctcctccgcc accgcagaag atatgccgga cgcttctttt 420
gccggtcagc aggaaacctt cctcaacgtt cagggttttg acgccgttct cgtggcagtg 480
aaacatgtat ttgcttctct gtttaacgat cgcgccatct cttatcgtgt gcaccagggt 540
tacgatcacc gtggtgtggc gctctccgcc ggtgttcaac ggatggtgcg ctctgacctc 600
gcatcatctg gcgtgatgtt ctccattgat accgaatccg gctttgacca ggtggtgttt 660
atcacttccg catggggcct tggtgagatg gtcgtgcagg gtgcggttaa cccggatgag 720
ttttacgtgc ataaaccgac actggcggcg aatcgcccgg ctatcgtgcg ccgcaccatg 780
gggtcgaaaa aaatccgcat ggtttacgcg ccgacccagg agcacggcaa gcaggttaaa 840
atcgaagacg taccgcagga acagcgtgac atcttctcgc tgaccaacga agaagtgcag 900
gaactggcaa aacaggccgt acaaattgag aaacactacg gtcgcccgat ggatattgag 960
tgggcgaaag atggccacac cggtaaactg ttcattgtgc aggcgcgtcc ggaaaccgtg 1020
cgctcacgcg gtcaggtcat ggagcgttat acgctgcatt cacagggtaa gattatcgcc 1080
gaaggccgtg ctatcggtca tcgcatcggt gcgggtccgg tgaaagtcat ccatgacatc 1140
agcgaaatga accgcatcga acctggcgac gtgctggtta ctgacatgac cgacccggac 1200
tgggaaccga tcatgaagaa agcatctgcc atcgtcacca accgtggcgg tcgtacctgt 1260
cacgcggcga tcatcgctcg tgaactgggc attccggcgg tagtgggctg tggagatgca 1320
acagaacgga tgaaagacgg tgagaacgtc actgtttctt gtgccgaagg tgataccggt 1380
tacgtctatg cggagttgct ggaatttagc gtgaaaagct ccagcgtaga aacgatgccg 1440
gatctgccgt tgaaagtgat gatgaacgtc ggtaacccgg accgtgcttt cgacttcgcc 1500
tgcctaccga acgaaggcgt gggccttgcg cgtctggaat ttatcatcaa ccgtatgatt 1560
ggcgtccacc cacgcgcact gcttgagttt gacgatcagg aaccgcagtt gcaaaacgaa 1620
atccgcgaga tgatgaaagg ttttgattct ccgcgtgaat tttacgttgg tcgtctgact 1680
gaagggatcg cgacgctggg tgccgcgttt tatccgaagc gcgtcattgt ccgtctctct 1740
gattttaaat cgaacgaata tgccaacctg gtcggtggtg agcgttacga gccagatgaa 1800
gagaacccga tgctcggctt ccgtggcgcg ggccgctatg tttccgacag cttccgcgac 1860
tgtttcgcgc tggagtgtga agcagtgaaa cgtgtgcgca acgacatggg actgaccaac 1920
gttgagatca tgatcccgtt cgtgcgtacc gtagatcagg cgaaagcggt ggttgaagaa 1980
ctggcgcgtc aggggctgaa acgtggcgag aacgggctga aaatcatcat gatgtgtgaa 2040
atcccgtcca acgccttgct ggccgagcag ttcctcgaat atttcgacgg cttctcaatt 2100
ggctcaaacg atatgacgca gctggcgctc ggtctggacc gtgactccgg cgtggtgtct 2160
gaattgttcg atgagcgcaa cgatgcggtg aaagcactgc tgtcgatggc tatccgtgcc 2220
gcgaagaaac agggcaaata tgtcgggatt tgcggtcagg gtccgtccga ccacgaagac 2280
tttgccgcat ggttgatgga agaggggatc gatagcctgt ctctgaaccc ggacaccgtg 2340
gtgcaaacct ggttaagcct ggctgaactg aagaaataa 2379
<210> 19
<211> 792
<212> PRT
<213> Escherichia coli W3110
<400> 19
Met Ser Asn Asn Gly Ser Ser Pro Leu Val Leu Trp Tyr Asn Gln Leu
1 5 10 15
Gly Met Asn Asp Val Asp Arg Val Gly Gly Lys Asn Ala Ser Leu Gly
20 25 30
Glu Met Ile Thr Asn Leu Ser Gly Met Gly Val Ser Val Pro Asn Gly
35 40 45
Phe Ala Thr Thr Ala Asp Ala Phe Asn Gln Phe Leu Asp Gln Ser Gly
50 55 60
Val Asn Gln Arg Ile Tyr Glu Leu Leu Asp Lys Thr Asp Ile Asp Asp
65 70 75 80
Val Thr Gln Leu Ala Lys Ala Gly Ala Gln Ile Arg Gln Trp Ile Ile
85 90 95
Asp Thr Pro Phe Gln Pro Glu Leu Glu Asn Ala Ile Arg Glu Ala Tyr
100 105 110
Ala Gln Leu Ser Ala Asp Asp Glu Asn Ala Ser Phe Ala Val Arg Ser
115 120 125
Ser Ala Thr Ala Glu Asp Met Pro Asp Ala Ser Phe Ala Gly Gln Gln
130 135 140
Glu Thr Phe Leu Asn Val Gln Gly Phe Asp Ala Val Leu Val Ala Val
145 150 155 160
Lys His Val Phe Ala Ser Leu Phe Asn Asp Arg Ala Ile Ser Tyr Arg
165 170 175
Val His Gln Gly Tyr Asp His Arg Gly Val Ala Leu Ser Ala Gly Val
180 185 190
Gln Arg Met Val Arg Ser Asp Leu Ala Ser Ser Gly Val Met Phe Ser
195 200 205
Ile Asp Thr Glu Ser Gly Phe Asp Gln Val Val Phe Ile Thr Ser Ala
210 215 220
Trp Gly Leu Gly Glu Met Val Val Gln Gly Ala Val Asn Pro Asp Glu
225 230 235 240
Phe Tyr Val His Lys Pro Thr Leu Ala Ala Asn Arg Pro Ala Ile Val
245 250 255
Arg Arg Thr Met Gly Ser Lys Lys Ile Arg Met Val Tyr Ala Pro Thr
260 265 270
Gln Glu His Gly Lys Gln Val Lys Ile Glu Asp Val Pro Gln Glu Gln
275 280 285
Arg Asp Ile Phe Ser Leu Thr Asn Glu Glu Val Gln Glu Leu Ala Lys
290 295 300
Gln Ala Val Gln Ile Glu Lys His Tyr Gly Arg Pro Met Asp Ile Glu
305 310 315 320
Trp Ala Lys Asp Gly His Thr Gly Lys Leu Phe Ile Val Gln Ala Arg
325 330 335
Pro Glu Thr Val Arg Ser Arg Gly Gln Val Met Glu Arg Tyr Thr Leu
340 345 350
His Ser Gln Gly Lys Ile Ile Ala Glu Gly Arg Ala Ile Gly His Arg
355 360 365
Ile Gly Ala Gly Pro Val Lys Val Ile His Asp Ile Ser Glu Met Asn
370 375 380
Arg Ile Glu Pro Gly Asp Val Leu Val Thr Asp Met Thr Asp Pro Asp
385 390 395 400
Trp Glu Pro Ile Met Lys Lys Ala Ser Ala Ile Val Thr Asn Arg Gly
405 410 415
Gly Arg Thr Cys His Ala Ala Ile Ile Ala Arg Glu Leu Gly Ile Pro
420 425 430
Ala Val Val Gly Cys Gly Asp Ala Thr Glu Arg Met Lys Asp Gly Glu
435 440 445
Asn Val Thr Val Ser Cys Ala Glu Gly Asp Thr Gly Tyr Val Tyr Ala
450 455 460
Glu Leu Leu Glu Phe Ser Val Lys Ser Ser Ser Val Glu Thr Met Pro
465 470 475 480
Asp Leu Pro Leu Lys Val Met Met Asn Val Gly Asn Pro Asp Arg Ala
485 490 495
Phe Asp Phe Ala Cys Leu Pro Asn Glu Gly Val Gly Leu Ala Arg Leu
500 505 510
Glu Phe Ile Ile Asn Arg Met Ile Gly Val His Pro Arg Ala Leu Leu
515 520 525
Glu Phe Asp Asp Gln Glu Pro Gln Leu Gln Asn Glu Ile Arg Glu Met
530 535 540
Met Lys Gly Phe Asp Ser Pro Arg Glu Phe Tyr Val Gly Arg Leu Thr
545 550 555 560
Glu Gly Ile Ala Thr Leu Gly Ala Ala Phe Tyr Pro Lys Arg Val Ile
565 570 575
Val Arg Leu Ser Asp Phe Lys Ser Asn Glu Tyr Ala Asn Leu Val Gly
580 585 590
Gly Glu Arg Tyr Glu Pro Asp Glu Glu Asn Pro Met Leu Gly Phe Arg
595 600 605
Gly Ala Gly Arg Tyr Val Ser Asp Ser Phe Arg Asp Cys Phe Ala Leu
610 615 620
Glu Cys Glu Ala Val Lys Arg Val Arg Asn Asp Met Gly Leu Thr Asn
625 630 635 640
Val Glu Ile Met Ile Pro Phe Val Arg Thr Val Asp Gln Ala Lys Ala
645 650 655
Val Val Glu Glu Leu Ala Arg Gln Gly Leu Lys Arg Gly Glu Asn Gly
660 665 670
Leu Lys Ile Ile Met Met Cys Glu Ile Pro Ser Asn Ala Leu Leu Ala
675 680 685
Glu Gln Phe Leu Glu Tyr Phe Asp Gly Phe Ser Ile Gly Ser Asn Asp
690 695 700
Met Thr Gln Leu Ala Leu Gly Leu Asp Arg Asp Ser Gly Val Val Ser
705 710 715 720
Glu Leu Phe Asp Glu Arg Asn Asp Ala Val Lys Ala Leu Leu Ser Met
725 730 735
Ala Ile Arg Ala Ala Lys Lys Gln Gly Lys Tyr Val Gly Ile Cys Gly
740 745 750
Gln Gly Pro Ser Asp His Glu Asp Phe Ala Ala Trp Leu Met Glu Glu
755 760 765
Gly Ile Asp Ser Leu Ser Leu Asn Pro Asp Thr Val Val Gln Thr Trp
770 775 780
Leu Ser Leu Ala Glu Leu Lys Lys
785 790
<210> 20
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn-pps_F
<400> 20
aaaggagctc actagttttg ttcttcccgt gatg 34
<210> 21
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn-pps_R
<400> 21
cagggtgtgg tcgatatctt atttcttcag ttcagc 36
<210> 22
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> pps_F
<400> 22
taggagatta acatatgtcc aacaatggct cg 32
<210> 23
<211> 1992
<212> DNA
<213> Escherichia coli W3110
<400> 23
atgtcctcac gtaaagagct tgccaatgct attcgtgcgc tgagcatgga cgcagtacag 60
aaagccaaat ccggtcaccc gggtgcccct atgggtatgg ctgacattgc cgaagtcctg 120
tggcgtgatt tcctgaaaca caacccgcag aatccgtcct gggctgaccg tgaccgcttc 180
gtgctgtcca acggccacgg ctccatgctg atctacagcc tgctgcacct caccggttac 240
gatctgccga tggaagaact gaaaaacttc cgtcagctgc actctaaaac tccgggtcac 300
ccggaagtgg gttacaccgc tggtgtggaa accaccaccg gtccgctggg tcagggtatt 360
gccaacgcag tcggtatggc gattgcagaa aaaacgctgg cggcgcagtt taaccgtccg 420
ggccacgaca ttgtcgacca ctacacctac gccttcatgg gcgacggctg catgatggaa 480
ggcatctccc acgaagtttg ctctctggcg ggtacgctga agctgggtaa actgattgca 540
ttctacgatg acaacggtat ttctatcgat ggtcacgttg aaggctggtt caccgacgac 600
accgcaatgc gtttcgaagc ttacggctgg cacgttattc gcgacatcga cggtcatgac 660
gcggcatcta tcaaacgcgc agtagaagaa gcgcgcgcag tgactgacaa accttccctg 720
ctgatgtgca aaaccatcat cggtttcggt tccccgaaca aagccggtac ccacgactcc 780
cacggtgcgc cgctgggcga cgctgaaatt gccctgaccc gcgaacaact gggctggaaa 840
tatgcgccgt tcgaaatccc gtctgaaatc tatgctcagt gggatgcgaa agaagcaggc 900
caggcgaaag aatccgcatg gaacgagaaa ttcgctgctt acgcgaaagc ttatccgcag 960
gaagccgctg aatttacccg ccgtatgaaa ggcgaaatgc cgtctgactt cgacgctaaa 1020
gcgaaagagt tcatcgctaa actgcaggct aatccggcga aaatcgccag ccgtaaagcg 1080
tctcagaatg ctatcgaagc gttcggtccg ctgttgccgg aattcctcgg cggttctgct 1140
gacctggcgc cgtctaacct gaccctgtgg tctggttcta aagcaatcaa cgaagatgct 1200
gcgggtaact acatccacta cggtgttcgc gagttcggta tgaccgcgat tgctaacggt 1260
atctccctgc acggtggctt cctgccgtac acctccacct tcctgatgtt cgtggaatac 1320
gcacgtaacg ccgtacgtat ggctgcgctg atgaaacagc gtcaggtgat ggtttacacc 1380
cacgactcca tcggtctggg cgaagacggc ccgactcacc agccggttga gcaggtcgct 1440
tctctgcgcg taaccccgaa catgtctaca tggcgtccgt gtgaccaggt tgaatccgcg 1500
gtcgcgtgga aatacggtgt tgagcgtcag gacggcccga ccgcactgat cctctcccgt 1560
cagaacctgg cgcagcagga acgaactgaa gagcaactgg caaacatcgc gcgcggtggt 1620
tatgtgctga aagactgcgc cggtcagccg gaactgattt tcatcgctac cggttcagaa 1680
gttgaactgg ctgttgctgc ctacgaaaaa ctgactgccg aaggcgtgaa agcgcgcgtg 1740
gtgtccatgc cgtctaccga cgcatttgac aagcaggatg ctgcttaccg tgaatccgta 1800
ctgccgaaag cggttactgc acgcgttgct gtagaagcgg gtattgctga ctactggtac 1860
aagtatgttg gcctgaacgg tgctatcgtc ggtatgacca ccttcggtga atctgctccg 1920
gcagagctgc tgtttgaaga gttcggcttc actgttgata acgttgttgc gaaagcaaaa 1980
gaactgctgt aa 1992
<210> 24
<211> 663
<212> PRT
<213> Escherichia coli W3110
<400> 24
Met Ser Ser Arg Lys Glu Leu Ala Asn Ala Ile Arg Ala Leu Ser Met
1 5 10 15
Asp Ala Val Gln Lys Ala Lys Ser Gly His Pro Gly Ala Pro Met Gly
20 25 30
Met Ala Asp Ile Ala Glu Val Leu Trp Arg Asp Phe Leu Lys His Asn
35 40 45
Pro Gln Asn Pro Ser Trp Ala Asp Arg Asp Arg Phe Val Leu Ser Asn
50 55 60
Gly His Gly Ser Met Leu Ile Tyr Ser Leu Leu His Leu Thr Gly Tyr
65 70 75 80
Asp Leu Pro Met Glu Glu Leu Lys Asn Phe Arg Gln Leu His Ser Lys
85 90 95
Thr Pro Gly His Pro Glu Val Gly Tyr Thr Ala Gly Val Glu Thr Thr
100 105 110
Thr Gly Pro Leu Gly Gln Gly Ile Ala Asn Ala Val Gly Met Ala Ile
115 120 125
Ala Glu Lys Thr Leu Ala Ala Gln Phe Asn Arg Pro Gly His Asp Ile
130 135 140
Val Asp His Tyr Thr Tyr Ala Phe Met Gly Asp Gly Cys Met Met Glu
145 150 155 160
Gly Ile Ser His Glu Val Cys Ser Leu Ala Gly Thr Leu Lys Leu Gly
165 170 175
Lys Leu Ile Ala Phe Tyr Asp Asp Asn Gly Ile Ser Ile Asp Gly His
180 185 190
Val Glu Gly Trp Phe Thr Asp Asp Thr Ala Met Arg Phe Glu Ala Tyr
195 200 205
Gly Trp His Val Ile Arg Asp Ile Asp Gly His Asp Ala Ala Ser Ile
210 215 220
Lys Arg Ala Val Glu Glu Ala Arg Ala Val Thr Asp Lys Pro Ser Leu
225 230 235 240
Leu Met Cys Lys Thr Ile Ile Gly Phe Gly Ser Pro Asn Lys Ala Gly
245 250 255
Thr His Asp Ser His Gly Ala Pro Leu Gly Asp Ala Glu Ile Ala Leu
260 265 270
Thr Arg Glu Gln Leu Gly Trp Lys Tyr Ala Pro Phe Glu Ile Pro Ser
275 280 285
Glu Ile Tyr Ala Gln Trp Asp Ala Lys Glu Ala Gly Gln Ala Lys Glu
290 295 300
Ser Ala Trp Asn Glu Lys Phe Ala Ala Tyr Ala Lys Ala Tyr Pro Gln
305 310 315 320
Glu Ala Ala Glu Phe Thr Arg Arg Met Lys Gly Glu Met Pro Ser Asp
325 330 335
Phe Asp Ala Lys Ala Lys Glu Phe Ile Ala Lys Leu Gln Ala Asn Pro
340 345 350
Ala Lys Ile Ala Ser Arg Lys Ala Ser Gln Asn Ala Ile Glu Ala Phe
355 360 365
Gly Pro Leu Leu Pro Glu Phe Leu Gly Gly Ser Ala Asp Leu Ala Pro
370 375 380
Ser Asn Leu Thr Leu Trp Ser Gly Ser Lys Ala Ile Asn Glu Asp Ala
385 390 395 400
Ala Gly Asn Tyr Ile His Tyr Gly Val Arg Glu Phe Gly Met Thr Ala
405 410 415
Ile Ala Asn Gly Ile Ser Leu His Gly Gly Phe Leu Pro Tyr Thr Ser
420 425 430
Thr Phe Leu Met Phe Val Glu Tyr Ala Arg Asn Ala Val Arg Met Ala
435 440 445
Ala Leu Met Lys Gln Arg Gln Val Met Val Tyr Thr His Asp Ser Ile
450 455 460
Gly Leu Gly Glu Asp Gly Pro Thr His Gln Pro Val Glu Gln Val Ala
465 470 475 480
Ser Leu Arg Val Thr Pro Asn Met Ser Thr Trp Arg Pro Cys Asp Gln
485 490 495
Val Glu Ser Ala Val Ala Trp Lys Tyr Gly Val Glu Arg Gln Asp Gly
500 505 510
Pro Thr Ala Leu Ile Leu Ser Arg Gln Asn Leu Ala Gln Gln Glu Arg
515 520 525
Thr Glu Glu Gln Leu Ala Asn Ile Ala Arg Gly Gly Tyr Val Leu Lys
530 535 540
Asp Cys Ala Gly Gln Pro Glu Leu Ile Phe Ile Ala Thr Gly Ser Glu
545 550 555 560
Val Glu Leu Ala Val Ala Ala Tyr Glu Lys Leu Thr Ala Glu Gly Val
565 570 575
Lys Ala Arg Val Val Ser Met Pro Ser Thr Asp Ala Phe Asp Lys Gln
580 585 590
Asp Ala Ala Tyr Arg Glu Ser Val Leu Pro Lys Ala Val Thr Ala Arg
595 600 605
Val Ala Val Glu Ala Gly Ile Ala Asp Tyr Trp Tyr Lys Tyr Val Gly
610 615 620
Leu Asn Gly Ala Ile Val Gly Met Thr Thr Phe Gly Glu Ser Ala Pro
625 630 635 640
Ala Glu Leu Leu Phe Glu Glu Phe Gly Phe Thr Val Asp Asn Val Val
645 650 655
Ala Lys Ala Lys Glu Leu Leu
660
<210> 25
<211> 34
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn-tktA_F
<400> 25
aaaggagctc actagtgttt tctcttccgc acaa 34
<210> 26
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn-tktA_R
<400> 26
cagggtgtgg tcgatatctt acagcagttc ttttgc 36
<210> 27
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> tktA_F
<400> 27
taggagatta acatatgtcc tcacgtaaag ag 32
<210> 28
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj1-aroG_R
<400> 28
cccgcggttt acccgcgacg cgcttt 26
<210> 29
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj1-pps_F
<400> 29
gcgggtaaac cgcgggctta ttccat 26
<210> 30
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj1-pps_R
<400> 30
cccgcggttt atttcttcag ttcagc 26
<210> 31
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj1-tktA_F
<400> 31
agaaataaac cgcgggctta ttccat 26
<210> 32
<211> 71
<212> DNA
<213> Artificial Sequence
<220>
<223> aroD deletion cassette_F
<400> 32
tcatggggtt cggtgcctga caggctgacc gcgtgcagaa agggtaaaaa gctggagctg 60
cttcgaagtt c 71
<210> 33
<211> 79
<212> DNA
<213> Artificial Sequence
<220>
<223> aroD deletion cassette_R
<400> 33
atatattttt tagttcggcg gggagggtgt tcccgccgaa atattattgc gccatggtcc 60
atatgaatat cctccttag 79
<210> 34
<211> 23
<212> DNA
<213> Artificial Sequence
<220>
<223> aroD deletion_F
<400> 34
caaagatttc cctctggaat atg 23
<210> 35
<211> 21
<212> DNA
<213> Artificial Sequence
<220>
<223> aroD deletion_R
<400> 35
cagatgtgat tttccctacg c 21
<210> 36
<211> 1389
<212> DNA
<213> Corynebacterium glutamicum ATCC13032
<400> 36
gtgagttgga cagttgatat ccctaaagaa gttctccctg atttgccacc attgccagaa 60
ggcatgcagc agcagttcga ggacaccatt tcccgtgacg ctaagcagca acctacgtgg 120
gatcgtgcac aggcagaaaa cgtgcgcaag atccttgagt cggttcctcc aatcgttgtt 180
gcccctgagg tacttgagct gaagcagaag cttgctgatg ttgccaacgg taaggccttc 240
ctcttgcagg gtggtgactg tgcggaaact ttcgagtcaa acactgagcc gcacattcgc 300
gccaacgtaa agactctgct gcagatggca gttgttttga cctacggtgc atccactcct 360
gtgatcaaga tggctcgtat tgctggtcag tacgcaaagc ctcgctcttc tgatctggat 420
ggaaatggtc tgccaaacta ccgtggcgat atcgtcaacg gtgtggaggc aaccccagag 480
gctcgtcgcc acgatcctgc ccgcatgatc cgtgcttacg ctaacgcttc tgctgcgatg 540
aacttggtgc gcgcgctcac cagctctggc accgctgatc tttaccgtct cagcgagtgg 600
aaccgcgagt tcgttgcgaa ctccccagct ggtgcacgct acgaggctct tgctcgtgag 660
atcgactccg gtctgcgctt catggaagca tgtggcgtgt ccgatgagtc cctgcgtgct 720
gcagatatct actgctccca cgaggctttg ctggtggatt acgagcgttc catgctgcgt 780
cttgcaaccg atgaggaagg caacgaggaa ctttacgatc tttcagctca ccagctgtgg 840
atcggcgagc gcacccgtgg catggatgat ttccatgtga acttcgcatc catgatctct 900
aacccaatcg gcatcaagat tggtcctggt atcacccctg aagaggctgt tgcatacgct 960
gacaagctcg atccgaactt cgagcctggc cgtttgacca tcgttgctcg catgggccac 1020
gacaaggttc gctccgtact tcctggtgtt atccaggctg ttgaggcatc cggacacaag 1080
gttatttggc agtccgatcc gatgcacggc aacactttca ccgcatccaa tggctacaag 1140
acccgtcact tcgacaaggt tatcgatgag gtccagggct tcttcgaggt ccaccgcgca 1200
ttgggcaccc acccaggcgg aatccacatt gagttcactg gtgaagatgt caccgagtgc 1260
ctcggtggcg ctgaagacat caccgatgtt gatctgccag gccgctacga gtccgcatgc 1320
gatcctcgcc tgaacactca gcagtctttg gagttggctt tcctcgttgc agaaatgctg 1380
cgtaactaa 1389
<210> 37
<211> 462
<212> PRT
<213> Corynebacterium glutamicum ATCC13032
<400> 37
Met Ser Trp Thr Val Asp Ile Pro Lys Glu Val Leu Pro Asp Leu Pro
1 5 10 15
Pro Leu Pro Glu Gly Met Gln Gln Gln Phe Glu Asp Thr Ile Ser Arg
20 25 30
Asp Ala Lys Gln Gln Pro Thr Trp Asp Arg Ala Gln Ala Glu Asn Val
35 40 45
Arg Lys Ile Leu Glu Ser Val Pro Pro Ile Val Val Ala Pro Glu Val
50 55 60
Leu Glu Leu Lys Gln Lys Leu Ala Asp Val Ala Asn Gly Lys Ala Phe
65 70 75 80
Leu Leu Gln Gly Gly Asp Cys Ala Glu Thr Phe Glu Ser Asn Thr Glu
85 90 95
Pro His Ile Arg Ala Asn Val Lys Thr Leu Leu Gln Met Ala Val Val
100 105 110
Leu Thr Tyr Gly Ala Ser Thr Pro Val Ile Lys Met Ala Arg Ile Ala
115 120 125
Gly Gln Tyr Ala Lys Pro Arg Ser Ser Asp Leu Asp Gly Asn Gly Leu
130 135 140
Pro Asn Tyr Arg Gly Asp Ile Val Asn Gly Val Glu Ala Thr Pro Glu
145 150 155 160
Ala Arg Arg His Asp Pro Ala Arg Met Ile Arg Ala Tyr Ala Asn Ala
165 170 175
Ser Ala Ala Met Asn Leu Val Arg Ala Leu Thr Ser Ser Gly Thr Ala
180 185 190
Asp Leu Tyr Arg Leu Ser Glu Trp Asn Arg Glu Phe Val Ala Asn Ser
195 200 205
Pro Ala Gly Ala Arg Tyr Glu Ala Leu Ala Arg Glu Ile Asp Ser Gly
210 215 220
Leu Arg Phe Met Glu Ala Cys Gly Val Ser Asp Glu Ser Leu Arg Ala
225 230 235 240
Ala Asp Ile Tyr Cys Ser His Glu Ala Leu Leu Val Asp Tyr Glu Arg
245 250 255
Ser Met Leu Arg Leu Ala Thr Asp Glu Glu Gly Asn Glu Glu Leu Tyr
260 265 270
Asp Leu Ser Ala His Gln Leu Trp Ile Gly Glu Arg Thr Arg Gly Met
275 280 285
Asp Asp Phe His Val Asn Phe Ala Ser Met Ile Ser Asn Pro Ile Gly
290 295 300
Ile Lys Ile Gly Pro Gly Ile Thr Pro Glu Glu Ala Val Ala Tyr Ala
305 310 315 320
Asp Lys Leu Asp Pro Asn Phe Glu Pro Gly Arg Leu Thr Ile Val Ala
325 330 335
Arg Met Gly His Asp Lys Val Arg Ser Val Leu Pro Gly Val Ile Gln
340 345 350
Ala Val Glu Ala Ser Gly His Lys Val Ile Trp Gln Ser Asp Pro Met
355 360 365
His Gly Asn Thr Phe Thr Ala Ser Asn Gly Tyr Lys Thr Arg His Phe
370 375 380
Asp Lys Val Ile Asp Glu Val Gln Gly Phe Phe Glu Val His Arg Ala
385 390 395 400
Leu Gly Thr His Pro Gly Gly Ile His Ile Glu Phe Thr Gly Glu Asp
405 410 415
Val Thr Glu Cys Leu Gly Gly Ala Glu Asp Ile Thr Asp Val Asp Leu
420 425 430
Pro Gly Arg Tyr Glu Ser Ala Cys Asp Pro Arg Leu Asn Thr Gln Gln
435 440 445
Ser Leu Glu Leu Ala Phe Leu Val Ala Glu Met Leu Arg Asn
450 455 460
<210> 38
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn-cgl aroG_F
<400> 38
ggtatcgata agcttgattc gatctgctcc cattcc 36
<210> 39
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn-cgl aroG_R
<400> 39
cggtggcggc cgctctagtt agttacgcag catttc 36
<210> 40
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-cgl aroG_F
<400> 40
cgaaaggaaa cactcgatgt gagttggaca gttgatatc 39
<210> 41
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-cgl aroG_R
<400> 41
ccgccaaaac agctctagtt tagttacgca gcatttc 37
<210> 42
<211> 60
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_ABCD_F
<400> 42
ccaaacacca acaaaaggct ctacccatat gatgagtatc gtccaagcaa agtttgaagc 60
60
<210> 43
<211> 60
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_ABCD_R
<400> 43
agagggtgac gctagtcaga actagtttat gaattatttt ccagacaatc ttgtaaacgc 60
60
<210> 44
<211> 54
<212> DNA
<213> Artificial Sequence
<220>
<223> O2 promoter_F
<400> 44
ccaaacacca acaaaaggct ctacccatat gcaataatcg tgaattttgg cagc 54
<210> 45
<211> 53
<212> DNA
<213> Artificial Sequence
<220>
<223> O2 promoter_R
<400> 45
caaactttgc ttggacgata ctcattgttt tgatctcctc caataatcta tgc 53
<210> 46
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn-cgl tkt_F
<400> 46
ggtatcgata agcttgataa gattgatcac acctttg 37
<210> 47
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pn-cgl tkt_R
<400> 47
cggtggcggc cgctctagtt aaccgttaat ggagtc 36
<210> 48
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-cgl tkt_F
<400> 48
cgaaaggaaa cactcgattt gaccaccttg acgctg 36
<210> 49
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-cgl tkt_R
<400> 49
ccgccaaaac agctctagtt taaccgttaa tggagtc 37
<210> 50
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Ptrc-cgl pps_F
<400> 50
tcacacagga aagatatcat gaccaacagt ttgaac 36
<210> 51
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Ptrc-cgl pps_R
<400> 51
ccgccaaaac agctctagtt tacttcgtgc cggtcattg 39
<210> 52
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-cgl pps_F
<400> 52
cgaaaggaaa cactcgatat gaccaacagt ttgaac 36
<210> 53
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-aroG_F
<400> 53
ttagaaatgg aacctaaaag aaacatccca gcgctac 37
<210> 54
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-aroG_R
<400> 54
tagcgctggg atgtttcttt agttacgcag catttc 36
<210> 55
<211> 36
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-tkt_F
<400> 55
gaaatgctgc gtaactaaag aaacatccca gcgcta 36
<210> 56
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-tkt_R
<400> 56
gtagcgctgg gatgtttctt taaccgttaa tggagtc 37
<210> 57
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-pps_F
<400> 57
gactccatta acggttaaag aaacatccca gcgctac 37
<210> 58
<211> 39
<212> DNA
<213> Artificial Sequence
<220>
<223> Pcj7-pps_R
<400> 58
ggagaatact gtcgttcatt tacttcgtgc cggtcattg 39
<210> 59
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_F
<400> 59
ggatccacag tttattcctg gcatcc 26
<210> 60
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_R
<400> 60
tttgcttgga cgatactcat ttgtttgttt atgtgtgttt 40
<210> 61
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_A_F
<400> 61
aaacacacat aaacaaacaa atgagtatcg tccaagcaaa 40
<210> 62
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_A_R
<400> 62
ggatgccagg aataaactgt ttatttaaca ctcccgatta 40
<210> 63
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_F
<400> 63
taatcgggag tgttaaataa acagtttatt cctggcatcc 40
<210> 64
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_R
<400> 64
tggacaatca catttgtcaa ttgtttgttt atgtgtgttt 40
<210> 65
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_B_F
<400> 65
aaacacacat aaacaaacaa ttgacaaatg tgattgtcca 40
<210> 66
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_B_R
<400> 66
gtcgacttaa ggttgtattc tgcgga 26
<210> 67
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_F
<400> 67
ggatccacag tttattcctg gcatcc 26
<210> 68
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_R
<400> 68
aaagagattg attgtgccat ttgtttgttt atgtgtgttt 40
<210> 69
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_C_F
<400> 69
aaacacacat aaacaaacaa atggcacaat caatctcttt 40
<210> 70
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_C_R
<400> 70
ggatgccagg aataaactgt ttagtcgccc cctaattcca 40
<210> 71
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_F
<400> 71
tggaattagg gggcgactaa acagtttatt cctggcatcc 40
<210> 72
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_R
<400> 72
aggatattaa gtactggcat ttgtttgttt atgtgtgttt 40
<210> 73
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_D_F
<400> 73
aaacacacat aaacaaacaa atgccagtac ttaatatcct 40
<210> 74
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> Ava_D_R
<400> 74
gtcgactcaa ttttgtaaca cctttt 26
<210> 75
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> TKL1_F
<400> 75
ggatccatga ctcaattcac tgacat 26
<210> 76
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> TKL1_R
<400> 76
gtcgacttag aaagcttttt tcaaag 26
<210> 77
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> ARO4_F
<400> 77
ggatccatga gtgaatctcc aatgtt 26
<210> 78
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> ARO4_R
<400> 78
gtcgacctat ttcttgttaa cttctc 26
<210> 79
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> pps_F
<400> 79
ggatccatgt ccaacaatgg ctcgtc 26
<210> 80
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> pps_R
<400> 80
gtcgacttat ttcttcagtt cagcca 26
<210> 81
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_F
<400> 81
ggatccacag tttattcctg gcatcc 26
<210> 82
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_R
<400> 82
atgtcagtga attgagtcat ttgtttgttt atgtgtgttt 40
<210> 83
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> TKL1_F
<400> 83
aaacacacat aaacaaacaa atgactcaat tcactgacat 40
<210> 84
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> TKL1_R
<400> 84
ggatgccagg aataaactgt ttagaaagct tttttcaaag 40
<210> 85
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_F
<400> 85
ctttgaaaaa agctttctaa acagtttatt cctggcatcc 40
<210> 86
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> pGPD_R
<400> 86
aacattggag attcactcat ttgtttgttt atgtgtgttt 40
<210> 87
<211> 40
<212> DNA
<213> Artificial Sequence
<220>
<223> ARO4_F
<400> 87
aaacacacat aaacaaacaa atgagtgaat ctccaatgtt 40
<210> 88
<211> 26
<212> DNA
<213> Artificial Sequence
<220>
<223> ARO4_R
<400> 88
gtcgacctat ttcttgttaa cttctc 26
<210> 89
<211> 438
<212> DNA
<213> Corynebacterium glutamicum ATCC13032
<400> 89
atgcctggaa aaattctcct cctcaacggc ccaaacctga acatgctggg caaacgcgag 60
cctgacattt acggacacga caccttggaa gacgtcgtcg cgctggcaac cgctgaggct 120
gcgaagcacg gccttgaggt tgaggcgctg cagagcaatc acgaaggtga gctaatcgat 180
gcgctgcaca acgctcgcgg cacccacatc ggttgcgtga ttaaccccgg cggcctgact 240
cacacttcgg tggcgctttt ggatgctgtg aaggcgtctg agcttcctac cgttgaggtg 300
cacatttcca atccgcatgc ccgtgaagag ttccgccacc attcttacat ttccctcgcc 360
gcggtctccg ttatcgctgg cgctggcatc cagggttacc gtttcgcggt cgatatcctg 420
gcaaatctca aaaagtag 438
<210> 90
<211> 145
<212> PRT
<213> Corynebacterium glutamicum ATCC13032
<400> 90
Met Pro Gly Lys Ile Leu Leu Leu Asn Gly Pro Asn Leu Asn Met Leu
1 5 10 15
Gly Lys Arg Glu Pro Asp Ile Tyr Gly His Asp Thr Leu Glu Asp Val
20 25 30
Val Ala Leu Ala Thr Ala Glu Ala Ala Lys His Gly Leu Glu Val Glu
35 40 45
Ala Leu Gln Ser Asn His Glu Gly Glu Leu Ile Asp Ala Leu His Asn
50 55 60
Ala Arg Gly Thr His Ile Gly Cys Val Ile Asn Pro Gly Gly Leu Thr
65 70 75 80
His Thr Ser Val Ala Leu Leu Asp Ala Val Lys Ala Ser Glu Leu Pro
85 90 95
Thr Val Glu Val His Ile Ser Asn Pro His Ala Arg Glu Glu Phe Arg
100 105 110
His His Ser Tyr Ile Ser Leu Ala Ala Val Ser Val Ile Ala Gly Ala
115 120 125
Gly Ile Gln Gly Tyr Arg Phe Ala Val Asp Ile Leu Ala Asn Leu Lys
130 135 140
Lys
145
<210> 91
<211> 32
<212> DNA
<213> Artificial Sequence
<220>
<223> aroD_F
<400> 91
gatcggatcc cactggacgt ttgggtgaga cc 32
<210> 92
<211> 37
<212> DNA
<213> Artificial Sequence
<220>
<223> aroD_R
<400> 92
gatcggatcc actagtttta ggttccattt ctaattg 37
<210> 93
<211> 29
<212> DNA
<213> Artificial Sequence
<220>
<223> aroD_F
<400> 93
gatcactagt atgaacgaca gtattctcc 29
<210> 94
<211> 30
<212> DNA
<213> Artificial Sequence
<220>
<223> aroD_D
<400> 94
gatcaagctt gttacatcct gacgttgtgg 30
<210> 95
<211> 2103
<212> DNA
<213> Corynebacterium glutamicum ATCC13032
<400> 95
ttgaccacct tgacgctgtc acctgaactt caggcgctca ctgtacgcaa ttacccctct 60
gattggtccg atgtggacac caaggctgta gacactgttc gtgtcctcgc tgcagacgct 120
gtagaaaact gtggctccgg ccacccaggc accgcaatga gcctggctcc ccttgcatac 180
accttgtacc agcgggttat gaacgtagat ccacaggaca ccaactgggc aggccgtgac 240
cgcttcgttc tttcttgtgg ccactcctct ttgacccagt acatccagct ttacttgggt 300
ggattcggcc ttgagatgga tgacctgaag gctctgcgca cctgggattc cttgacccca 360
ggacaccctg agtaccgcca caccaagggc gttgagatca ccactggccc tcttggccag 420
ggtcttgcat ctgcagttgg tatggccatg gctgctcgtc gtgagcgtgg cctattcgac 480
ccaaccgctg ctgagggcga atccccattc gaccaccaca tctacgtcat tgcttctgat 540
ggtgacctgc aggaaggtgt cacctctgag gcatcctcca tcgctggcac ccagcagctg 600
ggcaacctca tcgtgttctg ggatgacaac cgcatctcca tcgaagacaa cactgagatc 660
gctttcaacg aggacgttgt tgctcgttac aaggcttacg gctggcagac cattgaggtt 720
gaggctggcg aggacgttgc agcaatcgaa gctgcagtgg ctgaggctaa gaaggacacc 780
aagcgaccta ccttcatccg cgttcgcacc atcatcggct tcccagctcc aactatgatg 840
aacaccggtg ctgtgcacgg tgctgctctt ggcgcagctg aggttgcagc aaccaagact 900
gagcttggat tcgatcctga ggctcacttc gcgatcgacg atgaggttat cgctcacacc 960
cgctccctcg cagagcgcgc tgcacagaag aaggctgcat ggcaggtcaa gttcgatgag 1020
tgggcagctg ccaaccctga gaacaaggct ctgttcgatc gcctgaactc ccgtgagctt 1080
ccagcgggct acgctgacga gctcccaaca tgggatgcag atgagaaggg cgtcgcaact 1140
cgtaaggctt ccgaggctgc acttcaggca ctgggcaaga cccttcctga gctgtggggc 1200
ggttccgctg acctcgcagg ttccaacaac accgtgatca agggctcccc ttccttcggc 1260
cctgagtcca tctccaccga gacctggtct gctgagcctt acggccgtaa cctgcacttc 1320
ggtatccgtg agcacgctat gggatccatc ctcaacggca tttccctcca cggtggcacc 1380
cgcccatacg gcggaacctt cctcatcttc tccgactaca tgcgtcctgc agttcgtctt 1440
gcagctctca tggagaccga cgcttactac gtctggaccc acgactccat cggtctgggc 1500
gaagatggcc caacccacca gcctgttgaa accttggctg cactgcgcgc catcccaggt 1560
ctgtccgtcc tgcgtcctgc agatgcgaac gagaccgccc aggcttgggc tgcagcactt 1620
gagtacaagg aaggccctaa gggtcttgca ctgacccgcc agaacgttcc tgttctggaa 1680
ggcaccaagg agaaggctgc tgaaggcgtt cgccgcggtg gctacgtcct ggttgagggt 1740
tccaaggaaa ccccagatgt gatcctcatg ggctccggct ccgaggttca gcttgcagtt 1800
aacgctgcga aggctctgga agctgagggc gttgcagctc gcgttgtttc cgttccttgc 1860
atggattggt tccaggagca ggacgcagag tacatcgagt ccgttctgcc tgcagctgtg 1920
accgctcgtg tgtctgttga agctggcatc gcaatgcctt ggtaccgctt cttgggcacc 1980
cagggccgtg ctgtctccct tgagcacttc ggtgcttctg cggattacca gaccctgttt 2040
gagaagttcg gcatcaccac cgatgcagtc gtggcagcgg ccaaggactc cattaacggt 2100
taa 2103
<210> 96
<211> 700
<212> PRT
<213> Corynebacterium glutamicum ATCC13032
<400> 96
Met Thr Thr Leu Thr Leu Ser Pro Glu Leu Gln Ala Leu Thr Val Arg
1 5 10 15
Asn Tyr Pro Ser Asp Trp Ser Asp Val Asp Thr Lys Ala Val Asp Thr
20 25 30
Val Arg Val Leu Ala Ala Asp Ala Val Glu Asn Cys Gly Ser Gly His
35 40 45
Pro Gly Thr Ala Met Ser Leu Ala Pro Leu Ala Tyr Thr Leu Tyr Gln
50 55 60
Arg Val Met Asn Val Asp Pro Gln Asp Thr Asn Trp Ala Gly Arg Asp
65 70 75 80
Arg Phe Val Leu Ser Cys Gly His Ser Ser Leu Thr Gln Tyr Ile Gln
85 90 95
Leu Tyr Leu Gly Gly Phe Gly Leu Glu Met Asp Asp Leu Lys Ala Leu
100 105 110
Arg Thr Trp Asp Ser Leu Thr Pro Gly His Pro Glu Tyr Arg His Thr
115 120 125
Lys Gly Val Glu Ile Thr Thr Gly Pro Leu Gly Gln Gly Leu Ala Ser
130 135 140
Ala Val Gly Met Ala Met Ala Ala Arg Arg Glu Arg Gly Leu Phe Asp
145 150 155 160
Pro Thr Ala Ala Glu Gly Glu Ser Pro Phe Asp His His Ile Tyr Val
165 170 175
Ile Ala Ser Asp Gly Asp Leu Gln Glu Gly Val Thr Ser Glu Ala Ser
180 185 190
Ser Ile Ala Gly Thr Gln Gln Leu Gly Asn Leu Ile Val Phe Trp Asp
195 200 205
Asp Asn Arg Ile Ser Ile Glu Asp Asn Thr Glu Ile Ala Phe Asn Glu
210 215 220
Asp Val Val Ala Arg Tyr Lys Ala Tyr Gly Trp Gln Thr Ile Glu Val
225 230 235 240
Glu Ala Gly Glu Asp Val Ala Ala Ile Glu Ala Ala Val Ala Glu Ala
245 250 255
Lys Lys Asp Thr Lys Arg Pro Thr Phe Ile Arg Val Arg Thr Ile Ile
260 265 270
Gly Phe Pro Ala Pro Thr Met Met Asn Thr Gly Ala Val His Gly Ala
275 280 285
Ala Leu Gly Ala Ala Glu Val Ala Ala Thr Lys Thr Glu Leu Gly Phe
290 295 300
Asp Pro Glu Ala His Phe Ala Ile Asp Asp Glu Val Ile Ala His Thr
305 310 315 320
Arg Ser Leu Ala Glu Arg Ala Ala Gln Lys Lys Ala Ala Trp Gln Val
325 330 335
Lys Phe Asp Glu Trp Ala Ala Ala Asn Pro Glu Asn Lys Ala Leu Phe
340 345 350
Asp Arg Leu Asn Ser Arg Glu Leu Pro Ala Gly Tyr Ala Asp Glu Leu
355 360 365
Pro Thr Trp Asp Ala Asp Glu Lys Gly Val Ala Thr Arg Lys Ala Ser
370 375 380
Glu Ala Ala Leu Gln Ala Leu Gly Lys Thr Leu Pro Glu Leu Trp Gly
385 390 395 400
Gly Ser Ala Asp Leu Ala Gly Ser Asn Asn Thr Val Ile Lys Gly Ser
405 410 415
Pro Ser Phe Gly Pro Glu Ser Ile Ser Thr Glu Thr Trp Ser Ala Glu
420 425 430
Pro Tyr Gly Arg Asn Leu His Phe Gly Ile Arg Glu His Ala Met Gly
435 440 445
Ser Ile Leu Asn Gly Ile Ser Leu His Gly Gly Thr Arg Pro Tyr Gly
450 455 460
Gly Thr Phe Leu Ile Phe Ser Asp Tyr Met Arg Pro Ala Val Arg Leu
465 470 475 480
Ala Ala Leu Met Glu Thr Asp Ala Tyr Tyr Val Trp Thr His Asp Ser
485 490 495
Ile Gly Leu Gly Glu Asp Gly Pro Thr His Gln Pro Val Glu Thr Leu
500 505 510
Ala Ala Leu Arg Ala Ile Pro Gly Leu Ser Val Leu Arg Pro Ala Asp
515 520 525
Ala Asn Glu Thr Ala Gln Ala Trp Ala Ala Ala Leu Glu Tyr Lys Glu
530 535 540
Gly Pro Lys Gly Leu Ala Leu Thr Arg Gln Asn Val Pro Val Leu Glu
545 550 555 560
Gly Thr Lys Glu Lys Ala Ala Glu Gly Val Arg Arg Gly Gly Tyr Val
565 570 575
Leu Val Glu Gly Ser Lys Glu Thr Pro Asp Val Ile Leu Met Gly Ser
580 585 590
Gly Ser Glu Val Gln Leu Ala Val Asn Ala Ala Lys Ala Leu Glu Ala
595 600 605
Glu Gly Val Ala Ala Arg Val Val Ser Val Pro Cys Met Asp Trp Phe
610 615 620
Gln Glu Gln Asp Ala Glu Tyr Ile Glu Ser Val Leu Pro Ala Ala Val
625 630 635 640
Thr Ala Arg Val Ser Val Glu Ala Gly Ile Ala Met Pro Trp Tyr Arg
645 650 655
Phe Leu Gly Thr Gln Gly Arg Ala Val Ser Leu Glu His Phe Gly Ala
660 665 670
Ser Ala Asp Tyr Gln Thr Leu Phe Glu Lys Phe Gly Ile Thr Thr Asp
675 680 685
Ala Val Val Ala Ala Ala Lys Asp Ser Ile Asn Gly
690 695 700
<210> 97
<211> 1095
<212> DNA
<213> Corynebacterium glutamicum ATCC13032
<400> 97
atgaccaaca gtttgaacat cccgtttgtc cagcgcttcg atgaaggcct ggatcctgtt 60
ctagaagtac tcggtggcaa gggcgcttca ctagtcacca tgacagatgc tggaatgccc 120
gttccacctg gatttgtggt cactactgcc agctttgatg aattcatccg tgaagcaggg 180
gttgctgaac acatcgataa attcctaaac gatctcgatg cagaagatgt taaggaagtg 240
gatcgagttt ctgcgatcat ccgcgatgag ctgtgcagtc ttgacgttcc agagaatgct 300
cgtttcgcag tgcaccaggc ttatcgcgat ctcatggaac gatgcggtgg cgacgtcccg 360
gttgctgtcc ggtcatcggc cactgccgaa gatctgcccg atgcttcctt cgcagggcaa 420
caggacacct atctgtggca agtcggtttg agcgctgtca ctgaacacat ccgtaaatgc 480
tgggcttcgc tgttcacttc ccgtgccatt atctaccgtc tgaaaaacaa catccccaat 540
gagggcctct ccatggcggt agttgttcaa aaaatggtca actctcgtgt cgcaggcgtg 600
gcaatcacta tgaatccttc caacggcgac cgctcgaaga tcaccatcga ttcctcatgg 660
ggtgttggtg aaatggtggt ctcaggtgaa gtgacaccag acaatatctt gctggacaag 720
atcacgctgc aggttgtctc cgaacacatt ggaagcaaac acgctgaact catccccgat 780
gccaccagtg gaagcctcgt ggaaaagccc gttgatgaag aacgcgcaaa ccgccgcagt 840
ctgactgatg aggaaatgct cgctgtggca caaatggcta agcgtgcaga aaaacactac 900
aagtgcccac aagatatcga atgggcgctg gacgctgatc tgccagatgg agaaaacctt 960
ctgttattgc aatcccgccc ggaaactatc cactccaacg gtgtgaagaa ggaaacccca 1020
actccgcagg ctgccaaaac cataggcacc ttcgatttca gctcaatcac cgtcgcaatg 1080
accggcacga agtaa 1095
<210> 98
<211> 364
<212> PRT
<213> Corynebacterium glutamicum ATCC13032
<400> 98
Met Thr Asn Ser Leu Asn Ile Pro Phe Val Gln Arg Phe Asp Glu Gly
1 5 10 15
Leu Asp Pro Val Leu Glu Val Leu Gly Gly Lys Gly Ala Ser Leu Val
20 25 30
Thr Met Thr Asp Ala Gly Met Pro Val Pro Pro Gly Phe Val Val Thr
35 40 45
Thr Ala Ser Phe Asp Glu Phe Ile Arg Glu Ala Gly Val Ala Glu His
50 55 60
Ile Asp Lys Phe Leu Asn Asp Leu Asp Ala Glu Asp Val Lys Glu Val
65 70 75 80
Asp Arg Val Ser Ala Ile Ile Arg Asp Glu Leu Cys Ser Leu Asp Val
85 90 95
Pro Glu Asn Ala Arg Phe Ala Val His Gln Ala Tyr Arg Asp Leu Met
100 105 110
Glu Arg Cys Gly Gly Asp Val Pro Val Ala Val Arg Ser Ser Ala Thr
115 120 125
Ala Glu Asp Leu Pro Asp Ala Ser Phe Ala Gly Gln Gln Asp Thr Tyr
130 135 140
Leu Trp Gln Val Gly Leu Ser Ala Val Thr Glu His Ile Arg Lys Cys
145 150 155 160
Trp Ala Ser Leu Phe Thr Ser Arg Ala Ile Ile Tyr Arg Leu Lys Asn
165 170 175
Asn Ile Pro Asn Glu Gly Leu Ser Met Ala Val Val Val Gln Lys Met
180 185 190
Val Asn Ser Arg Val Ala Gly Val Ala Ile Thr Met Asn Pro Ser Asn
195 200 205
Gly Asp Arg Ser Lys Ile Thr Ile Asp Ser Ser Trp Gly Val Gly Glu
210 215 220
Met Val Val Ser Gly Glu Val Thr Pro Asp Asn Ile Leu Leu Asp Lys
225 230 235 240
Ile Thr Leu Gln Val Val Ser Glu His Ile Gly Ser Lys His Ala Glu
245 250 255
Leu Ile Pro Asp Ala Thr Ser Gly Ser Leu Val Glu Lys Pro Val Asp
260 265 270
Glu Glu Arg Ala Asn Arg Arg Ser Leu Thr Asp Glu Glu Met Leu Ala
275 280 285
Val Ala Gln Met Ala Lys Arg Ala Glu Lys His Tyr Lys Cys Pro Gln
290 295 300
Asp Ile Glu Trp Ala Leu Asp Ala Asp Leu Pro Asp Gly Glu Asn Leu
305 310 315 320
Leu Leu Leu Gln Ser Arg Pro Glu Thr Ile His Ser Asn Gly Val Lys
325 330 335
Lys Glu Thr Pro Thr Pro Gln Ala Ala Lys Thr Ile Gly Thr Phe Asp
340 345 350
Phe Ser Ser Ile Thr Val Ala Met Thr Gly Thr Lys
355 360
<210> 99
<211> 3926
<212> DNA
<213> Saccharomyces cerevisiae
<400> 99
ttggtcatgc ttatgaagct atactaaccc cacaagcatt acatggtgaa tgtgtgtcca 60
ttggtatggt taaagaggcg gaattatccc gttatttcgg tattctctcc cctacccaag 120
ttgcacgtct atccaagatt ttggttgcct acgggttgcc tgtttcgcct gatgagaaat 180
ggtttaaaga gctaacctta cataagaaaa caccattgga tatcttattg aagaaaatga 240
gtattgacaa gaaaaacgag ggttccaaaa agaaggtggt cattttagaa agtattggta 300
agtgctatgg tgactccgct caatttgtta gcgatgaaga cctgagattt attctaacag 360
atgaaaccct cgtttacccc ttcaaggaca tccctgctga tcaacagaaa gttgttatcc 420
cccctggttc taagtccatc tccaatcgtg ctttaattct tgctgccctc ggtgaaggtc 480
aatgtaaaat caagaactta ttacattctg atgatactaa acatatgtta accgctgttc 540
atgaattgaa aggtgctacg atatcatggg aagataatgg tgagacggta gtggtggaag 600
gacatggtgg ttccacattg tcagcttgtg ctgacccctt atatctaggt aatgcaggta 660
ctgcatctag atttttgact tccttggctg ccttggtcaa ttctacttca agccaaaagt 720
atatcgtttt aactggtaac gcaagaatgc aacaaagacc aattgctcct ttggtcgatt 780
ctttgcgtgc taatggtact aaaattgagt acttgaataa tgaaggttcc ctgccaatca 840
aagtttatac tgattcggta ttcaaaggtg gtagaattga attagctgct acagtttctt 900
ctcagtacgt atcctctatc ttgatgtgtg ccccatacgc tgaagaacct gtaactttgg 960
ctcttgttgg tggtaagcca atctctaaat tgtacgtcga tatgacaata aaaatgatgg 1020
aaaaattcgg tatcaatgtt gaaacttcta ctacagaacc ttacacttat tatattccaa 1080
agggacatta tattaaccca tcagaatacg tcattgaaag tgatgcctca agtgctacat 1140
acccattggc cttcgccgca atgactggta ctaccgtaac ggttccaaac attggttttg 1200
agtcgttaca aggtgatgcc agatttgcaa gagatgtctt gaaacctatg ggttgtaaaa 1260
taactcaaac ggcaacttca actactgttt cgggtcctcc tgtaggtact ttaaagccat 1320
taaaacatgt tgatatggag ccaatgactg atgcgttctt aactgcatgt gttgttgccg 1380
ctatttcgca cgacagtgat ccaaattctg caaatacaac caccattgaa ggtattgcaa 1440
accagcgtgt caaagagtgt aacagaattt tggccatggc tacagagctc gccaaatttg 1500
gcgtcaaaac tacagaatta ccagatggta ttcaagtcca tggtttaaac tcgataaaag 1560
atttgaaggt tccttccgac tcttctggac ctgtcggtgt atgcacatat gatgatcatc 1620
gtgtggccat gagtttctcg cttcttgcag gaatggtaaa ttctcaaaat gaacgtgacg 1680
aagttgctaa tcctgtaaga atacttgaaa gacattgtac tggtaaaacc tggcctggct 1740
ggtgggatgt gttacattcc gaactaggtg ccaaattaga tggtgcagaa cctttagagt 1800
gcacatccaa aaagaactca aagaaaagcg ttgtcattat tggcatgaga gcagctggca 1860
aaactactat aagtaaatgg tgcgcatccg ctctgggtta caaattagtt gacctagacg 1920
agctgtttga gcaacagcat aacaatcaaa gtgttaaaca atttgttgtg gagaacggtt 1980
gggagaagtt ccgtgaggaa gaaacaagaa ttttcaagga agttattcaa aattacggcg 2040
atgatggata tgttttctca acaggtggcg gtattgttga aagcgctgag tctagaaaag 2100
ccttaaaaga ttttgcctca tcaggtggat acgttttaca cttacatagg gatattgagg 2160
agacaattgt ctttttacaa agtgatcctt caagacctgc ctatgtggaa gaaattcgtg 2220
aagtttggaa cagaagggag gggtggtata aagaatgctc aaatttctct ttctttgctc 2280
ctcattgctc cgcagaagct gagttccaag ctctaagaag atcgtttagt aagtacattg 2340
caaccattac aggtgtcaga gaaatagaaa ttccaagcgg aagatctgcc tttgtgtgtt 2400
taacctttga tgacttaact gaacaaactg agaatttgac tccaatctgt tatggttgtg 2460
aggctgtaga ggtcagagta gaccatttgg ctaattactc tgctgatttc gtgagtaaac 2520
agttatctat attgcgtaaa gccactgaca gtattcctat catttttact gtgcgaacca 2580
tgaagcaagg tggcaacttt cctgatgaag agttcaaaac cttgagagag ctatacgata 2640
ttgccttgaa gaatggtgtt gaattccttg acttagaact aactttacct actgatatcc 2700
aatatgaggt tattaacaaa aggggcaaca ccaagatcat tggttcccat catgacttcc 2760
aaggattata ctcctgggac gacgctgaat gggaaaacag attcaatcaa gcgttaactc 2820
ttgatgtgga tgttgtaaaa tttgtgggta cggctgttaa tttcgaagat aatttgagac 2880
tggaacactt tagggataca cacaagaata agcctttaat tgcagttaat atgacttcta 2940
aaggtagcat ttctcgtgtt ttgaataatg ttttaacacc tgtgacatca gatttattgc 3000
ctaactccgc tgcccctggc caattgacag tagcacaaat taacaagatg tatacatcta 3060
tgggaggtat cgagcctaag gaactgtttg ttgttggaaa gccaattggc cactctagat 3120
cgccaatttt acataacact ggctatgaaa ttttaggttt acctcacaag ttcgataaat 3180
ttgaaactga atccgcacaa ttggtgaaag aaaaactttt ggacggaaac aagaactttg 3240
gcggtgctgc agtcacaatt cctctgaaat tagatataat gcagtacatg gatgaattga 3300
ctgatgctgc taaagttatt ggtgctgtaa acacagttat accattgggt aacaagaagt 3360
ttaagggtga taataccgac tggttaggta tccgtaatgc cttaattaac aatggcgttc 3420
ccgaatatgt tggtcatacc gctggtttgg ttatcggtgc aggtggcact tctagagccg 3480
ccctttacgc cttgcacagt ttaggttgca aaaagatctt cataatcaac aggacaactt 3540
cgaaattgaa gccattaata gagtcacttc catctgaatt caacattatt ggaatagagt 3600
ccactaaatc tatagaagag attaaggaac acgttggcgt tgctgtcagc tgtgtaccag 3660
ccgacaaacc attagatgac gaacttttaa gtaagctgga gagattcctt gtgaaaggtg 3720
cccatgctgc ttttgtacca accttattgg aagccgcata caaaccaagc gttactcccg 3780
ttatgacaat ttcacaagac aaatatcaat ggcacgttgt ccctggatca caaatgttag 3840
tacaccaagg tgtagctcag tttgaaaagt ggacaggatt caagggccct ttcaaggcca 3900
tttttgatgc cgttacgaaa gagtag 3926
<210> 100
<211> 2247
<212> DNA
<213> Anabaena variabilis ATCC29413
<400> 100
atgagtatcg tccaagcaaa gtttgaagct aaggaaacat cttttcatgt agaaggttac 60
gaaaagattg agtatgattt ggtgtatgta gatggtattt ttgaaatcca gaattctgca 120
ctagcagatg tatatcaagg ttttggacga tgcttggcga ttgtagatgc taacgtcagt 180
cggttgtatg gtaatcaaat tcaggcatat ttccagtatt atggtataga actgaggcta 240
tttcctatta ccattactga accagataag actattcaaa ctttcgagag agttatagat 300
gtctttgcag atttcaaatt agtccgcaaa gaaccagtat tagtcgtggg tggcggttta 360
attacagatg ttgtcggctt tgcttgttct acatatcgtc gcagcagcaa ttacatccgc 420
attcctacta cattgattgg attaattgat gccagtgtag caattaaggt agcagttaat 480
catcgcaaac tgaaaaaccg tttgggtgct tatcatgctt ctcgcaaagt atttttagat 540
ttctccttgt tgcgtactct ccctacagac caagtacgta acgggatggc ggaattggta 600
aaaatcgctg tagtagcgca tcaagaagtt tttgaattgt tggagaagta cggcgaagaa 660
ttactacgta ctcattttgg caatatagat gcaactccag agattaaaga aatagcccat 720
cgtttgactt acaaagctat ccataagatg ttggaattgg aagttcccaa cctgcatgag 780
ttagacctag atagggtgat tgcttacggt cacacttgga gtcccacctt ggaacttgcg 840
cctcgtctac ccatgttcca cggacacgcc gttaatgtag atatggcttt ctcggcaacg 900
atcgccgccc gtagaggata tattacaatt gcagaacgcg atcgtatttt aggattaatg 960
agtcgcgttg gtctatccct cgaccatccc atgttggata tagatatttt gtggcgtggt 1020
actgaatcta tcacattaac tcgtgatggt ttgttaagag ctgctatgcc aaaacccatt 1080
ggtgattgtg tcttcgtcaa tgacctgaca agagaagaat tagcagccgc attagctgac 1140
cacaaagaac tttgtaccag ttatccccgt ggtggtgaag gtgtggatgt gtatcccgtt 1200
tatcaaaaag aattaatcgg gagtgttaaa taatgacttt tttgaattca aaatgcaaaa 1260
tactccacgg atacactgcg cgagcgcggt agcatttctg ttcgcggagc gtcccgtagg 1320
gaaagagaag gctacgcaaa taatcggaca ctaattgtct ttaattttga attttgaatt 1380
ttgaattttg aattggagcg aagcgacttg acaaatgtga ttgtccaacc aacagctaga 1440
cctgttacac cattgggaat tttaaccaag cagttagaag ccatagtcca agaggttaag 1500
caacatccag atttacctgg ggaattgata gcaaacatcc atcaggcttg gcgtttagcc 1560
gcaggtatag acccttattt ggaagaatgc accactccag aatctcctga actcgctgca 1620
ttggcaaaaa ccacagccac cgaagcctgg ggagaacact tccacggagg tacaaccgtc 1680
cgtcctctag aacaagagat gctttctggt catatcgaag gacaaacctt aaagatgttt 1740
gttcacatga ccaaagctaa aaaagtctta gaaattggga tgtttaccgg ttattcggcg 1800
ctggcgatgg cggaagcatt accagaggat ggactgcttg tggcttgtga agttgaccct 1860
tacgcggcgg aaattggaca gaaagccttt caacaatctc cccacggtgg aaagattcgt 1920
gtggaattgg atgcagcctt agcaactctt gataagttag cagaagctgg ggagtctttt 1980
gacttggtat ttatcgacgc agataaaaaa gagtatgtag cctattttca caagttgcta 2040
ggtagcagtt tgttagcacc agatggcttt atttgtgtag ataacacctt attacaaggg 2100
gaagtttatc taccagcaga ggaacgtagc gtcaatggtg aagcgatcgc gcaatttaat 2160
catacagtag ctatagaccc ccgtgtagaa caggttttgt tgccgttgcg agatggttta 2220
acaattatcc gcagaataca accttaa 2247
<210> 101
<211> 3532
<212> DNA
<213> Anabaena variabilis ATCC29413
<400> 101
tccagaattc tgcactagca gatgtatatc aaggttttgg acgatgcttg gcgattgtag 60
atgctaacgt cagtcggttg tatggtaatc aaattcaggc atatttccag tattatggta 120
tagaactgag gctatttcct attaccatta ctgaaccaga taagactatt caaactttcg 180
agagagttat agatgtcttt gcagatttca aattagtccg caaagaacca gtattagtcg 240
tgggtggcgg tttaattaca gatgttgtcg gctttgcttg ttctacatat cgtcgcagca 300
gcaattacat ccgcattcct actacattga ttggattaat tgatgccagt gtagcaatta 360
aggtagcagt taatcatcgc aaactgaaaa accgtttggg tgcttatcat gcttctcgca 420
aagtattttt agatttctcc ttgttgcgta ctctccctac agaccaagta cgtaacggga 480
tggcggaatt ggtaaaaatc gctgtagtag cgcatcaaga agtttttgaa ttgttggaga 540
agtacggcga agaattacta cgtactcatt ttggcaatat agatgcaact ccagagatta 600
aagaaatagc ccatcgtttg acttacaaag ctatccataa gatgttggaa ttggaagttc 660
ccaacctgca tgagttagac ctagataggg tgattgctta cggtcacact tggagtccca 720
ccttggaact tgcgcctcgt ctacccatgt tccacggaca cgccgttaat gtagatatgg 780
ctttctcggc aacgatcgcc gcccgtagag gatatattac aattgcagaa cgcgatcgta 840
ttttaggatt aatgagtcgc gttggtctat ccctcgacca tcccatgttg gatatagata 900
ttttgtggcg tggtactgaa tctatcacat taactcgtga tggtttgtta agagctgcta 960
tgccaaaacc cattggtgat tgtgtcttcg tcaatgacct gacaagagaa gaattagcag 1020
ccgcattagc tgaccacaaa gaactttgta ccagttatcc ccgtggtggt gaaggtgtgg 1080
atgtgtatcc cgtttatcaa aaagaattaa tcgggagtgt taaataatga cttttttgaa 1140
ttcaaaatgc aaaatactcc acggatacac tgcgcgagcg cggtagcatt tctgttcgcg 1200
gagcgtcccg tagggaaaga gaaggctacg caaataatcg gacactaatt gtctttaatt 1260
ttgaattttg aattttgaat tttgaattgg agcgaagcga cttgacaaat gtgattgtcc 1320
aaccaacagc tagacctgtt acaccattgg gaattttaac caagcagtta gaagccatag 1380
tccaagaggt taagcaacat ccagatttac ctggggaatt gatagcaaac atccatcagg 1440
cttggcgttt agccgcaggt atagaccctt atttggaaga atgcaccact ccagaatctc 1500
ctgaactcgc tgcattggca aaaaccacag ccaccgaagc ctggggagaa cacttccacg 1560
gaggtacaac cgtccgtcct ctagaacaag agatgctttc tggtcatatc gaaggacaaa 1620
ccttaaagat gtttgttcac atgaccaaag ctaaaaaagt cttagaaatt gggatgttta 1680
ccggttattc ggcgctggcg atggcggaag cattaccaga ggatggactg cttgtggctt 1740
gtgaagttga cccttacgcg gcggaaattg gacagaaagc ctttcaacaa tctccccacg 1800
gtggaaagat tcgtgtggaa ttggatgcag ccttagcaac tcttgataag ttagcagaag 1860
ctggggagtc ttttgacttg gtatttatcg acgcagataa aaaagagtat gtagcctatt 1920
ttcacaagtt gctaggtagc agtttgttag caccagatgg ctttatttgt gtagataaca 1980
ccttattaca aggggaagtt tatctaccag cagaggaacg tagcgtcaat ggtgaagcga 2040
tcgcgcaatt taatcataca gtagctatag acccccgtgt agaacaggtt ttgttgccgt 2100
tgcgagatgg tttaacaatt atccgcagaa tacaacctta attgtccaat cgactatggc 2160
acaatccctt cccctttctt ccgcacctgc tacaccgtct cttccttccc agacgaaaat 2220
agccgcaatt atccaaaata tctgcacttt ggctttgtta ttactagcat tgcccattaa 2280
tgccaccatt gtttttatat ccttgttagt cttccgaccg caaaaggtca aagcagcaaa 2340
cccccaaacc attcttatca gtggcggtaa gatgaccaaa gctttacaac tagcaaggtc 2400
attccacgcg gctggacata gagttgtctt ggtggaaacc cataaatact ggttgactgg 2460
tcatcgtttt tcccaagcag tggataagtt ttacacagtc cccgcacccc aggacaatcc 2520
ccaagcttac attcaggctt tggtagatat cgtcaaacaa gaaaacatcg atgtttatat 2580
tcccgtcacc agtccagtgg gtagctacta cgactcatta gccaaaccag agttatccca 2640
ttattgcgaa gtgtttcact ttgacgcaga tattacccaa atgttggatg ataaatttgc 2700
gttgacacaa aaagcgcgat cgcttggttt atcagtaccc aaatccttta aaattacctc 2760
accagaacaa gtcatcaact tcgatttttc tggagagaca cgtaaataca tcctcaaaag 2820
cattccctac gactcagtgc ggcggttgga cttaaccaaa ctcccctgtg ctactccaga 2880
ggaaacagca gcattcgtca gaagtttgcc aattactccc gaaaaaccgt ggattatgca 2940
ggaatttatc cccggtaagg aattctgcac ccatagcacc gttcggaatg gggaactcag 3000
actgcattgc tgttgcgaat cttcagcctt ccaagttaat tatgagaatg taaataaccc 3060
gcaaattacc gaatgggtac agcattttgt caaggaactg aaactgacag gacagatttc 3120
ctttgacttt atccaagccg aagacggaac agtttacgcc atcgagtgta acccccgcac 3180
acattcagca attaccacat tttacgacca cccccaggta gcagaagcgt acttgagtca 3240
agcaccgacg actgaaacca tacaaccact aacgacaagc aagcctacct attggactta 3300
tcacgaagtt tggcgtttaa ctggtatccg ttctttcacc cagttgcaaa gatggctggg 3360
gaatatttgg cgcgggactg atgcgattta tcagccagat gaccccttac cgtttttgat 3420
ggtacatcat tggcaaattc ccctactgtt attgaataat ttgcgtcgtc ttaaaggttg 3480
gacgcggata gatttcaata ttgggaagtt ggtggaattg gggggagatt ag 3532
<210> 102
<211> 1233
<212> DNA
<213> Anabaena variabilis ATCC29413
<400> 102
atgagtatcg tccaagcaaa gtttgaagct aaggaaacat cttttcatgt agaaggttac 60
gaaaagattg agtatgattt ggtgtatgta gatggtattt ttgaaatcca gaattctgca 120
ctagcagatg tatatcaagg ttttggacga tgcttggcga ttgtagatgc taacgtcagt 180
cggttgtatg gtaatcaaat tcaggcatat ttccagtatt atggtataga actgaggcta 240
tttcctatta ccattactga accagataag actattcaaa ctttcgagag agttatagat 300
gtctttgcag atttcaaatt agtccgcaaa gaaccagtat tagtcgtggg tggcggttta 360
attacagatg ttgtcggctt tgcttgttct acatatcgtc gcagcagcaa ttacatccgc 420
attcctacta cattgattgg attaattgat gccagtgtag caattaaggt agcagttaat 480
catcgcaaac tgaaaaaccg tttgggtgct tatcatgctt ctcgcaaagt atttttagat 540
ttctccttgt tgcgtactct ccctacagac caagtacgta acgggatggc ggaattggta 600
aaaatcgctg tagtagcgca tcaagaagtt tttgaattgt tggagaagta cggcgaagaa 660
ttactacgta ctcattttgg caatatagat gcaactccag agattaaaga aatagcccat 720
cgtttgactt acaaagctat ccataagatg ttggaattgg aagttcccaa cctgcatgag 780
ttagacctag atagggtgat tgcttacggt cacacttgga gtcccacctt ggaacttgcg 840
cctcgtctac ccatgttcca cggacacgcc gttaatgtag atatggcttt ctcggcaacg 900
atcgccgccc gtagaggata tattacaatt gcagaacgcg atcgtatttt aggattaatg 960
agtcgcgttg gtctatccct cgaccatccc atgttggata tagatatttt gtggcgtggt 1020
actgaatcta tcacattaac tcgtgatggt ttgttaagag ctgctatgcc aaaacccatt 1080
ggtgattgtg tcttcgtcaa tgacctgaca agagaagaat tagcagccgc attagctgac 1140
cacaaagaac tttgtaccag ttatccccgt ggtggtgaag gtgtggatgt gtatcccgtt 1200
tatcaaaaag aattaatcgg gagtgttaaa taa 1233
<210> 103
<211> 840
<212> DNA
<213> Anabaena variabilis ATCC29413
<400> 103
ttgacaaatg tgattgtcca accaacagct agacctgtta caccattggg aattttaacc 60
aagcagttag aagccatagt ccaagaggtt aagcaacatc cagatttacc tggggaattg 120
atagcaaaca tccatcaggc ttggcgttta gccgcaggta tagaccctta tttggaagaa 180
tgcaccactc cagaatctcc tgaactcgct gcattggcaa aaaccacagc caccgaagcc 240
tggggagaac acttccacgg aggtacaacc gtccgtcctc tagaacaaga gatgctttct 300
ggtcatatcg aaggacaaac cttaaagatg tttgttcaca tgaccaaagc taaaaaagtc 360
ttagaaattg ggatgtttac cggttattcg gcgctggcga tggcggaagc attaccagag 420
gatggactgc ttgtggcttg tgaagttgac ccttacgcgg cggaaattgg acagaaagcc 480
tttcaacaat ctccccacgg tggaaagatt cgtgtggaat tggatgcagc cttagcaact 540
cttgataagt tagcagaagc tggggagtct tttgacttgg tatttatcga cgcagataaa 600
aaagagtatg tagcctattt tcacaagttg ctaggtagca gtttgttagc accagatggc 660
tttatttgtg tagataacac cttattacaa ggggaagttt atctaccagc agaggaacgt 720
agcgtcaatg gtgaagcgat cgcgcaattt aatcatacag tagctataga cccccgtgta 780
gaacaggttt tgttgccgtt gcgagatggt ttaacaatta tccgcagaat acaaccttaa 840
840
<210> 104
<211> 1377
<212> DNA
<213> Anabaena variabilis ATCC29413
<400> 104
atggcacaat cccttcccct ttcttccgca cctgctacac cgtctcttcc ttcccagacg 60
aaaatagccg caattatcca aaatatctgc actttggctt tgttattact agcattgccc 120
attaatgcca ccattgtttt tatatccttg ttagtcttcc gaccgcaaaa ggtcaaagca 180
gcaaaccccc aaaccattct tatcagtggc ggtaagatga ccaaagcttt acaactagca 240
aggtcattcc acgcggctgg acatagagtt gtcttggtgg aaacccataa atactggttg 300
actggtcatc gtttttccca agcagtggat aagttttaca cagtccccgc accccaggac 360
aatccccaag cttacattca ggctttggta gatatcgtca aacaagaaaa catcgatgtt 420
tatattcccg tcaccagtcc agtgggtagc tactacgact cattagccaa accagagtta 480
tcccattatt gcgaagtgtt tcactttgac gcagatatta cccaaatgtt ggatgataaa 540
tttgcgttga cacaaaaagc gcgatcgctt ggtttatcag tacccaaatc ctttaaaatt 600
acctcaccag aacaagtcat caacttcgat ttttctggag agacacgtaa atacatcctc 660
aaaagcattc cctacgactc agtgcggcgg ttggacttaa ccaaactccc ctgtgctact 720
ccagaggaaa cagcagcatt cgtcagaagt ttgccaatta ctcccgaaaa accgtggatt 780
atgcaggaat ttatccccgg taaggaattc tgcacccata gcaccgttcg gaatggggaa 840
ctcagactgc attgctgttg cgaatcttca gccttccaag ttaattatga gaatgtaaat 900
aacccgcaaa ttaccgaatg ggtacagcat tttgtcaagg aactgaaact gacaggacag 960
atttcctttg actttatcca agccgaagac ggaacagttt acgccatcga gtgtaacccc 1020
cgcacacatt cagcaattac cacattttac gaccaccccc aggtagcaga agcgtacttg 1080
agtcaagcac cgacgactga aaccatacaa ccactaacga caagcaagcc tacctattgg 1140
acttatcacg aagtttggcg tttaactggt atccgttctt tcacccagtt gcaaagatgg 1200
ctggggaata tttggcgcgg gactgatgcg atttatcagc cagatgaccc cttaccgttt 1260
ttgatggtac atcattggca aattccccta ctgttattga ataatttgcg tcgtcttaaa 1320
ggttggacgc ggatagattt caatattggg aagttggtgg aattgggggg agattag 1377
<210> 105
<211> 2358
<212> DNA
<213> Anabaena variabilis ATCC29413
<400> 105
gttcctattg accctgaata tcctcaagaa cgcatagctt atatgctaga agattctcag 60
gtgaaggtac tactaactca agaaaaatta ctcaatcaaa ttccccacca tcaagcacaa 120
actatctgtg tagataggga atgggagaaa atttccacac aagctaatac caatcccaaa 180
agtaatataa aaacggataa tcttgcttat gtaatttaca cctctggttc cactggtaaa 240
ccaaaaggtg caatgaacac ccacaaaggt atctgtaatc gcttattgtg gatgcaggaa 300
gcttatcaaa tcgattccac agatagcatt ttacaaaaaa ccccctttag ttttgatgtt 360
tccgtttggg agttcttttg gactttatta actggcgcac gtttggtaat agccaaacca 420
ggcggacata aagatagtgc ttacctcatc gatttaatta ctcaagaaca aatcactacg 480
ttgcattttg tcccctcaat gctgcaagtg tttttacaaa atcgccatgt aagcaaatgc 540
agctctctaa aaagagttat ttgtagcggt gaagctttat ctatagattt acaaaataga 600
tttttccagc atttgcaatg tgaattacat aacctctatg gcccgacaga agcagcaatt 660
gatgtcacat tttggcaatg tagaaaagat agtaatttaa agagtgtacc tattggtcgt 720
cccattgcta atactcaaat ttatattctt gatgccgatt tacaaccagt aaatattggt 780
gtcactggtg aaatttatat tggtggtgta ggggttgctc gtggttattt gaataaagaa 840
gaattgacca aagaaaaatt tattattaat ccctttccca attctgagtt taagcgactt 900
tataaaacag gtgatttagc tcgttattta cccgatggaa atattgaata tcttggtaga 960
acagattatc aagtaaaaat tcggggttat agaattgaaa ttggcgagat tgaaaatgtt 1020
ttatcttcac acccacaagt cagagaagct gtagtcatag cgcgggatga taacgctcaa 1080
gaaaaacaaa tcatcgctta tattacctat aactccatca aacctcagct tgataatctg 1140
cgtgatttcc taaaagcaag gctacctgat tttatgattc cagccgcttt tgtgatgctg 1200
gagcatcttc ctttaactcc cagtggtaaa gtagaccgta aggcattacc taagcctgat 1260
ttatttaatt atagtgaaca taattcctat gtagcgcctc ggaatgaagt tgaagaaaaa 1320
ttagtacaaa tctggtcgaa tattctgcat ttacctaaag taggtgtgac agaaaacttt 1380
ttcgctattg gtggtaattc cctcaaagct ctacatttaa tttctcaaat tgaagagtta 1440
tttgctaaag agatatcctt agcaacactt ttaacaaatc cagtaattgc agatttagcc 1500
aaggttattc aagcaaacaa ccaaatccat aattcacccc tagttccaat tcaaccacaa 1560
ggtaagcagc agcctttctt ttgtatacat cctgctggtg gtcatgtttt atgctatttt 1620
aaactcgcac aatatatagg aactgaccaa ccattttatg gcttacaagc tcaaggattt 1680
tatggagatg aagcaccctt gacgcgagtt gaagatatgg ctagtctcta cgtcaaaact 1740
attagagaat ttcaacccca agggccttat cgtgtcgggg ggtggtcatt tggtggagtc 1800
gtagcttatg aagtagcaca gcagttacat agacaaggac aagaagtatc tttactagca 1860
atattagatt cttacgtacc gattctgctg gataaacaaa aacccattga tgacgtttat 1920
ttagttggtg ttctctccag agtttttggc ggtatgtttg gtcaagataa tctagtcaca 1980
cctgaagaaa tagaaaattt aactgtagaa gaaaaaatta attacatcat tgataaagca 2040
cggagcgcta gaatattccc gcctggtgta gaacgtcaaa ataatcgccg tattcttgat 2100
gttttggtgg gaactttaaa agcaacttat tcctatataa gacaaccata tccaggaaaa 2160
gtcactgtat ttcgagccag ggaaaaacat attatggctc ctgacccgac cttagtttgg 2220
gtagaattat tttctgtaat ggcggctcaa gaaattaaga ttattgatgt ccctggaaac 2280
cattattcgt ttgttctaga accccatgta caggttttag cacagcgttt acaagattgt 2340
ctggaaaata attcataa 2358
<210> 106
<211> 1233
<212> DNA
<213> Nostoc punctiforme ATCC29133
<400> 106
atgagtaatg ttcaagcatc gtttgaagca acggaagctg aattccgcgt ggaaggttac 60
gaaaaaattg agtttagtct tgtctatgta aatggtgcat ttgatatcag taacagagaa 120
attgcagaca gctatgagaa gtttggccgt tgtctgactg tgattgatgc taatgtcaac 180
agattatatg gcaagcaaat caagtcatat tttagacact atggtattga tctgaccgta 240
gttcccattg tgattactga gcctactaaa acccttgcaa cctttgagaa aattgttgat 300
gctttttctg actttggttt aatccgcaag gaaccagtat tagtagtggg tggtggttta 360
accactgatg tagctgggtt tgcgtgtgct gcttaccgtc gtaagagtaa ctatattcgg 420
gttccgacaa cgctgattgg tctgattgat gcaggtgtag cgattaaggt tgcagtcaac 480
catcgcaagt taaaaaatcg cttgggtgca tatcatgctc ctttgaaagt catcctcgat 540
ttctccttct tgcaaacatt accaacggct caagttcgta atgggatggc agagttggtc 600
aaaattgctg ttgtggcgaa ctctgaagtc tttgaattgt tgtatgaata tggcgaagag 660
ttgctttcca ctcactttgg atatgtgaat ggtacaaagg aactgaaagc gatcgcacat 720
aaactcaatt acgaggcaat aaaaactatg ctggagttgg aaactccaaa cttgcatgag 780
ttagacctcg atcgcgtcat tgcctacggt cacacttgga gtccgacctt agaattagct 840
ccgatgatac cgttgtttca cggtcatgcc gtcaatatag acatggcttt gtctgcaact 900
attgcggcaa gacgtggcta cattacatca ggagaacgcg atcgcatttt gagcttgatg 960
agtcgtatag gtttatcaat cgatcatcct ctactagatg gcgatttgct ctggtatgct 1020
acccaatcta ttagcttgac gcgagacggg aaacaacgcg cagctatgcc taaacccatt 1080
ggtgagtgct tctttgtcaa cgatttcacc cgtgaagaac tagatgcagc tttagctgaa 1140
cacaaacgtc tgtgtgctac ataccctcgt ggtggagatg gcattgacgc ttacatagaa 1200
actcaagaag aatccaaact attgggagtg tga 1233
<210> 107
<211> 834
<212> DNA
<213> Nostoc punctiforme ATCC29133
<400> 107
atgaccagta ttttaggacg agataccgca agaccaataa cgccacatag cattctggta 60
gcacagctac aaaaaaccct cagaatggca gaggaaagta atattccttc agagatactg 120
acttctctgc gccaagggtt gcaattagca gcaggtttag atccctatct ggatgattgc 180
actacccctg aatcgaccgc attgacagca ctagcgcaga agacaagcat tgaagactgg 240
agtaaacgct tcagtgatgg tgaaacagtg cgtcaattag agcaagaaat gctctcagga 300
catcttgaag gacaaacact aaagatgttt gtgcatatca ctaaggctaa aagcatccta 360
gaagtgggaa tgttcacagg atattcagct ttggcaatgg cagaggcgtt accagatgat 420
gggcgactga ttgcttgtga agtagactcc tatgtggccg agtttgctca aacttgcttt 480
caagagtctc cccacggccg caagattgtt gtagaagtgg cacctgcact agagacgctg 540
cacaagctgg tggctaaaaa agaatccttt gatctgatct tcattgatgc ggataaaaag 600
gagtatatag aatacttcca aattatcttg gatagccatt tactagctcc cgacggatta 660
atctgtgtag ataatacttt gttgcaagga caagtttacc tgccatcaga acagcgtaca 720
gccaatggtg aagcgatcgc tcaattcaac cgcattgtcg ccgcagatcc tcgtgtagag 780
caagttctgt tacccatacg agatggtata accctgatta gacgcttggt ataa 834
<210> 108
<211> 1086
<212> DNA
<213> Nostoc punctiforme ATCC29133
<400> 108
tactggttat cagggcatag attctcaaat tctgtgagtc gtttttatac agttcctgca 60
ccacaagacg acccagaagg ctatacccaa gcgctattgg aaattgtcaa acgagagaag 120
attgacgttt atgtacccgt atgcagccct gtagctagtt attacgactc tttggcaaag 180
tctgcactat cagaatattg tgaggttttt cactttgatg ctgatataac caagatgctg 240
gatgataaat ttgcctttac cgatcgggcg cgatcgcttg gtttatcagc cccgaaatct 300
tttaaaatta ccgatcctga acaagttatc aacttcgatt ttagtaaaga gacgcgcaaa 360
tatattctta agagtatttc ttacgactca gttcgccgct taaatttaac caaacttcct 420
tgtgataccc cagaagagac agcagcgttt gtcaagagtt tacccatcag cccagaaaaa 480
ccttggatta tgcaagaatt tattcctggg aaagaattat gcacccatag cacagtccga 540
gacggcgaat taaggttgca ttgctgttca aattcttcag cgtttcagat taactatgaa 600
aatgtcgaaa atccccaaat tcaagaatgg gtacaacatt tcgtcaaaag tttacggctg 660
actggacaaa tatctcttga ctttatccaa gctgaagatg gtacagctta tgccattgaa 720
tgtaatcctc gcacccattc ggcgatcaca atgttctaca atcacccagg tgttgcagaa 780
gcctatcttg gtaaaactcc tctagctgca cctttggaac ctcttgcaga tagcaagccc 840
acttactgga tatatcacga aatctggcga ttgactggga ttcgctctgg acaacaatta 900
caaacttggt ttgggagatt agtcagaggt acagatgcca tttatcgcct ggacgatccg 960
ataccatttt taactttgca ccattggcag attactttac ttttgctaca aaatttgcaa 1020
cgactcaaag gttgggtaaa gatcgatttt aatatcggta aactcgtgga attagggggc 1080
gactaa 1086
<210> 109
<211> 1047
<212> DNA
<213> Nostoc punctiforme ATCC29133
<400> 109
atgccagtac ttaatatcct tcatttagtt gggtctgcac acgataagtt ttactgtgat 60
ttatcacgtc tttatgccca agactgttta gctgcaacag cagatccatc gctttataac 120
tttcaaattg catatatcac acccgatcgg cagtggcgat ttcctgactc tctcagtcga 180
gaagatattg ctcttaccaa accgattcct gtgtttgatg ccatacaatt tctaacaggc 240
caaaacattg acatgatgtt accacaaatg ttttgtattc ctggaatgac tcagtaccgt 300
gccctattcg atctgctcaa gatcccttat ataggaaata ccccagatat tatggcgatc 360
gcggcccaca aagccagagc caaagcaatt gtcgaagcag caggggtaaa agtgcctcgt 420
ggagaattgc ttcgccaagg agatattcca acaattacac ctccagcagt cgtcaaacct 480
gtaagttctg acaactcttt aggagtagtc ttagttaaag atgtgactga atatgatgct 540
gccttaaaga aagcatttga atatgcttcg gaggtcatcg tagaagcatt catcgaactt 600
ggtcgagaag tcagatgcgg catcattgta aaagacggtg agctaatagg tttacccctt 660
gaagagtatc tggtagaccc acacgataaa cctatccgta actatgctga taaactccaa 720
caaactgacg atggcgactt gcatttgact gctaaagata atatcaaggc ttggatttta 780
gaccctaacg acccaatcac ccaaaaggtt cagcaagtgg ctaaaaggtg tcatcaggct 840
ttgggttgtc gccactacag tttatttgac ttccgaatcg atccaaaggg acaaccttgg 900
ttcttagaag ctggattata ttgttctttt gcccccaaaa gtgtgatttc ttctatggcg 960
aaagcagccg gaatccctct aaatgattta ttaataaccg ctattaatga aacattgggt 1020
agtaataaaa aggtgttaca aaattga 1047
<210> 110
<211> 1626
<212> DNA
<213> Saccharomyces cerevisiae CEN.PK-1D
<400> 110
aacaagccgg gctttacctt gtctgacaac tacacctatg ttttcttggg tgacggttgt 60
ttgcaagaag gtatttcttc agaagcttcc tccttggctg gtcatttgaa attgggtaac 120
ttgattgcca tctacgatga caacaagatc actatcgatg gtgctaccag tatctcattc 180
gatgaagatg ttgctaagag atacgaagcc tacggttggg aagttttgta cgtagaaaat 240
ggtaacgaag atctagccgg tattgccaag gctattgctc aagctaagtt atccaaggac 300
aaaccaactt tgatcaaaat gaccacaacc attggttacg gttccttgca tgccggctct 360
cactctgtgc acggtgcccc attgaaagca gatgatgtta aacaactaaa gagcaaattc 420
ggtttcaacc cagacaagtc ctttgttgtt ccacaagaag tttacgacca ctaccaaaag 480
acaattttaa agccaggtgt cgaagccaac aacaagtgga acaagttgtt cagcgaatac 540
caaaagaaat tcccagaatt aggtgctgaa ttggctagaa gattgagcgg ccaactaccc 600
gcaaattggg aatctaagtt gccaacttac accgccaagg actctgccgt ggccactaga 660
aaattatcag aaactgttct tgaggatgtt tacaatcaat tgccagagtt gattggtggt 720
tctgccgatt taacaccttc taacttgacc agatggaagg aagcccttga cttccaacct 780
ccttcttccg gttcaggtaa ctactctggt agatacatta ggtacggtat tagagaacac 840
gctatgggtg ccataatgaa cggtatttca gctttcggtg ccaactacaa accatacggt 900
ggtactttct tgaacttcgt ttcttatgct gctggtgccg ttagattgtc cgctttgtct 960
ggccacccag ttatttgggt tgctacacat gactctatcg gtgtcggtga agatggtcca 1020
acacatcaac ctattgaaac tttagcacac ttcagatccc taccaaacat tcaagtttgg 1080
agaccagctg atggtaacga agtttctgcc gcctacaaga actctttaga atccaagcat 1140
actccaagta tcattgcttt gtccagacaa aacttgccac aattggaagg tagctctatt 1200
gaaagcgctt ctaagggtgg ttacgtacta caagatgttg ctaacccaga tattatttta 1260
gtggctactg gttccgaagt gtctttgagt gttgaagctg ctaagacttt ggccgcaaag 1320
aacatcaagg ctcgtgttgt ttctctacca gatttcttca cttttgacaa acaaccccta 1380
gaatacagac tatcagtctt accagacaac gttccaatca tgtctgttga agttttggct 1440
accacatgtt ggggcaaata cgctcatcaa tccttcggta ttgacagatt tggtgcctcc 1500
ggtaaggcac cagaagtctt caagttcttc ggtttcaccc cagaaggtgt tgctgaaaga 1560
gctcaaaaga ccattgcatt ctataagggt gacaagctaa tttctccttt gaaaaaagct 1620
ttctaa 1626
<210> 111
<211> 1113
<212> DNA
<213> Saccharomyces cerevisiae CEN.PK-1D
<400> 111
atgagtgaat ctccaatgtt cgctgccaac ggcatgccaa aggtaaatca aggtgctgaa 60
gaagatgtca gaattttagg ttacgaccca ttagcttctc cagctctcct tcaagtgcaa 120
atcccagcca caccaacttc tttggaaact gccaagagag gtagaagaga agctatagat 180
attattaccg gtaaagacga cagagttctt gtcattgtcg gtccttgttc catccatgat 240
ctagaagccg ctcaagaata cgctttgaga ttaaagaaat tgtcagatga attaaaaggt 300
gatttatcca tcattatgag agcatacttg gagaagccaa gaacaaccgt cggctggaaa 360
ggtctaatta atgaccctga tgttaacaac actttcaaca tcaacaaggg tttgcaatcc 420
gctagacaat tgtttgtcaa cttgacaaat atcggtttgc caattggttc tgaaatgctt 480
gataccattt ctcctcaata cttggctgat ttggtctcct tcggtgccat tggtgccaga 540
accaccgaat ctcaactgca cagagaattg gcctccggtt tgtctttccc agttggtttc 600
aagaacggta ccgatggtac cttaaatgtt gctgtggatg cttgtcaagc cgctgctcat 660
tctcaccatt tcatgggtgt tactaagcat ggtgttgctg ctatcaccac tactaagggt 720
aacgaacact gcttcgttat tctaagaggt ggtaaaaagg gtaccaacta cgacgctaag 780
tccgttgcag aagctaaggc tcaattgcct gccggttcca acggtctaat gattgactac 840
tctcacggta actccaataa ggatttcaga aaccaaccaa aggtcaatga cgttgtttgt 900
gagcaaatcg ctaacggtga aaacgccatt accggtgtca tgattgaatc aaacatcaac 960
gaaggtaacc aaggcatccc agccgaaggt aaagccggct tgaaatatgg tgtttccatc 1020
actgatgctt gtataggttg ggaaactact gaagacgtct tgaggaaatt ggctgctgct 1080
gtcagacaaa gaagagaagt taacaagaaa tag 1113
<210> 112
<211> 1588
<212> PRT
<213> Saccharomyces cerevisiae
<400> 112
Met Val Gln Leu Ala Lys Val Pro Ile Leu Gly Asn Asp Ile Ile His
1 5 10 15
Val Gly Tyr Asn Ile His Asp His Leu Val Glu Thr Ile Ile Lys His
20 25 30
Cys Pro Ser Ser Thr Tyr Val Ile Cys Asn Asp Thr Asn Leu Ser Lys
35 40 45
Val Pro Tyr Tyr Gln Gln Leu Val Leu Glu Phe Lys Ala Ser Leu Pro
50 55 60
Glu Gly Ser Arg Leu Leu Thr Tyr Val Val Lys Pro Gly Glu Thr Ser
65 70 75 80
Lys Ser Arg Glu Thr Lys Ala Gln Leu Glu Asp Tyr Leu Leu Val Glu
85 90 95
Gly Cys Thr Arg Asp Thr Val Met Val Ala Ile Gly Gly Gly Val Ile
100 105 110
Gly Asp Met Ile Gly Phe Val Ala Ser Thr Phe Met Arg Gly Val Arg
115 120 125
Val Val Gln Val Pro Thr Ser Leu Leu Ala Met Val Asp Ser Ser Ile
130 135 140
Gly Gly Lys Thr Ala Ile Asp Thr Pro Leu Gly Lys Asn Phe Ile Gly
145 150 155 160
Ala Phe Trp Gln Pro Lys Phe Val Leu Val Asp Ile Lys Trp Leu Glu
165 170 175
Thr Leu Ala Lys Arg Glu Phe Ile Asn Gly Met Ala Glu Val Ile Lys
180 185 190
Thr Ala Cys Ile Trp Asn Ala Asp Glu Phe Thr Arg Leu Glu Ser Asn
195 200 205
Ala Ser Leu Phe Leu Asn Val Val Asn Gly Ala Lys Asn Val Lys Val
210 215 220
Thr Asn Gln Leu Thr Asn Glu Ile Asp Glu Ile Ser Asn Thr Asp Ile
225 230 235 240
Glu Ala Met Leu Asp His Thr Tyr Lys Leu Val Leu Glu Ser Ile Lys
245 250 255
Val Lys Ala Glu Val Val Ser Ser Asp Glu Arg Glu Ser Ser Leu Arg
260 265 270
Asn Leu Leu Asn Phe Gly His Ser Ile Gly His Ala Tyr Glu Ala Ile
275 280 285
Leu Thr Pro Gln Ala Leu His Gly Glu Cys Val Ser Ile Gly Met Val
290 295 300
Lys Glu Ala Glu Leu Ser Arg Tyr Phe Gly Ile Leu Ser Pro Thr Gln
305 310 315 320
Val Ala Arg Leu Ser Lys Ile Leu Val Ala Tyr Gly Leu Pro Val Ser
325 330 335
Pro Asp Glu Lys Trp Phe Lys Glu Leu Thr Leu His Lys Lys Thr Pro
340 345 350
Leu Asp Ile Leu Leu Lys Lys Met Ser Ile Asp Lys Lys Asn Glu Gly
355 360 365
Ser Lys Lys Lys Val Val Ile Leu Glu Ser Ile Gly Lys Cys Tyr Gly
370 375 380
Asp Ser Ala Gln Phe Val Ser Asp Glu Asp Leu Arg Phe Ile Leu Thr
385 390 395 400
Asp Glu Thr Leu Val Tyr Pro Phe Lys Asp Ile Pro Ala Asp Gln Gln
405 410 415
Lys Val Val Ile Pro Pro Gly Ser Lys Ser Ile Ser Asn Arg Ala Leu
420 425 430
Ile Leu Ala Ala Leu Gly Glu Gly Gln Cys Lys Ile Lys Asn Leu Leu
435 440 445
His Ser Asp Asp Thr Lys His Met Leu Thr Ala Val His Glu Leu Lys
450 455 460
Gly Ala Thr Ile Ser Trp Glu Asp Asn Gly Glu Thr Val Val Val Glu
465 470 475 480
Gly His Gly Gly Ser Thr Leu Ser Ala Cys Ala Asp Pro Leu Tyr Leu
485 490 495
Gly Asn Ala Gly Thr Ala Ser Arg Phe Leu Thr Ser Leu Ala Ala Leu
500 505 510
Val Asn Ser Thr Ser Ser Gln Lys Tyr Ile Val Leu Thr Gly Asn Ala
515 520 525
Arg Met Gln Gln Arg Pro Ile Ala Pro Leu Val Asp Ser Leu Arg Ala
530 535 540
Asn Gly Thr Lys Ile Glu Tyr Leu Asn Asn Glu Gly Ser Leu Pro Ile
545 550 555 560
Lys Val Tyr Thr Asp Ser Val Phe Lys Gly Gly Arg Ile Glu Leu Ala
565 570 575
Ala Thr Val Ser Ser Gln Tyr Val Ser Ser Ile Leu Met Cys Ala Pro
580 585 590
Tyr Ala Glu Glu Pro Val Thr Leu Ala Leu Val Gly Gly Lys Pro Ile
595 600 605
Ser Lys Leu Tyr Val Asp Met Thr Ile Lys Met Met Glu Lys Phe Gly
610 615 620
Ile Asn Val Glu Thr Ser Thr Thr Glu Pro Tyr Thr Tyr Tyr Ile Pro
625 630 635 640
Lys Gly His Tyr Ile Asn Pro Ser Glu Tyr Val Ile Glu Ser Asp Ala
645 650 655
Ser Ser Ala Thr Tyr Pro Leu Ala Phe Ala Ala Met Thr Gly Thr Thr
660 665 670
Val Thr Val Pro Asn Ile Gly Phe Glu Ser Leu Gln Gly Asp Ala Arg
675 680 685
Phe Ala Arg Asp Val Leu Lys Pro Met Gly Cys Lys Ile Thr Gln Thr
690 695 700
Ala Thr Ser Thr Thr Val Ser Gly Pro Pro Val Gly Thr Leu Lys Pro
705 710 715 720
Leu Lys His Val Asp Met Glu Pro Met Thr Asp Ala Phe Leu Thr Ala
725 730 735
Cys Val Val Ala Ala Ile Ser His Asp Ser Asp Pro Asn Ser Ala Asn
740 745 750
Thr Thr Thr Ile Glu Gly Ile Ala Asn Gln Arg Val Lys Glu Cys Asn
755 760 765
Arg Ile Leu Ala Met Ala Thr Glu Leu Ala Lys Phe Gly Val Lys Thr
770 775 780
Thr Glu Leu Pro Asp Gly Ile Gln Val His Gly Leu Asn Ser Ile Lys
785 790 795 800
Asp Leu Lys Val Pro Ser Asp Ser Ser Gly Pro Val Gly Val Cys Thr
805 810 815
Tyr Asp Asp His Arg Val Ala Met Ser Phe Ser Leu Leu Ala Gly Met
820 825 830
Val Asn Ser Gln Asn Glu Arg Asp Glu Val Ala Asn Pro Val Arg Ile
835 840 845
Leu Glu Arg His Cys Thr Gly Lys Thr Trp Pro Gly Trp Trp Asp Val
850 855 860
Leu His Ser Glu Leu Gly Ala Lys Leu Asp Gly Ala Glu Pro Leu Glu
865 870 875 880
Cys Thr Ser Lys Lys Asn Ser Lys Lys Ser Val Val Ile Ile Gly Met
885 890 895
Arg Ala Ala Gly Lys Thr Thr Ile Ser Lys Trp Cys Ala Ser Ala Leu
900 905 910
Gly Tyr Lys Leu Val Asp Leu Asp Glu Leu Phe Glu Gln Gln His Asn
915 920 925
Asn Gln Ser Val Lys Gln Phe Val Val Glu Asn Gly Trp Glu Lys Phe
930 935 940
Arg Glu Glu Glu Thr Arg Ile Phe Lys Glu Val Ile Gln Asn Tyr Gly
945 950 955 960
Asp Asp Gly Tyr Val Phe Ser Thr Gly Gly Gly Ile Val Glu Ser Ala
965 970 975
Glu Ser Arg Lys Ala Leu Lys Asp Phe Ala Ser Ser Gly Gly Tyr Val
980 985 990
Leu His Leu His Arg Asp Ile Glu Glu Thr Ile Val Phe Leu Gln Ser
995 1000 1005
Asp Pro Ser Arg Pro Ala Tyr Val Glu Glu Ile Arg Glu Val Trp Asn
1010 1015 1020
Arg Arg Glu Gly Trp Tyr Lys Glu Cys Ser Asn Phe Ser Phe Phe Ala
1025 1030 1035 1040
Pro His Cys Ser Ala Glu Ala Glu Phe Gln Ala Leu Arg Arg Ser Phe
1045 1050 1055
Ser Lys Tyr Ile Ala Thr Ile Thr Gly Val Arg Glu Ile Glu Ile Pro
1060 1065 1070
Ser Gly Arg Ser Ala Phe Val Cys Leu Thr Phe Asp Asp Leu Thr Glu
1075 1080 1085
Gln Thr Glu Asn Leu Thr Pro Ile Cys Tyr Gly Cys Glu Ala Val Glu
1090 1095 1100
Val Arg Val Asp His Leu Ala Asn Tyr Ser Ala Asp Phe Val Ser Lys
1105 1110 1115 1120
Gln Leu Ser Ile Leu Arg Lys Ala Thr Asp Ser Ile Pro Ile Ile Phe
1125 1130 1135
Thr Val Arg Thr Met Lys Gln Gly Gly Asn Phe Pro Asp Glu Glu Phe
1140 1145 1150
Lys Thr Leu Arg Glu Leu Tyr Asp Ile Ala Leu Lys Asn Gly Val Glu
1155 1160 1165
Phe Leu Asp Leu Glu Leu Thr Leu Pro Thr Asp Ile Gln Tyr Glu Val
1170 1175 1180
Ile Asn Lys Arg Gly Asn Thr Lys Ile Ile Gly Ser His His Asp Phe
1185 1190 1195 1200
Gln Gly Leu Tyr Ser Trp Asp Asp Ala Glu Trp Glu Asn Arg Phe Asn
1205 1210 1215
Gln Ala Leu Thr Leu Asp Val Asp Val Val Lys Phe Val Gly Thr Ala
1220 1225 1230
Val Asn Phe Glu Asp Asn Leu Arg Leu Glu His Phe Arg Asp Thr His
1235 1240 1245
Lys Asn Lys Pro Leu Ile Ala Val Asn Met Thr Ser Lys Gly Ser Ile
1250 1255 1260
Ser Arg Val Leu Asn Asn Val Leu Thr Pro Val Thr Ser Asp Leu Leu
1265 1270 1275 1280
Pro Asn Ser Ala Ala Pro Gly Gln Leu Thr Val Ala Gln Ile Asn Lys
1285 1290 1295
Met Tyr Thr Ser Met Gly Gly Ile Glu Pro Lys Glu Leu Phe Val Val
1300 1305 1310
Gly Lys Pro Ile Gly His Ser Arg Ser Pro Ile Leu His Asn Thr Gly
1315 1320 1325
Tyr Glu Ile Leu Gly Leu Pro His Lys Phe Asp Lys Phe Glu Thr Glu
1330 1335 1340
Ser Ala Gln Leu Val Lys Glu Lys Leu Leu Asp Gly Asn Lys Asn Phe
1345 1350 1355 1360
Gly Gly Ala Ala Val Thr Ile Pro Leu Lys Leu Asp Ile Met Gln Tyr
1365 1370 1375
Met Asp Glu Leu Thr Asp Ala Ala Lys Val Ile Gly Ala Val Asn Thr
1380 1385 1390
Val Ile Pro Leu Gly Asn Lys Lys Phe Lys Gly Asp Asn Thr Asp Trp
1395 1400 1405
Leu Gly Ile Arg Asn Ala Leu Ile Asn Asn Gly Val Pro Glu Tyr Val
1410 1415 1420
Gly His Thr Ala Gly Leu Val Ile Gly Ala Gly Gly Thr Ser Arg Ala
1425 1430 1435 1440
Ala Leu Tyr Ala Leu His Ser Leu Gly Cys Lys Lys Ile Phe Ile Ile
1445 1450 1455
Asn Arg Thr Thr Ser Lys Leu Lys Pro Leu Ile Glu Ser Leu Pro Ser
1460 1465 1470
Glu Phe Asn Ile Ile Gly Ile Glu Ser Thr Lys Ser Ile Glu Glu Ile
1475 1480 1485
Lys Glu His Val Gly Val Ala Val Ser Cys Val Pro Ala Asp Lys Pro
1490 1495 1500
Leu Asp Asp Glu Leu Leu Ser Lys Leu Glu Arg Phe Leu Val Lys Gly
1505 1510 1515 1520
Ala His Ala Ala Phe Val Pro Thr Leu Leu Glu Ala Ala Tyr Lys Pro
1525 1530 1535
Ser Val Thr Pro Val Met Thr Ile Ser Gln Asp Lys Tyr Gln Trp His
1540 1545 1550
Val Val Pro Gly Ser Gln Met Leu Val His Gln Gly Val Ala Gln Phe
1555 1560 1565
Glu Lys Trp Thr Gly Phe Lys Gly Pro Phe Lys Ala Ile Phe Asp Ala
1570 1575 1580
Val Thr Lys Glu
1585
<210> 113
<211> 820
<212> PRT
<213> Anabaena variabilis ATCC29413
<400> 113
Met Ser Ile Val Gln Ala Lys Phe Glu Ala Lys Glu Thr Ser Phe His
1 5 10 15
Val Glu Gly Tyr Glu Lys Ile Glu Tyr Asp Leu Val Tyr Val Asp Gly
20 25 30
Ile Phe Glu Ile Gln Asn Ser Ala Leu Ala Asp Val Tyr Gln Gly Phe
35 40 45
Gly Arg Cys Leu Ala Ile Val Asp Ala Asn Val Ser Arg Leu Tyr Gly
50 55 60
Asn Gln Ile Gln Ala Tyr Phe Gln Tyr Tyr Gly Ile Glu Leu Arg Leu
65 70 75 80
Phe Pro Ile Thr Ile Thr Glu Pro Asp Lys Thr Ile Gln Thr Phe Glu
85 90 95
Arg Val Ile Asp Val Phe Ala Asp Phe Lys Leu Val Arg Lys Glu Pro
100 105 110
Val Leu Val Val Gly Gly Gly Leu Ile Thr Asp Val Val Gly Phe Ala
115 120 125
Cys Ser Thr Tyr Arg Arg Ser Ser Asn Tyr Ile Arg Ile Pro Thr Thr
130 135 140
Leu Ile Gly Leu Ile Asp Ala Ser Val Ala Ile Lys Val Ala Val Asn
145 150 155 160
His Arg Lys Leu Lys Asn Arg Leu Gly Ala Tyr His Ala Ser Arg Lys
165 170 175
Val Phe Leu Asp Phe Ser Leu Leu Arg Thr Leu Pro Thr Asp Gln Val
180 185 190
Arg Asn Gly Met Ala Glu Leu Val Lys Ile Ala Val Val Ala His Gln
195 200 205
Glu Val Phe Glu Leu Leu Glu Lys Tyr Gly Glu Glu Leu Leu Arg Thr
210 215 220
His Phe Gly Asn Ile Asp Ala Thr Pro Glu Ile Lys Glu Ile Ala His
225 230 235 240
Arg Leu Thr Tyr Lys Ala Ile His Lys Met Leu Glu Leu Glu Val Pro
245 250 255
Asn Leu His Glu Leu Asp Leu Asp Arg Val Ile Ala Tyr Gly His Thr
260 265 270
Trp Ser Pro Thr Leu Glu Leu Ala Pro Arg Leu Pro Met Phe His Gly
275 280 285
His Ala Val Asn Val Asp Met Ala Phe Ser Ala Thr Ile Ala Ala Arg
290 295 300
Arg Gly Tyr Ile Thr Ile Ala Glu Arg Asp Arg Ile Leu Gly Leu Met
305 310 315 320
Ser Arg Val Gly Leu Ser Leu Asp His Pro Met Leu Asp Ile Asp Ile
325 330 335
Leu Trp Arg Gly Thr Glu Ser Ile Thr Leu Thr Arg Asp Gly Leu Leu
340 345 350
Arg Ala Ala Met Pro Lys Pro Ile Gly Asp Cys Val Phe Val Asn Asp
355 360 365
Leu Thr Arg Glu Glu Leu Ala Ala Ala Leu Ala Asp His Lys Glu Leu
370 375 380
Cys Thr Ser Tyr Pro Arg Gly Gly Glu Gly Val Asp Val Tyr Pro Val
385 390 395 400
Tyr Gln Lys Glu Leu Ile Gly Ser Val Lys Met Ser Ile Val Gln Ala
405 410 415
Lys Phe Glu Ala Lys Glu Thr Ser Phe His Val Glu Gly Tyr Glu Lys
420 425 430
Ile Glu Tyr Asp Leu Val Tyr Val Asp Gly Ile Phe Glu Ile Gln Asn
435 440 445
Ser Ala Leu Ala Asp Val Tyr Gln Gly Phe Gly Arg Cys Leu Ala Ile
450 455 460
Val Asp Ala Asn Val Ser Arg Leu Tyr Gly Asn Gln Ile Gln Ala Tyr
465 470 475 480
Phe Gln Tyr Tyr Gly Ile Glu Leu Arg Leu Phe Pro Ile Thr Ile Thr
485 490 495
Glu Pro Asp Lys Thr Ile Gln Thr Phe Glu Arg Val Ile Asp Val Phe
500 505 510
Ala Asp Phe Lys Leu Val Arg Lys Glu Pro Val Leu Val Val Gly Gly
515 520 525
Gly Leu Ile Thr Asp Val Val Gly Phe Ala Cys Ser Thr Tyr Arg Arg
530 535 540
Ser Ser Asn Tyr Ile Arg Ile Pro Thr Thr Leu Ile Gly Leu Ile Asp
545 550 555 560
Ala Ser Val Ala Ile Lys Val Ala Val Asn His Arg Lys Leu Lys Asn
565 570 575
Arg Leu Gly Ala Tyr His Ala Ser Arg Lys Val Phe Leu Asp Phe Ser
580 585 590
Leu Leu Arg Thr Leu Pro Thr Asp Gln Val Arg Asn Gly Met Ala Glu
595 600 605
Leu Val Lys Ile Ala Val Val Ala His Gln Glu Val Phe Glu Leu Leu
610 615 620
Glu Lys Tyr Gly Glu Glu Leu Leu Arg Thr His Phe Gly Asn Ile Asp
625 630 635 640
Ala Thr Pro Glu Ile Lys Glu Ile Ala His Arg Leu Thr Tyr Lys Ala
645 650 655
Ile His Lys Met Leu Glu Leu Glu Val Pro Asn Leu His Glu Leu Asp
660 665 670
Leu Asp Arg Val Ile Ala Tyr Gly His Thr Trp Ser Pro Thr Leu Glu
675 680 685
Leu Ala Pro Arg Leu Pro Met Phe His Gly His Ala Val Asn Val Asp
690 695 700
Met Ala Phe Ser Ala Thr Ile Ala Ala Arg Arg Gly Tyr Ile Thr Ile
705 710 715 720
Ala Glu Arg Asp Arg Ile Leu Gly Leu Met Ser Arg Val Gly Leu Ser
725 730 735
Leu Asp His Pro Met Leu Asp Ile Asp Ile Leu Trp Arg Gly Thr Glu
740 745 750
Ser Ile Thr Leu Thr Arg Asp Gly Leu Leu Arg Ala Ala Met Pro Lys
755 760 765
Pro Ile Gly Asp Cys Val Phe Val Asn Asp Leu Thr Arg Glu Glu Leu
770 775 780
Ala Ala Ala Leu Ala Asp His Lys Glu Leu Cys Thr Ser Tyr Pro Arg
785 790 795 800
Gly Gly Glu Gly Val Asp Val Tyr Pro Val Tyr Gln Lys Glu Leu Ile
805 810 815
Gly Ser Val Lys
820
<210> 114
<211> 1099
<212> PRT
<213> Anabaena variabilis ATCC29413
<400> 114
Met Ser Ile Val Gln Ala Lys Phe Glu Ala Lys Glu Thr Ser Phe His
1 5 10 15
Val Glu Gly Tyr Glu Lys Ile Glu Tyr Asp Leu Val Tyr Val Asp Gly
20 25 30
Ile Phe Glu Ile Gln Asn Ser Ala Leu Ala Asp Val Tyr Gln Gly Phe
35 40 45
Gly Arg Cys Leu Ala Ile Val Asp Ala Asn Val Ser Arg Leu Tyr Gly
50 55 60
Asn Gln Ile Gln Ala Tyr Phe Gln Tyr Tyr Gly Ile Glu Leu Arg Leu
65 70 75 80
Phe Pro Ile Thr Ile Thr Glu Pro Asp Lys Thr Ile Gln Thr Phe Glu
85 90 95
Arg Val Ile Asp Val Phe Ala Asp Phe Lys Leu Val Arg Lys Glu Pro
100 105 110
Val Leu Val Val Gly Gly Gly Leu Ile Thr Asp Val Val Gly Phe Ala
115 120 125
Cys Ser Thr Tyr Arg Arg Ser Ser Asn Tyr Ile Arg Ile Pro Thr Thr
130 135 140
Leu Ile Gly Leu Ile Asp Ala Ser Val Ala Ile Lys Val Ala Val Asn
145 150 155 160
His Arg Lys Leu Lys Asn Arg Leu Gly Ala Tyr His Ala Ser Arg Lys
165 170 175
Val Phe Leu Asp Phe Ser Leu Leu Arg Thr Leu Pro Thr Asp Gln Val
180 185 190
Arg Asn Gly Met Ala Glu Leu Val Lys Ile Ala Val Val Ala His Gln
195 200 205
Glu Val Phe Glu Leu Leu Glu Lys Tyr Gly Glu Glu Leu Leu Arg Thr
210 215 220
His Phe Gly Asn Ile Asp Ala Thr Pro Glu Ile Lys Glu Ile Ala His
225 230 235 240
Arg Leu Thr Tyr Lys Ala Ile His Lys Met Leu Glu Leu Glu Val Pro
245 250 255
Asn Leu His Glu Leu Asp Leu Asp Arg Val Ile Ala Tyr Gly His Thr
260 265 270
Trp Ser Pro Thr Leu Glu Leu Ala Pro Arg Leu Pro Met Phe His Gly
275 280 285
His Ala Val Asn Val Asp Met Ala Phe Ser Ala Thr Ile Ala Ala Arg
290 295 300
Arg Gly Tyr Ile Thr Ile Ala Glu Arg Asp Arg Ile Leu Gly Leu Met
305 310 315 320
Ser Arg Val Gly Leu Ser Leu Asp His Pro Met Leu Asp Ile Asp Ile
325 330 335
Leu Trp Arg Gly Thr Glu Ser Ile Thr Leu Thr Arg Asp Gly Leu Leu
340 345 350
Arg Ala Ala Met Pro Lys Pro Ile Gly Asp Cys Val Phe Val Asn Asp
355 360 365
Leu Thr Arg Glu Glu Leu Ala Ala Ala Leu Ala Asp His Lys Glu Leu
370 375 380
Cys Thr Ser Tyr Pro Arg Gly Gly Glu Gly Val Asp Val Tyr Pro Val
385 390 395 400
Tyr Gln Lys Glu Leu Ile Gly Ser Val Lys Met Ser Ile Val Gln Ala
405 410 415
Lys Phe Glu Ala Lys Glu Thr Ser Phe His Val Glu Gly Tyr Glu Lys
420 425 430
Ile Glu Tyr Asp Leu Val Tyr Val Asp Gly Ile Phe Glu Ile Gln Asn
435 440 445
Ser Ala Leu Ala Asp Val Tyr Gln Gly Phe Gly Arg Cys Leu Ala Ile
450 455 460
Val Asp Ala Asn Val Ser Arg Leu Tyr Gly Asn Gln Ile Gln Ala Tyr
465 470 475 480
Phe Gln Tyr Tyr Gly Ile Glu Leu Arg Leu Phe Pro Ile Thr Ile Thr
485 490 495
Glu Pro Asp Lys Thr Ile Gln Thr Phe Glu Arg Val Ile Asp Val Phe
500 505 510
Ala Asp Phe Lys Leu Val Arg Lys Glu Pro Val Leu Val Val Gly Gly
515 520 525
Gly Leu Ile Thr Asp Val Val Gly Phe Ala Cys Ser Thr Tyr Arg Arg
530 535 540
Ser Ser Asn Tyr Ile Arg Ile Pro Thr Thr Leu Ile Gly Leu Ile Asp
545 550 555 560
Ala Ser Val Ala Ile Lys Val Ala Val Asn His Arg Lys Leu Lys Asn
565 570 575
Arg Leu Gly Ala Tyr His Ala Ser Arg Lys Val Phe Leu Asp Phe Ser
580 585 590
Leu Leu Arg Thr Leu Pro Thr Asp Gln Val Arg Asn Gly Met Ala Glu
595 600 605
Leu Val Lys Ile Ala Val Val Ala His Gln Glu Val Phe Glu Leu Leu
610 615 620
Glu Lys Tyr Gly Glu Glu Leu Leu Arg Thr His Phe Gly Asn Ile Asp
625 630 635 640
Ala Thr Pro Glu Ile Lys Glu Ile Ala His Arg Leu Thr Tyr Lys Ala
645 650 655
Ile His Lys Met Leu Glu Leu Glu Val Pro Asn Leu His Glu Leu Asp
660 665 670
Leu Asp Arg Val Ile Ala Tyr Gly His Thr Trp Ser Pro Thr Leu Glu
675 680 685
Leu Ala Pro Arg Leu Pro Met Phe His Gly His Ala Val Asn Val Asp
690 695 700
Met Ala Phe Ser Ala Thr Ile Ala Ala Arg Arg Gly Tyr Ile Thr Ile
705 710 715 720
Ala Glu Arg Asp Arg Ile Leu Gly Leu Met Ser Arg Val Gly Leu Ser
725 730 735
Leu Asp His Pro Met Leu Asp Ile Asp Ile Leu Trp Arg Gly Thr Glu
740 745 750
Ser Ile Thr Leu Thr Arg Asp Gly Leu Leu Arg Ala Ala Met Pro Lys
755 760 765
Pro Ile Gly Asp Cys Val Phe Val Asn Asp Leu Thr Arg Glu Glu Leu
770 775 780
Ala Ala Ala Leu Ala Asp His Lys Glu Leu Cys Thr Ser Tyr Pro Arg
785 790 795 800
Gly Gly Glu Gly Val Asp Val Tyr Pro Val Tyr Gln Lys Glu Leu Ile
805 810 815
Gly Ser Val Lys Met Thr Asn Val Ile Val Gln Pro Thr Ala Arg Pro
820 825 830
Val Thr Pro Leu Gly Ile Leu Thr Lys Gln Leu Glu Ala Ile Val Gln
835 840 845
Glu Val Lys Gln His Pro Asp Leu Pro Gly Glu Leu Ile Ala Asn Ile
850 855 860
His Gln Ala Trp Arg Leu Ala Ala Gly Ile Asp Pro Tyr Leu Glu Glu
865 870 875 880
Cys Thr Thr Pro Glu Ser Pro Glu Leu Ala Ala Leu Ala Lys Thr Thr
885 890 895
Ala Thr Glu Ala Trp Gly Glu His Phe His Gly Gly Thr Thr Val Arg
900 905 910
Pro Leu Glu Gln Glu Met Leu Ser Gly His Ile Glu Gly Gln Thr Leu
915 920 925
Lys Met Phe Val His Met Thr Lys Ala Lys Lys Val Leu Glu Ile Gly
930 935 940
Met Phe Thr Gly Tyr Ser Ala Leu Ala Met Ala Glu Ala Leu Pro Glu
945 950 955 960
Asp Gly Leu Leu Val Ala Cys Glu Val Asp Pro Tyr Ala Ala Glu Ile
965 970 975
Gly Gln Lys Ala Phe Gln Gln Ser Pro His Gly Gly Lys Ile Arg Val
980 985 990
Glu Leu Asp Ala Ala Leu Ala Thr Leu Asp Lys Leu Ala Glu Ala Gly
995 1000 1005
Glu Ser Phe Asp Leu Val Phe Ile Asp Ala Asp Lys Lys Glu Tyr Val
1010 1015 1020
Ala Tyr Phe His Lys Leu Leu Gly Ser Ser Leu Leu Ala Pro Asp Gly
1025 1030 1035 1040
Phe Ile Cys Val Asp Asn Thr Leu Leu Gln Gly Glu Val Tyr Leu Pro
1045 1050 1055
Ala Glu Glu Arg Ser Val Asn Gly Glu Ala Ile Ala Gln Phe Asn His
1060 1065 1070
Thr Val Ala Ile Asp Pro Arg Val Glu Gln Val Leu Leu Pro Leu Arg
1075 1080 1085
Asp Gly Leu Thr Ile Ile Arg Arg Ile Gln Pro
1090 1095
<210> 115
<211> 410
<212> PRT
<213> Anabaena variabilis ATCC29413
<400> 115
Met Ser Ile Val Gln Ala Lys Phe Glu Ala Lys Glu Thr Ser Phe His
1 5 10 15
Val Glu Gly Tyr Glu Lys Ile Glu Tyr Asp Leu Val Tyr Val Asp Gly
20 25 30
Ile Phe Glu Ile Gln Asn Ser Ala Leu Ala Asp Val Tyr Gln Gly Phe
35 40 45
Gly Arg Cys Leu Ala Ile Val Asp Ala Asn Val Ser Arg Leu Tyr Gly
50 55 60
Asn Gln Ile Gln Ala Tyr Phe Gln Tyr Tyr Gly Ile Glu Leu Arg Leu
65 70 75 80
Phe Pro Ile Thr Ile Thr Glu Pro Asp Lys Thr Ile Gln Thr Phe Glu
85 90 95
Arg Val Ile Asp Val Phe Ala Asp Phe Lys Leu Val Arg Lys Glu Pro
100 105 110
Val Leu Val Val Gly Gly Gly Leu Ile Thr Asp Val Val Gly Phe Ala
115 120 125
Cys Ser Thr Tyr Arg Arg Ser Ser Asn Tyr Ile Arg Ile Pro Thr Thr
130 135 140
Leu Ile Gly Leu Ile Asp Ala Ser Val Ala Ile Lys Val Ala Val Asn
145 150 155 160
His Arg Lys Leu Lys Asn Arg Leu Gly Ala Tyr His Ala Ser Arg Lys
165 170 175
Val Phe Leu Asp Phe Ser Leu Leu Arg Thr Leu Pro Thr Asp Gln Val
180 185 190
Arg Asn Gly Met Ala Glu Leu Val Lys Ile Ala Val Val Ala His Gln
195 200 205
Glu Val Phe Glu Leu Leu Glu Lys Tyr Gly Glu Glu Leu Leu Arg Thr
210 215 220
His Phe Gly Asn Ile Asp Ala Thr Pro Glu Ile Lys Glu Ile Ala His
225 230 235 240
Arg Leu Thr Tyr Lys Ala Ile His Lys Met Leu Glu Leu Glu Val Pro
245 250 255
Asn Leu His Glu Leu Asp Leu Asp Arg Val Ile Ala Tyr Gly His Thr
260 265 270
Trp Ser Pro Thr Leu Glu Leu Ala Pro Arg Leu Pro Met Phe His Gly
275 280 285
His Ala Val Asn Val Asp Met Ala Phe Ser Ala Thr Ile Ala Ala Arg
290 295 300
Arg Gly Tyr Ile Thr Ile Ala Glu Arg Asp Arg Ile Leu Gly Leu Met
305 310 315 320
Ser Arg Val Gly Leu Ser Leu Asp His Pro Met Leu Asp Ile Asp Ile
325 330 335
Leu Trp Arg Gly Thr Glu Ser Ile Thr Leu Thr Arg Asp Gly Leu Leu
340 345 350
Arg Ala Ala Met Pro Lys Pro Ile Gly Asp Cys Val Phe Val Asn Asp
355 360 365
Leu Thr Arg Glu Glu Leu Ala Ala Ala Leu Ala Asp His Lys Glu Leu
370 375 380
Cys Thr Ser Tyr Pro Arg Gly Gly Glu Gly Val Asp Val Tyr Pro Val
385 390 395 400
Tyr Gln Lys Glu Leu Ile Gly Ser Val Lys
405 410
<210> 116
<211> 410
<212> PRT
<213> Anabaena variabilis ATCC29413
<400> 116
Met Ser Ile Val Gln Ala Lys Phe Glu Ala Lys Glu Thr Ser Phe His
1 5 10 15
Val Glu Gly Tyr Glu Lys Ile Glu Tyr Asp Leu Val Tyr Val Asp Gly
20 25 30
Ile Phe Glu Ile Gln Asn Ser Ala Leu Ala Asp Val Tyr Gln Gly Phe
35 40 45
Gly Arg Cys Leu Ala Ile Val Asp Ala Asn Val Ser Arg Leu Tyr Gly
50 55 60
Asn Gln Ile Gln Ala Tyr Phe Gln Tyr Tyr Gly Ile Glu Leu Arg Leu
65 70 75 80
Phe Pro Ile Thr Ile Thr Glu Pro Asp Lys Thr Ile Gln Thr Phe Glu
85 90 95
Arg Val Ile Asp Val Phe Ala Asp Phe Lys Leu Val Arg Lys Glu Pro
100 105 110
Val Leu Val Val Gly Gly Gly Leu Ile Thr Asp Val Val Gly Phe Ala
115 120 125
Cys Ser Thr Tyr Arg Arg Ser Ser Asn Tyr Ile Arg Ile Pro Thr Thr
130 135 140
Leu Ile Gly Leu Ile Asp Ala Ser Val Ala Ile Lys Val Ala Val Asn
145 150 155 160
His Arg Lys Leu Lys Asn Arg Leu Gly Ala Tyr His Ala Ser Arg Lys
165 170 175
Val Phe Leu Asp Phe Ser Leu Leu Arg Thr Leu Pro Thr Asp Gln Val
180 185 190
Arg Asn Gly Met Ala Glu Leu Val Lys Ile Ala Val Val Ala His Gln
195 200 205
Glu Val Phe Glu Leu Leu Glu Lys Tyr Gly Glu Glu Leu Leu Arg Thr
210 215 220
His Phe Gly Asn Ile Asp Ala Thr Pro Glu Ile Lys Glu Ile Ala His
225 230 235 240
Arg Leu Thr Tyr Lys Ala Ile His Lys Met Leu Glu Leu Glu Val Pro
245 250 255
Asn Leu His Glu Leu Asp Leu Asp Arg Val Ile Ala Tyr Gly His Thr
260 265 270
Trp Ser Pro Thr Leu Glu Leu Ala Pro Arg Leu Pro Met Phe His Gly
275 280 285
His Ala Val Asn Val Asp Met Ala Phe Ser Ala Thr Ile Ala Ala Arg
290 295 300
Arg Gly Tyr Ile Thr Ile Ala Glu Arg Asp Arg Ile Leu Gly Leu Met
305 310 315 320
Ser Arg Val Gly Leu Ser Leu Asp His Pro Met Leu Asp Ile Asp Ile
325 330 335
Leu Trp Arg Gly Thr Glu Ser Ile Thr Leu Thr Arg Asp Gly Leu Leu
340 345 350
Arg Ala Ala Met Pro Lys Pro Ile Gly Asp Cys Val Phe Val Asn Asp
355 360 365
Leu Thr Arg Glu Glu Leu Ala Ala Ala Leu Ala Asp His Lys Glu Leu
370 375 380
Cys Thr Ser Tyr Pro Arg Gly Gly Glu Gly Val Asp Val Tyr Pro Val
385 390 395 400
Tyr Gln Lys Glu Leu Ile Gly Ser Val Lys
405 410
<210> 117
<211> 279
<212> PRT
<213> Anabaena variabilis ATCC29413
<400> 117
Met Thr Asn Val Ile Val Gln Pro Thr Ala Arg Pro Val Thr Pro Leu
1 5 10 15
Gly Ile Leu Thr Lys Gln Leu Glu Ala Ile Val Gln Glu Val Lys Gln
20 25 30
His Pro Asp Leu Pro Gly Glu Leu Ile Ala Asn Ile His Gln Ala Trp
35 40 45
Arg Leu Ala Ala Gly Ile Asp Pro Tyr Leu Glu Glu Cys Thr Thr Pro
50 55 60
Glu Ser Pro Glu Leu Ala Ala Leu Ala Lys Thr Thr Ala Thr Glu Ala
65 70 75 80
Trp Gly Glu His Phe His Gly Gly Thr Thr Val Arg Pro Leu Glu Gln
85 90 95
Glu Met Leu Ser Gly His Ile Glu Gly Gln Thr Leu Lys Met Phe Val
100 105 110
His Met Thr Lys Ala Lys Lys Val Leu Glu Ile Gly Met Phe Thr Gly
115 120 125
Tyr Ser Ala Leu Ala Met Ala Glu Ala Leu Pro Glu Asp Gly Leu Leu
130 135 140
Val Ala Cys Glu Val Asp Pro Tyr Ala Ala Glu Ile Gly Gln Lys Ala
145 150 155 160
Phe Gln Gln Ser Pro His Gly Gly Lys Ile Arg Val Glu Leu Asp Ala
165 170 175
Ala Leu Ala Thr Leu Asp Lys Leu Ala Glu Ala Gly Glu Ser Phe Asp
180 185 190
Leu Val Phe Ile Asp Ala Asp Lys Lys Glu Tyr Val Ala Tyr Phe His
195 200 205
Lys Leu Leu Gly Ser Ser Leu Leu Ala Pro Asp Gly Phe Ile Cys Val
210 215 220
Asp Asn Thr Leu Leu Gln Gly Glu Val Tyr Leu Pro Ala Glu Glu Arg
225 230 235 240
Ser Val Asn Gly Glu Ala Ile Ala Gln Phe Asn His Thr Val Ala Ile
245 250 255
Asp Pro Arg Val Glu Gln Val Leu Leu Pro Leu Arg Asp Gly Leu Thr
260 265 270
Ile Ile Arg Arg Ile Gln Pro
275
<210> 118
<211> 888
<212> PRT
<213> Anabaena variabilis ATCC29413
<400> 118
Met Gln Thr Ile Asp Phe Asn Ile Arg Lys Leu Leu Val Glu Trp Asn
1 5 10 15
Ala Thr His Arg Asp Tyr Asp Leu Ser Gln Ser Leu His Glu Leu Ile
20 25 30
Val Ala Gln Val Glu Arg Thr Pro Glu Ala Ile Ala Val Thr Phe Asp
35 40 45
Lys Gln Gln Leu Thr Tyr Gln Glu Leu Asn His Lys Ala Asn Gln Leu
50 55 60
Gly His Tyr Leu Gln Thr Leu Gly Val Gln Pro Glu Thr Leu Val Gly
65 70 75 80
Val Cys Leu Glu Arg Ser Leu Glu Met Val Ile Cys Leu Leu Gly Ile
85 90 95
Leu Lys Ala Gly Gly Ala Tyr Val Pro Ile Asp Pro Glu Tyr Pro Gln
100 105 110
Glu Arg Ile Ala Tyr Met Leu Glu Asp Ser Gln Val Lys Val Leu Leu
115 120 125
Thr Gln Glu Lys Leu Leu Asn Gln Ile Pro His His Gln Ala Gln Thr
130 135 140
Ile Cys Val Asp Arg Glu Trp Glu Lys Ile Ser Thr Gln Ala Asn Thr
145 150 155 160
Asn Pro Lys Ser Asn Ile Lys Thr Asp Asn Leu Ala Tyr Val Ile Tyr
165 170 175
Thr Ser Gly Ser Thr Gly Lys Pro Lys Gly Ala Met Asn Thr His Lys
180 185 190
Gly Ile Cys Asn Arg Leu Leu Trp Met Gln Glu Ala Tyr Gln Ile Asp
195 200 205
Ser Thr Asp Ser Ile Leu Gln Lys Thr Pro Phe Ser Phe Asp Val Ser
210 215 220
Val Trp Glu Phe Phe Trp Thr Leu Leu Thr Gly Ala Arg Leu Val Ile
225 230 235 240
Ala Lys Pro Gly Gly His Lys Asp Ser Ala Tyr Leu Ile Asp Leu Ile
245 250 255
Thr Gln Glu Gln Ile Thr Thr Leu His Phe Val Pro Ser Met Leu Gln
260 265 270
Val Phe Leu Gln Asn Arg His Val Ser Lys Cys Ser Ser Leu Lys Arg
275 280 285
Val Ile Cys Ser Gly Glu Ala Leu Ser Ile Asp Leu Gln Asn Arg Phe
290 295 300
Phe Gln His Leu Gln Cys Glu Leu His Asn Leu Tyr Gly Pro Thr Glu
305 310 315 320
Ala Ala Ile Asp Val Thr Phe Trp Gln Cys Arg Lys Asp Ser Asn Leu
325 330 335
Lys Ser Val Pro Ile Gly Arg Pro Ile Ala Asn Thr Gln Ile Tyr Ile
340 345 350
Leu Asp Ala Asp Leu Gln Pro Val Asn Ile Gly Val Thr Gly Glu Ile
355 360 365
Tyr Ile Gly Gly Val Gly Val Ala Arg Gly Tyr Leu Asn Lys Glu Glu
370 375 380
Leu Thr Lys Glu Lys Phe Ile Ile Asn Pro Phe Pro Asn Ser Glu Phe
385 390 395 400
Lys Arg Leu Tyr Lys Thr Gly Asp Leu Ala Arg Tyr Leu Pro Asp Gly
405 410 415
Asn Ile Glu Tyr Leu Gly Arg Thr Asp Tyr Gln Val Lys Ile Arg Gly
420 425 430
Tyr Arg Ile Glu Ile Gly Glu Ile Glu Asn Val Leu Ser Ser His Pro
435 440 445
Gln Val Arg Glu Ala Val Val Ile Ala Arg Asp Asp Asn Ala Gln Glu
450 455 460
Lys Gln Ile Ile Ala Tyr Ile Thr Tyr Asn Ser Ile Lys Pro Gln Leu
465 470 475 480
Asp Asn Leu Arg Asp Phe Leu Lys Ala Arg Leu Pro Asp Phe Met Ile
485 490 495
Pro Ala Ala Phe Val Met Leu Glu His Leu Pro Leu Thr Pro Ser Gly
500 505 510
Lys Val Asp Arg Lys Ala Leu Pro Lys Pro Asp Leu Phe Asn Tyr Ser
515 520 525
Glu His Asn Ser Tyr Val Ala Pro Arg Asn Glu Val Glu Glu Lys Leu
530 535 540
Val Gln Ile Trp Ser Asn Ile Leu His Leu Pro Lys Val Gly Val Thr
545 550 555 560
Glu Asn Phe Phe Ala Ile Gly Gly Asn Ser Leu Lys Ala Leu His Leu
565 570 575
Ile Ser Gln Ile Glu Glu Leu Phe Ala Lys Glu Ile Ser Leu Ala Thr
580 585 590
Leu Leu Thr Asn Pro Val Ile Ala Asp Leu Ala Lys Val Ile Gln Ala
595 600 605
Asn Asn Gln Ile His Asn Ser Pro Leu Val Pro Ile Gln Pro Gln Gly
610 615 620
Lys Gln Gln Pro Phe Phe Cys Ile His Pro Ala Gly Gly His Val Leu
625 630 635 640
Cys Tyr Phe Lys Leu Ala Gln Tyr Ile Gly Thr Asp Gln Pro Phe Tyr
645 650 655
Gly Leu Gln Ala Gln Gly Phe Tyr Gly Asp Glu Ala Pro Leu Thr Arg
660 665 670
Val Glu Asp Met Ala Ser Leu Tyr Val Lys Thr Ile Arg Glu Phe Gln
675 680 685
Pro Gln Gly Pro Tyr Arg Val Gly Gly Trp Ser Phe Gly Gly Val Val
690 695 700
Ala Tyr Glu Val Ala Gln Gln Leu His Arg Gln Gly Gln Glu Val Ser
705 710 715 720
Leu Leu Ala Ile Leu Asp Ser Tyr Val Pro Ile Leu Leu Asp Lys Gln
725 730 735
Lys Pro Ile Asp Asp Val Tyr Leu Val Gly Val Leu Ser Arg Val Phe
740 745 750
Gly Gly Met Phe Gly Gln Asp Asn Leu Val Thr Pro Glu Glu Ile Glu
755 760 765
Asn Leu Thr Val Glu Glu Lys Ile Asn Tyr Ile Ile Asp Lys Ala Arg
770 775 780
Ser Ala Arg Ile Phe Pro Pro Gly Val Glu Arg Gln Asn Asn Arg Arg
785 790 795 800
Ile Leu Asp Val Leu Val Gly Thr Leu Lys Ala Thr Tyr Ser Tyr Ile
805 810 815
Arg Gln Pro Tyr Pro Gly Lys Val Thr Val Phe Arg Ala Arg Glu Lys
820 825 830
His Ile Met Ala Pro Asp Pro Thr Leu Val Trp Val Glu Leu Phe Ser
835 840 845
Val Met Ala Ala Gln Glu Ile Lys Ile Ile Asp Val Pro Gly Asn His
850 855 860
Tyr Ser Phe Val Leu Glu Pro His Val Gln Val Leu Ala Gln Arg Leu
865 870 875 880
Gln Asp Cys Leu Glu Asn Asn Ser
885
<210> 119
<211> 410
<212> PRT
<213> Nostoc punctiforme ATCC 29133
<400> 119
Met Ser Asn Val Gln Ala Ser Phe Glu Ala Thr Glu Ala Glu Phe Arg
1 5 10 15
Val Glu Gly Tyr Glu Lys Ile Glu Phe Ser Leu Val Tyr Val Asn Gly
20 25 30
Ala Phe Asp Ile Ser Asn Arg Glu Ile Ala Asp Ser Tyr Glu Lys Phe
35 40 45
Gly Arg Cys Leu Thr Val Ile Asp Ala Asn Val Asn Arg Leu Tyr Gly
50 55 60
Lys Gln Ile Lys Ser Tyr Phe Arg His Tyr Gly Ile Asp Leu Thr Val
65 70 75 80
Val Pro Ile Val Ile Thr Glu Pro Thr Lys Thr Leu Ala Thr Phe Glu
85 90 95
Lys Ile Val Asp Ala Phe Ser Asp Phe Gly Leu Ile Arg Lys Glu Pro
100 105 110
Val Leu Val Val Gly Gly Gly Leu Thr Thr Asp Val Ala Gly Phe Ala
115 120 125
Cys Ala Ala Tyr Arg Arg Lys Ser Asn Tyr Ile Arg Val Pro Thr Thr
130 135 140
Leu Ile Gly Leu Ile Asp Ala Gly Val Ala Ile Lys Val Ala Val Asn
145 150 155 160
His Arg Lys Leu Lys Asn Arg Leu Gly Ala Tyr His Ala Pro Leu Lys
165 170 175
Val Ile Leu Asp Phe Ser Phe Leu Gln Thr Leu Pro Thr Ala Gln Val
180 185 190
Arg Asn Gly Met Ala Glu Leu Val Lys Ile Ala Val Val Ala Asn Ser
195 200 205
Glu Val Phe Glu Leu Leu Tyr Glu Tyr Gly Glu Glu Leu Leu Ser Thr
210 215 220
His Phe Gly Tyr Val Asn Gly Thr Lys Glu Leu Lys Ala Ile Ala His
225 230 235 240
Lys Leu Asn Tyr Glu Ala Ile Lys Thr Met Leu Glu Leu Glu Thr Pro
245 250 255
Asn Leu His Glu Leu Asp Leu Asp Arg Val Ile Ala Tyr Gly His Thr
260 265 270
Trp Ser Pro Thr Leu Glu Leu Ala Pro Met Ile Pro Leu Phe His Gly
275 280 285
His Ala Val Asn Ile Asp Met Ala Leu Ser Ala Thr Ile Ala Ala Arg
290 295 300
Arg Gly Tyr Ile Thr Ser Gly Glu Arg Asp Arg Ile Leu Ser Leu Met
305 310 315 320
Ser Arg Ile Gly Leu Ser Ile Asp His Pro Leu Leu Asp Gly Asp Leu
325 330 335
Leu Trp Tyr Ala Thr Gln Ser Ile Ser Leu Thr Arg Asp Gly Lys Gln
340 345 350
Arg Ala Ala Met Pro Lys Pro Ile Gly Glu Cys Phe Phe Val Asn Asp
355 360 365
Phe Thr Arg Glu Glu Leu Asp Ala Ala Leu Ala Glu His Lys Arg Leu
370 375 380
Cys Ala Thr Tyr Pro Arg Gly Gly Asp Gly Ile Asp Ala Tyr Ile Glu
385 390 395 400
Thr Gln Glu Glu Ser Lys Leu Leu Gly Val
405 410
<210> 120
<211> 277
<212> PRT
<213> Nostoc punctiforme ATCC 29133
<400> 120
Met Thr Ser Ile Leu Gly Arg Asp Thr Ala Arg Pro Ile Thr Pro His
1 5 10 15
Ser Ile Leu Val Ala Gln Leu Gln Lys Thr Leu Arg Met Ala Glu Glu
20 25 30
Ser Asn Ile Pro Ser Glu Ile Leu Thr Ser Leu Arg Gln Gly Leu Gln
35 40 45
Leu Ala Ala Gly Leu Asp Pro Tyr Leu Asp Asp Cys Thr Thr Pro Glu
50 55 60
Ser Thr Ala Leu Thr Ala Leu Ala Gln Lys Thr Ser Ile Glu Asp Trp
65 70 75 80
Ser Lys Arg Phe Ser Asp Gly Glu Thr Val Arg Gln Leu Glu Gln Glu
85 90 95
Met Leu Ser Gly His Leu Glu Gly Gln Thr Leu Lys Met Phe Val His
100 105 110
Ile Thr Lys Ala Lys Ser Ile Leu Glu Val Gly Met Phe Thr Gly Tyr
115 120 125
Ser Ala Leu Ala Met Ala Glu Ala Leu Pro Asp Asp Gly Arg Leu Ile
130 135 140
Ala Cys Glu Val Asp Ser Tyr Val Ala Glu Phe Ala Gln Thr Cys Phe
145 150 155 160
Gln Glu Ser Pro His Gly Arg Lys Ile Val Val Glu Val Ala Pro Ala
165 170 175
Leu Glu Thr Leu His Lys Leu Val Ala Lys Lys Glu Ser Phe Asp Leu
180 185 190
Ile Phe Ile Asp Ala Asp Lys Lys Glu Tyr Ile Glu Tyr Phe Gln Ile
195 200 205
Ile Leu Asp Ser His Leu Leu Ala Pro Asp Gly Leu Ile Cys Val Asp
210 215 220
Asn Thr Leu Leu Gln Gly Gln Val Tyr Leu Pro Ser Glu Gln Arg Thr
225 230 235 240
Ala Asn Gly Glu Ala Ile Ala Gln Phe Asn Arg Ile Val Ala Ala Asp
245 250 255
Pro Arg Val Glu Gln Val Leu Leu Pro Ile Arg Asp Gly Ile Thr Leu
260 265 270
Ile Arg Arg Leu Val
275
<210> 121
<211> 461
<212> PRT
<213> Nostoc punctiforme ATCC 29133
<400> 121
Met Ala Gln Ser Ile Ser Leu Ser Leu Pro Gln Ser Thr Thr Pro Ser
1 5 10 15
Lys Gly Val Arg Leu Lys Ile Ala Ala Leu Leu Lys Thr Ile Gly Thr
20 25 30
Leu Ile Leu Leu Leu Ile Ala Leu Pro Leu Asn Ala Leu Ile Val Leu
35 40 45
Ile Ser Leu Met Cys Arg Pro Phe Thr Lys Lys Pro Ala Val Ala Thr
50 55 60
His Pro Gln Asn Ile Leu Val Ser Gly Gly Lys Met Thr Lys Ala Leu
65 70 75 80
Gln Leu Ala Arg Ser Phe His Ala Ala Gly His Arg Val Ile Leu Ile
85 90 95
Glu Gly His Lys Tyr Trp Leu Ser Gly His Arg Phe Ser Asn Ser Val
100 105 110
Ser Arg Phe Tyr Thr Val Pro Ala Pro Gln Asp Asp Pro Glu Gly Tyr
115 120 125
Thr Gln Ala Leu Leu Glu Ile Val Lys Arg Glu Lys Ile Asp Val Tyr
130 135 140
Val Pro Val Cys Ser Pro Val Ala Ser Tyr Tyr Asp Ser Leu Ala Lys
145 150 155 160
Ser Ala Leu Ser Glu Tyr Cys Glu Val Phe His Phe Asp Ala Asp Ile
165 170 175
Thr Lys Met Leu Asp Asp Lys Phe Ala Phe Thr Asp Arg Ala Arg Ser
180 185 190
Leu Gly Leu Ser Ala Pro Lys Ser Phe Lys Ile Thr Asp Pro Glu Gln
195 200 205
Val Ile Asn Phe Asp Phe Ser Lys Glu Thr Arg Lys Tyr Ile Leu Lys
210 215 220
Ser Ile Ser Tyr Asp Ser Val Arg Arg Leu Asn Leu Thr Lys Leu Pro
225 230 235 240
Cys Asp Thr Pro Glu Glu Thr Ala Ala Phe Val Lys Ser Leu Pro Ile
245 250 255
Ser Pro Glu Lys Pro Trp Ile Met Gln Glu Phe Ile Pro Gly Lys Glu
260 265 270
Leu Cys Thr His Ser Thr Val Arg Asp Gly Glu Leu Arg Leu His Cys
275 280 285
Cys Ser Asn Ser Ser Ala Phe Gln Ile Asn Tyr Glu Asn Val Glu Asn
290 295 300
Pro Gln Ile Gln Glu Trp Val Gln His Phe Val Lys Ser Leu Arg Leu
305 310 315 320
Thr Gly Gln Ile Ser Leu Asp Phe Ile Gln Ala Glu Asp Gly Thr Ala
325 330 335
Tyr Ala Ile Glu Cys Asn Pro Arg Thr His Ser Ala Ile Thr Met Phe
340 345 350
Tyr Asn His Pro Gly Val Ala Glu Ala Tyr Leu Gly Lys Thr Pro Leu
355 360 365
Ala Ala Pro Leu Glu Pro Leu Ala Asp Ser Lys Pro Thr Tyr Trp Ile
370 375 380
Tyr His Glu Ile Trp Arg Leu Thr Gly Ile Arg Ser Gly Gln Gln Leu
385 390 395 400
Gln Thr Trp Phe Gly Arg Leu Val Arg Gly Thr Asp Ala Ile Tyr Arg
405 410 415
Leu Asp Asp Pro Ile Pro Phe Leu Thr Leu His His Trp Gln Ile Thr
420 425 430
Leu Leu Leu Leu Gln Asn Leu Gln Arg Leu Lys Gly Trp Val Lys Ile
435 440 445
Asp Phe Asn Ile Gly Lys Leu Val Glu Leu Gly Gly Asp
450 455 460
<210> 122
<211> 348
<212> PRT
<213> Nostoc punctiforme ATCC 29133
<400> 122
Met Pro Val Leu Asn Ile Leu His Leu Val Gly Ser Ala His Asp Lys
1 5 10 15
Phe Tyr Cys Asp Leu Ser Arg Leu Tyr Ala Gln Asp Cys Leu Ala Ala
20 25 30
Thr Ala Asp Pro Ser Leu Tyr Asn Phe Gln Ile Ala Tyr Ile Thr Pro
35 40 45
Asp Arg Gln Trp Arg Phe Pro Asp Ser Leu Ser Arg Glu Asp Ile Ala
50 55 60
Leu Thr Lys Pro Ile Pro Val Phe Asp Ala Ile Gln Phe Leu Thr Gly
65 70 75 80
Gln Asn Ile Asp Met Met Leu Pro Gln Met Phe Cys Ile Pro Gly Met
85 90 95
Thr Gln Tyr Arg Ala Leu Phe Asp Leu Leu Lys Ile Pro Tyr Ile Gly
100 105 110
Asn Thr Pro Asp Ile Met Ala Ile Ala Ala His Lys Ala Arg Ala Lys
115 120 125
Ala Ile Val Glu Ala Ala Gly Val Lys Val Pro Arg Gly Glu Leu Leu
130 135 140
Arg Gln Gly Asp Ile Pro Thr Ile Thr Pro Pro Ala Val Val Lys Pro
145 150 155 160
Val Ser Ser Asp Asn Ser Leu Gly Val Val Leu Val Lys Asp Val Thr
165 170 175
Glu Tyr Asp Ala Ala Leu Lys Lys Ala Phe Glu Tyr Ala Ser Glu Val
180 185 190
Ile Val Glu Ala Phe Ile Glu Leu Gly Arg Glu Val Arg Cys Gly Ile
195 200 205
Ile Val Lys Asp Gly Glu Leu Ile Gly Leu Pro Leu Glu Glu Tyr Leu
210 215 220
Val Asp Pro His Asp Lys Pro Ile Arg Asn Tyr Ala Asp Lys Leu Gln
225 230 235 240
Gln Thr Asp Asp Gly Asp Leu His Leu Thr Ala Lys Asp Asn Ile Lys
245 250 255
Ala Trp Ile Leu Asp Pro Asn Asp Pro Ile Thr Gln Lys Val Gln Gln
260 265 270
Val Ala Lys Arg Cys His Gln Ala Leu Gly Cys Arg His Tyr Ser Leu
275 280 285
Phe Asp Phe Arg Ile Asp Pro Lys Gly Gln Pro Trp Phe Leu Glu Ala
290 295 300
Gly Leu Tyr Cys Ser Phe Ala Pro Lys Ser Val Ile Ser Ser Met Ala
305 310 315 320
Lys Ala Ala Gly Ile Pro Leu Asn Asp Leu Leu Ile Thr Ala Ile Asn
325 330 335
Glu Thr Leu Gly Ser Asn Lys Lys Val Leu Gln Asn
340 345
<210> 123
<211> 680
<212> PRT
<213> Saccharomyces cerevisiae CEN.PK-1D
<400> 123
Met Thr Gln Phe Thr Asp Ile Asp Lys Leu Ala Val Ser Thr Ile Arg
1 5 10 15
Ile Leu Ala Val Asp Thr Val Ser Lys Ala Asn Ser Gly His Pro Gly
20 25 30
Ala Pro Leu Gly Met Ala Pro Ala Ala His Val Leu Trp Ser Gln Met
35 40 45
Arg Met Asn Pro Thr Asn Pro Asp Trp Ile Asn Arg Asp Arg Phe Val
50 55 60
Leu Ser Asn Gly His Ala Val Ala Leu Leu Tyr Ser Met Leu His Leu
65 70 75 80
Thr Gly Tyr Asp Leu Ser Ile Glu Asp Leu Lys Gln Phe Arg Gln Leu
85 90 95
Gly Ser Arg Thr Pro Gly His Pro Glu Phe Glu Leu Pro Gly Val Glu
100 105 110
Val Thr Thr Gly Pro Leu Gly Gln Gly Ile Ser Asn Ala Val Gly Met
115 120 125
Ala Met Ala Gln Ala Asn Leu Ala Ala Thr Tyr Asn Lys Pro Gly Phe
130 135 140
Thr Leu Ser Asp Asn Tyr Thr Tyr Val Phe Leu Gly Asp Gly Cys Leu
145 150 155 160
Gln Glu Gly Ile Ser Ser Glu Ala Ser Ser Leu Ala Gly His Leu Lys
165 170 175
Leu Gly Asn Leu Ile Ala Ile Tyr Asp Asp Asn Lys Ile Thr Ile Asp
180 185 190
Gly Ala Thr Ser Ile Ser Phe Asp Glu Asp Val Ala Lys Arg Tyr Glu
195 200 205
Ala Tyr Gly Trp Glu Val Leu Tyr Val Glu Asn Gly Asn Glu Asp Leu
210 215 220
Ala Gly Ile Ala Lys Ala Ile Ala Gln Ala Lys Leu Ser Lys Asp Lys
225 230 235 240
Pro Thr Leu Ile Lys Met Thr Thr Thr Ile Gly Tyr Gly Ser Leu His
245 250 255
Ala Gly Ser His Ser Val His Gly Ala Pro Leu Lys Ala Asp Asp Val
260 265 270
Lys Gln Leu Lys Ser Lys Phe Gly Phe Asn Pro Asp Lys Ser Phe Val
275 280 285
Val Pro Gln Glu Val Tyr Asp His Tyr Gln Lys Thr Ile Leu Lys Pro
290 295 300
Gly Val Glu Ala Asn Asn Lys Trp Asn Lys Leu Phe Ser Glu Tyr Gln
305 310 315 320
Lys Lys Phe Pro Glu Leu Gly Ala Glu Leu Ala Arg Arg Leu Ser Gly
325 330 335
Gln Leu Pro Ala Asn Trp Glu Ser Lys Leu Pro Thr Tyr Thr Ala Lys
340 345 350
Asp Ser Ala Val Ala Thr Arg Lys Leu Ser Glu Thr Val Leu Glu Asp
355 360 365
Val Tyr Asn Gln Leu Pro Glu Leu Ile Gly Gly Ser Ala Asp Leu Thr
370 375 380
Pro Ser Asn Leu Thr Arg Trp Lys Glu Ala Leu Asp Phe Gln Pro Pro
385 390 395 400
Ser Ser Gly Ser Gly Asn Tyr Ser Gly Arg Tyr Ile Arg Tyr Gly Ile
405 410 415
Arg Glu His Ala Met Gly Ala Ile Met Asn Gly Ile Ser Ala Phe Gly
420 425 430
Ala Asn Tyr Lys Pro Tyr Gly Gly Thr Phe Leu Asn Phe Val Ser Tyr
435 440 445
Ala Ala Gly Ala Val Arg Leu Ser Ala Leu Ser Gly His Pro Val Ile
450 455 460
Trp Val Ala Thr His Asp Ser Ile Gly Val Gly Glu Asp Gly Pro Thr
465 470 475 480
His Gln Pro Ile Glu Thr Leu Ala His Phe Arg Ser Leu Pro Asn Ile
485 490 495
Gln Val Trp Arg Pro Ala Asp Gly Asn Glu Val Ser Ala Ala Tyr Lys
500 505 510
Asn Ser Leu Glu Ser Lys His Thr Pro Ser Ile Ile Ala Leu Ser Arg
515 520 525
Gln Asn Leu Pro Gln Leu Glu Gly Ser Ser Ile Glu Ser Ala Ser Lys
530 535 540
Gly Gly Tyr Val Leu Gln Asp Val Ala Asn Pro Asp Ile Ile Leu Val
545 550 555 560
Ala Thr Gly Ser Glu Val Ser Leu Ser Val Glu Ala Ala Lys Thr Leu
565 570 575
Ala Ala Lys Asn Ile Lys Ala Arg Val Val Ser Leu Pro Asp Phe Phe
580 585 590
Thr Phe Asp Lys Gln Pro Leu Glu Tyr Arg Leu Ser Val Leu Pro Asp
595 600 605
Asn Val Pro Ile Met Ser Val Glu Val Leu Ala Thr Thr Cys Trp Gly
610 615 620
Lys Tyr Ala His Gln Ser Phe Gly Ile Asp Arg Phe Gly Ala Ser Gly
625 630 635 640
Lys Ala Pro Glu Val Phe Lys Phe Phe Gly Phe Thr Pro Glu Gly Val
645 650 655
Ala Glu Arg Ala Gln Lys Thr Ile Ala Phe Tyr Lys Gly Asp Lys Leu
660 665 670
Ile Ser Pro Leu Lys Lys Ala Phe
675 680
<210> 124
<211> 370
<212> PRT
<213> Saccharomyces cerevisiae CEN.PK-1D
<400> 124
Met Ser Glu Ser Pro Met Phe Ala Ala Asn Gly Met Pro Lys Val Asn
1 5 10 15
Gln Gly Ala Glu Glu Asp Val Arg Ile Leu Gly Tyr Asp Pro Leu Ala
20 25 30
Ser Pro Ala Leu Leu Gln Val Gln Ile Pro Ala Thr Pro Thr Ser Leu
35 40 45
Glu Thr Ala Lys Arg Gly Arg Arg Glu Ala Ile Asp Ile Ile Thr Gly
50 55 60
Lys Asp Asp Arg Val Leu Val Ile Val Gly Pro Cys Ser Ile His Asp
65 70 75 80
Leu Glu Ala Ala Gln Glu Tyr Ala Leu Arg Leu Lys Lys Leu Ser Asp
85 90 95
Glu Leu Lys Gly Asp Leu Ser Ile Ile Met Arg Ala Tyr Leu Glu Lys
100 105 110
Pro Arg Thr Thr Val Gly Trp Lys Gly Leu Ile Asn Asp Pro Asp Val
115 120 125
Asn Asn Thr Phe Asn Ile Asn Lys Gly Leu Gln Ser Ala Arg Gln Leu
130 135 140
Phe Val Asn Leu Thr Asn Ile Gly Leu Pro Ile Gly Ser Glu Met Leu
145 150 155 160
Asp Thr Ile Ser Pro Gln Tyr Leu Ala Asp Leu Val Ser Phe Gly Ala
165 170 175
Ile Gly Ala Arg Thr Thr Glu Ser Gln Leu His Arg Glu Leu Ala Ser
180 185 190
Gly Leu Ser Phe Pro Val Gly Phe Lys Asn Gly Thr Asp Gly Thr Leu
195 200 205
Asn Val Ala Val Asp Ala Cys Gln Ala Ala Ala His Ser His His Phe
210 215 220
Met Gly Val Thr Lys His Gly Val Ala Ala Ile Thr Thr Thr Lys Gly
225 230 235 240
Asn Glu His Cys Phe Val Ile Leu Arg Gly Gly Lys Lys Gly Thr Asn
245 250 255
Tyr Asp Ala Lys Ser Val Ala Glu Ala Lys Ala Gln Leu Pro Ala Gly
260 265 270
Ser Asn Gly Leu Met Ile Asp Tyr Ser His Gly Asn Ser Asn Lys Asp
275 280 285
Phe Arg Asn Gln Pro Lys Val Asn Asp Val Val Cys Glu Gln Ile Ala
290 295 300
Asn Gly Glu Asn Ala Ile Thr Gly Val Met Ile Glu Ser Asn Ile Asn
305 310 315 320
Glu Gly Asn Gln Gly Ile Pro Ala Glu Gly Lys Ala Gly Leu Lys Tyr
325 330 335
Gly Val Ser Ile Thr Asp Ala Cys Ile Gly Trp Glu Thr Thr Glu Asp
340 345 350
Val Leu Arg Lys Leu Ala Ala Ala Val Arg Gln Arg Arg Glu Val Asn
355 360 365
Lys Lys
370
<---
Группа изобретений относится к модифицированному микроорганизму, продуцирующему микоспорин-подобную аминокислоту (MAA), и способу получения MAA с использованием указанного микроорганизма. Предложен модифицированный микроорганизм, предназначенный для продуцирования микоспорин-подобной аминокислоты, где микоспорин-подобная аминокислота представляет собой соединение, которое имеет центральное кольцо циклогексанона или циклогексенимина. При этом микроорганизм экспрессирует кластер генов биосинтеза MAA и где активность по меньшей мере одного белка, выбранного из группы, состоящей из 2-дегидро-3-дезоксифосфогептонатальдолазы (AroG), фосфоенолпируватсинтетазы (Pps) и транскетолазы (TktA), модифицирована для усиления путем введения гена, кодирующего указанный по меньшей мере один белок, в данный микроорганизм, экспрессирующий кластер генов биосинтеза MAA. При этом микроорганизм демонстрирует увеличенную продукцию микоспорин-подобной аминокислоты по сравнению с родительским или немодифицированным микроорганизмом, не имеющим указанную модификацию и экспрессирующим только кластер генов биосинтеза MAA. Также предложен способ получения микоспорин-подобной аминокислоты, включающий культивирование указанного модифицированного микроорганизма в среде и выделение микоспорин-подобной аминокислоты из культивированного микроорганизма или среды. Группа изобретений может быть эффективно использована в получении микоспорин-подобной аминокислоты. 2 н. и 5 з.п. ф-лы, 27 табл., 19 пр.
1. Модифицированный микроорганизм для продуцирования микоспорин-подобной аминокислоты,
где микоспорин-подобная аминокислота представляет собой соединение, которое имеет центральное кольцо циклогексанона или циклогексенимина,
где микроорганизм экспрессирует кластер генов биосинтеза микоспорин-подобной аминокислоты (MAA),
где активность по меньшей мере одного белка, выбранного из группы, состоящей из 2-дегидро-3-дезоксифосфогептонатальдолазы (AroG), фосфоенолпируватсинтетазы (Pps) и транскетолазы (TktA), модифицирована для усиления путем введения гена, кодирующего указанный по меньшей мере один белок, в данный микроорганизм, экспрессирующий кластер генов биосинтеза MAA,
где микроорганизм демонстрирует увеличенную продукцию микоспорин-подобной аминокислоты по сравнению с родительским или немодифицированным микроорганизмом, не имеющим указанную модификацию и экспрессирующим только кластер генов биосинтеза микоспорин-подобной аминокислоты.
2. Модифицированный микроорганизм по п. 1, где генный кластер биосинтеза микоспорин-подобной аминокислоты содержит ген, кодирующий 2-деметил-4-дезоксигадузолсинтазу, и ген, кодирующий O-метилтрансферазу.
3. Модифицированный микроорганизм по п. 2, где генный кластер биосинтеза микоспорин-подобной аминокислоты дополнительно содержит ген, кодирующий C-N-лигазу.
4. Модифицированный микроорганизм по п. 2, где генный кластер биосинтеза микоспорин-подобной аминокислоты дополнительно содержит ген, кодирующий по меньшей мере один белок, выбранный из группы, состоящей из нерибосомальной пептидсинтетазы, фермента, подобного нерибосомальной пептидсинтетазе (NRPS-подобного фермента), и D-аланин- D-аланин-лигазы (D-Ala-D-Ala-лигазы).
5. Модифицированный микроорганизм по п. 1, где микроорганизм представляет собой микроорганизм рода Corynebacterium, или микроорганизм рода Escherichia, или дрожжи.
6. Модифицированный микроорганизм по п. 1, где микоспорин-подобная аминокислота по меньшей мере представляет собой микоспорин-подобную аминокислоту, выбранную из группы, состоящей из микоспорин-2-глицина, палитинола, палитеновой кислоты, дезоксигадузола, микоспорин-метиламин-треонина, микоспорин-глицин-валина, палитина, астерины-330 (asterina-330), шинорина, порфиры-334 (porphyra-334), эухалотеца-362 (euhalothece-362), микоспорин-глицина, микоспорин-орнитина, микоспорин-лизина, микоспорин-глутаминовая кислота-глицина, микоспорин-метиламин-серина, микоспорин-таурина, палитена, палитин-серина, палитин-серин-сульфата, палитинола и усуджирена (usujirene).
7. Способ получения микоспорин-подобной аминокислоты, включающий: культивирование модифицированного микроорганизма по любому из пп. 1-6 в среде; и выделение микоспорин-подобной аминокислоты из культивированного микроорганизма или среды.
| WO 2015174427 A1, 19.11.2015 | |||
| КОМПОЗИЦИИ И СПОСОБЫ ДЛЯ УЛУЧШЕНИЯ ОБОГАЩЕНИЯ БИБЛИОТЕК | 2019 |
|
RU2809771C2 |
| POPE M.A | |||
| ET AL | |||
| Приспособление для забивки свай | 1932 |
|
SU29413A1 |
| Chembiochem | |||
| Устройство для закрепления лыж на раме мотоциклов и велосипедов взамен переднего колеса | 1924 |
|
SU2015A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| ПРЕПАРАТ APHANIZOMENON FLOS AQUAE, ЭКСТРАКТЫ И ОЧИЩЕННЫЕ КОМПОНЕНТЫ ЭКСТРАКТОВ ДЛЯ ЛЕЧЕНИЯ НЕВРОЛОГИЧЕСКИХ, НЕЙРОДЕГЕНЕРАТИВНЫХ И АФФЕКТИВНЫХ РАССТРОЙСТВ | 2007 |
|
RU2441663C2 |
Авторы
Даты
2021-10-15—Публикация
2019-02-22—Подача