Изобретение относится к средствам молекулярной диагностики и может быть использовано в клинической иммунологии и медицине для оценки иммунного статуса пациента методом ПЦР в реальном времени. Настоящее изобретение представляет собой диагностическую тест-систему для количественной оценки уровней мРНК генов MxA, OAS1, EIF2AK2 человека в биологическом образце. Тест-система включает в себя две пробы (I и II), в которых одновременно проходят несколько реакций: в пробе I - определение уровней экспрессии генов MxA, OAS1, EIF2AK2 человека, в пробе II - определение уровня экспрессии гена GAPDH (ген нормировки).
С момента открытия интерферонов (ИФН) был достигнут значительный прогресс в описании природы самих цитокинов - сигнальных компонентов, которые управляют клеточным ответом, и их противовирусной активности. Выявлено четыре основных эффекторных пути ИФН-опосредованного противовирусного ответа: путь Mx GTPase, путь 2′-5′-олигоаденилатсинтетазы (OAS1), управляемый рибонуклеазой L, путь протеинкиназы R (эукариотический фактор инициации трансляции 2-альфа-киназа 2, который у человека кодируется геном EIF2AK2) и путь, подобный убиквитину ISG15. Данные эффекторные пути индивидуально блокируют вирусную транскрипцию, разрушают вирусную РНК, ингибируют трансляцию и модифицируют функцию белка, чтобы контролировать все этапы репликации вируса [1].
В ответ на инфицирование гриппом клетка вырабатывает врожденный иммунный ответ, ограничивающий размножение вируса. Основным драйвером этого ответа является группа цитокинов под общим названием ИФН I типа. Экспрессия ИФН требует выявления вирус-специфических паттернов (патоген-ассоциированных молекулярных паттернов [PAMPs]), которые распознаются клеточными сенсорами (рецепторами распознавания паттернов [PRRs]) как чужеродные, из которых RIG-I актуален во время инфицирования эпителиальных клеток вирусом гриппа. RIG-I активируется при распознавании двухцепочечной РНК (дцРНК), содержащей 5′-трифосфат, и передает сигнал ниже по пути через взаимодействие с митохондриальным белком IPS-1 / MAVS / VISA / Cardif. Следствием этой нижестоящей передачи сигналов является активация факторов транскрипции IRF3, NF-κB и ATF-2 / c-Jun, которые активируют транскрипцию ИФН. Секретируемые ИФН действуют аутокринным и паракринным образом, активируя рецептор ИФН. Сигнал передается по пути Jak / STAT в ядро, где он активирует транскрипцию сотен ИФН-зависимых генов (ISG), многие из которых обладают противовирусной активностью, включая EIF2AK2, MxA, OAS1, необходимой клетке для преодоления вирусной инфекции и ограничения распространения вируса на соседние неинфицированные клетки [2].
Индуцируемый интерфероном I типа MxA-белок обладает избирательной активностью против нескольких вирусов. Однако точный механизм противовирусного действия не выяснен. Связываясь с дцРНК, OAS-1 катализирует образование 20-50-связанного олигоаденилата и активирует РНКазу, которая расщепляет вирусную и клеточную РНК. EIF2AK2 также активируется дцРНК; это приводит к фосфорилированию его субстрата, eIF2a, который ингибирует фактор обмена гуанозиновых нуклеотидов, eIF2b, и останавливает репликацию вируса. EIF2AK2 может действовать путем отключения белка после инфицирования клетки и ограничивают передачу вируса неинфицированным клеткам [3].
Совместное определение уровня экспрессии генов MxA, OAS1, EIF2AK2 позволяет определить уровень активации врожденного иммунного ответа организма и дать оценку активации ИФН-опосредованного противовирусного ответа. Особенную актуальность данная оценка приобретает при анализе патогенеза острых респираторных вирусных инфекций, вызванных вирусами гриппа, пневмовирусами, аденовирусами, коронавирусами. Наиболее чувствительным и специфичным методом определения уровня экспрессии генов является ПЦР в реальном времени. В данной методике предполагается наличие двух праймеров и флуоресцентно меченого олигонуклеотидного зонда, специфических к интересующей мРНК.
В настоящее время не зарегистрировано тест-систем, которые можно использовать для одновременного определения экспрессии генов MxA, OAS1, EIF2AK2 методом ПЦР в реальном времени. Известен способ определения экспрессии большого числа генов (включая MxA, OAS1, EIF2AK2) методом микроэррей [4], который является более дорогостоящим методом по сравнению с методом ПЦР. Также известны методы определения экспрессии MxA с использованием мультиплексной тест-системы для количественного анализа экспрессии генов интерферона лямбда (IFNλ), противовирусного белка МхА и интерлейкина 23 (IL23) [5], а также двух генов MxA, OAS1 с использованием набора для диагностики или прогнозирования лихорадки денге, включающего средства для обнаружения изменения уровня экспрессии одного или нескольких белков, родственных интерферону [6].
Таким образом, актуальность предлагаемого изобретения обусловлена необходимостью определения уровня экспрессии ИФН-индуцируемых генов для более глубокого понимания патогенеза острых респираторных вирусных инфекций и определения дальнейших стратегий их лечения, а также определения уровня активации интерферонового ответа. Отсутствие аналогичных коммерческих тест-систем делает данное изобретение уникальным продуктом интеллектуального творчества.
Технический результат заключается в подобранных последовательностях праймеров и олигонуклеотидных TaqMan зондов, а также условиях проведения ПЦР в реальном времени (концентрации вносимых реагентов и температурном профиле проводимых реакций). Данный результат достигается тем, что тест-система для количественной диагностики мРНК генов MxA, OAS1, EIF2AK2 человека на основе ПЦР включает набор олигонуклеотидных праймеров и флуоресцентных зондов со следующей структурой:
прямой праймер на ген MxA - GAGACAATCGTGAAACAGCAAATCA (SEQ NO. 1)
обратный праймер на ген MxA - TATCGAAACATCTGTGAAAGCAAGC (SEQ NO. 2)
флуоресцентный зонд на ген MxA Fam- CACTGGAAGAGCCGGCTGTGGATATG-BHQ2
(SEQ NO. 3)
прямой праймер на ген OAS1 - CCAAGGTGGTAAAGGGTGGCT (SEQ NO. 4)
обратный праймер на ген OAS1 - CTGGACCTCAAACTTCACGGAAA (SEQ NO. 5)
флуоресцентный зонд на ген OAS1 - Rox-AGGCCGATCTGACGCTGACCTGGTTGT- BHQ3
(SEQ NO. 6)
прямой праймер на ген EIF2AK2 - GAAAGCGAACAAGGAGTAAGGGA (SEQ NO. 7)
обратный праймер на ген EIF2AK2 - CCATCCCGTAGGTCTGTGAAA (SEQ NO. 8)
флуоресцентный зонд на ген EIF2AK2 - Cy5-AGCCCCAAAGCGTAGAGGTCCACTTCC-BHQ1
(SEQ NO. 9).
С помощью указанной тест-системы осуществляют количественное определение уровня мРНК генов MxA, OAS1, EIF2AK2 человека, в ходе которого проводят предварительную реакцию обратной транскрипции, а далее полимеразную цепную реакцию в реальном времени по следующему температурному профилю: первичная денатурация в течение 5 мин при температуре 95°С, далее 40 циклов двухступенчатой реакции, состоящей из этапа денатурации в течение 10 с при температуре 95°С и этапа отжига праймеров и элонгации цепи в течение 30 с при температуре 61°С.
Описание фигур
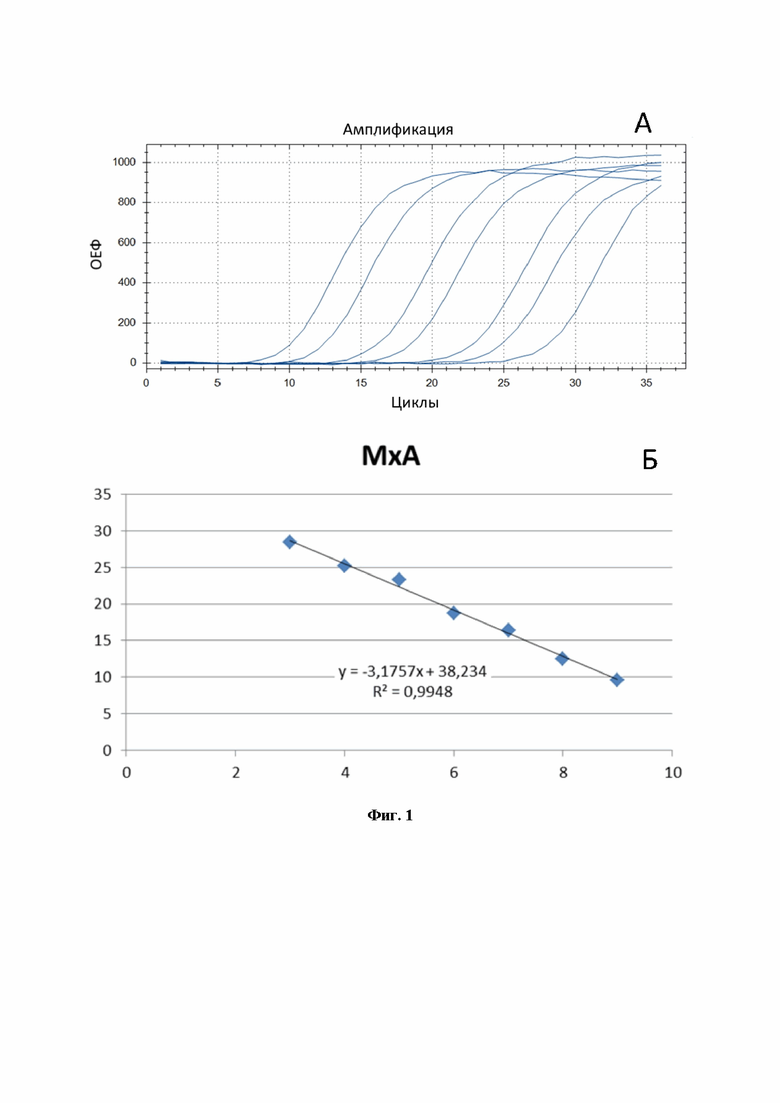
На фиг. 1 представлен расчёт эффективности ПЦР для количественной оценки мРНК гена MxA, проведённой в моноплексном режиме: A - кривые роста флуоресценции, полученные для линейки разведений образцов (от -9 до -3) при анализе флуоресценции на канале FAM в режиме реального времени; Б - построенная по полученным значениям Cq кривая, используемая для расчёта эффективности реакции.
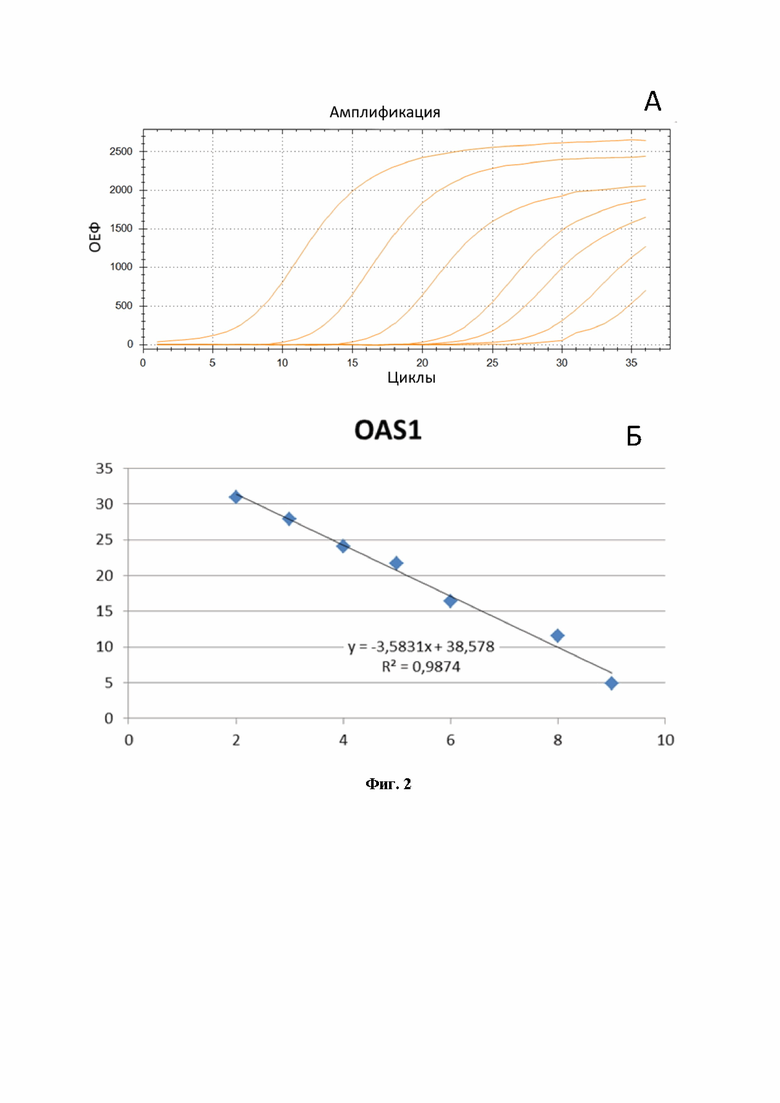
На фиг. 2 представлен расчёт эффективности реакции ПЦР для количественной оценки мРНК гена OAS1, проведённой в моноплексном режиме: A - кривые роста флуоресценции, полученные для линейки разведений образцов (от -9 до -2) при анализе флуоресценции на канале ROX в режиме реального времени; Б - построенная по полученным значениям Cq кривая, используемая для расчёта эффективности реакции.
На фиг. 3 представлен расчёт эффективности реакции ПЦР для количественной оценки мРНК гена EIF2AK2, проведённой в моноплексном режиме: A - кривые роста флуоресценции, полученные для линейки разведений образцов (от -9 до -2) при анализе флуоресценции на канале Cy5 в режиме реального времени; Б - построенная по полученным значениям Cq кривая, используемая для расчёта эффективности реакции.
На фиг. 4 представлено сравнение эффективностей реакций ПЦР в моноплексном и мультиплексном форматах: А - представлены стандартные калибровочные кривые, используемые для расчёта эффективностей амплификации MxA, OAS1 и EIF2AK2 в мультиплексном формате (одновременная детекция в одной пробирке); Б - стандартная калибровочная кривая, полученная в мультиплексном формате ПЦР в сравнении c калибровочной кривой при моноплексной постановке для гена MxA; В - стандартная калибровочная кривая, полученная в мультиплексном формате ПЦР в сравнении с калибровочной кривой при моноплексной постановке для гена OAS1; Г - стандартная калибровочная кривая, полученная в мультиплексном формате ПЦР, в сравнении c калибровочной кривой при моноплексной постановке для гена EIF2AK2.
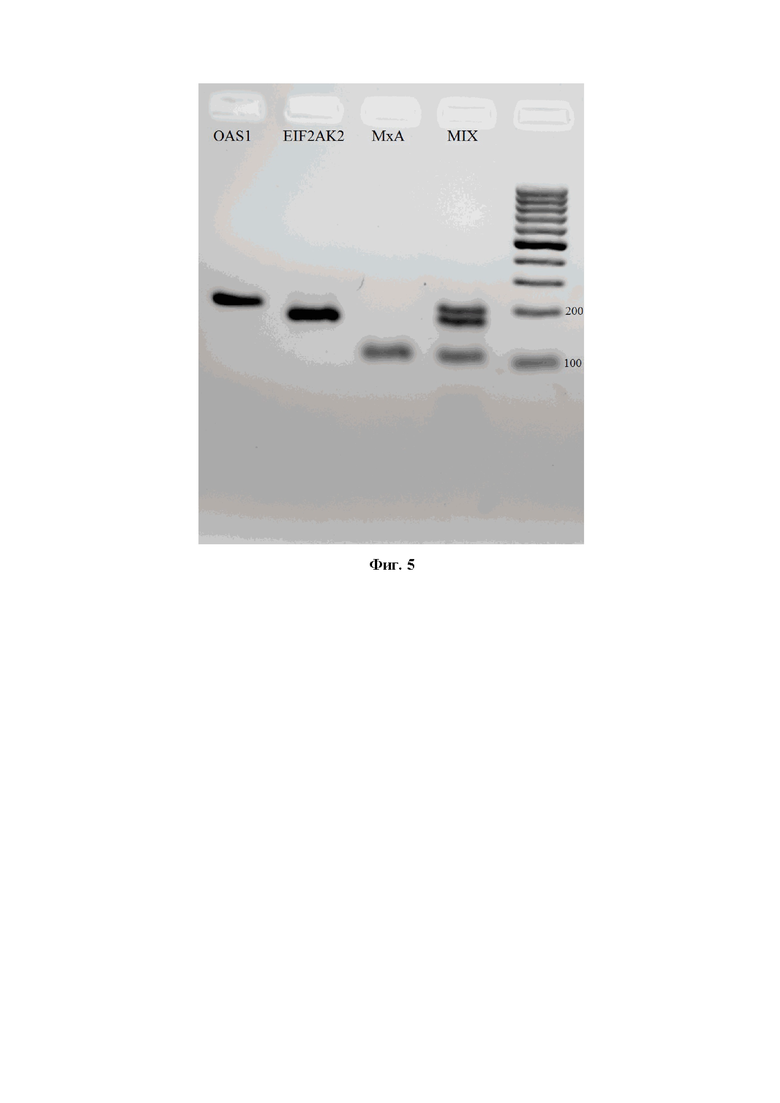
На фиг. 5 представлен результат электрофоретического разделения в агарозном геле продуктов, полученных в ходе проведения мультиплексной и моноплексных ПЦР, для оценки возможности образования в ходе реакции нежелательных неспецифических продуктов.
На фиг. 6 представлено определение уровня экспрессии генов MxA, OAS1 и EIF2AK2 в периферических мононуклеарных клетках крови пациентов с гриппозной инфекцией, новой коронавирусной инфекцией (COVID-19) и здоровых волонтеров. Достоверность различий определялась с использованием непараметрического критерия Краскела-Уоллиса с множественными сравнениями. * - P < 0,05; ** - P < 0,01; **** - P < 0,0001.
Для определения уровня экспрессии генов MxA, OAS1, EIF2AK2 был проведен дизайн праймеров и олигонуклеотидных зондов. Оригинальные последовательности всех праймеров и зондов для количественной оценки мРНК генов MxA, OAS, EIF2AK2 были подобраны к белок-кодирующей области генов таким образом, чтобы праймеры были разделены областью интрона, температуры их плавления были схожи, а длины ампликонов, образующихся в процессе ПЦР, не превышали 300 п.н. В качестве эндогенного контроля, используемого для нормировки экспрессии генов MxA, OAS1, EIF2AK2, был предложен ген глицеральдегид-3-фосфатдегидрогеназы (GAPDH) (табл.1).
NM_001256799.3 NM_001289745.3 NM_001289746.2 NM_001357943.2
Подобранные праймеры выявляют все транскрипционные варианты мРНК генов (3 транскрипционных варианта MxA, 4 транскрипционных варианта OAS1, 3 транскрипционных варианта EIF2AK2, 5 транскрипционных вариантов GAPDH).
Подбор и оптимизация условий ПЦР
На данном этапе работы была проведена оценка качества подобранных пар праймеров и зондов, а также возможность образования ими гетеро- и гомодимеров и неспецифических продуктов. В качестве матрицы для проведения данного исследования использовали образцы кДНК, полученные из препаратов тотальной РНК, выделенных из клеток A549, инфицированных вирусом гриппа A. Работа проводилась с использованием 2× реакционной смеси, содержащей Taq ДНК-полимеразу с «горячим» стартом, для проведения ПЦР с контролем по конечной точке (БиоМастер HS-Taq ПЦР (2×), Биолабмикс). Реакцию проводили в объёме 25 мкл, содержащем от 6,25 до 12,5 пмоль прямого и обратного праймеров и TaqMan зонда. Для определения оптимального термального профиля реакций были проведены ПЦР с температурным градиентом отжига праймеров: 1) 95°С - 300 c; 2) [95°С - 10 c; градиент температур 57,5°С - 63,5°С - 30 c; 72°С - 30 c] - 40 циклов.
Детекцию результатов осуществляли по росту флуоресценции, наличие неспецифических продуктов оценивали электрофоретическим разделением продуктов в агарозном геле.
С учётом полученных результатов в качестве оптимального температурного профиля проводимой мультиплексной ПЦР было предложено использовать следующий:
1) первичная денатурация 95°С - 5 мин,
далее 40 двухступенчатых циклов:
2) денатурация 95°С - 10 с;
3) отжиг праймеров и элонгация цепи 61°С - 30 c.
Расчет эффективностей ПЦР
Для корректного проведения относительного количественного анализа экспрессии ИФН-индуцируемых генов в мультиплексном формате значения эффективностей амплификации соответствующих мРНК должны быть идентичными или максимально приближенными друг к другу. Поэтому далее при подобранных оптимальных условиях были рассчитаны эффективности проводимых ПЦР.
Для определения эффективностей реакций использовали специфические очищенные ампликоны, полученные в неколичественных ПЦР (без использования TaqMan зонда). Расчёт проводили по углу наклона кривой, полученной в результате постановки qПЦР с серией последовательных разбавлений подготовленных ампликонов.
На Фиг. 1Б показаны результаты расчёта эффективности ПЦР при амплификации гена MxA в формате моноплексной ПЦР. Рассчитанная эффективность ПЦР на MxA (Фиг. 1Б) составила 106%, что является хорошим параметром амплификации. Аналогичным образом были получены эффективности амплификации ПЦР для OAS1 (Фиг. 2Б) и для EIF2AK2 (Фиг. 3Б). Таким образом, продемонстрировано, что в моноплексном формате эффективности амплификации продуктов трех генов MxA, OAS1 и EIF2AK2 составили: 106%, 90%, 118%, соответственно. Стандартные калибровочные кривые, представленные на Фиг. 1-3Б, имеют похожие углы наклона, т.е. параллельны.
При постановке мультиплексной ПЦР амплификация всех генов MxA, OAS1 и EIF2AK2 проводилась одновременно в одной пробирке. При этом в пробу ПЦР, содержащую фермент ДНК-зависимую ДНК-полимеразу и прилагаемый к ней буфер, также добавлялись все подобранные пары праймеров и олигонуклеотидные зонды, специфически выявляющие заявленные гены. Конечные концентрации праймеров и зондов, содержащиеся в пробе при проведении мультиплексной ПЦР, представлены в таблице 2.
Ввиду того, что во время реакции пары праймеров могут давать неспецифические химерные продукты, был проведен анализ продуктов мультиплексной и моноплексной ПЦР методом электрофоретического разделения в агарозном геле (Фиг. 5).
Результаты, представленные на Фиг. 5, свидетельствуют о том, что при проведении моноплексной ПЦР в пробирках амплифицировались только целевые продукты, соответствующие заявленным генам MxA, OAS1, EIF2AK2. Продукты ПЦР представлены яркими бэндами с длинами, соответствующими расчётным: 105 п.н., 200 п.н. и 175 п.н. для каждого анализируемого гена (MxA, OAS1, EIF2AK2, соответственно). При проведении мультиплексной ПЦР в пробирке одновременно выявлялись все три гена, имеющие аналогичные длины. Таким образом, в ходе мультиплексной ПЦР не образовывались какие-либо нежелательные неспецифические продукты.
Далее были рассчитаны эффективности амплификации каждого из заявленных генов при проведении мультиплексной ПЦР. Как продемонстрировано на Фиг. 4А, эффективности амплификации всех специфических продуктов MxA, OAS1 и EIF2AK2 при проведении ПЦР в одной пробирке составили: 102%, 93%, 113%, соответственно. Кроме того, мультиплексирование праймеров приводит к снижению амплификации не более чем на 3-5% по сравнению с амплификацией в моноплексном формате для каждого из специфических продуктов (Фиг. 4Б-Г).
Верификация тест-системы на клинических образцах
Разработанная тест-система применялась для одновременного измерения экспрессии генов MxA, OAS1 и EIF2AK2 в периферических мононуклеарных клетках крови пациентов с диагнозом COVID-19, гриппозной инфекцией, а также контрольной группы здоровых доноров. Диагностика инфекций была проведена ПЦР-наборами, сертифицированными для проведения подобных исследований.
В результате измерения было установлено, что у пациентов с COVID-19 происходило повышение экспрессии OAS1 практически на два порядка по сравнению с контрольной группой пациентов, тогда как у пациентов с гриппозной инфекцией экспрессия OAS1 была достоверно ниже, чем у пациентов с COVID-19, но достоверно выше, чем у контрольной группы. Экспрессия генов MxA и EIF2AK2 возрастала как в случае гриппозной инфекции, так и в случае заражения COVID-19.
Использование разработанной мультиплексной ПЦР тест-системы позволяет с высокой эффективностью проводить количественный анализ генов MxA, OAS1 и EIF2AK2 в клинических образцах.
Список литературы
1. Anthony J. Sadler and Bryan R. G. Williams. Interferon-inducible antiviral effectors// Nature Reviews Immunology. - 2008. - 8(7). - С. 559-568.
2. Haller, O. et al. Protective role of interferon-induced Mx GTPases against influenza viruses// Scientific and Technical Review. - 2009. - 28(1). С. 219-231.
3. Knapp, S. et al. Polymorphisms in interferon-induced genes and the outcome of hepatitis C virus infection: roles of MxA, OAS-1 and PKR// Genes & Immunity. - 2003. - 4(6). - С. 411-419.
4. Compositions and Methods for Diagnosing and Assessing Inflammatory Myopathies: patent application US2010190659A1, the United States of America, filed 07.07.2008, publ. 29.07.2010.
5. Test system for determination of interferon, IL23 interleukine and MXA anti-virus protein RNA: patent RU2627179, Russian Federation, appl. RU2016131205, filed 28.07.2016, publ. 03.08.2017.
6. Dengue diagnosis and treatment: patent application US2010068147, the United States of America, filed 05.10.2007, publ. 18.03.2010.
--->
<?xml version="1.0" encoding="UTF-8"?><!DOCTYPE ST26SequenceListing
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN"
"ST26SequenceListing_V1_3.dtd"><ST26SequenceListing
dtdVersion="V1_3" fileName="/Users/fedorivanov/Downloads/Seq.
list.xml" softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2023-03-15"> <ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText></ApplicationNumberText>
<FilingDate></FilingDate> </ApplicationIdentification>
<ApplicantFileReference>MxA, OAS1, EIF2AK2</ApplicantFileReference>
<ApplicantName languageCode="ru">Федеральное государственное
автономное образовательное учреждение высшего образования
"Санкт-Петербургский политехнический университет Петра
Великого" (ФГАОУ ВО "СПбПУ")</ApplicantName>
<ApplicantNameLatin>Federal State Autonomous Educational Institution
of Higher Education "Peter the Great St. Petersburg Polytechnic
University" (SPbPU)</ApplicantNameLatin> <InventionTitle
languageCode="ru">Тест-система для количественной диагностики мРНК
генов MxA, OAS1, EIF2AK2 человека на основе ПЦР</InventionTitle>
<SequenceTotalQuantity>9</SequenceTotalQuantity> <SequenceData
sequenceIDNumber="1"> <INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GAGACAATCGTGAAACAGCAAATCA</INSDSeq_sequence>
</INSDSeq> </SequenceData> <SequenceData sequenceIDNumber="2">
<INSDSeq> <INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TATCGAAACATCTGTGAAAGCAAGC</INSDSeq_sequence>
</INSDSeq> </SequenceData> <SequenceData sequenceIDNumber="3">
<INSDSeq> <INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CACTGGAAGAGCCGGCTGTGGATATG</INSDSeq_sequence>
</INSDSeq> </SequenceData> <SequenceData sequenceIDNumber="4">
<INSDSeq> <INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CCAAGGTGGTAAAGGGTGGCT</INSDSeq_sequence>
</INSDSeq> </SequenceData> <SequenceData sequenceIDNumber="5">
<INSDSeq> <INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CTGGACCTCAAACTTCACGGAAA</INSDSeq_sequence>
</INSDSeq> </SequenceData> <SequenceData sequenceIDNumber="6">
<INSDSeq> <INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AGGCCGATCTGACGCTGACCTGGTTGT</INSDSeq_sequence>
</INSDSeq> </SequenceData> <SequenceData sequenceIDNumber="7">
<INSDSeq> <INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GAAAGCGAACAAGGAGTAAGGGA</INSDSeq_sequence>
</INSDSeq> </SequenceData> <SequenceData sequenceIDNumber="8">
<INSDSeq> <INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CCATCCCGTAGGTCTGTGAAA</INSDSeq_sequence>
</INSDSeq> </SequenceData> <SequenceData sequenceIDNumber="9">
<INSDSeq> <INSDSeq_length>27</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division> <INSDSeq_feature-table>
<INSDFeature> <INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..27</INSDFeature_location>
<INSDFeature_quals> <INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier> <INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier> </INSDFeature_quals> </INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AGCCCCAAAGCGTAGAGGTCCACTTCC</INSDSeq_sequence>
</INSDSeq> </SequenceData></ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Способ диагностики инвазивного кандидоза и видовой идентификации его основных возбудителей методом ПЦР с гибридизационно-флуоресцентной детекцией в режиме реального времени | 2022 |
|
RU2809386C1 |
| Тест-система для количественной диагностики мРНК интерферонов I, II и III типов человека на основе ПЦР | 2020 |
|
RU2751791C1 |
| Способ выявления химерного транскрипта DNAJB1-PRKACA в клинических образцах ткани пациентов с фиброламеллярной карциномой печени методом полимеразной цепной реакции в реальном времени | 2023 |
|
RU2807306C1 |
| Многопараметрическая диагностическая тест-система для количественного определения уровня мРНК генов RIG-1, IFIT-1, IFIH-1 человека | 2021 |
|
RU2782428C1 |
| Тест-система для количественной диагностики мРНК генов OAS1a/g, IL-6, IRF-7, IFNA, IFNB, IFNL мыши методом ПЦР в режиме реального времени | 2022 |
|
RU2796522C1 |
| Способ генотипирования однонуклеотидного варианта rs13056243 (С>Т) гена ZNRF3 человека методом полимеразно-цепной реакции в режиме реального времени | 2023 |
|
RU2819836C1 |
| Способ генотипирования однонуклеотидного варианта rs2269578 (G>C) гена XBP1 человека методом полимеразно-цепной реакции в режиме реального времени | 2024 |
|
RU2831205C1 |
| Набор реагентов для определения малых рибонуклеиновых кислот (микроРНК) при диагностике множественной миеломы | 2024 |
|
RU2836054C1 |
| Нуклеиновая кислота для аллотопической экспрессии гена MT-ND4 | 2023 |
|
RU2809065C1 |
| НАБОР ОЛИГОНУКЛЕОТИДОВ ДЛЯ ВЫЯВЛЕНИЯ ДНК ГЕНЕТИЧЕСКИ МОДИФИЦИРОВАННОГО АТЛАНТИЧЕСКОГО ЛОСОСЯ МЕТОДОМ ПЦР В РЕЖИМЕ РЕАЛЬНОГО ВРЕМЕНИ | 2023 |
|
RU2808662C1 |



Изобретение относится к биотехнологии. Изобретение относится к средствам молекулярной диагностики и может быть использовано в клинической иммунологии и медицине для оценки иммунного статуса пациента методом ПЦР в реальном времени. Описана диагностическая тест-система для количественной оценки уровней мРНК генов MxA, OAS1, EIF2AK2 человека в биологическом образце. Тест-система включает набор олигонуклеотидных праймеров и флуоресцентных зондов: прямой праймер на ген MxA GAGACAATCGTGAAACAGCAAATCA, обратный праймер на ген MxA TATCGAAACATCTGTGAAAGCAAGC, флуоресцентный зонд на ген MxA Fam- CACTGGAAGAGCCGGCTGTGGATATG-BHQ2, прямой праймер на ген OAS1 CCAAGGTGGTAAAGGGTGGCT, обратный праймер на ген OAS1 CTGGACCTCAAACTTCACGGAAA, флуоресцентный зонд на ген OAS1 Rox-AGGCCGATCTGACGCTGACCTGGTTGT- BHQ3, прямой праймер на ген EIF2AK2 GAAAGCGAACAAGGAGTAAGGGA, обратный праймер на ген EIF2AK2 CCATCCCGTAGGTCTGTGAAA, флуоресцентный зонд на ген EIF2AK2 Cy5-AGCCCCAAAGCGTAGAGGTCCACTTCC-BHQ1. Технический результат состоит в обеспечении возможности проведения быстрой и точной количественной оценки уровней мРНК генов MxA, OAS1, EIF2AK2 человека. 6 ил., 2 табл.
Тест-система для количественной диагностики мРНК генов MxA, OAS1, EIF2AK2 человека на основе ПЦР, включающая набор олигонуклеотидных праймеров и флуоресцентных зондов:
прямой праймер на ген MxA GAGACAATCGTGAAACAGCAAATCA;
обратный праймер на ген MxA TATCGAAACATCTGTGAAAGCAAGC;
флуоресцентный зонд на ген MxA Fam-CACTGGAAGAGCCGGCTGTGGATATG-BHQ2;
прямой праймер на ген OAS1 CCAAGGTGGTAAAGGGTGGCT;
обратный праймер на ген OAS1 CTGGACCTCAAACTTCACGGAAA;
флуоресцентный зонд на ген OAS1 Rox-AGGCCGATCTGACGCTGACCTGGTTGT- BHQ3; прямой праймер на ген EIF2AK2 GAAAGCGAACAAGGAGTAAGGGA; обратный праймер на ген EIF2AK2 CCATCCCGTAGGTCTGTGAAA; флуоресцентный зонд на ген EIF2AK2 Cy5-AGCCCCAAAGCGTAGAGGTCCACTTCC-BHQ1, отличающаяся тем, что концентрация праймеров и зонда к гену MxA составляет 250 нМ и 100 нМ, соответственно; праймеров и зонда к гену OAS1 — 500 нМ и 200 нМ, соответственно; праймеров и зонда к гену EIF2AK2 — 500 нМ и 200 нМ, соответственно.
| Plotnikova M., Lozhkov A., Romanovskaya-Romanko E., Baranovskaya I., Sergeeva M., Kаа K., Klotchenko S., Vasin A | |||
| Печь для непрерывного получения сернистого натрия | 1921 |
|
SU1A1 |
| Viruses | |||
| Способ регенерирования сульфо-кислот, употребленных при гидролизе жиров | 1924 |
|
SU2021A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| PMID: 34452467; PMCID: PMC8402797 | |||
| Johansson, Mary | |||
| Пломбировальные щипцы | 1923 |
|
SU2006A1 |
| Choosing | |||
Авторы
Даты
2024-01-17—Публикация
2022-12-28—Подача