Изобретение относится к ветеринарной вирусологии, к средствам молекулярной диагностики, а именно к дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Калицивирусная инфекция кошек является высококонтагиозным вирусным заболеванием, характеризующимся поражением слизистой оболочки ротовой полости и верхних дыхательных путей [1, 2]. Вирус распространен повсеместно и поражает домашних кошек всех пород и возрастов, а также зоопарковых и диких представителей семейства кошачьих.
Вирионы калицивируса кошек имеют пулевидную форму длиной 100-300 нм, диаметром 45-100 нм. На наружной поверхности вирусной частицы имеются выступы в виде шипов длиной 10-12 нм, которые прикреплены к двуслойной липидной оболочке [1].
Возбудитель заболевания Feline calicivirus (FCV), относится к порядку Picornavirales, семейству Caliciviridae, роду Vesivirus. Нуклеиновая кислота вируса представлена одноцепочечной РНК(+) размером около 7690 н.о. 5’-NTR (не транслируемый регион) соответствует 1…19 н.о., ORF1 (2C, протеаза, РНК-полимераза) – 20…5311 н.о., ORF2 (структурный белок VP1) – 5314…7329 н.о., ORF3 (белок VP2 с неизвестной функцией) – 7617…7643 н.о. [2, 3].
Система мер для борьбы с калицивирозом кошек и его профилактика предусматривает иммунизацию домашних животных [4-7]. Для этой цели применяют вакцинные препараты, которые включают в свой состав штамм «Мира» калицивируса кошек [6-8].
Учитывая, что вакцинные штаммы калицивируса применяются для производства инактивированных культуральных препаратов, но при этом отличаются друг от друга по иммунобиологическим показателям, необходимо на этапе контроля вируссодержащего материала использовать способы, которые могут быстро проводить генотипирование вакцинного штамма «Мира» от других штаммов FCV [5]. Данный этап крайне необходим при контроле качества сырья при изготовлении вакцинных препаратов и подтверждения использования требуемого штамма для изготовления вакцин против калицивироза в рамках крупномасштабного процесса.
Учитывая, что в настоящее время для изготовления вакцин широко применяется штамм «Мира» FCV, целесообразно провести разработку способа дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек. Штамм «Мира» депонирован во Всероссийской государственной коллекции экзотических типов вируса ящура и других патогенов животных ФГБУ «ВНИИЗЖ» под регистрационным номером: №483 – деп / 23-33 ГКШМ ФГБУ «ВНИИЗЖ».
Методы молекулярно-биологического анализа находят широкое применение в диагностике различных инфекционных заболеваний, в том числе FCV. С помощью анализа однонуклеотидных полиморфизмов целевого участка гена возбудителя при наличии одной или нескольких уникальных замен можно проводить дифференциацию генетических форм вирусов друг от друга даже внутри одного вида [6-15].
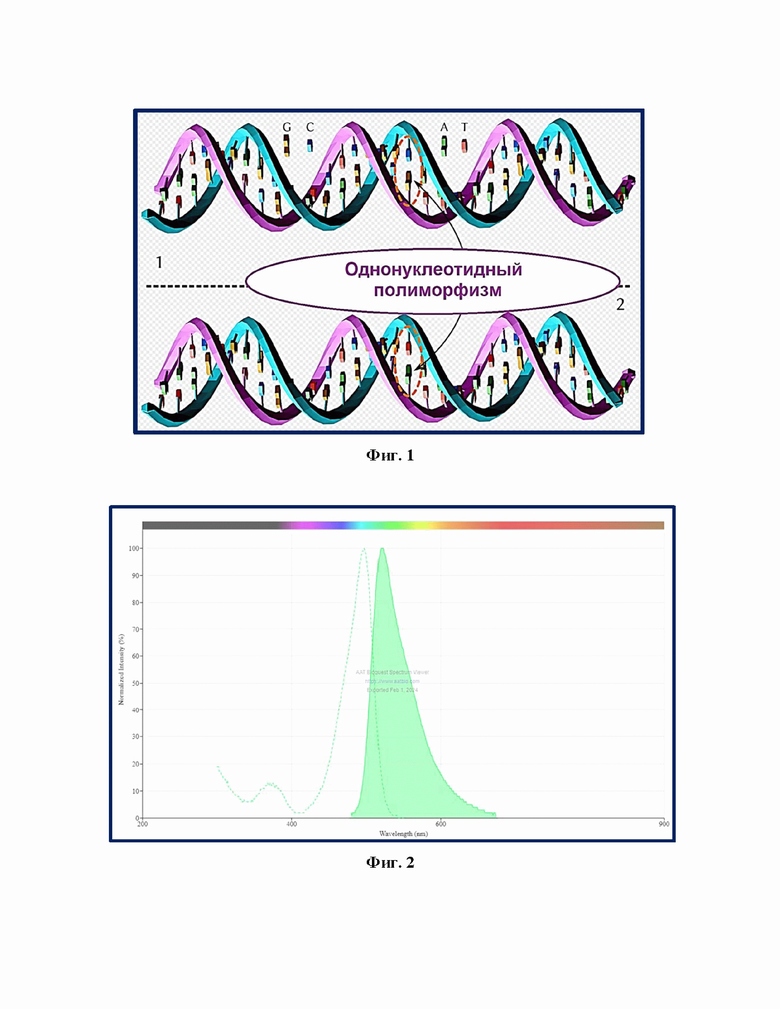
Однонуклеотидный полиморфизм представляет собой различие последовательностей ДНК в одинаковых участках генома на один нуклеотид (аденин (A), тимин (T), гуанин (G) или цитозин (С)). Если в последовательности гена имеется участок с заменой одного нуклеотида на другой, например, TAGGCT и TATGCT, то говорят о существовании двух аллелей данного гена - G-аллель и Т-аллель (фиг. 1).
Анализ максимальных значений температур плавления для различных штаммов вирусов и других микроорганизмов представляет собой исследование кривой плавления ампликонов, содержащих аллель-специфичные мутации, в широком диапазоне температур с узким шагом. Сочетание улучшенного инструментария количественной детекции ампликонов и флуоресцирующих красителей последнего поколения позволила выявлять генетические изменения в последовательностях нуклеиновых кислот путем контролируемого плавления двухцепочечных молекул кДНК [12, 14, 15].
Сущность представленного метода заключается в том, что после проведения ПЦР целевого участка гена с применением флуоресцирующего красителя смесь постепенно нагревают, и при достижении определённой температуры продукты реакции начинают разъединяться, флуорофор постепенно высвобождается, уровень флуоресценция (Fl), которая детектируется оптической системой термоциклера, снижается. Чувствительность анализа достигает одного нуклеотида по изменению пика температуры плавления кДНК, что позволяет достичь высокой общей точности разработанного способа.
Прототипным вариантом проведения дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек является секвенирование методом Фредерика Сенгера [9]. Указанный метод является дорогостоящим, трудоемким и продолжительным во времени проводимого исследования.
Задачей настоящего изобретения является разработка чувствительного и специфичного, быстрого способа дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1 с целью устранения вышеуказанных недостатков.
Данная задача решена благодаря разработке способа дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Сущность разработанного способа заключается в следующем:
1) создание дизайна высокоспецифичных олигонуклеотидных прямого и обратного праймеров для синтеза целевого фрагмента кДНК гена ORF1 с мутациями, уникальными для штамма «Мира» FCV; 2) проведение реакций обратной транскрипции и ПЦР с применением SYBR Green (табл. 3); 3) постепенное нагревание в широком диапазоне температур полученных ПЦР-продуктов. При достижении пика температуры плавления двуцепочечная молекула кДНК FCV распадается на отдельные цепи, что вызывает яркий флуоресцентный сигнал, специфичный для красителя SYBR Green. Для каждого варианта нуклеотидного состава характерна своя температура плавления. Анализируя кривые плавления ПЦР-продуктов, возможно проводить дифференциацию штамма «Мира» FCV от других штаммов калицивируса кошек.
Разработанный способ дает возможность: 1) сократить время проведения анализа до 2,5 ч; 2) применять SYBR Green, который не ингибирует реакцию и характеризующийся яркой флуоресценцией после встраивания в ПЦР-продукты, что позволяет достичь высокую точность и чувствительность анализа; 3) увеличить чувствительность и специфичность анализа за счет использования высокоспецифичных олигонуклеотидных праймеров, рассчитанных для целевого участка кДНК участка гена ORF1 FCV штамма «Мира» (размер ПЦР-продукта составляет 93 п.н.). Данная разработка позволит расширить арсенал средств для проведения контроля качества сырья при производстве вакцин против калицивироза.
Технический результат изобретения заключается в том, что разработанный способ дает возможность: 1) повысить чувствительность и специфичность за счет применения высокоспецифичных праймеров:
FCV-F291 с дизайном 5'-TCTCTGAGGACACCTCCGACGAGTTATG-3' и FCV-R359 с дизайном 5'-CCGGGTGGGACTGAGTGGATAGCGC-3', рассчитанных для амплификации ПЦР-продукта размером 93 п.н.; 2) повысить достоверность проводимого анализа благодаря применению SYBR Green; 3) с помощью анализа однонуклеотидных полиморфизмов гена ORF1 FCV и температуре плавления ПЦР-продуктов провести дифференциацию штамма «Мира» FCV от других штаммов калицивируса.
Сущность изобретения отражена на графических изображениях:
Фиг. 1 – Сущность анализа однонуклеотидных полиморфизмов.
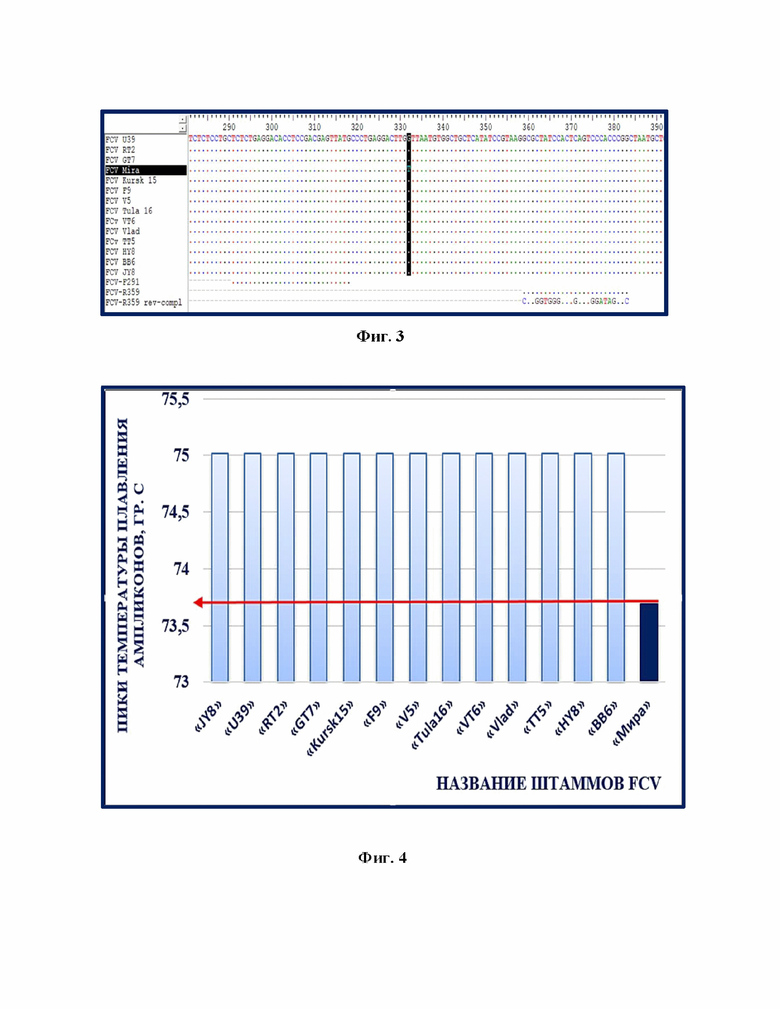
Фиг. 2 – Спектр красителя SYBR Green.
Фиг. 3 – Карта нуклеотидных замен для участка гена ORF1 FCV. Примечание: отражены нуклеотидные последовательности для следующих штаммов: «U39», «RT2», «GT7», «Mira» («Мира»), «Kursk15», «F9», «V5», «Tula16», «VT6», «Vlad», «TT5», «HY8», «BB6», «JY8», представлены последовательности прямого и обратного праймеров.
Фиг. 4 – Диаграмма максимумов температур плавления ПЦР-продуктов при анализе участка гена ORF1 FCV.
Фиг. 5 – Карта нуклеотидного состава олигонуклеотидных праймеров для амплификации участка гена ORF1 FCV. Примечание: А – для прямого праймера, Б – для обратного праймера.
Сущность изобретения пояснена в перечне последовательностей, в котором:
SEQ ID NO:1 представляет последовательность нуклеотидов гена ORF1 кДНК штамма «Мира» FCV;
SEQ ID NO:2 представляет последовательность аминокислот, кодируемых последовательностью нуклеотидов гена ORF1 кДНК FCV.
SEQ ID NO:3 представляет последовательность нуклеотидов прямого праймера FCV-F291;
SEQ ID NO:4 представляет последовательность нуклеотидов обратного праймера FCV-R359.
Заявляемый способ основан на проведении следующих этапов анализа: 1) экстрагирование РНК FCV из образца; 2) проведение обратной транскрипции и ПЦР специфического фрагмента кДНК генома FCV размером 93 п.н. с применением олигонуклеотидных высокоспецифичных праймеров FCV-F291 с дизайном 5'-TCTCTGAGGACACCTCCGACGAGTTATG-3' и FCV-R359 с дизайном 5'-CCGGGTGGGACTGAGTGGATAGCGC-3' и SYBR Green; 3) проведение дифференциации штамма «Мира» FCV от других штаммов возбудителя калицивироза с помощью анализа однонуклеотидных полиморфизмов гена ORF1 FCV.
По результатам анализа опубликованных в научной литературе данных, в настоящее время используется только прототипный способ дифференциации штаммов FCV с помощью секвенирования по Ф. Сенгеру [9], однако, как указано выше, он очень дорогостоящий, трудоемкий и продолжительный по времени проводимого исследования [10, 11]. При анализе публикаций способ дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1 не представлен. Таким образом, сведений об аналогах предлагаемого способа авторами не обнаружено.
Разработанный способ дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1 по сравнению с прототипом отличается экономической выгодой, легкостью и экспрессностью выполнения анализа, что позволяет быстро проводить анализ. SYBR Green более чувствителен своих аналогов, в частности, чем бромистый этидий, для обнаружения различных типов нуклеиновых кислот. Максимум возбуждения для красителя SYBR Green отмечается при длине волны 498 нм, а максимум излучения – при 522 нм (фиг. 2).
В отличие от прототипа разработанный способ позволяет провести анализ выравнивания множественных нуклеотидных последовательностей целевого участка кДНК генома FCV. Разработанный способ предусматривает проведение ПЦР специфического фрагмента гена ORF1 с применением олигонуклеотидных высокоспецифичных праймеров для амплификации фрагмента размером 93 п.н., дизайн которых представлен в таблице 1. Физические, термодинамические параметры и температура плавления данных олигонуклеотидных праймеров представлены в таблице 2. Разработанный способ предусматривает проведение плавления ПЦР-продуктов в представленном режиме (табл. 3) с учетов пиков температур плавления двуцепочечных кДНК и применение SYBR Green, а также детекцию результатов анализа с определением максимальных значений температур плавления для решения задачи по дифференциации штаммов штамма «Мира» FCV от других штаммов калицивирусов. Таким образом, разработан способ дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1. По результатам анализа длины, молекулярного веса и термодинамических показателей, представленных в таблице 2, видно, что олигонуклеотидные праймеры соответствуют требованиям [9, 11] и могут быть использованы в дальнейших исследованиях.
Обоснованием выбранного участка гена ORF1 возбудителя FCV является наличие уникальной аллель-специфичной мутации, характерной для штамма «Мира» FCV в сравнении с другими штаммами калицивируса, а именно в позиции 332 п.н. замена G на T (фиг. 3).
Ключевым элементом заявляемого способа является применение олигонуклеотидных праймеров FCV-F291 с дизайном 5'-TCTCTGAGGACACCTCCGACGAGTTATG-3' и FCV-R359 с дизайном 5'-CCGGGTGGGACTGAGTGGATAGCGC-3', и SYBR Green для проведения дифференциации штамма «Мира» FCV от других штаммов возбудителя калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Сопоставительный анализ с прототипом позволяет сделать вывод, что новизна и изобретательский уровень заявляемого изобретения заключаются в применении высокоспецифичных олигонуклеотидных праймеров FCV-F291 и FCV-R359, рассчитанных для амплификации целевого участка кДНК генома возбудителя FCV размером 93 п.н. и дифференциации штамма «Мира» FCV от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Сущность предлагаемого изобретения пояснена на графическом материале – «Диаграмма максимумов температур плавления ПЦР-продуктов при анализе участка гена ORF1 FCV» (фиг. 4).
На основном этапе исследования проводят выделение РНК из образцов с использованием набора «РНК-сорб» в соответствии с инструкцией производителя («Интерлабсервис», РФ).
На следующем этапе проводят обратную транскрипцию и ПЦР с применением высокоспецифичных олигонуклеотидных праймеров и SYBR Green для исследования стандартного образца и проб. Стандартным образцом является культуральная суспензия FCV штамма «Мира» FCV, которая изначально исследована с помощью секвенирования по Ф. Сенгеру для подтверждения наличия именно требуемого штамма в стандарте (n = 5). В качестве отрицательного контроля используют деионизированную воду без нуклеаз Milli Q.
Для постановки реакции готовят смесь, которая включает в свой состав следующие компоненты: PCR buffer 10х – 2,5 мкл, хлорид магния 25 мМ – 2,8 мкл, олигонуклеотидные праймеры – по 6 мкл (карта нуклеотидного состава праймеров отражена на фиг. 5), краситель SYBR Green – 2 мкл, Taq ДНК-полимераза – 1 е.а. (0,1 мкл), MMLV-ревертаза – 1 е.а. (0,1 мкл), элюат кДНК – 5 мкл, деионизированная вода – 0,5 мкл. Итоговый объем смеси для проведения одной реакции составляет 25 мкл.
Постановку ПЦР осуществляют в детектирующем термоциклере CFX-96 (Bio-Rad, США), или аналоге при температурных и временных параметрах, сведения о которых представлены в таблице 3.
Обратную транскрипцию проводят при температуре 42°С в течение 15 мин. Предварительную денатурацию проводят при температуре 95°С в течение 3 минут. ПЦР в режиме реального времени включает в себя 3 подэтапа: денатурацию, отжиг олигонуклеотидов, элонгацию. Денатурацию осуществляют при температуре 95°С в течение 10 секунд, отжиг олигонуклеотидов и элонгацию (совмещение двух этапов реакции) – при температуре 63°С в течение 35 секунд. Далее проводят плавление при температуре, начиная с 65 до 90°С. Увеличение температуры составляет - 0,1°С за каждый шаг. При этом после первого шага плавления требуется ждать 90 секунд при температуре первого шага. Для каждого последующего шага время ожидания составляет 2 секунды.
Исследование кривой плавления двуцепочечных фрагментов кДНК представленных выше генотипов возбудителя FCV для анализа данных и профилей плавления образцов выполняют с использованием программного обеспечения для сканирования с помощью детектирующего амплификатора. Проводят построение кривых плавления в виде параболы и детектируют пики температур плавления для полученных графиков. Выявляют, какие максимумы температур плавления характерны для штаммов вируса FCV, что позволяет проводить дифференциацию штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Пример 1. Разработка способа дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Для определения показателей, позволяющих проводить дифференциацию штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1, осуществляли этапы работы, представленные ниже.
На первом этапе исследования проводили выделение нуклеиновой кислоты из вируссодержащего образца, как указано выше.
На следующем этапе осуществляли обратную транскрипцию и ПЦР для исследования проб в соответствии с описанием, представленным выше. Для постановки реакции готовили реакционную смесь, как отражено выше. Проведение анализа осуществляли, как отражено выше.
Исследование профилей плавления образцов выполняли с использованием программного обеспечения CFX-96, или аналога. Проводили построение кривых плавления и детектировали максимумы температур плавления для полученных графиков. Тестировали кДНК калицивируса в 32 повторностях для каждого образца для получения результатов с высокой степенью достоверности следующих штаммов: «U39», «RT2», «GT7», «Mira» («Мира»), «Kursk15», «F9», «V5», «Tula16», «VT6», «Vlad», «TT5», «HY8», «BB6», «JY8» (табл. 4).
Выявили, что данная температура для указанных штаммов составила:
«Мира» - 73,69±0,01°С, а для «U39», «RT2», «GT7», «Kursk15», «F9», «V5», «Tula16», «VT6», «Vlad», «TT5», «HY8», «BB6», «JY8» - 75,02±0,010С (n=32, p<0,001) (фиг. 4, табл. 4).
Следовательно, для каждого из исследуемых штаммов возбудителя FCV характерны одинаковые значения пика температуры плавления. При этом значительно отличается именно штамм «Мира» FCV по анализируемому участку гена ORF1. Таким образом, разработан способ дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Пример 2. Исследование аналитической специфичности разработанного способа дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Специфичность анализа дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1 оценивали при исследовании суспензий вируса FCV штамма «Мира» и других, штамма «РВ-97» вируса бешенства, штамма «Рич» возбудителя коронавирусного энтерита собак, штамма «Rockborn» вируса чумы плотоядных животных. Количество инфекционных доз вирусов в суспензиях составлял не менее 6,0 lg ТЦД50/см3 или 6,0 lg ККИД50/см3. Исследования проводили в 5 повторностях.
В качестве положительного контроля использовали суспензию FCV штамма «Мира». Отрицательным контролем служила деионизированная вода. Для проведения исследования использовали детектирующий амплификатор марки CFX-96 (Bio-Rad, США).
В результате исследования обнаружили, что для проб, не содержащих нуклеиновую кислоту возбудителя FCV штамма «Мира», и отрицательного контроля не формировались графики плавления и не были обнаружены пиковые значения при анализе высокого разрешения. При исследовании положительного контроля получены следующие значения температур плавления для штамма «Мира» калицивируса кошек: 73,69±0,010С (n = 3, p<0,005). Результаты анализа контролей подтверждают стандартные данные, отраженные в примере 1.
Таким образом, полученные данные свидетельствовали о 100%-ной специфичности разработанного способа дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Пример 3. Определение диагностических показателей разработанного способа дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1.
Для определения диагностической чувствительности разработанного способа анализировали 388 заведомо положительных проб суспензий возбудителя FCV штамма «Мира» с титрами инфекционной активности не ниже 6,0 lg ТЦД50/см3.
Проведение анализа осуществляли, как отражено выше. С помощью разработанного способа (предлагаемое изобретение) определили, что из 388 исследуемых проб FCV все определены в качестве положительных и подтверждено наличие именно штамма «Мира» FCV, который содержался в анализируемых суспензиях.
Для исследования специфичности метода тестировали 205 суспензий возбудителя калицивируса, за исключением штамма «Мира» калицивируса кошек. В результате исследования с помощью разработанного способа (предлагаемое изобретение) определили, что все 205 проб содержали геном возбудителя FCV, но не геном штамма «Мира» калицивируса кошек. Пользуясь представленными выше статистическими методами анализа определили, что в 95%-ном доверительном интервале диагностическая чувствительность (DSe) составила 99,05-100,00%, диагностическая специфичность (DSp) – 98,22-100,0%, k-критерий – 1,000; общая точность (DAc) – 99,38-100,00% (табл. 5).
Основными преимуществами предлагаемого изобретения является возможность проводить за короткий промежуток времени (не более 2,5 ч) дифференциацию штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1. Выявили, что в целевом участке генома кДНК возбудителя калицивируса кошек имеется одна аллель-специфичная мутация в позиции 332 п.н., уникальная для штамма «Мира» калицивируса, что позволило использовать высокоспецифичные олигонуклеотидные праймеры для амплификации целевого участка кДНК генома возбудителя калицивируса размером 93 п.н. и с помощью анализа однонуклеотидных полиморфизмов гена ORF1 проводить дифференциацию.
Разработанный способ характеризуется 100%-ной аналитической специфичностью.
Источники информации, принятые во внимание при составлении описания изобретения к заявке на выдачу патента РФ на изобретение «Способ дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1»:
1. Abd-Eldaim MM, Wilkes RP, Thomas KV, Kennedy MA. Development and validation of a TaqMan real-time reverse transcription-PCR for rapid detection of feline calicivirus. Arch Virol. 2009;154(4):555-60. doi: 10.1007/s00705-009-0337-5. Epub 2009 Mar 1. PMID: 19253013; PMCID: PMC7086925.
2. Алипер Т. И., Непоклонов Е. А., Мухин А. Н. и др. Диагностика и профилактика инфекционных болезней кошек и кошек: руководство для практикующих ветеринарных врачей. Под ред. Т. И. Алипера. М.: ЗооВетКнига; 2017. 300 с.
3. Komina A, Krasnikov N, Kucheruk O, Zhukova E, Yuzhakov A, Gulyukin A. Distribution and genetic diversity of Feline calicivirus in Moscow metropolitan area. J Vet Sci. 2022 Nov;23(6):e92. doi: 10.4142/jvs.22182. PMID: 36448438; PMCID: PMC9715382.
4. Глотова Т.И., Семенова О.В., Никонова А.А., Глотов А.Г., Вяткин Ю.В., Бондарь А.А. Выделение и филогенетический анализ калицивируса кошек в Сибири. Вопросы вирусологии. 2018; 63(6):268-274. DOI: http://dx.doi.org/ 10.18821/0507-4088-2018-63-6-268-274.
5. Hou J, Sánchez-Vizcaíno F, McGahie D, Lesbros C, Almeras T, Howarth D, O'Hara V, Dawson S, Radford AD. European molecular epidemiology and strain diversity of feline calicivirus. Vet Rec. 2016 Jan 30;178(5):114-5. doi: 10.1136/vr.103446. Epub 2016 Jan 25. PMID: 26811440; PMCID: PMC4752659.
6. Рахманина М.М. Калицивирусная инфекция кошек: биологические свойства возбудителя, эпизоотология, специфические средства и методы профилактики: Автореф. диcс. … д-ра ветеринар. наук. М.; 2005.
7. Hofmann-Lehmann R, Hosie MJ, Hartmann K, Egberink H, Truyen U, Tasker S, Belák S, Boucraut-Baralon C, Frymus T, Lloret A, Marsilio F, Pennisi MG, Addie DD, Lutz H, Thiry E, Radford AD, Möstl K. Calicivirus Infection in Cats. Viruses. 2022 Apr 29;14(5):937. doi: 10.3390/v14050937. PMID: 35632680; PMCID: PMC9145992.
8. Вакцина против калицивироза, вирусного ринотрахеита и панлейкопении кошек Нобивак Tricat Trio. – URL: https://www.vetlek.ru/directions/?id=112 (Дата обращения 11.08.2023).
9. Estrada-Rivadeneyra D. Sanger sequencing. FEBS J. 2017 Dec;284(24):4174. doi: 10.1111/febs.14319. Epub 2017 Nov 24. PMID: 29171728.
10. Reid SM, Pierce KE, Mistry R, Bharya S, Dukes JP, Volpe C, Wangh LJ, King DP. Pan-serotypic detection of foot-and-mouth disease virus by RT linear-after-the-exponential PCR. Mol Cell Probes. 2010 Oct;24(5):250-5. doi: 10.1016/j.mcp.2010.04.004. Epub 2010 Apr 28. PMID: 20433917.
11. Liu YL, Ding YZ, Dai JF, Ma B, He JJ, Ma WM, Lv JL, Ma XY, Ou YW, Wang J, Liu YS, Chang HY, Wang YL, Zhang Q, Liu XT, Zhang YG, Zhang J. Development of a New RT-PCR with Multiple Primers for Detecting Southern African Territories Foot-and-mouth Disease Viruses. J Vet Res. 2018 Dec 31;62(4):431-437. doi: 10.2478/jvetres-2018-0064. PMID: 30729199; PMCID: PMC6364153.
Таблица 1
Дизайн специфичных олигонуклеотидных праймеров для дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1 (предлагаемое изобретение)
Таблица 2
Физические, термодинамические параметры и температура плавления олигонуклеотидных праймеров для дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1 (предлагаемое изобретение)
- статистический метод ближайших соседей
Примечание: термодинамические параметры разработанных олигонуклеотидных праймеров рассчитаны при условии: концентрация NaCl 1 М,
температура 250С,
водородный показатель (рН) 7,0.
Таблица 3
Временные и температурные режимы обратной транскрипции и ПЦР в режиме реального времени дифференциации штамма «Мира» FCV от других штаммов калицивируса кошек с помощью анализа однонуклеотидных полиморфизмов гена ORF1 (предлагаемое изобретение)
на шаге 1
Примечание: градиент температур составлен на этапе плавления с шагом 0,10С,
* - детекция сигнала.
Таблица 4
Средние показания температур плавления, при которых достигались пиковые значения для исследуемых ампликонов штаммов возбудителя FCV (предлагаемое изобретение)
(n = 32, p<0,001)
ампликона при использовании разработанного способа, 0С
Примечание: анализ проводили на приборе CFX-96 (Bio-Rad, США), отражены средние значения пиков температур и среднеквадратичное отклонение.
Таблица 5
Пределы диагностических возможностей способа дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1 (предлагаемое изобретение)
(95%-ный доверительный интервал)
Примечание: DSe – диагностическая чувствительность,
DSp – диагностическая специфичность,
k-критерий - индекс Каппа Коэна,
DAc - общая точность.
--->
<?xml version="1.0" encoding="ISO-8859-1"?>
<!DOCTYPE ST26SequenceListing SYSTEM "ST26SequenceListing_V1_3.dtd"
PUBLIC "-//WIPO//DTD Sequence Listing 1.3//EN">
<ST26SequenceListing productionDate="2025-02-06"
softwareVersion="2.1.2" softwareName="WIPO Sequence"
fileName="FCV-Mira-diff.xml" dtdVersion="V1_3">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2025-02-06</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>615</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2025-02-06</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБУ "Федеральный центр охраны
здоровья животных" (ФГБУ "ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin> Federal State-Financed Institution Federal
Centre for Animal Health (FGBI ARRIAH)</ApplicantNameLatin>
<InventorName languageCode="ru">Доронин Максим
Игоревич</InventorName>
<InventorNameLatin>Doronin Maksim Igorevich </InventorNameLatin>
<InventionTitle languageCode="ru">Способ дифференциации штамма «Мира»
калицивируса кошек от других штаммов с помощью анализа
однонуклеотидных полиморфизмов гена ORF1</InventionTitle>
<SequenceTotalQuantity>4</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>5289</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5289</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtctcaaactctgagcttcgtgcttaaaactcacagtgtccgaaaggact
ttgtgcactctgtcaagttaacacttgcacggaggcgcgatcttcagtatatttataacaagctctcacg
cactatacgtgctgaggcttgcccttcttgtgctagttacgacgtatgtcctaactgcacctctggtgac
gtcccggatgacgggtcttcgacaatgtcgattccatcatgggaggatgtcacaaagtcttcaacctact
ctctcctgctctctgaggacacctccgacgagttatgccctgaggacttggttaatgtggctgctcatat
ccgtaaggcgctatccactcagtcccacccggctaatgctgaaatgtgcaaagaacagctcactttctta
ttagtcatggctgaggcgatgctgccccaacgatcccgagcgtcaatcccactgcaccaacaacacacgg
ctgcacggttggaatggagggagaaattcttctctaaacctcttgactttctccttgaaagagttggtgt
gtcaaaagatattctccaaactactgcgatttggaaaattatcttggaaaaagcatgctattgtaaatcc
tatggagagcagtggttcactgctgccaaacaaaagttaagggaaatgaaaaactttgagagtgatacgc
taaaacctcttattggtggatttatagatggtctacggttcttgaccgtggacaacccaaacccgatggg
tttcctcccaaaactcatagggcttgtaaaacccctgaatttggcgatgattattgataatcatgaaaac
acaatatctggctggatcatcacattaaccgcaataatggagctatacaacatcaccgaatgcaccatag
atattataacatcagtcattacggctttctatgataaaattggcaaggcaaccaaattttacagttgtgt
taaggcgctgttcactggatttagatctgaggacgtagcgaattccttttggtacatggcagcagcgatt
ctatgttacctgatcactgggttaatcccaaacaacggcagattttcaaagataaaagcttgcctggccg
gagcgacaactcttgtatcaggtatagttgccacacaaaagctagctgcaatgttcgcaacatggaactc
tgagtccattgttaatgagttgtcagcaagaactgttgccctatcagagctaaacaatccaacaacaacg
tctgacacggattcggtagaacgactgctagaattggctaagatcttgcatgaggagatcaagattcata
ctctaaaccccatcatgcaatcatacaatccaatcttgagaaatttaatgtcaaccttggacggtgttat
aacatcatgcaacaagaggaaagctattgctcggaagagacaggtgccagtttgttacatattaactggc
ccacctggatgtgggaaaacaaccgcagctcaagcattagctaagaaattgtctgatcaagagccgtcgg
taattaaccttgatgttgatcatcatgacacatatactggaaatgaggtatgtatcattgatgagtttga
ttcgtctgacaaggtagactatgcaaattttgtgattgggatggtaaactctgctcctatggtgttaaat
tgtgacatgcttgagaacaagggaaagctcttcacctcaaagtatataattatgacttccaactctgaaa
ctccagtgaaaccctcttccaagcgcgcaggtgcattctaccgaagggttactatcatcgatgtaactaa
cccttttgtggagtcgcacaagcgtgcaaggcctgggacgtctgttcctcgtagctgctataagaaaaac
ttctcccatctctcacttgctaaacgaggggccgagtgctggtgcaaggaatacgttcttgaccctaagg
ggcttcaacaccaaagcatgaaggctccccctcctacctttcttaacattgactctttagctcagacgat
gaagcaagacttcttgctcaagaacatggcttttgaggctgaagatgggtgcgcagaacatcgatatggg
tttgtgtgtcaacaggaagaggttgaaactgttcgcaggctcctcaacgcggttagggcaaggatgaatg
ctaccttcactgtgtgtgttggacccgagacctcgcactcgattggttgcactgcgcacgtgttaactcc
caatgagacctttaatggaaagaagtttgttgtgtcacgatgtaacgaagcatcactttctgcccttgaa
ggtaactgtgtaaagtcagctttgggcgtgtgtatgtcagataaggatctcactcatttatgccacttca
ttaaagggaaaattgtcaatgacagtgttaggttggatgaactacccgccaatcagcatgtggtaaccgt
taattcggtgtttgatttggcctgggctgttcgtcgtcatctcacactggcagggcagtttcaagctatc
agagccgcatatgatgtgcttactgtccccgacaagatccctgcaatgttgcgtcactggatggatgaga
cctccttttctgatgaccacgttgtaacacaatttgtaacacctggtggcatagtcatcctggagtcttg
cggtggtgcacgcatctgggctttaggtcgcaacgtgatccgagcaggaggtgtcaccgcaaccccaacc
ggaggatgtgttaggttaatgggattatctgcgccaaccatgccatggtccgagatctttcgtgagctct
tctctctcttaggtagaatttggtcatctgtcaaagtatcagccctagtactaactgctcttggaatgta
cgcatctagatttagaccaaaatcggaagcaaaaggaaaaaccaaattgaagattgggacatacaggggt
cgcggtgtagcgctgactgatgacgagtacgatgagtggcgcgaacacaacgcctccagaaaattggatt
tgtcagtggaggatttcttaatgttgcgccatcgtgccgctctaggagccgacgacaatgatgcagtaaa
attccggtcgtggtggaactccagaaccaaaatggccaatgattatgaggatgtcaccgtaattggcaaa
ggtggcgtcaaacatgaaaagatcagaaccaataccctaaaagctgtggatcgtggttatgacgtcagct
ttgctgaggaatcaggaccaggcaccaagttccacaagaatgcaattggatctgtcacagatgtttgtgg
ggagcacaaaggctattgcatccacatgggccacggtgtttacgcatccgtagctcacgtggtgaaaggg
gattcatttttcttgggtgaaaggatttttgatcttaagactaatggtgaattttgctgctttcgcagca
cgaaaattctacctagtgctgcacctttcttttctgggaagcccactcgtgatccgtggggatcccccgt
ggcaactgagtggaagcctaaaatgtacacaacaacctctggaaagattctggggtgctttgcaacaact
tcaactgaaactcacccgggagactgtggcctcccatatattgatgacaacgggagggtgaccggcctcc
acactggctctgggggacccaaaaccccaagtgccaagttggtggtgccatatgtgcatattgacatgaa
gactaaatccgtcactgctcaaaagtatgacgtaacaaagcctgatataagttacaaaggcttaatttgt
aagcaattggatgagattaggattataccaaaaggcacacgtctccatgtctccccagcccacactgagg
attatcaagaatgctcacaccaacccgcatcacttggaagcggggatccccgctgtccaaaatctctcac
tgctatagttgttgattctctaaaaccatactgtgagaacgttgagggtcctccacatgatgttttgcac
agagttcaaaagatgcttatcgaccacctttcaggctttgtccctatgaacatttcctcggaaacctcta
tgctctcagctttccacaaactcaatcatgatacttcctgtggaccatacttgggtggcagaaagaaaga
tcacatggctaacggtgagccggacaagcagttattggatctcctgtctgcaaaatggaaattggcaacc
caaggcatagcactaccacatgagtacacaattgggctaaaggacgagttaaggcccgtggagaaggtta
gtgaagggaagagaaggatgatttggggttgtgatgttggcgtcgctactgtctgtgcagctgcgttcaa
gggtgttagcgatgccatcacagcaaaccaccagtacgggcctatacaggttggtatcaacatggatagc
cccagcgtcgaagcgctgttccaaaggatcaaaagcgcggccaaggtatttgcggtcgattattccaaat
gggattcgacccaatcgcctcgtgtcagtgcagcttcaattgacatccttcgttacttttccgatcgctc
tccaattgttgactcagcctctaacacactgaagagccctcctgttgcaatctttaatggtgttgctgta
aaagtgtcctctggcttaccatctggaatgcctcttacctcagtaatcaattcccttaatcattgtctgt
atgttgggtgtgccattcttcaatccctagaagctaaggccattcccgtcacttggaaccttttctcaac
ttttgatatcatgacttacggggatgatggtgtctacatgtttcctattatgtatgcaagtattagtgac
caaatttttggaaatctttcttcctatggcctgaaaccaactcgggttgacaagtccgttggagcaattg
agcctattgatcctgactctgttgttttcttgaagagaacaatcacaaggacacctcaggggataagggg
tttacttgatcgcagctctataataagacaattctattatattaaaggtgagaactccgatgactggaag
agccccccaaaacatattgacccaacatctcgagggcaacagctttggaatgcctgtctgtacgctagcc
aacatggcttggagtttttcaacaaggtttacaggctggccgagagggctgttgaatatgaagagctgca
ctttgaacccccaacatatgcttcggctttggatcattacaacagccagttcaatggcgtggaggcgcgg
tctgaccagatcgactcgagtggcatgaccgccctacactgtgatgtgttcgaagtt</INSDSeq_seq
uence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1763</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1763</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSQTLSFVLKTHSVRKDFVHSVKLTLARRRDLQYIYNKLSRTIRAEACPSCA
SYDVCPNCTSGDVPDDGSSTMSIPSWEDVTKSSTYSLLLSEDTSDELCPEDLVNVAAHIRKALSTQSHPA
NAEMCKEQLTFLLVMAEAMLPQRSRASIPLHQQHTAARLEWREKFFSKPLDFLLERVGVSKDILQTTAIW
KIILEKACYCKSYGEQWFTAAKQKLREMKNFESDTLKPLIGGFIDGLRFLTVDNPNPMGFLPKLIGLVKP
LNLAMIIDNHENTISGWIITLTAIMELYNITECTIDIITSVITAFYDKIGKATKFYSCVKALFTGFRSED
VANSFWYMAAAILCYLITGLIPNNGRFSKIKACLAGATTLVSGIVATQKLAAMFATWNSESIVNELSART
VALSELNNPTTTSDTDSVERLLELAKILHEEIKIHTLNPIMQSYNPILRNLMSTLDGVITSCNKRKAIAR
KRQVPVCYILTGPPGCGKTTAAQALAKKLSDQEPSVINLDVDHHDTYTGNEVCIIDEFDSSDKVDYANFV
IGMVNSAPMVLNCDMLENKGKLFTSKYIIMTSNSETPVKPSSKRAGAFYRRVTIIDVTNPFVESHKRARP
GTSVPRSCYKKNFSHLSLAKRGAECWCKEYVLDPKGLQHQSMKAPPPTFLNIDSLAQTMKQDFLLKNMAF
EAEDGCAEHRYGFVCQQEEVETVRRLLNAVRARMNATFTVCVGPETSHSIGCTAHVLTPNETFNGKKFVV
SRCNEASLSALEGNCVKSALGVCMSDKDLTHLCHFIKGKIVNDSVRLDELPANQHVVTVNSVFDLAWAVR
RHLTLAGQFQAIRAAYDVLTVPDKIPAMLRHWMDETSFSDDHVVTQFVTPGGIVILESCGGARIWALGRN
VIRAGGVTATPTGGCVRLMGLSAPTMPWSEIFRELFSLLGRIWSSVKVSALVLTALGMYASRFRPKSEAK
GKTKLKIGTYRGRGVALTDDEYDEWREHNASRKLDLSVEDFLMLRHRAALGADDNDAVKFRSWWNSRTKM
ANDYEDVTVIGKGGVKHEKIRTNTLKAVDRGYDVSFAEESGPGTKFHKNAIGSVTDVCGEHKGYCIHMGH
GVYASVAHVVKGDSFFLGERIFDLKTNGEFCCFRSTKILPSAAPFFSGKPTRDPWGSPVATEWKPKMYTT
TSGKILGCFATTSTETHPGDCGLPYIDDNGRVTGLHTGSGGPKTPSAKLVVPYVHIDMKTKSVTAQKYDV
TKPDISYKGLICKQLDEIRIIPKGTRLHVSPAHTEDYQECSHQPASLGSGDPRCPKSLTAIVVDSLKPYC
ENVEGPPHDVLHRVQKMLIDHLSGFVPMNISSETSMLSAFHKLNHDTSCGPYLGGRKKDHMANGEPDKQL
LDLLSAKWKLATQGIALPHEYTIGLKDELRPVEKVSEGKRRMIWGCDVGVATVCAAAFKGVSDAITANHQ
YGPIQVGINMDSPSVEALFQRIKSAAKVFAVDYSKWDSTQSPRVSAASIDILRYFSDRSPIVDSASNTLK
SPPVAIFNGVAVKVSSGLPSGMPLTSVINSLNHCLYVGCAILQSLEAKAIPVTWNLFSTFDIMTYGDDGV
YMFPIMYASISDQIFGNLSSYGLKPTRVDKSVGAIEPIDPDSVVFLKRTITRTPQGIRGLLDRSSIIRQF
YYIKGENSDDWKSPPKHIDPTSRGQQLWNACLYASQHGLEFFNKVYRLAERAVEYEELHFEPPTYASALD
HYNSQFNGVEARSDQIDSSGMTALHCDVFEV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>28</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..28</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tctctgaggacacctccgacgagttatg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>25</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..25</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ccgggtgggactgagtggatagcgc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к области молекулярной биологии. Описан способ дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1. Способ включает применение олигонуклеотидных праймеров, рассчитанных для амплификации ПЦР-продукта размером 93 п.н., и красителя SYBR Green, затем анализ однонуклеотидных полиморфизмов гена ORF1. Технический результат изобретения заключается в том, что разработанный способ дает возможность повысить чувствительность, специфичность и достоверность проводимого анализа. 1 з.п. ф-лы, 5 ил., 5 табл., 3 пр.
1. Способ дифференциации штамма «Мира» калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1, включающий:
- применение высокоспецифичных олигонуклеотидных праймеров FCV-F291 с дизайном 5'-TCTCTGAGGACACCTCCGACGAGTTATG-3' и FCV-R359 с дизайном 5'-CCGGGTGGGACTGAGTGGATAGCGC-3', рассчитанных для амплификации ПЦР-продукта размером 93 п.н., и красителя SYBR Green;
- анализ однонуклеотидных полиморфизмов гена ORF1, при проведении которого определено, что для штамма «Мира» калицивируса кошек пик температуры плавления составляет 73,69±0,01°С.
2. Способ по п. 1, где время проведения анализа составляет 2,5 ч, аналитическая специфичность составляет 100%, диагностическая чувствительность в 95%-ном доверительном интервале - 99,05-100,00%, диагностическая специфичность - 98,22-100,0%.
| CN 107699638 A, 16.02.2018 | |||
| Набор специфических олигодезоксирибонуклеотидных праймеров и флуоресцентно-меченого зонда к фрагменту гена ORF1 (Non structural Protein 1) РНК (кДНК) калицивируса кошек Feline calicivirus (FCV) для детекции калицивируса кошек Feline calicivirus (FCV). | 2022 |
|
RU2802065C1 |
| Глотова Т | |||
| И | |||
| и др | |||
| Распространение калицивируса среди кошек и его тропность к органам | |||
| Российский ветеринарный журнал | |||
| Мелкие домашние и дикие животные, 2013, N 4, С | |||
| Солесос | 1922 |
|
SU29A1 |
Авторы
Даты
2025-05-05—Публикация
2024-08-13—Подача