Изобретение относится к области биотехнологии и производству вакцин против калицивироза кошек, а именно к способу определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин с помощью расчета дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1.
Вирионы калицивируса кошек имеют пулевидную форму длиной 100-300 нм, диаметром 45-100 нм. На наружной поверхности вирусной частицы имеются выступы в виде шипов длиной 10-12 нм, которые прикреплены к двуслойной липидной оболочке (фиг. 1) [1, 2].
Возбудитель заболевания Feline calicivirus (FCV), относится к порядку Picornavirales, семейству Caliciviridae, роду Vesivirus. Нуклеиновая кислота вируса представлена одноцепочечной РНК (+) размером около 7690 н.о. 5'-NTR (нетранслируемый регион) соответствует 1…19 н.о., ORF1 (2С, протеаза, РНК-полимераза) - 20…5311 н.о. (размер гена - 5292 н.о.) (фиг. 2), ORF2 (структурный белок VP1) - 5314…7329 н.о., ORF3 (белок VP2 с неизвестной функцией) - 7617…7643 н.о. [3].
Кошачий калицивирус является одним из наиболее распространенных вирусных патогенов у домашних кошек во всем мире. Первое сообщение о данном заболевании относится к 1957 году, когда возбудитель был выделен из желудочно-кишечного тракта кошек в Новой Зеландии. В последующие годы калицивирус был признан причиной респираторных заболеваний у кошек, и в настоящее время ветеринарные врачи по всему миру ежедневно сталкиваются с кошками, страдающими данным заболеванием. Высокомутагенная природа калицивируса и его высокая генетическая пластичность позволяют вирусу успешно выживать в популяции кошек и представляют особую проблему с точки зрения диагностики, лечения и профилактики калицивироза кошек [2, 4].
Система мер для борьбы с калицивирозом кошек и его профилактики предусматривает иммунизацию домашних животных [5-11]. Для этой цели применяют вакцинные препараты. При изготовлении цельновирионных вакцин против данного заболевания вируссодержащее сырье до инактивации исследуют на определение концентрации важнейшего иммуногенного компонента вакцины - вирионов, каждый из которых содержит одну молекулу вирусной РНК.
Как правило, для определения концентрации вирионов FCV применяют спектрометрический анализ (FCV-SpA). Данный метод является биохимическим, количественным анализом вирионов, основанным на их способности связываться с химическими агентами, меняющими свою окраску при связывании с аминокислотами. Тест проводят путем последовательного разбавления вирусной суспензии и химического титрования [4] (прототип). Данный метод имеет серьезные недостатки: низкая чувствительность и специфичность анализа.
В связи с этим целесообразно провести поиск альтернативного способа определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин.
В настоящее время развиваются методы молекулярной биологии, в частности, варианты полимеразной цепной реакции (ПЦР) с использованием формул из алгебры и начала анализа данных реакции амплификации, в частности, расчета дифференциала второй степени для точки CP (crossing point - точка пересечения в переводе с английского языка) при амплификации таргетного участка кДНК. Разработанный способ на основе данного метода является гораздо чувствительнее и специфичнее.
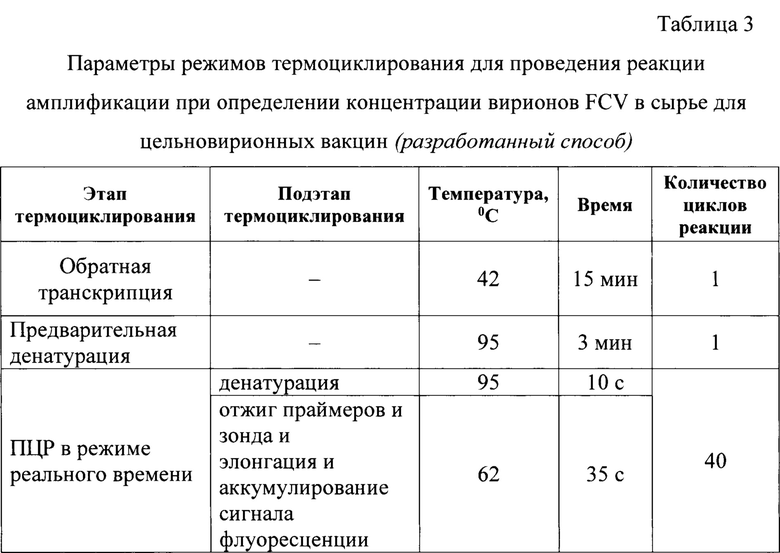
ПЦР - широко используемый метод амплификации ДНК, представляющий интерес во многих областях, таких как биомедицинские исследования, диагностическое тестирование и судебно-медицинская экспертиза. На результат ПЦР могут влиять многие факторы, такие как подготовка ДНК-матрицы и условия реакции, но разработка хорошей пары праймеров является решающим фактором. Общее требование заключается в том, что праймеры должны иметь схожие температуры плавления (Tm) и сбалансированное содержание G/C, не должны допускать самокомплементарности и структуры, напоминающей шпильку.
Задачей настоящего изобретения является разработка высокочувствительного и высокоспецифичного способа определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин с целью устранения вышеуказанных недостатков.
Данная задача решена благодаря разработке способа определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин с помощью расчета дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1.
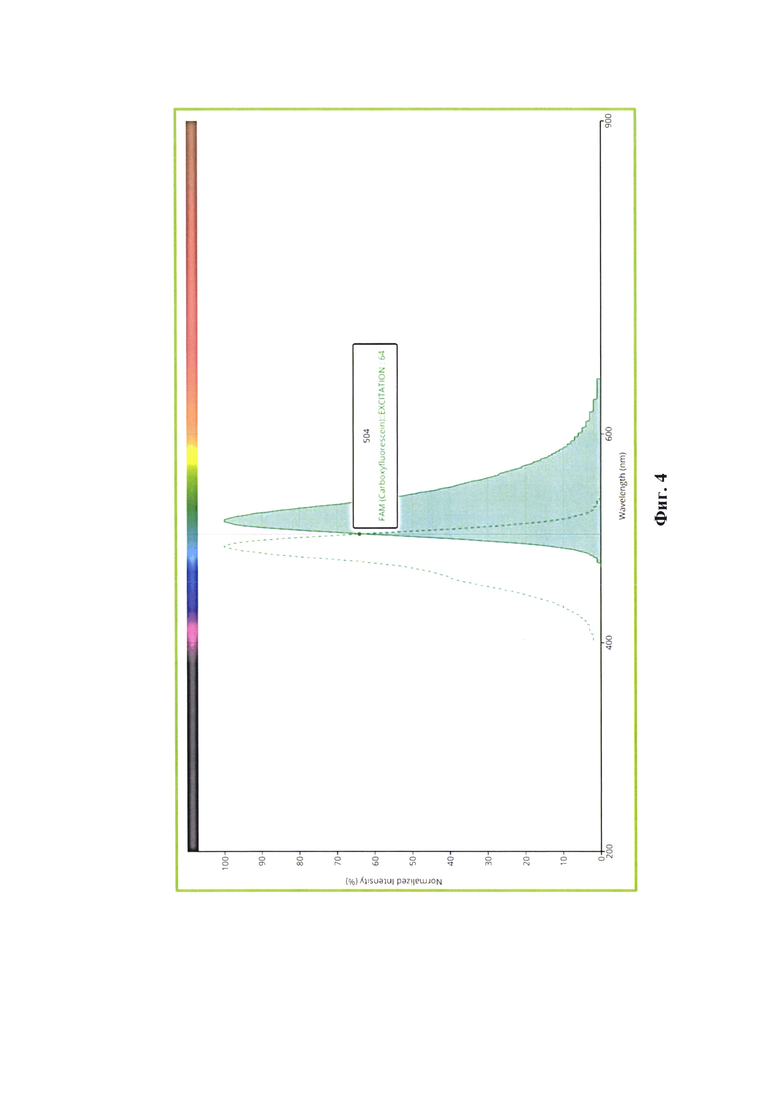
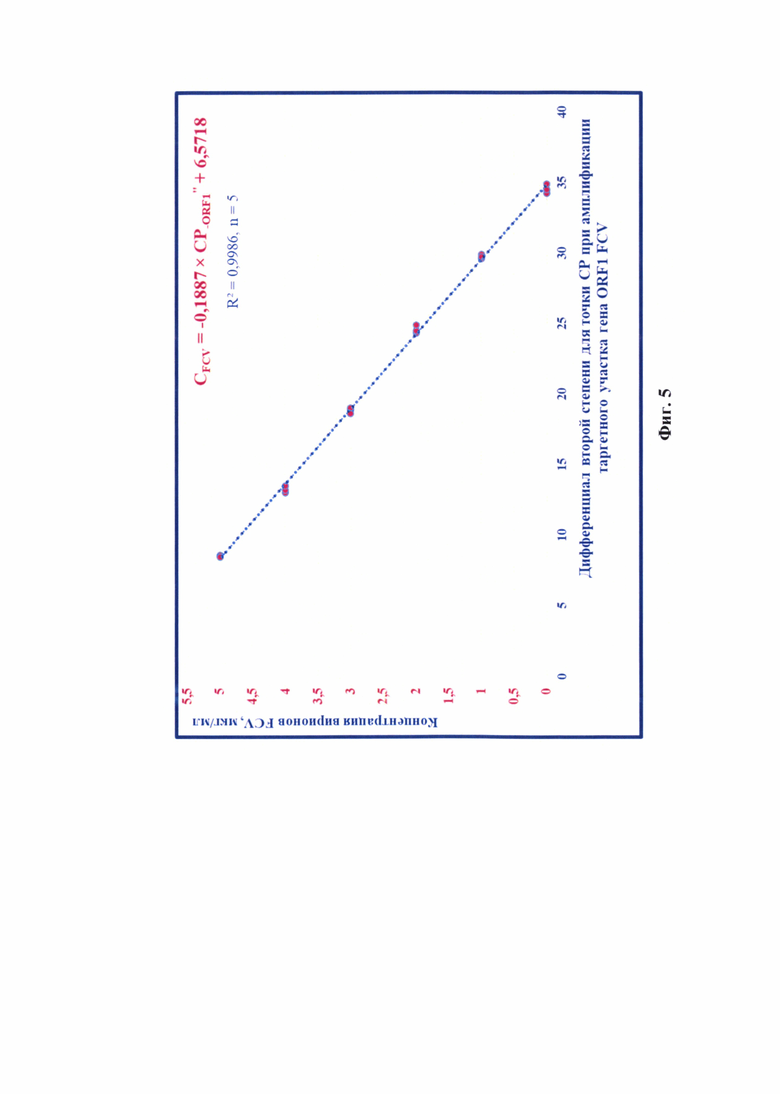
Технический результат изобретения заключается в том, что разработанный способ дает возможность: 1) повысить специфичность анализа проб за счет применения олигонуклеотидных праймеров и молекулярного зонда, рассчитанных на целевой участок гена ORF1 кДНК калицивируса кошек (фиг. 3); 2) увеличить чувствительность анализа за счет применения молекулярного зонда, конъюгированного с красителем карбоксифлуоресцеином (FAM), который представляет собой флуоресцентное соединение с пиком возбуждения при 493 нм и пиком излучения при 517 нм с точкой пересечения при 504 нм (фиг. 4), и гасителем флуоресценции GTQ1; 3) повысить достоверность проводимого анализа благодаря установлению зависимости между концентрацией вирионов FCV (CFCV) и расчета дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1 FCV (CP-ORF1ʺ), представленной в виде алгебраической формулы (фиг. 5):
CFCV=-0,1887 (CP-ORF1ʺ+6,5718)
с высокой достоверностью аппроксимации (R2=0,9986, n=5). Предложенная модель позволит опосредованно определять концентрацию вирионов калицивируса кошек в сырье для цельновирионных вакцин.
Сущность изобретения отражена на графических изображениях:
Фиг. 1 - Электронная фотография вирионов FCV.
Фиг. 2 - Модель строения гена ORF1 калицивируса кошек. Примечание: на общей модельной схеме ген ORF1 FCV обозначен «QKE71_gp1» зеленым маркером.
Фиг. 3 - Дизайн олигонуклеотидных праймеров и зонда для определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин с помощью расчета дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1. Примечание: А - позиция праймеров относительно генома FCV, отраженная с помощью программы Primer-BLAST, указана как «Primer5» красного цвета, Б - представлены прямой праймер, молекулярный зонд, обратный праймер, который дан в двух вариантах, а именно, в комплементарном прямом и в формате «rev-comp» - перевернутая комплементарная цепь с помощью программы BioEdit.
Фиг. 4 - Спектр с пиками возбуждения и излучения для флуоресцентного красителя FAM.
Фиг. 5 - График зависимости концентрации вирионов FCV (CFCV) и расчета дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1 (CP-ORF1ʺ) (n=5, р<0,005).
Сущность изобретения пояснена в перечне последовательностей, в котором:
SEQ ID NO: 1 представляет последовательность нуклеотидов гена ORF1 калицивируса кошек;
SEQ ID NO: 2 представляет последовательность аминокислот ORF1-белка калицивируса кошек;
SEQ ID NO: 3 представляет последовательность нуклеотидов прямого праймера FCV-F3145;
SEQ ID NO: 4 представляет последовательность нуклеотидов молекулярного зонда FCV-P3194 (FAM/GTQ1);
SEQ ID NO: 5 представляет последовательность нуклеотидов обратного праймера FCV-R3238.
Сущность изобретения заключается в подходе по определению концентрации вирионов FCV в неинактивированном сырье для цельновирионных вакцин. Заявляемый способ основан на получении элюата РНК, проведении реакции обратной транскрипции и амплификации таргетного участка гена ORF1 вирусной кДНК, проведении расчета дифференциала второй степени для точки CP при амплификации таргетного участка данного гена для определения по регрессионной формуле концентрации вирионов FCV в сырье для цельновирионных вакцин против калицивироза кошек.
В настоящее время применяется спектрометрический анализ (прототип), ограничения применения которого в производственном процессе отражены выше. Для решения поставленной задачи способ определения концентрации вирионов калицивируса кошек в сырье для вакцин не применялся и в мировой научной литературе не представлен.
В отличие от прототипа разработанный способ включает этапы сорбционного экстрагирования РНК, реакцию обратной транскрипции и амплификации кДНК с применением олигонуклеотидных праймеров и зонда; определение дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1 для расчета по математической формуле концентрации вирионов FCV в сырье для цельновирионных вакцин против калицивироза кошек.
Применение разработанного способа повышает достоверность анализа по определению концентрации вирионов FCV в пробах сырья для цельновирионных вакцин. Исходя из этого, актуально применять данный способ для определения концентрации вирионов калицивируса кошек в сырье для вакцин.
Ключевым элементом заявляемого способа является определение значений по оси абсцисс графика расчета дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1 и расчет по разработанной математической формуле концентрации вирионов FCV в сырье для цельновирионных вакцин против данного заболевания.
Сопоставительный анализ с прототипом позволяет сделать вывод, что новизна и изобретательский уровень заявляемого изобретения заключается в применении способа определения концентрации вирионов калицивируса кошек в сырье для вакцин.
Сведений о разработке предлагаемого способа определения концентрации вирионов калицивируса кошек в сырье для вакцин авторами не обнаружено.
На первом этапе исследования составляют контрольную панель готовых разведений стандарта с содержанием вирусной РНК, эквивалентными следующим концентрациям вирионов FCV (положительные контроли): 0,01; 1,0; 2,0; 3,0; 4,0; 5,0 мкг/мл. В рамках производственного технологического процесса наиболее часто получают суспензии с концентрациями вирионов FCV от 1 до 3 мкг/мл.
На следующем этапе анализа осуществляют выделение РНК из культуральных суспензий калицивируса кошек твердофазным сорбционным методом с применением набора «РНК-сорб» («Интерлабсервис», РФ) в соответствии с инструкцией производителя [12].
После получения экстракта РНК калицивируса кошек проводят совмещенные обратную транскрипцию и реакцию амплификации в режиме реального времени с использованием специфических олигонуклеотидных праймеров и зонда. Составляют реакционную смесь, рецептура приготовления которой представлена в таблице 1. Для разработки дизайна олигонуклеотидов использовали референтную полногеномную последовательность из GenBank с регистрационным номером М86379.1 [13].
В качестве гомологичных гену ORF1 FCV олигонуклеотидов используют:
FCV-F3145 с дизайном 5'-TGGCAAAGGTGGCGTCAAAC-3',
FCV-P3194 (FAM/GTQ1) с дизайном 5'-GCTGTGGATCGTGGTTATGACG-3',
FCV-R3238 с дизайном 5'-TGTGGAACTTGGTGCCTGGT-3((фиг.3А)
в диапазоне 3145…3257 п.н. (113 п.н.) (фиг. 3Б) в концентрации 5 пМ на реакцию. Для формирования нуклеотидных цепей продуктов реакции применяют дезоксирибонуклеозидтрифосфаты с концентрацией каждого в реакционной смеси по 2,6 мМ. В реакционную смесь добавляют 3,0 мМ хлорида магния и диметилсульфооксид в количестве 1,3% от общего объема без элюата РНК. В качестве катализатора обратной транскрипции используют фермент MMLV-ревертазу (2 е.а.), для реакции амплификации применяют Taq РНК-полимеразу (1 е.а.). Физические и термодинамические показатели для разработанных праймеров и зонда представлены в таблице 2, по которым они полностью удовлетворяют общим требованиям мировых стандартов, предъявляемых к олигонуклеотидам [12].
Объем реакционной смеси компонентов для проведения одной реакции составляет 20 мкл. Элюаты РНК каждого образца добавляют к смеси по 5 мкл. Общий объем смеси составляет 25 мкл.
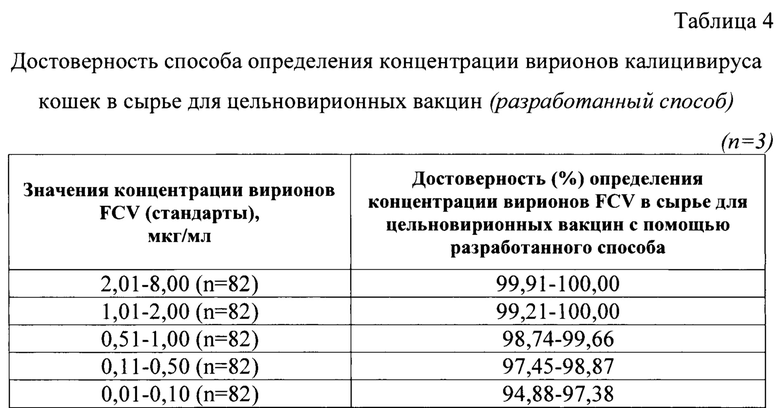
Постановку реакции осуществляют при температурных и временных параметрах, сведения о которых представлены в таблице 3. Обратную транскрипцию проводят при температуре 42°С в течение 15 минут. Предварительную денатурацию комплементарной РНК проводят при температуре 95°С за 3 мин в течение 1 цикла. Реакцию амплификации в режиме реального времени осуществляют в течение 40 циклов, каждый из которых складывается из 3 подэтапов: «денатурации», проводимой при температуре 95°С в течение 10 с, «отжига праймеров и зонда» и «элонгации и аккумулирования флуоресцентного сигнала» (совмещены для экономии времени, поскольку режимы одинаковы в данном случае) - при температуре 62°С в течение 35 с.
Результаты реакции анализируют, оценивая и сравнивая графики накопления флуоресцентного сигнала с помощью определения расчета дифференциала второй степени для точки CP при амплификации таргетного участка гена ORFT, определенных с помощью построения графика второй производной функции Fl=f×(CP-ORF1ʺ). Величина СР-ORF1(является важной характеристикой реакции, она прямо пропорциональна количеству копий исходной вирусной РНК и соответственно вирионов FCV в сырье для цельновирионных вакцин.
Используемый в данном случае метод имеет преимущество в связи с тем, что при умножении любой точки сигмоиды в экспоненциальном участке на любые множители положение наибольших значений функции не изменяется. Максимум на графике второй производной при его исследовании на максимумы располагается внутри экспоненциального участка сигмоиды, где эффективность реакции амплификации таргетного участка кДНК FCV является константой. Наибольшие значения первого дифференциала находятся, чаще всего, в зоне искажения значений эффективности амплификации, поэтому их не рекомендуется использовать для анализа. Графики третьей и четвертой производной дают менее точные результаты, поскольку их координаты во многом сопряжены с шумовыми значениями [14].
Вычислив значения CP-ORF1(для кривых, отражающих накопление флуоресцентного сигнала образцов с разными концентрациями вирионов калицивируса кошек, устанавливают зависимость между концентрацией вирионов FCV в неинактивированном сырье для цельновирионных вакцин и значением точки CP-ORF1ʺ. На основе разработанной модели рассчитывают значение концентрации вирионов FCV в неинактивированном сырье для цельновирионных вакцин.
Сущность предлагаемого изобретения пояснена примерами его использования, которые не ограничивают объем изобретения.
Пример 1. Выявление существования зависимости между концентрацией вирионов калицивируса кошек в неинактивированном сырье для вакцины и значением точки CP-ORF1ʺ.
На первом этапе исследования применяли панель готовых разведений стандарта, в качестве которого использовали не инактивированную суспензию культурального калицивируса кошек с количествами вирусной РНК, эквивалентными следующим концентрациям вирионов FCV (положительные контроли): 0,01; 1,0; 2,0; 3,0; 4,0; 5,0 мкг/мл.
Проводили выделение РНК из культуральных суспензий FCV твердофазным сорбционным методом в соответствии с процедурой, представленной выше.
Осуществляли постановку обратной транскрипции и реакции амплификации в режиме реального времени с использованием олигонуклеотидных праймеров и зонда для исследования контрольных образцов и проб. Для проведения реакции составляли реакционную смесь, рецептура которой представлена выше.
Постановку обратной транскрипции и реакции амплификации в режиме реального времени осуществляли, как описано выше. Результаты реакции анализировали, оценивая и сравнивая графики накопления флуоресцентного сигнала после определения значений точек CP-ORF1ʺ.
Полученные данные анализировали с помощью программного обеспечения «Rotor-Gene FRT-Manager», которое позволяет строить графики накопления флуоресцентного сигнала в режиме реального времени на протяжении заданного количества циклов амплификации. Применяя систему компьютерной алгебры «Maxima - GPL CAS based on DOE-MACSYMA Code» (версия 5.46.0), проводили построение графиков после вычисления расчета дифференциала второй степени для полученных элюатов РНК FCV каждого разведения стандарта с определенными значениями концентрации вирионов и рассчитывали средние значения CP-ORF1ʺ с проекцией на ось абсцисс O-CP-ORF1ʺ (n=5). При исследовании отрицательного контроля накопления флуоресцентного сигнала не наблюдалось, что подтверждает отсутствие калицивируса кошек в данном образце. В представленных исследованиях р-уровень значимости меньше 0,005, что подтверждает достоверность проводимых количественных исследований. Зависимость концентрации вирионов FCV и значений точек CP-ORF1(представлена на фиг. 4 и отражена в виде математической функции:
CFCV=-0,1887 × CP-ORF1ʺ+6,5718
с высокой достоверностью аппроксимации (R2=0,9986, n=5). Предложенная модель позволит определять концентрацию вирионов калицивируса кошек в сырье для цельновирионных вакцин.
Пример 2. Апробация способа опосредованного определения концентрации вирионов калицивируса кошек в сырье для вакцины.
В исследовании использовали 6 суспензий культурального вируса FCV со следующими концентрациями вирионов: 0,65; 1,35; 2,45; 3,12; 4,42; 4,98 мкг/мл, соответственно (пробы №1-6). В качестве положительного контроля применяли суспензию культурального вируса FCV с концентрацией вирионов 1,50 мкг/мл. В качестве отрицательных контролей применяли суспензию монослойной культуры клеток почки кошки (CrFK), не контаминированную микроорганизмами. Испытуемые пробы и контрольные образцы исследовали в пяти повторностях. Этапы выделения РНК, проведение обратной транскрипции и постановку ПЦР в режиме реального времени проводили, как описано в примере 1.
Средние значения CP-ORF1(для проб №1-6 составляли 31,33±0,01, 27,67±0,01, 21,47±0,02, 18,35±0,01, 11,40±0,01, 8,44±0,01, соответственно. Пользуясь разработанной математической функцией, представленной выше, рассчитали средние значения концентрации вирионов FCV для проб №1-6, которые составили 0,66, 1,35, 2,47, 3,11, 4,42, 4,98 мкг/мл, соответственно. Полученные значения соответствовали референтным данным. Для положительного контроля значение порогового цикла амплификации составило 26,88±0,01, что соответствовало концентрации вирионов FCV, равной 1,50 мкг/мл. Для отрицательных контролей экспоненциальные графики не были сформированы, что означало отсутствие возбудителя FCV в данных образцах. Таким образом, разработанный способ позволяет определять концентрацию вирионов FCV в неинактивированном сырье для цельновирионных вакцин.
Пример 3. Выявление степени достоверности определения концентрации вирионов калицивируса кошек в сырье для вакцин.
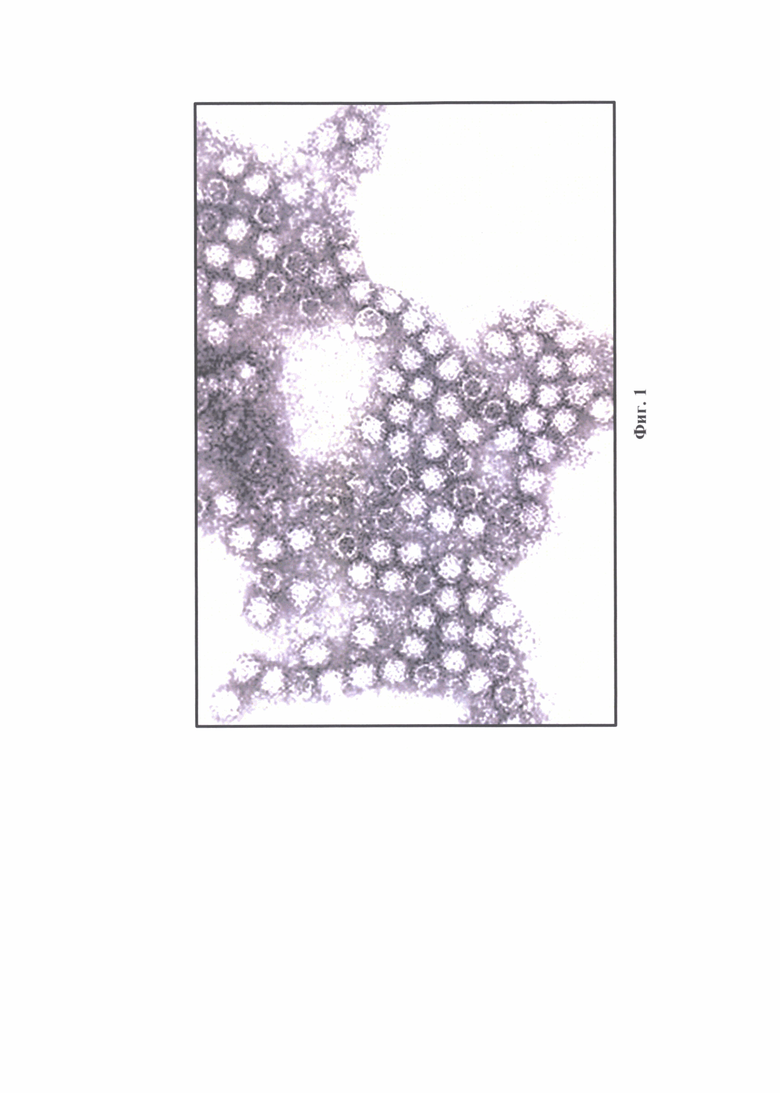
Для анализа использовали 410 суспензий культурального калицивируса кошек с концентрациями вирионов FCV от 0,01 до 5,00 мкг/мл. В качестве положительных и отрицательных контролей использовали те же образцы, которые представлены в примерах 1 и 2. Испытуемые пробы и контрольные образцы исследовали в трех повторностях. Этапы экстрагирования нуклеиновых кислот и проведение реакции амплификации в режиме реального времени осуществляли, как отражено в примере 1. Результаты анализа представлены в таблице 4.
Интерпретацию результатов проводили, пользуясь разработанной регрессионной функцией, с получением значений CP-ORF1(для каждой из 410 проб. Для положительного контроля значение порогового цикла амплификации составило 26,88±0,01, что соответствовало концентрации вирионов FCV, равной 1,50 мкг/мл. Для отрицательных контролей экспоненциальные графики не были сформированы, что означало отсутствие FCV в данных образцах.
Выявили, что данные, полученные с помощью разработанного способа, коррелировали с эталонными значениями на 99,91-100,00% для 2,01-8,00 мкг/мл (n=82), на 99,21-100,00% для 1,01-2,00 мкг/мл (n=82), на 98,74-99,66% для 0,51-1,00 (n=82), на 97,45-98,87% для 0,11-0,50 мкг/мл (n=82), на 94,88-97,38% для 0,01-0,10 (n=82). Каждый из 410 образцов исследовали в 3 повторностях. Полученные результаты свидетельствовали о высокой степени точности разработанного способа. Для концентрации 0,01 мкг/мл достоверность проводимого анализа с помощью разработанного способа составляет 94,88%, исходя из этого, аналитическая чувствительность разработанного способа составила не менее 0,10 мкг/мл с достоверностью не менее 97,38% (n=82, р<0,005). Таким образом, разработанный способ определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин является достоверным.
Пример 4. Исследование специфичности способа определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин.
При оценке специфичности способа (предлагаемое изобретение) исследовали суспензии возбудителей гепатита собак, бешенства, коронавирусной инфекции, чумы плотоядных животных и инфекционного ринотрахеита кошек. Количество инфекционных доз вирусов в суспензиях составлял не менее 6,0 lg ТЦД50/см3 или 6,0 lg ККИД50/см3. Исследования проводили в 5 повторностях.
Этапы элюирования нуклеиновых кислот и постановку реакции амплификации в режиме реального времени проводили, как описано в примере 1. Для проб, содержащих другие вирусы, не наблюдалось формирования графиков экспоненты, и они не выходили за пороговый уровень флуоресцентного сигнала (0,005 у.е.). Таким образом, разработанный способ является специфичным по отношению к возбудителю калицивироза кошек и может быть использован для определения концентрации вирионов FCV в неинактивированном сырье для цельновирионных вакцин.
Основным преимуществом предлагаемого изобретения является возможность определять концентрацию вирионов FCV в неинактивированном сырье для цельновирионных вакцин. Предлагаемое изобретение предполагает сочетание молекулярно-биологического исследования не инактивированной вирусной суспензии калицивируса кошек с методами алгебры и начала анализа данных, что повышает точность, специфичность и чувствительность способа. В предлагаемом изобретении зависимость концентрации вирионов FCV и значений точек CP-ORF1ʺ представлена в виде математической функции:
CFCV=-0,1887 (CP-ORF1ʺ+6,5718
с высокой достоверностью аппроксимации (R2=0,9986, n=5). Предложенная модель позволит определять концентрацию вирионов калицивируса кошек в сырье для цельновирионных вакцин.
Источники информации, принятые во внимание при составлении описания изобретения к заявке на выдачу патента РФ на изобретение «Способ определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин»:
1. Pesavento PA, Chang KO, Parker JS. Molecular virology of feline calicivirus. Vet Clin North Am Small Anim Pract. 2008 Jul; 38(4):775-86, vii. doi: 10.1016/j.cvsm.2008.03.002. PMID: 18501277.
2. Pedersen NC. Feline calicivirus-infectie [Feline calicivirus infection]. Tijdschr Diergeneeskd. 2005 May 1; 130(9):275-7. Dutch. PMID: 15906827.
3. Abd-Eldaim MM, Wilkes RP, Thomas KV, Kennedy MA. Development and validation of a TaqMan real-time reverse transcription-PCR for rapid detection of feline calicivirus. Arch Virol. 2009; 154(4):555-60. doi: 10.1007/s00705-009-0337-5. Epub 2009 Mar 1. PMID: 19253013; PMCID: PMC7086925.
4. Алипер Т.И., Непоклонов Е.А., Мухин А.Н. и др. Диагностика и профилактика инфекционных болезней кошек и кошек: руководство для практикующих ветеринарных врачей. Под ред. Т.И. Алипера. М.: ЗооВетКнига; 2017. 300 с.
5. Вакцина против калицивироза, вирусного ринотрахеита и панлейкопении кошек Нобивак Tricat Trio. - URL: https://www.vetlek.ru/directions/?id=112 (Дата обращения 12.10.2023)
6. Komina A, Krasnikov N, Kucheruk О, Zhukova E, Yuzhakov A, Gulyukin A. Distribution and genetic diversity of Feline calicivirus in Moscow metropolitan area. J Vet Sci. 2022 Nov; 23(6):e92. doi: 10.4142/jvs.22182. PMID: 36448438; PMCID: PMC9715382.
7. Radford AD, Dawson S, Coyne KP, Porter CJ, Gaskell RM. The challenge for the next generation of feline calicivirus vaccines. Vet Microbiol. 2006 Oct 5; 117(1):14-8. doi: 10.1016/j.vetmic.2006.04.004. Epub 2006 Apr 18. PMID: 16698199.
8. Глотова Т.И., Семенова O.B., Никонова A.A., Глотов А.Г., Вяткин Ю.В., Бондарь А.А. Выделение и филогенетический анализ калицивируса кошек в Сибири. Вопросы вирусологии. 2018; 63(6):268-274. DOI: http://dx.doi.org/10.18821/0507-4088-2018-63-6-268-274.
9. Hou J, Sanchez-Vizcaino F, McGahie D, Lesbros С, Almeras T, Howarth D, O'Hara V, Dawson S, Radford AD. European molecular epidemiology and strain diversity of feline calicivirus. Vet Rec. 2016 Jan 30; 178(5): 114-5. doi: 10.1136/vr. 103446. Epub 2016 Jan 25. PMID: 26811440; PMCID: PMC4752659.
10. Di Martino B, Lanave G, Di Profio F, Melegari I, Marsilio F, Camero M, Catella C, Capozza P, Banyai K, Barrs VR, Buonavoglia C, Martella V. Identification of feline calicivirus in cats with enteritis. Transbound Emerg Dis. 2020 Nov; 67(6):2579-2588. doi: 10.1111/tbed.13605. Epub 2020 May 14. PMID: 32359195.
11. Рахманина M.M. Калицивирусная инфекция кошек: биологические свойства возбудителя, эпизоотология, специфические средства и методы профилактики: Автореф. дисс. … д-ра ветеринар, наук. М.; 2005.
12. Sooknanan R., van Gemen В., Malek L. Nucleis acid sequence-based amplification // Molecular methods for virus detection-London: Academic press, 1995. - P. 261-285.
13. Feline calicivirus, complete genome URL: https://www.ncbi.nlm.nih.gov/nuccore/M86379.1 (Дата обращения: 12.11.2023).
14. Bar T, Kubista M, Tichopad A. Validation of kinetics similarity in qPCR. Nucleic Acids Res. 2012 Feb; 40(4): 1395-406. Doi: 10.1093/nar/gkr778. Epub 2011 Oct 19. PMID: 22013160; PMCID: PMC3287174.

--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="FCV-Cvirion.xml"
softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2024-07-31">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2024-01-07</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>566</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2024-01-07</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБУ "Федеральный центр охраны
здоровья животных" (ФГБУ "ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin> Federal State-Financed Institution Federal
Centre for Animal Health (FGBI ARRIAH)</ApplicantNameLatin>
<InventorName languageCode="ru">Доронин Максим
Игоревич</InventorName>
<InventorNameLatin>Doronin Maksim Igorevich </InventorNameLatin>
<InventionTitle languageCode="ru">Способ опосредованного определения
концентрации вирионов калицивируса кошек в сырье для цельновирионных
вакцин с помощью расчета дифференциала второй степени для точки СР
при амплификации таргетного участка гена ORF1</InventionTitle>
<SequenceTotalQuantity>5</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>5292</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5292</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtctcaaactctgagcttcgtgcttaaaactcacagtgtccgaaagg
actttgtgcactctgtcaagttaacacttgcacggaggcgcgatcttcagtatatttataacaagctctc
acgcactatacgtgctgaggcttgcccttcttgtgctagttacgacgtatgtcctaactgcacctctggt
gacgtcccggatgacgggtcttcgacaatgtcgattccatcatgggaggatgtcacaaagtcttcaacct
actctctcctgctctctgaggacacctccgacgagttatgccctgaggacttggttaatgtggctgctca
tatccgtaaggcgctatccactcagtcccacccggctaatgctgaaatgtgcaaagaacagctcactttc
ttattagtcatggctgaggcgatgctgccccaacgatcccgagcgtcaatcccactgcaccaacaacaca
cggctgcacggttggaatggagggagaaattcttctctaaacctcttgactttctccttgaaagagttgg
tgtgtcaaaagatattctccaaactactgcgatttggaaaattatcttggaaaaagcatgctattgtaaa
tcctatggagagcagtggttcactgctgccaaacaaaagttaagggaaatgaaaaactttgagagtgata
cgctaaaacctcttattggtggatttatagatggtctacggttcttgaccgtggacaacccaaacccgat
gggtttcctcccaaaactcatagggcttgtaaaacccctgaatttggcgatgattattgataatcatgaa
aacacaatatctggctggatcatcacattaaccgcaataatggagctatacaacatcaccgaatgcacca
tagatattataacatcagtcattacggctttctatgataaaattggcaaggcaaccaaattttacagttg
tgttaaggcgctgttcactggatttagatctgaggacgtagcgaattccttttggtacatggcagcagcg
attctatgttacctgatcactgggttaatcccaaacaacggcagattttcaaagataaaagcttgcctgg
ccggagcgacaactcttgtatcaggtatagttgccacacaaaagctagctgcaatgttcgcaacatggaa
ctctgagtccattgttaatgagttgtcagcaagaactgttgccctatcagagctaaacaatccaacaaca
acgtctgacacggattcggtagaacgactgctagaattggctaagatcttgcatgaggagatcaagattc
atactctaaaccccatcatgcaatcatacaatccaatcttgagaaatttaatgtcaaccttggacggtgt
tataacatcatgcaacaagaggaaagctattgctcggaagagacaggtgccagtttgttacatattaact
ggcccacctggatgtgggaaaacaaccgcagctcaagcattagctaagaaattgtctgatcaagagccgt
cggtaattaaccttgatgttgatcatcatgacacatatactggaaatgaggtatgtatcattgatgagtt
tgattcgtctgacaaggtagactatgcaaattttgtgattgggatggtaaactctgctcctatggtgtta
aattgtgacatgcttgagaacaagggaaagctcttcacctcaaagtatataattatgacttccaactctg
aaactccagtgaaaccctcttccaagcgcgcaggtgcattctaccgaagggttactatcatcgatgtaac
taacccttttgtggagtcgcacaagcgtgcaaggcctgggacgtctgttcctcgtagctgctataagaaa
aacttctcccatctctcacttgctaaacgaggggccgagtgctggtgcaaggaatacgttcttgacccta
aggggcttcaacaccaaagcatgaaggctccccctcctacctttcttaacattgactctttagctcagac
gatgaagcaagacttcttgctcaagaacatggcttttgaggctgaagatgggtgcgcagaacatcgatat
gggtttgtgtgtcaacaggaagaggttgaaactgttcgcaggctcctcaacgcggttagggcaaggatga
atgctaccttcactgtgtgtgttggacccgagacctcgcactcgattggttgcactgcgcacgtgttaac
tcccaatgagacctttaatggaaagaagtttgttgtgtcacgatgtaacgaagcatcactttctgccctt
gaaggtaactgtgtaaagtcagctttgggcgtgtgtatgtcagataaggatctcactcatttatgccact
tcattaaagggaaaattgtcaatgacagtgttaggttggatgaactacccgccaatcagcatgtggtaac
cgttaattcggtgtttgatttggcctgggctgttcgtcgtcatctcacactggcagggcagtttcaagct
atcagagccgcatatgatgtgcttactgtccccgacaagatccctgcaatgttgcgtcactggatggatg
agacctccttttctgatgaccacgttgtaacacaatttgtaacacctggtggcatagtcatcctggagtc
ttgcggtggtgcacgcatctgggctttaggtcgcaacgtgatccgagcaggaggtgtcaccgcaacccca
accggaggatgtgttaggttaatgggattatctgcgccaaccatgccatggtccgagatctttcgtgagc
tcttctctctcttaggtagaatttggtcatctgtcaaagtatcagccctagtactaactgctcttggaat
gtacgcatctagatttagaccaaaatcggaagcaaaaggaaaaaccaaattgaagattgggacatacagg
ggtcgcggtgtagcgctgactgatgacgagtacgatgagtggcgcgaacacaacgcctccagaaaattgg
atttgtcagtggaggatttcttaatgttgcgccatcgtgccgctctaggagccgacgacaatgatgcagt
aaaattccggtcgtggtggaactccagaaccaaaatggccaatgattatgaggatgtcaccgtaattggc
aaaggtggcgtcaaacatgaaaagatcagaaccaataccctaaaagctgtggatcgtggttatgacgtca
gctttgctgaggaatcaggaccaggcaccaagttccacaagaatgcaattggatctgtcacagatgtttg
tggggagcacaaaggctattgcatccacatgggccacggtgtttacgcatccgtagctcacgtggtgaaa
ggggattcatttttcttgggtgaaaggatttttgatcttaagactaatggtgaattttgctgctttcgca
gcacgaaaattctacctagtgctgcacctttcttttctgggaagcccactcgtgatccgtggggatcccc
cgtggcaactgagtggaagcctaaaatgtacacaacaacctctggaaagattctggggtgctttgcaaca
acttcaactgaaactcacccgggagactgtggcctcccatatattgatgacaacgggagggtgaccggcc
tccacactggctctgggggacccaaaaccccaagtgccaagttggtggtgccatatgtgcatattgacat
gaagactaaatccgtcactgctcaaaagtatgacgtaacaaagcctgatataagttacaaaggcttaatt
tgtaagcaattggatgagattaggattataccaaaaggcacacgtctccatgtctccccagcccacactg
aggattatcaagaatgctcacaccaacccgcatcacttggaagcggggatccccgctgtccaaaatctct
cactgctatagttgttgattctctaaaaccatactgtgagaacgttgagggtcctccacatgatgttttg
cacagagttcaaaagatgcttatcgaccacctttcaggctttgtccctatgaacatttcctcggaaacct
ctatgctctcagctttccacaaactcaatcatgatacttcctgtggaccatacttgggtggcagaaagaa
agatcacatggctaacggtgagccggacaagcagttattggatctcctgtctgcaaaatggaaattggca
acccaaggcatagcactaccacatgagtacacaattgggctaaaggacgagttaaggcccgtggagaagg
ttagtgaagggaagagaaggatgatttggggttgtgatgttggcgtcgctactgtctgtgcagctgcgtt
caagggtgttagcgatgccatcacagcaaaccaccagtacgggcctatacaggttggtatcaacatggat
agccccagcgtcgaagcgctgttccaaaggatcaaaagcgcggccaaggtatttgcggtcgattattcca
aatgggattcgacccaatcgcctcgtgtcagtgcagcttcaattgacatccttcgttacttttccgatcg
ctctccaattgttgactcagcctctaacacactgaagagccctcctgttgcaatctttaatggtgttgct
gtaaaagtgtcctctggcttaccatctggaatgcctcttacctcagtaatcaattcccttaatcattgtc
tgtatgttgggtgtgccattcttcaatccctagaagctaaggccattcccgtcacttggaaccttttctc
aacttttgatatcatgacttacggggatgatggtgtctacatgtttcctattatgtatgcaagtattagt
gaccaaatttttggaaatctttcttcctatggcctgaaaccaactcgggttgacaagtccgttggagcaa
ttgagcctattgatcctgactctgttgttttcttgaagagaacaatcacaaggacacctcaggggataag
gggtttacttgatcgcagctctataataagacaattctattatattaaaggtgagaactccgatgactgg
aagagccccccaaaacatattgacccaacatctcgagggcaacagctttggaatgcctgtctgtacgcta
gccaacatggcttggagtttttcaacaaggtttacaggctggccgagagggctgttgaatatgaagagct
gcactttgaacccccaacatatgcttcggctttggatcattacaacagccagttcaatggcgtggaggcg
cggtctgaccagatcgactcgagtggcatgaccgccctacactgtgatgtgttcgaagtttga</INSDS
eq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1764</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1764</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSQTLSFVLKTHSVRKDFVHSVKLTLARRRDLQYIYNKLSRTIRAEACP
SCASYDVCPNCTSGDVPDDGSSTMSIPSWEDVTKSSTYSLLLSEDTSDELCPEDLVNVAAHIRKALSTQS
HPANAEMCKEQLTFLLVMAEAMLPQRSRASIPLHQQHTAARLEWREKFFSKPLDFLLERVGVSKDILQTT
AIWKIILEKACYCKSYGEQWFTAAKQKLREMKNFESDTLKPLIGGFIDGLRFLTVDNPNPMGFLPKLIGL
VKPLNLAMIIDNHENTISGWIITLTAIMELYNITECTIDIITSVITAFYDKIGKATKFYSCVKALFTGFR
SEDVANSFWYMAAAILCYLITGLIPNNGRFSKIKACLAGATTLVSGIVATQKLAAMFATWNSESIVNELS
ARTVALSELNNPTTTSDTDSVERLLELAKILHEEIKIHTLNPIMQSYNPILRNLMSTLDGVITSCNKRKA
IARKRQVPVCYILTGPPGCGKTTAAQALAKKLSDQEPSVINLDVDHHDTYTGNEVCIIDEFDSSDKVDYA
NFVIGMVNSAPMVLNCDMLENKGKLFTSKYIIMTSNSETPVKPSSKRAGAFYRRVTIIDVTNPFVESHKR
ARPGTSVPRSCYKKNFSHLSLAKRGAECWCKEYVLDPKGLQHQSMKAPPPTFLNIDSLAQTMKQDFLLKN
MAFEAEDGCAEHRYGFVCQQEEVETVRRLLNAVRARMNATFTVCVGPETSHSIGCTAHVLTPNETFNGKK
FVVSRCNEASLSALEGNCVKSALGVCMSDKDLTHLCHFIKGKIVNDSVRLDELPANQHVVTVNSVFDLAW
AVRRHLTLAGQFQAIRAAYDVLTVPDKIPAMLRHWMDETSFSDDHVVTQFVTPGGIVILESCGGARIWAL
GRNVIRAGGVTATPTGGCVRLMGLSAPTMPWSEIFRELFSLLGRIWSSVKVSALVLTALGMYASRFRPKS
EAKGKTKLKIGTYRGRGVALTDDEYDEWREHNASRKLDLSVEDFLMLRHRAALGADDNDAVKFRSWWNSR
TKMANDYEDVTVIGKGGVKHEKIRTNTLKAVDRGYDVSFAEESGPGTKFHKNAIGSVTDVCGEHKGYCIH
MGHGVYASVAHVVKGDSFFLGERIFDLKTNGEFCCFRSTKILPSAAPFFSGKPTRDPWGSPVATEWKPKM
YTTTSGKILGCFATTSTETHPGDCGLPYIDDNGRVTGLHTGSGGPKTPSAKLVVPYVHIDMKTKSVTAQK
YDVTKPDISYKGLICKQLDEIRIIPKGTRLHVSPAHTEDYQECSHQPASLGSGDPRCPKSLTAIVVDSLK
PYCENVEGPPHDVLHRVQKMLIDHLSGFVPMNISSETSMLSAFHKLNHDTSCGPYLGGRKKDHMANGEPD
KQLLDLLSAKWKLATQGIALPHEYTIGLKDELRPVEKVSEGKRRMIWGCDVGVATVCAAAFKGVSDAITA
NHQYGPIQVGINMDSPSVEALFQRIKSAAKVFAVDYSKWDSTQSPRVSAASIDILRYFSDRSPIVDSASN
TLKSPPVAIFNGVAVKVSSGLPSGMPLTSVINSLNHCLYVGCAILQSLEAKAIPVTWNLFSTFDIMTYGD
DGVYMFPIMYASISDQIFGNLSSYGLKPTRVDKSVGAIEPIDPDSVVFLKRTITRTPQGIRGLLDRSSII
RQFYYIKGENSDDWKSPPKHIDPTSRGQQLWNACLYASQHGLEFFNKVYRLAERAVEYEELHFEPPTYAS
ALDHYNSQFNGVEARSDQIDSSGMTALHCDVFEVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tggcaaaggtggcgtcaaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gctgtggatcgtggttatgacg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>other DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtggaacttggtgcctggt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---


Изобретение относится к области биотехнологии. Способ определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин, включающий выделение РНК калицивируса кошек твердофазным методом; проведение реакции обратной транскрипции и амплификации таргетного участка гена ORF1 вирусной кДНК с использованием олигонуклеотидов: FCV-F3145 с дизайном 5'-TGGCAAAGGTGGCGTCAAAC-3', FCV-P3194 (FAM/GTQ1) с дизайном 5'-GCTGTGGATCGTGGTTATGACG-3', FCV-R3238 с дизайном 5'-TGTGGAACTTGGTGCCTGGT-3'. Олигонуклеотиды рассчитаны на целевой участок гена ORF1 калицивируса кошек в диапазоне 3145-3257 п.н. для амплификации фрагмента размером 113 п.н.; определение дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1 FCV, расчет концентрации вирионов FCV с помощью математической формулы. Технический результат изобретения заключается в том, что разработанный способ дает возможность повысить специфичность, увеличить чувствительность и повысить достоверность проводимого анализа. 1 з.п. ф-лы, 5 ил., 4 табл., 4 пр.
1. Способ определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин, включающий следующие этапы:
- выделение РНК калицивируса кошек твердофазным методом;
- проведение реакции обратной транскрипции и амплификации таргетного участка гена ORF1 вирусной кДНК с использованием олигонуклеотидов:
FCV-F3145 с дизайном 5'-TGGCAAAGGTGGCGTCAAAC-3',
FCV-P3194 (FAM/GTQ1) с дизайном 5'-GCTGTGGATCGTGGTTATGACG-3',
FCV-R3238 с дизайном 5'-TGTGGAACTTGGTGCCTGGT-3',
которые рассчитаны на целевой участок гена ORF1 калицивируса кошек в диапазоне 3145…3257 п.н. для амплификации фрагмента размером 113 п.н.;
- определение дифференциала второй степени для точки CP при амплификации таргетного участка гена ORF1 FCV - CP-ORF1ʺ;
- расчет концентрации вирионов FCV с помощью математической формулы
CFCV = -0,1887 × CP-ORF1ʺ + 6,5718
с высокой достоверностью аппроксимации 0,9986.
2. Способ по п. 1, где аналитическая чувствительность составляет не менее 0,1 мкг/мл с достоверностью анализа - не менее 97,38%.
| Способ опосредованного определения титра инфекционной активности калицивируса кошек в сырье для вакцины методом ПЦР в режиме реального времени | 2023 |
|
RU2809221C1 |
| Glenn M | |||
| et al | |||
| Nucleotide sequence of UK and Australian isolates of feline calicivirus (FCV) and phylogenetic analysis of FCVs | |||
| Veterinary microbiology, 1999, V.67(3), P | |||
| Ручной прибор для загибания кромок листового металла | 1921 |
|
SU175A1 |
| CN 108754019 A, 06.11.2018. | |||
Авторы
Даты
2025-04-11—Публикация
2024-08-13—Подача