Изобретение относится к области ветеринарной вирусологии и биотехнологии, может быть использовано для разработки и изготовления средств диагностики и специфической профилактики калицивирусной инфекции кошек.
Возбудитель калицивирусной инфекции кошек (калицивироза кошек) принадлежит к семейству Caliciviridae, роду Vesivirus и представляет собой безоболочечный вирус икосаэдрической формы, геном которого представлен одноцепочечной положительной РНК [1, 2]. Заболевание характеризуется поражением верхних дыхательных путей, конъюнктивитом, язвенным стоматитом, в более редких случаях артритами, пневмонией, энтеритами, отеками подкожной клетчатки и системной инфекцией с летальным исходом [3, 4]. Калицивирозом кошек болеют домашние и дикие представители семейства кошачьих [5, 6]. Восприимчивы к инфекции кошки любой породы и возраста, но тяжелее болезнь протекает у котят в возрасте до 1 года [5]. Распространенность калицивирусной инфекции в популяции кошек высока (50-90%), особенно среди животных, содержащихся в приютах и питомниках [3, 7, 8]. Вакцины против калицивирусной инфекции кошек применяются уже около пятидесяти лет, однако, калицивирус характеризуется высокой степенью изменчивости и большим антигенным разнообразием штаммов, что значительно снижает эффективность применяющихся вакцин. Изучение генетической структуры штаммов вируса калицивироза, циркулирующих на территории Российской Федерации, и использование этих штаммов при производстве вакцин, значительно облегчит разработку средств специфической профилактики, обеспечивающих надежную защиту от данной инфекции.
В настоящее время известны следующие штаммы вируса калицивироза кошек, которые применяются для диагностики калицивирусной инфекции, а также для производства вакцин против данного заболевания:
- штамм «F-9» [9];
- штамм G1 [7];
- штамм «Ларс» [10];
- штамм «Эшли» [11];
- штамм «Фауна» [12];
- штамм «Перс» [13].
Штаммы «F-9» и «G1» входят в состав импортных ассоциированных вакцин против инфекционных болезней кошек. Данные о штамме «F-9» представлены в GenBank, однако, исследования показывают, что с момента его выделения, вирус калицивироза претерпел значительные генетические изменения, и вакцины на основе штамма «F-9» (и альтернативного ему «F-255») не могут обеспечить надежную защиту от заражения новыми штаммами [14]. Кроме того, на территории РФ импортные препараты, изготовленные на основе этих штаммов имеют высокую стоимость.
Штамм калицивирусной инфекции «Эшли» запатентован в 2016 г. для изучения противовирусной активности препаратов в отношении возбудителя калицивироза кошек и не применяется для изготовления вакцин [11].
Штамм «Фауна» вируса калицивироза кошек запатентован в 2023 г. для изготовления биопрепаратов для диагностики и специфической профилактики калицивироза кошек [12].
Штамм «Перс» вируса калицивироза кошек запатентован в 2023 г. для изготовления биопрепаратов для диагностики и специфической профилактики калицивироза кошек [13].
Наиболее близким предполагаемому изобретению по совокупности существенных признаков является штамм «Ларс» калицивироза кошек, используемый для изготовления отечественной ассоциированной вакцины «Мультифел», однако, молекулярно-генетические свойства данного штамма не изучены [10].
Генетически штаммы вируса калицивирусной инфекции принадлежат к одной разнообразной геногруппе, однако, в Восточной Азии была описана вторая геногруппа. Филогенетический анализ обычно приводит к «звездообразной» филогении. Это генетическое разнообразие сопровождается антигенным разнообразием, хотя перекрестная реактивность достаточна, чтобы все изоляты считались принадлежащими к одному серотипу. При исследовании пространственного и временного распределения штаммов вируса калицивирусной инфекции была описана очень высокая генетическая и антигенная сложность штаммов вируса FCV, при этом в популяции кошек циркулирует множество различных штаммов вируса FCV, и ни один отдельный полевой штамм не преобладает в ней [3, 6].
Задача, на решение которой направлено настоящее изобретение, заключается в расширении арсенала производственных штаммов вируса калицивироза кошек, обладающих новыми биологическими и генетическими характеристиками, и пригодных для изготовления средств диагностики и специфической профилактики данного заболевания. Указанная задача решена в результате депонирования нового штамма «Мира» вируса калицивироза кошек, который может быть использован для проведения лабораторных исследований и изготовления средств вакцинопрофилактики.
Изолят вируса калицивироза кошек, послуживший источником для получения штамма «Мира» был выделен из клинического материала (смыв с ротовой полости), полученного от кота, принадлежащего частному владельцу (г. Москва), в 2022 году.
Штамм «Мира» депонирован во Всероссийской государственной коллекции экзотических типов вируса ящура и других патогенов животных (ГКШМ) ФГБУ «ВНИИЗЖ», под регистрационным номером: № 458 - деп / 23-33 - ГКШМ ФГБУ «ВНИИЗЖ».
Экспериментально подтверждена возможность использования штамма «Мира» вируса Feline calicivirus калицивироза кошек для изготовления средств диагностики и профилактики данного заболевания.
Для получения штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек, обладающего оптимальными биотехнологическими свойствами, использовали перевиваемую культуру клеток почки кошки (CRFK). В результате проведения серии последовательных пассажей на данной культуре клеток из ранее выделенного изолята был получен штамм «Мира» вируса Feline calicivirus калицивироза кошек, имеющий стабильные биотехнологические свойства и пригодный для использования при проведении лабораторных исследований, изготовлении диагностических препаратов и препаратов специфической профилактики данного заболевания. Штамм адаптирован к перевиваемой культуре клеток почки кошки (CRFK), что позволяет в промышленных масштабах получать вирусный материал штамма «Мира» вируса Feline calicivirus калицивироза кошек с титрами инфекционной активности от 6,5 до 7,5 lg ТЦД50/см3.
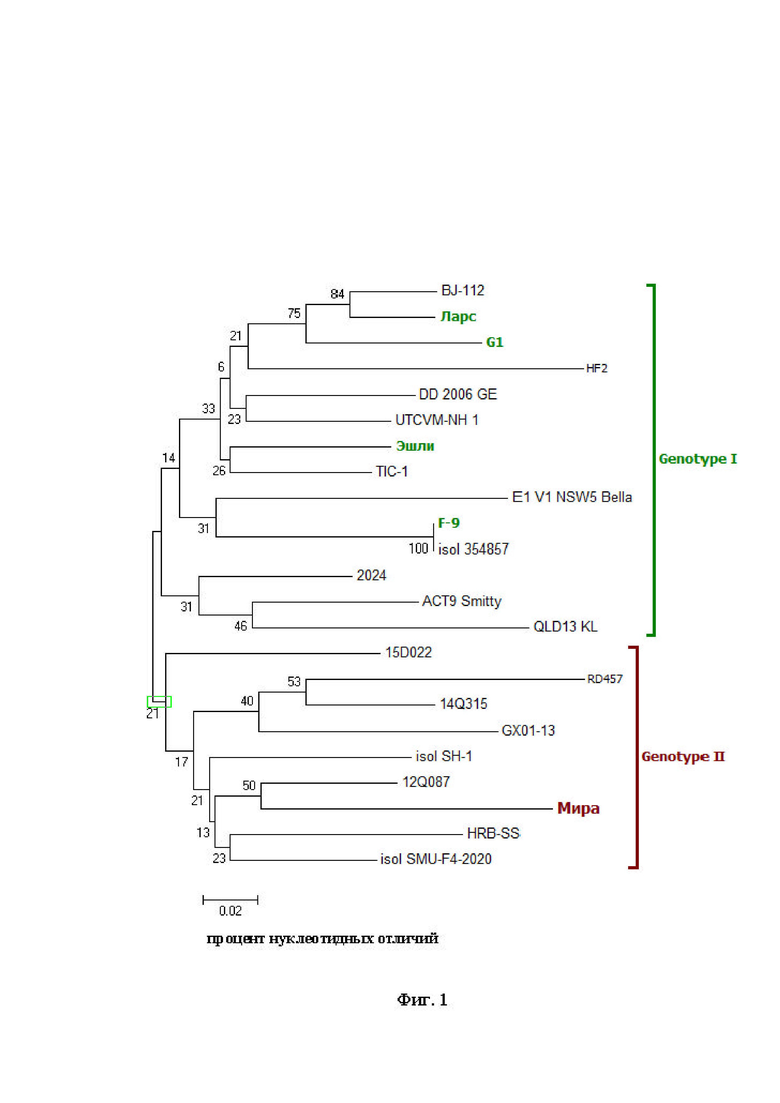
Идентификация и филогенетическое родство штамма «Мира» калицивируса кошек.
Проводили генетическую идентификацию полученного вируса и сравнительный анализ последовательности кДНК. Использовали обратную транскрипцию и полимеразную цепную реакцию (ОТ-ПЦР). Для идентификации и филогенетического анализа осуществляли секвенирование.
Редактор выравнивания последовательностей BioEdit использовался для анализа необработанных последовательностей. Выравнивания, содержащие полногеномные последовательности, были построены с использованием программы Clustal_W. Эволюционная история была выведена с использованием критерия максимального правдоподобия, основанного на 3 параметрах классической модели Tamura. Дерево было нарисовано в масштабе, с длиной ветвей, измеренной в количестве замен на сайт. Эволюционный анализ был проведен в MEGA6. Выравнивание аминокислотных последовательностей проводили с использованием Clustal X.
Дендрограмма основана на сравнении нуклеотидных последовательностей генных областей ORF2 (F-область, белок VP1) и ORF3 (белок VP2) кДНК вируса Feline calicivirus калицивироза кошек (FСV) (был взят участок длиной 324 п.н. - позиции в геноме 7253…7576 п.н.). Штамм «Мира» имеет большое количество нуклеотидных отличий от других заявленных штаммов, при этом относится к геногруппе II (Фиг. 1).
Сущность изобретения отражена на графических изображениях:
Фиг. 1 - Филогенетическое древо для вируса Feline calicivirus калицивирусной инфекции кошек штамма «Мира».
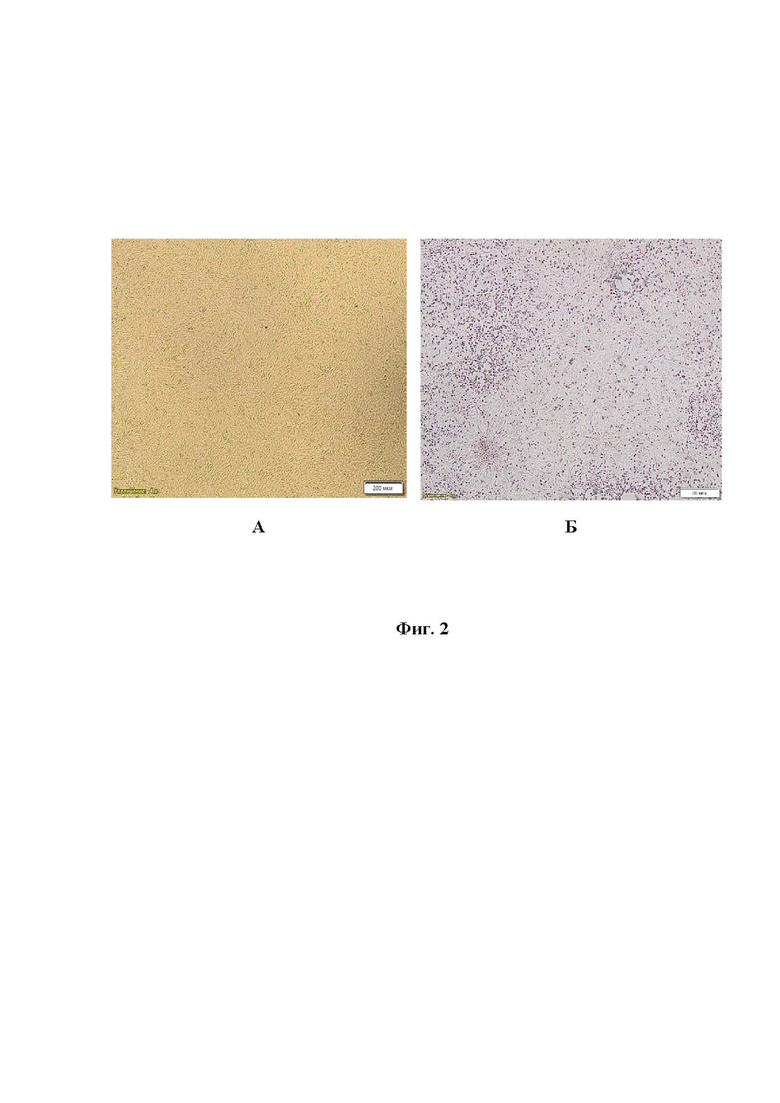
Фиг. 2 - Состояние монослоя клеточной линии CRFK до (А) и после (Б) репродукции вируса Feline calicivirus калицивирусной инфекции кошек штамма «Мира».
Сущность изобретения пояснена в перечне последовательностей, в котором:
SEQ ID NO:1 представляет последовательность нуклеотидов кДНК вируса Feline calicivirus калицивирусной инфекции кошек штамма «Мира»;
SEQ ID NO:2 представляет последовательность аминокислот белков вируса Feline calicivirus калицивирусной инфекции кошек штамма «Мира».
Штамм «Мира» вируса Feline calicivirus калицивирусной инфекции кошек характеризуется следующими признаками и свойствами.
Морфологические признаки.
Штамм «Мира» вируса калицивирусной инфекции кошек относится к семейству Caliciviridae, роду Vesivirus, виду Feline calicivirus и обладает морфологическими признаками, характерными для представителей калицивирусов: диаметр вирионов составляет 37-40 нм, вирионы без суперкапсидной оболочки, кубического типа симметрии, состоят из 32 капсомеров.
Антигенные свойства.
Все циркулирующие штаммы калицивируса представляют собой единый серотип. У переболевших кошек в сыворотках крови образуются антитела, выявляемые в реакции нейтрализации (РН). Вирус стабильно нейтрализуется гомологичной антисывороткой в РН в культуре клеток CRFK. Инокуляция в организм животного штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек сопровождается образованием вируснейтрализующих антител в крови в титре от 3,00±0,52 log2 SN50 у морских свинок.
Устойчивость к внешним факторам.
Вирус достаточно устойчив во внешней среде. На поверхностях при температуре (22±2)°С сохраняет активность в течение 24 ч, а при температуре 10°С и ниже - в течение 30 дней. Относительно стабилен в кислой среде при pH 4,0. Практически не чувствителен к эфиру, хлороформу. Чувствителен к формальдегиду, глутаровому альдегиду, хлорсодержащим дезинфицирующим средствам. Переносит двукратное размораживание и оттаивание.
Дополнительные признаки и свойства:
Патогенность - патогенен для естественно-восприимчивых животных.
Вирулентность - вирулентен для естественно-восприимчивых животных при интраназальном заражении.
Безвреден для кроликов, морских свинок и белых мышей при внутримышечном и подкожном заражении.
Стабильность - сохраняет исходные биологические свойства при пассировании в чувствительных биологических системах в течение 5 пассажей (срок наблюдения) в перевиваемой культуре клеток CRFK.
Контаминация бактериями, грибами, микоплазмами и посторонними вирусами - штамм «Мира» не контаминирован бактериями, грибами, микоплазмами и посторонними вирусами.
Условия хранения.
При хранении штамма в нативном состоянии при температуре -70±5°С допустимая длительность хранения без освежения составляет 12 мес., а при хранении в лиофилизированном состоянии при той же температуре - 10 лет.
Биотехнологические характеристики.
Штамм «Мира» вируса Feline calicivirus калицивирусной инфекции кошек репродуцируется в перевиваемой культуре клеток почки кошки (CRFK). Репродукция вируса в культуре клеток CRFK сопровождается специфическим цитопатическим действием, приводящим к округлению клеток и деструкции монослоя через 24-36 ч культивирования. Биологические свойства характеризовали путем определения инфекционной активности вируса каждого пассажа в перевиваемой культуре клеток CRFK. При культивировании вирус штамма «Мира» накапливается в титре не менее 6,50±0,25 lg ТЦД50/см3 и сохраняет исходные характеристики при пассировании в течение не менее 5 пассажей (срок наблюдения).
Гено- и хемотаксономические характеристики.
Штамм «Мира» калицивируса кошек относится к семейству Caliciviridae, роду Vesivirus, виду Feline calicivirus (FCV). Геном представляет собой 7700 н.о. одноцепочечной РНК положительной полярности с тремя открытыми рамками считывания (ORF).
5’-NTR (нетранслируемый регион) соответствует 1…19 н.о., ORF1 (ему соответствует продукт, который называется polyprotein precursor - предшественник полипротеина, из которого формируются 2C, протеаза, РНК-полимераза) - 20…5310 н.о., ORF2 (структурный белок VP1) - 5311…7320 н.о., ORF3 (белок VP2 с неизвестной функцией) - 7321…7637 н.о. ORF2 делится на шесть областей (A-F) и включает в себя различные нуклеотидные замены. Геномные области A, B, D и F в значительной степени консервативны, в то время как области C и E более изменчивы. Рекомбинация является распространенным механизмом эволюции FCV. Большинство зарегистрированных событий рекомбинации в геноме калицивирусов происходят в ORF1, ORF2. Область F ORF2 (7253-7329 п.н. и начальная область ORF3 (7465-7524 п.н.) консервативны и, следовательно, удобны для точной идентификации FCV. При этом большинство зарегистрированных событий рекомбинации в геноме FCV происходят в генном участке капсидной (ORF2) области, исходя из этого, для проведения филогенетического анализа использовали именно этот генный участок (5310…5674 н.о.).
Сущность предлагаемого изобретения пояснена примерами его использования, которые не ограничивают объем изобретения.
Пример 1. Биологические и вирусологические исследования штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек.
Биологические и вирусологические методы включали адаптацию вируса, выделенного из патологического материала, полученного от заболевшего кота, к культуре клеток CRFK. Для выделения вируса, вирусную суспензию вносили на освобожденный от ростовой среды монослой перевиваемой культуры клеток CRFK и экспонировали в течение 60 мин. при температуре (37,0±0,5)°С. Затем вносили поддерживающую среду ПСС с добавлением 2% фетальной сыворотки крови КРС и антибиотиков (стрептомицин 100 мкг/см3 и пенициллин 100 ЕД/см3). Инфицированную культуру инкубировали при температуре (37,0±0,5)°С до появления специфического цитопатического действия (ЦПД) через 12-16 ч, которое характеризовалось округлением клеток и деструкцией монослоя. При поражении не менее 80% площади монослоя (24-36 ч) культуральные флаконы промораживали при температуре минус (45,0±5,0)°С и после оттаивания при комнатной температуре производили сбор вируса с последующим отбором проб для исключения микробной контаминации и определения инфекционной активности вируса методом титрования в культуре клеток CRFK. Клеточный монослой до действия вируса представлен на фиг. 2А, после формирования ЦПД - на фиг. 2Б. Титрование вируса проводилось микрометодом по общепринятой методике в культуре клеток CRFK в 96-луночных планшетах. Инфекционная активность вируса в культуре клеток CRFK при репродукции в течение 5 пассажей составила 7,22±0,44 lg ТЦД50/см3. Полученные данные, свидетельствуют о хорошей адаптационной активности штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек к данной клеточной культуре. Первое проявление ЦПД вируса в культуре клеток CRFK наблюдается через 12-16 ч культивирования. Через 24-36 ч культивирования большая часть клеток округлялась, собиралась в агрегаты и отслаивалась от субстрата.
Пример 2. Контроль стабильности биологической активности штамма «Мира» вируса калицивирусной инфекции кошек.
Контроль стабильности биологической активности штамма определяли в течение 5 последовательных пассажей в культуре клеток CRFK. Результаты изучения стабильности штамма по его биологической активности в течение 5 пассажей представлены в таблице 1. В ходе проведенных исследований установлено, что штамм «Мира» вируса Feline calicivirus калицивирусной инфекции кошек в течение 5 последовательных пассажей в культуре клеток CRFK проявлял стабильную биологическую активность, которая находилась в пределах от 6,50±0,25 lg ТДЦ50/см3 до 7,50±0,25 lg ТДЦ50/см3.
Пример 3. Получение антигена вируса калицивирусной инфекции кошек штамма «Мира».
Для приготовления антигена в культуру клеток CRFK, выращенную в культуральных флаконах с площадью рабочей поверхности 300 см2, предварительно слив с них ростовую среду, вносили вируссодержащий материал в дозе 0,001 ТЦД50/кл. Флаконы помещали на один час в термостат при температуре (37,0±0,5)°С для контакта клеток культуры с вирусом. После этого вносили поддерживающую среду ПСС с добавлением 2% сыворотки крови КРС. Инфицированную культуру инкубировали при (37,0±0,5)°С до появления ЦПД вируса. При поражении площади монослоя не менее 80% культуральные флаконы подвергали замораживанию при температуре минус (45,0±0,5)°С. Стерильный инфицированный монослой дезагрегировали с рабочей поверхности флаконов путем его разморозки при температуре (20±2)°С и периодического встряхивания. По окончании разморозки интенсивным встряхиванием остатки монослоя удаляли с рабочей поверхности, затем соблюдая условия асептики инфицированную суспензию собирали в общую емкость. Из собранного материала отбирали пробу для контроля. Контроль вируссодержащего материала штамма «Мира» вируса калицивироза кошек осуществляли по показателю отсутствия контаминации грибами, бактериями и микоплазмами. Контроль контаминации грибами и бактериями вируссодержащего материала штамма «Мира» вируса калицивирусной инфекции кошек проводили по ГОСТ 28085 [15]. Контроль контаминации микоплазмами осуществляли в соответствии с требованиями ГФ XIV, том II, с. 2997-3008 (ОФС.1.7.2.0031.15) [16]. Нативный вируссодержащий материал с активностью не ниже 6,5 lg ТЦД50/см3 хранили при температуре не выше минус (20±2)°С и использовали для изготовления вакцинных и диагностических препаратов.
Пример 4. Получение гипериммунной сыворотки против антигена вируса калицивирусной инфекции кошек штамма «Мира».
Штамм «Мира» вируса Feline calicivirus калицивироза кошек использовали для получения высокоактивной гипериммунной сыворотки, предназначенной для оценки иммунобиологических свойств вируса калицивироза кошек. Для гипериммунизации животных применяли антиген вируса калицивирусной инфекции кошек штамма «Мира», полученный по методике, описанной в примере 3. Кроликов в количестве 4 голов иммунизировали в дозе 2,0 см3 внутримышечно двукратно через 21 день, отбор крови осуществляли на 14 сутки после последней иммунизации. Полученную сыворотку крови исследовали на наличие антител против антигена вируса калицивирусной инфекции кошек штамма «Мира». Титр антител определяли в реакции нейтрализации микрометодом (РМН). Данные, представленные в таблице 2, показывают, что получены диагностические сыворотки со специфической активностью 7,25-8,25 log2 SN50 в РМН.
Пример 5. Изучение антигенной активности штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек на морских свинках.
Для изучения антигенной активности штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек использовали вируссодержащий материал с биологической активностью равной 7,0 lg ТЦД50/см3, полученный по методике, описанной в примере 3. Подопытным морским свинкам в количестве 5 голов вводили вируссодержащий материал в область холки в дозе 1,0 см3 однократно. Морских свинок в количестве 5 голов оставили в качестве контроля. Титр антител к антигену вируса калицивироза кошек в сыворотках крови морских свинок определяли через 21 сутки после иммунизации в РМН по общепринятой методике.
Результаты исследований антигенной активности штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек показывают, что титр антител у иммунизированных морских свинок составил 3,00±0,52 log2 SN50. У контрольных животных антител к вирусу калицивирусной инфекции кошек не выявлено.
Пример 6. Определение патогенности для кошек штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек.
Патогенность штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек изучали на естественно восприимчивых животных (беспородные котята в возрасте 2-3 мес.). Животных разделили на две группы по 4 головы в каждой - подопытную и контрольную. Подопытным котятам инокулировали интраназально вируссодержащую культуральную жидкость штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек в объеме 1,0 см3 с титром инфекционной активности 7,0 lg ТЦД50/см3. Животным контрольной группы инокулировали интраназально 0,9% раствор хлорида натрия в объеме 1,0 см3. На 3-4 сутки после заражения у 3 из 4 животных подопытной группы наблюдали характерные клинические признаки калицивироза кошек: повышение температуры тела, отказ от корма, слюнотечение, катаральный ринит, язвенные поражения на слизистой ротовой полости и языка. Гибели животных не наблюдалось. Специфичность заболевания животных подтверждена исследованиями биоматериала методом ПЦР.
Таким образом, штамм «Мира» вируса Feline calicivirus калицивирусной инфекции кошек, полученный в соответствии с предлагаемым изобретением, обладает высокой биологической, антигенной активностью, пригоден для изготовления инактивированных вакцин, определения их иммуногенной активности, а также диагностических целей, и расширяет арсенал новых отечественных производственных штаммов вируса Feline calicivirus калицивироза кошек.
Источники информации, принятые во внимание при составлении описания изобретения к заявке на выдачу патента Российской Федерации на изобретение «Штамм «Мира» вируса Feline calicivirus калицивирусной инфекции кошек для изготовления биопрепаратов для диагностики и специфической профилактики калицивирусной инфекции кошек».
1.Guo J., Ding Y., Sun F., Zhou H., He P., Chen J., Guo J., Zeng H., Long J., Wei Z., Ouyang K., Huang W., Chen Y. Co-circulation and evolution of genogroups I and II of respiratory and enteric feline calicivirus isolates in cats. Transbound Emerg Dis. 2022 Sep;69(5):2924-2937. doi: 10.1111/tbed.14447. Epub 2022 Jan 17. PMID: 34982847; PMCID: PMC9787975.
2. Palombieri A., Sarchese V., Giordano M.V., Fruci P., Crisi P.E., Aste G., Bongiovanni L., Rinaldi V., Sposato A., Camero M., Lanave G., Martella V., Marsilio F., Di Martino B., Di Profio F. Detection and Characterization of Feline Calicivirus Associated with Paw and Mouth Disease. Animals (Basel). 2022 Dec 23;13(1):65. doi: 10.3390/ani13010065. PMID: 36611675; PMCID: PMC9818015.
3. Hofmann-Lehmann R., Hosie M.J., Hartmann K., Egberink H., Truyen U., Tasker S., Belák S., Boucraut-Baralon C., Frymus T., Lloret A., Marsilio F., Pennisi M.G., Addie D.D., Lutz H., Thiry E., Radford A.D., Möstl K. Calicivirus Infection in Cats. Viruses. 2022 Apr 29;14(5):937. doi: 10.3390/v14050937. PMID: 35632680; PMCID: PMC9145992.
4. Алипер Т. И., Непоклонов Е. А., Мухин А. Н. и др. Диагностика и профилактика инфекционных болезней собак и кошек: руководство для практикующих ветеринарных врачей. Под ред. Т. И. Алипера. М.: ЗооВетКнига; 2017. 300 с.
5.Рахманина М.М. Калицивирусная инфекция кошек: биологические свойства возбудителя, эпизоотология, специфические средства и методы профилактики: Автореф. диcс. … д-ра ветеринар. наук. М.; 2005.
6. Komina A., Krasnikov N., Kucheruk O., Zhukova E., Yuzhakov A., Gulyukin A. Distribution and genetic diversity of Feline calicivirus in Moscow metropolitan area. J Vet Sci. 2022 Nov;23(6):e92. doi: 10.4142/jvs.22182. PMID: 36448438; PMCID: PMC9715382.
7. Hou J., Sánchez-Vizcaíno F., McGahie D., Lesbros C., Almeras T., Howarth D., O'Hara V., Dawson S., Radford A.D. European molecular epidemiology and strain diversity of feline calicivirus. Vet Rec. 2016 Jan 30;178(5):114-5. doi: 10.1136/vr.103446. Epub 2016 Jan 25. PMID: 26811440; PMCID: PMC4752659.
8. Bergmann M., Speck S., Rieger A., Truyen U., Hartmann K. Antibody Response to Feline Calicivirus Vaccination in Healthy Adult Cats. Viruses. 2019 Jul 31;11(8):702. doi: 10.3390/v11080702. PMID: 31370359; PMCID: PMC6723298.
9. Вакцина против калицивироза, вирусного ринотрахеита и панлейкопении кошек Нобивак Tricat Trio. - URL: https://www.vetlek.ru/directions/?id=112 (дата обращения 29.05.23 г.).
10. Пат. 2 184 567 Российская Федерация, МПК A61K 39/125(2006.01), C12N 7/00(2006.01). Штамм feline calicivirus, используемый для контроля иммуногенной активности вакцин, изготовления специфических лечебно-профилактических и диагностических биопрепаратов [Текст]/ Рахманина М.М., Элизбарашвили Э.И., Уласов В.И.; заявитель и патентообладатель ФГБУ «ВГНКИ». - № 2001113087/13; заявл. 2001.05.16; опубл. 2002.07.10.
11. Пат. 2 628 196 Российская Федерация, МПК A61K 39/125(2006.01), C12N 7/00(2006.01). Штамм Эшли вируса калицивироза кошек для изучения противовирусной активности препаратов в отношении калицивироза кошек [Текст]/ Глотова Т.И., Семенова О.Э., Глотов А.Г.; заявитель и патентообладатель ФГБУ «ВГНКИ». - № 2016121721; заявл. 2016.06.01; опубл. 2017.08.14.
12. Пат. 2 804 623 Российская Федерация, МПК A61K 39/125(2006.01), C12N 7/00(2006.01). Штамм «Фауна» вируса калицивироза кошек Feline calicivirus для изготовления биопрепаратов для диагностики и специфической профилактики калицивироза кошек [Текст]/ Галкина Т.С., Комарова А.А., Доронин М.И., Киселев А.М.; заявитель и патентообладатель ФГБУ «ВНИИЗЖ». - № 2023113417; заявл. 2023.05.23; опубл. 2023.10.03.
13. Пат. 2 804 639 Российская Федерация, МПК A61K 39/125(2006.01), C12N 7/00(2006.01). Штамм «Перс» вируса Feline calicivirus калицивирусной инфекции кошек для изготовления биопрепаратов для диагностики и специфической профилактики калицивирусной инфекции кошек [Текст]/ Галкина Т.С., Комарова А.А., Доронин М.И.; заявитель и патентообладатель ФГБУ «ВНИИЗЖ». - № 2023112435; заявл. 2023.05.11; опубл. 2023.10.03.
14. Глотова Т.И., Семенова О.В., Никонова А.А., Глотов А.Г., Вяткин Ю.В., Бондарь А.А. Выделение и филогенетический анализ калицивируса кошек в Сибири. Вопросы вирусологии. 2018; 63(6):268-274. DOI: http://dx.doi.org/ 10.18821/0507-4088-2018-63-6-268-274.
15. ГОСТ 28085-13 «Средства лекарственные биологические для ветеринарного применения. Метод бактериологического контроля стерильности».
16. ГФ ХIV, т. 2, с. 2997-3008. Испытания на присутствие микоплазм (ОФС.1.7.2.0031.15).
Таблица 1
Стабильность штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек при репродукции в культуре клеток CRFK
(n=3, p<0,005)
вируса
Таблица 2
Специфическая активность штамма «Мира» вируса Feline calicivirus калицивирусной инфекции кошек
(n=3, p<0,005)
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="FCV Mira.xml"
softwareName="WIPO Sequence" softwareVersion="2.1.2"
productionDate="2023-06-17">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-06-17</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>514</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-06-17</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantNamelanguageCode="ru">ФГБУ"Федеральный центр охраны
здоровья животных " (ФГБУ"ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin>FGBI "ARRIAH"</ApplicantNameLatin>
<InventorNamelanguageCode="ru">ГалкинаТатьяна
Сергеевна</InventorName>
<InventorNameLatin>Galkina Tatiana Sergeevna</InventorNameLatin>
<InventionTitlelanguageCode="ru">Штамм «Мира» вируса Feline
Calicivirus калицивирусной инфекции кошек для изготовления
биопрепаратов для диагностики и специфической профилактики
калицивируснойинфекциикошек</InventionTitle>
<SequenceTotalQuantity>2</SequenceTotalQuantity>
<SequenceDatasequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>5289</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5289</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atgtctcaaactctgagcttcgtgcttaaaactcacagtgtccgaaagg
actttgtgcactctgtcaagttaacacttgcacggaggcgcgatcttcagtatatttataacaagctctc
acgcactatacgtgctgaggcttgcccttcttgtgctagttacgacgtatgtcctaactgcacctctggt
gacgtcccggatgacgggtcttcgacaatgtcgattccatcatgggaggatgtcacaaagtcttcaacct
actctctcctgctctctgaggacacctccgacgagttatgccctgaggacttggttaatgtggctgctca
tatccgtaaggcgctatccactcagtcccacccggctaatgctgaaatgtgcaaagaacagctcactttc
ttattagtcatggctgaggcgatgctgccccaacgatcccgagcgtcaatcccactgcaccaacaacaca
cggctgcacggttggaatggagggagaaattcttctctaaacctcttgactttctccttgaaagagttgg
tgtgtcaaaagatattctccaaactactgcgatttggaaaattatcttggaaaaagcatgctattgtaaa
tcctatggagagcagtggttcactgctgccaaacaaaagttaagggaaatgaaaaactttgagagtgata
cgctaaaacctcttattggtggatttatagatggtctacggttcttgaccgtggacaacccaaacccgat
gggtttcctcccaaaactcatagggcttgtaaaacccctgaatttggcgatgattattgataatcatgaa
aacacaatatctggctggatcatcacattaaccgcaataatggagctatacaacatcaccgaatgcacca
tagatattataacatcagtcattacggctttctatgataaaattggcaaggcaaccaaattttacagttg
tgttaaggcgctgttcactggatttagatctgaggacgtagcgaattccttttggtacatggcagcagcg
attctatgttacctgatcactgggttaatcccaaacaacggcagattttcaaagataaaagcttgcctgg
ccggagcgacaactcttgtatcaggtatagttgccacacaaaagctagctgcaatgttcgcaacatggaa
ctctgagtccattgttaatgagttgtcagcaagaactgttgccctatcagagctaaacaatccaacaaca
acgtctgacacggattcggtagaacgactgctagaattggctaagatcttgcatgaggagatcaagattc
atactctaaaccccatcatgcaatcatacaatccaatcttgagaaatttaatgtcaaccttggacggtgt
tataacatcatgcaacaagaggaaagctattgctcggaagagacaggtgccagtttgttacatattaact
ggcccacctggatgtgggaaaacaaccgcagctcaagcattagctaagaaattgtctgatcaagagccgt
cggtaattaaccttgatgttgatcatcatgacacatatactggaaatgaggtatgtatcattgatgagtt
tgattcgtctgacaaggtagactatgcaaattttgtgattgggatggtaaactctgctcctatggtgtta
aattgtgacatgcttgagaacaagggaaagctcttcacctcaaagtatataattatgacttccaactctg
aaactccagtgaaaccctcttccaagcgcgcaggtgcattctaccgaagggttactatcatcgatgtaac
taacccttttgtggagtcgcacaagcgtgcaaggcctgggacgtctgttcctcgtagctgctataagaaa
aacttctcccatctctcacttgctaaacgaggggccgagtgctggtgcaaggaatacgttcttgacccta
aggggcttcaacaccaaagcatgaaggctccccctcctacctttcttaacattgactctttagctcagac
gatgaagcaagacttcttgctcaagaacatggcttttgaggctgaagatgggtgcgcagaacatcgatat
gggtttgtgtgtcaacaggaagaggttgaaactgttcgcaggctcctcaacgcggttagggcaaggatga
atgctaccttcactgtgtgtgttggacccgagacctcgcactcgattggttgcactgcgcacgtgttaac
tcccaatgagacctttaatggaaagaagtttgttgtgtcacgatgtaacgaagcatcactttctgccctt
gaaggtaactgtgtaaagtcagctttgggcgtgtgtatgtcagataaggatctcactcatttatgccact
tcattaaagggaaaattgtcaatgacagtgttaggttggatgaactacccgccaatcagcatgtggtaac
cgttaattcggtgtttgatttggcctgggctgttcgtcgtcatctcacactggcagggcagtttcaagct
atcagagccgcatatgatgtgcttactgtccccgacaagatccctgcaatgttgcgtcactggatggatg
agacctccttttctgatgaccacgttgtaacacaatttgtaacacctggtggcatagtcatcctggagtc
ttgcggtggtgcacgcatctgggctttaggtcgcaacgtgatccgagcaggaggtgtcaccgcaacccca
accggaggatgtgttaggttaatgggattatctgcgccaaccatgccatggtccgagatctttcgtgagc
tcttctctctcttaggtagaatttggtcatctgtcaaagtatcagccctagtactaactgctcttggaat
gtacgcatctagatttagaccaaaatcggaagcaaaaggaaaaaccaaattgaagattgggacatacagg
ggtcgcggtgtagcgctgactgatgacgagtacgatgagtggcgcgaacacaacgcctccagaaaattgg
atttgtcagtggaggatttcttaatgttgcgccatcgtgccgctctaggagccgacgacaatgatgcagt
aaaattccggtcgtggtggaactccagaaccaaaatggccaatgattatgaggatgtcaccgtaattggc
aaaggtggcgtcaaacatgaaaagatcagaaccaataccctaaaagctgtggatcgtggttatgacgtca
gctttgctgaggaatcaggaccaggcaccaagttccacaagaatgcaattggatctgtcacagatgtttg
tggggagcacaaaggctattgcatccacatgggccacggtgtttacgcatccgtagctcacgtggtgaaa
ggggattcatttttcttgggtgaaaggatttttgatcttaagactaatggtgaattttgctgctttcgca
gcacgaaaattctacctagtgctgcacctttcttttctgggaagcccactcgtgatccgtggggatcccc
cgtggcaactgagtggaagcctaaaatgtacacaacaacctctggaaagattctggggtgctttgcaaca
acttcaactgaaactcacccgggagactgtggcctcccatatattgatgacaacgggagggtgaccggcc
tccacactggctctgggggacccaaaaccccaagtgccaagttggtggtgccatatgtgcatattgacat
gaagactaaatccgtcactgctcaaaagtatgacgtaacaaagcctgatataagttacaaaggcttaatt
tgtaagcaattggatgagattaggattataccaaaaggcacacgtctccatgtctccccagcccacactg
aggattatcaagaatgctcacaccaacccgcatcacttggaagcggggatccccgctgtccaaaatctct
cactgctatagttgttgattctctaaaaccatactgtgagaacgttgagggtcctccacatgatgttttg
cacagagttcaaaagatgcttatcgaccacctttcaggctttgtccctatgaacatttcctcggaaacct
ctatgctctcagctttccacaaactcaatcatgatacttcctgtggaccatacttgggtggcagaaagaa
agatcacatggctaacggtgagccggacaagcagttattggatctcctgtctgcaaaatggaaattggca
acccaaggcatagcactaccacatgagtacacaattgggctaaaggacgagttaaggcccgtggagaagg
ttagtgaagggaagagaaggatgatttggggttgtgatgttggcgtcgctactgtctgtgcagctgcgtt
caagggtgttagcgatgccatcacagcaaaccaccagtacgggcctatacaggttggtatcaacatggat
agccccagcgtcgaagcgctgttccaaaggatcaaaagcgcggccaaggtatttgcggtcgattattcca
aatgggattcgacccaatcgcctcgtgtcagtgcagcttcaattgacatccttcgttacttttccgatcg
ctctccaattgttgactcagcctctaacacactgaagagccctcctgttgcaatctttaatggtgttgct
gtaaaagtgtcctctggcttaccatctggaatgcctcttacctcagtaatcaattcccttaatcattgtc
tgtatgttgggtgtgccattcttcaatccctagaagctaaggccattcccgtcacttggaaccttttctc
aacttttgatatcatgacttacggggatgatggtgtctacatgtttcctattatgtatgcaagtattagt
gaccaaatttttggaaatctttcttcctatggcctgaaaccaactcgggttgacaagtccgttggagcaa
ttgagcctattgatcctgactctgttgttttcttgaagagaacaatcacaaggacacctcaggggataag
gggtttacttgatcgcagctctataataagacaattctattatattaaaggtgagaactccgatgactgg
aagagccccccaaaacatattgacccaacatctcgagggcaacagctttggaatgcctgtctgtacgcta
gccaacatggcttggagtttttcaacaaggtttacaggctggccgagagggctgttgaatatgaagagct
gcactttgaacccccaacatatgcttcggctttggatcattacaacagccagttcaatggcgtggaggcg
cggtctgaccagatcgactcgagtggcatgaccgccctacactgtgatgtgttcgaagtt</INSDSeq_
sequence>
</INSDSeq>
</SequenceData>
<SequenceDatasequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>1763</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..1763</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MSQTLSFVLKTHSVRKDFVHSVKLTLARRRDLQYIYNKLSRTIRAEACP
SCASYDVCPNCTSGDVPDDGSSTMSIPSWEDVTKSSTYSLLLSEDTSDELCPEDLVNVAAHIRKALSTQS
HPANAEMCKEQLTFLLVMAEAMLPQRSRASIPLHQQHTAARLEWREKFFSKPLDFLLERVGVSKDILQTT
AIWKIILEKACYCKSYGEQWFTAAKQKLREMKNFESDTLKPLIGGFIDGLRFLTVDNPNPMGFLPKLIGL
VKPLNLAMIIDNHENTISGWIITLTAIMELYNITECTIDIITSVITAFYDKIGKATKFYSCVKALFTGFR
SEDVANSFWYMAAAILCYLITGLIPNNGRFSKIKACLAGATTLVSGIVATQKLAAMFATWNSESIVNELS
ARTVALSELNNPTTTSDTDSVERLLELAKILHEEIKIHTLNPIMQSYNPILRNLMSTLDGVITSCNKRKA
IARKRQVPVCYILTGPPGCGKTTAAQALAKKLSDQEPSVINLDVDHHDTYTGNEVCIIDEFDSSDKVDYA
NFVIGMVNSAPMVLNCDMLENKGKLFTSKYIIMTSNSETPVKPSSKRAGAFYRRVTIIDVTNPFVESHKR
ARPGTSVPRSCYKKNFSHLSLAKRGAECWCKEYVLDPKGLQHQSMKAPPPTFLNIDSLAQTMKQDFLLKN
MAFEAEDGCAEHRYGFVCQQEEVETVRRLLNAVRARMNATFTVCVGPETSHSIGCTAHVLTPNETFNGKK
FVVSRCNEASLSALEGNCVKSALGVCMSDKDLTHLCHFIKGKIVNDSVRLDELPANQHVVTVNSVFDLAW
AVRRHLTLAGQFQAIRAAYDVLTVPDKIPAMLRHWMDETSFSDDHVVTQFVTPGGIVILESCGGARIWAL
GRNVIRAGGVTATPTGGCVRLMGLSAPTMPWSEIFRELFSLLGRIWSSVKVSALVLTALGMYASRFRPKS
EAKGKTKLKIGTYRGRGVALTDDEYDEWREHNASRKLDLSVEDFLMLRHRAALGADDNDAVKFRSWWNSR
TKMANDYEDVTVIGKGGVKHEKIRTNTLKAVDRGYDVSFAEESGPGTKFHKNAIGSVTDVCGEHKGYCIH
MGHGVYASVAHVVKGDSFFLGERIFDLKTNGEFCCFRSTKILPSAAPFFSGKPTRDPWGSPVATEWKPKM
YTTTSGKILGCFATTSTETHPGDCGLPYIDDNGRVTGLHTGSGGPKTPSAKLVVPYVHIDMKTKSVTAQK
YDVTKPDISYKGLICKQLDEIRIIPKGTRLHVSPAHTEDYQECSHQPASLGSGDPRCPKSLTAIVVDSLK
PYCENVEGPPHDVLHRVQKMLIDHLSGFVPMNISSETSMLSAFHKLNHDTSCGPYLGGRKKDHMANGEPD
KQLLDLLSAKWKLATQGIALPHEYTIGLKDELRPVEKVSEGKRRMIWGCDVGVATVCAAAFKGVSDAITA
NHQYGPIQVGINMDSPSVEALFQRIKSAAKVFAVDYSKWDSTQSPRVSAASIDILRYFSDRSPIVDSASN
TLKSPPVAIFNGVAVKVSSGLPSGMPLTSVINSLNHCLYVGCAILQSLEAKAIPVTWNLFSTFDIMTYGD
DGVYMFPIMYASISDQIFGNLSSYGLKPTRVDKSVGAIEPIDPDSVVFLKRTITRTPQGIRGLLDRSSII
RQFYYIKGENSDDWKSPPKHIDPTSRGQQLWNACLYASQHGLEFFNKVYRLAERAVEYEELHFEPPTYAS
ALDHYNSQFNGVEARSDQIDSSGMTALHCDVFEV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| Штамм "Перс" вируса Feline calicivirus калицивирусной инфекции кошек для изготовления биопрепаратов для диагностики и специфической профилактики калицивирусной инфекции кошек | 2023 |
|
RU2804639C1 |
| Штамм "Фауна" вируса калицивироза кошек Feline calicivirus для изготовления биопрепаратов для диагностики и специфической профилактики калицивироза кошек | 2023 |
|
RU2804623C1 |
| Ассоциированная вакцина против панлейкопении, калицивироза, вирусного ринотрахеита и бешенства кошек | 2024 |
|
RU2827230C1 |
| Ассоциированная вакцина против панлейкопении, калицивироза и вирусного ринотрахеита кошек | 2023 |
|
RU2812330C1 |
| Способ опосредованного определения титра инфекционной активности калицивируса кошек в сырье для вакцины методом ПЦР в режиме реального времени | 2023 |
|
RU2809221C1 |
| ШТАММ FELINE CALICIVIRUS, ИСПОЛЬЗУЕМЫЙ ДЛЯ КОНТРОЛЯ ИММУНОГЕННОЙ АКТИВНОСТИ ВАКЦИН, ИЗГОТОВЛЕНИЯ СПЕЦИФИЧЕСКИХ ЛЕЧЕБНО-ПРОФИЛАКТИЧЕСКИХ И ДИАГНОСТИЧЕСКИХ БИОПРЕПАРАТОВ | 2001 |
|
RU2184567C1 |
| Способ опосредованного определения титра инфекционной активности калицивируса кошек в неинактивированном сырье для культуральных вакцин с помощью количественного учета реакции амплификации участка ORF1-гена вирусной РНК | 2023 |
|
RU2821027C1 |
| Способ дифференциации штамма "Мира" калицивируса кошек от других штаммов калицивируса с помощью анализа однонуклеотидных полиморфизмов гена ORF1 | 2024 |
|
RU2839481C1 |
| Способ определения концентрации вирионов калицивируса кошек в сырье для цельновирионных вакцин | 2024 |
|
RU2838181C1 |
| Штамм "Лавр" вируса Alphaherpesvirus 1 инфекционного ринотрахеита кошек для изготовления биопрепаратов для диагностики и специфической профилактики инфекционного ринотрахеита кошек | 2023 |
|
RU2806603C1 |
Изобретение относится к области ветеринарной вирусологии и биотехнологии. Предложен штамм «Мира» вируса калицивирусной инфекции кошек семейства Caliciviridae, рода Vesivirus, вида Feline calicivirus, депонированный в коллекцию штаммов микроорганизмов ФГБУ «ВНИИЗЖ» под регистрационным номером №458 – деп / 23-33 – ГКШМ ФГБУ «ВНИИЗЖ». Представленный штамм репродуцируется в перевиваемой культуре клеток почки кошки (CRFK). В перевиваемой культуре клеток почки кошки CRFK в течение 36 ч инкубирования титр инфекционной активности вируса достигает значений 7,50±0,25 lg ТЦД50/см3, сохраняя исходные характеристики при пассировании в клеточной культуре на протяжении 5 пассажей. Представленный штамм может быть использован для изготовления биопрепаратов для диагностики и специфической профилактики калицивирусной инфекции кошек и для контроля антигенной активности вакцин против данного заболевания. 2 ил., 2 табл., 6 пр.
Штамм «Мира» вируса калицивирусной инфекции Feline calicivirus, депонированный во Всероссийской государственной коллекции экзотических типов вируса ящура и других патогенов животных (ГКШМ) ФГБУ «ВНИИЗЖ», под регистрационным номером № 458 - деп / 23-33 - ГКШМ ФГБУ «ВНИИЗЖ» для изготовления биопрепаратов для диагностики и специфической профилактики калицивирусной инфекции кошек.
Авторы
Даты
2024-10-16—Публикация
2024-04-22—Подача