Область изобретения
Настоящее изобретение относится к фармацевтической композиции для лечения рака, включающей в качестве активных ингредиентов слитый белок, содержащий белок CD80 и вариант IL-2, и NK-клетку (от англ. Natural killer cell -естественная клетка-киллер).
Предшествующий уровень техники
IL-2, также называемый фактором роста Т-клеток (TCGF - от англ. T-cell growth factor), представляет собой глобулярный гликопротеин, который играет центральную роль в продукции, выживаемости и гомеостазе лимфоцита. Белок IL-2 имеет размер 15,5 кДа - 16 кДа белка и состоит из 133 аминокислот.IL-2 опосредует разные иммунные ответы посредством связывания с рецептором IL-2, который имеет три отличающихся субъединицы. Кроме того, IL-2 синтезируется главным образом активированными Т-клетками, в частности, CD4+ хэлперными Т-клетками. IL-2 стимулирует пролиферацию и дифференцировку Т-клеток и индуцирует продукцию цитотоксических Т-лимфоцитов (CTL - от англ. cytotoxic Т lymphocyte) и дифференцировку лимфоцитов периферической крови в цитотоксические клетки и лимфокин-активированные клетки - киллеры (клетки LAK (от англ. lymphokine-activated killer)).
Тем не менее, CD80, также известный как В7-1, представляет собой член семейства В7 мембраносвязанных белков, которые участвуют в иммунной регуляции посредством связывания со своим лигандом посредством доставки костимулирующих ответов и коингибирующих ответов. CD80 представляет собой трансмембранный белок, экспрессируемый на поверхности Т-клеток, В-клеток, дендритных клеток и моноцитов. Известно, что CD80 связывает CD28, CTLA4 (CD152) и PD-L1. CD80, CD86, CTLA4 и CD28 участвуют в системе костимуляции-коингибирования. Например, они регулируют активность Т-клеток и участвуют в их пролиферации, дифференцировки и выживаемости.
Кроме того, известно, что естественные клетки-киллеры (ниже в данном документе, NK-клетки) демонстрируют противораковую активность за счет удаления раковых клеток (Loris Zamai et al., J. Immunol., 178:4011-4016, 2007). Активность NK-клеток регулируется балансом сигнализации на основе разных активирующих и ингибирующих рецепторов. Известно, что противораковая активность NK-клеток также достигается посредством распознавания раковых клеток за счет разных иммунных рецепторов, находящихся на поверхности. Благодаря главному комплексу гистосовместимости (МНС - от англ. histocompatibility complex) класса I, находящемуся в нормальных клетках, нормальные клетки распознаются ингибирующими рецепторами NK-клеток и, таким образом, не подвергаются атаке, но раковые клетки или некоторые инфицированные клетки устраняются NK-клетками, благодаря редуцированному МНС класса I или лигандам для активирующих рецепторов NK-клеток. NK-клетки могут устранять раковые стволовые клетки помимо раковых клеток и, таким образом, находятся в центре внимания в качестве источника терапевтических средств, которые не только могли бы ингибировать развитие, пролиферацию и метастазирование рака, но также уменьшать рецидив рака после полного выздоровления.
Подробное описание изобретения
Техническая проблема
Соответственно, в результате исследования для разработки безопасного и эффективного IL-2, авторы настоящего изобретения обнаружили, что совместное введение нового димера слитого белка, содержащего белок IL-2 и белок CD80 в одной молекуле, в комбинации с естественными клетками-киллерами демонстрирует превосходный противораковый эффект, и завершили настоящее изобретение.
Решение проблемы
Для достижения указанной выше цели в соответствии с одним аспектом настоящего изобретения предложено противораковое средство, включающее в качестве активных ингредиентов димер слитого белка, содержащий белок IL-2 и белок CD80, и естественную клетку-киллер.
Эффекты изобретения
Подтвердили, что димер слитого белка, содержащий белок IL-2 и белок CD80, может не только активировать клетки иммунной системы, но также демонстрировать синергические эффекты при введении в комбинации с естественными клетками-киллерами. Таким образом, такую комбинированную терапию можно полезным образом применять для лечения рака.
Краткое описание графических материалов
Фиг. 1 представляет собой схематичное изображение воплощения димера слитого белка, используемого в настоящем изобретении;
На Фиг. 2 показано изображение SDS-PAGE (от англ. sodium dodecyl sulphate-polyacrylamide gel electrophoresis - электрофорез в полиакриламидном геле в присутствии додецилсульфата натрия), подтверждающее полученный димер слитого белка (GI-101);
На Фиг. 3 показан анализ полученного димера слитого белка (GI-101) на основе эксклюзионной хроматографии (SEC - от англ. size exclusion chromatography);
На Фиг. 4 показано изображение SDS-PAGE, подтверждающее полученный слитый белок Fc-IL2v2;
На Фиг. 5 показан анализ полученного слитого белка Fc-IL2v2 на основе эксклюзионной хроматографии (SEC);
На Фиг. 6 показано изображение SDS-PAGE, подтверждающее полученный слитый белок hCD80-Fc;
На Фиг. 7 показан анализ полученного димера слитого белка hCD80-Fc на основе эксклюзионной хроматографии (SEC);
На Фиг. 8 показаны результаты жизнеспособности раковых клеток в соответствии с обработкой GI-101 или CD80-Fc+Fc-IL2v2 при культивировании линии K562 клеток отдельно без естественных-клеток-киллеров (отношение Е/Т равно 0/1). В данном случае, Е показывает NK-клетку в качестве эффекторной клетки, и Т показывает линию раковых клеток K562 в качестве клетки-мишени.
На Фиг. 9 показаны результаты жизнеспособности раковых клеток в соответствии с обработкой GI-101 или CD80-Fc+Fc-IL2v2 при культивировании линии клеток MDA-MB-231 отдельно без естественных клеток-киллеров (отношение Е/Т равно 0/1). В данном случае Е указывает на NK-клетку в качестве эффекторной клетки и Т указывает на линию раковых клеток MDA-MB-231 в качестве клетки-мишени.
На Фиг. 10 показаны результаты жизнеспособности раковых клеток в соответствии с обработкой GI-101 или CD80-Fc+Fc-IL2v2 при культивировании линии клеток НСТ-116 отдельно без естественных клеток-киллеров (отношение Е/Т равно 0/1). В данном случае Е указывает на NK-клетку в качестве эффекторной клетки, и Т указывает на линию раковых клеток НСТ-116 в качестве клетки-мишени.
На Фиг. 11 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой GI-101 или CD80-Fc+Fc-IL2v2 при культивировании линии клеток А549 отдельно без естественных клеток-киллеров (отношение Е/Т равно 0/1). В данном случае Е показывает NK-клетку в качестве эффекторной клетки и Т показывает линию раковых клеток А549 в качестве клетки-мишени.
На Фиг. 12 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании линии клеток K562 (клетка-мишень) с естественными клетками-киллерами (эффекторная клетка) в соотношении Е/Т, равном 1/3.
В данном документе, Y-ось=0 указывает на то, что интервал сигнала жизнеспособности (красный сигнал) раковых клеток в момент исходного посева установлен на уровне 0. Y-ось>0 представляет собой случай, когда интервал сигнала жизнеспособности раковых клеток, обнаруженного после момента посева, увеличивается, что указывает на то, что скорость пролиферации клеток быстрее, чем скорость гибели клеток (скорость пролиферации клеток больше скорости гибели клеток). То есть, видно увеличение числа раковых клеток. Однако, полагают, что чем ниже значение увеличения количества, тем больше ингибировалась пролиферация раковых клеток. Между тем, если измеренная площадь жизнеспособных раковых клеток меньше, чем площадь жизнеспособных раковых клеток, измеренная в момент исходного посева, она выражена как отрицательное значение, которое указывает на то, что скорость пролиферации клеток медленнее, чем скорость гибели клеток (скорость пролиферации клеток меньше скорости гибели клеток). А именно, уменьшение площади, по сравнению с площадью, измеренной в момент исходного посева, выражено отрицательным значением. Таким образом, полагают, что большее отрицательное значение может быть связано не только с ингибированием пролиферации раковых клеток, но также с гибелью раковых клеток.
На Фиг. 13 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании линии клеток K562 с естественными клетками-киллерами в соотношении Е/Т, равном 1/1.
На Фиг. 14 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании линии клеток K562 с естественными клетками-киллерами в соотношении Е/Т, равном 3/1.
На Фиг. 15 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании линии клеток K562 с естественными клетками-киллерами в соотношении Е/Т, равном 10/1.
На Фиг. 16 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии MDA-MB231 (клетка - мишень) с естественными клетками-киллерами (эффекторная клетка) в соотношении Е/Т, равном 1/3.
На Фиг. 17 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии MDA-MB231 с естественными клетками-киллерами в соотношении Е/Т, равном 1/1.
На Фиг. 18 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии MDA-MB231 с естественными клетками-киллерами в соотношении Е/Т, равном 3/1.
На Фиг. 19 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии MDA-MB231 с естественными клетками-киллерами в соотношении Е/Т, равном 10/1.
На Фиг. 20 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии НСТ-116 (клетка-мишень) с естественными клетками-киллерами (эффекторная клетка) в соотношении Е/Т, равном 1/3.
На Фиг. 21 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии НСТ-116 с естественными клетками-киллерами в соотношении Е/Т, равном 1/1.
На Фиг. 22 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии НСТ-116 с естественными клетками-киллерами в соотношении Е/Т, равном 3/1.
На Фиг. 23 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии НСТ-116 с естественными клетками-киллерами в соотношении Е/Т, равном 10/1.
На Фиг. 24 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии А549 (клетка-мишень) с естественными клетками-киллерами (эффекторная клетки) в соотношении Е/Т, равном 1/3.
На Фиг. 25 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии А549 с естественными клетками - киллерами в соотношении Е/Т, равном 1/1.
На Фиг. 26 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии А549 с естественными клетками-киллерами в соотношении Е/Т, равном 3/1.
На Фиг. 27 показаны результаты по жизнеспособности раковых клеток в соответствии с обработкой комбинацией материала GI-101 или CD80-Fc+Fc-IL2v2 при совместном культивировании клеточной линии А549 с естественными клетками-киллерами в соотношении Е/Т, равном 10/1.
Фиг. 28 представляет собой схематичное изображение, показывающее схему введения комбинированных терапий на основе mGI-101 и/или NK-клеток мышиной модели трансплантируемой карциномы СТ26.
На Фиг. 29 показаны результаты эффекта ингибирования роста опухоли в соответствии с совместным введением естественных клеток-киллеров и mGI-101 мышиной модели трансплантируемой карциномы СТ26.
На Фиг. 30 показан процент ингибирования роста опухоли в соответствии с совместным введением естественных клеток-киллеров и mGI-101 мышиной модели трансплантируемой карциномы СТ26.
На Фиг. 31 показаны измерения роста опухоли отдельных лабораторных животных в каждой экспериментальной группе (носитель, NK-клетка, mGI-101, NK-клетка + mGI-101) в мышиной модели трансплантируемой карциномы СТ26.
На Фиг. 32 показаны измерения роста опухоли отдельных лабораторных животных в группе носителя в мышиной модели трансплантируемой карциномы СТ26.
На Фиг. 33 показаны измерения роста опухоли отдельных лабораторных животных в группе обработки NK-клетками в мышиной модели трансплантируемой карциномы СТ26.
На Фиг. 34 показаны измерения роста опухоли отдельных лабораторных животных в группе обработки mGI-101 в мышиной модели трансплантируемой карциномы СТ26.
На Фиг. 35 показаны измерения роста опухоли отдельных лабораторных животных в группе совместного введения естественных клеток-киллеров и mGI-101 в мышиной модели трансплантируемой карциномы СТ26.
Наилучший способ осуществления изобретения
Согласно аспекту настоящего изобретения предложена фармацевтическая композиция, включающая в качестве активных ингредиентов слитый белок, содержащий белок CD80 и белок IL-2, и NK-клетки.
Согласно еще одному аспекту настоящего изобретения предложена фармацевтическая композиция для лечения рака, включающая в качестве активных ингредиентов слитый белок, содержащий белок CD80 и белок IL-2, и NK-клетки.
Естественная клетка-киллер
В том виде, в котором он используется в данном документе, термин «NK-клетка» относится к естественной клетке-киллеру (ниже в данном документе NK-клетка) и представляет собой одну из клеток врожденного иммунитета, которая непосредственно взаимодействует с разными макрофагами и Т-клетками или генерирует цитокины для регуляции иммунных ответов и, таким образом, играя важную роль в аутоиммунных заболеваниях. В настоящем изобретении NK-клетки могут быть выделены из селезенки или костного мозга, но не ограничиваются ими. В частности, NK-клетки могут быть получены из аутологичных или гетерологичных клеток.
Кроме того, NK-клетки могут происходить из млекопитающих или человека. Предпочтительно, они могут быть получены от индивида, который собирается получить лечение NK-клетками. В данном случае, NK-клетки можно непосредственно выделять из крови индивида и использовать, или незрелые NK-клетки или стволовые клетки, полученные от индивида, могут быть дифференцированы и использованы.
Кроме того, естественные клетки-киллеры могут быть получены посредством следующих стадий, включая i): отделение клеток, которые не экспрессируют CD3, от мононуклеарных клеток периферической крови (РВМС - от англ. peripheral blood mononuclear cell); ii) отделение клеток, которые экспрессируют CD56, от клеток, которые не экспрессируют CD3, выделенных на указанной выше стадии; и ii) культивирование выделенных клеток в присутствии димера слитого белка, содержащего IL-2 или его вариант и CD80 или его фрагмент.
Корме того, естественные клетки-киллеры могут быть получены посредством следующих стадий, включая: i) отделение клеток, которые не экспрессируют CD3, от РВМС; и ii) культивирование выделенных клеток в присутствии димера слитого белка, содержащего IL-2 или его вариант и CD80 или его фрагмент.
Кроме того, естественные клетки-киллеры могут быть получены посредством следующих стадий, включая: i) отделение клеток, которые не экспрессируют CD56, от РВМС; и ii) культивирование выделенных клеток в присутствии димера слитого белка, содержащего IL-2 или его вариант и CD80 или его фрагмент.
Димер слитого белка, содержащий белок IL-2 и белок CD80
Термин «IL-2» или «интерлейкин-2» в том виде, как он используется в данном документе, если не утверждается иное, относится к любому IL-2 дикого типа, полученному из любого источника-позвоночного, включая млекопитающих, например, приматов (таких как человек) и грызунов (таких как мыши и крысы). IL-2 может быть получен из животных клеток и также включает IL-2, полученный из рекомбинантных клеток, способных к продукции IL-2. Кроме того, IL-2 может представлять собой IL-2 дикого типа или его вариант.
В настоящем описании изобретения IL-2 или его вариант могут быть совместно выражены термином «белок IL-2» или «полипептид IL-2». IL-2, белок IL-2, полипептид IL-2 и вариант IL-2 специфично связываются, например, с рецептором IL-2. Это специфичное связывание может быть идентифицировано способами, известными специалистам в данной области.
В одном из воплощений IL-2 может иметь аминокислотную последовательность SEQ ID NO: 35 или SEQ ID NO: 36. Здесь IL-2 также может находиться в зрелой форме. В частности, зрелый IL-2 может не содержать сигнальную последовательность и может иметь аминокислотную последовательность SEQ ID NO: 10. Здесь IL-2 может использоваться согласно конфигурации, охватывающей фрагмент IL-2 дикого типа, в котором усечена часть N-конца или С-конца IL-2 дикого типа.
Кроме того, данный фрагмент IL-2 может находиться в форме, в которой 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 смежных аминокислот усечены с N-конца белка, имеющего аминокислотную последовательность SEQ ID NO: 35 или SEQ ID NO: 36. Кроме того, фрагмент IL-2 может находиться в форме, в которой 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 смежных аминокислот усечены с С-конца белка, имеющего аминокислотную последовательность SEQ ID NO: 35 или SEQ ID NO: 36.
Термин «вариант IL-2» в том виде, как он используется в данном документе, относится к форме, в которой часть аминокислот в полноразмерном IL-2 или вышеописанном фрагменте IL-2 заменена. То есть, вариант IL-2 может иметь отличную аминокислотную последовательность от IL-2 дикого типа или его фрагмента. Однако вариант IL-2 может иметь эквивалентную или аналогичную активность относительно IL-2 дикого типа. Здесь «активность IL-2» может, например, относиться к специфичному связыванию с рецептором IL-2, причем данное специфичное связывание можно измерять способами, известными специалистам в данной области.
В частности, вариант IL-2 можно получать заменой части аминокислот в IL-2 дикого типа. Одно воплощение варианта IL-2, полученного аминокислотной заменой, может быть получено заменой по меньшей мере одной из 38-ой, 42-ой, 45-ой, 61-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10.
В частности, вариант IL-2 может быть получен заменой по меньшей мере одной из 38-ой, 42-ой, 45-ой, 61-ой или 72-ой аминокислоты в аминокислотной последовательности SEQ ID NO: 10 другой аминокислотой. Кроме того, когда IL-2 находится в форме, в которой часть N-конца аминокислотной последовательности SEQ ID NO: 35 усечена, аминокислота в положении, комплементарно соответствующем положению в аминокислотной последовательности SEQ ID NO: 10, может быть заменена другой аминокислотой. Например, когда IL-2 имеет аминокислотную последовательность SEQ ID NO: 35, его вариант IL-2 может быть получен заменой по меньшей мере одной из 58-ой, 62-ой, 65-ой, 81-ой или 92-ой аминокислоты в аминокислотной последовательности SEQ ID NO: 35 другой аминокислотой. Данные аминокислотные остатки соответствуют 38-му, 42-му, 45-му, 61-му и 72-му аминокислотному остатку в аминокислотной последовательности SEQ ID NO: 10 соответственно. Согласно одному воплощению могут быть заменены одна, две, три, четыре, пять, шесть, семь, восемь, девять или десять аминокислот, при условии, что такой вариант IL-2 сохраняет активность IL-2. Согласно другому воплощению могут быть заменены от одной до пяти аминокислот.
В одном воплощении вариант IL-2 может находиться в форме, в которой заменены две аминокислоты. В частности, вариант IL-2 может быть получен заменой 38-ой и 42-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой и 45-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой и 61-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 42-ой и 45-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 42-ой и 61-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 42-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 45-ой и 61-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 45-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 61-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10.
Кроме того, вариант IL-2 может находится в форме, в которой заменены три аминокислоты. В частности, вариант IL-2 может быть получен заменой 38-ой, 42-ой и 45-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой, 42-ой и 61-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой, 42-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой, 45-ой и 61-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой, 45-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой, 61-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 42-ой, 45-ой и 61-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 42-ой, 45-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 45-ой, 61-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10.
Кроме того, вариант IL-2 может находится в форме, в которой заменены четыре аминокислоты. В частности, вариант IL-2 может быть получен заменой 38-ой, 42-ой, 45-ой и 61-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой, 42-ой, 45-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой, 45-ой, 61-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 38-ой, 42-ой, 61-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10. Кроме того, в одном воплощении вариант IL-2 может быть получен заменой 42-ой, 45-ой, 61-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10.
Кроме того, вариант IL-2 может находится в форме, в которой заменены пять аминокислот.В частности, вариант IL-2 может быть получен заменой каждой из 38-ой, 42-ой, 45-ой, 61-ой и 72-ой аминокислот в аминокислотной последовательности SEQ ID NO: 10 другой аминокислотой.
Здесь «другая аминокислота», вводимая заменой, может представлять собой любую аминокислоту, выбранную из группы, состоящей из аланина, аргинина, аспарагина, аспарагиновой кислоты, цистеина, глутаминовой кислоты, глутамина, гистидина, изолейцина, лейцина, лизина, метионина, фенилаланина, пролина, серина, треонина, триптофана, тирозина и валина. Однако относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 38-ая аминокислота не может быть заменена аргинином, 42-ая аминокислота не может быть заменена фенилаланином, 45-ая аминокислота не может быть заменена тирозином, 61-ая аминокислота не может быть заменена глутаминовой кислотой, и 72-ая аминокислота не может быть заменена лейцином.
Относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 38-ая аминокислота - аргинин - может быть заменена аминокислотой, отличной от аргинина. Предпочтительно относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 38-ая аминокислота - аргинин - может быть заменена аланином (R38A).
Относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 42-ая аминокислота - фенилаланин - может быть заменена аминокислотой, отличной от фенилаланина. Предпочтительно относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 42-ая аминокислота - фенилаланин - может быть заменена аланином (F42A).
Относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 45-ая аминокислота - тирозин - может быть заменена аминокислотой, отличной от тирозина. Предпочтительно относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 45-ая аминокислота - тирозин - может быть заменена аланином (Y45A).
Относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 61-ая аминокислота - глутаминовая кислота -может быть заменена аминокислотой, отличной от глутаминовой кислоты. Предпочтительно относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 61-ая аминокислота - глутаминовая кислота - может быть заменена аргинином (E61R).
Относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 72-ая аминокислота - лейцин - может быть заменена аминокислотой, отличной от лейцина. Предпочтительно относительно аминокислотной замены для варианта IL-2 в аминокислотной последовательности SEQ ID NO: 10, 72-ая аминокислота - лейцин - может быть заменена глицином (L72G).
В частности, вариант IL-2 может быть получен по меньшей мере одной заменой, выбранной из группы, состоящей из R38A, F42A, Y45A, E61R и L72G в аминокислотной последовательности SEQ ID NO: 10.
В частности, вариант IL-2 может быть получен аминокислотными заменами в двух, трех, четырех или пяти положениях среди положений, выбранных в группе, состоящей из R38A, F42A, Y45A, Е61 R и L72G.
Кроме того, вариант IL-2 может находиться в форме, в которой заменены две аминокислоты. В частности, вариант IL-2 может быть получен заменами R38A и F42A. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A и Y45A. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A и E61R. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами F42A и Y45A. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами F42A и E61R. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами F42A и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами E61R и L72G.
Кроме того, вариант IL-2 может находиться в форме, в которой заменены три аминокислоты. В частности, вариант IL-2 может быть получен заменами R38A, F42A и Y45A. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A, F42A и E61R. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A, F42A и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A, Y45A и E61R. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A, Y45A и E61R. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A, Y45A и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами F42A, Y45A и E61R. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами F42A, Y45A и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами F42A, E61R и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами Y45A, E61R и L72G.
Кроме того, вариант IL-2 может находиться в форме, в которой заменены четыре аминокислоты. В частности, вариант IL-2 может быть получен заменами R38A, F42A, Y45A и E61R. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A, F42A, Y45A и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A, F42A, E61R и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами R38A, Y45A, E61R и L72G. Кроме того, в одном воплощении вариант IL-2 может быть получен заменами F42A, Y45A, E61R и L72G.
Кроме того, вариант IL-2 может быть получен заменами R38A, F42A, Y45A, E61R и L72G.
Предпочтительно одно воплощение варианта IL-2 может содержать замену, которая представляет собой любую замену, выбранную из следующих комбинаций замен (a)-(d) в аминокислотной последовательности SEQ ID NO: 10:
(a) R38A/F42A
(b) R38A/F42A/Y45A
(c) R38A/F42A/E61 R
(d) R38A/F42A/L72G
Здесь, когда IL-2 имеет аминокислотную замену SEQ ID NO: 35, аминокислотная замена может присутствовать в положении, комплементарно соответствующем положению в аминокислотной последовательности SEQ ID NO: 10. Кроме того, даже когда IL-2 представляет собой фрагмент аминокислотной последовательности SEQ ID NO: 35, аминокислотная замена может присутствовать в положении, комплементарно соответствующем положению в аминокислотной последовательности SEQ ID NO: 10.
В частности, вариант IL-2 может иметь аминокислотную последовательность SEQ ID NO: 6, 22, 23 или 24.
Кроме того, вариант IL-2 может отличаться низкой токсичностью in vivo. Здесь низкая токсичность in vivo может представлять собой побочный эффект, вызванный связыванием IL-2 с альфа цепью рецептора IL-2 (IL-2Rα). Были разработаны разные варианты IL-2 для уменьшения интенсивности побочного эффекта, вызванного связыванием IL-2 с IL-2Rα, и такие варианты IL-2 могут представлять собой варианты, раскрытые в патенте США №5229109 и корейском патенте №1667096. В частности, варианты IL-2, описанные в настоящей заявке, имеют низкую аффинность связывания в отношении альфа цепи рецептора IL-2 (IL-2Rα) и, таким образом, имеют более низкую токсичность in vivo, чем IL-2 дикого типа.
Термин «CD80» в том виде, в котором он используется в данном документе, также именуемый «В7-1», относится к мембранному белку, присутствующему в дендритных клетках, активированных В-клетках и моноцитах. CD80 предоставляет костимулирующие сигналы, важные для активации и выживания Т-клеток. CD80 известен в качестве лиганда двух разных белков - CD28 и CTLA-4, присутствующих на поверхности Т-клеток. CD80 состоит из 288 аминокислот и может, в частности, иметь аминокислотную последовательность SEQ ID NO: 11. Кроме того, термин «белок CD80» в том виде, в котором он используется в данном документе, относится к полноразмерному CD80 или к фрагменту CD80.
Термин «фрагмент CD80» в том виде, в котором он используется в данном документе, относится к усеченной форме CD80. Кроме того, фрагмент CD80 может представлять собой внеклеточный домен CD80. Одно воплощение фрагмента CD80 может быть получено делецией с 1-ой по 34-ую аминокислоты с N-конца, которые представляют собой сигнальную последовательность CD80. В частности, одно воплощение фрагмента CD80 может представлять собой белок, состоящий из 35-ой - 288-ой аминокислот в SEQ ID NO: 11. Кроме того, одно воплощение фрагмента CD80 может представлять собой белок, состоящий из 35-ой - 242-ой аминокислот в SEQ ID NO: 11. Кроме того, одно воплощение фрагмента CD80 может представлять собой белок, состоящий из 35-ой - 232-ой аминокислот в SEQ ID NO: 11. Кроме того, одно воплощение фрагмента CD80 может представлять собой белок, состоящий из 35-ой - 139-ой аминокислот в SEQ ID NO: 11. Кроме того, одно воплощение фрагмента CD80 может представлять собой белок, состоящий из 142-ой - 242-ой аминокислот в SEQ ID NO: 11. В одном воплощении фрагмент CD80 может иметь аминокислотную последовательность SEQ ID NO: 2.
Кроме того, белок IL-2 и белок CD80 могут быть присоединены друг к другу через линкер или носитель. В частности, IL-2 или его вариант и CD80 (В7-1) или его фрагмент могут присоединяться друг к другу через линкер или носитель. В настоящем описании термины «линкер» и «носитель» могут использоваться взаимозаменяемо.
Линкер связывает два белка. Одно воплощение линкера может содержать от 1 до 50 аминокислот, альбумин или его фрагмент, Fc-домен иммуноглобулина или тому подобное. Здесь Fc-домен иммуноглобулина относится к белку, который содержит константную область 2 тяжелой цепи (СН2) и константную область 3 тяжелой цепи (СН3) иммуноглобулина, и не содержит вариабельные области тяжелой и легкой цепи, и константную область 1 легкой цепи (СН1) иммуноглобулина. Данный иммуноглобулин может представлять собой IgG, IgA, IgE, IgD или IgM и предпочтительно может представлять собой IgG4. Здесь Fc-домен иммуноглобулина G4 дикого типа может иметь аминокислотную последовательность SEQ ID NO: 4.
Кроме того, Fc-домен иммуноглобулина может представлять собой вариант Fc-домена, а также Fc-домен дикого типа. Кроме того, термин «вариант Fc-домена» в том виде, как он используется в данном документе, может относиться к форме, которая отличается от Fc-домена дикого типа в показателях картины гликозилирования, имеет высокое гликозилирование по сравнению с доменом Fc дикого типа или имеет низкое гликозилирование по сравнению с доменом Fc дикого типа, или представляет собой дегликозилированную форму. Кроме того, в данный документ включается негликозилированный Fc-домен. Fc-домен или его вариант может быть адаптирован для наличия скорректированного числа сиаловых кислот, фукозилирований или гликозилирований посредством культуральных условий или генетических манипуляций с хозяином.
Кроме того, гликозилирование Fc-домена иммуноглобулина может быть модифицировано традиционными способами, такими как химические способы, ферментативные способы и способы генной инженерии с использованием микроорганизмов. Кроме того, вариант Fc-домена может находиться в смешанной форме соответствующих областей Fc иммуноглобулинов - IgG, IgA, IgE, IgD и IgM. Кроме того, вариант Fc-домена может находиться в форме, в которой некоторые аминокислоты Fc-домена заменены другими аминокислотами. Одно воплощение варианта Fc-домена может иметь аминокислотную последовательность SEQ ID NO: 12.
Данный слитый белок может иметь структуру, в которой с использованием Fc-домена в качестве линкера (или носителя) белок CD80 и белок IL-2 или белок IL-2 и белок CD80 связываются с N-концом и С-концом линкера или носителя соответственно (Фиг. 1А). Связь между N-концом или С-концом Fc-домена и CD-80 или IL-2 возможно может достигаться посредством линкерного пептида.
В частности, слитый белок может состоять из следующей структурной формулы (I) или (II):
N'-X-[линкер (1)]n-Fc-домен-[линкер (2)]m-Y-C' (I)
N'-Y-[линкер (1)]n-Fc-домен-[линкер (2)]m-X-C' (II)
Здесь в структурных формулах (I) и (II)
N' представляет собой N-конец слитого белка,
С' представляет собой С-конец слитого белка,
X представляет собой белок CD80,
Y представляет собой белок IL-2,
линкеры (1) и (2) представляют собой пептидные линкеры и n и m каждый независимо равен 0 или 1.
Предпочтительно данный слитый белок может состоять из структурной формулы (I). Белок IL-2 является таким, как описано выше. Кроме того, белок CD80 является таким, как описано выше. Согласно одному воплощению белок IL-2 может представлять собой вариант IL-2 с одной-пятью аминокислотными заменами по сравнению с IL-2 дикого типа. Белок CD80 может представлять собой фрагмент, полученный усечением вплоть до примерно 34 смежных аминокислотных остатков от N-конца или С-конца CD80 дикого типа. В качестве альтернативы, белок CD может представлять собой внеклеточный иммуноглобулиноподобный домен, имеющий активность связывания с рецепторами поверхности Т-клеток CTLA-4 и CD28.
В частности, данный слитый белок может иметь аминокислотную последовательность SEQ ID NO: 9, 26, 28 или 30. Согласно другому воплощению данный слитый белок содержит полипептид, имеющий идентичность последовательности 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100% с аминокислотной последовательностью SEQ ID NO: 9, 26, 28 или 30. Здесь идентичность представляет собой, например, процент гомологии и может определяться посредством программы для сравнения гомологии, такой как программа BlastN Национального центра биотехнологической информации (NCBI).
Пептидный линкер (1) может содержаться между белком CD80 и доменом Fc. Пептидный линкер (1) может состоять из 5-80 смежных аминокислот, 20-60 смежных аминокислот, 25-50 смежных аминокислот или 30-40 смежных аминокислот. В одном воплощении пептидный линкер (1) может состоять из 30 аминокислот. Кроме того, пептидный линкер (1) может содержать по меньшей мере один цистеин. В частности, пептидный линкер (1) может содержать один, два или три цистеина. Кроме того, пептидный линкер (1) может происходить из шарнира иммуноглобулина. В одном воплощении пептидный линкер (1) может представлять собой пептидный линкер, состоящий из аминокислотной последовательности SEQ ID NO: 3.
Пептидный линкер (2) может состоять из 1-50 смежных аминокислот, 3-30 смежных аминокислот или 5-15 смежных аминокислот.В одном воплощении пептидный линкер (2) может представлять собой (G4S)n (где n представляет собой целое число от 1 до 10). Здесь в (G4S)n n может быть равен 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10. В одном воплощении пептидный линкер (2) может представлять собой пептидный линкер, состоящий из аминокислотной последовательности SEQ ID NO: 5.
В другом аспекте настоящего изобретения предложена фармацевтическая композиция, содержащая в качестве активного ингредиента димер, полученный путем связывания двух слитых белков, каждый из которых содержит белок IL-2 и белок CD80, и NK-клетку. Слитый белок, содержащий IL-2 или его вариант и CD80 или его фрагмент, является таким, как описано выше.
Здесь связывание между слитыми белками, составляющими димер, может быть достигнуто в том числе за счет дисульфидной связи, образованной цистеинами, присутствующими в линкере. Слитые белки, составляющие димер, могут быть одинаковыми или отличными друг от друга слитыми белками. Предпочтительно димер может быть гомодимером. Вариант осуществления слитого белка, составляющего димер, может представлять собой белок, имеющий аминокислотную последовательность SEQ ID NO: 9.
В настоящем изобретении рак может представлять собой любой рак, выбранный из группы, состоящей из рака желудка, рака печени, рака легкого, колоректального рака, рака молочной железы, рака предстательной железы, рака яичника, рака поджелудочной железы, рака шейки матки, рака щитовидной железы, рака гортани, острого лимфобластного лейкоза, опухоли головного мозга, нейробластомы, ретинобластомы, рака головы и шеи, рака слюнной железы и лимфомы.
Предпочтительная доза фармацевтической композиции варьирует в зависимости от состояния пациента и массы тела, тяжести заболевания, формы лекарственного средства, пути и продолжительности введения и может быть приблизительно выбрана специалистами в данной области. В фармацевтической композиции для лечения и предупреждения рака по настоящему изобретению активный ингредиент может содержаться в любом количестве (эффективном количестве) в зависимости от применения, использования, формы дозировки, цели смешивания и т.п., при условии, что активный ингредиент может демонстрировать противораковую активность. Его общепринятое эффективное количество будет определено в интервале от 0,001 масс. % до 20,0 масс. % в расчете на общую массу композиции. В данном документе, термин «эффективное количество» относится к количеству активного ингредиента, способному вызывать противораковое действие. Такое эффективное количество можно экспериментально определить в объеме общего знания специалистов в данной области.
В том виде, в котором он используется в данном документе, термин «лечение» может быть использован для обозначения как терапевтического, так и профилактического лечения. В данном документе, профилактика может использоваться для обозначения того, что патологическое состояние или заболевание индивида облегчено или ослаблено. В одном воплощении термин «лечение» включает как нанесение, так и любую форму введения для лечения заболевания у животного, включая человека. Кроме того, термин включает ингибирование или замедление заболевания или прогрессирования заболевания; и включает значения восстановления или исправления нарушенной или потерянной функции таким образом, что заболевание частично или полностью облегчено; стимуляцию неэффективных процессов; или облегчение серьезного заболевания.
В том виде, в котором он используется в данном документе, термин «эффективность» относится к способности, которую можно определить по одному из параметров, например, выживаемости или выживаемости без признаков заболевания на протяжении определенного периода времени, такого как один год, пять лет или десять лет.Кроме того, параметр может включать заболевание размером по меньшей мере в одну опухоль у индивида.
Фармакокинетические параметры, такие как биодоступность, и основополагающие параметры, такие как скорость клиренса, могут также влиять на эффективность. Таким образом, «усиленная эффективность» (например, улучшение в эффективности) может быть обусловлена усиленными фармакокинетическими параметрами и улучшенной эффективностью, которые могут быть измерены посредством сравнения скорости клиренса и роста опухоли у лабораторных животных или у субъектов, являющихся человеком, или посредством сравнения параметров, таких как выживаемость, возникновение рецидива или выживаемость без признаков заболевания.
В том виде, в котором он используется в данном документе, термин «терапевтически эффективное количество» или «фармацевтически эффективное количество» относится к количеству соединения или композиции, эффективному для предупреждения или лечения рассматриваемого заболевания, которое является достаточным для лечения заболевания при разумном соотношении польза/риск, применимом для медицинского лечения, и не вызывает негативных последствий. Уровень эффективного количества можно определять в зависимости от факторов, включая состояние здоровья пациента, виды и тяжесть заболевания, активность лекарственного средства, чувствительность пациента к лекарственному средству, способ введения, время введения, путь введения и скорость выведения, продолжительность лечения, состав или одновременно используемые лекарственные средства, и другие факторы, хорошо известные в области медицины. В одном воплощении терапевтически эффективное количество означает количество лекарственного средства, эффективное для лечения рака.
В данном документе, фармацевтическая композиция может дополнительно включать фармацевтически приемлемый носитель. Фармацевтически приемлемый носитель может представлять собой любой носитель, при условии, что носитель является нетоксичным веществом, подходящим для доставки пациенту. Дистиллированная вода, спирт, жир, воск и инертное твердое вещество могут содержаться в качестве носителя. Фармацевтически приемлемый адъювант (буфер, диспергирующее средство) может также содержаться в фармацевтической композиции.
В частности, посредством включения фармацевтически приемлемого носителя помимо активного ингредиента, фармацевтическая композиция может быть приготовлена в виде парентерального препарата, в зависимости от пути его введения с использованием традиционных способов, известных в данной области. В данном документе, термин «фармацевтически приемлемый» означает, что носитель не обладает токсичностью большей, чем токсичность, к которой субъект, предназначенный для применения (назначения), может адаптироваться, без одновременного ингибирования активности активного ингредиента.
Когда фармацевтическая композиция приготовлена в виде парентерального препарата, ее можно получить в виде препаратов в форме инъекций, трансдермальных пластырей, назальных средств для ингаляций или суппозиториев с подходящими носителями согласно способам, известным в данной области. В случае приготовления в виде инъекций, стерильную воду, этанол, полиол, такой как глицерин или пропиленгликоль или их смесь, можно использовать в качестве подходящего носителя; и предпочтительно можно использовать изотонический раствор, такой как раствор Рингера, фосфатно-солевой буферный раствор (PBS - от англ. phosphate buffered saline), содержащий триэтаноламин, или стерильную воду для инъекций, и 5%-ную декстрозу или т.п. Состав фармацевтических композиций известен в данной области, и ссылка может быть, в частности, сделана на Remington's Pharmaceutical Sciences (19th ed., 1995) и т.п.Данный документ считается частью настоящего описания.
Предпочтительная доза димера в фармацевтической композиции может находиться в интервале от 0,01 мкг/кг до 10 г/кг, или от 0,01 мг/кг до 1 г/кг, в сутки, в зависимости от состояния пациента, массы тела, пола, возраста, тяжести пациента и пути введения. Дозу можно вводить один раз в сутки или она может быть разделена на несколько раз в сутки. Такую дозу не следует считать ограничивающей объем настоящего изобретения в любом аспекте. Кроме того, NK-клетки в фармацевтической композиции могут быть введены в количестве 1×102 - 1×1013 клеток, от 1×107 до 1,5×1011 клеток, при регуляции соответствующим образом в интервале, показывающем фармакологический эффект.
Субъекты, для которых может быть применена (назначена) данная фармацевтическая композиция, представляют собой млекопитающих и человека, причем человек является особенно предпочтительным. Помимо активного ингредиента, фармацевтическая композиция по настоящей заявке может дополнительно включать любое соединение или природный экстракт, который уже проверен в отношении безопасности и, как известно, обладает противораковой активностью, таким образом, чтобы простимулировать или усилить противораковую активность.
В еще одном аспекте настоящего изобретения предложено применение димера слитого белка, содержащего белок IL-2 и белок CD80, и NK-клетки для лечения рака.
В еще одном аспекте настоящего изобретения предложено применение димера слитого белка, содержащего белок IL-2 и белок CD80, и NK-клеток для усиления терапевтического эффекта, оказываемого на рак.
В еще одном аспекте настоящего изобретения предложено применение димера слитого белка, содержащего белок IL-2 и белок CD80, и NK-клеток для изготовления лекарственного средства для лечения рака.
В еще одном аспекте настоящего изобретения предложен способ лечения рака и/или способ усиления терапевтического эффекта, оказываемого на рак, включающий введение субъекту слитого белка, содержащего белок IL-2 и белок CD80, или димер слитого белка, где связаны два слитых белка, и NK-клетки.
В данном документе, димер слитого белка и NK-клетки можно вводить одновременно или последовательно. В данном случае порядок введения может быть определен таким образом, что за введением димера слитого белка может следовать введение NK-клеток или за введением NK-клеток может следовать введение димера слитого белка.
Субъект может являться индивидом, страдающим раком или инфекционным заболеванием. Корме того, субъект может представлять собой млекопитающее, предпочтительно человека. Слитый белок, содержащий белок IL-2 и белок CD80, или димер слитого белка, где связаны два слитых белка, представляет собой такой, как описано выше.
Путь введения, доза и частота введения слитого белка или димера слитого белка и NK-клеток может варьировать в зависимости от состояния пациента и наличия или отсутствия побочных эффектов, и таким образом слитый белок или димер слитого белка можно вводить субъекту разными путями и в разных количествах. Оптимальный способ введения, доза и частота введения могут быть выбраны в соответствующем диапазоне специалистами в данной области. Кроме того, слитый белок или димер слитого белка можно вводить в комбинации с другими лекарственными средствами или физиологически активными веществами, чей терапевтический эффект известен в отношении заболевания, подлежащего лечению, или можно приготовить в форме комбинированных препаратов с другими лекарственными средствами.
Благодаря активности IL-2, слитый белок в воплощении настоящего изобретения может активировать клетки иммунной системы, такие как естественные клетки-киллеры. Таким образом, слитый белок может эффективно использоваться для раковых и инфекционных заболеваний. В частности, было идентифицировано, что, по сравнению с диким типом, вариант IL-2 с двумя-пятью заменами аминокислот, в частности, вариант IL-2, который содержит замены аминокислот в двух, трех, четырех или пяти положениях среди положений, выбранных из группы, состоящей из R38A, F42A, Y45A, E61R и L72G, имеет низкую способность к связыванию в отношении цепи альфа рецептора IL-2 и, таким образом, демонстрирует улучшенные характеристики в отношении фармакологических побочных эффектов общепринятого IL-2. Таким образом, такой вариант IL-2, при использовании отдельно или в форме слитого белка, может уменьшать встречаемость синдрома сосудистой (или капиллярной) утечки (VLS - от англ. vascular leakage syndrome), широко известной проблемы с IL-2.
Способ по изобретению
Ниже в данном документе, настоящее изобретение будет описано более подробно посредством следующих примеров. Однако, следующие примеры предназначены лишь для иллюстрации настоящего изобретения, и объем настоящего изобретения не ограничивается ими.
I. Получение димера слитого белка, содержащего IL-2 и CD80 Пример получения 1. Получение варианта hCD80-Fc-IL-2 (2М): GI-101 Для получения слитого белка, содержащего фрагмент человеческого CD80, Fc-домен и вариант IL-2, полинуклеотид, включающий нуклеотидную последовательность (SEQ ID NO: 8), кодирующую слитый белок, содержащий сигнальный пептид (SEQ ID NO: 1), фрагмент CD80 (SEQ ID NO: 2), шарнир Ig, конъюгированный с линкером (SEQ ID NO: 3), Fc-домен (SEQ ID NO: 4), линкер (SEQ ID NO: 5) и вариант IL-2 (2M), в котором две аминокислоты заменены (R38A, F42A) (SEQ ID NO: 6), в данном порядке от N-конца синтезировали посредством службы синтеза генов Invitrogen GeneArt ThermoFisher Scientific Inc. и клонировали в вектор pcDNA3_4. Кроме того, вектор вводили в клетки СНО (от англ. Chinese Hamster Ovary - яичник китайского хомяка) (EXPI-CHO™) для экспрессии слитого белка SEQ ID NO: 9. После введения вектора клетки СНО культивировали в окружающей среде с 37°С, 125 об./мин. и 8% CO2 в течение 7 суток и затем собирали для очистки слитого белка. Очищенный димер слитого белка был назван «GI-101».
Очистку проводили, используя хроматографию, включая смолу с белком А MabSelect SuRe. Слитый белок связывался в условиях 25 мМ Tris, 25 мМ NaCl и рН 7,4. Затем, его элюировали 100 мМ NaCl и 100 мМ уксусной кислотой при рН 3. После помещения 20% 1М Tris-HCI при рН 9 в пробирку для сбора собирали слитый белок. Собранный слитый белок подвергали диализу в буфер PBS в течение 16 часов для замены.
Затем, поглощение при длине волны 280 нм со временем измеряли посредством использования эксклюзионной хроматографии с помощью колонки TSKgel G3000SWXL (TOSOH Bioscience) с получением высокой концентрации слитого белка. При этом, выделенный и очищенный слитый белок подвергали SDS-PAGE в восстанавливающих (R) или невосстанавливающих (NR) условиях и окрашивали кумасси синим для подтверждения его чистоты (Фиг. 2). Подтверждали, что слитый белок содержался в концентрации 2,78 мг/мл, как выявлено с использованием NanoDrop.Также, результат, анализируемый с использованием эксклюзионной хроматографии, представляет собой такой, как показано на Фиг. 3.
Пример получения 2. Получение димера варианта Fc-IL-2 (2М): Fc-IL-2v2 Для получения слитого белка, содержащего Fc-домен и вариант IL-2, полинуклеотид, включающий нуклеотидную последовательность (SEQ ID NO: 45), кодирующую слитый белок, содержащий сигнальный пептид (SEQ ID NO: 1), шарнир Ig (SEQ ID NO: 38), Fc-домен (SEQ ID NO: 4), линкер (SEQ ID NO: 5) и вариант IL-2 (2M), в котором две аминокислоты заменены (R38A, F42A) (SEQ ID NO: 6), в данном порядке от N-конца синтезировали посредством службы синтеза генов Invitrogen GeneArt ThermoFisher Scientific Inc. и клонировали в вектор pcDNA3_4. Кроме того, вектор вводили в клетки СНО (EXPI-CHO™) для экспрессии слитого белка SEQ ID NO: 44. После введения вектора клетки СНО культивировали в окружающей среде с 37°С, 125 об./мин. и 8% CO2 в течение 7 суток и затем собирали для очистки димера слитого белка. Очищенный димер слитого белка был назван «Fc-IL2v2».
Очистку и сбор слитого белка осуществляли тем же образом, как в Примере получения 1. Выделенный и очищенный слитый белок подвергали SDS-PAGE в восстанавливающих (R) или невосстанавливающих (NR) условиях и окрашивали кумасси синим для подтверждения его чистоты (Фиг. 4). В результате, подтверждали, что слитый белок образует димер. Также, результат, анализируемый с использованием эксклюзионной хроматографии, представляет собой такой, как показано на Фиг. 5.
Пример получения 3. Получение димера hCD80-Fc: hCD80-Fc
Для получения слитого белка, содержащего фрагмент человеческого CD80 и Fc-домен, полинуклеотид (SEQ ID NO: 39), включающий нуклеотидную последовательность, кодирующую слитый белок, содержащий сигнальный пептид (SEQ ID NO: 1), фрагмент CD80 (SEQ ID NO: 2), шарнир Ig, связанный с линкером (SEQ ID NO: 3), и Fc-домен (SEQ ID NO: 4), в данном порядке от N-конца синтезировали посредством службы синтеза генов Invitrogen GeneArt ThermoFisher Scientific Inc. и клонировали в вектор pcDNA3_4. Кроме того, вектор вводили в клетки СНО (EXPI-CHO™) для экспрессии слитого белка SEQ ID NO: 40. После введения вектора клетки СНО культивировали в окружающей среде с 37°С, 125 об./мин. и 8% CO2 в течение 7 суток и затем собирали для очистки димера слитого белка. Очищенный димер слитого белка был назван «hCD80-Fc».
Очистку осуществляли с использованием хроматографии, включающей смолу с белком A MabSelect SuRe. Слитый белок связывался в условиях 25 мМ Tris, 25 мМ NaCl и рН 7,4. Затем, его элюировали 100 мМ NaCl и 100 мМ уксусной кислотой при рН 3. После помещения 20% 1М Tris-HCI при рН 9 в пробирку для сбора слитый белок собирали. Собранный слитый белок подвергали диализу в буфер PBS в течение 16 часов для замены.
Затем, поглощение при длине волны 280 нм со временем измеряли посредством использования эксклюзионной хроматографии с помощью колонки TSKgel G3000SWXL (TOSOH Bioscience) с получением высокой концентрации слитого белка. При этом, выделенный и очищенный слитый белок подвергали SDS-PAGE в восстанавливающих (R) или невосстанавливающих (NR) условиях и окрашивали кумасси синим для подтверждения его чистоты (Фиг. 6). В результате, подтверждали, что слитый белок образует димер. Также, результат, анализируемый с использованием эксклюзионной хроматографии, представляет собой такой, как показано на Фиг. 7.
Пример получения 4. Получение варианта mCD80-Fc-IL-2 (2М): mGI101 В соответствии с тем же способом, как в Примере получения 1, изготавливали GI-101 мышиного типа, содержащий CD80 мышиного происхождения и IL-2. В частности, для получения слитого белка, содержащего мышиный CD80, Fc-домен и вариант IL-2, полинуклеотид синтезировали посредством службы синтеза генов Invitrogen GeneArt ThermoFisher Scientific. Полинуклеотид включает нуклеотидную последовательность (SEQ ID NO: 14), которая кодирует слитый белок, который содержит сигнальный пептид (SEQ ID NO: 1), mCD80 (SEQ ID NO: 13), шарнир Ig, связанный с линкером (SEQ ID NO: 3), Fc-домен (SEQ ID NO: 4), линкер (SEQ ID NO: 5) и вариант IL-2 (2M) (R38A, F42A) (SEQ ID NO: 6) с двумя заменами аминокислот в данном порядке с N-конца. Полинуклеотид вставляли в вектор pcDNA3_4. Корме того, вектор вводили в клетки СНО (EXPI-CHO™) для экспрессии слитого белка SEQ ID NO: 47. После введения вектора, культивирование проводили в течение 7 суток в окружающей среде с 37°С, 125 об./мин. и 8% CO2. Затем, культуру собирали, и слитый белок очищали от нее. Очищенный слитый белок обозначали «mGI101».
II. Получение и культивирование NK-клеток
Пример получения 1. Выделение CD3(-)CD56(+) естественных клеток-киллеров, происходящих из мононуклеарных клеток периферической крови (РВМС)
Для получения CD3(-) клеток число РВМС (мононуклеарные клетки периферической крови, Zen-Bio. Inc, NC 27709, США, кат. №: SER-PBMC-200-F) подсчитывали с использованием автоматического счетчика клеток ADAM-MC2 (NanoEnTek, приобретенный у Cosmo Genetech Co., Ltd.). РВМС переносили в новую пробирку и затем центрифугировали при 300 х g в течение 5 минут при температуре 4°С. 0,5% (об./об.) бычьего сывороточного альбумина (BSA - от англ. bovine serum albumin) и 2 мМ ЭДТА (этилендиаминтетрауксусная кислота) включали в PBS для получения буфера MACS (рН 7,2). После завершения центрифугирования осадок клеток обрабатывали 80 мкл буфера MACS и 20 мкл магнитных шариков с CD3 (Miltenyi biotech, 130-050-101) на 1×107 клеток для суспендирования и затем приводили во взаимодействие при температуре 4°С в течение 15 минут.10 мл буфера MACS добавляли для промывки и центрифугировали при 300 х g в течение 10 минут при температуре 4°С, и затем осадок клеток ресуспендировали в 0,5 мл буфера MACS.
2 мл буфера MACS сначала наливали в колонку LD (Miltenyi Biotec, Bergisch Gladbach, Германия, кат. №: 130-042-901), и затем наливали клеточную суспензию. Затем, получали CD3(-) клетки, проходящие через колонку LD. При этом, CD3(-) клетки получали посредством протекания 2 мл буфера MACS три раза таким образом, чтобы можно было достаточно отделить клетки, остающиеся в колонке LD. Полученные CD3(-) клетки подсчитывали с использованием счетчика клеток и затем помещали в новую пробирку и центрифугировали при 300 х g в течение 5 минут при температуре 4°С. Затем, супернатант удаляли, и затем 80 мкл буфера MACS и 20 мкл магнитных шариков с CD56 (Miltenyi biotech, кат. №: 130-050-401) добавляли на 1×107 клеток с последующим взаимодействием при температуре 4°С в течение 15 минут. 10 мл буфера MACS добавляли для промывки и центрифугировали при 300 х g в течение 10 минут при температуре 4°С, и затем осадок клеток ресуспендировали в 0,5 мл буфера MACS.
Сначала 3 мл буфера MACS наливали в колонку LS (Miltenyi Biotec, Bergisch Gladbach, Германия, кат. №: 130-042-901), и затем наливали суспензию клеток. При этом, 2 мл буфера MACS три раза наливали таким образом, чтобы клетки, остающиеся в колонке LS, можно было достаточным образом отделить. Затем, после отделения колонки LS от магнитного держателя, добавляли 5 мл буфера MACS, и давление прикладывали посредством поршня с получением CD3(-)CD56(+) естественных клеток-киллеров. Полученные CD3(-)CD56(+) естественные клетки-киллеры помещали в новую пробирку и центрифугировали при 300 х g в течение 5 минут при температуре 4°С. После удаления супернатанта клетки суспендировали в культуральных средах, и число суспендированных клеток измеряли с использованием счетчика клеток.
Пример получения 2. Культивирование и получение CD3-CD56+ естественных клеток-киллеров 100 мкл CD335 (NKp46)-биотина и 100 мкл CD2-биотина, включенных в набор для активации/размножения NK-клеток (кат. №: 130-112-968) (Miltenyi Biotec, Bergisch Gladbach, Германия), помещали в микропробирку, объемом 1,5 мл, и перемешивали. Добавляли 500 мкл частиц с антителами против биотина MACSiBead и перемешивали. Затем, добавляли 300 мкл буфера MACS и перемешивали при 2°С - 8°С в течение 2 часов с использованием ротора для микропробирок.
Затем, 5 мкл шариков для активации NK на 1×106 клеток переносили в новую пробирку. 1 мл PBS добавляли и центрифугировали при 300 х g в течение 5 минут. После удаления супернатанта среду RPMI1640 с добавлением 5%-ной сыворотки АВ человека (кат.№: H4522)(Sigma, St. Louis, Missouri, США), подлежащую использованию, добавляли из расчета 5 мкл на 106 NK-клеток и суспендировали шарики с последующей инокуляцией в CD3(-)CD56(+) естественные клетки-киллеры, выделенные в Примере получения 1.
Далее, CD3-CD56+ естественные клетки-киллеры засевали в 24-луночный планшет, и туда добавляли среду RPMI1640, в которую добавляли 5%-ную сыворотку АВ человека (кат. №:Н4522) (Sigma, St. Louis, Missouri, США), и rhIL-2 (500 МЕд/мл) с последующей культивацией в условиях 37°С и 5% CO2. Затем, число клеток определяли каждые 2 суток для субкультивирования в следующем порядке: 12-луночный планшет, 6-луночный планшет и колба 25Т, когда клетки занимали 80% или больше сосуда для культивирования (конфлюентность), и, в конечном итоге, все клетки собирали в сутки 21.
Пример получения 3. Культивирование и сбор естественных клеток-киллеров мышиного происхождения
Пример получения 3.1. Получение клеток селезенки и костного мозга мыши
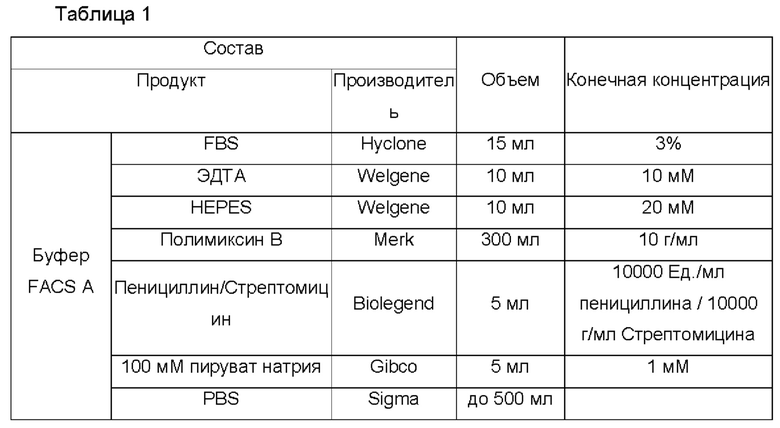
Для получения естественных клеток-киллеров мышиного происхождения сначала получали клетки селезенки и костный мозг мыши. В частности, селезенку и бедренную кость выделяли из самок Balb/c в возрасте 6 недель (ORIENT BIO Inc.), и удаляли жир и мышцы, насколько возможно, одновременно, стараясь не сломать бедренную кость. Выделенную бедренную кость помещали в 70%-ный этанол с последующим помещением в пробирку, объемом 50 мл, содержащую 5 мл PBS, и селезенку сразу переносили в пробирку, объемом 50 мл, содержащую 5 мл PBS, и затем хранили на льду. 70 мкм сетчатым фильтром перекрывали пробирку, объемом 50 мл, содержащую 5 мл буфера FACS (от англ. fluorescence-activated cell sorting - сортировка клеток с активированной флуоресценцией), и готовили для селезенки и костного мозга, соответственно. Состав буфера FACS А представляет собой такой, как описано ниже в Таблице 1.
После раздавливания ткани рукояткой поршня шприца 5 мл буфера FACS А добавляли для сбора клеток. Затем, проводили центрифугирование при 1300 об./мин. в течение 5 минут при 4°С, и супернатант удаляли. Селезенку растворяли посредством 3 мл лизирующего буфера АСК для удаления красных кровяных клеток и затем инкубировали на льду в течение 3 минут.Спустя 3 минуты, добавляли буфер FACS А с получением общего объема 20 мл. Полученный продукт перемешивали на вибромешалке и затем центрифугировали при 1300 об./мин. в течение 5 минут при 4°С, и супернатант удаляли. Красные клетки крови удаляли посредством повторения указанного выше процесса до тех пор, пока цвет осадка не станет розовым, и растворяли посредством 10 мл буфера FACS А для подсчета клеток.
Обе стороны бедренной кости на сетчатом фильтрее 70 мкм для костного мозга разрезали, и буфер FACS А заливали в отверстия кости с использованием иглы 1 мл шприца, заполненного 10 мл буфера FACS А, одновременно двигая назад и вперед, и затем также клетки собирали при одновременной заливки буфера FACS А в нарезанную ткань. Клетки собирали, и затем центрифугировали при 1300 об./мин. в течение 5 минут при 4°С. Затем, супернатант удаляли и растворяли 10 мл буфера FACS А с последующим подсчетом клеток.
Пример получения 3.2. Выделение и культивирование мышиных NK-клеток
Используя набор для выделения NK-клеток (#130-115-818), NK-клетки выделяли из селезенки и костного мозга, которые выделяли из самки мыши Balb/c 6-недельного возраста (ORIENT BIO Inc.) в Примере получения 3.1, указанном выше. После выделения проводили центрифугирование при 1300 х g при 4°С, и удаляли супернатант.
40 мкл буфера MACS на 107 клеток (600 мкл в случае селезенки, 200 мкл в случае костного мозга) добавляли для ресуспендирования осадка клеток, и добавляли 40 мкл смеси NK-клеток на 107 клеток (150 мкл для селезенки, 50 мкл для костного мозга). Однако, в данном случае, более чем 5×107 клеток демонстрировали явление агрегирования, таким образом, их разделяли и смешивали. Затем, проводили центрифугирование при 300 х g при 4°С, и удаляли супернатант.
Добавляли 2 мл промывочного буфера на 107 клеток (30 мл для селезенки, 10 мл для костного мозга), промывали, центрифугировали при 4°С при 300 х g, и удаляли супернатант. Затем, добавляли 80 мкл буфера MACS на 107 клеток (1,2 мл для селезенки, 400 мкл для костного мозга), и затем добавляли 20 мкл микрошариков с антителами против биотина на 107 клеток (300 мкл для селезенки, 100 мкл для костного мозга), смешивали и культивировали в холодильнике в течение 10 минут.
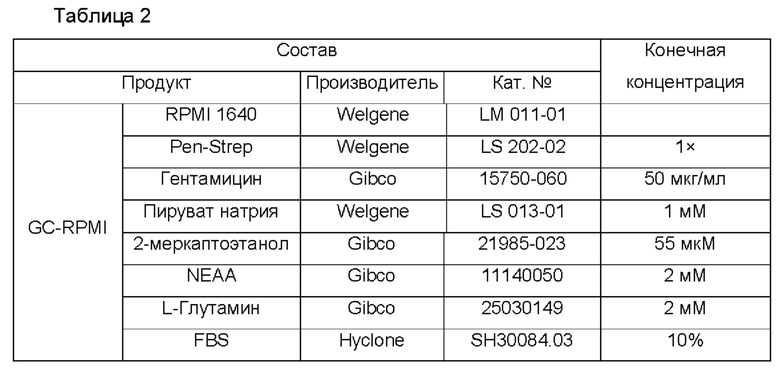
500 мкл клеток, смешанных с микрошариками с антителами против биотина, наливали в колонку MS с получением супернатанта, который проходил через колонку. Такой же процесс повторяли с получением супернатанта, который проходил через колонку, и центрифугировали при 4°С при 300 х g. Затем, супернатант удаляли. Клетки подсчитывали посредством ресуспендирования в среде GC-RPMI (1х) (селезенка: 6,95×105/мл с жизнеспособностью 59%, костный мозг: 1,58×106/мл с жизнеспособностью 64%). Состав среды GC-RPMI представляет собой такой, как описано в нижеуказанной Таблице 2.
После посева 3,5×105 NK-клеток, выделенных из селезенки и костного мозга в 60 мм сосуде для культивирования, 50 нг/мл rmIL-2 обрабатывали и культивировали в течение 14 суток.
III. Получение раковых клеток и конструирование мышиной модели Пример получения 4. Получение линии клеток карциномы, происходящей из человека, и среды для ее культивирования
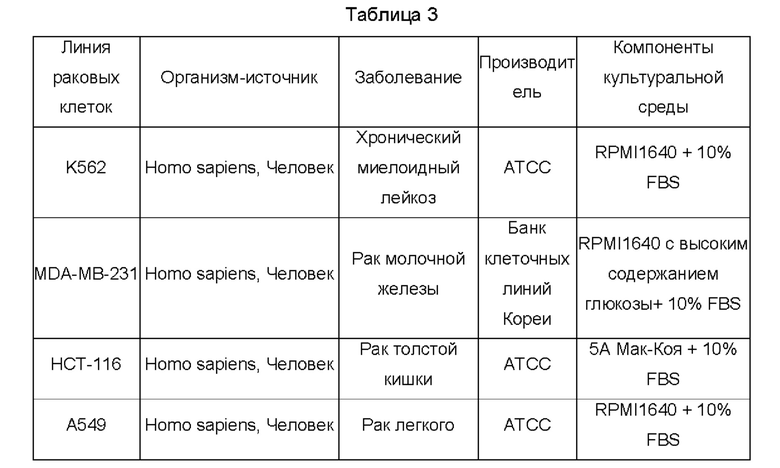
Культуральный раствор, подходящий для каждой линии раковых клеток, использовали со ссылкой на указанную ниже Таблицу 3.
В частности, 2×106 клеток линий раковых клеток ресуспендировали в 8 мл каждого культурального раствора и культивировали в колбе 25Т. При выделении клеток 1 мл трипсина-ЭДТА (0,25%) подвергали обработке и затем проводили реакцию в 5% CO2 в течение 2 минут в отношении клеток адгезивного типа. Затем, 5 мл культурального раствора добавляли для выделения клеток, которые отсоединялись от колбы, и центрифугировали при 300 x g течение 5 минут.
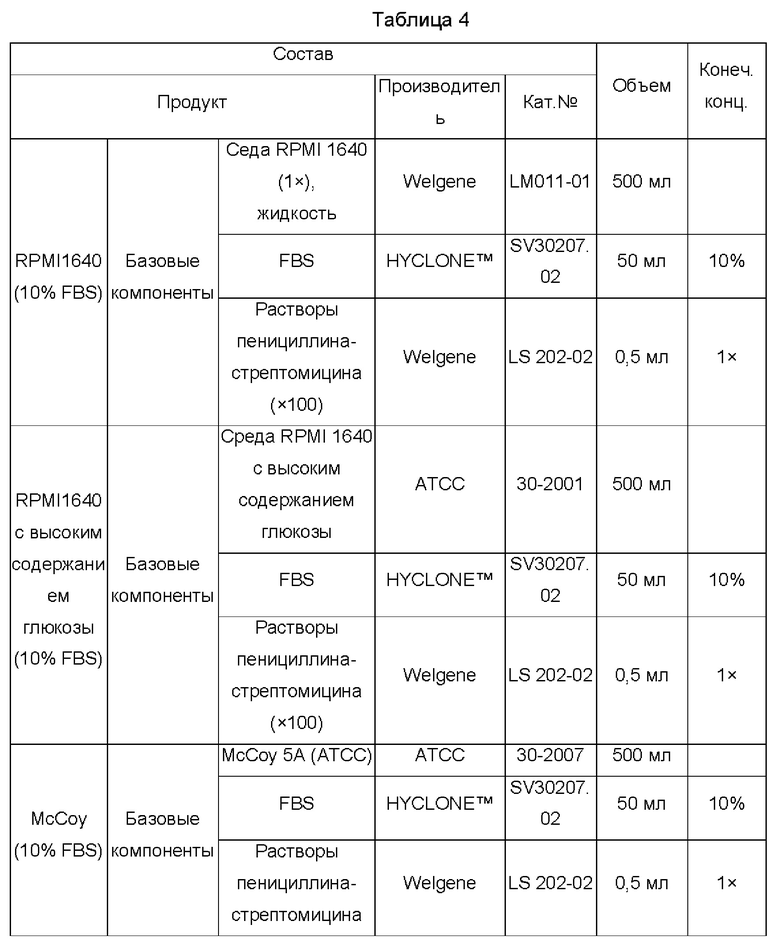
В указанной ниже Таблице 4 показаны конкретные составы культурального раствора для каждой линии раковых клеток.

Пример получения 5. Получение линии клеток карциномы мышиного происхождения и среды для ее культивирования
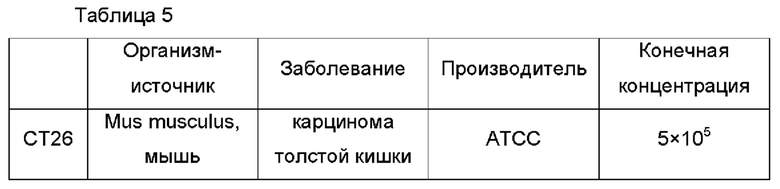
CT26.WT, клетки карциномы толстой кишки Mus musculus (домовая мышь) приобретали из АТСС (от англ. American Type Culture Collection - Американская коллекция типовых культур, США) (Таблица 5). Клетки карциномы, подлежащие использованию в эксперименте, размораживали, помещали в колбу для культивирования клеток и культивировали при 37°С, в инкубаторе 5% CO2 (МСО-170М, Panasonic, Япония). Клетки, культивируемые в сутки трансплантации линии клеток, помещали в центрифужную пробирку и собирали, и затем центрифугировали при 125 х g в течение 5 минут для удаления супернатанта. Затем, PBS добавляли для получения клеточной суспензии (5×107 клеток/мл), дозировали на аликвоты для 9 мышей и хранили на льду до введения.
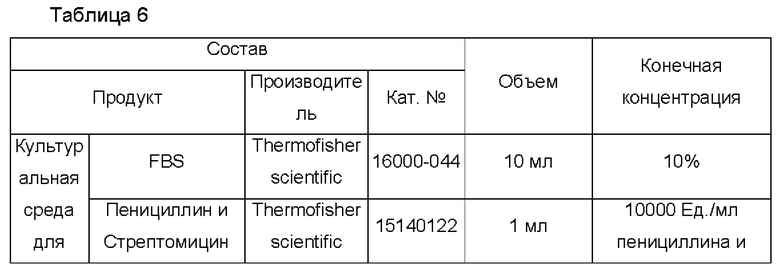
Фетальную телячью сыворотку (FBS; 16000-044, Thermofisher scientific, США), пенициллин-стрептомицин (10000 единиц/мл пенициллина и 10000 мкг/мл стрептомицина; 15140122, Thermofisher scientific, США) и RPMI1640 (А1049101, Thermofisher scientific, США) смешивали для получения композиции, описанной в нижеуказанной Таблице 6, на 100 мл и использовали в качестве среды для клеток карциномы.

Пример получения 6. Получение мышиной модели карциномы Пример получения 6.1. Карантин и процесс акклиматизации экспериментальной мыши
36 самок мышей BALB/c в возрасте 12 недель приобретали у ORIENT BIO Inc. Мышей приносили в лабораторию для животных и подвергали акклиматизации в течение 5 суток перед использованием в эксперименте. При получении мыши их оценивали в отношении внешнего вида и взвешивали для измерения массы тела. За общими симптомами наблюдали один раз в сутки на протяжении периода акклиматизации в течение 5 суток, и массу тела измеряли в конце периода акклиматизации, и затем общие симптомы и изменения массы проверяли для оценки для оценки состояния здоровья мышей. Мышей с патологией безболезненно умерщвляли при анестезии на основе газа CO2.
Информация о лабораторных мышах кратко изложена и показана ниже в Таблице 7.

Пример получения 6.2. Трансплантация линии клеток карциномы
Для модели ингибирования роста опухоли массу тела измеряли на следующие сутки после конца карантина и периода акклиматизации, и затем суспензию клеток СТ26 (5×105 клеток/0,1 мл), полученную для здоровых животных, дозировали, наполняли ими одноразовый шприц и вводили подкожно (0,1 мл/голова) в правую часть спины мыши для трансплантации. За общими симптомами наблюдали один раз в сутки на протяжении приживления трансплантата и периода роста после трансплантации линии клеток.
Пример получения 6.3. Распределение по группам мышиных моделей ингибирования роста опухоли
После определенного периода трансплантации клеток СТ26 объем опухоли и массу тела измеряли для мышей без патологического состояния здоровья и делили на 4 группы (9 мышей на группу) таким образом, что среднее каждой группы достигало 50 мм3.
IV. Определение противораковой активности NK-клеток и димера слитого белка: In vitro
Пример 1. Определение эффекта ингибирования роста раковых клеток NK-клетками и/или димером слитого белка в отношении разных карцином
Ссылаясь на схему планшета, используемую для 96-луночного планшета, 50 мкл 0,01%-ного раствора поли-L-орнитина (кат. №. Р4957) (Sigma Aldrich, США), соответственно, дозировали в лунку и осуществляли покрытие. Затем, его оставляли при комнатной температуре на 1 час. Спустя 1 час, дозированный 0,01%-ный раствор поли-L-орнитина удаляли и полностью сушили при комнатной температуре в течение 1 часа. 2 мкл темно-красного красителя CELLTRACKER™ (кат. №С34565) (Thermo Scientific, Waltham, MA, США) добавляли к раковым клеткам (клетка-мишень), полученными в количестве 4×105 клеток/мл, и давали взаимодействовать в течение 60 минут при 37°С и в условиях 5% CO2.
После реакции полученный в результате продукт центрифугировали при 300 x g в течение 5 минут.Супернатант удаляли, и затем растворяли в культуральном растворе RPMI1640 + 5% hABS до 4×105 клеток/мл. 50 мкл полученных раковых клеток дозировали в лунку в покрытом 96-луночном планшете, соответственно. Затем, поученный планшет помещали в систему анализа живых клеток INCUCYTE® (Satorius, Германия), и затем давали стабилизироваться в течение 10 минут.Культуральный раствор, содержащий тестируемый материал, получали в RPMI1640 + 5% hABS со ссылкой на указанную ниже Таблицу 8.

Естественные клетки-киллеры (эффекторные клетки) получали посредством суспендирования в культуральном растворе RPMI1640 + 5% hABS до 4×105 клеток/мл. Ссылаясь на указанную ниже Таблицу 9, естественные клетки-киллеры, полученные в примерах Получения 1 и 2, добавляли в каждую лунку в планшете, имеющую дозированные раковые клетки, и затем добавляли 100 мкл культурального раствора, обработанного INCUCYTE® CytoTox (250 нМ).

Затем, его помещали в систему анализа живых клеток INCUCYTE® и анализировали в течение 3 суток с временным интервалом 30 минут.
Пример 1.1. Определение эффективности димера слитого белка в отдельности в присутствии раковой клетки-мишени без NK-клетки
Как описано выше в Примере 1, наблюдали, что соответствующий тестируемый материал GI-101 и CD80-Fc+Fc-IL2v2 значимо не влиял на жизнеспособность раковых клеток при культивировании клеточных линий для разных типов рака (клеточные линии К562, MDA-MB-231, НСТ-116 и А549) в присутствии раковых клеток-мишеней в отдельности без NK-клеток (см. Таблица 9, отношение Е/Т равно 0/1) (Фиг. 8-11).
Пример 1.2. Эффект ингибирования пролиферации раковых клеток NK-клетками и димером слитого белка в отношении клетки K562 (лимфобласт)
Подтверждали способность уничтожения раковых клеток естественными клетками-киллерами в клеточной линии K562, линии раковых клеток лейкоза. В частности, подтверждали жизнеспособность раковых клеток в соответствии с обработкой тестируемым материалом GI-101 или CD80-Fc+Fc-IL2v2 в качестве материала комбинации при совместном культивировании линий клеток K562 в качестве клеток-мишеней (Т) и естественных клеток-киллеров в качестве эффекторных клеток (Е) при отношении Е/Т, равном 1/3, 1/1, 3/1 и 10/1, соответственно.
Результат показал, что при уничтожении раковых клеток K562, обработка GI-101 в качестве материала комбинации с естественной клеткой-киллером, который является формой слитого белка IL2-Fc-CD80, ингибировала жизнеспособность раковых клеток, более, чем обработка CD80-Fc+Fc-IL2v2 (Фиг. 16 - 19).
Пример 1.3. Эффект ингибирования пролиферации раковых клеток NK-клеткой и димером слитого белка в отношении клетки MDA-MB231
Подтверждали способность естественных клеток-киллеров уничтожать раковые клетки в линии клеток MDA-MB231, линии клеток рака молочной железы. В частности, подтверждали жизнеспособность раковых клеток в соответствии с обработкой тестируемым материалом GI-101 или CD80-Fc+Fc-IL2v2 в качестве материала комбинации при совместном культивировании линий клеток MDA-MB231 в качестве клеток-мишеней (Т) и естественных клеток-киллеров в качестве эффекторных клеток (Е) при отношении Е/Т, равном 1/3, 1/1, 3/1 и 10/1, соответственно.
Результат показал, что при уничтожении раковых клеток MDA-MB231 обработка GI-101 в качестве материала комбинации с естественной клеткой-киллером, который является формой слитого белка IL2-Fc-CD80, ингибировала жизнеспособность раковых клеток больше, чем обработка CD80-Fc+Fc-IL2v2 (Фиг. 16- 19).
Пример 1.4. Эффект ингибирования пролиферации раковых клеток NK-клеткой и димером слитого белка в отношении клетки НСТ-116
Подтверждали способность естественных клеток-киллеров уничтожать раковые клетки в линии клеток НСТ-116, линии клеток рака толстой кишки. В частности, подтверждали жизнеспособность раковых клеток в соответствии с обработкой тестируемым материалом GI-101 или CD80-Fc+Fc-IL2v2 в качестве материала комбинации при совместном культивировании линий клеток НСТ-116 в качестве клеток-мишеней (Т) и естественных клеток-киллеров в качестве эффекторных клеток (Е) при отношении Е/Т, равном 1/3, 1/1, 3/1 и 10/1, соответственно.
Результат показал, что при уничтожении раковых клеток НСТ-116 обработка GI-101 в качестве материала комбинации с естественной клеткой-киллером, который является формой слитого белка IL2-Fc-CD80, ингибировала жизнеспособность раковых клеток больше, чем обработка CD80-Fc+Fc-IL2v2 (Фиг. 20 - 23).
Пример 1.5. Эффект ингибирования пролиферации раковых клеток NK-клеткой и димером слитого белка в отношении клетки А549
Подтверждали способность естественных клеток-киллеров уничтожать раковые клетки в линии клеток А549, линии клеток рака толстой кишки. В частности, подтверждали жизнеспособность раковых клеток в соответствии с обработкой тестируемым материалом GI-101 или CD80-Fc+Fc-IL2v2 в качестве материала комбинации при совместном культивировании линий клеток А549 в качестве клеток-мишеней (Т) и естественных клеток-киллеров в качестве эффекторных клеток (Е) при отношении Е/Т, равном 1/3, 1/1, 3/1 и 10/1, соответственно.
Результат показал, что при уничтожении раковых клеток А549 обработка GI-101 в качестве материала комбинации с естественной клеткой-киллером, который является формой слитого белка IL2-Fc-CD80, ингибировала жизнеспособность раковых клеток больше, чем обработка CD80-Fc+Fc-IL2v2 (Фиг. 24 - 27).
V. Определение способности NK-клетки и димера слитого белка уничтожать раковые клетки в мышиной модели карциномы: In vivo
Пример 2. Введение mGI-101 и NK-клетки
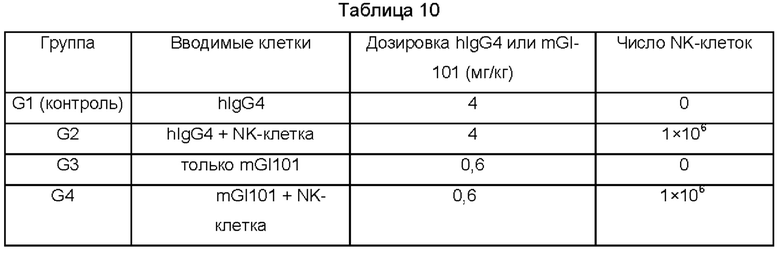
В случае мышиной модели карциномы, mGI-101, полученный в Примере получения 4, вводили внутрибрюшинным путем, и NK-клетки мышиного происхождения, полученные в Примере получения 3, вводили внутривенным путем. Введение проводили в общей сложности три раза один раз в сутки введения (сутки 6, сутки 10 и сутки 13 после трансплантации опухоли) с использованием одноразового шприца (31G, 1 мл). В случае модели ингибирования роста опухоли, как показано ниже в Таблице 10, клетки, подлежащие введению, и дозы введения четырех групп отличались (Фиг. 28).
Пример 3. Измерение объема опухоли мышиной модели карциномы
Максимальную длину (L) и перпендикулярную ширину (W) опухоли измеряли два раза в неделю с использованием цифрового штангенциркуля (mitutoyo, Япония) и применяли для Уравнения 1, указанного ниже, для расчета объема опухоли (TV-от англ. tumor volume).
Уравнение 1
TV (мм3)=W×W×L×0,5
Процент ингибирования роста опухоли рассчитывали посредством использования следующего уравнения 2:
Уравнение 2
Ингибирование роста опухоли (%)=(1-(Ti-T0)/(Vi-V0))×100
Ti = объем опухоли перед введением в опытной группе
Т0 = объем опухоли после введения в опытной группе
Vi = объем опухоли перед введением в контрольной группе
V0 = объем опухоли после введения в контрольной группе
Объем опухоли каждого индивида перед введением устанавливали как значение, измеренное в момент разделения на группы.
Пример 4. Определение эффекта ингибирования роста опухоли в мышиной модели трансплантируемой карциномы СТ26
Пример 4.1. Измерение объема опухоли
Суспензию клеток рака толстой и прямой кишки СТ26 (5×105 клеток/0,1 мл), полученную для здоровых мышей Balb/c, отмеряли, и полученным раствором наполняли одноразовый шприц и вводили подкожно (0,1 мл/голова) в правую часть спины животного для трансплантации. После трансплантации опухоли лекарственные средства, показанные в Таблице 10, вводили, соответственно. Затем, размер опухоли измеряли в сутки 10, сутки 13 и сутки 17. Рост опухоли был ингибирован в группах, обработанных естественными клетками-киллерами (NK-клетками) или только mGI-101, по сравнению с контрольной группой (носитель). Рост опухоли ингибировался в группе, обрабатываемой естественной клеткой-киллером (NK-клетка) в комбинации с mGI-101, по сравнению с контрольной группой (носитель). Рост опухоли группы, обрабатываемой естественными клетками-киллерами в комбинации с mGI-101, ингибировался, по сравнению с группами, обрабатываемыми естественными клетками-киллерами или mGI-101 в отдельности (Фиг. 29).
Пример 4.2. Анализ ингибирования роста опухоли
Степень ингибирования роста опухоли рассчитывали в конце эксперимента (после трансплантации опухоли, сутки 17), по сравнению с сутками обработки лекарственным средством 1 (после трансплантации опухоли, сутки 10). В контрольной группе (носитель) было 3 мыши, имеющих степень ингибирования роста опухоли 30% или больше, 3 мыши, имеющих степень ингибирования роста опухоли 50% или больше, и 2 мыши, имеющих степень ингибирования роста опухоли 80%. В группе обработки естественными клетками-киллерами было 6 мышей, имеющих степень ингибирования роста опухоли 30% или больше, 5 мышей, имеющих степень ингибирования роста опухоли 50% или больше, и 2 мыши, имеющих степень ингибирования роста опухоли 80%. В группе обработки mGI-101 было 5 мышей, имеющих степень ингибирования роста опухоли 30% или больше, 5 мышей, имеющих степень ингибирования роста опухоли 50% или больше, и 1 мышь, имеющая степень ингибирования роста опухоли 80%. В группе обработки комбинацией естественных клеток-киллеров и mGI-101, было 7 мышей, имеющих степень ингибирования роста опухоли 30% или больше, 6 мышей, имеющих степень ингибирования роста опухоли 50% или больше, и 3 мыши, имеющих степень ингибирования роста опухоли 80% (Фиг. 30). На Фиг. 30, черный столбец показывает степень ингибирования роста опухоли 30% или больше, светло-серый столбец показывает степень ингибирования роста опухоли 50% или больше и темно-серый столбец показывает степень ингибирования роста опухоли 80% или больше.
Пример 4.3. Измерение объема опухоли для отдельных лабораторных животных
В каждой экспериментальной группе определяли рост опухоли отдельных лабораторных животных. В частности, определяли рост опухоли отдельных лабораторных животных в контрольной группе (носитель), группах обработки естественными клетками-киллерами (NK-клетками), mGI-101 и естественными клетками-киллерами + mGI-101, и он показан на Фиг. 31. На Фиг. 31 пунктирная линия показывает размер опухоли 500 мм3 и сплошная линия показывает размер опухоли 250 мм3.
Более конкретно, определяли степень роста опухоли отдельных лабораторных животных контрольной группы, и она показана на Фиг. 32, и определяли степень роста опухоли отдельных лабораторных животных группы обработки естественными клетками-киллерами, и она показана на Фиг 33. Кроме того, определяли степень роста опухоли отдельных лабораторных животных группы обработки mGI-101, и она показана на Фиг. 34, и определяли степень роста опухоли отдельных лабораторных животных группы обработки комбинацией естественных клеток-киллеров и mGI-101, и она показана на Фиг. 35.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> GI CELL, INC.
<120> КОМПОЗИЦИЯ ДЛЯ ПРОТИВОРАКОВОГО ЛЕЧЕНИЯ, СОДЕРЖАЩАЯ NK-КЛЕТКИ И СЛИТЫЙ
БЕЛОК, КОТОРЫЙ СОДЕРЖИТ БЕЛОК IL-2 И БЕЛОК CD80
<130> PF-B2649
<140> PCT/KR2020/016949
<141> 2020-11-26
<150> 10-2019-0154631
<151> 2019-11-27
<150> 10-2020-0015802
<151> 2020-02-10
<160> 47
<170> PatentIn version 3.5
<210> 1
<211> 25
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> сигнальный пептид (TPA)
<400> 1
Met Asp Ala Met Leu Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 10 15
Ala Val Phe Val Ser Pro Ser His Ala
20 25
<210> 2
<211> 208
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> hB7-1:35-242
<400> 2
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro Asp Asn
195 200 205
<210> 3
<211> 30
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> шарнир с линкером
<400> 3
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
1 5 10 15
Ser Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
20 25 30
<210> 4
<211> 216
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> fc иммуноглобулина
<400> 4
Ala Pro Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys
1 5 10 15
Pro Lys Asp Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val
20 25 30
Val Val Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr
35 40 45
Val Asp Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu
50 55 60
Gln Phe Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His
65 70 75 80
Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys
85 90 95
Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln
100 105 110
Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met
115 120 125
Thr Lys Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro
130 135 140
Ser Asp Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn
145 150 155 160
Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu
165 170 175
Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val
180 185 190
Phe Ser Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln
195 200 205
Lys Ser Leu Ser Leu Ser Leu Gly
210 215
<210> 5
<211> 5
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> linker
<400> 5
Gly Gly Gly Gly Ser
1 5
<210> 6
<211> 133
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> hIL-2M
<400> 6
Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His
1 5 10 15
Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys
20 25 30
Asn Pro Lys Leu Thr Ala Met Leu Thr Ala Lys Phe Tyr Met Pro Lys
35 40 45
Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys
50 55 60
Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu
65 70 75 80
Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu
85 90 95
Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala
100 105 110
Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile
115 120 125
Ile Ser Thr Leu Thr
130
<210> 7
<211> 617
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок, содержащий варианты IL-2 и фрагменты CD80
<400> 7
Met Asp Ala Met Leu Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 10 15
Ala Val Phe Val Ser Pro Ser His Ala Val Ile His Val Thr Lys Glu
20 25 30
Val Lys Glu Val Ala Thr Leu Ser Cys Gly His Asn Val Ser Val Glu
35 40 45
Glu Leu Ala Gln Thr Arg Ile Tyr Trp Gln Lys Glu Lys Lys Met Val
50 55 60
Leu Thr Met Met Ser Gly Asp Met Asn Ile Trp Pro Glu Tyr Lys Asn
65 70 75 80
Arg Thr Ile Phe Asp Ile Thr Asn Asn Leu Ser Ile Val Ile Leu Ala
85 90 95
Leu Arg Pro Ser Asp Glu Gly Thr Tyr Glu Cys Val Val Leu Lys Tyr
100 105 110
Glu Lys Asp Ala Phe Lys Arg Glu His Leu Ala Glu Val Thr Leu Ser
115 120 125
Val Lys Ala Asp Phe Pro Thr Pro Ser Ile Ser Asp Phe Glu Ile Pro
130 135 140
Thr Ser Asn Ile Arg Arg Ile Ile Cys Ser Thr Ser Gly Gly Phe Pro
145 150 155 160
Glu Pro His Leu Ser Trp Leu Glu Asn Gly Glu Glu Leu Asn Ala Ile
165 170 175
Asn Thr Thr Val Ser Gln Asp Pro Glu Thr Glu Leu Tyr Ala Val Ser
180 185 190
Ser Lys Leu Asp Phe Asn Met Thr Thr Asn His Ser Phe Met Cys Leu
195 200 205
Ile Lys Tyr Gly His Leu Arg Val Asn Gln Thr Phe Asn Trp Asn Thr
210 215 220
Thr Lys Gln Glu His Phe Pro Asp Asn Gly Ser Gly Gly Gly Gly Ser
225 230 235 240
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Ala Glu Ser Lys Tyr Gly
245 250 255
Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
260 265 270
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Gln Leu Met Ile Ser Arg
275 280 285
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro
290 295 300
Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
305 310 315 320
Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val
325 330 335
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
340 345 350
Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr
355 360 365
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
370 375 380
Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
385 390 395 400
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
405 410 415
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
420 425 430
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser
435 440 445
Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Leu His Glu Ala
450 455 460
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Gly
465 470 475 480
Gly Gly Gly Ser Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu
485 490 495
Gln Leu Glu His Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile
500 505 510
Asn Asn Tyr Lys Asn Pro Lys Leu Thr Ala Met Leu Thr Ala Lys Phe
515 520 525
Tyr Met Pro Lys Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu
530 535 540
Glu Glu Leu Lys Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys
545 550 555 560
Asn Phe His Leu Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile
565 570 575
Val Leu Glu Leu Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala
580 585 590
Asp Glu Thr Ala Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe
595 600 605
Cys Gln Ser Ile Ile Ser Thr Leu Thr
610 615
<210> 8
<211> 1857
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (GI101)
<400> 8
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgat ccacgtgacc aaagaagtga aagaggtcgc cacactgtcc 120
tgcggccaca acgtttcagt ggaagaactg gcccagacca ggatctactg gcagaaagaa 180
aagaaaatgg tgctgaccat gatgtccggc gacatgaaca tctggcctga gtacaagaac 240
cggaccatct tcgacatcac caacaacctg tccatcgtga ttctggccct gaggccttct 300
gatgagggca cctatgagtg cgtggtgctg aagtacgaga aggacgcctt caagcgcgag 360
cacctggctg aagtgacact gtccgtgaag gccgactttc ccacaccttc catctccgac 420
ttcgagatcc ctacctccaa catccggcgg atcatctgtt ctacctctgg cggctttcct 480
gagcctcacc tgtcttggct ggaaaacggc gaggaactga acgccatcaa caccaccgtg 540
tctcaggacc ccgaaaccga gctgtacgct gtgtcctcca agctggactt caacatgacc 600
accaaccaca gcttcatgtg cctgattaag tacggccacc tgagagtgaa ccagaccttc 660
aactggaaca ccaccaagca agagcacttc cctgacaatg gatctggcgg cggaggttct 720
ggcggaggtg gaagcggagg cggaggatct gctgagtcta agtatggccc tccttgtcct 780
ccatgtcctg ctccagaagc tgctggcgga ccctctgtgt tcctgtttcc tccaaagcct 840
aaggaccagc tcatgatctc tcggacaccc gaagtgacct gcgtggtggt ggatgtgtct 900
caagaggacc ctgaggtgca gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 960
aagaccaagc ctagagagga acagttcaac tccacctaca gagtggtgtc cgtgctgacc 1020
gtgctgcacc aggattggct gaacggcaaa gagtacaagt gcaaggtgtc caacaagggc 1080
ctgccttcca gcatcgaaaa gaccatctcc aaggctaagg gccagcctag ggaaccccag 1140
gtttacaccc tgcctccaag ccaagaggaa atgaccaaga accaggtgtc cctgacctgc 1200
ctggtcaagg gcttctaccc ttccgacatt gccgtggaat gggagtccaa tggccagcct 1260
gagaacaact acaagaccac acctcctgtg ctggactccg acggctcctt ctttctgtac 1320
tctcgcctga ccgtggacaa gtctagatgg caagagggca acgtgttctc ctgctctgtg 1380
ctgcacgagg ccctgcacaa tcactacacc cagaagtccc tgtctctgtc tcttggaggt 1440
ggtggcggtt ctgcccctac cagctcctct accaagaaaa cccagctcca gttggagcat 1500
ctgctgctgg acctccagat gattctgaac gggatcaaca actataagaa ccccaagctg 1560
accgccatgc tgaccgctaa gttctacatg cccaagaagg ccaccgagct gaagcacctc 1620
cagtgcctgg aagaagaact gaagcccctg gaagaggtgc tgaatctggc ccagtccaag 1680
aacttccacc tgaggccacg ggacctgatc agcaacatca acgtgatcgt gctggaactg 1740
aagggctccg agacaacctt tatgtgcgag tacgccgacg agacagccac catcgtggaa 1800
tttctgaacc ggtggatcac cttctgccag agcatcatct ccacactgac ctgatga 1857
<210> 9
<211> 592
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (GI101)
<400> 9
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro Asp Asn
195 200 205
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser
450 455 460
Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu
465 470 475 480
Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr
485 490 495
Ala Met Leu Thr Ala Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu
500 505 510
Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val
515 520 525
Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu
530 535 540
Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr
545 550 555 560
Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe
565 570 575
Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
580 585 590
<210> 10
<211> 133
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> hIL-2
<400> 10
Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His
1 5 10 15
Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys
20 25 30
Asn Pro Lys Leu Thr Arg Met Leu Thr Phe Lys Phe Tyr Met Pro Lys
35 40 45
Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys
50 55 60
Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu
65 70 75 80
Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu
85 90 95
Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala
100 105 110
Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile
115 120 125
Ile Ser Thr Leu Thr
130
<210> 11
<211> 288
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CD80
<400> 11
Met Gly His Thr Arg Arg Gln Gly Thr Ser Pro Ser Lys Cys Pro Tyr
1 5 10 15
Leu Asn Phe Phe Gln Leu Leu Val Leu Ala Gly Leu Ser His Phe Cys
20 25 30
Ser Gly Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu
35 40 45
Ser Cys Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile
50 55 60
Tyr Trp Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp
65 70 75 80
Met Asn Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr
85 90 95
Asn Asn Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly
100 105 110
Thr Tyr Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg
115 120 125
Glu His Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr
130 135 140
Pro Ser Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile
145 150 155 160
Ile Cys Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu
165 170 175
Glu Asn Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp
180 185 190
Pro Glu Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met
195 200 205
Thr Thr Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg
210 215 220
Val Asn Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro
225 230 235 240
Asp Asn Leu Leu Pro Ser Trp Ala Ile Thr Leu Ile Ser Val Asn Gly
245 250 255
Ile Phe Val Ile Cys Cys Leu Thr Tyr Cys Phe Ala Pro Arg Cys Arg
260 265 270
Glu Arg Arg Arg Asn Glu Arg Leu Arg Arg Glu Ser Val Arg Pro Val
275 280 285
<210> 12
<211> 215
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> модифицированный Fc
<400> 12
Ser His Thr Gln Pro Leu Gly Val Phe Leu Phe Pro Pro Lys Pro Lys
1 5 10 15
Asp Thr Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
20 25 30
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
35 40 45
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
50 55 60
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
65 70 75 80
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
85 90 95
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
100 105 110
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
115 120 125
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
130 135 140
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
145 150 155 160
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
165 170 175
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
180 185 190
Cys Ser Val Met His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
195 200 205
Leu Ser Leu Ser Leu Gly Lys
210 215
<210> 13
<211> 306
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> mCD80
<400> 13
Met Ala Cys Asn Cys Gln Leu Met Gln Asp Thr Pro Leu Leu Lys Phe
1 5 10 15
Pro Cys Pro Arg Leu Ile Leu Leu Phe Val Leu Leu Ile Arg Leu Ser
20 25 30
Gln Val Ser Ser Asp Val Asp Glu Gln Leu Ser Lys Ser Val Lys Asp
35 40 45
Lys Val Leu Leu Pro Cys Arg Tyr Asn Ser Pro His Glu Asp Glu Ser
50 55 60
Glu Asp Arg Ile Tyr Trp Gln Lys His Asp Lys Val Val Leu Ser Val
65 70 75 80
Ile Ala Gly Lys Leu Lys Val Trp Pro Glu Tyr Lys Asn Arg Thr Leu
85 90 95
Tyr Asp Asn Thr Thr Tyr Ser Leu Ile Ile Leu Gly Leu Val Leu Ser
100 105 110
Asp Arg Gly Thr Tyr Ser Cys Val Val Gln Lys Lys Glu Arg Gly Thr
115 120 125
Tyr Glu Val Lys His Leu Ala Leu Val Lys Leu Ser Ile Lys Ala Asp
130 135 140
Phe Ser Thr Pro Asn Ile Thr Glu Ser Gly Asn Pro Ser Ala Asp Thr
145 150 155 160
Lys Arg Ile Thr Cys Phe Ala Ser Gly Gly Phe Pro Lys Pro Arg Phe
165 170 175
Ser Trp Leu Glu Asn Gly Arg Glu Leu Pro Gly Ile Asn Thr Thr Ile
180 185 190
Ser Gln Asp Pro Glu Ser Glu Leu Tyr Thr Ile Ser Ser Gln Leu Asp
195 200 205
Phe Asn Thr Thr Arg Asn His Thr Ile Lys Cys Leu Ile Lys Tyr Gly
210 215 220
Asp Ala His Val Ser Glu Asp Phe Thr Trp Glu Lys Pro Pro Glu Asp
225 230 235 240
Pro Pro Asp Ser Lys Asn Thr Leu Val Leu Phe Gly Ala Gly Phe Gly
245 250 255
Ala Val Ile Thr Val Val Val Ile Val Val Ile Ile Lys Cys Phe Cys
260 265 270
Lys His Arg Ser Cys Phe Arg Arg Asn Glu Ala Ser Arg Glu Thr Asn
275 280 285
Asn Ser Leu Thr Phe Gly Pro Glu Glu Ala Leu Ala Glu Gln Thr Val
290 295 300
Phe Leu
305
<210> 14
<211> 1848
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (mGI101)
<400> 14
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgga cgagcagctc tccaagtccg tgaaggataa ggtcctgctg 120
ccttgccggt acaactctcc tcacgaggac gagtctgagg accggatcta ctggcagaaa 180
cacgacaagg tggtgctgtc cgtgatcgcc ggaaagctga aagtgtggcc tgagtacaag 240
aacaggaccc tgtacgacaa caccacctac agcctgatca tcctgggcct cgtgctgagc 300
gatagaggca cctattcttg cgtggtgcag aagaaagagc ggggcaccta cgaagtgaag 360
cacctggctc tggtcaagct gtccatcaag gccgacttca gcacccctaa catcaccgag 420
tctggcaacc cttccgccga caccaagaga atcacctgtt tcgcctctgg cggcttccct 480
aagcctcggt tctcttggct ggaaaacggc agagagctgc ccggcatcaa taccaccatt 540
tctcaggacc cagagtccga gctgtacacc atctccagcc agctcgactt taacaccacc 600
agaaaccaca ccatcaagtg cctgattaag tacggcgacg cccacgtgtc cgaggacttt 660
acttgggaga aacctcctga ggaccctcct gactctggat ctggcggcgg aggttctggc 720
ggaggtggaa gcggaggcgg aggatctgct gagtctaagt atggccctcc ttgtcctcca 780
tgtcctgctc cagaagctgc tggcggaccc tctgtgttcc tgtttcctcc aaagcctaag 840
gaccagctca tgatctctcg gacccctgaa gtgacctgcg tggtggtgga tgtgtctcaa 900
gaggaccctg aggtgcagtt caattggtac gtggacggcg tggaagtgca caacgccaag 960
accaagccta gagaggaaca gttcaactcc acctatagag tggtgtccgt gctgaccgtg 1020
ctgcaccagg attggctgaa cggcaaagag tacaagtgca aggtgtccaa caagggcctg 1080
ccttccagca tcgaaaagac catcagcaag gctaagggcc agcctaggga accccaggtt 1140
tacaccctgc ctccaagcca agaggaaatg accaagaacc aggtgtccct gacctgcctg 1200
gtcaagggct tctacccttc cgacattgcc gtggaatggg agtccaatgg ccagcctgag 1260
aacaactaca agaccacacc tcctgtgctg gactccgacg gctccttctt tctgtactct 1320
cgcctgaccg tggacaagtc taggtggcaa gagggcaacg tgttctcctg ctctgtgctg 1380
cacgaggctc tgcacaacca ctacacccag aagtccctgt ctctgtctct tggaggtggt 1440
ggcggttctg cccctacctc cagctctacc aagaaaaccc agctccagtt ggagcatctg 1500
ctgctggacc tccagatgat cctgaatggc atcaacaatt acaagaaccc caagctgacc 1560
gccatgctga ccgctaagtt ctacatgccc aagaaggcca ccgagctgaa gcacttgcag 1620
tgcctggaag aggaactgaa gcccctggaa gaagtgctga atctggccca gtccaagaac 1680
ttccacctga ggcctaggga cctgatctcc aacatcaacg tgatcgtgct ggaactgaaa 1740
ggctccgaga caaccttcat gtgcgagtac gccgacgaga cagccaccat cgtggaattt 1800
ctgaaccggt ggatcacctt ctgccagagc atcatctcca cactgacc 1848
<210> 15
<211> 616
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (mGI101) с сигнальным пептидом
<400> 15
Met Asp Ala Met Leu Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 10 15
Ala Val Phe Val Ser Pro Ser His Ala Val Asp Glu Gln Leu Ser Lys
20 25 30
Ser Val Lys Asp Lys Val Leu Leu Pro Cys Arg Tyr Asn Ser Pro His
35 40 45
Glu Asp Glu Ser Glu Asp Arg Ile Tyr Trp Gln Lys His Asp Lys Val
50 55 60
Val Leu Ser Val Ile Ala Gly Lys Leu Lys Val Trp Pro Glu Tyr Lys
65 70 75 80
Asn Arg Thr Leu Tyr Asp Asn Thr Thr Tyr Ser Leu Ile Ile Leu Gly
85 90 95
Leu Val Leu Ser Asp Arg Gly Thr Tyr Ser Cys Val Val Gln Lys Lys
100 105 110
Glu Arg Gly Thr Tyr Glu Val Lys His Leu Ala Leu Val Lys Leu Ser
115 120 125
Ile Lys Ala Asp Phe Ser Thr Pro Asn Ile Thr Glu Ser Gly Asn Pro
130 135 140
Ser Ala Asp Thr Lys Arg Ile Thr Cys Phe Ala Ser Gly Gly Phe Pro
145 150 155 160
Lys Pro Arg Phe Ser Trp Leu Glu Asn Gly Arg Glu Leu Pro Gly Ile
165 170 175
Asn Thr Thr Ile Ser Gln Asp Pro Glu Ser Glu Leu Tyr Thr Ile Ser
180 185 190
Ser Gln Leu Asp Phe Asn Thr Thr Arg Asn His Thr Ile Lys Cys Leu
195 200 205
Ile Lys Tyr Gly Asp Ala His Val Ser Glu Asp Phe Thr Trp Glu Lys
210 215 220
Pro Pro Glu Asp Pro Pro Asp Ser Gly Ser Gly Gly Gly Gly Ser Gly
225 230 235 240
Gly Gly Gly Ser Gly Gly Gly Gly Ser Ala Glu Ser Lys Tyr Gly Pro
245 250 255
Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Gln Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
290 295 300
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
370 375 380
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Leu His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Gly Gly
465 470 475 480
Gly Gly Ser Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln
485 490 495
Leu Glu His Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn
500 505 510
Asn Tyr Lys Asn Pro Lys Leu Thr Ala Met Leu Thr Ala Lys Phe Tyr
515 520 525
Met Pro Lys Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu
530 535 540
Glu Leu Lys Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn
545 550 555 560
Phe His Leu Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val
565 570 575
Leu Glu Leu Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp
580 585 590
Glu Thr Ala Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys
595 600 605
Gln Ser Ile Ile Ser Thr Leu Thr
610 615
<210> 16
<211> 1437
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (GI101C1)
<400> 16
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgat ccacgtgacc aaagaagtga aagaggtcgc cacactgtcc 120
tgcggccaca acgtttcagt ggaagaactg gcccagacca ggatctactg gcagaaagaa 180
aagaaaatgg tgctgaccat gatgtccggc gacatgaaca tctggcctga gtacaagaac 240
cggaccatct tcgacatcac caacaacctg tccatcgtga ttctggccct gaggccttct 300
gatgagggca cctatgagtg cgtggtgctg aagtacgaga aggacgcctt caagcgcgag 360
cacctggctg aagtgacact gtccgtgaag gccgactttc ccacaccttc catctccgac 420
ttcgagatcc ctacctccaa catccggcgg atcatctgtt ctacctctgg cggctttcct 480
gagcctcacc tgtcttggct ggaaaacggc gaggaactga acgccatcaa caccaccgtg 540
tctcaggacc ccgaaaccga gctgtacgct gtgtcctcca agctggactt caacatgacc 600
accaaccaca gcttcatgtg cctgattaag tacggccacc tgagagtgaa ccagaccttc 660
aactggaaca ccaccaagca agagcacttc cctgacaatg gatctggcgg cggaggttct 720
ggcggaggtg gaagcggagg cggaggatct gctgagtcta agtatggccc tccttgtcct 780
ccatgtcctg ctccagaagc tgctggcgga ccctctgtgt tcctgtttcc tccaaagcct 840
aaggaccagc tcatgatctc tcggacaccc gaagtgacct gcgtggtggt ggatgtgtct 900
caagaggacc ctgaggtgca gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 960
aagaccaagc ctagagagga acagttcaac tccacctaca gagtggtgtc cgtgctgacc 1020
gtgctgcacc aggattggct gaacggcaaa gagtacaagt gcaaggtgtc caacaagggc 1080
ctgccttcca gcatcgaaaa gaccatctcc aaggctaagg gccagcctag ggaaccccag 1140
gtttacaccc tgcctccaag ccaagaggaa atgaccaaga accaggtgtc cctgacctgc 1200
ctggtcaagg gcttctaccc ttccgacatt gccgtggaat gggagtccaa tggccagcct 1260
gagaacaact acaagaccac acctcctgtg ctggactccg acggctcctt ctttctgtac 1320
tctcgcctga ccgtggacaa gtctaggtgg caagagggca acgtgttctc ctgctctgtg 1380
ctgcacgagg ccctgcacaa tcactacacc cagaagtccc tgtctctgtc cctgggc 1437
<210> 17
<211> 454
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (GI101C1)
<400> 17
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro Asp Asn
195 200 205
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Leu Gly
450
<210> 18
<211> 1176
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (GI101C2)
<400> 18
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccatctc acgccgctga gtctaagtac ggccctcctt gtcctccatg tcctgctcca 120
gaagctgctg gcggaccctc tgtgttcctg tttcctccaa agcctaagga ccagctcatg 180
atctctcgga cccctgaagt gacctgcgtg gtggtggatg tgtctcaaga ggaccctgag 240
gtgcagttca attggtacgt ggacggcgtg gaagtgcaca acgccaagac caagcctaga 300
gaggaacagt tcaactccac ctacagagtg gtgtccgtgc tgaccgtgct gcaccaggat 360
tggctgaacg gcaaagagta caagtgcaag gtgtccaaca agggcctgcc ttccagcatc 420
gaaaagacca tctccaaggc taagggccag cctagggaac cccaggttta caccctgcct 480
ccaagccaag aggaaatgac caagaaccag gtgtccctga cctgcctggt caagggcttc 540
tacccttccg acattgccgt ggaatgggag tccaatggcc agcctgagaa caactacaag 600
accacacctc ctgtgctgga ctccgacggc tccttctttc tgtactctcg cctgaccgtg 660
gacaagtcta ggtggcaaga gggcaacgtg ttctcctgct ctgtgctgca cgaggccctg 720
cacaatcact acacccagaa gtccctgtct ctgtctcttg gcggaggcgg aggatctgct 780
cctacctcca gctccaccaa gaaaacccag ctccagttgg agcatctgct gctggacctc 840
cagatgatcc tgaatggcat caacaattac aagaacccca agctgaccgc catgctgacc 900
gctaagttct acatgcccaa gaaggccacc gagctgaagc acctccagtg cctggaagag 960
gaactgaagc ccctggaaga agtgctgaat ctggcccagt ccaagaactt ccacctgagg 1020
cctagggacc tgatctccaa catcaacgtg atcgtgctgg aactgaaagg ctccgagaca 1080
accttcatgt gcgagtacgc cgacgagaca gccaccatcg tggaatttct gaaccggtgg 1140
atcaccttct gccagtccat catctccaca ctgacc 1176
<210> 19
<211> 367
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (GI101C2)
<400> 19
Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu
1 5 10 15
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
20 25 30
Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
35 40 45
Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
50 55 60
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
65 70 75 80
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
85 90 95
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
100 105 110
Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
115 120 125
Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn
130 135 140
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
145 150 155 160
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
165 170 175
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg
180 185 190
Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys
195 200 205
Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
210 215 220
Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser Ser
225 230 235 240
Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu Gln
245 250 255
Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr Ala
260 265 270
Met Leu Thr Ala Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu Lys
275 280 285
His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val Leu
290 295 300
Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu Ile
305 310 315 320
Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr Thr
325 330 335
Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe Leu
340 345 350
Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
355 360 365
<210> 20
<211> 1434
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (mGI101C1)
<400> 20
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgga cgagcagctc tccaagtccg tgaaggataa ggtcctgctg 120
ccttgccggt acaactctcc tcacgaggac gagtctgagg accggatcta ctggcagaaa 180
cacgacaagg tggtgctgtc cgtgatcgcc ggaaagctga aagtgtggcc tgagtacaag 240
aacaggaccc tgtacgacaa caccacctac agcctgatca tcctgggcct cgtgctgagc 300
gatagaggca cctattcttg cgtggtgcag aagaaagagc ggggcaccta cgaagtgaag 360
cacctggctc tggtcaagct gtccatcaag gccgacttca gcacccctaa catcaccgag 420
tctggcaacc cttccgccga caccaagaga atcacctgtt tcgcctctgg cggcttccct 480
aagcctcggt tctcttggct ggaaaacggc agagagctgc ccggcatcaa taccaccatt 540
tctcaggacc cagagtccga gctgtacacc atctccagcc agctcgactt taacaccacc 600
agaaaccaca ccatcaagtg cctgattaag tacggcgacg cccacgtgtc cgaggacttt 660
acttgggaga aacctcctga ggaccctcct gactctggat ctggcggcgg aggttctggc 720
ggaggtggaa gcggaggcgg aggatctgct gagtctaagt atggccctcc ttgtcctcca 780
tgtcctgctc cagaagctgc tggcggaccc tctgtgttcc tgtttcctcc aaagcctaag 840
gaccagctca tgatctctcg gacccctgaa gtgacctgcg tggtggtgga tgtgtctcaa 900
gaggaccctg aggtgcagtt caattggtac gtggacggcg tggaagtgca caacgccaag 960
accaagccta gagaggaaca gttcaactcc acctatagag tggtgtccgt gctgaccgtg 1020
ctgcaccagg attggctgaa cggcaaagag tacaagtgca aggtgtccaa caagggcctg 1080
ccttccagca tcgaaaagac catcagcaag gctaagggcc agcctaggga accccaggtt 1140
tacaccctgc ctccaagcca agaggaaatg accaagaacc aggtgtccct gacctgcctg 1200
gtcaagggct tctacccttc cgacattgcc gtggaatggg agtccaatgg ccagcctgag 1260
aacaactaca agaccacacc tcctgtgctg gactccgacg gctccttctt tctgtactct 1320
cgcctgaccg tggacaagtc taggtggcaa gagggcaacg tgttctcctg ctctgtgctg 1380
cacgaggctc tgcacaacca ctacacccag aagtccctgt ctctgtccct gggc 1434
<210> 21
<211> 478
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (mGI101C1)
<400> 21
Met Asp Ala Met Leu Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 10 15
Ala Val Phe Val Ser Pro Ser His Ala Val Asp Glu Gln Leu Ser Lys
20 25 30
Ser Val Lys Asp Lys Val Leu Leu Pro Cys Arg Tyr Asn Ser Pro His
35 40 45
Glu Asp Glu Ser Glu Asp Arg Ile Tyr Trp Gln Lys His Asp Lys Val
50 55 60
Val Leu Ser Val Ile Ala Gly Lys Leu Lys Val Trp Pro Glu Tyr Lys
65 70 75 80
Asn Arg Thr Leu Tyr Asp Asn Thr Thr Tyr Ser Leu Ile Ile Leu Gly
85 90 95
Leu Val Leu Ser Asp Arg Gly Thr Tyr Ser Cys Val Val Gln Lys Lys
100 105 110
Glu Arg Gly Thr Tyr Glu Val Lys His Leu Ala Leu Val Lys Leu Ser
115 120 125
Ile Lys Ala Asp Phe Ser Thr Pro Asn Ile Thr Glu Ser Gly Asn Pro
130 135 140
Ser Ala Asp Thr Lys Arg Ile Thr Cys Phe Ala Ser Gly Gly Phe Pro
145 150 155 160
Lys Pro Arg Phe Ser Trp Leu Glu Asn Gly Arg Glu Leu Pro Gly Ile
165 170 175
Asn Thr Thr Ile Ser Gln Asp Pro Glu Ser Glu Leu Tyr Thr Ile Ser
180 185 190
Ser Gln Leu Asp Phe Asn Thr Thr Arg Asn His Thr Ile Lys Cys Leu
195 200 205
Ile Lys Tyr Gly Asp Ala His Val Ser Glu Asp Phe Thr Trp Glu Lys
210 215 220
Pro Pro Glu Asp Pro Pro Asp Ser Gly Ser Gly Gly Gly Gly Ser Gly
225 230 235 240
Gly Gly Gly Ser Gly Gly Gly Gly Ser Ala Glu Ser Lys Tyr Gly Pro
245 250 255
Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Gln Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
290 295 300
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
370 375 380
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Leu His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
465 470 475
<210> 22
<211> 133
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> варианты IL-2 (3M, M45)
<400> 22
Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His
1 5 10 15
Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys
20 25 30
Asn Pro Lys Leu Thr Ala Met Leu Thr Ala Lys Phe Ala Met Pro Lys
35 40 45
Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys
50 55 60
Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu
65 70 75 80
Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu
85 90 95
Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala
100 105 110
Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile
115 120 125
Ile Ser Thr Leu Thr
130
<210> 23
<211> 133
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> варианты IL-2 (3M, M61)
<400> 23
Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His
1 5 10 15
Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys
20 25 30
Asn Pro Lys Leu Thr Ala Met Leu Thr Ala Lys Phe Tyr Met Pro Lys
35 40 45
Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Arg Glu Leu Lys
50 55 60
Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu
65 70 75 80
Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu
85 90 95
Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala
100 105 110
Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile
115 120 125
Ile Ser Thr Leu Thr
130
<210> 24
<211> 133
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> варианты IL-2 (3M, M72)
<400> 24
Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His
1 5 10 15
Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys
20 25 30
Asn Pro Lys Leu Thr Ala Met Leu Thr Ala Lys Phe Tyr Met Pro Lys
35 40 45
Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys
50 55 60
Pro Leu Glu Glu Val Leu Asn Gly Ala Gln Ser Lys Asn Phe His Leu
65 70 75 80
Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu
85 90 95
Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala
100 105 110
Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile
115 120 125
Ile Ser Thr Leu Thr
130
<210> 25
<211> 1851
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (GI102-M45)
<400> 25
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgat ccacgtgacc aaagaagtga aagaggtcgc cacactgtcc 120
tgcggccaca acgtttcagt ggaagaactg gcccagacca ggatctactg gcagaaagaa 180
aagaaaatgg tgctgaccat gatgtccggc gacatgaaca tctggcctga gtacaagaac 240
cggaccatct tcgacatcac caacaacctg tccatcgtga ttctggccct gaggccttct 300
gatgagggca cctatgagtg cgtggtgctg aagtacgaga aggacgcctt caagcgcgag 360
cacctggctg aagtgacact gtccgtgaag gccgactttc ccacaccttc catctccgac 420
ttcgagatcc ctacctccaa catccggcgg atcatctgtt ctacctctgg cggctttcct 480
gagcctcacc tgtcttggct ggaaaacggc gaggaactga acgccatcaa caccaccgtg 540
tctcaggacc ccgaaaccga gctgtacgct gtgtcctcca agctggactt caacatgacc 600
accaaccaca gcttcatgtg cctgattaag tacggccacc tgagagtgaa ccagaccttc 660
aactggaaca ccaccaagca agagcacttc cctgacaatg gatctggcgg cggaggttct 720
ggcggaggtg gaagcggagg cggaggatct gctgagtcta agtatggccc tccttgtcct 780
ccatgtcctg ctccagaagc tgctggcgga ccctctgtgt tcctgtttcc tccaaagcct 840
aaggaccagc tcatgatctc tcggacaccc gaagtgacct gcgtggtggt ggatgtgtct 900
caagaggacc ctgaggtgca gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 960
aagaccaagc ctagagagga acagttcaac tccacctaca gagtggtgtc cgtgctgacc 1020
gtgctgcacc aggattggct gaacggcaaa gagtacaagt gcaaggtgtc caacaagggc 1080
ctgccttcca gcatcgaaaa gaccatctcc aaggctaagg gccagcctag ggaaccccag 1140
gtttacaccc tgcctccaag ccaagaggaa atgaccaaga accaggtgtc cctgacctgc 1200
ctggtcaagg gcttctaccc ttccgacatt gccgtggaat gggagtccaa tggccagcct 1260
gagaacaact acaagaccac acctcctgtg ctggactccg acggctcctt ctttctgtac 1320
tctcgcctga ccgtggacaa gtctagatgg caagagggca acgtgttctc ctgctctgtg 1380
ctgcacgagg ccctgcacaa tcactacacc cagaagtccc tgtctctgtc tcttggaggt 1440
ggtggcggtt ctgcccctac cagctcctct accaagaaaa cccagctcca gttggagcat 1500
ctgctgctgg acctccagat gattctgaac gggatcaaca actataagaa ccccaagctg 1560
accgccatgc tgaccgctaa gttcgccatg cccaagaagg ccaccgagct gaagcacctc 1620
cagtgcctgg aagaagaact gaagcccctg gaagaggtgc tgaatctggc ccagtccaag 1680
aacttccacc tgaggccacg ggacctgatc agcaacatca acgtgatcgt gctggaactg 1740
aagggctccg agacaacctt tatgtgcgag tacgccgacg agacagccac catcgtggaa 1800
tttctgaacc ggtggatcac cttctgccag agcatcatct ccacactgac c 1851
<210> 26
<211> 592
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (GI102-M45)
<400> 26
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro Asp Asn
195 200 205
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser
450 455 460
Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu
465 470 475 480
Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr
485 490 495
Ala Met Leu Thr Ala Lys Phe Ala Met Pro Lys Lys Ala Thr Glu Leu
500 505 510
Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val
515 520 525
Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu
530 535 540
Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr
545 550 555 560
Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe
565 570 575
Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
580 585 590
<210> 27
<211> 1851
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (GI102-M61)
<400> 27
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgat ccacgtgacc aaagaagtga aagaggtcgc cacactgtcc 120
tgcggccaca acgtttcagt ggaagaactg gcccagacca ggatctactg gcagaaagaa 180
aagaaaatgg tgctgaccat gatgtccggc gacatgaaca tctggcctga gtacaagaac 240
cggaccatct tcgacatcac caacaacctg tccatcgtga ttctggccct gaggccttct 300
gatgagggca cctatgagtg cgtggtgctg aagtacgaga aggacgcctt caagcgcgag 360
cacctggctg aagtgacact gtccgtgaag gccgactttc ccacaccttc catctccgac 420
ttcgagatcc ctacctccaa catccggcgg atcatctgtt ctacctctgg cggctttcct 480
gagcctcacc tgtcttggct ggaaaacggc gaggaactga acgccatcaa caccaccgtg 540
tctcaggacc ccgaaaccga gctgtacgct gtgtcctcca agctggactt caacatgacc 600
accaaccaca gcttcatgtg cctgattaag tacggccacc tgagagtgaa ccagaccttc 660
aactggaaca ccaccaagca agagcacttc cctgacaatg gatctggcgg cggaggttct 720
ggcggaggtg gaagcggagg cggaggatct gctgagtcta agtatggccc tccttgtcct 780
ccatgtcctg ctccagaagc tgctggcgga ccctctgtgt tcctgtttcc tccaaagcct 840
aaggaccagc tcatgatctc tcggacaccc gaagtgacct gcgtggtggt ggatgtgtct 900
caagaggacc ctgaggtgca gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 960
aagaccaagc ctagagagga acagttcaac tccacctaca gagtggtgtc cgtgctgacc 1020
gtgctgcacc aggattggct gaacggcaaa gagtacaagt gcaaggtgtc caacaagggc 1080
ctgccttcca gcatcgaaaa gaccatctcc aaggctaagg gccagcctag ggaaccccag 1140
gtttacaccc tgcctccaag ccaagaggaa atgaccaaga accaggtgtc cctgacctgc 1200
ctggtcaagg gcttctaccc ttccgacatt gccgtggaat gggagtccaa tggccagcct 1260
gagaacaact acaagaccac acctcctgtg ctggactccg acggctcctt ctttctgtac 1320
tctcgcctga ccgtggacaa gtctagatgg caagagggca acgtgttctc ctgctctgtg 1380
ctgcacgagg ccctgcacaa tcactacacc cagaagtccc tgtctctgtc tcttggaggt 1440
ggtggcggtt ctgcccctac cagctcctct accaagaaaa cccagctcca gttggagcat 1500
ctgctgctgg acctccagat gattctgaac gggatcaaca actataagaa ccccaagctg 1560
accgccatgc tgaccgctaa gttctacatg cccaagaagg ccaccgagct gaagcacctc 1620
cagtgcctgg aaagggaact gaagcccctg gaagaggtgc tgaatctggc ccagtccaag 1680
aacttccacc tgaggccacg ggacctgatc agcaacatca acgtgatcgt gctggaactg 1740
aagggctccg agacaacctt tatgtgcgag tacgccgacg agacagccac catcgtggaa 1800
tttctgaacc ggtggatcac cttctgccag agcatcatct ccacactgac c 1851
<210> 28
<211> 592
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (GI102-M61)
<400> 28
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro Asp Asn
195 200 205
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser
450 455 460
Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu
465 470 475 480
Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr
485 490 495
Ala Met Leu Thr Ala Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu
500 505 510
Lys His Leu Gln Cys Leu Glu Arg Glu Leu Lys Pro Leu Glu Glu Val
515 520 525
Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu
530 535 540
Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr
545 550 555 560
Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe
565 570 575
Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
580 585 590
<210> 29
<211> 1857
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (GI102-M72)
<400> 29
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgat ccacgtgacc aaagaagtga aagaggtcgc cacactgtcc 120
tgcggccaca acgtttcagt ggaagaactg gcccagacca ggatctactg gcagaaagaa 180
aagaaaatgg tgctgaccat gatgtccggc gacatgaaca tctggcctga gtacaagaac 240
cggaccatct tcgacatcac caacaacctg tccatcgtga ttctggccct gaggccttct 300
gatgagggca cctatgagtg cgtggtgctg aagtacgaga aggacgcctt caagcgcgag 360
cacctggctg aagtgacact gtccgtgaag gccgactttc ccacaccttc catctccgac 420
ttcgagatcc ctacctccaa catccggcgg atcatctgtt ctacctctgg cggctttcct 480
gagcctcacc tgtcttggct ggaaaacggc gaggaactga acgccatcaa caccaccgtg 540
tctcaggacc ccgaaaccga gctgtacgct gtgtcctcca agctggactt caacatgacc 600
accaaccaca gcttcatgtg cctgattaag tacggccacc tgagagtgaa ccagaccttc 660
aactggaaca ccaccaagca agagcacttc cctgacaatg gatctggcgg cggaggttct 720
ggcggaggtg gaagcggagg cggaggatct gctgagtcta agtatggccc tccttgtcct 780
ccatgtcctg ctccagaagc tgctggcgga ccctctgtgt tcctgtttcc tccaaagcct 840
aaggaccagc tcatgatctc tcggacaccc gaagtgacct gcgtggtggt ggatgtgtct 900
caagaggacc ctgaggtgca gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 960
aagaccaagc ctagagagga acagttcaac tccacctaca gagtggtgtc cgtgctgacc 1020
gtgctgcacc aggattggct gaacggcaaa gagtacaagt gcaaggtgtc caacaagggc 1080
ctgccttcca gcatcgaaaa gaccatctcc aaggctaagg gccagcctag ggaaccccag 1140
gtttacaccc tgcctccaag ccaagaggaa atgaccaaga accaggtgtc cctgacctgc 1200
ctggtcaagg gcttctaccc ttccgacatt gccgtggaat gggagtccaa tggccagcct 1260
gagaacaact acaagaccac acctcctgtg ctggactccg acggctcctt ctttctgtac 1320
tctcgcctga ccgtggacaa gtctagatgg caagagggca acgtgttctc ctgctctgtg 1380
ctgcacgagg ccctgcacaa tcactacacc cagaagtccc tgtctctgtc tcttggaggt 1440
ggtggcggtt ctgcccctac cagctcctct accaagaaaa cccagctcca gttggagcat 1500
ctgctgctgg acctccagat gattctgaac gggatcaaca actataagaa ccccaagctg 1560
accgccatgc tgaccgctaa gttctacatg cccaagaagg ccaccgagct gaagcacctc 1620
cagtgcctgg aagaagaact gaagcccctg gaagaggtgc tgaatggggc ccagtccaag 1680
aacttccacc tgaggccacg ggacctgatc agcaacatca acgtgatcgt gctggaactg 1740
aagggctccg agacaacctt tatgtgcgag tacgccgacg agacagccac catcgtggaa 1800
tttctgaacc ggtggatcac cttctgccag agcatcatct ccacactgac ctgatga 1857
<210> 30
<211> 592
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (GI102-M72)
<400> 30
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro Asp Asn
195 200 205
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser
450 455 460
Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu
465 470 475 480
Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr
485 490 495
Ala Met Leu Thr Ala Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu
500 505 510
Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val
515 520 525
Leu Asn Gly Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu
530 535 540
Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr
545 550 555 560
Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe
565 570 575
Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
580 585 590
<210> 31
<211> 1851
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (GI101w)
<400> 31
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgat ccacgtgacc aaagaagtga aagaggtcgc cacactgtcc 120
tgcggccaca acgtttcagt ggaagaactg gcccagacca ggatctactg gcagaaagaa 180
aagaaaatgg tgctgaccat gatgtccggc gacatgaaca tctggcctga gtacaagaac 240
cggaccatct tcgacatcac caacaacctg tccatcgtga ttctggccct gaggccttct 300
gatgagggca cctatgagtg cgtggtgctg aagtacgaga aggacgcctt caagcgcgag 360
cacctggctg aagtgacact gtccgtgaag gccgactttc ccacaccttc catctccgac 420
ttcgagatcc ctacctccaa catccggcgg atcatctgtt ctacctctgg cggctttcct 480
gagcctcacc tgtcttggct ggaaaacggc gaggaactga acgccatcaa caccaccgtg 540
tctcaggacc ccgaaaccga gctgtacgct gtgtcctcca agctggactt caacatgacc 600
accaaccaca gcttcatgtg cctgattaag tacggccacc tgagagtgaa ccagaccttc 660
aactggaaca ccaccaagca agagcacttc cctgacaatg gatctggcgg cggaggttct 720
ggcggaggtg gaagcggagg cggaggatct gctgagtcta agtatggccc tccttgtcct 780
ccatgtcctg ctccagaagc tgctggcgga ccctctgtgt tcctgtttcc tccaaagcct 840
aaggaccagc tcatgatctc tcggacaccc gaagtgacct gcgtggtggt ggatgtgtct 900
caagaggacc ctgaggtgca gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 960
aagaccaagc ctagagagga acagttcaac tccacctaca gagtggtgtc cgtgctgacc 1020
gtgctgcacc aggattggct gaacggcaaa gagtacaagt gcaaggtgtc caacaagggc 1080
ctgccttcca gcatcgaaaa gaccatctcc aaggctaagg gccagcctag ggaaccccag 1140
gtttacaccc tgcctccaag ccaagaggaa atgaccaaga accaggtgtc cctgacctgc 1200
ctggtcaagg gcttctaccc ttccgacatt gccgtggaat gggagtccaa tggccagcct 1260
gagaacaact acaagaccac acctcctgtg ctggactccg acggctcctt ctttctgtac 1320
tctcgcctga ccgtggacaa gtctagatgg caagagggca acgtgttctc ctgctctgtg 1380
ctgcacgagg ccctgcacaa tcactacacc cagaagtccc tgtctctgtc tcttggaggt 1440
ggtggcggtt ctgcccctac cagctcctct accaagaaaa cccagctcca gttggagcat 1500
ctgctgctgg acctccagat gattctgaac gggatcaaca actataagaa ccccaagctg 1560
acccgcatgc tgacctttaa gttctacatg cccaagaagg ccaccgagct gaagcacctc 1620
cagtgcctgg aagaagaact gaagcccctg gaagaggtgc tgaatctggc ccagtccaag 1680
aacttccacc tgaggccacg ggacctgatc agcaacatca acgtgatcgt gctggaactg 1740
aagggctccg agacaacctt tatgtgcgag tacgccgacg agacagccac catcgtggaa 1800
tttctgaacc ggtggatcac cttctgccag agcatcatct ccacactgac c 1851
<210> 32
<211> 592
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (GI101w)
<400> 32
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro Asp Asn
195 200 205
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser
450 455 460
Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu
465 470 475 480
Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr
485 490 495
Arg Met Leu Thr Phe Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu
500 505 510
Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val
515 520 525
Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu
530 535 540
Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr
545 550 555 560
Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe
565 570 575
Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
580 585 590
<210> 33
<211> 1848
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (mGI102-M61)
<400> 33
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgga cgagcagctc tccaagtccg tgaaggataa ggtcctgctg 120
ccttgccggt acaactctcc tcacgaggac gagtctgagg accggatcta ctggcagaaa 180
cacgacaagg tggtgctgtc cgtgatcgcc ggaaagctga aagtgtggcc tgagtacaag 240
aacaggaccc tgtacgacaa caccacctac agcctgatca tcctgggcct cgtgctgagc 300
gatagaggca cctattcttg cgtggtgcag aagaaagagc ggggcaccta cgaagtgaag 360
cacctggctc tggtcaagct gtccatcaag gccgacttca gcacccctaa catcaccgag 420
tctggcaacc cttccgccga caccaagaga atcacctgtt tcgcctctgg cggcttccct 480
aagcctcggt tctcttggct ggaaaacggc agagagctgc ccggcatcaa taccaccatt 540
tctcaggacc cagagtccga gctgtacacc atctccagcc agctcgactt taacaccacc 600
agaaaccaca ccatcaagtg cctgattaag tacggcgacg cccacgtgtc cgaggacttt 660
acttgggaga aacctcctga ggaccctcct gactctggat ctggcggcgg aggttctggc 720
ggaggtggaa gcggaggcgg aggatctgct gagtctaagt atggccctcc ttgtcctcca 780
tgtcctgctc cagaagctgc tggcggaccc tctgtgttcc tgtttcctcc aaagcctaag 840
gaccagctca tgatctctcg gacccctgaa gtgacctgcg tggtggtgga tgtgtctcaa 900
gaggaccctg aggtgcagtt caattggtac gtggacggcg tggaagtgca caacgccaag 960
accaagccta gagaggaaca gttcaactcc acctatagag tggtgtccgt gctgaccgtg 1020
ctgcaccagg attggctgaa cggcaaagag tacaagtgca aggtgtccaa caagggcctg 1080
ccttccagca tcgaaaagac catcagcaag gctaagggcc agcctaggga accccaggtt 1140
tacaccctgc ctccaagcca agaggaaatg accaagaacc aggtgtccct gacctgcctg 1200
gtcaagggct tctacccttc cgacattgcc gtggaatggg agtccaatgg ccagcctgag 1260
aacaactaca agaccacacc tcctgtgctg gactccgacg gctccttctt tctgtactct 1320
cgcctgaccg tggacaagtc taggtggcaa gagggcaacg tgttctcctg ctctgtgctg 1380
cacgaggctc tgcacaacca ctacacccag aagtccctgt ctctgtctct tggaggtggt 1440
ggcggttctg cccctacctc cagctctacc aagaaaaccc agctccagtt ggagcatctg 1500
ctgctggacc tccagatgat cctgaatggc atcaacaatt acaagaaccc caagctgacc 1560
gccatgctga ccgctaagtt ctacatgccc aagaaggcca ccgagctgaa gcacttgcag 1620
tgcctggaaa gggaactgaa gcccctggaa gaagtgctga atctggccca gtccaagaac 1680
ttccacctga ggcctaggga cctgatctcc aacatcaacg tgatcgtgct ggaactgaaa 1740
ggctccgaga caaccttcat gtgcgagtac gccgacgaga cagccaccat cgtggaattt 1800
ctgaaccggt ggatcacctt ctgccagagc atcatctcca cactgacc 1848
<210> 34
<211> 616
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> слитый белок (mGI102-M61)
<400> 34
Met Asp Ala Met Leu Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 10 15
Ala Val Phe Val Ser Pro Ser His Ala Val Asp Glu Gln Leu Ser Lys
20 25 30
Ser Val Lys Asp Lys Val Leu Leu Pro Cys Arg Tyr Asn Ser Pro His
35 40 45
Glu Asp Glu Ser Glu Asp Arg Ile Tyr Trp Gln Lys His Asp Lys Val
50 55 60
Val Leu Ser Val Ile Ala Gly Lys Leu Lys Val Trp Pro Glu Tyr Lys
65 70 75 80
Asn Arg Thr Leu Tyr Asp Asn Thr Thr Tyr Ser Leu Ile Ile Leu Gly
85 90 95
Leu Val Leu Ser Asp Arg Gly Thr Tyr Ser Cys Val Val Gln Lys Lys
100 105 110
Glu Arg Gly Thr Tyr Glu Val Lys His Leu Ala Leu Val Lys Leu Ser
115 120 125
Ile Lys Ala Asp Phe Ser Thr Pro Asn Ile Thr Glu Ser Gly Asn Pro
130 135 140
Ser Ala Asp Thr Lys Arg Ile Thr Cys Phe Ala Ser Gly Gly Phe Pro
145 150 155 160
Lys Pro Arg Phe Ser Trp Leu Glu Asn Gly Arg Glu Leu Pro Gly Ile
165 170 175
Asn Thr Thr Ile Ser Gln Asp Pro Glu Ser Glu Leu Tyr Thr Ile Ser
180 185 190
Ser Gln Leu Asp Phe Asn Thr Thr Arg Asn His Thr Ile Lys Cys Leu
195 200 205
Ile Lys Tyr Gly Asp Ala His Val Ser Glu Asp Phe Thr Trp Glu Lys
210 215 220
Pro Pro Glu Asp Pro Pro Asp Ser Gly Ser Gly Gly Gly Gly Ser Gly
225 230 235 240
Gly Gly Gly Ser Gly Gly Gly Gly Ser Ala Glu Ser Lys Tyr Gly Pro
245 250 255
Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
260 265 270
Phe Leu Phe Pro Pro Lys Pro Lys Asp Gln Leu Met Ile Ser Arg Thr
275 280 285
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
290 295 300
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
305 310 315 320
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
325 330 335
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
340 345 350
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
355 360 365
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
370 375 380
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
385 390 395 400
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
405 410 415
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
420 425 430
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
435 440 445
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Leu His Glu Ala Leu
450 455 460
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Gly Gly
465 470 475 480
Gly Gly Ser Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln
485 490 495
Leu Glu His Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn
500 505 510
Asn Tyr Lys Asn Pro Lys Leu Thr Ala Met Leu Thr Ala Lys Phe Tyr
515 520 525
Met Pro Lys Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Arg
530 535 540
Glu Leu Lys Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn
545 550 555 560
Phe His Leu Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val
565 570 575
Leu Glu Leu Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp
580 585 590
Glu Thr Ala Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys
595 600 605
Gln Ser Ile Ile Ser Thr Leu Thr
610 615
<210> 35
<211> 153
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> hIL-2 дикого типа
<400> 35
Met Tyr Arg Met Gln Leu Leu Ser Cys Ile Ala Leu Ser Leu Ala Leu
1 5 10 15
Val Thr Asn Ser Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu
20 25 30
Gln Leu Glu His Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile
35 40 45
Asn Asn Tyr Lys Asn Pro Lys Leu Thr Arg Met Leu Thr Phe Lys Phe
50 55 60
Tyr Met Pro Lys Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu
65 70 75 80
Glu Glu Leu Lys Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys
85 90 95
Asn Phe His Leu Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile
100 105 110
Val Leu Glu Leu Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala
115 120 125
Asp Glu Thr Ala Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe
130 135 140
Cys Gln Ser Ile Ile Ser Thr Leu Thr
145 150
<210> 36
<211> 158
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> IL-2 с сигнальной последовательностью
<400> 36
Met Asp Ala Met Leu Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 10 15
Ala Val Phe Val Ser Pro Ser His Ala Ala Pro Thr Ser Ser Ser Thr
20 25 30
Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu Gln Met
35 40 45
Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr Arg Met
50 55 60
Leu Thr Phe Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu Lys His
65 70 75 80
Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val Leu Asn
85 90 95
Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu Ile Ser
100 105 110
Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr Thr Phe
115 120 125
Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe Leu Asn
130 135 140
Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
145 150 155
<210> 37
<211> 474
<212> ДНК
<213> Искусственная последовательность
<220>
<223> nucleotide sequence coding IL-2 с сигнальной последовательностью
<400> 37
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgcccc taccagctcc tctaccaaga aaacccagct ccagttggag 120
catctgctgc tggacctcca gatgattctg aacgggatca acaactataa gaaccccaag 180
ctgacccgca tgctgacctt taagttctac atgcccaaga aggccaccga gctgaagcac 240
ctccagtgcc tggaagaaga actgaagccc ctggaagagg tgctgaatct ggcccagtcc 300
aagaacttcc acctgaggcc acgggacctg atcagcaaca tcaacgtgat cgtgctggaa 360
ctgaagggct ccgagacaac ctttatgtgc gagtacgccg acgagacagc caccatcgtg 420
gaatttctga accggtggat caccttctgc cagagcatca tctccacact gacc 474
<210> 38
<211> 13
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> шарнир
<400> 38
Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro
1 5 10
<210> 39
<211> 1461
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие белок CD80-Fc
<400> 39
ggatccgcca ccatggatgc tatgctgaga ggcctgtgtt gcgtgctgct gctgtgtggc 60
gctgtgttcg tgtctccttc tcacgctgtg atccacgtga ccaaagaagt gaaagaggtc 120
gccacactgt cctgcggcca caacgtttca gtggaagaac tggcccagac caggatctac 180
tggcagaaag aaaagaaaat ggtgctgacc atgatgtccg gcgacatgaa catctggcct 240
gagtacaaga accggaccat cttcgacatc accaacaacc tgtccatcgt gattctggcc 300
ctgaggcctt ctgatgaggg cacctatgag tgcgtggtgc tgaagtacga gaaggacgcc 360
ttcaagcgcg agcacctggc tgaagtgaca ctgtccgtga aggccgactt tcccacacct 420
tccatctccg acttcgagat ccctacctcc aacatccggc ggatcatctg ttctacctct 480
ggcggctttc ctgagcctca cctgtcttgg ctggaaaacg gcgaggaact gaacgccatc 540
aacaccaccg tgtctcagga ccccgaaacc gagctgtacg ctgtgtcctc caagctggac 600
ttcaacatga ccaccaacca cagcttcatg tgcctgatta agtacggcca cctgagagtg 660
aaccagacct tcaactggaa caccaccaag caagagcact tccctgacaa tggatctggc 720
ggcggaggtt ctggcggagg tggaagcgga ggcggaggat ctgctgagtc taagtatggc 780
cctccttgtc ctccatgtcc tgctccagaa gctgctggcg gaccctctgt gttcctgttt 840
cctccaaagc ctaaggacca gctcatgatc tctcggacac ccgaagtgac ctgcgtggtg 900
gtggatgtgt ctcaagagga ccctgaggtg cagttcaatt ggtacgtgga cggcgtggaa 960
gtgcacaacg ccaagaccaa gcctagagag gaacagttca actccaccta cagagtggtg 1020
tccgtgctga ccgtgctgca ccaggattgg ctgaacggca aagagtacaa gtgcaaggtg 1080
tccaacaagg gcctgccttc cagcatcgaa aagaccatct ccaaggctaa gggccagcct 1140
agggaacccc aggtttacac cctgcctcca agccaagagg aaatgaccaa gaaccaggtg 1200
tccctgacct gcctggtcaa gggcttctac ccttccgaca ttgccgtgga atgggagtcc 1260
aatggccagc ctgagaacaa ctacaagacc acacctcctg tgctggactc cgacggctcc 1320
ttctttctgt actctcgcct gaccgtggac aagtctaggt ggcaagaggg caacgtgttc 1380
tcctgctctg tgctgcacga ggccctgcac aatcactaca cccagaagtc cctgtctctg 1440
tccctgggct gatgactcga g 1461
<210> 40
<211> 479
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CD80-Fc белок
<400> 40
Met Asp Ala Met Leu Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 10 15
Ala Val Phe Val Ser Pro Ser His Ala Val Ile His Val Thr Lys Glu
20 25 30
Val Lys Glu Val Ala Thr Leu Ser Cys Gly His Asn Val Ser Val Glu
35 40 45
Glu Leu Ala Gln Thr Arg Ile Tyr Trp Gln Lys Glu Lys Lys Met Val
50 55 60
Leu Thr Met Met Ser Gly Asp Met Asn Ile Trp Pro Glu Tyr Lys Asn
65 70 75 80
Arg Thr Ile Phe Asp Ile Thr Asn Asn Leu Ser Ile Val Ile Leu Ala
85 90 95
Leu Arg Pro Ser Asp Glu Gly Thr Tyr Glu Cys Val Val Leu Lys Tyr
100 105 110
Glu Lys Asp Ala Phe Lys Arg Glu His Leu Ala Glu Val Thr Leu Ser
115 120 125
Val Lys Ala Asp Phe Pro Thr Pro Ser Ile Ser Asp Phe Glu Ile Pro
130 135 140
Thr Ser Asn Ile Arg Arg Ile Ile Cys Ser Thr Ser Gly Gly Phe Pro
145 150 155 160
Glu Pro His Leu Ser Trp Leu Glu Asn Gly Glu Glu Leu Asn Ala Ile
165 170 175
Asn Thr Thr Val Ser Gln Asp Pro Glu Thr Glu Leu Tyr Ala Val Ser
180 185 190
Ser Lys Leu Asp Phe Asn Met Thr Thr Asn His Ser Phe Met Cys Leu
195 200 205
Ile Lys Tyr Gly His Leu Arg Val Asn Gln Thr Phe Asn Trp Asn Thr
210 215 220
Thr Lys Gln Glu His Phe Pro Asp Asn Gly Ser Gly Gly Gly Gly Ser
225 230 235 240
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Ala Glu Ser Lys Tyr Gly
245 250 255
Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser
260 265 270
Val Phe Leu Phe Pro Pro Lys Pro Lys Asp Gln Leu Met Ile Ser Arg
275 280 285
Thr Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro
290 295 300
Glu Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala
305 310 315 320
Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val
325 330 335
Ser Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr
340 345 350
Lys Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr
355 360 365
Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu
370 375 380
Pro Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys
385 390 395 400
Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser
405 410 415
Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp
420 425 430
Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser
435 440 445
Arg Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Leu His Glu Ala
450 455 460
Leu His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly
465 470 475
<210> 41
<211> 1851
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие слитый белок (hCD80-Fc-IL2wt)
<400> 41
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgtgat ccacgtgacc aaagaagtga aagaggtcgc cacactgtcc 120
tgcggccaca acgtttcagt ggaagaactg gcccagacca ggatctactg gcagaaagaa 180
aagaaaatgg tgctgaccat gatgtccggc gacatgaaca tctggcctga gtacaagaac 240
cggaccatct tcgacatcac caacaacctg tccatcgtga ttctggccct gaggccttct 300
gatgagggca cctatgagtg cgtggtgctg aagtacgaga aggacgcctt caagcgcgag 360
cacctggctg aagtgacact gtccgtgaag gccgactttc ccacaccttc catctccgac 420
ttcgagatcc ctacctccaa catccggcgg atcatctgtt ctacctctgg cggctttcct 480
gagcctcacc tgtcttggct ggaaaacggc gaggaactga acgccatcaa caccaccgtg 540
tctcaggacc ccgaaaccga gctgtacgct gtgtcctcca agctggactt caacatgacc 600
accaaccaca gcttcatgtg cctgattaag tacggccacc tgagagtgaa ccagaccttc 660
aactggaaca ccaccaagca agagcacttc cctgacaatg gatctggcgg cggaggttct 720
ggcggaggtg gaagcggagg cggaggatct gctgagtcta agtatggccc tccttgtcct 780
ccatgtcctg ctccagaagc tgctggcgga ccctctgtgt tcctgtttcc tccaaagcct 840
aaggaccagc tcatgatctc tcggacaccc gaagtgacct gcgtggtggt ggatgtgtct 900
caagaggacc ctgaggtgca gttcaattgg tacgtggacg gcgtggaagt gcacaacgcc 960
aagaccaagc ctagagagga acagttcaac tccacctaca gagtggtgtc cgtgctgacc 1020
gtgctgcacc aggattggct gaacggcaaa gagtacaagt gcaaggtgtc caacaagggc 1080
ctgccttcca gcatcgaaaa gaccatctcc aaggctaagg gccagcctag ggaaccccag 1140
gtttacaccc tgcctccaag ccaagaggaa atgaccaaga accaggtgtc cctgacctgc 1200
ctggtcaagg gcttctaccc ttccgacatt gccgtggaat gggagtccaa tggccagcct 1260
gagaacaact acaagaccac acctcctgtg ctggactccg acggctcctt ctttctgtac 1320
tctcgcctga ccgtggacaa gtctagatgg caagagggca acgtgttctc ctgctctgtg 1380
ctgcacgagg ccctgcacaa tcactacacc cagaagtccc tgtctctgtc tcttggaggt 1440
ggtggcggtt ctgcccctac cagctcctct accaagaaaa cccagctcca gttggagcat 1500
ctgctgctgg acctccagat gattctgaac gggatcaaca actataagaa ccccaagctg 1560
acccgcatgc tgacctttaa gttctacatg cccaagaagg ccaccgagct gaagcacctc 1620
cagtgcctgg aagaagaact gaagcccctg gaagaggtgc tgaatctggc ccagtccaag 1680
aacttccacc tgaggccacg ggacctgatc agcaacatca acgtgatcgt gctggaactg 1740
aagggctccg agacaacctt tatgtgcgag tacgccgacg agacagccac catcgtggaa 1800
tttctgaacc ggtggatcac cttctgccag agcatcatct ccacactgac c 1851
<210> 42
<211> 367
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Fc-IL2wt
<400> 42
Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu
1 5 10 15
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
20 25 30
Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
35 40 45
Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
50 55 60
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
65 70 75 80
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
85 90 95
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
100 105 110
Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
115 120 125
Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn
130 135 140
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
145 150 155 160
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
165 170 175
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg
180 185 190
Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys
195 200 205
Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
210 215 220
Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser Ser
225 230 235 240
Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu Gln
245 250 255
Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr Arg
260 265 270
Met Leu Thr Phe Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu Lys
275 280 285
His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val Leu
290 295 300
Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu Ile
305 310 315 320
Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr Thr
325 330 335
Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe Leu
340 345 350
Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
355 360 365
<210> 43
<211> 1176
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Fc-IL2wt
<400> 43
atggatgcta tgctgagagg cctgtgttgc gtgctgctgc tgtgtggcgc tgtgttcgtg 60
tctccttctc acgctgctga gtctaagtat ggccctcctt gtcctccatg tcctgctcca 120
gaagctgctg gcggaccctc tgtgttcctg tttcctccaa agcctaagga ccagctcatg 180
atctctcgga cacccgaagt gacctgcgtg gtggtggatg tgtctcaaga ggaccctgag 240
gtgcagttca attggtacgt ggacggcgtg gaagtgcaca acgccaagac caagcctaga 300
gaggaacagt tcaactccac ctacagagtg gtgtccgtgc tgaccgtgct gcaccaggat 360
tggctgaacg gcaaagagta caagtgcaag gtgtccaaca agggcctgcc ttccagcatc 420
gaaaagacca tctccaaggc taagggccag cctagggaac cccaggttta caccctgcct 480
ccaagccaag aggaaatgac caagaaccag gtgtccctga cctgcctggt caagggcttc 540
tacccttccg acattgccgt ggaatgggag tccaatggcc agcctgagaa caactacaag 600
accacacctc ctgtgctgga ctccgacggc tccttctttc tgtactctcg cctgaccgtg 660
gacaagtcta gatggcaaga gggcaacgtg ttctcctgct ctgtgctgca cgaggccctg 720
cacaatcact acacccagaa gtccctgtct ctgtctcttg gaggtggtgg cggttctgcc 780
cctaccagct cctctaccaa gaaaacccag ctccagttgg agcatctgct gctggacctc 840
cagatgattc tgaacgggat caacaactat aagaacccca agctgacccg catgctgacc 900
tttaagttct acatgcccaa gaaggccacc gagctgaagc acctccagtg cctggaagaa 960
gaactgaagc ccctggaaga ggtgctgaat ctggcccagt ccaagaactt ccacctgagg 1020
ccacgggacc tgatcagcaa catcaacgtg atcgtgctgg aactgaaggg ctccgagaca 1080
acctttatgt gcgagtacgc cgacgagaca gccaccatcg tggaatttct gaaccggtgg 1140
atcaccttct gccagagcat catctccaca ctgacc 1176
<210> 44
<211> 392
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> Fc-IL2v2
<400> 44
Met Asp Ala Met Leu Arg Gly Leu Cys Cys Val Leu Leu Leu Cys Gly
1 5 10 15
Ala Val Phe Val Ser Pro Ser His Ala Ala Glu Ser Lys Tyr Gly Pro
20 25 30
Pro Cys Pro Pro Cys Pro Ala Pro Glu Ala Ala Gly Gly Pro Ser Val
35 40 45
Phe Leu Phe Pro Pro Lys Pro Lys Asp Gln Leu Met Ile Ser Arg Thr
50 55 60
Pro Glu Val Thr Cys Val Val Val Asp Val Ser Gln Glu Asp Pro Glu
65 70 75 80
Val Gln Phe Asn Trp Tyr Val Asp Gly Val Glu Val His Asn Ala Lys
85 90 95
Thr Lys Pro Arg Glu Glu Gln Phe Asn Ser Thr Tyr Arg Val Val Ser
100 105 110
Val Leu Thr Val Leu His Gln Asp Trp Leu Asn Gly Lys Glu Tyr Lys
115 120 125
Cys Lys Val Ser Asn Lys Gly Leu Pro Ser Ser Ile Glu Lys Thr Ile
130 135 140
Ser Lys Ala Lys Gly Gln Pro Arg Glu Pro Gln Val Tyr Thr Leu Pro
145 150 155 160
Pro Ser Gln Glu Glu Met Thr Lys Asn Gln Val Ser Leu Thr Cys Leu
165 170 175
Val Lys Gly Phe Tyr Pro Ser Asp Ile Ala Val Glu Trp Glu Ser Asn
180 185 190
Gly Gln Pro Glu Asn Asn Tyr Lys Thr Thr Pro Pro Val Leu Asp Ser
195 200 205
Asp Gly Ser Phe Phe Leu Tyr Ser Arg Leu Thr Val Asp Lys Ser Arg
210 215 220
Trp Gln Glu Gly Asn Val Phe Ser Cys Ser Val Leu His Glu Ala Leu
225 230 235 240
His Asn His Tyr Thr Gln Lys Ser Leu Ser Leu Ser Leu Gly Gly Gly
245 250 255
Gly Gly Ser Ala Pro Thr Ser Ser Ser Thr Lys Lys Thr Gln Leu Gln
260 265 270
Leu Glu His Leu Leu Leu Asp Leu Gln Met Ile Leu Asn Gly Ile Asn
275 280 285
Asn Tyr Lys Asn Pro Lys Leu Thr Ala Met Leu Thr Ala Lys Phe Tyr
290 295 300
Met Pro Lys Lys Ala Thr Glu Leu Lys His Leu Gln Cys Leu Glu Glu
305 310 315 320
Glu Leu Lys Pro Leu Glu Glu Val Leu Asn Leu Ala Gln Ser Lys Asn
325 330 335
Phe His Leu Arg Pro Arg Asp Leu Ile Ser Asn Ile Asn Val Ile Val
340 345 350
Leu Glu Leu Lys Gly Ser Glu Thr Thr Phe Met Cys Glu Tyr Ala Asp
355 360 365
Glu Thr Ala Thr Ile Val Glu Phe Leu Asn Arg Trp Ile Thr Phe Cys
370 375 380
Gln Ser Ile Ile Ser Thr Leu Thr
385 390
<210> 45
<211> 1200
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотиды, кодирующие Fc-IL2v2
<400> 45
ggatccgcca ccatggatgc tatgctgaga ggcctgtgtt gcgtgctgct gctgtgtggc 60
gctgtgttcg tgtctccatc tcacgccgct gagtctaagt acggccctcc ttgtcctcca 120
tgtcctgctc cagaagctgc tggcggaccc tctgtgttcc tgtttcctcc aaagcctaag 180
gaccagctca tgatctctcg gacccctgaa gtgacctgcg tggtggtgga tgtgtctcaa 240
gaggaccctg aggtgcagtt caattggtac gtggacggcg tggaagtgca caacgccaag 300
accaagccta gagaggaaca gttcaactcc acctacagag tggtgtccgt gctgaccgtg 360
ctgcaccagg attggctgaa cggcaaagag tacaagtgca aggtgtccaa caagggcctg 420
ccttccagca tcgaaaagac catctccaag gctaagggcc agcctaggga accccaggtt 480
tacaccctgc ctccaagcca agaggaaatg accaagaacc aggtgtccct gacctgcctg 540
gtcaagggct tctacccttc cgacattgcc gtggaatggg agtccaatgg ccagcctgag 600
aacaactaca agaccacacc tcctgtgctg gactccgacg gctccttctt tctgtactct 660
cgcctgaccg tggacaagtc taggtggcaa gagggcaacg tgttctcctg ctctgtgctg 720
cacgaggccc tgcacaatca ctacacccag aagtccctgt ctctgtctct tggcggaggc 780
ggaggatctg ctcctacctc cagctccacc aagaaaaccc agctccagtt ggagcatctg 840
ctgctggacc tccagatgat cctgaatggc atcaacaatt acaagaaccc caagctgacc 900
gccatgctga ccgctaagtt ctacatgccc aagaaggcca ccgagctgaa gcacctccag 960
tgcctggaag aggaactgaa gcccctggaa gaagtgctga atctggccca gtccaagaac 1020
ttccacctga ggcctaggga cctgatctcc aacatcaacg tgatcgtgct ggaactgaaa 1080
ggctccgaga caaccttcat gtgcgagtac gccgacgaga cagccaccat cgtggaattt 1140
ctgaaccggt ggatcacctt ctgccagtcc atcatctcca cactgacctg atgactcgag 1200
<210> 46
<211> 592
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> CD80-Fc-IL2wt
<400> 46
Val Ile His Val Thr Lys Glu Val Lys Glu Val Ala Thr Leu Ser Cys
1 5 10 15
Gly His Asn Val Ser Val Glu Glu Leu Ala Gln Thr Arg Ile Tyr Trp
20 25 30
Gln Lys Glu Lys Lys Met Val Leu Thr Met Met Ser Gly Asp Met Asn
35 40 45
Ile Trp Pro Glu Tyr Lys Asn Arg Thr Ile Phe Asp Ile Thr Asn Asn
50 55 60
Leu Ser Ile Val Ile Leu Ala Leu Arg Pro Ser Asp Glu Gly Thr Tyr
65 70 75 80
Glu Cys Val Val Leu Lys Tyr Glu Lys Asp Ala Phe Lys Arg Glu His
85 90 95
Leu Ala Glu Val Thr Leu Ser Val Lys Ala Asp Phe Pro Thr Pro Ser
100 105 110
Ile Ser Asp Phe Glu Ile Pro Thr Ser Asn Ile Arg Arg Ile Ile Cys
115 120 125
Ser Thr Ser Gly Gly Phe Pro Glu Pro His Leu Ser Trp Leu Glu Asn
130 135 140
Gly Glu Glu Leu Asn Ala Ile Asn Thr Thr Val Ser Gln Asp Pro Glu
145 150 155 160
Thr Glu Leu Tyr Ala Val Ser Ser Lys Leu Asp Phe Asn Met Thr Thr
165 170 175
Asn His Ser Phe Met Cys Leu Ile Lys Tyr Gly His Leu Arg Val Asn
180 185 190
Gln Thr Phe Asn Trp Asn Thr Thr Lys Gln Glu His Phe Pro Asp Asn
195 200 205
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
210 215 220
Ser Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro
225 230 235 240
Glu Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys
245 250 255
Asp Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val
260 265 270
Asp Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp
275 280 285
Gly Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe
290 295 300
Asn Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp
305 310 315 320
Trp Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu
325 330 335
Pro Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg
340 345 350
Glu Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys
355 360 365
Asn Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp
370 375 380
Ile Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys
385 390 395 400
Thr Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser
405 410 415
Arg Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser
420 425 430
Cys Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser
435 440 445
Leu Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser
450 455 460
Ser Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu
465 470 475 480
Gln Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr
485 490 495
Arg Met Leu Thr Phe Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu
500 505 510
Lys His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val
515 520 525
Leu Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu
530 535 540
Ile Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr
545 550 555 560
Thr Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe
565 570 575
Leu Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
580 585 590
<210> 47
<211> 591
<212> ПРТ
<213> Искусственная последовательность
<220>
<223> mGI-101
<400> 47
Val Asp Glu Gln Leu Ser Lys Ser Val Lys Asp Lys Val Leu Leu Pro
1 5 10 15
Cys Arg Tyr Asn Ser Pro His Glu Asp Glu Ser Glu Asp Arg Ile Tyr
20 25 30
Trp Gln Lys His Asp Lys Val Val Leu Ser Val Ile Ala Gly Lys Leu
35 40 45
Lys Val Trp Pro Glu Tyr Lys Asn Arg Thr Leu Tyr Asp Asn Thr Thr
50 55 60
Tyr Ser Leu Ile Ile Leu Gly Leu Val Leu Ser Asp Arg Gly Thr Tyr
65 70 75 80
Ser Cys Val Val Gln Lys Lys Glu Arg Gly Thr Tyr Glu Val Lys His
85 90 95
Leu Ala Leu Val Lys Leu Ser Ile Lys Ala Asp Phe Ser Thr Pro Asn
100 105 110
Ile Thr Glu Ser Gly Asn Pro Ser Ala Asp Thr Lys Arg Ile Thr Cys
115 120 125
Phe Ala Ser Gly Gly Phe Pro Lys Pro Arg Phe Ser Trp Leu Glu Asn
130 135 140
Gly Arg Glu Leu Pro Gly Ile Asn Thr Thr Ile Ser Gln Asp Pro Glu
145 150 155 160
Ser Glu Leu Tyr Thr Ile Ser Ser Gln Leu Asp Phe Asn Thr Thr Arg
165 170 175
Asn His Thr Ile Lys Cys Leu Ile Lys Tyr Gly Asp Ala His Val Ser
180 185 190
Glu Asp Phe Thr Trp Glu Lys Pro Pro Glu Asp Pro Pro Asp Ser Gly
195 200 205
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
210 215 220
Ala Glu Ser Lys Tyr Gly Pro Pro Cys Pro Pro Cys Pro Ala Pro Glu
225 230 235 240
Ala Ala Gly Gly Pro Ser Val Phe Leu Phe Pro Pro Lys Pro Lys Asp
245 250 255
Gln Leu Met Ile Ser Arg Thr Pro Glu Val Thr Cys Val Val Val Asp
260 265 270
Val Ser Gln Glu Asp Pro Glu Val Gln Phe Asn Trp Tyr Val Asp Gly
275 280 285
Val Glu Val His Asn Ala Lys Thr Lys Pro Arg Glu Glu Gln Phe Asn
290 295 300
Ser Thr Tyr Arg Val Val Ser Val Leu Thr Val Leu His Gln Asp Trp
305 310 315 320
Leu Asn Gly Lys Glu Tyr Lys Cys Lys Val Ser Asn Lys Gly Leu Pro
325 330 335
Ser Ser Ile Glu Lys Thr Ile Ser Lys Ala Lys Gly Gln Pro Arg Glu
340 345 350
Pro Gln Val Tyr Thr Leu Pro Pro Ser Gln Glu Glu Met Thr Lys Asn
355 360 365
Gln Val Ser Leu Thr Cys Leu Val Lys Gly Phe Tyr Pro Ser Asp Ile
370 375 380
Ala Val Glu Trp Glu Ser Asn Gly Gln Pro Glu Asn Asn Tyr Lys Thr
385 390 395 400
Thr Pro Pro Val Leu Asp Ser Asp Gly Ser Phe Phe Leu Tyr Ser Arg
405 410 415
Leu Thr Val Asp Lys Ser Arg Trp Gln Glu Gly Asn Val Phe Ser Cys
420 425 430
Ser Val Leu His Glu Ala Leu His Asn His Tyr Thr Gln Lys Ser Leu
435 440 445
Ser Leu Ser Leu Gly Gly Gly Gly Gly Ser Ala Pro Thr Ser Ser Ser
450 455 460
Thr Lys Lys Thr Gln Leu Gln Leu Glu His Leu Leu Leu Asp Leu Gln
465 470 475 480
Met Ile Leu Asn Gly Ile Asn Asn Tyr Lys Asn Pro Lys Leu Thr Ala
485 490 495
Met Leu Thr Ala Lys Phe Tyr Met Pro Lys Lys Ala Thr Glu Leu Lys
500 505 510
His Leu Gln Cys Leu Glu Glu Glu Leu Lys Pro Leu Glu Glu Val Leu
515 520 525
Asn Leu Ala Gln Ser Lys Asn Phe His Leu Arg Pro Arg Asp Leu Ile
530 535 540
Ser Asn Ile Asn Val Ile Val Leu Glu Leu Lys Gly Ser Glu Thr Thr
545 550 555 560
Phe Met Cys Glu Tyr Ala Asp Glu Thr Ala Thr Ile Val Glu Phe Leu
565 570 575
Asn Arg Trp Ile Thr Phe Cys Gln Ser Ile Ile Ser Thr Leu Thr
580 585 590
<---
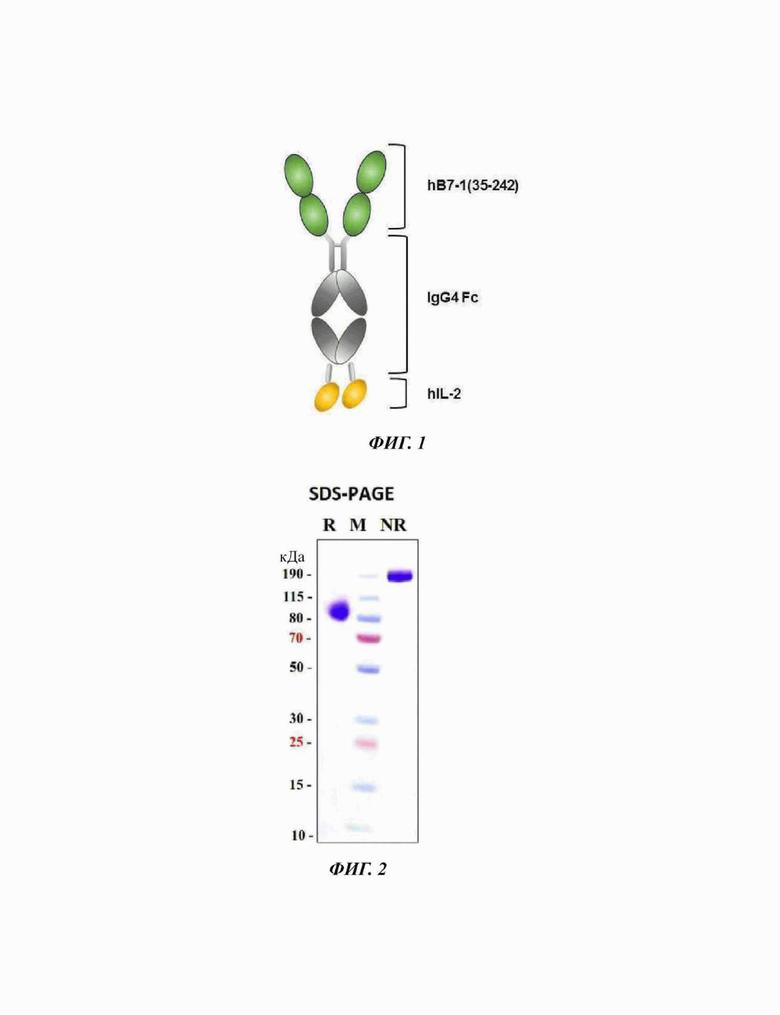
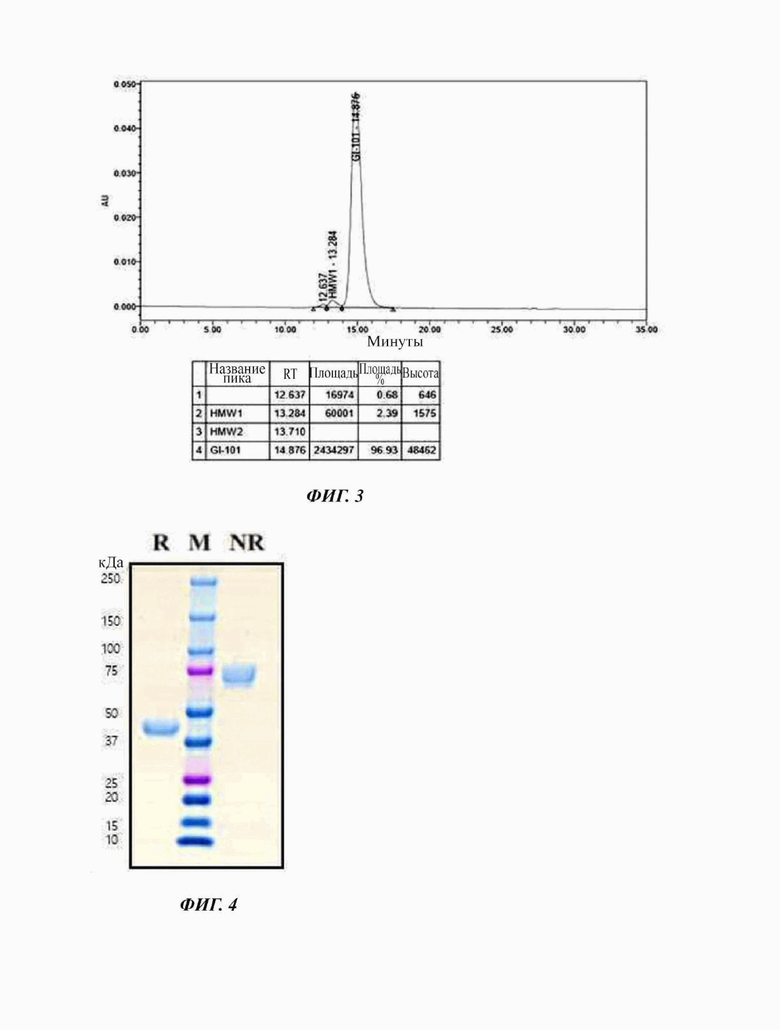
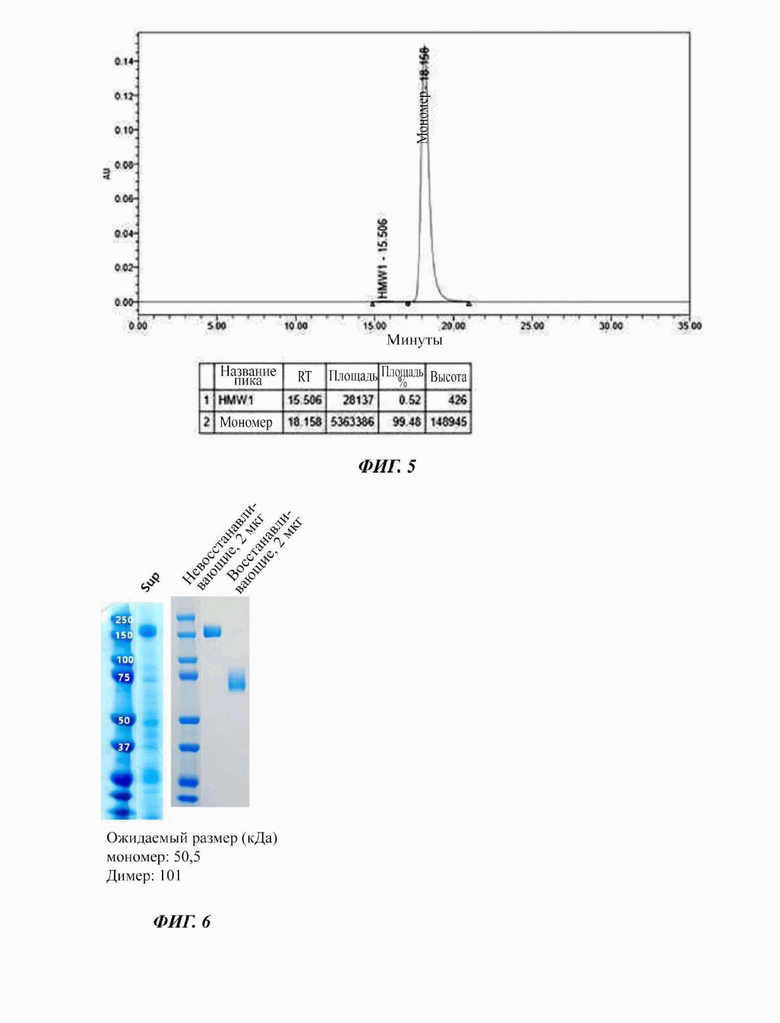
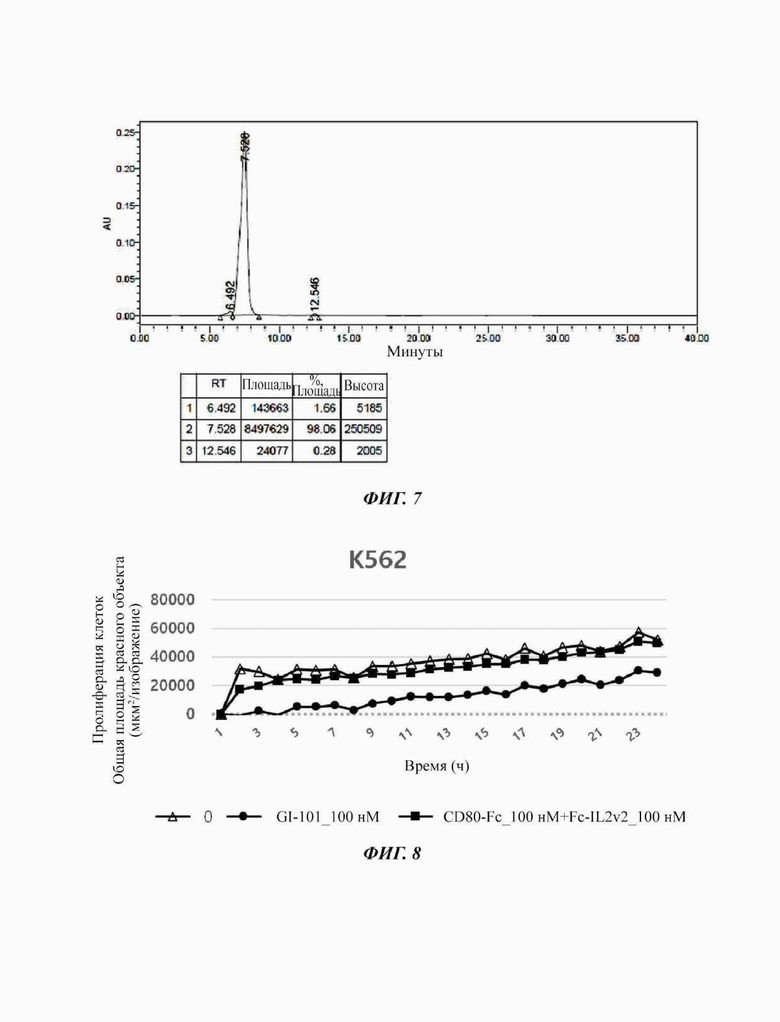
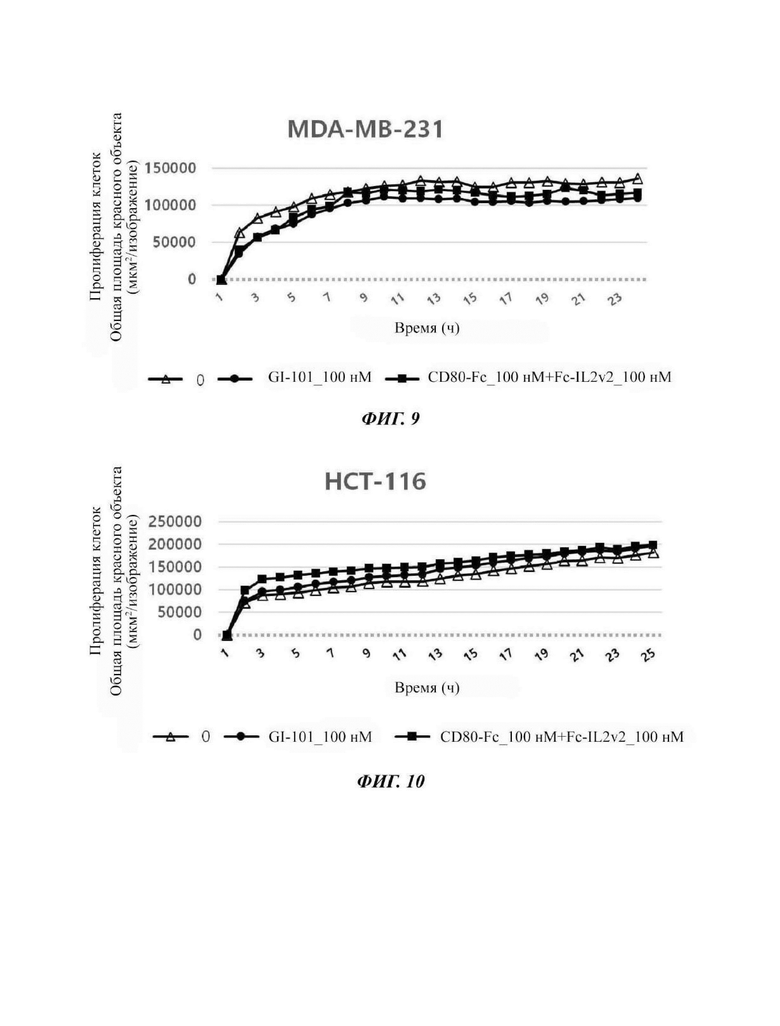
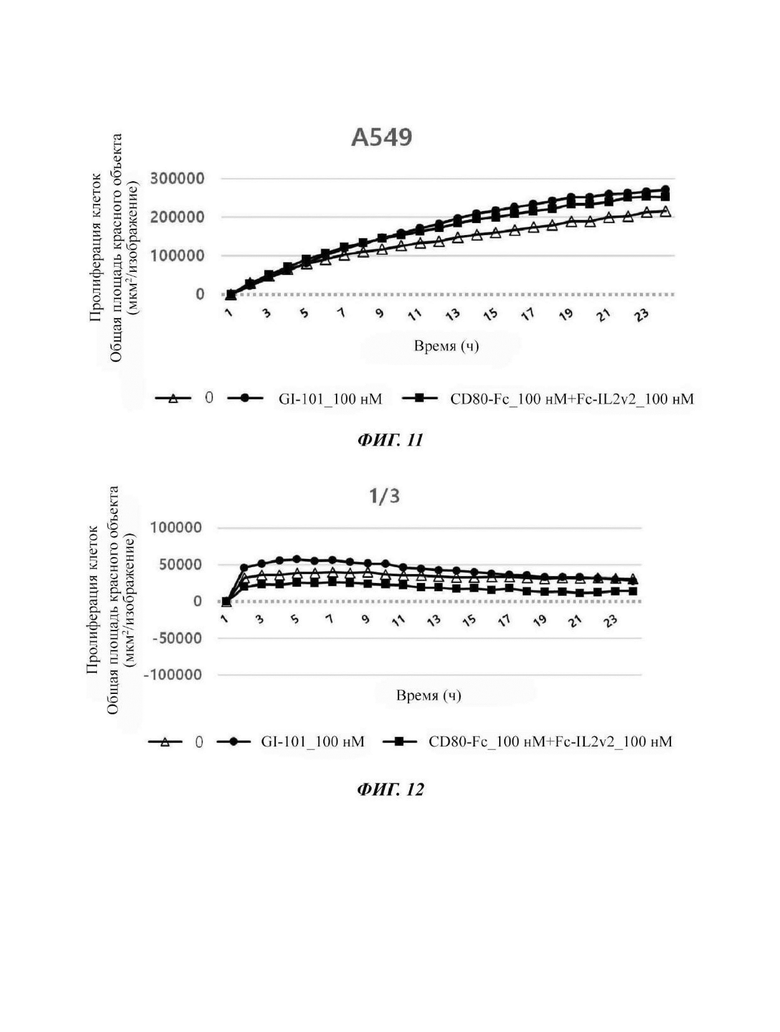
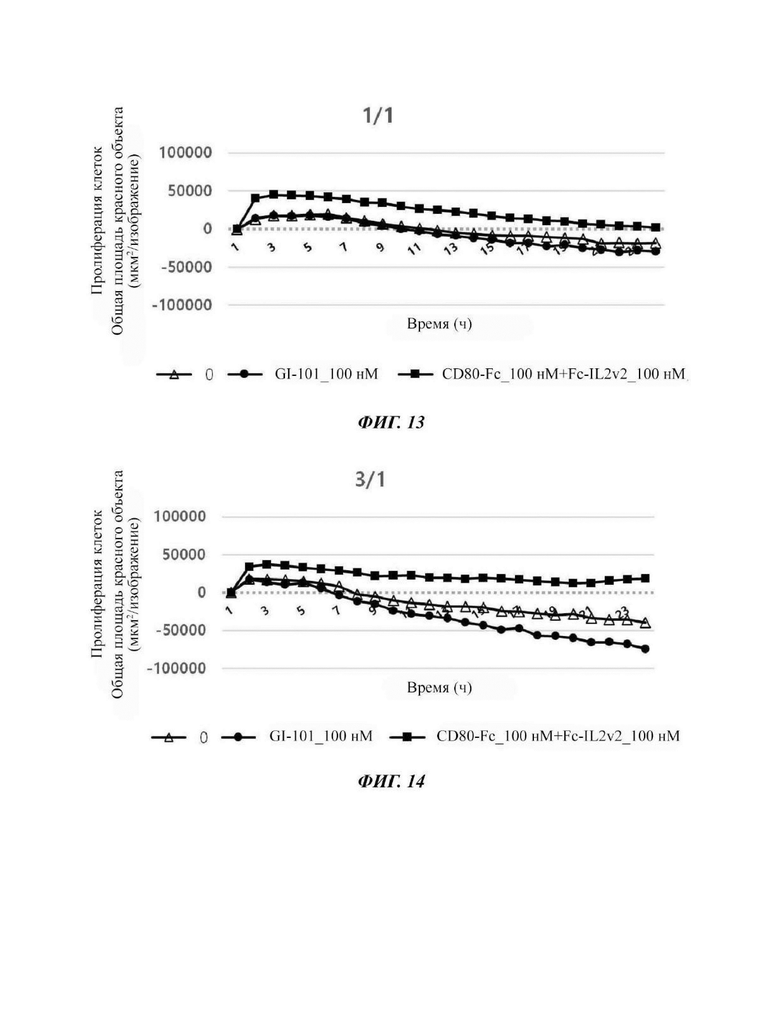
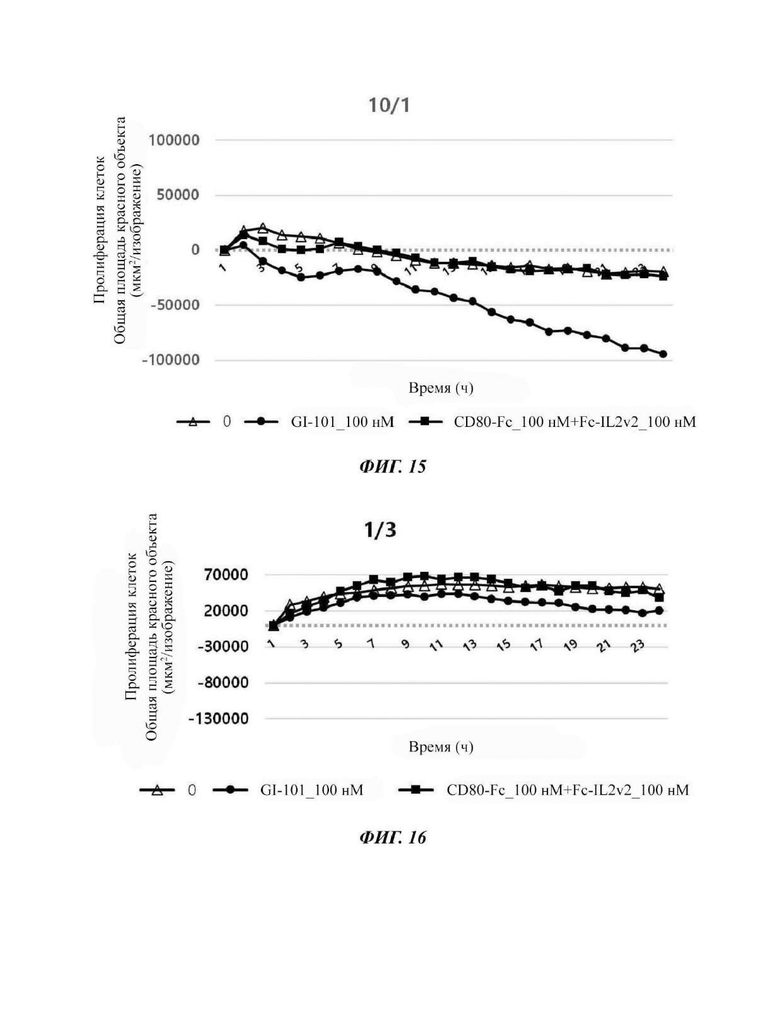
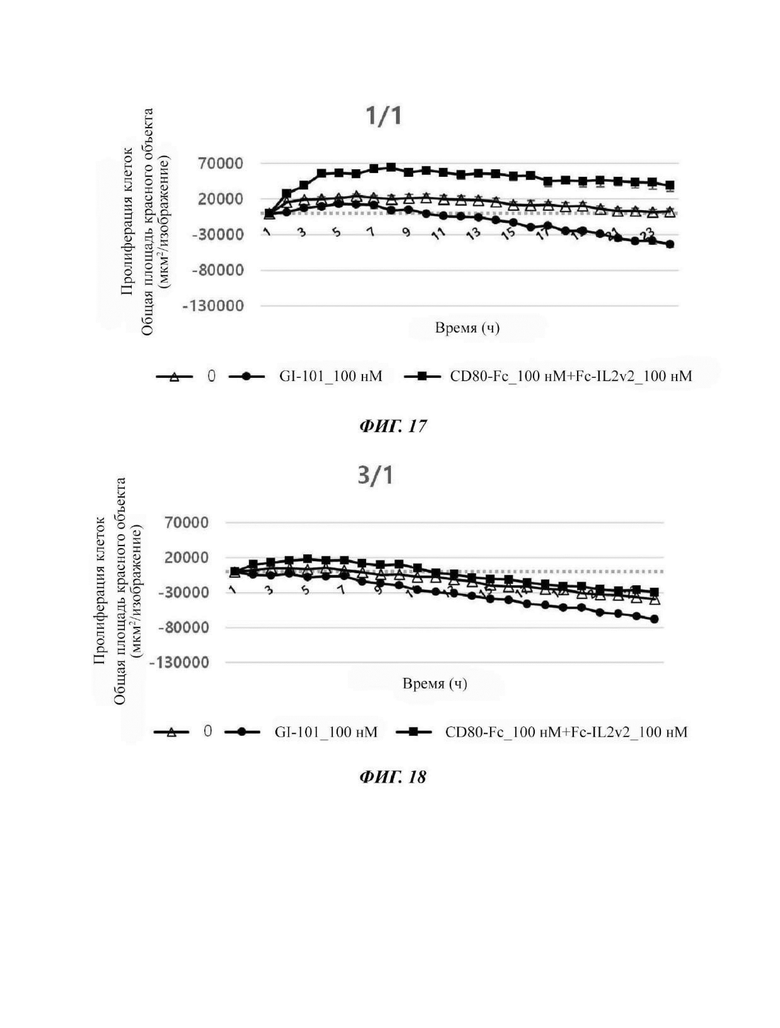
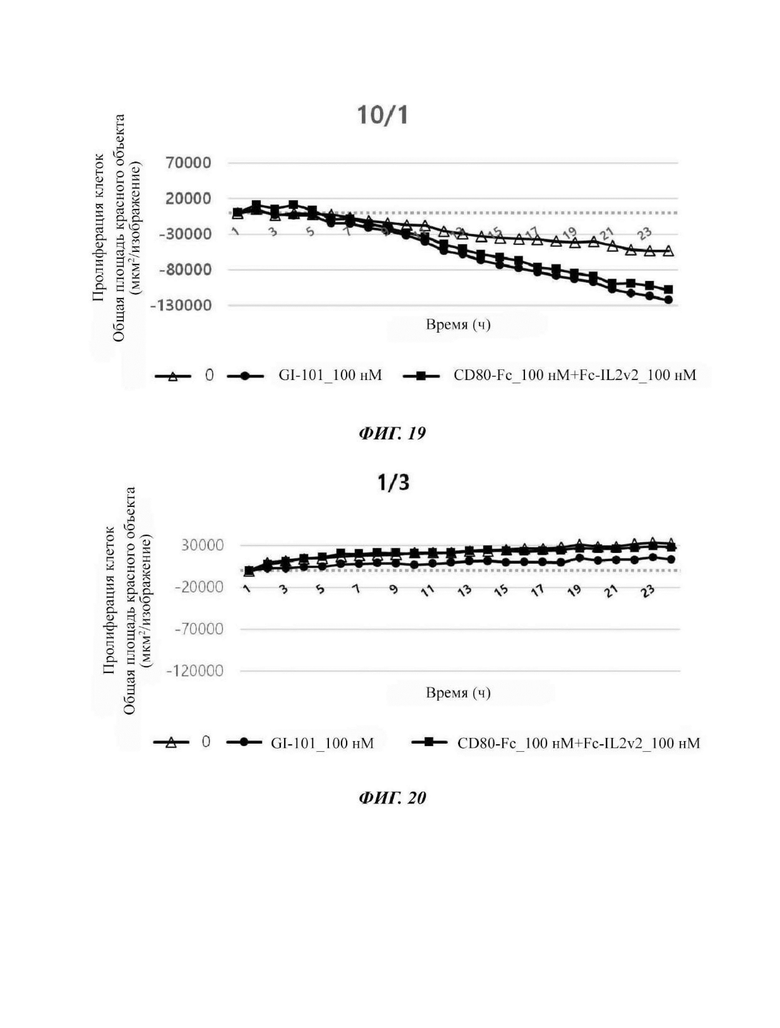
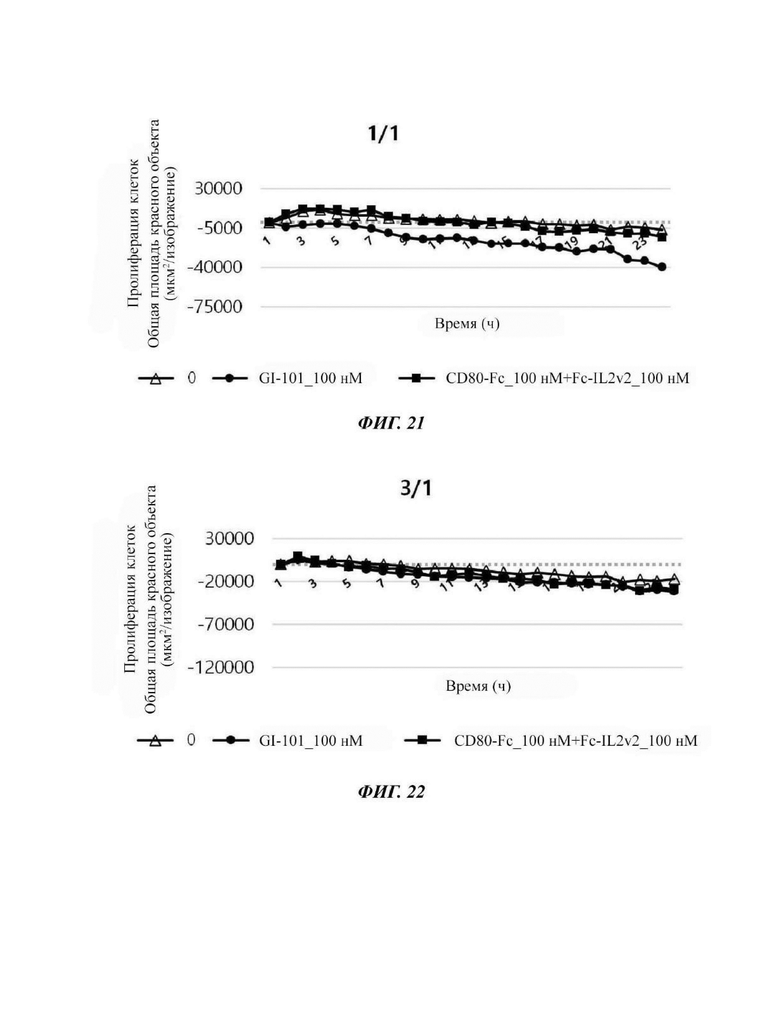
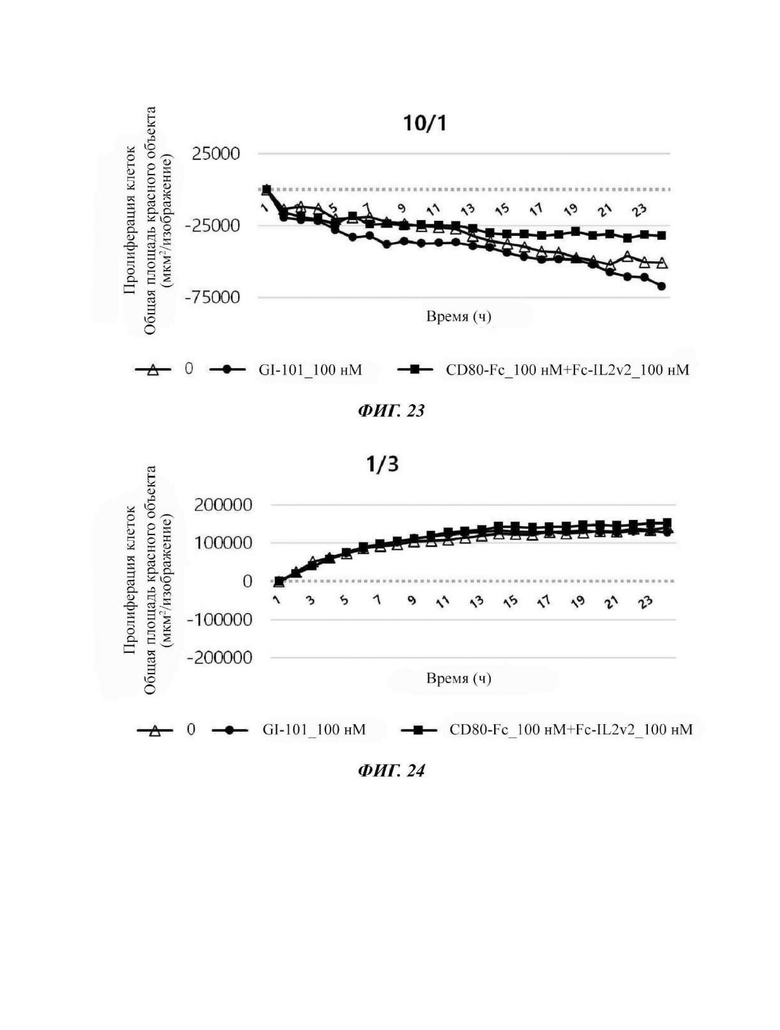
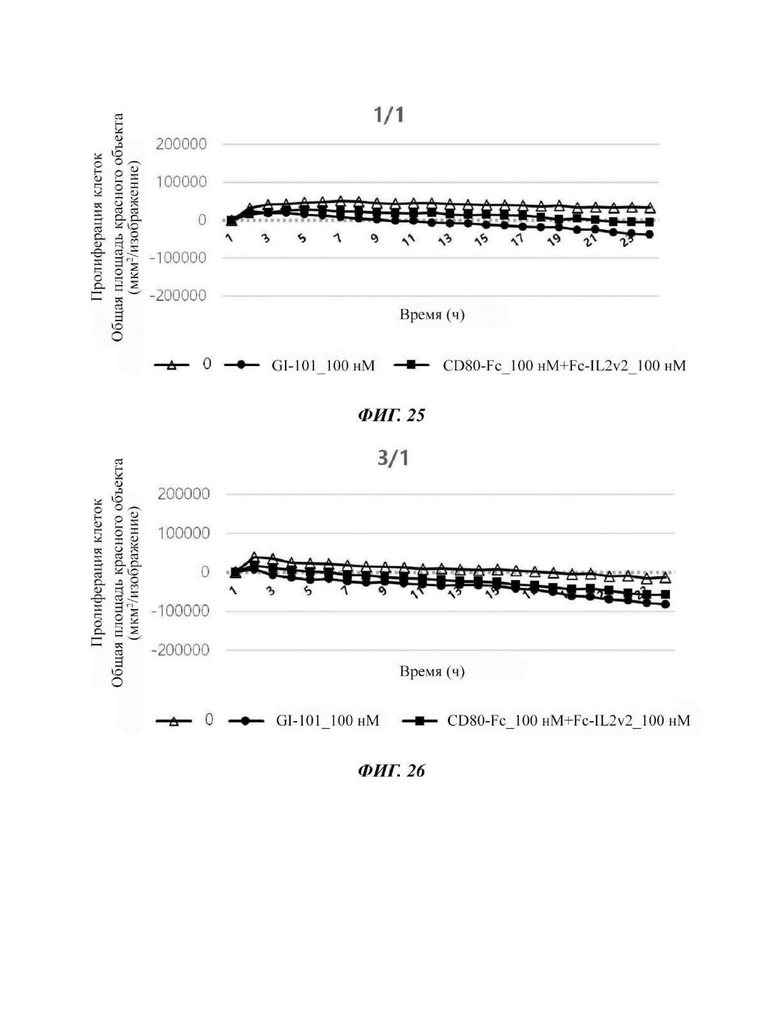
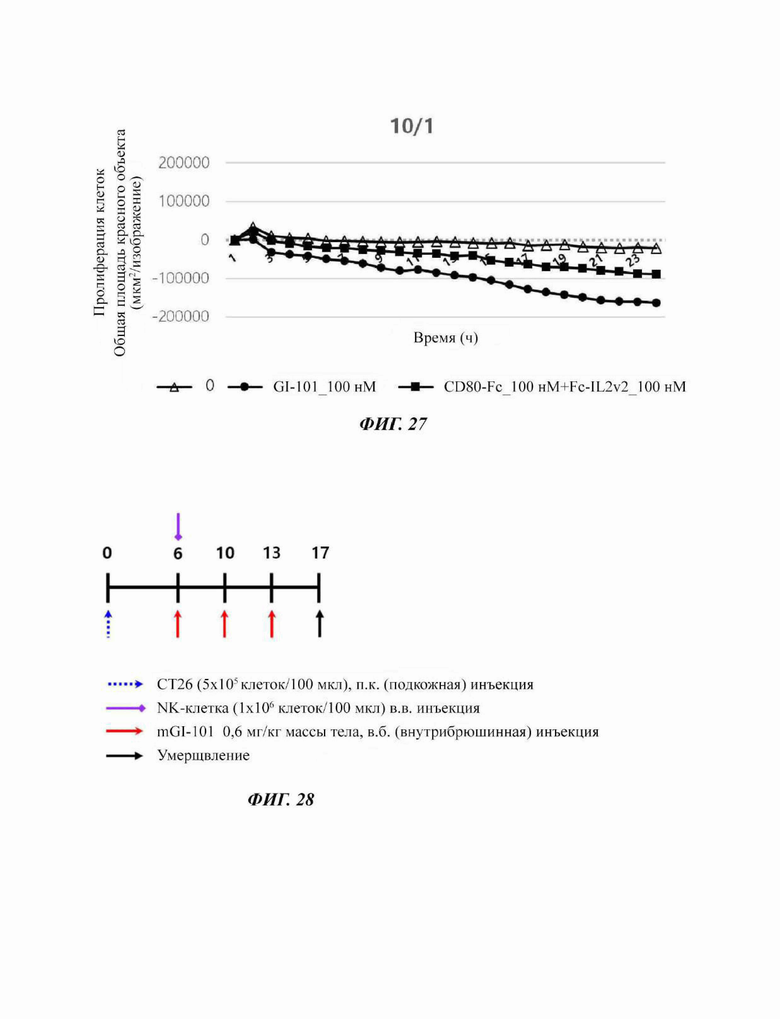
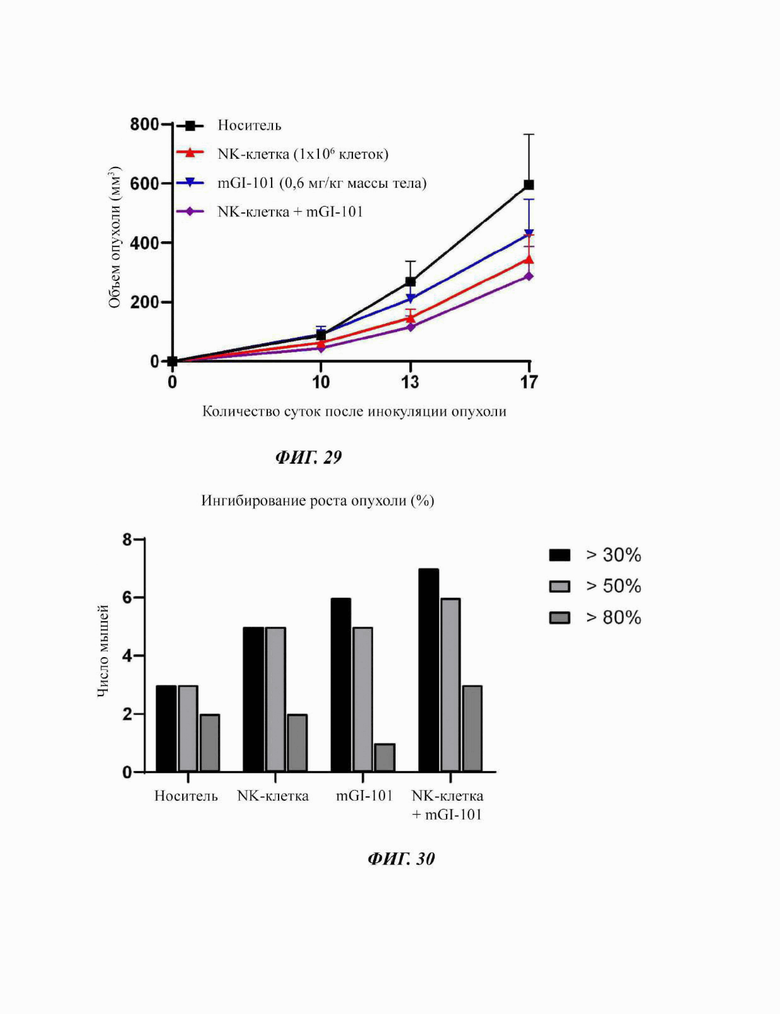
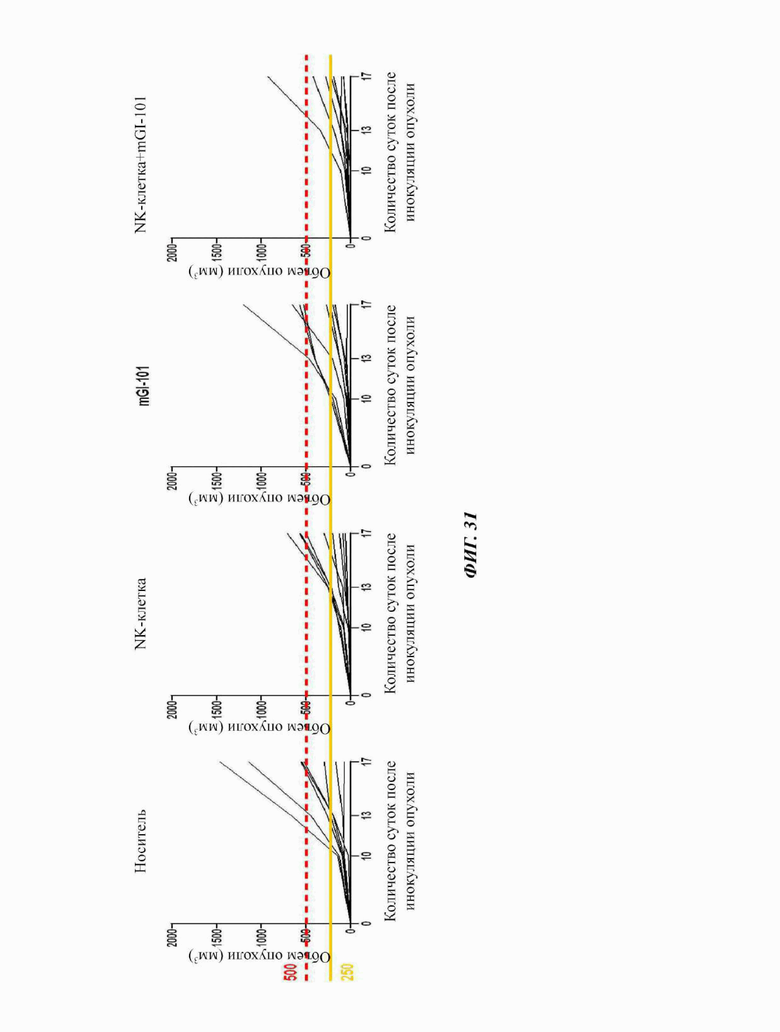
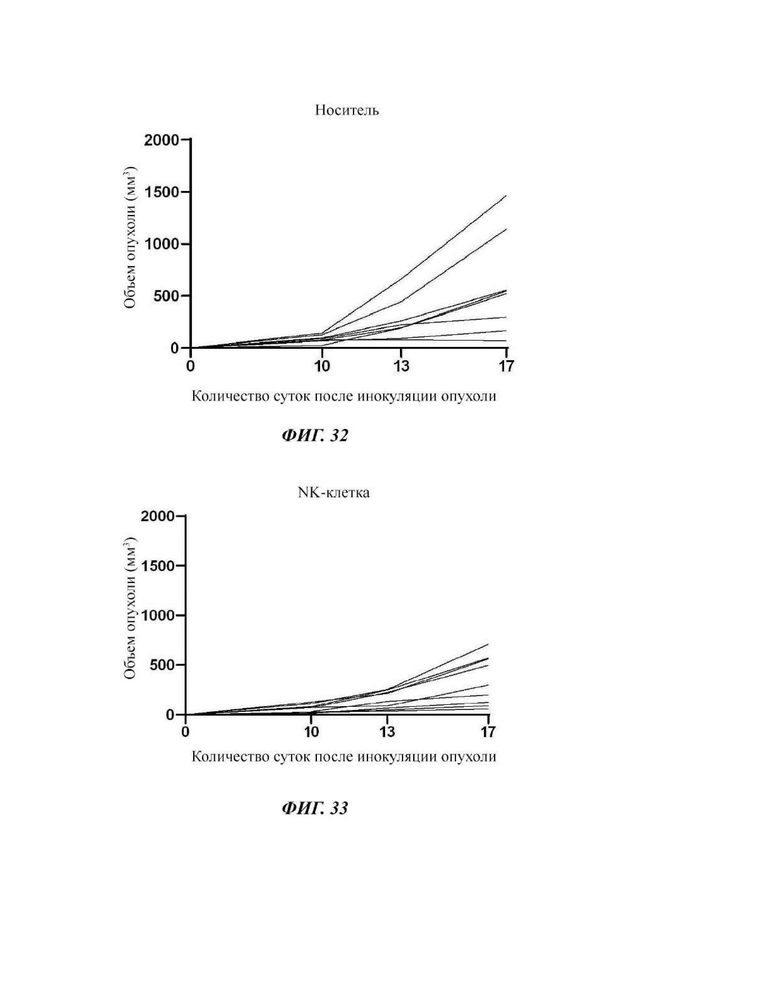
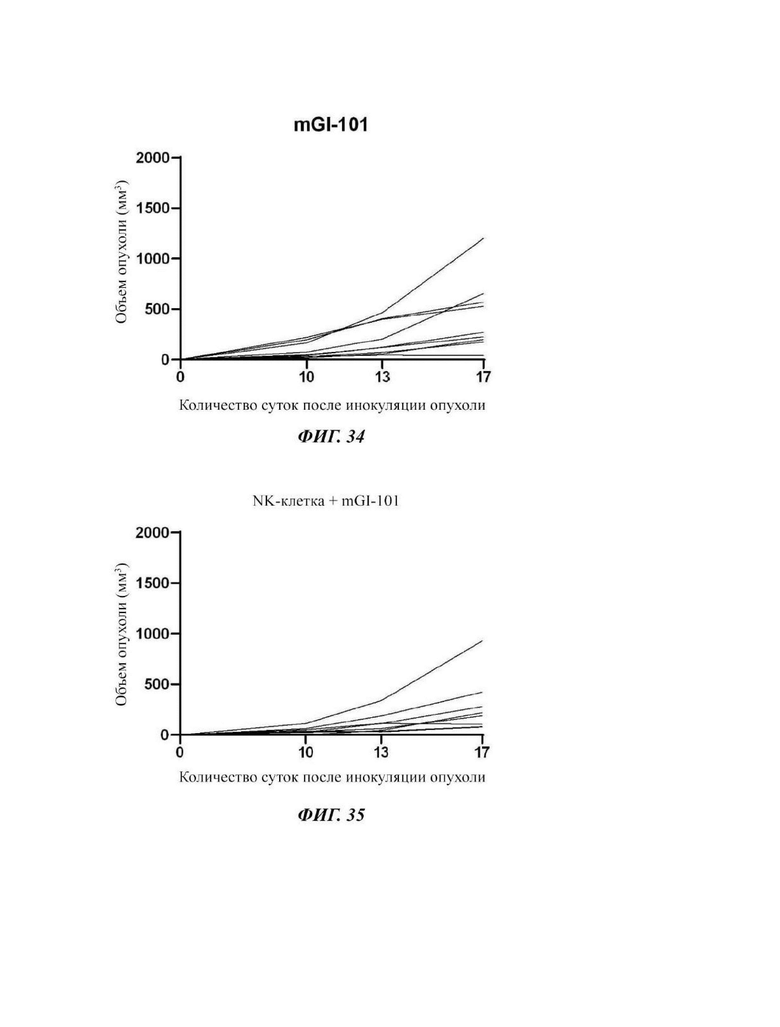
Изобретения относятся к области биотехнологии, конкретно к средству, содержащему в качестве активных ингредиентов NK-клетки и слитый белок, который содержит белок IL-2 и белок CD80, и может быть использовано в медицине для иммунотерапии рака. Предложены способ лечения рака, включающий введение димера слитого белка, содержащего аминокислотную последовательность SEQ ID NO: 9, 26, 28 или 30, в комбинации с естественными клетками-киллерами, и фармацевтическая композиция указанного димера слитого белка и естественных клеток-киллеров. Изобретения обеспечивают значительное ингибирование роста опухоли по сравнению с группами, обрабатываемыми естественными клетками-киллерами или слитым белком в отдельности. 2 н. и 1 з.п. ф-лы, 35 ил., 10 табл., 14 пр.
1. Способ лечения рака, включающий введение димера слитого белка в комбинации с естественными клетками-киллерами, где слитый белок содержит аминокислотную последовательность SEQ ID NO: 9, 26, 28 или 30.
2. Способ по п. 1, где рак представляет собой любой рак, выбранный из группы, состоящей из рака желудка, рака печени, рака легкого, колоректального рака, рака молочной железы, рака предстательной железы, рака яичника, рака поджелудочной железы, рака шейки матки, рака щитовидной железы, рака гортани, острого лимфобластного лейкоза, опухоли головного мозга, нейробластомы, ретинобластомы, рака головы и шеи, рака слюнной железы и лимфомы.
3. Фармацевтическая композиция для лечения рака, содержащая в качестве активного ингредиента димер слитого белка, естественные клетки-киллеры, где слитый белок содержит аминокислотную последовательность SEQ ID NO: 9, 26, 28 или 30.
| PARK J.C | |||
| Ветряный двигатель | 1925 |
|
SU3190A1 |
| Станок для придания концам круглых радиаторных трубок шестигранного сечения | 1924 |
|
SU2019A1 |
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
Авторы
Даты
2024-12-19—Публикация
2020-11-26—Подача