Настоящее изобретение относится к области терапии рака, более конкретно к иммунотерапевтическим способам. В частности настоящее изобретение обеспечивает различные пептиды, которые можно использовать в иммунотерапии рака, особенно для профилактики и лечения В-клеточного гемобластоза.
Среди всех В-клеточных гемобластозов, таких как В-клеточные лимфомы, неходжкинская лимфома (NHL) является седьмой ведущей причиной новых случаев рака и ответственна за примерно 3% смертей от рака в Соединенных Штатах. Среди всех NHL диффузная крупноклеточная В-клеточная лимфома (DLBCL) является наиболее распространенным подтипом лимфомы, составляющим 32,5% всех впервые диагностированных случаев, за ней следуют фолликулярная лимфома (FL) с 17,1% и мантийноклеточная лимфома (MCL), представляющая 3-5% случаев. Ежегодно в Соединенных Штатах диагностируют более 25 000 новых случаев DLBCL, что соответствует уровню заболеваемости 6,9 на 100 000 человек. Добавление моноклонального антитела к CD20, а именно ритуксимаба, к стандартной химиотерапии R-CHOP привело к значительному улучшению показателей полного ответа (CR), бессобытийной (EFS) и общей (OS) выживаемости при DLBCL. К сожалению, примерно в 30–40% случаев после R-CHOP отмечается рецидив или прогрессирование. Существуют определенные подгруппы пациентов, которые будут иметь плохие ответы и исходы при стандартной R-CHOP, такие как пациенты с MYC-реаранжированной DLBCL, В-клеточными лимфомами высокой степени злокачественности с MYC, BCL2 или BCL-реаранжировками, активированной B-клеточной (ABC) DLBCL, для которых могут быть полезными новые подходы (Chavez et al., CAR T-cell therapy for B-cell lymphomas: clinical trial results of available products; Ther Adv Hematol. 2019).
Среди новых подходов Т-клетки с химерным антигенным рецептором (CAR), такие как CAR Т-клетки, нацеленные на CD19, представляют собой новый стандарт лечения пациентов с DLBCL, рефрактерных как минимум к двум предшествующим линиям терапии. Два продукта CAR Т-клеток (аксикабтаген цилолейцел (акси-цел) (KTE-019) и тисагенлеклейцел (CTL019)) получили одобрение Управления по санитарному надзору за качеством пищевых продуктов и медикаментов США для лечения рефрактерной DLBCL после двух линий терапии. Хотя это представляет собой значительное дополнение к арсеналу средств лечения DLBCL, примерно в 50% случаев заболевание не поддается лечению. В результате будущие исследования должны быть сосредоточены на выявлении факторов, связанных с заболеванием, лечением или пациентом, которые могут помочь успешно предсказать результаты лечения.
Таким образом, вакцинация на основе опухолевых антигенов представляет собой уникальный подход к терапии рака, который вызвал значительный интерес, поскольку он позволяет задействовать собственную иммунную систему пациента для распознавания, атаки и уничтожения опухолей специфическим и устойчивым образом. Действительно, известно, что опухолевые клетки экспрессируют большое количество пептидных антигенов, восприимчивых к распознаванию иммунной системой. Таким образом, вакцины на основе таких антигенов предоставляют большие возможности не только для повышения общей выживаемости пациентов, но и для мониторинга иммунных реакций и получения продукта, соответствующего требованиям GMP, благодаря низкой токсичности и малой молекулярной массе опухолевых антигенов. Примеры опухолевых антигенов включают, помимо прочего, побочные продукты белков, транскрибируемых с обычно молчащих генов или сверхэкспрессированных генов, а также белков, экспрессируемых онковирусом (Kvistborg et al., Human Cancer Regression Antig. Curr Opin Immunol. 2013 Apr;25(2):284-90), и неоантигены, возникающие в результате точечных мутаций клеточных белков. Однако большинство опухоль-ассоциированных антигенов (TAA) и опухоль-специфических антигенов (TSA) являются (существующими) человеческими белками и, таким образом, считаются аутоантигенами. В процессе селекции тимусом Т-клетки, которые распознают комплексы пептид/ауто-МНС комплексы с достаточной аффинностью, клонально истощаются. Предлагая защиту от аутоиммунных заболеваний, этот механизм отбора репертуара Т-клеток также снижает возможность развития иммунитета против ТАА и TSA. Это подтверждается тем фактом, что онкореактивные TCR обычно имеют слабую аффинность. Кроме того, до настоящего времени в большинстве испытаний вакцин, проведенных с выбранными TAA и TSA с высокой аффинностью связывания с MHC, не было показано, что они вызывают сильный иммунитет, что, вероятно, отражает последствие тимусной селекции. Таким образом, мощный противоопухолевый ответ будет зависеть от презентации иммунореактивных пептидов и наличия достаточного количества реактивных клеток, «обученных» распознавать эти антигены. Таким образом, в данной области техники существует потребность в идентификации альтернативных антигенных пептидов, которые могут преодолеть ограничения, встречающиеся в этой области техники.
Изобретение направлено на удовлетворение вышеуказанных потребностей. Эта цель достигается с помощью объекта, изложенного ниже, в частности, в элементах, предусмотренных настоящим изобретением, и в прилагаемой формуле изобретения.
Изобретение описано более подробно ниже.
ОПРЕДЕЛЕНИЯ
Если дополнительно не оговаривается иное, научные и технические термины, используемые в настоящем описании, будут иметь значения, которые обычно хорошо известны специалистам в данной области техники. Кроме того, если иное не предусмотрено требованиями, используемые в описании условные обозначения, а также методики культивирования клеток и тканей хорошо известны специалистам в данной области техники.
Такие методики подробно описаны в литературе, например, у Owen et al. (Kuby Immunology, 7th, edition, 2013 – W. H. Freeman) и Sambrook et al. (Molecular cloning: A laboratory manual 4th edition, Cold Spring Harbor Laboratory Press - Cold Spring Harbor, NY, USA, 2012).
Тем не менее, при использовании различных терминов в настоящем описании более применимыми являются следующие определения.
Термины «пептид», «полипептид», «белок» и также различные варианты указанных терминов относятся к пептидам, олигопептидам, полипептидам или белкам, включающим по меньшей мере две аминокислоты, соединенные между собой предпочтительно обычной пептидной связью или, альтернативно, модифицированной пептидной связью, как, например, в случае изостерических пептидов. Термин «(поли)пептид» относится к пептиду и/или полипептиду. В частности, термины «пептид», «полипептид» и «белок» относятся к последовательной цепи аминокислот любой длины, причем аминокислоты соединены между собой пептидными связями (-NHCO-). Пептиды, полипептиды и белки могут играть структурную и/или функциональную роль в клетке in vitro и/или in vivo. Термины «пептид», «полипептид», «белок», предпочтительно объединяют аминокислотные цепи, длина которых составляет от 2 до по меньшей мере примерно 1000 аминокислотных остатков. Термин «пептид» предпочтительно объединяет аминокислотные цепи, длина которых составляет менее примерно 30 аминокислот, причем термины «полипептид» и «белок» предпочтительно объединяют аминокислотные цепи длиной по меньшей мере 30 аминокислот. Термины «полипептид» и «белок» используются в настоящем описании взаимозаменяемо. Согласно предпочтительному варианту осуществления изобретения, термины «пептид», «полипептид» и «белок» также включают «пептидомиметики», которые обозначаются как пептидные аналоги, содержащие непептидные структурные элементы, причем такие пептиды способны имитировать биологическую активность натурального исходного пептида или наоборот противодействовать ей. Пептидомиметики не обладают свойствами классических белков, такими как пептидные связи, расщепляемые ферментами. В частности, пептид, полипептид или белок может включать аминокислоты, которые отличаются от 20 аминокислот, зашифрованных генетическим кодом, либо он может полностью состоять из аминокислот, отличающихся от тех 20, что зашифрованы в генетическом коде. В частности, пептид, полипептид или белок в контексте настоящего изобретения могут в равной степени состоять из аминокислот, модифицированных естественными способами, такими как посттрансляционные процессы созревания или химические процессы, которые хорошо известны специалистам в данной области техники. Такие модификации подробно описываются в литературе. Такие модификации могут располагаться в любом месте полипептида: в пептидном каркасе, в аминокислотной цепи или даже на карбокси- или амино-концевых участках. В частности, пептид или полипептид может быть разветвленным после убиквитинирования, или быть циклическим, с разветвлением или без него. Такой тип модификации может быть результатом естественных или синтетических посттрансляционных процессов, которые хорошо известны специалистам в данной области техники. Термины «пептид», «полипептид», «белок» в контексте настоящего изобретения, в частности, также включают модифицированные пептиды, полипептиды и белки. Например модификации пептида, полипептида или белка могут включать ацетилирование, ацилирование, АДФ-рибозилирование, амидирование, ковалентное присоединение нуклеотида или нуклеотидного производного, ковалентное присоединение липида или липидного производного, ковалентное присоединение фосфатидилинозитола, ковалентную или нековалентную перекрестную сшивку, циклизацию, образование дисульфидных связей, деметилирование, гликозилирование, включая пегилирование, гидроксилирование, иодирование, метилирование, миристоилирование, окисление, протеолитические процессы, фосфорилирование, пренилирование, рацемизацию, селеноилирование, сульфатирование, добавление аминокислот, такое как аргинилирование или убиквитинирование. Такие модификации детально описаны в литературе (Proteins Structure and Molecular Properties (1993) 2nd Ed., T.E. Creighton, New York; Post-translational Covalent Modifications of Proteins (1983) B.C. Johnson, Ed., Academic Press, New York; Seifter et al. (1990) Analysis for protein modifications and nonprotein cofactors, Meth. Enzymol. 182: 626-646; и Rattan et al., (1992) Protein Synthesis: Post-translational Modifications and Aging, Ann NY Acad Sci, 663: 48-62). Соответственно, термины «пептид», «полипептид», «белок» предпочтительно также включают, например, липопептиды, липопротеины, гликопептиды, гликопротеины и подобные соединения.
Согласно предпочтительному варианту осуществления изобретения, (поли)пептид или белок представляет собой «классический» (поли)пептид или белок, причем «классический» (поли)пептид или белок обычно состоит из аминокислот, выбранных из 20 аминокислот, зашифрованных в генетическом коде, соединенных между собой обычной пептидной связью.
Как хорошо известно из области техники, пептиды, полипептиды и белки могут кодироваться нуклеиновыми кислотами. Термины «нуклеиновая кислота», «нуклеиновокислотная молекула», «нуклеиновокислотная последовательность», «полинуклеотид», «нуклеотидная последовательность» используются в настоящем описании взаимозаменяемо и обозначают точную последовательность натуральных нуклеотидов (например, A, T, G, C и U) или синтетических нуклеотидов, то есть цепь, состоящую по меньшей мере из двух нуклеотидов. В частности, термины «нуклеиновая кислота», «нуклеиновокислотная молекула», «нуклеиновокислотная последовательность», «полинуклеотид», «нуклеотидная последовательность» относятся к ДНК или РНК. Нуклеиновые кислоты, предпочтительно включают одноцепочечные, двухцепочечные или частично двухцепочечные ДНК или РНК, предпочтительно, выбранные из геномной ДНК (гДНК), комплементарной ДНК (кДНК), рибосомальной ДНК (рДНК), а также продукта транскрипции указанной ДНК, такого как РНК. Предпочтительными примерами нуклеиновых кислот являются рибосомальная РНК (рРНК), информационная РНК (мРНК); антисмысловая ДНК, антисмысловая РНК; комплементарные РНК и/или ДНК последовательности, рибозимы, (комплементарные) последовательности РНК/ДНК c элементами экспрессии и без них, вектор; мини-ген, фрагменты генов, регуляторные элементы, промоторы, а также их комбинации. Дополнительными предпочтительными примерами нуклеиновых кислот (молекул) и/или полинуклеотидов являются, например, рекомбинантный полинуклеотид, вектор, олигонуклеотид, молекула РНК, такая как рРНК, мРНК или транспортная РНК (тРНК), или молекула ДНК, как описано выше. Таким образом, предпочтительно, чтобы нуклеиновая кислота (молекула) представляла собой молекулу ДНК или молекулу РНК; предпочтительно, выбранную из группы гДНК; кДНК; рРНК; мРНК; антисмысловую ДНК; антисмысловую РНК; комплементарные РНК и/или ДНК последовательности; РНК и/или ДНК последовательности с элементами экспрессии или без них, регуляторные элементы, и/или промоторы; вектор; а также их комбинации. Специалисту в данной области техники не составляет труда определить нуклеотидную последовательность, которая может кодировать специфическую аминокислотную последовательность.
(Поли)пептиды и/или нуклеиновые кислоты в соответствии с настоящим изобретением могут быть получены при помощи любого способа, известного из области техники, включая без ограничений указанными, любой рекомбинантный метод, любой метод синтеза ex vivo и подобные методы, а также любые их комбинации. Такие методики подробно описаны в литературе, как уже упоминалось выше.
Термин «антигенный пептид», используемый в настоящем изобретении, относится к пептиду, который способен вызывать/индуцировать, усиливать, пролонгировать или поддерживать иммунный ответ у субъекта, у которого его применяют. Конкретнее, антигенный пептид представляет собой вариант последовательности (фрагмента/эпитопа) опухолевого антигена (человека). Другими словами, антигенный пептид предпочтительно отличается от (фрагмента/эпитопа) опухолевого антигена (человека), но предпочтительно имеет аминокислотное сходство с (фрагментом/эпитопом) опухолевого антигена (человека). Важно, что антигенный пептид имеет такую же коровую последовательность, что и коровая последовательность соответствующего (фрагмента/эпитопа) опухолевого антигена (человека). Предпочтительно, иммунный ответ, индуцированный/вызванный, усиленный, пролонгированный или поддерживаемый антигенным пептидом, (также) нацелен на соответствующий (фрагмент/эпитоп) опухолевого антигена (человека).
Используемый в настоящей заявке термин «опухолевой антиген» включает опухоль-специфические антигены (TSA) и опухоль-ассоциированные антигены (TAA). В общем, термин «опухолевой антиген» или «опухолевой белок» обозначает в настоящей заявке антигенное вещество, продуцируемое в опухолевых клетках, а иногда также и в нормальных клетках, и которое может вызывать иммунный ответ при введении субъекту. У людей они были классифицированы в соответствии с характером их экспрессии, функцией или генетическим происхождением и включают, помимо прочего, сверхэкспрессированные аутоантигены (такие как BIRC5); антигены рака яичек (CT) (такие как MAGE-1); мутационные антигены, также известные как неоантигены (такие как мутанты р53); тканеспецифические дифференцировочные антигены (такие как антигены меланомы Melan A/MART-1); вирусные антигены, которые экспрессируются онковирусами (такими как HPV, EBV); онкофетальные антигены (такие как альфа-фетопротеин AFP и карциноэмбриональный антиген СЕА); и универсальные антигены (теломераза).
Термин «В-клеточный опухолевый антиген», используемый в настоящей заявке, относится к антигену, ассоциированному и/или вовлеченному в этиологию В-клеточного гемобластоза, который, например, участвует или экспрессируется при В-клеточном гемобластозе. Другими словами, антиген экспрессируется В-клетками или на них, включая В-клетки человека, такой как любой из ряда известных маркеров В-клеток. Предпочтительно В-клеточный опухолевый антиген экспрессируется на высоком уровне (сверхэкспрессируется) в В-клеточных лимфомах, таких как CD19, CD20, CD22, CD37 или TNFRSF13C. Антигенный пептид, «полученный из» В-клеточного опухолевого антигена, обычно имеет ту же коровую последовательность, что и эпитоп («эталонный эпитоп») указанного В-клеточного опухолевого антигена.
Термин «коровая последовательность», используемый в настоящей заявке, относится к аминокислотам в середине последовательности (также называемым «центральными аминокислотами» последовательности), например, в середине антигенного пептида и/или (эталонного) эпитопа. Соответственно, коровая последовательность состоит из всех аминокислот, кроме двух наиболее крайних N-концевых и двух наиболее крайних C-концевых аминокислот. Например, в пептиде из девяти аминокислот (например, антигенном пептиде по настоящему изобретению или соответствующем (фрагменте/эпитопе) опухолевого антигена (человека), пять средних аминокислот представляют собой коровую последовательность, и изменения могут происходить только в любом из двух N-концевых и двух С-концевых аминокислотных положений. Соответственно, «общая коровая последовательность» (или «сохраненная» коровая последовательность) обычно означает, что мутации/различия разрешены только в двух наиболее крайних N-концевых и в двух наиболее крайних C-концевых аминокислотах (эталонного) эпитопа/ последовательности.
Термин «распространенность», используемый в настоящей заявке, относится к совокупной частоте каждого белка микробиоты человека, в котором коровая последовательность, общая с соответствующим (фрагментом/эпитопом) опухолевого антигена (человека), обнаружена и присутствует в антигенном пептиде. Действительно, интересующая коровая последовательность может присутствовать в одном или нескольких различных антигенных пептидах, каждый из таких антигенных пептидов может присутствовать в одном или разных белках, экспрессируемых в микробиоте человека. Соответственно, глобальная распространенность коровой последовательности определяется по частоте каждого белка микробиоты человека, в которой обнаружена коровая последовательность, общая с соответствующим (фрагментом/эпитопом) опухолевого антигена (человека), и с учетом частоты каждого белка микробиоты человека, где были обнаружены сходные пептиды (т.е. разные антигенные пептиды), имеющие одинаковую коровую последовательность.
Термин «микробиота», используемый в настоящем описании, относится к симбиотическим микроорганизмам, которые можно обнаружить во всех многоклеточных организмах, изученных на сегодняшний день, как растительных, так и животных. В частности, было установлено, что микробиота необходима для иммуногенного, гормонального и метаболического гомеостаза организма хозяина. Микробиота включает бактерии, археи, простейшие, грибы и вирусы. Соответственно, термин «вариант последовательности микробиоты» представляет собой вариант эталонной последовательности (человека) (в частности, эпитоп/фрагмент опухолевого антигена человека), который существует в микробиоте, такой как бактерии (например, он может содержаться в белке микробиоты, таком как бактериальный белок). Предпочтительно, антигенный пептид по изобретению представляет собой вариант последовательности микробиоты (эталонный эпитоп/фрагмент В-клеточного опухолевого антигена человека). Соответственно, антигенный пептид предпочтительно присутствует (например, входит в состав) по меньшей мере в одном белке, экспрессируемом микробиотой человека.
«Вариант последовательности», в частности по всей длине последовательности обычно по меньшей мере на 50% идентичен эталонной последовательности, конкретно фрагменту/эпитопу (эталонного) опухолевого антигена. Предпочтительно, вариант последовательности по меньшей мере на 60%, предпочтительно по меньшей мере на 70%, предпочтительно по меньшей мере на 75%, более предпочтительно по меньшей мере на 80%, еще более предпочтительно по меньшей мере на 85%, еще более предпочтительно по меньшей мере на 90%, особо предпочтительно по меньшей мере на 95% и наиболее предпочтительно по меньшей мере на 99% идентичен эталонной последовательности, конкретно, фрагменту/эпитопу (эталонного) опухолевого антигена. Идентичность последовательностей может быть рассчитана с помощью методов, хорошо известных из области техники, в частности, как описано ниже. Предпочтительно, вариант последовательности сохраняет специфическую функцию эталонной последовательности, например, функцию опухолевого эпитопа и/или способность вызывать или поддерживать иммунный ответ. Вариант последовательности микробиоты предпочтительно выбран из группы, включающей варианты последовательностей бактерий, варианты последовательностей археев, варианты последовательностей простейших, варианты последовательностей грибов и варианты последовательностей вирусов. Более предпочтительно вариант последовательности микробиоты представляет собой вариант последовательности бактерий.
Анатомически микробиота располагается на поверхности или внутри целого ряда тканей и биологических жидкостей, включая кожу, конъюнктиву, молочные железы, влагалище, плаценту, семенную жидкость, матку, фолликулы яичников, легкие, слюну, ротовую полость (в частности, слизистую оболочку ротовой полости) и желудочно-кишечный тракт, в частности, кишечник. В контексте настоящего изобретения вариант последовательности микробиоты предпочтительно представляет собой вариант последовательности микробиоты желудочно-кишечного тракта (микроорганизмов, населяющих желудочно-кишечный тракт), более предпочтительно, вариант последовательности микробиоты кишечника (микроорганизмов, населяющих кишечник). Соответственно, более предпочтительно вариант последовательности микробиоты представляет собой вариант последовательности кишечных бактерий (человека) (то есть вариант последовательности бактерий, населяющих кишечник (человека)).
Хотя микробиоту можно обнаружить во многих многоклеточных организмах (во всех многоклеточных организмах, изученных на сегодняшний день от растений до животных), в контексте настоящего изобретения предпочтительной считается микробиота, обнаруженная в организме человека и на его поверхности. Такая микробиота упоминается в настоящей заявке как «микробиота человека» (причем термин «человек» относится конкретно к локализации/месту расположения микробиоты). В контексте настоящего изобретения вариант последовательности микробиоты представляет собой вариант последовательности микробиоты человека.
Термин «иммуногенное соединение» относится к соединению, содержащему антигенный пептид, заявленный в соответствии с настоящим изобретением. «Иммуногенное соединение» способно индуцировать/вызывать, усиливать, пролонгировать или поддерживать иммунный ответ против указанного антигенного пептида у субъекта, у которого его применяют. Согласно некоторым вариантам осуществления изобретения, иммуногенные соединения содержат по меньшей мере один антигенный пептид, или альтернативно по меньшей мере одно соединение, содержащее такой антигенный пептид, соединенный с белком, таким как белок-носитель.
«Белок-носитель» обычно представляет собой белок, который способен переносить грузы, такие как антигенный пептид, заявленный в соответствии с настоящим изобретением. Например, белок-носитель может переносить свой груз через мембрану. В контексте настоящего изобретения белок-носитель, в частности (также) относится к белку или полипептиду, который обладает способностью вызывать иммунный ответ против антигенного пептида, с которым он связан. Белки-носители хорошо известны из данной области техники.
Альтернативно, такой пептид- или полипептид-носитель может быть введен совместно в форме иммунного адъюванта.
Предпочтительно, антигенный пептид, описанный в настоящей заявке, может быть введен одновременно или быть связанным, например, посредством ковалентной или нековалентной связи с белком/пептидом, обладающим иммуноадъювантными свойствами, такими как обеспечение стимуляции CD4+ Тh1-клеток. Принимая во внимание, что описанный в настоящей заявке антигенный пептид предпочтительно связывается с MHC I класса, CD4+ хелперные эпитопы могут дополнительно применяться для стимуляции эффективного иммунного ответа. Th1 хелперные клетки способны поддерживать эффективную активацию дендритных клеток (DC) и специфическую активацию CTL за счет секреции интерферона гамма (IFN-γ), фактора некроза опухолей альфа (TNF-α) и интерлейкина-2 (IL-2) и усиления экспрессии костимулирующего сигнала на дендритных клетках и T-клетках (Galaine et al., Interest of Tumor-Specific CD4 T Helper 1 Cells for Therapeutic Anticancer Vaccine. Vaccines (Basel). 2015 Jun 30; 3(3):490-502).
Например, адъювантный пептид/белок предпочтительно может отличаться от антигенного пептида, заявленного в соответствии с настоящим изобретением. Предпочтительно, адъювантный пептид/белок способен вызывать иммунную память или оказывать неспецифическую помощь, или представлять собой специфический хелперный белок. В литературе описано несколько хелперных пептидов для оказания помощи неспецифическим Т-клеткам, такие как хелперный белок столбняка, пептид гемоцианина лимфы улитки или PADRE пептид (Adotévi et al., Targeting antitumor CD4 helper T cells with universal tumor-reactive helper peptides derived from telomerase for cancer vaccine. Hum Vaccin Immunother. 2013 May; 9(5):1073-7, Slingluff CL, The present and future of peptide vaccines for cancer: single or multiple, long or short, alone or in combination? Cancer J. 2011 Sep-Oct; 17(5):343-50). Соответственно, хелперный белок столбняка, пептид гемоцианина лимфы улитки и PADRE пептид являются предпочтительными примерами таких адъювантных белков/пептидов. В частности, антигенный пептид, как описано в настоящей заявке, или полипептид, содержащий указанный антигенный пептид, могут быть связаны, например, ковалентной или нековалентной связью с HHD-DR3 пептидом с последовательностью MAKTIAYDEEARRGLERGLN (SEQ ID № 473). Этот пептид является другим примером хелперного пептида (обладающего иммуноадъювантными свойствами), который является предпочтительным в контексте настоящего изобретения. Другим предпочтительным примером является h-pAg T13L (последовательность: TPPAYRPPNAPIL; SEQ ID № 474; Bhasin M, Singh H, Raghava GP (2003) MHCBN: a comprehensive database of MHC binding and non-binding peptides. Bioinformatics 19: 665–666). Другими примерами предпочтительных хелперных белков являются UCP2 пептид (например, описанный в WO 2013/135553 A1 или у Dosset M, Godet Y, Vauchy C, Beziaud L, Lone YC, Sedlik C, Liard C, Levionnois E, Clerc B, Sandoval F, Daguindau E, Wain-Hobson S, Tartour E, Langlade-Demoyen P, Borg C, Adotévi O: Universal cancer peptide-based therapeutic vaccine breaks tolerance against telomerase and eradicates established tumor. Clin Cancer Res. 2012 Nov 15; 18(22):6284-95. doi: 10.1158/1078-0432.CCR-12-0896. Epub 2012 Oct 2) и пептид BIRC5 (например, как описано в EP2119726 A1 или в Widenmeyer M, Griesemann H, Stevanović S, Feyerabend S, Klein R, Attig S, Hennenlotter J, Wernet D, Kuprash DV, Sazykin AY, Pascolo S, Stenzl A, Gouttefangeas C, Rammensee HG: Promiscuous survivin peptide induces robust CD4+ T-cell responses in the majority of vaccinated cancer patients. Int J Cancer. 2012 Jul 1;131(1):140-9. doi: 10.1002/ijc.26365. Epub 2011 Sep 14). Наиболее предпочтительным хелперным пептидом является пептид UCP2 (аминокислотная последовательность: KSVWSKLQSIGIRQH; SEQ ID № 475, например, как описано в WO 2013/135553 A1 или у Dosset et al., Clin Cancer Res. 2012 Nov 15;18(22):6284-95). В частности, антигенный пептид, описанный в настоящей заявке, или полипептид, содержащий указанный антигенный пептид, может быть связан, например, ковалентной или нековалентной связью, с хелперным пептидом.
Используемый в настоящем изобретении термин «иммуногенная композиция» относится к композиции, которая обладает способностью вызывать, индуцировать, усиливать, пролонгировать или поддерживать иммунный ответ, в частности к композиции, которая вызывает, индуцирует, усиливает, пролонгирует или поддерживает иммунный ответ при введении млекопитающему, особенно при введении человеку. Предпочтительно, иммуногенная композиция дополнительно содержит одно или несколько иммуноадъювантных веществ.
Под термином «фармацевтически приемлемый наполнитель или носитель» в настоящем описании понимается соединение фармацевтического качества, которое улучшает доставку, стабильность или биодоступность активного ингредиента, и может метаболизироваться в организме субъекта, которому его вводят, не оказывая при этом токсического действия. Предпочтительными наполнителями или носителями в соответствии с настоящим изобретением являются любые наполнители или носители, обычно используемые в фармацевтических продуктах, такие как, например, вода, солевой раствор, фосфатно-буферный солевой раствор, декстроза, глицерин, этанол и подобные вещества, а также их комбинации. Во многих случаях будет предпочтительно использовать в композиции изотонические агенты, например, сахара, полиспирты, такие как маннитол, сорбитол или хлорид натрия. Фармацевтически приемлемые наполнители или носители могут дополнительно содержать минимальные количества вспомогательных соединений, таких как увлажняющие или эмульгирующие агенты, или консерванты.
Термин «вакцина» относится к композиции, способной стимулировать иммунную систему живого организма таким образом, чтобы обеспечить защиту против вредоносного антигена, либо посредством профилактики, либо посредством терапии. Профилактические вакцины являются предпочтительными. Предпочтительно, вакцина или вакцинная композиция дополнительно содержит одно или несколько иммунноадъювантных соединений.
Согласно различным аспектам и вариантам осуществления изобретения, описанным в настоящей заявке, термин «субъект» или «хозяин» предпочтительно относится к млекопитающему, и наиболее предпочтительно к человеку. Указанный субъект может уже иметь В-клеточный гемобластоз, обследоваться с подозрением на него или быть подверженным риску развития такого заболевания.
Термин «В-клеточный гемобластоз» относится к заболеваниям, связанным с трансформацией В-клеток. Он охватывает, среди прочего, В-клеточные лимфомы, острый лимфоцитарный (или лимфобластный) лейкоз (ALL), хронический лимфолейкоз (CLL, болезнь Рихтера). В контексте настоящего изобретения предпочтительными являются В-клеточные лимфомы, такие как неходжкинская лимфома (NHL). Например, NHL выбрана из группы, состоящей из индолентной (медленно растущей) NHL, агрессивной NHL, диффузной В-клеточной крупноклеточной лимфомы (DLBCL), NOS (de novo и трансформированной из индолентной), первичной медиастинальной В-крупноклеточной лимфомы (PMBCL), Т-клеточной/богатой гистоцитами В-клеточной крупноклеточной лимфомы (TCHRBCL), лимфомы Беркитта, мантийноклеточной лимфомы (MCL) и/или фолликулярной лимфомы (FL), при необходимости фолликулярной лимфомы степени 3B (FL3B).
Используемый в настоящем изобретении термин «профилактика», «предотвращение» или «предотвращать», «предупреждать» в целом означает действия, позволяющие избежать или свести к минимуму вероятность возникновения или развития заболевания или состояния до их возникновения, в то время как термин «лечение» или «лечить» означает уменьшение, облегчение или излечение заболевания или состояния (или симптомов заболевания или состояния) после его начала. Термин «профилактика» относится к «уменьшению вероятности возникновения» или к «уменьшению вероятности рецидива» заболевания.
Термин «эффективное количество» или «эффективная доза», используемый в настоящем описании, обозначает количество, которое обеспечивает достижение необходимого эффекта. Для терапевтических целей эффективное количество представляет собой количество, достаточное для достижения благоприятного или необходимого клинического результата. Предпочтительное эффективное количество для данного применения может быть легко определено специалистом в данной области техники с учетом, например, роста, массы тела и возраста субъекта, типа заболевания/расстройства, которое подлежит профилактике или лечению, а также времени, которое прошло с начала заболевания/расстройства. В контексте настоящего изобретения, что касается профилактики и лечения, эффективное количество композиции представляет собой количество, достаточное для того чтобы индуцировать гуморальный/или клеточно-опосредованный иммунный ответ, направленный против заболевания/расстройства.
Во всем настоящем описании и в представленной ниже формуле изобретения, если контекст не подразумевает другого, термин «включает» и его производные «включающий» и «включая» следует понимать, как определяющие включение обозначенных компонентов, целых чисел и этапов, но не исключение других необозначенных компонентов, целых чисел и этапов. Термин «состоять» относится к частному случаю термина «содержать», когда другие необозначенные компоненты, целые числа и этапы исключены. В контексте настоящего изобретения термин «содержит» включает термин «состоит». Термин «содержащий», таким образом, относится и к термину «включающий» так же как и к термину «состоящий», например, композиция, «содержащая» Х, может состоять исключительно из Х, а может включать какой-либо дополнительный элемент, например, X+Y.
Формы единственного числа и сходные обозначения, используемые в настоящем описании (особенно в контексте формулы изобретения), относятся как к единственному, так и к множественному числу, если специальным образом не указано иное, или иное явным образом не следует из контекста изобретения. Описание диапазона значений в настоящей заявке призвано выступать в качестве упрощенного метода обращения к каждому индивидуальному значению в пределах данного диапазона. За исключением случаев, когда иное указано в настоящем описании, каждое индивидуальное значение включено в описание таким образом, как если бы оно было указано в настоящем описании в индивидуальном порядке. Ни какие формулировки в данном описании не должны рассматриваться как указывающие на какой-либо незаявленный элемент, имеющий важное значение для практического применения данного изобретения.
Выражение «по существу» не исключает термина «полностью», то есть композиция, которая «по существу полностью» свободна от соединения Y, может быть полностью очищена от соединения Y. Там, где это необходимо, выражение «по существу» может быть исключено из описания изобретения.
Термин «примерно», используемый по отношению к численной величине x, означает x±10%.
Далее в описании представлены дополнительные определения.
Настоящее изобретение может быть более понятным благодаря ссылкам на следующее подробное описание, включающее предпочтительные варианты осуществления изобретения и включенные в него примеры.
Подробное описание изобретения
Несмотря на то, что настоящее изобретение подробно описано ниже, должно быть понятно, что настоящее изобретение не ограничивается конкретными методологиями, протоколами и реагентами, описанными в настоящей заявке, поскольку они могут меняться. Кроме того, должно быть понятно, что вся используемая терминология не предназначена для ограничения объема настоящего изобретения, который ограничивается лишь прилагаемой формулой изобретения. Если дополнительно не оговаривается иное, технические и научные термины, используемые в настоящем описании, имеют общепринятые значения, понятные любому специалисту в данной области техники.
Ниже представлено описание элементов настоящего изобретения. Указанные элементы перечислены в рамках конкретных вариантов осуществления изобретения, однако должно быть понятно, что они могут комбинироваться между собой любым образом и в любых количествах с получением дополнительных вариантов осуществления изобретения. Разнообразные описанные ниже примеры и предпочтительные варианты осуществления изобретения не должны толковаться как ограничивающие сущность настоящего изобретения только непосредственно описанными вариантами осуществления изобретения. Следует понимать, что описание предоставляет и охватывает варианты осуществления изобретения, которые объединяют конкретно изложенные варианты с любым количеством раскрытых и/или предпочтительных элементов. Кроме того, любые перестановки и комбинации всех описанных в настоящей заявке элементов изобретения должны рассматриваться как входящие в объем настоящего изобретения и раскрытые в настоящей заявке, если дополнительно не оговаривается иное.
Антигенные пептиды в соответствии с настоящим изобретением
В первом аспекте настоящее изобретение обеспечивает антигенный пептид, полученный из опухолевого антигена, особенно опухолевого антигена В-клеток, где антигенный пептид имеет ту же коровую последовательность, что и эталонный эпитоп опухолевого антигена, и где общая коровая последовательность имеет высокую распространенность в микробиоте человека.
Настоящее изобретение также относится к антигенному пептиду, содержащему или состоящему из аминокислотной последовательности, представленной в любой из SEQ ID №№ 316, 304-315, 317-472 и 501-509. Предпочтительно, антигенный пептид может содержать или состоять из аминокислотной последовательности, указанной в любой из SEQ ID №№ 316, 304–315 и 317–326.
Кроме того, настоящее изобретение относится к антигенному пептиду, содержащему или состоящему из аминокислотной последовательности, представленной в любой из SEQ ID №№ 1-257 и 476-500, где при необходимости один или два аминокислотных остатка могут быть заменены, удалены или добавлены. Предпочтительно, коровая последовательность (SEQ ID №№ 1-257 и 476-500) сохраняется, если один или два аминокислотных остатка заменены, удалены или добавлены. Более предпочтительно антигенный пептид содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 1-257 и 476-500 (без мутаций). Аминокислотные последовательности в соответствии с SEQ ID №№ 1-257 и 476-500 представляют собой варианты последовательности микробиоты (варианты микробиоты, в частности варианты бактериальной последовательности) эпитопов опухолей человека, в частности эталонных эпитопов/фрагментов В-клеточных опухолевых антигенов человека. Другими словами, аминокислотные последовательности в соответствии с SEQ ID №№ 1–257 и 476–500 могут быть обнаружены в бактериальных белках и демонстрируют сходство последовательностей с эталонными эпитопами/фрагментами В-клеточных опухолевых антигенов человека, как подробно показано в Таблице 1А.
Авторы настоящего изобретения идентифицировали набор антигенных пептидов, которые можно использовать для индукции специфического иммунного ответа против опухолевых клеток. Эти антигенные пептиды отличаются от (фрагментов) опухолевых антигенов человека, но имеют аминокислотное сходство с ними, особенно опухолевыми антигенами с высокой экспрессией в B-клеточных лимфомах, такими как CD19, CD20, CD22, CD37 или TNFRSF13C, как показано в Таблице 1A, Таблице 1B и Таблице 1C. Важно отметить, что антигенные пептиды по настоящему изобретению имеют коровую последовательность, идентичную коровой последовательности эпитопа (фрагмента) эталонного опухолевого антигена. Более того, коровая последовательность демонстрирует высокую распространенность в зависимости от частоты белков, присутствующих в микробиоте человека, где обнаружена коровая последовательность.
В частности, антигенные пептиды, заявленные в соответствии с настоящим изобретением, содержатся в полипептидах и белках, продуцируемых комменсальными бактериями из кишечника человека. Соответственно, антигенные пептиды, заявленные в соответствии с настоящим изобретением, не являются последовательностями человека, а представляют собой бактериальные последовательности. Не желая углубляться в какую-либо теорию, авторы изобретения считают, что иммунный репертуар человека включает клоны Т-клеток, которые обладают реактивностью по отношению к бактериальным пептидам (содержащимся в белках, продуцируемых комменсальными бактериями кишечника), которые имеют аминокислотную идентичность с фрагментами опухолевых антигенов человека. В частности, антигенные пептиды, заявленные в соответствии с настоящим изобретением, могут вызывать более сильный иммунный ответ, чем соответствующие пептиды человека, поскольку Т-клетки, обладающее способностью распознавать только человеческие пептиды, были истощены при распознавании аутоантигенов в процессе созревания, и это не относится к антигенным пептидам, заявленным в соответствии с настоящим изобретением. Это может объяснить, почему описанные в настоящей заявке антигенные пептиды способны индуцировать иммунный ответ, особенно Т-клеточный ответ, при введении в организм индивидуума (человека).
Соответственно, не углубляясь в какую-либо теорию, авторы настоящего изобретения считают, что белки, продуцируемые комменсальными бактериями кишечника, обладают способностью «имитировать» опухолевые антигены и могут применяться для стимуляции специфического иммунного ответа против опухолевых клеток. Эти данные являются дополнительным доказательством того, что комменсальные бактерии могут способствовать уничтожению опухолевых клеток.
Антигенные пептиды, описанные в настоящей заявке, могут быть получены при помощи широко известных методик. Например, пептиды могут быть получены синтетическим путем, при помощи технологий рекомбинантных ДНК или с помощью химического синтеза. Пептиды, раскрытые в настоящем изобретении, могут быть синтезированы индивидуально или в форме более длинных полипептидов, содержащих два и более пептида (например, два или более пептида, или пептид и не-пептид). Антигенные пептиды могут быть изолированы, то есть практически полностью очищены от других натуральных белков и фрагментов клеток-хозяев, например, очищены по меньшей мере на 70%, 80% или 90%. Предпочтительно, антигенные пептиды, заявленные в соответствии с настоящим изобретением, представляют собой изолированные антигенные пептиды.
Таким образом, коровая последовательность представляет собой главную особенность антигенных пептидов по настоящему изобретению. Таким образом, авторы изобретения идентифицировали коровые последовательности, представляющие большой интерес, с высокой распространенностью, поскольку они присутствуют в нескольких вариантах последовательностей фрагмента (эталонного) опухолевого антигена и/или в нескольких белках микробиоты человека с высокой распространенностью в значительной части общей человеческой популяции. Авторы изобретения выбрали антигенные пептиды по изобретению, способные вызывать наилучший перекрестно-реактивный опухолеспецифический цитотоксический Т-клеточный иммунный ответ для предотвращения и лечения В-клеточного гемобластоза.
В целом, общая коровая последовательность имеет высокую распространенность в микробиоте человека, при этом распространенность составляет выше 30%, предпочтительно выше 40%, предпочтительно выше 50%, более предпочтительно выше 60%, еще более предпочтительно выше 70%, еще более предпочтительно выше 80%, особо предпочтительно выше 90% и наиболее предпочтительно выше 95%.
Распространенность коровой последовательности определяют на основе частоты каждого белка микробиоты человека, в которой обнаружена коровая последовательность, общая с соответствующим (фрагментом/эпитопом) опухолевого антигена (человека), и с учетом частоты каждого белка микробиоты человека, где были обнаружены схожие пептиды (т.е. разные антигенные пептиды), имеющие одинаковую коровую последовательность. Например, чтобы оценить, имеет ли коровая последовательность высокую распространенность, частоту каждого белка, присутствующего в микробиоте человека, в которой обнаружена указанная коровая последовательность, рассчитывают из базы данных последовательностей микробиоты. Затем распространенность получают из кумулятивной частоты, рассчитанной для каждого белка микробиоты человека, где интересующая коровая последовательность обнаружена и присутствует в антигенном пептиде. Такая база данных может предпочтительно содержать данные о микробиоте (последовательности) множества индивидуумов (субъектов). Примером такой базы данных является «Интегрированный справочный каталог микробиома кишечника человека» (версия 1.0, март 2014; Li et al. MetaHIT Consortium. An integrated catalog of reference genes in the human gut microbiome. Nat Biotechnol. 2014 Aug; 32(8):834-41 URL: http://meta.genomics.cn/meta/home), который включает данные основных усилий по профилированию микробиома человека; Проект микробиома человека Американского национального института здоровья (NIH-HMP) и Европейская инициатива по метагеномике желудочно-кишечного тракта человека (MetaHIT).
Соответственно, изобретение относится к антигенным пептидам, имеющим аминокислотное сходство с опухолевым антигеном, поскольку они получены из этого опухолевого антигена (или опухолевого эпитопа). Выражение «имеющий аминокислотное сходство с опухолевым антигеном», используемое в настоящей заявке, относится, в частности, к варианту последовательности фрагментов (эталонного) опухолевого антигена человека, такого как CD22, или другим приведенным в качестве примера опухолевым антигенам человека, описанным ниже в Таблицах 1A, 1В и 1С. «Вариант последовательности» обычно имеет, в частности, по всей длине последовательности, по меньшей мере 50% идентичности последовательности с эталонной последовательностью, а именно фрагментом (эталонного) опухолевого антигена. Предпочтительно, вариант последовательности имеет по меньшей мере 55%, предпочтительно по меньшей мере 60%, предпочтительно 66%, предпочтительно по меньшей мере 70%, предпочтительно по меньшей мере 77%, более предпочтительно по меньшей мере 80%, еще более предпочтительно по меньшей мере 88%, еще более предпочтительно по меньшей мере 90% идентичность последовательности с эталонной последовательностью, а именно фрагментом (эталонного) опухолевого антигена. Идентичность последовательности можно рассчитать, как известно в данной области техники, в частности, как описано ниже. Предпочтительно вариант последовательности сохраняет специфическую функцию эталонной последовательности, например, ее функцию эпитопа опухоли и/или ее способность вызывать или поддерживать иммунный ответ. В частности, вариант аминокислотной последовательности имеет измененную последовательность, в которой одна или несколько аминокислот в эталонной последовательности мутированы, например, удалены или заменены, или одна или несколько аминокислот вставлены в последовательность эталонной аминокислотной последовательности. Например, вариантные последовательности, идентичные по меньшей мере на 90%, имеют не более 10 изменений, т. е. любую комбинацию делеций, вставок или замен, на 100 аминокислот эталонной последовательности.
Способы определения идентичности (сходства) двух и более последовательностей хорошо известны из области техники. Процент идентичности двух последовательностей можно определить, например, при помощи математического алгоритма. Предпочтительным, но не ограничивающим примером подходящего математического алгоритма, который может использоваться для этих целей, является алгоритм Karlin et al. (1993), PNAS USA, 90:5873-5877. Такой алгоритм интегрирован в пакет программ BLAST, например, программу BLAST или NBLAST (см также Altschul et al., 1990, J. Mol. Biol. 215, 403-410 или Altschul et al. (1997), Nucleic Acids Res, 25:3389-3402), которые доступны на домашней странице NCBI в мировой сети интернет ncbi.nlm.nih.gov) и FASTA (Pearson (1990), Methods Enzymol. 183, 63-98; Pearson and Lipman (1988), Proc. Natl. Acad. Sci. U. S. A 85, 2444-2448). Последовательности, которые идентичны другим последовательностям на определенном протяжении, могут быть идентифицированы с помощью этих программ. Кроме того, для определения процента идентичности между двумя полинуклеотидами и процента идентичности между двумя (поли)пептидными последовательностями могут быть использованы программы, доступные в пакете для анализа последовательностей Висконсин, версия 9.1 (Devereux et al., 1984, Nucleic Acids Res., 387-395), например, программы BESTFIT и GAP. BESTFIT использует алгоритм «локальной гомологии» Smith and Waterman (1981), J. Mol. Biol. 147, 195-197 и находит единственный участок с наилучшим сходством двух последовательностей.
В целом, антигенный пептид по настоящему изобретению связывается с молекулами MHC I класса (главного комплекса гистосовместимости I класса, MHC I).
Молекулы MHC I класса презентируют эпитопы Т-киллерам, также называемым цитотоксическими Т-лимфоцитами (CTL). CTL экспрессируют рецепторы CD8 в дополнение к TCR (Т-клеточным рецепторам). Когда рецептор CD8 CTL стыкуется с молекулой МНС I класса, если TCR CTL соответствует эпитопу внутри молекулы МНС I класса, CTL запускает запрограммированную гибель клетки путем апоптоза. Этот путь особенно полезен для профилактики и/или лечения рака, поскольку раковые клетки атакуются напрямую. У людей MHC I класса включает молекулы HLA-A, HLA-B и HLA-C. Обычно пептиды (эпитопы) длиной 8-10 аминокислот презентируются MHC I.
В целом, антигенный пептид, заявленный в соответствии с настоящим изобретением, может иметь любую длину. Предпочтительно, длина антигенного пептида в соответствии с настоящим изобретением не превышает 350 аминокислот. Например, максимальная длина антигенного пептида в соответствии с настоящим изобретением может составлять от 300 до 250 аминокислот. Более предпочтительно, максимальная длина антигенного пептида в соответствии с настоящим изобретением не превышает 200 аминокислот, например, не превышает 190, 180, 170, 160, 150, 140, 130, 120, 110, 100, 95, 90, 85, 80, 75, 70, 65, 60, 55, 50, 45, 40, 35, 30, 29, 28, 27, 26, 25, 24, 23, 22, 21, 20, 19, 18, 17, 16, 15, 14 или 13 аминокислот. В частности, длина антигенных пептидов в соответствии с настоящим изобретением предпочтительно составляет самое большее 30 или 25 аминокислот, более предпочтительно самое большее 20 или 15 аминокислот, еще более предпочтительно самое большее 10 аминокислот. Особо предпочтительно, антигенный пептид по настоящему изобретению содержит по меньшей мере 8 или 9 аминокислот, например, 10 аминокислот. Еще более предпочтительно, антигенный пептид имеет длину 9 или 10 аминокислот. В частности, антигенные пептиды не являются полноразмерными белками, продуцируемыми микробиотой человека (из которых могут быть получены антигенные пептиды). Другими словами, антигенный пептид по изобретению предпочтительно является фрагментом полноразмерного белка (продуцируемого микробиотой человека).
Точно так же «фрагмент/эпитоп» (эталонного) опухолевого антигена, который обычно служит эталонной последовательностью, предпочтительно содержит последовательные 9 аминокислот опухолевого антигена и, возможно, 10 аминокислот. Понятно, что «фрагмент/эпитоп» (эталонного) опухолевого антигена не является полноразмерным опухолевым антигеном (белком).
«Фрагмент» (белка или нуклеиновой кислоты (последовательности)), как применяется в настоящей заявке, предпочтительно имеет максимальную длину, которая составляет 95%, 90%, 85%, 80%, 75%, 70%, 65%, 60%, 55%, 50%, 45%, 40%, 35%, 30%, 25%, 20%, 19%, 18%, 17%, 16%, 15%, 14%, 13%, 12%, 11%, 10%, 9%, 8%, 7%, 6%, 5%, 4%, 3%, 2% или 1% oт длины полноразмерной (эталонной) последовательности белка/нуклеиновой кислоты. Согласно некоторым вариантам осуществления изобретения длина фрагмента не превышает 50% от длины (полноразмерного) (эталонного) белка/нуклеиновой кислоты. Согласно другим вариантам осуществления изобретения, длина фрагмента (эталонного) белка/нуклеиновой кислоты не превышает 20% или 10% от длины (полноразмерного) (эталонного) белка/нуклеиновой кислоты.
В более общем смысле, настоящее изобретение относится к антигенному пептиду, который содержит или состоит из варианта последовательности фрагмента/эпитопа человеческого (эталонного) опухолевого антигена, особенно варианта последовательности микробиоты фрагмента/эпитопа опухолевого антигена человека. Опухолевый антиген человека может быть выбран из группы, состоящей из CD19, CD20, CD22, CD37 и TNFRSF13C. Фрагмент/эпитоп человеческого (эталонного) опухолевого антигена может быть выбран из группы, состоящей из любой из SEQ ID №№ 258-280.
Предпочтительно антигенный пептид содержит или состоит из варианта микробиоты эталонного пептида человека в соответствии с любой из SEQ ID №№ 258-280. В частности, SEQ ID №№ 258-280 относятся к эпитопам опухоли человека, в частности к эталонным эпитопам/фрагментам опухолевых антигенов В-клеток человека. Примерами вариантов микробиоты эталонных пептидов человека в соответствии с любой из SEQ ID №№ 258-280 являются пептиды в соответствии с SEQ ID №№ 1-257 и 476-500 (как показано в Таблице 1A ниже). Предпочтительные варианты последовательности микробиоты (варианты микробиоты) представляют собой варианты последовательности бактерий. Другими словами, аминокислотные последовательности в соответствии с SEQ ID №№ 1–257 и 476–500 могут быть обнаружены в бактериальных белках и демонстрируют сходство последовательностей с эталонными эпитопами/фрагментами опухолевых антигенов В-клеток человека, как подробно показано в Таблице 1А. Более предпочтительно, антигенный пептид содержит или состоит из варианта микробиоты эталонного пептида человека в соответствии с любой из SEQ ID №№ 258, 260-266, 270, 271, 279 и 280. Еще более предпочтительно, антигенный пептид содержит или состоит из варианта микробиоты эталонного пептида человека в соответствии с любой из SEQ ID №№ 260, 264, 270, 271, 279 и 280. Еще более предпочтительно, антигенный пептид содержит или состоит из варианта микробиоты эталонного пептида человека в соответствии с любой из SEQ ID №№ 264, 270, 271 и 279.
Соответственно, настоящее изобретение также относится к антигенному пептиду, содержащему или состоящему из варианта микробиоты эталонного пептида человека в соответствии с любой из SEQ ID №№ 258-280; предпочтительно в соответствии с любой из SEQ ID №№ 258, 260-266, 270, 271, 279 и 280; более предпочтительно в соответствии с любой из SEQ ID №№ 260, 264, 270, 271, 279 и 280; еще более предпочтительно в соответствии с любой из SEQ ID №№ 264, 270, 271 и 279.
В конкретном варианте осуществления настоящее изобретение относится к антигенному пептиду, полученному из CD19, CD20, CD22, CD37 или TNFRSF13C, где антигенный пептид имеет ту же коровую последовательность, что и фрагмент/эпитоп (эталонного) опухолевого антигена; и при этом общая коровая последовательность широко распространена в микробиоте человека. Такая распространенность определяется частотой по меньшей мере одного белка, присутствующего в микробиоте человека, в котором обнаружена указанная коровая последовательность. Предпочтительно антигенный пептид по изобретению содержит или состоит из любой из SEQ ID №№ 304-326 (Консенсусные последовательности MHC I).
В одном варианте осуществления антигенный пептид умеренно, сильно или очень сильно связывается с молекулами MHC I класса (главного комплекса гистосовместимости I класса, MHC I).
Связывание по меньшей мере одного антигенного пептида с молекулами MHC I класса (главного комплекса гистосовместимости I класса) можно проверить с помощью тестов связывания MHC I in silico или in vitro, как описано в настоящей заявке. Соответственно, сильные и очень сильные связующие агенты могут быть выбраны, как описано выше. Предпочтительно, связывание с MHC I (in silico и/или in vitro, как описано в настоящей заявке) тестируют для по меньшей мере одного антигенного пептида с молекулами MHC I и, дополнительно, для (соответствующего эталонного) опухолевого антигена (фрагмента/эпитопа (эталонного) опухолевого антигена) с молекулами MHC I, а аффинность связывания предпочтительно получают для обоих из них (фрагмента/эпитопа опухолевого антигена и антигенного пептида).
После теста на связывание предпочтительно выбирают только такие антигенные пептиды, которые умеренно, сильно или очень сильно связываются с MHC I. Более предпочтительно, выбирают только сильные и очень сильные связывающие агенты и наиболее предпочтительно выбирают только такие антигенные пептиды, которые очень сильно связываются с MHC I. Более предпочтительно, выбирают только такие антигенные пептиды, которые сильно или очень сильно связываются с молекулой MHC I, и где эпитоп/фрагмент (эталонного) опухолевого антигена («соответствующая» последовательность опухолевого антигенного эпитопа) связывается слабее (например, слабо или умеренно) с молекулой MHC I. Еще более предпочтительно, выбирают только такие варианты последовательности микробиоты, которые очень сильно связываются с MHC I, и в которых эпитоп/фрагмент (эталонного) опухолевого антигена слабо связывается с MHC I.
Соответственно, антигенный пептид по изобретению предпочтительно связывается сильнее (т.е. имеет более высокую аффинность связывания) с молекулой MHC I, чем соответствующий эталонный человеческий эпитоп опухолевого антигена В-клеток (который имеет ту же коровую последовательность). Другими словами, эталонный эпитоп опухолевого антигена В-клеток человека, который имеет ту же коровую последовательность, связывается предпочтительно слабее с молекулами MHC I (т.е. имеет более низкую аффинность связывания), чем соответствующий антигенный пептид.
Прогноз связывания MHC I класса (тест связывания MHC in silico) может быть выполнен с использованием общедоступных инструментов, таких как «NetMHCpan», например, «NetMHCpan 3.0 Server» или «NetMHCpan 4.0 Server» (Центр анализа биологических последовательностей, Датский технический университет DTU; URL: http://www.cbs.dtu.dk/services/NetMHCpan/). Метод NetMHCpan, в частности NetMHCpan 3.0 или более поздняя версия, обучен более чем 180 000 количественным данным связывания, охватывающим 172 молекулы MHC человека (HLA-A, B, C, E) и других видов. В общем, аффинность можно прогнозировать, оставив пороговые значения по умолчанию для сильных и слабых связующих агентов. Например, для HLA-A*0201 расчетная аффинность ниже 50 нМ может указывать на «сильные связывающие агенты», а аффинность от 50 до 300 нМ может указывать на «умеренные связывающие агенты». В NetMHCpan, например, в NetMHCpan 3.0 или NetMHCpan 4.0, ранг прогнозируемой аффинности можно сравнить с набором из 400000 случайных натуральных пептидов, которые можно использовать в качестве меры % ранга аффинности связывания. На это значение не влияет присущее некоторым молекулам смещение в сторону более высокой или более низкой средней прогнозируемой аффинности. Например (к примеру, для HLA-A*0201), очень сильные связующие агенты могут быть определены как имеющие % ранга <0,5, сильные связующие агенты могут быть определены как имеющие % ранга <1,0, средние связующие агенты могут быть определены как имеющие % ранга от 1,0 до 2,0, а слабые связующие агенты могут быть определены как имеющее % ранга >2,0. Способ тестирования in vitro хорошо известен специалистам в данной области техники. Например, специалист в данной области техники может использовать экспериментальный протокол, утвержденный для пептидов, представленных HLA-A*0201, в Tourdot et al., A general strategy to enhance immunogenicity of low-affinity HLA-A2.1 - associated peptides: implication in the identification of cryptic tumor epitopes. Eur J Immunol. 2000 Dec; 30(12):341 1-21. В этом контексте в тесте можно дополнительно использовать эталонный пептид, такой как pol 589-597 ВИЧ. Это позволяет рассчитать аффинность in vitro относительно связывания, наблюдаемого с эталонным пептидом, например, следующим уравнением: Относительная аффинность = концентрация каждого пептида, индуцирующая 20% экспрессии HLA-A*0201 / концентрация эталонного пептида, индуцирующая 20% экспрессии HLA-A*0201 (где 100% — уровень экспрессии HLA-A*0201, выявленный с помощью эталонного пептида, например, pol 589-597 ВИЧ, используемого, например, в концентрации 100 мкМ). Например, пептид с относительной аффинностью ниже 1 можно рассматривать как «сильный связывающий агент», пептид с относительной аффинностью от 1 до 2 можно рассматривать как «умеренный связывающий агент», а пептид с относительной аффинностью более 3 можно рассматривать как «слабый связующий агент».
У человека существует три различных генетических локуса, которые кодируют молекулы MHC I класса (молекулы MHC человека также обозначаются как человеческие лейкоцитарные антигены (HLA)): HLA-A, HLA-B и HLA-C. HLA-A*01, HLA-A*02, HLA-A*24 и HLA-B*07 являются примерами различных аллелей MHC I класса, которые могут экспрессироваться из этих локусов. Например, антигенный пептид по изобретению может связываться с молекулами HLA-A*01, HLA-A*02, HLA-A*24 и HLA-B*07. В частности, антигенный пептид по изобретению связывается с HLA-A*02. В одном варианте осуществления антигенный пептид по изобретению представляет собой вариант последовательности микробиоты эпитопа опухолевого антигена человека.
В частности, вариант последовательности микробиоты эпитопа опухолевого антигена человека идентифицирован по меньшей мере в одном белке, экспрессированном в микробиоте человека. Предпочтительно, по меньшей мере один белок, присутствующий в микробиоте человека, секретируется или содержит трансмембранный домен.
Клеточную локализацию, в частности то, секретируется ли белок или содержит трансмембранный домен, можно протестировать in silico или in vitro способами, хорошо известными специалистам в данной области техники. Например, «SignalP 4.1 Server» (Центр анализа биологических последовательностей, Датский технический университет, DTU; URL: www.cbs.dtu.dk/services/SignalP) и/или «Phobius» (Комбинированный предиктор трансмембранной топологии и сигнальных пептидов, Стокгольмский центр биоинформатики; URL: phobius.sbc.su.se). Предпочтительно можно комбинировать два инструмента прогнозирования (например, SignalP 4.1 Server и Phobius).
Например, чтобы проверить, секретируется ли белок, можно оценить присутствие сигнального пептида. Сигнальные пептиды представляют собой повсеместно распространенные сигналы сортировки белков, которые нацелены на свой белок-пассажир (карго) для транслокации через цитоплазматическую мембрану у прокариот. Для проверки наличия сигнального пептида можно использовать, например, «SignalP 4.1 Server» (Центр анализа биологических последовательностей, Датский технический университет, DTU; URL: www.cbs.dtu.dk/services/SignalP) и/или «Phobius» (Комбинированный предиктор трансмембранной топологии и сигнальных пептидов, Стокгольмский центр биоинформатики; URL: phobius.sbc.su.se).). Предпочтительно можно комбинировать два инструмента прогнозирования (например, SignalP 4.1 Server и Phobius).
Далее, можно определить, содержит ли белок трансмембранный домен. И сигнальные пептиды, и трансмембранные домены являются гидрофобными, но трансмембранные спирали обычно имеют более длинные гидрофобные участки. Например, SignalP 4.1 Server и Phobius способны отличать сигнальные пептиды от трансмембранных доменов. Предпочтительно установить минимальное количество двух прогнозируемых трансмембранных спиралей, чтобы различать мембранные и цитоплазматические белки, чтобы получить окончательный консенсусный список.
В одном варианте осуществления вариант последовательности микробиоты фрагмента/эпитопа опухолевого антигена человека презентируется полностью после расщепления бактериального белка, экспрессируемого в микробиоте человека. В этом контексте термин «расщепление» относится к процессингу пептида для презентации MHC, в частности к процессингу MHC-I. Например, прогностический показатель расщепления и/или аффинность можно использовать для прогнозирования антигенного расщепления для надлежащего связывания MHC (для презентации CD8 Т-клеткам). Такая «оценка вероятности расщепления» может быть рассчитана с помощью программного обеспечения (предпочтительно такая оценка выше 70%, предпочтительно выше 80%, более предпочтительно выше 90%). Например, можно использовать «IEDB» (Базу данных и ресурс анализа иммунных эпитопов, Ресурс анализа IEDB, поддерживается контрактом с Национальным институтом аллергии и инфекционных заболеваний, компонентом Национальных институтов здравоохранения в Министерстве здравоохранения и социального обеспечения), которая обеспечивает, например, прогнозы обработки MHC-I (URL: http://tools.iedb.org/mhcnp/). В другом примере NetChop3 (The role of the proteasome in generating cytotoxic T cell epitopes: Insights obtained from improved predictions of proteasomal cleavage. M. Nielsen, C. Lundegaard, O. Lund, and C. Kesmir. Immunogenetics., 57(1-2):33-41, 2005; Prediction of proteasome cleavage motifs by neural networks. C. Kesmir, A. Nussbaum, Hansjorg Schild, Vincent Detours, and S. Brunak, Prot. Eng., 15(4): 287-296, 2002; URL: http://www.cbs.dtu.dk/services/NetChop/) можно использовать для прогнозирования расщепления. Этот алгоритм основан на нейронной сети, обученной на экспериментальных данных по расщеплению протеасом in vitro и известных лигандах HLA. Результатом сети была оценка от 0,0 до 1,0. Случайный предиктор будет иметь оценку 0,5, а идеальный предиктор - 1. Соответственно, высокая оценка для пептида (например, выше 0,7, предпочтительно выше 0,8, более предпочтительно выше 0,9, как описано выше) свидетельствует о том, что он эффективно расщепляется на его N- и/или С-конце (а не в его центре), тогда как низкий балл связан с пептидами, расщепленными в их центре. Таким образом, информация о протеасомном расщеплении, транспорте TAP и инструментах анализа MHC 1 класса может быть объединена для прогнозирования презентации пептида.
В одном варианте осуществления антигенный пептид индуцирует перекрестную реактивность Т-клеток против человеческого эпитопа (эталонного) опухолевого антигена. Т-клеточная перекрестная реактивность представляет собой феномен иммунной системы, определяемый как распознавание двух или более комплексов пептид-МНС (pMHC) Т-клеточным рецептором (TCR).
Мимикрия эпитопов относится к концепции сходства последовательности и структуры между чужеродными антигенами и аутоантигенами как пусковому механизму, вызывающему перекрестно-реактивный иммунный ответ против аутоантигенов. Интересно, что такая мимикрия эпитопов предлагает возможный способ обойти ограничение репертуара Т-клеток человека из-за клонального истощения Т-клеток, распознающих аутоантигены.
В частности, антигены (т.е. антигенные пептиды по настоящему изобретению), отличные от аутоантигенов (например, человеческого эпитопа опухолевого антигена), но обладающие сходством последовательностей с аутоантигеном, (i) все же могут быть распознаны благодаря перекрестной реактивности Т-клеточного рецептора и (ii) ожидается, что такие антигены распознаются Т-клетками/TCR, которые не были истощены в процессе обучения Т-клеток. Соответственно, такие антигены способны вызывать сильный иммунный ответ, приводящий к клональной экспансии Т-клеток, обладающих потенциальной перекрестной реактивностью с аутоантигенами.
Перекрёстную реактивность Т-клеточного рецептора с эпитопом человеческого (эталонного) опухолевого антигена можно измерить, как показано в разделе Примеры, с помощью анализа EL1SPOT-IFNγ. Вкратце, HLA-A2 трансгенных мышей (например, мышей HHD DR1, экспрессирующих человеческие HLA-A2 и HLA-DR1 MHC и лишенные мышиных H-2 MHC I и II классов, и/или мышей HHD DR3, экспрессирующих человеческие HLA-A2 и HLA-DR3 MHC) иммунизируют в день 0 (d0) первичной инъекцией и в день 14 бустерной инъекцией антигенного пептида по настоящему изобретению и эпитопа человеческого (эталонного) опухолевого антигена. Через семь дней после повторной инъекции (т.е. в день 21) мышей подвергают эвтаназии, и спленоциты стимулируют in vitro антигенным пептидом по настоящему изобретению для оценки их способности секретировать IFN-гамма по данным ELISPOT.
В частности, настоящее изобретение относится к антигенному пептиду, который представляет собой вариант последовательности микробиоты фрагмента/эпитопа опухолевого антигена человека, где эпитоп опухолевого антигена человека может включать или состоять из любой из SEQ ID №№ 1-257 и 476-500.
В приведенной ниже Таблице 1А представлен обзор антигенных пептидов согласно настоящему изобретению с их аминокислотными последовательностями и SEQ ID №, а также с соответствующим фрагментом/эпитопом человеческого опухолевого антигена (также называемого в настоящем документе «человеческим эталонным пептидом»). В Таблице 1А также представлена информация, к какому опухолевому антигену относится каждый антигенный пептид по настоящему изобретению. SEQ ID №№ 1-257 и 476-500 относятся к антигенным пептидам HLA-A*02 по настоящему изобретению.
Таблица 1А. HLA-A*02 Антигенные пептиды по изобретению.
В приведенной ниже Таблице 1В представлен обзор консенсусных последовательностей (консенсусной последовательности MHC I класса) антигенных пептидов согласно настоящему изобретению с их аминокислотными последовательностями и SEQ ID №, а также с соответствующим фрагментом/эпитопом опухолевого антигена человека (также называемого в настоящей заявке «эталонным пептидом человека»), а также их коровой последовательности.
Таблица 1В. Консенсусные последовательности МНС I класса антигенных пептидов по изобретению.
MHC I класса
В приведенной ниже Таблице 1С представлен обзор консенсусных последовательностей HLA-A*02 антигенных пептидов согласно настоящему изобретению с их аминокислотными последовательностями и SEQ ID №, а также с соответствующей коровой последовательностью опухолевого антигена человека. В Таблице 1С также представлена информация, к какой коровой последовательности и консенсусной последовательности HLA-A*02 относится каждый антигенный пептид по настоящему изобретению.
Соответственно, антигенный пептид может содержать или состоять из аминокислотной последовательности, как указано в любой из SEQ ID №№ 309-326. В некоторых вариантах осуществления антигенный пептид содержит или состоит из аминокислотной последовательности, как указано в любой из SEQ ID №№ 316, 304-315 и 317-472 и 501-509, предпочтительно, как указано в любой из SEQ ID №№ 316, 304-315 и 317-326.
Таблица 1С. Консенсусные последовательности HLA-A*02 антигенных пептидов по изобретению.
HLA-A*02
Как видно из Таблиц 1А-1С, антигенные пептиды по настоящему изобретению могут быть классифицированы по соответствующему опухолевому антигену человека, соответствующей коровой последовательности и соответствующим консенсусным последовательностям MHC I класса и/или HLA-A*02.
Соответственно, антигенный пептид может содержать (коровую) последовательность в соответствии с любой из SEQ ID № 281–303. Предпочтительно антигенный пептид содержит (коровую) последовательность в соответствии с любой из SEQ ID №№ 281, 283–289, 293, 294, 302 и 303. Более предпочтительно, антигенный пептид содержит (коровую) последовательность в соответствии с любой из SEQ ID №№ 283, 287, 293, 294, 302 и 303. Еще более предпочтительно, антигенный пептид содержит (коровую) последовательность в соответствии с любой из SEQ ID №№ 287, 293, 294 и 302.
В одном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD19 (эталонного пептида человека), такой как «FLLFLTPME» (SEQ ID № 258), «KLSLGLPGL» (SEQ ID № 259), «SLVGILHLQ» (SEQ ID № 260), «TLAYLIFCL» (SEQ ID № 261) или «QQMGGFYLC» (SEQ ID № 262). В предпочтительном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD19, такой как антигенные пептиды, имеющие коровую последовательность, состоящую из аминокислотной последовательности, представленной в любой из SEQ ID №№ 281-285. Соответственно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD19, содержащий или состоящий из консенсусной последовательности MHC I класса, как указано в любой из SEQ ID №№ 304-308. В конкретном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD19, который связывается с конкретной молекулой HLA (HLA-A2*02), такой как антигенный пептид, содержащий или состоящий из консенсусной последовательности HLA-A2*02, указанной в любой из SEQ ID №№ 327-346. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности микробиоты фрагмента опухолевого антигена CD19, такой как антигенные пептиды, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 1-40. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD19 эталонного пептида человека) «FLLFLTPME» (SEQ ID № 258), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как представлено в SEQ ID №№ 1-12, 304 и 327-333, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID № 10. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD19 (эталонный пептид человека) «KLSLGLPGL». (SEQ ID № 259), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 13-33, 305 и 334-343. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD19 (эталонный пептид человека) «SLVGILHLQ» (SEQ ID № 260), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID № 34-35, 306 и 344, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID №№ 34 или 35. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD19 (эталонный пептид человека) «TLAYLIFCL» (SEQ ID № 261), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 36-39, 307 и 345-346, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID № 39. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD19 (эталонный пептид человека) «QQMGGFYLC» (SEQ ID № 262), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 40 и 308, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID № 40.
В одном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD20 (MS4A1; эталонный пептид человека), такой как «IALGGLLMI» (SEQ ID № 263), «IMNSLSLFA» (SEQ ID № 264), «LMIPAGIYA» (SEQ ID № 265) или «SLFLGILSV» (SEQ ID № 266). В предпочтительном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD20, такой как антигенные пептиды, имеющие коровую последовательность, состоящую из аминокислотной последовательности, представленной в любой из SEQ ID №№ 286-289. Соответственно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD20, содержащий или состоящий из консенсусной последовательности MHC I класса, как указано в любой из SEQ ID №№ 309-312. В конкретном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD20, который связывается с конкретной молекулой HLA (HLA-A2*02), такой как антигенный пептид, содержащий или состоящий из консенсусной последовательности HLA-A2*02, указанной в любой из SEQ ID №№ 347-374. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности микробиоты фрагмента опухолевого антигена CD20, такой как антигенные пептиды, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 41-87. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD20 (эталонный пептид человека) «IALGGLLMI» (SEQ ID № 263), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как представлено в SEQ ID №№ 41-64, 309 и 347-360, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID № 61. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD20 (эталонный пептид человека) «IMNSLSLFA» (SEQ ID № 264), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 65-70, 310 и 361-364 и 476-484, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в любой из SEQ ID №№ 65, 68, 70 и 477. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD20 (эталонный пептид человека) «LMIPAGIYA» (SEQ ID № 265), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 71-80, 311 и 365-369, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID № 72. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD20 (эталонный пептид человека) «SLFLGILSV» (SEQ ID № 266), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 81-87, 312 и 370-374, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID № 86.
В одном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD22 (эталонный пептид человека), такой как «FLSNDTVQL» (SEQ ID № 267), «HLLGPWLLL» (SEQ ID № 268), «ILILAICGL» (SEQ ID № 269) или «WVFEHPETL» (SEQ ID № 270). В предпочтительном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD22, такой как антигенные пептиды, имеющие коровую последовательность, состоящую из аминокислотной последовательности, представленной в любой из SEQ ID №№ 290-293. Соответственно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD22, содержащий или состоящий из консенсусной последовательности MHC I класса, как указано в любой из SEQ ID №№ 313-316. В конкретном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD22, который связывается с конкретной молекулой HLA (HLA-A2*02), такой как антигенный пептид, содержащий или состоящий из консенсусной последовательности HLA-A2*02, указанной в любой из SEQ ID №№ 375-390. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности микробиоты фрагмента опухолевого антигена CD22, такой как антигенные пептиды, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 88-110. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD22 (эталонный пептид человека) FLSNDTVQL» (SEQ ID № 267), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 88-96, 313 и 375-380. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD22 (эталонный пептид человека) «HLLGPWLLL» (SEQ ID № 268), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 97-101, 314 и 381-383. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD22 (эталонный пептид человека) «ILILAICGL» (SEQ ID № 269), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 102-105, 315 и 384-386. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD22 (эталонный пептид человека) «WVFEHPETL» (SEQ ID № 270), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 106-110, 316 и 387-390 и 485-488, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в любой из SEQ ID №№ 107, 108, 109 и 110.
В одном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD37 (эталонный пептид человека), такой как «GLAFVPLQI» (SEQ ID № 271), «GLYFGMLLL» (SEQ ID № 272), «ILILAICGL» (SEQ ID № 273), «LLLLFATQI» (SEQ ID № 274), «SIVGICLGV» (SEQ ID № 275) или «SLIKYFLFV» (SEQ ID № 276). В предпочтительном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD37, такой как антигенные пептиды, имеющие коровую последовательность, состоящую из аминокислотной последовательности, представленной в любой из SEQ ID №№ 294-299. Соответственно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD37, содержащий или состоящий из консенсусной последовательности MHC I класса, как указано в любой из SEQ ID №№ 317-322. В конкретном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена CD37, который связывается с конкретной молекулой HLA (HLA-A2*02), такой как антигенный пептид, содержащий или состоящий из консенсусной последовательности HLA-A2*02, указанной в любой из SEQ ID №№ 391-425. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности микробиоты фрагмента опухолевого антигена CD37, такой как антигенные пептиды, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 111-162. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD37 (эталонный пептид человека) «GLAFVPLQI» (SEQ ID № 271), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 111-130, 317 и 391-402 и 489-493, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в любой из SEQ ID №№ 114, 117, 119, 120, 491 и 493. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD37 (эталонный пептид человека) «GLYFGMLLL» (SEQ ID № 272), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 131-136, 318 и 403-406. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD37 (эталонный пептид человека) «ILILAICGL» (SEQ ID № 273), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 137-147, 319 и 407-414. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD37 (эталонный пептид человека) «LLLLFATQI» (SEQ ID № 274), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 148-155, 320 и 415-420. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD37 (эталонный пептид человека) «SIVGICLGV» (SEQ ID № 275), такой как антигенный пептид, содержащий или состоящий из аминокислоты последовательности, представленной в SEQ ID №№ 156-157, 321 и 421. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента CD37 (эталонный пептид человека) «SLIKYFLFV» (SEQ ID № 276), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 158-162, 322 и 422-425.
В одном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена TNFRSF13C (эталонный пептид человека), такой как «GLAFVPLQI» (SEQ ID № 277), «GLALVLALV» (SEQ ID № 278), «LLFGAPALL» (SEQ ID № 279), или «PLPGLLFGA» (SEQ ID № 280). В предпочтительном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена TNFRSF13C, такой как антигенные пептиды, имеющие коровую последовательность, состоящую из аминокислотной последовательности, представленной в любой из SEQ ID №№ 300-303. Соответственно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена TNFRSF13C, содержащий или состоящий из консенсусной последовательности MHC I класса, как указано в любой из SEQ ID №№ 323-326. В конкретном варианте осуществления антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента опухолевого антигена TNFRSF13C, который связывается с конкретной молекулой HLA (HLA-A2*02), такой как антигенный пептид, содержащий или состоящий из консенсусной последовательности HLA-A2*02, указанной в любой из SEQ ID №№ 426-472. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности микробиоты фрагмента опухолевого антигена TNFRSF13C, такой как антигенные пептиды, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 163-257. Более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента TNFRSF13C (эталонный пептид человека) «GLAFVPLQI» (SEQ ID № 277), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как представлено в SEQ ID №№ 163-170, 323 и 426-428. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента TNFRSF13C (эталонный пептид человека) «GLALVLALV» (SEQ ID № 278), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 171-209, 324 и 429-446. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента TNFRSF13C (эталонный пептид человека) «LLFGAPALL» (SEQ ID № 279), такой как антигенный пептид, содержащий или состоящий из аминокислоты последовательность, как указано в SEQ ID №№ 210-225, 325 и 447-455 и 494-500, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в любой из SEQ ID №№ 212, 217, 220 и 224. Также более предпочтительно, антигенный пептид по настоящему изобретению представляет собой вариант последовательности фрагмента TNFRSF13C (эталонный пептид человека) «LLFGAPALL» (SEQ ID № 279), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID №№ 226-257, 326 и 456-472, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 227.
Предпочтительно антигенный пептид по настоящему изобретению содержит или состоит из консенсусной последовательности МНС I класса, представленной в любой из SEQ ID №№ 304-312, 315-316 и 325. Более предпочтительно, антигенный пептид по настоящему изобретению содержит или состоит из консенсусной последовательности HLA-A*02, как указано в любой из SEQ ID №№ 330, 332-333, 336, 341-344, 346, 354-355, 360, 363-364, 367, 371-372, 374, 387, 390, 392-393, 402 и 450.
В некоторых вариантах осуществления антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID №№ 1-12, 34-35, 36-39, 40, 41-64, 65-70, 476-484, 71-80, 81-87, 106-110, 485-488, 111-130, 489-493, 210-225, 494-500 и 226-257; предпочтительно антигенный пептид содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 34-35, 65-70, 476-484, 106-110, 485-488, 111-130, 489-493, 210-225, 494-500 и 226-257; более предпочтительно антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID №№ 65-70, 476-484, 106-110, 485-488, 111-130, 489-493, 210-225 и 494-500.
В некоторых вариантах осуществления антигенный пептид содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 10, 34, 35, 39, 40, 61, 68, 70, 72, 86, 107-110, 114, 117, 119, 120, 212, 217, 220, 224, 227, 231, 477, 491 и 493. Еще более предпочтительно, антигенный пептид по настоящему изобретению содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 61, 65, 68, 72, 86, 110, 114 и 220. Еще более предпочтительно, антигенный пептид по настоящему изобретению содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 110, 114 и 220. Наиболее предпочтительно, антигенный пептид по настоящему изобретению содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID № 10, 110, 114 и 220. Еще более предпочтительно антигенные пептиды по настоящему изобретению содержат или состоят из аминокислотной последовательности, указанной в любой из SEQ ID №№ 65, 110, 114 и 220. В некоторых вариантах осуществления антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в SEQ ID №№ 65. В некоторых вариантах осуществления антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в SEQ ID № 110. В некоторых вариантах осуществления антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в SEQ ID № 114. В некоторых вариантах осуществления антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в SEQ ID № 220.
Как показано в приведенных здесь примерах, специфические антигенные пептиды по настоящему изобретению позволяют вызывать сильный иммунный ответ против самих себя и, что наиболее важно, позволяют вызывать сильный иммунный ответ против пептидов, имеющих аминокислотное сходство с теми, которые входят в состав опухолевого антигена, даже если эталонные пептиды человека, содержащиеся в опухолевом антигене, могут быть толерогенными.
Предпочтительно, антигенные пептиды по настоящему изобретению могут быть в форме иммуногенных соединений, в частности, для применения в профилактике или лечении В-клеточного гемобластоза.
Иммуногенные соединения, содержащие антигенный пептид по изобретению
В еще одном аспекте настоящее изобретение также относится к иммуногенному соединению, содержащему антигенный пептид по настоящему изобретению, как описано выше. В частности, предпочтительные варианты антигенного пептида, как описано выше, также применимы для иммуногенного соединения по настоящему изобретению. Например, антигенный пептид, содержащийся в иммуногенном соединении, предпочтительно содержит или состоит из консенсусной последовательности MHC I класса, как указано в любой из SEQ ID №№ 304-326, такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указано в любой из SEQ ID №№ 1-247 и 476-500. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, указанной в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 61, 65, 68, 72, 86, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 10, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 65, 110, 114 и 220, являются еще более предпочтительными. Также предпочтительными являются их комбинации, а именно, иммуногенное соединение, содержащее различные антигенные пептиды по настоящему изобретению.
Используемый в настоящем изобретении термин «иммуногенное соединение» относится к соединению, которое обладает способностью индуцировать, усиливать, пролонгировать или поддерживать иммунный ответ, в частности, индуцировать, усиливать, пролонгировать или поддерживать иммунный ответ при введении млекопитающему, в особенности при введении человеку.
В целом, термин «иммуногенное соединение» относится ко всем типам соединений, включающим антигенный пептид по настоящему изобретению. Например, антигенный пептид по настоящему изобретению может быть связан с несущей молекулой, или антигенный пептид по настоящему изобретению может быть включен в состав полипептида или белка (причем такой полипептид или белок может существовать «отдельно», то есть не быть связанным с каким-либо другим соединением, или такой полипептид или белок, включающий антигенный пептид, может быть связан с несущей молекулой).
Предпочтительно иммуногенное соединение по настоящему изобретению включает антигенный пептид и несущую молекулу, в частности, где антигенный пептид (или полипептид или белок, включающий антигенный пептид) связан с несущей молекулой. Предпочтительной несущей молекулой является белок-носитель или пептид-носитель. Согласно предпочтительному варианту осуществления изобретения антигенный пептид, как раскрыто выше, или полипептид/белок, включающий указанный антигенный пептид, соединен с белком-носителем или пептидом-носителем, например, при помощи ковалентной или нековалентной связи. Альтернативно, такой белок-носитель или пептид-носитель, как раскрыто в настоящей заявке, можно вводить (отдельно) совместно в форме иммуностимулятора (то есть не как самостоятельное «иммуногенное соединение», но в составе комбинированной терапии, как раскрыто ниже).
Несущая молекула также может представлять собой липид или липидоподобную молекулу. В этом случае иммуногенное соединение может представлять собой липопептид. Используемый в настоящем изобретении термин «липопептид» относится к молекуле, которая включает липид или липидоподобную молекулу, ковалентно связанную с молекулой пептида. В целом «липид» растворим в неполярных растворителях, обычно «липид» не растворим (или плохо растворим) в воде. Примеры липидной или липидоподобной молекулы включают жирные кислоты, воски, стерины, моноглицериды, диглицериды, триглицериды и фосфолипиды, но не ограничиваются ими. Липид может представлять собой жирную кислоту, глицеролипид, глицерофосфолипид, сфинголипид, стероловый липид, преноловый липид, сахаролипид или поликетид. Предпочтительно липид представляет собой жирную кислоту или ее производное (включая моноглицериды, диглицериды, триглицериды и фосфолипиды). Жирные кислоты обычно включают углеводородную цепь, которая заканчивается карбоксильной группой. Жирные кислоты могут быть насыщенными или ненасыщенными. Жирные кислоты могут прикрепляться к функциональным группам, например, включающим кислород, галогены, азот или серу. Предпочтительные жирные кислоты представляют собой насыщенные или ненасыщенные длинноцепочечные жирные кислоты, такие как миристиновая кислота с 14 атомами углерода (CH3(CH2)12COOH) или пальмитиновая кислота с 16 атомами углерода (CH3(CH2)14COOH), а также фосфолипиды, такие как фосфатидилглицерин (PG).
Предпочтительно, антигенный пептид, как раскрыто в настоящем описании, или полипептид/белок, включающий антигенный пептид, может быть введен совместно или быть связанным, например, с помощью ковалентной или нековалентной связи, с белком/пептидом, обладающим иммуностимулирующими свойствами, такими как обеспечивающие стимуляцию CD4+ Th1 клеток. Хотя антигенный пептид, раскрытый в настоящей заявке, предпочтительно, связывается с MHC I класса, CD4+ хелперные эпитопы могут дополнительно использоваться для обеспечения эффективного иммунного ответа. Th1 хелперные клетки обладают способностью поддерживать эффективную активацию дендритных клеток (DC) и специфическую CTL активацию за счет секреции интерферона-гамма (IFN-γ), фактора некроза опухолей-альфа (TNF-α) и интерлейкина-2 (IL-2), а также усиления экспрессии костимулирующего сигнала на DC и Т-клетках (Galaine et al., Interest of Tumor-Specific CD4 T Helper 1 Cells for Therapeutic Anticancer Vaccine. Vaccines (Basel). 2015 Jun 30;3(3):490-502).
Например, адъювантный пептид/белок может предпочтительно представлять собой неопухолевый антиген, который оживляет иммунную память или оказывает неспецифическую помощь, либо представляет собой специфический хелперный белок опухолевого происхождения. В литературе описано несколько хелперных белков для обеспечения неспецифической помощи Т-клеткам, например, столбнячный хелперный белок, гемоцианин лимфы улитки или пептид PADRE (Adotévi et al., Targeting antitumor CD4 helper T cells with universal tumor-reactive helper peptides derived from telomerase for cancer vaccine. Hum Vaccin Immunother. 2013 May; 9(5):1073-7, Slingluff CL, The present and future of peptide vaccines for cancer: single or multiple, long or short, alone or in combination? Cancer J. 2011 Sep-Oct; 17(5):343-50). Таким образом, столбнячный хелперный пептид, гемоцианин лимфы улитки и пептид PADRE являются предпочтительными примерами таких адъювантных пептидов/белков. Кроме того, специфические хелперные белки опухолевого происхождения являются предпочтительными. Специфические хелперные пептиды опухолевого происхождения обычно презентируются MHC II класса, в частности, с помощью HLA-DR, HLA-DP или HLA-DQ. Специфические хелперные пептиды опухолевого происхождения могут представлять собой фрагменты последовательностей общих сверхэкспрессируемых опухолевых антигенов, таких как HER2, NY-ESO-1, hTERT или IL13RA2. Такие фрагменты имеют предпочтительную длину по меньшей мере 10 аминокислот, более предпочтительно по меньшей мере 11 аминокислот, еще более предпочтительно по меньшей мере 12 аминокислот и еще более предпочтительно по меньшей мере 13 аминокислот. В частности, фрагменты общих сверхэкспрессируемых опухолевых антигенов, такие как HER2, NY-ESO-1, hTERT или IL13RA2, имеющие длину от 13 до 24 аминокислот, являются предпочтительными. Предпочтительные фрагменты связываются с MHC II класса и могут, таким образом, быть идентифицированы с применением, например, средств прогнозирования IEDB (Базы данных и ресурс анализа иммунных эпитопов, поддерживается контрактом с Национальным институтом аллергии и инфекционных заболеваний, компонентом Национальных институтов здравоохранения в Министерстве здравоохранения и социального обеспечения; URL: http://tools.iedb.org/mhcnp/) для связывания с MHC II класса. Предпочтительно адъювантный пептид/белок может представлять собой пептид HHD-DR3 с последовательностью MAKTIAYDEEARRGLERGLN (SEQ ID № 473). Другим предпочтительным примером является h-pAg T13L (последовательность: TPPAYRPPNAPIL; SEQ ID № 474; Bhasin M, Singh H, Raghava GP (2003) MHCBN: a comprehensive database of MHC binding and non-binding peptides. Bioinformatics 19: 665–666). Другие примеры предпочтительных адъювантных пептидов/белков, в частности хелперных пептидов, включают UCP2 пептид (например, как раскрыто в WO 2013/135553 A1 или в Dosset et al. Clin Cancer Res. 2012 Nov 15; 18(22):6284-95) и BIRC5 пептид (например, как описано в EP2119726 A1 или в Widenmeyer et al. Int J Cancer. 2012 Jul 1; 131(1):140-9). Наиболее предпочтительным хелперным пептидом является UCP2 пептид (аминокислотная последовательность: KSVWSKLQSIGIRQH; SEQ ID № 475, например, как раскрыто в WO 2013/135553 A1 или в Dosset et al., Clin Cancer Res. 2012 Nov 15;18(22):6284-95).
Также предпочтительно иммуногенное соединение по настоящему изобретению представляет собой полипептид или белок, включающий антигенный пептид по настоящему изобретению. Предпочтительно, такой белок или полипептид представляет собой рекомбинантный белок или полипептид, например, гибридный белок. Термин «рекомбинантный» означает, что он не существует в природе.
Согласно предпочтительному варианту осуществления изобретения, иммуногенное соединение по настоящему изобретению состоит из полипептида формулы (I) или включает его:
PepNt-CORE-PepCt (I)
где:
- «PepNt» состоит из полипептида, имеющего длину от 0 до 500 аминокислотных остатков, и расположенного на N-конце полипептида формулы (I);
- «CORE» состоит из антигенного пептида по настоящему изобретению, как определено выше; и
- «PepCt» состоит из полипептида, имеющего длину от 0 до 500 аминокислотных остатков и расположенного на С-конце полипептида формулы (I).
Например, иммуногенное соединение может состоять из полипептида формулы (Ia) или (Ib), или включать их:
PepNt- CORE (Ia); или
CORE-PepCt (Ib);
где «PepNt» и «PepCt» и «CORE» являются такими, как определено выше.
Предпочтительно полипептид формулы (I), (Ia) или (Ib) представляет собой гибридный пептид или белок, в частности рекомбинантный гибридный пептид или белок.
Также предпочтительно, полипептид или иммуногенное соединение, как определено выше, содержит от 9 до 1000 аминокислот; что включает 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67? 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 250, 300, 350, 400, 450, 500, 600, 700, 800, 900 и 1000 аминокислот. Соответственно, длина «PepNt» и «PepCt», если необходимо, может быть определена соответствующим образом.
Таким образом, «PepNt» и «PepCt», как определено выше, могут содержать от 0 до 500 аминокислотных остатков; что включает 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45, 46, 47, 48, 49, 50, 51, 52, 53, 54, 55, 56, 57, 58, 59, 60, 61, 62, 63, 64, 65, 66, 67, 68, 69, 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, 100, 110, 120, 130, 140, 150, 160, 170, 180, 190, 200, 250, 300, 350, 400, 450, и 500 аминокислотных остатков.
Типы молекул-носителей, которые используются для создания иммуногенного соединения по изобретению, такого как иммуногенное соединение, включающее или состоящее из полипептида формулы (I), связанного с молекулой-носителем, хорошо известны специалисту в данной области техники. В частности, функция молекулы-носителя заключается в обеспечении цитокиновой помощи (или Т-клеточной помощи), для того чтобы усилить иммунный ответ против опухолевого антигена.
Предпочтительно, антигенный пептид соединен с молекулой-носителем, в частности с белком-носителем, предпочтительно посредством ковалентной или нековалентной связи. Молекула-носитель, с которой при необходимости связан пептид, может быть выбрана из большого разнообразия известных носителей. Примерами молекул-носителей, которые могут применяться для создания вакцин, являются белки, такие как человеческий или бычий сывороточный альбумин и гемоцианин лимфы улитки (KLH), а также жирные кислоты. Другие варианты осуществления молекул-носителей, с которыми может быть ковалентно связан антигенный пептид формулы (I), включают бактериальные токсины или анатоксины, такие как дифтерийный, холерный анатоксины, термолабильный анатоксин E. сoli или столбнячный анатоксин, белок наружной мембраны N. meningitidis (Европейская патентная заявка № EP0372501), синтетические пептиды (Европейские патентные заявки № EP0378881 и № EP0427347), белки теплового шока (заявка PCT № W093/17712), коклюшные белки (заявка PCT № W098/58668), белок D из H. influenzae (заявка PCT № WO00/56360) и токсин A или B из C. difficile (Международная патентная заявка WO00/61761).
Более предпочтительно, белок-носитель или пептид-носитель представляет собой белок/пептид, обладающий иммуностимулирующими свойствами, такими как обеспечение стимуляции CD4+ Th1 клеток, как раскрыто в настоящем описании. Предпочтительным примером таких молекул является неопухолевый антиген, который вызывает иммунную память или оказывает неспецифическую помощь, или представляет собой специфический хелперный пептид опухолевого происхождения, такой как столбнячный хелперный пептид, пептид из гемоцианина лимфы улитки или пептид PADRE. Другим предпочтительным примером является специфический хелперный пептид опухолевого происхождения, который может быть презентирован MHC II, в частности HLA-DR, HLA-DP или HLA-DQ, такой как фрагменты общих сверхэкспрессируемых опухолевых антигенов, например, HER2, NY-ESO-1 или hTERT. В предпочтительном варианте осуществления изобретения белок-носитель или пептид- носитель может представлять собой белок/пептид, обладающий иммуностимулирующими свойствами, например, пептид-носитель HHD-DR3 MAKTIAYDEEARRGLERGLN (SEQ ID № 473). В частности, «PepNt» и/или «PepCt» могут соответствовать белку-носителю или пептиду-носителю, такому как пептид-носитель HHD-DR3 MAKTIAYDEEARRGLERGLN (SEQ ID № 473). Другим предпочтительным примером является h-pAg T13L (последовательность: TPPAYRPPNAPIL; SEQ ID № 474; Bhasin M, Singh H, Raghava GP (2003) MHCBN: a comprehensive database of MHC binding and non-binding peptides. Bioinformatics 19: 665–666). Другими примерами предпочтительных белков/пептидов-носителей, в частности, хелперных пептидов являются UCP2 пептид (например, как раскрыто в WO 2013/135553 A1 или в Dosset et al., Clin Cancer Res. 2012 Nov 15;18(22):6284-95) и BIRC5 пептид (например, как раскрыто в EP2119726 A1 или в Widenmeyer et al., Int J Cancer. 2012 Jul 1;131(1):140-9). Наиболее предпочтительным хелперным пептидом является UCP2 пептид (аминокислотная последовательность: KSVWSKLQSIGIRQH; SEQ ID № 475).
Кроме того, в полипептиде формулы (I), (Ia) или (Ib) «PepNt» и/или «PepCt» могут предпочтительно соответствовать такому белку/пептиду, обладающему иммуноадъювантными свойствами, который обеспечивает стимуляцию CD4+ Th1 клеток, как раскрыто в настоящем описании.
Кроме того, иммуногенное соединение может включать или состоять из такого белка/пептида с иммуноадъювантными свойствами, такими как обеспечение стимуляции CD4+ Th1 клеток, как раскрыто в настоящем описании, который ковалентно связан с N-концом антигенного пептида по настоящему изобретению или N-концом полипептида/белка, включающего указанный антигенный пептид.
Предпочтительно, антигенный пептид по настоящему изобретению (или полипептид/белок, включающий указанный антигенный пептид) ковалентно связан с молекулой-носителем посредством линкерной молекулы.
Предпочтительные линкерные агенты включают линкерные агенты, обозначаемые как GMBS, сульфо-GMBS, SMPB и сульфо-SMPB.
Согласно некоторым вариантам осуществления иммуногенного соединения, как определено в настоящем описании, линкерный агент выбран из группы, включающей GMBS (N-[γ-малеимидобутирил-окси] сукцинимидный эфир), сульфо-GMBS (N-[γ-малеимидобутирил-окси] сульфосукцинимидный эфир), SMPB (сукцинимидил 4-[p-малеимидофенил]бутират) и сульфо-SMPB (сульфосукцинимидил 4-[p-малеимидофенил] бутират).
Способы конъюгации двух белков с линкерным агентом в целом и более конкретно с линкерным агентом, выбранным из группы, включающей GMBS, сульфо-GMBS, SMPB и сульфо-SMPB, хорошо известны специалисту в данной области техники. Например, такие протоколы раскрыты в листовках, которые публикуются компанией Pierce (Иллинойс, США). GMBS, сульфо-GMBS, SMPB и сульфо-SMPB состоят из гетеробифункциональных линкерных агентов, которые включают как эфирную группу N-гидроксисукцинимида (NHS), так и малеимидную группу. Конъюгация с использованием GMBS, сульфо-GMBS, SMPB или сульфо-SMPB обычно происходит в два этапа. На первом этапе аминосодержащий белок вступает в реакцию с линкерным агентом, молярное количество которого в несколько раз превышает количество белка, при рН 7-9 с формированием амидных связей с последующим удалением избытка не вступившего в реакцию линкерного агента, обычно при помощи обессоливания или диализа. На втором этапе добавляют молекулу, содержащую сульфгидрильную группу (например, пептид формулы (I)), для осуществления реакции с малеимидной группой, уже присоединенной к первому белку, при рН 6,5-7,5 для формирования стабильных тиоэфирных связей.
Применение SMPB или сульфо-SMPB в качестве линкерных агентов для ковалентного связывания антигенного пептида по настоящему изобретению (или полипептида/белка, включающего указанный антигенный пептид, такого как полипептид формулы (I)) c аминосодержащим белком-носителем приводит к образованию конъюгата формулы (II) ниже:
где:
- R1 состоит из одной реакционноспособной группы аминосодержащего белка- носителя, причем присоединенная к ней NH группа происходит из (i) альфа аминогруппы, расположенной на N-конце аминосодержащего белка-носителя, или (ii) аминогруппы боковой цепи аминокислотного остатка лизина (К) из аминосодержащего белка-носителя; и
- R2 состоит из антигенного пептида по настоящему изобретению (или полипептида/белка, включающего указанный антигенный пептид, такой как полипептид формулы (I)), причем прикрепленный к нему атом серы (S) происходит из сульфгидрильной группы (SH) цистеинового остатка, расположенного на N-конце или С-конце пептида формулы (I). Согласно некоторым вариантам осуществления изобретения сульфгидрильная молекула может быть частью ненатуральной аминокислоты, или любой другой молекулы, находящейся на конце пептида формулы (I).
Применение GMBS или сульфо-GMBS в качестве линкерных агентов для ковалентного связывания антигенного пептида по настоящему изобретению (или полипептида/белка, включающего указанный антигенный пептид, такого как полипептид формулы (I)) c аминосодержащим белком-носителем, в частности, с носителем CRM197, приводит к образованию конъюгата формулы (III) ниже:
где:
- R1 состоит из одной реакционноспособной группы аминосодержащего белка- носителя, причем прикрепленная к ней NH группа происходит из (i) альфа аминогруппы, расположенной на N-конце аминосодержащего белка-носителя, или (ii) аминогруппы боковой цепи аминокислотного остатка лизина (К) из аминосодержащего белка-носителя; и
- R2 состоит из антигенного пептида по настоящему изобретению (или полипептида/белка, включающего указанный антигенный пептид, такого как полипептид формулы (I)), причем прикрепленный к нему атом серы (S) происходит из сульфгидрильной группы (SH) цистеинового остатка, расположенного на N-конце или С-конце пептида формулы (I). Согласно некоторым вариантам осуществления изобретения сульфгидрильная молекула может быть частью ненатуральной аминокислоты или любой другой молекулы, находящейся на конце пептида формулы (I).
Мультимеры пептид–MHC (pMHC), включающие антигенный пептид
В дополнительном аспекте настоящее изобретение также относится к мультимеру пептид–MHC (pMHC), включающему антигенный пептид по настоящему изобретению.
Используемый в настоящем изобретении термин «мультимер пептид-MHC» (pMHC) относится к стабильному мультимерному комплексу, состоящему из белковых субъединиц основного комплекса гистосовместимости (MHC), нагруженных антигенным пептидом по изобретению. В целом, «мультимеры MHC» представляют собой олигомерные формы молекул MHC. Основной функцией молекулы MHC является связывание антигена. Согласно настоящему изобретению указанным антигеном является антигенный пептид по настоящему изобретению. Соответственно, комплекс белков МНС, «нагруженный» антигенным пептидом по изобретению, обычно означает, что антигенный пептид по изобретению связан с одним или несколькими белками MHC. «Мультимеры пептид–MHC» (pMHC) по изобретению включают димер, тример, тетрамер, пентамер, гексамер, гептамер или октамер пептид-MHC, но не ограничиваются ими. MHC тетрамеры и пентамеры являются предпочтительными. Термин «главный комплекс гистосовместимости (MHC)» - это общепринятое обозначение системы антигенов гистосовместимости, описанной для разных видов, включая антигены лейкоцитов человека (HLA). У людей существует три основных различных генетических локуса, которые кодируют молекулы MHC I класса: HLA-A, HLA-B, и HLA-C. HLA-A*01, HLA-A*02, и HLA-A*11 являются примерами аллелей различных MHC I класса, которые могут экспрессироваться с этих локусов.
Согласно одному варианту осуществления изобретения рMHC мультимер представляет собой мультимер пептида/молекулы MHC I класса. Согласно другому определенному варианту осуществления изобретения рMHC представляет собой мультимер HLA, соответствующего MHC I класса/пептида. Соответственно, pMHC мультимер может представлять собой мультимер HLA-пептид, выбранный из группы, включающей мультимер HLA-A-пептид, мультимер HLA-B-пептид, мультимер HLA-C-пептид, мультимер HLA-E-пептид, мультимер MICA-пептид и мультимер MICB-пептид.
Способы получения рMHC мультимеров хорошо известны из области техники и описаны, например, в WO96/26962 и WO01/18053, которые включены в настоящее описание в качестве ссылок.
Помимо молекулы MHC и антигенного пептида по изобретению, pMHC может включать другие компоненты, такие как агент для мультимеризации и/или метка (например, для визуализации). Примеры меток включают флуоресцентные метки, например, флуоресцентно-меченые белки, такие как стрептавидин, но не ограничиваются ими. Флуоресцентные метки включают аллофикоцианин (АРС), фикоэритрин (РЕ), R-фикоэритрин (R-PE) и флуоресцеин-изотиоцианат (FITC). Предпочтительной меткой является биотин.
В одном варианте осуществления изобретения указанный pMHC мультимер может применяться для визуализации популяции Т-клеток, которые являются специфичными для MHC I класса пептидного комплекса или комплексу HLA, соответствующий MHC I класса/пептид, как описано выше. Например, pMHC мультимер может представлять собой мультимер, в котором тяжелая цепь MHC является биотинилированной, что позволяет создавать тетрамерную комбинацию со стрептавидином. Такой рMHC тетрамер обладает повышенной авидностью по отношению к соответствующим TCR-несущим Т-лимфоцитам и, таким образом, может применяться для визуализации реактивных популяций с помощью иммунофлуоресценции. Согласно другому варианту осуществления изобретения указанный рMHC мультимер может применяться для детекции и/или выделения в скрининге (в проточной цитометрии или в иммуномагнитном скрининге) Т-клеточных популяций, специфичных для pMHC комплекса, как раскрыто в настоящем описании выше.
Специфичные к антигенному пептиду цитотоксические Т-лимфоциты (CTL)
В другом аспекте настоящее изобретение также обеспечивает цитотоксический Т-лимфоцит (CTL), специфичный для антигенного пептида согласно изобретению, в частности, активированный цитотоксический Т-лимфоцит (CTL), специфичный для антигенного пептида согласно изобретению.
Настоящее изобретение также обеспечивает способ получения цитотоксических Т-лимфоцитов (CTL), специфичных для антигенного пептида в соответствии с изобретением, в частности активированных цитотоксических Т-лимфоцитов (CTL), специфичных для антигенного пептида в соответствии с изобретением, включающий обеспечение контакта in vitro CTL с загруженной антигеном молекулой МНС I или II класса человека, экспрессируемой на поверхности антигенпрезентирующей клетки, или искусственной конструкцией, имитирующей антигенпрезентирующую клетку, где указанный антиген представляет собой антигенный пептид согласно изобретению. Предпочтительные антигенпрезентирующие клетки включают дендритные клетки. Искусственная конструкция, имитирующая антигенпрезентирующую клетку, может быть, например, мультимером пептид-MHC согласно изобретению. Стадия обеспечения контакта CTL с загруженной антигеном молекулой МНС человека класса I или II, экспрессируемой на поверхности антигенпрезентирующей клетки или искусственной конструкции, имитирующей антигенпрезентирующую клетку, может выполняться в течение периода времени, достаточного для активации указанного CTL антигенспецифичным образом. Предпочтительно, антигенный пептид представляет собой предпочтительный антигенный пептид, как описано выше, такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в любой из следующих SEQ ID № 65, 110, 114 и 220.
(Активированные) Т-клетки, которые направлены против антигенных пептидов по изобретению, полезны в терапии. В частности, активированные Т-клетки, которые получают вышеуказанным способом, избирательно распознают клетку, которая аберрантно экспрессирует полипептид, который содержит аминокислотную последовательность SEQ ID №№ 258-280 (т.е. опухолевый антиген), например, полипептид, который содержит аминокислотную последовательность, как указано в любой из следующих SEQ ID №№ 264, 270, 271 и 279.
Предпочтительно, (активированные) цитотоксические Т-лимфоциты (CTL) согласно настоящему изобретению, которые специфичны для антигенного пептида по изобретению, могут иметь (проявлять/экспрессировать) маркеры памяти. Такие маркеры памяти предпочтительно являются маркерами памяти кишечных клеток памяти, такими как CCR9, CXCR3, CD103, CX3CR1 и α4β7+.
(Активированные) цитотоксические Т-лимфоциты (CTL) согласно настоящему изобретению, которые специфичны для антигенного пептида по изобретению, предпочтительно в большей степени/сильнее амплифицируются после вакцинации антигенным пептидом по изобретению (полученным из последовательностей микробиоты человека) по сравнению с вакцинацией пептидами, не полученными из последовательностей микробиоты, такими как человеческая (эталонная) последовательность и/или синтетический пептид (например, включающий мутации, которые были, например, введены искусственно). Другими словами, вакцинация субъектов антигенным пептидом по изобретению предпочтительно увеличивает количество (активированных) цитотоксических Т-лимфоцитов (CTL) согласно настоящему изобретению, которые специфичны для указанного антигенного пептида по изобретению, в большей степени, чем вакцинация соответствующими человеческими пептидами или синтетическими пептидами (не полученными из микробиоты), которые относятся к одному и тому же эталонному эпитопу.
(Активированные) цитотоксические Т-лимфоциты (CTL) согласно настоящему изобретению, которые специфичны для антигенного пептида по изобретению, предпочтительно более/сильнее и/или быстрее амплифицируются после вакцинации у субъектов, имеющих указанный пептид в кишечнике (экспрессируемый микробиотой субъекта), например, пептид может быть обнаружен в образце кала субъекта, по сравнению с субъектами, не имеющими указанного пептида в кишечнике (не экспрессируемого микробиотой субъекта), например, субъектами, у которых указанный пептид не обнаруживается в образцах кала. В частности, субъекты, имеющие указанный пептид в кишечнике (экспрессируемый микробиотой субъекта), могут реагировать быстрее (проявлять более быструю экспансию Т-клеток) и/или иметь Т-клетки необходимого типа Tc1.
Клетки, нагруженные антигенным пептидом или иммуногенным соединением
Согласно дополнительному аспекту настоящее изобретение также относится к клеткам, нагруженным антигенным пептидом по настоящему изобретению или иммуногенным пептидом, включающим антигенный пептид по настоящему изобретению, как описано выше. В частности, предпочтительные варианты осуществления антигенного пептида, как описано выше, также применимы к таким клеткам по настоящему изобретению. Например, антигенный пептид, загруженный в клетку или включенный в иммуногенное соединение, загруженное в клетку, предпочтительно включает или состоит из консенсусной последовательности МНС I класса, как указано в любой из SEQ ID №№ 304-326, такой как антигенный пептид, включающий или состоящий из аминокислотной последовательности, как указано в любой из SEQ ID №№ 1-247 и 476-500. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 61, 65, 68, 72, 86, 110, 114 и 220, являются более предпочтительными. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 110, 114 и 220, являются более предпочтительными. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 65, 110, 114 и 220, являются наиболее предпочтительными. Также предпочтительными являются их комбинации, конкретнее - клетки, нагруженные разными антигенными пептидами по настоящему изобретению (или соответствующим иммуногенным соединением (соединениями)).
Предпочтительные клетки, нагруженные антигенным пептидом по настоящему изобретению или иммуногенным соединением по настоящему изобретению, представляют собой антигенпрезентирующие клетки (АРС), более предпочтительно дендритные клетки (DC).
АРС представляют наибольший интерес, поскольку их основная функция заключается в процессинге антигенов и презентации их на своей клеточной поверхности Т-клеткам иммунной системы таким образом, чтобы инициировать и модулировать Т-клеточные ответы in vivo. В контексте настоящего изобретения является предпочтительным, чтобы АПК были нагружены антигенным пептидом (пептидами) и/или иммунногенным соединением (соединениями) по настоящему изобретению. Это можно осуществить путем экспонирования АПК in vitro с указанным антигенным пептидом (пептидами) и/или иммуногенным соединением (соединениями) (как описано в Rizzo MM, Alaniz L, Mazzolini G. Ex vivo loading of autologous dendritic cells with tumor antigens. Methods Mol Biol. 2014; 1139:41-4; Rolinski J, Hus I. Breaking immunotolerance of tumors: a new perspective for dendritic cell therapy. J Immunotoxicol. 2014 Oct;11(4):311-8).
Предпочтительные АРС в соответствии с настоящим изобретением представляют собой дендритные клетки (DC). Действительно, может быть предпочтительно комбинировать по меньшей мере один антигенный пептид или иммуногенное соединение по настоящему изобретению с DC, поскольку они являются наиболее эффективными АРС и, как сообщалось, часто бывают функционально неполноценными у больных раком. DC могут быть легко выделены специалистом в данной области техники, как из организма здоровых совместимых доноров (то есть DC являются HLA-родственными), так и из организма самого пациента с условием, что они являются функциональными (то есть DC являются аутологичными), например, либо путем простого выделения из периферической крови, либо путем выведения из клеток периферической крови, таких как CD14+ моноциты или CD34+ гемопоэтические клетки-предшественники (Figdor CG, de Vries IJ, Lesterhuis WJ, Melief CJ. Dendritic cell immunotherapy: mapping the way. Nat Med. 2004 May; 10(5):475-80). DC действительно можно отличить от других клеток периферической крови по их поверхностным маркерам, таким как S100, p55, CD83 и/или OX62, и, таким образом, их можно выделить и очистить c помощью указанных маркеров с использованием методик культур клеток, хорошо известных в данной области техники.
Нуклеиновые кислоты, кодирующие антигенные пептиды, и клетки-хозяева, содержащие нуклеиновые кислоты
Согласно дополнительному аспекту настоящее изобретение также относится к нуклеиновой кислоте, кодирующей антигенный пептид по настоящему изобретению, полипептид формулы (I), как раскрыто выше, или иммуногенное соединение по настоящему изобретению, причем иммуногенное соединение представляет собой пептид или белок. В частности, предпочтительные варианты осуществления антигенного пептида, как описано выше, также применимы к нуклеиновым кислотам по настоящему изобретению. Например, антигенный пептид, кодируемый нуклеиновой кислотой, предпочтительно включает или состоит из консенсусной последовательности МНС I класса, как указано в любой из SEQ ID №№ 304-326, такой как антигенный пептид, включающий или состоящий из аминокислотной последовательности, указанной в любой из SEQ ID №№ 1-247 и 476-500. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 61, 65, 68, 72, 86, 110, 114 и 220, являются более предпочтительными. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 65, 110, 114 и 220, являются более предпочтительными. Также предпочтительными являются их комбинации, а именно, комбинации нуклеиновых кислот, кодирующих разные антигенные пептиды по изобретению.
Нуклеиновые кислоты предпочтительно представляют собой одноцепочечные, двухцепочечные или частично двухцепочечные нуклеиновые кислоты, предпочтительно выбранные из геномной ДНК, кДНК, РНК, антисмысловой ДНК, антисмысловой РНК, комплементарных РНК/ДНК последовательностей, как с элементами экспрессии, мини-генами, генными фрагментами, регуляторными элементами, промоторами, так и без них; а также их комбинации. Дополнительные предпочтительные примеры нуклеиновой кислоты (молекул) и/или полинуклеотидов включают, например, рекомбинантный полинуклеотид, вектор, олигонуклеотид, молекулу РНК, такую как рРНК, мРНК, или тРНК, или молекулу ДНК, как описано выше. Таким образом, предпочтительно нуклеиновая кислота (молекула) представляет собой молекулу ДНК или молекулу РНК; предпочтительно, выбранную из гДНК, кДНК, рРНК, мРНК; антисмысловой ДНК, антисмысловой РНК, комплементарных последовательностей РНК и/или ДНК, последовательностей РНК и/или ДНК с элементами экспрессии, регуляторными элементами и/или промоторами, или без них; вектор и их комбинации.
Возможность доставки нуклеиновой кислоты, например, рибонуклеиновой кислоты (РНК) внутрь клетки, in vitro, in vivo, in situ или ex vivo, с целью последующей внутриклеточной трансляции нуклеиновой кислоты и синтеза интересующего кодируемого пептида, представляет огромный интерес в сфере терапии, диагностики, реагентов и биологического анализа. Особое значение имеют доставка и функционирование неинтегративного полинуклеотида. Соответственно, предпочтительными являются нуклеиновые кислоты, которые не встраиваются в хромосомы хозяина, такие как мРНК. В целом нуклеиновые кислоты, такие как мРНК, могут быть оптимизированы для экспрессии антигенного пептида по изобретению, например, с помощью методов, известных из области техники, таких как оптимизация кодонов. Кроме того, нуклеиновая кислота может быть модифицирована, например, с целью повышения стабильности, увеличения продолжительности жизни и/или усиления экспрессии антигенного пептида по изобретению. Соответственно, оптимизированная или модифицированная мРНК (ммРНК), которая кодирует антигенный пептид по настоящему изобретению, является предпочтительной. ммРНК отличаются от мРНК дикого типа своими функциональными и/или структурными характеристиками, касающимися оптимальной доставки мРНК и/или оптимальной экспрессии антигенного пептида по изобретению (например, как раскрыто в WO 2013/151672 A2, WO 2013/101690 A1, WO2013/052523 A, которые включены в настоящее описание в качестве ссылок). В целом, нуклеиновые кислоты могут доставляться «депротеинизированными» или вместе с носителем, например, катионным носителем. Катионные носители (положительно заряженные) обычно легко соединяются с нуклеиновыми кислотами, которые заряжены отрицательно. Носитель может быть любого типа, включая, например, полимеры, белки, липиды и наночастицы. Катионные липиды и наночастицы (в частности, липидные наночастицы, LNP) являются предпочтительными для доставки нуклеиновых кислот. Таким образом, настоящее изобретение также относится к нуклеиновым кислотам, как раскрыто в настоящем описании, связанным с носителем (например, липидом, в частности с катионным липидом или LNP).
Согласно некоторым вариантам осуществления изобретения, нуклеиновокислотная молекула может представлять собой вектор. Термин «вектор», используемый в контексте настоящего изобретения, относится к нуклеиновокислотной молекуле, предпочтительно к искусственной нуклеиновокислотной молекуле, то есть к нуклеиновокислотной молекуле, которой не существует в природе. Вектор в контексте настоящего изобретения подходит для встраивания и сохранения необходимой нуклеиновокислотной последовательности. Такие векторы могут представлять собой векторы хранения, векторы экспрессии, клонирующие векторы, векторы переноса и т.д. Вектор хранения представляет собой вектор, который обеспечивает надлежащее хранение нуклеиновокислотной молекулы. Таким образом, вектор может включать последовательность, соответствующую, например, необходимому антигенному пептиду по настоящему изобретению. Вектор экспрессии может применяться для получения продуктов экспрессии, таких как РНК, например, мРНК или пептидов, полипептидов или белков. Например, вектор экспрессии может включать последовательности, необходимые для транскрипции удлинения последовательности вектора, например, промоторную последовательность. Клонирующий вектор обычно представляет собой вектор, который включает сайт клонирования, который может применяться для встраивания последовательностей нуклеиновых кислот в вектор. Клонирующий вектор может представлять собой, например, плазмидный вектор или бактериофаговый вектор. Вектор переноса может представлять собой вектор, подходящий для переноса молекул нуклеиновых кислот в клетки или организмы, например, вирусный вектор. Вектор в контексте настоящего изобретения может представлять собой, например, РНК вектор или ДНК вектор. Предпочтительно, вектор представляет собой молекулу ДНК. Например, вектор в рамках настоящей заявки включает сайт клонирования, селекционный маркер, такой как фактор устойчивости к антибиотикам, а также последовательность, обеспечивающую размножение вектора, такую как точка начала репликации. Предпочтительно, вектор в контексте настоящей заявки представляет собой плазмидный вектор. Предпочтительный вектор представляет собой вектор для экспрессии в бактериальных клетках. Более предпочтительно, вектор полезен для экспрессии в так называемых «живых бактериальных вакцинных векторах», в которых живые бактериальные клетки (такие как бактерии или их споры, например, эндоспоры, экзоспоры или микробные цисты) могут выполнять функцию вакцин. Предпочтительные примеры таких векторов описаны в da Silva et al., Live bacterial vaccine vectors: an overview; Braz J Microbiol. 2015 Mar 4; 45(4):1117-29.
Нуклеиновые кислоты, кодирующие антигенные пептиды по настоящему изобретению, могут быть в форме депротеинизированных нуклеиновых кислот или нуклеиновых кислот, клонированных в плазмиды или вирусные векторы (Tregoning and Kinnear, Using Plasmids as DNA Vaccines for Infectious Diseases. Microbiol Spectr. 2014 Dec; 2(6). doi: 10.1128/microbiolspec.PLAS-0028-2014), последние являются особо предпочтительными. Примеры подходящих вирусных векторов по настоящему изобретению включают ретровирусные, аденовирусные, аденоассоциированные вирусные векторы (AAV), герпесвирусные и поксвирусные векторы, но не ограничиваются ими. В компетенции специалиста в данной области техники находится клонирование нуклеиновой кислоты в плазмидном или вирусном векторе с применением стандартных рекомбинантных технологий.
Согласно дополнительному аспекту, настоящее изобретение также относится к клетке-хозяину, включающей нуклеиновую кислоту по настоящему изобретению. Также предпочтительными являются их комбинации, в частности клетки-хозяева, включающие различные нуклеиновые кислоты по настоящему изобретению, например, кодирующие различные антигенные пептиды по настоящему изобретению.
Предпочтительно, нуклеиновые кислоты, заключенные в клетке-хозяине, представляют собой вектор. Предпочтительно, клетка-хозяин представляет собой бактериальную клетку. Такая клетка-хозяин предпочтительно может быть применена для получения антигенного пептида по настоящему изобретению или иммуногенного пептида по настоящему изобретению. Более того, такая клетка-хозяин также может быть активным компонентом вакцины.
Предпочтительно, клетка-хозяин представляет собой бактериальную клетку, более предпочтительно, кишечную бактериальную клетку. Термин «кишечная бактериальная клетка» относится к бактериям, живущим в кишечнике (человека).
Такая бактериальная клетка-хозяин может выполнять роль «живого бактериального вакцинного вектора», причем живые бактериальные клетки (такие как бактерии или бактериальные споры, например, эндоспоры, экзоспоры или микробные цисты) могут служить в качестве вакцин. Предпочтительные примеры таких клеток описаны в da Silva et al., Live bacterial vaccine vectors: an overview; Braz J Microbiol. 2015 Mar 4;45(4):1117-29.
Бактериальные клетки (такие как бактерии или бактериальные споры, например, эндоспоры, экзоспоры или микробные цисты), в частности, виды бактерий, живущие в кишечнике (на всем протяжении), могут быть предпочтительными, поскольку обладают способностью к индукции более сильного иммунного ответа, чем (поли)пептиды или нуклеиновые кислоты, которые они содержат.
Альтернативно, бактериальные клетки, в частности, клетки кишечных бактерий по настоящему изобретению могут находиться в форме пробиотиков, то есть в форме живых кишечных бактерий, которые, таким образом, могут применяться в качестве пищевой добавки благодаря благоприятному влиянию на здоровье. Их можно, например, лиофилизировать в гранулы, пилюли или капсулы, или непосредственно смешивать с молочными продуктами перед употреблением.
Наночастицы, включающие антигенный пептид или иммуногенное соединение
Согласно дополнительному аспекту, настоящее изобретение также относится к наночастице, включающей, в частности, наночастицу, нагруженную:
- по меньшей мере одним антигенным пептидом по настоящему изобретению, или
- по меньшей мере одним иммуногенным соединением по настоящему изобретению;
и при необходимости, адъювантом.
В частности, предпочтительные варианты осуществления антигенного пептида, как раскрыто в настоящей заявке, также применимы для таких наночастиц по настоящему изобретению. Например, антигенный пептид, нагруженный на наночастицу или включенный в иммуногенное соединение, нагруженное на наночастицу, предпочтительно содержит или состоит из консенсусной последовательности МНС I класса, как указано в любой из SEQ ID №№ 304-326, такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в любой из SEQ ID №№ 1-247 и 476-500. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 61, 65, 68, 72, 86, 110, 114 и 220, являются более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№s 10, 21, 33, 35, 39, 40, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, включающие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 65, 110, 114 и 220, являются более предпочтительными. Также предпочтительными являются их комбинации, в частности наночастицы, нагруженные различными антигенными пептидами по настоящему изобретению (или соответствующим иммуногенным соединением (соединениями).
Наночастицы, в частности, предназначенные для применения в качестве вакцин, известны из области техники и описаны, например, в Shao et al., Nanoparticle-based immunotherapy for cancer, ACS Nano 2015, 9(1):16-30; Zhao et al., Nanoparticle vaccines, Vaccine 2014, 32(3):327-37; и Gregory et al., Vaccine delivery using nanoparticles, Front Cell Infect Microbiol. 2013, 3:13, doi: 10.3389/fcimb.2013.00013. eCollection 2013, Review. В частности, наночастица применяется для доставки антигенного пептида (или иммуногенного соединения/полипептида/белка/нуклеиновой кислоты, включающей антигенный пептид) и может при необходимости выполнять роль адъюванта. Антигенный пептид (иммуногенное соединение/полипептид/белок/нуклеиновая кислота, включающая антигенный пептид) обычно либо инкапсулирован в наночастицу, либо связан/присоединен (выложен на поверхности) к поверхности наночастицы («покрытие»). По сравнению с традиционными подходами, наночастицы могут защищать загруженное вещество (антиген/адъювант) от биологического окружения, увеличивают продолжительность жизни, минимизируют системное токсическое действие, усиливают доставку к АРС или даже напрямую запускают активацию TAA-специфических Т-клеток. Предпочтительно наночастицы имеют размер (диаметр) не выше 300 нм, более предпочтительно не выше 200 нм, и еще более предпочтительно не выше 100 нм. Такие наночастицы адекватным образом защищены от поглощения фагоцитами, обладают хорошей структурной целостностью в кровотоке и длительным временем циркуляции, способны накапливаться в местах роста опухоли и глубоко проникать в опухолевую ткань.
Примеры наночастицвключают полимерные наночастицы, такие как поли(этиленгликоль) (ПЭГ) и поли(D,L-лактид-ко-гликолевая кислота) (PLGA); неорганические наночастицы, такие как золотые наночастицы, железооксидные гранулы, железоксидные и цинкоксидные наночастицы, карбоновые нанотрубки и мезопористые кремниевые наночастицы; липосомы; такие как катионные липосомы; иммуностимулирующие комплексы (ISCOM); вирусподобные частицы (VLP); и самособирающиеся белки.
Полимерные наночастицы представляют собой наночастицы на основе полимеров/или содержащие их, такие как поли(D,L-лактид-ко-гликолид) (PLG), поли(D,L-лактид-ко-гликолевая кислота) (PLGA), поли(γ-глутаминовая кислота) (γ-PGA), поли(этиленгликоль) (PEG), и полистирол. Полимерные наночастицы могут захватывать антиген (например, антигенный пептид или (поли)пептид, включающий его) или связываться с антигеном или конъюгироваться с ним (например, с антигенным пептидом или (поли)пептидом, включающим его). Полимерные наночастицы могут применяться для доставки, например, в определенные клетки, или для замедленного высвобождения антигена благодаря медленной скорости их биодеградации. Например, g-PGA наночастицы могут применяться для инкапсулирования гидрофобных антигенов. Полистироловые наночастицы могут формировать конъюгаты с разнообразными антигенами, поскольку могут быть поверхностно-модифицированными при помощи различных функциональных групп. Полимеры, такие как поли(L-молочная кислота) (PLA), PLGA, PEG и натуральные полимеры, такие как полисахариды, также могут применяться для синтеза гидрогелевых наночастиц, которые представляют собой тип гидрофильных трехмерных полимерных наночастиц. Наногели имеют благоприятные свойства, включая гибкий размер ячеек, большую поверхность для многовалентной конъюгации, высокое содержание воды и также высокую нагрузочную способность для антигенов. Соответственно, предпочтительной наночастицей является наногель, такой как хитозановый наногель. Предпочтительными полимерными наночастицами являются наночастицы на основе PEG и PLGA, или содержащие их.
Неорганические наночастицы представляют собой наночастицы на основе неорганических соединений или включающие их; примерами таких наночастиц являются золотые наночастицы, железооксидные наночастицы, железооксидные-цинкооксидные наночастицы, углеродные наноастицы (например, углеродные нанотрубки) и мезопористые кремниевые наночастицы. Неорганические наночастицы обеспечивают жесткую структуру и контролируемый синтез. Например, золотые наночастицы могут быть синтезированы в виде различных форм, таких как сферы, палочки, кубы. Неорганические наночастицы могут быть поверхностно-модифицированными, например, с помощью углеводов. Углеродные наночастицы обеспечивают хорошую биосовместимость и могут быть получены, например, в виде нанотрубок или (микроспорических) сфер. Например, множественные копии антигенного пептида по настоящему изобретению (или (поли(пептид), включающий его) могут быть конъюгированы с углеродными наночастицами, например, углеродными нанотрубками. Мезопористые углеродные наночастицы являются предпочтительными для перорального применения. Наночастицы на основе кремния (SiNP) также являются предпочтительными. SiNP являются биосовместимыми и показывают прекрасные свойства в отношении селективного опухолевого таргетинга и доставки вакцин. Большое количество силанольных групп на поверхности SiNP может быть добавлено для дополнительной модификации и придания частице дополнительной функциональности, такой как способность распознавать клетки, абсорбция специфических биомолекул, улучшение взаимодействия с клетками, а также усиление захвата клетками. Мезопорические кварцевые наночастицы являются наиболее предпочтительными.
Липосомы обычно формируются фосфолипидами, такими как 1,2-диолеоил-3-триметиламмоний пропан (DOTAP). В целом катионные липосомы являются предпочтительными. Липосомы самостоятельно собираются из фосфолипидной двухслойной оболочки и водного ядра. Липосомы могут быть получены в форме униламеллярных везикул (имеющих один фосфолипидный бислой) или в форме мультиламеллярных везикул (имеющих несколько концентрических фосфолипидных слоев, разделенных слоями воды). Соответственно, антигены могут быть инкапсулированы в ядро или заключены между различными слоями/оболочками. Предпочтительные липосомные системы, одобренные для применения у человека, включают системы Inflexal® V и Epaxal®.
Иммуностимулирующие комплексы (ISCOM) являются частицами, напоминающими корзину, приблизительно 40 нм (в диаметре), представляющими собой коллоидные содержащие сапонин мицеллы, например, сформированные из сапонинового адъюванта Quil-A, холестерина, фосфолипидов и (поли)пептидного антигена (такого как антигенный пептид или полипептид, включающий его). Такие сферические частицы могут захватывать антигены за счет полярных взаимодействий. Описано два типа ISCOM, оба из которых включают холестерин, фосфолипид (обычно или фосфатидилэтаноламин, или фосфатидилхолин) и сапонин (такой как Quil-A).
Вирусоподобные частицы (VLP) представляют собой самособираемые наночастицы, формирующиеся в процессе самосборки биосовместимых капсидных белков. Благодаря оптимизированному естественным образом размеру частиц и повторяющемуся характеру структуры, вирусоподобные частицы могут вызывать сильный иммунный ответ. Вирусоподобные частицы могут быть получены на основе различных вирусов, размер которых варьирует от 20 нм до 800 нм и обычно находится в пределах 20-150 нм. Вирусоподобные частицы могут быть сконструированы для экспрессии дополнительных пептидов или белков либо путем слияния этих пептидов/ белков с частицей, либо путем экспрессии нескольких антигенов. Кроме того, антигены могут быть химически связаны с вирусной поверхностью с образованием биоконъюгата VLP.
Примеры самособирающихся белков включают ферритин и основной белок свода (MVP – абрревиатура от англ. Main Vault Protein). Ферритин представляет собой белок, который может самостоятельно собираться в напоминающую сферу структуру размером 10 нм. Девяносто шесть единиц MVP могут самостоятельно собираться в бочкообразную сводчатую наночастицу размером приблизительно 40 нм в ширину и 70 нм в длину. Антигены, генетически слитые с доменом минимального взаимодействия, могут быть упакованы в сводчатые наночастицы посредством процесса самосборки при смешивании с MVP. Соответственно, антиген (такой как антигенный пептид по-настоящему изобретению или полипептид, содержащий его) может быть слит с самособирающимся белком или его фрагментом/доменом, таким как домен минимального взаимодействия MVP. Соответственно, настоящее изобретение относится к гибридному белку, включающему самособирающийся белок (или его фрагмент/домен) и антигенный пептид по настоящему изобретению.
В целом предпочтительными примерами наночастиц (NP) являются железооксидные частицы, полистироловые микросферы, наночастицы на основе поли(γ-глутаминовой кислоты) (γ-PGA), железоксидные-цинкоксидные наночастицы, катионизированные желатиновые наночастицы, стабилизированные плюроником наночастицы на основе поли(пропиленсульфида) (PPS), PLGA NP, (катионные) липосомы, (рН-зависимые) полимерные мицеллы, PLGA, PLGA с покрытием из мембраны раковых клеток, липидно-кальциево-фосфатные наночастицы (LCP), липосомальные наночастицы на основе протамина и гиалуроновой кислоты (LPN), полистироловые латексные гранулы, магнитные гранулы, железо-декстрановые частицы и нанокристаллы - квантовые точки.
Предпочтительно, наночастицы дополнительно включают адъювант, например, агонист толл-подобного рецептора (TLR). Таким образом, антигенный пептид (или иммуногенное соединение/полипептид/белок/нуклеиновая кислота, включающие антигенный пептид) могут доставляться к антигенпрезентирующим клеткам (АРС), таким как дендритные клетки (DC) вместе с адъювантом. Адъювант может быть инкапсулирован в наночастицу или связан/конъюгирован с ее поверхностью, предпочтительно, аналогичным образом, что и антигенный пептид.
Наиболее предпочтительными адъювантами являются полинозиновая:полицитидиловая кислота (также обозначаемая как «поли I:C») и/или ее производное поли-ICLC. Поли I:C представляет собой несопряженную двухцепочечную РНК, в которой одна цепь представляет собой полимер инозиновой кислоты, а другая – полимер цитидиловой кислоты. Поли I:C представляет собой иммуностимулирующий агент, который, как известно, взаимодействует с толл-подобным рецептором (TLR3). Поли I:C структурно схожа с двухцепочечной РНК, которая является «натуральным» стимулятором TLR3. Соответственно, поли I:C может считаться синтетическим аналогом двухцепочечной РНК. Поли-ICLC представляет собой синтетический комплекс карбоксиметилцеллюлозы, полиинозиновой-полицитидиловой кислоты и поли-L-лизин двухцепочечной РНК. Аналогично поли I:C, поли-ICLC представляет собой лиганд для TLR3. Поли I:C и поли-ICLC обычно стимулируют высвобождение цитотоксических цитокинов. Предпочтительным примером поли-ICLC является Hiltonol®.
Фармацевтические композиции
В дополнительном аспекте настоящее изобретение также относится к фармацевтической композиции, включающей по меньшей мере одно из следующего:
- антигенный пептид по настоящему изобретению, как раскрыто в настоящем описании,
- иммуногенное соединение по настоящему изобретению, как раскрыто в настоящем описании,
- наночастицу по настоящему изобретению, как раскрыто в настоящем описании,
- клетку по настоящему изобретению, как раскрыто в настоящем описании,
- нуклеиновую кислоту по настоящему изобретению, как раскрыто в настоящем описании,
- клетку-хозяин по настоящему изобретению, как раскрыто в настоящем описании, и/или
- цитотоксический Т-лимфоцит по настоящему изобретению, как раскрыто в настоящем описании,
и при необходимости один или несколько фармацевтически приемлемых наполнителей или носителей.
Соответственно, настоящее изобретение обеспечивает фармацевтическую композицию, содержащую (по меньшей мере) один антигенный пептид по настоящему изобретению, как описано в настоящей заявке. Кроме того, настоящее изобретение также обеспечивает фармацевтическую композицию, содержащую (по меньшей мере) одно иммуногенное соединение по настоящему изобретению, как описано в настоящей заявке. Кроме того, настоящее изобретение также обеспечивает фармацевтическую композицию, содержащую (по меньшей мере) одну наночастицу по настоящему изобретению, как описано в настоящей заявке. Далее, настоящее изобретение также обеспечивает фармацевтическую композицию, содержащую (по меньшей мере) одну клетку по настоящему изобретению, как описано в настоящей заявке. Далее, настоящее изобретение также обеспечивает фармацевтическую композицию, содержащую (по меньшей мере) одну нуклеиновую кислоту по настоящему изобретению, как описано в настоящей заявке. Далее, настоящее изобретение также обеспечивает фармацевтическую композицию, содержащую (по меньшей мере) одну клетку-хозяина по настоящему изобретению, как описано в настоящей заявке. Далее, настоящее изобретение также обеспечивает фармацевтическую композицию, содержащую (по меньшей мере) один цитотоксический Т-лимфоцит по настоящему изобретению, как описано в настоящей заявке.
В частности, предпочтительные варианты осуществления антигенного пептида, как описано выше, также применимы к таким фармацевтическим композициям по настоящему изобретению. Например, антигенный пептид в составе фармацевтической композиции или антигенный пептид в составе иммуногенного соединения, наночастицы, клетки, нуклеиновой кислоты или клетки-хозяина, включенной в фармацевтическую композицию, предпочтительно содержит или состоит из аминокислотной последовательности, как указано в любой из SEQ ID №№ 1-247 и 476-500. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 61, 65, 68, 72, 86, 110, 114 и 220, являются более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 65, 110, 114 и 220, являются еще более предпочтительными.
Кроме того, предпочтительными являются их комбинации, в частности, фармацевтические композиции, содержащие различные антигенные пептиды по настоящему изобретению. Например, фармацевтическая композиция может содержать:
(i) по меньшей мере два различных антигенных пептида по настоящему изобретению;
(ii) по меньшей мере два различных иммуногенных соединения по настоящему изобретению;
(iii) по меньшей мере две различные наночастицы по настоящему изобретению;
(iv) по меньшей мере две различные нуклеиновые кислоты по настоящему изобретению; и/или
(v) по меньшей мере два различных цитотоксических Т-лимфоцита по настоящему изобретению.
Соответственно, фармацевтическая композиция может содержать по меньшей мере «два отдельных компонента» (фармацевтической композиции по настоящему изобретению), предпочтительно три или четыре отдельных компонента. Как правило, выражение «отдельные компоненты», используемое в настоящей заявке, означает:
(1) первый компонент, такой как антигенный пептид по настоящему изобретению, как описано в настоящей заявке, иммуногенное соединение по настоящему изобретению, как описано в настоящей заявке, наночастица по настоящему изобретению, как описано в настоящей заявке, клетка по настоящему изобретению, как описано в настоящей заявке, нуклеиновая кислота по настоящему изобретению, как описано в настоящей заявке, клетка-хозяин по настоящему изобретению, как описано в настоящей заявке, или цитотоксический Т-лимфоцит по настоящему изобретению, как описано в настоящей заявке; и
(2) по меньшей мере один другой компонент (который отличается от первого компонента, тогда как в случае более чем двух отдельных компонентов каждый компонент отличается от другого), такой как противораковый терапевтический агент, как описано выше, отдельный антигенный пептид по настоящему изобретению, как описано в настоящей заявке, отдельное иммуногенное соединение по настоящему изобретению, как описано в настоящей заявке, отдельную наночастицу по настоящему изобретению, как описано в настоящей заявке, отдельную клетку по настоящему изобретению, как описано в настоящей заявке, отдельную нуклеиновую кислоту по настоящему изобретению, как описано в настоящей заявке, отдельную клетку-хозяин по настоящему изобретению, как описано в настоящей заявке, отдельный цитотоксический Т-лимфоцит по настоящему изобретению, как описано в настоящей заявке, или один или несколько (фрагментов) опухолевых антигенов человека в любой форме («изолированных», в виде иммуногенного соединения, как описано в настоящей заявке, в виде наночастицы, как описано в настоящей заявке, в виде клетки (хозяина), как описано в настоящей заявке, или в виде нуклеиновой кислоты, как описано в настоящей заявке).
Соответственно, «отдельные компоненты» предпочтительно являются активными компонентами (как описано выше) в контексте заболевания (В-клеточного гемобластоза), которое необходимо предотвратить и/или лечить. Другими словами, каждый из отдельных компонентов также может быть полезен для профилактики и/или лечения указанного рака, если его вводить отдельно (не в комбинации, как описано в настоящей заявке), хотя комбинация (т.е. комбинированное введение) обычно усиливает их профилактическое и/или терапевтическое действие (такое как иммунный ответ), предпочтительно синергическим образом.
Предпочтительно «различные компоненты» относятся к одному и тому же типу (например, разные антигенные пептиды, разные иммуногенные соединения, разные наночастицы, разные клетки, разные нуклеиновые кислоты, разные клетки-хозяева или разные цитотоксические Т-лимфоциты) и отличаются друг от друга только в том, что они относятся к различным антигенным пептидам по настоящему изобретению, как описано в настоящей заявке.
Например, по меньшей мере три или четыре различных активных компонента предпочтительно относятся к одному типу, но отличаются (только) тем, что каждый из них относится к отдельному антигенному пептиду. Более предпочтительно:
- первый компонент относится к антигенному пептиду, содержащему или состоящему из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD22;
- (отдельный) второй компонент относится к антигенному пептиду, содержащему или состоящему из варианта последовательности (микробиоты) фрагмента опухолевого антигена CD37 человека;
- (отдельный) третий компонент относится к антигенному пептиду, включающему или состоящему из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека TNFRSF13C; и
- при необходимости, (отдельный) четвертый компонент относится к антигенному пептиду, содержащему или состоящему из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека MS4A1 (CD20).
Еще более предпочтительно:
- первый компонент относится к антигенному пептиду, содержащему или состоящему из варианта последовательности (микробиоты) SEQ ID № 270;
- (отдельный) второй компонент относится к антигенному пептиду, содержащему или состоящему из варианта последовательности (микробиоты) SEQ ID № 271;
- (отдельный) третий компонент относится к антигенному пептиду, содержащему или состоящему из варианта последовательности (микробиоты) SEQ ID № 279; и
- при необходимости, (отдельный) четвертый компонент относится к антигенному пептиду, содержащему или состоящему из варианта последовательности (микробиоты) SEQ ID № 264.
Еще более предпочтительно:
- первый компонент относится к антигенному пептиду, содержащему или состоящему из аминокислотной последовательности, указанной в SEQ ID № 110;
- (отдельный) второй компонент относится к антигенному пептиду, содержащему или состоящему из аминокислотной последовательности, указанной в SEQ ID № 114;
- (отдельный) третий компонент относится к антигенному пептиду, содержащему или состоящему из аминокислотной последовательности, указанной в SEQ ID № 220; и
- при необходимости, (отдельный) четвертый компонент относится к антигенному пептиду, содержащему или состоящему из аминокислотной последовательности, указанной в SEQ ID № 65.
Предпочтительно, фармацевтическая композиция содержит по меньшей мере два различных антигенных пептида по настоящему изобретению.
Предпочтительно, фармацевтическая композиция включает первый антигенный пептид по настоящему изобретению, который содержит вариант последовательности (микробиоты) фрагмента опухолевого антигена человека CD22 или состоит из него, и второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека TNFRSF13C. Предпочтительно, первый антигенный пептид содержит или состоит из варианта последовательности (микробиоты) фрагмента CD22 (эталонного пептида человека) «WVFEHPETL» (SEQ ID № 270), такого как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 106-110, 316 и 387-390 и 485-488, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в любой из SEQ ID № 107, 108, 109 и 110, а второй антигенный пептид содержит или состоит из варианта последовательности (микробиоты) фрагмента TNFRSF13C (эталонного пептида человека) «LLFGAPALL» (SEQ ID № 279), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 210-225, 325 и 447-455 и 494-500, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в любой из SEQ ID №№ 212, 217, 220 и 224, в частности, как указано в SEQ ID №№ 220, 325 и 450. Более предпочтительно, первый антигенный пептид содержит или состоит из аминокислотной последовательности, указанной в SEQ ID №№ 110, 387 и 390, а второй антигенный пептид содержит или состоит из аминокислотной последовательности, указанной в SEQ ID №№ 220 и 450. Еще более предпочтительно фармацевтическая композиция содержит антигенный пептид, содержащий или состоящий из SEQ ID № 110, и антигенный пептид, содержащий или состоящий из SEQ ID № 220.
Более предпочтительно, фармацевтическая композиция содержит по меньшей мере три различных антигенных пептида по настоящему изобретению.
В частности, фармацевтическая композиция может содержать первый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD22, второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека TNFRSF13C, и третьего антигенного пептида по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD37. Предпочтительно фармацевтическая композиция включает первый антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD22 (эталонного пептида человека) «WVFEHPETL» (SEQ ID № 270), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 106-110, 316 и 387-390 и 485-488, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в любой из SEQ ID №№ 107, 108, 109 и 110; второй антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента TNFRSF13C (эталонного пептида человека) «LLFGAPALL» (SEQ ID № 279), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 210-225, 325 и 447-455 и 494-500, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в любой из SEQ ID №№ 212, 217, 220 и 224, в частности, как указано в SEQ ID №№ 220, 325 и 450; и третий антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD37 (эталонного пептида человека) «GLAFVPLQI» (SEQ ID № 271), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 111-130, 317, 391-402 и 489-493, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в любой из SEQ ID №№ 114, 117, 119, 120, 491 и 493, в частности, как указано в SEQ ID №№ 113-116, 324 и 392-393.
Еще более предпочтительно, фармацевтическая композиция содержит по меньшей мере четыре различных антигенных пептида по настоящему изобретению.
В частности, фармацевтическая композиция может включать первый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD22, второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека TNFRSF13C, третий антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD37, четвертый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD19 или CD20. Предпочтительно фармацевтическая композиция включает первый антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD22 (эталонного пептида человека) «WVFEHPETL» (SEQ ID № 270), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 106-110, 316 и 387-390 и 485-488, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в любой из SEQ ID №№ 107, 108, 109 и 110; второй антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента TNFRSF13C (эталонного пептида человека) «LLFGAPALL» (SEQ ID № 279), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 210-225, 325 и 447-455 и 494-500, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в любой из SEQ ID №№ 212, 217, 220 и 224, в частности, как указано в SEQ ID №№ 220, 325 и 450; третий антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD37 (эталонного пептида человека) «GLAFVPLQI» (SEQ ID № 271), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 111-130, 317 и 391-402 и 489-493, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в любой из SEQ ID №№ 114, 117, 119, 120, 491 и 493, в частности, как указано в SEQ ID №№ 113-116, 324 и 392-393; и четвертый антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD20 (MS4A1) (эталонного пептида человека) «IMNSLSLFA» (SEQ ID № 264), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 65-70, 310 и 361-364 и 476-484, например, антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в любой из SEQ ID №№ 65, 68, 70 и 477.
Предпочтительно, фармацевтическая композиция включает:
- первый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD22;
- второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека TNFRSF13C;
- третий антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD37; и
- при необходимости, четвертый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD20.
Более предпочтительно, фармацевтическая композиция включает:
- первый антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD22 (эталонного пептида человека) «WVFEHPETL» (SEQ ID № 270), такого как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 106-110, 316 и 387-390;
- второй антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента TNFRSF13C (эталонного пептида человека) «LLFGAPALL» (SEQ ID № 279), такого как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID № 220, 325 и 450;
- третий антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD37 (эталонного пептида человека) «GLAFVPLQI» (SEQ ID № 271), такого как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID № 113-116, 324 и 392-393; и
- при необходимости, четвертый антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD20 (эталонного пептида человека) «IMNSLSLFA» (SEQ ID № 264), такого как антигенный пептид, содержащий или состоящий из аминокислотной последовательности как указано в SEQ ID №№ 65-70, 310 и 361-364.
Еще более предпочтительно, фармацевтическая композиция содержит антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, указанной в SEQ ID №№ 110, 114, 220 и, при необходимости, 65.
В некоторых вариантах осуществления фармацевтическая композиция не содержит дополнительных антигенных пептидов (в дополнение к антигенным пептидам по изобретению, как описано выше).
Понятно, что фармацевтическая композиция может также содержать вместо вышеописанных предпочтительных комбинаций антигенных пептидов соответствующую комбинацию иммуногенных соединений по изобретению, соответствующую комбинацию наночастиц по изобретению или соответствующую комбинацию нуклеиновых кислот по изобретению.
Предпочтительно, фармацевтическая композиция дополнительно содержит один или несколько фармацевтически приемлемых наполнителей или носителей.
Фармацевтическая композиция по изобретению может находиться в любой форме, подходящей для осуществления целей настоящего изобретения. Например, указанная композиция может находиться в форме, подходящей для парентерального, энтерального или местного введения, такой как жидкая суспензия, твердая лекарственная форма (гранулы, пилюли, капсулы или таблетки), или же паста или гель. Выбор подходящей формы композиции в зависимости от предполагаемой области применения находится в компетенции специалиста в данной области техники.
Композиция по настоящему изобретению может дополнительно содержать другие активные агенты, например, такие, которые могут усиливать эффекты антигенного пептида или иммуногенного соединения. Альтернативно, композиция может не содержать других активных агентов (то есть, отличных от антигенного пептида по настоящему изобретению, иммуногенного соединения по настоящему изобретению, наночастицы по настоящему изобретению, клетки по настоящему изобретению, и/или клетки-хозяина по настоящему изобретению).
Фармацевтическая композиция, как определено в настоящем описании, предпочтительно представляет собой иммуногенную композицию, то есть композицию, которая способна индуцировать, усиливать, пролонгировать и поддерживать иммунный ответ. Это может достигаться за счет антигенного пептида по настоящему изобретению или за счет иммуногенного соединения по настоящему изобретению, находящихся в составе указанной композиции. Предпочтительно, фармацевтическая композиция дополнительно содержит одно или несколько иммуноадъювантных соединений. Фармацевтическая композиция, конкретно, иммуногенная композиция, может в настоящем описании также обозначаться термином «вакцинная композиция».
Предпочтительно, фармацевтическая композиция также содержит по меньшей мере один иммуностимулирующий агент, в частности, способный усиливать, потенциировать, пролонгировать и поддерживать иммунный ответ, опосредованный антигенным пептидом. Предпочтительные иммуностимулирующие агенты по настоящему изобретению включают иммуноадъюванты, антигенпрезентирующие клетки, а также их комбинации, но не ограничиваются ими. Предпочтительно, иммуностимулирующий агент представляет собой иммуноадъювант или антигенпрезентирующую клетку (АРС).
Предпочтительно, иммуностимулирующий агент представляет собой иммуноадъювант. Некоторые иммуноадъюванты обладают способностью помогать и пролонгировать взаимодействие между антигеном и иммунной системой, в то время как другие способны к рекрутингу или активации клеток естественного иммунитета и таким образом индуцируют адаптивный иммунный ответ. Адъюванты, принадлежащие к первой категории, включают минеральные соединения, такие как сульфат алюминия, гидроксид алюминия, фосфат алюминия, кальций фосфат гидроксид; и масляные эмульсии, такие как парафиновое масло, растительное масло, полный/неполный адъювант Фрейнда (FCA/FIA), сапонины (например, из таких растений, как квиллайя, соя, истод сенега), но не ограничиваются ими. Адъюванты, принадлежащие к последней категории, включают, без ограничений указанными, иммуностимулирующие комплексы (ISCOM), такие как цитокины (например, GM-CSF; интерлейкины, такие как IL-1, IL-2, IL6, IL8, или IL12; факторы некроза опухолей (TNF), такие как TNFα или TNFβ; интерфероны (IFN), такие как IFNα, IFNβ, IFNγ или IFNδ, и т.д.); лиганды толл-подобных рецепторов (TLR), такие как имиквимод, резикмивод или MPL; экзосомы, такие как экзосомы, выделенные из дендритных клеток (DC) или из опухолевых клеток; бактериальные продукты, такие как белки теплового шока (HSP), такие как gp96, hsp90, hsp70, кальретикулин, hsp110, hsp170), патоген-ассоциированные молекулярные паттерны (PAMP), димиколат трегалозы, (TDM), мурамилдипептид (MDP), полисахариды (PLS), такие как полисахарид-К.
Более предпочтительно, иммуноадъювант представляет собой белок/пептид, обладающий иммуноадъювантными свойствами, такими как обеспечение стимуляции CD4+ Th1 клеток, как раскрыто в настоящей заявке («хелперные» пептиды). Предпочтительным примером таких соединений являются неопухолевый антиген, который вызывает иммунную память или оказывает неспецифическую помощь, или представляет собой специфический хелперный белок опухолевого происхождения, такой как столбнячный хелперный белок, пептид гемоцианин лимфы улитки и PADRE пептид, как раскрыто в настоящем описании. Другим предпочтительным примером является специфический хелперный пептид опухолевого происхождения, который может быть презентирован MHC II, в частности HLA-DR, HLA-DP или HLA-DQ, такой как фрагменты последовательностей родственных сверхэкспрессируемых опухолевых антигенов, таких как HER2, NY-ESO-1, hTERT или IL13RA2, как раскрыто в настоящем описании выше. В частности, иммуноадъювант может представлять собой HHD-DR3 пептид с последовательностью MAKTIAYDEEARRGLERGLN (SEQ ID № 473). Этот пептид является другим примером хелперного пептида (обладающего иммуноадъювантными свойствами), который является предпочтительным в контексте настоящего изобретения. Другим предпочтительным примером является h-pAg T13L (последовательность: TPPAYRPPNAPIL; SEQ ID № 474; Bhasin M, Singh H, Raghava GP (2003) MHCBN: a comprehensive database of MHC binding and non-binding peptides. Bioinformatics 19: 665–666). Другие примеры предпочтительных иммуноадъювантов, в частности хелперных пептидов, включают UCP2 пептид (например, как раскрыто в WO 2013/135553 A1 или в Dosset et al., Clin Cancer Res. 2012 Nov 15;18(22):6284-95) и BIRC5 пептид (например, как раскрыто в EP2119726 A1 или в Widenmeyer et al., Int J Cancer. 2012 Jul 1;131(1):140-9). Наиболее предпочтительным хелперным пептидом является UCP2 (аминокислотная последовательность: KSVWSKLQSIGIRQH; SEQ ID № 475).
Предпочтительно, фармацевтическая композиция содержит по меньшей мере два различных антигенных пептида по настоящему изобретению и хелперный пептид, предпочтительно, UCP2 пептид (SEQ ID № 475).
В частности, фармацевтическая композиция может содержать первый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности фрагмента опухолевого антигена человека CD22, второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности фрагмента опухолевого антигена человека TNFRSF13C, и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475). Предпочтительно фармацевтическая композиция включает первый антигенный пептид, содержащий или состоящий из варианта последовательности фрагмента CD22 (эталонный пептид человека) «WVFEHPETL» (SEQ ID № 270), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности как указано в SEQ ID №№ 106-110, 316 и 387-390, второй антигенный пептид, содержащий или состоящий из варианта последовательности фрагмента TNFRSF13C (эталонный пептид человека) «LLFGAPALL» (SEQ ID № 279), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID №№ 220, 325 и 450, и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475). Более предпочтительно, первый антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в SEQ ID №№ 110, 387 и 390, а второй антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в SEQ ID №№ 220 и 450. Еще более предпочтительно, фармацевтическая композиция содержит антигенный пептид, содержащий или состоящий из SEQ ID № 110, антигенный пептид, содержащий или состоящий из SEQ ID № 220, и хелперный пептид UCP2 (SEQ ID № 475).
Еще более предпочтительно, фармацевтическая композиция содержит по меньшей мере три различных антигенных пептида по настоящему изобретению и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475).
В частности, фармацевтическая композиция может включать первый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD22, второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека TNFRSF13C, третий антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD37, и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475). Предпочтительно, фармацевтическая композиция включает первый антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD22 (эталонного пептида человека) «WVFEHPETL» (SEQ ID № 270), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 106-110, 316 и 387-390, второй антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента TNFRSF13C (эталонного пептида человека) «LLFGAPALL» (SEQ ID № 279), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 220, 325 и 450, третий антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD37 (эталонного пептида человека) «GLAFVPLQI» (SEQ ID № 271), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 113-116, 324 и 392-393, и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475).
Наиболее предпочтительно фармацевтическая композиция содержит по меньшей мере четыре различных антигенных пептида по настоящему изобретению и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475).
В частности, фармацевтическая композиция может включать первый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD22, второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека TNFRSF13C, третьего антигенного пептида по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD37, четвертый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD19 или CD20, и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475).
Предпочтительно, фармацевтическая композиция включает первый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD22, второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека TNFRSF13C, третий антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD37, (при необходимости) четвертый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности (микробиоты) фрагмента опухолевого антигена человека CD20, и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475). Более предпочтительно, фармацевтическая композиция включает первый антигенный пептид, который содержит или состоит из варианта последовательности (микробиоты) фрагмента CD22 (эталонного пептида человека) «WVFEHPETL» (SEQ ID № 270), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 106-110, 316 и 387-390, второй антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента TNFRSF13C (эталонный пептид человека) «LLFGAPALL» (SEQ ID № 279), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, указанной в SEQ ID №№ 220, 325 и 450, третий антигенный пептид, содержащий или состоящий из варианта последовательности (микробиоты) фрагмента CD37 (эталонный пептид человека) «GLAFVPLQI» (SEQ ID № 271), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 113-116, 324 и 392-393, (при необходимости) четвертый антигенный пептид, содержащий или ко состоящий из варианта последовательности (микробиоты) фрагмента CD20 (эталонный пептид человека) «IMNSSLSLFA» (SEQ ID № 264), такой как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, представленной в SEQ ID №№ 65-70, 310 и 361-364, и хелперный пептид, предпочтительно пептид UCP2 (SEQ ID № 475).
Особо предпочтительными иммуноадъювантами являются полинозиновая:полицитидиловая кислота (также обозначаемая как «поли I:C») и/или ее производное поли-ICLC. Поли I:C представляет собой несопряженную двухцепочечную РНК, в которой одна цепь представляет собой полимер инозиновой кислоты, а другая – полимер цитидиловой кислоты. Поли I:C представляет собой иммуностимулирующий агент, который, как известно, взаимодействует с толл-подобным рецептором 3 (TLR3). Поли I:C структурно схожа с двуцепочечной РНК, которая является «натуральным» стимулятором TLR3. Соответственно, поли I:C может считаться синтетическим аналогом двухцепочечной РНК. Поли-ICLC представляет собой синтетический комплекс карбоксиметилцеллюлозы, полиинозиновой-полицитидиловой кислоты и поли-L-лизин двухцепочечной РНК. Аналогично поли I:C, поли: ICLC представляет собой лиганд для TLR3. Поли I:C и поли-ICLC обычно стимулируют высвобождение цитотоксических цитокинов. Предпочтительным примером поли-ICLC является Hiltonol®.
Наиболее предпочтительно, адъювант представляет собой Монтанид, такой как Монтанид ISA VG и/или Монтанид ISA 720 VG. Такие адъюванты образуют стандартные эмульсии вода-в-масле при смешивании с антигенной средой на водной основе. Монтанид ISA 51 VG основан на смеси сурфактанта моноолеата маннида и минерального масла, тогда как в Монтаниде ISA 720 VG используется неминеральное масло (Aucouturier J, Dupuis L, Deville S, Ascarateil S, Ganne V. Montanide ISA 720 and 51: a new generation of water in oil emulsions as adjuvants for human vaccines. Expert Rev Vaccines. 2002 Jun;1(1):111-8; Ascarateil S, Puget A, Koziol M-E. Safety data of Montanide ISA 51 VG and Montanide ISA 720 VG, two adjuvants dedicated to human therapeutic vaccines. Journal for Immunotherapy of Cancer. 2015;3(Suppl 2): P428. doi:10.1186/2051-1426-3-S2-P428).
Также предпочтительно, иммуностимулирующий агент представляет собой антигенпрезентирующую клетку (АРС). АРС вызывают особый интерес, поскольку их основной функцией является процессинг антигенов и презентация их на клеточной поверхности Т-клеток иммунной системы, чтобы инициировать и модулировать Т-клеточные ответы in vivo. В представленной композиции предпочтительно АРС нагружены антигенным пептидом (пептидами) и/или иммуногенным соединением (соединениями) по настоящему изобретению, что может быть осуществлено путем взаимодействия АРС in vitro с указанным антигенным пептидом (пептидами) и/или иммуногенным соединением (соединениями) (Rizzo et al., Methods Mol Biol. 2014; 1139:41-4; Rolinski and Hus, J Immunotoxicol. 2014 Oct; 11(4):311-8).
Предпочтительно, АРС представляет собой дендритную клетку (DC). DC являются наиболее эффективными АРС и, как сообщается, часто являются функционально неполноценными у онкологических больных. DC могут быть легко выделены специалистом в данной области техники, как из организма здоровых совместимых доноров (то есть DC являются HLA-родственными), так и из организма самого пациента с условием, что они являются функциональными (то есть DC являются аутологичными), например, либо путем простого выделения из периферической крови, либо путем выведения из клеток периферической крови, таких как CD14+ моноциты или CD34+ гемопоэтические клетки-предшественники (Emens et al., 2008). DC действительно можно отличить от других клеток периферической крови по их поверхностным маркерам, таким как S100, p55, CD83 и/или OX62, и, таким образом, их можно выделить и очистить c помощью указанных маркеров с использованием методов культур клеток, хорошо известных в данной области техники.
Согласно предпочтительному варианту осуществления изобретения, фармацевтическая композиция может дополнительно содержать по меньшей мере один противораковый терапевтический агент. Таким образом, указанный терапевтический агент предпочтительно способен предотвращать и/или лечить тот же тип рака, что и антигенный пептид по настоящему изобретению. Предпочтительно, противораковый терапевтический агент выбран из группы, включающей антитела, CAR Т-клетки, лизаты опухолевых клеток, химиотерапевтические агенты, радиотерапевтические агенты, модуляторы иммунных контрольных точек, и их комбинации.
Антитела являются особо предпочтительными для лечения рака, поскольку они могут как связываться со специфическими антигенами на поверхности раковых клеток, тем самым «направляя» терапию непосредственно в опухоль (такие антитела обозначаются как противоопухолевые таргетные антитела), так и блокировать иммунные контрольные точки, регуляция которых нарушена при раке (такие антитела обозначаются как иммуномодулирующие антитела). Целью последнего типа антител является подавление иммунной резистентности к раковым клеткам, которая может наблюдаться против Т-клеток, которые являются специфическими по отношению к опухолевым антигенам. Действительно, из области техники хорошо известно, что в естественных физиологических условиях иммунные контрольные точки играют ключевую роль в поддержании аутотолерантности (то есть предотвращают аутоиммунные реакции) и защите тканей от повреждений в тех случаях, когда иммунная система борется с патогенной инфекцией. Однако, при раковых заболеваниях регуляция экспрессии иммунных контрольных точек может нарушаться, что является важным механизмом в развитии иммунной резистентности. Такая резистентность наблюдается при меланоме, раке яичников, раке легких, глиобластоме, раке молочной железы и раке поджелудочной железы, и связана с контрольной точкой PD-L1 (Konishi et al., B7-H1 expression on non-small cell lung cancer cells and its relationship with tumor-infiltrating lymphocytes and their PD-1 expression. Clin Cancer Res. 2004 Aug 1;10(15):5094-100; Ghebeh et al., The B7-H1 (PD-L1) T lymphocyte-inhibitory molecule is expressed in breast cancer patients with infiltrating ductal carcinoma: correlation with important high-risk prognostic factors. Neoplasia. 2006 Mar;8(3):190-8; Hino et al., Tumor cell expression of programmed cell death-1 ligand 1 is a prognostic factor for malignant melanoma. Cancer. 2010 Apr 1;116(7):1757-66). Другими примерами иммунных контрольных точек являются, без ограничений указанными, PD-L2, PD-1, CD80, CD86, CTLA-4, B7H3, B7H4, PVR, TIGIT, GAL9, LAG-3, GITR, CD137, TIM3, VISTA, VISTA-R (Pico de Coaña et al., Checkpoint blockade for cancer therapy: revitalizing a suppressed immune system. Trends Mol Med. 2015 Aug;21(8):482-91; Pardoll DM. The blockade of immune checkpoints in cancer immunotherapy. Nat Rev Cancer. 2012 Mar 22; 12(4):252-64).
Для описанных выше целей антитела применяют как в форме изолированных моноклональных антител (то есть, неконъюгированных), так и в форме антител, конъюгированных с другими молекулами, которые могут быть токсичными для клеток или радиоактивными.
Примерами хорошо известных моноклональных противоопухолевых таргетных антител, которые применяются в иммунотерапии рака, являются, без ограничений указанными, алемтузумаб (хронический лимфоцитарный лейкоз), бевацизумаб (колоректальный рак, мультиформная глиобластома, рак шейки матки, рак легких, рак почек), брентуксимаб/ведотин (лимфомы), блинатумумаб (острый лимфобластный лейкоз), катумаксомаб (злокачественный асцит при EPCAM+ опухолях), цетуксимаб (рак головы и шеи, колоректальный рак), деносумаб (рак молочной железы, рак простаты и рак костей), гемтузумаб/озогамицин (острый миелоидный лейкоз), ибритумомаб/тиуксетан (неходжкинская лимфома), панитумумаб (колоректальный рак), пертузумаб (рак молочной железы), обинутузумаб (хронический лимфоцитарный лейкоз), офатумумаб (хронический лимфоцитарный лейкоз), опилимумаб (меланома), рамуцирумаб (рак желудка и гастроэзофагеальный рак), ритуксимаб (хронический лимфоцитарный лейкоз и неходжкинская лимфома), силтуксимаб (многоочаговая болезнь Кастлемана), тозитумомаб (неходжкинская лимфома) и трастузумаб (рак молочной железы, рак желудка и рак пищевода); в то время как примеры иммуномодулирующих антител включают, без ограничений указанными, ипилимумаб (меланома), который блокирует CTLA4-зависимую иммунную контрольную точку, ниволумаб (меланома, рак легкого) и пембролизубмаб (меланома), которые оба блокируют PDCD1-зависимую иммунную контрольную точку, а также MPDL3280A, MEDI4736, MEDI0680 и MSB0010718C, которые все блокируют PD-L1-зависимую иммунную контрольную точку (Sharma and Allison, The future of immune checkpoint therapy. Science. 2015 Apr 3; 348(6230):56-61).
Другие антитела для иммунотерапии рака был описаны Buqué et al., Trial Watch: Immunomodulatory monoclonal antibodies for oncological indications. Oncoimmunology. 2015 Mar 2; 4(4):e1008814. eCollection 2015 Apr; Redman et al., Mechanisms of action of therapeutic antibodies for cancer. Mol Immunol. 2015 Oct; 67(2 Pt A):28-45; Simpson and Caballero, Monoclonal antibodies for the therapy of cancer MC Proc. 2014; 8(Suppl 4): O6; а также на веб-сайте, где представлена вся совокупность антител (список терапевтических моноклональных антител, одобренных или находящихся на рассмотрении в Европейском Союзе или США, доступен по веб-ссылке http://www.antibodysociety.org/news/approved_mabs.php).
Адоптивная клеточная иммунотерапия Т-клетками с химерным антигенным рецептором (CAR) изменила подходы к лечению В-клеточной неходжкинской лимфомы (NHL), особенно агрессивных В-клеточных лимфом. Например, CAR Т-клетки, нацеленные на CD19, представляют собой новый стандарт лечения пациентов с DLBCL, которые не поддаются как минимум двум предшествующим линиям терапии. Два продукта CAR Т-клеток аксикабтаген цилолейцел (акси-цел) (KTE-019) (YESCARTA™) и тисагенлеклейцел (CTL019) (KYMRIAH™) получили одобрение Управления по санитарному надзору за качеством пищевых продуктов и медикаментов США для лечения рефрактерной DLBCL после двух линий терапии. Третий продукт, лизокабтаген маралейцел (liso-cel) (JCAR017), в настоящее время проходит клинические испытания. Другие CAR T-клетки включают CD20-CAR T-клетки.
Лизаты опухолевых клеток также могут быть комбинированы с антигенным пептидом (пептидами) по настоящему изобретению. Опухолевые клетки действительно способны инициировать иммунный ответ за счет презентации комплексов эндогенных пептидов-MHC, а также посредством дендритных клеток (DC) хозяина, которые могут процессировать и презентировать антиген, доставляемый такими лизатами. Таким образом, увеличивается спектр антигенов, против которых может быть индуцирован иммунный ответ. Лизаты опухолевых клеток могут быть легко получены при обработке опухолевых клеток тепловым шоком/или с помощью химической обработки, кроме того, они могут быть аутологичными (то есть выделенными из организма пациента) или аллогенными (то есть выделенными из организма другого субъекта).
Стандартные химиотерапевтические препараты и радиотерапевтические агенты не нуждаются в дополнительном описании здесь, поскольку они подробно описаны в литературе, а именно у Baskar и соавт. (Baskar et al., Cancer and radiation therapy: current advances and future directions. Int J Med Sci. 2012;9(3):193-9), Paci et al., (Paci et al., Review of therapeutic drug monitoring of anticancer drugs part 1--cytotoxics. Eur J Cancer. 2014 Aug; 50(12): 2010-9) и Widmer и соавт. (Widmer et al., Review of therapeutic drug monitoring of anticancer drugs part two--targeted therapies. Eur J Cancer. 2014 Aug; 50(12):2020-36). Список таких лекарств и агентов также доступен на сайте cancer.gov (http://www.cancer.gov/about-cancer/treatment/drugs).
Предпочтительно, модулятор иммунных контрольных точек, который может применяться в комбинации с антигенным пептидом, как раскрыто в настоящем описании, представляет собой активатор или ингибитор одной или нескольких молекул иммунной контрольной точки, выбранной из группы, включающей CD27, CD28, CD40, CD122, CD137, OX40, GITR, ICOS, A2AR, B7-H3, B7-H4, BTLA, CD40, CTLA-4, IDO, KIR, LAG3, PD-1, TIM-3, VISTA, CEACAM1, GARP, PS, CSF1R, CD94/NKG2A, TDO, GITR, TNFR и/или FasR/DcR3; или активатор или ингибитор одного или нескольких их лигандов.
Более предпочтительно, модулятор иммунной контрольной точки представляет собой активатор (ко)стимулирующей контрольной молекулы или ингибитор ингибирующей молекулы контрольной точки, или их комбинацию. Соответственно, модулятор иммунной контрольной точки более предпочтительно представляет собой (i) активатор CD27, CD28, CD40, CD122, CD137, OX40, GITR и/или ICOS; или (ii) ингибитор A2AR, B7-H3, B7-H4, BTLA, CD40, CTLA-4, IDO, KIR, LAG3, PD-1, PDL-1, PD-L2, TIM-3, VISTA, CEACAM1, GARP, PS, CSF1R, CD94/NKG2A, TDO, TNFR и/или FasR/DcR3.
Еще более предпочтительно, модулятор иммунной контрольной точки представляет собой ингибитор ингибирующей молекулы контрольной точки (но, предпочтительно, не является ингибитором стимулирующей молекулы контрольной точки). Соответственно, модулятор иммунной контрольной точки более предпочтительно представляет собой ингибитор A2AR, B7-H3, B7-H4, BTLA, CTLA-4, IDO, KIR, LAG3, PD-1, PDL-1, PD-L2, TIM-3, VISTA, CEACAM1, GARP, PS, CSF1R, CD94/NKG2A, TDO, TNFR и/или DcR3 или их лиганда.
Также предпочтительно, модулятор иммунной контрольной точки представляет собой активатор стимулирующей или костимулирующей молекулы контрольной точки (но предпочтительно не является активатором ингибирующей молекулы контрольной точки). Соответственно, модулятор иммунной контрольной точки более предпочтительно представляет собой активатор CD27, CD28, CD40, CD122, CD137, OX40, GITR и/или ICOS или их лиганда.
Еще более предпочтительно, модулятор иммунной контрольной точки представляет собой модулятор CD40 каскада, или IDO каскада, или LAG3 каскада, или CTLA-4 каскада и/или PD-1 каскада. В частности, модулятор иммунной контрольной точки предпочтительно представляет собой модулятор CD40, LAG3, CTLA-4, PD-L1, PD-L2, PD-1 и/или IDO, более предпочтительно, модулятор иммунной контрольной точки представляет собой ингибитор CTLA-4, PD-L1, PD-L2, PD-1, LAG3, и/или IDO или активатор CD40, еще более предпочтительно, модулятор иммунной контрольной точки представляет собой ингибитор CTLA-4, PD-L1, PD-1, LAG3 и/или IDO, еще более предпочтительно, модулятор иммунной контрольной точки представляет собой ингибитор LAG3, CTLA-4 и/или PD-1, и наиболее предпочтительно, модулятор иммунной контрольной точки представляет собой ингибитор CTLA-4 и/или PD-1.
Таким образом, модулятор контрольной точки для комбинации с антигенным пептидом может быть выбран из известных модуляторов CTLA-4 каскада или PD-1 каскада. Предпочтительно, модулятор контрольной точки для комбинации с антигенным пептидом, как раскрыто в настоящем описании, может быть выбран из известных модуляторов CTLA-4 каскада или PD-1 каскада. Особо предпочтительно, модулятор иммунной контрольной точки представляет собой ингибитор PD-1. Предпочтительные ингибиторы CTLA-4 каскада или PD-1 каскада включают моноклональные антитела Yervoy® (Ипилимумаб; Bristol Myers Squibb) и Тремелимумаб (Pfizer/MedImmune), а также Opdivo® (Ниволумаб; Bristol Myers Squibb), Keytruda® (Пембролизумаб, также известный как Ламбролизумаб или MK-3475; Merck), Imfinzi® (Дурвалумаб, также известный как MEDI4736; MedImmune/AstraZeneca), Tecentriq® (Атезолизумаб, также известный как MPDL3280A; Roche/Genentech), Пидилизумаб (CT-011; CureTech), MEDI0680 (AMP-514; AstraZeneca), Bavencio® (Авелумаб; Merck KGaA/Pfizer, также известный как MSB-0010718C), MIH1 (Affymetrix), LY3300054 (Eli Lilly) и Спартализумаб (также известный как PDR001; Novartis). Более предпочтительные ингибиторы контрольных точек включают ингибиторы CTLA-4, такие как Yervoy® (Ипилимумаб; Bristol Myers Squibb) и Тремелимумаб (Pfizer/MedImmune), а также ингибиторы PD-1 Opdivo® (Ниволумаб; Bristol Myers Squibb), Keytruda® (Пембролизумаб; Merck), Пидилизумаб (CT-011; CureTech), MEDI0680 (AMP-514; AstraZeneca), AMP-224 (PD-L2 Fc гибридный белок; MedImmune).
Также предпочтительно, модулятор иммунной контрольной точки для комбинации с антигенным пептидом, как раскрыто в настоящем описании, выбран из группы, включающей Пембролизумаб, Ипилимумаб, Ниволумаб, Атезолизумаб, Дурвалумаб, Тремелимумаб, Авелумаб, Спартализумаб, LAG525 (анти-LAG-3 моноклональное антитело), Эпакадостат (также известный как INCB24360; ингибитор IDO), Варлилумаб (анти-CD27 моноклональное антитело), Урелумаб (анти-CD137 моноклональное антитело), AMP-224 и CM-24 (анти-CEACAM1 моноклональное антитело).
Выбор подходящего иммунного противоракового лекарственного агента для целей настоящего изобретения находится в компетенции специалиста в данной области техники. Например, если целью является профилактика или лечение меланомы, предпочтительно использовать лизат клеток меланомы и/или антитело Ипилимумаб вместе с подходящим антигенным пептидом. Подходящие антигенные пептиды могут быть выбраны путем (i) подбора подходящего опухолевого антигена для определенного типа рака, как известно из области техники и/или как раскрыто в настоящем описании в Таблице 1B; и (ii) подбора подходящего антигенного пептида по изобретению для выбранного опухолевого антигена, как описано ранее, например, в Таблице 1А.
Противораковый терапевтический агент также можно применять в комбинации с композицией по настоящему изобретению, как одновременно, так раздельно или последовательно. Если композицию и лекарственный агент применяют раздельно или последовательно, их можно вводить в различных лекарственных формах.
Таким образом, согласно другому аспекту, изобретение относится к композиции по изобретению и по меньшей мере одному противораковому терапевтическому агенту, как описано выше, как к комбинированной лекарственной форме для одновременного, раздельного или последовательного введения. Другими словами, изобретение предлагает комбинированное применение композиции по изобретению и по меньшей мере одного противоракового терапевтического агента, как описано выше, для одновременного, раздельного или последовательного введения.
Набор из частей
В другом аспекте настоящее изобретение также относится к набору из частей (который также обозначается в настоящем описании как «набор»), который содержит по меньшей мере один их следующих элементов:
- антигенный пептид по настоящему изобретению, как раскрыто в настоящем описании,
- иммуногенное соединение по настоящему изобретению, как раскрыто в настоящем описании,
- наночастицу по настоящему изобретению, как раскрыто в настоящем описании,
- клетку по настоящему изобретению, как раскрыто в настоящем описании,
- нуклеиновую кислоту по настоящему изобретению, как раскрыто в настоящем описании,
- клетку-хозяин по настоящему изобретению, как раскрыто в настоящем описании,
- цитотоксический Т-лимфоцит по настоящему изобретению, как раскрыто в настоящем описании, и/или
- фармацевтическую композиция по настоящему изобретению, как раскрыто в настоящем описании.
В частности, предпочтительные варианты осуществления антигенного пептида, как описано выше, также применимы для такого набора по настоящему изобретению. Например, антигенный пептид, содержащийся в наборе, или антигенный пептид, содержащийся в любом из иммуногенного соединения, наночастицы, клетки, нуклеиновой кислоты, клетки-хозяина или фармацевтической композиции, включенной в набор, предпочтительно содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 1-247 и 476-500. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, указанной в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 61, 65, 68, 72, 86, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 10, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, представленной в любой из SEQ ID №№ 65, 110, 114 и 220, являются еще более предпочтительными.
Также предпочтительными являются их комбинации, а именно, наборы, содержащие различные антигенные пептиды по настоящему изобретению. В частности, набор из частей по настоящему изобретению может содержать более одного описанного выше компонента, например, 2, 3, 4, 5, 6, 7, 8, 9, или 10 различных компонентов. Например, набор из частей по настоящему изобретению может содержать по меньшей мере два (например, 2, 3, 4, 5, 6, 7, 8, 9 или 10) различных иммуногенных соединения, по меньшей мере два (например, 2, 3, 4, 5, 6, 7, 8, 9 или 10) различных антигенных пептида, по меньшей мере две (например, 2, 3, 4, 5, 6, 7, 8, 9 или 10) различные наночастицы, по меньшей мере две (например, 2, 3, 4, 5, 6, 7, 8, 9 или 10) различные клетки, по меньшей мере две (например, 2, 3, 4, 5, 6, 7, 8, 9 или 10) различные нуклеиновые кислоты, по меньшей мере две (например, 2, 3, 4, 5, 6, 7, 8, 9 или 10) различные клетки-хозяева, и/или по меньшей мере две (например, 2, 3, 4, 5, 6, 7, 8, 9 или 10) различные фармацевтические композиции. Предпочтительно, такие различные компоненты в составе набора из частей, как описано выше, различаются по антигенным пептидам, заявленным в соответствии с настоящим изобретением, например, один компонент относится к первому антигенному пептиду, а другой компонент относится ко второму антигенному пептиду (отличному от первого антигенного пептида). Например, набор может содержать по меньшей мере два различных иммуногенных соединения по настоящему изобретению. Например, набор может содержать по меньшей мере два различных антигенных пептида по настоящему изобретению. Например, набор может содержать по меньшей мере две различных наночастицы по настоящему изобретению. Например, набор может содержать по меньшей мере две различные нуклеиновые кислоты по настоящему изобретению. Например, набор может содержать по меньшей мере два различных цитотоксических Т-лимфоцита по настоящему изобретению.
Предпочтительные комбинации компонентов, таких как антигенные пептиды по настоящему изобретению, в составе набора соответствуют предпочтительным комбинациям компонентов, таких как антигенные пептиды по настоящему изобретению, включенные в фармацевтическую композицию, как описано выше.
Таким образом, настоящее изобретение обеспечивает набор, содержащий (по меньшей мере один) антигенный пептид по настоящему изобретению, как раскрыто в настоящем описании. Кроме того, настоящее изобретение также обеспечивает набор, содержащий (по меньшей мере одно) иммуногенное соединение по настоящему изобретению, как раскрыто в настоящем описании. Кроме того, настоящее изобретение также обеспечивает набор, содержащий (по меньшей мере одну) наночастицу по настоящему изобретению, как раскрыто в настоящем описании. Кроме того, настоящее изобретение также обеспечивает набор, содержащему (по меньшей мере одну) клетку по настоящему изобретению, как раскрыто в настоящем описании. Кроме того, настоящее изобретение также обеспечивает набор, содержащий (по меньшей мере одну) нуклеиновую кислоту по настоящему изобретению, как раскрыто в настоящем описании. Кроме того, настоящее изобретение также обеспечивает набор, содержащий (по меньшей мере одну) клетку-хозяин по настоящему изобретению, как раскрыто в настоящем описании.
Различные компоненты набора частей могут быть упакованы в один или несколько контейнеров. Описанные выше компоненты могут быть представлены в лиофилизированной или сухой форме, или быть растворены в подходящем буфере. Набор также может содержать дополнительные реагенты, включая, например, консерванты, питательные среды и/или буферы для хранения и/или восстановления описанных выше компонентов, промывающие растворы и тому подобные.
Таким образом, настоящее изобретение относится к набору, содержащему по меньшей мере два, предпочтительно три различных антигенных пептида по настоящему изобретению, как раскрыто в настоящем описании (или иммуногенных соединения, наночастицы, нуклеиновых кислоты, клетки и т.д., как описано выше, которые отличаются по антигенному пептиду), и при необходимости, хелперный пептид, такой как UCP2 пептид, и/или адъювант, такой как MONTANIDE ISA 51. Различные антигенные пептиды (или иммуногенных соединения, наночастицы, нуклеиновые кислоты, клетки и т.д., как описано выше, которые отличаются по антигенному пептиду) могут быть упакованы в один или в несколько контейнеров. Например, набор может включать (один) контейнер, содержащий первый антигенный пептид, как раскрыто в настоящем описании, и второй антигенный пептид, как раскрыто в настоящем описании. Указанный (один) контейнер может также дополнительно содержать хелперный пептид, такой как UCP2 пептид. При необходимости первый и второй антигенные пептиды (и при необходимости хелперный пептид) в составе (одного) контейнера могут находиться в составе одной лекарственной формы, например, быть растворенными в воде для инъекций и/или в диметилсульфоксиде (DMSO). Кроме того, набор может содержать дополнительный контейнер (отличный от контейнера, содержащего антигенные пептиды), который включает адъювант, например, MONTANIDE ISA 51.
Таким образом, предпочтительно, контейнер содержит:
(i) первый флакон, содержащий один или несколько антигенных пептидов по настоящему изобретению (например, по меньшей мере 200 или 300 мкг каждого антигенного пептида) и, при необходимости, хелперный пептид, такой как UCP2 (например, по меньшей мере 200 или 300 мкг каждого антигенного пептида), при необходимости в составе воды для инъекций и диметилсульфоксида (DMSO); и
(ii) второй флакон, содержащий MONTANIDE ISA 51 (например, по меньшей мере 0.4 или 0.5 мл).
Кроме того, набор может содержать один или несколько (например, 2 или 3) шприцев, например, шприцев, не содержащих силикона и латекса. Набор также может содержать коннектор, такой как I-коннектор.
Неограничивающими примерами таких коннекторов являются:
- I-коннектор, разработанный компанией Green Peptide (Япония),
- коннектор, обозначенный как DIDRACDLLFT от Didanorm (Франция),
- I-коннектор (кат.№: ODG0015ST) от Promepla (Монако), и
- I-коннектор (кат.№: MX494) от Smiths medical (США).
Шприцы предпочтительно являются пригодными для введения MONTANIDE, т.е. не содержат силикона и резины (то есть не содержат резинового колпачка на кончике поршня) и, предпочтительно, также не содержат латекса. Неограничивающими примерами таких шприцев являются:
- 2 мл INKJET (Ref: 4606701V от B-Braun, Германия),
- 5 мл INKJET (Ref: 4606710V от B-Braun, Германия),
- 2 мл Norm-Ject (Ref: 4020.000V0 от Henke Sass Wolf GMBH, Германия), и
- 5 мл Norm-Ject (Ref: 4050.000V0 от Henke Sass Wolf GMBH, Германия).
Например, набор может содержать (i) первый флакон, включающий по меньшей мере 300 мкг антигенного пептида по изобретению (или два или три антигенных пептида по меньшей мере 300 мкг каждого) и при необходимости по меньшей мере 300 мкг UCP2, растворенного в воде для инъекций и диметилсульфоксиде (DMSO); (ii) второй флакон, включающий по меньшей мере 0,5 мл MONTANIDE ISA 51; (iii) два не содержащих силикона и резины шприца; и (iv) I-коннектор.
При необходимости, набор также может включать флакон воды для инъекций и/или адаптер для флакона. Также может прилагаться стерильная игла, например, для вакцинации пациента после получения эмульсии. Объем шприцев в составе набора может составлять, например, 2 мл.
Кроме того, набор из частей по настоящему изобретению может при необходимости содержать инструкцию по применению. Соответственно, предпочтительно набор содержит листок-вкладыш или инструкцию по применению с указаниями по профилактике и лечению В-клеточного гемобластоза путем применения иммуногенного соединения по настоящему изобретению, антигенного пептида по настоящему изобретению, наночастицы по настоящему изобретению, клетки по настоящему изобретению, нуклеиновой кислоты по настоящему изобретению, клетки-хозяина по настоящему изобретению или фармацевтической композиции по настоящему изобретению.
Также предпочтительно, в дополнение к любому из компонентов, описанных выше, набор содержит противораковый терапевтический агент, как раскрыто в настоящем описании.
Кроме того, настоящее изобретение также относится к вакцинному набору для лечения, профилактики и/или стабилизации В-клеточного гемобластоза, включающему фармацевтическую композицию или вакцину, как раскрыто в настоящей заявке, а также инструкции по применению указанной фармацевтической композиции или указанной вакцины для профилактики и/или лечения В-клеточного гемобластоза.
Медицинское применение и лечение
Как раскрыто выше, композиция по настоящему изобретению может быть особенно полезна для терапевтических целей (в качестве медикамента), главным образом, для запуска специфического иммунного ответа против определенного опухолевого антигена/белка, например, для профилактики или лечения В-клеточного гемобластоза у пациента, нуждающегося в таком лечении.
С учетом этого, настоящее изобретение предлагает:
- антигенный пептид по настоящему изобретению, как раскрыто в настоящем описании,
- иммуногенное соединение по настоящему изобретению, как раскрыто в настоящем описании,
- наночастицу по настоящему изобретению, как раскрыто в настоящем описании,
- цитотоксический Т-лимфоцит (CTL) по настоящему изобретению, как раскрыто в настоящем описании,
- клетку по настоящему изобретению, как раскрыто в настоящем описании,
- нуклеиновую кислоту по настоящему изобретению, как раскрыто в настоящем описании,
- клетку-хозяин по настоящему изобретению, как раскрыто в настоящем описании,
- фармацевтическую композицию по настоящему изобретению, как раскрыто в настоящем описании,
- набор по настоящему изобретению, как раскрыто в настоящем описании, или
- комбинацию по настоящему изобретению, как раскрыто в настоящем описании,
для применения в медицине, в частности, в профилактике и/или лечении В-клеточного гемобластоза.
В частности, предпочтительные варианты осуществления антигенного пептида, как описано выше, также применимы для использования в соответствии с настоящим изобретением для профилактики и/или лечения В-клеточного гемобластоза. Например, антигенный пептид, используемый для профилактики и/или лечения В-клеточного гемобластоза, или антигенный пептид, содержащийся в любом из иммуногенного соединения, наночастицы, клетки, нуклеиновой кислоты, клетки-хозяина или фармацевтической композиции, используемых для профилактики и/или лечения рака, предпочтительно содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 1-257 и 476-500. В некоторых вариантах осуществления антигенные пептиды по настоящему изобретению содержат или состоят из аминокислотной последовательности, указанной в любой из SEQ ID №№ 1-12, 34-35, 36-39, 40, 41-64, 65-70, 476-484, 71-80, 81- 87, 106-110, 485-488, 111-130, 489-493, 210-225, 494-500 и 226-257; предпочтительно антигенный пептид содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 34-35, 65-70, 476-484, 106-110, 485-488, 111-130, 489-493, 210-225, 494-500 и 226-257; более предпочтительно, антигенный пептид содержит или состоит из аминокислотной последовательности, представленной в любой из SEQ ID №№ 65-70, 476-484, 106-110, 485-488, 111-130, 489-493, 210-225. и 494-500. Более предпочтительно, антигенный пептид содержит или состоит из аминокислотной последовательности, указанной в любой из SEQ ID №№ 10, 34, 35, 39, 40, 61, 68, 70, 72, 86, 107-110, 114, 117, 119, 120, 212, 217, 220, 224, 227, 231, 477, 491 и 493. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, как указано в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 61, 65, 68, 72, 86, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, указанной в любой из SEQ ID №№ 10, 21, 33, 35, 39, 40, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, указанной в любой из SEQ ID №№ 10, 110, 114 и 220, являются еще более предпочтительными. Например, антигенные пептиды по настоящему изобретению, содержащие или состоящие из аминокислотной последовательности, указанной в любой из SEQ ID №№ 65, 110, 114 и 220, являются еще более предпочтительными.
Предпочтительными являются также их комбинации, а именно отдельные антигенные пептиды по настоящему изобретению для применения в профилактике и/или лечении В-клеточного гемобластоза. В частности, более чем один из вышеописанных компонентов можно использовать для профилактики и/или лечения В-клеточного гемобластоза. Например, по меньшей мере два разных антигенных пептида, по меньшей мере два разных иммуногенных соединения, по меньшей мере две разные наночастицы, по меньшей мере две разные клетки, по меньшей мере две разные нуклеиновые кислоты, по меньшей мере две разные клетки-хозяева и/или по меньшей мере две разные фармацевтические композиции могут быть использованы для профилактики и/или лечения В-клеточного гемобластоза. Предпочтительно такие различные компоненты, используемые для профилактики и/или лечения В-клеточного гемобластоза, как описано выше, различаются в антигенных пептидах по настоящему изобретению, например, один компонент относится к первому антигенному пептиду, а другой компонент относится к второму антигенному пептиду (отличному от первого антигенного пептида). Например, по меньшей мере два различных иммуногенных соединения по настоящему изобретению можно использовать для профилактики и/или лечения В-клеточного гемобластоза. Например, по меньшей мере два различных антигенных пептида по настоящему изобретению можно использовать для профилактики и/или лечения В-клеточного гемобластоза. Например, по меньшей мере две различные наночастицы по настоящему изобретению можно использовать для профилактики и/или лечения В-клеточного гемобластоза. Например, по меньшей мере две различные нуклеиновые кислоты по настоящему изобретению можно использовать для профилактики и/или лечения В-клеточного гемобластоза.
Соответственно, настоящее изобретение обеспечивает (по меньшей мере один) антигенный пептид по настоящему изобретению, как описано здесь, для применения в профилактике и/или лечении В-клеточного гемобластоза. Далее, настоящее изобретение также обеспечивает (по меньшей мере одно) иммуногенное соединение по настоящему изобретению, как описано здесь, для применения в профилактике и/или лечении В-клеточного гемобластоза. Далее, настоящее изобретение также обеспечивает (по меньшей мере одну) наночастицу согласно настоящему изобретению, как описано здесь, для применения в профилактике и/или лечении В-клеточного гемобластоза. Далее, настоящее изобретение также обеспечивает (по меньшей мере одну) клетку согласно настоящему изобретению, как описано здесь, для применения в профилактике и/или лечении В-клеточного гемобластоза. Далее, настоящее изобретение также обеспечивает (по меньшей мере одну) нуклеиновую кислоту по настоящему изобретению, как описано здесь, для применения в профилактике и/или лечении В-клеточного гемобластоза. Далее, настоящее изобретение также обеспечивает (по меньшей мере одну) клетку-хозяин согласно настоящему изобретению, как описано здесь, для применения в профилактике и/или лечении В-клеточного гемобластоза. Далее, настоящее изобретение также обеспечивает (по меньшей мере одну) фармацевтическую композицию по настоящему изобретению, как описано здесь, для применения для профилактики и/или лечения В-клеточного гемобластоза. Далее, настоящее изобретение также обеспечивает набор согласно настоящему изобретению, как описано здесь, для применения для профилактики и/или лечения В-клеточного гемобластоза.
Соответственно, настоящее изобретение также относится к способу предотвращения (уменьшения частоты) и/или лечения В-клеточного гемобластоза, или инициации, усиления или продления противоопухолевого ответа против В-клеточного гемобластоза у субъекта, нуждающегося в этом, включающему применение у субъекта:
- антигенного пептида по настоящему изобретению,
- иммуногенного соединения по настоящему изобретению,
- наночастицы по настоящему изобретению,
- клетки по настоящему изобретению,
- цитотоксического Т-лимфоцита (CTL) по изобретению, как описано в настоящей заявке,
- нуклеиновой кислоты по настоящему изобретению,
- клетки-хозяина по настоящему изобретению,
- фармацевтической композиции по настоящему изобретению,
- набора по настоящему изобретению, или
- комбинации согласно настоящему изобретению, как описано в настоящей заявке.
Предпочтительно подлежащие лечению В-клеточные гемобластозы включают лейкоз и лимфому, например, острый миелоидный (или миелогенный) лейкоз (AML), хронический миелоидный (или миелогенный) лейкоз (CML), острый лимфоцитарный (или лимфобластный) лейкоз (ALL), хронический В-лимфолейкоз (BCLL), хронический лимфолейкоз (CLL, болезнь Рихтера), волосатоклеточный лейкоз (HCL), лимфоплазмоцитарную лимфому (LPC) или макроглобулинемию Вальденстрема, пролимфоцитарный лейкоз (PLL), малую лимфоцитарную лимфому (SLL), мантийноклеточную лимфому (MCL), лимфому маргинальной зоны (MZL), лимфому Беркитта (BL), лимфому Ходжкина (HL), неходжкинскую лимфому (NHL), анапластическую крупноклеточную лимфому (ALCL), фолликулярную лимфому (FL), рефрактерную фолликулярную лимфому, диффузную крупноклеточную В-клеточную лимфому (DLBCL) и множественную миелому (ММ). В некоторых вариантах осуществления В-клеточный гемобластоз выбран из острого лимфобластного лейкоза (OLL), OLL взрослых, хронического лимфобластного лейкоза (CLL), неходжкинской лимфомы (NHL) и диффузной крупноклеточной В-клеточной лимфомы (DLBCL). В некоторых вариантах осуществления заболевание или состояние представляет собой NHL, и NHL выбрана из группы, состоящей из индолентной (медленно растущей) NHL, агрессивной NHL, диффузной крупноклеточной В-клеточной лимфомы (DLBCL), NOS (de novo и трансформированной из индолентной), первичной медиастинальной крупноклеточной В-клеточной лимфомы (PMBCL), T-клеточной/богатой гистоцитами крупноклеточной B-клеточной лимфомы (TCHRBCL), лимфомы Беркитта, мантийноклеточной лимфомы (MCL) и/или фолликулярной лимфомы (FL), при необходимости, фолликулярной лимфомы степени 3В (FL3B).
Кроме того, настоящее изобретение обеспечивает способ индукции или улучшения у субъекта иммунного ответа против одного или нескольких эпитопов, зависящих от CD8+ цитотоксических Т-клеток, включающий применение у указанного субъекта любого из следующих компонентов:
- антигенного пептида по настоящему изобретению,
- иммуногенного соединения по настоящему изобретению,
- наночастицы по настоящему изобретению,
- цитотоксического Т-лимфоцита (CTL) по настоящему изобретению,
- клетки по настоящему изобретению,
- нуклеиновой кислоты по настоящему изобретению,
- клетки-хозяина по настоящему изобретению,
- фармацевтической композиции по настоящему изобретению,
- набора по настоящему изобретению, или
- комбинации по настоящему изобретению, как раскрыто в настоящем описании.
Иммунный ответ, который зависит от CD8+ ответа, можно определить путем оценки воспалительного ответа, провоспалительного цитокинового ответа, включая увеличение экспрессии одного или нескольких из мРНК или белка IFN-γ, TNF-α и IL-2 относительно уровня до применения соединений по настоящему изобретению. Он также может быть оценен путем измерения частоты или абсолютного количества антигенспецифических Т-клеток после применения соединений по настоящему изобретению, оцениваемого с помощью окрашивания HLA-пептидного мультимера, анализа ELISPOT и тестов гиперчувствительности замедленного типа. Его также можно опосредованно оценить по повышению антиген-специфических антител в сыворотке, которые зависят от антиген-специфических Т-хелперных клеток.
Настоящее изобретение также обеспечивает способ индукции или улучшения у субъекта иммунного ответа против одного или нескольких эпитопов, рестриктированных множественными молекулами MHC I класса, включающий введение указанному субъекту любого из следующих компонентов:
- антигенного пептида по настоящему изобретению,
- иммуногенного соединения по настоящему изобретению,
- наночастицы по настоящему изобретению,
- цитотоксического Т-лимфоцита по настоящему изобретению,
- клетки по настоящему изобретению,
- нуклеиновой кислоты по настоящему изобретению,
- клетки-хозяина по настоящему изобретению,
- фармацевтической композиции по настоящему изобретению,
- набора по настоящему изобретению, или
- комбинации по настоящему изобретению, как раскрыто в настоящем описании.
Способ индукции или улучшения у субъекта иммунного ответа против множественных эпитопов, как раскрыто в настоящем описании, которые ограничены множественными молекулами MHC I класса, можно оценить путем измерения цитокинового ответа, включая усиление экспрессии одного или нескольких из мРНК или белка IFN-γ, TNF-α и IL-2 по сравнению c исходным уровнем до применения соединений по настоящему изобретению после стимуляции Т-клеток in vitro индивидуальными пептидами, связывающимися с различными молекулами MHC I класса на поверхности антигенпрезентирующих клеток. Рестрикция молекулами MHC I класса может быть подтверждена за счет использования антигенпрезентирующих клеток, экспрессирующих молекулы MHC I класса, или за счет использования антител, блокирующих MHC I класса. Его также можно измерить по увеличению числа или абсолютного количества антиген-специфических Т-клеток после применения соединений по изобретению, которое измеряют при помощи окрашивания HLA-пептидных мультимеров с применением мультимеров, объединенных с молекулами MHC I класса.
Таким образом, согласно другому аспекту, настоящее изобретение также обеспечивает:
- антигенный пептид по настоящему изобретению,
- иммуногенное соединение по настоящему изобретению,
- наночастицу по настоящему изобретению,
- цитотоксический Т-лимфоцит (CTL) по настоящему изобретению,
- клетку по настоящему изобретению,
- нуклеиновую кислоту по настоящему изобретению,
- клетку-хозяин по настоящему изобретению,
- фармацевтическую композицию по настоящему изобретению,
- набор по настоящему изобретению, или
- комбинацию по настоящему изобретению, как раскрыто в настоящем описании,
для применения в качестве лекарственного средства.
Настоящее изобретение более конкретно относится к композиции, как раскрыто выше, предназначенной для применения в качестве вакцины для иммунотерапии. Кроме того,
- антигенный пептид по настоящему изобретению,
- иммуногенное соединение по настоящему изобретению,
- наночастицу по настоящему изобретению,
- цитотоксический Т-лимфоцит (CTL) по настоящему изобретению,
- клетку по настоящему изобретению,
- нуклеиновую кислоту по настоящему изобретению,
- клетку-хозяин по настоящему изобретению,
- фармацевтическую композицию по настоящему изобретению,
- набор по настоящему изобретению, или
- комбинацию по настоящему изобретению, как раскрыто в настоящем описании,
можно применять в качестве вакцины, в частности, для иммунотерапии (рака).
Используемый в контексте настоящего изобретения термин «вакцина» относится к (биологическому) препарату, который обеспечивает врожденный и/или приобретенный иммунитет к определенному заболеванию, предпочтительно, В-клеточному гемобластозу. Таким образом, вакцина поддерживает, в частности, врожденный и/или приобретенный иммунный ответ иммунной системы у субъекта, который получает лечение. Например, антигенный пептид по настоящему изобретению обычно приводит к появлению приобретенного иммунного ответа у пациента, получающего лечение, или поддерживает такой иммунный ответ.
В контексте настоящего изобретения вакцина (композиция) может индуцировать специфический иммунный ответ против опухолевого антигена и, таким образом, предпочтительно применяется для профилактики и лечения В-клеточного гемобластоза.
Таким образом, в предпочтительном варианте осуществления настоящее изобретение относится к композиции, как раскрыто выше, для применения с целью профилактики и/или лечения рака у субъекта, нуждающегося в этом. Более предпочтительно, настоящее изобретение относится к применению композиции по настоящему изобретению для производства лекарственного средства, предназначенного для профилактики или лечения рака у субъекта, нуждающегося в этом. Другими словами, настоящее изобретение относится к способу профилактики и лечения рака у субъекта, нуждающегося в этом, который заключается в применении эффективного количества композиции по изобретению у указанного субъекта.
Предпочтительно, рак, профилактика и/или лечение которого проводят
- антигенным пептидом по настоящему изобретению,
- иммуногенным соединением по настоящему изобретению,
- наночастицей по настоящему изобретению,
- цитотоксическим Т-лимфоцитом (CTL) по настоящему изобретению,
- клеткой по настоящему изобретению,
- нуклеиновой кислотой по настоящему изобретению,
- клеткой-хозяином по настоящему изобретению,
- фармацевтической композицией по настоящему изобретению,
- набором по настоящему изобретению или
- комбинацией по настоящему изобретению, как раскрыто в настоящем описании,
связан с (эталонным) опухолевым антигеном, как раскрыто в настоящем описании. Конкретно, подходящие антигенные пептиды могут быть выбраны (i) путем подбора подходящего опухолевого антигена для определенного типа рака, как известно из области техники, и (ii) подбора подходящего антигенного пептида по настоящему изобретению для отобранного опухолевого антигена, как описано выше, например, в Таблице 1А. Специалисту в данной области будет очевидно, что антигенный пептид по настоящему изобретению может быть выбран, исходя из природы В-клеточного гемобластоза, профилактику и/или лечение которого проводят, и/или на основании человеческого гена/опухолевого антигена, участвующего в патогенезе этого В-клеточного гемобластоза.
В целом, антигенные пептиды по настоящему изобретению могут быть введены «изолированными» или в форме иммуногенных соединений по настоящему изобретению, клеток, нагруженных такими пептидами по настоящему изобретению, наночастиц по настоящему изобретению, нуклеиновых кислот по настоящему изобретению, клеток-хозяев по настоящему изобретению и/или фармацевтических композиций по настоящему изобретению.
Согласно предпочтительному варианту осуществления изобретения, они также могут быть введены в составе микроорганизма, такого как бактерия микрофлоры кишечника. Полноценные бактерии микрофлоры кишечника могут также иметь преимущество, поскольку обладают способностью инициировать более мощный иммунный ответ чем (поли)пептиды или нуклеиновые кислоты, которые они содержат. Альтернативно, бактерии микрофлоры кишечника по настоящему изобретению могут находиться в форме пробиотиков, то есть живых бактерий микрофлоры кишечника, которые, соответственно, могут применяться как пищевые добавки, благодаря благоприятному влиянию на здоровье, которые они оказывают. Они могут быть, например, лиофилизированы в гранулы, пилюли или капсулы, или непосредственно смешаны с молочными продуктами перед употреблением.
Способы применения хорошо известны специалисту в данной области техники. В зависимости от типа композиции по настоящему изобретению, она может быть введена напрямую субъекту, в пораженный орган (то есть путем местного введения) или системно (то есть путем энтерального или парентерального введения), или даже применяться ex vivo на клетках, полученных от субъекта, или человеческой клеточной линии, которую затем вновь вводят субъекту, или даже применяться in vitro для отбора субпопуляции иммунных клеток, выделенных от субъекта, которые затем повторно вводят этому субъекту. Энтеральные способы применения включают пероральное и ректальное введение, а также введение посредством гастральных зондов, дуоденальных зондов или гастростомы, в то время как парентеральные способы применения включают, среди прочего, подкожные, внутривенные, внутримышечные, внутриартериальные, внутрикожные, внутрикостные, внутримозговые и интратекальные инъекции. Способ применения чаще всего будет зависеть от антигенного пептида (пептидов) и/или иммуногенного соединения (соединений) в составе композиции, а также типа рака, подлежащего лечению, а также присутствия других активных ингредиентов в составе композиции. Например, введение предпочтительно осуществляют посредством внутримышечной или интрадермальной инъекции в том случае, если иммуногенное соединение представляет собой нуклеиновую кислоту, как раскрыто выше, пероральное/назальное введение является наиболее предпочтительным в том случае, если указанная нуклеиновая кислота клонирована в вирусном векторе. Альтернативно, в том случае, если антигенный пептид и/или иммуногенное соединение представляет собой (поли)пептид, как раскрыто ранее, или если он нагружен в/на наночастицу, как раскрыто ранее, предпочтительным является внутримышечное, интрадермальное или пероральное введение. Также альтернативно, в том случае если антигенный пептид и/или иммуногенное соединение доставляют в форме бактерий микрофлоры кишечника, как раскрыто выше, особенно если бактерии микрофлоры кишечника находятся в составе пробиотиков, предпочтительным является пероральное введение.
Антигенные пептиды, иммуногенные соединения и нуклеиновые кислоты по настоящему изобретению могут быть дополнительно инкапсулированы, чтобы облегчить их введение субъекту, нуждающемуся в этом. Например, они могут быть инкапсулированы в пептидные нано-носители (предпочтительно в том случае, если иммуногенное представляет собой нуклеиновую кислоту или (поли)пептид), в виросомы (предпочтительно, если иммуногенное соединение представляет собой нуклеиновую кислоту или (поли)пептид), или систему-носитель на липидной основе, такую как липосомный-поликатионный-ДНК комплекс (предпочтительно, если иммуноген представляет собой нуклеиновую кислоту или (поли)пептид) (Trovato M, De Berardinis P. Novel antigen delivery systems. World J Virol. 2015 Aug 12; 4(3):156-68; Saade F, Petrovsky N. Technologies for enhanced efficacy of DNA vaccines. Expert Rev Vaccines. 2012 Feb; 11(2):189-209; Li et al., Peptide Vaccine: Progress and Challenges. Vaccines (Basel). 2014 Jul 2; 2(3):515-36).
Композиция также может быть введена более чем один раз для достижения необходимого эффекта. Согласно предпочтительному варианту осуществления изобретения указанную композицию вводят повторно, по меньшей мере дважды, предпочтительно более чем дважды. Введение можно осуществлять в течение продолжительного периода времени, например, еженедельно, каждые две недели, каждый месяц, каждый год или даже каждые несколько лет после самого первого введения для обеспечения правильной иммунизации субъекта.
Комбинированная терапия
Применение антигенного пептида по настоящему изобретению, иммуногенного соединения по настоящему изобретению, наночастицы по настоящему изобретению, клетки по настоящему изобретению, нуклеиновой кислоты по настоящему изобретению, клетки-хозяина по настоящему изобретению, фармацевтической композиции по настоящему изобретению, в частности, согласно способам и применениям по настоящему изобретению, может быть осуществлено по отдельности или в комбинации с другим с агентом для совместного применения, полезным для лечения и/или профилактики рака, таким как противораковый терапевтический агент.
Таким образом, указанный терапевтический агент предпочтительно обладает способностью к профилактике и/или лечению того же типа рака, для которого применяют антигенный пептид по настоящему изобретению. Особо предпочтительные противораковые терапевтические агенты согласно настоящему изобретению, без ограничений указанными, включают антитела, CAR-T-клетки, лизаты опухолевых клеток, химиотерапевтические агенты, радиотерапевтические агенты, модуляторы иммунных контрольных точек, а также их комбинации.
Антитела являются особо предпочтительными для терапии рака, поскольку они могут либо связываться со специфическими антигенами на поверхности раковых клеток, тем самым «направляя» терапию непосредственно в опухоль (такие антитела обозначаются как противоопухолевые таргетные антитела), либо блокировать иммунные контрольные точки, регуляция которых нарушена при раке (такие антитела обозначаются как иммуномодулирующие антитела). Целью последнего типа антител является подавление иммунной резистентности к раковым клеткам, которая может наблюдаться против Т-клеток, которые являются специфическими по отношению к опухолевым антигенам. Действительно, из области техники хорошо известно, что в нормальных физиологических условиях иммунные контрольные точки играют ключевую роль в поддержании аутотолерантности (то есть предотвращают аутоиммунные реакции) и защите тканей от повреждений в тех случаях, когда иммунная система борется с патогенной инфекцией. Однако при раке регуляция экспрессии иммунных контрольных точек может нарушаться, что является важным механизмом иммунной резистентности. Такая резистентность наблюдается при меланоме, раке яичников, раке легких, глиобластоме, раке молочной железы и раке поджелудочной железы, и связана с PD-L1 контрольной точкой (Konishi et al., B7-H1 expression on non-small cell lung cancer cells and its relationship with tumor-infiltrating lymphocytes and their PD-1 expression. Clin Cancer Res. 2004 Aug 1;10(15):5094-100; Ghebeh et al., The B7-H1 (PD-L1) T lymphocyte-inhibitory molecule is expressed in breast cancer patients with infiltrating ductal carcinoma: correlation with important high-risk prognostic factors. Neoplasia. 2006 Mar;8(3):190-8; Hino et al., Tumor cell expression of programmed cell death-1 ligand 1 is a prognostic factor for malignant melanoma. Cancer. 2010 Apr 1;116(7):1757-66). Другие примеры иммунных контрольных точек включают, без ограничений указанными, PD-L2, PD-1, CD80, CD86, CTLA4, B7H3, B7H4, PVR, TIGIT, GAL9, LAG-3, GITR, CD137, TIM3, VISTA, VISTA-R (Pico de Coaña et al., Checkpoint blockade for cancer therapy: revitalizing a suppressed immune system. Trends Mol Med. 2015 Aug;21(8):482-91; Pardoll DM1. The blockade of immune checkpoints in cancer immunotherapy. Nat Rev Cancer. 2012 Mar 22;12(4):252-64).
Для описанных выше целей антитела применяют как в форме изолированных моноклональных антител (т.е. неконъюгированных), так и в форме антител, конъюгированных с другими молекулами, которые могут быть токсичными для клеток или радиоактивными.
Примерами хорошо известных моноклональных противоопухолевых таргетных антител, которые применяются в иммунотерапии рака, являются, без ограничений указанными, алемтузумаб (хронический лимфоцитарный лейкоз), бевацизумаб (колоректальный рак, мультиформная глиобластома, рак шейки матки, рак легких, рак почек), брентуксимаб/ведотин (лимфомы), блинатумумаб (острый лимфобластный лейкоз), катумаксомаб (злокачественный асцит при EPCAM+ опухолях), цетуксимаб (рак головы и шеи, колоректальный рак), деносумаб (рак молочной железы, рак простаты и рак костей), гемтузумаб/озогамицин (острый миелоидный лейкоз), ибритумомаб/тиуксетан (неходжкинская лимфома), панитумумаб (колоректальный рак), пертузумаб (рак молочной железы) обинутузумаб (хронический лимфоцитарный лейкоз), офатумумаб (хронический лимфоцитарный лейкоз), опилимумаб (меланома), рамуцирумаб (рак желудка и гастроэзофагеальный рак), ритуксимаб (хронический лимфоцитарный лейкоз и неходжкинская лимфома), силтуксимаб (многоочаговая болезнь Кастлемана), тозитумомаб (неходжкинская лимфома) и трастузумаб (рак молочной железы, рак желудка и рак пищевода); в то время как примеры иммуномодулирующих антител включают, без ограничений указанными, ипилимумаб (меланома), который блокирует CTLA4-зависимую иммунную контрольную точку, ниволумаб (меланома, рак легкого) и пембролизубмаб (меланома), которые оба блокируют PDCD1-зависимую иммунную контрольную точку, а также MPDL3280A, MEDI4736, MEDI0680, и MSB0010718C, все из которых блокируют PD-L1-зависимую иммунную контрольную точку (Sharma and Allison, The future of immune checkpoint therapy. Science. 2015 Apr 3;348(6230):56-61).
Другие антитела для иммунотерапии рака были описаны Buqué et al. (Buqué et al., Trial Watch: Immunomodulatory monoclonal antibodies for oncological indications. Oncoimmunology. 2015 Mar 2; 4(4):e1008814. eCollection 2015 Apr), Redman et al. (Redman et al., Mechanisms of action of therapeutic antibodies for cancer. Mol Immunol. 2015 Oct;67(2 Pt A):28-45); Simpson and Caballero, Monoclonal antibodies for the therapy of cancer MC Proc. 2014; 8(Suppl 4): О6, а также на веб-сайте, где представлена совокупность антител (список терапевтических моноклональных антител, одобренных или находящихся на рассмотрении в Европейском Союзе или США, доступном по веб-ссылке http://www.antibodysociety.org/news/approved_mabs.php).
Адоптивная клеточная иммунотерапия Т-клетками с химерным антигенным рецептором (CAR) изменила подходы к лечению В-клеточной неходжкинской лимфомы (NHL), особенно агрессивных В-клеточных лимфом. Например, CAR Т-клетки, нацеленные на CD19, представляют собой новый стандарт лечения пациентов с DLBCL, которые не поддаются как минимум двум предшествующим линиям терапии. Два продукта CAR Т-клеток аксикабтаген цилолейцел (акси-цел) (KTE-019) (YESCARTA™) и тисагенлеклейцел (CTL019) (KYMRIAH™) получили одобрение Управления по санитарному надзору за качеством пищевых продуктов и медикаментов США для лечения рефрактерного DLBCL после двух линий терапии. Третий продукт, лизокабтаген маралейцел (лизо-цел) (JCAR017) в настоящее время проходит клинические испытания. Другие CAR T-клетки включают CD20-CAR-T клетки.
Лизаты опухолевых клеток также могут быть комбинированы с антигенным пептидом (пептидами) по настоящему изобретению. Опухолевые клетки действительно способны инициировать иммунный ответ за счет презентации комплексов эндогенных пептидов-MHC, а также посредством дендритных клеток (DC) хозяина, которые могут процессировать и презентировать антиген, доставляемый такими лизатами. Таким образом, увеличивается спектр антигенов, против которых может быть индуцирован иммунный ответ. Лизаты опухолевых клеток могут быть легко получены при обработке опухолевых клеток тепловым шоком/или с помощью химической обработки, кроме того, они могут быть аутологичными (то есть выделенными из организма пациента) или аллогенными (то есть выделенными из организма другого субъекта).
Стандартные химиотерапевтические препараты и радиотерапевтические агенты не нуждаются в дополнительном описании в настоящей заявке, поскольку они подробно описаны в литературе, а именно у Baskar и соавт. (Baskar et al., Cancer and radiation therapy: current advances and future directions. Int J Med Sci. 2012;9(3):193-9), Paci и соавт., (Paci et al., Review of therapeutic drug monitoring of anticancer drugs part 1--cytotoxics. Eur J Cancer. 2014 Aug; 50(12):2010-9) и Widmer и соавт. (Widmer et al., Review of therapeutic drug monitoring of anticancer drugs part two--targeted therapies. Eur J Cancer. 2014 Aug; 50(12):2020-36). Список таких лекарств и агентов также доступен на сайте cancer.gov (http://www.cancer.gov/about-cancer/treatment/drugs).
Предпочтительно, модулятор иммунной контрольной точки, который может применяться в комбинации с антигенным пептидом, как раскрыто в настоящем описании, представляет собой активатор или ингибитор одной или нескольких молекул иммунной контрольной точки, выбранных из CD27, CD28, CD40, CD122, CD137, OX40, GITR, ICOS, A2AR, B7-H3, B7-H4, BTLA, CD40, CTLA-4, IDO, KIR, LAG3, PD-1, TIM-3, VISTA, CEACAM1, GARP, PS, CSF1R, CD94/NKG2A, TDO, GITR, TNFR и/или FasR/DcR3; или активатор или ингибитор одного и нескольких их лигандов.
Более предпочтительно, модулятор иммунной контрольной точки представляет собой активатор (ко)стимулирующей молекулы контрольной точки или ингибитор ингибирующей молекулы контрольной точки, или их комбинацию. Соответственно, модулятор иммунной контрольной точки более предпочтительно представляет собой (i) активатор CD27, CD28, CD40, CD122, CD137, OX40, GITR и/или ICOS, или (ii) ингибитор A2AR, B7-H3, B7-H4, BTLA, CD40, CTLA-4, IDO, KIR, LAG3, PD-1, PDL-1, PD-L2, TIM-3, VISTA, CEACAM1, GARP, PS, CSF1R, CD94/NKG2A, TDO, TNFR и/или FasR/DcR3.
Еще более предпочтительно, модулятор иммунной контрольной точки представляет собой ингибитор ингибирующей молекулы контрольной точки (но предпочтительно не является ингибитором стимулирующей молекулы контрольной точки). Соответственно, модулятор иммунной контрольной точки более предпочтительно представляет собой ингибитор A2AR, B7-H3, B7-H4, BTLA, CTLA-4, IDO, KIR, LAG3, PD-1, PDL-1, PD-L2, TIM-3, VISTA, CEACAM1, GARP, PS, CSF1R, CD94/NKG2A, TDO, TNFR и/или DcR3, или их лиганда.
Также предпочтительно, модулятор иммунной контрольной точки является активатором стимулирующей или костимулирующей молекулы контрольной точки (но предпочтительно не являлся активатором ингибиторной молекулы контрольной точки). Соответственно, модулятор иммунной контрольной точки более предпочтительно представляет собой активатор CD27, CD28, CD40, CD122, CD137, OX40, GITR и/или ICOS, или их лигандов.
Еще более предпочтительно модулятор иммунной контрольной точки является модулятором CD40 каскада, IDO каскада, LAG3 каскада, CTLA-4 каскада и/или PD-1 каскада. В частности, модулятор иммунной контрольной точки предпочтительно представляет собой модулятор CD40, LAG3, CTLA-4, PD-L1, PD-L2, PD-1 и/или IDO, более предпочтительно модулятор иммунной контрольной точки представляет собой ингибитор CTLA-4, PD-L1, PD-L2, PD-1, LAG3 и/или IDO или активатор CD40, еще более предпочтительно модулятор иммунной контрольной точки представляет собой ингибитор CTLA-4, PD-L1, PD-1, LAG3 и/или IDO, еще более предпочтительно модулятор иммунной контрольной точки представляет собой ингибитор LAG3, CTLA-4 и/или PD-1, и наиболее предпочтительно модулятор иммунной контрольной точки представляет собой ингибитор CTLA-4 и/или PD-1.
Таким образом, модулятор контрольной точки для комбинации с антигенным пептидом может быть выбран из известных модуляторов CTLA-4 каскада или PD-1 каскада. Предпочтительно модулятор контрольной точки для комбинации с антигенным пептидом, как раскрыто в настоящем описании, может быть выбран из известных модуляторов CTLA-4 каскада или PD-1 каскада. Особо предпочтительно модулятор иммунной контрольной точки представляет собой ингибитор PD-1. Предпочтительные ингибиторы CTLA-4 каскада или PD-1 каскада включают моноклональные антитела Yervoy® (Ипилимумаб; Bristol Myers Squibb) и Тремелимумаб (Pfizer/MedImmune) а также Opdivo® (Ниволумаб; Bristol Myers Squibb), Keytruda® (Пембролизумаб; Merck), Пидилизумаб (CT-011; CureTech), MEDI0680 (AMP-514; AstraZeneca), Bavencio® (Авелумаб; Merck KGaA/Pfizer, также известный как MSB-0010718C), MIH1 (Affymetrix), LY3300054 (Eli Lilly) и Спартализумаб (также известный как PDR001; Novartis). Более предпочтительными ингибиторами контрольных точек являются ингибиторы CTLA-4, такие как Yervoy® (Ипилимумаб; Bristol Myers Squibb) и Тремелимумаб (Pfizer/MedImmune), а также ингибиторы PD-1 Opdivo® (Ниволумаб; Bristol Myers Squibb), Keytruda® (Пембролизумаб; Merck), Пидилизумаб (CT-011; CureTech), MEDI0680 (AMP-514; AstraZeneca), AMP-224 (PD-L2 Fc гибридный белок; MedImmune).
Также предпочтительно модулятор иммунной контрольной точки для комбинации с антигенным пептидом, как определено в настоящем описании, выбирают из группы, состоящей из Пембролизумаба, Ипилимумаба, Ниволумаба, Атезолизумаба, MEDI4736, Тремелимумаба, Авелумаба, Спартализумаба, LAG525 (анти-AG-3 моноклонального антитела), Эпакадостата (также известного как INCB24360; ингибитор IDO), Варлилумаба (анти-CD27 моноклонального антитела), Урелумаба (анти-CD137 моноклонального антитела), AMP-224 и CM-24 (анти-CEACAM1 моноклонального антитела).
Выбор подходящего иммунного противоракового терапевтического агента для целей настоящего изобретения находится в компетенции специалиста в данной области техники.
Противораковый терапевтический агент также может быть введен вместе с антигенным пептидом по настоящему изобретению, иммуногенным соединением по настоящему изобретению, наночастицей по настоящему изобретению, клеткой по настоящему изобретению, нуклеиновой кислотой по настоящему изобретению, клеткой-хозяином по настоящему изобретению или фармацевтической композицией по настоящему изобретению, как в одно и то же время, так и последовательно, как раскрыто в настоящей заявке, а также в составе как одной, так и нескольких разных фармацевтических форм. Таким образом, изобретение предлагает комбинированное применение заявленной композиции и по меньшей мере одного противоракового терапевтического агента, как раскрыто выше, для одновременного, отдельного или последовательного введения, как раскрыто в настоящем описании.
Кроме того, настоящее изобретение также относится к комбинации по меньшей мере двух различных антигенных пептидов по настоящему изобретению, например, для применения с целью профилактики и лечения В-клеточного гемобластоза. Кроме того, настоящее изобретение также относится к комбинации по меньшей мере двух различных иммуногенных соединений по настоящему изобретению, например, для применения с целью профилактики и/или лечения В-клеточного гемобластоза. Кроме того, настоящее изобретение также относится к комбинации по меньшей мере двух различных наночастиц по настоящему изобретению, например, для применения с целью профилактики и/или лечения В-клеточного гемобластоза. Кроме того, настоящее изобретение также относится к комбинации по меньшей мере двух различных нуклеиновых кислот по настоящему изобретению, например, для применения с целью профилактики и/или лечения В-клеточного гемобластоза.
Таким образом, согласно предпочтительному варианту осуществления изобретения по меньшей мере два различных антигенных пептида по настоящему изобретению могут применяться в комбинации, например, в составе одной фармацевтической композиции. Например, по меньшей мере 3 антигенных пептида, по меньшей мере 4 антигенных пептида, по меньшей мере 5 антигенных пептидов, по меньшей мере 6 антигенных пептидов, по меньшей мере 7 антигенных пептидов, по меньшей мере 8 антигенных пептидов, по меньшей мере 9 антигенных пептидов, по меньшей мере 10 антигенных пептидов, по меньшей мере 11 антигенных пептидов, по меньшей мере 12 антигенных пептидов, по меньшей мере 13 антигенных пептидов, по меньшей мере 14 антигенных пептидов, по меньшей мере 15 антигенных пептидов, по меньшей мере 20 антигенных пептидов, по меньшей мере 25 антигенных пептидов, по меньшей мере 50 антигенных пептидов, по меньшей мере 100 антигенных пептидов применяют в комбинации, например, в составе одной и той же фармацевтической композиции. В рамках компетенции специалиста в данной области находится выбор комбинации антигенных пептидов, которая является подходящей для предусмотренной цели.
В особо предпочтительном варианте осуществления изобретения два различных антигенных пептида по настоящему изобретению (например, имеющих отношение к одному и тому же типу В-клеточного гемобластоза и/или одному и тому же эталонному антигену) применяют в комбинации. Например,
(i) по меньшей мере два различных иммуногенных соединения по настоящему изобретению;
(ii) по меньшей мере два различных антигенных пептида по настоящему изобретению;
(iii) по меньшей мере два различных наночастицы по настоящему изобретению; или
(iv) по меньшей мере две различных нуклеиновых кислоты по настоящему изобретению
могут применяться в комбинации.
Например, настоящее изобретение относится к комбинации по меньшей мере двух различных антигенных пептидов по настоящему изобретению, в частности:
(i) первого антигенного пептида по настоящему изобретению, и
(ii) второго антигенного пептида по настоящему изобретению (отличного от первого), предпочтительно для применения с целью профилактики и/или лечения В-клеточного гемобластоза.
Например, настоящее изобретение обеспечивает комбинацию по меньшей мере двух различных иммуногенных соединений по настоящему изобретению, в частности
(i) иммуногенного соединения по настоящему изобретению, включающего первый антигенный пептид по настоящему изобретению, и
(ii) иммуногенного соединения по настоящему изобретению, включающего второй антигенный пептид по настоящему изобретению (отличный от первого),
предпочтительно для применения с целью профилактики и/или лечения В-клеточного гемобластоза.
Например, настоящее изобретение обеспечивает комбинацию по меньшей мере двух различных наночастиц по настоящему изобретению, в частности
(i) наночастицы по настоящему изобретению, включающей первый антигенный пептид по настоящему изобретению, и
(ii) наночастицы по настоящему изобретению, включающей второй антигенный пептид по настоящему изобретению (отличный от первого),
предпочтительно для применения с целью профилактики и/или лечения В-клеточного гемобластоза.
Например, настоящее изобретение обеспечивает комбинацию по меньшей мере двух различных нуклеиновых кислот по настоящему изобретению, в частности
(i) нуклеиновой кислоты по настоящему изобретению, включающей полинуклеотид, кодирующей первый антигенный пептид по настоящему изобретению, и
(ii) нуклеиновой кислоты по настоящему изобретению, включающей полинуклеотид, кодирующий второй антигенный пептид по настоящему изобретению (отличный от первого),
предпочтительно для применения с целью профилактики и/или лечения В-клеточного гемобластоза.
Например, настоящее изобретение обеспечивает комбинацию по меньшей мере двух различных цитотоксических Т-лимфоцитов по настоящему изобретению, в частности
(i) цитотоксического Т-лимфоцита по настоящему изобретению, специфичного к первому антигенному пептиду по настоящему изобретению, и
(ii) цитотоксического Т-лимфоцита по настоящему изобретению, специфичного ко второму антигенному пептиду по настоящему изобретению (отличному от первого),
предпочтительно для применения в профилактике и/или лечении В-клеточного гемобластоза.
В комбинациях по настоящему изобретению предпочтительными являются такие комбинации компонентов, как антигенные пептиды по настоящему изобретению, которые соответствуют предпочтительным комбинациям компонентов, таких как антигенные пептиды по настоящему изобретению, входящие в состав фармацевтической композиции, как описано выше.
Кроме того, антигенный пептид по настоящему изобретению также можно комбинировать с соответствующим (человеческим) эпитопом опухолевого антигена (как описано выше в отношении «семейств» пептидов). Таким образом, достигается/поддерживается отбор клонов Т-клеток, которые очень эффективны против опухоли. В частности, антигенный пептид по настоящему изобретению и соответствующий (человеческий) эпитоп опухолевого антигена можно вводить совместно. Такое совместное введение может быть осуществлено примерно в одно и то же время (одновременно) или последовательно, при этом при последовательном введении предпочтительно вводить вначале антигенный пептид по настоящему изобретению, а затем вводить соответствующий (человеческий) эпитоп опухолевого антигена. В частности, антигенный пептид по настоящему изобретению может быть введен первым, а соответствующий эпитоп опухолевого антигена (человека) может быть использован в качестве повторной стимуляции. Например, антигенный пептид в соответствии с SEQ ID № 10 можно комбинировать с эталонным пептидом в соответствии с SEQ ID № 258. В другом примере антигенный пептид в соответствии с SEQ ID № 110 можно комбинировать с эталонным пептидом в соответствии с SEQ ID № 270. В другом примере антигенный пептид в соответствии с SEQ ID № 114 может быть объединен с эталонным пептидом в соответствии с SEQ ID № 271. В другом примере антигенный пептид в соответствии с SEQ ID № 220 может быть объединен с эталонным пептидом в соответствии с SEQ ID № 279.
Пептиды, которые можно комбинировать, такие как (а) антигенный пептид по настоящему изобретению и соответствующий эпитоп (человеческого) опухолевого антигена, или (b) два различных антигенных пептида по настоящему изобретению, можно применять:
- в одном иммуногенном соединении по настоящему изобретению или в различных иммуногенных соединениях по настоящему изобретению,
- (нагруженными) на одну наночастицу по настоящему изобретению или на различные наночастицы по настоящему изобретению,
- (нагруженными) в одну клетку по настоящему изобретению или в различные клетки по настоящему изобретению,
- (закодированными) в одной молекуле нуклеиновой кислоты по настоящему изобретению или в различных молекулах нуклеиновой кислоты по настоящему изобретению,
- (экспрессируемыми) одной клеткой-хозяином по настоящему изобретению или различными клетками-хозяевами по настоящему изобретению, или
- (включенными) в одну фармацевтическую композицию по настоящему изобретению или в различные фармацевтические композиции по настоящему изобретению.
Как правило, выражение «два отдельных компонента» в контексте комбинации, т.е. для применения по настоящему изобретению (комбинированной терапии) определяется, как указано выше (в контексте фармацевтической композиции). В частности, это относится к:
(1) первому компоненту, такому как антигенный пептид по настоящему изобретению, как описано в настоящей заявке, иммуногенное соединение по настоящему изобретению, как описано в настоящей заявке, наночастица по настоящему изобретению, как описано в настоящей заявке, клетка согласно настоящему изобретению, как описано в настоящей заявке, нуклеиновая кислота по настоящему изобретению, как описано в настоящей заявке, клетка-хозяин по настоящему изобретению, как описано в настоящей заявке, или фармацевтическая композиция в соответствии с настоящим изобретением, как описано в настоящей заявке; и
(2) второму компоненту (отличному от первого компонента), такому как противораковый терапевтический агент, как описано выше, отдельный антигенный пептид по настоящему изобретению, как описано в настоящей заявке, отдельное иммуногенное соединение по настоящему изобретению, как описано в настоящей заявке, отдельная наночастица по настоящему изобретению, как описано в настоящей заявке, отдельная клетка по настоящему изобретению, как описано в настоящей заявке, отдельная нуклеиновая кислота по настоящему изобретению, как описано в настоящей заявке, отдельная клетка-хозяин по настоящему изобретению, как описано в настоящей заявке, отдельная фармацевтическая композиция по настоящему изобретению, как описано в настоящей заявке, или один или несколько (фрагментов) опухолевых антигенов человека в любой форме («изолированных», в качестве иммуногенного соединения, как описано в настоящей заявке, в виде наночастицы, как описано в настоящей заявке, в виде клетки (хозяина), как описано в настоящей заявке, в виде нуклеиновой кислоты, как описано в настоящей заявке, или в виде фармацевтической композиции, как описано в настоящей заявке).
Соответственно, «два отдельных компонента», как упоминается в настоящей заявке в контексте комбинации для применения в соответствии с настоящим изобретением (комбинированная терапия), предпочтительно являются активными компонентами в контексте заболевания (В-клеточного гемобластоза), которое необходимо предотвратить и/или лечить. Другими словами, каждый из по меньшей мере двух отдельных компонентов также может быть полезен для профилактики и/или лечения указанного рака, если его вводить по отдельности (не в комбинации, как описано в настоящей заявке), хотя комбинация (т.е. комбинированное введение) обычно усиливает их профилактический и /или терапевтический эффект (такой как иммунный ответ), в частности, синергическим образом.
Соответственно, настоящее изобретение также обеспечивает комбинацию (по меньшей мере) двух различных антигенных пептидов по настоящему изобретению, как описано в настоящей заявке. В этом контексте (по меньшей мере) два различных антигенных пептида могут быть в любой форме, например, «изолированными», состоять из иммуногенных соединений, наночастиц, (фармацевтических) композиций или клеток, нагруженных ими, или кодироваться нуклеиновыми кислотами (например, векторами). Соответственно, (по меньшей мере) два различных антигенных пептида могут состоять (по меньшей мере) из двух отдельных компонентов (которые должны быть объединены). В предпочтительном варианте осуществления по меньшей мере два отдельных компонента комбинации по настоящему изобретению представляют собой по меньшей мере разные антигенные пептиды по настоящему изобретению (в любой форме, например, содержащиеся в иммуногенных соединениях, наночастицах, клетках, фармацевтических композициях, кодируемые нуклеиновыми кислотами и др.).
Предпочтительно, по меньшей мере два различных компонента комбинации для применения по настоящему изобретению относятся к одному и тому же типу рака, например, к одним и тем же или различным антигенам, ассоциированным с этим раком, и/или к одним и тем же или различным (эталонным) эпитопам в пределах антигена, ассоциированного с этим раком. Более предпочтительно по меньшей мере два различных компонента относятся к одному и тому же опухолевому антигену.
В некоторых вариантах осуществления по меньшей мере два отдельных компонента комбинации для применения в соответствии с настоящим изобретением входят в состав одной и той же или разных композиций. В некоторых вариантах осуществления по меньшей мере два отдельных компонента комбинации для применения согласно настоящему изобретению вводят одним и тем же или разными путями введения. В некоторых вариантах осуществления по меньшей мере два отдельных компонента комбинации для применения согласно настоящему изобретению вводят примерно в одно и то же время (одновременно) или последовательно.
Предпочтительно, по меньшей мере два отдельных компонента комбинации для применения по настоящему изобретению вводят примерно в одно и то же время. В более общем плане предпочтительно, чтобы первый компонент был введен примерно в то же время, что и второй компонент, при этом по меньшей мере два различных компонента комбинации для применения согласно настоящему изобретению предпочтительно вводят в одной и той же форме (т.е. в составе того же типа, например, в виде наночастиц, в виде фармацевтических композиций и т.д.).
Выражение «примерно в одно и то же время», как используется в настоящей заявке, означает, в частности, одновременное введение или то, что сразу после введения первого компонента вводят второй компонент, или сразу после введения второго компонента вводят первый компонент. Специалисту в данной области техники понятно, что «непосредственно после» включает время, необходимое для подготовки второго введения, в частности время, необходимое для обнажения и дезинфекции места для второго введения, а также для соответствующей подготовки «устройства для введения» (например, шприца, насоса и др.). Одновременное введение также включает варианты, если периоды введения первого компонента и второго компонента перекрываются или если, например, один компонент вводят в течение более длительного периода времени, такого как 30 минут, 1 час, 2 часа или даже более, например, путем инфузии, а другой компонент вводят в какое-то время в течение такого длительного периода. Введение первого компонента и второго компонента примерно в одно и то же время является особо предпочтительным, если используют разные пути введения и/или разные места введения.
Также предпочтительно по меньшей мере два отдельных компонента комбинации для применения согласно настоящему изобретению вводят последовательно. В более общем плане предпочтительно первый компонент и второй компонент вводят последовательно, при этом по меньшей мере два различных компонента комбинации для применения согласно настоящему изобретению предпочтительно вводят в одной и той же форме (т.е. в виде одного и того же типа состава, например, в виде наночастиц, в виде фармацевтических композиций и т. д.).
Это означает, что первый компонент применяют до или после второго компонента. При последовательном введении время между введением первого компонента и введением второго компонента предпочтительно составляет не более одной недели, более предпочтительно не более 3 дней, еще более предпочтительно не более 2 дней и наиболее предпочтительно не более 24 часов. Особо предпочтительно, первый компонент и второй компонент вводят в один и тот же день, при этом время между введением первого компонента и введением второго компонента предпочтительно составляет не более 6 часов, более предпочтительно не более 3 часов, еще более предпочтительно не более 2 часов, и наиболее предпочтительно не более 1 часа.
Предпочтительно, первый компонент и второй компонент применяют одним и тем же путем введения. В более общем плане предпочтительно, первый компонент и второй компонент вводят одним и тем же путем введения, при этом по меньшей мере два различных компонента комбинации для применения согласно настоящему изобретению предпочтительно вводят в одной и той же форме (т.е. в составе того же типа, например, в виде наночастиц, в виде фармацевтических композиций и т.д.).
Также предпочтительно, по меньшей мере два различных компонента комбинации для применения согласно настоящему изобретению вводят различными путями введения. В более общем плане предпочтительно первый компонент и второй компонент вводят различными путями введения, при этом по меньшей мере два различных компонента комбинации для применения согласно настоящему изобретению предпочтительно вводят в одной и той же форме (т.е. в составе того же типа, например, в виде наночастиц, в виде фармацевтических композиций и т.д.).
Предпочтительно, по меньшей мере два отдельных компонента комбинации для применения согласно настоящему изобретению содержатся в одной и той же композиции. В более общем плане предпочтительно, первый компонент и второй компонент содержатся в одной и той же композиции, при этом по меньшей мере два различных компонента комбинации для применения согласно настоящему изобретению предпочтительно вводят в одной и той же форме (т.е. в составе того же типа, например, в виде наночастиц и т. д.).
Также предпочтительно, по меньшей мере два отдельных компонента комбинации для применения согласно настоящему изобретению содержатся в отдельных композициях. В более общем плане предпочтительно, первый компонент и второй компонент содержатся в различных композициях, при этом по меньшей мере два различных компонента комбинации для применения согласно настоящему изобретению предпочтительно вводят в одной и той же форме (т.е. в одном и том же типе препарата, например, в виде наночастиц и т.д.).
В частности, настоящее изобретение обеспечивает фармацевтическую композицию, содержащую первый антигенный пептид по настоящему изобретению, который включает или состоит из варианта последовательности фрагмента опухолевого антигена человека.
В частности, настоящее изобретение обеспечивает комбинацию, например, для применения в профилактике и/или лечении В-клеточного гемобластоза, включающую первый антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности микробиоты фрагмента опухолевого антигена CD22 человека, и второй антигенный пептид по настоящему изобретению, который содержит или состоит из варианта последовательности фрагмента опухолевого антигена человека TNFRSF13C. Предпочтительно, первый антигенный пептид содержит или состоит из варианта последовательности фрагмента CD22 (эталонного пептида человека) «WVFEHPETL» (SEQ ID № 270), такого как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№: 106-110, 316 и 387-390, а второй антигенный пептид содержит или состоит из варианта последовательности фрагмента TNFRSF13C (эталонного пептида человека) «LLFGAPALL» (SEQ ID № 279), такого как антигенный пептид, содержащий или состоящий из аминокислотной последовательности, как указано в SEQ ID №№ 220, 325 и 450. Более предпочтительно, первый антигенный пептид содержит или состоит из аминокислотной последовательности, как указано в SEQ ID №№ 110, 387 и 390, а второй антигенный пептид содержит или состоит из аминокислотной последовательности, указанной в SEQ ID №№ 220 и 450. Еще более предпочтительно, фармацевтическая композиция включает антигенный пептид, содержащий или состоящий из SEQ ID № 110, и антигенный пептид, содержащий или состоящий из SEQ ID № 220.
Более предпочтительно, комбинация по настоящему изобретению (например, для применения для профилактики и/или лечения В-клеточного гемобластоза) содержит по меньшей мере три различных компонента, как описано выше, в частности по меньшей мере три различных антигенных пептида по настоящему изобретению. Приведенное выше описание комбинации двух отдельных компонентов применимо, соответственно, к трем отдельным компонентам.
Наиболее предпочтительно, комбинация по настоящему изобретению (например, для применения для профилактики и/или лечения В-клеточного гемобластоза) содержит по меньшей мере четыре различных компонента, как описано выше, в частности по меньшей мере четыре различных антигенных пептида по настоящему изобретению. Приведенное выше описание комбинации двух отдельных компонентов применимо, соответственно, к четырем отдельным компонентам.
Понятно, что комбинация, например, для применения в профилактике и/или лечении В-клеточного гемобластоза, может также содержать вместо вышеописанных предпочтительных комбинаций антигенных пептидов соответствующую комбинацию иммуногенных соединений по изобретению, соответствующую комбинацию наночастиц по изобретению или соответствующую комбинацию нуклеиновых кислот по изобретению.
Краткое описание чертежей
Далее представлено краткое описание прилагаемых к заявке фигур. Фигуры предназначены для более детальной иллюстрации настоящего изобретения. Однако они ни коим образом не ограничивают сущность изобретения.
Фигура 1 показывает для Примера 1 аффинность in vitro к антигенному пептиду CD22-B1 по сравнению с соответствующим эпитопом человеческого CD22 CD22-H1.
Фигура 2 показывает для Примера 1 аффинность in vitro для антигенного пептида CD37-B1 по сравнению с соответствующим эпитопом человеческого CD37 CD37-H1.
Фигура 3 показывает для Примера 1 аффинность in vitro для антигенного пептида CD19-B1 по сравнению с соответствующим эпитопом человеческого CD19 CD19-H1.
Фигура 4 показывает для Примера 1 аффинность in vitro для антигенного пептида CD19-B2 по сравнению с соответствующим эпитопом человеческого CD19 CD19-H2.
Фигура 5 показывает для Примера 1 аффинность in vitro для антигенного пептида TNFRSF13C-B1 по сравнению с соответствующим эпитопом человеческого TNFRSF13C TNFRSF13C-H1.
Фигура 6 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR1 HLA-A2, вакцинированных антигенным пептидом CD22-B1, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом CD22-H1. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 7 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR1 HLA-A2, вакцинированных антигенным пептидом TNFRSF13C-B1, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом TNFRSF13C-H1. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 8 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR1 HLA-A2, вакцинированных антигенным пептидом CD37-B1, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом CD37-H1. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 9 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR1 HLA-A2, вакцинированных антигенным пептидом CD19-B2, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом CD19-H2. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 10 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR1 HLA-A2, вакцинированных антигенным пептидом CD19-B1, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом CD19-H1. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 11 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR3 HLA-A2, вакцинированных антигенным пептидом CD22-B1, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом CD22-H1. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 12 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR3 HLA-A2, вакцинированных антигенным пептидом TNFRSF13C-B1, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом TNFRSF13C-H1. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 13: показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR3 HLA-A2, вакцинированных антигенным пептидом CD37-B1, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом CD37-H1. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 14 показывает для Примера 1 аффинность in vitro для антигенного пептида MS4A1-B4 по сравнению с соответствующим эпитопом MS4A1 человека MS4A1-H4.
Фигура 15 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR1 HLA-A2, вакцинированных антигенным пептидом MS4A1-B4, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом MS4A1-H4. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 16 показывает для Примера 2 результаты ELISPOT для трансгенных мышей HHD DR3 HLA-A2, вакцинированных антигенным пептидом MS4A1-B4, как показано на фигуре, и перекрестную реактивность с соответствующим человеческим пептидом MS4A1-H4. Данные были представлены как количество пятен на 1х106 общих Т-клеток.
Фигура 17 показывает для Примера 3 детекцию CD8+ T-клеток, специфичных к пептиду CD22-B1, CD37B1, TNFRSF13C-B1 и MS4A1-B4, обнаруженных в периферической крови здоровых доноров (HLA-A2-позитивных).
Фигура 18 показывает для Примера 3 цитотоксическую способность CD22-B1, CD37B1, TNFRSF13C-B1 и MS4A1-B4 пептид-специфического клона Т-клеток человека, размноженных in vitro путем стимуляции пептидом, полученным из микробиома. Т-клетки, специфичные к пептидам CD22-B1, CD37B1, TNFRSF13C-B1 и MS4A1-B4, обладают способностью убивать Т-клетки, нагруженные бактериальными или человеческими пептидами.
Фигура 19 показывает для Примера 4 аффинность in vitro для антигенных пептидов CD22-B1, CD22-B12 и CD22-B13 по сравнению с соответствующим эпитопом CD22 CD22-H1 человека.
Фигура 20 показывает для Примера 4 аффинность in vitro для антигенных пептидов CD37-B1, CD37-B12, CD37-B13, CD37-B14 и CD37-B15 по сравнению с соответствующим эпитопом CD37 CD37-H1 человека.
Фигура 21 показывает для Примера 4 аффинность in vitro для антигенных пептидов TNFRSF13C-B1, TNFRSF13C-B11, TNFRSF13C-B12 и TNFRSF13C-B13 по сравнению с соответствующим эпитопом человеческого TNFRSF13C TNFRSF13C-H1.
Фигура 22 показывает для Примера 4 аффинность in vitro для антигенных пептидов MS4A1-B4, MS4A1-B42 и MS4A1-B43 по сравнению с соответствующим эпитопом MS4A1 человека MS4A1-H4.
Примеры
Далее представлены конкретные примеры, иллюстрирующие различные варианты осуществления и аспекты изобретения. Однако объем настоящего изобретения не должен ограничиваться конкретными вариантами осуществления, описанными в настоящей заявке. Следующие препараты и примеры даны для того, чтобы специалисты в данной области техники могли более четко понять и применить на практике настоящее изобретение. Однако объем настоящего изобретения не ограничивается приведенными в качестве примеров вариантами осуществления, которые предназначены только для иллюстрации отдельных аспектов изобретения, и способы, которые функционально эквивалентны, входят в объем изобретения. Действительно, различные модификации изобретения в дополнение к тем, которые описаны здесь, станут очевидными для специалистов в данной области техники из приведенного выше описания, сопроводительных фигур и приведенных ниже примеров. Все такие модификации входят в объем прилагаемой формулы изобретения.
ПРИМЕР 1: Антигенные пептиды обладают превосходной аффинностью к аллелю HLA-A*0201.
Затем аффинность связывания различных выбранных антигенных пептидов и соответствующих фрагментов опухолевых антигенов человека (эталонных пептидов человека) с аллелем HLA-A*0201 была подтверждена in vitro. А именно, антигенный пептид с последовательностью SEQ ID № 110 («YIFEHPELL», также обозначаемый здесь как CD22-B1) сравнивали с соответствующими эталонными пептидами человека, полученными из CD22 («WVFEHPETL», SEQ ID № 270, также обозначаемыми здесь как CD22-H1). Кроме того, антигенный пептид с последовательностью SEQ ID № 109 («YVFEHPELL», также обозначаемый здесь как CD22-B11) сравнивали с соответствующим эталонным пептидом человека, полученным из CD22 («WVFEHPETL», SEQ ID № 270, также обозначаемым здесь как CD22-H1). Кроме того, антигенный пептид с последовательностью SEQ ID № 114 («FLAFVPLQL», также обозначаемый здесь как CD37-B1) сравнивали с соответствующим эталонным пептидом человека, полученным из CD37 («GLAFVPLQI», SEQ ID № 271, также обозначаемым здесь как CD37-H1). Далее, антигенный пептид с последовательностью SEQ ID № 117 («GMAFVPLLL», также обозначаемый здесь как CD37-B11) сравнивали с соответствующим эталонным пептидом человека, полученным из CD37 («GLAFVPLQI», SEQ ID № 271, также обозначаемым здесь как CD37-H1). Далее, антигенный пептид с последовательностью SEQ ID № 34 («LLVGILHLV», также обозначаемый здесь как CD19-B1) сравнивали с соответствующим эталонным пептидом человека, полученным из CD19 («SLVGILHLQ», SEQ ID № 260, также обозначаемым здесь как CD19-H1). Далее, антигенный пептид с последовательностью SEQ ID № 10 («TLLFLTPML», также обозначаемый здесь как CD19-B2) сравнивали с соответствующим эталонным пептидом человека, полученным из CD19 («FLLFLTPME», SEQ ID № 258, также обозначаемым здесь как CD19-H2). Далее, антигенный пептид с последовательностью SEQ ID № 39 («YLAYLIFEL», также обозначаемый здесь как CD19-B6) сравнивали с соответствующим эталонным пептидом человека, полученным из CD19 («TLAYLIFCL», SEQ ID № 261, также обозначаемым здесь как CD19-H6). Далее, антигенный пептид с последовательностью SEQ ID № 40 («LQMGGFYLL», также обозначаемый здесь как CD19-B7) сравнивали с соответствующим эталонным пептидом человека, полученным из CD19 («QQMGGFYLC», SEQ ID № 262, также обозначаемым здесь как CD19-H7). Далее, антигенный пептид с последовательностью SEQ ID № 220 («LMFGAPALV», также обозначаемый здесь как TNFRSF13C-B1) сравнивали с соответствующим эталонным пептидом человека, полученным из TNFRSF13C («LLFGAPALL», SEQ ID № 279, также обозначаемым здесь как TNFRSF13C-H1). Далее, антигенный пептид с последовательностью SEQ ID № 231 («ILPGLLFGL», также обозначаемый здесь как TNFRSF13C-B31) сравнивали с соответствующим эталонным пептидом человека, полученным из TNFRSF13C («PLPGLLFGA», SEQ ID № 280, также обозначаемым здесь как TNFRSF13C-H3). Далее, антигенный пептид с последовательностью SEQ ID № 227 («FMPGLLFGA», также обозначаемый здесь как TNFRSF13C-B33) сравнивали с соответствующим эталонным пептидом человека, полученным из TNFRSF13C («PLPGLLFGA», SEQ ID № 280, также обозначаемым здесь как TNFRSF13C-H3). Далее, антигенный пептид с последовательностью SEQ ID № 61 («YILGGLLMV», также обозначаемый здесь как MS4A1-B12) сравнивали с соответствующим эталонным пептидом человека, полученным из MS4A1 (также известным как CD20) («IALGGLLMI», SEQ ID №: 263, также обозначаемый здесь как MS4A1-H1). Далее, антигенный пептид с последовательностью SEQ ID № 72 («ILIPAGIYL», также обозначаемый здесь как MS4A1-B3) сравнивали с соответствующим эталонным пептидом человека, полученным из MS4A1 (также известным как CD20) («LMIPAGIYA», SEQ ID № 265, также обозначаемый здесь как MS4A1-H3). Далее, антигенный пептид с последовательностью SEQ ID № 65 («AMNSLSLYI», также обозначаемый здесь как MS4A1-B4) сравнивали с соответствующим эталонным пептидом человека, полученным из MS4A1 (также известным как CD20) («IMNSLSLFA», SEQ ID № 264, также обозначаемым здесь как MS4A1-H4). Далее, антигенный пептид с последовательностью SEQ ID № 86 («YLFLGILSL», также обозначаемый здесь как MS4A1-B5) сравнивали с соответствующим эталонным пептидом человека, полученным из MS4A1 (также известным как CD20) («SLFLGILSV», SEQ ID № 266, также обозначаемым здесь как MS4A1-H5).
А. Материалы и методы
А1. Измерение аффинности пептида к клеточной линии Т2.
Экспериментальный протокол аналогичен тому, который был утвержден для пептидов, представленных HLA-A*0201 (Tourdot et al., A general strategy to enhance immunogenicity of low-affinity HLA-A2.1-associated peptides: implication in the identification of cryptic tumor epitopes. Eur J Immunol. 2000 Dec; 30(12):3411-21). Измерение аффинности пептидов достигается с помощью опухолевых клеток человека Т2, которые экспрессируют молекулу HLA-A*0201, но являются TAP1/2-отрицательными и неспособными презентировать эндогенные пептиды.
Клетки T2 (5х104 клеток на лунку) инкубировали с уменьшающимися концентрациями пептидов от 100 мкМ до 0,1 мкМ (4 концентрации: 100 мкМ, 10 мкМ, 1 мкМ, 0,1 мкМ) в бессывороточной среде (TexMacs) с добавлением 100 нг/мкл β2-микроглобулина при 37°С в течение 16 часов. Затем клетки дважды промывали и метили анти-HLA-A2-антителом, связанным с PE (клон BB7.2, BD Pharmagen).
Анализ осуществляли с помощью FACS (анализатор Macsquant 10-Miltenyi).
Для каждой концентрации пептида среднее геометрическое значение метки, связанной с интересующим пептидом, вычитали из фонового значения и представляли в виде процента от среднего геометрического мечения HLA-A*0202, полученного для эталонного пептида HIV pol 589-597 при концентрации 100 мкМ. Затем относительную аффинность определяли следующим образом:
Относительная аффинность = концентрация каждого пептида, индуцирующая 20% экспрессии HLA-A*0201/концентрация эталонного пептида, индуцирующая 20% экспрессии HLA-A*0201.
А2. Солюбилизация пептидов
Каждый пептид солюбилизирован с учетом аминокислотного состава. Для пептидов, не содержащих цистеин, метионин или триптофан, возможно добавление ДМСО до 10% от общего объема. Другие пептиды ресуспендировали в воде или PBS с pH 7,4.
В. Результаты
Средние значения относительной интенсивности флуоресценции (данные нормированы на среднюю флуоресценцию пептида ВИЧ, т.е. значение 100 равно наилучшему связыванию, наблюдаемому с пептидом ВИЧ) клеток Т2, полученные для различных концентраций каждого пептида, показаны в Таблице 2 ниже.
Таблица 2
В приведенной ниже Таблице 3 для каждого тестируемого пептида приведены концентрации, необходимые для индукции 20% экспрессии HLA-A2, и аффинность связывания in vitro (* нормализовано по отношению к концентрации пептида HIV-pol, вызывающей 20% экспрессии HLA-A2, проведенной в ходе того же эксперимента).
Таблица 3
ND – не определяется
Кроме того, на Фигурах 1-5 и 14 показаны результаты для выбранных примеров, а именно для антигенного пептида CD22-B1 в сравнении с соответствующим фрагментом CD22 человека CD22-H1 (Фигура 1), для антигенного пептида CD37-B1 в сравнении с соответствующим фрагментом CD37 человека CD37-H1 (Фигура 2), для антигенного пептида CD19-B1 в сравнении с соответствующим фрагментом CD19 человека CD19-H1 (Фигура 3), для антигенного пептида CD19-B2 в сравнении с соответствующим фрагментом CD19 CD19-H2 человека (Фигура 4), для антигенного пептида TNFRSF13C-B1 в сравнении с соответствующим фрагментом TNFRSF13C человека TNFRSF13C-H1 (Фигура 5), и для антигенного пептида MS4A1-B4 в сравнении с соответствующим фрагментом MS4A1 человека MS4A1-B4 (Фигура 14).
Таким образом, результаты показывают, что антигенные пептиды по настоящему изобретению демонстрируют по меньшей мере такую же аффинность связывания с HLA-A*0201, как и соответствующие фрагменты опухолевого антигена человека. В большинстве случаев аффинность связывания, наблюдаемая для антигенных пептидов по настоящему изобретению, была выше, чем у соответствующих человеческих эпитопов. Не углубляясь в какую-либо теорию, можно предположить, что такая сильная аффинность связывания антигенных пептидов по настоящему изобретению отражает их способность вызывать иммунный ответ (т.е. их иммуногенность).
ПРИМЕР 2: Иммуногенность CD22-B1, CD19B1, CD19-B2, CD37B1, TNFRSF13C-B1 и MS4A1-B4 у трансгенных мышей HLA-A2 и перекрестная реактивность с соответствующим пептидом человека.
А. Материалы и методы
A.1 Мышиная модель
Вкратце, гуманизированных мышей HLA-A2 HHD-DR1 (C57BL/6JB2mtm1UncIAb-/-Tg(HLA-DRA,HLA-DRB1*0101)#GjhTg(HLA-A/H2-D/B2M)1Bpe) или гуманизированных мышей HHD-DR3 (C57BL/6JB2mtm1UncIAb-/-Tg(HLA-DRA,HLA-DRB1*0301)#GjhTg(HLA-A/H2-D/B2M)1Bpe) случайным образом (в зависимости от пола и возраста мышей) распределяли по экспериментальным группам, где каждую группу иммунизировали специфическим вакцинным пептидом (vacc-pAg), объединенным с общим хелперным пептидом (h-pAg UCP2; последовательность: KSVWSKLQSIGIRQH; SEQ ID № 475; для мышей HHD DR1 или h-pAg DR3; последовательность MAKTIAYDEEARRGLERGLN; SEQ ID № 473; для мышей HHD DR3) (как указано в Таблице 4 ниже).
Таблица 4. Состав экспериментальной группы. h-pAg: «хелперный» пептид; vacc-pAg: вакцинный пептид. В скобках указано количество бустерных инъекций.
(vacc-pAg)
(h-Ag)
Пептиды были предоставлены следующим образом:
• vacc-pAg: CD22-B1, CD19-B1, CD19-B2, CD37-B1, TNFRSF13C-B1 и MS4A1-B4, все производятся и поставляются в концентрации 4 мг/мл (4 мМ);
• h-pAg: DR3 или UCP2 ресуспендированы в чистой дистиллированной воде в концентрации 10 мг/мл.
Пептидный состав для инъекций (эмульсию) готовили свежим в каждый день инъекции и для каждой группы. Смесь для 10 животных была приготовлена с использованием люэровских шприцев объемом 2 мл (4606701V, B BRAUN) и люэровских коннекторов (Cole-Parmer, 45502-22): 500 мкл смеси пептидов в шприце 1 эмульгировали с 500 мкл неполного адъюванта Фрейнда, находящегося в шприце 2, как можно быстрее до образования густой (белой пены) эмульсии. Каждая эмульсия была приготовлена в избытке, чтобы компенсировать мертвые объемы при инъекции.
Животных иммунизировали на 0-й день (d0) первичной инъекцией и на 14-й день повторной инъекцией. Каждой мыши вводили подкожно в основание хвоста 100 мкл эмульсии на масляной основе, которая содержала:
• 60 нМоль vacc-pAg; 105 нМоль хелперного пептида UCP2 (для мышей HHD-DR1) или 65 нМоль хелперного пептида DR3 (для мышей HHD-DR3)
• 10 мкл фосфатно-солевого буферного раствора до достижения общего объема 50 мкл (на мышь);
• Добавляли неполный адъювант Фрейнда (IFA) в соотношении 1:1 (об:об) (50 мкл на мышь).
А.2. Анализ
Через семь дней после повторной инъекции (т.е. в день 21) животных подвергали эвтаназии и собирали селезенку. Спленоциты получали механическим разрушением органа с последующей фильтрацией через 70 мкм и очисткой в градиенте плотности фиколла.
Суспензии клеток далее использовали в анализе ELISPOT-IFNɣ (Таблица 5). Клетки культивировали в 200 мкл полной Т-клеточной среды. Экспериментальные условия (дубликаты) были следующими: всего 2×105 клеток на лунку при культивировании в присутствии различных pAg (10 мкМ) или только в среде; и 2×104 общих клеток при культивировании в присутствии гранул, нагруженных CD3/CD28 (набор для активации/экспансии Т-клеток, 130-093-627, Miltenyi) (соотношение гранул к клеткам 1:1). Культуры оценивали на их способность секретировать IFNɣ (Diaclone Kit Murine IFNɣ ELISpot, 862.031-005PC) в соответствии с инструкциями производителя (время инкубации ~16-18 часов перед проведением анализа). Пептиды, используемые для повторной стимуляции, описаны в Таблице 5.
Таблица 5. Параметры анализа ELISPOT-IFNγ.
Пятна подсчитывали на считывающем устройстве CTL ELISpot. Построение графиков и статистический анализ проводили с помощью программного обеспечения Prism-5 (GraphPad Software Inc.).
В. Результаты
Все мыши были в возрасте от 8 до 13 недель на дату начала эксперимента. В исследовании использовали как самцов, так и самок. Животных содержали группами максимум по 6 в клетке. Во время умерщвления популяцию Т-клеток селезенки анализировали с помощью проточной цитометрии, показывая, что подавляющее большинство принадлежало к субпопуляции CD4+ Т-клеток.
После посева и инкубации с соответствующими стимулами выявляли и подсчитывали клетки, продуцирующие IFNγ. Данные были представлены как количество пятен на 1х106 общих Т-клеток. Индивидуальные средние значения (полученные из трех повторов) затем использовали для построения графика средних значений группы. Статистический анализ для сравнения (со средним состоянием) проводили с использованием непарного непараметрического критерия (Манна-Уитни) (**: р<0,01; *: р<0,05).
В целом, вакцинация антигенными пептидами по настоящему изобретению (CD19-B1, CD19-B2, CD22-B1, CD37-B1, TNFRSF13C-B1 и MS4A1-B4) индуцировала значительный Т-клеточный ответ в анализе ELISPOT-IFNγ у мышей HHD DR1 (Фигуры 6-10). Иммуногенность CD22-B1, CD37-B1, TNFRSF13C-B1 и MS4A1-B4 была подтверждена на мышах HHD DR1 (Фигуры 11-13).
Результаты (Фигура 6) показывают, что иммунизация мышей HHD-DR1 CD22-B1 позволяет индуцировать Т-клетки, которые способны проявлять сильную реакцию после иммунизации либо CD22-B1, либо соответствующим пептидом человека CD22-H1. Таким образом, CD22-B2 является сильно иммуногенным и способен вызывать эффективный иммунный ответ против соответствующего пептида человека.
Эти результаты были подтверждены на мышах HHD DR3, экспрессирующих человеческие HLA-A2 и HLA-DR3 MHC и лишенных мышиных H-2 MHC I и II класса (Фигура 11).
Результаты (Фигура 7) показывают, что иммунизация мышей HHD-DR1 TNFRSF13C-B1 позволяет индуцировать Т-клетки, которые способны проявлять сильную реакцию после иммунизации либо TNFRSF13C-B1, либо соответствующим пептидом человека TNFRSF13-H1. Таким образом, TNFRSF13C-B1 является сильно иммуногенным и способен вызывать эффективный иммунный ответ против соответствующего пептида человека.
Эти результаты были подтверждены на мышах HHD DR3, экспрессирующих человеческие HLA-A2 и HLA-DR3 MHC и лишенных мышиных H-2 MHC I и II класса (Фигура 12).
Результаты (Фигура 8) показывают, что иммунизация мышей HHD-DR1 CD37-B1 позволяет индуцировать Т-клетки, которые способны проявлять сильную реакцию после иммунизации либо CD37-B1, либо соответствующим пептидом человека CD37-H1. Таким образом, CD37-B2 является сильно иммуногенным и способен вызывать эффективный иммунный ответ против соответствующего пептида человека.
Эти результаты были подтверждены на мышах HHD DR3, экспрессирующих человеческие HLA-A2 и HLA-DR3 MHC и лишенных мышиных H-2 MHC I и II класса (Фигура 13).
Результаты (Фигура 9) показывают, что иммунизация мышей HHD-DR1 CD19-B2 позволяет индуцировать Т-клетки, которые способны проявлять сильную реакцию после иммунизации либо CD19-B2, либо соответствующим пептидом человека CD19-H2. Таким образом, CD19-B2 является сильно иммуногенным и способен вызывать эффективный иммунный ответ против соответствующего пептида человека.
Результаты (Фигура 10) показывают, что иммунизация мышей HHD-DR1 CD19-B1 позволяет индуцировать Т-клетки, которые способны проявлять сильную реакцию после иммунизации либо CD19-B1, либо соответствующим пептидом человека CD19-H1. Таким образом, CD19-B1 является сильно иммуногенным и способен вызывать эффективный иммунный ответ против соответствующего пептида человека.
Результаты (Фигура 15) показывают, что иммунизация мышей HHD-DR1 MS4A1-B4 позволяет индуцировать Т-клетки, которые способны проявлять сильную реакцию после иммунизации либо MS4A1-B4, либо соответствующим пептидом человека MS4A1-H4. Таким образом, MS4A1-B4 является сильно иммуногенным и способен вызывать эффективный иммунный ответ против соответствующего пептида человека.
Эти результаты были подтверждены на мышах HHD DR3, экспрессирующих человеческие HLA-A2 и HLA-DR3 MHC и лишенных мышиных H-2 MHC I и II класса (Фигура 16).
В целом эти исследования иммуногенности, описанные в Примере 2, проведенные на мышах HHD DR3 и HHD DR1, показали, что 6 антигенных пептидов по изобретению, CD19-B1, CD19-B2, CD22-B1, CD37-B1, TNFRSF13C-B1 и MS4A1-B4 вызывали сильные иммунные реакции. Перекрестная реактивность Т-клеток, генерируемых против CD19-B1, CD19-B2, CD22-B1, CD37-B1, TNFRSF13C-B1 и MS4A1-B4 для соответствующих человеческих пептидов, была показана на мышах HHD DR3 и HHD DR1.
Соответственно, эти результаты предоставляют экспериментальные доказательства того, что иммунотерапия на основе антигенов способна улучшать Т-клеточный ответ in vivo и что антигенные пептиды по настоящему изобретению особенно эффективны для этой цели.
ПРИМЕР 3: Цитотоксические эффекты ex vivo CD22-B1, CD37B1, TNFRSF13C-B1 и MS4A1-B4 специфических CD8 Т-клеток человека.
Многочисленные исследования подтверждают представление о наличии репертуара Т-клеток, специфичных против микробных пептидов. Ожидается, что количество Т-клеток, специфичных против микробных пептидов, будет низким, но достаточным для повторной активации при иммунизации вакциной.
Для идентификации и функциональной характеристики циркулирующих Т-клеток, специфичных против CD22-B1, CD37B1, TNFRSF13C-B1 и MS4A1-B4 у людей был разработан протокол амплификации in vitro для выявления Т-клеток, специфичных для каждого антигенного пептида, и исследования их цитотоксической способности.
3.1 Идентификация CD8 Т-клеток, специфичных к антигенному пептиду, у человека
Метод амплификации in vitro и специфические мультимеры pMHC использовали для идентификации Т-клеток, специфичных к CD22-B1, CD37-B1, TNFRSF13C-B1 и MS4A1-B4. Мультимеры pMHC были созданы для всех бактериальных пептидов и их соответствующих человеческих аналогов. РВМС от нескольких здоровых доноров HLA-A*02 (до 19 доноров) собирали, обогащали после селекции CD137 и CD8 и подвергали множественным циклам амплификации in vitro с Т2-клетками, нагруженными пептидами EO2463, для увеличения числа специфических клонов Т-клеток. Детекцию специфичных к пептиду OMP CD8 T-клеток с помощью цитометрического анализа с флуоресцентным мультимером проводили на обогащенных популяциях CD8 T-клеток.
Фигура 17 иллюстрирует результаты, полученные с одним здоровым донором HLA-A2. Для этого донора амплификация клеток позволяет обнаруживать клетки, специфичные для MS4A1-B4 (19,7%), клетки, специфичные для TNFRSF13C-B1 (13%), клетки, специфичные для CD22-B1 (4,6%), и клетки, специфичные для CD37-B1 (2,5%).
В заключение, эти результаты демонстрируют присутствие в крови здоровых доноров HLA-A2 CD8 Т-клеток, которые могут распознавать пептиды, происходящие из микробиома, и, что важно, также соответствующие пептиды человека.
3.2 Цитотоксические функции CD8 T-клеток, специфичных к антигенному пептиду.
CD8+ Т-клетки, размноженные, как указано выше, использовали для проведения цитотоксических анализов в присутствии различных соотношений клеток-мишеней и эффекторных клеток для оценки их цитотоксической способности с использованием показаний проточной цитометрии. Клетки-мишени представляли собой клеточные линии Т2, нагруженные бактериальным пептидом или соответствующим пептидом человека. Отрицательный контроль представлял собой ненагруженные клетки Т2 и клетки Т2, нагруженные нерелевантным пептидом. Как показано на Фигуре 18, специфический к антигенному пептиду клон Т-клеток человека, размноженный in vitro, обладает способностью убивать клетки Т2, нагруженные всеми бактериальными пептидами, CD22-B1, CD37B1, TNFRSF13C-B1 и MS4A1-B4. Что еще более важно, клоны Т-клеток человека, специфичные для CD22-B1, CD37B1, TNFRSF13C-B1 и MS4A1-B4, размноженные in vitro, были способны убивать клетки T2, нагруженные пептидом TNFR13C (BAFF-R) или MS4A1 (CD20) человека, где наблюдалась перекрестная реактивность при окрашивании (Фигура 17).
В целом эти результаты демонстрируют наличие клонов Т-клеток у здоровых добровольцев, способных распознавать микробные пептиды и уничтожать мишени с микробными пептидами и соответствующими человеческими пептидами. Эти данные особенно обнадеживают, поскольку клоны Т-клеток были получены у здоровых доноров, поэтому можно ожидать, что специфические клоны Т-клеток могут быть эффективно амплифицированы у пациентов, подвергшихся иммунизации антигенными пептидами по изобретению.
ПРИМЕР 4: Другие антигенные пептиды имеют более высокую аффинность к аллелю HLA-A*0201.
Затем аффинность связывания дополнительных выбранных антигенных пептидов и соответствующих фрагментов опухолевых антигенов человека (эталонных пептидов человека) с аллелем HLA-A*0201 подтверждали in vitro.
А именно, антигенные пептиды с последовательностью SEQ ID № 110 («YIFEHPELL», также обозначаемой здесь как CD22-B1), последовательностью SEQ ID № 107 («LIFEHPERV», также обозначаемой здесь как CD22-B12) и последовательностью SEQ ID № 108 («RVFEHPELV», также обозначаемый здесь как CD22-B13) сравнивали с соответствующим эталонным пептидом человека, полученным из CD22 («WVFEHPETL», SEQ ID № 270, также обозначаемым здесь как CD22-H1).
Кроме того, антигенные пептиды с последовательностью SEQ ID № 114 («FLAFVPLQL», также обозначаемой здесь как CD37-B1), с последовательностью SEQ ID № 119 («ILAFVPLYL», также обозначаемой здесь как CD37-B12), с последовательностью SEQ ID № 120 («IMAFVPLAV», также обозначаемой здесь как CD37-B13), последовательностью SEQ ID № 491 («FLAFVPLDV», также обозначаемой здесь как CD37-B14) и последовательности SEQ ID № 493 («VLAFVPLGV», также обозначаемой здесь как CD37-B15), сравнивали с соответствующим эталонным пептидом человека, полученным из CD37 («GLAFVPLQI», SEQ ID № 271, также обозначаемым здесь как CD37-H1).
Кроме того, антигенные пептиды с последовательностью SEQ ID № 220 («LMFGAPALV», также обозначаемой здесь как TNFRSF13C-B1), с последовательностью SEQ ID № 212 («FLFGAPASA», также обозначаемой здесь как TNFRSF13C-B11), с последовательностью SEQ ID № 217 («LLFGAPAGV», также обозначаемой здесь как TNFRSF13C-B12), и последовательностью SEQ ID № 224 («VLFGAPAYL», также обозначаемой здесь как TNFRSF13C-B13) сравнивали с соответствующим эталонным пептидом человека, полученным из TNFRSF13C ( «LLFGAPALL», SEQ ID № 279, также обозначаемой здесь как TNFRSF13C-H1).
Кроме того, антигенные пептиды с последовательностью SEQ ID № 65 («AMNSLSLYI», также обозначаемой здесь как MS4A1-B4), с последовательностью SEQ ID № 70 («YMNSLSLAL», также обозначаемой здесь как MS4A1-B42) и последовательностью SEQ ID № 477 («AMNSLSLTV», также обозначаемый здесь как MS4A1-B43), сравнивали с соответствующим эталонным пептидом человека, полученным из MS4A1 (также известным как CD20) («IMNSLSLFA», SEQ ID № 264, также обозначаемым здесь как MS4A1-Н4).
А. Материалы и методы
А1. Измерение аффинности пептида к клеточной линии Т2.
Экспериментальный протокол аналогичен тому, который был утвержден для пептидов, презентированных HLA-A*0201 (Tourdot et al., A general strategy to enhance immunogenicity of low-affinity HLA-A2.1-associated peptides: implication in the identification of cryptic tumor epitopes. Eur J Immunol. 2000 Dec; 30(12):3411-21). Измерение аффинности пептидов достигается с помощью опухолевых клеток человека Т2, которые экспрессируют молекулу HLA-A*0201, но являются TAP1/2-отрицательными и неспособными презентировать эндогенные пептиды.
Клетки T2 (5х104 клеток на лунку) инкубировали с уменьшающимися концентрациями пептидов от 100 мкМ до 0,1 мкМ (4 концентрации: 100 мкМ, 10 мкМ, 1 мкМ, 0,1 мкМ) в бессывороточной среде (TexMacs) с добавлением 100 нг/мкл β2-микроглобулина при 37°С в течение 16 часов. Затем клетки дважды промывали и метили анти-HLA-A2-антителом, связанным с фикоэритрином (клон BB7.2, BD Pharmagen).
Анализ осуществляли с помощью FACS (анализатор Macsquant 10-Miltenyi).
Для каждой концентрации пептида среднее геометрическое значение метки, связанной с интересующим пептидом, вычитали из фонового значения и представляли в виде процента от среднего геометрического значения метки HLA-A*0202, полученного для эталонного пептида ВИЧ pol 589-597 при концентрации 100 мкМ.
А2. Солюбилизация пептидов
Каждый пептид солюбилизирован с учетом аминокислотного состава. Для пептидов, не содержащих цистеин, метионин или триптофан, возможно добавление ДМСО до 10% от общего объема. Другие пептиды ресуспендировали в воде или фосфатно-солевом буфере с pH 7,4.
В. Результаты
Результаты показаны на Фигурах 19-22. Для каждого из протестированных человеческих эталонных эпитопов CD22-H1 (Фигура 19), CD37-H1 (Фигура 20), TNFRSF13C-H1 (Фигура 21) и MS4A1-H4 (Фигура 22) соответствующие антигенные пептиды по настоящему изобретению проявляют более сильную аффинность связывания с HLA-A*0201.
Таким образом, результаты показывают, что антигенные пептиды по настоящему изобретению проявляют более сильную аффинность связывания с HLA-A*0201, чем соответствующие фрагменты опухолевого антигена человека. Как указано выше, не углубляясь в какую-либо теорию, можно предположить, что такая сильная аффинность связывания антигенных пептидов по настоящему изобретению отражает их способность вызывать иммунный ответ (т.е. их иммуногенность).
--->
<ST26SequenceListing dtdVersion="V1_3" fileName="B384554PCT D44797
WIPO Sequence.xml" softwareName="WIPO Sequence"
softwareVersion="2.3.0" productionDate="2024-07-15">
<ApplicationIdentification>
<IPOfficeCode>EP</IPOfficeCode>
<ApplicationNumberText>2020082101</ApplicationNumberText>
<FilingDate>2020-11-13</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>B384554PCT D44797</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>EP</IPOfficeCode>
<ApplicationNumberText>19306475</ApplicationNumberText>
<FilingDate>2019-11-15</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="fr">ENTEROME</ApplicantName>
<ApplicantNameLatin>ENTEROME</ApplicantNameLatin>
<InventionTitle languageCode="en">ANTIGENIC PEPTIDES FOR PREVENTION
AND TREATMENT OF B-CELL MALIGNANCY</InventionTitle>
<SequenceTotalQuantity>509</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLLFLTPIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q3">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLLFLTPLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q5">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FMLFLTPRI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q7">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLLFLTPLA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q9">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLLFLTPLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q11">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLLFLTPLM</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q13">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILLFLTPLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q15">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLLFLTPLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q17">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLLFLTPLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q19">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLLFLTPML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q21">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLLFLTPML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q23">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLLFLTPVL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q25">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALSLGLPGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q27">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALSLGLPLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q29">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALSLGLPML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q31">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALSLGLPQL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q33">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALSLGLPRL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q35">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AMSLGLPCL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q37">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AMSLGLPML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q39">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSLGLPIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q41">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSLGLPKL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q43">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILSLGLPIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q45">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLSLGLPVL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q47">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLSLGLPFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q49">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLSLGLPGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q51">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLSLGLPIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q53">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RLSLGLPGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="28">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q55">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q56">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLSLGLPIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="29">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q57">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q58">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLSLGLPKL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="30">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q59">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q60">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLSLGLPLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="31">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q61">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q62">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLSLGLPTA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="32">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q63">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q64">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLSLGLPTV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="33">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q65">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q66">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLSLGLPIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="34">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q67">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q68">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLVGILHLV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="35">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q69">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q70">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLVGILHII</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="36">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q71">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q72">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLAYLIFGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="37">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q73">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q74">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLAYLIFTL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="38">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q75">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q76">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RLAYLIFLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="39">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q77">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q78">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLAYLIFEL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="40">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q79">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q80">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LQMGGFYLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="41">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q81">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q82">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AILGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="42">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q83">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q84">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="43">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q85">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q86">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALLGGLLML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="44">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q87">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q88">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FALGGLLTV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="45">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q89">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q90">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="46">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q91">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q92">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLLGGLLMV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="47">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q93">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q94">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GMLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="48">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q95">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q96">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GMLGGLLML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="49">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q97">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q98">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>HILGGLLMV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="50">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q99">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IILGGLLVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="51">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q101">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q102">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="52">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q103">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q104">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="53">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q105">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLLGGLLMI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="54">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q107">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q108">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>NLLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="55">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q109">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q110">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SILGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="56">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q111">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="57">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q113">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q114">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLLGGLLML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="58">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q115">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q116">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SMLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="59">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q117">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLLGGLLMI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="60">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q119">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q120">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YALGGLLEV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="61">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q121">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q122">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YILGGLLMV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="62">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q123">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YMLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="63">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q125">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q126">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YVLGGLLMI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="64">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q127">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q128">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YVLGGLLMV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="65">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q129">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q130">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AMNSLSLYI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="66">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q131">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q132">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILNSLSLKI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="67">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q133">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q134">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILNSLSLKL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="68">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q135">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q136">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILNSLSLLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="69">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q137">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>Artificial</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q138">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLNSLSLFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="70">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q139">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q140">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YMNSLSLAL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="71">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q141">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q142">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLIPAGIFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="72">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q143">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q144">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILIPAGIYL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="73">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q145">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q146">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLIPAGIAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="74">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q147">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q148">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLIPAGIEL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="75">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q149">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLIPAGIGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="76">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q151">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q152">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLIPAGILI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="77">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q153">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q154">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLIPAGILL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="78">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q155">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q156">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLIPAGIPA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="79">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q157">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q158">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MMIPAGIAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="80">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q159">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q160">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VMIPAGIFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="81">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q161">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q162">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLFLGILGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="82">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q163">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q164">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLFLGILPL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="83">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q165">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q166">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLFLGILSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="84">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q167">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q168">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLFLGILGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="85">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q169">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q170">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLFLGILGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="86">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q171">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q172">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLFLGILSL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="87">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q173">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q174">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLFLGILYL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="88">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q175">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q176">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSNDTVLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="89">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q177">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q178">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSNDTVPL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="90">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q179">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q180">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSNDTVSA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="91">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q181">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q182">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FMSNDTVKV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="92">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q183">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q184">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILSNDTVWL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="93">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q185">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q186">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KMSNDTVVL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="94">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q187">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q188">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RLSNDTVGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="95">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q189">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q190">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RMSNDTVEI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="96">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q191">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q192">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLSNDTVWL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="97">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q193">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q194">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALLGPWLIV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="98">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q195">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q196">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLLGPWLCL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="99">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q197">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q198">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLLGPWLSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="100">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q199">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q200">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLLGPWLLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="101">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q201">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q202">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLLGPWLLV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="102">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q203">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q204">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILILAICGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="103">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q205">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q206">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMILAICLV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="104">
...
</SequenceData>
<SequenceData sequenceIDNumber="105">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q209">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q210">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLILAICGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="106">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q211">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q212">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KIFEHPELL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="107">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q213">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q214">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LIFEHPERV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="108">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q215">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q216">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RVFEHPELV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="109">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q217">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q218">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YVFEHPELL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="110">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q219">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q220">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YIFEHPETA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="111">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q221">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q222">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALAFVPLAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="112">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q223">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q224">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALAFVPLSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="113">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q225">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q226">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLAFVPLIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="114">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q227">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q228">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLAFVPLQL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="115">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q229">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q230">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLAFVPLVL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="116">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q231">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q232">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FMAFVPLQL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="117">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q233">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q234">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GMAFVPLLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="118">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q235">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q236">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>HLAFVPLLV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="119">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q237">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q238">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILAFVPLYL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="120">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q239">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q240">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMAFVPLAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="121">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q241">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q242">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMAFVPLIV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="122">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q243">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q244">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMAFVPLVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="123">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q245">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q246">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLAFVPLAL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="124">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q247">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q248">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLAFVPLDV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="125">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q249">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q250">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLAFVPLML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="126">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q251">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q252">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLAFVPLSL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="127">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q253">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q254">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LMAFVPLTL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="128">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q255">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q256">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLAFVPLAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="129">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q257">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q258">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLAFVPLLV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="130">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q259">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q260">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VMAFVPLVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="131">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q261">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q262">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLYFGMLLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="132">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q263">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q264">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLYFGMLHM</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="133">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q265">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q266">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLYFGMLGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="134">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q267">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q268">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLYFGMLLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="135">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q269">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q270">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TMYFGMLYL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="136">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q271">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q272">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLYFGMLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="137">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q273">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q274">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>CLIDKTSVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="138">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q275">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q276">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLIDKTSAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="139">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q277">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q278">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILIDKTSGA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="140">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q279">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q280">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILIDKTSGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="141">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q281">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q282">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMIDKTSTV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="142">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q283">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q284">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VIIDKTSSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="143">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q285">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q286">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLIDKTSQL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="144">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q287">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q288">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLIDKTSSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="145">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q289">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q290">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLIDKTSNI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="146">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q291">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q292">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLIDKTSNL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="147">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q293">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q294">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLIDKTSTV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="148">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q295">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q296">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FILLFATHV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="149">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q297">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q298">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLLLFATSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="150">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q299">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q300">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLLLFATAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="151">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q301">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q302">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLLLFATSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="152">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q303">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q304">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LMLLFATSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="153">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q305">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q306">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MMLLFATLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="154">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q307">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q308">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLLLFATNV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="155">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q309">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q310">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLLLFATYL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="156">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q311">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q312">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILVGICLGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="157">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q313">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q314">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SIVGICLNV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="158">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q315">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q316">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLIKYFLKV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="159">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q317">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q318">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLIKYFLVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="160">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q319">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q320">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MMIKYFLCV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="161">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q321">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q322">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>NLIKYFLYV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="162">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q323">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q324">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLIKYFLSI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="163">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q325">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q326">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALVLALVGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="164">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q327">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q328">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALVLALVPV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="165">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q329">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q330">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLVLALVGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="166">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q331">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q332">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>NLVLALVEV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="167">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q333">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q334">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLVLALVGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="168">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q335">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q336">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLVLALVSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="169">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q337">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q338">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLVLALVYV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="170">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q339">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q340">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLVLALVSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="171">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q341">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q342">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALALVLALL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="172">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q343">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q344">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALALVLAML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="173">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q345">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q346">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALALVLATV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="174">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q347">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q348">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLAAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="175">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q349">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q350">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLAAL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="176">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q351">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q352">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLAGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="177">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q353">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q354">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLAGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="178">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q355">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q356">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLALL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="179">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q357">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q358">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLAML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="180">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q359">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q360">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLATL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="181">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q361">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q362">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLAAI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="182">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q363">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q364">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLAAL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="183">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q365">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q366">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLAAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="184">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q367">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q368">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLALL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="185">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q369">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q370">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLALV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="186">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q371">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q372">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLANI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="187">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q373">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q374">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLATL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="188">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q375">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q376">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLATV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="189">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q377">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q378">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLAVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="190">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q379">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q380">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GMALVLAAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="191">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q381">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q382">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILALVLAMV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="192">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q383">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q384">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILALVLARV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="193">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q385">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q386">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILALVLAYL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="194">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q387">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q388">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLALVLAML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="195">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q389">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q390">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLALVLAEV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="196">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q391">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q392">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLALVLAGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="197">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q393">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q394">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLALVLALL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="198">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q395">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q396">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLALVLALV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="199">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q397">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q398">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLALVLAML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="200">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q399">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q400">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALVLAEL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="201">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q401">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q402">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALVLAEV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="202">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q403">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q404">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALVLAGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="203">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q405">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q406">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALVLALV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="204">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q407">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q408">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALVLAMV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="205">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q409">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q410">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALVLATV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="206">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q411">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q412">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLALVLAFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="207">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q413">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q414">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLALVLALI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="208">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q415">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q416">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLALVLALL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="209">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q417">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q418">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLALVLAML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="210">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q419">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q420">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALFGAPAAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="211">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q421">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q422">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALFGAPAKL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="212">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q423">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q424">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLFGAPASA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="213">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q425">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q426">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLFGAPAFI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="214">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q427">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q428">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFGAPAAL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="215">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q429">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q430">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFGAPAGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="216">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q431">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q432">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFGAPAGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="217">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q433">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q434">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFGAPAGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="218">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q435">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q436">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFGAPALL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="219">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q437">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q438">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LMFGAPAFV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="220">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q439">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q440">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LMFGAPALV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="221">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q441">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q442">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLFGAPAEA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="222">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q443">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q444">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SMFGAPAHV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="223">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q445">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q446">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLFGAPANL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="224">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q447">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q448">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLFGAPAYL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="225">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q449">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q450">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLFGAPAAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="226">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q451">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q452">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLPGLLFSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="227">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q453">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q454">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FMPGLLFGA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="228">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q455">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q456">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FVPGLLFGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="229">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q457">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q458">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLPGLLFEL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="230">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q459">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q460">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFAI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="231">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q461">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q462">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="232">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q463">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q464">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="233">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q465">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q466">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFSL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="234">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q467">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q468">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFYI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="235">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q469">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q470">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFYM</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="236">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q471">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q472">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMPGLLFHI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="237">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q473">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q474">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMPGLLFQV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="238">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q475">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q476">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMPGLLFYV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="239">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q477">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q478">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLPGLLFHA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="240">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q479">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q480">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLPGLLFSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="241">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q481">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q482">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLPGLLFGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="242">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q483">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q484">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLPGLLFTL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="243">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q485">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q486">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLPGLLFVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="244">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q487">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q488">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLPGLLFAL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="245">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q489">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q490">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLPGLLFGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="246">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q491">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q492">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLPGLLFKV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="247">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q493">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q494">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLPGLLFVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="248">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q495">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q496">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PLPGLLFSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="249">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q497">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q498">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RMPGLLFKV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="250">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q499">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q500">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLPGLLFFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="251">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q501">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q502">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLPGLLFTL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="252">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q503">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q504">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLPGLLFVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="253">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q505">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q506">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLPGLLFPA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="254">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q507">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q508">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLPGLLFPV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="255">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q509">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q510">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLPGLLFGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="256">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q511">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q512">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLPGLLFYV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="257">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q513">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q514">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLPGLLFLM</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="258">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q515">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLLFLTPME</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="259">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q516">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLSLGLPGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="260">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q517">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLVGILHLQ</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="261">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q518">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLAYLIFCL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="262">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q519">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QQMGGFYLC</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="263">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q520">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IALGGLLMI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="264">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q521">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMNSLSLFA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="265">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q522">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LMIPAGIYA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="266">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q523">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLFLGILSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="267">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q524">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSNDTVQL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="268">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q525">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>HLLGPWLLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="269">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q526">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILILAICGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="270">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q527">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>WVFEHPETL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="271">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q528">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLAFVPLQI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="272">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q529">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLYFGMLLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="273">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q530">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILIDKTSFV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="274">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q531">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLLLFATQI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="275">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q532">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SIVGICLGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="276">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q533">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SIVGICLGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="277">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q534">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALVLALVLV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="278">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q535">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLALV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="279">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q536">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFGAPALL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="280">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q537">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PLPGLLFGA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="281">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q538">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LFLTP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="282">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q539">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLGLP</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="283">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q540">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VGILH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="284">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q541">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AYLIF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="285">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q542">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MGGFY</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="286">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q543">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LGGLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="287">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q544">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>NSLSL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="288">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q545">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IPAGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="289">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q546">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLGIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="290">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q547">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SNDTV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="291">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q548">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LGPWL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="292">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q549">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILAIC</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="293">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q550">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FEHPE</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="294">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q551">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AFVPL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="295">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q552">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YFGML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="296">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q553">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IDKTS</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="297">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q554">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFAT</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="298">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q555">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VGICL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="299">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q556">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IKYFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="300">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q557">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="301">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q558">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALVLA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="302">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q559">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FGAPA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="303">
<INSDSeq>
<INSDSeq_length>5</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..5</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q560">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>Homo sapiens</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>PGLLF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="304">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q561">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q562">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q563">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q564">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLFLTPXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="305">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q565">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q566">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q567">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q568">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXSLGLPXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="306">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q569">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q570">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q571">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q572">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXVGILHXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="307">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q573">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q574">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q575">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q576">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXAYLIFXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="308">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q577">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q578">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q579">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q580">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXMGGFYXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="309">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q581">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q582">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q583">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q584">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLGGLLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="310">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q585">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q586">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q587">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q588">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXNSLSLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="311">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q589">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q590">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q591">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q592">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXIPAGIXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="312">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q593">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q594">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q595">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q596">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXFLGILXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="313">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q597">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q598">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q599">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q600">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXSNDTVXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="314">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q601">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>XXLGPWLXX</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q602">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q603">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q604">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLGPWLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="315">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q605">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q606">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q607">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q608">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXILAICXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="316">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q609">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q610">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q611">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q612">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXFEHPEXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="317">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q613">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q614">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q615">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q616">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXAFVPLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="318">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q617">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q618">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q619">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q620">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXYFGMLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="319">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q621">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q622">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q623">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q624">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXIDKTSXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="320">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q625">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q626">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q627">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q628">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLLFATXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="321">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q629">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q630">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q631">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q632">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXVGICLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="322">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q633">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q634">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q635">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q636">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXIKYFLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="323">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q637">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q638">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q639">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q640">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXVLALVXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="324">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q641">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q642">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q643">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q644">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXALVLAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="325">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q645">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q646">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q647">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q648">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXFGAPAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="326">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q649">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q650">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q651">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q652">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXPGLLFXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="327">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q653">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q654">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q655">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLLFLTPXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="328">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q656">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q657">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q658">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q659">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FXLFLTPXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="329">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q660">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q661">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q662">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLLFLTPXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="330">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q663">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q664">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q665">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLLFLTPXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="331">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q666">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q667">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q668">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLLFLTPLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="332">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q669">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q670">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q671">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLLFLTPML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="333">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q672">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q673">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q674">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q675">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLLFLTPXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="334">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q676">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q677">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q678">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALSLGLPXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="335">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q679">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q680">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q681">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AMSLGLPXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="336">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q682">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q683">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q684">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSLGLPXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="337">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q685">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q686">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q687">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLSLGLPXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="338">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q688">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q689">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q690">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLSLGLPXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="339">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q691">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q692">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q693">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLSLGLPXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="340">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q694">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q695">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q696">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLSLGLPGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="341">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q697">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q698">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q699">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLSLGLPIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="342">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q700">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q701">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q702">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLSLGLPKL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="343">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q703">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q704">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q705">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q706">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLSLGLPXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="344">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q707">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q708">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q709">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q710">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLVGILHXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="345">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q711">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q712">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q713">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLAYLIFXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="346">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q714">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q715">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q716">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q717">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLAYLIFXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="347">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q718">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q719">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q720">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALLGGLLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="348">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q721">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q722">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q723">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AXLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="349">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q724">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q725">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q726">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q727">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FXLGGLLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="350">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q728">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q729">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q730">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GMLGGLLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="351">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q731">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q732">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q733">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q734">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLLGGLLXI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="352">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q735">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q736">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q737">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLLGGLLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="353">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q738">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q739">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q740">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SXLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="354">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q741">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q742">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q743">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YXLGGLLMV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="355">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q744">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q745">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q746">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q747">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YXLGGLLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="356">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q748">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q749">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q750">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="357">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q751">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q752">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q753">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMLGGLLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="358">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q754">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q755">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q756">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLGGLLMI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="359">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q757">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q758">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q759">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLGGLLML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="360">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q760">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q761">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q762">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLGGLLMV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="361">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q763">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q764">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q765">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILNSLSLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="362">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q766">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q767">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q768">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q769">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLNSLSLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="363">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q770">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q771">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q772">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q773">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMNSLSLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="364">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q774">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q775">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q776">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q777">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXNSLSLXI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="365">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q778">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q779">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q780">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLIPAGIXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="366">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q781">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q782">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q783">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q784">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MXIPAGIXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="367">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q785">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q786">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q787">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q788">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLIPAGIXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="368">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q789">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q790">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q791">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q792">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMIPAGIXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="369">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q793">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q794">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q795">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXIPAGIAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="370">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q796">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q797">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q798">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLFLGILXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="371">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q799">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q800">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q801">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLFLGILXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="372">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q802">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q803">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q804">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLFLGILXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="373">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q805">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q806">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q807">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLFLGILGL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="374">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q808">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q809">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q810">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q811">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLFLGILSX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="375">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q812">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q813">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q814">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSNDTVXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="376">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q815">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q816">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q817">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLSNDTVXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="377">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q818">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q819">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q820">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q821">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FXSNDTVXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="378">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q822">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q823">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q824">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q825">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMSNDTVXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="379">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q826">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q827">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q828">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLSNDTVWL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="380">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q829">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q830">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q831">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q832">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLSNDTVXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="381">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q833">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q834">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q835">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q836">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLLGPWLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="382">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q837">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q838">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q839">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q840">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLLGPWLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="383">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q841">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q842">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q843">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q844">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLLGPWLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="384">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q845">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q846">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q847">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q848">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IXILAICXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="385">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q849">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q850">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q851">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLILAICGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="386">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q852">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q853">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q854">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q855">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLILAICGX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="387">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q856">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q857">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q858">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q859">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XIFEHPEXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="388">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q860">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q861">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q862">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXFEHPELL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="389">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q863">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q864">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q865">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q866">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXFEHPELX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="390">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q867">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q868">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q869">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q870">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXFEHPEXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="391">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q871">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q872">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q873">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALAFVPLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="392">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q874">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q875">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q876">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLAFVPLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="393">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q877">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q878">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q879">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FXAFVPLQL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="394">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q880">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q881">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q882">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMAFVPLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="395">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q883">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q884">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q885">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLAFVPLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="396">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q886">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q887">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q888">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLAFVPLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="397">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q889">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q890">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q891">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q892">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMAFVPLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="398">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q893">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q894">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q895">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q896">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMAFVPLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="399">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q897">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q898">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q899">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLAFVPLAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="400">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q900">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q901">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q902">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q903">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLAFVPLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="401">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q904">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q905">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q906">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXAFVPLAV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="402">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q907">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q908">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q909">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q910">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXAFVPLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="403">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q911">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q912">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q913">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLYFGMLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="404">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q914">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q915">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q916">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q917">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLYFGMLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="405">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q918">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q919">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q920">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q921">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLYFGMLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="406">
<INSDSeq>
<INSDSeq_length>10</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q922">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q923">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q924">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..10</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q925">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLYFGMLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="407">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q926">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q927">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q928">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILIDKTSGX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="408">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q929">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q930">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q931">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q932">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IXIDKTSXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="409">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q933">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q934">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q935">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLIDKTSXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="410">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q936">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1234">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VXIDKTSSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="411">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q938">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q939">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q940">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLIDKTSXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="412">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q941">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q942">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q943">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q944">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLIDKTSXA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="413">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q945">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q946">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q947">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q948">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLIDKTSXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="414">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q949">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q950">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q951">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q952">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLIDKTSXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="415">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q953">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q954">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q955">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q956">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FXLLFATXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="416">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q957">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q958">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q959">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLLLFATXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="417">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q960">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q961">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q962">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q963">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LXLLFATXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="418">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q964">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q965">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q966">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLLLFATXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="419">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q967">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q968">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q969">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXLLFATSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="420">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q970">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q971">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q972">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q973">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMLLFATXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="421">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q974">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q975">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q976">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q977">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXVGICLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="422">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q978">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q979">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q980">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLIKYFLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="423">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q981">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q982">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q983">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q984">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLIKYFLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="424">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q985">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q986">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q987">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q988">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLIKYFLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="425">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q989">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q990">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q991">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q992">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXIKYFLXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="426">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q993">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q994">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q995">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALVLALVXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="427">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q996">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q997">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q998">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLVLALVXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="428">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q999">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1000">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1001">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1002">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLVLALVXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="429">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1003">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1004">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1005">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALALVLAXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="430">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1006">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1007">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1008">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALALVLAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="431">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1009">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1010">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1011">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLAAX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="432">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1012">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1013">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1014">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLALVLAXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="433">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1015">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1016">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1017">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLAXI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="434">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1018">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1019">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1020">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLAXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="435">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1021">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1022">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1023">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GLALVLAXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="436">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1024">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1025">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1026">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GXALVLAVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="437">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1027">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1028">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1029">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLALVLALX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="438">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1030">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1031">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1032">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALVLAEX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="439">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1033">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1034">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1035">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLALVLAXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="440">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1036">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1037">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1038">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YLALVLAXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="441">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1039">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1040">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1041">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLALVLAEV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="442">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1042">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1043">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1044">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLALVLALL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="443">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1045">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1046">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1047">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLALVLAML</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="444">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1048">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1049">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1050">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1051">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLALVLAXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="445">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1052">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1053">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1054">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1055">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLALVLAXI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="446">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1056">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1057">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1058">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1059">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLALVLAXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="447">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1060">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1061">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1062">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALFGAPAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="448">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1063">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1064">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1065">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFGAPAXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="449">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1066">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1067">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1068">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLFGAPAGX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="450">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1069">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1070">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1071">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LMFGAPAXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="451">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1072">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1073">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1074">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLFGAPAXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="452">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1075">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1076">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1077">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1078">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLFGAPAXA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="453">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1079">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1080">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1081">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1082">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLFGAPAXI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="454">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1083">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1084">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1085">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1086">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMFGAPAXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="455">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1087">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1088">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1089">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1090">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XXFGAPAXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="456">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1091">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1092">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1093">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1094">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FXPGLLFXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="457">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1095">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1096">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1097">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFYX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="458">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1098">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1099">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1100">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFXI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="459">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1101">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1102">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1103">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILPGLLFXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="460">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1104">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1105">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1106">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>IMPGLLFXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="461">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1107">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1108">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1109">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KLPGLLFXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="462">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1110">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1111">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1112">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLPGLLFXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="463">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1113">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1114">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1115">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MLPGLLFXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="464">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1116">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1117">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1118">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLPGLLFXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="465">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1119">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1120">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1121">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLPGLLFPX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="466">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1122">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1123">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1124">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLPGLLFXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="467">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1125">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1126">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1127">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1128">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMPGLLFXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="468">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1129">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1130">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1131">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLPGLLFSV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="469">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1132">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1133">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1134">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLPGLLFVV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="470">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1135">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1136">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1137">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1138">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLPGLLFXM</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="471">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1139">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1140">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1141">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1142">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLPGLLFXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="472">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1143">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1144">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1145">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>misc_feature - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1146">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLPGLLFXV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="473">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1147">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1148">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>MAKTIAYDEEARRGLERGLN</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="474">
<INSDSeq>
<INSDSeq_length>13</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1149">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..13</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1150">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TPPAYRPPNAPIL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="475">
<INSDSeq>
<INSDSeq_length>15</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1151">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..15</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1152">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>KSVWSKLQSIGIRQH</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="476">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1153">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1154">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AMNSLSLLL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="477">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1155">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1156">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AMNSLSLTV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="478">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1157">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1158">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>GMNSLSLLV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="479">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1159">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1160">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILNSLSLDI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="480">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1161">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1162">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILNSLSLTA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="481">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1163">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1164">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>QMNSLSLFL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="482">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1165">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1166">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLNSLSLYA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="483">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1167">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1168">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VMNSLSLLI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="484">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1169">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1170">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YINSLSLFI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="485">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1171">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1172">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLFEHPETF</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="486">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1173">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1174">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>HIFEHPEHL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="487">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1175">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1176">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>RVFEHPERV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="488">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1177">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1178">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YTFEHPETI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="489">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1179">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1180">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALAFVPLWL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="490">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1181">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1182">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AMAFVPLAL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="491">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1183">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1184">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>FLAFVPLDV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="492">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1185">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1186">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LLAFVPLPL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="493">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1187">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1188">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLAFVPLGV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="494">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1189">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1190">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALFGAPAAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="495">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1191">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1192">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ILFGAPAGA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="496">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1193">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1194">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>NMFGAPAQV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="497">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1195">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1196">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>SLFGAPATA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="498">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1197">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1198">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>TLFGAPAAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="499">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1199">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1200">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VIFGAPALV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="500">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1201">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1202">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLFGAPAGI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="501">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1203">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1204">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1205">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>AMNSLSLXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="502">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1206">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1207">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1208">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1209">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMNSLSLLX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="503">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1210">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1211">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1212">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1213">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMNSLSLXI</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="504">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1214">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1215">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>8</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1216">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1217">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMNSLSLXL</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="505">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1218">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>2</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1219">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1220">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1221">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>YXFEHPETX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="506">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1222">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1223">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1224">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ALFGAPAAX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="507">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1225">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>8..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1226">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1227">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>VLFGAPAXX</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="508">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1228">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1229">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1230">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XLFGAPAAA</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="509">
<INSDSeq>
<INSDSeq_length>9</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>REGION</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1231">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>peptide</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>SITE</INSDFeature_key>
<INSDFeature_location>1</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier id="q1232">
<INSDQualifier_name>note</INSDQualifier_name>
<INSDQualifier_value>MISC_FEATURE - Xaa can be any naturally
occurring amino acid</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..9</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1233">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>synthetic construct</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>XMFGAPALV</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ПЕПТИД, ПРИМЕНЯЕМЫЙ ДЛЯ ИММУНОТЕРАПЕВТИЧЕСКИХ СРЕДСТВ | 2021 |
|
RU2836172C1 |
| БИСПЕЦИФИЧЕСКИЕ АНТИТЕЛА И ИХ ПРИМЕНЕНИЯ | 2022 |
|
RU2821577C2 |
| ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2018 |
|
RU2841528C2 |
| АНТИТЕЛА ПРОТИВ ПРОПЕРДИНА И ИХ ПОЛУЧЕНИЕ | 2021 |
|
RU2830308C1 |
| ХИМЕРНЫЕ ЦИТОКИНОВЫЕ РЕЦЕПТОРЫ, НЕСУЩИЕ ЭКТОДОМЕН PD-1 | 2020 |
|
RU2828214C2 |
| Штамм гибридных культивируемых клеток животных Mus musculus 5B9 - продуцент мышиного моноклонального антитела 5B9, антитело моноклональное мышиное 5В9 и антитело рекомбинантное химерное (мышь-человек) xi5В9, нейтрализующие рицин Ricinus communis | 2022 |
|
RU2802436C1 |
| Вариант LysE и способ получения L-аргинина с его применением | 2022 |
|
RU2840267C2 |
| АНТИТЕЛА ПРОТИВ IDE И ИХ ПРИМЕНЕНИЕ | 2020 |
|
RU2839026C1 |
| МИКРООРГАНИЗМ РОДА Corynebacterium, ПРОДУЦИРУЮЩИЙ L-АРГИНИН, И СПОСОБ ПОЛУЧЕНИЯ L-АРГИНИНА C ПРИМЕНЕНИЕМ ЭТОГО МИКРООРГАНИЗМА | 2022 |
|
RU2837670C2 |
| Микроорганизм, имеющий ДНК-связывающий регулятор транскрипции семейства LacI с ослабленной активностью, и способ получения L-глутаминовой кислоты с его использованием | 2022 |
|
RU2833333C2 |

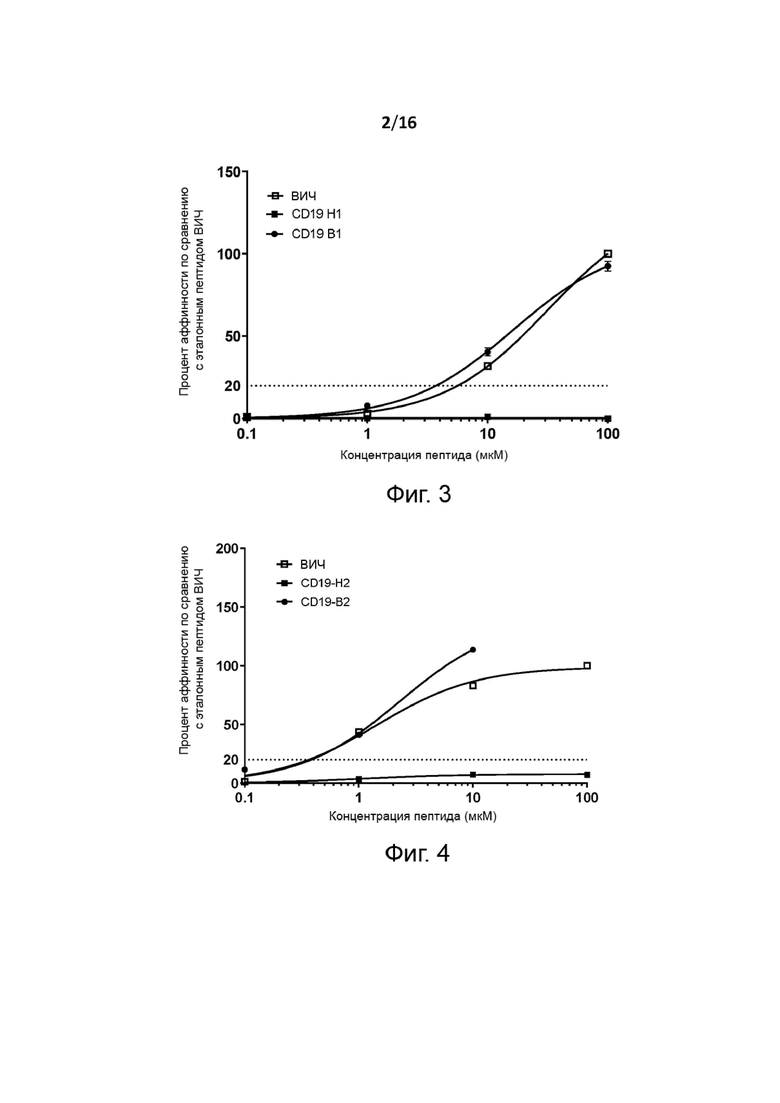
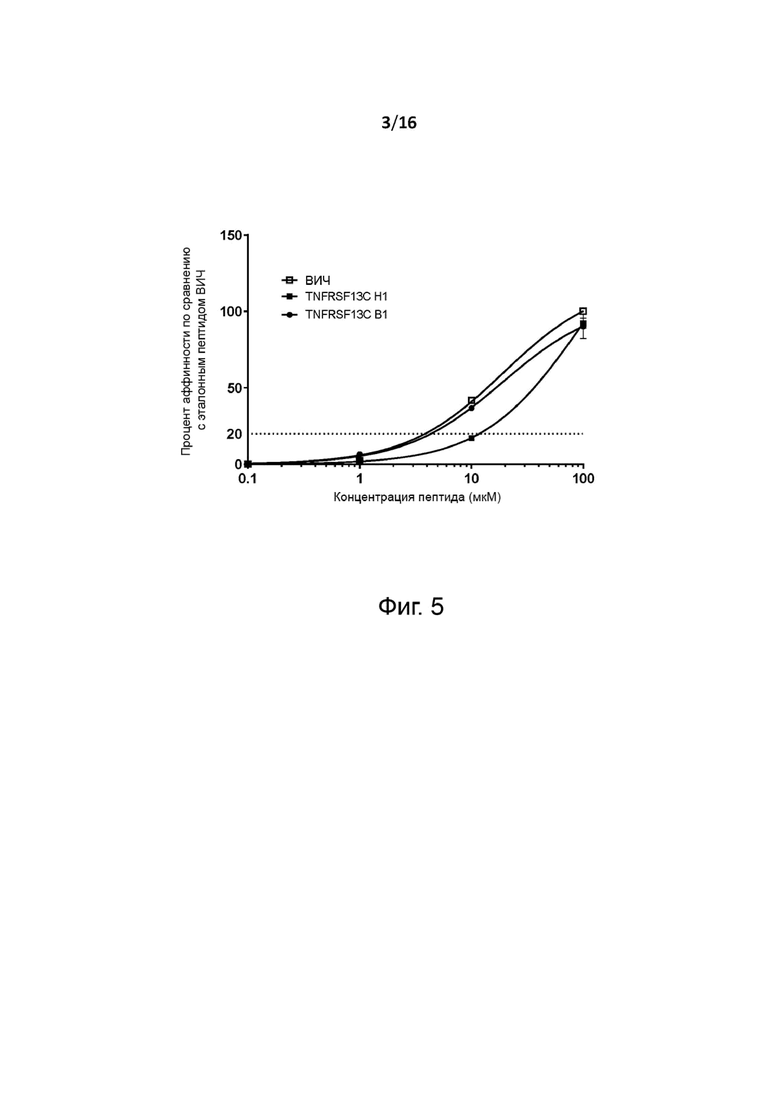
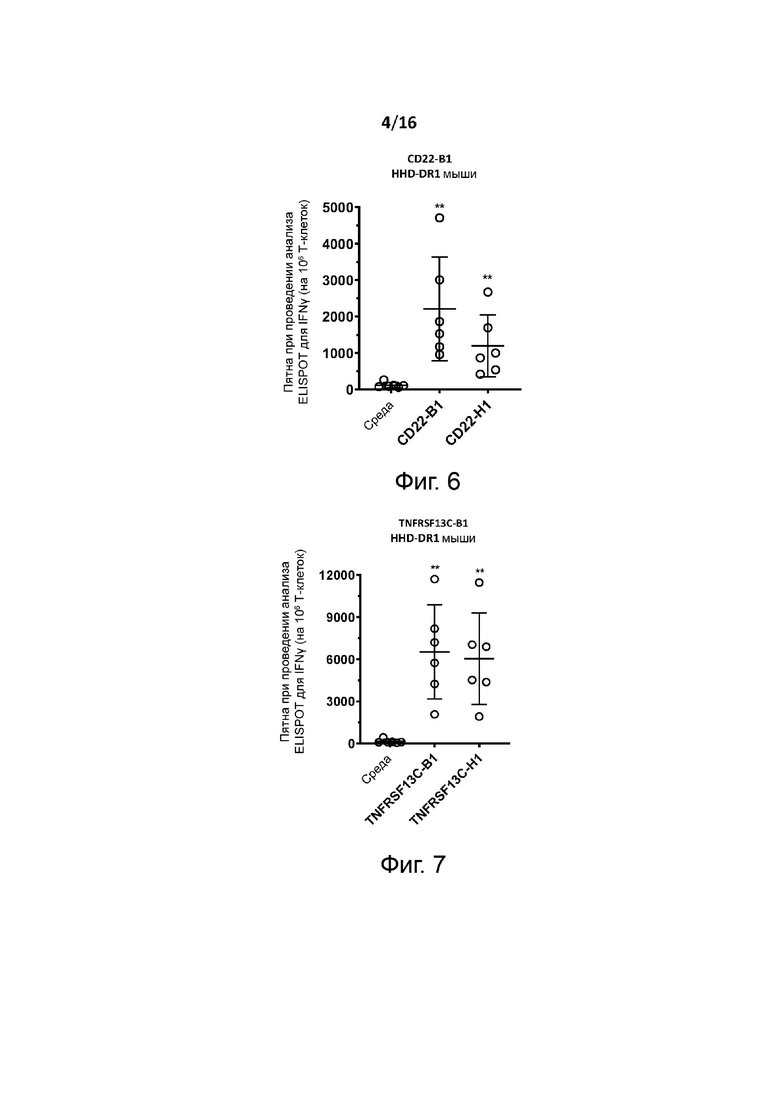
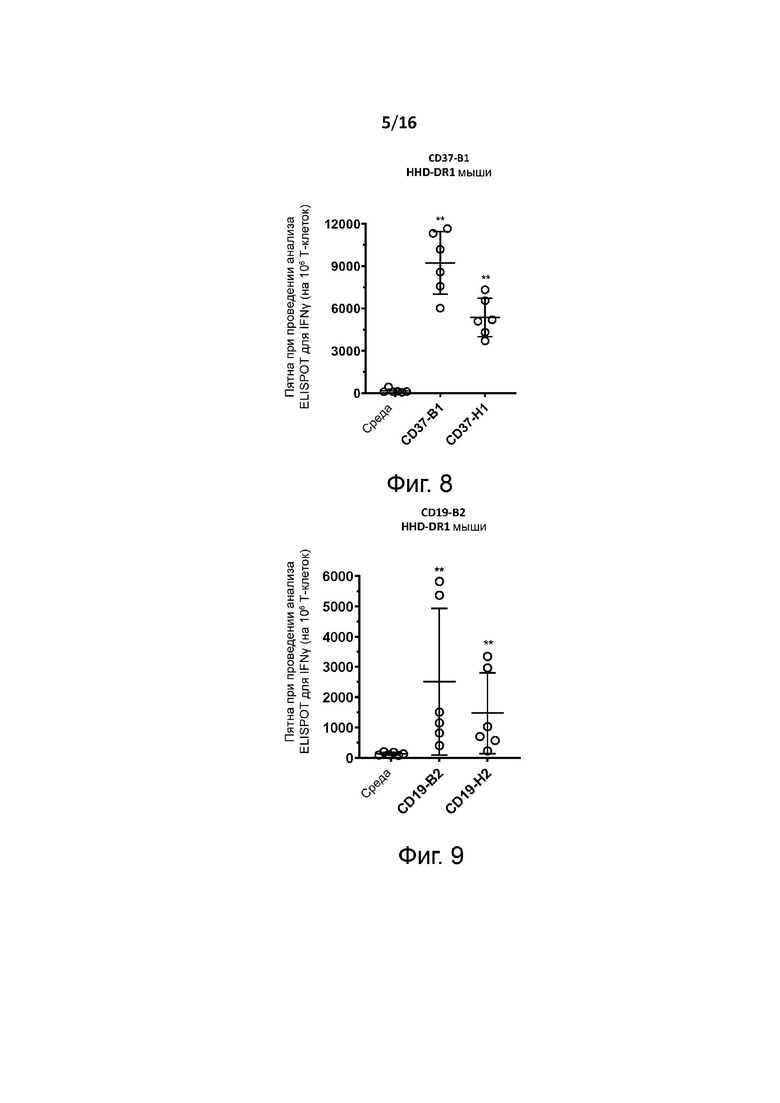
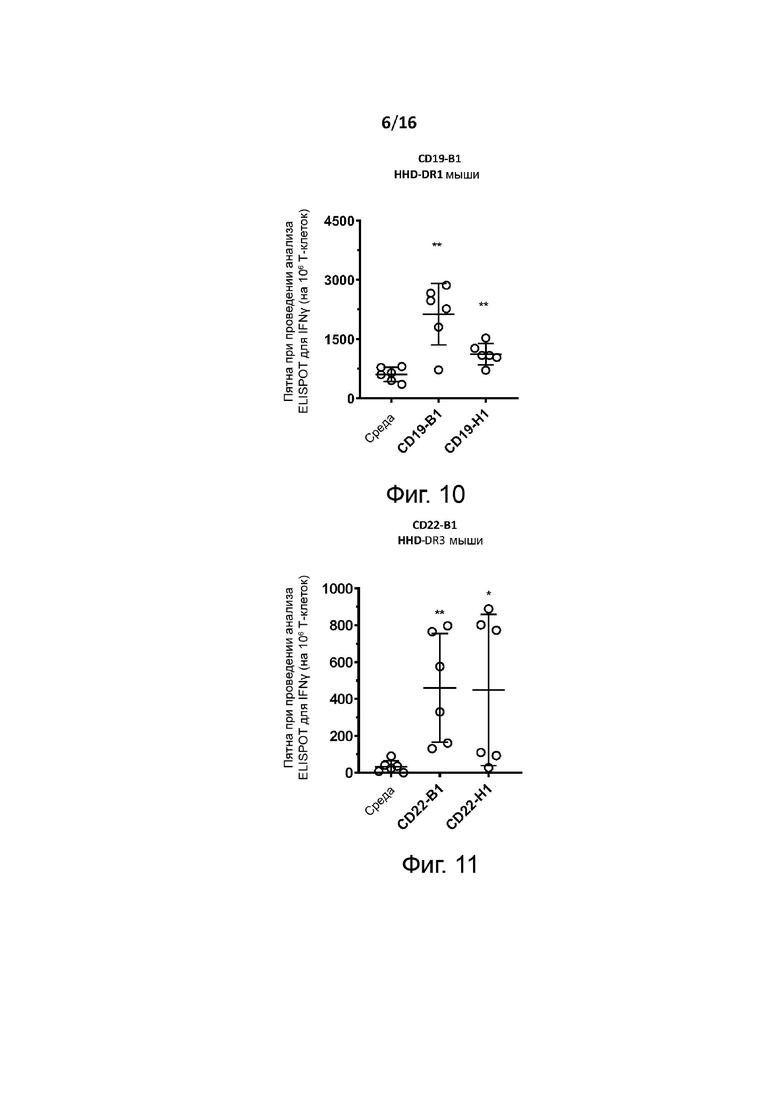
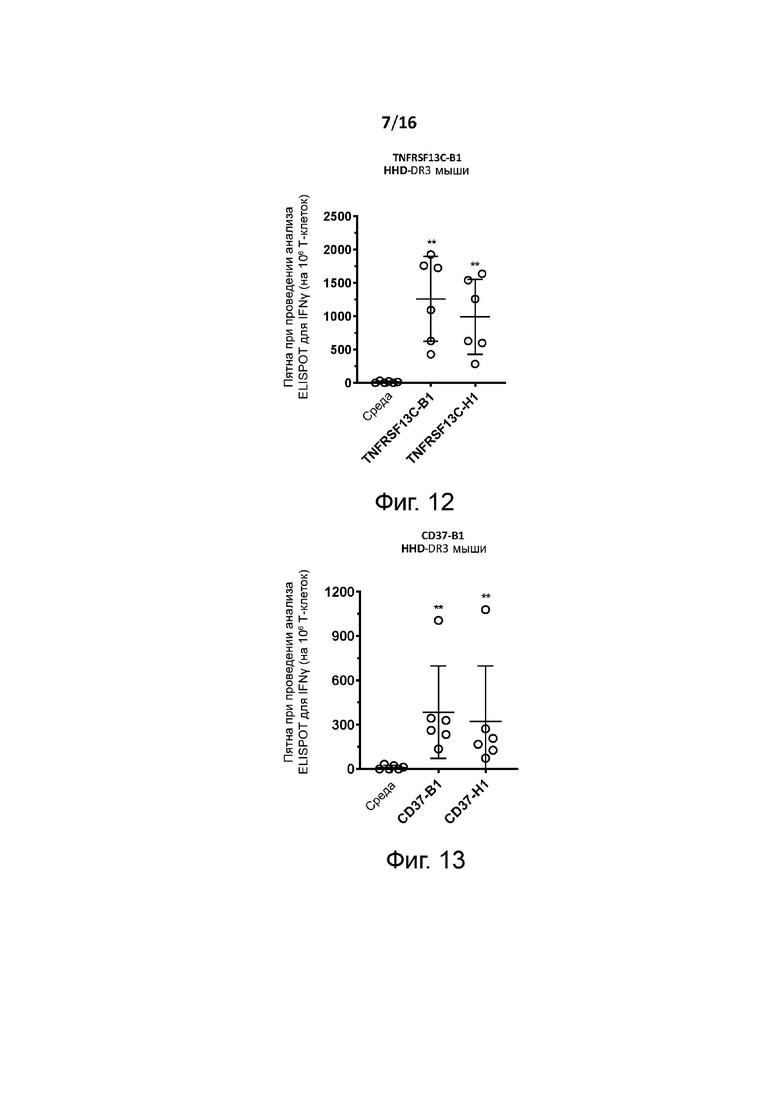
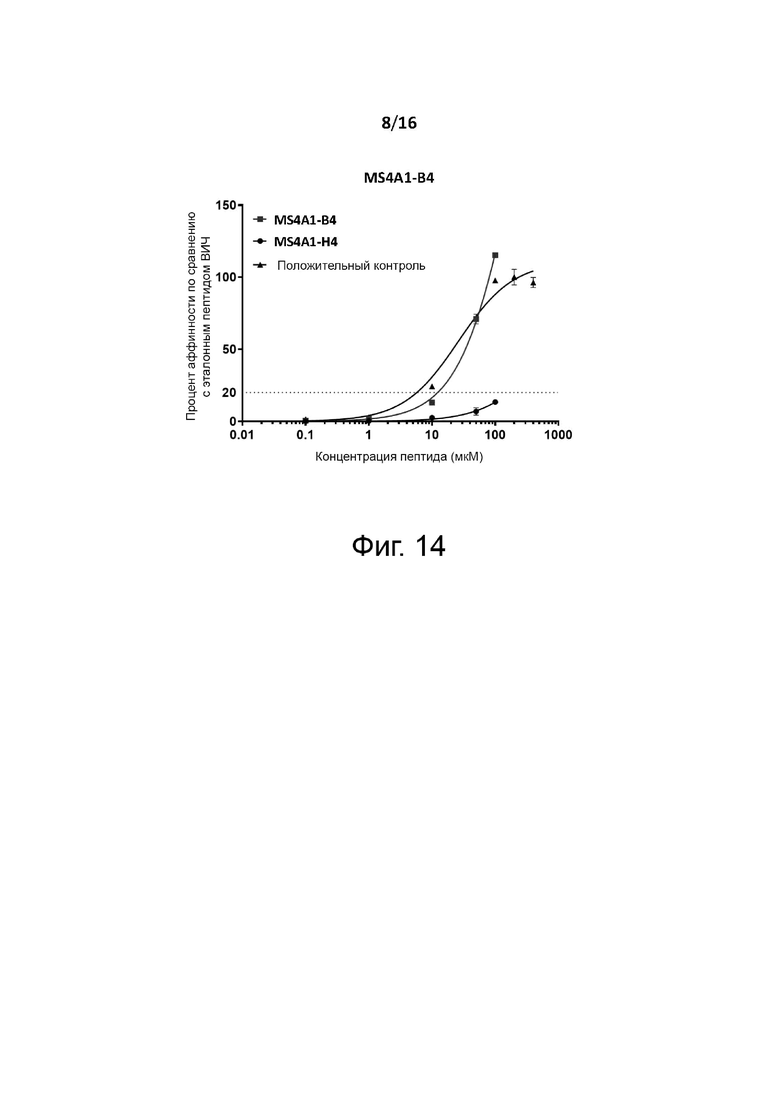
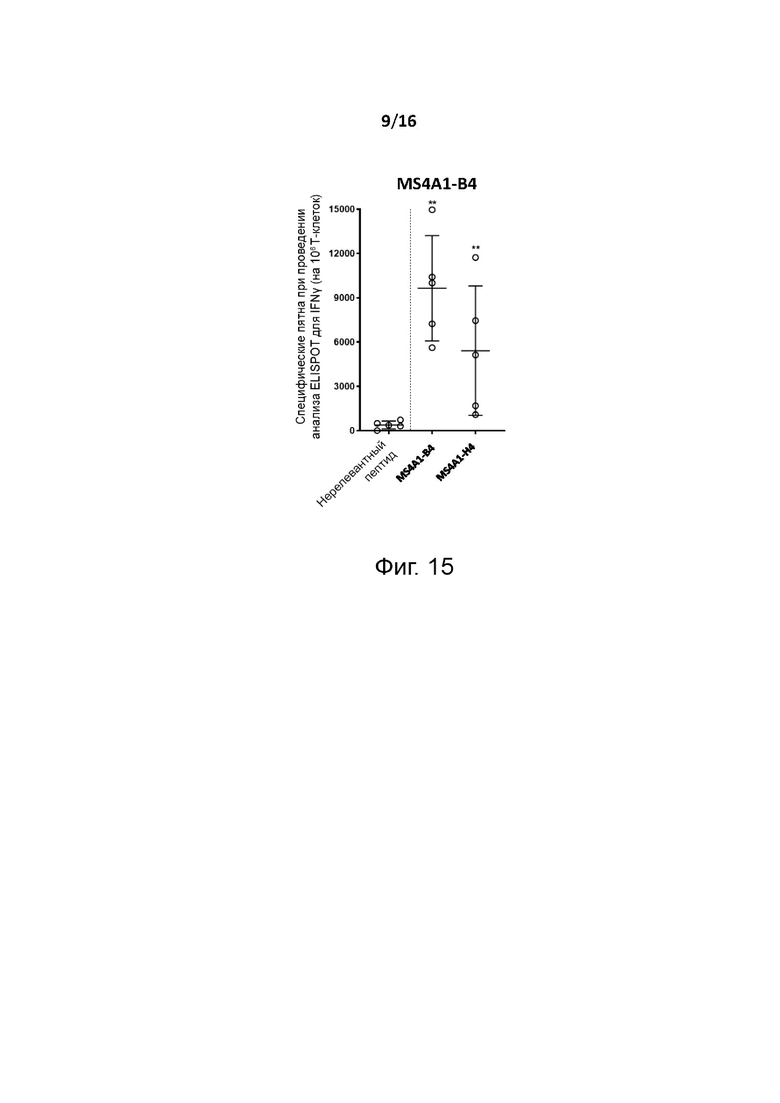
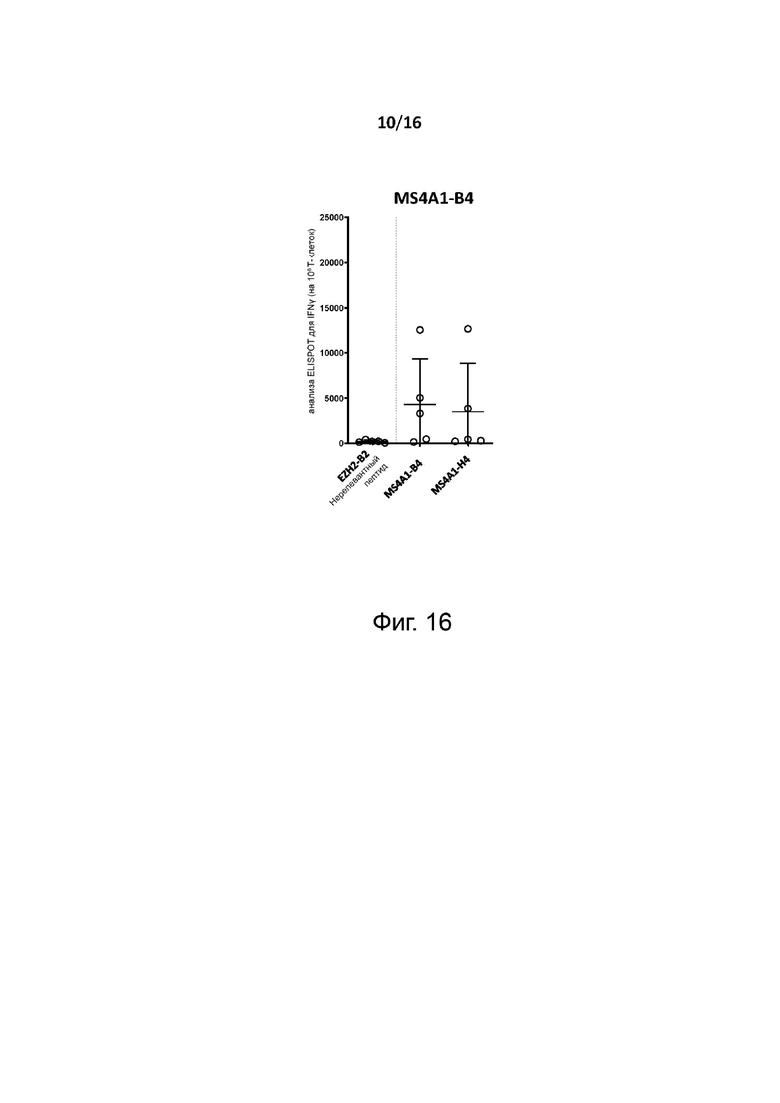
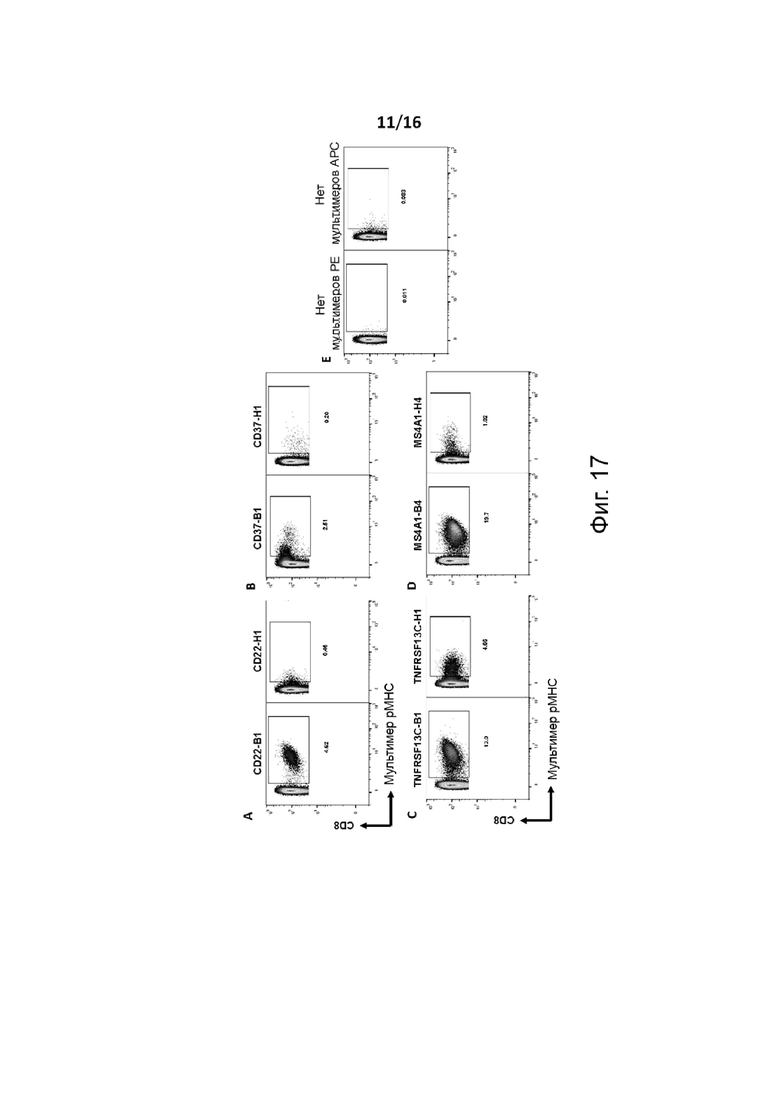
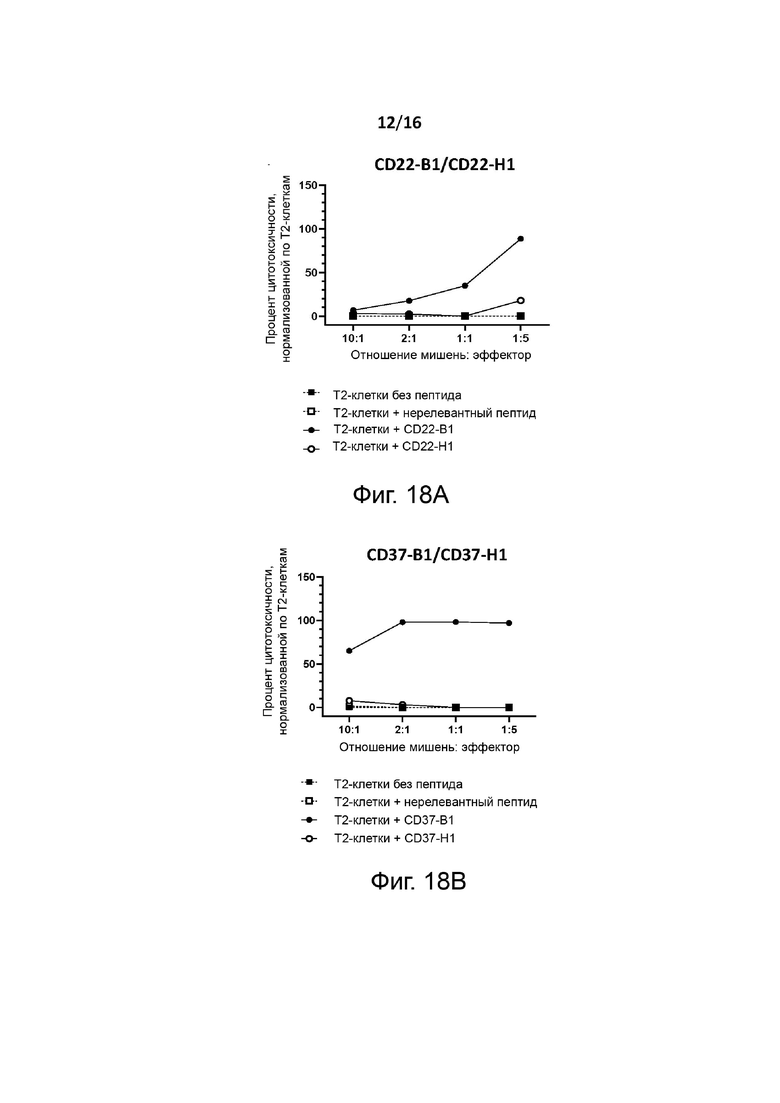
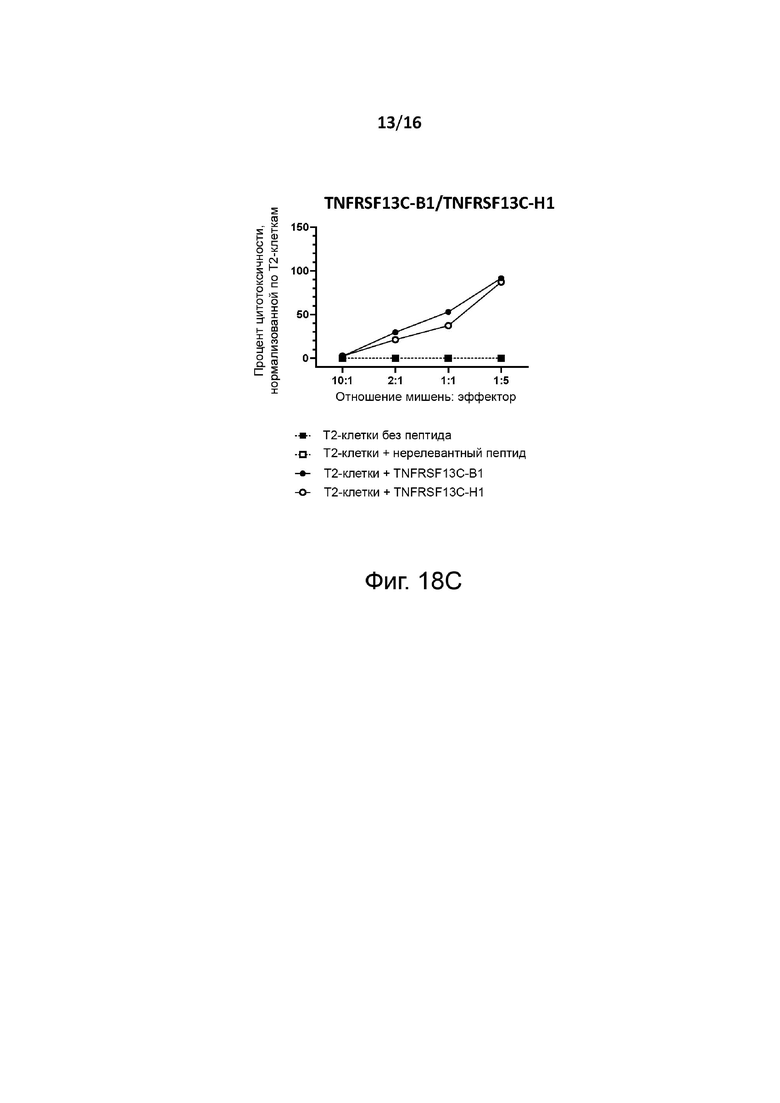
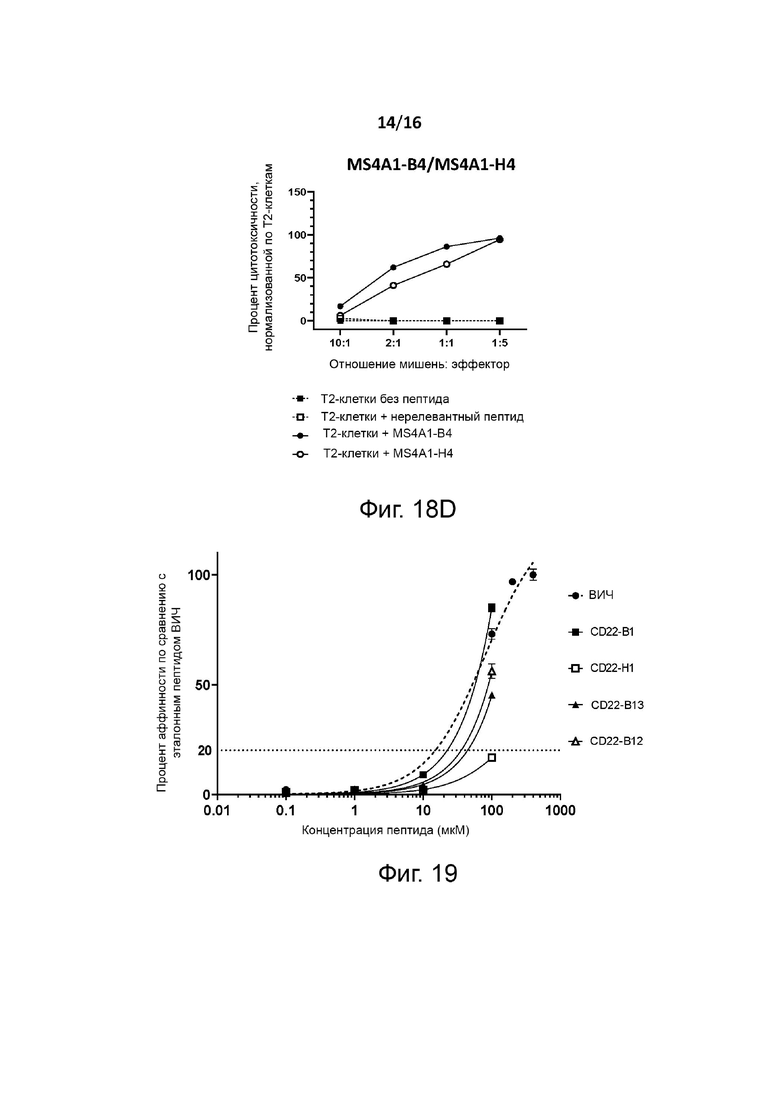
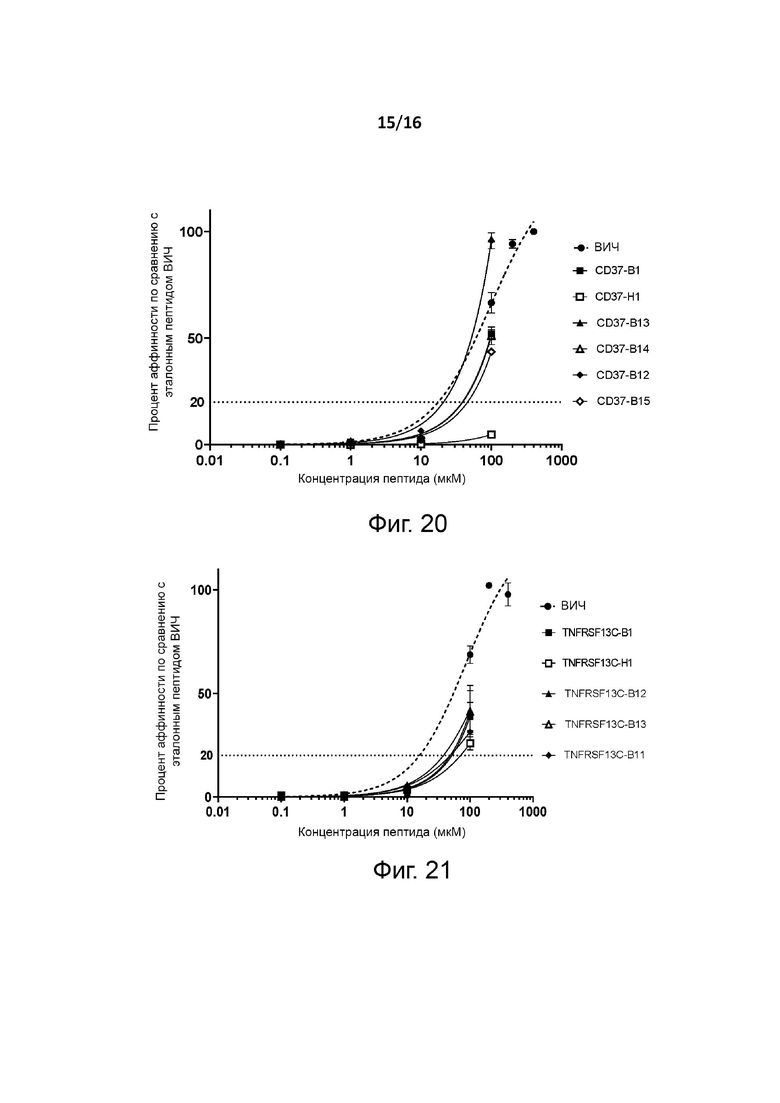
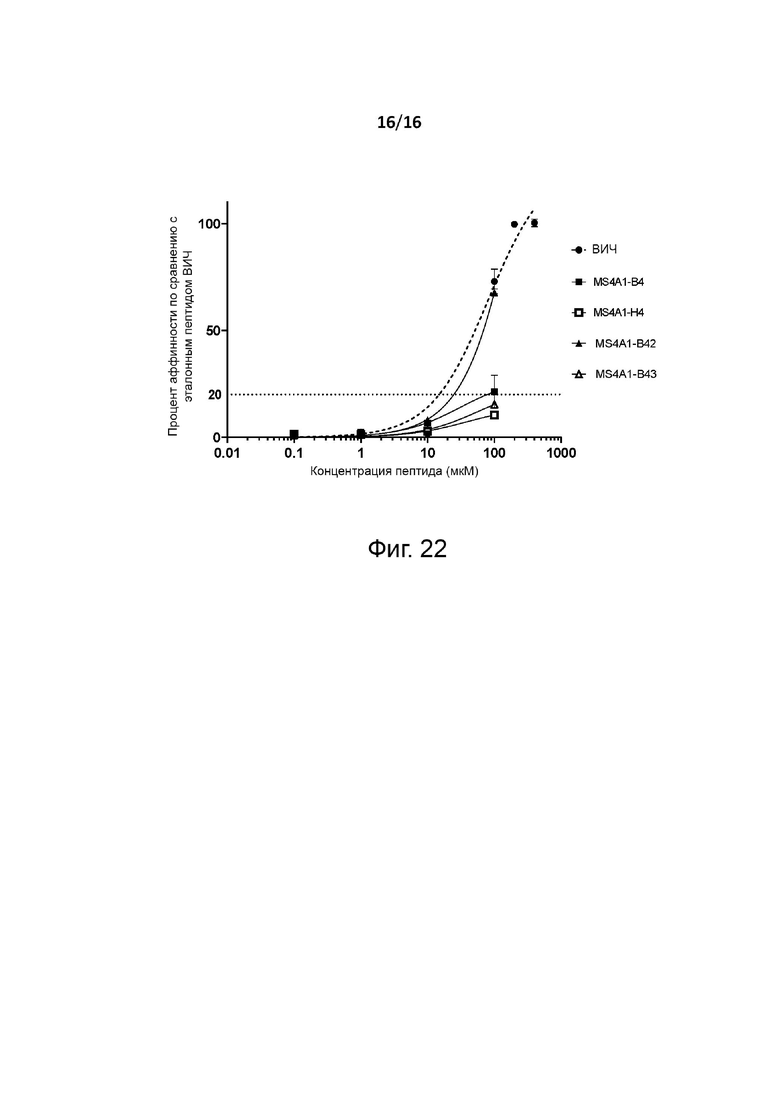
Настоящее изобретение относится к медицинской биотехнологии, а именно к иммунотерапии на основе антигенов, в частности к иммунотерапии рака. Предложен антигенный пептид для лечения В-клеточной злокачественной опухоли, состоящий из аминокислотной последовательности, представленной в SEQ ID NO 65; иммуногенное соединение для лечения В-клеточной злокачественной опухоли, содержащее указанный антигенный пептид. Предложена антигенпрезентирующая клетка для лечения В-клеточной злокачественной опухоли, где антигенпрезентирующая клетка нагружена указанным антигенным пептидом или иммуногенным соединением; нуклеиновая кислота, кодирующая антигенный пептид, иммуногенное соединение, где иммуногенное соединение представляет собой пептид или белок. Предложены фармацевтическая композиция и комбинация для лечения В-клеточной злокачественной опухоли; применение антигенного пептида для лечения В-клеточной злокачественной опухоли; способ лечения В-клеточной злокачественной опухоли у субъекта, нуждающегося в этом, и мультимер пептид-МНС (рМНС) для обнаружения антигенспецифических Т-клеток. Заявленный антигенный пептид можно использовать для индукции специфического иммунного ответа против известных опухолевых антигенов. 10 н. и 10 з.п. ф-лы, 25 ил., 7 табл., 4 пр.
1. Антигенный пептид для лечения В-клеточной злокачественной опухоли, состоящий из аминокислотной последовательности, представленной в SEQ ID NO 65.
2. Иммуногенное соединение для лечения В-клеточной злокачественной опухоли, содержащее антигенный пептид по п. 1.
3. Иммуногенное соединение по п. 2, где антигенный пептид связан с молекулой-носителем.
4. Иммуногенное соединение по п. 3, где молекула-носитель представляет собой белок-носитель или пептид-носитель.
5. Антигенпрезентирующая клетка для лечения В-клеточной злокачественной опухоли, где антигенпрезентирующая клетка нагружена антигенным пептидом по п. 1 или иммуногенным соединением по пп. 2-4.
6. Клетка по п. 5, где клетка является дендритной клеткой.
7. Нуклеиновая кислота для лечения В-клеточной злокачественной опухоли, кодирующая антигенный пептид по п. 1, иммуногенное соединение по любому из пп. 2-4, где иммуногенное соединение представляет собой пептид или белок.
8. Нуклеиновая кислота по п. 7, где нуклеиновая кислота является молекулой ДНК или молекулой РНК; предпочтительно выбранной из геномной ДНК; кДНК; мРНК; антисмысловой ДНК; антисмысловой РНК; комплементарной последовательности РНК и/или ДНК; и их комбинаций.
9. Клетка-хозяин для лечения В-клеточной злокачественной опухоли, содержащая нуклеиновую кислоту по п. 7 или 8.
10. Фармацевтическая композиция для лечения В-клеточной злокачественной опухоли, включающая эффективное количество
- антигенного пептида по п. 1,
- иммуногенного соединения по любому из пп. 2-4,
- клетки по п. 5 или 6,
- нуклеиновой кислоты по п. 7 или 8,
- клетки-хозяина по п. 9.
11. Фармацевтическая композиция по п. 10, дополнительно содержащая один или более иммуностимулирующих агентов.
12. Фармацевтическая композиция по п. 11, в которой иммуностимулирующий агент выбирается из группы, содержащей иммуноадъюванты и антигенпрезентирующие клетки.
13. Фармацевтическая композиция по п. 12, включающая по меньшей мере три или четыре различных компонента, где по меньшей мере три или четыре различных активных компонента относятся к
- антигенному пептиду, состоящему из аминокислотной последовательности, указанной в SEQ ID NO 65;
- антигенному пептиду, состоящему из аминокислотной последовательности, указанной в SEQ ID NO 110;
- антигенному пептиду, состоящему из аминокислотной последовательности, указанной в SEQ ID NO 114;
- антигенному пептиду, состоящему из аминокислотной последовательности, указанной в SEQ ID NO 220.
14. Фармацевтическая композиция по любому из пп. 10-13, дополнительно включающая хелперный пептид, предпочтительно пептид, содержащий или состоящий из аминокислотной последовательности в соответствии с SEQ ID NO 475.
15. Комбинация для лечения В-клеточной злокачественной опухоли, содержащая антигенный пептид по п. 1, и по меньшей мере один антигенный пептид выбран из группы, состоящей из:
- антигенного пептида, состоящего из аминокислотной последовательности, указанной в SEQ ID NO 110;
- антигенного пептида, состоящего из аминокислотной последовательности, указанной в SEQ ID NO 114;
- антигенного пептида, состоящего из аминокислотной последовательности, указанной в SEQ ID NO 220.
16. Применение антигенного пептида по п. 1 для лечения В-клеточной злокачественной опухоли.
17. Применение по п. 16, где В-клеточная злокачественная опухоль представляет собой В-клеточную лимфому, выбранную из группы, состоящей из неходжкинской лимфомы (NHL), диффузной крупноклеточной В-клеточной лимфомы (DLBCL), NOS (de novo и трансформированной из индолентной), первичной медиастинальной крупноклеточной В-клеточной лимфомы (PMBCL), Т-клеточной/богатой гистоцитами крупноклеточной В-клеточной лимфомы (TCHRBCL), лимфомы Беркитта, мантийноклеточной лимфомы (MCL) и фолликулярной лимфомы (FL).
18. Способ лечения В-клеточной злокачественной опухоли у субъекта, нуждающегося в этом, включающий введение субъекту
- антигенного пептида по п. 1,
- иммуногенного соединения по любому из пп. 2-4,
- клетки по п. 5 или 6,
- нуклеиновой кислоты по п. 7 или 8,
- клетки-хозяина по п. 9,
- фармацевтической композиции по любому из пп. 10-14,
- комбинации по п. 15.
19. Способ по п. 18, где В-клеточная злокачественная опухоль выбрана из В-клеточной лимфомы, выбранной из группы, состоящей из неходжкинской лимфомы (NHL), диффузной крупноклеточной В-клеточной лимфомы (DLBCL), NOS (de novo и трансформированной из индолентной), первичной медиастинальной крупноклеточной В-клеточной лимфомы (PMBCL), Т-клеточной/богатой гистоцитами крупноклеточной В-клеточной лимфомы (TCHRBCL), лимфомы Беркитта, мантийноклеточной лимфомы (MCL) и фолликулярной лимфомы (FL).
20. Мультимер пептид-МНС (рМНС) для обнаружения антигенспецифических Т-клеток, где рМНС содержит антигенный пептид по п. 1.
| Светоэлектрический измеритель длин и площадей | 1919 |
|
SU106A1 |
| Способ изготовления гибких металлических волноводов методом электролиза | 1948 |
|
SU74153A1 |
| Приспособление для суммирования отрезков прямых линий | 1923 |
|
SU2010A1 |
| Способ обработки светочувствительных бумаг | 1947 |
|
SU77384A1 |
| Предохранительное приспособление к маятниковым и балансирным круглым пилам для дерева | 1930 |
|
SU22253A1 |
| Способ и приспособление для нанесения предохранительного слоя на внутреннюю поверхность стеклянной оболочки газосветных ламп | 1934 |
|
SU49091A1 |
Авторы
Даты
2025-01-28—Публикация
2020-11-13—Подача