Изобретение относится к ветеринарной вирусологии, к средствам молекулярной диагностики, и предназначено для определения типовой таксономической принадлежности (генотипирования) изолятов и штаммов калицивируса кошек посредством филогенетического анализа после секвенирования с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Важной задачей при проведении генетических исследований является осуществление достоверного филогенетического анализа. В этом случае важно проводить анализ полногеномных последовательностей нуклеиновых кислот исследуемых организмов и выявлять те области, которые характеризуются высоким уровнем вариабельности нуклеотидов [1].
Отдельное внимание вирусологи и молекулярные биологи в области ветеринарной вирусологии уделяют калицивирусу кошек (FCV), который вызывает высококонтагиозное заболевание, характеризующееся преимущественно поражением слизистой ротовой полости и верхних дыхательных путей [2]. Следует отметить, что возбудитель калицивироза имеет широкое распространение в мире, поражает домашних кошек всех пород и возрастов, а также зоопарковых и диких представителей семейства кошачьих. Калицивирус выделяют от зараженных животных в России, США, Австралии, Бразилии, некоторых странах Европы (Великобритания, Италия, Германия) и Азии (Южная Корея, Япония, Китай) [3].
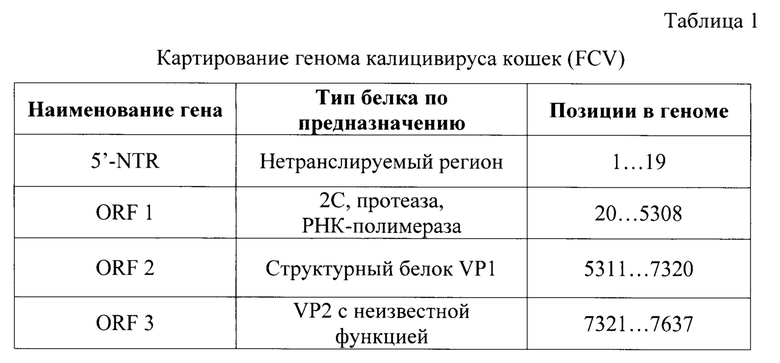
Возбудитель заболевания Feline calicivirus (FCV) относится к порядку Picornavirales, семейству Caliciviridae, роду Vesivirus. Нуклеиновая кислота вируса представлена одноцепочечной РНК (+) размером около 7700 н.о. с тремя открытыми рамками считывания (ORF). Картирование генома FCV представлено в таблице 1, из которой видно, что 5'-NTR (нетранслируемый регион) соответствует 1…19 н.о., ORF1 (ему соответствует продукт, который называется polyprotein precursor - предшественник полипротеина, из которого формируются 2С, протеаза, РНК-полимераза) - 20…5310 н.о., ORF2 (структурный белок VP1) - 5311…7320 н.о., ORF3 (белок VP2 с неизвестной функцией) - 7321…7637 н.о. [4]. ORF2 делится на шесть областей (A-F) и включает в себя различные нуклеотидные замены. Геномные области А, В, D и F в значительной степени консервативны, в то время как области С и Е более изменчивы. Рекомбинация является распространенным механизмом эволюции FCV. Большинство зарегистрированных событий рекомбинации в геноме калицивирусов происходят в ORF1, ORF2 [5].
Калицивирус характеризуется высокой степенью изменчивости и большим антигенным разнообразием штаммов, что значительно снижает эффективность существующих вакцин. Генетический анализ калицивирусов обычно приводит к звездообразному филогенетическому дереву с небольшой статистической достоверностью, что не позволяет корректно идентифицировать штамм FCV [1]. В настоящее время Pereira J.J. et al. (2018) пришли к выводу, что для калицивирусов кошек характерно наличие двух генетических групп: I и II, при этом внутри генотипа I выделяют разделение на более мелкие единицы (кластеры): кластер 1 (например, штаммы BJ-112, BJ-DH), кластер 2 (например, штамм DD 2006 GE) [6].
Изучение генетической структуры штаммов калицивирусов, циркулирующих на территории РФ, значительно облегчит разработку средств специфической профилактики, обеспечивающих надежную защиту от данной инфекции. Исходя из этого актуально определять таксономическую принадлежность изолятов и штаммов калицивирусов с точностью до гена (генотипирование), т.е. осуществлять их генотипирование по средством филогенетического анализа с последующим их использованием при создании эффективных вакцинных препаратов относительно возбудителя того или иного генотипа, циркулирующего на данной территории.
В связи с этим целесообразно провести разработку способа генотипирования изолятов и штаммов калицивирусов кошек. Для решения поставленной задачи требуется осуществить молекулярно-биологический анализ генома калицивирусов кошек, что даст возможность использовать при секвенировании разработанные оригинальные олигонуклеотидные праймеры для высоковариабельного участка генов ORF2-ORF3 и проведение филогенетического анализа результатов генотипирования изолятов и штаммов калицивируса кошек.
В настоящее время методы молекулярно-биологического анализа находят широкое применение в диагностике различных инфекционных заболеваний, в частности, обратная транскрипция, полимеразная цепная реакция и секвенирование для FCV [6, 7, 8].
Прототипной является тест-система для филогенетического анализа калицивирусов в реакции амплификации участка гена ORF1 (размер 420 п. н.) с последующим секвенированием (тест-система разработана Afonso М.М. et al.) [9]. Однако, ампликоны, которые получают в результате применения указанной тест-системы включают в себя невысокое количество нуклеотидных замен (участок не является высоко вариабельным и содержит в среднем 35-88 замен на 420 п. н., что составляет не более 8,30-20,95%). Для проведения достоверного филогенетического анализа требуется определить участок генома калицивируса в высокой степенью вариабельности и разработать такие олигонуклеотидные праймеры для ПЦР и секвенирования, которые позволят амплифицировать данный продукт. Исходя из этого необходимо разработать альтернативный способ генотипирования изолятов и вакцинных штаммов калицивируса кошек методом секвенирования с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генома возбудителя калицивироза.
Задачей настоящего изобретения является разработка способа генотипирования с применением оригинальных олигонуклеотидных праймеров для проведения достоверного филогенетического анализа изолятов и вакцинных штаммов калицивируса кошек на основе молекулярно-биологического метода с целью устранения вышеуказанных недостатков.
Данная задача решена благодаря разработке способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3. Разработанный способ дает возможность: 1) амплифицировать высоковариабельный участок генов ORF2-ORF3 кДНК FCV размером 324 п. н.; 2) проводить достоверный филогенетический анализ изолятов и штаммов калицивируса кошек, поскольку в получаемом ампликоне среднее количество замен равно 85-102 нуклеотида на 324 нуклеотида, что составляет 26,23-31,48% и на 5,28-23,18% выше в сравнении с прототипом. Данная разработка позволит проводить дифференциацию изолятов и штаммов калицивирусов и осуществлять их филогенетический анализ для создания эффективных вакцинных препаратов относительно возбудителя того или иного генотипа и диагностических гомологичных тест-систем.
Сущность изобретения отражена на графических изображениях:
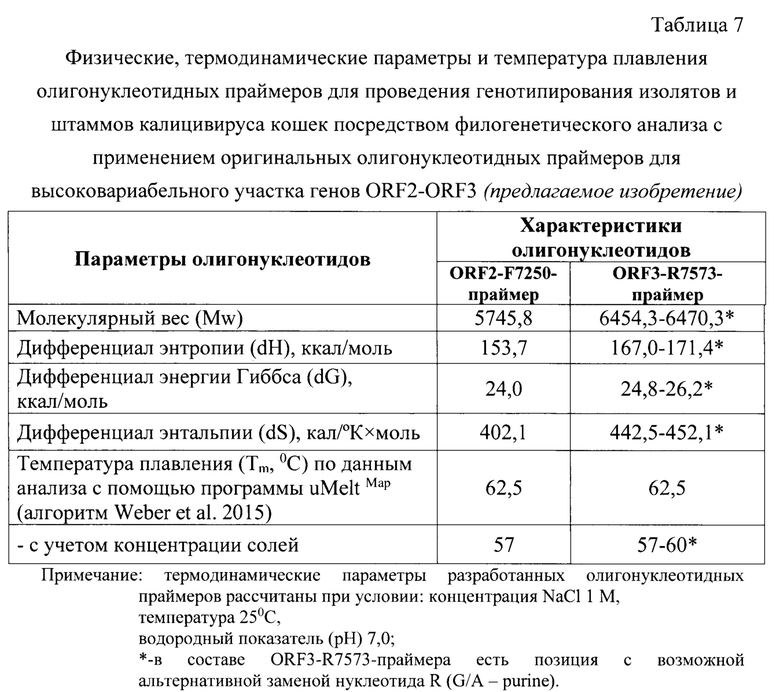
Фиг. 1 - Электрофореграмма ПЦР-продуктов после проведения реакции амплификации для разных вакцинных штаммов калицивируса кошек. Примечание: А - 1 - штамм «Мира», 2 - «Элис», 3 - «Перс», 4 - положительный контроль на этапе выделения РНК, 5 - положительный контроль на этапе ПЦР, 6 - отрицательный контроль на этапе выделения РНК, 7 - отрицательный контроль на этапе ПЦР, М - маркер молекулярного веса; Б - 1 - штамм «Фауна», 2 - «F-9», 3 - положительный контроль на этапе выделения РНК, 4 - положительный контроль на этапе ПЦР, 5 - отрицательный контроль на этапе выделения РНК, 6 - отрицательный контроль на этапе ПЦР, М - маркер молекулярного веса.
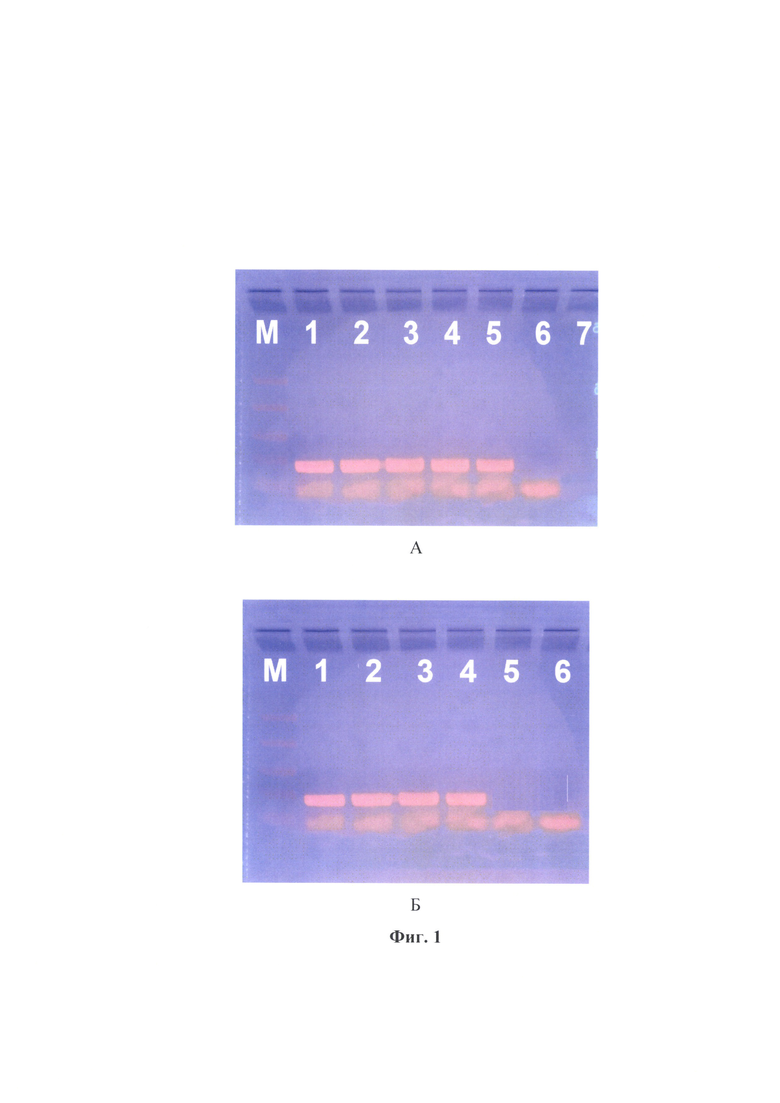
Фиг. 2 - Электрофореграмма ПЦР-продуктов после очистки для разных вакцинных штаммов калицивируса кошек. Примечание: 1 - штамм «Мира», «Элис», «Перс», «Фауна», «F-9», М - маркер молекулярного веса. Примечание: А - 1 - штамм «Мира», 2 - «Элис», 3 - «Перс», 4 - положительный контроль на этапе выделения РНК, 5 - положительный контроль на этапе ПЦР, 6 - отрицательный контроль на этапе выделения РНК, 7 - отрицательный контроль на этапе ПЦР, М - маркер молекулярного веса; Б - 1 - штамм «Фауна», 2 - «F-9», 3 - положительный контроль на этапе выделения РНК, 4 - положительный контроль на этапе ПЦР, 5 -отрицательный контроль на этапе выделения РНК, 6 - отрицательный контроль на этапе ПЦР, М - маркер молекулярного веса.
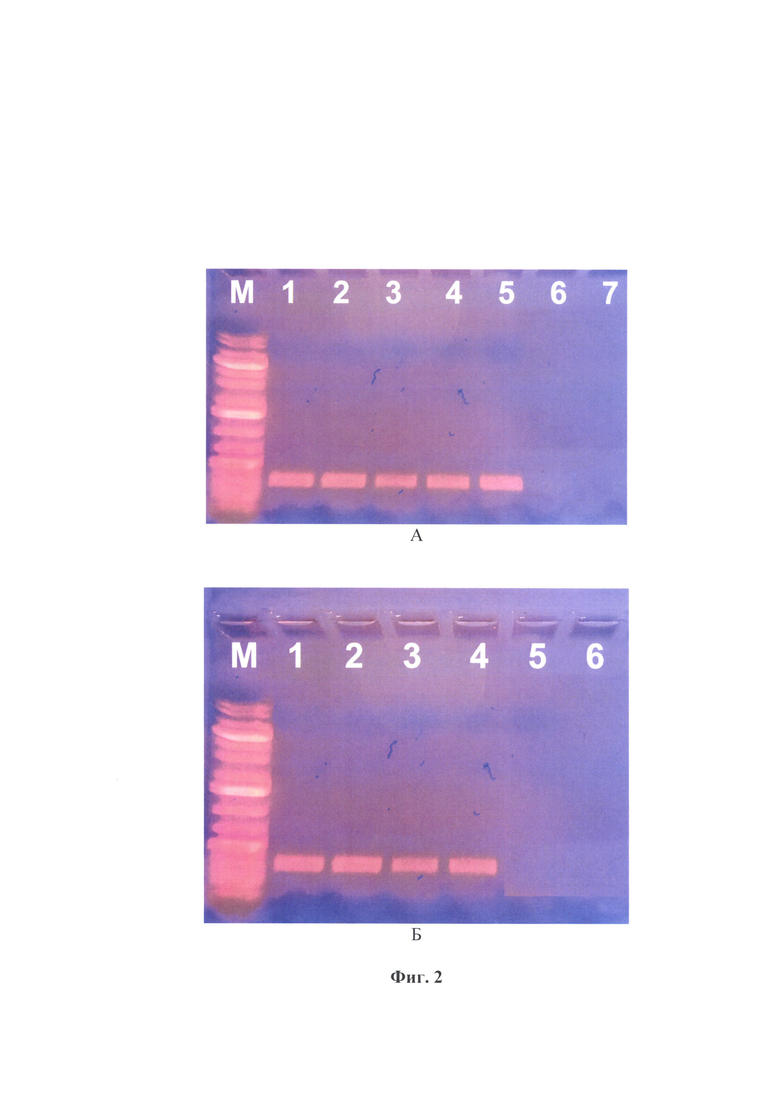
Фиг. 3 - Картирование прямого и обратного оригинальных праймеров, разработанных для проведения филогенетического анализа штаммов калицивируса кошек методом секвенирования с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генома.
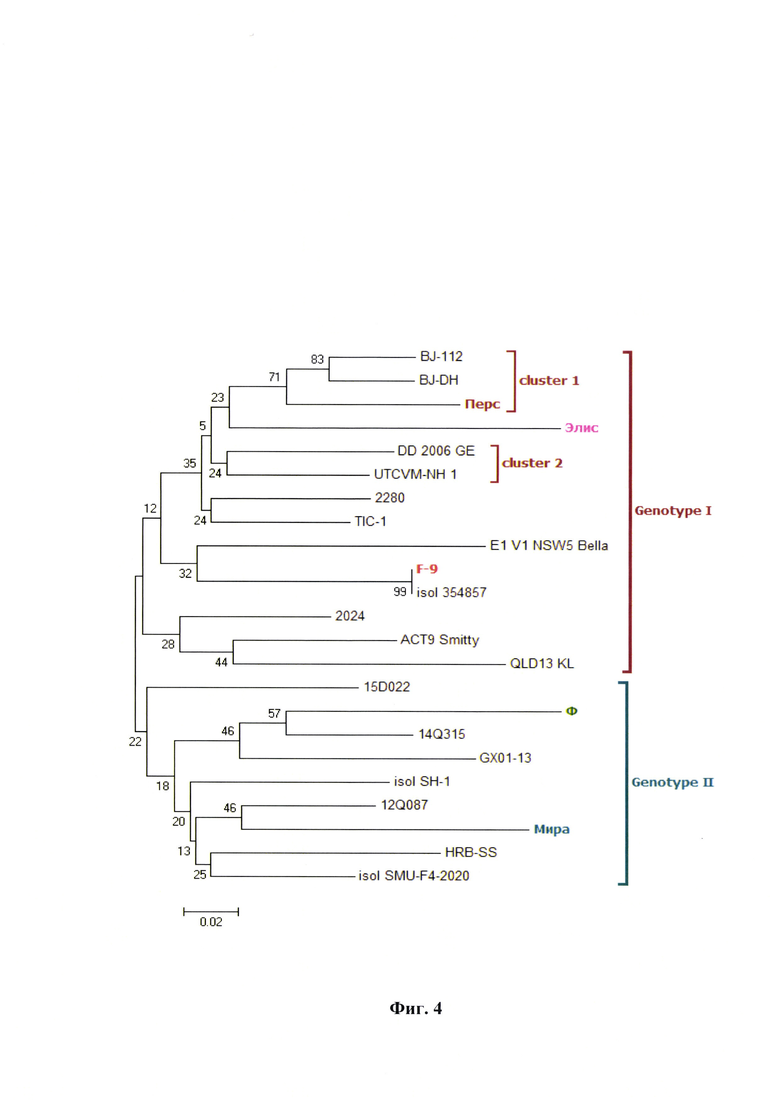
Фиг. 4 - Филогенетическое древо для некоторых изолятов/штаммов калицивируса кошек, построенное по данным, полученным с помощью разработанной тест-системы. Примечание: количество повторов 1000, алгоритм для построения Neighbor Joining.
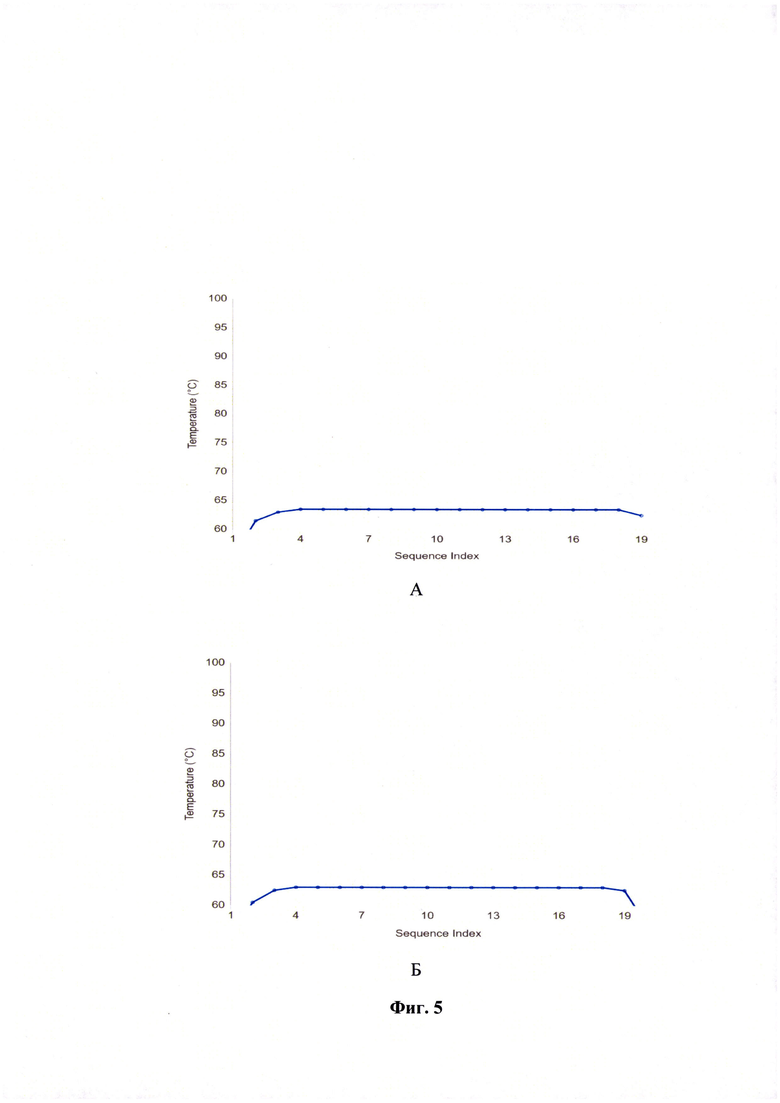
Фиг. 5 - График температуры плавления прямого и обратного праймеров, разработанных для дифференциации штаммов калицивируса кошек (с помощью программы uMeltMap (алгоритм Weber et al. 2015).
Сущность изобретения пояснена в перечне последовательностей, в котором:
SEQ ID N0:1 представляет последовательность нуклеотидов генов ORF2-ORF3 кДНК калицивируса кошек штамма «Мира»;
SEQ ID NO:2 представляет последовательность аминокислот, соответствующих участку генов ORF2-ORF3 кДНК калицивируса кошек штамма «Мира»;
SEQ ID NO:3 представляет последовательность нуклеотидов генов ORF2-ORF3 кДНК калицивируса кошек штамма «Элис»;
SEQ ID N0:4 представляет последовательность аминокислот, соответствующих участку генов ORF2-ORF3 кДНК калицивируса кошек штамма «Элис»;
SEQ ID NO:5 представляет последовательность нуклеотидов генов ORF2-ORF3 кДНК калицивируса кошек штамма «Перс»;
SEQ ID N0:6 представляет последовательность аминокислот, соответствующих участку генов ORF2-ORF3 кДНК калицивируса кошек штамма «Перс»;
SEQ ID N0:7 представляет последовательность нуклеотидов генов ORF2-ORF3 кДНК калицивируса кошек штамма «Фауна»;
SEQ ID N0:8 представляет последовательность аминокислот, соответствующих участку генов ORF2-ORF3 кДНК калицивируса кошек штамма «Фауна»;
SEQ ID NO:9 представляет последовательность нуклеотидов генов ORF2-ORF3 кДНК калицивируса кошек штамма «F-9»;
SEQ ID NO:10 представляет последовательность аминокислот, соответствующих участку генов ORF2-ORF3 кДНК калицивируса кошек штамма «F-9».
Сущность изобретения заключается в создании способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3. Заявляемый способ основан на: 1) элюировании РНК калицивируса кошек из исследуемого биологического материала; 2) проведении обратной транскрипции PFIK; 3) осуществлении амплификации специфического фрагмента кДНК калицивируса в диапазоне 7250…7573 п. н. с применением разработанных оригинальных олигонуклеотидных специфических праймеров FCV-F7250 и FCV-R7573 (длина ампликона 324 п. н.); 3) проведении очистки продуктов ПЦР; 4) секвенирование ампликона с применением разработанных оригинальных олигонуклеотидных специфических праймеров FCV-F7250 и FCV-R7573; 5) проведение филогенетического анализа с применением алгоритма Neighbor Joining и заключение о принадлежности штамма к определенному генотипу калицивируса кошек.
В публикациях многих авторов показана необходимость проведения дифференциации вакцинных штаммов калицивируса. По данным проведенного анализа литературных данных, в настоящее время подобный анализ проводят с помощью ПЦР и секвенирования участка гена ORF1, однако он содержит невысокое для проведения глубокого филогенетического анализа количество замен [2]. При анализе публикаций способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3 в диапазоне 7250…7573 п. н. (размер ПЦР-продукта 324 п. н.) не представлен. Таким образом, сведений об аналогах предлагаемого способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3 авторами не обнаружено.
Разработанный способ генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3 по сравнению с прототипом отличается высокой достоверностью, что позволяет точно определить принадлежность исследуемого изолята и/или штамма FCV к тому или иному генотипу.
В отличие от прототипа разработанный способ позволяет провести анализ выравнивания множественных нуклеотидных последовательностей геномов штаммов и изолятов калицивируса кошек и идентифицировать область генов ORF2-ORF3 с высоким количеством нуклеотидных замен (в среднем они составляют 26,23-31,48%). Данный способ предусматривает проведение реакции амплификации специфического фрагмента кДНК калицивируса кошек в диапазоне 7250…7573 п. н. с применением разработанных оригинальных олигонуклеотидных специфических праймеров ORF2-F7250 и ORF3-R7573 для амплификации фрагмента размером 324 п. н., содержащего нуклеотидные замены, позволяющие провести точный филогенетический анализ. Таким образом, актуально применять предложенный способ генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3 в диапазоне 7250…7573 п. н. кДНК.
Ключевым элементом заявляемого способа является амплификация участка кДНК FCV генов ORF2-ORF3 в диапазоне 7250…7573 п. н. с высоким количеством нуклеотидных замен с последующим секвенированием ампликонов и проведением детального филогенетического анализа штаммов калицивируса кошек.
Сопоставительный анализ с прототипом позволяет сделать вывод, что новизна и изобретательский уровень заявляемого изобретения заключается в применении способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3 в диапазоне 7250…7573 п.н. кДНК.
Технический результат заключается в расширении арсенала технических средств для проведения филогенетического анализа изолятов и штаммов калицивируса кошек методом секвенирования с применением разработанных оригинальных олигонуклеотидных специфических праймеров для высоковариабельного участка генов ORF2-ORF3, что позволит определять генетическую принадлежность исследуемых изолятов и штаммов и на основе этого составлять композицию вакцинных препаратов.
Сущность предлагаемого изобретения пояснена на графическом материале - Картирование прямого и обратного оригинальных праймеров, разработанных для проведения филогенетического анализа вакцинных штаммов калицивируса кошек методом секвенирования с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генома (фиг. 3А и 3Б) и Филогенетическое древо для некоторых изолятов/штаммов калицивируса кошек, построенное по данным, полученным с помощью разработанного способа (фиг. 4).
На первом этапе исследования проводят выделение нуклеиновой кислоты из анализируемых образцов, содержащих калицивирус кошек с применением твердофазного способа экстракции на стекловолокнистых фильтрах GF/F с использованием 5М раствора гуанидинтиоциата в качестве сильного детергента и смеси 80% пропанола-2 и 70% этанола, взятых в соотношении 4:1, для отмывания остатков балластных компонентов из пробы. Элюирование осуществляют в деионизированной воде объемом 40 мкл.
На этапе выделения нуклеиновой кислоты применяют положительный контроль (РНК FCV) и отрицательный контроль (деионизированная вода без нуклеаз MilliQ вместо ДНК-матрицы).
Полученный образец, содержащий РНК, исследуют с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) или аналога для определения степени чистоты РНК и концентрации нуклеиновой кислоты. Элюат РНК считали свободным от примесей белка и карболовой кислоты, если OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,8-2,2 и оптимально составлял примерно 2,0. Более низкие значения R1 указывали на наличие ДНК, протеина и остатков карболовой кислоты в экстракте. Более высокие значения коэффициента Ri свидетельствовали о деградации РНК и наличии свободных рибонуклеотидов. Экстракт нуклеиновой кислоты вируса считали незагрязненным полисахаридами, если OD260-262/OD235 (коэффициент экстинкции R2) приближен к значению 2,000. При замещении 1% РНК на полисахаридные составляющие R2 снижался на 0,002. Значения коэффициента R2 большие 2,000 могли указывать на деградацию молекул РНК. Отсутствие взвеси крупных частиц в элюате подтверждалось, если OD320 приближена к нулевому значению.
По итогам предварительно проведенной работы определили, что допустимая концентрация РНК в образце для последующих этапов анализа должна быть не ниже 3 нг/мкл.
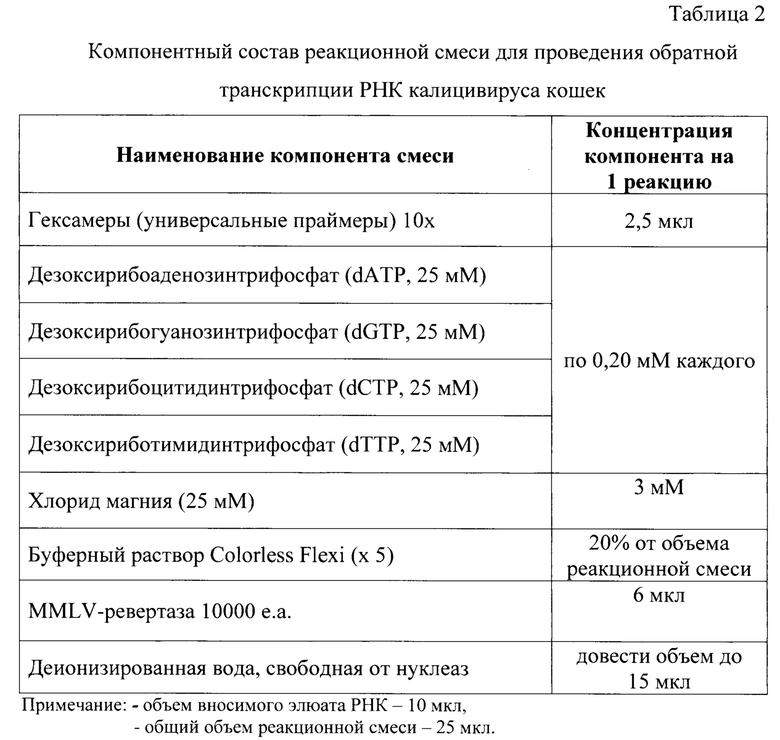
На следующем этапе проводят обратную транскрипцию и ПЦР с последующим гель-электрофорезом в 1% геле агарозы. Обратную транскрипцию проводят с применением реагентов, информация о которых отражена в таблице 2. В качестве универсальных праймеров для реверсии РНК используют гексамеры 10х в количестве 2,5 мкл, дезоксирибонуклеозидтрифосфаты по 0,2 мМ каждого, хлорид магния в концентрации 3 мМ, MMLV-ревертазу (1:10 000 е.а.) в объеме 6 мкл, буферный раствор бесцветный для ПЦР 5х в количестве 20% от общего объема. До 15 мкл объем доводят с помощью дистиллированной воды. Объем вносимого элюата РНК составляет 10 мкл. Итоговый объем реакционной смеси равен 25 мкл. Термоциклирование осуществляют при температуре 37°С в течение 30 мин.
На следующем этапе исследования проводят ПЦР в соответствии с прописью, которая отражена в таблице 3. Для анализа на 1 реакцию используют по 10 пМ прямого и обратного праймера, дезоксирибонуклеозидтрифосфаты по 0,2 мМ каждого, хлорид магния в концентрации 3 мМ, буферный раствор зеленого цвета (5х) - 20% от объема реакционной смеси, Taq ДНК-полимераза - 1 ед. Объем вносимого образца кДНК FCV - 5 мкл, общий объем реакционной смеси составляет 25 мкл.
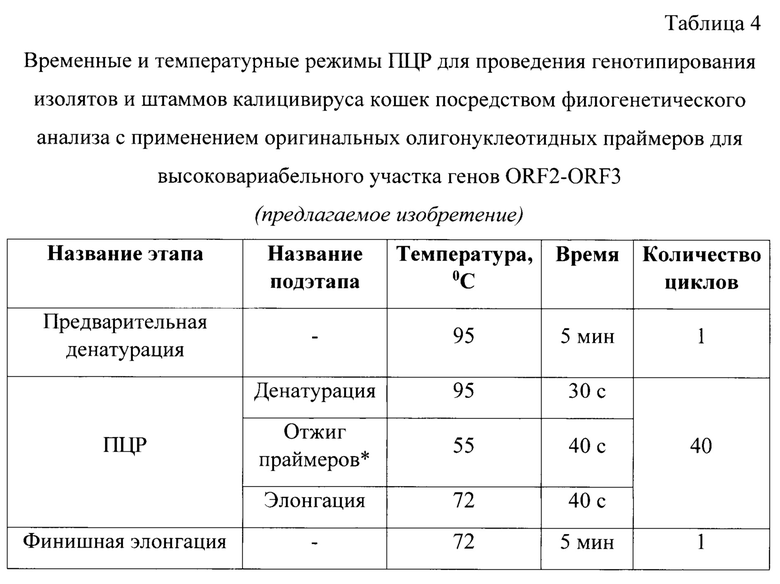
Режимы термоциклирования реакции амплификации отражены в таблице 4, из которой следует, что предварительную денатурацию осуществляют при температуре 95°С в течение 5 мин (1 цикл). ПЦР проводят в течение 40 циклов, каждый из которых складывается из трех подэтапов: денатурация - при температуре 95°С в течение 30 с, отжиг праймеров - при температуре 55°С в течение 40 с, элонгация - при температуре 72°С в течение 40 с. Финишную элонгацию осуществляют при температуре 72°С в течение 5 мин за 1 цикл.
При проведении ПЦР применяют положительный контроль (кДНК FCV) и отрицательный контроль (деионизированная вода без нуклеаз MilliQ вместо ДНК-матрицы).
Для детекции ПЦР-продуктов проводят горизонтальный гель-электрофорез в 1% агарозном геле по стандартной методике, внося в лунку геля весь объем исследуемой пробы. Оценивают наличие ампликонов. Критерии правильности проведенного исследования: для положительного контроля на этапе выделения нуклеиновой кислоты и положительного контроля на этапе проведения реакции амплификации должно быть наличие ПЦР-продукта с размером 324 п. н., что можно оценить с помощью маркера молекулярного веса для фрагментов ДНК. Для отрицательного контроля на этапе выделения нуклеиновой кислоты и постановки ПЦР ампликоны не обнаруживаются. При соблюдении данных условий можно детектировать промежуточные результаты реакции амплификации: при наличии светящихся бендов делают вывод о наличии специфического ПЦР-продукта (проба положительная, ее исследуют далее на следующих этапах работы), в случае отсутствия полос - ампликонов нет (проба отрицательная).
Проводят очистку ПЦР-продуктов из агарозного геля по стандартной методике. Готовят 4М раствор ГТЦ и 80%-ный раствор изопропилового спирта. Выставляют 3 ряда пробирок на 1,5 мл по количеству проб. В первый ряд пробирок вносят по 400 мкл 4М ГТЦ и добавляют пробы. Полученные смеси вносят на фильтр прибора Manifold, включают вакуумный насос и промывают фильтры 3 раза по 700 мкл пропанолом-2. Выключают насос. Переносят колонки в пробирки второго ряда и центрифугируют 1 минуту при скорости 13 400 об/мин. Колонки переносят в пробирки третьего ряда, вносят по 40 мкл деионизированной воды, инкубируют 1 минуту и центрифугируют при 13 400 об/мин. Полученные пробы исследуют с помощью гель-электрофореза для подтверждения очистки и наличия ампликонов. Очищенные ПЦР-продукты исследуют с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) или аналога для определения степени чистоты ампликонов и концентрации нуклеиновой кислоты (достаточное содержание ПЦР-продуктов составляет 20 нг/мкл).
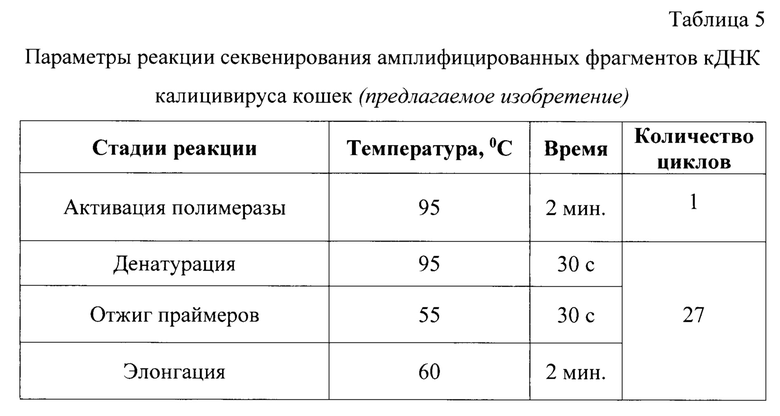
Проводят секвенирование ПЦР-продуктов. Определение нуклеотидных последовательностей полученных фрагментов генома калицивируса кошек осуществляют с использованием наборов производства Applied Biosystems (США) на автоматическом секвенаторе ABI Prism 3130/3130x1 (Applied Biosystems, США) согласно инструкциям производителя. Реакцию секвенирования осуществляют в программируемом амплификаторе с использованием набора Big Dye Terminator Kit версия 1.1 или 3.1 (Applied Biosystems, США). Перед началом сбора смеси необходимо разморозить все необходимые компоненты реакции, встряхнуть на шейкере, затем центрифугировать несколько секунд на низкоскоростной центрифуге.
Для реакции секвенирования амплифицированного фрагмента кДНК реакционная смесь собирается отдельно с каждым праймером ORF2-F7250 и ORF3-R7573. Реакционная смесь общим объемом 17,5 мкл на одну реакцию включает в себя следующие компоненты: Big Dye Terminator Kit - 8,0 мкл, буфер 5х для секвенирования - 8,0 мкл, раствор праймера (10 пмоль/мкл) - 1,0 мкл, Dream Taq ДНК-полимераза - 0,5 мкл.
Приготовленную реакционную смесь перемешивают, центрифугируют и вносят по 17,5 мкл в соответствующее количество пробирок объемом 0,2 мл. В пробирки с реакционной смесью добавляют 5 мкл очищенного фрагмента кДНК FCV. Общий объем реакции составляет 22,5 мкл.
Поверх смеси наслаивают 15 мкл минерального масла, чтобы исключить возможность ее испарения, даже в случае использования амплификатора с подогреваемой крышкой. Центрифугируют пробирки.
Приготовленные пробирки устанавливают в амплификатор и проводят реакцию в соответствии с температурно-временным режимом, представленном в таблице 5. Активацию ДНК-полимеразы проводят при температуре 95°С в течение 2 мин в течение 1 цикла. Секвенирование осуществляют за 27 циклов, каждый из которых состоит из трех подэтапов: денатурация - при температуре 95°С в течение 30 с, отжиг праймеров - при температуре 55°С в течение 30 с, элонгация - при температуре 60°С в течение 2 мин.
Анализируют сиквенсы, полученные в результате исследования. Проводят выравнивания последовательностей нуклеотидов участка генов ORF2-ORF3 в диапазоне 7250…7573 п. н. (размер ПЦР-продукта 324 п. н.) кДНК FCV. С помощью программы MEGA7 и алгоритма Neighbor Joining делают заключение о принадлежности штамма к определенному генотипу калицивируса кошек. Проводят построение филогенетического древа с обозначением месторасположения на нем интересующего штамма FCV.
Предлагаемый разработанный способ является новым и промышленно применимым. Способ создан путем подбора дизайна праймеров и других компонентов реакции, лабораторной оценки основных характеристик и совместного использования сведений, содержащихся в уровне техники, а также общих знаний специалиста в области биотехнологии.
Сущность изобретения поясняется на следующих примерах, полученных при апробации предлагаемого способа.
Пример 1. Разработка оригинальных олигонуклеотидных праймеров для способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа.
Для проведения генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа разрабатывали оригинальные специфичные для высоковариабельного участка кДНК FCV олигонуклеотидные праймеры. Так, для выявления регионов кДНК калицивируса кошек с высоким количеством различий между изолятами и штаммами FCV проводили выравнивание полногеномных последовательностей изолятов и штаммов, опубликованных в GenBank [10]. Для анализа из Genbank были взяты последовательности наиболее часто применяемые 19 следующих изолятов и штаммов калицивируса кошек: 2280, TIC-1, UTCVM-NH 1, DD 2006 GE, BJ-112, BJ-DH, АСТ9 Smitty, QLD 13 KL, 2024, E1 V1 NSW5 Bella, F-9, isol 354857 (генотип I); 12Q087, isol SMU-F-4-2020, HRB-SS, isol SH-1, GX01-13, 14Q315, 15D022 (генотип II).
В результате сравнительного анализа был выбран регион в диапазоне 7250…7573 п. н., поскольку именно данная область имеет наибольшее количество нуклеотидных замен в геноме FCV. Для данного участка разрабатывали прямой и обратный олигонуклеотидные праймеры, которые были гомологичны выбранным 19 последовательностям и позволили проводить амплификацию фрагмента размером 324 п. н., содержащего высокое количество нуклеотидных замен. Дизайн разработанных олигонуклеотидов представлен в таблице 6 и имеет следующий вид: ORF2-F7250-праймер - 5'-TTTCTGCCTCCTACATGGG-3' и ORF3-R7573-праймер -5'-TATGAGTAAGGGTCRACCCTA-3'.
Вышеупомянутые праймеры были отобраны, синтезированы и очищены. Специфичность последовательностей праймеров проверяли с помощью Базового инструмента поиска локального выравнивания (NCBI/Primer-BLAST). Определены температуры плавления (Tm) для каждого олигонуклеотида, сведения о которых представлены на фиг. 5. Термодинамические характеристики праймеров отражены в таблице 7, из которой следует, что температуры плавления разработанных олигонуклеотидов составляют около 62°С, учитывая метод uMelt, 57-60°С по данным метода учета концентрации солей, а прогнозируемые температуры их отжига - около 57°С, учитывая метод uMelt, и 52-55°С по данным метода учета концентрации солей. Проводя эксперименты по использованию данных праймеров выявили, что экспериментальная температура отжига составила 57°С.
При анализе нуклеотидных последовательностей олигонуклеотидов установили, что для праймеров не характерно образование «шпилек», а также не выявлено 3'-комплементарности и сайтов, отжигающих сами на себя. Расчет вероятности образования «шпилек» и димеров олигонуклеотидов проводили при условии, что минимальное количество пар оснований, необходимых для димеризации, - 5, а для образования «шпилек», - 4. Таким образом, были разработаны оригинальные специфичные олигонуклеотидные праймеры, которые позволили использовать для проведения филогенетического анализа изолятов и штаммов калицивируса кошек методом секвенирования с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Пример 2. Применение способа генотипирования вакцинного штамма «Мира» калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Проводили выделение нуклеиновой кислоты из суспензии калицивируса кошек штамма «Мира» с применением твердофазного способа экстракции на стекловолокнистых фильтрах GF/F с использованием 5М раствора гуанидинтиоциата в качестве сильного детергента и смеси 80% пропанола-2 и 70% этанола, взятых в соотношении 4:1, для отмывания остатков балластных компонентов из пробы. Элюирование осуществляли в деионизированной воде объемом 40 мкл. На этапе выделения нуклеиновой кислоты применяли положительный контроль (РНК FCV) и отрицательный контроль (деионизированная вода без нуклеаз MilliQ вместо ДНК-матрицы).
Полученный образец, содержащий РНК калицивируса кошек штамма «Мира», исследовали с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) для определения степени чистоты РНК и концентрации нуклеиновой кислоты. Элюат РНК считали свободным от примесей белка и карболовой кислоты, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 1,99. Экстракт нуклеиновой кислоты вируса считали незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) приближен к значению 2,000 и составил 2,001. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,001. Концентрация РНК калицивируса кошек составила 38 нг/мкл, что является допустимым и высоким значением для получения надежного результата.
На следующем этапе проводили обратную транскрипцию и ПЦР с последующим гель-электрофорезом в 1% геле агарозы. Для обратной транскрипции в качестве универсальных праймеров для реверсии РНК использовали гексамеры 10х в количестве 2,5 мкл, дезоксирибонуклеозидтрифосфаты по 0,2 мМ каждого, хлорид магния в концентрации 3 мМ, MMLV-ревертазу (1:10 000 е.а.) в объеме 6 мкл, буферный раствор бесцветный для ПЦР 5х в количестве 20% от общего объема. До 15 мкл объем доводили с помощью дистиллированной воды. Объем вносимого элюата РНК составил 10 мкл. Итоговый объем реакционной смеси равен 25 мкл. Термоциклирование осуществляли при температуре 37°С в течение 30 мин.
На следующем этапе исследования проводили ПЦР. Для анализа на 1 реакцию использовали по 10 пМ прямого и обратного праймера, дезоксирибонуклеозидтрифосфаты по 0,2 мМ каждого, хлорид магния в концентрации 3 мМ, буферный раствор зеленого цвета (5х) - 20% от объема реакционной смеси, Taq ДНК-полимераза - 1 ед. Объем вносимого образца кДНК FCV штамма «Мира» - 5 мкл, общий объем реакционной смеси составляет 25 мкл.
Режимы термоциклирования реакции амплификации отражены в таблице 4. При проведении ПНР применяли положительный контроль (кДНК FCV) и отрицательный контроль (деионизированная вода без нуклеаз MilliQ вместо ДНК-матрицы).
Для детекции ПЦР-продуктов проводили горизонтальный гель-электрофорез в 1% агарозном геле по стандартной методике, внося в лунку геля весь объем исследуемой пробы. Оценивали наличие ампликонов. Критерии правильности проведенного исследования отражены выше. В результате электрофореза обнаружили наличие бэнда размером 324 п. н. для положительного контроля на этапе выделения РНК, положительного контроля на этапе постановки ПЦР и для исследуемой пробы (фиг.1А и 1Б).
Проводили очистку ПЦР-продуктов из агарозного геля по стандартной методике, как описано выше. Очищенные ПЦР-продукты исследовали с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) для определения степени чистоты ампликонов и концентрации нуклеиновой кислоты. По результатам исследования выявили, что ампликоны имели высокую степень чистоты. Элюаты считали свободным от примесей белка и аминокислот, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 2,00. ПЦР-продукты были незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) составил 2,000. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,000. Концентрация ПЦР-продукта составила 45 нг/мкл, что является допустимым и высоким значением для получения надежного результата (более 20 нг/мкл). После очистки ПЦР-продуктов проводили гель-электрофорез, результаты которого были удовлетворительными (фиг. 2А и 2Б), а именно, ампликоны необходимого размера присутствовали на электрофореграмме.
Проводили секвенирование ПЦР-продукта для штамма «Мира» FCV (было изначально получено 8 одинаковых проб для повышения достоверности анализа с помощью секвенирования), как описано выше. Анализировали сиквенс, полученный в результате исследования. Осуществляли выравнивание последовательностей нуклеотидов участка генов ORF2-ORF3 в диапазоне 7250…7573 п. н. (размер ПЦР-продукта 324 п. н.) кДНК FCV штамма «Мира» со следующими штаммами/изолятами (представлены в GenBank и являются наиболее часто используемыми для филогенетического анализа исследователями в мире): 2280, TIC-1, UTCVM-NH 1, DD 2006 GE, BJ-112, BJ-DH, АСТ9 Smitty, QLD 13 KL, 2024, E1 V1 NSW5 Bella, F-9, isol 354857 (генотип I); 12Q087, isol SMU-F-4-2020, HRB-SS, isol SH-1, GX01-13, 14Q315, 15D022 (генотип II). С помощью программы MEGA7 и алгоритма Neighbor Joining сделали заключение о принадлежности штамма «Мира» к генотипу II калицивируса кошек. Выяснили, что он наиболее близок штамму 12Q087. Результаты анализа представили на филогенетическом древе с обозначением месторасположения на нем интересующего штамма «Мира» FCV (фиг. 4).
Пример 3. Применение способа генотипирования вакцинного штамма «Элис» калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Проводили выделение нуклеиновой кислоты из суспензии калицивируса кошек штамма «Элис» с применением твердофазного способа экстракции на стекловолокнистых фильтрах GF/F, как описано выше.
Полученный образец, содержащий РНК калицивируса кошек штамма «Элис», исследовали с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) для определения степени чистоты РНК и концентрации нуклеиновой кислоты. Элюат РНК считали свободным от примесей белка и карболовой кислоты, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 1,997. Экстракт нуклеиновой кислоты вируса считали незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) приближен к значению 2,000 и составил 1,999. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,001. Концентрация РНК калицивируса кошек составила 35 нг/мкл, что является допустимым и высоким значением для получения надежного результата.
На следующем этапе проводили обратную транскрипцию и ПЦР с последующим гель-электрофорезом в 1% геле агарозы. Анализ проводили, как описано выше. Данные гель-электрофореза отражены на фиг.1А и 1Б.
Проводили очистку ПЦР-продуктов из агарозного геля по стандартной методике, как описано выше. Очищенные ПЦР-продукты исследовали с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) для определения степени чистоты ампликонов и концентрации нуклеиновой кислоты. По результатам исследования выявили, что ампликоны имели высокую степень чистоты. Элюаты считали свободным от примесей белка и аминокислот, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 2,00. ПЦР-продукты были незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) составил 2,000. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,000. Концентрация ГЩР-продукта составила 47 нг/мкл, что является допустимым и высоким значением для получения надежного результата (более 20 нг/мкл).
Проводили секвенирование ПЦР-продукта для штамма «Элис» FCV (было изначально получено 8 одинаковых проб для повышения достоверности анализа с помощью секвенирования), как описано выше. Анализировали сиквенс, полученный в результате исследования. Осуществляли выравнивание последовательностей нуклеотидов участка генов ORF2-ORF3 в диапазоне 7250…7573 п. н. (размер ПЦР-продукта 324 п. н.) кДНК FCV штамма «Элис» со следующими штаммами/изолятами (представлены в GenBank и являются наиболее часто используемыми для филогенетического анализа исследователями в мире): 2280, TIC-1, UTCVM-NH 1, DD 2006 GE, BJ-112, BJ-DH, АСТ9 Smitty, QLD 13 KL, 2024, E1 V1 NSW5 Bella, F-9, isol 354857 (генотип I); 12Q087, isol SMU-F-4-2020, HRB-SS, isol SH-1, GX01-13, 14Q315, 15D022 (генотип II). С помощью программы MEGA7 и алгоритма Neighbor Joining сделали заключение о принадлежности штамма «Элис» к генотипу I калицивируса кошек. Результаты анализа представили на филогенетическом древе с обозначением месторасположения на нем интересующего штамма «Элис» FCV (фиг. 4).
Пример 4. Применение способа генотипирования вакцинного штамма «Перс» калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Проводили выделение нуклеиновой кислоты из суспензии калицивируса кошек штамма «Перс» с применением твердофазного способа экстракции на стекловолокнистых фильтрах GF/F, как описано выше.
Полученный образец, содержащий РНК калицивируса кошек штамма «Перс», исследовали с помощью спектрофотометра Implen NanoPhotometer Р-Class (Р-360) для определения степени чистоты РНК и концентрации нуклеиновой кислоты. Элюат РНК считали свободным от примесей белка и карболовой кислоты, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 1,998. Экстракт нуклеиновой кислоты вируса считали незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) приближен к значению 2,000 и составил 2,001. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,002. Концентрация РНК калицивируса кошек составила 33 нг/мкл, что является допустимым и высоким значением для получения надежного результата.
На следующем этапе проводили обратную транскрипцию и ПЦР с последующим гель-электрофорезом в 1% геле агарозы. Анализ проводили, как описано выше. Данные гель-электрофореза отражены на фиг. 1А и 1Б.
Проводили очистку ПЦР-продуктов из агарозного геля по стандартной методике, как описано выше. Очищенные ПЦР-продукты исследовали с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) для определения степени чистоты ампликонов и концентрации нуклеиновой кислоты. По результатам исследования выявили, что ампликоны имели высокую степень чистоты. Элюаты считали свободным от примесей белка и аминокислот, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 2,00. ПЦР-продукты были незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) составил 2,000. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,000. Концентрация ПЦР-продукта составила 44 нг/мкл, что является допустимым и высоким значением для получения надежного результата (более 20 нг/мкл).
Проводили секвенирование ПЦР-продукта для штамма «Перс» FCV (было изначально получено 8 одинаковых проб для повышения достоверности анализа с помощью секвенирования), как описано выше. Анализировали сиквенс, полученный в результате исследования. Осуществляли выравнивание последовательностей нуклеотидов участка генов ORF2-ORF3 в диапазоне 7250…7573 п. н. (размер ПЦР-продукта 324 п. н.) кДНК FCV штамма «Перс» со следующими штаммами/изолятами (представлены в GenBank и являются наиболее часто используемыми для филогенетического анализа исследователями в мире): 2280, TIC-1, UTCVM-NH 1, DD 2006 GE, BJ-112, BJ-DH, АСТ9 Smitty, QLD 13 KL, 2024, E1 V1 NSW5 Bella, F-9, isol 354857 (генотип I); 12Q087, isol SMU-F-4-2020, HRB-SS, isol SH-1, GX01-13, 14Q315, 15D022 (генотип II). С помощью программы MEGA7 и алгоритма Neighbor Joining сделали заключение о принадлежности штамма «Перс» к генотипу I кластеру 1 калицивируса кошек. Результаты анализа представили на филогенетическом древе с обозначением месторасположения на нем интересующего штамма «Перс» FCV (фиг. 4).
Пример 5. Применение способа генотипирования вакцинного штамма «Ф» калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Проводили выделение нуклеиновой кислоты из суспензии калицивируса кошек штамма «Ф» с применением твердофазного способа экстракции на стекловолокнистых фильтрах GF/F, как описано выше.
Полученный образец, содержащий РНК калицивируса кошек штамма «Ф», исследовали с помощью спектрофотометра Implen NanoPhotometer Р-Class (Р-360) для определения степени чистоты РНК и концентрации нуклеиновой кислоты. Элюат РНК считали свободным от примесей белка и карболовой кислоты, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 1,999. Экстракт нуклеиновой кислоты вируса считали незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) приближен к значению 2,000 и составил 2,000. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,002. Концентрация РНК калицивируса кошек составила 38 нг/мкл, что является допустимым и высоким значением для получения надежного результата.
На следующем этапе проводили обратную транскрипцию и ПЦР с последующим гель-электрофорезом в 1% геле агарозы. Анализ проводили, как описано выше. Данные гель-электрофореза отражены на фиг.1А и 1Б.
Проводили очистку ПЦР-продуктов из агарозного геля по стандартной методике, как описано выше. Очищенные ПЦР-продукты исследовали с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) для определения степени чистоты ампликонов и концентрации нуклеиновой кислоты. По результатам исследования выявили, что ампликоны имели высокую степень чистоты. Элюаты считали свободным от примесей белка и аминокислот, поскольку OD260-262/OD28O (коэффициент экстинкции Р1) находился в пределах 1,80-2,20 и составил 1,999. ПЦР-продукты были незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) составил 2,000. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,000. Концентрация ПЦР-продукта составила 41 нг/мкл, что является допустимым и высоким значением для получения надежного результата (более 20 нг/мкл).
Проводили секвенирование ПЦР-продукта для штамма «Ф» FCV (было изначально получено 8 одинаковых проб для повышения достоверности анализа с помощью секвенирования), как описано выше. Анализировали сиквенс, полученный в результате исследования. Осуществляли выравнивание последовательностей нуклеотидов участка генов ORF2-ORF3 в диапазоне 7250...7573 п. н. (размер ПЦР-продукта 324 п. н.) кДНК FCV штамма «Ф» со следующими штаммами/изолятами (представлены в GenBank и являются наиболее часто используемыми для филогенетического анализа исследователями в мире): 2280, TIC-1, UTCVM-NH 1, DD 2006 GE, BJ-112, BJ-DH, АСТ9 Smitty, QLD 13 KL, 2024, E1 V1 NSW5 Bella, F-9, isol 354857 (генотип I); 12Q087, isol SMU-F-4-2020, HRB-SS, isol SH-1, GX01-13, 14Q315, 15D022 (генотип И). С помощью программы MEGA7 и алгоритма Neighbor Joining сделали заключение о принадлежности штамма «Ф» к генотипу II калицивируса кошек. Результаты анализа представили на филогенетическом древе с обозначением месторасположения на нем интересующего штамма «Ф» FCV (фиг. 4).
Пример 6. Пргшенение способа генотипирования вакцинного штамма «F-9» калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Проводили выделение нуклеиновой кислоты из суспензии калицивируса кошек штамма «F-9» с применением твердофазного способа экстракции на стекловолокнистых фильтрах GF/F, как описано выше.
Полученный образец, содержащий РНК калицивируса кошек штамма «F-9», исследовали с помощью спектрофотометра Implen NanoPhotometer Р-Class (Р-360) для определения степени чистоты РНК и концентрации нуклеиновой кислоты. Элюат РНК считали свободным от примесей белка и карболовой кислоты, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 1,996. Экстракт нуклеиновой кислоты вируса считали незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) приближен к значению 2,000 и составил 2,002. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,001. Концентрация РНК калицивируса кошек составила 40 нг/мкл, что является допустимым и высоким значением для получения надежного результата.
На следующем этапе проводили обратную транскрипцию и ПЦР с последующим гель-электрофорезом в 1% геле агарозы. Анализ проводили, как описано выше. Данные гель-электрофореза отражены на фиг.1А и 1Б.
Проводили очистку ПЦР-продуктов из агарозного геля по стандартной методике, как описано выше. Очищенные ПЦР-продукты исследовали с помощью спектрофотометра Implen NanoPhotometer P-Class (Р-360) для определения степени чистоты ампликонов и концентрации нуклеиновой кислоты. По результатам исследования выявили, что ампликоны имели высокую степень чистоты. Элюаты считали свободным от примесей белка и аминокислот, поскольку OD260-262/OD280 (коэффициент экстинкции R1) находился в пределах 1,80-2,20 и составил 2,000. ПЦР-продукты были незагрязненным полисахаридами, поскольку OD260-262/OD235 (коэффициент экстинкции R2) составил 2,000. Отсутствие взвеси крупных частиц в элюате подтверждалось, поскольку OD320 составила 0,000. Концентрация ПЦР-продукта составила 44 нг/мкл, что является допустимым и высоким значением для получения надежного результата (более 20 нг/мкл).
Проводили секвенирование ПЦР-продукта для штамма «F-9» FCV (было изначально получено 8 одинаковых проб для повышения достоверности анализа с помощью секвенирования), как описано выше. Анализировали сиквенс, полученный в результате исследования. Осуществляли выравнивание последовательностей нуклеотидов участка генов ORF2-ORF3 в диапазоне 7250…7573 п. н. (размер ПЦР-продукта 324 п. н.) кДНК FCV штамма «F-9» со следующими штаммами/изолятами (представлены в GenBank и являются наиболее часто используемыми для филогенетического анализа исследователями в мире): 2280, TIC-1, UTCVM-NH 1, DD 2006 GE, BJ-112, BJ-DH, АСТ9 Smitty, QLD 13 KL, 2024, E1 V1 NSW5 Bella, F-9, isol 354857 (генотип I); 12Q087, isol SMU-F-4-2020, HRB-SS, isol SH-1, GX01-13, 14Q315, 15D022 (генотип II). С помощью программы MEGA7 и алгоритма Neighbor Joining сделали заключение о принадлежности штамма «F-9» к генотипу I калицивируса кошек. Результаты анализа представили на филогенетическом древе с обозначением месторасположения на нем интересующего штамма «F-9» FCV (фиг. 4).
Пример 7. Исследование аналитической специфичности разработанного способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Специфичность разработанного способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3 оценивали путем тестирования 8 образцов от других патогенов мелких домашних животных (штамм «Рокборн» вируса чумы плотоядных, штамм «Грей» возбудителя парвовирусного энтерита, штамм «Рич» возбудителя коронавирусного энтерита собак, штамм «ВГНКИ» аденовируса 1 серотипа). Анализ проводили, как представлено выше. В результате исследования амплификация с неспецифичными нуклеиновыми кислотами других представленных инфекционных агентов не была обнаружена (по данным гель-электрофореза ампликоны отсутствовали), то есть для проб, не содержащих нуклеиновую кислоту калицивируса кошек, и отрицательного контроля ПЦР-продукты не амплифицировались. При исследовании положительного контроля получены ампликоны размером 324 п. н. (n=3). Таким образом, полученные результаты свидетельствовали о 100%-ной специфичности разработанного способа генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3.
Основными преимуществами предлагаемого изобретения является возможность проведения филогенетического анализа изолятов и штаммов калицивируса кошек (FCV) посредством филогенетического анализа с применением разработанных оригинальных специфических олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3. Разработанный способ генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа впервые применили для решения данной задачи. Выявили, что в анализируемом участке кДНК калицивируса кошек в диапазоне 7250…7573 п. н. имеется максимальное количество нуклеотидных замен (среднее значение замен составляет 26,23-31,48% и на 5,28-23,18% выше в сравнении с прототипом). Это позволило разработать оригинальные специфические олигонуклеотидные праймеры ORF2-F7250 и ORF3-R7573 для детекции данного участка генома размером 324 п. н. с помощью обратной транскрипции и ПЦР. Впервые представлена идентификация нуклеотидных последовательностей данного участка генома вакцинных штаммов калицивируса кошек для проведения филогенетического анализа. Разработанный способ является специфичным по отношению к геному калицивируса кошек, что подтверждено исследованиями.
Данная разработка позволила проводить дифференциацию изолятов и штаммов калицивирусов и осуществлять их филогенетический анализ для создания эффективных вакцинных препаратов относительно возбудителя того или иного генотипа и диагностических гомологичных тест-систем.
Источники информации, принятые во внимание при составлении описания изобретения к заявке на выдачу патента РФ на изобретение «Способ генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3»:
1. Henzel A, Brum MC, Lautert C, Martins M, Lovato LT, Weiblen R. Isolation and identification of feline calicivirus and feline herpesvirus in Southern Brazil. Braz J Microbiol. 2012;43(2):560-568.
2. Gao J, Li Y, Xie Q, Al-Zaban MI, Al-Saeed FA, Shati AA, Al-Doaiss AA, Ahmed AE, Nawaz S, Ebrahem H, Irshad I, Kulyar MF, Li J. Epidemiological Investigation of Feline Upper Respiratory Tract Infection Encourages a Geographically Specific FCV Vaccine. Vet Sci. 2023 Jan 9;10(1):46. doi: 10.3390/vetsci10010046. PMID: 36669047; PMCID: PMC9864582.
3. Bordicchia M, Fumian TM, Van Brussel K, Russo AG, Carrai M, Le SJ, Pesavento PA, Holmes EC, Martella V, White P, Beatty JA, Shi M, Barrs VR. Feline Calicivirus Virulent Systemic Disease: Clinical Epidemiology, Analysis of Viral Isolates and In Vitro Efficacy of Novel Antivirals in Australian Outbreaks. Viruses. 2021 Oct 9;13(10):2040. doi: 10.3390/v13102040. PMID: 34696470; PMCID: PMC8537534.
4. Radford AD, Coyne KP, Dawson S, Porter CJ, Gaskell RM. Feline calicivirus. Vet Res. 2007 Mar-Apr;38(2):319-35. doi: 10.105l/vetres:2006056. Epub 2007 Feb 13. PMID: 17296159.
5. Hofmann-Lehmann R, Hosie MJ, Hartmann K, Egberink H, Truyen U, Tasker S, Belak S, Boucraut-Baralon C, Frymus T, Lloret A, Marsilio F, Pennisi MG, Addie DD, Lutz H, Thiry E, Radford AD, Möstl K. Calicivirus Infection in Cats. Viruses. 2022 Apr 29;14(5):937. doi: 10.3390/vl4050937. PMID: 35632680; PMCID: PMC9145992.
6. Meli ML, Berger A, Willi B, Spiri AM, Riond B, Hofmann-Lehmann R. Molecular detection of feline calicivirus in clinical samples: a study comparing its detection by RT-qPCR directly from swabs and after virus isolation. J Virol Methods. 2018;251:54-60.
7. Spiri AM. An Update on Feline Calicivirus. Schweiz Arch Tierheilkd. 2022 Mar;164(3):225-241. English, doi: 10.17236/sat00346. PMID: 35232714.
8. Radford AD, Addie D, Belak S, Boucraut-Baralon C, Egberink H, Frymus T, et al. Feline calicivirus infection. ABCD guidelines on prevention and management. J Feline Med Surg. 2009; 11(7):556-564.
9. Afonso MM, Pinchbeck GL, Smith SL, Daly JM, Gaskell RM, Dawson S, Radford AD. A multi-national European cross-sectional study of feline calicivirus epidemiology, diversity and vaccine cross-reactivity. Vaccine. 2017 May 9;35(20):2753-2760. doi: 10.1016/j.vaccine.2017.03.030. Epub 2017 Apr 5. PMID: 28389099.
10. GenBank. Feline calicivirus URL: https://www.ncbi.nlm.nih.gov/nuccore/?term=Feline+calicivirus+(Дата обращения: 16.05.2023).


--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="FCV phylogeni
_1685013195378.xml" softwareName="WIPO Sequence"
softwareVersion="2.1.2" productionDate="2023-05-25">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-05-25</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>506</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>0</ApplicationNumberText>
<FilingDate>2023-05-25</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">ФГБУ "Федеральный центр охраны
здоровья животных" (ФГБУ "ВНИИЗЖ")</ApplicantName>
<ApplicantNameLatin>FGBI "ARRIAH"</ApplicantNameLatin>
<InventorName languageCode="ru">Доронин Максим
Игоревич</InventorName>
<InventorNameLatin>Doronin Maksim Igorevich </InventorNameLatin>
<InventionTitle languageCode="ru">Тест-система для проведения
филогенетического анализа вакцинных штаммов калицивируса кошек
методом секвенирования с применением оригинальных олигонуклеотидных
праймеров для высоковариабельного участка генов ORF2-ORF3
</InventionTitle>
<SequenceTotalQuantity>10</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>324</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..324</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q1">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgcctcctacatgggaattcagctggcaaagcttcgtcttgctctata
cattaggagttccatgactaaaatttgaatccaatattgggtctcatagatactgtgactaacacaattg
gaagggctcagcaaattggattggacaaagctgcacttggtcaaagtcgcgagttggcattgaaacgcat
aaatcttgaccagcaagcattaaacaatcaagttgagcaatttaacaaattacttgagcagagggtacaa
ggcccagtccagtcggtgcgatttgcgcgcgcggctggatttagggttgacccttactcatacac</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q3">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LPPTWEFSWQSFVLLYTLGVPOLKFESNIGSHRYCDOHNWKGSANWIGQ
SCTWSKSRVGIETHKSOPASIKQSSOAIOQITOAEGTRPSPVGAICARGWIOGOPLLIH</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>324</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..324</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q5">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgcctcctacatgggaattcagctggcaaagcctcatctccccctcca
agttaggggttcaataagtaagatatgaattcaattttgggcttaattgttactgttacaaatacaatta
gaaaggcacaaagaattgaattggacaaggctgcgcttggtcagcaacgagaactggcactgcaacggat
gaacttggatcgccaggcgttgaacaaccaagtggagcaatttaacaaaattcttgagcagagggtacac
ggccccatccagtcagtacgatttgcgcgcgcggctggatttagggttgacccttactcatacac</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LPPTWEFSWQSLISPSKLGVQOVRYEFNFGLNCYCYKYNOKGTKNOIGQ
GCAWSATRTGTATDELGSPGVEQPSGAIOQNSOAEGTRPHPVSTICARGWIOGOPLLIH</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>324</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..324</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q7">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgcctcctacatgggaattcagctggcaaagattcgtcctccctcaaa
tattaggagcacaatgattagattatgaattcaatattgggtctaatcgacactgttactaacacaattg
gaaaagcccagcaaattgagttagataaagctgcccttggccaacagcgtgatctggcactccagcgaat
gaacctcgaccgccaggcgttgaacaaccaagttgagcagtttaacaagattcttgagcaaagggtacac
ggccccatccagtctgtgcggcttgcgcgcgcggctggatttagggttgacccttactcatacac</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LPPTWEFSWQRFVLPQILGAQOLDYEFNIGSNRHCYOHNWKSPANOVRO
SCPWPTAOSGTPANEPRPPGVEQPSOAVOQDSOAKGTRPHPVCAACARGWIOGOPLLIH</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>324</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..324</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q9">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgcctcctacatgggaattcagctggcaaaccttcgcttccctctata
cattaggagtcaaatgactaaattatgaatttcatattaggccttactgacactgtaacctgcacagtaa
gcaaagcccagcaaattgaattggataaggctgcacttggccaaaatagagagcttgcactcaaaagaat
taacttagatcagcaggcactcaaccaccaagtggagcaatttaacaaaattcttgagcaaagggtacaa
ggccctatccaatctgtacgacttgcgcgcgcggctggatttagggttgacccttactcatacac</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LPPTWEFSWQTFASLYTLGVKOLNYEFHIRPYOHCNLHSKQSPANOIGO
GCTWPKORACTQKNOLRSAGTQPPSGAIOQNSOAKGTRPYPICTTCARGWIOGOPLLIH</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>324</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..324</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>genomic DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q11">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctgcctcctacatgggaatacaattggcaaagatccggcttgcctctaa
cattaggagtcccatgactaagttatgaattcaatattaggcctgattgatactgttactaacactattg
gtaaagctcagcagattgaattggacaaagctgcacttggtcaacagcgtgaattggctctccagcgcat
tggcttggaccgccaagccttaaacaaccaagttgagcagtttaacaaaattcttgagcaaagggtacaa
ggcccaatccaatccgttcgccttgcgcgcgcggctggatttagggttgacccttactcatacac</INS
DSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>108</INSDSeq_length>
<INSDSeq_moltype>AA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..108</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>protein</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>FCV</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>LPPTWEYNWQRSGLPLTLGVPOLSYEFNIRPDOYCYOHYWOSSADOIGQ
SCTWSTAOIGSPAHWLGPPSLKQPSOAVOQNSOAKGTRPNPIRSPCARGWIOGOPLLIH</INSDSeq_s
equence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---
Изобретение относится к ветеринарной вирусологии, к средствам молекулярной диагностики. Предложен способ генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3. Способ предполагает использование разработанных оригинальных олигонуклеотидных праймеров со следующим дизайном: ORF2-F7250-праймер - 5'- TTTCTGCCTCCTACATGGG-3' и ORF3-R7573-праймер -5 - TATGAGTAAGGGTCRACCCTA-3' и включает проведение следующих этапов анализа и использование следующих компонентов реакции: - экстрагирование РНК из положительного и отрицательного контролей и исследуемых проб на стекловолокнистых фильтрах GF/F с использованием 5М раствора гуанидинтиоциата и смеси 80% пропанола-2 и 70% этанола, взятых в соотношении 4:1, проведение оценки степени чистоты элюатов и контроль концентрации РНК в образце; проведение обратной транскрипции и реакции амплификации в режиме реального времени Q с разработанными оригинальными олигонуклеотидными праймерами и температурой отжига, равной 55°С; детекция ПЦР-продуктов с помощью гель-электрофореза; очистка ампликонов, определение степени чистоты ПЦР-продуктов и определение концентрации амплифицированных фрагментов кДНК; секвенирование образцов и проведение филогенетического анализа на основе данных о нуклеотидных последовательностях исследуемых образцов штаммов калицивируса кошек. Разработанный способ является специфичным по отношению к геному калицивируса кошек. Способ позволил проводить дифференциацию изолятов и штаммов калицивирусов для создания эффективных вакцинных препаратов относительно возбудителя того или иного генотипа и диагностических гомологичных тест-систем. 2 з.п. ф-лы, 5 ил., 7 табл., 7 пр.
1. Способ генотипирования изолятов и штаммов калицивируса кошек посредством филогенетического анализа с применением оригинальных олигонуклеотидных праймеров для высоковариабельного участка генов ORF2-ORF3, отличающийся тем, что предполагает использование разработанных оригинальных олигонуклеотидных праймеров со следующим дизайном:
ORF2-F7250-npafiMep - 5 - TTTCTGCCTCCTACATGGG-3' и
ORF3-R7573-npaftMep -5'- TATGAGTAAGGGTCRACCCTA-3, и включающий проведение следующих этапов анализа и использование следующих компонентов реакции:
- экстрагирование РНК из положительного и отрицательного контролей и исследуемых проб на стекловолокнистых фильтрах GF/F с использованием 5М раствора гуанидинтиоциата и смеси 80% пропанола-2 и 70% этанола, взятых в соотношении 4:1, проведение оценки степени чистоты элюатов и контроль концентрации РНК в образце;
- проведение обратной транскрипции и реакции амплификации в режиме реального времени Q с разработанными оригинальными олигонуклеотидными праймерами и температурой отжига, равной 55°С;
- детекция ПГДР-продуктов с помощью гель-электрофореза;
- очистка ампликонов, определение степени чистоты ПЦР-продуктов и определение концентрации амплифицированных фрагментов кДНК;
- секвенирование образцов и проведение филогенетического анализа на основе данных о нуклеотидных последовательностях исследуемых образцов штаммов калицивируса кошек.
2. Способ по п. 1, отличающийся тем, что ПЦР проводят с соблюдением следующих режимов:
- предварительная денатурация: температура 95°С в течение 5 мин;
- ПЦР: денатурация - температура 95°С в течение 30 с, отжиг олигонуклеотидов - температура 55°С в течение 40 с, элонгация - температура 72°С в течение 40 с.
- финишная элонгация - температура 72°С в течение 5 с.
3. Способ по п. 1, отличающийся тем, что секвенирование амплифицированных фрагментов кДНК проводят с соблюдением следующих режимов:
- активация полимеразы - температура 95°С в течение 5 мин;
- денатурация - температура 95°С в течение 30 с;
- отжиг праймеров - температура 55°С в течение 30 с;
- элонгация - температура 60°С в течение 2 мин.
| Глотова Т.И | |||
| и др., ВЫДЕЛЕНИЕ И ФИЛОГЕНЕТИЧЕСКИЙ АНАЛИЗ КАЛИЦИВИРУСА КОШЕК В СИБИРИ, Problems of Virology | |||
| Способ получения цианистых соединений | 1924 |
|
SU2018A1 |
| Способ генотипирования энтеровирусов методом секвенирования 1A-1B участка генома | 2019 |
|
RU2701145C1 |
| WO 2011011431 A1, 27.01.2011. | |||
Авторы
Даты
2024-07-02—Публикация
2023-07-31—Подача