Область техники, к которой относится изобретение
Настоящее изобретение относится, к связывающим белкам, содержащим, по меньшей мере, две повторяющиеся области, специфически связывающие рецептор эпидермального фактора роста человека 2-го типа (HER2), а также к нуклеиновым кислотам, кодирующим такие связывающие HER2 белки, фармацевтическим композициям, содержащим такие белки, и применению таких белков при лечении болезней.
Предпосылки создания изобретения
Рецептор эпидермального фактора роста человека 2-го типа (HER2, зарегистрированный под номером Р04626 в базе данных UniProtKB/Swiss-Prot), также известный как ErbB2, является белком, который в организме человека кодируется геном ERBB2. Поскольку доказано, что амплификация или сверхэкспрессия этого гена играет важную роль в патогенезе и развитии рака определенных типов, за последние годы он превратился в важный биомаркер и мишень при лечении болезней. HER2 является трансмембранной рецепторной тирозинкиназой (RTK) и относится к более обширному семейству рецепторов ErbB (Bublil, Е.М. и Yarden, Y. Curr. Opin. Cell Biol. 19(2), 124-34, 2007). Рецепторы семейства ErbB встречается у позвоночных, и в него также входит рецептор-основатель семейства ErbBl (также называемый рецептором эпидермального фактора роста (EGFR) или HER1; зарегистрирован под номером Р00533 в базе данных UniProKB/Swiss-Prot белков человека) и обнаруженные в последнее время рецептор HER3 (также называемый ЕrbB3; зарегистрирован под номером Р21860 в базе данных UniProKB/Swiss-Prot белков человека) рецептор HER4 (также называемый ЕrbВ4; зарегистрирован под номером Q15303 в базе данных UniProKB/Swiss-Prot белков человека). Все рецепторы семейства ErbB имеют общие обширные гомологии последовательностей и областей и образуют функциональные гомодимеры (например, ErbBl-ErbBl, HER2-HER2 и HER4-HER4) и гетеродимеры во всех сочетаниях. Гомо- и гетеродимеризация рецепторов происходит после связывания связывание лиганда или сверхэкспрессии рецепторов, что в свою очередь вызывает активацию внутриклеточных областей рецепторов киназы путем автофосфорилирования. Затем инициируется внутриклеточный нисходящий сигнальный путь и биологические реакции. В отличие от остальных рецепторов ErbB, HER2 не имеет известного лиганда и способен образовывать димеры, что ярко проявляется после его сверхэкспрессии, в результате чего он активируется без предшествующего связывания лиганда. Что важно, HER3 не имеет активной внутриклеточной области киназы, и активируется посредством гетеродимеризации с другими рецепторами семейства ErbB, что приводит к передаче очень мощных сигналов по нисходящему пути. Такая гетеродимеризация и активация HER3 происходит после связывания с ним лиганда или в случае сильной сверхэкспрессии рецептора-партнера, такого как HER2.
HER2, а также все остальные рецепторы семейства ErbB состоят из четырех внеклеточных областей, которые последовательно именуются областями I, II, III и IV, при этом область IV является ближайшей к клеточной мембране, а область I наиболее удаленной от нее. В условиях отсутствия лигандов в областях I и III рецепторов ErbB происходят общее внутримолекулярное взаимодействие, которое закрывает область II. В результате этого предотвращается гомо-/гетеродимеризация рецепторов и передача сигналов, поскольку для димеризации требуется взаимодействие между областями II двух соседних рецепторов ErbB (Burguess A.W. и др., Mol. Cell 12(3), 541-552, 2003). Связывание лиганда нарушает взаимодействие между областями I и III, в результате чего происходит конформационное изменение рецепторов из связанной формы в растянутую, и область II остается открытой. Это заставляет рецептор делать случайный выбор при димеризации с другими расширенными рецепторами ErbB и инициировать передачу сигналов. Интересно, что, поскольку HER2 является единственным конститутивным рецептором семейства ErbB при растянутой конформации, область II постоянно открыта и доступна для гомо- и гетеродимеризации.
Димеризация и автофосфорилирование рецепторов ErbB приводит к активации множества ключевых молекул в нисходящем сигнальном пути, вовлеченных как в нормальные физиологические процессы, так и патологические процессы. Природа таких активируемых сигнальных молекул до некоторой степени зависит от состава димеризованных активных рецепторов ErbB. Например, гомодимеры HER1-HER1 и HER2-HER2 предпочтительно активируют нисходящий сигнальный путь и пролиферацию регулируемой внеклеточными сигналами киназы (ERK), тогда как гетеродимеры HER2-HER3 также активируют сигнальный путь PI3K (включая активацию киназы АКТ нисходящего сигнального пути) и тем самым выживание клеток. По существу, активация АКТ посредством сигнального пути HER2-HER3 в опухолевых клетках способствует их выживанию и делает опухолевые клетки резистентными к лекарствам, нацеленным на HER2, таким как моноклональное. антитело трастузумаб (Berns К. и др., Cancer Cell 12, 395-402, 2007). Интересно, что ингибирование опосредованного HER2-HER3 сигнального пути PI3K-AKT в этих клетках становится ограничивающим выживаемость фактором и приводит к некрозу клеток. Помимо пролиферации и выживаемости клеток сигнальный путь HER2 также участвует в других процессах, таких как развитие кровеносных сосудов и миграция.
Приблизительно в 20% случаев рака молочной железы всех типов наблюдается сверхэкспрессия HER2. С учетом своей клинической значимости HER2 стал первым RTK, против которого была разработан нацеленный биологический препарат, а именно, трастузумаб (Herceptin®; Genentech). Это антитело связывает область IV HER2 и ингибирует сигнальный путь HER2 посредством нескольких механизмов, которые еще не до конца поняты. В их число входит индуцирование интернализации рецепторов в опухолевых клетках, что вызывает снижение уровней экспрессии и подавление сигнального пути HER2 и приводит к ослаблению опухолеобразующего фенотипа. Трастузумаб продлил жизнь и повысил качество жизни десятков тысяч женщин с раком молочной железы. Тем не менее, поскольку трастузумаб в основном противодействует пролиферации клеток, опухоли на поздних стадиях заболевания могут не поддаваться такому лечению. В попытке создания более эффективных средств лечения было получено новое антитело, которое распознает область II или HER2, а именно, пертузумаб (Omnitarg®, Perjeta®; Genentech). В отличие от трастузумаба это антитело имеет целью не снижение уровней мембранной экспрессии HER2, а противодействие образованию гомо- и гетеродимеров HER2 путем связывания и закрытия области II димеризации рецептора. Эффективность лечения пертузумабом в качестве единственного средства является неожиданно низкой in vitro и in vivo, но его сочетание с трастузумабом обладает синергетическим эффектом. Соответственно, сочетание обоих антител может стать стандартной схемой терапии при лечении пациентов с раком молочной железы (Capelan M. и др., Ann. Oncol., 24, 273-82, 2013).
Успех доклинических и клинических испытаний сочетания трастузумаба и пертузумаба дал толчок идее, согласно которой для достижения более высокой противоопухолевой эффективности требуется двойное нацеливание на области II и IV в HER2. Эта идея подтверждается другими молекулами, созданными в последнее время с целью одновременного нацеливания на области II и IV в HER2. Например, датской компанией Symphogen ведется разработка смешанных антител к областям II и IV HER2, которые продемонстрировали несколько более высокую эффективность (т.е. более высокую, чем у трастузумаба в отдельности) при доклинических испытаниях на моделированных на мышах опухолях.
Аналогичным образом, из US 2011/033460 известно, что сочетание антител, связывающих область I и область IV HER2, обладает синергетическим эффектом в отношении синтеза ДНК и жизнеспособности клеток ВТ474. Кроме того, в US 2011/033460 также описаны биспецифические антитела, которые связывают два различных эпитопа HER2, один из которых находится в области IHER2, а другой - в области IV HER2.
В WO 2009/068625 описана разработка бипаратопных моделей антител, содержащих первую область, которая конкурирует с трастузумабом за связывание HER2, и вторую область, которая связывает отличающийся эпитоп или часть HER2. Интересно, что, некоторые модели являются антагонистами пролиферации клеток SKBR3, а другие модели обладают агонистическим действием. В частности, в WO 2009/068625 описана разработка бипаратопных моделей антител, содержащих первую область, которая конкурирует с трастузумабом за связывание HER2 (т.е. связывание области IV Неr2), и вторую область, которая конкурирует с пертузумабом за связывание HER2 (т.е. связывание области II HER2). Модели, в которых связывающая область IV область антитела клонирована как N-конец связывающей область II области антитела, блокируют активацию МАР-киназы, тогда как такое блокирование не наблюдается при другой ориентации (т.е. при нахождении связывающей область II области антитела на N-конце). В целом, в WO 2009/068625 описаны разнообразные модели бипаратопных антител, нацеленных на HER2, которые обладают различными степенями действия (агонистического или антагонистического) на пролиферации или сигнальный путь клеток SKBR3, но не описано цитотоксическое или апоптическое действие.
Также известны бивалентные связывающие белки, такие как бивалентные молекулы димерных или аффинных антител, нацеленные на HER2 (Nielsen, U.B. и др., Cancer Res., 60, 6434-6440, 2000; Steffen, А-С, Cancer Biother. Radiopharmaceut. 20, 239-248, 2005). Поскольку в таких молекулах дважды повторена одна связывающая область, они отличаются от бипаратопных молекул, которые содержат две связывающие области, каждая из которых связывает отличающийся эпитоп в одной молекуле-мишени.
В качестве альтернативы средствам на основе антител и SMI, известны новые связывающие белки или связывающие области, которые могут применяться для специфического связывания молекулы-мишени (например, Binz, Н.К., Amstutz, Р. и Plückthun, Α., Nat. Biotechnol. 23, 1257-1268, 2005) и тем самым действовать как антагонист. Основой связывающих белков или связывающих области одного такого нового класса, не обладающих Fс, являются сконструированные повторяющиеся белки или сконструированные повторяющиеся области (WO 2002/020565; Binz, Н.К., Amstutz, P., Kohl, Α., Stumpp, M.T., Briand, С, Forrer, P., Grütter, M.G. и Plückthun, Α., Nat. Biotechnol. 22, 575-582, 2004; Stumpp, M.T., Binz, H.K и Amstutz, P., Drug Discov. Today 13, 695-701,2008).
В WO 2002/020565 описано создание крупных библиотек повторяющихся белков и их общее применение. В таких сконструированных повторяющихся областях используется модульная природа повторяющихся белков, и они могут обладать N-концевыми и С-концевыми кэппирующими модулями для предотвращения агрегации повторяющихся областей путем экранирования гидрофобной сердцевины области (Forrer, P., Stumpp, М.Т., Binz, H.K. и Plückthun, Α., FEBS letters 539, 2-6, 2003). В этот новый класс связывающих белков входят сконструированные анкириновые повторы (DARPins). В литературе описано моноспецифическое связывание HER2 посредством DARPins (например, Steiner, D., Forrer, Р. и Plückthun, Α., J. Mol. Biol. 382, 1211-1227, 2008; Zahnd, С, Pecorari, F., Straumann, Ν., Wyler, Ε. и Plückthun, Α., J. Biol. Chem. 281(46), 35167-35175, 2006).
Недавно описан биспецифический сконструированный анкириновый повтор, нацеленный на HER2 (Jost, Ch. и др., Structure 21, 1-13, 2013). Авторами показано, что при связывании двумя областями анкириновых повторов, соединенными коротким линкером (более длинные линкеры неэффективны), одна из которых нацелена на область I Her2, а другая на область IV Her2, обеспечивается более сильное цитотоксическое действие на клетки ВТ474, чем в случае только трастузумаба, который нацелен на область IV Her2. Этот бипаратопный повторяющийся белок действует путем внутримолекулярного сшивания двух молекул Her2, т.е. он соединяет две мембраносвязанные молекулы HER2, деформируя их таким образом, что лишить их способности образовывать обеспечивающие сигнальный путь димеры с каким-либо рецептором семейства EGFR, что предотвращает димеризацию киназы и тем самым обеспечивает наблюдаемое цитотоксическое действие.
Несмотря на то, что из уровня техники известны выгоды нацеливания на HER2 при лечении таких болезней, как рак, существует явная потребность с создании связывающих белков, с более высокой эффективностью нацеленных на HER2.
Задача изобретения
В основу настоящего изобретения положена задача создания новых антагонистов Her2.
Другой задачей настоящего изобретения является создание нового механизма ингибирования связанного с HER2 клеточного сигнального пути.
Еще одной задачей настоящего изобретения является создание нового подхода к ингибированию опосредованной HER2 пролиферации клеток и/или индуцированию апоптоза в клетке (например, опухолевой клетке), ткани, органе или организме пациента.
Еще одной задачей настоящего изобретения является создание монотерапии, нацеленной на две области Her2, путем использования бипаратопных повторяющихся белков.
Еще одной задачей настоящего изобретения является создание новых возможностей лечения рака.
Еще одной задачей настоящего изобретения является создание средства лечения неоплазий с высокой эффективностью и/или незначительными побочными эффектами.
Еще одной задачей настоящего изобретения является создание альтернативного средства лечения неоплазий, которые не реагируют (или только частично реагируют) или являются резистентными к известным из уровня техники терапиям.
Краткое изложение сущности изобретения
Решение этих задач достигается за счет объекта независимых пунктов формулы изобретения, при этом в зависимых пунктах и в описании раскрыты дополнительные предпочтительные варианты осуществления.
Хотя изобретение проиллюстрировано на чертежах и подробно описано далее, такие чертежи и описание следует считать иллюстрирующими или поясняющими, а не ограничивающими изобретение, которое не ограничено раскрытыми вариантами осуществления. При практическом применении заявленного изобретения специалисты в данной области техники, изучившие чертежи, описание и прилагаемую формулу изобретения, могут предложить и реализовать другие разновидности раскрытых вариантов осуществления. Термин "содержащий" в формуле изобретения не исключает другие элементы или стадии, а неопределенный артикль не исключает множественного числа. Тот факт, что некоторые признаки содержатся в различных пунктах формулы изобретения, не означает невозможность выгодного применения сочетания этих признаков. Приведенные в формуле изобретения ссылочные позиции не следует трактовать как ограничение ее объема.
Краткое описание чертежей
Фиг. 1
Связывание HER2 областями DARPin
Испытали связывание моновалентными DARPins внеклеточной области HER2 (областей I-IV) методом конкурентного твердофазного иммуноферментного анализа (ИФА) с использованием областей очищенного HER2 (области I, областей III-IV или областей I-III) в качестве конкурентов, как показано на фиг. 1А и 1В. В присутствии 500 нМ области I Her2 DARPin # 51 и DARPin # 52 больше не способны связывать HER2 (область I-IV), что говорит о том, что они связывают эпитоп в области I. DARPin # 7, DARPin # 53 и DARPin # 54 связывают область II, поскольку ни 500 нМ области I Her2, ни 500 нМ областей III-IV Her2 не способны предотвратить связывание ими Her2 (областей I-IV) по всей длине. На фиг. 1С показано, что моновалентные DARPins способны связывать предварительно сформированный комплекс НЕR2-пертузумаб и, соответственно, эпитоп, который они связывают, отличается от эпитопа, который связывает пертузумаб в области II HER2. Смотри далее определения DARPins. OD означает оптическую плотность при концентрации 450 нМ минус OD при концентрации 620 нм; С означает контрольный DARPin, который не связывает HER2; d1 означает область I HER2; d1-3 означает области I-III HER2; d3-4 означает области III-IV HER2.
Фиг. 2
Ингибирование пролиферации клеток ВТ474 моновалентными и бипаратопными связывающими белками
Определили ингибирование пролиферации клеток ВТ474 моновалентными DARPins (т.е. DARPin # 1 и DARPin # 18) в виде нековалентной смеси этих моновалентных DARPins и бипаратопных связывающих белков, содержащих эти моновалентные DARPins с различными ориентациями (DARPin # 41 и DARPin # 49). На фиг. 2А показано ингибирование пролиферации клеток бипаратопными DARPins в различных концентрациях, и представлены соответствующие подогнанные кривые ингибирования для отдельного единичного эксперимента. Затем вычислили показатель IC50 для DARPin #41, который составил около 2 нм. В Таблице 2 приведены показатели IC50 для отдельных DARPins. На диаграмме на фиг. 2А показана оптическая плотность (OD) при концентрации 450 мн минус OD при концентрации 620 нм в зависимости от концентрации (С) DARPins в нМ. По шкале X отложена логарифмическая шкала. На фиг. 2В показано ингибирование пролиферации бипаратопными DARPins в виде смеси как моновалентных DARPins, так и отдельных соответствующих моновалентных DARPins в концентрации 100 нМ. По оси Y отложена OD. Ингибирование пролиферации отобржено низкой OD. Смотри далее определения DARPins. #41 означает DARPin # 41; # 49 означает DARPin # 49; # 18 означает DARPin # 18; # 1 означает DARPin # 1; п. с. означает отрицательный контроль.
Фиг. 3
Ингибирование пролиферации клеток ВТ474 различными бипаратопными DARPins
Показано ингибирование пролиферации клеток ВТ474 подмножеством бипаратопных DARPins (# 23, # 24, # 33, # 37, # 43, # 44 и # 41), содержащих различные N-концевые и/или С-концевые области анкириновых повторов. Показаны кривые ингибирования пролиферации DARPins в различных концентрациях и соответствующие подогнанные кривые ингибирования для каждого отдельного единичного эксперимента. В Таблице 2 приведены показатели IC50 для отдельных DARPins. На фиг. 3А показано ингибирование бипаратопными DARPins с DARPin # 15 на N-конце, а на фиг. 3В показано ингибирование бипаратопными DARPins с DARPin # 18 на С-конце. На фиг. 3С показано ингибирование бипаратопными DARPins с DARPin # 51 на N-конце и DARPin # 18 на С-конце, а на фиг. 3D показано ингибирование бипаратопными DARPins с DARPin #51 на N-конце и DARPin # 21 на С-конце. На диаграмме показана оптическая плотность (OD) при концентрации 450 мн минус OD при концентрации 620 нм в зависимости от концентрации (С) DARPins в нМ. По шкале X отложена логарифмическая шкала. Смотри далее определения DARPins. # 23 означает DARPin # 23; # 24 означает DARPin # 24; # 33 означает DARPin # 33; # 37 означает DARPin # 37; # 41 означает DARPin # 41; # 43 означает DARPin # 43; # 44 означает DARPin # 44.
Фиг. 4
Ингибирование пролиферации клеток различных линий бипаратопными DARPin #41
Определили ингибирование пролиферации клеток NCI-N87 (фиг. 4А), ZR75-30 (фиг. 4В) и MDA-MB175 (фиг. 4С) DARPin # 41 и трастузумабом. Показано ингибирование пролиферации DARPins в различных концентрациях и соответствующие подогнанные кривые ингибирования для каждого отдельного единичного эксперимента. В Таблице 3 приведены показатели IC50 для отдельных линий клеток. На диаграмме показана оптическая плотность (OD) при концентрации 450 мн минус OD при концентрации 620 нм в зависимости от концентрации (С) DARPins в нМ. По шкале X отложена логарифмическая шкала. Смотри далее определения DARPins и эталонных молекул. #41 означает DARPin # 41; Τ означает трастузумаб.
Фиг. 5
Индуцирование апоптоза бипаратопными DARPin # 41 в различных линиях клеток.
Определили индуцирование апоптоза в клетках ВТ474 (фиг. 5А), NCI-N87 (фиг. 5 В) и MDA-MB175 (фиг. 5С) DARPin # 41 и трастузумабом. Показано индуцирование апоптоза DARPins в различных концентрациях и соответствующие подогнанные кривые ингибирования для каждого отдельного единичного эксперимента. В Таблице 3 приведены показатели IC50 для отдельных линий клеток. В диаграмме на фиг. 5А показана оптическая плотность (OD) при концентрации 450 нМ минус OD при концентрации 490 нМ в зависимости от концентрации (С) DARPins или трастузумаба в нМ. В диаграмме на фиг. 5В и 5С показаны относительные световые единицы (RLU) в зависимости от концентрации (С) DARPins или трастузумаба в нМ. По шкале X отложена логарифмическая шкала. Смотри далее определения DARPins. Τ означает трастузумаб; #41 означает DARPin #41.
Фиг. 6
Сравнение эффективности DARPin # 41 с эталонами при ингибировании пролиферации клеток и индуцировании апоптоза
Определили ингибирование пролиферации (фиг. 6А) и индуцирование апоптоза (фиг. 6В) в клетках ВТ474 для DARPin #41, используемых в качестве эталонов трастузумаба и пертузумаба и сочетания 100 нМ трастузумаба и титрованного пертузумаба. На фиг. 6А показано ингибирование пролиферации клеток DARPin в различных концентрациях и эталонами в соответствующих концентрациях, и показаны соответствующие подогнанные кривые ингибирования для каждого отдельного единичного эксперимента. В Таблице 3 приведены показатели IC50 для отдельных линий клеток. На диаграмме показана оптическая плотность (OD) при концентрации 450 нМ минус OD при концентрации 620 нМ в зависимости от концентрации (С) DARPin/эталонов в нМ. По шкале X отложена логарифмическая шкала. На фиг. 6 В показано индуцирование апоптоза DARPin в различных концентрациях, эталонами в соответствующих концентрациях, и показаны соответствующие подогнанные кривые активации для каждого отдельного единичного эксперимента. В Таблице 3 приведены показатели IC50 для отдельных линий клеток. На диаграмме показаны относительные световые единицы (RLU) в зависимости от концентрации (С) DARPin/эталоном в нМ. По шкале X отложена логарифмическая шкала. Смотри далее определения DARPins. Τ означает трастузумаб; Ρ означает пертузумаб; # 41 означает DARPin # 41.
Фиг. 7
Ингибирование пролиферации клеток ВТ474 бипаратопными связывающими белками различных форматов
Показано ингибирование пролиферации клеток ВТ474 бипаратопными DARPins различных форматов с DARPin # 1 на N-конце и DARPin # 18 на С-конце. На фиг. 7А показано ингибирование пролиферации бипаратопными DARPins в различных концентрациях с длительным периодом полураспада в сыворотке, и показаны соответствующие подогнанные кривые ингибирования для отдельного единичного эксперимента. Бипаратопный DARPin # 63 имеет пэгилированный цистеиновый остаток на С-конце, а бипаратопные DARPins # 64 и # 65 содержат область анкиринового повтора, связывающую сывороточный альбумин. На фиг. 7 В показано ингибирование пролиферации бипаратопными DARPins в различных концентрациях, содержащими отличающиеся линкеры между повторяющимися областями, связывающими HER2, и показаны соответствующие подогнанные кривые ингибирования для отдельного единичного эксперимента. В Таблице 2 приведены показатели IC50 для DARPins. На диаграмме показана оптическая плотность (OD) при концентрации 450 мН минус OD при концентрации 620 нм в зависимости от концентрации (С) DARPins в нМ. По шкале X отложена логарифмическая шкала. Смотри далее определения DARPins. # 66 означает DARPin # 66, который содержит короткий GS-линкер длиной в две аминокислоты между двумя повторяющимися областями; # 67 означает DARPin # 67, который содержит GS-линкер длиной в пять аминокислот между двумя повторяющимися областями; #41 означает DARPin #41, который содержит GS-линкер длиной в десять аминокислот между двумя повторяющимися областями; # 68 означает DARPin # 68, который содержит РТ-линкер длиной в 24 аминокислоты между двумя повторяющимися областями.
Подробное описание изобретения
В одном из вариантов осуществления изобретения предложен рекомбинантный связывающий белок, содержащий, по меньшей мере, первую и вторую повторяющиеся области, каждая из которых связывает внеклеточную область HER2, при этом повторяющиеся области ковалентно связаны.
Было неожиданно обнаружено, что связывание внеклеточной части HER2 рекомбинантным связывающим белком, содержащим, по меньшей мере, два ковалентно связанные повторяющиеся области, каждая из которых обладает специфичностью к внеклеточной области HER2, оказывает выгодное и неожиданное действие по сравнению с рассмотренными выше известными из техники подходами, согласно которым HER2 связывают отдельные связующие (например, сочетание трастузумаба и пертузумаба; фиг. 6).
HER2 человека состоит из 1255 аминокислот с 21 аминокислотной сигнальной последовательностью, 631 аминокислотной внеклеточной областью (например, эктообластью, содержащей области I-IV), 23 аминокислотными трансмембранными областями и 580 аминокислотными цитоплазматическими областями.
Упомянутое связывание внеклеточной области HER2 рекомбинантным связывающим белком предпочтительно представляет собой синхронное или одновременное связывание повторяющимися областями внеклеточной области HER2. Кроме того, повторяющиеся области предпочтительно связывают два различных эпитопа внеклеточной области HER2. Помимо этого, повторяющиеся области предпочтительно связывают два различных и не перекрывающих друг друга эпитопа внеклеточной области HER2.
Эта повышенная эффективности может объясняться тем, что рекомбинантный связывающий белок согласно изобретению индуцирует до сих пор не описанную связанную конформацию внеклеточной области HER2, которое, вероятно, является следствием внутримолекулярного взаимодействия бипаратопного связывающего белка согласно изобретению с двумя различными эпитопами во внеклеточной области HER2 (Пример 8); т.е. обе повторяющиеся области связывающего белка, вероятно, одновременно связывают различные эпитопы в одной и той же молекуле HER2 и тем самым побуждают внеклеточную область HER2 перейти к этой новой связанной конформации. Такая связанная конформация не известна из техники. Важно, что эти две повторяющиеся области должны быть связаны за счет присутствия в одном и том же связывающем белке, т.е. простая смесь двух повторяющихся областей неэффективна (фиг. 2 В). Кроме того, бивалентное связывание внеклеточной области HER2 таким связывающим белком может создавать синергетический эффект связывания за счет повышения авидности, т.е. совокупной силы синхронного связывания различных эпитопов мишени. Авидность отличается от сродства, которое соответствует силе отдельного связывающего взаимодействия. В целом, этим специфическим взаимодействием связывающего белка с HER2 может объясняться высокоэффективное ингибирование пролиферации и индуцирования апоптоза такими молекулами, как показаны в примерах.
Согласно этой теории две повторяющиеся области в одном и том же белке взаимно усиливают связывание своего соответствующего эпитопа и тем самым обеспечивают усиление общего сродства к мишени.
В результате связывания первой повторяющейся областью своего эпитопа в HER2 вторая повторяющаяся область занимает энергетически и/или пространственно выгодное положение, которое облегчает связывание ей своего соответствующего эпитопа в HER2.
Как показано в примерах, ковалентная связь первой и второй повторяющихся областей, вероятно, усиливает их биологическую активность.
В одном из предпочтительных вариантов осуществления рекомбинантного связывающего белка согласно изобретению первая повторяющаяся область связывает область II HER2, а вторая повторяющаяся область связывает область IV HER2.
Важно понимать, что фраза "связывает область II" означает, что соответствующая повторяющаяся область связывает в основном область II HER2. Тем не менее, это определение не исключает того, что части упомянутой повторяющейся области могут связывать или перекрывать другие области. То же самое относится к выражению "связывает область IV".
Одновременное нацеливание на области II и IV HER2 бипаратопного связывающего белка согласно настоящему изобретению обеспечивает конкретные неожиданные эффекты, не известные из уровня техники. Реакция клеток на ингибирование пролиферации и индуцирования апоптоза такими связывающими белками является значительно более выраженной, чем при использовании известных антител. Например, степень такой реакции оказалась большей, чем при использовании эталонных клинических антител, таких как сочетание трастузумаба и пертузумаба, нацеленное на области IV и II HER2, соответственно (фиг. 4, 5 и 6). Интересно, что, некоторые бипаратопные белки, связывающие область I и область IV HER2, не обладают таким неожиданным действием (фиг. 3С и 3D).
Способы идентификации внеклеточной области HER2, которую связывает повторяющаяся область, например, как показано в Примере 3, хорошо известны специалистам в данной области техники (например, Jost и др., в процитированном месте).
Полученные заявителем данные имеют большое значение для лечения обусловленного HER2 рака у людей в том смысле, что нацеливание бипаратопного связывающего белка согласно настоящему изобретению одновременно на области II и IV HER2 могло бы стать более эффективной альтернативой существующим подходам к нацеливанию антител.
Соответственно, связывающий белок согласно настоящему изобретению предпочтительно является бипаратопным связывающим белком, т.е. содержит две области повтора антигена, распознающие два различных эпитопа или области (например, области II и IV) в одном и том же белке-мишени (а именно, HER2). Тем не менее, в объем изобретения входят полипептиды, которые являются мультипаратопными, т.е. содержащими области повтора антигена, распознающие три, четыре или более эпитопов в одном и том же белке-мишени, а также полипептиды, которые являются би- или мультипаратопными и многовалентными, т.е. также содержат области повтора антигена, распознающие один или несколько других белков-мишеней.
Используемым термином HER2 обозначается рецептор эпидермального фактора роста человека 2-го типа, также известный как Neu, ErbB-2, CD340 (группа дифференциации 340) или р185. HER2 входит в семейство рецепторов эпидермального фактора роста (EGFR/ErbB). В организме человека HER2 кодируется ERBB2, известным протоонкогеном, который содержится в длинном плече хромосомы 17 (17ql2) человека. HER2 зарегистрирован под номером Р04626 в базе данных UniProtKB/Swiss-Prot.
В одном из предпочтительных вариантов осуществления изобретения первая и вторая повторяющиеся области находятся в одном и том же полипептиде, при этом повторяющаяся область, нацеленная на область II HER2, находится на N-конце повторяющейся области, нацеленной на область IV HER2.
Эти варианты осуществления проиллюстрированы, например, на фиг. 2А и в соответствующем описании. Авторами изобретения неожиданно обнаружено, что связывающий белок, у которого повторяющаяся область, нацеленная на область II HER2, находится на С-конце повторяющейся области, нацеленной на область IV HER2, значительно менее эффективен, чем связывающий белок, у которого повторяющаяся область, нацеленная на область II HER2, находится на N-конце повторяющейся области, нацеленной на область IV HER2.
Первая повторяющаяся область, связывающая область II HER2, предпочтительно не конкурирует с пертузумабом за связывание HER2. Такие повторяющиеся области, которые не конкурирует с пертузумабом за связывание HER2, показаны, например, на фиг. 1С. Аналогичным образом, вторая повторяющаяся область, связывающая IV HER2, не конкурирует с трастузумабом за связывание HER2. Например, повторяющиеся области с DARPins # 18-20 не конкурирует с трастузумабом за связывание HER2. Специалистам в данной области техники хорошо известно, как определять, конкурирует ли повторяющаяся область с трастузумабом или пертузумабом за связывание HER2, например, как показано в Примере 3.
Это означает, что в первом предпочтительном варианте осуществления первая повторяющаяся область и пертузумаб связывают не один и тот же эпитоп области II HER2. Аналогичным образом, во втором предпочтительном варианте осуществления вторая повторяющаяся область и трастузумаб связывают не один и тот же эпитоп области IV HER2. Вне связи с теорией авторы объясняют этим, по меньшей мере, часть эффектов, продемонстрированных в экспериментальном разделе.
В другом предпочтительном варианте осуществления изобретения первой повторяющейся областью является область анкиринового повтора или сконструированная область анкиринового повтора, и второй повторяющейся областью является область анкиринового повтора или сконструированная область анкиринового повтора.
Области анкириновых повторов или сконструированные области анкириновых повторов предпочтительно содержат от 70 до 300 аминокислот, в частности, от 90 до 200 аминокислот.
Повторяющейся областью согласно изобретению также предпочтительно является область анкиринового повтора сконструированная область анкиринового повтора, описанная в WO 2002/020565. Примеры сконструированных областей анкириновых повторов со специфичностью бипаратопного связывания различных областей Her2 приведены в Примерах.
В одном из предпочтительных вариантов осуществления изобретения первая повторяющаяся область связывает внеклеточную область HER2 в PBS при Kd менее 10-7 M, а вторая повторяющаяся область связывает внеклеточную область HER2 в PBS при Kd менее 10-7 М.
Kd означает константу диссоциации, которая пояснена далее в тексте. Kd менее 10-7 M требуется для обеспечения достаточного сродства повторяющейся области к ее мишени. Повторяющиеся области предпочтительно связывают свои области-мишени в PBS при Kd менее 10-8М, 10-9М, 10-10М, наиболее предпочтительно менее 10-11М.
Рекомбинантные связывающие белки, представляющие собой белки, связывающие область II и/или область IV Her2 в PBS при Kd менее 10-7М, показаны в Примере 2.
В одном из предпочтительных вариантов осуществления связывающий белок ингибирует стимулированную пролиферацию клеток ВТ474 при показателе концентрации полумаксимального ингибирования (IC50) менее 100 нМ. Связывающий белок предпочтительно ингибирует стимулированную пролиферацию клеток ВТ474 при показателе IC50 менее 90, 80, 70, 60, 50, 40, 30, 20 или 10 нМ. Кроме того, связывающий белок предпочтительно ингибирует стимулированную пролиферацию клеток ВТ474, по меньшей мере, на 100%, 90%, 80%, 70%, 60%, 50%, 40%, 30%, 20% или 10%.
Для определения функциональной способности связывающих белков согласно изобретению ингибировать пролиферацию стандартными средствами, известными специалистам в данной области техники, например, как показано в Примере 4 могут использоваться клетки ВТ474. Для определения функциональной способности соединений согласно изобретению ингибировать пролиферацию предпочтительно могут использоваться клетки ВТ474, SKBR-3, NCI-N87, ZR75-30, НСС1419 или MDA-MB175, например, как показано в Примере 5.
Рекомбинантные связывающие белки, которые ингибируют стимулированную пролиферацию клеток ВТ474 при показателе IC50 менее 100 нМ, описаны и рассмотрены в Примере 4.
В другом предпочтительном варианте осуществления связывающий белок индуцирует апоптоз в клетках ВТ474 при показателе полумаксимальной эффективной концентрации (ЕС50) менее 100 нМ. Связывающий белок предпочтительно индуцирует апоптоз в клетках ВТ474 при показателе ЕС50 менее 90, 80, 70, 60, 50, 40, 30, 20 или 10 нМ.
Для определения функциональной способности связывающих белков согласно изобретению индуцировать апоптоз стандартными средствами, известными специалистам в данной области техники, например, как показано в Примере 5, могут использоваться клетки ВТ474. Для определения функциональной способности соединений согласно изобретению индуцировать апоптоз предпочтительно могут использоваться клетки ВТ474, SKBR-3, NCI-N87, ZR75-30, НСС1419 или MDA-MB175, например, как показано в Примере 5.
Рекомбинантные связывающие белки, которые вызывают апоптоз в клетках ВТ474 при показателе ЕС50 менее 100 нМ, описаны и рассмотрены в Примере 5.
В одном из предпочтительных вариантов осуществления первая и вторая повторяющиеся области соединены полипептидным линкером.
Такой полипептидный линкер может, например, быть реализован путем простого генетического слияния кодирующих кДНК соответствующих областей для слияния. В варианте осуществления такого типа слитым пептидом считается белок с двумя различными повторяющимися областями.
Линкер может состоять, например, из олигопептида, содержащего аминокислоты G и S или Ρ и Т, соответственно, согласно последовательностям SEQ ID 7-12. В другом предпочтительном варианте осуществления может использоваться описанный далее "способный к мультимеризации фрагмент". В качестве альтернативы, две повторяющиеся области могут быть связаны друг с другом, например, посредством химических линкеров не пептидной основе.
Рекомбинантный связывающий белок и/или повторяющаяся область предпочтительно имеет среднюю температуру денатурации (Tm) выше 45°С, более предпочтительно выше 50°С, еще более предпочтительно выше 55°С, наиболее предпочтительно выше 60°С после теплового разворачивания в PBS при рН 7,4. Связывающий белок или повторяющаяся область согласно изобретению обладает заданной вторичной и третичной структурой в физиологических условиях. В результате теплового разворачивания такой полипептид теряет свою вторичную и третичную структуру, после чего могут осуществляться, например, измерения циркулярного дихроизма (CD). Средняя температура денатурации связывающего белка или повторяющейся области после теплового разворачивания соответствует температуре в срединной точке кооперативного перехода в физиологическом буфером после тепловой денатурации упомянутого белка или области при медленном повышении температуры от 10°С до около 100°С. Определение средней температуры денатурации после теплового разворачивания хорошо известно специалистам в данной области техники. Эта средняя температура денатурации связывающего белка или повторяющейся области после теплового разворачивания является показателем термостабильности полипептида.
Также предпочтительным является рекомбинантный связывающий белок и/или область анкиринового повтора, образующая менее 5% (по весу) нерастворимых агрегатов в концентрациях до 20 г/л, предпочтительно до 40 г/л, более предпочтительно до 60 г/л, еще более предпочтительно до 80 г/л, наиболее предпочтительно до 100 г/л при инкубации в течение более 5 суток, предпочтительно более 10 суток, более предпочтительно более 20 суток, еще более предпочтительно более 40 суток, наиболее предпочтительно более 100 суток при 37°С в PBS. Образование нерастворимых агрегатов может быть обнаружено на основании внешнего вида видимого осадка, гель-фильтрации или динамического рассеяния света, которое значительно усиливается после образования нерастворимых агрегатов. Нерастворимые агрегаты могут удаляться из образца белка путем центрифугирования со скоростью 10000 × g в течение 10 минут. Рекомбинантный связывающий белок и/или область анкиринового повтора предпочтительно образует менее 2%, более предпочтительно менее 1%, 0,5%, 0,2%, 0,1%, наиболее предпочтительно менее 0,05% (по весу) нерастворимых агрегатов в упомяных условиях инкубации при 37°С в PBS. Процентное содержание нерастворимых агрегатов может определяться путем отделения нерастворимых агрегатов от растворимого белка с последующим определением количеств белка в растворимой и нерастворимой фракциях стандартными методами определения количества.
Также предпочтительным является рекомбинантный связывающий белок и/или область анкиринового повтора, которая не теряет своей нативной трехмерной структуры после инкубации в PBS, содержащем 100 мм дитиотреитола (DTT), в течение 1-10 часов при 37°С.
В одном из частных вариантов осуществления изобретения предложен рекомбинантный связывающий белок, содержащий две области анкириновых повторов, специфически связывающих HER2 и имеющих указанную или предпочтительную среднюю температуру денатурации и описанную способность предотвращать агрегацию.
В других предпочтительных вариантах осуществления изобретения, предусмотрено, что
первая повторяющаяся область конкурирует за связывание HER2 с областью анкиринового повтора, выбранной из группы, включающей SEQ ID 62-68, 72 и 114-121 и/или
вторая повторяющаяся область конкурирует за связывание HER2 с областью анкиринового повтора, выбранной из группы, включающей SEQ ID 74-82.
Авторы изобретения имеют данные, согласно которым первая из этих повторяющихся областей связывает область II HER2, а вторая повторяющаяся область связывает область IV HER2.
Первая повторяющаяся область предпочтительно конкурирует за связывание HER2 с областью анкиринового повтора, выбранной из группы, включающей SEQ ID 62-67 и 115-121. Первая повторяющаяся область более предпочтительно конкурирует за связывание HER2 с областью анкиринового повтора, выбранной из группы, включающей SEQ ID 62, 115, 120 и 121, в частности SEQ ID 115 и 120. Первая повторяющаяся область также предпочтительно конкурирует за связывание HER2 со связывающим белком, выбранным из группы DARPins # 1-6 и 54-60; более предпочтительно со связывающим белком из группы DARPins # 1, 54, 59 и 60; в частности, со связывающим белком из группы DARPins # 54 и 60.
Кроме того, вторая повторяющаяся область предпочтительно конкурирует за связывание HER2 с областью анкиринового повтора, выбранной из группы, включающей SEQ ID 79-81, в частности SEQ ID 80 и 81. Вторая повторяющаяся область также предпочтительно конкурирует за связывание HER2 со связывающим белком, выбранным из группы DARPins # 18-20; в частности, со связывающим белком из группы DARPins # 19 и 20.
В других предпочтительных вариантах осуществления изобретения предусмотрено, что
первая повторяющаяся область содержит аминокислотную последовательность, которая, по меньшей мере, на 70% идентична одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 62-68, 72 и 114-121,
вторая повторяющаяся область содержит аминокислотную последовательность, которая, по меньшей мере, на 70% идентична одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 74-82,
при этом дополнительно
G в положении 1 и/или S в положении 2 упомянутой области анкиринового повтора необязательно отсутствует; и
L в предпоследнем положении и/или N в последнем положении упомянутой области анкиринового повтора необязательно заменены А.
Первая повторяющаяся область предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70% идентична одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 62-67 и 115-121. Первая повторяющаяся область более предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70% идентична одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 62, 115, 120 и 121, в частности SEQ ID 115 и 120. Кроме того, первая повторяющаяся область предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70% идентична связывающему белку, выбранному из группы, включающей DARPins # 1-6 и 54-60; более предпочтительно связывающему белку из группы DARPins # 1, 54, 59 и 60; в частности, связывающему белку из группы DARPins # 54 и 60.
Кроме того, вторая повторяющаяся область предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70% идентична одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 79-81, в частности SEQ ID 80 и 81. Кроме того, вторая повторяющаяся область предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70% идентична связывающему белку из группы, включающей DARPins # 18-20; в частности, связывающему белку из группы DARPins # 19 и 20.
Первая область анкиринового повтора предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, или 100% идентична одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 62-68, 72 и 114-121.
Вторая область анкиринового повтора предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, или 100% идентична одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 74-82.
Кроме того, первая область анкиринового повтора предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, или 100% идентична одному, двум или трем модулям анкиринового повтора, присутствующим между N-концевым и С-концевым кэппирующими модулями области анкиринового повтора, выбранной из группы, включающей SEQ ID 62-68, 72 и 114-121.
Вторая область анкиринового повтора также предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99, или 100% идентична одному, двум или трем модулям анкиринового повтора, присутствующим между N-концевым и С-концевым кэппирующими модулями области анкиринового повтора, выбранной из группы, включающей SEQ ID 74-82.
В других предпочтительных вариантах осуществления изобретения, предусмотрено, что
первая повторяющаяся область выбрана из группы, включающей SEQ ID 62-68, 72 и 114-121,
вторая повторяющаяся область выбрана из группы, включающей SEQ ID 74-82,
при этом дополнительно
G в положении 1 и/или S в положении 2 упомянутой области анкиринового повтора необязательно отсутствуют; и
L в предпоследнем положении и/или N в последнем положении упомянутой области анкиринового повтора необязательно заменены А.
Первая область анкиринового повтора предпочтительно выбрана из группы, включающей SEQ ID 62-67 и 115-121; более предпочтительно SEQ ID 115, 120 и 121; в частности, SEQ ID 115-120.
Вторая область анкиринового повтора предпочтительно выбрана из группы, включающей SEQ ID 79-81, в частности SEQ ID 80-81.
В других предпочтительных вариантах осуществления изобретения, предусмотрено, что
первая повторяющаяся область содержит собой модуль анкиринового повтора, имеющий аминокислотную последовательность, выбранную из группы, включающей SEQ ID 15-18, 21-23, 37, 38, 125, 126, 129, 130, 133 и 134 и последовательности, в которых до 9 аминокислотных остатков в SEQ ID 15-18, 21-23, 37, 38, 125, 126, 129, 130, 133 и 134 заменены любыми другими аминокислотными остатками, и/или
вторая повторяющаяся область содержит модуль анкиринового повтора, имеющий аминокислотную последовательность, выбранную из группы, включающей SEQ ID 46, 47, 51, 52, 55 и 56 и последовательности, в которых до 9 аминокислотных остатков в SEQ ID 46, 47, 51, 52, 55 и 56 заменены любыми другими аминокислотными остатками.
Такой модуль анкиринового повтора первой области анкиринового повтора предпочтительно выбран из группы, включающей SEQ ID 15-18, 125, 126, 129, 130, 133 и 134; более предпочтительно 15, 125, 129 и 133; еще более предпочтительно 125 и 133.
Такой модуль анкиринового повтора второй области анкиринового повтора предпочтительно выбран из группы, включающей SEQ ID 46, 47, 55 и 56; более предпочтительно 55 и 56.
В модулях SEQ ID 15-18,21-23, 37, 38, 46, 47, 51, 52, 55, 56, 125, 126, 129, 130, 133 и 134 до 8 аминокислот, более предпочтительно до 7 аминокислот, более предпочтительно до 6 аминокислот, более предпочтительно до 5 аминокислот, еще более предпочтительно до 4 аминокислот, более предпочтительно до 3 аминокислот, более предпочтительно до 2 аминокислот, наиболее предпочтительно 1 аминокислота также предпочтительно заменена другой аминокислотой.
При замене аминокислот в кэппирующих модулях, модулях повтора, повторяющихся областях или связывающих белках эти аминокислоты заменены аминокислотой, выбранной из группы, включающей A, D, Е, F, Н, I, K, L, М, N, Q, R, S, Т, V, W и Y; более предпочтительно из группы, включающей A, D, Е, Н, I, K, L, Q, R, S, Т, V и Y. Аминокислота также предпочтительно заменена гомологичной аминокислотой; т.е. аминокислота заменена аминокислотой, имеющей боковую цепь с аналогичными биофизическими свойствами. Например, отрицательно заряженная аминокислота D может быть заменена отрицательно заряженной аминокислотой Е, а гидрофобная аминокислота, такая как L, может быть заменена аминокислотой А, I или V. Методы замены одной аминокислоты другой аминокислотой в полипептиде хорошо известны специалистам в данной области техники.
Модуль повтора согласно изобретению предпочтительно содержит аминокислотную последовательность, выбранную из группы, включающей KDFQGITPLHIAATSGHLEIVEVLLKAGADVNA (SEQ ID 16) и последовательности, в которых до 9 аминокислотных остатков в SEQ ID 16 заменены любыми другими аминокислотными остатками, и в которых
F в положении 3 необязательно заменена А;
Q в положении 4 необязательно заменена Е;
G в положении 5 необязательно заменена S;
I в положении 6 необязательно заменена V;
I в положении 11 необязательно заменена L;
Т в положении 14 необязательно заменена Q; и/или
S в положении 15 необязательно заменена аминокислотой, выбранной из группы, включающей N и W.
Один из предпочтительных модулей повтора этой группы содержит аминокислотную последовательность, выбранную из группы, включающей KDFQGVTPLHIAAQSGHLEIVEVLLKAGADVNA (SEQ ID 125), SEQ ID 129 и SEQ ID 133.
Модуль анкиринового повтора согласно изобретению также предпочтительно содержит аминокислотную последовательность, выбранную из группы, включающей KDITGETPLHHAADSGHLEIVEVLLKAGADVNA (SEQ ID
18) и последовательности, в которых до 9 аминокислотных остатков в SEQ ID 18 заменены любыми другими аминокислотными остатками, и в которых
I в положении 3 необязательно заменена V;
Ε в положении 6 необязательно заменена D;
Η в положении 11 необязательно заменена L;
D в положении 14 необязательно заменена Q;
S в положении 15 необязательно заменена Н; и/или
Ε в положении 19 необязательно заменена V.
Один из предпочтительных модулей повтора этой группы содержит аминокислотную последовательность, выбранную из группы, включающей KDVTGDTPLHLAAQHGHLEIVEVLLKAGADVNA (SEQ ID 126), SEQ ID 130 или SEQ ID 134.
Модуль анкиринового повтора согласно изобретению также предпочтительно содержит аминокислотную последовательность, выбранную из группы, включающей KDWEGTTPLHLAAHTGHLEIVEVLLKAGADVNA (SEQ ID 21) и последовательности, в которых до 9 аминокислотных остатков в SEQ ID 21 заменены любыми другими аминокислотными остатками, и в которых
W в положении 3 необязательно заменена F;
W в положении 4 необязательно заменена Q;
Τ в положении 6 необязательно заменена аминокислотой, выбранной из группы, включающей I, Y и V, предпочтительно Т;
L в положении 11 необязательно заменена аминокислотой, выбранной из группы, включающей I и V, предпочтительно I и V;
H в положении 14 необязательно заменена аминокислотой, выбранной из группы, включающей H, Q, Y и W, предпочтительно Н; и/или
Τ в положении 15 необязательно исключена или заменена аминокислотой, выбранной из группы, включающей А и D.
Модуль анкиринового повтора согласно изобретению также предпочтительно содержит аминокислотную последовательность, выбранную из группы, включающей KDTVGTTPLHYAAEDGHLEIVEVLLKAGADVNA (SEQ ID 22) и последовательности, в которых до 9 аминокислотных остатков in SEQ ID 22 заменены любыми другими аминокислотными остатками, и в которых
Τ в положении 3 необязательно заменена аминокислотой, выбранной из группы, включающей S, К, Ε и I; при равном распределении аминокислот;
V в положении 4 необязательно заменена аминокислотой, выбранной из группы, включающей Q, I и Υ, предпочтительно Υ;
Τ в положении 6 необязательно заменена аминокислотой, выбранной из группы, включающей Q, F, R и W;
Y в положении 11 необязательно заменена аминокислотой, выбранной из группы, включающей L, Ε и S, предпочтительно S;
Ε в положении 14 необязательно заменена аминокислотой, выбранной из группы, включающей S, Q, Y и V; и/или
D в положении 15 необязательно заменена аминокислотой, выбранной из группы, включающей S, F и Y.
G в положении 16 необязательно заменена D.
Модуль анкиринового повтора согласно изобретению также предпочтительно содержит аминокислотную последовательность, выбранную из группы, включающей KDVEGWTPLHYAASSGHLEIVEVLLKAGADVNA (SEQ ID 38) и последовательности, в которых до 9 аминокислотных остатков in SEQ ID 38 заменены любыми другими аминокислотными остатками, и в которых
W в положении 6 необязательно заменена Q;
Y в положении 11 необязательно заменена L; и/или
S в положении 15 необязательно заменена Y.
Модуль анкиринового повтора согласно изобретению также предпочтительно содержит аминокислотную последовательность, выбранную из группы, включающей KDWRGFTPLHYAAYLGHLEIVEVLLKAGADVNA (SEQ ID 46) и последовательности, в которых до 9 аминокислотных остатков SEQ ID 46 заменены любыми другими аминокислотными остатками, и в которых
W в положении 3 необязательно заменена аминокислотой, выбранной из группы, включающей W, Τ, V и R, предпочтительно Τ и R;
R в положении 4 необязательно заменена аминокислотой, выбранной из группы, включающей R, Τ и I, предпочтительно I;
F в положении 6 необязательно заменена F или Н, предпочтительно F;
Y в положении 11 необязательно заменена R;
Y в положении 14 необязательно заменена F;
L в положении 15 необязательно заменена V; и/или
H в положении 17 необязательно заменена Q.
9, 8, 7, 6, 5, 4, 3, 2 или 1 аминокислотный остаток в SEQ ID 16, 18, 28, 31. 21, 22, 38 и/или 46 предпочтительно заменен любыми другими аминокислотными остатками.
Кроме того, особо предпочтительно, чтобы связывающий белок содержал полипептид, который содержит первую и вторую области анкириновых повторов, и в котором аминокислотная последовательность, по меньшей мере, на 70% идентична полипептиду, выбранному из группы, включающей SEQ ID 83-98, 102, 103, 122, 123 и 136-141.
Полипептид предпочтительно содержит аминокислотную последовательность, которая, по меньшей мере, на 70, 71, 72, 73, 74, 75, 76, 77, 78, 79, 80, 81, 82, 83, 84, 85, 86, 87, 88, 89, 90, 91, 92, 93, 94, 95, 96, 97, 98, 99 или 100% идентична полипептиду, выбранному из группы, включающей SEQ ID 83-98, 102, 103, 122, 123 и 136-141.
Такой полипептид также предпочтительно выбран из группы, включающей SEQ ID 84, 85, 86, 87, 90, 91, 92, 98, 102, 103, 122 и 123, более предпочтительно 85, 86, 87, 90, 91, 92, 102, 103, 122 и 123, еще более предпочтительно 86, 87, 91 и 92, и наиболее предпочтительно 86 и 87.
В еще одном предпочтительном варианте осуществления один или несколько аминокислотных остатков модулей анкириновых повторов первой и второй области анкириновых повторов заменены аминокислотным остатком, находящимся в соответствующем положении с выравниванием по анкириновому повтору.
В другом варианте осуществления изобретения предложена молекула нуклеиновой кислоты, кодирующая, по меньшей мере, один связывающий белок или конкретную область анкиринового повтора согласно изобретению. Кроме того, предусмотрен вектор, содержащий упомянутую молекулу нуклеиновой кислоты.
Не все связывающие композиции согласно настоящему изобретению содержат полипептиды или белки. Последний вариант осуществления относится только к тем композициям, которые их содержат. Заявитель не приводит в описании все способные их кодировать молекулы нуклеиновой кислоты, поскольку вследствие вырожденности генетического кода один и тот полипептид или белок может кодироваться множеством молекул нуклеиновой кислоты.
Тем не менее, можно безошибочно и однозначно определять, кодирует ли заданная нуклеиновая кислота заданный полипептид или белок. Соответственно, этот вариант осуществления ясен специалистам в данной области техники, и его объем легко определим.
В другом варианте осуществления изобретения предложено применение описанного связывающего белка с целью ингибирования, по меньшей мере, одного из следующего:
димеризации рецепторов HER2,
гетеродимеризации HER2/HER3,
автофосфорилирования рецепторов HER2,
опосредованной рецепторами HER сигнальной трансдукции,
индуцированного лигандами рецепторов HER3 фосфорилирования и/или
опосредованной рецепторами HER3 сигнальной трансдукции.
Димеризация рецепторов HER2 (также называемая "гомодимеризацией") происходит в тканях с независимой от лиганда сверхэкспрессией HER2. Гомодимеризация приводит к внутриклеточному автофосфорилированию, которое способно впоследствии вызывать, например, увеличение пролиферации клеток.
Поскольку у HER3 отсутствует собственная активность киназы, HER3 фосфорилирует в клетках рака молочной железы со сверхэкспрессией HER2 после образования гетеродимеров HER2/HER3, что может впоследствии вызывать, например, ингибирование апоптоза.
Упомянутое применение возможно in vitro или in vivo. Как указано выше, все эти процессы могут иметь патогенные последствия, а именно, путем активации соответствующих путей сигнальной трансдукции. Пути сигнальной трансдукции, активируемые димеризацией HER2 и/или гетеродимеризацией HER2/HER3, включают путь митоген-активируемой протеинкиназы (МАРК), фосфоинозитид-3-киназы (PI3K/Akt), фосфолипазы С γ, протеинкиназы С (РКС), сигнальной трансдукции и активации транскрипции (STAT), Ras-MAP-киназы и путь mTOR.
Например, путь фосфоинозитид-3-киназы (PI3K/Akt) считается одним из важнейших путей, который обеспечивает выживание клеток путем блокирования апоптоза. Соответственно, его патологическая активация, например, путем гетеродимеризации HER2/HER3 может приводить к пролиферации злокачественных клеток (например, смотри Примеры).
Патологическая активация HER2, например, путем гомодимеризации HER2 может приводить к миграции, инвазии или пролиферации злокачественных клеток (например, смотри Примеры; Hynes NE. и Lane НА., Nat. Rev. Cancer., 5,341-54, 2005).
В еще одном варианте осуществления изобретения предложен фармацевтический состав, содержащий связывающий белок или композицию согласно приведенному выше описанию и необязательно фармацевтически приемлемый носитель и/или разбавитель.
Фармацевтически приемлемые носители и/или разбавители известны специалистам в данной области техники и подробнее описаны далее. Кроме того, предложена диагностическая композиция, содержащая один или несколько упомянутых рекомбинантных связывающих белков, в частности, связывающих белков, содержащих повторяющиеся области.
Фармацевтический состав содержит описанные выше рекомбинантные связывающие белки и фармацевтически приемлемый носитель, наполнитель или стабилизатор, например, как описано в Remington's Pharmaceutical Sciences, 16-е издание под редакцией Osol, А. [1980]. Применимыми носителями, наполнителями или стабилизаторами, известными специалистам в данной области техники, являются физиологический раствор, раствор Рингера, раствор декстрозы, раствор Хэнка, нелетучие масла, этилолеат, 5% декстроза в физиологическом растворе, вещества, способствующие изотоничности и химической устойчивости, буферы и консерванты. Другие применимые носители включают любой носитель, который сам по себе не вызывает синтеза антител, вредных для организма человека, принимающего композицию, такой как белки, полисахариды, полимолочные кислоты, полигликолевые кислоты, полиаминокислоты и сополимеры аминокислот.
Составы, применимые для введения in vivo, должны являться асептическими или стерильными. Это легко достигается путем фильтрации через стерильные фильтрационные мембраны. Фармацевтический состав может вводиться любым применимым путем, известным специалистам в данной области техники.
Кроме того, в другом варианте осуществления настоящего изобретения предложено применение, по меньшей мере, одного связывающего белка, композиции или фармацевтического состава согласно изобретению в качестве лекарственного средства. Аналогичным образом, предложен способ, включающий введение пациенту связывающего белка, композиции или фармацевтического состава согласно изобретению. В обоих случаях болезнью предпочтительно является неоплазия, предпочтительно рак.
В каждом случае с целью лечения болезни пациенту предпочтительно вводят эффективное количество связывающего белка, композиции или фармацевтического состава согласно изобретению.
Используемым термином "неоплазия" обозначается аномальное состояние клеток или ткани, характеризующееся быстро пролиферирующим ростом клеток или новообразования. В более точном смысле этим термин обозначаются злокачественные процессы, например, опухоли и/или лейкозы.
Связывающие белки согласно изобретению продемонстрировали апоптический и антипролиферативный эффекты (смотри экспериментальный раздел). Поскольку неоплазий частот характеризуются подавлением апоптоза и/или увеличенной пролиферацией, из проведенных экспериментов, вероятно, следует, что связывающие белки согласно настоящему изобретению могут применяться для лечения неоплазий.
Неоплазией предпочтительно является болезнь, характеризуемая, по меньшей мере, одним фактором, выбранным из группы, включающей
амплификацию кодирующего HER2 гена,
сверхэкспрессию кодирующего HER2 гена,
экспрессию мутантного кодирующего HER2 гена и/или
сверхэкспрессию кодирующего HER3 гена в резистентных к трастузумабу опухолях.
В организме человеке HER2 кодируется геном ERBB2. Перечисленные факторы могут объясняться мутациями в гене ERBB2, которые могут обнаруживаться средствами современной молекулярной диагностики, предлагаемыми в настоящее время на рынке.
Используемый термин "экспрессия кодирующего HER2 гена" относится к клеткам, тканям или органам, в которых экспрессирует белок рецептора HER2, при обнаружении, например, методами иммуногистохимии (IHC). Используемым термином "амплификация или сверхэкспрессия кодирующего HER2 гена" обозначается аномальный уровень экспрессии белка рецептора HER2 в клетке, ткани или органе относительно уровня экспрессии в нормальной клетке, ткани или органе, при обнаружении, например, методами иммуногистохимии (IHC).
Такие исследования методами IHC известны из техники и включают Clinical Trial Assay (СТА), предлагаемый на рынке тест LabCorp 4D5 и предлагаемый на рынке DAKO HercepTest® (DAKO, Карпинтерия, шт. Калифорния, США). В последнем случае применяется особа шкала показателей окрашивания клеток от 0 до 3+ (0 соответствует нормальной экспрессии, 3+ соответствует наибольшую положительную экспрессию) с целью идентификации рака со сверхэкспрессией белка HER2. Соответственно, способы лечения согласно настоящему изобретению могли принести пользу пациентам, страдающим раком, который характеризуется сверхэкспрессией белка HER2 с показателем 1+, 2+ или 3+, предпочтительно 2+ или 3+, более предпочтительно 3+.
В качестве альтернативы, показатели экспрессии и/или сверхэкспрессии Her2 также могут определяться методом цитологической гибридизации (ISH), RT-РСТ и другими методами.
В одном из особо предпочтительных вариантов осуществления неоплазией является, по меньшей мере, болезнь, выбранная из группы включающей
рак молочной железы,
рак яичников,
рак желудочно-кишечного тракта,
рак желудка,
рак матки и/или
колоректальный рак.
Кроме того, упомянутое применение предпочтительно скоординированным образом дополнено введением, по меньшей мере, одного действующего вещества, выбранного из группы, включающей
противоопухолевое средство
эндокринное лекарство,
противоопухолевую вакцину,
иммунотерапию и/или
клеточную терапию.
Используемым термином "скоординированным образом дополнено" обозначается совместное введение, которое осуществляется согласно заданной схеме. Оно включает синхронное введение различных соединений, а также введение различных соединений со сдвигом во времени (например, один раз вводится соединение А, после чего несколько раз вводится соединение В, или наоборот, или оба соединения вводятся синхронно, при этом одно из них также вводится позднее).
Используемым термином "противоопухолевое средство" обозначается лекарство или сочетание лекарств, обладающих противоопухолевым или противораковым действием. Это относится, прежде всего, к химиотерапевтическим средствам, которые действуют, ослабляя митоз, эффективно нацеливаясь на быстро делящиеся клетки или вызывая апоптоз клеток. Большинство химиотерапевтических средств могут подразделяться на алкилирующие средства, антиметаболиты, антрациклины, растительные алкалоиды, ингибиторы топоизомеразы и другие противоопухолевые средства.
Предпочтительными противоопухолевыми средства являются 5-фторурацил, актиномицин, адриамицин, амсакрин, антрациклины, азатиоприн, бендамустин, блеомицин, карбоплатин, хлорамбуцил, цисплатин, циклофосфамид, даунорубицин, доцетаксел, доксорубицин, эпирубицин, этопозид, идарубицин, ифосфамид, иринотекан, мехлорэтамин, меркаптопурин, метотрексат, митомицин, оксалиплатин, паклитаксел, пликамицин, подофиллотоксин, тенипозид, топотекан, валрубицин, винбластин, винкристин, виндезин и/или винорелбин.
Иммунотерапия предусматривает выделение белков из раковых клеток и последующую иммунизацию раковых пациентов против этих белков в расчете на стимуляцию иммунной реакции, которая уничтожила бы раковые клетки. Другим подходом к противораковой вакцинации является генерирование у пациента иммунного ответа in situ. Тем самым усиливается противоопухолевый иммунный ответ на опухолевые антигены, которые высвобождаются после литической репликации вируса, в результате чего получают in situ специфическую для пациента противоопухолевую вакцину. Еще один подход предусматривает иммунизацию пациента соединением, которое играет физиологическую роль в канцерогенезе, с тем, чтобы организм человека уничтожал это соединение.
Нацеленные лекарства являются лекарственными средствами, которые блокируют рост раковых клеток путем противодействия конкретным молекулам-мишеням, необходимым для канцерогенеза и роста опухоли, а не только путем противодействия быстро делящимся клеткам (например, как традиционные химиотерапевтические средства). Основными категориями средств нацеленной терапии являются малые молекулы и моноклональные антитела.
Малые молекулы, подпадающие под это определение, включают без ограничения лапатиниб, нератиниб, афатиниб, иматиниб, гефитиниб, эрлотиниб, бортезомиб, ингибиторы Bc1-2 (например, обатоклакс, АВТ-263 и госсипол), ингибиторы PARP (например, инипариб, олапариб), ингибиторы янус-киназ, ингибиторы PI3K, апатиниб, ингибиторы mTOR (эверолимус), AN-152, ингибиторы АКТ, ингибиторы HDAC, ингибиторы протеасом, доксорубицин, связанный с [D-Lys(6)]-LHRH, пегаптаниб, сунитиниб, сорафениб, тивозаниб и пазопаниб. Моноклональные антитела, подпадающие под это определение, включают без ограничения ритуксимаб, трастузумаб, трастузумаб-TDM1, пертузумаб, цетуксимаб и бевацизумаб.
Используемым термином "эндокринные лекарства" обозначаются лекарства, которые являются антагонистами гормонов или рецепторов гормонов и, соответственно, противодействуют раку тех типов, в которых требуется рост гормонов. Одним из примеров такого эндокринного лекарства служит тамоксифен, который является антагонистом рецептора эстрогена в ткани молочной железы.
Используемым термином "клеточная терапия" обозначаются клеточные терапии, такие как адоптивный перенос модифицированных или немодифицированньгх цитотоксичных лимфоцитов или дендритов.
Используемым термином "противоопухолевая вакцина" обозначаются вакцины, которые а) предотвращают инфицирование вызывающими рак вирусами (принцип их действия является таким же, как у других вакцин против вирусных инфекций), б) лечат существующий рак (терапевтические противораковые вакцины) или в) предотвращают развитие рака, или ослабляют его последствия (профилактические противораковые вакцины).
Дополнительно или в качестве альтернативы, упомянутое применение предпочтительно скоординированным образом дополнено, по меньшей мере, одной другой терапией, выбранной из группы, включающей
лучевую терапию,
оперативное вмешательство и/или
лазерную абляцию.
Кроме того, предложен способ лечения людей или животных, включающий описанное выше применение. Способ лечения предпочтительно относится к описанным выше показаниям. Способ включает введение нуждающемуся в этом человеку или животному терапевтически эффективного количества рекомбинантного связывающего белка согласно изобретению.
Рекомбинантный связывающий белок или область анкиринового повтора согласно изобретению может быть получена и/или дополнительно выделена несколькими способами, такими как дисплей на поверхности бактериофагов (WO 1990/002809, WO 2007/006665) или бактериальных клеток (WO 1993/ 010214), рибосомный дисплей (WO 1998/048008), плазмидный дисплей (WO 1993/008278) или путем использования ковалентных гибридных белковых конструкций на основе повторов РНК (WO 2000/032823) или внутриклеточной экспрессии и отбора/скрининга, такого как комплементационный анализ белков (WO 1998/341120). Такие способы известны специалистам в данной области техники.
Согласно протоколам, известным специалистам в данной области техники (WO 2002/020565, Binz, H.K. и др., J. Mol. Biol., 332, 489-503, 2003 и Binz и др., 2004, в процитированном месте), может быть получена библиотека анкириновых повторов, используемых для отбора/скрининга рекомбинантного связывающего белка или области анкиринового повтора согласно изобретению. Применение таких библиотек для отбора областей анкириновых повторов со специфичностью к внеклеточной области HER2 проиллюстрировано в Примере 1. Кроме того, области анкириновых повторов согласно настоящему изобретению могут быть скомпонованы из модулей анкириновых повторов согласно настоящему изобретению и соответствующих кэппирующих модулей или кэппирующих повторов (Forrer, Р. и др., FEBS letters 539, 2-6, 2003) с использованием стандартных технологий рекомбинантных ДНК (например, WO 2002/020565, Binz и др., 2003, в процитированном месте и Binz и др., 2004, в процитированном месте).
Изобретение не ограничено конкретными вариантами осуществления, описанными в Примерах. Могут использоваться и обрабатываться другие источники согласно общей процедуре, описанной далее.
Определения
Термином "белок" обозначается полипептид, по меньшей мере, часть которого имеет или способна образовывать заданную трехмерную структуру путем формирования вторичной, третичной или четвертичной структур внутри и/или между полипептидной цепью(-ями). Если белок содержит два или более полипептидов, отдельные полипептидные цепи могут быть нековалентно или ковалентно связаны, например, дисульфидным мостиком между двумя полипептидами. Часть белка, которая в отдельности имеет или способна образовывать заданную трехмерную структуру путем формирования вторичной или третичной структур, именуется "белковым доменом". Такие белковые домены хорошо известны специалистам в данной области техники.
Термин "рекомбинантный" применительно к белку, белковому домену, связывающему белку и т.п. означает, что упомянутые полипептиды получены с применением технологий рекомбинантных ДНК, хорошо известных специалистам в данной области техники. Например, молекула рекомбинантной ДНК (например, полученная путем синтеза генов), кодирующей полипептид, может быть клонирована в в экспрессирующую бактериальную плазмиду (например, pQE30, Qiagen), экспрессирующую дрожжевую плазмиду дрожжей или экспрессирующую плазмиду млекопитающих. Когда, например, такую сконструированную рекомбинантную экспрессирующую бактериальную плазмиду вводят в соответствующие бактерии (например, Escherichia coli), эти бактерии способны вырабатывать полипептид, закодированный этой рекомбинантной ДНК. Соответствующим образом выработанный полипептид называется рекомбинантным полипептидом.
Термином "полипептид" в контексте настоящего изобретения обозначается молекула, состоящая из одной или нескольких цепей из множества, т.е. двух или более аминокислот, связанных пептидными связями. Полипептид предпочтительно состоит из более восьми аминокислот, связанных пептидными связями.
Термином "полипептидная метка" обозначается присоединенная к полипептиду/белку аминокислотная последовательность, применимая для очистки, обнаружения или нацеливания полипептида/белка или улучшающая физико-химическое поведение полипептида/белка или обладающая эффекторной функцией. Отдельные полипептидные метки, фрагменты и/или области связывающего белка могут быть соединены друг с другом непосредственно или полипептидными линкерами. Эти полипептидные метки хорошо известны из техники и целиком доступны специалистам в данной области техники. Примерами полипептидных меток являются малые полипептидные последовательности, например, His (например, His-метка SEQ ID 6), myc, FLAG, или Strep-метки или фрагменты, такие как ферменты (например, такие ферменты, как щелочная фосфатаза), которые позволяют обнаруживать полипептид/белок, или фрагменты, которые могут применяться для нацеливания (такие как иммуноглобулины или их фрагменты) и/или в качестве эффекторных молекул.
Термином "полипептидный линкер" обозначается аминокислотная последовательность, которая способна связывать, например, два белковых домена, полипептидную метку и белковый домен, белковый домен неполипептидный фрагмент, такой как полиэтиленгликоль, или две метки последовательностей. Такие дополнительные домены, метки, неполипептидные фрагменты и линкеры известны специалистам в соответствующей области техники. Перечень их примеров приведен в описании патентной заявки WO 2002/020565. Конкретными примерами таких линкеров служат глицин-сериновые линкеры и пролин-треониновые линкеры различной длины, предпочтительно длиной от 2 до 24 аминокислот, более предпочтительно длиной от 2 до 16 аминокислот. Примерами глицин-сериновых линкеров служат последовательности SEQ ID 7-10, а примерами пролин-треониновых линкеров служат последовательности SEQ ID 11 и 12. Пролин-треониновому линкеру SEQ ID 11 предшествует GS, и/или за ним следует GS.
Термином "полимерный фрагмент" обозначается белковый полимерный фрагмент небелковый полимерный фрагмент.Белковым полимерным фрагментом предпочтительно является полипептид, который не образует устойчивой третичной структуры. Примерами белковых полимерных фрагментов являются полипептиды XTEN® (зарегистрированный товарный знак компании Amunix; WO 2007/103515) или полипептиды, содержащие пролиновые, аланиновые и сериновые остатки, как описано в WO 2008/155134. Такие белковые полимерные фрагменты могут быть ковалентно связаны, например, с повторяющейся областью согласно изобретению путем генетического слияния полипептидов с использованием стандартных технологий клонирования ДНК с их последующей стандартной экспрессией и очисткой. Небелковым полимерным фрагментом является полимерный фрагмент, не состоящий из полипептидов. Примерами небелковых полимерных фрагментов служат гидроксиэтилкрахмал (HES), полиэтиленгликоль (PEG), полипропиленгликоль или полиоксиалкилен. Термин "пэгилированный" означает, что фрагмент PEG ковалентно связан, например, с полипептидом согласно изобретению. Молекулярная масса полимерного фрагмента согласно изобретению может значительно варьировать. Полимерный фрагмент предпочтительно соединен полипептидным линкером с повторяющейся областью.
В одном из конкретных вариантов осуществления фрагмент PEG или любой другой небелковый полимер может быть, например, связан с тиоловой группой цистеина посредством малеимидного линкера, при этом цистеин посредством пептидного линкера связан с N- или С-концом описанной в изобретении повторяющейся области.
Термином "связывающий белок" обозначается белок, содержащий одну или несколько связывающих областей, одно или несколько биологически активных соединений и один или несколько полимерных фрагментов, как дополнительно пояснено далее. Связывающий белок предпочтительно содержит до четырех связывающих областей. Кроме того, любой такой связывающий белок может содержать дополнительные белковые домены, не являющиеся связывающими областями, способные к мультимеризации фрагменты, полипептидные метки, полипептидные линкеры и/или один цистеиновый остаток.
Примерами "способных к мультимеризации фрагментов" служат константные области тяжелых цепей иммуноглобулина, пары которых образуют функциональные Fc-области иммуноглобулина, и лейциновые "молнии" или полипептиды, содержащие свободный тиол, которые образует межмолекулярный дисульфидный мостик двумя такими полипептидами. Один цистеиновый остаток может использоваться для конъюгации других фрагментов с полипептидом, например, путем использования химических свойств малеимидов, известных специалистам в данной области техники. Связывающим белком предпочтительно является рекомбинантный связывающий белок. Связывающие области связывающего белка также предпочтительно обладают различной специфичностью к мишеням.
Термином "конкурировать за связывание" обозначается неспособность двух различных связывающих областей согласно изобретению одновременно связывать одну и ту же мишень, при этом каждая из них способна связывать ее по отдельности. Соответственно, такие две связывающие области конкурируют за связывание мишени. Упомянутые две конкурирующие связывающие области предпочтительно связывают перекрывающийся или один и тот же эпитоп мишени. Специалистам в данной области техники хорошо известны методы, позволяющие определять, конкурируют ли две связывающие области за связывание мишени, такие как конкурентный твердофазный иммуноферментный анализ (ИФА) или конкурентный анализ поверхностного плазмонного резонанса (SPR) (например, путем использования прибора Proteon производства компании BioRad).
Термином "мультипаратопный связывающий белок" обозначается связывающий белок, нацеленный на два или более различных эпитопа в одном и том же белке-мишени. Например, мультипаратопный связывающий белок, нацеленный на HER2, содержит, по меньшей мере, первую связывающую область, нацеленную на первый эпитоп в HER2, вторую связывающую область, нацеленную на отличающийся второй эпитоп в HER2, и необязательно дополнительную связывающую область, нацеленную на дополнительные эпитопы в HER2.
Термином "бипаратопный связывающий белок" обозначается связывающий белок, нацеленный на два различных эпитопа в одном и том же белке-мишени. Например, бипаратопный связывающий белок, нацеленный на HER2, содержит, по меньшей мере, первую связывающую область, нацеленную на первый эпитоп в HER2, и вторую связывающую область, нацеленную на отличающийся второй эпитоп в HER2. Соответственно, "бипаратопный DARPin" содержит первую связывающую область, нацеленную на первый эпитоп, и вторую связывающую область, нацеленную на отличающийся второй эпитоп в той же самой молекуле-мишени.
Термином "биологически активное соединение" обозначается соединение, которое модифицирует болезнь при введении страдающему этой болезнью млекопитающему. Биологически активное соединение может обладать свойствами антагониста или агониста и может являться белковое биологически активное соединение или небелковое биологически активное соединение. Такие белковые биологически активные соединения могут быть ковалентно связаны, например, со связывающей областью согласно изобретению путем генетического слияния полипептидов с использованием стандартных технологий клонирования ДНК с их последующей стандартной экспрессией и очисткой. Такие небелковые биологически активные соединения могут быть ковалентно связаны, например, со связывающей областью согласно изобретению химическим путем, например, путем связывания с тиоловой группой цистеина посредством малеимидного линкера, при этом цистеин посредством пептидного линкера связан с N- или С-концом описанной в изобретении связывающей области. Примерами белковых биологически активных соединений служат связывающие области с индивидуальной специфичностью к мишеням (например, нейтрализующие фактор роста путем его связывания), цитокины (например, интерлейкины), факторы роста (например, гормон роста человека), антитела и их фрагменты, гормоны (например, GLP-1) и любое возможное белковое лекарство. Примерами небелковых биологически активных соединений служат токсины (например, DM1 производства компании ImmunoGen), малые молекулы, нацеленные на GPCR, антибиотики и любое возможное небелковое лекарство.
Термином "связывающая область" обозначается белковый домен, имеющий "укладку" (трехмерную структуру) как у белкового каркаса и заданные свойства, определенные далее. Такая связывающая область может быть получена методами рациональной или чаще всего комбинаторной белковой инженерии, которые известны из техники (Binz и др., 2005, в процитированном месте). Например, связывающая область с заданными свойствами может быть получена способом, включающим стадии (а) использования разнообразного набора белковых доменов с укладкой как у белкового каркаса, дополнительно определенной далее; и (б) скрининга разнообразного набора и/или отбора из разнообразного набора с целью получения, по меньшей мере, одного белкового домена с заданным свойством. Разнообразный набор белковых доменов может использоваться различным способами в соответствии с используемой системой скрининга и/или отбора, включая способы, хорошо известные специалистам в данной области техники, такие как фаговый дисплей или рибосомный дисплей. Связывающей областью предпочтительно является рекомбинантная связывающая область. Связывающей областью также предпочтительно является повторяющийся белок или сконструированный повторяющийся белок.
Соответственно, используемым термином "связывает" обозначается связывающая область, которая распознает и связывает заданную мишень, но преимущественно не распознает и не связывает другие мишени. Чтобы считаться связывающей областью в смысле настоящего изобретения, для области-кандидата требуется константа диссоциации в PBS менее ю-7 м.
Термином "Kd" обозначается константа диссоциации, которая является константой равновесия специфического типа, служащей показателем предрасположения более крупного объекта к необратимому разделению (диссоциации) на меньшие компоненты, как в случае распада комплекса на составляющие его молекулы.
Способы определения констант диссоциации взаимодействий белок-белок, такие как технологии на основе поверхностного плазмонного резонанса (SPR) (например, анализ равновесия SPR) или изотермическая титрационная калориметрия (ITC), хорошо известны специалистам в данной области техники. Полученные величины Kd конкретного взаимодействия белок-белок могут варьировать в случае измерений в различных условиях (например, концентрации солей, рН). Соответственно, величины Kd предпочтительно измеряют с использованием стандартных растворов белка и стандартного буфера, такого как PBS.
Термином "PBS" обозначается забуференный фосфатом водный раствор, содержащий 137 мМ NaCl, 10 мМ фосфтата и 2,7 мМ KCl и имеющий рН 7,4.
Термином "белковый каркас" обозначается белок с открытыми поверхностями с высокой переносимостью вставок, замен или делеций аминокислот. Примерам белковых каркасов, которые могут использоваться для генерирования связывающих областей согласно настоящему изобретению, служат антитела или их фрагменты, такие как фрагменты одноцепочечных Fv или Fab, белок А из Staphylococcus aureus, билин-связывающий белок из Pieris brassicae или других липокалинов, анкириновые повторы или другие повторяющиеся белки и фибронектин человека. Белковые каркасы известны специалистам в данной области техники (Binz и др., 2005, в процитированном месте; Binz и др., 2004, в процитированном месте).
Термином "мишень" обозначается отдельная молекула, такая как молекула нуклеиновой кислоты, полипептид или белок, углевод или любая другая встречающаяся в природе молекула, включая любую часть такой отдельной молекулы или комплексы из двух или более таких молекул. Мишенью также может являться целая клетка или образец ткани, или любая не встречающаяся в природе молекула или фрагмент. Мишенью предпочтительно является встречающийся или не встречающийся в природе, полипептид или полипептид, содержащий химические модификации, например, модифицированный путем натурального или ненатурального фосфорилирования, ацетилирования или метилирования. В частном случае применения настоящего изобретения мишенью является внеклеточная область HER2.
Термином "заданное свойство" обозначается свойство, такое как связывание мишени, блокирование мишени, активация опосредованной мишенью реакции, ферментативная активность и сопутствующие дополнительные свойства. Специалисты в данной области техники смогут определить формат и необходимые шаги для осуществления скрининга и/или отбора связывающей области с желаемым свойством в зависимости от типа желаемого свойства. Заданным свойством предпочтительно является связывание мишени.
Приведенные далее определения повторяющихся белков основаны на определениях из патентной заявки WO 2002/020565. В патентной заявке WO 2002/020565 дополнительно дано общее описание признаков, методов получения и применений повторяющихся белков.
Термином "повторяющийся белок" обозначается белок, содержащий одну или несколько повторяющихся областей. Каждый из повторяющихся белков предпочтительно содержит до четырех повторяющихся областей. Каждый из повторяющихся белков более предпочтительно содержит до двух повторяющихся областей. Каждый из повторяющихся белков наиболее предпочтительно содержит только одну повторяющуюся область. Кроме того, повторяющийся белок может содержать дополнительные неповторяющиеся белковые домены, полипептидные метки и/или полипептидные линкеры.
Термином "повторяющаяся область" обозначается белковый домен, содержащий два или более последовательных повторяющихся звеньев (модулей) в качестве структурных единиц, которые имеют одинаковую плотную укладку и образуют сверхспиральную структуру с общей гидрофобной сердцевиной. Повторяющаяся область предпочтительно дополнительно содержит N-концевое и/или а С-концевое кэппирующее звено (или модуль). Еще более предпочтительно N-концевыми и/или С-концевыми кэппирующими звеньями (или модулями) являются кэппирующие повторы.
Термином "сконструированный повторяющийся белок" и "сконструированная повторяющаяся область" обозначается, соответственно, повторяющийся белок или повторяющаяся область, полученная способом по изобретению, описанному в патентной заявке WO 2002/020565. Сконструированные повторяющиеся белки и сконструированные повторяющиеся области являются синтетическими и не встречаются в природе. Они являются искусственными белками или областями, соответственно, полученными путем экспрессии соответствующим образом сконструированных нуклеиновых кислот. Экспрессия предпочтительно осуществляется в эукариотах или прокариотах, таких как бактериальные клетки, или путем использования бесклеточной системы экспрессии in vitro. Таким образом, сконструированный анкириновый повтор (т.е. DARPin) соответствует рекомбинантному связывающему белку согласно изобретению, содержащему, по меньшей мере, одну область анкиринового повтора.
Термином "структурная единица" обозначается локально упорядоченная часть полипептида, сформированная в результате трехмерных взаимодействий между двумя или более сегментами вторичной структуры, которые находятся вблизи друг друга на протяжении полипептидной цепи. Такая структурная единица обладает структурным мотивом. Термином "структурный мотив" обозначается трехмерная структура из элементов вторичной структуры, присутствующих, по меньшей мере, в одной структурной единице. Структурные мотивы хорошо известны специалистам в данной области техники. Структурные единицы как таковые не способны приобретать заданную трехмерную структуру, однако, в результате их последовательного расположения, например, в виде модулей повтора в повторяющейся области происходит взаимная стабилизация соседних единиц, и образуется сверхспиральная структура.
Термином "повторяющееся звено" обозначаются аминокислотные последовательности, содержащие повторные мотивы из одного или нескольких встречающихся в природе повторяющихся белков, при этом "повторяющиеся звенья" существуют в нескольких экземплярах и обладают заданной топологией укладки, общей для всех мотивов, определяющих укладку белка. Такие повторяющиеся звенья соответствуют "повторяющимся звеньям (повторам)" повторяющихся белков, описанных Forrer и др., 2003, в процитированном месте, или "последовательным гомологичным структурным единицам (повторам)" повторяющихся белков, описанных Binz и др. 2004, в процитированном месте. Такие повторяющиеся звенья содержат каркасные остатки и остатки взаимодействия. Примерами таких повторяющихся звеньев служат повторяющиеся звенья типа "броненосец", богатые лейцином повторяющиеся звенья, анкириновые повторы, тетратрикопептидные повторяющиеся звенья, повторяющиеся звенья HEAT и богатые лейцином вариантные повторяющиеся звенья. Встречающиеся в природе белки, содержащие два или более таких повторяющихся звеньев, называют "встречающимися в природе повторяющимися белками". Аминокислотные последовательности отдельных повторяющихся звеньев повторяющегося белка могут иметь значительное число мутаций, замен, добавлений и/или делеций при сравнении друг с другом, при этом они преимущественно сохраняют общий характер или мотив из повторяющихся звеньев.
Соответственно, термином "анкириновый повтор" обозначается повторяющееся звено, которым является анкириновый повтор, описанный, например, Forrer и др., 2003, в процитированном месте. Анкириновые повторы хорошо известны специалистам в данной области техники. Термином "область анкиринового повтора" обозначается а повторяющаяся область, содержащая два или более последовательных анкириновых повторов (модулей) в качестве структурных единиц и предпочтительно N-концевое и/или С-концевое кэппирующее звено (или модуль).
Термином "каркасные остатки" обозначается аминокислотные остатки повторяющихся звеньев или соответствующие аминокислотные остатки модулей повтора, которые содействуют топологии укладки, т.е. содействуют укладке повторяющегося звена (или модуля) или способствуют взаимодействию с соседним звеном (или модулем). Таким содействием может являться взаимодействие с другими остатками в повторяющемся звене (модуле) или влияние на конформацию полипептидного остова в α-спирали или β-листах или на участки аминокислот, образующие линейные полипептиды или петли.
Термином "остатки взаимодействия с мишенью" обозначаются аминокислотные остатки повторяющихся звеньев или соответствующие аминокислотные остатки модулей повтора, которые содействуют взаимодействию с веществами-мишенями. Таким содействием может являться прямое взаимодействие с веществами-мишенями или влияние на другие непосредственно взаимодействующие остатки, например, путем стабилизации конформации полипептида повторяющегося звена (или модуля), чтобы обеспечить или усилить взаимодействие непосредственно взаимодействующих остатков с мишенью. Такие остатки взаимодействия каркаса и мишени могут быть идентифицированы путем анализа структурных данных, полученных физико-химическими методами, такими как рентгеновская кристаллография, ЯМР- и/или CD-спектроскопия, или путем сравнения с известной и родственной структурной информацией, хорошо известной специалистам в области структурной биологии и/или биоинформатики.
Повторяющиеся звенья, используемые для вывода повторного мотива последовательности, предпочтительно представляют собой гомологичные повторяющиеся звенья, в которых содержится одинаковый структурный мотив, и более 70% каркасных остатков которых гомологичны друг другу. Предпочтительно более 80% каркасных остатков повторяющихся звеньев являются гомологичными. Наиболее предпочтительно более 90% каркасных остатков повторяющихся звеньев являются гомологичными. Специалистам в данной области известны компьютерные программы для определения степени гомологии полипептидов, такие как Fasta, Blast или Gap. Кроме того, повторяющиеся звенья, используемые для вывода повторного мотива последовательности, предпочтительно представляют собой гомологичные повторяющиеся звенья, полученные из повторяющихся областей, выбранных в заданной мишени.
Термином "повторный мотив последовательности" обозначается аминокислотная последовательность, которая выводится из одного или нескольких повторяющихся звеньев или модулей повтора. Повторяющиеся звенья или модули повтора предпочтительно получают из повторяющихся областей, специфически связывающих одну и ту же мишень. Такие повторные мотивы содержат положения каркасных остатков и положения остатков взаимодействий с мишенью. Положения каркасных остатков соответствуют положениям остатков взаимодействий с мишенью повторяющихся звеньев (или модулей). Кроме того, указанные положения остатков взаимодействий с мишенью соответствуют положениям остатков взаимодействий с мишенью повторяющихся звеньев (или модулей). Повторные мотивы последовательности содержат фиксированные положения и рандомизированные положения. Термином "фиксированное положение" обозначается положение аминокислоты в повторном мотиве последовательности, установленное для конкретной аминокислоты. Чаще всего такие фиксированные положения соответствуют положениям каркасных остатков и/или положениям остатков взаимодействия с мишенью, которые являются специфическими для определенной мишени. Термином "рандомизированное положение" обозначается положение аминокислоты в повторном мотиве последовательности, в котором могут находиться две или более аминокислот, например, в котором может находиться любая из обычных встречающихся в природе двадцати аминокислот или большинство из двадцати встречающихся в природе аминокислот, таких как аминокислоты помимо цистеина или аминокислоты помимо глицина, цистеина и пролина. Чаще всего такие рандомизированные положения соответствуют положениям остатков взаимодействия с мишенью. Тем не менее, некоторые положения каркасных остатков также могут быть рандомизированы.
Термином "топология укладки" обозначается третичная структура повторяющихся звеньев или модулей повтора. Топология укладки определяется участками аминокислот, образующими, по меньшей мере, части α-спиралей или β-листов, или участками аминокислот, образующими линейные полипептиды или петли или любое сочетание α-спиралей, β-листов и/или линейных полипептидов/петель. Например, анкириновый повтор/модуль состоит из β-витка, за которым следуют две антипараллельные α-спирали и петля, достигающая изгиба следующего повторяющегося звена/модуля.
Термином "последовательный" обозначается расположение, в котором повторяющиеся звенья или модули повтора образуют тандем. В сконструированных повторяющихся белках содержится, по меньшей мере, 2, обычно от 2 до 6, в частности, по меньшей мере, около 6, часто 20 или более повторяющихся звеньев (или модулей). В большинстве случаев повторяющиеся звенья (или модули) повторяющейся области обладают высокой степенью идентичности последовательностей (содержат одинаковые аминокислотные остатки в соответствующих положениях) или сходством последовательностей (аминокислотные остатки различаются, но имеют сходные физико-химические свойства), а некоторые из аминокислотных остатков могут являться строго консервативными ключевыми остатками. Тем не менее, в результате вставок и/или делеций и/или замен аминокислот возможна высокая степень изменчивости последовательностей в различных повторяющихся звеньях (или модулях) повторяющейся области при условии сохранения общей топология укладки повторяющихся звеньев (или модулей).
Специалистам в данной области техники хорошо известны методы прямого определения топологии укладки повторных белков физико-химическими средствами, такими как рентгеновская кристаллография, ЯМР-спектроскопия или CD-спектроскопия. В биоинформатике хорошо отработаны, и специалистам в данной области техники хорошо известны методы идентификации и определения повторяющихся звеньев или повторных мотивов последовательности или идентификации семейств родственных белков, содержащих такие повторяющиеся звенья или мотивы, такие как поиск гомологии (BLAST и др.). Стадия очистки начального повторного мотива последовательность может предусматривать итеративный процесс.
Термин "модули повтора" обозначаются повторы аминокислотных последовательностей сконструированных повторяющихся областей, которые изначально получены из повторяющихся звеньев встречающихся в природе повторяющихся белков. Каждый модуль повтора, содержащийся в повторяющейся области, получен из одного или нескольких повторяющихся звеньев семейства или подсемейства встречающихся в природе повторяющихся белков, например, семейства повторяющихся белков типа "броненосец" или анкириновых повторов. Кроме того, каждый модуль повтора, предпочтительно содержащийся в повторяющейся области, содержит повторный мотив последовательности, выведенный из гомологичных повторяющихся звеньев, полученных из повторяющихся областей, выбранных в мишени, например, как описано в Примере 1, и имеющих одинаковую специфичность к мишени.
Соответственно, термином "модуль анкиринового повтора" обозначается модуль повтора, который изначально получен из повторяющихся звеньев встречающихся в природе анкириновых повторов. Анкириновые повторы хорошо известны специалистам в данной области техники.
Модули повтора могут содержать положения с аминокислотными остатками, присутствующими во всех копиях соответствующих модулей повтора ("фиксированные положения"), и положения с различными или "рандомизированными" аминокислотными остатками ("рандомизированные положения").
Термином "кэппирующий модуль" обозначается полипептид, слитый с N-или С-концевым модулем повтора повторяющейся области, при этом кэппирующий модуль формирует жесткие третичные взаимодействия (т.е. взаимодействия третичной структуру) с модулем повтора и тем самым обеспечивает защиту от растворителя гидрофобной сердцевины модуля повтора со стороны, не контактирующей с последовательным модулем повтора. N- и/или С-концевой кэппирующий модуль может являться или может быть получен из кэппирующего звена или другой структурной единицы встречающегося в природе повторяющегося белка, смежной с повторяющимся звеном. Термином "кэппирующее звено" обозначается встречающиеся в природе уложенный полипептид, определяющий конкретную структурную единицу, N- или С-конец которой слит с повторяющимся звеном, при этом полипептид формирует жесткие третичные взаимодействия с повторяющимся звеном и тем самым обеспечивает защиту от растворителя гидрофобной сердцевины повторяющегося звена с одной стороны. Кэппирующими модулями или кэппирующими звеньями предпочтительно являются кэппирующие повторы. Термином "кэппирующий повтор" обозначается кэппирующий модуль или кэппирующее звено, имеющее сходную или такую же укладку как у упомянутого смежного повторяющегося звена (или модуля), и/или с последовательности, сходные с упомянутым повторяющимся звеном (или модулем). Кэппирующие модули и кэппирующие повторы описаны в WO 2002/020565 и Interlandi и др., 2008 (в процитированном месте).
Примерами N-концевых анкириновых кэппирующих модулей (т.е. N-концевых кэппирующих повторов) служат последовательности SEQ ID 1, 2, 3, 13, 14, 20, 26, 27 36, 40, 44, 45, 50, 54, 124, 128 и 132, а примерами С-концевых анкириновых кэппирующих модулей (т.е. С-концевых кэппирующих повторов) служат последовательности SEQ ID 4, 5, 19, 24, 25, 33, 34, 35, 39, 43, 48, 49, 53, 57, 127, 131 и 135.
Например, N-концевой анкириновый кэппирующий модуль с последовательностью SEQ ID 13 кодируется аминокислотами из положений 1-32, а С-концевой кэппирующий модуль с последовательностью SEQ ID 19 кодируется аминокислотами из положений 99-126.
Рекомбинантный связывающий белок согласно изобретению содержит, по меньшей мере, одну область анкиринового повтора, специфически связывающую внеклеточную область HER2 млекопитающих.
Термин "специфичность связывания мишени", "специфически связывающий мишень" или "специфичность к мишени" и т.п. означает, что связывающий белок или связывающая областью связывает мишень в PBS с меньшей константой диссоциации, чем неродственный белок, такой как связывающий мальтозу белок (МВР) Е. coli. Константа диссоциации в PBS для мишени является, по меньшей мере, в 10, более предпочтительно, по меньшей мере, в 102, еще более предпочтительно, по меньшей мере, в 103, наиболее предпочтительно, по меньшей мере, в 104 раз меньшей, чем соответствующая константа диссоциации для МВР.
Термином "консенсусная последовательность" обозначается аминокислотная последовательность, которая получена путем выравнивания структур и/или последовательностей множества повторяющихся звеньев. Путем использования двух или более повторяющихся звеньев с выравниванием структур и/или последовательностей с учетом разрывов при выравнивании можно определять наиболее часто встречающиеся аминокислотные остатки в каждом положении. Консенсусная последовательность представляет собой последовательность, содержащую аминокислоты, которые чаще всего встречаются в каждом положении. Если встречаемость двух или более аминокислот в одном положении превышает среднюю встречаемость, консенсусная последовательность может содержать подгруппу этих аминокислот. Упомянутые два или несколько повторяющихся звеньев могут быть взяты из повторяющихся звеньев, входящих в один повторяющийся белок или в два или более различных повторяющихся белков.
Консенсусные последовательности и способы их определения хорошо известны специалистам в данной области техники.
"Консенсусным аминокислотным остатком" является аминокислота в определенном положении в консенсусной последовательности. Если два или более, например, три, четыре или пять аминокислотных остатков имеют с одинаковой вероятностью встречаются в двух или более повторяющихся звеньев, консенсусной аминокислотой может являться одна из наиболее часто встречаемых аминокислот или сочетание упомянутых двух или более аминокислотных остатков.
Предпочтительными также являются не встречающиеся в природе кэппирующие модули, модули повтора, связывающие белки или связывающие области.
Термин "не встречающийся в природе" означает синтетический или искусственный, более точно, являющийся продуктом человеческой деятельности. Термин "не встречающийся в природе связывающий белок" или "не встречающаяся в природе связывающая область" означает, что такой связывающий белок или такая связывающая область является синтетической (т.е. полученной из аминокислот путем химического синтеза) или рекомбинантной, а не натуральной. Не встречающимся в природе связывающим белком или не встречающейся в природе связывающей областью является искусственный белок или область, соответственно, полученная путем экспрессии соответствующим образом разработанных нуклеиновых кислот. Экспрессия предпочтительно осуществляется в эукариотах или бактериальных клетках или путем использования бесклеточной системы экспрессии in vitro. Этот термин дополнительно означает, что последовательность связывающего белка или связывающей областью отсутствует в базе данных неискусственных последовательностей, например, GenBank, EMBL-Bank или Swiss-Prot. Эти и аналогичные базы данных последовательностей хорошо известны специалистам в данной области техники.
Далее описаны общие модификации и производные областей анкириновых повторов согласно изобретению, в частности, модулей анкиринового повтора и кэппирующих модулей.
Предпочтительным также является N-концевой или С-концевой анкириновый кэппирующий модуль, содержащий N-концевой или С-концевой анкириновый кэппирующий повтор, соответственно, в котором один или несколько аминокислотных остатков заменены аминокислотным остатком, встречающимся в соответствующем положении с выравниванием по соответствующему анкириновому кэппирующему звену или анкириновому повтору.
Аминокислоты могут быть заменены любой из 20 наиболее часто встречающихся в природе аминокислот, предпочтительно выбранных из группы, включающей A, D, Е, F, H, I, К, L, M, N, Q, R, S, Τ, V, W и Y; более предпочтительно из группы, включающей A, D, Ε, H, I, К, L, Q, R, S, Τ, V и Y. Аминокислоты также могут быть предпочтительно заменены гомологичной аминокислотой, т.е. аминокислота заменяется аминокислотой, имеющей боковую цепь со сходными биофизическими свойствами. Например, отрицательно заряженная аминокислота D может быть заменена отрицательно заряженной аминокислотой Е, или гидрофобная аминокислота, такая как L, может быть заменена аминокислотой А, I или V. Замена аминокислоты гомологичной аминокислотой хорошо известна специалистам в данной области техники.
Также предпочтительным является С-концевой анкириновый кэппирующий модуль, содержащий аминокислоту А в положении 27 и 28, из любых упомянутых С-концевых кэппирующих модулей на основе SEQ ID 4, 5, 19, 24, 25, 33, 34, 35, 39, 43, 48, 49, 53, 57,127, 131 или 135.
Также предпочтительным является С-концевой кэппирующий модуль, содержащий аминокислоты в положениях 1-26 или 1-27, из любых упомянутых С-концевых кэппирующих модулей на основе SEQ ID 4, 5, 19, 24, 25, 33, 34, 35, 39, 43, 48, 49, 53, 57, 127, 131 135.
Из N-концевых анкириновых кэппирующих модулей могут быть без видимого влияния на их свойства удалены аминокислоты G в положении 1 и/или S в положении 2 SEQ ID 1, 2, 3, 13, 14, 20, 26, 27, 36, 40, 44, 45, 50, 54, 124, 128 или 132. Эти две аминокислоты служат линкерами для соединения области анкиринового повтора с дополнительными аминокислотами и белками. В изобретении также предусмотрены такие области анкириновых повторов, содержащие N-концевые анкириновые кэппирующие модули, у которых удалены аминокислота G в положении 1 и/или аминокислота S в положении 2. Подразумевается, что указанные в описании положения аминокислот (например, "положение 33") в области анкиринового повтора соответствующим образом адаптированы с учетом сдвига числа, и например, "положение 33" становится "положением 32", если отсутствует одна аминокислота, или "положение 33" становится "положением 31", если отсутствуют две аминокислоты.
Анкириновый кэппирующий модуль области анкиринового повтора согласно изобретению может быть заменен анкириновым кэппирующим модулем методами комбинирования, такими как выравнивание аминокислотных последовательностей, мутагенез и синтез генов, известными специалистам в данной области техники. Например, С-концевой кэппирующий повтор SEQ ID 79 может быть заменен С-концевым кэппирующим повтором SEQ ID 5 путем (i) обнаружения С-концевого кэппирующего повтора SEQ ID 79 (т.е. положений 99-126) посредством выравнивания последовательности с SEQ ID 5, (ii) замены последовательности обнаруженного С-концевого кэппирующего повтора SEQ ID 79 последовательностью SEQ ID 5, (iii) генерирования гена, кодирующего повторяющуюся область, кодирующую замененный С-концевой кэппирующий модуль, (iv) экспрессии модифицированной повторяющейся области в цитоплазме Е. coli и (v) очистки модифицированной повторяющейся области стандартными средствами. В качестве дополнительно примера, N-концевой кэппирующий повтор SEQ ID 79 может быть заменен N-концевым кэппирующим повтором SEQ ID 3 путем (i) обнаружения N-концевого кэппирующего повтора SEQ ID 79 (т.е. положений 1-32) посредством выравнивания последовательности с SEQ ID 3, (ii) замены последовательности обнаруженного N-концевого кэппирующего повтора SEQ ID 79 последовательностью SEQ ID 3, (iii) генерирования гена, кодирующего повторяющуюся область, кодирующую замененный N-концевой кэппирующий модуль, (iv) экспрессии модифицированной повторяющейся области в цитоплазме Е. coli и (ν) очистки модифицированной повторяющейся области стандартными средствами.
Кроме того, область анкиринового повтора согласно изобретению может быть генетически сконструирована путем синтеза генов с целью компоновки Ν-концевого анкиринового каптирующего модуля (например, N-концевого кэппирующего повтора SEQ ID 3), за которым следует один или несколько модулей повтора (например, два модуля анкиринового повтора, содержащие аминокислотные остатки в положениях 33-99 SEQ ID 79) и С-концевой кэппирующий модуль (например, С-концевой кэппирующий повтор SEQ ID 5). Затем ген генетически скомпонованной повторяющейся области может быть экспрессирован в Е. Coli, как описано выше.
Также предпочтительным является а рекомбинантный связывающий белок, повторяющаяся область, модуль повтора, N-концевой кэппирующий модуль или С-концевой кэппирующий модуль, имеющий аминокислотную последовательность, лишенную аминокислот С, M или N.
Также предпочтительным является рекомбинантный связывающий белок, повторяющаяся область, модуль повтора, N-концевой кэппирующий модуль или С-концевой кэппирующий модуль, имеющий аминокислотную последовательность, лишенную аминокислоты N, за которой следует G.
Также предпочтительным является рекомбинантный связывающий белок или повторяющаяся область, содержащая любой такой N-концевой или С-концевой кэппирующий модуль.
В одном из дополнительных предпочтительных вариантов осуществления рекомбинантного связывающего белка, содержащего область анкиринового повтора согласно настоящему изобретению, один или несколько аминокислотных остатков N-концевого кэппирующего модуля повторяющейся области заменен аминокислотным остатком, находящимся в соответствующем положении с выравниванием по N-концевому кэппирующему звену. Предпочтительно заменено до 30%, более предпочтительно до 20%, еще более предпочтительно до 10% аминокислотных остатков. Наиболее предпочтительно таким N-концевым кэппирующим звеном является встречающееся в природе N-концевое кэппирующее звено.
В одном из дополнительных предпочтительных вариантов осуществления рекомбинантного связывающего белка, содержащего область анкиринового повтора согласно настоящему изобретению, один или несколько аминокислотных остатков С- концевого кэппирующего модуля повторяющейся области заменен аминокислотным остатком, находящимся в соответствующем положении с выравниванием по С-концевому кэппирующему звену. Предпочтительно заменено до 30%, более предпочтительно до 20%, еще более предпочтительно до 10% аминокислотных остатков. Наиболее предпочтительно таким С-концевым кэппирующим звеном является встречающееся в природе С-концевое кэппирующее звено.
В еще одном частном варианте осуществления до 30%, более предпочтительно, до 20%, еще более предпочтительно до 10% аминокислотных остатков заменено аминокислотами, которые не находятся в соответствующих положениях повторяющихся звеньев, N-концевых кэппирующих блоков или С-концевых кэппирующих блоков.
В одном из дополнительных предпочтительных вариантов осуществления рекомбинантного связывающего белка, содержащего область анкиринового повтора согласно настоящему изобретению, один или несколько аминокислотных остатков модулей повтора упомянутой области анкиринового повтора заменены аминокислотным остатком, находящимся в соответствующем положении с выравниванием по повторяющемуся звену. Предпочтительно заменено до 30%, более предпочтительно до 20%, еще более предпочтительно до 10% аминокислотных остатков. Наиболее предпочтительно таким повторяющимся звеном является встречающееся в природе повторяющееся звено.
В еще одном частном варианте осуществления до 30%, более предпочтительно до 20%, еще более предпочтительно до 10% аминокислотных остатков заменено аминокислотами, которые не находятся в соответствующих положениях повторяющихся звеньев.
В дополнительных вариантах осуществления любые описанные в изобретении связывающие HER2 рекомбинантные белки или области могут ковалентно связывать один или несколько дополнительных фрагментов, включая, например, фрагмент, который связывает другую мишень, с целью создания связывающего средства с двойной специфичностью, биологически активного соединения, фрагмента для мечения (например, флуоресцентной метки, такой как флуоресцеин, или радиоактивный индикатор), фрагмента, который облегчает очистку белка (например, метки на основе малых пептидов, такой His- или strep-метка), фрагмента, который обеспечивает эффекторные функции с целью повышения эффективности терапии (например, Fc-части антитела для придания антитело-зависимой клеточно-опосредованной цитотоксичности, фрагмента токсичного белка, такого как экзотоксин A (ETA) Pseudomonas aeruginosa, или маломолекулярного токсина, такого как майтансиноиды или алкилирующие ДНК средства), или фрагмента, который обеспечивает улучшение фармакокинетики. Улучшение фармакокинетики может оцениваться в соответствии с ощущаемой терапевтической потребностью. Часто желательным является повышение биологической доступности и/или увеличение времени между дозами, возможно, путем увеличения времени, в течение которого белок остается доступным в сыворотке после введения дозы. В некоторых случаях желательным является повышение постоянства концентрации белка в сыворотке с течением времени (например, уменьшения различий в концентрации белка в сыворотке вскоре после введения и незадолго перед следующим введением). Фрагменты, которые имеют тенденцию замедлять выведение белка из крови, включают гидроксиэтилкрахмал (HES), полиэтиленгликоль (PEG), сахара (например, сиаловую кислоту), хорошо переносимые фрагменты белков (например, Fc-фрагменты или сывороточный альбумин) и связывающие области или пептиды со специфичностью и сродством к распространенным сывороточным белкам, таким как Fc-фрагменты антител или сывороточный альбумин. Примеры таких связывающих областей или повторяющихся областей со сродством к сывороточному альбумину приведены в WO 2012/069654. Рекомбинантный связывающий белок согласно изобретению может быть связан с фрагментом, который снижает скорость выведения полипептидов у млекопитающего (например, мыши, крысы или человека) в три с лишним раза по сравнению с немодифицированными полипептидами.
В одном из частных вариантов осуществления предложен рекомбинантный связывающий белок, содержащий первую повторяющуюся область, связывающую HER2, вторую повторяющуюся область, связывающую HER2, и дополнительно содержащий одну или несколько областей анкириновых повторов, специфически связывающих сывороточный альбумин человека. Примеры повторяющихся областей со специфичностью к HER2 приведены в описании, а примеры областей анкириновых повторов со специфичностью к сывороточному альбумину описаны в WO 2012/069654. Такие области могут быть генетическим средствами связаны полипептидным линкером способами, известными специалистам в данной области техники.
В другом предпочтительном варианте осуществления предложен рекомбинантный связывающий белок, в котором первой повторяющейся областью и второй повторяющейся областью являются области анкириновых повторов, специфически связывающие HER2 и содержащие один, два, три или более внутренних модулей повтора, которые участвуют в связывании HER2. Такие области анкириновых повторов предпочтительно содержат N-концевой кэппирующий модуль, от одного до четырех внутренних модулей повтора и С-концевой кэппирующий модуль. Кэппирующими модулями предпочтительно являются кэппирующие повторы. Кэппирующими модулями также предпочтительно участвуют в связывании HER2.
Кроме того, любая их упомянутых фармацевтических композиций рассчитана на лечение заболевания.
В изобретении дополнительно предложен способ лечения. Способ включает введение нуждающемуся в этом пациенту терапевтически эффективного количества рекомбинантного связывающего белка согласно изобретению.
Кроме того, предложен способ лечения патологического состояния у млекопитающего, в том числе человека, включающий введение нуждающемуся в этом пациенту эффективного количества упомянутой фармацевтической композиции.
Примеры
Все указанные далее исходные материалы и реагенты известны специалистам в данной области техники и предлагаются на рынке или могут быть получены с использованием известных методов.
Материалы
Химические вещества приобретены у компании Fluka (Швейцария). Олигонуклеотиды получены от компании Microsynth (Швейцария). Если не указано иное, ДНК-полимеразы, рестриктазы и буферы получены от компаний New England Biolabs (США) или Fermentas (Литва). В качестве штамма для клонирования и синтеза белка использован штамм E.coli XL I-blue (производства компании Stratagene, США) или BL21 (производства компании Novagen, США). Эктообласть рекомбинантного HER2 человека (ErbB2 S22-N530-Flag и ErbB2 S22-E645-Flag, синтезированные в клетках СНО стандартными средствами) приобретена у компании CSIRO Enquiries (Австралия). Эктообласть биотинилированного Her2 получена химическим способом путем связывания биотинового фрагмента с первичными аминами белка с использованием стандартных реагентов и способов биотинилирования (производства компании Pierce, США). Линии клеток приобретены у компаний LGC/ATCC (Франция/США; номера по каталогу: ВТ474 - НТВ-20, SKBR-3 - НТВ-30, NCI-N87 - CRL5822, ZR75-30 - CRL1504, НСС1419 - CRL2326, MDA-MB175 VII - НТВ-25). Питательные среды для культур клеток получены от компаний Invitrogen/Lubio (Швейцария). Фетальная телячья сыворотка получена от компании РАА. Химический реактив для обнаружения пролиферации клеток методом ИФА BrdU (колориметрический) (номер по каталогу 1164722900) получен от компании Roche, (Швейцария), химический реактив для обнаружения апоптоза Caspase Glo 3/7 (номер по каталогу G8091) получен от компании Promega (Швейцария), а система обнаружения некроза клеток ELISAPLUS (11 774 425 001) получена от компании Roche (Швейцария). Реактив для трансфекции клеток Lipofectamin 2000 (11668027) получен от компании Invitrogen (Швейцария). Анализ методом FACS осуществлялся с использованием системы FACS Canto II производства компании Becton-Dickinson (Швейцария). Для обнаружения связывания Her2 DARPins использовали конъюгат анти-Penta-His Alexa Fluor 647 (номер по каталогу А21445; производства компании Lubio (Швейцария)). Использована аккутаза (номер по каталогу L-11-007) производства компании РАА. Трастузумаб приобретен в кантональной аптеке Цюриха, а пертузумаб синтезирован компанией Evitra (Швейцария). Вектор экспрессии GFP-меченного Her2 (номер по каталогу RG212583) получен от компании Origene (США).
Молекулярная биология
Если не указано иное, процедуры осуществлялись в соответствии с описанными протоколами (Sambrook J., Fritsch Ε.F. и Maniatis Т., Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory 1989, New York).
Анализ пролиферации
Определили воздействие DARPins на пролиферацию клеток путем измерения синтеза ДНК с использованием мечения BrdU (BrdU, Cell Proliferation ELISA, производства компании Roche). Вкратце, высеяли по 10000 клеток ВТ474 на лунку 96-луночного планшета в 100 мкл полной среды и инкубировали в течение 24 ч. Добавили DARPins и эталоны в течение еще 72 часов. Добавили BrdU с целью мечения клеток в течение последних 24 часов. Детектировали меченые (пролиферирующие) клетки согласно протоколу производителя. Проанализировали данные с использованием программного обеспечения GraphPad, и построили график зависимости логарифма [с] по оси х от OD450-602 нМ по оси у. Для подбора данных использовали метод нелинейной регрессии (логарифм (антагонист) в зависимости от ответа - переменный наклон (четыре параметра)).
Анализ апоптоза
Определили индуцирование апоптоза DARPins путем измерения активации Caspase3/7 с использованием систем Caspase 3/7-Glo (производства компании Promega, Швейцария). Вкратце, высеяли по 10000 клеток ВТ474 на лунку 96-луночного планшета в 100 мкл полной среды и инкубировали в течение 24 ч. Добавили DARPins и эталоны в течение еще 24 часов. Добавили реактив Caspase Glo согласно протоколу производителя в течение 1 часа. Контролировали активацию Caspase 3/7 путем измерения активности люциферазы.
В качестве альтернативы, определили индуцирование апоптоза с использованием системы обнаружения некроза клеток ELISAPLUS (производства компании Roche, (Швейцария)). Осуществили анализ согласно протоколу производителя. Использовали такое же число клеток и длительность инкубации, как при обнаружении с помощью Caspase Glo.
Проанализировали данные с использованием программного обеспечения GraphPad, и построили график зависимости концентрации по оси х от OD450-602 нМ или RLU по оси у. Для подбора данных использовали метод нелинейной регрессии (логарифм (агонист) в зависимости от ответа - переменный наклон (четыре параметра)).
Библиотеки сконструированных анкириновых повторов
Методы генерирования библиотек сконструированных анкириновых повторов известны из техники (WO 2002/020565; Binz и др. 2003, в процитированном месте; Binz и др. 2004, в процитированном месте). Такими методами могут создаваться библиотеки сконструированных анкириновых повторов, содержащие рандомизированные модули анкириновых повторов и/или рандомизированные кэппирующие модули. Например, такие библиотеки могут соответствующим образом компоноваться на основе фиксированного N-концевого кэппирующего модуля (например, N-концевого кэппирующего модуля SEQ ID 2) или рандомизированного N-концевого кэппирующего модуля согласно мотиву последовательности SEQ ID 60, одного или нескольких рандомизированных модулей повтора согласно мотиву последовательности SEQ ID 58 или 59 и фиксированного С-концевого кэппирующего модуля (например, С-концевого кэппирующего модуля SEQ ID 5) или рандомизированного С-концевого кэппирующего модуля согласно мотиву последовательности SEQ ID 61. Такие библиотеки предпочтительно компонуются таким образом, чтобы в них не входили аминокислоты С, G, Μ, N (перед остатком G) или Ρ в рандомизированных положениях модулей повтора или кэппирующих модулях. Кроме того, рандомизированные модули повтора согласно мотиву последовательности SEQ ID 58 или 59 могут быть дополнительно рандомизированы в положении 10 и/или положении 17; рандомизированный N-концевой кэппирующий модуль согласно мотиву последовательности SEQ ID 60 может быть дополнительно рандомизирован в положении 7 и/или положении 9; и рандомизированные С-концевые кэппирующие модули согласно мотиву последовательности SEQ ID 61 могут быть дополнительно рандомизированы в положениях 10, 11 и/или 17.
Кроме того, такие рандомизированные модули в таких библиотеках могут содержать дополнительные петлевые вставки в полипептидные цепи с рандомизированными положениями аминокислот. Примерами таких петлевых вставок в полипептидные цепи служат библиотеки содержащих петлевые комплемент-определяющие области (CDR) антител или библиотеки вновь генерированных пептидов. Например, такая вставка в петлю может быть сконструирована с использованием структуры N-концевой области анкиринового повтора рибонуклеазы L человека (Tanaka, Ν., Nakanishi, M, Kusakabe, Y, Goto, Y., Kitade, Y, Nakamura, K.T., EMBO J. 23(30), 3929-3938, 2004) в качестве ориентира. По аналогии с этой областью анкиринового повтора со вставкой из 10 аминокислот в бета-изгиб вблизи более границы двух анкириновых повторов, в библиотеках анкириновых повторах могут содержатся рандомизированные петли (с фиксированными и рандомизированными положении) переменной длины (например, от 1 20 аминокислот), вставленные в один или несколько бета-изгибов области анкиринового повтора.
Любой такой N-концевой кэппирующий модуль из библиотеки анкириновых повторов предпочтительно содержит мотив RELLKA или RILKAA вместо мотива RILLAA (например, в положениях 21-26 в SEQ ID 65), а любой такой С-концевой кэппирующий модуль из библиотеки анкириновых повторов предпочтительно содержит мотив КАА или мотив KLA вместо мотива KLN (например, последние три аминокислоты в SEQ ID 65).
В качестве ориентира при конструировании такой библиотеки анкириновых повторов могут использоваться известные структуры области анкиринового повтора, взаимодействующей с мишенью. Примеры таких структур, идентифицируемых по их уникальным кодам доступа или идентификации (PDB-ID) в банке данных белков, служат 1WDY, 3V31, 3V30, 3V2X, 3V20, 3UXG, 3TWQ-3TWX, 1N11, 1S70 и 2ZGD.
В литературе описаны примеры библиотек сконструированных анкириновых повторов, таких как библиотеки сконструированных анкириновых повторов N2C и N3C (WO 2002/020565; Binz и др., 2003, в процитированном месте; Binz и др. 2004, в процитированном месте). Индексами N2C и N3C обозначается номер рандомизированного модуля повтора между N-концевыми С-концевыми кэппирующими модулями.
Для определения положений внутри повторяющихся звеньев и модулей используется номенклатура, предложенная Binz и др. (2004, в процитированном месте), за исключением того, что границы модулей анкиринового повтора и анкириновых повторов сдвинуты на одно положение аминокислоты. Например, положение 1 модуля анкиринового повтора у Binz и др. (2004, в процитированном месте) соответствует положению 2 модуля анкиринового повтора согласно настоящему изобретению, и, следовательно, положение 33 модуля анкиринового повтора у Binz и др. (2004, в процитированном месте) соответствует положению 1 следующего модуля анкиринового повтора согласно настоящему изобретению.
Все последовательности ДНК были подтверждены путем секвенирования, а вычисленная молекулярная масса всех описанных белков была подтверждена путем масс-спектрометрии.
Пример 1
Отбор, связывающих белков, содержащих области анкириновых повторов, специфически связывающие HER2
Отобрали множество сконструированных анкириновых повторов (DARPins), специфически связывающих эктообласть HER2, из библиотек DARPin, как описано у Binz и др. (2004, в процитированном месте) с использованием рибосомного дисплея (Hanes, J. и Plückthun, Α., PNAS 94, 4937-42, 1997). Оценили их специфичность связывания методом ИФА суммарного экстракта (смотри далее), что означало, что были отобраны сотни специфически связывающих HER2 белков. Определили HER2- специфическое ингибирование пролиферации и индуцирования апоптоза отобранных клонов путем тестирования способности бипаратопных DARPins ингибировать пролиферацию клеток ВТ474.
Например, области анкириновых повторов SEQ ID 62-82, 112-121 образуют аминокислотные последовательности отобранных связывающих белков, содержащих область анкиринового повтора, специфически связывающую HER2. Отдельные модули анкиринового повтора таких областей анкириновых повторов, специфически связывающих HER2, содержатся в SEQ ID 15-18, 21-23, 28-32, 37, 38, 41, 42, 46, 47, 51, 52, 55, 56, 125, 126, 129, 130, 133 и 134. Отдельные кэппирующие модули таких областей анкириновых повторов, специфически связывающих HER2, содержатся в SEQ ID 13, 14, 19, 20, 24-27, 33-36, 39, 40, 43-45, 48-50, 53, 54, 57, 124, 127, 128, 131, 132 и 135.
Отбор HER2-специфических анкириновых повторов с помощью рибосомного дисплея
Осуществили отбор HER2-специфических анкириновых повторов с помощью рибосомного дисплея (Hanes и Plückthun, в процитированном месте) с использованием HER2 человека в качестве белков-мишеней, описанных выше библиотек сконструированных анкириновьгх повторов и принятых протоколов (Zahnd, С, Amstutz, Р. и Plückthun, Α., Nat. Methods 4, 69-79, 2007). Число циклов обратной транскрипции (ОТ) - полимеразной цепной реакции (ПЦР) после каждого раунда отбора постоянно снижалось с 45 до 30 в соответствии с выходом вследствие накопления связывающих белков. В первых четырех раундах осуществляли стандартный отбор с использованием рибосомного дисплея со снижением концентрации мишени и ужесточением строгости промывания с целью повышения давление отбора от раунда 1 к раунду 4 (Binz и др., 2004, в процитированном месте). С целью накопления DARPins с высоким сродством к HER2 использовали DARPins, отобранные стандартным путем в четвертом раунде с использованием рибосомного дисплея, для отбора на основании скорости диссоциации с повышенной строгостью отбора (Zahnd, 2007, в процитированном месте). Осуществили окончательный раунд стандартного отбора, чтобы амплифицировать и восстановить связывающие белки, отобранные на основании скорости диссоциации.
Специфическое связывание HER2 отобранными клонами, определенное методом ИФА суммарного экстракта
Идентифицировали отдельные отобранные DARPins, специфически связывающие эктообласть HER2, методом твердофазного иммуноферментного анализа (ИФА) с использованием суммарных экстрактов Escherichia coli экспрессирующих DARPin клеток согласно стандартным протоколам. Клонировали DARPins, отобранные с использованием рибосомного дисплея, в вектор pQE30 (Qiagen) экспрессии, преобразовали в XL 1-Blue (Stratagene) Ε. Coli и затем культивировали в течение ночи при 37°С на 96-луночном планшете с глубокими лунками (каждый клон в отдельной лунке), содержащем 1 мл среды для выращивания (2YT, содержащей 1% глюкозу и 100 мкг/мл ампициллина). Засеяли 100 мкл выращенной в течение ночи культуры в свежий 96-луночный планшет с глубокими лунками с 1 мл свежей 2YT, содержащей 50 мкг/мл ампициллина. После инкубации в течение 2 часов при 37°С с помощью IPTG (с конечной концентрацией 1 мМ) индуцировали экспрессию, которая продолжалась в течение 3 часов. Собрали клетки, повторно суспендировали в 100 мкл B-PERII (Pierce) и инкубировали со встряхиванием в течение 15 минут при комнатной температуре. Затем добавили 900 мкл PBS-TG (PBS с добавлением 0,25% гидролизата казеина, 0,1% Tween 20®, рН 7,4), и удалили клеточные остатки путем центрифугирования. Поместили 100 мкл каждого лизированного клона в лунку покрытого нейтравидином планшета MaxiSorp, содержащего HER2 или неродственные МВР, иммобилизованные посредством из биотинового фрагмента и инкубированный в течение 1 часа при комнатной температуре. После обильного промывания планшета с помощью PBS-T (PBS с добавлением 0,1% Tween 20®, рН 7,4) осуществили стандартные процедуры ИФА с использованием меченного пероксидазой хрена моноклонального антитела к RGS(His)4 (34650, Qiagen). Затем детектировали связывание субстратом POD (Roche). Измерили проявление цвета при концентрации 405 нМ. В результате скрининга нескольких сотен клонов таким методом ИФА суммарного экстракта было обнаружено более ста различных DARPins со специфичностью к HER2. Эти связывающие белки были отобраны для дальнейшего анализа. Примеры аминокислотных последовательностей отобранных областей анкириновых повторов, которые специфически связывают эктообласть HER2, служат SEQ ID 62-82 и 112-121.
Клонировали эти области анкириновых повторов, специфически связывающие HER2, и область анкиринового повтора, специфически не связывающие HER2, в качестве отрицательного контроля (т.е. SEQ ID 111) в вектор экспрессии на основе pQE (QIAgen, Германия), обеспечивающий N-концевую His-метку для облегчения очистки белка, как описано далее. Соответственно, сконструировали векторы экспрессии, кодирующие следующие DARPins:
DARPin # 1 (SEQ ID 62 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 2 (SEQ ID 63 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 3 (SEQ ID 64 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 5 (SEQ ID 66 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 6 (SEQ ID 67 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 7 (SEQ ID 68 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 8 (SEQ ID 69 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 9 (SEQ ID 70 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 10 (SEQ ID 71 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 11 (SEQ ID 72 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 12 (SEQ ID 73 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 13 (SEQ ID 74 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 14 (SEQ ID 75 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 15 (SEQ ID 76 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 16 (SEQ ID 77 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 17 (SEQ ID 78 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 18 (SEQ ID 79 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 19 (SEQ ID 80 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 20 (SEQ ID 81 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 21 (SEQ ID 82 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin #50 (SEQ ID 111 с His-меткой (SEQ ID 6), слитой с ее N-концом).
DARPin # 51 (SEQ ID 112 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 52 (SEQ ID 113 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 53 (SEQ ID 114 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 54 (SEQ ID 115 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 55 (SEQ ID 116 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 56 (SEQ ID 117 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 57 (SEQ ID 118 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin #58 (SEQ ID 119 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 59 (SEQ ID 120 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 60 (SEQ ID 121 с His-меткой (SEQ ID 6), слитой с ее N-концом).
Примерами аминокислотных последовательностей отобранных бипаратопных анкириновых повторов служат SEQ ID 83-110, 122, 123 и 136-141. Клонировали эти бипаратопные DARPins в вектор экспрессии на основе pQE (QIAgen, Германия), обеспечивающий N-концевую His-метку для облегчения очистки белка, как описано далее. Соответственно, сконструировали векторы экспрессии, кодирующие следующие DARPins:
DARPin # 22 (SEQ ID 83 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 23 (SEQ ID 84 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 24 (SEQ ID 85 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 25 (SEQ ID 86 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 26 (SEQ ID 87 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 27 (SEQ ID 88 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 28 (SEQ ID 89 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 29 (SEQ ID 90 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 30 (SEQ ID 91 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 31 (SEQ ID 92 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 32 (SEQ ID 93 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 33 (SEQ ID 94 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 34 (SEQ ID 95 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin #35 (SEQ ID 96 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 36 (SEQ ID 97 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 37 (SEQ ID 98 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin #38 (SEQ ID 99 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin #39 (SEQ ID 100 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 40 (SEQ ID 101 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 41 (SEQ ID 102 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 42 (SEQ ID 103 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 43 (SEQ ID 104 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 44 (SEQ ID 105 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 45 (SEQ ID 106 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 46 (SEQ ID 107 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 47 (SEQ ID 108 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 48 (SEQ ID 109 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 49 (SEQ ID 110 с His-меткой (SEQ ID 6), слитой с ее N-концом)
DARPin # 61 (SEQ ID 122 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 62 (SEQ ID 123 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 63 (SEQ ID 136 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 64 (SEQ ID 137 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 65 (SEQ ID 138 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 66 (SEQ ID 139 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 67 (SEQ ID 140 с His-меткой (SEQ ID 6), слитой с ее N-концом);
DARPin # 68 (SEQ ID 141 с His-меткой (SEQ ID 6), слитой с ее N-концом).
Высокоуровневая экспрессия моновалентных DARPins в растворимой форме
С целью дальнейшего анализа экспрессировали DARPins # 1-50 в BL21 Е. coli или клетках XL1-Blue и очистили с использованием их His-метки согласно стандартным протоколам. Использовали 25 мл культивированных в течение ночи стационарных культур (LB, 1% глюкозы, 100 мг/л ампициллина; 37°С), чтобы засеять 1 литр культур (такую же среду). При оптической плотности 0,7 на волне 600 нм индуцировали культуры 0,5 мМ IPTG и инкубировали при 37°С в течение 4-5 часов. Центрифугировали культуры, повторно суспендировали полученные гранулы в 40 мл TBS500 (50 мМ трис-НС1, 500 мМ NaCl, рН 8) и подвергли действию ультразвука. Повторно центрифугировали лизат, и добавили в полученную надосадочную жидкость глицерин (с конечной концентрацией 10% по объему) и имидазол (с конечной концентрацией 20 мМ). Очистили белки в колонне с Ni-нитрилтрехуксусной кислотой (объемом 2,5 мл) согласно указаниям производителя (QIAgen, Германия). В качестве альтернативы, очистили DARPins или отобранные повторяющиеся области, лишенные 6xHis-мeтки, путем анионообменной хроматографии с последующей гель-хроматографией согласно стандартным протоколам, известным специалистам в данной области техники. Из одного литра культуры Е. coli может быть получеро до 200 мг очищенных высокорастворимых DARPins, специфически связывающих HER2, со степенью чистоты >95% по оценке методом SDS-15% PAGE. Использовали такие очищенные DARPins для дальнейшего определения характеристик.
Пример 2
Определение характеристик DARPins, специфически связывающих HER2, методом анализа поверхностного плазмонного резонанса
Исследовали кинетику связывания белка интересующих очищенных DARPins, связывающих HER2, методом анализа поверхностного плазмонного резонанса (SPR) с помощью системы ProteOn (BioRad) с использованием настройки, при которой биотинилированный HER2 человека был иммобилизован посредством нейтравидина, и было измерено взаимодействие путем добавления свободного моновалентного DARPin. Определили показатели Kd согласно стандартным процедурам.
Иммобилизовали биотинилированную эктообласть молекулы HER2 человека в проточной кювете путем связывания стрептавидина с покрытием, и проанализировали взаимодействие с различным отобранными DARPins.
Анализ поверхностного плазмонного резонанса (SPR)
Определили SPR с помощью прибора ProteOn (BioRad) согласно стандартным процедурам, известным специалистам в данной области техники. В качестве подвижного буфера использовали PBS (рН 7,4), содержащий 0,005% Tween 20®. Ковалентно иммобилизовали нейтравидин на чипе GLC (BioRad) до уровня около 8000 резонансных единиц (RU). Затем осуществили иммобилизацию HER2 на покрытом нейтравидином чипе. Измерили взаимодействие DARPin и HER2 путем инъекции 100 мкл подвижного буфера (PBS, содержащего 0,005% Tween®), содержащего последовательные разведения DARPins в концентрации 50, 25, 12,5, 6,25 и 3,125 нМ (для измерения скорости ассоциации), а затем использовали поток подвижного буфера в течение от 10 минут до 3 часов с постоянной скоростью 100 мкл/мин (для измерения скорости диссоциации). Вычли сигналы (т.е. величины резонансных единиц (RU)) эталонной клетки без покрытия и эталонной инъекции (т.е. инъекции только подвижного буфера) из следов RU, полученных после инъекции HER2 (двойное сравнение с эталоном). На основании следов SRP, полученных путем измерения скорости ассоциации и диссоциации, могут быть определены скорости ассоциации и диссоциации при соответствующем взаимодействии DARPin и HER2.
Полученные результаты представлены в Таблице 1. На основании расчетных скоростей ассоциации и диссоциации вычислили константы диссоциации (Kd) с использованием стандартных процедур, известных специалистам в данной области техники.
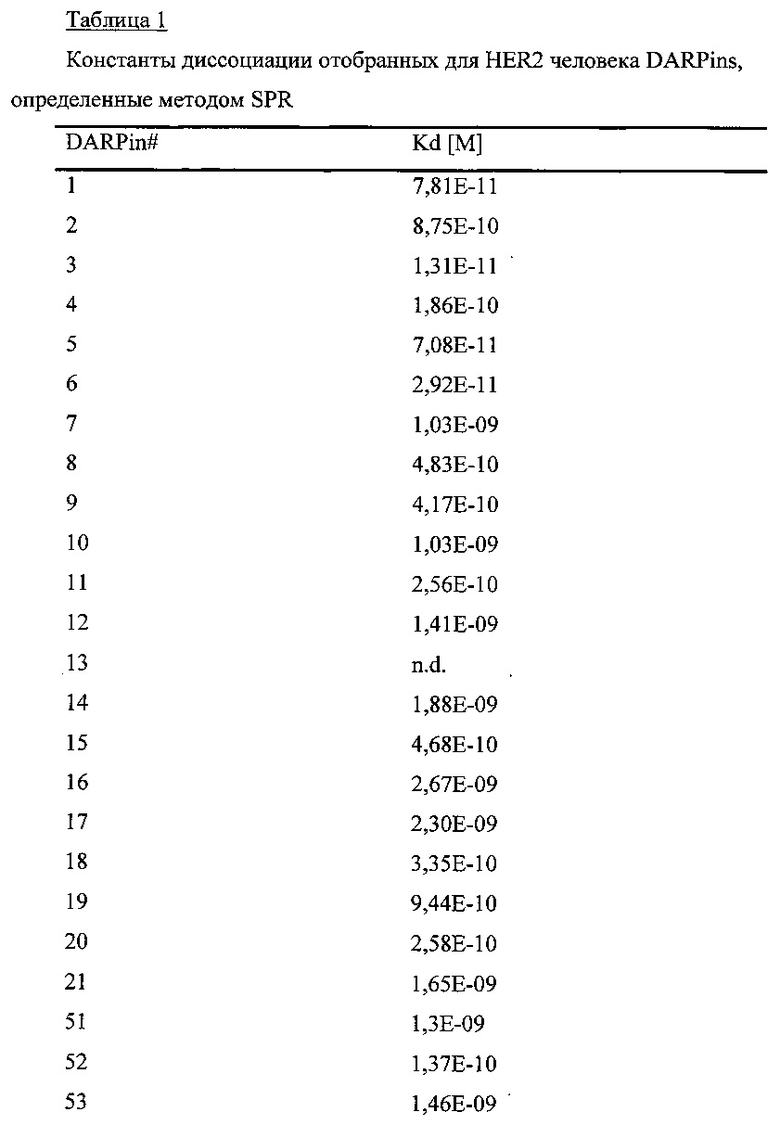

Пример 3
Картирование повторяющейся области, специфически связывающей внеклеточные эпитопы HER2
Проанализировали взаимодействие повторяющихся областей с внеклеточными областями HER2 стандартными методами, известными специалистам в данной области техники, такими как анализ четвертичной структуры комплексов путем рентгеновской кристаллографии или ЯМР-спектроскопии или картирование эпитопов путем использования аланинового мутагенеза потенциальных остатков взаимодействия или путем использования масс-спектрометрии и ковалентного мечения. Кроме того, провели различные конкурентные анализы, такие как методом конкурентного твердофазного иммуноферментного анализа (ИФА), известные специалистам в данной области техники, чтобы идентифицировать внеклеточные области, связываемые отобранным повторяющимся белком, или определить наличие у них эпитопов во внеклеточных областях HER2, перекрывающихся с другими связывающими белками, например, антителами, такими как трастузумаб или пертузумаб.
Внеклеточные области HER2 приобретены или получены, как описано у Jost и др. (в процитированном месте).
Исследовали конкуренцию интересующих очищенных связывающих HER2 DARPins методом анализа поверхностного плазмонного резонанса (SPR) с помощью системы ProteOn (BioRad) с использованием настройки, при которой S22-N530 и ErbB2 S22-E645 биотинилированного HER2 человека были иммобилизованы посредством нейтравидина, и была измерена конкуренция путем добавления первого моновалентного DARPin в состоянии насыщения (1 мкМ), а затем смеси первого и второго DARPin (по 100 нМ каждого) в соотношении 1:1. Если у второго DARPin наблюдалось связывание, несмотря на присутствие первого DARPin, считалось, что второй DARPin связывает другой эпитоп.
Например, по данным конкурентного ИФА (фиг. 1А и 1В) предполагается, что DARPin # 54 связывает область II Her2, a DARPin #51 связывает область I HER2. Ранее было продемонстрировано, что DARPin #18 связывает область IV HER2 (Jost и др., в процитированном месте). Предварительно инкубировали DARPins (20 нМ) с областью I, областью I-III или областью III-IV HER2 (в каждом случае при концентрации области 500 нМ) в PBS в течение 45 минут при комнатной температуре. Добавили смесь в 20 нМ непроцессированного Her2, нанесенного на планшет с F96 MaxiSorb (производства компании Nunc, номер по каталогу 442404). Для обнаружения специфически связывающих DARPins использовали моноклональное антитело мыши к RGS-His (Qiagen, номер по каталогу 34650) в качестве первичного антитело и меченное пероксидазой хрена антитело мыши (Pierce, номер по каталогу 31438) в качестве вторичного антитела. Для проведения ИФА, проиллюстрированного на фиг. 1В, заменили первичное антитело (антитело мыши к RGS-His) моноклональным антителом мыши к DARPin антитело.
Осуществили считывание данных на волне 450 нм. Осуществили все стадии инкубации в PBS (рН 7,4), содержащем 0,1% Tween 20® и 0,25% казеина при комнатной температуре в течение 2 часов в шейкере Heidolph Titramax 1000 со скоростью 450 об/мин за исключением покрытия лунок планшета, которое осуществили в течение ночи при 4°С с использованием PBS (рН 7,4).
Эти факты были подтверждены путем проточного цитометрического анализа (FACS) конкурирующего связывания этими DARPins клеток ВТ474 со сверхэкспрессией рекомбинантной области I, области I-II-III и области III-IV HER2. Предварительно инкубировали DARPins (100 нМ) с отдельными конструкциями Her2 (1 мкМ) при 25°С в течение 30 минут. На 20 минут поместили смесь на клетки (100000 клеток в 100 мкл) на льду. Контролировали связывание клеток DARPin с использованием меченного Alexa 647 антитела к Penta-His (Qiagen, номер по каталогу 35370). Путем анализа подтвердили связывание DARPin #51 области I HER2, связывание DARPin # 1 области II HER2 и связывание DARPin# 18 области IV HER2.
Также исследовали конкуренцию DARPin #1 с пертузумабом и DARPin #18 с трастузумабом путем проточного цитометрического анализа. С этой целью предварительно инкубировали клетки ВТ474, соответственно, с пертузумабом и трастузумабом (по 1 мкМ в каждом случае) до инкубации с соответствующим DARPin (1 мкМ). Контролировали связывание клеток DARPin с использованием меченного Alexa 647 антитела к Penta-His (Qiagen, номер по каталогу 35370) и связывание клеток пертузумабом или трастузумабом с использованием меченного Alexa 546 антитела к IgG человека (Invitrogen, номер по каталогу А-21089). Эксперименты показали, что ни один из DARPins не конкурирует с пертузумабом или трастузумабом за связывание HER2, который экспрессируют клетки ВТ474.
Это также наблюдалось при осуществлении ИФА (фиг. 1 С), когда предварительно инкубировали пертузумаб (нанесенный на планшет с F96 MaxiSorb (производства компании Nunc, номер по каталогу 442404) в концентрации 20 нМ) с 20 нМ Her2 (области I-IIII) до инкубации с соответствующими DARPins (20 нМ). Детектировали специфическое связывание DARPin комплекса Her2-пертузумаб с использованием моноклонального антитела мыши к RGS-His (Qiagen, номер по каталогу 34650) и меченного пероксидазой хрена антитела мыши (Pierce, номер по каталогу 31438) (предварительно перемешанных в течение 45 минут при комнатной температуре). Осуществили все стадии инкубации при комнатной температуре в течение 2 часов в шейкере Heidolph Titramax 1000 со скоростью 450 об/мин за исключением покрытия лунок планшета, которое осуществили в течение ночи при 4°С. В качестве блокирующего средства использовали PBS (рН 7,4), содержащий 0,1% Tween 20® и 0,25% казеина. Все исследованные N-концевые DARPins (DARPin # 7, DARPin # 52, DARPin # 53 и DARPin # 54) связывали Her2 в присутствии пертузумаба, что доказывает, что все они связывают не тот же самый эпитоп, который связывает антитело.
В целом, такие эксперименты показали, что моновалентные повторяющиеся области, кодируемые SEQ ID 62-68, 72 и 114-121, связывают область II HER2, моновалентные повторяющиеся области, кодируемые SEQ ID 69-71, 73, 112 и 113, связывают область IHER2, а моновалентные повторяющиеся области, кодируемые SEQ ID 74-82, связывают область IV HER2. Ни одна из моновалентных повторяющихся областей, связывающих область II HER2 (SEQ ID 62-68, 72 и 114-121) не конкурирует с пертузумабом за связывание HER2. Из моновалентных повторяющихся областей, связывающих область IV HER2, повторяющиеся области, кодируемые SEQ ID 77, 78 и 82 конкурируют с трастузумабом за связывание HER2, а повторяющиеся области, кодируемые SEC ID 74-76 и 79-81, не конкурируют с трастузумабом.
Пример 4
Блокирование роста опухолевых клеток со сверхэкспрессией HER2 бипаратопными связывающими Her2 DARPins
Исследовали ингибирование пролиферации клеток ВТ474 моновалентными DARPins, смесями DARPins и бипаратопными связывающими Her2 DARPins. На фиг. 2 показано, что моновалентные DARPins и смеси моновалентных DARPins не способны блокировать пролиферацию клеток ВТ474. В отличие от этого, поднабор бипаратопных DARPins индуцирует ингибирование пролиферации (фиг. 2 и Таблица 2). Интересно, что DARPins, связывающие область IV HER2, должны находиться на С-конце молекулы (фиг. 2). Ингибирующие пролиферацию бипаратопные DARPins были получены из множества сочетаний моновалентных DARPins в бипаратопном формате. Тем не менее, не все сочетания способны на 90-100% блокировать пролиферацию клеток ВТ474 (фиг. 3), что позволяет ранжировать некоторые сочетания DARPin. Эти данные показывают, что ключевым фактором достижения связывающей активности является нацеливание в бипаратопном формате на особый поднабор некоторых эпитопов в HER2. Индуцируемой трастузумабом интернализации и распада HER2 недостаточно, чтобы индуцировать мощное ингибирование пролиферации опухолевых клеток (фиг. 3 и 5). Как DARPin # 41, так и DARPin # 43 индуцируют распад HER2 аналогично трастузумабу, но только DARPins, такие как DARPin #41, ингибируют пролиферацию опухолевых клеток.
Были проведены эксперименты, описанные в разделе "Эксперименты", как описано разделе "Методы". Полученные результаты приведены в Таблице 2. На основании титрационных кривых, полученных, как описано, вычислили показатели IC50 с использованием стандартных процедур, известных специалистам в данной области техники. На фиг. 2 и 3 приведены примеры титрационных кривых для DARPin #41.
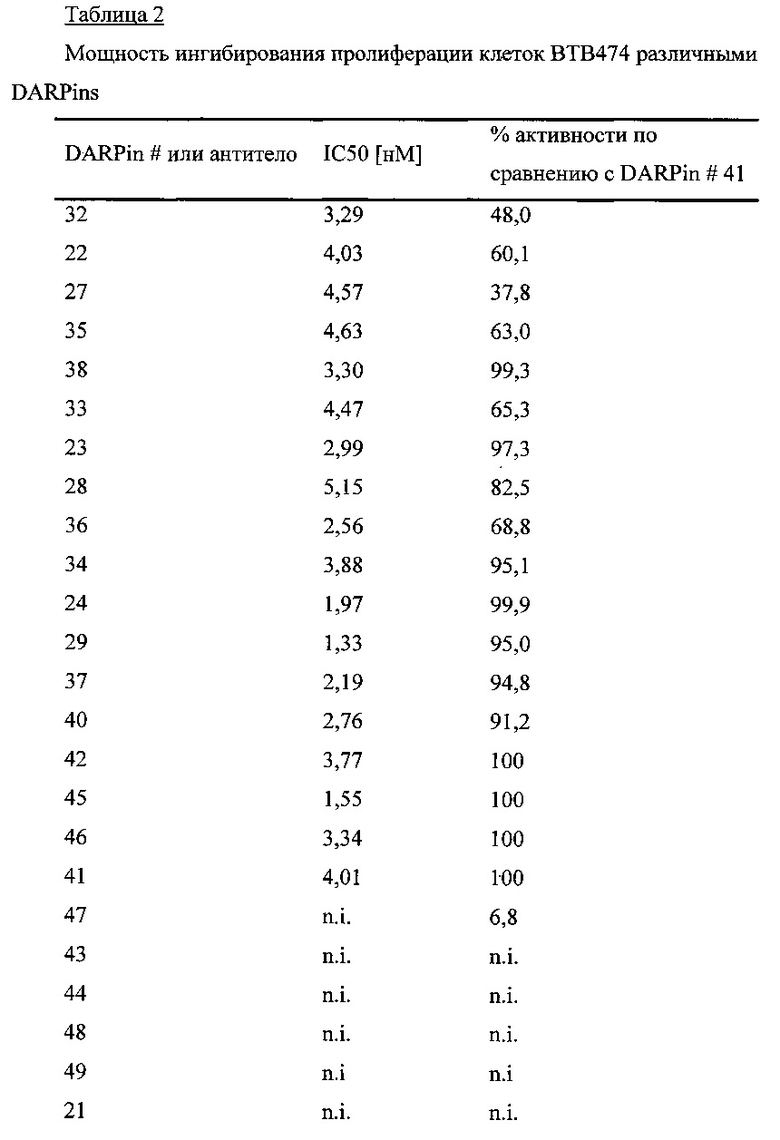

Пример 5
Ингибирование нацеленными на Her2 бипаратопными DARPins пролиферации и индуцирования апоптоза клеток различных линий со сверхэкспрессией Her2
Исследовали активность бипаратопного DARPin #41. DARPin ингибировал пролиферацию клеток со сверхэкспрессией Her2 в интервале от IHC 3+ до 1+ и не ингибировал пролиферацию клеток, экспрессирующих HER2 дикого типа (фиг. 4; Таблица 3). Кроме того, DARPin устойчиво индуцирует апоптоз клеток перечисленных линий в течение 24 часов инкубации (фиг. 5, Таблица 3).
Были проведены эксперименты, как описано в разделе "Методы". Полученные результаты приведены в Таблице 3. На основании титрационных кривых, полученных, как описано, вычислили показатели IC50 и ЕС50 с использованием стандартных процедур, известных специалистам в данной области техники. На фиг. 4 и 5 приведены примеры титрационных кривых для DARPin #41 при использовании клеток трех различных линий. Показатели IC50 и ЕС50 находятся в интервале 0,2-10 нМ в зависимости от используемого DARPin и линии клеток. Например, продемонстрировано, что DARPin # 41, # 45 и # 46 индуцируют апоптоз клеток ВТ474, MDA-MB175 и NCI-N87 (Таблица 3). При использовании других бипаратопных связывающих белков согласно изобретению были получены сходные результаты.
Пример 6
Ингибирование бипаратопными нацеленными на Her2 DARPins пролиферации и индуцирования апоптоза клеток ВТ474 в отличие от существующих стандартных терапий
Сравнили активность бипаратопного DARPin # 41 и трастузумаба и пертузумаба в качестве лекарств, одобренных для лечения HER2-позитивного рака молочной железы. DARPin эффективно ингибирует пролиферация и индуцирует апоптоз в отличие от трастузумаба, пертузумаба или сочетания трастузумаба и пертузумаба (фиг. 6).
Были проведены эксперименты, как описано в разделе "Методы". Полученные результаты приведены в Таблице 6. На основании титрационных кривых, полученных, как описано, вычислили показатели IC50 и ЕС50 (Таблица 3) с использованием стандартных процедур, известных специалистам в данной области техники. При использовании других бипаратопных связывающих белков согласно изобретению были получены сходные результаты.
Пример 7
Генерирование DARPin различных форматов
В качестве одного из примеров сравнили активность различных форматов бипаратопного DARPin # 41 и DARPin #41 при ингибировании пролиферации клеток ВТ474 (фиг. 7, Таблица 2). Пэгилирование или слияние связывающего сывороточный альбумин человека DARPin (DARPin # 41, # 63, # 64, # 65) с Ν- или С-концом не повлияло на его активность (фиг. 7А). Кроме того, на активность не повлияло варьирование линкеров между фрагментами DARPin (фиг. 7В). Показатели IC50 находились в интервале 1,5-5,5 нМ. Соответствующие результаты были получены при использовании других форматов бипаратопных DARPins #41, # 66, # 6, 7# 68. В целом, это ясно свидетельствует о том, что бипаратопные DARPins могут быть модифицированы (способами, известными специалистам в данной области техники, такими как пэгилирование или слияние со связывающими сывороточный альбумин областями) с целью увеличения их периода полураспада in vivo без потери активности. Кроме того, эти эксперименты предполагают, что в бипаратопной конструкции линкер между двумя повторяющимися областями, связывающими HER2, может варьировать в интервале, по меньшей мере, от 2 до 24 аминокислот без значительно влияния на эффективность бипаратопной конструкции.
Пример 8
Картирование взаимодействия DARPin/Her2
Дополнительно исследовали взаимодействие бипаратопных DARPins согласно изобретению с эктообластью HER2 путем химического перекрестного сшивания комплекса, образуемого этими двумя молекулами в растворе (т.е. в PBS с рН 7,4), а затем расщепления комплекса с помощью протеазы и анализа полученных пептидов путем масс-спектроскопии. В таком эксперименте области DARPin могут ковалентно перекрестно сшиваться только с областями HER2 в непосредственной близости от них. Обнаружение путем масс-спектроскопии пептидов DARPin, ковалентно перекрестно сшитых с соответствующим пептидом HER2, указывает на то, что эти пептиды находятся в непосредственной близости друг от друга в комплексе HER2/DRAPin. Такие методы анализ близости хорошо известны специалистам в данной области техники (например, Birch, С.и др., Anal. Chem., 82, 172-179, 2010) и в качестве услуги предлагаются различными компаниями (например, CovalX AG, Цюрих, Швейцария).
Например, путем таких экспериментов было обнаружено, что бипаратопный DARPin #41, который связывает область II и область IV HER2, способен образовывать с HER2 комплекс 1:1. К удивлению, наблюдались ковалентные поперечные связи между С-концевой повторяющейся областью (связывающей область IV HER2) и областью I HER2, что говорит о непосредственной близости этой повторяющейся области и области I HER2 в комплексе, несмотря даже на то, что она связывает область IV. Такие поперечные связи, вероятно, не существовали бы при конформации HER2, описанной в технике (например, у Bublil и Yarden, в процитированном месте). Важно, что, когда эктообласть HER2 была исследована в комплексе с этой С-концевой повторяющейся областью, связывающей только область IV, такие поперечные связи с областью I HER2 не наблюдались, что говорит о том, что в случае комплекса, образованного HER2 и мономерной повторяющейся областью, связывающей область IV, отсутствует близость этой повторяющейся области и области I. Соответственно, расположение трехмерных областей HER2 в комплексе с бипаратопным связывающим белком согласно изобретению и в комплексе с отдельной повторяющейся областью, связывающей область IV HER2, должно различаться.
Интересно, что, известные структуры эктообласти HER2 не допускают одновременного связывания обеими повторяющимися областями бипаратопного связывающего белка согласно изобретению с одной и той же молекулы HER2 при использовании коротких линкеров от 2 до 24 аминокислот между двумя повторяющимися областями. Это говорит о том, что HER2 может иметь еще неизвестную конформацию, допускающую одновременное связывание обеими повторяющимися областями.
В целом, такие эксперименты демонстрируют, что бипаратопные связывающие белки согласно изобретению могут быть способны к внутримолекулярному взаимодействию с эктообластью HER2, и что тем самым они фиксируют эктообласть HER2 в новой конформации, не известной из техники, а именно, придают области I и области IV пространственное расположение, которое допускает образование наблюдавшихся поперечных связей между повторяющейся областью (связывающей область IV HER2) и областью I. Соответственно, бипаратопный связывающий белок согласно изобретению, по-видимому, стабилизирует эту новую конформацию HER2 путем одновременного связывания области II и области IV HER2 внутримолекулярным путем.
SEQUENCE LISTING
<110> Molecular Partners
<120> Binding proteins comprising at least two binding domains against
HER2
<130> MD41080
<160> 141
<170> PatentIn version 3.5
<210> 1
<211> 126
<212> PRT
<213> Artificial
<220>
<223> Capping module
<400> 1
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Lys Asp Gly Tyr Thr Pro Leu His Leu Ala Ala Arg Glu Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Lys Asp Gly Tyr Thr Pro Leu His Leu Ala Ala Arg Glu
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Asp
100 105 110
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
115 120 125
<210> 2
<211> 32
<212> PRT
<213> Artificial
<220>
<223> Capping module
<400> 2
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 3
<211> 32
<212> PRT
<213> Artificial
<220>
<223> Capping module
<400> 3
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 4
<211> 28
<212> PRT
<213> Artificial
<220>
<223> Capping module
<400> 4
Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Asp Asn Gly
1 5 10 15
Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
20 25
<210> 5
<211> 28
<212> PRT
<213> Artificial
<220>
<223> Capping module
<400> 5
Gln Asp Lys Ser Gly Lys Thr Pro Ala Asp Leu Ala Ala Asp Ala Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 6
<211> 10
<212> PRT
<213> Artificial
<220>
<223> His-tag
<400> 6
Met Arg Gly Ser His His His His His His
1 5 10
<210> 7
<211> 2
<212> PRT
<213> Artificial
<220>
<223> GS-linker
<400> 7
Gly Ser
1
<210> 8
<211> 5
<212> PRT
<213> Artificial
<220>
<223> GS-linker
<400> 8
Gly Gly Gly Gly Ser
1 5
<210> 9
<211> 10
<212> PRT
<213> Artificial
<220>
<223> GS-linker
<400> 9
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
1 5 10
<210> 10
<211> 20
<212> PRT
<213> Artificial
<220>
<223> GS-linker
<400> 10
Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
1 5 10 15
Gly Gly Gly Ser
20
<210> 11
<211> 5
<212> PRT
<213> Artificial
<220>
<223> PT-linker
<400> 11
Pro Thr Pro Thr Pro
1 5
<210> 12
<211> 20
<212> PRT
<213> Artificial
<220>
<223> PT-linker
<400> 12
Pro Thr Pro Thr Pro Thr Thr Pro Thr Pro Thr Pro Thr Thr Pro Thr
1 5 10 15
Pro Thr Pro Thr
20
<210> 13
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 13
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Ser Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 14
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 14
Gly Ser Asp Leu Gly Val Asn Leu Leu Trp Ala Ala Thr Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 15
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 15
Lys Asp Phe Gln Ser Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 16
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 16
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Ser Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 17
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 17
Lys Asp Phe Glu Gly Val Thr Pro Leu His Leu Ala Ala Gln Trp Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 18
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 18
Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 19
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 19
Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala Trp Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 20
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 20
Gly Ser Asp Leu Gly Trp Lys Leu Leu Trp Ala Ala Ala His Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 21
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 21
Lys Asp Trp Glu Gly Thr Thr Pro Leu His Leu Ala Ala His Thr Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 22
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 22
Lys Asp Thr Val Gly Thr Thr Pro Leu His Tyr Ala Ala Glu Asp Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 23
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 23
Lys Asp Glu Tyr Gly Phe Thr Pro Leu His Leu Ala Ala Gln Phe Asp
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 24
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 24
Gln Asp Trp Val Gly Gln Thr Pro Ala Asp Leu Ala Ala Ala Trp Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 25
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 25
Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 26
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 26
Gly Ser Asp Leu Gly His Lys Leu Leu Glu Ala Ala Val Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 27
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 27
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ser His Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 28
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 28
Lys Asp Trp Tyr Gly Lys Thr Pro Leu His Phe Ala Ala Gly Leu Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 29
<211> 32
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 29
Lys Asp Phe Phe Gly Ile Thr Pro Leu His Gln Ala Ala Trp Gly His
1 5 10 15
Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 30
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 30
Lys Asp Asp Phe Gly Thr Thr Pro Leu His Ala Ala Ala Asp Tyr Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 31
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 31
Lys Asp Glu Asp Gly Gln Thr Pro Leu His Leu Ala Ala Ala Tyr Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 32
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 32
Lys Glu Glu Asp Gly Thr Thr Pro Leu His Leu Ala Ala Thr His Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 33
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 33
Gln Asp Tyr Thr Gly His Thr Pro Ala Asp Leu Ala Ala Val Tyr Gly
1 5 10 15
His Glu Asp Ile Ala Ala Val Leu Gln Lys Leu Asn
20 25
<210> 34
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 34
Gln Asp Asn Asp Gly Phe Thr Pro Ala Asp Leu Ala Ala Asp Ser Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 35
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 35
Gln Asp Trp Tyr Gly Thr Thr Pro Ala Asp Leu Ala Ala Trp Trp Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 36
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 36
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ser Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 37
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 37
Lys Asp Phe Glu Gly Ile Thr Pro Leu His Ala Ala Ala Arg Ser Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 38
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 38
Lys Asp Val Glu Gly Trp Thr Pro Leu His Tyr Ala Ala Ser Ser Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 39
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 39
Gln Asp Asn His Gly Ala Thr Pro Ala Asp Leu Ala Ala Gln Trp Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 40
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 40
Gly Ser Asp Leu Gly Asn Lys Leu Leu Ile Ala Ala Ser Val Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 41
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 41
Lys Asp Glu Thr Gly Trp Thr Pro Leu His Leu Ala Ala Ala Trp Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 42
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 42
Lys Asp Val Lys Gly Gln Thr Pro Leu His Leu Ala Ala Ala Tyr Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 43
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 43
Gln Asp Asn Asp Gly Tyr Thr Pro Ala Asp Leu Ala Ala Arg Tyr Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 44
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 44
Gly Ser Asp Leu Gly Lys Lys Leu Leu Asn Ala Ala Val Cys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Val Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 45
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 45
Gly Ser Asp Leu Gly Thr Lys Leu Leu Asp Ala Ala Thr Tyr Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 46
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 46
Lys Asp Trp Arg Gly Phe Thr Pro Leu His Tyr Ala Ala Tyr Leu Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 47
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 47
Lys Asp Thr Ile Gly His Thr Pro Leu His Arg Ala Ala Phe Val Gly
1 5 10 15
Gln Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 48
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 48
Gln Asp Thr Ala Gly Tyr Thr Pro Ala Asp Leu Ala Ala Trp Thr Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 49
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 49
Gln Asp Asp Tyr Gly Trp Thr Pro Ala Asp Leu Ala Ala Asn Ser Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 50
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (Nr)
<400> 50
Gly Ser Asp Leu Gly Ile Lys Leu Leu Gln Ala Ala Asn Leu Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Thr Gly Ala Asp Val Asn Ala
20 25 30
<210> 51
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 51
Lys Asp Ser Ile Gly Gln Thr Pro Leu His Trp Ala Ala Arg Arg Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 52
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (M1.1b)
<400> 52
Lys Asp Glu Tyr Gly Val Thr Pro Leu His Leu Ala Ala Ser Leu Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 53
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (Cr)
<400> 53
Gln Asp Thr Ala Gly Gln Thr Pro Ala Asp Leu Ala Ala Asp Asp Gly
1 5 10 15
His Glu Asp Ile Ala Val Val Leu Gln Lys Leu Asn
20 25
<210> 54
<211> 32
<212> PRT
<213> Artificial
<220>
<223> N-Cap module (old)
<400> 54
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 55
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (old)
<400> 55
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 56
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR module (old)
<400> 56
Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 57
<211> 28
<212> PRT
<213> Artificial
<220>
<223> C-Cap module (old)
<400> 57
Gln Asp Lys Ser Gly Lys Thr Pro Ala Asp Leu Ala Ala Gly Ala Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 58
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR sequence motif
<220>
<221> misc_feature
<222> (1)..(1)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (3)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (14)..(15)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (27)..(27)
<223> Xaa can be any naturally occurring amino acid
<400> 58
Xaa Asp Xaa Xaa Gly Xaa Thr Pro Leu His Leu Ala Ala Xaa Xaa Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Xaa Gly Ala Asp Val Asn
20 25 30
Ala
<210> 59
<211> 33
<212> PRT
<213> Artificial
<220>
<223> AR sequence motif
<220>
<221> misc_feature
<222> (3)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (11)..(11)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (14)..(15)
<223> Xaa can be any naturally occurring amino acid
<400> 59
Lys Asp Xaa Xaa Gly Xaa Thr Pro Leu His Xaa Ala Ala Xaa Xaa Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 60
<211> 32
<212> PRT
<213> Artificial
<220>
<223> AR sequence motif
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (10)..(10)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (13)..(14)
<223> Xaa can be any naturally occurring amino acid
<400> 60
Gly Ser Asp Leu Gly Xaa Lys Leu Leu Xaa Ala Ala Xaa Xaa Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 61
<211> 28
<212> PRT
<213> Artificial
<220>
<223> AR sequence motif
<220>
<221> misc_feature
<222> (3)..(4)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa can be any naturally occurring amino acid
<220>
<221> misc_feature
<222> (14)..(15)
<223> Xaa can be any naturally occurring amino acid
<400> 61
Gln Asp Xaa Xaa Gly Xaa Thr Pro Ala Asp Leu Ala Ala Xaa Xaa Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 62
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 62
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Val Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 63
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 63
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 64
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 64
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Ser Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Ser Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 65
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 65
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Lys Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 66
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 66
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Asn Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 67
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 67
Gly Ser Asp Leu Gly Val Asn Leu Leu Trp Ala Ala Thr Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Val Thr Pro Leu His Leu Ala Ala Gln Trp Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 68
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 68
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Tyr Thr Pro Leu His Val Ala Ala Tyr Asp Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ser Gln Gly Arg Thr Pro Leu His Glu Ala Ala Tyr Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asp Ala Gly Glu Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 69
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 69
Gly Ser Asp Leu Gly Ile Lys Leu Leu Trp Ala Ala Ala His Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Asp Ala Asp Val Asn Ala
20 25 30
Lys Asp Trp Tyr Gly Thr Thr Pro Leu His Ile Ala Ala Val Ala Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Asp Phe Gly Thr Thr Pro Leu His Leu Ala Ala Tyr His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Trp Gln Gly Gln Thr Pro Ala Asp Leu Ala Ala Gln
100 105 110
Asp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 70
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 70
Gly Ser Asp Leu Gly His Lys Leu Leu Glu Ala Ala Val Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Trp Tyr Gly Lys Thr Pro Leu His Phe Ala Ala Gly Leu Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Glu Asp Gly Gln Thr Pro Leu His Leu Ala Ala Ala Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn Asp Gly Phe Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Ser Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 71
<211> 125
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 71
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ser His Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Phe Gly Ile Thr Pro Leu His Gln Ala Ala Trp Gly His
35 40 45
Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
50 55 60
Lys Glu Glu Asp Gly Thr Thr Pro Leu His Leu Ala Ala Thr His Gly
65 70 75 80
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
85 90 95
Ala Gln Asp Trp Tyr Gly Thr Thr Pro Ala Asp Leu Ala Ala Trp Trp
100 105 110
Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 72
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 72
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ser Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Ile Thr Pro Leu His Ala Ala Ala Arg Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Glu Gly Trp Thr Pro Leu His Tyr Ala Ala Ser Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn His Gly Ala Thr Pro Ala Asp Leu Ala Ala Gln
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 73
<211> 159
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 73
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Tyr Gly Ile Thr Pro Leu His Leu Ala Ala Ala Tyr Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys His Gly Ala Asp Val Asn
50 55 60
Ala His Asp Trp Asn Gly Trp Thr Pro Leu His Leu Ala Ala Lys Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys His Gly Ala Asp Val
85 90 95
Asn Ala Ile Asp Asn Ala Gly Lys Thr Pro Leu His Leu Ala Ala Ala
100 105 110
His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Tyr Gly Ala Asp
115 120 125
Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile
130 135 140
Asp Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
145 150 155
<210> 74
<211> 93
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 74
Gly Ser Asp Leu Gly Lys Lys Leu Leu Asn Ala Ala Val Cys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Val Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Trp Arg Gly Phe Thr Pro Leu His Tyr Ala Ala Tyr Leu Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Gln Asp Thr Ala Gly Tyr Thr Pro Ala Asp Leu Ala Ala Trp Thr
65 70 75 80
Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
85 90
<210> 75
<211> 93
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 75
Gly Ser Asp Leu Gly Ala Lys Leu Leu Ile Ala Ala Thr Val Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Thr Ile Gly His Thr Pro Leu His Arg Ala Ala Phe Val Gly
35 40 45
Gln Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Gln Asp Asp Tyr Gly Trp Thr Pro Ala Asp Leu Ala Ala Asn Ser
65 70 75 80
Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
85 90
<210> 76
<211> 93
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 76
Gly Ser Asp Leu Gly Ala Lys Leu Leu Val Ala Ala Thr Ser Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Arg Ile Gly Phe Thr Pro Leu His Arg Ala Ala Phe Val Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Gln Asp Asp Phe Gly His Thr Pro Ala Asp Leu Ala Ala Ser Leu
65 70 75 80
Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
85 90
<210> 77
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 77
Gly Ser Asp Leu Gly Ile Lys Leu Leu Gln Ala Ala Asn Leu Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Ser Ile Gly Gln Thr Pro Leu His Trp Ala Ala Arg Arg Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Glu Tyr Gly Val Thr Pro Leu His Leu Ala Ala Ser Leu
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Ser Gly Glu Thr Pro Ala Asp Leu Ala Ala Leu
100 105 110
His Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 78
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 78
Gly Ser Asp Leu Gly Leu Lys Leu Leu Gln Ala Ala Asn Leu Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Ser Ile Gly Gln Thr Pro Leu His Trp Ala Ala Arg Arg Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Glu Tyr Gly Val Thr Pro Leu His Leu Ala Ala Ser Leu
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Thr Ala Gly Gln Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Asp Gly His Glu Asp Ile Ala Val Val Leu Gln Lys Leu Asn
115 120 125
<210> 79
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 79
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
50 55 60
Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile
65 70 75 80
Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly
100 105 110
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
115 120 125
<210> 80
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 80
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
50 55 60
Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile
65 70 75 80
Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ser Gly Lys Thr Pro Ala Asp Leu Ala Ala Gly
100 105 110
Ala Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 81
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 81
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile
65 70 75 80
Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Phe Gly Lys Thr Pro Ala Asp Ile Ala Ala Gly
100 105 110
Ala Gly Asn Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 82
<211> 159
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain)
<400> 82
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Thr Asp Ile His Gly His Thr Pro Leu His Leu Ala Ala Ala Met Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
50 55 60
Ala Asn Asp Trp Arg Gly Phe Thr Pro Leu His Leu Ala Ala Leu Asn
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val
85 90 95
Asn Ala Thr Asp Thr Ala Gly Asn Thr Pro Leu His Leu Ala Ala Trp
100 105 110
Phe Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp
115 120 125
Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile
130 135 140
Asp Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
145 150 155
<210> 83
<211> 270
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 83
Gly Ser Asp Leu Gly Asp Lys Leu Leu Gln Ser Asp Leu Gly Ile Lys
1 5 10 15
Leu Leu Phe Ala Ala Ala Lys Ser Gln Asp Asp Glu Val Arg Ile Leu
20 25 30
Leu Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Ser Val Thr
35 40 45
Pro Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val
50 55 60
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp
65 70 75 80
Thr Pro Leu His Leu Ala Ala Gln His Gly His Leu Glu Ile Val Glu
85 90 95
Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly
100 105 110
Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala
115 120 125
Glu Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Arg Ser Asp Leu Gly Ile Lys Leu Leu Gln Ala Ala Asn Leu Gly Gln
145 150 155 160
Asp Asp Glu Val Arg Ile Leu Leu Ala Thr Gly Ala Asp Val Asn Ala
165 170 175
Lys Asp Ser Ile Gly Gln Thr Pro Leu His Trp Ala Ala Arg Arg Gly
180 185 190
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
195 200 205
Ala Lys Asp Glu Tyr Gly Val Thr Pro Leu His Leu Ala Ala Ser Leu
210 215 220
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
225 230 235 240
Asn Ala Gln Asp Glu Ser Gly Glu Thr Pro Ala Asp Leu Ala Ala Leu
245 250 255
His Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
260 265 270
<210> 84
<211> 237
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 84
Gly Ser Asp Leu Gly Asp Lys Leu Leu Gln Ser Asp Leu Gly Ile Lys
1 5 10 15
Leu Leu Phe Ala Ala Ala Lys Ser Gln Asp Asp Glu Val Arg Ile Leu
20 25 30
Leu Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Ser Val Thr
35 40 45
Pro Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val
50 55 60
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp
65 70 75 80
Thr Pro Leu His Leu Ala Ala Gln His Gly His Leu Glu Ile Val Glu
85 90 95
Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly
100 105 110
Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala
115 120 125
Glu Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Arg Ser Asp Leu Gly Ala Lys Leu Leu Val Ala Ala Thr Ser Gly Gln
145 150 155 160
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
165 170 175
Lys Asp Arg Ile Gly Phe Thr Pro Leu His Arg Ala Ala Phe Val Gly
180 185 190
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
195 200 205
Ala Gln Asp Asp Phe Gly His Thr Pro Ala Asp Leu Ala Ala Ser Leu
210 215 220
Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
225 230 235
<210> 85
<211> 270
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 85
Gly Ser Asp Leu Gly Asp Lys Leu Leu Gln Ser Asp Leu Gly Ile Lys
1 5 10 15
Leu Leu Phe Ala Ala Ala Lys Ser Gln Asp Asp Glu Val Arg Ile Leu
20 25 30
Leu Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Ser Val Thr
35 40 45
Pro Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val
50 55 60
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp
65 70 75 80
Thr Pro Leu His Leu Ala Ala Gln His Gly His Leu Glu Ile Val Glu
85 90 95
Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly
100 105 110
Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala
115 120 125
Glu Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Arg Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
145 150 155 160
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
165 170 175
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
180 185 190
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
195 200 205
Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile
210 215 220
Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val
225 230 235 240
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly
245 250 255
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
260 265 270
<210> 86
<211> 272
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 86
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Ala Ala Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala
145 150 155 160
Gly Gln Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val
165 170 175
Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala
180 185 190
His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp
195 200 205
Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala
210 215 220
Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys Ala Gly Ala
225 230 235 240
Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Pro Ala Asp Ile Ala
245 250 255
Ala Gly Ala Gly Asn Glu Asp Ile Ala Glu Val Leu Gln Lys Ala Ala
260 265 270
<210> 87
<211> 274
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 87
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Ala Ala Gly Ser
115 120 125
Pro Thr Pro Thr Pro Thr Thr Pro Thr Pro Thr Pro Thr Thr Pro Thr
130 135 140
Pro Thr Pro Thr Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala
145 150 155 160
Arg Ala Gly Gln Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala
165 170 175
Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala
180 185 190
Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly
195 200 205
Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu
210 215 220
Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys Ala
225 230 235 240
Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Pro Ala Asp
245 250 255
Ile Ala Ala Gly Ala Gly Asn Glu Asp Ile Ala Glu Val Leu Gln Lys
260 265 270
Ala Ala
<210> 88
<211> 261
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 88
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Asn Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ile Lys Leu Leu
130 135 140
Gln Ala Ala Asn Leu Gly Gln Asp Asp Glu Val Arg Ile Leu Leu Ala
145 150 155 160
Thr Gly Ala Asp Val Asn Ala Lys Asp Ser Ile Gly Gln Thr Pro Leu
165 170 175
His Trp Ala Ala Arg Arg Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Val Thr Pro
195 200 205
Leu His Leu Ala Ala Ser Leu Gly His Leu Glu Ile Val Glu Val Leu
210 215 220
Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Ser Gly Glu Thr
225 230 235 240
Pro Ala Asp Leu Ala Ala Leu His Gly His Glu Asp Ile Ala Glu Val
245 250 255
Leu Gln Lys Leu Asn
260
<210> 89
<211> 228
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 89
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Asn Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ala Lys Leu Leu
130 135 140
Val Ala Ala Thr Ser Gly Gln Asp Asp Glu Val Arg Ile Leu Leu Ala
145 150 155 160
Ala Gly Ala Asp Val Asn Ala Lys Asp Arg Ile Gly Phe Thr Pro Leu
165 170 175
His Arg Ala Ala Phe Val Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Asp Phe Gly His Thr Pro
195 200 205
Ala Asp Leu Ala Ala Ser Leu Gly His Glu Asp Ile Ala Glu Val Leu
210 215 220
Gln Lys Leu Asn
225
<210> 90
<211> 261
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 90
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Asn Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Lys Lys Leu Leu
130 135 140
Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met Ala
145 150 155 160
Asn Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu
165 170 175
Tyr Leu Ala Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Asn Gly Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro
195 200 205
Leu His Leu Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu
210 215 220
Leu Lys His Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr
225 230 235 240
Ala Phe Asp Ile Ser Ile Gly Asn Gly Asn Glu Asp Leu Ala Glu Ile
245 250 255
Leu Gln Lys Leu Asn
260
<210> 91
<211> 272
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 91
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Asn Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Ala Ala Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly
130 135 140
Gly Ser Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala
145 150 155 160
Gly Gln Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val
165 170 175
Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala
180 185 190
His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp
195 200 205
Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala
210 215 220
Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys Ala Gly Ala
225 230 235 240
Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Pro Ala Asp Ile Ala
245 250 255
Ala Gly Ala Gly Asn Glu Asp Ile Ala Glu Val Leu Gln Lys Ala Ala
260 265 270
<210> 92
<211> 274
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 92
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Asn Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Ala Ala Gly Ser
115 120 125
Pro Thr Pro Thr Pro Thr Thr Pro Thr Pro Thr Pro Thr Thr Pro Thr
130 135 140
Pro Thr Pro Thr Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala
145 150 155 160
Arg Ala Gly Gln Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala
165 170 175
Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala
180 185 190
Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly
195 200 205
Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu
210 215 220
Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys Ala
225 230 235 240
Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Pro Ala Asp
245 250 255
Ile Ala Ala Gly Ala Gly Asn Glu Asp Ile Ala Glu Val Leu Gln Lys
260 265 270
Ala Ala
<210> 93
<211> 261
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 93
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ser Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Ile Thr Pro Leu His Ala Ala Ala Arg Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Glu Gly Trp Thr Pro Leu His Tyr Ala Ala Ser Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn His Gly Ala Thr Pro Ala Asp Leu Ala Ala Gln
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ile Lys Leu Leu
130 135 140
Gln Ala Ala Asn Leu Gly Gln Asp Asp Glu Val Arg Ile Leu Leu Ala
145 150 155 160
Thr Gly Ala Asp Val Asn Ala Lys Asp Ser Ile Gly Gln Thr Pro Leu
165 170 175
His Trp Ala Ala Arg Arg Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Val Thr Pro
195 200 205
Leu His Leu Ala Ala Ser Leu Gly His Leu Glu Ile Val Glu Val Leu
210 215 220
Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Ser Gly Glu Thr
225 230 235 240
Pro Ala Asp Leu Ala Ala Leu His Gly His Glu Asp Ile Ala Glu Val
245 250 255
Leu Gln Lys Leu Asn
260
<210> 94
<211> 228
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 94
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ser Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Ile Thr Pro Leu His Ala Ala Ala Arg Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Glu Gly Trp Thr Pro Leu His Tyr Ala Ala Ser Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn His Gly Ala Thr Pro Ala Asp Leu Ala Ala Gln
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ala Lys Leu Leu
130 135 140
Val Ala Ala Thr Ser Gly Gln Asp Asp Glu Val Arg Ile Leu Leu Ala
145 150 155 160
Ala Gly Ala Asp Val Asn Ala Lys Asp Arg Ile Gly Phe Thr Pro Leu
165 170 175
His Arg Ala Ala Phe Val Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Asp Phe Gly His Thr Pro
195 200 205
Ala Asp Leu Ala Ala Ser Leu Gly His Glu Asp Ile Ala Glu Val Leu
210 215 220
Gln Lys Leu Asn
225
<210> 95
<211> 261
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 95
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ser Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Ile Thr Pro Leu His Ala Ala Ala Arg Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Glu Gly Trp Thr Pro Leu His Tyr Ala Ala Ser Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn His Gly Ala Thr Pro Ala Asp Leu Ala Ala Gln
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Lys Lys Leu Leu
130 135 140
Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met Ala
145 150 155 160
Asn Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu
165 170 175
Tyr Leu Ala Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Asn Gly Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro
195 200 205
Leu His Leu Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu
210 215 220
Leu Lys His Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr
225 230 235 240
Ala Phe Asp Ile Ser Ile Gly Asn Gly Asn Glu Asp Leu Ala Glu Ile
245 250 255
Leu Gln Lys Leu Asn
260
<210> 96
<211> 261
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 96
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Tyr Thr Pro Leu His Val Ala Ala Tyr Asp Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ser Gln Gly Arg Thr Pro Leu His Glu Ala Ala Tyr Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asp Ala Gly Glu Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ile Lys Leu Leu
130 135 140
Gln Ala Ala Asn Leu Gly Gln Asp Asp Glu Val Arg Ile Leu Leu Ala
145 150 155 160
Thr Gly Ala Asp Val Asn Ala Lys Asp Ser Ile Gly Gln Thr Pro Leu
165 170 175
His Trp Ala Ala Arg Arg Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Val Thr Pro
195 200 205
Leu His Leu Ala Ala Ser Leu Gly His Leu Glu Ile Val Glu Val Leu
210 215 220
Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Ser Gly Glu Thr
225 230 235 240
Pro Ala Asp Leu Ala Ala Leu His Gly His Glu Asp Ile Ala Glu Val
245 250 255
Leu Gln Lys Leu Asn
260
<210> 97
<211> 228
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 97
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Tyr Thr Pro Leu His Val Ala Ala Tyr Asp Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ser Gln Gly Arg Thr Pro Leu His Glu Ala Ala Tyr Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asp Ala Gly Glu Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ala Lys Leu Leu
130 135 140
Val Ala Ala Thr Ser Gly Gln Asp Asp Glu Val Arg Ile Leu Leu Ala
145 150 155 160
Ala Gly Ala Asp Val Asn Ala Lys Asp Arg Ile Gly Phe Thr Pro Leu
165 170 175
His Arg Ala Ala Phe Val Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Asp Phe Gly His Thr Pro
195 200 205
Ala Asp Leu Ala Ala Ser Leu Gly His Glu Asp Ile Ala Glu Val Leu
210 215 220
Gln Lys Leu Asn
225
<210> 98
<211> 261
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 98
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Glu Gly Tyr Thr Pro Leu His Val Ala Ala Tyr Asp Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ser Gln Gly Arg Thr Pro Leu His Glu Ala Ala Tyr Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asp Ala Gly Glu Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Lys Lys Leu Leu
130 135 140
Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met Ala
145 150 155 160
Asn Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu
165 170 175
Tyr Leu Ala Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Asn Gly Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro
195 200 205
Leu His Leu Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu
210 215 220
Leu Lys His Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr
225 230 235 240
Ala Phe Asp Ile Ser Ile Gly Asn Gly Asn Glu Asp Leu Ala Glu Ile
245 250 255
Leu Gln Lys Leu Asn
260
<210> 99
<211> 261
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 99
Gly Ser Asp Leu Gly His Lys Leu Leu Glu Ala Ala Val Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Trp Tyr Gly Lys Thr Pro Leu His Phe Ala Ala Gly Leu Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Glu Asp Gly Gln Thr Pro Leu His Leu Ala Ala Ala Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn Asp Gly Phe Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Ser Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ile Lys Leu Leu
130 135 140
Gln Ala Ala Asn Leu Gly Gln Asp Asp Glu Val Arg Ile Leu Leu Ala
145 150 155 160
Thr Gly Ala Asp Val Asn Ala Lys Asp Ser Ile Gly Gln Thr Pro Leu
165 170 175
His Trp Ala Ala Arg Arg Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Val Thr Pro
195 200 205
Leu His Leu Ala Ala Ser Leu Gly His Leu Glu Ile Val Glu Val Leu
210 215 220
Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Ser Gly Glu Thr
225 230 235 240
Pro Ala Asp Leu Ala Ala Leu His Gly His Glu Asp Ile Ala Glu Val
245 250 255
Leu Gln Lys Leu Asn
260
<210> 100
<211> 228
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 100
Gly Ser Asp Leu Gly His Lys Leu Leu Glu Ala Ala Val Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Trp Tyr Gly Lys Thr Pro Leu His Phe Ala Ala Gly Leu Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Glu Asp Gly Gln Thr Pro Leu His Leu Ala Ala Ala Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn Asp Gly Phe Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Ser Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ala Lys Leu Leu
130 135 140
Val Ala Ala Thr Ser Gly Gln Asp Asp Glu Val Arg Ile Leu Leu Ala
145 150 155 160
Ala Gly Ala Asp Val Asn Ala Lys Asp Arg Ile Gly Phe Thr Pro Leu
165 170 175
His Arg Ala Ala Phe Val Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Asp Phe Gly His Thr Pro
195 200 205
Ala Asp Leu Ala Ala Ser Leu Gly His Glu Asp Ile Ala Glu Val Leu
210 215 220
Gln Lys Leu Asn
225
<210> 101
<211> 261
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 101
Gly Ser Asp Leu Gly His Lys Leu Leu Glu Ala Ala Val Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Trp Tyr Gly Lys Thr Pro Leu His Phe Ala Ala Gly Leu Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Glu Asp Gly Gln Thr Pro Leu His Leu Ala Ala Ala Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn Asp Gly Phe Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Ser Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Lys Lys Leu Leu
130 135 140
Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met Ala
145 150 155 160
Asn Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu
165 170 175
Tyr Leu Ala Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys Asn Gly Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro
195 200 205
Leu His Leu Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu
210 215 220
Leu Lys His Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr
225 230 235 240
Ala Phe Asp Ile Ser Ile Gly Asn Gly Asn Glu Asp Leu Ala Glu Ile
245 250 255
Leu Gln Lys Leu Asn
260
<210> 102
<211> 269
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 102
Gly Ser Asp Leu Gly Ala Lys Leu Leu Ser Asp Leu Gly Val Lys Leu
1 5 10 15
Leu Trp Ala Ala Ala Arg Gly Gln Asp Asp Glu Val Arg Ile Leu Leu
20 25 30
Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Gly Ile Thr Pro
35 40 45
Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val Leu
50 55 60
Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp Thr
65 70 75 80
Pro Leu His Leu Ala Ala Gln His Gly His Leu Val Ile Val Glu Val
85 90 95
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly Trp
100 105 110
Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala Glu
115 120 125
Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Arg
130 135 140
Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln Asp
145 150 155 160
Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala Lys
165 170 175
Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly His
180 185 190
Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn Ala
195 200 205
Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile Gly
210 215 220
His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val Asn
225 230 235 240
Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly Asn
245 250 255
Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
260 265
<210> 103
<211> 302
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 103
Gly Ser Asp Leu Gly Ala Lys Leu Leu Ser Asp Leu Gly Val Lys Leu
1 5 10 15
Leu Trp Ala Ala Ala Arg Gly Gln Asp Asp Glu Val Arg Ile Leu Leu
20 25 30
Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Gly Ile Thr Pro
35 40 45
Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val Leu
50 55 60
Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp Thr
65 70 75 80
Pro Leu His Leu Ala Ala Gln His Gly His Leu Val Ile Val Glu Val
85 90 95
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly Trp
100 105 110
Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala Glu
115 120 125
Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Arg
130 135 140
Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln Asp
145 150 155 160
Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala Thr
165 170 175
Asp Ile His Gly His Thr Pro Leu His Leu Ala Ala Ala Met Gly His
180 185 190
Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn Ala
195 200 205
Asn Asp Trp Arg Gly Phe Thr Pro Leu His Leu Ala Ala Leu Asn Gly
210 215 220
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
225 230 235 240
Ala Thr Asp Thr Ala Gly Asn Thr Pro Leu His Leu Ala Ala Trp Phe
245 250 255
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val
260 265 270
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Asp
275 280 285
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
290 295 300
<210> 104
<211> 270
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 104
Gly Ser Asp Leu Gly Asp Lys Leu Leu Gln Ser Asp Leu Gly Asn Lys
1 5 10 15
Leu Leu Ile Ala Ala Ser Val Gly Gln Asp Asp Glu Val Arg Ile Leu
20 25 30
Leu Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Glu Thr Gly Trp Thr
35 40 45
Pro Leu His Leu Ala Ala Ala Trp Gly His Leu Glu Ile Val Glu Val
50 55 60
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Lys Gly Gln
65 70 75 80
Thr Pro Leu His Leu Ala Ala Ala Tyr Gly His Leu Glu Ile Val Glu
85 90 95
Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Asn Asp Gly
100 105 110
Tyr Thr Pro Ala Asp Leu Ala Ala Arg Tyr Gly His Glu Asp Ile Ala
115 120 125
Glu Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Arg Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
145 150 155 160
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
165 170 175
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
180 185 190
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
195 200 205
Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile
210 215 220
Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val
225 230 235 240
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly
245 250 255
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
260 265 270
<210> 105
<211> 303
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 105
Gly Ser Asp Leu Gly Asp Lys Leu Leu Gln Ser Asp Leu Gly Asn Lys
1 5 10 15
Leu Leu Ile Ala Ala Ser Val Gly Gln Asp Asp Glu Val Arg Ile Leu
20 25 30
Leu Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Glu Thr Gly Trp Thr
35 40 45
Pro Leu His Leu Ala Ala Ala Trp Gly His Leu Glu Ile Val Glu Val
50 55 60
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Lys Gly Gln
65 70 75 80
Thr Pro Leu His Leu Ala Ala Ala Tyr Gly His Leu Glu Ile Val Glu
85 90 95
Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Asn Asp Gly
100 105 110
Tyr Thr Pro Ala Asp Leu Ala Ala Arg Tyr Gly His Glu Asp Ile Ala
115 120 125
Glu Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Arg Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
145 150 155 160
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
165 170 175
Thr Asp Ile His Gly His Thr Pro Leu His Leu Ala Ala Ala Met Gly
180 185 190
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
195 200 205
Ala Asn Asp Trp Arg Gly Phe Thr Pro Leu His Leu Ala Ala Leu Asn
210 215 220
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val
225 230 235 240
Asn Ala Thr Asp Thr Ala Gly Asn Thr Pro Leu His Leu Ala Ala Trp
245 250 255
Phe Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp
260 265 270
Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile
275 280 285
Asp Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
290 295 300
<210> 106
<211> 294
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 106
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Tyr Gly Ile Thr Pro Leu His Leu Ala Ala Ala Tyr Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys His Gly Ala Asp Val Asn
50 55 60
Ala His Asp Trp Asn Gly Trp Thr Pro Leu His Leu Ala Ala Lys Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys His Gly Ala Asp Val
85 90 95
Asn Ala Ile Asp Asn Ala Gly Lys Thr Pro Leu His Leu Ala Ala Ala
100 105 110
His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Tyr Gly Ala Asp
115 120 125
Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile
130 135 140
Asp Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Gly Gly
145 150 155 160
Gly Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Lys Lys Leu
165 170 175
Leu Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met
180 185 190
Ala Asn Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro
195 200 205
Leu Tyr Leu Ala Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu
210 215 220
Leu Lys Asn Gly Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr
225 230 235 240
Pro Leu His Leu Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val
245 250 255
Leu Leu Lys His Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys
260 265 270
Thr Ala Phe Asp Ile Ser Ile Gly Asn Gly Asn Glu Asp Leu Ala Glu
275 280 285
Ile Leu Gln Lys Leu Asn
290
<210> 107
<211> 327
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 107
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Tyr Gly Ile Thr Pro Leu His Leu Ala Ala Ala Tyr Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys His Gly Ala Asp Val Asn
50 55 60
Ala His Asp Trp Asn Gly Trp Thr Pro Leu His Leu Ala Ala Lys Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys His Gly Ala Asp Val
85 90 95
Asn Ala Ile Asp Asn Ala Gly Lys Thr Pro Leu His Leu Ala Ala Ala
100 105 110
His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Tyr Gly Ala Asp
115 120 125
Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile
130 135 140
Asp Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Gly Gly
145 150 155 160
Gly Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Lys Lys Leu
165 170 175
Leu Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met
180 185 190
Ala Asn Gly Ala Asp Val Asn Ala Thr Asp Ile His Gly His Thr Pro
195 200 205
Leu His Leu Ala Ala Ala Met Gly His Leu Glu Ile Val Glu Val Leu
210 215 220
Leu Lys Asn Gly Ala Asp Val Asn Ala Asn Asp Trp Arg Gly Phe Thr
225 230 235 240
Pro Leu His Leu Ala Ala Leu Asn Gly His Leu Glu Ile Val Glu Val
245 250 255
Leu Leu Lys Asn Gly Ala Asp Val Asn Ala Thr Asp Thr Ala Gly Asn
260 265 270
Thr Pro Leu His Leu Ala Ala Trp Phe Gly His Leu Glu Ile Val Glu
275 280 285
Val Leu Leu Lys Asn Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly
290 295 300
Lys Thr Ala Phe Asp Ile Ser Ile Asp Asn Gly Asn Glu Asp Leu Ala
305 310 315 320
Glu Ile Leu Gln Lys Leu Asn
325
<210> 108
<211> 270
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 108
Gly Ser Asp Leu Gly Asp Lys Leu Leu Gln Ser Asp Leu Gly Ile Lys
1 5 10 15
Leu Leu Phe Ala Ala Ala Lys Ser Gln Asp Asp Glu Val Arg Ile Leu
20 25 30
Leu Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Ser Val Thr
35 40 45
Pro Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val
50 55 60
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp
65 70 75 80
Thr Pro Leu His Leu Ala Ala Gln His Gly His Leu Glu Ile Val Glu
85 90 95
Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly
100 105 110
Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala
115 120 125
Glu Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
130 135 140
Arg Ser Asp Leu Gly Ile Lys Leu Leu Val Ala Ala Ala Gln Gly Gln
145 150 155 160
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
165 170 175
Lys Asp Gln Gln Gly Ala Thr Pro Leu His Leu Ala Ala Trp Lys Gly
180 185 190
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
195 200 205
Ala Lys Asp Leu Ser Gly Asp Thr Pro Leu His Ile Ala Ala Trp Phe
210 215 220
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
225 230 235 240
Asn Ala Gln Asp Thr Glu Gly Tyr Thr Pro Ala Asp Leu Ala Ala Leu
245 250 255
Tyr Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
260 265 270
<210> 109
<211> 294
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 109
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
50 55 60
Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile
65 70 75 80
Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly
100 105 110
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Lys Lys Leu Leu
130 135 140
Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met Ala
145 150 155 160
Asn Gly Ala Asp Val Asn Ala Lys Asp Phe Tyr Gly Ile Thr Pro Leu
165 170 175
His Leu Ala Ala Ala Tyr Gly His Leu Glu Ile Val Glu Val Leu Leu
180 185 190
Lys His Gly Ala Asp Val Asn Ala His Asp Trp Asn Gly Trp Thr Pro
195 200 205
Leu His Leu Ala Ala Lys Tyr Gly His Leu Glu Ile Val Glu Val Leu
210 215 220
Leu Lys His Gly Ala Asp Val Asn Ala Ile Asp Asn Ala Gly Lys Thr
225 230 235 240
Pro Leu His Leu Ala Ala Ala His Gly His Leu Glu Ile Val Glu Val
245 250 255
Leu Leu Lys Tyr Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys
260 265 270
Thr Ala Phe Asp Ile Ser Ile Asp Asn Gly Asn Glu Asp Leu Ala Glu
275 280 285
Ile Leu Gln Lys Leu Asn
290
<210> 110
<211> 269
<212> PRT
<213> Artificial
<220>
<223> AR domain (two-domain)
<400> 110
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
50 55 60
Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile
65 70 75 80
Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly
100 105 110
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Arg Ser Asp Leu Gly Ala Lys Leu Leu
130 135 140
Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln Asp
145 150 155 160
Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala Lys
165 170 175
Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Gln Ser Gly His
180 185 190
Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
195 200 205
Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His Gly
210 215 220
His Leu Val Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
225 230 235 240
Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp
245 250 255
Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
260 265
<210> 111
<211> 126
<212> PRT
<213> Artificial
<220>
<223> AR domain (one-domain) Negative Control
<400> 111
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Lys Asp Gly Tyr Thr Pro Leu His Leu Ala Ala Arg Glu Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Lys Asp Gly Tyr Thr Pro Leu His Leu Ala Ala Arg Glu
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Asp
100 105 110
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
115 120 125
<210> 112
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 112
Gly Ser Asp Leu Gly Asn Lys Leu Leu Ile Ala Ala Ser Val Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Glu Thr Gly Trp Thr Pro Leu His Leu Ala Ala Ala Trp Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Lys Gly Gln Thr Pro Leu His Leu Ala Ala Ala Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Asn Asp Gly Tyr Thr Pro Ala Asp Leu Ala Ala Arg
100 105 110
Tyr Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 113
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 113
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala His Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Asp Ala Asp Val Asn Ala
20 25 30
Lys Asp Trp Tyr Gly Thr Thr Pro Leu His Ile Ala Ala Val Ala Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Asp Phe Gly Thr Thr Pro Leu His Ala Ala Ala Asp Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Tyr Thr Gly His Thr Pro Ala Asp Leu Ala Ala Val
100 105 110
Tyr Gly His Glu Asp Ile Ala Ala Val Leu Gln Lys Leu Asn
115 120 125
<210> 114
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 114
Gly Ser Asp Leu Gly Ala Lys Leu Leu Trp Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Trp Glu Gly Val Thr Pro Leu His Ile Ala Ala His Ala Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Ile Gly Trp Thr Pro Leu His Ser Ala Ala Val Tyr
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Trp Tyr Gly Thr Thr Pro Ala Asp Leu Ala Ala Trp
100 105 110
Trp Gly His Glu Asp Ile Ala Val Val Leu Gln Lys Leu Asn
115 120 125
<210> 115
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 115
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 116
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR doamin (one-domain)
<400> 116
Gly Ser Asp Leu Gly Ile Lys Leu Leu Ile Ala Ala Ser His Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asn Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 117
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 117
Gly Ser Asp Leu Gly Gln Lys Leu Leu Ile Ala Ala Ser Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Thr Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 118
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 118
Gly Ser Asp Leu Gly Ile Lys Leu Leu Trp Ala Ala Ala Gln Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Val Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 119
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 119
Gly Ser Asp Leu Gly Phe Lys Leu Leu Phe Ala Ala Ala Lys Ser Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Leu Ala Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Val Thr Ser Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 120
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 120
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Asn Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 121
<211> 126
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (one-domain)
<400> 121
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
115 120 125
<210> 122
<211> 274
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (two-domain)
<400> 122
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Ala Ala Gly Ser
115 120 125
Pro Thr Pro Thr Pro Thr Thr Pro Thr Pro Thr Pro Thr Thr Pro Thr
130 135 140
Pro Thr Pro Thr Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala
145 150 155 160
Arg Ala Gly Gln Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala
165 170 175
Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala
180 185 190
Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly
195 200 205
Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu
210 215 220
Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys Ala
225 230 235 240
Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Pro Ala Asp
245 250 255
Ile Ala Ala Gly Ala Gly Asn Glu Asp Ile Ala Glu Val Leu Gln Lys
260 265 270
Ala Ala
<210> 123
<211> 274
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (two-domain)
<400> 123
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Gln Ser Gly
35 40 45
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Glu Arg Gly Lys Thr Pro Ala Asp Leu Ala Ala Asp
100 105 110
Trp Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Ala Ala Gly Ser
115 120 125
Pro Thr Pro Thr Pro Thr Thr Pro Thr Pro Thr Pro Thr Thr Pro Thr
130 135 140
Pro Thr Pro Thr Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala
145 150 155 160
Arg Ala Gly Gln Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala
165 170 175
Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala
180 185 190
Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly
195 200 205
Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu
210 215 220
Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys Ala
225 230 235 240
Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Pro Ala Asp
245 250 255
Ile Ala Ala Gly Ala Gly Asn Glu Asp Ile Ala Glu Val Leu Gln Lys
260 265 270
Ala Ala
<210> 124
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> N-Cap module (Nr)
<400> 124
Gly Ser Asp Leu Gly Ile Lys Leu Leu Phe Ala Ala Ala Lys Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 125
<211> 33
<212> PRT
<213> Artificial Sequence
<220>
<223> AR module (M1.1b)
<400> 125
Lys Asp Phe Gln Gly Val Thr Pro Leu His Ile Ala Ala Gln Ser Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 126
<211> 33
<212> PRT
<213> Artificial Sequence
<220>
<223> AR module (M1.1b)
<400> 126
Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 127
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<223> C-Cap module (Cr)
<400> 127
Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 128
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> N-Cap module (Nr)
<400> 128
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 129
<211> 33
<212> PRT
<213> Artificial Sequence
<220>
<223> AR module (M1.1b)
<400> 129
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Thr Asn Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 130
<211> 33
<212> PRT
<213> Artificial Sequence
<220>
<223> AR module (M1.1b)
<400> 130
Lys Asp Ile Thr Gly Glu Thr Pro Leu His His Ala Ala Asp Ser Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 131
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<223> C-Cap module (Cr)
<400> 131
Gln Asp Lys Ala Gly Val Thr Pro Ala Asp Leu Ala Ala Ala Trp Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 132
<211> 32
<212> PRT
<213> Artificial Sequence
<220>
<223> N-Cap module (Nr)
<400> 132
Gly Ser Asp Leu Gly Val Lys Leu Leu Trp Ala Ala Ala Arg Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Glu Leu Leu Lys Ala Gly Ala Asp Val Asn Ala
20 25 30
<210> 133
<211> 33
<212> PRT
<213> Artificial Sequence
<220>
<223> AR module (M1.1b)
<400> 133
Lys Asp Phe Gln Gly Ile Thr Pro Leu His Ile Ala Ala Gln Ser Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 134
<211> 33
<212> PRT
<213> Artificial Sequence
<220>
<223> AR module (M1.1b)
<400> 134
Lys Asp Val Thr Gly Asp Thr Pro Leu His Leu Ala Ala Gln His Gly
1 5 10 15
His Leu Glu Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
20 25 30
Ala
<210> 135
<211> 28
<212> PRT
<213> Artificial Sequence
<220>
<223> C-Cap module (Cr)
<400> 135
Gln Asp Glu Arg Gly Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly
1 5 10 15
His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Asn
20 25
<210> 136
<211> 278
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (two-domain)
<400> 136
Gly Ser Asp Leu Gly Ala Lys Leu Leu Ser Asp Leu Gly Val Lys Leu
1 5 10 15
Leu Trp Ala Ala Ala Arg Gly Gln Asp Asp Glu Val Arg Ile Leu Leu
20 25 30
Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Gly Ile Thr Pro
35 40 45
Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val Leu
50 55 60
Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp Thr
65 70 75 80
Pro Leu His Leu Ala Ala Gln His Gly His Leu Val Ile Val Glu Val
85 90 95
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly Trp
100 105 110
Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala Glu
115 120 125
Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Arg
130 135 140
Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln Asp
145 150 155 160
Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala Lys
165 170 175
Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly His
180 185 190
Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn Ala
195 200 205
Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile Gly
210 215 220
His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val Asn
225 230 235 240
Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly Asn
245 250 255
Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Ala Ala Gly Gly Gly
260 265 270
Ser Gly Gly Gly Ser Cys
275
<210> 137
<211> 414
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (three-domain)
<400> 137
Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
1 5 10 15
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
20 25 30
Lys Asp Tyr Phe Ser His Thr Pro Leu His Leu Ala Ala Arg Asn Gly
35 40 45
His Leu Lys Ile Val Glu Val Leu Leu Lys Ala Gly Ala Asp Val Asn
50 55 60
Ala Lys Asp Phe Ala Gly Lys Thr Pro Leu His Leu Ala Ala Asn Asp
65 70 75 80
Gly His Leu Glu Ile Val Glu Val Leu Leu Lys His Gly Ala Asp Val
85 90 95
Asn Ala Gln Asp Ile Phe Gly Lys Thr Pro Ala Asp Ile Ala Ala Asp
100 105 110
Ala Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu Gly Gly Gly
115 120 125
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly
130 135 140
Ser Arg Ser Asp Leu Gly Ala Lys Leu Leu Ser Asp Leu Gly Val Lys
145 150 155 160
Leu Leu Trp Ala Ala Ala Arg Gly Gln Asp Asp Glu Val Arg Ile Leu
165 170 175
Leu Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Gly Ile Thr
180 185 190
Pro Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val
195 200 205
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp
210 215 220
Thr Pro Leu His Leu Ala Ala Gln His Gly His Leu Val Ile Val Glu
225 230 235 240
Val Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly
245 250 255
Trp Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala
260 265 270
Glu Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser
275 280 285
Arg Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln
290 295 300
Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala
305 310 315 320
Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly
325 330 335
His Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn
340 345 350
Ala Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile
355 360 365
Gly His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val
370 375 380
Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly
385 390 395 400
Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn
405 410
<210> 138
<211> 417
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (three-domain)
<400> 138
Gly Ser Asp Leu Gly Ala Lys Leu Leu Ser Asp Leu Gly Val Lys Leu
1 5 10 15
Leu Trp Ala Ala Ala Arg Gly Gln Asp Asp Glu Val Arg Ile Leu Leu
20 25 30
Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Gly Ile Thr Pro
35 40 45
Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val Leu
50 55 60
Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp Thr
65 70 75 80
Pro Leu His Leu Ala Ala Gln His Gly His Leu Val Ile Val Glu Val
85 90 95
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly Trp
100 105 110
Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala Glu
115 120 125
Val Leu Gln Lys Leu Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Arg
130 135 140
Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln Asp
145 150 155 160
Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala Lys
165 170 175
Asp Glu Tyr Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly His
180 185 190
Leu Glu Ile Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn Ala
195 200 205
Val Asp Ala Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile Gly
210 215 220
His Leu Glu Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val Asn
225 230 235 240
Ala Gln Asp Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly Asn
245 250 255
Gly Asn Glu Asp Leu Ala Glu Ile Leu Gln Lys Leu Asn Lys Leu Gly
260 265 270
Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly
275 280 285
Gly Gly Ser Arg Ser Asp Leu Gly Lys Lys Leu Leu Glu Ala Ala Arg
290 295 300
Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met Ala Asn Gly Ala Asp
305 310 315 320
Val Asn Ala Lys Asp Tyr Phe Ser His Thr Pro Leu His Leu Ala Ala
325 330 335
Arg Asn Gly His Leu Lys Ile Val Glu Val Leu Leu Lys Ala Gly Ala
340 345 350
Asp Val Asn Ala Lys Asp Phe Ala Gly Lys Thr Pro Leu His Leu Ala
355 360 365
Ala Asn Asp Gly His Leu Glu Ile Val Glu Val Leu Leu Lys His Gly
370 375 380
Ala Asp Val Asn Ala Gln Asp Ile Phe Gly Lys Thr Pro Ala Asp Ile
385 390 395 400
Ala Ala Asp Ala Gly His Glu Asp Ile Ala Glu Val Leu Gln Lys Leu
405 410 415
Asn
<210> 139
<211> 260
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (two-domain)
<400> 139
Gly Ser Asp Leu Gly Ala Lys Leu Leu Ser Asp Leu Gly Val Lys Leu
1 5 10 15
Leu Trp Ala Ala Ala Arg Gly Gln Asp Asp Glu Val Arg Ile Leu Leu
20 25 30
Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Gly Ile Thr Pro
35 40 45
Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val Leu
50 55 60
Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp Thr
65 70 75 80
Pro Leu His Leu Ala Ala Gln His Gly His Leu Val Ile Val Glu Val
85 90 95
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly Trp
100 105 110
Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala Glu
115 120 125
Val Leu Gln Lys Ala Ala Gly Ser Asp Leu Gly Lys Lys Leu Leu Glu
130 135 140
Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu Met Ala Asn
145 150 155 160
Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr Pro Leu Tyr
165 170 175
Leu Ala Thr Ala His Gly His Leu Glu Ile Val Glu Val Leu Leu Lys
180 185 190
Asn Gly Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe Thr Pro Leu
195 200 205
His Leu Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu Val Leu Leu
210 215 220
Lys His Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly Lys Thr Ala
225 230 235 240
Phe Asp Ile Ser Ile Gly Asn Gly Asn Glu Asp Leu Ala Glu Ile Leu
245 250 255
Gln Lys Ala Ala
260
<210> 140
<211> 263
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (two-domain)
<400> 140
Gly Ser Asp Leu Gly Ala Lys Leu Leu Ser Asp Leu Gly Val Lys Leu
1 5 10 15
Leu Trp Ala Ala Ala Arg Gly Gln Asp Asp Glu Val Arg Ile Leu Leu
20 25 30
Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Gly Ile Thr Pro
35 40 45
Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val Leu
50 55 60
Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp Thr
65 70 75 80
Pro Leu His Leu Ala Ala Gln His Gly His Leu Val Ile Val Glu Val
85 90 95
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly Trp
100 105 110
Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala Glu
115 120 125
Val Leu Gln Lys Ala Ala Gly Gly Gly Gly Ser Asp Leu Gly Lys Lys
130 135 140
Leu Leu Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val Arg Ile Leu
145 150 155 160
Met Ala Asn Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr Gly Leu Thr
165 170 175
Pro Leu Tyr Leu Ala Thr Ala His Gly His Leu Glu Ile Val Glu Val
180 185 190
Leu Leu Lys Asn Gly Ala Asp Val Asn Ala Val Asp Ala Ile Gly Phe
195 200 205
Thr Pro Leu His Leu Ala Ala Phe Ile Gly His Leu Glu Ile Ala Glu
210 215 220
Val Leu Leu Lys His Gly Ala Asp Val Asn Ala Gln Asp Lys Phe Gly
225 230 235 240
Lys Thr Ala Phe Asp Ile Ser Ile Gly Asn Gly Asn Glu Asp Leu Ala
245 250 255
Glu Ile Leu Gln Lys Ala Ala
260
<210> 141
<211> 282
<212> PRT
<213> Artificial Sequence
<220>
<223> AR domain (two-domain)
<400> 141
Gly Ser Asp Leu Gly Ala Lys Leu Leu Ser Asp Leu Gly Val Lys Leu
1 5 10 15
Leu Trp Ala Ala Ala Arg Gly Gln Asp Asp Glu Val Arg Ile Leu Leu
20 25 30
Ala Ala Gly Ala Asp Val Asn Ala Lys Asp Phe Gln Gly Ile Thr Pro
35 40 45
Leu His Ile Ala Ala Gln Ser Gly His Leu Glu Ile Val Glu Val Leu
50 55 60
Leu Lys Ala Gly Ala Asp Val Asn Ala Lys Asp Val Thr Gly Asp Thr
65 70 75 80
Pro Leu His Leu Ala Ala Gln His Gly His Leu Val Ile Val Glu Val
85 90 95
Leu Leu Lys Ala Gly Ala Asp Val Asn Ala Gln Asp Glu Arg Gly Trp
100 105 110
Thr Pro Ala Asp Leu Ala Ala Asp Trp Gly His Glu Asp Ile Ala Glu
115 120 125
Val Leu Gln Lys Ala Ala Gly Ser Pro Thr Pro Thr Pro Thr Thr Pro
130 135 140
Thr Pro Thr Pro Thr Thr Pro Thr Pro Thr Pro Thr Gly Ser Asp Leu
145 150 155 160
Gly Lys Lys Leu Leu Glu Ala Ala Arg Ala Gly Gln Asp Asp Glu Val
165 170 175
Arg Ile Leu Met Ala Asn Gly Ala Asp Val Asn Ala Lys Asp Glu Tyr
180 185 190
Gly Leu Thr Pro Leu Tyr Leu Ala Thr Ala His Gly His Leu Glu Ile
195 200 205
Val Glu Val Leu Leu Lys Asn Gly Ala Asp Val Asn Ala Val Asp Ala
210 215 220
Ile Gly Phe Thr Pro Leu His Leu Ala Ala Phe Ile Gly His Leu Glu
225 230 235 240
Ile Ala Glu Val Leu Leu Lys His Gly Ala Asp Val Asn Ala Gln Asp
245 250 255
Lys Phe Gly Lys Thr Ala Phe Asp Ile Ser Ile Gly Asn Gly Asn Glu
260 265 270
Asp Leu Ala Glu Ile Leu Gln Lys Ala Ala
275 280
| название | год | авторы | номер документа |
|---|---|---|---|
| РЕКОМБИНАНТНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ И ИХ ПРИМЕНЕНИЕ | 2017 |
|
RU2778346C2 |
| Биспецифическое антитело анти-HER2/анти-4-1BB и его применение | 2020 |
|
RU2831838C2 |
| РЕКОМБИНАНТНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ И ИХ ПРИМЕНЕНИЕ | 2016 |
|
RU2769470C2 |
| HER-2-НАПРАВЛЕННЫЕ АНТИГЕНСВЯЗЫВАЮЩИЕ МОЛЕКУЛЫ, СОДЕРЖАЩИЕ 4-1BBL | 2019 |
|
RU2815451C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИГЕНСВЯЗЫВАЮЩИЕ ПОЛИПЕПТИДЫ | 2015 |
|
RU2723940C2 |
| ВВЕДЕНИЕ СКОНСТРУИРОВАННЫХ Т-КЛЕТОК ДЛЯ ЛЕЧЕНИЯ РАКА ЦЕНТРАЛЬНОЙ НЕРВНОЙ СИСТЕМЫ | 2017 |
|
RU2757308C2 |
| ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2018 |
|
RU2815452C2 |
| БИСПЕЦИФИЧЕСКИЕ АНТИГЕНСВЯЗЫВАЮЩИЕ КОНСТРУКЦИИ ПРОТИВ HER2 | 2014 |
|
RU2737882C2 |
| АНТИТЕЛА ПРОТИВ БЕЛКА-1 ЗАПРОГРАММИРОВАННОЙ КЛЕТОЧНОЙ СМЕРТИ (PD-1) И ИХ ПРИМЕНЕНИЕ | 2017 |
|
RU2725950C1 |
| УЛУЧШЕННЫЕ ОДИНОЧНЫЕ ВАРИАБЕЛЬНЫЕ ДОМЕНЫ ИММУНОГЛОБУЛИНА, СВЯЗЫВАЮЩИЕСЯ С СЫВОРОТОЧНЫМ АЛЬБУМИНОМ | 2017 |
|
RU2765384C2 |

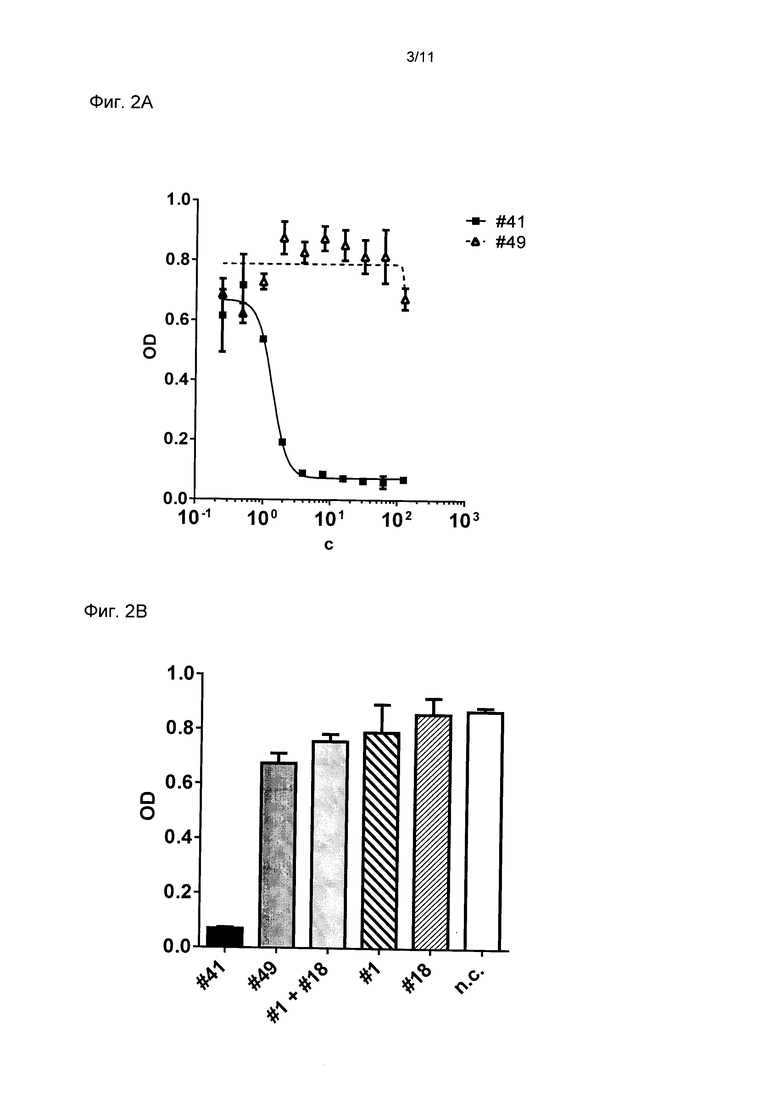
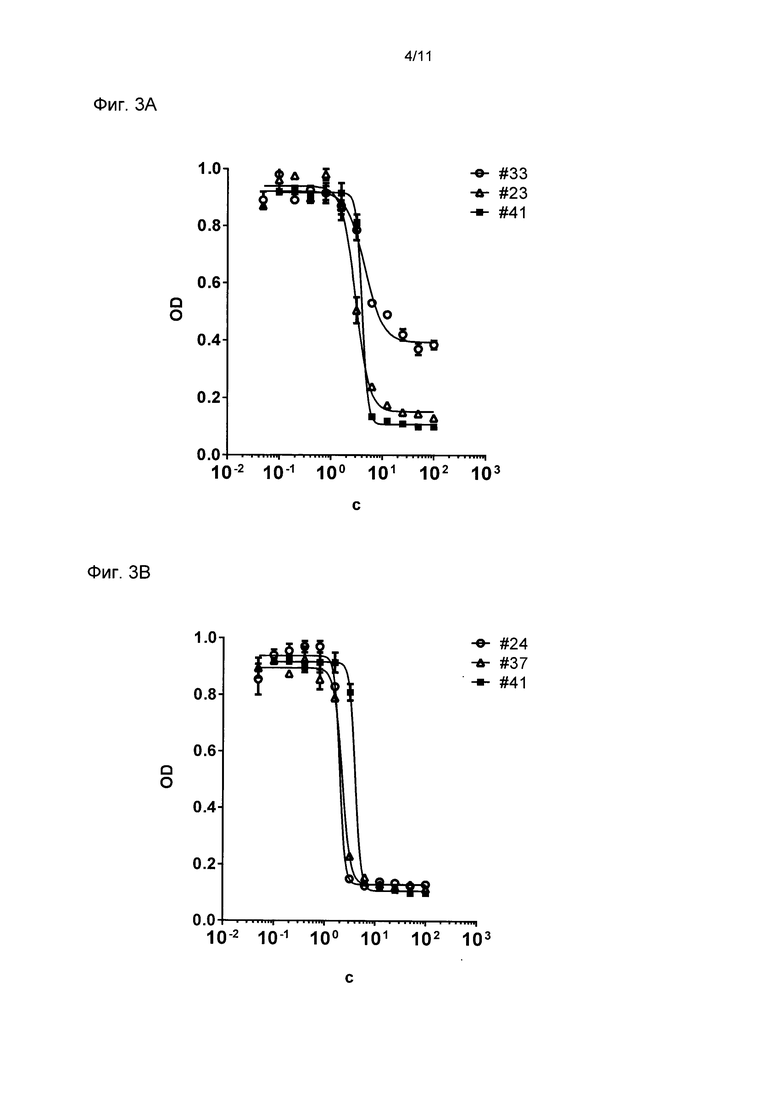
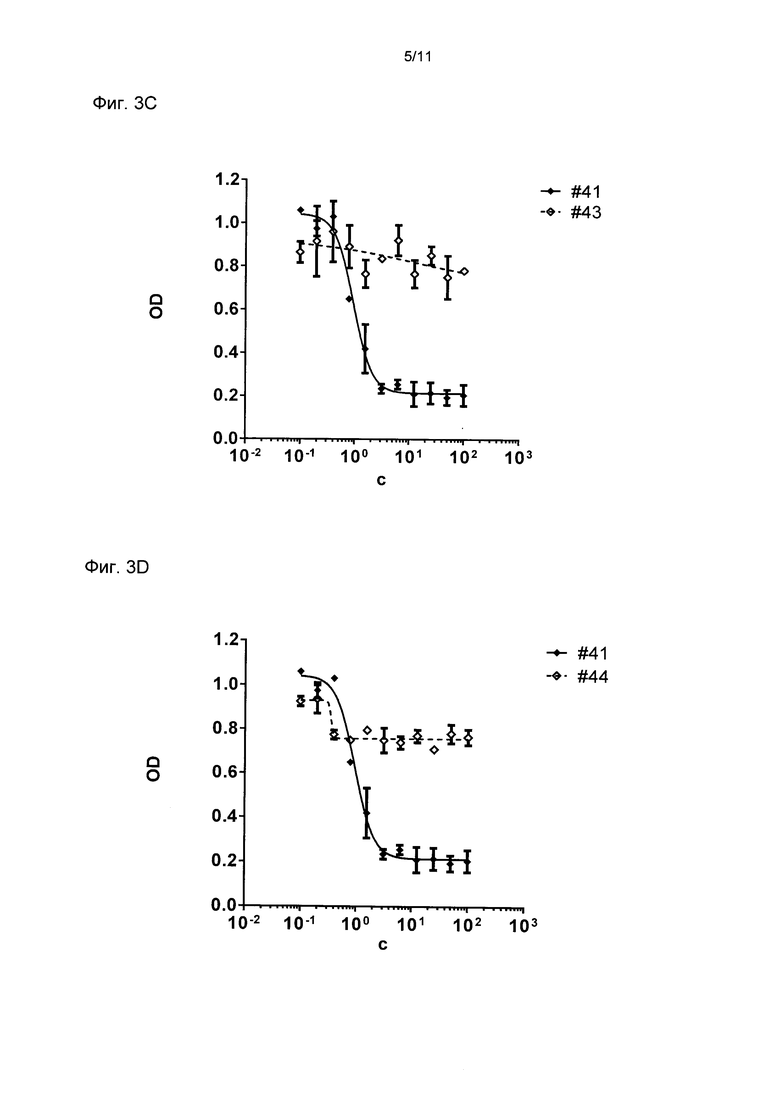
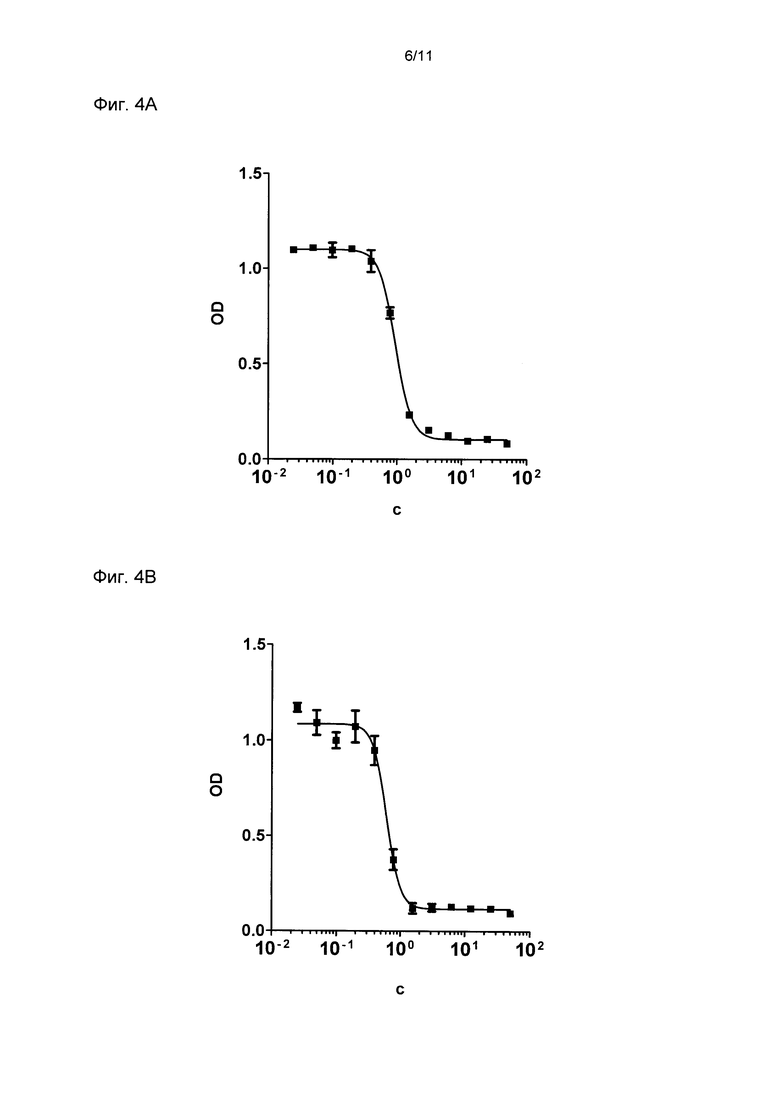
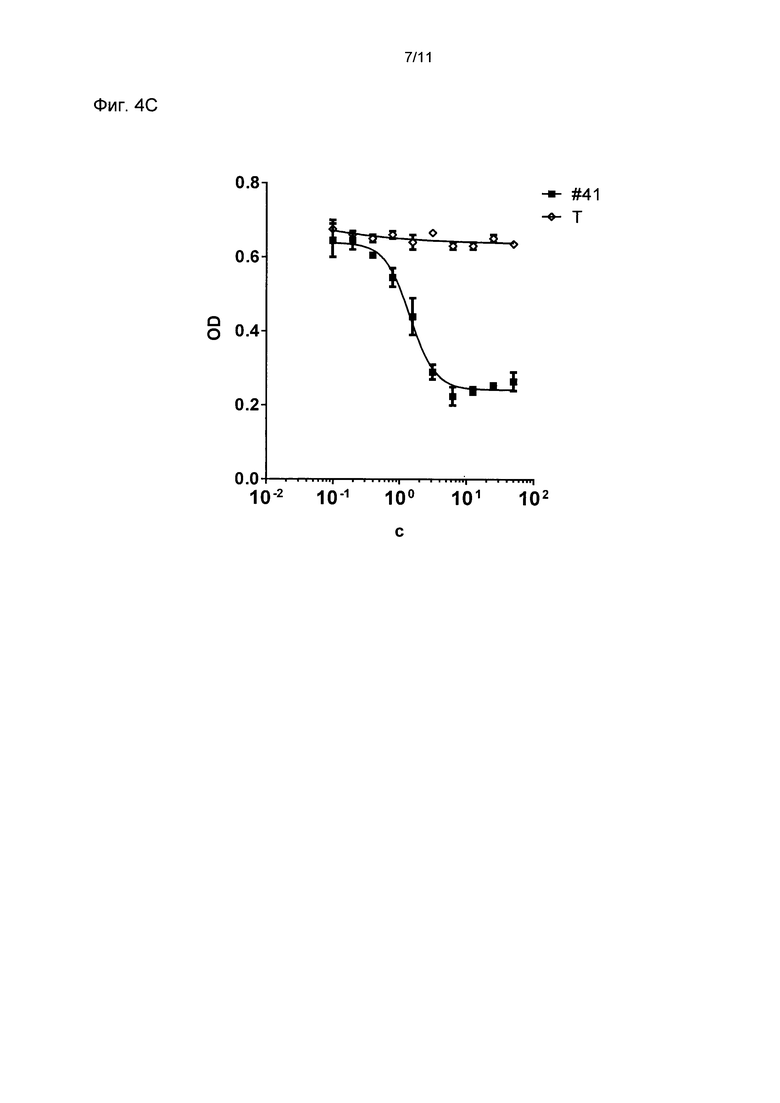
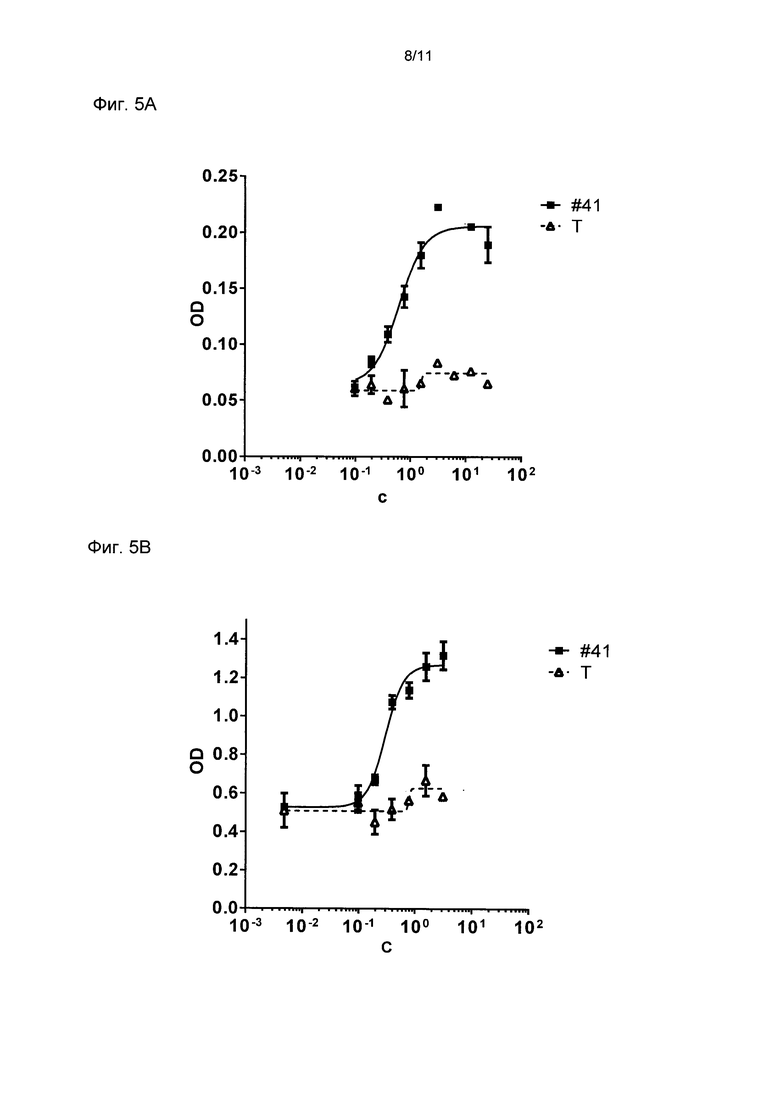
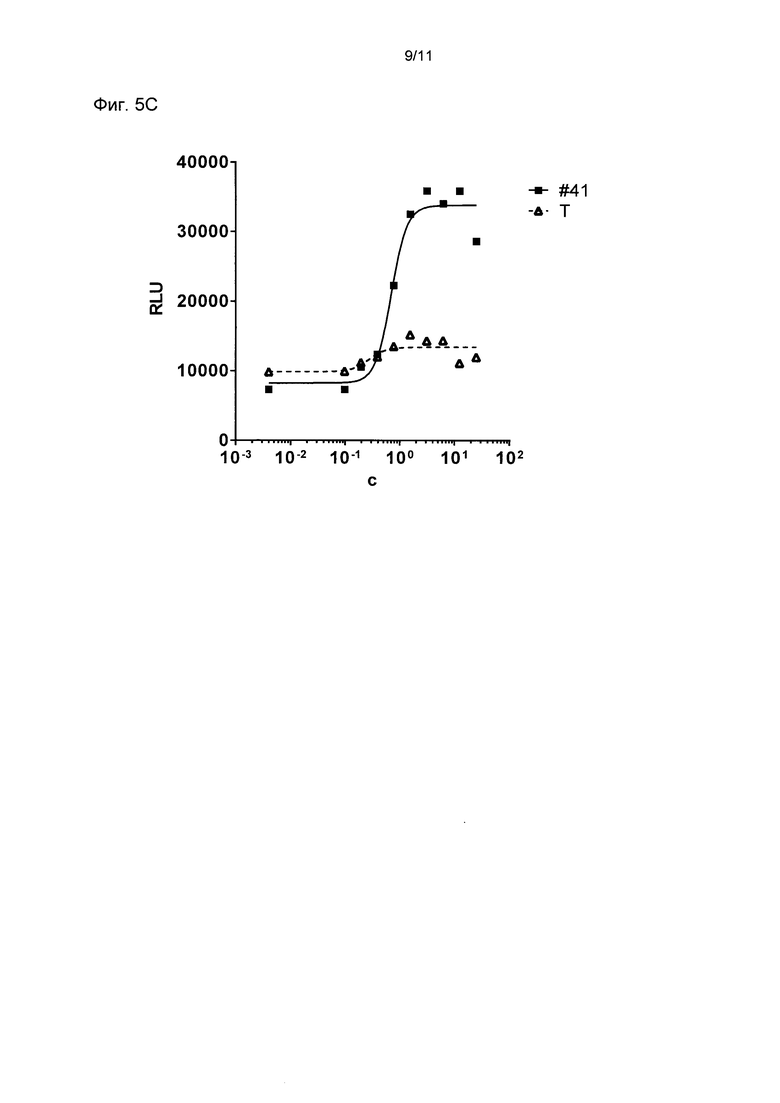

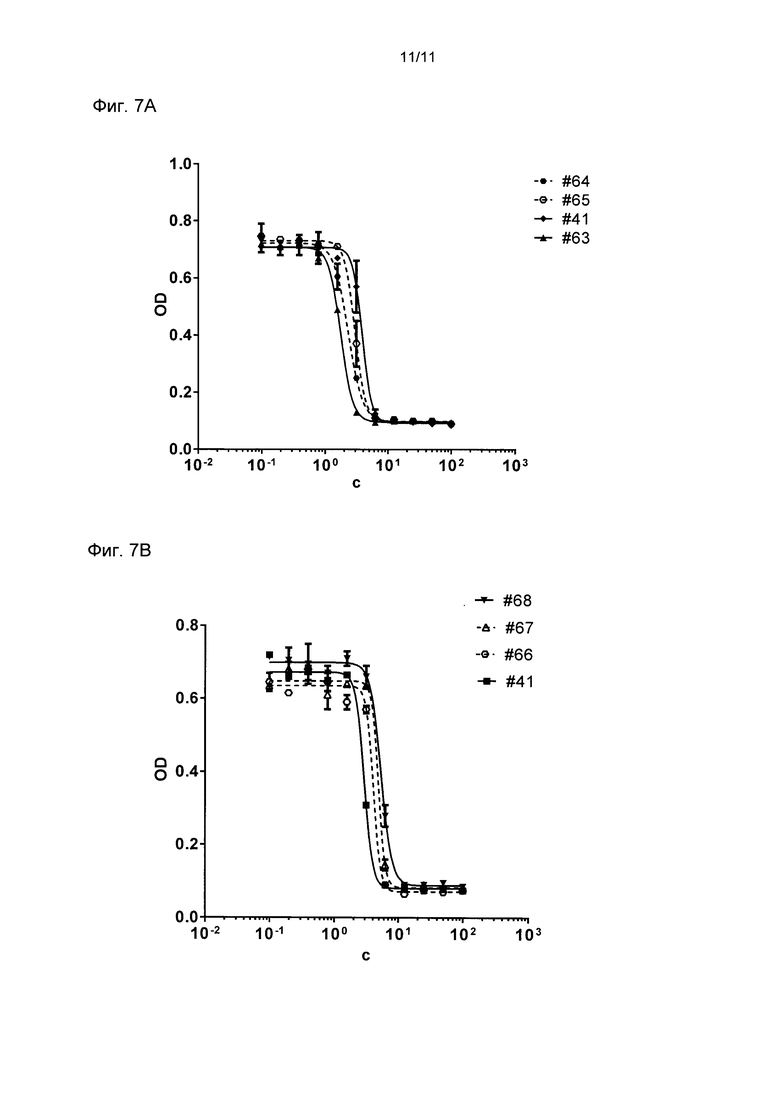
Изобретение относится к области биохимии, в частности к рекомбинантному связывающему HER2 белку, содержащему по меньшей мере первую и вторую области анкириновых повторов, каждая из которых связывает внеклеточную область HER2. При этом первая область анкиринового повтора связывает область II HER2, а вторая область анкиринового повтора связывает область IV HER2, первая и вторая области анкиринового повтора находятся в одном и том же полипептиде, первая область анкиринового повтора находится на N-конце второй области анкиринового повтора. Также раскрыта нуклеиновая кислота, кодирующая вышеуказанный связывающий белок. Изобретение также относится к фармацевтической композиции, содержащей вышеуказанный связывающий белок, а также к способу лечения рака, характеризующегося амплификацией кодирующего HER2 гена и/или сверхэкспрессией кодирующего HER2 гена, с ее использованием. Изобретение позволяет эффективно лечить рак, характеризующийся амплификацией кодирующего HER2 гена и/или сверхэкспрессией кодирующего HER2 гена. 6 н. и 26 з.п. ф-лы, 7 ил., 3 табл., 8 пр.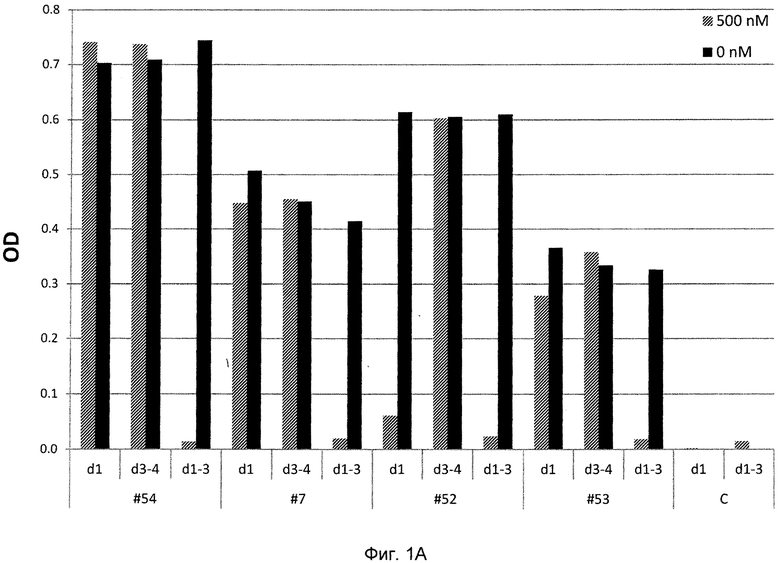
1. Рекомбинантный белок для связывания HER2, содержащий по меньшей мере первую и вторую области анкириновых повторов, каждая из которых связывает внеклеточную область HER2, при этом первая область анкиринового повтора связывает область II HER2, а вторая область анкиринового повтора связывает область IV HER2, первая и вторая области анкиринового повтора находятся в одном и том же полипептиде, первая область анкиринового повтора находится на N-конце второй области анкиринового повтора, и
(а) первая область анкиринового повтора содержит аминокислотную последовательность, которая по меньшей мере на 91% идентична аминокислотной последовательности области анкиринового повтора, выбранной из группы, включающей SEQ ID 62-68, 72 и 114-121, и
(б) вторая область анкиринового повтора содержит аминокислотную последовательность, которая по меньшей мере на 91% идентична аминокислотной последовательности области анкиринового повтора, выбранной из группы, включающей SEQ ID с 74 по 82.
2. Рекомбинантный белок по п. 1, в котором первая область анкиринового повтора не конкурирует с пертузумабом за связывание HER2.
3. Рекомбинантный белок по п. 1, в котором вторая область анкиринового повтора не конкурирует с трастузумабом за связывание HER2.
4. Рекомбинантный белок по п. 1, который ингибирует стимулированную пролиферацию клеток ВТ474 при показателе IC50 менее 100 нМ.
5. Рекомбинантный белок по п. 1, который индуцирует апоптоз в клетках ВТ474 при показателе ЕС50 менее 100 нМ.
6. Рекомбинантный белок по п. 1, в котором первая и вторая области анкиринового повтора соединены полипептидным линкером, который содержит аминокислотную последовательность SEQ ID 11.
7. Рекомбинантный белок по п. 1, в котором
а) первая область анкиринового повтора содержит аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 62-68, 72 и 114-121, и/или
б) вторая область анкиринового повтора содержит аминокислотную последовательность, которая по меньшей мере на 95% идентична аминокислотной последовательности одной области анкиринового повтора, выбранной из группы, включающей SEQ ID 74-82.
8. Рекомбинантный белок по п. 1, в котором
а) первая область анкиринового повтора конкурирует за связывание HER2 с областью анкиринового повтора SEQ ID 115, и/или
б) вторая область анкиринового повтора конкурирует за связывание HER2 с областью анкиринового повтора SEQ ID 81.
9. Рекомбинантный белок по п. 1, в котором одна или несколько аминокислот изменены следующим образом:
а) G в положении 1 и/или S в положении 2 упомянутых областей анкиринового повтора отсутствуют; и/или
б) L в предпоследнем положении и/или N в последнем положении упомянутых областей анкиринового повтора заменены на А.
10. Рекомбинантный белок по п. 1, в котором
а) первая область анкиринового повтора выбрана из группы, включающей SEQ ID 62-68, 72 и 114-121, и/или
б) вторая область анкиринового повтора выбрана из группы, включающей SEQ ID 74-82,
и в котором дополнительно
а) G в положении 1 и/или S в положении 2 упомянутых областей анкиринового повтора необязательно отсутствуют; и
б) L в предпоследнем положении и/или N в последнем положении упомянутых областей анкиринового повтора необязательно заменены на А.
11. Рекомбинантный белок по п. 1, в котором первая область анкиринового повтора содержит аминокислотную последовательность SEQ ID 115, в которой G в положении 1 и/или S в положении 2 первой области анкиринового повтора необязательно отсутствуют; и в которой L в предпоследнем положении и/или N в последнем положении первой области анкиринового повтора необязательно заменены на А.
12. Рекомбинантный белок по п. 1, в котором вторая область анкиринового повтора содержит аминокислотную последовательность SEQ ID 81, в которой G в положении 1 и/или S в положении 2 второй области анкиринового повтора необязательно отсутствуют; и в которой L в предпоследнем положении и/или N в последнем положении второй области анкиринового повтора необязательно заменены на А.
13. Рекомбинантный белок по п. 1, в котором
а) первая область анкиринового повтора содержит модуль анкиринового повтора, имеющий аминокислотную последовательность, выбранную из группы, включающей (a) SEQ ID 15-18, 21-23, 37, 38, 125, 126, 129, 130, 133 и 134 и (б) последовательности, в которых до 9 аминокислотных остатков в SEQ ID 15-18, 21-23, 37, 38, 125, 126, 129, 130, 133 и 134 заменены любыми другими аминокислотными остатками, и/или
б) вторая область анкиринового повтора содержит модуль анкиринового повтора, имеющий аминокислотную последовательность, выбранную из группы, включающей (a) SEQ ID 46, 47, 51, 52, 55 и 56 и (б) последовательности, в которых до 4 аминокислотных остатков в SEQ ID 46, 47, 51, 52, 55 и 56 заменены любыми другими аминокислотными остатками.
14. Рекомбинантный белок по п. 1, в котором первая область анкиринового повтора содержит модуль анкиринового повтора, имеющий аминокислотную последовательность, выбранную из группы, включающей (а)
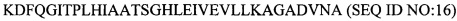 и (б)
и (б)
последовательности, в которых по меньшей мере один аминокислотный остаток в SEQ ID 16 изменен следующим образом:
а) F в положении 3 необязательно заменена А
б) Q в положении 4 необязательно заменена Е;
в) G в положении 5 необязательно заменена S;
г) I в положении 6 необязательно заменена V;
д) I в положении 11 необязательно заменена L;
е) Т в положении 14 необязательно заменена Q; и/или
ж) S в положении 15 заменена аминокислотой, выбранной из группы, включающей N и W.
15. Рекомбинантный белок по п. 1, в котором первая область анкиринового повтора содержит модуль анкиринового повтора, имеющий аминокислотную последовательность, выбранную из группы, включающей (а)
 и (б)
и (б)
последовательности, в которых по меньшей мере один аминокислотный остаток в SEQ ID 18 изменен следующим образом:
а) I в положении 3 необязательно заменена V;
б) Е в положении 6 необязательно заменена D;
в) Н в положении 11 необязательно заменена L;
г) D в положении 14 необязательно заменена Q;
д) S в положении 15 необязательно заменена Н; и/или
е) Е в положении 19 необязательно заменена V.
16. Рекомбинантный белок по п. 1, в котором первая область анкиринового повтора содержит модуль анкиринового повтора, имеющий аминокислотную последовательность, выбранную из группы, включающей SEQ ID 125, и последовательности, в которых до 4 аминокислотных остатков в SEQ ID 125 заменены любыми другими аминокислотными остатками.
17. Рекомбинантный белок по п. 1, в котором первая область анкиринового повтора содержит модуль анкиринового повтора, имеющий аминокислотную последовательность, выбранную из группы, включающей SEQ ID 126, и последовательности, в которых до 4 аминокислотных остатков в SEQ ID 126 заменены любыми другими аминокислотными остатками.
18. Рекомбинантный белок по п. 1, в котором вторая область анкиринового повтора содержит модуль анкиринового повтора, имеющий аминокислотную последовательность SEQ ID 55, и/или содержит модуль анкиринового повтора, имеющий аминокислотную последовательность SEQ ID 56.
19. Рекомбинантный белок для связывания HER2, содержащий по меньшей мере первую и вторую области анкиринового повтора, каждая из которых связывает внеклеточную область HER2, при этом первая область анкиринового повтора связывает область II HER2, а вторая область анкиринового повтора связывает область IV HER2, первая и вторая области анкиринового повтора находятся в одном и том же полипептиде, первая область анкиринового повтора находится на N-конце второй области анкиринового повтора, и рекомбинантный белок содержит аминокислотную последовательность, которая по меньшей мере на 90% идентична аминокислотной последовательности полипептида, выбранной из группы, включающей SEQ ID 83-98, 102, 103, 122, 123 и 136-141.
20. Рекомбинантный белок по п. 19, в котором аминокислотная последовательность по меньшей мере на 95% идентична аминокислотной последовательности полипептида, выбранной из группы, включающей SEQ ID 83-98, 102, 103, 122, 123 и 136-141.
21. Рекомбинантный белок по п. 19, в котором аминокислотная последовательность по меньшей мере на 90% идентична аминокислотной последовательности полипептида SEQ ID 87.
22. Рекомбинантный белок по п. 19, в котором аминокислотная последовательность идентична аминокислотной последовательности SEQ ID 87.
23. Рекомбинантный белок по п. 1, в котором первая и вторая области анкиринового повтора связывают внеклеточную область HER2 в PBS при Kd менее 10-7 М, при этом PBS представляет собой забуференный фосфатом водный раствор, содержащий 137 мМ NaCl, 10 мМ фосфтата и 2,7 мМ КСl и имеющий рН 7,4.
24. Рекомбинантный белок по п. 19, в котором первая и вторая области анкиринового повтора связывают внеклеточную область HER2 в PBS при Кd менее 10-7 М, при этом PBS представляет собой забуференный фосфатом водный раствор, содержащий 137 мМ NaCl, 10 мМ фосфтата и 2,7 мМ КСl и имеющий рН 7,4.
25. Рекомбинантный белок для ингибирования HER2, содержащий по меньшей мере первую и вторую области анкириновых повторов, каждая из которых связывает внеклеточную область HER2, при этом первая область анкиринового повтора связывает область II HER2, а вторая область анкиринового повтора связывает область IV HER2, первая и вторая области анкиринового повтора находятся в одном и том же полипептиде, первая область анкиринового повтора находится на N-конце второй области анкиринового повтора, и рекомбинантный белок содержит аминокислотную последовательность, которая по меньшей мере на 90% идентична аминокислотной последовательности полипептида, выбранной из группы, включающей SEQ ID: 83-85, 87-90, 93-99, 101-103, 106-107 и 136-141.
26. Рекомбинантный белок по п. 25, в котором аминокислотная последовательность по меньшей мере на 95% идентична аминокислотной последовательности полипептида, выбранной из группы, включающей SEQ ID: 83-85, 87-90, 93-99, 101-103, 106-107 и 136-141.
27. Рекомбинантный белок по п. 25, в котором аминокислотная последовательность по меньшей мере на 90% идентична аминокислотной последовательности полипептида SEQ ID NO: 87.
28. Рекомбинантный белок по п. 25, в котором аминокислотная последовательность по меньшей мере на 95% идентична аминокислотной последовательности полипептида SEQ ID NO: 87.
29. Рекомбинантный белок по п. 25, в котором аминокислотная последовательность идентична аминокислотной последовательности SEQ ID NO: 87.
30. Нуклеиновая кислота, кодирующая рекомбинантный белок по п. 25.
31. Фармацевтическая композиция для лечения рака, содержащая терапевтически эффективное количество рекомбинантный белка по любому из пп. 4-5 и 25-29 и фармацевтически приемлемый носитель, при этом рак характеризуется по меньшей мере одним фактором, выбранным из группы, включающей:
(а) амплификацию кодирующего HER2 гена и
(б) сверхэкспрессию кодирующего HER2 гена.
32. Способ лечения рака у пациента, включающий введение эффективного количества фармацевтической композиции по п. 31 нуждающемуся в этом субъекту, страдающему раком, при этом рак характеризуется по меньшей мере одним фактором, выбранным из группы, включающей:
(а) амплификацию кодирующего HER2 гена и
(б) сверхэкспрессию кодирующего HER2 гена.
33. Способ по п. 32, в котором рак выбран из группы, включающей
(а) рак молочной железы,
(б) рак яичников,
(в) рак желудочно-кишечного тракта,
(г) рак желудка,
(д) рак матки и
(е) колоректальный рак.
| NIELSEN U.B | |||
| et al | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Способ получения молочной кислоты | 1922 |
|
SU60A1 |
| Машина для добывания торфа и т.п. | 1922 |
|
SU22A1 |
| Способ повышения способности скольжения фильмы | 1926 |
|
SU6434A1 |
| ZAHND et al | |||
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Прибор для сжигания нефти | 1921 |
|
SU369A1 |
| Очаг для массовой варки пищи, выпечки хлеба и кипячения воды | 1921 |
|
SU4A1 |
| ГАЛЬВАНИЧЕСКИЙ ЭЛЕМЕНТ | 1923 |
|
SU1015A1 |
| STUMPP MT et al | |||
| DARPins: a true alternative to antibodies, CURRENT OPINION IN DRUG DISCOVERY AND DEVELOPMENT, CURRENT DRUGS, 2007, vol | |||
| Печь-кухня, могущая работать, как самостоятельно, так и в комбинации с разного рода нагревательными приборами | 1921 |
|
SU10A1 |
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Паровозный золотник (байпас) | 1921 |
|
SU153A1 |
| КОМПОЗИЦИЯ АНТИТЕЛ К HER2 | 2005 |
|
RU2361880C2 |
Авторы
Даты
2018-08-17—Публикация
2013-12-02—Подача