ПЕРЕКРЕСТНАЯ ССЫЛКА НА РОДСТВЕННЫЕ ЗАЯВКИ
[0001] Данная заявка заявляет приоритет по предварительной заявке США № 62/305 092, поданной 8 марта 2016 года, которая полностью включена посредством ссылки во всей ее полноте для всех целей.
УРОВЕНЬ ТЕХНИКИ
[0002] Селективное уничтожение отдельной клетки или определенного типа клеток часто желательно в различных клинических условиях. Например, основной целью терапии рака является специфическое уничтожение опухолевых клеток, и в то же время сохранение здоровых клеток и тканей целыми и неповрежденными, насколько это возможно. Одним из таких способов является индукция иммунного ответа против опухоли для того, чтобы заставить иммунные эффекторные клетки, такие как натуральные клетки-киллеры (NK) или цитотоксические Т-лимфоциты (ЦТЛ), атаковать и уничтожать опухолевые клетки.
[0003] Использование интактных моноклональных антител (мкАТ), которые обеспечивают превосходную специфичность и аффинность связывания к/с опухолеассоциированному антигену, успешно применяются в области лечения и диагностики рака. Тем не менее большой размер интактных мкАТ, их плохое биораспределение и продолжительное время жизни в пуле крови ограничивают их применения в медицинской практике. Например, интактные антитела могут проявлять специфическое накопление в области опухоли. В исследованиях биораспределения при точном исследовании опухоли отмечается неоднородное распределение антител с первичным накоплением в периферических областях. Из-за некроза опухолей, неоднородного распределения антигена и увеличения внутритканевого давления невозможно достичь центральных участков опухоли с помощью конструкций интактных антител. Напротив, более мелкие фрагменты антител быстро демонстрируют локализацию опухоли, проникают глубже в опухоль, а также удаляются относительно быстро из кровотока.
[0004] Одноцепочечные фрагменты (scFv), полученные из малого связывающего домена исходного мкАТ, обеспечивают лучшее биораспределение, чем интактные мкАТ для применения в медицинской практике, и могут более эффективно нацеливаться на опухолевые клетки. Одноцепочечные фрагменты могут быть эффективно сконструированы из бактерий, однако большинство сконструированных scFv имеют моновалентную структуру и демонстрируют снижение накопления внутри опухоли, например, короткое время пребывания в опухолевой клетке, и специфичность по сравнению с их исходным мкАТ ((C(c), D) из-за недостаточности авидности, которую проявляют двухвалентные соединения.
[0005] Несмотря на благоприятные свойства scFv, некоторые функции препятствуют их полноценному клиническому применению в химиотерапии рака. Особо следует отметить их перекрестную реактивность между пораженной и здоровой тканью из-за нацеленности этих агентов на рецепторы клеточной поверхности, общие как для пораженной, так и для здоровой ткани. ScFv с улучшенным терапевтическим индексом могли бы значительно повысить клиническую полезность этих агентов. Данное изобретение обеспечивает такие улучшенные scFv и способы их изготовления и применения. Улучшенные scFv согласно изобретению имеют неожиданное преимущество для преодоления недостаточности авидности, проявляемой одной единицей, путем образования димерного соединения.
СУЩНОСТЬ ИЗОБРЕТЕНИЯ
[0006] В различных вариантах осуществления данное изобретение относится к состоящим из двух частей полипептидам. Со ссылкой на фиг. 53, в иллюстративных вариантах осуществления, две области полипептида соединены региональным линкером scFv (RL), находящимся в диапазоне от одной связи до более крупного полипептидного домена, который может содержать один или более расщепляемых линкеров (CL) с одним или более сайтами расщепления, что обеспечивает разделение этих двух областей при расщеплении. Каждая из двух областей полипептида содержит один или более болезнь-нацеленных доменов (например, домены, связывающие целевой антиген, которые могут представлять собой любой формат одноцепочечного связывающего домена, включая scFv, sdAb (однодоменное антитело), домены клеточных рецепторов, лектины и т.п.), связанные посредством по меньшей мере одного нерасщепляемого линкера (NCL1 и NCL2) с инактивированным scFv, нацеленным на белок активации Т-клеток (αCD3, αCD16, αTCRα, αTCRβ, αCD28 и т.п.). ScFv, нацеленные на домены активации Т-клеток, инактивируются или в их VH- или VL-сегментах, и два сегмента каждого scFv соединены с помощью расщепляемого линкера (CL1 и CL2), который восприимчив к расщеплению в больной ткани.
[0007] Конструкции антигенсвязывающего полипептида, описанные в данном документе, придают множественные терапевтические преимущества по сравнению со стандартными моноклональными антителами и другими более мелкими биспецифическими молекулами. Особо следует отметить условную активацию полипептидных конструкций согласно данному изобретению. Конструкции по существу способны связывать свои целевые антигены, однако активность сигналинга CD3 зависит от уникальной стадии деградации полипептида, запрограммированной в структуре самого полипептида. Таким образом, специфическая активность к непораженной, здоровой ткани иллюстративных полипептидов согласно изобретению значительно снижается по сравнению с таковой аналогичных антител и фрагментов антител. Способность полипептидов «включаться» в желаемом месте их действия, оставаясь «молчаливым» во время их продвижения к этому месту, является заметным достижением в области специфически связывающихся полипептидных терапевтических средств, обладающих потенциалом сильнодействующих и специфических лекарственных препаратов в легко оформляемом и представимом лекарственном формате.
[0008] Как правило, эффективность рекомбинантных полипептидных фармацевтических препаратов часто ограничивается собственной быстрой фармакокинетикой самого полипептида, что приводит к быстрому клиренсу полипептида. Дополнительным преимуществом, обеспечиваемым иллюстративными антигенсвязывающими полипептидами согласно изобретению, является увеличенный фармакокинетический период полувыведения из-за наличия домена увеличения периода полувыведения, например, связывающего домена, специфически связывающегося с ЧСА (человеческий сывороточный альбумин). В этом отношении иллюстративные антигенсвязывающие полипептиды согласно изобретению имеют увеличенный период полувыведения в сыворотке. Иллюстративные полипептидные конструкции данного мотива имеют периоды полувыведения около двух, трех, около пяти, около семи, около десяти, около двенадцати или около четырнадцати суток в некоторых вариантах осуществления. Это выгодно контрастирует с другими связывающими белками, такими как молекулы BiTE (биспецифические активаторы T-клеток) или DART (переориентирующееся антитело с двойной аффинностью), которые имеют относительно более короткие периоды полувыведения. Например, BiTE CD19 × CD3-биспецифическая scFv-scFv молекула слияния требует доставка лекарственного средства путем непрерывной внутривенной инфузии (в/в) из-за ее короткого периода полувыведения. Более длительные собственные периоды полувыведения иллюстративных антигенсвязывающих полипептидов согласно изобретению устраняют этот недостаток, тем самым позволяя увеличить терапевтический потенциал, например, низкодозированных фармацевтических препаратов, уменьшенное периодическое введение и/или новые фармацевтические композиции, содержащие соединения согласно изобретению.
[0009] Иллюстративные антигенсвязывающие полипептиды согласно изобретению также имеют оптимальный размер для улучшенного проникновения и распределения в ткани и уменьшения почечного клиренса при первом прохождении. Поскольку почка обычно отфильтровывает молекулы меньше около 50 кДа, усилия по уменьшению клиренса в дизайне белковых терапевтических средств направлены на увеличение молекулярного размера посредством слияния белков, гликозилирования или добавления полимеров полиэтиленгликоля (т.е. ПЭГ). Однако несмотря на то, что увеличение размера белкового терапевтического средства может предотвратить почечный клиренс, больший размер также предотвращает проникновение молекулы в целевые ткани. Иллюстративные антигенсвязывающие полипептиды, описанные в данном документе, избегают этого путем связывания с альбумином, который предотвращает быстрый почечный клиренс, и в то же время имеющие малый размер, обеспечивающий повышенное проникновение и распределение в ткани и оптимальную эффективность. В различных вариантах осуществления домен увеличения периода полувыведения помещается в положение в молекуле, в котором он отделен от терапевтически активного компонента расщепляемым линкером. Так, например, при достижении желаемой мишени, в которой агент расщепляет линкер (например, протеаза, эстераза, восстановительное или окислительное микроокружение), домен увеличения периода полувыведения отщепляется от терапевтически активного компонента, уменьшая размер терапевтического компонента и способствуя его проникновению в ткани или поглощению клетками. В других вариантах осуществления домен увеличения периода полувыведения будет помещен между антигенсвязывающим доменом и активным анти-CD-3 доменом.
[0010] Таким образом, в иллюстративном варианте осуществления данное изобретение относится к одноцепочечному полипептиду scFv, направленному на антиген CD-3. Полипептид scFv содержит первый домен scFv и второй домен scFv, связанный через расщепляемый линкер scFv. Первый домен scFv содержит первый домен VH1 и первый домен VL1, соединенный через расщепляемый первый линкерный фрагмент scFv. Один из VL или VH является неактивным, как этот термин определен в данном документе (то есть VL1i, VH1i). Первый домен VH и первый домен VL взаимодействуют с образованием первого scFv, однако из-за неактивного родства scFv не связывает специфически CD-3. Первый линкерный фрагмент scFv (например, CL1) содержит первый сайт расщепления протеазой между первым VH1- и первым VL1- доменом. При протеазном расщеплении первого scFv-линкера в сайте расщепления протеазой неактивный VHi- или неактивный VLi-домен отделяется от его партнера по связыванию VL или VH, который затем соединяется с его активным родственным вариантом, обеспечивая правильно спаренный анти-CD-3 для образования и связывания антигена CD-3. Домен, связывающий целевой антиген, связан через линкер с активной родственной парой VH/VL.
[0011] В иллюстративном варианте осуществления первый домен scFv соединяется через первый линкерный фрагмент, необязательно содержащий второй сайт расщепления (например, сайт расщепления протеазой) ко второму домену scFv. Второй домен scFv структурирован подобно первому домену и содержит второй домен VH и второй домен VL, соединенный через второй линкерный фрагмент scFv. Второй линкерный фрагмент scFv необязательно содержит третий сайт расщепления протеазой между вторым доменом VH и вторым доменом VL. Второй домен VH и второй домен VL взаимодействуют с образованием второй пары VH/VL. Как и в случае первой пары VH/VL, описанной выше, один из второго домена VH и второго VL является неактивным, так что второй домен scFv не связывает специфически антиген CD-3, и комплекс между первым и вторым scFv-связывающими доменами. Второй домен scFv соединен через второй доменный линкер со вторым доменом, связывающим целевой антиген. Этот второй доменный линкер соединяет член, выбранный из первого домена VH и указанного первого домена VL, со вторым доменом, связывающим целевой антиген. Домен, связывающий целевой антиген, связан через линкер с активной родственной парой VH/VL.
[0012] Полипептидная конструкция согласно изобретению расщепляется в расщепляемых линкерах и образуется активный CD-3-связывающий домен, отображающий в присутствии клетки антиген CD-3, связывается с антигеном CD-3. Аналогично, домены, связывающие целевой антиген, связываются с целевым антигеном.
[0013] В иллюстративном варианте осуществления изобретение обеспечивает одноцепочечный полипептид scFv, имеющий единственный домен scFv, который направлен на антиген CD-3. Полипептид scFv содержит первый домен scFv, содержащий первый домен VH и первый домен VL, присоединенный через первый линкерный фрагмент scFv. Этот первый линкерный фрагмент scFv содержит первый сайт расщепления, например, сайт расщепления протеазой, между первым VH и первым доменом VL. Первый домен VH и первый домен VL взаимодействуют с образованием первой пары VH/VL, в которой один из первого домена VH и первого домена VL является неактивным. Соответственно, первый домен scFv не способен специфически связывать антиген CD-3. Первый полипептид scFv соединяют с помощью первого линкерного фрагмента домена с первым доменом, связывающим целевой антиген. Этот первый доменный линкер соединяет элемент, выбранный из первого домена VH и первого домена VL, с первым доменом, связывающим целевой антиген. Первый домен, связывающий целевой антиген, не связывается с неактивным VL или неактивным VH.
[0014] В иллюстративном варианте осуществления предложена пара однодоменных scFv-конструкций, описанных выше. Пара конструкций взаимодействует с антигеном CD-3 через их спаренные CD-3-связывающие домены. Связывание с антигеном CD-3 спаренных сайтов CD-3 отдельных молекул scFv пары облегчается, усиливается и/или управляется связыванием домена, связывающего целевой антиген, каждого члена пары с его родственным антигеном.
[0015] В некоторых вариантах осуществления обеспечивается антигенсвязывающий полипептид, содержащий единственную полипептидную цепь, содержащую два или более обратимо неактивных CD3-связывающих домена, два или более доменов, связывающих целевой антиген, необязательно один или более доменов увеличения периода полувыведения, и один или более доменов расщепления протеазой; причем при протеазном расщеплении домена расщепления протеазой, CD3-связывающий домен становится активным и связывается с CD3. В иллюстративном варианте осуществления CD3-связывающий домен становится активным и способен связываться с CD3 после расщепления сайта расщепления протеазой. В различных вариантах осуществления CD3-связывающий домен становится активным после расщепления сайта расщепления протеазой и связывания целевого антигена(ов) при помощи домена, связывающего целевой антиген. В некоторых вариантах осуществления связывание с CD3 активирует Т-клетку, которая, в свою очередь, разрушает пораженную (например, раковую) клетку.
[0016] В иллюстративном варианте осуществления полипептидные конструкции согласно изобретению включают scFv, содержащую связывающий домен, селективно связывающийся с CD3. CD3-связывающий домен включает VH или VL, который способен селективно связываться с CD3. Этот VH или VL сопряжен с VL или VH, соответственно.
[0017] Полипептиды согласно изобретению проиллюстрированы в данном документе ссылкой на условный CD3-связывающий полипептид, содержащий scFv, содержащий CD3-связывающий домен(ы) и сайт(ы) расщепления протеазой, который после расщепления протеазой отделяет неактивные VL или VH от их сопряженного активного VH или VL, соответственно, активируя CD3-связывающий домен(ы) и обеспечивая его(их) связывание с CD3. Репрезентативный scFv содержит домен VH и домен VL, связанный через полипептидный линкер, содержащий сайт расщепления протеазой. CD3-связывающий домен обратимо неактивен и поэтому практически не способен связываться с CD3 до протеазного расщепления сайта расщепления протеазой. Репрезентативная протеаза, способная расщеплять сайт расщепления протеазой, представляет собой протеазу, экспрессируемую раковой клеткой или локализованной в микроокружении опухоли. В иллюстративном варианте осуществления полипептид согласно изобретению дополнительно содержит по меньшей мере один целевой антигенсвязывающий сайт. Репрезентативный целевой антиген представляет собой антиген, обнаруженный на поверхности раковой клетки, например, EGFR.
[0018] В некоторых вариантах осуществления домен расщепления протеазой расщепляется до того, как домены, связывающие целевой антиген(ы), связываются с целевым антигеном(ами). В некоторых вариантах осуществления домен расщепления протеазой расщепляется после присоединения антигенсвязывающего домена(ов) с целевыми антигенами. В некоторых вариантах осуществления полипептид содержит два или более доменов, связывающих целевой антиген. Два или более антигенсвязывающих домена имеют одинаковую или различную полипептидную последовательность. В различных вариантах осуществления два или более антигенсвязывающих домена имеют одинаковую или различную полипептидную последовательность и связывают один и тот же целевой антиген. В иллюстративном варианте осуществления полипептидная последовательность двух или более антигенсвязывающих доменов различается и два или более доменов связываются с одним и тем же целевым антигеном или с другим целевым антигеном. В некоторых вариантах осуществления каждый из двух или более доменов, связывающих целевой антиген, связывается с целевыми антигенами различной последовательности или структуры в одной и той же клетке. В иллюстративном варианте осуществления каждый из двух или более доменов, связывающих целевой антиген, связывается с антигенами различной последовательности на каждой из двух или более клеток. В различных вариантах осуществления каждый из двух или более антигенсвязывающих доменов связывается с антигеном той же последовательности или с другой последовательностью на каждой из двух или более клеток.
[0019] В данном документе описаны условно связывающие антигенсвязывающие полипептиды, их фармацевтические композиции, а также нуклеиновые кислоты, рекомбинантные векторы экспрессии и клетки-хозяева для получения таких антигенсвязывающих полипептидов и способы лечения заболеваний, расстройств или патологических состояний с использованием антигенсвязывающих полипептидов согласно изобретению.
[0020] Другие цели, варианты осуществления и преимущества данного изобретения очевидны в подробном описании ниже.
КРАТКОЕ ОПИСАНИЕ ЧЕРТЕЖЕЙ
[0021] Новые признаки изобретения подробно изложены в прилагаемой формуле изобретения. Более полное понимание особенностей и преимуществ данного изобретения будет получено со ссылкой на следующее подробное описание, в котором представлены иллюстративные варианты осуществления, в которых используются принципы изобретения, и сопровождающие их графические материалы:
[0022] На фиг. 1А изображены профили ДСН-ПААг-электрофореза транзиентно экспрессированных Prodent 1-4.
[0023] На фиг. 1B изображены уровни экспрессии Pro1-4, рассчитанные по результатам измерений после диализа.
[0024] На фиг. 2А изображена аналитическая эксклюзионная хроматография очищенных белков.
[0025] На фиг.2В изображена аналитическая эксклюзионная хроматография очищенных белков.
[0026] На фиг. 2С изображена аналитическая эксклюзионная хроматография очищенных белков.
[0027] На фиг. 2D изображена аналитическая эксклюзионная хроматография очищенных белков.
[0028] На фиг. 3 изображена Pro5: Prodent Платформа 2.
[0029] На фиг. 4 изображены Pro6 и Pro7: бифункциональные партнеры. На фиг. 4 подтверждено, что введение сайта расщепления ЭК (энтерокиназа) в CDR2 VH или VL в анти-CD3 scFv нейтрализуют связывание и активность CD-3.
[0030] На фиг. 5 изображен Pro8: положительный контроль. На фиг. 5 подтверждено, что вставка ЭК сайта в линкер scFv не мешает фолдингу scFv и связыванию CD-3.
[0031] На фиг. 6 изображены Prodent 5-8 - транзиентная экспрессия в Expi293.
[0032] На фиг. 7 представлены данные, демонстрирующие, что очищенные Prodent 5-8 демонстрируют мономерные профили в ЭХ. На фиг. 7А изображен Pro 5 - G8: (I2ci) x2:D12::His6.
[0033] На фиг. 7 представлены данные, демонстрирующие, что очищенные Prodent 5-8 демонстрируют мономерные профили в ЭХ. На фиг. 7B изображен Pro 6 - G8 (sdAb): I2Ci::His6.
[0034] На фиг. 7 изображены очищенные Prodent 5-8, демонстрирующие мономерные профили в ЭХ. На фиг. 7C изображен Pro 7 - I2Ci: D12 (sdAb)::His6.
[0035] На фиг. 7 представлены данные, демонстрирующие, что очищенные Prodent 5-8 демонстрируют мономерные профили в ЭХ. На фиг. 7D изображен Pro 8 - G8 (sdAb): I2Cflag::His6.
[0036] На фиг. 8 изображены белки платформы 2 на ДСН-ПААГ-электрофорезе, очищенные Ni-excel.
[0037] На фиг. 9 изображены четыре типа анализа связывания/активности.
[0038] На фиг. 10А изображены Prodent платформы 2, связывающиеся с hEGFR. На фиг. 10А изображено, что Prodent связываются с EGFR-ELISA (rhEGFR-Fc, анти-His-HRP определение.
[0039] На фиг. 10B изображены Prodent платформы 2 связывается с hEGFR. На фиг. 10B изображены Prodent с EGFR-FACS OVCAR8 анти-His ФИТЦ-определение.
[0040] На фиг. 11A изображены неактивные Prodent платформы 2, которые не связываются с CD3. На фиг. 11А изображено, что Prodent связываются с CD3-ELISA (cyCD3-Flag-Fc, анти-His-HRP определение.
[0041] На фиг. 11B изображены неактивные Prodent платформы 2, которые не связываются с CD3. На фиг. 11A изображено, что Prodent, связывающиеся с CD3, определяются с помощью FACS jurkat анти-His-ФИТЦ обнаружения.
[0042] На фиг. 12 изображены Pro6 и Pro7: активация связывания CD3 путем протеазного расщепления.
[0043] На фиг. 13 изображено расщепление Prodent при помощи рекомбинантной энтерокиназы.
[0044] На фиг. 14А изображен формат анализа методом ИФА для исследования связывания prodent с CD3 после расщепления ЭК (сэндвич-ИФА).
[0045] На фиг. 14B изображено, что Pro 6 не связывается с CD3 после расщепления ЭК (сэндвич-ИФА). На фиг. 14B изображено связывание Pro6 с rEGFR::huFC, обнаруженное с помощью биотин-cyCD3::Flag::huFC, SAV-HRP.
[0046] На фиг. 14C изображено, что Pro7 не связывается с CD3 после расщепления ЭК (сэндвич-ИФА). На фиг. 14C изображено связывание Pro7 с rEGFR::huFC, обнаруженное с помощью биотин-cyCD3::Flag::huFC, SAV-HRP.
[0047] На фиг. 14D изображено, что Pro 6+Pro7 cовместно связываются с CD3 после расщепления ЭК (сэндвич-ИФА). На фиг. 14D изображено связывание Pro6+Pro7 с rEGFR::huFC, обнаруженное с помощью биотин-cyCD3::Flag::huFC, SAV-HRP.
[0048] На фиг. 14E изображено, что Pro 6+Pro7 cовместно связываются с CD3 после расщепления ЭК (сэндвич-ИФА).
[0049] На фиг. 15А изображен формат анализа методом проточной цитометрии для исследования связывания с CD3 после расщепления ЭК на поверхности EGFR-экспрессирующих клеток (сэндвич-FACS).
[0050] На фиг. 15B изображено, что Pro 6 не связывается с CD3 после расщепления ЭК (сэндвич-FACS). На фиг. 15B изображено связывание ЭК-расщепленного Pro6 с OVCAR-8, обнаруженное с помощью A488-cyCD3::Flag::hFC.
[0051] На фиг. 15C изображено, что Pro7 не связывается с CD3 после расщепления ЭК (сэндвич-FACS). На фиг. 15C изображено связывание ЭК-расщепленного Pro7 с OVCAR-8, обнаруженное с помощью A488-cyCD3::Flag::hFC.
[0052] На фиг. 15D изображено, что Pro 6+Pro7 cовместно связываются с CD3 после расщепления ЭК (сэндвич-FACS). На фиг. 15D изображено связывание ЭК-расщепленного Pro6+Pro7 с OVCAR-8, обнаруженное с помощью A488-cyCD3::Flag::huFC.
[0053] На фиг. 15E изображено, что Pro 6+Pro7 cовместно связываются с CD3 после расщепления ЭК (сэндвич-FACS).
[0054] На фиг. 16 изображено, что связывание CD3 с помощью Pro 5 активируется после протеолитического расщепления при помощи ЭК. Связывание CD3 с помощью Pro 5 активируется после протеолитического расщепления при помощи ЭК. На фиг. 16 изображено связывание ЭК-расщепленного Pro5 с OVCAR-8, обнаруженное с помощью A488-cyCD3::Flag::huFC.
[0055] На фиг. 17 изображен Pro8: контрольная молекула. На фиг. 17 подтверждено, что вставка ЭК сайта в линкер scFv не мешает фолдингу scFv и связыванию CD3.
[0056] На фиг. 18A изображен Pro8: контрольная молекула. На фиг. 18A подтверждено, что вставка ЭК сайта в линкер scFv не мешает фолдингу scFv и связыванию CD3. На фиг. 18A изображено связывание Pro8 с rhEGFR::hFC, обнаруженное с помощью биотин-cyCD3::Flag::huFC, SAV-HRP.
[0057] На фиг. 18B изображен Pro8: контрольная молекула. На фиг. 18B подтверждено, что вставка ЭК сайта в линкер scFv не мешает фолдингу scFv и связыванию CD3. На фиг. 18B изображено связывание ЭК-расщепленного Pro8 с OVCAR-8, обнаруженное с помощью A488-cyCD3::flag::hFC.
[0058] На фиг. 18C изображен Pro8: молекула положительного контроля.
[0059] На фиг. 19 изображено, что расщепление ЭК совместно активирует Т-клеточное уничтожение EGFR+целевых клеток с помощью Pro6+Pro7, но уменьшает цитолиз с помощью Pro8. На фиг. 19А изображены результаты для Pro6.
[0060] На фиг. 19 изображено, что расщепление ЭК совместно активирует Т-клеточное уничтожение EGFR+целевых клеток с помощью Pro6+Pro7, но уменьшает цитолиз с помощью Pro8. На фиг. 19B изображены результаты для Pro7.
[0061] На фиг. 19 изображено, что расщепление ЭК совместно активирует Т-клеточное уничтожение EGFR+целевых клеток с помощью Pro6+Pro7, но уменьшает цитолиз с помощью Pro8. На фиг. 19C изображены результаты для Pro6+Pro7.
[0062] На фиг. 19 изображено, что расщепление ЭК совместно активирует Т-клеточное уничтожение EGFR+целевых клеток с помощью Pro6+Pro7, но уменьшает цитолиз с помощью Pro8. На фиг. 19D изображены результаты для Pro8.
[0063] На фиг. 20A изображен Pro25.
[0064] На фиг. 20B изображен Pro26.
[0065] На фиг. 20C изображен Pro27.
[0066] На фиг. 21 изображено, что генерация активного CD3-связывающего домена зависит от целевого связывания обоих плеч. ЗФБ не экспрессируется на поверхности клеток OvCar8. На фиг. 21А изображено связывание Pro6+Pro7 с rhEGFR, обнаруженное с помощью b-cyCD3::Flag::hFC, SAV-HRP.
[0067] На фиг. 21 изображено, что генерация активного CD3-связывающего домена зависит от целевого связывания обоих плеч. ЗФБ не экспрессируется на поверхности клеток OvCar8. На фиг. 21B изображено связывание Pro6+Pro9 с rhEGFR, обнаруженное с помощью b-cyCD4::Flag::hFC, SAV-HRP.
[0068] На фиг. 21 изображено, что генерация активного CD3-связывающего домена зависит от целевого связывания обоих плеч. ЗФБ не экспрессируется на поверхности клеток OvCar8. На фиг. 21C изображено связывание Pro6+Pro26 с rhEGFR, обнаруженное с помощью b-cyCD3::Flag::hFC, SAV-HRP.
[0069] На фиг. 21 изображено, что генерация активного CD3-связывающего домена зависит от целевого связывания обоих плеч. ЗФБ не экспрессируется на поверхности клеток OvCar8. На фиг. 21D изображено связывание Pro6+Pro27 с rhEGFR, обнаруженное с помощью b-cyCD3::Flag::hFC, SAV-HRP.
[0070] На фиг. 21 изображено, что генерация активного CD3-связывающего домена зависит от целевого связывания обоих плеч. ЗФБ не экспрессируется на поверхности клеток OvCar8. На фиг. 21E изображено, что связывание Pro7+Pro25 с rhEGFR обнаруживается с помощью b-cyCD3::Flag::hFC, SAV-HRP.
[0071] На фиг. 21 изображено, что генерация активного CD3-связывающего домена зависит от целевого связывания обоих плеч. ЗФБ не экспрессируется на поверхности клеток OvCar8. На фиг. 21F изображено связывание Pro9+Pro25 с rhEGFR, обнаруженное с помощью b-cyCD3::Flag::huFC, SAV-HRP.
[0072] На фиг. 22 изображено, что Pro8 с сайтом расщепления матрипазой (M) и продукты расщепленного Pro8, взаимодействующего с раковой клеткой после расщепления исходного Pro8. На фиг. 22 изображено, что αCD3 scFv-линкер может быть модифицирован для вариации длин и специфичностей протеаз.
[0073] На фиг. 23А изображены данные сэндвич-ELISA по связыванию Pro8 с rhEGFR::hFC, обнаруженного до и после расщепления ЭК с помощью биотин-cyCD3E::Flag::huFC, SAV-HRP.
[0074] На фиг. 23B изображены данные из сэндвич-ИФА по связыванию Pro8 MS (линкер 14aa) с rhEGFR::hFC, обнаруженного до и после матрипазного расщепления с помощью биотин-cyCD3E::Flag::huFC, SAV-HRP.
[0075] На фиг. 23C изображены данные сэндвич-ELISA по связыванию Pro8 ML (линкер 24aa) с rhEGFR::hFC, обнаруженного до и после ST14 расщепления с помощью биотин-cyCD3E::Flag::huFC, SAV-HRP.
[0076] На фиг. 24A представлены данные проточной цитометрии для связывания Pro8, до и после расщепления при помощи ЭК, с OvCAR8, обнаруженного с помощью AF488-cyCD3::Flag::hFC.
[0077] На фиг. 24B представлены данные проточной цитометрии для связывания Pro8 MS (линкер 14aa), до и после расщепления при помощи ST14, с OvCAR8, обнаруженного с помощью AF488-cyCD3::Flag::hFC.
[0078] На фиг. 24C представлены данные проточной цитометрии для связывания Pro8 ML (линкер 24aa), до и после расщепления при помощи ST14, с OvCAR8, обнаруженного с помощью AF488-cyCD3::Flag::hFC.
[0079] На фиг. 25 изображены дополнительные репрезентативные схемы Prodent. На фиг. 25 изображены полностью активные αCD3 scFv I2C (Pro8, Pro11) и OKT3 (Pro15).
[0080] На фиг. 26 изображены иллюстративные неполные комбинации αCD3 Prodent, в которых отсутствует активный сайт связывания CD3.
[0081] На фиг. 27A изображено связывание Pro6+Pro10 с rhEGFR, обнаруженное с помощью биотин-cyCD4::Flag::FC, SAV-HRP.
[0082] На фиг. 27B изображено связывание Pro6+Pro14 с rhEGFR, обнаруженное с помощью биотин-cyCD4::Flag::FC, SAV-HRP.
[0083] На фиг. 27C изображено связывание Pro7+Pro9 с rhEGFR, обнаруженное с помощью биотин-cyCD4::Flag::FC, SAV-HRP.
[0084] На фиг. 27D изображено связывание Pro7+Pro12 с rhEGFR, обнаруженное с помощью биотин-cyCD4::Flag::FC, SAV-HRP.
[0085] На фиг. 27E изображено связывание Pro9+Pro12 с rhEGFR, обнаруженное с помощью биотин-cyCD4::Flag::FC, SAV-HRP.
[0086] На фиг. 27F изображено связывание Pro10+Pro14 с rhEGFR, обнаруженное с помощью биотин-cyCD4::Flag::FC, SAV-HRP.
[0087] На фиг. 28 изображены репрезентативные структуры Pro с изменением в расположении нацеливающего домена от N-конца до C-конца, и влияние ориентации домена Pro на связывание CD3.
[0088] На фиг. 29 изображено, что С-концевые по сравнению с N-концевыми мишень-связывающими доменами имеют сходную активность. На фиг. 29А изображены данные проточной цитометрии по связыванию Pro6+Pro9 с OVCAR8.
[0089] На фиг. 29 изображено, что С-концевые по сравнению с N-концевыми мишень-связывающими доменами имеют сходную активность. На фиг. 29B изображено связывание ЭК расщепленного Pro6+Pro7 с OVCAR-8, обнаруженное с помощью A488-cyCD3::Flag::huFC.
[0090] На фиг. 30 изображены репрезентативные структуры Pro, используемые для исследования влияния моноспецифических нацеливающих доменов по сравнению с двойными.
[0091] Фиг. 31. Двойное нацеливание возможно с помощью sdAb, которые должны связывать отдельные целевые молекулы. На фиг. 31A изображены данные проточной цитометрии для связывания Pro9+Pro14 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[0092] Фиг. 31. Двойное нацеливание возможно с помощью sdAb, которые должны связывать отдельные целевые молекулы. На фиг. 31B изображено связывание ЭК расщепленного Pro6+Pro7 с OVCAR-8, обнаруженное с помощью AF488-cyCD3::Flag::huFC.
[0093] На фиг. 32A изображены комбинации иллюстративных Prodent с комплементарными доменами αCD3.
[0094] На фиг. 32B изображены комбинации иллюстративных Prodent с комплементарными доменами αCD3, то есть Pro6+Pro9 (одиночное-цис+ двойное -транс-молекулярное нацеливание).
[0095] На фиг. 32C изображены комбинации иллюстративных Prodent с комплементарными доменами αCD3, то есть Pro9+Pro14 (двойное молекулярное- транс-нацеливание).
[0096] На фиг. 33A изображены данные проточной цитометрии для связывания Pro6+Pro7 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[0097] На фиг. 33B изображены данные проточной цитометрии для связывания Pro9+Pro10 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[0098] На фиг. 33C изображены данные проточной цитометрии для связывания Pro12+Pro14 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[0099] На фиг. 33D изображены данные проточной цитометрии для связывания Pro7+Pro10 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[00100] На фиг. 33E изображены данные проточной цитометрии для связывания Pro6+Pro9 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[00101] На фиг. 34А изображены данные из сэндвич-FACS (только транс-связывание) для связывания Pro6+Pro12 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[00102] На фиг. 34B изображены данные из сэндвич-FACS (только транс-связывание) для связывания Pro7+Pro14 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[00103] На фиг. 34C изображены данные из сэндвич-FACS (только транс-связывание) для связывания Pro9+Pro14 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[00104] На фиг. 34D изображены данные из сэндвич-FACS (только транс-связывание) для связывания Pro10+Pro12 с OVCAR8, обнаруженного с помощью AF488-cyCD3.
[00105] На фиг. 35А изображены TDCC: цис+транс и только транс активности являются аналогичными. На фиг. 35A изображены TDCC OVCAR8 LucB цис-связывание Prodent, расщепленных и нерасщепленных. (Pro6+Pro7, Pro6+Pro9, Pro7+Pro10).
[00106] На фиг. 35B изображены TDCC: цис+транс и только транс активности, которые аналогичны. На фиг. 35B изображены расщепленные и нерасщепленные TDCC OVCAR8 LucB транс-связывающие Prodent. (Pro9+Pro14; Pro6+Pro18)
[00107] На фиг. 36 изображены TDCC - Prodent положительного контроля, теряющие активность после расщепления ЭК: данные TDCC по уничтожению клеток OVCAR8 LucB с использованием расщепленных и нерасщепленных Prodent 8, 11, 15.
[00108] На фиг. 37 изображена стабильная экспрессия ЭК-His6 в клетках OVCAR8-lux, один пик представляет собой окрашивание на экспрессию ЭК в нетрансфицированных клетках, а расширенная кривая представляет собой окрашивание на экспрессию ЭК в клетках, стабильно трансфицированных экспрессионным вектором ЭК.
[00109] На фиг. 38 изображены ЭК-экспрессирующие клоны OVCAR8 (высокая, средняя и низкая экспрессия).
[00110] На фиг. 39 изображена зависимая от дозы активация Prodent с помощью ЭК-экспрессирующих клеток OVCAR8. На фиг. 39А изображено связывание нерасщепленного Pro6+Pro9 с ЭК-экспрессирующими клонами OVCAR-8, обнаруженные с использованием меченых cyCD3ε.
[00111] На фиг. 39 изображена зависимая от дозы активация Prodent с помощью ЭК-экспрессирующих клеток OVCAR8. На фиг. 39В изображены данные проточной цитометрии для связывания нерасщепленного Pro6+Pro9 с ЭК-экспрессирующими клонами OVCAR-8, обнаруженные с использованием флуоресцентно меченных cyCD3ε.
[00112] На фиг. 40А изображены данные TDCC по уничтожению клеток OVCAR8 с помощью Pro6+Pro9 с и без ЭК.
[00113] На фиг. 40B изображены данные по TDCC для ЭК-экспрессирующего клона OVCAR8 при помощи Pro6+Pro9.
[00114] На фиг. 41А изображена структурная модель, используемая для идентификации инактивации изменений CDR в VH и VL αCD3: гомологическое моделирование αCD3e scFv, демонстрирующая гомологическую модель, Swiss-Model с использованием 5fxc.pdb; scFv-SM3, 69% идентичности GMQE 0.77 QMEAN -1.11.
[00115] На фиг. 41B изображена структурная модель, используемая для идентификации изменений, которые инактивируют CDR в VH и VL αCD3: гомологическое моделирование αCD3e scFv, демонстрирующее гомологическую модель, выровненную с 1xiw.pdb, humanCD3-e/d димер с ScFv.
[00116] На фиг. 42А изображены репрезентативные последовательности для связывания CD3e (домен VH) и области для мутации с образованием неактивных вариантов, выровненных с ближайшими последовательностями зародышевой линии человека.
[00117] На фиг. 42В изображены иллюстративные последовательности для связывания неактивного варианта CD3e (домен VH) в иллюстративных Prodent согласно изобретению.
[00118] На фиг. 43 изображены иллюстративные последовательности для связывания CD3e (домен VL), и области для мутации, чтобы сформировать неактивные варианты, а также иллюстративные аминокислотные сайты для формирования неактивных вариантов, выровненных с ближайшими последовательностями зародышевой линии человека.
[00119] На фиг. 44А изображены иллюстративные Prodent для применения в анализе Octet для связывания.
[00120] На фиг. 44В изображены активности связывания выбранных Prodent - анализ Octet.
[00121] На фиг. 45A изображено Pro23 - расщепление в сыворотке на две половины, демонстрируя, что Pro23 чувствителен к расщеплению при помощи ЭК и тромбина.
[00122] На фиг. 45B изображено Pro24 - расщепление в опухоли на две половины, демонстрирующие сайты расщепления ЭК-активными протеазами.
[00123] На фиг. 46 изображены данные из ДСН-ПААГ электрофореза, демонстрирующие расщепление Pro23 при помощи (1) ЭК (2), ЭК и тромбина (3) и только тромбина (4); расщепление Pro24 при помощи (5) ЭК (6).
[00124] На фиг. 47А изображены данные TDCC для уничтожения OVCAR8 при помощи расщепленного или нерасщепленного Pro23.
[00125] На фиг. 47В изображены данные TDCC для уничтожения OVCAR8 с помощью расщепленного и нерасщепленного Pro24.
[00126] На фиг. 48 представлены последовательности репрезентативных scFv и линкеров доменов, используемых в полипептидных конструкциях согласно изобретению, и данные о расщеплении этих линкеров. На фиг. 48А изображено расщепление пептидных субстратов MMP9 с помощью MMP9 (матриксная металлопротеиназа 9) при 1 нМ, субстраты Dabcyl-Edans. На фиг. 48B изображено расщепление субстратов MMP9 в сыворотке мыши. На фиг. 48C изображено расщепление субстратов MMP9 в сыворотке человека. На фиг. 48D изображено расщепление субстратов MMP9 в сыворотке яванского макака.
[00127] На фиг. 49 представлен перечень различных иллюстративных полипептидных последовательностей для репрезентативных линкеров, используемых в полипептидных конструкциях согласно изобретению. На фиг. 49A изображено расщепление пептидных субстратов при помощи 3 нМ меприна 1a (мембраносвязанная металлоэндопептидаза). На фиг. 49B изображено расщепление пептидных субстратов при помощи 3 нМ меприна 1b. На фиг. 49C изображено расщепление пептидных субстратов при помощи 3 нМ меприна 1a. На фиг. 49D изображено расщепление пептидных субстратов в сыворотке человека. На фиг. 49E изображено расщепление пептидных субстратов в сыворотке мыши. На фиг. 49F изображено расщепление пептидных субстратов в сыворотке яванского макака.
[00128] На фиг. 50 представлен перечень различных иллюстративных полипептидных последовательностей для репрезентативных линкеров, используемых в полипептидных конструкциях согласно изобретению. На фиг. 50A изображено расщепление пептидных субстратов при помощи матриптазы ST14. На фиг. 50B изображено расщепление пептидных субстратов в сыворотке мыши. На фиг. 50C изображено расщепление пептидных субстратов в сыворотке человека. На фиг. 50D изображено расщепление пептидных субстратов в сыворотке яванского макака.
[00129] На фиг. 51А изображены иллюстративные линкерные последовательности, используемые в полипептидных конструкциях согласно изобретению, которые расщепляются протеазами крови. На фиг. 51А изображены иллюстративные пептидные субстраты, расщепленные тромбином. На фиг. 51В изображены пептидные субстраты, расщепленные тромбином. На фиг. 51С изображены пептидные субстраты, расщепленные нейтрофильной эластазой.
[00130] На фиг. 52 изображены иллюстративные линкерные последовательности, используемые в полипептидных конструкциях согласно изобретению, которые расщепляются протеазами сыворотки. На фиг. 52A изображено расщепление пептидных субстратов в сыворотке человека. На фиг. 52B изображено расщепление пептидных субстратов в сыворотке мыши. На фиг. 52C изображено расщепление пептидных субстратов в сыворотке яванского макака.
[00131] На фиг. 53 продемонстрирована гибкость расположения составных частей иллюстративных полипептидных конструкций согласно изобретению.
[00132] На фиг. 53A изображен первый формат, в котором, считывая с N- к C-концу, первый домен, связывающий целевой антиген, (α-T1) связывается через доменный линкер с первым CD-3 VL-связывающим доменом, который, в свою очередь, связан с первым неактивным CD-3 VHi-связывающим доменом через расщепляемый доменный линкер (CL1). CL1 также связан с первым доменом увеличения периода полувыведения (HED), который связан посредством доменного линкера со вторым доменом, связывающим целевой антиген, (α-T2), который сам связан со вторым CD-3 VH-связывающим доменом. Второй CD-3 VH-связывающий домен связан через расщепляемый доменный линкер (CL2) со вторым CD-3 VL-связывающим доменом (VLi), который неактивен, который также связан со вторым доменом увеличения периода полувыведения. С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00133] На фиг. 53B изображен второй формат, в котором, считывая с N- к C-концу, второй домен, связывающий целевой антиген, (α-T2) связывается с первым CD-3 VL-связывающим доменом, который, в свою очередь, связан с первым неактивным CD-3 VHi-связывающим доменом через расщепляемый доменный линкер (CL1). CD-3 VHi также связан с первым доменом увеличения периода полувыведения, который связан посредством доменного линкера с первым доменом, связывающим целевым антигеном, (α-T1), который сам связан со вторым CD-3 VH-связывающим доменом. Второй CD-3 VH-связывающий связан через расщепляемый линкер (CL2) со вторым CD-3 VL-связывающим доменом (VLi), который неактивен, который также связан со вторым доменом увеличения периода полувыведения. С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00134] На фиг. 53C изображен третий формат, в котором, считывая с N- к C-концу, первый домен, связывающий целевой антиген, (α-T1) связывается с первым CD-3 VH-связывающим доменом, который, в свою очередь, связан с первым неактивным CD-3 VLi-связывающим доменом через расщепляемый доменный линкер (CL1). CD3 VLi также связан с первым доменом увеличения периода полувыведения, который связан посредством доменного линкера со вторым доменом, связывающим целевой антиген, (α-T2), который сам связан со вторым CD-3 VL-связывающим доменом. Второй CD-3 VL-связывающий связан через расщепляемый линкер (CL2) со вторым CD-3 VH-связывающим доменом (VHi), который неактивен, который также связан со вторым доменом увеличения периода полувыведения. С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00135] На фиг. 53D изображен четвертый формат, в котором, считывая с N- к C-концу, второй домен, связывающий целевой антиген, (α-T2) связывается с первым CD-3 VH-связывающим доменом, который, в свою очередь, связан с первым неактивным CD-3 VLi-связывающим доменом через расщепляемый линкер (CL1). CD3 VLi также связан с первым доменом увеличения периода полувыведения, который связан посредством доменного линкера с первым доменом, связывающим целевой антиген (α-T1), который сам связан со вторым CD-3 VL-связывающим доменом. Второй CD-3 VH-связывающий связан через расщепляемый доменный линкер (CL2) со вторым CD-3 VH-связывающим доменом (VHi), который неактивен, который также связан со вторым доменом увеличения периода полувыведения. С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00136] На фиг. 53E изображен пятый формат, в котором, считывая с N- к C-концу, первый домен увеличения периода полувыведения связан с первым CD-3 VH-связывающим доменом (V H i), который неактивен и который связан через первый расщепляемый линкер (CL1) с первым CD-3 V L-связывающим доменом, связанным с первым доменом, связывающим целевой антиген, (α-T1). Первый домен, связывающий целевой антиген, связывается через доменный линкер со вторым доменом увеличения периода полувыведения, который связан со вторым CD-3 VL-связывающим доменом (VLi), который неактивен и связан через второй расщепляемый доменный линкер (CL2) со вторым доменом CD-3 VH, который сам связан со вторым доменом, связывающим целевой антиген (α-T2). С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00137] На фиг. 53F изображен иллюстративный формат полипептидной конструкции согласно изобретению, в которой, считывая с N- к С-концу, домен увеличения периода полувыведения связывается с первым CD-3 VH -связывающим доменом (VHi), который является неактивным, и который связан через первый расщепляемый линкер (CL1) с первым CD-3 VL-связывающим доменом, связанным со вторым доменом, связывающим целевой антиген, (α-T2). Второй домен, связывающий целевой антиген, связывается через доменный линкер со вторым доменом увеличения периода полувыведения, который связан со вторым CD-3 VL-связывающим доменом (VLi), который неактивен и связан через второй расщепляемый доменный линкер (CL2) со вторым доменом CD-3 VH, который сам связан с первым доменом, связывающим целевой антиген, (α-T1). С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00138] На фиг. 53G изображен седьмой иллюстративный формат полипептидной конструкции согласно изобретению, в которой, считывая с N- к С-концу, домен увеличения периода полувыведения связывается с первым CD-3 VL-связывающим доменом (VLi), который является неактивным, и который связан через первый расщепляемый линкер (CL1) с первым CD-3 VH -связывающим доменом, связанным с первым доменом, связывающим целевой антиген, (α-T1). Первый домен, связывающий целевой антиген, связывается через доменный линкер со вторым доменом увеличения периода полувыведения, который связан с CD 3 VH-связывающим доменом (VHi), который неактивен и связан через второй расщепляемый линкер (CL2) со вторым доменом CD-3 VL, который сам связан со вторым доменом, связывающим целевой антиген, (α-T2). С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00139] На фиг. 53H изображен восьмой иллюстративный формат полипептидной конструкции согласно изобретению, в которой, считывая с N- к С-концу, домен увеличения периода полувыведения связывается с первым CD-3 VL -связывающим доменом (VLi), который является неактивным, и который связан через первый расщепляемый доменный линкер (CL1) с первым CD-3 VH -связывающим доменом, связанным со вторым доменом, связывающим целевой антиген, (α-T2). Второй домен, связывающий целевой антиген, связывается через доменный линкер со вторым доменом увеличения периода полувыведения, который связан со вторым CD 3 VH -связывающим доменом (VHi), который неактивен и связан через второй расщепляемый линкер (CL2) со вторым доменом CD-3 VL, который сам связан с первым доменом, связывающим целевой антиген, (α-T2). С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00140] На фиг. 53I изображен еще один иллюстративный формат полипептидных конъюгатов согласно изобретению, в котором, считывая с N- к С-концу, первый домен, связывающий целевой антиген, (α-T1) связан с первым доменом CD-3 VL, который связан через первый расщепляемый линкер (CL1) с первым доменом CD-3 VH (VHi), который неактивен и который связан с первым доменом увеличения периода полувыведения, который связан через доменный линкер со вторым доменом увеличения периода полувыведения. Второй домен увеличения периода полувыведения связан со вторым доменом CD-3 VL (VLi), который является неактивным и связан через второй расщепляемый линкер (CL2) со вторым доменом CD-3 VH, который связан со вторым доменом, связывающим целевой антиген, (α-T2). С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00141] На фиг. 53J изображен еще один иллюстративный формат полипептидных конъюгатов согласно изобретению, в котором, считывая с N- к С-концу, второй домен, связывающий целевой антиген, (α-T2) связан с первым доменом CD-3 VL, который связан через первый расщепляемый линкер (CL1) с первым доменом CD-3 VH (VHi), который неактивен и который связан с первым доменом увеличения периода полувыведения, который связан через доменный линкер со вторым доменом увеличения периода полувыведения. Второй домен увеличения периода полувыведения связан с scFv и вторым доменом CD-3 VL (VLi), который является неактивным и связан через второй расщепляемый линкер (CL2) со вторым доменом CD-3 VH, который связан с первым доменом, связывающим целевой антиген, (α-T1). С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00142] На фиг. 53K изображен еще один иллюстративный формат полипептидных конъюгатов согласно изобретению, в котором, считывая с N- к С-концу, первый домен, связывающий целевой антиген, (α-T1) связан с первым доменом CD-3 VH, который связан через первый расщепляемый линкер (CL1) с первым доменом CD-3 VL (VLi), который неактивен и который связан с первым доменом увеличения периода полувыведения, который связан через доменный линкер со вторым доменом увеличения периода полувыведения. Второй домен увеличения периода полувыведения связан со вторым доменом CD-3 VH (VHi), который является неактивным и связан через второй расщепляемый линкер (CL2) со вторым доменом CD-3 VL, который связан со вторым доменом, связывающим целевой антиген, (α-T2). С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00143] На фиг. 53L изображен еще один иллюстративный формат полипептидных конъюгатов согласно изобретению, в котором, считывая с N- к С-концу, второй домен, связывающий целевой антиген, (α-T2) связан с первым доменом CD-3 VH, который связан через первый расщепляемый линкер (CL1) с первым доменом CD-3 VL (VLi), который неактивен и который связан с первым доменом увеличения периода полувыведения, который связан через доменный линкер со вторым доменом увеличения периода полувыведения. Второй домен увеличения периода полувыведения связан со вторым доменом CD-3 VH (VHi), который является неактивным и связан через второй расщепляемый линкер (CL2) со вторым доменом CD-3 VL, который связан с первым доменом, связывающим целевой антиген, (α-T1). С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9 или более аминокислот, например His6.
[00144] На фиг. 53M изображен другой иллюстративный формат полипептидных конъюгатов согласно изобретению, в котором, считывая с N- к С-концу, первый домен увеличения периода полувыведения связан с первым доменом CD-3 VH (VHi), который является неактивным и который связан через первый расщепляемый линкер (CL1) с первым доменом CD-3 VL. Домен CD-3 VL связан с первым доменом, связывающим целевой антиген, (α-T1), который связан через доменный линкер со вторым доменом, связывающим целевой антиген (α-T2), который связан со вторым доменом CD-3 VH, связанным через второй расщепляемый доменный линкер со вторым доменом CD3-VL (VLi), который неактивен и который связан со вторым доменом увеличения периода полувыведения. С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00145] На фиг. 53N изображен другой иллюстративный формат полипептидных конъюгатов согласно изобретению, в котором, считывая с N- к С-концу, первый домен увеличения периода полувыведения связан с первым доменом CD-3 VH (VHi), который является неактивным и который связан через первый расщепляемый линкер (CL1) с первым доменом CD-3 VL. Домен CD-3 VL связан со вторым антигенсвязывающим доменом (α-T2), который связан через доменный линкер с первым антигенсвязывающим доменом (α-T1), который связан со вторым доменом CD-3 VH, связанным через второй расщепляемый доменный линкер (CL2) со вторым доменом CD3-VL (VLi), который неактивен и который связан со вторым доменом увеличения периода полувыведения. С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00146] На фиг. 53O изображен другой иллюстративный формат полипептидных конъюгатов согласно изобретению, в котором, считывая с N- к С-концу, первый домен увеличения периода полувыведения связан с первым доменом CD-3 VL (VLi), который является неактивным и который связан через первый расщепляемый линкер (CL1) с первым доменом CD-3 VH. Домен CD-3 VH связан с первым доменом, связывающим целевой антиген, (α-T1), который связан через доменный линкер со вторым доменом, связывающим целевой антиген, (α-T2), который связан со вторым доменом CD-3 VL, связанным со вторым доменом CD3 VH (VHi), который неактивен и который связан со вторым доменом увеличения периода полувыведения. С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00147] На фиг. 53P изображен другой иллюстративный формат полипептидных конъюгатов согласно изобретению, в котором, считывая с N- к С-концу, первый домен увеличения периода полувыведения связан с первым доменом CD-3 VL (VLi), который является неактивным и который связан через первый расщепляемый линкер (CL1) с первым доменом CD-3 VH. Первый домен CD-3 VH связан со вторым доменом, связывающим целевой антиген, (α-T2), который связан через доменный линкер с первым доменом, связывающим целевой антиген (α-T1), который связан со вторым доменом CD-3 VL, который связан через второй расщепляемый линкер (CL2) со вторым доменом CD3 VH (VHi), который неактивен и который связан со вторым доменом увеличения периода полувыведения. С-конец полипептида необязательно содержит блокирующую группу из 1, 2, 3, 4, 5, 6, 7, 8, 9, 10 или более аминокислот, например, His6.
[00148] На фиг. 54 представлены репрезентативные последовательности нуклеиновых кислот и полипептидов для иллюстративных полипептидных конструкций согласно изобретению. Иллюстративные полипептиды, используемые в изобретении, блокируются на С-конце, поэтому последовательности, продемонстрированные с помощью Hisx C-концевых меток, могут быть использованы с этими метками, короткими или более длинными версиями этих меток или без меток.
[00149] На фиг. 55 представлено соответствие SEQ ID NO для различных полипептидных конструкций согласно изобретению и сокращенная номенклатура для этих конструкций.
[00150] На фиг. 56 представлены иллюстративные последовательности линкеров для применения в вариантах осуществления изобретения.
ПОДРОБНОЕ ОПИСАНИЕ СУЩНОСТИ ИЗОБРЕТЕНИЯ
Введение
[00151] В данном документе описаны условно активируемые антигенсвязывающие полипептиды. Иллюстративные полипептиды согласно изобретению содержат по меньшей мере один домен, связывающий целевой антиген, по меньшей мере один scFv против CD-3 с по меньшей мере одной парой VH/VL, в которой по меньшей мере один из VH и VL является неактивным по отношению к специфическому связыванию с CD-3, различные scFv- и доменные линкеры, ковалентно связывающие компоненты полипептидной конструкции, и сайты расщепления в одном или более доменных и/или scFv-линкерах и, необязательно, с одним или более доменами увеличения периода полувыведения Иллюстративный расщепляемый сайт расщепляется ферментом сыворотки (например, эстеразой) или деградирующим ферментом (например, протеазой), расположенным или сконцентрированным в микроокружении опухоли, против которой направлена полипептидная конструкция. Иллюстративный деградирующий фермент представляет собой протеазу, экспрессируемую опухолью или в пределах микроокружения опухоли. При расщеплении по меньшей мере одного расщепляемого сайта в линкере конструкции неактивный член(ы) пары scFv, удаленный из конструкции, и активный член(ы) пары scFv взаимодействует с его активным родственным вариантом (например, VH1/VLi1 становится VH1, VHi2/VL2 становится VL2, а VH1 и VL2 взаимодействуют, образуя функциональный scFv, специфически связывающийся с CD-3. Конструкция также специфически связывается с выбранным целевым антигеном через домен, связывающий целевой антиген. В иллюстративном варианте осуществления VH1 и VL2 остаются присоединенными линкером домена, дополнительно связывающим домен, связывающий целевой антиген, с VH1 и VL2. Некоторые полипептидные конструкции согласно изобретению также содержат один или более доменов увеличения периода полувыведения, которые увеличивают период полувыведения полипептида после его введения субъекту, нуждающемуся в этом. Иллюстративный домен увеличения периода полувыведения представляет собой антитело или фрагмент антитела, направленный против циркулирующего плазменного белка, например, ЧСА. Домен(ы) увеличения периода полувыведения может быть включен в полипептидную последовательность с одним или более расщепляемыми линкерами между ним и остальной частью конструкции, так что домен увеличения периода полувыведения отделяется от конструкции после достижения цели, например, доставки конструкции в опухоль или завершения желаемого in vivo периода полувыведения из крови. Домен(ы) увеличения периода полувыведения может быть включен в полипептидную последовательность без расщепляемых линкеров между ней и остальной частью конструкции, так что домен увеличения периода полувыведения сохраняется в полипептиде после активации CD3-связывающего домена. Прилагаемые фигуры содержат структуры многих иллюстративных мотивов полипептидных конструкций согласно изобретению.
[00152] Полипептидные конструкции согласно изобретению, имеющие более одной пары VH/VL, существуют как единый объект. В различных вариантах осуществления соединения согласно изобретению содержат одну пару VH/VL. В данных вариантах осуществления полипептидные конструкции обычно используются в парах, в которых один член пары содержит VH/VLi и другой - VHi/VL, так что при расщеплении неактивного члена пары VH/VL могут соединяться и связываться с CD-3.
[00153] В иллюстративных вариантах осуществления полипептидных конструкций согласно изобретению домен нацеливания на больную клетку связывается с анти-Т-клеточным связывающим сегментом при помощи нерасщепляемого линкера (NCL1 и NCL2). Активные и неактивные анти-Т-клеточные scFv-сегменты связываются при помощи расщепляемого линкера, который чувствителен к микроокружению больной ткани (CL1 и CL2). Две полумолекулы или белковые области связываются при помощи другого деградируемого линкера (RL). Фиг. 53.
[00154] В различных вариантах осуществления исходные конструкции состоят из двух полипептидных областей, которые могут быть разделены путем расщепления в региональном линкере (RL) после инъекции в организм. Домены, связывающие болезнь-мишень, активны в начале и могут связывать свою мишень, тем самым обогащая неактивные белки на поверхности больных клеток. Затем расщепляемые линкеры могут быть расщеплены в микроокружении пораженной ткани, и активные сегменты связывания Т-клеток (которые связаны с пораженными клетками при помощи доменов нацеливания и нерасщепляемых линкеров) можно затем рекомбинировать для создания активных scFv, связывающих Т-клетки, на поверхности больных клеток. Эта рекомбинация создает активные связывающие Т-клетки scFv, взаимодействующие с Т-клетками для связывания пораженной клетки и ее уничтожения. Домены увеличения периода полувыведения 1-2 продлевают периоды полувыведения молекул из крови до того, как они достигают пораженной клетки, и удаляются с неактивным анти-Т-клеточным доменом (VLi или VHi), чтобы ограничить период полувыведения расщепленных/активированных молекул, если они покидают пораженную ткань. Один домен увеличения периода полувыведения является желательным, если региональный линкер расщепляется в опухоли, и два являются желательными в полноразмерной молекуле, если областной линкер расщепляется в крови, чтобы гарантировать, что обе половины молекулы/белковые области имеют достаточное количество периодов полувыведения для накопления в пораженных клетках. Полипептидные конструкции, включая линкеры, расщепляемые соответствующими опухолевыми и относящимися к крови ферментами, входят в объем изобретения.
[00155] Кроме того, согласно изобретению предлагаются фармацевтические композиции полипептидных конструкций, а также нуклеиновые кислоты, рекомбинантные векторы экспрессии и клетки-хозяева для изготовления этих конструкций. Также представлены способы применения раскрытых полипептидов в профилактике и/или лечении заболеваний, патологических состояний и расстройств.
Определения
[00156] Для того, что изобретение может быть более полно понято, несколько определений приведены ниже. Такие определения предназначены для охвата грамматических эквивалентов.
[00157] Под «аминокислотой» и «аминокислотной идентичностью», как используется в данном документе, подразумевают одну из 20 встречающихся в природе аминокислот или любых неприродных аналогов, которые могут присутствовать в конкретном, определенном положении. Под «белком» в данном документе подразумевают по меньшей мере две ковалентно присоединенные аминокислоты, которые включают белки, полипептиды, олигопептиды и пептиды. Белок может состоять из природных аминокислот и пептидных связей или синтетических пептидомиметических структур, то есть «аналогов», такие как пептоиды (см. Simon et al., PNAS USA 89(20):9367 (1992)), особенно когда пептиды LC (легкая цепь) должны вводиться пациенту. Таким образом, «аминокислота» или «пептидный остаток», как используется в данном документе, означает как природные, так и синтетические аминокислоты. Например, гомофенилаланин, цитруллин и норлейцин считаются аминокислотами для целей изобретения. «Аминокислота» также включает иминокислотные остатки, такие как пролин и гидроксипролин. Боковая цепь может находиться в конфигурации или (R) или (S). В предпочтительном варианте осуществления аминокислоты находятся в (S) или L-конфигурации. Если используются не встречающиеся в природе боковые цепи, могут быть использованы неаминокислотные заместители, например, для предотвращения или замедления деградации in vivo.
[00158] Под «аминокислотной модификацией» в данном документе подразумевают аминокислотную замену, вставку и/или делецию в полипептидной последовательности или изменение фрагмента, химически связанного с белком. Например, модификация может быть измененной углеводной или ПЭГ-структурой, присоединенной к белку. Под «аминокислотной модификацией» в данном документе подразумевают аминокислотную замену, вставку и/или делецию в полипептидной последовательности. Для ясности, если не указано иное, аминокислотная модификация всегда соответствует аминокислоте, кодируемой ДНК, например, 20 аминокислотам, которые имеют кодоны в ДНК и РНК. Предпочтительная аминокислотная модификация в данном документе представляет собой замену.
[00159] Под «аминокислотной заменой» или «заменой» в данном документе подразумевают замену аминокислоты в определенном положении в исходной полипептидной последовательности на другую аминокислоту. В частности, в некоторых вариантах осуществления замена представляет собой аминокислоту, которая не встречается в природе в конкретном положении, как не встречающаяся в природе в организме, так и в любом организме. Например, замена E272Y относится к варианту полипептида, в данном случае варианту Fc, в котором глутаминовая кислота в положении 272 заменена тирозином. Для ясности, белок, который был сконструирован для изменения кодирующей последовательности нуклеиновой кислоты, но не меняет исходную аминокислоту (например, обмен CGG (кодирующий аргинин) на CGA (все еще кодирующий аргинин) для увеличения уровней экспрессии в организме-хозяине) не является "аминокислотной заменой"; то есть, несмотря на создание нового гена, кодирующего один и тот же белок, если белок имеет ту же самую аминокислоту в том месте, в котором она начиналась, это не является аминокислотной заменой.
[00160] Под «вставкой аминокислоты» или «вставкой», как используется в данном документе, подразумевают добавление аминокислотной последовательности в определенном положении в исходной полипептидной последовательности. Например, -233E или 233E обозначает введение глутаминовой кислоты после положения 233 и до положения 234. Кроме того, -233ADE или A233ADE обозначает вставку AlaAspGlu после положения 233 и до положения 234.
[00161] Под «делецией аминокислоты» или «делецией», как используется в данном документе, подразумевают удаление аминокислотной последовательности в определенном положении в исходной полипептидной последовательности. Например, E233- или E233#, E233() или E233del обозначает делецию глутаминовой кислоты в положении 233. Кроме того, EDA233- или EDA233# обозначает удаление последовательности GluAspAla, которая начинается в положении 233.
[00162] Используемый в данном документе термин «полипептид» означает по меньшей мере две ковалентно присоединенные аминокислоты, которые включают белки, полипептиды, олигопептиды и пептиды. Пептидильная группа может содержать природные аминокислоты и пептидные связи или синтетические пептидомиметические структуры, т.е. «аналоги», такие как пептоиды (см. Simon et al., PNAS USA 89(20):9367 (1992), полностью включенную посредством ссылки). Аминокислоты могут быть или природными, или синтетическими (например, не аминокислотой, кодируемой ДНК); как будет понятно специалистам в данной области техники. Например, гомофенилаланин, цитруллин, орнитин и норлейцин считаются синтетическими аминокислотами для целей изобретения, и могут быть использованы как D-, так и L- (R или S) аминокислоты. Варианты согласно данному изобретению могут содержать модификации, которые включают использование синтетических аминокислот, включенных с использованием, например, технологий, разработанных Шульцем и его коллегами, включая, но не ограничиваясь способами, описанными Cropp & Shultz, 2004, Trends Genet. 20(12):625-30, Anderson et al., 2004, Proc Natl Acad Sci USA 101(2):7566-71, Zhang et al., 2003, 303(5656):371-3, и Chin et al., 2003, Science 301(5635):964-7, все полностью включены посредством ссылки. Кроме того, полипептиды могут включать синтетическую дериватизацию одной или более боковых цепей или концов, гликозилирование, пегилирование, циклическую перестановку, циклизацию, линкеры с другими молекулами, слияние с белками или белковыми доменами и добавление пептидных меток или маркеров.
[00163] Полипептиды согласно изобретению специфически связываются с CD3 и целевыми клеточными рецепторами, как описано в данном документе. Термин «специфически связывает» в данном описании означает, что полипептиды имеют константы связывания в диапазоне по меньшей мере 10-4 -10-6 М-1, с предпочтительным диапазоном 10-7 -10-9 М-1.
[00164] В частности, в определение «полипептиды» включены агликозилированные полипептиды. Под «агликозилированным полипептидом», используемым в данном документе, подразумевают полипептид, который не содержит углеводов, прикрепленных в положении 297 области Fc, причем нумерация соответствует системе ЕС, как в Кабате. Агликозилированный полипептид может представлять собой дегликозилированный полипептид, то есть антитело или фрагмент антитела, из которого удаляется углевод Fc, например, химически или ферментативно. Альтернативно, агликозилированный полипептид может быть негликозилированным или негликозилированным антителом или его фрагментом, экспрессированным без углевода Fc, например, путем мутации одного или более остатков, которые кодируют профиль гликозилирования или экспрессией в организме, которые не присоединяет углеводы к белкам, например, бактерии.
[00165] Под «исходным полипептидом» или «полипептидом-предшественником» (включая исходный или предшественники Fc), как используется в данном документе, подразумевают полипептид, который впоследствии модифицируют для получения варианта. Указанный исходный полипептид может представлять собой природный полипептид или вариант, или модифицированный вариант природного полипептида. Исходный полипептид может относиться к самому полипептиду, композиции, которые содержат исходный полипептид, или аминокислотной последовательности, которая кодирует его. Соответственно, под «исходным полипептидом Fc», как используется в данном документе, подразумевают немодифицированный полипептид Fc, который модифицирован для генерации варианта, а под «исходным антителом», как используется в данном документе, подразумевается немодифицированное антитело, которое модифицировано для получения варианта антитела.
[00166] Под «положением», как используется в данном документе, подразумевают положение в последовательности белка. Положения могут быть пронумерованы последовательно или в соответствии с установленным форматом, например, индексом ЕС для нумерации антител.
[00167] Под «целевым антигеном», как используется в данном документе, подразумевают молекулу, которая специфически связана с вариабельной областью данного антитела. Целевой антиген может представлять собой белок, углевод, липид или другое химическое соединение. Ряд подходящих иллюстративных целевых антигенов описан в данном документе.
[00168] Под «целевой клеткой», как используется в данном документе, подразумевают клетку, которая экспрессирует целевой антиген.
[00169] Под «антителом» в данном документе подразумевается белок, состоящий из одного или более полипептидов, по существу, кодируемых всеми или частью распознанных генов иммуноглобулина. Распознанные гены иммуноглобулина, например, у людей, включают каппа (κ), лямбда (λ) и генетические локусы тяжелой цепи, которые вместе содержат большое число генов вариабельных областей и генов константных областей мю (μ), дельта (δ), гамма (γ), сигма (ε) и альфа (α), которые кодируют изотипы IgM, IgD, IgG, IgE и IgA, соответственно. В данном изобретении подразумевается, что антитело включает полноразмерные антитела и фрагменты антител и может относиться к природному антителу из любого организма, сконструированного антитела или антитела, получаемого рекомбинантно для экспериментальных, терапевтических или других целей, как дополнительно определено ниже. Таким образом, «антитело» включает как поликлональное, так и моноклональное антитело (мкАт). Способы получения и очистки моноклональных и поликлональных антител известны в данной области техники и, например, описаны в Harlow and Lane, Antibodies: A Laboratory Manual (New York: Cold Spring Harbor Laboratory Press, 1988). Как указано в данном документе, «антитело» конкретно включает варианты Fc, описанные в данном документе, «полноразмерные» антитела, включая описанные в данном документе фрагменты варианта Fc, и слития вариантов Fc с другими белками, как описано в данном документе.
[00170] Термин «антитело» включает фрагменты антител, как известно в данной области техники, такие как Fab, Fab', F(ab')2, Fc или другие антигенсвязывающие подпоследовательности антител, такие как одноцепочечные антитела (например, scFv), химерные антитела и т. д., или продуцируемые путем модификации полных антител, или синтезируемые de novo с использованием технологий рекомбинантных ДНК. Термин «антитело» дополнительно включает поликлональные антитела и мкАт, которые могут быть агонистами или антагонистами.
[00171] В частности, в определение «антитело» включены полноразмерные антитела, которые содержат часть варианта Fc. Под «полноразмерным антителом» в данном документе подразумевается структура, которая представляет собой естественную биологическую форму антитела, включая вариабельные и константные области. Например, у большинства млекопитающих, включая людей и мышей, полноразмерное антитело класса IgG представляет собой тетрамер и состоит из двух идентичных пар двух цепей иммуноглобулина, каждая из которых имеет одну легкую и одну тяжелую цепь, каждая легкая цепь содержит иммуноглобулиноподобные домены VL и CL, и каждая тяжелая цепь содержит иммуноглобулиновые домены VH, Cγ1, Cγ2 и Cγ3. У некоторых млекопитающих, например, у верблюдов и лам антитела IgG могут состоять только из двух тяжелых цепей, причем каждая тяжелая цепь содержит вариабельный домен, присоединенный к области Fc. Под «IgG», как используется в данном документе, подразумевается полипептид, относящийся к классу антител, которые, по существу, кодируются распознанным геном иммуноглобулина гамма. У людей этот класс включает IgG1, IgG2, IgG3 и IgG4. У мышей этот класс включает IgG1, IgG2a, IgG2b, IgG3.
[00172] В предпочтительном варианте осуществления антитела согласно изобретению являются гуманизированными. Используя современную технологию моноклональных антител, можно продуцировать гуманизированное антитело практически для любого целевого антигена, который можно идентифицировать [Stein, Trends Biotechnol. 15: 88-90 (1997)]. Гуманизированные формы нечеловеческих (например, мышиных) антител представляют собой химерные молекулы иммуноглобулинов, цепей иммуноглобулина или их фрагментов (таких как Fv, Fc, Fab, Fab', F (ab')2 или другие антигенсвязывающие подпоследовательности антител), которые содержат минимальную последовательность, полученную из нечеловеческого иммуноглобулина. Гуманизированные антитела включают человеческие иммуноглобулины (реципиентное антитело), в которых остатки, образующие определяющую комплементарность область (CDR) реципиента, заменяют остатками CDR нечеловеческих видов (донорское антитело), таких как мышь, крыса или кролик, с желательной специфичностью, аффинностью и способностью. В некоторых случаях каркасные остатки Fv человека заменены соответствующими нечеловеческими остатками. Гуманизированные антитела могут также содержать остатки, которые не обнаруживаются ни в реципиентном антителе, ни в введенные CDR или каркасных последовательностях. В общем, гуманизированное антитело будет содержать по существу все из по меньшей мере одного, и, как правило, двух вариабельных доменов, в которых все или по существу все области CDR соответствуют таковым из нечеловеческого иммуноглобулина, и все или по существ, все области FR представляют собой в том числе консенсусную последовательность человеческого иммуноглобулина. Гуманизированное антитело оптимально также будет содержать по меньшей мере часть константной области иммуноглобулина (Fc), как правило, иммуноглобулина человека [Jones et al., Nature 321:522-525 (1986); Riechmann et al., Nature 332:323-329 (1988); и Presta, Curr. Op. Struct. Biol. 2: 593-596 (1992)].
[00173] Способы гуманизации нечеловеческих антител хорошо известны в данной области техники. Как правило, гуманизированное антитело имеет один или более аминокислотных остатков, введенных в него из источника, который не является человеческим. Эти нечеловеческие аминокислотные остатки часто упоминаются как введенные остатки, которые обычно берутся из введенного вариабельного домена. Гуманизация может быть, по существу, выполнена по методу Винтер и ее коллег [Jones et al., supra; Riechmann et al., выше; и Verhoeyen et al., Science, 239:1534-1536 (1988)], путем замены CDR или последовательностей CDR грызунов соответствующими последовательностями человеческого антитела. Дополнительные примеры гуманизированных мышиных моноклональных антител также известны в данной области техники, например, антитела, связывающие человеческий белок C [O'Connor et al., Protein Eng. 11:321-8 (1998)], рецептор интерлейкина-2 [Queen et al., Proc. Natl. Acad. Sci., U.S.A. 86:10029-33 (1989)) и рецептор человеческого эпидермального фактора роста 2 типа [Carter et al., Proc. Natl. Acad. Sci. U.S.A. 89:4285-9 (1992)]. Соответственно, такие гуманизированные антитела представляют собой химерные антитела (патент США № 4816567), причем существенно меньше, чем интактный вариабельный домен человека, замененный соответствующей последовательностью из нечеловеческих видов. На практике гуманизированные антитела обычно представляют собой человеческие антитела, в которых некоторые остатки CDR и, возможно, некоторые остатки FR замещены остатками из аналогичных сайтов антител грызунов.
[00174] В предпочтительном варианте осуществления полипептиды согласно изобретению основаны на человеческих последовательностях и, следовательно, человеческие последовательности используются в качестве «базовых» последовательностей, с которыми сравниваются другие последовательности, такие как последовательности крысы, мыши и обезьяны. Для установления гомологии с первичной последовательностью или структурой, аминокислотная последовательность предшественника или исходного антитела, или scFv непосредственно сравнивается с соответствующей человеческой последовательностью. После выравнивания последовательностей, c использованием одной или более программ для выравнивания областей гомологии, описанных в данном документе (например, с использованием консервативных остатков для разных видов), которые делают возможными необходимые вставки и делеции для поддержания выравнивания (т.е. избегая удаления консервативных остатков посредством произвольной делеци и вставки), определяются остатки, эквивалентные определенным аминокислотам в первичной последовательности полипептида человека. Выравнивание консервативных остатков предпочтительно должно сохранять 100% таких остатков. Однако выравнивание более 75% или всего лишь 50% консервативных остатков также является достаточным для определения эквивалентных остатков (иногда называемых в данном документе «соответствующими остатками»).
[00175] Под «остатком», как используется в данном документе, подразумевается положение в белке и его ассоциированная аминокислотная идентичность. Например, аспарагин 297 (также называемый Asn297 или N297) представляет собой остаток в положении 297 в человеческом антителе IgG1.
[00176] Эквивалентные остатки также могут быть определены путем определения гомологии на уровне третичной структуры для фрагмента scFv, третичная структура которого определена при помощи рентгеноструктурной кристаллографии. Эквивалентные остатки определяются как те, для которых атомные координаты двух или более атомов основной цепи определенного аминокислотного остатка исходной последовательности или предшественника (N на N, CA на CA, C на C и O на O) находятся в пределах 0,13 нм и предпочтительно 0,1 нм после выравнивания. Выравнивание достигается после того, как лучшая модель была ориентирована и позиционирована так, чтобы обеспечить максимальное перекрытие атомных координат отличных от водорода атомов белка фрагмента scFv.
[00177] Под «Fv» или «фрагментом Fv» или «областью Fv», как используется в данном документе, подразумевают полипептид, который содержит VL- и VH-домены одного антитела. Как будет понятно специалистам в данной области техники, они обычно состоят из двух цепей или могут быть объединены (как правило, с линкером, как обсуждалось в данном документе) с образованием scFv.
[00178] Под «одноцепочечным Fv» или «scFv» в данном документе подразумевают вариабельный домен тяжелой цепи (VH), ковалентно связанный с вариабельным доменом легкой цепи (VL), как правило, с использованием scFv-линкер, как обсуждалось в данном документе, с образованием scFv или домена scFv. Домен scFv может быть в любой ориентации от N- до С-конца (VH-линкер-VL или VL-линкер-VH).
[00179] Под «вариабельной областью», как используется в данном документе, понимается область иммуноглобулина, которая содержит один или более доменов Ig, которые, по существу, кодируются любым из генов Vκ, Vλ, VL и/или VH, которые составляют каппа, лямбда и генетические локусы иммуноглобулина тяжелой и легкой цепи, соответственно.
[00180] Как используется в данном документе, «неактивный VH» и «неактивный VL» относятся к компонентам scFv, которые при соединении с их родственными партнерами VL или VH образуют соответственно пару VH/VL, которая не специфически связывается с антигеном, с которым «активный» VH или «активный» VL будет связываться, были связаны с аналогичным VL или VH, который не был «неактивным». Иллюстративные «неактивные VH» и «неактивные VL»-домены формируются при помощи мутации последовательности VH или VL дикого типа. Иллюстративные мутации находятся в CDR1, CDR2, или CDR3 VH, или VL. Иллюстративная мутация включает размещение доменного линкера в CDR2, тем самым образуя «неактивный VH» или «неактивный VL»-домен. Напротив, «активный VH» или «активный VL» является тем, который при спаривании со своим «активным» родственным партнером, то есть VL или VH, соответственно, способен специфически связываться с его целевым антигеном.
[00181] Напротив, как используется в данном документе, термин «активный» относится к CD-3-связывающему домену, который способен специфически связываться с CD-3. Этот термин используется в двух контекстах: (a) при обращении к одному члену scFv-связывающей пары (то есть к VH или VL), которая имеет последовательность, способную соединяться со своим родственным партнером и специфически связываться с CD- 3; и (b) парой когнатов (то есть VH и VL) последовательности, способной специфически связываться с CD-3. Иллюстративный «активный» VH, VL или пара VH/VL представляет собой последовательность дикого или исходного типа.
[00182] «CD- x» относится к белку кластера дифференцировки (CD). В иллюстративных вариантах осуществления CD-x выбирают из тех белков CD, которые играют роль в рекрутировании или активации Т-клеток у субъекта, которому была введена полипептидная конструкция согласно изобретению. В иллюстративном варианте осуществления CD-x представляет собой CD3.
[00183] Термин «связывающий домен» характеризует, в связи с данным изобретением, домен, который (в частности) связывается/взаимодействует с /распознает данный целевой эпитоп или данный целевой сайт на целевых молекулах (антигенах), например: EGFR и CD-3, соответственно. Структура и функция домена, связывающего целевой антиген, (распознающего EGFR), а предпочтительно и структура и/или функция CD-3-связывающего домена (распознающего CD3), основана(ы) на структуре и/или функции антитела, например, полноразмерной или полной иммуноглобулиновой молекулы. Согласно изобретению домен, связывающий целевой антиген, характеризуется наличием трех CDR легкой цепи (т.е. CDR1, CDR2 и CDR3 области VL) и/или трех CDR тяжелой цепи (то есть CDR1, CDR2 и CDR3 области VH). CD-3 связывающий домен предпочтительно также содержит по меньшей мере минимальные структурные требования к антителу, которые допускают целевое связывание. Более предпочтительно, чтобы CD-3-связывающий домен содержал по меньшей мере три CDR легкой цепи (т.е. CDR1, CDR2 и CDR3 области VL) и/или три CDR тяжелой цепи (т.е. CDR1, CDR2 и CDR3 области VH). Предполагается, что в иллюстративных вариантах осуществления целевой антиген и/или CD-3-связывающий домен получают путем или могут быть получены с помощью методов фагового дисплея или скрининга библиотеки.
[00184] Термин «Fc», «область Fc», «полипептид FC» и т.д., как используется в данном документе, означает антитело, как определено в данном документе, которое включает полипептиды, содержащие константную область антитела, за исключением первой константной области иммуноглобулина. Таким образом, Fc относится к последним двум иммуноглобулиновым доменам константной области IgA, IgD и IgG и последним трем иммуноглобулиновым доменам константной области IgE и IgM и гибкому шарнирому N-концу к этим доменам. Для IgA и IgM Fc может содержать J-цепь. Для IgG Fc содержит иммуноглобулиновые домены Cγ2 и Cγ3 и шарнир между Cγ1 и Cγ2. Хотя границы области Fc могут изменяться, область Fc тяжелой цепи человеческого IgG обычно определяется как содержащая остатки C226 или P230 на ее карбоксильном конце, причем нумерация соответствует индексу ЕС, как в Кабате. Fc может относиться к этой области изолированно или к этой области в контексте антитела, фрагмента антитела или Fc-слития. Fc может представлять собой антитело, Fc-слитие или белок, или белковый домен, который содержит Fc. Особенно предпочтительными являются варианты Fc, которые являются неприродными вариантами Fc.
[00185] Под «IgG», как используется в данном документе, подразумевается полипептид, относящийся к классу антител, которые, по существу, кодируются распознанным геном иммуноглобулина гамма. У людей этот класс включает IgG1, IgG2, IgG3 и IgG4. У мышей этот класс включает IgG1, IgG2a, IgG2b, IgG3. Под «иммуноглобулином (Ig)» в данном документе подразумевается белок, состоящий из одного или более полипептидов, по существу, кодированных генами иммуноглобулина. Иммуноглобулины включают, но не ограничиваясь антителами. Иммуноглобулины могут иметь ряд структурных форм, включая, но не ограничиваясь полноразмерными антителами, фрагментами антител и отдельными иммуноглобулиновыми доменами. Под «иммуноглобулином (Ig)» в данном документе подразумевается область иммуноглобулина, которая существует как отдельная структурная структура, как установлено специалистом в области структуры белка. Домены Ig обычно имеют характерную сэндвич-свернутую топологию. Известными доменами Ig в классе IgG антител являются VH, Cγ1, Cγ2, Cγ3, VL и CL.
[00186] Под «диким типом или ДТ» в данном документе подразумевается аминокислотная последовательность или нуклеотидная последовательность, которая находится в природе, включая аллельные вариации. Белок ДТ имеет аминокислотную последовательность или нуклеотидную последовательность, которая не была намеренно модифицирована.
[00187] Под «вариантным полипептидом», как используется в данном документе, понимается полипептидная последовательность, которая отличается от исходной полипептидной последовательности в силу по меньшей мере одной аминокислотной модификации. Модификации могут включать замены, удаления и дополнения. Вариантный полипептид может относиться к самому полипептиду, композиции, содержащей полипептид, или к аминокислотной последовательности, которая его кодирует. Предпочтительно вариантный полипептида имеет по меньшей мере одну аминокислотную модификацию по сравнению с исходным полипептидом, например, от около одной до десяти аминокислотных модификаций и предпочтительно от около одной до пяти аминокислотных модификаций по сравнению с исходным. Вариантная полипептидная последовательность в данном документе предпочтительно будет иметь по меньшей мере около 80% гомологии с исходной полипептидной последовательностью и наиболее предпочтительно по меньшей мере около 90% гомологии, более предпочтительно по меньшей мере около 95% гомологии. Соответственно, согласно «варианту Fc», как используется в данном документе, подразумевается последовательность Fc, которая отличается от исходной последовательности Fc в силу по меньшей мере одной аминокислотной модификации. Аналогичным образом иллюстративный «неактивный домен VL» или неактивный домен VH» представляет собой вариант исходного полипептида VL или VH.
[00188] В некоторых вариантах осуществления полипептидные конструкции согласно изобретению представляют собой «выделенные» или «по существу чистые» полипептидные конструкции. «Выделенный» или «по существу чистый», когда он используется для описания описанных в данном документе полипептидных конструкций, означает полипептидную конструкцию, которая была идентифицирована, разделена и/или выделена из компонента ее среды, в которой она была продуцирована. Предпочтительно, полипептидная конструкция свободна или практически не связана со всеми другими компонентами из ее среды, в которой она была продуцирована. Загрязняющие компоненты ее среды, в которой они продуцируются, такие, которые возникают из рекомбинантных трансфицированных клеток, являются вещества, обычно препятствуют диагностическому или терапевтическому применению полипептида и могут включать ферменты, гормоны и другие белковые или небелковые растворенные вещества. Желаемая полипептидная конструкция в среде для продуцирования может составлять по меньшей мере около 5%, по меньшей мере около 25% или по меньшей мере около 50% от общего веса полипептида в среде.
[00189] Иллюстративные выделенные полипептидные конструкции согласно изобретению по существу или практически не содержат компонентов, которые используются для получения материала. Для пептидных конъюгатов согласно изобретению термин «выделенный» относится к материалу, который по существу или практически не содержит компонентов, которые обычно сопровождают материал в смеси, используемой для получения пептидного конъюгата. «Выделенная» и «чистая» используются взаимозаменяемо. Как правило, выделенные полипептидные конструкции согласно изобретению имеют уровень чистоты, предпочтительно выраженный в диапазоне. Нижний предел диапазона чистоты для полипептидных конструкций составляет около 60%, около 70% или около 80%, а верхний предел диапазона чистоты составляет около 70%, около 80%, около 90% или более чем около 90%.
[00190] Когда полипептидные конструкции имеют более чем 90%-ную чистоту, их чистота также предпочтительно выражается как диапазон. Нижний предел диапазона чистоты составляет около 90%, около 92%, около 94%, около 96% или около 98%. Верхний предел диапазона чистоты составляет около 92%, около 94%, около 96%, около 98% или около 100% чистоты.
[00191] Чистота определяется любым признанным в данной области техники методом анализа (например, интенсивностью окрашивания полосы в окрашенном серебром геле, электрофорезом в полиакриламидном геле, ВЭЖХ или аналогичным способом).
[00192] В соответствии с данным изобретением связывающие домены находятся в форме одного или более полипептидов. Такие полипептиды могут содержать белковые части и небелковые части (например, химические линкеры или химические перекрестносшивающие агенты, такие как глутаровый альдегид). Полипептиды (включая их фрагменты, предпочтительно биологически активные фрагменты и пептиды, обычно имеющие более 30 аминокислот) содержат две или более аминокислот, связанных друг с другом посредством ковалентной пептидной связи (приводящей к образованию цепи аминокислот).
[00193] Взаимодействие между связывающим доменом и эпитопом или областью, содержащей эпитоп, подразумевает, что связывающий домен проявляет поддающуюся определению аффинность к эпитопу/области, содержащей эпитоп, на конкретном белке или антигене (например, EGFR и CD3, соответственно) и, как правило, не проявляет значительной реактивности с белками или антигенами, отличными от EGFR или CD3. «Поддающаяся определению аффинность» включает связывание с аффинностью около 10-6 М (KD) или более сильной. Предпочтительно связывание считается специфическим, если аффинность связывания составляет от 10-12 до 10-8 М, от 10-12 до 10-9 М, от 10-12 до 10-10 М, от 10-11 до 10-8 М, предпочтительно от около 10-11 до 10-8 М. Можно легко определить специфически взаимодействует ли или связывается ли связывающий домен с мишенью, в частности, путем сравнения реакции указанного связывающего домена с целевым белком или антигеном с реакцией указанного связывающего домена с белками или антигенами, отличными от EGFR или CD3. Предпочтительно, чтобы связывающий домен согласно изобретению фактически не содержал или по существу не связывался специфически с белками или антигенами, отличными от EGFR или CD3 (т.е. первый связывающий домен не связывается специфически с белками, отличными от EGFR, а второй связывающий домен не связывается специфически с белками, отличными от CD3).
[00194] Считается, что специфическое связывание обусловлено специфическими мотивами в аминокислотной последовательности связывающего домена и антигена. Таким образом, связывание достигается в результате их первичной, вторичной и/или третичной структуры, а также результатом вторичных модификаций указанных структур. Специфическое взаимодействие сайта взаимодействия c антигеном с его специфическим антигеном может привести к простому связыванию указанного сайта с антигеном. Более того, специфическое взаимодействие сайта взаимодействия с антигеном с его специфическим антигеном может альтернативно или дополнительно приводить к инициированию сигнала, например, из-за индукции изменения конформации антигена, олигомеризации антигена и т.д.
[00195] Термины «по существу не связываются специфически», «существенно не связываются специфически » или «не способны связываться специфически», используются взаимозаменяемо и означают, что связывающий домен согласно данному изобретению не связывает белок или антиген, отличный от EGFR, или CD3, т.е. не проявляет реакционную способность более чем 30%, предпочтительно не более чем 20%, более предпочтительно не более чем 10%, более предпочтительно не более чем 9%, 8%, 7%, 6% или 5% с белками или антигенами, отличными от EGFR или CD3, при этом связывание с EGFR или CD3, соответственно, установлено равным 100%. Данные термины также используются в отношении антигенсвязывающих свойств пары VH/VLi или VL/VHi.
[00196] Используемый в данном документе термин «биспецифический» относится к полипептидной конструкции согласно изобретению («Pro» или «Prodent»), которая является «по меньшей мере биспецифической», то есть она содержит по меньшей мере первый связывающий домен (например, целевой антиген, например, EGFR) и второй связывающий домен (например, CD-3, например, CD3), при этом первый связывающий домен связывается с одним антигеном или мишенью, а второй связывающий домен связывается с другим антигеном или мишенью. Соответственно, полипептидные конструкции согласно изобретению имеют специфичности по меньшей мере для двух разных антигенов или мишеней. Термин «биспецифическая полипептидная конструкция» согласно изобретению также охватывает мультиспецифические полипептидные конструкции, такие как триспецифические полипептидные конструкции, последние из которых включают три связывающих домена или конструкции, имеющие более трех (например, четыре, пять) специфичностей.
[00197] Учитывая, что полипептидные конструкции в соответствии с изобретением являются (по меньшей мере) биспецифическими, они не встречаются естественным образом, и они заметно отличаются от природных продуктов. Таким образом, «биспецифическая» полипептидная конструкция представляет собой искусственный гибридный полипептид, имеющий по меньшей мере два различных сайта связывания с различными специфичностями. Биспецифические полипептидные конструкции могут быть получены различными способами. См., например, Songsivilai & Lachmann, Clin. Exp. Immunol. 79:315-321 (1990).
[00198] По меньшей мере два связывающих домена и вариабельных домена полипептидной конструкции согласно данному изобретению могут содержать или не содержать пептидные линкеры (спейсерные пептиды). Термин «пептидный линкер» включает в соответствии с данным изобретением аминокислотную последовательность, посредством которой аминокислотные последовательности одного (вариабельного и/или связывающего) домена и другого (вариабельного и/или связывающего) домена конструкции антитела согласно изобретению связаны друг с другом. Существенной технической особенностью такого пептидного линкера является то, что он не имеет никакой полимеризационной активности. Среди подходящих пептидных линкеров есть те, которые описаны в патенте США No. №№ 4751180 и 4935233 или WO 88/09344. Пептидные линкеры также могут быть использованы для присоединения других доменов или модулей, или областей (например, доменов увеличения периода полувыведения) к конструкции антитела согласно изобретению.
[00199] В тех вариантах осуществления, в которых используется линкер, этот линкер предпочтительно имеет длину и последовательность, достаточные для обеспечения того, чтобы каждый из целевых антигенов и CD-3-связывающих доменов мог независимо друг от друга сохранять свои различающиеся специфичности связывания. Для пептидных линкеров, соединяющих по меньшей мере два связывающих домена (или два вариабельных домена) в конструкции антитела согласно изобретению, эти пептидные линкеры являются предпочтительными, которые содержат оптимизированное количество аминокислотных остатков, например, 12 аминокислотных остатков или менее. Таким образом, используют пептидные линкеры из 12, 11, 10, 9, 8, 7, 6 или 5 аминокислотных остатков. Предполагаемый пептидный линкер с менее чем 5 аминокислотами содержит 4, 3, 2 или одну аминокислоту(ы), причем Gly-богатые линкеры являются предпочтительными. Особенно предпочтительной «единственной» аминокислотой в контексте указанного «пептидного линкера» является Gly. Соответственно, указанный пептидный линкер может состоять из одной аминокислоты Gly. Другой предпочтительный вариант осуществления пептидного линкера характеризуется аминокислотной последовательностью Gly-Gly-Gly-Gly-Ser, то есть Gly4Ser (SEQ ID NO: 1), или их полимерами, т.е. (Gly4Ser)x, где x представляет собой целое число, равное 1 или более (например, 2 или 3). Характеристики упомянутого пептидного линкера, которые включают отсутствие стимулирования вторичных структур, известны в данной области техники и описаны, например, в Dall'Acqua et al. (Biochem. (1998) 37, 9266-9273), Cheadle et al. (Mol Immunol (1992) 29, 21-30) и Raag and Whitlow (FASEB (1995) 9(1), 73-80). Также полезны пептидные линкеры, которые, кроме того, не способствуют образованию каких-либо вторичных структур. Связывание указанных доменов друг с другом может быть обеспечено, например, с помощью генной инженерии, как описано в примерах. Способы получения слитых и функционально связанных биспецифических одноцепочечных конструкций и экспрессии их в клетках млекопитающих или бактериях хорошо известны в данной области техники (например, WO 99/54440 или Sambrook et al., Molecular Cloning: A Laboratory Manual, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, N.Y., 2001).
[00200] Иллюстративные варианты осуществления изобретения включают по меньшей мере один домен scFv, который, хотя и не встречается в природе, обычно содержит вариабельный домен тяжелой цепи и вариабельный домен легкой цепи, соединенные вместе с scFv-линкером. Как указано в данном документе, в то время как домен scFv обычно находится от N- к С-концу, ориентированного как VH -scFv-линкер-VL, это может быть отменено для любого из доменов scFv (или построенных с использованием последовательностей VH и VL из Fab), к VL-scFv-линкер-VH, с необязательными линкерами на одном или обоих концах в зависимости от формата. Как правило, один из VL или VH является «неактивным».
[00201] Как показано в данном документе, существует ряд подходящих scFv-линкеров, которые могут быть использованы, включая стандартные пептидные связи, генерируемые рекомбинантными методами. Линкерный пептид может преимущественно содержать следующие аминокислотные остатки: Gly, Ser, Ala или Thr. Линкерный пептид должен иметь длину, достаточную для связывания двух молекул таким образом, чтобы они принимали правильную конформацию относительно друг друга, чтобы сохранить желаемую активность. В одном варианте осуществления линкер имеет длину приблизительно от 1 до 50 аминокислот, предпочтительно от 1 до 30 аминокислот в длину. В одном варианте осуществления могут быть использованы линкеры длиной от 1 до 20 аминокислот, причем в некоторых вариантах осуществления используют от около 5 до около 10 аминокислот. Полезные линкеры включают глицин-сериновые полимеры, включая, например, (GS)n, (GSGGS)n, (GGGGS)n и (GGGS)n, где n представляет собой целое число по меньшей мере равное единице (и, как правило, от 3 до 4) глицин-аланиновые полимеры, аланин-сериновые полимеры и другие гибкие линкеры. Альтернативно, различные небелковые полимеры, включая, но не ограничиваясь следующими: полиэтиленгликоль (ПЭГ), полипропиленгликоль, полиоксиалкилены или сополимеры полиэтиленгликоля и полипропиленгликоля, могут найти применение в качестве линкеров, то есть могут найти применение в качестве линкеров.
[00202] Другие линкерные последовательности могут содержать любую последовательность любой длины домена CL/ CH1, но не все остатки домена CL/CH1; например, первые 5-12 аминокислотных остатков доменов CL/CH1. Линкеры могут быть получены из легкой цепи иммуноглобулина, например Cκ или Cλ. Линкеры могут быть получены из тяжелых цепей иммуноглобулина любого изотипа, включая, например, Cγ1, Cγ2, Cγ3, Cγ4, Cα1, Cα2, Cδ, Cε, и Cμ. Линкерные последовательности также могут быть получены из других белков, таких как Ig-подобные белки (например, TCR, FcR, KIR), последовательности, полученные из шарнирной области, и другие природные последовательности из других белков.
[00203] В некоторых вариантах осуществления линкер является «линкером домена», который используется для связывания любых двух доменов, как описано в данном документе. Хотя может быть использован любой подходящий линкер, во многих вариантах используют глицин-сериновый полимер, включая, например, (GS)n, (GSGGS)n, (GGGGS)n и (GGGS)n, где n представляет собой целое число равное по меньшей мере единице (и обычно от 3 до 4, до 5), а также любую пептидную последовательность, которая обеспечивает рекомбинантное присоединение двух доменов с достаточной длиной и гибкостью, чтобы позволить каждому домену сохранять свою биологическую функцию. В некоторых случаях и с уделением особого внимания «цепочечности», как описано ниже, могут использоваться заряженные доменные линкеры, используемые в некоторых вариантах осуществления scFv-линкеров. Иллюстративными линкерами доменов являются нерасщепляемые линкеры, которые по существу не расщепляются в условиях, в которых используются полипептидные конструкции, например, при физиологически значимых значениях рН и температуры в течение периода полувыведения in vivo полипептидных конструкций. Доменные линкеры могут содержать один или более расщепляемых фрагментов в их остове.
[00204] В некоторых вариантах осуществления scFv-линкер или домен представляет собой заряженный scFv-линкер или доменный линкер.
[00205] Под «методом компьютерного скрининга» в данном документе подразумевается любой способ конструирования одной или более полипептидных конструкций согласно изобретению, включая мутации в компоненте (например, VH, VL) конструкции, причем указанный способ использует компьютер для оценки энергий взаимодействий между возможными заменами боковых цепей аминокислот друг с другом и/или с остальной частью белка. Как будет понятно специалистам в данной области техники, оценка энергий, называемая вычислением энергии, относится к некоторому способу подсчета одной или более аминокислотных модификаций. Указанный способ может включать физический или химический энергетический термин или может включат энергетические термины, основанные на знаниях, статистике, последовательности и тому подобное. Расчеты, которые составляют метод компьютерного скрининга, в дальнейшем называются как «расчеты компьютерного скрининга».
Варианты осуществления
Полипептидные конструкции
[00206] Согласно предпочтительному варианту осуществления и как описано в прилагаемых примерах, иллюстративная полипептидная конструкция согласно изобретению представляет собой «биспецифическую одноцепочечную полипептидную конструкцию», более предпочтительно «одноцепочечную Fv» (scFv), содержащую по меньшей мере один домен, связывающий целевой антиген, и, необязательно, дополнительно связанный с по меньшей мере одним доменом увеличения периода полувыведения. Хотя два домена Fv-фрагмента VL и VH кодируются отдельными генами, их можно объединить, используя рекомбинантные методы, синтетическим линкером, как описано выше, что позволяет их делать в виде одиночной белковой цепи, в которой пары VL и VH образуют пару неактивных VL/VH ; см., например, Huston et al. (1988) Proc. Natl. Acad. Sci USA 85:5879-5883). Эти полипептидные фрагменты получают с использованием общепринятых методов, известных специалистам в данной области техники, и фрагменты оценивают на функции таким же образом, как и полные или полноразмерные антитела. Одноцепочечный вариабельный фрагмент (scFv) является, следовательно, слитым белком вариабельной области тяжелой цепи (VH) и легкой цепи (VL) иммуноглобулинов, обычно связанной коротким линкерным пептидом от около десяти до около 25 аминокислот, предпочтительно от около 15 до 20 аминокислот. Линкер обычно богат глицином для гибкости, а также серином или треонином для растворимости и может или соединять N-конец VH с C-концом VL, или наоборот. Часть полипептида в «неактивном» состоянии, по существу, сохраняет специфичность исходного иммуноглобулина, несмотря на удаление константных областей и введение линкера.
[00207] Таким образом, в иллюстративном варианте осуществления данное изобретение относится к одноцепочечному полипептиду scFv, направленному на антиген CD-3. Полипептид scFv содержит первый домен scFv, содержащий первый домен VH и первый домен VL, соединенные через первый линкерный фрагмент (например, scFv-линкер). Первый линкерный фрагмент необязательно содержит первый сайт расщепления протеазой между первым VH и первым доменом VL. Первый домен VH и первый домен VL взаимодействуют с образованием первой пары VH/VL. Чтобы обеспечить полипептид способностью при определенных условиях связывать свою цель CD-3, один из первого домена VH и первого домена VL должен быть неактивным так, как этот термин определен в данном документе (то есть VHi или VLi). Соответственно, иллюстративный первый домен scFv не связывает специфически антиген CD-3. При расщеплении протеазы первого scFv-линкера в сайте расщепления протеазой неактивный VH или неактивный домен VL отделяется от его активного VL или активного VH- связывающего партнера, который затем может соединяться с его активным родственным вариантом, обеспечивая правильно спаренный анти-CD-3 для образования и связывания антигена CD-3. В иллюстративном варианте осуществления два активных родственных варианта находятся в одной и той же полипептидной цепи. В различных вариантах осуществления два активных родственных варианта находятся в отдельных полипептидных цепях, которые объединяются и взаимодействуют на поверхности клетки с образованием активного CD-3-связывающего домена, который специфически связывает CD-3.
[00208] Первый полипептид scFv соединяют с помощью первого линкерного фрагмента домена, необязательно содержащего второй сайт расщепления протеазой, со вторым доменом scFv. Второй домен scFv содержит второй домен VH и второй домен VL, соединенный через второй линкерный фрагмент scFv. Второй линкерный фрагмент scFv содержит третий сайт расщепления протеазой между вторым доменом VH и вторым доменом VL. Второй домен VH и второй домен VL взаимодействуют с образованием второй пары VH/VL. Как и в случае первой пары VH/VL, описанной выше, один из указанного второго домена VH и указанного второго V L является неактивным, так что указанный второй домен scFv не связывает специфически антиген CD-3. Первый домен scFv соединяется через второй доменный линкер с первым доменом, связывающим целевой антиген. Этот второй доменный линкер соединяет элемент, выбранный из первого домена VH и указанного первого домена VL, с первым доменом, связывающим целевой антиген. Второй домен scFv соединен через третий доменный линкер со вторым доменом, связывающим целевой антиген. Этот третий доменный линкер соединяет член, выбранный из второго домена VH, и второго домена VL, со вторым доменом, связывающим целевой антиген.
[00209] В иллюстративном варианте осуществления изобретение относится к одноцепочечному полипептиду scFv, направленному на антиген CD-3. Полипептид scFv содержит первый домен scFv, содержащий первый домен VH и первый домен VL, присоединенный через первый линкерный фрагмент scFv. Первый линкерный фрагмент scFv содержит первый сайт расщепления протеазой между первым VH и указанным первым VL- доменом. Как указано выше, первый домен VH и первый домен VL взаимодействуют с образованием первой пары VH/VL, в которой один из первого домена VH и первого домена VL является неактивным первым доменом VH или неактивным первым доменом VL. Таким образом, первый домен scFv не связывает специфически антиген CD-3. Первый полипептид scFv соединяют с помощью первого линкерного фрагмента домена, необязательно содержащего второй сайт расщепления протеазой со вторым доменом scFv, содержащим второй домен VH и второй домен VL, соединенный через второй линкерный фрагмент scFv, содержащий третий сайт расщепления протеазой между вторым доменом VH и вторым доменом VL. Второй домен VH и указанный второй домен VL взаимодействуют с образованием второй пары VH/VL. Как описано выше, один из второго домена VH и второго VL является неактивным вторым VH или неактивным вторым VL-доменом, а второй домен scFv не связывает специфически указанный антиген CD-3.
[00210] Первый домен scFv данного полипептида соединен через второй доменный линкер с первым доменом, связывающим целевой антиген, причем указанный второй доменный линкер соединяет член, выбранный из первого домена VH и первого домена VL, с первым доменом, связывающим целевой антиген. Второй домен scFv соединен через третий доменный линкер со вторым доменом, связывающим целевой антиген. Третий доменный линкер соединяет член, выбранный из второго домена VH, и второго домена VL, со вторым доменом, связывающим целевой антиген. После приведения в контакт одноцепочечной scFv с первой протеазой, способной расщеплять первый сайт расщепления протеазой первого линкерного фрагмента scFv, неактивный первый домен VH или неактивный первый домен VL отделяют от одноцепочечного полипептида scFv. Аналогичным образом, когда полипептид взаимодействует со второй протеазой, способной расщеплять второй сайт расщепления протеазой второго линкерного фрагмента scFv, неактивный второй домен V H или неактивный второй домен VL отделяют от одноцепочечного полипептида scFv. Разделение неактивных доменов от активных доменов полипептида образует активный одноцепочечный Fv, способный специфически связывать антиген CD-3.
[00211] В иллюстративном варианте осуществления изобретение относится к одноцепочечному полипептиду scFv, направленному на антиген CD-3. Полипептид scFv содержит первый домен scFv, содержащий первый домен VH и первый домен VL, присоединенный через первый линкерный фрагмент scFv. Первый линкерный фрагмент содержит первый сайт расщепления протеазой между первым VH и первым доменом VL. Первый домен VH и указанный первый VL-домен взаимодействуют с образованием первой пары VH/VL, в которой один из первого домена VH и первого домена VL является неактивным. Соответственно, первый домен scFv не связывает специфически антиген CD-3. Первый полипептид scFv соединяют с помощью первого линкерного фрагмента домена, необязательно содержащего второй сайт расщепления протеазой, с первым доменом, связывающим целевой антиген. Этот первый доменный линкер соединяет элемент, выбранный из первого домена VH и первого домена VL, с первым доменом, связывающим целевой антиген.
[00212] В иллюстративном варианте осуществления предлагается пара таких конструкций ScFv, как описано выше. Пара из конструкций взаимодействует с антигеном CD-3 через их спаренные CD-3-связывающие домены. Связывание с антигеном CD-3 спаренных сайтов CD-3 отдельных молекул scFv пары облегчается, усиливается и/или управляется связыванием домена, связывающего целевой антиген, каждого члена пары с его родственным антигеном.
[00213] Полипептидные конструкции способны специфически связываться с одним или более целевыми антигенами, а также CD3, и, необязательно, доменом увеличения периода полувыведения, таким как ЧСА-связывающий домен. Связывание с CD3 возможно только после активации протеазой и связывания с целевым антигеном(ами). Следует понимать, что в некоторых вариантах осуществления протеазное расщепление домена расщепления протеазой происходит до связывания домена, связывающего целевой антиген, с целевым антигеном. Также следует понимать, что в некоторых вариантах осуществления протеазное расщепление домена расщепления протеазой происходит после связывания домена, связывающего целевой антиген, с целевым антигеном.
[00214] В некоторых вариантах осуществления полипептид scFv дополнительно содержит два или более доменов расщепления протеазой. В некоторых вариантах осуществления один или более СD3-связывающих доменов содержат полипептид, полученный из одноцепочечного вариабельного фрагмента (scFv), специфического для человеческого CD3. В иллюстративном варианте осуществления данный CD3-связывающий домен содержит VL- и VH-фрагмент, связанный линкером, в котором есть домен расщепления протеазой. В этом CD3-связывающем домене или VL, или VH делают неактивным (то есть практически неспособным специфически связывать CD3) при помощи известного способа, например, мутации в одном или более сайтах в VL или VH, делеции CDR, и т. д. Этот мутированный VL или VH является возможно спаренным и является спаренным с соответствующими VH или VL, соответственно, которые при отсутствии спаривания с мутированной последовательностью (или спаривания с соответствующей родственной последовательностью) способен по существу избирательно связывать CD3. Объединение неактивного VL или VH с соответствующим CD3-связывающим VH или VL делает CD3-связывающий VH или VL неактивным до тех пор, пока партнеры не будут разделены протеазным расщеплением домена расщепления протеазой в линкере. При расщеплении домена расщепления протеазой активные CD3-связывающие типы и его неактивный партнер являются «неспаренными», что позволяет CD3-связывающему домену по существу специфически связывать CD3 в паре с его активным комплементарным CD-3-связывающим доменом. В различных вариантах осуществления неактивный партнер оказывается неактивным из-за мутации одной или более аминокислот в CD3-связывающем VL или VH, причем мутация существенно разрушает способность партнера связываться с CD3 определенным образом, оставляя способность мутированных видов спариваться с CD3-связывающим доменом, тем самым существенно инактивируя CD3-связывающие характеристики CD3-связывающего домена до тех пор, пока партнеры не будут разделены расщеплением домена расщепления протеазой.
[00215] Как показано в примерах ниже, изобретатели обнаружили, что активность и эффективность полипептидных конструкций согласно изобретению мало зависят от ориентации различных доменов. Таким образом, отсчитывая от N-конца до С-конца, VL может находиться выше VHi или наоборот. Кроме того, VLi может находиться выше VH или наоборот. Домен удлинения полувыведения может быть присоединена к одному из неактивных CD-3-связывающих доменов или к домену, связывающему целеовой антиген. В иллюстративном варианте осуществления домен увеличения периода полувыведения соединяется с компонентом полипептидной конструкции, которая отделяется от активного полипептида при расщеплении scFv-линкера. Так, например, домен увеличения периода полувыведения соединяется с VLi или VHi.
[00216] В некоторых вариантах осуществления, один или более доменов увеличения периода полувыведения содержат связывающий домен для человеческого сывороточного альбумина. В некоторых вариантах осуществления один или более доменов увеличения периода полувыведения содержат scFv, вариабельный домен тяжелой цепи (VH), вариабельный домен легкой цепи (VL), наноантитело, пептид, лиганд или малую молекулу. В некоторых вариантах осуществления один или более доменов увеличения периода полувыведения содержит scFv. В некоторых вариантах осуществления один или более доменов увеличения периода полувыведения содержит домен Fc. В некоторых вариантах осуществления домен увеличения периода полувыведения находится на N-конце полипептида до расщепления протеазой. В некоторых вариантах осуществления домен увеличения периода полувыведения находится на C-конце полипептида до расщепления протеазой. В некоторых вариантах осуществления домен увеличения периода полувыведения не находится на C-конце или N-конце полипептида до расщепления протеазой.
[00217] Домен увеличения периода полувыведения позволяет изменять размер полипептидной конструкции практически по любому желаемому размеру для достижения подходящих фармакокинетических параметров. Соответственно, полипептидные конструкции, описанные в данном документе, в некоторых вариантах осуществления изобретения, имеют размер от около 50 кДа до около 150 кДа, от 50 кДа до около 100 кДа, от 50 кДа до около 80 кДа, от около 50 кДа до около 75 кДа, от около 50 кДа до около 70 кДа, или от около 50 кДа до около 65 кДа. Таким образом, размер антигенсвязывающих полипептидов выгоден по сравнению с антителами IgG, которые около 150 кДа, и молекул диател BiTE и DART, которые около 55 кДа, но не продлевают период полувыведения и поэтому быстро очищаются через почки. Другая особенность антигенсвязывающих полипептидов, описанных в данном документе, заключается в том, что они представляют собой однополипептидный дизайн с гибкой связью их доменов. Это позволяет легко производить и изготавливать полипептидные конструкции, поскольку они могут быть закодированы одной молекулой кДНК, которые легко могут быть включены в вектор. Кроме того, поскольку описанные в данном документе антигенсвязывающие полипептиды являются мономерной одиночной полипептидной цепью, нет проблем со спариванием цепей или требованием для димеризации. Предполагается, что описанные в данном документе антигенсвязывающие полипептиды имеют пониженную тенденцию к агрегации в отличие от других описанных молекул, таких как биспецифические белки BiTE.
[00218] Биспецифические одноцепочечные молекулы известны в данной области техники и описаны в WO 99/54440, Mack, J. Immunol. (1997), 158, 3965-3970, Mack, PNAS, (1995), 92, 7021-7025, Kufer, Cancer Immunol. Immunother., (1997), 45, 193-197, Loffler, Blood, (2000), 95, 6, 2098-2103, Bruhl, Immunol., (2001), 166, 2420-2426, Kipriyanov, J. Mol. Biol., (1999), 293, 41-56. Способы, описанные для производства одноцепочечных антител (см., в частности, патент США № 4946778) могут быть адаптированы для получения одноцепочечных полипептидных конструкций, специфически распознающих выбранную цель(и).
[00219] Полипептиды с более высокой валентностью, аналогичные двухвалентным антителам, также входят в объем данного изобретения. Например, в изобретение включена полипептидная конструкция, связывающаяся с двумя CD-3, например, 3 молекулами или двумя субъединицами CD3 в Т-клеточном рецепторе. Аналогично, полипептидные конструкции согласно изобретению могут содержать два или более доменов, связывающих целевой антиген. Таким образом, конструкции согласно изобретению могут содержать два или более идентичных, или два или более разных домена анти-EGFR-связывающих домена. Такие молекулы можно рассматривать как аналогичные двухвалентным (также называемым бивалентным) или биспецифическим одноцепочечным вариабельных фрагментам (bi-scFvs или di-scFv, имеющим формат (scFv)2. Такие конструкции могут быть сконструированы путем связывания двух молекул scFv (например, с линкерами, как описано выше). Если эти две молекулы scFv имеют одинаковую специфичность связывания, то полученная молекула (scFv)2 обычно известна как «двухвалентная» (т.е. имеет две валентности для одного и того же целевого эпитопа). Если две молекулы scFv имеют разную специфичность связывания, то полученную молекулу (scFv)2 обычно называют биспецифической. Связывание может быть осуществлено путем создания единственной пептидной цепи с двумя областями VH и двумя областями VL, с получением тандемных scFv (см., например, Kufer P. et al., (2004) Trends in Biotechnology 22(5):238-244).
[00220] Антигенсвязывающие полипептиды scFv, описанные в данном документе, предназначены для обеспечения специфического нацеливания клеток, экспрессирующих целевой антиген, путем рекрутинга цитотоксических Т-клеток. CD3-связывающий домен остается неактивным до тех пор, пока активируется протеазным расщеплением сайта расщепления протеазой, расположенного между или VH и VLi или VHi и VL, в котором «i» обозначает субъединицу, инактивированную мутацией исходной полипептидной последовательности из или VH, или VL. Это улучшает специфичность по сравнению с биспецифическими лекарственными средствами, привлекающими T-клетки, которые связываются с CD3 и целевым антигеном, который может или не может быть экспрессирован целевой клеткой, такой как опухоль или раковая клетка. В противоположность этому, путем активации связывания CD3 специфически в микроокружении целевой клетки, где целевой антиген и протеазы экспрессируются на высоком уровне, полипептидные конструкции могут сшивать цитотоксические Т-клетки с клетками, экспрессирующими целевой антиген, с высокой специфичностью, тем самым направляя цитотоксический потенциал Т-клетки к целевой клетке. Описанные в данном документе полипептидные конструкции взаимодействуют с цитотоксическими Т-клетками посредством протеаз-активированного связывания с поверхностно-экспрессируемым CD3, который является частью рецепторного комплекса Т-клеток. Одновременное связывание нескольких полипептидных конструкций с CD3 и целевым антигеном, экспрессируемым на поверхности конкретных клеток, вызывает активацию Т-клеток и опосредует последующий лизис конкретной клетки, экспрессирующей целевой антиген. Таким образом, предполагается, что полипептидные конструкции демонстрируют сильное, специфическое и эффективное уничтожение целевых клеток.
[00221] В некоторых вариантах осуществления описанные в данном документе полипептидные конструкции стимулируют уничтожение целевых клеток цитотоксическими Т-клетками для элиминации патогенных клеток в богатом протеазой микроокружении (например, опухолевые клетки, клетки, инфицированные вирусом или бактериями, аутореактивные Т-клетки и т.д.). В некоторых из таких вариантов осуществления клетки избирательно элиминируются, тем самым снижая вероятность токсических побочных эффектов. В других вариантах осуществления те же самые полипептиды могут быть использованы для усиления элиминации эндогенных клеток для терапевтического эффекта, таких как B или Т-лимфоциты при аутоиммунном заболевании, или гемопоэтических стволовых клеток (ГСК) для трансплантации стволовых клеток. Протеазы, которые, как известно, связаны с пораженными клетками или тканями, включают, но не ограничиваются следующими: сериновые протеазы, цистеиновые протеазы, аспартильные протеазы, треониновые протеазы, глутаминовые протеазы, металлопротеазы, аспарагин-пептид-лиазы, сывороточные протеазы, катепсины, катепсин B, катепсин C, катепсин D, катепсин E, катепсин K, катепсин L, калликрин, hK1, hK10, hK15, плазмин, коллагеназу, коллагеназу IV типа, стромелизин, фактор Xa, химотрипсинподобную протеазу, трипсиноподобную протеазу, эластазоподобную протеазу, субтилизин-подобную протеазу, актинидаин, бромелайн, кальпаин, каспазы, каспазу-3, Mir1-CP, папаин, протеазу ВИЧ-1, протеазу ВПГ, протеазу ЦМВ, химозин, ренин, пепсин, матриптазу, лемумин, плазмепсин, непентезин, металлоэкзопептидазы, металлоэндопептидазы, матричные металлопротеазы (MMP), MMP1, MMP2, MMP3, MMP8, MMP9, MMP13, MMP11, MMP14, урокиназный активатор плазминогена (uPA), энтерокиназу, простатический специфический антиген (PSA, hK3), интерлейкин-1β-превращающий фермент, тромбин, FAP (FAP-α), дипептидилпептидазу, меприны, гранзимы и дипептидилпептидазу IV (DPPIV/CD26).
[00222] Антигенсвязывающие полипептиды, описанные в данном документе, придают дополнительные терапевтические преимущества более известных моноклональных антител и других небольших молекул биспецифических. Биспецифические молекулы предназначены для связывания с целевой клеткой посредством клеточноспецифического маркера, связанного с патогенной клеткой. Токсичность возможна, если в некоторых случаях здоровые клетки или ткани экспрессируют один тот же маркер, что и патогенные клетки. Одним из преимуществ конструкции антигенсвязывающего полипептида согласно изобретению является то, что связывание с CD-3 зависит от активации протеазой, экспрессируемой целевой клеткой, такой как опухолевая клетка, и связыванием антигенсвязывающих доменов с одним или более целевыми антигенами, например, опухолевым антигеном. Полипептидные конструкции содержат неактивный CD-3-связывающий домен, содержащий домены VH и VLi или VHi и VL, разделенные одним или более сайтами расщепления протеазой. В богатой протеазой среде целевой клетки, сайты расщепления протеазой расщепляются, разделяя VH и VLi или VHi и VL и обеспечивая взаимодействие VH и VL или VL и VH с образованием активного CD-3-связывающего домена и связыванием конструкции с CD-3 при связывании одного или более целевых антигенов. В отсутствие расщепления протеазой CD-3-связывающий домен неактивен и не может связываться с CD-3.
[00223] Также представлены полипептидные конструкции, которые являются отдельными молекулами, которые образуют пару с активным анти-CD-3 scFv после протеазного расщепления. Таким образом, полипептидная конструкция, которая является первым членом пары, содержит VH/VLi или VHi/VL-домен, а VLi или VHi расщепляется протеазой из первого члена пары. Соответствующий VLi или VHi отщепляется от второго члена пары, что позволяет образовывать VL/VH- домен путем спаривания двух отдельных молекул на CD-3-мишени. В одном аспекте эти молекулы «полу-Pro» используются в качестве средства для для создания молекул «полного Pro», позволяя легко изменять две молекулы, образующие пару, и позволяя перевод информации, полученной из этих экспериментов, в разработку и подготовку соответствующей конструкции полипептида «полного Pro». В различных вариантах осуществления молекулы «полу-Pro» должны быть связаны как с CD-3-мишенью, так и с целевым антигеном для образования функциональной пары анти-CD-3 VL/VH.
[00224] Таким образом, также в данном документе предложена полипептидная конструкция, причем белок содержит одну полипептидную цепь, содержащую домен расщепления протеазой (P), разделяющий цепь на первую и вторую CD-3-связывающей области; причем первая область содержит анти-CD3 VH-связывающий домен (CVH) и домен, связывающий целевой антиген, (T1), а вторая область содержит анти-CD3 VL-связывающий домен (CVL) и домен, связывающий целевой антиген, (T2); причем белок необязательно содержит домен увеличения периода полувыведения (H) в первой или второй области и при этом после активации путем протеазного расщепления Р и связывания целевого антигена посредством Т1 и Т2, первая и вторая области объединяются с образованием полного анти-CD3 VL/VH-связывающего домена, который связывает CD3.
[00225] Также в некоторых аспектах в данном документе представлена полипептидная конструкция, причем белок содержит одну полипептидную цепь, содержащую домен расщепления протеазой (Р), разделяющий цепь на первую и вторую области; при этом первая область содержит анти-CD-3 VL- связывающий домен (CVL) и домен, связывающий целевой антиген, (T1), а вторая область содержит анти-CD3 VH-связывающий домен (CVH) и домен, связывающий целевой антиген, (T2); причем белок необязательно содержит домен увеличения периода полувыведения (H) в первой или второй области и при этом после активации путем протеазного расщепления Р и связывания целевого антигена посредством Т1 и Т2, первая и вторая области объединяются с образованием полного анти-CD3 VL/VH-связывающего домена, который связывает CD3.
[00226] Также в некоторых аспектах в данном документе представлена полипептидная конструкция, причем белок содержит одну полипептидную цепь, содержащую первую и вторую области; причем первая область содержит анти-CD3 VH-связывающий домен (CVH), неактивный анти-CD3 VL-связывающий домен (CVLi), который связывается с CVH и доменом, связывающим целевой антиген, (T1); при этом вторая область содержит анти-CD3 VL-связывающий домен (CVL); неактивный анти-CD3 VH-связывающий домен (CVHI), который объединяется с CVL и доменом, связывающим целевой антиген, (Т2); причем белок необязательно содержит домен увеличения периода полувыведения (H) в первой и/или второй области и при этом каждый из CVLi и CVHi содержит по меньшей мере один домен расщепления протеазой; и при этом после активации протеазным расщеплением домены расщепления протеазой и связывания целевого антигена посредством Т1 и Т2, первая и вторая области объединяются с образованием полного анти-CD3 VL/VH-связывающего домена, который связывает CD3.
[00227] Также в некоторых аспектах в данном документе представлена полипептидная конструкция, причем белок содержит одну полипептидную цепь, содержащую первую и вторую области; причем первая область содержит анти-CD3 VL-связывающий домен (CVL), неактивный анти-CD3 VH-связывающий домен (CVHi), который связывается с CVL и доменом, связывающим целевой антиген, (T1); причем вторая область содержит анти-CD3 VH-связывающий домен (CVH); неактивный анти-CD3 VL-связывающий домен (CVLi), который объединяется с CVH и доменом, связывающим целевой антиген, (T2); причем белок необязательно содержит домен увеличения периода полувыведения (H) в первой и/или второй области и при этом каждый из CVLi и CVHi содержит по меньшей мере один домен расщепления протеазой; и причем после активации протеазным расщеплением домены расщепления протеазой и связывания целевого антигена посредством Т1 и Т2, первая и вторая области объединяются с образованием полного анти-CD3 VL/VH-связывающего домена, который связывает CD3.
[00228] В одном аспекте антигенсвязывающие белки, в предварительно активированной форме, содержат одну полипептидную цепь, содержащую первый домен, содержащий по меньшей мере один анти-CD3-связывающий домен и вторую область, содержащую по меньшей мере один анти-мишень-связывающий домен. Первая область и вторая область разделены полипептидным линкером, который необязательно содержит одну или более расщепляемых фрагментов в своей последовательности, например, по меньшей мере один домен расщепления протеазой (Р). В иллюстративном варианте осуществления первая область содержит анти-CD3 VH-связывающий домен (CVH) и домен, связывающий целевой антиген, (T1). В одном варианте осуществления вторая область содержит анти-CD3 VL-связывающий домен (CVL) и целевой домен, связывающий целевой антиген, (T2). В одном варианте осуществления антигенсвязывающий домен необязательно содержит домен увеличения периода полувыведения (H) в первой области. В одном варианте осуществления антигенсвязывающий домен необязательно содержит домен увеличения периода полувыведения (H) во второй области. Однажды, активированный протеазным расщеплением домен расщепления протеазой (Р) и связывающие целевой антиген(ы) домены Т1 и Т2, связывающие целевые антигены, анти-CD3-связывающие домены CVH и CVL активируются для связывания с CD3 на T- клетке. Предполагается, что домены в антигенсвязывающем белке будут расположены в любом порядке в каждой области с доменом расщепления протеазой (Р) в центре предварительно активированного полипептида. Кроме того, каждая область может находиться в любом порядке внутри предварительно активированного полипептида. Таким образом, только в качестве примера предполагается, что иллюстративный порядок доменов полипептидных конструкций включает, но не ограничивается следующими:
a) CVH-T1-P-T2-CVL,
b) T1-CVH-P-T2-CVL,
c) CVH-T1-P-CVL-T2,
d) T1-CVH-P-CVL-T2,
e) H-CVH-T1-P-T2-CVL,
f) CVH-H-T1-P-T2-CVL,
g) CVH-T1-H-P-T2-CVL,
h) CVH-T1-P-H-T2-CVL,
i) CVH-T1-P-T2-H-CVL,
j) CVH-T1-P-T2-CVL-H,
k) H-T1-CVH-P-T2-CVL,
l) T1-H-CVH-P-T2-CVL,
m) T1-CVH-H-P-T2-CVL,
n) T1-CVH-P-H-T2-CVL,
o) T1-CVH-P-T2-H-CVL,
p) T1-CVH-P-T2-CVL-H,
q) H-CVH-T1-P-CVL-T2,
r) CVH-H-T1-P-CVL-T2,
s) CVH-T1-H-P-CVL-T2,
t) CVH-T1-P-H-CVL-T2,
u) CVH-T1-P-CVL-H-T2,
v) CVH-T1-P -CVL-T2-H,
w) H-T1-CVH-P-CVL-T2,
x) T1-H-CVH-P-CVL-T2,
y) T1-CVH-H-P-CVL-T2,
z) T1-CVH-P-H-CVL-T2,
aa) T1-CVH-P-CVL-H-T2, и
bb) T1-CVH-P-CVL-T2-H.
[00229] Как будет понятно специалистам в данной области техники, в каждом из a-bb, выше, один из CVH и CVL является неактивным, то есть CVHi или CVLi. Порядок отдельных компонентов в a-bb имеет отношение как к молекулам «полу-Pro», так и к молекулам «полный Pro». Для молекул «полный Pro» число «Т» и других фрагментов можно варьировать по желанию, чтобы сформировать полезную полипептидную конструкцию согласно изобретению. Обычно предпочтительно, чтобы H был связан с неактивным вариантом CVL или CVH. Как изображено на фиг. 53, компоненты полипептидных конструкций согласно изобретению могут быть связаны в диапазоне порядков и с линкерами различных свойств, расположенных между ними.
[00230] В различных вариантах осуществления данное изобретение относится к полипептидной конструкции (или вектору нуклеиновой кислоты, направляющей экспрессию такого полипептида), которая является пролекарством. Таким образом, обеспечивается пролекарственная композиция, содержащая: i) первую полипептидную последовательность, кодирующую CD-3-связывающий домен, содержащий первый домен scFv, содержащий первый домен VH и первый домен VL, соединенный через первый линкерный фрагмент scFv, содержащий первый сайт расщепления протеазой, так что указанный первый домен scFv не специфически связывается с CD-3; ii) вторую полипептидную последовательность, кодирующую домен, связывающий опухолевый антиген, содержащий второй домен scFv, содержащий второй домен VH, и второй домен VL, соединенный через второй линкерный фрагмент scFv, содержащий второй сайт расщепления протеазой, так что указанный второй домен scFv не связывается специфически с опухолевым антигеном; и iii) необязательно по меньшей мере один домен увеличения периода полувыведения.
[00231] В иллюстративном варианте осуществления в пролекарственной композиции первая полипептидная последовательность и вторая полипептидная последовательность функционально связаны с первым доменным линкерным фрагментом, необязательно содержащим сайт расщепления протеазой.
[00232] В различных вариантах осуществления предложена пролекарственная композиция, содержащая: i) первую полипептидную последовательность, содержащую а) первый CD-3-связывающий домен, содержащий первый домен scFv, содержащий первый домен VH и первый домен VL, соединенный через первый линкерный фрагмент scFv, содержащий первый сайт расщепления протеазой, причем указанный первый домен scFv не связывается специфически с CD-3 и b) первый домен, связывающий опухолевый антиген; ii) вторую полипептидную последовательность, содержащую a) второй CD-3-связывающий домен, содержащий второй домен scFv, содержащий второй домен VH и второй домен VL, присоединенный через второй линкерный фрагмент scFv, содержащий второй сайт расщепления протеазой, причем указанный второй домен scFv не связывается специфически с CD-3 и b) второй домен, связывающий опухолевый антиген; и iii) необязательно по меньшей мере один домен увеличения периода полувыведения. В иллюстративном варианте осуществления первый домен VH и второй домен VL специфически связываются с CD-3 и/или вторым доменом VH, а первый домен VL специфически связывается с CD-3.
[00233] В пролекарственной композиции первый домен, связывающий опухолевый антиген, и второй домен, связывающий опухолевый антиген, связываются с одним и тем же опухолевым антигеном. В различных вариантах осуществления первый домен, связывающий опухолевый антиген, и второй домен, связывающий опухолевый антиген, связываются с различными неоантигенными белками. В различных вариантах осуществления первый домен, связывающий опухолевый антиген, связывает первый опухолевый антиген, присутствующий в первой опухолевой клетке, и второй домен, связывающий опухолевый антиген, связывается со вторым опухолевым антигеном, присутствующим в первой опухолевой клетке.
CD-3-связывающий домен
[00234] Специфичность ответа Т-клеток опосредуется путем распознавания антигена (отображается в контексте главного комплекса гистосовместимости, ГКГС) с помощью комплекса Т-клеточного рецептора. В составе комплекса рецепторов Т-клеток, CD-3 представляет собой белковый комплекс, который содержит CD-3γ (гамма)-цепь, CD-3δ (дельта)-цепь и две CD-3ε (эпсилон)-цепи, которые присутствуют на клеточной поверхности. CD-3 объединяются с α (альфа)- и β (бета)-цепями Т-клеточного рецептора (TCR), также, как и CD-3 ζ (дзета), в целом для включения в комплекс Т-клеточного рецептора. Кластеризация CD-3 на Т-клетках, например, иммобилизованными анти-CD-3 антителами, приводит к активации Т-клеток, сходной с вовлечением рецептора Т-клеток, но не зависящей от его клонотипической специфичности. В некоторых вариантах осуществления связывание анти-CD-3 антитела с CD-3 регулируется доменом расщепления протеазой, который ограничивает связывание CD-3 антитела с CD-3 только в микроокружении пораженной клетки или ткани с повышенными уровнями протеаз, например, в микроокружении опухоли.
[00235] В одном аспекте полипептидные конструкции, описанные в данном документе, содержат домен, которые специфически связывается с CD-3 при активации протеазой. В одном аспекте описанные в данном документе полипептидные конструкции содержат два или более домена, которые, когда активированы протеазой, специфически связываются с человеческим CD-3. В некоторых вариантах осуществления описанные в данном документе полипептидные конструкции содержат два или более домена, которые при активации протеазой, которые специфически связывается с CD-3γ. В некоторых вариантах осуществления описанные в данном документе полипептидные конструкции содержат два или более домена, которые при активации протеазой специфически связываются с CD-3δ. В некоторых вариантах осуществления описанные в данном документе полипептидные конструкции содержат два или более домена, которые при активации протеазой специфически связываются с CD-3ε.
[00236] В некоторых вариантах осуществления сайт расщепления протеазой находится между анти-CD-3 VH- и VL-доменов и удерживает их от сворачивания и связывания с CD-3 на Т-клетке. После того как сайт расщепления протеазой расщепляется протеазой, присутствующей в целевой клетке, анти-CD-3 VH- и VL-домены способны сворачиваться и связываться с CD-3 на Т-клетке. В альтернативном варианте осуществления сайт расщепления протеазой сконструирован в домен VL и VH, несвязывающийся с CD-3, который связывается с анти-CD-3 VH- и VL- доменами. Расщепление сайта расщепления протеазой при помощи протеазы, присутствующей в целевой клетке, удаляет VL- и VH-домен, несвязывающий с CD-3, и позволяет анти-CD-3 VH- и VL-домену сворачиваться и связывать CD-3 на Т-клетке.
[00237] Антигенсвязывающие белки, описанные в данном документе, содержат домен, который специфический связывается с CD-3 при активации протеазой. В одном варианте осуществления домен, который специфически связывается с CD-3, содержит домен VH и домен VL, разделенный по меньшей мере одним сайтом расщепления протеазой. Когда сайт расщепления протеазой расщепляется, домен VH и домен VL способны сворачиваться и, следовательно, связываться с CD-3. В некоторых вариантах осуществления сайт расщепления протеазой находится в области петли. В некоторых вариантах осуществления сайт расщепления протеазой находится в доменах VH и/или VL, и сайты расщепления протеазой расщепляются, выявляя VH- и/или VL,-домены, что позволяет им сворачиваться и, следовательно, связываться с CD-3.
[00238] В других вариантах осуществления полипептидные конструкции, описанные в данном документе, содержат два или более доменов, которые при активации протеазой специфически связываются с Т-клеточным рецептором (TCR). В некоторых случаях полипептидные конструкции, описанные в данном документе, содержат два или более доменов, которые при активации протеазой специфически связывают цепь TCR. В некоторых случаях полипептидные конструкции, описанные в данном документе, содержат два или более доменов, которые при активации протеазой специфически связывают β-цепь TCR.
[00239] В некоторых вариантах осуществления CD-3-связывающий домен полипептидных конструкций, описанными в данном документе, демонстрирует не только сильные аффинности связывания CD-3 с человеческим CD-3, но демонстрирует также превосходную перекрестную реактивность с соответствующими белками CD-3 яванского макака. В некоторых случаях CD-3-связывающий домен полипептидных конструкций является перекрестно-реактивным с CD-3 от яванского макака. В некоторых случаях, соотношения KD человека: яванский макака для CD-3 составляют от 5 до 0,2.
[00240] В некоторых вариантах осуществления CD-3-связывающий домен антигенсвязывающего белка может быть любым доменом, который связывается с CD-3, включая, но не ограничиваясь, домены из моноклонального антитела, поликлонального антитела, рекомбинантного антитела, человеческого антитела, гуманизированного антитела. В некоторых случаях полезно, чтобы CD-3-связывающий домен был получен из того же вида, в котором в конечном счете будет использоваться антигенсвязывающий белок. Так, например, для применения у людей, это может быть полезным для CD-3-связывающего домена антигенсвязывающего белка, чтобы содержать человеческие или гуманизированные остатки из антигенсвязывающего домена антитела или фрагмента антитела.
[00241] Таким образом, в одном аспекте, антигенсвязывающий домен содержит гуманизированное или человеческое антитело или фрагмент антитела, или мышиное антитело или фрагмент антитела. В одном варианте осуществления гуманизированный или человеческий анти-CD-3-связывающий домен содержит одну или более (например, все три) из определяющей комплементарность области 1 легкой цепи (LC CDR1), определяющей комплементарность области 2 легкой цепи (LC CDR2) и определяющей комплементарность области 3 легкой цепи (LC CDR3) гуманизированного или человеческого анти-CD-3-связывающего домена, описанного в данного документе, и/или одну или более (например, всех трех) из определяющей комплементарность области 1 тяжелой цепи (HC CDR1), определяющей комплементарность области 2 тяжелой цепи (HC CDR2) и определяющей комплементарность области 3 тяжелой цепи (HC CDR3) гуманизированного или человеческого анти-CD-3-связывающего домена, описанного в данном документе, например, гуманизированного или человеческого анти-CD-3-связывающего домена, содержащего одну или более, например, все три, LC CDR и одну или более, например, все три HC CDR.
[00242] В некоторых вариантах осуществления гуманизированный или человеческий анти-CD-3-связывающий домен содержит гуманизированную или человеческую вариабельную область легкой цепи, специфическую для CD-3, причем вариабельная область легкой цепи, специфическая для CD-3, содержит человеческую или нечеловеческую CDR легкой цепи в каркасной области легкой цепи человека. В некоторых случаях каркасная область легкой цепи представляет собой каркасную область легкой цепи λ (лямбда). В других случаях каркасная область легкой цепи представляет собой каркасную обасть легкой цепи κ (каппа).
[00243] В некоторых вариантах осуществления один или более CD-3-связывающих домена являются специфическими для CD-3ε (эпсилон). В некоторых вариантах осуществления один или более CD-3-связывающих доменов являются специфическими для CD-3δ (дельта). В некоторых вариантах осуществления один или более CD-3-связывающих доменов являются специфическими для CD-3γ (гамма).
[00244] В некоторых вариантах осуществления, один или более CD-3-связывающих доменом являются гуманизированными или полностью человеческими. В некоторых вариантах осуществления один или более активированных CD-3-связывающих доменов имеют KD связывания 1000 нМ или менее с CD-3 на CD-3-экспрессирующих клетках. В некоторых вариантах осуществления один или более активированных CD-3-связывающих доменов имеют KD связывания 100 нМ или менее с CD-3 на CD-3-экспрессирующих клетках. В некоторых вариантах осуществления один или более активированных CD-3-связывающих доменов имеют KD связывания 10 нМ или менее с CD-3 на CD-3-экспрессирующих клетках. В некоторых вариантах осуществления один или более CD-3-связывающих доменов имеют перекрестную реактивность с CD-3 яванского макака. В некоторых вариантах осуществления один или более CD-3-связывающих доменов содержат аминокислотную последовательность, представленную в данном документе.
[00245] В некоторых вариантах осуществления гуманизированный или человеческий анти-CD-3-связывающий домен содержит гуманизированную или человеческую вариабельную область тяжелой цепи, специфическую для CD-3, причем вариабельная область тяжелой цепи, специфическая для CD-3, содержит человеческую или нечеловеческую CDR тяжелой цепи в каркасной области тяжелой цепи человека.
[00246] В некоторых случаях определяющие комплементарность области тяжелой цепи и/или легкой цепи получают из известных анти-CD-3 антител, таких как, например, муромонаб-CD-3 (OKT3), отеликсизумаб (TRX4), теплизумаб (MGA031), висилизумаб (Nuvion), SP34 или I2C, TR-66 или X35-3, VIT3, BMA030 (BW264/56), CLB-T3/3, CRIS7, YTH12.5, F111-409, CLB-T3.4.2, TR-66, WT32, SPv-T3b, 11D8, XIII-141, XIII-46, XIII-87, 12F6, T3/RW2-8C8, T3/RW2-4B6, OKT3D, M-T301, SMC2, F101.01, UCHT-1 и WT-31.
[00247] В одном варианте осуществления анти-CD-3-связывающий домен представляет собой одноцепочечный вариабельный фрагмент (ScFv), содержащий легкую цепь и тяжелую цепь из аминокислотной последовательности, представленной в данном документе. В одном варианте осуществления связывающий домен анти-CD-3 содержит: вариабельную область легкой цепи, содержащую аминокислотную последовательность, имеющую по меньшей мере одну, две или три модификации (например, замены), но не более 30, 20 или 10 модификаций (например, замен) аминокислотной последовательности вариабельной области легкой цепи, представленной в данном документе, или последовательность с идентичностью 95-99% с аминокислотной последовательностью, представленной в данном документе; и/или вариабельную область тяжелой цепи, содержащую аминокислотную последовательность, имеющую по меньшей мере одну, две или три модификации (например, замены), но не более 30, 20 или 10 модификаций (например, замен) аминокислотной последовательности вариабельной области тяжелой цепи, представленной в данном документе, или последовательность с 95-99% идентичностью с аминокислотной последовательностью, представленной в данном документе. В одном варианте осуществления гуманизированный или человеческий анти-CD-3-связывающий домен представляет собой scFv, и вариабельную область легкой цепи, содержащую аминокислотную последовательность, описанную в данном документе, присоединенную к вариабельной области тяжелой цепи, содержащей аминокислотную последовательность, описанную в данном документе, посредством scFv-линкера. Вариабельная область легкой цепи и вариабельная область тяжелой цепи scFv могут быть, например, в любой из следующих ориентаций: вариабельная область легкой цепи - scFv-линкер - вариабельная область тяжелой цепи или вариабельная область тяжелой цепи- scFv-линкер - вариабельная область тяжелой цепи.
[00248] В некоторых вариантах осуществления CD-3-связывающий домен антигенсвязывающего белка имеет аффинность CD-3 на CD-3-экспрессирующих клетках с KD 1000 нМ или менее, 100 нМ или менее, 50 нМ или менее, 20 нМ или менее, 10 нМ или менее, 5 нМ или менее, 1 нМ или менее или 0,5 нМ или менее. В некоторых вариантах осуществления CD-3-связывающий домен антигенсвязывающего белка имеет аффинность CD-3ε, γ, или δ с KD 1000 нМ или менее, 100 нМ или менее, 50 нМ или менее, 20 нМ или менее, 10 нМ или менее, 5 нМ или менее, 1 нМ или менее или 0,5 нМ или менее. В других вариантах осуществления CD-3-связывающий домен антигенсвязывающего белка имеет низкую аффинность с CD-3, то есть около 100 нМ или выше.
[00249] Аффинность связывания к CD-3 можно определить, например, по способности самого антигенсвязывающего антигена или его CD-3-связывающего домена связываться с CD-3, которым покрыт аналитический планшет; отображаемого на поверхности микробной клетки; в растворе; и т.д. Активность связывания самого антигенсвязывающего белка или его CD-3-связывающего домена согласно данному изобретению с CD-3 может быть проанализирована путем иммобилизации лиганда (например, CD-3) или самого антигенсвязывающего белка или его CD-3, к грануле, субстрату, клетке и т. д. Агенты могут быть добавлены в соответствующий буфер, а партнеры по связыванию инкубированы в течение определенного периода времени при данной температуре. После промывки для удаления несвязанного материала связанный белок может быть высвобожден, например, с помощью ДСН, буферов с высоким рН и тому подобного и проанализирован, например, при помощи поверхностного плазмонного резонанса (SPR).
Линкеры
[00250] Эти два домена соединены вместе при помощи линкера, который необязательно является расщепляемым. Иллюстративными сайтами расщепления являются сайты расщепления протеазой. Иллюстративные протеазы, расщепляющие междоменный линкер, включают те, которые обнаружены в плазме, например, тромбин, и те, которые сверхэкспрессируются в микроокружении опухоли.
[00251] В антигенсвязывающих полипептидах, описанных в данном документе, домены связывают линкерами доменов, например, L1, L2, L3, и L4, где L1 связывает первый и второй домен полипептидной конструкции, L2 связывает второй и третий домены полипептидной конструкции, L3 связывает третий и четвертый домены полипептидной конструкции, а L4 связывает четвертый и пятый домены протеаз-активированной полипептидной конструкции. Линкеры, например, L1, L2, L3 и L4 имеют оптимизированную длину и/или аминокислотный состав. В некоторых вариантах осуществления линкеры, например, L1, L2, L3, и L4, имеют одинаковую длину и аминокислотный состав. В других вариантах осуществления линкеры, например, L1, L2, L3, и L4, различны. В некоторых вариантах осуществления внутренние линкеры L1, L2, L3 и/или L4 являются «короткими», то есть состоят из 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12 аминокислотных остатков. Таким образом, в некоторых случаях внутренние линкеры состоят из около 12 или менее аминокислотных остатков. В случае 0 аминокислотных остатков доменный линкер представляет собой пептидную связь. В некоторых вариантах осуществления доменные линкеры L1, L2, L3 и/или L4 являются «длинными», то есть состоят из 15, 20 или 25 аминокислотных остатков. В некоторых вариантах осуществления эти доменные линкеры состоят из от около 3 до около 15, например, 8, 9 или 10 смежных аминокислотных остатков. Что касается аминокислотного состава доменные линкеры, пептиды выбирают со свойствами, которые придают гибкость полипептидной конструкции, не мешают связыванию доменов и, необязательно, препятствуют расщеплению протеазами. Например, глициновые и сериновые остатки обычно обеспечивают устойчивость к действию протеазы. Примеры внутренних линкеров, подходящих для связывания доменов в полипептидах согласно изобретению, включают, но не ограничиваются следующими: (GS)n, (GGS)n, (GGGS)n, (GGSG)n, (GGSGG)n, или (GGGGS)n, где n представляет собой 1, 2, 3, 4, 5, 6, 7, 8, 9, или 10. В одном варианте осуществления внутренний линкера L1, L2, и/или L3 представляет собой (GGGGS)4 или (GGGGS)3.
[00252] В некоторых случаях scFv, которые связываются с CD3, получают в соответствии с известными способами. Например, молекулы scFv могут быть получены путем связывания областей VH и VL вместе с использованием гибких полипептидных линкеров. Молекулы ScFv содержат ScFv-линкер (например, серин-глициновый линкер) с оптимизированной длиной и/или аминокислотным составом. Соответственно, в некоторых вариантах осуществления, длина ScFv-линкер является такой, что VH- или VL-домен можно межмолекулярно объединить с другой вариабельной областью с образованием CD-3-связывающего сайта. В некоторых вариантах осуществления такие scFv-линкеры являются «короткими», т.е. состоят из 0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11 или 12 аминокислотных остатков. Таким образом, в некоторых случаях scFv-линкеры состоят из около 12 или менее аминокислотных остатков. В случае 0 аминокислотных остатков scFv-линкер представляет собой пептидную связь. В некоторых вариантах осуществления эти ScFv-линкеры состоят из от около 3 до около 15, например, 8, 9 или 10 смежных аминокислотных остатков. Например, scFv-линкеры, содержащие глициновые и сериновые остатки, обычно обеспечивают устойчивость к действию протеазы. В некоторых вариантах осуществления линкеры в scFv содержат глициновые и сериновые остатки. Аминокислотную последовательность scFv-линкеров можно оптимизировать, например, методами фагового дисплея для улучшения связывания CD-3 и выхода продукта scFv. Примеры пептидных scFv-линкеров, подходящих для связывания вариабельного домена легкой цепи и вариабельного домена тяжелой цепи в scFv, включают, но не ограничиваются следующими: (GS)n, (GGS)n, (GGGS)n, (GGSG)n, (GGSGG)n, или (GGGGS)n,, где n представляет собой 1, 2, 3, 4, 5, 6, 7, 8, 9 или 10. В одном варианте осуществления scFv-линкер может быть (GGGGS)4 или (GGGGS)3. Изменение длины линкера может сохранять или усиливать активность, что приводит к повышению эффективности в исследованиях активности.
[00253] Дополнительные иллюстративные доменные и scFv-линкеры представлены на фиг. 56.
Домены расщепления протеазой
[00254] Описанные в данном документе антигенсвязывающих полипептиды содержат по меньшей мере один сайт расщепления протеазой, содержащий аминокислотную последовательность, которая расщепляется по меньшей мере одной протеазой. В некоторых случаях описанные в данном документе антигенсвязывающие белки содержат 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20 или более сайтов расщепления протеазой, которые расщепляются по меньшей мере одной протеазой. В некоторых случаях сайт расщепления протеазой, содержащий аминокислотную последовательность, распознаваемую протеазой, представляет собой сайт MMP9-расщепления, содержащий полипептид, имеющий аминокислотную последовательность LEATA.
[00255] Домены расщепления протеазой являются полипептидами, имеющими последовательность, распознаваемую и расщепляемую специфическим для последовательности образом. Предложенные в данном документе антигенсвязывающие белки, в некоторых случаях, содержат домен расщепления протеазой, распознаваемый специфическим для последовательности образом пи помощи матричной металлопротеазы (MMP), например, MMP9. В некоторых случаях домен расщепления протеазой, распознаваемый MMP9, содержит полипептид, имеющий аминокислотную последовательность PR(S/T)(L/I)(S/T). В некоторых случаях домен расщепления протеазой, распознаваемый MMP9, содержит полипептид, имеющий аминокислотную последовательность LEATA. В некоторых случаях домен расщепления протеазой распознается специфическим для последовательности образом с помощью MMP11. В некоторых случаях домен расщепления протеазой, распознаваемый MMP11, содержит полипептид, имеющий аминокислотную последовательность GGAANLVRGG. В некоторых случаях домен расщепления протеазой распознается протеазой, раскрытой в таблице 1. В некоторых случаях домен расщепления протеазой, распознаваемый протеазой, раскрытой в таблице 1, содержит полипептид, имеющий аминокислотную последовательность, выбранную из последовательности, раскрытой в таблице 1.
[00256] Протеазы представляют собой белки, которые расщепляют белки, в некоторых случаях, специфическим для последовательности образом. Протеазы включают, но не ограничиваются следующими: сериновые протеазы, цистеиновые протеазы, аспартильные протеазы, треониновые протеазы, глутаминовые протеазы, металлопротеазы, аспарагин-пептид-лиазы, сывороточные протеазы, катепсины, катепсин B, катепсин C, катепсин D, катепсин E, катепсин K, катепсин L, калликрин, hK1, hK10, hK15, плазмин, коллагеназу, коллагеназу IV типа, стромелизин, фактор Xa, химотрипсинподобную протеазу, трипсиноподобную протеазу, эластазоподобную протеазу, субтилизин-подобную протеазу, актинидаин, бромелайн, кальпаин, каспазы, каспазу-3, Mir1-CP, папаин, протеазу ВИЧ-1, протеазу ВПГ, протеазу ЦМВ, химозин, ренин, пепсин, матриптазу, лемумин, плазмепсин, непентезин, металлоэкзопептидазы, металлоэндопептидазы, матричные металлопротеазы (MMP), MMP1, MMP2, MMP3, MMP8, MMP9, MMP13, MMP11, MMP14, урокиназный активатор плазминогена (uPA), энтерокиназу, простатический специфический антиген (PSA, hK3), интерлейкин-1β-превращающий фермент, тромбин, FAP (FAP-α), дипептидилпептидазу, и дипептидилпептидазу IV (DPPIV/CD26).
Таблица 1: Иллюстративные протеазы и последовательности доменов расщепления протеазой
[00257] Известно, что протеазы секретируются некоторыми пораженными клетками и тканями, например, опухолевыми или раковыми клетками, создавая микроокружение, которая богата на протеазы, или богатое протеазой микроокружение. В некоторых случаях кровь субъекта богата на протеазы. В некоторых случаях клетки, окружающие опухоль, выделяют протеазы в микроокружении опухоли. Клетки, окружающие протеазы, секретирующие опухоль, включают, но не ограничиваются следующими: стромальные клетки опухоли, миофибробласты, клетки крови, тучные клетки, В-клетки, NK-клетки, регуляторные Т-клетки, макрофаги, цитотоксические Т-лимфоциты, дендритные клетки, мезенхимальные стволовые клетки, полиморфноядерные клетки, и другие клетки. В некоторых случаях протеазы присутствуют в крови субъекта, например, протеазы, которые нацелены на аминокислотные последовательности, обнаруженные в микробных пептидах. Эта особенность позволяют направленные лекарственные препараты, такие как антигенсвязывающие белки, иметь дополнительную специфичность, потому что Т-клетки не будут связываться с антигенсвязывающим белком, за исключением богатого протеазой микроокружения в клетках или тканях.
[00258] В иллюстративных вариантах осуществления полипептидные конструкции содержат один или более сайтов расщепления протеазой, например, сайты, указанные в таблице 1 выше. Эти сайты в различных вариантах осуществления находятся в scFv и расположены между одним или более VLi или VHi и одним или более VH или VL, так что VLi отделен от VH и/или VHi отделен от VL при расщеплении сайта соответствующей протеазой. Как будет понятно специалистам в данной области техники, сайт расщепления протеазой может содержать полную последовательность связывающего полипептидного scFv-линкера между доменами VL и VH или, альтернативно, сайт расщепления может быть фланкирован на одном или обоих концах дополнительным аминокислотными или пептидными последовательностями.
[00259] В некоторых вариантах осуществления домен расщепления протеазой находится в домене увеличения периода полувыведения или в CD3-связывающем домене. В некоторых вариантах осуществления домен расщепления протеазой не находится в домене увеличения периода полувыведения или в CD3-связывающем домене.
Домен увеличения периода полувыведения
[00260] В данном документе рассмотрены домены, которые продлевают период полувыведения антигенсвязывающего домена. Предполагается, что такие домены включают, но не ограничиваются, ЧСА-связывающие домены, домены Fc, малые молекулы и другие домены увеличения периода полувыведения, известные в данной области техники.
[00261] Человеческий сывороточный альбумин (ЧСА) (молекулярная масса ~67 кДа) является наиболее распространенным белком в плазме, присутствующим в концентрации около 50 мг/мл (600 мкМ) и имеет период полувыведения около 20 суток у людей. ЧСА служит для поддержания рН в плазме, способствует коллоидному артериальному давлению, функционирует как носитель многих метаболитов и жирных кислот и служит основным транспортным белком лекарственных средств в плазме.
[00262] Нековалентная связь с альбумином продлевает период полувыведения короткоживущих белков. Например, рекомбинантное слитие альбуминсвязывающего домена с Fab-фрагментом приводило к уменьшению in vivo клиренса в 25 и 58 раз и периода полувыведения в 26 и 37 раз при внутривенном введении мышам и кроликам, соответственно, по сравнению с введением только Fab-фрагмента. В другом примере, когда инсулин ацилируют жирными кислотами, чтобы способствовать ассоциации с альбумином, наблюдается длительный эффект при подкожном введении у кроликов или свиней. Вместе эти исследования демонстрируют связь между связыванием альбумина и продленным действием.
[00263] В одном аспекте описанные в данном документе антигенсвязывающие белки содержат домен увеличения периода полувыведения, например, домен, который специфически связывается с ЧСА. В некоторых вариантах осуществления ЧСА-связывающий домен антигенсвязывающего белка может быть любым доменом, который связывается с ЧСА, включая, но не ограничиваясь, домены из моноклонального антитела, поликлонального антитела, рекомбинантного антитела, человеческого антитела, гуманизированного антитела. В некоторых вариантах осуществления ЧСА-связывающий домен представляет собой одноцепочечные вариабельные фрагменты (ScFv), однодоменными антитела, такие как вариабельный домен тяжелой цепи (VH), вариабельный домен легкой цепи (VL) и вариабельный домен (VHH) наноантитела, полученного из верблюдовых, пептид, лиганд или малая молекула, специфическая для ЧСА. В некоторых вариантах осуществления ЧСА-связывающий домен представляет собой однодоменное антитело. В других вариантах осуществления ЧСА-связывающий домен представляет собой пептид. В других вариантах осуществления ЧСА-связывающий домен представляет собой малую молекулу. Предполагается, что ЧСА-связывающий домен антигенсвязывающего белка довольно мал и не более 25 кДа, не более 20 кДа, не более 15 кДа или не более 10 кД в некоторых вариантах осуществления. В некоторых случаях ЧСА-связывающий составляет 5 кДа или менее, если это пептид или небольшая молекула.
[00264] Домен увеличения периода полувыведения антигенсвязывающего белка обеспечивает измененную фармакодинамику и фармакокинетику самого антигенсвязывающего белка. Как и выше, домен увеличения периода полувыведения продлевает период полувыведения. Домен увеличения периода полувыведения также изменяет фармакодинамические свойства, включая изменение распределения в ткани, проникновения и диффузии антигенсвязывающего белка. В некоторых вариантах осуществления домен увеличения периода полувыведения обеспечивает улучшение нацеливания на ткань (включая опухоль), проникновение в ткани, распределение в ткани, диффузию внутри ткани и повышенную эффективность по сравнению с белком без домена увеличения периода полувыведения. В одном варианте осуществления терапевтические способы результативно и эффективно используют уменьшенное количество антигенсвязывающего белка, приводящее к уменьшенным побочным эффектам, таким как уменьшение цитотоксичности для неопухолевых клеток.
[00265] Кроме того, характеристики домена увеличения периода полувыведения, например, ЧСА-связывающего домена, включают аффинность связывания ЧСА-связывающего домена для ЧСА. Аффинность указанного ЧСА-связывающего домена может быть выбрана так, чтобы нацеливаться на конкретный период полувыведения в конкретной полипептидной конструкции. Таким образом, в некоторых вариантах осуществления ЧСА-связывающий домен обладает высокой аффинностью связывания. В других вариантах осуществления ЧСА-связывающий домен имеет среднюю аффинность связывания. В других вариантах осуществления ЧСА-связывающий домен имеет низкую или минимальную аффинность связывания. Иллюстративные аффинности связывания включают концентрации KD при 10 нМ или менее (высокая), между 10 нМ и 100 нМ (средняя) и более 100 нМ (низкая). Как указано выше, аффинности связывания к ЧСА определяют известными способами, такими как поверхностный плазмонный резонанс (SPR).
Домен, связывающий целевой антиген
[00266] В дополнение к описанным выше CD3 и доменам увеличения периода полувыведения, описанные в данном документе полипептидные конструкции также содержат по меньшей мере один или по меньшей мере два или более домена, которые связываются с одним или более целевыми антигенами, или одной или более областями на одном целевом антигене. Предполагается, что описанная в данном документе полипептидная конструкция согласно изобретению расщепляется, например, в болезнь-специфическом микроокружении или в крови субъекта в домене расщепления протеазой и что каждый домен, связывающий целевой антиген, будет связываться с целевым антигеном на целевой клетке, тем самым активируя CD3-связывающий домен для связывания Т-клетки. По меньшей мере один целевой антиген участвует и/или связан с заболеванием, расстройством или патологическим состоянием. Иллюстративные целевые антигены включают те, которые связаны с пролиферативным заболеванием, опухолевидным заболеванием, воспалительным заболеванием, иммунологическим расстройством, аутоиммунным заболеванием, инфекционным заболеванием, вирусным заболеванием, аллергической реакцией, реакцией на паразита, болезнью «трансплантат против хозяина» или болезнью «хозяин против трансплантата». В некоторых вариантах осуществления целевой антиген представляет собой опухолевый антиген, экспрессируемый на опухолевой клетке. Альтернативно, в некоторых вариантах осуществления целевой антиген ассоциирован с патогеном, таким как вирус или бактерия. По меньшей мере один целевой антиген также может быть направлен против здоровой ткани.
[00267] В некоторых вариантах осуществления целевым антигеном является молекула клеточной поверхности, такая как белок, липид или полисахарид. В некоторых вариантах осуществления целевой антиген представляет собой опухолевую клетку, инфицированную вирусом клетку, инфицированную бактерией клетку, поврежденный эритроцит, тромбоцит или клетку фиброзной ткани. Предполагается, что при связывании более одного целевого антигена два неактивных CD3-связывающих домена локализуются и образуют активный CD3-связывающий домен на поверхности целевой клетки. В некоторых вариантах осуществления антигенсвязывающий белок содержит более одного домена, связывающего целевой антиген, для активации неактивного CD3-связывающего домена в антигенсвязывающем белке. В некоторых вариантах осуществления антигенсвязывающий белок содержит более одного домена, связывающего целевой антиген, для усиления прочности связывания с целевой клеткой. В некоторых вариантах осуществления антигенсвязывающий белок содержит более одного домена, связывающего целевой антиген, для усиления прочности связывания с целевой клеткой. В некоторых вариантах осуществления более чем один антигенсвязывающий домен содержит тот же антигенсвязывающий домен. В некоторых вариантах осуществления более чем один антигенсвязывающий домен содержит различные антигенсвязывающие домены. Например, два разных антигенсвязывающих домена, которые, как известно, были дважды экспрессированы в пораженной клетке или ткани, например, опухолевой или раковой клетке, могут усиливать связывание или селективность антигенсвязывающего белка для мишени.
[00268] Предлагаемые в данном документе полипептидные конструкции содержат по меньшей мере один антигенсвязывающий домен, причем антигенсвязывающий домен связывается по меньшей мере с одним целевым антигеном. Целевые антигены, в некоторых случаях, экспрессируются на поверхности пораженной клетки или ткани, например, опухолевой или раковой клетки. Целевые антигены включают, но не ограничиваются следующими: EpCAM, EGFR, HER-2, HER-3, c-Met, FoIR и CEA. Полипептидные конструкции, описанные в данном документе, также включают белки, содержащие два антигенсвязывающих домена, которые связываются с двумя различными целевыми антигенами, которые, как известно, экспрессируются на пораженной клетке или ткани. Иллюстративные пары антигенсвязывающих доменов включают, но не ограничиваются следующими: EGFR/CEA, EpCAM/CEA и HER-2/HER-3.
[00269] Конструкция описанных в данном документе полипептидных конструкций позволяет связывающему домену к одному или более целевым антигенам быть гибким в том, что связывающий домен к целевому антигену может быть любым типом связывающего домена, включая, но не ограничиваясь доменами из моноклонального антитела, поликлонального антитела, рекомбинантного антитела, человеческого антитела, гуманизированного антитела. В некоторых вариантах осуществления связывающий домен к целевому антигену представляет собой одноцепочечный вариабельный фрагмент (ScFv), однодоменное антитело, такие как вариабельный домен тяжелой цепи (VH), вариабельный домен легкой цепи (VL) и вариабельный домен (VHH) наноантитела, полученного из верблюдовых. В других вариантах осуществления домен связывания с целевым антигеном представляет собой домен, связывающий не Ig, то есть антитело, такое как антикалины, аффилины, молекулы аффител, аффимеры, аффитины, альфа-антитела, авимеры, DARPin (сконструированный белок с анкириновым повтором), финомеры, пептиды домена Куница, и монотела. В других вариантах осуществления связывающий домен к одному или более целевым антигенам представляет собой лиганд, рецепторный домен, лектин или пептид, который связывается с одним или более целевыми антигенами или ассоциируется с ними.
[00270] В некоторых вариантах осуществления целевые клеточные антигенсвязывающие домены независимо содержат scFv, домен VH, домен VL, не-Ig домен или лиганд, который специфически связывается с целевым антигеном. В некоторых вариантах осуществления домены, связывающие целевой антиген(ы), специфически связываются с молекулой клеточной поверхности. В некоторых вариантах осуществления домены, связывающие целевой антиген(ы), специфически связываются с опухолевым антигеном. В некоторых вариантах осуществлениядомены, связывающие целевой антиген(ы), специфически и независимо связываются с антигеном, выбранным из по меньшей мере одного из EpCAM, EGFR, HER-2, HER-3, cMet, CEA и FoIR. В некоторых вариантах осуществления домены, связывающие целевой антиген(ы), специфически и независимо связываются с двумя различными антигенами, причем по меньшей мере один из антигенов выбран из одного из EpCAM, EGFR, HER-2, HER-3, cMet, CEA и FoIR. В некоторых вариантах осуществления белок перед расщеплением домена расщепления протеазой составляет менее около 100 кДа. В некоторых вариантах осуществления белок после расщепления домена расщепления протеазой составляет от около 25 до около 75 кДа. В некоторых вариантах осуществления белок перед расщеплением протеазой имеет размер, который выше почечного порога для клиренса при первом прохождении. В некоторых вариантах осуществления белок до расщепления протеазой имеет период полувыведения, составляющий по меньшей мере около 50 часов. В некоторых вариантах осуществления белок перед расщеплением протеазой имеет период полувыведения по меньшей мере около 100 часов. В некоторых вариантах осуществления белок увеличивает проникновение в ткани по сравнению с IgG с тем же целевым антигеном. В некоторых вариантах осуществления белок увеличивает распределение в ткани по сравнению с IgG с одним и тем же целевым антигеном.
Фармокинетика полипептидной конструкции
[00271] Описанные в данном документе полипептидные конструкции имеют определенные преимущества, которые признаны специалистом в данной области техники. Например, описанные в данном документе полипептидные конструкции улучшают фармакокинетику по сравнению со стандартными терапевтическими средствами на основе антител. Улучшенная фармакокинетика полипептидных конструкций в данном документе относится к по меньшей мере домену увеличения периода полувыведения и к CD3-связывающему домену. Домены увеличения периода полувыведения, как раскрыто в данном документе, включают различные полипептиды, включая, но не ограничиваясь Fc-доменами и полипептидами, связывающимися с ЧСА. CD3-связывающие домены в данном документе имеют уникальные свойства, которые обеспечивают превосходную фармакокинетику. CD3-связывающие домены в данном документе не связываются с CD3 до тех пор, пока они не активируются по меньшей мере расщеплением по меньшей мере одного домена расщепления протеазой и связывания антигенсвязывающих доменов с целевыми антигенами. Следовательно, усиленная фармакокинетика антигенсвязывающих белков в данном документе объясняется по меньшей мере частично уменьшением или устранением опосредованного мишенью лекарственного средства путем связывания CD3 в кровотоке человека. Улучшенная фармакокинетика включает по меньшей мере одну пологую альфа-фазу и более высокую экспозицию в бета-фазе. Антигенсвязывающие белки, описанные в данном документе, таким образом, имеют более широкое терапевтическое окно с меньшими различиями между максимальной и минимальной концентрациями в равновесном состоянии при экспозиции по сравнению со стандартными терапевтическими средствами на основе антител.
Модификации полипептидных конструкций
[00272] Описанные в данном документе полипептидные конструкции охватывают производные или аналоги, в которых (i) аминокислота замещена аминокислотным остатком, который не кодируется генетическим кодом, (ii) зрелый полипептид слит с другим соединением, таким как полиэтиленгликоль, или (iii) дополнительные аминокислоты слиты с белком, таким как лидерная или секреторная последовательность или последовательность для очистки белка.
[00273] Иллюстративные модификации включают, но не ограничиваются следующими: ацетилирование, ацилирование, АДФ-рибозилирование, амидирование, ковалентное присоединение флавина, ковалентное присоединение фрагмента гема, ковалентное присоединение нуклеотида или нуклеотидного производного, ковалентное присоединение липида или липидного производного, ковалентное присоединение фосфатидилинозитола, сшивание, циклизация, образование дисульфидных связей, деметилирование, образование ковалентных поперечных связей, образование цистина, образование пироглутамата, формилирование, гамма-карбоксилирование, гликозилирование, образование ГФИ-якоря, гидроксилирование, иодирование, метилирование, ацилирование остатком миристиновой кислоты, окисление, протеолитический процессинг, фосфорилирование, пренилирование, рацемизацию, селеноилирование, сульфатирование, опосредуемую транспортной РНК вставку аминокислоты в белок, такую как аргинилирование и убиквитинирование.
[00274] Модификации выполняются в любом месте в описанных в данном документе полипептидных конструкциях, включая пептидный остов, боковые цепи аминокислот и амино- или карбоксильные концы. Некоторые общие пептидные модификации, которые применимы для модификации полипептидных конструкций, включают гликозилирование, присоединение липидов, сульфатирование, гамма-карбоксилирование остатков глутаминовой кислоты, гидроксилирование, блокирование аминогруппы или карбоксильной группы в полипептиде или и то, и другое путем ковалентной модификации и АДФ-рибозилирования.
Полинуклеотиды, кодирующие антигенсвязывающие белки
[00275] Кроме того, в некоторых вариантах осуществления представлены полинуклеотидные молекулы, кодирующие антигенсвязывающий белок, описанный в данном документе. В некоторых вариантах осуществления полинуклеотидные молекулы представлены в виде ДНК-конструкции. В других вариантах осуществления молекулы полинуклеотидов представлены в виде транскрипта матричной РНК.
[00276] Полинуклеотидные молекулы сконструированы с помощью известных методов, например, путем комбинирования генов, кодирующих три связывающих доменов или разделенных пептидными линкерами, или, в других вариантах осуществления, непосредственно связанных пептидной связью, в единую генетическую конструкцию, функционально связанную с подходящим промотором и необязательно подходящим терминатор транскрипции, и экспрессии ее в бактериях или другой подходящей системе экспрессии, такой как, например, клетки СНО. В вариантах осуществления, где мишень-связывающий домен является малой молекулой, полинуклеотиды содержат гены, кодирующие домены, которые связываются с CD-3 и ЧСА. В вариантах осуществления, где домен увеличения периода полувыведения представляет собой малую молекулу, полинуклеотиды содержат гены, кодирующие домены, которые связываются с CD-3 и целевым антигеном. В зависимости от используемой векторной системы и хозяина может использоваться любое количество подходящих элементов транскрипции и трансляции, включая конститутивные и индуцибельные промоторы. Промотор выбирают таким образом, чтобы он управлял экспрессией полинуклеотида в соответствующей клетке-хозяине.
[00277] В некоторых вариантах осуществления полинуклеотид вставляется в вектор, предпочтительно экспрессионный вектор, который представляет собой дополнительный вариант осуществления. Этот рекомбинантный вектор может быть сконструирован в соответствии с известными способами. Векторы, представляющие особый интерес, включают плазмиды, фагмиды, производные фага, вирусы (например, ретровирусы, аденовирусы, аденоассоциированные вирусы, вирусы герпеса, лентивирусы и тому подобные), и космиды.
[00278] Для того чтобы содержать и экспрессировать полинуклеотид, кодирующий полипептид описанной полипептидной конструкции, можно использовать множество систем экспрессионный вектор/хозяин. Примерами векторов экспрессии для экспрессии в E.coli являются pSKK (Le Gall et al., J Immunol Methods. (2004)285(1):111-27) или pcDNA5 (Invitrogen) для экспрессии в клетках млекопитающих.
[00279] Таким образом, полипептидные конструкции, как описано в данном документе, в некоторых вариантах осуществления получают путем введения вектора, кодирующего белок, как описано выше, в клетку-хозяина и культивирование указанной клетки-хозяина в условиях, при которых экспрессируются белковые домены, которые могут быть выделены и, необязательно, дополнительно очищены.
Фармацевтические композиции
[00280] В некоторых вариантах осуществления также предложены фармацевтические композиции, содержащие антигенсвязывающий белок, описанный в данном документе, вектор, содержащий полинуклеотид, кодирующий полипептид полипептидных конструкций, или клетки-хозяева, трансформированные этим вектором, и по меньшей мере один фармацевтически приемлемый носитель. Термин «фармацевтически приемлемый носитель» включает, но не ограничивается любым носителем, который не влияет на эффективность биологической активности ингредиентов и не токсичен для пациента, которому он вводится. Примеры подходящих фармацевтических носителей хорошо известны в данной области техники и включают фосфатно-солевые буферные растворы, воду, эмульсии, такие как масляные эмульсии в воде, различные типы смачивающих агентов, стерильные растворы и т. д. Такие носители могут быть приготовлены обычными способами и могут быть введены субъекту в подходящей дозе. Предпочтительно композиции являются стерильными. Данные композиции могут также содержать адъюванты, такие как консервант, эмульгирующие агенты и диспергирующие агенты. Предотвращение действия микроорганизмов может быть обеспечено за счет включения различных антибактериальных и противогрибковых средств.
[00281] В некоторых вариантах осуществления фармацевтических композиций антигенсвязывающий белок, описанный в данном документе, инкапсулируют в наночастицы. В некоторых вариантах осуществления наночастицы представляют собой фуллерены, жидкие кристаллы, липосому, квантовые точки, суперпарамагнитные наночастицы, дендримеры или наностержни. В других вариантах осуществления фармацевтических композиций антигенсвязывающий белок присоединяют к липосомам. В некоторых случаях антигенсвязывающий белок конъюгируют с поверхностью липосом. В некоторых случаях антигенсвязывающий белок инкапсулируют в оболочку липосомы. В некоторых случаях липосома представляет собой катионную липосому.
[00282] Полипептидные конструкции, описанные в данном документе, предназначены для использования в качестве лекарственного средства. Введение осуществляют различными способами, например, внутривенным, внутрибрюшинным, подкожным, внутримышечным введением, местным применением или внутрикожным введением. В некоторых вариантах осуществления способ введения зависит от вида терапии и вида соединения, содержащегося в фармацевтической композиции. Схема лечения будет определяться лечащим врачом и другими клиническими факторами. Частота приема для любого пациента зависит от многих факторов, включая размер пациента, площадь поверхности тела, возраст, пол, вводимое соединение, время и путь введения, вид терапии, общее состояние здоровья и другие лекарственные средства, которые вводятся параллельно. «Эффективная доза» относится к количествам активного ингредиента, которые являются достаточными для воздействия на течение и тяжесть заболевания, что приводит к уменьшению или ремиссии такой патологии и может быть определено с использованием известных методов.
Способы лечения
[00283] Также в некоторых вариантах осуществления в данном документе предлагаются способы и варианты использования для стимуляции иммунной системы нуждающегося в ней индивидуума, включающие введение описанного в данном документе антигенсвязывающего белка. В некоторых случаях введение описанного в данном документе антигенсвязывающего белка индуцирует и/или поддерживает цитотоксичность по отношению к клетке, экспрессирующей целевой антиген, причем клетка, экспрессирующая целевой антиген, находится в микроокружении с повышенными уровнями протеазной активности. В некоторых случаях клетка, экспрессирующая целевой антиген, представляет собой раковую или опухолевую клетку, инфицированную вирусом клетку, инфицированную бактерией клетку, аутореактивную Т- или В-клетку, поврежденные эритроциты, артериальные бляшки или фиброзную ткань. В некоторых случаях кровь субъекта богата на протеазы.
[00284] В данном документе также предложены способы и варианты использования для лечения заболевания, расстройства или патологического состояния, связанные с целевым антигеном, включающие введение индивидууму, нуждающемуся в этом, антигенсвязывающего белка, описанного в данном документе. Заболевания, расстройства или патологические состояния, связанные с целевым антигеном, включают, но не ограничиваются вирусной инфекцией, бактериальной инфекцией, аутоиммунным заболеванием, отторжением трансплантата, атеросклерозом или фиброзом. В других вариантах осуществления заболевание, расстройство или патологическое состояние, связанное с целевым антигеном, преставляет собой пролиферативное заболевание, опухолеподобное заболевание, воспалительное заболевание, иммунологическое расстройство, аутоиммунное заболевание, инфекционное заболевание, вирусное заболевание, аллергическую реакцию, реакцию на паразита, болезнь «трансплантат против хозяина» или болезнь «хозяин против трансплантата» В одном варианте осуществления заболевание, расстройство или патологическое состояние, связанное с целевым антигеном, представляет собой рак. В одном случае рак представляет собой гемобластоз. В другом случае рак представляет собой солидную опухоль.
[00285] Как используется в данном документе, в некоторых вариантах осуществления «лечение» или «лечить» или «подвергнутый лечению» относится к терапевтическому воздействию, в котором объектом является замедление (уменьшение) нежелательного физиологического состояния, расстройства или заболевания, или получение полезных или желаемых клинических результатов. Для целей, описанных в данном документе, полезные или желаемые клинические результаты включают, но не ограничиваются следующими: облегчение симптомов; уменьшение степени патологического состояния, расстройства или болезни; стабилизация (т. е. не прогессирование) течения патологического состояния, расстройства или заболевания; задержка начала или замедления прогрессирования патологического состояния, расстройства или заболевания; улучшение течения патологического состояния, расстройства или болезни; и ремиссии (независимо от того, являются ли они частичными или тотальными), независимо от того, являются они определяемыми или неопределяемыми, или усиливают или улучшают патологическое состояние, расстройство или заболевание. Лечение включает выявление клинически значимого ответа без повышенного уровня побочных эффектов. Лечение также включает увеличение выживаемости по сравнению с ожидаемой выживаемостью, если не получают лечение. В других вариантах осуществления «лечение» или «лечить» или «получающий лечение» относится к профилактическим мерам, причем объект получает задержку начала или уменьшения тяжести нежелательного физиологического состояния, расстройства или заболевания, так, например, когда человек предрасположен к заболеванию (например, человек, который несет генетический маркер заболевания, такого как рак молочной железы).
[00286] В способах согласно изобретению терапия используется для обеспечения положительного терапевтического ответа в отношении заболевания или патологического состояния. Под «положительным терапевтическим ответом» подразумевается улучшение в заболевании или патологическом состоянии и/или улучшение в симптомах, связанных с заболеванием или патологическим состоянием. Например, положительный терапевтический ответ будет относиться к одному или более из следующих улучшений в болезни: (1) уменьшение числа неопластических клеток; (2) увеличение смертности неопластических клеток; (3) ингибирование выживаемости неопластических клеток; (5) ингибирование (то есть замедление до некоторой степени, предпочтительно прекращение) роста опухоли; (6) повышенная выживаемость пациентов; и (7) некоторое облегчение одного или более симптомов, связанных с заболеванием или патологическим состоянием.
[00287] Положительные терапевтические реакции при любом заболевании или патологическом состоянии могут определяться стандартизованными критериями ответа, специфичными для этого заболевания или патологического состояния Ответ опухоли может быть оценен по изменениям морфологии опухоли (то есть общей опухолевой нагрузки, размера опухоли и т.п.) с использованием методов скрининга, таких как сканирование магнитно-резонансной томографии (МРТ), рентгенография, компьютерно-томографическое (КТ) сканирование, остеосцинтиграфия, эндоскопия и забор материала на биопсию опухоли, включая аспирацию костного мозга (BMA) и подсчет опухолевых клеток в циркулирующей крови.
[00288] В дополнение к этим положительным терапевтическим ответам субъект, проходящий лечение, может испытать положительный эффект улучшения симптомов, связанных с этим заболеванием.
[00289] Лечение в соответствии с данным изобретением включает «терапевтически эффективное количество» используемых лекарственных препаратов. «Терапевтически эффективное количество» относится к количеству, эффективному в дозах и в течение периодов времени, необходимых для достижения желаемого терапевтического результата.
[00290] Терапевтически эффективное количество может варьироваться в зависимости от таких факторов, как течение заболевания, возраст, пол и вес человека, а также способность лекарственных препаратов вызывать желаемый ответ у индивидуума. Терапевтически эффективное количество является таким, для которого любые токсические или вредные эффекты лечения антителом или частью антитела перевешиваются терапевтически благоприятными эффектами.
[00291] «Терапевтически эффективное количество» для терапии опухолей также может быть измерено его способностью стабилизировать прогрессирование заболевания. Способность соединения ингибировать рак может быть оценена в животной модельной системе, прогнозирующей эффективность в опухолях человека.
[00292] Альтернативно, это свойство композиции может быть оценено путем изучения способности соединения ингибировать рост клеток или индуцировать апоптоз с помощью анализов in vitro, известных практикующему специалисту. Терапевтически эффективное количество терапевтического соединения может уменьшать размер опухоли или иным образом ослаблять симптомы у субъекта. Специалист в данной области техники может определить такие количества на основе таких факторов, как размер субъекта, степень тяжести симптомов субъекта и выбранная композиция или способ введения.
[00293] Схему лечения корректируют для обеспечения оптимального желаемого ответа (например, терапевтического ответа). Например, можно вводить один болюс, можно вводить несколько разделенных доз с течением времени или дозу можно пропорционально уменьшать или увеличивать, в соответствии с конкретной терапевтической ситуацией. Парентеральные композиции могут быть составлены в виде единичной дозированной формы для удобства применения и однородности дозы. Используемая в данном документе единичная дозированная форма относится к физически дискретным единицам, подходящим в качестве единичных доз для субъектов, подлежащих лечению; причем каждая единица содержит заданное количество активного соединения, рассчитанное для получения желаемого терапевтического эффекта в комбинации с необходимым фармацевтическим носителем.
[00294] Спецификация для единичной дозированной формы согласно данному изобретению определяется и непосредственно зависит от (a) уникальных характеристик активного соединения и конкретного терапевтического эффекта, который должен быть достигнут, и (b) ограничений, присущих процессу смешивания такого активного соединения для лечения чувствительности у индивидуумов.
[00295] Эффективные дозы и схемы приема для биспецифических антител, используемых в данном изобретении, зависят от заболевания или патологического состояния, подлежащего лечению, и могут быть определены специалистами в данной области техники.
[00296] Иллюстративный неограничивающий диапазон для терапевтически эффективного количества биспецифического антитела, используемого в данном изобретении, составляет около 0,1-100 мг/кг.
[00297] В некоторых вариантах осуществления описанных в данном документе способов полипептидные конструкции вводят в комбинации с агентом для лечения конкретного заболевания, расстройства или патологического состояния. Агенты включают, но не ограничиваются терапией, включающей антитела, малые молекулы (например, химиотерапевтические средства), гормоны (стероиды, пептиды и т.п.), радиотерапии (γ-лучи, рентгеновские лучи и/или направленную доставку радиоизотопов, микроволновое радиоизлучение, УФ-излучение и т. п.), генотерапии (например, антисмысловая, ретровирусная терапия и тому подобное) и другие иммунотерапии. В некоторых вариантах осуществления полипептидные конструкции вводят в комбинации с противодиарейными средствами, противорвотными средствами, анальгетиками, опиоидами и/или нестероидными противовоспалительными агентами. В некоторых вариантах осуществления полипептидные конструкции вводят до, во время или после операции.
[00298] Все цитируемые ссылки в данном документе полностью включены в качестве ссылки во всей их полноте.
ПРИМЕРЫ
Материалы и способы
[00299] Клонирование ДНК-экспрессирующих конструкций, кодирующих полипептидную конструкцию: анти-CD-3 scFv с доменами сайта расщепления протеазой используются для конструирования антигенсвязывающего белка в комбинации с доменом анти-CD-3 scFv и доменом увеличения периода полувыведения (например, ЧСА-связывающий пептид или домен VH), с доменами, организованными, как изображено на фиг. 53. Для экспрессии антигенсвязывающего белка в клетках СНО кодирующие последовательности всех белковых доменов клонируют в векторную систему экспрессии млекопитающих. Короче говоря, последовательности генов, кодирующие CD3-связывающий домен, домен увеличения периода полувыведения и CD-3-связывающий домен вместе с пептидными линкерами L1 и L2, отдельно синтезируются и субклонируются. Полученные конструкции затем лигируют вместе в порядке мишень-связывающий домен - L1 - VH CD-3-связывающий домен - L2 - домен расщепления протеазой - L3 - VLi CD-3-связывающий домен - L4 - мишень-связывающий домен - L5 - VL CD-3-связывающий домен - L6 - домен расщепления протеазой - L7 - VHi CD-3-связывающий домен - L8 - домен увеличения периода полувыведения, чтобы получить окончательную конструкцию. Все экспрессирующие конструкции сконструированы так, чтобы содержать кодирующие последовательности для N-концевого сигнального пептида и C-концевого гекса- или дека-гистидиновой (6x- или 10x-His) метки для облегчения секреции и очистки белка, соответственно.
[00300] Экспрессия полипептидных конструкций в стабильно трансфицированных клетках СНО: использовали систему экспрессии клеток CHO (Flp-In®, Life Technologies), производная линии клеток яичника китайского хомяка CHO-K1 (ATCC, CCL-61) (Kao and Puck, Proc. Natl. Acad Sci USA 1968;60(4):1275-81). Адгезивные клетки субкультивируются в соответствии со стандартными протоколами культуры клеток, предоставленными Life Technologies.
[00301] Для адаптации к росту суспензии клетки отделяют от колб для тканевой культуры и помещают в бессывороточную питательную среду. Клетки, адаптированные к суспензии, криоконсервируют в среде с 10% ДМСО.
[00302] Рекомбинантные клеточные линии СНО, стабильно экспрессирующие секретируемые полипептидные конструкции, получают путем трансфекции адаптированных к суспензии клеток. В течение селекции с использованием антибиотика гигромицин В плотности жизнеспособных клеток измеряют два раза в неделю и клетки центрифугируют и ресуспендируют в свежей среде для селекции при максимальной плотности 0,1×106 жизнеспособных клеток/мл. Пулы клеток, которые стабильно экспрессируют полипептидные конструкции, выделяют после 2-3 недель отбора, при этом точечные клетки переносят в стандартную культуральную среду во встряхиваемых колбах. Экспрессия рекомбинантных секретируемых белков подтверждается проведением электрофореза в белковых гель-элекрофореза или проточной цитометрии. Стабильные клеточные пулы криоконсервируют в среде, содержащей ДМСО.
[00303] Полипептидные конструкции получают в 10-суточных культурах с подпиткой стабильно трансфицированных клеточных линий СНО путем секреции в супернатанте клеточной культуры. Супернатанты клеточной культуры собирают через 10 суток при жизнеспособности культуры, как правило, >75%. Образцы собирают из производственных культур каждый день и оценивают плотность и жизнеспособность клеток. В день сбора урожая супернатанты клеточной культуры очищают при помощи центрифугирования и вакуумной фильтрации перед дальнейшим использованием.
[00304] Титры экспрессированных белков и целостность продукта в супернатантах клеточной культуры анализируют с помощью ДСН-ПААГ-электрофореза.
[00305] Очистка полипептидных конструкций: Полипептидные конструкции очищают от супернатантов культуры клеток СНО в двухстадийной процедуре. Конструкции подвергают аффинной хроматографии на первой стадии, после чего проводят препаративной гель-эксклюзионной хроматографии (ЭХ) на Superdex 200 на второй стадии. Образцы подвергают буферному обмену и концентрируют при помощи ультрафильтрации до типичной концентрации >1 мг/мл. Чистоту и однородность (как правило >90%) конечных образцов оценивают с помощью ДСН-ПААГ-электрофорезу в восстанавливающих и невосстанавливающих условиях с последующим иммуноблоттингом с использованием анти-ЧСА или антиидиотипическое антитело, а также аналитической ЭХ, соответственно. Очищенные белки хранят в аликвотах при -80°С до использования.
[00306] Сэндвич-ИФА, демонстрирующий связывание CD3: 96-луночные планшеты EIA покрывали EGFR макака резус::hFC в концентрации 1 мкг/мл в PBS (фосфатно-солевой буфер) и инкубировали в течение ночи при 4°C. Затем планшеты трижды промывали PBS, содержащим 0,05% Tween-20, и блокировали SuperBlock (PBS) в течение 1 часа при комнатной температуре. После трех дополнительных промывок в соответствующие лунки добавляли серийно разведенные Prodent и инкубировали в течение 1 часа при комнатной температуре. Планшеты снова промывали и биотин-конъюгированный CD3E яванского макака::hFC добавляли до конечной концентрации 1 мкг/мл и инкубировали в течение 1 часа при комнатной температуре. После промывки планшетов еще три раза HRP-конъюгированный стрептавидин добавляли в концентрации 0,1 мкг/мл и инкубировали в течение 30 минут. Наконец, планшеты снова промывали и удерживали в течение 5 минут с помощью однокомпонентным субстратом TMB. Реакцию останавливали с помощью останавливающего раствора Surmodics 650, и планшеты считывали при 650 нм.
[00307] Сэндвич-FACS, демонстрирующий связывание с CD3: клетки OvCAR8, выращенные до около 80% конфлюентности, отделяли с помощщью 20 нМ ЭДТА в PBS. Клетки затем блокировали PBS, содержащим 10% FBS, и помещали в 96-луночный круглодонный планшет при 2×105 клеток/лунку. Все дальнейшие шаги выполнялись на льду. Планшеты центрифугировали при 800×g в течение 5 минут для осаждения клеток. Супернатант отбрасывали и клетки ресуспендировали в серийно разведенных Prodent. После инкубации Prodent в течение 1 часа на льду клетки трижды промывали с использованием PBS, содержащим 1% FBS. Затем добавляли AF488, меченное CD3E яванского макака::hFC, в концентрации 0,5 мкг/1×106 клеток и инкубировали на льду и в темноте в течение 30 минут. Клетки промывали еще три раза, ресуспендировали в 150 мкл PBS, содержащем 1% FBS и 0,5 мкг/мл йодида пропидия, и анализировали на проточном цитометре.
[00308] Анализ TDCC: трансдуцированные люциферазой клетки OvCAR8 выращивали примерно до 80% конфлюэнтности и отделяли с помощью TrypLE Express. Клетки центрифугировали и ресуспендировали в средах до 1×106/мл. Очищенные человеческие пан-Т-клетки оттаивали, центрифугировали и ресуспендировали в средах. Наконец, совместную культуру клеток OvCAR8 и Т-клеток добавляли к 384-луночным планшетам для культивирования клеток. Затем к совместной культуре добавляли последовательно разбавленные продукты и инкубировали в течение 48 часов. Наконец, равный объем реагента для люциферазного анализа SteadyGlo добавляли к планшетам и инкубировали в течение 20 минут. Планшеты считывали и записывали общую люминесценцию.
[00309] ДСН-ПААГ-электрофорез для ЭК-расщепления: Prodent заменяли буфер на HBS, содержащий 2 мМ CaCl2, и расщепляли рекомбинантной энтерокиназой (NEB, P8070L) в двух концентрациях. Реакцию расщепления проводили в течение 2 часов при комнатной температуре и останавливали избытком бензамидина-сефарозы. Продукты расщепления проводили в 4-20% трис-глициновом геле и окрашивали Coomassie G-250.
[00310] ДСН-ПААГ-электрофорез для неочищенных белков: для определения уровней экспрессии кондиционированные среды из временно трансфицированных клеток Expi293 оценивали с помощью ДСН-ПААГ-электрофорезу. 10 мкл супернатанта из каждой трансфекции проводили в восстанавливающих и невосстанавливающих условиях на 10-20% трис-глициновом геле. Гель окрашивали Coomassie G-250, и ожидаемые полосы наблюдалиси в границах соответствующих молекулярных масс.
[00311] ДСН-ПААГ-электрофорез для очищенных белков: после очистки 2 мкг каждого Prodent проводили в невосстанавливающих условиях на 10-20% трис-глициновом геле для оценки чистоты и стабильности. Гель окрашивали Coomassie G-250, и ожидаемые полосы наблюдались в границах соответствующих молекулярных масс.
[00312] Непрямой ИФА - связывание Prodent с EGFR или CD3: 96-луночный планшет EIA покрывали антигеном захвата- или EGFR макака резус:: hFC, или CD3E яванского макака::Flag::hFC в концентрации 1 мкг/мл в PBS и инкубировали в течение ночи при 4 °C. Затем планшеты трижды промывали PBS, содержащим 0,05% Tween-20, и блокировали SuperBlock (PBS) в течение 1 часа при комнатной температуре. После трех дополнительных промывок в соответствующие лунки добавляли серийно разведенные Prodent и инкубировали в течение 1 часа при комнатной температуре. Планшеты снова промывали и HRP -конъюгированное анти-6x His Tag антитело в концентрации 1 мкг/мл и инкубировали в течение 1 часа при комнатной температуре. Наконец, планшеты снова промывали и удерживали в течение 5 минут с помощью однокомпонентного субстрата TMB. Реакцию останавливали с помощью останавливающего раствора Surmodics 650, и планшеты считывали при 650 нм.
[00313] FACS - связывание Prodent с OvCAR8 или Jurkat: нерасщепленные Prodent оценивали с использованием FACS для подтверждения связывания EGFR с клетками OvCAR8 и связывания CD3 с Jurkat. Клетки блокировали PBS, содержащим 10% FBS, и помещали в 96-луночный круглодонный планшет при 2×105 клеток/лунку. Все дальнейшие шаги выполнялись на льду. Планшеты центрифугировали при 800×g в течение 5 минут для осаждения клеток. Супернатант отбрасывали и клетки ресуспендировали в серийно разведенных Prodent. После инкубации Prodent в течение 1 часа на льду клетки трижды промывали PBS, содержащим 1% FBS. Клетки ресуспендировали в ФИТЦ-меченом анти-6x His Tag антителе в концентрации 0,5 мкг/мл и инкубировали в течение 30 минут. Клетки промывали еще три раза, ресуспендировали в 150 мкл PBS, содержащем 1% FBS и 0,5 мкг/мл йодида пропидия, и анализировали на проточном цитометре.
[00314] FACS и MSD - Расщепление Prodent при помощи клеток, трансфицированных ЭK: Расщепление Prodent клонами OvCAR8, трансфицированными ЭК, оценивали при помощи FACS и MSD. Клетки выращивали примерно до 80% конфлюэнтности и отделяли с помощью 20 нМ ЭДТА в PBS. Для MSD 2×104 клеток были иммобилизовано в каждой лунке 96-луночного планшета Sector MSD в течение 2 часов при 37°C. Затем лунки блокировали PBS, содержащим 10% FBS в течение 1 часа при комнатной температуре. Планшет промывали три раза аналитическим буфером (PBS, содержащим 1% ФБС (фетальная бычья сыворотка)). Добавляли серийно разведенные нерасщепленные продукты и инкубировали в течение 1 часа при комнатной температуре. Планшет промывали еще три раза и Sulfo-Tag-меченное CD3E яванского макака::Flag::hFC добавляли до конечной концентрации 1 мкг/мл и инкубировали в течение 1 часа при комнатной температуре. Планшет промывали дополнительно три раза. Добавляли буфер для считывания Т без поверхностно-активных веществ и немедленно измеряли общую люминесценцию.
[00315] Для FACS клетки блокировали с использованием PBS, содержащего 10% ФБС, и помещали в 96-луночный круглодонный планшет для культуры клеток при 2×105 клеток/лунка. Все дальнейшие шаги выполнялись на льду. Планшеты центрифугировали при 800×g в течение 5 минут для осаждения клеток. Супернатант отбрасывали и клетки ресуспендировали в серийно разведенных нерасщепленных Prodent. После инкубации Prodent в течение 1 часа на льду клетки трижды промывали PBS, содержащим 1% FBS. Затем добавляли AF488, меченное CD3E яванского макака::hFC, в концентрации 0,5 мкг/1×106 клеток и инкубировали на льду и в темноте в течение 30 минут. Клетки промывали еще три раза, ресуспендировали в 150 мкл PBS, содержащем 1% FBS и 0,5 мкг/мл йодида пропидия, и анализировали на проточном цитометре.
[00316] FACS - Генерация ЭК-экспрессирующих клеток OvCar8: клетки, трансфецированные вектором, кодирующим энтерокиназу с внеклеточным 6xHis Tag, выращивали в условиях селекции. Клоны собирали и анализировали с помощью FACS для определения относительных уровней экспрессии ЭК. Клетки выращивали примерно до 80% конфлюэнтности, отделяли с использованием 20 нМ ЭДТА в PBS и блокировали PBS, содержащим 10% ФБС. Все дальнейшие шаги выполнялись на льду. Каждый клон окрашивали в двух экземплярах с помощью ФИТЦ-меченого мышиного IgG1 анти-6x His Tag антитела в концентрации 0,5 мкг/мл. В качестве отрицательного окрашивания использовали ФИТЦ- меченый изотипический контроль мышиного IgG1. Нетрансфицированные клетки OvCAR8 также окрашивались обоими антителами в качестве отрицательного контроля. После 1-часовой инкубации на льду клетки промывали три раза и ресуспендировали в 150 мкл PBS, содержащем 1% ФБС и 0,5 мкг/мл йодида пропидия. Клоны анализировали на проточном цитометре и ранжировали по экспрессии ЭК.
[00317] Аффинности связывания выбранных prodent с помощью Octet: Конфигурация анализа Octet для измерения аффинности: биосенсор захвата античеловеческого IgG (AHC) ---> huEGFR.huFc или hCD3e.flag. hFc--->Prodent.6his. Стадии анализа Octet: исходный уровень - 60 секунд, загрузка - 120 секунд, исходный уровень 2-60 секунд, ассоциация - 180 секунд, диссоциация - 300 секунд. Загрузите 100 нМ белка huEGFR.huFc или hCD3e.flag. hFc на кончик датчика AHC. Концентрация Prodent составляла 100 нМ. Буфер: 0,25% казеина в буфере PBS, это было использовано для гидратации сенсора, разбавления образцов и всех этапов исходного уровня и диссоциации. Температура - 30°C. Скорость шейкера - 1000 об/мин. Положительный контроль анти-huEGFR мкАт от BD Pharmingen, кат. № 555996, и анти-hCD3e мкАт от BD Pharmingen, кат. № 551916. Отрицательный контроль: мышиный IgG2b, IgG1 и Enbrel. Для получения данных использовался инструмент Octet RED96
[00318] Конфигурация количественного анализа с белком А: биосенсор с белком A ---> Prodent. Стадии анализа Octet: погрузите в образец на 120 секунд сенсо с белком А, регенерируйте х 3 раза и повторите для всех образцов. Буфер: 0,25% казеина в буфере PBS или среде для экспрессии, тот же буфер для гидратации сенсора и разведения образцов. Температура - 30°C. Скорость шейкера - 400 об/мин. Диапазон стандартных кривых 100, 50, 25, 12,5, 6,25, 3,125, 1,56, 0,78 мкг/мл очищенного Prodent.
[00319] Реакция расщепления матриптазой: для протеолитической реакции к 69 мкМ Pro8MS и к 81 мкМ Pro8ML образцов до конечной концентрации 0,3 мкМ добавляли рекомбинантный каталитический домен матрипазы ST14 (R&D, кат. № 3946-SE), белок 26 кДа. Реакционную смесь оставляли на 24 часа при комнатной температуре и останавливали избытком бензамидин-сефарозы. Образцы анализировали с помощью ДСН-ПААГ-электрофореза (10-20% трис/глициновый гель, Invitrogen, невосстанавливающие условия). Оказалось, что расщепление завершено на >95%. Необработанные образцы Pro8MS и Pro8ML сохраняли при комнатной температуре в то же самое время, что и обработанные образцы. Реакцию проводили в буфере, содержащем 25 мМ цитрата натрия, 75 мМ L-аргинина, 75 мМ хлорида натрия, 4% сахарозный буфер (рН 7,0).
[00320] Профили ЭХ Prodent: аналитическую эксклюзионную хроматографию проводили с использованием колонки Yarra 3 мкм SEC-2000 (Phenomenex) в системе ВЭЖХ (Ailient Technologies 1290 Infinity II). Препаративную эксклюзионную хроматографию проводили с использованием колонки HiLoad 26/600 Superdex 200 (GE) на чистой хроматографической системе AKTA (GE) в буфере из 31,25 мМ цитрата натрия, 94 мМ L-аргинина, 94 мМ NaCl (pH 7,0).
[00321] Анализы протеазной активности: протеолитическая активность коммерческих рекомбинантных или очищенных протеаз и сывороток человека, мыши и яванского макака измеряли с использованием пептидов, меченых флуорофорной парой, (пептиды FRET) в качестве субстратов. Флуоресценцию пептидов, меченых Abz-Dnp, измеряли при длине волны возбуждения/излучения 320 и 420 нм, соответственно. Флуоресценцию пептидов, меченых Dabcyl-EDANS, измеряли при длине волны возбуждения/излучения 340 и 490 нм, соответственно. Пептиды добавляли из 20 мМ раствора в ДМСО в реакционную лунку, содержащую или специфический для протеазы буфер, или сыворотку, до конечной концентрации 3-120 мкМ. Концентрация добавленной протеазы составляла 1-10 нМ. Флуоресценцию регистрировали с использованием ридера 96-луночного планшета в диапазоне линейной чувствительности флуоресценции.
Пример 1: Подготовка и характеристика начальной платформы PRO
[00322] Целью этого исследования была разработка «условно активного» привлекающего T-клетки активатора, в котором активация Т-клеток и цитотоксичность усиливаются в микроокружении опухоли. Стратегия: заключалась в том, чтобы вставить опухолеспецифические сайты расщепления протеазой в патентованные молекулы αX/αCD3 так, чтоб результаты расщепления и связывания опухоли приводили к образованию активной молекулы. αX является связывающим доменом для 1 или предпочтительно 2 опухолевых антигенов. Молекулярный дизайн использует сайты расщепления протеазой, расположенные в scFv-линкерах пары неактивных анти-CD3 scFv, которые содержат комплементарные активные анти-CD3 домены (VH и VL). В принципе, после связывания двух противоопухолевых связывающих доменов с поверхностью опухолевой клетки, два связанных функциональных анти-CD3-связывающих домена могут связываться с образованием активного CD3-связывающего домена и инициировать опосредованное T-клеткой уничтожение опухолевой клетки.
Платформа 1 (неспаренные αCD3 scFv)
Pro1 - αEGFR G8 sdAb - I2C VH - His10
Pro2 - I2C VL - αEGFR D12 sdAb - His10
Pro3 - αEGFR G8 sdAb - I2C scFv (VH -(GS)3-VL) -αEGFR D12 sdAb- His10
Pro4 - αEGFR G8 sdAb - I2C VH - Flag* - I2C VL - αEGFR D12 sdAb- His10
(короткий scFv-линкер предотвращает образование aCD3 пар VH и VL)
Flag* представляет собой 8-аминокислотный сайт расщепления протеазой - энтерокиназой (ЭК)
[00323] Конструкции Платформы 1 были подготовлены следующим образом. Гены, кодирующие Prodent 1-4, клонировали в экспрессионный вектор млекопитающих и получали плазмидную ДНК. Белки транзиентно экспрессировались в линиях клеток HEK293 и CHO-S в 25 мл среды для роста во встряхиваемых колбах. Каждый поли-His-меченый белок очищали с использованием Ni - excel смолы. Результаты изображены на фиг. 1А и фиг. 1B.
Генерация scFv CD3-связывающего домена
[00324] Каноническая последовательность CD3ε-цепи человека - № доступа в Uniprot P07766. Каноническая последовательность CD3γ-цепи человека - № доступа в Uniprot P09693. Каноническая последовательность CD3δ-цепи человека - № доступа в Uniprot P043234. Антитела против CD3ε, CD3γ или CD3δ получают с помощью известных технологий, таких как созревание аффинности. В тех случаях, когда в качестве исходного материала используются мышиные анти-CD3 антитела, для клинических условий необходима гуманизация мышиных анти-CD3 антител, где остатки, специфические для мыши, могут индуцировать ответ человеческого антимышиного антитела (HAMA) у субъектов, которые получают лечение с описанным в данном документе антигенсвязывающим белком. Гуманизацию осуществляют путем замены CDR-областей из мышиного анти-CD3 антитела к соответствующим акцепторным каркасам человеческой зародышевой линии, необязательно включая другие модификации CDR и/или каркасных областей. Как указано в данном документе, нумерация остатков антител и фрагментов антител согласно Кабату (Kabat E.A. et al, 1991; Chothia et al., 1987).
[00325] Таким образом, человеческие или гуманизированные анти-CD3 антитела используются для создания последовательностей scFv для CD3-связывающих доменов полипептидной конструкции. Получают последовательности ДНК, кодирующие человеческие или гуманизированные домены VL и VH, и кодоны для конструкций, необязательно, оптимизированы для экспрессии в клетках Homo sapiens. Сайт расщепления протеазой включают между доменами VH и VL. Порядок, в котором появляются домены VL и VH в scFv, варьируется (т.е. ориентация VL-VH или VH-VL) и три копии субъединицы «G4S» или «G4S» (G4S)3 соединяют вариабельные домены для создания домена scFv. Плазмидные конструкции анти-CD3 scFv могут иметь необязательный Flag, His или другие аффинные метки, и их вводят при помощи электропорации в HEK293 или другие подходящие клеточные линии человека или млекопитающих, а белки экспрессируют и очищают. Подтверждающие анализы включают анализ связывания при помощи проточной цитометрии, кинетический анализ с использованием Proteon и окрашивание клеток, экспрессирующих CD-3 или мишень.
[00326] Экспрессированные полипептиды подвергали эксклюзионной хроматографии, и было установлено, что они образуют агрегаты, возможно, диатела. Фиг. 2.
[00327] Приведенные выше эксперименты дали следующие результаты и выводы. Наблюдали экспрессию каждого из 4 поли-His-меченых белков prodent, за исключением Pro1 в клетках HEK293. Таким образом, полипептиды могут быть экспрессированы. Для очистки полипептидов из экспрессирующих сред использовалась Ni- excel смола. Затем образцы диализовали против PBS, а концентрацию полипептида определяли с помощью A280, а уровни экспрессии расчитывали путем обратных вычислений. Каждый очищенный поли-His-меченый белок имел ожидаемую молекулярную массу при прогоне на ДСН-ПААГ геле. Аналитическая ЭХ была выполнена на диализованных Niexcel элюированных образцах, однако Pro1 и Pro2 показали сильную тенденцию к агрегации. В анализах на основе ИФА Pro4 с ограниченным αCD3 scFv-линкером связывал с белком CD3ε, эквивалентно белку положительного контроля Pro3, поэтому ограничение линкера не создавало условно активного привлекающего T-клетки активатора.
Пример 2: Подготовка и характеристика платформы PRO второго поколения
[00328] Полипептиды платформы PRO второго поколения были сконструированы так, чтобы иметь домен VL или VH, который оказывается неактивным (то есть, по существу, не связывается с CD3) путем изменения полипептидной последовательности этого VL или VH домена Иллюстративные полипептиды Pro второго поколения представлены ниже.
Платформа 2 (инактивированные αCD3 scFv)
Pro5 - αEGFR G8 sdAb - I2C VH - Flag - I2CVLi - Flag -
I2CVHi - Flag - I2CVL - αEGFR D12 sdAb - His6
Pro6 - αEGFR G8 sdAb - I2C VH - Flag - I2 CVL - His6
Pro7 - I2CVHi - Flag - I2CVL - αEGFR D12 sdAb - His6
Pro8 - αEGFR G8 sdAb - I2C VH - Flag - I2CVL - His6
[00329] Структура Pro 5 изображена на фиг. 3. Ожидалось, что для Pro 5 нерасщепленные полипептиды будут хорошо связывать EGFR, не будут связываться с CD3 и не будут активны в анализе Т-зависимой цитотоксичности (TDCC). После расщепления было ожидаемо, что обе половины активного анти-CD-3 scFv будут связаны с раковой клеткой путем связывания с EGFR. Два активных домена scFv будут взаимодействовать с формированием активного CD-3-связывающего scFv с конструкцией, демонстрирующей активность в анализе TDCC.
[00330] Структуры бифункциональных партнеров Pro 6 и Pro 7 изображены на фиг. 4. Описанные в данном документе эксперименты продемонстрировали, что введение модельного сайта расщепления протеазой (сайт расщепления ЭК) в CDR2 VH или VL в анти-CD3scFv отменяет связывание и активность CD3. Ожидалось, что нерасщепленные молекулы будут связывать EGFR, не будут связывать CD3 и не будут активны в анализе TDCC. После расщепления Pro 6 и Pro 7 будут продуцировать активные молекулы, поскольку интактные VH и VL связываются с раковой клеткой через EGFR.
[00331] Для того, чтобы получить анти-CD3e ScFv с неактивным VH, были сделаны следующие мутации: в Pro21 (N30S, K31G, Y32S, A49G, Y55A, N57S, Y61A, D64A, N97K, N100K, S110A, Y111F); в Pro29 (Y32S, Y61A, D64A, S110A, Y111F); в Pro30 (Y32S, Y61A, S110T, Y111F); в Pro31 (N30S, K31G, Y55A, N57S, Y61E, D64A, F104A, Y108A); в Pro 32 (N30S, K31G, Y32H, Y55A, N57S, N103A, F104N). Мутации были помещены в CDR-области VH: в CDR1-N30S, K31G, Y32S, Y32H; в CDR2 - A49G, Y55A, N57S, Y61A, D64A, Y55A, N57S, Y61E; в CDR3 - N97K, N100K, N103A, F104N, F104A, Y108A, S110A, S110T, Y111F. Мутации N30S, K31G, Y32S, Y32H, A49G, Y55A, N57S, Y61A, D64A, Y55A, N57S, Y61E были выбраны на основе появления остатков в последовательностях зародышевой линии человека и их возможного положения в интерфейсе, при связывании с CD3 в комплексе. Мутации N103A, F104N, F104A, Y108A в области CDR3 были выбраны так, что они были на части поверхности CDR3, которая экспонирована, на расстоянии от потенциального интерфейса VH-VL и на потенциальном интерфейсе с CD3e-взаимодействиями. Мутации S110A, S110T, Y111F были выбраны для того, чтобы мягко дестабилизировать потенциальный интерфейс VH-VL, чтобы вызвать незначительную перестройку области.
[00332] При экспрессии, Pro29-32 образуют стабильные белки с Tm 53-55°С, как измерено с помощью DSF. Pro21 не экспрессируется хорошо.
[00333] Для того, чтобы продуцировать анти-CD3e ScFv с неактивным VL, следующие мутации были внесены в Pro20 (N32H, K54S, F55N, L56K, A57H, P58S, G59W, W94G, N96R). Мутации были помещены в области CDR VL: в CDR1 - N32H; в CDR2 - K54S, F55N, L56K, A57H, P58S, G59W; в CDR3 - W94G, N96R. Мутации N32H, K54S, F55N, L56K, A57H, P58S, G59W были выбраны на основе появления остатков в последовательностях зародышевой линии человека и их потенциального положения на интерфейсе, неблагоприятно влияющих на связывание CD3 в комплексе. Мутации W94G, N96R в области CDR3 были выбраны так, что они были на части поверхности CDR3, которая была экспонирована, на расстоянии от потенциального интерфейса VH -VL.
[00334] При экспрессии Pro20 продуцирует стабильный белок.
[00335] Pro8 является положительным контролем. Pro8 использовали для подтверждения того, что введение модельного сайта расщепления протеазой (сайт расщепления ЭК) в scFv-линкере не мешает сворачиванию scFv и связыванию CD3. Фиг. 5. Нерасщепленные молекулы Pro8 должны связывать EGFR, связывать CD3 и быть активными в анализе TDCC. После расщепления Pro8 должно потерять способность связывания CD3 из-за разделения VH и VL, вызванного отсутствием совместного влияния каждой половины расщепленной молекулы, связанной с поверхностью клетки посредством связывания с EGFR.
[00336] С полипептидами было проведено четыре типа анализа связывания/активности. Иллюстративные анализы изображены на фиг. 9.
[00337] Модельная протеаза, энтерокиназа, была использована для расщепления конструкций согласно изобретению в сайте расщепления протеазой между активными и неактивными доменами VH и VL.
Результаты
[00338] На фиг. 6 изображен ДСН-ПААГ-электрофоре неочищенных полипептидов согласно изобретению и различных контролей. Как показано с помощью ПААГ-электрофореза, полипептиды хорошо экспрессируются. Эксклюзионная хроматография Pro 5-8 демонстрирует отсутствие агрегации, что подтверждает, что эти структуры Pro имеют тенденцию образовывать мономерные типы. Фиг. 7. Полипептиды очищали хроматографией на основе Ni-excel, и каждый полипептид обеспечивал, по существу, одну полосу на ДСН-ПААГ-электрофорезе. В таблице на фиг. 8 показаны результаты экспрессии белка и его очистки.
[00339] Анализы EGFR-ИФА продемонстрировали, что полипептиды Платформы 2 способны связываться с EGFR в анализе ИФА (фиг. 10А) и с EGFR на клетке (фиг. 10В). Неактивные (т.е. нерасщепленные) полипептиды Платформы 2 не связываются с CD3, как это подтверждено при помощи CD3-ИФА и CD3-FACS на клетках Jurkat. Фиг. 11А и фиг. 11B.
[00340] Показано, что Pro 6 и Pro 7 активируются путем расщепления протеазой, разделяя неактивные VLi Pro6 и неактивные VHi Pro7 от их соответствующих VH- и VL-партнеров в конструкции. Нерасщепленные молекулы, связанные с EGFR, не связывают CD3 и не активны в анализе TDCC. После расщепления смесь Pro6 и Pro 7 продуцирует активный анти-CD3 домен, так как интактные VH и VL вместе связываются с раковой клеткой посредством связывания с EGFR. Фиг. 12.
[00341] Было показано, что энтерокиназа (ЭК) расщепляет Pro 5-8, как показано при помощи ДСН-ПААГ-электрофореза на Фиг. 13. С помощью ИФА было показано, что Pro6 и Pro7 совместно связываются с CD3 после расщепления ЭК. Фиг. 14. На фиг. 14В и фиг. 14C изображено минимальное связывание с CD3 отдельных Pro 6 и PRO 7, соответственно. При использовании вместе в анализе, Pro 6 и Pro 7 совместно связываются с CD3, образуя активный CD3-связывающий домен после расщепления ЭК (фиг. 14D). Сценарий схематично изображен на фиг. 14Е. Также с помощью сэндвич-FACS, что Pro 6 и Pro 7 совместно связываются с CD3 после расщепления ЭК. Таким образом, на фиг. 15В и фиг. 15C изображено, что отдельные конструкции Pro не связываются с CD3, однако, когда они объединяются и образуют активный CD3-связывающий домен на поверхности EGFR-экспрессирующих OvCar8-клеток, они способны совместно связывать CD3 (фиг. 15D).
[00342] CD3-связывание полноразмерной конструкцией Pro5 активируется после протеолитического расщепления конструкции при помощи ЭК. Фиг. 16.
[00343] Pro 8 представляет собой положительную контрольную модель, имеющую единый мишень-связывающий домен (анти-EGFR). Таким образом, когда эта конструкция расщепляется на сайте расщепления протеазой, она теряет способность связываться с CD3, потому что активный CD3-связывающий домен не образуется: фрагмент VL, который не связан с мишень-связывающим доменом, не связывается с клеткой способом, достаточно эффективным для получения совместного взаимодействия между VH и VL для получения активного CD3-связывающего домена. До расщепления Pro8 связывает EGFR через единственный EGFR-связывающий домен, связывает CD3 с активным CD3-связывающим доменом и, следовательно, активен в анализе TDCC. После расщепления расщепленная конструкция теряет способность связывать CD3 из-за слабого взаимодействия между компонентами scFv. Фиг. 17. Этот результат показан на фиг. 18A и фиг. 18B.
[00344] Анализ TDCC Pro6, Pro7 и Pro8 показан на фиг. 19 (A-D). На фиг. 19А и фиг. 19B показаны результаты анализа TDCC дл Pro6 и Pro7 по отдельности. По существу, отсуствует опосредованная T-клетками цитотоксичность, вызванная этими отдельными конструкциями после расщепления ЭК. И наоборот, когда Pro6 и Pro7 объединяются и расщепляются, как изображено на фиг. 19C, наблюдается существенная Т-клеточная цитотоксичность. Напротив, когда Pro8 расщепляется с помощью ЭК, цитотоксичность снижается (фиг. 19D).
Пример 3: Оценка зависимости связывания от множества мишень-связывающих доменов
[00345] Эксперимент был разработан для оценки важности более чем одного мишень-связывающего домена на способность конструкций связываться с CD3. Pro25-27 были сконструированы без доменов, связывающих EGFR-мишени, причем эти домены заменяются доменами, связывающими зеленый флуоресцентный белок (ЗФБ). Фиг. 20. Часть мотивации для использования анти-ЗФБ-связывающих доменов заключалась в том, что ЗФБ не экспрессируется на поверхности клеток OvCar8. Конструкции PRO, содержащие анти-GFP, объединяли с Pro6 и Pro7 и подвергали протеазному расщеплению с помощью ЭК. Как изображено на фиг. 21C (Pro6+Pro26), фиг. 21D (Pro6+Pro27), фиг. 21E (фиг. 7+25) и фиг. 21F (Pro9+25) по существу отсутствует связывание CD3 этими конструкциями после расщепления ЭК. Таким образом, каждый компонент Pro должен содержать по меньшей мере один мишень-связывающий домен для расщепленной конструкции для связывания и образования активного CD3-связывающего домена.
Пример 4: Оценка альтернативных протеаз и сайт расщепления
[00346] Чтобы подтвердить то, что описанные выше явления не зависят исключительно от ЭК и ее консенсусных сайтов расщепления, были сконструированы конструкции Pro с сайтами расщепления протеазой для альтернативных протеаз, включая матриптазу. Pro8 MS и Pro8ML содержат чувствительный к матрипазе линкер из 14 аминокислот и чувствительный к матрипазе линкер из 24 аминокислот, соответственно. Линкеры находятся между доменами VH и VL конструкции. Используя способы, изложенные в предыдущих примерах, было показано, что Pro8, Pro8 MS и Pro8 ML имеют эквивалентные характеристики связывания до и после расщепления с соответствующей линкер-специфической протеазой. Таким образом, перед расщеплением каждая из конструкций связывает EGFR, связывает CD3 и активна в анализе TDCC. После расщепления CD3-связывающая активность и активность в анализе TDCC теряются из-за слабого взаимодействия scFv. Результаты этого эксперимента приведены на фиг. 23, которая демонстрируют результаты анализов сэндвич-ИФА, на фиг. 24, которая демонстрируют результаты анализов FACS.
[00347] Результаты, рассмотренные выше, демонстрируют, что конструкции согласно изобретению хорошо экспрессируются в эукариотической платформе. Вставка иллюстративного сайта расщепления протеазой (например, ЭК) (Flag) в CDR (например, CDR2) α-CD3 scFv (VH или VL) эффективно инактивирует α-CD3 scFv. Расщепление на сайте расщепления протеазой приводит к образованию функционального CD3-связывающего сайта. В иллюстративной паре Pro (Pro6 и Pro7) CD3-связывающий сайт формируется только тогда, когда Pro6 и Pro7 находятся в непосредственной близости. Эти результаты были получены с использованием покрытых целевым антигеном планшетов для ИФА и раковых клеток, экспрессирующих целевой антиген (на основе данных ИФА, FACS и TDCC).
Пример 5: Исследование влияние ориентации Pro на связывание
[00348] С использованием дополнительных мотивов Pro исследовали то, влияет ли ориентация Pro (порядок доменов от N- до C-конца) на способность связывания Pro (фиг. 25). На этой фиг. Pro 10 является инвертированным аналогом Pro6, а Pro9 является инвертированным аналогом Pro 7. Pro8, Pro 11 и Pro15 (OKT3) являются полностью активными α-CD3 scFv. На фиг. 25 представлена таблица, демонстрирующая комбинации Pro6, Pro7, Pro9, Pro10, Pro12 и Pro14, которые являются неполными, связываются с EGFR, но не с CD3. Отсутствие CD3-связывания неполных пар CD3 было продемонстрировано при помощи сэндвич-ИФА (фиг. 26).
[00349] Если Pro6 и Pro9 объединяются и подвергаются протеазному расщеплению, они образуют функциональный CD3-связывающий домен (фиг. 27). Pro6+Pro9 демонстрируют эквивалентные характеристики связывания по сравнению с Pro6+Pro7 (фиг. 28, 29).
[00350] Была также исследована актуальность моноспецифических и двойных нацеливающих доменов конструкций Pro. Pro9 и Pro14 объединяли и расщепляли, каждый с тем же самым EGFR-связывающим доменом (фиг. 30). На фиг. 31А изображены данные FACS для нерасщепленных и расщепленных ЭК Pro9+Pro14 и на фиг. 31B изображены аналогичные данные для Pro6+Pro7.
[00351] Также были получены и испытаны пары конструкции Pro, в которых каждый Pro имеет другой EGFR-связывающий домен. На фиг. 32A представлена таблица, в которой выложены пары Pro с EGFR- и CD3-связывающими доменами. Также был подготовлен и расщеплен первый набор пар Pro, в которых каждый член пары имеет другой EGFR-связывающий домен. Полагают, что члены этой пары подвергаются связыванию с той же молекулой EGFR («цис»-связывание) через различные связывающие домены и связыванию с различными молекулами EGFR («транс»-связывание) через различные связывающие домены (фиг. 32В). Был собран второй набор конструкций Pro, отображающих один и тот же EGFR-связывающий домен для каждого члена пары. В этом случае члены пары должны связываться с другой молекулой EGFR («транс»-связывание), поскольку мишень-связывающий сайт на сайте EGFR занят EGFR-связывающим доменом одного члена пары. Фиг. 32C. Сэндвич-ИФА на этих парах показали оба цис+транс-связывания для PRO6+Pro7 (фиг. 33A), Pro9+PRO10 (фиг. 33B), Pro12+Pro14 (фиг. 33C), Pro7+PRO10 (фиг. 33D) и Pro6+Pro9 (фиг. 33Е). В противоположность этому, только транс связывание было продемонстрировано для Pro6+Pro12 (фиг. 34А), Pro7+Pro14 (фиг. 34В), Pro9+Pro14 (фиг. 34 C) и Pro10+Pro12 (фиг. 34D). Интересно отметить, что после расщепления активности пар Pro, связывающих цис+транс, и тех, которые связывают только транс, являются сходными. Результаты анализа TDCC изображены на фиг. 35. Фиг. 35А (цис+транс), фиг. 35B (только транс). Как показано на фиг. 36, конструкции Pro положительного контроля теряют активность после расщепления ЭК, вероятно, из-за того, что они не способны образовать функциональный CD3-связывающий сайт без того, чтобы каждый член пары, имеющий функциональный EGFR-связывающий сайт, который сближает достаточно близко два компонента CD3-связывающего домена.
Пример 6: Расщепление клетками, экспрессирующими протеазу
[00352] В этом примере вектор, экспрессирующий ЭК, трансфицировали в клетки люцифераза+OVCAR8, и выбирали клоны, стабильно экспрессирующие белок. Было выбрано 100 клонов, и положительные результаты были подтверждены при помощи FACS (α-His6-ФИТЦ). Сохраняли образцы клеток, которые имели клетки с высокой, средней и низкой экспрессией. Эти образцы клеток тестировали на отдельные полипептидные конструкции согласно изобретению с использованием сэндвич-FACS, сэндвич-MSD и TDCC.
[00353] На фиг. 37 продемонстрирована стабильная экспрессия EK-His6 в клетках OvCar8-lux. Были идентифицированы колонии с высокой, средней и низкой экспрессией. Фиг. 38. Неактивированные конструкции Pro согласно изобретению приводили в контакт с клеткой, которая, как было показано, оказывает зависимую от дозы активацию конструкций Pro (фиг. 39). Результаты MSD (фиг. 39А) и FACS (фиг. 39В) являются сравнимыми. FACS-ранжирование экспрессии EK является прогностическим для расщепления Pro.
[00354] Было показано, что TDCC с использованием ЭК-сверхэкспрессирующих клеток OvCAR8 активирует Т-клеточную цитотоксичность в присутствии нерасщепленных конструкций Pro. Фиг. 40. Клетки OVCAR8 дикого типа, которые не сверхэкспрессируют ЭК, не активируют в заметной степени конструкции Pro и дают минимальную опосредованную Т-клетками цитотоксичность с использованием нерасщепленных белков (фиг. 40A). В противоположность этому, клетки OvCAR8, сверхэкспрессирующие ЭК, демонстрируют опосредованную Т-клетками цитотоксичность с использованием нерасщепленных белков (фиг. 40B).
Пример 7: Инактивация α-CD3 VH и VL
[00355] На фиг. 41 изображены гомологические модели α-CD3 scFv. Последовательность исходного полипептида VH и его общее выравнивание с наиболее гомологическими последовательностями зародышевой линии изображены на фиг. 42А. Иллюстративные варианты, предназначенные для инактивации связывания этого полипептида с CD3, приведены на фиг. 42B. Аналогично на фиг. 43 изображена последовательность исходного VL-полипептида CD3 и его общее выравнивание с наиболее гомологическими последовательностями зародышевой линии и представлены иллюстративные варианты последовательности, предназначенные для того, чтобы сделать полипептид неактивным по отношению к его связыванию с CD3.
[00356] На фиг. 44А схематически изображены некоторые конструкции полипептида Pro согласно изобретению, содержащие EGFR-связывающий домен, домены VL и VH, один из которых инактивирован (то есть VLi, VHi), домен увеличения периода полувыведения (α -HAS) и сайт Flag расщепления протеазой между доменами VH и VL. На фиг. 44B представлена таблица, в которой представлены связывающие активности этих иллюстративных видов Pro. Pro 22 представляет собой положительный контроль, который не имеет неактивного домена VH или VL. Этот Pro связывается с EGFR и CD3 перед активацией. Как указано в таблице, ни один из других видов Pro не связывается с CD3 до активации протеазой.
[00357] На фиг. 45 изображены схематические диаграммы Pro23 (фиг. 45А) и Pro24 (фиг. 45В), каждый из которых содержит более одного сайта Flag расщепления ЭК. Pro23 также содержит сайт расщепления тромбином, что делает его восприимчивым к расщеплению в плазме. Каждое «плечо» Pro23 содержит активный и неактивный CD3-связывающий домен, разделенный сайтом Flag, расщепляемым протеазой. Каждое «плечо» также содержит домен увеличения периода полувыведения, например, α-ЧСА. Как видно из Pro24, сайт расщепления тромбином может быть заменен другим сайтом расщепления, например, сайтом расщепления ЭК. На фиг. 46 представлены данные ДСН-ПААГ-электрофореза по протеазному расщеплению Pro23 и Pro24. Данные об активности Pro23 и Pro24 представлены на фиг. 47. На фиг. 47А изображено, что активность TDCC Pro23 активируется путем расщепления ЭК, но не тромбином, что подтверждает то, что отделение активного CD3-связывающего домена от его неактивного партнера является условием для связывания с полипептидом CD3. Аналогично, Pro24 активируется путем расщепления ЭК (фиг. 47B).
Пример 8: Активация путем расщепления с использованием протеаз, отличных от ЭК
[00358] Для того, чтобы подтвердить, что явления расщепления/связывания, которые наблюдаются для полипептидов Pro, не ограничивается расщеплением ЭК, были разработаны и испытаны дополнительные виды Pro виды с сайтами не-ЭК-расщепления. Исследуемые соединения были сконструированы таким образом, чтобы содержать пары переноса энергии флуоресценции, которые могли бы давать сигнал при расщеплении полипептида в сайте расщепления протеазой. На фиг. 48-52 приведены данные этого исследования. Известно, что протеаза MMP9 сверхэкспрессируется в опухолевых клетках. Пептиды были сконструированы таким образом, чтобы содержать сайты расщепления MMP9. Пептиды GPSGPAGLKGAPG и GPPGPAGMKGLPG стабильны в сыворотке и расщепляются рекомбинантным MMP9, не расщепляются рекомбинантной матриптазой ST14, TACE (ADAM17), очищенными катепсинами B и D (фиг. 48).
[00359] Были также разработаны и испытаны дополнительные пептиды, содержащие сайт расщепления протеазой Meprin. Фиг. 49. Пептиды GYVADAPK и KKLADEPE стабильны в сыворотке и расщепляются рекомбинантными Mep1A и Mep1B, не расщепляемыми рекомбинантным MMP9, TACE (ADAM17), катепсином B. Пептид GGSRPAHLRDSGK стабилен в человеческой сыворотке и, тем более, в сыворотках мыши и яванского макака, расщепляемых рекомбинантным Mep1A, частично расщепляемым рекомбинантным MMP9, но не ADAM17, катепсином B, матриптазой ST14.
[00360] Также были разработаны и испытаны пептиды, чувствительные к расщеплению матрипазой. Как изображено на фиг. 50, ни один из пептидов не является стабильным в сыворотке. Пептиды SFTQARVVGG и LSGRSDNH расщепляются рекомбинантной матрипатизой ST14, но не MMP9, TACE (ADAM17), катепсином B.
[00361] Полипептиды, чувствительные к расщеплению протеазами крови (тромбин, нейстрофильная эластаза и фурин), были разработаны и испытаны (фиг. 51). Пептидный субстрат для тромбина-1 очень эффективно расщепляется тромбином (очищенным от плазмы человека) (с низким Km и высоким Vmax). Пептидный субстрат для эластазы-1 очень эффективно расщепляется рекомбинантной нейтрофильной эластазой. На фиг. 52 изображены данные по расщеплению пептидных субстратов протеазами крови в сыворотке. Расщепление пептидов тромбином-1, тромбином-2 и фурином-2 было наиболее эффективным в человеческой сыворотке. Расщепление субстратов нейтрофильной эластазой не наблюдалось из-за отсутствия нейтрофилов, несущих активную протеазу в сыворотке.
Хотя иллюстративные варианты осуществления данного изобретения были показаны и описаны в данном документе, для специалистов в данной области техники будет очевидно, что такие варианты осуществления изобретения приведены только в качестве примера. Для специалистов в данной области техники будут очевидны многочисленные варианты, изменения и замены, не отступая от сущности изобретения. Следует понимать, что различные альтернативы вариантов осуществления изобретения, описанные в данном документе, могут быть использованы при осуществлении изобретения. Предполагается, что нижеследующая формула изобретения определяет объем изобретения, и что таким образом охватываются способы и структуры, входящие в объем этой формулы и их эквивалентов.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> MAVERICK THERAPEUTICS, INC.
<120> ИНДУЦИБЕЛЬНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ И СПОСОБЫ ИСПОЛЬЗОВАНИЯ
<130> 118459-5001-WO
<140> PCT/US2017/021435
<141> 2017-03-08
<160> 127
<170> PatentIn версии 3.5
<210> 1
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Пептидный scFv-линкер
<400> 1
Gly Gly Gly Gly Ser
1 5
<210> 2
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP7
<400> 2
Lys Arg Ala Leu Gly Leu Pro Gly
1 5
<210> 3
<211> 12
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP7
<400> 3
Asp Glu Arg Pro Leu Ala Leu Trp Arg Ser Asp Arg
1 5 10
<210> 4
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP9
<400> 4
Pro Arg Ser Thr Leu Ile Ser Thr
1 5
<210> 5
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP9
<400> 5
Leu Glu Ala Thr Ala
1 5
<210> 6
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP11
<400> 6
Gly Gly Ala Ala Asn Leu Val Arg Gly Gly
1 5 10
<210> 7
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP14
<400> 7
Ser Gly Arg Ile Gly Phe Leu Arg Thr Ala
1 5 10
<210> 8
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP
<400> 8
Pro Leu Gly Leu Ala Gly
1 5
<210> 9
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP
<220>
<221> misc_feature
<222> (6)..(6)
<223> Xaa может быть любой природной аминокислотой
<400> 9
Pro Leu Gly Leu Ala Xaa
1 5
<210> 10
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP
<400> 10
Pro Leu Gly Cys Ala Gly
1 5
<210> 11
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP
<400> 11
Glu Ser Pro Ala Tyr Tyr Thr Ala
1 5
<210> 12
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP
<400> 12
Arg Leu Gln Leu Lys Leu
1 5
<210> 13
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP
<400> 13
Arg Leu Gln Leu Lys Ala Cys
1 5
<210> 14
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой MMP2, MMP9, MMP14
<400> 14
Glu Pro Gly His Tyr Leu
1 5
<210> 15
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой урокиназный
активатор плазминогена (uPA)
<400> 15
Ser Gly Arg Ser Ala
1 5
<210> 16
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой урокиназный
активатор плазминогена (uPA)
<400> 16
Asp Ala Phe Lys
1
<210> 17
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой урокиназный
активатор плазминогена (uPA)
<400> 17
Gly Gly Gly Arg Arg
1 5
<210> 18
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой лизосомальный
фермент
<400> 18
Gly Phe Leu Gly
1
<210> 19
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой лизосомальный
фермент
<400> 19
Ala Leu Ala Leu
1
<210> 20
<211> 2
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой лизосомальный
фермент
<400> 20
Phe Lys
1
<210> 21
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой катепсин B
<400> 21
Asn Leu Leu
1
<210> 22
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой катепсин D
<400> 22
Pro Ile Cys Phe Phe
1 5
<210> 23
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой катепсин K
<400> 23
Gly Gly Pro Arg Gly Leu Pro Gly
1 5
<210> 24
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой простатический
специфический антиген
<400> 24
His Ser Ser Lys Leu Gln
1 5
<210> 25
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой простатический
специфический антиген
<400> 25
His Ser Ser Lys Leu Gln Leu
1 5
<210> 26
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой простатический
специфический антиген
<400> 26
His Ser Ser Lys Leu Gln Glu Asp Ala
1 5
<210> 27
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой вируса простого
герпеса
<400> 27
Leu Val Leu Ala Ser Ser Ser Phe Gly Tyr
1 5 10
<210> 28
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой ВИЧ
<400> 28
Gly Val Ser Gln Asn Tyr Pro Ile Val Gly
1 5 10
<210> 29
<211> 10
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой ЦМВ
<400> 29
Gly Val Val Gln Ala Ser Cys Arg Leu Ala
1 5 10
<210> 30
<211> 3
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой тромбин
<400> 30
Phe Arg Ser
1
<210> 31
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой тромбин
<400> 31
Asp Pro Arg Ser Phe Leu
1 5
<210> 32
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой тромбин
<400> 32
Pro Pro Arg Ser Phe Leu
1 5
<210> 33
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой каспаза-3
<400> 33
Asp Glu Val Asp
1
<210> 34
<211> 5
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой каспаза-3
<400> 34
Asp Glu Val Asp Pro
1 5
<210> 35
<211> 8
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой каспаза-3
<400> 35
Lys Gly Ser Gly Asp Val Glu Gly
1 5
<210> 36
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой интерлейкин-1-
бета-превращающий фермент
<400> 36
Gly Trp Glu His Asp Gly
1 5
<210> 37
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой энтерокиназа
<400> 37
Glu Asp Asp Asp Asp Lys Ala
1 5
<210> 38
<211> 9
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой FAP
<400> 38
Lys Gln Glu Gln Asn Pro Gly Ser Thr
1 5
<210> 39
<211> 6
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой калликреин 2
<400> 39
Gly Lys Ala Phe Arg Arg
1 5
<210> 40
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой плазмин
<400> 40
Asp Ala Phe Lys
1
<210> 41
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой плазмин
<400> 41
Asp Val Leu Lys
1
<210> 42
<211> 4
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой плазмин
<400> 42
Asp Ala Phe Lys
1
<210> 43
<211> 7
<212> Белок
<213> Искусственная последовательность
<220>
<223> Последовательность домена расщепления протеазой TOP
<400> 43
Ala Leu Leu Leu Ala Leu Leu
1 5
<210> 44
<211> 271
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 1
<400> 44
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser His His His His His His His His His His
260 265 270
<210> 45
<211> 813
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Prodent 1 - репрезентативная нуклеиновая кислота
<400> 45
gaagtccaac tggtggaatc gggcggtgga ctcgtgcagg ccggaggttc cctgcgcctt 60
tcctgtgcgg cttcgggaag aaccttctcc tcgtatgcca tgggatggtt ccgccaagcc 120
cctggaaaag agcgggaatt cgtcgtggcg atcaattgga gctccggttc gacttactac 180
gccgactccg tgaagggccg gttcaccatt agccgggaca atgctaagaa caccatgtat 240
ctgcagatga actcactgaa acctgaggac acggcggtgt actactgcgc cgctgggtac 300
cagatcaatt ccggaaacta caacttcaag gactacgagt acgactactg gggtcagggc 360
acccaggtca ccgtgtccag cggaggaggt ggaagcggag gcggttccga agtgcagctc 420
gtcgagtccg ggggtggact ggtccaaccg ggcggatcac tgaagctgag ctgcgcagcc 480
tccggattca ccttcaacaa gtacgccatg aactgggtca gacaggcacc cgggaaggga 540
ctggaatggg tggcccggat caggtccaag tacaacaact acgccaccta ctacgcggac 600
agcgtgaagg ataggttcac catctcccgg gacgacagca agaacactgc ctacctccaa 660
atgaacaacc tcaagaccga agatactgcg gtgtattact gcgtgcgcca cgggaacttc 720
ggaaacagct acatcagcta ctgggcctac tggggccagg gcactctggt gaccgtgtca 780
tcccaccatc accaccacca tcatcaccat cac 813
<210> 46
<211> 252
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 2
<400> 46
Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly
20 25 30
Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly
35 40 45
Leu Ile Gly Gly Thr Lys Phe Leu Ala Pro Gly Thr Pro Ala Arg Phe
50 55 60
Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val
65 70 75 80
Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser Asn
85 90 95
Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly
100 105 110
Gly Ser Gly Gly Gly Ser Gln Val Lys Leu Glu Glu Ser Gly Gly Gly
115 120 125
Ser Val Gln Thr Gly Gly Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly
130 135 140
Arg Thr Ser Arg Ser Tyr Gly Met Gly Trp Phe Arg Gln Ala Pro Gly
145 150 155 160
Lys Glu Arg Glu Phe Val Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr
165 170 175
Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn
180 185 190
Ala Lys Asn Thr Val Asp Leu Gln Met Asn Ser Leu Lys Pro Glu Asp
195 200 205
Thr Ala Ile Tyr Tyr Cys Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly
210 215 220
Thr Leu Tyr Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val
225 230 235 240
Ser Ser His His His His His His His His His His
245 250
<210> 47
<211> 756
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 2
<400> 47
cagaccgtgg tgacccagga accctcactg accgtgtccc caggaggaac cgtgaccctt 60
acctgtggct cctcgaccgg tgccgtgacg tccgggaact accccaactg ggtccagcaa 120
aagccgggac aagcccctcg gggactgatc ggggggacta agttcctggc ccctggcact 180
cctgcccgct tcagcggcag cctcctggga ggaaaagcgg ccctgacact ctcgggggtg 240
cagcctgaag atgaggccga atactactgc gtgctgtggt actccaatcg ctgggtgttt 300
ggagggggca ccaagctgac cgtgctggga ggaggaggaa gcggcggagg ttcccaggtc 360
aagctggagg aatcgggtgg aggctcagtg cagacaggag gtagcctccg gctcacttgc 420
gccgcttccg gaaggacttc ccggagctac gggatgggct ggtttcggca agcccccgga 480
aaggagagag aattcgtgtc cggaattagc tggaggggcg actcaactgg atacgcggac 540
tccgtcaagg gcagattcac tatctctcgg gacaacgcca agaacaccgt ggacttgcaa 600
atgaattccc tgaagccgga ggacactgcc atctactact gtgctgcggc agcaggatct 660
gcctggtacg gcacccttta tgaatacgat tactggggac agggaaccca ggtcacggtg 720
tcgtcccacc atcaccacca ccatcatcac catcac 756
<210> 48
<211> 528
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 3
<400> 48
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Gly
260 265 270
Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val
275 280 285
Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala
290 295 300
Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln
305 310 315 320
Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Ala Pro Gly Thr
325 330 335
Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr
340 345 350
Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu
355 360 365
Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val
370 375 380
Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Gln Val Lys Leu Glu Glu
385 390 395 400
Ser Gly Gly Gly Ser Val Gln Thr Gly Gly Ser Leu Arg Leu Thr Cys
405 410 415
Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr Gly Met Gly Trp Phe Arg
420 425 430
Gln Ala Pro Gly Lys Glu Arg Glu Phe Val Ser Gly Ile Ser Trp Arg
435 440 445
Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile
450 455 460
Ser Arg Asp Asn Ala Lys Asn Thr Val Asp Leu Gln Met Asn Ser Leu
465 470 475 480
Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys Ala Ala Ala Ala Gly Ser
485 490 495
Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp Tyr Trp Gly Gln Gly Thr
500 505 510
Gln Val Thr Val Ser Ser His His His His His His His His His His
515 520 525
<210> 49
<211> 1584
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 3
<400> 49
gaagtccaac tggtggaatc gggcggtgga ctcgtgcagg ccggaggttc cctgcgcctt 60
tcctgtgcgg cttcgggaag aaccttctcc tcgtatgcca tgggatggtt ccgccaagcc 120
cctggaaaag agcgggaatt cgtcgtggcg atcaattgga gctccggttc gacttactac 180
gccgactccg tgaagggccg gttcaccatt agccgggaca atgctaagaa caccatgtat 240
ctgcagatga actcactgaa acctgaggac acggcggtgt actactgcgc cgctgggtac 300
cagatcaatt ccggaaacta caacttcaag gactacgagt acgactactg gggtcagggc 360
acccaggtca ccgtgtccag cggaggaggt ggaagcggag gcggttccga agtgcagctc 420
gtcgagtccg ggggtggact ggtccaaccg ggcggatcac tgaagctgag ctgcgcagcc 480
tccggattca ccttcaacaa gtacgccatg aactgggtca gacaggcacc cgggaaggga 540
ctggaatggg tggcccggat caggtccaag tacaacaact acgccaccta ctacgcggac 600
agcgtgaagg ataggttcac catctcccgg gacgacagca agaacactgc ctacctccaa 660
atgaacaacc tcaagaccga agatactgcg gtgtattact gcgtgcgcca cgggaacttc 720
ggaaacagct acatcagcta ctgggcctac tggggccagg gcactctggt gaccgtgagc 780
tcaggaggag gcggctccgg aggcggaggc tcagggggag gaggttcgca gaccgtggtg 840
acccaggaac cctcactgac cgtgtcccca ggaggaaccg tgacccttac ctgtggctcc 900
tcgaccggtg ccgtgacgtc cgggaactac cccaactggg tccagcaaaa gccgggacaa 960
gcccctcggg gactgatcgg ggggactaag ttcctggccc ctggcactcc tgcccgcttc 1020
agcggcagcc tcctgggagg aaaagcggcc ctgacactct cgggggtgca gcctgaagat 1080
gaggccgaat actactgcgt gctgtggtac tccaatcgct gggtgtttgg agggggcacc 1140
aagctgaccg tgctgggagg aggaggaagc ggcggaggtt cccaggtcaa gctggaggaa 1200
tcgggtggag gctcagtgca gacaggaggt agcctccggc tcacttgcgc cgcttccgga 1260
aggacttccc ggagctacgg gatgggctgg tttcggcaag cccccggaaa ggagagagaa 1320
ttcgtgtccg gaattagctg gaggggcgac tcaactggat acgcggactc cgtcaagggc 1380
agattcacta tctctcggga caacgccaag aacaccgtgg acttgcaaat gaattccctg 1440
aagccggagg acactgccat ctactactgt gctgcggcag caggatctgc ctggtacggc 1500
accctttatg aatacgatta ctggggacag ggaacccagg tcacggtgtc gtcccaccat 1560
caccaccacc atcatcacca tcac 1584
<210> 50
<211> 523
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 4
<400> 50
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly Gln
260 265 270
Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr
275 280 285
Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly Asn
290 295 300
Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly Leu
305 310 315 320
Ile Gly Gly Thr Lys Phe Leu Ala Pro Gly Thr Pro Ala Arg Phe Ser
325 330 335
Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln
340 345 350
Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser Asn Arg
355 360 365
Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly
370 375 380
Ser Gly Gly Gly Ser Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser
385 390 395 400
Val Gln Thr Gly Gly Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg
405 410 415
Thr Ser Arg Ser Tyr Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys
420 425 430
Glu Arg Glu Phe Val Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly
435 440 445
Tyr Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala
450 455 460
Lys Asn Thr Val Asp Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr
465 470 475 480
Ala Ile Tyr Tyr Cys Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr
485 490 495
Leu Tyr Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser
500 505 510
Ser His His His His His His His His His His
515 520
<210> 51
<211> 1569
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Prodent 4 - репрезентативная нуклеиновая кислота
<400> 51
gaagtccaac tggtggaatc gggcggtgga ctcgtgcagg ccggaggttc cctgcgcctt 60
tcctgtgcgg cttcgggaag aaccttctcc tcgtatgcca tgggatggtt ccgccaagcc 120
cctggaaaag agcgggaatt cgtcgtggcg atcaattgga gctccggttc gacttactac 180
gccgactccg tgaagggccg gttcaccatt agccgggaca atgctaagaa caccatgtat 240
ctgcagatga actcactgaa acctgaggac acggcggtgt actactgcgc cgctgggtac 300
cagatcaatt ccggaaacta caacttcaag gactacgagt acgactactg gggtcagggc 360
acccaggtca ccgtgtccag cggaggaggt ggaagcggag gcggttccga agtgcagctc 420
gtcgagtccg ggggtggact ggtccaaccg ggcggatcac tgaagctgag ctgcgcagcc 480
tccggattca ccttcaacaa gtacgccatg aactgggtca gacaggcacc cgggaaggga 540
ctggaatggg tggcccggat caggtccaag tacaacaact acgccaccta ctacgcggac 600
agcgtgaagg ataggttcac catctcccgg gacgacagca agaacactgc ctacctccaa 660
atgaacaacc tcaagaccga agatactgcg gtgtattact gcgtgcgcca cgggaacttc 720
ggaaacagct acatcagcta ctgggcctac tggggccagg gcactctggt gaccgtgtcc 780
tccgactaca aggacgatga cgataagggc ggccagaccg tggtgaccca ggaaccctca 840
ctgaccgtgt ccccaggagg aaccgtgacc cttacctgtg gctcctcgac cggtgccgtg 900
acgtccggga actaccccaa ctgggtccag caaaagccgg gacaagcccc tcggggactg 960
atcgggggga ctaagttcct ggcccctggc actcctgccc gcttcagcgg cagcctcctg 1020
ggaggaaaag cggccctgac actctcgggg gtgcagcctg aagatgaggc cgaatactac 1080
tgcgtgctgt ggtactccaa tcgctgggtg tttggagggg gcaccaagct gaccgtgctg 1140
ggaggaggag gaagcggcgg aggttcccag gtcaagctgg aggaatcggg tggaggctca 1200
gtgcagacag gaggtagcct ccggctcact tgcgccgctt ccggaaggac ttcccggagc 1260
tacgggatgg gctggtttcg gcaagccccc ggaaaggaga gagaattcgt gtccggaatt 1320
agctggaggg gcgactcaac tggatacgcg gactccgtca agggcagatt cactatctct 1380
cgggacaacg ccaagaacac cgtggacttg caaatgaatt ccctgaagcc ggaggacact 1440
gccatctact actgtgctgc ggcagcagga tctgcctggt acggcaccct ttatgaatac 1500
gattactggg gacagggaac ccaggtcacg gtgtcgtccc accatcacca ccaccatcat 1560
caccatcac 1569
<210> 52
<211> 786
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 5
<400> 52
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
260 265 270
Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
275 280 285
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
290 295 300
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
305 310 315 320
Gln Ala Pro Arg Gly Leu Ile Gly Asp Tyr Lys Asp Asp Asp Asp Lys
325 330 335
Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala
340 345 350
Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys
355 360 365
Val Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu
370 375 380
Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu
385 390 395 400
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu
405 410 415
Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp
420 425 430
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg
435 440 445
Ser Lys Tyr Asp Tyr Lys Asp Asp Asp Asp Lys Ala Asp Ser Val Lys
450 455 460
Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu
465 470 475 480
Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val
485 490 495
Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp
500 505 510
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr
515 520 525
Lys Asp Asp Asp Asp Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln
530 535 540
Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys
545 550 555 560
Gly Ser Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val
565 570 575
Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys
580 585 590
Phe Leu Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly
595 600 605
Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala
610 615 620
Glu Tyr Tyr Cys Val Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly
625 630 635 640
Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser
645 650 655
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
660 665 670
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
675 680 685
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
690 695 700
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
705 710 715 720
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
725 730 735
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
740 745 750
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
755 760 765
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser His His His His
770 775 780
His His
785
<210> 53
<211> 2358
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 5
<400> 53
gaagtgcagc tcgttgagtc aggcgggggt ctcgttcagg cgggtggtag tctccgcttg 60
agctgcgcag ctagcggccg aaccttctca tcttacgcaa tgggttggtt tagacaggcc 120
cctggaaagg aaagagaatt cgtggttgca attaactgga gcagcggctc aacttactat 180
gccgattcag tgaagggcag gttcaccata agccgagaca atgccaaaaa caccatgtac 240
cttcaaatga atagcctcaa acctgaagat accgccgttt actactgtgc agctggctat 300
caaataaact cagggaatta taattttaag gactatgagt acgattactg gggtcaaggc 360
acccaagtaa ctgtaagttc cggtggggga ggcagtggtg gagggagcga agtacagttg 420
gtcgagtctg gcggggggtt ggttcaacca ggtggttctc ttaaacttag ttgcgcggca 480
tccggtttca ctttcaacaa atatgcaatg aattgggtta ggcaagcccc cgggaagggc 540
ctcgaatggg tagctaggat tagatcaaaa tacaacaact atgctactta ttacgcggac 600
agtgtaaagg acaggtttac catctcccgc gatgactcta aaaacactgc gtatctgcaa 660
atgaataacc ttaagaccga agatacggcc gtctactatt gtgtccggca tggtaatttt 720
ggcaactcat acataagcta ttgggcatat tggggccaag gtactctggt taccgtaagc 780
agcggaggag gcggcgacta caaagacgat gacgataaag gaggtggaag tcagacggtg 840
gtgacacagg agccttccct gacggtatcc ccgggaggta ctgttactct tacttgtgga 900
tcaagcacag gggcagtaac ctctggcaac tacccaaact gggtacaaca gaagccaggt 960
caggcaccgc gaggcttgat aggagattac aaagacgacg acgacaaggg cactccagca 1020
agattttcag ggagcctgct cggcggtaaa gcagcgctga ccctgagcgg agtccaaccc 1080
gaagatgaag cggaatatta ctgtgtcttg tggtattcta atcggtgggt attcggtggt 1140
ggaaccaagc ttaccgtgct gggtggcggt ggtagcggtg gcgggagtga ggttcagctt 1200
gttgaatcag ggggaggtct ggtacagcca ggcggaagtt tgaaactgag ttgtgcagct 1260
tctggattta cgttcaacaa atacgccatg aattgggtga gacaggcacc gggcaagggg 1320
cttgaatggg tcgcaaggat ccggtccaag tacgactaca aggacgatga cgataaggct 1380
gactctgtaa aagaccgatt tacaatatcc agagacgatt caaaaaacac tgcgtatctc 1440
cagatgaaca atttgaaaac agaggatact gcggtttact attgtgtgag acacggcaac 1500
ttcggcaaca gctacatcag ctattgggcc tattggggac agggcactct cgtaacggtt 1560
tcatccgggg gaggaggaga ctacaaggac gatgacgata agggcggagg ctctcagacg 1620
gtcgtaactc aggagccatc tctcactgtt agcccgggcg gaactgttac tctcacctgt 1680
gggagcagta ctggggcggt tacttccggc aactacccta actgggttca acagaagcca 1740
ggtcaggcac caagaggtct gataggcgga actaaattcc tcgcccctgg tacccctgca 1800
cgattcagcg gatccctttt gggcggcaaa gcggctctta cactttctgg agtccaaccg 1860
gaagatgagg cggaatacta ttgtgtactt tggtatagta atcgctgggt attcggcggc 1920
ggcaccaaac tcactgtcct tggaggagga ggaagcggcg gaggttccca ggtcaagctg 1980
gaggaatcgg gtggaggctc agtgcagaca ggaggtagcc tccggctcac ttgcgccgct 2040
tccggaagga cttcccggag ctacgggatg ggctggtttc ggcaagcccc cggaaaggag 2100
agagaattcg tgtccggaat tagctggagg ggcgactcaa ctggatacgc ggactccgtc 2160
aagggcagat tcactatctc tcgggacaac gccaagaaca ccgtggactt gcaaatgaat 2220
tccctgaagc cggaggacac tgccatctac tactgtgctg cggcagcagg atctgcctgg 2280
tacggcaccc tttatgaata cgattactgg ggacagggaa cccaggtcac ggtctcgagt 2340
caccaccacc atcaccac 2358
<210> 54
<211> 393
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 6
<400> 54
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
260 265 270
Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
275 280 285
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
290 295 300
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
305 310 315 320
Gln Ala Pro Arg Gly Leu Ile Gly Asp Tyr Lys Asp Asp Asp Asp Lys
325 330 335
Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala
340 345 350
Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys
355 360 365
Val Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu
370 375 380
Thr Val Leu His His His His His His
385 390
<210> 55
<211> 1179
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 6
<400> 55
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatg aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
agtgtaaagg accgctttac tatcagtaga gatgacagta agaacacggc ttatttgcaa 660
atgaacaact tgaagacaga agatacggcg gtctattatt gtgtacgaca cggtaatttt 720
gggaattcat atataagcta ttgggcatac tggggtcaag gaacccttgt tacggtgagc 780
agcgggggcg gtggtgacta taaggacgac gatgacaaag gcggcggctc ccagactgtg 840
gtaacacagg aaccatcttt gacagtaagt cctggaggta cggtcacgct cacttgtggg 900
tcctcaaccg gggctgtaac gtcaggcaat taccctaact gggtccaaca gaagcctgga 960
caagctccca ggggtctgat aggcgattac aaagatgatg atgataaggg cactccagcg 1020
cgctttagcg gttctcttct gggtggaaaa gcagccctca ctctgagtgg agtacaaccc 1080
gaggatgagg cggaatatta ttgcgtgctc tggtattcaa accgctgggt cttcggtggc 1140
ggtacgaaac ttactgtact gcatcatcat catcaccac 1179
<210> 56
<211> 390
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 7
<400> 56
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Arg Ile Arg Ser Lys Tyr Asp Tyr Lys Asp Asp Asp Asp Lys Ala
50 55 60
Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn
65 70 75 80
Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser Tyr
100 105 110
Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly Gly Ser Gln Thr
130 135 140
Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val
145 150 155 160
Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr
165 170 175
Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile
180 185 190
Gly Gly Thr Lys Phe Leu Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly
195 200 205
Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro
210 215 220
Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser Asn Arg Trp
225 230 235 240
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser
245 250 255
Gly Gly Gly Ser Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val
260 265 270
Gln Thr Gly Gly Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr
275 280 285
Ser Arg Ser Tyr Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu
290 295 300
Arg Glu Phe Val Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr
305 310 315 320
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys
325 330 335
Asn Thr Val Asp Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala
340 345 350
Ile Tyr Tyr Cys Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu
355 360 365
Tyr Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser
370 375 380
His His His His His His
385 390
<210> 57
<211> 1170
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота
Prodent 7
<400> 57
gaggttcagc ttgttgaatc agggggaggt ctggtacagc caggcggaag tttgaaactg 60
agttgtgcag cttctggatt tacgttcaac aaatacgcca tgaattgggt gagacaggca 120
ccgggcaagg ggcttgaatg ggtcgcaagg atccggtcca agtacgacta caaggacgat 180
gacgataagg ctgactctgt aaaagaccga tttacaatat ccagagacga ttcaaaaaac 240
actgcgtatc tccagatgaa caatttgaaa acagaggata ctgcggttta ctattgtgtg 300
agacacggca acttcggcaa cagctacatc agctattggg cctattgggg acagggcact 360
ctcgtaacgg tttcatccgg gggaggagga gactacaagg acgatgacga taagggcgga 420
ggctctcaga cggtcgtaac tcaggagcca tctctcactg ttagcccggg cggaactgtt 480
actctcacct gtgggagcag tactggggcg gttacttccg gcaactaccc taactgggtt 540
caacagaagc caggtcaggc accaagaggt ctgataggcg gaactaaatt cctcgcccct 600
ggtacccctg cacgattcag cggatccctt ttgggcggca aagcggctct tacactttct 660
ggagtccaac cggaagatga ggcggaatac tattgtgtac tttggtatag taatcgctgg 720
gtattcggcg gcggcaccaa actcactgtc cttggaggag gaggaagcgg cggaggttcc 780
caggtcaagc tggaggaatc gggtggaggc tcagtgcaga caggaggtag cctccggctc 840
acttgcgccg cttccggaag gacttcccgg agctacggga tgggctggtt tcggcaagcc 900
cccggaaagg agagagaatt cgtgtccgga attagctgga ggggcgactc aactggatac 960
gcggactccg tcaagggcag attcactatc tctcgggaca acgccaagaa caccgtggac 1020
ttgcaaatga attccctgaa gccggaggac actgccatct actactgtgc tgcggcagca 1080
ggatctgcct ggtacggcac cctttatgaa tacgattact ggggacaggg aacccaggtc 1140
acggtctcga gtcaccacca ccatcaccac 1170
<210> 58
<211> 392
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 8
<400> 58
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
260 265 270
Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
275 280 285
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
290 295 300
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
305 310 315 320
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Ala Pro Gly
325 330 335
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
340 345 350
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val
355 360 365
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
370 375 380
Val Leu His His His His His His
385 390
<210> 59
<211> 1176
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 8
<400> 59
gaagtccaac tggtggaatc gggcggtgga ctcgtgcagg ccggaggttc cctgcgcctt 60
tcctgtgcgg cttcgggaag aaccttctcc tcgtatgcca tgggatggtt ccgccaagcc 120
cctggaaaag agcgggaatt cgtcgtggcg atcaattgga gttccggttc gacttactac 180
gccgactccg tgaagggccg gttcaccatt agccgggaca atgctaagaa caccatgtat 240
ctgcagatga actcactgaa acctgaggac acggcggtgt actactgcgc cgctgggtac 300
cagatcaatt ccggaaacta caacttcaag gactacgagt acgactactg gggtcagggc 360
acccaggtca ccgtgtccag cggaggaggt ggaagcggag gcggttccga agtgcagctc 420
gtcgagtccg ggggtggact ggtccaaccg ggcggatcac tgaagctgag ctgcgcagcc 480
tccggattca ccttcaacaa gtacgccatg aactgggtca gacaggcacc cgggaaggga 540
ctggaatggg tggcccggat caggtccaag tacaacaact acgccaccta ctacgcggac 600
agcgtgaagg ataggttcac catctcccgg gacgacagca agaacactgc ctacctccaa 660
atgaacaacc tcaagaccga agatactgcg gtgtattact gcgtgcgcca cgggaacttc 720
ggaaacagct acatcagcta ctgggcctac tggggccagg gcactctggt gaccgtgagt 780
tcaggaggag gcggcgacta caaggacgat gacgataagg gaggaggttc gcagaccgtg 840
gtgacccagg aaccctcact gaccgtgtcc ccaggaggaa ccgtgaccct tacctgtggc 900
tcctcgaccg gtgccgtgac gtccgggaac taccccaact gggtccagca aaagccggga 960
caagcccctc ggggactgat cgggggaact aaattcctcg cccctggcac tcctgcccgc 1020
ttcagcggca gcctcctggg aggaaaagcg gccctgacac tctcgggggt gcagcctgaa 1080
gatgaggccg aatactactg cgtgctgtgg tactccaatc gctgggtgtt tggagggggc 1140
accaagctta ccgtgctgca ccaccaccat caccac 1176
<210> 60
<211> 390
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 8MS
<400> 60
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Leu Ser Gly Arg Ser Asp Asn His
260 265 270
Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser
275 280 285
Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val
290 295 300
Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala
305 310 315 320
Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Ala Pro Gly Thr Pro
325 330 335
Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu
340 345 350
Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp
355 360 365
Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
370 375 380
His His His His His His
385 390
<210> 61
<211> 1170
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 8MS
<400> 61
gaggttcaac tggtggaatc aggaggcggt ttggttcagg caggagggtc attgcgattg 60
tcatgtgcgg cgtccgggcg gactttcagt tcttacgcga tgggttggtt taggcaagcg 120
ccgggcaaag agagggagtt tgtagtggca attaactgga gttctggatc aacttattac 180
gctgattccg tcaagggacg ctttacgatt agccgggata atgcaaaaaa cactatgtac 240
cttcaaatga actctctgaa accggaggac accgccgtct actattgcgc ggctggttat 300
cagatcaact ctgggaatta taatttcaaa gactatgaat atgattattg gggtcaaggc 360
acgcaagtta cagttagcag cggaggcggg gggtcaggtg gtgggagtga agtgcaattg 420
gtcgaatctg gaggcggcct ggtccaaccc gggggctcat tgaagttgtc ctgtgccgca 480
agtggattta cgttcaataa gtacgctatg aactgggtga gacaagcccc tggaaaggga 540
ctggaatggg tggcgcgcat aaggtcaaaa tacaataact acgcaaccta ctatgccgat 600
agtgtaaaag acagattcac catttctcga gacgattcta aaaataccgc gtatcttcaa 660
atgaataatt tgaagaccga ggatacagca gtgtattact gtgttagaca tggcaacttt 720
gggaactctt acatatctta ttgggcgtat tggggacaag ggaccctggt gacagtaagt 780
agcggaggcg gactgtccgg gcgaagcgac aaccatgggg gcagtcagac agtggtaacg 840
caagaaccga gtctcactgt atcaccagga ggtacagtga ccctcacatg cggatcctcc 900
acgggggcag tcacatctgg taattatcca aattgggttc agcagaagcc aggacaagct 960
ccacgaggat tgattggcgg gacaaaattt ctggccccag gaacgccggc caggtttagt 1020
ggtagcctgc ttggaggtaa agcggcgctg acgctttccg gcgtacaacc tgaagacgag 1080
gcagagtact actgtgtact ctggtactct aacaggtggg tgttcggagg tggaaccaaa 1140
ctcacggttt tgcatcacca tcatcatcat 1170
<210> 62
<211> 400
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 8ML
<400> 62
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Ser Gly Gly Ser Gly Leu Ser Gly
260 265 270
Arg Ser Asp Asn His Gly Ser Gly Gly Ser Gly Gly Ser Gln Thr Val
275 280 285
Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr
290 295 300
Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr Pro
305 310 315 320
Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile Gly
325 330 335
Gly Thr Lys Phe Leu Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser
340 345 350
Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu
355 360 365
Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser Asn Arg Trp Val
370 375 380
Phe Gly Gly Gly Thr Lys Leu Thr Val Leu His His His His His His
385 390 395 400
<210> 63
<211> 1200
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 8ML
<400> 63
gaggttcaac tggtggaatc aggaggcggt ttggttcagg caggagggtc attgcgattg 60
tcatgtgcgg cgtccgggcg gactttcagt tcttacgcga tgggttggtt taggcaagcg 120
ccgggcaaag agagggagtt tgtagtggca attaactgga gttctggatc aacttattac 180
gctgattccg tcaagggacg ctttacgatt agccgggata atgcaaaaaa cactatgtac 240
cttcaaatga actctctgaa accggaggac accgccgtct actattgcgc ggctggttat 300
cagatcaact ctgggaatta taatttcaaa gactatgaat atgattattg gggtcaaggc 360
acgcaagtta cagttagcag cggaggcggg gggtcaggtg gtgggagtga agtgcaattg 420
gtcgaatctg gaggcggcct ggtccaaccc gggggctcat tgaagttgtc ctgtgccgca 480
agtggattta cgttcaataa gtacgctatg aactgggtga gacaagcccc tggaaaggga 540
ctggaatggg tggcgcgcat aaggtcaaaa tacaataact acgcaaccta ctatgccgat 600
agtgtaaaag acagattcac catttctcga gacgattcta aaaataccgc gtatcttcaa 660
atgaataatt tgaagaccga ggatacagca gtgtattact gtgttagaca tggcaacttt 720
gggaactctt acatatctta ttgggcgtat tggggacaag ggaccctggt gacagtaagt 780
agcggaggcg ggagcggggg atctggactt agtggccggt cagataatca tggaagcggc 840
ggatcagggg gcagtcagac agtggtaacg caagaaccga gtctcactgt atcaccagga 900
ggtacagtga ccctcacatg cggatcctcc acgggggcag tcacatctgg taattatcca 960
aattgggttc agcagaagcc aggacaagct ccacgaggat tgattggcgg gacaaaattt 1020
ctggccccag gaacgccggc caggtttagt ggtagcctgc ttggaggtaa agcggcgctg 1080
acgctttccg gcgtacaacc tgaagacgag gcagagtact actgtgtact ctggtactct 1140
aacaggtggg tgttcggagg tggaaccaaa ctcacggttt tgcatcacca tcatcatcat 1200
<210> 64
<211> 390
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 9
<400> 64
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Ala Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn
275 280 285
Lys Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Ala Arg Ile Arg Ser Lys Tyr Asp Tyr Lys Asp Asp Asp Asp
305 310 315 320
Lys Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser
325 330 335
Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr
340 345 350
Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile
355 360 365
Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
370 375 380
His His His His His His
385 390
<210> 65
<211> 1170
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 9
<400> 65
caagtcaaac ttgaagaaag tggtggtgga tccgtgcaaa caggcggatc cctgcgcctg 60
acgtgtgcgg cgtcaggaag gacttctagg tcatacggta tgggttggtt caggcaagcc 120
cctgggaagg agagggagtt cgtttcaggc atcagctgga ggggagactc tactggctac 180
gcagacagcg tcaaaggcag atttacaatc agcagagaca atgcgaagaa cactgttgac 240
ctgcaaatga acagcttgaa accagaagat acagctatct actattgcgc tgccgcagcc 300
ggatcagcct ggtacggcac gctgtatgag tatgattatt ggggacaagg cacgcaggta 360
acagtcagct ctggcggtgg ggggagcggg ggtggaagtc aaaccgtcgt tactcaggaa 420
ccatcactga ctgtgtctcc tgggggcact gtaactctta cgtgtggttc atctacaggc 480
gctgtcacca gtggcaacta tcctaactgg gtccagcaga agcctggtca ggctcctcgg 540
gggcttattg gaggtacaaa gttccttgct ccgggcacac ccgcaaggtt tagcgggtca 600
ttgcttggag gcaaggctgc cctcactctt tccggcgtgc aaccagaaga tgaagccgaa 660
tattattgcg tgctgtggta ctccaatcga tgggtctttg gtggtgggac taagctgaca 720
gtccttgggg gcggcgggga ctataaagat gatgatgata aggggggtgg gtccgaggtg 780
cagcttgttg aatctggcgg gggccttgtg caacctgggg gttccctgaa gctcagctgt 840
gccgcttcag gtttcacatt caataagtac gccatgaact gggtgcggca ggccccaggt 900
aagggtcttg aatgggtcgc tagaatacgc agtaagtacg actacaaaga cgatgacgac 960
aaagcggact cagtaaaaga ccgctttacg ataagtcgcg acgattccaa gaacacggcg 1020
tatctccaaa tgaacaatct taaaacggag gacacagcag tctactactg tgtccgccac 1080
ggcaatttcg gtaatagtta catcagttat tgggcctact ggggccaggg tactctcgtg 1140
actgtctcat cacatcacca ccaccatcac 1170
<210> 66
<211> 393
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 10
<400> 66
Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly
20 25 30
Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly
35 40 45
Leu Ile Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Thr Pro Ala Arg
50 55 60
Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly
65 70 75 80
Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser
85 90 95
Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly
100 105 110
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly Gly Ser Glu Val
115 120 125
Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu
130 135 140
Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met
145 150 155 160
Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg
165 170 175
Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val
180 185 190
Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr
195 200 205
Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr
225 230 235 240
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
245 250 255
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
260 265 270
Gln Ala Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr
275 280 285
Phe Ser Ser Tyr Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu
290 295 300
Arg Glu Phe Val Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr
305 310 315 320
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys
325 330 335
Asn Thr Met Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn
355 360 365
Phe Lys Asp Tyr Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr
370 375 380
Val Ser Ser His His His His His His
385 390
<210> 67
<211> 1179
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 10
<400> 67
cagacggtgg tcactcaaga accttccttg actgtatctc cgggcgggac agtcactctt 60
acgtgtggat caagcactgg cgcggttact agtggcaact accctaattg ggtacagcag 120
aaaccgggcc aagcgccgag aggtctgatt ggggattata aggatgacga cgacaagggt 180
acgccagcac gcttttctgg gtccttgctc ggtggaaagg cagccctgac tctcagtggc 240
gttcagccgg aggacgaggc tgaatattat tgcgtcttgt ggtactccaa caggtgggtc 300
ttcgggggcg gtacaaagtt gaccgtcctc gggggcggag gcgactataa agacgatgat 360
gacaaaggtg gtggttcaga agtgcagctt gtggagagcg ggggtggtct ggtgcaaccg 420
ggaggctctc tcaagctcag ttgcgcagca tctgggttta ctttcaacaa atacgcgatg 480
aactgggtta ggcaggctcc gggtaagggg ctcgaatggg ttgccagaat ccggtctaag 540
tataacaact atgctactta ttacgctgac agtgtaaagg atcgctttac tatctcccga 600
gatgattcca agaacacggc gtatttgcag atgaacaatt tgaagacgga ggataccgct 660
gtttactatt gtgttcggca tgggaatttc ggaaactcct atataagtta ctgggcatac 720
tggggtcaag gcacactcgt gactgtaagt tctgggggcg gtggaagcgg agggggatca 780
gaggtgcaac tcgttgagag cggcgggggc ttggtacagg caggggggtc actcaggctc 840
tcttgtgcgg cctcagggag aactttcagt tcatatgcga tgggttggtt taggcaggct 900
cctggtaaag aaagagaatt tgtcgtggca atcaattgga gttccggttc cacgtattat 960
gcggatagcg ttaagggcag attcacgata agtagggata acgcgaaaaa caccatgtac 1020
ctgcaaatga attctcttaa accggaggac acagcggttt attactgtgc ggccggatac 1080
caaatcaaca gcggtaatta taacttcaaa gactacgaat atgattactg ggggcagggg 1140
actcaggtaa ctgttagttc acaccatcac catcatcat 1179
<210> 68
<211> 389
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 11
<400> 68
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Ala Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn
275 280 285
Lys Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr
305 310 315 320
Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys
325 330 335
Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser
355 360 365
Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser His
370 375 380
His His His His His
385
<210> 69
<211> 1167
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 11
<400> 69
caggtcaaac tcgaagagtc tggaggagga agtgtgcaga cgggcggtag cctgcgcctt 60
acttgcgcgg cttcaggccg aacatccaga tcatacggaa tgggatggtt tagacaagcg 120
cctggtaaag agcgggagtt cgtttcagga atatcatggc ggggagattc aacagggtat 180
gccgacagcg tcaaaggacg cttcactatt agcagagaca atgcaaaaaa tactgtagac 240
cttcagatga attccctgaa gccggaggat acggctattt actattgcgc ggctgctgcc 300
gggtcagcct ggtacgggac attgtatgaa tatgattatt gggggcaagg aacccaagtt 360
acagttagca gtgggggtgg gggcagtgga ggtggttccc aaacggtggt gactcaagaa 420
ccatccctga ctgttagtcc gggagggacc gtaactctca cttgtggttc atccacagga 480
gccgtgacgt ccggtaacta tccgaactgg gtacaacaaa agccgggcca agcaccccga 540
ggtctgattg gtgggacaaa gtttctggcc cctgggacac ccgctcggtt ctcagggtcc 600
ctcctgggcg gaaaggccgc gcttacgttg tccggcgtgc agcctgaaga tgaggcagaa 660
tactattgtg tgctttggta ctctaatagg tgggtttttg gtgggggtac caagttgact 720
gtcctgggtg gagggggaga ctataaagac gatgacgaca aaggtggagg aagtgaggtg 780
caactcgtag aaagtggggg cggacttgtt caaccagggg gcagcctgaa gctgtcttgt 840
gcagcaagtg ggttcacctt taataaatac gcaatgaatt gggtgagaca ggccccaggc 900
aagggccttg agtgggtcgc gcgaatacga agcaagtaca ataactatgc tacatactat 960
gcggactctg ttaaggaccg attcaccatc agtcgagatg actctaaaaa tacggcgtac 1020
ctccaaatga ataacctcaa aacggaagac acggcggtgt attactgtgt taggcatggc 1080
aactttggta atagctacat tagctactgg gcttactggg gccaaggcac cttggttact 1140
gttagttccc atcaccatca tcatcac 1167
<210> 70
<211> 393
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 12
<400> 70
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Arg Ile Arg Ser Lys Tyr Asp Tyr Lys Asp Asp Asp Asp Lys Ala
50 55 60
Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn
65 70 75 80
Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser Tyr
100 105 110
Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly Gly Ser Gln Thr
130 135 140
Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val
145 150 155 160
Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr
165 170 175
Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile
180 185 190
Gly Gly Thr Lys Phe Leu Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly
195 200 205
Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro
210 215 220
Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser Asn Arg Trp
225 230 235 240
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser
245 250 255
Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val
260 265 270
Gln Ala Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr
275 280 285
Phe Ser Ser Tyr Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu
290 295 300
Arg Glu Phe Val Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr
305 310 315 320
Ala Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys
325 330 335
Asn Thr Met Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn
355 360 365
Phe Lys Asp Tyr Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr
370 375 380
Val Ser Ser His His His His His His
385 390
<210> 71
<211> 1179
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 12
<400> 71
gaagtgcagc tggtagagag cggtggtggg ttggtgcagc ctggtggtag cttgaaattg 60
tcatgtgcgg catctgggtt tacttttaat aagtacgcca tgaactgggt gcgccaagcg 120
cctggtaaag gtcttgagtg ggtcgccaga atacggtcta aatatgatta caaagatgac 180
gacgacaagg ccgacagcgt gaaagaccgc tttacaataa gtagggatga cagtaaaaac 240
accgcttatt tgcaaatgaa taaccttaag acggaggaca ctgctgtata ttattgtgta 300
aggcatggca acttcgggaa ttcatacatt tcatattggg catactgggg tcaaggcacg 360
ctcgtaacgg tcagttccgg cggcggggga gactataagg atgatgacga caagggcgga 420
ggttcccaga cagtcgtcac gcaagaaccc agccttacag tttctcctgg cggtacagta 480
acattgacct gtggcagcag cactggtgcg gtgacatctg gtaattaccc aaactgggtt 540
cagcaaaagc ctggccaagc cccaagagga ctgattggtg gaaccaagtt cctggcccct 600
ggcacaccgg cgagattttc cgggtcattg ttggggggta aagctgcgct gactttgtct 660
ggtgttcaac ctgaagatga agccgaatat tattgtgtct tgtggtacag taatagatgg 720
gtgtttggtg gggggactaa gctaacggtc cttggcggag ggggatctgg tggaggatct 780
gaggtgcaac ttgttgagag cggcggagga cttgttcagg ccggaggctc acttcgcctt 840
agctgtgctg ctagtggaag aacgttcagt tcttacgcta tgggatggtt tagacaagct 900
ccaggaaaag aaagggagtt cgtcgtggct ataaattggt cttccgggag tacatattac 960
gccgacagcg tcaaagggag atttacgatc tctcgggaca acgctaaaaa cacgatgtac 1020
ctgcaaatga atagcttgaa acccgaggat accgctgtgt actactgcgc cgccgggtat 1080
cagatcaaca gtggtaacta taacttcaag gactacgagt acgactactg gggccaggga 1140
actcaggtca ccgtgagttc tcatcaccac catcaccac 1179
<210> 72
<211> 390
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 14
<400> 72
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
130 135 140
Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe
145 150 155 160
Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys
165 170 175
Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala
180 185 190
Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp
195 200 205
Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu
210 215 220
Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser
225 230 235 240
Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val
245 250 255
Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
260 265 270
Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro
275 280 285
Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr
290 295 300
Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro
305 310 315 320
Arg Gly Leu Ile Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Thr Pro
325 330 335
Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu
340 345 350
Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp
355 360 365
Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu
370 375 380
His His His His His His
385 390
<210> 73
<211> 1170
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 14
<400> 73
caagtcaagt tggaagagtc cggtggtggt tcagtacaga ccggcgggtc tctccgactt 60
acgtgtgccg caagcggacg aacatccagg tcctatggca tgggttggtt tcgccaggct 120
ccagggaagg aacgcgagtt cgtcagtggg attagttggc gaggtgactc cactgggtac 180
gcagattcag ttaaaggccg cttcaccatc tcacgagaca atgctaagaa tacagttgat 240
ctccaaatga atagtctcaa acccgaagat acagctatct attattgtgc ggctgccgca 300
gggtcagcct ggtatggaac tttgtatgaa tacgactatt gggggcaggg gacgcaagtc 360
acagtttcct ccggtggagg tggatcaggg ggaggctccg aggtgcaact cgtagagtcc 420
ggtggcggac tcgtccagcc tggcggatca ctgaagttgt catgcgcggc tagtggtttc 480
actttcaata aatacgccat gaattgggta cgccaagcgc ctgggaaggg acttgaatgg 540
gtggcgagaa tccgctccaa atataataac tacgctacgt attatgcaga ctctgtcaag 600
gatcggttca caatatccag ggacgacagt aaaaacaccg cttaccttca gatgaacaat 660
ttgaagacgg aagataccgc ggtgtattat tgtgtacgcc atggtaattt tggtaactcc 720
tatatttctt actgggccta ctggggacaa ggaactctgg tcactgtgtc atctggtggg 780
ggaggcgact acaaagatga tgatgacaaa ggaggaggaa gccaaacagt agtaacccag 840
gaacctagtc ttactgtcag tcctggtggt acggtaacct tgacgtgtgg ttccagcacg 900
ggagcagtga cttcaggcaa ctatcctaac tgggtacaac agaaacccgg acaagcacca 960
cgaggattga ttggtgacta caaagacgac gacgataaag gcacccccgc taggttctct 1020
ggtagtcttt tgggaggcaa ggcagcgttg acactctcag gggtgcaacc cgaggatgag 1080
gcagaatatt actgtgtact gtggtactca aatagatggg tgtttggcgg gggcacaaaa 1140
cttactgtat tgcatcacca tcaccaccac 1170
<210> 74
<211> 384
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 15
<400> 74
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Gln Val Gln Leu Val Gln Ser Gly
130 135 140
Gly Gly Val Val Gln Pro Gly Arg Ser Leu Arg Leu Ser Cys Lys Ala
145 150 155 160
Ser Gly Tyr Thr Phe Thr Arg Tyr Thr Met His Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Ile Gly Tyr Ile Asn Pro Ser Arg Gly
180 185 190
Tyr Thr Asn Tyr Asn Gln Lys Val Lys Asp Arg Phe Thr Ile Ser Arg
195 200 205
Asp Asn Ser Lys Asn Thr Ala Phe Leu Gln Met Asp Ser Leu Arg Pro
210 215 220
Glu Asp Thr Gly Val Tyr Phe Cys Ala Arg Tyr Tyr Asp Asp His Tyr
225 230 235 240
Cys Leu Asp Tyr Trp Gly Gln Gly Thr Pro Val Thr Val Ser Ser Gly
245 250 255
Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly Gly Ser Asp
260 265 270
Ile Gln Met Thr Gln Ser Pro Ser Ser Leu Ser Ala Ser Val Gly Asp
275 280 285
Arg Val Thr Ile Thr Cys Ser Ala Ser Ser Ser Val Ser Tyr Met Asn
290 295 300
Trp Tyr Gln Gln Thr Pro Gly Lys Ala Pro Lys Arg Trp Ile Tyr Asp
305 310 315 320
Thr Ser Lys Leu Ala Ser Gly Val Pro Ser Arg Phe Ser Gly Ser Gly
325 330 335
Ser Gly Thr Asp Tyr Thr Phe Thr Ile Ser Ser Leu Gln Pro Glu Asp
340 345 350
Ile Ala Thr Tyr Tyr Cys Gln Gln Trp Ser Ser Asn Pro Phe Thr Phe
355 360 365
Gly Gln Gly Thr Lys Leu Gln Ile Thr Arg His His His His His His
370 375 380
<210> 75
<211> 1152
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 15
<400> 75
gaggttcagt tggtggagtc aggtgggggc ttggttcaag caggtggaag tctgcggctt 60
tcctgtgccg ctagtggtcg gaccttcagt tcatatgcta tgggatggtt ccggcaagcc 120
ccgggcaagg agcgcgagtt tgtcgtagcg attaattggt catcagggtc tacgtattac 180
gcggattccg ttaagggcag gttcacaata tcccgggaca acgccaagaa taccatgtat 240
cttcaaatga actcccttaa accagaggat actgctgttt attactgcgc ggctgggtat 300
caaataaaca gcgggaacta caacttcaaa gactatgagt acgactactg gggtcaggga 360
acccaagtca ctgtgagttc aggtggaggc ggaagcggag gcggttccca agtgcaactg 420
gttcagtccg gaggaggcgt ggttcagccc gggcgaagcc tcaggctgtc ttgtaaagca 480
tccggatata cattcaccag gtacaccatg cactgggtga gacaagcacc tggtaagggc 540
cttgagtgga tcggatacat aaacccaagt cgaggataca ccaattacaa tcaaaaggtc 600
aaagacaggt tcacgatctc acgagataat tcaaaaaaca ctgcgttcct gcaaatggat 660
agcctgcggc ctgaggatac gggtgtgtac ttctgtgcac gctactatga tgaccactac 720
tgtcttgatt actggggaca agggaccccg gtgacggtat cctccggggg aggcggcgac 780
tacaaagatg acgacgataa agggggaggc tccgacattc aaatgaccca atctcccagt 840
agtctgagtg cttccgtcgg tgaccgggtt acaataacgt gctcagcgtc ctcttctgtc 900
tcttacatga attggtacca gcaaaccccc ggcaaagccc ctaagagatg gatctatgat 960
acctccaaat tggcgtctgg cgtgccctcc cgattcagtg ggtctggatc aggtacggac 1020
tacaccttca caatttcctc attgcagcca gaagatatag caacctacta ctgtcaacaa 1080
tggtcctcaa atccctttac cttcgggcaa ggaaccaagc tccaaatcac gcggcaccac 1140
caccatcacc ac 1152
<210> 76
<211> 517
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 16
<400> 76
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
260 265 270
Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
275 280 285
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
290 295 300
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
305 310 315 320
Gln Ala Pro Arg Gly Leu Ile Gly Asp Tyr Lys Asp Asp Asp Asp Lys
325 330 335
Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala
340 345 350
Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys
355 360 365
Val Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu
370 375 380
Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu
385 390 395 400
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu
405 410 415
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp
420 425 430
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser
435 440 445
Gly Ser Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe
450 455 460
Thr Ile Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn
465 470 475 480
Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly
485 490 495
Ser Leu Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His
500 505 510
His His His His His
515
<210> 77
<211> 1551
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 16
<400> 77
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatc aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
caagtaaagg accgctttac tatcagtaga gatgacagta agaacacggc ttatttgcaa 660
atgaacaact tgaagacaga agatacggcg gtctattatt gtgtacgaca cgcaaatttt 720
gggaattcat atataagcta ttgggcatac tggggtcaag gaacccttgt tacggtgagc 780
agcgggggcg gtggtgacta taaggacgac gatgacaaag gcggcggctc ccagactgtg 840
gtaacacagg aaccatcttt gacagtaagt cctggaggta cggtcacgct cacttgtggg 900
tcctcaaccg gggctgtaac gtcaggcaat taccctaact gggtccaaca gaagcctgga 960
caagctccca ggggtctgat aggcgattac aaagatgatg atgataaggg cactccagcg 1020
cgctttagcg gatcccttct gggtggaaaa gcagccctca ctctgagtgg agtacaaccc 1080
gaggatgagg cggaatatta ttgcgtgctc tggtattcaa accgctgggt cttcggtggc 1140
ggtacgaaac ttactgtact ggggggaggc ggctcaggcg gcggatcaga agtgcagctt 1200
gttgaatctg gcggaggtct ggtccagcca ggtaacagct tgagactgtc ctgtgctgct 1260
agcggcttta ccttctctaa attcggtatg agttgggttc ggcaagcccc tggaaagggt 1320
ttggaatggg tatcaagcat tagtggttct gggcgagata cactctatgc cgaatcagtg 1380
aagggccgct ttaccattag tagggataac gctaaaacta ctctgtatct gcaaatgaat 1440
agtctgagac cagaagatac tgccgtttac tactgcacaa tagggggatc tctgagcgtt 1500
tcatctcaag gtacacttgt gactgttagc agtcatcatc atcatcacca c 1551
<210> 78
<211> 514
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 17
<400> 78
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Asn
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe
20 25 30
Gly Met Ser Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ser Ser Ile Ser Gly Ser Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Thr Ile Gly Gly Ser Leu Ser Val Ser Ser Gln Gly Thr Leu Val Thr
100 105 110
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu
115 120 125
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu
130 135 140
Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp
145 150 155 160
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg
165 170 175
Ser Lys Tyr Asp Tyr Lys Asp Asp Asp Asp Lys Ala Asp Ser Val Lys
180 185 190
Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu
195 200 205
Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val
210 215 220
Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp
225 230 235 240
Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr
245 250 255
Lys Asp Asp Asp Asp Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln
260 265 270
Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys
275 280 285
Ala Ser Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val
290 295 300
Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys
305 310 315 320
Phe Leu Val Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly
325 330 335
Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala
340 345 350
Glu Tyr Tyr Cys Thr Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly
355 360 365
Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser
370 375 380
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
385 390 395 400
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
405 410 415
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
420 425 430
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
435 440 445
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
450 455 460
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
465 470 475 480
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
485 490 495
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser His His His His
500 505 510
His His
<210> 79
<211> 1542
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 17
<400> 79
gaagtgcagc ttgttgaatc tggcggaggt ctggtccagc caggtaacag cttgagactg 60
tcctgtgctg ctagcggctt taccttctct aaattcggta tgagttgggt tcggcaagcc 120
cctggaaagg gtttggaatg ggtatcaagc attagtggtt ctgggcgaga tacactctat 180
gccgaatcag tgaagggccg ctttaccatt agtagggata acgctaaaac tactctgtat 240
ctgcaaatga atagtctgag accagaagat actgccgttt actactgcac aataggggga 300
tctctgagcg tttcatctca aggtacactt gtgactgtta gcagtggggg aggcggctca 360
ggcggcggat cagaggttca gcttgttgaa tcagggggag gtctggtaca gccaggcgga 420
agtttgaaac tgagttgtgc agcttctgga tttacgttca acaaatacgc catgaattgg 480
gtgagacagg caccgggcaa ggggcttgaa tgggtcgcaa ggatccggtc caagtacgac 540
tacaaggacg atgacgataa ggctgactct gtaaaagacc gatttacaat atccagagac 600
gattcaaaaa acactgcgta tctccagatg aacaatttga aaacagagga tactgcggtt 660
tactattgtg tgagacacgg caacttcggc aacagctaca tcagctattg ggcctattgg 720
ggacagggca ctctcgtaac ggtttcatcc gggggaggag gagactacaa ggacgatgac 780
gataagggcg gaggctctca gacggtcgta actcaggagc catctctcac tgttagcccg 840
ggcggaactg ttactctcac ctgtgctagc agtactgggg cggttacttc cggcaactac 900
cctaactggg ttcaacagaa gccaggtcag gcaccaagag gtctgatagg cggaactaaa 960
ttcctcgtcc ctggtacccc tgcacgattc agcggttccc ttttgggcgg caaagcggct 1020
cttacacttt ctggagtcca accggaagat gaggcggaat actattgtac cctttggtat 1080
agtaatcgct gggtattcgg cggcggcacc aaactcactg tccttggagg aggaggaagc 1140
ggcggaggtt cccaggtcaa gctggaggaa tcgggtggag gctcagtgca gacaggaggt 1200
agcctccggc tcacttgcgc cgcttccgga aggacttccc ggagctacgg gatgggctgg 1260
tttcggcaag cccccggaaa ggagagagaa ttcgtgtccg gaattagctg gaggggcgac 1320
tcaactggat acgcggactc cgtcaagggc agattcacta tctctcggga caacgccaag 1380
aacaccgtgg acttgcaaat gaattccctg aagccggagg acactgccat ctactactgt 1440
gctgcggcag caggatctgc ctggtacggc accctttatg aatacgatta ctggggacag 1500
ggaacccagg tcacggtctc gagtcaccac caccatcacc ac 1542
<210> 80
<211> 393
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 18
<400> 80
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro
130 135 140
Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser
145 150 155 160
Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln
165 170 175
Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu
180 185 190
Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys
195 200 205
Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr
210 215 220
Tyr Cys Val Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr
225 230 235 240
Lys Leu Thr Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
245 250 255
Lys Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
260 265 270
Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe
275 280 285
Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys
290 295 300
Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asp Tyr Lys Asp
305 310 315 320
Asp Asp Asp Lys Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg
325 330 335
Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr
340 345 350
Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn
355 360 365
Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
370 375 380
Val Ser Ser His His His His His His
385 390
<210> 81
<211> 1179
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 18
<400> 81
gaggtccaac ttgttgagag cggtggggga cttgtacagg ccggagggtc cttgaggctg 60
agttgtgctg cgtccggacg caccttcagt agttatgcga tgggatggtt ccggcaagcg 120
ccggggaaag agagagaatt tgttgtcgct atcaattggt ccagtgggag cacttattac 180
gctgactctg taaaaggaag atttactata tctcgagata atgctaagaa caccatgtat 240
cttcagatga actctctgaa accagaggat actgctgtgt attactgtgc tgcgggatat 300
cagataaatt caggtaatta taactttaaa gactatgagt atgactactg gggacagggg 360
actcaagtta cggtgagttc aggcggcggg ggttctgggg gtggaagcca gaccgtcgtg 420
acccaggaac catctcttac agtctccccg ggaggcactg taacccttac ctgcgggtca 480
tcaactggcg ctgtaacgtc cggcaactac cccaactggg tgcagcagaa accgggacag 540
gcgccacgag gcctgatagg tgggactaag tttcttgctc caggaactcc tgctcgattt 600
tctggcagtc tgttgggcgg caaggcagcc ctgacacttt ctggtgtgca accggaggac 660
gaggctgagt actactgcgt actctggtat tcaaatcgat gggtttttgg gggtggaacg 720
aaattgaccg ttctcggagg tggtggtgac tataaagatg atgacgacaa aggtggtgga 780
tccgaagtcc aactcgtcga gtccggggga ggacttgtcc aacctggagg atcattgaaa 840
ctcagttgtg cagcctccgg gttcacattt aataaatacg ccatgaattg ggtccggcaa 900
gccccaggca aggggcttga gtgggttgcg cggatccgat caaagtacga ctataaagac 960
gatgacgata aggctgattc agtcaaagac aggtttacca taagtcgcga tgacagcaag 1020
aacaccgctt accttcaaat gaacaatctg aaaacagaag acaccgcagt atactactgc 1080
gtgcgccacg gcaattttgg taacagttac atttcctatt gggcgtattg ggggcaggga 1140
actctcgtca cggtaagttc ccatcatcac catcatcat 1179
<210> 82
<211> 514
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 19
<400> 82
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn
275 280 285
Lys Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Ala Arg Ile Arg Ser Lys Tyr Asp Tyr Lys Asp Asp Asp Asp
305 310 315 320
Lys Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser
325 330 335
Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr
340 345 350
Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile
355 360 365
Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser
370 375 380
Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser
385 390 395 400
Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala
405 410 415
Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln
420 425 430
Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly
435 440 445
Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser
450 455 460
Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg
465 470 475 480
Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser
485 490 495
Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His
500 505 510
His His
<210> 83
<211> 1542
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 19
<400> 83
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtcttcg gaggagggac gaaacttact 720
gtacttggag gcggcggtga ctacaaggac gacgatgaca aaggcggcgg cagcgaggtc 780
cagttggtag aatccggagg tggattggtt caaccgggag gaagccttaa gctttcatgc 840
gccgcatccg gattcacctt caataagtac gcaatgaatt gggttagaca ggcaccaggt 900
aaagggttgg aatgggtggc acgcattagg tctaaatacg attacaagga cgacgacgac 960
aaagctgaca gcgtaaaaga ccgatttacg ataagccggg atgattctaa gaacactgct 1020
tatttgcaga tgaataattt gaagaccgag gatactgctg tctattattg cgtccgccac 1080
ggtaattttg gtaactctta cattagctat tgggcgtatt gggggcaggg cactctggtc 1140
accgtctcat ctggcggagg gggcagtggc ggcgggtcag aggttcaact tgtcgagtct 1200
ggaggcggtc tcgtacaacc ggggaatagt ctccgactct cttgcgctgc gtccgggttc 1260
acgttctcaa agtttgggat gtcttgggtt aggcaagccc caggtaaggg actcgaatgg 1320
gtcagcagca tctcaggctc cggcagagac acgttgtatg ccgaaagtgt caaagggagg 1380
ttcacaatct ctcgggacaa tgcaaaaacc accttgtatc tccaaatgaa ctcactccgg 1440
cctgaggaca cagcagttta ctactgtacg ataggagggt cccttagcgt atcttctcag 1500
ggaactttgg taacggtcag ctcccaccac catcatcatc ac 1542
<210> 84
<211> 513
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 19 CD3plus
<400> 84
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn
275 280 285
Lys Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr
305 310 315 320
Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys
325 330 335
Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser
355 360 365
Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
370 375 380
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
385 390 395 400
Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala
405 410 415
Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala
420 425 430
Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg
435 440 445
Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
450 455 460
Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro
465 470 475 480
Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val
485 490 495
Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His
500 505 510
His
<210> 85
<211> 1539
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 19 CD3plus
<400> 85
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtcttcg gaggagggac gaaacttact 720
gtacttggag gcggcggtga ctacaaggac gacgatgaca aaggcggcgg cagcgaggtc 780
cagttggtag aatccggagg tggattggtt caaccgggag gaagccttaa gctttcatgc 840
gccgcatccg gattcacctt caataagtac gcaatgaatt gggttagaca ggcaccaggt 900
aaagggttgg aatgggtggc acgcattagg tccaagtaca acaactacgc cacctactac 960
gcggacagcg taaaagaccg atttacgata agccgggatg attctaagaa cactgcttat 1020
ttgcagatga ataatttgaa gaccgaggat actgctgtct attattgcgt ccgccacggt 1080
aattttggta actcttacat tagctattgg gcgtattggg ggcagggcac tctggtcacc 1140
gtctcatctg gcggaggggg cagtggcggc gggtcagagg ttcaacttgt cgagtctgga 1200
ggcggtctcg tacaaccggg gaatagtctc cgactctctt gcgctgcgtc cgggttcacg 1260
ttctcaaagt ttgggatgtc ttgggttagg caagccccag gtaagggact cgaatgggtc 1320
agcagcatct caggctccgg cagagacacg ttgtatgccg aaagtgtcaa agggaggttc 1380
acaatctctc gggacaatgc aaaaaccacc ttgtatctcc aaatgaactc actccggcct 1440
gaggacacag cagtttacta ctgtacgata ggagggtccc ttagcgtatc ttctcaggga 1500
actttggtaa cggtcagctc ccaccaccat catcatcac 1539
<210> 86
<211> 516
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 20
<400> 86
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
260 265 270
Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
275 280 285
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
290 295 300
Ala Val Thr Ser Gly His Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
305 310 315 320
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Ser Asn Lys His Ser Trp
325 330 335
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
340 345 350
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val
355 360 365
Leu Trp Gly Ser Arg Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
370 375 380
Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val
385 390 395 400
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser
405 410 415
Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val
420 425 430
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly
435 440 445
Ser Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr
450 455 460
Ile Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser
465 470 475 480
Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser
485 490 495
Leu Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His
500 505 510
His His His His
515
<210> 87
<211> 1548
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 20
<400> 87
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatc aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
caagtaaagg accgctttac tatctctcga gatgacagta agaacacggc ttatttgcaa 660
atgaacaact tgaagacaga agatacggcg gtctattatt gtgtacgaca cgcaaatttt 720
gggaattcat atataagcta ttgggcatac tggggtcaag gaacccttgt tacggtgagc 780
agcgggggcg gtggtgacta taaggacgac gatgacaaag gcggcggatc ccagactgtg 840
gtaacacagg aaccatcttt gacagtaagt cctggaggta cggtcacgct cacttgtggg 900
tcctcaaccg gggctgtaac gtcaggccat taccctaact gggtccaaca gaagcctgga 960
caagctccca ggggtctgat aggcggaact tcaaacaagc actcttggac tccagcgcgc 1020
tttagcggtt cccttctggg tggaaaagca gccctcactc tgagtggagt acaacccgag 1080
gatgaggcgg aatattattg cgtgctctgg ggttcacgcc gctgggtctt cggtggcggt 1140
acgaaactta ctgtactggg gggaggcggc tcaggcggcg gatcagaagt gcagcttgtt 1200
gaatctggcg gaggtctggt ccagccaggt aacagcttga gactgtcctg tgctgcaagc 1260
ggctttacct tctctaaatt cggtatgagt tgggttcggc aagcccctgg aaagggtttg 1320
gaatgggtat caagcattag tggttctggg cgagatacac tctatgccga atcagtgaag 1380
ggccgcttta ccattagtag ggataacgct aaaactactc tgtatctgca aatgaatagt 1440
ctgagaccag aagatactgc cgtttactac tgcacaatag ggggatctct gagcgtttca 1500
tctcaaggta cacttgtgac tgttagcagt catcatcatc atcaccac 1548
<210> 88
<211> 513
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 21
<400> 88
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser
275 280 285
Gly Ser Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Gly Arg Ile Arg Ser Lys Ala Asn Ser Tyr Ala Thr Ala Tyr
305 310 315 320
Ala Ala Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys
325 330 335
Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Val Arg His Gly Lys Phe Gly Lys Ser Tyr Ile Ala
355 360 365
Phe Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
370 375 380
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
385 390 395 400
Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala
405 410 415
Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala
420 425 430
Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg
435 440 445
Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
450 455 460
Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro
465 470 475 480
Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val
485 490 495
Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His
500 505 510
His
<210> 89
<211> 1539
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 21
<400> 89
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtcttcg gaggagggac gaaacttact 720
gtacttggag gcggcggtga ctacaaggac gacgatgaca aaggcggcgg cagcgaggtc 780
cagttggtag aatccggagg tggattggtt caaccgggag gaagccttaa gctttcatgc 840
gccgcatccg gattcacctt ctctggttcc gcaatgaatt gggttagaca ggcaccaggt 900
aaagggttgg aatgggtggg aagaatacgc agtaaagcca attcttatgc gactgcttat 960
gccgctagtg taaaggaccg atttacgata agccgggatg attctaagaa cactgcttat 1020
ttgcagatga ataatttgaa gaccgaggat actgctgtct attattgcgt ccgccacggt 1080
aaatttggta agtcttacat tgccttttgg gcgtattggg ggcagggcac tctggtcacc 1140
gtctcatctg gcggaggggg cagtggcggc gggtcagagg ttcaacttgt cgagtctgga 1200
ggcggtctcg tacaaccggg gaatagtctc cgactctctt gcgctgcgtc cgggttcacg 1260
ttctcaaagt ttgggatgtc ttgggttagg caagccccag gtaagggact cgaatgggtc 1320
agcagcatct caggctccgg cagagacacg ttgtatgccg aaagtgtcaa agggaggttc 1380
acaatctctc gggacaatgc aaaaaccacc ttgtatctcc aaatgaactc actccggcct 1440
gaggacacag cagtttacta ctgtacgata ggagggtccc ttagcgtatc ttctcaggga 1500
actttggtaa cggtcagctc ccaccaccat catcatcac 1539
<210> 90
<211> 516
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 22
<400> 90
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
260 265 270
Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
275 280 285
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
290 295 300
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
305 310 315 320
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
325 330 335
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
340 345 350
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
355 360 365
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
370 375 380
Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val
385 390 395 400
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser
405 410 415
Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val
420 425 430
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly
435 440 445
Ser Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr
450 455 460
Ile Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser
465 470 475 480
Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser
485 490 495
Leu Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His
500 505 510
His His His His
515
<210> 91
<211> 1548
<212> Белок
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 22
<400> 91
Gly Ala Ala Gly Thr Thr Cys Ala Ala Cys Thr Gly Gly Thr Thr Gly
1 5 10 15
Ala Ala Thr Cys Cys Gly Gly Thr Gly Gly Thr Gly Gly Cys Cys Thr
20 25 30
Thr Gly Thr Cys Cys Ala Gly Gly Cys Gly Gly Gly Ala Gly Gly Cys
35 40 45
Thr Cys Cys Cys Thr Thr Cys Gly Ala Cys Thr Gly Thr Cys Thr Thr
50 55 60
Gly Thr Gly Cys Cys Gly Cys Cys Ala Gly Cys Gly Gly Thr Cys Gly
65 70 75 80
Cys Ala Cys Ala Thr Thr Cys Ala Gly Cys Ala Gly Thr Thr Ala Thr
85 90 95
Gly Cys Cys Ala Thr Gly Gly Gly Ala Thr Gly Gly Thr Thr Cys Cys
100 105 110
Gly Gly Cys Ala Gly Gly Cys Cys Cys Cys Thr Gly Gly Thr Ala Ala
115 120 125
Ala Gly Ala Gly Cys Gly Gly Gly Ala Gly Thr Thr Cys Gly Thr Cys
130 135 140
Gly Thr Thr Gly Cys Gly Ala Thr Cys Ala Ala Thr Thr Gly Gly Ala
145 150 155 160
Gly Thr Ala Gly Cys Gly Gly Thr Thr Cys Cys Ala Cys Gly Thr Ala
165 170 175
Thr Thr Ala Thr Gly Cys Gly Gly Ala Thr Thr Cys Thr Gly Thr Ala
180 185 190
Ala Ala Gly Gly Gly Cys Ala Gly Gly Thr Thr Cys Ala Cys Thr Ala
195 200 205
Thr Cys Thr Cys Ala Cys Gly Cys Gly Ala Thr Ala Ala Thr Gly Cys
210 215 220
Ala Ala Ala Ala Ala Ala Thr Ala Cys Cys Ala Thr Gly Thr Ala Thr
225 230 235 240
Cys Thr Thr Cys Ala Gly Ala Thr Gly Ala Ala Cys Thr Cys Ala Cys
245 250 255
Thr Gly Ala Ala Gly Cys Cys Cys Gly Ala Gly Gly Ala Cys Ala Cys
260 265 270
Gly Gly Cys Ala Gly Thr Thr Thr Ala Thr Thr Ala Cys Thr Gly Thr
275 280 285
Gly Cys Thr Gly Cys Cys Gly Gly Thr Thr Ala Cys Cys Ala Gly Ala
290 295 300
Thr Cys Ala Ala Thr Thr Cys Cys Gly Gly Ala Ala Ala Thr Thr Ala
305 310 315 320
Cys Ala Ala Thr Thr Thr Cys Ala Ala Gly Gly Ala Cys Thr Ala Cys
325 330 335
Gly Ala Gly Thr Ala Cys Gly Ala Thr Thr Ala Thr Thr Gly Gly Gly
340 345 350
Gly Thr Cys Ala Gly Gly Gly Cys Ala Cys Cys Cys Ala Gly Gly Thr
355 360 365
Ala Ala Cys Cys Gly Thr Cys Ala Gly Cys Ala Gly Cys Gly Gly Gly
370 375 380
Gly Gly Ala Gly Gly Cys Gly Gly Ala Thr Cys Ala Gly Gly Ala Gly
385 390 395 400
Gly Cys Gly Gly Thr Thr Cys Ala Gly Ala Gly Gly Thr Thr Cys Ala
405 410 415
Gly Cys Thr Cys Gly Thr Thr Gly Ala Gly Ala Gly Thr Gly Gly Thr
420 425 430
Gly Gly Ala Gly Gly Gly Cys Thr Gly Gly Thr Thr Cys Ala Gly Cys
435 440 445
Cys Ala Gly Gly Gly Gly Gly Ala Ala Gly Thr Thr Thr Gly Ala Ala
450 455 460
Gly Cys Thr Thr Thr Cys Cys Thr Gly Thr Gly Cys Gly Gly Cys Cys
465 470 475 480
Thr Cys Thr Gly Gly Thr Thr Thr Cys Ala Cys Cys Thr Thr Thr Ala
485 490 495
Ala Cys Ala Ala Ala Thr Ala Cys Gly Cys Thr Ala Thr Cys Ala Ala
500 505 510
Cys Thr Gly Gly Gly Thr Ala Cys Gly Ala Cys Ala Ala Gly Cys Cys
515 520 525
Cys Cys Cys Gly Gly Thr Ala Ala Ala Gly Gly Gly Cys Thr Thr Gly
530 535 540
Ala Ala Thr Gly Gly Gly Thr Thr Gly Cys Ala Ala Gly Ala Ala Thr
545 550 555 560
Ala Cys Gly Cys Ala Gly Thr Ala Ala Ala Thr Ala Cys Ala Ala Thr
565 570 575
Ala Ala Thr Thr Ala Thr Gly Cys Gly Ala Cys Thr Thr Ala Thr Thr
580 585 590
Ala Thr Gly Cys Cys Gly Ala Thr Cys Ala Ala Gly Thr Ala Ala Ala
595 600 605
Gly Gly Ala Cys Cys Gly Cys Thr Thr Thr Ala Cys Thr Ala Thr Cys
610 615 620
Thr Cys Thr Cys Gly Ala Gly Ala Thr Gly Ala Cys Ala Gly Thr Ala
625 630 635 640
Ala Gly Ala Ala Cys Ala Cys Gly Gly Cys Thr Thr Ala Thr Thr Thr
645 650 655
Gly Cys Ala Ala Ala Thr Gly Ala Ala Cys Ala Ala Cys Thr Thr Gly
660 665 670
Ala Ala Gly Ala Cys Ala Gly Ala Ala Gly Ala Thr Ala Cys Gly Gly
675 680 685
Cys Gly Gly Thr Cys Thr Ala Thr Thr Ala Thr Thr Gly Thr Gly Thr
690 695 700
Ala Cys Gly Ala Cys Ala Cys Gly Cys Ala Ala Ala Thr Thr Thr Thr
705 710 715 720
Gly Gly Gly Ala Ala Thr Thr Cys Ala Thr Ala Thr Ala Thr Ala Ala
725 730 735
Gly Cys Thr Ala Thr Thr Gly Gly Gly Cys Ala Thr Ala Cys Thr Gly
740 745 750
Gly Gly Gly Thr Cys Ala Ala Gly Gly Ala Ala Cys Cys Cys Thr Thr
755 760 765
Gly Thr Thr Ala Cys Gly Gly Thr Gly Ala Gly Cys Ala Gly Cys Gly
770 775 780
Gly Gly Gly Gly Cys Gly Gly Thr Gly Gly Thr Gly Ala Cys Thr Ala
785 790 795 800
Thr Ala Ala Gly Gly Ala Cys Gly Ala Cys Gly Ala Thr Gly Ala Cys
805 810 815
Ala Ala Ala Gly Gly Cys Gly Gly Cys Gly Gly Ala Thr Cys Cys Cys
820 825 830
Ala Ala Ala Cys Ala Gly Thr Thr Gly Thr Thr Ala Cys Thr Cys Ala
835 840 845
Gly Gly Ala Gly Cys Cys Ala Thr Cys Cys Thr Thr Gly Ala Cys Ala
850 855 860
Gly Thr Ala Thr Cys Cys Cys Cys Cys Gly Gly Thr Gly Gly Ala Ala
865 870 875 880
Cys Gly Gly Thr Gly Ala Cys Cys Cys Thr Gly Ala Cys Ala Thr Gly
885 890 895
Cys Gly Cys Thr Thr Cys Ala Ala Gly Thr Ala Cys Ala Gly Gly Thr
900 905 910
Gly Cys Thr Gly Thr Ala Ala Cys Cys Thr Cys Ala Gly Gly Thr Ala
915 920 925
Ala Cys Thr Ala Cys Cys Cys Gly Ala Ala Thr Thr Gly Gly Gly Thr
930 935 940
Gly Cys Ala Gly Cys Ala Ala Ala Ala Ala Cys Cys Thr Gly Gly Ala
945 950 955 960
Cys Ala Ala Gly Cys Ala Cys Cys Cys Cys Gly Gly Gly Gly Thr Cys
965 970 975
Thr Thr Ala Thr Cys Gly Gly Gly Gly Gly Gly Ala Cys Gly Ala Ala
980 985 990
Gly Thr Thr Cys Thr Thr Gly Gly Thr Ala Cys Cys Gly Gly Gly Thr
995 1000 1005
Ala Cys Cys Cys Cys Thr Gly Cys Gly Cys Gly Cys Thr Thr Cys
1010 1015 1020
Ala Gly Cys Gly Gly Ala Ala Gly Thr Cys Thr Thr Cys Thr Gly
1025 1030 1035
Gly Gly Thr Gly Gly Ala Ala Ala Ala Gly Cys Cys Gly Cys Cys
1040 1045 1050
Thr Thr Gly Ala Cys Cys Thr Thr Gly Thr Cys Ala Gly Gly Cys
1055 1060 1065
Gly Thr Thr Cys Ala Gly Cys Cys Cys Gly Ala Ala Gly Ala Thr
1070 1075 1080
Gly Ala Gly Gly Cys Cys Gly Ala Ala Thr Ala Thr Thr Ala Thr
1085 1090 1095
Thr Gly Cys Ala Cys Gly Cys Thr Gly Thr Gly Gly Thr Ala Thr
1100 1105 1110
Thr Cys Thr Ala Ala Cys Cys Gly Gly Thr Gly Gly Gly Thr Cys
1115 1120 1125
Thr Thr Cys Gly Gly Ala Gly Gly Ala Gly Gly Gly Ala Cys Gly
1130 1135 1140
Ala Ala Ala Cys Thr Thr Ala Cys Thr Gly Thr Ala Cys Thr Thr
1145 1150 1155
Gly Gly Gly Gly Gly Ala Gly Gly Cys Gly Gly Cys Thr Cys Ala
1160 1165 1170
Gly Gly Cys Gly Gly Cys Gly Gly Ala Thr Cys Ala Gly Ala Ala
1175 1180 1185
Gly Thr Gly Cys Ala Gly Cys Thr Thr Gly Thr Thr Gly Ala Ala
1190 1195 1200
Thr Cys Thr Gly Gly Cys Gly Gly Ala Gly Gly Thr Cys Thr Gly
1205 1210 1215
Gly Thr Cys Cys Ala Gly Cys Cys Ala Gly Gly Thr Ala Ala Cys
1220 1225 1230
Ala Gly Cys Thr Thr Gly Ala Gly Ala Cys Thr Gly Thr Cys Cys
1235 1240 1245
Thr Gly Thr Gly Cys Thr Gly Cys Ala Ala Gly Cys Gly Gly Cys
1250 1255 1260
Thr Thr Thr Ala Cys Cys Thr Thr Cys Thr Cys Thr Ala Ala Ala
1265 1270 1275
Thr Thr Cys Gly Gly Thr Ala Thr Gly Ala Gly Thr Thr Gly Gly
1280 1285 1290
Gly Thr Thr Cys Gly Gly Cys Ala Ala Gly Cys Cys Cys Cys Thr
1295 1300 1305
Gly Gly Ala Ala Ala Gly Gly Gly Thr Thr Thr Gly Gly Ala Ala
1310 1315 1320
Thr Gly Gly Gly Thr Ala Thr Cys Ala Ala Gly Cys Ala Thr Thr
1325 1330 1335
Ala Gly Thr Gly Gly Thr Thr Cys Thr Gly Gly Gly Cys Gly Ala
1340 1345 1350
Gly Ala Thr Ala Cys Ala Cys Thr Cys Thr Ala Thr Gly Cys Cys
1355 1360 1365
Gly Ala Ala Thr Cys Ala Gly Thr Gly Ala Ala Gly Gly Gly Cys
1370 1375 1380
Cys Gly Cys Thr Thr Thr Ala Cys Cys Ala Thr Thr Ala Gly Thr
1385 1390 1395
Ala Gly Gly Gly Ala Thr Ala Ala Cys Gly Cys Thr Ala Ala Ala
1400 1405 1410
Ala Cys Thr Ala Cys Thr Cys Thr Gly Thr Ala Thr Cys Thr Gly
1415 1420 1425
Cys Ala Ala Ala Thr Gly Ala Ala Thr Ala Gly Thr Cys Thr Gly
1430 1435 1440
Ala Gly Ala Cys Cys Ala Gly Ala Ala Gly Ala Thr Ala Cys Thr
1445 1450 1455
Gly Cys Cys Gly Thr Thr Thr Ala Cys Thr Ala Cys Thr Gly Cys
1460 1465 1470
Ala Cys Ala Ala Thr Ala Gly Gly Gly Gly Gly Ala Thr Cys Thr
1475 1480 1485
Cys Thr Gly Ala Gly Cys Gly Thr Thr Thr Cys Ala Thr Cys Thr
1490 1495 1500
Cys Ala Ala Gly Gly Thr Ala Cys Ala Cys Thr Thr Gly Thr Gly
1505 1510 1515
Ala Cys Thr Gly Thr Thr Ala Gly Cys Ala Gly Thr Cys Ala Thr
1520 1525 1530
Cys Ala Thr Cys Ala Thr Cys Ala Thr Cys Ala Cys Cys Ala Cys
1535 1540 1545
<210> 92
<211> 1041
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 23
<400> 92
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
260 265 270
Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
275 280 285
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
290 295 300
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
305 310 315 320
Gln Ala Pro Arg Gly Leu Ile Gly Asp Tyr Lys Asp Asp Asp Asp Lys
325 330 335
Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala
340 345 350
Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys
355 360 365
Val Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu
370 375 380
Thr Val Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu
385 390 395 400
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu
405 410 415
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp
420 425 430
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser
435 440 445
Gly Ser Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe
450 455 460
Thr Ile Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn
465 470 475 480
Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly
485 490 495
Ser Leu Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser Gly
500 505 510
Gly Gly Gly Leu Val Pro Arg Gly Ser Leu Gly Gly Gly Gly Ser Gln
515 520 525
Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly Ser
530 535 540
Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr Gly
545 550 555 560
Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val Ser
565 570 575
Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val Lys
580 585 590
Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp Leu
595 600 605
Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys Ala
610 615 620
Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp Tyr
625 630 635 640
Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly Ser
645 650 655
Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val
660 665 670
Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly Ala
675 680 685
Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln
690 695 700
Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly Thr
705 710 715 720
Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr
725 730 735
Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr Leu
740 745 750
Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val
755 760 765
Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly Gly
770 775 780
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
785 790 795 800
Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys
805 810 815
Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
820 825 830
Val Ala Arg Ile Arg Ser Lys Tyr Asp Tyr Lys Asp Asp Asp Asp Lys
835 840 845
Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys
850 855 860
Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
865 870 875 880
Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser
885 890 895
Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
900 905 910
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
915 920 925
Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala
930 935 940
Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala
945 950 955 960
Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg
965 970 975
Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
980 985 990
Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro
995 1000 1005
Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser
1010 1015 1020
Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His
1025 1030 1035
His His His
1040
<210> 93
<211> 3123
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 23
<400> 93
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatc aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
caagtaaagg accgctttac tatcagtaga gatgacagta agaacacggc ttatttgcaa 660
atgaacaact tgaagacaga agatacggcg gtctattatt gtgtacgaca cgcaaatttt 720
gggaattcat atataagcta ttgggcatac tggggtcaag gaacccttgt tacggtgagc 780
agcgggggcg gtggtgacta taaggacgac gatgacaaag gcggcggctc ccagactgtg 840
gtaacacagg aaccatcttt gacagtaagt cctggaggta cggtcacgct cacttgtggg 900
tcctcaaccg gggctgtaac gtcaggcaat taccctaact gggtccaaca gaagcctgga 960
caagctccca ggggtctgat aggcgattac aaagatgatg atgataaggg cactccagcg 1020
cgctttagcg gatcccttct gggtggaaaa gcagccctca ctctgagtgg agtacaaccc 1080
gaggatgagg cggaatatta ttgcgtgctc tggtattcaa accgctgggt cttcggtggc 1140
ggtacgaaac ttactgtact ggggggaggc ggctcaggcg gcggatcaga agtgcagctt 1200
gttgaatctg gcggaggtct ggtccagcca ggtaacagct tgagactgtc ctgtgctgct 1260
agcggcttta ccttctctaa attcggtatg agttgggttc ggcaagcccc tggaaagggt 1320
ttggaatggg tatcaagcat tagtggttct gggcgagata cactctatgc cgaatcagtg 1380
aagggccgct ttaccattag tagggataac gctaaaacta ctctgtatct gcaaatgaat 1440
agtctgagac cagaagatac tgccgtttac tactgcacaa tagggggatc tctgagcgtt 1500
tcatctcaag gtacacttgt gactgttagc agtggtggcg gaggacttgt acctcgaggt 1560
agcttgggtg gagggggatc ccaagtcaaa cttgaggaaa gcgggggagg tagcgtacag 1620
actggtggat ctctgaggtt gacttgcgcc gccagtggcc gaacatccag aagttacggg 1680
atgggttggt ttcgacaggc tccgggaaaa gagcgggagt ttgtatctgg cataagctgg 1740
aggggcgact ccactggtta cgcagattcc gtcaaagggc ggtttacgat ctctcgggat 1800
aacgcgaaga ataccgttga tctccaaatg aactctctta aacccgagga tacagcaata 1860
tactattgcg ccgccgctgc ggggtcagcc tggtatggca cattgtacga atatgactat 1920
tggggtcaag gtacccaagt aacggtcagt tccggtggtg gggggtctgg tggtggatcc 1980
caaacagttg ttactcagga gccatccttg acagtatccc ccggtggaac ggtgaccctg 2040
acatgcgctt caagtacagg tgctgtaacc tcaggtaact acccgaattg ggtgcagcaa 2100
aaacctggac aagcaccccg gggtcttatc ggggggacga agttcttggt accgggtacc 2160
cctgcgcgct tcagcggaag tcttctgggt ggaaaagccg ccttgacctt gtcaggcgtt 2220
cagcccgaag atgaggccga atattattgc acgctgtggt attctaaccg gtgggtcttc 2280
ggaggaggga cgaaacttac tgtacttgga ggcggcggtg actacaagga cgacgatgac 2340
aaaggcggcg gcagcgaggt ccagttggta gaatccggag gtggattggt tcaaccggga 2400
ggaagcctta agctttcatg cgccgcatcc ggattcacct tcaataagta cgcaatgaat 2460
tgggttagac aggcaccagg taaagggttg gaatgggtgg cacgcattag gtctaaatac 2520
gattacaagg acgacgacga caaagctgac agcgtaaaag accgatttac gataagccgg 2580
gatgattcta agaacactgc ttatttgcag atgaataatt tgaagaccga ggatactgct 2640
gtctattatt gcgtccgcca cggtaatttt ggtaactctt acattagcta ttgggcgtat 2700
tgggggcagg gcactctggt caccgtctca tctggcggag ggggcagtgg cggcgggtca 2760
gaggttcaac ttgtcgagtc tggaggcggt ctcgtacaac cggggaatag tctccgactc 2820
tcttgcgctg cgtccgggtt cacgttctca aagtttggga tgtcttgggt taggcaagcc 2880
ccaggtaagg gactcgaatg ggtcagcagc atctcaggct ccggcagaga cacgttgtat 2940
gccgaaagtg tcaaagggag gttcacaatc tctcgggaca atgcaaaaac caccttgtat 3000
ctccaaatga actcactccg gcctgaggac acagcagttt actactgtac gataggaggg 3060
tcccttagcg tatcttctca gggaactttg gtaacggtca gctcccacca ccatcatcat 3120
cac 3123
<210> 94
<211> 917
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 24
<400> 94
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
260 265 270
Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
275 280 285
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly
290 295 300
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
305 310 315 320
Gln Ala Pro Arg Gly Leu Ile Gly Asp Tyr Lys Asp Asp Asp Asp Lys
325 330 335
Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala
340 345 350
Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys
355 360 365
Val Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu
370 375 380
Thr Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly
385 390 395 400
Gly Gly Ser Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln
405 410 415
Thr Gly Gly Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser
420 425 430
Arg Ser Tyr Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg
435 440 445
Glu Phe Val Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala
450 455 460
Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn
465 470 475 480
Thr Val Asp Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile
485 490 495
Tyr Tyr Cys Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr
500 505 510
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
515 520 525
Gly Gly Gly Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro
530 535 540
Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser
545 550 555 560
Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln
565 570 575
Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu
580 585 590
Val Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys
595 600 605
Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr
610 615 620
Tyr Cys Thr Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr
625 630 635 640
Lys Leu Thr Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp
645 650 655
Lys Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu
660 665 670
Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe
675 680 685
Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys
690 695 700
Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asp Tyr Lys Asp
705 710 715 720
Asp Asp Asp Lys Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg
725 730 735
Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr
740 745 750
Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn
755 760 765
Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr
770 775 780
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu
785 790 795 800
Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu
805 810 815
Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp
820 825 830
Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser
835 840 845
Gly Ser Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe
850 855 860
Thr Ile Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn
865 870 875 880
Ser Leu Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly
885 890 895
Ser Leu Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His
900 905 910
His His His His His
915
<210> 95
<211> 2751
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 24
<400> 95
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatc aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
caagtaaagg accgctttac tatcagtaga gatgacagta agaacacggc ttatttgcaa 660
atgaacaact tgaagacaga agatacggcg gtctattatt gtgtacgaca cgcaaatttt 720
gggaattcat atataagcta ttgggcatac tggggtcaag gaacccttgt tacggtgagc 780
agcgggggcg gtggtgacta taaggacgac gatgacaaag gcggcggctc ccagactgtg 840
gtaacacagg aaccatcttt gacagtaagt cctggaggta cggtcacgct cacttgtggg 900
tcctcaaccg gggctgtaac gtcaggcaat taccctaact gggtccaaca gaagcctgga 960
caagctccca ggggtctgat aggcgattac aaagatgatg atgataaggg cactccagcg 1020
cgctttagcg gatcccttct gggtggaaaa gcagccctca ctctgagtgg agtacaaccc 1080
gaggatgagg cggaatatta ttgcgtgctc tggtattcaa accgctgggt cttcggtggc 1140
ggtacgaaac ttactgtact gggtggaggt ggtgactaca aggatgacga cgacaagggg 1200
ggcgggagtc aagtcaaact tgaggaaagc gggggaggta gcgtacagac tggtggatct 1260
ctgaggttga cttgcgccgc cagtggccga acatccagaa gttacgggat gggttggttt 1320
cgacaggctc cgggaaaaga gcgggagttt gtatctggca taagctggag gggcgactcc 1380
actggttacg cagattccgt caaagggcgg tttacgatct ctcgggataa cgcgaagaat 1440
accgttgatc tccaaatgaa ctctcttaaa cccgaggata cagcaatata ctattgcgcc 1500
gccgctgcgg ggtcagcctg gtatggcaca ttgtacgaat atgactattg gggtcaaggt 1560
acccaagtaa cggtcagttc cggtggtggg gggtctggtg gtggatccca aacagttgtt 1620
actcaggagc catccttgac agtatccccc ggtggaacgg tgaccctgac atgcgcttca 1680
agtacaggtg ctgtaacctc aggtaactac ccgaattggg tgcagcaaaa acctggacaa 1740
gcaccccggg gtcttatcgg ggggacgaag ttcttggtac cgggtacccc tgcgcgcttc 1800
agcggaagtc ttctgggtgg aaaagccgcc ttgaccttgt caggcgttca gcccgaagat 1860
gaggccgaat attattgcac gctgtggtat tctaaccggt gggtcttcgg aggagggacg 1920
aaacttactg tacttggagg cggcggtgac tacaaggacg acgatgacaa aggcggcggc 1980
agcgaggtcc agttggtaga atccggaggt ggattggttc aaccgggagg aagccttaag 2040
ctttcatgcg ccgcatccgg attcaccttc aataagtacg caatgaattg ggttagacag 2100
gcaccaggta aagggttgga atgggtggca cgcattaggt ctaaatacga ttacaaggac 2160
gacgacgaca aagctgacag cgtaaaagac cgatttacga taagccggga tgattctaag 2220
aacactgctt atttgcagat gaataatttg aagaccgagg atactgctgt ctattattgc 2280
gtccgccacg gtaattttgg taactcttac attagctatt gggcgtattg ggggcagggc 2340
actctggtca ccgtctcatc tggcggaggg ggcagtggcg gcgggtcaga ggttcaactt 2400
gtcgagtctg gaggcggtct cgtacaaccg gggaatagtc tccgactctc ttgcgctgcg 2460
tccgggttca cgttctcaaa gtttgggatg tcttgggtta ggcaagcccc aggtaaggga 2520
ctcgaatggg tcagcagcat ctcaggctcc ggcagagaca cgttgtatgc cgaaagtgtc 2580
aaagggaggt tcacaatctc tcgggacaat gcaaaaacca ccttgtatct ccaaatgaac 2640
tcactccggc ctgaggacac agcagtttac tactgtacga taggagggtc ccttagcgta 2700
tcttctcagg gaactttggt aacggtcagc tcccaccacc atcatcatca c 2751
<210> 96
<211> 380
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 25
<400> 96
Gln Val Gln Leu Val Glu Ser Gly Gly Ala Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro Val Asn Arg Tyr
20 25 30
Ser Met Arg Trp Tyr Arg Gln Ala Pro Gly Lys Glu Arg Glu Trp Val
35 40 45
Ala Gly Met Ser Ser Ala Gly Asp Arg Ser Ser Tyr Glu Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ala Arg Asn Thr Val Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Asn Val Asn Val Gly Phe Glu Tyr Trp Gly Gln Gly Thr Gln Val Thr
100 105 110
Val Ser Ser Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val
115 120 125
Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser
130 135 140
Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val
145 150 155 160
Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser
165 170 175
Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val Lys Asp Arg
180 185 190
Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met
195 200 205
Asn Asn Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His
210 215 220
Gly Asn Phe Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln
225 230 235 240
Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Asp Tyr Lys Asp
245 250 255
Asp Asp Asp Lys Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro
260 265 270
Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser
275 280 285
Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln
290 295 300
Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile Gly Asp Tyr Lys Asp Asp
305 310 315 320
Asp Asp Lys Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly
325 330 335
Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu
340 345 350
Tyr Tyr Cys Val Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly
355 360 365
Thr Lys Leu Thr Val Leu His His His His His His
370 375 380
<210> 97
<211> 1140
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 25
<400> 97
caggtacagt tggtagaaag cggcggtgca ttggtacaac ccggcggctc cttgaggctc 60
agctgcgcgg cgtccggctt tccggttaac cgctattcta tgagatggta caggcaagca 120
ccgggcaaag aacgcgagtg ggtcgcaggt atgagcagcg cgggagaccg atcctcctat 180
gaagactccg ttaaaggtag gtttaccatt agtcgagacg atgctagaaa cacggtgtac 240
cttcagatga atagtttgaa accagaagat acggccgtgt attattgtaa tgtgaacgta 300
gggttcgagt actgggggca aggaacacag gttaccgtga gcagcggagg cggatctggc 360
ggcggttctg aggttcaact tgttgaaagt ggaggaggtc tcgttcaacc cggtggttct 420
cttaaactga gctgcgcagc cagtgggttc acattcaata aatatgcgat gaactgggtg 480
cggcaggctc caggcaaggg cttggaatgg gtcgcccgga tcaggtctaa atacaataat 540
tatgccacct attatgccga tagtgtcaaa gatcgcttca cgatatctag agatgactca 600
aagaacacag cgtacctgca aatgaataac ctgaaaacag aagacacagc tgtatattat 660
tgtgtcagac atggtaattt tgggaacagt tacatcagct actgggctta ttggggacaa 720
ggaaccctcg tgactgtgtc ctctggagga ggcggagatt acaaagacga cgatgacaag 780
ggcggcggct cacaaactgt cgtcacacag gaaccttccc tcactgttag cccgggcggg 840
acagtcacac ttacttgtgg gagtagcacc ggagccgtaa cctccggaaa ctatcctaat 900
tgggtacagc agaaaccggg ccaggctcct agaggcttga ttggcgatta taaggatgat 960
gatgataagg gcacgccggc taggttctct gggtccttgc tgggcgggaa ggccgctctt 1020
acgttgtctg gggtgcagcc tgaggacgaa gcagaatatt actgtgtgct ctggtattca 1080
aatcgatggg tgtttggtgg aggaacaaag ttgactgtac tgcaccacca ccatcaccat 1140
<210> 98
<211> 381
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 26
<400> 98
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys Tyr
20 25 30
Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val
35 40 45
Ala Arg Ile Arg Ser Lys Tyr Asp Tyr Lys Asp Asp Asp Asp Lys Ala
50 55 60
Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn
65 70 75 80
Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val
85 90 95
Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser Tyr
100 105 110
Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
115 120 125
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly Gly Ser Gln Thr
130 135 140
Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val
145 150 155 160
Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr
165 170 175
Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile
180 185 190
Gly Gly Thr Lys Phe Leu Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly
195 200 205
Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro
210 215 220
Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser Asn Arg Trp
225 230 235 240
Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Ser
245 250 255
Gly Gly Gly Ser Gln Val Gln Leu Val Glu Ser Gly Gly Ala Leu Val
260 265 270
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro
275 280 285
Val Asn Arg Tyr Ser Met Arg Trp Tyr Arg Gln Ala Pro Gly Lys Glu
290 295 300
Arg Glu Trp Val Ala Gly Met Ser Ser Ala Gly Asp Arg Ser Ser Tyr
305 310 315 320
Glu Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ala Arg
325 330 335
Asn Thr Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Asn Val Asn Val Gly Phe Glu Tyr Trp Gly Gln Gly
355 360 365
Thr Gln Val Thr Val Ser Ser His His His His His His
370 375 380
<210> 99
<211> 1143
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 26
<400> 99
gaagtacagc tcgttgagtc cggaggcggc ctcgttcagc ctggaggttc tcttaagctt 60
tcttgtgcag catctggatt tacgtttaat aaatatgcga tgaactgggt gcgccaagca 120
cccggaaagg ggctcgaatg ggtagcacga atcaggagca agtatgacta caaagatgac 180
gacgacaaag ctgactccgt taaagacaga tttacgattt cacgagacga cagcaagaat 240
acggcgtacc ttcaaatgaa taatcttaaa acggaggata ccgcagttta ttattgcgtg 300
aggcatggta acttcgggaa ctcttacatt agctactggg cttattgggg tcaagggaca 360
ttggtgacgg tctcctccgg tggcggcgga gactataaag atgacgacga caaggggggc 420
gggtctcaga cagtagttac acaggagcct agtcttaccg tatcacccgg tgggaccgta 480
actcttacct gcggttcttc tacgggcgca gtaacgtccg ggaattaccc gaactgggtt 540
cagcaaaaac cgggacaagc tccgaggggc ctcattggtg gtactaaatt cctcgcacct 600
ggcacacctg cgcggttttc agggagcttg ctcggaggca aggcggccct gacattgtcc 660
ggcgttcaac cggaggacga agctgagtac tattgcgtac tgtggtatag caataggtgg 720
gtatttggtg gtggtacgaa gctcacggtc ttggggggag gcggctctgg gggagggagc 780
caggtgcagc ttgttgaatc tggtggtgcc cttgtccagc ccggaggaag tcttcgactc 840
agttgcgcag catctggctt tccggtgaat cgatattcca tgcggtggta ccgacaggcg 900
cctggaaaag aacgcgagtg ggttgcaggt atgagttctg ctggcgatcg gagttcctac 960
gaggatagtg tgaagggcag atttactatt agtcgagatg atgcgcggaa tacggtgtac 1020
ttgcagatga acagcctgaa gccggaggat acagcagttt attattgtaa tgtcaacgtc 1080
ggatttgaat actgggggca ggggacacaa gttactgtaa gctcacatca tcatcatcac 1140
cac 1143
<210> 100
<211> 381
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 27
<400> 100
Gln Val Gln Leu Val Glu Ser Gly Gly Ala Leu Val Gln Pro Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro Val Asn Arg Tyr
20 25 30
Ser Met Arg Trp Tyr Arg Gln Ala Pro Gly Lys Glu Arg Glu Trp Val
35 40 45
Ala Gly Met Ser Ser Ala Gly Asp Arg Ser Ser Tyr Glu Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ala Arg Asn Thr Val Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Asn Val Asn Val Gly Phe Glu Tyr Trp Gly Gln Gly Thr Gln Val Thr
100 105 110
Val Ser Ser Gly Gly Gly Gly Ser Gly Gly Gly Ser Gln Thr Val Val
115 120 125
Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly Thr Val Thr Leu
130 135 140
Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly Asn Tyr Pro Asn
145 150 155 160
Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly Leu Ile Gly Gly
165 170 175
Thr Lys Phe Leu Ala Pro Gly Thr Pro Ala Arg Phe Ser Gly Ser Leu
180 185 190
Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly Val Gln Pro Glu Asp
195 200 205
Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser Asn Arg Trp Val Phe
210 215 220
Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly Gly Gly Asp Tyr Lys
225 230 235 240
Asp Asp Asp Asp Lys Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser
245 250 255
Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala
260 265 270
Ala Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met Asn Trp Val Arg Gln
275 280 285
Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr
290 295 300
Asp Tyr Lys Asp Asp Asp Asp Lys Ala Asp Ser Val Lys Asp Arg Phe
305 310 315 320
Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn
325 330 335
Asn Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Gly
340 345 350
Asn Phe Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly
355 360 365
Thr Leu Val Thr Val Ser Ser His His His His His His
370 375 380
<210> 101
<211> 1143
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 27
<400> 101
caggtacaac tggtcgagtc aggcggggca cttgtacagc ctggtggctc tcttcggctt 60
tcttgcgccg cttccggatt cccagtgaac cggtactcta tgcgctggta caggcaggcc 120
ccggggaagg agcgcgaatg ggttgcggga atgtccagtg cgggggatcg aagcagttat 180
gaagacagcg tcaagggtcg gttcactatt agtagagatg acgcgcgaaa cacggtttac 240
ttgcaaatga atagtctgaa gccagaggat actgccgttt actactgtaa tgtaaacgta 300
ggatttgaat attggggaca agggacgcaa gtaaccgtct cctccggcgg aggaggaagc 360
gggggtggtt ctcaaactgt tgttacgcag gagccctcac ttaccgtgag tcccggcggg 420
actgtcacgc tcacctgtgg ttccagtaca ggggccgtca cctccggaaa ttacccaaat 480
tgggtacaac aaaagccagg acaagccccc agaggcctta taggaggaac caagttcctc 540
gcgcctggta ctccagcccg cttttctggt tccttgttgg ggggtaaagc agcgcttact 600
ctgtccggcg ttcagcctga ggatgaagcg gagtactatt gcgtactctg gtacagcaac 660
cgctgggtgt tcggtggggg gactaaactt actgtgctcg ggggcggggg cgactacaag 720
gacgacgacg ataagggtgg gggctcagaa gtccaacttg ttgaatctgg cggtgggttg 780
gttcagccag ggggatccct gaagctcagc tgcgccgcaa gtggatttac attcaataag 840
tacgcaatga actgggtgag gcaagcgcca ggaaagggac ttgaatgggt tgctagaatc 900
cgatccaaat atgactacaa agatgacgac gacaaagcgg attccgtgaa agacagattc 960
accatctctc gcgatgattc aaaaaacact gcgtacctcc aaatgaataa tctgaaaaca 1020
gaggacacag cagtctatta ttgtgttcgg cacggaaact ttggcaatag ttacatctca 1080
tattgggcat attggggcca gggtacactc gtcacagtca gtagccatca tcatcaccat 1140
cac 1143
<210> 102
<211> 381
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 28
<400> 102
Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val Ser Pro Gly Gly
1 5 10 15
Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala Val Thr Ser Gly
20 25 30
Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln Ala Pro Arg Gly
35 40 45
Leu Ile Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Thr Pro Ala Arg
50 55 60
Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr Leu Ser Gly
65 70 75 80
Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu Trp Tyr Ser
85 90 95
Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val Leu Gly Gly
100 105 110
Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly Gly Ser Glu Val
115 120 125
Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly Gly Ser Leu
130 135 140
Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn Lys Tyr Ala Met
145 150 155 160
Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ala Arg
165 170 175
Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Tyr Tyr Ala Asp Ser Val
180 185 190
Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr
195 200 205
Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys
210 215 220
Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr
225 230 235 240
Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly Gly Gly Ser
245 250 255
Gly Gly Gly Ser Gln Val Gln Leu Val Glu Ser Gly Gly Ala Leu Val
260 265 270
Gln Pro Gly Gly Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Phe Pro
275 280 285
Val Asn Arg Tyr Ser Met Arg Trp Tyr Arg Gln Ala Pro Gly Lys Glu
290 295 300
Arg Glu Trp Val Ala Gly Met Ser Ser Ala Gly Asp Arg Ser Ser Tyr
305 310 315 320
Glu Asp Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp Asp Ala Arg
325 330 335
Asn Thr Val Tyr Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Asn Val Asn Val Gly Phe Glu Tyr Trp Gly Gln Gly
355 360 365
Thr Gln Val Thr Val Ser Ser His His His His His His
370 375 380
<210> 103
<211> 1143
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 28
<400> 103
caaaccgttg ttacccaaga gcctagtttg acagtttctc ccggtgggac agtcactttg 60
acttgtggat catccacggg agctgtgacc tcagggaact acccgaattg ggttcagcag 120
aagcctggac aagcaccacg aggtttgata ggagattaca aagatgacga tgataaaggc 180
acccccgctc gattcagtgg gtctctcctt ggcgggaagg ctgctctcac cttgagcggg 240
gtgcagccag aggatgaagc tgagtactac tgcgttctgt ggtatagcaa ccggtgggtt 300
ttcggagggg gtacgaaatt gactgtcctc ggcggtgggg gtgactataa ggatgacgac 360
gataagggcg gtgggtctga ggtccaactc gtggaaagtg gaggaggtct tgtacaaccg 420
ggcgggtcac ttaagctcag ttgcgcggcc tcaggcttca ctttcaataa gtatgccatg 480
aactgggtga gacaggctcc cggaaaggga cttgaatggg tcgcccgaat taggtctaag 540
tataacaatt acgcaaccta ttacgctgat tcagtaaaag accggtttac aatttcacgc 600
gacgatagta agaacaccgc atatctccag atgaataact tgaagaccga ggatactgcc 660
gtatattatt gtgttcgaca tggcaatttc ggaaactcat atataagcta ctgggcgtac 720
tgggggcagg gtactcttgt taccgtatct tctgggggtg gaggttcagg gggcggttcc 780
caggttcaac tggtagaaag cgggggtgct ttggtgcaac ccggtggctc actgcgattg 840
tcctgtgccg cttcaggttt ccccgtaaac cggtactcca tgagatggta tcgacaggcg 900
ccgggcaagg aacgcgagtg ggttgcaggg atgtctagcg ccggtgatcg gtcttcttac 960
gaagattcag tcaaaggacg attcaccatc tcccgcgatg acgcgaggaa cactgtatac 1020
ctgcaaatga actctcttaa gcccgaagac accgctgtct actactgtaa cgtaaatgtc 1080
gggttcgagt actggggcca aggcacccaa gtgacggttt ccagtcacca ccaccatcat 1140
cac 1143
<210> 104
<211> 513
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 29
<400> 104
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn
275 280 285
Lys Ser Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Ala Tyr
305 310 315 320
Ala Ala Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys
325 330 335
Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Ala
355 360 365
Phe Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
370 375 380
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
385 390 395 400
Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala
405 410 415
Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala
420 425 430
Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg
435 440 445
Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
450 455 460
Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro
465 470 475 480
Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val
485 490 495
Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His
500 505 510
His
<210> 105
<211> 1539
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 29
<400> 105
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtcttcg gaggagggac gaaacttact 720
gtacttggag gcggcggtga ctacaaggac gacgatgaca aaggcggcgg cagcgaggtc 780
cagttggtag aatccggagg tggattggtt caaccgggag gaagccttaa gctttcatgc 840
gccgcatccg gattcacctt caataagtcc gcaatgaatt gggttagaca ggcaccaggt 900
aaagggttgg aatgggtggc acgcattagg tccaagtaca acaactacgc caccgcctac 960
gcggccagcg taaaagaccg atttacgata agccgggatg attctaagaa cactgcttat 1020
ttgcagatga ataatttgaa gaccgaggat actgctgtct attattgcgt ccgccacggt 1080
aattttggta actcttacat tgccttttgg gcgtattggg ggcagggcac tctggtcacc 1140
gtctcatctg gcggaggggg cagtggcggc gggtcagagg ttcaacttgt cgagtctgga 1200
ggcggtctcg tacaaccggg gaatagtctc cgactctctt gcgctgcgtc cgggttcacg 1260
ttctcaaagt ttgggatgtc ttgggttagg caagccccag gtaagggact cgaatgggtc 1320
agcagcatct caggctccgg cagagacacg ttgtatgccg aaagtgtcaa agggaggttc 1380
acaatctctc gggacaatgc aaaaaccacc ttgtatctcc aaatgaactc actccggcct 1440
gaggacacag cagtttacta ctgtacgata ggagggtccc ttagcgtatc ttctcaggga 1500
actttggtaa cggtcagctc ccaccaccat catcatcac 1539
<210> 106
<211> 513
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 30
<400> 106
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Asn
275 280 285
Lys Ser Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn Asn Tyr Ala Thr Ala Tyr
305 310 315 320
Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys
325 330 335
Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Val Arg His Gly Asn Phe Gly Asn Ser Tyr Ile Thr
355 360 365
Phe Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
370 375 380
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
385 390 395 400
Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala
405 410 415
Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala
420 425 430
Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg
435 440 445
Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
450 455 460
Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro
465 470 475 480
Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val
485 490 495
Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His
500 505 510
His
<210> 107
<211> 1539
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 30
<400> 107
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtcttcg gaggagggac gaaacttact 720
gtacttggag gcggcggtga ctacaaggac gacgatgaca aaggcggcgg cagcgaggtc 780
cagttggtag aatccggagg tggattggtt caaccgggag gaagccttaa gctttcatgc 840
gccgcatccg gattcacctt caataagtcc gcaatgaatt gggttagaca ggcaccaggt 900
aaagggttgg aatgggtggc acgcattagg tccaagtaca acaactacgc caccgcctac 960
gcggacagcg taaaagaccg atttacgata agccgggatg attctaagaa cactgcttat 1020
ttgcagatga ataatttgaa gaccgaggat actgctgtct attattgcgt ccgccacggt 1080
aattttggta actcttacat taccttttgg gcgtattggg ggcagggcac tctggtcacc 1140
gtctcatctg gcggaggggg cagtggcggc gggtcagagg ttcaacttgt cgagtctgga 1200
ggcggtctcg tacaaccggg gaatagtctc cgactctctt gcgctgcgtc cgggttcacg 1260
ttctcaaagt ttgggatgtc ttgggttagg caagccccag gtaagggact cgaatgggtc 1320
agcagcatct caggctccgg cagagacacg ttgtatgccg aaagtgtcaa agggaggttc 1380
acaatctctc gggacaatgc aaaaaccacc ttgtatctcc aaatgaactc actccggcct 1440
gaggacacag cagtttacta ctgtacgata ggagggtccc ttagcgtatc ttctcaggga 1500
actttggtaa cggtcagctc ccaccaccat catcatcac 1539
<210> 108
<211> 513
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 31
<400> 108
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser
275 280 285
Gly Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Ala Arg Ile Arg Ser Lys Ala Asn Ser Tyr Ala Thr Glu Tyr
305 310 315 320
Ala Ala Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys
325 330 335
Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Val Arg His Gly Asn Ala Gly Asn Ser Ala Ile Ser
355 360 365
Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
370 375 380
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
385 390 395 400
Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala
405 410 415
Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala
420 425 430
Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg
435 440 445
Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
450 455 460
Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro
465 470 475 480
Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val
485 490 495
Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His
500 505 510
His
<210> 109
<211> 1539
<212> Белок
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 31
<400> 109
Cys Ala Ala Gly Thr Cys Ala Ala Ala Cys Thr Thr Gly Ala Gly Gly
1 5 10 15
Ala Ala Ala Gly Cys Gly Gly Gly Gly Gly Ala Gly Gly Thr Ala Gly
20 25 30
Cys Gly Thr Ala Cys Ala Gly Ala Cys Thr Gly Gly Thr Gly Gly Ala
35 40 45
Thr Cys Thr Cys Thr Gly Ala Gly Gly Thr Thr Gly Ala Cys Thr Thr
50 55 60
Gly Cys Gly Cys Cys Gly Cys Cys Ala Gly Thr Gly Gly Cys Cys Gly
65 70 75 80
Ala Ala Cys Ala Thr Cys Cys Ala Gly Ala Ala Gly Thr Thr Ala Cys
85 90 95
Gly Gly Gly Ala Thr Gly Gly Gly Thr Thr Gly Gly Thr Thr Thr Cys
100 105 110
Gly Ala Cys Ala Gly Gly Cys Thr Cys Cys Gly Gly Gly Ala Ala Ala
115 120 125
Ala Gly Ala Gly Cys Gly Gly Gly Ala Gly Thr Thr Thr Gly Thr Ala
130 135 140
Thr Cys Thr Gly Gly Cys Ala Thr Ala Ala Gly Cys Thr Gly Gly Ala
145 150 155 160
Gly Gly Gly Gly Cys Gly Ala Cys Thr Cys Cys Ala Cys Thr Gly Gly
165 170 175
Thr Thr Ala Cys Gly Cys Ala Gly Ala Thr Thr Cys Cys Gly Thr Cys
180 185 190
Ala Ala Ala Gly Gly Gly Cys Gly Gly Thr Thr Thr Ala Cys Gly Ala
195 200 205
Thr Cys Thr Cys Thr Cys Gly Gly Gly Ala Thr Ala Ala Cys Gly Cys
210 215 220
Gly Ala Ala Gly Ala Ala Thr Ala Cys Cys Gly Thr Thr Gly Ala Thr
225 230 235 240
Cys Thr Cys Cys Ala Ala Ala Thr Gly Ala Ala Cys Thr Cys Thr Cys
245 250 255
Thr Thr Ala Ala Ala Cys Cys Cys Gly Ala Gly Gly Ala Thr Ala Cys
260 265 270
Ala Gly Cys Ala Ala Thr Ala Thr Ala Cys Thr Ala Thr Thr Gly Cys
275 280 285
Gly Cys Cys Gly Cys Cys Gly Cys Thr Gly Cys Gly Gly Gly Gly Thr
290 295 300
Cys Ala Gly Cys Cys Thr Gly Gly Thr Ala Thr Gly Gly Cys Ala Cys
305 310 315 320
Ala Thr Thr Gly Thr Ala Cys Gly Ala Ala Thr Ala Thr Gly Ala Cys
325 330 335
Thr Ala Thr Thr Gly Gly Gly Gly Thr Cys Ala Ala Gly Gly Thr Ala
340 345 350
Cys Cys Cys Ala Ala Gly Thr Ala Ala Cys Gly Gly Thr Cys Ala Gly
355 360 365
Thr Thr Cys Cys Gly Gly Thr Gly Gly Thr Gly Gly Gly Gly Gly Gly
370 375 380
Thr Cys Thr Gly Gly Thr Gly Gly Thr Gly Gly Ala Thr Cys Cys Cys
385 390 395 400
Ala Ala Ala Cys Ala Gly Thr Thr Gly Thr Thr Ala Cys Thr Cys Ala
405 410 415
Gly Gly Ala Gly Cys Cys Ala Thr Cys Cys Thr Thr Gly Ala Cys Ala
420 425 430
Gly Thr Ala Thr Cys Cys Cys Cys Cys Gly Gly Thr Gly Gly Ala Ala
435 440 445
Cys Gly Gly Thr Gly Ala Cys Cys Cys Thr Gly Ala Cys Ala Thr Gly
450 455 460
Cys Gly Cys Thr Thr Cys Ala Ala Gly Thr Ala Cys Ala Gly Gly Thr
465 470 475 480
Gly Cys Thr Gly Thr Ala Ala Cys Cys Thr Cys Ala Gly Gly Thr Ala
485 490 495
Ala Cys Thr Ala Cys Cys Cys Gly Ala Ala Thr Thr Gly Gly Gly Thr
500 505 510
Gly Cys Ala Gly Cys Ala Ala Ala Ala Ala Cys Cys Thr Gly Gly Ala
515 520 525
Cys Ala Ala Gly Cys Ala Cys Cys Cys Cys Gly Gly Gly Gly Thr Cys
530 535 540
Thr Thr Ala Thr Cys Gly Gly Gly Gly Gly Gly Ala Cys Gly Ala Ala
545 550 555 560
Gly Thr Thr Cys Thr Thr Gly Gly Thr Ala Cys Cys Gly Gly Gly Thr
565 570 575
Ala Cys Cys Cys Cys Thr Gly Cys Gly Cys Gly Cys Thr Thr Cys Ala
580 585 590
Gly Cys Gly Gly Ala Ala Gly Thr Cys Thr Thr Cys Thr Gly Gly Gly
595 600 605
Thr Gly Gly Ala Ala Ala Ala Gly Cys Cys Gly Cys Cys Thr Thr Gly
610 615 620
Ala Cys Cys Thr Thr Gly Thr Cys Ala Gly Gly Cys Gly Thr Thr Cys
625 630 635 640
Ala Gly Cys Cys Cys Gly Ala Ala Gly Ala Thr Gly Ala Gly Gly Cys
645 650 655
Cys Gly Ala Ala Thr Ala Thr Thr Ala Thr Thr Gly Cys Ala Cys Gly
660 665 670
Cys Thr Gly Thr Gly Gly Thr Ala Thr Thr Cys Thr Ala Ala Cys Cys
675 680 685
Gly Gly Thr Gly Gly Gly Thr Cys Thr Thr Cys Gly Gly Ala Gly Gly
690 695 700
Ala Gly Gly Gly Ala Cys Gly Ala Ala Ala Cys Thr Thr Ala Cys Thr
705 710 715 720
Gly Thr Ala Cys Thr Thr Gly Gly Ala Gly Gly Cys Gly Gly Cys Gly
725 730 735
Gly Thr Gly Ala Cys Thr Ala Cys Ala Ala Gly Gly Ala Cys Gly Ala
740 745 750
Cys Gly Ala Thr Gly Ala Cys Ala Ala Ala Gly Gly Cys Gly Gly Cys
755 760 765
Gly Gly Cys Ala Gly Cys Gly Ala Gly Gly Thr Cys Cys Ala Gly Thr
770 775 780
Thr Gly Gly Thr Ala Gly Ala Ala Thr Cys Cys Gly Gly Ala Gly Gly
785 790 795 800
Thr Gly Gly Ala Thr Thr Gly Gly Thr Thr Cys Ala Ala Cys Cys Gly
805 810 815
Gly Gly Ala Gly Gly Ala Ala Gly Cys Cys Thr Thr Ala Ala Gly Cys
820 825 830
Thr Thr Thr Cys Ala Thr Gly Cys Gly Cys Cys Gly Cys Ala Thr Cys
835 840 845
Cys Gly Gly Ala Thr Thr Cys Ala Cys Cys Thr Thr Cys Ala Gly Thr
850 855 860
Gly Gly Gly Thr Ala Cys Gly Cys Ala Ala Thr Gly Ala Ala Thr Thr
865 870 875 880
Gly Gly Gly Thr Thr Ala Gly Ala Cys Ala Gly Gly Cys Ala Cys Cys
885 890 895
Ala Gly Gly Thr Ala Ala Ala Gly Gly Gly Thr Thr Gly Gly Ala Ala
900 905 910
Thr Gly Gly Gly Thr Gly Gly Cys Ala Cys Gly Cys Ala Thr Thr Ala
915 920 925
Gly Gly Thr Cys Cys Ala Ala Gly Gly Cys Cys Ala Ala Cys Ala Gly
930 935 940
Cys Thr Ala Cys Gly Cys Cys Ala Cys Cys Gly Ala Gly Thr Ala Cys
945 950 955 960
Gly Cys Gly Gly Cys Cys Ala Gly Cys Gly Thr Ala Ala Ala Ala Gly
965 970 975
Ala Cys Cys Gly Ala Thr Thr Thr Ala Cys Gly Ala Thr Ala Ala Gly
980 985 990
Cys Cys Gly Gly Gly Ala Thr Gly Ala Thr Thr Cys Thr Ala Ala Gly
995 1000 1005
Ala Ala Cys Ala Cys Thr Gly Cys Thr Thr Ala Thr Thr Thr Gly
1010 1015 1020
Cys Ala Gly Ala Thr Gly Ala Ala Thr Ala Ala Thr Thr Thr Gly
1025 1030 1035
Ala Ala Gly Ala Cys Cys Gly Ala Gly Gly Ala Thr Ala Cys Thr
1040 1045 1050
Gly Cys Thr Gly Thr Cys Thr Ala Thr Thr Ala Thr Thr Gly Cys
1055 1060 1065
Gly Thr Cys Cys Gly Cys Cys Ala Cys Gly Gly Thr Ala Ala Thr
1070 1075 1080
Gly Cys Thr Gly Gly Thr Ala Ala Cys Thr Cys Thr Gly Cys Cys
1085 1090 1095
Ala Thr Thr Ala Gly Cys Thr Ala Thr Thr Gly Gly Gly Cys Gly
1100 1105 1110
Thr Ala Thr Thr Gly Gly Gly Gly Gly Cys Ala Gly Gly Gly Cys
1115 1120 1125
Ala Cys Thr Cys Thr Gly Gly Thr Cys Ala Cys Cys Gly Thr Cys
1130 1135 1140
Thr Cys Ala Thr Cys Thr Gly Gly Cys Gly Gly Ala Gly Gly Gly
1145 1150 1155
Gly Gly Cys Ala Gly Thr Gly Gly Cys Gly Gly Cys Gly Gly Gly
1160 1165 1170
Thr Cys Ala Gly Ala Gly Gly Thr Thr Cys Ala Ala Cys Thr Thr
1175 1180 1185
Gly Thr Cys Gly Ala Gly Thr Cys Thr Gly Gly Ala Gly Gly Cys
1190 1195 1200
Gly Gly Thr Cys Thr Cys Gly Thr Ala Cys Ala Ala Cys Cys Gly
1205 1210 1215
Gly Gly Gly Ala Ala Thr Ala Gly Thr Cys Thr Cys Cys Gly Ala
1220 1225 1230
Cys Thr Cys Thr Cys Thr Thr Gly Cys Gly Cys Thr Gly Cys Gly
1235 1240 1245
Thr Cys Cys Gly Gly Gly Thr Thr Cys Ala Cys Gly Thr Thr Cys
1250 1255 1260
Thr Cys Ala Ala Ala Gly Thr Thr Thr Gly Gly Gly Ala Thr Gly
1265 1270 1275
Thr Cys Thr Thr Gly Gly Gly Thr Thr Ala Gly Gly Cys Ala Ala
1280 1285 1290
Gly Cys Cys Cys Cys Ala Gly Gly Thr Ala Ala Gly Gly Gly Ala
1295 1300 1305
Cys Thr Cys Gly Ala Ala Thr Gly Gly Gly Thr Cys Ala Gly Cys
1310 1315 1320
Ala Gly Cys Ala Thr Cys Thr Cys Ala Gly Gly Cys Thr Cys Cys
1325 1330 1335
Gly Gly Cys Ala Gly Ala Gly Ala Cys Ala Cys Gly Thr Thr Gly
1340 1345 1350
Thr Ala Thr Gly Cys Cys Gly Ala Ala Ala Gly Thr Gly Thr Cys
1355 1360 1365
Ala Ala Ala Gly Gly Gly Ala Gly Gly Thr Thr Cys Ala Cys Ala
1370 1375 1380
Ala Thr Cys Thr Cys Thr Cys Gly Gly Gly Ala Cys Ala Ala Thr
1385 1390 1395
Gly Cys Ala Ala Ala Ala Ala Cys Cys Ala Cys Cys Thr Thr Gly
1400 1405 1410
Thr Ala Thr Cys Thr Cys Cys Ala Ala Ala Thr Gly Ala Ala Cys
1415 1420 1425
Thr Cys Ala Cys Thr Cys Cys Gly Gly Cys Cys Thr Gly Ala Gly
1430 1435 1440
Gly Ala Cys Ala Cys Ala Gly Cys Ala Gly Thr Thr Thr Ala Cys
1445 1450 1455
Thr Ala Cys Thr Gly Thr Ala Cys Gly Ala Thr Ala Gly Gly Ala
1460 1465 1470
Gly Gly Gly Thr Cys Cys Cys Thr Thr Ala Gly Cys Gly Thr Ala
1475 1480 1485
Thr Cys Thr Thr Cys Thr Cys Ala Gly Gly Gly Ala Ala Cys Thr
1490 1495 1500
Thr Thr Gly Gly Thr Ala Ala Cys Gly Gly Thr Cys Ala Gly Cys
1505 1510 1515
Thr Cys Cys Cys Ala Cys Cys Ala Cys Cys Ala Thr Cys Ala Thr
1520 1525 1530
Cys Ala Thr Cys Ala Cys
1535
<210> 110
<211> 513
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 32
<400> 110
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Gly Gly Asp Tyr Lys Asp Asp Asp Asp Lys Gly Gly
245 250 255
Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro
260 265 270
Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser
275 280 285
Gly His Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu
290 295 300
Trp Val Ala Arg Ile Arg Ser Lys Ala Asn Ser Tyr Ala Thr Tyr Tyr
305 310 315 320
Ala Asp Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys
325 330 335
Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala
340 345 350
Val Tyr Tyr Cys Val Arg His Gly Ala Asn Gly Asn Ser Tyr Ile Ser
355 360 365
Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly
370 375 380
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
385 390 395 400
Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala
405 410 415
Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala
420 425 430
Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg
435 440 445
Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg
450 455 460
Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro
465 470 475 480
Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val
485 490 495
Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His
500 505 510
His
<210> 111
<211> 1539
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 32
<400> 111
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtcttcg gaggagggac gaaacttact 720
gtacttggag gcggcggtga ctacaaggac gacgatgaca aaggcggcgg cagcgaggtc 780
cagttggtag aatccggagg tggattggtt caaccgggag gaagccttaa gctttcatgc 840
gccgcatccg gattcacctt cagtgggcac gcaatgaatt gggttagaca ggcaccaggt 900
aaagggttgg aatgggtggc acgcattagg tccaaggcca acagctacgc cacctactac 960
gcggacagcg taaaagaccg atttacgata agccgggatg attctaagaa cactgcttat 1020
ttgcagatga ataatttgaa gaccgaggat actgctgtct attattgcgt ccgccacggt 1080
gctaatggta actcttacat tagctattgg gcgtattggg ggcagggcac tctggtcacc 1140
gtctcatctg gcggaggggg cagtggcggc gggtcagagg ttcaacttgt cgagtctgga 1200
ggcggtctcg tacaaccggg gaatagtctc cgactctctt gcgctgcgtc cgggttcacg 1260
ttctcaaagt ttgggatgtc ttgggttagg caagccccag gtaagggact cgaatgggtc 1320
agcagcatct caggctccgg cagagacacg ttgtatgccg aaagtgtcaa agggaggttc 1380
acaatctctc gggacaatgc aaaaaccacc ttgtatctcc aaatgaactc actccggcct 1440
gaggacacag cagtttacta ctgtacgata ggagggtccc ttagcgtatc ttctcaggga 1500
actttggtaa cggtcagctc ccaccaccat catcatcac 1539
<210> 112
<211> 515
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 39(MMP9)
<400> 112
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Ser Gly Gly Pro Gly Pro Ala Gly Met Lys Gly
260 265 270
Leu Pro Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val
275 280 285
Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala
290 295 300
Val Thr Ser Gly His Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln
305 310 315 320
Ala Pro Arg Gly Leu Ile Gly Gly Thr Ser Asn Lys His Ser Trp Thr
325 330 335
Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr
340 345 350
Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu
355 360 365
Trp Gly Ser Arg Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val
370 375 380
Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu
385 390 395 400
Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys
405 410 415
Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg
420 425 430
Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser
435 440 445
Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile
450 455 460
Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu
465 470 475 480
Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu
485 490 495
Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His
500 505 510
His His His
515
<210> 113
<211> 1545
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 39(MMP9)
<400> 113
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatc aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
caagtaaagg accgctttac tatctctcga gatgactcta agaacactgc ctatttgcag 660
atgaacaatc ttaaaacaga ggacacagcg gtgtactatt gtgtaagaca tgccaacttt 720
ggaaacagct atattagcta ttgggcttac tgggggcagg gcactctggt caccgtcagt 780
tcctctgggg ggccagggcc agcgggcatg aaaggccttc cgggatccca gactgtggta 840
acacaggaac catctttgac agtaagtcct ggaggtacgg tcacgctcac ttgtgggtcc 900
tcaaccgggg ctgtaacgtc aggccattac cctaactggg tccaacagaa gcctggacaa 960
gctcccaggg gtctgatagg cggaacttca aacaagcact cttggactcc agcgcgcttt 1020
agcggttccc ttctgggtgg aaaagcagcc ctcactctga gtggagtaca acccgaggat 1080
gaggcggaat attattgcgt gctctggggt tcacgccgct gggtcttcgg tggcggtacg 1140
aaacttactg tactgggggg aggcggctca ggcggcggat cagaagtgca gcttgttgaa 1200
tctggcggag gtctggtcca gccaggtaac agcttgagac tgtcctgtgc tgcaagcggc 1260
tttaccttct ctaaattcgg tatgagttgg gttcggcaag cccctggaaa gggtttggaa 1320
tgggtatcaa gcattagtgg ttctgggcga gatacactct atgccgaatc agtgaagggc 1380
cgctttacca ttagtaggga taacgctaaa actactctgt atctgcaaat gaatagtctg 1440
agaccagaag atactgccgt ttactactgc acaatagggg gatctctgag cgtttcatct 1500
caaggtacac ttgtgactgt tagcagtcat catcatcatc accac 1545
<210> 114
<211> 512
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 40(MMP9)
<400> 114
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Ser Gly Gly Pro Gly Pro Ala Gly Met Lys Gly Leu Pro Gly
245 250 255
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
260 265 270
Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Gly
275 280 285
Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
290 295 300
Val Ala Arg Ile Arg Ser Lys Ala Asn Ser Tyr Ala Thr Glu Tyr Ala
305 310 315 320
Ala Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn
325 330 335
Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val
340 345 350
Tyr Tyr Cys Val Arg His Gly Asn Ala Gly Asn Ser Ala Ile Ser Tyr
355 360 365
Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
370 375 380
Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
385 390 395 400
Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala Ser
405 410 415
Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala Pro
420 425 430
Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg Asp
435 440 445
Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
450 455 460
Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro Glu
465 470 475 480
Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val Ser
485 490 495
Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His His
500 505 510
<210> 115
<211> 1536
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 40(MMP9)
<400> 115
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtctttg ggggtggtac gaagttgacc 720
gttctcagcg gtgggccagg accagcaggt atgaaggggt tgcccggctc agaagtccag 780
ttggtagaat ccgggggggg actggttcaa ccaggaggta gtttgaagct ttcatgcgcc 840
gcatccggat tcaccttcag tgggtacgca atgaattggg ttagacaggc accaggtaaa 900
gggttggaat gggtggcacg cattaggtcc aaggccaaca gctacgccac cgagtacgcg 960
gccagcgtaa aagaccgatt tacgataagc cgggatgatt ctaagaacac tgcttatttg 1020
cagatgaata atttgaagac cgaggatact gctgtctatt attgcgtccg ccacggtaat 1080
gctggtaact ctgccattag ctattgggcg tattgggggc agggcactct ggtcaccgtc 1140
tcatctggcg gagggggcag tggcggcggg tcagaggttc aacttgtcga gtctggaggc 1200
ggtctcgtac aaccggggaa tagtctccga ctctcttgcg ctgcgtccgg gttcacgttc 1260
tcaaagtttg ggatgtcttg ggttaggcaa gccccaggta agggactcga atgggtcagc 1320
agcatctcag gctccggcag agacacgttg tatgccgaaa gtgtcaaagg gaggttcaca 1380
atctctcggg acaatgcaaa aaccaccttg tatctccaaa tgaactcact ccggcctgag 1440
gacacagcag tttactactg tacgatagga gggtccctta gcgtatcttc tcagggaact 1500
ttggtaacgg tcagctccca ccaccatcat catcac 1536
<210> 116
<211> 515
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 41 (Mep)
<400> 116
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Ser Gly Gly Gly Lys Lys Leu Ala Asp Glu Pro
260 265 270
Glu Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val
275 280 285
Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala
290 295 300
Val Thr Ser Gly His Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln
305 310 315 320
Ala Pro Arg Gly Leu Ile Gly Gly Thr Ser Asn Lys His Ser Trp Thr
325 330 335
Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr
340 345 350
Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu
355 360 365
Trp Gly Ser Arg Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val
370 375 380
Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu
385 390 395 400
Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys
405 410 415
Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg
420 425 430
Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser
435 440 445
Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile
450 455 460
Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu
465 470 475 480
Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu
485 490 495
Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His
500 505 510
His His His
515
<210> 117
<211> 1545
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 41 (Mep)
<400> 117
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatc aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
caagtaaagg accgctttac tatctctcga gatgattcta aaaacaccgc atatttgcag 660
atgaacaatc ttaaaactga ggacaccgca gtgtattact gcgtgcgaca tgcgaacttc 720
ggtaactctt acatttccta ctgggcgtat tggggccagg gcacgcttgt gacggttagt 780
tctagcggag gtggtaaaaa gctcgctgac gagccagagg gaggatccca gactgtggta 840
acacaggaac catctttgac agtaagtcct ggaggtacgg tcacgctcac ttgtgggtcc 900
tcaaccgggg ctgtaacgtc aggccattac cctaactggg tccaacagaa gcctggacaa 960
gctcccaggg gtctgatagg cggaacttca aacaagcact cttggactcc agcgcgcttt 1020
agcggttccc ttctgggtgg aaaagcagcc ctcactctga gtggagtaca acccgaggat 1080
gaggcggaat attattgcgt gctctggggt tcacgccgct gggtcttcgg tggcggtacg 1140
aaacttactg tactgggggg aggcggctca ggcggcggat cagaagtgca gcttgttgaa 1200
tctggcggag gtctggtcca gccaggtaac agcttgagac tgtcctgtgc tgcaagcggc 1260
tttaccttct ctaaattcgg tatgagttgg gttcggcaag cccctggaaa gggtttggaa 1320
tgggtatcaa gcattagtgg ttctgggcga gatacactct atgccgaatc agtgaagggc 1380
cgctttacca ttagtaggga taacgctaaa actactctgt atctgcaaat gaatagtctg 1440
agaccagaag atactgccgt ttactactgc acaatagggg gatctctgag cgtttcatct 1500
caaggtacac ttgtgactgt tagcagtcat catcatcatc accac 1545
<210> 118
<211> 512
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 42 (Mep)
<400> 118
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Ser Gly Gly Gly Lys Lys Leu Ala Asp Glu Pro Glu Gly Gly
245 250 255
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
260 265 270
Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Gly
275 280 285
Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
290 295 300
Val Ala Arg Ile Arg Ser Lys Ala Asn Ser Tyr Ala Thr Glu Tyr Ala
305 310 315 320
Ala Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn
325 330 335
Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val
340 345 350
Tyr Tyr Cys Val Arg His Gly Asn Ala Gly Asn Ser Ala Ile Ser Tyr
355 360 365
Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
370 375 380
Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
385 390 395 400
Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala Ser
405 410 415
Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala Pro
420 425 430
Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg Asp
435 440 445
Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
450 455 460
Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro Glu
465 470 475 480
Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val Ser
485 490 495
Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His His
500 505 510
<210> 119
<211> 1536
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 42 (Mep)
<400> 119
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtgtttg gtgggggtac aaaattgacg 720
gtcttgtcag ggggtggaaa gaaactggca gatgaacctg agggcggttc tgaggttcag 780
cttgttgaga gcggtggcgg tcttgtgcaa cccggaggct cactcaagct ttcatgcgcc 840
gcatccggat tcaccttcag tgggtacgca atgaattggg ttagacaggc accaggtaaa 900
gggttggaat gggtggcacg cattaggtcc aaggccaaca gctacgccac cgagtacgcg 960
gccagcgtaa aagaccgatt tacgataagc cgggatgatt ctaagaacac tgcttatttg 1020
cagatgaata atttgaagac cgaggatact gctgtctatt attgcgtccg ccacggtaat 1080
gctggtaact ctgccattag ctattgggcg tattgggggc agggcactct ggtcaccgtc 1140
tcatctggcg gagggggcag tggcggcggg tcagaggttc aacttgtcga gtctggaggc 1200
ggtctcgtac aaccggggaa tagtctccga ctctcttgcg ctgcgtccgg gttcacgttc 1260
tcaaagtttg ggatgtcttg ggttaggcaa gccccaggta agggactcga atgggtcagc 1320
agcatctcag gctccggcag agacacgttg tatgccgaaa gtgtcaaagg gaggttcaca 1380
atctctcggg acaatgcaaa aaccaccttg tatctccaaa tgaactcact ccggcctgag 1440
gacacagcag tttactactg tacgatagga gggtccctta gcgtatcttc tcagggaact 1500
ttggtaacgg tcagctccca ccaccatcat catcac 1536
<210> 120
<211> 515
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 43
<400> 120
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Gly Gly Ser Phe Thr Arg Gln Ala Arg Val Val
260 265 270
Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val
275 280 285
Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala
290 295 300
Val Thr Ser Gly His Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln
305 310 315 320
Ala Pro Arg Gly Leu Ile Gly Gly Thr Ser Asn Lys His Ser Trp Thr
325 330 335
Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr
340 345 350
Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu
355 360 365
Trp Gly Ser Arg Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val
370 375 380
Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu
385 390 395 400
Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys
405 410 415
Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg
420 425 430
Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser
435 440 445
Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile
450 455 460
Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu
465 470 475 480
Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu
485 490 495
Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His
500 505 510
His His His
515
<210> 121
<211> 1545
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 43
<400> 121
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatc aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
caagtaaagg accgctttac tatctctcga gatgactcca agaatacagc ttaccttcaa 660
atgaataatc tgaagacaga ggataccgcc gtgtattact gcgtccgaca tgcgaacttt 720
ggtaattctt atatctctta ttgggcctat tggggtcagg gcacgttggt taccgtttct 780
tctggaggtt cattcacccg ccaggcgcga gttgtcggtg gaggatccca gactgtggta 840
acacaggaac catctttgac agtaagtcct ggaggtacgg tcacgctcac ttgtgggtcc 900
tcaaccgggg ctgtaacgtc aggccattac cctaactggg tccaacagaa gcctggacaa 960
gctcccaggg gtctgatagg cggaacttca aacaagcact cttggactcc agcgcgcttt 1020
agcggttccc ttctgggtgg aaaagcagcc ctcactctga gtggagtaca acccgaggat 1080
gaggcggaat attattgcgt gctctggggt tcacgccgct gggtcttcgg tggcggtacg 1140
aaacttactg tactgggggg aggcggctca ggcggcggat cagaagtgca gcttgttgaa 1200
tctggcggag gtctggtcca gccaggtaac agcttgagac tgtcctgtgc tgcaagcggc 1260
tttaccttct ctaaattcgg tatgagttgg gttcggcaag cccctggaaa gggtttggaa 1320
tgggtatcaa gcattagtgg ttctgggcga gatacactct atgccgaatc agtgaagggc 1380
cgctttacca ttagtaggga taacgctaaa actactctgt atctgcaaat gaatagtctg 1440
agaccagaag atactgccgt ttactactgc acaatagggg gatctctgag cgtttcatct 1500
caaggtacac ttgtgactgt tagcagtcat catcatcatc accac 1545
<210> 122
<211> 512
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 44
<400> 122
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Gly Gly Ser Phe Thr Arg Gln Ala Arg Val Val Gly Gly Gly
245 250 255
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
260 265 270
Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Gly
275 280 285
Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
290 295 300
Val Ala Arg Ile Arg Ser Lys Ala Asn Ser Tyr Ala Thr Glu Tyr Ala
305 310 315 320
Ala Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn
325 330 335
Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val
340 345 350
Tyr Tyr Cys Val Arg His Gly Asn Ala Gly Asn Ser Ala Ile Ser Tyr
355 360 365
Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
370 375 380
Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
385 390 395 400
Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala Ser
405 410 415
Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala Pro
420 425 430
Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg Asp
435 440 445
Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
450 455 460
Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro Glu
465 470 475 480
Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val Ser
485 490 495
Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His His
500 505 510
<210> 123
<211> 1536
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 44
<400> 123
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtgtttg gtggaggcac gaaactgacg 720
gtattggggg gatcatttac gcgccaagct agagtcgtgg gaggtggatc agaggtccag 780
ttggtcgaga gcgggggggg tctggtccaa ccagggggta gtctcaagct ttcatgcgcc 840
gcatccggat tcaccttcag tgggtacgca atgaattggg ttagacaggc accaggtaaa 900
gggttggaat gggtggcacg cattaggtcc aaggccaaca gctacgccac cgagtacgcg 960
gccagcgtaa aagaccgatt tacgataagc cgggatgatt ctaagaacac tgcttatttg 1020
cagatgaata atttgaagac cgaggatact gctgtctatt attgcgtccg ccacggtaat 1080
gctggtaact ctgccattag ctattgggcg tattgggggc agggcactct ggtcaccgtc 1140
tcatctggcg gagggggcag tggcggcggg tcagaggttc aacttgtcga gtctggaggc 1200
ggtctcgtac aaccggggaa tagtctccga ctctcttgcg ctgcgtccgg gttcacgttc 1260
tcaaagtttg ggatgtcttg ggttaggcaa gccccaggta agggactcga atgggtcagc 1320
agcatctcag gctccggcag agacacgttg tatgccgaaa gtgtcaaagg gaggttcaca 1380
atctctcggg acaatgcaaa aaccaccttg tatctccaaa tgaactcact ccggcctgag 1440
gacacagcag tttactactg tacgatagga gggtccctta gcgtatcttc tcagggaact 1500
ttggtaacgg tcagctccca ccaccatcat catcac 1536
<210> 124
<211> 515
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 45 (Thb)
<400> 124
Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Ala Gly Gly
1 5 10 15
Ser Leu Arg Leu Ser Cys Ala Ala Ser Gly Arg Thr Phe Ser Ser Tyr
20 25 30
Ala Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Val Ala Ile Asn Trp Ser Ser Gly Ser Thr Tyr Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Met Tyr
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Val Tyr Tyr Cys
85 90 95
Ala Ala Gly Tyr Gln Ile Asn Ser Gly Asn Tyr Asn Phe Lys Asp Tyr
100 105 110
Glu Tyr Asp Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly
115 120 125
Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly
130 135 140
Gly Gly Leu Val Gln Pro Gly Gly Ser Leu Lys Leu Ser Cys Ala Ala
145 150 155 160
Ser Gly Phe Thr Phe Asn Lys Tyr Ala Ile Asn Trp Val Arg Gln Ala
165 170 175
Pro Gly Lys Gly Leu Glu Trp Val Ala Arg Ile Arg Ser Lys Tyr Asn
180 185 190
Asn Tyr Ala Thr Tyr Tyr Ala Asp Gln Val Lys Asp Arg Phe Thr Ile
195 200 205
Ser Arg Asp Asp Ser Lys Asn Thr Ala Tyr Leu Gln Met Asn Asn Leu
210 215 220
Lys Thr Glu Asp Thr Ala Val Tyr Tyr Cys Val Arg His Ala Asn Phe
225 230 235 240
Gly Asn Ser Tyr Ile Ser Tyr Trp Ala Tyr Trp Gly Gln Gly Thr Leu
245 250 255
Val Thr Val Ser Ser Ser Ser Gly Gly Gly Met Pro Arg Ser Phe Arg
260 265 270
Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr Val
275 280 285
Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Gly Ser Ser Thr Gly Ala
290 295 300
Val Thr Ser Gly His Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly Gln
305 310 315 320
Ala Pro Arg Gly Leu Ile Gly Gly Thr Ser Asn Lys His Ser Trp Thr
325 330 335
Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu Thr
340 345 350
Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Val Leu
355 360 365
Trp Gly Ser Arg Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr Val
370 375 380
Leu Gly Gly Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu
385 390 395 400
Ser Gly Gly Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys
405 410 415
Ala Ala Ser Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg
420 425 430
Gln Ala Pro Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser
435 440 445
Gly Arg Asp Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile
450 455 460
Ser Arg Asp Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu
465 470 475 480
Arg Pro Glu Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu
485 490 495
Ser Val Ser Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His
500 505 510
His His His
515
<210> 125
<211> 1545
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 45 (Thb)
<400> 125
gaagttcaac tggttgaatc cggtggtggc cttgtccagg cgggaggctc ccttcgactg 60
tcttgtgccg ccagcggtcg cacattcagc agttatgcca tgggatggtt ccggcaggcc 120
cctggtaaag agcgggagtt cgtcgttgcg atcaattgga gtagcggttc cacgtattat 180
gcggattctg taaagggcag gttcactatc tcacgcgata atgcaaaaaa taccatgtat 240
cttcagatga actcactgaa gcccgaggac acggcagttt attactgtgc tgccggttac 300
cagatcaatt ccggaaatta caatttcaag gactacgagt acgattattg gggtcagggc 360
acccaggtaa ccgtcagcag cgggggaggc ggatcaggag gcggttcaga ggttcagctc 420
gttgagagtg gtggagggct ggttcagcca gggggaagtt tgaagctttc ctgtgcggcc 480
tctggtttca cctttaacaa atacgctatc aactgggtac gacaagcccc cggtaaaggg 540
cttgaatggg ttgcaagaat acgcagtaaa tacaataatt atgcgactta ttatgccgat 600
caagtaaagg accgctttac tatctctcga gatgactcaa agaatacagc atatctgcaa 660
atgaacaatt tgaaaacaga agacacggca gtttattact gcgttaggca cgctaacttc 720
ggtaattcat acatatcata ttgggcctac tggggccaag ggactttggt cacagtatcc 780
tccagctcag ggggtggtat gcctcgctct ttcagggggg gcggatccca gactgtggta 840
acacaggaac catctttgac agtaagtcct ggaggtacgg tcacgctcac ttgtgggtcc 900
tcaaccgggg ctgtaacgtc aggccattac cctaactggg tccaacagaa gcctggacaa 960
gctcccaggg gtctgatagg cggaacttca aacaagcact cttggactcc agcgcgcttt 1020
agcggttccc ttctgggtgg aaaagcagcc ctcactctga gtggagtaca acccgaggat 1080
gaggcggaat attattgcgt gctctggggt tcacgccgct gggtcttcgg tggcggtacg 1140
aaacttactg tactgggggg aggcggctca ggcggcggat cagaagtgca gcttgttgaa 1200
tctggcggag gtctggtcca gccaggtaac agcttgagac tgtcctgtgc tgcaagcggc 1260
tttaccttct ctaaattcgg tatgagttgg gttcggcaag cccctggaaa gggtttggaa 1320
tgggtatcaa gcattagtgg ttctgggcga gatacactct atgccgaatc agtgaagggc 1380
cgctttacca ttagtaggga taacgctaaa actactctgt atctgcaaat gaatagtctg 1440
agaccagaag atactgccgt ttactactgc acaatagggg gatctctgag cgtttcatct 1500
caaggtacac ttgtgactgt tagcagtcat catcatcatc accac 1545
<210> 126
<211> 512
<212> Белок
<213> Искусственная последовательность
<220>
<223> Полипептидный конструкт Prodent 46 (Thb)
<400> 126
Gln Val Lys Leu Glu Glu Ser Gly Gly Gly Ser Val Gln Thr Gly Gly
1 5 10 15
Ser Leu Arg Leu Thr Cys Ala Ala Ser Gly Arg Thr Ser Arg Ser Tyr
20 25 30
Gly Met Gly Trp Phe Arg Gln Ala Pro Gly Lys Glu Arg Glu Phe Val
35 40 45
Ser Gly Ile Ser Trp Arg Gly Asp Ser Thr Gly Tyr Ala Asp Ser Val
50 55 60
Lys Gly Arg Phe Thr Ile Ser Arg Asp Asn Ala Lys Asn Thr Val Asp
65 70 75 80
Leu Gln Met Asn Ser Leu Lys Pro Glu Asp Thr Ala Ile Tyr Tyr Cys
85 90 95
Ala Ala Ala Ala Gly Ser Ala Trp Tyr Gly Thr Leu Tyr Glu Tyr Asp
100 105 110
Tyr Trp Gly Gln Gly Thr Gln Val Thr Val Ser Ser Gly Gly Gly Gly
115 120 125
Ser Gly Gly Gly Ser Gln Thr Val Val Thr Gln Glu Pro Ser Leu Thr
130 135 140
Val Ser Pro Gly Gly Thr Val Thr Leu Thr Cys Ala Ser Ser Thr Gly
145 150 155 160
Ala Val Thr Ser Gly Asn Tyr Pro Asn Trp Val Gln Gln Lys Pro Gly
165 170 175
Gln Ala Pro Arg Gly Leu Ile Gly Gly Thr Lys Phe Leu Val Pro Gly
180 185 190
Thr Pro Ala Arg Phe Ser Gly Ser Leu Leu Gly Gly Lys Ala Ala Leu
195 200 205
Thr Leu Ser Gly Val Gln Pro Glu Asp Glu Ala Glu Tyr Tyr Cys Thr
210 215 220
Leu Trp Tyr Ser Asn Arg Trp Val Phe Gly Gly Gly Thr Lys Leu Thr
225 230 235 240
Val Leu Ser Ser Gly Gly Gly Met Pro Arg Ser Phe Arg Gly Gly Gly
245 250 255
Ser Glu Val Gln Leu Val Glu Ser Gly Gly Gly Leu Val Gln Pro Gly
260 265 270
Gly Ser Leu Lys Leu Ser Cys Ala Ala Ser Gly Phe Thr Phe Ser Gly
275 280 285
Tyr Ala Met Asn Trp Val Arg Gln Ala Pro Gly Lys Gly Leu Glu Trp
290 295 300
Val Ala Arg Ile Arg Ser Lys Ala Asn Ser Tyr Ala Thr Glu Tyr Ala
305 310 315 320
Ala Ser Val Lys Asp Arg Phe Thr Ile Ser Arg Asp Asp Ser Lys Asn
325 330 335
Thr Ala Tyr Leu Gln Met Asn Asn Leu Lys Thr Glu Asp Thr Ala Val
340 345 350
Tyr Tyr Cys Val Arg His Gly Asn Ala Gly Asn Ser Ala Ile Ser Tyr
355 360 365
Trp Ala Tyr Trp Gly Gln Gly Thr Leu Val Thr Val Ser Ser Gly Gly
370 375 380
Gly Gly Ser Gly Gly Gly Ser Glu Val Gln Leu Val Glu Ser Gly Gly
385 390 395 400
Gly Leu Val Gln Pro Gly Asn Ser Leu Arg Leu Ser Cys Ala Ala Ser
405 410 415
Gly Phe Thr Phe Ser Lys Phe Gly Met Ser Trp Val Arg Gln Ala Pro
420 425 430
Gly Lys Gly Leu Glu Trp Val Ser Ser Ile Ser Gly Ser Gly Arg Asp
435 440 445
Thr Leu Tyr Ala Glu Ser Val Lys Gly Arg Phe Thr Ile Ser Arg Asp
450 455 460
Asn Ala Lys Thr Thr Leu Tyr Leu Gln Met Asn Ser Leu Arg Pro Glu
465 470 475 480
Asp Thr Ala Val Tyr Tyr Cys Thr Ile Gly Gly Ser Leu Ser Val Ser
485 490 495
Ser Gln Gly Thr Leu Val Thr Val Ser Ser His His His His His His
500 505 510
<210> 127
<211> 1536
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Репрезентативная нуклеиновая кислота Prodent 46 (Thb)
<400> 127
caagtcaaac ttgaggaaag cgggggaggt agcgtacaga ctggtggatc tctgaggttg 60
acttgcgccg ccagtggccg aacatccaga agttacggga tgggttggtt tcgacaggct 120
ccgggaaaag agcgggagtt tgtatctggc ataagctgga ggggcgactc cactggttac 180
gcagattccg tcaaagggcg gtttacgatc tctcgggata acgcgaagaa taccgttgat 240
ctccaaatga actctcttaa acccgaggat acagcaatat actattgcgc cgccgctgcg 300
gggtcagcct ggtatggcac attgtacgaa tatgactatt ggggtcaagg tacccaagta 360
acggtcagtt ccggtggtgg ggggtctggt ggtggatccc aaacagttgt tactcaggag 420
ccatccttga cagtatcccc cggtggaacg gtgaccctga catgcgcttc aagtacaggt 480
gctgtaacct caggtaacta cccgaattgg gtgcagcaaa aacctggaca agcaccccgg 540
ggtcttatcg gggggacgaa gttcttggta ccgggtaccc ctgcgcgctt cagcggaagt 600
cttctgggtg gaaaagccgc cttgaccttg tcaggcgttc agcccgaaga tgaggccgaa 660
tattattgca cgctgtggta ttctaaccgg tgggtcttcg gagggggtac caagctgacg 720
gtgttgtcat ctggcggagg tatgccaagg agctttcgcg gtggaggctc agaagtacaa 780
cttgtagaaa gcgggggggg tctggtccag ccaggcggaa gcctcaagct ttcatgcgcc 840
gcatccggat tcaccttcag tgggtacgca atgaattggg ttagacaggc accaggtaaa 900
gggttggaat gggtggcacg cattaggtcc aaggccaaca gctacgccac cgagtacgcg 960
gccagcgtaa aagaccgatt tacgataagc cgggatgatt ctaagaacac tgcttatttg 1020
cagatgaata atttgaagac cgaggatact gctgtctatt attgcgtccg ccacggtaat 1080
gctggtaact ctgccattag ctattgggcg tattgggggc agggcactct ggtcaccgtc 1140
tcatctggcg gagggggcag tggcggcggg tcagaggttc aacttgtcga gtctggaggc 1200
ggtctcgtac aaccggggaa tagtctccga ctctcttgcg ctgcgtccgg gttcacgttc 1260
tcaaagtttg ggatgtcttg ggttaggcaa gccccaggta agggactcga atgggtcagc 1320
agcatctcag gctccggcag agacacgttg tatgccgaaa gtgtcaaagg gaggttcaca 1380
atctctcggg acaatgcaaa aaccaccttg tatctccaaa tgaactcact ccggcctgag 1440
gacacagcag tttactactg tacgatagga gggtccctta gcgtatcttc tcagggaact 1500
ttggtaacgg tcagctccca ccaccatcat catcac 1536
<---
| название | год | авторы | номер документа |
|---|---|---|---|
| ПОЛИПЕПТИД, СОДЕРЖАЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2017 |
|
RU2827545C2 |
| ПОЛИПЕПТИД, ВКЛЮЧАЮЩИЙ АНТИГЕНСВЯЗЫВАЮЩИЙ ДОМЕН И ТРАНСПОРТИРУЮЩИЙ СЕГМЕНТ | 2018 |
|
RU2815452C2 |
| ХИМЕРНЫЙ АНТИГЕННЫЙ РЕЦЕПТОР | 2019 |
|
RU2810092C2 |
| МУЛЬТИВАЛЕНТНЫЕ FV-АНТИТЕЛА | 2016 |
|
RU2785766C2 |
| АНТИТЕЛО-ПОДОБНЫЕ СВЯЗЫВАЮЩИЕ БЕЛКИ С ДВОЙНЫМИ ВАРИАБЕЛЬНЫМИ ОБЛАСТЯМИ, ИМЕЮЩИЕ ОРИЕНТАЦИЮ СВЯЗЫВАЮЩИХ ОБЛАСТЕЙ КРЕСТ-НАКРЕСТ | 2012 |
|
RU2823693C2 |
| МУЛЬТИСПЕЦИФИЧЕСКИЕ СВЯЗЫВАЮЩИЕ БЕЛКИ НА ОСНОВЕ ПСЕВДО-FAB | 2019 |
|
RU2820254C2 |
| АКТИВИРУЕМЫЕ ПОЛИПЕПТИДЫ ИНТЕРЛЕЙКИНА 12 И СПОСОБЫ ИХ ПРИМЕНЕНИЯ | 2019 |
|
RU2826183C2 |
| СВЯЗЫВАЮЩИЙ ДОМЕН | 2019 |
|
RU2829934C2 |
| ХИМЕРНЫЙ ЦИТОКИНОВЫЙ РЕЦЕПТОР | 2016 |
|
RU2826122C1 |
| АНТИТЕЛА, СОДЕРЖАЩИЕ ПОЛИПЕПТИД, ВСТРОЕННЫЙ В УЧАСТОК КАРКАСНОЙ ОБЛАСТИ 3 | 2019 |
|
RU2796254C2 |
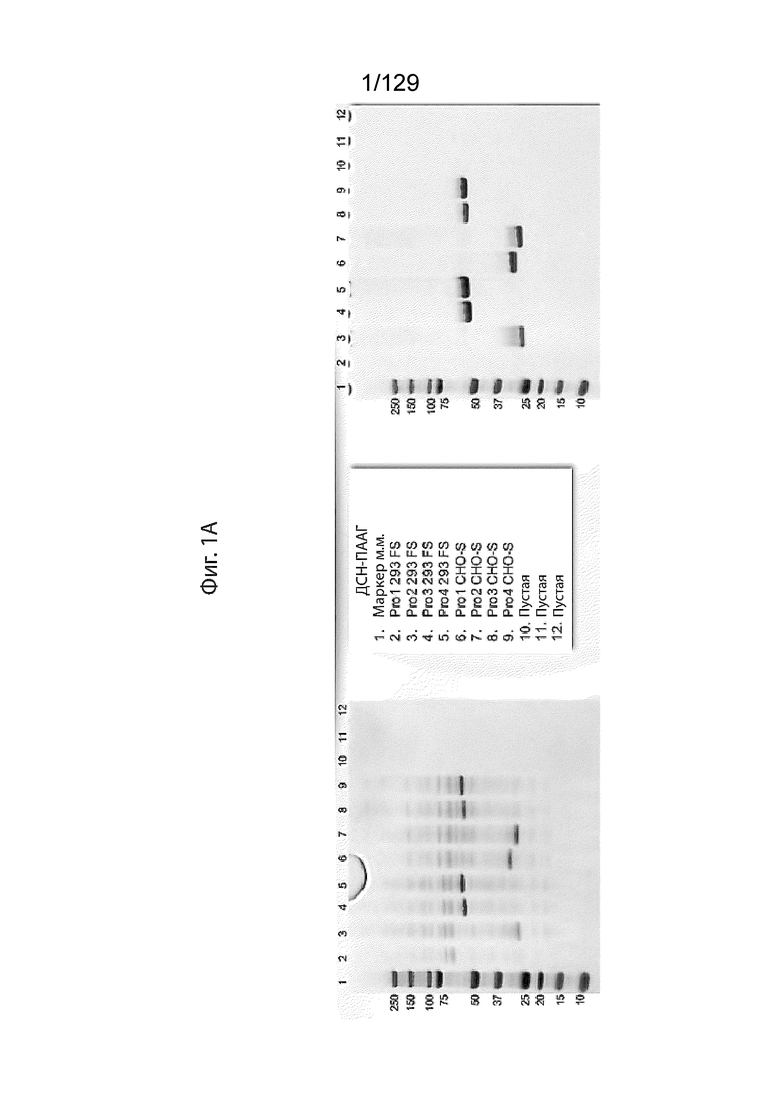

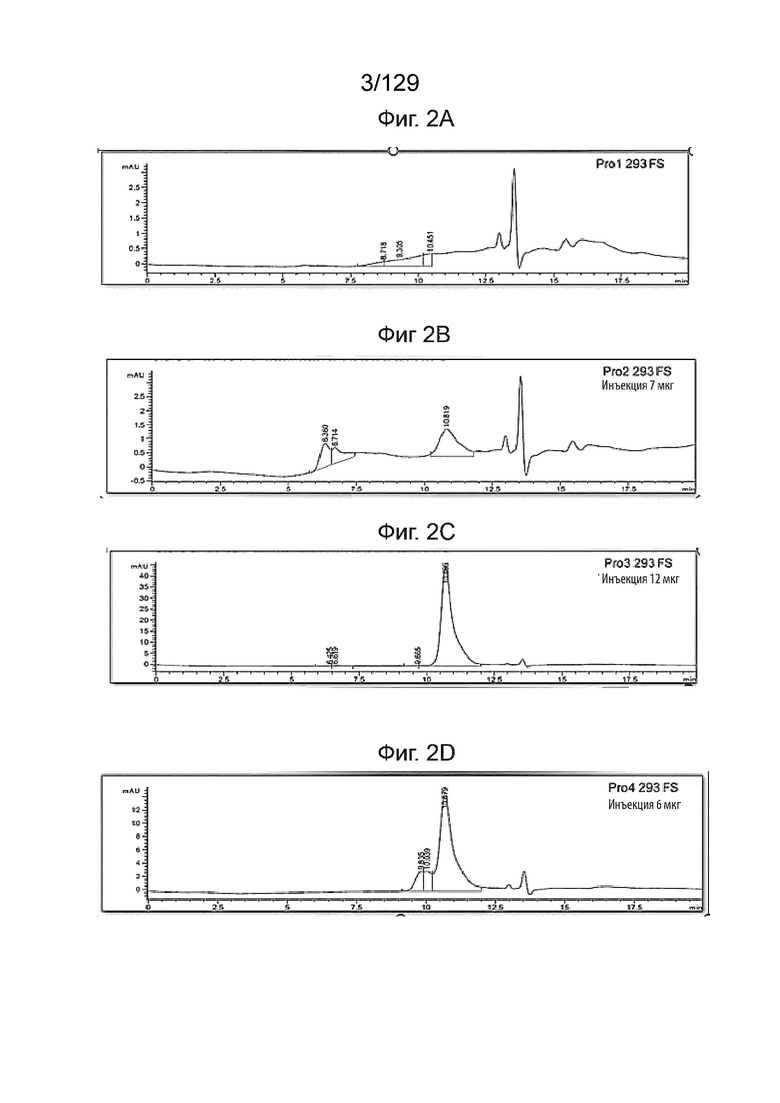
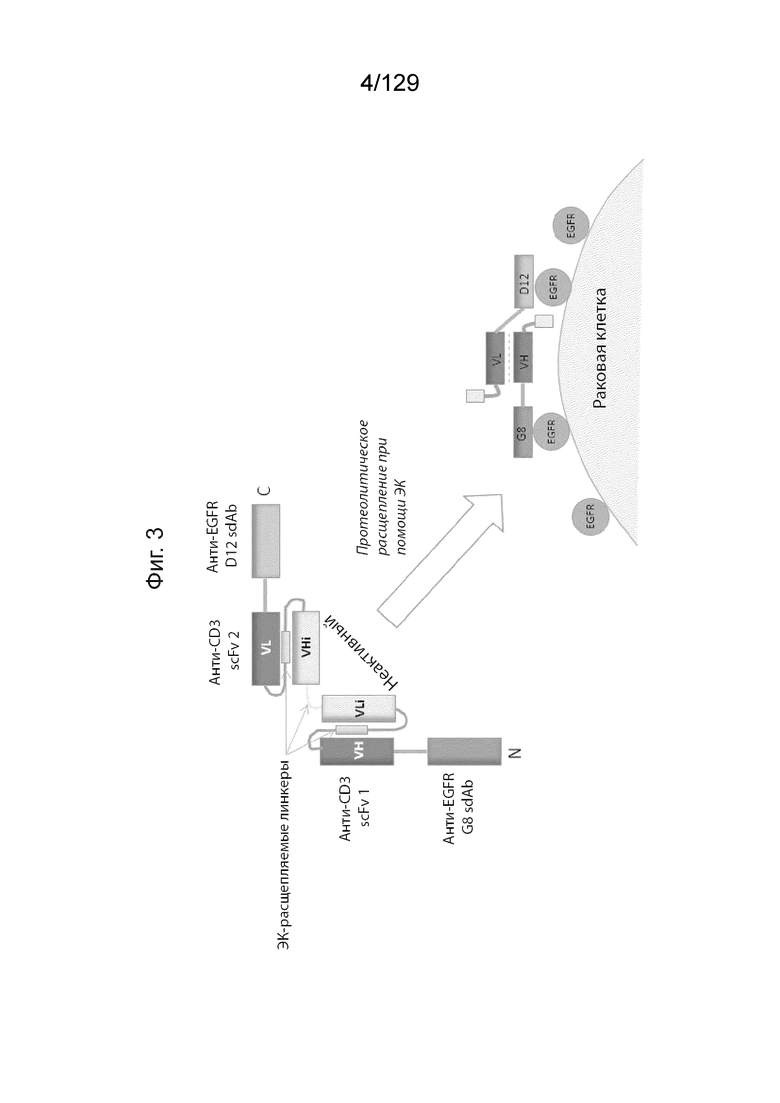
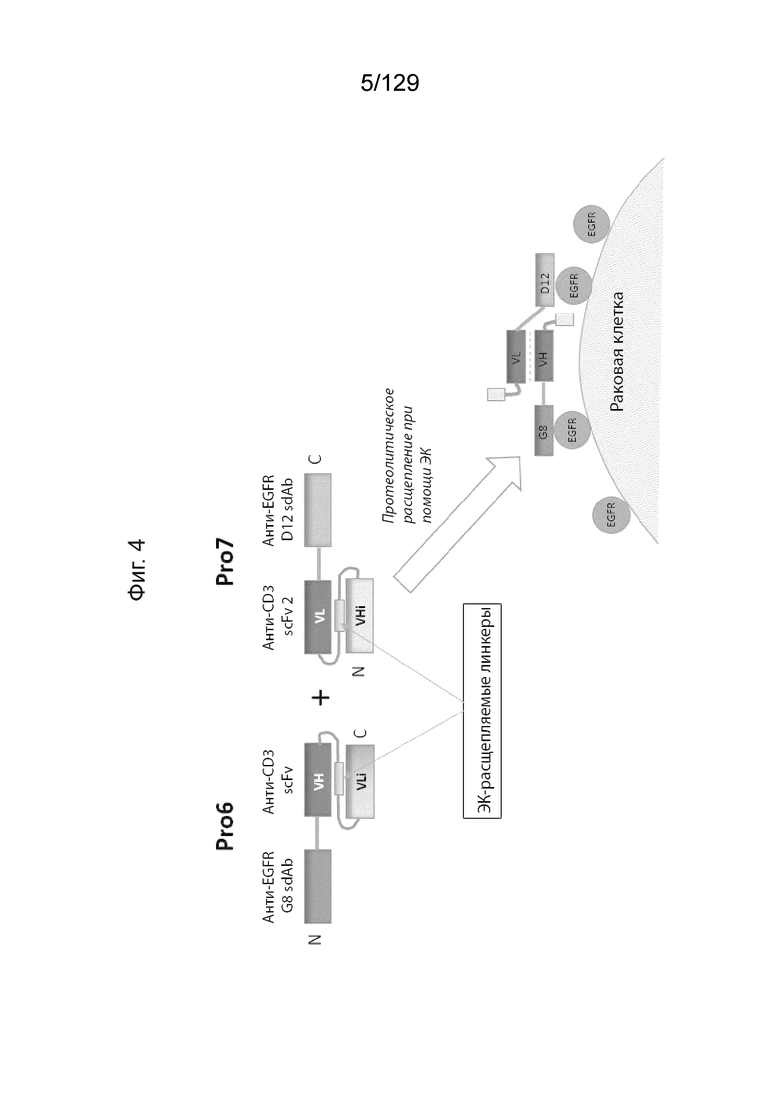
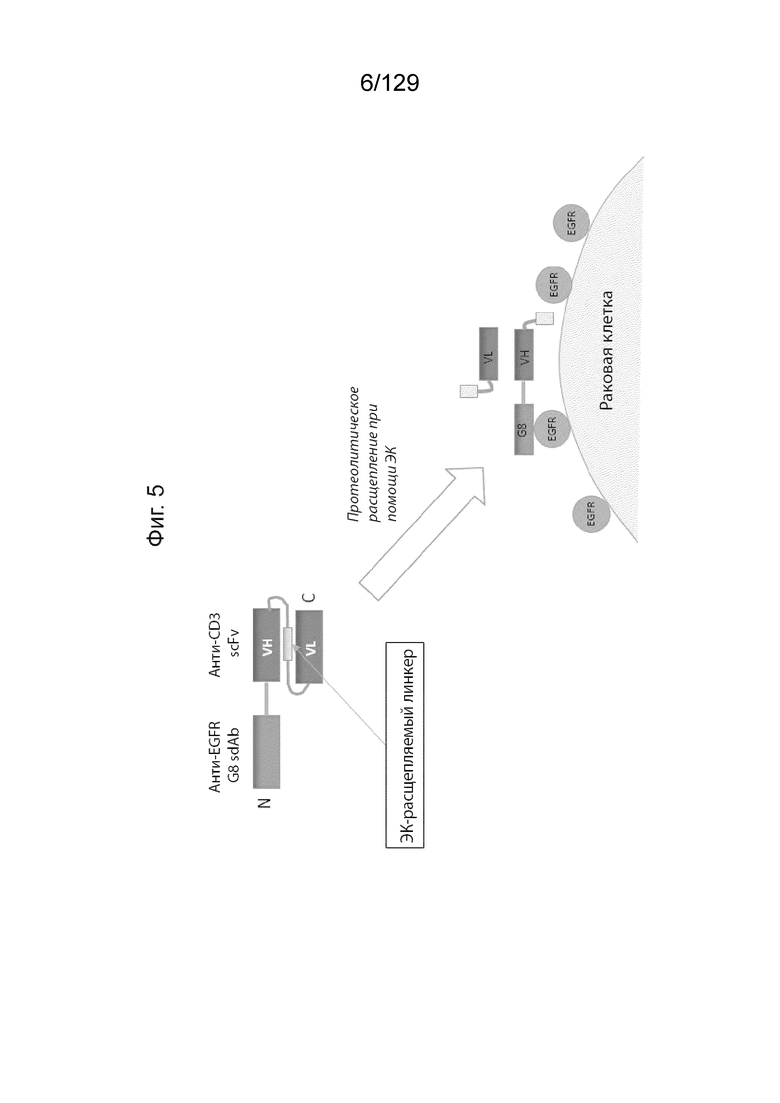

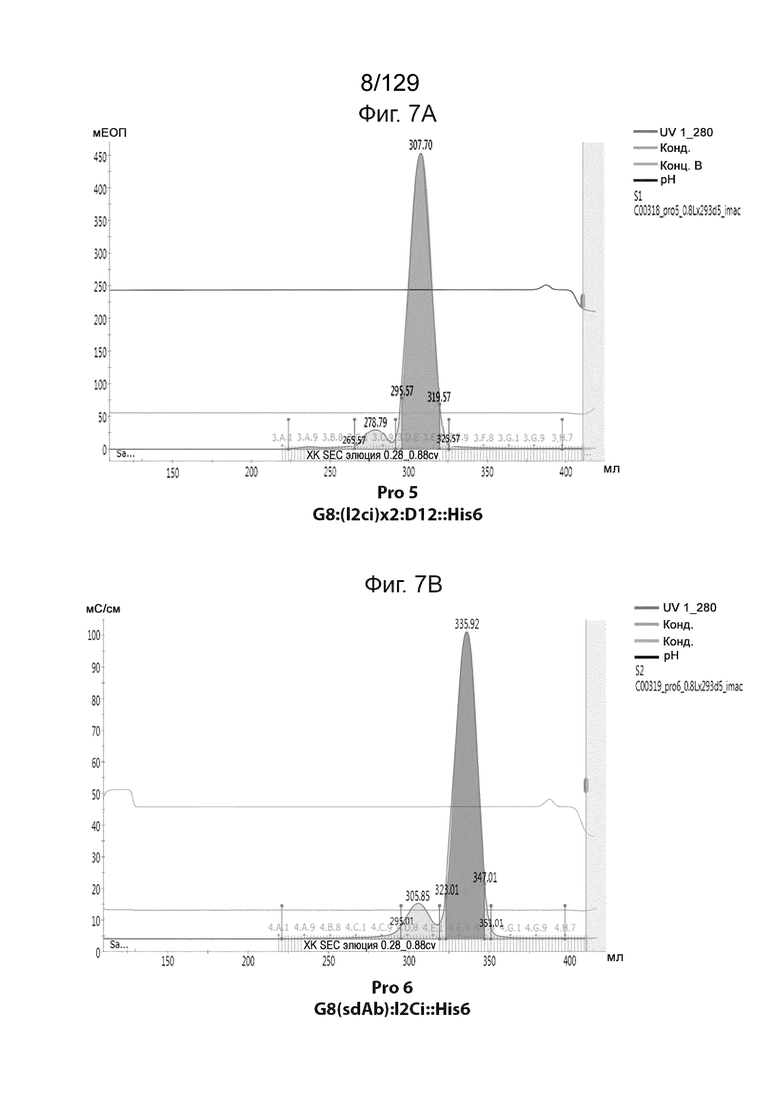
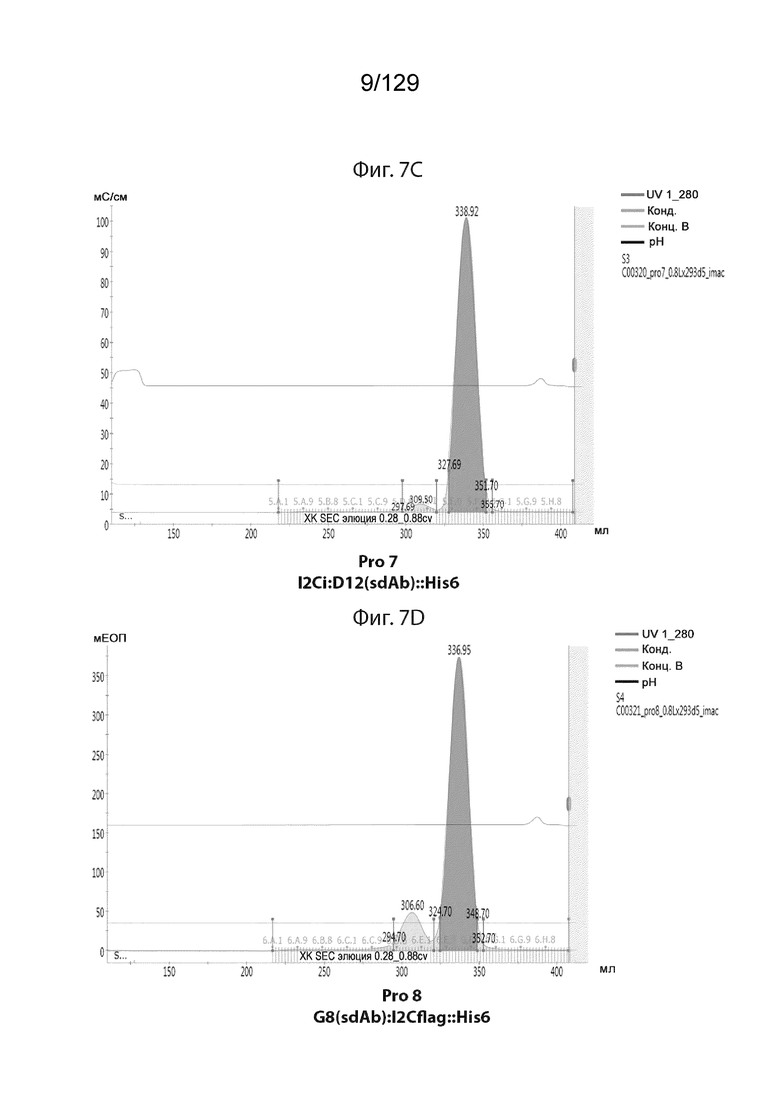

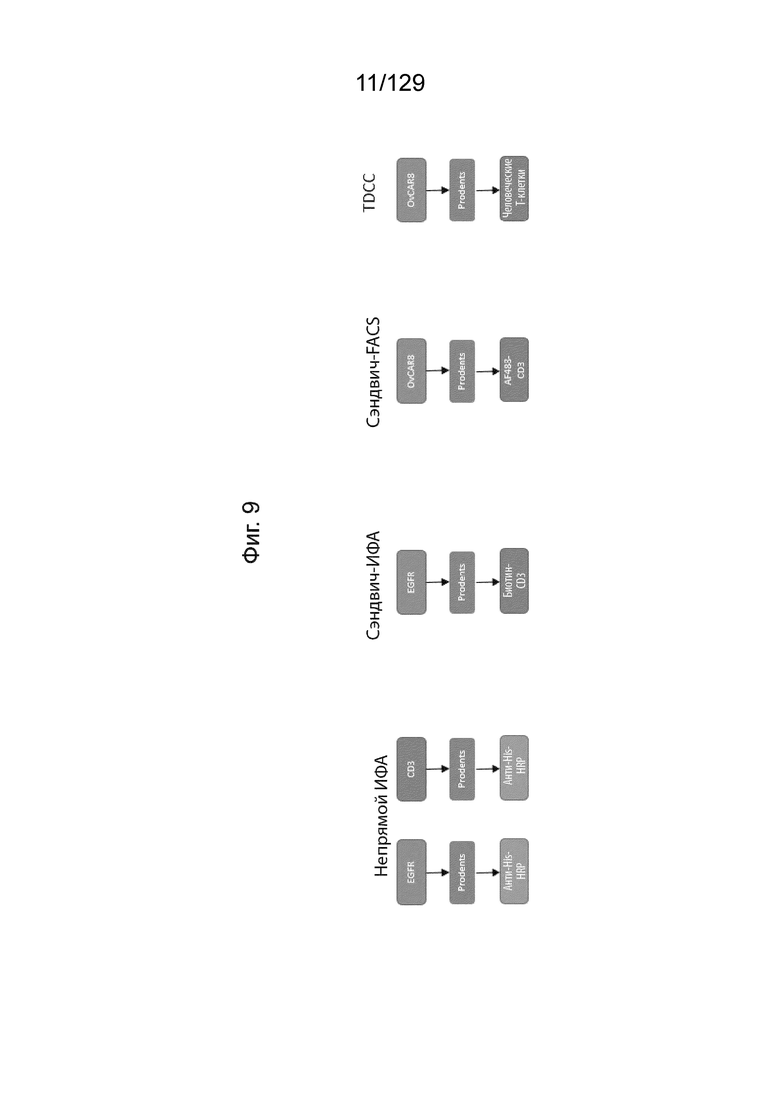
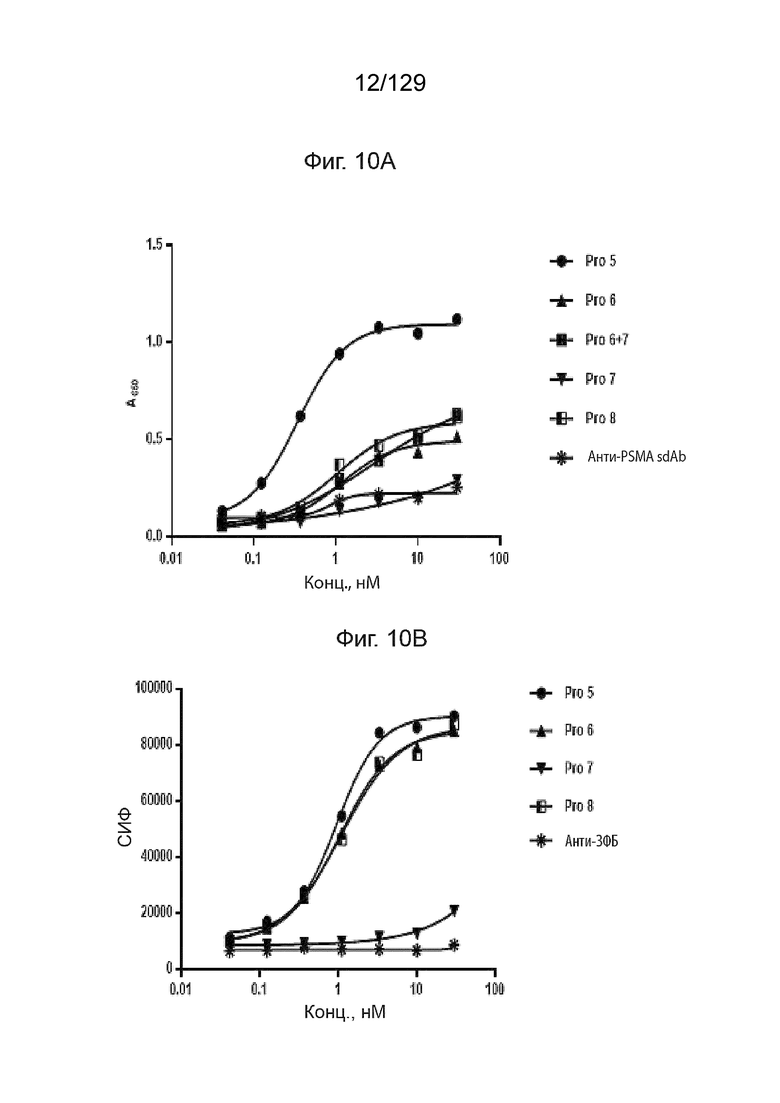
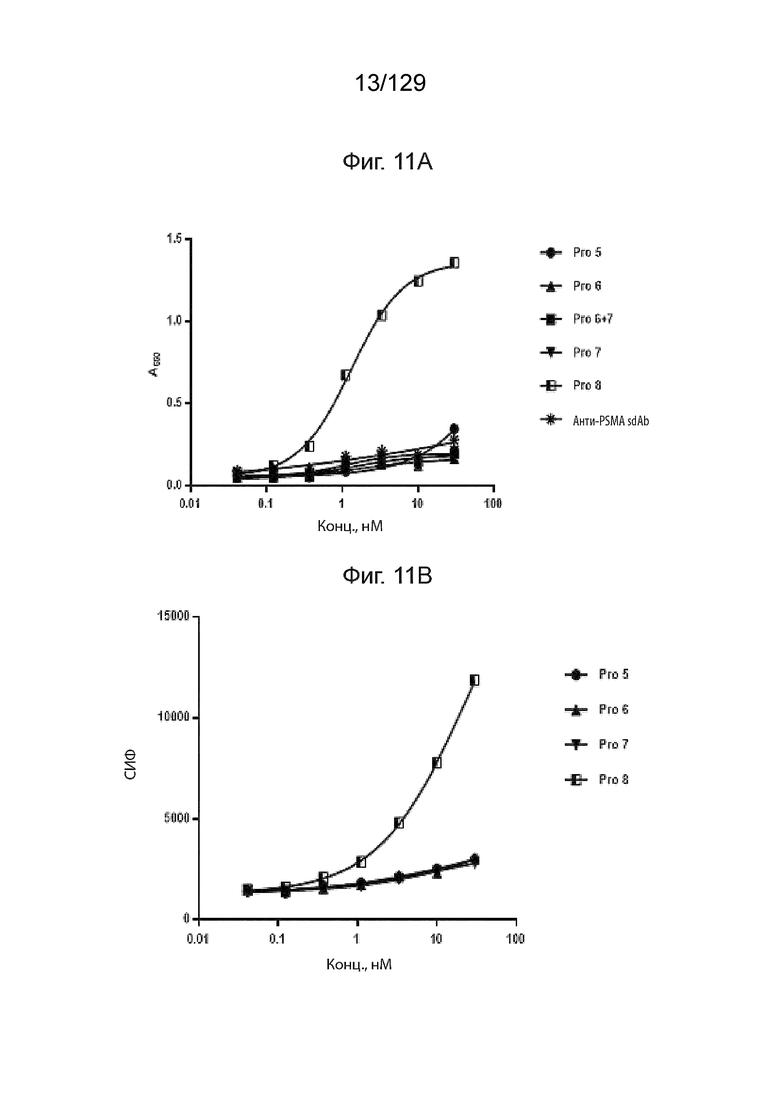
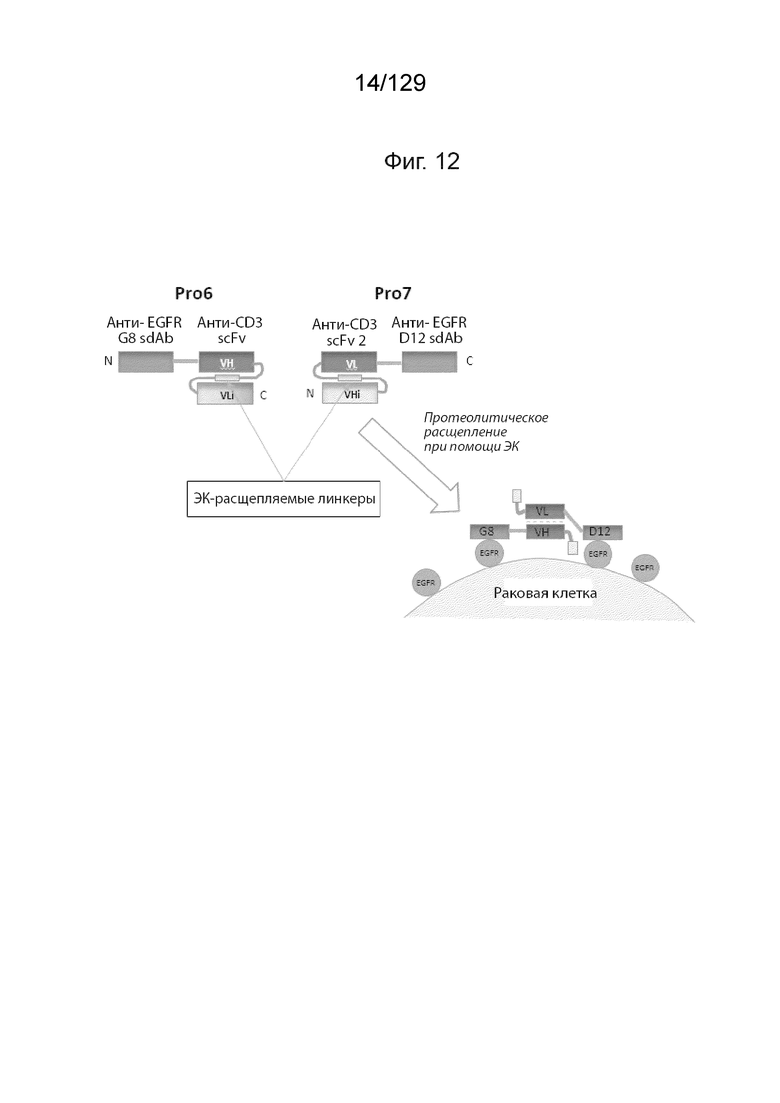

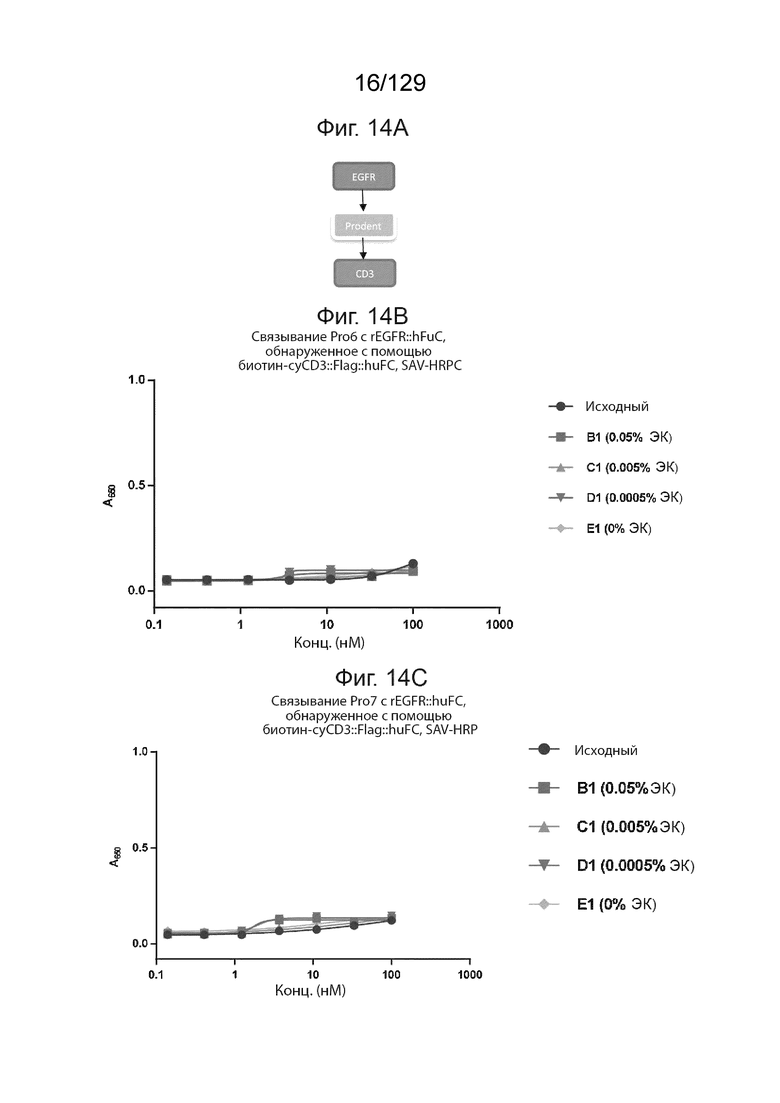
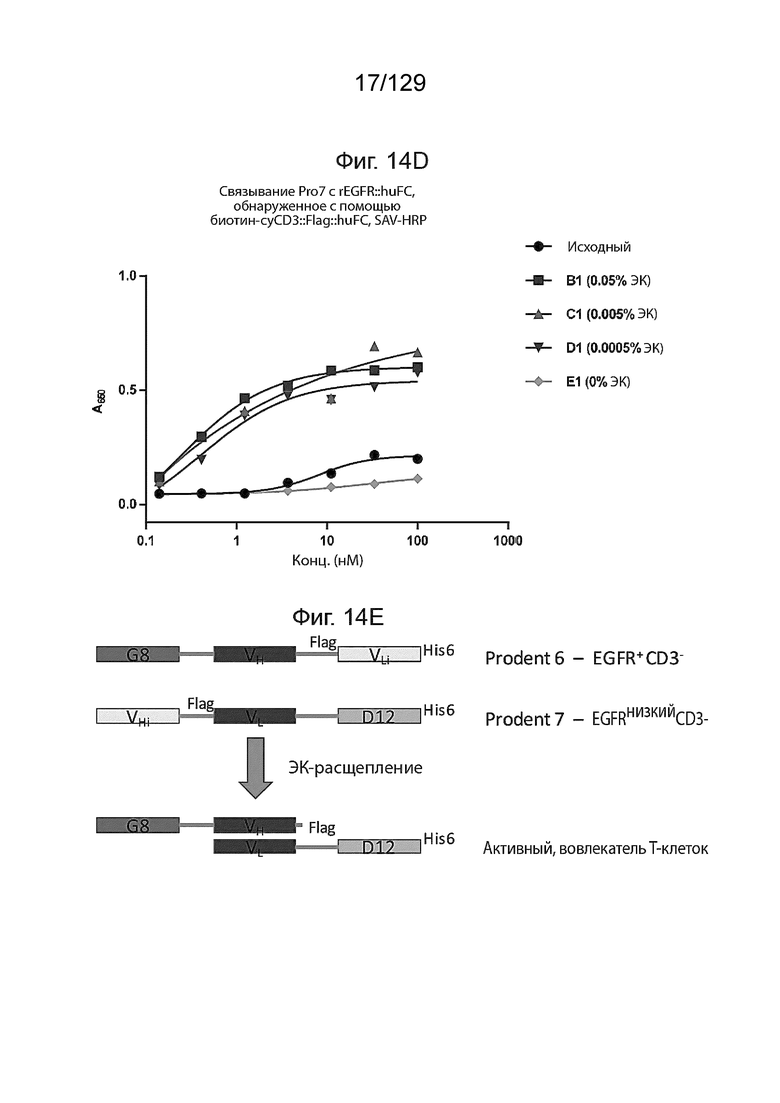
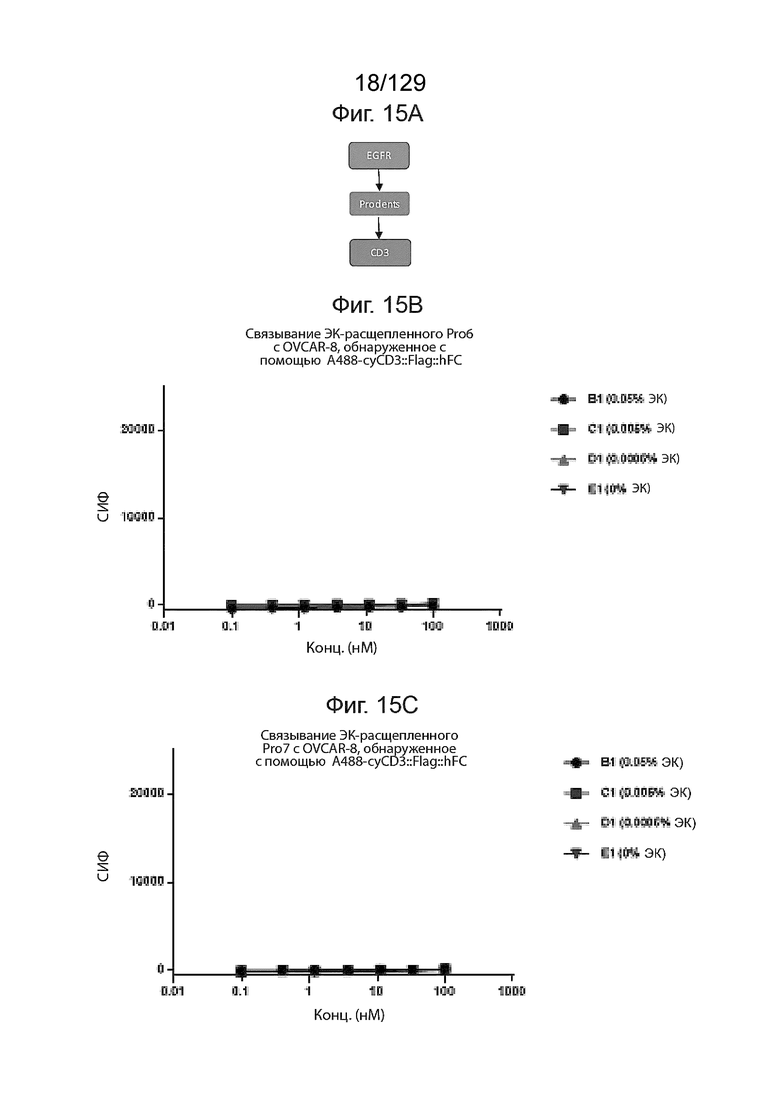
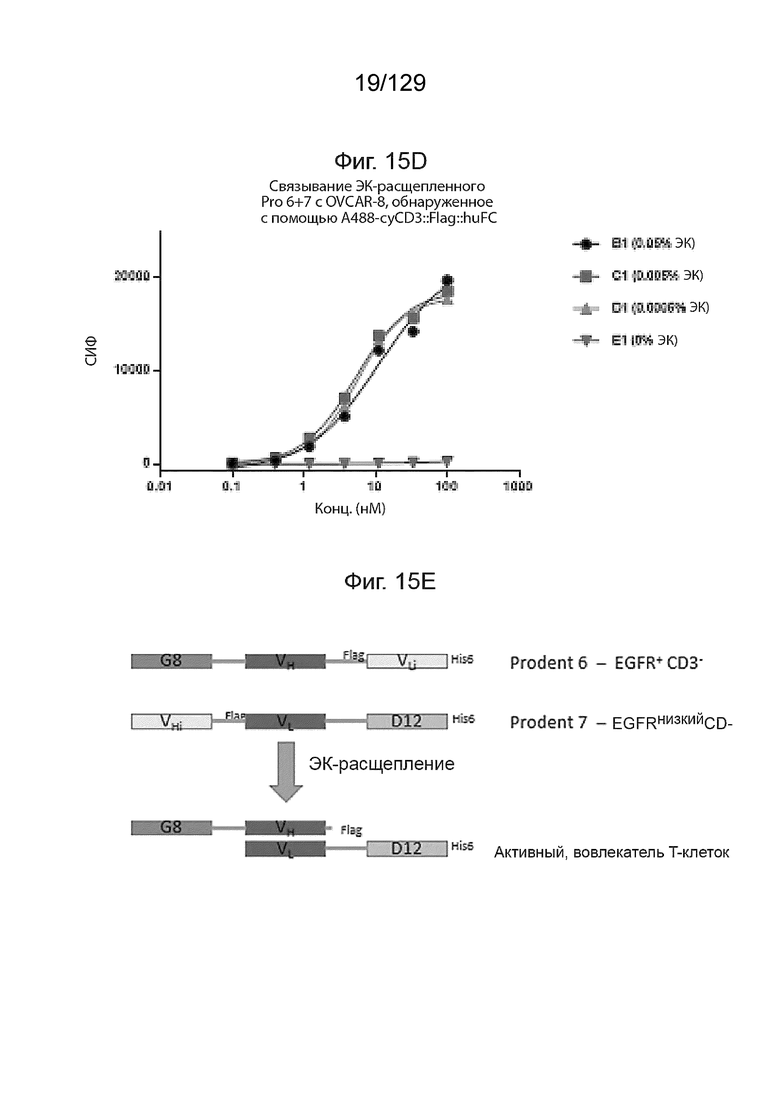
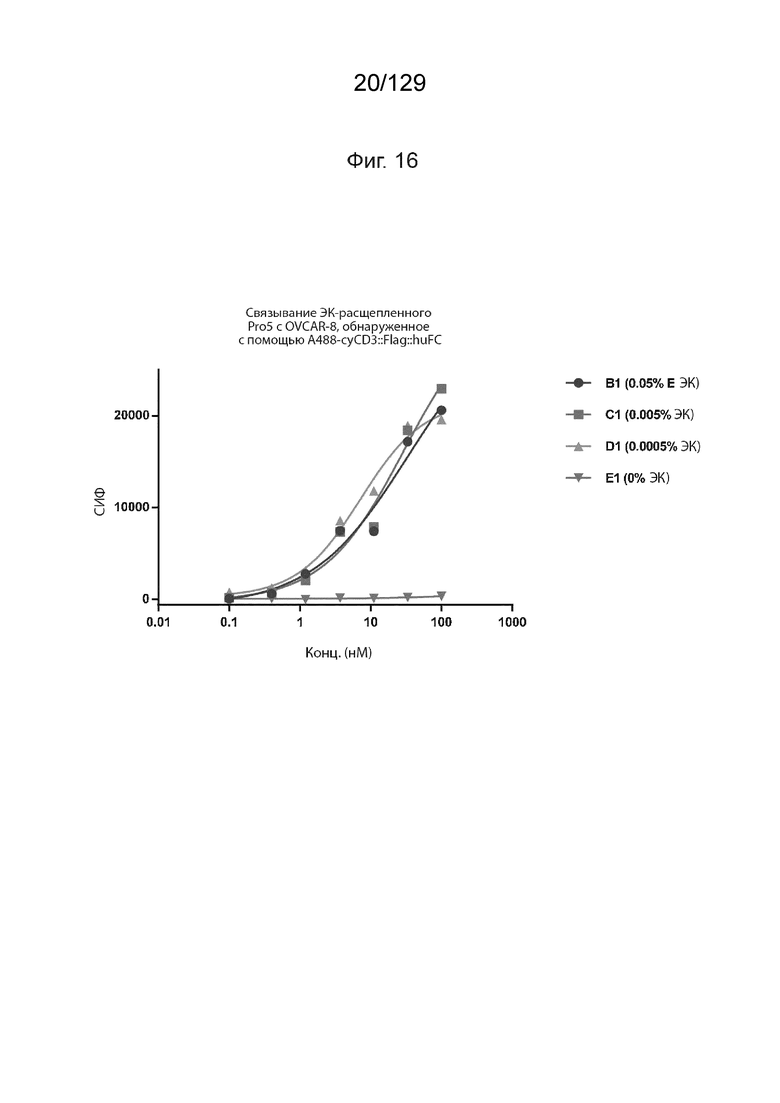
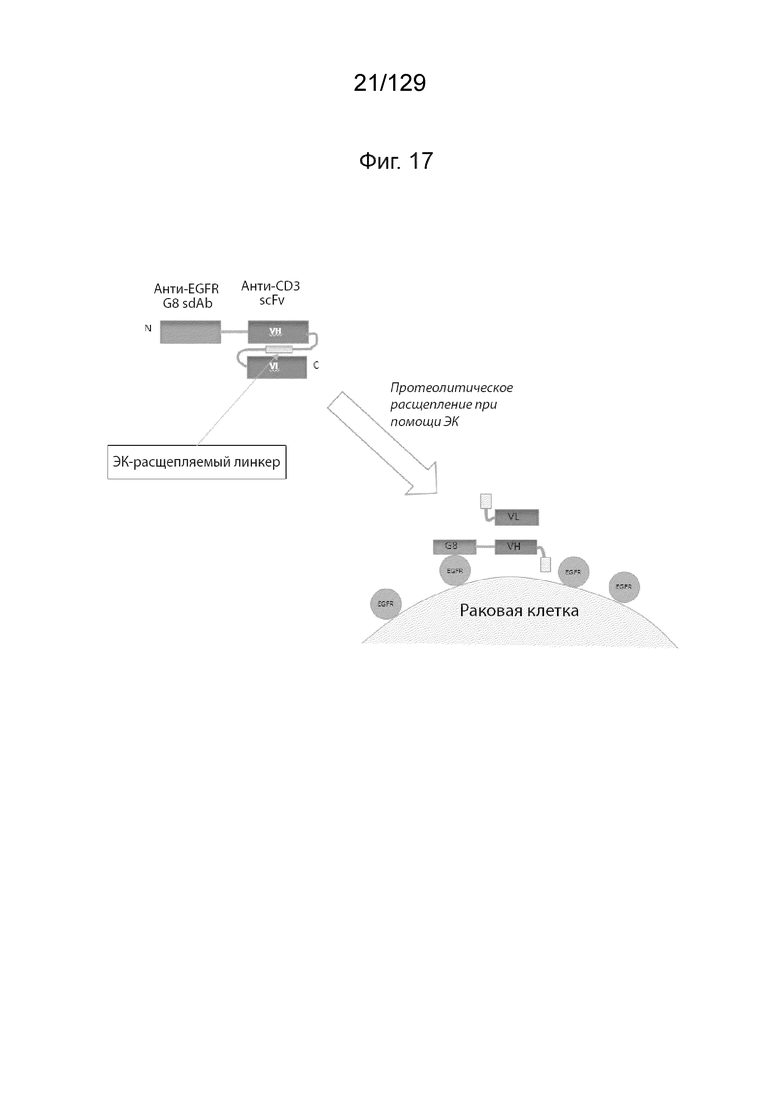
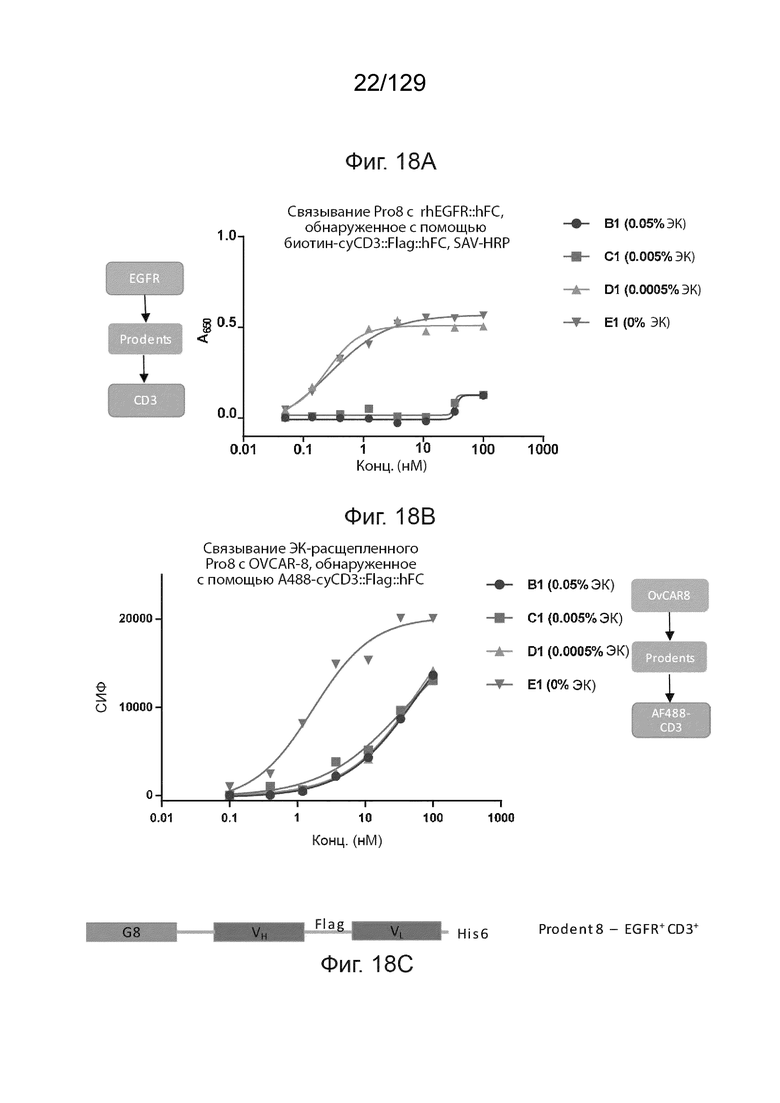
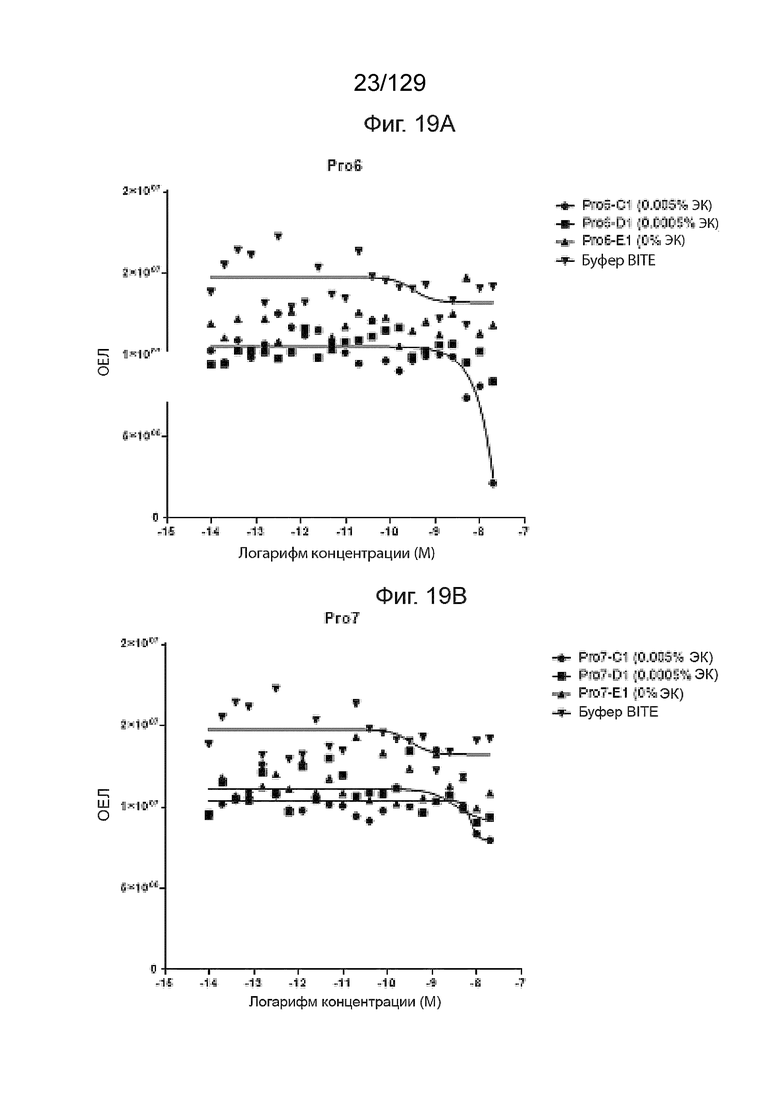
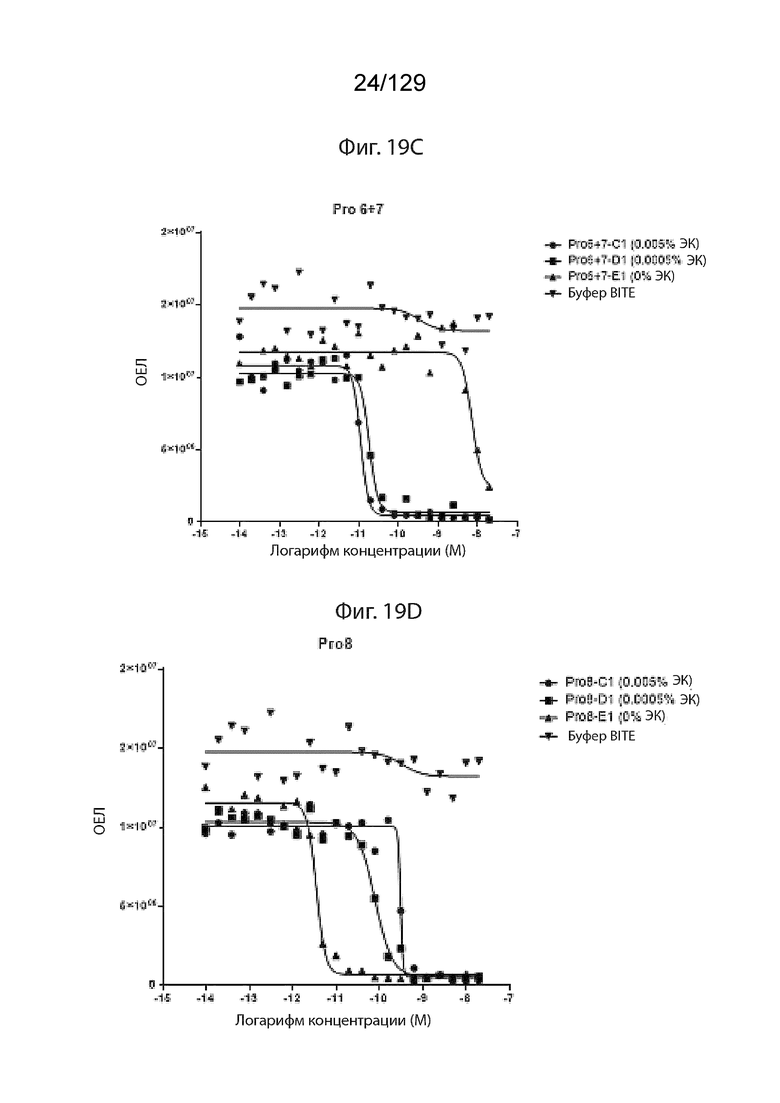
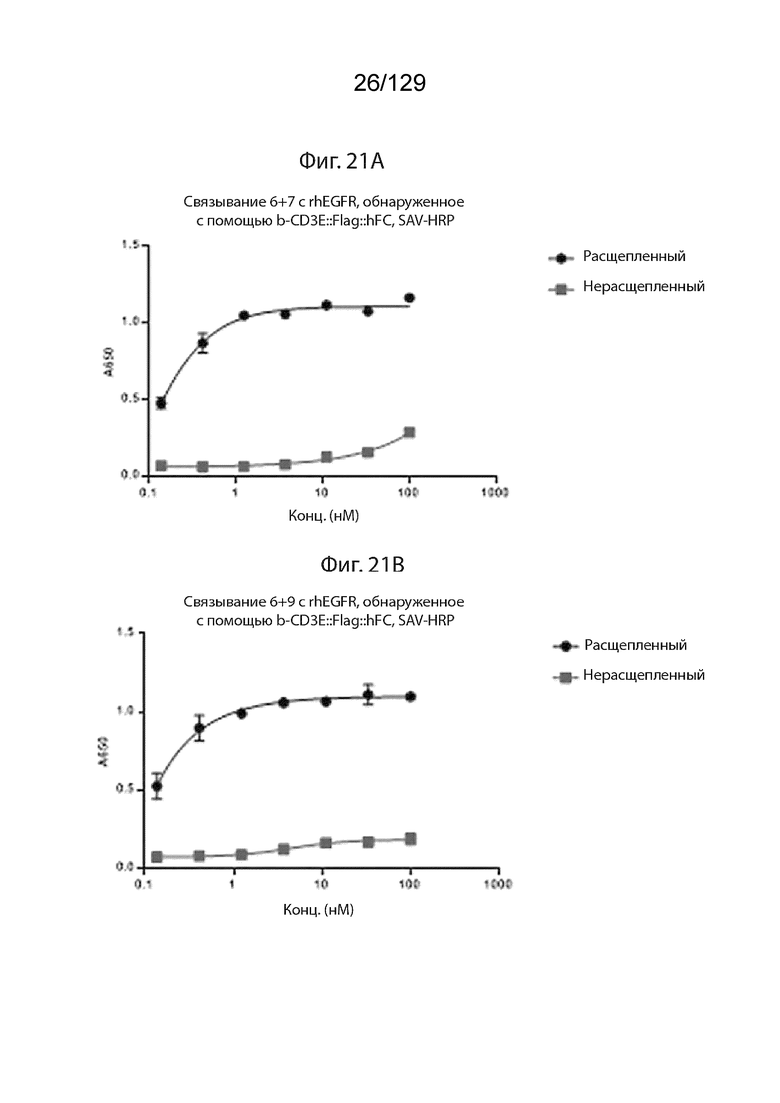
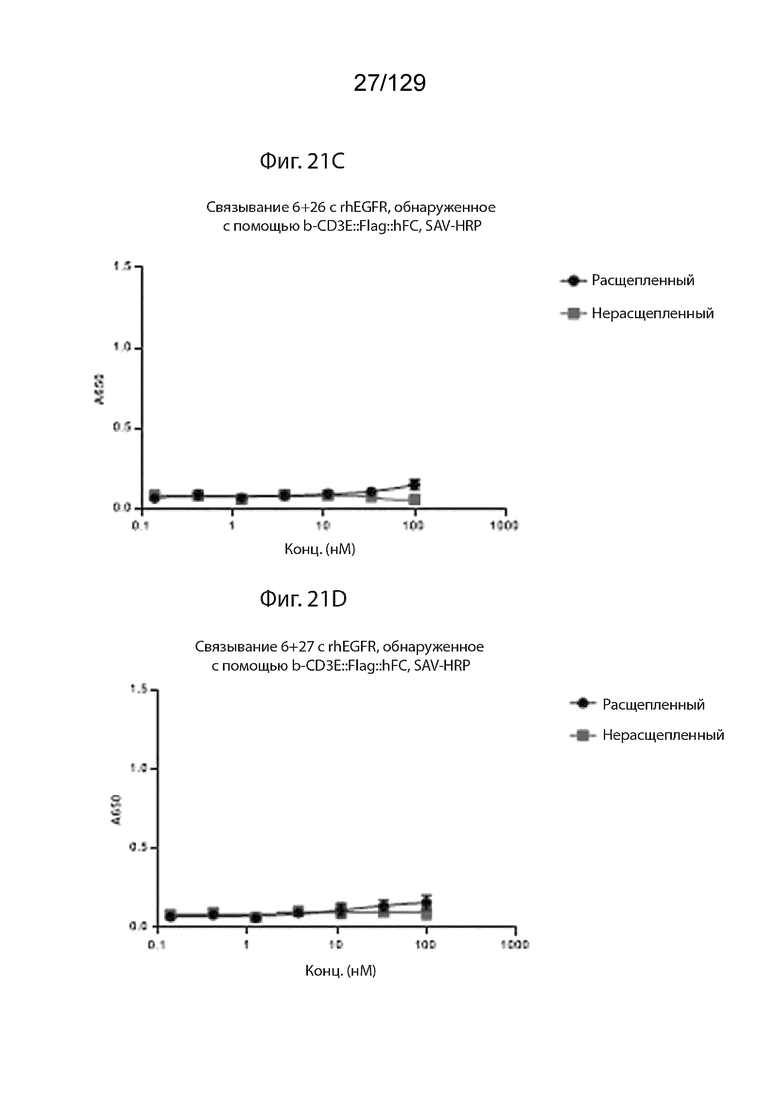
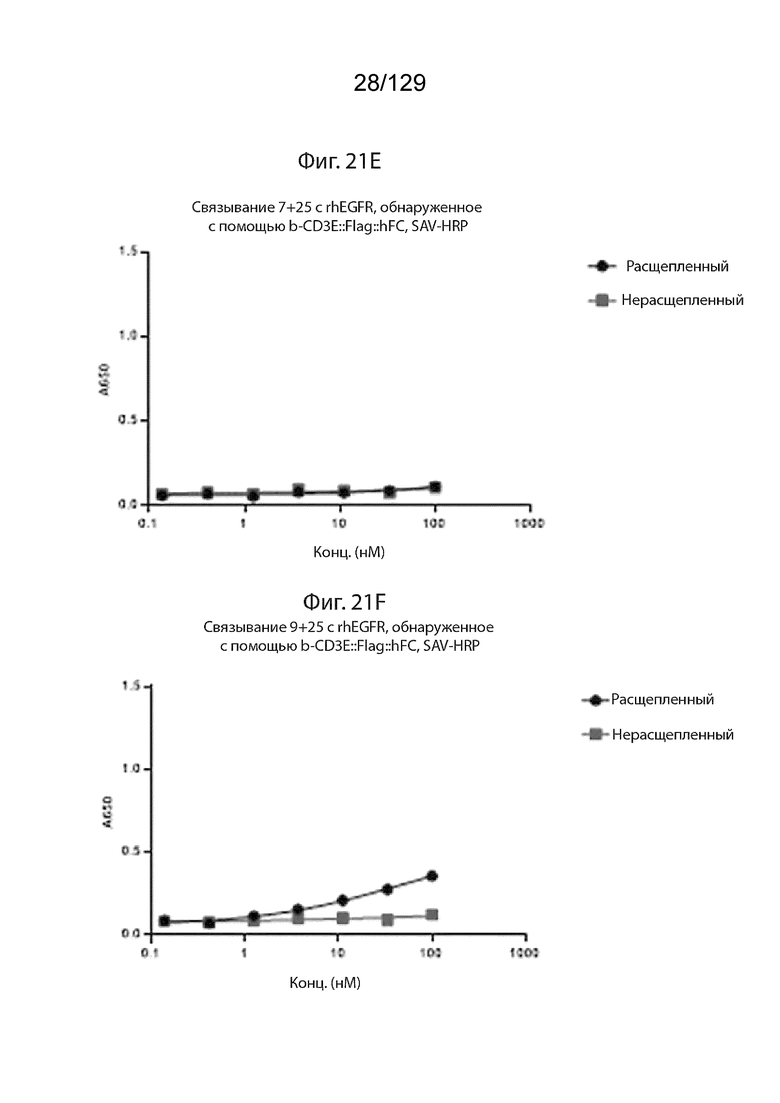
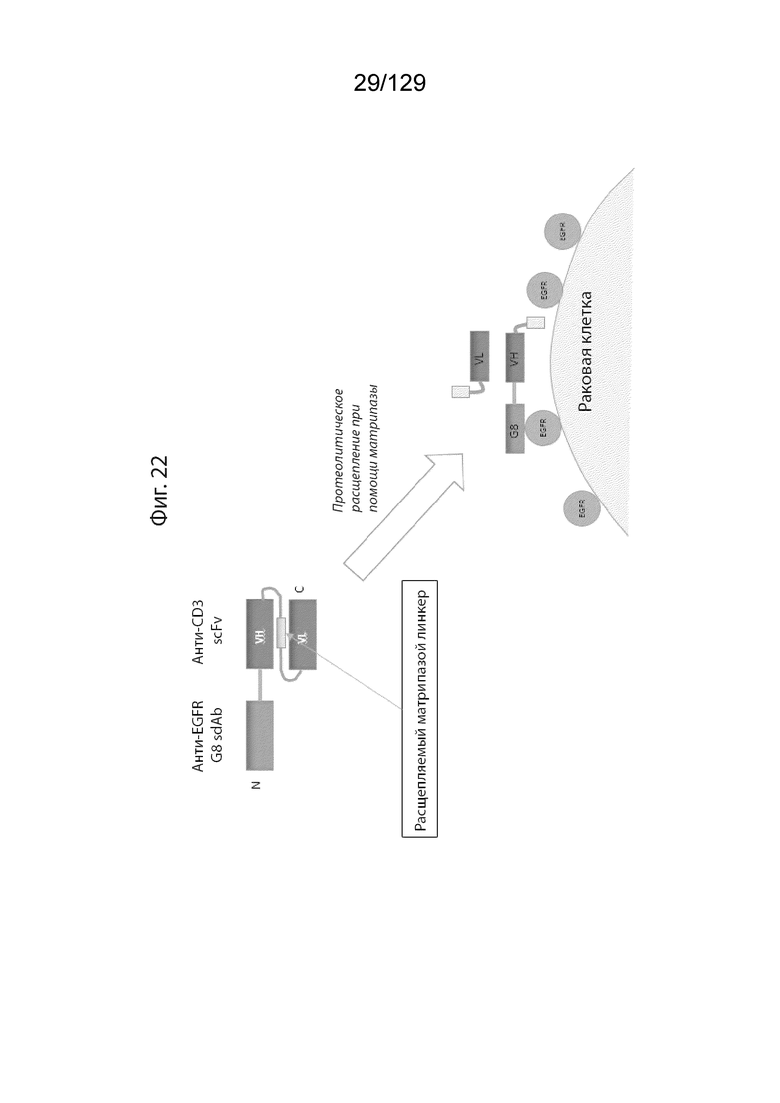
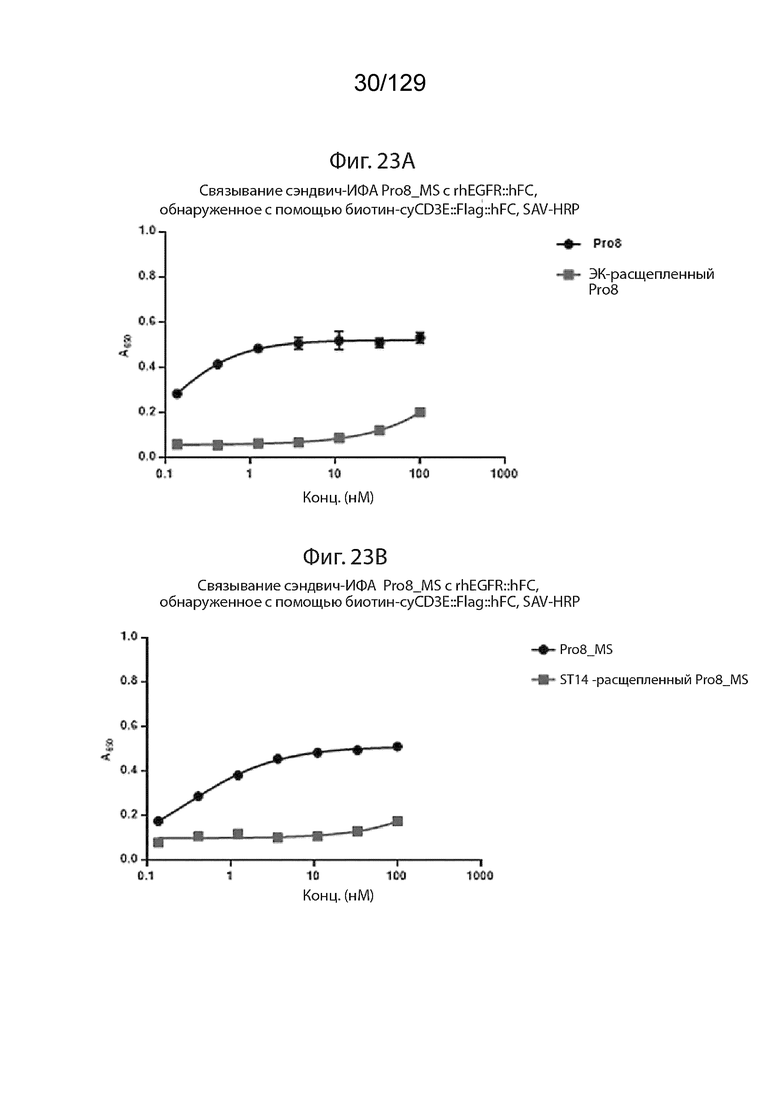
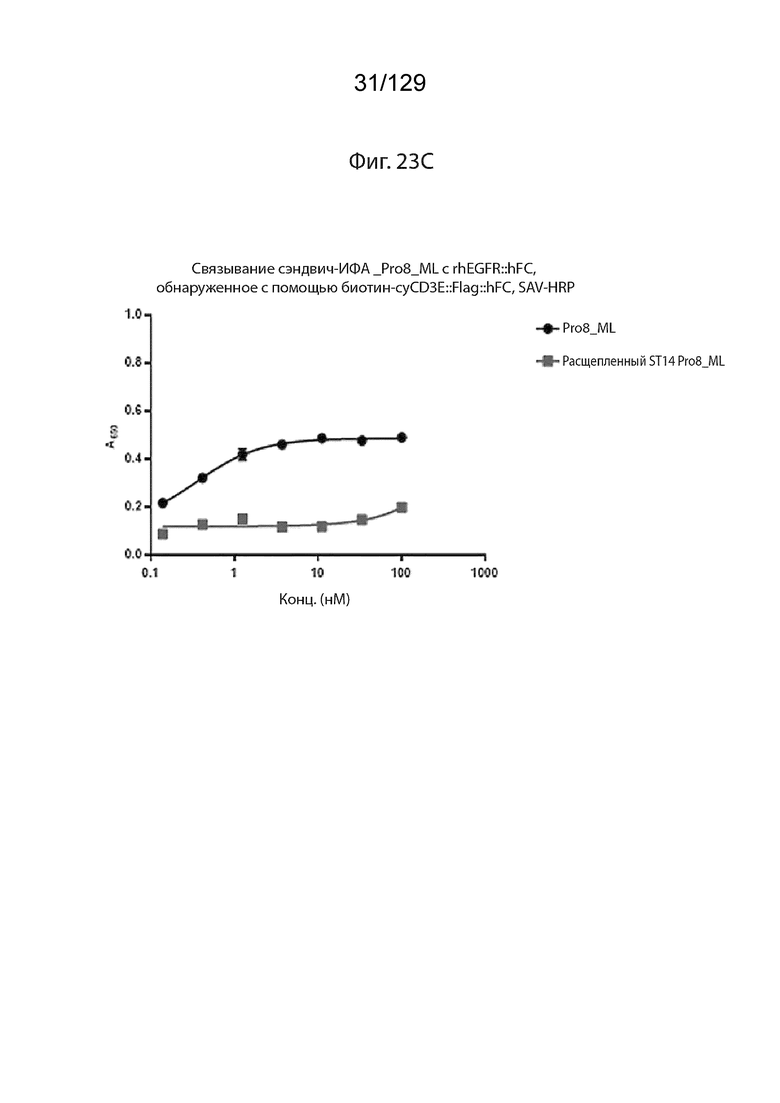
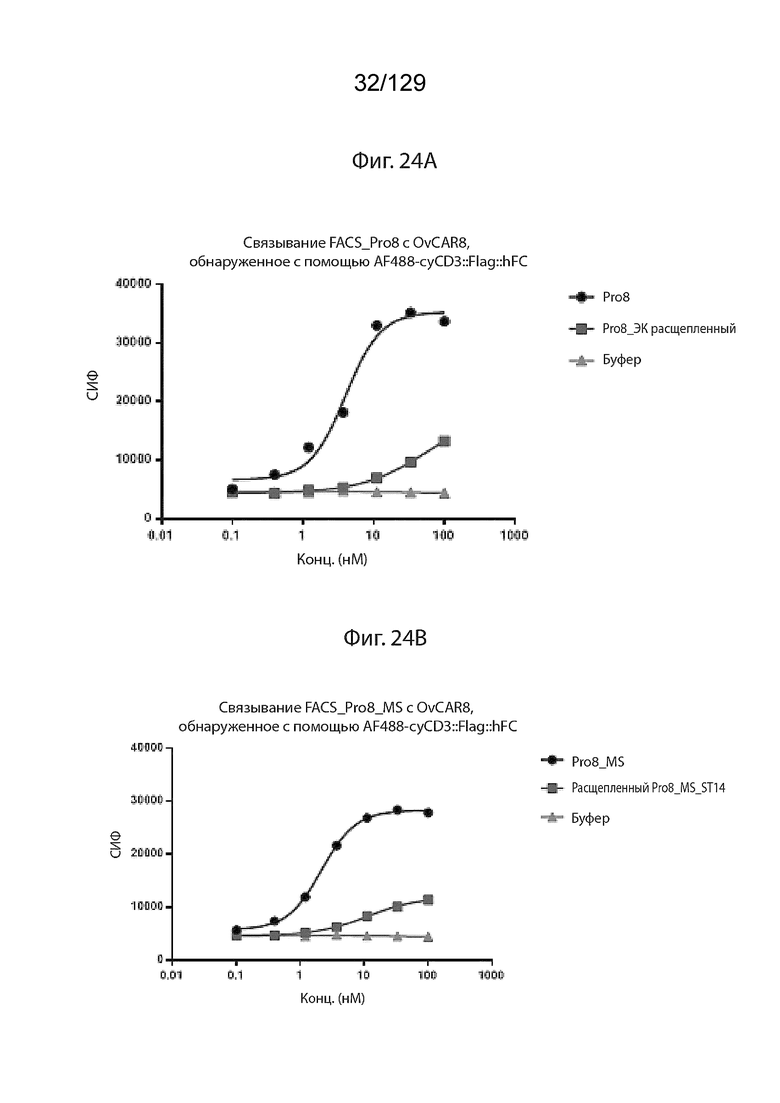
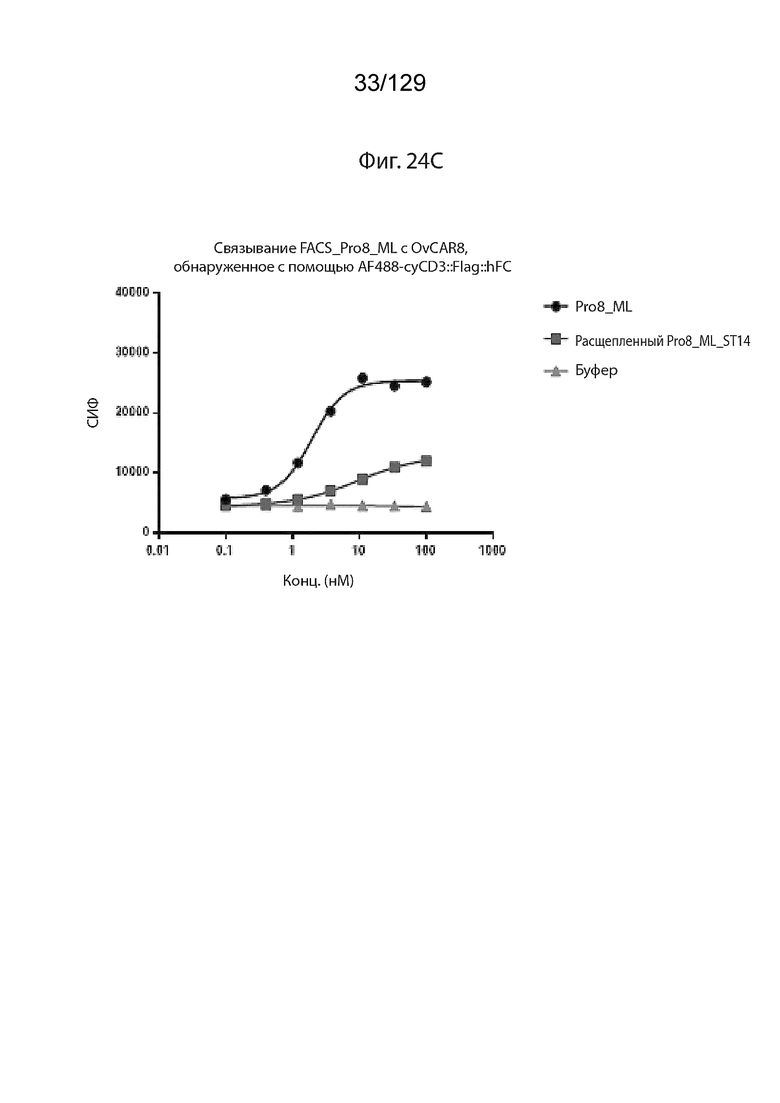
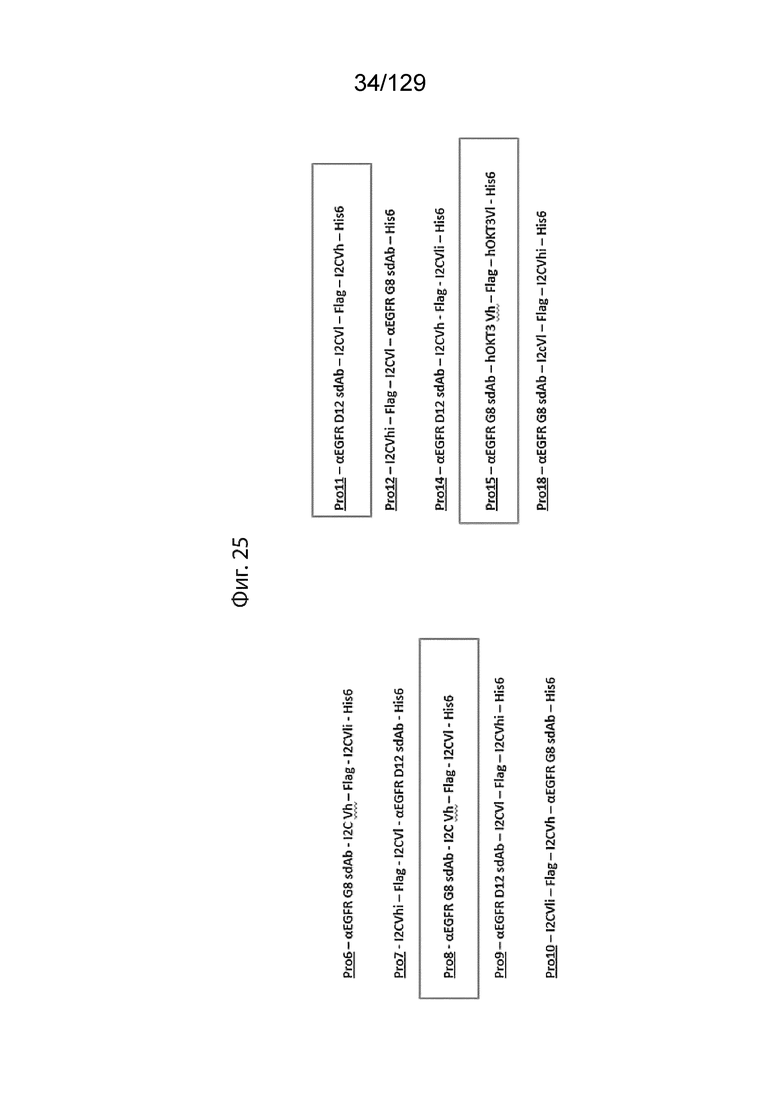
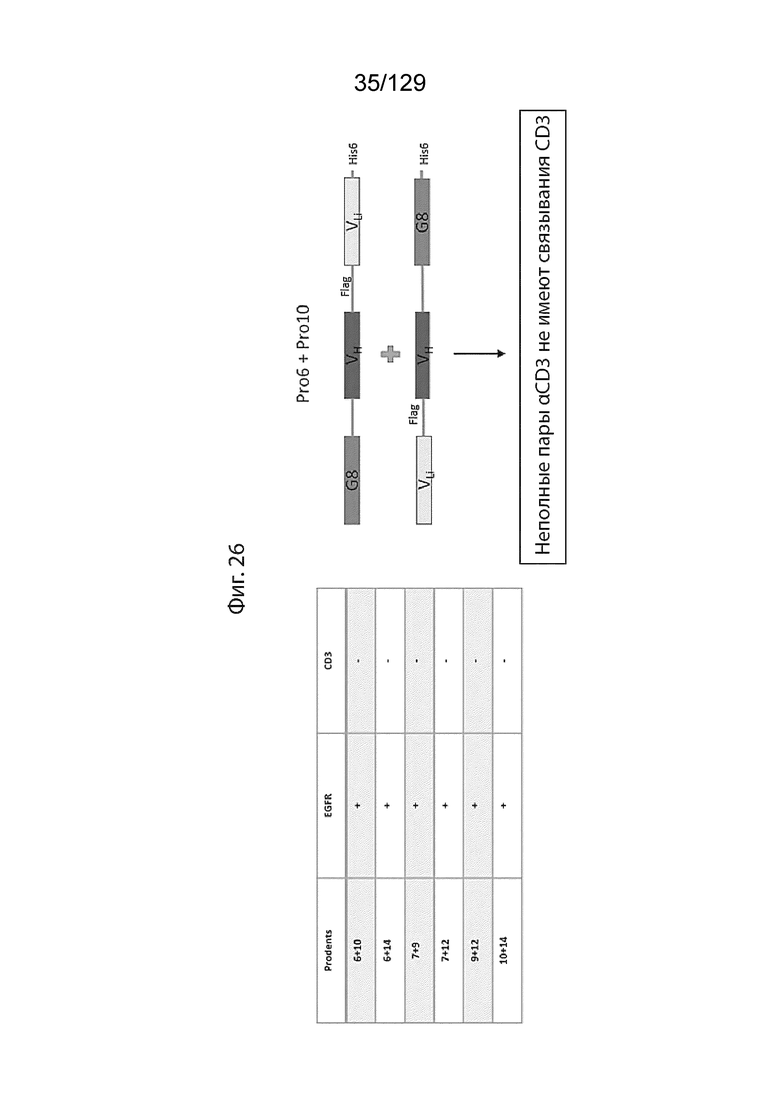
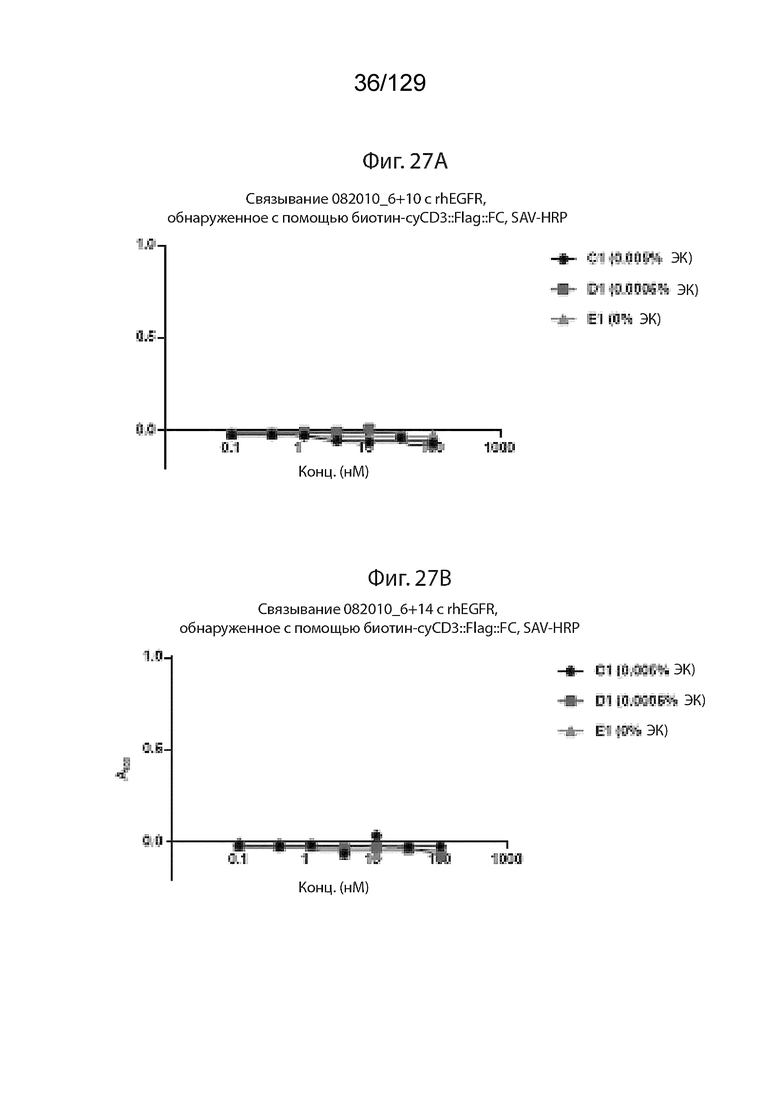
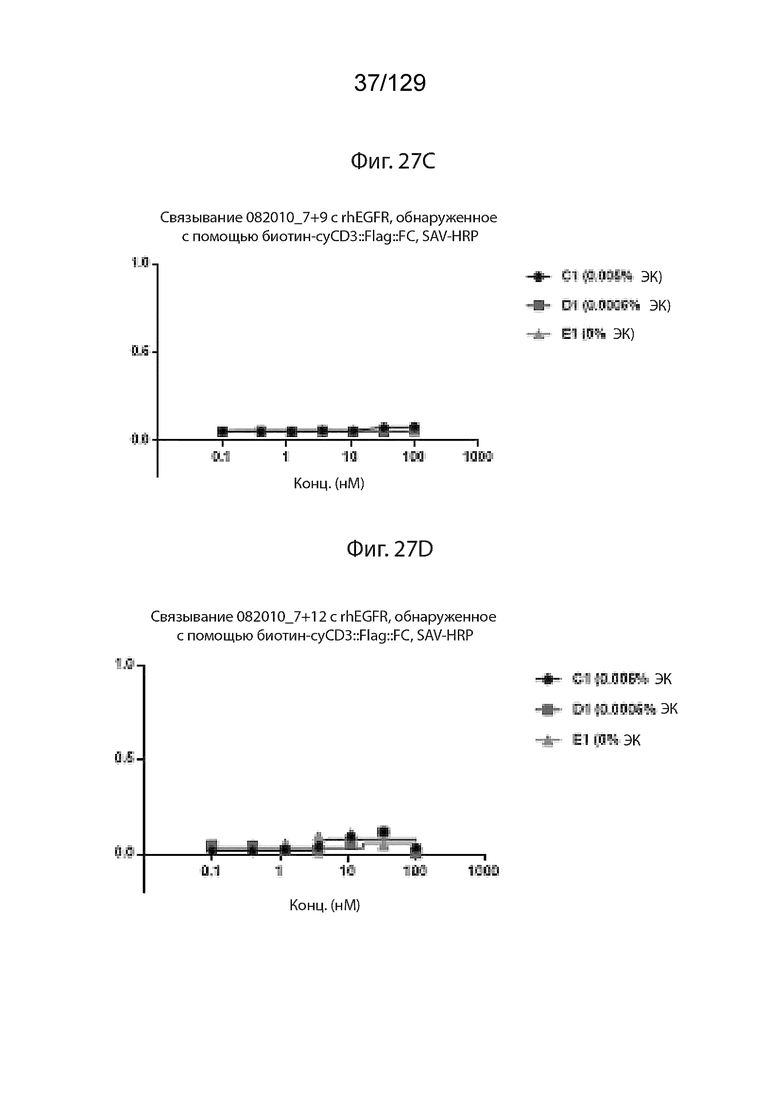
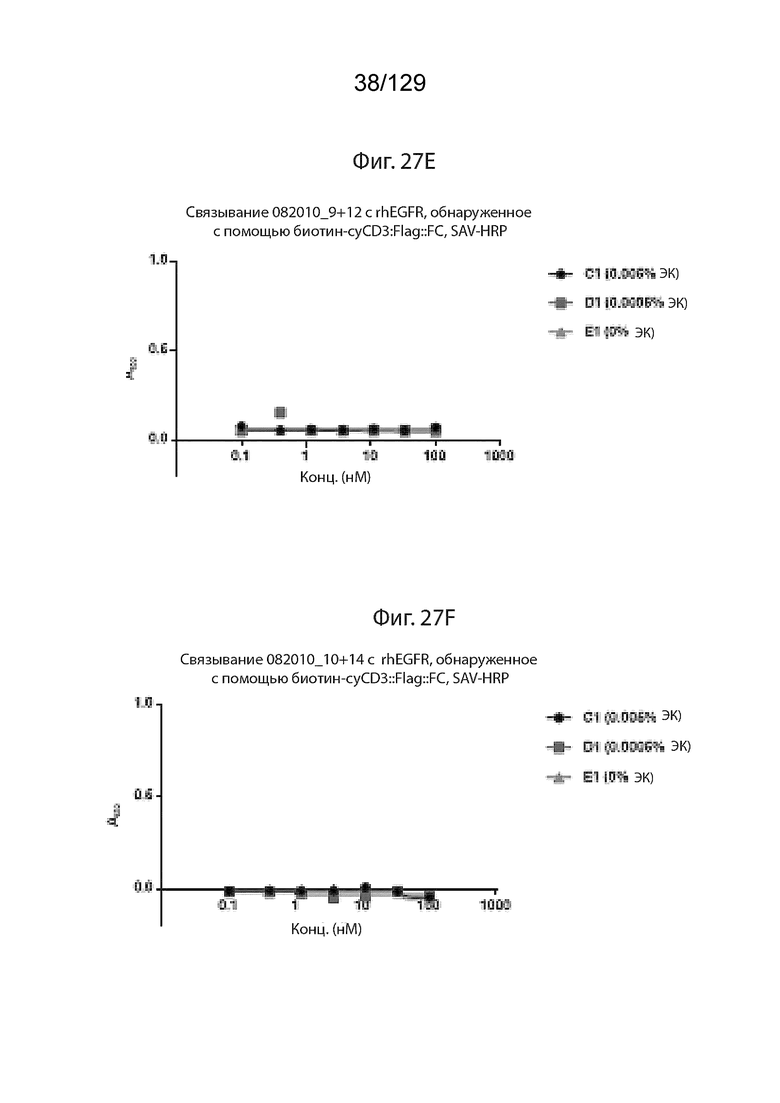
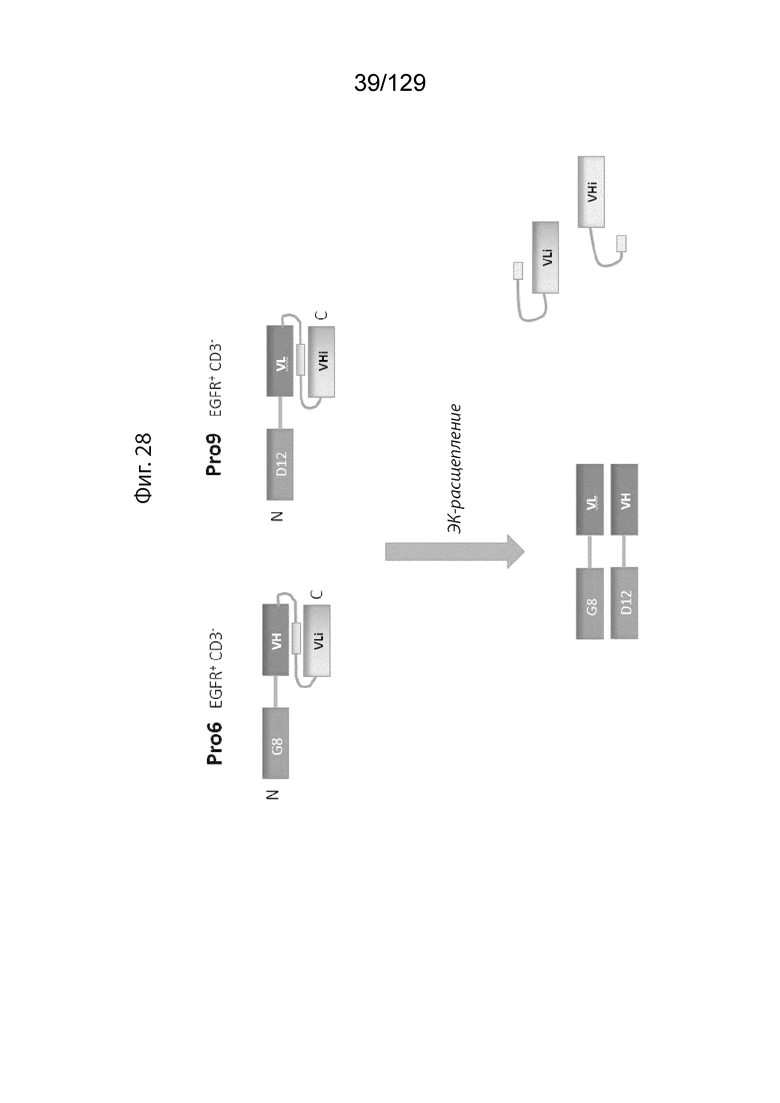
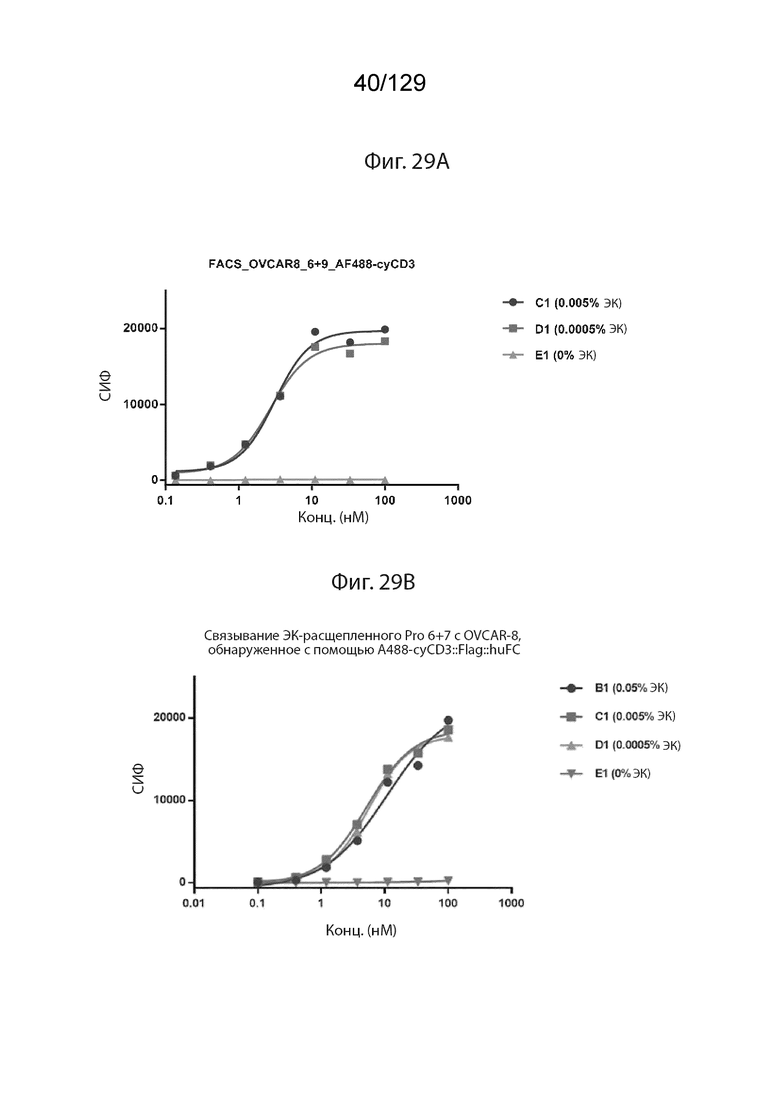
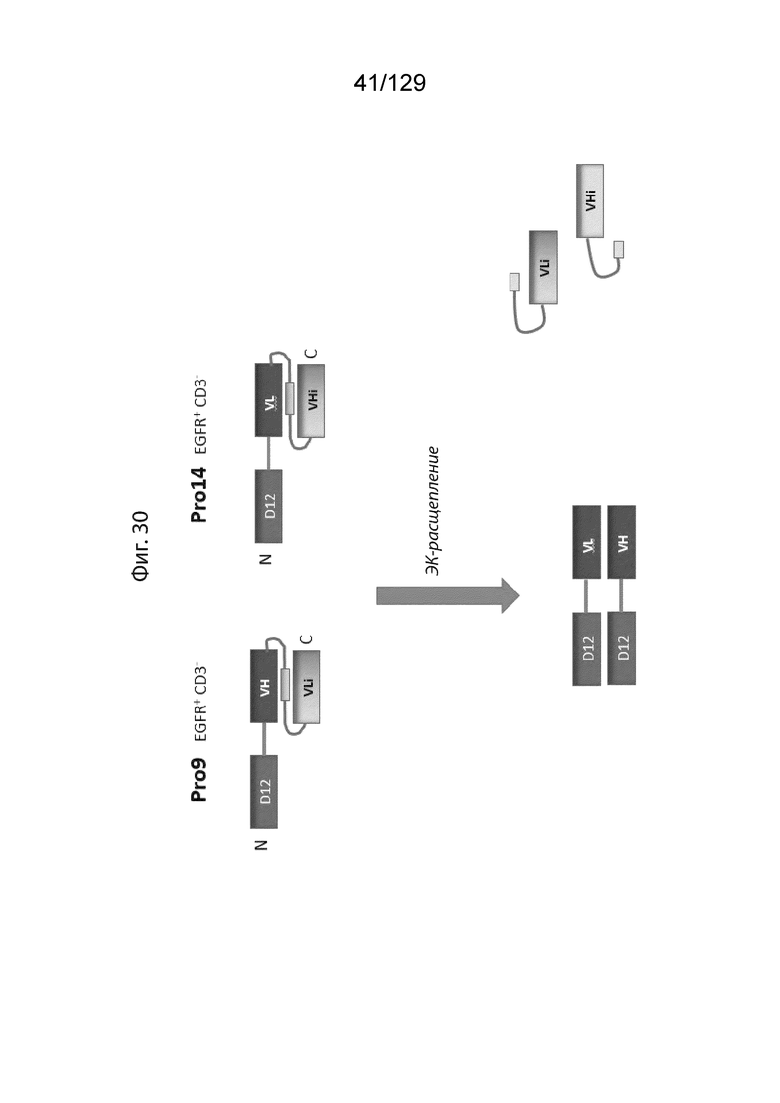
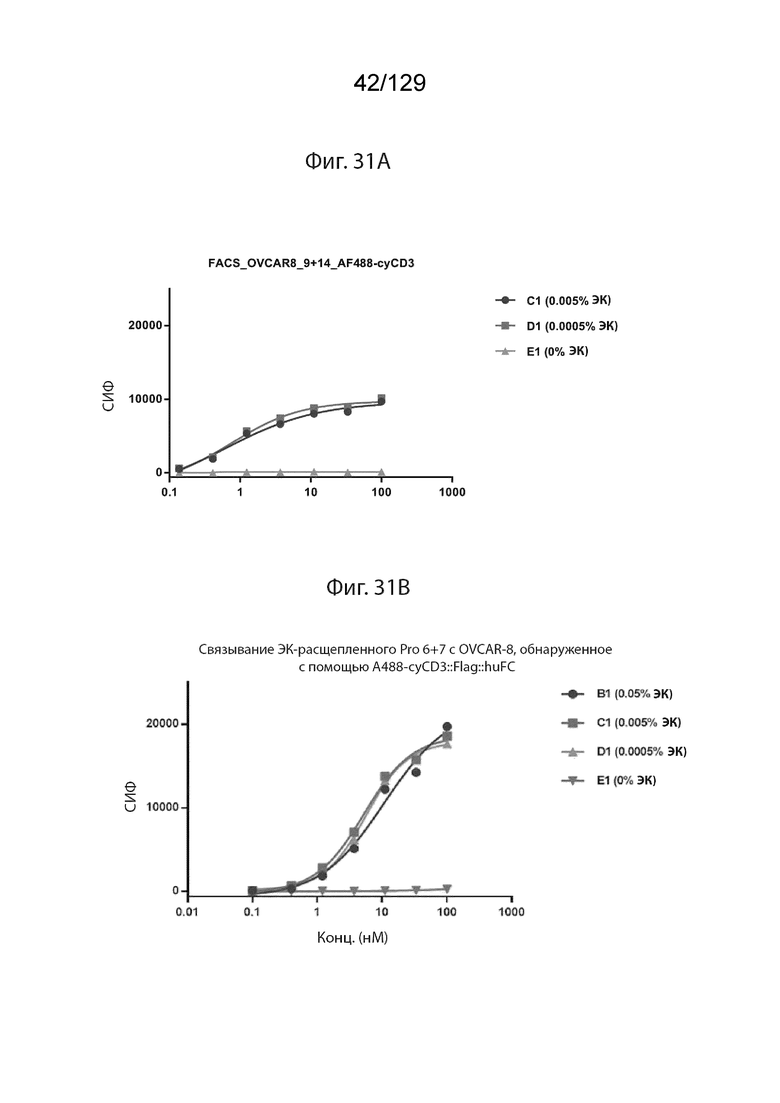




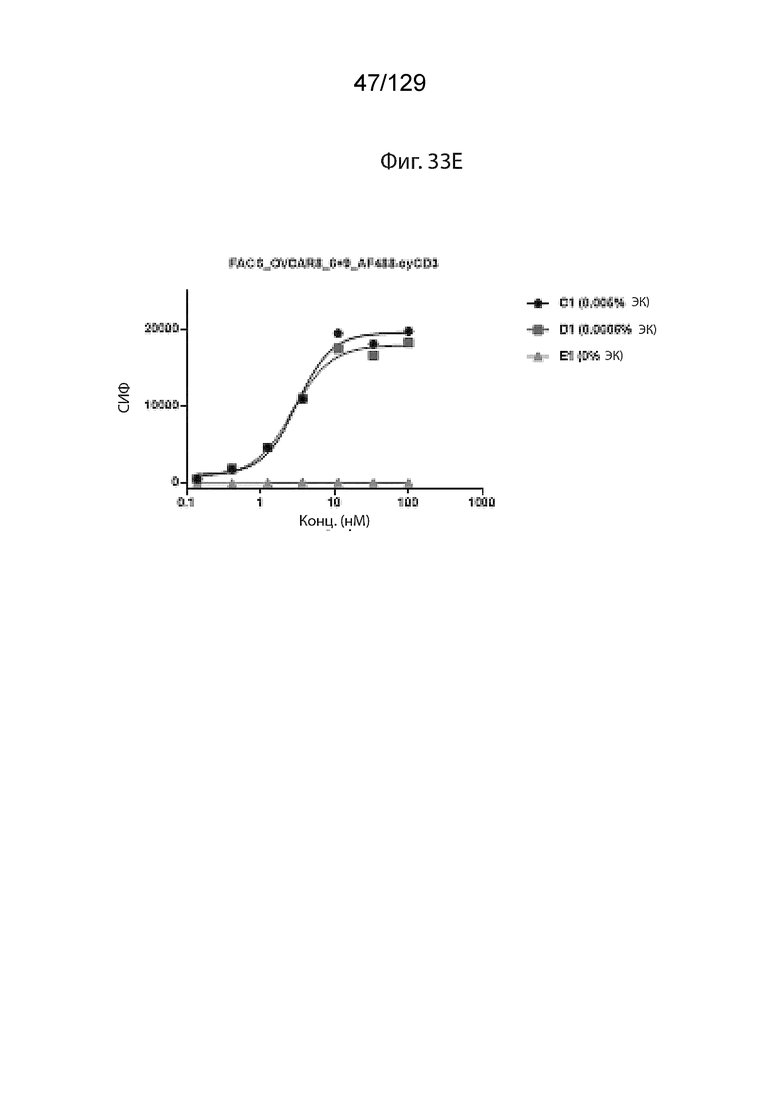

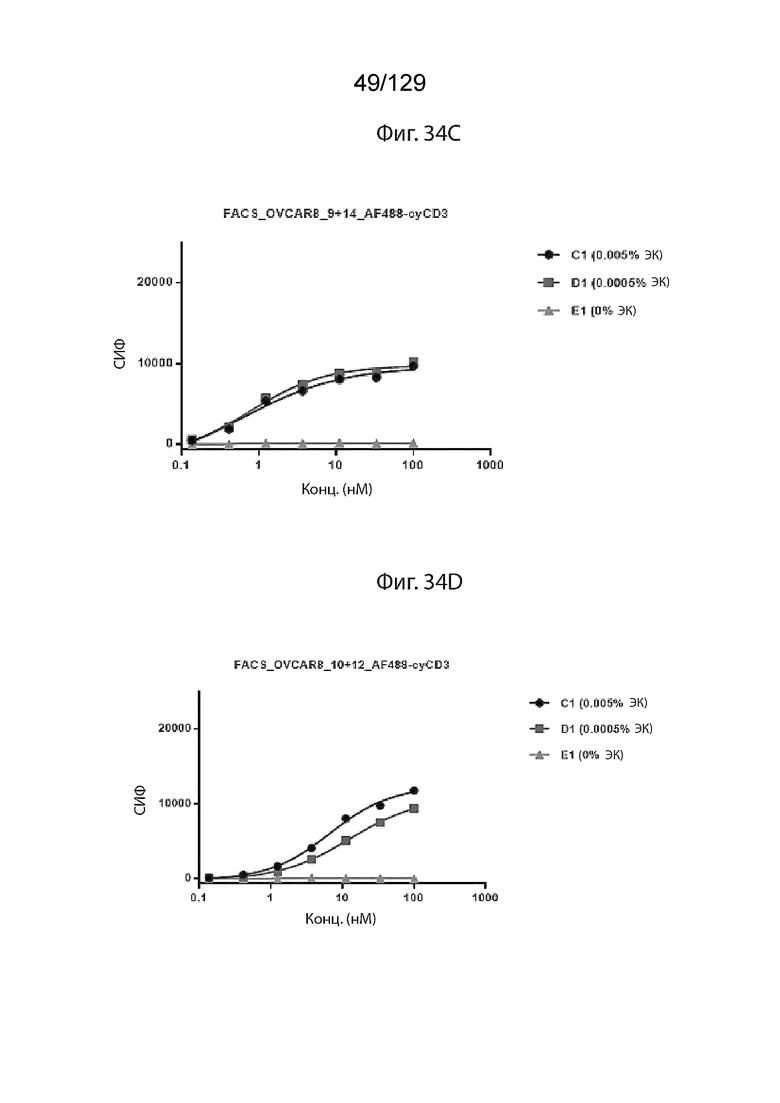
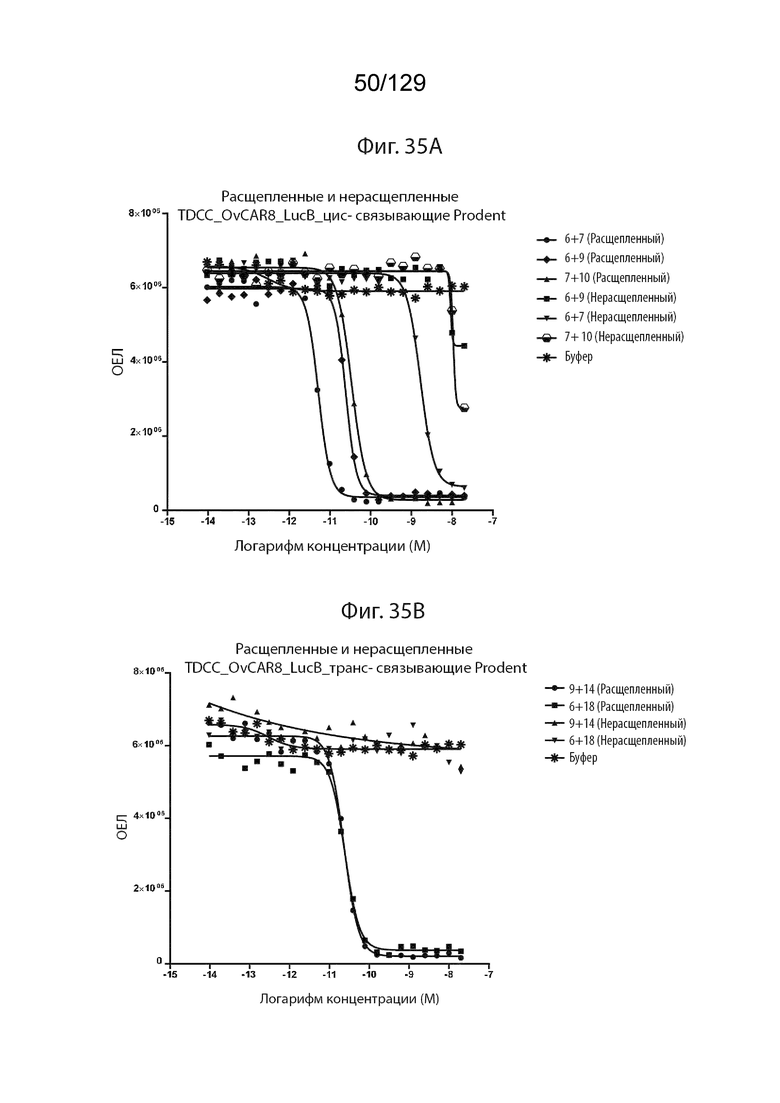


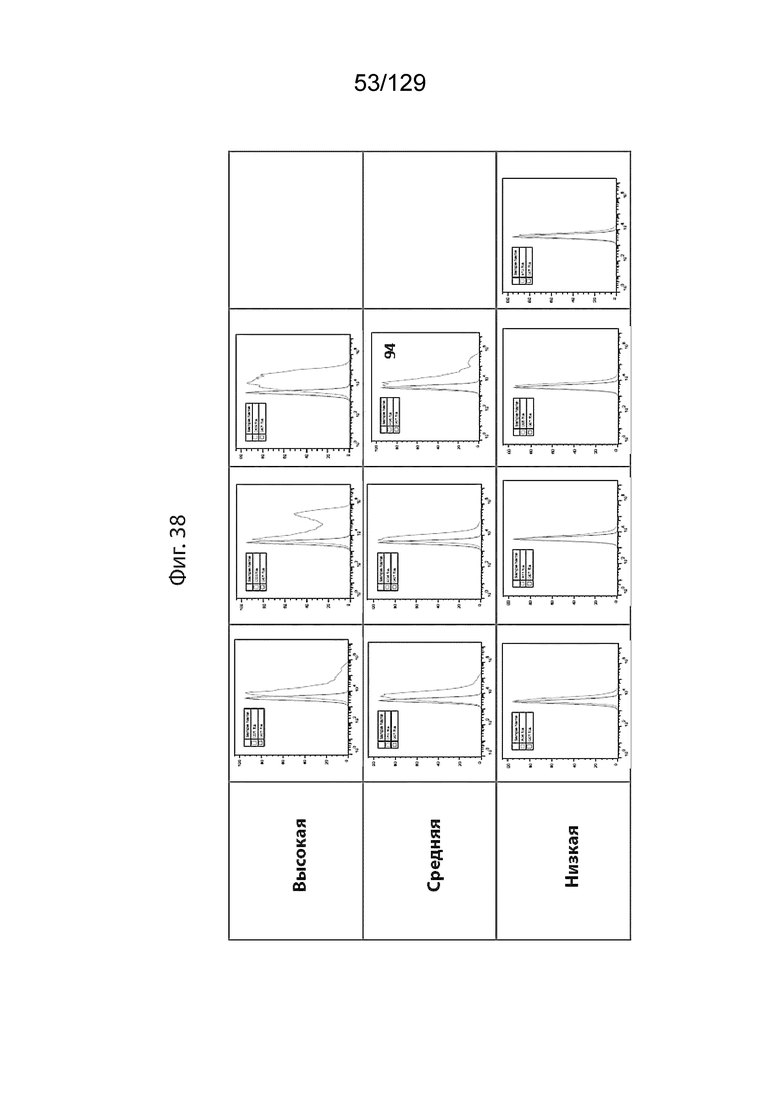
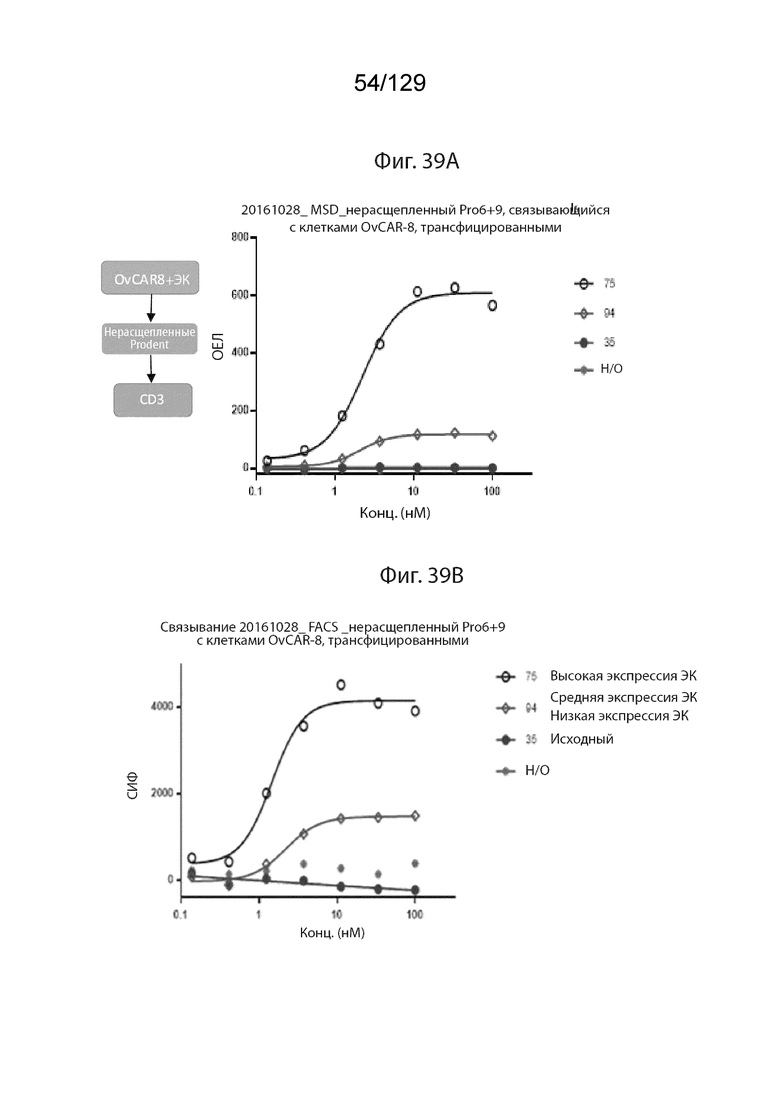

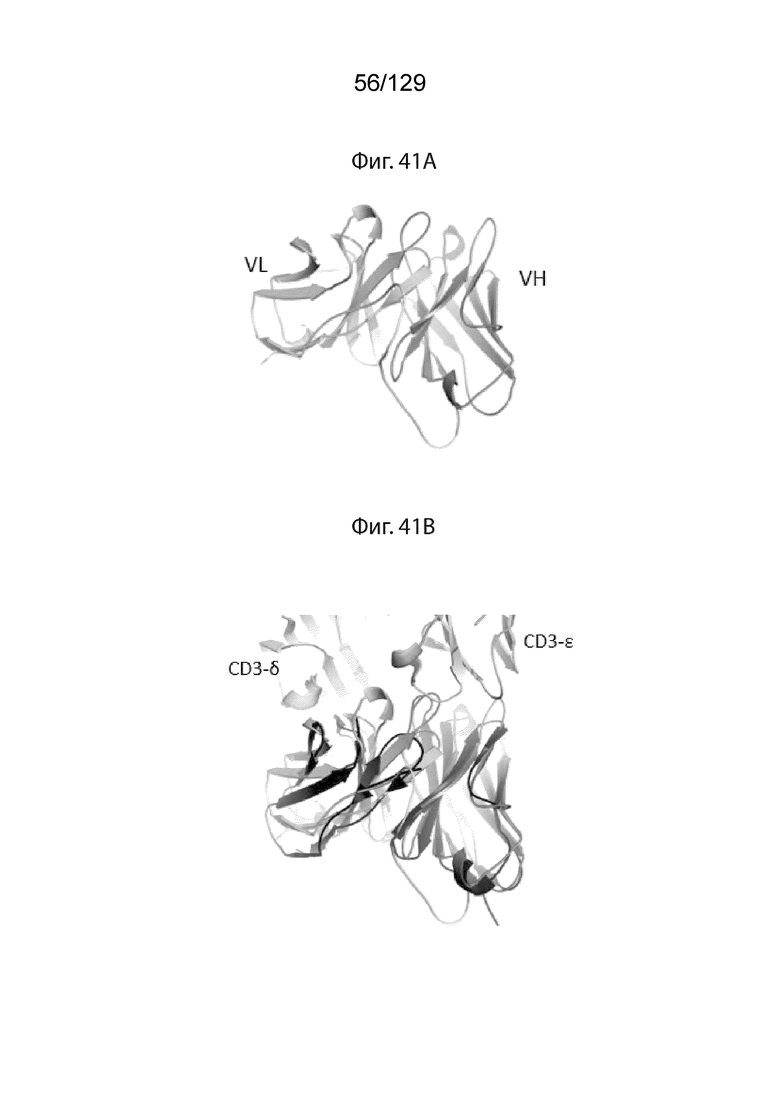


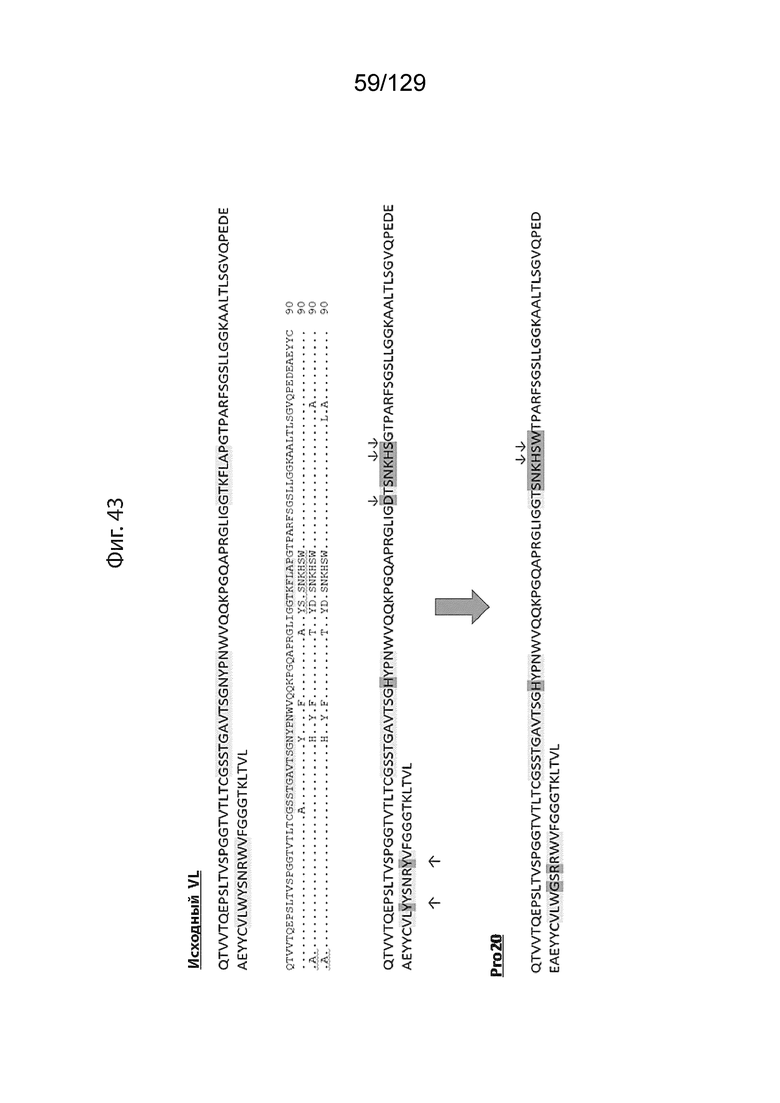


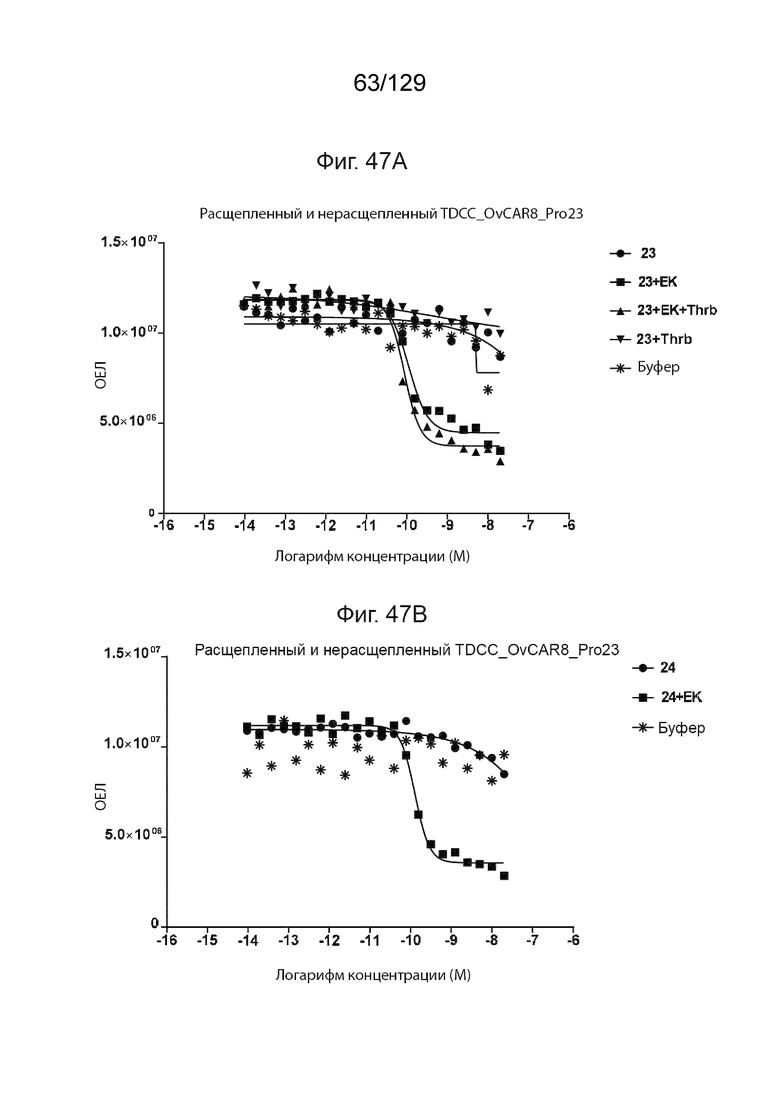
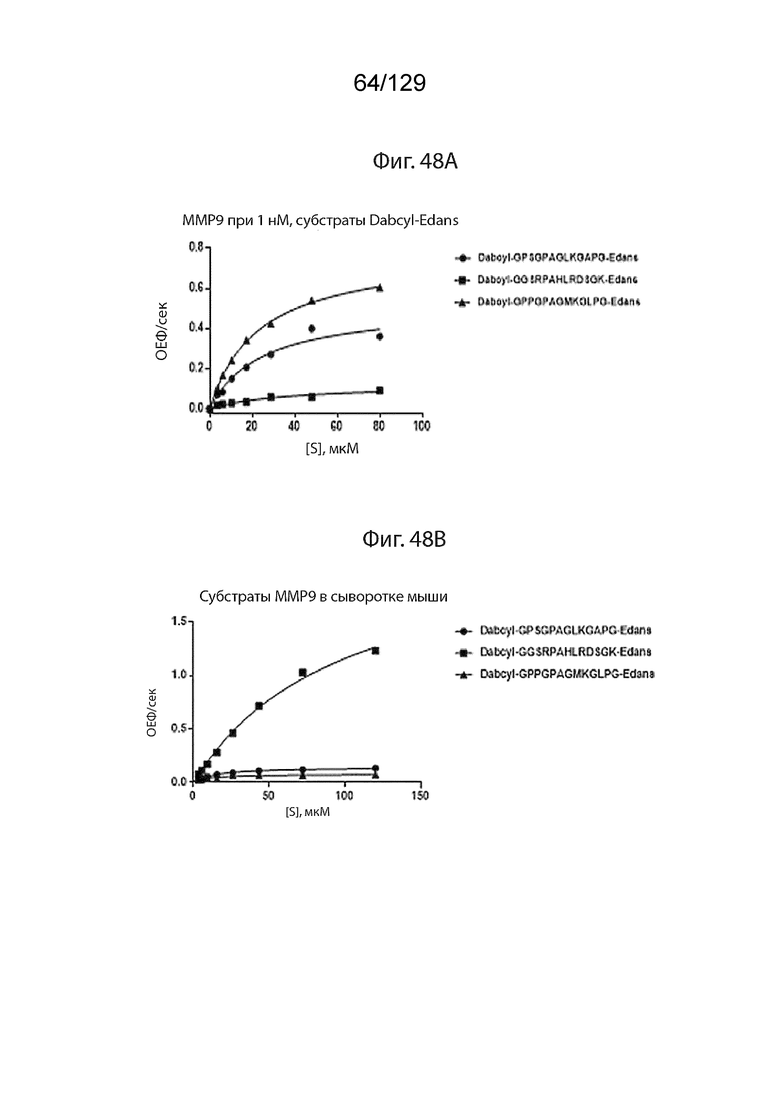
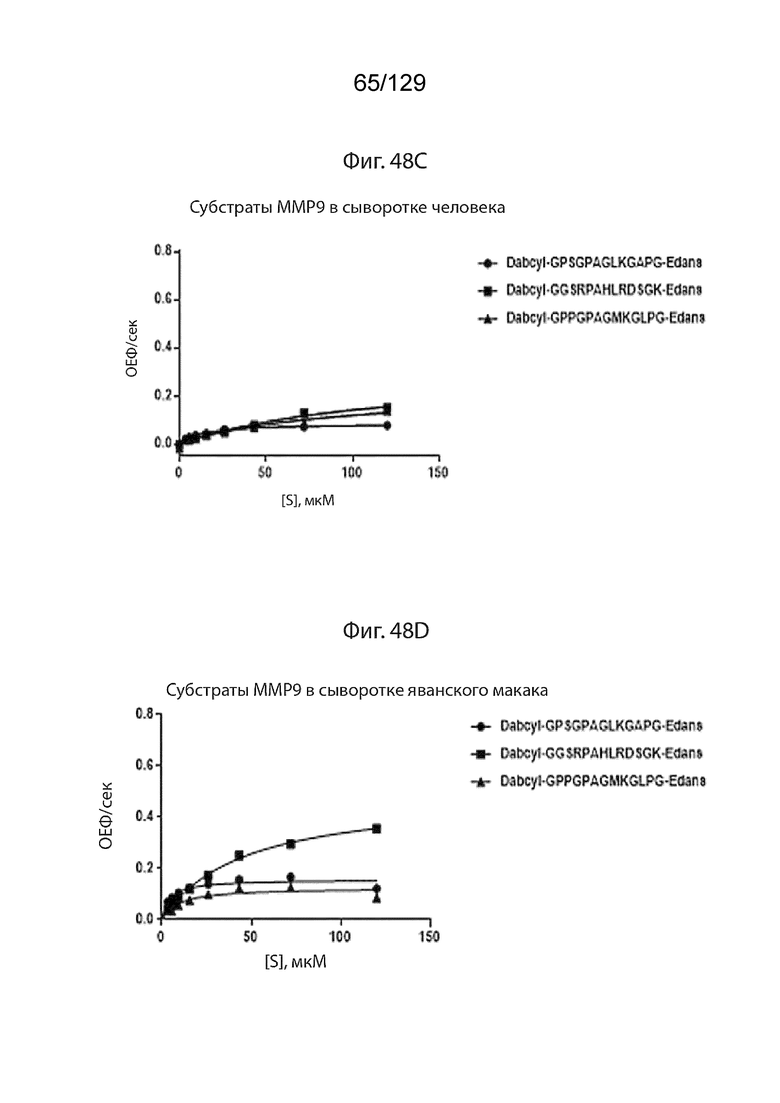
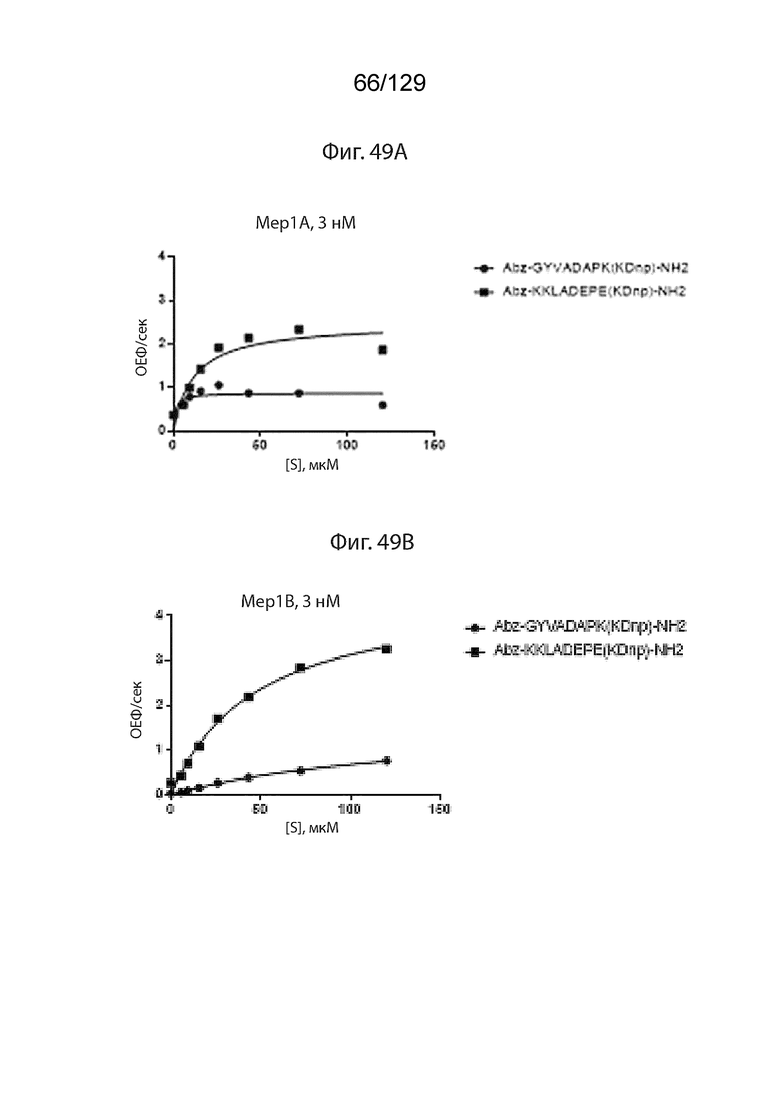
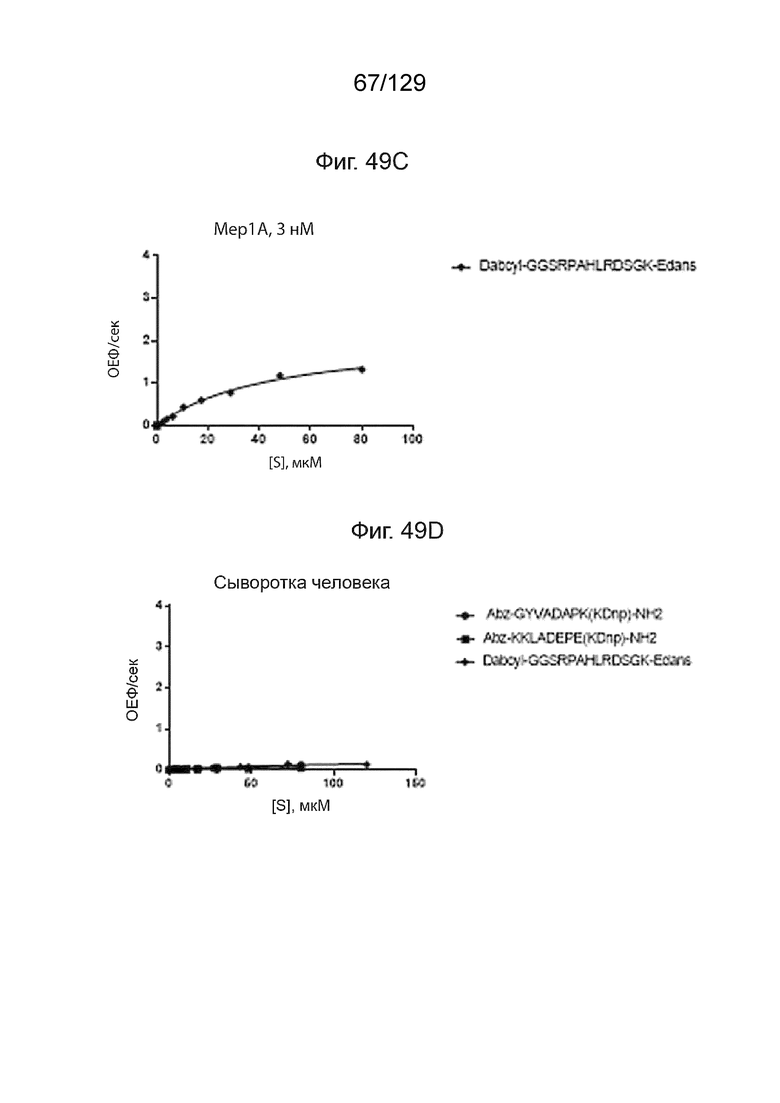

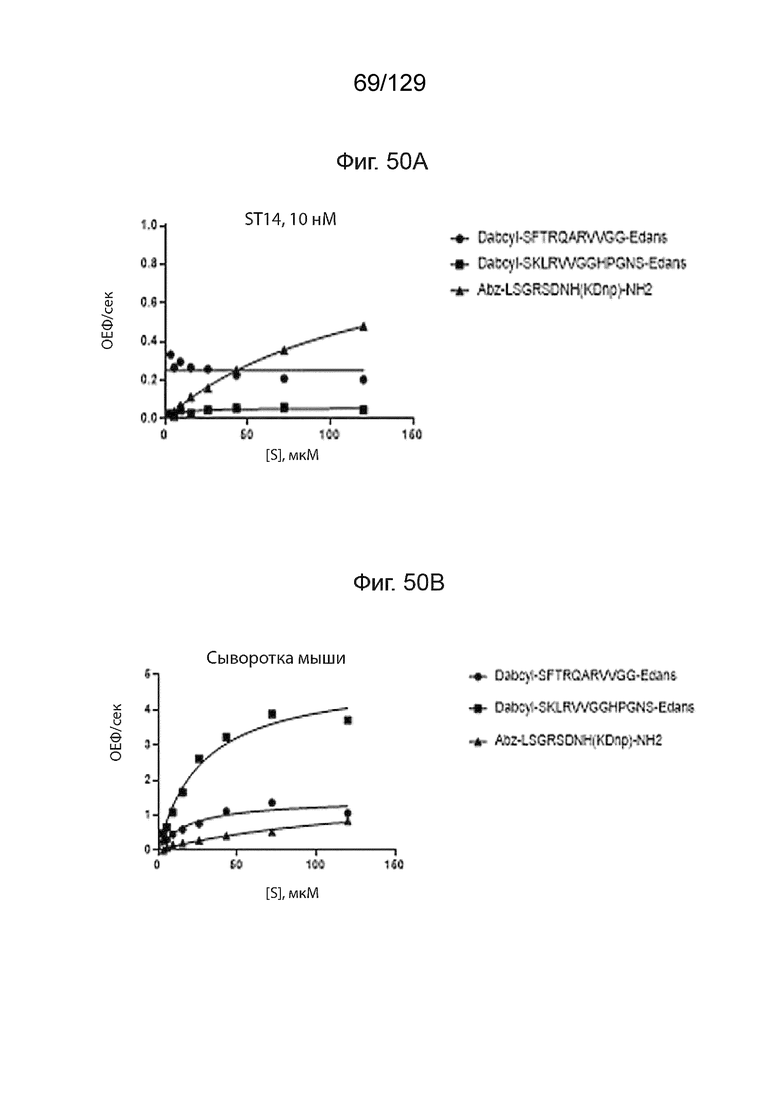
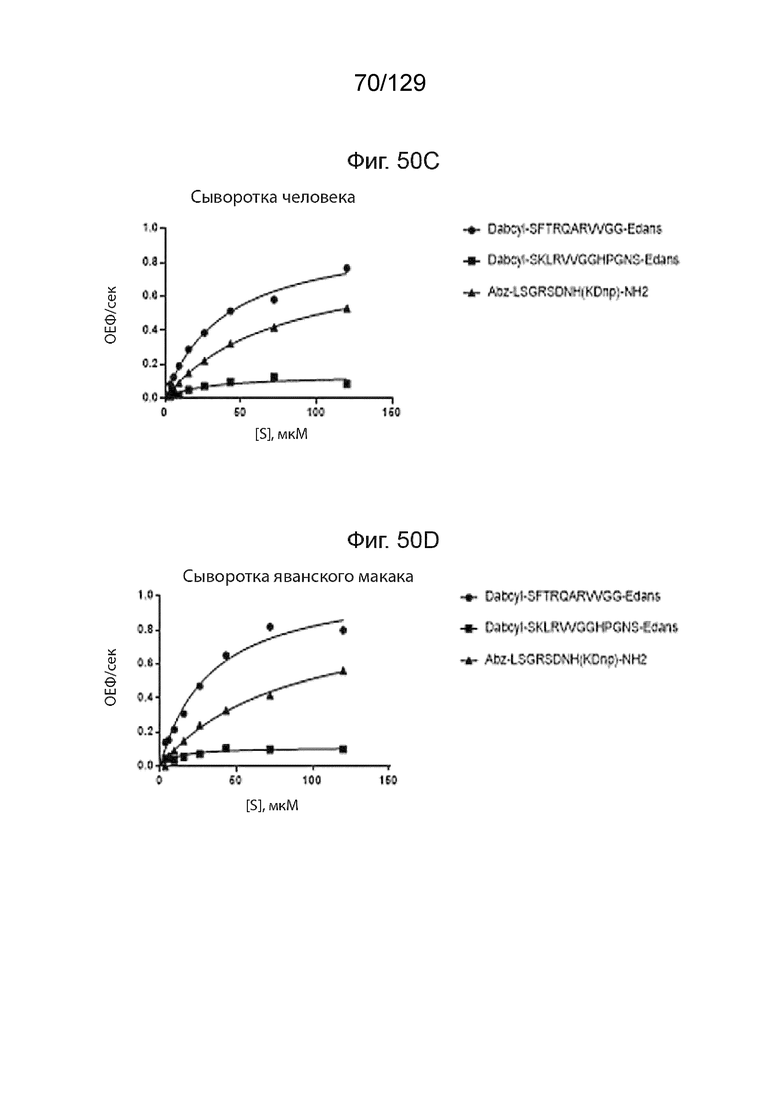
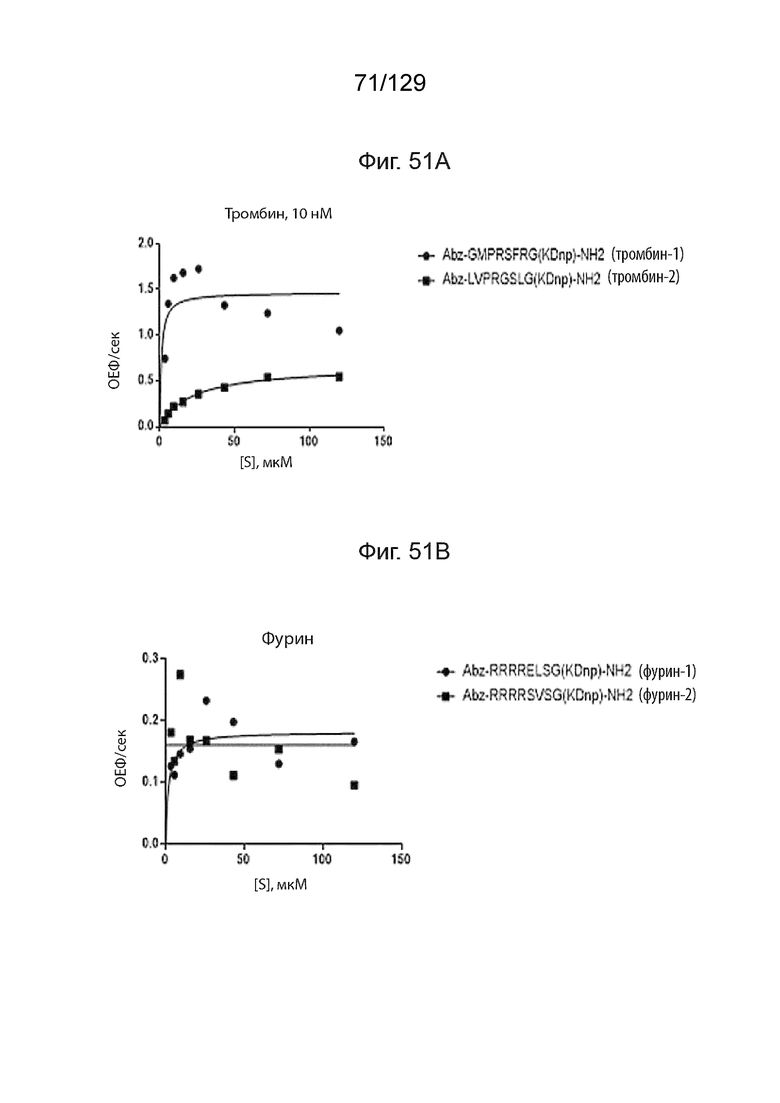
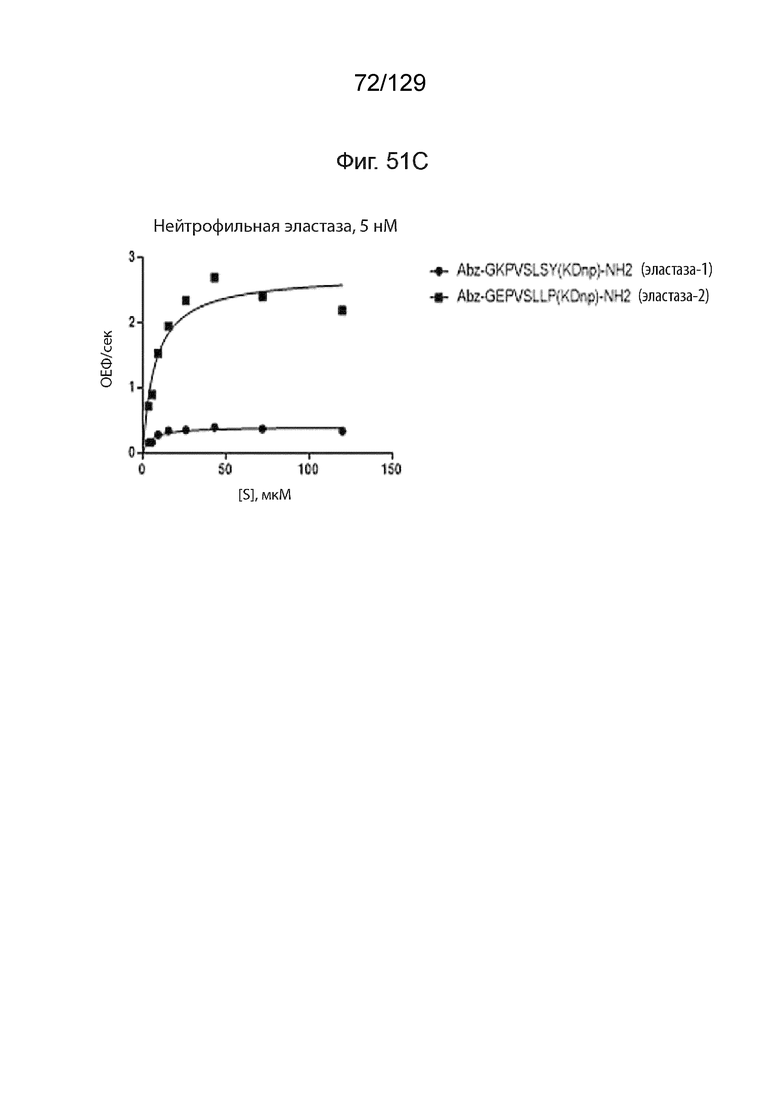
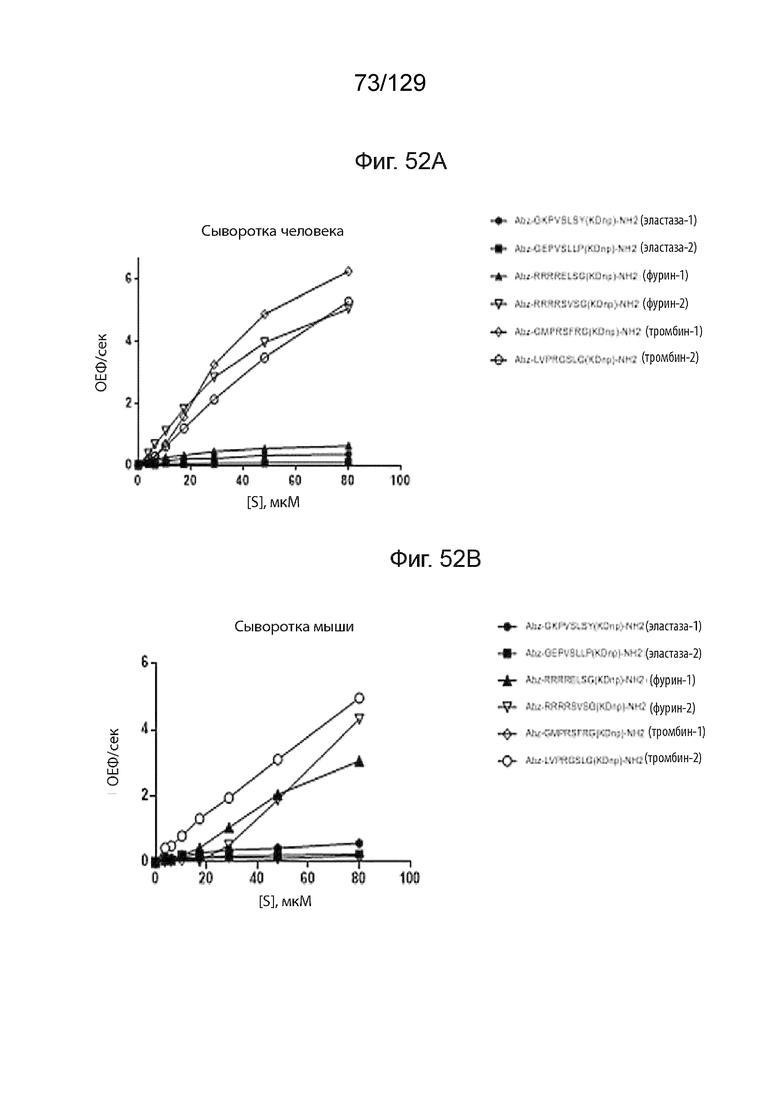
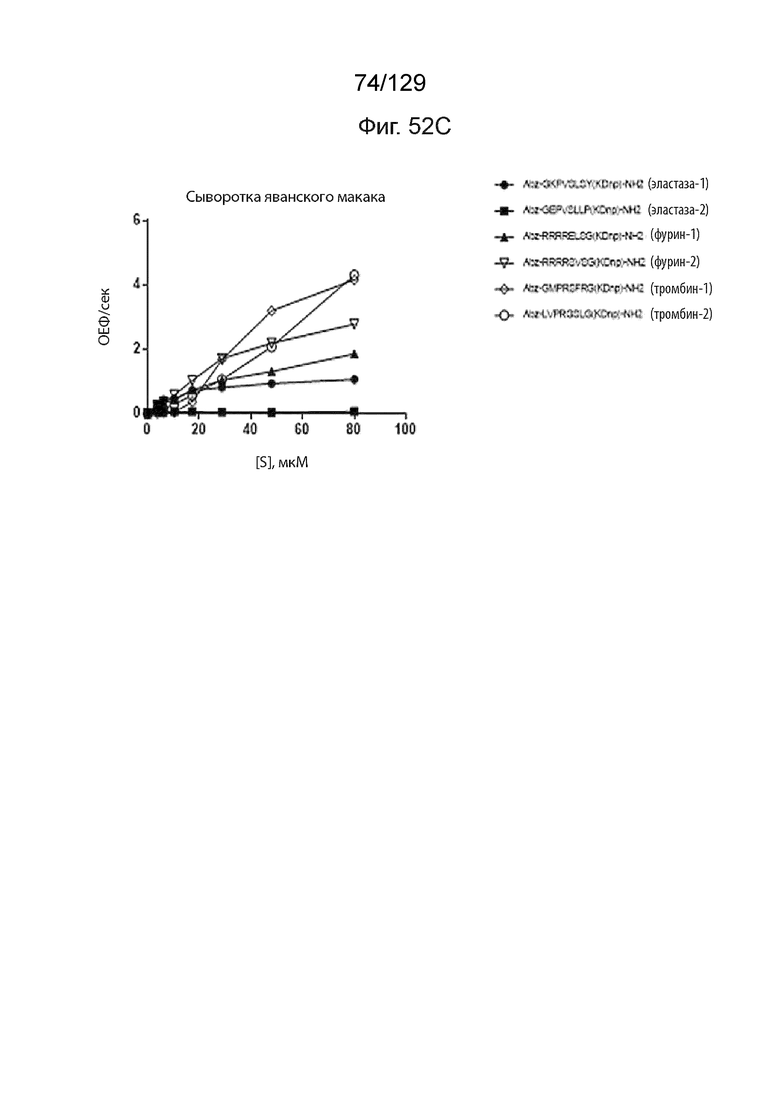




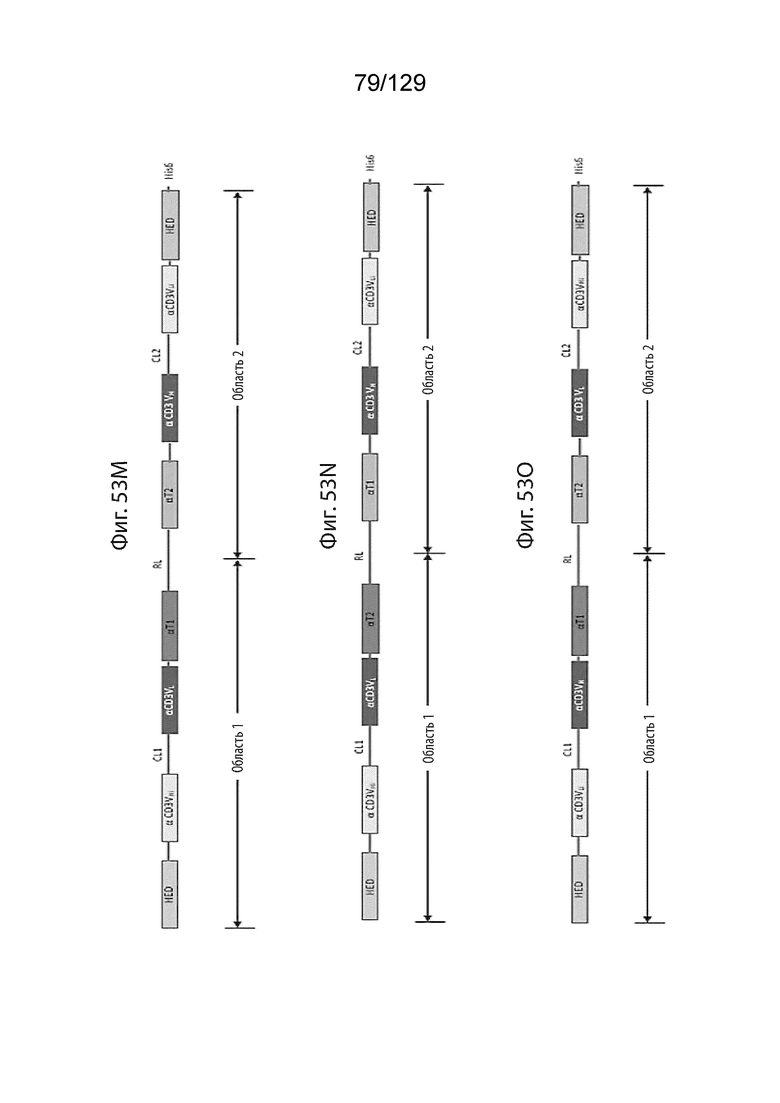
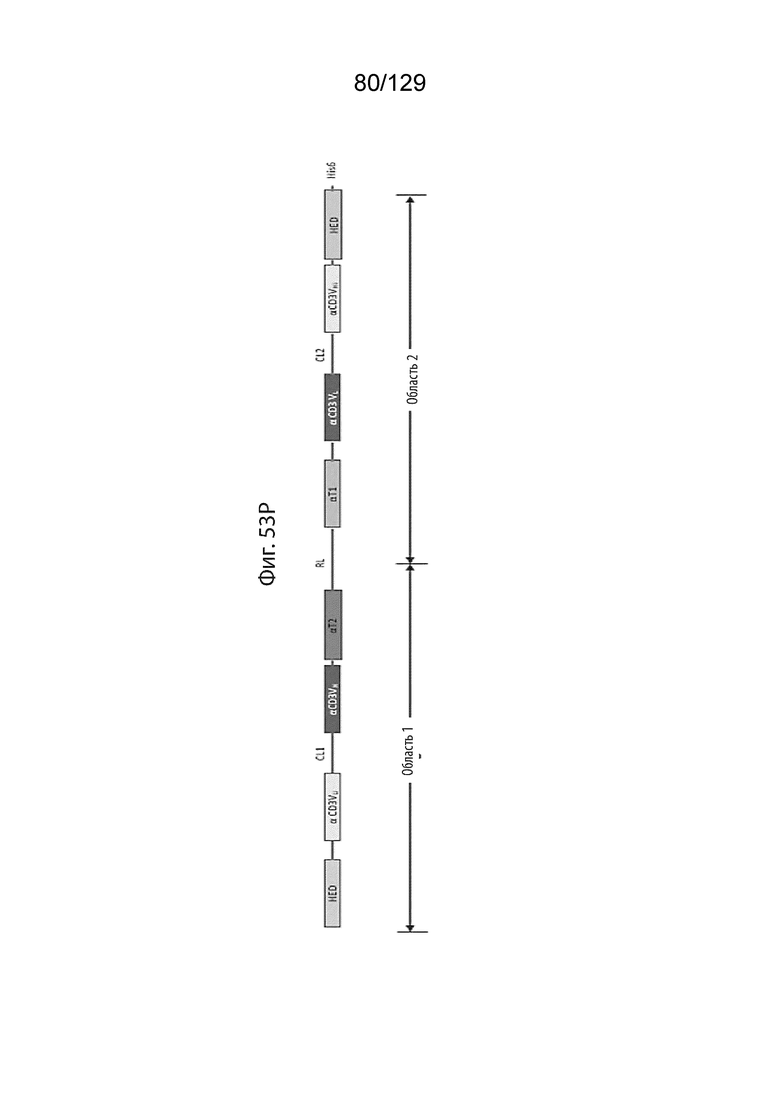













































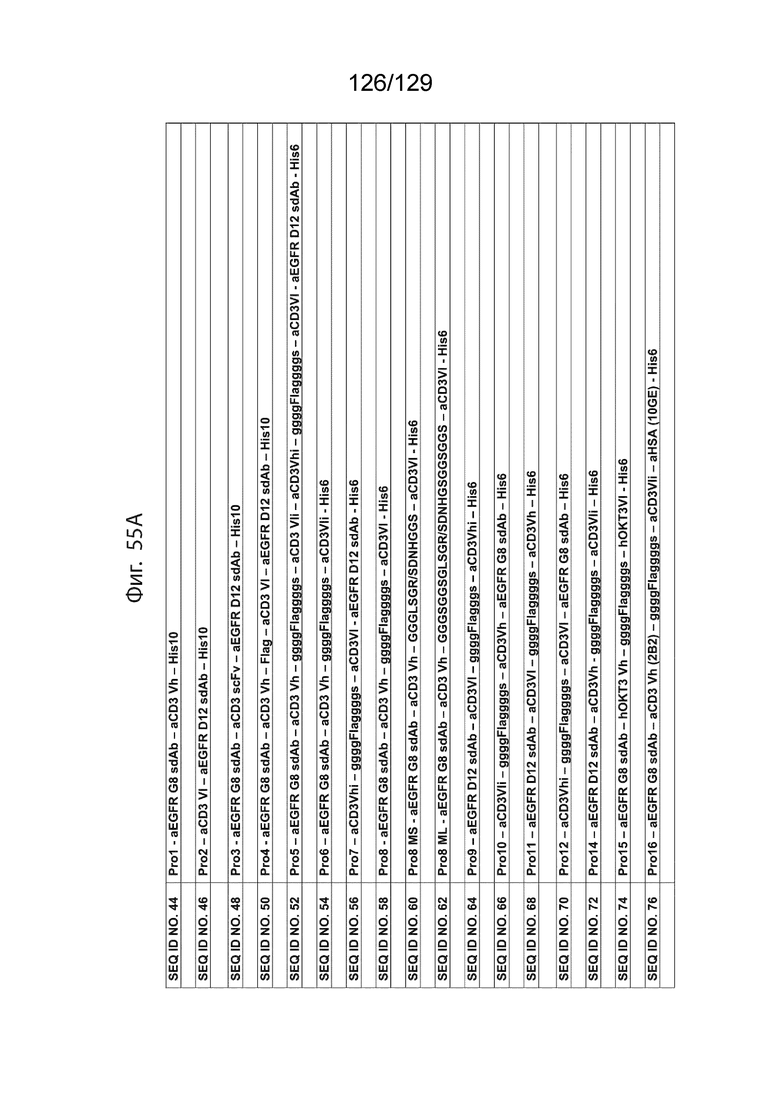



Группа изобретений относится к биотехнологии. Представлены: композиция для активации Т-клеток, содержащая пару полипептидных конструкций, вектор экспрессии, клетка-хозяин для получения композиции и фармацевтическая композиция для активации Т-клеток. Изобретения используются для лечения рака. 4 н. и 15 з.п. ф-лы, 1 табл., 129 ил., 8 пр.
1. Композиция для активации Т-клеток, содержащая пару полипептидных конструкций, содержащих:
a) первый полипептид, содержащий в порядке от N-конца к C-концу:
i) первый домен, связывающий целевой антиген, который связывает первый целевой антиген на раковой клетке;
ii) первый доменный линкер;
iii) первый домен scFv, направленный на антиген CD-3, содержащий:
1) первый активный домен VL, который способен спариваться с активным доменом VH после активации, чтобы специфически связывать антиген CD-3;
2) линкер, расщепляемый первой протеазой; и
3) первый неактивный домен VH, где первый активный домен VL и первый неактивный домен VH первого домена scFv спариваются вместе таким образом, что первый домен scFv не связывает указанный антиген CD-3 до активации; и
iv) первый домен увеличения периода полувыведения; и
b) второй полипептид, содержащий в порядке от N- до C-конца:
i) второй домен, связывающий целевой антиген, который связывает второй целевой антиген на раковой клетке;
ii) второй доменный линкер;
iii) второй домен scFv, направленный на указанный антиген CD-3, содержащий:
1) первый активный домен VH, который способен спариваться с активным доменом VL после активации, чтобы специфически связывать антиген CD-3;
2) линкер, расщепляемый второй протеазой; и
3) первый неактивный домен VL, где указанный первый активный домен VH и первый неактивный домен VL второго домена scFv спариваются вместе таким образом, что второй домен scFv не связывает указанный антиген CD-3 до активации; и
iv) второй домен увеличения периода полувыведения,
где активация включает расщепление протеазой указанных линкеров, расщепляемых первой и второй протеазами, где при таком расщеплении протеазой:
(a) первый неактивный домен VH первого домена scFv и первый домен увеличения периода полувыведения отщепляются от первого полипептида,
(b) первый неактивный домен VL второго домена scFv и второй домен увеличения периода полувыведения отщепляются от второго полипептида,
(c) первый активный домен VL первого домена scFv и первый активный домен VH второго домена scFv образуют пару активный VH/активный VL, чтобы специфически связывать указанный антиген CD-3, и
(d) первый домен, связывающий целевой антиген, и второй домен, связывающий целевой антиген, связывают первый целевой антиген и второй целевой антиген, соответственно.
2. Композиция по п. 1, где указанный первый и/или второй домен увеличения периода полувыведения связывается с ЧСА.
3. Композиция по любому из предшествующих пунктов, где указанный первый и/или второй домен увеличения периода полувыведения имеет последовательность EVQLVESGGGLVQPGNSLRLSCAASGFTFSKFGMSWVRQAPGKGLEWVSSISGSGRDTLYAESVKGRFTISRDNAKTTLYLQMNSLRPEDTAVYYCTIGGSLSVSSQGTLVTVSS (аминокислоты 397 - 511 SEQ ID NO: 76).
4. Композиция по п. 1, где указанный первый и/или второй домен увеличения периода полувыведения имеет последовательность EVQLVESGGGLVQPGNSLRLSCAASGFTFSKFGMSWVRQAPGKGLEWVSSISGSGRDTLYAESVKGRFTISRDNAKTTLYLQMNSLRPEDTAVYYCTIGGSLSVSSQGTLVTVSSHHHHHH (аминокислоты 397 - 517 SEQ ID NO: 76).
5. Композиция по любому из пп. 1, 2 или 4, где указанный первый и второй целевые антигены выбирают из группы, состоящей из EpCAM, EGFR и FOLR1.
6. Композиция по любому из пп. 1, 2 или 4, где указанный линкер, расщепляемый первой и/или второй протеазами и расщепляемый протеазой человека, выбранной из группы, состоящей из MMP2, MMP9, меприна, матриптазы, тромбина, uPA и катепсина.
7. Композиция по п. 1 или 2, где указанный первый полипептид и указанный второй полипептид каждый содержат дополнительный домен, связывающий целевой антиген.
8. Композиция по пп. 1, 2 или 4, где указанные первый и второй домены, связывающие целевой антиген, являются одинаковыми.
9. Композиция по любому из пп. 1, 2 или 4, где указанные первый и второй домены, связывающие целевой антиген, являются разными.
10. Композиция по любому из пп. 1, 2 или 4, где каждый из дополнительных доменов, связывающих целевой антиген, являются N-концевыми к первому домену, связывающему целевой антиген, первого полипептида и второму домену, связывающему целевой антиген, второго полипептида.
11. Композиция по любому из пп. 1, 2 или 4, где указанный первый активный домен VH содержит последовательность EVQLVESGGGLVQPGGSLKLSCAASGFTFNKYAMNWVRQAPGKGLEWVARIRSKYNNYATYYADSVKDRFTISRDDSKNTAYLQMNNLKTEDTAVYYCVRHGNFGNSYISYWAYWGQGTLVTVSS (аминокислоты 137-261 SEQ ID NO: 44) и указанный первый активный домен VL содержит QTVVTQEPSLTVSPGGTVTLTCGSSTGAVTSGNYPNWVQQKPGQAPRGLIGGTKFLAPGTPARFSGSLLGGKAALTLSGVQPEDEAEYYCVLWYSNRWVFGGGTKLTVL (аминокислоты 134-242 SEQ ID NO: 66).
12. Композиция по любому из пп. 1, 2 или 4, где указанный первый активный домен VH содержит последовательность EVQLVESGGGLVQPGGSLKLSCAASGFTFNKYAINWVRQAPGKGLEWVARIRSKYNNYATYYADQVKDRFTISRDDSKNTAYLQMNNLKTEDTAVYYCVRHANFGNSYISYWAYWGQGTLVTVSS (аминокислоты 137-261 SEQ ID NO: 76), второй активный домен VL содержит последовательность QTVVTQEPSLTVSPGGTVTLTCASSTGAVTSGNYPNWVQQKPGQAPRGLIGGTKFLVPGTPARFSGSLLGGKAALTLSGVQPEDEAEYYCTLWYSNRWVFGGGTKLTVL (аминокислоты 134-241 SEQ ID NO: 82).
13. Композиция по любому из пп. 1, 2 или 4, где указанный первый домен, связывающий целевой антиген, представляет собой sdAb, содержащий последовательность аминокислот 1-127 SEQ ID NO: 76 или последовательность аминокислот 1-124 SEQ ID NO: 82.
14. Композиция по любому из пп. 1, 2 или 4, где указанный второй домен, связывающий целевой антиген, представляет собой sdAb, содержащий последовательность аминокислот 1-127 SEQ ID NO: 76 или последовательность аминокислот 1-124 SEQ ID NO: 82.
15. Вектор экспрессии, содержащий первую нуклеиновую кислоту, кодирующую указанный первый полипептид, охарактеризованный в любом из пп. 1-14, и вторую нуклеиновую кислоту, кодирующую указанный второй полипептид, охарактеризованный в любом из пп. 1-14.
16. Клетка-хозяин для получения композиции по любому из пп. 1-14, содержащая вектор экспрессии по п. 15.
17. Фармацевтическая композиция для активации Т-клеток, содержащая эффективное количество:
(а) композиции по любому из пп. 1-14,
(b) вектора экспрессии по п. 15 или
(с) клетки-хозяина по п. 16.
18. Фармацевтическая композиция по п. 17, где фармацевтическая композиция предназначена для лечения рака.
19. Фармацевтическая композиция по п. 18, где целевым раковым антигеном является EGFR.
| US 2007123479 A1, 31.05.2007 | |||
| ПОЛИСПЕЦИФИЧЕСКИЕ ДЕИММУНИЗИРУЮЩИЕ CD3-СВЯЗУЮЩИЕ | 2004 |
|
RU2401843C2 |
| CHENG M | |||
| et al., Structural design of disialoganglioside GD2 and CD3‐bispecific antibodies to redirect T cells for tumor therapy, International Journal of cancer, 2015, V | |||
| Регулятор для ветряного двигателя в ветроэлектрических установках | 1921 |
|
SU136A1 |
| Аппарат для очищения воды при помощи химических реактивов | 1917 |
|
SU2A1 |
| Рыболовная приманка | 1924 |
|
SU8799A1 |
Авторы
Даты
2023-07-14—Публикация
2017-03-08—Подача